一种胚系基因大片段重排的检测方法
文献发布时间:2023-06-19 19:30:30
技术领域
本发明涉及一种胚系RB1大片段重排的检测分析方法,特别是涉及了一种基于高通量测序数据检测大片段重排的技术,属于生物信息学技术领域。
背景技术
视网膜母细胞瘤基因(Retinoblastomal transcriptional corepressor 1,RB1)位于人类13号染色体,是人类第一个发现的抑癌基因,该基因与视网膜母细胞瘤(RB)的发生相关。视网膜母细胞瘤是儿童中最常见的眼内癌,其中90%以上的双侧病例受试者和10-25%的单侧病例具有遗传性RB1突变[1]。遗传性RB则与种系突变有关,种系变异是由父母遗传或胎儿发育过程中获得的,在新生儿中Rb的发病率为1/20000[2],大约40%的RB受试者是主要是由遗传易感基因RB1基因的胚系突变所致[3],一般判断为家族性(遗传性)视网膜母细胞瘤,遗传方式是常染色体的显性遗传。首都医科大学附属北京儿童医院于2014年2月至2020年8月期间,纳入263名无血缘关系的单侧RB受试者,使用NGS和ddPCR技术分别检测RB1基因的点突变及大片段缺失,在39名(14.8%)受试者中检出RB1种系突变,其中11名(28.2%)受试者有错义突变,10名(25.6%)有无义突变,2名(5.1%)有移码突变,1名(2.6%)有同义突变,7名(17.9%)有大片段缺失,2名(5.1%)有剪切位点突变,6名(15.4%)有不确定意义的变异[1]。在39名胚系RB1突变的受试者中,7名(17.9%)大片段缺失受试者均为致病突变,说明胚系RB1大片段重排的突变与肿瘤的发生密切相关。因此,开发一种NGS检测大片段重排技术,对具有家族遗传性疾病的早期诊断,早治疗,以防止肿瘤进一步进展和转移至关重要。
胚系突变中大片段重排(Large genomic rearrangements,LGR)指数百至数百万个碱基片段的重复或缺失,常累及一个或多个外显子。与常规点突变或小片段的插入缺失相比,检测难度较大。目前用于检测大片段重排的技术主要包括多重连接探针扩增(Multiplex Ligation Probe amplification,MLPA)、多重扩增子定量技术(MultiplexAmplicon Quantification,MAQ)即多重PCR技术、NGS-CNV。
多重连接探针扩增(MLPA)方法是检测LGR的金标准,它是一种基于多重聚合酶链反应(基于PCR技术)的方法,可检测基因拷贝数的变化、点突变和DNA甲基化。它可以通过使用极少量DNA在一次反应中同时对多达50种MLPA探针进行半定量扩增,其扩增产物的相对量反映了靶序列的相对拷贝数。由于商业化的探针试剂盒比较昂贵,越来越多的实验室使用自制的MLPA探针混合物,需要设计多达50种MLPA探针的全新的混合物,且MLPA探针的制备流程包括探针设计、探针合成、探针库优化、性能验证等环节,耗时长[4],且对实验室条件要求高等缺陷,难以在临床应用中普及。此外,MLPA探针只能检测已知的点突变,会遗漏未知且频率较低的点突变。
多重扩增子定量(MAQ)即多重PCR技术,是一种基于PCR的低成本及高通量的新技术,一次扩增反应可确定多种基因的拷贝数变化,与MLPA方法相比,MAQ可以可靠的检测重要的基因组畸变,具有广泛的运用前景[5]。该技术也存在一些局限性,如多重PCR检测重数增加,易形成二聚体甚至多聚体,非特异性扩增出现,扩增效率的降低,检测灵敏度和特异性降低;严重时非特异性扩增占主导,靶标扩增失败,造成假阳性或者假阴性,影响检测结果准确性。MAQ技术同样也只能用于已知的点突变的检测。
参考文献:
1.Fang X,Chen J,Wang Y,et al.Gallie BL.RB1 germline mutation spectrumand clinical features in patients with unilateral retinoblastomas.OphthalmicGenet.2021Oct;42(5):593-599.
2.Fernandes AG,Pollock BD,Rabito FA.Retinoblastoma in the UnitedStates:A40-Year Incidence and Survival Analysis.J Pediatr OphthalmolStrabismus.2018May 1;55(3):182-188.
3.Kamihara J,Bourdeaut F,Foulkes WD,et al.Retinoblastoma andNeuroblastoma Predisposition and Surveillance.Clin Cancer Res.2017Jul 1;23(13):e98-e106.
4.
5.Kumps C,Van Roy N,Heyrman L,et al.Multiplex Amplicon Quantification(MAQ),a fast and efficient method for the simultaneous detection of copynumber alterations in neuroblastoma.BMC Genomics.2010May 12;11:298.
发明内容
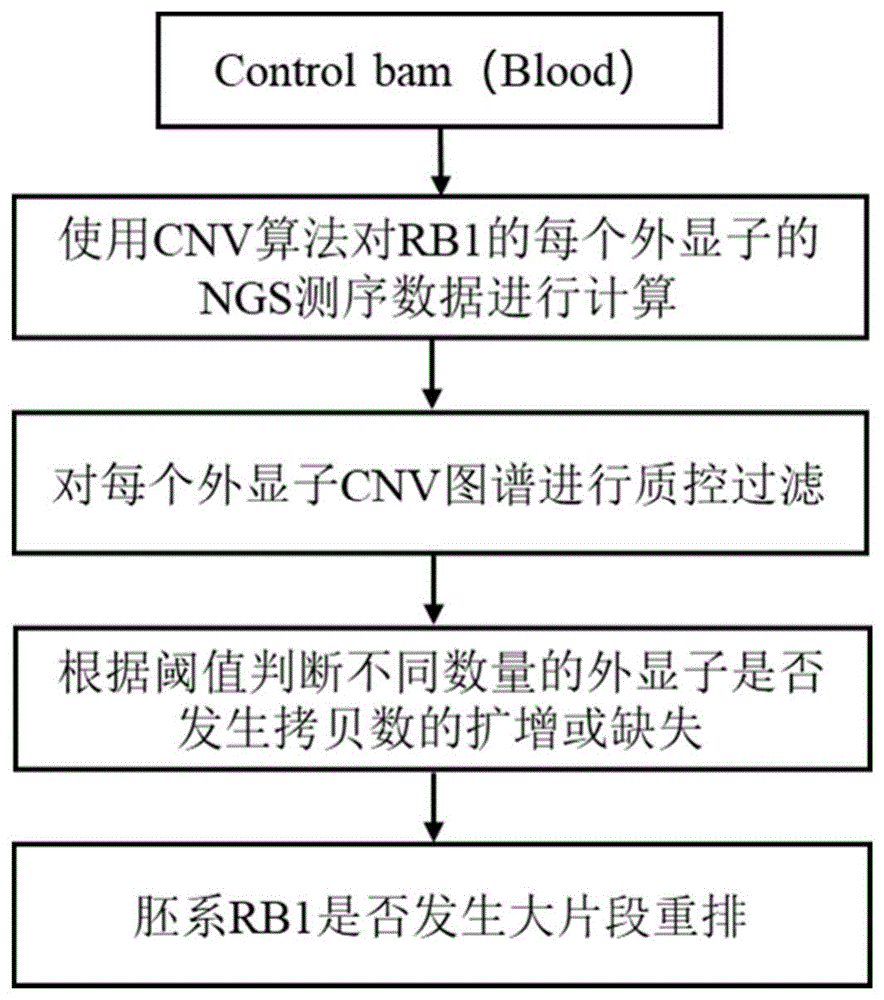
本发明所要解决的问题是目前多重扩增子定量(MAQ)和多重连接探针扩增(MLPA)难以推测大片段重排的基因变异问题,提出了一种检测胚系RB1大片段重排的方法。本发明是来自待测血液样本RB1基因的NGS测序数据,以RB1外显子为单位,通过使用CNV算法对测序数据进行处理,对待测样本(全血或白细胞)的RB1每个外显子的CNV图谱进行质控过滤,筛选出候选的外显子,根据扩增或缺失阈值过滤掉一些低证据的候选外显子,最后得到待测样本RB1外显子发生的大片段重排的情况。
一种胚系基因大片段重排的检测方法,包括如下步骤:
步骤1,通过引物或者探针对于目标基因的外显子区域进行扩增或者捕获,并获得用于高通量测序的文库;
步骤2,对文库进行高通量测序,获得RB1全部外显子reads的测序深度数据,计算出每个外显子的Zscore值;
步骤3,筛选得到噪音强度小于设定值的样本数据;
步骤4,进行以下判定:
步骤4-1,当需要判定RB1外显子缺失时,必须满足目标区间的SNP均为纯合型;
步骤4-2,当存在外显子区域的Zscore达到小于设定阈值时,将该外显子归入第一外显子集合;
步骤4-3,根据reads数据找到外显子区域上的两个断点,并获得断点在参考基因组上的位置,若在参考基因组上两个断点外侧的序列经拼接后能够与测序数据相同或互补,则将这两个断点之间的全部外显子归入第二外显子集合;
步骤4-4,对第一外显子集合和第二外显子集合进行并集处理,得到存在缺失的外显子。
所述的步骤1中,测序深度数据需要经过均一化、GC矫正或者降噪处理。
所述的Zscore值通过如下步骤计算得到:
Z=(x-μ)/σ;
X为单个外显子的Log
所述的第3步中,筛选过程包括如下步骤:
计算VP值,
log2ratio(exon)是指某个外显子log
且需要满足:对于血液样本,VP≤0.5;对于FFPE、新鲜组织样本或者除血液样本外的其他体液样本,VP≤0.8。
所述的步骤3中,杂合SNP的判定条件是:0.4 所述的步骤3中,纯合SNP的判定条件是:Vaf<0.1或Vaf>0.9。 所述的步骤4-2中,Zscore达到设定阈值是指:当1个外显子表现为缺失时,需要满足的条件为该外显子Zscore<-6且CNV ratio<0.70;当2-8个外显子表现为缺失时,需要满足的条件为目标区间的Zscore≤-6;当≥9个外显子表现为缺失时,需要满足的条件为目标区间的Zscore≤-4。 上述的方法应用于非治疗与诊断过程。 一种胚系基因大片段重排的检测装置,包括: 测序模块,用于对文库进行高通量测序,获得RB1全部外显子reads的测序深度数据,计算出每个外显子的Zscore值;所述的文库是通过引物或者探针对于目标基因的外显子区域进行扩增或者捕获后进行处理得到; 去噪声模块,用于根据测序模块得到的reads数据筛选得到噪音强度小于设定值的样本数据; SNP筛选模块,用于筛选出SNP均为纯合型的目标区间; 第一外显子缺失判定模块,用于判定当存在外显子区域的Zscore达到小于设定阈值时,将该外显子归入第一外显子集合; 第二外显子缺失判定模块,用于根据reads数据找到外显子区域上的两个断点,并获得断点在参考基因组上的位置,若在参考基因组上两个断点外侧的序列经拼接后能够与测序数据相同或互补,则将这两个断点之间的全部外显子归入第二外显子集合; 并集处理模块,用于对第一外显子集合和第二外显子集合进行并集处理,得到存在缺失的外显子。 去噪声模块的筛选过程包括如下步骤: 计算VP值, log2ratio(exon)是指某个外显子log 且需要满足:对于血液样本,VP≤0.5;对于FFPE、新鲜组织样本或者除血液样本外的其他体液样本,VP≤0.8。 一种计算机可读取介质,其记载有可以运行上述的胚系基因大片段重排的检测方法的计算机指令。 有益效果 本发明提供了基于NGS数据检测胚系RB1大片段重排的生物信息分析方法,可用于家族遗传性视网膜母细胞瘤受试者的基因检测。 相比于现有的技术,NGS检测RB1基因突变类型包括点突变、小片段的插入缺失、大片段的重排。NGS检测用于家族遗传性肿瘤受试者的检测,表现为更加全面的突变类型及较高的灵敏度和特异性。在遗传性视网膜母细胞瘤的常规诊断中一份样本可以同时得到更加全面的RB1突变信息,节约检测时间及降低检测成本。 NGS-CNV技术不仅为RB受试者检测各类胚系RB1提供早期的诊断和治疗提供服务,NGS检测同样可扩展至其他癌种中遗传易感基因RB1基因LGR的致病性胚系突变,甚至应用于其他胚系易感基因的LGR检测。 附图说明 图1是Zscore不同阈值条件下AUC曲线:A是单个外显子缺失Zscore在不同阈值下的AUC曲线,B是2-8个外显子缺失Zscore在不同阈值下的AUC曲线,C是≥9个外显子缺失Zscore在不同阈值下的AUC曲线; 图2是NGS检测RB1基因大片段缺失的通过断点序列配对分析方法的IGV示例; 图3是样本降噪前后CNV结果图; 图4是VP较高样本IGV示例; 图5是NGS检测RB1基因大片段缺失的IGV示例; 图6是视网膜母细胞瘤胚系RB1基因的突变类型扇形图; 图7是其他癌种发生胚系RB1大片段重排的受试者例数分布图; 图8是本方法流程图。 具体实施方式 下述实施方案中NGS检测胚系RB1基因大片段重排的检测,采用世和基因自主研发的世和一号panel进行基因检测获得,该测序panel全面覆盖RB1基因的全外显子及重要内含子区域,世和一号panel具体基因列表见表1。总的来说,本发明所述方法可以通过世和一号panel得到的NGS测序数据,可检测肿瘤易感基因胚系RB1基因大片段重排,用于视网膜母细胞瘤及其他泛实体瘤的早诊检测。 表1:425panel基因列表 /> 以上的检测过程采用的基因panel为示例,其仅仅用于获得RB1基因的测序数据,并非意指需要特定这种基因panel进行检测。 本发明中,RB1的LGR多表现为一个或多个外显子的杂合性拷贝数缺失。 NGS的检测过程涉及到样本的提取、文库的制备、上机测序等过程,这些可以通过现有的高通量测序中涉及的实验方法进行处理。本发明不作特定限定。对于下机数据的进行处理时,基于以下的步骤: 获得CNV分析数据: 基于CNV算法对测序数据进行处理,得到CNV图谱,该算法说明为:计算RB1外显子的read count,对read count进行均一化,计算待测样本的平均测序深度,进行GC矫正后重定位到二倍体状态,计算log 每个外显子的Zscore值,其公式为: Z=(x-μ)/σ; X为单个外显子的Log 接下来,需要进行VP质控,所述的VP质控用于显示样本CNV图谱的噪音强度,具体描述为:每个外显子到前一个外显子的log 如果VP值过高,则认为该样本存在过高噪音,质控不合格,会导致样本拷贝数变异存在假阳性或假阴性结果。 exon 本发明基于RB1外显子的CNV图谱进行质控过滤,样本的阈值需满足下列要求,质控才表现为合格:对于血液样本,VP≤0.5;对于FFPE、新鲜组织样本或者其他体液样本,VP≤0.8。 当血液样本VP≤0.5时,说明该样本外显子的质控合格; 当FFPE、新鲜组织样本或者其他体液样本VP≤0.8时,说明该样本外显子的质控合格; 接下来需要结合缺失阈值,对质控合格的候选外显子进行过滤筛选,去除一些低证据的外显子: 筛选出质控合格的外显子后,引入SNP作为辅助判断的依据,用来排除假阳性。所选SNP位点为在1000g/ExAC/gnomAD等数据库中总人群或东亚人群的突变频率≥1%的SNP位点,即高频胚系突变位点,见表2。 表2:RB1基因候选SNP位点 /> 若目标区间的SNP均为纯合型,则可以进一步根据Zscore阈值判断是否发生CNV缺失;若目标区间的SNP为杂合型或者偏移SNP,则判断其发生缺失的可能性很小;由于发生大片段缺失时,杂合型的SNP会存在一条链的丢失,较大的可能性无法检测得到,因此通过该校验步骤可以使提高准确性。进行校验时采用以下的判定规则:所述纯合SNP和偏移SNP的丰度范围为:纯合SNP(Vaf<0.1或Vaf>0.9),杂合SNP(0.4 具体而言: 缺失阈值判断的必要条件为SNP的类型属于纯合型,当1个外显子表现为缺失时,需要满足的条件为该外显子Zscore<-6且该外显子CNV ratio<0.70;当2-8个外显子表现为缺失时,需要满足的条件为每个目标区间的Zscore≤-6;当≥9个外显子表现为缺失时,需要满足的条件为每个目标区间的Zscore≤-4。 其中,CNV ratio=2 上述步骤Zscore阈值的合理性,通过135例经ddPCR验证RB1基因LGR突变状态的患者,分别去计算单外显子缺失,Zscore值在-2,-3,-4,-5,-6,-7,-8条件下的AUC曲线,见图1的A,Zscore阈值为-6时,检测准确性更高。2-8个外显子缺失,Zscore值在-2,-3,-4,-5,-6,-7,-8条件下的AUC曲线,见图1的B,Zscore阈值为-6时,检测准确性更高。≥9个外显子缺失,Zscore值在-2,-3,-4,-5,-6条件下的AUC曲线,见图1的C,Zscore阈值为-4时,检测准确性更高。 此外,上述的步骤中,通过对测序read深度进行判定,以外显子为单位判定是否存在缺失,由于个别外显子可能由于GC含量、DNA质量、探针覆盖度、探针特异性等原因,使得Zscore或CNV ratio略高于阈值,可能导致该外显子出现假阴性/假阳性。为了进一步提高检测灵敏度,本专利中采用断点分析的方式进行检测结果的补充。 目标区间断点序列分析中,首先根据测序数据对全部的外显子区域内进行断点分析,可以采用现有的手段获得,例如采用Delly软件进行分析。该软件是采用两种算法检测基因结构变异,discordantly mapped read-pairs以及split-read (https://github.com/dellytools/delly.git)。 首先,计算read-pair的方向,以及insert size分布。基于这些数据来确定所有discordantly mapped read-pairs是否存在非正常的mapping方向或者是insert size比预计的大。接着,split-read分析可以帮助找到单碱基精度的融合断点信息,其分析过程可以分成多个步骤:1)检索候选的split-read;2)提取结构变异区域的参考基因;3)索引和k-mer计算;4)找到最好的断点支持;5)split-read的一致性计算;6)split-read的一致序列mapped到融合参考基因区域,生成候选RB1基因结构变异的表格,根据所述RB1内部断点染色体位置,通过IGV加载样本bam格式原始数据文件,进一步核对两处断点的序列是否相互匹配,如图2示意图,如果RB1基因内两处断点的序列相互匹配(序列相同或者互补),我们认为该目标区间存在大片段缺失,该方法可以辅助判别种系大片段缺失发生的具体染色体位置。 以下的测试过程基于以上的数据统计方法,更具体而言: 对测序数据进行处理进行拷贝数变异(CNV)的检测,首先计算Panel覆盖区域readcount,并对read count进行均一化处理,计算待测样本的平均测序深度,进行GC矫正后重定位到二倍体状态,计算log 纳入6例均为视网膜母细胞瘤受试者的血液样本,该样本LGR的RB1基因状态已经ddPCR验证,将ddPCR验证结果作为标准。这6例样本用表1所示的世和一号大panel进行高通量测序,我们首先测试是否纳入目标区域断点序列配对关系,对测序结果的性能影响。根据生信分析结果,于IGV查看发生大片段缺失的RB1基因,RB1大片段缺失均位于13号染色体-短臂,图5中显示V1样本RB1大片段缺失发生在染色体上两侧的位置分别为:13:49,034,823和13:49,053,867,前后断点序列之间互相匹配,进一步说明e21-e26存在大片段缺失,与Zscore分析结果一致。同样的图2显示V5样本中RB1大片段缺失发生在染色体上两侧的位置分别为:13:48,948,963和13:48,954,246,前后断点序列之间也是互相匹配的关系,进一步说明e13-16存在大片段缺失,然而该样本Zscore分析结果只显示e13-14的缺失,增加断点序列的匹配分析,可以提高检测的灵敏度。 最后我们将测序数据分别经本发明分析流程和Atlas-CNV(PMID:30890783)软件对RB1基因状态进行分析,以检出的外显子为单位计算本方法检测RB1基因LGR的灵敏度和特异性等指标。本发明所述方法检测RB1基因LGR具体信息如表3所示: 表3:不同分析流程方法下样本RB1大片段重排分析的结果(n=6) 注:ID代表每个样本名称;ddPCR-RB1代表ddPCR检测RB1基因LGR;本发明流程-RB1代表通过本发明生信分析流程检测分析RB1基因LGR结果;Atlas-CNV-RB1代表通过本Atlas-CNV(PMID:30890783)软件检测分析RB1基因LGR结果;del代表基因拷贝数缺失。 以ddPCR方法验证确认的胚系RB1基因LGR变异结果为标准,以发生缺失的RB1外显子(exon)为单位,分析两种高通量LGR分析方法的性能,见表4。结果表明:两种方法特异性均达到100%,本发明方法相较于Atlas-CNV方法灵敏度提升59%,准确性提升25%。从分析性能看,本发明分析流程性能更优。 表4:不同分析流程下样本RB1大片段重排分析性能(以外显子为单位n=162) 其他样本集的检测和验证 收集了188例根据全血样本检测到视网膜母细胞瘤胚系RB1突变的受试者,对每位受试者的突变类型进行整理,结果表明:其中130名(69%)受试者存在错义突变,13名(7%)受试者存在大片段的缺失,31名(17%)受试者存在小片段缺失导致的移码突变,14名(7%)受试者存在小片段的重复导致的移码突变,如图6所示。 筛选了从2020年11月至2022年2月送检世和一号的受试者样本,其中15例发现胚系RB1基因大片段重排,均为致病的或可能致病的变异,其中6例(40%)为卵巢癌,5例(33%)为肺癌,3例(20%)为乳腺癌,1例(6.7%)为胰腺癌,如图7所示。
