一种基因组瘢痕模型的建立方法
文献发布时间:2023-06-19 09:23:00
技术领域
本发明属于基因检测技术领域,具体涉及一种基因组瘢痕模型的建立方法。
背景技术
同源重组修复(Homologous Recombination Repair,HRR)是DNA双链损伤的重要修复方式,常见于细胞精确修复双链DNA上的有害断裂。HRR是一条涉及到多个步骤的复杂的信号通路,其中乳腺癌易感基因(BRCA1/2)作为重要的同源重组功能相关基因。如果BRCA基因出现突变导致BRCA1和BRCA2蛋白失去功能,就会引起HRR功能异常,常被称为同源重组缺陷(Homologous Recombination Deficiency,HRD),HRD作为肿瘤驱动事件广泛的存在于乳腺癌、卵巢癌、前列腺癌和胰腺癌中。通常携带BRCA1/2突变或表达异常的肿瘤表现为对铂类化疗药物和聚[ADP-核糖]聚合酶抑制剂(PARPi)敏感。因此对BRCA1/2基因突变检测作为此类疾病的临床分类与用药指导都有着突出的作用。
然而随着研究深入,BRCA基因突变检测逐渐不能满足现有的临床需求,BRCA基因突变检测富集的用药有效人群偏低,会遗漏部分治疗受益人群。例如,在三阴性乳腺癌中,20%的患者携带BRCA基因突变,然而患者人群对铂类药物整体响应率约为30%。同时在高级别浆液性卵巢癌中,30%的患者携带BRCA基因突变,然而患者人群对PARPi药物整体响应率约为50%,表明一部分BRCA检测阴性患者对铂类或PARPi仍有应答,因此BRCA检测会遗漏部分治疗受益人群。遗漏的原因主要包括:首先,BRCA基因突变检测相对局限。HRR功能相关基因众多,BRCA1/2是其中突变频率较高的两个。从药物作用原理上分析,只要能和铂类以及PARPi形成合成致死效应的HRR基因都值的关注(统称BRCAness事件),如PROfound临床研究结果表明HRR相关基因ATM缺陷对PRAPi奥拉帕利治疗前列腺癌具有效果,因此越来越多临床实验由关注BRCA转向其他HRR相关基因。其次,BRCA基因突变检测,不能覆盖所有引起HRR功能缺失的基因组异常类型。除了基因突变,BCRA1启动子区域的甲基化和BRCA基因区域内部的杂合性缺失(Loss of Heterozygosity,LoH)也是引起HR功能缺陷的主因。最后,BRCA基因突变检测结果解读复杂,容易遗漏,临床应用门槛较高。目前有很多权威机构如美国医学遗传学与基因组学学会(ACMG)和欧洲分子基因诊断质量联盟(EMQN)都推出了遗传性乳腺癌/卵巢癌分子遗传分析最佳实践指南。对BRCA突变分类:致病性,可能致病性,意义未明,可能良性和良性,不同指南引用证据等级也略有不同,这会给临床应用造成很大障碍。
基于以上原因,新型的可以简单定量评估细胞同源重组修复缺陷的临床分子标记物需求极为迫切。通过寻找因HRR缺陷引发的下游基因组突变(包括突变、拷贝数变异(copynumber variation,CNV),基因表达异常)特征的分子标记物是目前主要的研究方向。2009年,Olafur等人发现CNV突变特征和BRCAness紧密相关,2012年,Abkevich等人发现全基因组中LoH的个数和BRCAness事件显著相关。同年,Popova等人发现基因中大片段转移事件(Large-scale state transitions,LST)和BRCA1/2基因失活相关,而Birkbak等人也发现端粒不平衡(Telomeric allelic imbalance,TAI)在三阴性乳腺癌中与BRCAness事件相关,并在铂类治疗敏感的人群中显著富集。2016年,美国Myriad公司通过统计全基因组LoH、LST和TAI发生次数,定量计算HRD分数(HRD评分),该统计指标可以准确地预测BRCAness事件,同时也可以有效地富集出对铂类和PARPi治疗敏感的患者人群。HRD评分相比于单独检测BRCA基因可以多筛选出40%的潜在受益患者。此外,2017年Davies等人,发现全基因组单碱基突变模式,长短片段重排模式以及插入缺失模式都和HRR缺陷紧密相关,并且将这几种模式联合HRD评分,通过罗杰斯特回归可以准确预测BRCAness事件。然而以上方法都有各自的局限性,例如,HRD评分只是笼统将LoH,LST和TAI简单相加,事实上TAI和LoH在某些情况是有重叠,从而导致重复计数,此外某些CNV类型也未被HRD评分考虑在内。而Davies的模型虽然考虑全面,但是该模型需要全基因组测序统计各种突变类型的模式,从而导致检测成本极为昂贵。
发明内容
本发明的目的在于克服现有技术缺陷,提供一种基因组瘢痕模型的建立方法。
本发明的另一目的在于提供上述建立方法所建立的基因组瘢痕模型的应用
本发明的技术方案如下:
一种基因组瘢痕模型的建立方法,包括如下步骤:
(1)收集已知的阳性样本和阴性样本,组成训练集;
(2)对上述训练集中的CNV的情况进行分析,确定CNV的类型及相应的数量;
(3)确定BRCAness阳性事件和BRCAness阴性事件;
(4)通过机器学习方法,根据训练集中的BRCAness阳性事件、BRCAness阴性事件,训练获得步骤(2)所确定的不同类型的CNV的权重,然后将不同类型的CNV带权重累加,得到用以计算GSS(基因组瘢痕分数)的基因组瘢痕模型;
(5)另外收集已知的阳性样本和阴性样本,组成测试集,并按照步骤(2)获得测试集中CNV的类型及相应的数量;
(6)将步骤(5)所获得的结果代入步骤(4)得到的基因组瘢痕模型,以计算测试集的GSS,并根据GSS的分数验证上述基因组瘢痕模型,即成。
在本发明的一个优选实施方案中,步骤(2)中,对步骤(1)所获得的训练集进行测序分析,计算上述测序分析结果中的CNV情况,同时将相邻且拷贝数变异相同的区域连接成片段,以避免重复计算,确定CNV的类型及相应的数量;
进一步优选的,上述测序分析基于全基因组,全外显子组,靶向捕获测序或拷贝数变异芯片。
在本发明的一个优选实施方案中,所述BRCAness阳性事件包括:BRCA1/2中任意之一中,一个等位基因发生致病或疑致病性突变,另一个等位基因发生杂合性缺失;BRCA1/2中任意之一中,发生两个致病或疑似致病性突变;BCRA1中一个等位基因发生杂合性缺失,另一个等位基因启动子区域发生甲基化。
在本发明的一个优选实施方案中,所述BRCAness阴性事件为:HRR相关基因为野生型,并且对应等位基因没有发生杂合性缺失,或其启动子区域没有甲基化。
在本发明的一个优选实施方案中,所述步骤(2)中的CNV的类型根据变异片段长度、变异片段类型以及变异片段在基因组的位置而确定。
进一步优选的,所述变异片段的长度分为5-10M的短片段、大于10M且小于等于15M的中片段和大于15M的长片段。
进一步优选的,所述变异片段类型包括杂合性缺失、变异片段链不平衡性扩增和变异片段链平衡性扩增。
进一步优选的,所述变异片段在基因组上的位置包括变异片段位于端粒侧、变异片段位于着丝粒区域内以及变异片段位于除端粒侧和着丝粒区域内以外的位置。
本发明的另一技术方案之一:
上述建立方法所建立的基因组瘢痕模型在富集HRR突变相关人群中的应用。
本发明的另一技术方案之二:
上述建立方法所建立的基因组瘢痕模型在富集铂类用药敏感性人群中的应用。
本发明的另一技术方案之三:
上述建立方法所建立的基因组瘢痕模型在富集PARPi用药敏感性人群中的应用。
本发明的有益效果是:
1、相比BRCA基因突变检测,本发明可以不用检测BRCA1启动子区域的甲基化,以及BRCA基因的杂合性缺失,准确预测待测样本BRCAness状态。此外,相对于BRCA突变的复杂解读,本发明可以直接根据基因组瘢痕分数直接给出判读结果。
2、相比BRCA基因突变检测,本发明可以富集HRR相关基因突变患者。
3、相比BRCA基因突变检测,本发明可以富集更多的铂类用药敏感性患者。
4、相比BRCA基因突变检测,本发明可以富集更多的PARPi用药敏感性患者。
附图说明
图1为本发明实施例3的实验结果图。
图2为本发明实施例4的实验结果图之一。
图3为本发明实施例4的实验结果图之二。
具体实施方式
以下通过具体实施方式结合附图对本发明的技术方案进行进一步的说明和描述。
实施例1
分别收集110和18例检测样本类型为卵巢癌患者的FFPE样本和对照血液样本,作为构建基因组瘢痕模型的训练集和测试集。后采用厦门艾德生物医药科技股份有限公司同源重组缺陷(HRD)检测试剂盒进行文库构建和捕获,此试剂盒包含35个HRR相关基因以及7万个snp位点作为捕获区域。所捕获和富集的DNA最后在Illumina Novaseq测序仪上进行测序。
原始下机数据通过BWA(Li H.and Durbin R.2009),比对到人类参考基因组序列(版本号hg19)。生成比对好的BAM文件,作为突变和拷贝数变异的输入文件。其中突变检测利用Varscan流程(Koboldt,D.2012),链特异性的拷贝数变异利用sequenza(FaveroF.2015)流程。
BRCAness样本确认。选择目前最为常见的BRCAness事件作为阳性样本标签,具体包括a.BRCA1/2任意一个基因,一个等位基因发生致病或疑致病性突变,另一个等位基因发生杂合性缺失;b.BRCA1/2任意一个基因,发生两个致病或疑似致病性突变,即功能性缺失;c.BCRA1一个等位基因发生杂合性缺失,另一个等位基因启动子区域发生甲基化,其中BCRA1启动子区域类甲基化利用焦磷酸测序技术获得。
BRCAness阴性样本确认,HRR相关突变基因为野生型,并且对应基因没有发生LoH。厦门艾德生物HRD检测试剂包括HRR相关基因如下:ATM,FAM175A,FANCI,NBN,RAD51C,ATR,FANCA,FANCL,PALB2,RAD51D,ATRX,FANCC,FANCM,RAD50,RAD52,BAP1,FANCD2,KMT2D,RAD51,RAD54L,BARD1,FANCE,MDC1,RAD51B,SLX4,BLM,FANCF,MRE11A,WRN,XRCC2,BRCA1,FANCG,BRCA2,BRIP1,EMSY。
本实施例拷贝数变异片段按着变异长度分类,包括短片段(5-10M)、中型片段(大于10且小于等于15M)和长片段(>15M)。拷贝数变异片段也可以按着变异类型分类,包括杂合性缺失(Loss ofheterozygosity,LOH),变异片段链不平衡性扩增(Allele specificCNV,ASCNV),变异片段链平衡性扩增(Balance CNV,BCNV)。拷贝数变异片段还可以按着所处基因组位置分类,包括变异片段位于端粒侧,变异片段位于着丝粒区域内和剩余其他区域。最终拷贝数变异片段被分为27种类型(即长度分类×变异类型分类×所处基因组位置分类=27)
按上述处理,训练集中BRCAness样本68例,阴性样本42例,测试集中BRCAness样本10例,阴性样本8例。在训练过程中为了防止过拟合,只保留拷贝数变异片段在训练样本中出现次数大于训练集样本数目的类型。后利用逻辑斯蒂回归根据样本BRCAness类型训练拷贝数变异片段类型权重,从而构建基因组瘢痕模型。基因组瘢痕模型计算待测样本GSS分数小于0.5样本判定为BRCAness阴性样本,GSS分数大于0.5样本判定为BRCAness阳性样本。
在测试集中,利用基因组瘢痕模型计算待测样本GSS,判定待测样本的BRCAness状态。之后和测试集中样本的之前标记好BRCAness状态进行比较,其中10例BRCAness阳性样本,基因组瘢痕模型能准确判为阳性,8例BRCAness阴性样本,基因组瘢痕模型能准确判为阴性。即基因组瘢痕模型的GSS可准确预测样本BRCAness状态,其灵敏度为100%,特异性为100%,准确性为100%。
实施例2
收集191例检测样本类型为卵巢癌患者的FFPE样本和对照血液样本。后采用厦门艾德生物医药科技股份有限公司同源重组缺陷(HRD)检测试剂盒进行文库构建和捕获,并在Illumina Novaseq测序仪上进行测序,后采用实施例1中已训练好的基因组瘢痕模型计算待测样本GSS。
统计GSS高分组和HRR突变人群的关系,如下表
通过超几何分布检验,GSS高分组显著富集HRR相关基因突变的患者,P=0.0003。此外,相比于HRR相关基因,实施例1获得的基因组瘢痕模型的GSS可以富集更多基因组不稳患者。
实施例3
收集44例检测样本类型为卵巢癌患者的FFPE样本和对照血液样本,这些患者术后首次治疗方案为铂类化疗。后采用厦门艾德生物医药科技股份有限公司同源重组缺陷(HRD)检测试剂盒进行文库构建和捕获,并在Illumina Novaseq测序仪上进行测序,后采用实施例1中已训练好的基因组瘢痕模型计算待测样本GSS。
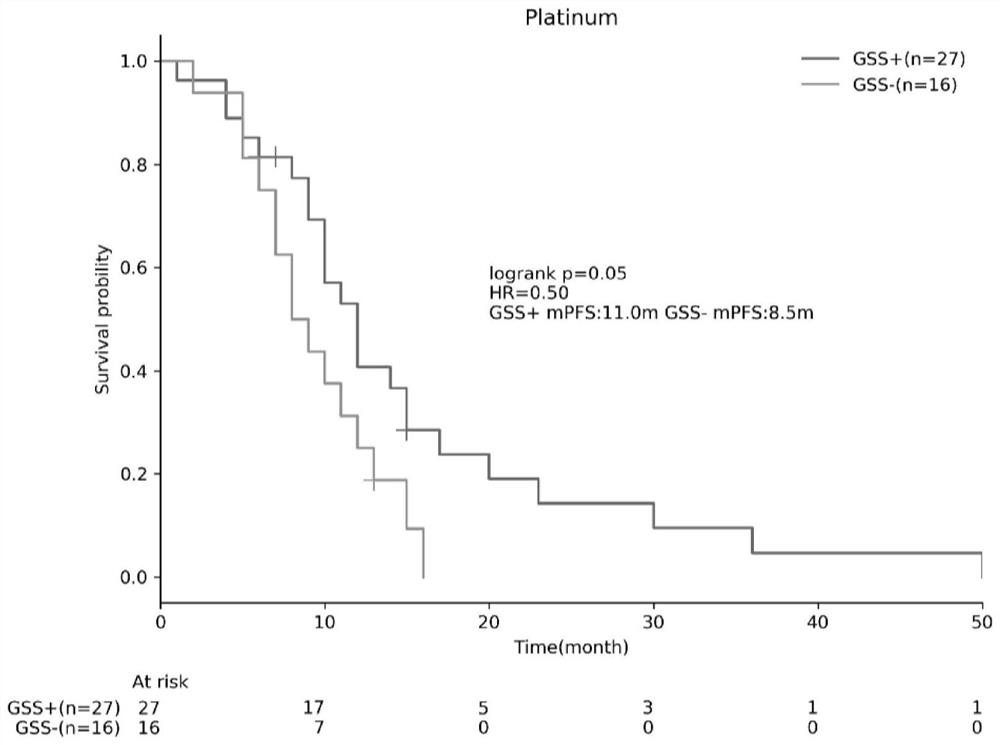
通过比较GSS高分组和GSS低分组患者的无进展生存期(progression-freesurvival,PFS),评估GSS富集铂类用药敏感人群的作用。结果如图1所示。如图所示,GSS状态即GSS高分(GSS+)和GSS低分(GSS-)分组,患者无进展生存期有显著差异,GSS高分组中患者采用铂类治疗无进展生存期显著较长(P=0.05),其中GSS高分组患者中位PFS=11个月,GSS低分组患者中位PFS=8.5个月。
实施例4
收集14和20例检测样本类型为卵巢癌患者的FFPE样本和对照血液样本,这些患者分别接受一线维持治疗和后线治疗方案为PARPi。后采用厦门艾德生物医药科技股份有限公司同源重组缺陷(HRD)检测试剂盒进行文库构建和捕获,并在Illumina Novaseq测序仪上进行测序,后采用实施例1中已训练好的基因组瘢痕模型计算待测样本GSS。
在接受PARPi一线维持治疗的卵巢癌患者中,如图2所示,GSS高分(GSS+)和GSS低分(GSS-)分组,患者无进展生存期有显著差异,GSS高分组中患者采用PARPi治疗无进展生存期显著较长(P=0.03),其中GSS高分组患者中位PFS=10.5个月,GSS低分组患者中位PFS=7个月
在PARPi后线治疗卵巢癌患者中,GSS高分组中客观缓解率(Objective ResponseRate,ORR)为38.5%(5/13),相对于HRD高分组(35.7%)和BRCA突变组(33.3%)中的客观缓解率最高,而GSS低分组中ORR为14.3%(1/7),相对于HRD低分组(16.7%)和BRCA野生型组(21.4%)中的客观缓解率最低,结果如图3所示,即实施例1获得的基因组瘢痕模型的GSS可富集PARPi类用药敏感人群。
以上所述,仅为本发明的较佳实施例而已,故不能依此限定本发明实施的范围,即依本发明专利范围及说明书内容所作的等效变化与修饰,皆应仍属本发明涵盖的范围内。
- 一种基因组瘢痕模型的建立方法
- 一种基因组瘢痕模型的建立方法
