烃的生产
文献发布时间:2023-06-19 09:44:49
本申请要求于2018年4月20日提交的英国专利申请1806483.2的优先权,本申请通过引用上述英国专利申请的全文并入本发明。
技术领域
本发明涉及烃的酶促生产方法以及适用于所述方法的酶、构建体和细胞。具体地,本发明涉及从生物质原料酶促生产轻链烷烃,例如丁烷、丙烷和异丁烷的材料和方法。
背景技术
对经济上可行的微生物生物燃料的深入研究反映了全球对减少碳排放、减少化石燃料供应的担忧,以及开发可再生和可持续的替代性能源战略的必要性[1,2]。对能源安全和气候变化的担忧,推动了限制温室气体的排放、加大废弃生物材料回收利用的政府决策,并驱动以生态为基础的经济发展[3,4]。这促进了最近对利用多种微生物宿主进行的生物燃料生产(如生物酒精)的广泛研究[5]。
当前交通业的化石燃料(例如无铅汽油)包含30多种脂肪族和芳族烃类,主要是戊烷、丙烷和壬烷[6]。化石燃料的燃烧是一种主要的污染源,其导致温室气体排放、酸雨和气候变化。相比而言,丙烷是一种高效、清洁的燃料,碳排放量低;与原油相比,其一氧化碳和烃的排放量显著减少。丙烷是液化石油气(LPG)的主要成分,来源于天然气或石油精炼[7]。它是全球第三大广泛使用的交通燃料(每年2000万吨),也是家庭供热、能源和环境友好型制冷剂和气溶胶推进剂的常见来源[7,8]。它与其他替代性气体燃料相比具有优势,因为其易于与碳氢化合污染物分离,并且液化和存储所需的能源较低[8]。
由于缺乏天然可以代谢产生丙烷的已知微生物,丙烷的生物制备途径受到限制。然而,从海洋原绿球藻(Procholorococcus marinus)st.MIT9313中发现的蓝藻脂肪醛去甲酰加氧酶(cyanobacterial aldehyde deformylating enzyme,ADO)展现出了微生物制备烃的可能性[9]。该酶催化铁氧还蛋白和主要长链脂肪醛的氧依赖性脱羰基化,生成烷烃和甲酸盐(图1A)[9]。利用基于结构的对ADO底物接入通道的工程改造,产生了具备丙烷生产能力增强的A134F变体[10]。现有技术公开了使用ADO在大肠杆菌中生产微生物来源丙烷的合成生物学途径,其中,通过脂肪酸生物合成[11]、反向β-氧化[6]、缬氨酸生物合成(异丁醛前体)[7]或梭菌丁醇通路[8]制备前体丁醛。然而,上述方法都存在ADO转换率极低的瓶颈(约3-5h
最近描述了一种新型的光能酶,其催化游离脂肪酸的蓝光依赖性脱羧生成正构烷烃或烯烃[13]。来自变异小球藻(Chlorella variabilis)NC64A的这种脂肪酸光脱羧酶或光烷烃合成酶(PAS或FAP)是葡萄糖-甲醇-胆碱氧化还原酶家族的成员,并包含结合的光激发FAD辅因子,其量子产率>80%(0.86±0.13s
本文记载了丙烷的体内生物生产工艺,该工艺使用了变异小球藻(C.variabilis)的脂肪酸脱羧酶(CvPAS或CvFAP)。我们对底物接入通道进行定点突变,得到了突变型G462V(G402V成熟序列),其对丁酸的催化性能有所提高。本发明研究是在嗜盐单胞菌(Halomonas)菌株中进行的,该菌株是已知的稳定低成本生产聚羟基脂肪酸酯生物塑料的宿主[14,15]。这种生物能够在高pH和高盐浓度的非无菌环境中生长,而没有明显的微生物污染[14]。据我们所知,这是首次报道将嗜盐单胞菌用作气态生物燃料生产宿主的例子,并且将其与增强型的生产丙烷的CvPAS变体相结合是实现商业可行化生物丙烷生产的关键进展。
发明概要
本发明公开的第一方面是一种脂肪酸脱羧酶,所述脂肪酸脱羧酶包括与SEQ IDNO:1或2的参照序列具有至少40%的序列同一性的序列,并且在对应于SEQ ID NO:1的G462位具有氨基酸取代。
在一些实施方案中,所述脂肪酸脱羧酶包含选自G462V、G462F、G462I、G462L、G462A、G462Y、G462C、G462H、G462N、G462Q和G462W的氨基酸取代。任选地,所述取代是G462V或G462I。
在本文公开的一些实施方案中,所述脂肪酸脱羧酶还包含至少一种个对应于以下位点上的氨基酸取代:SEQ ID NO:1的V453、SEQ ID NO:1的G455、SEQ ID NO:1的A457、SEQID NO:1的Y466或SEQ ID NO:1的T484。
在一些实施方案中,所述脂肪酸脱羧酶包含选自V453F、V453I、V453L、V453W、V453E和V453A的氨基酸取代。所述取代可以优选为V453F、V453I、V453L和V453W中的一个。任选地,所述取代可以为V453I。
在一些实施方案中,所述脂肪酸脱羧酶包含选自G455F、G455I、G455V、G455W、G455L、G455E和G455A的氨基酸取代。所述取代可以优选为G455F、G455I、G455V、G455W和G455L中的一个。
在一些实施方案中,所述脂肪酸脱羧酶包含选自A457F、A457I、A457L、A457V、A457W和A457E的氨基酸取代。所述取代可以优选为A457F、A457I、A457L和457V中的一个。
在一些实施方案中,所述脂肪酸脱羧酶包含选自Y466W、Y466F、Y466I、Y466V、Y466L、Y466E和Y466A的氨基酸取代。所述取代可以优选为Y466W。
在一些实施方案中,所述脂肪酸脱羧酶包含选自以下的一个或多个氨基酸取代:T484A、T484E、T484I、T484L、T484F、T484V和T484W。所述取代可以优选为T484A、T484E、T484I和T484L中的一个。
在一实施方案中,所述脂肪酸脱羧酶包含氨基酸取代G462V和V453I。
在一实施方案中,所述脂肪酸脱羧酶包含氨基酸取代G462I和V453I。
在一些实施方案中,所述脂肪酸脱羧酶包含与SEQ ID NO:1的425-429位残基共有的氨基酸序列,其中所述共有序列包含与SEQ ID NO:3具有至少70%的序列同一性的序列。
在一些实施方案中,所述脂肪酸脱羧酶包含在对应于SEQ ID NO:1的398-575位残基具有活性位点的氨基酸序列,所述活性位点与选自SEQ ID NO:4-7的一个或多个序列具有至少70%的同一性。
在一些实施方案中,所述脂肪酸脱羧酶包含与SEQ ID NO:1或2具有至少70%序列同一性的氨基酸序列。
在一些实施方案中,所述脂肪酸脱羧酶的底物为链长等于或小于8个碳的脂肪酸。优选地,所述脂肪酸脱羧酶的底物为链长2-5个碳的脂肪酸。
在一些实施方案中,在对应于SEQ ID NO:1的G462位的氨基酸取代在空间上阻碍链长等于或大于8个碳的脂肪酸与所述脂肪酸脱羧酶结合。
在一些实施方案中,与在SEQ ID NO:1的G462位无取代的脂肪酸脱羧酶相比,所述脂肪酸脱羧酶在以C
在一些实施方案中,所述脂肪酸脱羧酶与包含SEQ ID NO:1的野生型脂肪酸脱羧酶相比,具有更高的丁酸至丙烷的脱羧酶活性。
在一些实施方案中,所述脂肪酸脱羧酶具有光依赖性脱羧酶活性。在一些实施方案中,所述脂肪酸脱羧酶具有蓝光依赖性脱羧酶活性。在一些实施方案中,所述活性依赖于波长为400-520nm的光。
本发明公开的另一方面为一种脂肪酸脱羧酶,所述脂肪酸脱羧酶包含与SEQ IDNO:1或2的参照序列具有至少40%的序列同一性的序列,并且包含至少一个对应于以下位点上的氨基酸取代:SEQ ID NO:1的V453、SEQ ID NO:1的G455、SEQ ID NO:1的A457、SEQ IDNO:1的Y466或SEQ ID NO:1的T484。
在一些实施方案中,所述脂肪酸脱羧酶包含选自V453F、V453I、V453L、V453W、V453E和V453A的氨基酸取代。所述取代可以优选为V453F、V453I、V453L和V453W中的一个。任选地,所述取代可以为V453I。
在一些实施方案中,所述脂肪酸脱羧酶包含选自G455F、G455I、G455V、G455W、G455L、G455E和G455A的氨基酸取代。所述取代可以优选为G455F、G455I、G455V、G455W和G455L中的一个。
在一些实施方案中,所述脂肪酸脱羧酶包含选自A457F、A457I、A457L、A457V、A457W和A457E的氨基酸取代。所述取代可以优选为A457F、A457I、A457L和457V中的一个。
在一些实施方案中,所述脂肪酸脱羧酶包含选自Y466W、Y466F、Y466I、Y466V、Y466L、Y466E和Y466A的氨基酸取代。所述取代可以优选为Y466W。
在一些实施方案中,所述脂肪酸脱羧酶包含选自以下的一个或多个氨基酸取代:T484A、T484E、T484I、T484L、T484F、T484V和T484W。所述取代可以优选为T484A、T484E、T484I和T484L中的一个。
本公开还提供了包含本文所述脂肪酸脱羧酶的细胞。在一些实施方案中,所述细胞是细菌细胞。在一个优选的实施方案中,所述细胞是嗜盐单胞菌细胞。
本文还提供了一种方法,其包括使用脂肪酸脱羧酶催化C
在一些实施方案中,所述C
在一些实施方案中,所述脂肪酸脱羧酶为如本文所述的脂肪酸脱羧酶或其片段、变体或同源物。在一些实施方案中,所述脂肪酸脱羧酶包含在如权利要求11所述的细胞内。在一些实施方案中,所述方法包括使C
在一些实施方案中,所述方法包括回收丙烷的步骤。
本文还提供了一种生产C
在一些实施方案中,所述C
在一些实施方案中,所述生产C
在一些实施方案中,所述脂肪酸脱羧酶为如本文所述的脂肪酸脱羧酶或其片段、变体或同源物。在一些实施方案中,所述脂肪酸脱羧酶包含在细胞内,所述细胞包含编码如本文所述的脂肪酸脱羧酶的核酸。在一些实施方案中,所述方法包括使C
在一些实施方案中,所述方法包括回收所述烷烃/烯烃的步骤。
本发明还提供了一种脂肪酸脱羧酶在本文所述的生产烷烃/烯烃方法中的用途。
在另一方面,本公开提供了一种方法,所述方法包括:
使用醛脱氢酶催化丁酰-CoA转化为丁醛。
本公开还提供了一种方法,所述方法包括:使用脂肪醛去甲酰加氧酶催化丁醛转化为丙烷。
本公开还提供了一种方法,所述方法包括:
使用醛脱氢酶催化丁酰-CoA转化为丁醛,再使用脂肪醛去甲酰加氧酶催化丁醛转化为丙烷。
本公开还提供了一种生产丙烷的方法,所述方法包括使用醛脱氢酶催化丁酰-CoA转化为丁醛的步骤。
本公开还提供了一种生产丙烷的方法,所述方法包括:
a.使用醛脱氢酶催化丁酰-CoA转化为丁醛,和
b.使用脂肪醛去甲酰加氧酶催化丁醛转化为丙烷。
在一些实施方案中,所述方法包括回收丙烷的步骤。
在一些实施方案中,所述醛脱氢酶为来自拜氏梭菌的醛脱氢酶,或其片段、变体或同源物。
在一些实施方案中,所述醛脱氢酶包含与SEQ ID NO:20的具有至少40%的序列同一性的序列。
在一些实施方案中,所述醛脱氢酶包含与SEQ ID NO:21的具有至少70%的序列同一性的序列。
在一些实施方案中,所述方法包括在使用醛脱氢酶催化丁酰-CoA转化为丁醛之前,使用丁酸-乙酰乙酰CoA转移酶催化丁酸转化为丁酰-CoA的步骤。
在一些实施方案中,所述丁酸-乙酰乙酰CoA转移酶包含与SEQ ID NO:22的具有至少70%的序列同一性的序列。
本文还提供了包含编码醛脱氢酶和脂肪醛去甲酰加氧酶的核酸序列的多核苷酸序列。
本文还提供了包含编码异源醛脱氢酶的核酸的细胞。在一些实施方案中,所述细胞包含编码脂肪醛去甲酰加氧酶的核酸。
在一些实施方案中,所述细胞包含编码醛脱氢酶和脂肪醛去甲酰加氧酶的核酸。在一些实施方案中,所述细胞包含表达载体(如本文所定义),所述表达载体包含编码醛脱氢酶的核酸和编码脂肪醛去甲酰加氧酶的核酸。在一些实施方案中,所述表达包含与编码醛脱氢酶的核酸和/或编码脂肪醛去甲酰加氧酶的核酸可操作地连接的一个或多个启动子。
在一些实施方案中,所述醛脱氢酶包含与SEQ ID NO:20具有至少70%的序列同一性的序列。
在一些实施方案中,所述醛脱氢酶包含与SEQ ID NO:21具有至少70%的序列同一性的序列。
在一些实施方案中,所述方法包括以下步骤:使丁酰-CoA或包含丁酰-CoA的组合物与包含编码异源醛脱氢酶核酸的细胞或包含编码异源醛脱氢酶和脂肪醛去甲酰加氧酶核酸的细胞接触,前述细胞如本文所述。
本文还提供了醛脱氢酶在本文所述的生产丁烷方法中的用途。
本发明包括本发明所描述的方面和优选特征的组合,除非该组合是明显不允许的或明确应避免的。
序列
附图说明
现在将结合附图讨论示例了本发明原理的实施方案和实验:
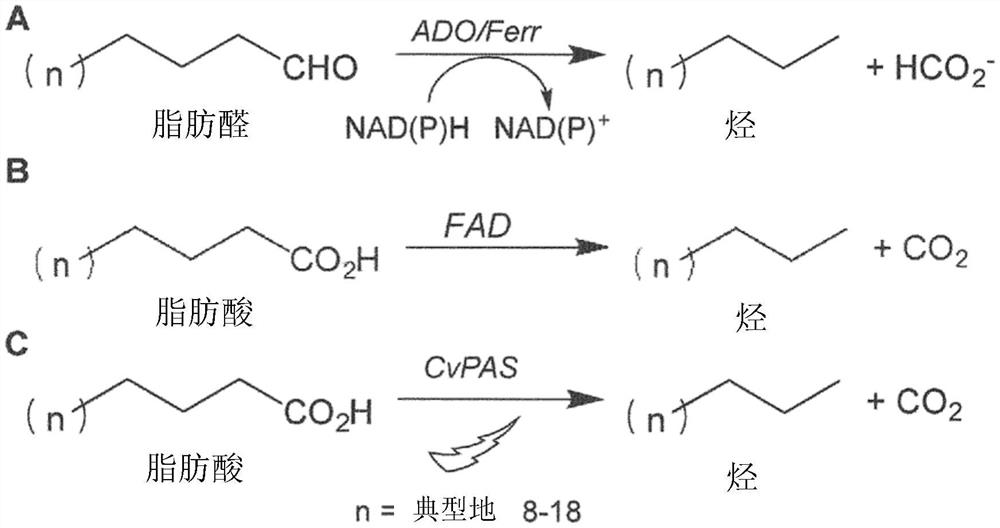
图1.酶促形成烷烃的途径。(A)利用海洋原绿球藻(P.marinus)st.MIT9313的脂肪醛去甲酰加氧酶(ADO)催化脂肪醛的NAD(P)H依赖性脱羧。(B)脂肪酸脱羧酶(FAD)催化脂肪酸脱羧的常规方案(C)利用变异小球藻NC64A的光依赖性脂肪酸脱羧酶光烷烃合成酶(CvPAS)使脂肪酸脱羧。
图2.棕榈酸结合的CvPAS
图3.脂肪酸脱羧酶CvPAS G462V的示例性反应途径:(A)CvPAS G462V催化丁酸转化为丙烷;可选步骤为利用YciA将丁酰-CoA转化为丁酸的预处理。(B)CvPAS G462V催化戊酸转化为丁烷。(C)CvPAS G462V催化异戊酸转化为异丁烷。
图4.不同物种的脂肪酸脱羧酶的分子比对:(从上到下)长尾团藻Volvox carterif.nagariensis(SEQ ID NO:19)、莱茵衣藻Chlamydomonas reinhardtii(SEQ ID NO:12)、胸状盘藻Gonium pectorale(SEQ ID NO:14)、变异小球藻Chlorella variabilis(Cva-APS;SEQ ID NO:1)、球隐孢子虫Coccomyxa subellipsoidea(SEQ ID NO:13)、褐潮藻Aureococcus anophagefferens(SEQ ID NO:8)、三角褐指藻Phaeodactylum tricornutum(SEQ ID NO:15)、嗜铬绿藻Chrysochromulina spp.(SEQ ID NO:10)、赫氏圆石藻Emiliania huxleyi(SEQ ID NO:16)、皱波角藻Chondrus crispus(SEQ ID NO:9)和红藻Cyanidioschyzon merolae(SEQ ID NO:11)。粗体和下划线表示SEQ ID NO:1的G462位相对应的位点。下划线表示与SEQ ID NO:1的V453相对应的位点。双下划线表示与SEQ ID NO:1的G455相对应的位点。点划线表示与SEQ ID NO:1的A457相对应的位点。粗体和点划线表示与SEQ ID NO:1的Y466相对应的位点。粗体和双下划线表示与SEQ ID NO:1的T484相对应的位点。点下划线表示与共有序列SEQ ID NO:3相对应的位点。高亮粗体表示与活性位点序列SEQ ID NO:4相对应的位点。
图5.过表达野生型CvPAS和G462V变体CvPAS的大肠杆菌细胞裂解液的SDS PAGE分析。方框表示CvPAS表达条带(63kDa)。
图6.图表显示了在恒定光照条件下丁酸盐浓度对野生型CvPAS和G462V变体CvPAS活性的影响。反应体系(200μL)为在密封的玻璃气相色谱瓶中的无细胞裂解液(180μL)和丁酸(0.36-4.5mM)。在蓝色LED(455nm)照射下,将反应液在30℃和180rpm下孵育24小时。使用Micro GC(100ms进样)分析液上气体的丙烷含量。
图7.通过丁醛中间体,利用醛脱氢酶(BALDH)和脂肪醛去甲酰加氧酶(ADO,及电子转移伴侣蛋白铁:氧还蛋白)从丁酰CoA生成丙烷的反应途径。利用乙酰乙酸丁酸酯CoA转移酶(CoAT)的作用,该过程也可以从丁酸起始。
图8.通过YciA/CAR/SFP/ADO的作用,经丁酸和丁醛,从葡萄糖生成丙烷的反应途径(Menon等,2015年)[8]。
图9.通过CvPAS的作用,经丁酸,从葡萄糖生成丙烷的反应途径。
图10.通过BALDH/ADO的作用,经丁醛,从葡萄糖生成丙烷的反应途径。
图11.生物液化石油气方案的图解概述,将新的光生物反应器设计与现有废料、运输和供应的基础设施相结合。
图12.图表显示了CvFAP变体在体外可比的丙烷生产。培养物(20mL)在含有卡那霉素(30μg/mL)的LB培养基中37℃培养至OD
图13A和B.a)野生型CvFAP和b)CvFAP的G462V变体的活性位点中的丁酸盐和棕榈酸盐的图解模型。结晶测定棕榈酸在野生型酶中的位置(PDB:5NCC)。其余配体的位置由Autodock Vina确定[16],使用SwissPDBViewer 4.10进行定点突变(G462V)[48]。在两个图中,蛋白质均显示为带有二级结构阴影的卡通。虚线显示了棕榈酸盐和野生型酶之间的氢键。高亮虚线显示了建模中配体-G/V462残基距离。
图14A和B.图表显示了在大肠杆菌BL21(DE3)ΔyqhD/ΔyjgB中,野生型CvFAP和CvFAP变体内产生的气态烃含量。a)CvFAP-pETM11变体
图15A和B.代谢工程改造的图解说明,其通过体内丁酸产量的上调使CO
图16A-C.嗜盐单胞菌XV12表达CvFAP构建体生产丙烷。a)基因表达构建体的示意图。b)图表显示了嗜盐单胞菌构建体的丙烷生产。c)图表显示丁酸浓度对pHal2
图17A和B.在平板光生物反应器中发酵表达pHal2-FAP
图18.显微照片和表格显示了CvFAP
图19.图表显示了丁酸浓度对无磷酸盐缓冲剂情况下大肠杆菌中体内生产丙烷的影响。将培养物(20-100mL)在含有卡那霉素(50μg/mL)和过夜起始培养液(1%体积;相同培养基)的LB培养基中于37℃和180rpm下培养6h。用IPTG(0.1mM)诱导重组蛋白表达,并在培养物中添加丁酸(1-25mM)。将一式三份的培养物(5mL)密封在20mL玻璃小瓶中,在30℃下以200rpm孵育16-18h,并用蓝色LED面板连续照射。使用配有Al
图20A-D。图表显示了a)pHal1-FAP
图21.图表显示在平板光生物反应器表达pHal2-FAP
图22.表S1:推定的FAP同源物在大肠杆菌中的表达和活性。
图23.表S2:在大肠杆菌中表达的CvFAP
图24.表S3:CvPAS野生型和其变体与丁酸盐和棕榈酸盐的分子对接模拟。
图25.表S4:在大肠杆菌BL21(DE3)中CvFAP
图26.表S5:在大肠杆菌中表达的pET21b中的N-His
图27.表S6:在短链有机酸的存在下,在大肠杆菌中CvFAP变体在体内产生气态烃的产量。
图28.表S7:丁酸/戊酸混合物对大肠杆菌BL21(DE3)ΔyqhD/ΔyjgB中野生型CvFAP产生气态烃产量的影响。
图29.表S8:嗜盐单胞菌XV12中CvFAP
图30.表S9:大肠杆菌和嗜盐单胞菌中的寡核苷酸和其他DNA序列。
图31.表S11:用于DNA组装的前连接子和后连接子。
图32.表S12:前连接子和后连接子。
发明详述
以下将结合附图讨论本发明的各方面和实施方案,在这基础之上的进一步的方面和实施方案对于本领域技术人员而言是显而易见的。本文中提及的所有文件均通过引用并入本文。
本发明是基于发明人对一种酶的鉴定,所述酶能够以短链脂肪酸和辅酶A活化的脂肪酸为原料产生短链烃,如丁烷、异丁烷和丙烷。由于短链脂肪酸通常产生于生物质的微生物分解过程中,因此本发明提供了一种能够以容易获得的原料生产生物燃料短链烃的方法。
如本文所用,蛋白质的“片段”、“变体”或“同源物”可以任选地表征为与参照蛋白的氨基酸序列具有至少50%,优选为60%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%之一的氨基酸序列同一性。参照蛋白的片段、变体、同种型和同源物可以通过执行参照蛋白之功能的能力来表征。
本领域技术人员可通过各种已知方式进行成对和多序列比对,以确定两个或多个氨基酸或核酸序列之间的同一性百分比。例如,使用诸如ClustalOmega((
“片段”通常是指参照蛋白的一部分。“变体”通常是指一类蛋白质,该类蛋白质的序列相对于参照蛋白的氨基酸序列具有一个或多个氨基酸的取代、插入、缺失或其他修饰,但其保留与参照蛋白的氨基酸序列相当程度的序列同一性(例如,至少60%)。“同种型”通常是指由与表达该参照蛋白的物种相同的物种所表达的参照蛋白变体。“同源物”通常是指由与表达参照蛋白的物种不同的物种产生的参照蛋白变体。
参照蛋白的“片段”可以具有任何长度(以氨基酸数量计),但应当任选地为参照蛋白(即片段所源自的蛋白)的长度的至少25%,其最大长度可以是参照蛋白长度的50%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%之一。
多肽片段的最小长度可以是10、20、30、40、50、60、70、80、90或100个氨基酸之一,最大长度可以是15、20、25、30、40、50、100、110、120或130个氨基酸之一。
如本文所用,“烃”指包含由氢和碳组成的主链的有机化合物。烃包括烷烃、烯烃、芳烃、环烷烃和炔烃。烷烃是通式为C
如本文所用,“CX”烃或脂肪酸是总碳数为X的烃或脂肪酸。例如,丁烷和异丁烷都是C4烃。“CX”碳是第X位的碳。例如,硬脂酸的C5碳是第5位的碳。
如本文所用,“链长”是指最长连续碳链的碳数。例如,正戊烷的链长为5,正丁烷的链长为4,而异丁烷和丙烷的链长为3。
如本文所用,“脂肪酸”是指具有脂肪族烃链的羧酸(-COOH)分子。“脂肪酸”包括脂肪酸的盐和其离子。例如,脂肪酸“丁酸”包括游离丁酸以及丁酸盐等。除非另有说明,否则本文所用的“短链”脂肪酸是指碳链长度为2-8的脂肪酸。所述短链脂肪酸的链长可以是2、3、4、5、6、7或8个碳,例如长度为2-7、2-6、2-5、2-4、2-3或2个碳原子。“长链”脂肪酸是指链长长于短链脂肪酸的脂肪酸。例如,长链脂肪酸可以指链长为13或更长的脂肪酸,其链长优选为13-21、13-20、13-19、13-18、13-17、13-16、13-15、13-14、14-21、14-20、14-19、14-18、14-17、14-16、14-15、14、15-21、15-20、15-19、15-18、15-17、15-16、15、16-21、16-20、16-19、16-18、16-17、16、17-21、17-20、17-19、17-18、17、18-21、18-20、18-19、18、19-21、19-20、19、20-21、20或21个碳。
本文所述“脂肪酸脱羧酶”是指具有脂肪酸脱羧酶活性的酶,即能够催化正脂肪酸脱去羧基生成正构烷烃或-烯烃,特别是直接脱去而无需经醛中间体,也不会引入末端不饱和键。该反应方案如图1C所示。脂肪酸脱羧酶的活性可以使用本领域技术人员可用的任何方法来测量。
示例性的脂肪酸脱羧酶由SEQ ID NO:1、2和8-19之一的氨基酸序列的多肽编码。特别地,SEQ ID NO:1编码变异小球藻NC64A(CvPAS)的光烷烃合成酶,该酶能够使游离脂肪酸蓝光依赖性地脱羧成正构烷烃或烯烃(图1C)[13]。CvPAS是葡萄糖-甲醇-胆碱氧化还原酶家族的成员,包含结合的光激发性FAD辅因子,量子产率>80%(0.86±0.13s
654个氨基酸的SEQ ID NO:1对应于CvPAS的全长多肽序列(UniProt:A0A248QE08)。该序列如下所示:
SEQ ID NO:1的G462位以粗体突出显示,并用下划线标出。
其天然序列在N末端包含由61个氨基酸组成的叶绿体靶向序列(虚线下划线),其在转运过程中被切除。该序列被去除并由甲硫氨酸残基代替,得到594个氨基酸的SEQ IDNO:2:
对应于SEQ ID NO:1的G462位的SEQ ID NO:2的G402位以粗体突出并用下划线标出。
在本说明书中,“SEQ ID NO:1的G462位”也指“SEQ ID NO:2的G402位”。“SEQ IDNO:1的G462位”应被视为等同于“SEQ ID NO:2的G402位”并且两者可以互换。
类似地,SEQ ID NO:1的V453位、G455位、A457位、Y466位和T484位可以互换地描述为SEQ ID NO:2的V393位、G395位、A397位、Y406位和T424位。
SEQ ID NO:8-19分别为在褐潮藻Aureococcus anophagefferens(SEQ ID NO:8)、皱波角藻Chondrus crispus(SEQ ID NO:9)、嗜铬绿藻Chrysochromulina spp.(SEQ IDNO:10)、红藻Cyanidioschyzon merolae(SEQ ID NO:11)、莱茵衣藻Chlamydomonasreinhardtii(SEQ ID NO:12)、球隐孢子虫Coccomyxa subellipsoidea(SEQ ID NO:13)、胸状盘藻Gonium pectorale(SEQ ID NO:14)、三角褐指藻Phaeodactylum tricornutum(SEQID NO:15)、赫氏圆石藻Emiliania huxleyi(SEQ ID NO:16和17)、甘南芥Gannanchloropsis gaditana(SEQ ID NO:18)和长尾团藻Volvox carteri f.nagariensi(SEQ ID NO:19)中发现的SEQ ID NO:1的同源物。技术人员通过序列比对能够确定哪些残基对应于SEQ ID NO:1的G462位。这些残基在图4中高亮显示。
在本说明书中,“脂肪酸脱羧酶”是指来自任何物种的脂肪酸脱羧酶,并且包括来自任何物种的脂肪酸脱羧酶的同种型、片段、变体或同源物同源物。所述同源物包括直系同源物。在一些实施方案中,所述脂肪酸脱羧酶是原核脂肪酸脱羧酶,例如细菌脂肪酸脱羧酶。在一些实施方案中,所述脂肪酸脱羧酶来自或衍生自微藻,例如在团藻属(Volvox)、衣藻属(Chlamydomonas)、盘藻属(Gonium)、小球藻属(Chlorella)、胶球藻属(Coccomyxa)、褐潮藻属(Aureococcus)、褐指藻属(Phaeodactylum)、金色藻属(Chrysochromulina)、圆石藻属(Emiliania)、角叉菜属(Chondrus)或红藻属(Cyanidioschyzon)中。示例性的脂肪酸脱羧酶如在长尾团藻、莱茵衣藻、胸状盘藻、变异小球藻、球隐孢子虫、褐潮藻、三角褐指藻、嗜铬绿藻、赫氏圆石藻、皱波角藻或红藻Cyanidioschyzon merolae中发现的脂肪酸脱羧酶。
本文提供的脂肪酸脱羧酶为野生型脂肪酸脱羧酶的变体,其氨基酸序列与天然存在的野生型酶不同。如此,它们可以被称为“突变的”、“非天然存在的”或“修饰的”。具有修饰对象酶(例如SEQ ID NO:1或SEQ ID NO:2的序列)作用以产生另一个野生型酶序列(如SEQ ID NO:8-19中任一同源物,或其缺乏叶绿体靶向序列的成熟氨基酸序列)的任何氨基酸取代,均任选地排除出本公开,并且可以任选地从本发明要求保护的范围中排除。
脂肪酸脱羧酶的片段、变体、同种型和同源物同源物可以任选地通过催化脂肪酸转化为烷烃或烯烃的能力进行表征,特别是以将短链脂肪酸转化为短链烯烃或烷烃的能力进行表征。
可以根据与参照脂肪酸脱羧酶的相似性来描述本发明各方面所述的脂肪酸脱羧酶。例如,所述脂肪酸脱羧酶可与参照序列具有至少40%的序列同一性。本发明各方面所述的脂肪酸脱羧酶可包含与参照序列至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的序列同一性。所述参照序列可以是任何脂肪酸脱羧酶,并且可以包含SEQ ID NO:1、SEQ ID NO:2或SEQ ID NO:8-19中任何一氨基酸序列。在优选的实施方案中,所述参照序列选自SEQ ID NO:1或SEQ ID NO:2。与参照脂肪酸脱羧酶相比,本发明所述的脂肪酸脱羧酶可以具有改善的脱羧酶活性和/或更高的产率。
在一些实施方案中,相对于参照脂肪酸脱羧酶的氨基酸序列,所述脂肪酸脱羧酶包含一个或多个(例如,具有SEQ ID NO:1或2氨基酸序列的脂肪酸脱羧酶)的氨基酸取代。在一些实施方案中,相对于参照脂肪酸脱羧酶的氨基酸序列,所述脂肪酸脱羧酶包含例如1、2、3、4、5、6、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20个或更多氨基酸的取代。在一些实施方案中,相对于参照脂肪酸脱羧酶的氨基酸序列,所述脂肪酸脱羧酶包含例如1-3、1-5、1-10、1-15或1-20个氨基酸取代。
一般认为,野生型脂肪酸脱羧酶对棕榈酸(C16)的更高偏好是因短疏水链的稳定性,因为其包裹在弯曲的、暴露于溶剂的狭窄底物结合通道周围(图2)。因此,脂肪酸脱羧酶对短链脂肪酸的偏好可以通过下述工程改造实现:在等同于G462的位点引入取代,以空间位阻进入溶剂暴露的底物结合通道。
如上所述,本发明所述的脂肪酸脱羧酶在对应于SEQ ID NO:1的G462位上包含氨基酸取代。可以通过用侧链较大的氨基酸取代G462侧链相对较小的甘氨酸来引入空间位阻。可以用在野生型序列中该位点上未发现的任何其他氨基酸取代SEQ ID NO:1的G462位。本公开中包括的方法中,是用在本文所述野生型序列中该位点上未发现的任何其他氨基酸取代SEQ ID NO:1的G462位。
优选地,所述取代用侧链可以空间位阻进入溶剂暴露的底物结合通道的氨基酸,替换对应于SEQ ID NO:1的G462位的氨基酸,例如在物理上阻塞通道以阻绝本文所述的长链脂肪酸的结合。可以用具有大的、体积大的、不带电荷的和/或非极性的侧链的氨基酸进行取代,以引入空间位阻。例如,可以用具有大的、体积大的、不带电荷的和/或非极性侧链的氨基酸取代SEQ ID NO:1的G462位侧链(H)相对小的甘氨酸来引入空间位阻。
在一些实施方案中,对应于SEQ ID NO:1的G462位氨基酸被选自具有大侧链的V、F、I、L、A、Y、C、H、N、Q和W残基取代。在一些实施方案中,对应于SEQ ID NO:1的G462位氨基酸被选自具有不带电荷侧链的V、F、I、L、A、Y、C、N、Q和W残基取代。在一些实施方案中,对应于SEQ ID NO:1的G462位氨基酸被选自具有非极性侧链的V、F、I、L、A和W残基取代。
在一些实施方案中,对应于SEQ ID NO:1的G462位的氨基酸取代是G462V。在一些实施方案中,所述氨基酸取代是G462F。在一些实施方案中,所述氨基酸取代是G462I。在一些实施方案中,所述氨基酸取代是G462L。在一些实施方案中,所述氨基酸取代是G462A。在一些实施方案中,所述氨基酸取代是和G462W。在一些实施方案中,所述氨基酸取代是G462Y。在一些实施方案中,所述氨基酸取代是G462C。在一些实施方案中,所述氨基酸取代是G462H。在一些实施方案中,所述氨基酸取代是G462N。在一些实施方案中,所述氨基酸取代是G462Q。
本领域技术人员能够很好地识别脂肪酸脱羧酶中与参照序列SEQ ID NO:1所示位点不同的对应位点。对对应位点的识别,可以通过例如将给定的脂肪酸脱羧酶氨基酸序列与SEQ ID NO:1的氨基酸序列进行比对。为此目的的序列比对可以通过本领域技术人员已知的各种方式来实现,例如,使用公开可获得的计算机软件,如ClustalOmega的(
在一些实施方案中,所述脂肪酸脱羧酶进一步包含下述共有序列:
G-X1-L-X2-X3-X4-X5-C-[D/E]-X6-G-[A/G]-F-X7-[K/R](SEQ ID NO:3),
其中X可以是任意氨基酸。
优选地,X1可以选自P、L和G构成的组。优选地,X2可以选自T和A构成的组。优选地,X3可以选自T、S和C构成的组。优选地,X4可以选自P、T和A构成的组。优选地,X5可以选自G和A构成的组。优选地,X6可以选自H、N和R构成的组。X7可以是疏水性氨基酸,特别是选自L、V、A和F构成的组。
在一些实施方案中,所述共有序列可以具有与SEQ ID NO:3至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的序列同一性。所述共有序列可包含0、1、2、3、4、5、6、7或8个相对于SEQ ID NO:3的取代、插入或缺失。在一个优选的实施方案中,所述共有序列可以包含0、1或2个相对于SEQ ID NO:3的取代、插入或缺失。
所述共有序列可以在对应于SEQ ID NO:1的425-429位残基的位置,或者可以在远离此位置最多20、最多15、最多10、最多5、最多3、最多5个、最多2个或最多1个残基范围内的位置。
在一些实施方案中,本发明所述的脂肪酸脱羧酶包含下述活性位点序列:
[I/L/K/D]X
其中X是任意氨基酸,并且[Z]是在对应于SEQ ID NO:1的G462位氨基酸取代的残基。在一个优选的实施方案中,所述活性位点序列包含SEQ ID NO:5或SEQ ID NO:6中的任一个,其中X是任意氨基酸,并且[Z]是在对应于SEQ ID NO:1的G462位氨基酸取代的残基。在一个具体的实施方案中,所述活性位点包含以下序列:
IAISDHIYNEKGQIRKRAIASYLLGGRGGLTSTGCDRGAFVRTAGQALPDLQVRFVPGMALDPD[Z]VSTYVRFAKFQSQGLKWPSGITMQLIACRPQSTGSVGLKSADPFAPPKLSPGYLTDKDGADLATLRKGIHWARDVARSSALSEYLDGELFPGSGVVSDDQIDEYIRRSIHSSN(SEQ ID NO:7)
其中[Z]是对应于SEQ ID NO:1的G462位氨基酸取代的残基。
所述活性位点序列可以在对应于SEQ ID NO:1的393-575位残基的位置,或者可以在远离此位置最多20、最多15、最多10、最多5、最多3、最多5个、最多2个或最多1个残基范围内的位置。
如本文所述,可以通过在对应于SEQ ID NO:1的V453位、G455位、A457位、Y466位或T484位中的一个或多个位点上进行氨基酸取代,以进一步修饰活性位点序列。
在一些实施方案中,所述脂肪酸脱羧酶包含活性位点,所述活性位点具有与选自SEQ ID NO:4-7的一个或多个共有序列具有至少40%的序列同一性,优选至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的序列同一性。
本文所述的脂肪酸脱羧酶能够催化脂肪酸转化为烷烃或烯烃,特别是短链脂肪酸转化为短链烯烃或烷烃。例如,本发明所述的脂肪酸脱羧酶能够催化丁酸转化为丙烷(图3A)、戊酸转化为丁烷(图3B)和/或异戊酸转化为异丁烷(图3B)中的一种、两种或全部三种。在一些实施方案中,本发明所述的脂肪酸脱羧酶能够催化丁酸转化为丙烷以及戊酸转化为丁烷。在一个优选的实施方案中,本发明所述的脂肪酸脱羧酶能够催化丁酸转化为丙烷。
本发明所述的脂肪酸脱羧酶接受脂肪酸作为底物。被接受为底物的脂肪酸是一种能够与脂肪酸脱羧酶结合并被催化而转化为烃的脂肪酸。此外,将本发明所述的脂肪酸脱羧酶被工程改造为以短链脂肪酸为底物。这种工程改造使得所述脂肪酸脱羧酶偏好性地产生短链烯烃/烷烃,包括挥发性的烯烃/烷烃如丁烷、异丁烷和丙烷。
在一些实施方案中,所述脂肪酸脱羧酶接受短链脂肪酸作为底物,例如C2、C3、C4、C5、C6、C7和/或C8脂肪酸。在一些实施方案中,所述脂肪酸脱羧酶接受C3-C5脂肪酸。在一些实施方案中,所述脂肪酸脱羧酶接受C 3和/或C4脂肪酸。在一些实施方案中,所述脂肪酸脱羧酶可以接受链长为10个碳或以下、9个或以下、8个或以下、7个或以下、6个或以下、5个或以下、4个或以下、3个或以下或2个或以下的脂肪酸作为底物。在一些实施方案中,所述脂肪酸脱羧酶能够接受C1-C8、C1-C7、C1-C6、C1-C5、C1-C4或C1-C3脂肪酸。在一些实施方案中,所述脂肪酸脱羧酶能够接受C2-C8、C2-C7、C2-C6、C2-C5、C2-C4或C2-C3脂肪酸。
在一些实施方案中,相比于长链脂肪酸,所述脂肪酸脱羧酶可偏好选择短链脂肪酸。在一些实施方案中,实施脂肪酸脱羧酶可能几乎或完全不能接受短链脂肪酸以外的脂肪酸作为底物。在一些实施方案中,所述脂肪酸脱羧酶可能几乎或完全不能接受链长高于某一阈值的脂肪酸作为底物。这种偏好和/或不能可能是由于在对应于SEQ ID NO:1的G462位的氨基酸取代与链长大于8个碳的脂肪酸产生空间位阻并阻碍其结合。
例如,本发明所述的脂肪酸脱羧酶不能接受长链脂肪酸作为底物,例如链长和/或总碳原子数为5个或以上、6个或以上、7个或以上、8个或以上、9个或以上、10个或以上、11个或以上、12个或以上、12个或以上、13个或以上、14个或以上、15个或以上、16个或以上、17个或以上、18个或以上、19个或以上、20个或以上、21个或以上或22个或以上碳原子。
本发明所述脂肪酸脱羧酶的性质可以参考参照脂肪酸脱羧酶来描述。如本文所用,“参照脂肪酸脱羧酶”可以是在对应于SEQ ID NO:1的G462位上不包含氨基酸取代的任何脂肪酸脱羧酶。所述参考脂肪酸脱羧酶可以是野生型脂肪酸脱羧酶。所述参照脂肪酸脱羧酶可以是在对应于SEQ ID NO:1的G462位上没有取代的等价脂肪酸脱羧酶,其在其他方面与本发明所述的脂肪酸脱羧酶相同。所述参照脂肪酸脱羧酶包括SEQ ID NO:1、2和8-19,及其同源物、变体和/或活性片段。
所述脂肪酸脱羧酶可以将短链脂肪酸脱羧,从而高产率地产生短链烷烃/烯烃。在一些实施方案中,通过脂肪酸脱羧酶活性的可比性测定可知,与使用参照脂肪酸脱羧酶获得相同烷烃/烯烃的产率相比,以短链脂肪酸为底物时,包含上述取代的脂肪酸脱羧酶得到的短链烷烃/烯烃产率更高。在脂肪酸脱羧酶活性的可比性测定中,以短链脂肪酸为底物时,包含取代的脂肪酸脱羧酶获得的短链烷烃/烯烃产率可以是参照脂肪酸脱羧酶获得相同烷烃/烯烃的产率的≥5倍、≥4.5倍、≥4倍、≥3.5倍、≥3倍、≥2.5倍、≥2倍、≥1.9倍、≥1.8倍、≥1.7倍、≥1.6倍、≥1.5倍、≥1.4倍、≥1.3倍、≥1.2倍或≥1.1倍。
在一些实施方案中,产率较高的是C
在一些实施方案中,与长链脂肪酸相比,本发明所述的脂肪酸脱羧酶对短链脂肪酸具有底物偏好。在一些实施方案中,相对于长链脂肪酸,本发明所述的脂肪酸脱羧酶对短链脂肪酸的底物偏好,比参照脂肪酸脱羧酶的底物偏好更高。底物偏好可以参考特异性常数(k
在一些实施方案中,本发明所述的脂肪酸脱羧酶得到的烯烃/烷烃产率可以为未取代的等价脂肪酸脱羧酶获得产率的1倍以上,例如超过1.1倍、1.2倍、1.3倍、1.4倍、1.5倍、1.6倍、1.7倍、1.8倍、1.9倍、2.0倍、2.1倍、2.2倍、2.3倍、2.4倍、2.5倍、2.6倍、2.7倍、2.8倍、2.9倍、3.0倍、3.5倍、4.0倍、5.0倍、6.0倍、7.0倍、8.0倍、9.0倍或超过10.0倍。
在一些实施方案中,与参照蛋白相比,每单位有取代的脂肪酸脱羧酶在单位时间内产生的烯烃/烷烃的量更多。在一些实施方案中,经可比性测定,相比于未取代的等价脂肪酸脱羧酶,脂肪酸脱羧酶转化脂肪酸为烯烃/烷烃(例如以nmol.min
合适的可比性测定包括一种或多种测定法,其选自:实施例3中所述的无细胞测定、实施例4中所述的大肠杆菌表达测定和/或实施例6中所述的嗜盐单胞菌属测定。
在一些实施方案中,有取代的脂肪酸脱羧酶在测定每升反应产生烯烃/烷烃的产率,例如细胞培养物包含本发明得到的脂肪脱羧酶,相当于0.01mg/L、0.02mg/L、0.03mg/L、0.04mg/L、0.05mg/L、0.06mg/L、0.07mg/L、0.08mg/L、0.09mg/L、0.1mg/L、0.2mg/L、0.3mg/L、0.4mg/L、0.5mg/L、0.6mg/L、0.7mg/L、0.8mg/L、0.9mg/L、1mg/L、2mg/L、3mg/L、4mg/L、5mg/L、6mg/L、7mg/L、8mg/L、9mg/L、10mg/L、11mg/L、12mg/L、13mg/L、14mg/L、15mg/L、16mg/L、17mg/L、18mg/L、19mg/L、20mg/L或更高。在一些实施方案中,所述产率可以相对于所述脂肪酸脱羧酶的总重或包含所述脂肪酸脱羧酶的细胞的总重进行归一化。在一些实施方案中,所述反应在最佳条件(例如温度、pH、盐度,以及饱和浓度的底物、辅因子、光等)下进行。在一些实施方案中,所述测定如实施例2-4和/或6-8中所述。
脂肪酸脱羧酶可能是光依赖性的,即其活性的发挥需要其暴露于光。例如,变异小球藻NC64A含有葡萄糖-甲醇-胆碱氧化还原酶,其在蓝光照射下催化游离脂肪酸脱羧成正构烷烃或正构烯烃。所述光可以是任何波长和/或颜色或它们的组合(例如白光),但优选可为蓝光(400-520nm)或红光(620-750nm波长),最优选蓝光。在一优选的实施方案中,所述光的波长为400-520nm,优选为450nm-495nm,更优选约450nm-470nm。所提供的光的量可以是例如10-3000μmol.光子.m
可以按本发明的方法操作,以使上述脂肪酸脱羧酶或产生其的细胞暴露于适当波长的光。
如本文所用,“醛脱氢酶”是指能够催化酰基-CoA转化为醛的酶,例如催化丁酰-CoA转化为丁醛的酶。
一些醛脱氢酶是双功能酶,例如丙酮丁醇梭菌(C.acetobutylicum)ATCC 824酶AdhE2(醛/醇脱氢酶;GenBank ID:Q9ANR5),除了可将丁酰-CoA转化为丁酸外,还可将丁酰-CoA转化为丁醇,丁醇属于有毒的不良副产物。因此,在一些实施方案中,本发明使用的醛脱氢酶不催化醇生成反应。
一种示例性的醛脱氢酶是来自拜氏梭菌(Clostridium beijerinckii)的醛脱氢酶(BALDH),其氨基酸序列如SEQ ID NO:20所示。其他的醛脱氢酶包括包含选自如SEQ IDNO:32-37所示氨基酸序列的多肽。在一些实施方案中,所示丁醛脱氢酶包含SEQ ID NO:20或32-37中任一项的氨基酸序列或具有与SEQ ID NO:20或32-37中任一氨基酸序列至少30%,优选至少35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
醛脱氢酶的片段、变体、亚型和同源物可以任选地用催化丁酰-CoA转化为丁醛的能力来表征。
如本文所用,“脂肪醛去甲酰加氧酶”是指能够催化脂肪醛脱甲酰以进行烷烃生物合成的酶(图1A)。特别地,本发明所用的脂肪醛去甲酰加氧酶是指能够催化丁醛脱甲酰生成丙烷的酶。
一种示例性的脂肪醛去甲酰加氧酶是来自海洋原绿球藻st.MIT9313的脂肪醛去甲酰加氧酶(ADO),如Menon N等人在Biotechnol Biofuels 2015;8:61-12中所述,其全部内容通过引用并入本文。该酶催化铁氧还蛋白和主要的长链(C17-C19)脂肪醛的氧依赖性脱羰基生成烷烃(C15-C17)和甲酸酯的过程[9]。基于结构的对ADO底物接入通道的工程改造产生了丙烷产量提高的ADO变体A134F和V41Y[10]。
ADO的氨基酸序列如SEQ ID NO:21所示。在一些实施方案中,脂肪醛去甲酰加氧酶包含SEQ ID NO:21的氨基酸序列或具有与SEQ ID NO:21至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。在一些实施方案中,脂肪醛去甲酰加氧酶在对应于SEQ ID NO:21的A134位包含氨基酸取代。在一些实施方案中,所述氨基酸取代是A134F。在一些实施方案中,脂肪醛去甲酰加氧酶在对应于SEQ ID NO:21的V41位包含氨基酸取代。在一些实施方案中,所述氨基酸取代是V41Y。
所述脂肪醛去甲酰加氧酶可以从丁醛的脱甲酰化得到更高产率的丙烷。一些实施方案中,通过脂肪醛去甲酰加氧酶活性的可比性测定可知,与使用包含SEQ ID NO:21多肽的脂肪醛去甲酰加氧酶相比,以丁醛为底物时,包含上述取代的脂肪醛去甲酰加氧酶得到的丙烷产率更高。在脂肪醛去甲酰加氧酶活性的可比性测定中,以丁醛为底物时,包含上述取代的脂肪醛去甲酰加氧酶得到的丙烷产率可以是使用包含SEQ ID NO:21多肽的脂肪醛去甲酰加氧酶的丙烷产率的≥5倍、≥4.5倍、≥4倍、≥3.5倍、≥3倍、≥2.5倍、≥2倍、≥1.9倍、≥1.8倍、≥1.7倍、≥1.6倍、≥1.5倍、≥1.4倍、≥1.3倍、≥1.2倍或≥1.1倍。
脂肪醛去甲酰加氧酶的片段、变体、同种型和同源物可以任选地用催化丁醛转化为丁烷的能力来表征。
在一些实施方案中,所述脂肪醛去甲酰加氧酶可以使用电子传递伴侣蛋白,例如铁氧还蛋白。在一些实施方案中,所述脂肪醛去甲酰加氧酶可以使用辅酶,例如烟酰胺腺嘌呤二核苷酸(NAD
如本文所用,“乙酰-CoA乙酰转移酶”是指能够催化乙酰CoA转化为乙酰乙酰-CoA的酶。在一些实施方案中,所述乙酰-CoA乙酰转移酶包含如SEQ ID NO:28所示的氨基酸序列或具有与SEQ ID NO:28所示氨基酸序列至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
如本文所用,“乙酰乙酰CoA合酶”是指能够催化丙二酰-CoA转化为乙酰乙酰-CoA的酶。在一些实施方案中,所述乙酰乙酰CoA合酶包含如SEQ ID NO:24所示的氨基酸序列或具有与SEQ ID NO:24所示氨基酸序列至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
如本文所用,“梭菌3-羟基丁酰-CoA脱氢酶”是指能够催化乙酰乙酰-CoA转化为3-羟基丁酰-CoA的酶。在一些实施方案中,所述梭菌3-羟基丁酰-CoA脱氢酶包含如SEQ IDNO:29所示的氨基酸序列或具有与SEQ ID NO:29所示氨基酸序列至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
如本文所用,“3-羟基丁酰-CoA脱水酶”是指能够催化3-羟基丁酰-CoA转化为巴豆酰-CoA的酶。在一些实施方案中,所述梭菌3-羟基丁酰-CoA脱氢酶包含SEQ ID NO:30所示的氨基酸序列或具有与SEQ ID NO:30所示氨基酸序列至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
如本文所用,“反式-烯酰-CoA还原酶”是指能够催化巴豆酰基-CoA转化为丁酰基-CoA的酶。在一些实施方案中,所述梭菌3-羟基丁酰-CoA脱氢酶包含如SEQ ID NO:31所示的氨基酸序列或具有与SEQ ID NO:31所示氨基酸序列至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
如本文所用,“酰基-CoA硫酯水解酶”是指能够催化丁酰辅酶A转化为丁酸的酶。在一些实施方案中,所述梭菌3-羟基丁酰-CoA脱氢酶包含如SEQ ID NO:23所示的氨基酸序列或具有与SEQ ID NO:23所示氨基酸序列至少70%,优选为80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%氨基酸序列同一性的氨基酸序列,或由上述氨基酸序列组成。
除了编码本文所述酶的氨基酸序列之外,本公开提供了编码所述氨基酸序列的核酸。
在一些实施方案中,所述核酸是DNA。在一些实施方案中,所述核酸是RNA。所述核酸可以是单链的或双链的。所述核酸可以分离/纯化的形式呈现或为在宿主细胞内的形式呈现。
编码如本文所述的酶的核酸可以可操作地连接至异源调节性序列,例如启动子,如组成型或诱导型启动子。
在本说明书中,术语“可操作地连接”可以包括如下情形:所选择的核苷酸序列和调节性核苷酸序列(例如启动子和/或增强子)通过共价连接的方式以使该核苷酸序列的表达受到所述调节性核苷酸影响或控制(从而形成表达框)。因此,如果调节序列能够影响所述核苷酸序列的转录,则调节序列为可操作地连接至所选择的核苷酸序列。然后在适当情况下,可以将所得的转录物翻译成所需的蛋白质或多肽。
“启动子”是指天然的、改造的或合成的核苷酸序列,其指导编码序列的起始和转录速率。许多合适的启动子是本领域已知的,并且可以依本发明需要使用。所述启动子包含RNA聚合酶结合位点,并且还包含结合其他调控元件(例如转录因子)的位点。典型情形下,启动子包括最小启动子以及用于确定转录起始位点的其他序列,其中所述最小启动子是由普里布诺框(Pribnow box,TATAAT)组成的短DNA序列;在该启动子基础上添加调节元件以控制其表达。
编码所述酶的核酸可以包含在核酸构建体或载体上,特别是表达载体上。本文所用之“载体”是作为媒介以将外来遗传物质转移到细胞中的寡核苷酸分子(DNA或RNA)。载体包括双链或单链线性或环状形式的,尤其是任何质粒、粘粒和噬菌体等,其可以是能自主传染的或自主可移动的,或不能自主传染的或自主可移动的,并且可以转化原核或真核宿主,特别是可将所述载体整合入细胞基因组或使其存在于染色体外(例如具有复制起点的自主复制质粒)的细菌宿主。
所述载体可以是用于在细胞中表达外源遗传物质的表达载体。这样的载体可以包含与编码要表达序列的核苷酸序列可操作地连接的启动子和/或核糖体结合位点(RBS)序列。所述载体还可以包含终止密码子和表达增强子。此类表达载体是分子生物学领域中常规构建的,并且可以涉及例如质粒DNA和合适的起始因子、启动子、RBS、增强子和其他元件的使用,例如多聚腺苷酸信号,它们可能是必需的并且被以正确的方向定位以允许蛋白质表达。
所述载体可用于在相容的宿主细胞中复制核酸。因此,可以通过将多核苷酸插入可复制的载体、将该载体转入相容的宿主细胞并在促使载体复制的条件下培养宿主细胞的方式,来产生本发明所述的核酸。
载体可以包括与编码要表达基因序列的核苷酸序列可操作地连接的启动子序列。载体还可以包括终止密码子和表达增强子。本领域已知的任何合适的载体、启动子、增强子和终止密码子可用于表达本发明所述载体编码的酶。合适的载体包括质粒、双载体、病毒载体和人工染色体(例如酵母人工染色体)。
尤其当该载体用于将核酸导入细胞并重组进基因组时,包含上述核酸的构建体或载体不需要包含启动子或其他调控序列。
构建体和载体可以进一步包含选择基因标记,该选择基因标记由赋予细胞可选表型(例如对抗生素的抗性,诸如卡那霉素、潮霉素、膦丝菌素、氯磺隆、甲氨蝶呤、庆大霉素、大观霉素、氯霉素、氨苄青霉素等)的基因组成。
本领域技术人员可以构建用于重组基因表达的载体和设计方案,例如在微生物细胞中。本领域技术人员可以选择或构建合适的载体,其包含合适的调控序列,包括启动子序列、终止子片段、多聚腺苷酸序列、增强子序列、标记基因和其他合适的序列。更多细节请参见如《分子克隆实验指南:第三版》(Molecular Cloning:a Laboratory Manual:3rdedition),Sambrook等,2001,冷泉港实验室出版社,以及《分子生物学技术》(Protocols inMolecular Biology),第二版,Ausubel等,英语方言协会,约翰·威利父子(John Wiley&Sons),1992年。
本领域已知的任何合适的载体、启动子、增强子和终止密码子可用于表达本发明所述载体表达的多肽。在一些实施方案中,所述载体可以是质粒、噬菌体、MAC和病毒等。
在一些实施方案中,所述载体可以是真核表达载体。在一些实施方案中,所述载体可以为如下真核表达载体,例如含有在真核细胞中从该载体表达蛋白质所必需的元件的载体。在一些实施方案中,所述载体可以是哺乳动物表达载体,例如包含巨细胞病毒(CMV)或SV40启动子以驱动蛋白质表达的哺乳动物表达载体。
其他合适的载体对于本领域技术人员也是显而易见的。这方面进一步的例子可参考Sambrook等,2001,《分子克隆实验指南:第三版》(Molecular Cloning:a LaboratoryManual:3rd edition),冷港实验室出版社。
术语“可操作地连接”可以包括如下情形:所选择的核苷酸序列和调节性核苷酸序列(例如启动子和/或增强子)通过共价连接的方式以使该核苷酸序列的表达受到所述调节性核苷酸影响或控制(从而形成表达框)。因此,如果调节序列能够影响所述核苷酸序列的转录,则调节序列为可操作地连接至所选择的核苷酸序列。然后在适当情况下,可以将所得的转录物翻译成所需的蛋白质或多肽。然后可以将所得的转录物翻译成所需的蛋白质或多肽。所述启动子可以是T7启动子。
在一些实施方案中,所述载体可包含用于促进从构建体转录的mRNA翻译出编码蛋白的元件。例如,所述构建体可以在起始密码子上游包含核糖体结合位点(RBS),例如Shine-Delgarno(SD)序列。在一些实施方案中,可以设计RBS序列以提供对编码蛋白不同水平的表达。
在一些实施方案中,所述载体可以编码一种或多种用于调节编码蛋白表达的应答元件。在一些实施方案中,所述应答元件可以引起基因或蛋白质表达上调的元件,该上调是响应于用特定试剂的处理。例如,所述试剂可以在包含所述试剂的响应元件的载体中,诱导编码蛋白质的DNA的转录。在一些实施方案中,所述试剂可以是异丙基-β-D-硫代半乳糖苷(IPTG),并且所述载体可以包含乳糖操纵子。其他诱导试剂/响应元件的组合均是本领域已知的。
在一些实施方案中,所述载体可以编码一种或多种用于编码蛋白的组成型表达应答元件,使得蛋白表达不需要诱导。
在一些实施方案中,所述载体可以在编码一种或多种目的蛋白质的序列下游包含转录终止子序列。在一些实施方案中,所述终止子可以是T7终止子序列。在一些实施方案中,所述载体可以包含与编码目的蛋白的序列在同一阅读框的编码可检测标记的序列,以促进对蛋白质表达的检测和/或对蛋白质的纯化或分离(例如一个His(也可为6×His)、Myc、GST、MBP、FLAG、HA、E或生物素标签,可以选择位于N-或C-末端)。
可以通过本领域技术人员所知的任何合适方式将核酸/表达载体导入细胞。在一些实施方案中,通过转化、转导、接合、转染或电穿孔将所述核酸/表达载体导入细胞。
特别优选的是T7和T7样启动子系统。T7 RNA聚合酶是本领域众所周知的。它是一种非常活跃的酶,可以以大肠杆菌RNA聚合酶几倍的高速率合成RNA。此外,它具有较低的终止频率,并且其转录可以绕过质粒,从而产生RNA,其大小是质粒长度的几倍。T7 RNA聚合酶对其自身的启动子序列也具有很高的选择性,并且对抑制大肠杆菌RNA聚合酶的抗生素(如利福平)具有抗性。
“T7样”系统是IPTG诱导型系统,其工作方式类似于病毒聚合酶T7(IPTG诱导型;发现于pET系统载体中),但兼容于其他细菌物种(例嗜盐单胞菌)。MmP1基因属于T7样启动子,其可促使IPTG诱导重组蛋白在嗜盐单胞菌中表达(Zhao H et al 2017Novel T7-likeexpression systems used for Halomonas.Metabolic Engineering 39:p.128-140,其全文通过引用并入本文)。优选地,所述嗜盐单胞菌菌株包含整合于染色体或在载体或质粒上的MmP1基因。
在一些实施方案中,所述诱导型系统可以使用T7样的MmP1或类似系统(例如K1F、VP4或RiboJ),或基于proD的组成型系统。
以下是可在嗜盐单胞菌中编码酶多肽核酸上游使用的T7样启动子序列:
>MmP1_高表达(Ih)(SEQ ID NO:38)
ATATTTGTGGCATTATAGAATTGTGAGCGCTCACAATTAGCTGTCACCGGATGTGCTTTCCGGTCTGATGAGTCCGTGAGGACGAAACAG
>MmP1_中等表达(Im)(SEQ ID NO:39)
ATATTTGTGGCATTAGGGAATTGTGAGCGCTCACAATTAGCTGTCACCGGATGTGCTTTCCGGTCTGATGAGTCCGTGAGGACGAAACAG
>MmP1_低表达(Il)(SEQ ID NO:40)
ATATTTGTGGCATACTTGAATTGTGAGCGCTCACAATTAGCTGTCACCGGATGTGCTTTCCGGTCTGATGAGTCCGTGAGGACGAAACAG
上述序列需要在嗜盐单胞菌菌株中具有MmP1基因,其中MmP1基因整合进染色体或载体或质粒。
启动子也可能是组成型的。“组成型”表示不需要诱导(添加特定化学试剂,例如IPTG)即可表达所述重组蛋白。下列启动子序列可被用于嗜盐单胞菌中编码酶多肽的核酸上游,并提供不同的表达水平。
>P40-1_低表达(Cl)(SEQ ID NO:41)
TTTTTCTATTGCGTCCGTGTATTCTTTTGTATAGAGTTTGAGAC
>P40_中等表达(Cm)(SEQ ID NO:42)
TTTTTCTATTGCGTTCACTGGAATCCCAGTATAGAGTTTGAGAC
>P40-9_高表达(Ch)(SEQ ID NO:43)
TTTTTCTATTGCGTGAAAACAAGGATTTGTATAGAGTTTGAGAC
>P40-58_非常高表达(Cvh)(SEQ ID NO:44)
TTTTTCTATTGCGTCAAAACATTTATTTGTATAGAGTTTGAGAC
本公开还涉及包含编码本文所述酶的核酸的细胞。所述细胞可以表达本文所述的酶。所述细胞可以产生、包含或分泌本文所述的酶。
所述细胞可以是分离的形式和/或在培养中的形式。所述细胞可以处于体外。
包含本发明所述酶的细胞可以通过从已经引入细胞中的本发明所述核酸/表达载体的表达来实现。
预期可用于本发明的细胞包括原核细胞和真核细胞。例如,所述原核细胞可以是细菌或古细菌,真核微生物可以是真菌、原生生物、微动物或微植物等生物。在一些实施方案中,所述细胞是分离自多细胞生物的细胞。
在优选的方面,所述细胞是细菌。在一些实施方案中,所述细菌可以是革兰氏阳性细菌。革兰氏阳性细菌包括芽孢杆菌属的细菌、李斯特菌属或梭菌属[例如艰难梭菌(C.difficile)]的细菌,或如葡萄球菌(例如金黄色葡萄球菌)或链球菌的球菌。在一些实施方案中,所述细菌可以是革兰氏阴性细菌。革兰氏阴性菌可定义为一类不保留革兰氏染色细菌分化方法中使用的结晶紫染色的细菌,从而使阳性鉴定成为可能。所述革兰氏阴性细菌包括变形菌门,或如大肠杆菌、沙门氏菌、志贺氏菌的肠杆菌科细菌,或选自假单胞菌属(Pseudomonas)、螺杆菌属(Helicobacter)、奈瑟菌属(Neisseria)、军团菌属(Legionella)、嗜盐单胞菌属(Halomonas)、克雷伯菌属(Klebsiella)或耶尔森氏菌属(Yersinia)的细菌。
本发明可以使用任何细菌,例如实验室菌株(例如大肠杆菌或枯草芽孢杆菌)或野生菌株。优选的细菌为有机营养的细菌,例如化能异养菌,其能够将生物质或由其衍生的化合物用作能源。
优选的细菌为强力细菌,例如土壤细菌和/或嗜极性细菌。所述嗜极性细菌包括轻度嗜盐菌(能够在1.7至4.8%的NaCl中生长)、中度嗜盐菌(能够在4.7至20%NaCl中生长)、极度嗜盐菌(能够在20%至30%NaCl中生长)、嗜酸性菌(能够在低pH值中生长,例如低于pH5.0,例如pH 2或更低)、嗜碱性菌(能够在pH 8.5或更高的条件下生长)、耐金属细菌(能够在含有高浓度溶解重金属的环境中生存)、嗜热菌[最佳生长温度在约41至122℃之间,例如热解纤维素果汁杆菌属(Caldicellulosiruptor)、热袍菌属(Thermotoga)、嗜热厌氧菌属(Thermoanaerobacterium)、火球菌属(Pyrococcus)和气火菌属(Aeropyrum)]或多嗜极菌(具有两个或多个嗜极性的细菌)。这样的细菌在生物质发酵罐的细菌培养生产烃类中可以发挥独特效用。
优选嗜盐细菌。其能够在开放的非无菌条件下生长。由于这些菌株具有耐盐性,因此只要环境中盐含量高,它们就不会竞争淘汰。此外,添加高盐的缓冲液(例如至少3%的盐溶液)可用于控制其他的竞争细菌。嗜盐细菌包括嗜盐单胞菌属的细菌。现有技术已经记载了嗜盐单胞菌的示例性物种,包括H.alimentaria,H.alkaliantarctica,H.alkaliphila,H.almeriensis,H.andesensis,H.anticariensis,H.aquamarina,H.arcis,H.axialensis,H.beimenensis,H.bluephagenesis,H.boliviensis,H.campaniensis,H.campisalis,H.caseinilytica,H.cerina,H.cibimaris,H.cupida,H.daqiaonensis,H.daqingensis,H.denitrificans,H.desiderata,H.elongata,H.eurihalina,H.flava,H.fontilapidosi,H.garicola,H.gomseomensis,H.gudaonensis,H.halmophila,H.halocynthiae,H.halodenitrificans,halophila,H.hamiltonii,H.heilongjiangensis,H.huangheensis,H.hydrothermalis,H.ilicicola,H.janggokensis,H.jeotgali,H.johnsoniae,H.kenyensis,H.koreensis,H.korlensis,H.kribbensis,H.lutea,H.lutescence,H.magadiensis,H.maura,H.meridian,H.mongoliensis,H.muralis,H.nanhaiensis,H.neptunia,H.nitroreducens,H.olivaria,H.organivorans,H.pacifica,H.pantelleriensis,H.qiaohouensis,H.qijiaojingensis,H.ramblicola,H.rifensis,H.sabkhae,H.saccharevitans,H.salicampi,H.salifodinae,H.salina,H.sediminicola,H.shengliensis,H.sinaiensis,H.smyrnensis,H.songnenensis,H.stenophila,H.stevensii,H.subglaciescola,H.subterranean,H.sulfidaeris,H.taeanensis,H.titanicae,H.urumqiensis,H.variabilis,H.ventosae,H.venusta,H.vilamensis,H.xianhensis,H.xinjiangensis,H.zhangjiangensis,andH.zincidurans。
优选的嗜盐单胞菌属菌株包括嗜盐单胞菌st.TQ10和嗜盐单胞菌st.TD01。TQ10菌株是TD01菌株的基因改造版本,其中编码MmP1的基因通过染色体整合导入到细菌中。MmP1基因是T7样启动子,其使IPTG诱导重组蛋白在嗜盐单胞菌中表达(Zhao H et al2017Novel T7-like expression systems used for Halomonas.Metabolic Engineering39:p.128-140,其全文通过引用并入本文)。优选地,所述嗜盐单胞菌属菌株包含整合进染色体、载体或质粒上的MmP1基因。
本发明所用的细菌能够从生物质原料生产脂肪酸,例如丁酸等短链脂肪酸。例如,细菌能够将葡萄糖或纤维素转化为脂肪酸,和将脂肪酸转化为烃,如图9所示。细菌可能具有将葡萄糖或纤维素转化为脂肪酸所需的内源基因,或者此功能可以由一个或多个异源基因赋予。能够产生脂肪酸的细菌可以包含编码任何数目的如SEQ ID NO:23-31所示多肽的核苷酸序列。
在特定的实施方案中,如大肠杆菌(E.coli)的埃希氏菌属、如酿酒酵母(S.cerevisiae)的酵母属和蓝细菌均可预期适用于本发明。
在一些实施方案中,所述多肽可以通过无细胞蛋白质合成(CFPS),例如使用Zemella et al.Chembiochem(2015)16(17):2420-2431中所述的系统,在此以引用的方式将其全文并入本发明。
本发明还提供了一种生产如本发明所述组合物的方法,该方法包括(i)在适合于编码蛋白表达的条件下培养本发明所述的细胞。在一些实施方案中,该方法进一步包括(ii)分离所述被表达的蛋白。本发明还包括通过该方法生产的组合物。
本发明还提供了包含如本发明所述细胞、核酸、表达载体和酶/酶组合的组合物。所述组合物可用于例如本发明所述烃类合成的方法中。
本发明还提供了包含异源脂肪酸脱羧酶的细胞,例如细菌细胞。
如本发明所述的编码酶的多肽的制备可以按照本领域技术人员已知的用于重组蛋白生产的方法进行。适用于重组生产的分子生物学技术是本领域众所周知的,例如Green和Sambrook在2012年冷泉港出版社出版的《分子克隆实验指南:第四版》(MolecularCloning:A Laboratory Manual(4th Edition))中提出的那些方法,该文献全文通过引用结合到本文中。
表达可以来自核酸序列和/或表达载体,例如本发明所述的核酸序列或表达载体。本领域已知的任何合适的载体、启动子、增强子和终止密码子均可用于表达如本发明所述的表达载体的肽或多肽。可以在如本发明实施的细胞中进行表达。任何适合于表达编码酶的多肽的细胞均可用于所述表达。
生产可能涉及经修饰以表达相关多肽的细胞的培养或发酵。所述培养或发酵可以在提供有适当养分、空气/氧气和/或生长因子的生物反应器中进行。分泌的蛋白质可通过下述方式收集:将培养基/发酵液于细胞分离、从中提取蛋白质成分并分离各种蛋白质以分离分泌或表达的肽或多肽。培养、发酵和分离技术是本领域技术人员众所周知的,并且例如在Green和Sambrook的《分子克隆实验指南:第四版》(通过引用并入本文中)中所记载的。
生物反应器包括一个或多个可以在其中培养细胞的容器。所述生物反应器中的培养可以持续进行:反应物持续流入反应器,和培养的细胞连续从反应器流出。或者,培养可以分批进行。所述生物反应器监测和控制环境条件,例如pH、氧气、光的波长和强度、进出容器的流速以及在容器内的搅拌,从而为培养的细胞提供最佳条件。
培养后,可以分离表达目的多肽的细胞。蛋白质可以用本领域已知的从细胞中分离的任何合适方法。为了分离多肽,可能有必要将细胞与营养培养基分离。
如果多肽是由细胞分泌出来的,则可以通过离心将细胞与含有被分泌的目的多肽的培养基分离。
如果目的多肽积累在细胞内,则蛋白质的分离可包括从细胞培养基中离心分离出细胞、用裂解液处理细胞沉淀和裂解细胞,例如使用超声处理、快速冻融或渗透裂解。
由于上清液或营养培养基可能含有其他蛋白质和非蛋白质成分,其后可能需要从上清液或营养培养基中分离目的多肽。
从上清液或培养基中分离蛋白质组分的一种方法是通过沉淀。不同溶解度的蛋白质会在不同浓度的沉淀剂(例如硫酸铵)中沉淀。例如,可在低浓度的沉淀剂中提取水溶性蛋白质。因此,通过添加不同浓度的沉淀剂,可以分离具有不同溶解度的蛋白质。随后可用透析以从分离的蛋白质中去除硫酸铵。分离蛋白质组分的其他方法包括离子交换层析和尺寸排阻层析。这些可以作为沉淀的替代方法,或者可以在沉淀之后进行。
从培养物中分离出目的多肽后,可能需要或必须浓缩所述肽或多肽。浓缩蛋白质的许多方法是本领域已知的,例如超滤和冻干。
应当理解,本发明所述编码酶的多肽可作为编码酶或多肽复合物的较大多肽的组分。例如,本文所述编码酶的多肽可以以编码酶的融合多肽形式呈现。在一些实施方案中,所述编码酶的多肽可以包含一个或多个氨基酸序列,以促进表达、折叠、运输、加工或纯化,这些氨基酸序列例如His(如6×His)、Myc GST、MBP、FLAG、HA、E或生物素标签,并且可选地位于N或C末端。
本公开还提供了包括使用一种或多种酶催化原料转化为烯烃和/或烷烃的方法。这些方法可以为制备用作燃料的短链烷烃如丙烷、丁烷和异丁烷的方法。
在一些实施方案中,所述方法包括为转化提供底物。所述方法可以包括将底物或代谢前体添加到含有酶的容器中。在一些实施方案中,所述方法还包括回收转化产物。在一些实施方案中,可以根据随后的方法步骤将算是产物回收并用作转化的底物。在一些实施方案中,所述产物作为该方法的最终产物被回收。回收的产物可被分离/纯化。反应产物可被分析,例如通过气相色谱、高效液相色谱和/或质谱分析。
包含催化底物的任何合适的物质都可以用作原料。在一个优选的实施方案中,所述方法使用生物质衍生的原料,例如通过微生物消化/发酵对生物质进行微生物加工的产物。该原料通常富含丁酸、异戊酸和/或戊酸以及上述物质的前体。
还将理解的是,“一种包括使用酶Z催化底物X转化为产物Y的方法”暗含包括在适合于将底物X转化为产物Y的条件下,使底物X与酶Z接触。所述方法还可包括孵育的步骤。
在一些实施方案中,所述酶是按顺序提供的。在一些实施方案中,所述酶是同时(即,一起)提供。在一些实施方案中,所述酶通过依次添加酶(即一个接一个)来同时提供。
与按本发明所述方法进行的一种或多种转化有关的条件相关的因素,包括一种或多种酶、一种或多种底物、一种或多种酶的活性、一种或多种酶的浓度、一种或多种底物浓度、一种或多种酶辅因子浓度/可用性、温度、盐度、pH、搅拌、二氧化碳水平、氧气水平、营养物可用性、反应体积和可见光(尤其是蓝光,如470nm左右的蓝光)的存在,等等。
按本发明所述方法所给定的转化或转化组合,适合其的条件可以容易地由技术人员根据目标反应产物,参考例如本申请的实验实施例和本文中提供的参考文献来确定。
在一些实施方案中,可在反应中添加一种或多种辅因子或电子转移伴侣。一些实施方案提供了辅因子或电子转移伴侣的一种或多种来源。一些实施方案提供了用于产生/再循环一种或多种辅因子/电子转移伴侣系统,或辅因子/电子转移伴侣的来源系统。在一些实施方案中,辅助因子或电子转移伴侣选自NADH、NAD
本发明所述的方法可以进行于或本发明所述的产品可以存在于体外、离体或体内。术语“体外”旨在涵盖在实验室条件下或在培养物中用材料、生物物质、细胞和/或组织进行的实验,而术语“体内”旨在涵盖完整的多细胞生物的实验和程序。“离体”是指在生物体外部存在或发生的某些事物,例如在人体或动物体外,其可能在组织(例如整个器官)或从生物体获取的细胞中。
本发明所述的方法可以在人或动物体外进行。例如,所述方法可以在容器、生物反应器、发酵罐或类似设备中进行。这时本发明所述的方法可以进行于或本发明所述的产品可以存在于体外或离体情形。术语“体外”旨在涵盖在实验室条件下或在培养物中用材料、生物物质、细胞和/或组织进行的实验,而术语“体内”旨在涵盖完整的多细胞生物的实验和程序。“离体”是指在生物体外部存在或发生的某些事物,例如在人体或动物体外,其可能在组织(例如整个器官)或从生物体获取的细胞中。
在一些实施方案中,本发明所述的方法在体外进行。在一些实施方案中,本发明所述的方法使用分离/纯化的酶进行。在一些实施方案中,所述酶来自商购途径。在一些实施方案中,一种或多种所述酶可以是重组表达的或已经被重组表达,并且随后被分离/纯化,如本文所记载的。在一些实施方案中,一种或多种所述酶可以来自表达该一种或多种酶的生物(例如微生物)或多细胞生物的细胞、组织或器官。在一些实施方案中,所述酶可来自表达所述酶的多细胞生物的生物体或细胞、组织或器官,该表达源自所述生物的内源性核酸表达。在一些实施方案中,由于表达非该生物体内源的异源核酸(例如,如本发明所述的核酸/表达载体),所述酶可以来自表达一种或多种所述酶的所述生物体。在一些实施方案中,表达一种或多种所述酶的生物体或细胞、组织或器官可以分泌一种或多种所述酶。在一些实施方案中,所述酶的获取可包括从生物体(例如微生物)或表达所述酶的多细胞生物的细胞、组织或器官或其分泌物中,分离/纯化所述酶。
在一些实施方案中,本发明所述的方法是使用生物体(例如微生物)的提取物或表达一种或多种所述酶的多细胞生物体的细胞、组织或器官来进行的。制备提取物以使一种或多种所述酶保留相关活性。在一些实施方案中,一种或多种所述提取物可以用表达一种或多种所述酶的多细胞生物的生物体或细胞、组织或器官制备,该提取物源自所述生物的内源性核酸表达。在一些实施方案中,由于表达非该生物体内源的异源核酸(例如,如本发明所述的核酸/表达载体),一种或多种所述提取物可以来自表达一种或多种所述酶的所述生物体。提取物的制备可以包括以下一种或多种:使器官/组织/细胞均质化(例如通过细胞破碎或超声处理)、裂解细胞(例如用裂解缓冲液)、去除细胞碎片等。
在一些实施方案中,本发明所述的方法使用活细胞或全细胞(例如完整的呼吸细胞)。在一些实施方案中,一种或多种所述酶和一种或多种所述底物可以在细胞内互相接触。在一些实施方案中,所述底物可以由细胞产生。在一些实施方案中,所述底物/其前体可以扩散到细胞中。在一些实施方案中,所述底物/其前体可以被细胞吸收,例如通过跨膜的主动转运。在一些实施方案中,一种或多种所述酶可以由活细胞分泌,并且一种或多种所述酶和一种或多种上述底物可以在细胞外部互相接触。在一些实施方案中,所述方法在非无菌条件下进行。
在需要多个催化步骤的所述方法中,提供多种酶的组合物是可取的。因此,本公开内容提供了包含多种酶的组合物,例如包括给定方法列出的所有酶。本文还提供了编码给定方法所需的多种酶的核酸。这些被编码的酶可以包含于单个表达框,例如在一个启动子的统一控制下。本文还提供了编码给定方法列出的所有酶的多个核酸,其中一个或每个单独的核酸可以不编码给定方法列出的酶,或编码其中的一个或多个酶。一个优选的实施方案提供了一些细胞,例如细菌细胞,其包含编码给定方法列出的多种酶的核酸。本文还提供了包含多个细胞的组合物,所述组合物包含给定方法列出的多种酶,其中一个或每个细胞可以不包含编码给定方法列出的酶的核酸,或包含其中的一个或多个的核酸。所述组合物可以包含携带编码一种或多种所列酶的不同核酸的多种菌株。
本文提供的方法适用于将生物质原料生物转化为烃,特别是气态燃料。在这些方法中,本发明所述的细菌细胞或其培养物可被添加到生物质原料中。所述细菌细胞可以单独将所述原料中的葡萄糖和/或纤维素转化成烃,例如它能够进行图8、9或10所示的整个反应方案。或者,所述细胞仅能够将葡萄糖/纤维素下游的中间体加工成烃。所述方法可包括将生物质原料转化为本发明所述细菌细胞所接受中间体的额外步骤。该步骤可以同时或在所述中间体转化为烃之前进行。可以通过一种或多种其他细菌菌株进行转化。所述方法可以进一步包括杀死有害细菌的步骤,例如进行高压灭菌或添加对有害细菌有毒但被本发明所述细胞耐受的物质。在一些实施方案中,上述有毒物质为抗生素、盐溶液、过氧化氢溶液和/或改变pH的试剂。
在一些实施方案中,所述编码酶的多肽可以通过无细胞蛋白质合成(CFPS),例如使用Zemella et al.ChemBioChem(2015)16(17):2420-2431中所述的系统,在此以引用的方式将其全文并入本发明。
本文提供的方法包括使用脂肪酸脱羧酶催化脂肪酸转化为烷烃和/或烯烃的方法。优选地,所述方法包括使用脂肪酸脱羧酶催化短链脂肪酸转化为烷烃和/或烯烃。在一些实施方案中,所述方法包括使用脂肪酸脱羧酶催化C
所述方法可以使用多种底物。例如,所述脂肪酸底物可以是丁酸、戊酸或异戊酸,分别产生丙烷、丁烷和异丁烷。然而,所述方法可以利用能被脂肪酸脱羧酶转化的底物的混合物。例如,可以给脂肪酸脱羧酶提供丁酸、戊酸和异戊酸的混合物,从而产生丙烷、丁烷和异丁烷的混合物。可以给脂肪酸脱羧酶提供丁酸和异戊酸的混合物,从而产生丙烷和异丁烷的混合物。可以给脂肪酸脱羧酶提供丁酸和戊酸的混合物,从而产生丙烷和丁烷的混合物。可以给脂肪酸脱羧酶提供戊酸和异戊酸的混合物,从而产生丁烷和异丁烷的混合物。
在一些实施方案中,所述方法包括催化底物转化为烯烃/烷烃之前的步骤。
例如,所述方法可以包括以下步骤:使用脂肪酸脱羧酶催化脂肪酸转化为烯烃/烷烃之前,使用酰基-CoA硫酯水解酶催化酰基辅酶A(酰基-CoA)转化为脂肪酸的步骤。这可使更多的脂肪酸用于转化,致使烯烃/烷烃的产率增加。所述酰基辅酶A(酰基-CoA)可以是例如产生丁酸的丁酰-CoA,但也可以是链长为1、2、3、4或5个碳的任何其他酰基-CoA。优选的酰基-CoA硫酯水解酶是YciA(SEQ ID NO:23)或其衍生物、片段或变体(例如包含与SEQ IDNO:23具有至少5%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性的序列)。
本文所述的方法还包括使用醛脱氢酶(如丁醛脱氢酶)催化丁酰-CoA转化为丁醛,随后使用脂肪醛去甲酰加氧酶催化丁醛转化为丙烷。所述方法可以是生产丙烷的方法。
所述方法可以利用来自拜氏梭状芽胞杆菌(Clostridium beijerinckii)的醛脱氢酶或其片段、变体或同源物。所述方法可以利用如本文所述的如SEQ ID NO:20所示的醛脱氢酶或其变体、片段或同源物。所述方法可以利用如本文所述的如SEQ ID NO:21所示的脂肪醛去甲酰加氧酶或其变体、片段或同源物。可以为所述脂肪醛去甲酰加氧酶提供辅因子和电子转移伴侣,例如铁氧还蛋白和/或NADH/NADPH。
在一些实施方案中,所述方法包括预富集步骤,所述步骤包括将丁酸转化为丁酰-CoA。由于许多合适的原料中丁酸含量高,因此该步骤可增加该酶促途径的总产量。可以使用乙酰乙酸丁酸酯CoA转移酶对丁酸转化为丁酰-CoA进行催化。然后如前所述,用醛脱氢酶将丁酰-CoA转化为丁醛。示例性的乙酰乙酸丁酸酯CoA转移酶包括如SEQ ID NO:22所示的酶,或其变体、片段或同源物。
由于所述方法需要多个步骤,因此提供多种酶的组合物是是可取的。因此,本公开内容提供了包含醛脱氢酶、脂肪醛去甲酰加氧酶和(任选的)乙酰乙酸丁酸酯CoA转移酶的组合物。本文还提供了编码所述醛脱氢酶和脂肪醛去甲酰加氧酶的核酸。这两种被编码的酶可以包含于单个表达框,例如在一个启动子的统一控制下。或者,一个表达框可包含一个以上的启动子,例如每个被表达的酶最多可对应一个启动子,这样一个“表达框”可包含多个酶,并且最多可包含与酶相同数量的启动子。核酸可以任选地进一步包含乙酰乙酸丁酸酯CoA转移酶,其也可以在同一表达框中。
本文还提供了多个编码醛脱氢酶、脂肪醛去甲酰加氧酶和(任选的)乙酰乙酸丁酸酯CoA转移酶的核酸。上述多个核酸中的单个核酸可以不包含上述酶,或包含其中的一种、两种或所有三种酶。
一个优选的实施方案在细胞例如细菌细胞中提供所述酶。例如,可以提供包含编码异源醛脱氢酶核酸的细胞。所述细胞可以进一步包含编码脂肪醛去甲酰加氧酶的核酸和/或编码乙酰乙酸丁酸酯CoA转移酶的核酸。本文还提供了一种包含多个细胞的组合物,所述组合物包含至少一个含有编码醛脱氢酶核酸的细胞,至少一个含有编码脂肪醛去甲酰加氧酶核酸的细胞,以及任选地至少一个含有编码乙酰乙酸丁酸酯CoA转移酶核酸的细胞。至少一个细胞可以包含编码两种或三种所述酶的核酸。或者,编码所述酶的核酸可以在不同细胞中。
在一些实施方案中,所述方法包括将葡萄糖转化为丙烷。该反应的已知途径是通过丁酸和丁醛从丁酰-CoA到丙烷(图8)。但是,该反应途径的产率低。此外,其他天然酶通过CAR/SFP作用对丁醛的富集浓度起作用的同时,会产生副产物丁醇。本文提供的方法以低量的、可忽略的丁醇产生获得较高的丙烷产率。
一种这样的方法通过丁酸/脂肪酸脱羧酶途径进行。反应方案见于图9。该途径可通过糖酵解将葡萄糖转化为乙酰-CoA和/或丙二酰-CoA。随后,所述方法可以包括按顺序执行的以下步骤:
(a)使用乙酰-CoA乙酰转移酶(例如SEQ ID NO:28,或其片段、其变体的同源物)催化乙酰CoA转化为乙酰乙酰-CoA,和/或使用乙酰乙酰CoA合酶(例如SEQ ID NO:24,或其片段、其变体的同源物)催化丙二酰-CoA转化为乙酰乙酰-CoA,
(b)使用梭菌3-羟基丁酰-CoA脱氢酶(例如SEQ ID NO:29,或其片段、其变体的同源物)催化乙酰乙酰-CoA转化为3-羟基丁酰-CoA,
(c)使用3-羟基丁酰-CoA脱水酶(例如SEQ ID NO:30,或其片段、其变体的同源物)催化催化3-羟基丁酰-CoA转化为巴豆酰-CoA,
(d)使用反式-烯酰-CoA还原酶(例如SEQ ID NO:31,或其片段、其变体的同源物)催化巴豆酰基-CoA转化为丁酰基-CoA,
(e)使用酰基-CoA硫酯水解酶(例如SEQ ID NO:23,或其片段、其变体的同源物)催化丁酰辅酶A转化为丁酸,和
(f)使用如上所述的脂肪酸羧化酶催化丁酸转化为丙烷以产生丙烷。
在一些实施方案中,通过丁醛和醛脱氢酶/脂肪醛去甲酰加氧酶的作用将葡萄糖转化为丙烷。葡萄糖可通过糖酵解转化为乙酰CoA和/或丙二酰-CoA。该反应方案见图10。
随后,所述方法可以包括按顺序执行的以下步骤:
(a)使用乙酰-CoA乙酰转移酶(例如SEQ ID NO:28,或其片段、其变体的同源物)催化乙酰CoA转化为乙酰乙酰-CoA,和/或试用乙酰乙酰CoA合酶(例如SEQ ID NO:24,或其片段、其变体的同源物)催化丙二酰-CoA转化为乙酰乙酰-CoA,
(b)使用梭菌3-羟基丁酰-CoA脱氢酶(例如SEQ ID NO:29,或其片段、其变体的同源物)催化乙酰乙酰-CoA转化为3-羟基丁酰-CoA,
(c)使用3-羟基丁酰-CoA脱水酶(例如SEQ ID NO:30,或其片段、其变体的同源物)催化3-羟基丁酰-CoA转化为巴豆酰-CoA,
(d)使用反式-烯酰-CoA还原酶(例如SEQ ID NO:31,或其片段、其变体的同源物)催化巴豆酰基-CoA转化为丁酰基-CoA,
(e)使用如上所述的醛脱氢酶催化丁酰-CoA转化为丁醛,和
(f)如上所述,使用脂肪醛去甲酰加氧酶催化丁醛转化为丙烷。
应当理解,在催化途径中所述酶顺序起作用的情况下,所述方法可以包括提供第一步的底物和催化后续转化的酶。所述方法可以从步骤(a)-(f)中的任何步骤开始,并提供正确的底物和酶,然后依次进行其余步骤。这些步骤可以顺序地或同时进行。上述反应方案所需的酶可以以单一组合物或多种组合物的形式提供,例如在单个细胞中,该细胞包含编码从任何给定步骤开始的方法所指定的所有酶的核酸,或在包含多个核酸的多个细胞中,每个核酸编码一种或多种从任何给定步骤开始的方法所指定的酶。
本发明所述的特征可以以适当的方式(如分离的形式或任何组合的形式)被用于实现本发明的各种形式,所述特征包括公开于前述说明中、在以下权利要求中、在附图中的、以其特定形式或用于执行所公开功能的手段表达的或者用于得到所公开结果的方法或过程中的。
尽管已经结合上述示例性实施方案描述了本发明,但是当得到本公开时,许多等同的修改和变化对于本领域技术人员将是显而易见的。因此,以上阐述的本发明的示例性实施方案应当是说明性的而非对本发明的限制。在不偏离本发明的精神和范围的情况下,可以对本发明记载的实施方案进行各种改变。
为了避免任何疑问,本文提供的任何理论解释仅仅是为了使读者更好理解本发明。发明人并不被上述理论解释限制。
本文使用的任何章节标题仅用于组织文章目的,并且不应解释为对所描述主题的限制。
在整个说明书中(包括所附的权利要求书),除非另有规定,否则词语“包括”和“包含”以及诸如“由…组成”、“含有”和“包括…在内”等变体将被理解为意指包括所述整数、步骤或一组整数或一组步骤,但不排除任何其他整数、步骤或一组整数或一组步骤。
必须注意的是,除非另有明确规定,如说明书和所附权利要求书中所使用的单数形式的“一个”、“一种”和“所述”包括复数对象。范围可在本文中表示为从“大约”一个特定值和/或到“大约”另一特定值。当表达这样的范围时,另一实施方案包括从一个特定值和/或至另一特定值。类似地,先行词“约”被用于将数值表示为近似值,本领域技术人员可以知晓由特定值构成其他实施例。相对于数值的术语“约”是可选的,并且是指例如+/-10%。
实施例
所有化学试剂和溶剂均为商购,并且其纯度均为分析纯或更高。丙烷气标准品(99.95%)获自Sigma Aldrich。培养基组分获自Formedium(Norfolk,UK)。基因测序和寡核苷酸合成均由Eurofins MWG(Ebersberg,Germany)实施。嗜盐单胞菌(Halomonas)株TD01[17]和TQ10以及质粒p321和p341由Guo-Qiang Chen教授(清华大学,中国北京)友好提供。已安装的Hi-Power蓝色LED和LED驱动器来自Thorlabs(New Jersey,USA),波长分别为455nm(输出功率1020mW)和470nm(253mW)。使用的白光LED(集成)具有25W的功率(2060流明)。
GeneArt(Germany)设计并合成了编码来自变异小球藻(Chlorella variabilis)NC64A(Genbank ID:A0A248QE08;N末端61个氨基酸截短)的成熟CvPAS序列的基因[13],结合密码子优化技术,将稀有密码子去除以实现在E.coli中的最佳表达。该基因被亚克隆(NcoI-XhoI)到pETM11中,并掺入了一个包含六组氨酸序列的78bp的N末端标签,用于蛋白质快速纯化。根据生产商的说明书,将构建体(CvPAS
N末端截短(成熟)和His
采用Stratagene QuickChange全质粒合成方案,通过定点诱变产生变体CvPAS
E.coli菌株BL21(DE3)中的野生型和CvPAS变体的培养物在含有卡那霉素(30μg/mL)和过夜起子培养物培养液(1%vol)的LB培养基中于37℃,200rpm下生长,直到OD
将细胞沉淀重悬于裂解缓冲液(25mL;50mM Tris,pH为8,含有300mM NaCl、10mM咪唑、10%甘油、0.25mg/mL溶菌酶、10μg/mL DNase I和1×蛋白酶抑制剂)中,超声处理20分钟(开启20秒,关闭60秒;振幅30%)。通过在4℃下以26600×g离心30分钟来制备无细胞裂解物。使用Precision Plus非预染蛋白质分子量标准(Bio-Rad)在300V下通过SDS PAGE(12%Mini-PROTEAN-TGX免染胶;Bio-Rad)20分钟分析裂解物样品的重组蛋白表达。使用Gel Doc EZ成像仪(Bio-Rad)可视化蛋白质含量。
丙烷生产的体外反应(200μL)由置于密封的玻璃GC瓶中的无细胞裂解物(180μL)和丁酸(0.36-4.5mM)组成。在蓝色LED(455nm)存在或不存在(对照样品)的情况下,将反应液于30℃在180rpm下孵育24小时。使用配有Al
使用Agilent 490Micro GC手动瓶顶空气进样以测定丙烷含量,其包含Al
野生型CvPAS的丙烷产量适中(1.65±0.61mg/L裂解物),但与使用替代ADO脱羰酶的体内产量水平相当(0.4-3.4mg/L)。使用变体CvPAS.G462V对无细胞裂解物进行生物转化显示丙烷产量明显增长了4倍(6.45±1.4mg/L裂解物)。
有趣的是,初步的浓度依赖性研究表明,与野生型酶相比,在4.5mM时不存在饱和,可见该突变影响了丁酸的结合(图6)。需要使用纯化的酶进行进一步动力学比较研究,以更全面地研究G462V突变对活性的影响。
通过以下方案在E.coli中进行CvPAS
该实验的结果如表1所示。两个数据集之间的不一致是由于后诱导时间和与光源的距离不同而引起的,但是很明显,该变体在瓶顶空气产生的丙烷明显多于野生型。较高浓度的丁酸(10mM)显示丙烷产量增加,但100mM浓度时溶解细胞。
表1野生型和变体CvPAS的体内丙烷生产
接下来,研究各种添加剂对丙烷生产的影响。
将培养物(20-100mL)在含有卡那霉素(50μg/mL)和过夜起子培养物(1%体积;相同培养基)的LB培养基中以180rpm、37℃培养6小时。用IPTG(0.1mM)诱导重组蛋白表达,并在培养物中添加丁酸(1-25mM)±Triton X-100(1%)、蔗糖(1%)和/或乙酰乙酸乙酯(0-30mM)。将培养物(5mL)密封在20mL玻璃小瓶中,一式三份,以200rpm、30℃孵育16-18小时,并用LED(白色或蓝色(455nm或470nm))连续照射。使用配有Al
这些实验的结果如表1和2所示。已知在细菌培养物中添加Triton X-100和蔗糖可以使细胞通透,从而有效使它们成为更“有漏洞的”[20]。这将增加CvPAS从细胞漏到培养物上清液中的可能性,从而增加其暴露于光下的机会,并导致更高的丙烷产量。结果表明,对于CvPAS变体G462V,同时添加Triton X-100和蔗糖(未被E.coli代谢)导致丙烷产量大幅增加。但是,野生型酶未见该效果。
表2野生型和变异CvPAS的体内丙烷生产
另外的实验用添加剂乙酰乙酸乙酯进行,乙酰乙酸乙酯被认为是E.coli中丁酸转运体的一种刺激因子[21]。在这种情况下,添加了10mM乙酰乙酸乙酯的CvPAS的G462V变体生成32.0±3.2mg/L丙烷。
工艺的商业应用通常是最大化产品产量和最小化成本之间的平衡。对于CvPAS介导的丙烷生产,这需要使用体内生物工厂而非纯化的酶。这将影响丙烷的产率,因为体内的光通路将受到损害,而高浓度丁酸的存在可能会影响培养物的pH值和细胞活力。因此,选择了可靠的嗜盐单胞菌(Halomonas)作为生物工厂宿主。
使用无缝克隆(In-Fusion cloning)(Clontech),将编码CvPAS
通过对CvPAS质粒和pSEVA431的变体形式进行限制性NcoI/XhoI双重消化生成构建体pHPAS
将E.coli菌株S17-1中的pHPAS
嗜盐单胞菌(Halomonas)菌株中的丙烷生产可通过如下修改的E.coli通用方案来进行:将培养物在含有大观霉素(50μg/mL)和较大接种量(5%;OD600=0.1-0.3)的YTN6培养基中,以180rpm、37℃生长5小时。用IPTG(0.1mM)诱导重组蛋白表达,其细胞密度高于E.coli培养物(OD~1.6)。体内丙烷生产过程的其余部分如上所述进行。
表3提供了该实验的结果。
表3嗜盐单胞菌(Halomonas)st.XV12中CvPAS野生型和G462V变体的体内丙烷生产
尽管E.coli中G462V变体最初产生的丙烷水平低于野生型产生的丙烷水平,但G462V变体的产量仍显著较高。
研究了变化的光强对变体酶的丙烷产量的影响。
起子培养物(5mL)在含有大观霉素(50μg/mL)的YTN6(0.5%酵母提取物+1%胰蛋白胨+6%NaCl,pH为9.0)上37℃、200rpm生长过夜。将培养物(20mL)在含有相同抗生素和发酵剂(5%;OD600=0.1-0.3)的YTN6中以200rpm、30℃生长5小时(OD~1.6)。加入IPTG(0.1mM)和丁酸(1mM),将等分试样(5mL)密封在20mL反应瓶中,在具有不同程度的遮蔽的明亮的白光存在下以200rpm、30℃孵育18小时(平放在摇床上)。使用Al
该实验的结果提供在表4中。丙烷的产生似乎强烈地依赖于光强,但是所使用的光的水平似乎未使酶的表达水平饱和。预期更高水平的光将会得到更高的产量。
表4嗜盐单胞菌(Halomonas)在不同光强下的体内丙烷生产
研究了不同丁酸盐利用率对变体酶的丙烷产量的影响。
起子培养物(5mL)在含有大观霉素(50μg/mL)的YTN6(0.5%酵母提取物+1%胰蛋白胨+6%NaCl,pH为9.0)上37℃、200rpm生长过夜。将培养物(20mL)在含有相同抗生素和起子培养物(5%;OD
结果如表5所示。丙烷的产生强烈依赖于丁酸盐的浓度,最大值约为20mM。在1M丁酸存在下生长的培养物发生细胞裂解。
表5不同丁酸盐浓度下的体内丙烷生产
开发经济上可行的微生物生物燃料
丙烷是一种理想的生物燃料。这种烃气体是一种高效且清洁燃烧的燃料,具有低碳排放量。目前,它的来源是天然气和石油提炼
ADO不佳的催化性能刺激了替代生物催化剂的研究。最近报道了一种新型的脂肪酸光脱羧酶(FAP)类光酶,该酶催化蓝光依赖的脂肪酸脱羧成正构烷烃或正构烯烃
描述了利用工程化光敏FAP变体进行生物丙烷生产技术的设计和实施,作为“实地”理念的一个证明。该技术使用生物材料废料和可以在海水中非无菌条件下繁殖的微生物产气底盘。这些功能是本地化、低成本“实地”生产所期望的。我们着手进行酶的重新设计、底盘选择以及工艺开发和优化,以提高生产率,并降低资金成本和运营成本。我们的方法还扩展到生产生物丁烷和其他气态烃,以显示如何将完整的生物路线设计成生物生产丙烷和可调合混合燃料(bio-LPG)。生物基燃料最终可以替代石化衍生的丙烷、丁烷和液化石油气(LPG)混合物。本地燃料生产可能取代石化和精炼厂提供的液化石油气供应,在乡村和/或干旱地区中将特别具有吸引力。
用于Bio-LPG生产的光活化生物催化剂
制备表达来自Chlorella variabilis NC64A(CvFAP)和其他7种蓝细菌同系物的可诱导的、成熟的N端His
CvFAP
最初的靶标是残基G462,该残基突变为10个其他氨基酸(V、N、W、L、C、I、F、A、H和Y)。用表达不同变体的生长的细胞进行丙烷生产研究。在相同条件下,显示与野生型相比,CvFAP
使用Autodock Vina
最终,规模化的生产设备将需要使用在发酵条件下的活细胞,并且确定物理参数(例如所用生物催化剂的光强和波长)的范围很重要。由于FAP被光激活,暴露于酶的光的波长和范围会影响气体的产生。在玻璃小瓶中也研究了E.coli活细胞中各种光源对丙烷生产的影响。使用的光源包括冷白色(25W;2060流明)和蓝色LED(1020mW时455nm或253mW时470nm)。在这些活细胞测定中,尽管与455nm(0.13±0.03mg/L)和白色LED(0.07±0.01mg/L)相比光强低,但在470nm处检测到最高的丙烷含量(0.32±0.10mg/L培养物)。这与CvFAP
在活细胞培养中,产气的潜在限制是细胞内CvFAP
有趣的是,观察到的质粒主链(pETM11与pET21b),His
用CvFAP
bio-LPG策略的制定
LPG混合物中最常见的气体是丙烷和正丁烷,但混合物中也可能含有异丁烷、乙烷、乙烯、丙烯、丁烯和异丁烯。LPG气体的确切成分因国家而异,并且可能因季节而异
碳中性光合生物丙烷生产
理想的能源战略是开发可持续的碳中性燃料,从而将其燃烧排放物(CO
天然(微生物)碳捕集解决方案是利用蓝细菌的光合能力将CO
为了检测由CO
规模化的气态烃的光合生物途径将产生可观的废弃生物质,这些废弃生物质又可以用作生产生物燃料(乙醇)的原料。但是,目前的评估表明,后一种处理在经济上并不合理,因为能量投资回报率(Energy return on energy invested,EROEI)不理想
可靠的异养底盘开发
目前市售丙烷的价格相对较低(2018年1月1日约为3.34美元/加仑),因此任何商业生物策略的成功都取决于大幅提高的产量以及降低资金和运营成本。来自自养CO
微生物衍生(生物)化学品生产的主要成本密集型因素是资金成本(例如,具有复杂监控系统的钢基生物反应器)、防止微生物污染(灭菌设备和无菌条件)和运行成本(高能耗曝气、混合和下游处理)。废物处理/处置以及大量清洁水的要求也存在环境问题。为了解决这些问题,我们选择了嗜盐单胞菌(Halomonas)细菌作为下一代生物丙烷底盘,因为事实证明它可以在没有微生物污染的非无菌条件下生长
使用多种生物体特异性的pSEVA质粒制备嗜盐单胞菌(Halomonas)兼容的构建体(pHal1-3)
令人意外的是,与细胞通透性或atoB转运体激动剂的比较研究表明,与在E.coli中的生产相反,丙烷的产量没有显著影响(表S8(图29))。前者可能与在嗜盐条件下生长的细胞壁和磷脂的适应性有关
与E.coli相比,嗜盐单胞菌(Halomonas)培养物对丁酸的耐受性相对较高,在存在缓冲盐的情况下,其最佳浓度为80mM(157.1±17.1mg/L培养物;图16c)。这是迄今为止报道的最高的生物丙烷产量,通过CvFAP
可再生、可持续的饲料库存和可扩展性
商业上可行的微生物生物工艺发展的关键是具有成本效益的可再生和可持续的原料的来源,并证明该工艺的可扩展性。嗜盐单胞菌(Halomonas)菌株的丙烷生产需要在含有高盐的矿物质、维生素和丁酸中的简单碳源(例如糖、甘油)中有氧生长。海水是具有成本效益的天然矿物质和盐汤(3.5%),而澄清的废水流为内陆站点提供了富足的选择。在高碱度下进一步补充海盐到所需的盐度和矿物质含量将有效地对培养基进行消毒,而无需进行灭菌。可以通过自裂解的废啤酒酵母(一种丰富的废品)来提供维生素。具有成本效益的碳源是原始生物柴油废料
我们利用蓝细菌平板光生物反应器(400mL)开发了一种实验室规模的原型策略,其中嗜盐单胞菌(Halomonas)表达了性能最高的pHal2-FAP
令人鼓舞的是,海水和生物柴油废品杂质的存在对培养物的生长仅产生较小的负面影响(图17a),并且在48小时时,两种培养物都达到了大约相同的细胞密度。在粗介质中,丙烷的最大生产速率只有小幅下降(82vs 100mg/g细胞/天;图17b),在诱导后的8-13小时达到峰值产量。因此,鉴于丙烷生产之间只有很小的差异(1.2倍),因此在设计用于可再生生物丙烷生产的大型生物反应器时,我们可以利用廉价的大量废弃生物材料和海水的成本优势。
微生物底盘和/或生物处理的可靠性还取决于长期产量的维持。一旦用质粒携带的可诱导构建体实现了原理证明,该方法的商业潜力将通过开发稳定的、染色体整合的和组成型表达的底盘而得以增强,该底盘能够在延长的发酵时间内维持稳定的丙烷产量。在这项研究中,我们利用了质粒携带的IPTG诱导型构建体,该构建体显示了丙烷产量随时间的一般性稳定下降(图17b)。因此,该生物丙烷/bio-LPG策略的未来应用将需要组成型表达的CvFAP变体的基因组整合,从而消除了对抗生素和蛋白质诱导剂的需求。此外,将这种发酵策略与现有的瓶顶空气提取和丙烷液化“drop-in”技术以及分销基础设施相结合,将加强对气态烃燃料生产具有潜在改变意义的技术的实施。
我们已经证明了CvFAP变体能够利用光而不是烟酰胺辅助因子,从容易获得的脂肪酸中生产各种bio-LPG烃。通过用单一的光活化酶促步骤代替冗长的基于ADO的途径的策略,打开了气态生物燃料生产领域,并具有通过简单地改变光强来严格调节烃产量的优势。从实验室标准E.coli成功过渡到功能强大且经济高效的嗜盐单胞菌(Halomonas)工业底盘,证明了这种新型bio-LPG策略的潜在可扩展性。
光生物反应器战略的“drop-in”性质取决于天然气的内在稳健性和有效的工程能力,使其能够在具有可变碳和/或VFA来源的各种废水中生长。这将能够在任何选定的位置调整光生物催化工艺的设计,从而能够从现有的区域性来源的材料中高效生产挥发性烃。这有效地将我们带入了第五代生物燃料的开发,从而利用现有生产实践中产生的废物流进行了局部的、可靠的非无菌发酵。
体内丙烷生产
平均培养物曝光量由诸如光的“强度”(例如,以mol光子/秒/m
为了使培养物曝光标准化,我们组装了一个定制的LED阵列光源,该光源由480个单独的蓝色LED组成,具有396cm
丁酸利用率
与传统的酶动力学研究不同,这种优化的变体表征方法没有考虑到体内每种变体蛋白的溶解度和辅助因子含量可能存在的差异。但是,这种方法可以洞察总体上哪些不同的变体在发酵条件下显示出最佳性能。
用CvFAP
嗜盐单胞菌(Halomonas)中的体内丙烷生产
有趣的是,尽管在每种构建体中表达了相同的酶,发现嗜盐单胞菌(Halomonas)构建体的培养基中最佳丁酸盐浓度约为80mM(157.1±17.1mg/L培养物;表S4),而使用E.coli构建体时为10mM(17.51±0.98mg/L)。最佳底物浓度的明显差异可能部分归因于培养基中含有磷酸酯缓冲盐,从而抵消了与诱导时加入丁酸有关的pH值变化。在不存在磷酸酯的情况下进行的比较研究表明,最佳浓度约为40mM,这可能是由于在较高的丁酸浓度下,培养液的pH值降至≤5.0。可能影响的其他因素可能是两种生物之间的丁酸盐耐受性(IC50)和摄取率的差异。
材料和设备
所有化学试剂和溶剂均为商购,并且其纯度均为分析纯或更高。培养基组分获自Formedium(Norfolk,UK)。基因测序和寡核苷酸合成均由Eurofins MWG(Ebersberg,Germany)实施。寡核苷酸的序列如表S9(图30)所示。已安装的Hi-Power蓝色LED和LED驱动器来自Thorlabs(New Jersey,USA),波长分别为455nm(输出功率1020mW)和470nm(253mW)。国产白光LED(集成)的功率为25W(2060流明)。光生物反应器是恒温平板FMT 150(400mL;Photon Systems Instruments,Czech Republic),具有整合的培养监控(OD 680nm)、pH和进料控制以及LED蓝光面板(465-470nm;最大PPFD=1648μE光子)。
如前所述,通过两个醛还原酶基因yqhD和ahr/yjgB(BL21(DE3)ΔyqhD/ΔyjgB/Kan
基因合成、亚克隆和诱变
由GeneArt(Thermo Fisher)进行以下N末端截短(ΔN)FAP酶的密码子优化基因合成:来自Chlorella variabilis NC64A12的CvFAP
使用QuikChange完整质粒合成方案(Stratagene)与CloneAmp HiFi PCR预混液(Clontech),通过在pETM11中对野生型构建体进行定点诱变来生成变体CvFAP
分子建模
底物棕榈酸和丁酸使用Autodock vina对接在与棕榈酸结合的CvPAS结构5NCC的晶体结构的A链中
E.coli多酶构建体的制备
双基因构建体CvFAP
集胞囊藻(Synechcocystis)构建体制备
通过使用如前所述的模板pJET-FAP(来自Chlamydomonas reinhardtii的CrFAP)制备编码CvFAP
将质粒转化到含有pRL623动员质粒的E.coliHB101辅助/运载菌株中。使用如前所述的三亲结合方法
嗜盐单胞菌(Halomonas)构建体制备
在每个嗜盐单胞菌(Halomonas)兼容性构建体中,IPTG诱导型P
使用改良的偶联方案将E.coli衍生的质粒插入嗜盐单胞菌(Halomonas)st XV12中
蛋白质表达和裂解物产生
将E.coli st.BL21(DE3)中的野生型FAP-pETM11同源物于含有30μg/mL卡那霉素的LB Broth Miller(500mL;Formedium)中以180rpm、37℃振荡培养,直至OD
将细胞沉淀重悬于裂解缓冲液(1.2-1.7mL/g沉淀;50mM Tris pH为8,其中包含300mM NaCl、10mM咪唑、10%甘油、0.25g/mL溶菌酶、10μg/mL DNase I和1×蛋白酶抑制剂)并超声处理20分钟(开启20s,关闭60s;幅度30%)。通过在4℃下以48000×g离心30分钟来制备无细胞裂解物。使用Precision Plus非预染蛋白质分子量标准(Bio-Rad)在300V下通过SDS PAGE(12%Mini-PROTEAN-TGX免染胶;Bio-Rad)分析裂解物样品的重组蛋白表达。使用EZ Gel Doc(Bio-Rad)可视化蛋白质含量。
烃生产
丙烷生产的体外反应(200μL)由置于密封的玻璃GC瓶中的无细胞裂解物(180μL)和丁酸(0.36-4.5mM)组成。在照射(蓝色LED;455nm)的情况下,将反应液于30℃在180rpm下孵育24小时。使用微型气相色谱仪(Micro GC)分析瓶顶空气的丙烷含量。
通过以下通用方案在E.coli中制备包含pETM11和pET21b体内丙烷生产的CvFAP
为了测定集胞囊藻(Synechcocystis)中的CvFAP
嗜盐单胞菌(Halomonas)菌株中的丙烷生产可通过如下改良的E.coli通用方案进行:将培养物在含有大观霉素(pHal2-FAPG462V;50g/mL)或氯霉素(pHal1-和pHal3构建体;34μg/mL)的磷酸酯缓冲液的YTN6培养基(50mM K
嗜盐单胞菌(Halomonas)发酵
将光生物反应器以高盐甘油培养基在pH 6.8下分批模式设置(5g/L酵母提取物、1g/L甘油、60g/L NaCl、50μg/mL大观霉素和0.2mL/L消泡剂;400mL),以最大搅拌速率于30℃预平衡。加入pHal2-FAP
集胞囊藻(Synechcocystis)发酵
将光生物反应器(400mL)设置为分批模式,将起子培养物在pH 8.0
分析技术
通过使用Agilent 490微型气相色谱仪手动瓶顶空气进样来测定丙烷含量,该色谱仪配Al
使用具有1260ALS自动进样器、TCC SL柱加热器和1260折射率检测器(AID)的Agilent 1260Infinity HPLC,通过HPLC分析水培养代谢物(甘油和丁酸)。在Agilent Hi-Plex H色谱柱(300×7.7mm;5mM H
参考文献:
为了更全面地描述和公开本发明以及本发明所属的技术状况,以上引用了一些出版物。这些参考文献的完整引文如下。所有这些参考文献都包含在本文中。
1.Bugg TDH,Resch MG.Editorial overview:Energy:Prospects for fuels andchemicals from a biomass-based biorefinery using post-genomic chemicalbiology tools.Curr Opin Chem Biol.2015;29:v-vii.
2.Chen R,Dou J.Biofuels and bio-based chemicals from lignocellulose:Metabolic engineering strategies in strain development.Biotechnol Lett.2016;38:213-21.
3.Cheon S,Kim HM,Gustavsson M,Lee SY.Recent trends in metabolicengineering of microorganisms for the production of advanced biofuels.CurrOpin Chem Biol.2016;35:10-21.
4.de Jong E,Jungmeier G.In Industrial biorefineries&whitebiotechnology,eds.Pandey A,Hofer R,Larroche C,Taherzadeh M and NampoothiriM.Place:Elsevier B.V.;2015.pp.3-33.
5.Toogood HS,Scrutton NS.Retooling microorganisms for thefermentative production of alcohols.Curr Opin Biotechnol.2018;50:1-10.
6.Sheppard MJ,Kunjapur AM,Prather KLJ.Modular and selectivebiosynthesis of gasoline-range alkanes.Metab Eng.2016;33:28-40.
7.Zhang L,Liang Y,Wu W,Tan X,Lu X.Microbial synthesis of propane byengineering valine pathway and aldehyde-deformylating oxygenase.BiotechnolBiofuels.2016;9:80.
8.Menon N,Pásztor A,Menon BR,Kallio P,Fisher K,Akhtar MK,et al.Amicrobial platform for renewable propane synthesis based on a fermentativebutanol pathway.Biotechnol Biofuels.2015;8:61-12.
9.Schirmer A,Rude M,Li X,Popova E,del Cardayre S.Microbialbiosynthesis of alkanes.Science.2010;329:559-62.
10.Khara B,Menon N,Levy C,Mansell D,Das D,Marsh ENG,et al.Productionof propane and other short-chain alkanes by structure-based engineering ofligand specificity in aldehyde-deformylating oxygenase.ChemBioChem.2013;14:1204-8.
11.Kallio P,Pásztor A,Thiel K,Akhtar MK,Jones PR.An engineeredpathway for the biosynthesis of renewable propane.Nat Commun.2014;5:4731.
12.Li N,Chang WC,Warui DM,Booker SJ.Biochemistry.2012;51:7908-16.
13.SoriguéD,Légeret B,CuinéS,Blangy S,Moulin S,Billon E,et al.Analgal photoenzyme converts fatty acids to hydrocarbons.Science.2017;357:903-7.
14.Chen X,Yin J,Ye J,Zhang H,Che X,Ma Y,et al.Engineering Halomonasbluephagenesis TD01 for non-sterile production of poly(3-hydroxybutyrate-co-4-hydroxybutyrate).Bioresour Technol.2017;244:534-41.
15.Tan D,Wu Q,Chen J-C,Chen G-Q.Engineering Halomonas TD01 for thelow-cost production of polyhydroxyalkanoates.Metab Eng.2014;26:34-47.
16.Sun CQ,O'Connor CJ,Turner SJ,Lewis GD,Stanley RA,Roberton AM.Theeffect of pH on the inhibition of bacterial growth by physiologicalconcentrations of butyric acid:Implications for neonates fed on suckledmilk.Chem Biol Interact.1998;113:117-31.
17.Tao W,Lv L,Chen G-Q.Engineering Halomonas species TD01 forenhanced poly-hydroxyalkanoates synthesis via CRISPRi.Microb Cell Fact.2017,DOI:10.1186/s12934-017-0655-3:1-11.
18.Zhao H,Zhang HM,Chen X,Li T,Wu Q,Ouyang Q,et al.Novel T7-likeexpression systems used for Halomonas.Metab Eng.2017;39:128-4
19.Lan et al 2013Oxygen-tolerant coenzyme A-acylating aldehydedehydrogenase facilitates efficient photosynthetic n-butanol biosynthesis incyanobacteria.Energy Environ Sci.6(9):2672-2681
20.Hettwer,D.J.and Wang,H.Y.1986Protein release from chemicallypermeabilized Escherichia coli.In Separation,Recovery,and Purification inBiotechnology.ACS Symposium Series,Vol.314pp 2-8.
21.Jenkins,L.S.and Nunn,W.D.1987Genetic and molecularcharacterization of the genes involved in short-chain fatty acid degradationin Escherichia coli:the ato system.J Bacteriol.169(1):42-52
22.Silva-Rocha,R.et al 2013.The Standard European Vector Architecture(SEVA):a coherent platform for the analysis and deployment of complexprokaryotic phenotypes.Nucleic Acids Res.41:D666-D675
23.Zhao,H.et al 2017.Novel T7-like expression systems used forHalomonas.Metab Eng.39:128-140.
24.Simon R,Priefer U,Puhler A(1983)A broad host range mobilizationsystem for in vivo genetic-engineering-transposon mutagenesis in gram-negative bacteria.Bio-Technol 1:784–791.
1.Toogood,H.S.&Scrutton,N.S.Retooling microorganisms for thefermentative production of alcohols.Curr Opin Biotechnol 50,1-10,(2018).
2.Bugg,T.D.H.&Resch,M.G.Editorial overview:Energy:Prospects for fuelsand chemicals from a biomass-based biorefinery using post-genomic chemicalbiology tools.Curr Opin Chem Biol 29,v-vii,(2015).
3.Chen,R.&Dou,J.Biofuels and bio-based chemicals from lignocellulose:metabolic engineering strategies in strain development.Biotechnol Lett 38,213-221,(2016).
4.Cheon,S.,Kim,H.M.,Gustavsson,M.&Lee,S.Y.Recent trends in metabolicengineering of microorganisms for the production of advanced biofuels.CurrOpin Chem Biol 35,10-21,(2016).
5.de Jong,E.&Jungmeier,G.in Industrial Biorefineries&WhiteBiotechnology(eds A.Pandey et al.)Ch.1,3-33(Elsevier B.V.,2015).
6.Zhang,L.,Liang,Y.,Wu,W.,Tan,X.&Lu,X.Microbial synthesis of propaneby engineering valine pathway and aldehyde-deformylating oxygenase.BiotechnolBiofuels 9,80,(2016).
7.Menon,N.et al.A microbial platform for renewable propane synthesisbased on a fermentative butanol pathway.Biotechnol Biofuels 8,61-12,(2015).
8.Zakaria,Z.Y.,Mohamad,N.F.&Amin,N.A.S.Catalysts screening forcatalytic conversion of glycerol to olefins.J Appl Sci 10,1166-1170,(2010).
9.Schirmer,A.,Rude,M.,Li,X.,Popova,E.&del Cardayre,S.Microbialbiosynthesis of alkanes.Science 329,559-562,(2010).
10.Kallio,P.,Pásztor,A.,Thiel,K.,Akhtar,M.K.&Jones,P.R.An engineeredpathway for the biosynthesis of renewable propane.Nat Commun 5,4731,(2014).
11.Sheppard,M.J.,Kunjapur,A.M.&Prather,K.L.J.Modular and selectivebiosynthesis of gasoline-range alkanes.Metab Eng 33,28-40,(2016).
12.Sorigué,D.et al.An algal photoenzyme converts fatty acids tohydrocarbons.Science 357,903-907,(2017).
13.Zhang,W.et al.Hydrocarbon synthesis via photoenzymaticdecarboxylation of carboxylic acids.J Am Chem Soc,jacs.8b12282,(2019).
14.Huijbers,M.M.E.,Zhang,W.,Tonin,F.&Hollmann,F.Light-drivenenzymatic decarboxylation of fatty acids.Angew Chem Int Ed Engl 57,13648-13651,(2018).
15.Khara,B.et al.Production of propane and other short-chain alkanesby structure-based engineering of ligand specificity in aldehyde-deformylating oxygenase.ChemBioChem 14,1204-1208,(2013).
16.Trott,O.&Olson,A.J.AutoDock Vina:Improving the speed and accuracyof docking with a new scoring function,efficient optimization andmultithreading.J.Comput.Chem.31,455–461,(2010).
17.Bao,R.-M.,Yang,H.-M.,Yu,C.-M.,Zhang,W.-F.&Tang,J.-B.An efficientprotocol to enhance the extracellular production of recombinant protein fromEscherichia coli by the synergistic effects of sucrose,glycine,and Triton X-100.Protein Expression Purif 126,9-15,(2016).
18.Matta,M.K.,Lioliou,E.E.,Panagiotidis,C.H.,Kyriakidis,D.A.&Panagiotidis,C.A.Interactions of the antizyme AtoC with regulatory elementsof the Escherichia coli atoDAEB operon.J Bacteriol 189,6324-6332,(2007).
19.Jenkins,L.S.&Nunn,W.D.Genetic and molecular characterization ofthe genes involved in short-chain fatty acid degradation in Escherichia coli:the ato system.J Bacteriol 169,42-52,(1987).
20.Elgas.LPG Gas Mixture of Propane&Butane:Which Gas is Present inLPG,
21.Agency,I.E.Energy Technology Perspectives.48(OECD,2006).
22.Liberton,M.et al.Organization and flexibility of cyanobacterialthylakoid membranes examined by neutron scattering.J.Biol.Chem.288,3632–3640,(2013).
23.Kufryk,G.I.,Sachet,M.,Schmetterer,G.&Vermaas,W.F.J.Transformationof the Cyanobacterium Synechocystis Sp.PCC 6803 as a Tool for GeneticMapping:Optimization of Efficiency.FEMS Microbiol.Lett.206,215-219,(2002).
24.Zang,X.,Liu,B.,Liu,S.,Arunakumara,K.K.I.U.&Zhang,X.OptimumConditions for Transformation of Synechocystis Sp.PCC 6803.J.Microbiol.SeoulKorea 45,241–245,(2007).
25.
26.Jahn,M.et al.Growth of Cyanobacteria Is Constrained by theAbundance of Light and Carbon Assimilation Proteins.Cell Rep.25,478-486,(2018).
27.van Alphen,P.,Abedini Najafabadi,H.,Branco Dos Santos,F.&Hellingwerf,K.J.Increasing the Photoautotrophic Growth Rate of SynechocystisSp.PCC 6803 by Identifying the Limitations of ItsCultivation.Biotechnol.J.13,e1700764,(2018).
28.Atsumi,S.,Higashide,W.&Liao,J.C.Direct photosynthetic recycling ofcarbon dioxide to isobutyraldehyde.Nat.Biotechnol.27,1177–1180,(2009).
29.Yoshikawa,K.,Toya,Y.&Shimizu,H.Metabolic engineering ofSynechocystis sp.PCC 6803 for enhanced ethanol production based on fluxbalance analysis.Bioprocess Biosyst.Eng.40,791–796,(2017).
30.Gao,Z.,Zhao,H.,Li,Z.,Tan,X.&Lu,X.Photosynthetic Production ofEthanol from Carbon Dioxide in Genetically Engineered Cyanobacteria.EnergyEnv.Sci.5,9857–9865,(2012).
31.Yunus,I.S.&Jones,P.R.Photosynthesis-dependant biosynthesis ofmedium chain-length fatty acids and alcohols.Metabolic Engineering 44,81-88,(2018).
32.Yunus,I.S.et al.Synthetic metabolic pathways for photobiologicalconversion of CO2 into hydrocarbon fuel.Metabolic Engineering 49,201-211,(2018).
33.Lehtinen,T.,Virtanen,H.,Santala,S.&Santala,V.Production of Alkanesfrom CO2 by Engineered Bacteria.Biotechnol.Biofuels 11,228,(2018).
34.Cotton,C.A.R.et al.Photosynthetic Constraints on Fuel from Microbes.Front.Bioeng.Biotechnol.3,36,(2015).
35.Tan,D.,Xue,Y.-S.,Aibaidula,G.&Chen,G.-Q.Unsterile and continuousproduction of polyhydroxybutyrate by Halomonas TD01.Bioresour Technol 102,8130-8136,(2011).
36.Tao,W.,Lv,L.&Chen,G.-Q.Engineering Halomonas species TD01 forenhanced polyhydroxyalkanoates synthesis via CRISPRi.Microb Cell Fact,1-11,(2017).
37.Quillaguamán,J.,Guzmán,H.,Van-Thuoc,D.&Hatti-Kaul,R.Synthesis andproduction of polyhydroxyalkanoates by halophiles:current potential andfuture prospects.Appl Microbiol Biotechnol 85,1687-1696,(2009).
38.Ye,J.et al.Pilot scale-up of poly(3-hydroxybutyrate-co-4-hydroxybutyrate)production by Halomonas bluephagenesis via cell growthadapted optimization process.Biotechnol J 13,1800074-1800010,(2018).
39.Silva-Rocha,R.et al.The Standard European Vector Architecture(SEVA):a coherent platform for the analysis and deployment of complexprokaryotic phenotypes.Nucleic Acids Res 41,D666-D675,(2012).
40.Tan,D.,Wu,Q.,Chen,J.-C.&Chen,G.-Q.Engineering Halomonas TD01 forthe low-cost production of polyhydroxyalkanoates.Metab Eng 26,34-47,(2014).
41.Li,T.et al.Engineering of core promoter regions enables theconstruction of constitutive and inducible promoters in Halomonassp.Biotechnol J 11,219-227,(2016).
42.Zhao,H.et al.Novel T7-like expression systems used forHalomonas.Metab Eng 39,128-140,(2017).
43.Vreeland,R.H.,Anderson,R.&Murray,R.G.Cell wall and phospholipidcomposition and their contribution to the salt tolerance of Halomonaselongata.J Bacteriol 160,879-883,(1984).
44.Chozhavendhan,S.et al.in Waste to Wealth.Energy,Environment,andSustainability Vol.41(eds R Singhania,R Agarwal,R Kumar,&R Sukumaran)65-82(Springer Singapore,2017).
45.Quispe,C.A.G.,Coronado,C.J.R.&Carvalho Jr,J.A.Glycerol:Production,consumption,prices,characterization and new trends in combustion.RenewableSustainable Energy Rev 27,475-493,(2013).
46.Kallio,P.,Pásztor,A.,Thiel,K.,Akhtar,M.K.&Jones,P.R.An engineeredpathway for the biosynthesis of renewable propane.Nat.Commun.5,4731,(2014).
47.Datsenko,K.A.&Wanner,B.L.One-step inactivation of chromosomalgenes in Escherichia coli K-12using PCR products.Proc.Natl.Acad.Sci.U.S.A.97,6640-6645,(2000).
48.Guex,N.&Peitsch,M.C.SWISS-MODEL and the Swiss-PdbViewer:Anenvironment for comparative protein modeling.Electrophoresis 18,2714-2723,(1997).
49.Lee,T.S.et al.Biobrick vectors and datasheets:A synthetic biologyplatform for metabolic engineering.Abstracts of Papers of the AmericanChemical Society 239,(2010).
50.Storch,M.et al.BASIC:a new biopart assembly standard foridempotent cloning provides accurate,single-tier DNA assembly for syntheticbiology.ACS Synth.Biol.4,781–787,(2015).
51.J.,E.,A.,V.,M.,M.-P.A.,E.,F.&P.,W.C.Reduction of conjugal transferefficiency by three restriction activities of Anabaena sp.strain PCC7120.J.Bacteriol.179,1998-2005,(1997).
52.R.Y.,S.,R.,K.,M.,M.&G.,C.-B.Purification and properties ofunicellular blue-green algae(Order Chroococcales).Bacteriol.Rev.35,171-205,(1971).
53.Lou,C.,Stanton,B.,Chen,Y.-J.,Munsky,B.&Voigt,C.A.Ribozyme-basedinsulator parts buffer synthetic circuits from genetic context.Nat Biotechnol30,1137-1142,(2012).
54.Strand,T.A.,Lale,R.,Degnes,K.F.,Lando,M.&Valla,S.A new andimproved host-independent plasmid system for RK2-based conjugal transfer.PLoSOne 9,e90372,(2014).
分子克隆实验指南第三版,参见Sambrook,J.,Russel,D.W.Molecular Cloning,ALaboratory Manual.3ed.2001,Cold Spring Harbor,New York:Cold Spring HarborLaboratory Press。
- 由烃井物流生产烃产品物流的系统和方法及烃井物流分离罐
- 由烃井物流生产烃产品物流的系统和方法及烃井物流分离罐
