肽-MHC comPACT
文献发布时间:2023-06-19 09:47:53
交叉引用
本申请要求2018年4月2日提交的U.S.临时申请No.62/651,639的优先权,出于所有目的,其通过引用整体并入本文。
序列表
本申请包含序列表,所述序列表已通过EFS-Web提交,并在此通过引用整体并入本文。在20XX年XX月创建的所述ASCII副本被命名为 XXXXXUS_sequencelisting.txt,且大小为X,XXX,XXX字节。
背景技术
T细胞为适应性免疫的主要介导者。在每个T细胞的独特T细胞受体(TCR)的特异性的指导下,T细胞调节自身免疫,帮助激活B细胞和固有效应子,并以精确靶向方式直接杀死已感染的和癌性细胞。每个TCR识别在靶细胞上由主要的组织相容性复合体(MHC)分子呈递的配体。相关肽 -MHC复合物配体的鉴定在理解对肿瘤和病原体的免疫应答中发挥作用。 MHC复合体配体对于理解对自身和食物性抗原的反应也具有价值。该理解能实现临床有益的免疫疗法(例如,TCR基因转移和疫苗),所述疗法启动,放大或减弱对靶抗原的免疫应答。
突变的“新表位”是对癌症的内源性和工程改造的免疫应答的重要靶标。新表位-反应性TIL存在于内源性谱系(repertoire)中,并在过继转移时消退肿瘤。同样,肿瘤突变负荷预测了CTLA-4或PD-1阻断的临床有效性,表明这些检查点抑制战略通过释放新表位-反应性T细胞而影响肿瘤消退。因为新表位是在肿瘤细胞中的体细胞突变的结果,因此它们通常不由胸腺上皮细胞呈递以诱导中枢耐受。因此,针对这些新表位的T细胞应答是肿瘤特异性的,有可能是高亲和力的,且患者特异性的(即个人化)。从临床立场而言,这提出了一个机会和挑战:新表位是免疫疗法的优秀靶标,但TCR分离方法应是充分高通量的,使其能够以临床有用的规模用于治疗。
对于基础和转化研究两者,存在对快速和稳健的TCR配体发现技术的尚未解决的需要。肽-MHC多聚体使得能够根据它们的TCR的抗原性特异性实现T细胞分选,所述分选是分离用于基因疗法的肿瘤-特异性TCR 中的重要步骤。当前典型的肽-MHC产生方案以目标肽配体的固相合成开始。并行地,在大肠杆菌中异源表达通用β2-微球蛋白和相关MHC I类分子,收获错误折叠的包涵体。将每一肽添加到含有β2M和相关MHC I分子的重折叠反应中。最后,正确重折叠的三重复合体的部分可进行纯化和配制,用于肽-MHC多聚体生产。为了促进平行产生具有许多不同肽配体的特定MHC分子,Schumacher和同事设计了作为条件配体与特定的MHC 分子结合的可光切割的肽。进行了单一重折叠反应,以生成结合至其条件配体的该MHC分子。当暴露于UV光时,条件配体被切割和交换为过量存在的所需肽。能平行进行许多这样的交换反应,实现构建对于该特定的 MHC等位基因的pMHC文库。即使如此,该最先进的技术仍具有挑战性的局限性。首先,在大肠杆菌包涵体中表达的MHC分子的产生,纯化和重折叠是费力的,且产生出较低产量的适当折叠的肽-MHC复合物。其次,在针对患者特异性新表位的个性化按需TCR基因疗法的情况下,商业性肽合成的周转时间(以周计)与最佳时间量程是不协调的。第三,许多预测的配体不能用于通过这种途径筛选T细胞,原因在于肽的生物物理特性(例如疏水性)阻碍了其合成或交换。第四,交换效率通常较差(对大多数预测的HLA-结合肽而言为<50%的交换效率)。正确折叠的交换MHC与错误折叠的未连接MHC的所得混合物导致了多聚体染色具有较低信噪比,当用肽-MHC试剂的多重池筛选T细胞时,问题变得更加严重。第五,对于每一种新MHC等位基因的条件配体的设计和验证是费力的且不稳健的任务。由于MHC基因座是人基因组中最多的等位基因座,因此这是在多种MHC 单倍型患者中实施新表位靶向基因疗法的主要阻碍。总而言之,这些局限性强调了对该领域新技术的需求。本发明公开解决这些局限性的用于产生肽-MHC多聚体的各种组合物和方法。
发明内容
本文描述的方法和组合物能够快速鉴定被免疫应答靶向的抗原,以及分离T细胞,从而分离介导该应答的TCR序列。该快速鉴定是在传染病,肿瘤免疫,自身免疫和免疫疗法领域中研究和药物开发的目标。组合物可以应用来鉴定和分离抗原特异性T细胞,以及它们编码的MHC I限制性 TCR而用于药品开发。抗原特异性T细胞的鉴定和表征也可应用于治疗性处理(前和后)背景下的免疫应答和疾病的诊断,监测和预后中。comPACT 途径可也应用于鉴定MHC II类限制性T细胞应答的靶标。这种反应对自身免疫疾病是重要的,并正在成为癌症免疫疗法的重要组分。此外,由本文描述的方法生成的哺乳动物细胞的comPACT蛋白分泌效率表达数据潜在地可用作适当蛋白折叠的替代度量,并因此用作每个新表位配体对其同源MHC分子的亲和力的替代度量。然后,本技术可提供独特的能力,以针对研究较少的MHC等位基因完善MHC结合算法,从而改进其他靶向免疫疗法,如新表位疫苗。因此,本文公开的组合物将广泛应用于人类健康并具有明显的商业潜力。
在一个方面,本文公开了多核苷酸分子,沿5’至3’方向,其包含(i) 第一通用靶序列,(ii)编码抗原性肽的核苷酸序列,(iii)第二通用靶序列,其与第一通用靶序列不同,(iv)β2微球蛋白(β2M)序列,和(v)多种组织相容性复合体(MHC)等位基因序列。在一个实施方案中,多核苷酸分子沿5’至3’方向包含(i)启动子序列,(ii)第一通用靶序列,其包含如SEQID NO:3 所示的序列,(iii)编码抗原性肽的核苷酸序列,其中,抗原性肽为肿瘤新抗原,(iv)第二通用靶序列,其包含如SEQ ID NO:4所示的序列,(v)β2M 序列,其包含如SEQ IDNO:106所示的序列,(vi)MHC等位基因序列,其包含选自由如SEQ ID NO:109-174所示的序列组成的组的序列,(vii)第一亲和标签序列,其包含如SEQ ID NO:29所示的序列,(viii)蛋白酶切割位点序列,其包含如SEQ ID NO:31所示的序列,和(ix)第二亲和标签序列,其包含如SEQ ID NO:35所示的序列。
在一个实施方案中,抗原性肽选自由肿瘤抗原,新抗原,肿瘤新抗原,病毒抗原,细菌抗原,膦抗原和微生物抗原组成的组。在其它实施方案中,抗原性肽为新抗原。
在一些实施方案中,新抗原是通过分析来自受试者的肿瘤测序数据以鉴定一种或多种体细胞突变而选择的。在一个实施方案中,分析是使用计算机预测算法进行的。在另一个实施方案中,预测算法进一步包括MHC 结合算法,以预测在新抗原与MHC等位基因之间的结合。
在一些实施方案中,MHC等位基因为哺乳动物MHC等位基因。在一些实施方案中,MHC等位基因为人MHC等位基因。在一些实施方案中, MHC等位基因为I类HLA等位基因。在其他的实施方案中,HLA等位基因包括HLA-A,HLA-B或HLA-C等位基因。HLA等位基因为受试者的HLA等位基因。
在一些实施方案中,HLA等位基因选自由HLA-A*01:01, HLA-A*02:01,HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01, HLA-A*32:01,HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01, HLA-A*30:01,HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02, HLA-A*68:02,HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02, HLA-B*39:01,HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01, HLA-B*57:01,HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03, HLA-B*35:01,HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01, HLA-B*53:01,HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03, HLA-B*37:01,HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01, HLA-B*52:01,HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01, HLA-C*07:01,HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02, HLA-C*16:01,HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02, HLA-C*12:02,HLA-C*12:03,HLA-C*14:02,HLA-C*15:02和 HLA-C*17:01组成的组。
在一些实施方案中,HLA等位基因包括选自由SEQ ID NO:109-174 组成的组的序列。
在一些实施方案中,β2M等位基因为哺乳动物β2M等位基因。在一些实施方案中,β2M等位基因为人β2M等位基因。在一些实施方案中,β2M 等位基因包括如SEQ ID NO:106所示的序列。
在一些实施方案中,编码抗原性肽的核苷酸序列的长度在20-60,20-30, 25-35,20-45,30-45,40-60或45-60个核苷酸之间。在一个实施方案中,编码抗原性肽的核苷酸序列的长度在20-30个核苷酸之间。
在一些实施方案中,第一通用靶序列的长度在4-50,4-15,15-40,15-35, 15-30,20-40,25-40或30-40个核苷酸之间。在一个实施方案中,第一通用靶序列的长度在25-35个核苷酸之间。在另一实施方案中,第一通用靶序列的长度为至少约15个核苷酸。在另一实施方案中,其中,第一通用靶序列的长度在4-6个核苷酸之间。在一些实施方案中,第二通用靶序列的长度在4-50,4-15,15-40,15-35,15-30,20-40,25-40或30-40个核苷酸之间。在一个实施方案中,第二通用靶序列的长度在25-35个核苷酸之间。在另一实施方案中,第二通用靶序列的长度为至少约15个核苷酸。在另一实施方案中,第二通用靶序列的长度在4-6个核苷酸之间。
在一些实施方案中,第一和第二通用靶序列包含聚合酶链式反应(PCR) 引物靶序列。在其他的实施例中,第一和第二通用靶序列包含限制酶切割位点。
在一些实施方案中,第一通用靶序列包括如SEQ ID NO:3所示的序列。在一些实施方案中,第二通用靶序列包括如SEQ ID NO:4所示的序列。
在一些实施方案中,第一通用靶序列进一步包括信号序列。在一些实施方案中,信号序列编码包含人生长激素信号序列,hIG1 Kappa轻链信号序列,β2微球蛋白,信号序列或IL2信号序列的信号序列。在一些实施方案中,信号序列包括包含如SEQ ID NO:1,24,26或28所示的序列的序列。在一个实施方案中,信号序列编码人生长激素(HGH)。在一些实施方案中,信号序列包括如SEQ ID NO:1所示的序列。在一个实施方案中,信号序列的长度在40-90,40-60,45-70,50-80,60-90,55-70,60-80或70-80 个核苷酸之间。
在一些实施方案中,多核苷酸序列的3'端,沿5’至3’方向,进一步包含纯化簇序列,所述纯化簇序列包含(i)第一亲和标签序列,(ii)蛋白酶切割位点序列,和(iii)第二亲和标签序列。在一些实施方案中,第一和第二亲和标签选自由Avi标签,链霉亲和素-标签,多组氨酸(His6)标签,FLAG- 标签,HA-标签和Myc-标签组成的组。在一些实施方案中,第一亲和标签包括包含如SEQ ID NO:29所示的序列的序列,而第二亲和标签包括包含如SEQ ID NO:33所示的序列的序列。
在一些实施方案中,蛋白酶切割位点序列选自由TEV切割位点序列,凝血酶切割位点序列,因子Xa切割位点序列,肠肽酶切割位点序列和鼻病毒3C蛋白酶切割位点序列组成的组。在一个实施方案中,蛋白酶切割位点序列包括如SEQ ID NO:31所示的TEV切割位点序列。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码TEV 切割位点,而第二亲和标签编码His6肽。在一个实施方案中,第一亲和标签序列包括如SEQ ID NO:29所示的序列,蛋白酶切割位点包括如SEQ ID NO:31所示的序列,而第二亲和标签包括如SEQ ID NO:33所示的序列。
在一些实施方案中,第二通用靶序列进一步包含第一接头序列。在一个实施方案中,第一接头序列包含如SEQ ID NO:10,14,16或18所示的序列。
在一些实施方案中,多核苷酸进一步包含在β2M序列和MHC等位基因序列之间的第二接头序列。在一个实施方案中,第二接头序列包含如 SEQ ID NO:10或20所示的序列。
在一些实施方案中,多核苷酸进一步包含在MHC等位基因序列和第一亲和标签之间的第三接头序列。在一个实施方案中,第三接头序列包含如SEQ ID NO:12或22所示的序列。
多核苷酸序列的5’端可进一步包含连接至第一通用靶标的5’端的启动子序列。在一些实施方案中,启动子序列为CMV,EF1α或SV40启动子。在一个实施方案中,启动子序列为CMV启动子。在一个实施方案中,启动子序列为CMV启动子序列。
多核苷酸序列的3'端可进一步包含polyA序列。在一些实施方案中, polyA序列为SV40,hGH,BHG或rbGlob polyA序列。在一个实施方案中,polyA序列包含如SEQ ID NO:179所示的bGH polyA序列。
在一个实施方案中,多核苷酸分子沿5’至3’方向包含(i)启动子序列, (ii)第一通用靶序列,(iii)编码抗原性肽的核苷酸序列,(iv)第二通用靶序列,其与第一通用靶序列不同,(v)β2M序列,(v)MHC等位基因序列,(vi)第一亲和标签序列,(vii)蛋白酶切割位点序列,和(viii)第二亲和标签序列。
在另一个方面,本文公开了表达构建体,其包含如本文所述的多核苷酸分子。在一个实施方案中,表达构建体包括质粒或病毒载体。
在另一个方面,本发明描述了宿主细胞,其包含如前描述的多核苷酸分子或表达构建体。在一个实施方案中,多核苷酸被整合到细胞基因组中。在另一个实施方案中,多核苷酸位于染色体外。在一些实施方案中,宿主细胞为哺乳动物细胞。在一些实施方案中,宿主细胞为人细胞。在一个实施方案中,细胞为干细胞,肿瘤细胞,无限增殖化细胞或胎儿细胞。在一些实施方案中,宿主细胞为原核细胞。在一些实施方案中,细胞为大肠杆菌细胞。在一些实施方案中,细胞表达BirA蛋白或其片段。
在又另一个方面,本发明描述了文库,其包含如本文所述的多核苷酸分子或表达构建体,其中,文库包含大于或等于两种不同的多核苷酸分子,其中,每一种不同的多核苷酸分子包括(i)第一通用序列,(ii)编码抗原性肽的核苷酸序列,其中,对于大于或等于两种多核苷酸分子的每一种而言,核苷酸序列不相同,(iii)第二通用靶序列,(iv)β2M序列,和(v)MHC等位基因序列。在一个实施方案中,对于大于或等于两种多核苷酸分子的每一种而言,MHC等位基因序列不相同。在另一个实施方案中,文库包括 20~500种不同的多核苷酸分子。在一些实施方案中,文库包括至少66种不同的多核苷酸分子。
在一些实施方案中,文库包括至少HLA-A*01:01,HLA-A*02:01, HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01,HLA-A*32:01, HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01,HLA-A*30:01, HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02,HLA-A*68:02, HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02,HLA-B*39:01,HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01,HLA-B*57:01, HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03,HLA-B*35:01, HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01,HLA-B*53:01, HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03,HLA-B*37:01, HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01,HLA-B*52:01,HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01,HLA-C*07:01, HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02,HLA-C*16:01,HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02,HLA-C*12:02, HLA-C*12:03,HLA-C*14:02,HLA-C*15:02和HLA-C*17:01等位基因。在一些实施方案中,文库包括至少如SEQ ID NO:109-174所示的序列。
在另一个方面,本发明描述了多肽,沿氨基至羧基末端方向,其包含 (i)第一通用靶肽,(ii)抗原性肽,(iii)与第一通用靶肽不同的第二通用靶肽, (iv)β2M肽,和(v)MHC肽。在一些实施方案中,抗原性肽选自由肿瘤抗原,新抗原,肿瘤新抗原,病毒抗原,细菌抗原,膦抗原和微生物抗原组成的组。在一个实施方案中,抗原性肽为新抗原。
在一些实施方案中,多肽沿5’至3’方向包含(i)启动子肽,其包含如 SEQ ID NO:2所示的序列,(ii)第一通用靶肽,(iii)编码抗原性肽的核苷酸序列,其中,抗原性肽为肿瘤新抗原,(iv)第二通用靶肽,其包含如SEQ ID NO:15或17所示的序列,(v)β2M肽,其包含如SEQID NO:105所示的序列,(vi)MHC肽,其包含如SEQ ID NO:38-103所示的序列,(vii)第一亲和肽,其包含如SEQ ID NO:30所示的序列,(viii)蛋白酶切割位点肽,其包含如SEQ ID NO:32所示的序列,和(ix)第二亲和肽,其包含如SEQ ID NO:36所示的序列。
在一些实施方案中,新抗原是通过分析来自受试者的肿瘤测序数据以鉴定一种或多种体细胞突变而选择的。在一个实施方案中,分析是使用计算机预测算法进行的。在另一个实施方案中,预测算法进一步包括MHC 结合算法,以预测在新抗原与MHC肽之间的结合。
在一个实施方案中,抗原性肽的长度为7-15个氨基酸,7-10,8-9,7, 8,9,10,11,12,13,14或15个氨基酸。
在一些实施方案中,MHC肽为哺乳动物MHC肽。在一些实施方案中, MHC肽为人MHC肽。在一些实施方案中,MHC肽为I类HLA肽。在另一个的实施例中,HLA肽包括HLA-A,HLA-B或HLA-C肽。在一个实施方案中,HLA肽为受试者的HLA肽。在一个实施方案中,HLA肽包括 Y84A或Y84C突变。
在一些实施方案中,β2M肽为哺乳动物β2M肽。在一些实施方案中,β2M肽为人β2M肽。在一些实施方案中,β2M肽包含如SEQ ID NO:105 所示的序列。在另一个实施方案中,β2M肽包括S88C突变。在一些实施方案中,β2M肽包含如SEQ ID NO:107所示的序列,所述序列在第88位氨基酸处包含S88C突变。
在一些实施方案中,HLA肽包括HLA-A*01:01,HLA-A*02:01, HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01,HLA-A*32:01, HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01,HLA-A*30:01, HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02,HLA-A*68:02, HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02,HLA-B*39:01, HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01,HLA-B*57:01, HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03,HLA-B*35:01, HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01,HLA-B*53:01, HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03,HLA-B*37:01, HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01,HLA-B*52:01, HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01,HLA-C*07:01, HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02,HLA-C*16:01, HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02,HLA-C*12:02, HLA-C*12:03,HLA-C*14:02,HLA-C*15:02或HLA-C*17:01。在一些实施方案中,HLA肽包含选自由SEQ ID NO:38-103组成的组的序列。
在一些实施方案中,第一通用靶肽进一步包括信号肽。在一个实施方案中,信号序列的长度在15-45,15-30,20-45,20-30或30-45个氨基酸之间。在一个实施方案中,信号肽包括人生长激素。在一些实施方案中,信号肽包含人生长激素信号肽,hIG1 Kappa轻链信号肽,β2微球蛋白信号肽或IL2信号肽。在一些实施方案中,信号肽包括包含如SEQ ID NO:2, 23,25或27所示的序列的序列。在一些实施方案中,信号肽包括如SEQ ID NO:2所示的人生长激素(HGH)信号肽序列。在一个实施方案中,第二通用靶肽包括序列GGGGSGGGGSGGGGS。在一个实施方案中,第二通用靶肽包含如SEQ ID NO:15或17所示的序列。
在一些实施方案中,多肽的羧基末端在沿氨基至羧基末端方向,可进一步包含纯化簇,所述纯化簇包含(i)第一亲和肽,(ii)蛋白酶切割位点,和 (iii)第二亲和肽。在一些实施方案中,第一和第二亲和肽选自由Avi标签, strep-标签,多组氨酸(His6)标签,FLAG-标签,HA-标签和/或Myc-标签组成的组。在一些实施方案中,第一亲和肽包括包含SEQ ID NO:30的序列,而第二亲和肽包括包含SEQ ID NO:34的序列。
在一些实施方案中,蛋白酶切割位点为TEV切割位点,凝血酶切割位点,因子Xa切割位点,肠肽酶切割位点或鼻病毒3C蛋白酶切割位点。在一些实施方案中,纯化簇包含Avi标签表位,TEV切割位点和His6表位。在一些实施方案中,蛋白酶切割位点包括如SEQ ID NO:32所示的TEV 切割位点序列。在一些实施方案中,纯化簇包括如SEQ ID NO:30所示的 Avi标签肽序列,如SEQ ID NO:32所示的TEV切割位点序列和如SEQ ID NO:34或36所示的His6肽序列。在一些实施方案中,纯化簇包括如SEQ ID NO:34所示的His6肽序列的2个以上拷贝。在一些实施方案中,纯化簇包含如SEQ ID NO:37所示的序列。
在一些实施方案中,第二通用靶肽进一步包含接头,所述接头包含如 SEQ ID NO:9,11,15或17所示的序列。
在一些实施方案中,多肽包含在β2M序列和MHC等位基因序列之间的第二接头序列。在一些实施方案中,第二接头序列包含如SEQ ID NO:9 或19所示的序列。在一些实施方案中,多肽进一步包含在MHC等位基因序列和第一亲和标签之间的第三接头序列。在一些实施方案中,第三接头序列包含如SEQ ID NO:13或21所示的序列。在一些实施方案中,多肽为生物素化的。
在一个实施方案中,多肽在沿氨基至羧基末端方向,包含(i)第一通用靶肽,(ii)抗原性肽,(iii)第二通用靶肽,(iv)β2M肽,(v)MHC肽,(vi)第一亲和标签肽,(vii)蛋白酶切割位点,和(viii)第二亲和标签肽。在一些实施方案中,多肽为生物素化的。
在另一个方面,本文公开了文库,所述文库包含大于或等于两种不同的多肽分子,其中,不同的多肽分子包括(i)第一通用靶肽,(ii)抗原性肽,其中,对于大于或等于两种多肽分子的每一种而言,抗原性肽不相同,(iii) 与第一通用靶肽不同的第二通用靶肽,(iv)β2M肽,和(v)MHC肽。在一个实施方案中,对于大于或等于两种多肽分子的每一种而言,MHC肽不相同。在其它实施方案中,文库包括20~500种不同的多肽分子。在一些实施方案中,文库包括至少66种不同的多肽。
在一些实施方案中,文库包括HLA-A*01:01,HLA-A*02:01, HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01,HLA-A*32:01, HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01,HLA-A*30:01, HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02,HLA-A*68:02, HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02,HLA-B*39:01, HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01,HLA-B*57:01, HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03,HLA-B*35:01, HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01,HLA-B*53:01, HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03,HLA-B*37:01, HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01,HLA-B*52:01, HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01,HLA-C*07:01, HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02,HLA-C*16:01, HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02,HLA-C*12:02, HLA-C*12:03,HLA-C*14:02,HLA-C*15:02和HLA-C*17:01多肽。
在一些实施方案中,多肽附着至颗粒,其中,颗粒为表面,纳米颗粒,珠子或聚合物。在一个实施方案中,多肽经由接头附着至颗粒。在一个实施方案中,颗粒为纳米颗粒,且纳米颗粒为磁性纳米颗粒或聚苯乙烯纳米颗粒。在一个实施方案中,磁性纳米颗粒包括磁性氧化铁。在另一个实施方案中,珠子为琼脂糖(agarose)珠或琼脂糖凝胶(sepharose)珠。在一些实施方案中,附着至颗粒的多肽进一步包含荧光团。在一个实施方案中,荧光团利用或不利用接头地附着至颗粒。
在一些实施方案中,多肽的文库附着至颗粒,其中,文库包含大于或等于两种不同的多肽分子,其中,对于大于或等于两种多肽分子的每一种而言,抗原性肽不相同,且其中,每种不同的多肽附着至颗粒。在一个实施方案中,对于大于或等于两种多肽分子的每一种而言,MHC肽不相同。在一个实施方案中,文库进一步包含独特定义的条形码序列,所述条形码序列与每种不同的多肽的标识可操作地关联。在另一个实施方案中,文库包括20~500种不同的多肽。在另一个实施方案中,文库包括至少66种不同的多肽。
在另一方面,本发明描述了试剂盒,其包含上述实施方案中任一种的组合物和使用说明。
在另一方面,本文公开了多核苷酸分子的制备方法,包括如下步骤:(a) 获得第一多核苷酸序列,沿5’至3’方向,所述第一多核苷酸序列包含(i) 包含限制位点的第一通用靶序列,(ii)与第一通用靶序列不相同的,包含限制位点的第二通用靶序列,(iii)β2M序列,和(iv)MHC等位基因序列;(b) 获得第二多核苷酸,沿5’至3’方向,所述第二多核苷酸包含(i)第一通用靶序列的一部分,(i)编码抗原性肽的序列,和(iii)第二通用靶序列的一部分;(c)获得第三多核苷酸,沿5’至3’方向,所述第三多核苷酸包含(i)第二通用靶序列的一部分的反向互补序列,(ii)编码抗原性肽的序列的反向互补序列,和(iii)第一通用靶序列的一部分的反向互补序列;(d)将第二和第三多核苷酸混合在一起,使得互补序列退火;(e)利用至少一种限制酶对第一多核苷酸序列进行限制性消化;和(f)通过将经消化的第一多核苷酸和经退火的第二和第三多核苷酸与DNA连接反应试剂混合在一起,将第一和第二多核苷酸连接到一起。
在一些实施方案中,方法进一步包括在互补序列退火后将第二多核苷酸和第三多核苷酸的5’核苷酸进行磷酸化,其中,磷酸化包括将第一或第二和第三多核苷酸与酶孵育。在一些实施方案中,酶为T4激酶。在一些实施方案中,第一多核苷酸为表达构建体。在一些实施方案中,包括将制备的多核苷酸插入到表达构建体中。
在一些实施方案中,第一多核苷酸进一步包含启动子序列。在一个实施方案中,启动子序列为CMV,EF1α或SV40启动子。
在另一个实施方案中,第二多核苷酸序列的3’端,沿5’至3’方向,进一步包含纯化簇序列,所述纯化簇序列包含(i)第一亲和标签序列,(ii)蛋白酶切割位点序列,和(iii)第二亲和标签序列。
在一个实施方案中,第二多核苷酸序列的3’端进一步包含polyA序列。
在一些实施方案中,获得第三和第四多核苷酸序列的步骤进一步包括获得来自受试者的肿瘤测序数据的预测数据集。在一个实施方案中,获得第三和第四多核苷酸序列的步骤进一步包括从肿瘤测序数据预测编码抗原性肽的序列。在另一个实施方案中,获得第三和第四多核苷酸序列的步骤进一步包括基于预测的序列合成编码抗原性肽的多核苷酸。
在一些实施方案中,方法进一步包括制备文库,所述文库包含大于或等于两种不同的多核苷酸分子,其中,不同的多核苷酸分子包括(i)第一通用靶序列,(ii)编码抗原性肽的核苷酸序列,其中,对于大于或等于两种多肽分子的每一种而言,抗原性肽不相同,(iii)第二通用靶序列,其与第一通用靶序列不同,(iv)β2M肽,和(v)MHC等位基因序列。在一个实施方案中,对于大于或等于两种多核苷酸分子的每一种而言,MHC等位基因不相同。在另一个实施方案中,文库包括40~500种不同的多核苷酸分子。
在一些实施方案中,方法进一步包括将制备的多核苷酸插入到表达构建体中。在一个实施方案中,方法进一步包括制备文库,所述文库包含大于或等于两种包含不同的多核苷酸分子的表达构建体,其中,不同的多核苷酸分子包括(i)第一通用靶序列,(ii)编码抗原性肽的核苷酸序列,其中,对于大于或等于两种多核苷酸分子的每一种而言,核苷酸序列不相同,(iii) 第二通用靶序列,其与第一通用靶序列不同,(iv)β2M序列,和(v)MHC 等位基因序列。在一个实施方案中,对于大于或等于两种多核苷酸分子的每一种而言,MHC等位基因不相同。在另一个实施方案中,文库包括 20~500种不同的表达构建体。在一些实施方案中,文库包括至少66种不同的多核苷酸分子。在一些实施方案中,MHC等位基因选自由SEQID NO:109-174所示的序列组成的组。
在一些实施方案中,方法进一步包括从多核苷酸分子表达多蛋白分子。在一些实施方案中,多核苷酸被转染或转导入细胞。在一个实施方案中,多核苷酸被整合到细胞基因组中。在另一个实施方案中,多核苷酸在细胞中保持在染色体外。在一些实施方案中,宿主细胞为哺乳动物细胞或人细胞。在一个实施方案中,细胞为干细胞,肿瘤细胞,无限增殖化细胞或胎儿细胞。在一些实施方案中,细胞为原核细胞。在一些实施方案中,细胞为大肠杆菌细胞。
在一个实施方案中,方法进一步包括定量多蛋白的表达。在一些实施方案中,表达和非表达多蛋白序列用于完善所述方法的抗原性肽预测分析步骤。在一个实施方案中,方法进一步包括使所述多蛋白生物素化。
在一些实施方案中,方法包括使多蛋白纯化。纯化步骤可包含亲和色谱。在一个实施方案中,亲和色谱包括固定化金属亲和色谱,包含支持物,螯合剂和二价金属。用于亲和色谱的螯合剂可以为次氮基三乙酸 (nitrolotriacetic acid)(NTA)或亚氨基二乙酸(iminidiacetic acid)(IDA)。在一些实施方案中,二价金属选自由镍(Ni),钴(Co),铜(Cu)和铁(Fe)组成的组。在一个实施方案中,螯合剂为NTA且二价金属为Ni。在其他的实施例中,支持物选自由琼脂糖(agarose)珠子,琼脂糖凝胶珠子和磁珠组成的组。在一些实施方案中,方法进一步包括定量经纯化的多蛋白的生物素化水平。
在一些实施方案中,方法进一步包括使多肽附着至颗粒,其中,颗粒为表面,纳米颗粒,珠子或聚合物。在一个实施方案中,多肽经由接头附着至颗粒。在一个实施方案中,颗粒为纳米颗粒,且纳米颗粒为磁性纳米颗粒或聚苯乙烯纳米颗粒。在一个实施方案中,磁性纳米颗粒包括磁性氧化铁。在另一个实施方案中,珠子为琼脂糖珠或琼脂糖凝胶珠。在一些实施方案中,附着至颗粒的多肽进一步包含荧光团。在一个实施方案中,荧光团利用或不利用接头附着至颗粒。
在另一个实施方案中,方法进一步包括产生文库,所述文库包含大于或等于两种不同的多蛋白,所述多蛋白附着至至少一个颗粒,其中,不同的多肽包括(i)第一通用靶肽,(ii)抗原性肽,其中,对于大于或等于两种多肽分子的每一种而言,抗原性肽不相同,(iii)与第一通用靶肽不同的第二通用靶肽,(iv)β2M肽,和(v)MHC肽。在一个实施方案中,对于大于或等于两种多肽分子的每一种而言,MHC肽不相同。在另一个实施方案中,文库包括20~500种不同的单一多蛋白。在另一个实施方案中,文库包括至少66种不同的单一多蛋白。在另一个实施方案中,MHC肽选自由SEQ ID NO:39-104组成的组所示的序列。
在另一个方面,本文公开了多核苷酸分子的制备方法,包括如下步骤:(a)获得第一多核苷酸序列,沿5’至3’方向,所述第一多核苷酸序列包含(i)包含限制位点的第一通用靶序列,(ii)与第一通用靶序列不相同的,包含限制位点的第二通用靶序列,(iii)β2M序列,和(iv)MHC等位基因序列; (b)获得第二多核苷酸,沿5’至3’方向,所述第二多核苷酸包含(i)第一通用靶序列,(i)编码抗原性肽的序列,和(iii)第二通用靶序列;(c)利用至少一种限制酶对第一多核苷酸序列施行限制性消化;(d)利用至少一种限制酶对第二多核苷酸序列施行限制性消化;和(e)通过将经消化的第一和第二多核苷酸与DNA连接反应试剂混合在一起,将第一多核苷酸与第二多核苷酸连接到一起。
在另一个方面,本发明描述了多核苷酸分子的制备方法,包括如下步骤:(a)获得第一多核苷酸序列,其包含第一通用靶序列;(b)获得第二多核苷酸序列,沿5’至3’方向,所述第二多核苷酸序列包含(i)第二通用靶序列, (ii)β2M序列,和(iii)MHC等位基因序列;(c)获得第三多核苷酸序列,沿 5’至3’方向,所述第三多核苷酸序列包含编码抗原性肽的序列和第二通用靶序列;(d)获得第四多核苷酸序列,沿5’至3’方向,所述第四多核苷酸序列包含抗原性肽的反向互补序列和第一通用靶序列的反向互补序列;(e) 将第一,第二,第三和第四多核苷酸合并在溶液中;(f)添加聚合酶链式反应(PCR)试剂;和(g)进行PCR反应,其中,在第二多核苷酸和第三多核苷酸中的第二通用靶序列的互补区彼此退火,并且在第一多核苷酸和第四多核苷酸中的第一通用靶序列的互补区彼此退火;并且其中,经退火的序列提供用于PCR延伸和扩增反应引发(priming)序列。
本文还公开了用于分离抗原特异性T细胞的方法,所述方法包括如下步骤:(a)提供多肽,沿氨基末端至羧基末端方向,所述多肽包含(i)第一通用靶肽,(ii)抗原性肽,(iii)与第一通用靶肽不同的第二通用靶肽,(iv)β2M 肽,和(v)MHC肽,其中,多肽连接至一种颗粒;(b)提供已知或怀疑包含一种或多种T细胞的样品;(c)将多肽与样品接触,其中,接触包括提供足够使单个T细胞与附着至颗粒的多肽进行结合的条件,和(d)分离与颗粒结合的单个T细胞。
附图说明
结合如下说明和附图,将更好地理解所公开的组合物和方法的这些和其他特征,方面和优点,其中:
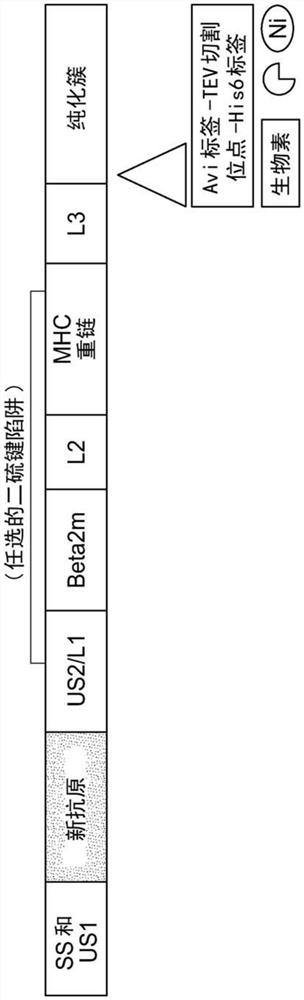
图1示出示例性的comPACT小基因的设计。SS指任选的信号序列; US1指第一通用靶位点;新抗原指抗原性肽序列位点;US2指第二通用靶位点;L1指任选的第一接头序列;β2M指β-2-微球蛋白结构域序列;L2 指任选的第二接头序列;MHC重链指MHC重链等位基因;L3指任选的第三接头序列;以及纯化簇指任选的纯化簇,所述纯化簇具有生物素化序列,蛋白酶切割位点和亲和标签序列。
图2示出示例性模块化的、现成的平台的图,所述平台用于迅速装配与选定的MHC等位基因复合的抗原性肽配体的文库。
图3为示例性限制性消化克隆反应的图,所述反应利用选定的新表位序列替换在MHC模板中的虚拟插入物。虚拟插入物(下划线,粗体)包含在不同框中的四个终止密码子和独特限制位点,所述独特限制位点用于破坏未切割或重连接的模板。将所述插入物的任一侧的限制位点显示在盒中。
图4为示例性限制性消化克隆反应的图,所述反应在MHC模板中插入选定的新表位序列。新表位序列(下划线,粗体)被合成为侧翼为两个不同限制位点(带框)的引物。在PCR反应中使用了通用引物以形成新表位序列的双链引物二聚体,所述通用引物具有3’限制位点的反向互补序列。新表位和MCH模板载体两者的限制性消化允许将选定的新表位序列插入至 MHC模板序列中的连接反应。将连接反应转化到大肠杆菌中,并将从转化的大肠杆菌制备的质粒用于哺乳动物生产细胞转染反应中。
图5为限制性消化克隆反应的示例性可替换形式的图,以将选定的新表位序列插入至MHC模板中。合成两个互补NeoE-编码引物,所述引物具有5’和3’限制位点的一部分。这些引物经退火并模拟来自限制性消化的突出端。然后将预切割载体(在其突出端关键保留5’磷酸酯)与退火的NeoE 插入物进行连接,并将连接产物转化入大肠杆菌中,用于产生质粒。
图6为示例性的基于PCR的方法的图,以在MHC模板中插入选定的新表位序列。合成了两个互补NeoE-编码引物,正向引物具有用于在MHC 模板中的第二通用位点的3’序列;且反向引物具有用于在MHC模板中第一通用位点的互补序列的3’序列。这些引物与具有第一通用序列位点的 MHC模板的5’片段,以及具有第二通用位点的MHC模板的第二片段和 comPACT小基因的剩余物混合。第一PCR扩增循环产生两个核苷酸片段,一个片段编码具有下游新表位的第一通用位点区,另一个编码新表位然后是comPACT基因的剩余物。然后,在独特新表位序列重叠的这两个片段被组装,扩增并清理完整的组装体用于转染。
图7示出了在7天的时间过程中,用comPACT基因(Neo12)转染的30 mL哺乳动物细胞中的总蛋白表达,以及使用检测His-标签的NTA-HRP 试剂的蛋白质印迹。
图8示出Neo12 comPACT蛋白的Ni-NTA亲和色谱纯化的凝胶。Pre 代表粗裂解物,FT代表流通,W代表洗涤且E代表洗脱。
图9示出经纯化的Neo12蛋白的尺寸排阻层析图谱。主峰为Neo12 蛋白,次峰为在生物素化步骤期间添加的ATP。
图10示出与在图8中示出的相似的纯化试验,使用了0.7细胞培养体积。
图11示出8种不同的NeoE comPACT蛋白质的粗品和纯化蛋白,所述蛋白质每一种具有不同的抗原性序列。
图12示出图11的8种NeoE comPACT蛋白的尺寸排阻层析图谱。
图13A示出了与从质粒生产的NeoE comPACT蛋白相比,使用在图6 中描述的PCR组装方法(线性扩增子)生产的NeoE comPACT蛋白。图13B 示出通过PCR组装方法生产的线性扩增子的DNA凝胶。每一泳道包含具有不同的新表位序列的comPACT小基因。
图14示出用于测试comPACT蛋白的完全生物素化的链霉亲和素珠子下拉(pulldown)分析。
图15示出在粗细胞裂解物中的不同comPACT蛋白质的生物素化,使用链霉亲和素-HRP通过蛋白质印迹进行可视化。
图16A-C示出在大肠杆菌中的BirA酶(16B和16C)和TEV蛋白酶(16A) 的产生和纯化。
图17示出使用BirA(泳道2)的comPACT蛋白的生物素化,和使用TEV 蛋白酶(泳道3)的His6标签切割。将未经处理的comPACT蛋白示于泳道1 中。
图18示出基于V5表达的,利用BirA和V5转导的细胞的细胞分选。
图19示出了使用多聚化的comPACT蛋白的T细胞的抗原特异性捕获。
图20示出了使用S88Cβ2M comPACT蛋白的comPACT NTAmer产生。
图21示出Cy5与S88C comPACT蛋白单体的偶联。
图22A示出制备comPACT多核苷酸的克隆策略的示例性图。图22B 提供了从824个单独的comPACT多核苷酸获得的序列验证统计数据。
图23示出用于comPACT蛋白质的代表性选择的蛋白表达和纯化。
图24提供了制备comPACT多核苷酸和蛋白质的工作流程的示例性图。
图25示出了使用BirA的EBV和MART-1 comPACT蛋白的生物素化。
图26A-B示出相对于comPACT HLA谱系大小,被在美国中最高的 HLA I等位基因覆盖的患者百分比。
图27A-C示出了对于comPACT蛋白质的代表性选择而言,comPACT 蛋白单分散性,产量和表达。
图28A-C示出用Cy5和His标签修饰的neo12 comPACT分子与neo12 TCR编辑的T细胞结合。
图29A-D示出Neo12 NTAmer以抗原特异性方式结合T细胞,以及咪唑的存在阻止NTAmer结合。
图30A-D示出MART-1NTAmer以抗原特异性方式结合T细胞。
图31A-C示出了F5 TCR编辑的T细胞在咪唑破坏NTAmer后显示快速的Cy5信号衰减。
详细描述
定义
除非另有说明,权利要求和说明书中使用的术语定义如下。
本文公开的组合物和方法的实施方案包括重组抗原-MHC复合物,其能够与同源T细胞配对。如本文所用,“抗原-MHC,”“抗原-MHC复合物,”“重组抗原-MHC复合物,”“肽MHC,”和“p/MHC,”可互换地用于指在 MHC的抗原结合槽中带有肽的主要组织相容性复合体。
如本文所用,"抗原"包括任何包括患者特异性新抗原的抗原。“抗原性肽”指能够结合MHC分子的肽或肽片段。“新抗原”指具有至少一个改变的抗原,所述改变使得所述新抗原或新抗原的呈递与其相应的野生型抗原不同,例如在多肽序列中的突变,翻译后修饰的差异,或表达水平的差异。“肿瘤新抗原”指衍生自肿瘤或癌,例如,来自患者的肿瘤的新抗原。
如本文所用,“多核苷酸”可指ssDNA,dsDNA,ssRNA,dsRNA或 mRNA。本领域技术人员能够例如基于多核苷酸使用的背景来理解所指的是哪种形式。
术语“体内”指发生在活的生物体(包括细胞)中的过程。
如本文所用,术语“哺乳动物”包括人类和非人类两者,包括但不限于人类,非人灵长类,犬科,猫科,鼠科,牛,马和猪。
在2个以上的核酸或多肽序列的背景下,术语百分比“同一性”指2 个以上的序列或子序列,当对于最大对应(correspondence)进行比较和比对时,它们具有相同核苷酸或氨基酸残基的指定百分比,如使用下述序列比较算法之一(例如,BLASTP和BLASTN或本领域技术人员可获得的其他算法)或通过目检所测量的。取决于应用,百分比"同一性"能够存在于进行比较的序列的区上,例如,功能结构域上,或者,存在于将要进行比较的两种序列的全长上。
对于序列比较,通常以一个序列作为参考序列,将测试序列与其进行比较。当使用序列比较算法时,将测试和参考序列输入计算机,如有必要,子序列坐标是指定的,且序列算法程序参数是指定的。然后,基于指定的程序参数,序列比较算法计算测试序列相对于参考序列的百分比序列同一性。
用于比较的序列的最佳比对能够例如通过以下来进行:例如,通过 Smith&Waterman,Adv.Appl.Math.2:482(1981)的局部同源性算法,通过 Needleman&Wunsch,J.Mol.Biol.48:443(1970)同源性比对算法,通过 Pearson&Lipman,Proc.Nat'l.Acad.Sci.USA85:2444(1988)的寻找相似性方法,通过这些算法(GAP,BESTFIT,FASTA和TFASTA在Wisconsin Genetics软件包中,遗传计算机组,575Science Dr.,Madison,Wis.)的计算机化执行,或通过目检(通常见Ausubel等.,infra)。
适于确定百分比序列同一性和序列相似性的算法的一个实例是描述在Altschul等.,J.Mol.Biol.215:403-410(1990)中的BLAST算法。对用于施行BLAST分析的软件,可通过国家生物技术信息中心(National Center for Biotechnology Information)(www.ncbi.nlm.nih.gov/)公开获得。
必须注意的是,如说明书和所附权利要求中所用,除非上下文明确另有指定,单数形式"a"、"an"和"the"均包括复数引用。
其他解释性约定
本文所列举的范围应理解为在该范围内所有值的简写形式,包括所列举的端点。例如,在1~50的范围理解为包括来自1,2,3,4,5,6,7, 8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23,24, 25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40, 41,42,43,44,45,46,47,48,49和50组成的组中的任一数字,数字的组合,或子范围。例如,1~50的子范围能够包括2-40,5-25和10-20。
核苷酸和肽组合物
T细胞介导的免疫能通过抗原特异性细胞毒性T细胞的活化而被表征,所述T细胞能够诱导在细胞表面上的主要组织相容性复合体(MHC)中展示抗原的细胞发生死亡。这些展示出载有抗原的MHC复合体的细胞包括:病毒感染的细胞,具有胞内细菌的细胞,具有内在化的或吞噬的胞外来源蛋白的细胞,和显示肿瘤抗原的癌细胞。
天然I类MHC重链包括约350个氨基酸;天然β2-微球蛋白包括约 100个氨基酸;且I类抗原肽通常具有约7~约15个氨基酸的长度。I类重链由主要组织相容性复合体的基因编码,在人类中指定为HLA-A,B和C,在小鼠中为H-2K,D和L。I类重链和β2-微球蛋白分别在不同的染色体上编码。抗原肽通常由蛋白质来源的细胞(例如,病毒,细菌或癌细胞)加工。对于由人HLA-A,B和C MHC基因,以及鼠H-2K,D和L MHC基因编码的多肽,已经鉴定了多种变体。
本文公开的方法的实施方案涉及制备单分子的方法,其中,选择的抗原(例如,新抗原)连接至包含β2-微球蛋白(β2M)和MHC重链的MHC复合体。不同的MHC重链能连接至β2M分子,以形成不同数量的MHC模板。本文公开的经由限制性消化或基于PCR的组装,通过利用与抗原插入位点侧接的通用靶序列而将抗原插入至MHC模板中的方法,导致能够以高通量方法构建不同的抗原-MHC复合物的文库,例如对给定的患者是个体化的。这些示例性复合物包括在术语“comPACT”中,并然后可以例如连接至颗粒,条形码(barcoded)的颗粒或表面,用于分离和鉴定靶向于患者特异性新抗原的患者特异性T细胞群体。连接抗原-MHC复合物的方法和这样的复合物的使用公开在2018年3月8日递交的PCT/US2018/021611 中,其整体通过引用并入本文。
肽-MHC复合物
简而言之,如本文所用,“comPACT”指单一多肽融合体,其包括通用靶序列,抗原肽,第二通用靶序列,β2-微球蛋白和MHC I类重链,其中,所述MHC I类重链包含例如,形成MHC展示部分的α1,α2和α3结构域。MHC展示部分可包括重组MHC分子。在某些实施方案中,comPACT能够包含如US公开No.2009/0117153和US公开No.2008/0219947中所述的二硫键陷阱;将其中的每一个通过引用并入本文。通过comPACT形成的抗原-MHC复合物导致抗原的展示,使得它们能够被同源TCR分子识别。在一些实施方案中,MHC复合体可以为与CD8-阳性(CD8+)T“杀伤”细胞配对的MHC I类(MHC I)复合体。在一些实施方案中,MHC复合体可以为与CD4-阳性(CD4+)T细胞配对的MHC II类(MHC II)复合体。每一个 comPACT中编码的MHC等位基因能被容易地换出(swap out)为其他MHC I或II类等位基因,使能够对来自任何MHC单倍型患者的T细胞的抗原询问。
在一些实施方案中,comPACT的MHC I类重链序列可以包括一个或多个氨基酸置换,添加和/或缺失,如用除了脯氨酸之外的非芳族氨基酸置换tyr-84。在这些实施方案中,氨基酸置换可以为通过标准遗传密码子编码的氨基酸,如亮氨酸,异亮氨酸,缬氨酸,丝氨酸,苏氨酸,丙氨酸,组氨酸,谷氨酰胺,天冬酰胺,赖氨酸,天冬氨酸,谷氨酸,半胱氨酸,精氨酸,丝氨酸或甘氨酸,或可以为修饰的或不寻常的氨基酸。在一个实施方案中,comPACT的MHC I类重链序列包含酪氨酸-84到丙氨酸的置换。在另一个实施方案中,comPACT的MHC I类重链序列包括酪氨酸-84 到半胱氨酸的置换。
在一些实施方案中,MHC等位基因缺乏跨膜结构域。在一些实施方案中,MHC等位基因缺乏细胞质结构域。在一些实施方案中,MHC等位基因缺乏跨膜和细胞质结构域。在一些实施方案中,HLA等位基因缺乏跨膜结构域。在一些实施方案中,HLA等位基因缺乏细胞质结构域。在一些实施方案中,HLA等位基因缺乏跨膜和细胞质结构域。
任何MHC或HLA等位基因可以使用在本文描述的comPACT中。示例性HLA等位基因包括但不限于,HLA-A*01:01,HLA-A*02:01, HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01,HLA-A*32:01, HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01,HLA-A*30:01,HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02,HLA-A*68:02, HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02,HLA-B*39:01, HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01,HLA-B*57:01, HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03,HLA-B*35:01, HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01,HLA-B*53:01,HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03,HLA-B*37:01, HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01,HLA-B*52:01, HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01,HLA-C*07:01, HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02,HLA-C*16:01, HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02,HLA-C*12:02,HLA-C*12:03,HLA-C*14:02,HLA-C*15:02和HLA-C*17:01。任何本领域已知的其他合适HLA等位基因可以使用在本文描述的comPACT中。
β2-微球蛋白(β2M)可包括重组β2M分子。在一些实施方案中,β2M 序列能够包括一个或多个上述的氨基酸置换,添加和/或缺失。在一个实施方案中,该置换包括丝氨酸-88到半胱氨酸的置换。
将具有hGH信号序列,虚拟新抗原插入,HLA*A02:01等位基因, Avi标签肽,TEV切割位点和串接的His标签的示例性comPACT蛋白的氨基酸和核苷酸序列示于下表1中。
在一些实施方案中,多核苷酸分子沿5’至3’方向包含(i)启动子序列, (ii)第一通用靶序列,其包含如SEQ ID NO:3所示的序列,(iii)编码抗原性肽的核苷酸序列,其中,抗原性肽为肿瘤新抗原,(iv)第二通用靶序列,其包含如SEQ ID NO:4所示的序列,(v)β2M序列,其包含如SEQ ID NO:106所示的序列,(vi)MHC等位基因序列,其包含选自由如SEQ ID NO:109-174所示的序列组成的组的序列,(vii)第一亲和标签序列,其包含如SEQ ID NO:29所示的序列,(viii)蛋白酶切割位点序列,其包含如SEQ ID NO:31所示的序列,和(ix)第二亲和标签序列,其包含如SEQ ID NO:35所示的序列。在一些实施方案中,多核苷酸分子沿5’至3’方向包含(i)启动子序列,(ii)第一通用靶序列,其包含如SEQ ID NO:3所示的序列,(iii)编码抗原性肽的核苷酸序列,其中,抗原性肽为肿瘤新抗原,(iv)第二通用靶序列,其包含如SEQ ID NO:4所示的序列,(v)β2M序列,其包含如SEQ ID NO:106所示的序列,(vi)MHC等位基因序列,其包含选自由如SEQ ID NO:109-174所示的序列组成的组的序列。在一些实施方案中,多核苷酸分子沿5’至3’方向包含(i)启动子序列,(ii)第一通用靶序列,其包含如SEQ ID NO:3所示的序列,(iii)编码抗原性肽的核苷酸序列,其中,抗原性肽为肿瘤新抗原,(iv)第二通用靶序列,其包含如SEQ ID NO:4所示的序列, (v)β2M序列,(vi)MHC等位基因序列,其包含选自由如SEQ ID NO:109-174 所示的序列组成的组的序列,(vii)第一亲和标签序列,(viii)蛋白酶切割位点序列,和(ix)第二亲和标签。
在一些实施方案中,多肽沿5’至3’方向包含(i)启动子肽,其包含如 SEQ ID NO:2所示的序列,(ii)第一通用靶肽,(iii)编码抗原性肽的核苷酸序列,其中,抗原性肽为肿瘤新抗原,(iv)第二通用靶肽,(v)β2M肽,其包含如SEQ ID NO:105所示的序列,和(vi)MHC肽,其包含如SEQ ID NO:38-103所示的序列。
通用序列
抗原性肽通常被通用靶序列或其部分侧接。这些序列允许快速,高通量的方法,用于在多核苷酸MHC模板中替换或插入编码抗原性肽的核苷酸。通用序列可包含用于基于限制性消化的克隆的限制位点。示例性限制位点包括但不限于NotI,BamHI,BlpI,BspEI,BstBI,Xbal,HindIII, EcoRI,ApaI,NotI,及其的任意组合。在某些方面中,一个或多个通用靶序列不存在于被操作的遗传材料中,例如,以减少或消除脱靶效果和/ 或增加特异性。
通用靶序列的长度可以为4-100,在4-15之间,15-40之间,15-35之间,15-30之间,20-40之间,25-40之间,30-40之间,15-75之间,50-100 之间,50-75之间,75-100之间,25-50之间,40-50之间,50-60之间, 60-70之间,70-80之间,80-90之间或在90-100个核苷酸之间。通用序列的长度可以在至少4,至少10,至少15,至少20,至少25,至少30,至少35,至少40,至少45,至少50,至少60,至少70,至少80,至少90 或至少100个核苷酸。在一些实施方案中,通用靶序列的长度为4-8个核苷酸,例如,4,5,6,7或8个。在其他的实施方案中,通用靶序列的长度在25-35个核苷酸之间,例如,25,26,27,28,29,30,31,32,33, 34或35个核苷酸。在其他的实施方案中,通用靶序列的长度在35-75个核苷酸之间,例如,35,36,37,38,39,40,41,42,43,44,45,46, 47,48,49,50,51,52,53,54,55,56,57,58,59,60,61,62, 63,64,65,66,67,68,69,70,71,72,73,74或75个核苷酸。在其他的实施方案中,通用靶序列的长度为至少约15个核苷酸。在一些实施方案中,多核苷酸包括至少两个不相同的通用靶序列。在一些实施方案中,多核苷酸包括至少两个相同的通用靶序列。
示例性通用靶序列示于表2:
在一些实施方案中,通用靶序列包含聚合酶链式反应(PCR)引物靶序列或引物结合位点。本领域已知的通用引物序列可以使用于本文公开的组合物和方法中,或所述序列可以与之前描述的通用引物序列不同。
在一些实施方案中,第一和/或第二通用靶序列包含限制酶切割位点。
接头
在各种实施方案中,comPACT能够包含介于抗原性肽区段和β2-微球蛋白区段之间的第一柔性接头。这样的接头能够从抗原性肽区段的羧基末端延伸并连接到β2-微球蛋白区段的氨基末端,或反之亦然。在不受理论限制的情况下,当表达comPACT时,连接的肽配体能够折叠进入结合槽中,从而产生功能性comPACT蛋白。在各种实施方案中,该接头可以包含至少约4个氨基酸,多达约20个氨基酸,例如,4,5,6,7,8,9, 10,11,12,13,14,15,16,17,18,19或20个氨基酸。
在各种实施方案中,comPACT能够包含介于β2-微球蛋白区段和MHC 重链区段之间的第二柔性接头。这样的接头能够从β2-微球蛋白区段的羧基末端延伸并连接到重链区段的氨基末端,或反之亦然。在不受理论限制的情况下,当表达comPACT时,β2-微球蛋白和重链能够折叠进入结合槽中,从而产生能够在促进T细胞扩增中发挥功能的分子。在各种实施方案中,该接头可以包含至少约4个氨基酸,多达约20个氨基酸,例如,4, 5,6,7,8,9,10,11,12,13,14,15,16,17,18,19或20个氨基酸。
在各种实施方案中,comPACT能够包含介于MHC重链区段和纯化簇之间的第三柔性接头。这样的接头能够从重链区段的羧基末端延伸并连接纯化簇的氨基末端,或反之亦然。
可以使用本领域已知的任何适当柔性接头序列。这样的接头序列包括但不限于包含GS,SG,GGGGS(G
接头也可以包含用于在接头序列和MHC等位基因序列之间的二硫键的半胱氨酸残基,使得所述半胱氨酸残基形成二硫键陷阱。在一些实施方案中,接头序列或第二通用靶肽包括半胱氨酸残基。在一些实施方案中,接头序列或第二通用靶肽包含与MHC等位基因形成二硫键的半胱氨酸残基。
信号序列
在各种实施方案中,comPACT多核苷酸和多肽可包含信号序列,所述信号序列在例如多核苷酸的情况下编码信号肽。信号序列可以为分泌信号序列。分泌信号序列在哺乳动物细胞中指导经翻译的蛋白质通过分泌通路,并确保经翻译的蛋白质受到细胞质量控制。包含分泌信号能够确保 comPACT蛋白质被分泌进入细胞介质,使得它们被均匀而良好地折叠,并更容易从介质或澄清的上清液中分离。
在一个实施方案中,信号序列为来自人生长激素(hGH)的信号序列。在另一个实施方案中,信号序列为来自hIG1 Kappa轻链,β2微球蛋白(β2M) 或IL2信号序列的信号序列。也可使用额外的信号序列,包括来自β2M(例如,人β2M)的信号序列或本领域已知的任何其他真核或原核信号序列。
信号序列的长度可以在10-100,10-20,10-50,10-40,20-40,20-60, 40-90,40-60,45-70,50-80,60-90,55-70,60-80,70-80,50-100,60-100, 70-100,80-100或90-100个核苷酸之间。信号肽的长度可以在3-33,3-10, 10-30,10-20,15-30或20-30个氨基酸之间。
将示例性信号序列示于表3:
在一些实施方案中,信号序列包括包含如SEQ ID NO:1,24,26或 28所示的序列的序列。在一些实施方案中,信号序列包括包含如SEQ ID NO:2,23,25或27所示的序列的序列。在一个实施方案中,信号序列编码人生长激素(HGH)信号序列。在另一个实施方案中,信号序列包括如SEQ ID NO:1所示的序列。在另一个实施方案中,信号序列包括如SEQ ID NO:2所示的序列。
启动子
comPACT多核苷酸组合物可进一步包含启动子,例如,用于能被宿主细胞翻译的mRNA转录物的转录。启动子的起源可以为原核的或真核的(例如,哺乳动物)。可以使用用于在细胞中基因转录的任何适当启动子,如EF1α,巨细胞病毒(CMV)或SV40。在一些实施方案中,comPACT多核苷酸包含CMV启动子。
在各种实施方案中,多核苷酸序列的5’端进一步包含连接至第一通用靶序列的5’端的启动子序列。在一些实施方案中,启动子序列选自由CMV, EF1α和SV40启动子组成的组。在一个实施方案中,启动子序列为CMV 启动子序列。在一个实施方案中,启动子序列包括包含如SEQ ID NO:37 所示的序列的序列。
亲和标签
comPACT多核苷酸组合物可进一步包含至少一个编码亲和标签或肽的序列。在一些实施方案中,comPACT多核苷酸包括至少两个亲和标签或肽序列。
任何适当亲和标签或肽可以使用于comPACT多核苷酸或多肽中。这样的肽包括但不限于,Avi标签,链霉亲和素-标签,多组氨酸(His6)标签, FLAG-标签,HA-标签和Myc-标签。在多核苷酸comPACT基因中的序列通常被翻译成在comPACT多肽中的肽。这些表位可用于亲和色谱纯化或表达的comPACT多肽的定量。比如,His6标签可用于经由Ni-NTA亲和色谱而纯化comPACT蛋白。在一些实施方案中,Hs6标签可以是串接的 His6标签,其包含多个His6单元,具有任选的接头序列。第一和第二亲和标签可以是相同的,即都是His6标签,或都是HA标签,或它们可以为不同标签,即链霉亲和素-标签和His6标签。
此外,Avi标签编码被BirA酶识别的生物素化位点。在蛋白中包含这种肽序列从而允许序列的生物素化,所述生物素化利用BirA经由酶修饰。因此,包含Avi标签序列和His6标签的comPACT多肽可以被生物素化,利用His6标签经由Ni-NTA亲和色谱进行纯化,并利用链霉亲和素或其他亲和素试剂经由生物素可视化,从而评估经纯化的蛋白的纯度或数量。
在一些实施方案中,comPACT多核苷酸包含Avi标签序列。在一些实施方案中,comPACT多肽包含Avi标签肽。在一些实施方案中,comPACT 多核苷酸包含His6序列。在一些实施方案中,comPACT多肽包含His6肽。在一些实施方案中,comPACT多肽包括串接的His6肽。在一些实施方案中,comPACT多核苷酸包含Avi标签序列和His6序列。在一些实施方案中,comPACT多核苷酸包含Avi标签序列和串接的His6序列。在一些实施方案中,comPACT多肽包含Avi标签肽和His6肽。在一些实施方案中, comPACT多肽包含Avi标签肽和串接的His6肽。
在一些实施方案中,第一和第二亲和标签选自由Avi标签,链霉亲和素-标签,多组氨酸(His6)标签,FLAG-标签,HA-标签和Myc-标签组成的组。
在一些实施方案中,comPACT多核苷酸包含FLAG序列。在一些实施方案中,comPACT多肽包含FLAG肽。在一些实施方案中,comPACT 多核苷酸包含HA序列。在一些实施方案中,comPACT多肽包含HA肽。在一些实施方案中,comPACT多核苷酸包含Myc序列。在一些实施方案中,comPACT多肽包含Myc肽。在一些实施方案中,comPACT多核苷酸包含链霉亲和素序列。在一些实施方案中,comPACT多肽包含链霉亲和素肽。在一些实施方案中,comPACT多核苷酸包含Avi标签序列和FLAG 序列。在一些实施方案中,comPACT多肽包含Avi标签肽和FLAG肽。在一些实施方案中,comPACT多核苷酸包含Avi标签序列和HA序列。在一些实施方案中,comPACT多肽包含Avi标签肽和HA肽。在一些实施方案中,comPACT多核苷酸包含Avi标签序列和链霉亲和素序列。在一些实施方案中,comPACT多肽包含Avi标签肽和链霉亲和素肽。
在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码TEV切割位点,且第二亲和标签编码FLAG肽。在一些实施方案中,纯化簇包含Avi标签肽,TEV切割位点和FLAG肽。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码 TEV切割位点,且第二亲和标签编码HA肽。在一些实施方案中,纯化簇包含Avi标签肽,TEV切割位点和HA表位。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码TEV切割位点,且第二亲和标签编码Myc肽。在一些实施方案中,纯化簇包含Avi标签肽, TEV切割位点和Myc肽。在一些实施方案中,第一亲和标签序列编码Avi 标签肽,蛋白酶切割位点序列编码TEV切割位点,且第二亲和标签编码链霉亲和素肽。在一些实施方案中,纯化簇包含Avi标签肽,TEV切割位点和链霉亲和素肽。
在一些实施方案中,第一亲和标签序列编码His肽。在一些实施方案中,第一亲和标签序列编码FLAG肽。在一些实施方案中,第一亲和标签序列编码HA肽。在一些实施方案中,第一亲和标签序列编码Myc肽。在一些实施方案中,第一亲和标签序列编码链霉亲和素肽。在一些实施方案中,纯化簇包含His肽。在一些实施方案中,纯化簇包含FLAG肽。在一些实施方案中,纯化簇包含HA肽。在一些实施方案中,纯化簇包含Myc 肽。在一些实施方案中,纯化簇包含链霉亲和素肽。
蛋白酶切割位点
comPACT多核苷酸组合物可进一步包含序列,所述序列编码例如在纯化簇中的蛋白酶切割位点。该切割位点可以被编码在第一和第二亲和标签序列之间,并一旦comPACT被表达并经历一轮纯化时允许从comPACT 蛋白中切割第二亲和标签。可以使用本领域已知的任何适当蛋白酶切割位点,包括但不限于被TEV,凝血酶,因子Xa,肠肽酶和鼻病毒3C蛋白酶等识别的切割位点。
在一个实施方案中,蛋白酶切割位点序列为TEV切割位点序列,凝血酶切割位点序列,因子Xa切割位点序列,肠肽酶切割位点序列和/或鼻病毒3C蛋白酶切割位点序列。在一个实施方案中,蛋白酶切割位点核苷酸序列编码TEV切割位点。在另一个实施方案中,comPACT多肽包含TEV 蛋白酶切割位点ENLYFQG。在一个实施方案中,蛋白酶切割位点核苷酸序列编码因子Xa切割位点。在一个实施方案中,蛋白酶切割位点核苷酸序列编码鼻病毒3C切割位点。在一个实施方案中,蛋白酶切割位点核苷酸序列编码肠肽酶切割位点。在一个实施方案中,蛋白酶切割位点核苷酸序列编码凝血酶切割位点。
在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码TEV切割位点,且第二亲和标签编码His6肽。在一些实施方案中,纯化簇包含Avi标签表位,TEV切割位点和His6表位。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码凝血酶切割位点,且第二亲和标签编码His6肽。在一些实施方案中,纯化簇包含Avi标签表位,凝血酶切割位点和His6表位。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码因子Xa切割位点,且第二亲和标签编码His6肽。在一些实施方案中,纯化簇包含Avi标签表位,因子Xa切割位点和His6表位。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码肠肽酶切割位点,且第二亲和标签编码His6肽。在一些实施方案中,纯化簇包含Avi 标签表位,肠肽酶切割位点和His6表位。在一些实施方案中,第一亲和标签序列编码Avi标签肽,蛋白酶切割位点序列编码鼻病毒3C切割位点,且第二亲和标签编码His6肽。在一些实施方案中,纯化簇包含Avi标签表位,鼻病毒3C切割位点和His6表位。
PolyA尾
comPACT多核苷酸组合物可进一步包含聚腺苷酸化(polyA)尾。可以使用哺乳动物,真核或原核polyA序列基序。当comPACT多核苷酸经由 PCR组装用于直接转染入宿主细胞(例如,不在表达构建体或载体背景下) 时,可以包含该序列。任何适当polyA尾巴和序列基序可以用于comPACT 多核苷酸中,包括但不限于SV40,hGH,bGH和rbGlob序列。这样的序列包括RNA序列基序:AAUAA。在一个实施方案中,polyA序列包含bGH polyA序列。在一个实施方案中,polyA序列选自bGH polyA序列,SV40 polyA序列,hGH polyA序列和rbGlob polyA序列。在一个实施方案中, polyA序列包含SV40 polyA序列。在一个实施方案中,polyA序列包含hGH polyA序列。在一个实施方案中,polyA序列包含rbGlob polyA序列。
抗原性序列
抗原性序列的长度可以在5-100之间,5-10之间,10-20之间,10-30 之间,10-40之间,10-50之间,10-60之间,10-70之间,10-80之间,10-90 之间,10-100之间,20-100之间,30-100之间,40-100之间,50-100之间,60-100之间,70-100之间,80-100之间,90-100之间,20-40之间, 20-50之间,20-60之间,20-70之间,20-80之间,20-19之间,20-100之间,20-30之间,25-35之间,20-45之间,30-45之间,30-50之间,30-60 之间,30-70之间,30-80之间,30-90之间,30-100之间,40-50之间, 40-60之间,45-60之间,40-70之间,40-80之间,40-90之间,40-100之间,50-60之间,50-70之间,50-80之间,50-90之间,50-100之间,60-70 之间,60-80之间,60-90之间,60-100之间,70-80之间,80-90之间,80-100之间,或在90-100个核苷酸之间。抗原性序列的长度可以为至少5, 10,15,20,25,30,35,40,45,50,55,60,65,70,75,80,85, 90,95或100个核苷酸。抗原性肽的长度可以在3-50之间,3-10之间, 5-15之间,7-15之间,5-20之间,7-20之间,10-15之间,10-20之间, 15-20之间,20-25之间,20-30之间,25-35之间,30-40之间,或在40-50 个氨基酸之间。抗原性肽的长度可以为5,6,7,8 9,10,11,12,13, 14,15,16,17,18,19或20个氨基酸。抗原性肽可包括肿瘤抗原,新抗原,肿瘤新抗原,病毒抗原,细菌抗原,膦抗原或微生物抗原。在一个实施方案中,抗原性肽为新抗原。抗原性肽可以选自患者数据,以选择具有一个或多个体细胞突变的抗原。抗原性肽的预测可包括预测性算法,并预测抗原性肽或新抗原与HMC等位基因的结合。下面进一步讨论抗原性肽的预测。
在一些实施方案中,编码抗原性肽的核苷酸序列的长度在20-60之间, 20-30之间,25-35之间,20-45之间,30-45之间,40-60之间或在45-60 个核苷酸之间。在其他的实施方案中,编码抗原性肽的核苷酸序列的长度在20-30个核苷酸之间。在一些实施方案中,抗原性肽的长度为7-15个氨基酸,7-10,8-9,7,8,9,10,11,12,13,14或15个氨基酸。在一些实施方案中,抗原性肽的长度为5,6,7,8,9,10,11,12,13,14, 15,16,17,18,19,20,21,22,23,24,25,26,27,28,29或20 个氨基酸。
生物素化
本文所述的comPACT蛋白质可进一步地经由任何适当方法被生物素化。一种这样的方法利用了BirA生物素-蛋白连接酶,其可商购获得。在目标蛋白中编码特异性氨基酸序列,已知称为Avti标签序列 (GLNDIFEAQKIEWHE)。将BirA连接酶,d-生物素和ATP添加到含有目标蛋白的反应混合物中。BirA将生物素与Avi标签序列中的赖氨酸共价连接,因此使目标蛋白生物素化。然后,新的生物素化的蛋白能够被纯化并用于下游应用。也可采用本领域已知的使蛋白质生物素化的其他方法。
在一些实施方案中,comPACT蛋白质在蛋白纯化后用纯化的BirA蛋白进行生物素化。在一些实施方案中,comPACT蛋白质在蛋白纯化期间,在细胞裂解物中用纯化的BirA蛋白进行生物素化。使用纯化的BirA,以及经纯化的或部分经纯化的comPACT蛋白质的这种方法称为“体外”生物素化。在一些实施方案中,comPACT蛋白质在蛋白产生期间在细胞中通过细胞表达的BirA蛋白进行生物素化,所述细胞表达的BirA蛋白在细胞质中,在细胞表面上,或分泌至细胞培养基中。使用细胞表达的BirA,以及未纯化的comPACT蛋白质的这种方法被称为“体内”生物素化。
表达构建体和载体
comPACT多核苷酸分子可以插入至表达构建体或载体中,例如,用于生产质粒和蛋白。表达构建体或载体可以为质粒或病毒载体。可以使用本领域已知的任何合适表达构建体或载体,包括细菌表达质粒,如大肠杆菌或枯草芽胞杆菌(Bacillus subtilis)质粒;真核表达载体,如巴斯德毕赤酵母(Pichia pastoris)表达载体;或病毒载体,如慢病毒载体,痘苗病毒载体或杆状病毒载体。也可考虑用于培养的哺乳动物细胞系,如中国仓鼠卵巢 (CHO),HEK293,Expi293或任何其他合适哺乳动物细胞系中的哺乳动物表达载体。此外,表达构建体或载体可包含核苷酸条形码。核苷酸条形码可以是对于每一个表达构建体或载体而言独特的。在一些实施方案中,可以将编码信号序列,β-2-微球蛋白和MHC等位基因的核苷酸序列连接到具有非编码或虚拟抗原插入物的表达构建体或载体中。然后,可以通过适当的克隆技术(例如限制性消化)去除该非编码抗原插入物,并将所需的抗原序列经由连接或任何其他适当克隆技术进行插入。
在一些方面中,本文提供文库,所述文库包含编码不同的MHC等位基因的大于或等于两种不同的载体。
宿主细胞
在另一个方面中,本文提供宿主细胞,其包含如本文所述的多核苷酸分子或表达构建体。宿主细胞可以为本领域已知的任何合适宿主细胞,包括但不限于细菌细胞如大肠杆菌或枯草芽胞杆菌,或真核宿主细胞如中国仓鼠卵巢(CHO),HEK293,Expi293,HeLa,昆虫细胞系如Sf9或Sf12,或酵母细胞如巴斯德毕赤酵母。宿主细胞也可以稳定表达生物素化酶BirA。宿主细胞可以为原代细胞或无限增殖化细胞。
在一些实施方案中,多核苷酸被整合到细胞基因组中。在一些实施方案中,多核苷酸位于染色体外。在一些实施方案中,宿主细胞为哺乳动物细胞。在一些实施方案中,细胞为人细胞。在一些实施方案中,细胞选自由干细胞,肿瘤细胞,无限增殖化细胞和胎儿细胞组成的组。在一些实施方案中,宿主细胞为原核细胞。在一些实施方案中,细胞为大肠杆菌细胞。在一些实施方案中,细胞表达BirA蛋白或其片段。
文库
也考虑了文库,所述文库包含大于或等于两种不同的comPACT多核苷酸分子,多肽分子或附着至颗粒的多肽分子。文库可包含2~1000个分子。在一些实施方案中,文库包括在2-900,2-800,2-700,2-600,2-500, 2-480,2-400,2-300,2-200,2-100,2-50,2-66,2-48,2-30,2-20,2-19, 10-1000,10-900,10-800,10-700,10-600,10-500,10-480,10-400,10-300, 10-200,10-100,10-50,10-66,10-48,10-30,10-20,20-1000,20-900, 20-800,20-700,20-600,20-500,20-480,20-400,20-300,20-200,20-100, 20-50,20-50,20-66,20-48,20-30,30-1000,30-900,30-800,30-700, 30-600,30-500,30-480,30-400,30-300,30-200,30-100,30-50,30-50, 30-66,30-48,30-40,40-1000,40-900,40-800,40-700,40-600,40-500, 40-480,40-400,40-300,40-200,40-100,40-60,40-50,40-66,40-48, 50-1000,50-900,50-800,50-700,50-600,50-500,50-480,50-400,50-300, 50-200,50-100,50-60,50-66,60-1000,60-900,60-800,60-700,60-600, 60-500,60-480,60-400,60-300,60-200,60-100,70-1000,70-900,70-800, 70-700,70-600,70-500,70-480,70-400,70-300,70-200,70-100,70-80, 70-90,80-1000,80-900,80-800,80-700,80-600,80-500,80-480,80-400, 80-300,80-200,80-100个之间的多核苷酸或多肽分子。在一些实施方案中,文库包括在2-19之间,48-480,48-66之间,66-480之间,220-240 之间,40-60之间,48-66之间,50-70之间,或在60-80个之间的多核苷酸或多肽分子。在一些实施方案中,文库包括至少2,5,10,15,20,25, 30,35,40,45,48,50,55,60,65,66,70,75,80,85,90,100, 110,120,130,140,150,160,170,180,190,200,225,250,275, 300,325,350,375,400,425,450,475,500,525,550,600,562,650,675,700,725,750,775,800,825,850,875,900,925,950, 975或1000个comPACT多核苷酸或多肽分子。在一些实施方案中,文库包括2,10,15,20,24,48,66,100,200,300,400,500,600,700, 800,900或1000个comPACT多核苷酸或多肽分子。分子可以为多核苷酸,多肽或附着至颗粒的多肽。在一些实施方案中,大于或等于两种多核苷酸或多肽分子具有不同的抗原性肽序列。在一些实施方案中,大于或等于两种分子具有不同的抗原性肽序列和不同的MHC分子。
在一些实施方案中,文库包含大于或等于两种不同的多核苷酸分子,其中,每一种不同的多核苷酸分子包括(i)第一通用序列,(ii)编码抗原性肽的核苷酸序列,其中,对于大于或等于两种多核苷酸分子的每一种而言,核苷酸序列不相同,(iii)第二通用靶序列,(iv)β2M序列,和(v)MHC等位基因序列。在一些实施方案中,对于大于或等于两种多核苷酸分子的每一种而言,MHC等位基因序列不相同。
在一个实施方案中,文库包括至少2个或多个的HLA-A*01:01, HLA-A*02:01,HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01, HLA-A*32:01,HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01, HLA-A*30:01,HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02, HLA-A*68:02,HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02,HLA-B*39:01,HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01, HLA-B*57:01,HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03, HLA-B*35:01,HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01, HLA-B*53:01,HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03, HLA-B*37:01,HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01,HLA-B*52:01,HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01, HLA-C*07:01,HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02, HLA-C*16:01,HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02, HLA-C*12:02,HLA-C*12:03,HLA-C*14:02,HLA-C*15:02和 HLA-C*17:01等位基因。在一个实施方案中,文库包括至少HLA-A*01:01, HLA-A*02:01,HLA-A*03:01,HLA-A*24:02,HLA-A*30:02,HLA-A*31:01, HLA-A*32:01,HLA-A*33:01,HLA-A*68:01,HLA-A*11:01,HLA-A*23:01,HLA-A*30:01,HLA-A*33:03,HLA-A*25:01,HLA-A*26:01,HLA-A*29:02, HLA-A*68:02,HLA-B*07:02,HLA-B*14:02,HLA-B*18:01,HLA-B*27:02, HLA-B*39:01,HLA-B*40:01,HLA-B*44:02,HLA-B*46:01,HLA-B*50:01, HLA-B*57:01,HLA-B*58:01,HLA-B*08:01,HLA-B*15:01,HLA-B*15:03, HLA-B*35:01,HLA-B*40:02,HLA-B*42:01,HLA-B*44:03,HLA-B*51:01, HLA-B*53:01,HLA-B*13:02,HLA-B*15:07,HLA-B*27:05,HLA-B*35:03, HLA-B*37:01,HLA-B*38:01,HLA-B*41:02,HLA-B*44:05,HLA-B*49:01, HLA-B*52:01,HLA-B*55:01,HLA-C*02:02,HLA-C*03:04,HLA-C*05:01, HLA-C*07:01,HLA-C*01:02,HLA-C*04:01,HLA-C*06:02,HLA-C*07:02, HLA-C*16:01,HLA-C*03:03,HLA-C*07:04,HLA-C*08:01,HLA-C*08:02, HLA-C*12:02,HLA-C*12:03,HLA-C*14:02,HLA-C*15:02和 HLA-C*17:01等位基因。
在一些实施方案中,文库包含大于或等于两种不同的多肽分子,其中,对于大于或等于两种多肽分子的每一种而言,抗原性肽不相同,且其中,每种不同的多肽附着至颗粒。在一些实施方案中,文库进一步包含独特定义的条形码序列,其与每种不同的多肽的标识可操作地关联。
实施方案可以包括条形码化的多核苷酸,其包含定义的条形码序列。条形码化的多核苷酸可以是提供独特抗原特异性序列的多核苷酸,所述独特抗原特异性序列用于在T细胞分离后的鉴定。因此,每一个独特的 comPACT附着至具有独特定义的条形码序列的颗粒。这允许在给定的抗原和给定的条形码之间进行有效的缔合,所述给定的条形码对所述配对而言是独特的。
条形码化的多核苷酸可以为ssDNA或dsDNA。包含条形码的多核苷酸可以在它们的5’端进行修饰,以包含用于附着至颗粒的附着部分。例如,将包含条形码序列的多核苷酸缀合至生物素分子,以结合至附着于颗粒(例如葡聚糖)的链霉亲和素-核。然而,可以使用任何合适的附着部分用于将多核苷酸与颗粒附着。如本文所述,以及作为本领域技术人员理解的,合适的附着部分对是本领域已知的。附着部分的非限制性实施例包括硫醇,马来酰亚胺,金刚烷,环糊精,胺,羧基,叠氮和炔烃。
颗粒
如本文所用,“纳米颗粒”,或可替换地“颗粒”指能够被特异性分选或分离并且可附着其他实体的基质。在一些实施方案中,纳米颗粒为磁性,例如,用于使用磁体分离。在一些实施方案中,磁性纳米颗粒包括磁性氧化铁。磁性颗粒的实例包括,但不限于Dynabeads
根据某些实施方案,纳米颗粒被附着部分所修饰,所述附着部分用于附着另外的分子。纳米颗粒的修饰包含附着部分,所述附着部分能够与附着至多核苷酸的相应同源(例如,互补)附着部分进行配对(例如,共价结合)。可以使用任何合适的附着部分对,以对纳米颗粒和多核苷酸检测标签进行修饰而用于附着。附着部分对的非限制性实例包括:链霉亲和素/生物素系统,硫醇基(例如,半胱氨酸)和马来酰亚胺,金刚烷和环糊精,氨基和羧基,以及叠氮基和炔基。在一些实施方案中,附着部分能够包含切割部分。在其他的实施方案中,结合至互补同源附着部分的附着部分可以是可逆的,如可还原的硫醇基。在示例性实施方案中,经修饰的纳米颗粒为链霉亲和素包被的磁性纳米颗粒,如1μm纳米颗粒(例如,Dynabeads
颗粒可以为葡聚糖,如经生物素化的葡聚糖或链霉亲和素包被的葡聚糖。经修饰的葡聚糖在Bethune等.,BioTechniques 62:123-130Mar.2017和美国公开号No.2015/0329617中进一步地详细描述,其整体通过引用并入本文。生物素化的comPACT可以被附着至链霉亲和素包被的葡聚糖。
comPACT也能够被组装到四聚体中,所述四聚体包含结合至链霉亲和素核的1,2,3或4个经生物素化的comPACT蛋白质。四聚体也可以包含结合至链霉亲和素核的荧光团,如藻红蛋白(PE)或别藻蓝蛋白(APC)。 MHC I类和II类的四聚体是本领域熟知的。MHC I类四聚体在Burrow SR 等,J Immunol December 1,2000,165(11)6229-6234中进一步地详细描述,而MHC II类四聚体在Nepom GT,J Immunol March 15,2012, 188(6)2477-2482中进一步地详细描述,两者均通过引用整体并入本文。
ComPACT蛋白质也能够组装到多聚体中。在一些实施方案中, comPACT蛋白多聚体可以为二聚体,三聚体、四聚体、五聚体、六聚体,或更高阶的多聚体。在一些实施方案中,多聚体可以包含至少2个或更多个的comPACT蛋白质。在一些实施方案中,多聚体可以包含至少2,3, 4,5,6,7,8,9或10个comPACT蛋白质。
生产方法
抗原预测
为了制备comPACT,初始步骤之一可以包括患者的肿瘤特异性抗原 (例如,新抗原)的鉴定。然后,通过该方法生产的组合物可用于T-细胞介导的免疫过程中,例如用于患者特异性癌症免疫疗法。为了鉴定患者的推定的新抗原(肿瘤或病原体),可以采用计算机预测算法程序,所述程序分析包括全基因组,全外显子组或转录组测序数据的肿瘤,病毒或细菌测序数据,以鉴定相应于推定表达的新抗原的一个或多个突变。此外,能从患者的肿瘤或血液样品确定人白细胞抗原(HLA)分型,并且该HLA信息能与经鉴定的推定的新抗原肽序列一起采用于MHC结合的预测算法中,如由 Fritsch等.,2014,Cancer Immunol Res.,2:522-529所验证的,其全部内容通过引用并入本文。人群中经常发现的HLA也可以包括在新抗原预测算法中,例如在高加索人中,HLA-A*02,24,01;HLA-B*35,44,51; DRB1*11,13,07;在非洲裔巴西人中,HLA-A*02,03,30;HLA-B*35, 15,44;DRB1*13,11,03,和在亚洲人中,HLA-A*24,02,26;HLA-B*40, 51,52;DRB1*04,15,09。也能够使用HLA等位基因的特异性配对。在人群中发现的常见等位基因进一步地描述在Bardi等.(Rev Bras Hematol Hemoter.2012;34(1):25–30.)中。
鉴定新抗原的方法的另外的实例包括:将测序与质谱法和MHC呈递预测(例如,美国公开号No.2017/0199961)组合,和将测序与MHC结合亲和力预测(例如,已公布的美国专利9,115,402)组合。此外,用于鉴定新抗原特异性T细胞是否存在于患者样品中的方法可以与这里所述的方法(例如,如美国公开号No.2017/0003288和PCT/US17/59598中所述的方法,其整体通过引用并入本文)组合使用。这些分析产生了患者候选新抗原肽的排序列表,所述新抗原肽能容易地通过使用用于筛选同源抗原特异性T细胞的常规方法来合成。
引物退火和限制性消化组装
一般来说,comPACT多核苷酸的制备能通过本文公开的步骤和通过公认的重组DNA技术完成,例如,质粒DNA的制备,利用限制性酶的 DNA的切割,DNA的连接,宿主的转化或转染,宿主培养,以及表达融合复合体的分离和纯化。这样的步骤通常是已知的,并公开在标准参考文献如上文的Sambrook等中。
在一些方面中,编码MHC I类重链的DNA能从合适的细胞系,例如人淋巴母细胞获得。在各种构型中,编码I类重链的基因或cDNA可以通过聚合酶链式反应(PCR)或本领域已知的其他手段扩增。在一些方面中, PCR产物也可以包含编码接头的序列,和/或用于连接这样的序列的一种或多种限制性酶位点。
在一些实施方案中,编码comPACT多核苷酸的载体可以通过将编码 MHC重链和β2-微球蛋白的序列连接至编码抗原肽的序列而制备。编码抗原肽的DNA可以通过从天然来源分离DNA或通过已知的合成方法(例如,磷酸三酯方法)获得。参见,例如,寡核苷酸合成(Oligonucleotide Synthesis), IRL Press(M.Gait,ed.,1984)。合成的寡核苷酸也可以使用可商购获得的自动寡核苷酸合成仪制备。编码本文讨论的通用靶序列的DNA序列可以置于编码信号序列的序列和编码抗原性肽的序列之间,而第二通用靶序列可以置于编码抗原肽区段的序列和编码β2-微球蛋白区段的序列之间。在一些实施方案中,所述区段(segment)可以使用连接酶连接。在一些实施方案中,编码抗原肽的序列可以利用合适的多核苷酸激酶进行磷酸化。在一些实施方案中,多核苷酸激酶为T4多核苷酸激酶。可以使用本领域已知的任何适当多核苷酸激酶,包括但不限于T4多核苷酸激酶,其也称为T7 多核苷酸激酶。
PCR组装
在一些方面中,comPACT可以经由聚合酶链式反应(PCR)扩增进行组装。与限制性消化方法相似,编码MHC重链和β2-微球蛋白的DNA可以从合适来源获得。编码选定的信号序列的第二DNA片段也可以从合适来源获得。DNA的片段两者可具有不同的通用靶序列,使得针对一种通用序列的引物不与第二通用序列退火。编码选定的抗原性肽的两种序列可以是合成的;一种正向引物,其在5’端具有抗原性序列,且在3’端具有MHC DNA片段上的通用引物序列的互补序列;和一种反向引物,其在5’端具有选定的抗原性序列的反向互补序列,且在3’端具有来自信号序列片段的通用引物的反向互补序列。利用所有四种DNA片段以及针对5’端的信号序列片段和3’端的MHC等位基因片段的引物的PCR反应将导致两种 DNA片段的扩增,一种在3’端具备信号序列并在5’端具备抗原性序列,以及一种在3’端具有抗原性序列并在3’端具备MHC等位基因。进一步的 PCR扩增循环将允许重叠的抗原性肽序列发生退火并得到单一全长DNA 片段。在一些实施方案中,信号肽片段进一步包含启动子序列。在一些实施方案中,MHC片段进一步包含纯化簇和/或polyA尾巴。
宿主细胞的转染,转导和遗传修饰
comPACT多核苷酸可以经由已知的适当方法插入到宿主细胞中,包括但不限于转染,转导,电穿孔,脂质转染,声孔效应,机械破坏或病毒载体。示例性的转染试剂包括但不限于Expifectamine,Lipofectamine,聚乙烯亚胺(PEI)或Fugene。在一些实例中,Expifectamine用于利用comPACT 多核苷酸来转染哺乳动物细胞。
comPACT多核苷酸可以在宿主细胞中瞬时或稳定表达。在一些实施方案中,comPACT多核苷酸被整合到宿主基因组中。在其他的实施例中, comPACT多核苷酸保持在染色体外。也可以使用本领域已知的任何适当的遗传编辑技术来利用comPACT多核苷酸修饰宿主细胞,所述技术包括 CRISPR/Cas9,锌指核酸酶或TALEN核酸酶。
表达
可以使用许多策略来表达comPACT多蛋白。例如,可以通过已知方法(例如,通过使用限制酶和连接酶)(参见,例如,Sambrook等,同上)将 comPACT掺入合适的载体中。可以基于与克隆方案相关的因素来选择载体。例如,载体能与所使用的宿主相容并具有对宿主而言适当的复制子。合适的宿主细胞包括真核和原核细胞,并可以为能够容易转化并在培养基中表现快速生长的细胞。宿主细胞的实例包括原核生物如大肠杆菌和枯草芽胞杆菌,以及真核生物如动物细胞和酵母,如例如,哺乳动物细胞和人细胞。作为能用作宿主而表达comPACT的哺乳动物细胞的非限制性实例包括J558,NSO,SP2-O,293T,Expi293和CHO。可能的宿主的其他实例包括昆虫细胞如Sf9或Sf12,其能使用常规培养条件生长。见Sambrook等,同上。在各种实施方案中,能使用已知的方法鉴定表达comPACT多肽的细胞。例如,comPACT多肽的表达能通过ELISA或蛋白质印迹测定,所述ELISA或蛋白质印迹使用了针对comPACT的MHC重链部分的抗体探针,或针对亲和标签(如His6,或者如果comPACT已被生物素化,则为链霉亲和素试剂)的抗体。
在一些方面中,comPACT在哺乳动物细胞中表达。在哺乳动物细胞而不是在大肠杆菌细胞中表达蛋白质的优势是多重的。在大肠杆菌细胞中表达的蛋白必须被仔细纯化以除去脂多糖(LPS),在哺乳动物细胞中表达蛋白质不会对经纯化的蛋白质造成LPS污染。此外,哺乳动物细胞更有可能正确折叠哺乳动物蛋白质,因为哺乳动物细胞生产具有正确折叠所需要的正确的翻译后修饰的蛋白质,包括二硫键的正确形成。此外,哺乳动物细胞提供了正确的伴侣蛋白来协助蛋白在内质网或高尔基体中折叠。与在大肠杆菌细胞中表达的蛋白质相比,这导致均匀而良好折叠的蛋白质的纯化增加。
comPACT能够基本上不包含LPS。comPACT能够不包含LPS,例如, comPACT能够不具有可检测的LPS,如使用本领域已知的LPS检测方法所测量的。comPACT可以被糖基化。comPACT可以经由在真核或哺乳动物细胞中表达而被修饰,例如,经由一种或多种翻译后修饰,如糖基化。 comPACT能够包含一种或多种翻译后修饰。comPACT可以(1)基本上不包含LPS,或不包含LPS;并且(2)被糖基化。
示例性的ComPACT工作流程
图24示出comPACT蛋白的组装和表达的示例性概要示意。编码目标新抗原肽序列的正义和反义寡核苷酸是合成的,并退火以形成在5’和3’端具有突出端的双链寡核苷酸,然后其可以被连接到含有β2M基因和 MHC等位基因的质粒中。全长comPACT寡核苷酸可以被扩增到双链扩增子中,并转染到细胞中用于蛋白表达和任选的生物素化。comPACT蛋白可以通过SDS-PAGE评估。然后,可选择ComPACT用于在大肠杆菌中扩大质粒生产。将蛋白生产细胞用选择的质粒进行转染,并从所述生产细胞纯化comPACT用于功能分析。
纯化(色谱法)
表达的comPACT多肽可以通过已知的方法进行分离和纯化。例如,包含His6亲和标签的comPACT可以经由亲和色谱通过通常已知的和公开的步骤在Ni-NTA柱上纯化。此外,含有人HLA序列的comPACT可以通过亲和色谱通过通常已知的和公开的步骤在单克隆抗体-琼脂糖柱上纯化。
T细胞分离
在另一个方面,本文提供分离抗原特异性T细胞的方法,所述方法包括如下步骤:(a)提供多肽,沿氨基末端至羧基末端方向,所述多肽包含(i) 第一通用靶肽,(ii)抗原性肽,(iii)与第一通用靶肽不同的第二通用靶肽, (iv)β2M肽,和(v)MHC肽,其中,多肽连接至一种颗粒;(b)提供已知或怀疑包含一种或多种T细胞的样品;(c)将多肽与样品接触,其中,接触包括提供足够使单个T细胞与附着至颗粒的多肽进行结合的条件,和(d)分离与颗粒结合的单个T细胞。
使用如本文所述的comPACT分离和鉴定患者来源的和抗原特异性的 T细胞可以包括将comPACT蛋白与患者来源T细胞孵育。在一些实施方案中,可以将包含至少两种comPACT的文库与患者来源的T细胞孵育。 T细胞可以使用标准方法制备,所述方法从组织(如血液,淋巴结或肿瘤) 开始。
患者来源的T细胞可以从患者的外周血单核细胞(PBMC)或肿瘤浸润淋巴细胞(TIL)分离。例如,CD4+和CD8+T细胞两者都能使用抗CD4和抗CD8荧光抗体进行标记并从PBMC或TIL中分选出来,带有使用荧光激活细胞分选(FACS)所分选的CD4+和CD8+单阳性细胞的活群体,从而仅分离CD4+或CD8+细胞。在一些实施方案中,对于CD4和CD8两者均为阳性的T细胞可以使用抗CD3荧光抗体接着用FACS进行分离。本领域技术人员能够确定T细胞的类型,以针对所使用的comPACT蛋白的一种或多种类型进行分离。
comPACT或comPACT文库与T细胞悬浮液的孵育允许了颗粒-结合抗原完全地并彻底地暴露于各种T细胞受体。该方法可包括摇动或旋转细胞。在一些实施方案中,comPACT与颗粒结合。
在comPACT或comPACT文库和T细胞孵育后,选择性地分离或选择性地收集结合的comPACT-T细胞复合体。T细胞将有可能结合至相同 comPACT文库元件的许多相同的拷贝,并能够基于这些相互作用进行分离。例如,如果comPACT包括荧光团或附着至具有荧光团的颗粒,则可以将荧光相关细胞分选(FACS)(包括单细胞分选)用于选择性分离T细胞。如果comPACT是附着至磁性颗粒的,则对悬浮液应用磁体能够允许分离与抗原配对的T细胞复合的颗粒,并除去未配对的T细胞。备选地,如果颗粒为聚苯乙烯颗粒,则未配对的T细胞可以通过重力(例如,离心)分离。在除去未配对的T细胞后,在一些实施方案中,将分离的结合颗粒洗涤至少一次,以除去任何非特异性结合的T细胞。
ComPACT-结合T细胞也可以通过FACS分离到单独收集容器例如多孔板中。单独收集容器可以为单细胞反应容器。例如,可以将用于下游处理和分析的成分添加到每一个单细胞反应容器中。comPACT-结合T细胞可以通过FACS分离到批收集容器中(例如,将每个分离的T细胞收集到相同容器中)。
还可以使用液滴产生微流体装置(即,“液滴发生器”)将ComPACT-结合T细胞各自分离到液滴中。用于封装单细胞的液滴产生装置对本领域技术人员是已知的,例如,如美国公开号No.2006/0079583,美国公开号 No.2006/0079584,美国公开号No.2010/0021984,美国公开号 No.2015/0376609,美国公开号No.2009/0235990和美国公开号 No.2004/0180346中所述。
在将comPACT-结合T细胞分离到单细胞反应容器中(例如,分离到单独的孔或液滴中)后,comPACT-结合T细胞的核酸可以进一步加工用于下游分析。具体地,表达的TCRα和TCRβmRNA转录物可以首先通过逆转录转换为cDNA,并将cDNA扩增以用于本领域技术人员已知的下一代测序(next generation sequencing,NGS)方法,包括但不限于通过合成技术(Illumina)进行测序。
实施例
实施例1:经由限制性消化克隆的comPACT小基因的设计及克隆
用于限制性消化的comPACT小基因的结构:
comPACT小基因的基本示例性成分为指导蛋白分泌的信号序列,通用靶序列如限制性位点或引物结合位点,抗原性肽(或新抗原,NeoE),第二通用靶位点,恒定的β2M,MHC等位基因的细胞外结构域和纯化簇,所述纯化簇能够实现酶促修饰(例如,生物素化)和经由亲和标签的 comPACT纯化。簇也可以包含在肽成分之间的蛋白酶切割位点和接头序列。小基因也可以包含用作二硫键陷阱的半胱氨酸突变。comPACT小基因的图示于图1中。MHC重链序列上游和下游的额外的限制性位点能够用于插入其他MHC等位基因,从而构建不同的MHC模板并建立MHC 模板的文库(图2)。编码信号序列,通用靶序列,恒定的β2M和MHC等位基因的胞外域的DNA片段为基本MHC模板。
对于限制性消化克隆方法,每一个comPACT DNA构建体为具有虚拟抗原性序列插入物的基本MHC模板,所述插入物含有三个框内的终止密码子和用于破坏未切割或重连接的模板的独特限制位点(图3),所述构建体能用作现成的平台的一部分,用于迅速装配与所述MHC等位基因复合的抗原性肽的文库。MHC等位基因也可以被修饰或被突变(例如,Y84A 或Y84C)以改善折叠或增加抗原性肽与MHC蛋白的结合。此外,β2M蛋白也可以被突变(例如,S88C)以允许其结合硫醇染料。
在本实施例中,comPACT小基因显示为具有如下结构:NotI限制位点在5’端;来自人生长激素,hGH的信号序列示于表3中;抗原性肽区上游的限制位点Blp1和抗原性肽序列下游的BamHI限制位点示于表2中;主导性甘氨酸和丝氨酸残基的接头序列(即Gly-Ser接头);β2M序列;具有BspI限制位点的第二Gly-Ser接头序列;MHC重链;具有BstBI限制位点的第三Gly-Ser接头序列;以及具有Avi标签序列,TEV切割位点和串接的组氨酸标签的纯化簇。
comPACT小基因的限制性消化克隆和组装
本文描述了经由限制性消化插入新抗原的三个不同方法。在第一种中,如在图4中的图所示,抗原性肽(NeoE)编码引物在5’端跨第一限制位点(在本实施例中为BlpI)并在3’端跨第二限制位点(在本实施例中为BamHI)。该引物扩增编码第二限制位点的通用反向引物,收获~70bp的引物二聚体。
在第二种方法中,抗原性肽编码引物跨第二限制位点作为5’端,并且是抗原的反向互补序列。该引物从编码信号序列的模板DNA以反向进行引发。与跨第一限制位点序列的正向引物进行配对,该反应收获70bp产物,或如果使用跨抗原位点更远的上游的限制位点的正向引物,则收获~140bp产物。
在第一和第二种方法两者中,插入物在商品柱上净化,用适当的限制性酶消化,再次在商品柱上净化,然后与在载体中的预消化的MHC模板连接。连接反应物被转化到大肠杆菌中,并且将从转化的大肠杆菌制备的质粒使用在哺乳动物生产细胞转染反应中。
实施例2:经由引物退火的comPACT小基因的设计和克隆
在MHC模板载体连接的第三种变化中,通过将两个反向互补的新抗原编码引物退火而绕过了PCR和限制性消化。这些引物被设计为具有开始并终止于互补序列中的5’和3’端,所述互补序列模拟来自限制性消化的突出端(图5)。将正义和反义引物与T4多核苷酸激酶和ATP进行孵育,以使5’端磷酸化(图22A)。当这些引物彼此退火时,它们形成了双链寡核苷酸序列,所述序列具有如同已用限制性酶消化的突出的核苷酸。经磷酸化的新抗原插入物被连接到在载体中的预切割MHC模板中。comPACT小基因具有与实施例1中所述的相同的结构。然后,使用书夹(bookend)通用引物,将连接产物用于线性comPACT扩增子的PCR扩增,以扩增完整的 comPACT基因并测序。使用该方法制作了具有独特新抗原序列的824个 comPACT,其中,大于99%的生成的comPACT具有正确的新抗原序列(图 22B)。
接下来,用连接产物质粒转化大肠杆菌,并铺板在含有氨苄青霉素的选择性琼脂平板上。拾取单独菌落并生长过夜用于质粒纯化,并测序用于全基因验证。在测序验证后,将质粒批次归档并繁殖为更大数量。
备选地,如果预切割的MHC模板载体在其突出端上保持5’磷酸盐,则不使用T4激酶。然后,可以将退火的抗原插入物与切割的MHC载体连接,并将连接产物转化入大肠杆菌中用于质粒生产。
实施例3:经由PCR装配的comPACT小基因的设计和克隆
用于PCR装配的comPACT小基因的结构:
也可以使用插入新抗原的第四种方法。在本方法中,经由聚合酶链式反应将新抗原插入到MHC模板中,以形成2.5kb的小基因,其中所述 MHC模板由上游启动子和下游聚腺苷酸化信号侧接。PCR装配反应图示于图6中。
在本实施例中,comPACT小基因显示为具有如下结构:5’端的启动子;具有第一通用靶序列的信号序列;抗原性肽;具有主导性甘氨酸和丝氨酸残基的接头序列(即GlySer接头)的第二通用靶序列;β2M序列;第二 Gly-Ser接头序列;MHC重链等位基因;第三Gly-Ser接头序列;纯化簇;和polyA序列。在该方法中通用靶序列是不相同的。
comPACT小基因的PCR组装:
在本方法中,合成了具有选定的新抗原序列的两种引物(<60nt)。第一引物在5’端具有新抗原序列,接着是在3’端的第二通用靶序列段。第二引物在5’端具有新抗原序列的反向互补序列,并在3’端具有第一通用序列的反向互补序列。将这些引物与编码启动子区、信号序列和第一通用靶序列的DNA片段,以及编码第二通用靶序列、β2M序列、MHC等位基因、纯化簇和polyA序列的另一个DNA片段进行混合。将每个抗原性肽引物与其互补序列退火,并运行PCR反应,所述PCR反应将新抗原序列扩增到启动子片段或MHC等位基因片段上。这两种新合成的片段现在各自具有新抗原序列。进一步的PCR反应,连同启动子序列5’端和polyA序列3’端的引物,可使新抗原序列彼此退火,并引发全长线性comPACT扩增子的组装。
然后,清理完全组装的线性comPACT多核苷酸,用于直接转染到哺乳动物生产细胞中,完全绕过使用大肠杆菌和质粒生产的步骤。
实施例4:从质粒表达和纯化comPACT蛋白质
蛋白质的表达
经由NotI和BamHI限制位点的限制性消化和如前所述的连接,将新抗原12(neo12)连接到HLA-A2模板序列中,并插入到表达质粒 (pPACT0010)中。
在第-1天,用与Expifectamine转染试剂一起孵育的pPACT0010转染 30mL摇瓶体积中的Expi293哺乳动物生产细胞。在第0天,添加 Expifectamine转染试剂盒中含有的增强子。在第1天至第7天从细胞上清中收集样品,并经由SDS-PAGE和使用Safestain(ThermoFisher)的总蛋白染色评估分泌的蛋白。分泌的comPACT蛋白水平增加,直到第3天,在该点,蛋白分泌水平平缓(图7)。分泌的comPACT蛋白最初通过其表观分子量(=53kDa)进行识别,并通过使用NTA-HRP以检测His6亲和标签的蛋白质印迹证实。
蛋白质的纯化
经由His6亲和标签的结合,通过Ni-NTA亲和色谱将第7天收集的 Neo12 comPACT蛋白纯化。经由SDS-PAGE和Safestain而对样品测定了总蛋白。在亲和柱的流通(FT)级分中缺乏comPACT蛋白,这证实了在表达和纯化期间His6标签没有被切割(图8)。经纯化的产量为每L培养体积>400mg。
将Neo12 comPACT蛋白进行生物素化(下面在实施例6中讨论),并进一步地通过尺寸排阻色谱法纯化。观察到单一的主峰,表明蛋白被正确地折叠并且为单体的,具有很少的聚集(图9)。第二个峰为ATP,其是用于 BirA催化的生物素化反应而添加的。
生产量和平行生产的优化
comPACT的生产从在摇瓶中30mL的培养体积,规模缩减到在96深孔摇动板中的0.7mL。Expi293哺乳动物生产细胞用含有pPACT0010质粒的质粒DNA进行转染,并且分泌的Neo12comPACT蛋白如前描述地进行纯化。从0.7mL孔体积收集437mg/L纯化的Neo12 comPACT蛋白,而之前描述的从30mL纯化实验中得出的产量>400mg/L(图10)。来自0.7 mL试验的蛋白产量对应于>300微克蛋白质,或比典型流式细胞仪试验通常需要的多约1000倍。
接下来,评估了多个comPACT构建体的平行表达。8个不同的具有不同新抗原(新抗原10,15,64,65,66,67,80和83)的comPACT构建体被表达在30mL摇瓶中,作为中通量测定(图11)。每个comPACT构建体如前描述地转染入细胞,其中表达comPACT蛋白并将其分泌到细胞上清中。将表达蛋白如前描述地纯化,浓缩,并标准化。如前描述,测定粗上清和浓缩蛋白质的样品中的总蛋白。comPACT蛋白质经由尺寸排阻层析纯化(图12)。对于每种蛋白质,观察到含有2-20mg蛋白的单峰,这也表明comPACT蛋白质正确折叠并且是单体。
实施例5:comPACT蛋白质从线性扩增子的表达和纯化
在之前的实施例中,由转染到哺乳动物生产细胞中的质粒表达 comPACT蛋白。作为可替换的途径,将由启动子序列和polyA序列侧接的neo12 comPACT小基因的线性扩增子(将新抗原12组装入具有HLA-A2 模板序列的小基因中),转染到在96深孔板中的0.7mL的生产细胞中。作为对照,也将pPACT0010质粒转染到单独的生产细胞中。来自两个样品的蛋白如前描述地进行了表达,纯化并测定了总蛋白。线性扩增子和质粒两者均产生了相似水平的表达蛋白质(图13A),表明可以不使用质粒中间体即可产生comPACT小基因编码的蛋白。生产了多种不同的具有不同 neo-表位序列的comPACT小基因(图13B),用于直接转染生产细胞。
使用在实施例2中描述的退火和磷酸化工作流程,制备了具有不同 HLA等位基因的额外的comPACT。使用书夹(bookend)PCR和通用引物从表达载体衍生了线性扩增子,并转染到Expi293F细胞中用于comPACT蛋白产生。图23示出了使用退火克隆工作流程制备的代表性comPACT蛋白质的蛋白凝胶。ComPACT蛋白质在SDS-PAGE凝胶上跑胶,并用Safestain(Thermofisher)染色。
实施例6:comPACT蛋白质的生物素化
用分离的BirA酶进行comPACT的体外生物素化
comPACT纯化簇包括用于生物素化的BirA识别序列(Avi标签)。纯化的comPACT蛋白质是非生物素化(无BirA处理)的,或根据制造商的说明用商业BirA蛋白进行了生物素化(经BirA处理)的。BirA酶促处理过夜后,将样品结合至两种不同种类的磁性链霉亲和素珠子(C1和T1)并孵育,以允许经生物素化的蛋白结合至链霉亲和素珠子。上清(SN)和珠子(“沉淀”P) 经由SDS-PAGE分离。样品用Safestain测定了总蛋白,及经由蛋白质印迹用NTA-HRP测定了comPACT蛋白的存在(图14)。在未处理的样品中, comPACT蛋白主要在SN级分中发现,确认其为非生物素化的。在经生物素化的样品中,在C1和T1链霉亲和素珠子两者的沉淀样品中发现了 comPACT蛋白,尽管在生物素化的蛋白质和C1链霉亲和素珠子之间的相互作用最完全。在C1链霉亲和素珠子-耗尽的上清中,经由蛋白质印迹没有检测到生物素化的comPACT蛋白,表明~100%的comPACT蛋白被生物素化。
在纯化前,ComPACT蛋白质也可以在澄清的上清中进行生物素化。如前所述,在生产细胞中表达了多种comPACT蛋白质。收集细胞培养上清,并经由离心澄清。根据制造商的说明,用商业BirA蛋白处理经澄清的上清,然后经由Ni-NTA亲和色谱进行纯化,并经由蛋白质印迹评估生物素化(图15)。所有试验的comPACT蛋白质均使用这方法进行生物素化,表明在澄清的细胞上清液中,comPACT蛋白质的生物素化是有效的。
为了生产足够的BirA用于comPACT蛋白质的高通量生物素化,在大肠杆菌细胞中表达了具有His6标签的BirA蛋白。该His6标记的BirA经由Ni-NTA亲和色谱纯化(图16B),并可用于使comPACT蛋白质生物素化。也表达了具有TEV-可切割的His6标签的第二版本的BirA-His6,并经由 Ni-NTA亲和色谱纯化(图16C)。该BirA-TEV-His6蛋白可以经由Ni-NTA 纯化,所述His6标签经由TEV切割而除去,然后,无标签的BirA用于使 comPACT蛋白质生物素化。在将comPACT蛋白质生物素化后,然后可以将无标签的BirA蛋白经由Ni-NTA亲和色谱而纯化。此外,将TEV蛋白酶异源表达在大肠杆菌中,用于与BirA-TEV-His6(图16A)使用而用于生物素化comPACT蛋白的生产。
也评估了在生物素化后在comPACT蛋白质上的His6标签的切割,将结果示于图17中。如前描述地将ComPACT蛋白质用BirA处理以使它们生物素化,或未处理(图17的泳道1和2)。将comPACT蛋白的第三样品用BirA处理,然后用TEV来切割存在于蛋白上的His6标签(泳道3)。通过SDS-PAGE分离样品,并经由Safestain评估总蛋白。所有的三个样品具有等量的comPACT蛋白。经由蛋白质印迹评估了comPACT蛋白质的生物素化和His6标签的切割,对于生物素信号使用SA-HRP试剂,对于 His6标签使用NTA-HRP试剂。非生物素化的样品没有显示生物素信号,但具有His6信号,而生物素化的和未切割的样品具有这两种信号。生物素化的和TEV切割的样品只具有生物素信号,表明His6标签已成功地从 comPACT蛋白切割下。
在表达BirA的细胞中,comPACT的体内生物素化
用于使comPACT生物素化的第三种途径为在Expi293生产细胞中表达BirA,并在纯化前在体内使comPACT生物素化。用慢病毒载体转导 Expi293细胞,以共表达由V5侧接的BirA,所述V5作为细胞表面转导标记。分选为V5+的转导细胞也表达BirA(图18)。这些细胞能够用于在 comPACT蛋白纯化前在体内生产生物素化的comPACT。生成了两种细胞系,其分泌BirA到培养基中(Expi293v0223),或在细胞表面表达BirA (Expi293v0263)。也制备了表达由内质网(ER)保留序列KDEL侧接的BirA 的第三细胞系。
为了评估在体外或在体内生物素化方法的生物素标记效率,进行了链霉亲和素下拉(pull down)分析。通过用BirA酶和适当的反应成分(酶促,体外)处理经澄清的培养基,或通过共表达comPACT与BirA(在体内)并与逐渐增加浓度的链霉亲和素包被的珠子进行孵育,将ComPACT蛋白(EBV 或MART-1)生物素化。将用链霉亲和素包被的磁珠(Dynabeads,Thermo Fisher)添加到20μg的comPACT蛋白样品中,并在室温孵育30min。通过磁化,在管的或孔的底部分离磁珠,上清的蛋白含量通过SDS-PAGE测定。图25示出了SDS-PAGE凝胶结果。通过在纯化期间添加BirA酶(体外)或通过共表达BirA酶和comPACT蛋白(在体内),能实现接近完全的生物素化。缺乏链霉亲和素珠子的样品在上清中具有显著量的生物素化的蛋白,而添加逐渐增加量的链霉亲和素珠子则导致几乎所有的蛋白从上清中除去,表明comPACT蛋白的生物素化。
实施例7:使用comPACT蛋白质的抗原特异性T细胞染色和亲和力评价
为了比较使用comPACT与常规肽-MHC的抗原特异性T细胞染色,根据已发表的方案(Bethune,M.T.等.BioTechniques 62,123-130, doi:10.2144/000114525(2017))制备了comPACT dextramer。T细胞被改造以表达A2/neo12-特异性TCR,并用HLA-A2/neo12肽-MHCdextramer或 HLA-A2/neo12肽comPACT dextramer染色。用comPACT dextramer染色至少与肽-MHC dextramer同样有效(图19)。该数据提示comPACT dextramer能够用于分选用于TCR测序的抗原特异性T细胞。
实施例8:功能性T细胞分析
NTAmer结合分析
材料和方法
除了T细胞的抗原特异性捕获外,comPACT的模块化设计和易于生产有利于它们在功能性T细胞分析中的使用。例如,β2M的突变版本(S88C) 的掺入使comPACT能够被马来酰亚胺-染料偶联物标记,组装成NTAmer,并用于测量TCRcomPACT结合的动力学参数。比如,NTAmer能够用于解决活细胞中的单价TCR-MHC I结合事件。CD8结合和在免疫突触处的多重TCR-MHC I相互作用允许T细胞和抗原呈递细胞之间的接触延长。将荧光染料偶联的ComPACTNTAmer与细胞孵育,以允许comPACT与 TCR结合。通过添加咪唑使NTAmer的Ni-PE颗粒组分从comPACT解离,并随着时间推移测量了荧光comPACT的释放。
染料偶联
构建了S88C突变型comPACT蛋白质,并以~150mg/L表达。这些突变型comPACT呈现与未发生突变的comPACT相似的纯度和洗脱曲线(图 20)。其他染料,如Cy5,也可以被缀合至S88C comPACT。comPACT88C-HisTag单体用三(2-羰基乙基)膦盐酸盐(TCEP)还原,并与马来酰亚胺-Cy5偶联,得到comPACT88c-Cy5标记的单体。图21示出了用 Cy5标记的comPACT的A280,A260和A650定量。染料与comPACT的比为67%。
NTAmer组装
通过用镍(Ni)加载(charge)生物素-NTA(生物素-NiNTA),然后通过用生物素-NiNTA和PE-链霉亲和素(SA)装配comPACT-Cy5,从而将生物素化的comPACT结合到PE-生物素-NTA珠子。通过将5mg生物素-NTA与1mL 镍加载溶液(50mM NiSO4在100mM HEPES中,pH7.5)混合而生成生物素-NiNTA,以收获Ni2+NTA-生物素(7nM)。溶液用HBS稀释以收获70uM Ni2+NTA-生物素溶液。将Ni2+NTA-生物素溶液与SA-PE(300kDa)组合,以5次进行添加,每次加入之间间隔5min。然后将20uM Cy5-S88C comPACT添加到NTAmer核,在室温在黑暗中孵育10min,并储存于4C。 Neo12和MART1 comPACT两者被制备和组装入NTAmer。在comPACT 蛋白上的His标签与NiNTA结合,NiNTA本身与PE-链霉亲和素结合。每个NTAmer包括comPACT单体的多个拷贝,所述comPACT单体结合至具有荧光团的链霉亲和素核。NTAmer的组装在Schmidt等,J.Biol Chem, December 2,2011,286(48)41723-41735和Schmidt等,Front Immunol,July 30,2013doi:10.3389/fimmu.2013.00218中进一步地讨论,两者整体通过引用并入本文。
生物素化的Neo12和MART1 comPACT蛋白质也被组装成PE标记的四聚体和dextramer。具有neo12抗原性肽的肽结合的重折叠MHC I分子也被组装成PE标记的dextramer作为对照。将肽-MHC蛋白质装配成四聚体和dextramer的方法通常是本领域已知的。
T细胞结合
将表达与Neo12或MART1新抗原结合的TCR的经基因编辑的T细胞重悬在1x染色缓冲液(BD牛血清白蛋白染色缓冲液,BD554657) 中,密度在1-2x10
结果
首先,评估了生物素化的comPACT-Cy5 NTAmer与它们的同源TCR 结合的能力。将Neo12肽-结合的重折叠MHC I分子,neo12 comPACT分子或neo12 NTAmer组装成dextramer,并与表达neo12 TCR的T细胞孵育。测定了每一种dextramer分子与细胞的结合。图28A显示,证实了表达neo12 TCR的基因编辑的T细胞与neo12肽-结合的重折叠MHC I分子dextramer 进行特异性结合(下图,x轴为dextramer PE信号),且这种结合对应于在它们的表面上表达CD3的T细胞群(顶部图,x轴为PE-Cy3信号)。图28B 显示,证实了表达neo12 TCR的基因编辑的T细胞与neo12 comPACT分子dextramer进行特异性结合(下图,x轴为dextramer PE信号),且这种结合对应于在它们的表面上表达CD3的T细胞群(顶部图,x轴为PE-Cy3 信号)。图28C显示,证实了表达neo12 TCR的基因编辑的T细胞与neo12 comPACT NTAmer dextramer进行特异性结合(下图,x轴为dextramer PE 信号),且这种结合对应于在它们的表面上表达CD3的T细胞群(顶部图, x轴为PE-Cy3信号)。
接下来,证实了生物素化的comPACT-Cy5 NTAmer以抗原特异性方式结合TCR的能力。将Neo12或F5 TCR T细胞与具有neo12抗原的 NTAmer孵育。F5 TCR T细胞用作阴性对照。Neo12 TCR T细胞也与由 neo12 comPACT蛋白质组成的dextramer孵育。然后,如前所述将结合的 T细胞与咪唑孵育,并如前所述地评估了NTAmer和dextramer的结合。添加咪唑抑制了neo12-ComPACT NTAmer结合至neo12 T细胞的能力(图 29A-C)。Neo12 TCR T细胞显示出与neo12 NTAmer试剂特异性结合(图 29A,NTAmer Cy5信号为y轴)。neo12-ComPACT NTAmer特异于Neo12 T细胞,且不结合至表达F5 TCR的T细胞(图29B)。Neo12 comPACT dextramer也与neo12 TCR T细胞结合(图29D)。当在咪唑的存在下孵育时, Neo12 TCR T细胞显示不与neo12 NTAmer试剂结合(图29C),这说明需要多聚体化试剂来观察来自经S88C修饰的comPACT分子的Cy5信号。
也使用了MART-1NTAmer,用于显示经编辑以表达F5 TCR的T细胞的抗原特异性结合(图30A-D)。图30A示出MART-1 comPACT dextramer 与F5 TCR T细胞的结合。图30B示出MART-1comPACT dextramer与M1W TCR T细胞的结合。图30C示出MART-1 comPACT NTAmer与F5 TCRT 细胞的结合。图30D示出MART-1 comPACT NTAmer与M1W TCR T细胞的结合。MART-1comPACT NTAmer与M1W T细胞的低水平结合可能归因于M1W TCR对同源抗原:MHC I蛋白的低亲和力。
通过测量针对相同肽:MHC I复合体(HLA:A02+MART-1)具有差异TCR:MHC I结合亲和力的两种TCR,MART-1F5和M1W的Cy5信号衰减而评估了单体TCR:pMHC-Cy5的解离。在不存在咪唑诱导的NTAmer 破坏情况下,MART1 NTAmer与表达F5 TCR的T细胞的结合是经时稳定的(图31A)。然而,用咪唑破坏多聚的MART-1NTAmer导致单体MART-1 comPACT分子与F5 TCR的时间依赖性解离(图31B)。图31C示出了来自图31B的时间数据的子集的放大图。
总之,Neo12和F5 T细胞在NTAmer和等价物comPACT dextramer 之间表明了相似的结合水平,并且comPACT NTAmer能够以抗原特异性方式结合新抗原T细胞。对于neo12和MART-1 comPACT两者,Neo12 和F5 T细胞在NTAmer和comPACT dextramer之间表明了相似的结合水平。此外,NTAmer复合体可以通过添加咪唑被破坏,并可以通过在活T 细胞上的comPACT-Cy5信号的衰减来测量单价TCR:MHC I解离速率。该衰减发生于数十秒的时间尺度。
实施例9:comPACT文库生产
通过生物信息学从等位基因频率Net数据库(www.allelefrequencies.net) 分析了美国人类群体的HLA等位基因多样性,以鉴定包括在HLA谱系中等位基因的最佳数目,以实现对受试者HLA频率的高覆盖。分析了9736 个等位基因。图26A示出了来自每个HLA A,B和C基因座的一个或两个等位基因被66个HLA等位基因的文库覆盖的患者百分比分析。实线表明1个等位基因被覆盖,而虚线表明两个等位基因均被覆盖。66个等位基因实现了在>95%的总群体中的每个患者覆盖至少6个HLA等位基因中的 4个,和在>80%的群体中覆盖6/6个等位基因(图26B)。最常见的的HLA-I 等位基因为HLA-A02:01,在美国具有~50%的流行率。因此,本文首次显示的HLA文库允许了对全球和多种多样的人群广泛实施个体化neoTCR-T 细胞疗法的最大潜力。
接下来,制备了具有不同的新表位和选择的HLA等位基因的 comPACT蛋白质的文库。新表位候选物选自免疫表位数据库 (www.iedb.org)。从IMGT数据库获得了66个HLA-I等位基因的每一种在谱系中的完整序列,并对其进行修饰以包括Y84C突变。对所有的克隆进行了序列验证并存入数据库和试剂清单中。从IEDB数据库选取10个新表位肽,并插入到一组36个HLA等位基因中。将选择的新表位的ComPACT 和HLA等位基因进行表达,并根据制造商的说明书经由连接到Agilent Infinity II HPLC系统的尺寸排阻层析柱(Agilent Sec Bio300)(SEC-HPLC) 进行纯化。结果示于图27A-C中。如通过测量单体峰曲线下面积除以整个色谱下的面积并经由SEC-HPLC所评估的那样,将comPACT纯化为单分散多肽(图27A和27B)。大多数comPACT以高滴定度表达(图27C)。所述的每个HLA等位基因的至少一个comPACT蛋白已通过HPLC纯化并表征,表明comPACT平台是稳健的,并可用于许多等位基因。
虽然已经参照优选实施方案和各种备选实施方案特别展示和描述了本发明,但相关领域技术人员将理解,在不背离本发明的精神和范围的情况下,可以对其中的形式和细节进行各种变化。
出于所有目的,在本说明书正文内引用的所有参考文献、已发布的专利和专利申请在此均通过引用以它们的整体并入本文。
- 一种高效筛选T细胞抗原表达肽,制备MHC-肽多聚体(MHC tetramer)的体系
- 分离的结合MHCⅠ类和MHCⅡ类分子的同NY-ESO-1的氨基酸序列相关的肽及其应用
