一种可特异性识别增殖状态内皮细胞的启动分子及工程细胞
文献发布时间:2023-06-19 10:13:22
技术领域
本发明属于生物工程领域,具体涉及一种可特异性识别增殖状态内皮细胞的启动分子及工程细胞。
背景技术
创伤性脑损伤(Traumatic brain injury,TBI)对大脑造成的损伤被分为了两个阶段:直接损伤和继发性损伤。直接损伤即由外部机械力对脑组织造成的破坏,导致组织结构被破坏,细胞受到毁灭性打击而直接死亡,这其中也包括血管损伤,出血等。继发性损伤则是由于脑组织的稳态被打破,特别是脑出血,血脑屏障破坏,导致外周的免疫细胞聚集,继发炎症,最后导致损伤蔓延,组织坏死的面积扩大。
血脑屏障的功能障碍将会引起多种蛋白质和电解质的泄漏,这破坏了细胞的微环境,并可能引发下游的连锁反应,例如小胶质细胞的激活和募集。继发性损伤的持续时间比原先设想的要长得多,即使在原发性损伤后间隔了40年之久,这种继发性损伤依然可能导致神经病变。
在TBI幸存者中,血脑屏障的恢复速率是大不相同的,在恢复期较长的患者中,有的需要一年以上,甚至在伤愈后11年依然有患者存在血脑屏障分解的情况。在脑外伤患者中,由继发性损伤带来的危害甚至超过了直接损伤,而越快修复血脑屏障,避免炎性细胞的聚集,就可以将继发性损伤带来的危害大大降低。在直接损伤造成后,血管会进行无序地再生,这类新生血管内皮功能不完善,血脑屏障尚未建立,加速新生血管成熟及血脑屏障形成是脑外伤修复的一大目标。
通过合成生物学方法合成融合蛋白是开发新生物学工具的常用方法之一,经过合理的设计,融合蛋白可以具备多种特性。经过融合蛋白修饰的工程细胞会受到融合蛋白的影响,进而发生细胞行为的改变,这种改变包括细胞的识别行为与应答行为。
新生血管内皮具备某些在成熟血管内皮中不具备的分子标志物,如爱帕琳(Apelin)/Apj。尽管在发育过程中的内皮细胞高度表达爱帕琳,但在成熟的内皮细胞中其表达量极低,只有在病理条件下,如肿瘤、脑外伤、脑卒中等情况下,内皮细胞才会重新表达爱帕琳。
Apj作为爱帕琳的内源性受体,是一种跨内皮细胞膜的G蛋白偶联受体,在胚胎时期它在各种组织中大量表达,但在成体组织中,Apj仅在出芽的血管中表达,可作为一种内皮细胞增殖的标志物。因此,爱帕琳/Apj这一对相互结合的膜蛋白分子可以作为靶向新生血管及后续治疗的靶点,两者敏感度高,结合紧密。
合成Notch系统是一种嵌合型蛋白受体工具,通过对天然的Notch蛋白加以改造,可使其调控特定的细胞信号通路。内源性Notch系统是哺乳动物重要的信号调节系统,内源性Notch1受体包含三个结构域,即细胞外识别结构域,跨膜结构域和细胞内部的转录调控因子,其中,细胞外识别结构域和转录调控因子结构域均可被其他结构取代,形成新的合成受体蛋白,从而实现细胞靶向调控和下游目的信号响应。
现有技术中,利用合成Notch系统识别新生血管的研究未见报道。
发明内容
本发明的目的在于提供一种可特异性识别增殖状态内皮细胞的启动分子及工程细胞,经该启动分子修饰的工程细胞可与Apj受体高效稳定地结合,形成细胞-细胞接触,特异性识别新生血管,并调控下游信号通路中设定基因的表达;可用于示踪增殖的内皮细胞,指示成体内出现血管重塑的情况,同时也可以作为治疗手段,实现血脑屏障的修复。
为了达到上述目的,本发明提供如下技术方案:
一种可特异性识别增殖状态内皮细胞的启动分子,其为一种融合蛋白,其特征在于,包含了人源爱帕琳蛋白、膜内可水解多肽以及效应因子,三者由膜内可水解多肽中的两个蛋白水解切割位点相连;其中,所述膜内可水解多肽为天然Notch的最小跨膜核心结构域,所述膜内水解多肽在静息状态时被邻近的爱帕琳和效应因子部分覆盖或完全覆盖。
优选地,所述爱帕琳蛋白的氨基酸序列如SEQ ID NO.1所示;所述膜内可水解多肽的氨基酸序列如SEQ ID NO.2所示。
又,所述效应因子选自四环素转录激活蛋白或Cre重组酶的结构域。
优选地,所述启动分子的效应因子为Mfsd2a转录启动因子,所述启动分子的氨基酸序列如SEQ ID NO.3所示。
本发明提供含有所述可特异性识别增殖状态内皮细胞的启动分子的工程细胞。
进一步,所述工程细胞是将所述启动分子通过DNA重组、DNA注射、质粒转染或病毒转染方式导入真核细胞获得的。
优选地,所述真核细胞为内皮祖细胞、T淋巴细胞、神经干细胞或胶质细胞。
一种synNotch工程化内皮祖细胞的制备方法,包括以下步骤:
1)制备内皮祖细胞;
2)构建含融合蛋白的慢病毒
设计含上述启动分子的上下游特异性PCR扩增引物,同时引入酶切位点,利用重叠延伸PCR进行扩增,从cDNA质粒或者文库模板中调取启动分子基因CDS区以连入T载体;将CDS区从T载体上切下,装入慢病毒过表达质粒载体;合成siRNA对应的DNA颈环结构,退火后接入慢病毒干扰质粒载体;
制备慢病毒穿梭质粒及其辅助包装载体质粒;
分别提取上述慢病毒过表达质粒载体、慢病毒干扰质粒载体、慢病毒穿梭质粒后共转染至293T细胞,得到含启动分子的慢病毒;
3)转染至真核细胞
将慢病毒转染内皮祖细胞,同时转染荧光报告基因,得到经所述融合蛋白修饰的工程化内皮祖细胞。
进一步,步骤3)中,转染慢病毒后的内皮祖细胞用EGM-2培养基进行扩增,待细胞量占培养瓶80-90%时,用荧光显微镜观察标记荧光蛋白的表达情况,采用流式细胞仪对转染的细胞群体进行标志物鉴定,检测工程细胞的激活情况。
本发明所述工程化细胞用于靶向新生血管、小鼠肿瘤模型或脑外伤模型的构建、对细胞进行重编程、进行细胞损伤修复、修复血脑屏障或者细胞再生。
进一步,所述靶向新生血管是指在亚急性期脑外伤中靶向新生血管。
本发明通过改造核酸序列,用爱帕琳替代了Notch受体的胞外段,将人源爱帕琳、膜内可水解多肽以及效应因子对应的脱氧核糖核酸(DNA)序列融合,得到了膜内水解多肽在静息状态时被邻近的爱帕琳和效应因子部分覆盖或完全覆盖的融合蛋白,将其称为新生血管识别启动分子,简称启动分子,这种特殊的分子结构降低了启动分子对Apj的敏感程度,提高了启动分子的特异性。
本发明获得的融合蛋白具有功能高度模块化的特点,其中的人源爱帕琳与新生血管表面分子标志物Apj受体结合,继而切割位点被识别水解,特异性地识别新生血管,天然Notch的最小跨膜核心结构域介导膜内段水解作用发挥信号传导功能,进而调控下游信号通路,调控设定基因的表达,根据下游效应基因的不同,启动分子工程细胞可以产生不同的细胞行为。
由于本发明的启动分子具备靶向新生血管的特点,通过利用包括但不限于DNA重组、DNA注射、质粒转染或病毒转染等方式将目的序列导入真核细胞,得到工程化细胞。
本发明的工程细胞中,启动分子分布于细胞膜上,启动分子横跨整个细胞膜,其中细胞膜外段为识别域,可以与增殖中的内皮细胞表面分子标志物Apj蛋白结合,从而赋予了启动分子工程细胞特异性识别新生血管的能力。启动分子与Apj结合,导致启动分子工程细胞与增殖状态的内皮细胞黏连,由于机械力的牵拉暴露出启动分子的可水解肽段,可水解肽段被水解后效应因子与膜内段的连接被破坏,效应因子从细胞膜上脱落,进入细胞核,激活下游效应基因,实现启动分子的特异性响应。
启动分子需要借助细胞间的拉力,这决定了工程细胞需要接触病变区域才可以发挥效果,极大地提高了精确性,有效避免了非特异性表达,由启动分子修饰的工程细胞具备与新生血管的内皮细胞结合的能力,并导致工程细胞启动下游效应基因。
由于本发明的融合蛋白具备靶向新生血管的特点,通过利用包括但不限于DNA重组、DNA注射、质粒转染或病毒转染等方式将目的序列导入真核细胞,得到工程化细胞。经本发明启动分子修饰的工程细胞存在多种用途,包括但不限于抑制肿瘤生长,检测肿瘤发生,修复血脑屏障,对细胞进行重编程,实现细胞损伤修复或者细胞再生。
S1P为促进血脑屏障关闭的分子,可由内皮祖细胞分泌,当融合蛋白的细胞外段结构域为人源爱帕琳蛋白,细胞内段的效应因子为Mfsd2a转录启动因子时,构建出可修复血脑屏障的Apelin-synNotch受体启动分子,在颅脑损伤模型中,采用该启动分子修饰内皮祖细胞,并对该细胞转染S1P相关的基因Mfsd2a,通过启动分子系统,启动分子的细胞外段可与Apj蛋白结合,继而切割位点被识别水解,Mfsd2a转录启动因子入细胞核,调控Mfsd2a基因的表达,重启了Mfsd2a的表达,促进S1P分泌到细胞外基质,成功地在损伤区提升S1P的浓度,促进血脑屏障关闭,实现血脑屏障的修复。
成年人体内的血管处于静息状态,而一旦体内出现肿瘤、外伤、炎症、中风等病理状态,在病灶区会出现缺氧状态,导致内皮细胞应激产生VEGF(血管内皮生长因子),刺激内皮细胞进入增殖状态,继而启动血管发芽过程,病灶区血管发生重塑。因此,内皮增殖可以作为区分正常组织与病变组织的一个标志。
内皮祖细胞是一种血液中游离的祖细胞,可以在血管中自由运输,内皮祖细胞会影响中风后梗死区域以及周围区域的神经祖细胞的迁移,这对于损伤后的神经再生有积极的意义,与此同时,内皮祖细胞也可以参与自然的损伤修复中,在损伤部位的缺血性中风和TBI模型中都报道了从骨髓和外周血中动员内皮祖细胞的方法。内皮祖细胞自身可以表达Spns2并分泌S1P,因此选择内皮祖细胞作为待改造的工程细胞具有极大优势。
本发明至少具有如下有益效果:
本发明调整了synNotch膜内结构域的氨基酸序列,根据氨基酸序列构建了多种蛋白结构,选出其中对水解肽段覆盖率较高的蛋白结构,使启动分子的核心调控区经过机械力的拉动会暴露出来,在紧邻跨膜区的核心调控区内,蛋白酶进行识别并切割,释放细胞内的效应域,进入细胞核并调控下游通路,降低了启动分子对Apj的敏感程度,显著提高了启动分子的特异性。
本发明通过synNotch工程化,利用爱帕琳替代了Notch受体的胞外段,获得了融合蛋白,作为嵌合型蛋白工具,其能特异性精确识别Apelin受体,可以调控细胞信号通路,调控下游基因的表达,其具备靶向新生血管的特点,将其导入真核细胞,可得到可特异性识别新生血管的工程化细胞。
本发明的工程化细胞可以特异性识别新生血管,存在多种用途:如通过血管注射方法,可以在成体内识别新生血管,检测肿瘤发生;或可以靶向肿瘤新生血管,利用工程细胞的下游响应基因发挥靶向抗肿瘤效应,抑制肿瘤生长;又或者可以定位到脑外伤病灶区,调控Mfsd2a基因表达,控制血脑屏障的闭合,修复血脑屏障;对细胞进行重编程,实现细胞损伤修复或者细胞再生。
附图说明
图1为本发明之一的启动分子的基因构成。
图2为本发明实施例1中筛选出的6种不同蛋白结构,其中,1:爱帕琳,2:天然Notch的最小跨膜核心结构域,3:效应因子。
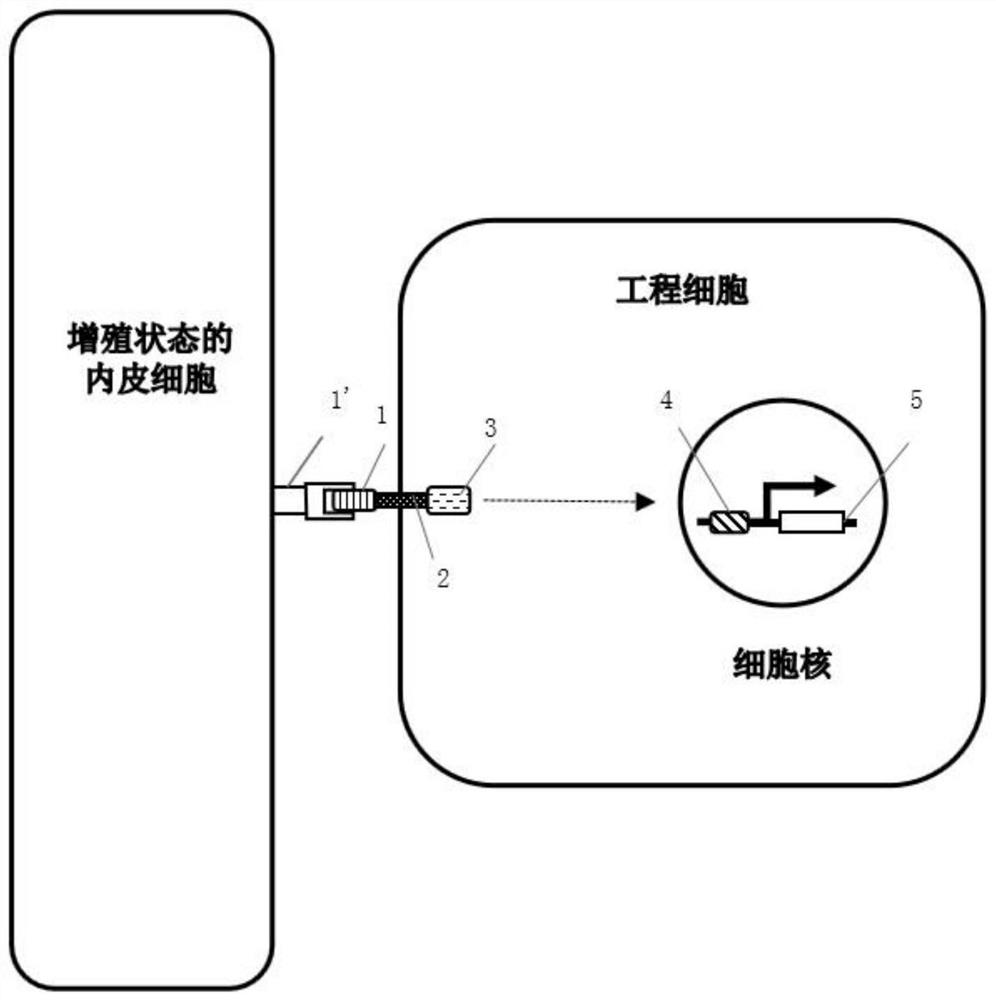
图3为本发明实施例1中含启动分子的工程细胞的工作原理示意图,其中,其中,1:爱帕琳,1’:增殖状态的内皮细胞表面分子标志物Apj蛋白,2:天然Notch的最小跨膜核心结构域,3:效应因子,4:响应元件,5:靶向基因。
图4为本发明实施例2中内皮细胞激发后,工程细胞的Sphk1和Mfsd2a的表达水平以及工程细胞被激活后,S1P的分泌量在不同细胞中的变化情况。
图5为本发明实施例2中S1P的分泌量在不同细胞中的变化情况。
图6为本发明实施例3中颅脑损伤4天后脑血管内皮细胞的排列模式,同时展示了启动分子工程细胞修复血脑屏障的基本原理。
图7本发明实施例4中小鼠脑外伤后注射启动分子修饰的内皮祖细胞后细胞外基质中S1P的浓度的变化情况。
具体实施方式
以下结合具体实施例对本发明作进一步说明。
本发明中出现的术语“新生血管识别启动分子”简称启动分子,即为可特异性识别增殖状态内皮细胞的融合蛋白;出现的术语“工程细胞”、“工程化细胞”指将所述启动分子通过DNA重组、DNA注射、质粒转染或病毒转染方式导入真核细胞获得的细胞。
S1P被认为是为内皮细胞提供屏障功能的关键因子,对血脑屏障的维持起着重要作用。血脑屏障的完整度是S1P浓度依赖性的,即在一定范围内(10nM-1μM),S1P浓度越高,血脑屏障完整性越高。脑中的内皮细胞表达S1P的转运蛋白Spns2,当这一转运蛋白功能障碍时,细胞外基质中的S1P明显下降,血管内皮的通透性会增加,紧密连接也遭到了破坏,而补充外源性的S1P可以使血管屏障渗漏得到缓解。作为内皮细胞中S1P的主要转运蛋白,Spns2将S1P从细胞内转运到细胞外。在现在已有的研究中,Spns2并非是S1P的唯一转运蛋白,与Spns2同属于MFS家族蛋白的Mfsd2a可以显著提高S1P的转运效率,并且特异性地表达于脑内皮细胞中。Mfsd2a无法作为S1P的直接载体,但可以显著提高S1P的转运效率。在脑内皮细胞中,当Mfsd2a与Spns2同时存在时,细胞外基质中的S1P浓度最高(600nM~1μM),而缺少了Mfsd2a之后,S1P浓度会下降至100nM到300nM之间(这一范围促进转胞吞作用,但不影响紧密连接)。Spns2和Mfsd2a支持的高效转运维持了细胞外基质中S1P的高浓度,这对于血脑屏障的形成和维持至关重要。
TBI模型的病灶区的内皮细胞会启动Apj的表达,本发明的启动分子可以通过与Apj结合形成细胞-细胞接触,这种配体与受体的结合非常牢固,这为工程细胞改变靶细胞的微环境中S1P浓度提供了空间上的条件。并且启动分子的下游响应程序可以在二十四小时之内完成,工程细胞从靶细胞上脱离需要至少二十四小时,这样就为改变S1P浓度提供了时间条件。
本发明通过重叠延伸PCR构建融合基因,通过慢病毒转染细胞表达出启动分子蛋白,同时转染荧光报告基因,得到启动分子修饰的工程细胞,在体外,将内皮细胞与工程细胞共培养检测工程细胞是否被激活;在体内,构建疾病模型,如脑外伤,检测工程细胞在体内的激活状态。通过免疫荧光染色和流式细胞术分析工程细胞的状态。通过尾静脉注射将工程细胞输送至模型小鼠体内,检测工程细胞发挥的作用。
本发明中,以工程化内皮祖细胞的制备及其在血脑屏障修复中的应用为范例详细描述,T淋巴细胞工程细胞,神经干细胞工程细胞,胶质细胞工程细胞的制备与应用与之类似。
本发明提供一种由Apelin-synNotch受体启动分子修饰的内皮祖细胞,Apelin-synNotch受体启动分子由识别新生血管的Apelin胞外段(其氨基酸序列如SEQ ID NO.1所示,核苷酸序列如SEQ ID NO.4所示)、膜内段天然Notch的最小跨膜核心结构域(简称Notch核心,其氨基酸序列如SEQ ID NO.2所示,核苷酸序列如SEQ ID NO.5所示),胞内段转录调控因子串联构成,该Apelin-synNotch受体的成熟蛋白氨基酸序列如SEQ ID NO.3所示,结构如图1所示,蛋白质翻译后在细胞内粗面型内质网切除信号肽后成为成熟Notch受体蛋白,分泌输出后并定位于内皮祖细胞的细胞膜上。
本发明的实施例中所用的试剂如下:
磷酸缓冲盐溶液(Phosphate buffer saline,PBS),pH7.4,购于Gibco公司,货号#10010049;双蒸水,购于Sigma-Aldrich公司,货号#DENWAT3MSDS;4%多聚甲醛溶液,购于Biosharp公司,货号#BL539A;蔗糖,购于Sigma-Aldrich公司,货号#S9378MSDS;10-kDadextran-tetramethylrhodamine,购于Thermo Fisher公司,货号#D3312;IsolectinGS-IB4 From Griffonia simplicifolia,Alexa Fluor
实施例1一种synNotch工程化内皮祖细胞的制备方法,包括以下步骤:
1)制备内皮祖细胞
取孕期14~18天的C57/BL6J鼠,受孕子宫放入含有500~1000U青霉素和链霉素双抗的预冷PBS中,浸泡3~5min,取出胎鼠,PBS洗涤2次;显微镜下取出胎肺组织放入PBS并剪碎至直径1mm左右小方块。
向胎肺组织加入0.25%胰酶消化,10%FBS的DMEM培养基培养16h-24h,1500rpm离心5分钟,弃上清,取其沉淀,用5ml含生长因子和5%FBS的血清的EBM-2培养基悬浮细胞,以10
内皮祖细胞表面标志物鉴定:1)免疫荧光:制备细胞爬片,4%多聚甲醛固定10min,PBS漂洗3次;加入0.5%的Triton X-100通透15min;加入山羊血清封闭30min;CD31,CD34,CD133,VEGFR2一抗4℃孵育过夜,PBS漂洗3次;加入荧光二抗避光孵育30min,PBS漂洗3次;加入DAPI染核,PBS漂洗3次;封片剂封片;共聚焦显微镜下观察并拍摄图像。2)流式细胞术:每管总量约5×105细胞,加入抗体,避光孵育30min,PBS漂洗3次,流式仪检测。
2)构建启动分子慢病毒
参见图2-图3,设计上下游特异性扩增引物,同时引入酶切位点,PCR(采用高保真KOD酶,3K内突变率为0%)从模板中(CDNA质粒或者文库)调取目的基因CDS区(codingsequence)连入T载体,将CDS区从T载体上切下,装入慢病毒过表达质粒载体;其中,所述目的基因为编码所述可特异性识别增殖状态内皮细胞的启动分子的基因,其膜内水解多肽天然Notch的最小跨膜核心结构域2在静息状态时被邻近的爱帕琳1和效应因子3部分覆盖或完全覆盖,其中的6种蛋白结构参见图2。
合成siRNA对应的DNA颈环结构,退火后连入慢病毒干扰质粒载体,制备慢病毒穿梭质粒及其辅助包装原件载体质粒,分别对慢病毒过表达质粒载体、慢病毒干扰质粒载体、慢病毒穿梭质粒进行高纯度无内毒素抽提,之后共转染293T细胞,转染后6h更换为完全培养基,培养24和48h后,分别收集富含慢病毒颗粒的细胞上清液,病毒上清液通过超离心浓缩病毒,获得启动分子慢病毒。
具体步骤如下:
慢病毒包装采用293T细胞,选取处于对数生长期的293T细胞,胰酶消化,接种于T25cm2培养瓶,37℃,5%二氧化碳培养箱内培养;细胞密度达到60%~70%时进行转染,将DNA和磷酸钙混合液转移至培养基中,混匀,培养12h后弃去旧的培养液,PBS漂洗3遍;每瓶细胞中加入新鲜DMEM培养基5mL,继续培养48h;收集转染72h后的293T细胞上清液;将收集的上清液于4℃,4000g离心10min,收集上清液;将各种不同RNA干扰质粒上清液过滤;于40mL超速离心管中,4℃,25000r/min离心20min;冰PBS液,分别溶于500ul DMEM和500ulPBS重旋病毒沉淀,4℃溶解过夜。
3)启动分子修饰内皮祖细胞
取1×10
具体步骤如下:
(1)慢病毒转染前18-24小时,用0.25%胰酶将内皮祖细胞消化,离心后加入EGM-2培养液重悬制成单细胞悬液并行细胞计数,将细胞悬液以1×10
(2)接种细胞24h后,弃掉旧的培养基,更换为含5μg/ml polybrene的2ml新鲜无血清培养基,计算MOL值为10时所需要的加入的病毒悬液量,加入到培养基中轻轻摇晃混合均匀,置于37℃、5%CO
(3)4小时后加入2ml新鲜培养基。
(4)继续培养24小时,更换为新鲜的无病毒的完全培养基。
(5)转染3-4天后在完全培养基中加入嘌呤霉素,嘌呤霉素终浓度为5ug/ml,以筛选稳定转染细胞株,获得含所述启动分子的工程细胞。
含所述启动分子的工程细胞可以特异性识别新生血管,调控细胞信号通路,调控下游基因的表达,工作原理如图3所示,构建的工程细胞中,启动分子分布于细胞膜上,启动分子横跨整个细胞膜,其中细胞膜外段为识别域,可以与增殖状态的内皮细胞表面分子标志物Apj蛋白1’结合,从而赋予了启动分子工程细胞特异性识别新生血管的能力。启动分子上的爱帕琳1与Apj结合,导致启动分子工程细胞与增殖状态的内皮细胞黏连,由于机械力的牵拉暴露出启动分子的可水解肽段天然Notch的最小跨膜核心结构域2,可水解肽段被水解后效应因子3与膜内段的连接被破坏,效应因子3从细胞膜上脱落,进入细胞核,激活下游响应元件4及靶向基因5,实现启动分子的特异性响应。
实施例2工程细胞与Apj阳性的内皮细胞共培养
1.分离培养内皮细胞
取产后小鼠的新鲜脐带,手术剪剪下长0.5厘米长的一段;PBS液注入脐静脉中,洗净残余血液;向脐静脉中注满浓度为0.1%的胶原酶,室温消化5分钟;将含有内皮细胞的消化液转移至15ml离心管中,1000rpm离心5分钟,丢弃上清,加入足量的1640培养液重悬,接种至T25培养瓶中培养2~3天,细胞长至80-90%融合度时传代,获得内皮细胞。
2.转染及共培养
将CMV启动分子、TRE和Sphk1-Mfsd2a基因转染至准备好的内皮祖细胞中,当启动分子与Apj结合后,tTA便会从细胞上脱离下来进入细胞核,与反应元件TRE结合,从而启动Sphk1和Mfsd2a的表达。
将消化好的内皮细胞与工程细胞用EGM-2完全培养基调节至细胞密度为1×10
如图4所示,Sphk1和Mfsd2a的mRNA水平显著提高,这代表呈递细胞可以激活工程细胞的下游响应程序,同时ELISA检测培养基中S1P浓度,结果如图5所示,培养基的S1P浓度也显著升高。因此该工程细胞可以用于新生血管中的血脑屏障修复。
实施例3工程细胞识别脑外伤后新生血管
(1)构建脑外伤模型
腹腔注射戊巴比妥钠(0.4mg/10g)麻醉小鼠,将小鼠固定在立体定位注射仪上,其后在右顶叶皮层使用颅钻打开一个直径约5毫米的孔,与中央缝线相邻。然后将直径为2.5毫米的换能金属棒放置在颅骨开孔上,并从15厘米的高度上释放一枚5克的重力锤使其自由落体,垂直撞击换能金属棒。
(2)将启动分子与转录响应元件TRE、红色荧光蛋白RFP同时转染进内皮祖细胞;
(3)TBI小鼠模型构建成功后第3天,尾静脉注射由内皮祖细胞改造的工程细胞,并于24小时后取小鼠的脑组织进行观察,通过报告基因的表达分析工程细胞在损伤区域激活的情况。
颅脑损伤4天后脑血管内皮细胞的排列模式参见图6,同时,展示了启动分子工程细胞修复血脑屏障的基本原理。
实施例4工程细胞对血脑屏障的修复作用评价
(1)注射工程细胞
用Mfsd2a-CreER、Rosa26-RFP小鼠构建TBI模型,在TBI小鼠模型构建成功后的第3天通过尾静脉注射1×10
(2)ELISA检测细胞外基质中S1P水平
按照ELISA试剂盒说明书进行检测试验,如图7所示,ELISA检测试验证明,与注射普通内皮祖细胞相比,工程细胞在损伤区被激活后,细胞外基质中S1P的浓度升高约3.4倍,显著提高。
(3)检测工程细胞响应情况
用qPCR法检测工程细胞S1P合成酶Sphk1与S1P转运蛋白Mfsd2a的表达情况,qPCR检测说明在内皮细胞激发后,工程细胞的Sphk1和Mfsd2a基因的表达水平显著提高,分别升高约3.4倍和10.2倍。
(4)检测血脑屏障恢复情况
使用10kDa dextran tracer检测血脑屏障,注射10kDa dextran tracer:异氟烷麻醉小鼠后,固定小鼠,切开胸前皮肤、真皮,暴露出心脏,向心尖部位注射50μl的10kDadextran tracer(2mg/ml),15分钟后使用过量异氟烷使小鼠死亡,剥取完整的脑组织,浸泡在4%PFA溶液中,于4℃冰箱中固定3-4小时。之后用30%蔗糖溶液4℃浸泡过夜脱水,并用OCT包埋剂包埋,冰冻切片后进行免疫荧光染色,结果显示,工程细胞治疗后血脑屏障提前关闭。
本发明开发的启动分子嵌合蛋白,不仅将工程细胞精确地靶向至损伤区,而且在TBI模型中提前关闭了血脑屏障,该启动分子不限于嵌入内皮祖细胞细胞膜上,也能在T淋巴细胞,神经干细胞,胶质细胞甚至更多的细胞表面表达,其胞外爱帕琳识别结构域可被其他多种潜在的配体分子所取代,意味着启动分子有十分广泛的应用范围;同时胞内的调控因子结构域可以根据需要替换成不同的反应元件,激活不同的下游响应程序,以此实现启动分子的功能多样化。
序列表
<110> 复旦大学附属华山医院
<120> 一种可特异性识别增殖状态内皮细胞的启动分子及工程细胞
<130> 2011245
<160> 5
<170> SIPOSequenceListing 1.0
<210> 1
<211> 77
<212> PRT
<213> 人(Homo sapiens )
<400> 1
Met Asn Leu Arg Leu Cys Val Gln Ala Leu Leu Leu Leu Trp Leu Ser
1 5 10 15
Leu Thr Ala Val Cys Gly Val Pro Leu Met Leu Pro Pro Asp Gly Thr
20 25 30
Gly Leu Glu Glu Gly Ser Met Arg Tyr Leu Val Lys Pro Arg Thr Ser
35 40 45
Arg Thr Gly Pro Gly Ala Trp Gln Gly Gly Arg Arg Lys Phe Arg Arg
50 55 60
Gln Arg Pro Arg Leu Ser His Lys Gly Pro Met Pro Phe
65 70 75
<210> 2
<211> 713
<212> PRT
<213> 人(Homo sapiens )
<400> 2
Pro Cys Val Gly Ser Asn Pro Cys Tyr Asn Gln Gly Thr Cys Glu Pro
1 5 10 15
Thr Ser Glu Asn Pro Phe Tyr Arg Cys Leu Cys Pro Ala Lys Phe Asn
20 25 30
Gly Leu Leu Cys His Ile Leu Asp Tyr Ser Phe Thr Gly Gly Ala Gly
35 40 45
Arg Asp Ile Pro Pro Pro Gln Ile Glu Glu Ala Cys Glu Leu Pro Glu
50 55 60
Cys Gln Val Asp Ala Gly Asn Lys Val Cys Asn Leu Gln Cys Asn Asn
65 70 75 80
His Ala Cys Gly Trp Asp Gly Gly Asp Cys Ser Leu Asn Phe Asn Asp
85 90 95
Pro Trp Lys Asn Cys Thr Gln Ser Leu Gln Cys Trp Lys Tyr Phe Ser
100 105 110
Asp Gly His Cys Asp Ser Gln Cys Asn Ser Ala Gly Cys Leu Phe Asp
115 120 125
Gly Phe Asp Cys Gln Leu Thr Glu Gly Gln Cys Asn Pro Leu Tyr Asp
130 135 140
Gln Tyr Cys Lys Asp His Phe Ser Asp Gly His Cys Asp Gln Gly Cys
145 150 155 160
Asn Ser Ala Glu Cys Glu Trp Asp Gly Leu Asp Cys Ala Glu His Val
165 170 175
Pro Glu Arg Leu Ala Ala Gly Thr Leu Val Leu Val Val Leu Leu Pro
180 185 190
Pro Asp Gln Leu Arg Asn Asn Ser Phe His Phe Leu Arg Glu Leu Ser
195 200 205
His Val Leu His Thr Asn Val Val Phe Lys Arg Asp Ala Gln Gly Gln
210 215 220
Gln Met Ile Phe Pro Tyr Tyr Gly His Glu Glu Glu Leu Arg Lys His
225 230 235 240
Pro Ile Lys Arg Ser Thr Val Gly Trp Ala Thr Ser Ser Leu Leu Pro
245 250 255
Gly Thr Ser Gly Gly Arg Gln Arg Arg Glu Leu Asp Pro Met Asp Ile
260 265 270
Arg Gly Ser Ile Val Tyr Leu Glu Ile Asp Asn Arg Gln Cys Val Gln
275 280 285
Ser Ser Ser Gln Cys Phe Gln Ser Ala Thr Asp Val Ala Ala Phe Leu
290 295 300
Gly Ala Leu Ala Ser Leu Gly Ser Leu Asn Ile Pro Tyr Lys Ile Glu
305 310 315 320
Ala Val Lys Ser Glu Pro Val Glu Pro Pro Leu Pro Ser Gln Leu His
325 330 335
Leu Met Tyr Val Ala Ala Ala Ala Phe Val Leu Leu Phe Phe Val Gly
340 345 350
Cys Gly Val Leu Leu Ser Arg Lys Arg Arg Arg Gln Leu Cys Ile Gln
355 360 365
Lys Leu Met Ser Asn Leu Leu Thr Val His Gln Asn Leu Pro Ala Leu
370 375 380
Pro Val Asp Ala Thr Ser Asp Glu Val Arg Lys Asn Leu Met Asp Met
385 390 395 400
Phe Arg Asp Arg Gln Ala Phe Ser Glu His Thr Trp Lys Met Leu Leu
405 410 415
Ser Val Cys Arg Ser Trp Ala Ala Trp Cys Lys Leu Asn Asn Arg Lys
420 425 430
Trp Phe Pro Ala Glu Pro Glu Asp Val Arg Asp Tyr Leu Leu Tyr Leu
435 440 445
Gln Ala Arg Gly Leu Ala Val Lys Thr Ile Gln Gln His Leu Gly Gln
450 455 460
Leu Asn Met Leu His Arg Arg Ser Gly Leu Pro Arg Pro Ser Asp Ser
465 470 475 480
Asn Ala Val Ser Leu Val Met Arg Arg Ile Arg Lys Glu Asn Val Asp
485 490 495
Ala Gly Glu Arg Ala Lys Gln Ala Leu Ala Phe Glu Arg Thr Asp Phe
500 505 510
Asp Gln Val Arg Ser Leu Met Glu Asn Ser Asp Arg Cys Gln Asp Ile
515 520 525
Arg Asn Leu Ala Phe Leu Gly Ile Ala Tyr Asn Thr Leu Leu Arg Ile
530 535 540
Ala Glu Ile Ala Arg Ile Arg Val Lys Asp Ile Ser Arg Thr Asp Gly
545 550 555 560
Gly Arg Met Leu Ile His Ile Gly Arg Thr Lys Thr Leu Val Ser Thr
565 570 575
Ala Gly Val Glu Lys Ala Leu Ser Leu Gly Val Thr Lys Leu Val Glu
580 585 590
Arg Trp Ile Ser Val Ser Gly Val Ala Asp Asp Pro Asn Asn Tyr Leu
595 600 605
Phe Cys Arg Val Arg Lys Asn Gly Val Ala Ala Pro Ser Ala Thr Ser
610 615 620
Gln Leu Ser Thr Arg Ala Leu Glu Gly Ile Phe Glu Ala Thr His Arg
625 630 635 640
Leu Ile Tyr Gly Ala Lys Asp Asp Ser Gly Gln Arg Tyr Leu Ala Trp
645 650 655
Ser Gly His Ser Ala Arg Val Gly Ala Ala Arg Asp Met Ala Arg Ala
660 665 670
Gly Val Ser Ile Pro Glu Ile Met Gln Ala Gly Gly Trp Thr Asn Val
675 680 685
Asn Ile Val Met Asn Tyr Ile Arg Asn Leu Asp Ser Glu Thr Gly Ala
690 695 700
Met Val Arg Leu Leu Glu Asp Gly Asp
705 710
<210> 3
<211> 790
<212> PRT
<213> 人(Homo sapiens )
<400> 3
Met Asn Leu Arg Leu Cys Val Gln Ala Leu Leu Leu Leu Trp Leu Ser
1 5 10 15
Leu Thr Ala Val Cys Gly Val Pro Leu Met Leu Pro Pro Asp Gly Thr
20 25 30
Gly Leu Glu Glu Gly Ser Met Arg Tyr Leu Val Lys Pro Arg Thr Ser
35 40 45
Arg Thr Gly Pro Gly Ala Trp Gln Gly Gly Arg Arg Lys Phe Arg Arg
50 55 60
Gln Arg Pro Arg Leu Ser His Lys Gly Pro Met Pro Phe Pro Cys Val
65 70 75 80
Gly Ser Asn Pro Cys Tyr Asn Gln Gly Thr Cys Glu Pro Thr Ser Glu
85 90 95
Asn Pro Phe Tyr Arg Cys Leu Cys Pro Ala Lys Phe Asn Gly Leu Leu
100 105 110
Cys His Ile Leu Asp Tyr Ser Phe Thr Gly Gly Ala Gly Arg Asp Ile
115 120 125
Pro Pro Pro Gln Ile Glu Glu Ala Cys Glu Leu Pro Glu Cys Gln Val
130 135 140
Asp Ala Gly Asn Lys Val Cys Asn Leu Gln Cys Asn Asn His Ala Cys
145 150 155 160
Gly Trp Asp Gly Gly Asp Cys Ser Leu Asn Phe Asn Asp Pro Trp Lys
165 170 175
Asn Cys Thr Gln Ser Leu Gln Cys Trp Lys Tyr Phe Ser Asp Gly His
180 185 190
Cys Asp Ser Gln Cys Asn Ser Ala Gly Cys Leu Phe Asp Gly Phe Asp
195 200 205
Cys Gln Leu Thr Glu Gly Gln Cys Asn Pro Leu Tyr Asp Gln Tyr Cys
210 215 220
Lys Asp His Phe Ser Asp Gly His Cys Asp Gln Gly Cys Asn Ser Ala
225 230 235 240
Glu Cys Glu Trp Asp Gly Leu Asp Cys Ala Glu His Val Pro Glu Arg
245 250 255
Leu Ala Ala Gly Thr Leu Val Leu Val Val Leu Leu Pro Pro Asp Gln
260 265 270
Leu Arg Asn Asn Ser Phe His Phe Leu Arg Glu Leu Ser His Val Leu
275 280 285
His Thr Asn Val Val Phe Lys Arg Asp Ala Gln Gly Gln Gln Met Ile
290 295 300
Phe Pro Tyr Tyr Gly His Glu Glu Glu Leu Arg Lys His Pro Ile Lys
305 310 315 320
Arg Ser Thr Val Gly Trp Ala Thr Ser Ser Leu Leu Pro Gly Thr Ser
325 330 335
Gly Gly Arg Gln Arg Arg Glu Leu Asp Pro Met Asp Ile Arg Gly Ser
340 345 350
Ile Val Tyr Leu Glu Ile Asp Asn Arg Gln Cys Val Gln Ser Ser Ser
355 360 365
Gln Cys Phe Gln Ser Ala Thr Asp Val Ala Ala Phe Leu Gly Ala Leu
370 375 380
Ala Ser Leu Gly Ser Leu Asn Ile Pro Tyr Lys Ile Glu Ala Val Lys
385 390 395 400
Ser Glu Pro Val Glu Pro Pro Leu Pro Ser Gln Leu His Leu Met Tyr
405 410 415
Val Ala Ala Ala Ala Phe Val Leu Leu Phe Phe Val Gly Cys Gly Val
420 425 430
Leu Leu Ser Arg Lys Arg Arg Arg Gln Leu Cys Ile Gln Lys Leu Met
435 440 445
Ser Asn Leu Leu Thr Val His Gln Asn Leu Pro Ala Leu Pro Val Asp
450 455 460
Ala Thr Ser Asp Glu Val Arg Lys Asn Leu Met Asp Met Phe Arg Asp
465 470 475 480
Arg Gln Ala Phe Ser Glu His Thr Trp Lys Met Leu Leu Ser Val Cys
485 490 495
Arg Ser Trp Ala Ala Trp Cys Lys Leu Asn Asn Arg Lys Trp Phe Pro
500 505 510
Ala Glu Pro Glu Asp Val Arg Asp Tyr Leu Leu Tyr Leu Gln Ala Arg
515 520 525
Gly Leu Ala Val Lys Thr Ile Gln Gln His Leu Gly Gln Leu Asn Met
530 535 540
Leu His Arg Arg Ser Gly Leu Pro Arg Pro Ser Asp Ser Asn Ala Val
545 550 555 560
Ser Leu Val Met Arg Arg Ile Arg Lys Glu Asn Val Asp Ala Gly Glu
565 570 575
Arg Ala Lys Gln Ala Leu Ala Phe Glu Arg Thr Asp Phe Asp Gln Val
580 585 590
Arg Ser Leu Met Glu Asn Ser Asp Arg Cys Gln Asp Ile Arg Asn Leu
595 600 605
Ala Phe Leu Gly Ile Ala Tyr Asn Thr Leu Leu Arg Ile Ala Glu Ile
610 615 620
Ala Arg Ile Arg Val Lys Asp Ile Ser Arg Thr Asp Gly Gly Arg Met
625 630 635 640
Leu Ile His Ile Gly Arg Thr Lys Thr Leu Val Ser Thr Ala Gly Val
645 650 655
Glu Lys Ala Leu Ser Leu Gly Val Thr Lys Leu Val Glu Arg Trp Ile
660 665 670
Ser Val Ser Gly Val Ala Asp Asp Pro Asn Asn Tyr Leu Phe Cys Arg
675 680 685
Val Arg Lys Asn Gly Val Ala Ala Pro Ser Ala Thr Ser Gln Leu Ser
690 695 700
Thr Arg Ala Leu Glu Gly Ile Phe Glu Ala Thr His Arg Leu Ile Tyr
705 710 715 720
Gly Ala Lys Asp Asp Ser Gly Gln Arg Tyr Leu Ala Trp Ser Gly His
725 730 735
Ser Ala Arg Val Gly Ala Ala Arg Asp Met Ala Arg Ala Gly Val Ser
740 745 750
Ile Pro Glu Ile Met Gln Ala Gly Gly Trp Thr Asn Val Asn Ile Val
755 760 765
Met Asn Tyr Ile Arg Asn Leu Asp Ser Glu Thr Gly Ala Met Val Arg
770 775 780
Leu Leu Glu Asp Gly Asp
785 790
<210> 4
<211> 291
<212> DNA
<213> 人(Homo sapiens )
<400> 4
atggccttac cagtgaccgc cttgctcctg ccgctggcct tgctgctcca cgccgccagg 60
ccgaatctga ggctctgcgt gcaggcgctg ctgctgctct ggctctcctt gactgcagtt 120
tgtggagtgc cactgatgtt gcctccagat ggaacaggac tagaagaagg aagcatgcgc 180
tacctggtga agcccagaac ttcgaggact ggaccaggag cctggcaggg aggcaggagg 240
aaatttcgca gacagcgccc ccggctctcc cataagggcc ccatgccttt c 291
<210> 5
<211> 1757
<212> DNA
<213> 人(Homo sapiens )
<400> 5
ccctgtgtgg gtagcaaccc ctgctacaat cagggcacct gtgagcccac atccgagaac 60
cctttctacc gctgtctatg ccctgccaaa ttcaacgggc tactgtgcca catcctggac 120
tacagcttca caggtggcgc tgggcgcgac attcccccac cgcagattga ggaggcctgt 180
gagctgcctg agtgccaggt ggatgcaggc aataaggtct gcaacctgca gtgtaataat 240
cacgcatgtg gctgggatgg tggcgactgc tccctcaact tcaatgaccc ctggaagaac 300
tgcacgcagt ctctacagtg ctggaagtat tttagcgacg gccactgtga cagccagtgc 360
aactcggccg gctgcctctt tgatggcttc gactgccagc tcaccgaggg acagtgcaac 420
cccctgtatg accagtactg caaggaccac ttcagtgatg gccactgcga ccagggctgt 480
aacagtgccg aatgtgagtg ggatggccta gactgtgctg agcatgtacc cgagcggctg 540
gcagccggca ccctggtgct ggtggtgctg cttccacccg accagctacg gaacaactcc 600
ttccactttc tgcgggagct cagccacgtg ctgcacacca acgtggtctt caagcgtgat 660
gcgcaaggcc agcagatgat cttcccgtac tatggccacg aggaagagct gcgcaagcac 720
ccaatcaagc gctctacagt gggttgggcc acctcttcac tgcttcctgg taccagtggt 780
gggcgccagc gcagggagct ggaccccatg gacatccgtg gctccattgt ctacctggag 840
atcgacaacc ggcaatgtgt gcagtcatcc tcgcagtgct tccagagtgc caccgatgtg 900
gctgccttcc taggtgctct tgcgtcactt ggcagcctca atattcctta caagattgag 960
gccgtgaaga gtgagccggt ggagcctccg ctgccctcgc agctgcacct catgtacgtg 1020
gcagcggccg ccttcgtgct cctgttcttt gtgggctgtg gggtgctgct gtcccgcaag 1080
cgccggcggt ctagactgga caagagcaaa gtcataaact ctgctctgga attactcaat 1140
gaagtcggta tcgaaggcct gacgacaagg aaactcgctc aaaagctggg agttgagcag 1200
cctaccctgt actggcacgt gaagaacaag cgggccctgc tcgatgccct ggcaatcgag 1260
atgctggaca ggcatcatac ccacttctgc cccctggaag gcgagtcatg gcaagacttt 1320
ctgcggaaca acgccaagtc attccgctgt gctctcctct cacatcgcga cggggctaaa 1380
gtgcatctcg gcacccgccc aacagagaaa cagtacgaaa ccctggaaaa tcagctcgcg 1440
ttcctgtgtc agcaaggctt ctccctggag aacgcactgt acgctctgtc cgccgtgggc 1500
cactttacac tgggctgcgt attggaggat caggagcatc aagtagcaaa agaggaaaga 1560
gagacaccta ccaccgattc tatgccccca cttctgagac aagcaattga gctgttcgac 1620
catcagggag ccgaacctgc cttccttttc ggcctggaac taatcatatg tggcctggag 1680
aaacagctaa agtgcgaaag cggcgggccg gccgacgccc ttgacgattt tgacttagac 1740
atgctcccag ccgatgc 1757
- 一种可特异性识别增殖状态内皮细胞的启动分子及工程细胞
- 一种促进内皮细胞增殖的合欢皮木脂素苷类化合物及应用
