一种鼓槌石斛SSR分子标记引物组合物及其应用
文献发布时间:2023-06-19 10:49:34
技术领域
本发明涉及分子生物学技术领域,具体涉及一种鼓槌石斛SSR分子标记引物组合物及其应用。
背景技术
鼓槌石斛(Dendrobiumchrysotoxum)为兰科石斛属多年附生草本植物,是集观赏、食、药为一体且栽培较广的石斛种类之一,被收录于2020年版《中国药典》。其茎、花晾干后泡水喝,具有美容明目、安神补血、抗氧化和增强免疫力等功效。同时,鼓槌石斛茎肉质纺锤形,叶四季常绿,花金黄色,花量大,稍带香气,花期长达1个月,具有较高的观赏价值,可作大小盆栽、垂直悬挂、附生石块上或树枝上等多样栽培和应用。鼓槌石斛原产于我国西南地区,生于海拔500-1600m,阳光充足的常绿阔叶林中树干上或疏林下岩石上,国外分布于印度东北部、缅甸、泰国、老挝和越南。
SSR分子标记具有使用简便、快速、稳定性高、等位基因多样性高以及可做共显性的等位基因分析等特点,目前已成为一种主要的分子标记技术,被广泛应用于植物遗传多样性分析、群体结构分析、品种鉴定和种质资源亲缘关系分析等多方面研究领域。如沈颖等采用ISSR-PCR的方法对石斛属9种植物进行鉴别,包括美花石斛、束花石斛、叠鞘石斛、鼓槌石斛、细叶石斛、罗河石斛、球花石斛、玫瑰石斛和铁皮石斛(沈颖,徐程,万小凤,等.ISSR-PCR在石斛种间鉴别中的应用[J].中草药,2005,36(3):423-427.)。专利CN104313162A则公开了一种用于鉴别鼓槌石斛和球花石斛的分子特异性标记引物及其方法。
近年来,在铁皮石斛、金钗石斛等石斛属植物上开发了大量的SSR引物,但同属的鼓槌石斛则尚未有开发的SSR引物的报道,其遗传变异分析仍采用RAPD等分析标记,例如Tikendra等(2019)利用12对RAPD引物和11对ISSR引物评估了组培过程中鼓槌石斛克隆苗的遗传稳定性,发现克隆苗与母株间较高的遗传单态性(96.3%)和较低的多态性(3.6%)。鼓槌石斛作为一种观赏兼药用的植物,目前生产上尚未见通过审定且广泛栽培的鼓槌石斛品种,生产上主要利用其野生资源或组培无菌播种获得的实生苗或克隆苗,而对这些鼓槌石斛种质资源进行鉴定、保育及开发利用也越来越受到各界的重视。
因此,寻找并开发适用于鼓槌石斛遗传研究的SSR引物是进一步开展鼓槌石斛种质资源管理、遗传、保护等研究的前提基础。
发明内容
针对上述不足,本发明提供了一种鼓槌石斛SSR分子标记引物组合物及其应用。本发明筛选出在鼓槌石斛中扩增效果好,多态性高的25对SSR分子标记引物,采用所述的SSR分子标记引物,可对鼓槌石斛群体的遗传多样性及亲缘关系进行分析,也可对鼓槌石斛无性系或品种进行种质鉴定和亲缘关系分析,具有高效、准确、实用、不受季节影响等优点,为后续鼓槌石斛种质资源管理、育种、遗传保护、群体间和群体内的遗传多样性分析等奠定了基础。
为了实现上述发明目的,本发明的技术方案如下:
一方面,本发明提供了一种鼓槌石斛SSR分子标记引物组合物。
具体地,所述的引物组合物如下表1所示:
表1.引物组合物
另一方面,本发明提供了一种鼓槌石斛种质鉴定、遗传多样性或亲缘关系分析的产品,所述的产品包含上述SSR分子标记引物组合物。
具体地,所述的产品为独立试剂或试剂盒。
又一方面,本发明提供了上述SSR分子标记引物组合物在制备鼓槌石斛种质鉴定、遗传多样性或亲缘关系分析产品中的应用。
又一方面,本发明提供了上述SSR分子标记引物组合物或产品在鼓槌石斛种质鉴定、遗传多样性或亲缘关系分析中的应用。
又一方面,本发明提供了一种鼓槌石斛种质鉴定、遗传多样性或亲缘关系分析的方法,所述的方法包括以下步骤:
(1)提取待测鼓槌石斛样本的基因组DNA;
(2)以步骤(1)提取的基因组DNA为模板进行PCR扩增,用于PCR扩增的引物对的上游引物采用生物荧光基团进行修饰;
(3)将步骤(2)获得的PCR扩增产物进行毛细管荧光电泳检测,收集数据;
(4)根据步骤(3)获得的数据统计分析鼓槌石斛群体的遗传多样性或聚类及多态信息含量。
具体地,步骤(2)中所述的PCR扩增体系为:反应体系总体积为10μL,包括1.0μLDNA模板(50ng/μL),1.0μL 10×BufferΙ缓冲液,0.1μL TAKARA HS Taq酶(5U/μL),引物(5μM)0.6μL,0.8μL2.5mM dNTP,去离子水补足至10μL。
进一步具体地,所述的PCR扩增体系的引物为SEQ ID NO:1-SEQ ID NO:50所示的引物。
进一步具体地,所述的PCR扩增反应程序:95℃预变性5min;95℃变性30s,依据引物的退火温度复性30s,72℃延伸30s,40个循环;60℃延伸10min。
又一方面,本发明还提供了上述SSR分子标记扩增引物组合物或产品在鼓槌石斛分子标记辅助育种中的应用。
与现有技术相比,本发明的积极和有益效果在于:
1、本发明提供了一种鼓槌石斛SSR分子标记及引物组合物,可对鼓槌石斛进行种质鉴定、遗传多样性或亲缘关系分析,为鼓槌石斛种质资源管理、育种、遗传保护、鼓槌石斛群体间和群体内的遗传多样性分析等奠定了基础。
2、本发明采用所述的鼓槌石斛SSR分子标记及引物组合物,在鼓槌石斛中扩增效果好,多态性高,可准确、高效、稳定地鉴定鼓槌石斛种质资源,操作简便。
3、本发明所述的鼓槌石斛SSR分子标记可用于辅助选择育种,从而加快鼓槌石斛的育种进程。
附图说明
图1为SSR分子标记引物DC 11在7个鼓槌石斛植株中的扩增位点图。
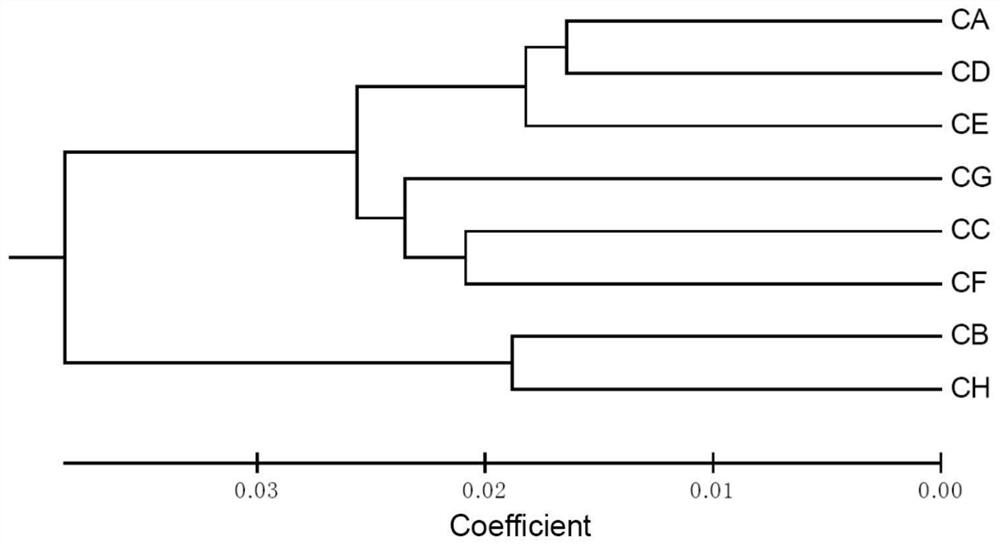
图2为8个鼓槌石斛群体的聚类分析图。
图3为12个鼓槌石斛无性系的聚类分析图。
具体实施方式
下面结合具体实施例,对本发明作进一步详细的阐述,下述实施例不用于限制本发明,仅用于说明本发明。以下实施例中所使用的实验方法如无特殊说明,实施例中未注明具体条件的实验方法,通常按照常规条件,下述实施例中所使用的材料、试剂等,如无特殊说明,均可从商业途径得到。
除非另外定义,否则本文中所用的全部技术与科学用语均具有本领域技术人员通常理解的含义。
实施例1鼓槌石斛SSR引物筛选
1.本发明利用鼓槌石斛转录组序列,设计并筛选得到100对SSR引物,在生工生物工程(上海)股份有限公司中合成后作为鼓槌石斛候选引物,且每对引物的上游引物分别采用FAM(蓝)、HEX(绿)2种生物荧光基团进行修饰。
2.随机选取7棵鼓槌石斛植株(实生苗)作为试验材料,剪取其嫩叶,利用DP305试剂盒(中国天根)提取其基因组DNA,分别采用1.2%琼脂糖凝胶电泳和NanoDrop分光光度计检测DNA的质量和浓度。
3.以样品DNA为模板,以荧光修饰引物及下游引物进行PCR扩增,PCR扩增反应体系为:反应体系总体积为10μL,包括1.0μL DNA模板(50ng/μL),1.0μL 10×BufferΙ缓冲液,0.1μL TAKARA HS Taq酶(5U/μL),引物(5μM)0.6μL,0.8μL 2.5mM dNTP,去离子水补足至10μL。PCR扩增程序为:95℃预变性5min;95℃变性30s,依据引物的退火温度复性30s,72℃延伸30s,40个循环;60℃延伸10min,从而得到扩增产物。
4.将PCR扩增产物进行毛细管荧光电泳检测,即将扩增产物利用ABI 3730xl DNAAnalyzer基因分析仪进行检测,并利用GeneMarker 2.2.0软件读取结果,统计每对引物扩增的特征性谱带与多态性谱带大小,并筛选具有多态性的SSR分子标记引物。
5.最终筛选得到以下25对SSR分子标记引物(表2)。以引物DC 11为例,该SSR引物对在7棵鼓槌石斛植株中扩增的毛细管电泳检测图见图1。
表2.SSR引物
实施例2SSR引物组合物在鼓槌石斛群体遗传多样性分析中的应用
1.试验材料为不同来源的8个鼓槌石斛群体(表3),每个群体采样8-14株,每棵植株单独编号,分别标为CA1-CA12、CB1-CB12、CC1-CC10、CD1-CD14、CE1-CE8、CF1-CF8、CG1-CG12、CH1-CH8,共采样84棵单株。
表3. 8个鼓槌石斛群体试验材料
2.剪取每棵植株的嫩叶,利用DP305试剂盒(中国天根)提取鼓槌石斛基因组DNA,分别采用1.2%琼脂糖凝胶电泳和NanoDrop分光光度计检测DNA的质量和浓度。
3.在生工生物工程(上海)股份有限公司中合成表2中的25对SSR引物,且每对引物的上游引物分别采用FAM(蓝)、HEX(绿)2种生物荧光基团进行修饰。利用所述引物及退火温度进行PCR扩增。
4.将扩增产物利用ABI 3730xl DNA Analyzer基因分析仪进行检测,并利用GeneMarker 2.2.0软件读取结果,统计每对引物扩增的特征性谱带、多态性谱带大小。利用软件POPGENE 1.32计算每个SSR位点的等位基因数量(Na)、有效等位基因数(Ne)、观察杂合度(Ho)、期望杂合度(He)、群体内繁育系数(F
表4.25个SSR位点的遗传分析结果
5.参考步骤4,利用软件POPGENE 1.32计算各群体的等位基因数量(Na)、有效等位基因数(Ne)、观察杂合度(Ho)、期望杂合度(He)、群体内繁育系数(F
表5.鼓槌石斛群体的遗传多样性
6.利用软件Arlequin 3.5.2.2计算成对的赖特固定指数(F
表6.鼓槌石斛群体的成对赖特固定指数F
注:**表示显著性(P<0.01)。
表7.25个SSR位点的分子方差分析
实施例3不同鼓槌石斛无性系的鉴定与亲缘关系分析
1.由于目前市场上还没有通过审定的广泛栽培的鼓槌石斛品种,生产上鼓槌石斛主要以播种繁殖(实生苗)、分株或组织培养繁育种苗。本申请经筛选得到了12个鼓槌石斛优良单株,利用分株和组培的方式构建了12个鼓槌石斛无性系,分别编号为Line 1-Line12,其主要形态特征见表8。以上述的12个无性系为供试材料,剪取每个无性系的嫩叶,利用DP305试剂盒(中国天根)提取鼓槌石斛基因组DNA,分别采用1.2%琼脂糖凝胶电泳和NanoDrop分光光度计检测DNA的质量和浓度。
2.利用实施例1筛选出的25对SSR引物及其退火温度进行PCR扩增。
3.将扩增产物利用ABI 3730xl DNA Analyzer基因分析仪进行检测,并利用GeneMarker 2.2.0软件读取结果,统计每对引物扩增的特征性谱带、多态性谱带大小。
4.利用NTSYS pc Version 2.10e软件进行聚类分析,结果见图3。由图3可知,25对SSR引物能够将上述12个供试的鼓槌石斛无性系完全鉴定和区分开来。在相对距离0.763处,12个供试的鼓槌石斛无性系可以分为4个类群,无性系Line 1、4、5、7、6、8、9聚为一类,无性系Line 2、11、12聚为一类,而无性系Line 3为一类,无性系Line 10为一类,这也说明无性系Line 1和4的亲缘关系最近,而无性系Line 3和10与其他10个无性系的亲缘关系要远一些。
表8.12个鼓槌石斛无性系的主要形态特征统计
实验例1准确性及重复性检测
根据本发明所述的SSR分子标记引物组合物,选取实施例2的8个鼓槌石斛群体各50个样品进行遗传多样性分析,与实施例2的遗传多样性结果进行比较,对其一致性与准确性进行检测,检测结果如下表9所示。
表9.准确性检测结果
由表9可知,采用本发明所述的SSR分子标记引物组合物,可准确鉴定鼓槌石斛群体中的单株,准确率及重复性为100%。
本发明所述的SSR分子标记可用于辅助选择育种,从而加快鼓槌石斛的育种进程。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110> 广东省农业科学院环境园艺研究所
<120> 一种鼓槌石斛SSR分子标记引物组合物及其应用
<130> 20210218
<160> 50
<170> SIPOSequenceListing 1.0
<210> 1
<211> 19
<212> DNA
<213> 人工序列(artificial sequence)
<400> 1
cagtggaggt ctgtttgat 19
<210> 2
<211> 18
<212> DNA
<213> 人工序列(artificial sequence)
<400> 2
aggagcagcg agtttagt 18
<210> 3
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 3
agttcttgtg gagccctgtg 20
<210> 4
<211> 18
<212> DNA
<213> 人工序列(artificial sequence)
<400> 4
gccatcacca accaccct 18
<210> 5
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 5
aaagcgtcaa taagggaact 20
<210> 6
<211> 19
<212> DNA
<213> 人工序列(artificial sequence)
<400> 6
agcaatggag cgaagatag 19
<210> 7
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 7
aagcagcggg aggagcagaa 20
<210> 8
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 8
acatcgcagt catcaatgta a 21
<210> 9
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 9
caggatgctc taagccagta 20
<210> 10
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 10
aaagaacaat gaaaggcaag 20
<210> 11
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 11
tccgtgtcct tgtttcttac 20
<210> 12
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 12
tcttccctct ttcctcttct 20
<210> 13
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 13
tggagcaaga cttgtctaag c 21
<210> 14
<211> 22
<212> DNA
<213> 人工序列(artificial sequence)
<400> 14
acttgagatt agcaaacagc ac 22
<210> 15
<211> 22
<212> DNA
<213> 人工序列(artificial sequence)
<400> 15
tcatggctca catcatattg tc 22
<210> 16
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 16
tacgctctta caggacttga c 21
<210> 17
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 17
gatgattggt aaagtggagt g 21
<210> 18
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 18
aacctacaaa ttcaaccctc t 21
<210> 19
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 19
gaataatgct tgagaggagg t 21
<210> 20
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 20
caacatttac atagcaaagc a 21
<210> 21
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 21
ggccacttac ctttctttct a 21
<210> 22
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 22
gaagagaagc gagagaagat t 21
<210> 23
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 23
gacattctcc gtcaggtact a 21
<210> 24
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 24
ctcacgccac taaaatcttc 20
<210> 25
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 25
tgatgatata atcctcgcaa t 21
<210> 26
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 26
tgcagttcta tcgtaccagt t 21
<210> 27
<211> 18
<212> DNA
<213> 人工序列(artificial sequence)
<400> 27
cggtggtcgt acagagag 18
<210> 28
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 28
cttgtgctca tgaatcttct c 21
<210> 29
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 29
tctttcctct cttcctccta a 21
<210> 30
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 30
cttctcgttc acgtaaacac t 21
<210> 31
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 31
agtggttacg gtcacttacc t 21
<210> 32
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 32
gagaagattc caaccatatc c 21
<210> 33
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 33
gatgtggtgt tgtgctagat t 21
<210> 34
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 34
tagattgctt gttcaggcta c 21
<210> 35
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 35
taactagaag cctttgctca g 21
<210> 36
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 36
gaagctccat gttaatcaca g 21
<210> 37
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 37
agtcgcctcc tatattactg g 21
<210> 38
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 38
cacacgacaa gacttcacat a 21
<210> 39
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 39
tagcttatcg tgagagattg g 21
<210> 40
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 40
gtcaggcaca tcacaaataa t 21
<210> 41
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 41
ttcagagttc ttggtgacat t 21
<210> 42
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 42
cagacccaaa ctctacctgt t 21
<210> 43
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 43
ctctacctca ccgtgttccg 20
<210> 44
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 44
ctcttgccat tgctctgctt 20
<210> 45
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 45
atgacgccat gtaccactcc 20
<210> 46
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 46
acaacgattc gcaccagttc 20
<210> 47
<211> 21
<212> DNA
<213> 人工序列(artificial sequence)
<400> 47
ctttctacca ccaggttgtc a 21
<210> 48
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 48
ccataaagtc ggtccatagc 20
<210> 49
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 49
tttgttttcc actatttctg 20
<210> 50
<211> 20
<212> DNA
<213> 人工序列(artificial sequence)
<400> 50
tctaacatta caagctctgc 20
- 一种鼓槌石斛SSR分子标记引物组合物及其应用
- 一种兰科植物通用型SSR分子标记引物组合物及其应用
