一种染色体非整倍体数目异常的扩增组合物及其应用
文献发布时间:2023-06-19 11:40:48
技术领域
本发明属于基因检测领域,具体涉及一种染色体非整倍体数目异常的扩增组合物及其应用。
背景技术
常见的常染色体非整倍体疾病包括21三体综合征(Down syndrome)、18三体综合征(Edwards syndrome)、13三体综合征(Patau syndrome)、以及一些性染色体畸变,如Klinefelter综合征(47,XXY)和Turner综合征(45,X)等。目前,临床上以染色体核型分析为诊断金标准,然而该技术有很大局限性。需要进行羊膜穿刺取羊水,然后培养羊水中胎儿细胞,再进行核型分析。需要的羊水样本量大,一次羊水细胞培养需要20mL左右羊水样本,分析周期时间长,一般在2-3周左右,给孕妇及其家庭在等待结果过程中带来很大的精神压力,且操作复杂,需要有经验的检测人员操作。另外近几年应用的比较多的技术为荧光原位杂交(FISH),这一方法速度稍快,无需细胞培养,为利用已知核酸序列作为探针,以荧光素直接标记或以非放射性物质标记后与靶DNA进行杂交。再通过免疫细胞化学过程连接上荧光素标记物,最后在荧光显微镜下观察杂交信号从而对标本中待测核酸进行定性、定位和定量分析。该方法成本较高,周期长,对操作人员要求较高的特点,同时结果为图片,需要人工判读或干预,难以实现自动化和高通量分析。
QF-PCR技术是通过荧光引物对样本DNA的不同区域进行PCR扩增,采用毛细管电泳对扩增片段进行分离,通过荧光检测系统确定扩增片段的长度并实现对多态性位点的分型,通过对扫描软件对预期大小的扩增产物对应等面积定量,实验对原始模板的定量,可应用于非整倍体疾病检测。QF-PCR中同时扩增多个13、18、21、X和Y染色体上短串联重复序列(Short Tandem Repeats,STR)基因座,利用STR基因座的多态性来判断染色体的数目。非整倍体数目异常是由染色体数目增多或减少一条引起的疾病,因此对染色体上STR基因座经过QF-PCR扩增、检测会得到1:1:1的三峰或2:1的双峰或一个峰,其中前两种情况是三体综合征样本相对于正常样本所特有。STR位点多态性越高则出现单一峰的几率越小,出现前两种个情况的比例越大。对多个特定染色体上STR位点进行检测,综合考量各个位点峰数量和峰面积即可判定检测样本是否存在三体综合征。且QF-PCR技术检测周期短,获得样本后5小时就可以得到结果。采用羊水细胞提取的基因组DNA进行扩增,扩增加检测时间共计5小时,大大加快了速度。
现有技术中已有基于QF-PCR的13、18、21、X和Y染色体非整倍体数目异常的扩增,比如申请人早期的专利CN104651488A,不过实践中发现该方法存在一定的检测效率和灵敏度问题,不能满足一些实际应用。
有鉴于此,提出本发明。
发明内容
本发明的首要目的是寻求一种检测效率和灵敏度显著提高的染色体非整倍体数目异常的扩增或检测方法。
本发明的另一目的是寻求一种检测效率和灵敏度显著提高的的染色体非整倍体数目异常的扩增组合物及其检测试剂盒。
为实现上述目的,本发明在申请人早期专利CN104651488A检测染色体非整倍体数目异常的扩增组合物及快速检测试剂盒的基础上,进行技术改进:对STR基因座的排布与位点选择进行改进,改进后的试剂盒不仅扩增检测效率更高,还整合了尿嘧啶DNA糖基化酶(UDG)防污染体系,可在PCR扩增之前消化可能存在的含有尿嘧啶的扩增产物,从而避免产物污染,检测结果准确性更高。
具体的,本发明首先提供一种扩增或检测染色体非整倍体数目异常的扩增组合物,其特征在于,所述扩增组合物包括如下引物:
所述引物分别针对D13S256、D13S797、DXS6809、DXS9895、D21S2052、TAF9L、D18S535、D13S317、D21S1411、D18S1002、D13S305、AMEL、D18S877、LFG21、ZFXY、D18S851、D21S1435、D18S391、D13S800、D21S1246、XHPRT、D13S325、GATA165B12、D21S1446、D21S11和SRY。
在一些实施方式中,所述引物序列如SEQ ID NO.1-52所示。
进一步的,针对D13S256、D13S797、DXS6809、DXS9895、D21S2052、TAF9L、D18S535、D13S317、D21S1411、D18S1002、D13S305、AMEL、D18S877、LFG21、ZFXY、D18S851、D21S1435、D18S391、D13S800、D21S1246、XHPRT、D13S325、GATA165B12、D21S1446、D21S11和SRY的引物浓度或用量比如表5中所示,或如下所示:1:0.35:0.65:1.2:0.35:0.3:3.1:0.35:1.5:1.3:0.65:0.75:0.85:0.7:0.75:1.15:0.5:1.65:0.35:3:0.45:0.65:0.6:0.6:0.9:0.4。
进一步的,所述引物可分为五组:
第一组为D13S256、D13S797、DXS6809、DXS9895、D21S2052;第二组为TAF9L、D18S535、D13S317、D21S1411、D18S1002、D13S305;第三组为AMEL、D18S877、LFG21、ZFXY、D18S851、D21S1435;第四组为D18S391、D13S800、D21S1246、XHPRT、D13S325;第五组为GATA165B12、D21S1446、D21S11、SRY;在一些实施方式中,不同组引物带有不同的荧光标记物;
优选的,第一组用FAM标记,第二组用HEX标记,第三组用L552标记,第四组用LR600标记,第五组用TET592标记。
本发明还提供了上述扩增组合物的如下任一用途:
1)在检测染色体非整倍体数目异常中的用途;
2)在制备检测染色体非整倍体数目异常的试剂或试剂盒中的用途;
3)在诊断染色体非整倍体异常相关疾病中的用途;
4)在制备诊断染色体非整倍体异常相关疾病诊断试剂或试剂盒中的用途。
在一些实施方式中,在具体的检测或诊断用途中,通过以下标准去进行结果分析:
所述扩增检测产物经毛细管电泳后数据分析,分析结果的判断标准如下:
正常位点表现为双峰,峰面积比值在0.8-1.4之间;异常位点表现为三峰,峰面积比值接近于1:1:1,或表现为双峰,峰面积比值在0.45-0.65或1.8-2.4之间;无效位点峰面积比值介于正常位点和异常位点区间之外;
在一些优选的实施方式中,具体判断标准如下:
1)正常结果判定:
至少两个STR位点为正常位点,表现为双峰,前后峰面积比值在0.8-1.4之间,其余位点为无效位点(峰面积比值介于正常位点和异常位点区间之外);
2)13、18、21染色体非整倍体结果判定:
13、18或21号染色体上至少两个STR遗传位点为异常位点表现为三峰,峰面积比值接近于1:1:1,或表现为双峰,峰面积比值在0.45-0.65或1.8-2.4之间,其余位点为无效位点峰面积比值介于正常位点和异常位点区间之外。
本发明还提供一种检测染色体非整倍体数目异常的试剂盒,其特征在于,所述试剂盒包括上述的检测染色体非整倍体数目异常的扩增组合物。
在一些实施方式中,所述试剂盒中进一步包括PCR反应液、酶混合液及质控品;
在一些实施方式中,所述酶混合液包括DNA聚合酶和UDG酶;
在一些实施方式中,所述PCR反应液中包含dUTP,优选的,所述dUTP/(dTTP+dUTP)为0.1-0.5;
在另一些实施方式中,所述质控品还包括21三体阳性质控品等。
本发明还提供一种染色体非整倍体数目异常的扩增或检测方法,其特征在于,所述方法包括采用上述扩增组合物或试剂盒进行扩增检测。
进一步的,所述扩增检测产物经毛细管电泳后数据分析,分析结果的判断标准如下:
正常位点表现为双峰,峰面积比值在0.8-1.4之间;异常位点表现为三峰,峰面积比值接近于1:1:1,或表现为双峰,峰面积比值在0.45-0.65或1.8-2.4之间;无效位点峰面积比值介于正常位点和异常位点区间之外;
在一些优选的实施方式中,具体判断标准如下:
1)正常结果判定:
至少两个STR位点为正常位点,表现为双峰,前后峰面积比值在0.8-1.4之间,其余位点为无效位点(峰面积比值介于正常位点和异常位点区间之外);
2)13、18、21染色体非整倍体结果判定:
13、18或21号染色体上至少两个STR遗传位点为异常位点表现为三峰,峰面积比值接近于1:1:1,或表现为双峰,峰面积比值在0.45-0.65或1.8-2.4之间,其余位点为无效位点峰面积比值介于正常位点和异常位点区间之外。
相比于现有技术,本发明至少具有如下有益技术效果:
(1)相比于申请人早期专利,本发明通过优化调整检测位点、引物序列、分组、用量以及反应体系等,显著提高了扩增检测的效率、灵敏度以及准确性。小的PCR产物比大的更容易被有效的扩增,本发明PCR扩增产物均在400bp以内,且使用了能量传递引物使扩增检测效率更高。
(2)相较于传统方法,本发明灵敏度更高,每次检测只需纳克级别的DNA,需要的样本量少,0.5-1mL的羊水即可满足需求;准确性高,与荧光原位杂交(FISH)相比,本技术对人员操作要求较低,发生污染的几率更小。由于采用了可靠的PCR技术和遗传分析仪检测,能够实现自动化和数字化分析数据,无需人工干预,结果更加客观和准确;检测周期短,获得样本后扩增加检测时间最快5小时就可以得到结果。
(3)本发明还整合了尿嘧啶DNA糖基化酶(UDG)防污染体系,可在PCR扩增之前消化可能存在的含有尿嘧啶的扩增产物,从而避免产物污染,检测结果准确性更高;可清晰判断样本母源细胞污染情况。
附图说明
为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
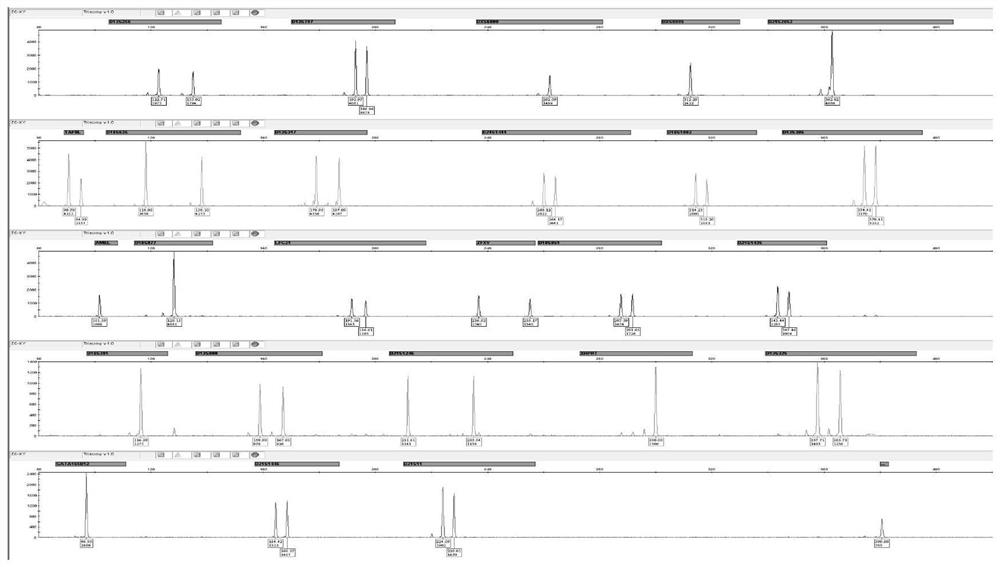
图1正常男性检测结果图;
图2正常女性检测结果图;
图3 13三体综合征检测结果图;
图4 18三体综合征检测结果图;
图5 21三体综合征检测结果图;
图6XXY综合征检测结果图;
图7XYY综合征检测结果图;
图8X单体综合征检测结果图;
图9XXX综合征检测结果图;
图10专利CN104651488A 13三体综合征1ng/μL检测结果图
图11专利CN104651488A 13三体综合征2ng/μL检测结果图
图12本发明13三体综合征1ng/μL检测结果图
图13本发明13三体综合征2ng/μL检测结果图
图14本发明与专利CN104651488A 13三体综合征对比峰高图,其中,上图为CN104651488A峰图,下图为本专利峰图。
图15本发明与专利CN104651488A XXY综合征对比峰高图,其中,上图为CN104651488A峰图,下图为本专利峰图。
具体实施方式
下面将结合附图对本发明的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
以下术语或定义仅仅是为了帮助理解本发明而提供。这些定义不应被理解为具有小于本领域技术人员所理解的范围。
除非在下文中另有定义,本发明具体实施方式中所用的所有技术术语和科学术语的含义意图与本领域技术人员通常所理解的相同。虽然相信以下术语对于本领域技术人员很好理解,但仍然阐述以下定义以更好地解释本发明。
如本发明中所使用,术语“包括”、“包含”、“具有”、“含有”或“涉及”为包含性的(inclusive)或开放式的,且不排除其它未列举的元素或方法步骤。术语“由…组成”被认为是术语“包含”的优选实施方案。如果在下文中某一组被定义为包含至少一定数目的实施方案,这也应被理解为揭示了一个优选地仅由这些实施方案组成的组。
在提及单数形式名词时使用的不定冠词或定冠词例如“一个”或“一种”,“所述”,包括该名词的复数形式。
本发明中的术语“大约”、“大体”表示本领域技术人员能够理解的仍可保证论及特征的技术效果的准确度区间。该术语通常表示偏离指示数值的±10%,优选±5%。
此外,说明书和权利要求书中的术语第一、第二、第三、(a)、(b)、(c)以及诸如此类,是用于区分相似的元素,不是描述顺序或时间次序必须的。应理解,如此应用的术语在适当的环境下可互换,并且本发明描述的实施方案能以不同于本发明描述或举例说明的其它顺序实施。
本发明中的术语“核酸”或“核酸序列”指包含核糖核酸、脱氧核糖核酸或其类似物单元的任何分子、优选聚合分子。所述核酸可为单链的或双链的。单链核酸可为变性双链DNA的一条链的核酸。或者,单链核酸可为不来源于任何双链DNA的单链核酸。
本文所使用的术语“互补”涉及核苷酸碱基G、A、T、C和U之间的氢键碱基配对,以使得当两种给定的多核苷酸或多核苷酸序列彼此退火时,在DNA中A与T配对、G与C配对,在RNA中G与C配对、A与U配对。
实施例1本发明反应体系优化
本实施例针对早先专利CN104651488A进行参数调整和优化改良。
1)位点选择优化
专利CN104651488A中的D18S51位点重复序列为(AGAA),重复次数为7-27。该基因座已报道的等位基因有50余个。Penta D位点重复序列(AAAGA),重复次数2.2-17。D18S51和PentaD位点重复次数相差较大,D18S51位点两个等位基因峰最大间距可达80bp,PentaD位点两个等位基因峰最大间距可达71.5bp,间距较大,染色体非整倍体数目异常检测需要依赖等位基因峰峰面积比值,小片段的优势扩增可使在正常人中出现等位基因失衡的情况而干扰判型。因此,本发明尝试删除D18S51和PentaD位点,并调整体系寻求效果提升。
另外,在专利CN104651488A中,X染色体上特异性STR位点只有3个,鉴于检测准确性考量,本发明进一步尝试增加X染色体上的特异性STR位点。本专利调查了16个X染色体上STR位点在人群中遗传多态性,经优化筛选确定在体系中增加GATA165B12位点进行X染色体数目异常的检测,效果最优。
2)引物序列优化
考虑到DNA样品含有血色素等抑制剂时,扩增结果会表现出大片段STR等位基因丢失,甚至所有基因座的扩增失败。寻求减小STR扩增子的大小可能帮助从一个受抑制样本中获得信息。
因此,本专利将CN104651488A PCR扩增产物大于400bp的位点进行引物修改,在引物修改过程中,由于扩增体系中引物变化,新引物与其他位点引物发生相互作用,导致引物出现扩增效率降低或非特异性扩增,PCR扩增产物小于400bp的部分位点也同时需要进行修改。引物修改优化过程中初设计引物序列见表1:
表1引物修改过程中引物序列
将上表中引物进行单位点扩增确认引物特异性,引物特异性扩增结果见表2:
表2引物特异性扩增结果
根据引物特异性扩增结果,综合选择引物特异、PCR扩增产物小于400bp且引物扩增效率(峰高)较好的引物进行26个位点的复合扩增引物,最后综合选定的引物序列如SEQID NO.1-52所示。
根据STR位点重复结构前后序列在进行引物设计时的难易程度,尽量保证原位点引物序列不变的条件下,对引物进行了重新分组,最后优化后引物分为五组,每组引物带有不同的荧光标记物,第一组为D13S256、D13S797、DXS6809、DXS9895、D21S2052,用FAM标记;第二组为TAF9L、D18S535、D13S317、D21S1411、D18S1002、D13S305,用HEX标记;第三组为AMEL、D18S877、LFG21、ZFXY、D18S851、D21S1435,用L552标记;第四组为D18S391、D13S800、D21S1246、XHPRT、D13S325,用LR600标记;第五组为GATA165B12、D21S1446、D21S11、SRY,用TET592标记。第三组、第四组和第五组使用能量传递引物增强PCR。
3)引物用量优化
引物比例会影响峰面积和峰面积比值,从而影响检测结果,因此,需要对扩增体系中引物的用量进行优化。表3为经过了初步优化后的引物混合液终浓组,按照表中引物在体系中终浓度进行引物混合液配制后进行PCR扩增,根据扩增结果进行调整,最终调整结果应满足同颜色组内均衡性30%-100%,颜色组间均衡性20%-100%。
表3引物混合液终浓度
以实验中1个样本为例,3个配比的引物混合液终浓度扩增样本的均衡性见表4,引物混合液终浓度3满足同颜色组内均衡性30%-100%,颜色组间均衡性20%-100%。
表4均衡性
经过引物序列优化及引物用量优化,确定引物序列及引物用量比见表5
表5引物序列及用量比
4)扩增体系改良
本发明还进一步整合了尿嘧啶DNA糖基化酶(UDG)防污染体系,在PCR扩增之前消化可能存在的含有尿嘧啶的扩增产物,从而避免产物污染。对防污染体系中dUTP的使用量、UDG酶定的使用量和孵育时间进行摸索,结果显示dUTP/(dTTP+dUTP)在0.1-0.5时,37℃孵育5-10min均能获得较好的防污染效果。
实施例2、本发明的检测试剂盒制备及检测方法建立
根据上述优化的参数体系,将引物、酶、缓冲液等组装本成本发明的试剂盒,具体的,所述试剂盒包括PCR反应液、引物混合液、酶混合液及质控品等,具体的,组分如下:
该试剂盒的检测方法建立如下:
1.DNA提取
采用基因组DNA提取试剂盒进行DNA提取,操作步骤依照试剂盒说明书,DNA提取完毕后用紫外分光光度计进行定量,并稀释成2-10ng/μl
2.多重PCR扩增
2.1按照下表进行PCR反应体系配制,涡旋振荡混匀后短暂离心,使液体聚集在管底。
2.2PCR反应条件:
2.3毛细管电泳检测
在检测前,对产物进行变性处理并与分子量内标混合,1μl产物+0.5μl分子量内标+8.5μl的HIDI(去离子甲酰胺),混匀后短暂离心,95℃变性5min后立即冰浴10min,用ABI系列遗传分析仪进行电泳检测。
2.4质控标准
(1)无核酸酶纯水检测结果为无PCR产物扩增。
(2)21三体阳性质控品检测结果为21三体阳性。
以上(1)和(2)同时满足时,表示PCR扩增成功,否则需要重新实验。
3.结果分析
毛细管电泳得到的数据,使用GeneMapper软件进行数据分析,由于扩增引物标记了荧光,可以通过峰面积表示扩增产物量的多少,通过STR位点出峰个数,峰面积比值判断样本是否存在21、18、13和性别染色体数目异常。判断标准如下:正常位点(表现为双峰,峰面积比值在0.8-1.4之间);异常位点(表现为三峰,峰面积比值接近于1:1:1,或表现为双峰,峰面积比值在0.45-0.65或1.8-2.4之间);无效位点(峰面积比值介于正常位点和异常位点区间之外)。
3.1正常结果判定:
至少两个STR位点为正常位点(表现为双峰,前后峰面积比值在0.8-1.4之间),其余位点为无效位点(峰面积比值介于正常位点和异常位点区间之外)。如图1-图2。
3.2 13、18、21染色体非整倍体结果判定:
13、18或21号染色体上至少两个STR遗传位点为异常位点(表现为三峰,峰面积比值接近于1:1:1,或表现为双峰,峰面积比值在0.45-0.65或1.8-2.4之间),其余位点为无效位点(峰面积比值介于正常位点和异常位点区间之外),如图3-图5。
3.3性别染色体结果判定:如图6-图9。
实施例3本发明与CN104651488A效果比较
将1例21三体综合征、1例18三体综合征、1例13三体综合征、1例X单体综合征、1例XXX综合征、1例XXY综合征和1例XYY综合征样本同时使用专利CN104651488A和本发明扩增体系进行检测对比。
与专利CN104651488A比较,本发明具有显著优势,具体如下:
1)检测灵敏度更高
将1例13三体综合征基因组DNA,梯度稀释至1ng/μL、2ng/μL和4μL/μL,本发明与专利CN104651488A同时对梯度稀释的DNA进行检测,重复检测3次。检测结果见表6。
表6灵敏度检测结果
专利CN104651488A检测灵敏度为2ng/μL,本发明检测灵敏度为1ng/μL,本发明检测灵敏度更高。专利CN104651488A与本发明1ng/μL和2ng/μL检测结果图谱见图10-图13。
2)扩增检测效率更好
将1例21三体综合征、1例18三体综合征、1例13三体综合征、1例X单体综合征、1例XXX综合征、1例XXY综合征和1例XYY综合征样本稀释至2ng/μL和4μL/μL。本发明与专利CN104651488A同时对2ng/μL和4μL/μL的DNA模板进行检测,比较检测峰高平均值,检测峰高平均值见表7。
表7检测峰高平均值
本发明与专利CN104651488A检出率均为100%,检测峰高值越高,检测效率越好,同时对2ng/μL和4μL/μL的DNA模板进行检测,本发明检测峰高平均值优于专利CN104651488A,本发明检测效率更优。以13三体综合征和XXY综合征样本2ng/μL的1个颜色组为例,纵坐标代表峰高值,将纵坐标调整到同一数值,通过峰的高度进行比较。本发明与专利CN104651488A对比峰高图见图14-图15。
3)准确度更高:本发明检测灵敏度和检测效率均优于专利CN104651488A,当检测模板量偏低时,本发明能更好的获得检测结果,从而保证检测结果的准确性。
上述对比实验可见,本发明在检测灵敏度、检测效率和准确度方面都显著优于专利CN104651488A,更适于产业应用。
前述对本发明的具体示例性实施方案的描述是为了说明和例证的目的。这些描述并非想将本发明限定为所公开的精确形式,并且很显然,根据上述教导,可以进行很多改变和变化。对示例性实施例进行选择和描述的目的在于解释本发明的特定原理及其实际应用,从而使得本领域的技术人员能够实现并利用本发明的各种不同的示例性实施方案以及各种不同的选择和改变。本发明的范围意在由权利要求书及其等同形式所限定。
序列表
<110> 北京阅微基因技术股份有限公司
<120> 一种染色体非整倍体数目异常的扩增组合物及其应用
<160> 52
<170> SIPOSequenceListing 1.0
<210> 1
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
aagagcaaaa ctccatctcg atag 24
<210> 2
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
tacttataag cagagagaca taa 23
<210> 3
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ttttggtttg ctggcatctg 20
<210> 4
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
ttgtctggag gcttttcagt c 21
<210> 5
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
tgtttccatc tttctctgaa c 21
<210> 6
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
gaatccaatt ttgctttagg c 21
<210> 7
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
ccatgattca aattatctcc cacc 24
<210> 8
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
ccatcatttg ccttgagaaa a 21
<210> 9
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
actgtacaga ggttctccgg gca 23
<210> 10
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
catgtcttga gccttccagc tctct 25
<210> 11
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
tttgacaggt agttttgggt ca 22
<210> 12
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
ctacagcatc tctgttaaat ttaga 25
<210> 13
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
acaaaagcca cacccataac 20
<210> 14
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
gaaatataga tgagaatgca gaga 24
<210> 15
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
tatcacagaa gtctgggatg 20
<210> 16
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
aaaaagacag acagaaagat 20
<210> 17
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
aatatgatga atgcatagat gg 22
<210> 18
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 18
cccactccca gccttctaaa tat 23
<210> 19
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 19
aggaagctat ctatacaaag agtg 24
<210> 20
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 20
aagatgtgag tgtgcttttc aggag 25
<210> 21
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 21
actgtttgag gacctgtcgt tacg 24
<210> 22
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 22
gcctcccaag tagttgagat ta 22
<210> 23
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 23
caccagccaa acctccctcc gc 22
<210> 24
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 24
gctgcatggg gtgcacaggt g 21
<210> 25
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 25
atagatgata gagatggcac atga 24
<210> 26
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 26
gttcttcata catgctttat catgc 25
<210> 27
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 27
ttggcgaatc atgacactaa 20
<210> 28
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 28
acttgatgga acagaaaaag agg 23
<210> 29
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 29
tggactcaga tgtaactgaa gaagt 25
<210> 30
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 30
taccaatact tcttctgaaa ctacg 25
<210> 31
<211> 27
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 31
tctctctgtc ctctaggctc atttagc 27
<210> 32
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 32
tgagatagtt taaatagttt gcc 23
<210> 33
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 33
ctcagcacat tctcctctag attta 25
<210> 34
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 34
aaagcaagag atttcagtgc catc 24
<210> 35
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 35
ttgaggaaga gagaaataga gaga 24
<210> 36
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 36
ctattcccat ctgagtcact cagc 24
<210> 37
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 37
aggtaggtag gtaaatagct agat 24
<210> 38
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 38
actggctttc ctcttccttc ctta 24
<210> 39
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 39
aaagtagaca ggtaaacata ca 22
<210> 40
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 40
tcatccatcc atccacctat ct 22
<210> 41
<211> 26
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 41
ttgaggtata cttttctctc cagaat 26
<210> 42
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 42
taatacacat ccccattcct gcc 23
<210> 43
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 43
atagattctc aatttccccg gttac 25
<210> 44
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 44
gtcagtgaag acagctgtgc tatct 25
<210> 45
<211> 29
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 45
atctatgggg catccatgat ctattcgta 29
<210> 46
<211> 26
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 46
gaagttggct gtggatcctg gtttat 26
<210> 47
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 47
atgaccatct tactggttta tgtat 25
<210> 48
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 48
caaaggacct gctcgaggct att 23
<210> 49
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 49
cgactttttc agtctccata aata 24
<210> 50
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 50
gacagacgaa taccaggtag atag 24
<210> 51
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 51
ttcctttgca ctgaaagctg ta 22
<210> 52
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 52
tgtccagttg cacttcgctg c 21
- 一种染色体非整倍体数目异常的扩增组合物及其应用
- 检测染色体非整倍体数目异常的PCR扩增组合物及检测试剂盒
