细胞状态评估模型的建立方法
文献发布时间:2023-06-19 12:13:22
技术领域
本发明关于一种细胞状态评估方法,特别是关于一种细胞状态评估模型的建立方法。
背景技术
生物学上发现,生物的真实年龄与生理年龄并非一致。由于生物个体之间的差异或是生活环境、生活习惯上的不同,会造成生物体各种功能性上的低落,例如:消化系统作用不良、免疫系统反应较慢等状况,就会被判定其生理年龄的相较于真实年龄更为老化。
另一方向,也有利用细胞分泌或细胞能量上的评估,来判定细胞的年龄。从而推知其生理年龄与真实年龄上是否有差异。
只是不论是从系统功能上或是细胞分泌状态上的评估,所必须耗费的评估时间及评估费用都较高,并且难以实际上的精准量化。
发明内容
在一些实施例中,细胞状态评估模型的建立方法。接收复数第一个体的生理数据,其中各生理数据包括第一个体的实际年龄及第一个体的血液检测资讯,血液检测资讯包括至少一致老化项目的相关基因资讯,并且各致老化项目的相关基因资讯包括复数细胞状态基因的表现量。接着,以各第一个体的实际年龄作为第一个体的细胞年龄。再以复数第一个体的复数细胞年龄及复数血液检测资讯建立至少一训练资料集,其中至少一训练资料集分别对应至少一致老化项目,并且各训练资料集包括复数第一个体的复数细胞年龄及复数第一个体的复数对应的致老化项目的相关基因资讯。而后,以至少一训练资料集个别训练类神经网路模型以分别形成基于至少一致老化项目的年龄预测模型,其中各类神经网路模型是以机器学习算法实现,并且各年龄预测模型用以基于对应的致老化项目的相关基因资讯预测细胞年龄。
在一些实施例中,至少一致老化项目的相关基因资讯的复数细胞状态基因的表现量包括TCP1次单元2的伴随蛋白基因的表现量、含有TCP1次单元5 的伴随蛋白基因的表现量、含有TCP1次单元6A的伴随蛋白基因的表现量、含有TCP1次单元7的伴随蛋白基因的表现量、含有TCP1次单元8的伴随蛋白基因的表现量、叉头盒蛋白质O基因的表现量、沉默调节蛋白1基因的表现量、PTEN诱发激酶基因的表现量、Parkin基因的表现量、自噬-相关蛋白质1基因的表现量、自噬-相关蛋白质8基因的表现量、聚ADP-核糖聚合酶1基因的表现量、聚ADP-核糖聚合酶2基因的表现量、榖胺酰胺-依赖型NAD+合成酶基因的表现量、粒线体核醣体蛋白质S5基因的表现量、泛素样蛋白质5基因的表现量、超氧化物歧化酶3基因的表现量、端粒酶反转录酶基因的表现量、端粒酶RNA组分基因的表现量及端粒延长解旋酶的调节子1基因的表现量。
在一些实施例中,至少一致老化项目包括抗老回春分类、粒线体活性分类、以及端粒酶活性分类。
在一些实施例中,抗老回春基因分类的相关基因资讯的复数细胞状态基因的表现量包括TCP1次单元2的伴随蛋白基因的表现量、含有TCP1次单元5 的伴随蛋白基因的表现量、含有TCP1次单元6A的伴随蛋白基因的表现量、含有TCP1次单元7的伴随蛋白基因的表现量、含有TCP1次单元8的伴随蛋白基因的表现量、叉头盒蛋白质O基因的表现量及沉默调节蛋白1基因的表现量。
在一些实施例中,粒线体活性分类的相关基因资讯的复数细胞状态基因的表现量包括PTEN诱发激酶基因的表现量、Parkin基因的表现量、自噬-相关蛋白质1基因的表现量、自噬-相关蛋白质8基因的表现量、聚ADP-核糖聚合酶 1基因的表现量、聚ADP-核糖聚合酶2基因的表现量、榖胺酰胺-依赖型NAD+ 合成酶基因的表现量、粒线体核醣体蛋白质S5基因的表现量、泛素样蛋白质5 基因的表现量及超氧化物歧化酶3基因的表现量。
在一些实施例中,端粒酶活性分类的相关基因资讯的复数细胞状态基因的表现量包括端粒酶反转录酶基因的表现量、端粒酶RNA组分基因的表现量及端粒延长解旋酶的调节子1基因的表现量。
在一些实施例中,至少一年龄预测模型包括基于抗老回春分类的相关基因资讯预测细胞年龄的第一预测模型、基于粒线体活性分类的相关基因资讯预测细胞年龄的第二预测模型以及基于端粒酶活性分类的相关基因资讯预测细胞年龄的第三预测模型。
在一些实施例中,细胞状态评估模型的建立方法更包括:耦接第一预测模型的输出、第二预测模型的输出及第三预测模型的输出至衰老判断模型,其中衰老判断模型用以基于检测个体的实际年龄与各年龄预测模型所预测的细胞年龄判定致老化项目的细胞衰老状态。
在一些实施例中,细胞状态评估模式的建立方法更包括:耦接至少一年龄预测模型基于抗老回春分类的相关基因资讯输出第一预测值,至少一年龄预测模型基于粒线体活性分类的相关基因资讯输出第二预测值,至少一年龄预测模型基于端粒酶活性分类的相关基因资讯输出第三预测值至衰老判断模型,其中衰老判断模型用以基于检测个体的实际年龄与各年龄预测模型所预测的细胞年龄判定致老化项目的细胞衰老状态。
在一些实施例中,衰老判断模型更用以基于至少一年龄预测模型所预测的至少一细胞年龄估算最终细胞年龄。
在一些实施例中,细胞状态评估模型的建立方法更包括:接收复数第二个体的生理数据,其中各生理数据包括第二个体的实际年龄及第二个体的血液检测资讯。接着,输入第二个体的各致老化项目的相关基因资讯至对应的年龄预测模型,以由年龄预测模型基于相关基因资讯预测第二个体的细胞年龄。再基于至少一年龄预测模型所预测的第二个体的至少一细胞年龄估算最终细胞年龄。之后,根据复数第二个体的复数实际年龄与复数最终细胞年龄设定衰老判断模型的判断条件。最后,耦接至少一年龄预测模型的输出至衰老判断模型的输入,其中衰老判断模型用以基于实际年龄、各年龄预测模型所预测的细胞年龄与判断条件判定对应的致老化项目的细胞衰老状态。
在一些实施例中,细胞状态评估模型的建立方法中,根据复数第二个体的复数实际年龄与复数最终细胞年龄设定衰老判断模型的判断条件的步骤包括:根据复数第二个体的复数实际年龄将复数第二个体的复数细胞年龄分为复数年龄群组,其中复数年龄群组的数量小于复数第二个体的数量,且各年龄群组是由复数细胞年龄的复数者所构成。接着,计算各年龄群组中的复数最终细胞年龄的标准差。之后,根据复数年龄群组的复数标准差决定一数值区间。以及以数值区间作为判断条件。
在一些实施例中,数值区间是由第一阈值与第二阈值所构成,以及根据复数年龄群组的复数标准差决定数值区间的步骤包括:基于复数年龄群组的复数标准差产生设定值,其中设定值大于复数标准差的最小整数;以设定值作为第一阈值;以及以设定值的相反数作为第二阈值。
在一些实施例中,机器学习演算法包括随机森林演算法、线性回归演算法及支持向量机演算法其中一种。在一些实施例中,机器学习演算法为随机森林演算法。
在一些实施例中,细胞状态评估模型的建立方法更包括:接收各第一个体的血液样本;从血液样本中提取出遗传物质;以及以遗传物质进行各细胞状态基因的检测以生成细胞状态基因的表现量。
综上所述,根据任一实施例的可以在短时间内获得精确的细胞年龄评估。根据任一实施例的可以所得到细胞年龄评估可以是简单的量化数值,或简化的理想/非理想的评估。
以下结合附图和具体实施例对本发明进行详细描述,但不作为对本发明的限定。
附图说明
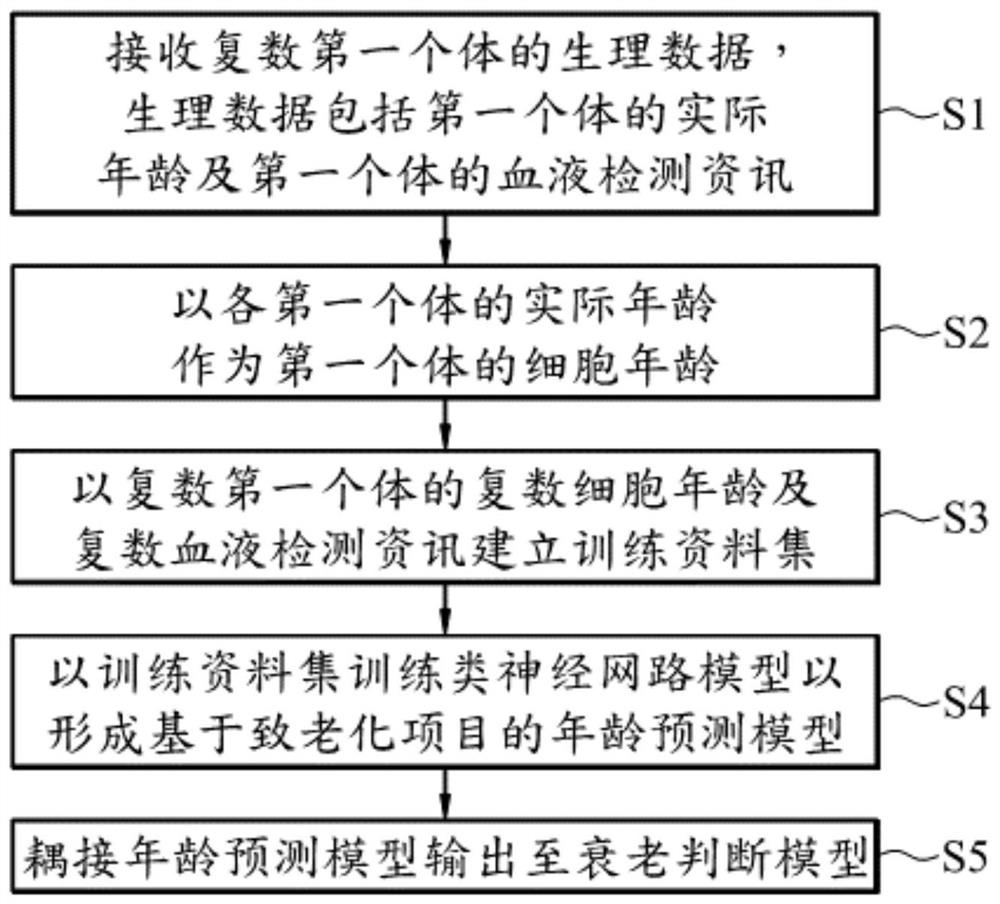
图1是一实施例细胞状态评估模型的建立方法的流程图。
图2是血液检测资讯取得一实施例的流程图。
图3是一实施例的细胞状态评估系统方块图。
图4是另一实施例细胞状态评估模型的建立方法的流程图。
图5是另一实施例的细胞状态评估系统方块图。
图6是再一实施例细胞状态评估模型的建立方法的流程图。
图7是一实施例判断条件建立的流程图。
图8是实际年龄与细胞年龄关系的散布图。
具体实施方式
参考图1。在一些实施例中,细胞状态评估模型的建立方法,其包括,接收复数第一个体的生理数据,其中生理数据包括第一个体的实际年龄及第一个体的血液检测资讯(步骤S1)。其中,血液检测资讯包括至少一致老化项目的相关基因资讯,并且各致老化项目的相关基因资讯包括复数细胞状态基因的表现量。
参考图2。在一些实施例中,细胞状态评估模型的建立方法在步骤S1之前更包括接收各第一个体的血液样本(步骤S010)。接着,从血液样本中提取出遗传物质(步骤S011)。之后,以遗传物质进行各细胞状态基因的检测以生成细胞状态基因的表现量(步骤S012)。在一些实施例中,步骤S011的遗传物质可以是RNA。在一些实施例中,步骤S012是经由定量即时聚合酶链反应(quantitative real time polymerase chain reaction,qRT-PCR)仪器测量遗传物质表达的相对定量为其表现量。
在一些实施例中,血液样本是指离体的血液样本。在一些实施例中,步骤 S010到S012可以利用生物芯片(BIOCHIP)来实现。
续参考图1。在步骤S1之后,以各第一个体的实际年龄作为第一个体的细胞年龄(步骤S2)。于此,第一个体可以是健康的成年人,意即非处于疾病状态下的成年人。在一些实施例中,成年人是指实际年龄为20到50岁之间的人。意即假设在健康的情况下,成年人的实际年龄应该与细胞年龄相等。
在步骤S2之后,再以复数第一个体的复数细胞年龄及复数血液检测资讯建立训练资料集(步骤S3),其中训练资料集分别对应于致老化项目,并且训练资料集包括复数第一个体的复数细胞年龄及复数第一个体的复数对应的致老化项目的相关基因资讯。
在一些实施例中,血液检测资料是指至少一致老化项目的相关基因资讯的复数细胞状态基因的表现量包括TCP1次单元2的伴随蛋白基因的表现量、含有TCP1次单元5的伴随蛋白基因的表现量、含有TCP1次单元6A的伴随蛋白基因的表现量、含有TCP1次单元7的伴随蛋白基因的表现量、含有TCP1次单元8的伴随蛋白基因的表现量、叉头盒蛋白质O基因的表现量、沉默调节蛋白1基因的表现量、PTEN诱发激酶基因的表现量、Parkin基因的表现量、自噬-相关蛋白质1基因的表现量、自噬-相关蛋白质8基因的表现量、聚ADP- 核糖聚合酶1基因的表现量、聚ADP-核糖聚合酶2基因的表现量、榖胺酰胺- 依赖型NAD+合成酶基因的表现量、粒线体核醣体蛋白质S5基因的表现量、泛素样蛋白质5基因的表现量、超氧化物歧化酶3基因的表现量、端粒酶反转录酶基因的表现量、端粒酶RNA组分基因的表现量及端粒延长解旋酶的调节子1基因的表现量。
续参考图1。在步骤S3之后,以训练资料集训练类神经网路模型以形成基于致老化项目的年龄预测模型20(步骤S4)。其中,各类神经网路模型是以机器学习演算法实现,并且年龄预测模型20用以基于检测个体的血液资讯来预测细胞年龄。也就是说,当输入检测个体的血液资讯内的至少一老化项目的相关基因资讯进入年龄预测模型20后,年龄预测模型20可以输出检测个体的细胞年龄预测值。
在一些实施例中,在步骤S04之后更包括,耦接年龄预测模型的输出至衰老判断模型50(步骤S5)。也就是说,年龄预测模型20于产生细胞年龄预测值后会进一步将细胞年龄预测值输入至衰老判断模型50,以供衰老判断模型50 基于检测个体的实际年龄与细胞年龄预测值判定细胞衰老状态。
参考图3。在使用状态下,依据细胞状态评估模型的建立方法所建立的细胞状态评估系统内包括输入模块10、年龄预测模型20。输入模块10耦接到年龄预测模型20以将检测个体的生理资讯输入到年龄预测模型20,年龄预测模型20依据生理资讯内的血液检测资讯的至少一致老化项目的相关基因资讯输出细胞年龄预测值。
在一些实施例中,细胞状态评估系统更包括衰老判断模型50及输出模块 70。年龄预测模型20耦接到衰老判断模型50以将所预测的细胞年龄预测值输入到衰老判断模型50,衰老判断模型50依据接收到的细胞年龄预测值及从输入模块10接收的实际年龄以判定细胞衰老状态,衰老判断模型50将细胞衰老状态传送到的输出模块70,输出模块70据以输出细胞衰老状态。
举例而言,衰老判断模型50可以将检测个体的实际年龄与细胞年龄预测值相减,依据所得数值的正负来判定细胞衰老状态。也就是说,当细胞年龄预测值大于实际年龄时判定细胞衰老状态为不理想,当细胞年龄预测值小于实际年龄时判定细胞衰老状态为理想,当细胞年龄预测值等于实际年龄时判定细胞衰老状态为标准。
在另一些实施例中,至少一致老化项目包括抗老回春分类、粒线体活性分类、以及端粒酶活性分类。
在一些实施例中,抗老回春基因分类的相关基因资讯的复数细胞状态基因的表现量包括TCP1次单元2的伴随蛋白基因的表现量、含有TCP1次单元5 的伴随蛋白基因的表现量、含有TCP1次单元6A的伴随蛋白基因的表现量、含有TCP1次单元7的伴随蛋白基因的表现量、含有TCP1次单元8的伴随蛋白基因的表现量、叉头盒蛋白质O基因的表现量及沉默调节蛋白1基因的表现量。
在一些实施例中,粒线体活性分类的相关基因资讯的复数细胞状态基因的表现量包括PTEN诱发激酶基因的表现量、Parkin基因的表现量、自噬-相关蛋白质1基因的表现量、自噬-相关蛋白质8基因的表现量、聚ADP-核糖聚合酶 1基因的表现量、聚ADP-核糖聚合酶2基因的表现量、榖胺酰胺-依赖型NAD+ 合成酶基因的表现量、粒线体核醣体蛋白质S5基因的表现量、泛素样蛋白质5 基因的表现量及超氧化物歧化酶3基因的表现量。
在一些实施例中,端粒酶活性分类的相关基因资讯的复数细胞状态基因的表现量包括端粒酶反转录酶基因的表现量、端粒酶RNA组分基因的表现量及端粒延长解旋酶的调节子1基因的表现量。
其中,TCP1次单元2的伴随蛋白(chaperonin containing TCP1 subunit 2,CCT2)基因的GeneID为10576,后续简称为CCT2基因。TCP1次单元5的伴随蛋白(chaperonincontaining TCP1 subunit 5,CCT5)基因的GeneID为22948,后续简称为CCT5基因。TCP1次单元6A的伴随蛋白(chaperonin containing TCP1 subunit 6A,CCT6A)基因的GeneID为908,后续简称为CCT6A基因。TCP1次单元7的伴随蛋白(chaperonin containing TCP1subunit 7,CCT7)基因的GeneID为 10574,后续简称为CCT7基因。CCT2基因、CCT5基因、CCT6A基因及CCT7 基因为组成TCP1环状复合物TRiC,复合物TRiC主要协助蛋白质进行折迭,其中包括ATP水解酶,同时也会协助折迭WRAP53/TCAB1,以调控端粒长度的维持。
其中,TCP1次单元8的伴随蛋白(chaperonin containing TCP1 subunit 8,CCT8)基因的GeneID为10694,后续简称为CCT8基因,在老化过程中生物维持体内蛋白质质量的能力会逐渐下降,受损蛋白质和错误折迭的蛋白质的累积起来会造成细胞死亡和细胞功能故障。CCT8基因可以调控TRiC/CCT复合体,而这个复合体负责折迭大约生物体内10%的细胞蛋白质。
其中,叉头盒蛋白质O 3(forkhead box protein O,FOXO3)基因的GeneID为2309,后续简称为FOXO3基因,FOXO3基因的表现量提升可以恢复粒线体活性以使细胞保持青春状态。在一些科学实验中曾发现可以延长线虫寿命接近二倍。
其中,沉默调节蛋白1(Sirtuin 1,SIRT1)基因的GeneID为23411,后续简称为SIRT1基因,SIRT1基因的表现量提升可以协助粒线体修护受损DNA以推迟老化的发生。
其中,PTEN诱发激酶(PTEN-induced kinase,Pink)基因的GeneID为65018,后续简称为Pink1基因,Pink1基因和保护细胞免受氧化压力及促进粒线体健康有关。
其中,Parkin基因的GeneID为5071,Parkin基因可以标记受损蛋白,以使细胞在其产生毒性前降解受损蛋白,并且Parkin基因可以去除细胞内受损的线粒体。在一些研究中发现提高果蝇细胞中parkin基因的表现量时,果蝇的寿命相比于对照组延长了25%。
其中,自噬-相关蛋白质1(Autophagy-related protein 1,Atg1)基因的GeneID为8408,后续简称为Atg1基因。自噬-相关蛋白质8(Autophagy-related protein 8,Atg8)基因的GeneID为11345,后续简称为Atg8基因。Atg1基因及Atg8基因与自噬作用(autophagy)相关,自噬作用是清除入侵微生物及毒性蛋白质聚合物的重要细胞机制,因此,Atg1基因及Atg8基因在感染、老化、及人类疾病的发病机制上扮演重要的角色。
其中,聚ADP-核糖聚合酶1(Poly(ADP-Ribose)Polymerase 1,PARP1)基因的GeneID为142,后续简称为PARP1基因,是一种DNA修复酶,当DNA发生断裂时,PARP1基因会识别并结合到断裂部位并吸引其他DNA修复酶到达受损处进行修复。
其中,聚ADP-核糖聚合酶2(Poly(ADP-Ribose)Polymerase 2,PARP2)基因的GeneID为10038,后续简称为PARP2基因,
其中,榖胺酰胺-依赖型NAD+合成酶(Glutamine-dependent NAD(+)synthetase,NADSYN)基因的GeneID为55191,后续简称为NADSYN基因,协助合成NAD+提供血液粒线体能量来源。
其中,粒线体核醣体蛋白质S5(mitochondrial ribosomal protein S5,MRPS5)基因的GeneID为64969,后续简称为MRPS5基因,MRPS5基因所编码的蛋白与粒腺体合成有重要的关系。
其中,泛素样蛋白质5(Ubiquitin-like protein 5,Ubl-5)基因的GeneID为59286,后续简称为Ubl-5基因,可以调节烟碱酰胺腺嘌呤二核苷酸NAD
其中,超氧化物歧化酶3(superoxide dismutase 3,SOD3)基因的GeneID 6649为,后续简称为SOD3基因,SOD3基因的表现是提升能够清除细胞内的超氧化物,保护细胞免受氧化损伤。
其中,端粒酶反转录酶(telomerase reverse transcriptase,TERT)基因的GeneID 7015后续简称TERT基因,有助延长端粒长度。
其中,端粒酶RNA组分(Telomerase RNA Component,TERC)基因的GeneID 为7012,后续简称为TERC基因,端粒酶是一种由TERT(端粒酶逆转录酶) 基因编码的逆转录酶蛋白和端粒酶RNA组分二者所组成的复合物。
其中,端粒延长解旋酶的调节子1(Regulator of telomere elongationhelicase 1,RTEL1)基因GeneID为51750,后续简称为RTEL1基因,端粒延长解旋酶的调节子可以用来协助端粒的延长作用。
在一些实施例中,步骤S5可以由下述步骤取代,耦接年龄预测模型基于抗老回春分类的相关基因资讯输出的第一预测值,年龄预测模型基于粒线体活性分类的相关基因资讯输出的第二预测值以及年龄预测模型基于端粒酶活性分类的相关基因资讯输出的第三预测值至衰老判断模型50。也就是说,当输入检测个体的相关基因资讯进入年龄预测模型50时,会依据抗老回春分类、粒线体活性分类以及端粒酶活性分类分别得到第一预测值、第二预测值及第三预测值。于此,衰老判断模型用以基于检测个体的实际年龄与第一预测值、第二预测值及第三预测值来判定细胞衰老状态。
续参考图3。在使用状态下,输入模块10将检测个体的生理资讯内致少一老化项目区分为抗老回春分类、粒线体活性分类、以及端粒酶活性分类。再将抗老回春分类的相关基因资讯传送到年龄预测模型20得到第一预测值,并输出第一预测值到衰老判断模型50。将粒线体活性分类的相关基因资讯传送到年龄预测模型20得到第二预测值,并输出第二预测值到衰老判断模型50。将端粒酶活性分类的相关基因资讯传送到年龄预测模型20得到第三预测值,并输出第三预测值到衰老判断模型50。换言之,在建立年龄预测模型20时是基于全部的老化项目相关基因资讯,而在使用状态下,则会将检测个体的老化项目相关基因资讯分为三类,再分别输入各类的相关基因资讯进入年龄预测模型20以得到三个预测值。
在一些实施例中,输出模块70输出的细胞衰老状态包括理想、标准及不理想三种分类。
举例而言,衰老判断模型50可以将检测个体的实际年龄分别与第一预测值、第二预测值及第三预测值相减,依据所得数值的正负来判定细胞衰老状态。也就是说,细胞衰老状态会分别标识出检测个体在抗老回春分类的细胞衰老状态是否理想,在粒线体活性分类的细胞衰老状态是否理想以及在端粒酶活性分类的细胞衰老状态是否理想。
在一些实施例中,输出模块70依据细胞衰老状态的三种分类输出保健建议。
举例而言,当检测个体被标识出端粒酶活性分类的细胞衰老状态为非理想的情况时,可以进一步据此给出保健健议中的饮食补充建议,例如:应补充 omega-3、补充维生素D,并且采用地中海饮食等,以改善检测个体的端粒酶活性。
也就是说,依据抗老回春分类的细胞衰老状态是否理想,在粒线体活性分类的细胞衰老状态是否理想以及在端粒酶活性分类的细胞衰老状态是否理想分别提供不同的保健建议(例如:饮食补充建议、生活作息建议等等)。
在另一些实施例中,训练资料集分别对应于抗老回春分类、粒线体活性分类、以及端粒酶活性分类。也就是说,分别具有抗老回春训练资料集、粒线体活性训练资料集,以及端粒酶活性训练资料集。抗老回春训练资料集包括复数第一个体的复数细胞年龄及复数第一个体的复数对应的抗老回春分类的相关基因资讯,粒线体活性训练资料集包括复数第一个体的复数细胞年龄及复数第一个体的复数对应的粒线体活性分类的相关基因资讯,端粒酶活性训练资料集包括复数第一个体的复数细胞年龄及复数第一个体的复数对应的端粒酶活性分类的相关基因资讯。
参考图4。在步骤S3的一实施例中,步骤S3可以是以至少一致老化项目的老回春分类、粒线体活性分类、以及端粒酶活性分类将复数第一个体的复数细胞年龄、复数血液检测资讯分别建立对应的训练资料集(步骤S031)。
而在步骤S4的一实施例中,步骤S4可以由步骤S41到步骤S43来实现。步骤S41是以抗老回春训练资料集训练类神经网路模型以形成第一预测模型 21,步骤S42是以粒线体活性训练资料集训练类神经网路模型以以形成第二预测模型22,以及步骤S43是以端粒酶活性训练资料集训练资料集个别训练类神经网路模型以形成第三预测模型23。于此,上述步骤S41到步骤S43不用依照顺序执行,可以随意调换顺序,也可以同时执行。
参考图5。年龄预测模型20包括基于抗老回春分类的相关基因资讯预测细胞年龄的第一预测模型21、基于粒线体活性分类的相关基因资讯预测细胞年龄的第二预测模型22以及基于端粒酶活性分类的相关基因资讯预测细胞年龄的第三预测模型23。
在一些实施例中,步骤S41到步骤S43之后的步骤是耦接第一预测模型21 的输出的第一预测值、第二预测模型22的输出的第二预测值及第三预测模型 23的输出的第三预测值至衰老判断模型(步骤S51)。也就是说,当输入检测个体的相关基因资讯时,会依据第一预测型21、第二预测模型22以及第三预测模型23分别得到第一预测值、第二预测值及第三预测值。于此,衰老判断模型 50用以基于检测个体的实际年龄与第一预测值、第二预测值及第三预测值来判定细胞衰老状态。
续参考图5。在使用状态下,输入模块10将检测个体的生理资讯内致少一老化项目区分为抗老回春分类、粒线体活性分类、以及端粒酶活性分类,再将抗老回春分类的相关基因资讯传送到第一预测模型21,粒线体活性分类的相关基因资讯传送到第二预测模型22,端粒酶活性分类的相关基因资讯传送到第三预测模型23,之后第一预测模型21输出的第一预测值、第二预测模型22输出的第二预测值及第三预测模型23输出的第三预测值都传送到衰老判断模型50。年龄预测模型20耦接到衰老判断模型50以将得到的第一预测值、第二预测值及第三预测值输入到衰老判断模型50,衰老判断模型50依据接收到的第一预测值、第二预测值及第三预测值,以及从输入模块10接收的实际年龄以判定细胞衰老状态,衰老判断模型50将细胞衰老状态传送到的输出模块70,输出模块70据以输出细胞衰老状态。
在一些实施例中,衰老判断模型50更用以基于至少一年龄预测模型20所预测的至少一细胞年龄估算最终细胞年龄。举例而言,可以将第一预测值、第二预则值及第三预测值的平均值作为最终细胞年龄。再举例而言,可以将第一预测值、第二预则值及第三预测值的依据不同的权重估算出最终细胞年龄。
在一些实施例中,衰老判断模型50还用以基于检测个体的实际年龄与最终细胞年龄预测值判定细胞衰老状态。举例而言,衰老判断模型50可以将检测个体的实际年龄与最终细胞年龄预测值相减,依据所得数值的正负来判定细胞衰老状态。也就是说,当最终细胞年龄预测值大于实际年龄时判定细胞衰老状态为不理想,当最终细胞年龄预测值小于实际年龄时判定细胞衰老状态为理想,当最终细胞年龄预测值等于实际年龄时判定细胞衰老状态为标准。
参考图5。在一些实施例中,细胞状态评估系统更包括判断条件设定模块 60。判断条件设定模块60耦接衰老判断模型50、输入模块10及年龄预测模型 20,并用以决定判断条件。
参考图6。在一些实施例中,细胞状态评估模型的建立方法更包括输入模块10接收复数第二个体的生理数据,其中各生理数据包括第二个体的实际年龄及第二个体的血液检测资讯(步骤S61)。
接着,输入第二个体的各致老化项目的相关基因资讯至对应的年龄预测模型20,以由年龄预测模型20基于相关基因资讯预测第二个体的细胞年龄(步骤 S62)。在一些实施例中,致老化项目的相关基因资讯的取得可以参考步骤S1 及步骤S010到步骤S012。
步骤S62之后,再基于至少一年龄预测模型20所预测的第二个体的至少一细胞年龄估算最终细胞年龄(步骤S63)。在一些实施例中,最终细胞年龄可以是输入第二个体的血液检测资讯的至少一老化项目的相关基因资讯进入年龄预测模型所得到细胞年龄预测值。在一些实施例中,的方式可以参考步骤S5及步骤S51的方式来取得。
之后,根据复数第二个体的复数实际年龄与复数最终细胞年龄设定衰老判断模型50的判断条件(步骤S64)。
在一些实施例中,复数第二个体可以是与复数第一个体具有不同特征的健康成年人,基此可以经由步骤S61到步骤S65以消除不同人种之间可能产生的差异。举例而言,复数第二个体可以是与复数第一个体不同人种的健康成年人。故而可以进一步提高本发明细胞状态评估模型的建立方法的准确性与适用性。
参考图7。在步骤S64的一实施例中,步骤S64可以由步骤S641到步骤 S644来实现。根据复数第二个体的复数实际年龄与复数最终细胞年龄设定衰老判断模型50的判断条件的步骤包括:根据复数第二个体的复数实际年龄将复数第二个体的复数细胞年龄分为复数年龄群组,其中复数年龄群组的数量小于复数第二个体的数量,且各年龄群组是由复数细胞年龄的复数者所构成(步骤 S641)。举例而言,可以将复数第二个体区分为实际年龄在20~29岁之间为A 组,实际年龄在30~39岁之间为B组,实际年龄在40~50岁之间为C组。
接着,计算各年龄群组中的复数最终细胞年龄的标准差(步骤S642)。之后,根据复数年龄群组的复数标准差决定一数值区间(步骤S643)。以及以数值区间作为判断条件(步骤S644)。
于此,采用的标准差公式为
在一些实施例中,数值区间是由第一阈值与第二阈值所构成。于此,根据复数年龄群组的复数标准差决定数值区间的步骤包括:基于复数年龄群组的复数标准差产生设定值,其中设定值大于复数标准差的最小整数;以设定值作为第一阈值;以及以设定值的相反数作为第二阈值。
最后,耦接至少一年龄预测模型的输出至衰老判断模型50的输入,其中衰老判断模型50用以基于实际年龄、各年龄预测模型所预测的细胞年龄与判断条件判定对应的致老化项目的细胞衰老状态(步骤S65)。
举例而言,衰老判断模型50可以将检测个体的实际年龄与细胞年龄预测值相减,依据所得数值以及该数值的正负对比于判断条件来判定细胞衰老状态。也就是说,当细胞年龄预测值减去实际年龄所得数值大于等于3时判定细胞衰老状态为不理想,当细胞年龄预测值减去实际年龄所得数值小于等于-3时判定细胞衰老状态为理想,当细胞年龄预测值减去实际年龄所得数值介于2到-2之间时判定细胞衰老状态为标准。
在步骤S64的另一实施例中,步骤S64可以由步骤S642到步骤S644来实现。根据复数第二个体的复数实际年龄与复数最终细胞年龄设定衰老判断模型 50的判断条件的步骤包括:计算复数第二个体的复数最终细胞年龄的标准差(步骤S642)。之后,根据复数年龄群组的复数标准差决定一数值区间(步骤S643)。以及以数值区间作为判断条件(步骤S644)并以数值区间作为判断条件(步骤S645)。
续参考图5。进行判断条件的计算的状态下,当年龄预测模型20先连接到判断条件设定模块60以进行判断条件的计算时,年龄预测模型20会先断开与衰老判断模型50的连接。待判断条件设定模块60以完成判断条件的计算,并将判断条件传送到衰老判断模型50。使用状态下,年龄预测模型20会断开与判断条件设定模块60的连接,以使得年龄预测模型20的预测结果传送到衰老判断模型50。
在一些实施例中,机器学习演算法包括随机森林演算法(random forest)、线性回归演算法(linear regression)及支持向量机演算法(support vector machine)其中一种。在一些实施例中,机器学习演算法包括随机森林演算法。
在一些实施例中,年龄预测模型20、第一预测模型21、第二预测模型22、第三预测模型23、衰老判断模型50及判断条件设定模块60可由一个或多个程序代码来实现。在一些实施例中,程序代码通过编码器、处理器等来执行。
综上所述,根据任一实施例的可以在短时间内获得精确的细胞年龄评估。根据任一实施例的可以所得到细胞年龄评估可以是简单的量化数值,或简化的理想/非理想的评估。
例1:年龄预测模型精准度测试
首先,依据图1的步骤S1到S3建立训练资料集。接下来,在步骤S4中分别采用森林演算法产生年龄预测模型A。
测试中采用统计学中的回归分析的决定系数R-squared(coefficient ofdetermination)愈接近1.0者为佳,据此判断年龄预测模型的准确度。
将训练资料集内的复数第一个体血的液检测资讯输入年龄预测模型A以得到对应各第一个体的细胞年龄预测值,再将年龄预测模型A所预测的细胞年龄值与真实年龄进行回归分析,得到其R-squared为0.952379,代表其准确度非常高。
参考图8,可见代表实际年龄的黑点与代表细胞年龄预测值的白点彼此分布位置十分接近,可知,以随机森林演算法所建新的年龄预测模型具有相当的准确度。
由上述可知,采用森林演算法所产生年龄预测模型A在统计学的验证具有非常高的准确率。
例2:老化项目的相关基因资讯的复数细胞状态基因的表现量的取得
抗老回春
衰老的生物学作用复杂,但在大部分的研究中可以查觉,老化过程中生物体内的蛋白质、粒线体(mitochondrion)及端粒酶(Telomerase)可以观察到对应的各种变化,基此本测试选择下列表一的20种基因的表现量作为观察的对象。
2.1材料、仪器及溶液配置
实验材料:从复数第一个体采集得到的离体血液样本。
试剂:RNA萃取试剂套组(购自Geneaid公司,中国台湾,Lot No.FC24015-G)、 KAPA
反转录酶:采用
检测仪器:ABI StepOnePlusTM实时PCR系统(Real-Time PCR system,购自ThermoFisher Scientific公司,美国)。
2.2.测试流程
使用RNA萃取试剂套组收集血液样本内之RNA。接着,每组取2000奈克 (ng)所萃取出的RNA为模板,藉由反转录酶以表一中之引子黏合进行反转录作用产生相应之cDNA。后续利用实时PCR系统,以及qPCR试剂组将二组反转录后产物分别以表二之组合引子进行定量实时反转录聚合酶连锁反应 (quantitative real-time reverse transcriptionpolymerase chain reaction)以观察实验组和控制组的单核球细胞的基因的表现量。定量实时反转录聚合酶连锁反应的仪器设定条件为95℃反应1秒,60℃反应20秒,总共40个循环,并使用2-ΔCt方法进行基因定量。
表一
于此,藉由cDNA进行定量实时反转录聚合酶连锁反应可间接定量各基因的mRNA表现量,进而推断各基因编码的蛋白质的表现量。
例3:数值区间的取得范例
下面以简化后的数值为说明:
表二
如同上表二,第二个体01的实际年龄为29岁,第二个体02的实际年龄为 28,第二个体03的实际年龄为28,故而被分类到A组。第二个体04的实际年龄为38岁,第二个体05的实际年龄为38,第二个体06的实际年龄为32,故而被分类到B组。第二个体07的实际年龄为46岁,第二个体08的实际年龄为42,第二个体09的实际年龄为49,故而被分类到C组(参考步骤S641)。
再依据标准差公式,意即以标准差公式计算A组的最终细胞年龄之间的标准差得到2.516,以标准差公式计算B组的最终细胞年龄之间的标准差得到 2.886,以标准差公式计算C组的最终细胞年龄之间的标准差得到2.645(参考步骤S642)。
于此,采用的标准差公式为
由上表二可知各组的标准差位在2.5~2.88之间以为数值区间(参考步骤 S643)。
延续示范例所用的数值来举例,由于各组的标准差皆在2.5~2.88之间,采用大于复数标准差的最小整数为设定值,因此以“3”作为数值区间以产生设定值。并且以设定值3为第一阈值,而以设定值3的反数-3为第二阈值。
虽然本发明的技术内容已经以较佳实施例揭露如上,然其并非用以限定本发明,任何熟习此技艺者,在不脱离本发明之精神所作些许之更动与润饰,皆应涵盖于本发明的范畴内,因此本发明之保护范围当视后附之申请专利范围所界定者为准。
当然,本发明还可有其它多种实施例,在不背离本发明精神及其实质的情况下,熟悉本领域的技术人员可根据本发明作出各种相应的改变和变形,但这些相应的改变和变形都应属于本发明权利要求的保护范围。
【序列表】
<110> 大江基因医学股份有限公司
<120> 细胞状态评估模型的建立方法
<130> NA
<150> TW109201532
<151> 2020-02-12
<160> 40
<170> PatentIn version 3.5
<210> 1
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT2-F
<400> 1
aagccacgaa ggctgcaa 18
<210> 2
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT2-R
<400> 2
tcatcggaac catgatcaac tg 22
<210> 3
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT5-F
<400> 3
cggataagtg ccccacctta 20
<210> 4
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT5-R
<400> 4
tccagtgcgt cggcaaa 17
<210> 5
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT6A-F
<400> 5
tggccagaac atctcttcgt act 23
<210> 6
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT6A-R
<400> 6
agtccactac agcctctgtt aagaca 26
<210> 7
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT7-F
<400> 7
gtggcatgga caagcttatt gtag 24
<210> 8
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT7-R
<400> 8
cagaattgtg gccccatca 19
<210> 9
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT8-F
<400> 9
acccggaggt ggagcaa 17
<210> 10
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> CCT8-R
<400> 10
ggacatgtct ctccatatga tgtga 25
<210> 11
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> FOXO3-F
<400> 11
cggacaaacg gctcactct 19
<210> 12
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> FOXO3-R
<400> 12
ggacccgcat gaatcgacta t 21
<210> 13
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> SIRT1-F
<400> 13
tagccttgtc agataaggaa gga 23
<210> 14
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> SIRT1-R
<400> 14
acagcttcac agtcaacttt gt 22
<210> 15
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> pink1-F
<400> 15
ctgtggtggc tagtgctcct 20
<210> 16
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> pink1-R
<400> 16
tccagacgtg agacagttgg 20
<210> 17
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Parkin-F
<400> 17
gcagagaccg tggagaaaag 20
<210> 18
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Parkin-R
<400> 18
cttttctcca cggtctctgc 20
<210> 19
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Atg1-F
<400> 19
caggaggacg agaacacggt gtc 23
<210> 20
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> Atg1-R
<400> 20
ggaaggttct ttggcaccag cac 23
<210> 21
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> Atg8-F
<400> 21
tatccagacc gtgtgcccgt c 21
<210> 22
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Atg8-R
<400> 22
gtggatgcgc ttgcgaatga gg 22
<210> 23
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> PARP1-F
<400> 23
agcgtgtttc taggtcgtgg 20
<210> 24
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> PARP1-R
<400> 24
catcaaacat gggcgactgc 20
<210> 25
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> PARP2-F
<400> 25
agcaagatga atctgtgaag gc 22
<210> 26
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> PARP2-R
<400> 26
cactgaagtt cctctgggca 20
<210> 27
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> NADSYN-F
<400> 27
gcaaaatgtg caggctcgaa 20
<210> 28
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> NADSYN-R
<400> 28
gcactggagc agtcgtactt 20
<210> 29
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> MRP5-F
<400> 29
gtccggacag tccctcac 18
<210> 30
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> MRP5-R
<400> 30
cccaataaat gacctgccgt c 21
<210> 31
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> UBL5-F
<400> 31
tcctagcgtt aactgcgacc 20
<210> 32
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> UBL5-R
<400> 32
ctagctggag ctcgaatcgc 20
<210> 33
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> SOD3-F
<400> 33
agctggaaag gtgcccga 18
<210> 34
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> SOD3-R
<400> 34
cttggcgtac atgtctcgga t 21
<210> 35
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> TERT-F
<400> 35
ggagcaagtt gcaaagcatt g 21
<210> 36
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> TERT-R
<400> 36
tcccacgacg tagtccatgt t 21
<210> 37
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> TERC-F
<400> 37
aagagttggg ctctgtcagc 20
<210> 38
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> TERC-R
<400> 38
gactcgctcc gttcctcttc 20
<210> 39
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> RTEL1-F
<400> 39
ctgagtgggg cctctaggat 20
<210> 40
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> RTEL1-R
<400> 40
gctggcctat tctgttgggt 20
- 细胞状态评估模型的建立方法
- 风机齿轮箱状态在线评估模型建立方法及在线评估方法
