用于靶向整合的方法
文献发布时间:2023-06-19 18:27:32
技术领域
本发明涉及用于生产重组蛋白质的优化表达系统领域。更具体而言,它涉及利用供体载体靶向整合到真核宿主细胞基因组的特定预定基因组位置内的基于细胞的方法,其中所述载体和宿主细胞包含核酸组分,其使得能够选择性地选择已将供体载体整合到宿主细胞基因组的预定基因组位置内的那些细胞,并且检测且去除已经历了在基因组的其它部分内的任何另外随机整合事件的细胞。
背景技术
在过去的30年期间,重组蛋白质治疗剂已从新奇事物进化为上市药物中的主导地位。治疗性蛋白质的重组生产已超过每年1000亿$的市场规模,并且在全球经济以及先进医疗保健中发挥重要作用。治疗性蛋白质类别包括替代蛋白质(胰岛素、生长因子、细胞因子和血液因子)、疫苗(抗原、VLP)和单克隆抗体。迄今为止的主导形式是单克隆抗体。一些重组蛋白质可以在简单的微生物细胞如大肠杆菌(
当今行业内生成高性能治疗性蛋白质生产细胞系的主导方法是经由随机整合方法将重组蛋白质基因引入宿主CHO细胞系的基因组内,并且选择/筛选个别细胞,所述细胞已在活性基因组位点处以产生足够高转录的拷贝数整合基因,并且同时具有能够支持高蛋白质翻译和分泌的表型。这是高度工作密集和耗时的过程,具有很大的固有不确定性和生物学变异。典型的过程持续时间跨越3-12个月,取决于宿主细胞的生长、实施的自动化水平和终点(例如,如果包括长期克隆稳定性的评价)。
与随机整合方法相关的一个基本局限性是转染细胞池中的细胞多样性的低采样。仅0.1-1%左右的转染细胞整合了重组DNA。进一步地,该亚群在整合位置、拷贝数和整合DNA的完整性方面是高度异质性的。加上CHO细胞固有的全局表型变异(由于高基因组和表观基因组可塑性,其对于CHO细胞是固有的),使得找到高产克隆像大海捞针一样。这也解释了为何一般观察到来自非克隆稳定池的蛋白质生产中的高变异(表型多样性的随机抽样)。
这种采样不足和高生物噪声也使得难以比较用于优化表达的治疗性蛋白质候选者的不同基因盒设计。经由平行生成稳定池的多重变体比较是高度工作密集的,并且高生物噪声将使得结果不可靠。变体文库的同时转染的使用受到以下事实的阻碍:随机整合通常导致表达载体的多重拷贝的整合,并且因此通过此类工作流程生成的任何细胞通常含有来自多于一种基因盒设计的整合拷贝。通过基于效应基因的随机整合的细胞系改造策略来改善蛋白质表达也受到同样原因的阻碍。
对所有上述局限性的一个潜在地重大改善是利用目的基因(GOI)的靶向整合(定点整合;SDI)。在此类情形下,已知支持高稳定转录的预先鉴定的基因组位置用作GOI的靶终点。使用预先引入的序列和载体设计的智能组合,包括使用共转染的核酸酶如核酸酶或重组酶,将促进靶向插入并确保培养中的所有细胞都含有正确插入的GOI,且因此具有高转录率。这将显著减少用于细胞系开发(CLD)的筛选活动中所需的克隆数目,并且减少在细胞系改造努力或基因盒设计的比较中的生物噪声。在本领域[3-6]中描述了用于靶向整合的多重技术方案。然而,尽管如此,挑战依然存在。
利用靶向整合的所有策略的一般挑战是生成足够高的GOI表达,因为典型的解决方案导致单个GOI拷贝的整合。下文概述了本领域中描述的可用解决方案的其它挑战和局限性。
用于靶向整合的Flp-In系统(基于Flp/翻转酶重组酶,也称为翻转酶重组酶) [7]是利用与其重组酶组合的单个重组酶识别序列,以允许在预定基因组位置处的靶向整合的解决方案的实例。在重组酶的作用之后,完整的表达载体在重组酶识别序列处整合。可以选择具有正确整合事件的细胞,因为在重组酶识别位点处的整合使一种选择标记物失活,并且激活第二选择标记物。这种解决方案的主要缺点是(i)不存在检测或去除已通过随机整合事件整合了表达载体的另外拷贝的细胞的机制,(ii)不存在去除序列区域例如质粒骨架序列和活性选择标记物基因的机制,所述序列区域可能对GOI的表达是负面的,并且(iii)该方法在选择标记物的选择方面具有可疑的灵活性,因为在整合过程中选择标记物的激活导致在N末端处的额外氨基酸的融合,这可能影响其功能性。
为了避免在靶向整合之后存在对GOI表达具有潜在负面影响的序列,本领域已描述了用于在预定基因组位置处的盒式交换反应的不同解决方案[3-6]。此类解决方案的实例已通过Rentschler [8]得到公开。预定基因组位置利用侧翼为两个正交重组酶识别序列的活性选择标记物基因(GFP),所述两个正交重组酶识别序列均为相同重组酶的靶。表达载体中的GOI依次地侧翼为与基因组中存在的两个匹配的两个重组酶识别序列。在重组酶的作用下,可以发生在选择标记物盒和GOI盒之间的盒式交换。已经历盒式交换的细胞可以通过不存在GFP表达进行选择。这种解决方案的缺点是(i)不存在检测或去除已通过随机整合事件整合了表达载体的另外拷贝的细胞的机制,(ii)因为已经历盒式交换的细胞的选择基于最初活性的基因产物的不存在,所以必须延迟用于选择的时间点以允许GFP的降解/稀释。
Haghighat-Khah RE等人公开了昆虫中的两步位点特异性盒式交换系统,所述昆虫即埃及伊蚊(
Yuan, Y等人公开了基于重组酶的方法,以利用PhiC31介导的基因递送到靶向细胞的基因组中天然存在的假attP序列内,来产生无选择标记物和载体骨架的转基因细胞[10]。经由在表达载体中存在活性eGFP表达盒来实现其中已发生整合的细胞的选择,并且使用在靶向整合后变得失活的att-B-TK融合基因作为阴性选择标记物,以在第二选择步骤中消除随机整合事件。随后通过使用另外两种重组酶Cre和Dre来切除选择系统和质粒细菌骨架。通过Yuan, Y等人公开的方法用于基于整合到一个预定基因组位置内的重组蛋白质生产应用的严重缺陷在于:(i)该方法并未提供区分仅在预定位置处已经历整合的细胞与在预定位点和随机的假attP位点两者处已经历整合的细胞的手段,因为无活性TK基因将起因于这两种情形,(ii)直到选择标记物的瞬时表达消失才能执行第一选择步骤,这增加了时间,(iii)第一选择步骤并不区分所需的整合、在假attP位点处的整合或随机整合事件。
相应地,本领域仍需要鉴定用于生产重组蛋白质的改善的表达系统。在这方面,本领域需要改善现有的SDI系统,以将下述期望特征组合到单个解决方案内:
(a)支持GOI表达水平的能力,与随机整合解决方案配对,
(b)已经历了在预定基因组位置处的整合的细胞的快速和特异性选择,
(c)检测且去除已经历了另外的不期望整合事件的细胞的手段,
(d)避免可以对分离细胞中的GOI表达具有负面作用的序列存在的手段。
发明内容
上文提到的问题现在已通过提供本文进一步呈现的方法和手段得到解决或至少减轻。
本公开内容提供了利用供体载体的单个拷贝定点整合(SDI)到分离的真核宿主细胞的预定基因组位置内,用于重组蛋白质生产的新型解决方案。本公开内容的基于SDI的系统基于充分确立的核酸组分的独特和创造性的组合,用于将供体载体有效整合到宿主细胞的专用靶位点内。该方法提供了已将供体载体整合到专用的预定基因组位置内的宿主细胞的特异性阳性选择。该方法还提供了通过阴性选择检测且任选地去除在宿主细胞基因组的其它位置处已发生不期望的整合事件的任何细胞。这种两步选择方法是独特的,并且在重组蛋白质生产领域中将是非常有用的。
如本文先前提到的,用于改善的细胞系开发、灵活的细胞系改造和高级应用(例如基因构建体文库的同时探测)的实现的关键组分是重组基因整合到宿主细胞系内的增加控制以及对其拷贝数的更佳控制。这现在由本公开内容提供。
最初,中国仓鼠卵巢(CHO)细胞用于建立本文呈现的方法,因为假定的热点位置已得到鉴定,但SDI系统应该适用于任何真核细胞系统,包括哺乳动物细胞,例如人细胞。
相应地,在第一个方面,本公开内容涉及用于将供体载体靶向整合到真核细胞的预定基因组位置内的方法,所述方法包括:
i)提供包含预定基因组位置的真核细胞,所述预定位置包括:
a. 包含第一DNA酶的识别位点的核酸序列I1;
b. 包含第二DNA酶的识别位点的核酸序列E1;和
c. 启动子核酸序列P1;
ii)提供供体载体,其包含:
a. 核酸序列I2;
b. 目的核酸序列;
c. 包含所述第二DNA酶的识别位点的核酸序列E2;
d. 编码第一选择标记物的核酸序列;和
e. 任选地编码第二选择标记物的表达盒;
iii)在第一DNA酶的存在下使供体载体与细胞接触,其中所述第一DNA酶的存在使得供体载体的核酸序列I2和细胞的预定基因组位置中存在的核酸序列I1之间能够进行重组;
iv)通过检测细胞中的第一选择标记物的表达,来选择具有在预定基因组位置处整合的供体载体的细胞,其中所述第一选择标记物的表达通过在预定基因组位置处的启动子核酸序列P1激活;和
v)分离在上一步中选择的细胞。
在一个进一步方面,本公开内容涉及可通过如本文所述的方法获得的分离的真核细胞。
在再一个进一步的方面,本公开内容涉及可通过如本文所述的方法获得的分离的真核细胞用于生产重组蛋白质的用途。
在再一个进一步的方面,本公开内容涉及用于生产重组蛋白质的方法,所述方法包括:
i)通过执行如本文公开的方法获得包含在预定基因组位置处整合的供体载体的分离的真核细胞,所述供体载体包含一种或多种目的核酸序列,其中至少一种目的核酸序列包含至少一个表达盒,所述表达盒包含编码目的蛋白质的基因;
ii)在步骤i)的所述细胞中,产生由目的基因编码的蛋白质;和
iii)分离步骤ii)的蛋白质。
附图说明
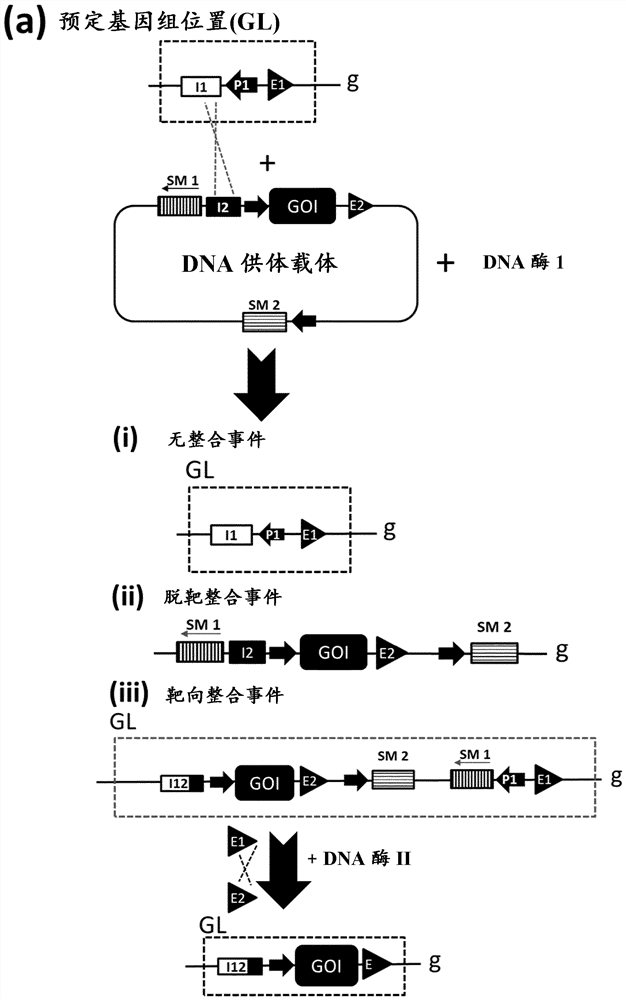
图1显示了经由使用具有正交特异性的至少两种DNA酶,用于将供体载体靶向整合到宿主细胞的预定基因组位置内的方法的一般概念的示意图。
图2显示了关于HyClone LP1P1和HyClone LP2P2细胞系的着陆垫(Landing Pad)设计(宿主细胞基因组的预定基因组位置中存在的核酸序列)和匹配供体载体的示意图。
图3说明了与未转染的对照(NC)相比,在转染后的第7天的流式细胞术图的实例。通过交替的黑色区域和白色区域(每个区域中的总细胞的20%)显现了具有集中的主要群体的图中的细胞密度。上面行显示了关于HyClone CHO细胞的非转染对照(NC)培养物的FACS数据。中间行显示了基于仅用供体载体B (不含PhiC31)转染的HyClone CHO细胞(缺乏LP),来自随机整合对照(RI)的FACS数据。下面行显示了关于用PhiC31和供体载体B (SDI)转染的HyClone CHO LP2P2细胞系的FACS数据。每行的中图中的门(B、D、F)基于未转染的对照进行设定,并且报告了已激活选择标记物超过背景的细胞百分比。
图4显示了所使用的着陆垫细胞系和供体载体,以及预计通过PhiC31 (1)和Cre(2)的活性发生的在着陆垫处的改变的示意图。
图5显示了与阴性模拟转染对照相比,在Cre重组酶变体转染后的第7天SDI群体的流式细胞术图。通过交替的黑色区域和白色区域(每个区域中的总细胞的20%)显现了具有集中的主要群体的图中的细胞密度。上图显示了关于缺乏Cre重组酶编码核酸分子的模拟转染的图,中图显示了用Cre重组酶表达质粒转染的群体的图,并且下图显示了用合成Cre重组酶mRNA转染的群体的图。
图6显示了所使用的着陆垫细胞系和供体载体,以及预计通过PhiC31重组酶(1)和Cre重组酶(2)的活性发生的在着陆垫处的改变的示意图。
图7显示了来自根据图6执行的步骤的流式细胞术图。通过交替的黑色区域和白色区域(每个区域中的总细胞的20%)显现了具有集中的主要群体的图中的细胞密度。上图中的左图显示了在第二eGFP (绿色荧光蛋白)阳性分选之后的群体。上图中的中图显示了Cre重组酶转染后7天的群体。上图中的右图显示了在Cre重组酶转染之后,根据门E执行的eGFP阴性分选后的群体。下图显示了用DNA供体载体B转染步骤2分选细胞后7天的群体的图。
图8显示了所使用的着陆垫细胞系和供体载体的示意图。GSx=谷氨酰胺合成酶基因变体。
图9显示了来自使用供体载体的SDI的细胞群体生成的流式细胞术图。通过交替的黑色区域和白色区域(每个区域中的总细胞的20%)显现了具有集中的主要群体的图中的细胞密度。上图显示了已经历了G418选择、RFP (红色荧光蛋白)阳性FACS分选、以及用合成Cre重组酶mRNA转染的群体的图。显示了关于RFP阴性亚群(对应于在着陆垫处的整合,伴随Cre重组酶介导的TagRFP-T切除)和RFP阳性亚群(对应于失败的Cre重组酶介导的TagRFP-T切除,其可以通过在着陆垫处的脱靶整合或截断整合引起)两者的eGFP直方图。下图显示了使用来自上图的RFP阴性/GFP阳性细胞的FACS分选生成的最终SDI池的图。
图10显示了经由IRES元件,与编码所述第二DNA酶的基因连接的所述供体载体的第一选择标记物。第一选择标记物和第二DNA酶两者均在预定基因组位置处的整合后得到激活。
图11显示了当预定基因组区域还包含所述第一DNA酶的表达盒时,所述表达盒这样定位使得当供体载体在预定基因组位置处整合后,它变得侧翼为所述第二DNA酶的识别位点,并且因此可以在所述第二DNA酶的存在下去除。
图12例示了关于靶向整合的变体,其中基因编辑酶用于催化供体载体整合到宿主细胞基因组的预定基因组位置内。
图13显示了关于靶向整合的变体,其中重组酶介导的盒式交换(RMCE)用于催化在宿主细胞基因组的预定基因组位置处的整合。
图14显示了关于靶向整合的变体,其中单个重组酶识别位点对用于催化供体载体在宿主细胞的预定基因组位置处的整合。
图15显示了使用单个重组酶识别位点对,以催化在预定基因组位置处的整合,并且在预定基因组位置处存在的启动子P1与断裂内含子的5'部分在功能上融合。
具体实施方式
本公开内容现在将与附图和一些非限制性实例结合更密切地进行描述。
本公开内容的细节在下文进行阐述。尽管现在描述了优选的材料和方法,但与本文所述的那些类似或等价的任何材料和方法都可以用于本发明的实践或测试中。本文中使用的所有词语和术语都应该被视为具有由本领域技术人员通常给予其的相同含义,除非另一种含义根据上下文是显而易见的。
“包含”一个或多个所述元素的组合物还可以包括未具体叙述的其它元素。
单数“一个”和“一种”应该解释为还包括复数。
“表达”用于意指自基因的蛋白质生产,并且在本文中指的是并包含“中心法则”的步骤,即转录、翻译和蛋白质折叠的相继作用,以达到蛋白质的活性状态。
如本文定义的“表达载体”是包含核酸序列的载体,以在存在于宿主细胞中时实现自载体的蛋白质表达。本文中的表达载体用于例如将特定的目的基因引入细胞内,以其后指导用于蛋白质合成的细胞机制,以产生由目的基因编码的目的蛋白质。表达载体可以含有“表达盒”,所述表达盒含有核酸序列以促进蛋白质表达。另外,载体可以含有其它核酸序列元件或组分。
如本文提及的“供体载体”是载体,优选DNA载体,其包含用于促进载体整合到分离的真核宿主细胞的预定基因组位置内的核酸元件或组分。供体载体携带促进与宿主细胞的预定基因组位置中存在的核酸序列的重组事件的核酸序列、任选地编码目的蛋白质的目的核酸序列、第二DNA酶的识别位点和编码第一选择标记物的核酸序列。任选地,它还可以含有第二选择标记物的表达盒。“供体载体”在本文中有时也可以简称为“载体”。“供体载体”有时可以是表达载体的形式,例如当供体载体包含编码第二选择标记物的表达盒时。更具体而言,本文所述的供体载体至少含有用于与真核细胞的预定基因组位置中存在的I1重组的核酸序列I2。另外,它包含目的核酸序列,如果所述目的核酸编码目的蛋白质,则在本文中也被称为目的基因(“GOI”)。它还包括包含第二DNA酶的识别位点的核酸序列E2,其使得一旦供体载体的稳定整合已在宿主细胞的预定基因组位置中发生,就能够切除载体骨架的部分。它还含有编码第一选择标记物(SM1)的核酸序列,仅当供体载体已整合到宿主细胞的预定基因组位置中的正确位置内时,所述核酸序列的表达才被激活。最后,供体载体任选地包含编码第二选择标记物(SM2)的表达盒。在第二DNA酶的作用之后,仅当载体的随机整合事件已发生并且用于本方法的第二轮选择中时,第二选择标记物才在细胞中表达并且可能检测到。供体载体优选为DNA供体载体,但不限于此。DNA供体载体有时缩写为“DDV”。
“表达盒”是构成表达载体的部分的核酸组分,其含有目的蛋白质的转录和翻译启动所需的所有元件。编码目的蛋白质的目的基因也构成表达盒的部分。表达盒含有例如对于转录启动必需的启动子,以及促进转录的其它序列例如增强子序列。有时在本文中使用术语“整合盒”,其对应于来自供体载体的核酸序列,其在第二DNA酶的作用后保留在预定基因组位置处。“整合盒”可以包含“表达盒”。
在本文中,“一种”目的基因指产生目的蛋白质所需的核酸组分,并且由于目的蛋白质可以包含多重多肽链,因此也可以指存在于相同表达盒中的多重目的基因。含有多重目的基因的表达盒可以利用个别启动子来实现个别基因的转录,或者两种或更多种基因可以作为共同mRNA进行转录,其中个别基因由例如IRES元件分开。这与本文一致,每当使用“一个/种”时,这也可以指复数。当表达盒包含多于一种目的基因时的实例是当待由目的基因表达抗体时,例如其中轻链和重链抗体组分作为分开的基因存在于表达盒中。
“内含子”是基因的核酸序列,其在转录后和最终RNA产物的生产过程中通过RNA剪接去除。内含子是RNA转录物或编码其的DNA的非编码区,其在翻译前通过剪接得到消除。
在本文中,与断裂内含子的5'部分在功能上融合的启动子意指断裂内含子的5'部分的转录由所述启动子驱动。在本文中,断裂内含子的5'部分定义为包含剪接供体位点序列(例如GT)。在本文中,断裂内含子的3'部分可以定义为包含(i)剪接分支位点序列,(ii)富含Py的序列区域和(iii)剪接受体位点序列(例如AG)。
转录包含通过细胞机制的DNA至RNA的转换。“转录调控序列”是能够增加或降低特定基因的最终表达的核酸序列的区段,即通过所述序列能够调控所述基因的转录。转录调控序列的实例是启动子、增强子和类似元件。
非翻译区(“UTR”)指在mRNA链上的编码序列的每一侧上的两个区段中的任一个。在5'侧上,它被称为5' UTR,在3'侧上,它被称为3' UTR。
如本文提及的,上游开放读码框(uORF)是mRNA分子的5'非翻译区(5'UTR)内的开放读码框(ORF)。uORF一般涉及真核基因表达的调控。uORF的翻译通常抑制初级ORF (开放读码框)的下游表达,相应地当存在时,这些导致蛋白质表达的减少。约一半的人基因含有这些区域。
内部核糖体进入位点(“IRES”)是允许以非帽依赖性方式的翻译启动的RNA元件。它们经常被称为能够将真核核糖体募集到mRNA的RNA分子的不同区域。IRES元件的定位经常在5' UTR区中,但它也可以在mRNA中的其它地方出现。
“质粒”是一种小的环状染色体外DNA分子,其可以不依赖于细胞进行复制,并且在细菌中发现。质粒经常用作用于分子克隆的载体,即将所选择的DNA转移且引入宿主细胞。质粒由特定和必要的元件构建,并且可能含有可以与细菌宿主细胞同源或异源的基因。质粒例如始终含有细菌的复制起点,且最经常含有特定抗生素抗性基因。
如本文提及的,“目的核酸序列”可以定义为希望整合到细胞内以影响所述细胞的功能性的核酸序列。它可以包含编码目的蛋白质的目的基因(“GOI”)。
如本文提到的,“重组”蛋白质意指由通过表达载体引入细胞内的表达盒制造的蛋白质。用于生产重组蛋白质的技术是本领域技术人员众所周知的。
“启动子”是在RNA聚合酶与其结合后启动基因转录的DNA区域。启动子定位于基因的转录起始位点附近。
如本文提及的,“宿主细胞”涉及预期或已被如本文公开的供体载体转化的真核细胞。
“分离的细胞”、“分离的宿主细胞”或“分离的真核宿主细胞”指已从其自然环境中分离的细胞,这意指它不含可能在自然界中出现的任何另外组分,并且它不再是其自然环境的任何部分。
在本文中,“预定基因组位置”有时也被称为“着陆垫”(缩写为“LP”),更确切地说被称为包含着陆垫序列的预定基因组位置,预期指特征在于宿主细胞基因组中的特定核酸序列的定位或核酸位置。预定基因组位置在本文中也可以被称为“安全港位点”和/或“重组位点”。在宿主细胞的预定基因组位置处,将发生通过第一DNA酶的存在促进的在核酸序列l1和l2之间的重组事件,启动第一选择标记物的表达并指示成功的整合事件。基本上,预定基因组位置包括包含第一DNA酶的识别位点的核酸序列、包含第二DNA酶的识别位点的核酸序列和启动子核酸序列。
在本文中,当提及“靶向整合”时,它预期意指将核酸序列元件或组分整合或引入另一种核酸元件或组分内,促进此类序列之间的重组事件,从而由原始序列生成杂合序列。此类整合事件通过在构成关于重组的基础的任何一种或几种核酸序列元件或组分中存在的酶识别核酸序列得到触发。
“酶的识别位点”指核酸序列中的核苷酸的特定组合,所述组合通过促进酶与其结合的特定酶识别,并且其中所述酶其后将启动在识别位点处的作用,例如在两个序列之间的重组事件。
本文提及的术语“DNA酶”定义为作用于DNA的酶,例如切割DNA的小片或切割DNA并将其整合到另一种DNA序列内。该术语包括酶如Crisps/Cas9、重组酶、整合酶、核酸酶等,但本公开内容并不限于此。
本文提及的“第一DNA酶”可以在功能上定义为在本文公开的方法中,负责供体载体在宿主细胞的预定基因组位置处的整合的酶。第一DNA酶的功能是将核酸序列引入预定基因组区域内而不是去除。当用于本文公开的方法中时,第一DNA酶可以是一种特定酶,或者它可以是不同的酶。例如如果供体载体的整合是序贯的,并且从而重复多次,则这是将目的核酸序列/供体载体的多重拷贝/变体引入宿主细胞的预定基因组位置内,或者如果执行目的核酸序列的可逆整合。用于在本方法的上下文中使用的“第一DNA酶”的实例在本文中的其它地方给出。
本文提及的“第二DNA酶”可以在功能上定义为在本文公开的方法中,负责从已整合了供体载体的预定基因组位置处切除核酸序列区域的酶,其中所述核酸序列区域的侧翼为由第二DNA酶识别的特定序列。当第二DNA酶识别序列时,它将切掉这些序列之间的核酸序列组分。用于在本方法的上下文中使用的“第二DNA酶”的实例在本文中的其它地方给出。
“在第一DNA酶的存在下”和/或“在第二DNA酶的存在下”意指第一DNA酶和/或第二DNA酶以如本文所述的任何形式提供,例如作为由供体载体、分开的表达载体、细胞的基因组中存在的表达盒、合成的mRNA等表达的蛋白质。“在……的存在下”预期指第一DNA酶和/或第二DNA酶的功能以本文公开的任何合适的方式提供。
本文提及的“选择标记物”是可以指示特定事件已发生的标记物,例如,在本上下文中,供体载体的整合已在宿主细胞的预定基因组位置处发生(第一选择标记物)。选择标记物经常是荧光蛋白,一旦供体载体已在宿主细胞基因组的正确位点处整合,所述荧光蛋白就由宿主细胞表达。荧光蛋白的表达可以例如通过FACS (荧光激活细胞分选)进行检测。其它可能的选择标记物在本文中的其它地方提到。
当存在于供体载体中时,“第一选择标记物”,在本文中也缩写为“SM1”,可以定义为沉默的、无活性的或无启动子的选择标记物。第一选择标记物含有与预定基因组位置中存在的启动子相容的非编码段。一旦已将供体载体整合到预定基因组位置中的正确位置内,第一选择标记物就可以被表达,因为它现在具有启动转录的启动子。一旦选择标记物被表达,表达第一选择标记物的细胞群体就可以选择为对于供体载体在预定基因组位置处的稳定整合呈阳性。第一选择标记物在本文中也可以被称为“报告分子”。合适的第一选择标记物的实例在本文中的其它地方提供。
当存在于供体载体中时,其是本文公开的供体载体的任选特征的“第二选择标记物”,在本文中也缩写为“SM2”,可以定义为非沉默的、活性的和/或功能性的选择标记物。选择标记物作为表达盒的部分进行编码,即选择标记物将在进入细胞内后瞬时表达,并且稍后促进稳定表达,而不依赖于它在基因组中引入的位置。在本文呈现的方法的大多数方面,第二选择标记物是阴性选择标记物,其意指表达该标记物的细胞优选地并不用于重组蛋白质生产,因为这些细胞(也)已在除预定基因组位置外的其它地方整合了供体载体。
本文提供了利用特定定点整合(SDI)系统用于将供体载体靶向和可检测的整合到分离的真核细胞的预定基因组位置内的方法。另外,该方法允许鉴定供体载体在真核宿主细胞基因组的其它部分而不是预定位置内的随机整合事件。
这组合地提供了已在宿主细胞基因组的靶位点(预定基因组位置)处阳性整合了供体载体的细胞群体的“双重”选择,优选地在宿主细胞基因组的其它位置处的供体载体的另外随机整合的不存在下,从而提供了用于后续重组蛋白质表达的优化系统。该方法使用基于细胞群体的阳性(在预定基因组位置处的整合)和后续阴性(随机整合事件的不存在)选择的组合选择策略。
总体解决方案基于所谓的“着陆垫”(LP)序列在预定基因组位置处的整合,所述预定基因组位置因其支持高转录及其长期稳定性的能力进行选择。着陆垫连同匹配的供体载体一起进行设计,其允许受控整合到预定位点内、以及直接选择其中仅已发生所需整合的细胞。在本文中,预定基因组位置和着陆垫/着陆垫序列可以互换使用。
SDI系统的基本设计使用两类DNA酶识别序列连同两种不同DNA酶例如特异性重组酶的组合,以允许(i)包含目的核酸序列的供体载体在真核宿主细胞的预靶向基因组位置处的整合,(ii)使用至少一个或可能两个正交选择步骤,来选择已在预定基因组位置处整合了供体载体的单个拷贝的细胞,以及(iii)任选地去除在预定基因组位置处来自供体载体的不期望序列。
该方法的一般实施方式在图1中进行概述。所述分离的真核细胞的预定基因组位置包括(i)包含第一DNA酶的识别位点的核酸序列I1,(ii)包含第二DNA酶的识别位点的核酸序列E1,以及(iii)包含起始转录位点的启动子核酸序列P1。I1、E2和P1以两个对称的5'-3'序列取向中的任一个进行配置:O1 = [I1,具有3’-5’方向性的P1,E1]或O2 = [E1,具有5’-3’ 方向性的P1,I1]。供体载体包含(i)在所述第一DNA酶的存在下促进与I1的重组的核酸序列I2,(ii)缺乏启动子的第一选择标记物基因(SM1),(iii)所述第二DNA酶的识别位点E2,(iv)整合盒IC以及任选地(v)第二选择标记物基因(SM2)的活性表达盒。SM1、SM2 (当存在时)、E2和IC以两个对称的顺时针取向中的任一个进行配置:O3 = [I2、IC、E2、SM2、SM1(具有逆时针方向性)]或O4 = [I2、SM1(具有顺时针方向性)、SM2、E2、IC]。存在于预定基因组位置和供体载体中的核酸序列元件始终以两个匹配取向(a) O1/O3或(b) O2/O4中的任一个进行配置。
通过在第一DNA酶的存在下将供体载体引入细胞内,来实现完整的供体载体或供体载体的部分整合到所述分离的真核细胞的预定基因组位置内,其中所述第一DNA酶的存在使得供体载体的核酸序列I2和细胞的预定基因组位置处存在的核酸序列I1之间能够进行重组。
在预定基因组位置处整合定位了SM1基因,使得P1可以实现SM1基因的转录、以及因此SM1基因产物的表达。相应地,可以通过使用SM1的表达作为阳性选择的标准,来选择且分离已在预定基因组位置处整合了完整的供体载体或供体载体的部分的细胞。
任选地,可以潜在地负面影响分离细胞的预期功能性的不期望序列可以在补充步骤中从预定基因组位置处特异性地去除,仅留下整合盒(IC)以及来自I1、I2、E1和E2的残留序列。
完整的供体载体或供体载体的部分在预定基因组位置处整合后,质粒骨架序列(即用于细菌中的质粒繁殖的序列)以及SM1和SM2的表达盒(如果存在的话)变得侧翼为两个核酸序列E1和E2 (参见图1)。在所述第二DNA酶的存在下,经由作用于E1和E2的第二DNA酶,从预定基因组位置处切除侧翼为E1和E2的该序列区域。可以在基于SM1表达的不存在(如果SM2不存在于原始供体载体中)和/或SM2表达的不存在(如果SM2存在于原始供体载体中)的阴性选择步骤中,选择且分离已切除了侧翼为E1和E2的区域的细胞。除了实现不期望序列的去除之外,这种补充选择步骤始终增加了细胞分离中的特异性,所述细胞已在预定基因组位置处整合了完整的供体载体或供体载体的部分,因为在预定基因组位置之外的整合后已实现SM1的激活(通过非特异性机制)的任何细胞,将不具有侧翼为E1和E2的SM1,并且因此不在基于SM1表达的阴性选择步骤中被选择。
具有在供体载体中存在的SM2,可以在所述第二DNA酶的作用之后执行具有改善功能性的选择步骤。由于SM2作为活性表达盒提供,因此在不期望的基因组位置处整合的供体载体的任何拷贝将导致SM2的表达。然而,重要的是,此类整合事件并不导致侧翼为E1和E2的SM2表达盒,因为E1仅存在于预定基因组位置处。因此,在所述第二DNA酶的作用导致侧翼为E1和E2的序列区域切除之后,可以在基于SM2表达的不存在的阴性选择步骤中,选择且分离已在且仅在预定基因组位置处整合了整合盒(IC)的单个拷贝的细胞。
整合盒(IC)通常包含目的基因(GOI)的表达盒,但该方法的应用并不限于此。
一般方法的具体实施方式和进一步实例一般将使用在预定基因组位置处以及在供体载体中存在的关键序列元件的两种可能的对称取向中的仅一种进行例示,但不限于此。
图4中概述了设计概念的一种具体实施方式,其特征为着陆垫(LP1P1)和DNA供体载体。基于该实施方式执行的实验的结果也在实施例2的实验部分中进一步说明且讨论。该实施方式仅仅是执行本发明的一种方式的实例,但并不预期限于此。
相应地,在如图4中所示的一种实施方式中,真核宿主细胞系含有在预定基因组位置中的重组酶PhiC31重组酶的第一重组酶识别序列(
PhiC31重组酶是衍生自链霉菌属(
匹配的DNA供体载体包括缺乏以逆时针取向编码的启动子的第一选择标记物(此处通过RFP,红色荧光蛋白进行例示)、匹配的PhiC31重组酶识别序列(
将DNA供体载体和用于表达PhiC31的载体共转染到包含着陆垫(LP)序列的预定基因组位置的真核宿主细胞内,将导致对于一小部分的转染细胞,经由PhiC31介导的
由于预定基因组位置含有
本文公开的一般SDI系统的一些关键共同理论益处在于:
(1)允许选择步骤使以下的可能性降到最低:分离的细胞不同于具有在且仅在预定基因组位置处整合的例如目的基因(GOI)的单个拷贝的期望结果。这在CLD活动中很重要,因为它减少了生物变异并且因此减少了筛选需要。它还改善了从CLD活动中分离的细胞在平台培养过程中表现良好的可能性。对于基于包含表达盒文库设计的供体载体混合物的转染的表达盒设计优化,这是关键特征,因为需要在细胞表型和单个相应基因盒设计之间存在一对一的相关性。
(2)仅保留了促成细胞系的生产率的序列。没有细胞资源浪费在选择标记物蛋白的表达或截短的GOI版本的表达上,就像基于随机整合(RI)的细胞系开发(CLD)的情况一样。可以避免存在对GOI的长期表达稳定性具有潜在的负面影响的细菌起源的序列。
(3)在选择标记物的选择中存在灵活性,因为选择标记物不是预定基因组位置序列的部分。可以基于应用来选择最佳选择标记物。
(4)所需的整合事件激活第一选择标记物的表达,允许具有在预定基因组位置处的整合的细胞以高特异性的阳性选择。使用选择标记物例如荧光蛋白或细胞表面标记物,这允许在转染和阳性整合体的选择之间非常短的时间段(使用例如FACS或MACS)。两到三天应该可以获得结果。这缩短了CLD活动所需的时间。另外,已经历了在所需位置处的整合的细胞与已经历了不期望的整合事件或无整合的细胞的早期分离可以具有进一步的益处,因为它使期望的细胞被不期望的细胞生长超过的风险降到最低。因此,与缺乏该特征的方法相比,该方法的效率和性能可以得到改善。
(5)该方法允许在同一基因组位置处的序贯整合,而无不需要的序列的积累。这可以通过将第二次整合事件所需的新序列置于第一GOI (或目的核酸序列)下游的预定基因组位置处来实现,如图6中对于用作第一DNA酶的PhiC31所示的。这个特征还允许生成具有多重着陆垫的宿主真核细胞系,所述多重着陆垫可以个别地进行寻址。这允许GOI的多重拷贝在一个或几个预定基因组位置处整合,以实现相应的目的蛋白质(POI)的表达增加。可替代地,它可以用于经由细胞效应蛋白的受控整合改善表达,来允许蛋白质和克隆特异性细胞系改造。
利用丝氨酸重组酶如PhiC31或Bxb1作为第一DNA酶与单个匹配重组酶识别序列对(即
因此,本公开内容提供了将目的核酸序列(例如编码目的蛋白质)有效和选择性靶向整合到宿主细胞内的新型和改善的方式。已选择性地整合了包含目的核酸序列的供体载体的单个拷贝的分离的宿主细胞将呈现用于重组蛋白质生产的极佳系统,其可用于许多不同的应用领域中。
相应地,在第一个方面,本公开内容涉及用于将供体载体靶向整合到分离的真核细胞的预定基因组位置内的方法,所述方法包括:
i)提供包含预定基因组位置的分离的真核细胞,所述预定位置包括:
a. 包含第一DNA酶的识别位点的核酸序列I1;
b. 包含第二DNA酶的识别位点的核酸序列E1;和
c. 启动子核酸序列P1;
ii)提供供体载体,其包含:
a. 核酸序列I2;
b. 目的核酸序列;
c. 包含所述第二DNA酶的识别位点的核酸序列E2;
d. 编码第一选择标记物的核酸序列;e. 任选地编码第二选择标记物的表达盒;
iii)在第一DNA酶的存在下使供体载体与细胞接触,其中所述第一DNA酶的存在使得供体载体的核酸序列I2和细胞的预定基因组位置中存在的核酸序列I1之间能够进行重组;
iv)通过检测细胞中的第一选择标记物的表达,来选择具有在预定基因组位置处整合的供体载体的细胞,其中所述第一选择标记物的表达通过在预定基因组位置处的启动子核酸序列P1激活;和
v)分离在上一步中选择的细胞。
第一选择标记物在本文中也可以被缩写并称为“SM1”。
第二选择标记物在本文中也可以被缩写并称为“SM2”。
第一DNA酶的非限制性实例是DNA重组酶,例如PhiC31或Bxb1重组酶,并且如本文其它地方所述的。当用作第一DNA酶时,重组酶的表征特征是它将核酸序列区域引入预定基因组区域内而不是去除。
第二DNA酶的非限制性实例是DNA重组酶,例如PhiC31重组酶、Bxb1重组酶、Cre重组酶和Dre重组酶,并且如本文其它地方所述的。当用作第二DNA酶时,重组酶的表征特征是它从预定基因组区域处去除核酸序列区域而不是引入。
包含第一DNA酶的识别位点的核酸序列I1可以是所述预定基因组位置中存在的PhiC31或Bxb1重组酶的
核酸序列I2可以是所述供体载体中存在的PhiC31或Bxb1重组酶的
核酸序列E1可以是Cre重组酶的
核酸序列E2可以是Cre重组酶的
如果第一DNA酶是PhiC31重组酶,则第二DNA酶不是PhiC31重组酶。这同样适用于任何其它第一DNA酶和第二DNA酶,即第一DNA酶和第二DNA酶在同一SDI系统中绝不等同。
在本文中,所述供体载体的第一选择标记物(SM1)可以经由IRES元件与编码所述第二DNA酶的基因连接,或SM1的氨基酸序列和所述第二DNA酶通过自剪切肽这样进行融合,使得第一选择标记物和第二DNA酶两者均在预定基因组位置处的整合后得到激活。这在图10中进行说明。一旦供体载体已整合到预定基因组位置内,这就确保了第二DNA酶的存在,并且不需要进一步引入核酸载体来继续进行该方法的步骤。SM1的表达可以继续进行,直到第二DNA酶的细胞内浓度已达到足够高的值,以促进侧翼为E1和E2的序列区域的核定位和切除。通过阳性选择步骤的适当时机,已经历了在预定基因组位置处的整合的细胞将含有允许阳性选择的SM1水平。
在本文中,预定基因组位置还可以包含所述第一DNA酶的表达盒,其这样定位使得当供体载体在预定基因组位置处整合后,它变得侧翼为所述第二DNA酶的识别位点,并且经由所述第二DNA酶的作用从预定基因组区域处切除。这在图11中进行说明。这进一步简化了方法,并且应该提高了高整合效率的可能性。由于表达盒在该方法的较后步骤过程中去除,因此没有细胞资源浪费在最终分离的细胞中的第一DNA酶的表达上,并且避免了第一DNA酶的长期存在的任何负面后果。
相应地,第一DNA酶可以通过由预定基因组位置表达或通过以任何形式引入细胞内进行提供,在所述细胞中产生所述第一DNA酶的瞬时存在。这包括引入分离的蛋白质本身、引入包含所述第一DNA酶的表达盒的分开的表达质粒、在所述供体载体中存在所述第一DNA酶的活性表达盒或引入编码所述第一DNA酶的合成mRNA。
如本文先前提到的,本公开内容的所有方面都允许在选择标记物的选择方面的灵活性,而不必对预定基因组位置进行任何改变。SM1可以选自(i)抗生素抗性基因,(ii)代谢酶基因如GS或DHFR,(iii)荧光蛋白基因,或(iv)细胞表面标记物如CD4或CD10。SM2可以选自(i)生成有毒产物的酶如TK,(ii)荧光蛋白基因,或(iii)细胞表面标记物如CD4或CD10。
优选地,两种选择标记物均选自(i)荧光蛋白基因,或(ii)细胞表面标记物,允许经由方法如FACS或MACS的快速选择步骤。
如果选择标记物是荧光蛋白,则可以例如通过使用FACS来检测第一选择标记物或第二选择标记物的表达。如果选择标记物是抗生素抗性基因,则可以通过在相应抗生素的存在下培养细胞来检测整合。如果细胞在已向其添加抗生素的培养基中存活,则供体载体已成功地整合。
本文还提供了进一步包括步骤vi)的方法,所述步骤vi)包括在第二DNA酶的存在下,从本文方法的步骤v)中分离的细胞的预定基因组位置处切除侧翼为核酸序列E1和E2的核酸序列,其中所述第二DNA酶的存在使得核酸序列E1和E2之间能够进行重组,其中在所述细胞中侧翼为核酸序列E1和E2的核酸序列的存在是供体载体稳定整合到细胞的预定基因组位置内的指示。
如本文提到的,重组酶可用于切除宿主细胞基因组中的侧翼为适当的核酸区域(E1和E2)的核酸序列。这是主要在宿主细胞基因组中“整理”的步骤,因为一旦已进行整合和选择,引入预定基因组位置内的核酸序列的一些部分将是多余的。它们的存在也可能消耗细胞能量。核酸序列的切除意指通过结合核苷酸的特定组合,即E1和E2,第二DNA酶能够从宿主细胞基因组中切割且去除核酸序列部分。核酸序列E1和E2在预定基因组位置处的存在是供体载体的稳定整合已发生的原理证明。
在本文中,步骤vi)可以构成步骤iii)的部分,或者可以在步骤iii)之后执行,例如在所述方法的步骤iv)之后或在步骤v)之后执行。步骤vi)也可以在步骤v)之前执行。
还提供了一种方法,其中步骤ii)的供体载体进一步包含e)编码第二选择标记物的表达盒,并且其中步骤v)中分离的细胞另外已基于其不表达第二选择标记物进行选择,其中所述第二选择标记物的表达表示供体载体已在与细胞的预定基因组位置不同的位置处整合。
本文还提供了一种方法,其中步骤ii)的供体载体进一步包含e)编码第二选择标记物的表达盒,并且其中步骤vii)中分离的细胞已基于其不表达第二选择标记物进行选择,其中所述第二选择标记物的表达表示供体载体已在与细胞的预定基因组位置不同的位置处整合。
编码第二选择标记物的所述表达盒这样置于所述供体载体中,使得在预定基因组位置处的整合后,它变得侧翼为E1和E2序列。然而,如果供体载体在预定基因组位置之外整合,则编码第二选择标记物的所述表达盒将不由E1和E2侧接。
相应地,如果在第二DNA酶的作用之后,例如通过FACS检测到细胞群体中的细胞中的第二选择标记物(SM2)表达是可能的,则这意指供体载体的不期望的整合事件已在细胞中的另一个位置处发生。可以去除此类细胞,以(通过阴性选择)选择其中供体载体的整合已仅在预定基因组位置处发生的细胞。
还提供了一种方法,其中步骤vi)在步骤v)之后执行,并且所述方法进一步包括在步骤vi)之后执行的步骤vii),其包括分离其中侧翼为核酸序列E1和E2的所述核酸序列已从步骤vi)中分离的细胞的预定基因组位置处切除的细胞。
第二DNA酶可以作为分离的蛋白质本身提供,它可以由分开的表达载体或质粒上的表达盒表达,或者可以由编码所述第二DNA酶的合成mRNA表达。如先前所述的,一旦整合到预定基因组位置内,它就也可以由供体载体表达。
本文还提供了一种方法,其中步骤ii)的供体载体的目的核酸序列包含至少一个表达盒,所述表达盒包含编码目的蛋白质的基因。目的蛋白质可以是使用者希望表达的任何类型的重组蛋白质,例如抗体或其它治疗性蛋白质。
本文还提供了一种方法,其中切除的核酸序列缺乏至少一个表达盒,所述表达盒含有编码目的蛋白质的基因。
基于随机整合(RI)的细胞系开发通常导致具有整合的靶基因的多重拷贝的顶部克隆。为了确保本公开内容的方法还可以生成竞争性蛋白质表达水平所需的转录水平(每个细胞的mRNA拷贝#),具有以受控方式增加靶基因拷贝数的能力将是有利的。由于表达质粒的大小成为细菌扩增效率、转染效率、CHO细胞中的质粒稳定性和整合效率的问题,因此一次性整合多重拷贝可能是成问题的。
因此,本文还提供了用于将目的核酸的多重拷贝序贯整合到宿主细胞基因组内的方法。除了减少表达质粒的大小之外,以序贯方式插入拷贝还提供了逐渐增加置于细胞上的重组表达负荷的潜在附加特征。由于逐渐适应新应激环境的可能性,这依次又可以改善分离高产表型的可能性。如先前提及的,在预定基因组位置处的重复整合也可以用于允许通过以下的蛋白质和克隆特异性细胞系改造:首先引入目的蛋白质的表达盒,随后引入可以改善后续整合步骤中的目的蛋白质表达的细胞效应基因。
本文呈现的基于PhiC31/
相应地,为了提供多重目的核酸序列的序贯整合,本文还提供了包括如本文其它地方定义的步骤的方法,其中本文呈现的方法的步骤ii)的供体载体进一步包括:
f.包含第一DNA酶的识别位点的核酸序列I3;和
g. 启动子核酸序列P2。
如上文解释的,在所述供体载体中存在进一步的识别位点I3提供了两种或更多种供体载体在相同的预定遗传位置处的重复靶向整合。一旦第一次重组事件已在核酸序列对I1/I2 (例如
相应地,本文提供了用于将n种另外的供体载体序贯靶向整合到真核细胞的预定基因组位置内的方法,其中除本文先前提到的组分之外,第一供体载体进一步包括:f.包含第一DNA酶的识别位点的核酸序列I3;和g. 启动子核酸序列P2。
除以任何合适的次序执行至少步骤i)至iv)和任选地v)、vi)和/或vii)之外,用于将n种另外的供体载体序贯靶向整合到真核细胞的预定基因组位置内的方法,还包括执行n种另外的供体载体序贯靶向整合到分离的真核细胞的预定基因组位置内,
其中n为≥ 1的整数,例如2、3、4、5、6、7、8、9、10或任何其它数目;
该方法包括:
(A)将第一种另外的供体载体整合到细胞的预定基因组位置内,其包括:
I. 提供步骤v)中分离的或步骤vii)中分离的细胞;
II. 提供供体载体,其包含:
A. 核酸序列I4,其在所述第一DNA酶的存在下,能够与上一步提供的细胞中存在的相应核酸序列I3重组;
B. 目的核酸序列;
C. 核酸序列E2;
D. 编码第一选择标记物的核酸序列;和
E. 任选地编码第二选择标记物的表达盒;
F. 任选地包含第一DNA酶的识别位点的核酸序列I1和启动子核酸序列P1;
III. 在第一DNA酶的存在下将步骤II)的供体载体引入细胞内,其中所述第一DNA酶的存在使得供体载体的核酸序列I4和细胞的预定基因组位置中存在的核酸序列I3之间能够进行重组;
IV. 通过检测细胞中的第一选择标记物的表达,来选择具有在预定基因组位置处整合的供体载体的细胞,其中所述第一选择标记物的表达通过在细胞的预定基因组位置处的启动子核酸序列P2激活;
V. 分离上一步中选择的细胞;
VI. 在第二DNA酶的存在下,从步骤V中分离的细胞的预定基因组位置处切除侧翼为核酸序列E1和E2的核酸序列,其中所述第二DNA酶的存在使得核酸序列E1和E2之间能够进行重组,其中在所述细胞中侧翼为核酸序列E1和E2的核酸序列的存在是供体载体稳定整合到细胞的预定基因组位置内的指示;
VII. 分离其中侧翼为序列E1和E2的所述核酸序列已从步骤V中分离的细胞的所述预定基因组位置处切除的细胞;
(B)如果n大于在上一步中分离的细胞的预定基因组位置处整合的另外供体载体的数目,则将另外的供体载体整合到细胞的预定基因组位置内,其包括:
I. 提供在上一步中分离的细胞,所述细胞包含在预定基因组位置处整合的核酸序列I1;
II. 执行步骤ii)、步骤iii)-iv)、步骤v)、步骤vi)和步骤vii);
(C)如果n大于在上一步中分离的细胞的预定基因组位置处整合的另外供体载体的数目,则将另外的供体载体整合到细胞的预定基因组位置内,其包括:
I. 提供通过执行上一步(B)获得的细胞,所述细胞包含在预定基因组位置处整合的核酸序列I3;
II. 重复步骤(A)II至(A)IX和步骤(B),直到细胞具有在预定基因组位置处整合的n种另外的供体载体。
如本文先前提到的,供体载体中的识别位点I4提供了与已经存在于宿主细胞的预定基因组位置处的识别位点I3 (即,其在前一轮整合中进行整合)的重组。因而,通过经由供体载体随后引入识别位点变体,可以在预定基因组位置处引入目的核酸的第二个和进一步的一个/多个拷贝。用于在引入的供体载体和预定基因组区域中存在的核酸序列 (即,第一DNA酶识别位点)之间重组的特定识别位点变体(对),可以在多轮重组事件自始至终重复使用,只要在每一轮整合之间存在一对不同的识别位点。选择标记物也是如此。
具体地,提供了仅基于第一重组酶的两对正交识别序列和所述第一选择标记物的两种变体,用于将任何所需数目的供体载体拷贝整合到所述真核细胞的预定基因组位置内的迭代方法,其中:
(i)所述第一重组酶选自丝氨酸重组酶如PhiC31或Bxb1或其突变变体;
(ii)所述两种选择标记物变体选自(a)荧光蛋白或(b)异源细胞表面标记物;
(iii)奇数整合步骤中使用的供体载体包含:
(a)所述第一选择标记物的第一版本。
(b)来自第一对识别序列的第一重组酶识别序列。
(c)来自第二对正交识别序列的第一重组酶识别序列;
(iv)偶数整合步骤中使用的供体载体包含:
(a)所述第一选择标记物的第二版本。
(b)来自所述第二对正交识别序列的第二重组酶识别序列。
(c)来自第一对识别序列的第二重组酶识别序列;
(v)奇数整合步骤中的整合通过来自所述第一对重组酶识别序列的重组酶识别序列之间的重组得到促进;
(vi)偶数整合步骤中的整合通过来自所述正交的第二对重组酶识别序列的重组酶识别序列之间的重组得到促进;
(vii)在奇数整合步骤的迭代之前,通过作用于重组酶识别序列E1和E2的所述第二重组酶的存在,从预定基因组位置处切除第一选择标记物的所述第一版本;
(viii)在偶数整合步骤的迭代之前,通过作用于重组酶识别序列E1和E2的第二重组酶的存在,从预定基因组位置处切除第一选择标记物的所述第二版本;
(ix)所述第二重组酶选自酪氨酸重组酶如Cre、Dre或Flp;
(x) E1 = E2。
进一步提供了一种方法,其中:
- 步骤i)的细胞包含n个预定基因组位置,其各自分别包含核酸序列I,I1
- 步骤ii)包括提供1至n种供体载体,所述1至n种供体载体各自分别包含核酸序列I2
- 步骤iv)包括将1至n种供体载体引入细胞内;
- 步骤v)包括通过检测细胞中不同的第一选择标记物SM
其中n为≥ 2的整数。
还提供了一种方法,其中步骤ii)的供体载体进一步包括:
f.包含第一DNA酶的识别位点的核酸序列I3;和
g. 启动子核酸序列P2,和
其中切除的序列包含含有编码目的蛋白质的基因的表达盒。
切除的序列包含含有编码目的蛋白质的基因的表达盒的事实意指已存在可逆整合,其中第二轮重组可以在预定基因组位置处引入编码目的蛋白质的新的和“第一”目的核酸序列。因此,将不存在预定基因组位置中存在的相同目的基因的多重拷贝,其是本文先前提到的序贯整合的目的。这可以用于重复使用高性能克隆以表达另一种目的蛋白质。
在图8和实施例4中,说明了使用两个连续选择步骤生成SDI细胞池,即其中在整合后添加第二DNA酶,以去除不再在细胞中实现目的的核酸序列。在这个实例中,抗生素抗性基因用作第一选择标记物(SM1)。使用Cre重组酶执行第二轮选择,以切除在每端中的侧翼为
如本文先前提到的,所述第一DNA酶可以是重组酶。第一DNA酶可以是不同DNA酶例如重组酶的混合物,只要第一DNA酶的任何DNA酶与第二DNA酶不同。
在本文中,对于供体载体中存在的所述第一DNA酶,可能存在多于一个识别位点,例如两个或更多个识别位点。这意指对于预定基因组位置中存在的所述第一DNA酶,还存在多于一个识别位点,例如两个或更多个识别位点。图13中显示了利用重组酶的此类系统的实例。图13显示了重组酶介导的盒式交换(RMCE),以催化在预定基因组位置处的整合。根据图1-2、图9-11和图15修饰的其变体也由本公开内容包含。
因此,在图13的实例中,预定基因组位置按5'-3'序列次序包含:(i)第一重组酶的第一识别位点(I1a);(ii)所述第一重组酶的第二识别位点(I1b),(iii)具有3'-5'方向性的启动子P1,以及(v)第二重组酶的识别位点E1。
在本实例中,供体载体按5'-3'序列次序包含:(i)所述第一重组酶的第三识别位点(I2a),(ii)整合盒(IC),此处通过目的基因(GOI)的表达盒进行例示,(iii)所述第二重组酶的识别位点E2,(iv)第二选择标记物(SM2)的表达盒,(v)以3'-5'方向性编码的第一选择标记物(SM1)的基因,以及(vi)所述第一重组酶的第四识别位点(I2b)。
将供体载体和第一重组酶引入细胞群体内导致:(a)对于一小部分细胞,供体载体中的整合盒(即,侧翼为所述第三重组酶识别位点和第四重组酶识别位点的序列区域)在预定基因组位置处的整合(参见图13b,小图(ii)),以及(b)对于一小部分细胞,供体载体的脱靶基因组整合(在预定基因组位置之外) (参见图13b,小图(iii))。
在预定基因组位置处的整合导致形成SM1的活性表达盒(参见图13b,小图(ii))。进一步地,在预定基因组位置处的整合之后,SM1和SM2两者的侧翼为所述第二重组酶的两个识别位点。
通过脱靶事件的整合(参见图13b,小图(iii))通常并不导致SM1的激活,但的确整合了侧翼并非所述第二重组酶的两个识别位点的活性SM2。
已经历了在预定基因组位置处的整合的细胞不同于没有整合事件的细胞(参见图13b,小图(i)),以及仅已经历了通过SM1活性的脱靶整合事件的细胞。因此,SM1的活性可以用于选择已经历了在LP处的整合的细胞。
为了去除除在预定基因组位置处的整合之外已经历了脱靶整合事件的细胞,将所述第二重组酶的重组酶活性引入对于SM1活性进行选择的细胞内。对于在LP处的整合,这导致SM1和SM2两者的切除,以及因此其相应活性的切除。对于脱靶整合事件,这种反应无法发生,并且SM2活性保留。结果,可以从通过SM2活性的不存在已经历了多重整合事件的细胞中选择仅已经历了在预定基因组位置处的所需靶向整合事件的细胞。
最终选择的细胞的预定基因组位置(参见图13c)并不含有SM2的表达盒,也不含有SM1的激活表达盒或来自供体载体的任何残留序列,除了通过E1和E2 (E)的重组产生的序列之外。
所述第一重组酶可以选自(i)丝氨酸重组酶或(ii)酪氨酸重组酶。
所述第一重组酶识别位点至第四重组酶识别位点可以根据以下进行选择:
(a) I1a=I1b和I2a=I2b,使用丝氨酸重组酶的一对匹配的识别位点,例如PhiC31[I1a=I1b=
(b)使用丝氨酸重组酶如PhiC31的突变识别对,来选择两对不同的匹配识别位点(I1a和I1b = 不同的
(c) I1a=I2a和I1b=I2b,对于酪氨酸重组酶,存在突变的识别位点变体对,例如Cre (选自可用的突变
所述第二重组酶不同于所述第一重组酶,并且可以选自(i)丝氨酸重组酶或(ii)酪氨酸重组酶。
所述第二重组酶的识别位点E1和E2可以在序列上等同,如通过以下例示的:(i)E1=E2=
所述第二重组酶的识别位点E1和E2可以具有不同的序列,如通过以下例示的:(i)E1=
第一DNA酶的进一步的识别位点在本文中可以被称为I1的变体,即I1a和I1b,以及I2的变体,即I2a和I2b,以此类推。
相应地,本文还提供了一种方法,其中
(a) I1包含两种重组酶识别位点变体I1a和I1b;和
(b) I2包含两种重组酶识别位点变体I2a和I2b;和
(c)在所述第一DNA酶的存在下,I1a能够与I2a重组并且I1b能够与I2b重组。
有时,I1a与I2a等同并且I1b与I2b等同。I1a、I1b、I2a和I2b可以分别选自
在本文中,还提供了一种方法,其中:
(a) I1包含单个重组酶识别位点;和
(b) I2包含单个重组酶识别位点;和
(c) I1和I2能够在所述第一DNA酶的存在下重组。
由I1包含的重组酶识别位点也可以在序列上不同于由I2包含的重组酶识别位点。本文提供的重组酶识别位点可以选自
识别位点/DNA酶的任何变体或突变体将是其功能上等价的变体或突变体。本领域技术人员将构建且生产此类功能上等价的变体或突变体。
图14中显示了使用单个重组酶识别位点对以催化在预定基因组位置处的整合的实例。根据图1、图9-11和图15修饰的变体也由本公开内容包含。
在本实例中,预定基因组位置按5'-3'序列次序包含:(i)第一重组酶的第一识别位点(I1);(ii)具有3'-5'方向性的启动子P1,以及(iii)第二重组酶的第一识别位点E1。
在本实例中,供体载体按5'-3'序列次序包含:(i)所述第一重组酶的第二识别位点(I2),(ii)整合盒(IC),此处通过目的基因(GOI)的表达盒进行例示,(iii)所述第二重组酶的第二识别位点E2,(iv)第二选择标记物(SM2)的表达盒,以及(v)以3'-5'方向性编码的第一选择标记物(SM1)的基因。
将供体载体和第一重组酶引入LP细胞群体内导致:(a)对于一小部分LP细胞,供体载体在LP处的整合(参见图14b,小图(ii)),以及(b)对于一小部分细胞,供体载体的脱靶基因组整合(在预定基因组位置之外) (参见图14b,小图(iii))。
在预定基因组位置处的整合导致形成SM1的活性表达盒(参见图14b,小图(ii))。进一步地,在预定基因组位置处的整合之后,SM1和SM2两者的侧翼为所述第二重组酶的两个识别位点E1和E2。
通过脱靶事件的整合(参见图14b,小图(iii))通常并不导致SM1的激活,但的确整合了侧翼并非所述第二重组酶的识别位点的活性SM2。
已经历了在预定基因组位置处的整合的细胞不同于没有整合事件的细胞(参见图14b,小图(i))、以及仅已经历了通过SM1活性的脱靶整合事件的细胞。因此,SM1的活性可以用于选择已经历了在预定基因组位置处的整合的细胞。
为了去除除在预定基因组位置处的整合之外已经历了脱靶整合事件的细胞,将所述第二重组酶的重组酶活性引入对于SM1活性进行选择的细胞内。对于在预定基因组位置处的整合,这导致SM1和SM2两者的切除,以及因此其相应活性的切除。对于脱靶整合事件,这种反应无法发生,并且SM2活性保留。结果,可以从通过SM2活性的不存在已经历了多重整合事件的细胞中选择仅已经历了在LP处的所需靶向整合事件的LP细胞。
最终选择的细胞的预定基因组位置(参见图14c)并不含有SM2的表达盒,也不含有SM1的激活表达盒或来自供体载体的任何残留序列,除了通过I1和I2 (I12)以及E1和E2(E)的重组产生的序列之外。
所述第一重组酶可以选自丝氨酸重组酶如PhiC31和Bxb1。所述第一重组酶识别位点和第二重组酶识别位点(I1和I2)可以选择为具有匹配根据以下选择的重组酶的识别位点:(a) I1=
所述第二重组酶不同于所述第一重组酶,并且可以选自(i)丝氨酸重组酶或(ii)酪氨酸重组酶。
所述第二重组酶的识别位点E1和E2可以在序列上等同,如通过以下例示的:(i)E1=E2=
所述第二重组酶的识别位点E1和E2可以具有不同的序列,如通过以下例示的:(i)E1=
还提供了如图15中说明的一种方法,其例示了使用单个重组酶识别位点对,以催化在预定基因组位置处的整合,并且其中在预定基因组位置处存在的启动子P1在功能上融合到断裂内含子的5'部分。较早描述但根据图15修饰的方法(即使用断裂内含子设计)也由本公开内容包含。
在本实例中,预定基因组位置进一步包含具有3'-5'方向性的内含子的5'部分,以及在所述第一重组酶的所述第一识别位点和具有3'-5'方向性的所述启动子P1之间、具有3'-5'方向性的功能序列区域F1。
在本实例中,供体载体进一步包含定位于具有3'-5'方向性的所述第一选择标记物SM1和所述第一重组酶的所述第二识别位点之间的序列区域。所述序列区域按5'-3'序列次序包含:(a)具有3'-5'方向性的功能序列区域F3和(b)具有3'-5'方向性的内含子的3'部分,其进一步包含在剪接受体位点序列下游的功能序列区域F2。
在预定基因组位置处的整合后(参见图15b,上图),形成用于所述第一选择标记物SM1的完整表达盒,包括功能性内含子。因此,SM1的表达被激活。
在脱靶整合事件之后(参见图15b,下图),整合了SM1表达盒的截短版本。
作为偶然事件可以发生截短的SM1盒的转录。这可以是由于(a)启动子拯救,其中供体载体以此类方式进行整合,使得截短的SM1盒变得与细胞基因组中存在的天然启动子符合读框地定位,或(b)供体载体的切割和连环化,使得存在于供体载体中的启动子变得与截短的SM1盒符合读框地重新定向,随后为所得到的连环体的整合。
此类偶然事件可以减少基于SM1的细胞选择中的特异性,所述细胞已在预定基因组位置处整合了供体载体。通过使用功能序列区域F1-F3的特定组合,可以实现改善的特异性(参见图15b)。
在F1-F3的第一种设计中:(a) SM1 (当存在于供体载体中时)缺乏ATG起始密码子,并且与3'-内含子直接融合,(b) F1由(从3'-5')都具有3'-5'方向性的起始转录位点(TSS)、第一5'-UTR区、Kozak/翻译起始位点和ATG起始密码子构成。在脱靶整合事件之后,这意指整合的任何SM1基因都缺乏起始密码子,并且因此并不生成功能性SM1蛋白的表达。然而,在预定基因组位置处的整合后,将形成功能性表达盒。在内含子剪接后,ATG起始密码子将与SM1直接融合,导致SM1蛋白的适当表达。
在F1-F3的第二种设计中:(a) SM1含有ATG起始密码子,(b) F3由(从3'-5')都具有3'-5'方向性的第二5'-UTR区和Kozak/翻译起始位点构成,(c) F2包含具有3'-5方向性的至少一个短上游开放读码框(uORF),并且(d) F1由起始转录位点(TSS)和第一5'-UTR区构成。在脱靶整合事件之后,截短的SM1盒通常保留一个或多个uORF。这些uORF将减少在预期SM1起始密码子处的启动,从而改善基于脱靶整合的SM1激活和基于在预定基因组位置处的整合的SM1激活之间的区别。优选地,使用串联的多重uORF,并且置于与SM1起始密码子的最小距离处(内含子剪接分支位点的直接下游)。
断裂内含子设计的使用也改善了激活的SM1的表达,因为最佳的5'-UTR序列可以用于SM1。在缺乏断裂内含子的设计中,通过I1和I2的重组生成的序列(参见图15)将由SM15'-UTR包含。这导致具有潜在地非最佳序列组成的扩展的5'-UTR,其可以减少可获得的SM1表达水平(影响基于SM1的阳性选择步骤中的特异性)。经由使用如本文所述的断裂内含子,在预定基因组位置处的整合后,I1/I2重组产物变得掺入完全形成的内含子中(参见图15)。在通过细胞生成成熟的SM1 mRNA后,内含子被剪接掉,并且相应的SM1 5'-UTR完全由F1和F2限定。相应地,SM1 5'-UTR可以设计为具有完全控制,以对于预期目的优化SM1表达。F2-F3的设计中的变化进一步提供了在LP处的整合后的SM1表达水平中的灵活性。增加F3的5'-UTR区的长度减少SM1的表达,并且在F2中添加转录增强子元件可以增加SM1的表达高于仅用最佳5'-UTR可以达到的表达。
最后,断裂内含子设计的使用可以改善在预定基因组位置处的I1和I2之间的重组效率,如实验部分,实施例1中所示。对观察到的整合效率改善的一个潜在解释是在预定基因组位置处的断裂内含子的5'部分充当关键间隔区,其可以避免/减少在起始转录位点周围结合的RNA聚合酶起始复合物与通过I1的结合和操作来执行其功能的第一DNA酶(例如PhiC31)的拷贝之间的空间干扰。为了增加整合效率,断裂内含子的所述5'部分可以设计为具有至少50 bp、至少100 bp或至少300 bp的长度。
图12说明了一种方法,其中基因编辑酶用于催化供体载体在预定基因组位置的整合(第一DNA酶是基因编辑酶)。根据图1、图9-11和图15对其的修饰也由本公开内容包含。
在本实例中,预定基因组位置按5'-3'序列次序包含:(i)左同源臂(LHA),(ii)基因编辑酶的识别位点/切割位点(CS),(iii)右同源臂(RHA),其也充当具有3'-5'方向性的内含子的5’部分(即在最接近启动子的端部处具有剪接供体位点),(iv)具有3'-5'方向性的启动子P1,以及(v)第二DNA酶的识别位点E1。
在本实例中,供体载体按5'-3'序列次序包含:(i)所述左同源臂(LHA),(ii)整合盒(IC),此处通过目的基因(GOI)的表达盒进行例示,(iii)所述第二DNA酶的识别位点E2,(iv)第二选择标记物(SM2)的表达盒,(v)以3'-5'方向性编码的第一选择标记物(SM1)的基因,(vi)具有3'-5'方向性的内含子的3'部分(即在最接近SM1的端部处具有剪接分支位点和剪接受体位点),以及(vii)所述右同源臂(RHA),其也充当具有3'-5'方向性的内含子的5'部分。
将供体载体以及对于CS具有切割特异性的基因编辑酶引入真核细胞群体内导致:(a)对于一小部分的真核细胞,在预定基因组位置中的CS处的双链断裂,(b)对于在CS处具有双链断裂的一小部分的真核细胞,通过同源性指导的DNA修复,侧翼为LHA和RHA的供体载体区域的整合,(c)对于一小部分的LP细胞,供体载体的脱靶基因组整合(在预定基因组区域之外)。
在预定基因组位置处的整合导致形成SM1的活性表达盒(参见图12b,小图(ii))。由于整合事件进一步产生了在启动子P1和SM1之间的完全功能性的内含子,因此SM1的成熟mRNA并不包含RHA。进一步地,在LP处的整合之后,SM1和SM2两者的侧翼为所述第二DNA酶的两个识别位点。
通过脱靶事件的整合(参见图12b,小图(iii))通常并不导致SM1的激活,但的确整合了侧翼并非所述第二DNA酶的两个识别位点的活性SM2。
已经历了在预定基因组位置处的整合的真核细胞不同于没有整合事件的细胞(参见图12b,小图(i))、以及仅已经历了通过SM1活性的脱靶整合事件的细胞。因此,SM1的活性可以用于选择已经历了在预定基因组位置处的整合的细胞。
为了去除除在预定基因组位置处的整合之外已经历了脱靶整合事件的细胞,将能够重组E1和E2的重组酶活性(第二DNA酶)引入对于SM1活性进行选择的细胞内。对于在LP处的整合,这导致SM1和SM2两者的切除,以及因此其相应活性的切除。对于脱靶整合事件,这种反应无法发生,并且SM2活性保留。结果,可以从通过SM2活性的不存在已经历了多重整合事件的细胞中选择仅已经历了在预定基因组位置处的所需靶向整合事件的细胞。
最终选择的细胞的预定基因组位置(参见图12c)并不含有SM2的表达盒,也不含有SM1的激活表达盒或来自供体载体的任何残留序列,除了通过E1和E2 (E)的重组产生的序列之外。
基因编辑酶可以选自(i)锌指核酸酶(ZFN);归巢核酸内切酶,例如大范围核酸酶;(iii) TALEN或(iv) DNA或RNA引导的核酸酶,例如CRISPR/Cas9,但它并不限于此。
所述第二DNA酶具有重组酶活性,并且可以选自(i)丝氨酸重组酶或(ii)酪氨酸重组酶。
E1和E2可以在序列上等同,如通过以下例示的:(i) E1=E2=
E1和E2可以具有不同的序列,如通过以下例示的:(i) E1=
相应地,本文提供了一种方法,其中所述第一DNA酶是基因编辑酶,例如基因编辑核酸酶。因此,提供了一种方法,其中:(a) I1包含所述基因编辑核酸酶的切割位点以及两个序列区域LHA1和RHA1;并且(b) I2包含与LHA1和LHA2同源的两个序列区域LHA2和RHA2;并且(c) I1和I2能够在所述第一DNA酶的存在下重组。
如先前提到的,提供了一种方法,其中所述基因编辑酶选自(i)锌指核酸酶(ZFN);(ii)归巢核酸内切酶,例如大范围核酸酶;(iii) TALENS和(iv) DNA或RNA引导的核酸酶,例如CRISPR/Cas 9,但本公开内容不限于此。
核酸序列E1和E2可以分别是等同的重组酶识别位点,例如
所述第二DNA酶可以选自Cre重组酶、Dre重组酶和FLP重组酶,条件是第一DNA酶不是Cre重组酶、Dre重组酶或FLP重组酶。
在本文提供的方法中,当在所述预定基因组位置处整合时,启动子核酸序列P1和/或P2可以与断裂内含子的5'部分在功能上融合。这在本文先前讨论的图15中进行说明。在预定基因组位置中的启动子P1 (或P2)和I1 (或其变体)之间引入断裂内含子提供了“间隔区”,其使可能由于从聚合酶到启动子的阻断而发生的空间位阻降到最低。如实验部分,实施例1中所示,该间隔区的存在提供了第一选择标记物(SM1)的改善表达。
相应地,本文提供了一种方法,其中所述预定基因组位置进一步包含具有3'-5'方向性的内含子的5'部分,以及在所述第一重组酶的所述第一识别位点和具有3'-5'方向性的所述启动子P1之间、具有3'-5'方向性的功能序列区域F1,并且其中所述供体载体进一步包含定位于具有3'-5'方向性的所述第一选择标记物SM1和所述第一重组酶的所述第二识别位点之间的序列区域。所述序列区域按5'-3'序列次序包含:(a)具有3'-5'方向性的功能序列区域F3和(b)具有3'-5'方向性的内含子的3'部分,其进一步包含在剪接受体位点下游的功能序列区域F2。
本文还提供了一种方法,其中如本文先前公开的,所述切除的核酸序列包含:
(a)编码第一选择标记物的核酸序列;
(b)启动子核酸序列P1或P2;和/或
(c)编码第二选择标记物的表达盒。
上文提到的切除的核酸序列的设计提供了基于第二选择标记物(SM2)的表达,选择并未在除预定基因组位置外的其它位置中随机整合了供体载体的细胞。这意指使用此类设计,在所述第二DNA酶的作用之后,SM2的表达仅对于已在预定基因组位置之外整合了供体载体的细胞是阳性的。相应地,第二轮选择可以使用基于SM2表达的阴性选择步骤,以去除已在预定基因组位置之外整合了供体载体的细胞。编码第二选择标记物的表达盒的去除也是对该方法的改善,因为这将对于可以取而代之用于生产目的蛋白质的细胞节省能量。
除本文其它地方已提到的之外,第一选择标记物可以选自(i)荧光蛋白和(ii)异源细胞表面标记物。荧光蛋白或细胞表面标记物作为选择标记物的使用提供了特别的优点,因为只要第一选择标记物的浓度已增加高于一定限度(允许在FACS中检测到高于背景的荧光,并允许在MACS中与磁珠的有效结合),就可以使用快速和直接的分离方法(即,基于FACS或MACS)执行选择。这与基于代谢酶或抗生素抗性基因的选择标记物形成对比,其需要基于具有活化选择标记物的细胞缓慢地生长超过缺乏活性选择标记物的细胞的长期和间接的分离策略。
第一DNA酶可以以质粒、mRNA或纯化蛋白质的形式提供,任选地其中所述第一DNA酶可以由所述供体载体编码并由其表达。第一DNA酶也可以由编码所述第一DNA酶的表达盒表达,所述表达盒存在于本文公开的方法的步骤i)的细胞的预定基因组位置中。
如本文先前提到的,步骤ii)的供体载体可以进一步包含编码第二DNA酶的表达盒,当所述供体载体已整合到本文公开的方法中的步骤i)的细胞的预定基因组位置内时,所述第二DNA酶的表达得到激活。
第二DNA酶也可以以质粒、mRNA或纯化蛋白质的形式提供。
用于本文呈现的方法中的真核细胞可以选自酵母细胞、丝状真菌细胞、植物细胞、昆虫细胞或哺乳动物细胞。哺乳动物细胞可以是人、猴、啮齿类动物或小鼠细胞,但不限于此。真核细胞是如本文先前提到的分离的真核细胞。分离的细胞是已从其自然环境中分离或取出的细胞。
可以基于在生物反应器中用于生产重组蛋白质的适合性,具体选择用于本文呈现的方法中的真核细胞。合适的细胞可以选自CHO或HEK细胞系。
可以基于与哺乳动物物种例如人中存在的细胞类型的相似性,具体选择用于本文呈现的方法中的真核细胞。
用于本文呈现的方法中的真核细胞可以选自能够在悬浮培养中生长的细胞系。
在另一个方面,还提供了可通过如本文公开的方法获得的分离的真核细胞。可通过本文公开的方法获得的分离的真核细胞含有整合到预定基因组位置内的一种或多种目的核酸序列。优选地,所获得的分离细胞并不含有已在宿主细胞基因组中的其它位置处而不是在预定基因组位置内整合的任何供体载体。
在再一个方面,还提供了可通过本文公开的方法获得的分离的真核细胞用于生产重组蛋白质的用途。
因此,在另一个方面,还提供了用于生产重组蛋白质的方法,所述方法包括:
i)通过执行如本文公开的方法,获得包含在预定基因组位置处整合的一种或多种目的核酸序列的分离的真核细胞,其中至少一种目的核酸序列包含至少一个表达盒,所述表达盒包含编码目的蛋白质的基因;
ii)在步骤i)的所述细胞中,产生由目的基因编码的蛋白质;和
iii)分离步骤ii)的蛋白质。
在一个进一步方面,提供了供体载体,其包括:
a. 包含第一DNA酶的识别位点的核酸序列;
b. 目的核酸序列;
c. 包含第二DNA酶的识别位点的核酸序列;
在再一个进一步的方面,提供了包含预定基因组位置的分离的真核细胞,所述预定位置包括:
a. 包含第一DNA酶的识别位点的核酸序列I1;
b. 包含第二DNA酶的识别位点的核酸序列E1;和
c. 启动子核酸序列P1;
d. 编码第一选择标记物的核酸序列;
e. 任选地编码第二选择标记物的表达盒;
在再一个进一步的方面,提供了用于将目的核酸序列靶向整合到宿主细胞内的重组表达系统,所述表达系统包括:
供体载体,其包括:
a. 包含第一DNA酶的识别位点的核酸序列;
b. 目的核酸序列;
c. 包含第二DNA酶的识别位点的核酸序列;和
包含预定基因组位置的分离的真核细胞,所述预定位置包括:
a. 包含第一DNA酶的识别位点的核酸序列I1;
b. 包含第二DNA酶的识别位点的核酸序列E1;和
c. 启动子核酸序列P1;
d. 编码第一选择标记物的核酸序列;
e. 任选地编码第二选择标记物的表达盒;
本公开内容现在将通过下述实验部分进行说明,而不限于其中提供的实例。实验部分仅仅说明了执行本发明的不同方式。
LP = 着陆垫
LP1P1 = 包含attP1的着陆垫1
LP2P2 = 包含attP2和断裂内含子的着陆垫2
CHO = 中国仓鼠卵巢
FC-eGFP = 与来自IgG1的FC融合的增强型绿色荧光蛋白
TagBFP2 = 蓝色荧光蛋白变体
TagRFP-T = 红色荧光蛋白变体
G418 = 也称为遗传霉素,一种广谱抗生素,其选择表达新霉素抗性基因(NeoR)的哺乳动物细胞。
下文序列用于实验部分中,但本公开内容并不限于这些序列。相应地,还设想了其变体,其中所述序列变体的功能保持与原始序列基本上相同。
GTGCCCCAACTGGGGTAACCTTTGAGTTCTCTCAGTTGGGGGCGTAG
CTCGAAGCCGCGGTGCGGGTGCCAGGGCGTGCCCTTGGGCTCCCCGGGCGCGTACTCCACCTCACCCATC
GTGCCCCAACTGGGGTAACCTAAGAGTTCTCTCAGTTGGGGGCGTAG
CTCGAAGCCGCGGTGCGGGTGCCAGGGCGTGCCCAAGGGCTCCCCGGGCGCGTACTCCACCTCACCCATC
ATGACCATGATTACCCCATCTGCCCAGCTGACCCTGACAAAGGGCAATAAGAGCTGGTCTAGCCTGGTGACAGCTGCTTCTGTGCTGGAGTTTGCCACCATGATCCAAGGGGTCGCTGGGGAAGTGACTTATGCCGGGGCGTACGACCGTCAGTCTCGGGAGCGCGAGAACTCTAGCGCGGCGTCTCCGGCCACTCAGCGTAGCGCTAACGAGGCCAAAGCCGCCGCTCTCCAGCGCGAGATCGAGCGCGCCGGGGGCCGGTTTCGTTTCGTCGGTCACTTCAGCGAGGCCCCCGGCACATCTGCCTTCGGTACAGCCGAGCGCCCTGAGTTCGAACGCATTCTGAACGAATGCCGCGCCGGTCGGCTGAACATGATTATCGTGTATGACGTGTCTCGCTTCTCTCGCCTGAAGGTTATGGACGCCATCCCTATCGTGTCAGAATTACTGGCCCTGGGCGTGACAATCGTCTCTACGCAGGAAGGCGTGTTCAGACAAGGGAACGTTATGGACCTGATCCACCTGATCATGCGGCTGGACGCCTCTCACAAAGAAAGCTCTCTGAAGTCTGCCAAGATCCTGGACACAAAGAACCTCCAGCGCGAACTTGGCGGTTACGTGGGCGGGAAGGCCCCCTACGGCTTCGAGCTTGTCAGCGAGACAAAGGAGATTACACGCAACGGACGTATGGTCAATGTGGTTATCAACAAGCTCGCCCACTCTACCACGCCTCTCACCGGACCTTTCGAGTTCGAGCCAGACGTAATTCGGTGGTGGTGGCGTGAGATCAAGACACACAAACACCTCCCTTTCAAGCCTGGCAGTCAAGCCGCCATCCACCCTGGCTCTATTACCGGACTCTGTAAGCGCATGGACGCGGACGCCGTGCCTACCAGAGGCGAGACAATCGGGAAGAAGACCGCGTCGTCTGCCTGGGACCCTGCGACCGTCATGCGTATTCTCAGAGACCCTCGTATCGCCGGGTTCGCTGCGGAGGTGATTTACAAGAAGAAGCCAGACGGCACACCTACCACAAAGATCGAGGGATACCGCATCCAGCGCGACCCTATTACTCTGCGGCCTGTGGAGCTTGATTGCGGTCCTATTATCGAGCCTGCGGAGTGGTATGAGCTTCAGGCCTGGTTGGACGGACGTGGTCGCGGCAAGGGTCTCTCTCGGGGTCAAGCCATCCTGTCTGCTATGGACAAGCTGTACTGCGAGTGTGGCGCCGTTATGACGAGCAAGCGCGGGGAAGAATCTATCAAGGACAGTTACCGCTGCCGTCGCAGAAAGGTGGTGGACCCTTCTGCGCCCGGTCAGCACGAAGGCACTTGCAACGTCTCTATGGCCGCGCTGGACAAGTTCGTCGCCGAACGCATTTTCAACAAGATCCGTCACGCCGAAGGCGACGAAGAGACACTTGCCCTCCTGTGGGAAGCCGCCCGTCGCTTCGGCAAGCTCACGGAGGCCCCCGAGAAGTCTGGCGAAAGAGCCAACCTCGTCGCCGAGCGCGCCGACGCCCTGAACGCCCTCGAAGAGCTGTACGAAGACCGCGCTGCGGGCGCCTACGACGGTCCTGTCGGACGAAAGCACTTCAGAAAGCAACAGGCGGCCCTGACTCTGCGCCAGCAAGGTGCCGAAGAGAGACTCGCCGAACTCGAAGCCGCCGAAGCCCCAAAGCTCCCTCTCGACCAATGGTTCCCAGAAGACGCCGACGCGGACCCTACCGGCCCCAAGTCTTGGTGGGGTCGCGCCTCGGTAGACGACAAGCGCGTGTTCGTGGGTCTGTTCGTAGACAAGATTGTCGTTACAAAGTCTACGACAGGCCGTGGGCAGGGGACACCTATCGAGAAGCGCGCGTCTATTACTTGGGCCAAGCCTCCTACCGACGACGACGAAGACGACGCCCAGGACGGCACAGAAGACGTAGCTGCTTGATAA
ATAACTTCGTATAGGATACTTTATACGAAGTTAT
ATGTCAAACCTTCTCACCGTCCACCAAAACCTCCCCGCACTCCCCGTTGACGCCACCTCCGACGAGGTCAGAAAAAACCTCATGGACATGTTCCGGGACCGCCAGGCCTTTTCCGAACACACTTGGAAAATGCTTCTCAGCGTTTGCCGTAGTTGGGCCGCTTGGTGTAAACTCAACAACCGCAAGTGGTTCCCCGCCGAACCCGAGGACGTCCGCGATTACCTTCTGTATTTGCAAGCGCGAGGACTGGCCGTGAAAACCATCCAGCAACATCTGGGTCAGCTTAACATGTTGCACCGGAGGAGCGGCCTGCCACGGCCTAGCGACTCCAACGCGGTGTCCCTCGTGATGAGGAGAATCCGCAAGGAGAATGTGGACGCCGGAGAAAGAGCAAAGCAGGCCCTGGCCTTCGAGAGGACTGACTTCGACCAAGTCCGGTCGCTGATGGAGAACTCGGACCGATGTCAGGACATCAGGAACCTCGCATTCTCGGCATTGCCTACAACACCCTGCTGAGAATTGCAGAGATCGCCCGCATCCGCGTCAAGGACATTTCGAGAACCGACGGAGGGCGGATGCTGATTCACATCGGCAGGACTAAGACCCTCGTGTCAACCGCCGGAGTGGAAAAGGCCCTCAGCCTGGGAGTGACAAAGCTCGTGGAGCGCTGGATCTCCGTGTCGGGGGTGGCCGACGATCCGAACAATTACCTGTTCTGCCGGGTCCGCAAAAATGGGGTGGCCGCCCCGTCTGCTACAAGCCAGTTGTCCACTCGCGCCCTGGAAGGAATCTTCGAGGCCACGCACCGCCTGATCTATGGGGCAAAGGACGATTCCGGCCAGAGGTATCTCGCGTGGTCCGGTCACTCCGCGCGCGTGGGCGCGGCCCGGGACATGGCCCGGGCTGGAGTGTCCATCCCTGAAATCATGCAGGCCGGTGGATGGACCAACGTGAACATCGTGATGAACTACATTCGGAACCTGGACAGCGAAACTGGTGCTATGGTCCGCCTGCTGGAGGACGGAGATTGA
GACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAACTCCTGGGGGGACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCACGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTCTACACCCTGCCCCCATCCCGGGAGGAGATGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTACAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCACGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTAAAGGTTCCTCCAGTTCCGGCAGCTCCAGTTCCGGTATGAGTAAAGGAGAGGAACTCTTCACCGGAGTCGTCCCGATACTCGTCGAGCTAGACGGAGACGTCAACGGCCACAAATTCTCCGTCTCCGGCGAGGGGGAGGGGGACGCCACCTACGGAAAACTCACCCTTAAGTTTATTTGCACTACCGGAAAACTCCCCGTCCCTTGGCCAACCCTAGTCACCACGCTGACATACGGAGTCCAATGTTTCTCGCGGTATCCCGACCACATGAAGCAGCATGACTTTTTCAAATCCGCGATGCCTGAGGGCTACGTGCAGGAACGCACCATCTTCTTCAAGGACGACGGGAATTACAAGACTAGAGCCGAGGTCAAGTTTGAAGGAGACACCCTCGTGAATCGCATCGAGCTTAAGGGCATTGACTTCAAGGAGGACGGCAACATCCTGGGTCACAAGCTGGAGTACAACTACAACTCGCATAACGTCTACATCATGGCCGACAAGCAAAAGAACGGTATCAAGGTCAACTTCAAGATTAGGCACAACATTGAGGATGGGTCCGTCCAACTGGCCGACCACTACCAGCAGAACACCCCCATCGGCGACGGACCTGTGCTCCTGCCTGATAACCACTATCTCAGCACTCAGAGCGCACTGTCCAAGGACCCTAACGAAAAACGGGACCACATGGTCTTGCTGGAGTTCGTGACAGCCGCTGGTATTACCCTGGGCATGGATGAACTGTATAAG
GACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAACTCCTGGGGGGACCGTCAGTCTTCCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTGGACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCAAGACAAAGCCACGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCAGGACTGGCTGAATGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAAACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTCTACACCCTGCCCCCATCCCGGGAGGAGATGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTGGGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTCTTCCTCTACAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGATGCACGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTAAAGGTTCCTCCAGTTCCGGCAGCTCCAGTTCCGGTATGGTGTCGAAGGGAGAGGAGCTGATTAAGGAGAACATGCACATGAAGCTGTATATGGAAGGGACGGTGGACAACCACCACTTCAAGTGCACCAGCGAAGGAGAAGGAAAGCCTTACGAAGGCACTCAAACTATGCGGATCAAAGTGGTGGAAGGCGGTCCTCTTCCGTTCGCCTTCGACATCTTGGCCACCTCCTTCCTCTACGGCTCCAAGACCTTTATCAACCACACCCAGGGAATCCCGGACTTCTTTAAGCAGAGCTTCCCTGAGGGCTTCACCTGGGAAAGAGTGACAACCTACGAGGACGGTGGCGTCCTGACCGCGACCCAGGACACCTCCCTGCAAGACGGCTGCCTGATCTACAACGTCAAGATTCGCGGCGTGAACTTCACCTCCAATGGTCCAGTGATGCAGAAGAAAACTCTGGGATGGGAGGCCTTCACTGAAACTCTGTACCCCGCCGATGGAGGACTGGAGGGGAGGAACGATATGGCTTTGAAGCTCGTGGGGGGATCGCACCTGATTGCGAATGCCAAGACCACCTACAGATCCAAGAAACCCGCCAAGAACCTCAAGATGCCCGGAGTCTACTACGTGGACTATAGACTGGAACGGATCAAGGAAGCCAACAACGAGACTTACGTGGAACAGCACGAGGTCGCTGTGGCACGCTACTGTGATCTGCCGTCAAAGCTCGGGCATAAGCTCAACTGATAA
ATGGTGTCAAAGGGAGAGGAACTGATTAAGGAGAATATGCACATGAAACTCTACATGGAGGGGACCGTGAACAACCACCACTTCAAGTGCACCTCCGAGGGCGAAGGGAAGCCGTACGAGGGAACTCAGACCATGCGGATTAAGGTCGTCGAAGGGGGTCCTCTGCCATTCGCCTTCGACATCCTCGCCACATCCTTTATGTACGGATCGCGGACCTTCATCAACCACACTCAGGGTATCCCCGACTTCTTCAAGCAATCGTTCCCGGAAGGCTTTACTTGGGAGCGCGTGACCACCTACGAGGATGGAGGGGTGCTGACGGCCACTCAGGACACCAGCCTGCAAGACGGCTGTCTTATCTACAACGTGAAGATTCGCGGCGTGAACTTCCCTAGCAACGGTCCGGTCATGCAGAAAAAGACCCTGGGTTGGGAGGCTAACACCGAAATGCTCTATCCTGCGGACGGAGGATTGGAAGGCCGGACTGACATGGCCCTGAAACTTGTGGGCGGCGGACATCTGATCTGCAATTTCAAGACCACTTACCGCTCCAAGAAGCCCGCCAAGAACCTGAAGATGCCTGGAGTGTACTACGTGGACCACAGACTCGAAAGGATCAAGGAGGCGGATAAGGAAACCTACGTGGAACAGCATGAAGTGGCAGTGGCCAGATACTGCGATCTGCCGTCCAAGCTCGGCCACAAGCTGAACGGAATGGACGAGCTGTATAAGTGATAA
ATGAGTAAAGGAGAGGAACTCTTCACCGGAGTCGTCCCGATACTCGTCGAGCTAGACGGAGACGTCAACGGCCACAAATTCTCCGTCTCCGGCGAGGGGGAGGGGGACGCCACCTACGGAAAACTCACCCTTAAGTTTATTTGCACTACCGGAAAACTCCCCGTCCCTTGGCCAACCCTAGTCACCACGCTGACATACGGAGTCCAATGTTTCTCGCGGTATCCCGACCACATGAAGCAGCATGACTTTTTCAAATCCGCGATGCCTGAGGGCTACGTGCAGGAACGCACCATCTTCTTCAAGGACGACGGGAATTACAAGACTAGAGCCGAGGTCAAGTTTGAAGGAGACACCCTCGTGAATCGCATCGAGCTTAAGGGCATTGACTTCAAGGAGGACGGCAACATCCTGGGTCACAAGCTGGAGTACAACTACAACTCGCATAACGTCTACATCATGGCCGACAAGCAAAAGAACGGTATCAAGGTCAACTTCAAGATTAGGCACAACATTGAGGATGGGTCCGTCCAACTGGCCGACCACTACCAGCAGAACACCCCCATCGGCGACGGACCTGTGCTCCTGCCTGATAACCACTATCTCAGCACTCAGAGCGCACTGTCCAAGGACCCTAACGAAAAACGGGACCACATGGTCTTGCTGGAGTTCGTGACAGCCGCTGGTATTACCCTGGGCATGGATGAACTGTATAAG
ATGGTGTCGAAGGGAGAGGAGCTGATTAAGGAGAACATGCACATGAAGCTGTATATGGAAGGGACGGTGGACAACCACCACTTCAAGTGCACCAGCGAAGGAGAAGGAAAGCCTTACGAAGGCACTCAAACTATGCGGATCAAAGTGGTGGAAGGCGGTCCTCTTCCGTTCGCCTTCGACATCTTGGCCACCTCCTTCCTCTACGGCTCCAAGACCTTTATCAACCACACCCAGGGAATCCCGGACTTCTTTAAGCAGAGCTTCCCTGAGGGCTTCACCTGGGAAAGAGTGACAACCTACGAGGACGGTGGCGTCCTGACCGCGACCCAGGACACCTCCCTGCAAGACGGCTGCCTGATCTACAACGTCAAGATTCGCGGCGTGAACTTCACCTCCAATGGTCCAGTGATGCAGAAGAAAACTCTGGGATGGGAGGCCTTCACTGAAACTCTGTACCCCGCCGATGGAGGACTGGAGGGGAGGAACGATATGGCTTTGAAGCTCGTGGGGGGATCGCACCTGATTGCGAATGCCAAGACCACCTACAGATCCAAGAAACCCGCCAAGAACCTCAAGATGCCCGGAGTCTACTACGTGGACTATAGACTGGAACGGATCAAGGAAGCCAACAACGAGACTTACGTGGAACAGCACGAGGTCGCTGTGGCACGCTACTGTGATCTGCCGTCAAAGCTCGGGCATAAGCTCAACTGATAA
ATGATTGAACAAGATGGATTGCACGCAGGTTCTCCGGCCGCTTGGGTGGAGAGGCTATTCGGCTATGACTGGGCACAACAGACAATCGGCTGCTCTGATGCCGCCGTGTTCCGGCTGTCAGCGCAGGGGCGCCCGGTTCTTTTTGTCAAGACCGACCTGTCCGGTGCCCTGAATGAACTGCAGGACGAGGCAGCGCGGCTATCGTGGCTGGCCACGACGGGCGTTCCTTGCGCAGCTGTGCTCGACGTTGTCACTGAAGCGGGAAGGGACTGGCTGCTATTGGGCGAAGTGCCGGGGCAGGATCTCCTGTCATCTCACCTTGCTCCTGCCGAGAAAGTATCCATCATGGCTGATGCAATGCGGCGGCTGCATACGCTTGATCCGGCTACCTGCCCATTCGACCACCAAGCGAAACATCGCATCGAGCGAGCACGTACTCGGATGGAAGCCGGTCTTGTCGATCAGGATGATCTGGACGAAGAGCATCAGGGGCTCGCGCCAGCCGAACTGTTCGCCAGGCTCAAGGCGCGCATGCCCGACGGCGAGGATCTCGTCGTGACCCATGGCGATGCCTGCTTGCCGAATATCATGGTGGAAAATGGCCGCTTTTCTGGATTCATCGACTGTGGCCGGCTGGGTGTGGCGGACCGCTATCAGGACATAGCGTTGGCTACCCGTGATATTGCTGAAGAGCTTGGCGGCGAATGGGCTGACCGCTTCCTCGTGCTTTACGGTATCGCCGCTCCCGATTCGCAGCGCATCGCCTTCTATCGCCTTCTTGACGAGTTCTTC
为了调查整合效率,HyClone CHO LP细胞和非LP HyClone CHO对照细胞用PhiC31重组酶表达质粒和供体载体A或B中的任一的组合进行转染(图2)。供体载体含有FC-eGFP和FC-TagBFP2的表达盒以及无启动子的TagRFP-T基因,其这样定位使得它在LP细胞中的LP处整合后激活。
调查了两种HyClone-CHO LP变体和匹配的供体载体,参见图2。在HyClone-CHOLP1P1中,LP中的启动子直接置于
在图3中,所生成的流式细胞术数据的实例对于HyClone CHO LP2P2与对照相比较示出。结果的完整集合概括于表1中。对于LP1P1,仅使用未转染的对照,而对于LP2P2,执行随机整合对照(RI对照,仅供体载体)和假att整合对照(在缺乏LP的CHO细胞系中的供体载体 + PhiC31)。根据数据,两种LP变体均是有功能的,但利用断裂内含子设计的LP2P2变体给出了优异的整合效率。
HyClone CHO LP1P1细胞使用PhiC31表达质粒和供体载体进行转染,所述供体载体含有FC-eGFP和FC-TagBFP2的表达盒以及无启动子的TagRFP-T基因,其这样定位使得它在LP细胞中的LP处整合后激活(图4)。通过对于高于背景的Tag-RFP-T信号进行门控的几个FACS分选步骤、以及FC-eGFP和FC-TagBFP2两者的平衡表达,来富集已在着陆垫(LP)处整合了供体载体的细胞。然后使用(a) Cre重组酶表达质粒、(b)编码Cre的合成mRNA、或(c)缺乏任何Cre重组酶编码核酸分子的模拟转染溶液,对所得到的分选且扩增的细胞池进行第二次转染。第二次转染后7天,通过流式细胞术分析所有细胞群体,以评估侧翼为两个
来自在Cre重组酶转染之后的流式细胞术分析的图可以在图5中看到。数据显示了,与模拟对照相比,对于两个Cre重组酶处理的池,并不表达FC-TagBFP2的细胞的增加。这依次又清楚地指示了供体载体在LP处的正确整合,使得FC-TagBFP2的侧翼为Cre重组酶可以作用于其的两个
HyClone CHO LP1P1细胞使用PhiC31表达质粒和供体载体进行转染,所述供体载体含有
为了验证改变的着陆垫的功能性,在最终分选(图7,步骤2分选)后获得的eGFP阴性池使用DNA供体载体B进行转染(图6,步骤3),并且在转染后7天通过流式细胞术进行分析(图7,下图)。数据指示了新着陆垫的功能性。最后,来自eGFP阴性池的细胞通过FACS使用单细胞分选进行克隆,并且通过PCR扩增其基因组的着陆垫区域并测序。通过测序(新着陆垫区域的完全覆盖)对于多重克隆确认了着陆垫的正确改变,显示了图7中概述的改变已成功地实现。
HyClone CHO LP2P2细胞(根据实施例3生成的克隆)使用PhiC31重组酶表达质粒和根据图8构建的供体载体进行转染。
在转染后两天开始,在G418的存在下培养细胞,以选择已在着陆垫处整合了供体载体并且从而激活新霉素抗性基因(Neo
来自FACS/流式细胞术的数据可以在图9中看到。在Cre重组酶转染之后的另外选择步骤减少了池中的异质性,如通过TagRFP-T阳性(不正确整合)和TagRFP-T阴性(正确整合)细胞的eGFP信号的平均值和CV指示的。
参考文献
[1] Kunert, R等人; Advances in recombinant antibody manufacturing;Appl Microbiol Biotechnol (2016) 100:3451–3461.
[2] Ecker, DM等人; The therapeutic monoclonal antibody market; mAbs7:1, 9-14; January/February 2015.
[3] Meinke, G等人; Cre Recombinase and Other Tyrosine Recombinases;Chem. Rev. 2016, 116, 12785−12820.
[4] Merrick, CA等人; Serine Integrases: Advancing Synthetic Biology;ACS Synth. Biol. 2018, 7, 299−310.
[5] Xu, Z等人; Accuracy and efficiency define Bxb1 integrase as thebest of fifteen candidate serine recombinases for the integration of DNA intothe human genome; BMC Biotechnology 2013, 13:87.
[6] Lee, JS等人; Accelerated Homology-Directed Targeted Integrationof Transgenes in Chinese Hamster Ovary Cells Via CRISPR/Cas9 and FluorescentEnrichment; Biotechnol. Bioeng. 2016;9999: 1–6.
[7] Invitrogen; Flp-In system for generating stable mammalianexpression cell lines by Flp recombinase-mediated integration; InvitrogenInstruction Manual 2001; Invitrogen, Carlsbad CA.
[8] Muller, D; Accelerating Time to Clinical Manufacturing FollowingaTargeted Gene Integration Approach; Bioprocess International Conference,Boston; Oct 28, 2015.
[9] Haghighat-Khah, RE等人; Site-Specific Cassette Exchange Systemsin the Aedes aegypti Mosquito and the Plutella xylostella Moth; PLOS ONE |DOI:10.1371/journal.pone.0121097 April 1, 2015.
[10] Yuan, Y等人; Improved site-specific recombinase-based method toproduce selectable marker- and vector-backbone-free transgenic cells;SCIENTIFIC REPORTS | 4 : 4240 | DOI: 10.1038/srep04240。
序列表
<110> GE HEALTHCARE BIO-SCIENCES AB
<120> 用于靶向整合的方法
<130> 600572-GB-1
<160> 13
<170> PatentIn version 3.5
<210> 1
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然attP序列
<400> 1
gtgccccaac tggggtaacc tttgagttct ctcagttggg ggcgtag 47
<210> 2
<211> 70
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然attB序列
<400> 2
ctcgaagccg cggtgcgggt gccagggcgt gcccttgggc tccccgggcg cgtactccac 60
ctcacccatc 70
<210> 3
<211> 47
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然attP序列
<400> 3
gtgccccaac tggggtaacc taagagttct ctcagttggg ggcgtag 47
<210> 4
<211> 70
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然attB序列
<400> 4
ctcgaagccg cggtgcgggt gccagggcgt gcccaagggc tccccgggcg cgtactccac 60
ctcacccatc 70
<210> 5
<211> 1941
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然PhiC31基因
<400> 5
atgaccatga ttaccccatc tgcccagctg accctgacaa agggcaataa gagctggtct 60
agcctggtga cagctgcttc tgtgctggag tttgccacca tgatccaagg ggtcgctggg 120
gaagtgactt atgccggggc gtacgaccgt cagtctcggg agcgcgagaa ctctagcgcg 180
gcgtctccgg ccactcagcg tagcgctaac gaggccaaag ccgccgctct ccagcgcgag 240
atcgagcgcg ccgggggccg gtttcgtttc gtcggtcact tcagcgaggc ccccggcaca 300
tctgccttcg gtacagccga gcgccctgag ttcgaacgca ttctgaacga atgccgcgcc 360
ggtcggctga acatgattat cgtgtatgac gtgtctcgct tctctcgcct gaaggttatg 420
gacgccatcc ctatcgtgtc agaattactg gccctgggcg tgacaatcgt ctctacgcag 480
gaaggcgtgt tcagacaagg gaacgttatg gacctgatcc acctgatcat gcggctggac 540
gcctctcaca aagaaagctc tctgaagtct gccaagatcc tggacacaaa gaacctccag 600
cgcgaacttg gcggttacgt gggcgggaag gccccctacg gcttcgagct tgtcagcgag 660
acaaaggaga ttacacgcaa cggacgtatg gtcaatgtgg ttatcaacaa gctcgcccac 720
tctaccacgc ctctcaccgg acctttcgag ttcgagccag acgtaattcg gtggtggtgg 780
cgtgagatca agacacacaa acacctccct ttcaagcctg gcagtcaagc cgccatccac 840
cctggctcta ttaccggact ctgtaagcgc atggacgcgg acgccgtgcc taccagaggc 900
gagacaatcg ggaagaagac cgcgtcgtct gcctgggacc ctgcgaccgt catgcgtatt 960
ctcagagacc ctcgtatcgc cgggttcgct gcggaggtga tttacaagaa gaagccagac 1020
ggcacaccta ccacaaagat cgagggatac cgcatccagc gcgaccctat tactctgcgg 1080
cctgtggagc ttgattgcgg tcctattatc gagcctgcgg agtggtatga gcttcaggcc 1140
tggttggacg gacgtggtcg cggcaagggt ctctctcggg gtcaagccat cctgtctgct 1200
atggacaagc tgtactgcga gtgtggcgcc gttatgacga gcaagcgcgg ggaagaatct 1260
atcaaggaca gttaccgctg ccgtcgcaga aaggtggtgg acccttctgc gcccggtcag 1320
cacgaaggca cttgcaacgt ctctatggcc gcgctggaca agttcgtcgc cgaacgcatt 1380
ttcaacaaga tccgtcacgc cgaaggcgac gaagagacac ttgccctcct gtgggaagcc 1440
gcccgtcgct tcggcaagct cacggaggcc cccgagaagt ctggcgaaag agccaacctc 1500
gtcgccgagc gcgccgacgc cctgaacgcc ctcgaagagc tgtacgaaga ccgcgctgcg 1560
ggcgcctacg acggtcctgt cggacgaaag cacttcagaa agcaacaggc ggccctgact 1620
ctgcgccagc aaggtgccga agagagactc gccgaactcg aagccgccga agccccaaag 1680
ctccctctcg accaatggtt cccagaagac gccgacgcgg accctaccgg ccccaagtct 1740
tggtggggtc gcgcctcggt agacgacaag cgcgtgttcg tgggtctgtt cgtagacaag 1800
attgtcgtta caaagtctac gacaggccgt gggcagggga cacctatcga gaagcgcgcg 1860
tctattactt gggccaagcc tcctaccgac gacgacgaag acgacgccca ggacggcaca 1920
gaagacgtag ctgcttgata a 1941
<210> 6
<211> 34
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然lox基因
<400> 6
ataacttcgt ataggatact ttatacgaag ttat 34
<210> 7
<211> 1031
<212> DNA
<213> 人工序列
<220>
<223> 部分修饰的天然Cre基因
<400> 7
atgtcaaacc ttctcaccgt ccaccaaaac ctccccgcac tccccgttga cgccacctcc 60
gacgaggtca gaaaaaacct catggacatg ttccgggacc gccaggcctt ttccgaacac 120
acttggaaaa tgcttctcag cgtttgccgt agttgggccg cttggtgtaa actcaacaac 180
cgcaagtggt tccccgccga acccgaggac gtccgcgatt accttctgta tttgcaagcg 240
cgaggactgg ccgtgaaaac catccagcaa catctgggtc agcttaacat gttgcaccgg 300
aggagcggcc tgccacggcc tagcgactcc aacgcggtgt ccctcgtgat gaggagaatc 360
cgcaaggaga atgtggacgc cggagaaaga gcaaagcagg ccctggcctt cgagaggact 420
gacttcgacc aagtccggtc gctgatggag aactcggacc gatgtcagga catcaggaac 480
ctcgcattct cggcattgcc tacaacaccc tgctgagaat tgcagagatc gcccgcatcc 540
gcgtcaagga catttcgaga accgacggag ggcggatgct gattcacatc ggcaggacta 600
agaccctcgt gtcaaccgcc ggagtggaaa aggccctcag cctgggagtg acaaagctcg 660
tggagcgctg gatctccgtg tcgggggtgg ccgacgatcc gaacaattac ctgttctgcc 720
gggtccgcaa aaatggggtg gccgccccgt ctgctacaag ccagttgtcc actcgcgccc 780
tggaaggaat cttcgaggcc acgcaccgcc tgatctatgg ggcaaaggac gattccggcc 840
agaggtatct cgcgtggtcc ggtcactccg cgcgcgtggg cgcggcccgg gacatggccc 900
gggctggagt gtccatccct gaaatcatgc aggccggtgg atggaccaac gtgaacatcg 960
tgatgaacta cattcggaac ctggacagcg aaactggtgc tatggtccgc ctgctggagg 1020
acggagattg a 1031
<210> 8
<211> 1428
<212> DNA
<213> 人工序列
<220>
<223> 与来自IgG1的FC融合的增强型绿色荧光蛋白(FC-eGFP基因)
<400> 8
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 60
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 120
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 180
ggcgtggagg tgcataatgc caagacaaag ccacgggagg agcagtacaa cagcacgtac 240
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 300
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 360
gggcagcccc gagaaccaca ggtctacacc ctgcccccat cccgggagga gatgaccaag 420
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 480
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 540
gacggctcct tcttcctcta cagcaagctc accgtggaca agagcaggtg gcagcagggg 600
aacgtcttct catgctccgt gatgcacgag gctctgcaca accactacac gcagaagagc 660
ctctccctgt ctccgggtaa aggttcctcc agttccggca gctccagttc cggtatgagt 720
aaaggagagg aactcttcac cggagtcgtc ccgatactcg tcgagctaga cggagacgtc 780
aacggccaca aattctccgt ctccggcgag ggggaggggg acgccaccta cggaaaactc 840
acccttaagt ttatttgcac taccggaaaa ctccccgtcc cttggccaac cctagtcacc 900
acgctgacat acggagtcca atgtttctcg cggtatcccg accacatgaa gcagcatgac 960
tttttcaaat ccgcgatgcc tgagggctac gtgcaggaac gcaccatctt cttcaaggac 1020
gacgggaatt acaagactag agccgaggtc aagtttgaag gagacaccct cgtgaatcgc 1080
atcgagctta agggcattga cttcaaggag gacggcaaca tcctgggtca caagctggag 1140
tacaactaca actcgcataa cgtctacatc atggccgaca agcaaaagaa cggtatcaag 1200
gtcaacttca agattaggca caacattgag gatgggtccg tccaactggc cgaccactac 1260
cagcagaaca cccccatcgg cgacggacct gtgctcctgc ctgataacca ctatctcagc 1320
actcagagcg cactgtccaa ggaccctaac gaaaaacggg accacatggt cttgctggag 1380
ttcgtgacag ccgctggtat taccctgggc atggatgaac tgtataag 1428
<210> 9
<211> 1431
<212> DNA
<213> 人工序列
<220>
<223> 与来自IgG1的FC融合的蓝色荧光蛋白变体(FC-TagBFP2基因)
<400> 9
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 60
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 120
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 180
ggcgtggagg tgcataatgc caagacaaag ccacgggagg agcagtacaa cagcacgtac 240
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 300
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 360
gggcagcccc gagaaccaca ggtctacacc ctgcccccat cccgggagga gatgaccaag 420
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 480
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 540
gacggctcct tcttcctcta cagcaagctc accgtggaca agagcaggtg gcagcagggg 600
aacgtcttct catgctccgt gatgcacgag gctctgcaca accactacac gcagaagagc 660
ctctccctgt ctccgggtaa aggttcctcc agttccggca gctccagttc cggtatggtg 720
tcgaagggag aggagctgat taaggagaac atgcacatga agctgtatat ggaagggacg 780
gtggacaacc accacttcaa gtgcaccagc gaaggagaag gaaagcctta cgaaggcact 840
caaactatgc ggatcaaagt ggtggaaggc ggtcctcttc cgttcgcctt cgacatcttg 900
gccacctcct tcctctacgg ctccaagacc tttatcaacc acacccaggg aatcccggac 960
ttctttaagc agagcttccc tgagggcttc acctgggaaa gagtgacaac ctacgaggac 1020
ggtggcgtcc tgaccgcgac ccaggacacc tccctgcaag acggctgcct gatctacaac 1080
gtcaagattc gcggcgtgaa cttcacctcc aatggtccag tgatgcagaa gaaaactctg 1140
ggatgggagg ccttcactga aactctgtac cccgccgatg gaggactgga ggggaggaac 1200
gatatggctt tgaagctcgt ggggggatcg cacctgattg cgaatgccaa gaccacctac 1260
agatccaaga aacccgccaa gaacctcaag atgcccggag tctactacgt ggactataga 1320
ctggaacgga tcaaggaagc caacaacgag acttacgtgg aacagcacga ggtcgctgtg 1380
gcacgctact gtgatctgcc gtcaaagctc gggcataagc tcaactgata a 1431
<210> 10
<211> 738
<212> DNA
<213> 人工序列
<220>
<223> 红色荧光蛋白变体(TagRFP-T基因)
<400> 10
atggtgtcaa agggagagga actgattaag gagaatatgc acatgaaact ctacatggag 60
gggaccgtga acaaccacca cttcaagtgc acctccgagg gcgaagggaa gccgtacgag 120
ggaactcaga ccatgcggat taaggtcgtc gaagggggtc ctctgccatt cgccttcgac 180
atcctcgcca catcctttat gtacggatcg cggaccttca tcaaccacac tcagggtatc 240
cccgacttct tcaagcaatc gttcccggaa ggctttactt gggagcgcgt gaccacctac 300
gaggatggag gggtgctgac ggccactcag gacaccagcc tgcaagacgg ctgtcttatc 360
tacaacgtga agattcgcgg cgtgaacttc cctagcaacg gtccggtcat gcagaaaaag 420
accctgggtt gggaggctaa caccgaaatg ctctatcctg cggacggagg attggaaggc 480
cggactgaca tggccctgaa acttgtgggc ggcggacatc tgatctgcaa tttcaagacc 540
acttaccgct ccaagaagcc cgccaagaac ctgaagatgc ctggagtgta ctacgtggac 600
cacagactcg aaaggatcaa ggaggcggat aaggaaacct acgtggaaca gcatgaagtg 660
gcagtggcca gatactgcga tctgccgtcc aagctcggcc acaagctgaa cggaatggac 720
gagctgtata agtgataa 738
<210> 11
<211> 714
<212> DNA
<213> 人工序列
<220>
<223> 增强型绿色荧光蛋白(eGFP基因)
<400> 11
atgagtaaag gagaggaact cttcaccgga gtcgtcccga tactcgtcga gctagacgga 60
gacgtcaacg gccacaaatt ctccgtctcc ggcgaggggg agggggacgc cacctacgga 120
aaactcaccc ttaagtttat ttgcactacc ggaaaactcc ccgtcccttg gccaacccta 180
gtcaccacgc tgacatacgg agtccaatgt ttctcgcggt atcccgacca catgaagcag 240
catgactttt tcaaatccgc gatgcctgag ggctacgtgc aggaacgcac catcttcttc 300
aaggacgacg ggaattacaa gactagagcc gaggtcaagt ttgaaggaga caccctcgtg 360
aatcgcatcg agcttaaggg cattgacttc aaggaggacg gcaacatcct gggtcacaag 420
ctggagtaca actacaactc gcataacgtc tacatcatgg ccgacaagca aaagaacggt 480
atcaaggtca acttcaagat taggcacaac attgaggatg ggtccgtcca actggccgac 540
cactaccagc agaacacccc catcggcgac ggacctgtgc tcctgcctga taaccactat 600
ctcagcactc agagcgcact gtccaaggac cctaacgaaa aacgggacca catggtcttg 660
ctggagttcg tgacagccgc tggtattacc ctgggcatgg atgaactgta taag 714
<210> 12
<211> 717
<212> DNA
<213> 人工序列
<220>
<223> 蓝色荧光蛋白变体(TagBFP2基因)
<400> 12
atggtgtcga agggagagga gctgattaag gagaacatgc acatgaagct gtatatggaa 60
gggacggtgg acaaccacca cttcaagtgc accagcgaag gagaaggaaa gccttacgaa 120
ggcactcaaa ctatgcggat caaagtggtg gaaggcggtc ctcttccgtt cgccttcgac 180
atcttggcca cctccttcct ctacggctcc aagaccttta tcaaccacac ccagggaatc 240
ccggacttct ttaagcagag cttccctgag ggcttcacct gggaaagagt gacaacctac 300
gaggacggtg gcgtcctgac cgcgacccag gacacctccc tgcaagacgg ctgcctgatc 360
tacaacgtca agattcgcgg cgtgaacttc acctccaatg gtccagtgat gcagaagaaa 420
actctgggat gggaggcctt cactgaaact ctgtaccccg ccgatggagg actggagggg 480
aggaacgata tggctttgaa gctcgtgggg ggatcgcacc tgattgcgaa tgccaagacc 540
acctacagat ccaagaaacc cgccaagaac ctcaagatgc ccggagtcta ctacgtggac 600
tatagactgg aacggatcaa ggaagccaac aacgagactt acgtggaaca gcacgaggtc 660
gctgtggcac gctactgtga tctgccgtca aagctcgggc ataagctcaa ctgataa 717
<210> 13
<211> 792
<212> DNA
<213> 人工序列
<220>
<223> 新霉素抗性基因(NeoR基因)
<400> 13
atgattgaac aagatggatt gcacgcaggt tctccggccg cttgggtgga gaggctattc 60
ggctatgact gggcacaaca gacaatcggc tgctctgatg ccgccgtgtt ccggctgtca 120
gcgcaggggc gcccggttct ttttgtcaag accgacctgt ccggtgccct gaatgaactg 180
caggacgagg cagcgcggct atcgtggctg gccacgacgg gcgttccttg cgcagctgtg 240
ctcgacgttg tcactgaagc gggaagggac tggctgctat tgggcgaagt gccggggcag 300
gatctcctgt catctcacct tgctcctgcc gagaaagtat ccatcatggc tgatgcaatg 360
cggcggctgc atacgcttga tccggctacc tgcccattcg accaccaagc gaaacatcgc 420
atcgagcgag cacgtactcg gatggaagcc ggtcttgtcg atcaggatga tctggacgaa 480
gagcatcagg ggctcgcgcc agccgaactg ttcgccaggc tcaaggcgcg catgcccgac 540
ggcgaggatc tcgtcgtgac ccatggcgat gcctgcttgc cgaatatcat ggtggaaaat 600
ggccgctttt ctggattcat cgactgtggc cggctgggtg tggcggaccg ctatcaggac 660
atagcgttgg ctacccgtga tattgctgaa gagcttggcg gcgaatgggc tgaccgcttc 720
ctcgtgcttt acggtatcgc cgctcccgat tcgcagcgca tcgccttcta tcgccttctt 780
gacgagttct tc 792
- 用于靶向病原体基因RNA的gRNA及基于C2c2的病原体基因的检测方法、检测试剂盒
- 用于靶向单链切割和靶向整合的方法和组合物
- 用于靶向单链切割和靶向整合的方法和组合物
