一种植物GS3基因或蛋白在调控植物耐盐碱中的应用
文献发布时间:2023-06-19 19:38:38
技术领域
本发明属于植物基因工程技术领域,具体涉及一种植物GS3基因或蛋白在调控植物耐盐碱中的应用。
背景技术
气候干旱、地下水位提高、地势低洼没有排水出路,这些都是土地盐碱化的原因。同时由于大量施用化肥可能会造成在不远的未来使50%的良田变为盐碱地,严重威胁粮食生产。由于普通作物很难在盐碱地上成活,因此盐碱地难以成为作物种植的耕地,除非经过非常复杂繁琐的去盐碱化处理。
实际上,植物耐盐和耐碱是两个不同的性状,根据FAO在2015年的统计,在调查的大于100万公顷盐碱地中,有60%实际可定义为由Na
虽然现在克隆到的耐盐基因很多,但是具有耐碱功能的基因相对来讲还比较匮乏,仅有野生大豆中的GsCHX19、GsPPCK等基因报道有抗碱功能(杨浩,朱延明.耐碱GsCHX19基因的克隆及对苜蓿的遗传转化[J].作物杂志,2016,(3):37-44;王家佳.GsPPCK基因对苜蓿的遗传转化及转基因株系的耐碱性分析[D].塔里木大学,2014年)因此,为了进一步拓宽培育耐碱作物所需的基因资源,更多的与耐碱有关的基因亟待鉴定。
水稻基因GS3为粒长负调控基因,对粒长、粒厚和长宽比有显著影响,常常作为鉴定水稻粒形标记开发的靶基因,目前还未有关于水稻基因GS3在耐碱相关功能的报道。
发明内容
有鉴于此,本发明的目的在于提供一种植物GS3基因或蛋白在调控植物耐盐碱中的应用,植物GS3基因或蛋白通过负调控达到植物抗碱性的目的。
本发明提供了一种植物GS3基因或蛋白在调控植物耐盐碱中的应用。
优选的,植物GS3蛋白的氨基酸序列如SEQ ID NO:1~SEQ ID NO:3中至少一项所示。
优选的,植物GS3基因的核苷酸序列如SEQ ID NO:4~SEQ ID NO:8中至少一项所示。
优选的,所述植物GS3基因或蛋白负调控植物耐盐碱。
本发明提供了一种提高植物耐碱性的方法,敲除或抑制植物中GS3基因或蛋白表达或抑制植物中GS3蛋白活性。
优选的,所述敲除或抑制植物中GS3基因或蛋白表达或抑制植物中GS3蛋白活性的方法包括基因编辑、RNA干扰和T-DNA插入中至少一种。
优选的,所述基因编辑的方法为利用CRISPR/Cas9技术敲除植物基因组GS3基因中如SEQ ID NO:9~SEQ ID NO:11任意一个所示的靶标区域。
本发明提供了一种提高植物耐碱性的试剂盒,包括至少以下分子中的一种:
(1)识别所述应用中植物GS3基因的RNA分子;所述RNA分子的序列如SEQ ID NO:12~SEQ ID NO:14中任意一个所示;
(2)编码(1)中所述RNA分子的DNA分子;
(3)表达(2)中所述DNA分子的载体。
本发明提供了所述试剂盒在提高植物耐盐碱或制备耐盐碱转基因植物中的应用。
本发明提供了一种植物突变基因或蛋白在提高植物耐盐碱中的应用,所述植物突变基因序列为SEQ ID NO:15或SEQ ID NO:16所示;所述植物突变蛋白的氨基酸序列如SEQID NO:17或SEQ ID NO:18所示。
本发明提供了一种植物GS3基因或蛋白在调控植物耐盐碱中的应用。本发明首次证实,水稻粒长负调控基因GS3同时也是碱耐性的负调控因子。本发明在水稻和玉米中验证了该基因在调控耐碱性中的功能,本发明通过敲除或抑制植株中GS3基因或蛋白的表达和/或抑制GS3蛋白活性,碱胁迫处理后,植株表现为植株的耐碱性提高,而超表达GS3基因或蛋白后不仅无法实现转基因植株的耐碱能力的提高,而且还表现为耐碱能力降低。因此,本发明植物GS3基因或蛋白作为负调控因子影响植物的耐盐碱。
附图说明
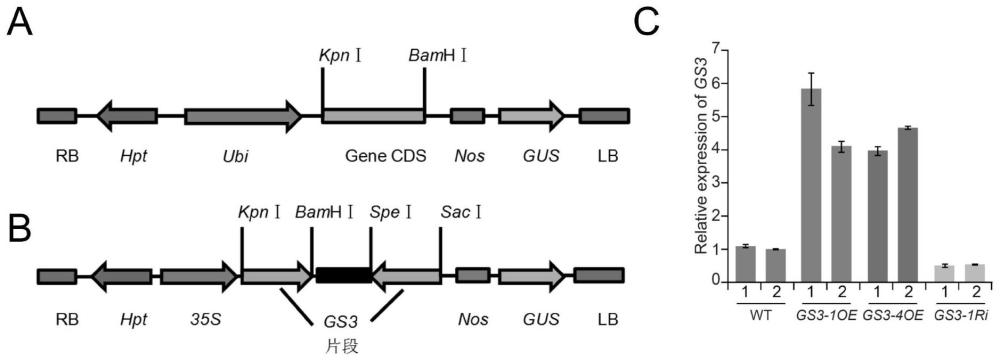
图1为超量表达水稻GS3-1或者GS3-4以及GS3抑制表达转基因植株的构建结果,A:超表达载体示意图;B:抑制表达载体示意图;C:超表达以及抑制表达转基因植株表达量检测结果;
图2为水稻野生型中花11植株(WT)与GS3-1OE、GS3-4OE以及GS3-1Ri抑制敲除植株的耐碱性比较,A:苗期碱处理生长势比较结果;B:苗期碱处理成活率比较结果;
图3为水稻GS3 CRISPR敲除载体pYL-Cas9-gRNA-OsGS3构建过程示意图,A:水稻GS3基因的结构以及CRISPR靶点位置示意图;B:两个靶点T1以及T2分别通过PCR的方法插入到pYL-OsU3-gRNA以及pYL-OsU6a-gRNA,获得pYL-OsU3-T1-gRNA以及pYL-OsU6a-T2-gRNA的过程;C:表达载体pYL-Cas9-gRNA-OsGS3结构示意图;
图4为水稻GS3 CRISPR敲除转基因植株T
图5为玉米ZmGS3 CRISPR敲除载体pYL-Cas9-gRNA-ZmGS3构建过程示意图,A:玉米ZmGS3基因的结构以及CRISPR靶点示意图;B:玉米ZmGS3 CRISPR敲除转基因植株T
图6为野生型玉米KN555与ZmGS3基因编辑植株的耐碱性比较,A:苗期碱处理生长势比较;B:苗期碱处理成活率比较;
具体实施方式
本发明提供了一种植物GS3基因或蛋白在调控植物耐盐碱中的应用。
在本发明中,植物GS3蛋白的氨基酸序列优选如SEQ ID NO:1~SEQ ID NO:3中至少一项所示。其中SEQ ID NO:1所示的GS3蛋白为水稻来源的等位基因型GS3-1,SEQ ID NO:2所示的GS3蛋白为水稻来源的等位基因型GS3-4。SEQ ID NO:3所示的植物GS3蛋白为玉米来源的GS3蛋白。
在本发明中,植物GS3基因的核苷酸序列如SEQ ID NO:4~SEQ ID NO:8中至少一项所示。SEQ ID NO:4所示的GS3基因来源于水稻基因组。SEQ ID NO:5所示的GS3基因为来源于水稻基因组的等位基因型GS3-4。SEQ ID NO:6所示的GS3基因为来源于水稻基因组的等位基因型GS3-4。SEQ ID NO:7所示的GS3基因来源于玉米基因组。SEQ ID NO:8所示的GS3基因为来源于玉米基因组的GS3基因的编码序列。
在本发明中,通过敲除或抑制植物基因组的GS3基因的表达,得到的植株经过碱胁迫处理发现在耐碱性方面有显著提高,而过表达GS3基因,得到的转基因植株表现为耐碱性降低。由此说明,所述植物GS3基因或蛋白优选通过负调控方法调控植物耐盐碱。所述植物优选包括单子叶植物。在本发明实施例中,所述单子叶植物以水稻和玉米为例,加以说明GS3基因调控耐碱性功能。
本发明提供了一种提高植物耐碱性的方法,敲除或抑制植物中GS3基因或蛋白表达或抑制植物中GS3蛋白活性。
在本发明中,所述敲除或抑制植物中GS3基因或蛋白表达或抑制植物中GS3蛋白活性的方法优选包括基因编辑、RNA干扰和T-DNA插入中至少一种。
在本发明中,所述基因编辑的方法优选为利用CRISPR/Cas9技术敲除植物基因组GS3基因中如SEQ ID NO:9~SEQ ID NO:11任意一个所示的靶标区域。其中SEQ ID NO:9和SEQ ID NO:10所示的靶标区域是水稻GS3基因的第一个外显子。SEQ ID NO:11所示的靶标区域是玉米GS3基因的第一外显子。所述敲除的方法优选构建基因编辑载体,将所述基因编辑载体导入植物中,筛选,得到含有基因编辑载体的植株。
下面以植物为水稻为例,说明基因编辑的方法。所述基因编辑载体的构建方法,优选为扩增含OsU6a启动子的GS3基因20bp T1靶序列,扩增GS3基因T1靶序列和gRNA-polyT形成的片段,将两片段进行融合,得到融合片段1,同时扩增含OsU6a启动子的GS3基因20bp T1靶序列,扩增GS3基因T1靶序列和gRNA-polyT形成的片段,将两片段进行融合,得到融合片段2,将两个融合片段同时插入基因编辑载体中,得到基因编辑重组载体。扩增含OsU6a启动子的GS3基因20bp T1靶序列的引物优选为引物B1’和T1R。含OsU6a启动子的GS3基因20bpT1靶序列优选以pYL-U6a-gRNA质粒为模板。扩增OsU6a-T1-gRNA-polyT片段的引物为T1F和B2,模板为pYL-U3-gRNA。所述融合的方法优选为PCR扩增,所述PCR扩增的引物优选为引物B1’和B2,以含OsU6a启动子的GS3基因20bp T1靶序列和OsU6a-T1-gRNA-polyT片段扩增产物为模板。扩增含OsU6b启动子的GS3基因20bp T2靶序列的引物优选为B2’和T2R,模板为pYL-U6a-gRNA质粒。扩增GS3基因T2靶序列和gRNA-polyT形成的序列的引物优选为T2F和BL。所述融合的方法,优选采用B2’和BL扩增,以GS3基因T2靶序列和gRNA-polyT形成的序列和GS3基因T2靶序列和gRNA-polyT形成的序列为模板。将两个融合片段同时插入基因编辑载体中的方法优选为酶切连接方法。所述酶切用酶优选为BsaI。所述连接优选为T7连接酶。所述基因编辑载体的种类优选为pYLCRISPR/Cas9-MT载体。pYLsgRNA-OsU3、pYLsgRNA-OsU6a以及pYLCRISPR/Cas-MT载体由华南农业大学刘耀光教授惠赠,其已经公开发表:Maet al.,ARobust CRISPR/Cas9 System for Convenient,High-Efficiency MultiplexGenome Editing inMonocot and DicotPlants.MolecularPlant,2015,8:1274–1284)。利用优化过的农杆菌介导的转基因方法(Lin and Zhang,Optimising the tissue cultureconditions for high efficiency transformation of indica rice.Plant Cell Rep,2005,23:540-548)将敲除载体pYL-Cas9-gRNA-GS3导入水稻受体中花11(由中国农科院作物所1979年选育,现在是水稻科研的常见基本材料),获得转化植株。
所述转化植株突变情况筛选的方法优选为采用PCR扩增的方法进行。所述PCR扩增优选采用GS3CRJCF/GS3CRJCR引物对进行。
在本发明中,还提供了利用CRISPR/Cas9技术敲除玉米基因组GS3基因的方法,优选利用识别玉米GS3基因第一外显子的sgRNA构建基因编辑重组载体,将所述基因编辑重组载体转染玉米,经过遗传转化,验证靶位点序列,得到玉米GS3突变基因。
在本发明中,利用RNA干扰敲降植物中GS3基因或蛋白表达的方法,优选为将植物GS3基因的编码序列分别按照正向和反向插入至pDS1301,得到的重组载体转化植物,得到GS3基因或蛋白表达量降低的植株。向所述pDS1301载体中插入GS3基因的方法优选采用酶切方式完成。正向插入时,采用KpnⅠ和BamHⅠ双酶切和连接。反向插入时,采用SacⅠ和SpeⅠ双酶切和连接。所述GS3基因的编码序列正向插入时,核苷酸序列为SEQ ID NO:19所示,所述GS3基因的编码序列反向插入时,核苷酸序列为SEQ ID NO:20所示。其中,pDS1301载体是本实验室改造而来(参见现有技术:Yuan B,Shen X,Li X,Xu C,Wang S(2007)Mitogen-activated protein kinase OsMPK6negatively regulates rice disease resistanceto bacterial pathogens.Planta226:953–960);其基本骨架是澳大利亚CAMBIA实验室(http://www.cambia.org/daisy/cambia/materials/overview.html)的pCAMBIA1301,通过加入35S启动子实现对转化基因的表达调控。实验证明通过碱胁迫处理GS3基因表达量降低的植物发现,提高耐碱能力,而超表达GS3基因的植物,耐碱能力降低。这表明GS3基因是一个以前未曾发现的碱负调控基因。因此抑制GS3蛋白表达可以提高植物的耐碱性。
本发明提供了一种提高植物耐碱性的试剂盒,包括至少以下分子中的一种:
(1)识别所述植物GS3基因的RNA分子;所述RNA分子的序列如SEQ ID NO:12~SEQID NO:14中任意一个所示;
(2)编码(1)中所述RNA分子的DNA分子;
(3)表达(2)中所述DNA分子的载体。
在本发明中,SEQ ID NO:12和SEQ ID NO:13所示的RNA分子特异性识别水稻基因组中GS3基因中SEQ ID NO:9和SEQ ID NO:10所示的靶标区域。SEQ ID NO:14所示的RNA分子特异性识别玉米基因组中GS3基因中SEQ ID NO:11所示的靶标区域。
本发明提供了所述试剂盒在提高植物耐盐碱或制备耐盐碱转基因植物中的应用。所述试剂盒通过敲降植物GS3基因的表达,达到抑制GS3基因或蛋白的活性,从而达到提高植物耐盐碱的目的。
本发明提供了一种植物突变基因或蛋白在提高植物耐盐碱中的应用,所述植物突变基因序列为SEQ ID NO:15或SEQ ID NO:16所示。
在本发明中,所述SEQ ID NO:15所示的突变基因对应利用基因编辑方法处理水稻后得到的GS3突变基因,对应的蛋白为SEQ ID NO:17。所述SEQ ID NO:16所示的突变基因对应利用基因编辑方法处理玉米后得到的GS3突变基因,对应的蛋白为SEQ ID NO:18。在本发明实施例中,可以通过基因编辑手段获得植物突变基因或蛋白,或利用基因工程手段用植物突变基因序列替换植物中GS3基因。与野生型相比,含有所述突变基因的植物在耐碱性方面具有显著提高。
提供以下定义和方法用以更好地界定本申请以及在本申请实践中指导本领域普通技术人员。除非另作说明,术语按照相关领域普通技术人员的常规用法理解。本文所引用的所有专利文献、学术论文、行业标准及其他公开出版物等,其中的全部内容整体并入本文作为参考。
如本文所用,“植物”包括对整株植物、植物器官、植物组织、种子和植物细胞以及它们的子代的标引。植物细胞包括但不限于来自种子、悬浮培养物、胚芽、分生区域、愈伤组织、叶、根、苗、配子体、孢子体、花粉和小孢子的细胞。“子代”包含植物的任何后续世代。“水稻”或“玉米”是任何水稻或玉米植物并包括可以与水稻或玉米育种的所有植物品种,包括整株植物、植物细胞、植物器官、植物原生质体、植物可以从中再生的植物细胞组织培养物、植物愈伤组织、植物或植物部分中完整的植物细胞,所述植物部分例如胚、花粉、胚珠、种子、叶、花、枝、果实、茎杆、根、根尖、花药等。
在本申请中,将词语“包括”、“包含”或其变体应理解为除所描述的元素、数或步骤外,还包含其它元素、数或步骤。
除非另有所指,核酸以5’至3’方向从左向右书写;氨基酸序列以氨基至羧基方向从左向右书写。氨基酸在本文可以用其通常所知的三字母符号或IUPAC-IUB生物化学命名委员会推荐的单字母符号来表示。同样地,可以用通常接受的单字母码表示核苷酸。数字范围包括限定该范围的数字。如本文所用,“核酸”包括涉及单链或双链形式的脱氧核糖核苷酸或核糖核苷酸多聚物,并且除非另有限制,包括具有天然核苷酸基本性质的已知类似物(例如,肽核酸),所述类似物以与天然存在的核苷酸类似的方式与单链核酸杂交。如本文所用,术语“编码”或“所编码的”用于特定核酸的上下文时,指该核酸包含指导该核苷酸序列翻译成特定蛋白的必需信息。使用密码子表示编码蛋白的信息。如本文所用,涉及特定多核苷酸或其所编码的蛋白的“全长序列”指具有天然(非合成)内源序列的整个核酸序列或整个氨基酸序列。全长多核苷酸编码该特定蛋白的全长、催化活性形式。本文可互换地使用术语“多肽”、“多肽”和“蛋白”,以指氨基酸残基的多聚物。该术语用于氨基酸多聚物,其中一个或多个氨基酸残基是相应天然存在的氨基酸的人工化学类似物。该术语还用于天然存在的氨基酸多聚物。本文可互换地使用术语“残基”或“氨基酸残基”或“氨基酸”,以指被并入蛋白、多肽或肽(统称“蛋白”)的氨基酸。氨基酸可以是天然存在的氨基酸,并且除非另有限制,可以包括天然氨基酸的已知类似物,所述类似物可以与天然存在的氨基酸相似的方式起作用。
在一些实施方案中,可以对本申请的核苷酸序列进行改变,以进行保守氨基酸替换。保守氨基酸替换的原则和实例在下文中进一步描述。在某些实施方案中,可以依照公开的单子叶密码子偏好性对本申请的核苷酸序列进行不改变氨基酸序列的替换,例如可以用单子叶植物偏好的密码子替换编码同一氨基酸序列的密码子,而不改变该核苷酸序列所编码的氨基酸序列。在一些实施方案中,以编码同一氨基酸序列的不同密码子替换本申请中的部分核苷酸序列,从而在改变核苷酸序列的同时不改变其编码的氨基酸序列。保守变体包括由于遗传密码子简并性而编码实施方案的蛋白中的一种的氨基酸序列的那些序列。在一些实施方案中,根据单子叶植物偏好密码子替换本申请中的部分核苷酸序列。本领域技术人员会认识到氨基酸添加和/或取代通常基于氨基酸侧链取代基的相对相似性,例如,所述取代基的疏水性、电荷、大小等等。具有各种前述所考虑性质的示例性氨基酸取代基团为本领域技术人员所公知,并且包括精氨酸与赖氨酸;谷氨酸和天门冬氨酸;丝氨酸和苏氨酸;谷氨酰胺和天冬酰胺;以及缬氨酸、亮氨酸和异亮氨酸。关于不影响目的蛋白生物学活性的适当氨基酸取代的指南可以在Dayhoff等人(1978)Atlas ofProtein Sequence andStructure(蛋白序列和结构图集)(Natl.Biomed.Res.Found.,Washington,D.C)(通过引用并入本文)的模型中找到。可以进行诸如将一个氨基酸换作具有相似性质的另一个氨基酸的保守性取代。序列一致性的鉴定包括杂交技术。例如,将已知核苷酸序列的全部或部分用作与其它相应核苷酸序列选择性杂交的探针,所述其它相应核苷酸序列存在于来自所选生物体的已克隆基因组DNA片段或cDNA片段群(即基因组文库或cDNA文库)。所述杂交探针可以是基因组DNA片段、cDNA片段、RNA片段或其它寡核苷酸,并且可以用诸如32P的可检测基团或其它可检测标志物来标记。因而,例如,可以通过标记基于实施方案序列的合成寡核苷酸制备杂交探针。制备杂交探针和构建cDNA及基因组文库的方法通常为本领域已知。可以在严紧条件下进行所述序列的杂交。如本文所用,术语“严紧条件”或“严紧杂交条件”表示如下条件,即在该条件下,相对于与其它序列杂交,探针将以可检测的更大程度(例如,背景的至少2倍、5倍或10倍)与其靶序列杂交。严紧条件是序列依赖性的并且在不同环境中有所不同。通过控制杂交严紧性和/或控制清洗条件,可以鉴定与所述探针100%互补的靶序列(同源探针法)。可选择地,可以调节严紧条件,以允许一些序列错配,以便检测较低的相似度(异源探针法)。通常,探针长度少于约1000或500个核苷酸。通常,严紧条件是如下的条件,即在该条件中,盐浓度为pH 7.0至8.3下,少于约1.5M Na离子,通常约0.01M至1.0M Na离子浓度(或其它盐),并且温度条件为:当用于短探针时(例如10到50个核苷酸),至少约30℃;当用于长探针时(例如大于50个核苷酸),至少约60℃。还可以通过添加诸如甲酰胺的去稳定剂来实现严紧条件。示例性的低严紧条件包括37℃下使用30%至35%的甲酰胺缓冲液、1M NaCl、1%SDS(十二烷基硫酸钠)杂交,50℃至55℃下在1×至2×SSC(20×SSC=3.0MNaCl/0.3M柠檬酸三钠)中清洗。示例性的中度严紧条件包括37℃下在40%至45%甲酰胺、1.0MNaCl、1%SDS中杂交,55℃至60℃下在0.5×至1×SSC中清洗。示例性的高严紧条件包括37℃下在50%甲酰胺、1M NaCl、1%SDS中杂交,60℃至65℃下在0.1×SSC中最后清洗至少约20分钟。任选地,清洗缓冲液可以包含约0.1%至约1%SDS。杂交持续时间通常少于约24小时,通常为约4小时至约12小时。特异性通常依赖杂交后的清洗,关键因素在于最后清洗溶液的离子强度和温度。DNA-DNA杂合体的Tm(热力学熔点)可以近似自Meinkoth andWahl(1984)Anal.Biochem.138:267-284的公式:Tm=81.5℃+16.6(logM)+0.41(%GC)-0.61(%甲酰胺)-500/L;其中M是一价阳离子的克分子浓度,%GC是DNA中鸟苷和胞嘧啶核苷酸的百分数,“甲酰胺%”是杂交溶液的甲酰胺百分数,而L是杂合体的碱基对长度。Tm是(确定的离子强度和pH下)50%的互补靶序列与完全匹配的探针杂交时的温度。通常将清洗至少进行至达到平衡,并且达到低的杂交背景水平,诸如进行2小时、1小时或30分钟。每1%的错配对应使Tm降低约1℃;因而,可以调节Tm、杂交和/或清洗条件,从而与所需一致性的序列杂交。例如,如果需要≥90%一致性的序列,可以将Tm降低10℃。通常,将严紧条件选择为比确定离子强度和pH下的特异序列及其互补序列的Tm低约5℃。然而,在非常严紧的条件下,可以在比所述Tm低4℃下进行杂交和/或清洗;在中度严紧条件下,可以在比所述Tm低6℃下进行杂交和/或清洗;在低严紧条件下,可以在比所述Tm低11℃下进行杂交和/或清洗。
除非另外指明,本说明书和权利要求书中使用的表示成分的量、反应条件等的所有数字应被理解为在所有情况下用术语“约”来修饰。如本文所使用的术语“约”,当指代可测量的值例如质量、重量、时间、体积、浓度或百分比的量时,意味着涵盖在一些实施例中与规定量相比±20%的变化、在一些实施例中与规定量相比±10%的变化、在一些实施例中与规定量相比±5%的变化、在一些实施例中与规定量相比±1%的变化、在一些实施例中与规定量相比±0.5%的变化、以及在一些实施例中与规定量相比±0.1%的变化,因为此类变化适合于执行所披露的方法和/或使用所披露的组合物、核酸、多肽等。因此,除非相反地指出,在本说明书和所附权利要求书中所列出的数值参数是可以取决于试图通过本申请披露的主题获得的期望特性而变化的近似值。
以下实施例用于说明本发明,但不用来限制本发明的范围。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本申请的范围。若无特别指明,实施例按照常规实验条件,如Sambrook等人的分子克隆实验手册(SambrookJ&Russell D W,Molecular cloning:a laboratory manual,2001),或按照制造厂商说明书建议的条件。若未特别指明,实施例中所用的化学试剂均为常规市售试剂,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
下面结合实施例对本发明提供的一种植物GS3基因或蛋白在调控植物耐盐碱中的应用进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
GS3基因调控耐碱性的功能研究
1.过表达GS3基因的转基因植株的构建
根据水稻基因组注释信息(GS3基因组序列如SEQ ID NO:4所示)设计引物,以水稻品种广陆矮的GS3全长cDNA(Osigcea013f09t3)为模板,分别扩增GS3的2种等位基因型GS3-1以及GS3-4,大小分别为696bp(如SEQ ID NO:5所示)和450bp(如SEQ ID NO:6所示)。
为了扩增GS3基因编码区的cDNA,本发明设计如下引物:
GS3OEF的正向引物(SEQ ID NO:21):
5'-
GS3-1OER的反向引物(SEQ ID NO:22):
5'-
GS3-4OER的反向引物(SEQ ID NO:23):
5'-
分别用引物组合GS3OEF、GS3-1OER和GS3OEF、GS3-4OER进行PCR扩增,获得GS3-1与GS3-4的目标片段。PCR反应总体积为50μl,包含cDNA模板2μl,2×GC I buffer 25μl,10mMdNTP 5μl,10mM引物GS3OEF和GS3OER各1μl,ExTaq酶1μl,加去离子水至50μl(所用到的2×GC I buffer、dNTP、rTaq酶等均购自宝生物工程大连有限公司)。PCR反应条件如下:①94℃4min,②94℃30s,③58℃30s,④72℃1min,⑤从②-④循环33次,⑥72℃7min,⑦25℃保存。PCR产物在1%(质量/体积)的TBE琼脂糖凝胶上电泳检测,回收长度为696bp的GS3-1(681bp的目标DNA区段加上引物上附加的两个限制性酶切位点15bp)与462bp的GS3-4(447bp的目标DNA区段加上引物上附加的两个限制性酶切位点15bp)的DNA片段。将它们分别构建到常用的超量表达载体pCAMBIA1301U上[该载体是本实验室改造而来:其基本骨架是澳大利亚CAMBIA实验室的(http://www.cambia.org/daisy/cambia/materials/overview.html)的pCAMBIA1301,通过加入Ubi启动子实现对转化基因的表达调控],转化水稻中花11获得超量表达植株GS3-1OE和GS3-4OE。
载体结构如图1中A所示。
2.RNAi干扰的植株的构建
构建GS3的表达抑制RNAi载体,分两步进行。首先将含有GS3全长cDNA(osigcea013f09t3)的质粒用BamHI和KpnI进行双酶切消化,连到经改造过的dsRNAi1301,得到含有第一链(正向)GS3的载体;同时将BamHI和KpnI消化产物连到中间载体GZ-1(本室丁新华博士提供)上,然后用SacI和SpeI双酶切消化,再将产物连到已经含有第一链的dsRNAi1301(也用SacI和SpeI双酶切消化)上,使得两个相同序列的片段方向相反,由35S启动子驱动,转化中花11,获得抑制表达转基因植株GS3-1RNAi。载体结构示意图如图1中B所示。
3.超量表达以及抑制表达转基因植株的表达量检测
(1)抽提GS3-1OE、GS3-4OE和GS3-1RNAi以及野生型植株幼穗分化期1cm长的幼穗的RNA,RNA抽提用的试剂购采用Invitrogen公司生产的Trizol抽提试剂盒(具体操作步骤按照试剂盒提供的说明书操作)。
(2)反转录合成cDNA第一链
步骤如下:
①取抽提的总RNA3μg,加入DNaseI 1μl,10×DNaseIbuffer 1μl,加DEPC(焦碳酸二乙酯,RNA酶的强烈抑制剂,工作浓度为0.01%)处理过的水到10μl,混匀后室温放置15min以去除残留的基因组DNA;
②加入0.2M EDTA 1μl,并于65℃水浴中孵育10min以去除DNaseI的活性
③加入oligo(dT)
④加入5×first strandbuffer4μl,0.1M DTT(巯基乙醇)2μl,10mM dNTPmixture1μl,反转录酶1μl,混匀后置于42℃水浴锅内温浴1.5h;
⑤反应结束后将反转录产物置于85℃干浴10min以灭活反转录酶;
⑥向反转录产物中加入80μl水,混匀后-20℃保存反应最终产物。反应中用到的试剂全部购自Invitrogen公司。
(3)对得到的反转录产物用Real-time PCR的检测,GS3基因检测引物为GS3QRT-F和GS3QRT-R,并且用Ubiquitin基因(LOC_Os03g13170)作为内参(引物组合为UbiQRT-F和UbiQRT-R),序列如下所示:
GS3QRT-F:5'-CCGCGAGATCGGATTCC-3'(SEQ ID NO:24);
GS3QRT-R:5'-CGTGGATCCCTTCGATTGA-3'(SEQ ID NO:25);
UbiQRT-F:5'-AACCAGCTGAGGCCCAAGA-3'(SEQ ID NO:26);
UbiQRT-R:5'-ACGATTGATTTAACCAGTCCATGA-3'(SEQ ID NO:27)。
在10μL体系中进行,其中含有1μL反转录产物、0.3μL的正向、反向引物和5μL的FastStartUniversal SYBR GreenMaster,补水至10μL。反应程序为:①95℃10min,②95℃10s,③60℃30s④从③-②循环40次。根据CT值计算相对表达量。
Real-time PCR检测结果表明,GS3-1OE以及GS3-4OE相对于野生型ZH11而言,其表达量明显上升。GS3RNAi植株相对于野生型ZH11而言,其表达量明显下降,如图1中C所示。
4.GS3基因耐碱性调控功能实验
将上述制备的过表达GS3基因的转基因植株和GS3表达量降低的植株分别进行碱处理,并比较主要农艺性状与非转基因受体的差异。具体如下:利用相同条件上生长,同一时间收获以及相同条件储存下的种子,进行了碱处理的实验。
将种子播种到等体积比的蛭石和营养土形成的土壤基质中,每穴播种12粒种子,再覆2cm左右的土层,与盘面铺平,设置重复2次。接着,用处理液(水或者75mM碱溶液)浇灌至土壤饱和作为处理,用清水浇灌的作为对照,置于平地上使其充分均匀吸收到土壤中,再将其置于模式植物玻璃温室中(幼苗生长环境如下:昼夜温度控制为28℃/22℃,光照/黑暗时间为16h/8h,相对湿度为60%至70%),后期用清水补齐,每组处理重复三次。待种子的胚芽顶出土壤表层时,就认为该种子已经发芽。按照公式I和公式II统计植株的存活率和相对存活率。
存活率=成苗种子粒数(14天)/供试种子数×100%公式I
相对存活率=碱处理存活率/相应对照存活率×100%公式II。
数据统计方法如下:首先,将Excel 2016进行数据整理与图表制作,利用DPS 7.5软件进行数据统计,采用最小显著差异法(LSD)和单因素方差分析(ANOVA)对结果进行显著性方差分析。若P<0.05,标为小写字母,差异显著;若P<0.01,标为大写字母,差异极显著。
表型观察结果表明:正常水处理的情况下,野生型ZH11、GS3基因过表达株系GS3-1OE、GS3-4OE以及GS3抑制表达植株GS3RNAi之间存活率无显著差异,而在75mM碱溶液处理下,GS3-1OE、GS3-4OE转基因植株相对于ZH11,存活率分别下降12.5%和26.4%,相反GS3RNAi存活率上升7.4%(图2),以上的结果均表明:水稻的GS3是一个负调控耐碱程度的重要基因。
结果显示,碱胁迫处理后,超表达转基因水稻的耐碱能力降低了,而抑制GS3显著提高耐碱能力,表明GS3是一个以前未曾发现的碱负调控基因。因此抑制蛋白表达有可能可以提高植物的耐碱性。
实施例2利用CRISPR/Cas9基因编辑手段抑制水稻中的GS3蛋白的表达
针对水稻GS3基因设计基于CRISPR/Cas9的靶点,合成sgRNA序列,将含有编码所述sgRNA序列的DNA片段连接到携带CRISPR/Cas的载体上(载体信息参见:Ma X,Zhang Q,ZhuQ,et al.A Robust CRISPR/Cas9System for Convenient,High-Efficiency MultiplexGenome Editing in Monocot and Dicot Plants.Mol Plant,2015,8(8):1274-84)中。
设计了2个靶点(如SEQ ID NO:9和SEQ ID NO:10所示),均位于GS3的第1个外显子上(图3中A)。所述基因编辑载体pYL-Cas9-gRNA-GS3的具体构建方法如下:
(1)OsU6a-T1-gRNA-polyT和OsU6b-T2-gRNA-polyT片段的构建
第一轮PCR以pYL-U6a-gRNA质粒为模板,用引物B1’和T1R扩增OsU6a启动子与GS3基因20bp T1靶序列;同样以及pYL-U3-gRNA为模板,用引物T1F和B2扩增GS3基因T1靶序列与gRNA-polyT;第二轮PCR是以第一轮PCR产物为模板,用引物B1’和B2扩增得到OsU6a-T1-gRNA-polyT片段;同样的方法获得OsU6a-T2-gRNA-polyT的片段。第一轮PCR以pYL-U6a-gRNA质粒为模板,用引物B2’和T2R扩增OsU6b启动子与GS3基因20bp T2靶序列;同样以pYL-U6b-gRNA为模板,用引物T2F和BL扩增GS3基因T2靶序列与gRNA-polyT;第二轮PCR是以第一轮PCR产物为模板,用引物B2’和BL扩增得到OsU6a-T2-gRNA-polyT片段(图3中B)。
其中步骤1)中使用的引物序列如下:
B1’:TTCAGA
B2:AGCGTG
B2’:TTCAGA
BL:AGCGTG
T1F:
T1R:
T2F:
T2R:
PCR反应总体积为50μl,包含cDNA模板2μl,2×GC I buffer 25μl,10mM dNTP 5μl,10mM引物GS3OEF和GS3OER各1μl,ExTaq酶1μl,加去离子水至50μl(所用到的2×GC Ibuffer、dNTP、ExTaq酶等均购自宝生物工程大连有限公司);PCR反应条件如下:①94℃4min,②94℃30s,③58℃30s,④72℃1min,⑤从②-④循环33次,⑥72℃7min,⑦25℃保存。
(2)pYL-Cas9-gRNA-OsGS3的构建
将OsU6a-T1-gRNA-polyT,OsU6b-T2-gRNA-polyT的PCR片段以及pYLCRISPR/Cas9-MT载体经过BsaI通过边切边连的方法,将OsU6-T1-gRNA-polyT,OsU6-T2-gRNA-polyT连接到pYLCRISPR/Cas9-MT载体中(图3中C),获得pYL-Cas9-gRNA-OsGS3载体,转化中花11获得转基因植株OsGS3
实施例3
OsGS3
在靶点上游111bp,下游72bp处设计引物,对实施例2得到的转基因植株OsGS3
GS3CRJCF:TACATAGCTGCTGCACCGTC(SEQ ID NO:36);
GS3CRJCR:GAAGCAAGATCGAAGGAGTATG(SEQ ID NO:37)。
PCR反应总体积为20μl,包含DNA模板2μl,2×GC I buffer 20μl,2mM dNTP 2μl,10mM引物GS3OEF和GS3OER各0.2μl,rTaq酶0.2μl,加去离子水至20μl(所用到的2×GC Ibuffer、dNTP、rTaq酶等均购自宝生物工程大连有限公司);PCR反应条件如下:①94℃4min,②94℃30s,③58℃30s,④72℃30s,⑤从②-④循环33次,⑥72℃7min,⑦25℃保存。测序结果显示,本发明得到一个纯和突变体OsGS3
利用类似的方法抑制了玉米自交系KN5585(品种权申请号20191002444)中的GS3蛋白(SEQ ID NO:3)。其中玉米GS3基因组序列如SEQ ID NO:7所示,cDNA序列如SEQ ID NO:8所示。靶点也是设计在第一外显子处,序列如SEQ ID NO:11所示,合成的sgRNA序列如SEQID NO:14所示。编辑载体构建和玉米遗传转化完成后,在靶点上游118bp,下游113bp处设计引物,PCR扩增和测序,判断靶位点突变情况。鉴定ZmGS3突变效果的引物序列如下,片段大小为255bp。
ZmGS36F:ACTATAACAATCGACGACGTG(SEQ ID NO:38);
ZmGS36R:AGCAGTGCAGCGTAATCGAT(SEQ ID NO:39)。
PCR反应总体积为20μl,包含DNA模板2μl,2×GC I buffer 20μl,2mM dNTP 2μl,10mM引物ZmGS36F和ZmGS36R各0.2μl,rTaq酶0.2μl,加去离子水至20μl(所用到的2×GC Ibuffer、dNTP、rTaq酶等均购自宝生物工程大连有限公司);PCR反应条件如下:①94℃4min,②94℃30s,③58℃30s,④72℃1min,⑤从②-④循环33次,⑥72℃7min,⑦25℃保存。根据测序结果可知,本实验获得了一个纯和敲除突变体,记为ZmGS3
实施例4
将实施例2获得的OsGS3
利用75mM碱溶液对水稻ZH11以及OsGS3
这些突变基因或突变蛋白可以通过常规杂交授粉的方式转育到其他水稻或玉米或其他可杂交的材料中,从而培育新的耐碱品系。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- WRKY20蛋白质及其编码基因在调控植物抗逆性中的应用
- ZmDRR206蛋白质及其编码基因在调控植物抗病性与生长发育中的应用
- ZmPht1;10蛋白及其编码基因在调控植物耐盐碱中的应用
- ZmPht1;10蛋白及其编码基因在调控植物耐盐碱中的应用
