CircRNA-miRNA关联预测方法、装置及介质
文献发布时间:2024-01-17 01:26:37
技术领域
本发明属于生物智能处理方法技术领域,更具体地,涉及一种CircRNA-miRNA关联预测方法、装置及介质。
背景技术
在20世纪70年代初,环状核糖核酸(circRNAs)就已经被发现,但在人类生理学中,它们并没有被认为是核糖核酸(RNA)表达分析的关键元素。2013年以后,由于高分辨率和高通量RNA-seq数据,特别是端到端读取和深度测序,新一批circRNAs被发现并被量化。根据已发表的文献,circRNA有三种类型的识别方法:从头(abinitio)预测方法、基于RNA-seq比对工具如(如segemehl),以及专门为寻找circRNA而设计的算法和工具(如CIRI)。这些研究证明,circRNA是一种内源性单链非编码RNA(ncRNA)分子,在RNA转录物的下游5'端剪接位点和上游3'位点产生共价闭环结构,具有重要的生物学功能,通过识别circRNA和疾病关联从而对疾病的治疗起到了至关重要的作用。大量实验分析表明,miRNA是一种ncRNA,是真核生物基因调控不可或缺的一部分。截至目前,越来越多的生物学实验验证表明circRNAs是miRNAs海绵并且circRNAs可作为一类新的生物标志物。为了减轻通过一些湿实验室实验探索circRNA-miRNA相互作用的劳动密集型的问题,许多计算机方法被用于circRNA-miRNA关联关系(CMAs)的鉴定。
近年来的研究表明,迅速涌现的计算机算法模型为CMAs预测不仅仅提供了良好的解决方案,而且在机器学习技术中快速处理海量数据的同时,也实现了更高的精度。例如,Lan等人开发了一种NECMA模型,使用内积和邻域正则化逻辑矩阵分解来预测CMAs。Qian等提出了一种基于奇异值分解和图变分自编码器的CMIVGSD模型来预测miRNA相关的circRNA。Guo等人设计了WSCD,该WSCD基于结构深度神经网络嵌入,使用卷积神经网络和深度神经网络对CMAs进行预测。Wang等通过多模态信息整合节点相似性特征融合,推断miRNA相关circRNA之间的关联关系得分。
此外,大多数现有的CMAs预测模型忽略了其序列中包含的完全相关的生物学信息和它们对miRNA功能的影响以及与circRNA的复杂关联。因此,以下有一明确值得考虑解决的问题:
(1)合理的特征融合模型融合多模态异构信息,提取circRNAs和miRNAs之间的关联关系;
(2)考虑适当的序列和交互信息策略来学习良好的特征;
(3)需要解决实验交叉反应产生的噪声影响。
发明内容
提供了本发明以解决现有技术中存在的上述问题。因此,需要一种CircRNA-miRNA关联预测方法、装置及介质,基于图表示学习(Graph Representation learning)预测CMAs的机器学习框架CMALF,该框架利用了深度学习同构嵌入融合模型和分解方法。具体而言,我们首先从基于circRNA序列的词嵌入(一种自然语言处理方法)中提取潜在的生物属性特征,word2vec[24]。其次,多源的特征信息包含其对circRNA-miRNA相互作用关系的影响,通过验证的相互作用对和相同数量的未标记样本得到异构图,构建可靠的分子关联网络。因此,将网络输入到大规模信息网络嵌入和图分解的融合模型中,用于低维嵌入向量生成。最后,利用梯度增强决策树分类器有效地推断出潜在的CMAs。
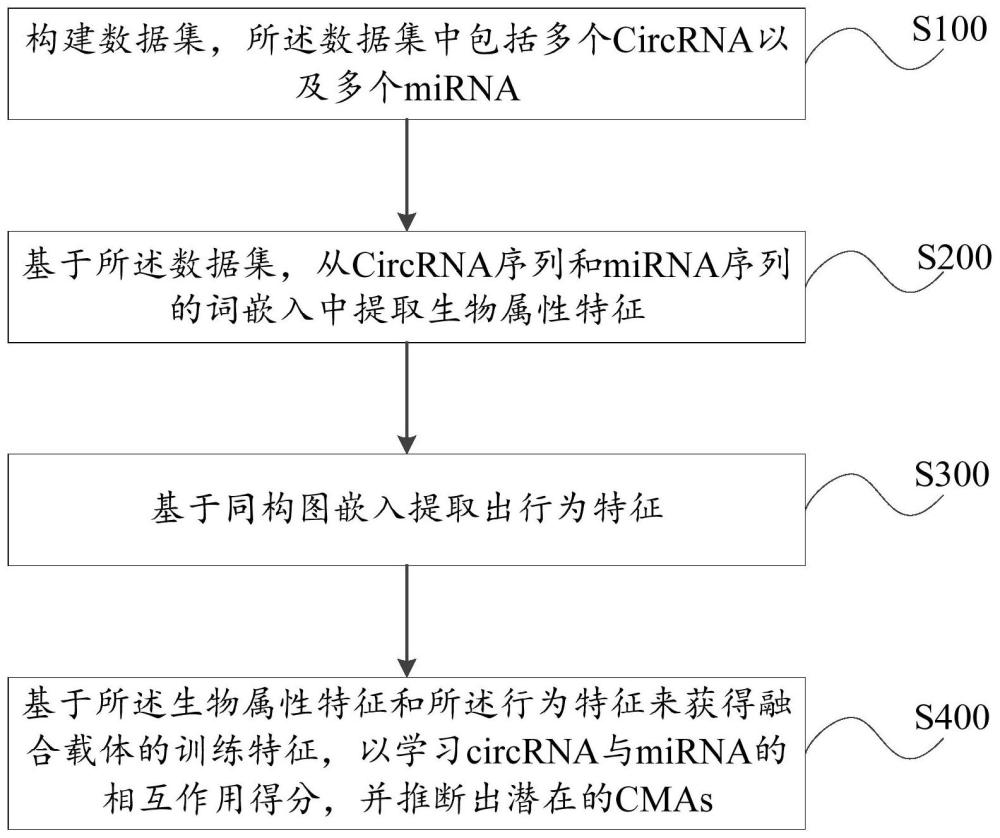
根据本发明的第一方案,提供了一种CircRNA-miRNA关联预测方法,所述方法包括:
构建数据集,所述数据集中包括多个CircRNA以及多个miRNA;
基于所述数据集,从CircRNA序列和miRNA序列的词嵌入中提取生物属性特征;
基于同构图嵌入提取出行为特征;
基于所述生物属性特征和所述行为特征来获得融合载体的训练特征,以学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs。
进一步地,通过公式(1)描述构建的数据集:
D=D
式中,D表示元素的并集,D
进一步地,构建维数为m×n的数据集的邻接矩阵,其中m表示CircRNA的数量,n表示miRNA的数量;
若miRNA(m(i))与circRNA(c(j))不相关,则DM中的DM(i,j)被设置为0;否则,DM中的DM(i,j)的值为1,其中miRNA(m(i))表示邻接矩阵中的第i个miRNA,circRNA(c(j))表示邻接矩阵中的第j个circRNA,DM(i,j)表示在邻接矩阵中第i个miRNA与第j个circRNA的相关性。
进一步地,所述基于所述数据集,从CircRNA序列和miRNA序列的词嵌入中提取生物属性特征,具体包括:
提供提取模型来实现生物属性特征的提取;
所述提取模型的输出值的目标函数表示为:
式中,ω表示权重矩阵,ω
进一步地,所述基于同构图嵌入提取出行为特征,具体包括:
利用多层感知器用来保持一阶与二阶相似度来学习低维密集向量,其中一阶与二阶的目标函数表示为:
式中,Q
对图的邻接矩阵进行因子分解,因子分解的目标函数表示为:
式中,Y,Z和λ分别表示权重邻接矩阵、因子矩阵和正则化参数;E表示邻接矩阵的大小,Z
进一步地,所述基于同构图嵌入提取出行为特征,具体包括:
提供融合图嵌入模型来提取出行为特征;
所述融合图嵌入模型的目标函数表示为:
式中,Y表示图的嵌入,Y
进一步地,所述基于所述生物属性特征和所述行为特征来获得融合载体的训练特征,以学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs,具体包括:
提供分类器来学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs。
进一步地,提供弱分类器,所述弱分类器用于多次迭代和先前分类器的残差;
所述弱分类器的目标函数表示为:
式中,F
根据本发明的第二技术方案,提供一种CircRNA-miRNA关联预测装置,所述装置包括:
构建模块,被配置为构建数据集,所述数据集中包括多个CircRNA以及多个miRNA;
第一提取模块,被配置为基于所述数据集,从CircRNA序列和miRNA序列的词嵌入中提取生物属性特征;
第二提取模块,被配置为基于同构图嵌入提取出行为特征;
预测模块,被配置为基于所述生物属性特征和所述行为特征来获得融合载体的训练特征,以学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs。
根据本发明的第三技术方案,提供一种可读存储介质,所述可读存储介质存储有一个或者多个程序,所述一个或者多个程序可被一个或者多个处理器执行,以实现如上所述的方法。
本发明至少具有以下有益效果:
根据本发明,可以构建出CMALF,这是一种基于融合模型的图表示学习框架,结合了两种同构图嵌入方法来预测CMAs。
对构建的数据集的总体评估证实,CMALF在CMAs预测方面优于其他优秀的模型。为了评估本文的最佳性能,我们在相同的数据集上进行了几个消融实验,包括CMALF-LINE模型、CMALF-GF和LAP模型的比较、不同的实验分类器模型、关联预测模型性能与其他最先进模型的比较。对构建的数据集的总体评估证实,CMALF在CMAs预测方面优于其他高级模型。通过最近文献中的案例研究,对大量实验结果对的预测获得了更高的关联分数。通过对结果的仔细分析表明,本发明实现了对学术研究潜在CMAs的出色预测。
本发明具有优异预测性能。特别是,本发明考虑了circRNA和miRNA序列提供的属性特征和行为特征,而以前的方法较少关注这些信息的充分应用。更具体地说,1.与以前侧重于CMAs特征提取信息的方法相比,使用序列信息可以用来增强特征表达。2.它可以执行全基因组定量测试和分析,几乎每个阅读片段都可以很好地表征其相关RNA序列,并且没有实验交叉反应和噪声影响。此外,将序列信息与交互信息相结合,以获得非常可靠的行为属性的特征向量,并提高特征提取的质量。
附图说明
图1为一种CircRNA-miRNA关联预测方法的流程图。
图2为CMALF模型的运行流程图,其中:A.通过预处理异构关联图来构建节点和节点拓扑特征;B.通过词嵌入和图嵌入模型学习circRNA和miRNA节点序列和拓扑结构的低维表示特征;C.GBDT预测的流程图。
图3为一个同质嵌入融合模型的运行流程图。
图4为通过CMALF实现的AUC和AUPR值的示意图,其中:A.AUC是通过对绘制ROC曲线的面板A下的面积求和而获得的;B.AUPR是指精确度和召回率所包围的曲线下的面积。
图5为不同策略的预测性能比较示意图。
图6为曲线图,其中:A.使用五折交叉验证的数据集上的LAP结果生成的ROC曲线;B.使用五折交叉验证的数据集的结果生成的AUPR。
图7为不同分类器模型获得的5倍CV的结果。
图8为一种CircRNA-miRNA关联预测装置的结构图。
具体实施方式
为使本领域技术人员更好的理解本发明的技术方案,下面结合附图和具体实施方式对本发明作详细说明。下面结合附图和具体实施例对本发明的实施例作进一步详细描述,但不作为对本发明的限定。本文中所描述的各个步骤,如果彼此之间没有前后关系的必要性,则本文中作为示例对其进行描述的次序不应视为限制,本领域技术人员应知道可以对其进行顺序调整,只要不破坏其彼此之间的逻辑性导致整个流程无法实现即可。
实施例1:
本发明实施例提供一种CircRNA-miRNA关联预测方法,如图1和图2所示,该方法包括如下步骤:
步骤S100,构建数据集,所述数据集中包括多个CircRNA以及多个miRNA。
示例性的,为了收集circRNA和miRNA的高可靠性序列信息以评估CMALF模型的性能,我们利用目前可用的实验验证的CircBank数据库和miRbase数据库作为高质量的选择数据集来评估上述模型的质量。因此,我们可以将构建的数据集描述如下:
D=D
在本实施例中,首先从构建的数据集中赛选了20208对实验验证的CMAs,涉及3569个circRNA和1152个miRNA。这里D
步骤S200,基于所述数据集,从CircRNA序列和miRNA序列的词嵌入中提取生物属性特征。
示例性的,可以基于Word2vec来进行生物属性特征的提取。
Word2Vec是由谷歌提出的。流行的word2vec是一个单词嵌入模型,它使用句子中出现的单词之间的关系来获得机器学习中从高维到低维空间的单词向量。类似地,节点被用作单词,节点序列被用作句子,如word2vec算法对circRNA和miRNA节点表示的序列分析中所述。
在这里,word2vec利用在依赖于CBOW(bag-of-words)模型的载体空间中提取circRNA和miRNA序列特征,该模型用于表征本研究中的节点特征。由CBOW确定的最优目标字在目标字之前和之后执行次预测,其中CBOW的输出值的目标函数如下:
这里ω是权重矩阵,ω
在本实施例中,通过使用word2vec模型的gensim-python软件包,训练circRNA和miRNA序列特征,可以获得64维的目标特征。
步骤S300,基于同构图嵌入提取出行为特征。
同构图嵌入是一种图表示学习方法,旨在学习顶点的低维表示时保留图拓扑。它也被公认为网络嵌入或非属性图嵌入,包含11种基于随机行走、基于深度学习和基于矩阵分解的方法。因此,本实施例中选择了矩阵分解中的GF、LAP和深度学习中的LINE。
在构建的数据集中,多源的特征信息包含其对circRNA-miRNA相互作用关系的影响,通过验证的相互作用对和相同数量的未标记样本得到异构图,构建可靠的分子关联网络,如图2中右上角的图片所示,基于该分子关联网络利用同构图嵌入提取出行为特征。
具体的,通过使用多层感知器用来保持一阶与二阶相似度来学习低维密集向量。用一阶与二阶的目标函数,分别如下所示
在这里,设置V={v
为了解决下游分类器的存储和构造问题,选择GF对图的邻接矩阵进行因子分解。该目标函数描述为
其中Y,Z和λ分别表示权重邻接矩阵、因子矩阵和正则化参数。
在一些实施例中,在行为特征提取过程中,结合深度学习和因子分解的优势,提出了一种基于集成LINE和GF的融合图嵌入模型(LPA),如图3所示。
本实施例提出了LAP模型,这是一种矩阵分解,旨在分解高维矩阵以获得嵌入。特别地,通过拉普拉斯特征映射来最小化所获得的目标函数。
其中Y
本文将所建立的open-NE库将应用于图嵌入模型的行为特征提取。在训练上述模型之后,获得了每个节点具有64个维度的向量。
步骤S400,基于所述生物属性特征和所述行为特征来获得融合载体的训练特征,以学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs。
该步骤是一个预测过程,可以基于决策树方法进行实现。
示例性的,提出一种分类器,可用于分类或回归或过滤特征。在这种策略下,再设置一个弱分类器,通过多次迭代和先前分类器的残差。本实施例将弱分类器的损失函数表示为
这里F
实施例2:CMALF模型的预测性能
CMALF模型是基于实施例1所述方法所构建的一个模型,其可以实现CircRNA-miRNA关联预测,本实施例的目的在于说明该模型的预测性能,以说明本发明的可行性和进步性。
对于在机器学习中进行的评估,不同的常用评估指标来衡量所提出的CMALF模型的预测性能,包括Spe.(specificity)、Pre.(Precision)、Sen.(Sensitivity)、MCC(Matthews correlation coefficient)和Acc.(Accuracy)。这些评价指标的定义如下:
这里,评估标准的缩写包括,TP表示真阳性,TN表示真阴性,FP表示假阳性,FN表示假阴性。此外,我们通过计算CMALF生成的TP率和FP率,可视化了ROC(Receiver-Operating-Characteristic)曲线,并计算了它们的平均AUC(ROC下面积)和AUPR(Area UnderPrecision-Recall(PR)),以考虑不平衡。此外,可靠的5倍交叉验证(CV)。
在评估中,我们使用我们构建的数据集,使用5倍CV方法来访问CMALF模型在Acc.、Sen.、Spe.、Pre.、MCC、AUC和AUPR方面预测潜在CMAs的能力。所有实验结果和黑体标粗的平均预测值如表1所示。CMALF获得了0.9075的AUC平均值和0.0016的标准差,其中5倍交叉验证实验的AUC值分别为0.9067、0.9103、0.9076、0.9070和0.9061。在图4中,AUC可以通过对绘制ROC曲线的面板A下的面积求和来获得,AUPR是指面板B中精度和召回率包围的PR曲线下的面积,分别到达曲线下面积较大的图像左上角和右上角。总体而言,上述统计数据证明,该模型具有最先进的性能,它可以通过有效预测潜在的CMAs,并且也为深入理解circRNA-miRNA的关系提供有力的证据。
表1 CMALF进行的五折交叉验证实验所得到的结果
实施例3:对比不同特征提取策略
为了确认两种不同的图嵌入方法的有效性是否比所提出的模型对模型的性能更好,我们将测试LINE和GF模型分别与CMALF进行了比较,分别命名为CMALF-LINE和CMALF-GF。CMALF-LINE和CMALF-GF与CMALF在同一数据集上进行的相同五折交叉验证实验的结果显示在图3的直方图中,其具体值如表2和表3所示。根据表1和表2中的数据,从CMALF-LINE模型获得的Acc.、Sen.、Spe.、Pre.、MCC和AUC的平均值分别比本文模型中提出的模型低3.65%、3.32%、5.77%、5.33%、9.1%和0.0342。与表1相比,表3显示CMALF-GF模型的平均值小于构建的模型。同样,图5也通过预测性能的比较显示了CMALF的优势。总之,CMALF的特征提取效果优于两种单方面信息特征提取策略。
表2 CMALF-LINE通过五折交叉验证实验所获得的结果
表3 CMALF-GF通过五折交叉验证实验所获得的结果
实施例4:与拉普拉斯特征映射模型的比较
为了推断所提出的模型利用生物属性特征和多源行为特征作为模型属性来优化该模型性能的有效性,我们将其与LAP方法生成的行为拓扑特征向量进行了比较。为了公平性和一致性,我们在体验过程中使用了LAP生成的低维嵌入向量的方法来代替所提出的融合模型的图嵌入,模型的其他部分保持不变。使用我们构建的数据集,我们使用五折交叉验证实验来和LAP方法训练模型,其结果的值呈现表4中。从表4可以看出,CMALF获得了更好的结果,其预测Acc.、Spe.、Pre.、MCC和AUC分别比LAP模型高8.91%、22.6%、14.4%、16.18%和0.0814。这一结果表明,所提出的模型使用的LINE和GF相结合的融合模型可以有效地建立向量的特征并训练计算机模型,这有助于改进模型并实现最有潜力的预测性能。CMALF的AUPR和ROC曲线优势可以从图5和图6的比较中看出。
表4通过LAP和CMALF获得的5倍CV的结果
实施例5:不同分类器模型的比较
为了确保通过特征提取选择最佳分类方法,我们比较了不同的分类器模型,以评估本研究中CMALF对特征和性能的影响。具体而言,我们保留了不变的生物属性特征和行为拓扑特征提取方法,仅用KNN(K-Nearest Neighbour)、LR(Logistic Regression)、RF(Rotation Forest)、SVM(support vector machine)和AdaBoost算法等五种不同的分类器代替GBDT模型进行研究。表5列出了上述模型在同一数据集上实现的五折交叉验证实验的平均值,如柱状图5所示。从表5可以得出,AdaBoost在Acc.、Sen.、Spe.、Pre.、MCC和AUC中排名第二,但比最佳结果低1.54%、1.26%、1.82%、1.76%、3.09%和0.0189,图7中的比较也表明CMALF模型是最佳结果。简而言之,这些结果表明,我们的GFBDT分类器CMALF模型优于其他分类器模型。
表5通过不同的分类器模型得到的平均结果
实施例6:与其他先进方法的比较
着近年来对CMAs的研究不断深入,许多著名学者提出了不同的CMAs预测方法。将CMALF与上述方法进行更公平的比较,以评估其预测性能。由于这里有一个精确的比较考虑,我们计算了先前模型中产生的AUC和AUPR分数,这些结果值列在表6中,其中包含了我们的模型和在CMAs预测新研究领域中的几篇新发表的论文的CMIVGSD、WSCD、KGDCMI[20]、SGCNCMI模型。该表显示,CMALF实现了最高的AUC和AUPR评分,分别优于第二好的SGCNCMI模型的为0.0133和0.0233,与其他三种方法的平均值相比大约超过了0.0198和0.0372。因此,上述比较可以得出结论,CMALF可以为进一步的学术研究提供最具竞争力的理论指导。
表6各种模型获得的AUC和AUPR得分
实施例7:案例研究
为了进一步研究CMALF识别新的miRNA候选circRNA的有效性,我们通过用已知的miRNA相关circRNA对训练模型进行了案例研究,并用训练的模型预测了所有未知的CMAs。然后,根据较高的分数对未知关联关系对的候选者进行排名,并通过查找相关研究文献或相关实验来确保预测有效性与正确性。结论模型预测结果如表7所示,从中我们可以看出,在最近的文献中,前30个miRNA相关的circRNA对中只有7个没有得到验证。总体而言,该案例研究表明,CMALF在预测潜在CMAs方面具有优越的预测性能,并且这些用于miRNA研究的有价值的circRNA候选物可能会被选择用于额外的湿实验室实验研究,以减少手动错误的不足。
表7 CMALF预测的前30对CMA
结合实施例1-7,本文提出了CMALF,这是一种基于融合模型的图表示学习框架,结合了两种同构图嵌入方法来预测CMAs。我们首先通过词嵌入构建了基于序列信息的低维嵌入向量生成,然后基于LINE和GF相结合的融合模型构建了低维表示,同时保留了图的拓扑结构和节点的属性。接下来,我们通过结合生物属性特征和多源行为特征信息来获得融合载体的训练特征,以学习circRNA与miRNA的相互作用得分。对构建的数据集的总体评估证实,CMALF在CMAs预测方面优于其他优秀的模型。为了评估本文的最佳性能,我们在相同的数据集上进行了几个消融实验,包括CMALF-LINE模型、CMALF-GF和LAP模型的比较、不同的实验分类器模型、关联预测模型性能与其他最先进模型的比较。对构建的数据集的总体评估证实,CMALF在CMAs预测方面优于其他高级模型。通过最近文献中的案例研究,对大量实验结果对的预测获得了更高的关联分数。通过对结果的仔细分析表明,我们提出的模型实现了对学术研究潜在CMAs的出色预测。
实施例8:CircRNA-miRNA关联预测方法装置
本发明实施例还提供一种CircRNA-miRNA关联预测方法装置,如图8所示,所述装置800包括:
构建模块801,被配置为构建数据集,所述数据集中包括多个CircRNA以及多个miRNA;
第一提取模块802,被配置为基于所述数据集,从CircRNA序列和miRNA序列的词嵌入中提取生物属性特征;
第二提取模块803,被配置为基于同构图嵌入提取出行为特征;
预测模块804,被配置为基于所述生物属性特征和所述行为特征来获得融合载体的训练特征,以学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs。
在一些实施例中,所述构建模块被进一步配置为通过公式(1)描述构建的数据集:
D=D
式中,D表示元素的并集,D
在一些实施例中,构建维数为m×n的数据集的邻接矩阵,其中m表示CircRNA的数量,n表示miRNA的数量;
若miRNA(m(i))与circRNA(c(j))不相关,则DM中的DM(i,j)被设置为0;否则,DM中的DM(i,j)的值为1,其中miRNA(m(i))表示邻接矩阵中的第i个miRNA,circRNA(c(j))表示邻接矩阵中的第j个circRNA,DM(i,j)表示在邻接矩阵中第i个miRNA与第j个circRNA的相关性。
在一些实施例中,所述第一提取模块被进一步配置为:
提供提取模型来实现生物属性特征的提取;
所述提取模型的输出值的目标函数表示为:
式中,ω表示权重矩阵,ω
在一些实施例中,所述第二提取模块被进一步配置为:
利用多层感知器用来保持一阶与二阶相似度来学习低维密集向量,其中一阶与二阶的目标函数表示为:
式中,Q
对图的邻接矩阵进行因子分解,因子分解的目标函数表示为:
式中,Y,Z和λ分别表示权重邻接矩阵、因子矩阵和正则化参数;E表示邻接矩阵的大小,Z
在一些实施例中,所述第二提取模块被进一步配置为:
提供融合图嵌入模型来提取出行为特征;
所述融合图嵌入模型的目标函数表示为:
式中,Y表示图的嵌入,Y
在一些实施例中,所述预测模块被进一步配置为:
提供分类器来学习circRNA与miRNA的相互作用得分,并推断出潜在的CMAs。
在一些实施例中,所述预测模块被进一步配置为提供弱分类器,所述弱分类器用于多次迭代和先前分类器的残差;
所述弱分类器的目标函数表示为:
式中,F
需要说明的是,本实施例所提出的装置与在先阐述的方法属于同一技术思路,其局有限相同的工作原理,并能起到同样的有益效果,此处不赘述。
本发明实施例最后还提供一种可读存储介质,所述可读存储介质存储有一个或者多个程序,所述一个或者多个程序可被一个或者多个处理器执行,以实现如上任一实施例所述的CircRNA-miRNA关联预测方法。
此外,尽管已经在本文中描述了示例性实施例,其范围包括任何和所有基于本发明的具有等同元件、修改、省略、组合(例如,各种实施例交叉的方案)、改编或改变的实施例。权利要求书中的元件将被基于权利要求中采用的语言宽泛地解释,并不限于在本说明书中或本申请的实施期间所描述的示例,其示例将被解释为非排他性的。因此,本说明书和示例旨在仅被认为是示例,真正的范围和精神由以下权利要求以及其等同物的全部范围所指示。
以上描述旨在是说明性的而不是限制性的。例如,上述示例(或其一个或更多方案)可以彼此组合使用。例如本领域普通技术人员在阅读上述描述时可以使用其它实施例。另外,在上述具体实施方式中,各种特征可以被分组在一起以简单化本发明。这不应解释为一种不要求保护的发明的特征对于任一权利要求是必要的意图。相反,本发明的主题可以少于特定的发明的实施例的全部特征。从而,以下权利要求书作为示例或实施例在此并入具体实施方式中,其中每个权利要求独立地作为单独的实施例,并且考虑这些实施例可以以各种组合或排列彼此组合。本发明的范围应参照所附权利要求以及这些权利要求赋权的等同形式的全部范围来确定。
- 基于预测模型的年龄预测方法、装置、服务器及存储介质
- 行为数据的处理方法、行为预测方法、装置、设备及介质
- 企业关联关系分析方法、装置及计算机可读存储介质
- 数据表关联的方法、装置、计算机设备和存储介质
- 行业关联度、风险确定方法及装置、存储介质和电子设备
- 基于关联性预测模型的新闻关联推荐方法、装置及介质
- 关联词登记装置、信息处理装置、关联词登记方法、关联词登记装置用程序、记录介质以及关联词登记系统
