产生功能性微生物聚生体
文献发布时间:2024-01-17 01:28:27
本申请要求于2020年12月8日提交的美国临时专利申请序列号63/122,889的优先权,其全部内容通过引用并入本文。
技术领域
本文提供的技术涉及鉴定和分离具有靶向功能的微生物,特别地但不排他地,涉及用于筛选和/或选择提供特定功能的个体微生物或微生物聚生体的方法、组合物和系统。
背景技术
各种植物群(flora)和/或动物群(fauna)可在称为生物群系的局部、自我维持的生态系统中存在并相互作用。包含植物群和/或动物群群落的生物群系的运行可能会影响局部环境或生态系统。在特定环境中,植物群和/或动物群可以包含微生物。尽管物理尺寸很小,但微生物在环境中的运行可能会产生重大影响。例如,在封闭环境中以糖为周围环境运行的酵母可能会产生醇,至在所述封闭系统中不再能产生更多醇的时刻。这可能通过糖的耗尽或通过阻碍产生更多醇的醇量而发生。在更大范围内,生物群系中植物群和/或动物群的运行可能会产生超越局部生态系统的全球影响。例如,Rothschild和Mancinelli等人假设微生物垫和叠层石有助于CO
人们希望不仅搜索单个动物群或植物群的运行,而且搜索相互协同的动物群和/或植物群的运行以优化对环境变量的影响。然而,用于鉴定和/或分离微生物生物体的传统技术专注于个体分离微生物的特定表型,并且大多数传统技术低效且缓慢。因此,需要筛选微生物聚生体以优化与对环境或生态系统的影响相关的变量。
发明内容
术语“生物采矿”是指搜索符合预定标准的生物体,例如,使用包括生物体的筛选和/或选择的方法。在这种情况下,如本文使用的术语“生物采矿”不应与描述使用生物体提取金属的其他领域中该术语的使用相混淆。常规生物采矿方法是从具有已知的期望特性的一组已知生物体(例如,微生物)开始的过程。鉴定一组新微生物,其中新微生物与已知生物体具有相似性,例如,具有与最初组的已知微生物相似的表型的微生物。然后针对特定应用测试新组。例如,在农业中,目标物种可能是用于覆盖作物的豆科植物,并且应用是在豆科植物茎中固定氮。
相反,本文提供的技术涉及“特定应用生物采矿”,其中生物采矿过程与上述常规生物采矿相反。具体而言,不是如常规生物采矿中那样从一组已知微生物开始,如本文所述的特定应用生物采矿鉴定将经受微生物生物体应用的目标(例如,物种、环境、生态系统等),例如,用于在功能上修饰(例如,改善)所述目标。然后针对一组微生物群体测试所述目标,这些微生物群体包含许多可用微生物,其可以是已知或未知的。在一些情况下,在测试之前可以对该组微生物群体进行最少的预过滤或预选择。如本文所用,术语“生物采矿”是指如上文和本文所述的“特定应用生物采矿”,除非上下文明确指出术语“生物采矿”是指常规生物采矿。
例如,在各种实施方式中,培养来自整个微生物群体组的一组微生物群体并应用于目标物种用于测试。应用于测试的该组微生物群体可以包括整个微生物群体的一部分。在针对该组微生物群体的一项或多项测试显示出关于一个或多个被测试变量的期望结果的趋势的情况下,选择该组微生物群体并进行传代培养,从而专注于使最有可能导致预期结果的微生物群体生长。这个过程可以迭代,直到期望的致病生物体被鉴定和分离。
以应用为中心的生物采矿有很多好处。首先,常规生物采矿从已知微生物开始,然后基于认为的“相似性”而不是有条不紊的测试操作来添加新微生物用于分析。在许多情况下,个体微生物可能不会对目标产生实质性的期望效果,而是包含两种或更多种协同作用的微生物的组(称为“微生物聚生体”)产生期望效果。因此,通过从预选的微生物组开始,使用常规生物采矿的研究者可能会无意中忽略与其他微生物协同提供期望效果的微生物。
相反,本文提供的以应用为中心的生物采矿的实施方式专注于要实现的应用和/或功能结果,例如,通过观察被测试一个或多个变量而测量的期望效果。因此,减少或消除了具有相似表型的微生物会导致相似的期望结果的潜在缺陷假设。另一个好处是,在特定应用生物采矿中,筛选过程可能会更快并且更有效。例如,在一项比较试验中,相对于常规生物采矿,使用特定应用生物采矿使用八分之一的员工发现期望微生物聚生体所需的时间的量减少了一半(例如,减少到十六分之一的人时),成本相应减少。
因此,本文提供了方法的实施方式,包括:获得多个环境样品,所述环境样品包括用于微生物生物采矿的有机物质;将所述多个环境样品混合成混合环境样品的组合;基于一个或多个选择标准选择所述混合环境样品中的特定混合环境样品用于测试;在包括一种或多种环境条件的环境中培养所选择的所述特定混合环境样品;以及响应于基于由所述培养产生的一个或多个变量测量确定所述特定混合环境样品产生了成功的微生物生物采矿结果,获得存在于所述特定混合环境样品的相应微生物聚生体中的微生物的识别信息。在一些实施方式中,方法还包括响应于基于由所述培养产生的一个或多个变量测量确定所述特定混合环境样品产生了不成功的微生物生物采矿结果,基于所述一个或多个选择标准选择额外混合环境样品用于测试。在一些实施方式中,方法还包括基于所述一个或多个选择标准选择所述混合环境样品中的额外混合环境样品用于测试;在包括一种或多种环境条件的环境中培养所选择的所述额外混合环境样品;以及响应于基于由所述培养产生的一个或多个变量测量确定所述额外混合环境样品产生了额外成功的微生物生物采矿结果,获得存在于所述额外混合环境样品的额外相应微生物聚生体中的额外微生物的额外识别信息。在一些实施方式中,方法还包括将所述特定混合环境样品的相应微生物聚生体培养成微生物培养物;使所述微生物培养物的选定培养物部分在包括一种或多种环境条件的环境中生长;以及响应于基于所述选定培养物部分的一个或多个变量测量确定所述选定培养物部分产生了成功的微生物生物采矿结果,获得存在于所述选定培养物部分的额外相应微生物聚生体中的额外微生物的额外识别信息。在一些实施方式中,方法还包括响应于基于所述选定培养物部分的一个或多个变量测量确定所述培养物部分产生了不成功的微生物生物采矿结果,选择所述微生物培养物的额外培养物部分用于测试。在一些实施方式中,方法还包括使所述微生物培养物的额外选定培养物部分在包括一种或多种环境条件的环境中生长;以及响应于基于所述额外选定培养物部分的一个或多个变量测量确定所述选定培养物部分产生了成功的微生物生物采矿结果,获得存在于所述额外选定培养物部分的另外相应微生物聚生体中的另外微生物的另外识别信息。
在一些实施方式中,方法还包括生成机器学习模型,其基于包括所述识别信息和所述额外识别信息的训练数据。在一些实施方式中,所述机器学习模型至少将所述多个环境样品中的一个或多个环境样品变量值与存在于所述多个环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联。在一些实施方式中,方法还包括接收对与一个或多个变量值相关的信息的请求,以及将所述机器学习模型应用于所述一个或多个变量值以用于以下中的至少一种:鉴定与所述一个或多个变量值相关联的一个或多个微生物物种,鉴定与所述一个或多个变量值相关联的一个或多个环境特征,或鉴定与所述一个或多个变量值相关联的至少一个微生物聚生体。在一些实施方式中,所述一个或多个变量值可包括以下中的至少一种:微生物的表型、期望的固氮量、期望的碳封存量、一个或多个环境样品特征、或一个或多个变量(例如,一个或多个气候变化变量)。在一些实施方式中,所述一个或多个环境特征包括环境源位置和环境组成。在一些实施方式中,所述机器学习模型进一步将一个或多个环境样品变量值和微生物变量值与一个或多个变量(例如,气候变化变量值)相关联,并且其中所述一个或多个变量值(例如,气候变化变量值)至少包括:被生物质封存的CO
在一些实施方式中,该技术提供了一种或多种存储计算机可执行指令的非暂时性计算机可读介质,所述指令在实行时使一个或多个处理器执行包括以下的动作:生成机器学习模型,其基于包括一种或多种微生物的识别信息的训练数据,所述机器学习模型至少将多个环境样品中的一个或多个环境样品变量值与存在于所述多个环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联;接收对与一个或多个变量值相关的信息的请求;以及将所述机器学习模型应用于所述一个或多个变量值以用于以下中的至少一种:鉴定与所述一个或多个变量值相关联的一个或多个微生物物种,鉴定与所述一个或多个变量值相关联的一个或多个环境特征,或鉴定与所述一个或多个变量值相关联的至少一个微生物聚生体。在一些实施方式中,所述一个或多个变量值可包括以下中的至少一种:微生物的表型、期望的固氮量、期望的碳封存量、一个或多个环境样品特征、或一个或多个变量(例如,气候变化变量)。在一些实施方式中,所述机器学习模型进一步将一个或多个环境样品变量值和微生物变量值与一个或多个变量值(例如,气候变化变量值)相关联,并且其中所述一个或多个变量值(例如,气候变化变量值)至少包括:被生物质封存的CO
在一些实施方式中,该技术提供了计算设备,其包括:一个或多个处理器;和存储器,所述存储器包括由所述一个或多个处理器可执行以执行多个动作的多个计算机可执行组件,所述多个动作包括:生成机器学习模型,其基于包括一种或多种微生物的识别信息的训练数据,所述机器学习模型至少将多个环境样品中的一个或多个环境样品变量值与存在于所述多个环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联;接收对与一个或多个变量值相关的信息的请求;以及将所述机器学习模型应用于所述一个或多个变量值以用于以下中的至少一种:鉴定与所述一个或多个变量值相关联的一个或多个微生物物种,鉴定与所述一个或多个变量值相关联的一个或多个环境特征,或鉴定与所述一个或多个变量值相关联的至少一个微生物聚生体。
在一些实施方式中,该技术提供了方法,其包括:获得环境样品,所述环境样品包括用于微生物生物采矿的有机物质;将所述环境样品均质化以产生输入样品;在包括一种或多种环境条件的环境中培养所述输入样品;以及响应于基于由所述培养产生的一个或多个变量测量确定所述输入样品产生了成功的微生物生物采矿结果,获得存在于所述输入样品的相应微生物聚生体中的微生物的识别信息。在一些实施方式中,方法包括:获得多个环境样品,所述多个环境样品包括用于微生物生物采矿的有机物质;将每个环境样品均质化以产生多个输入样品;以及从所述多个输入样品中选择输入样品。在一些实施方式中,方法还包括响应于基于由所述培养产生的一个或多个变量测量确定所述输入样品产生了不成功的微生物生物采矿结果,基于所述一个或多个选择标准产生第二输入样品用于测试。
在一些实施方式中,方法还包括基于所述一个或多个选择标准产生第二输入样品用于测试;在包括一种或多种环境条件的环境中培养所选择的所述第二输入样品;以及响应于基于由所述培养产生的一个或多个变量测量确定所述第二输入样品产生了额外成功的微生物生物采矿结果,获得存在于所述第二输入样品的第二相应微生物聚生体中的额外微生物的额外识别信息。在一些实施方式中,方法还包括将所述输入样品的相应微生物聚生体培养成微生物培养物;使所述微生物培养物的选定培养物部分在包括一种或多种环境条件的环境中生长;以及响应于基于所述选定培养物部分的一个或多个变量测量确定所述选定培养物部分产生了成功的微生物生物采矿结果,获得存在于所述选定培养物部分的额外相应微生物聚生体中的额外微生物的额外识别信息。在一些实施方式中,方法还包括响应于基于所述选定培养物部分的一个或多个变量测量确定所述培养物部分产生了不成功的微生物生物采矿结果,选择所述微生物培养物的额外培养物部分用于测试。在一些实施方式中,方法还包括使所述微生物培养物的额外选定培养物部分在包括一种或多种环境条件的环境中生长;以及响应于基于所述额外选定培养物部分的一个或多个变量测量确定所述选定培养物部分产生了成功的微生物生物采矿结果,获得存在于所述额外选定培养物部分的另外相应微生物聚生体中的另外微生物的另外识别信息。
在一些实施方式中,方法还包括生成机器学习模型,其基于包括所述识别信息和所述额外识别信息的训练数据。在一些实施方式中,所述机器学习模型至少将所述环境样品的一个或多个环境样品变量值与存在于所述环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联。在一些实施方式中,方法还包括接收对与一个或多个变量值相关的信息的请求,以及将所述机器学习模型应用于所述一个或多个变量值以用于以下中的至少一种:鉴定与所述一个或多个变量值相关联的一个或多个微生物物种;鉴定与所述一个或多个变量值相关联的一个或多个环境特征;和/或鉴定与所述一个或多个变量值相关联的至少一个微生物聚生体。在一些实施方式中,所述一个或多个变量值可包括以下中的至少一种:微生物的表型、期望的固氮量、期望的碳封存量、一个或多个环境样品特征、和/或一个或多个变量。在一些实施方式中,所述一个或多个环境特征包括环境源位置和环境组成。在一些实施方式中,所述机器学习模型进一步将一个或多个环境样品变量值和微生物变量值与一个或多个变量值相关联,并且其中所述一个或多个变量值至少包括:被生物质封存的CO
在一些实施方式中,该技术提供了一种或多种存储计算机可执行指令的非暂时性计算机可读介质,所述指令在实行时使一个或多个处理器执行包括以下的动作:生成机器学习模型,其基于包括一种或多种微生物的识别信息的训练数据,所述机器学习模型至少将环境样品的一个或多个环境样品变量值与存在于所述环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联;接收对与一个或多个变量值相关的信息的请求;以及将所述机器学习模型应用于所述一个或多个变量值以用于以下中的至少一种:鉴定与所述一个或多个变量值相关联的一个或多个微生物物种;鉴定与所述一个或多个变量值相关联的一个或多个环境特征;和/或鉴定与所述一个或多个变量值相关联的至少一个微生物聚生体。在一些实施方式中,所述一个或多个变量值可包括以下中的至少一种:微生物的表型、期望的固氮量、期望的碳封存量、一个或多个环境样品特征、或一个或多个变量。在一些实施方式中,所述机器学习模型进一步将一个或多个环境样品变量值和微生物变量值与一个或多个变量值相关联,并且其中所述一个或多个变量值至少包括:被生物质封存的CO
在一些实施方式中,该技术提供了计算设备,其包括:一个或多个处理器;和存储器,所述存储器包括由所述一个或多个处理器可执行以执行多个动作的多个计算机可执行组件,所述多个动作包括:生成机器学习模型,其基于包括一种或多种微生物的识别信息的训练数据,所述机器学习模型至少将环境样品的一个或多个环境样品变量值与存在于所述环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联;接收对与一个或多个变量值相关的信息的请求;以及将所述机器学习模型应用于所述一个或多个变量值以用于以下中的至少一种:鉴定与所述一个或多个变量值相关联的一个或多个微生物物种,鉴定与所述一个或多个变量值相关联的一个或多个环境特征,或鉴定与所述一个或多个变量值相关联的至少一个微生物聚生体。
此外,在一些实施方式中,该技术提供了用于产生执行特定功能(指定功能)的微生物聚生体的方法。例如,在一些实施方式中,方法包括:提供包含多种微生物的样品;用所述样品的一部分接种第一体积的生长培养基以提供第一培养物;使所述第一培养物在一组选择性条件下生长;在所述第一培养物中产生微生物的第一分类学分类;用所述第一培养物的一部分接种第二体积的生长培养基以提供第二培养物;使所述第二培养物在所述组选择性条件下生长;在所述第二培养物中产生微生物的第二分类学分类;以及使用所述第二分类学分类和所述第一分类学分类得出所述第二培养物相对于所述第一培养物的微生物群落稳定性的量度。
在一些实施方式中,该技术提供迭代和/或递归方法,其中重复步骤直到监测的测量特征达到特定值和/或达到平台。例如,在一些实施方式中,该技术提供了用于产生执行特定功能的微生物聚生体的方法,所述方法包括:提供包含多种微生物的样品;用所述样品的一部分接种第N体积的生长培养基以提供第N培养物;使所述第N培养物在一组选择性条件下生长;在所述第N培养物中产生微生物的第N分类学分类;用所述第N培养物的一部分接种第N+1体积的生长培养基;使第N+1培养物在所述组选择性条件下生长;在所述第N+1培养物中产生微生物的第N+1分类学分类;使用所述第N+1分类学分类和所述第N分类学分类得出所述第N+1培养物相对于所述第N培养物的微生物群落稳定性的量度;使用所述第N+1培养物作为所述第N培养物迭代地重复用第N培养物的一部分接种第N+1体积的生长培养基的步骤;使第N+1培养物在所述组选择性条件下生长;在所述第N+1培养物中产生微生物的第N+1分类学分类;以及使用所述第N+1分类学分类和所述第N分类学分类得出所述第N+1培养物相对于所述第N培养物的微生物群落稳定性的量度;以及提供稳定的第N+1培养物作为包含执行特定功能的微生物聚生体。在一些实施方式中,所述样品是环境样品。在一些实施方式中,所述环境样品是土壤或水样品。在一些实施方式中,所述生长培养基和/或选择性条件选择所述特定功能。在一些实施方式中,产生分类学分类包括获得培养物的宏基因组核苷酸序列数据以及使用所述宏基因组核苷酸序列数据的分析鉴定存在于所述培养物中的分类单元。在一些实施方式中,所述微生物聚生体包含为至少2、3、4、5或6个的分类单元数量。在一些实施方式中,具有少于所述微生物聚生体的分类单元数量的分类单元数量的微生物群落不执行所述特定功能。在一些实施方式中,任何一个分类单元单独不执行所述特定功能。在一些实施方式中,所述微生物群落稳定性的量度包括丰富度、多样性、丰度和/或成员资格的量度。在一些实施方式中,所述生长发生在经验确定的用于生长到指数期结束的时间内。在一些实施方式中,所述方法还包括测量第N或第N+1培养物的生长速率。在一些实施方式中,通过测量作为时间函数的细胞质量来确定生长速率。在一些实施方式中,所述分类单元中的至少一个在所述选择性条件下不作为纯培养物在所述培养基中生长。在一些实施方式中,包含至少两个且少于所述微生物聚生体的分类单元数量的分类单元数量的微生物群落在所述选择性条件下不在所述培养基中生长。
本说明书的一些部分根据信息操作的算法和符号表示来描述该技术的实施方式。这些算法描述和表示通常被数据处理领域的技术人员用来将他们工作的实质有效地传达给本领域的其他技术人员。这些操作虽然在功能上、计算上或逻辑上进行了描述,但应理解为由计算机程序或等效电路、微代码等来实现。此外,在不失一般性的情况下,有时将这些操作安排称为模块也被证明很方便。所描述的操作及其相关联的模块可以体现在软件、固件、硬件或其任何组合中。
本文描述的某些步骤、操作或过程可以用一个或多个硬件或软件模块单独或与其他设备组合来执行或实现。在一些实施方式中,软件模块是用计算机程序产品实现的,该计算机程序产品包括含有计算机程序代码的计算机可读介质,该计算机程序代码可以由计算机处理器实行以执行所描述的任何或所有步骤、操作或过程。
在一些实施方式中,系统包括虚拟提供的计算机和/或数据存储(例如,作为云计算资源)。在特定实施方式中,该技术包括使用云计算来提供虚拟计算机系统,该虚拟计算机系统包括如本文所述的组件和/或执行如本文所述的计算机的功能。因此,在一些实施方式中,云计算通过网络和/或经互联网提供如本文所述的基础设施、应用程序和软件。在一些实施方式中,计算资源(例如,数据分析、计算、数据存储、应用程序、文件存储等)是通过网络(例如,互联网;和/或蜂窝网络)远程提供的。
该技术的实施方式还可以涉及用于执行本文的操作的装置。该装置可以为所需目的而专门构造和/或它可以包括通用计算设备,该通用计算设备由存储在计算机中的计算机程序选择性地激活或重新配置。这样的计算机程序可以存储在非暂时性、有形计算机可读存储介质或适合于存储电子指令的任何类型的介质中,其可以耦合到计算机系统总线。此外,说明书中提及的任何计算系统可以包括单个处理器或者可以是采用多处理器设计以增加计算能力的架构。
基于本文包含的教导,其他实施方式对于相关领域的技术人员将是显而易见的。
附图说明
参考以下附图将更好地理解本技术的这些和其他特征、方面和优点。
图1例示了用于以应用为中心的微生物筛选的示例环境。
图2是显示支持关于以应用为中心的微生物筛选使用机器学习技术的一个或多个说明性计算设备的各种组件的框图。
图3a和图3b例示了用于执行以应用为中心的微生物筛选的示例过程的流程图。
图4是使用机器学习技术来鉴定微生物物种和与一个或多个变量相关的其他信息的示例过程的流程图。
图5是用于产生执行特定功能的微生物聚生体的示例过程的流程图。
应当理解,附图不一定按比例绘制,图中的对象也不一定相对于彼此按比例绘制。附图是旨在使本文公开的装置、系统和方法的各种实施方式清晰和理解的描述。在可能的情况下,贯穿附图将使用相同的附图标记来指代相同或相似的部分。此外,应当理解,附图不旨在以任何方式限制本教导的范围。
具体实施方式
本文提供的技术涉及鉴定和分离具有靶向功能的微生物,特别地但不排他地,涉及用于筛选和/或选择提供特定功能的个体微生物或微生物聚生体的方法、组合物和系统。
在各种实施方式的详细描述中,出于解释的目的,阐述了许多具体细节以提供对所公开的实施方式的透彻理解。然而,本领域技术人员将理解,可以在有或没有这些具体细节的情况下实践这些不同的实施方式。在另一些情况下,结构和设备以框图形式显示。此外,本领域的技术人员可以容易地理解,呈现和执行方法的特定顺序是说明性的,并且预期顺序可以变化并且仍然保持在本文公开的各种实施方式的精神和范围内。
本申请中引用的所有文献和类似材料,包括但不限于专利、专利申请、文章、书籍、论文和互联网网页,均出于任何目的明确通过引用整体并入。除非另有定义,本文使用的所有技术和科学术语具有本文描述的各种实施方式所属领域的普通技术人员普遍理解的相同含义。当并入的参考文献中的术语定义与本教导中提供的定义不同时,以本教导中提供的定义为准。本文使用的章节标题仅用于组织目的,不应被解释为以任何方式限制所描述的主题。
定义
为了促进对本技术的理解,下面定义了若干术语和短语。在整个详细描述中阐述了额外的定义。
在整个说明书和权利要求书中,以下术语具有与本文明确关联的含义,除非上下文另有明确规定。如本文所用,短语“在一种实施方式中”不一定指代相同的实施方式,尽管它可能指代相同的实施方式。此外,如本文所用,短语“在另一实施方式中”不一定指代不同的实施方式,尽管它可能指代不同的实施方式。因此,如下所述,在不脱离本发明的范围或精神的情况下,可以容易地组合本发明的各种实施方式。
此外,如本文所用,术语“或”是包含性的“或”运算符并且等同于术语“和/或”,除非上下文另有明确规定。术语“基于”不是排他性的并且允许基于未描述的额外因素,除非上下文另有明确规定。此外,在整个说明书中,“一个”、“一种”和“该”的含义包括复数引用。“在……内”的含义包括“在……内”和“在……上”。
如本文所用,术语“约”、“大约”、“基本上”和“显著”为本领域普通技术人员所理解,并且将在一定程度上根据它们所使用的上下文而变化。如果这些术语的使用在考虑使用它们的上下文的情况下对于本领域普通技术人员来说仍不清楚,则“约”和“大约”是指加上或减去特定术语的小于或等于10%,并且“基本上”和“显著”是指加上或减去特定术语的大于10%。
如本文所用,范围的公开包括对整个范围内的所有值和进一步划分的范围(包括为范围给出的端点和子范围)的公开。如本文所用,数值范围的公开包括端点和其间具有相同精确度的每个中间数字。例如,对于6-9的范围,除了6和9之外,还预期了数字7和8,对于6.0-7.0的范围,明确预期了数字6.0、6.1、6.2、6.3、6.4、6.5、6.6、6.7、6.8、6.9和7.0。
如本文所用,术语“无”是指省略了术语“无”所描述的特征的技术的实施方式。即,本文所用的术语“无X”是指“没有X”,其中X是“无X”技术中省略的技术特征。例如,“无钙”组合物不包含钙,“无混合”方法不包括混合步骤等。
尽管术语“第一”、“第二”、“第三”等可在本文中用于描述各种步骤、元件、组合物、组件、区域、层和/或部分,但这些步骤、元件、组合物、组件、区域、层和/或部分不应受这些术语的限制,除非另有说明。这些术语用于将一个步骤、元件、组合物、组件、区域、层和/或部分与另一步骤、元件、组合物、组件、区域、层和/或部分区分开来。除非上下文明确指出,否则本文使用的诸如“第一”、“第二”的术语和其他数字术语并不暗示序列或顺序。因此,在不脱离技术的情况下,本文讨论的第一步骤、元件、组合物、组件、区域、层或部分可以称为第二步骤、元件、组合物、组件、区域、层或部分。
如本文所用,词语“存在的”或“不存在的”(或可替代地,“存在”或“不存在”)在相对意义上用于描述特定实体(例如,组分、作用、元素)的量或水平。例如,当一个实体被称为“存在”时,这意味着该实体的水平或数量高于预定阈值;相反,当一个实体被称为“不存在”时,这意味着该实体的水平或数量低于预定阈值。预定阈值可以是与用于检测实体的特定测试相关联的可检测性阈值或任何其他阈值。当实体被“检测到”时,它就“存在”;当实体“未被检测到”时,它就“不存在”。
如本文所用,“增加”或“减少”分别指变量的值相对于先前测量的变量值、相对于预先确定的值和/或相对于标准对照的值的可检测(例如,测量的)正或负变化。增加是相对于先前测量的变量值、预先确定的值和/或标准对照的值正向变化优选至少10%,更优选50%,还更优选2倍,甚至更优选至少5倍,最优选至少10倍。类似地,减少是相对于先前测量的变量值、预先确定的值和/或标准对照的值负向变化优选至少10%,更优选50%,还更优选至少80%,最优选至少90%。其他表示数量变化或差异的术语,诸如“更多”或“更少”,在本文中以与上述相同的方式使用。
如本文所用,“系统”是指为了共同目的一起操作的多个真实和/或抽象组件。在一些实施方式中,“系统”是硬件和/或软件组件的集成组装体。在一些实施方式中,系统的每个组件与一个或多个其他组件交互和/或与一个或多个其他组件相关。在一些实施方式中,系统指的是用于控制和指导方法的组件和软件的组合。例如,“系统”或“子系统”可包括以下中的一种或多种或任意组合:机械设备、硬件、硬件的组件、电路、线路、逻辑设计、逻辑组件、软件、软件模块、软件或软件模块的组件、软件过程、软件指令、软件例程、软件对象、软件功能、软件类、软件程序、包含软件的文件等,以执行系统或子系统的功能。因此,实施方式的方法和装置,或其某些方面或部分,可以采用体现在有形介质例如软盘、CD-ROM、硬盘驱动器、闪存或任何其他机器可读存储介质中的程序代码(例如,指令)的形式,其中当程序代码被加载到机器诸如计算机中并由其实行时,该机器成为实践实施方式的装置。在程序代码在可编程计算机上实行的情况下,计算设备通常包括处理器、处理器可读的存储介质(例如,易失性和非易失性存储器和/或存储元件)、至少一个输入设备和至少一个输出设备。一个或多个程序可以实现或利用结合实施方式描述的过程,例如,通过使用应用程序编程接口(API)、可重用控件等。这样的程序优选地以高级过程或面向对象的编程语言来实现以与计算机系统通信。然而,如果需要,一个或多个程序可以用汇编语言或机器语言来实现。在任何情况下,该语言都可以是编译或解释语言,并与硬件实现相结合。
除非本文另有定义,与本技术相关的科学和技术术语应具有本领域普通技术人员通常理解的含义。此外,除非上下文另有要求,单数术语应包括复数,复数术语应包括单数。通常,与本文所述的细胞和组织培养、分子生物学、免疫学、微生物学、遗传学以及蛋白质和核酸化学和杂交相关使用的命名法和技术是本领域熟知和常用的那些。除非另有说明,否则本技术的方法和技术通常根据本领域众所周知的常规方法进行,并且如在整个本说明书中引用和讨论的各种一般和更具体的参考文献中所描述的那样。参见,例如,Sambrook etal.,Molecular Cloning:A Laboratory Manual,2ded.,Cold Spring Harbor LaboratoryPress,Cold Spring Harbor,N.Y.(1989);Sambrook et al.,Molecular Cloning:ALaboratory Manual,3d ed.,Cold Spring Harbor Laboratory Press,Cold SpringHarbor,N.Y.(2000);Ausubel et al.,Current Protocols in Molecular Biology,Greene Publishing Associates(1992and Supplements to 2000);Ausubel et al.,Short Protocols in Molecular Biology:A Compendium of Methods from CurrentProtocols in Molecular Biology,4thed.,Wiley&Sons(1999);Harlow和Lane,Antibodies:A Laboratory Manual,Cold Spring Harbor Laboratory Press,ColdSpring Harbor,N.Y.(1990);Harlow和Lane,Using Antibodies:A Laboratory Manual,Cold Spring Harbor Laboratory Press,Cold Spring Harbor,N.Y.(1998);以及T.Kieser et al.,Practical Streptomyces Genetics,John Innes Foundation,Norwich(2000);其中每一篇都通过引用整体并入本文。
如本文所用,术语“可培养生物体”是指可在实验室中维持和生长的活生物体。在一些实施方式中,可培养生物体可能无法在实验室中以不含其他生物体的纯培养物维持和生长,因此就作为纯培养物生长而言,其可被称为“不可培养的生物体”。然而,在一些实施方式中,这样的生物体可以在实验室中在包含至少一种其他生物体的微生物聚生体中生长,因此就该聚生体而言可以是“可培养的生物体”并且就在没有聚生体的其他一个或多个成员的情况下以纯培养物生长而言,其也是“不可培养的生物体”。
如本文所用,术语“选定环境”、“条件”或“多种条件”是指其中特定生物体或微生物群落的微生物聚生体比一种或多种其他生物体或微生物群落的聚生体更有效地生长(例如,更快,达到更高量或浓度,具有更高的存活率等)的任何外部特性。示例性“条件”或“环境”包括但不限于特定培养基、体积、容器、温度、混合、通气、重力、电磁场、细胞密度、pH、营养素、磷酸盐源、氮源,与一种或多种生物体共生,和/或与单一生物体的物种或多种生物体的物种(例如,混合群体)的相互作用。还包括为“条件”或“环境”的是可能对一种或多种生物体或微生物群落的聚生体有毒的物质,诸如重金属、抗生素和氯化化合物。应该理解,时间也可以被认为是一种“条件”,因为生物体不是静态实体。因此,在延长的时间段(例如,数天、数周、数月、数年)内生长的培养物可能产生包含特定生物体或聚生体的培养物,所述特定生物体或聚生体在培养物中的比例高于在生长该时间段之前培养物中特定生物体或聚生体的相对量。
如本文所用,术语“选择”是指通过有意或自发地以比其他类型更多地去除或富集某些类型的个体,从而增加群体中不同“类型”个体的频率。“类型”的性质可以通过以下来定义:遗传特征(例如,基因或核苷酸序列);功能特征(例如,酶促、代谢能力);分类学特征(例如,品系、亚种、种、属、科或基于核苷酸序列相似性或差异的操作分类单元(OTU));或物理特征。此外,一个类型可以包括一个或多个个体。选择的一个典型例子包括但不限于生长速率选择,其中生长和繁殖更快的个体在群体中变得更加普遍。进行选择的一个重要考虑因素是确定“因为什么选择”或什么是“被选择”,也就是说,在特定环境中有利或不利的遗传、功能和/或物理差异。生长速率选择用于选择生长速率比群体中其他个体快并且可以从亲本细胞传给其后代的生物体。
如本文所用,术语“富集”是指一种过程,其中感兴趣的一种或多种生物体、一种或多种功能能力、一种或多种基因或基因产物、或一种或多种核苷酸序列的丰度(例如,以绝对和/或相对术语表达)相对于一种或多种其他生物体、一种或多种其他功能能力、一种或多种其他基因或基因产物、或一种或多种其他核苷酸序列的丰度是增加的。例如,在一些实施方式中,术语“富集”是指增加存在于培养物中的一种或多种微生物的数量(例如,绝对和/或相对数量)的过程,例如,通过在选择性条件下在合适的培养基中培养。
如本文所用,术语“培养基(medium)”或“培养基(media)”是指生物体经受或提供进入的化学环境。生物体可以浸没在培养基中或与其物理接近(例如,物理接触)。培养基通常包含水以及可能有助于生物体生长或维持的其他额外营养素和/或化学物质。这些成分可能是纯化学物质(例如,“确定的”培养基)或复杂的、未表征的化学物质混合物,诸如由牛奶或血液制成的提取物。标准化培养基广泛用于实验室。用于细菌生长的培养基的实例包括但不限于LB和M9基本培养基。在提及培养基时,术语“基本”是指支持生物体生长但仅由最简单的可能化学化合物组成的培养基。例如,M9基本培养基可由溶解在水中并经过灭菌的以下成分组成:48mM Na
如本文所用,术语“培养物”是指在具有至少一种细胞或活生物体个体的容器或外壳中的培养基,通常是该生物体可以在其中生长的培养基。如本文所用,术语“连续培养”意指以与去除培养基的速率相等的某个速率向其中添加新培养基的液体培养。相反,如本文所用,“分批培养”意指不添加或去除新培养基的固定大小或体积的培养。
如本文所用,术语“遗传基础”是指特定观察的潜在遗传或基因组原因。
如本文所用,术语“遗传”是指在DNA核苷酸序列中编码的可遗传信息。因此,术语“遗传特征”旨在表示DNA中编码的信息的测序、基因分型、比较、作图谱或其他测定。
如本文所用,术语“遗传物质”是指生物体内从一代传给下一代的DNA。通常,遗传物质是指生物体的基因组。染色体外元件,诸如细胞器或质粒DNA,也可以是决定生物体特性的遗传物质的一部分。
如本文所用,术语“遗传改变”或“遗传适应”是指生物体基因组内的一个或多个突变。如本文所用,术语“突变”是指两个相关生物的DNA核苷酸序列的差异,包括例如置换、缺失、插入和重排,或移动遗传元件的运动。
如本文所用,术语“评估”意指对生物体的可观察表型的观察或测量。评估通常包括分析、解释和/或与另一种生物体的表型进行比较。应当理解,可以在遗传水平(例如,关于核苷酸序列)和基因产物水平评估表型。此外,可以根据生物体在环境中的行为和/或个体分子或分子组在生物体中的行为来评估表型。这种比较可用于确定由遗传适应引起的突变产物的详细功能。
如本文所用,术语“逐步”旨在表示在时间上一个接一个的一系列事件的方式。如本文所用,术语“同时”旨在表示同时发生。
如本文所用,术语“微生物(microbial)”、“微生物生物体”和“微生物(microorganism)”是指作为微观细胞存在的生物体,其包括在三域系统中的古菌、细菌或真核生物域中(参见Woese(1990)Proc Natl Acad Sci U S A 87:4576–79,通过引用并入本文),后者包括酵母和丝状真菌、原生动物、藻类或高等原生生物。因此,该术语旨在涵盖具有微观大小的原核或真核细胞或生物体,并且包括所有物种的细菌、古菌和真细菌以及真核微生物诸如酵母和真菌。还包括可以培养以用于生产化学物质的任何物种的细胞培养物。
如本文所述,在一些实施方式中,微生物是原核微生物。在一些实施方式中,原核微生物是细菌。“细菌”或“真细菌”是指原核生物体的域。细菌包括如下的至少十一个不同的组:(1)革兰氏阳性(革兰氏+)菌,其中有两个主要亚门:(1)高G+C组(放线菌、分枝杆菌、微球菌等),(2)低G+C组(芽孢杆菌、梭菌、乳杆菌、葡萄球菌、链球菌、支原体);(2)变形菌,例如紫色光合+非光合革兰氏阴性菌(包括最“常见”的革兰氏阴性菌);(3)蓝细菌,例如,生氧性光养生物;(4)螺旋体及相关物种;(5)浮霉状菌;(6)拟杆菌、黄杆菌;(7)衣原体;(8)绿色硫细菌;(9)绿色非硫细菌(亦称厌氧光养生物);(10)耐辐射微球菌及亲缘类群;(11)栖热袍菌和嗜热热袍菌(Thermosipho thermophiles)。“革兰氏阴性菌”包括球菌、非肠杆菌和肠杆菌。革兰氏阴性菌的属包括,例如,奈瑟氏菌属、螺旋菌属、巴斯德菌属、布鲁氏菌属、耶尔森氏菌属、弗朗西丝氏菌属、嗜血杆菌属、鲍特菌属、埃希菌属、沙门菌属、志贺氏菌属、克雷伯菌属、变形杆菌属、弧菌属、假单胞菌属、拟杆菌属、醋杆菌属、气杆菌属、农杆菌属、固氮菌属、螺形菌属、沙雷菌属、弧菌属、根瘤菌属、衣原体属、立克次氏体、密螺旋体属和梭杆菌属。“革兰氏阳性菌”包括球菌、非孢子杆菌和孢子杆菌。革兰氏阳性菌的属包括例如放线菌属、芽孢杆菌属、梭菌属、棒状杆菌属、丹毒丝菌属、乳杆菌属、李斯特菌属、分枝杆菌属、粘球菌属、诺卡菌属、葡萄球菌属、链球菌属和链霉菌属。
如本文所用,术语“天然存在”在用于提及微生物时意指在自然界中发现的微生物。例如,天然存在的生物体可以从自然界的来源中分离,并且没有在实验室中被人有意修饰。
如本文所用,应用于微生物的术语“非天然存在的”是指包含至少一种在天然存在的微生物中通常不存在的遗传改变的微生物。遗传改变包括,例如,引入编码代谢多肽的可表达核酸的修饰、其他核酸添加、核酸缺失和/或微生物遗传物质的其他功能破坏。这样的修饰包括例如参考物种的异源、同源、或异源和同源两种多肽的编码区及其功能片段。额外修饰包括,例如,非编码调节区,其中修饰改变基因或操纵子的表达。
如本文所用,术语“微生物聚生体(microbial consortium)”(复数“微生物聚生体(microbial consortia)”)是指一组微生物物种或物种的菌株,其可被描述为执行共同功能,或可被描述为参与或导致可识别的参数或表型性状或与之相关。聚生体可以包含两个或更多个微生物分类单元(例如,科、属、种或种的菌株)。在一些情况下,微生物在群落内共生共存。微生物聚生体可以通过描述聚生体中存在的分类单元(例如,若干菌株、亚种、种、属、科或基于核苷酸序列相似性或差异的操作分类单元(OTU));通过描述聚生体中存在的基因;通过描述聚生体中存在的核苷酸序列;或通过描述聚生体中存在和/或提供的功能来描述。微生物聚生体可以是在微生物群落中发现的生物体的子集。
如本文所用,术语“微生物群落”是指一组微生物,其包含两个或更多个微生物分类单元(例如,科、属、种或种的菌株)。与微生物聚生体不同,微生物群落不一定一致行动以进行共同功能,或者不必参与或导致可识别的参数或表型性状或与之相关。
如本文所用,术语“宏基因组”定义为“给定栖息地中存在的所有微生物的集体基因组”(Handelsman et al.,(1998)Chem.Biol.5:R245-R249)。该术语还旨在包括从微生物群落或微生物聚生体(例如,从环境样品)中提取的核酸,其作为微生物群落或微生物聚生体的代表,而无论是否提取了微生物群落或微生物聚生体的所有基因组核酸。
如本文所用,术语“分类单元”是被认为足够相似以被视为单独单元的一组生物体。分类单元可包括科、属、种或种内的群体(例如菌株),但不限于此。
如本文所用,术语“操作分类单元”(OTU)是指被认为足够相似以被视为单独单元的一组微生物。OTU可以包括分类学科、属或种,但不限于此。OTU通常是通过比较生物体之间的核苷酸序列来定义的。在某些情况下,OTU可以包括基于例如区分生物标志物(诸如,16S rRNA基因)的至少一部分之间≥97%、≥95%、≥90%、≥80%或≥70%的序列同一性被视为一个单元的一组微生物。
根据细菌和古菌分类纲要(Garrity et al.(2007)The Taxonomic Outline ofBacteria and Archaea.TOBA Release 7.7,March 2007.Michigan State UniversityBoard of Trustees),术语“属”可以定义为相关物种的分类群。术语“物种”可以定义为密切相关的生物体的集合,其具有大于97%的16S核糖体RNA序列同源性和大于70%的基因组杂交,并且与所有其他生物体有足够的差异从而被认为是一个独特的分类单元。
如本文所用,术语“相对丰度”是指第一生物样品中特定分类单元(例如,OTU)的微生物丰度与一个或其他(例如,第二)样品中相应分类单元的微生物丰度的比较。“相对丰度”可以反映在例如对应于分类单元的分离物种的数量或分类单元特异性的生物标志物(例如,核苷酸序列)在给定样品中存在或表达的程度。样品中特定分类单元的相对丰度可以使用本领域熟知的基于培养的方法或基于非培养的方法来确定。基于非培养的方法包括分类单元特异性的扩增多核苷酸的序列分析或样品中基于蛋白质组学的图谱的比较,反映了样品中存在的一种或多种分类单元的基于多肽、基于脂质、基于多糖或基于碳水化合物的生物标志物的数量和程度。参考检测到的所有分类单元/OTU,或参考一些组不变的分类单元/OTU,可以计算分类单元或OTU的相对丰度或丰度。在一些实施方式中,使用基于序列的方法鉴定分类单元,如在例如Wood(2014)“Kraken:ultrafast metagenomic sequenceclassification using exact alignments”Genome Biology 15:R46和Wood(2019)“Improved metagenomic analysis with Kraken 2”Genome Biology 20:257中描述的,每一篇都通过引用并入本文。
如本文所用,术语“显著改变的相对丰度”是指与样品中的总微生物或与另一样品中存在的相应分类单元的微生物的数量相比,特定分类单元的微生物数量的相对丰度在统计学上显著增加或减少。在一些实施方式中,相对丰度的“显著增加”或“显著减少”定义为相对于参考值的统计学上显著增加或统计学上显著减少。在一些实施方式中,统计学上显著增加或统计学上显著减少是相对丰度的标准偏差的两倍、三倍或四倍的增加或减少。在一些实施方式中,统计学上显著增加或统计学上显著减少是P值等于或小于0.1、0.05、0.01或0.005的增加或减少。
在一些实施方式中,相对丰度的“显著减少”或“显著增加”是指使用非参数统计测试(诸如符号秩测试),一个或多个指示物种或分类单元相互比较或与参考物种或分类单元相比的统计学上显著学差异。在一些实施方式中,相对丰度的“显著减少”或“显著增加”是使用采用贝叶斯推理和相关方法的模型来确定的。
在某些实施方式中,相对丰度的增加反映了相对于参考值增加10%、20%、30%、40%、50%、60%、70%、80%、90%、100%或更多。在一些实施方式中,相对丰度的增加反映了比参考值增加2倍、3倍、5倍、10倍、20倍、50倍或100倍。
如本文所用,“分离”、“分离的”、“分离的微生物”等术语旨在表示一种或多种微生物已经从至少一种与它在特定环境(例如,土壤、水或高等多细胞生物体)中相关联的材料中分离出来。因此,“分离的微生物”不存在于其自然发生的环境中;相反,通过本文所述的各种技术,微生物已从其自然环境中移出并置于非自然发生的存在状态中。因此,分离的菌株可以作为例如生物学上纯的培养物或作为与载体组合物结合的孢子(或其他形式的菌株)存在。在本公开的某些方面,分离的微生物作为分离的和生物学上纯的培养物存在。本领域技术人员将理解,特定微生物的分离的和生物学上纯的培养物表示所述培养物基本上不含(在科学原因内)其他活生物体并且仅包含所提及的个体微生物。培养物可以包含不同浓度的所述微生物,并且分离的和生物学上纯的微生物通常必然不同于不太纯或不纯的材料。此外,在一些方面,本公开提供了在分离的和生物学上纯的微生物培养物中发现的浓度或纯度限制的某些定量测量。在某些实施方式中,这些纯度值的存在是将目前公开的微生物与那些以自然状态存在的微生物区分开来的进一步属性。
如本文所用,术语“改善的”是指与对照环境相比或与所提及的特征相关联的已知平均量相比改善环境的特征。例如,“改善的”土壤可以指相对于未用有益微生物或微生物聚生体处理的土壤产生的植物生物质而言,在向土壤应用有益微生物或微生物聚生体后增加植物生物质生产的土壤,并且就对植物生物质生产的影响而言,其他土壤特征实质上和/或基本上相同。替代地,可以将在向土壤应用有益微生物或微生物聚生体后植物生物质的生产与土壤通常产生的平均生物质进行比较,如本领域技术人员已知的科学或农业出版物中所述。如本文所用,“改善的”不一定要求数据具有统计学上显著性(例如,p<0.05);相反,表明一个值(例如平均处理值)与另一个值(例如平均对照值)不同的任何可量化差异都可以上升到“改善的”水平。
如本文所用,术语“表型”是指单个细胞、细胞培养物、生物体或生物体群(例如,微生物聚生体)的可观察特征,其由单个细胞、细胞培养物、生物体或生物体群的基因组成(例如基因型)与环境之间的相互作用导致。
在一些实施方式中,微生物可以是环境的“内源性”。如本文所用,如果微生物源自其来源的环境,则该微生物被认为是环境的“内源性”。即,如果微生物被自然地发现与所述环境相关联,则该微生物对是环境内源性的。在将内源性微生物应用于环境的实施方式中,内源性微生物的应用量不同于特定自然环境中的水平。因此,如果微生物以非自然发生的水平存在于环境中和/或如果将微生物与对环境来说外源性和/或对环境来说内源性但以非自然发生的水平存在的其他生物体一起应用于环境,则给定环境内源性的微生物仍然可以改善环境。
在一些实施方式中,微生物对于环境可以是“外源的”(也称为“异源的”)。如本文所用,如果微生物不是源自其来源的环境,则认为该微生物对环境是“外源性的”。即,如果微生物不是自然被发现与环境相关联,则该微生物对是环境外源性的。例如,通常与第一环境相关联的微生物可被认为对于天然缺乏所述微生物的第二环境是外源性的。
如本文所用,“环境样品”是指从环境的任何部分(例如,生态系统、生态位、栖息地等)采集或获得的样品。环境样品可包括来自河流、湖泊、池塘、海洋、冰川、冰山、雨水、雪水、污水、水库、自来水、饮用水等的液体样品;来自土壤、堆肥、沙子、岩石、混凝土、木材、砖块、污物等的固体样品;以及来自空气、水下散热器、工业废气、车辆尾气等的气态样品。通常,非液态样品在用本方法分析样品之前被转化为液态。
描述
本文提供的技术涉及鉴定和分离具有靶向功能的微生物,特别地但不排他地,涉及用于筛选和/或选择提供特定功能的个体微生物或微生物聚生体的方法、组合物和系统。在某些方面,该技术提出了与微生物群落所需功能相关的问题,其中群落成员的生存或流行率增加取决于群落中的一个或多个成员对功能解决方案的响应。解决方案的遗传基础并不重要,重要的是响应生物体或微生物聚生体中一个或多个成员的相关特性。因此,选择不会偏向于特定组的基因,也不依赖于当前的知识。
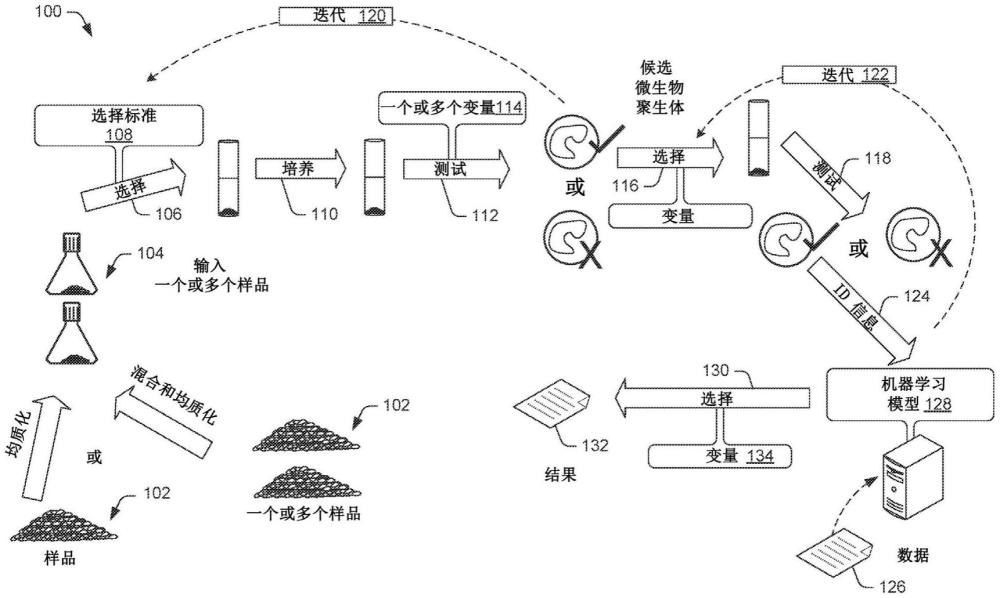
例如,如图1所示,该技术的实施方式涉及包括用于生物采矿的微生物筛选的方法。特别地,图1例示了用于以应用为中心的微生物筛选(例如,用于有效的气候变化变量和生物采矿)的示例环境100。因为以应用为中心的生物采矿专注于通过被测试变量测量的应用(例如功能)结果,而不是单个微生物表型,因此期望的应用结果不必局限于局部生态系统。因此,以应用为中心的生物采矿可用于识别可能导致更具体和/或更一般的环境(包括但不限于微环境、物种相关微生物组、生态系统、局部环境和全球环境)发生变化的微生物聚生体。
一类应用涉及影响气候变化变量。科学界众所周知,自工业革命以来,大气CO
有许多方法可以解决全球变暖问题,每种方法都有一组变量,可以在以应用为中心的生物采矿中测试这些变量以获得期望的结果。此类与全球变暖或气候变化相关的变量在本文中称为气候变化变量。一个具体的应用是使用微生物聚生体来最大限度地封存生物质中的CO
因为期望的结果是特定应用的,所以被测试的变量(例如与气候变化相关的或其他)不需要是生物学或化学因素的测量。期望的结果可能是经济的。在一个实例中,应用可能正在最大化用于执行CO
可能需要测量一些气候变化变量,以便为有关CO
在各种实施方式中,可以基于一个或多个气候变化变量来执行以应用为中心的生物采矿。例如,气候变化变量可以是对CO
在以应用为中心的生物采矿期间,可以收集一个或多个环境样品102(例如,有机物质含量高的环境样品)。如果收集单环境样品102,则方法包括将环境样品均质化以提供用于以应用为中心的生物采矿的输入样品(参见例如图1)。如果收集多个环境样品102,则方法包括将多个环境样品混合以提供混合环境样品并且将混合环境样品均质化以提供用于以应用为中心的生物采矿的输入样品(参见例如图1)。
在包括使用多个环境样品来产生输入样品的实施方式中,收集和混合多个环境样品不仅可以用来最大化要筛选的微生物的统计样品空间,而且可以用于最大化微生物聚生体中存在的微生物组合,这些微生物聚生体是使用本文描述的应用于输入样品的技术鉴定和/或产生的。此外,收集和混合多个环境样品以产生应用本文所述技术的输入样品可以通过组合在自然界中通常不生活在相同环境中的微生物来产生自然界中不存在的新型微生物聚生体。在一些实施方式中,可以混合来自不同地理区域的各种环境样品以进一步增加微生物聚生体组合的统计样品空间。例如,实施方式提供了可以获得多个环境样品,其中每个环境样品取自不同的生态系统、栖息地和/或生态位。实施方式进一步提供了,多个环境样品可从彼此分开1m、10m、100m、1000m、10,000m或超过10,000m的地点获得。在一些实施方式中,样品是从地球上任何地方的两个或更多个点获得的,包括地球陆地和水域表面之上和之下。
在一些情况下,可以在收集期间产生多个输入样品104。多个输入样品104中的每个输入样品可以包括混合在一起的单个环境样品的不同组合。例如,可以混合环境样品A、B和C(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A和B、B和C、或A和C的输入样品。作为进一步的实例,可以混合环境样品A、B、C和D(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A、B和C;A、B和D;A、C和D;或B、C和D的输入样品。作为另一个实例,可以混合环境样品A、B、C、D和E(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A和B;A和C;A和D;A和E;B和C;B和D;B和E;C和D;C和E;D和E;A、B和C;A、B和D;A、B和E;A、C和D;A、C和E;A、D和E;B、C和D;B、C和E;B、D和E;C、D和E;A、B、C和D;A、B、C和E;A、B、D和E;A、C、D和E;B、C、D和E;或A、B、C、D和E的输入样品。多个输入样品104中的每个输入样品可以包括混合在一起以提供输入样品的多个单个样品中的任意两个单个环境样品的分数组成的范围。例如,可以将任何两个单个环境样品混合在一起以提供输入样品,其包含范围从0.01至0.99的第一环境样品的分数组成(例如,包含0.01、0.05、0.10、0.20、0.30、0.40、0.50、0.60、0.70、0.80、0.90、0.95或0.99的第一环境样品)并且包含范围从0.99到0.01的第二环境样品的分数组成(例如,包含0.99、0.95、0.90、0.80、0.70、0.60、0.50、0.40、0.30、0.20、0.10、0.05或0.01的第二个环境样品)。
可以使用混合的环境样品的量和类型的变化来分离和开发输入样品104。这是因为人们认识到,微生物的组合可能不仅有益,而且还可能导致单个微生物变得不那么有效或被来自外来环境样品的微生物所支配。此外,该技术的实施方式包括使用被均质化以提供输入样品的单环境样品。本领域普通技术人员理解单环境样品可包含多个单个生态系统或生态位,它们在自然界中未混合但当单一样品被均质化时变得混合。例如,环境样品可能包含多个单独的子样品,这些子样品作为单独的地层、层或子群落存在,例如,圆柱形土壤核心样品的地层、微生物垫样品的地层、水柱样品的地层、包含生物膜的微生物群落的子群落等。
因此,本文提供的方法的实施方式包括使用被均质化以提供输入样品104的单环境样品和/或包括使用被混合和均质化以提供输入样品104的多个环境样品。
在一些实施方式中,例如,如图1所示,可以执行基于一个或多个标准108的输入样品(例如,环境样品或多个混合环境样品的混合环境样品)104的选择106。在一些情况下,至少最初的关注可能是环境样品中已知的固定碳和氮的微生物聚生体,诸如前面提到的蓝细菌和固氮菌(Azotobacter)。对特定微生物聚生体的关注可能是由目标物种驱动的。在另一个实例中,如果目标物种是豆科植物,关注可能是葡糖醋杆菌属(Gluconacetobacter)和草螺菌属(Herbaspirillium),因为它们在叶和茎中更有效地固氮,固氮螺菌属(Azospirillium),因为其也能有效地在茎和根中固氮,以及固氮菌属(Azotobacter)和拜叶林克氏菌属(Beijerinckia),因为它们能有效地在豆科植物根际中固氮。
输入样品的培养110可以在一种或多种环境条件下进行。在一些情况下,输入样品可以存储在允许光合作用的柱形物中。在一些实施方式中,提供的培养基不含氮或碳(例如,无氮和无碳培养基或“无C/N培养基”),其会干扰微生物聚生体对任何测量到的氮或碳吸收负责的确定。通过从环境浓度中供应氮或通过在缺氧N
在培养一段时间之后,可以基于一个或多个变量114执行培养物的测试112。在一些情况下,测试与期望的生物采矿结果相关的气候变化变量。在这种情况下,可以测试输入样品中碳和氮的增加或驻留微生物组固定CO
在一些情况下,可以应用添加剂来促进环境(例如土壤)或培养基对微生物聚生体的吸收。例如,微生物聚生体可能需要碳、能量、氮、微量营养素和还原当量。作为一个具体实例,水和葡萄糖喷雾可以促进环境或培养基中的大肠杆菌产生还原当量和用于CO
在一些实施方式中,统计模型和计算方法(包括机器学习)有助于选择微生物和微生物聚生体以进一步测试。机器学习模型中包含的统计模型可用于指导以应用为中心的生物采矿和传统生物采矿中的微生物选择。例如,在实验和迭代过程中,特定环境样品源位置、环境样品组成、微生物及其相关遗传生物标志物、微生物聚生体周围的数据可与各种变量(例如气候变化或其他)的结果相关联。在机器学习模型补充传统生物采矿的某些情况下,数据还可以补充有捕获微生物表型的信息。
在达到数据126的临界质量后,可以将数据开发成机器学习模型128,该机器学习模型将微生物和生物标志物以及微生物组合与被测试变量相关联。对于以应用为中心的生物采矿,用于初始测试的微生物聚生体的选择130和/或环境样品特征的选择可以由机器学习模型128建议为基于被测试变量134的结果132。对于传统的生物采矿,可以输入期望表型以及关于被测试变量134的期望结果132,并且机器学习模型可以建议相关微生物用于进一步测试。
在一些实施方式中,例如,如图2所示,计算设备支持关于以应用为中心的微生物筛选的机器学习技术用于有效变量和生物采矿。计算设备200可以提供通信接口202、一个或多个处理器204、存储器206和设备硬件208。通信接口202可以包括无线和/或有线通信组件,这些组件使设备能够向其他联网设备传输数据和从其他联网设备接收数据。设备硬件208可以包括额外的接口、数据通信或数据存储硬件。例如,硬件接口可以包括数据输出设备(例如,视觉显示器、音频扬声器)和一个或多个数据输入设备。数据输入设备可以包括但不限于小键盘、键盘、鼠标设备、接受手势的触摸屏、麦克风、语音或语音识别设备以及任何其他合适的设备中的一种或多种的组合。
存储器206可以使用计算机可读介质诸如计算机存储介质来实现。计算机可读介质至少包括两种类型的计算机可读介质,即计算机存储介质和通信介质。计算机存储介质包括以任何方法或技术实现的易失性和非易失性、可移动和不可移动介质,用于存储信息,诸如计算机可读指令、数据结构、程序模块或其他数据。计算机存储介质包括但不限于RAM、ROM、EEPROM、闪存或其他存储技术、CD-ROM、数字通用光盘(DVD)、高清多媒体/数据存储盘或其他光学存储、磁性盒式磁带(magnetic cassette)、磁带、磁盘存储器或其他磁性存储设备,或任何其他可用于存储信息以供计算设备访问的非传输介质。计算设备200也可以是虚拟机或虚拟机上的容器的形式,诸如通过Kubernetes或Docker提供的。在这种情况下,虚拟机托管在物理计算机平台上,并通过管理程序提供服务。通俗地说,虚拟机配置可称为“云”。
计算设备200的处理器204和存储器206可以实现操作系统210。反过来,操作系统210可以为机器学习平台212提供实行环境。操作系统210可以包括使计算设备200能够经由各种接口(例如,用户控制、通信接口和/或存储器输入/输出设备)接收和传输数据以及使用处理器204处理数据以生成输出的组件。操作系统210可以包括呈现输出(例如,在电子显示器上显示数据、将数据存储在存储器中、将数据传输到另一个电子设备等)的呈现组件。另外,操作系统210可以包括执行通常与操作系统相关联的各种附加功能的其他组件。
机器学习平台212可以包括数据输入模块214、模型生成模块216和选择模块218。这些模块可以包括执行特定任务或实现特定抽象数据类型的例程、程序指令、对象和/或数据结构。存储器206还可以包括由机器学习平台212使用的数据存储220。
数据输入模块214可以从各种来源接收数据,诸如数据库或经由用户界面输入的数据。数据输入模块214可以使用数据适配器从数据源的数据库中检索数据。例如,数据输入模块214可以使用与数据无关的数据适配器来访问非结构化数据库,和/或使用特定于数据库的数据适配器来访问结构化数据库。接收到的数据可能包括微生物的识别信息,诸如DNA生物标志物、表型信息、环境变量(例如,营养素类型、CO
模型生成模块216可以通过模型训练算法来训练机器学习模型,诸如机器学习模型128。模型训练算法可以实现训练数据输入期、特征工程期和模型生成期。在训练数据输入期,模型训练算法可以接收训练数据,诸如经由数据输入模块214接收的数据。在特征工程期期间,模型训练算法可以精确定位训练数据中的特征。因此,模型训练算法可以使用特征工程来找出训练数据中有助于机器学习模型区分不同数据类别的重要属性和关系。在模型生成期期间,模型训练算法可以选择初始类型的机器学习算法来使用训练数据训练机器学习模型。在将选定的机器学习算法应用于训练数据之后,模型训练算法可以确定机器学习模型的训练误差测量。如果训练误差测量超过训练误差阈值,则模型训练算法可以使用规则引擎基于训练误差测量的量级来选择不同类型的机器学习算法。不同类型的机器学习算法可以包括贝叶斯算法、决策树算法、支持向量机(SVM)算法、集成树算法(例如,随机森林和梯度提升树)、人工神经网络和/或等等。一般重复训练过程,直到训练结果低于训练误差阈值,并且生成经训练的机器学习模型。经训练的机器学习模型128可以存储在数据存储220中。
选择模块218可以将经训练的机器学习模型128应用于一个或多个查询变量值以生成用于生物采矿的查询结果。在一些情况下,通过选择模块218将机器学习模型128应用于查询变量值可以建议用于启动测试的微生物聚生体的选择和/或环境样品特征的选择。在其他情况下,可以输入期望的表型以及被测试变量(例如,固氮量、微生物的存活时间和/或等等)的期望结果(例如,期望的CO
因此,利用统计学上显著的数据量,可以开发机器学习模型来帮助选择微生物和微生物聚生体。如果机器学习模型补充有组成微生物的表型数据,机器学习模型也可以增强传统的生物采矿。
在一些实施方式中,例如,如图3a和图3b所示,该技术提供了用于执行以应用为中心的微生物筛选方法的过程(例如,过程300)。在示例过程300中描述操作的顺序不旨在被解释为限制,并且可以以任何顺序和/或并行地组合任何数量的所描述的框以实现该过程。此外,在一些实施方式中,可以通过获得若干环境样品302并将环境样品混合成混合样品的组合304以提供若干输入样品(例如,混合样品)在步骤306处选择来执行过程300。在一些实施方式中,单环境样品可以被均质化以提供在步骤306处选择用于输入的输入样品。在一些实施方式中,若干单环境样品和/或若干混合环境样品可以提供多个输入样品用于在步骤306处选择。因此,虽然本文描述的和图3a和图3b中的方法是在获得若干环境样品302并混合多个环境样品304,并且选择混合环境样品306用于在步骤308处培养方面来描述的,但该技术不限于包括混合多个环境样品的方法,并且包括其中将单环境样品均质化并作为选定样品提供用于在步骤308处培养的实施方式。此外,实施方式包括产生和提供多个均质化的单环境样品,并从多个均质化的单环境样品中选择均质化的单环境样品用于在步骤308处培养。
因此,用于执行以应用为中心的微生物筛选方法的过程(例如,过程300)的步骤(例如,步骤302、304和306)应被理解为包括提供若干(例如,一个或多个)通过混合和均质化多个环境样品产生的混合环境样品的步骤,或理解为包括提供若干(例如,一个或多个)均质化的单环境样品的步骤,用于在步骤308处培养。在方法的整个描述中提及混合环境样品应理解为指的是通过混合和均质化多个环境样品而产生的混合环境样品,或指的是均质化的单环境样品。在方法的整个描述中提及多个混合环境样品应被理解为指的是多个混合环境样品,其中多个混合环境样品中的每个混合环境样品是通过混合和均质化多个环境样品而产生的和/或是均质化的单环境样品。
在一些实施方式中,例如,在框302,可以获得包括有机物质的多个环境样品用于生物采矿。在框304,可以将多个环境样品混合成混合环境样品的组合。例如,多个环境样品可能来自不同的地理区域,使得环境样品包含不同的微生物聚生体。混合304可以通过环境样品的量和类型的变化来执行以用于最大化微生物组合。例如,可以混合(例如,在框304)环境样品A、B和C(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A和B、B和C、或A和C的输入样品。作为进一步的实例,可以混合(例如,在框304)环境样品A、B、C和D(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A、B和C;A、B和D;A、C和D;或B、C和D的输入样品。作为另一个实例,可以混合(例如,在框304)环境样品A、B、C、D和E(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A和B;A和C;A和D;A和E;B和C;B和D;B和E;C和D;C和E;D和E;A、B和C;A、B和D;A、B和E;A、C和D;A、C和E;A、D和E;B、C和D;B、C和E;B、D和E;C、D和E;A、B、C和D;A、B、C和E;A、B、D和E;A、C、D和E;B、C、D和E;或A、B、C、D和E的输入样品。多个输入样品中的每个输入样品可以包括混合在一起以提供输入样品的多个单独样品中的任意两个单独环境样品的分数组成的范围。例如,可以将任何两个单个环境样品混合在一起以提供输入样品,其包含范围从0.01至0.99的第一环境样品的分数组成(例如,包含0.01、0.05、0.10、0.20、0.30、0.40、0.50、0.60、0.70、0.80、0.90、0.95或0.99的第一环境样品)并且包含范围从0.99到0.01的第二环境样品的分数组成(例如,包含0.99、0.95、0.90、0.80、0.70、0.60、0.50、0.40、0.30、0.20、0.10、0.05或0.01的第二个环境样品)。
在框306,基于一个或多个选择标准可以选择所述混合环境样品中的特定混合环境样品用于测试。例如,在一些情况下,可以基于混合环境样品是否至少包含某些微生物物种和/或是否表现出某些特性(例如,功能)诸如固定一定量的氮或固定一定量的碳的能力来选择混合环境样品。在框308,可以在包括一种或多种环境条件的环境中培养所选择的混合环境样品。例如,环境条件可以包括特定浓度的N
在框310,可以基于从所选择的混合环境样品的培养产生的一个或多个变量测量来确定特定混合环境样品是否产生了成功的生物采矿结果。在各种实施方式中,变量可以包括变量(例如,气候变化变量),诸如培养物中生物质封存的CO
在框314,可以获得存在于所选择的混合环境样品的相应微生物聚生体中的微生物的识别信息。例如,包含相应微生物聚生体的微生物的DNA可以被分离和测序(例如,如本文所述)。在每种微生物的DNA中,可以鉴定生物标志物,诸如16SrRNA或GroEL。
在框316,可以将所选择的混合环境样品的相应微生物聚生体培养成微生物培养物。在框318,可以选择微生物培养物的培养物部分用于测试。在一些情况下,培养物部分可以是随机选择的培养物部分。在其他情况下,可以基于培养物部分是否至少包含某些微生物物种和/或是否能够展示某些特性(例如,功能)诸如固定一定量的氮、固定一定量的碳的能力和/或具有一定的生存时间/持久性来选择培养物部分。在框320,可以使微生物培养物的所选择的培养物部分在包括一种或多种环境条件的环境中生长。例如,环境条件可以包括特定浓度的N
在框322,可以基于所选择的培养物部分的一个或多个变量测量来确定所选择的培养物部分是否产生了成功的微生物生物采矿结果。在各种实施方式中,变量可以包括变量(例如,气候变化变量),诸如所选择的培养物部分的生物质封存的CO
在决策框324,如果所选择的培养物部分产生了成功的生物采矿结果(决策框324的“是”),则过程300可以进行到框326。在框326,可以获得存在于所选择的培养物部分的相应微生物聚生体中的微生物的识别信息。例如,包含相应微生物聚生体的微生物的DNA可以被分离和测序。在每种微生物的DNA中,可以鉴定生物标志物,诸如16S rRNA或GroEL。
随后,过程300可以进行到决策框328。返回到决策框324,如果所选择的培养物部分没有产生成功的生物采矿结果(在决策框324处为“否”),则过程300可以直接进行到决策框328。在决策框328,如果培养物中有更多的培养物部分要测试(决策框328处的“是”),则过程300可以进行到框330。例如,如果选择用于测试的微生物培养物的培养物部分的数量尚未达到阈值测试数量,如果微生物培养物的成功生物采矿结果的数量尚未达到成功阈值数量,或者如果仍有一部分微生物培养物用于测试,则可能有更多的培养物部分要测试。在框330,可以选择微生物培养物的额外培养物部分用于测试。随后,过程300可以返回到框320。
然而,如果培养物中没有更多的培养物部分要测试(决策框328处的“否”),则过程300可以进行到决策框332。在决策框332,如果有更多的混合环境样品要测试(决策框332的“是”),过程300可以进行到框334。在框334,可以基于一个或多个选择标准来选择额外混合环境样品用于测试。然而,如果没有更多的混合环境样品要测试(决策框332处的“否”),则过程300可以在框334处终止,使得测试结束。例如,如果选择用于测试的混合环境样品的数量尚未达到阈值测试数量,如果混合环境样品的组合的成功生物采矿结果的数量尚未达到成功阈值数量,或者如果仍有混合环境样品用于测试,则可能有更多的混合环境样品要测试。
返回到决策框312,如果所选择的混合环境样品的培养没有产生成功的结果(决策框312处的“否”),则过程300可以直接进行到决策框332。在一些替代实施方式中,过程300可以直接从框314进行到决策框332,而不是在进行到决策框332之前进行通过框316-330。
在一些实施方式中,例如,如图4所示,该技术提供机器学习技术来鉴定微生物物种和与一个或多个变量相关的其他信息。示例过程400被例示为逻辑流程图中的框的集合,其表示可以在硬件、软件或其组合中实现的操作顺序。在软件的上下文中,框表示计算机可执行指令,当由一个或多个处理器实行时,这些指令执行所述操作。通常,计算机可执行指令可以包括执行特定功能或实现特定抽象数据类型的例程、代码段、程序、对象、组件、数据结构等。描述操作的顺序不旨在被解释为限制,并且可以以任何顺序和/或并行地组合任何数量的所描述的框以实现该过程。
在框402,机器学习平台可以生成机器学习模型,该机器学习模型至少将环境样品的一个或多个环境样品变量值与存在于一个或多个环境样品中的一个或多个微生物物种和一个或多个微生物聚生体的微生物变量值相关联。在各种实施方式中,机器学习模型可以进一步将这样的变量值与环境变量(例如,气候变化变量)的值相关联。例如,在微生物识别过程(诸如过程300)的实验和迭代过程中,与特定环境样品源位置、环境样品组成、微生物及其相关的遗传生物标志物、微生物聚生体相关的数据可能与各种变量(例如,气候变化或其他)的结果相关联并用作生成机器学习模型的训练数据。其他相关变量可能包括生物质与固定氮的绝对量的质量比、源自生物质封存CO
在框404,机器学习平台可以接收一个或多个变量值的输入。例如,变量值可以包括微生物的表型、期望的固氮量、期望的碳封存量、环境样品特征、一个或多个气候变化变量,和/或等等。在框406,机器学习平台可以包括对与一个或多个变量值相关的信息的请求。在决策框408,如果所请求的信息包括与一个或多个变量值相关的微生物物种,则过程400可以进行到框410。在框410,机器学习平台可以将机器学习模型应用于一个或多个变量以鉴定与一个或多个变量值相关联的一个或多个微生物物种。在一些情况下,机器学习模型建议的微生物物种可用于进一步测试微生物生物采矿。
返回到决策框408,如果所请求的信息包括与一个或多个变量相关的环境特征,则过程400可以进行到框412。在框412,机器学习平台可以将机器学习模型应用于一个或多个变量以鉴定与一个或多个变量值相关联的一个或多个环境特征。
返回到决策框408,如果所请求的信息包括与一个或多个变量相关的微生物聚生体,则过程400可以进行到框414。在框414,机器学习平台可以将机器学习模块应用于一个或多个变量值以鉴定与一个或多个变量值相关联的至少一个微生物聚生体。在一些情况下,机器学习模型建议的微生物聚生体可用于进一步测试微生物生物采矿。
与常规生物采矿相比,本文描述的以应用为中心的生物采矿技术从大量微生物样品(例如,来自一个或多个环境样品)和微生物聚生体(例如,包含来自自然聚生体的一种或多种微生物和/或来自不同环境、生态系统、栖息地和/或生态位的一种或多种微生物)开始,并产生包含协同作用的微生物的新组合的新聚生体。通过测试特定于应用的变量,提供期望结果的微生物和微生物聚生体然后可以被测序,然后传代培养直到鉴定和/或分离期望的微生物和微生物聚生体。利用统计上学上显著的数据量,可以开发机器学习模型来帮助选择微生物和微生物聚生体。如果机器学习模型补充有组成微生物的表型数据,机器学习模型也可以增强传统的生物采矿。
如本文所述,以应用为中心的生物采矿专注于被测试变量而不是微生物的潜在表型。被测试变量可能是感兴趣的直接变量,诸如碳封存的测量和相关的因变量(诸如固氮)的测量。变量在本质上可以是生物学的,诸如微生物的存活时间和/或持久性。由于被测试变量与微生物的表型或其他未直接相关,因此被测试变量可以是全球性的,诸如对全球气候变化和/或全球食物生产的影响。此外,被测试变量在本质上不必是生物或化学的。被测试变量可以是经济的,诸如碳封存的收益,以及食物生产与碳封存的比较。
在与农业和土壤相关的特定实例中,这种对被测试变量的解耦能够对农业进行广范围的更细粒度分析。过去,一个农场的影响可以通过土壤管理技术和作物管理技术来区分。然而,特定于应用的生物采矿提供了选择变量并找到微生物聚生体以最大化期望结果的能力。由于这些结果不必是农业变量,特定于应用的生物采矿提高了将农业绩效和生产与任意变量诸如农业经济和气候变化联系起来的能力。
在一些实施方式中,该技术提供了用于选择提供特定功能的微生物聚生体的额外方法。在一些实施方式中,该技术提供了用于筛选微生物群落、微生物聚生体和/或多种微生物以产生和/或鉴定提供特定功能的微生物聚生体的方法。在一些实施方式中,该技术通过组合来自不同环境、生态位和/或栖息地的微生物(例如,在自然界中不一起存在的微生物)来产生在自然界中不存在的微生物聚生体。
在一些实施方式中,例如,如图5所示,该技术提供了产生具有特定功能的微生物聚生体的方法。方法包括提供(501)包含多种微生物的样品;用样品的一部分接种(502)第N体积的生长培养基以提供第N培养物;使第N培养物在一组选择性条件下生长(503);在第N培养物中产生(504)微生物的第N分类学分类;用第N培养物的一部分接种(505)第N+1体积的生长培养基;使第N+1培养物在该组选择性条件下生长(506);在第N+1培养物中产生(507)微生物的第N+1分类学分类;以及使用第N+1分类学分类和第N分类学分类得出(508)第N+1培养物相对于第N培养物的微生物群落稳定性的量度。监测微生物群落稳定性的量度以鉴定微生物群落稳定性的量度已达到平台值。如果微生物群落稳定性的量度没有达到平台值(509),则通过提供(510)第N+1样品作为步骤505处的第N样品再次执行步骤505-508。如果微生物群落稳定性的量度已达到平台值(509),该方法包括提供(511)稳定的第N+1培养物作为包含执行特定功能的微生物聚生体的培养物。在一些实施方式中,步骤505-508重复2、3、4、5、6、7、8、9或10次或更多次。
在一些实施方式中,方法还包括在纯培养物中分离稳定微生物聚生体的每种微生物。在一些实施方式中,方法还包括获得纯培养物中稳定微生物聚生体的每种微生物的基因组序列。在一些实施方式中,方法还包括储存稳定的微生物聚生体和/或稳定的微生物聚生体的每种微生物(例如,通过冷冻(例如,在–80C))。在一些实施方式中,方法还包括使用测试底物和测量功能输出的方法测量稳定的微生物聚生体的特定功能。
该技术不限于用作对其执行如本文所述的方法(例如,选择微生物聚生体的方法和/或筛选以鉴定微生物聚生体的方法)的起始材料(例如输入样品)的包含微生物的样品(例如环境样品)的类型。在一些实施方式中,所使用的输入样品可以是来自任何来源的环境样品,例如,天然存在的或人造的大气、水系统和来源、土壤或任何其他感兴趣的样品。在一些实施方式中,环境样品可获自例如室内或室外空气或大气颗粒收集系统;室内表面和机器、设备或仪器的表面。在一些实施方式中,对生态系统进行采样(例如,在一些实施方式中,样品是从生态系统中取得的环境样品)。生态系统可以是陆地的,包括所有已知的陆地环境,包括但不限于土壤、地表和地表以上环境。生态系统包括美国林业局(United StatesForest Service)开发的粮食及农业组织土地覆盖分类系统(LCCS)和森林范围环境研究生态系统(FRES)中分类的生态系统。示例性的生态系统包括森林,诸如热带雨林、温带雨林、温带阔叶林、北方森林、针叶林和山地针叶林;草地,包括稀树草原和草原;沙漠;湿地,包括草本沼泽(泥潭)、沼泽、泥炭沼泽(泥塘)、河口和泥沼;河岸生态系统、高山和苔原生态系统。生态系统还包括与水生环境相关联的生态系统,诸如湖泊、河流、泉水、珊瑚礁、海滩、河口、海山、海沟和潮间带。生态系统还包括土壤、腐殖质、矿质土壤和含水层。生态系统还涵盖地下环境,诸如矿山、油田、洞穴、断层和断裂带、地热带和含水层。生态系统还包括与植物、动物和人类相关联的微生物组。示例性植物相关联的微生物组包括在根、树皮、树干、树叶和花中或附近发现的微生物组。动物和人类相关联的微生物组包括在胃肠道、呼吸系统、鼻孔、泌尿生殖道、乳腺、口腔、耳道、粪便、尿液和皮肤中发现的微生物组。在一些实施方式中,样品可以是任何种类的临床或医学样品。例如,样品可以来自哺乳动物的血液、尿液、粪便、鼻孔、肺或肠道。
例如,在一些实施方式中,收集一个或多个环境样品。如果收集单环境样品,则方法包括使环境样品均质化以提供输入样品(例如,在框501处)。如果收集了多个环境样品,则方法包括混合多个环境样品以提供混合环境样品以及使混合环境样品均质化以提供输入样品(例如,在框501处)。
在包括使用多个环境样品来产生输入样品的实施方式中,收集和混合多个环境样品不仅可以用来最大化要筛选的微生物的统计样品空间,而且可以用于最大化微生物聚生体中存在的微生物组合,这些微生物聚生体是使用本文描述的应用于输入样品的技术鉴定和/或产生的。此外,收集和混合多个环境样品以产生应用本文所述技术的输入样品可以通过组合在自然界中通常不生活在相同环境中的微生物来产生自然界中不存在的新型微生物聚生体。在一些实施方式中,可以混合来自不同地理区域的各种环境样品以进一步增加微生物聚生体组合的统计样品空间。例如,实施方式提供了可以获得多个环境样品,其中每个环境样品取自不同的生态系统、栖息地和/或生态位。实施方式进一步提供了,多个环境样品可从彼此分开1m、10m、100m、1000m、10,000m或超过10,000m的地点获得。在一些实施方式中,样品是从地球上任何地方的两个或更多个点获得的,包括地球陆地和水域表面之上和之下。
在一些情况下,可以在收集期间产生多个输入样品。多个输入样品中的每个输入样品可以包括混合在一起的单个环境样品的不同组合。例如,可以混合环境样品A、B和C(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A和B、B和C、或A和C的输入样品。作为进一步的实例,可以混合环境样品A、B、C和D(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A、B和C;A、B和D;A、C和D;或B、C和D的输入样品。作为另一个实例,可以混合环境样品A、B、C、D和E(来自一个或多个不同的生态系统、栖息地和/或生态位)以提供包含A和B;A和C;A和D;A和E;B和C;B和D;B和E;C和D;C和E;D和E;A、B和C;A、B和D;A、B和E;A、C和D;A、C和E;A、D和E;B、C和D;B、C和E;B、D和E;C、D和E;A、B、C和D;A、B、C和E;A、B、D和E;A、C、D和E;B、C、D和E;或A、B、C、D和E的输入样品。多个输入样品中的每个输入样品可以包括混合在一起以提供输入样品的多个单个样品中的任意两个单个环境样品的分数组成的范围。例如,可以将任何两个单个环境样品混合在一起以提供输入样品,其包含范围从0.01至0.99的第一环境样品的分数组成(例如,包含0.01、0.05、0.10、0.20、0.30、0.40、0.50、0.60、0.70、0.80、0.90、0.95或0.99的第一环境样品)并且包含范围从0.99到0.01的第二环境样品的分数组成(例如,包含0.99、0.95、0.90、0.80、0.70、0.60、0.50、0.40、0.30、0.20、0.10、0.05或0.01的第二个环境样品)。
可以使用混合的环境样品的量和类型的变化来分离和开发输入样品。这是因为人们认识到,微生物的组合可能不仅有益,而且还可能导致单个微生物变得不那么有效或被来自外来环境样品的微生物所支配。此外,该技术的实施方式包括使用被均质化以提供输入样品的单环境样品。本领域普通技术人员理解单环境样品可包含多个单个生态系统或生态位,它们在自然界中未混合但当单一样品被均质化时变得混合。例如,环境样品可能包含多个单独的子样品,这些子样品作为单独的地层、层或子群落存在,例如,圆柱形土壤核心样品的地层、微生物垫样品的地层、水柱样品的地层、包含生物膜的微生物群落的子群落等。
因此,本文提供的方法的实施方式包括使用被均质化以在步骤501处提供输入样品的单环境样品和/或包括使用被混合和均质化以在步骤501处提供输入样品的多个环境样品。
该技术提供了用于降低微生物群落(例如,存在于环境样品中)的复杂性同时选择执行特定功能的微生物聚生体和/或鉴定执行特定功能的微生物聚生体的方法。可以选择和/或鉴定微生物聚生体的示例性功能包括例如生物降解、发酵、化学前体的产生、生物传感、固氮和碳固定。
在一些实施方式中,将环境样品用于接种培养基并且接种的培养基在培养基提供的选择性条件(例如,碳源的存在、不存在或类型;氮源的存在、不存在或类型;辅因子、矿物质、维生素或其他营养素的存在、不存在或类型;阳离子和/或阴离子的存在、不存在或类型;微量矿物质、阳离子和/或阴离子的存在、不存在或类型;固体生长基质(诸如沙子或其他固体基质)的存在、不存在或类型)或通过生长培养基外部提供的选择性条件(例如,温度;湿度;光的存在、不存在、波长和/或强度;光/暗周期;压力;培养体积;培养体积材料、尺寸或几何形状;培养物搅动的存在、不存在、类型或强度;提供的气体的存在、不存在和/或类型)下生长。
在一些实施方式中,将培养物接种(例如,在步骤502和/或步骤505)并生长(例如,在步骤503和/或步骤506)一段时间,例如30至60分钟(例如,30.0、30.5、31.0、31.5、32.0、32.5、33.0、33.5、34.0、34.5、35.0、35.5、36.0、36.5、37.0、37.5、38.0、38.5、39.0、39.5、40.0、40.5、41.0、41.5、42.0、42.5、43.0、43.5、44.0、44.5、45.0、45.5、46.0、46.5、47.0、47.5、48.0、48.5、49.0、49.5、50.0、50.5、51.0、51.5、52.0、52.5、53.0、53.5、54.0、54.5、55.0、55.5、56.0、56.5、57.0、57.5、58.0、58.5、59.0、59.5或60.0分钟);1至24小时(例如,1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5、10.0、10.5、11.0、11.5、12.0、12.5、13.0、13.5、14.0、14.5、15.0、15.5、16.0、16.5、17.0、17.5、18.0、18.5、19.0、19.5、20.0、20.5、21.0、21.5、22.0、22.5、23.0、23.5或24.0小时);1至30天(例如,1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5、10.0、10.5、11.0、11.5、12.0、12.5、13.0、13.5、14.0、14.5、15.0、15.5、16.0、16.5、17.0、17.5、18.0、18.5、19.0、19.5、20.0、20.5、21.0、21.5、22.0、22.5、23.0、23.5、24.0、24.5、25.0、25.5、26.0、26.5、27.0、27.5、28.0、28.5、29.0、29.5或30.0天);和/或1至10周(例如,1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5或10.0周)。
在一些实施方式中,测量生长速率、指数生长期的时间、培养物饱和的时间或其他培养物生长特征的经验测量以鉴定用于培养物生长的时间长度。在一些实施方式中,选择在指数生长期结束或接近结束时提供培养物的生长时间,以提供具有稳健类型和数量的微生物的培养物用于进一步表征和/或选择。在一些实施方式中,使用确定的培养物体积中微生物的绝对或相对数量的测量来定量和/或定性地测量生长。在一些实施方式中,通过使用光散射,测量从培养物中分离的固体(例如细胞)的干质量或湿质量,使用培养物的一部分对在固体培养基上生长的菌落进行计数,或测量培养物或其一部分的与培养物中的微生物数量具有相关或因果关系的一些其他特征,来测量一定体积培养物中微生物的绝对或相对数量。在一些实施方式中,生长的特征在于确定生长曲线;在一些实施方式中,生长的特征在于确定倍增时间和/或半饱和时间。在一些实施方式中,使用经验数据(例如,使用对数增长模型)对生长速率进行建模。
在一些实施方式中,培养物中的微生物通过鸟枪法宏基因组测序来表征(例如,在步骤507)。从样品(诸如环境或临床样品)中的多种生物体获得基因序列的技术和系统是本领域技术人员众所周知的。例如,Zhou等人(Appl.Environ.Microbiol.(1996)62:316-322)提供了稳健的核酸提取和纯化。该方案也可以根据实验目标和环境样品类型(诸如土壤、沉积物和地下水)进行修改。也可以使用许多市售的DNA提取和纯化试剂盒。具有低于2pg的纯化DNA的样品可能需要扩增,这可以使用本领域已知的常规技术诸如全群落基因组扩增(WCGA)方法(Wu et al.,Appl.Environ.Microbiol.(2006)72,4931-4941)进行。用于从环境样品中获得纯化的RNA的技术和系统也是本领域技术人员众所周知的。例如,可以使用Hurt等人(Appl.Environ.Microbiol.(2001)67:4495-4503)描述的方法。这种方法可以在同一样品中同时分离DNA和RNA。凝胶电泳方法也可用于分离群落RNA(McGrath et al.,J.Microbiol.Methods(2008)75:172-176)。具有低于5pg的纯化RNA的样品可能需要扩增,这可以使用本领域已知的常规技术例如全群落RNA扩增方法(WCRA)(Gao et al.,Appl.Environ.Microbiol.(2007)73:563-571)进行以获得cDNA。在一些实施方式中,环境采样和DNA提取如前所述进行(DeSantis et al.,Microbial Ecology,53(3):371-383,2007)。
可以对分离的核酸(例如,宏基因组DNA)进行测序方法以获得宏基因组测序数据。测序方法可大致分为通常使用模板扩增的方法和不使用模板扩增的那些。需要扩增的方法包括由罗氏作为454技术平台(例如GS 20和GS FLX)商业化的焦磷酸测序、LifeTechnologies/Ion Torrent、由Illumina商业化的Solexa平台、GnuBio以及由AppliedBiosystems商业化的Supported Oligonucleotide Ligation and Detection(SOLiD)平台。非扩增方法,也称为单分子测序,例如由Helicos BioSciences商业化的HeliScope平台,以及分别由VisiGen、Oxford Nanopore Technologies Ltd.和PacificBiosciences商业化的新兴平台。因此,在一些实施方式中,宏基因组鸟枪法测序包括焦磷酸测序、连接测序、单分子测序、边合成边测序(SBS)、半导体测序、纳米孔测序、大规模平行克隆、大规模平行单分子SBS、大规模平行单分子实时、大规模平行单分子实时纳米孔技术等。Morozova和Marra在Genomics,92:255(2008)中提供了一些此类技术的综述,其全部内容通过引用并入本文。本领域普通技术人员会认识到,由于RNA在细胞中稳定性较差并且更容易受到核酸酶的攻击,在实验中RNA通常在测序前逆转录为DNA。
一些DNA测序技术的具体描述包括基于荧光的测序方法(参见,例如,Birren etal.,Genome Analysis:Analyzing DNA,1,Cold Spring Harbor,N.Y.;其通过引用整体并入本文);自动化测序技术;分区扩增子的平行测序(Kevin McKernan等人的PCT公开号:WO2006084132,其通过引用整体并入本文);以及通过平行寡核苷酸延伸的测序(参见,例如,Macevicz等人的美国专利号5,750,341和Macevicz等人的美国专利号6,306,597,两者均通过引用整体并入本文)。测序技术的其他描述包括Church polony技术(Mitra et al.,2003,Analytical Biochemistry 320,55-65;Shendure et al.,2005Science 309,1728-1732;美国专利号6,432,360、美国专利号6,485,944、美国专利号6,511,803;它们通过引用整体并入本文),454皮滴定焦磷酸测序技术(Margulies et al.,2005Nature 437,376-380;US 20050130173;它们通过引用整体并入本文),Solexa单碱基添加技术(Bennett etal.,2005,Pharmacogenomics,6,373-382;美国专利号6,787,308;美国专利号6,833,246;它们通过引用整体并入本文),Lynx大规模平行签名测序技术(Brenner et al.(2000).Nat.Biotechnol.18:630-634;美国专利号5,695,934;美国专利号5,714,330;它们通过引用整体并入本文),以及Adessi PCR菌落技术(Adessi et al.(2000).Nucleic AcidRes.28,E87;WO00018957;其通过引用整体并入本文)。还参见,例如,Voelkerding et al.,Clinical Chem.,55:641-658,2009;MacLean et al.,Nature Rev.Microbiol.,7:287-296;每一个都通过引用整体并入本文)。
可以分析宏基因组核苷酸序列数据以表征从其获得宏基因组核酸的微生物群落(例如,微生物聚生体)(例如,在步骤507)。例如,在一些实施方式中,通过从微生物群落获得宏基因组核苷酸序列数据并使用将宏基因组核苷酸序列数据中的短基因组子串(k-mers)与最低共同祖先(LCA)分类群相关联的算法(例如,使用精选数据库),对微生物群落中的分类单元在分类学上分类和/或鉴定。参见,例如,Wood(2014)“Kraken:ultrafastmetagenomic sequence classification using exact alignments”Genome Biology 15:R46和Wood(2019)“Improved metagenomic analysis with Kraken 2”Genome Biology20:257,每一篇都通过引用并入本文。在一些实施方式中,BLAST用于鉴定存在于微生物群落(例如,微生物聚生体)中的微生物分类单元。参见,例如,Altschul(1990)“Basic localalignment search tool”J Mol Biol215:403–410,通过引用并入本文。用于使用来自微生物群落的宏基因组序列数据鉴定微生物群落中的分类单元的其他工具包括,例如,MEGAN(参见,例如,Huson(2007)“MEGAN analysis of metagenomic data”Genome Res 17:377–386,通过引用并入本文);PhymmBL(参见,例如,Brady(2009)“Phymm and PhymmBL:metagenomic phylogenetic classification with interpolated Markov models”NatMethods 6:673–676;和Brady(2011)“PhymmBL expanded:confidence scores,customdatabases,parallelization and more”Nat Methods 8:367,每一篇都通过引用并入本文);和朴素贝叶斯分类器(NBC)(参见,例如,Rosen(2008)“Metagenome fragmentclassification using N-mer frequency profiles”Adv Bioinformatics 2008:1–12,通过引用并入本文)。
在一些实施方式中,表征微生物群落包括以绝对和/或相对术语鉴定微生物群落中存在的生物体的分类单元(例如,菌株、亚种、种、属、科)。在一些实施方式中,表征微生物群落包括例如以相对术语鉴定相对于先前传代或初始环境样品在特定传代中富集的生物体的分类单元(例如菌株、亚种、种、属、科)。
在一些实施方式中,该技术提供迭代方法(例如,方法500包括步骤505至510的迭代),其中第一培养物的一部分用于接种第二体积的新鲜培养基。因此,在一些实施方式中,第一培养物(例如,通过用环境样品接种选择性生长培养基产生的培养物)的一部分被用于接种第二培养物(例如,包含与第一样品中相同或不同的生长培养基)。在一些实施方式中,第二培养物的一部分用于接种第三培养物。在一些实施方式中,第三培养物的一部分用于接种第四培养物。在一些实施方式中,第四培养物的一部分用于接种第五培养物。在一些实施方式中,第五培养物的一部分用于接种第六培养物。在一些实施方式中,第六培养物的一部分用于接种第七培养物。在一些实施方式中,第七培养物的一部分用于接种第八培养物。在一些实施方式中,第N培养物的一部分用于接种第N+1培养物。在一些实施方式中,第N培养物是使用环境样品的至少一部分接种的第一培养物。在一些实施方式中,第N种培养物是使用先前培养物(例如,分别为第一、第二、第三、第四、第五、第六或第七培养物)接种培养物的至少一部分接种的第二、第三、第四、第五、第六、第七、第八等培养物。如本文所用,通过使用第N培养物的一部分接种第N+1培养物的迭代培养过程称为培养物的“传代”。
此外,直接从环境样品接种的培养物在本文中可称为P0(零)培养物;第一传代包括使用P0培养物的一部分接种新鲜培养基以产生P1培养物;第二传代包括使用P1培养物的一部分接种新鲜培养基产生P2培养物;第三传代包括使用P2培养物的一部分接种新鲜培养基产生P3培养物;第四传代包括使用P3培养物的一部分接种新鲜培养基产生P4培养物;第五传代包括使用P4培养物的一部分接种新鲜培养基产生P5培养物;第六传代包括使用P5培养物的一部分接种新鲜培养基产生P6培养物;第七传代包括使用P6培养物的一部分接种新鲜培养基产生P7培养物;第八传代包括使用P7培养物的一部分接种新鲜培养基产生P8培养物;以及第N传代包括使用P(N-1)培养物的一部分产生PN培养物。如本文所用,术语“传代数”是指由数字指示的特定传代,例如,传代数1是指第一传代,传代数2是指第二传代等。
在一些实施方式中,用于接种第N+1(例如,第二)培养物的第N(例如,第一)培养物的一部分的体积为100μl至100L或更多,这取决于培养过程的规模(例如,从研究规模到中试规模再到商业生产规模)。因此,实施方式提供从一种培养物中去除100μl至100L(例如,100、150、200、250、300、350、400、450、500、550、600、650、700、750、800、850、900、950或1000μl;1、2、5、10、20、50、100、150、200、250、300、350、400、450、500、550、600、650、700、750、800、850、900、950或1000mL;或1、2、5、10、20、50或100L)的体积并将该体积添加到新鲜培养基中。在一些实施方式中,接种体积与新鲜培养基的体积之比为大约1:10至1:1000。因此,在一些实施方式中,新鲜培养基的体积为1ml至100,000L(例如,1;2;5;10;20;50;100;200;500;或1000mL;1;2;5;10;20;50;100;200;500;1000;2000;5000;10,000;20,000;50,000;或100,000L)。
在一些实施方式中,测量微生物群落和/或微生物聚生体的稳定性(例如,在步骤508),例如,通过得出第一培养物和使用第一培养物的一部分接种的第二培养物之间的相似性(或相异性)的量度,并且,任选地,遵循作为随后接种的函数的相似性的量度。在一些实施方式中,微生物群落中生物体的分类学分类和/或鉴定(例如,如通过上文描述的分类学分类器(例如,Kraken2)所提供的)可以为此类稳定性测量提供输入。在一些实施方式中,由微生物群落提供和/或存在于微生物群落中的功能能力或功能(例如,基因、基因产物、功能能力和/或活动)为稳定性测量提供输入。
可以使用各种措施来比较微生物群落的相似性(或相异性),包括估计微生物群落丰富度和多样性(参见,例如,Hughes(2001)“Counting the uncountable:statisticalapproaches to estimating microbial diversity”Appl.Environ.Microbiol.67:4399-4406;和Ley(2005)“Obesity alters gut microbial ecology”Proc.Natl.Acad.Sci.USA102:11070-11075,每一篇都通过引用并入本文)和估计α或β多样性,例如Bray-Curtis相异性指数(Bray and Curtis(1957)“An Ordination of the Upland Forest Communitiesof Southern Wisconsin”Ecol.Monogr.27:325–349,通过引用并入本文)。可以使用ecodist包中的bcdist函数计算Bray-Curtis距离(Goslee(2007)“The ecodist packagefor dissimilarity-based analysis of ecological data”J Stat Softw 22:1-19,通过引用并入本文)。群落数据、地理距离和环境变量的Bray-Curtis距离矩阵之间的相关性可以使用vegan包中的mantel函数来计算(Oksanen,vegan:Community Ecology Package forR);参见,例如,Legendre,P.和Legendre,L.(2012)Numerical Ecology.第3版英文版.Elsevier,通过引用并入本文)。
若干种工具可用,其提供微生物群落结构的这些和其他估计(例如,描述群落成员的丰度)。参见,例如,LIBSHUFF(Schloss(2004)“Integration of microbial ecology andstatistics:a test to compare gene libraries”Appl.Environ.Microbiol.70:5485-5492;以及Singleton(2001)“Quantitative comparisons of16S rRNA gene sequencelibraries from environmental samples”Appl.Environ.Microbiol.67:4374-4376,每一篇都通过引用并入本文)、TreeClimber(Martin(2002)“Phylogenetic approaches fordescribing and comparing the diversity of microbial communities”Appl.Environ.Microbiol.68:3673-3682;以及Schloss(2006)“IntroducingTreeClimber,a test to compare microbial community structures”Appl.Environ.Microbiol.72:2379-2384,每一篇都通过引用并入本文)、UniFrac(Lozupone(2005)“UniFrac:a new phylogenetic method for comparing microbialcommunities”Appl.Environ.Microbiol.71:8228-8235,通过引用并入本文),和分子方差分析(AMOVA)(Excoffier(1992)“Analysis of molecular variance inferred frommetric distances among DNA haplotypes:application to human mitochondrial DNArestriction data”Genetics131:479-491;以及Martin(2002)“Phylogenetic approachesfor describing and comparing the diversity of microbial communities”Appl.Environ.Microbiol.68:3673-3682,每一篇都通过引用并入本文);DOTUR(Schloss(2005)“Introducing DOTUR,a computer program for defining operationaltaxonomic units and estimating species richness”Appl.Environ.Microbiol.71:1501-1506,通过引用并入本文);以及SONS(Schloss(2006)“Introducing SONS,a Toolfor Operational Taxonomic Unit-Based Comparisons of Microbial CommunityMemberships and Structures”Appl Environ Microbiol.72:6773–6779,通过引用并入本文),其提供了若干种量度,包括成员资格(例如,基于发生率的
如本文所用,术语“稳定”在用于提及微生物群落(例如,微生物群落、微生物聚生体、微生物培养物或微生物的其他组、集或集合)时,指的是当第一培养物的一部分用于接种培养基以产生第二培养物并且当第一培养物和第二培养物的培养条件(包括外部因素(光、营养素、温度、通气等))相同时,从第一培养物到第二培养物没有显著变化(例如,通过上面讨论的相似性测量来测量)的微生物群落。因此,如本文所用,术语“稳定性”在用于提及微生物群落(例如,“微生物群落稳定性”)时,指的是当第一培养物的一部分用于接种培养基以产生第二培养物并且当第一培养物和第二培养物的培养条件(包括外部因素(光、营养素、温度、通气等))相同时,从第一培养物到第二培养物的微生物群落(例如,微生物群落、微生物聚生体、微生物培养物或微生物的其他组、集或集合)中变化的定性或定量指标或测量(例如,通过上面讨论的相似性测量来测量)。
因此,作为传代数的函数监测培养物、微生物群落和/或微生物群落的相似性测量(例如,在步骤508、509和511中)提供了培养物、微生物群落和/或微生物聚生体在来自传代过程的培养物中的稳定性的测量。作为传代数函数的相似性测量的变化率下降表明培养物、微生物群落和/或微生物聚生体的稳定性增加。作为传代数函数的相似性测量的平台或稳定表明培养物、微生物群落和/或微生物聚生体处于或接近最大稳定性(例如,在步骤509和511)。例如,在一些实施方式中,当稳定性量度在先前稳定性量度的10%至20%(例如,10%、11%、12%、13%、14%、15%、16%、17%、18%、19%或20%)以内时,稳定性量度达到平台。在一些实施方式中,当多次传代的(例如,2、3、4、5、6、7或8次传代的)稳定性量度在先前稳定性量度的10%至20%(例如,10%、11%、12%、13%、14%、15%、16%、17%、18%、19%或20%)以内时,稳定性量度达到平台。在一些实施方式中,当作为传代数函数拟合稳定性量度的线的斜率为零、基本为零或有效地为零时,稳定性量度达到平台。
此外,如本文所用,术语“稳定”在用于提及由微生物群落(例如,微生物群落、微生物聚生体、微生物培养物或微生物的其他组、集或集合)提供和/或执行的一种或多种功能时,指的是当第一培养物的一部分用于接种培养基以产生第二培养物并且当第一培养物和第二培养物的培养条件(包括外部因素(光、营养素、温度、通气等))相同时,从第一培养物到第二培养物没有显著变化的一个或多个功能(例如,通过检查宏基因组序列和/或通过从中推断功能来测量)。因此,如本文所用,术语“稳定性”在用于提及由微生物群落提供的一种或多种功能(例如,“功能稳定性”)时,指的是当第一培养物的一部分用于接种培养基以产生第二培养物并且当第一培养物和第二培养物的培养条件(包括外部因素(光、营养素、温度、通气等))相同时,从第一培养物到第二培养物的微生物群落(例如,微生物群落、微生物聚生体、微生物培养物或微生物的其他组、集或集合)提供的一种或多种功能中变化的定性或定量指标或测量(例如,通过上面讨论的相似性测量来测量)。因此,功能稳定性和微生物稳定性可能是独立的,使得微生物群落可能在功能上是稳定的,但具有不断变化的成员资格和/或成员丰度,使得微生物群落不具有微生物群落稳定性。因此,微生物群落可能具有功能稳定性和微生物群落稳定性二者;微生物群落可能既没有功能稳定性也没有微生物群落稳定性;微生物群落可能具有功能稳定性(例如,无论微生物群落稳定性的状态如何);微生物群落可能具有微生物群落稳定性(例如,无论功能稳定性的状态如何)。
尽管本文的公开涉及某些例示的实施方式,但应理解这些实施方式是以示例的方式而非以限制的方式呈现的。出于所有目的,上述说明书中提及的所有出版物和专利均通过引用整体并入本文。在不脱离所描述的技术的范围和精神的情况下,所描述的组合物、方法和技术的使用的各种修改和变化对于本领域技术人员来说将是显而易见的。尽管已经结合特定示例性实施方式描述了该技术,但是应当理解所要求保护的本发明不应不适当地限制于这些特定实施方式。实际上,对本领域技术人员显而易见的用于实施本发明的所述模式的各种修改旨在落入所附权利要求的范围内。
- 产生吡啶二羧酸的微生物聚生体和选择与运载体共配制的微生物的方法
- 用于使用高丙三醇浓度产生1,3-丙二醇的微生物聚生体
