血浆游离DNA甲基化特异性位点及DMR区段的检测方法
文献发布时间:2024-04-18 19:58:21
技术领域
本发明涉及基因工程技术领域,具体而言,涉及一种血浆游离DNA甲基化特异性位点及DMR区段的检测方法。
背景技术
癌症严重威胁人类健康,患者的5年整体生存率较低,其主要原因在于大部分癌症患者在确诊时已属中晚期,中晚期癌症的5年生存率极低。癌症的早筛早诊是提高患者生存率的关键,但目前癌症筛查方法存在操作复杂、可及性及依从性低等问题,并且很多癌症(如胰腺癌、卵巢癌等)尚未出现有效的筛查方式。癌细胞在生长、坏死、凋亡的过程中,会释放一些癌症信号到体液(例如血液)中,因此,通过检测体液中的癌症信号可以发现早期癌症。
近年来,表观遗传学、液体活检和生物标记物等检测技术的进步,使癌症早筛成为了可能。DNA甲基化异常是驱动癌症发生发展的重要表观遗传修饰之一。研究发现,在不同的肿瘤阶段,DNA甲基化位置和甲基化水平存在差异,而异常的DNA甲基化是肿瘤的特征之一。近年来,随着DNA甲基化分析技术和临床转化研究的突破,DNA甲基化检测可以作为一种肿瘤早筛和早诊的新技术。目前,临床常用的甲基化检测技术主要是通过亚硫酸氢盐法测序或PCR扩增等,但亚硫酸氢盐的浓度、反应温度、反应环境的PH值、反应时间及DNA模板量等都对核酸修饰是否完全或过量产生严重影响,因此实验条件比较苛刻,可造成DNA降解、DNA损伤、DNA丢失以及不完全转化且很难避免,不太容易把握,时常会对检测结果产生不利影响。这种处理方法对样本DNA的起始含量要求比较高,通常外周血痕量ctDNA难以满足检测要求,即使增大外周血的采样量也很难满意。荧光PCR对DNA甲基化区段的检测只能给出定性结果,仅仅反应靶基因区段甲基化是否存在异常情况,不能给出靶区段甲基化率及甲基化位置信息,而这些信息更能反应肿瘤存在风险及肿瘤临床进展情况。NGS测序不仅对起始样本量的要求更高,往往使得ctDNA含量不高的患者无法获得想要的结果,而且操作复杂,流程繁琐,仪器昂贵,检测价格极高。
因此,发明一种适合外周血微量ctDNA甲基化检测,并且检测特异度高,灵敏度高,可组织溯源,高性价比,操作简便的实验方法,已成为急需解决的问题。
发明内容
本发明解决了现有痕量游离DNA甲基化检测操作复杂的技术问题,实现了提高检测特异度和灵敏度,降低检测成本且操作简便的技术效果。
为解决上述问题,本发明提供一种血浆游离DNA甲基化特异性位点及DMR区段的检测方法,包括以下步骤:S10:提取血浆样品的cfDNA,采用甲基化修饰依赖型核酸内切酶对cfDNA进行酶切,得到酶切产物;S20:将包被有特异性接头序列的磁珠与酶切产物混合,磁珠包被的特异性接头序列在连接酶的催化下与酶切产物的酶切片段粘性末端3'端进行特异性连接,得到磁珠-酶切产物;S30:收集磁珠-酶切产物中的磁珠;S40:将连接有特异性探针的流式编码微球及反向引物与S30得到的磁珠进行混合,混合后进行PCR扩增,得到PCR产物;S50:使用核酸双链染料对PCR产物进行染色,染色后,使用流式细胞仪对流式编码微球逐颗进行分析,并检测流式编码微球荧光信号和核酸双链染料荧光信号,将流式编码微球荧光信号和核酸双链染料荧光信号转化为定量检测结果,计算位点甲基化信息与DMR区段的甲基化率。
与现有技术相比,采用该技术方案所达到的技术效果:现有的用于甲基化检测的方法在实际使用中存在灵敏度低,反应条件不易控制,操作繁琐等缺点。为了解决这些缺点,本发明通过使用甲基化修饰依赖型核酸内切酶对所有含有甲基化位点的序列进行切割,酶切后的序列与接头序列连接,以此对甲基化序列加上标签与未甲基化的同源序列相区别。通过PCR扩增的方式,对含有标签的序列进行特异性扩增,以此有针对性地增大特异甲基化序列的含量,使信号放大,并被流式编码微球捕获。经流式细胞仪检测,可快速分析甲基化位点的具体信息以及DMR甲基化率。由于在不同的肿瘤阶段,DNA甲基化位置和甲基化水平存在差异,因此,将核酸双链荧光信号转化为定量检测结果,更为直观,计算得到的位点甲基化信息与DMR区段甲基化率能够起到较好的提示作用,可以作为初筛手段进行肿瘤早期筛查。同时本发明提供的检测方法能够避免DNA损失过度,提高检测特异度和灵敏度,检测成本低且操作简便。
在本发明的一个实例中,甲基化修饰依赖型核酸内切酶为MspJI家族成员,包括MspJI,FspEI,LpnPI,AspBHI,RIaI,SgrTI中的任意一种或多种。
与现有技术相比,采用该技术方案所达到的技术效果:使用了MspJI家族的甲基化修饰依赖型核酸内切酶,该家族成员可识别近似100%的甲基化位点。FspEI限制性核酸内切酶用于表观遗传学分析,底物DNA的两条链中均包含CpG甲基化位点,FspEI限制性核酸内切酶能够对所有含有甲基化位点的序列进行切割。
在本发明的一个实例中,酶切产物的5'末端带有4个碱基的突出,4个碱基的突出可与磁珠带有的4个特异性碱基的特异性接头序列进行特异性连接。
与现有技术相比,采用该技术方案所达到的技术效果:切割后的含有甲基化位点的序列能够与磁珠特异性连接,进而能够区别甲基化序列与未甲基化的同源序列。
在本发明的一个实例中,收集磁珠-酶切产物中的磁珠采用磁吸法分离。
与现有技术相比,采用该技术方案所达到的技术效果:磁珠表面特异性连接有酶切产物,采用磁吸法收集磁珠,效率高,且不会破坏结构。
在本发明的一个实例中,流式编码微球由两种荧光染料按不同浓度配比,形成不同能级的荧光编码微球阵列,每种荧光编码微球的表面连接有一种甲基化位点特异性结合的正向引物。
与现有技术相比,采用该技术方案所达到的技术效果:每种荧光微球对应一个甲基化位点,即检测靶标,并且不同种类编码微球区分度高,进而能够区分不同甲基化位点,检测特异性高。
在本发明的一个实例中,每种荧光编码微球的数量为1000至5000颗。
与现有技术相比,采用该技术方案所达到的技术效果:每种荧光编码微球对应一个甲基化位点,因此,甲基化位点越多,则荧光编码微球的数量越多,便于流式细胞仪捕获荧光信号。
在本发明的一个实例中,核酸双链染料包括SYBR Green I,EVAGREEN,EB,CelRed,CelGreen中的任一种。
与现有技术相比,采用该技术方案所达到的技术效果:SYBR Green I是一种结合于所有dsDNA双螺旋小沟区域的具有绿色激发波长的染料。在游离状态下,SYBRGreen I发出微弱的荧光,但一旦与双链DNA结合后,荧光大大增强。因此,SYBR Green I的荧光信号强度与双链DNA的数量相关,可以根据荧光信号检测出PCR体系存在的双链DNA数量。PCR扩增后的DNA双链产物会连接在微球上,SYBR Green I镶嵌在DNA双链中在激光激发下会发出强烈的荧光,进而能够被流式细胞仪捕获到微量甲基化DNA和识别特异性甲基化位点。EVAGREEN核酸染料是一种用于实时定量PCR(qPCR)的DNA结合染料,EVAGREEN的稳定性极强,降低了细胞膜透性,安全性好。溴化乙锭(EB),是用于凝胶中DNA和RNA检测的最常用染料。CelRed、CelGreen核酸染料是结合于双链DNA双螺旋小沟区域的具有独特设计的荧光染料,可用于电泳前染色,对分子生物学中常用酶如Taq酶、逆转录酶、T4连接酶等无抑制作用,且安全无毒。
在本发明的一个实例中,DMR区段甲基化率根据检测到的染料荧光的位点数除以DMR区段所有位点数计算得到。
与现有技术相比,采用该技术方案所达到的技术效果:通过上述计算方式,计算得到DMR区段甲基化率,能够直观反映DNA甲基化水平。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
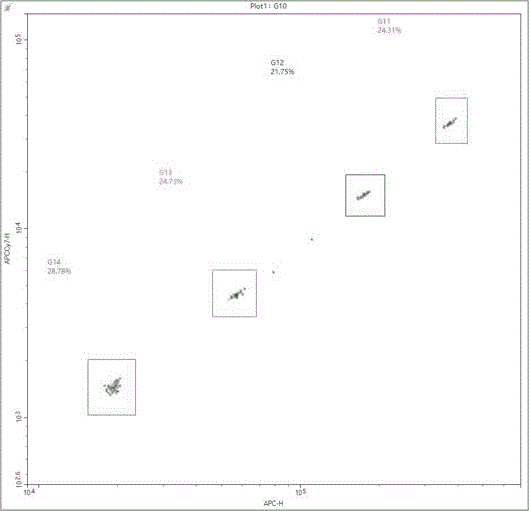
图1为本发明实施例提供的流式编码微球的流式检测结果图。
具体实施方式
为使本发明的上述目的、特征和优点能够更为明显易懂,下面对本发明的具体实施例做详细的说明。
实施例一:
本发明提供一种血浆游离DNA甲基化特异性位点及DMR区段的检测方法,包括以下步骤:
S10:提取血浆样品的cfDNA,采用甲基化修饰依赖型核酸内切酶对cfDNA进行酶切,得到酶切产物;
S20:将包被有特异性接头序列的磁珠与酶切产物混合,磁珠包被的特异性接头序列在连接酶的催化下与酶切产物的酶切片段粘性末端3'端进行特异性连接,得到磁珠-酶切产物;
S30:收集磁珠-酶切产物中的磁珠;
S40:将连接有特异性探针的流式编码微球及反向引物与S30得到的磁珠进行混合,混合后进行PCR扩增,得到PCR产物;
S50:使用核酸双链染料对PCR产物进行染色,染色后,使用流式细胞仪对流式编码微球逐颗进行分析,并检测流式编码微球荧光信号和核酸双链染料荧光信号,将流式编码微球荧光信号和核酸双链染料荧光信号转化为定量检测结果,计算位点甲基化信息与DMR区段的甲基化率。
具体的,现有的用于甲基化检测的方法在实际使用中存在灵敏度低,反应条件不易控制,操作繁琐等缺点。为了解决这些缺点,本发明通过使用甲基化修饰依赖型核酸内切酶对所有含有甲基化位点的序列进行切割,酶切后的序列与接头序列连接,以此对甲基化序列加上标签与未甲基化的同源序列相区别。通过PCR扩增的方式,对含有标签的序列进行特异性扩增,以此有针对性地增大特异甲基化序列的含量,使信号放大,并被流式编码微球捕获。经流式细胞仪检测,可快速分析甲基化位点的具体信息以及DMR甲基化率。由于在不同的肿瘤阶段,DNA甲基化位置和甲基化水平存在差异,因此,将核酸双链荧光信号转化为定量检测结果,更为直观,计算得到的位点甲基化信息与DMR区段甲基化率能够起到较好的提示作用,可以作为初筛手段进行肿瘤早期筛查。同时本发明提供的检测方法能够提高检测特异度和灵敏度,且操作简便。
举例来说,连接酶为T7DNA连接酶。
进一步的,甲基化修饰依赖型核酸内切酶为MspJI家族成员,包括MspJI,FspEI,LpnPI,AspBHI,RIaI,SgrTI中的任意一种或多种。
具体的,使用了MspJI家族的甲基化修饰依赖型核酸内切酶,该家族成员可识别近似100%的甲基化位点。FspEI限制性核酸内切酶用于表观遗传学分析,底物DNA的两条链中均包含CpG甲基化位点,被FspEI酶切后产生包含CpG甲基化位点的32bp片段。上述甲基化修饰依赖型核酸内切酶能够对所有含有甲基化位点的序列进行切割,可以单独使用,也可以组合使用。
进一步的,酶切产物的5'末端带有4个碱基的突出,4个碱基的突出可与磁珠带有的4个特异性碱基的特异性接头序列进行特异性连接。
具体的,切割后的含有甲基化位点的序列能够与磁珠特异性连接,进而能够区别甲基化序列与未甲基化的同源序列。
进一步的,收集磁珠-酶切产物中的磁珠采用磁吸法分离。
具体的,磁珠表面特异性连接有酶切产物,采用磁吸法收集磁珠,效率高,且不会破坏结构。
进一步的,流式编码微球由两种荧光染料按不同浓度配比,形成不同能级的荧光编码微球阵列,每种荧光编码微球的表面连接有一种甲基化位点特异性结合的正向引物。
举例来说,微球被两种荧光染料按10×10的浓度梯度染色编码,形成100种不同特征荧光谱的微球。由于在同一体系中,不同编码微球各自反应、互不干扰,因此每种微球可共价交联任意蛋白或核酸探针,实现一次检测获得100项结果的高通量联合检测,也因此可进行新项目的开发及个体化检测试剂的定制。微球逐颗经过检测仪器时,红色激光识别微球编码确定项目类型,绿色激光读取检测物信号值进行定量或定性分析。
具体的,每种荧光编码微球对应一个甲基化位点,即检测靶标,并且不同种类编码微球区分度高,进而能够区分不同甲基化位点,检测特异性高。
进一步的,每种荧光编码微球的数量为1000至5000颗。
具体的,每种荧光编码微球对应一个甲基化位点,因此,甲基化位点越多,则荧光编码微球的数量越多,便于流式细胞仪捕获荧光信号。
进一步的,核酸双链染料包括SYBR Green I,EVAGREEN,EB,CelRed,CelGreen中的任一种。
具体的,SYBR Green I是一种结合于所有dsDNA双螺旋小沟区域的具有绿色激发波长的染料。在游离状态下,SYBR Green I发出微弱的荧光,但一旦与双链DNA结合后,荧光大大增强。因此,SYBR Green I的荧光信号强度与双链DNA的数量相关,可以根据荧光信号检测出PCR体系存在的双链DNA数量。PCR扩增后的DNA双链产物会连接在微球上,SYBRGreen I镶嵌在DNA双链中在激光激发下会发出强烈的荧光,进而能够被流式细胞仪捕获到微量甲基化DNA和识别特异性甲基化位点。EVAGREEN核酸染料是一种用于实时定量 PCR(qPCR)的DNA结合染料,EVAGREEN的稳定性极强,降低了细胞膜透性,安全性好。溴化乙锭(EB),是用于凝胶中DNA和RNA检测的最常用染料。CelRed、CelGreen核酸染料是结合于双链DNA双螺旋小沟区域的具有独特设计的荧光染料,可用于电泳前染色,对分子生物学中常用酶如Taq酶、逆转录酶、T4连接酶等无抑制作用,且安全无毒。
进一步的,DMR区段甲基化率根据检测到的染料荧光的位点数除以DMR区段所有位点数计算得到。
具体的,通过上述计算方式,计算得到DMR区段甲基化率,能够直观反映DNA甲基化水平。
实施例二:
在一个具体实施例中,本发明以chr6:26020672-26021147区段为例,共包含4个差异甲基化位点,涉及的HIST1H4A基因是经TCGA数据库甲基化分析得到的差异甲基化基因,相应序列是根据人类全基因组h19获得。
1. 材料与试剂:
使用商品化流式编码微球及配套引物偶联试剂盒。MspJI、FspE甲基化修饰依赖型核酸内切酶从百奥莱博购买。5'端修饰NH2-C12的正向引物,3'端修饰NH2-C12的接头序列以及反向引物均从生工生物合成购买。cfDNA提取试剂盒和PCR扩增试剂盒均从QIAGEN公司购买。本发明实施例使用的DNA序列参见表1。
表1 DNA序列
2. 检测方法的具体步骤如下:
步骤1:使用引物偶联试剂盒将流式编码微球分别与4种正向引物偶联,按照说明书要求进行实验,偶联产物放于4℃避光保存。此步骤可替换成其他同类型试剂盒;
步骤2:使用cfDNA提取试剂盒提取血浆游离DNA,按照试剂盒说明书要求进行实验,提取到的cfDNA放于-20℃保存。此步骤可替换成其他同类型试剂盒;
步骤3:使用甲基化修饰依赖型核酸内切酶MspJI、FspEI消化cfDNA,得到酶切产物,反应总体积为50ul,具体步骤为:加入cfDNA 2μl,两种限制酶各1μl,CutFast Buffer 5μl,酶激活溶液1.6μl和无核酸酶水,37℃反应10分钟,然后在80℃加热20分钟,使酶失活,终止消化;
步骤4:将包被有特异性接头序列的磁珠与步骤3得到的酶切产物混合,同时在T7DNA连接酶的催化下,磁珠的特异性接头序列与酶切产物的酶切片段粘性末端3'端进行特异性连接,再利用磁吸法收集磁珠;
步骤5:PCR特异性扩增目的序列:反应体系50μl,将步骤4中收集得到的磁珠,偶联特异性探针的4种流式编码微球以及反向引物加到合适的PCR体系中,对目的序列进行特异性扩增,得到PCR产物,按照试剂盒说明书要求进行实验,此步骤可替换成其他同类型试剂盒。得到的PCR扩增产物放于4℃保存;
步骤6:用SYBR Green I对步骤5得到的PCR产物进行染色,使用流式细胞仪对流式编码微球逐颗进行分析,同时检测流式编码微球荧光和SYBR Green I荧光,将荧光信号转化为定量检测结果,并计算位点甲基化信息与DMR区段的甲基化率。
本实施例中使用的流式编码微球的流式检测结果如图1所示,可以看出不同种类编码微球区分度高,且同种编码微球聚集度高,表明编码微球质地均匀,且荧光编码效果较好,可以用于区分不同甲基化位点。DMR区段甲基化率计算方法:检测到SYBR Green I荧光的位点数除以DMR区段所有位点数,得到该DMR区段的甲基化率。
虽然本发明披露如上,但本发明并非限定于此。任何本领域技术人员,在不脱离本发明的精神和范围内,均可作各种更动与修改,因此本发明的保护范围应当以权利要求所限定的范围为准。
- 一种用于血浆游离DNA基因甲基化检测的超灵敏检测方法
- 肺癌血浆游离DNA甲基化检测用的多重PCR引物系统、检测方法及应用
