基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法
文献发布时间:2023-06-19 10:10:17
技术领域
本发明涉及驴肌肉发育相关基因挖掘技术领域,具体而言,涉及一种基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法。
背景技术
德州驴作为大型驴代表,具有耐粗饲、抵抗力高、抗病力强等特点。随着德州驴役用价值的弱化,如何加快养殖模式的变革,提高驴肉的生产成为重点。
骨骼肌是畜禽肉类的重要的组成的部分,肌纤维的数量和体积是影响肉质的关键因素,而骨骼肌的生长发育也是肉驴生产关注的问题。越来越多的研究表明非编码RNA(non-coding RNA,ncRNA)在调控生物体的胚胎发育、组织分化、器官形成等生命活动扮演着重要的角色,并且对肌肉发育起重要的调控作用。
因此,探究驴肌肉组织的非编码RNA的表达谱、骨骼肌发育分子机制对德州驴役用性能转为肉用性能和专门化肉用新品种开发具有重要的指导意义。
发明内容
本发明旨在至少解决现有技术或相关技术中存在的技术问题之一。
为此,本发明的一个目的在于提供一种基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法,基于RNAseq技术,结合实时定量PCR技术、双荧光素酶基因报告系统等方法确定具有重要功能的靶基因,以便为肉用驴的选育提供理论依据,对德州驴役用性能转为肉用性能和专门化肉用新品种开发具有重要的指导意义。
为了实现上述目的,本发明的技术方案提供了一种基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法,包括以下步骤:
S1:选取遗传背景一致的2月龄、24月龄健康公驴各3头,屠宰后取背最长肌组织样本;
S2:基于RNAseq技术,从所述背最长肌组织样本中提取RNA,并对这些组织样品单细胞所有RNA进行测序整理分析获得转录组数据;
S3:采用HISAT2分层次索引比对工具,将总的待分析数据clean Reads比对到参考基因组上进行转录组测序质量评价,若转录组测序的质量较高,则可以进行后续的生物信息分析;
S4:对mRNAs、lncRNAs、small RNA与参考基因组进行比对,采用GO和KEGG数据库对差异表达基因进行分析,筛选获得得到一定数目的上调差异表达基因或者下调差异表达基因,绘制lncRNA-mRNA互作网络图,构建miRNA-mRNA互作网络图;
S5:采用qRT-PCR验证对差异表达基因lncRNA和mRNA进行验证,从预测的miRNA-mRNA互作网络图中,随机地挑选几组进行验证,若DEGs的表达模式与转录组测序结果一致,说明此次实验测序结果是可靠的;
S6:预测并筛选miRNA的靶基因,通过双荧光素酶报告试验验证miRNA与其预测靶基因的关系,确定出TPM3和ZZZ3基因是miR-1的靶基因。
优选地,步骤S4中,采用GO和KEGG数据库对差异表达基因进行分析,筛选获得得到一定数目的上调差异表达基因或者下调差异表达基因,具体包括以下步骤:
采用GO和KEGG数据库对差异表达基因进行分析,筛选出P值小于0.05的GO和基因通路,GO注释说明基因的基本功能,KEGG说明基因所参与的代谢通路;
设定差异表达基因的筛选条件,所述差异表达基因的筛选条件包括筛选指标FDR小于0.01,Log2FC大于1,获得得到一定数目的上调差异表达基因或者下调差异表达基因,其中,对不同时期差异的mRNA和lncRNA进行筛选的筛选标准为P<0.05,|Fold change|>2,采取DESeq2(Love M I et al.2014)对差异miRNA进行定量分析,标准为p<0.05。
优选地,步骤S4中,绘制lncRNA-mRNA互作网络图,构建miRNA-mRNA互作网络图,具体包括:
lncRNA的功能预测:选择lncRNA上下游100kb间隔的编码基因作为顺式作用的靶基因,并对靶基因进行功能富集,以预测lncRNA的功能;
miRNA靶基因的预测:采用miRanda(Enright A J et al.2003)、PITA(Kertesz Met al.2007)和RNAhybrid(Kruger JRehmsmeier M 2006)对miRNA进行靶基因预测分析;
对选取差异的mRMA、miRNA、lncRNA的靶基因分别进行GO和KEGG富集分析;
lncRNA-mRNA互作分析:采用顺式作用进行靶基因预测,发现有差异lncRNA对应的靶基因,之后在肌肉发育相关信号通路上,找寻相关的编码基因,然后将靶基因与编码基因作集合,将重复筛选的靶基因与差异mRNA进行比对,得到的靶基因与肌肉发育显著相关,构建lncRNA-mRNA网络互作图;
miRNA-mRNA互作分析:将得到的差异mRNA与miRNA的靶基因进行对比,构建miRNA-mRNA网络互作图。
优选地,所述肌肉发育相关信号通路包括mTOR信号通路、wnt信号通路、AMPK信号通路中的一种或者几种。
优选地,步骤S5中,采用qRT-PCR验证对差异表达基因lncRNA和mRNA进行验证中,定量引物通过Primer 5设计,再由NCBI Primer-BLAST验证,而且对lncRNA-mRNA靶向关系也进行定量验证;选择GAPDH作为内参基因,设计定量引物;依照2
优选地,lncRNA的qRT-PCR引物序列为:
优选地,mRNA的qRT-PCR引物序列为:
优选地,步骤S6中预测并筛选miRNA的靶基因,通过双荧光素酶报告试验验证miRNA与其预测靶基因的关系,具体包括:
以P<0.05和|Fold change|>2为筛选标准预测并筛选miRNA的靶基因;
选到6个miR-1靶基因,之后构建IGF-1、TMSB4X、EFS1、TPM3、PLXDC2和ZZZ3这6个预测靶基因野生型及突变型双荧光素酶报告基因载体,并进行双荧光素酶试验。
优选地,步骤S1中屠宰后取背最长肌组织样本具体包括:
屠宰后取背最长肌,经磷酸缓冲液PBS清洗三次,装入1ml冻存管内,经液氮速冻后,于超低温冰箱中-80℃冷冻保存,用于后续RNA提取。
本发明提出的基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法具有以下有益技术效果:
(1)以德州驴背最长肌内在的遗传基因作为起点,基于转录组对幼龄和成年种公驴背最长肌基因进行分析,并通过差异表达基因的qRT-PCR验证和293T细胞双荧光素酶报告试验验证,筛选出与miR-1靶向的与肌肉发育相关的靶基因,为肉用驴的选育提供理论依据。
(2)lncRNA的qRT-PCR引物序列、mRNA的qRT-PCR引物序列设计合理,实验测序结果是可靠的,确定出TPM3和ZZZ3基因是miR-1的靶基因,为进一步挖掘德州驴种质资源和培育生长速度快的新品种提供支撑。
本发明的附加方面和优点将在下面的描述部分中给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施例的描述中将变得明显和容易理解,其中:
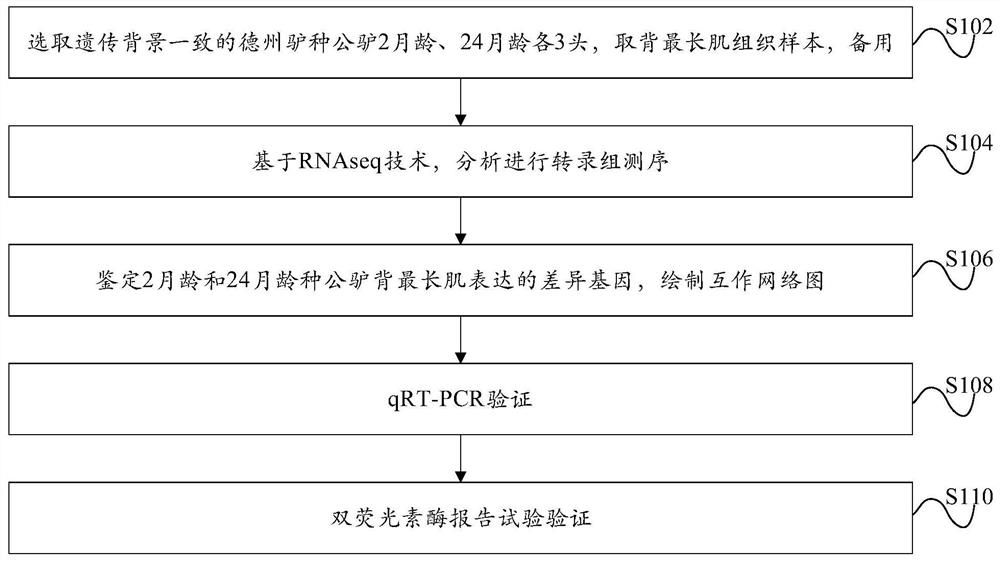
图1示出了根据本发明的一个实施例的基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法的流程示意图;
图2示出了根据本发明的一个实施例的lncRNA靶基因的功能分析结果图;
图3示出了根据本发明的一个实施例的mRNA靶基因的功能分析结果图;
图4示出了根据本发明的一个实施例的lncRNA-mRNA网络互作图;
图5示出了根据本发明的一个实施例的miRNA靶基因的功能分析结果图;
图6示出了根据本发明的一个实施例的miRNA-mRNA网络互作图;
图7示出了根据本发明的一个实施例的miRNA-mRNA互作分析图。
具体实施方式
本发明公开了一种基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法,本领域技术人员可以借鉴本文内容,适当改进工艺参数实现。特别需要指出的是,所有类似的替换和改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明的方法及应用已经通过较佳实施例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文所述的方法和应用进行改动或适当变更与组合,来实现和应用本发明技术。
下面结合实施例,进一步阐述本发明:
实施例1
如图1所示,根据本发明的实施例的一种基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法,包括以下步骤:
S102,选取遗传背景一致的德州驴种公驴2月龄、24月龄各3头,取背最长肌组织样本,备用;
S104,基于RNAseq技术,分析进行转录组测序;
S106,鉴定2月龄和24月龄种公驴背最长肌表达的差异基因,绘制互作网络图;
S108,qRT-PCR验证;
S110,双荧光素酶报告试验验证。
以德州驴背最长肌内在的遗传基因作为起点,基于转录组对幼龄和成年种公驴背最长肌基因进行分析,并通过差异表达基因的qRT-PCR验证和293T细胞双荧光素酶报告试验验证,筛选出与miR-1靶向的与肌肉发育相关的靶基因,确定出TPM3和ZZZ3基因是miR-1的靶基因,为肉用驴的选育提供理论依据。
实施例2
S202,选取遗传背景一致的2月龄、24月龄健康公驴各3头,屠宰后取背最长肌组织样本;
S204,对组织样本进行转录组测序:从背最长肌中提取RNA,然后对这些样品单细胞所有RNA进行测序获得结果,最后整理分析转录组数据,并对照参考基因组进行分析;
S206,转录组测序质量评价:质量评价结果可以反映本转录组测序的质量高低,若本转录组测序的质量较高,则可以进行后续的生物信息分析;
S208,幼龄和成年驴肌肉基因差异表达:采用GO和KEGG数据库对差异表达基因进行分析,筛选出P值小于0.05的GO和基因通路,GO注释说明基因的基本功能,KEGG说明基因所参与的代谢通路,设定差异表达基因的筛选条件,所述差异表达基因的筛选条件包括筛选指标FDR小于0.01,Log2FC大于1,获得得到一定数目的上调差异表达基因或者下调差异表达基因;
S210,对总的差异表达基因的KEGG和GO功能分析:对差异表达基因进行注释可进一步了解基因所参与的功能或代谢通路;绘制成lncRNA-mRNA互作网络图,构建miRNA-mRNA互作网络图;
S212,差异表达基因的qRT-PCR验证:从预测的miRNA-mRNA网络图中,随机地挑选几组进行验证,荧光定量试验结果与测序结果表现为大体一致,表明了测序结果的可靠性;
S214,双荧光素酶报告试验验证miRNA与其预测靶基因的关系,确定出TPM3和ZZZ3基因是miR-1的靶基因。
筛选出与miR-1靶向的与肌肉发育相关的靶基因,为肉用驴的选育提供了理论依据。
实施例3
S302,以德州驴为对象,选取2月龄和24月龄不同发育阶段种公驴各3头,屠宰后取背最长肌,经PBS清洗三次,装入1ml冻存管内,经液氮速冻后,于超低温冰箱中-80℃冷冻保存,用于后续RNA提取;
S304,进行mRNA和非编码RNA(lncRNAs、miRNA)RNA-seq测序:从背最长肌中提取RNA,然后对这些组织样品单细胞所有RNA进行测序获得结果;
S306,转录组测序质量评价:采用HISAT2软件将总的clean Reads比对到参考基因组上,若比对率,Q30满足标准,样品间的GC含量接近,则说明本试验碱基读取的准确率高;
S308,对mRNAs、lncRNAs、small RNA与参考基因组进行比对;
S310,不同发育阶段基因差异表达:对不同时期差异的mRNA和lncRNA进行筛选,筛选标准为P<0.05,|Fold change|>2,获得得到一定数目的上调差异表达基因或者下调差异表达基因,采取DESeq2(Love M I et al.2014)对差异miRNA进行定量分析,标准为p<0.05;
S312,lncRNA的功能预测:选择lncRNA上下游100kb间隔的编码基因作为顺式作用的靶基因,并对靶基因进行功能富集,以预测lncRNA的功能;
S314,miRNA靶基因的预测:为了解差异表达miRNA的生物学功能,采用miRanda(Enright A J et al.2003)、PITA(Kertesz M et al.2007)和RNAhybrid(KrugerJRehmsmeier M 2006)对miRNA进行靶基因预测分析;
S316,对选取差异的mRMA、miRNA、lncRNA的靶基因分别进行GO和KEGG富集分析,分析结果如图3、图5和图2所示,图2为lncRNA靶基因的功能分析结果图,图2中(A)为GO功能富集分析,图2中(B)为KEGG功能富集分析,图3为mRNA靶基因的功能分析结果图,图3中(A)为GO功能富集分析,图3中(B)为KEGG功能富集分析,图5为miRNA靶基因的功能分析结果图,图5中(A)为GO功能富集分析,图5中(B)为KEGG功能富集分析;
S318,lncRNA-mRNA互作分析:采用顺式作用进行靶基因预测,发现有差异lncRNA对应的靶基因,随后在肌肉发育相关信号通路,mTOR信号通路、wnt信号通路、AMPK信号通路等等,找寻相关的编码基因,然后将靶基因与编码基因作集合,将重复筛选的靶基因与差异mRNA进行比对,得到的靶基因与肌肉发育显著相关,构建lncRNA-mRNA网络互作图,如图4所示,其中,无填充的为上调,有点填充的为下调,矩形代表mRNA,椭圆形代表IncRNA;
S320,miRNA-mRNA互作分析:将得到的差异mRNA与miRNA的靶基因进行对比,构建miRNA-mRNA网络互作图,具体如图6、图7所示,图6中,无填充的为上调,有点填充的为下调,矩形代表mRNA,椭圆形代表miRNA;
S322,差异表达基因的qRT-PCR验证:随机选取几个差异的表达基因进行qRT-PCR验证,若DEGs的表达模式与转录组测序结果一致,说明此次实验测序结果是可靠的;
采用qRT-PCR对差异的lncRNA和mRNA进行验证。定量引物通过Primer 5设计,再由NCBI Primer-BLAST验证,保证引物的准确性。由生工生物工程(上海)公司合成,而且对lncRNA-mRNA靶向关系也进行定量验证。选择GAPDH作为内参基因,引物信息如下表1、表2所示。依照2-△△ct(Livak K JSchmittgen T D 2001)进行计算。
表1lncRNA的qRT-PCR引物序列
表2mRNA的qRT-PCR引物序列
S324,miR-1靶基因的预测和验证:通过miRanda等软件预测并筛选miRNA的靶基因(以P<0.05和|Fold change|>2为标准),再通过双荧光素酶报告试验验证miRNA与其预测靶基因的关系;
S326,选到6个miR-1靶基因,之后构建IGF-1、TMSB4X、EFS1、TPM3、PLXDC2和ZZZ3这6个预测靶基因野生型及突变型双荧光素酶报告基因载体,并进行双荧光素酶试验,结果发现,TPM3和ZZZ3基因是miR-1的靶基因。
本发明以德州驴种公驴肌肉内在的遗传基因作为起点,基于转录组对幼龄和成年种公驴背最长肌基因进行分析,筛选出与德州驴肌肉发育相关的靶基因,为进一步挖掘德州驴种质资源和培育生长速度快的新品种提供支撑。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
- 基于RNAseq技术的驴肌肉发育相关基因挖掘调控方法
- 一种基于MeRIP-Seq技术挖掘藏绵羊高原低氧适应性相关基因的方法
