一种斑马鱼胞嘧啶碱基编辑器及其构建方法与应用
文献发布时间:2024-04-18 19:58:21
技术领域
本发明涉及分子生物学技术领域,具体涉及一种斑马鱼胞嘧啶碱基编辑器及其构建方法与应用。
背景技术
胞嘧啶碱基编辑器(Cytosine Base Editors,CBE)是David R.Liu团队在2016开发的、能够在基因组中实现CG碱基对到TA碱基对转换的基因组编辑工具,可以有效应用于单碱基疾病的动物模型构建和治疗研究。该工具由Cas9切口酶(Cas9 nickase,nCas9)、胞嘧啶脱氨酶APOBEC1以及尿嘧啶糖基化酶抑制剂(Uracil glycosylase inhibitor,UGI)三部分组成。其工作原理为:sgRNA引导nCas9结合基因组靶标位点,靶标位点的DNA双链解开,与sgRNA互补的DNA单链与nCas9形成R环复合物,并被nCas9切割产生切口。游离出的DNA单链被nCas9与胞苷脱氨酶结合,其一定区域内的C被胞苷脱氨酶催化转变为U。这时细胞会启动两种DNA修复途径,一种是将U修复为C,另外一种是将G修复为A。前者的修复路径被UGI阻断,后者因为G所在的DNA单链存在切口而被增强,故而在DNA双链上出现了CG到UA的碱基对替换。UA碱基对在后续的修复过程中,被修复为TA碱基对,从而在不引入DNA双链断裂的前提下,对基因组序列实现从CG碱基对到TA碱基对的编辑。
70%的人类基因可以在斑马鱼中找到直系同源基因,因此,斑马鱼可以用来构建单碱基人类疾病模型。目前,应用于斑马鱼的主流CBE工具主要是AncBE4max和CBE4max-spry两种,其胞苷脱氨酶成分分别为AncAPOBEC1和APOBEC1,但这两种胞苷脱氨酶均存在TC序列的偏好性,难以有效编辑GC序列,且有效编辑窗口一般局限于C3-C8之间,从而使其在斑马鱼上的使用价值不高。而且,现有技术中也缺乏能够实际用于斑马鱼的胞嘧啶碱基编辑器(存在诸如:仅能编辑gRNA序列的C3-C8;编辑效率低;GC和CC序列难以有效编辑等影响实际应用的缺陷)。
因此,开发一种能够突破GC和CC编辑限制、突破PAM限制、降低indel生成效率且具有较窄的编辑窗口的,可用于斑马鱼的碱基编辑器,对于斑马鱼人类疾病模型的进一步开发及研究具有极为重要的意义。
发明内容
本发明旨在至少解决现有技术中存在的上述技术问题之一。为此,本发明的目的在于提供一种斑马鱼胞嘧啶碱基编辑器及其构建方法与应用。本发明中的斑马鱼胞嘧啶碱基编辑器填补了目前现有技术中在斑马鱼中能够有效编辑GC序列的编辑器的空白,实现了在斑马鱼中不受PAM限制的GC序列编辑,且在有效位点数量、编辑效率均显著优于现有编辑器(其与现有编辑器的编辑窗口不同),具有极高的实用价值,为斑马鱼领域中的基因编辑提供了有利的技术支持。
本发明的第一个方面,提供一种斑马鱼胞嘧啶碱基编辑器,所述斑马鱼胞嘧啶碱基编辑器包括evoCDA1脱氨酶、SpRYCas9蛋白和BE4max元件。
在本发明的一些实施方式中,所述斑马鱼胞嘧啶碱基编辑器经过斑马鱼密码子优化。
在本发明的一些实施方式中,所述BE4max元件包括UGI和入核序列。
在本发明的一些实施方式中,所述BE4max元件为2个UGI(2×UGI)和bpNLS序列(双粒子核定位信号序列)。
在本发明的一些实施方式中,所述evoCDA1脱氨酶连接于所述SpRYCas9的N端,2个UGI和bpNLS序列元件连接于所述SpRYCas9的C端。
在本发明的一些实施方式中,所述evoCDA1脱氨酶包括截短的evoCDA1脱氨酶变体。
在本发明的一些实施方式中,所述截短的evoCDA1脱氨酶变体包括在evoCDA1脱氨酶NES(核外运信号)部分进行截短处理的evoCDA1脱氨酶。
当然,本领域技术人员也可以根据实际使用需求,合理选择在其他部分进行截短处理且仍保有相关活性的evoCDA1脱氨酶作为替代,或使用在NES部分截短长度不同的evoCDA1脱氨酶作为替代。
在本发明的一些实施方式中,所述evoCDA1脱氨酶和所述SpRYCas9之间还存在linker。
在本发明的一些实施方式中,所述linker的长度为30~35个氨基酸残基。
在本发明的一些实施方式中,所述linker的长度为32个氨基酸残基。
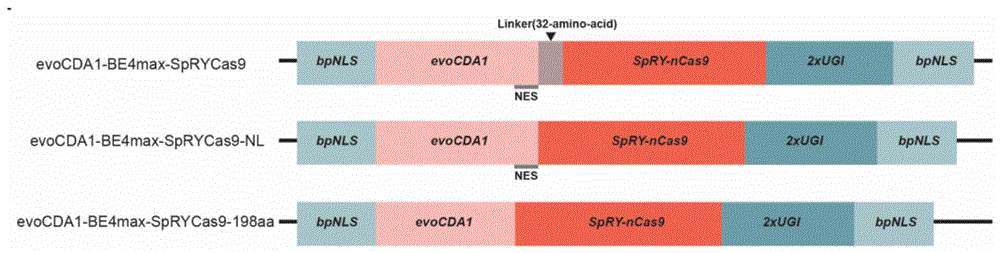
在本发明的一些实施方式中,所述斑马鱼胞嘧啶碱基编辑器的mRNA结构示意图如图1所示。
在本发明的一些实施方式中,所述斑马鱼胞嘧啶碱基编辑器含有如下(1)~(4)中任一项所述的氨基酸序列:
(1)SEQ ID NO:1;
(2)SEQ ID NO:3;
(3)SEQ ID NO:5;
(4)SEQ ID NO:1、3或5中任一项经过氨基酸替换、删减或增加得到变体序列,且所述变体序列保有对斑马鱼的GC序列编辑活性。
在本发明的一些实施方式中,所述变体序列与原氨基酸序列具有90%以上的同一性。
在本发明的一些实施方式中,所述变体序列与原氨基酸序列具有95%以上的同一性。
在本发明的一些实施方式中,所述变体序列与原氨基酸序列具有99%以上的同一性。
本发明的第二个方面,提供编码本发明第一个方面所述的斑马鱼胞嘧啶碱基编辑器的核酸分子。
在本发明的一些实施方式中,所述核酸分子包括如下(1)~(3)中任一个:
(1)SEQ ID NO:2;
(2)SEQ ID NO:4;
(3)SEQ ID NO:6。
本发明的第三个方面,提供含有本发明第二个方面所述核酸分子的表达子,所述表达子包括质粒。
在本发明的一些实施方式中,所述表达子还包括本领域中其他常规用于表达的构建体。
在本发明的一些实施方式中,所述质粒包括本领域中常规的质粒载体。
在本发明的一些实施方式中,所述表达子采用本领域常规方式构建得到。
本发明的第三个方面,提供含有本发明第二个方面所述核酸分子和/或本发明第三个方面所述表达子的转化子,所述转化子包括病毒、细菌和动植物细胞。
在本发明的一些实施方式中,所述转化子采用本领域常规方式构建得到。
本发明的第四个方面,提供本发明第一个方面所述的斑马鱼胞嘧啶碱基编辑器在斑马鱼基因编辑中的应用。
在本发明的一些实施方式中,所述斑马鱼基因编辑包括人类疾病模型的构建、靶标位点的机理研究等等。
在本发明的一些实施方式中,所述斑马鱼胞嘧啶碱基编辑器需配合靶标sgRNA使用。
本发明的有益效果是:
1.本发明中的斑马鱼胞嘧啶碱基编辑器可以高效编辑GC序列,且能够突破PAM限制,编辑效果优于常规的碱基编辑器。
2.本发明中还进一步提供了斑马鱼胞嘧啶碱基编辑器的变体,可以分别将编辑窗口缩短到sgRNA N20序列的第-1到第8位和第-1到第5位,且具有较低的脱靶效率。
附图说明
图1为本发明实施例中得到的三种斑马鱼胞嘧啶碱基编辑器的mRNA结构示意图。
图2为evoCDA1-BE4max-SpCas9的GC位点编辑效果。
图3为evoCDA1-BE4max-SpRY和CBE4max-SpRY对NTN和NCN PAM位点的编辑效果对比。
图4为evoCDA1-BE4max-SpRY和CBE4max-SpRY对NGN和NAN PAM位点的编辑效果对比。
图5为evoCDA1-BE4max-SpRY的indel效率(A)和总编辑效率(B);其中,*P≤0.05,**P≤0.01,***P≤0.001,****P≤0.0001。
图6为人类和斑马鱼的slc24a5编码蛋白序列比对图。
图7为斑马鱼slc24a5基因的结构模式图。
图8为使用本发明实施例中的编辑器编辑后的F
图9为evoCDA1-BE4max-SpRY(whole)和evoCDA1-BE4max-SpRY-No Linker(NL)以及evoCDA1-BE4max-SpRY-198aa(198)的编辑效果比较。
图10为斑马鱼和人类的pitx2基因比对。
图11为evoCDA1-BE4max-SpRY(A)、evoCDA1-BE4max-SpRY-No linker(B)、evoCDA1-BE4max-SpRY-198aa(C)对pitx2 W92*的编辑效果。
图12为斑马鱼pitx2基因结构图和靶标位点位置。
图13为No linker版本编辑效果一代测序峰图。
图14为斑马鱼pitx2 W92*F
具体实施方式
以下通过具体的实施例对本发明的内容作进一步详细的说明。实施例和对比例中所用的原料、试剂或装置如无特殊说明,均可从常规商业途径得到,或者可以通过现有技术方法得到。除非特别说明,试验或测试方法均为本领域的常规方法。
斑马鱼胞嘧啶碱基编辑器的构建
在本实施例中,发明人共构建得到了三种斑马鱼胞嘧啶碱基编辑器,包括evoCDA1-BE4max-SpRY、evoCDA1-BE4max-SpRY-No linker(或称evoCDA1-BE4max-SpRY-NL)和evoCDA1-BE4max-SpRY-198aa。
具体构建方法及详细信息如下:
分别选取evoCDA1脱氨酶、SpRYCas9蛋白以及BE4max其他组分(如UGI和入核序列),并对所选择的序列进行斑马鱼密码子选择性优化,使其高效编辑窗口为sgRNA序列(N20)的第-1到第10个碱基,得到evoCDA1-BE4max-SpRY。
具体地,evoCDA1-BE4max-SpRY是在SpRYCas9的N端融合了evoCDA1,在其C端融合了2个UGI蛋白,得到的融合蛋白的两端再加入经斑马鱼密码子优化后的入核序列bpNLS后得到的。组装完成后再一次对所有组分进行了斑马鱼密码子优化,得到evoCDA1-BE4max-SpRY。如此得到的evoCDA1-BE4max-SpRY是一种在斑马鱼中不受PAM限制、有效编辑GC序列、实现高效CG到TA的胞嘧啶碱基编辑器。
其中,得到的evoCDA1-BE4max-SpRY的氨基酸序列为:SEQ ID NO:1。
对应的核苷酸序列为:SEQ ID NO:2。
进一步在evoCDA1-BE4max-SpRY的基础上,对其进行进一步截短优化,从而得到evoCDA1-BE4max-SpRY-No linker和evoCDA1-BE4max-SpRY-198aa两个变体版本,并进一步使其编辑窗口从sgRNA N20序列的第-1到第10位缩短到了第-1到第8位以及第-1到第5位。
得到的evoCDA1-BE4max-SpRY-No linker的氨基酸序列为:SEQ ID NO:3。
对应的核苷酸序列为:SEQ ID NO:4。
得到的evoCDA1-BE4max-SpRY-198aa的氨基酸序列为:SEQ ID NO:5。
对应的核苷酸序列为:SEQ ID NO:6。
本实施例中得到的三种斑马鱼胞嘧啶碱基编辑器的mRNA结构示意图如图1所示。
斑马鱼胞嘧啶碱基编辑器的编辑效果验证
(1)evoCDA1在斑马鱼上的使用效果:
为了能够充分说明evoCDA1在斑马鱼上的使用效果,在本实施例中,发明人以evoCDA1-BE4max-SpCas9胞嘧啶碱基编辑器(即将上述实施例中的evoCDA1-BE4max-SpRY中的SpRY替换为SpCas9,其他元件无差异)作为实验组,以现有技术中的胞嘧啶碱基编辑器作为对照组,将其应用于斑马鱼上,以对比验证说明现有技术中的胞嘧啶碱基编辑器尚不可在斑马鱼中达到有效编辑GC序列的效果。
在本实施例中,选择本领域中常规的AncBE4max胞嘧啶碱基编辑器(参考Zhao Y,Shang D,Ying R,et al.An optimized base editor with efficient C-to-T baseediting in zebrafish[J].BMC Biology,2020,18(1).DOI:10.1186/s12915-020-00923-z.中的胞嘧啶碱基编辑器)作为对照用的现有技术中的胞嘧啶碱基编辑器。
以本领域中的常规位点作为靶标位点(参考ZHAO Y,SHANG D,YING R,et al.Anoptimized base editor with efficient C-to-T base editing in zebrafish[J].BMCBiol,2020,18(1):190.),测试AncBE4max和evoCDA1-BE4max-SpCas9胞嘧啶碱基编辑器的编辑效果。其中,括号内为PAM序列。
其中,具体gRNA为:
twist2 g1:5’-G
twist2 g2:5’-G
slc22a7a:5’-GG
其中,加粗其标记下划线的碱基为目标碱基。
按照本领域常规技术,将AncBE4max和evoCDA1-BE4max-SpCas9胞嘧啶碱基编辑器分别注入不同斑马鱼鱼卵中,选择注射后2dpf的斑马鱼鱼卵,6枚一组,每条gRNA设置3组重复,以碱裂解法获取基因组,PCR扩增后送至北京擎科生物科技有限公司进行一代测序,将测序峰图上传至EditR进行编辑效率分析。其中,注射时,碱基编辑器的浓度控制在400ng/μL左右,靶标sgRNA的浓度控制在200ng/μL左右,每枚受精卵注射体积为2nL。其他种属鱼类的注射用量可参考斑马鱼注射方案进行相应调整。
结果如图2所示。
可以发现,对于现有的胞嘧啶碱基编辑器而言,以上三个位点的GC效率为零,而evoCDA1-BE4max-SpCas9胞嘧啶碱基编辑器却能够在斑马鱼中分别以37%、36%和20%的效率,有效编辑这些位点。这证明了本发明实施例中的碱基编辑器中使用的evoCDA1胞苷脱氨酶可以有效编辑GC位点。
(2)evoCDA1-BE4max-SpRY在斑马鱼上的使用效果:
进一步,以现有技术中的CBE4max-SpRY(参考Liang F,Zhang Y,Li L,et al.SpGand SpRY variants expand the CRISPR toolbox for genome editing in zebrafish[J].Nature Communications,2022,13.DOI:10.1038/s41467-022-31034-8.)为对照,以evoCDA1-BE4max-SpRY为实验组,测试两者的碱基编辑效果差异。测试方法同上述实施例,区别在于,在本实施例中,使用的gRNA是针对NAN、NGN、NCN、NTN四个PAM位点。
具体gRNA为:
rpl9-NGG:5’-GGCCGCACAGTTACCGTGAA(SEQ ID NO:10)-(GGG)-3’;
rpl9-NGA:5’-CAAGGGCCGCACAGTTACCG(SEQ ID NO:11)-(TGA)-3’;
rpl17-NGT:5’-GCTAACAAGTACCTGAAGGA(SEQ ID NO:12)-(TGT)-3’;
rpl17-NGC:5’-ACATCCTTCAGGTACTTGTT(SEQ ID NO:13)-(AGC)-3’;
rpl17-NTG:5’-GGCTAACAAGTACCTGAAGG(SEQ ID NO:14)-(ATG)-3’;
rps16-NTA:5’-ATCAAGGATATCTTAATTCA(SEQ ID NO:15)-(GTA)-3’;
rps16-NTT:5’-AGTACGACAGGACCCTGCTG(SEQ ID NO:16)-(GTT)-3’;
rpl17-NTC:5’-ACAAGTACCTGAAGGATGTG(SEQ ID NO:17)-(ATC)-3’;
rps16-NAG:5’-ATCCCCGCCGCTGCGAGTCC(SEQ ID NO:18)-(AAG)-3’;
rpl9-NAA:5’-AAGGGCCGCACAGTTACCGT(SEQ ID NO:19)-(GAA)-3’;
rpl9-NAT:5’-AGACAGTGGACATCCCTGAC(SEQ ID NO:20)-(AAT)-3’;
rpl17-NAC:5’-ACATCCGCAAGGCTAACAAG(SEQ ID NO:21)-(TAC)-3’;
rpl17-NCG:5’-CAAGTACCTGAAGGATGTGA(SEQ ID NO:22)-(TCG)-3’;
rpl17-NCA:5’-CATGCACATCCGCAAGGCTA(SEQ ID NO:23)-(ACA)-3’;
rpl17-NCT:5’-ATCCGCAAGGCTAACAAGTA(SEQ ID NO:24)-(CCT)-3’;
rpl9-NCC:5’-CCCTCAAGGGCCGCACAGTT(SEQ ID NO:25)-(ACC)-3’;
结果如图3和图4所示。
可以发现,与CBE4max-SpRY对比,evoCDA1-BE4max-SpRYCas9展示出来更加高效的编辑效率和更广的有效编辑范围(如rpl17-NTC、rpl17-NTG、rpl9-NCC、rpl17-NCG、rpl7-NGC、rpl9-NGG和rpl17-NAC、rpl16-NAG等位点),其编辑能力远高于CBE4max-SpRY。
(3)本发明实施例中的胞嘧啶碱基编辑器的实际indel效率:
按照上述实施例中的方法,分别以evoCDA1-BE4max-SpCas9和evoCDA1-BE4max-SpRYCas9对rpl9-NGG、tyr g3和oca2三个位点进行编辑,并对其进行NGS测序分析,衡量它们的编辑效果差异。
具体gRNA为:
Oca2:5’-
tyr g3:5’-TGAAG
rpl9-NGG:5’-GG
其中,加粗其标记下划线的碱基为可被编辑的碱基。
结果如图5所示。
可以发现,相对于evoCDA1-BE4max-SpCas9,evoCDA1-BE4max-SpRYCas9编辑的位点,其indel效率大幅降低,且编辑效率并无显著差异。
(4)本发明实施例中的胞嘧啶碱基编辑器在斑马鱼模型构建中的实际使用效果:
由于上述实施例中已经充分证实了evoCDA1-BE4max-SpRY具有更好的编辑效果,对GC位点的编辑效率完胜常规的胞嘧啶碱基编辑器。因此,发明人以其作为编辑工具,构建斑马鱼模型,并观察构建效果。
在本领域中,基因slc24a5是引起眼皮肤白化病(oculocutaneous albinism,OCA)的致病基因之一,有报道指出,slc24a5的Q62*突变会导致白化病。基于该信息,发明人对人和斑马鱼slc24a5的氨基酸序列进行比对(如图6所示),选择了slc24a5的Q62*在斑马鱼中的同源位点Q74*作为碱基编辑的靶标位点(图7),设计了gRNA,其中红色字体代表目的碱基。
slc24a5 Q74*:5’-GTG
其中,加粗其标记下划线的碱基为目标碱基。
以不经编辑的斑马鱼野生型(WT)作为对照。
结果如图8所示。
图8为F
(5)本发明实施例中的胞嘧啶碱基编辑器的编辑窗口宽度:
基于上述实施例中对于evoCDA1-BE4max-SpRY的测试,发明人发现evoCDA1-BE4max-SpRY的编辑窗口最宽可达13bp,相对较宽(容易使编辑窗口内除目标位点之外的其他C也发生改变,从而造成不够精确的胞嘧啶碱基编辑),因此,发明人将其与得到的另外两种变体进行了对比验证。
首先,以evoCDA1-BE4max-SpRY(表示为whole)、evoCDA1-BE4max-SpRY-No Linker(表示为NL)以及evoCDA1-BE4max-SpRY-198aa作为测试对象,选择了4个窗口内含有多个C的gRNA位点(rpl17-NGC、rpl9-NTT、rpl9-NCC和rps16-NAA),以evoCDA1-BE4max-SpRY作为对照进行注射测试(方法同上述实施例),选取的4个gRNA序列如下:
rpl17-NGC:5’-ACATCCTTCAGGTACTTGTT(SEQ ID NO:29)-(AGC)-3’;
rpl9-NTT:5’-TGTCCCTCAAGGGCCGCACA(SEQ ID NO:30)-(GTT)-3’;
rpl9-NCC:SEQ ID NO:25;
rps16-NAA:5’-CCCCGCCGCTGCGAGTCCAA(SEQ ID NO:31)-(GAA)-3’;
结果如图9所示。
可以发现,以rpl7-NGC、rpl9-NTT和rpl9-NCC、rpl16-NAA位点为例,分别使用evoCDA1-BE4max-SpRY、evoCDA1-BE4max-SpRY-No Linker以及evoCDA1-BE4max-SpRY-198aa进行编辑,结果显示evoCDA1-BE4max-SpRY-No Linker可以将编辑窗口缩短到C-1-C8,evoCDA1-BE4max-SpRY-198aa可以将编辑窗口缩短到C-1-C5。
(5)三种胞嘧啶碱基编辑器的编辑效果对比:
为了体现出本发明实施例中的三种胞嘧啶碱基编辑器的实际编辑效果,发明人分别以三种胞嘧啶碱基编辑器为工具进行实际编辑测试。
在本实施中,发明人以三种胞嘧啶碱基编辑器为工具构建阿克森费尔德-里格尔综合征(Axenfeld-Rieger syndrome,ARS)斑马鱼模型。阿克森费尔德-里格尔综合征是一种常染色体显性疾病,其主要特征是眼前节发育不全,有资料证明,pitx2基因的W94*会导致阿克森费尔德-里格尔综合征。因此,发明人根据斑马鱼和人类的pitx2基因比对(如图10所示),找到了斑马鱼在人类W94*部分的同源位点,设计了pitx2 W92*gRNA,并分别使用本发明实施例中的三种胞嘧啶碱基编辑器进行模型构建。
其中,pitx2 W92*gRNA的核苷酸序列为:
pitx2 W92*:5’-CATTTTGCCCGTCGATTCTT(SEQ ID NO:32)-(GAA)-3’。
结果如图11所示。
evoCDA1-BE4max-SpRY全长版本对pitx2 W92*的编辑效果如图11中的A所示,Nolinker版本对pitx2W92*的编辑效果如图11中的B所示,可以看到,相对于全长版本的编辑情况,No linker版本没有产生旁观者效应,即靶位点附近的C被编辑。198aa版本对pitx2W92*的编辑效果如图11中的C所示,同样的,也没有产生旁观者效应。说明No linker版本和198aa版本在编辑精确度上要优于全长版本。
进一步以No linker版本为代表,观测其编辑处理后的模型构建情况。其中,斑马鱼pitx2基因结构图和靶标位点位置,以及No linker版本编辑效果一代测序峰图如图12和13所示。斑马鱼pitx2 W92*F
结果证实,No linker版本实现了对于靶标位点的有效编辑,成功构建了阿克森费尔德-里格尔综合征斑马鱼模型(模型组相对于对照组(野生型)有显著的眼前节发育不全的情况)。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
- 一种基因敲除选育pdlim5b基因缺失型斑马鱼的方法
- 一种植物高效胞嘧啶单碱基编辑器及其构建与应用
- 一种植物高效胞嘧啶单碱基编辑器及其构建与应用
