一种迭代更新的生物学高维数据集的知识推理方法及系统
文献发布时间:2024-04-18 20:01:30
技术领域
本发明属于基于特定计算模型的计算机系统领域,尤其涉及一种迭代更新的生物学高维数据集的知识推理方法及系统。
背景技术
知识推理是通过运用已有知识或信息,从中得出新的结论或信息的过程。这一过程通常基于逻辑、推理规则和先前获得的知识。知识推理从数据中提取信息、模式、关系和知识,通过分析数据来获取洞察力,用于预测未来事件或对数据进行分类。知识推理的主要类型包括:演绎推理:从一般规则或原理中推断出特定情境的结论。归纳推理:从具体的事实、案例或观察中推断出一般的规律或原则。例如,观察到多个案例中的共同特征,推断出一个普遍的规律。维度推理:从结果中推断可能的原因。当给定一些观察结果时,维度推理找到一个可能解释这些结果的原因。模糊推理:在处理模糊或不确定信息时进行推理。模糊推理使用模糊逻辑来处理不精确或模糊的知识。推理规则引擎:基于一系列预定义的规则,通过匹配规则、条件和执行相应的操作来进行推理。
在生物学研究中,生物学高维数据集是指获得的数据具有大量特征或维度的特征集合。在生物学高维数据集的计算中,由于组合数的急剧增加而导致计算复杂性迅速上升,引起组合爆炸问题,因此,穷举法难以适用于鉴定所有的条件变量的组合。
现有技术中,公开号CN114822698A公开了一种基于知识推理的生物学大样本数据集分析方法及系统,该方法采用逻辑数学的方法分析多个变量之间的协同作用对结果产生的影响,有效地发现隐含在数据中的特定的关系,推测数据的发展趋势。公开号CN116230091A公开了一种迭代更新的生物学大样本数据集的知识推理方法及系统,方法包括:收集m个案例的第一序列信息;定义条件变量和结果变量,数据集编码;计算单个条件变量的必要性指数;筛选条件变量,构建新的生物学大样本数据集;最小化推理及组合解释力计算;模型提取;模型统计学计算;变量排除,形成新的用于迭代计算的数据集。该方法的改进在于简化了编码,采用筛选的方法预先处理数据集,采用了变量迭代排除的方法,并且在条件变量的组合的基础上进行模型提取。生物学数据集中有高维和低维之分,上述两种方法的计算内核均为最小化推理,其是一种自上而下的推理方式,其需要对案例进行分组并构建新表,在计算生物学低维数据集时,能获得较好的效果,但是面对生物学高维数据集时,由于组合爆炸,最小化推理占用的计算资源非常大,不仅耗时长,甚至由于计算资源不足无法完成计算。
发明内容
为了解决现有技术中存在的问题,本发明提供了一种迭代更新的生物学高维数据集的知识推理方法及系统,其将条件变量采用三元编码,将结果变量采用二元编码,采用迭代更新的方法,首先随机初始化两个和声库,其次依据新和声产生方法分别产生两个新和声,再次依据和声库更新方法更新两个和声库,如此迭代更新直至达到最大迭代次数,最后进行统计学计算,分析二维变量组合的对结果产生的影响,快速从生物学高维数据集中发现隐含的、有价值的、分类明确的二维条件变量组合的模型。
为解决现有技术的问题,本发明提出了一种迭代更新的生物学高维数据集的知识推理方法,包括:
步骤1:根据待分析的问题,收集m个案例的第一序列信息,所述第一序列信息采样于对应案例的同一位置的基因片段;
步骤2:定义条件变量、结果变量和数据集编码;从所述基因片段中选取n个等位基因定义为条件变量,将待分析的问题定义结果变量,并对每个案例的所述条件变量按照1、2、3进行编码,编码具体为将野生型纯合子基因型编码为1,将杂合子基因型编码为2,将突变纯合子基因型编码为3;对每个案例的所述结果变量按照1、2进行编码,编码具体为将显性结果编码为1,将隐性结果编码为2;每一行代表一个案例,由此构建以数据矩阵表示的生物学高维数据集;
步骤3:初始化迭代更新参数,包括和声库大小HMS,记忆库取值概率HMCR,微调概率PAR,最大迭代次数max_iterations,音调微调带宽 bw;将条件变量两两组合后加上结果变量构成解空间,每个条件变量取值为1,2或3,结果变量取值为1或2;
步骤4:根据步骤3的迭代更新参数和解空间随机初始化产生两个大小均为HMS的和声库HM1和HM2;
步骤5:依据新和声产生方法分别产生两个新和声Hnew1和Hnew2;
步骤6:依据和声库更新方法更新和声库HM1和HM2;
步骤7:构建模型集合MS,对和声库HM1中的和声按必要性指数Nec进行降序排列,对和声库HM2中的和声按组合解释力Exp进行降序排列,扫描和声库HM1和HM2,选出HM1和HM2中都包含的和声构建模型集合MS;
步骤8:模型的统计学计算;将模型集合MS中的和声分别进行统计学计算,统计学计算采用皮尔森卡方检验,将模型集合MS中的和声及其对应的必要性指数Nec、组合解释力Exp、统计学计算得到的P值依次添加至结果集合中,最后得到结果集合。
作为本发明的进一步限定,所述收集m个案例的第一序列信息步骤中,每个案例的第一序列信息收集的步骤包括:
将每平方厘米点阵密度高于400的探针分子固定于支持物上后与标记的样品分子进行杂交,检测每个探针分子的杂交信号强度获取样品分子的序列信息。
作为本发明的进一步限定,所述的和声库HM1和HM2具体为:
HM1将必要性指数Nec作为目标函数,根据二维条件变量的组合的状态Sa×b和结果变量的状态Sy计算必要性指数Nec:Nec=Num(Sa×b,Sy)/Num(Sa×b),其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sa×b)表示条件变量的状态为Sa×b的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种;
HM2将组合解释力Exp作为目标函数,根据二维条件变量的组合的状态Sa×b和结果变量的状态Sy计算组合解释力Exp:Exp=Num(Sa×b,Sy)/Num(Sy),其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sy) 表示结果变量的状态为Sy的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种。
作为本发明的进一步限定,所述新和声产生方法为:在0和1之间随机的产生一个变量rand1,并且将rand1与记忆库取值概率HMCR进行比较,如果rand1小于HMCR,则在和声库中随机抽取一个和声作为预选新和声,如果rand1大于等于HMCR,则在解空间随机抽取一个作为新和声;在0和1之间随机的产生一个变量rand2,并且将rand2与微调概率PAR进行比较,如果rand2小于PAR,则根据微调带宽bw来对预选新和声进行调整,得到一个新和声,如果rand2大于等于PAR,则预选新和声不做调整直接作为新和声。
作为本发明的进一步限定,所述和声库更新方法为:将得到的新和声Hnew1计算必要性指数Nec,如果Hnew1比上面初始化的和声库HM1中最差的组合具有更大的必要性指数Nec,则将新的和声Hnew1替换掉HM1中最差的和声;将得到的新和声Hnew2计算组合解释力Exp,如果Hnew2比上面初始化的和声库HM2中最差的组合具有更大的组合解释力Exp,则将这新的和声Hnew2替换掉HM2中最差的和声;如此迭代,直至达到最大迭代次数max_iterations为止。
本发明的另一方面还提供了一种迭代更新的生物学高维数据集的知识推理系统,其包括序列检测模块,编码模块,必要性指数计算模块,组合解释力计算模块,模型统计学计算模块,迭代更新搜索模块;
所述序列检测模块用于针对待分析的问题,收集m个案例的第一序列信息,所述第一序列信息采样于所述案例的同一位置的基因片段;
所述编码模块用于从所述基因片段中选取n个等位基因定义为条件变量,将待分析的问题定义结果变量,并对每个案例的所述条件变量按照1、2、3进行编码,所述的编码具体为将野生型纯合子基因型编码为1,将杂合子基因型编码为2,将突变纯合子基因型编码为3;对每个案例的所述结果变量按照1、2进行编码,所述的编码具体为将显性结果编码为1,将隐性结果编码为2;每一行代表一个案例,由此构建以数据矩阵表示的生物学高维数据集;
所述必要性指数计算模块用于计算二维条件变量的组合的状态Sa×b相对于结果变量的状态Sy的必要性指数Nec;
所述组合解释力计算模块用于计算二维条件变量的组合的状态Sa×b相对于结果变量的状态Sy的组合解释力Exp;
所述模型统计学计算模块采用皮尔森卡方检验,得到P值;
所述迭代更新搜索模块用于完成迭代更新搜索,迭代更新搜索的步骤包括:首先,初始化迭代更新参数,包括和声库大小HMS,记忆库取值概率HMCR,微调概率PAR,最大迭代次数max_iterations,音调微调带宽 bw;将条件变量两两组合后加上结果变量构成解空间,每个条件变量取值为1,2或3,结果变量取值为1或2;其次,根据迭代更新参数和解空间随机初始化产生两个大小均为HMS的和声库HM1和HM2;HM1将必要性指数Nec作为目标函数,HM2将组合解释力Exp作为目标函数;再次,依据新和声产生方法分别产生两个新和声Hnew1和Hnew2;接着,依据和声库更新方法更新和声库HM1和HM2;然后,构建模型集合MS,对和声库HM1中的和声按必要性指数Nec进行降序排列,对和声库HM2中的和声按组合解释力Exp进行降序排列,扫描和声库HM1和HM2,选出HM1和HM2中都包含的和声构建模型集合MS;最后,模型的统计学计算;将模型集合MS中的和声分别进行统计学计算,统计学计算采用皮尔森卡方检验,将模型集合MS中的和声及其对应的必要性指数Nec、组合解释力Exp、统计学计算得到的P值依次添加至结果集合中,得到结果集合。
作为本发明的进一步限定,所述必要性指数Nec具体为:Nec=Num(Sa×b,Sy)/Num(Sa×b),其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sa×b) 表示条件变量的状态为Sa×b的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种。
作为本发明的进一步限定,所述组合解释力Exp具体为:Exp=Num(Sa×b,Sy)/Num(Sy),其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sy) 表示结果变量的状态为Sy的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种。
作为本发明的进一步限定,所述新和声产生方法为:在0和1之间随机的产生一个变量rand1,并且将rand1与记忆库取值概率HMCR进行比较,如果rand1小于HMCR,则在和声库中随机抽取一个和声作为预选新和声,如果rand1大于等于HMCR,则在解空间随机抽取一个作为新和声;在0和1之间随机的产生一个变量rand2,并且将rand2与微调概率PAR进行比较,如果rand2小于PAR,则根据微调带宽bw来对预选新和声进行调整,得到一个新和声,如果rand2大于等于PAR,则预选新和声不做调整直接作为新和声。
作为本发明的进一步限定,所述和声库更新方法为:将得到的新和声Hnew1计算必要性指数Nec,如果Hnew1比上面初始化的和声库HM1中最差的组合具有更大的必要性指数Nec,则将新的和声Hnew1替换掉HM1中最差的和声;将得到的新和声Hnew2计算组合解释力Exp,如果Hnew2比上面初始化的和声库HM2中最差的组合具有更大的组合解释力Exp,则将这新的和声Hnew2替换掉HM2中最差的和声;如此迭代,直至达到最大迭代次数max_iterations为止。
本发明相比于现有技术具有如下有益效果:
1、本发明采用了自下而上的推理方式,与现有技术中的计算内核(最小化推理,其是一种自上而下的推理方式,对案例进行分组并构建新表,然后逐步消去某些条件变量)相比,本申请将条件变量自下而上两两组合,计算其必要性指数Nec和组合解释力Exp,使得到的条件变量的组合更加简约,不需要进行模型提取,提高了运算速度,适用于生物学高维数据集的计算。
2、本发明采用了迭代更新的方法,设计了两个和声库,这两个和声库分别以必要性指数和解释力作为目标函数,通过和声库更新方法,迭代对和声库进行更新,最后提取两个和声库的交集,做统计学检验。与现有技术中的迭代删除源数据集中的案例或者条件变量相比,不需要删除案例或者条件变量构建新的数据集,降低了计算机算力的要求,改善了面对生物学高维数据集的计算的组合爆炸问题。
3、本发明的编码方案将条件变量编码为1或2或3,结果变量编码为1或2,与现有技术中的编码相比,既适用于卡方检验及多重校验,也可以直接对应基因分型。
4、本发明通过生物学高维数据集所得到的解作为计算提取到的模型的集合,该集合可以作为性状研究的基础材料数据,也可作为生物学网络的推理及构建的参考。
针对上述方案,本发明通过以下参照附图对提供的示例性实施例作详细描述,亦使本发明实施例的其它特征及其优点清楚。
附图说明
为了更清楚地说明本发明实施例的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,应当理解,以下附图仅示出了本发明的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
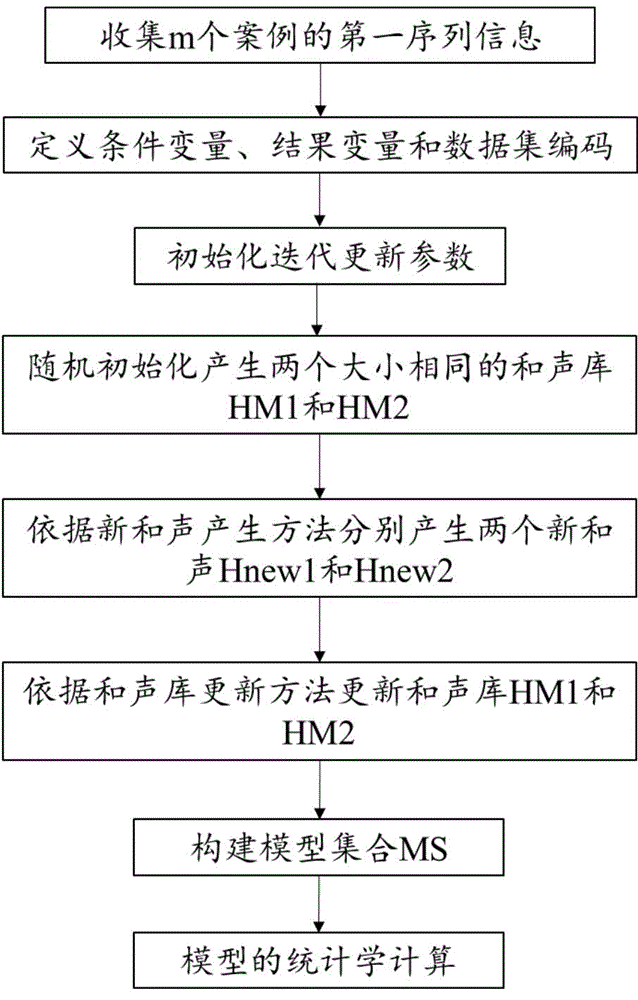
图1为本发明提供的迭代更新的生物学高维数据集的知识推理方法的流程图。
图2为本发明提供的迭代更新的生物学高维数据集的知识推理系统的结构图。
具体实施方式
下面将结合实施例对本发明的技术方案进行清楚、完整地描述。
除非特别说明,否则,本发明中的“第一”、“第二”等描述均用来区分不同的对象,并不用来表示大小或时序等含义,且不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”等的特征可以明示或者隐含地包括一个或者更多个该特征。在本发明创造的描述中,除非另有说明,“多个”的含义是两个或两个以上。
本发明中的术语“和/或”,仅是一种描述关联对象的关联关系,表示可以存在三种关系,例如,A和/或B,可以表示:单独存在A,单独存在B,和同时存在A和B这三种情况。显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
根据本发明的示范性实施例,图1示出了一种迭代更新的生物学高维数据集的知识推理方法的流程图,本发明的迭代更新的生物学高维数据集的知识推理方法包括:
步骤1:根据待分析的问题,收集m个案例的第一序列信息,所述第一序列信息采样于所述案例的同一位置的基因片段,将每平方厘米点阵密度高于400的探针分子固定于支持物上后与标记的样品分子进行杂交,检测每个探针分子的杂交信号强度获取样品分子的序列信息。
步骤2:定义条件变量和结果变量,数据集编码;从所述基因片段中选取n个等位基因定义为条件变量,将待分析的问题定义结果变量,并对每个案例的所述条件变量按照1、2、3进行编码,所述的编码具体为将野生型纯合子基因型编码为1,将杂合子基因型编码为2,将突变纯合子基因型编码为3;对每个案例的所述结果变量按照1、2进行编码,所述的编码具体为将显性结果编码为1,将隐性结果编码为2;每一行代表一个案例,由此构建以数据矩阵表示的生物学高维数据集;
步骤3:初始化迭代更新参数,包括和声库大小HMS,记忆库取值概率HMCR,微调概率PAR,最大迭代次数max_iterations,音调微调带宽 bw;将条件变量两两组合后加上结果变量构成解空间,每个条件变量取值为1,2或3,结果变量取值为1或2;
步骤4:根据步骤3的迭代更新参数和解空间随机初始化产生两个大小均为HMS的和声库HM1和HM2;HM1将必要性指数Nec作为目标函数,根据二维条件变量的组合的状态Sa×b和结果变量的状态Sy计算必要性指数Nec:Nec=Num(Sa×b,Sy)/Num(Sa×b);
其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sa×b) 表示条件变量的状态为Sa×b的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种;HM2将组合解释力Exp作为目标函数,根据二维条件变量的组合的状态Sa×b和结果变量的状态Sy计算组合解释力Exp:Exp=Num(Sa×b,Sy)/Num(Sy);
其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sy) 表示结果变量的状态为Sy的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种;
步骤5:依据新和声产生方法分别产生两个新和声Hnew1和Hnew2; 在0和1之间随机的产生一个变量rand1,并且将rand1与记忆库取值概率HMCR进行比较,如果rand1小于HMCR,则在和声库中随机抽取一个和声作为预选新和声,如果rand1大于等于HMCR,则在解空间随机抽取一个作为新和声;在0和1之间随机的产生一个变量rand2,并且将rand2与微调概率PAR进行比较,如果rand2小于PAR,则根据微调带宽bw来对预选新和声进行调整,得到一个新和声,如果rand2大于等于PAR,则预选新和声不做调整直接作为新和声。
步骤6:依据和声库更新方法更新和声库HM1和HM2; 将得到的新和声Hnew1计算必要性指数Nec,如果Hnew1比上面初始化的和声库HM1中最差的组合具有更大的必要性指数Nec,那么将这新的和声Hnew1替换掉HM1中那个最差的和声;将得到的新和声Hnew2计算组合解释力Exp,如果Hnew2比上面初始化的和声库HM2中最差的组合具有更大的组合解释力Exp,那么将这新的和声Hnew2替换掉HM2中那个最差的和声;如此迭代,直至达到最大迭代次数max_iterations为止。
步骤7:构建模型集合MS,对和声库HM1中的和声按必要性指数Nec进行降序排列,对和声库HM2中的和声按组合解释力Exp进行降序排列,扫描和声库HM1和HM2,选出HM1和HM2中都包含的和声构建模型集合MS;
步骤8:模型的统计学计算;将模型集合MS中的和声分别进行统计学计算,统计学计算采用皮尔森卡方检验,将模型集合MS中的和声及其对应的必要性指数Nec、组合解释力Exp、统计学计算得到的P值依次添加至结果集合中,最后得到结果集合。
根据本发明的示范性实施例,图2为本发明提供的迭代更新的生物学高维数据集的知识推理系统的结构图,本发明的迭代更新的生物学高维数据集的知识推理系统包括:包括序列检测模块,编码模块,必要性指数计算模块,组合解释力计算模块,模型统计学计算模块,迭代更新搜索模块;
所述序列检测模块用于针对待分析的问题,收集m个案例的第一序列信息,所述第一序列信息采样于所述案例的同一位置的基因片段;
所述编码模块用于从所述基因片段中选取n个等位基因定义为条件变量,将待分析的问题定义结果变量,并对每个案例的所述条件变量按照1、2、3进行编码,所述的编码具体为将野生型纯合子基因型编码为1,将杂合子基因型编码为2,将突变纯合子基因型编码为3;对每个案例的所述结果变量按照1、2进行编码,所述的编码具体为将显性结果编码为1,将隐性结果编码为2;每一行代表一个案例,由此构建以数据矩阵表示的生物学高维数据集;
所述必要性指数计算模块用于计算二维条件变量的组合的状态Sa×b相对于结果变量的状态Sy的必要性指数Nec:Nec=Num(Sa×b,Sy)/Num(Sa×b);
其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sa×b) 表示条件变量的状态为Sa×b的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种;
所述组合解释力计算模块用于计算二维条件变量的组合的状态Sa×b相对于结果变量的状态Sy的组合解释力Exp:Exp=Num(Sa×b,Sy)/Num(Sy),其中,Num(Sa×b,Sy)表示条件变量的组合的状态为Sa×b且结果变量的状态为Sy的案例的数量,Num(Sy) 表示结果变量的状态为Sy的案例的数量,二维条件变量的组合的状态包括(a=1×b=1)、(a=1×b=2)、(a=1×b=3)、(a=2×b=1)、(a=2×b=2)、(a=2×b=3)、(a=3×b=1)、(a=3×b=2)、(a=3×b=3)共9种,结果变量的状态包括(y=1)、(y=2)共2种;
所述模型统计学计算模块采用皮尔森卡方检验,得到P值;
所述迭代更新搜索模块用于完成迭代更新搜索,迭代更新搜索的步骤包括:
首先,初始化迭代更新参数,包括和声库大小HMS,记忆库取值概率HMCR,微调概率PAR,最大迭代次数max_iterations,音调微调带宽 bw;将条件变量两两组合后加上结果变量构成解空间,每个条件变量取值为1,2或3,结果变量取值为1或2;
然后,根据迭代更新参数和解空间随机初始化产生两个大小均为HMS的和声库HM1和HM2;HM1将必要性指数Nec作为目标函数,HM2将组合解释力Exp作为目标函数;再次,依据新和声产生方法分别产生两个新和声Hnew1和Hnew2。
需要说明的是,上述的新和声产生方法为:在0和1之间随机的产生一个变量rand1,并且将rand1与记忆库取值概率HMCR进行比较,如果rand1小于HMCR,则在和声库中随机抽取一个和声作为预选新和声,如果rand1大于等于HMCR,则在解空间随机抽取一个作为新和声;在0和1之间随机的产生一个变量rand2,并且将rand2与微调概率PAR进行比较,如果rand2小于PAR,则根据微调带宽bw来对预选新和声进行调整,得到一个新和声,如果rand2大于等于PAR,则预选新和声不做调整直接作为新和声。
接续前面的依据新和声产生方法分别产生两个新和声Hnew1和Hnew2的步骤,再依据和声库更新方法更新和声库HM1和HM2;
最后,进行模型的统计学计算;将模型集合MS中的和声分别进行统计学计算,统计学计算采用皮尔森卡方检验,将模型集合MS中的和声及其对应的必要性指数Nec、组合解释力Exp、统计学计算得到的P值依次添加至结果集合中,得到结果集合。
在一些具体的实施例中,上述的和声库更新方法为:
将得到的新和声Hnew1计算必要性指数Nec,如果Hnew1比上面初始化的和声库HM1中最差的组合具有更大的必要性指数Nec,那么将这新的和声Hnew1替换掉HM1中最差的和声;
将得到的新和声Hnew2计算组合解释力Exp,如果Hnew2比上面初始化的和声库HM2中最差的组合具有更大的组合解释力Exp,那么将这新的和声Hnew2替换掉HM2中最差的和声;如此迭代,直至达到最大迭代次数max_iterations为止;然后,构建模型集合MS,对和声库HM1中的和声按必要性指数Nec进行降序排列,对和声库HM2中的和声按组合解释力Exp进行降序排列,扫描和声库HM1和HM2,选出HM1和HM2中都包含的和声构建模型集合MS。
根据本发明的示范性实施例,本发明还提供了一种迭代更新的生物学高维数据集的知识推理的装置,其特征在于,包括:
测序仪,用于基因测序;
存储器,用于存储程序;
处理器,用于加载程序,以执行如上文所述的迭代更新的生物学高维数据集的知识推理方法。
将本申请中的方法简称为方法一,将现有技术1(公开号CN116230091A)的方案简称为方法二,将现有技术2(公开号CN114822698A)的方案简称为方法三。
通过将三种方法进行编程,分析分析同一数据集,比较得到的结果、已公开文献对结果的支持率及运算时长。第i个条件变量记为CV
实施例1
数据集1包含499个案例,其中结果变量为显性的257例,结果变量为隐性的242例,条件变量个数为8。经过计算,比较结果见表1。
表1 数据集1的分析结果比较
方法一、方法二和方法三均计算得到了1个二维组合,均得到已公开文献的支持,方法一运算时长为方法二的26.9%,为方法三的4.1%。在计算生物学大样本数据集时,方法一的运算速度更快,效率更高。
实施例2
数据集2包含146个案例,其中结果变量为显性的96例,结果变量为隐性的50例,条件变量个数为103611。该数据集为生物学高维数据集,经过计算,比较结果见表2。
表2 数据集2的分析结果比较
方法一合计得到了597个二维组合,已公开文献支持率为65.8%,方法二、方法三由于计算机算力不足,没有得到结果。方法一运算时长为22.58小时。在计算生物学高维数据集时,方法一能够节省计算机算力,获得结果,方法二和方法三无法完成生物学高维数据集的计算。
应当注意的是:因为本发明解决了现有分析方法的需要对案例进行分组并构建新表,在计算生物学低维数据集时,能获得较好的效果,但是面对生物学高维数据集时,由于组合爆炸,最小化推理占用的计算资源非常大,不仅耗时长,甚至由于计算资源不足无法完成计算的问题。
而,本发明的方案采用了自下而上的推理方式,将条件变量自下而上两两组合,计算其必要性指数Nec和组合解释力Exp,使得到的条件变量的组合更加简约,不需要进行模型提取,提高了运算速度,适用于生物学高维数据集的计算;并且,本发明设计了两个和声库,这两个和声库分别以必要性指数和解释力作为目标函数,通过和声库更新方法,迭代对和声库进行更新,最后提取两个和声库的交集,做统计学检验。
并且,与现有技术中的迭代删除源数据集中的案例或者条件变量相比,不需要删除案例或者条件变量构建新的数据集,降低了计算机算力的要求,改善了面对生物学高维数据集的计算的组合爆炸问题。条件变量编码为1或2或3,结果变量编码为1或2,与现有技术中的编码相比,既适用于卡方检验及多重校验,也可以直接对应基因分型。生物学高维数据集数据所得到的解为计算提取到的模型的集合,该集合可以作为性状研究的基础材料数据,也可作为生物学网络的推理及构建的参考。本发明的上述方案能够有效地发现隐含在数据中的某些特定的关系,推测数据的发展趋势的有益技术效果,所以在所附权利要求中要求保护的方案属于专利法意义上的技术方案。
以上所述,仅为本发明的较佳的具体实施方式,但本发明的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本发明揭露的技术范围内,可轻易想到的变化或替换,都应包涵在本发明的保护范围之内。除非以其他方式明确陈述,否则公开的每个特征仅是一般系列的等效或类似特征的一个示例。因此,本发明的保护范围应该以权利要求书的保护范围为准。
- 一种基于服务器的负载均衡方法的智能家居控制系统
- 基于可编程数据平面的服务器负载均衡方法
- 基于可编程数据平面的智能网内负载均衡方法与装置
