一种预测肿瘤间质化机制及治疗敏感性的方法
文献发布时间:2023-06-19 11:06:50
技术领域
本发明涉及一种预测肿瘤间质化机制及治疗敏感性的方法,其属于肿瘤间质化机制分析技术领域。
背景技术
肿瘤是现代医学中导致死亡的主要原因之一。在肿瘤发展过程中,大多数上皮肿瘤将经历间质转变。发生间质转变后肿瘤具有高迁移和侵袭能力,拥有永生增殖并会逃避免疫监视。在临床上,间质转化是提示早期转移,复发以及耐药的指标之一,常与肿瘤的不良预后有关。促癌通路的过度活化是肿瘤间质化的经典机制。近年来,肿瘤微环境对于肿瘤的作用被认为是促进肿瘤发生间质化的另一重要机制。两种机制所导致的间质化所产生的临床结局不同,例如促癌通路导致的间质化常与永生增殖有关,而微环境导致的间质化常与耐药或逃避免疫监视有关。因此,两种机制导致的间质化应该采用不同的治疗方法。然而,目前评估间质化的方法并不能够识别导致肿瘤间质化的机制。本发明涉及了一种能够预测肿瘤间质化机制并分析对应治疗敏感性的方法。
发明内容
为了解决现有技术中存在的问题,本发明提供一种预测肿瘤间质化机制及治疗敏感性的方法。
为实现发明目的,本发明采用如下技术方案:一种预测肿瘤间质化机制及治疗敏感性的方法,该方法包含以下步骤:
步骤一:获取训练样本数据集,从肿瘤基因组图谱TCGA数据库获得训练样本数据集,所述训练样本数据集包括24类上皮肿瘤类型共9415例数据,所述9415例数据包含每例样本的基因突变数据、基因拷贝数变异数据、基因表达count数据和临床信息数据。
步骤二:构建识别间质化机制特征的基因集,从分子特种数据库MsigDB下载四个与肿瘤间质化相关的基因集,所述四个与肿瘤间质化相关的基因集包含①肿瘤间质化,GOID:001837;②正性调控肿瘤间质化,GO ID:0010718;③逆肿瘤间质化,GO ID:0060231和④负性调控肿瘤间质化,GO ID:0010719,去掉基因集之间的重叠数据,识别出只参与肿瘤间质化正性过程的103个基因,利用在线工具蛋白互作分析和在线工具基因共表达分析分析对所述103个基因分析,构建用于识别间质化机制特征的基因集MTCG。
步骤三:根据基因集MTCG特征对TCGA肿瘤数据进行分类,从步骤一的基因表达count数据中提取TCGA肿瘤数据MTCG基因的表达数据,利用K-均值聚类,对训练样本数据集进行分类。
步骤四:四类间质化程度强弱确定,利用ssGSEA算法以步骤三得到的MTCG基因的表达数据为输入,计算每例样本的间质化活性评分,根据表达相对强弱,把样本定义为一类-间质化
步骤五:确定三类-间质化
步骤六:靶向治疗敏感性分析,从肿瘤药敏数据库GDSC获得细胞系数据的基因表达数据和药敏筛选数据,利用R语言预编译包sva包对GDSC数据库的基因表达数据和TCGA数据库中的基因表达count数据进行去批次,提取GDSC数据库中的MTCG基因表达数据,利用MTCG基因表达数据,将细胞系分类到步骤四所述的四类间质化中去,比较每种靶向药的IC50值在四类间质化中的大小,确定三类-间质化
步骤七:免疫治疗敏感性分析,从免疫治疗数据库IMvigor210CoreBiologies获得样本的基因表达和免疫治疗反应性数据,利用R语言预编译包sva包对IMvigor210CoreBiologies数据库的基因表达数据和TCGA数据库中的基因表达count数据进行去批次,提取IMvigor210CoreBiologies数据库中的MTCG基因表达数据,利用MTCG基因表达数据,将IMvigor210CoreBiologies数据库中的患者分类到步骤四所述的四类间质化中,比较四类间质化的免疫治疗反应情况,确定四类-间质化
步骤八:待测样本的间质化分析,获得待测样本的基因表达数据,利用R语言预编译包sva包对待测样本的基因表达数据和TCGA数据库中的基因表达count数据进行去批次,提取待测样本的MTCG基因表达数据,通过计算待测样本与TCGA数据四个聚类中心点的欧几里得距离,把待测样本划归到欧几里得距离最短的间质化类型中,确定待测样本的间质化类型。
所述步骤二中利用在线工具蛋白互作分析对所述103个基因分析,识别出58个节点基因,利用在线工具基因共表达分析对所述103个基因分析,识别出50个共表达基因,对58个节点基因和50个共表达基因取交集,构建用于识别间质化机制特征的包含35个识别基因的基因集MTCG。
所述步骤三中K-均值聚类的参数设置为:模拟次数=100,距离=欧几里得距离,连接方式=平均,聚类后根据一致性数据将聚类数设定为4。
所述步骤五通过比较三类-间质化
所述步骤六通过计算GDSC数据库中的样本与TCGA数据四个聚类中心点的欧几里得距离,并把GDSC数据中的样本划归到欧几里得距离最短的间质化类型中去,分别比较每种靶向药的IC50值在四类间质化中的大小,确定三类-间质化
本发明的有益效果是:一种预测肿瘤间质化机制及治疗敏感性的方法,该方法通过样本训练数据集和识别间质化机制特征的基因集建立模型将将上皮来源肿瘤分成一类-间质化
附图说明
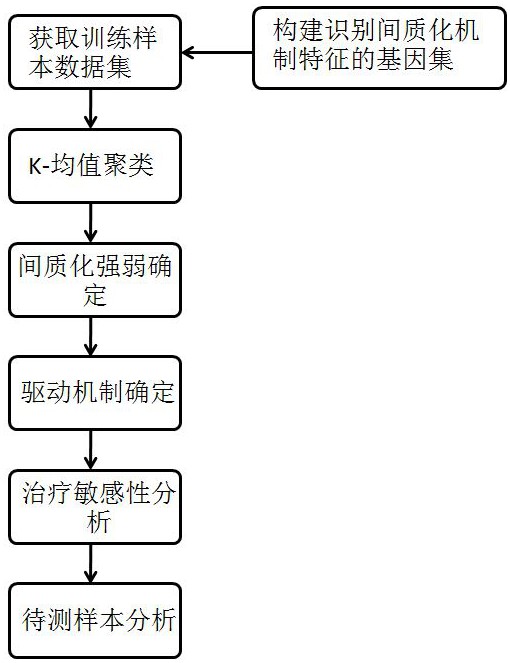
图1是一种预测肿瘤间质化机制及治疗敏感性的方法的流程示意图。
图2是鼠胶质瘤免疫检查点阻断治疗效果对比图。
图3是中国人脑胶质母细胞瘤患者间质化分类预测图。
具体实施方式
为使本发明的技术方案更加清楚,下面将结合本发明的实施例,对实施例中的技术方案进行清楚、完整地描述,以下实施例用于说明本发明,但不用来限制本发明的范围。
图1示出了一种预测肿瘤间质化机制及治疗敏感性的方法的流程示意图。图中,这种预测肿瘤间质化机制及治疗敏感性的方法包含以下步骤:
步骤一:从肿瘤基因组图谱(TCGA)图谱计划数据库获(https://portal.gdc.cancer.gov)得训练样本数据集,该训练集由各个训练样本的基因突变数据、基因拷贝数变异数据、基因表达count数据和临床信息构成。该训练数据集共包括24类上皮肿瘤类型共9415例数据。
步骤二:构建识别间质化机制特征的基因集。从分子特种数据库MsigDB(https://www.gsea-msigdb.org/gsea/index.jsp)下载四个与肿瘤间质化相关的基因集(①肿瘤间质化,GO ID:001837;②正性调控肿瘤间质化,GO ID:0010718;③逆肿瘤间质化,GO ID:0060231;④负性调控肿瘤间质化,GO ID:0010719),四个基因集共包括155个基因。通过韦恩图,识别出103个基因只参与肿瘤间质化的正性过程(即肿瘤间质化和正性调控肿瘤间质化过程)。利用在线工具蛋白互作分析(http://string-db.org)对103个基因分析,构建出蛋白互作网络,利用cytoscape软件对蛋白互作网络中的combined score进行分析,识别出58个节点基因(节点的度在整个网络中排前2/3);利用在线工具基因共表达分析(https://www.coexpedia.org)对103个基因分析,识别出50个共表达基因(评分在前2/3)。对58个节点基因和50个共表达基因取交集,从而构建出用于识别间质化机制特征的基因集,该基因集包括35个基因(MTCG)。
步骤三:根据MTCG特征对TCGA肿瘤数据进行分类。从基因表达count数据中提取TCGA肿瘤数据35个MTCG基因的表达情况。利用K-均值聚类,对步骤一中的训练样本进行聚类,参数如下:模拟次数=100;距离=欧几里得距离;连接方式=平均,输入数据为35个基因的表达情况。聚类后根据一致性数据将聚类数定为4。对35个基因进行PCA分析发现,聚类数为4时,可以很好地把各类患者分开。
步骤四:四类间质化程度强弱确定。利用ssGSEA算法以35个基因表达量为输入,计算每个患者的间质化活性评分。根据表达相对强弱,把患者定义为,一类-间质化
步骤五:三类-间质化
步骤六:靶向治疗敏感性敏感性分析。从肿瘤药敏数据库GDSC(https://www.cancerrxgene.org)获得细胞系数据的基因表达数据和药敏筛选数据。利用R语言预编译包sva包对GDSC数据库的基因表达数据TCGA数据库中的基因表达数据进行去批次。提取GDSC数据库数据中35个MTCG基因的表达情况。利用35个MTCG基因的表达数据,计算GDSC数据库中的样本与TCGA数据四个聚类中心点的欧几里得距离,并把GDSC数据中的样本分别划归到欧几里得距离最短的间质化类型中去。分别比较每种靶向药的IC50值在每类中的大小,从而确定三类-间质化
步骤七:免疫治疗敏感性分析。从免疫治疗数据库IMvigor210CoreBiologies(http://research-pub.gene.com/IMvigor210CoreBiologies/packageVersions/)获得患者样本的基因表达和免疫治疗反应性情况。利用R语言预编译包sva包对IMvigor210CoreBiologies数据库的基因表达数据TCGA数据库中的基因表达数据进行去批次。提取IMvigor210CoreBiologies数据库数据中35个MTCG基因的表达情况。利用35个MTCG基因的表达数据,计算IMvigor210CoreBiologies数据库中的样本与TCGA数据四个聚类中心点的欧几里得距离,并把IMvigor210CoreBiologies数据中的样本分别划归到欧几里得距离最短的那间质化类型中去。比较每类中免疫治疗的反应性情况,从而确定四类-间质化
步骤八:待测样本的间质化分析,获得待测样本的基因表达数据,利用R语言预编译包sva包对待测样本的基因表达数据和TCGA数据库中的基因表达count数据进行去批次,提取待测样本的MTCG基因表达数据,通过计算待测样本与TCGA数据四个聚类中心点的欧几里得距离,把待测样本划归到欧几里得距离最短的间质化类型中,确定待测样本的间质化类型。
实施例1
对鼠胶质瘤细胞系GL261进行测序,获得其基因表达谱,按照具体实施方式中的步骤对鼠胶质瘤细胞系GL261的基因表达谱进行分析,确定鼠胶质瘤细胞系GL261属于四类-间质化
图2是鼠胶质瘤免疫检查点阻断治疗效果对比图,其中,图A是经治疗后生存期对比图,图B是治疗后肿瘤大小对比图,图中,NC是不经任何处理的对照组,MK2206是经AKT通路抑制剂治疗组,PD-L1是免疫检查点治疗组,PD-L1+MK2206是免疫检查点联合AKT通路抑制剂治疗组。从图中可以看出,四类-间质化
实施例2
通过中国脑胶质瘤基因组图谱计划CGGA数据库(http://www.cgga.org.cn)下载388例中国人脑胶质母细胞瘤基因表达数据,通过具体实施方式中的步骤八对其进行分析,将388例中国人脑胶质母细胞瘤分成四种间质化类型。同时,按照步骤四和步骤五对分类后的四组间质化类型进行分析。
图3是中国人脑胶质母细胞瘤患者间质化分类预测图,其中,A是间质化分类结果图,B是四种间质化的间质化评分图,C是四种间质化的YAP通路评分图,D是四种间质化的AKT通路评分图。根据图3,中国人脑胶质母细胞瘤患者被分为四种间质化类型,比较四组的间质化评分,发现三类和四类符合高间质化的特点。比较四组的YAP通路评分,发现三类符合YAP通路激活的特点。比较四组的AKT通路评分,发现四类符合AKT通路激活的特点。可以看出,388例中国人脑胶质母细胞瘤基因数据特征符合一类-间质化
GDSC是药敏数据库,从细胞系的表达谱数据,就可以提取35个基因的表达,然后将其分为四种间质化类型,比较每种药物在四种不同间质化类型中的敏感性情况,从而得到治疗敏感性。
IMvigor210CoreBiologies是免疫治疗数据库,从接受免疫治疗的患者的表达谱中提取35个基因的表达,将其分类,然后比较每类患者中,免疫治疗的反应率,从而得到每类间质化类型的免疫治疗(免疫检查点阻断治疗)敏感或抵抗。之后任意患者样本的数据代入模型中,得到分类。然后根据GDSC和IMvigor210CoreBiologies数据库分析出来的特性,来推定这个患者样本的特性。
以上所述仅是本发明的较佳实施例而已,并非对本发明做任何形式上的限制,任何熟悉本发明的技术人员在不脱离本发明技术范围内,当可利用上述提示的技术内容做出些许变动或修饰为等同变化的等效实施例,但凡是未脱离本发明技术方案的内容,依据本发明的技术实质对以上实施例所做的任何简单修改、等同变化与修饰,均仍属于本发明方案的范围内。
- 一种预测肿瘤间质化机制及治疗敏感性的方法
- 用于确定肿瘤对抗肿瘤药剂的治疗的敏感性的方法
