一种用于生产香草醛的基因片段、酿酒酵母工程菌及其构建方法
文献发布时间:2023-06-19 11:19:16
技术领域
本发明涉及生物工程技术领域,具体涉及一种用于生产香草醛的基因片段、酿酒酵母工程菌及其构建方法。
背景技术
香草醛,又名香兰素(3-甲氧基-4-羟基苯甲醛),是全球最重要香料物质之一,在自然界中存在于一些植物和水果中,由于其特殊的香味,经常被用于食品添加香味制剂。香草醛是中国规定允许使用的食用香料,可作定香剂,是配制香草型香精的主要原料。也可直接用于饼干、糕点、糖果、饮料等食品的加香。香草醛也因特殊的化学和医药性质,被应用到医疗、制药、日化用品、电镀工业等多个行业,因此香草醛全球年需求量非常大。香草醛结构如下:
目前,化学合成香草醛仍然占据大部分市场,但是,化学制剂的危害性迫使人们不断寻找新的获取方法;天然植物中提取香草醛,是一种主要的获取方法,获取的产品香味纯正、安全健康,但是加剧了人类对环境的破坏,对土地资源的浪费,也存在成本高,产量低,价格昂贵等问题。因此微生物合成法便受到人们青睐。
微生物合成法,具有反应条件温和、周期短、成本低、安全性高、与天然香草醛同源性好,产生得到的物质均属于天然产物。目前在微生物异源合成香草醛的研究中,利用最多的底盘宿主菌是大肠杆菌(E.coli)和酿酒酵母(Saccharomyces cerevisae)。Ni,Jun等人通过使用微生物基因模拟植物的天然途径,在大肠杆菌中开发了一条新的代谢途径,用于合成香草醛,代谢工程菌株从左旋酪氨酸产生97.2mg/L香兰素,从葡萄糖产生19.3mg/L香兰素,从木糖产生13.3mg/L香兰素,从甘油产生24.7mg/L香兰素。Hansen等人在2009年,构建了一个真正的从头生物合成途径,可以从粟酒裂殖酵母(也称为裂变酵母或非洲啤酒酵母)以及面包酵母中的葡萄糖生产香兰素。分别导入三个和四个异源基因后,生产率分别为65mg/L和45mg/L。常用的微生物异源基因表达系统分为两种,(1)将多个外源基因放在同一载体质粒上,导入微生物体内,进行诱导表达,这种方法方便快捷,简单易操作。(2)利用DNA同源重组的原理导入微生物基因组上,外源基因位于基因组上,传代稳定,不易丢失,表达效果好。
由于酿酒酵母常作为植物天然产物高效合成的优质宿主平台,因此,开发一组用于生产香草醛的基因片段,并利用该组片段构建可代谢葡萄糖直接生成香草醛的酿酒酵母工程菌,实现天然产物香草醛的酿酒酵母人工合成是本领域需要解决的技术问题之一。
发明内容
为了解决现有技术中存在的问题,本发明的目的在于提供一组用于生产香草醛的基因片段,可用于构建生产香草醛的酿酒酵母工程菌,实现了天然产物香草醛的酿酒酵母人工合成,并经过简单提取即可获得香草醛。
为了实现上述发明目的,本发明提供了以下技术方案:
本发明提供了一组用于生产香草醛的基因片段,包括基因片段1~8;
所述基因片段1含有上游同源序列delta15-up,营养缺陷型标签,同源片段L1;
所述基因片段2含有同源片段L1,基因表达盒TDH3-Sam8-TPI1,基因表达盒ADH1-Sam5-GIT,同源片段L2;
所述基因片段3含有同源片段L2,基因表达盒TEF2-Comt-CYC1,同源片段L3;
所述基因片段4含有同源片段L3,下游同源序列delta15-down;
所述基因片段5含有上游同源序列delta17-up,抗性筛选标签,同源片段L4;
所述基因片段6含有同源片段L4,基因表达盒ENO2-fcs-LRP,同源片段L5;
所述基因片段7含有同源片段L5,基因表达盒ACT1-ech-MDM35,同源片段L6;
所述基因片段8含有同源片段L6,下游同源序列delta17-down。
优选的,所述基因片段1、4、5或8的组装采用OE-PCR方法构建。
优选的,所述基因片段2的组装采用Golden gate方法构建,基因Sam8和基因Sam5的启动子设计“头对头”的方式,终止子则是“脚并脚”的方式,每个连接处插入Bsa I的酶切位点。
优选的,所述基因片段3、6或7的组装采用酶切连接方法构建。
优选的,所述启动子TDH3、ADH1、TEF2、ENO2、ACT1和终止子TPI1、PGI、CYC1、LRP1、MDM35来源于酿酒酵母CEN.PK2-1C基因组。
优选的,所述基因Sam8序列如SEQ ID NO:1所示;基因Sam5序列如SEQ ID NO:2所示;基因Comt序列如SEQ ID NO:3所示;基因fcs序列如SEQ ID NO:4;基因ech序列如SEQ IDNO:5所示。
本发明还提供了一种生产香草醛的酿酒酵母工程菌,所述工程菌基因组包含上述基因片段1~8。
优选的,所述酿酒酵母菌株为CEN.PK2-1C。
本发明还提供了上述酿酒酵母工程菌的构建方法,包括:将基因片段1、2、3、4整合到酿酒酵母基因组上,筛选得到阳性菌株;再将基因片段5、6、7、8整合到所述阳性菌株基因组上,筛选得到生产香草醛的酵母菌株。
本发明还提供了上述基因片段或酿酒酵母工程菌在生产、提取、浓缩香草醛及中间产物上的应用。
与现有技术相比,本发明的技术方案的有益效果如下:
本发明利用香草醛途径中关键酶的基因,经化学合成密码子优化后与酿酒酵母启动子、终止子构建得到基因表达盒,并得到了用于生产香草醛的基因片段,可导入宿主菌中异源表达生产香草醛。
本发明获得了稳定生产香草醛的酿酒酵母菌株,香草醛的合成与酿酒酵母生长相结合,通过酵母自身的莽草酸途径合成所需要的酪氨酸为起点,加入外源基因从酪氨酸开始,逐步合成对香豆酸、咖啡酸、阿魏酸及最终产物香草醛。虽然前提物酪氨酸在酵母体内有分支途径和反馈调节,但是经过人工干预,可以大大提高前提物酪氨酸的产量,进而提升香草醛的产量,相比于大肠杆菌有明显的低毒高产优势。
本发明利用得到的酿酒酵母工程菌提取香草醛及其中间产物的方法简单易操作,香草醛提取效果好,为微生物生产提取香草醛奠定了基础,具有十分广阔的应用前景。
附图说明
图1为香草醛合成路径图;
图2为基因表达模块构建示意图;
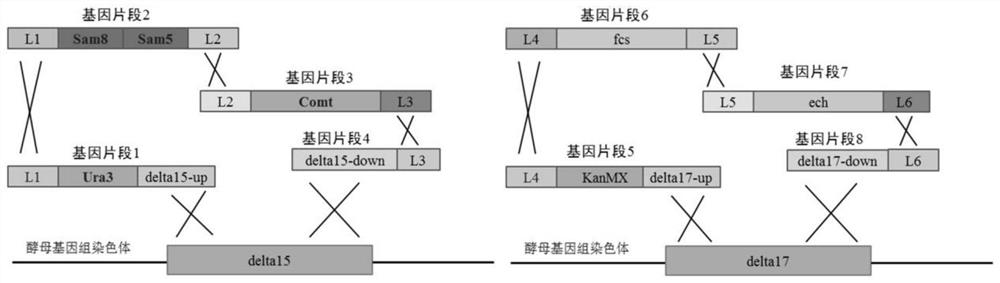
图3为基因片段在酵母基因组delta15和delta17位点的组装原理图;
图4为酿酒酵母工程菌M1的生长曲线;
图5为对香豆酸、咖啡酸、阿魏酸、香草醛的UPLC色谱图;
图6为香草醛的LC-MS的鉴定图;
图7为香草醛定量标准曲线。
具体实施方式
本发明提供了用于生产香草醛的基因片段,包括基因片段1~8。
基因片段1含有上游同源序列delta15-up,营养缺陷型标签,同源片段L1;其中营养缺陷型标签优选为营养缺陷型标签Ura3。
基因片段2含有同源片段L1,基因表达盒TDH3-Sam8-TPI1,基因表达盒ADH1-Sam5-GIT,同源片段L2;其中基因Sam8编码的是酪氨酸脱氨酶(TAL),可将酪氨酸的氨基基团裂解,生成碳碳双键形成对香豆酸;基因Sam5编码的是4-香豆酸-3羟化酶,可裂解对香豆酸苯环上3位点的羟基,形成咖啡酸。
基因片段3含有同源片段L2,基因表达盒TEF2-Comt-CYC1,同源片段L3;其中基因Comt编码的是咖啡酸-O-甲基转移酶,反应在咖啡酸苯环3位点生成甲氧基,形成阿魏酸。
基因片段4含有同源片段L3,下游同源序列delta15-down;
本发明基因片段1~4中的上游同源序列delta15-up、下游同源序列delta15-down和同源片段为宿主菌delta15位点的对应序列拷贝得到。
基因片段5含有上游同源序列delta17-up,抗性筛选标签,同源片段L4;其中抗性筛选标签优选为抗性筛选标签KanMX。
基因片段6含有同源片段L4,基因表达盒ENO2-fcs-LRP,同源片段L5;其中基因fcs编码的是反阿魏酰辅酶A合成酶,可将阿魏酸反应生成阿魏酰辅酶A。
基因片段7含有同源片段L5,基因表达盒ACT1-ech-MDM35,同源片段L6;其中基因ech编码的是烯酰辅酶A水合酶/醛缩酶,可将阿魏酰辅酶A经过两步反应最终生成香草醛。
所述基因片段8含有同源片段L6,下游同源序列delta17-down。
本发明基因片段5~8中的上游同源序列delta17-up、下游同源序列delta17-down和同源片段为宿主菌delta17位点的对应序列拷贝得到。
本发明中基因Sam8来源于放线菌西班牙酵母(Actinomycete Saccharothrixespanaensis),基因Sam5来源于光合细菌球形红杆菌(Rhodobactersphacroides),基因Comt来源于拟南芥(Arabidopsis thaliana),基因fcs和ech来源于链霉菌(Streptomycetaceae);上述五种基因经过网站http://www.jcat.de/进行酿酒酵母密码子优化,规避常用的限制性酶切位点,提交苏州金唯智公司合成获得。
优化后的基因Sam8序列如SEQ ID NO:1所示;优化后的基因Sam5序列如SEQ IDNO:2所示;优化后的基因Comt序列如SEQ ID NO:3所示;优化后的基因fcs序列如SEQ IDNO:4;优化后的基因ech序列如SEQ ID NO:5所示。
本发明中启动子TDH3、ADH1、TEF2、ENO2、ACT1和终止子TPI1、PGI、CYC1、LRP1、MDM35均来源于酿酒酵母基因组,是以酿酒酵母基因组为模板,通过PCR扩增获得的。
本发明基因片段1、4、5或8的组装均采用OE-PCR方法构建。
本发明基因片段2的组装采用Golden gate方法构建,基因Sam8和基因Sam5的启动子设计“头对头”的方式,终止子则是“脚并脚”的方式,每个连接处插入Bsa I的酶切位点,切开后的粘性末端的接口碱基序列也不相同,以防止自连。
本发明基因片段3、6或7的组装采用酶切连接方法构建。
本发明中基因片段1~8的组装方法也可采用其他方法进行替换。
本发明还提供了一种生产香草醛的酿酒酵母工程菌,所述工程菌基因组包含上述基因片段1~8。本发明将基因片段1~4整合到酿酒酵母delta15位点,将基因片段5~8整合到酿酒酵母delta17位点。
酵母delta15位点:上游同源序列delta15-up、带有营养缺陷型标签、同源臂L1、基因表达盒TDH3-Sam8-TPI1、基因表达盒ADH1-Sam5-GIT、同源臂L2、基因表达盒TEF2-Comt-CYC1、同源臂L3、酵母delta15位点下游同源序列delta15-down。
酵母delta17位点:上游同源序列delta17-up、带有抗性筛选标记、同源臂L4、基因表达盒ENO2-fcs-LRP、同源臂L5、基因表达盒ACT1-ech-MDM35、同源臂L6、酵母delta17位点下游同源序列delta17-down。
本发明中酿酒酵母为酿酒酵母菌株优选为CEN.PK2-1C。
本发明还提供了一种生产香草醛的酿酒酵母工程菌的构建方法,包括:将基因片段1、2、3、4整合到酿酒酵母基因组上,筛选得到阳性菌株;再将基因片段5、6、7、8整合到筛选得到的阳性菌株基因组上,筛选得到生产香草醛的酵母菌株。优选的,所述整合方法为醋酸锂方法。
本发明将基因片段1、2、3、4通过同源臂L1、L2、L3,以及delta15位点上下游同源部分delta15-up和delta15-down成功转化外源基因进入酵母基因组上,以尿嘧啶URA3为筛选标记,筛选得到菌株H1。
本发明菌株H1的筛选培养基为SD-Ura3液体培养基,所述SD-Ura3液体培养基包括0.67%YNP,2%葡萄糖,0.06%的四缺的氨基酸混合物drop-out(-His-Leu-Ura-Met),0.06%的三种氨基酸混合物(His-Leu-Trp)。
本发明在H1菌株的基础上,将基因片段5、6、7、8通过之间同源臂L4、L5、L6,以及delta17位点上下游同源部分delta17-up和delta17-down成功转化外源基因进入酵母基因组上,以KanMX为筛选标记,筛选得到菌株M1,M1即为生产香草醛的酵母菌株。
本发明菌株M1的筛选培养基为YPD-G418液体培养基,所述YPD-G418液体培养基包括:酵母浸出粉20g/L,蛋白胨10g/L,葡萄糖20g/L,G418抗生素500mg/L。
本发明将基因片段1~8整合到酿酒酵母基因组上,可成功构建出能合成香草醛的酿酒酵母菌株,并通过验证外源基因能够成功转录翻译,实现生产香草醛的功能。
本发明还提供了上述基因片段或酿酒酵母工程菌在生产、提取、浓缩香草醛及中间产物上的应用。
本发明通过对酿酒酵母工程菌发酵培养后,分离提取香草醛及中间产物。本发明对具体的发酵步骤不作限定。作为一种可选的实施方式,采用YPD培养基对酿酒酵母工程菌进行发酵培养,发酵条件为28~30℃,200~220r/min摇床培养90~96h。
本发明对具体的分离提取步骤不作限定。作为一种可选的实施方式,将发酵液加入等体积的乙酸乙酯,充分震荡萃取30~35min,用台式离心机以4800~5000r/min离心8~10min,将上层乙酸乙酯取出;重复萃取一次,获得两倍发酵液体积的乙酸乙酯萃取液;将萃取液放入旋转蒸发瓶中,用旋转蒸发仪进行浓缩,水浴温度为28~30℃,旋转转速为50~60r/min,将乙酸乙酯完全旋蒸干净,最后加入甲醇溶解,获得高浓度的香草醛及中间产物。
在本发明具体实施过程中,所述材料、试剂等如无特殊说明,均可从商业途径得到。
下面将结合本发明中的实施例,对本发明中的技术方案进行清楚、完整地描述。显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1
本实施例给出了外源基因及基因元件的获取方法。
1、外源基因的获取
香草醛途径的关键基因Sam8、Sam5、Comt、fcs、ech在NCBI数据库中获得基因序列,使用网站http://www.jcat.de/进行酿酒酵母密码子优化,并且规避常用限制性酶切位点,获得可在酿酒酵母体内转录翻译的基因序列,提交苏州金唯智公司经过化学合成获得香草醛的五个关键基因(构建载体为PUC57)。香草醛合成途径如图1所示。
优化后的基因Sam8序列如SEQ ID NO:1所示;优化后的基因Sam5序列如SEQ IDNO:2所示;优化后的基因Comt序列如SEQ ID NO:3所示;优化后的基因fcs序列如SEQ IDNO:4;优化后的基因ech序列如SEQ ID NO:5所示。
2、基因元件的获取
本发明所用到的启动子模块TDH3-ADH1,终止子模块及同源臂L1-TPI1-PGI-L2,启动子模块TEF2-PGK1,终止子模块及同源臂L2-ADH1-CYC1-L3,启动子ENO2-PFY1,终止子及同源臂L4-HOG1-LRP1-L5,启动子GMP1-ACT1,终止子及同源臂L5-SSD1-MDM35-L6,均为实验室(石河子大学化学化工学院生化组冷藏室510)保存,保存名称分别为:P1、T1、P2、T2、P3、T3、P4、T4。
以CEN.PK2-1C基因组为模板进行同源序列的扩增:设计引物15Site1-F和15Site1-R扩增delta15位点上游同源序列delta15-up;设计引物15Site2-F和15Site2-R扩增酵母delta15位点下游同源序列delta15-down;设计引物17Site1-F和17Site1-R扩增酵母delta17位点上游同源序列delta17-up;设计引物17Site2-F和17Site2-R扩增酵母delta17位点下游同源序列delta17-down。引物序列如表1所示。
表1 同源序列扩增引物序列
表1中15Site1-F序列如SEQ ID NO:6所示;15Site1-R序列如SEQ ID NO:7所示;15Site2-F序列如SEQ ID NO:8所示;15Site2-R序列如SEQ ID NO:9所示;17Site2-F序列如SEQ ID NO:10所示;17Site2-R序列如SEQ ID NO:11所示;17Site1-F序列如SEQ ID NO:12所示;17Site1-R序列如SEQ ID NO:13所示。
实施例2
本实施例给出了基因片段的构建方法。
1、基因表达模块的构建
1.1、本发明涉及Sam8、Sam5基因表达模块采用Golden gate的方法进行设计构建,运用酶切的方法,引入Bsa I酶切位点进行基因表达模块的组装。具体实验操作如下:
(1)启动子模块是TDH3-ADH1两个启动子“头对头”进行设计。由中间向两端开始识别,启动基因进行转录翻译。启动子两端加上了Bsa I的限制性酶切位点,并在两端酶切位点之后加上了四个不同序列的碱基对,以便于酶切连接,如图2所示。设计引物P1-F(TDH3-ADH1)和P1-R(TDH3-ADH1),以实验室保存的基因片段为模板,进行扩增获得两端含有Bsa I酶切位点的双启动子模块。引物序列如表2所示。
(2)终止子模块是在一个PUC57的质粒上,两个终止子TPI1-PGI朝识别的相反方向紧密相连,两个终止子之间,插入了两个Bsa I的酶切位点,并且方向相反。终止子末端连接有同源臂L1和L2,如图2所示。设计引物T1-F(TPI1-PGI)和T1-R(TPI1-PGI),以实验室保存的质粒为模板,进行扩增获得两端含有Bsa I酶切位点的双终止子模块,也是含有双终止子的线性化的PUC57的质粒。引物序列如表2所示。
(3)两个外源基因Sam8、Sam5,通过Golden gate方法可同时构建两个基因表达盒,高效快速。设计引物Sam8-F、Sam8-R和Sam5-F、Sam5-R,两端添加上Bsa I的酶切位点和四个碱基对,这四个碱基对在酶切之后,是能与启动子终止子相识别的粘性末端。并以化学合成的外源基因Sam8、Sam5为模板进行扩增,获取基因片段。引物序列如表2所示。
(4)将基因Sam8、Sam5,双启动子TDH3-ADH1和含有双终止子的线性化的质粒进行酶切连接,使用Bsa I进行酶切,使用T7连接酶进行连接,在37℃反应1h,25℃条件下反应1h,导入大肠杆菌DH5α感受态细胞中,在冰上孵育20min,42℃热激45s,再次在冰上孵育3min,加入没有300μL任何抗性的LB培养液,在37℃摇床中220rpm培养45min,进行细胞复苏。之后用涂布器将300μL的菌液涂布在固体LB培养基上,放在37℃培养箱中培养12h。进行菌落PCR验证以及测序验证,以确保基因表达模块构建正确且碱基序列为发生突变。此模块即为基因片段2。
1.2、本发明涉及的Comt、fcs、ech基因表达模块运用单酶切的方法,在基因、启动子和终止子两端引入Bsa I的酶切位点,进行酶切连接构建基因表达模块。具体实验操作如下:
(1)Comt、fcs、ech外源基因所用的启动子,均在实验室保存,设计引物P2-F-1、P2-R、P3-1-F2、P3-1-R2、P4-1-F和P4-1-R,两端引入Bsa I的酶切位点,酶切位点前面加上保护碱基CTCT,酶切位点后面加上酶切部位的碱基对序列。以实验室保存的启动子为模板,进行扩增。引物序列如表2所示。
(2)Comt、fcs、ech外源基因所用的终止子,均在实验室保存,设计引物T2-F、T2-R-1、T3-1-F2、T3-1-R、T4-1-F和T4-1-R,设计的引物两端引入Bsa I的酶切位点,酶切位点前面加上保护碱基CTCT,酶切位点后面加上酶切部位的碱基对序列。通过PCR扩增获得的终止子模块,是在PUC57的质粒上,包含有amp抗性序列,同源臂L2、L3、L4、L5、L6。以实验室保存的终止子质粒为模板,进行终止子模块的扩增。引物序列如表2所示。
(3)Comt、fcs、ech外源基因通过化学合成获得,设计引物Comt-F、Comt-R、fcs-F、fcs-R、ech-F和ech-R,进行基因表达盒的构建。同上述引物设计的方式一样,引物两端加上Bsa I的酶切位点,酶切位点前面加上保护碱基CTCT,酶切位点后面加上酶切部位的碱基对序列。以化学合成获得的基因Comt、fcs、ech为模板进行扩增,获得目的基因片段。引物序列如表2所示。
(4)Comt、fcs、ech三个基因表达模块的构建方法与实施例2中1.1-(4)的酶切方法一样,成功构建出三个基因表达模块,分别为基因片段3、基因片段6、基因片段7。
1.3、基因组delta15和delta17位点同源臂的构建
delta15-up上游序列、Ura3营养缺陷型序列和同源臂L1,通过OE-PCR的方法连接起来,为基因片段1;
delta15-down下游序列和同源臂L3,通过OE-PCR的方法连接起来,为基因片段4;
delta17-up上游序列、抗性基因KanMX序列和同源臂L4,通过OE-PCR的方法连接起来,为基因片段5;
delta17-down下游序列和同源臂L6,通过OE-PCR的方法连接起来,为基因片段8。
表2 基因片段序列扩增引物序列
表2中Sam8-F序列如SEQ ID NO:14所示;Sam8-R序列如SEQ ID NO:15所示;Sam5-F序列如SEQ ID NO:16所示;Sam5-R序列如SEQ ID NO:17所示;P1-F序列如SEQ ID NO:18所示;P1-R序列如SEQ ID NO:19所示;T1-F序列如SEQ ID NO:20所示;T1-R序列如SEQ ID NO:21所示;Comt-F序列如SEQ ID NO:22所示;Comt-R序列如SEQ ID NO:23所示;P2-F-1序列如SEQ ID NO:24所示;P2-R序列如SEQ ID NO:25所示;T2-F序列如SEQ ID NO:26所示;T2-R-1序列如SEQ ID NO:27所示;fcs-F2序列如SEQ ID NO:28所示;fcs-R2序列如SEQ ID NO:29所示;P3-1-F序列如SEQ ID NO:30所示;P3-1-R2序列如SEQ ID NO:31所示;T3-1-F2序列如SEQ ID NO:32所示;T3-1-R序列如SEQ ID NO:33所示;ech-F序列如SEQ ID NO:34所示;ech-R序列如SEQ ID NO:35所示;P4-1-F序列如SEQ ID NO:36所示;P4-1-R序列如SEQ IDNO:37所示;T4-1-F序列如SEQ ID NO:38所示;T4-1-R序列如SEQ ID NO:39所示。
实施例3
本实施例给出了基因片段1~8整合构建到酵母基因组的方法。
醋酸锂酵母转化法步骤如下:
1.制备酵母感受态细胞
(1)在超级工作台中,转接单克隆酵母菌株到无抗生素的20mL YPD培养基中,放入30℃、220r/min的摇床中培养36h。
(2)吸取10%体积的培养36h的酵母菌液,转接至不含抗生素的新鲜YPD培养液中,放入30℃、220r/min的摇床中培养7h。
(3)吸取1.5mL培养的新鲜菌液于已灭菌的离心管中,使用离心机5000r/min,离心5min,弃去上清液。
(4)吸取1mL无菌水清洗酵母细胞,使用离心机5000r/min,离心1min,弃去上清液。
(5)吸取1mL浓度为100mmol/L的LiAc溶液清洗细胞,静置5min,使用离心机4000r/min,离心5min,使用移液器吸取900μL上清液,剩余100μL充分与酵母细胞混匀,即可得到酵母感受态细胞,并放置于冰上以待使用。
2.转化外源基因
(1)吸取40μL的鲑鱼精放置于1.5mL的离心管中,置于沸水5min使其变性,迅速放入冰中以待使用。
(2)在已灭菌的离心管中依次加入外源基因的片段、鲑鱼精、50%w/v的PEG3350溶液480μL和浓度为1M的LiAc溶液72μL。在涡旋震荡仪中震荡1min,充分震荡混匀,30℃温育30min。
(3)吸取72μL的二甲基亚砜加入离心管中,充分混匀,42℃温育10min,使用离心机4000r/min,离心5min,弃去上清液。
(4)吸取200μL浓度为5mM的CaCl
(5)吸取1mL无菌水加入离心管中清洗细胞,温和的混合均匀,使用离心机4000r/min,离心1min,弃去上清液。
(6)重复步骤(5)
(7)加入100μL的无菌水充分混匀,并在超净工作台中,利用涂布器将其涂布在含有抗生素的固体培养基上。放入30℃培养箱,培养3天,观察细胞生长情况,筛选验证获得目的菌株。
将基因片段1~4通过醋酸锂的方法转到酿酒酵母CEN.PK2-1C中,基因片段1、2、3、4之间有同源臂L1、L2、L3通过重组连接在一起,基因片段1和基因片段4中的delta15位点的上、下游同源序列与酵母基因组上delta15位点发生重组而整合到基因组上,以Ura3营养缺陷培养基进行筛选,菌落PCR验证及测序验证,确保连接成功,从而获得重组菌株H1。
再将基因片段5~8通过醋酸锂的方法转到酿酒酵母重组菌株H1中,基因片段5、6、7、8之间有同源臂L4、L5、L6通过重组连接在一起,基因片段5和基因片段8中的delta17位点的上、下游同源序列与酵母基因组上delta17位点发生重组而整合到基因组上,以G418抗性培养基进行筛选,菌落PCR验证及测序验证,确保连接成功,从而获得重组菌株M1。如图3所示。
实施例4
本实施例给出了摇瓶发酵培养重组菌株M1的方法。
发酵培养基:20g/L葡萄糖,20g/L蛋白胨,10g/L酵母浸粉,其余为超纯水。
将菌株M1接种于20mLYPD种子培养基中,在30℃、220rpm的摇床中培养36h,以初始菌体浓度OD600=0.2接种到新鲜的50mL YPD培养基中,培养条件为30℃、220rpm培养96h,在发酵过程中,每12h测一次菌体的生长状态,生长曲线如图4所示。酵母在发酵72h,不添加任何补料的情况下,达到菌体浓度最大值OD600=13.26。
根据测定酵母菌株发酵96h的生长曲线可知,在不添加任何营养物质的前提下,重组酵母菌株在YPD培养基中培养72h达到生长的最大值,之后菌体开始死亡,测得OD600开始呈现缓慢下降趋势。
本发明中OD600值采用UV-5100紫外可见分光光度计(上海元析仪器有限公司)测得。
实施例5
本实施例给出了发酵液提取香草醛及中间产物的方法。
(1)将50mL发酵液加入等体积的乙酸乙酯中,充分震荡萃取30min,台式离心机进行离心5000rpm,将上层乙酸乙酯取出;重复萃取一次,获得100mL乙酸乙酯萃取液。
(2)将100mL萃取液放入旋转蒸发瓶中,用旋转蒸发仪进行浓缩,水浴温度为30℃,旋转转速为5rpm,将乙酸乙酯完全旋蒸干净,最后加入4mL甲醇溶解,获得高浓度的香草醛及中间产物。
本发明采用UPLC-MS检测发酵液提取物
(1)仪器:ACQUITY UPLC超高效液相色谱仪、XEVO TQ-S三重四级杆串联质谱仪、MassLynx工作站(美国Waters公司)。
(2)色谱条件:WatersACQUITYUPLC BEH C18柱(50mm×2.1mm,1.7μm);流速:0.3mL/min;进样量:1μL;柱温箱:30℃。
流动相:A(0.1%甲酸-水)、B(乙腈),梯度洗脱程序如表3所示。
表3 流动相梯度洗脱程序
(3)分别配制对香豆酸、咖啡酸、阿魏酸、香草醛的标准品,用UPLC液相测得各中间产物及香草醛的出峰位置及保留时间,对香豆酸、咖啡酸、阿魏酸、香草醛的保留时间分别是1.94、1.15、2.45、1.93。对酵母菌株M1的发酵提取物进行检测,并测到对香豆酸、咖啡酸、阿魏酸、香草醛四种产物的生成,表明酵母菌株M1的香草醛途径构建成功,如图5所示。
(4)进一步验证终产物香草醛在UPLC中检测的准确性,用LC-MS对香草醛的标准品进行质谱检测。检测条件:电压为6V,碰撞能量为22V。图6下部分为香草醛标准品的质谱图,打碎的母离子质荷比(m/z)为150.8及子离子m/z为135.8。再对酵母菌株M1的发酵提取物进行检测,得到图6上部分的质谱图,做出来的母离子m/z及子离子m/z完全照应,表明酵母菌株可以成功合成香草醛。
(5)配制香草醛的标准品,用甲醇定容成质量浓度为100、250、500、750、1000ng/mL香草醛对照溶液,进样1μL,以定量离子峰面积y为纵坐标,质量浓度(x,ng/mL)为横坐标绘制工作曲线,如图7所示。相关系数R
采用此标准曲线方程计算LC-MS测得的香草醛及中间产物含量得到:在不添加任何前提物的情况下,香草醛的产量达到了50.2μg/L。在添加1mmoL阿魏酸为前提物,通过发酵测得香草醛的产量达到了10.05mg/L的产量。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110> 石河子大学
<120> 一种用于生产香草醛的基因片段、酿酒酵母工程菌及其构建方法
<160> 39
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1533
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
atgactcaag ttgttgaaag acaagctgac agattgtctt ctagagaata cttggctaga 60
gttgttagat ctgctggttg ggacgctggt ttgacttctt gtactgacga agaaatcgtt 120
agaatgggtg cttctgctag aactatcgaa gaatacttga agtctgacaa gccaatctac 180
ggtttgactc aaggtttcgg tccattggtt ttgttcgacg ctgactctga attggaacaa 240
ggcggcagct tgatctctca cctcggtact ggtcaaggtg ctccattggc tccagaagtt 300
tctagattga tcttgtggtt gagaatccaa aacatgagaa agggttactc tgctgtttct 360
ccagttttct ggcaaaagtt ggctgacctc tggaacaagg gcttcactcc agctatacca 420
agacacggta ctgtttctgc ttctggtgac ttgcaaccat tggctcacgc tgctttggct 480
ttcactggtg ttggtgaagc ttggactaga gacgctgacg gtagatggtc tactgttcca 540
gctgttgacg ctttggctgc tttgggtgct gaaccattcg actggccagt tagagaagct 600
ttggctttcg ttaacggtac tggtgcttct ttggctgttg ctgttttgaa ccacagatct 660
gctttgagat tggttagagc ttgtgctgtt ttgtctgcta gactcgcgac attgttgggt 720
gctaatccag aacactacga cgttggtcac ggtgttgcta gaggtcaagt tggtcaattg 780
actgctgctg aatggatcag acaaggtttg ccaagaggta tggttagaga cggttctaga 840
ccattgcaag aaccatactc tttgagatgt gctccacaag ttttgggtgc tgttttggac 900
caattggacg gtgctggtga cgttttggct agagaagttg acggttgtca agacaaccca 960
atcacttacg aaggtgaatt gttgcacggt ggtaacttcc acgctatgcc agttggtttc 1020
gcttctgacc aaatcggttt ggctatgcac atggctgctt acttggctga aagacaattg 1080
ggtttgttgg tttctccagt tactaacggt gacttgccac caatgttgac tccaagagct 1140
ggtagaggtg ctggtttggc tggtgttcaa atctctgcta cttctttcgt ttctagaatc 1200
agacaattgg ttttcccagc ttctttgact actttgccaa ctaacggttg gaaccaagac 1260
cacgttccaa tggctttgaa cggtgctaac tctgttttcg aagctttgga attgggttgg 1320
ttgactgttg gttctttggc tgttggtgtt gctcaattgg ctgctatgac tggtcacgct 1380
gctgaaggtg tttgggctga attggctggt atctgtccac cattggacgc tgacagacca 1440
ttgggtgctg aagttagagc tgctagagac ttgttgtctg ctcacgctga ccaattgttg 1500
gttgacgaag ctgacggtaa ggacttcggt taa 1533
<210> 2
<211> 1539
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
atgactatca cttctccagc tccagctggt agattgaaca acgttagacc aatgactggt 60
gaagaatact tggaatcttt gagagacggt agagaagttt acatctacgg tgaaagagtt 120
gacgacgtta ctactcactt ggctttcaga aactctgtta gatctatcgc tagattgtac 180
gacgttttgc acgacccagc ttcggagggt gtgctcagag ttccaactga cactggcaat 240
ggtggcttca ctcatccatt cttcaagacc gctagatctt ctgaagactt ggttgctgct 300
agagaagcta tcgttggttg gcaaagattg gtttacggtt ggatgggtag aactccagat 360
tacaaggctg cgttcttcgg tactctcgac gctaacgctg aattctacgg tccattcgaa 420
gctaacgcta gaagatggta cagagacgct caagaaagag ttttgtactt caaccacgct 480
atcgttcacc caccagttga cagagacaga ccagctgaca gaactgctga catctgtgtt 540
cacgttgaag aagaaactga ctctggtttg atcgtttctg gtgctaaggt tgttgctact 600
ggttctgcta tgactaacgc taacttgatc gctcactacg gtttgccagt tagagacaag 660
aagttcggtt tggttttcac tgttccaatg aactctccag gtttgaagtt gatctgtaga 720
acttcttacg aattgatggt tgctactcaa ggttctccat tcgactaccc attgtcttct 780
agattggacg aaaacgactc tatcatgatc ttcgacagag ttttggttcc atgggaaaac 840
gttttcatgt acgacgctgg tgctgctaac tctttcgcta ctggttctgg tttcttggaa 900
agattcactt tccacggttg tactagattg gctgttaagt tggacttcat cgctggttgt 960
gttatgaagg ctgttgaagt tactggtact actcacttca gaggtgttca agctcaagtt 1020
ggtgaagttt tgaactggag agacgttttc tggggtttgt ctgacgctat ggctaagtct 1080
ccaaactctt gggttggtgg ttctgttcaa ccaaacttga actacggttt ggcttacaga 1140
actttcatgg gtgttggtta cccaagaatc aaggaaatca tccaacaaac tttgggttct 1200
ggtttgatct acttgaactc ttctgctgct gactggaaga acccagacgt tagaccatac 1260
ttggacagat acttgagagg ttctagaggt atccaagcta tcgacagagt taagttgttg 1320
aagttgttgt gggacgctgt tggtactgaa ttcgctggta gacacgaatt gtacgaaaga 1380
aactacggtg gtgaccacga aggtatcaga gttcaaactt tgcaagctta ccaagctaac 1440
ggtcaagctg ctgctttgaa gggtttcgct gaacaatgta tgtctgaata cgacttggac 1500
ggttggacta gaccagactt gatcaaccca ggtacttaa 1539
<210> 3
<211> 1092
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
atgggttcta ctgctgaaac tcaattgact ccagttcaag ttactgacga cgaagctgct 60
ttgttcgcta tgcaattggc ttctgcttct gttttgccaa tggctttgaa gtctgctttg 120
gaattggact tgttggaaat catggctaag aacggttctc caatgtctcc aactgaaatc 180
gcttctaagt tgccaactaa gaacccagaa gctccagtta tgttggacag aatcttgaga 240
ttgttgactt cttactctgt tttgacttgt tctaacagaa agttgtctgg tgacggtgtt 300
gaaagaatct acggtttggg tccagtttgt aagtacttga ctaagaacga agacggtgtt 360
tctatcgctg ctttgtgttt gatgaaccaa gacaaggttt tgatggaatc ttggtaccac 420
ttgaaggacg ctatcttgga cggtggtatc ccattcaaca aggcttacgg tatgtctgct 480
ttcgaatacc acggtactga cccaagattc aacaaggttt tcaacaacgg tatgtctaac 540
cactctacta tcactatgaa gaagatcttg gaaacttaca agggtttcga aggtttgact 600
tctttggttg acgttggtgg tggtatcggt gctactttga agatgatcgt ttctaagtac 660
ccaaacttga agggtatcaa cttcgacttg ccacacgtta tcgaagacgc tccatctcac 720
ccaggtatcg aacacgttgg tggtgacatg ttcgtttctg ttccaaaggg tgacgctatc 780
ttcatgaagt ggatctgtca cgactggtct gacgaacact gtgttaagtt cttgaagaac 840
tgttacgaat ctttgccaga agacggtaag gttatcttgg ctgaatgtat cttgccagaa 900
actccagact cttctttgtc tactaagcaa gttgttcacg ttgactgtat catgttggct 960
cacaacccag gtggtaagga aagaactgaa aaggaattcg aagctttggc taaggcttct 1020
ggtttcaagg gtatcaaggt tgtttgtgac gctttcggtg ttaacttgat cgaattgttg 1080
aagaagttgt aa 1092
<210> 4
<211> 1476
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
atgagaaacc aaggtttggg ttcttggcca gttagaagag ctagaatgtc tccacacgct 60
actgctgtta gacacggcgg cactgctctc acttacgctg agttgtctag aagagttgct 120
agattggcta acggtttgag agctgctggt gttagaccag gtgacagagt tgcttacttg 180
ggtccaaacc acccagctta cctcgaaaca ttgttcgctt gtggccaagc tggtgctgtt 240
ttcgttccat tgaacttcag attgggtgtt ccagagctcg accacgctct cgctgactct 300
ggcgcttctg ttttgatcca cactccagaa cacgctgaaa ctgttgctgc tttggctgct 360
ggtagattgt tgagagttcc agctggtgaa ttggacgctg ctgacgacga accaccagac 420
ttgccagttg gtttggacga cgtttgtttg ttgatgtaca cttctggttc tactggtaga 480
ccaaagggtg ctatgttgac tcacggtaac ttgacttgga actgtgttaa cgttttggtt 540
gaaactgact tggcttctga cgaaagagct ttggttgctg ctccattgtt ccacgctgct 600
gctttgggta tggtttgttt gccaactttg ttgaagggtg gtactgttat cttgcactct 660
gctttcgacc caggtgctgt tttgtctgct gttgaacaag aaagagttac tttggttttc 720
ggtgttccaa ctatgtacca agctatcgct gctcacccaa gatggagatc tgctgacttg 780
tcttctttga gaactttgtt gtgtggtggt gctccagttc cagctgactt ggcttctaga 840
tacttggaca gaggtttggc tttcgttcaa ggttacggta tgactgaagc tgcgccaggt 900
gtactcgttt tggacagagc tcacgttgct gaaaagatcg gttctgctgg tgttccaagc 960
ttcttcactg acgttagatt ggctggtcca tctggtgaac cagttccacc aggtgaaaag 1020
ggtgaaatcg ttgtttctgg tccaaacgtt atgaagggtt actggggtag accagaagct 1080
actgcggaag tcctcagaga cggttggttc cactctggtg acgttgctac tgttgacggt 1140
gacggttact tccacgttgt tgacagattg aaggacatga tcatctctgg tggtgaaaac 1200
atctacccag ctgaagttga aaacgaattg tacggttacc caggtgttga agcttgtgct 1260
gttatcggtg ttccagaccc aagatggggt gaagttggta aggctgttgt tgttccagct 1320
gacggttcta gaatcgacgg tgacgaattg ttggcttggt tgagaactag attggctggt 1380
tacaaggttc caaagtctgt tgaattcact gacagattgc caactactgg ttctggtaag 1440
atcttgaagg gtgaagttag aagaagattc ggttaa 1476
<210> 5
<211> 864
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
atgtctactg ctgttggtaa cggtagagtt agaactgaac catggggtga aactgttttg 60
gttgaattcg acgaaggtat cgcttgggtt atgttgaaca gaccagacaa gagaaacgct 120
atgaacccaa ctttgaacga cgaaatggtt agagttttgg accacttgga aggtgacgac 180
agatgtagag ttttggtttt gactggtgct ggtgaatctt tctctgctgg tatggacttg 240
aaggaatact tcagagaagt tgacgctact ggttctactg ctgttcaaat caaggttaga 300
agagcttctg ctgaatggca atggaagaga ttggctaact ggtctaagcc aactatcgct 360
atggttaacg gttggtgttt cggtggtgct ttcactccat tggttgcttg tgacttggct 420
ttcgctgacg aagacgctag attcggtttg tctgaagtta actggggtat cccaccaggc 480
ggcgttgtaa gcagagcttt ggctgctact gttccacaaa gagacgcttt gtactacatc 540
atgactggtg aaccattcga cggtagaaga gctgctgaaa tgagattggt taacgaagct 600
ttgccagctg acagattgag agaaagaact agagaagttg ctttgaagtt ggcttctatg 660
aaccaagttg ttttgcacgc tgctaagact ggttacaaga tcgctcaaga aatgccatgg 720
gaacaagctg aagactactt gtacgctaag ttggaccaat ctcaattcgc tgacaaggct 780
ggtgctagag ctaagggttt gactcaattc ttggaccaaa agtcttaccg tccaggcttg 840
tctgctttcg acccagaaaa gtaa 864
<210> 6
<211> 29
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
gccaggcgcc tttatatcat ataattaag 29
<210> 7
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
ctcgagaggc aattttagag ggg 23
<210> 8
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
tagacgccaa ctacgctgac 20
<210> 9
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
ataaagcagc cgctaccaaa c 21
<210> 10
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
attggacgag ttctacctga c 21
<210> 11
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
aaagctggct ccccttag 18
<210> 12
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
actgaaatta gagacaactg ttatc 25
<210> 13
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
aggttccaac tgctcttact g 21
<210> 14
<211> 37
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
ctctggtctc agaagatgac tcaagttgtt gaaagac 37
<210> 15
<211> 36
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
ctctggtctc aaggtttaac cgaagtcctt accgtc 36
<210> 16
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
ctctggtctc agaatatgac tatcacttct ccagc 35
<210> 17
<211> 37
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
ctctggtctc acgatttaag tacctgggtt gatcaag 37
<210> 18
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 18
ctctggtctc acttctttgt ttgtttatgt gtgtttattc 40
<210> 19
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 19
ctctggtctc aattctgtat atgagatagt tgattgtatg 40
<210> 20
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 20
ctctggtctc aatcgaacaa atcgctctta aatatatacc 40
<210> 21
<211> 44
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 21
ctctggtctc aacctgatta atataattat ataaaaatat tatc 44
<210> 22
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 22
ctctggtctc agaatatggg ttctactgct gaaac 35
<210> 23
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 23
ctctggtctc acgatttaca acttcttcaa caattcgatc 40
<210> 24
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 24
ctctggtctc acttcatctg tgcgttatac ttacatatag 40
<210> 25
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 25
ctctggtctc aattcggtac tagtgtttag ttaattatag 40
<210> 26
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 26
ctctggtctc aatcgtcatg taattagtta tgtcacgc 38
<210> 27
<211> 37
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 27
ctctggtctc agaagcatgc cgatagtccg cgagttg 37
<210> 28
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 28
ctctggtctc aattcatgag aaaccaaggt ttggg 35
<210> 29
<211> 36
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 29
ctctggtctc acgatttaac cgaatcttct tctaac 36
<210> 30
<211> 45
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 30
ctctggtctc aattcaggag acgttacttt gtttatatat attag 45
<210> 31
<211> 33
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 31
ctctggtctc agaattatta ttgtatgtta tag 33
<210> 32
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 32
ctctggtctc aatcgaggtc gacgtatact ggtac 35
<210> 33
<211> 34
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 33
ctctggtctc agaataggtt ccaactgctc ttac 34
<210> 34
<211> 37
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 34
ctctggtctc agaatatgtc tactgctgtt ggtaacg 37
<210> 35
<211> 36
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 35
ctctggtctc acgatttact tttctgggtc gaaagc 36
<210> 36
<211> 31
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 36
ctctggtctc acttcacaag cgcgcctcta c 31
<210> 37
<211> 43
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 37
ctctggtctc aattctgtta attcagtaaa ttttcgatct tgg 43
<210> 38
<211> 39
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 38
ctctggtctc aatcgtttag cacagaatgt gcattattc 39
<210> 39
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 39
ctctggtctc agaagcgacg aacgagatac gatag 35
- 一种用于生产香草醛的基因片段、酿酒酵母工程菌及其构建方法
- 一种酿酒酵母基因工程菌株以及此菌株的构建方法和生产圣草酚或五羟黄酮的方法
