一种缺血性脑损伤差异蛋白组合及非诊断用的MCAO大鼠差异蛋白组学分析方法
文献发布时间:2023-06-19 11:29:13
技术领域
本发明属于生物医学检测领域,具体涉及脑缺血病的差异蛋白及非治病用的MCAO大鼠差异蛋白组学分析方法。
背景技术
缺血性脑卒中又称“中风”,是一种急性脑血管疾病,具有发病率高和死亡率高等特点,是威胁人类健康的主要疾病之一。缺血性脑卒中的病理机制仍有许多未知之处。目前缺血性脑卒中的诊断主要依赖于临床诊断和影响学检查。但在缺血早期,脑CT对梗死部位不敏感,而磁共振成像价格较为昂贵。寻找理想的脑卒中标记物,对于脑卒中的诊断治疗具有非常重要的意义。近年来,蛋白组学的快速发展促进了脑卒中生物标记物的研究。这种方法通过考察蛋白质性质和丰度揭示蛋白功能,从而找到与脑卒中相关的差异蛋白,使其成为疾病发生的生物标记物或药物筛选的靶标。
广泛被采用的是基于二维凝胶电泳技术的蛋白组学方法,但这种方法操作耗时,难以自动化,且动态范围有限。液质联用的蛋白组学操作简单,节省了工作时间,在分析微量样品中有更大优势,规避了二维凝胶电泳的缺点,成为一种必然的发展趋势。通过蛋白标志物来进行脑缺血病的早期检测具有十分重要的价值和意义。
发明内容
为了克服现有技术上的问题,本发明提供一种缺血性脑损伤差异蛋白组合及非诊断用的MCAO 大鼠差异蛋白组学分析方法。
本发明提供以下技术方案:
一种缺血性脑损伤的差异蛋白组合,所述蛋白组合包括以下17种蛋白:
一种缺血性脑损伤的差异蛋白组合所述蛋白组合包括以下25种蛋白:
一种缺血性脑损伤的差异蛋白组合,所述蛋白组合包括以下7种蛋白:
进一步地,所述蛋白组合来自大鼠模型动物。
缺血性脑损伤的差异蛋白组合在检测脑缺血病的药物中的应用。
进一步地,其中,NADH:ubiquinone oxidoreductase subunit A1(Ndufa1),mitochondrial ribosomal protein S9(Mrps9),cytochrome c oxidase subunit VIIapolypeptide 2(Cox7a2), NADH:ubiquinone oxidoreductase subunit A4(Ndufa4),ribosomal protein S7(Rps7),mitochondrial ribosomal protein L21(Mrpl21),mitochondrial ribosomal protein S17(Mrps17),mitochondrial ribosomal proteinS5(Mrps5),mitochondrial ribosomal protein L3(Mrpl3),cytochrome c oxidasesubunit 4i1(Cox4i1),cytochrome c oxidase subunit 6C(Cox6c),ribosomal proteinL27(Rpl27), cytochrome c oxidase subunit 7C(Cox7c),ATP synthase,H+transporting,mitochondrial F1 complex,O subunit(Atp5o),mitochondrialribosomal protein L23(Mrpl23),mitochondrial ribosomal protein S18A(Mrps18a)蛋白表达水平下调;BCL2 associated X,apoptosis regulator(Bax)蛋白表达水平上调。
进一步地,其中,serpin family A member 1(Serpina1),protein kinase C,beta(Prkcb), ceruloplasmin(Cp),small nuclear ribonucleoprotein polypeptide G(Snrpg),biliverdin reductase B(Blvrb),Aly/REF export factor(Alyref),serpinfamily E member 1(Serpine1),kininogen 1(Kng1), Complement C3(C3),fibrinogengamma chain(Fgg),fibrinogen alpha chain(Fga),heme oxygenase 1(Hmox1),heatshock 70kD protein 1A(Hspa1a),transferrin(Tf),fibrinogen beta chain(Fgb),hexokinase 2(Hk2),TIMP metallopeptidase inhibitor 1(Timp1),Complement C1q Bchain(C1qb), RNA binding motif protein,X chromosome retrogene-like(Rbmxrtl),plasminogen(Plg),nuclear cap binding protein subunit 1(Ncbp1),uroporphyrinogen III synthase(Uros),serine and arginine rich splicing factor2(Srsf2),heterogeneous nuclear ribonucleoprotein A3(Hnrnpa3))蛋白表达水平上调; calcium/calmodulin-dependent protein kinase II gamma(Camk2g)蛋白表达水平下调。
进一步地,其中,3-phosphoinositide dependent protein kinase-1(Pdpk1),apolipoprotein A1(Apoa1),apolipoprotein A2(Apoa2),calcium/calmodulin-dependent protein kinase I(Camk1), calcium/calmodulin-dependent proteinkinase ID(Camk1d)蛋白表达水平上调;diazepam binding inhibitor,acyl-CoA bindingprotein(Dbi),calcium voltage-gated channel subunit alpha1 G(Cacna1g)蛋白表达水平下调。
一种非诊断用的MCAO大鼠的差异蛋白组合的蛋白质组学分析方法,
1)建立假手术组(Sham组)及大脑中动脉阻塞(MCAO组)大鼠模型,对模型进行神经功能学评分;
2)TTC染色,测量和分析脑梗死面积;
3)得到大鼠脑组织,分为左侧和右侧脑组织;
4)从左侧脑组织中提取蛋白质;
5)对蛋白质依次进行酶解、脱盐;
6)进行肽段iTRAQ标记、肽段分离和LC-MS质谱分析;
7)将所述步骤6)中得到的质谱鉴定结果分别进行数据搜库、定量分析,获取MCAO大鼠组和Sham组大鼠的脑组织的差异蛋白质。
进一步地,所述肽段分离时,将iTRAQ标记后的多肽样品重溶于A液(98%ddH2O,2%乙腈,氨水调pH至10.0)中。经离线
进一步地,在所述LC-MS质谱分析时,将多肽组分重溶于30μL,0.1%FA溶液中,经过U3000 液相,低pH反相C18毛细管色谱(150μm×150mm,1.9μm)分离,A相为0.1%FA的水溶液,B 相为80%乙腈,0.1%FA的水溶液,有效洗脱梯度为6%-40%的B相,总洗脱时间100min,流速 0.6μL/min。液相条件如下表:
进一步地,使用Orbitrap Q-Exactive HF-X质谱仪分析检测多肽混合物,扫描时间100min。质谱采集模式为高灵敏度模式,仪器参数设置:每个全扫描为高速信号依赖扫描,一级全扫描分辨率120000,AGC 3e6,扫描范围350-1500m/z;每一个一级扫描后筛选前20个离子进入碰撞池打碎,二级扫描分辨率45000,AGC 5e4,扫描范围350-1500m/z,碰撞能量32%,电荷状态筛选 (包含+2至+5电荷的前体),动态消除15s。
采用上述技术方案,本发明具有如下有益效果:
1、脑缺血时,脑组织最先受到影响。线粒体作为能量代谢的工厂,也是最易受到影响的细胞器。差异蛋白组一是和线粒体氧化磷酸化、核糖体通路相关的蛋白。脑缺血发生时,这些蛋白首先受到影响,通过测定其蛋白表达的变化,可表征脑缺血的发生。差异蛋白组二是和补体系统, HIF-1信号通路相关的蛋白,差异蛋白组三是和PPAR信号通路相关的蛋白。脑缺血发生时,补体系统和HIF-1信号通路被激活,产生大量的活性物质,增加了血管的通透性,可表征脑缺血发生的特征。脑缺血发生时,PPAR信号通路也被激活,对脑组织的神经元起保护作用。这三组差异蛋白组合均能作为生物标志物,对缺血性脑损伤、脑缺血病进行早期检测,灵敏度高、精度强。
2、脑缺血发生时,生命活动的执行者-蛋白不可避免的发生各种变化。研究蛋白的方法包括经典的生物学方法和质谱方法。经典的生物手段只能同时表征几个蛋白的变化,抗体成本较高,而本发明的蛋白组学方法以蛋白组为研究对象,可同时表征大量蛋白的表达变化,不需要抗体,适宜作为评价脑缺血发生的一种工具。
附图说明
图1是本发明实施例中神经功能评分结果图;
图2是本发明实施例中TTC染色结果图;
图3是本发明实施例中SDS-PAGE结果图;
图4是本发明实施例中标记强度分布及定量权重的图谱;
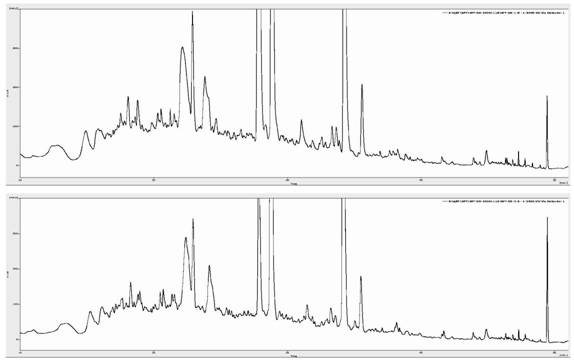
图5本发明实施例中色谱图谱;
图6是本发明实施例中差异表达火山图。
具体实施方式
为了使本发明的目的、技术方案及优点更加清楚明白,下面结合附图及实施例,对本发明进行进一步详细说明。应当理解,此处所描述的结构图及具体实施例仅用以解释本发明,并不用于限定本发明。
实施例1
MCAO大鼠模型,神经行为损伤评分。
将大鼠分为假手术组和脑缺血组,每组大鼠10只。采用大鼠中动脉栓塞(MCAO)模型,假手术组只进行术前麻醉以及血管分离,不进行线栓,其余各组均进行MCAO模型。采用10%水合氯醛腹腔注射(400mg/kg)对大鼠进行麻醉,将大鼠仰卧固定,75%酒精消毒颈部皮肤,于正中切口,分离并暴露右侧颈总动脉,并在近心端夹闭颈总动脉,继续分离出颈外和颈内动脉,结扎颈外动脉,在颈总动脉距分叉处1.5cm处剪一小口,用栓线沿颈总动脉插入颈内动脉。当进入颈动脉分叉处以上的栓线长度为2cm时,停止并结扎开口上下的颈总动脉,整个手术过程中室温度保持在(22±2)℃。术后12h,进行神经功能评分及取材。
参照Bederson五级评分法(0-4分),将动物于处死前进行神经行为学观察和打分,分数越高,说明其神经行为损伤越严重。大鼠置于地面,观察其行走情况。分数越高,说明其神经行为损伤越严重。
(1)行为正常者,记0分;
(2)将大鼠提离地面,手术对侧前肢内旋、内收者,记1分;
(3)用手挤压大鼠两侧检查其抗力,MCAO手术对侧抗力下降者,记2分;
(4)观察大鼠地面行走,围绕手术对侧转圈者,记3分;
(5)损伤极其严重,无法自主活动者,记4分。
大鼠行MCAO手术12h后,模型组神经行为学评分均显著高于假手术组,出现了明显的神经行为学损伤。表明模型成功复现。
TTC染色,测量和分析脑梗死面积。
生理盐水灌注后获取整个大脑,转至-20℃冰冻切片机中做厚度约为2mm切片,然后将切片放入2,3,5-氯化三苯基四氮唑染液(2%)中,在37℃的温箱中避光孵育30min。再用4%多聚甲醛固定,并对整个大脑切片并拍照,最后用Image J测量和分析脑梗死面积(缺血梗死面积比=各白色缺血区面积之和/各脑片面积之和*100%)见图2。
TTC染色结果可知,模型组的脑梗死面积大于假手术组,出现了明显的神经行为学损伤。表明模型成功复现。
得到大鼠脑组织,从左侧脑组织中提取蛋白质。
采用10%水合氯醛对大鼠进行麻醉,大鼠麻醉后经左心室插入灌流针心脏灌流后迅速断头取脑,称重,将其分为左侧和右侧,将左侧大脑置于预冷的小烧杯中,用小剪刀将脑组织剪成碎块。将脑组织转入玻璃匀浆器中,加入分离缓冲液10mL/g组织,手动匀浆10次。将匀浆液转移到1.5 mL的离心管中,超声破碎,功率设定为28%(210W),超声1s,停1s,共超声1min。将离心管配平后,放入普通的4℃离心机,12,000rpm(13,400g)离心10min,取上清,沉淀用等量的匀浆缓冲液悬起,重复以上操作。所得上清液为脑组织蛋白。
Bradford方法测定蛋白浓度,制备浓度梯度的BSA标准品溶液,测定吸光度,根据标准品的浓度及相应的吸光度绘制浓度/吸光度标准曲线,并得到根据吸光度计算蛋白浓度的公式,计算各样品蛋白浓度。其原理采用考马斯亮蓝G-250染料与蛋白结合后由红色变为蓝色,在595nm处有最大吸收,通过测定595nm处光吸收的增加量可知与其结合蛋白质的量,从而根据标准品汇出的标准曲线,测定样品中蛋白的含量。
蛋白标线为y=0.0008x+0.11767,R
实施例2
蛋白质变性,膜辅助溶液内蛋白酶切。
1)蛋白质的还原:每个样品分别取等量蛋白加入10kDa的超滤管中,加入0.25MDTT(使 DTT的终浓度为25mM),涡旋混匀,放入水浴锅中37℃水浴1h,取出放至室温待冷却。2) 蛋白质的烷基化:加入0.5M IAA(使IAA的终浓度为50mM),涡旋混匀,避光室温放置30min。 3)清洗样品:加入5倍稀释的TEAB 300μL,12,000rpm(13,400g)离心10min,弃掉收集管底部溶液,重复3次,再加入2倍稀释的TEAB 300μL,12,000rpm(13,400g)离心10min。4)膜辅助溶液内酶切蛋白:更换新的收集管,在超滤管中加入胰蛋白酶,总量与蛋白质量比1:50,体积50μL,37℃反应过夜。5)多肽的收集:次日,加入2倍稀释的TEAB 100μL,12,000rpm(13,400 g)离心10min,重复3次,酶解消化后的肽段溶液离心于收集管底部。
iTRAQ标记定量、肽段分离和LC-MS质谱分析。
iTRAQ标记定量,1)从冰箱中取出iTRAQ试剂,平衡到室温,将iTRAQ试剂离心至管底。 2)向每管iTRAQ试剂中加入80μL异丙醇,涡旋振荡,离心至管底。3)加入30μL 2倍稀释的TEAB复溶样本,涡旋振荡,离心至管底。4)将iTRAQ试剂按照标记顺序(见表1)填加到样品中,涡旋振荡,离心至管底,室温反应2.5h。5)为了检测标记效率及定量准确性,从标记样品中各取出5μL混合,将混合样本及标记完成的样本真空冷冻离心干燥,抽干后的样品冷冻保存待用,混合样本用Ziptip脱盐后进行Orbitrap Q-Exactive plus检测,确认标记反应良好。
标记强度分布及定量权重如图4所示,紫色线表示每个强度水平下标记强度的标准偏差,第绿色线表示每个强度水平下的定量权重。本次实验SD值大部分为30%左右,定量准确性可靠。
表1 iTRAQ标记顺序
样品脱盐-HLB固相萃取柱除盐,1)HLB固相萃取柱活化:加入2mL 100%ACN,依靠重力使液体通过小柱,重复两次。2)HLB固相萃取柱平衡:加入2mL 0.1%FA,依靠重力使液体通过小柱,重复两次。3)上样:将标记好的多肽样本复溶混合,终体积为1mL,注入固相萃取小柱,依靠重力使液体通过小柱。4)清洗脱盐:吸取2mL 0.1%FA,清洗样品中的盐,重复三次。 5)洗脱:吸取900μL 70%ACN加入柱子中,用EP管收集洗脱液,再加入250μL 100%ACN,重复2次,收集在同一EP管中。6)将收集的洗脱液置于旋转真空干燥仪中,在不加热的条件下,真空干燥后,置于-80℃冰箱保存备用。
离线高pH反相高效液相色谱分离-iTRAQ样品。1)将iTRAQ标记后的多肽样品重溶于A液 (98%ddH2O,2%乙腈,氨水调pH至10.0)中。2)经离线
表2高pH反相色谱梯度
LC-MS/MS分析。
1)将多肽组分重溶于30μL,0.1%FA溶液中。2)经过U3000液相,低pH反相C18毛细管色谱(150μm×150mm,1.9μm)分离,A相为0.1%FA的水溶液,B相为80%乙腈,0.1%FA的水溶液,有效洗脱梯度为6%-40%的B相,总洗脱时间100min,流速0.6μL/min。液相条件如表3:
表3液相梯度
3)使用Orbitrap Q-Exactive HF-X质谱仪分析检测多肽混合物。扫描时间100min。质谱采集模式为高灵敏度模式,仪器参数设置:每个全扫描为高速信号依赖扫描,一级全扫描分辨率120000, AGC 3e6,扫描范围350-1500m/z;每一个一级扫描后筛选前20个离子进入碰撞池打碎,二级扫描分辨率45000,AGC 5e4,扫描范围350-1500m/z,碰撞能量32%,电荷状态筛选(包含+2至+5 电荷的前体),动态消除15s。
数据搜库、定量分析,获取MCAO大鼠组和Sham组大鼠的脑组织的差异蛋白质。
1)所得质谱数据经过Mascot(version 2.5.1)检索。2)检索参数为:相应物种的蛋白数据库,胰蛋白酶,最多2个漏切位点,母离子和碎片离子质量误差分别为10ppm和0.02Da,固定修饰为Carbamidomethy(C),可变修饰为Oxidation(M),Acetyl(N-terminal)。3)蛋白数据库:选择适当的蛋白质序列数据库是对质谱数据进行蛋白质定性分析的基础和关键步骤。通常数据库的来源主要有以下几种:a.综合性蛋白质数据库,如NCBInr、UniProt等;b.由转录组测序结果翻译而成的蛋白质序列数据。本发明使用数据库为:uniprot_rat_36135_20200211.fasta(序列总数: 36135)。4)数据质控和定量经过Scaffold Q+(version4.6.2)处理,质控参数为:多肽的假发现率 (FDR)<1.0%,蛋白的假发现率(FDR)<1.0%,每个蛋白至少鉴定到1个特异性多肽,通过中值数据归一化。
2)变化倍数的确定
基于样本技术重复间蛋白定量比值的分布,确定筛选差异蛋白ratio的阈值,1±2*SD和1± 3*SD分别为其95%和99%置信度区间,即没有考虑生物学因素影响条件下,95%和99%蛋白定量的比值将分布于1±2*SD和1±3*SD区间。因此选取2组样本间蛋白ratio≤1-2SD或ratio≥1+2SD 做为筛选差异蛋白ratio的阈值。
3)统计学分析
对需要比较的两组样本的蛋白定量数据进行成组t检验,筛选P value≤0.05的蛋白质。
差异候选蛋白的选取,需要根据变化倍数、统计学显著性共同判断。在数据分析中,选取2 组样本间ratio≤(1-2*SD)或ratio≥(1+2*SD),并且P value≤0.05的蛋白作为候选差异蛋白。
差异蛋白质分析结果如下:
表4蛋白组学分析结果
差异表达火山图见图6,图6中的每一个点表示一个蛋白,横坐标为两组间蛋白ratio差异表达火山图中的每一个点表示一个蛋白,横坐标为两组间蛋白ratio的log2对数值;纵坐标为两组间 P-Value的-log 10对数值。横坐标的绝对值越大,说明蛋白在两组样品间的表达量差异越大;纵坐标值越大,表明差异表达越显著,筛选得到的差异表达蛋白越可靠。图中绿色的点代表下调差异表达蛋白,红色的点代表上调差异表达蛋白,黑色的代表非差异表达蛋白。
表5差异蛋白组合一
表5中所列蛋白是和线粒体氧化磷酸化通路、线粒体自身的核糖体蛋白,转录因子有关的。线粒体是氧化磷酸化生成能量的重要部位,严重缺氧可降低线粒体功能,使ATP的生成减少,线粒体可出现肿胀、嵴崩解、外膜碎裂和基质外溢。脑缺血动物模型及体外研究证实,多数情况下,严重脑缺血后,线粒体可通过坏死机制引起神经细胞的死亡。一方面,线粒体功能障碍、氧化磷酸化受抑制或电子传递链脱偶联,ATP减少;另一方面,ATP磷酸化障碍使氧自由基产生增加。自由基可能作用于线粒体,抑制细胞色素C氧化酶mRNA的转录,使电子传递DNA链必需蛋白的合成减少,从而破坏电子传递,最终致ATP产生停止,细胞坏死。
此外,当脑缺血发生时,脑组织中的线粒体最先受到影响,线粒体核糖体蛋白L21、L3、L27、 L23、S17、S5、S18A表达下调,表明负责完成线粒体翻译的过程受到阻碍,线粒体合成受到影响,从而最终影响到ATP生成。这些差异蛋白作为检测缺血性脑损伤的蛋白标志物,具有可靠性和灵敏性。
表6差异蛋白组合二
表6中所列蛋白是和补体系统,HIF-1通路相关的蛋白。补体系统是由30多种性质不同的液相或细胞膜蛋白组成的多分子系统,分为补体固有成分,补体调节因子和补体受体。补体可由经典途径或旁路替代途径激活,通过补体系统各成分之间相互作用形成级联反应,产生多种重要的生物活性物质,具有溶解靶细胞,扩大炎症反应以及促进巨噬细胞吞噬等多种功能,对机体的防御功能,免疫系统功能的调节等都发挥重要作用。动脉粥样硬化是缺血性脑血管病的主要病因,而补体系统激活并参与了动脉粥样硬化的发生发展以及血栓的形成,在缺血性脑血管疾病的发病机制中起重要作用。活化的补体系统产生多种生物活性物质,如C3a、C5a等过敏毒素增加血管通透性,促进脂质沉积,促进动脉粥样硬化;C3a、C5a、C567等作为趋化因子吸引单核细胞、中性粒细胞到局部,并通过一系列反应促进白细胞附壁,白细胞附壁后继续移行至内皮细胞下,通过定期释放胞浆素使血管内皮细胞由抗凝变为促凝,最终导致血栓形成。
激活补体系统的主要有效途径有3条:经典途径、凝集素途径和旁路途径,活化的补体系统产生多种生物活性物质,这些补体成分能直接或间接通过促进炎性反应而加重缺血-再灌注损伤。如C3a、C5a等过敏毒素增加血管通透性,C3a、C5a、C567等作为趋化因子吸引单核细胞、中性粒细胞到局部,并通过一系列反应促进白细胞附壁,白细胞附壁后继续移行至内皮细胞下,通过定期释放胞浆素使血管内皮细胞由抗凝变为促凝,最终导致血栓形成等。
脑缺血后,补体系统的激活参与了脑损害的病理生理过程。脑组织缺血后补体C1q和C3d蛋白表达上调,参与了脑组织损伤的全过程,并与脑组织损伤程度密切相关。
HIF-1信号通路与细胞凋亡有着密不可分的关系。在严重缺血缺氧时,HIF-1a可通过与p53 结合诱导细胞调亡,或诱导bcl2家族中的调亡前基因BNIP3表达等途径激活凋亡途径。研究发现栀子苷可以抑制局灶性脑缺血损伤引起的的HIF-1α过渡表达,减少HIF-1依赖性凋亡相关基因的表达,减少神经元凋亡,发挥脑保护作用。这些差异蛋白作为检测缺血性脑损伤的蛋白标志物,具有可靠性和灵敏性。
表7差异蛋白组合三
表7中所列蛋白是和PPAR信号转导通路相关的蛋白。脑缺血时,流向脑组织的血液被打断,使脑组织失去氧气和外部代谢物的供应,导致乳酸、琥珀酸的堆积。PPAR通路可参与脂肪细胞分化和能量平衡、调节脂质代谢与糖代谢等生理过程,对短暂的脑缺血发挥保护作用。这些差异蛋白作为检测缺血性脑损伤的蛋白标志物,具有可靠性和灵敏性。
验证实验
上述质谱实验结果本身能够证明本发明要求保护的三组差异蛋白质组合能够作为检测缺血性脑损伤的早期蛋白标志物,也可采用酶联免疫技术对49个差异蛋白进行逐个检测,或者采用蛋白芯片技术对49个蛋白逐个检测,或者采用RT-PCR技术对49个差异蛋白进行逐个检测,确定检测到的脑组织差异蛋白的浓度变化。
mRNA提取,质量检测及逆转录反应
1)取约100mg假手术组和正常组大鼠脑组织,加入1ml Trizol溶液后匀浆,匀浆后室温静置5min。12000g 4℃离心5min,将上清液转移至新的1.5ml离心管中。
2)加入200μl氯仿,剧烈震荡15s后室温静置5min。12000g 4℃再次离心15min后分为三层,mRNA主要溶解在最上层溶液中,小心吸取最上层溶液至新的离心管中。
3)加入500μl异丙醇后室温静置10min,12000g 4℃离心10min,倾倒离心上清液,留取沉淀。
4)用1ml 75%乙醇清洗沉淀3遍,弃上清留取沉淀,将离心管置于超净台边缘,待沉淀自然风干。
5)用适量的DEPC水溶解沉淀,适当稀释,使用NanoDrop2000超微量分光光度计测定RNA 浓度,A260/A280比值范围在1.8-2.0表示纯度较好。
6)此外,变性琼脂糖凝胶电泳对提取的RNA进行再次验证。取3μgRNA,加入适量的DNA 上样缓冲液,混匀。将样品加入上样孔,100V恒压模式下电泳25-35min至溴酚蓝指示剂剧胶孔至少2-3cm止。将凝胶取出,置于紫外投射光下观察并拍照。
实时定量PCR
1)反应体如下表所示:
首先分别将①⑥预混,②到⑤预混,充分混匀,使用移液枪分别加入96孔板中。
2)反应条件设置:
预变性95℃30s,循环反应40次(95℃5s,60℃30s),溶解曲线反应(60℃开始,以0.7℃每次递增至95℃)
3)引物设计与合成
所有引物进行primer blast验证其特异性。具体引物序列如下:
与假手术组相比,采用RT-PCR技术得到的己糖激酶mRNA的表达水平升高,约为假手术组的1.3倍。
己糖激酶对应hexokinase 2(Hk2)肽端,从而证明PCR结果与质谱结果一致,上述3组缺血性脑损伤的差异蛋白组合,可作为检测缺血性脑损伤的早期蛋白标志物应用在检测脑缺血病中。与现有的检测方法(CT或者核磁)相比,本发明检测方法特异性、灵敏度和准确性高。
以上所述实施例仅表达了本发明的实施方式,其描述较为具体和详细,但并不能因此而理解为对本发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110> 中国中医科学院中药研究所
<120> 一种缺血性脑损伤差异蛋白组合及非诊断用的MCAO大鼠差异蛋白组学分析方法
<160> 49
<170> SIPOSequenceListing 1.0
<210> 1
<211> 10
<212> PRT
<213> 多肽(人工序列)
<400> 1
Val His Tyr Gln Trp Tyr Leu Met Glu Arg
1 5 10
<210> 2
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 2
His Asp Val Thr Cys Thr Val Ser Gly Gly Gly Arg
1 5 10
<210> 3
<211> 10
<212> PRT
<213> 多肽(人工序列)
<400> 3
Gly Gly Thr Ser Asp Ala Leu Leu Tyr Arg
1 5 10
<210> 4
<211> 10
<212> PRT
<213> 多肽(人工序列)
<400> 4
Phe Tyr Ser Val Asn Val Asp Tyr Ser Lys
1 5 10
<210> 5
<211> 8
<212> PRT
<213> 多肽(人工序列)
<400> 5
Glu Leu Asn Ile Thr Ala Ala Lys
1 5
<210> 6
<211> 10
<212> PRT
<213> 多肽(人工序列)
<400> 6
Ile Ile Val Asn Pro Gln Thr Ile Leu Arg
1 5 10
<210> 7
<211> 8
<212> PRT
<213> 多肽(人工序列)
<400> 7
Val Ile Gly Thr Ala Met Val Lys
1 5
<210> 8
<211> 11
<212> PRT
<213> 多肽(人工序列)
<400> 8
Gly Ala Leu Ala Glu Thr Gly Ala Gly Ala Arg
1 5 10
<210> 9
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 9
Arg Pro Gly Ala Ile Ser Thr Gly Asp Ile Ala Arg
1 5 10
<210> 10
<211> 20
<212> PRT
<213> 多肽(人工序列)
<400> 10
Met Ala Gly Glu Thr Pro Glu Leu Thr Leu Glu Gln Pro Pro Gln Asp
1 5 10 15
Ala Ser Thr Lys
20
<210> 11
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 11
Ser Glu Asp Tyr Ala Leu Pro Ser Tyr Val Asp Arg
1 5 10
<210> 12
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 12
Asn Tyr Asp Ser Met Lys Asp Phe Glu Glu Met Arg
1 5 10
<210> 13
<211> 13
<212> PRT
<213> 多肽(人工序列)
<400> 13
Asn Ser Glu Ala Val Asn Leu Val Thr Ser Leu Pro Lys
1 5 10
<210> 14
<211> 9
<212> PRT
<213> 多肽(人工序列)
<400> 14
Asn Leu Pro Phe Ser Val Glu Asn Lys
1 5
<210> 15
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 15
Val Glu Glu Pro Glu Thr Leu Val Glu Leu Gln Lys
1 5 10
<210> 16
<211> 15
<212> PRT
<213> 多肽(人工序列)
<400> 16
Asn Tyr Leu Glu Gln Ile Tyr Asn Val Pro Val Ala Ala Val Arg
1 5 10 15
<210> 17
<211> 11
<212> PRT
<213> 多肽(人工序列)
<400> 17
Val Gly Met Ala Val Gly Ser Pro Leu Leu Lys
1 5 10
<210> 18
<211> 8
<212> PRT
<213> 多肽(人工序列)
<400> 18
Ala Val Leu Thr Leu Asp Glu Arg
1 5
<210> 19
<211> 8
<212> PRT
<213> 多肽(人工序列)
<400> 19
Ala Gly Val Asp Gly Trp Phe Lys
1 5
<210> 20
<211> 11
<212> PRT
<213> 多肽(人工序列)
<400> 20
Asp Ile Phe Thr Gly Leu Ile Gly Pro Met Lys
1 5 10
<210> 21
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 21
Gly Asn Ser Ile Ile Met Leu Glu Ala Leu Glu Arg
1 5 10
<210> 22
<211> 11
<212> PRT
<213> 多肽(人工序列)
<400> 22
Cys Leu Thr Thr His Glu Tyr Asp Gly Gln Lys
1 5 10
<210> 23
<211> 11
<212> PRT
<213> 多肽(人工序列)
<400> 23
Ser Leu Gly Thr Ala Asp Val His Phe Glu Arg
1 5 10
<210> 24
<211> 13
<212> PRT
<213> 多肽(人工序列)
<400> 24
Asn Glu Ile Ser Thr Ala Asp Ala Ile Phe Val Gln Arg
1 5 10
<210> 25
<211> 10
<212> PRT
<213> 多肽(人工序列)
<400> 25
Ala Thr Ser Gln Val Val Ala Gly Thr Lys
1 5 10
<210> 26
<211> 14
<212> PRT
<213> 多肽(人工序列)
<400> 26
Val Leu Ile Glu Asp Gly Ser Gly Glu Ala Val Leu Ser Arg
1 5 10
<210> 27
<211> 13
<212> PRT
<213> 多肽(人工序列)
<400> 27
Val Ala Gln Leu Glu Ala Gln Cys Gln Glu Pro Cys Lys
1 5 10
<210> 28
<211> 9
<212> PRT
<213> 多肽(人工序列)
<400> 28
Ala Gln Gln Ile Gln Val Leu Gln Lys
1 5
<210> 29
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 29
Tyr Leu Gly Asp Leu Ser Gly Gly Gln Val Leu Lys
1 5 10
<210> 30
<211> 13
<212> PRT
<213> 多肽(人工序列)
<400> 30
Gly Ala Val Leu Phe Gly Gln Ala Pro Gly Val Val Arg
1 5 10
<210> 31
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 31
Leu Thr Gln Tyr Ile Asp Gly Gln Gly Arg Pro Arg
1 5 10
<210> 32
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 32
Ala Ser Asp Ser Ser Ile Asn Trp Asn Asn Leu Lys
1 5 10
<210> 33
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 33
Gly Phe Gly Asn Ile Ala Thr Asn Glu Asp Thr Lys
1 5 10
<210> 34
<211> 8
<212> PRT
<213> 多肽(人工序列)
<400> 34
Ala Glu Asn Thr Gln Phe Val Lys
1 5
<210> 35
<211> 15
<212> PRT
<213> 多肽(人工序列)
<400> 35
Phe Met Gly Ser Pro Glu Ile Ile Glu Thr Thr Leu Tyr Gln Arg
1 5 10 15
<210> 36
<211> 12
<212> PRT
<213> 多肽(人工序列)
<400> 36
Val Ile Thr Asn Val Asn Asp Asn Tyr Glu Pro Arg
1 5 10
<210> 37
<211> 13
<212> PRT
<213> 多肽(人工序列)
<400> 37
Leu Phe Ile Gly Gly Leu Asn Thr Glu Thr Asn Glu Lys
1 5 10
<210> 38
<211> 9
<212> PRT
<213> 多肽(人工序列)
<400> 38
Val Ile Leu Gly Ala His Glu Glu Arg
1 5
<210> 39
<211> 11
<212> PRT
<213> 多肽(人工序列)
<400> 39
Ala Thr Asn Asp Glu Ile Phe Ser Ile Leu Lys
1 5 10
<210> 40
<211> 16
<212> PRT
<213> 多肽(人工序列)
<400> 40
Leu Ser His Pro Glu Gly Phe Gly Gly Leu Ile Phe Thr Ser Pro Arg
1 5 10 15
<210> 41
<211> 8
<212> PRT
<213> 多肽(人工序列)
<400> 41
Val Gly Asp Val Tyr Ile Pro Arg
1 5
<210> 42
<211> 15
<212> PRT
<213> 多肽(人工序列)
<400> 42
Gly Phe Ala Phe Val Thr Phe Asp Asp His Asp Thr Val Asp Lys
1 5 10 15
<210> 43
<211> 19
<212> PRT
<213> 多肽(人工序列)
<400> 43
Ala Asn Ser Phe Val Gly Thr Ala Gln Tyr Val Ser Pro Glu Leu Leu
1 5 10 15
Thr Glu Lys
<210> 44
<211> 13
<212> PRT
<213> 多肽(人工序列)
<400> 44
Val Lys Asp Phe Ala Thr Val Tyr Val Asp Ala Val Lys
1 5 10
<210> 45
<211> 9
<212> PRT
<213> 多肽(人工序列)
<400> 45
Ala Tyr Phe Gln Asn Ala Gln Glu Arg
1 5
<210> 46
<211> 18
<212> PRT
<213> 多肽(人工序列)
<400> 46
Gln Ala Thr Val Gly Asp Val Asn Thr Asp Arg Pro Gly Leu Leu Asp
1 5 10 15
Leu Lys
<210> 47
<211> 15
<212> PRT
<213> 多肽(人工序列)
<400> 47
Ser Ser Ser Thr Gly Val Gly Glu Ala Leu Gly Ser Gly Ser Arg
1 5 10 15
<210> 48
<211> 16
<212> PRT
<213> 多肽(人工序列)
<400> 48
Asp Ile Lys Pro Ser Asn Leu Leu Leu Gly Asp Asp Gly His Val Lys
1 5 10 15
<210> 49
<211> 17
<212> PRT
<213> 多肽(人工序列)
<400> 49
Glu Thr Leu Gly Thr Gly Ala Phe Ser Glu Val Val Leu Ala Glu Glu
1 5 10 15
Lys
- 一种缺血性脑损伤差异蛋白组合及非诊断用的MCAO大鼠差异蛋白组学分析方法
- 一种基于iTRAQ联合LC、MALDI的差异蛋白组学
