基于细胞的组合疗法
文献发布时间:2023-06-19 11:57:35
相关申请的交叉引用
本申请要求在2018年10月1日提交的美国临时专利申请号62/739,814和在2019年2月20日提交的美国临时专利申请号62/807,783的优先权和权益,其全部内容通过引用以其全文并入本文。
技术领域
本公开涉及使用具有疫苗(例如,gp96-Ig)的细胞和具有T细胞共刺激分子的细胞进行治疗的方法。
电子提交的文本文件的说明
一并电子提交的文本文件的内容通过引用以其全文并入本文:序列表的计算机可读格式的副本(文件名:HTB-030_ST25.txt;记录日期:2019年9月30日;文件大小:79.2KB)。
背景技术
癌症的特征在于逐渐获得导致细胞生长和死亡的内在失调的基因突变。一旦细胞获得足够的突变(通常认为是至少六个),它将不再响应会抑制其生长或触发凋亡的内在或外在信号。由于肿瘤产生自宿主细胞,因此身体免疫系统最初对这些细胞耐受。宿主免疫系统可以寻找并破坏获得免疫原性突变的细胞,这一过程称为免疫监视。靶向T细胞中的调节途径以增强抗肿瘤免疫应答的免疫检查点治疗剂,已引起重大的临床进展,并提供了新的针对癌症的防御。此外,疫苗也可以通过增强抗肿瘤免疫应答来促进这种防御。因此,包括一种或多种检查点抑制剂、一种或多种疫苗和一种或多种T细胞共刺激分子的组合或亚组合的组合疗法有可能扩大能够受益于免疫疗法的癌症患者群体。
发明内容
旨在包括一种或多种疫苗、一种或多种T细胞共刺激分子和一种或多种检查点抑制剂的组合的免疫疗法,可扩大能够受益于此类疗法的癌症患者群体。疫苗可以通过增加肿瘤抗原特异性CD8+T细胞的频率以及被这些CD8+T细胞识别的肿瘤抗原数量,来促进这种应答。T细胞共刺激分子可通过进一步增加肿瘤抗原特异性T细胞的频率和/或增强其激活,并且还通过增加CD8+T细胞的肿瘤杀伤性效应分子的表达,来增强应答。当与检查点抑制剂组合使用时,可能产生广范围的高度活化的CD8+T细胞,其将能够浸润肿瘤,且一旦发生浸润则将不会被各种检查点途径所抑制。本公开至少部分基于以下发现:疫苗接种(例如gp96-Ig疫苗接种)与T细胞共刺激(使用OX40、ICOS、4-1BB、TNFRSF25、CD40、CD27和/或GITR等的一种或多种激动剂)的组合提供了协同抗肿瘤作用。临床前模型已经评估了gp96-Ig疫苗与靶向OX40、ICOS、4-1BB和TNFRSF25的激动性抗体组合的独立组合物,并证明了对机械性和抗肿瘤互补性的可变作用。本文所述的方法提供了第一细胞,其包含含有编码可分泌型疫苗蛋白(例如,gp96-Ig)的核苷酸序列的表达载体,其中所述患者正在经历利用第二细胞的治疗,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,以提供T细胞共刺激,所述T细胞共刺激融合蛋白包括但不限于融合蛋白例如ICOSL-Ig、4-1BBL-Ig、TL1A-Ig、OX40L-Ig、CD40L-Ig、CD70-Ig或GITRL-Ig。
在一些实施方案中,本文所述的方法分泌协同作用的融合蛋白。局部分泌的T细胞共刺激融合蛋白(即OX40L-Ig)的作用尤其不同于与可分泌型疫苗蛋白(例如,gp96-Ig)组合的全身施用。不希望受到理论的束缚,当比较疫苗蛋白(例如,gp96-Ig)和逐步增加剂量的全身OX40激动抗体(agonist antibody)时,仅分泌疫苗蛋白(例如,gp96-Ig)的细胞的作用和与逐步增加剂量的分泌T细胞共刺激融合蛋白(即OX40L-Ig)的细胞组合的作用不同。
在一些实施方案中,可分泌型疫苗蛋白(例如,gp96-Ig)的分泌量高于T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达。在一些实施方案中,疫苗蛋白(例如,gp96-Ig)分泌与T细胞共刺激融合蛋白(例如,OX40L-Ig)表达的比例为约1:10、1:25、1:50、1:100、1:200、1:300、1:400、1:500、1:600、1:700、1:800、1:900或1:1000(包含所有端点)。
在一些实施方案中,疫苗蛋白(例如,gp96-Ig)的分泌量低于T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达。在一些实施方案中,疫苗蛋白(例如,gp96-Ig)分泌与T细胞共刺激融合蛋白(例如,OX40L-Ig)表达的比例为约10:1、25:1、50:1、100:1、200:1、300:1、400:1、500:1、600:1、700:1、800:1、900:1或1000:1(包含所有端点)。
在一些实施方案中,T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达量高于疫苗蛋白(例如,gp96-Ig)的分泌。在一些实施方案中,T细胞共刺激融合蛋白(例如OX40L-Ig)表达与疫苗蛋白(例如gp96-Ig)分泌的比例为约1:10、1:25、1:50、1:100、1:200、1:300、1:400、1:500、1:600、1:700、1:800、1:900或1:1000(包含所有端点)。
在一些实施方案中,T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达量低于疫苗蛋白(例如,gp96-Ig)的分泌。在一些实施方案中,T细胞共刺激融合蛋白(例如,OX40L-Ig)表达与疫苗蛋白(例如,gp96-Ig)分泌的比例为约10:1、25:1、50:1、100:1、200:1、300:1、400:1、500:1、600:1、700:1、800:1、900:1或1000:1(包含所有端点)。
在一些实施方案中,可分泌型疫苗蛋白(例如,gp96-Ig)的分泌量与T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达几乎相同。在一些实施方案中,疫苗蛋白(例如,gp96-Ig)分泌与T细胞共刺激融合蛋白(例如,OX40L-Ig)表达的比例为约1:1。在一些实施方案中,疫苗蛋白(例如,gp96-Ig)分泌与T细胞共刺激融合蛋白(例如OX40L-Ig)表达的比例为约1:1.3。
在一些实施方案中,可分泌型疫苗蛋白(例如,gp96-Ig)的表达量与T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达几乎相同。在一些实施方案中,疫苗蛋白(例如,gp96-Ig)表达与T细胞共刺激融合蛋白(例如,OX40L-Ig)表达的比例为约1:1。在一些实施方案中,疫苗蛋白(例如,gp96-Ig)表达与T细胞共刺激融合蛋白(例如OX40L-Ig)表达的比例为约1:1.3。
在一些实施方案中,T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达量与疫苗蛋白(例如,gp96-Ig)的分泌几乎相同。在一些实施方案中,T细胞共刺激融合蛋白(例如,OX40L-Ig)表达与疫苗蛋白(例如,gp96-Ig)分泌的比例为约1:1。
在一些实施方案中,分泌gp96-Ig的细胞数高于分泌OX40L-Ig的细胞数。在一些实施方案中,分泌gp96-Ig的细胞数与分泌OX40L-Ig的细胞数的比例为约1:0.01、约1:0.1、约1:1、约1:10、约1:25、约1:50、约1:100、约1:200、约1:300、约1:400、约1:500、约1:600、约1:700、约1:800、约1:900或约1:1000(包含所有端点)。
在一些实施方案中,分泌gp96-Ig的细胞数低于分泌OX40L-Ig的细胞数。在一些实施方案中,分泌gp96-Ig的细胞数与分泌OX40L-Ig的细胞数的比例为约0.01:1、约0.1:1、约1:1、约1:1.3、约10:1、约25:1、约50:1、约100:1、约200:1、约300:1、约400:1、约500:1、约600:1、约700:1、约800:1、约900:1或约1000:1(包含所有端点)。
在一些实施方案中,gp96-Ig的表达高于OX40L-Ig的表达。在一些实施方案中,gp96-Ig的表达与OX40L-Ig的表达的比例为约1:0.01、约1:0.1、约1:1、约1:10、约1:25、约1:50、约1:100、约1:200、约1:300、约1:400、约1:500、约1:600、约1:700、约1:800、约1:900或约1:1000(包含所有端点)。
在一些实施方案中,gp96-Ig的表达低于OX40L-Ig的表达。在一些实施方案中,gp96-Ig的表达与OX40L-Ig的表达的比例为约0.01:1、约0.1:1、约1:1、约1:3、约10:1、约25:1、约50:1、约100:1、约200:1、约300:1、约400:1、约500:1、约600:1、约700:1、约800:1、约900:1或约1000:1(包含所有端点)。
在一些实施方案中,诱导型启动子可用于诱导疫苗蛋白(例如,gp96-Ig)的表达。在一些实施方案中,gp96-Ig处于强诱导型启动子作用下。在一些实施方案中,gp96-Ig处于中等诱导型启动子作用下。在一些实施方案中,gp96-Ig处于弱诱导型启动子作用下。
在一些实施方案中,诱导型启动子可用于诱导T细胞共刺激融合蛋白(例如,OX40L-Ig)的表达。在一些实施方案中,OX40L-Ig处于强诱导型启动子作用下。在一些实施方案中,OX40L-Ig处于中等诱导型启动子作用下。在一些实施方案中,OX40L-Ig处于弱诱导型启动子作用下。
在一些实施方案中,疫苗蛋白(例如,gp96-Ig)和/或T细胞共刺激融合蛋白(例如,OX40L-Ig)在宿主细胞(例如,哺乳动物细胞)中表达。在一些实施方案中,gp96-Ig和/或OX40L-Ig的表达和/或分泌可以通过本领域已知的技术,例如体外细胞培养方法或蛋白检测测定法,容易地检测和定量。在一些实施方案中,蛋白检测测定法包括酶联免疫吸附测定法(ELISA)、免疫沉淀和基于荧光的方法。
在一些实施方案中,细胞分泌的gp96-Ig的量高于细胞分泌的OX40L-Ig的量。在一些实施方案中,细胞分泌的gp96-Ig与细胞分泌的OX40L-Ig的比例为约1:0.01、约1:0.1、约1:1、约1:10、约1:25、约1:50、约1:100、约1:200、约1:300、约1:400、约1:500、约1:600、约1:700、约1:800、约1:900或约1:1000(包含所有端点)。
在一些实施方案中,细胞分泌的gp96-Ig的量低于细胞分泌的OX40L-Ig的量。在一些实施方案中,细胞分泌的gp96-Ig与细胞分泌的OX40L-Ig的比例为约0.01:1、约0.1:1、约1:1、约1:1.3、约10:1、约25:1、约50:1、约100:1、约200:1、约300:1、约400:1、约500:1、约600:1、约700:1、约800:1、约900:1或约1000:1(包含所有端点)。
在一个方面,本公开提供了一种治疗患者的方法,其包括向患者施用有效量的第一细胞,所述第一细胞包含含有编码可分泌型疫苗蛋白的核苷酸序列的表达载体,其中患者正在经历利用第二细胞的治疗,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,并且其中当施用给受试者时,T细胞共刺激融合蛋白增强抗原特异性T细胞的激活。
在一个方面,本公开提供了一种治疗患者的方法,其包括向患者施用有效量的第二细胞,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,其中当施用给受试者时,T细胞共刺激融合蛋白增强抗原特异性T细胞的激活,并且其中患者正在经历利用第一细胞的治疗,所述第一细胞包含含有编码可分泌型疫苗蛋白的核苷酸序列的表达载体。
在一个方面,本公开提供了一种治疗患者的方法,其包括向患者施用有效量的(a)第一细胞,所述第一细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,和(b)第二细胞,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,并且其中当施用给受试者时,T细胞共刺激融合蛋白增强抗原特异性T细胞的激活。
在一些实施方案中,可分泌型疫苗蛋白是可分泌型gp96-Ig融合蛋白,其任选缺少gp96 KDEL(SEQ ID NO:3)序列。在一些实施方案中,gp96-Ig融合蛋白中的Ig标签包含人IgG1、IgG2、IgG3、IgG4、IgM、IgA或IgE的Fc区。
在一些实施方案中,T细胞共刺激融合蛋白是OX40L-Ig或其与OX40结合的一部分。在一些实施方案中,T细胞共刺激融合蛋白是ICOSL-Ig或其与ICOS结合的部分。在一些实施方案中,T细胞共刺激融合蛋白是4-1BBL-Ig或其与4-1BBR结合的部分。在一些实施方案中,T细胞共刺激融合蛋白是TL1A-Ig或其与TNFRSF25结合的部分。在一些实施方案中,T细胞共刺激融合蛋白是GITRL-Ig或其与GITR结合的部分。在一些实施方案中,T细胞共刺激融合蛋白是CD40L-Ig或其与CD40结合的部分。在一些实施方案中,T细胞共刺激融合蛋白是CD70-Ig或其与CD27结合的部分。在一些实施方案中,T细胞共刺激融合蛋白中的Ig标签包含人IgG1、IgG2、IgG3、IgG4、IgM、IgA或IgE的Fc区。
在一些实施方案中,将表达载体引入病毒或病毒样颗粒中。在一些实施方案中,将表达载体引入人肿瘤细胞中。在一些实施方案中,患者是人类癌症患者。在一些实施方案中,对人类患者的施用增加了患者中肿瘤抗原特异性T细胞的激活或增殖。
在一些实施方案中,与施用前患者中肿瘤抗原特异性T细胞的激活或增殖水平相比,患者中肿瘤抗原特异性T细胞的激活或增殖增加了至少25%。
在一些实施方案中,与抑制由肿瘤细胞产生的免疫抑制性分子的药剂组合施用。在一些实施方案中,药剂是针对PD-1的抗体。在一些实施方案中,针对PD-1的抗体选自纳武单抗(Nivolumab)、派姆单抗(Pembrolizumab)、匹地利珠单抗(Pidilizumab)、西米普利单抗(Cemiplimab)、AGEN2034、AMP-224、AMP-514、PDR001。
在一些实施方案中,患者是患有急性或慢性感染的人。在一些实施方案中,急性或慢性感染是丙型肝炎病毒、乙型肝炎病毒、人免疫缺陷病毒或疟疾的感染。
在一些实施方案中,对人类患者的施用刺激了病原性抗原特异性T细胞的激活或增殖。
在一些实施方案中,与单独的gp96-Ig疫苗接种相比,T细胞共刺激分子增强受试者中抗原特异性T细胞的激活至更大水平。
附图简要说明
图1是pcDNA3.4 OX40L-Ig的质粒载体图谱。
图2显示了小鼠和人OX40L对Jurkat细胞中人OX40受体的激活。
图3的图像显示了在一定剂量范围内小鼠HS-110(B16F10-OVA-gp96)和小鼠HS-130(B16F10-OVA-OX40L)免疫接种,以将CD8+T细胞扩增与肿瘤生长延迟关联。
图4显示了初次疫苗接种后第7天的流式细胞图、点图和设门策略(外周血)。将100万个细胞(290ng的gp96-Ig)固定剂量的小鼠mHS-110和不同比例的mHS-130(0.1、0.3、1、3、10)注射给接受者小鼠。1:1的比例是290ng的gp96(100万个mHS-110细胞)比290ng的OX40L。图4显示了使用FlowJo版本10(2018)的流式细胞术设门策略,其通过在血液中对单个细胞(singlet)和CD3+T细胞进行设门(gate),然后是CD8+OT-I GFP+T细胞。在第7天进行样品分析,且代表性点图中的数字表示设门群体内CD8+OT-I GFP+阳性细胞的百分比。这些图显示了表示所选日的峰扩增(peak expansion)的代表性小鼠个体。
图5显示了加强疫苗接种后第21天的流式细胞图、点图和设门策略(外周血)。将100万个细胞(290ng的gp96-Ig)固定剂量的小鼠mHS-110和不同比例的mHS-130(0.1、0.3、1、3、10)注射给接受者小鼠。1:1的比例是290ng的gp96(100万个mHS-110细胞)比290ng的OX40L。图5显示了使用FlowJo版本10(2018)的流式细胞术设门策略,其通过在血液中对单个细胞和CD3+T细胞进行设门,然后是CD8+OT-I GFP+T细胞。在第14天进行加强免疫后,在第21天进行样品分析,且代表性点图中的数字表示设门群体内CD8+OT-I GFP+阳性细胞的百分比。这些图显示了表示所选日的峰扩增的代表性小鼠个体。
图6A和图6B显示了在肿瘤激发(tumor challenge)之前和之后,在使用设定剂量的mHS-110和不同比例的mHS-130进行初次和二次疫苗接种后,OT-I CD8+T细胞的百分比(外周血)。将100万个细胞(290ng的gp96-Ig)固定剂量的小鼠mHS-110和不同比例的mHS-130注射给接受者小鼠。疫苗接种后,在疫苗接种后的第0至53天在血液中分析OT-I GFP+CD8+T细胞。然后在第14天使用与初次阶段相同比例的mHS110和mHS130加强小鼠,并且在激发后的第17、19、21、24、28、33、38、41天在血液中分析OT-I GFP+CD8+T细胞。数据代表来自n=5只小鼠的平均总数±SEM。*p<0.05**p<0.01(仅mHS-110相比于不同比例的mHS-130)。(图6A):无重叠的线图;(图6B):仅将小鼠离群值移出至第41天的线图。
图7A、图7B、图7C、图7D和图7E显示了在第54天研究结束(End-of-Study)时对疫苗接种的内源性应答。图7A显示了在第54天研究结束时,对疫苗接种的内源性脾应答(百分比),以FSC-H和FSC-A设门从而对双联体细胞(doublet)进行活/死设门,然后以CD45和SSC设门,然后是CD3+CD8+双阳性细胞。图7A显示了通过非参数统计学检验Mann-Whitney的平均值±SEM,与mHS-110相比*p<0.05。图7B是流式细胞图,图7C显示了在第54天研究结束时,对疫苗接种的内源性脾应答和用pg100肽进行离体刺激以用于细胞内细胞因子染色。图中显示了百分比。事件以FSC-H和FSC-A设门从而对双联体细胞进行活/死设门,然后以CD45和SSC设门,然后是IFN-γCD8+双阳性细胞。图7C显示了通过非参数统计检验Mann-Whitney的平均值±SEM,与mHS-110相比*p<0.05;“ns”表示p>0.05,不显著。图7D显示了通过IFN-γELISPOT测量的内源性脾免疫应答。图7D显示了每百万个脾细胞的IFN-γ斑点(spot)平均值±SEM,通过非参数统计学检验Mann-Whitney,与mHS-110相比,**p<0.01,*p<0.05;“ns”表示p>0.05,不显著。最右显示使用机器计数的代表性ELISPOT孔。不从画图的数据集中减去背景(仅培养基)孔。阳性对照孔起作用(数据未显示)。图7E是在第54天研究结束时,对疫苗接种的内源性应答(百分比),以FSC-H和FSC-A设门从而对双联体细胞进行活/死设门,然后以CD45和SSC设门,然后是CD3+CD4+双阳性细胞。图7E显示了平均值±SEM,通过非参数统计学检验Mann-Whitney,与mHS-110相比,**p<0.01。
图8A是流式细胞图,图8B显示了CD8+肿瘤浸润淋巴细胞(TIL)。图8A显示了在第54天研究结束时,对疫苗接种的内源性TIL应答(百分比),以FSC-H和FSC-A设门从而对双联体细胞进行活/死设门,然后以CD45和SSC设门,然后是CD3+CD8+双阳性细胞。MACS MiltenylBiotec肿瘤分离试剂盒用于这个步骤(目录号130-096-730)。图8B显示了平均值±SEM,通过非参数统计学检验Mann-Whitney,与mHS-110相比,*p<0.05。
图9显示了研究结束时个体小鼠的最终肿瘤质量,以克为单位。使用毫克敏感秤称量每只动物的肿瘤质量(湿重)。图9显示了平均值±SEM。所进行的统计是非参数Mann-Whitney,“ns”表示不显著(p>0.05)的值,*p<0.05;**p<0.01。
图10A和图10B显示了随时间的肿瘤体积,肿瘤平均大小和对个体动物的单独的图。图10A显示了所有肿瘤体积随时间的平均值±SEM。图10B显示了个体动物在每个测量时间点的平均值。收集肿瘤移植物黑色素瘤B16F10细胞,并以5x10
图11A和图11B显示了第54天的CD8+OT-1+T细胞百分比(脾)和流式图设门策略。图11A显示了第54天研究结束时,对疫苗接种的内源性应答(百分比),以FSC-H和FSC-A设门从而对双联体细胞进行活/死设门,然后以CD45和SSC设门,然后是GFP-OT-1CD8+双阳性细胞。图11B显示了平均值±SEM,通过非参数统计学检验Mann-Whitney,与mHS-110相比*p<0.05。
图12A和图12B显示了第54天的CD8+PD-1+T细胞百分比(脾)和流式设门策略。图12A显示了第54天研究结束时,对疫苗接种的内源性应答(百分比),以FSC-H和FSC-A设门从而对双联体细胞进行活/死设门,然后以CD3和SSC设门,然后是PD-1+CD8+双阳性细胞。图12B显示了平均值±SEM,通过非参数统计学检验Mann-Whitney,与mHS-110相比,*p<0.05。
图13是gp96-Ig(mHS-110,B16F10-OVA-gp96)与OX40L-Ig(mHS-130,B16F10-OVA-OX40L)剂量比例的研究设计的非限制性示意图,以将CD8+T-细胞扩增与肿瘤生长延迟关联。
图14显示了使用图13研究中不同比例和剂量组合的mHS-110和mHS-130的初次和加强免疫的抗肿瘤CD8+OT-I T细胞扩增(外周血中)。利用mHS-110和mHS-130以不同比例和剂量的gp96-Ig比OX40L-Ig注射给接受者小鼠。在疫苗接种后的第0至54天,在血液中分析OT-I GFP+CD8+T细胞。在第14天使用与初始阶段相同比例的mHS110和mHS130加强小鼠,并在激发后的天数中在血液中分析OT-I GFP+CD8+T细胞。数据代表平均百分数±SEM。
图15A至图15D显示了流式细胞术设门策略和使用图13研究中的mHS-110/130免疫接种下CD8+OT-I T细胞随时间的扩增。图15A显示了对所测试的比例和mHS-110/130免疫接种剂量,CD8+OT-I T细胞随时间的流式细胞术设门策略。图15B的条形图显示了第7天和第17天的CD8+OT-I T细胞扩增。图15C的条形图显示了第19、21、24、26、28、33、38和41天的CD8+OT-I T细胞扩增。图15D的条形图显示了第45、48和54天的CD8+OT-I T细胞扩增。数据代表平均百分数±SEM。统计学分析是Mann-Whitney,*p<0.05,**p<0.01,***p<0.001;“ns”表示p>0.05或“不显著”。
图16、17和18显示了在图13的研究的第7天,外周血中针对SLEC、MPEC、激活的/CD44
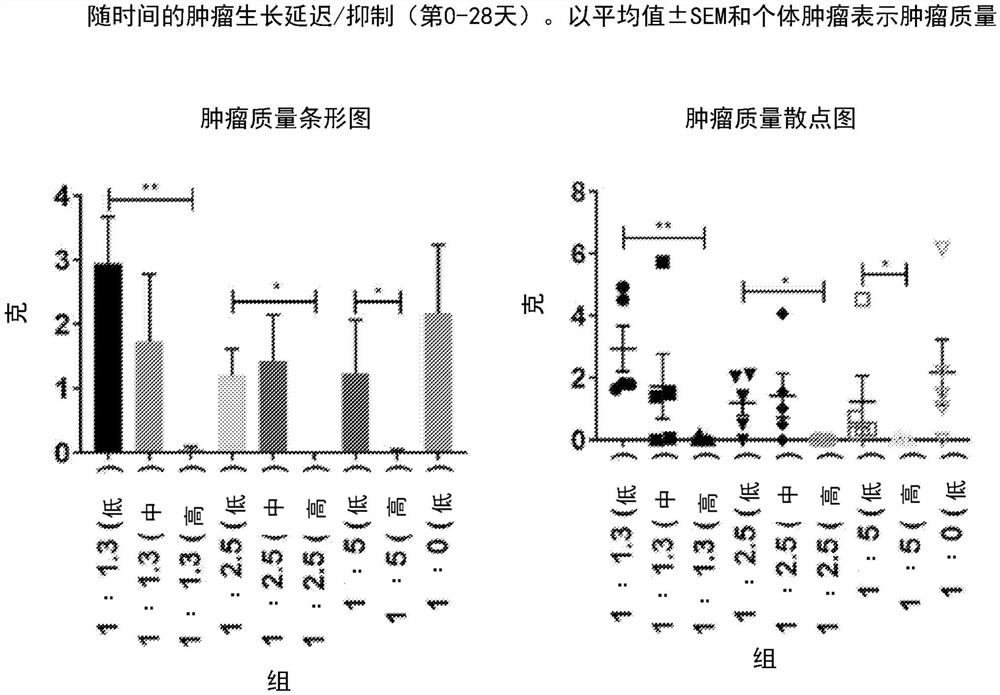
图19和20显示了图13研究中随时间的肿瘤生长延迟/抑制。图19显示了每个剂量比例组第0至28天的肿瘤直径(mm
图21的条形图显示了在图13研究的第55天,脾中CD3+CD8+四聚体-TRP2+T细胞的百分比。显示了设门样品的作图值,并且代表平均百分比±SEM。进行的统计学分析是Mann-Whitney,*p<0.05,**p<0.01;“ns”表示p>0.05或“不显著”。
图22的条形图显示了在图13研究的第55天,脾和血液中CD3+CD8+eGFP/OT-1+T细胞的百分比。显示了血液和脾中设门的CD8+eGFP/OT-1+T细胞,并且代表平均百分数±SEM。进行的统计学分析是Mann-Whitney。
图23的条形图显示了第55天的脾细胞表型。数据显示了在图13研究的第55天,脾中CD3+CD4+PD-1+T细胞的百分比。数据代表平均百分数±SEM。进行的统计学分析是Mann-Whitney,“ns”表示p>0.05或“不显著”。
图24的条形图显示了在图13研究的第55天,脾中CD3+CD4+CD44/CD62L中央记忆T细胞的百分比。数据代表平均百分数±SEM。进行的统计学分析是Mann-Whitney,*p<0.05,**p<0.01。
图25的条形图显示了肿瘤浸润淋巴细胞(TIL)表型。在图13研究的第55天显示了CD8+TIL(%CD8+CD3+T细胞)。数据代表平均百分数±SEM。进行的统计学分析为Mann-Whitney,*p<0.05,“ns”表示p>0.05或“不显著”。
图26的条形图显示了肿瘤浸润淋巴细胞(TIL)表型。在图13研究的第55天显示了CD4+TIL(%CD4+CD3+T细胞)。数据代表平均百分数±SEM。进行的统计学分析是Mann-Whitney,*p<0.05;“ns”表示p>0.05或“不显著”。
具体实施方式
各种可分泌型蛋白,即本文所述的疫苗蛋白,可用于在体内刺激免疫应答。例如,基于可分泌型热休克蛋白gp96-Ig的同种异基因细胞疫苗可通过体内抗原交叉致敏(antigen cross-priming)实现对飞摩尔浓度的肿瘤抗原的高频多克隆CD8+T细胞应答(Oizumi et al.,J Immunol 2007,179(4):2310-2317)。然而,由已形成的肿瘤产生的多种免疫抑制机制能够抑制这种疫苗方法的活性。在针对疾病晚期的患者的组合免疫疗法中,在早已建立的B16-F10黑色素瘤的小鼠模型中对PD-1、PD-L1、CTLA-4和LAG-3阻断抗体的系统性比较表明,gp96-Ig疫苗接种和PD-1阻断的组合与其他检查点相比要好得多。协同抗肿瘤优势可归因于gp96-Ig疫苗接种、PD-1阻断和T细胞共刺激的三重组合,使用以下一种或多种产生所述T细胞共刺激:OX40的激动剂(例如OX40配体-Ig(OX40L-Ig)融合蛋白或其与OX40结合的片段)、诱导型T细胞共刺激因子(ICOS)的激动剂(例如,ICOS配体-Ig(ICOSL-Ig)融合蛋白或其与ICOS结合的片段)、CD40的激动剂(例如,CD40L-Ig融合蛋白或其片段)、CD27的激动剂(例如,CD70-Ig融合蛋白或其片段)、4-1BB的激动剂(例如,4-1BB配体-Ig(4-1BBL-Ig)融合蛋白或其与4-1BB结合的片段)、TNFRSF25的激动剂(例如,TL1A-Ig融合蛋白或其与TNFRSF25结合的片段)或糖皮质激素诱导的肿瘤坏死因子受体(GITR)的激动剂(例如,GITR配体-Ig(GITRL-Ig)融合蛋白或其与GITF结合的片段)。同种异基因细胞系分泌的gp96-Ig和这些共刺激融合蛋白增强了抗原特异性CD8+T细胞的激活。尽管任何理论,局部分泌的T细胞共刺激融合蛋白(即OX40L-Ig)的作用尤其不同于与可分泌型疫苗蛋白(例如gp96-Ig)组合的全身施用。
疫苗蛋白
疫苗蛋白可以诱导在本发明中有用的免疫应答。在一些实施方案中,本公开提供了基于细胞的治疗剂,其包括第一细胞,所述第一细胞包含含有编码可分泌型疫苗蛋白的核苷酸序列的表达载体,以及第二细胞,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体。还提供了可用于本发明的基于细胞的治疗剂中的组合物。在多个实施方案中,这样的组合物用于治疗受试者的方法中,以刺激受试者中免疫应答,包括增强受试者中抗原特异性T细胞的激活。本发明的组合物可用于治疗各种疾病,包括癌症。
位于内质网(ER)中的热休克蛋白(hsp)gp96,在各种多肽通向I类和II类MHC分子的路径中作为肽的伴侣。从肿瘤细胞获得并用作疫苗的gp96能诱导特异性肿瘤免疫,推测是通过将肿瘤特异性肽运送至抗原呈递细胞(APC)(J Immunol 1999,163(10):5178-5182)。例如,与gp96相关的肽通过树突细胞(DC)交叉呈递至CD8细胞。
通过将gp96-Ig G1-Fc融合蛋白转染到肿瘤细胞中,导致gp96-Ig与伴侣性肿瘤肽的复合体的分泌,从而开发了一种用于抗肿瘤治疗的疫苗接种系统(参见,J Immunother2008,31(4):394-401,以及其中引用的参考文献)。胃肠外施用分泌gp96-Ig的肿瘤细胞会触发稳健的抗原特异性CD8细胞毒性T淋巴细胞(CTL)扩增,并激活先天免疫系统。肿瘤分泌的gp96导致DC和自然杀伤(NK)细胞募集到gp96分泌部位,并介导DC激活。此外,gp96及其伴侣性肽的内吞摄取会引起通过主要I类MHC的肽交叉呈递,以及独立于CD4细胞的强同源CD8激活。
本文提供的基于细胞的治疗剂涉及编码gp96-Ig融合蛋白的第一核苷酸序列。人gp96的编码区长度为2,412个碱基(SEQ ID NO:1),并编码803个氨基酸的蛋白(SEQ ID NO:2),所述蛋白包括在氨基末端的21个氨基酸的信号肽、富含疏水残基的潜在跨膜区以及在羧基末端的ER保留肽序列(
atgagggccctgtgggtgctgggcctctgctgcgtcctgctgaccttcgggtcggtcagagctgacgatgaagttgatgtggatggtacagtagaagaggatctgggtaaaagtagagaaggatcaaggacggatgatgaagtagtacagagagaggaagaagctattcagttggatggattaaatgcatcacaaataagagaacttagagagaagtcggaaaagtttgccttccaagccgaagttaacagaatgatgaaacttatcatcaattcattgtataaaaataaagagattttcctgagagaactgatttcaaatgcttctgatgctttagataagataaggctaatatcactgactgatgaaaatgctctttctggaaatgaggaactaacagtcaaaattaagtgtgataaggagaagaacctgctgcatgtcacagacaccggtgtaggaatgaccagagaagagttggttaaaaaccttggtaccatagccaaatctgggacaagcgagtttttaaacaaaatgactgaagcacaggaagatggccagtcaacttctgaattgattggccagtttggtgtcggtttctattccgccttccttgtagcagataaggttattgtcacttcaaaacacaacaacgatacccagcacatctgggagtctgactccaatgaattttctgtaattgctgacccaagaggaaacactctaggacggggaacgacaattacccttgtcttaaaagaagaagcatctgattaccttgaattggatacaattaaaaatctcgtcaaaaaatattcacagttcataaactttcctatttatgtatggagcagcaagactgaaactgttgaggagcccatggaggaagaagaagcagccaaagaagagaaagaagaatctgatgatgaagctgcagtagaggaagaagaagaagaaaagaaaccaaagactaaaaaagttgaaaaaactgtctgggactgggaacttatgaatgatatcaaaccaatatggcagagaccatcaaaagaagtagaagaagatgaatacaaagctttctacaaatcattttcaaaggaaagtgatgaccccatggcttatattcactttactgctgaaggggaagttaccttcaaatcaattttatttgtacccacatctgctccacgtggtctgtttgacgaatatggatctaaaaagagcgattacattaagctctatgtgcgccgtgtattcatcacagacgacttccatgatatgatgcctaaatacctcaattttgtcaagggtgtggtggactcagatgatctccccttgaatgtttcccgcgagactcttcagcaacataaactgcttaaggtgattaggaagaagcttgttcgtaaaacgctggacatgatcaagaagattgctgatgataaatacaatgatactttttggaaagaatttggtaccaacatcaagcttggtgtgattgaagaccactcgaatcgaacacgtcttgctaaacttcttaggttccagtcttctcatcatccaactgacattactagcctagaccagtatgtggaaagaatgaaggaaaaacaagacaaaatctacttcatggctgggtccagcagaaaagaggctgaatcttctccatttgttgagcgacttctgaaaaagggctatgaagttatttacctcacagaacctgtggatgaatactgtattcaggcccttcccgaatttgatgggaagaggttccagaatgttgccaaggaaggagtgaagttcgatgaaagtgagaaaactaaggagagtcgtgaagcagttgagaaagaatttgagcctctgctgaattggatgaaagataaagcccttaaggacaagattgaaaaggctgtggtgtctcagcgcctgacagaatctccgtgtgctttggtggccagccagtacggatggtctggcaacatggagagaatcatgaaagcacaagcgtaccaaacgggcaaggacatctctacaaattactatgcgagtcagaagaaaacatttgaaattaatcccagacacccgctgatcagagacatgcttcgacgaattaaggaagatgaagatgataaaacagttttggatcttgctgtggttttgtttgaaacagcaacgcttcggtcagggtatcttttaccagacactaaagcatatggagatagaatagaaagaatgcttcgcctcagtttgaacattgaccctgatgcaaaggtggaagaagagcccgaagaagaacctgaagagacagcagaagacacaacagaagacacagagcaagacgaagatgaagaaatggatgtgggaacagatgaagaagaagaaacagcaaaggaatctacagctgaaaaagatgaattgtaa(SEQ ID NO:1)
MRALWVLGLCCVLLTFGSVRADDEVDVDGTVEEDLGKSREGSRTDDEVVQREEEAIQLDGLNASQIRELREKSEKFAFQAEVNRMMKLIINSLYKNKEIFLRELISNASDALDKIRLISLTDENALSGNEELTVKIKCDKEKNLLHVTDTGVGMTREELVKNLGTIAKSGTSEFLNKMTEAQEDGQSTSELIGQFGVGFYSAFLVADKVIVTSKHNNDTQHIWESDSNEFSVIADPRGNTLGRGTTITLVLKEEASDYLELDTIKNLVKKYSQFINFPIYVWSSKTETVEEPMEEEEAAKEEKEESDDEAAVEEEEEEKKPKTKKVEKTVWDWELMNDIKPIWQRPSKEVEEDEYKAFYKSFSKESDDPMAYIHFTAEGEVTFKSILFVPTSAPRGLFDEYGSKKSDYIKLYVRRVFITDDFHDMMPKYLNFVKGVVDSDDLPLNVSRETLQQHKLLKVIRKKLVRKTLDMIKKIADDKYNDTFWKEFGTNIKLGVIEDHSNRTRLAKLLRFQSSHHPTDITSLDQYVERMKEKQDKIYFMAGSSRKEAESSPFVERLLKKGYEVIYLTEPVDEYCIQALPEFDGKRFQNVAKEGVKFDESEKTKESREAVEKEFEPLLNWMKDKALKDKIEKAVVSQRLTESPCALVASQYGWSGNMERIMKAQAYQTGKDISTNYYASQKKTFEINPRHPLIRDMLRRIKEDEDDKTVLDLAVVLFETATLRSGYLLPDTKAYGDRIERMLRLSLNIDPDAKVEEEPEEEPEETAEDTTEDTEQDEDEEMDVGTDEEEETAKESTAEKDEL(SEQ ID NO:2).
可以使用美国专利号8,685,384(通过引用以其全文并入本文)中描述的方法生产编码gp96-Ig融合序列的核酸。在一些实施方案中,gp96-Ig融合蛋白的gp96部分可含有野生型gp96序列(例如,SEQ ID NO:2所示的人序列)的全部或部分。例如,可分泌型gp96-Ig融合蛋白可包括SEQ ID NO:2的前799个氨基酸,使得其缺乏C末端KDEL(SEQ ID NO:3)序列。或者,融合蛋白的gp96部分可以具有与野生型gp96序列的前799个氨基酸相比含有一个或多个取代、缺失或添加的氨基酸序列,使得其与野生型多肽具有至少90%(例如至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%)的序列同一性。
如本公开全文所用,如下地确定特定核酸或氨基酸序列与由特定序列识别号所表示的序列之间的序列同一性百分比。首先,使用来自含有BLASTN版本2.0.14和BLASTP版本2.0.14的BLASTZ独立版本的BLAST 2Sequences(Bl2seq)程序,将核酸或氨基酸序列与特定序列识别号中所示的序列进行比较。可以从fr.com/blast或ncbi.nlm.nih.gov在线获得BLASTZ的独立版本。可以在BLASTZ随附的自述文件中找到解释如何使用Bl2seq程序的说明。Bl2seq使用BLASTN或BLASTP算法进行两个序列间的比较。BLASTN用于比较核酸序列,而BLASTP用于比较氨基酸序列。为了比较两个核酸序列,选项设置如下:-i设置为含有待比较的第一核酸序列的文件(例如C:\seq1.txt);-j设置为含有待比较的第二核酸序列的文件(例如C:\seq2.txt);-p设置为blastn;-o设置为任何希望的文件名(例如C:\output.txt);-q设置为-1;-r设置为2;且其他所有选项均保留其默认设置。例如,以下命令可用于生成含有两个序列之间的比较的输出文件:C:\Bl2seq-i c:\seq1.txt-j c:\seq2.txt-p blastn-o c:\output.txt-q-1-r 2。为了比较两个氨基酸序列,Bl2seq的选项设置如下:-i设置为含有待比较的第一氨基酸序列的文件(例如C:\seq1.txt);-j设置为含有待比较的第二氨基酸序列的文件(例如C:\seq2.txt);-p设置为blastp;-o设置为任何希望的文件名(例如C:\output.txt);且其他所有选项均保留其默认设置。例如,以下命令可用于生成含有两个氨基酸序列之间的比较的输出文件:C:\Bl2seq-i c:\seq1.txt-j c:\seq2.txt-p blastp-o c:\output.txt。如果两个比较的序列具有同源性,则指定的输出文件将这些同源性区域呈现为比对序列。如果两个比较的序列不具有同源性,则指定的输出文件将不会呈现比对序列。
一旦比对,通过计数两个序列中存在相同核苷酸或氨基酸残基的位置的数目来确定匹配数。序列同一性百分比是通过以下确定的:将匹配数除以所鉴定序列(例如SEQ IDNO:1)中所示的序列长度,或除以铰接长度(例如,来自所鉴定序列中所示的序列的100个连续核苷酸或氨基酸残基),然后将所得数值乘以100。例如,与SEQ ID NO:1中所示的序列比对时具有2,200个匹配的核酸序列,与SEQ ID NO:1中所示的序列91.2%同一(即,2,000÷2,412x100=91.2)。应注意,序列同一性百分比值四舍五入到小数点后一位。例如,将75.11、75.12、75.13和75.14四舍五入为75.1,而将75.15、75.16、75.17、75.18和75.19四舍五入为75.2。还应注意,长度值将始终为整数。
因此,在一些实施方案中,编码gp96-Ig融合多肽的核酸的gp96部分可编码在一个或多个氨基酸位置与野生型gp96多肽不同的氨基酸序列,使得其含有一个或多个保守性取代、非保守性取代、剪接变体、同工型(isoform)、其他物种的同源物以及多态性。
如本文所定义,“保守性取代”是指氨基酸残基被另一个生物学上相似的残基所替换。通常,如上所述,生物学相似性反映了野生型序列上用保守性氨基酸的取代。例如,预期保守性氨基酸取代对生物学活性几乎没有影响或没有影响,尤其是如果它们占多肽或蛋白中残基总数的不到10%。可以例如基于所涉及的氨基酸残基的极性、电荷、大小、溶解度、疏水性、亲水性和/或两亲性质的相似性,来进行保守性取代。20种天然存在的氨基酸可分为以下六个标准氨基酸组:(1)疏水性:Met、Ala、Val、Leu、Ile;(2)中性亲水性:Cys、Ser、Thr;Asn、Gln;(3)酸性:Asp、Glu;(4)碱性:His、Lys、Arg;(5)影响链取向的残基:Gly、Pro;和(6)芳香族:Trp、Tyr、Phe。因此,可以通过将氨基酸与上述六个标准氨基酸组的同一组中列出的另一个氨基酸进行交换来实现保守性取代。例如,将Asp交换为Glu在如此修饰的多肽中保留一个负电荷。另外,甘氨酸和脯氨酸基于它们破坏α-螺旋的能力而可以彼此取代。保守性氨基酸取代的其他实例包括但不限于,一个疏水性残基取代为另一个,例如异亮氨酸、缬氨酸、亮氨酸或甲硫氨酸,或一个极性残基取代为另一个,例如精氨酸取代赖氨酸,谷氨酸取代天冬氨酸,或谷氨酰胺取代天冬酰胺等。术语“保守性取代”还包括使用取代的氨基酸残基代替未取代的亲本氨基酸残基,条件是针对取代的多肽产生的抗体也对未取代的多肽有免疫反应。
如本文所用,“非保守性取代”定义为氨基酸与上述六个标准氨基酸组(1)至(6)的不同组中列出的另一个氨基酸的交换。
在多个实施方案中,取代还可以包括非经典氨基酸(例如、硒代半胱氨酸、吡咯赖氨酸、N-甲酰甲硫氨酸β-丙氨酸、GABA和δ-氨基乙酰丙酸、4-氨基苯甲酸(PABA)、常见氨基酸的左旋体、2,4-二氨基丁酸、α-氨基异丁酸、4-氨基丁酸、Abu、2-氨基丁酸、γ-Abu、ε-Ahx、6-氨基己酸、Aib、2-氨基异丁酸、3-氨基丙酸、鸟氨酸、正亮氨酸、正缬氨酸、羟脯氨酸、sarcosme、瓜氨酸、高瓜氨酸、半胱氨酸、叔丁基甘氨酸、叔丁基丙氨酸、苯基甘氨酸、环己基丙氨酸、β-丙氨酸、氟代氨基酸、设计者氨基酸如β甲基氨基酸、Cα-甲基氨基酸、Nα-甲基氨基酸以及一般的氨基酸类似物。
还可以通过参考遗传密码,包括考虑密码子简并性,来对本发明的融合蛋白的核苷酸序列进行突变。
gp96-Ig融合蛋白的Ig部分(“标签”)可含有例如免疫球蛋白分子(例如IgG1、IgG2、IgG3、IgG4、IgM、IgA或IgE分子)的非可变部分。通常,这样的部分含有至少免疫球蛋白重链恒定区的功能性CH2和CH3结构域。也可以使用恒定结构域Fc部分的羧基末端,或紧接重链或轻链CH1氨基末端的区域进行融合。Ig标签可以来自哺乳动物(例如人、小鼠、猴子或大鼠)免疫球蛋白,但当gp96-Ig融合蛋白旨在用于人的体内用途时,人免疫球蛋白可以尤其有用。
编码免疫球蛋白轻或重链恒定区的DNA是已知的或容易从cDNA文库获得的。参见,例如,Adams et al.,Biochemistry 1980,19:2711-2719;Gough et al.,Biochemistry1980 19:2702-2710;Dolby et al.,Proc Natl Acad Sci USA 1980,77:6027-6031;Riceet al.,Proc Natl Acad Sci USA 1982,79:7862-7865;Falkner et al.,Nature 1982,298:286-288;和Morrison et al.,Ann Rev Immunol 1984,2:239-256。由于许多免疫学试剂和标记系统可用于免疫球蛋白检测,因此可以通过本领域已知的各种免疫学技术(例如酶联免疫吸附测定法(ELISA)、免疫沉淀和荧光激活细胞分选(FACS))容易地检测和定量gp96-Ig融合蛋白。同样,如果肽标签是具有容易获得的抗体的表位,则可以将此类试剂与上述技术一起使用,以检测、定量和分离gp96-Ig融合蛋白。
在多个实施方案中,gp96-Ig融合蛋白和/或共刺激分子融合蛋白包含接头。在多个实施方案中,接头可以衍生自天然存在的多结构域蛋白或经验性接头,如例如以下中描述:Chichili et al.,(2013),Protein Sci.22(2):153-167,Chen et al.,(2013),AdvDrug Deliv Rev.65(10):1357-1369,其通过引用以其全文并入本文。在一些实施方案中,可以使用接头设计数据库和计算机程序(例如描述在Chen et al.,(2013),Adv DrugDeliv Rev.65(10):1357-1369和Crasto et.al.,(2000),Protein Eng.13(5):309-312中的那些,其通过引用以其全文并入本文)来设计接头。
在一些实施方案中,接头是合成接头,例如PEG。
在其他实施方案中,接头是多肽。在一些实施方案中,接头的长度小于约100个氨基酸。例如,接头的长度可以小于约100个、约95个、约90个、约85个、约80个、约75个、约70个、约65个、约60个、约55个、约50个、约45个、约40个、约35个、约30个、约25个、约20个、约19个、约18个、约17个、约16个、约15个、约14个、约13个、约12个、约11个、约10个、约9个、约8个、约7个、约6个、约5个、约4个、约3个或约2个氨基酸。在一些实施方案中,接头是柔性的。在另一个实施方案中,接头是刚性的。在多个实施方案中,接头基本上包含甘氨酸和丝氨酸残基(例如约30%,或约40%,或约50%,或约60%,或约70%,或约80%,或约90%,或约95%,或约97%的甘氨酸和丝氨酸)。
在多个实施方案中,接头是抗体(例如IgG、IgA、IgD和IgE,包括亚类(例如IgG1、IgG2、IgG3和IgG4,以及IgA1和IgA2))的铰链区。在IgG、IgA、IgD和IgE类抗体中发现的铰链区作为柔性间隔区,允许Fab部分在空间中自由移动。与恒定区相比,铰链结构域在结构上是多样的,其序列和长度在免疫球蛋白类别和亚类之间均变化。例如,铰链区的长度和柔性在IgG亚类之间变化。IgG1的铰链区包含氨基酸216-231,并且由于它是自由柔性的,Fab片段能够绕其对称轴旋转并在以两个重链间二硫键的第一个为中心的球内移动。IgG2的铰链比IgG1短,具有12个氨基酸残基和4个二硫键。IgG2的铰链区缺少甘氨酸残基,相对较短,并含有刚性的多脯氨酸双螺旋,其由额外的重链间二硫键来稳定。这些性质限制了IgG2分子的柔性。IgG3与其他亚类的不同之处在于其独特的延伸铰链区(约为IgG1铰链的四倍),含有62个氨基酸(包括21个脯氨酸和11个半胱氨酸),形成非柔性的多脯氨酸双螺旋。在IgG3中,Fab片段距离Fc片段相对较远,赋予了分子更大的柔性。IgG3中的延长铰链还导致其相比于其他亚类较高的分子量。IgG4的铰链区比IgG1的短,并且其柔性在IgG1和IgG2的铰链区之间。据报告,铰链区的柔性以IgG3>IgG1>IgG4>IgG2的顺序降低。
其他示例性的接头包括但不限于具有以下序列的接头:LE、GGGGS(SEQ ID NO:14)、(GGGGS)n(n=1-4)(SEQ ID NO:15)、(Gly)8(SEQ ID NO:16)、(Gly)6(SEQ ID NO:17)、(EAAAK)n(n=1-3)(SEQ ID NO:18)、A(EAAAK)nA(n=2-5)(SEQ ID NO:19)、AEAAAKEAAAKA(SEQ ID NO:20)、A(EAAAK)4ALEA(EAAAK)4A(SEQ ID NO:21)、PAPAP(SEQ ID NO:22)、KESGSVSSEQLAQFRSLD(SEQ ID NO:23)、EGKSSGSGSESKST(SEQ ID NO:24)、GSAGSAAGSGEF(SEQ ID NO:25)和(XP)n,其中X表示任意氨基酸,例如Ala、Lys或Glu。
在多个实施方案中,接头可以是功能性的。例如但不限于,接头可以起到改善本发明组合物的折叠和/或稳定性、改善表达、改善药物动力学和/或改善生物活性的作用。在另一个实例中,接头可以起到使组合物靶向特定细胞类型或位置的作用。
在一些实施方案中,可以将gp96肽融合至鼠IgG1的铰链、CH2和CH3结构域(Bowenet al.,J Immunol 1996,156:442-449)。IgG1分子的这个区域含有三个半胱氨酸残基,其通常与Ig分子中其他半胱氨酸一起参与二硫键合。由于肽不需要任何半胱氨酸发挥其作为标签的功能,因此这些半胱氨酸残基的一个或多个可以被另一氨基酸残基例如丝氨酸所取代。
本领域已知的多种前导序列也可以用于从细菌和哺乳动物细胞中有效分泌gp96-Ig融合蛋白(参见,von Heijne,J Mol Biol 1985,184:99-105)。前导肽可以基于目标宿主细胞进行选择,并且可以包括细菌、酵母、病毒、动物和哺乳动物序列。例如,疱疹病毒糖蛋白D前导肽适合用于多种哺乳动物细胞中。可用于哺乳动物细胞中的另一前导肽可以从小鼠免疫球蛋白κ链的V-J2-C区获得(Bernard et al.,Proc Natl Acad Sci USA1981,78:5812-5816)。编码肽标签或前导肽的DNA序列是已知的或容易从文库或商业供应商获得的,并且适用于本文所述的融合蛋白。
此外,在多个实施方案中,可以用一种或多种疫苗蛋白取代本公开的gp96。例如,疫苗蛋白中包括多种热休克蛋白。在多个实施方案中,热休克蛋白是以下的一个或多个:小hsp、hsp40、hsp60、hsp70、hsp90和hsp110家族成员,包括其片段、变体、突变体、衍生物或组合(Hickey,et al.,1989,Mol.Cell.Biol.9:2615-2626;Jindal,1989,Mol.Cell.Biol.9:2279-2283)。
T细胞共刺激
使用本文提供的表达载体的基于细胞的治疗剂可以编码一种或多种生物应答修饰子(modifier)。在多个实施方案中,基于细胞的治疗剂可以编码一种或多种T细胞共刺激分子。
在多个实施方案中,基于细胞的治疗剂允许稳健的抗原特异性CD8细胞毒性T淋巴细胞(CTL)扩增。在多个实施方案中,基于细胞的治疗剂选择性增强CD8细胞毒性T淋巴细胞(CTL),并且基本上不增强可以是促肿瘤(pro-tumor)的T细胞类型,并且其包括但不限于Treg、表达一种或多种检查点抑制性受体的CD4+和/或CD8+T细胞、Th2细胞和Th17细胞。检查点抑制性受体是指在免疫细胞上表达的可防止或抑制不受控制的免疫应答的受体(例如CTLA-4、B7-H3、B7-H4、TIM-3)。例如,本发明的基于细胞的治疗剂基本上不增强FOXP3+调节性T细胞。在一些实施方案中,这种选择性CD8 T细胞增强相比于通过gp-96融合蛋白和针对T细胞共刺激分子的抗体的组合疗法观察到的非特异性T细胞增强。
例如,基于细胞的治疗剂包含OX40的激动剂(例如OX40配体-Ig(OX40L-Ig)融合蛋白或其与OX40结合的片段)、诱导型T细胞共刺激因子(ICOS)的激动剂(例如,ICOS配体-Ig(ICOSL-Ig)融合蛋白或其与ICOS结合的片段)、CD40的激动剂(例如,CD40L-Ig融合蛋白或其片段)、CD27的激动剂(例如,CD70-Ig融合蛋白或其片段)、4-1BB的激动剂(例如,4-1BB配体-Ig(4-1BBL-Ig)融合蛋白或其与4-1BB结合的片段)。在一些实施方案中,基于细胞的治疗剂包含编码以下的载体:TNFRSF25的激动剂(例如,TL1A-Ig融合蛋白或其与TNFRSF25结合的片段),或糖皮质激素诱导的肿瘤坏死因子受体(GITR)的激动剂(例如,GITR配体-Ig(GITRL-Ig)融合蛋白或其与GITF结合的片段),或CD40的激动剂(例如,CD40配体-Ig(CD40L-Ig)融合蛋白或其与CD40结合的片段);或CD27的激动剂(例如CD27配体-Ig(例如CD70L-Ig)融合蛋白或其与CD4 0结合的片段)。
ICOS是可诱导型T细胞共刺激受体分子,其与CD28和CTLA-4具有一些同源性,并且与抗原呈递细胞表面上表达的B7-H2相互作用。ICOS已经参与调节细胞介导的免疫应答和体液免疫应答。
4-1BB是属于TNF超家族的2型跨膜糖蛋白,并在激活的T淋巴细胞上表达。
OX40(也称为CD134或TNFRSF4)是由OX40L接合的T细胞共刺激分子,并经常在抗原呈递细胞和其他细胞类型中被诱导。已知OX40可增强细胞因子表达和效应T细胞的存活。
GITR(TNFRSF18)是由GITRL接合的T细胞共刺激分子,并优选在FoxP3+调节性T细胞中表达。GITR在肿瘤微环境内Treg的维持和功能中起重要作用。
TNFRSF25是在抗原刺激后优选在CD4+和CD8+T细胞中表达的T细胞共刺激分子。经由TNFRSF25的信号传导由TL1A提供,并以依赖于同源性抗原的方式增强T细胞对IL-2受体介导的增殖的敏感性。
CD40是在多种抗原呈递细胞上发现且在其激活中起作用的共刺激蛋白。TH细胞上CD40L(CD154)与CD40的结合激活了抗原呈递细胞并诱导多种下游效应。
CD27是属于TNF超家族的T细胞共刺激分子,其在T细胞免疫的产生和长期维持中起作用。它在多种免疫学过程中与配体CD70结合。
可用于本发明的其他共刺激分子包括但不限于HVEM、CD28、CD30、CD30L、CD40、CD70、LIGHT(CD258)、B7-1和B7-2。
对于gp96-Ig融合蛋白,T细胞共刺激融合蛋白的Ig部分(“标签”)可含有免疫球蛋白分子(例如IgG1、IgG2、IgG3、IgG4、IgM、IgA或IgE分子)的非可变部分。如上所述,这样的部分通常至少含有免疫球蛋白重链恒定区的功能性CH2和CH3结构域。在一些实施方案中,可以将T细胞共刺激肽与鼠IgG1的铰链、CH2和CH3结构域融合(Bowen et al.,J Immunol1996,156:442-449)。Ig标签可以来自哺乳动物(例如人、小鼠、猴子或大鼠)免疫球蛋白,但当融合蛋白旨在用于人的体内用途时,人免疫球蛋白可以尤其有用。同样,编码免疫球蛋白轻或重链恒定区的DNA是已知的或容易从cDNA文库获得的。如上所述的多种前导序列也可以用于从细菌和哺乳动物细胞分泌T细胞共刺激融合蛋白。
提供了编码与Ig融合的人ICOSL的细胞外结构域的代表性核苷酸序列(SEQ IDNO:4),以及所编码的融合蛋白的氨基酸序列(SEQ ID NO:5):
ATGAGACTGGGAAGCCCTGGCCTGCTGTTTCTGCTGTTCAGCAGCCTGAGAGCCGACACCCAGGAAAAAGAAGTGCGGGCCATGGTGGGAAGCGACGTGGAACTGAGCTGCGCCTGTCCTGAGGGCAGCAGATTCGACCTGAACGACGTGTACGTGTACTGGCAGACCAGCGAGAGCAAGACCGTCGTGACCTACCACATCCCCCAGAACAGCTCCCTGGAAAACGTGGACAGCCGGTACAGAAACCGGGCCCTGATGTCTCCTGCCGGCATGCTGAGAGGCGACTTCAGCCTGCGGCTGTTCAACGTGACCCCCCAGGACGAGCAGAAATTCCACTGCCTGGTGCTGAGCCAGAGCCTGGGCTTCCAGGAAGTGCTGAGCGTGGAAGTGACCCTGCACGTGGCCGCCAATTTCAGCGTGCCAGTGGTGTCTGCCCCCCACAGCCCTTCTCAGGATGAGCTGACCTTCACCTGTACCAGCATCAACGGCTACCCCAGACCCAATGTGTACTGGATCAACAAGACCGACAACAGCCTGCTGGACCAGGCCCTGCAGAACGATACCGTGTTCCTGAACATGCGGGGCCTGTACGACGTGGTGTCCGTGCTGAGAATCGCCAGAACCCCCAGCGTGAACATCGGCTGCTGCATCGAGAACGTGCTGCTGCAGCAGAACCTGACCGTGGGCAGCCAGACCGGCAACGACATCGGCGAGAGAGACAAGATCACCGAGAACCCCGTGTCCACCGGCGAGAAGAATGCCGCCACCTCTAAGTACGGCCCTCCCTGCCCTTCTTGCCCAGCCCCTGAATTTCTGGGCGGACCCTCCGTGTTTCTGTTCCCCCCAAAGCCCAAGGACACCCTGATGATCAGCCGGACCCCCGAAGTGACCTGCGTGGTGGTGGATGTGTCCCAGGAAGATCCCGAGGTGCAGTTCAATTGGTACGTGGACGGGGTGGAAGTGCACAACGCCAAGACCAAGCCCAGAGAGGAACAGTTCAACAGCACCTACCGGGTGGTGTCTGTGCTGACCGTGCTGCACCAGGATTGGCTGAGCGGCAAAGAGTACAAGTGCAAGGTGTCCAGCAAGGGCCTGCCCAGCAGCATCGAAAAGACCATCAGCAACGCCACCGGCCAGCCCAGGGAACCCCAGGTGTACACACTGCCCCCTAGCCAGGAAGAGATGACCAAGAACCAGGTGTCCCTGACCTGTCTCGTGAAGGGCTTCTACCCCTCCGATATCGCCGTGGAATGGGAGAGCAACGGCCAGCCAGAGAACAACTACAAGACCACCCCCCCAGTGCTGGACAGCGACGGCTCATTCTTCCTGTACTCCCGGCTGACAGTGGACAAGAGCAGCTGGCAGGAAGGCAACGTGTTCAGCTGCAGCGTGATGCACGAAGCCCTGCACAACCACTACACCCAGAAGTCCCTGTCTCTGTCCCTGGGCAAATGA(SEQ ID NO:4).
MRLGSPGLLFLLFSSLRADTQEKEVRAMVGSDVELSCACPEGSRFDLNDVYVYWQTSESKTVVTYHIPQNSSLENVDSRYRNRALMSPAGMLRGDFSLRLFNVTPQDEQKFHCLVLSQSLGFQEVLSVEVTLHVAANFSVPVVSAPHSPSQDELTFTCTSINGYPRPNVYWINKTDNSLLDQALQNDTVFLNMRGLYDVVSVLRIARTPSVNIGCCIENVLLQQNLTVGSQTGNDIGERDKITENPVSTGEKNAATSKYGPPCPSCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLSGKEYKCKVSSKGLPSSIEKTISNATGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSSWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK(SEQ ID NO:5).
提供了编码与Ig融合的人4-1BBL的细胞外结构域的代表性核苷酸序列(SEQ IDNO:6),以及所编码的氨基酸序列(SEQ ID NO:7):
ATGTCTAAGTACGGCCCTCCCTGCCCTAGCTGCCCTGCCCCTGAATTTCTGGGCGGACCCAGCGTGTTCCTGTTCCCCCCAAAGCCCAAGGACACCCTGATGATCAGCCGGACCCCCGAAGTGACCTGCGTGGTGGTGGATGTGTCCCAGGAAGATCCCGAGGTGCAGTTCAATTGGTACGTGGACGGCGTGGAAGTGCACAACGCCAAGACCAAGCCCAGAGAGGAACAGTTCAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAGCGGCAAAGAGTACAAGTGCAAGGTGTCCAGCAAGGGCCTGCCCAGCAGCATCGAGAAAACCATCAGCAACGCCACCGGCCAGCCCAGGGAACCCCAGGTGTACACACTGCCCCCTAGCCAGGAAGAGATGACCAAGAACCAGGTGTCCCTGACCTGTCTCGTGAAGGGCTTCTACCCCTCCGATATCGCCGTGGAATGGGAGAGCAACGGCCAGCCTGAGAACAACTACAAGACCACCCCCCCAGTGCTGGACAGCGACGGCTCATTCTTCCTGTACAGCAGACTGACCGTGGACAAGAGCAGCTGGCAGGAAGGCAACGTGTTCAGCTGCAGCGTGATGCACGAGGCCCTGCACAACCACTACACCCAGAAGTCCCTGTCTCTGAGCCTGGGCAAGGCCTGTCCATGGGCTGTGTCTGGCGCTAGAGCCTCTCCTGGATCTGCCGCCAGCCCCAGACTGAGAGAGGGACCTGAGCTGAGCCCCGATGATCCTGCCGGACTGCTGGATCTGAGACAGGGCATGTTCGCCCAGCTGGTGGCCCAGAACGTGCTGCTGATCGATGGCCCCCTGAGCTGGTACAGCGATCCTGGACTGGCTGGCGTGTCACTGACAGGCGGCCTGAGCTACAAAGAGGACACCAAAGAACTGGTGGTGGCCAAGGCCGGCGTGTACTACGTGTTCTTTCAGCTGGAACTGCGGAGAGTGGTGGCCGGCGAAGGATCCGGCTCTGTGTCTCTGGCTCTGCATCTGCAGCCCCTGAGATCTGCTGCTGGCGCTGCTGCTCTGGCCCTGACAGTGGACCTGCCTCCTGCCTCTAGCGAGGCCAGAAACAGCGCATTCGGGTTTCAAGGCAGACTGCTGCACCTGTCTGCCGGCCAGAGACTGGGAGTGCATCTGCACACAGAGGCCAGAGCCAGGCACGCCTGGCAGCTGACTCAGGGCGCTACAGTGCTGGGCCTGTTCAGAGTGACCCCCGAGATTCCAGCCGGCCTGCCTAGCCCCAGATCCGAATGA(SEQ ID NO:6)
MSKYGPPCPSCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLSGKEYKCKVSSKGLPSSIEKTISNATGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSSWQEGNVFSCSVMHEALHNHYTQKSLSLSLGKACPWAVSGARASPGSAASPRLREGPELSPDDPAGLLDLRQGMFAQLVAQNVLLIDGPLSWYSDPGLAGVSLTGGLSYKEDTKELVVAKAGVYYVFFQLELRRVVAGEGSGSVSLALHLQPLRSAAGAAALALTVDLPPASSEARNSAFGFQGRLLHLSAGQRLGVHLHTEARARHAWQLTQGATVLGLFRVTPEIPAGLPSPRSE(SEQ ID NO:7).
提供了编码与Ig融合的人TL1A的细胞外结构域的代表性核苷酸序列(SEQ ID NO:8),以及所编码的氨基酸序列(SEQ ID NO:9):
ATGTCTAAGTACGGCCCTCCCTGCCCTAGCTGCCCTGCCCCTGAATTTCTGGGCGGACCCAGCGTGTTCCTGTTCCCCCCAAAGCCCAAGGACACCCTGATGATCAGCCGGACCCCCGAAGTGACCTGCGTGGTGGTGGATGTGTCCCAGGAAGATCCCGAGGTGCAGTTCAATTGGTACGTGGACGGCGTGGAAGTGCACAACGCCAAGACCAAGCCCAGAGAGGAACAGTTCAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAGCGGCAAAGAGTACAAGTGCAAGGTGTCCAGCAAGGGCCTGCCCAGCAGCATCGAGAAAACCATCAGCAACGCCACCGGCCAGCCCAGGGAACCCCAGGTGTACACACTGCCCCCTAGCCAGGAAGAGATGACCAAGAACCAGGTGTCCCTGACCTGTCTCGTGAAGGGCTTCTACCCCTCCGATATCGCCGTGGAATGGGAGAGCAACGGCCAGCCTGAGAACAACTACAAGACCACCCCCCCAGTGCTGGACAGCGACGGCTCATTCTTCCTGTACAGCAGACTGACCGTGGACAAGAGCAGCTGGCAGGAAGGCAACGTGTTCAGCTGCAGCGTGATGCACGAGGCCCTGCACAACCACTACACCCAGAAGTCCCTGTCTCTGAGCCTGGGCAAGATCGAGGGCCGGATGGATAGAGCCCAGGGCGAAGCCTGCGTGCAGTTCCAGGCTCTGAAGGGCCAGGAATTCGCCCCCAGCCACCAGCAGGTGTACGCCCCTCTGAGAGCCGACGGCGATAAGCCTAGAGCCCACCTGACAGTCGTGCGGCAGACCCCTACCCAGCACTTCAAGAATCAGTTCCCCGCCCTGCACTGGGAGCACGAACTGGGCCTGGCCTTCACCAAGAACAGAATGAACTACACCAACAAGTTTCTGCTGATCCCCGAGAGCGGCGACTACTTCATCTACAGCCAAGTGACCTTCCGGGGCATGACCAGCGAGTGCAGCGAGATCAGACAGGCCGGCAGACCTAACAAGCCCGACAGCATCACCGTCGTGATCACCAAAGTGACCGACAGCTACCCCGAGCCCACCCAGCTGCTGATGGGCACCAAGAGCGTGTGCGAAGTGGGCAGCAACTGGTTCCAGCCCATCTACCTGGGCGCCATGTTTAGTCTGCAAGAGGGCGACAAGCTGATGGTCAACGTGTCCGACATCAGCCTGGTGGATTACACCAAAGAGGACAAGACCTTCTTCGGCGCCTTTCTGCTCTGA(SEQ ID NO:8)
MSKYGPPCPSCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLSGKEYKCKVSSKGLPSSIEKTISNATGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSSWQEGNVFSCSVMHEALHNHYTQKSLSLSLGKIEGRMDRAQGEACVQFQALKGQEFAPSHQQVYAPLRADGDKPRAHLTVVRQTPTQHFKNQFPALHWEHELGLAFTKNRMNYTNKFLLIPESGDYFIYSQVTFRGMTSECSEIRQAGRPNKPDSITVVITKVTDSYPEPTQLLMGTKSVCEVGSNWFQPIYLGAMFSLQEGDKLMVNVSDISLVDYTKEDKTFFGAFLL(SEQ ID NO:9).
提供了编码人OX40L-Ig的代表性核苷酸序列(SEQ ID NO:10),以及所编码的氨基酸序列(SEQ ID NO:11):
ATGTCTAAGTACGGCCCTCCCTGCCCTAGCTGCCCTGCCCCTGAATTTCTGGGCGGACCCAGCGTGTTCCTGTTCCCCCCAAAGCCCAAGGACACCCTGATGATCAGCCGGACCCCCGAAGTGACCTGCGTGGTGGTGGATGTGTCCCAGGAAGATCCCGAGGTGCAGTTCAATTGGTACGTGGACGGCGTGGAAGTGCACAACGCCAAGACCAAGCCCAGAGAGGAACAGTTCAACAGCACCTACCGGGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAGCGGCAAAGAGTACAAGTGCAAGGTGTCCAGCAAGGGCCTGCCCAGCAGCATCGAGAAAACCATCAGCAACGCCACCGGCCAGCCCAGGGAACCCCAGGTGTACACACTGCCCCCTAGCCAGGAAGAGATGACCAAGAACCAGGTGTCCCTGACCTGTCTCGTGAAGGGCTTCTACCCCTCCGATATCGCCGTGGAATGGGAGAGCAACGGCCAGCCTGAGAACAACTACAAGACCACCCCCCCAGTGCTGGACAGCGACGGCTCATTCTTCCTGTACAGCAGACTGACCGTGGACAAGAGCAGCTGGCAGGAAGGCAACGTGTTCAGCTGCAGCGTGATGCACGAGGCCCTGCACAACCACTACACCCAGAAGTCCCTGTCTCTGAGCCTGGGCAAGATCGAGGGCCGGATGGATCAGGTGTCACACAGATACCCCCGGATCCAGAGCATCAAAGTGCAGTTTACCGAGTACAAGAAAGAGAAGGGCTTTATCCTGACCAGCCAGAAAGAGGACGAGATCATGAAGGTGCAGAACAACAGCGTGATCATCAACTGCGACGGGTTCTACCTGATCAGCCTGAAGGGCTACTTCAGTCAGGAAGTGAACATCAGCCTGCACTACCAGAAGGACGAGGAACCCCTGTTCCAGCTGAAGAAAGTGCGGAGCGTGAACAGCCTGATGGTGGCCTCTCTGACCTACAAGGACAAGGTGTACCTGAACGTGACCACCGACAACACCAGCCTGGACGACTTCCACGTGAACGGCGGCGAGCTGATCCTGATTCACCAGAACCCCGGCGAGTTCTGCGTGCTCTGA(SEQ ID NO:10)
MSKYGPPCPSCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLSGKEYKCKVSSKGLPSSIEKTISNATGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSSWQEGNVFSCSVMHEALHNHYTQKSLSLSLGKIEGRMDQVSHRYPRIQSIKVQFTEYKKEKGFILTSQKEDEIMKVQNNSVIINCDGFYLISLKGYFSQEVNISLHYQKDEEPLFQLKKVRSVNSLMVASLTYKDKVYLNVTTDNTSLDDFHVNGGELILIHQNPGEFCVL(SEQID NO:11).
人TL1A的代表性核苷酸和氨基酸序列分别如SEQ ID NO:12和SEQ ID NO:13中所示:
TCCCAAGTAGCTGGGACTACAGGAGCCCACCACCACCCCCGGCTAATTTTTTGTATTTTTAGTAGAGACGGGGTTTCACCGTGTTAGCCAAGATGGTCTTGATCACCTGACCTCGTGATCCACCCGCCTTGGCCTCCCAAAGTGCTGGGATTACAGGCATGAGCCACCGCGCCCGGCCTCCATTCAAGTCTTTATTGAATATCTGCTATGTTCTACACACTGTTCTAGGTGCTGGGGATGCAACAGGGGACAAAATAGGCAAAATCCCTGTCCTTTTGGGGTTGACATTCTAGTGACTCTTCATGTAGTCTAGAAGAAGCTCAGTGAATAGTGTCTGTGGTTGTTACCAGGGACACAATGACAGGAACATTCTTGGGTAGAGTGAGAGGCCTGGGGAGGGAAGGGTCTCTAGGATGGAGCAGATGCTGGGCAGTCTTAGGGAGCCCCTCCTGGCATGCACCCCCTCATCCCTCAGGCCACCCCCGTCCCTTGCAGGAGCACCCTGGGGAGCTGTCCAGAGCGCTGTGCCGCTGTCTGTGGCTGGAGGCAGAGTAGGTGGTGTGCTGGGAATGCGAGTGGGAGAACTGGGATGGACCGAGGGGAGGCGGGTGAGGAGGGGGGCAACCACCCAACACCCACCAGCTGCTTTCAGTGTTCTGGGTCCAGGTGCTCCTGGCTGGCCTTGTGGTCCCCCTCCTGCTTGGGGCCACCCTGACCTACACATACCGCCACTGCTGGCCTCACAAGCCCCTGGTTACTGCAGATGAAGCTGGGATGGAGGCTCTGACCCCACCACCGGCCACCCATCTGTCACCCTTGGACAGCGCCCACACCCTTCTAGCACCTCCTGACAGCAGTGAGAAGATCTGCACCGTCCAGTTGGTGGGTAACAGCTGGACCCCTGGCTACCCCGAGACCCAGGAGGCGCTCTGCCCGCAGGTGACATGGTCCTGGGACCAGTTGCCCAGCAGAGCTCTTGGCCCCGCTGCTGCGCCCACACTCTCGCCAGAGTCCCCAGCCGGCTCGCCAGCCATGATGCTGCAGCCGGGCCCGCAGCTCTACGACGTGATGGACGCGGTCCCAGCGCGGCGCTGGAAGGAGTTCGTGCGCACGCTGGGGCTGCGCGAGGCAGAGATCGAAGCCGTGGAGGTGGAGATCGGCCGCTTCCGAGACCAGCAGTACGAGATGCTCAAGCGCTGGCGCCAGCAGCAGCCCGCGGGCCTCGGAGCCGTTTACGCGGCCCTGGAGCGCATGGGGCTGGACGGCTGCGTGGAAGACTTGCGCAGCCGCCTGCAGCGCGGCCCGTGACACGGCGCCCACTTGCCACCTAGGCGCTCTGGTGGCCCTTGCAGAAGCCCTAAGTACGGTTACTTATGCGTGTAGACATTTTATGTCACTTATTAAGCCGCTGGCACGGCCCTGCGTAGCAGCACCAGCCGGCCCCACCCCTGCTCGCCCCTATCGCTCCAGCCAAGGCGAAGAAGCACGAACGAATGTCGAGAGGGGGTGAAGACATTTCTCAACTTCTCGGCCGGAGTTTGGCTGAGATCGCGGTATTAAATCTGTGAAAGAAAACAAAACAAAACAA(SEQ ID NO:12)
MEQRPRGCAAVAAALLLVLLGARAQGGTRSPRCDCAGDFHKKIGLFCCRGCPAGHYLKAPCTEPCGNSTCLVCPQDTFLAWENHHNSECARCQACDEQASQVALENCSAVADTRCGCKPGWFVECQVSQCVSSSPFYCQPCLDCGALHRHTRLLCSRRDTDCGTCLPGFYEHGDGCVSCPTPPPSLAGAPWGAVQSAVPLSVAGGRVGVFWVQVLLAGLVVPLLLGATLTYTYRHCWPHKPLVTADEAGMEALTPPPATHLSPLDSAHTLLAPPDSSEKICTVQLVGNSWTPGYPETQEALCPQVTWSWDQLPSRALGPAAAPTLSPESPAGSPAMMLQPGPQLYDVMDAVPARRWKEFVRTLGLREAEIEAVEVEIGRFRDQQYEMLKRWRQQQPAGLGAVYAALERMGLDGCVEDLRSRLQRGP(SEQ ID NO:13).
人HVEM的代表性核苷酸和氨基酸序列分别如SEQ ID NO:26(登录号CR456909)和SEQ ID NO:27(登录号CR456909)中所示:
ATGGAGCCTCCTGGAGACTGGGGGCCTCCTCCCTGGAGATCCACCCCCAAAACCGACGTCTTGAGGCTGGTGCTGTATCTCACCTTCCTGGGAGCCCCCTGCTACGCCCCAGCTCTGCCGTCCTGCAAGGAGGACGAGTACCCAGTGGGCTCCGAGTGCTGCCCCAAGTGCAGTCCAGGTTATCGTGTGAAGGAGGCCTGCGGGGAGCTGACGGGCACAGTGTGTGAACCCTGCCCTCCAGGCACCTACATTGCCCACCTCAATGGCCTAAGCAAGTGTCTGCAGTGCCAAATGTGTGACCCAGCCATGGGCCTGCGCGCGAGCCGGAACTGCTCCAGGACAGAGAACGCCGTGTGTGGCTGCAGCCCAGGCCACTTCTGCATCGTCCAGGACGGGGACCACTGCGCCGCGTGCCGCGCTTACGCCACCTCCAGCCCGGGCCAGAGGGTGCAGAAGGGAGGCACCGAGAGTCAGGACACCCTGTGTCAGAACTGCCCCCCGGGGACCTTCTCTCCCAATGGGACCCTGGAGGAATGTCAGCACCAGACCAAGTGCAGCTGGCTGGTGACGAAGGCCGGAGCTGGGACCAGCAGCTCCCACTGGGTATGGTGGTTTCTCTCAGGGAGCCTCGTCATCGTCATTGTTTGCTCCACAGTTGGCCTAATCATATGTGTGAAAAGAAGAAAGCCAAGGGGTGATGTAGTCAAGGTGATCGTCTCCGTCCAGCGGAAAAGACAGGAGGCAGAAGGTGAGGCCACAGTCATTGAGGCCCTGCAGGCCCCTCCGGACGTCACCACGGTGGCCGTGGAGGAGACAATACCCTCATTCACGGGGAGGAGCCCAAACCATTAA(SEQ ID NO:26)
MEPPGDWGPPPWRSTPKTDVLRLVLYLTFLGAPCYAPALPSCKEDEYPVGSECCPKCSPGYRVKEACGELTGTVCEPCPPGTYIAHLNGLSKCLQCQMCDPAMGLRASRNCSRTENAVCGCSPGHFCIVQDGDHCAACRAYATSSPGQRVQKGGTESQDTLCQNCPPGTFSPNGTLEECQHQTKCSWLVTKAGAGTSSSHWVWWFLSGSLVIVIVCSTVGLIICVKRRKPRGDVVKVIVSVQRKRQEAEGEATVIEALQAPPDVTTVAVEETIPSFTGRSPNH(SEQ ID NO:27).
人CD28的代表性核苷酸和氨基酸序列分别如SEQ ID NO:28(登录号NM_006139)和SEQ ID NO:29中所示:
TAAAGTCATCAAAACAACGTTATATCCTGTGTGAAATGCTGCAGTCAGGATGCCTTGTGGTTTGAGTGCCTTGATCATGTGCCCTAAGGGGATGGTGGCGGTGGTGGTGGCCGTGGATGACGGAGACTCTCAGGCCTTGGCAGGTGCGTCTTTCAGTTCCCCTCACACTTCGGGTTCCTCGGGGAGGAGGGGCTGGAACCCTAGCCCATCGTCAGGACAAAGATGCTCAGGCTGCTCTTGGCTCTCAACTTATTCCCTTCAATTCAAGTAACAGGAAACAAGATTTTGGTGAAGCAGTCGCCCATGCTTGTAGCGTACGACAATGCGGTCAACCTTAGCTGCAAGTATTCCTACAATCTCTTCTCAAGGGAGTTCCGGGCATCCCTTCACAAAGGACTGGATAGTGCTGTGGAAGTCTGTGTTGTATATGGGAATTACTCCCAGCAGCTTCAGGTTTACTCAAAAACGGGGTTCAACTGTGATGGGAAATTGGGCAATGAATCAGTGACATTCTACCTCCAGAATTTGTATGTTAACCAAACAGATATTTACTTCTGCAAAATTGAAGTTATGTATCCTCCTCCTTACCTAGACAATGAGAAGAGCAATGGAACCATTATCCATGTGAAAGGGAAACACCTTTGTCCAAGTCCCCTATTTCCCGGACCTTCTAAGCCCTTTTGGGTGCTGGTGGTGGTTGGTGGAGTCCTGGCTTGCTATAGCTTGCTAGTAACAGTGGCCTTTATTATTTTCTGGGTGAGGAGTAAGAGGAGCAGGCTCCTGCACAGTGACTACATGAACATGACTCCCCGCCGCCCCGGGCCCACCCGCAAGCATTACCAGCCCTATGCCCCACCACGCGACTTCGCAGCCTATCGCTCCTGACACGGACGCCTATCCAGAAGCCAGCCGGCTGGCAGCCCCCATCTGCTCAATATCACTGCTCTGGATAGGAAATGACCGCCATCTCCAGCCGGCCACCTCAGGCCCCTGTTGGGCCACCAATGCCAATTTTTCTCGAGTGACTAGACCAAATATCAAGATCATTTTGAGACTCTGAAATGAAGTAAAAGAGATTTCCTGTGACAGGCCAAGTCTTACAGTGCCATGGCCCACATTCCAACTTACCATGTACTTAGTGACTTGACTGAGAAGTTAGGGTAGAAAACAAAAAGGGAGTGGATTCTGGGAGCCTCTTCCCTTTCTCACTCACCTGCACATCTCAGTCAAGCAAAGTGTGGTATCCACAGACATTTTAGTTGCAGAAGAAAGGCTAGGAAATCATTCCTTTTGGTTAAATGGGTGTTTAATCTTTTGGTTAGTGGGTTAAACGGGGTAAGTTAGAGTAGGGGGAGGGATAGGAAGACATATTTAAAAACCATTAAAACACTGTCTCCCACTCATGAAATGAGCCACGTAGTTCCTATTTAATGCTGTTTTCCTTTAGTTTAGAAATACATAGACATTGTCTTTTATGAATTCTGATCATATTTAGTCATTTTGACCAAATGAGGGATTTGGTCAAATGAGGGATTCCCTCAAAGCAATATCAGGTAAACCAAGTTGCTTTCCTCACTCCCTGTCATGAGACTTCAGTGTTAATGTTCACAATATACTTTCGAAAGAATAAAATAGTTCTCCTACATGAAGAAAGAATATGTCAGGAAATAAGGTCACTTTATGTCAAAATTATTTGAGTACTATGGGACCTGGCGCAGTGGCTCATGCTTGTAATCCCAGCACTTTGGGAGGCCGAGGTGGGCAGATCACTTGAGATCAGGACCAGCCTGGTCAAGATGGTGAAACTCCGTCTGTACTAAAAATACAAAATTTAGCTTGGCCTGGTGGCAGGCACCTGTAATCCCAGCTGCCCAAGAGGCTGAGGCATGAGAATCGCTTGAACCTGGCAGGCGGAGGTTGCAGTGAGCCGAGATAGTGCCACAGCTCTCCAGCCTGGGCGACAGAGTGAGACTCCATCTCAAACAACAACAACAACAACAACAACAACAACAAACCACAAAATTATTTGAGTACTGTGAAGGATTATTTGTCTAACAGTTCATTCCAATCAGACCAGGTAGGAGCTTTCCTGTTTCATATGTTTCAGGGTTGCACAGTTGGTCTCTTTAATGTCGGTGTGGAGATCCAAAGTGGGTTGTGGAAAGAGCGTCCATAGGAGAAGTGAGAATACTGTGAAAAAGGGATGTTAGCATTCATTAGAGTATGAGGATGAGTCCCAAGAAGGTTCTTTGGAAGGAGGACGAATAGAATGGAGTAATGAAATTCTTGCCATGTGCTGAGGAGATAGCCAGCATTAGGTGACAATCTTCCAGAAGTGGTCAGGCAGAAGGTGCCCTGGTGAGAGCTCCTTTACAGGGACTTTATGTGGTTTAGGGCTCAGAGCTCCAAAACTCTGGGCTCAGCTGCTCCTGTACCTTGGAGGTCCATTCACATGGGAAAGTATTTTGGAATGTGTCTTTTGAAGAGAGCATCAGAGTTCTTAAGGGACTGGGTAAGGCCTGACCCTGAAATGACCATGGATATTTTTCTACCTACAGTTTGAGTCAACTAGAATATGCCTGGGGACCTTGAAGAATGGCCCTTCAGTGGCCCTCACCATTTGTTCATGCTTCAGTTAATTCAGGTGTTGAAGGAGCTTAGGTTTTAGAGGCACGTAGACTTGGTTCAAGTCTCGTTAGTAGTTGAATAGCCTCAGGCAAGTCACTGCCCACCTAAGATGATGGTTCTTCAACTATAAAATGGAGATAATGGTTACAAATGTCTCTTCCTATAGTATAATCTCCATAAGGGCATGGCCCAAGTCTGTCTTTGACTCTGCCTATCCCTGACATTTAGTAGCATGCCCGACATACAATGTTAGCTATTGGTATTATTGCCATATAGATAAATTATGTATAAAAATTAAACTGGGCAATAGCCTAAGAAGGGGGGAATATTGTAACACAAATTTAAACCCACTACGCAGGGATGAGGTGCTATAATATGAGGACCTTTTAACTTCCATCATTTTCCTGTTTCTTGAAATAGTTTATCTTGTAATGAAATATAAGGCACCTCCCACTTTTATGTATAGAAAGAGGTCTTTTAATTTTTTTTTAATGTGAGAAGGAAGGGAGGAGTAGGAATCTTGAGATTCCAGATCGAAAATACTGTACTTTGGTTGATTTTTAAGTGGGCTTCCATTCCATGGATTTAATCAGTCCCAAGAAGATCAAACTCAGCAGTACTTGGGTGCTGAAGAACTGTTGGATTTACCCTGGCACGTGTGCCACTTGCCAGCTTCTTGGGCACACAGAGTTCTTCAATCCAAGTTATCAGATTGTATTTGAAAATGACAGAGCTGGAGAGTTTTTTGAAATGGCAGTGGCAAATAAATAAATACTTTTTTTTAAATGGAAAGACTTGATCTATGGTAATAAATGATTTTGTTTTCTGACTGGAAAAATAGGCCTACTAAAGATGAATCACACTTGAGATGTTTCTTACTCACTCTGCACAGAAACAAAGAAGAAATGTTATACAGGGAAGTCCGTTTTCACTATTAGTATGAACCAAGAAATGGTTCAAAAACAGTGGTAGGAGCAATGCTTTCATAGTTTCAGATATGGTAGTTATGAAGAAAACAATGTCATTTGCTGCTATTATTGTAAGAGTCTTATAATTAATGGTACTCCTATAATTTTTGATTGTGAGCTCACCTATTTGGGTTAAGCATGCCAATTTAAAGAGACCAAGTGTATGTACATTATGTTCTACATATTCAGTGATAAAATTACTAAACTACTATATGTCTGCTTTAAATTTGTACTTTAATATTGTCTTTTGGTATTAAGAAAGATATGCTTTCAGAATAGATATGCTTCGCTTTGGCAAGGAATTTGGATAGAACTTGCTATTTAAAAGAGGTGTGGGGTAAATCCTTGTATAAATCTCCAGTTTAGCCTTTTTTGAAAAAGCTAGACTTTCAAATACTAATTTCACTTCAAGCAGGGTACGTTTCTGGTTTGTTTGCTTGACTTCAGTCACAATTTCTTATCAGACCAATGGCTGACCTCTTTGAGATGTCAGGCTAGGCTTACCTATGTGTTCTGTGTCATGTGAATGCTGAGAAGTTTGACAGAGATCCAACTTCAGCCTTGACCCCATCAGTCCCTCGGGTTAACTAACTGAGCCACCGGTCCTCATGGCTATTTTAATGAGGGTATTGATGGTTAAATGCATGTCTGATCCCTTATCCCAGCCATTTGCACTGCCAGCTGGGAACTATACCAGACCTGGATACTGATCCCAAAGTGTTAAATTCAACTACATGCTGGAGATTAGAGATGGTGCCAATAAAGGACCCAGAACCAGGATCTTGATTGCTATAGACTTATTAATAATCCAGGTCAAAGAGAGTGACACACACTCTCTCAAGACCTGGGGTGAGGGAGTCTGTGTTATCTGCAAGGCCATTTGAGGCTCAGAAAGTCTCTCTTTCCTATAGATATATGCATACTTTCTGACATATAGGAATGTATCAGGAATACTCAACCATCACAGGCATGTTCCTACCTCAGGGCCTTTACATGTCCTGTTTACTCTGTCTAGAATGTCCTTCTGTAGATGACCTGGCTTGCCTCGTCACCCTTCAGGTCCTTGCTCAAGTGTCATCTTCTCCCCTAGTTAAACTACCCCACACCCTGTCTGCTTTCCTTGCTTATTTTTCTCCATAGCATTTTACCATCTCTTACATTAGACATTTTTCTTATTTATTTGTAGTTTATAAGCTTCATGAGGCAAGTAACTTTGCTTTGTTTCTTGCTGTATCTCCAGTGCCCAGAGCAGTGCCTGGTATATAATAAATATTTATTGACTGAGTGAAAAAAAAAAAAAAAAA(SEQ ID NO:28)
MLRLLLALNLFPSIQVTGNKILVKQSPMLVAYDNAVNLSCKYSYNLFSREFRASLHKGLDSAVEVCVVYGNYSQQLQVYSKTGFNCDGKLGNESVTFYLQNLYVNQTDIYFCKIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKPFWVLVVVGGVLACYSLLVTVAFIIFWVRSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS(SEQ ID NO:29).
人CD30L的代表性核苷酸和氨基酸序列分别如SEQ ID NO:30(登录号L09753)和SEQ ID NO:31中所示:
CCAAGTCACATGATTCAGGATTCAGGGGGAGAATCCTTCTTGGAACAGAGATGGGCCCAGAACTGAATCAGATGAAGAGAGATAAGGTGTGATGTGGGGAAGACTATATAAAGAATGGACCCAGGGCTGCAGCAAGCACTCAACGGAATGGCCCCTCCTGGAGACACAGCCATGCATGTGCCGGCGGGCTCCGTGGCCAGCCACCTGGGGACCACGAGCCGCAGCTATTTCTATTTGACCACAGCCACTCTGGCTCTGTGCCTTGTCTTCACGGTGGCCACTATTATGGTGTTGGTCGTTCAGAGGACGGACTCCATTCCCAACTCACCTGACAACGTCCCCCTCAAAGGAGGAAATTGCTCAGAAGACCTCTTATGTATCCTGAAAAGAGCTCCATTCAAGAAGTCATGGGCCTACCTCCAAGTGGCAAAGCATCTAAACAAAACCAAGTTGTCTTGGAACAAAGATGGCATTCTCCATGGAGTCAGATATCAGGATGGGAATCTGGTGATCCAATTCCCTGGTTTGTACTTCATCATTTGCCAACTGCAGTTTCTTGTACAATGCCCAAATAATTCTGTCGATCTGAAGTTGGAGCTTCTCATCAACAAGCATATCAAAAAACAGGCCCTGGTGACAGTGTGTGAGTCTGGAATGCAAACGAAACACGTATACCAGAATCTCTCTCAATTCTTGCTGGATTACCTGCAGGTCAACACCACCATATCAGTCAATGTGGATACATTCCAGTACATAGATACAAGCACCTTTCCTCTTGAGAATGTGTTGTCCATCTTCTTATACAGTAATTCAGACTGAACAGTTTCTCTTGGCCTTCAGGAAGAAAGCGCCTCTCTACCATACAGTATTTCATCCCTCCAAACACTTGGGCAAAAAGAAAACTTTAGACCAAGACAAACTACACAGGGTATTAAATAGTATACTTCTCCTTCTGTCTCTTGGAAAGATACAGCTCCAGGGTTAAAAAGAGAGTTTTTAGTGAAGTATCTTTCAGATAGCAGGCAGGGAAGCAATGTAGTGTGGTGGGCAGAGCCCCACACAGAATCAGAAGGGATGAATGGATGTCCCAGCCCAACCACTAATTCACTGTATGGTCTTGATCTATTTCTTCTGTTTTGAGAGCCTCCAGTTAAAATGGGGCTTCAGTACCAGAGCAGCTAGCAACTCTGCCCTAATGGGAAATGAAGGGGAGCTGGGTGTGAGTGTTTACACTGTGCCCTTCACGGGATACTTCTTTTATCTGCAGATGGCCTAATGCTTAGTTGTCCAAGTCGCGATCAAGGACTCTCTCACACAGGAAACTTCCCTATACTGGCAGATACACTTGTGACTGAACCATGCCCAGTTTATGCCTGTCTGACTGTCACTCTGGCACTAGGAGGCTGATCTTGTACTCCATATGACCCCACCCCTAGGAACCCCCAGGGAAAACCAGGCTCGGACAGCCCCCTGTTCCTGAGATGGAAAGCACAAATTTAATACACCACCACAATGGAAAACAAGTTCAAAGACTTTTACTTACAGATCCTGGACAGAAAGGGCATAATGAGTCTGAAGGGCAGTCCTCCTTCTCCAGGTTACATGAGGCAGGAATAAGAAGTCAGACAGAGACAGCAAGACAGTTAACAACGTAGGTAAAGAAATAGGGTGTGGTCACTCTCAATTCACTGGCAAATGCCTGAATGGTCTGTCTGAAGGAAGCAACAGAGAAGTGGGGAATCCAGTCTGCTAGGCAGGAAAGATGCCTCTAAGTTCTTGTCTCTGGCCAGAGGTGTGGTATAGAACCAGAAACCCATATCAAGGGTGACTAAGCCCGGCTTCCGGTATGAGAAATTAAACTTGTATACAAAATGGTTGCCAAGGCAACATAAAATTATAAGAATTC(SEQ ID NO:30)
MDPGLQQALNGMAPPGDTAMHVPAGSVASHLGTTSRSYFYLTTATLALCLVFTVATIMVLVVQRTDSIPNSPDNVPLKGGNCSEDLLCILKRAPFKKSWAYLQVAKHLNKTKLSWNKDGILHGVRYQDGNLVIQFPGLYFIICQLQFLVQCPNNSVDLKLELLINKHIKKQALVTVCESGMQTKHVYQNLSQFLLDYLQVNTTISVNVDTFQYIDTSTFPLENVLSIFLYSNSD(SEQ ID NO:31).
人CD40的代表性核苷酸和氨基酸序列分别如SEQ ID NO:32(登录号NM_001250)和SEQ ID NO:33中所示:
TTTCCTGGGCGGGGCCAAGGCTGGGGCAGGGGAGTCAGCAGAGGCCTCGCTCGGGCGCCCAGTGGTCCTGCCGCCTGGTCTCACCTCGCTATGGTTCGTCTGCCTCTGCAGTGCGTCCTCTGGGGCTGCTTGCTGACCGCTGTCCATCCAGAACCACCCACTGCATGCAGAGAAAAACAGTACCTAATAAACAGTCAGTGCTGTTCTTTGTGCCAGCCAGGACAGAAACTGGTGAGTGACTGCACAGAGTTCACTGAAACGGAATGCCTTCCTTGCGGTGAAAGCGAATTCCTAGACACCTGGAACAGAGAGACACACTGCCACCAGCACAAATACTGCGACCCCAACCTAGGGCTTCGGGTCCAGCAGAAGGGCACCTCAGAAACAGACACCATCTGCACCTGTGAAGAAGGCTGGCACTGTACGAGTGAGGCCTGTGAGAGCTGTGTCCTGCACCGCTCATGCTCGCCCGGCTTTGGGGTCAAGCAGATTGCTACAGGGGTTTCTGATACCATCTGCGAGCCCTGCCCAGTCGGCTTCTTCTCCAATGTGTCATCTGCTTTCGAAAAATGTCACCCTTGGACAAGCTGTGAGACCAAAGACCTGGTTGTGCAACAGGCAGGCACAAACAAGACTGATGTTGTCTGTGGTCCCCAGGATCGGCTGAGAGCCCTGGTGGTGATCCCCATCATCTTCGGGATCCTGTTTGCCATCCTCTTGGTGCTGGTCTTTATCAAAAAGGTGGCCAAGAAGCCAACCAATAAGGCCCCCCACCCCAAGCAGGAACCCCAGGAGATCAATTTTCCCGACGATCTTCCTGGCTCCAACACTGCTGCTCCAGTGCAGGAGACTTTACATGGATGCCAACCGGTCACCCAGGAGGATGGCAAAGAGAGTCGCATCTCAGTGCAGGAGAGACAGTGAGGCTGCACCCACCCAGGAGTGTGGCCACGTGGGCAAACAGGCAGTTGGCCAGAGAGCCTGGTGCTGCTGCTGCTGTGGCGTGAGGGTGAGGGGCTGGCACTGACTGGGCATAGCTCCCCGCTTCTGCCTGCACCCCTGCAGTTTGAGACAGGAGACCTGGCACTGGATGCAGAAACAGTTCACCTTGAAGAACCTCTCACTTCACCCTGGAGCCCATCCAGTCTCCCAACTTGTATTAAAGACAGAGGCAGAAGTTTGGTGGTGGTGGTGTTGGGGTATGGTTTAGTAATATCCACCAGACCTTCCGATCCAGCAGTTTGGTGCCCAGAGAGGCATCATGGTGGCTTCCCTGCGCCCAGGAAGCCATATACACAGATGCCCATTGCAGCATTGTTTGTGATAGTGAACAACTGGAAGCTGCTTAACTGTCCATCAGCAGGAGACTGGCTAAATAAAATTAGAATATATTTATACAACAGAATCTCAAAAACACTGTTGAGTAAGGAAAAAAAGGCATGCTGCTGAATGATGGGTATGGAACTTTTTAAAAAAGTACATGCTTTTATGTATGTATATTGCCTATGGATATATGTATAAATACAATATGCATCATATATTGATATAACAAGGGTTCTGGAAGGGTACACAGAAAACCCACAGCTCGAAGAGTGGTGACGTCTGGGGTGGGGAAGAAGGGTCTGGGGG(SEQ ID NO:32)
MVRLPLQCVLWGCLLTAVHPEPPTACREKQYLINSQCCSLCQPGQKLVSDCTEFTETECLPCGESEFLDTWNRETHCHQHKYCDPNLGLRVQQKGTSETDTICTCEEGWHCTSEACESCVLHRSCSPGFGVKQIATGVSDTICEPCPVGFFSNVSSAFEKCHPWTSCETKDLVVQQAGTNKTDVVCGPQDRLRALVVIPIIFGILFAILLVLVFIKKVAKKPTNKAPHPKQEPQEINFPDDLPGSNTAAPVQETLHGCQPVTQEDGKESRISVQERQ(SEQ ID NO:33).
人CD70的代表性核苷酸和氨基酸序列分别如SEQ ID NO:34(登录号NM_001252)和SEQ ID NO:35中所示:
CCAGAGAGGGGCAGGCTGGTCCCCTGACAGGTTGAAGCAAGTAGACGCCCAGGAGCCCCGGGAGGGGGCTGCAGTTTCCTTCCTTCCTTCTCGGCAGCGCTCCGCGCCCCCATCGCCCCTCCTGCGCTAGCGGAGGTGATCGCCGCGGCGATGCCGGAGGAGGGTTCGGGCTGCTCGGTGCGGCGCAGGCCCTATGGGTGCGTCCTGCGGGCTGCTTTGGTCCCATTGGTCGCGGGCTTGGTGATCTGCCTCGTGGTGTGCATCCAGCGCTTCGCACAGGCTCAGCAGCAGCTGCCGCTCGAGTCACTTGGGTGGGACGTAGCTGAGCTGCAGCTGAATCACACAGGACCTCAGCAGGACCCCAGGCTATACTGGCAGGGGGGCCCAGCACTGGGCCGCTCCTTCCTGCATGGACCAGAGCTGGACAAGGGGCAGCTACGTATCCATCGTGATGGCATCTACATGGTACACATCCAGGTGACGCTGGCCATCTGCTCCTCCACGACGGCCTCCAGGCACCACCCCACCACCCTGGCCGTGGGAATCTGCTCTCCCGCCTCCCGTAGCATCAGCCTGCTGCGTCTCAGCTTCCACCAAGGTTGTACCATTGCCTCCCAGCGCCTGACGCCCCTGGCCCGAGGGGACACACTCTGCACCAACCTCACTGGGACACTTTTGCCTTCCCGAAACACTGATGAGACCTTCTTTGGAGTGCAGTGGGTGCGCCCCTGACCACTGCTGCTGATTAGGGTTTTTTAAATTTTATTTTATTTTATTTAAGTTCAAGAGAAAAAGTGTACACACAGGGGCCACCCGGGGTTGGGGTGGGAGTGTGGTGGGGGGTAGTGGTGGCAGGACAAGAGAAGGCATTGAGCTTTTTCTTTCATTTTCCTATTAAAAAATACAAAAATCA(SEQ ID NO:34)
MPEEGSGCSVRRRPYGCVLRAALVPLVAGLVICLVVCIQRFAQAQQQLPLESLGWDVAELQLNHTGPQQDPRLYWQGGPALGRSFLHGPELDKGQLRIHRDGIYMVHIQVTLAICSSTTASRHHPTTLAVGICSPASRSISLLRLSFHQGCTIASQRLTPLARGDTLCTNLTGTLLPSRNTDETFFGVQWVRP(SEQ ID NO:35).
人LIGHT的代表性核苷酸和氨基酸序列分别如SEQ ID NO:36(登录号CR541854)和SEQ ID NO:37中所示:
ATGGAGGAGAGTGTCGTACGGCCCTCAGTGTTTGTGGTGGATGGACAGACCGACATCCCATTCACGAGGCTGGGACGAAGCCACCGGAGACAGTCGTGCAGTGTGGCCCGGGTGGGTCTGGGTCTCTTGCTGTTGCTGATGGGGGCCGGGCTGGCCGTCCAAGGCTGGTTCCTCCTGCAGCTGCACTGGCGTCTAGGAGAGATGGTCACCCGCCTGCCTGACGGACCTGCAGGCTCCTGGGAGCAGCTGATACAAGAGCGAAGGTCTCACGAGGTCAACCCAGCAGCGCATCTCACAGGGGCCAACTCCAGCTTGACCGGCAGCGGGGGGCCGCTGTTATGGGAGACTCAGCTGGGCCTGGCCTTCCTGAGGGGCCTCAGCTACCACGATGGGGCCCTTGTGGTCACCAAAGCTGGCTACTACTACATCTACTCCAAGGTGCAGCTGGGCGGTGTGGGCTGCCCGCTGGGCCTGGCCAGCACCATCACCCACGGCCTCTACAAGCGCACACCCCGCTACCCCGAGGAGCTGGAGCTGTTGGTCAGCCAGCAGTCACCCTGCGGACGGGCCACCAGCAGCTCCCGGGTCTGGTGGGACAGCAGCTTCCTGGGTGGTGTGGTACACCTGGAGGCTGGGGAGGAGGTGGTCGTCCGTGTGCTGGATGAACGCCTGGTTCGACTGCGTGATGGTACCCGGTCTTACTTCGGGGCTTTCATGGTGTGA(SEQ ID NO:36)
MEESVVRPSVFVVDGQTDIPFTRLGRSHRRQSCSVARVGLGLLLLLMGAGLAVQGWFLLQLHWRLGEMVTRLPDGPAGSWEQLIQERRSHEVNPAAHLTGANSSLTGSGGPLLWETQLGLAFLRGLSYHDGALVVTKAGYYYIYSKVQLGGVGCPLGLASTITHGLYKRTPRYPEELELLVSQQSPCGRATSSSRVWWDSSFLGGVVHLEAGEEVVVRVLDERLVRLRDGTRSYFGAFMV(SEQ ID NO:37).
在多个实施方案中,本发明提供了包含本文所述的任意序列的变体,例如,与本文公开的任意序列(例如,SEQ ID NOS:1-13和26-37)具有以下序列同一性的序列:至少约60%,或至少约61%,或至少约62%,或至少约63%,或至少约64%,或至少约65%,或至少约66%,或至少约67%,或至少约68%,或至少约69%,或至少约70%,或至少约71%,或至少约72%,或至少约73%,或至少约74%,或至少约75%,或至少约76%,或至少约77%,或至少约78%,或至少约79%,或至少约80%,或至少约81%,或至少约82%,或至少约83%,或至少约84%,或至少约85%,或至少约86%,或至少约87%,或至少约88%,或至少约89%,或至少约90%,或至少约91%,或至少约92%,或至少约93%,或至少约94%,或至少约95%,或至少约96%,或至少约97%,或至少约98%,或至少约99%的序列同一性。
在多个实施方案中,本发明提供了相对于本文所述的任意蛋白序列具有一个或多个氨基酸突变的氨基酸序列。在一些实施方案中,一个或多个氨基酸突变可独立地选自本文所述的保守性或非保守性取代、插入、缺失和截短。
检查点阻断/肿瘤免疫抑制的阻断
某些人类肿瘤可由患者的免疫系统消除。例如,施用靶向免疫“检查点”分子的单克隆抗体可导致完全应答和肿瘤缓解。此类抗体的作用方式是通过抑制已被肿瘤选为保护其免受抗肿瘤免疫应答的免疫调节分子。通过抑制这些“检查点”分子(例如,使用拮抗性抗体),可以使患者的CD8+T细胞增殖并破坏肿瘤细胞。例如,施用靶向例如但不限于CTLA-4或PD-1的单克隆抗体可导致完全应答和肿瘤缓解。此类抗体的作用方式是通过抑制已被肿瘤选为保护其免受抗肿瘤免疫应答的CTLA-4或PD-1。通过抑制这些“检查点”分子(例如,使用拮抗性抗体),可以使患者的CD8+T细胞增殖并破坏肿瘤细胞。
因此,本文提供的基于细胞的治疗剂(therapy)可以与靶向免疫“检查点”分子的一种或多种阻断抗体组合使用。例如,在一些实施方案中,本文提供的基于细胞的治疗剂可以与靶向例如CTLA-4或PD-1的分子的一种或多种阻断抗体组合使用。例如,本文提供的基于细胞的治疗剂可以与阻断、减少和/或抑制PD-1和PD-L1或PD-L2和/或PD-1与PD-L1或PD-L2结合的药剂组合使用(作为非限制性实例,所述药剂为以下的一种或多种:纳武单抗(ONO-4538/BMS-936558,MDX1106,OPDIVO,BRISTOL MYERS SQUIBB)、派姆单抗(KEYTRUDA,Merck)、匹地利珠单抗(CT-011,CURE TECH)、MK-3475(MERCK)、BMS 936559(BRISTOL MYERSSQUIBB)、MPDL328OA(ROCHE))。在一个实施方案中,本文提供的基于细胞的治疗剂可以与阻断、降低和/或抑制CTLA-4活性和/或CTLA-4与一种或多种受体(例如CD80、CD86、AP2M1、SHP-2和PPP2R5A)结合的药剂组合使用。例如,在一些实施方案中,免疫调节剂是抗体,例如但不限于伊匹木单抗(ipilimumab)(MDI-010,MDX-101,Yervoy,BMS)和/或曲美木单抗(tremelimumab)(Pfizer)。针对这些分子的阻断抗体可以获得自例如Bristol MyersSquibb(纽约,NY)、Merck(Kenilworth,NJ)、MedImmune(Gaithersburg,MD)和Pfizer(纽约,NY)。
此外,本文提供的基于细胞的治疗剂可以与靶向免疫“检查点”分子的一种或多种阻断抗体组合使用,所述免疫“检查点”分子例如BTLA、HVEM、TIM3、GAL9、LAG3、VISTA、KIR、2B4、CD160(也称为BY55)、CGEN-15049、CHK 1和CHK2激酶、A2aR、CEACAM(例如CEACAM-1、CEACAM-3和/或CEACAM-5)、GITR、GITRL、半乳糖凝集素-9(galectin-9)、CD244、CD160、TIGIT、SIRPα、ICOS、CD172a和TMIGD2以及各种B-7家族配体(包括但不限于B7-1、B7-2、B7-DC、B7-H1、B7-H2、B7-H3、B7-H4、B7-H5、B7-H6和B7-H7)。
基于细胞的治疗剂
本公开提供了基于细胞的治疗剂,其包含第一细胞,所述第一细胞包含含有编码可分泌型疫苗蛋白(例如,gp96-Ig融合蛋白)的核苷酸序列的表达载体,和第二细胞,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,所述T细胞共刺激融合蛋白例如OX40L-Ig或其与OX40特异性结合的部分、ICOSL-Ig或其与ICOS特异性结合的部分、4-1BBL-Ig或其与4-1BBR特异性结合的部分、CD40L-Ig或其与CD40特异性结合的部分、CD70-Ig或其与CD27特异性结合的部分、TL1A-Ig或其与TNFRSF25特异性结合的部分或GITRL-Ig或其与GITR特异性结合的部分。另外,本公开提供了制备本文所述的基于细胞的治疗剂的方法,以及施用基于细胞的治疗剂的方法。通常,本文提供的方法包括向患者施用有效量的第一细胞,所述第一细胞包含含有编码可分泌型疫苗蛋白的核苷酸序列的表达载体,其中所述患者正在经历使用第二细胞的治疗,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,并且其中当施用给受试者时,T细胞共刺激融合蛋白增强抗原特异性T细胞的激活。
在一些实施方案中,本文提供的方法包括向患者施用有效量的第二细胞,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,其中当施用给受试者时,T细胞共刺激融合蛋白增强抗原特异性T细胞的激活,且其中患者正在经历使用第一细胞的治疗,所述第一细胞包含含有编码可分泌型疫苗蛋白的核苷酸序列的表达载体。
在一些实施方案中,本文提供的方法包括向患者施用有效量的第一细胞,所述第一细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,和第二细胞,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,且其中当施用给受试者时,T细胞共刺激融合蛋白增强抗原特异性T细胞的激活。
在一些实施方案中,可以产生基于gp96-Ig的疫苗以刺激针对由猿猴免疫缺陷病毒、人免疫缺陷病毒、丙型肝炎病毒和疟疾表达的个体抗原的抗原特异性免疫应答。通过T细胞共刺激融合蛋白的基于细胞的治疗剂和gp96-Ig的基于细胞的治疗剂,可以增强对这些疫苗的免疫应答。
可以使用常规DNA克隆和诱变方法、DNA扩增方法和/或合成方法,获得(和修饰,如需要)编码疫苗蛋白融合蛋白(例如,gp96-Ig融合蛋白)和T细胞共刺激融合蛋白的cDNA或DNA序列。通常,可以将编码疫苗蛋白融合蛋白(例如,gp96-Ig融合蛋白)和/或T细胞共刺激融合蛋白的序列在表达之前插入克隆载体中以用于遗传修饰和复制目的。每个编码序列可以有效连接至调控元件,例如启动子,以在合适的宿主细胞中体外和体内表达编码的蛋白。
可以施用基于细胞的治疗剂以产生可分泌型疫苗蛋白(例如,gp96-Ig)和T细胞共刺激融合蛋白。例如,可以对细胞进行体外培养或基因工程化。宿主细胞可以从正常或患病的受试者中获得,包括健康人、癌症患者和患有传染病的患者、私人实验室存储、公共培养物保藏机构(例如American Type Culture Collection)或商业供应商。可用于体内产生和分泌gp96-Ig融合蛋白和T细胞共刺激融合蛋白的细胞包括但不限于上皮细胞、内皮细胞、角质形成细胞、成纤维细胞、肌肉细胞、肝细胞;血细胞例如T淋巴细胞、B淋巴细胞、单核细胞、巨噬细胞、嗜中性粒细胞、嗜酸性粒细胞、巨核细胞或粒细胞、各种干细胞或祖细胞例如造血干细胞或祖细胞(例如,从骨髓获得)、脐带血、外周血、胎肝等,以及肿瘤细胞(例如人肿瘤细胞)。细胞类型的选择取决于所治疗或预防的肿瘤或传染性疾病的类型,并且可以由本领域技术人员确定。
不同的宿主细胞具有特征性的具体机制用于蛋白的翻译后加工和修饰。可选择宿主细胞,其以类似于接受者加工其热休克蛋白(hsp)的特定方式来修饰和加工所表达的基因产物。为了产生大量的gp96-Ig,可优选的是宿主细胞的类型已用于表达异源基因,并且因此被很好地表征和开发用于大规模生产过程。在一些实施方案中,宿主细胞对于随后向其施用本发明的融合蛋白或分泌本发明的融合蛋白的重组细胞的患者是自体的。
在一些实施方案中,可以将本文提供的基于细胞的治疗剂引入到抗原性细胞中。如本文所用,抗原性细胞可以包括被致癌传染剂例如病毒感染但尚未形成肿瘤的前肿瘤细胞,或已经暴露于诱变剂或致癌剂例如DNA破坏剂或辐射的抗原性细胞。可以使用的其他细胞是处于从正常到赘生物形式的转换(以形态或生理或生化功能为表征)中的前肿瘤细胞。
通常,本文提供的方法中使用的癌细胞和前肿瘤细胞是哺乳动物来源的。所考虑的哺乳动物包括人、陪伴动物(例如狗和猫)、家畜动物(例如绵羊、牛、山羊、猪和马)、实验动物(例如小鼠、大鼠和兔子)和圈养的或自由的野生动物。
在一些实施方案中,癌细胞(例如人肿瘤细胞)可以用于本文所述的方法中。癌细胞提供成为与所表达的gp96-Ig融合蛋白非共价结合的抗原性肽。也可以使用衍生自肿瘤前病变、癌组织或癌细胞的细胞系,前提是所述细胞系的细胞具有与靶癌细胞上的抗原相同的至少一个或多个抗原决定簇。可以使用癌组织、癌细胞、被致癌剂感染的细胞、其他前肿瘤细胞和人来源的细胞系。从最终要向其施用融合蛋白的患者中切除的癌细胞可以尤其有用,尽管也可以使用同种异基因细胞。在一些实施方案中,癌细胞可以来自已建立的肿瘤细胞系,例如但不限于已建立的非小细胞肺癌(NSCLC)、膀胱癌、黑色素瘤、卵巢癌、肾细胞癌、前列腺癌、肉瘤、乳腺癌、鳞状细胞癌、头颈癌、肝细胞癌、胰腺癌或结肠癌细胞系。
在多个实施方案中,本发明的融合蛋白允许共刺激T细胞和多种肿瘤细胞抗原的呈递。例如,在一些实施方案中,本发明的疫苗蛋白融合蛋白(例如gp96融合蛋白)伴护多种这些肿瘤抗原。在多个实施方案中,肿瘤细胞分泌多种抗原。可被分泌的示例性而非限制性抗原是:ACRBP、ACTL8、ADAM2、ADAM29、AKAP3、AKAP4、ANKRD45、ARMC3、ARX、ATAD2、BAGE、BAGE2、BAGE3、BAGE4、BAGE5、BRDT、C15ORF60、C21ORF99、CABYR、CAGE1、CALR3、CASC5、CCDC110、CCDC33、CCDC36、CCDC62、CCDC83、CDCA1、CEP290、CEP55、COX6B2、CPXCR1、CRISP2、CSAG1、CSAG2、CSAG3B、CT16.2、CT45A1、CT45A2、CT45A3、CT45A4、CT45A5、CT45A6、CT47A1、CT47A10、CT47A11、CT47A2、CT47A3、CT47A4、CT47A5、CT47A6、CT47A7、CT47A8、CT47A9、CT47B1、CT66、AA884595、CT69、BC040308、CT70、BI818097、CTAG1A、CTAG1B、CTAG2、CTAGE-2、CTAGE1、CTAGE5、CTCFL、CTNNA2、CXORF48、CXORF61、CYCLIN A1、DCAF12、DDX43、DDX53、DKKL1、DMRT1、DNAJB8、DPPA2、DSCR8、EDAG、NDR、ELOVL4、FAM133A、FAM46D、FATE1、FBXO39、FMR1NB、FTHL17、GAGE1、GAGE12B、GAGE12C、GAGE12D、GAGE12E、GAGE12F、GAGE12G、GAGE12H、GAGE12I、GAGE12J、GAGE13、GAGE2A、GAGE3、GAGE4、GAGE5、GAGE6、GAGE7、GAGE8、GOLGAGL2FA、GPAT2、GPATCH2、HIWI、MIWI、PIWI、HORMAD1、HORMAD2、HSPB9、IGSF11、IL13RA2、IMP-3、JARID1B、KIAA0100、LAGE-1B、LDHC、LEMD1、LIPI、LOC130576、LOC196993、LOC348120、LOC440934、LOC647107、LOC728137、LUZP4、LY6K、MAEL、MAGEA1、MAGEA10、MAGEA11、MAGEA12、MAGEA2、MAGEA2B、MAGEA3、MAGEA4、MAGEA5、MAGEA6、MAGEA8、MAGEA9、MAGEA9B、LOC728269、MAGEB1、MAGEB2、MAGEB3、MAGEB4、MAGEB5、MAGEB6、MAGEC1、MAGEC2、MAGEC3、MCAK、MMA1B、MORC1、MPHOSPH1、NLRP4、NOL4、NR6A1、NXF2、NXF2B、NY-ESO-1、ODF1、ODF2、ODF3、ODF4、OIP5、OTOA、PAGE1、PAGE2、PAGE2B、PAGE3、PAGE4、PAGE5、PASD1、PBK、PEPP2、PIWIL2、PLAC1、POTEA、POTEB、POTEC、POTED、POTEE、POTEG、POTEH、PRAME、PRM1、PRM2、PRSS54、PRSS55、PTPN20A、RBM46、RGS22、ROPN1、RQCD1、SAGE1、SEMG1、SLCO6A1、SPA17、SPACA3、SPAG1、SPAG17、SPAG4、SPAG6、SPAG8、SPAG9、SPANXA1、SPANXA2、SPANXB1、SPANXB2、SPANXC、SPANXD、SPANXE、SPANXN1、SPANXN2、SPANXN3、SPANXN4、SPANXN5、SPATA19、SPEF2、SPINLW1、SPO11、SSX1、SSX2、SSX2B、SSX3、SSX4、SSX4B、SSX5、SSX6、SSX7、SSX9、SYCE1、SYCP1、TAF7L、TAG、TDRD1、TDRD4、TDRD6、TEKT5、TEX101、TEX14、TEX15、TFDP3、THEG、TMEFF1、TMEFF2、TMEM108、TMPRSS12、TPPP2、TPTE、TSGA10、TSP50、TSPY1D、TSPY1E、TSPY1F、TSPY1G、TSPY1H、TSPY1I、TSPY2、TSPY3、TSSK6、TTK、TULP2、VENTXP1、XAGE-3B、XAGE-4、RP11-167P23.2、XAGE1、XAGE1B、XAGE1C、XAGE1D、XAGE1E、XAGE2、XAGE2B、CTD-2267G17.3、XAGE3、XAGE5、ZNF165、ZNF645、MART-1/Melan-A、gp100、二肽基肽酶IV(DPPIV)、腺苷脱氨酶结合蛋白(ADAbp)、亲环蛋白b、结直肠相关抗原(CRC)-0017-1A/GA733、癌胚抗原(CEA)及其免疫原性表位CAP-1和CAP-2、etv6、aml1、前列腺特异性抗原(PSA)及其免疫原性表位PSA-1、PSA-2和PSA-3、前列腺特异性膜抗原(PSMA)、T细胞受体/CD3-ζ链、RAGE、NAG、GnT-V、MUM-1、CDK4、酪氨酸酶、p53、MUC家族、HER2/neu、p21ras、RCAS1、甲胎蛋白、E-钙黏着蛋白、α-连环蛋白(α-catenin)、β-连环蛋白(β-catenin)和γ-连环蛋白(γ-catenin)、p120ctn、gp100Pmel117、cdc27、腺瘤性结肠息肉蛋白(adenomatous polyposis coli protein,APC)、胞衬蛋白(fodrin)、连接蛋白37(Connexin 37)、Ig独特型、p15、gp75、GM2和GD2神经节苷脂、病毒产物例如人乳头瘤病毒蛋白、Smad家族的肿瘤抗原、lmp-1、NA、EBV编码的核抗原(EBNA)-1、脑糖原磷酸化酶、SCP-1CT-7、c-erbB-2、CD19、CD20、CD22、CD30、CD33、CD37、CD56、CD70、CD74、CD138、AGS16、MUC1、GPNMB、Ep-CAM、PD-L1、PD-L2、PMSA。在一些实施方案中,抗原是人内源性逆转录病毒抗原。示例性抗原还可以包括来自人内源性逆转录病毒的抗原,其包括但不限于衍生自Gag的至少一部分、Tat的至少一部分、Rev的至少一部分、Nef的至少一部分和gp160的至少一部分的表位。
此外,在一些实施方案中,本发明的疫苗蛋白融合蛋白(例如,gp96融合蛋白)提供佐剂作用,其在用于本文所述的各种方法中时进一步允许患者的免疫系统针对目标疾病被激活。
原核和真核载体均可用于在本文提供的基于细胞的治疗方法中表达疫苗蛋白(例如,gp96-Ig)和T细胞共刺激融合蛋白。原核载体包括基于大肠杆菌序列的构建体(参见,例如,Makrides,Microbiol Rev 1996,60:512-538)。可用于在大肠杆菌中表达的调节区的非限制性实例包括lac、trp、lpp、phoA、recA、tac、T3、T7和λPL。原核表达载体的非限制性实例可包括λgt载体系列例如λgt11(Huynh et al.,in“DNACloning Techniques,Vol.I:APractical Approach,”1984,(D.Glover,ed.),pp.49-78,IRL Press,Oxford),以及pET载体系列(Studier et al.,Methods Enzymol 1990,185:60-89)。然而,原核宿主-载体系统不能进行哺乳动物细胞的许多翻译后加工。因此,真核宿主-载体系统可以尤其有用。
多种调控区可用于在哺乳动物宿主细胞中表达疫苗蛋白(例如,gp96-Ig)和T细胞共刺激融合蛋白。例如,可以使用SV40早期和晚期启动子、巨细胞病毒(CMV)即早期启动子和劳斯肉瘤病毒长末端重复(RSV-LTR)启动子。可用于哺乳动物细胞的诱导型启动子包括但不限于与以下相关的启动子:金属硫蛋白II基因、小鼠乳腺肿瘤病毒糖皮质激素响应性长末端重复序列(MMTV-LTR)、β-干扰素基因和hsp70基因(参见Williams et al.,CancerRes 1989,49:2735-42;and Taylor et al.,Mol Cell Biol 1990,10:165-75)。热休克启动子或应激启动子对于驱动融合蛋白在重组宿主细胞中的表达也可以是有利的。
在一些实施方案中,本发明包括使用能够响应于信号而产生高水平瞬时表达的诱导型启动子。示例性的诱导型表达控制区包括那些包含由信号(例如小分子化合物)刺激的诱导型启动子的诱导型表达控制区。具体实例可以在例如美国专利号5,989,910、5,935,934、6,015,709和6,004,941中找到,其每一个均通过引用以其全文并入本文。
表现组织特异性并且已用于转基因动物中的动物调节区也可以用于特定组织类型的肿瘤细胞:在胰腺腺泡细胞中有活性的弹性蛋白酶I基因控制区(Swift et al.,Cell1984,38:639-646;Ornitz et al.,Cold Spring Harbor Symp Quant Biol 1986,50:399-409;和MacDonald,Hepatology 1987,7:425-515);在胰岛β细胞中有活性的胰岛素基因控制区(Hanahan,Nature 1985,315:115-122),在淋巴样细胞中有活性的免疫球蛋白基因控制区(Grosschedl et al.,Cell 1984,38:647-658;Adames et al.,Nature 1985,318:533-538;和Alexander et al.,Mol Cell Biol 1987,7:1436-1444),在睾丸、乳腺、淋巴和肥大细胞中有活性的小鼠乳腺肿瘤病毒控制区(Leder et al.,Cell 1986,45:485-495),在肝中有活性的白蛋白基因控制区(Pinkert et al.,Genes Devel,1987,1:268-276),在肝中有活性的甲胎蛋白基因控制区(Krumlauf et al.,Mol Cell Biol 1985,5:1639-1648;and Hammer et al.,Science 1987,235:53-58);在肝中有活性的α1-抗胰蛋白酶基因控制区(Kelsey et al.,Genes Devel 1987,1:161-171),在髓样细胞中有活性的β-珠蛋白基因控制区(Mogram et al.,Nature 1985,315:338-340;and Kollias et al.,Cell1986,46:89-94);在大脑的少突胶质细胞中有活性的髓磷脂碱性蛋白基因控制区(Readhead et al.,Cell 1987,48:703-712);在骨骼肌中有活性的肌球蛋白轻链-2基因控制区(Sani,Nature 1985,314:283-286)和在下丘脑中有活性的促性腺激素释放激素基因控制区(Mason et al.,Science 1986,234:1372-1378)。
表达载体还可以包含转录增强子元件,例如在SV40病毒、乙型肝炎病毒、巨细胞病毒、免疫球蛋白基因、金属硫蛋白和β-肌动蛋白中发现的那些(参见,Bittner et al.,MethEnzymol 1987,153:516-544;和Gorman,Curr Op Biotechnol 1990,1:36-47)。另外,表达载体可以含有允许载体在一种以上类型的宿主细胞中保持和复制,或允许载体整合到宿主染色体中的序列。这样的序列包括但不限于复制起点、自主复制序列(ARS)、着丝粒DNA和端粒DNA。
另外,表达载体可以含有一个或多个可选择的或可筛选的标记基因,用于初始分离、鉴定或追踪含有编码本文所述的融合蛋白的DNA的宿主细胞。对于gp96-Ig和T细胞共刺激融合蛋白的长期高产量生产,在哺乳动物细胞中的稳定表达可以是有用的。许多选择系统可用于哺乳动物细胞。例如,单纯疱疹病毒胸苷激酶(Wigler et al.,Cell 1977,11:223)、次黄嘌呤-鸟嘌呤磷酸核糖基转移酶(Szybalski and Szybalski,Proc Natl AcadSci USA 1962,48:2026)和腺嘌呤磷酸核糖基转移酶(Lowy et al.,Cell 1980,22:817)基因可分别用于tk-、hgprt-或aprt-细胞。另外,抗代谢物抗性可用作选择二氢叶酸还原酶(dhfr)的基础,其赋予对甲氨蝶呤的抗性(Wigler et al.,Proc Natl Acad Sci USA1980,77:3567;O’Hare et al.,Proc Natl Acad Sci USA 1981,78:1527);gpt,其赋予对麦考酚酸(mycophenolic acid)的抗性(Mulligan和Berg,Proc Natl Acad Sci USA 1981,78:2072);新霉素磷酸转移酶(neo),其赋予对氨基糖苷G-418的抗性(Colberre-Garapinet al.,J Mol Biol 1981,150:1);和潮霉素磷酸转移酶(hyg),其赋予对潮霉素的抗性(Santerre et al.,Gene 1984,30:147)。也可以使用其他可选择的标记,例如组氨醇和Zeocin
有用的哺乳动物宿主细胞包括但不限于衍生自人、猴子和啮齿动物的细胞(参见,例如,Kriegler in“Gene Transfer and Expression:A Laboratory Manual,”1990,NewYork,Freeman&Co.)。这些包括通过SV40转化的猴肾细胞系(例如COS-7、ATCC CRL 1651);人胚胎肾细胞系(例如293、293-EBNA或经亚克隆以在悬浮培养中生长的293细胞,Grahamet al.,J Gen Virol 1977,36:59);幼仓鼠肾细胞(例如BHK、ATCC CCL 10);中国仓鼠卵巢细胞-DHFR(例如CHO、Urlaub和Chasin,Proc Natl Acad Sci USA 1980,77:4216);小鼠塞尔托利氏细胞(sertoli cell)(Mather,Biol Reprod 1980,23:243-251);小鼠成纤维细胞(例如NIH-3T3)、猴肾细胞(例如CV1 ATCC CCL 70);非洲绿猴肾细胞(例如VERO-76、ATCCCRL-1587);人宫颈癌细胞(例如HELA、ATCC CCL 2);犬肾细胞(例如MDCK、ATCC CCL34);水牛大鼠肝细胞(例如BRL 3A、ATCC CRL 1442);人肺细胞(例如W138、ATCC CCL 75);人肝细胞(例如Hep G2、HB 8065);和小鼠乳腺肿瘤细胞(例如MMT 060562、ATCC CCL51)。用于表达本文所述的融合蛋白的示例性癌细胞类型包括小鼠成纤维细胞系、NIH3T3、小鼠Lewis肺癌细胞系、LLC、小鼠肥大细胞瘤细胞系、P815、小鼠淋巴瘤细胞系、EL4及其卵白蛋白转染子、E.G7、小鼠黑色素瘤细胞系、B16F10、小鼠纤维肉瘤细胞系、MC57、人小细胞肺癌细胞系、SCLC#2和SCLC#7、人肺腺癌细胞系例如AD100,和人前列腺癌细胞系例如PC-3。
许多基于病毒的表达系统也可以与哺乳动物细胞一起使用,以生产gp96-Ig和T细胞共刺激融合蛋白。使用DNA病毒主链的载体已衍生自猿猴病毒40(SV40)(Hamer et al.,Cell 1979,17:725)、腺病毒(Van Doren et al.,Mol Cell Biol 1984,4:1653)、腺相关病毒(McLaughlin et al.,J Virol 1988,62:1963)和牛乳头瘤病毒(Zinn et al.,ProcNatl Acad Sci USA 1982,79:4897)。当腺病毒用作表达载体时,可将供体DNA序列连接至腺病毒转录/翻译控制复合体,例如晚期启动子和三联体前导序列(tripartite leadersequence)。然后可以通过体外或体内重组将这个融合基因插入腺病毒基因组中。插入病毒基因组的非关键区域(例如,区域E1或E3)可以产生在被感染的宿主中存活并能够表达异源产物的重组病毒(参见,例如,Logan and Shenk,Proc Natl Acad Sci USA 1984,81:3655-3659)。
牛乳头瘤病毒(BPV)可以感染许多高等脊椎动物,包括人类,并且其DNA以附加体(episome)复制。已经开发出许多用于重组基因表达的穿梭载体,其在哺乳动物细胞中以稳定的多拷贝(20-300个拷贝/细胞)染色体外元件存在。通常,这些载体含有BPV DNA(整个基因组或69%的转化片段)的区段、具有广泛宿主范围的启动子、聚腺苷酸化信号、剪接信号、可选择标记和“无毒”质粒序列,其允许载体在大肠杆菌中增殖。在细菌中构建和扩增后,通过例如磷酸钙共沉淀将表达基因构建体转染到培养的哺乳动物细胞中。对于那些不显现转化表型的宿主细胞,通过使用显性可选择标记(dominant selectable marker),例如组氨醇和G418抗性来进行转化子的选择。
可选地,可以使用牛痘7.5K启动子。(参见,例如,Mackett et al.,Proc NatlAcad Sci USA 1982,79:7415-7419;Mackett et al.,J Virol 1984,49:857-864;和Panicali et al.,Proc Natl Acad Sci USA 1982,79:4927-4931.)在使用人宿主细胞的情况下,可以使用基于爱泼斯坦-巴尔病毒(EBV)起源(OriP)和EBV核抗原1(EBNA-1;反式作用复制因子)的载体。这样的载体可以与广范围的人宿主细胞一起使用,例如EBO-pCD(Spickofsky et al.,DNA Prot Eng Tech 1990,2:14-18);pDR2和λDR2(可从ClontechLaboratories获得)。
Gp96-Ig和T细胞共刺激融合蛋白也可以用基于逆转录病毒的表达系统制备。可以使用逆转录病毒,例如莫洛尼氏鼠白血病病毒,因为可以将大多数病毒基因序列移除并用外源编码序列替代,而缺失的病毒功能可以以反式提供。与转染相比,逆转录病毒可以有效地感染基因并将其转移到广范围的细胞类型中,包括例如原代造血细胞。此外,可以通过选择用于载体包装的包膜,来控制逆转录病毒载体感染的宿主范围。
例如,逆转录病毒载体可包含5’长末端重复(LTR)、3’LTR、包装信号、细菌复制起点和可选择标记。例如,可以将gp96-Ig融合蛋白编码序列插入5’LTR和3’LTR之间的位置,使得从5’LTR启动子的转录能转录克隆的DNA。5’LTR依次含有启动子(例如LTR启动子)、R区域、U5区域和引物结合位点。这些LTR元件的核苷酸序列是本领域公知的。异源启动子以及多种药物选择标记也可以包括在表达载体中,以促进选择经感染的细胞。参见,McLauchlinet al.,Prog Nucleic Acid Res Mol Biol 1990,38:91-135;Morgenstern et al.,Nucleic Acid Res 1990,18:3587-3596;Choulika et al.,J Virol 1996,70:1792-1798;Boesen et al.,Biotherapy 1994,6:291-302;Salmons and Gunzberg,Human Gene Ther1993,4:129-141;和Grossman and Wilson,Curr Opin Genet Devel 1993,3:110-114。
可以使用本领域已知的技术从已知的DNA序列合成和组装本文所述的任意克隆和表达载体。调节区和增强子元件可以是多种天然和合成来源。一些载体和宿主细胞可商购获得。可用载体的非限制性实例描述于以下:Current Protocols in Molecular Biology,1988,ed.Ausubel et al.,Greene Publish.Assoc.&Wiley Interscience的附录5,其通过引用并入本文;和商业供应商如Clontech Laboratories、Stratagene Inc.和Invitrogen,Inc的目录。
治疗方法
基于细胞的治疗剂可用于施用给受试者(例如,具有临床状况例如癌症或感染的研究动物或哺乳动物,例如人)。例如,可以将基于细胞的治疗剂施用给受试者以治疗癌症或感染。因此,本申请提供了使用本文提供的表达载体治疗临床状况如癌症或感染的方法。感染可以是例如急性感染或慢性感染。在一些实施方案中,感染可以是丙型肝炎病毒、乙型肝炎病毒、人免疫缺陷病毒或疟疾的感染。所述方法可包括在以治疗性方式降低受试者中临床状况的进展或症状的情况下,向受试者施用基于细胞的治疗剂。
在多个实施方案中,本发明涉及癌症和/或肿瘤;例如,治疗或预防癌症和/或肿瘤。癌症或肿瘤是指细胞不受控制的生长和/或细胞存活的异常增加和/或凋亡的抑制,其干扰了身体器官和系统的正常功能。包括良性和恶性癌症、息肉、增生以及休眠的肿瘤或微转移。还包括具有不受免疫系统阻碍的异常增殖的细胞(例如,病毒感染的细胞)。癌症可以是原发性癌症或转移性癌症。原发性癌症可以是在临床上可检测到的发源部位处的癌细胞区域,并且可以是原发性肿瘤。相比之下,转移性癌症可以是疾病从一个器官或部分扩散到另一个非临近器官或部分。转移性癌症可以由获得穿透并浸润局部区域内的周围正常组织从而形成新肿瘤(其可以是局部转移)的能力的癌细胞引起。癌症也可以由获得穿透淋巴管壁和/或血管壁并此后癌细胞能够通过血流循环(从而成为循环肿瘤细胞)到达体内的其他部位和组织的能力的癌细胞引起。癌症可以归因于例如淋巴或血行扩散的过程。癌症也可以由在另一个位置停留,重新穿透血管或壁,继续增殖,并最终形成另一临床上可检测的肿瘤的肿瘤细胞引起。癌症可以是这个新肿瘤,其可以是转移性(或继发性)肿瘤。
癌症可以由已经转移的肿瘤细胞引起,其可以是继发性或转移性肿瘤。所述肿瘤的细胞可以与原肿瘤中的细胞相似。例如,如果乳腺癌或结肠癌转移至肝,则继发性肿瘤虽然存在于肝中,但由异常的乳腺癌或结肠癌细胞组成,而不是异常的肝细胞。因此,肝中的肿瘤可以是转移性乳腺癌或转移性结肠癌,而不是肝脏癌症。
癌症可以发源于任何组织。癌症可以发源于黑色素瘤、结肠、乳腺或前列腺,并因此可以由最初分别是皮肤、结肠、乳腺或前列腺的细胞组成。癌症也可以是血液恶性肿瘤,其可以是淋巴瘤。癌症可以侵入例如肝、肺、膀胱或肠的组织。
可以治疗的示例性癌症包括但不限于癌,例如各种亚型,包括例如腺癌、基底细胞癌、鳞状细胞癌和移行细胞癌)、肉瘤(包括例如骨和软组织)、白血病(包括例如急性髓样白血病、急性淋巴母细胞白血病、慢性髓样白血病、慢性淋巴细胞白血病和毛细胞白血病)、淋巴瘤和骨髓瘤(包括例如霍奇金淋巴瘤和非霍奇金淋巴瘤、轻链非分泌性MGUS和浆细胞瘤)以及中枢神经系统癌症(包括例如脑癌(例如神经胶质瘤(例如星形细胞瘤、少突胶质细胞瘤和室管膜瘤)、脑膜瘤、垂体腺瘤和神经瘤,以及脊髓肿瘤(例如脑膜瘤和神经纤维瘤)。
本发明的代表性癌症和/或肿瘤包括但不限于基底细胞癌、胆管癌;膀胱癌;骨癌;脑和中枢神经系统癌症;乳腺癌;腹膜癌;宫颈癌;绒毛膜癌;结肠和直肠癌;结缔组织癌;消化系统癌症;子宫内膜癌;食道癌;眼癌;头颈癌;胃癌(包括胃肠道癌);胶质母细胞瘤;肝癌(hepatic carcinoma);肝细胞瘤(hepatoma);上皮内赘生物;肾脏或肾癌;喉癌;白血病;肝脏癌症(liver cancer);肺癌(例如小细胞肺癌、非小细胞肺癌、肺腺癌和肺鳞癌);黑色素瘤;骨髓瘤;成神经细胞瘤;口腔癌(唇、舌、口和咽);卵巢癌;胰腺癌;前列腺癌;视网膜母细胞瘤;横纹肌肉瘤;直肠癌;呼吸系统癌症;唾液腺癌;肉瘤;皮肤癌;鳞状细胞癌;胃癌;睾丸癌;甲状腺癌;子宫或子宫内膜癌;泌尿系统癌症;外阴癌;淋巴瘤,包括霍奇金淋巴瘤和非霍奇金淋巴瘤,以及B细胞淋巴瘤(包括低度/滤泡性非霍奇金淋巴瘤(NHL);小淋巴细胞(SL)NHL;中度/滤泡性NHL;中度弥漫性NHL;高度免疫母细胞性NHL;高度淋巴母细胞性NHL;高度小无裂细胞NHL;大包块疾病NHL(bulky disease NHL);套细胞淋巴瘤;AIDS相关淋巴瘤;和华氏巨球蛋白血症(Waldenstrom's Macroglobulinemia);慢性淋巴细胞性白血病(CLL);急性淋巴细胞性白血病(ALL);毛细胞白血病;慢性粒细胞性白血病;以及其他癌和肉瘤;和移植后淋巴组织增生性病症(PTLD),以及与斑痣性错构瘤病(phakomatoses)、水肿(例如与脑肿瘤相关的水肿)和Meigs综合征相关的异常血管增殖。
在一些方面,本发明的融合蛋白用于消除细胞内病原体。在一些方面,本发明的融合蛋白用于治疗一种或多种感染。在一些实施方案中,本发明的融合蛋白用于治疗病毒感染(包括例如HIV和HCV)、寄生虫感染(包括例如疟疾)和细菌感染的方法中。在多个实施方案中,感染诱导免疫抑制。例如,HIV感染常常导致感染受试者中的免疫抑制。因此,如本文别处所述,在多个实施方案中,此类感染的治疗可包括用本发明的融合蛋白调节免疫系统,以使免疫刺激优于免疫抑制。另外,本发明提供了治疗诱导免疫激活的感染的方法。例如,肠道蠕虫感染已与慢性免疫激活相关。在这些实施方案中,此类感染的治疗可包括用本发明的融合蛋白调节免疫系统,以使免疫抑制优于免疫刺激。
在多个实施方案中,本发明提供了治疗病毒感染的方法,所述病毒感染包括但不限于急性或慢性病毒感染,例如呼吸道病毒感染、乳头瘤病毒感染、单纯疱疹病毒(HSV)感染、人免疫缺陷病毒(HIV)感染以及内部器官的病毒感染,例如肝炎病毒感染。在一些实施方案中,病毒感染由黄病毒属的病毒引起。在一些实施方案中,黄病毒属的病毒选自黄热病毒、西尼罗河病毒、登革热病毒、日本脑炎病毒、圣路易斯脑炎病毒和丙型肝炎病毒。在其他实施方案中,病毒感染由Picornaviridae家族的病毒,例如脊髓灰质炎病毒、鼻病毒、柯萨奇病毒引起。在其他实施方案中,病毒感染由正粘病毒科的成员,例如流感病毒引起。在其他实施方案中,病毒感染由逆转录病毒科的成员,例如慢病毒引起。在其他实施方案中,病毒感染由副粘病毒科的成员,例如呼吸道合胞病毒、人副流感病毒、风疹病毒(例如腮腺炎病毒)、麻疹病毒和人偏肺病毒(human metapneumovirus)引起。在其他实施方案中,病毒感染由布尼亚病毒科的成员,例如汉坦病毒引起。在其他实施方案中,病毒感染由呼肠孤病毒科的成员,例如轮状病毒引起。
在多个实施方案中,本发明提供了治疗寄生虫感染如原生动物或蠕虫感染的方法。在一些实施方案中,寄生虫感染由原生动物寄生虫引起。在一些实施方案中,oritiziab寄生虫选自肠原生动物、组织原生动物或血液原生动物。示例性的原生动物寄生虫包括但不限于痢疾阿米巴(Entamoeba hystolytica)、兰氏贾第鞭毛虫(Giardia lamblia)、鼠隐孢子虫(Cryptosporidium muris)、冈比亚锥虫(Trypanosomatida gambiense)、Trypanosomatida rhodesiense、Trypanosomatida crusi、墨西哥利什曼原虫(Leishmaniamexicana)、巴西利什曼原虫(Leishmania braziliensis)、热带利什曼原虫(Leishmaniatropica)、杜氏利什曼原虫(Leishmania donovani)、刚地弓形虫(Toxoplasma gondii)、间日疟原虫(Plasmodium vivax)、卵形疟原虫(Plasmodium ovale)、三日疟原虫(Plasmodiummalariae)、恶性疟原虫(Plasmodium falciparum)、阴道毛滴虫(Trichomonas vaginalis)和组织滴虫(Histomonas meleagridis)。在一些实施方案中,寄生虫感染由蠕虫类寄生虫例如线虫(例如,有腺纲(Adenophorea))引起。在一些实施方案中,寄生虫选自侧尾腺口纲(Secementea)(例如毛首鞭虫(Trichuris trichiura)、人蛔虫(Ascaris lumbricoides)、蛲虫(Enterobius vermicularis)、十二指肠钩虫(Ancylostoma duodenale)、美洲钩虫(Necator americanus)、粪类圆线虫(Strongyloides stercoralis)、班氏线虫(Wuchereria bancrofti)、麦地那龙线虫(Dracunculus medinensis))。在一些实施方案中,寄生虫选自吸虫(例如血吸虫、肝吸虫、肠吸虫和肺吸虫)。在一些实施方案中,寄生虫选自:曼氏血吸虫(Schistosoma mansoni)、埃及血吸虫(Schistosoma haematobium)、日本血吸虫(Schistosoma japonicum)、肝片吸虫(Fasciola hepatica)、巨片吸虫(Fasciolagigantica)、异形吸虫(Heterophyes heterophyes)、卫氏并殖吸虫(Paragonimuswestermani)。在一些实施方案中,寄生虫选自绦虫(例如猪肉绦虫(Taenia solium)、牛肉绦虫(Taenia saginata)、微膜壳绦虫(Hymenolepis nana)、细粒棘球绦虫(Echinococcusgranulosus))。
在多个实施方案中,本发明提供了治疗细菌感染的方法。在多个实施方案中,细菌感染由革兰氏阳性菌、革兰氏阴性菌、需氧和/或厌氧细菌引起。在多个实施方案中,细菌选自但不限于葡萄球菌、乳酸杆菌、链球菌、八叠球菌(Sarcina)、埃希氏菌(Escherichia)、肠杆菌、克雷伯菌(Klebsiella)、假单胞菌、不动杆菌、分枝杆菌、变形杆菌(Proteus)、弯曲杆菌、柠檬酸杆菌、奈瑟氏菌、芽孢杆菌、拟杆菌、消化球菌、梭菌、沙门氏菌、志贺氏菌、沙雷氏菌、嗜血杆菌、布鲁氏菌及其他生物体。在一些实施方案中,细菌选自但不限于铜绿假单胞菌、荧光假单胞菌、食酸假单胞菌(Pseudomonas acidovorans)、产碱假单胞菌、恶臭假单胞菌、嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia)、洋葱伯克氏菌(Burkholderiacepacia)、嗜水气单胞菌(Aeromonas hydrophilia)、埃希氏大肠杆菌、弗氏柠檬酸杆菌(Citrobacter freundii)、鼠伤寒沙门氏菌(Salmonella typhimurium)、伤寒沙门氏菌(Salmonella typhi)、副伤寒沙门氏菌(Salmonella paratyphi)、肠炎沙门氏菌(Salmonella enteritidis)、痢疾志贺氏菌(Shigella dysenteriae)、弗氏志贺氏菌(Shigella flexneri)、宋内志贺氏菌(Shigella sonnei)、阴沟肠杆菌(Enterobactercloacae)、产气肠杆菌(Enterobacter aerogenes)、肺炎克雷伯菌(Klebsiellapneumoniae)、产酸克雷伯菌(Klebsiella oxytoca)、粘质沙雷氏菌(Serratiamarcescens)、土拉弗朗西斯菌(Francisella tularensis)、摩氏摩根菌(Morganellamorganii)、奇异变形杆菌(Proteus mirabilis)、普通变形杆菌(Proteus vulgaris)、产碱普罗威登斯菌(Providencia alcalifaciens)、雷氏普罗威登斯菌(Providenciarettgeri)、斯氏普罗威登斯菌(Providencia stuartii)、鲍曼不动杆菌(Acinetobacterbaumannii)、乙酸钙不动杆菌(Acinetobacter calcoaceticus)、溶血不动杆菌(Acinetobacter haemolyticus)、结肠炎耶尔森菌(Yersinia enterocolitica)、鼠疫杆菌(Yersinia pestis)、假结核耶尔森菌(Yersinia pseudotuberculosis)、中间耶尔森菌(Yersinia intermedia)、百日咳杆菌(Bordetella pertussis)、副百日咳博德特氏菌(Bordetella parapertussis)、支气管炎博德特氏菌(Bordetella bronchiseptica)、流感嗜血杆菌(Haemophilus influenzae)、副流感嗜血杆菌(Haemophilus parainfluenzae)、溶血性嗜血杆菌(Haemophilus haemolyticus)、副溶血性嗜血杆菌(Haemophilusparahaemolyticus)、杜克雷嗜血杆菌(Haemophilus ducreyi)、多杀巴斯德氏菌(Pasteurella multocida)、溶血性巴斯德氏菌(Pasteurella haemolytica)、卡他氏杆菌(Branhamella catarrhalis)、幽门螺杆菌(Helicobacter pylori)、胎儿弯曲菌(Campylobacter fetus)、空肠弯曲菌(Campylobacter jejuni)、大肠弯曲杆菌(Campylobacter coli)、伯氏疏螺旋体(Borrelia burgdorferi)、霍乱弧菌(Vibriocholerae)、副溶血性弧菌(Vibrio parahaemolyticus)、嗜肺军团菌(Legionellapneumophila)、单核细胞增生性李斯特氏菌(Listeria monocytogenes)、淋病奈瑟氏菌(Neisseria gonorrhoeae)、脑膜炎奈瑟氏菌(Neisseria meningitidis)、金氏菌(Kingella)、莫拉氏菌(Moraxella)、阴道加德纳氏菌(Gardnerella vaginalis)、脆弱拟杆菌(Bacteroides fragilis)、吉氏拟杆菌(Bacteroides distasonis)、拟杆菌3452A同源组(Bacteroides 3452A homology group)、普通拟杆菌(Bacteroides vulgatus)、卵形拟杆菌(Bacteroides ovalus)、多形拟杆菌(Bacteroides thetaiotaomicron)、单形拟杆菌(Bacteroides uniformis)、埃氏拟杆菌(Bacteroides eggerthii)、内脏拟杆菌(Bacteroides splanchnicus)、艰难梭状芽胞杆菌(Clostridium difficile)、结核分枝杆菌(Mycobacterium tuberculosis)、鸟分枝杆菌(Mycobacterium avium)、胞内分枝杆菌(Mycobacterium intracellulare)、麻风分枝杆菌(Mycobacterium leprae)、白喉棒状杆菌(Corynebacterium diphtheriae)、溃疡棒状杆菌(Corynebacterium ulcerans)、肺炎链球菌、无乳链球菌、酿脓链球菌、粪肠球菌(Enterococcus faecalis)、屎肠球菌(Enterococcus faecium)、金黄色葡萄球菌、表皮葡萄球菌(Staphylococcusepidermidis)、腐生葡萄球菌(Staphylococcus saprophyticus)、中间葡萄球菌(Staphylococcus intermedius)、猪葡萄球菌hyicus亚种(Staphylococcus hyicussubsp.hyicus)、溶血性葡萄球菌、人葡萄球菌(Staphylococcus hominis)或解糖葡萄球菌(Staphylococcus saccharolyticus)。可以将待施用的基于细胞的治疗剂与其他分子、分子结构或化合物的混合物例如脂质体、受体或细胞靶向分子,或口服、局部或其他制剂混合、封装、结合或以其他方式关联,以协助摄取、分配和/或吸收。待施用的基于细胞的治疗剂可以与药学上可接受的载体组合。
因此,本公开还提供了含有本文所述的基于细胞的治疗剂与生理学和药学上可接受的载体组合的组合物。生理和药学上可接受的载体可以包括任何可用于免疫的熟知组分。载体可以促进或增强对疫苗中施用的抗原的免疫应答。细胞制剂可以含有缓冲液以维持优选的pH范围、盐或其他组分,其将抗原在组合物中呈递给个体,刺激针对该抗原的免疫应答。生理上可接受的载体还可以含有一种或多种增强对抗原的免疫应答的佐剂。药学上可接受的载体包括例如药学上可接受的溶剂、悬浮剂或任何其他用于将化合物递送至受试者的药学上惰性的媒介物。药学上可接受的载体可以是液体或固体,并且可以与所计划的施用方式一并选择,以便在与一种或多种治疗性化合物和所给药物组合物的任意其他组分组合时,能提供所需的体积、稠度和其他相关的运输和化学性质。典型的药学上可接受的载体包括但不限于:水、盐溶液、结合剂(例如聚乙烯吡咯烷酮或羟丙基甲基纤维素);填充剂(例如乳糖或右旋糖以及其他糖、明胶或硫酸钙)、润滑剂(例如淀粉、聚乙二醇或乙酸钠)、崩解剂(例如淀粉或淀粉羟乙酸钠)和润湿剂(例如月桂基硫酸钠)。可以配制组合物用于皮下、肌内或皮内施用,或以任意免疫所接受的方式。
佐剂是指当添加至免疫原性剂如表达分泌的疫苗蛋白(例如gp96-Ig)和T细胞共刺激融合多肽的肿瘤细胞时,非特异性增强或加强在暴露于混合物后接受者宿主中针对该药剂的免疫应答的物质。佐剂可包括例如水包油乳剂、油包水乳剂、明矾(铝盐)、脂质体和微粒,例如聚苯乙烯、淀粉,聚磷腈和聚丙交酯/多糖苷。
佐剂还可包括例如角鲨烯混合物(SAF-1)、胞壁肽、皂苷衍生物、分枝杆菌细胞壁制备物、单磷酰基脂质A、霉菌酸衍生物、非离子嵌段共聚物表面活性剂、Quill A、霍乱毒素B亚基、多磷腈及衍生物,以及免疫刺激复合物(ISCOM),例如描述在Takahashi et al.,Nature 1990,344:873-875中的那些。为了兽医用途和在动物中生产抗体,可以使用弗氏佐剂(Freund's adjuvant)的促有丝分裂组分(完全和不完全)。在人体中,弗氏不完全佐剂(IFA)是有用的佐剂。各种合适的佐剂是本领域公知的(参见例如Warren和Chedid,CRCCritical Reviews in Immunology 1988,8:83;以及Allison和Byars,in Vaccines:NewApproaches to Immunological Problems,1992,Ellis,ed.,Butterworth-Heinemann,Boston)。其他佐剂包括例如bacille Calmett-Guerin(BCG)、DETOX(含有分枝杆菌的细胞壁骨架(CWS)和来自明尼苏达沙门氏菌(Salmonella minnesota)的单磷酰脂质(MPL)A)等(参见例如Hoover et al.,J Clin Oncol 1993,11:390;and Woodlock et al.,JImmunother 1999,22:251-259)。
在一些实施方案中,可以将分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞按以下施用给受试者:约100,000个细胞、约150,000个细胞、约200,000个细胞、约250,000个细胞、约300,000个细胞、约350,000个细胞、约400,000个细胞、约450,000个细胞、约500,000个细胞、约550,000个细胞、约600,000个细胞、约650,000个细胞、约700,000个细胞、约750,000个细胞、约800,000个细胞、约850,000个细胞、约900,000个细胞、约950,000个细胞、约100万个细胞、约150万个细胞、约200万个细胞、约250万个细胞、约300万个细胞、约350万个细胞、约400万个细胞、约450万个细胞、约600万个细胞、约650万个细胞、约700万个细胞、约750万个细胞、约800万个细胞、约850万个细胞、约900万个细胞、约950万个细胞或约1000万个细胞。
在一些实施方案中,可以将分泌疫苗蛋白(例如,gp96-Ig)的细胞按以下施用给受试者:约100,000个细胞、约150,000个细胞、约200,000个细胞、约250,000个细胞、约300,000个细胞、约350,000个细胞、约400,000个细胞、约450,000个细胞、约500,000个细胞、约550,000个细胞、约600,000个细胞、约650,000个细胞、约700,000个细胞、约750,000个细胞、约800,000个细胞、约850,000个细胞、约900,000个细胞、约950,000个细胞、约100万个细胞、约150万个细胞、约200万个细胞、约250万个细胞、约300万个细胞、约350万个细胞、约400万个细胞、约450万个细胞、约600万个细胞、约650万个细胞、约700万个细胞、约750万个细胞、约800万个细胞、约850万个细胞、约900万个细胞、约950万个细胞或约1000万个细胞。
在一些实施方案中,分泌疫苗蛋白的细胞的固定剂量为1x10
在一些实施方案中,分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞的固定剂量为1x10
在一些实施方案中,可以将分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞按以下施用给受试者:约0.1-1.1x10
在一些实施方案中,可以将分泌疫苗蛋白(例如,gp96-Ig)的细胞按以下施用给受试者:约0.1-1.1x10
在一些实施方案中,可以将基于细胞的治疗剂施用给受试者一次或多次(例如一次、两次、两至四次、三至五次、五至八次、六至十次、八至十二次或超过十二次)。可以施用本文提供的基于细胞的治疗剂每天一次或多次、每周一次或多次、隔一周一次、每月一次或多次、每两至三个月一次、每三至六个月一次或每六至十二个月一次。基于细胞的治疗剂可以在任意合适的时间段内施用,例如从约1天至约12个月的时间段。在一些实施方案中,例如,施用期可以是从约1天至90天;从约1天至60天;从约1天至30天;从约1天至20天;从约1天至10天;从约1天至7天。在一些实施方案中,施用期可以是从约1周至50周;从约1周至50周;从约1周至40周;从约1周至30周;从约1周至24周;从约1周至20周;从约1周至16周;从约1周至12周;从约1周至8周;从约1周至4周;从约1周至3周;从约1周至2周;从约2周至3周;从约2周至4周;从约2周至6周;从约2周至8周;从约3周至8周;从约3周至12周;或约4周至20周或之间的任意周数增量。
在一些实施方案中,在已经施用了基于细胞的治疗剂的初始剂量(有时称为“初免(priming)”剂量)并且已经达到最大的抗原特异性免疫应答之后,可以施用本文提供的基于细胞的治疗剂的一个或多个加强剂量。例如,可以在初免剂量后的约10至30天、约15至35天、约20至40天、约25至45天或约30至50天施用加强剂量。
在一些实施方案中,按以下施用可分泌型疫苗蛋白(例如,gp96-Ig)和T细胞共刺激融合蛋白(例如,分泌OX40L-Ig的细胞):间隔1分钟、间隔10分钟、间隔30分钟、间隔少于1小时、间隔1小时、间隔1小时至2小时、间隔2小时至3小时、间隔3小时至4小时、间隔4小时至5小时、间隔5小时至6小时、间隔6小时至7小时、间隔7小时至8小时、间隔8小时至9小时、间隔9小时至10小时、间隔10小时至11小时、间隔11小时至12小时、间隔1天、间隔2天、间隔3天、间隔4天、间隔5天、间隔6天、间隔1周、间隔2周、间隔3周或间隔4周。
在一些实施方案中,提供了一种方案,其中施用分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞的第一治疗,并随后施用分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞的第二治疗,并施用分泌疫苗蛋白(例如,gp96-Ig)的细胞。例如,在一些实施方案中,第一治疗和第二治疗间隔约3天,或约5天,或约1周,或约2周或约3周。
在一些实施方案中,第一和第二治疗间隔约两周。
在一些实施方案中,施用分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞的单个固定剂量,并且在施用分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞的第一剂量后,施用第二剂量并施用分泌疫苗蛋白(例如,gp96-Ig)的细胞的固定剂量。
在一些实施方案中,分泌可分泌型疫苗蛋白(例如,gp96-Ig)的细胞的单个固定剂量与分泌T细胞共刺激融合蛋白(例如,OX40L-Ig)的细胞的递增剂量一起施用。
在一些实施方案中,本文提供的方法可用于控制实体瘤的生长(例如乳腺、前列腺、黑素色瘤、肾、结肠或宫颈肿瘤的生长)和/或转移。所述方法可以包括向有此需要的受试者施用有效量的本文所述的基于细胞的治疗剂。在一些实施方案中,受试者是哺乳动物(例如人)。
本文提供的基于细胞的治疗剂和方法可用于刺激针对肿瘤的免疫应答。这样的免疫应答可用于治疗或减轻与肿瘤相关的迹象(sign)或症状。如本文所用,“治疗”是指与未经治疗的个体的症状相比,减轻、阻止和/或逆转了已被施用本文所述的基于细胞的治疗剂的个体的症状。从业人员将意识到,本文所述的方法将与熟练的从业人员(医师或兽医)进行的连续临床评估一同使用,以确定后续疗法。这样的评估将有助于评估是否增加、减少或继续特定的治疗剂量、施用方式等并告知。
因此,本文提供的方法可用于治疗肿瘤,包括例如癌症。所述方法可用于例如通过阻止进一步的肿瘤生长、通过减慢肿瘤生长或通过引起肿瘤消退来抑制肿瘤的生长。因此,所述方法可以用于例如治疗癌症,例如肺癌。可以理解的是,向其施用化合物的受试者不必遭受特定的创伤状态。实际上,本文所述的基于细胞的治疗剂可以在症状发展之前(例如,癌症缓解的患者)预防性地施用。术语“治疗性的”和“治疗上的”以及这些术语的变换用于涵盖治疗性、缓和性和预防性用途。因此,如本文所用,“治疗或减轻症状”是指与未接受这样的施用的个体的症状相比,减轻、阻止和/或逆转已被施用治疗上有效量的组合物的个体的症状。
如本文所用,术语“有效量”和“治疗上有效的量(治疗有效量)”是指足以在受试者(例如,诊断为患有癌症或感染的人)中提供所需治疗(例如抗癌、抗肿瘤或抗感染)效果的量。抗肿瘤和抗癌效果包括但不限于调节肿瘤生长(例如,肿瘤生长延迟)、肿瘤大小或转移、与特定抗癌剂相关的毒性和副作用的减少、癌症的临床损害或症状的改善或最小化、将受试者的存活期延长超出没有此类治疗的情况下所期望的存活期,以及预防在施用前没有肿瘤形成的动物中的肿瘤生长,即预防性施用。在一些实施方案中,施用有效量的基于细胞的治疗剂可以增加受试者中肿瘤抗原特异性T细胞的激活或增殖。例如,与施用前受试者中肿瘤抗原特异性T细胞的激活或增殖水平相比,受试者中肿瘤抗原特异性T细胞的激活或增殖可以增加至少10%(例如,至少25%、至少50%或至少75%)。
抗感染作用包括例如减少感染剂(例如病毒或细菌)的量。当受试者中要治疗的临床状况是感染时,施用本文提供的基于细胞的治疗剂能刺激受试者中病原性抗原特异性T细胞的激活或增殖。例如,施用基于细胞的治疗剂能导致受试者中抗原特异性T细胞的激活水平高于通过单独的gp96-Ig疫苗接种所达到的水平。
本领域技术人员将理解,可以通过微调和/或通过施用多于一个剂量(例如,通过同时施用两种不同的基因修饰的肿瘤细胞,或通过施用基于细胞的治疗剂与另一种剂(例如PD-1拮抗剂)以增强治疗效果(例如,协同地),来降低或增加基于细胞的治疗剂的有效量。因此,本公开提供了一种针对给定哺乳动物特有的具体紧急情况来定制施用/治疗的方法。治疗有效量可以通过例如从相对较低的量开始并逐步增加量并同时评估有益效果来确定。因此,本文提供的方法可以单独使用或与其他公知的肿瘤疗法组合使用,以治疗患有肿瘤的患者。本领域技术人员将容易理解,本文提供的基于细胞的治疗剂和方法的有利用途,例如,在延长癌症患者的预期寿命和/或改善癌症患者(例如,肺癌患者)的生活质量方面。
组合疗法和结合(conjugation)
在一些实施方案中,本发明提供了进一步包括向受试者施用额外的药剂的方法。在一些实施方案中,本发明涉及共同施用和/或共同配制。
在一些实施方案中,向患者施用第一细胞,所述第一细胞包含含有编码可分泌型疫苗蛋白的核苷酸序列的表达载体,所述患者正在经历第二细胞的治疗,所述第二细胞包含含有编码T细胞共刺激融合蛋白的核苷酸序列的表达载体,这种施用在与另一药剂共同施用时协同作用。
在一些实施方案中,疫苗蛋白(例如,gp96-Ig)和一种或多种共刺激分子的施用在与另一药剂共同施用时协同作用,并且以低于当此类药剂用作单一疗法施用时通常采用的剂量的剂量施用。
在一些实施方案中,包括但不限于癌症应用,本发明涉及以化疗剂作为额外的药剂。化疗剂的实例包括但不限于烷化剂,例如噻替派和CYTOXAN环磷酰胺;烷基磺酸盐,例如白消安、英丙舒凡(improsulfan)和哌泊舒凡(piposulfan);氮丙啶,例如benzodopa、卡波醌、美妥替哌(meturedopa)和乌瑞替哌(uredopa);乙烯亚胺和甲基三聚氰胺(methylamelamine),包括六甲蜜胺,三乙烯蜜胺,三乙烯磷酰胺(trietylenephosphoramide),三乙烯硫代磷酰胺(triethiylenethiophosphoramide)和三甲基三聚氰胺(trimethylolomelamine);多聚乙酰(acetogenin)(例如,布洛他辛(bullatacin)和布洛他辛酮(bullatacinone));喜树碱(包括合成类似物拓扑替康);苔藓抑素(bryostatin);cally statin;CC-1065(包括其阿多来新(adozelesin)、卡折来新(carzelesin)和比折来新(bizelesin)合成类似物);念珠藻素(例如念珠藻素1和念珠藻素8);尾海兔素(dolastatin);倍癌霉素(duocarmycin)(包括合成类似物,KW-2189和CB 1-TM1);软珊瑚醇(eleutherobin);水鬼蕉碱(pancratistatin);匍枝珊瑚醇(sarcodictyin);海绵抑制素(spongistatin);氮芥,例如苯丁酸氮芥(chlorambucil)、萘氮芥(chlornaphazine)、氯磷酰胺(cholophosphamide)、雌莫司汀(estramustine)、异环磷酰胺(ifosfamide)、二氯甲基二乙胺(mechlorethamine)、盐酸甲氧氮芥(mechlorethamineoxide hydrochloride)、美法仑、新恩比兴(novembichin)、苯芥胆甾醇(phenesterine)、泼尼莫司汀(prednimustine)、曲磷胺(trofosfamide)、乌拉莫司汀(uracil mustard);nitrosurea,例如卡莫司汀、氯脲霉素(chlorozotocin)、福莫司汀(fotemustine)、洛莫斯汀(lomustine)、尼莫斯汀(nimustine)和雷莫斯汀(ranimnustine);抗生素,例如烯二炔类抗生素(例如卡奇霉素(calicheamicin),尤其是卡奇霉素gammall和卡奇霉素omegall(参见例如Agnew,Chem.Intl.Ed.Engl.,33:183-186(1994));达内霉素(dynemicin),包括达内霉素A;双膦酸盐,例如氯膦酸盐;埃斯佩拉霉素(esperamicin);以及新制癌菌素发色团(neocarzinostatin chromophore)和相关的色蛋白烯二炔抗生素发色团)、阿克拉霉素(aclacinomysin)、放线菌素、安曲霉素(authramycin)、重氨丝氨酸(azaserine)、博来霉素(bleomycin)、放线菌素(cactinomycin)、卡拉比星(carabicin)、caminomycin、嗜癌素(carzinophilin)、色霉素(chromomycinis)、更生霉素(dactinomycin)、柔红霉素(daunorubicin)、地托比星(detorubicin)、6-重氮-5-氧代-L-正亮氨酸(6-diazo-5-oxo-L-norleucine),ADRIAMYCIN阿霉素(ADRIAMYCIN doxorubicin)(包括吗啉代阿霉素、cyanomorpholino-阿霉素,2-吡咯啉-阿霉素和脱氧阿霉素)、表柔比星(epirubicin)、依索比星(esorubicin)、伊达比星(idarubicin)、麻西罗霉素(marcellomycin)、丝裂霉素例如丝裂霉素C、霉酚酸(mycophenolic acid)、诺加霉素(nogalamycin)、橄榄霉素(olivomycin)、培洛霉素(peplomycin)、potfiromycin、嘌呤霉素(puromycin)、quelamycin、罗多比星(rodorubicin)、链霉黑素(streptonigrin)、链脲菌素(streptozocin)、杀结核菌素(tubercidin)、乌苯美司(ubenimex)、净司他丁(zinostatin)、佐柔比星(zorubicin);抗代谢物,例如甲氨蝶呤和5-氟尿嘧啶(5-FU);叶酸类似物,例如denopterin、甲氨蝶呤、蝶罗呤(pteropterin)、三甲曲沙(trimetrexate);嘌呤类似物,例如氟达拉滨(fludarabine)、6-巯基嘌呤、硫咪嘌啉(thiamiprine)、硫鸟嘌呤;嘧啶类似物,例如安西他滨、阿扎胞苷、6-氮杂尿苷、卡莫氟、阿糖胞苷、双脱氧尿苷、去氧氟尿苷(doxifluridine)、依诺他滨(enocitabine)、氟尿苷;雄激素,例如卡普睾酮(calusterone)、屈他雄酮丙酸酯(dromostanolone propionate)、环硫雄醇(epitiostanol)、美雄烷(mepitiostane)、睾内酯(testolactone);抗肾上腺素,如minoglutethimide、米托坦(mitotane)、曲洛司坦(trilostane);叶酸补充剂,例如叶酸;乙酰葡醛酯(aceglatone);醛磷酰胺糖苷(aldophosphamide glycoside);氨基乙酰丙酸;恩尿嘧啶(eniluracil);安吖啶(amsacrine);bestrabucil;比生群(bisantrene);依达曲沙(edatraxate);地美可辛(demecolcine);地吖醌(diaziquone);elformithine;依利醋铵(elliptinium acetate);埃博霉素(epothilone);乙环氧啶(etoglucid);硝酸镓;羟基脲;香菇多糖(lentinan);洛尼达宁(lonidainine);美登素类(maytansinoid),例如美登素(maytansine)和安丝菌素(ansamitocin);米托胍腙(mitoguazone);米托蒽醌(mitoxantrone);mopidanmol;二胺硝吖啶(nitraerine);喷司他丁(pentostatin);蛋氨氮芥(phenamet);吡柔比星(pirarubicin);洛索蒽醌(losoxantrone);鬼臼酸(podophyllinic acid);2-乙基肼(2-ethylhydrazide);丙卡巴肼(procarbazine);PSK多糖复合物(JHS Natural Products,Eugene,Oreg.);雷佐生(razoxane);根霉素(rhizoxin);sizofuran;锗螺胺(spirogermanium);细交链孢菌酮酸(tenuazonic acid);三亚胺醌(triaziquone);2,2',2"-三氯三乙基胺(2,2',2"-trichlorotriethylamine);单端孢霉烯(trichothecene)(例如T-2毒素、verracurin A、漆斑菌素A(roridin A)和蛇形菌素(anguidine));尿烷(urethan);长春地辛;达卡巴嗪;甘露司汀(mannomustine);二溴甘露醇(mitobronitol);二溴卫矛醇(mitolactol);哌泊溴烷(pipobroman);gacytosine;阿糖苷(“Ara-C”);环磷酰胺;噻替派;紫杉烷类,例如TAXOL紫杉醇(Bristol-Myers SquibbOncology,Princeton,N.J.)、ABRAXANE不含氢化蓖麻油(ABRAXANE Cremophor-free)、白蛋白工程化的紫杉醇纳米颗粒制剂(American Pharmaceutical Partners,Schaumberg,111.)和TAXOTERE doxetaxel(Rhone-Poulenc Rorer,Antony,France);苯丁酸氮芥;GEMZAR吉西他滨;6-硫鸟嘌呤;巯嘌呤;甲氨蝶呤;铂类似物,例如顺铂、奥沙利铂和卡铂;长春碱;铂;依托泊苷(VP-16);异环磷酰胺;米托蒽醌;长春新碱;NAVELBINE.长春瑞滨;novantrone;替尼泊苷;依达曲沙;道诺霉素(daunomycin);氨蝶呤;希罗达(xeloda);伊班膦酸(ibandronate);伊立替康(Camptosar,CPT-11)(包括伊立替康组合5-FU和亚叶酸的治疗方案);拓扑异构酶抑制剂RFS 2000;二氟甲基鸟氨酸(DMFO);类维生素A,例如视黄酸;卡培他滨;康普瑞汀(combretastatin);亚叶酸(LV);奥沙利铂,包括奥沙利铂治疗方案(FOLFOX);拉帕替尼(TYKERB);降低细胞增殖的PKC-α、Raf、H-Ras、EGFR(例如埃罗替尼(Tarceva))和VEGF-A的抑制剂,以及以上任意的药学上可接受的盐、酸或衍生物。另外,治疗方法可进一步包括使用辐射。另外,治疗方法可进一步包括使用光动力疗法。
在一些实施方案中,包括但不限于感染性疾病应用,本发明涉及抗感染剂作为额外的药剂。在一些实施方案中,抗感染剂是抗病毒剂,包括但不限于阿巴卡韦(Abacavir)、阿昔洛韦(Acyclovir)、阿德福韦(Adefovir)、安普那韦(Amprenavir)、阿扎那韦(Atazanavir)、西多福韦(Cidofovir)、达鲁那韦(Darunavir)、地拉韦啶(Delavirdine)、地达诺新(Didanosine)、二十二醇(Docosanol)、依法韦伦(Efavirenz)、埃替格韦(Elvitegravir)、恩曲他滨、恩夫韦地(Enfuvirtide)、依曲韦林(Etravirine)、泛昔洛韦(Famciclovir)和膦甲酸(Foscarnet)。在一些实施方案中,抗感染剂是抗细菌剂,包括但不限于头孢菌素类抗生素(头孢氨苄、头孢呋辛、头孢羟氨苄、头孢唑啉、头孢菌素、头孢克洛、头孢孟多酯(cefamandole)、头孢西丁、头孢丙烯和头孢吡普(ceftobiprole));氟喹诺酮类抗生素(环丙沙星(cipro)、左氧氟沙星(Levaquin)、氧氟沙星(floxin)、加替沙星(tequin)、avelox和诺氟沙星(norflox));四环素类抗生素(四环素、米诺环素、土霉素和强力霉素);青霉素类抗生素(阿莫西林、氨苄青霉素、青霉素V、双氯西林、羧苄青霉素、万古霉素和甲氧西林);单菌霉素类抗生素(氨曲南);和碳青霉烯类抗生素(厄他培南、多利培南、亚胺培南/西司他丁和美罗培南)。在一些实施方案中,抗感染剂包括抗疟疾剂(例如氯喹、奎宁、甲氟喹、伯氨喹、强力霉素、蒿甲醚/苯芴醇、阿托伐醌/氯胍(proguanil)和磺胺多辛/乙胺嘧啶)、甲硝唑、替硝唑、伊维菌素、双羟萘酸噻嘧啶(pyrantel pamoate)和阿苯达唑(albendazole)。
本文别处描述了其他的额外的药剂,包括靶向免疫“检查点”分子的阻断抗体。
在一些实施方案中,所述方法包括与抑制由肿瘤细胞产生的免疫抑制分子的药剂组合施用。在一些实施方案中,所述药剂是针对PD-1的抗体。在一些实施方案中,针对PD-1的抗体选自纳武单抗、派姆单抗、匹地利珠单抗、西米普利单抗、AGEN2034、AMP-224、AMP-514、PDR001。
在一些实施方案中,所述药剂是针对PD-L1的抗体。在一些实施方案中,针对PD-L1的抗体选自阿特朱单抗(Atezolizumab,Tecentriq)、阿维单抗(Avelumab,Bavencio)、德瓦鲁单抗(Durvalumab,Imfinzi)、BMS-936559和CK-301。
在一些实施方案中,所述药剂是针对CTLA-4的抗体。在一些实施方案中,针对CTLA-4的抗体选自伊匹单抗(ipilimumab)、tremelimumab、AGEN1884和RG2077。
在一些实施方案中,所述药剂是针对OX40的抗体。在一些实施方案中,针对OX40的抗体选自PF-04518600、BMS-986178、INCAGN01949、MEDI0562、GSK1795091和GSK3174998。
受试者和/或动物
本文所述的方法旨在用于可以产生这些方法的益处的任意受试者。因此,“受试者”、“患者”和“个体”(可互换使用)包括人以及非人受试者,尤其是家养动物。
在一些实施方案中,受试者和/或动物是哺乳动物,例如人、小鼠、大鼠、豚鼠、狗、猫、马、牛、猪、兔、绵羊或非人灵长类动物,例如猴子、黑猩猩或狒狒。在其他实施方案中,受试者和/或动物是非哺乳动物,例如斑马鱼。在一些实施方案中,受试者和/或动物可以包含荧光标记的细胞(使用例如GFP)。在一些实施方案中,受试者和/或动物是包含荧光细胞的转基因动物。
在一些实施方案中,受试者和/或动物是人。在一些实施方案中,人是儿科人类。在其他实施方案中,人是成年人。在其他实施方案中,人是老年人。在其他实施方案中,人可以称为患者。
在某些实施方案中,人的年龄为约0个月至约6个月、约6至约12个月、约6至约18个月、约18至约36个月、约1至约5岁、约5至约10岁、约10至约15岁、约15至约20岁、约20至约25岁、约25至约30岁、约30至约35岁、约35至约40岁、约40至约45岁、约45至约50岁、约50至约55岁、约55至约60岁、约60至约65岁、约65至约70岁、约70至约75岁、约75至约80岁、约80至约85岁、约85至约90岁、约90至约95岁或约95至约100岁。
在其他实施方案中,受试者是非人动物,且因此本发明涉及兽医用途。在一个具体的实施方案中,非人动物是家养宠物。在另一个具体的实施方案中,非人动物是家畜动物。在某些实施方案中,受试者是不能接受化疗的人类癌症患者,例如,患者对化疗无反应或病得太重而没有合适的化疗治疗窗口(例如,经历太多限制剂量或方案的副作用)。在某些实施方案中,受试者是患有晚期和/或转移性疾病的人类癌症患者。
如本文所用,“同种异基因细胞”是指并非衍生自要被施用细胞的个体的细胞,即,具有与所述个体不同的遗传构成。同种异基因细胞通常从与要被施用细胞的个体相同的物种获得。例如,同种异基因细胞可以是人细胞,如本文所公开,用于施用给人类患者例如癌症患者。如本文所用,“同种异基因肿瘤细胞”是指并非衍生自要被施用同种异基因细胞的个体的肿瘤细胞。通常,同种异基因肿瘤细胞表达一种或多种肿瘤抗原,其可以刺激要被施用细胞的个体中针对肿瘤的免疫应答。如本文所用,“同种异基因癌细胞”,例如肺癌细胞,是指并不衍生自要被施用同种异基因细胞的个体的癌细胞。
如本文所用,“遗传修饰的细胞”是指已经经遗传修饰(例如通过转染或转导)以表达外源核酸的细胞。
除非另外定义,否则本文所用的技术和科学术语具有本发明所属领域的技术人员通常理解的含义。
如本文所用,单数形式“a”、“an”和“the”具体还涵盖它们所指代的术语的复数形式,除非内容另有明确规定。如本文所用,除非另有明确指示,否则词语“或”以“和/或”的“包含”含义使用,而不是“两者其一/或”的“排他”含义。在说明书和所附权利要求书中,单数形式包括复数对象,除非上下文另外明确指出。
本文所用的术语“约”意指大约,在附近,大致上或大约。当术语“约”与数值范围结合使用时,它通过扩展所述数值上方和下方的边界来修改此范围。通常,术语“约”在本文中用于修饰数值低于或高于所述值的20%的差异。如在本说明书中所用,无论是在过渡性短语中还是在权利要求主体中,术语“包括”和“包含”均应解释为具有开放式含义。即,所述术语要与短语“具有至少”或“包括至少”同义地解释。当用于一个过程(方法)的上下文中时,术语“包含”意指所述过程(方法)包括至少所列举的步骤,但可以包括额外的步骤。当用于化合物或组合物的上下文中时,术语“包含”意指所述化合物或组合物至少包含所列举的特征或组分,但也可以包含额外的特征或组分。在以下实施例中将进一步描述本发明,所述实施例不限制权利要求中描述的本发明的范围。
实施例
为了可以更有效地理解本文所公开的发明,以下提供实施例。应当理解,这些实施例仅用于说明性目的,而不应以任何方式解释为对本发明的限制。
实施例1:小鼠中的计划剂量范围发现研究以支持临床给药
以下非临床研究使用替代(对待测的动物物种特异)的HS-130(分泌OX40L-Ig融合蛋白的基因工程化的人肺腺癌细胞系,也称为mHS-130(B16F10))和HS-110(称为mHS-110(B16F10),由B16F10-ova细胞系产生)的基于细胞的疫苗在小鼠中进行,以确定与固定剂量的mHS-110共同施用时mHS-130的剂量。人类中HS-130的起始剂量基于在小鼠模型中产生最小反应(MABEL)的mHS-130剂量,其基于小鼠中mHS-110相比于人类中HS-110的当前剂量的比例使用。
在先前的研究中,小鼠中的鼠HS-110(mHS-110)剂量确定为1x10
实验设计
将野生型C57BL/6小鼠用10
表1:小鼠中剂量范围发现研究的研究方案
在人OX40受体信号传导测定中比较鼠和人OX40L-Ig的信号传导强度的体外测定:
为了表征衍生自小鼠和人物种的OX40L对在人Jurkat细胞系中转染的人OX40受体的信号传导活性,以确定跨物种信号传导的等价性。使用基于Jurkat/OX40细胞的系统,通过人OX40受体结合对NFκB信号诱导的muOX40L-mIgG1(小鼠衍生的OX40L)和huOX40L-hIgG4(人衍生的OX40L)进行并排比较。用muOX40L-mIgG1或huOX40L-hIgG4以1μg/ml的最大浓度刺激细胞。在同一平板上一式两份对每个配体进行检测。5小时后,使用来自Promega的Bio-Glo试剂测量荧光素酶的激活,并使用光度计测量相对发光。使用四参数逻辑曲线分析计算EC
实施例2:1期临床试验、剂型、施用途径和给药方案
药物产品是活的全细胞疫苗,其已经经辐射以使其在表达共刺激融合蛋白OX40L-Ig期间不能进行细胞复制,所述OX40L-Ig是OX40受体(TNF-受体超家族的成员)的配体。所述药物产品是衍生自人肺腺癌细胞系的活的全细胞疫苗。用稳定表达OX40L-Ig的cDNA的7192bp质粒cDNA“pcDNA3.4OX40L-Ig”转染细胞系,以开发如图1所示的经辐射的全细胞疫苗。HS-110是指(viagenpumatucel-L);分泌gp96-Ig融合蛋白的基因工程化的人肺腺癌细胞系,HS-130是指分泌OX40L-Ig融合蛋白的基因工程化的人肺腺癌细胞系AD100。
进行了首次在人类中的I期剂量递增研究,以评估仅HS-130和与HS-110组合在用标准治疗难治的实体瘤患者中的安全性和免疫剂量。
主要目的:仅HS-130和与HS-110组合在用标准治疗(SOC)难治的实体瘤患者中的安全性和耐受性。
次要目的:确定在用SOC难治的实体瘤患者中,与固定剂量的HS-110组合施用的HS-130的免疫剂量。通过使用流式细胞仪确定外周血单核细胞(PBMC)总数和PBMC亚群的激活状态,来研究由HS-130与HS-110组合产生的免疫学作用。
探索目标
1)通过RNA-seq评估档案组织与HS-110共有的抗原表达;2)使用IFNγ、颗粒酶B(gzB)的产生作为功能性读数,通过ELISPOT评估免疫再激活应答;3)确定响应于HS-130和HS-110组合治疗的特定细胞因子标签物的存在;和4)确定对HS-130和HS-110组合治疗的临床应答。
方法:
这是在用SOC难治的晚期实体瘤患者的混合群体中进行的仅HS-130和与HS-110组合的首次人类中的I期试验。HS-130和HS-110均是经过遗传修饰的不能复制的活的癌细胞,设计为当施用至皮肤内皮层中时会刺激免疫应答。研究的目的是研究与治疗有关的安全性和免疫应答。在这项研究中,符合纳入/排除标准的患者接受使用3+3设计逐步增加剂量的HS-130细胞。基于在动物模型中建立的最低预期生物学效应水平(MABEL),第一组以单一固定剂量的HS-130开始治疗。在首剂HS-130之后2周的安全评估间隔后,向患者施用相同剂量的HS-130以及固定剂量的HS-110细胞(1x10
每个剂量组招募三名患者。在第一名患者之后,第二位和第三名患者在第一患者给药后有1周的交错延迟。一旦全部3名患者均已完成第一个给药周期(即4周,其中第1天一剂HS-130,且第15天一剂组合剂量),研究者/主办者(Investigator/Sponsor)剂量递增委员会则对患者组中施用的治疗的安全性和耐受性进行审查。如果没有出现剂量限制毒性(DLT),则剂量递增委员会可以建议登记到下一个更高的剂量水平。如果有一名患者发生DLT,则最多招募3名其他患者并以剂量水平进行治疗。如果六名患者中少于或等于一名患者经历了DLT,则剂量递增委员会建议登记到下一个更高的剂量水平。每个后续剂量组的进行和完成均遵循类似的方式,直到在某一剂量水平下,在总共最多6名所治疗的患者中有2名患者发生DLT。
研究中患者治疗方案的示意图:
阴影部分代表每名患者的4周DLT期
如果在达到MTD之前鉴定到免疫活性剂量,则主办者可以决定停止进一步的剂量递增。替换掉任何未完成第一个治疗周期(即至少一个组合剂量)的患者。预计研究4个HS-130剂量水平,并且试验招募了12至24名患者。通过流式细胞术监测所有患者以进行广泛安全性评估,包括血清细胞因子/趋化因子和免疫细胞亚群的免疫表型谱分析。使用HS-110裂解物和HS-110特异性肽(用于产生IFNγ和颗粒酶B)通过ELISPOT评估免疫应答。通过不良事件(AE)的频率,对临床实验室参数(血液学和生物化学)、体重、生命体征、心电图(ECG)、表现状态、体检(PE)的评估,以及并发疾病/治疗和不良事件的记录,来评估安全性。使用CTCAE版本5对所有毒性分级。
患者数量:总共招募多达12-24名患者。
纳入标准:患者必须符合所有以下纳入标准,才被允许参加试验:患者具有所选实体瘤类型(定义为具有CTA过表达重叠于由HS-110过表达的至少10个CTA),他们对于现有的标准疗法是失败的或不是标准疗法的候选者,并且研究者认为,对于他们来说HS-110+HS-130的实验疗法可能是有益的。年龄≥18岁。具有可接受的器官功能。东部合作肿瘤小组(Eastern Cooperative Oncology Group,ECOG)表现状态为0或1。预期寿命至少三个月。在筛查时可获得新鲜的或归档的肿瘤组织。具有生育/繁育潜力的女性和男性患者必须同意在试验期间以及最后一次用HS-130和/或HS-110治疗后六个月内使用合适的避孕措施。患者必须签署知情同意书。
排除标准:如果以下任何一项适用,则患者不得进入试验:临床上显著的心脏病、充血性心力衰竭和/或不受控制的高血压。已知或临床上怀疑有软脑膜疾病。只要认为稳定(通过CT或MR)并且不需要全身性皮质类固醇,则允许脑或脊髓中的稳定的、先前已经治疗的转移。≥3级过敏反应病史,以及对这个试验、活细胞疗法或活疫苗过程中施与的任何药剂已知或怀疑过敏或不耐受。疑似细胞因子释放综合征(CRS)的病史。已知的免疫缺陷病症。进行中或当前的自身免疫疾病(如已解决,则允许检查点抑制剂免疫相关不良事件的病史)。任何需要同时进行全身免疫抑制疗法的病况。首次IMP施用前四周内进行过大手术。任何正在进行的抗癌治疗,包括:小分子、免疫疗法、化疗、单克隆抗体或任何其他实验药物。必须在研究的首次输注前四周内,或5个半衰期,或所研究产品的生物学作用持续时间的两倍(以最短者为准),停止先前的治疗。允许对先前的乳腺癌或前列腺癌进行辅助抗激素治疗。已知的目前的恶性肿瘤而非纳入诊断。允许完全缓解超过2年的先前的可治愈癌症。任何其他正在进行的显著的不受控制的医学状况。在首个研究药物剂量前的30天内,接受了活疫苗。临床上显著的活性病毒、细菌或真菌感染需要:在首个剂量前不到两周完成抗菌疗法的静脉治疗,或在首个剂量前不到一周完成抗菌疗法的口服治疗。允许预防性治疗(例如拔牙)。已知有对人免疫缺陷病毒(HIV)、乙型肝炎病毒或丙型肝炎病毒的阳性血清学(除了治愈感染后的免疫情形)。可能干扰患者参与试验或试验结果评估的药物滥用、医疗、心理或社会状况。怀孕或哺乳中的女性。剂量和施用方式:第1天向每名患者施用皮内注射HS-130,并在第15天施用相同剂量的HS-130和固定剂量的HS-110(1x10
剂量限制毒性:DLT定义为在研究治疗的前28天内观察到的任何不可接受的(如下所定义)与治疗相关的毒性(即不是由于活动性疾病、正在调查的与疾病相关的过程或并发疾病)。在研究过程中,对所有组均审查了第1周期后正在进行的安全事件,以帮助指导剂量递增决策。
DLT包括:血液毒性≥CTCAEv5 3级。非血液毒性≥CTCAEv5 3级。考虑为临床上显著的和/或不可接受的,以及对支持性护理无反应并导致超过14天的给药计划中断的任何其他毒性(大于基线)。
DLT不包括:3级自我限制或医学上可控制的毒性(例如,没有大于3级中性粒细胞减少、恶心、呕吐、腹泻、疲劳的发热)。使用补充疗法可将其控制在1级或以下的电解质紊乱。
数据监测委员会:数据监测委员会(DMC)评估了在每个剂量水平下获得的数据,并建议是否应按照方案将剂量逐步增加、修改为较低水平或中间水平、完全停止或在同一剂量水平需要更多患者以评估安全性。在每次DMC会议之后,会召开主办人安全委员会会议,以讨论和确认DMC建议采取的行动。
治疗持续时间:在没有疾病进展或不能接受的毒性的情况下,在第一个治疗周期(即4周,第1天一个HS-130剂量,且第15天一个与HS-110组合的剂量)完成后,可以继续每两周一次以相同剂量的HS-130+HS-110组合治疗患者,持续6个月,或直到疾病进展、死亡、患者同意撤回、研究者决定移除患者或无法忍受的毒性,以先发生者为准。
评估标准:
主要终点
·安全性和耐受性:在接受至少1个研究药物剂量的患者中治疗紧急不良事件(TEAE)/严重不良事件(SAE)的频率来衡量,包括临床上显著(CS)的异常实验室参数、ECG、PE和生命体征。
·最大耐受剂量(MTD;最高剂量水平,其中在第一个治疗周期中至少6名患者中少于三分之一在此经历了DLT),或定义为使进一步的剂量递增变得多余的免疫活性剂量(即免疫标记物的平稳期或免疫抑制的触发)。
次要终点
·代表T细胞激活和增殖的表面标志物CD3、CD4、CD8、CD19、CD25、CD39、CD45、CD56、CD73、FoxP3、Ki-67和ICAM-1的外周血IR分析。
探索终点
·与历史基因表达数据比较生成的RNA-seq数据的生物信息学分析。
·在减去合适的患者对照后,计算治疗过程中的响应细胞数。
·通过Luminex多重面板进行分析,以检测在用HS130和HS110治疗之前和之后促炎性血清细胞因子的变化。
·根据机构标准,通过临床和放射学评估来监测患者疾病状态。根据研究者评估,将患者的临床应答评估为完全应答、部分应答、稳定疾病或进行性疾病。
安全性:所有接受任意研究治疗组分的患者均认为可评估安全性。通过身体检查、体重、生命体征、表现状态、实验室评估(血液学,生物化学,包括细胞因子和C反应蛋白)、心电图(ECG)的方式以及记录并发疾病/治疗和不良事件,来评估安全性。使用CTCAE版本5对所有毒性进行分级。监视所有相关不良事件直至解决。在整个研究中,对患者的安全性和伴随用药进行监控。在每个剂量后监测一次细胞因子(例如IFNγ、IL-1β、IL-2、IL-4、IL-6、IL-10、IL-12p70、IL-17A、TNFα),并根据药物注射后临床上观察到的情况进行重复。
统计方法:所有分析都是描述性的。分类变量以数字和(如果有意义的话)百分比表示。连续变量由n、平均值、中位数、标准偏差和范围(最小和最大)合适地表示。以各剂量组表示。
功效:这是一项探索性试验,且因此未进行样本大小计算。
安全性群体:评估所有接受至少1个研究药物剂量的患者的安全性。实施例3:以“窄”剂量范围免疫小鼠HS-110(B16F10-OVA-gp96)和小鼠HS-130(B16F10-OVA-OX40L),以将CD8+T细胞扩增和肿瘤生长相关联
这个实施例的实验证明了产生第一和第二肿瘤特异性CD8+T细胞应答的小鼠HS-110(mHS-110;B16F10-OVA-gp96-Ig)和小鼠HS-130(mHS-130;B16F10-OVA-OX40L)的最佳比例,并证明体内的抗肿瘤T细胞扩增如何与肿瘤生长控制相关。
如在前实施例中所公开,HS-110是肺腺癌细胞系。HS-110分泌gp96-Ig,其呈递抗原以引发和扩展CD8+T细胞应答。临床前研究已表明,在表达特定抗原的细胞系统中添加分泌的T细胞共刺激分子OX40L-Ig与Gp96-Ig的组合,增强了T细胞免疫并导致肿瘤消除(Fromm G et al.,Cancer Immunol Res.2016Sep 2;4(9):766-78.)。因此,设计了仅分泌OX40L-Ig的小鼠细胞系,本文中称为“小鼠HS-130”或“mHS-130”,其与mHS-110组合使用(参见上述实施例1)。在以下实验中研究了用于生成第一和第二CD8+或CD4+T细胞池的mHS-110和mHS-130的最佳比例。这个实施例的实验进一步包括肿瘤激发臂,以更好地理解CD8+T细胞的体内扩增如何与抗肿瘤免疫应答和肿瘤生长控制相关联。
实验设计和方法
显示完整研究设计的图可以在图3中找到。
OT-1纯化、过继T细胞转移、mHS-110/mHS-130给药和流式细胞术染色:使用EasySep Mouse CD8 T细胞分离试剂盒(目录号19853A)从OT-I-GFP繁殖的小鼠中分离出T细胞受体(TCR)转基因小鼠CD8+(OT-I)细胞,并通过侧尾静脉静脉内(i.v)注射到每只C57BL/6小鼠中,100万个OT-I细胞悬浮于HBSS(GIBCO 14175-095)中。注射OT-I后两天,从所有小鼠尾部抽血作为基线,并在4小时后,分别用10μg/mL丝裂霉素-C(Sigma-Aldrich目录号M0503)处理mHS-110(B16F10-OVA-gp96-Ig)细胞和mHS-130(B16F10-OVA-OX40L-Ig)3小时,并相应地腹膜内(i.p.)施与各组。将小鼠分成7组,每组5只小鼠,除了亲本组有3只小鼠。根据图3和下表2中所示的设计,基于gp96-Ig或OX40L-Ig的纳克表达水平(每24小时每10
表2:动物给药设计
在免疫后第3、5、7、10和14天,以及在加强免疫后第17、19、21、24、28、33、38和41天,从小鼠尾部连续抽血至肝素化PBS(10个单位/ml)中,并使用ACK裂解缓冲液(150mMNH
细胞内细胞因子染色和流式细胞术:将脾细胞(1×10
B16F10-OVA肿瘤激发和体积计算:收获黑色素瘤B16F10细胞,并以5×10
肿瘤浸润淋巴细胞(TIL)的肿瘤组织消化:MACS Miltenyl Biotec肿瘤分离试剂盒用于这个步骤(目录号130096-730)。
ELISPOT测定法:收获脾细胞,并使用红细胞裂解缓冲液(目录号36858500,Roche)消除红细胞。将细胞在IMDM培养基中洗涤并沉淀。将所计数的细胞重悬于含10%FBS的IMDM中。每个ELISPOT孔接收总体积为200μl的100万个细胞。治疗包括使用10倍稀释的B16F10-OVA亲本系裂解物(1000万个细胞的小瓶;冻融3次),B16F10肿瘤的免疫优势表位:“gp100/pmel”(EGSRNQDWL(SEQ ID NO:38))、“TRP-1/gp75”(TWHRYHLL(SEQ ID NO:39))、TRP-2(SVYDFFVWL(SEQ ID NO:40)),以及OVA的H2
如先前所公布(Klinman DM et al.,Current Protocols inImmunology.1994June(1):6.19.1-6.19.8)地进行干扰素-γ捕获ELISPOT测定。简而言之,用在PBS盐水中的10μg/mL的抗小鼠IFN-γ纯化单克隆抗体(克隆AN18)以50μl/孔在4℃下包被MAIP N45 Milipore 96孔滤板过夜。第二天,将细胞用200μl/孔的IMEM培养基加10%FBS洗涤,洗涤3次,然后在25℃下封闭1小时。如上所述地处理细胞,并将100万或100,000个脾细胞添加到每个孔中,并按照以上描述用各种肽刺激进行处理。将每个平板在37℃、5%CO2下于非振动培养箱中孵育16小时或过夜。孵育结束时,将细胞轻轻拍打至水槽中以停止培养,并用PBS+0.1%Tween-20洗涤3次,并在纸巾上用力吸干以除去在最后一次洗涤后残留的液体。生物素化二抗以每孔50μl添加,4μg/mL抗IFN-γ(克隆AN18),在25℃下孵育2小时。然后将板用PBS+0.1%Tween-20再洗涤3次。然后添加来自杰克逊研究实验室的过氧化物酶偶联的链霉亲和素,在PBS+0.1%Tween-20+2%BSA中从储备液浓度稀释1至1000倍,每孔50μl,在4℃下孵育30分钟。然后用PBS+0.1%Tween洗涤平板3次。去除ELISPOT平板的塑料背衬,并将整个平板在25℃下浸没并浸泡在PBS+0.1%Tween-20中1小时。然后将平板在PBS中洗涤以去除tween,然后每个孔中添加在pH 8.2的100mM TRIS缓冲液中的100μl AEC底物(Vector试剂盒),以允许斑点显色。在孵育结束时,10-20分钟,所有平板孵育相同时间,通过用自来水冲洗平板并风干来停止反应。
在AID Autoimmun Diagnostika GMBH iSpot单色ELISPOT读取器(2018)中扫描和计数平板。每个平板的计数参数在所有一起显色的平板上保持一致,以防止偏差,因为斑点的显色、密度和量级受操作员、试剂批号和最终底物显色时间的影响。
实验结果
将表达Vα2/Vβ5并且对H-2K
疫苗接种后,在第0、3、5、7、10和14天(代表CD8+T细胞应答的急性扩增期和收缩期)在血液中分析OT-I GFP+CD8+T细胞。基于转移细胞的积累,发现主要效应池的生成在第7天达到峰值(图4,图6A,图6B),并在疫苗接种后不晚于第14天收缩(图6A,图6B)。第7天,在1:1、1:3和1:10比例的mHS-110和mHS-130中发现显著增加(图6A,图6B)(*p<0.05;**p<0.01)。此外,疫苗接种后第10天和第14天,1:1比例具有显著增加(*p<0.05)。
在这个分析后,在第14天施与加强免疫接种,并在第17、19、21、24和28天(代表CD8+T细胞应答的第二扩增期和收缩期)在血液中分析OT-I GFP+CD8+T细胞(图5,图6A,图6B)。基于转移细胞的积累,发现记忆效应池的生成达到峰值并在激发后不晚于第21天以百分比增加,而在激发后不晚于第28天收缩。在第21、24和28天,在1:1比例的mHS-110和mHS-130中发现显著增加(图6A,图6B)(*p<0.05)。
随着mHS-110和mHS-130的每次后续加强,抗原特异性T细胞的扩增变少。在第28天以每只小鼠500,000个B16F10-OVA细胞s.c.施与肿瘤,并在第31天施与另一加强的mHS-110和mHS-130。这导致抗原特异性CD8+T细胞的非显著增加(图6A,图6B),然而在未去除离群值的情况下,1:1比例提供了最佳和最一致的对疫苗再激发的应答(图6B)。
对于内源性应答,在第54天研究结束时,CD8+T细胞百分比仅在那些显示最大抗肿瘤应答的比例组(1:1和1:10)中升高(图7A),与仅mHS-110相比,*p<0.05。用B16F10、H2K
使用来自脾细胞的研究结束时的ELISPOT检查了对疫苗接种和肿瘤负荷的内源性免疫应答。与所报告的流式细胞结果和肿瘤生长相一致,对肿瘤裂解物、各种B16F10免疫优势肽和OVA的应答均遵循所预测的比例,并且似乎依赖于mHS-130的存在(图7D)。通过Mann-Whitney非参数统计检验,与仅mHS-110相比,对各种刺激剂的应答是显著的。
关于流式细胞图,内源性CD4+T细胞以1:1和1:10比例剂量组的频率扩增,表明需要Th1帮助以在体内产生抗肿瘤细胞毒性T淋巴细胞(图7E)。还测量了效应(CD62L
在第54天研究结束时,对每只动物的每个剩余的肿瘤块进行肿瘤浸润淋巴细胞(TIL)的定量。1:1和1:10组中的CD8+TIL的占比分别是mHS-110和mHS-13以01:0.1疫苗接种比例的51倍和118倍(图8A,图8B)。1:1组有2只动物未建立稳定的肿瘤(完全的肿瘤生长抑制),并且1:10组有3只动物具有完全的肿瘤生长抑制。由于这个原因,使用少于三个样本无法进行对TIL百分比的合适统计,因此1:10组没有资格进行统计学分析。然而,将1:1组与仅mHS-110进行比较(图8A,图8B;*p<0.05),观察到在这些肿瘤中发现显著更多的CD8+TIL,并且与1:0.1组比较时也是如此。在这两种情况下,CD8+TIL的占比呈剂量依赖性增加,其中疫苗接种比例为1:1和1:10比例时产生最佳应答。
从肿瘤细胞接种时间点到研究结束,随时间测量肿瘤体积,共25天的对数生长。由于这是一项保护性、预防性研究,故测量肿瘤延迟,而不是对已建立肿瘤的治疗性应答。如图10A所示,仅有两个比例,1:1(290ng的gp96-Ig比290ng OX40L-Ig)和1:10(290ng的gp96-Ig比2900ng的OX40L-Ig)的mHS-110和mHS-130,与仅mHS-110组相比具有一致且显著的肿瘤生长抑制,其中在第21-25天观察到最大分离(**p<0.01)。图10B中显示了个体肿瘤生长曲线,以显示个体动物差异。测量研究结束时的肿瘤质量证实了肿瘤体积已证明的,即与仅mHS-110相比,1:1和1:10比例均给出最佳应答(图9)。对于肿瘤体积,1:1和1:10比例无显著不同。
在1:1剂量比例组中观察到,肿瘤生长抑制、离体抗肿瘤T细胞应答和TIL浸润,与CD8+T细胞上的PD-1+表达减少相关(图12A,图12B;*p<0.05)。尽管与仅mHS-110组无显著不同,但1:10比例也显示出PD-1表达的下降趋势。PD-1的表达在初始T细胞激活期间发生,但在持续的重新刺激下或同源抗原存在下,PD-1由耗竭的T细胞表达(Simon S.et al.,Oncoimmunology.2018September 7(1):e1364828)。在那些具有最大肿瘤负荷的组(1:0.1、仅mHS-110和仅亲本B16F10-OVA)中,可见PD-1表达升高,这表明细胞已经因持续的抗肿瘤斗争而耗竭,然而那些已通过mHS-130疫苗接种接受了适当的OX40L刺激的细胞,则显示出较低的PD-1表达和较少的肿瘤负担。这可以解释为何1:1和1:10比例剂量组在脾中显示最低的PD-1表达。还通过观察TIL测量了PD-1在肿瘤中的表达,但频率低于定量的限度,使得分析变得困难(数据未显示)。
在研究结束时,测量了剩余组在第54天的脾OT-1频率和绝对计数。仅1:1比例产生的长期存活的循环OT-1细胞超出所有其他所检测的组(图11A,图11B;*p<0.05)。这个数据有力地表明,1:1比例的gp96-Ig和OX40L-Ig对于即时和长期免疫应答来说均产生最佳的抗肿瘤T细胞扩增应答,并且这与体内肿瘤生长抑制直接有关。
实施例4:gp96-Ig(mHS-110)和OX40L-Ig(mHS-130)的剂量比例的研究
在这个实施例中,进行实验以确定产生CD8+T细胞扩增和肿瘤生长抑制的最佳gp96-Ig(mHS-110)和OX40L-Ig(mHS-130)剂量比例。HS-110是分泌gp96-Ig的肺腺癌细胞系,其呈递抗原以引发和扩展CD8+T细胞应答。在本实施例中,开发了仅分泌OX40L-Ig的小鼠细胞系(mHS-130)。
在本研究中,将mHS-130(分泌gp96-Ig融合蛋白)与mHS-110(分泌OX40L-Ig融合蛋白)组合使用。研究目的是确定最适合产生第一和第二CD8+或CD4+T细胞池的mHS-110与mHS-130的比例。因此,这个研究以剂量递增的方式在荷瘤动物中测试了可变比例,以确定导致最有效的CD8+T细胞扩增和肿瘤生长抑制组合的比例和剂量。
实验设计和方法
图13显示了本研究的设计。
OT-1纯化、过继T细胞转移、mHS-110/mHS-130给药和流式细胞术染色
使用Easy Sep Mouse CD8 T细胞分离试剂盒(目录号19853A)从内部繁殖的OT-I-GFP小鼠中分离出T细胞受体(TCR)转基因小鼠CD8+(OT-I)细胞,并通过侧尾静脉静脉内(i.v.)注射到每只C57BL/6小鼠中,100万个OT-I细胞悬浮于HBSS(GIBCO 14175-095)中。注射OT-I后两天,从所有小鼠尾部抽血作为基线,并在4小时后,分别用10μg/mL丝裂霉素-C(Sigma-Aldrich目录号M0503)处理mHS-110(B16F10-OVA-gp96-Ig)细胞和mHS-130(B16F10-OVA-OX40L-Ig)3小时,并相应地腹膜内(i.p.)施予各组。将小鼠分为10组,每组5只小鼠。在每个比例组内提供剂量递增的三个不同的治疗比例,并全部与仅mHS-110(gp96-Ig)的对照组(比例1:0)进行比较。基于gp96-Ig或OX40L-Ig的纳克表达水平(以ng/10
mHS-110和mHS-130的细胞系蛋白表达数据
通过ELISA确定由mHS-110细胞表达的鼠gp96蛋白的量。对于每个待测样品,将一百万个B16F10-Ova9亲本细胞和mHS-110细胞以每个1ml的总体积接种在6孔组织培养平板中。将细胞在37℃和5%CO
为了进行ELISA,用在碳酸盐-碳酸氢盐缓冲液中的2μg/ml绵羊抗gp96(R&DSystems,目录号AF7606)包被96孔板(Corning,目录号9018)。将平板密封并储存在4℃下过夜。然后将平板用1X TBST(VWR,目录号K873)洗涤4次,然后在室温下用1X酪蛋白溶液(Sigma-Aldrich,目录号B6429)封闭1小时。然后将平板用1X TBST洗涤4次,并在含10%FBS(Gibco,目录号10082-147)的IMDM(Gibco,目录号12440-053)中制备人gp96-小鼠Fc标准品(Thermo Fisher Scientific,批号2065447)。制备2000ng/ml的人gp96-mFc标准溶液,并进行2倍连续稀释至1.95ng/ml。将样品上清液上样到ELISA平板上,开始于1:2稀释,然后进行2倍连续稀释至最高稀释度1:16。将平板密封并在室温下孵育1小时,并然后用1X TBST洗涤4次。将检测抗体山羊抗小鼠IgG(Fc)-HRP(Jackson Immunoresearch,目录号115-036-008)在1X TBST中1:5,000稀释,并添加到ELISA平板。然后将平板密封并在室温下于黑暗中孵育1小时。用1X TBST将平板洗涤4次后,将TMB底物(SeraCare,目录号5120-0076)添加到每个孔中,并在室温下于黑暗中孵育15分钟。然后用1N硫酸终止反应,并在Biotek ELx800读板仪上读取平板。然后根据标准曲线确定从每个样品表达的gp96浓度。
还通过ELISA确定了由mHS-130细胞表达的小鼠OX40L蛋白的量。对于每个待测样品,将一百万个B16F10-Ova9亲本细胞和mHS-130细胞以每个1ml的总体积接种在6孔组织培养平板中。将细胞在37℃和5%CO
为了进行ELISA,用在PBS中的2.5ug/ml His标记的小鼠OX40蛋白(AcroBiosystems,目录号OX0-M5228)包被96孔板。将平板密封并储存在4℃下过夜。然后将平板用1X TBST洗涤4次,然后在室温下用1%BSA(Sigma-Aldrich,目录号A2153)封闭1小时。然后将平板用1X TBST洗涤4次,并在含10%FBS的IMDM中制备小鼠IgG1小鼠OX40L标准品(Thermo Fisher Scientific,批号2214217)。制备2000ng/ml的mIgG1-mOX40L标准溶液,并进行2倍连续稀释至1.95ng/ml。将样品上清液上样到ELISA平板上,并进行2倍连续稀释。将平板密封并在37℃下孵育90分钟,并然后用1X TBST洗涤4次。将检测抗体山羊抗小鼠IgG(Fc)-HRP(Jackson Immunoresearch,目录号115-036-008)在1X PBS/0.05%Tween 20/0.1%BSA中1:5000稀释并添加到ELISA平板中。然后将平板密封并在室温下于黑暗中孵育1小时。用1X TBST将平板洗涤4次后,将TMB底物添加到每个孔中,并在室温下于黑暗中孵育10分钟。然后用1N硫酸终止反应,并在Biotek ELx800读板仪上读取平板。然后根据标准曲线确定从每个样品表达的OX40L浓度。这个规程基于人OX40L规程。
表3:分别来自mHS-110和mHS-130的小鼠gp96-Ig和OX40L-Ig的表达,24小时内每百万个细胞
小鼠gp96-Ig(24小时中每百万个细胞的纳克/mL)
小鼠OX40L-Ig(24小时中每百万个细胞的纳克/mL)
表4:每组中每种细胞类型的活性生物蛋白的表达水平和所用比例
在免疫后第3、5、7、10、12和14天以及第17、19、21、24、26、28、33、38、41、45、48和54天,从小鼠尾部连续抽血至肝素化PBS(10个单位/ml)中,并使用ACK裂解缓冲液(150mMNH
细胞内细胞因子染色和流式细胞术:将脾细胞(1×10
B16F10-OVA的肿瘤激发和体积计算:收获黑色素瘤B16F10细胞,并以5×10
肿瘤浸润淋巴细胞(TIL)的肿瘤组织消化:MACS Miltenyl Biotec肿瘤分离试剂盒用于这个步骤(目录号130096-730)。
ELISPOT测定法:收获脾细胞,并使用红细胞裂解缓冲液(目录号36858500,Roche)消除红细胞。将细胞在IMDM培养基中洗涤并沉淀。将所计数的细胞重悬于含10%FBS的IMDM中。每个ELISPOT孔接收总体积为200μl的100万个细胞。治疗包括使用10倍稀释的B16F10-OVA亲本系裂解物(1000万个细胞的小瓶;冻融3次),B16F10肿瘤的免疫优势表位:“gp100/pmel”(EGSRNQDWL(SEQ ID NO:38))、“TRP-1/gp75”(TWHRYHLL(SEQ ID NO:39))、TRP-2(SVYDFFVWL(SEQ ID NO:40)),以及OVA的H2
如先前所公布(Klinman DM et al.,Current Protocols inImmunology.1994June(1):6.19.1-6.19.8)地进行干扰素-γ捕获ELISPOT测定。简而言之,用在PBS盐水中的10μg/mL的抗小鼠IFN-γ纯化单克隆抗体(克隆AN18)以50μl/孔在4℃下包被MAIP N45 Milipore 96孔滤板过夜。第二天,将细胞用200μl/孔的含10%FBS的IMEM培养基洗涤,洗涤3次,然后在25℃下封闭1小时。如上所述地处理细胞,并将100万或100,000个脾细胞添加到每个孔中,并按照以上描述用各种肽刺激进行处理。将每个平板在37℃、5%CO
在AID Autoimmun Diagnostika GMBH iSpot单色ELISPOT读取器(2018)中扫描和计数平板。每个平板的计数参数在所有一起显色的平板上保持一致,以防止偏差,因为斑点的显色、密度和量级受操作员、试剂批号和最终底物显色时间的影响。
实验结果
如图14、15A、15B、15C和15D所示,用mHS-110(gp96-Ig)和mHS-130(OX40L-Ig)进行引发产生了第一免疫应答和OT-1细胞(抗OVA,CD8+TCR转基因T细胞)的剂量和比例依赖性的细胞扩增。
图14显示了使用mHS-110和mHS-130的不同比例和剂量组合的初始和加强免疫的外周血中的抗肿瘤CD8+OT-I T细胞扩增。向接受者小鼠注射不同gp96-Ig:OX40L-Ig比例和剂量的mHS-110和mHS-130。在疫苗接种后第0-54天,在血液中分析OT-I GFP+CD8+T细胞。在第14天用与初始阶段相同的mHS-110和mHS-130比例加强小鼠,并在激发后的日子在血液中分析OT-I GFP+CD8+T细胞。如所示,1:1.3比例的mHS-110与mHS-130(高剂量)在第7天达到CD8+OT-I T细胞扩增的峰值,并且直到第54天仍保持高于其他剂量比例。
图15A显示了图13研究的流式细胞实验的设门策略。向接受者小鼠注射不同gp96-Ig:OX40L-Ig比例和剂量的mHS-110和mHS-130。疫苗接种后第0-54天,在血液中分析了OT-IGFP+CD8+T细胞。在第14天和第31天用与初始阶段相同比例的mHS-110和mHS-130加强小鼠,并在激发后的日子在血液中分析OT-I GFP+CD8+T细胞。在第28天提供肿瘤。
图15B显示了OT-1细胞在第7天和第17天的扩增,显示剂量比例1:1.3提供了最佳应答,其中339ng gp96比441ng OX40L的高剂量可见最大扩增。尤其是在初次免疫后,将OX40L添加到gp96(38ng gp96相比于50ng的组;比例1:1.3)后,与仅mHS-110(38ng gp96)相比,OT-1细胞显著增加近三倍,如图15B所示。如图15B中的第7天所示,OT-1细胞的扩增存在减少趋势,这与OX40L-Ig:gp96-Ig的增加间接相关。在其他天数(从第7天到第26天)观察到这个趋势,但在第7天的初次免疫峰最为明显。在第14天的加强导致细胞扩增增加,并且在所研究的剂量比例组中,对于1:1.3剂量,观察到的扩增不显著(图15B,第17天)。刚好在第14天加强之前含有最多OT-1细胞的组,在第17天至第26天显示出最佳、最快的应答,其中在比例和组之间具有相同剂量趋势,尤其是其与OX40L-I相关联,如图15C所示。1:1.3比例的gp96-Ig与OX40L-Ig维持了CD8+OT-I T细胞的最佳扩增,直至研究结束,如图15D所示,显示了第45、48和54天的结果。
在本研究中,还对所研究的外周血群体测量了激活和关键记忆标志物。对记忆标志物KLRG1和IL-7R的流式细胞设门策略显示在图16中。另外,如图17所示,在第7天在内源性细胞群体中观察到CD8+记忆前体效应细胞(MPEC)(KLRG1
另外,与图15B-15D中观察到的OT-1T细胞扩增类似,更多OX40L比更少gp96刺激产生了更稳健的细胞应答,导致更多的SLEC形成(图17,第7天,内源性CD8+T细胞)。SLEC的形成与激活增加相关,如内源性CD8+T细胞群体上粘附分子CD44的上调所示(图18,第7天)。出现在抗原刺激的T细胞上以允许进入靶组织的CD44表达在内源性群体上大幅升高,并且随着OX40L剂量水平相对于gp96的增加而呈现趋势。
在第28天,皮下施与B16F10-OVA肿瘤,并开始肿瘤延迟激发。每个所测试的组的肿瘤生长抑制在图19中显示。观察到,通过单向ANOVA统计学检验测量,与仅mHS-110(38nggp96-Ig)相比,339ng:441ng组(1:1.3,高,gp96:OX40L)、339ng:848ng(1:2.5,高,gp96:OX40L)和339ng:1695ng(1:5,高,gp96:OX40L)中存在显著差异(图19,****p<0.0001)。研究结束(第55天)时,分组(图20,左)和个体(图20,右)的肿瘤质量均与卡尺测量(图19)相一致。
肿瘤特异性TRP2四聚阳性CD8+T细胞在治疗下以剂量依赖性方式增加,并且显示出显著增加超出用仅mHS-110的38ng的gp96-Ig的治疗(图21)。对于第55天的转移的CD8+OT-1+eGFP细胞,仅1:1.3比例的339ng gp96-Ig:441ng OX40L-Ig的剂量(高剂量)显示出第55天在脾和血液中均存在扩增的亚群,如图22所示。
图23显示了在第55天,脾中对PD-1染色呈阳性的耗竭的CD4+T细胞百分比(CD3+CD4+PD-1+T细胞的百分比)增加。如图24所示,所治疗的小鼠和荷瘤小鼠的脾中效应记忆CD4+T细胞百分比随接种以OX40L剂量依赖性方式变化。这种激活的CD4+T细胞百分比的增加与浸润肿瘤的CD4+T细胞(TIL)百分比的增加相关联,并且是剂量依赖性的(参见图26)。
本研究证明1)CD4+和CD8+T细胞亚群均在gp96/OX40L-Ig共同接种下扩增,这与增加的TIL百分比和肿瘤生长抑制相关联;2)肿瘤特异性CD8+T细胞的长期存活和扩增的最佳剂量组合是1:1.3比例的339ng的gp96-Ig:441ng的OX40L-Ig的剂量;和3)剂量决定肿瘤生长抑制,在小鼠中,38ng的gp96-Ig:50ng的OX40L-Ig未观察到作用水平(NOEL),且113nggp96-Ig:147ng OX40L-Ig是这个剂量组合的最小活性生物学效应水平(MABEL)。
其他实施方案
应当理解,尽管已经结合本公开的详细描述描述了本公开,但在前描述旨在说明而不是限制本公开的范围,本公开的范围由所附权利要求的范围定义。其他方面、优点和修改在以下权利要求的范围内。
参考文献的并入
本文引用的所有专利和出版物均通过引用以其全文并入本文。仅提供其公开先于本申请的提交日期的出版物用于本文讨论。本文中的任何内容均不应解释为承认本发明无权凭借在先发明而早于此类出版物。如本文所用,所有标题仅用于组织,而不旨在以任何方式限制本公开。
序列表
<110> 热生物制品有限公司(HEAT BIOLOGICS, INC.)
<120> 基于细胞的组合疗法
<130> 114358-5016
<150> 62/807,783
<151> 2019-02-20
<150> 62/739,814
<151> 2018-10-01
<160> 41
<170> PatentIn version 3.5
<210> 1
<211> 2412
<212> DNA
<213> 人(Homo sapiens)
<400> 1
atgagggccc tgtgggtgct gggcctctgc tgcgtcctgc tgaccttcgg gtcggtcaga 60
gctgacgatg aagttgatgt ggatggtaca gtagaagagg atctgggtaa aagtagagaa 120
ggatcaagga cggatgatga agtagtacag agagaggaag aagctattca gttggatgga 180
ttaaatgcat cacaaataag agaacttaga gagaagtcgg aaaagtttgc cttccaagcc 240
gaagttaaca gaatgatgaa acttatcatc aattcattgt ataaaaataa agagattttc 300
ctgagagaac tgatttcaaa tgcttctgat gctttagata agataaggct aatatcactg 360
actgatgaaa atgctctttc tggaaatgag gaactaacag tcaaaattaa gtgtgataag 420
gagaagaacc tgctgcatgt cacagacacc ggtgtaggaa tgaccagaga agagttggtt 480
aaaaaccttg gtaccatagc caaatctggg acaagcgagt ttttaaacaa aatgactgaa 540
gcacaggaag atggccagtc aacttctgaa ttgattggcc agtttggtgt cggtttctat 600
tccgccttcc ttgtagcaga taaggttatt gtcacttcaa aacacaacaa cgatacccag 660
cacatctggg agtctgactc caatgaattt tctgtaattg ctgacccaag aggaaacact 720
ctaggacggg gaacgacaat tacccttgtc ttaaaagaag aagcatctga ttaccttgaa 780
ttggatacaa ttaaaaatct cgtcaaaaaa tattcacagt tcataaactt tcctatttat 840
gtatggagca gcaagactga aactgttgag gagcccatgg aggaagaaga agcagccaaa 900
gaagagaaag aagaatctga tgatgaagct gcagtagagg aagaagaaga agaaaagaaa 960
ccaaagacta aaaaagttga aaaaactgtc tgggactggg aacttatgaa tgatatcaaa 1020
ccaatatggc agagaccatc aaaagaagta gaagaagatg aatacaaagc tttctacaaa 1080
tcattttcaa aggaaagtga tgaccccatg gcttatattc actttactgc tgaaggggaa 1140
gttaccttca aatcaatttt atttgtaccc acatctgctc cacgtggtct gtttgacgaa 1200
tatggatcta aaaagagcga ttacattaag ctctatgtgc gccgtgtatt catcacagac 1260
gacttccatg atatgatgcc taaatacctc aattttgtca agggtgtggt ggactcagat 1320
gatctcccct tgaatgtttc ccgcgagact cttcagcaac ataaactgct taaggtgatt 1380
aggaagaagc ttgttcgtaa aacgctggac atgatcaaga agattgctga tgataaatac 1440
aatgatactt tttggaaaga atttggtacc aacatcaagc ttggtgtgat tgaagaccac 1500
tcgaatcgaa cacgtcttgc taaacttctt aggttccagt cttctcatca tccaactgac 1560
attactagcc tagaccagta tgtggaaaga atgaaggaaa aacaagacaa aatctacttc 1620
atggctgggt ccagcagaaa agaggctgaa tcttctccat ttgttgagcg acttctgaaa 1680
aagggctatg aagttattta cctcacagaa cctgtggatg aatactgtat tcaggccctt 1740
cccgaatttg atgggaagag gttccagaat gttgccaagg aaggagtgaa gttcgatgaa 1800
agtgagaaaa ctaaggagag tcgtgaagca gttgagaaag aatttgagcc tctgctgaat 1860
tggatgaaag ataaagccct taaggacaag attgaaaagg ctgtggtgtc tcagcgcctg 1920
acagaatctc cgtgtgcttt ggtggccagc cagtacggat ggtctggcaa catggagaga 1980
atcatgaaag cacaagcgta ccaaacgggc aaggacatct ctacaaatta ctatgcgagt 2040
cagaagaaaa catttgaaat taatcccaga cacccgctga tcagagacat gcttcgacga 2100
attaaggaag atgaagatga taaaacagtt ttggatcttg ctgtggtttt gtttgaaaca 2160
gcaacgcttc ggtcagggta tcttttacca gacactaaag catatggaga tagaatagaa 2220
agaatgcttc gcctcagttt gaacattgac cctgatgcaa aggtggaaga agagcccgaa 2280
gaagaacctg aagagacagc agaagacaca acagaagaca cagagcaaga cgaagatgaa 2340
gaaatggatg tgggaacaga tgaagaagaa gaaacagcaa aggaatctac agctgaaaaa 2400
gatgaattgt aa 2412
<210> 2
<211> 803
<212> PRT
<213> 人(Homo sapiens)
<400> 2
Met Arg Ala Leu Trp Val Leu Gly Leu Cys Cys Val Leu Leu Thr Phe
1 5 10 15
Gly Ser Val Arg Ala Asp Asp Glu Val Asp Val Asp Gly Thr Val Glu
20 25 30
Glu Asp Leu Gly Lys Ser Arg Glu Gly Ser Arg Thr Asp Asp Glu Val
35 40 45
Val Gln Arg Glu Glu Glu Ala Ile Gln Leu Asp Gly Leu Asn Ala Ser
50 55 60
Gln Ile Arg Glu Leu Arg Glu Lys Ser Glu Lys Phe Ala Phe Gln Ala
65 70 75 80
Glu Val Asn Arg Met Met Lys Leu Ile Ile Asn Ser Leu Tyr Lys Asn
85 90 95
Lys Glu Ile Phe Leu Arg Glu Leu Ile Ser Asn Ala Ser Asp Ala Leu
100 105 110
Asp Lys Ile Arg Leu Ile Ser Leu Thr Asp Glu Asn Ala Leu Ser Gly
115 120 125
Asn Glu Glu Leu Thr Val Lys Ile Lys Cys Asp Lys Glu Lys Asn Leu
130 135 140
Leu His Val Thr Asp Thr Gly Val Gly Met Thr Arg Glu Glu Leu Val
145 150 155 160
Lys Asn Leu Gly Thr Ile Ala Lys Ser Gly Thr Ser Glu Phe Leu Asn
165 170 175
Lys Met Thr Glu Ala Gln Glu Asp Gly Gln Ser Thr Ser Glu Leu Ile
180 185 190
Gly Gln Phe Gly Val Gly Phe Tyr Ser Ala Phe Leu Val Ala Asp Lys
195 200 205
Val Ile Val Thr Ser Lys His Asn Asn Asp Thr Gln His Ile Trp Glu
210 215 220
Ser Asp Ser Asn Glu Phe Ser Val Ile Ala Asp Pro Arg Gly Asn Thr
225 230 235 240
Leu Gly Arg Gly Thr Thr Ile Thr Leu Val Leu Lys Glu Glu Ala Ser
245 250 255
Asp Tyr Leu Glu Leu Asp Thr Ile Lys Asn Leu Val Lys Lys Tyr Ser
260 265 270
Gln Phe Ile Asn Phe Pro Ile Tyr Val Trp Ser Ser Lys Thr Glu Thr
275 280 285
Val Glu Glu Pro Met Glu Glu Glu Glu Ala Ala Lys Glu Glu Lys Glu
290 295 300
Glu Ser Asp Asp Glu Ala Ala Val Glu Glu Glu Glu Glu Glu Lys Lys
305 310 315 320
Pro Lys Thr Lys Lys Val Glu Lys Thr Val Trp Asp Trp Glu Leu Met
325 330 335
Asn Asp Ile Lys Pro Ile Trp Gln Arg Pro Ser Lys Glu Val Glu Glu
340 345 350
Asp Glu Tyr Lys Ala Phe Tyr Lys Ser Phe Ser Lys Glu Ser Asp Asp
355 360 365
Pro Met Ala Tyr Ile His Phe Thr Ala Glu Gly Glu Val Thr Phe Lys
370 375 380
Ser Ile Leu Phe Val Pro Thr Ser Ala Pro Arg Gly Leu Phe Asp Glu
385 390 395 400
Tyr Gly Ser Lys Lys Ser Asp Tyr Ile Lys Leu Tyr Val Arg Arg Val
405 410 415
Phe Ile Thr Asp Asp Phe His Asp Met Met Pro Lys Tyr Leu Asn Phe
420 425 430
Val Lys Gly Val Val Asp Ser Asp Asp Leu Pro Leu Asn Val Ser Arg
435 440 445
Glu Thr Leu Gln Gln His Lys Leu Leu Lys Val Ile Arg Lys Lys Leu
450 455 460
Val Arg Lys Thr Leu Asp Met Ile Lys Lys Ile Ala Asp Asp Lys Tyr
465 470 475 480
Asn Asp Thr Phe Trp Lys Glu Phe Gly Thr Asn Ile Lys Leu Gly Val
485 490 495
Ile Glu Asp His Ser Asn Arg Thr Arg Leu Ala Lys Leu Leu Arg Phe
500 505 510
Gln Ser Ser His His Pro Thr Asp Ile Thr Ser Leu Asp Gln Tyr Val
515 520 525
Glu Arg Met Lys Glu Lys Gln Asp Lys Ile Tyr Phe Met Ala Gly Ser
530 535 540
Ser Arg Lys Glu Ala Glu Ser Ser Pro Phe Val Glu Arg Leu Leu Lys
545 550 555 560
Lys Gly Tyr Glu Val Ile Tyr Leu Thr Glu Pro Val Asp Glu Tyr Cys
565 570 575
Ile Gln Ala Leu Pro Glu Phe Asp Gly Lys Arg Phe Gln Asn Val Ala
580 585 590
Lys Glu Gly Val Lys Phe Asp Glu Ser Glu Lys Thr Lys Glu Ser Arg
595 600 605
Glu Ala Val Glu Lys Glu Phe Glu Pro Leu Leu Asn Trp Met Lys Asp
610 615 620
Lys Ala Leu Lys Asp Lys Ile Glu Lys Ala Val Val Ser Gln Arg Leu
625 630 635 640
Thr Glu Ser Pro Cys Ala Leu Val Ala Ser Gln Tyr Gly Trp Ser Gly
645 650 655
Asn Met Glu Arg Ile Met Lys Ala Gln Ala Tyr Gln Thr Gly Lys Asp
660 665 670
Ile Ser Thr Asn Tyr Tyr Ala Ser Gln Lys Lys Thr Phe Glu Ile Asn
675 680 685
Pro Arg His Pro Leu Ile Arg Asp Met Leu Arg Arg Ile Lys Glu Asp
690 695 700
Glu Asp Asp Lys Thr Val Leu Asp Leu Ala Val Val Leu Phe Glu Thr
705 710 715 720
Ala Thr Leu Arg Ser Gly Tyr Leu Leu Pro Asp Thr Lys Ala Tyr Gly
725 730 735
Asp Arg Ile Glu Arg Met Leu Arg Leu Ser Leu Asn Ile Asp Pro Asp
740 745 750
Ala Lys Val Glu Glu Glu Pro Glu Glu Glu Pro Glu Glu Thr Ala Glu
755 760 765
Asp Thr Thr Glu Asp Thr Glu Gln Asp Glu Asp Glu Glu Met Asp Val
770 775 780
Gly Thr Asp Glu Glu Glu Glu Thr Ala Lys Glu Ser Thr Ala Glu Lys
785 790 795 800
Asp Glu Leu
<210> 3
<211> 4
<212> PRT
<213> 人(Homo sapiens)
<400> 3
Lys Asp Glu Leu
1
<210> 4
<211> 1455
<212> DNA
<213> 人(Homo sapiens)
<400> 4
atgagactgg gaagccctgg cctgctgttt ctgctgttca gcagcctgag agccgacacc 60
caggaaaaag aagtgcgggc catggtggga agcgacgtgg aactgagctg cgcctgtcct 120
gagggcagca gattcgacct gaacgacgtg tacgtgtact ggcagaccag cgagagcaag 180
accgtcgtga cctaccacat cccccagaac agctccctgg aaaacgtgga cagccggtac 240
agaaaccggg ccctgatgtc tcctgccggc atgctgagag gcgacttcag cctgcggctg 300
ttcaacgtga ccccccagga cgagcagaaa ttccactgcc tggtgctgag ccagagcctg 360
ggcttccagg aagtgctgag cgtggaagtg accctgcacg tggccgccaa tttcagcgtg 420
ccagtggtgt ctgcccccca cagcccttct caggatgagc tgaccttcac ctgtaccagc 480
atcaacggct accccagacc caatgtgtac tggatcaaca agaccgacaa cagcctgctg 540
gaccaggccc tgcagaacga taccgtgttc ctgaacatgc ggggcctgta cgacgtggtg 600
tccgtgctga gaatcgccag aacccccagc gtgaacatcg gctgctgcat cgagaacgtg 660
ctgctgcagc agaacctgac cgtgggcagc cagaccggca acgacatcgg cgagagagac 720
aagatcaccg agaaccccgt gtccaccggc gagaagaatg ccgccacctc taagtacggc 780
cctccctgcc cttcttgccc agcccctgaa tttctgggcg gaccctccgt gtttctgttc 840
cccccaaagc ccaaggacac cctgatgatc agccggaccc ccgaagtgac ctgcgtggtg 900
gtggatgtgt cccaggaaga tcccgaggtg cagttcaatt ggtacgtgga cggggtggaa 960
gtgcacaacg ccaagaccaa gcccagagag gaacagttca acagcaccta ccgggtggtg 1020
tctgtgctga ccgtgctgca ccaggattgg ctgagcggca aagagtacaa gtgcaaggtg 1080
tccagcaagg gcctgcccag cagcatcgaa aagaccatca gcaacgccac cggccagccc 1140
agggaacccc aggtgtacac actgccccct agccaggaag agatgaccaa gaaccaggtg 1200
tccctgacct gtctcgtgaa gggcttctac ccctccgata tcgccgtgga atgggagagc 1260
aacggccagc cagagaacaa ctacaagacc acccccccag tgctggacag cgacggctca 1320
ttcttcctgt actcccggct gacagtggac aagagcagct ggcaggaagg caacgtgttc 1380
agctgcagcg tgatgcacga agccctgcac aaccactaca cccagaagtc cctgtctctg 1440
tccctgggca aatga 1455
<210> 5
<211> 484
<212> PRT
<213> 人(Homo sapiens)
<400> 5
Met Arg Leu Gly Ser Pro Gly Leu Leu Phe Leu Leu Phe Ser Ser Leu
1 5 10 15
Arg Ala Asp Thr Gln Glu Lys Glu Val Arg Ala Met Val Gly Ser Asp
20 25 30
Val Glu Leu Ser Cys Ala Cys Pro Glu Gly Ser Arg Phe Asp Leu Asn
35 40 45
Asp Val Tyr Val Tyr Trp Gln Thr Ser Glu Ser Lys Thr Val Val Thr
50 55 60
Tyr His Ile Pro Gln Asn Ser Ser Leu Glu Asn Val Asp Ser Arg Tyr
65 70 75 80
Arg Asn Arg Ala Leu Met Ser Pro Ala Gly Met Leu Arg Gly Asp Phe
85 90 95
Ser Leu Arg Leu Phe Asn Val Thr Pro Gln Asp Glu Gln Lys Phe His
100 105 110
Cys Leu Val Leu Ser Gln Ser Leu Gly Phe Gln Glu Val Leu Ser Val
115 120 125
Glu Val Thr Leu His Val Ala Ala Asn Phe Ser Val Pro Val Val Ser
130 135 140
Ala Pro His Ser Pro Ser Gln Asp Glu Leu Thr Phe Thr Cys Thr Ser
145 150 155 160
Ile Asn Gly Tyr Pro Arg Pro Asn Val Tyr Trp Ile Asn Lys Thr Asp
165 170 175
Asn Ser Leu Leu Asp Gln Ala Leu Gln Asn Asp Thr Val Phe Leu Asn
180 185 190
Met Arg Gly Leu Tyr Asp Val Val Ser Val Leu Arg Ile Ala Arg Thr
195 200 205
Pro Ser Val Asn Ile Gly Cys Cys Ile Glu Asn Val Leu Leu Gln Gln
210 215 220
Asn Leu Thr Val Gly Ser Gln Thr Gly Asn Asp Ile Gly Glu Arg Asp
225 230 235 240
Lys Ile Thr Glu Asn Pro Val Ser Thr Gly Glu Lys Asn Ala Ala Thr
245 250 255
Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe Leu
260 265 270
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
275 280 285
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
290 295 300
Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu
305 310 315 320
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr
325 330 335
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Ser
340 345 350
Gly Lys Glu Tyr Lys Cys Lys Val Ser Ser Lys Gly Leu Pro Ser Ser
355 360 365
Ile Glu Lys Thr Ile Ser Asn Ala Thr Gly Gln Pro Arg Glu Pro Gln
370 375 380
Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val
385 390 395 400
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
405 410 415
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
420 425 430
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr
435 440 445
Val Asp Lys Ser Ser Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val
450 455 460
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
465 470 475 480
Ser Leu Gly Lys
<210> 6
<211> 1305
<212> DNA
<213> 人(Homo sapiens)
<400> 6
atgtctaagt acggccctcc ctgccctagc tgccctgccc ctgaatttct gggcggaccc 60
agcgtgttcc tgttcccccc aaagcccaag gacaccctga tgatcagccg gacccccgaa 120
gtgacctgcg tggtggtgga tgtgtcccag gaagatcccg aggtgcagtt caattggtac 180
gtggacggcg tggaagtgca caacgccaag accaagccca gagaggaaca gttcaacagc 240
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgag cggcaaagag 300
tacaagtgca aggtgtccag caagggcctg cccagcagca tcgagaaaac catcagcaac 360
gccaccggcc agcccaggga accccaggtg tacacactgc cccctagcca ggaagagatg 420
accaagaacc aggtgtccct gacctgtctc gtgaagggct tctacccctc cgatatcgcc 480
gtggaatggg agagcaacgg ccagcctgag aacaactaca agaccacccc cccagtgctg 540
gacagcgacg gctcattctt cctgtacagc agactgaccg tggacaagag cagctggcag 600
gaaggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 660
aagtccctgt ctctgagcct gggcaaggcc tgtccatggg ctgtgtctgg cgctagagcc 720
tctcctggat ctgccgccag ccccagactg agagagggac ctgagctgag ccccgatgat 780
cctgccggac tgctggatct gagacagggc atgttcgccc agctggtggc ccagaacgtg 840
ctgctgatcg atggccccct gagctggtac agcgatcctg gactggctgg cgtgtcactg 900
acaggcggcc tgagctacaa agaggacacc aaagaactgg tggtggccaa ggccggcgtg 960
tactacgtgt tctttcagct ggaactgcgg agagtggtgg ccggcgaagg atccggctct 1020
gtgtctctgg ctctgcatct gcagcccctg agatctgctg ctggcgctgc tgctctggcc 1080
ctgacagtgg acctgcctcc tgcctctagc gaggccagaa acagcgcatt cgggtttcaa 1140
ggcagactgc tgcacctgtc tgccggccag agactgggag tgcatctgca cacagaggcc 1200
agagccaggc acgcctggca gctgactcag ggcgctacag tgctgggcct gttcagagtg 1260
acccccgaga ttccagccgg cctgcctagc cccagatccg aatga 1305
<210> 7
<211> 434
<212> PRT
<213> 人(Homo sapiens)
<400> 7
Met Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Ser Gly Lys Glu Tyr Lys Cys Lys Val Ser Ser Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Asn Ala Thr Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Ser Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys Ala Cys Pro Trp Ala Val Ser Gly Ala Arg Ala
225 230 235 240
Ser Pro Gly Ser Ala Ala Ser Pro Arg Leu Arg Glu Gly Pro Glu Leu
245 250 255
Ser Pro Asp Asp Pro Ala Gly Leu Leu Asp Leu Arg Gln Gly Met Phe
260 265 270
Ala Gln Leu Val Ala Gln Asn Val Leu Leu Ile Asp Gly Pro Leu Ser
275 280 285
Trp Tyr Ser Asp Pro Gly Leu Ala Gly Val Ser Leu Thr Gly Gly Leu
290 295 300
Ser Tyr Lys Glu Asp Thr Lys Glu Leu Val Val Ala Lys Ala Gly Val
305 310 315 320
Tyr Tyr Val Phe Phe Gln Leu Glu Leu Arg Arg Val Val Ala Gly Glu
325 330 335
Gly Ser Gly Ser Val Ser Leu Ala Leu His Leu Gln Pro Leu Arg Ser
340 345 350
Ala Ala Gly Ala Ala Ala Leu Ala Leu Thr Val Asp Leu Pro Pro Ala
355 360 365
Ser Ser Glu Ala Arg Asn Ser Ala Phe Gly Phe Gln Gly Arg Leu Leu
370 375 380
His Leu Ser Ala Gly Gln Arg Leu Gly Val His Leu His Thr Glu Ala
385 390 395 400
Arg Ala Arg His Ala Trp Gln Leu Thr Gln Gly Ala Thr Val Leu Gly
405 410 415
Leu Phe Arg Val Thr Pro Glu Ile Pro Ala Gly Leu Pro Ser Pro Arg
420 425 430
Ser Glu
<210> 8
<211> 1284
<212> DNA
<213> 人(Homo sapiens)
<400> 8
atgtctaagt acggccctcc ctgccctagc tgccctgccc ctgaatttct gggcggaccc 60
agcgtgttcc tgttcccccc aaagcccaag gacaccctga tgatcagccg gacccccgaa 120
gtgacctgcg tggtggtgga tgtgtcccag gaagatcccg aggtgcagtt caattggtac 180
gtggacggcg tggaagtgca caacgccaag accaagccca gagaggaaca gttcaacagc 240
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgag cggcaaagag 300
tacaagtgca aggtgtccag caagggcctg cccagcagca tcgagaaaac catcagcaac 360
gccaccggcc agcccaggga accccaggtg tacacactgc cccctagcca ggaagagatg 420
accaagaacc aggtgtccct gacctgtctc gtgaagggct tctacccctc cgatatcgcc 480
gtggaatggg agagcaacgg ccagcctgag aacaactaca agaccacccc cccagtgctg 540
gacagcgacg gctcattctt cctgtacagc agactgaccg tggacaagag cagctggcag 600
gaaggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 660
aagtccctgt ctctgagcct gggcaagatc gagggccgga tggatagagc ccagggcgaa 720
gcctgcgtgc agttccaggc tctgaagggc caggaattcg cccccagcca ccagcaggtg 780
tacgcccctc tgagagccga cggcgataag cctagagccc acctgacagt cgtgcggcag 840
acccctaccc agcacttcaa gaatcagttc cccgccctgc actgggagca cgaactgggc 900
ctggccttca ccaagaacag aatgaactac accaacaagt ttctgctgat ccccgagagc 960
ggcgactact tcatctacag ccaagtgacc ttccggggca tgaccagcga gtgcagcgag 1020
atcagacagg ccggcagacc taacaagccc gacagcatca ccgtcgtgat caccaaagtg 1080
accgacagct accccgagcc cacccagctg ctgatgggca ccaagagcgt gtgcgaagtg 1140
ggcagcaact ggttccagcc catctacctg ggcgccatgt ttagtctgca agagggcgac 1200
aagctgatgg tcaacgtgtc cgacatcagc ctggtggatt acaccaaaga ggacaagacc 1260
ttcttcggcg cctttctgct ctga 1284
<210> 9
<211> 427
<212> PRT
<213> 人(Homo sapiens)
<400> 9
Met Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Ser Gly Lys Glu Tyr Lys Cys Lys Val Ser Ser Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Asn Ala Thr Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Ser Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys Ile Glu Gly Arg Met Asp Arg Ala Gln Gly Glu
225 230 235 240
Ala Cys Val Gln Phe Gln Ala Leu Lys Gly Gln Glu Phe Ala Pro Ser
245 250 255
His Gln Gln Val Tyr Ala Pro Leu Arg Ala Asp Gly Asp Lys Pro Arg
260 265 270
Ala His Leu Thr Val Val Arg Gln Thr Pro Thr Gln His Phe Lys Asn
275 280 285
Gln Phe Pro Ala Leu His Trp Glu His Glu Leu Gly Leu Ala Phe Thr
290 295 300
Lys Asn Arg Met Asn Tyr Thr Asn Lys Phe Leu Leu Ile Pro Glu Ser
305 310 315 320
Gly Asp Tyr Phe Ile Tyr Ser Gln Val Thr Phe Arg Gly Met Thr Ser
325 330 335
Glu Cys Ser Glu Ile Arg Gln Ala Gly Arg Pro Asn Lys Pro Asp Ser
340 345 350
Ile Thr Val Val Ile Thr Lys Val Thr Asp Ser Tyr Pro Glu Pro Thr
355 360 365
Gln Leu Leu Met Gly Thr Lys Ser Val Cys Glu Val Gly Ser Asn Trp
370 375 380
Phe Gln Pro Ile Tyr Leu Gly Ala Met Phe Ser Leu Gln Glu Gly Asp
385 390 395 400
Lys Leu Met Val Asn Val Ser Asp Ile Ser Leu Val Asp Tyr Thr Lys
405 410 415
Glu Asp Lys Thr Phe Phe Gly Ala Phe Leu Leu
420 425
<210> 10
<211> 1107
<212> DNA
<213> 人(Homo sapiens)
<400> 10
atgtctaagt acggccctcc ctgccctagc tgccctgccc ctgaatttct gggcggaccc 60
agcgtgttcc tgttcccccc aaagcccaag gacaccctga tgatcagccg gacccccgaa 120
gtgacctgcg tggtggtgga tgtgtcccag gaagatcccg aggtgcagtt caattggtac 180
gtggacggcg tggaagtgca caacgccaag accaagccca gagaggaaca gttcaacagc 240
acctaccggg tggtgtccgt gctgaccgtg ctgcaccagg attggctgag cggcaaagag 300
tacaagtgca aggtgtccag caagggcctg cccagcagca tcgagaaaac catcagcaac 360
gccaccggcc agcccaggga accccaggtg tacacactgc cccctagcca ggaagagatg 420
accaagaacc aggtgtccct gacctgtctc gtgaagggct tctacccctc cgatatcgcc 480
gtggaatggg agagcaacgg ccagcctgag aacaactaca agaccacccc cccagtgctg 540
gacagcgacg gctcattctt cctgtacagc agactgaccg tggacaagag cagctggcag 600
gaaggcaacg tgttcagctg cagcgtgatg cacgaggccc tgcacaacca ctacacccag 660
aagtccctgt ctctgagcct gggcaagatc gagggccgga tggatcaggt gtcacacaga 720
tacccccgga tccagagcat caaagtgcag tttaccgagt acaagaaaga gaagggcttt 780
atcctgacca gccagaaaga ggacgagatc atgaaggtgc agaacaacag cgtgatcatc 840
aactgcgacg ggttctacct gatcagcctg aagggctact tcagtcagga agtgaacatc 900
agcctgcact accagaagga cgaggaaccc ctgttccagc tgaagaaagt gcggagcgtg 960
aacagcctga tggtggcctc tctgacctac aaggacaagg tgtacctgaa cgtgaccacc 1020
gacaacacca gcctggacga cttccacgtg aacggcggcg agctgatcct gattcaccag 1080
aaccccggcg agttctgcgt gctctga 1107
<210> 11
<211> 368
<212> PRT
<213> 人(Homo sapiens)
<400> 11
Met Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro Glu Phe
1 5 10 15
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
20 25 30
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
35 40 45
Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val
50 55 60
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser
65 70 75 80
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
85 90 95
Ser Gly Lys Glu Tyr Lys Cys Lys Val Ser Ser Lys Gly Leu Pro Ser
100 105 110
Ser Ile Glu Lys Thr Ile Ser Asn Ala Thr Gly Gln Pro Arg Glu Pro
115 120 125
Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln
130 135 140
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
145 150 155 160
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
165 170 175
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu
180 185 190
Thr Val Asp Lys Ser Ser Trp Gln Glu Gly Asn Val Phe Ser Cys Ser
195 200 205
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
210 215 220
Leu Ser Leu Gly Lys Ile Glu Gly Arg Met Asp Gln Val Ser His Arg
225 230 235 240
Tyr Pro Arg Ile Gln Ser Ile Lys Val Gln Phe Thr Glu Tyr Lys Lys
245 250 255
Glu Lys Gly Phe Ile Leu Thr Ser Gln Lys Glu Asp Glu Ile Met Lys
260 265 270
Val Gln Asn Asn Ser Val Ile Ile Asn Cys Asp Gly Phe Tyr Leu Ile
275 280 285
Ser Leu Lys Gly Tyr Phe Ser Gln Glu Val Asn Ile Ser Leu His Tyr
290 295 300
Gln Lys Asp Glu Glu Pro Leu Phe Gln Leu Lys Lys Val Arg Ser Val
305 310 315 320
Asn Ser Leu Met Val Ala Ser Leu Thr Tyr Lys Asp Lys Val Tyr Leu
325 330 335
Asn Val Thr Thr Asp Asn Thr Ser Leu Asp Asp Phe His Val Asn Gly
340 345 350
Gly Glu Leu Ile Leu Ile His Gln Asn Pro Gly Glu Phe Cys Val Leu
355 360 365
<210> 12
<211> 1588
<212> PRT
<213> 人(Homo sapiens)
<400> 12
Thr Cys Cys Cys Ala Ala Gly Thr Ala Gly Cys Thr Gly Gly Gly Ala
1 5 10 15
Cys Thr Ala Cys Ala Gly Gly Ala Gly Cys Cys Cys Ala Cys Cys Ala
20 25 30
Cys Cys Ala Cys Cys Cys Cys Cys Gly Gly Cys Thr Ala Ala Thr Thr
35 40 45
Thr Thr Thr Thr Gly Thr Ala Thr Thr Thr Thr Thr Ala Gly Thr Ala
50 55 60
Gly Ala Gly Ala Cys Gly Gly Gly Gly Thr Thr Thr Cys Ala Cys Cys
65 70 75 80
Gly Thr Gly Thr Thr Ala Gly Cys Cys Ala Ala Gly Ala Thr Gly Gly
85 90 95
Thr Cys Thr Thr Gly Ala Thr Cys Ala Cys Cys Thr Gly Ala Cys Cys
100 105 110
Thr Cys Gly Thr Gly Ala Thr Cys Cys Ala Cys Cys Cys Gly Cys Cys
115 120 125
Thr Thr Gly Gly Cys Cys Thr Cys Cys Cys Ala Ala Ala Gly Thr Gly
130 135 140
Cys Thr Gly Gly Gly Ala Thr Thr Ala Cys Ala Gly Gly Cys Ala Thr
145 150 155 160
Gly Ala Gly Cys Cys Ala Cys Cys Gly Cys Gly Cys Cys Cys Gly Gly
165 170 175
Cys Cys Thr Cys Cys Ala Thr Thr Cys Ala Ala Gly Thr Cys Thr Thr
180 185 190
Thr Ala Thr Thr Gly Ala Ala Thr Ala Thr Cys Thr Gly Cys Thr Ala
195 200 205
Thr Gly Thr Thr Cys Thr Ala Cys Ala Cys Ala Cys Thr Gly Thr Thr
210 215 220
Cys Thr Ala Gly Gly Thr Gly Cys Thr Gly Gly Gly Gly Ala Thr Gly
225 230 235 240
Cys Ala Ala Cys Ala Gly Gly Gly Gly Ala Cys Ala Ala Ala Ala Thr
245 250 255
Ala Gly Gly Cys Ala Ala Ala Ala Thr Cys Cys Cys Thr Gly Thr Cys
260 265 270
Cys Thr Thr Thr Thr Gly Gly Gly Gly Thr Thr Gly Ala Cys Ala Thr
275 280 285
Thr Cys Thr Ala Gly Thr Gly Ala Cys Thr Cys Thr Thr Cys Ala Thr
290 295 300
Gly Thr Ala Gly Thr Cys Thr Ala Gly Ala Ala Gly Ala Ala Gly Cys
305 310 315 320
Thr Cys Ala Gly Thr Gly Ala Ala Thr Ala Gly Thr Gly Thr Cys Thr
325 330 335
Gly Thr Gly Gly Thr Thr Gly Thr Thr Ala Cys Cys Ala Gly Gly Gly
340 345 350
Ala Cys Ala Cys Ala Ala Thr Gly Ala Cys Ala Gly Gly Ala Ala Cys
355 360 365
Ala Thr Thr Cys Thr Thr Gly Gly Gly Thr Ala Gly Ala Gly Thr Gly
370 375 380
Ala Gly Ala Gly Gly Cys Cys Thr Gly Gly Gly Gly Ala Gly Gly Gly
385 390 395 400
Ala Ala Gly Gly Gly Thr Cys Thr Cys Thr Ala Gly Gly Ala Thr Gly
405 410 415
Gly Ala Gly Cys Ala Gly Ala Thr Gly Cys Thr Gly Gly Gly Cys Ala
420 425 430
Gly Thr Cys Thr Thr Ala Gly Gly Gly Ala Gly Cys Cys Cys Cys Thr
435 440 445
Cys Cys Thr Gly Gly Cys Ala Thr Gly Cys Ala Cys Cys Cys Cys Cys
450 455 460
Thr Cys Ala Thr Cys Cys Cys Thr Cys Ala Gly Gly Cys Cys Ala Cys
465 470 475 480
Cys Cys Cys Cys Gly Thr Cys Cys Cys Thr Thr Gly Cys Ala Gly Gly
485 490 495
Ala Gly Cys Ala Cys Cys Cys Thr Gly Gly Gly Gly Ala Gly Cys Thr
500 505 510
Gly Thr Cys Cys Ala Gly Ala Gly Cys Gly Cys Thr Gly Thr Gly Cys
515 520 525
Cys Gly Cys Thr Gly Thr Cys Thr Gly Thr Gly Gly Cys Thr Gly Gly
530 535 540
Ala Gly Gly Cys Ala Gly Ala Gly Thr Ala Gly Gly Thr Gly Gly Thr
545 550 555 560
Gly Thr Gly Cys Thr Gly Gly Gly Ala Ala Thr Gly Cys Gly Ala Gly
565 570 575
Thr Gly Gly Gly Ala Gly Ala Ala Cys Thr Gly Gly Gly Ala Thr Gly
580 585 590
Gly Ala Cys Cys Gly Ala Gly Gly Gly Gly Ala Gly Gly Cys Gly Gly
595 600 605
Gly Thr Gly Ala Gly Gly Ala Gly Gly Gly Gly Gly Gly Cys Ala Ala
610 615 620
Cys Cys Ala Cys Cys Cys Ala Ala Cys Ala Cys Cys Cys Ala Cys Cys
625 630 635 640
Ala Gly Cys Thr Gly Cys Thr Thr Thr Cys Ala Gly Thr Gly Thr Thr
645 650 655
Cys Thr Gly Gly Gly Thr Cys Cys Ala Gly Gly Thr Gly Cys Thr Cys
660 665 670
Cys Thr Gly Gly Cys Thr Gly Gly Cys Cys Thr Thr Gly Thr Gly Gly
675 680 685
Thr Cys Cys Cys Cys Cys Thr Cys Cys Thr Gly Cys Thr Thr Gly Gly
690 695 700
Gly Gly Cys Cys Ala Cys Cys Cys Thr Gly Ala Cys Cys Thr Ala Cys
705 710 715 720
Ala Cys Ala Thr Ala Cys Cys Gly Cys Cys Ala Cys Thr Gly Cys Thr
725 730 735
Gly Gly Cys Cys Thr Cys Ala Cys Ala Ala Gly Cys Cys Cys Cys Thr
740 745 750
Gly Gly Thr Thr Ala Cys Thr Gly Cys Ala Gly Ala Thr Gly Ala Ala
755 760 765
Gly Cys Thr Gly Gly Gly Ala Thr Gly Gly Ala Gly Gly Cys Thr Cys
770 775 780
Thr Gly Ala Cys Cys Cys Cys Ala Cys Cys Ala Cys Cys Gly Gly Cys
785 790 795 800
Cys Ala Cys Cys Cys Ala Thr Cys Thr Gly Thr Cys Ala Cys Cys Cys
805 810 815
Thr Thr Gly Gly Ala Cys Ala Gly Cys Gly Cys Cys Cys Ala Cys Ala
820 825 830
Cys Cys Cys Thr Thr Cys Thr Ala Gly Cys Ala Cys Cys Thr Cys Cys
835 840 845
Thr Gly Ala Cys Ala Gly Cys Ala Gly Thr Gly Ala Gly Ala Ala Gly
850 855 860
Ala Thr Cys Thr Gly Cys Ala Cys Cys Gly Thr Cys Cys Ala Gly Thr
865 870 875 880
Thr Gly Gly Thr Gly Gly Gly Thr Ala Ala Cys Ala Gly Cys Thr Gly
885 890 895
Gly Ala Cys Cys Cys Cys Thr Gly Gly Cys Thr Ala Cys Cys Cys Cys
900 905 910
Gly Ala Gly Ala Cys Cys Cys Ala Gly Gly Ala Gly Gly Cys Gly Cys
915 920 925
Thr Cys Thr Gly Cys Cys Cys Gly Cys Ala Gly Gly Thr Gly Ala Cys
930 935 940
Ala Thr Gly Gly Thr Cys Cys Thr Gly Gly Gly Ala Cys Cys Ala Gly
945 950 955 960
Thr Thr Gly Cys Cys Cys Ala Gly Cys Ala Gly Ala Gly Cys Thr Cys
965 970 975
Thr Thr Gly Gly Cys Cys Cys Cys Gly Cys Thr Gly Cys Thr Gly Cys
980 985 990
Gly Cys Cys Cys Ala Cys Ala Cys Thr Cys Thr Cys Gly Cys Cys Ala
995 1000 1005
Gly Ala Gly Thr Cys Cys Cys Cys Ala Gly Cys Cys Gly Gly Cys
1010 1015 1020
Thr Cys Gly Cys Cys Ala Gly Cys Cys Ala Thr Gly Ala Thr Gly
1025 1030 1035
Cys Thr Gly Cys Ala Gly Cys Cys Gly Gly Gly Cys Cys Cys Gly
1040 1045 1050
Cys Ala Gly Cys Thr Cys Thr Ala Cys Gly Ala Cys Gly Thr Gly
1055 1060 1065
Ala Thr Gly Gly Ala Cys Gly Cys Gly Gly Thr Cys Cys Cys Ala
1070 1075 1080
Gly Cys Gly Cys Gly Gly Cys Gly Cys Thr Gly Gly Ala Ala Gly
1085 1090 1095
Gly Ala Gly Thr Thr Cys Gly Thr Gly Cys Gly Cys Ala Cys Gly
1100 1105 1110
Cys Thr Gly Gly Gly Gly Cys Thr Gly Cys Gly Cys Gly Ala Gly
1115 1120 1125
Gly Cys Ala Gly Ala Gly Ala Thr Cys Gly Ala Ala Gly Cys Cys
1130 1135 1140
Gly Thr Gly Gly Ala Gly Gly Thr Gly Gly Ala Gly Ala Thr Cys
1145 1150 1155
Gly Gly Cys Cys Gly Cys Thr Thr Cys Cys Gly Ala Gly Ala Cys
1160 1165 1170
Cys Ala Gly Cys Ala Gly Thr Ala Cys Gly Ala Gly Ala Thr Gly
1175 1180 1185
Cys Thr Cys Ala Ala Gly Cys Gly Cys Thr Gly Gly Cys Gly Cys
1190 1195 1200
Cys Ala Gly Cys Ala Gly Cys Ala Gly Cys Cys Cys Gly Cys Gly
1205 1210 1215
Gly Gly Cys Cys Thr Cys Gly Gly Ala Gly Cys Cys Gly Thr Thr
1220 1225 1230
Thr Ala Cys Gly Cys Gly Gly Cys Cys Cys Thr Gly Gly Ala Gly
1235 1240 1245
Cys Gly Cys Ala Thr Gly Gly Gly Gly Cys Thr Gly Gly Ala Cys
1250 1255 1260
Gly Gly Cys Thr Gly Cys Gly Thr Gly Gly Ala Ala Gly Ala Cys
1265 1270 1275
Thr Thr Gly Cys Gly Cys Ala Gly Cys Cys Gly Cys Cys Thr Gly
1280 1285 1290
Cys Ala Gly Cys Gly Cys Gly Gly Cys Cys Cys Gly Thr Gly Ala
1295 1300 1305
Cys Ala Cys Gly Gly Cys Gly Cys Cys Cys Ala Cys Thr Thr Gly
1310 1315 1320
Cys Cys Ala Cys Cys Thr Ala Gly Gly Cys Gly Cys Thr Cys Thr
1325 1330 1335
Gly Gly Thr Gly Gly Cys Cys Cys Thr Thr Gly Cys Ala Gly Ala
1340 1345 1350
Ala Gly Cys Cys Cys Thr Ala Ala Gly Thr Ala Cys Gly Gly Thr
1355 1360 1365
Thr Ala Cys Thr Thr Ala Thr Gly Cys Gly Thr Gly Thr Ala Gly
1370 1375 1380
Ala Cys Ala Thr Thr Thr Thr Ala Thr Gly Thr Cys Ala Cys Thr
1385 1390 1395
Thr Ala Thr Thr Ala Ala Gly Cys Cys Gly Cys Thr Gly Gly Cys
1400 1405 1410
Ala Cys Gly Gly Cys Cys Cys Thr Gly Cys Gly Thr Ala Gly Cys
1415 1420 1425
Ala Gly Cys Ala Cys Cys Ala Gly Cys Cys Gly Gly Cys Cys Cys
1430 1435 1440
Cys Ala Cys Cys Cys Cys Thr Gly Cys Thr Cys Gly Cys Cys Cys
1445 1450 1455
Cys Thr Ala Thr Cys Gly Cys Thr Cys Cys Ala Gly Cys Cys Ala
1460 1465 1470
Ala Gly Gly Cys Gly Ala Ala Gly Ala Ala Gly Cys Ala Cys Gly
1475 1480 1485
Ala Ala Cys Gly Ala Ala Thr Gly Thr Cys Gly Ala Gly Ala Gly
1490 1495 1500
Gly Gly Gly Gly Thr Gly Ala Ala Gly Ala Cys Ala Thr Thr Thr
1505 1510 1515
Cys Thr Cys Ala Ala Cys Thr Thr Cys Thr Cys Gly Gly Cys Cys
1520 1525 1530
Gly Gly Ala Gly Thr Thr Thr Gly Gly Cys Thr Gly Ala Gly Ala
1535 1540 1545
Thr Cys Gly Cys Gly Gly Thr Ala Thr Thr Ala Ala Ala Thr Cys
1550 1555 1560
Thr Gly Thr Gly Ala Ala Ala Gly Ala Ala Ala Ala Cys Ala Ala
1565 1570 1575
Ala Ala Cys Ala Ala Ala Ala Cys Ala Ala
1580 1585
<210> 13
<211> 426
<212> PRT
<213> 人(Homo sapiens)
<400> 13
Met Glu Gln Arg Pro Arg Gly Cys Ala Ala Val Ala Ala Ala Leu Leu
1 5 10 15
Leu Val Leu Leu Gly Ala Arg Ala Gln Gly Gly Thr Arg Ser Pro Arg
20 25 30
Cys Asp Cys Ala Gly Asp Phe His Lys Lys Ile Gly Leu Phe Cys Cys
35 40 45
Arg Gly Cys Pro Ala Gly His Tyr Leu Lys Ala Pro Cys Thr Glu Pro
50 55 60
Cys Gly Asn Ser Thr Cys Leu Val Cys Pro Gln Asp Thr Phe Leu Ala
65 70 75 80
Trp Glu Asn His His Asn Ser Glu Cys Ala Arg Cys Gln Ala Cys Asp
85 90 95
Glu Gln Ala Ser Gln Val Ala Leu Glu Asn Cys Ser Ala Val Ala Asp
100 105 110
Thr Arg Cys Gly Cys Lys Pro Gly Trp Phe Val Glu Cys Gln Val Ser
115 120 125
Gln Cys Val Ser Ser Ser Pro Phe Tyr Cys Gln Pro Cys Leu Asp Cys
130 135 140
Gly Ala Leu His Arg His Thr Arg Leu Leu Cys Ser Arg Arg Asp Thr
145 150 155 160
Asp Cys Gly Thr Cys Leu Pro Gly Phe Tyr Glu His Gly Asp Gly Cys
165 170 175
Val Ser Cys Pro Thr Pro Pro Pro Ser Leu Ala Gly Ala Pro Trp Gly
180 185 190
Ala Val Gln Ser Ala Val Pro Leu Ser Val Ala Gly Gly Arg Val Gly
195 200 205
Val Phe Trp Val Gln Val Leu Leu Ala Gly Leu Val Val Pro Leu Leu
210 215 220
Leu Gly Ala Thr Leu Thr Tyr Thr Tyr Arg His Cys Trp Pro His Lys
225 230 235 240
Pro Leu Val Thr Ala Asp Glu Ala Gly Met Glu Ala Leu Thr Pro Pro
245 250 255
Pro Ala Thr His Leu Ser Pro Leu Asp Ser Ala His Thr Leu Leu Ala
260 265 270
Pro Pro Asp Ser Ser Glu Lys Ile Cys Thr Val Gln Leu Val Gly Asn
275 280 285
Ser Trp Thr Pro Gly Tyr Pro Glu Thr Gln Glu Ala Leu Cys Pro Gln
290 295 300
Val Thr Trp Ser Trp Asp Gln Leu Pro Ser Arg Ala Leu Gly Pro Ala
305 310 315 320
Ala Ala Pro Thr Leu Ser Pro Glu Ser Pro Ala Gly Ser Pro Ala Met
325 330 335
Met Leu Gln Pro Gly Pro Gln Leu Tyr Asp Val Met Asp Ala Val Pro
340 345 350
Ala Arg Arg Trp Lys Glu Phe Val Arg Thr Leu Gly Leu Arg Glu Ala
355 360 365
Glu Ile Glu Ala Val Glu Val Glu Ile Gly Arg Phe Arg Asp Gln Gln
370 375 380
Tyr Glu Met Leu Lys Arg Trp Arg Gln Gln Gln Pro Ala Gly Leu Gly
385 390 395 400
Ala Val Tyr Ala Ala Leu Glu Arg Met Gly Leu Asp Gly Cys Val Glu
405 410 415
Asp Leu Arg Ser Arg Leu Gln Arg Gly Pro
420 425
<210> 14
<211> 5
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 14
Gly Gly Gly Gly Ser
1 5
<210> 15
<211> 5
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 15
Gly Gly Gly Gly Ser
1 5
<210> 16
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 16
Gly Gly Gly Gly Gly Gly Gly Gly
1 5
<210> 17
<211> 6
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 17
Gly Gly Gly Gly Gly Gly
1 5
<210> 18
<211> 5
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 18
Glu Ala Ala Ala Lys
1 5
<210> 19
<211> 12
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 19
Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala
1 5 10
<210> 20
<211> 12
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 20
Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala
1 5 10
<210> 21
<211> 46
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 21
Ala Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys
1 5 10 15
Glu Ala Ala Ala Lys Ala Leu Glu Ala Glu Ala Ala Ala Lys Glu Ala
20 25 30
Ala Ala Lys Glu Ala Ala Ala Lys Glu Ala Ala Ala Lys Ala
35 40 45
<210> 22
<211> 5
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 22
Pro Ala Pro Ala Pro
1 5
<210> 23
<211> 18
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 23
Lys Glu Ser Gly Ser Val Ser Ser Glu Gln Leu Ala Gln Phe Arg Ser
1 5 10 15
Leu Asp
<210> 24
<211> 14
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 24
Glu Gly Lys Ser Ser Gly Ser Gly Ser Glu Ser Lys Ser Thr
1 5 10
<210> 25
<211> 12
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 25
Gly Ser Ala Gly Ser Ala Ala Gly Ser Gly Glu Phe
1 5 10
<210> 26
<211> 852
<212> DNA
<213> 人(Homo sapiens)
<400> 26
atggagcctc ctggagactg ggggcctcct ccctggagat ccacccccaa aaccgacgtc 60
ttgaggctgg tgctgtatct caccttcctg ggagccccct gctacgcccc agctctgccg 120
tcctgcaagg aggacgagta cccagtgggc tccgagtgct gccccaagtg cagtccaggt 180
tatcgtgtga aggaggcctg cggggagctg acgggcacag tgtgtgaacc ctgccctcca 240
ggcacctaca ttgcccacct caatggccta agcaagtgtc tgcagtgcca aatgtgtgac 300
ccagccatgg gcctgcgcgc gagccggaac tgctccagga cagagaacgc cgtgtgtggc 360
tgcagcccag gccacttctg catcgtccag gacggggacc actgcgccgc gtgccgcgct 420
tacgccacct ccagcccggg ccagagggtg cagaagggag gcaccgagag tcaggacacc 480
ctgtgtcaga actgcccccc ggggaccttc tctcccaatg ggaccctgga ggaatgtcag 540
caccagacca agtgcagctg gctggtgacg aaggccggag ctgggaccag cagctcccac 600
tgggtatggt ggtttctctc agggagcctc gtcatcgtca ttgtttgctc cacagttggc 660
ctaatcatat gtgtgaaaag aagaaagcca aggggtgatg tagtcaaggt gatcgtctcc 720
gtccagcgga aaagacagga ggcagaaggt gaggccacag tcattgaggc cctgcaggcc 780
cctccggacg tcaccacggt ggccgtggag gagacaatac cctcattcac ggggaggagc 840
ccaaaccatt aa 852
<210> 27
<211> 283
<212> PRT
<213> 人(Homo sapiens)
<400> 27
Met Glu Pro Pro Gly Asp Trp Gly Pro Pro Pro Trp Arg Ser Thr Pro
1 5 10 15
Lys Thr Asp Val Leu Arg Leu Val Leu Tyr Leu Thr Phe Leu Gly Ala
20 25 30
Pro Cys Tyr Ala Pro Ala Leu Pro Ser Cys Lys Glu Asp Glu Tyr Pro
35 40 45
Val Gly Ser Glu Cys Cys Pro Lys Cys Ser Pro Gly Tyr Arg Val Lys
50 55 60
Glu Ala Cys Gly Glu Leu Thr Gly Thr Val Cys Glu Pro Cys Pro Pro
65 70 75 80
Gly Thr Tyr Ile Ala His Leu Asn Gly Leu Ser Lys Cys Leu Gln Cys
85 90 95
Gln Met Cys Asp Pro Ala Met Gly Leu Arg Ala Ser Arg Asn Cys Ser
100 105 110
Arg Thr Glu Asn Ala Val Cys Gly Cys Ser Pro Gly His Phe Cys Ile
115 120 125
Val Gln Asp Gly Asp His Cys Ala Ala Cys Arg Ala Tyr Ala Thr Ser
130 135 140
Ser Pro Gly Gln Arg Val Gln Lys Gly Gly Thr Glu Ser Gln Asp Thr
145 150 155 160
Leu Cys Gln Asn Cys Pro Pro Gly Thr Phe Ser Pro Asn Gly Thr Leu
165 170 175
Glu Glu Cys Gln His Gln Thr Lys Cys Ser Trp Leu Val Thr Lys Ala
180 185 190
Gly Ala Gly Thr Ser Ser Ser His Trp Val Trp Trp Phe Leu Ser Gly
195 200 205
Ser Leu Val Ile Val Ile Val Cys Ser Thr Val Gly Leu Ile Ile Cys
210 215 220
Val Lys Arg Arg Lys Pro Arg Gly Asp Val Val Lys Val Ile Val Ser
225 230 235 240
Val Gln Arg Lys Arg Gln Glu Ala Glu Gly Glu Ala Thr Val Ile Glu
245 250 255
Ala Leu Gln Ala Pro Pro Asp Val Thr Thr Val Ala Val Glu Glu Thr
260 265 270
Ile Pro Ser Phe Thr Gly Arg Ser Pro Asn His
275 280
<210> 28
<211> 4900
<212> DNA
<213> 人(Homo sapiens)
<400> 28
taaagtcatc aaaacaacgt tatatcctgt gtgaaatgct gcagtcagga tgccttgtgg 60
tttgagtgcc ttgatcatgt gccctaaggg gatggtggcg gtggtggtgg ccgtggatga 120
cggagactct caggccttgg caggtgcgtc tttcagttcc cctcacactt cgggttcctc 180
ggggaggagg ggctggaacc ctagcccatc gtcaggacaa agatgctcag gctgctcttg 240
gctctcaact tattcccttc aattcaagta acaggaaaca agattttggt gaagcagtcg 300
cccatgcttg tagcgtacga caatgcggtc aaccttagct gcaagtattc ctacaatctc 360
ttctcaaggg agttccgggc atcccttcac aaaggactgg atagtgctgt ggaagtctgt 420
gttgtatatg ggaattactc ccagcagctt caggtttact caaaaacggg gttcaactgt 480
gatgggaaat tgggcaatga atcagtgaca ttctacctcc agaatttgta tgttaaccaa 540
acagatattt acttctgcaa aattgaagtt atgtatcctc ctccttacct agacaatgag 600
aagagcaatg gaaccattat ccatgtgaaa gggaaacacc tttgtccaag tcccctattt 660
cccggacctt ctaagccctt ttgggtgctg gtggtggttg gtggagtcct ggcttgctat 720
agcttgctag taacagtggc ctttattatt ttctgggtga ggagtaagag gagcaggctc 780
ctgcacagtg actacatgaa catgactccc cgccgccccg ggcccacccg caagcattac 840
cagccctatg ccccaccacg cgacttcgca gcctatcgct cctgacacgg acgcctatcc 900
agaagccagc cggctggcag cccccatctg ctcaatatca ctgctctgga taggaaatga 960
ccgccatctc cagccggcca cctcaggccc ctgttgggcc accaatgcca atttttctcg 1020
agtgactaga ccaaatatca agatcatttt gagactctga aatgaagtaa aagagatttc 1080
ctgtgacagg ccaagtctta cagtgccatg gcccacattc caacttacca tgtacttagt 1140
gacttgactg agaagttagg gtagaaaaca aaaagggagt ggattctggg agcctcttcc 1200
ctttctcact cacctgcaca tctcagtcaa gcaaagtgtg gtatccacag acattttagt 1260
tgcagaagaa aggctaggaa atcattcctt ttggttaaat gggtgtttaa tcttttggtt 1320
agtgggttaa acggggtaag ttagagtagg gggagggata ggaagacata tttaaaaacc 1380
attaaaacac tgtctcccac tcatgaaatg agccacgtag ttcctattta atgctgtttt 1440
cctttagttt agaaatacat agacattgtc ttttatgaat tctgatcata tttagtcatt 1500
ttgaccaaat gagggatttg gtcaaatgag ggattccctc aaagcaatat caggtaaacc 1560
aagttgcttt cctcactccc tgtcatgaga cttcagtgtt aatgttcaca atatactttc 1620
gaaagaataa aatagttctc ctacatgaag aaagaatatg tcaggaaata aggtcacttt 1680
atgtcaaaat tatttgagta ctatgggacc tggcgcagtg gctcatgctt gtaatcccag 1740
cactttggga ggccgaggtg ggcagatcac ttgagatcag gaccagcctg gtcaagatgg 1800
tgaaactccg tctgtactaa aaatacaaaa tttagcttgg cctggtggca ggcacctgta 1860
atcccagctg cccaagaggc tgaggcatga gaatcgcttg aacctggcag gcggaggttg 1920
cagtgagccg agatagtgcc acagctctcc agcctgggcg acagagtgag actccatctc 1980
aaacaacaac aacaacaaca acaacaacaa caaaccacaa aattatttga gtactgtgaa 2040
ggattatttg tctaacagtt cattccaatc agaccaggta ggagctttcc tgtttcatat 2100
gtttcagggt tgcacagttg gtctctttaa tgtcggtgtg gagatccaaa gtgggttgtg 2160
gaaagagcgt ccataggaga agtgagaata ctgtgaaaaa gggatgttag cattcattag 2220
agtatgagga tgagtcccaa gaaggttctt tggaaggagg acgaatagaa tggagtaatg 2280
aaattcttgc catgtgctga ggagatagcc agcattaggt gacaatcttc cagaagtggt 2340
caggcagaag gtgccctggt gagagctcct ttacagggac tttatgtggt ttagggctca 2400
gagctccaaa actctgggct cagctgctcc tgtaccttgg aggtccattc acatgggaaa 2460
gtattttgga atgtgtcttt tgaagagagc atcagagttc ttaagggact gggtaaggcc 2520
tgaccctgaa atgaccatgg atatttttct acctacagtt tgagtcaact agaatatgcc 2580
tggggacctt gaagaatggc ccttcagtgg ccctcaccat ttgttcatgc ttcagttaat 2640
tcaggtgttg aaggagctta ggttttagag gcacgtagac ttggttcaag tctcgttagt 2700
agttgaatag cctcaggcaa gtcactgccc acctaagatg atggttcttc aactataaaa 2760
tggagataat ggttacaaat gtctcttcct atagtataat ctccataagg gcatggccca 2820
agtctgtctt tgactctgcc tatccctgac atttagtagc atgcccgaca tacaatgtta 2880
gctattggta ttattgccat atagataaat tatgtataaa aattaaactg ggcaatagcc 2940
taagaagggg ggaatattgt aacacaaatt taaacccact acgcagggat gaggtgctat 3000
aatatgagga ccttttaact tccatcattt tcctgtttct tgaaatagtt tatcttgtaa 3060
tgaaatataa ggcacctccc acttttatgt atagaaagag gtcttttaat ttttttttaa 3120
tgtgagaagg aagggaggag taggaatctt gagattccag atcgaaaata ctgtactttg 3180
gttgattttt aagtgggctt ccattccatg gatttaatca gtcccaagaa gatcaaactc 3240
agcagtactt gggtgctgaa gaactgttgg atttaccctg gcacgtgtgc cacttgccag 3300
cttcttgggc acacagagtt cttcaatcca agttatcaga ttgtatttga aaatgacaga 3360
gctggagagt tttttgaaat ggcagtggca aataaataaa tacttttttt taaatggaaa 3420
gacttgatct atggtaataa atgattttgt tttctgactg gaaaaatagg cctactaaag 3480
atgaatcaca cttgagatgt ttcttactca ctctgcacag aaacaaagaa gaaatgttat 3540
acagggaagt ccgttttcac tattagtatg aaccaagaaa tggttcaaaa acagtggtag 3600
gagcaatgct ttcatagttt cagatatggt agttatgaag aaaacaatgt catttgctgc 3660
tattattgta agagtcttat aattaatggt actcctataa tttttgattg tgagctcacc 3720
tatttgggtt aagcatgcca atttaaagag accaagtgta tgtacattat gttctacata 3780
ttcagtgata aaattactaa actactatat gtctgcttta aatttgtact ttaatattgt 3840
cttttggtat taagaaagat atgctttcag aatagatatg cttcgctttg gcaaggaatt 3900
tggatagaac ttgctattta aaagaggtgt ggggtaaatc cttgtataaa tctccagttt 3960
agcctttttt gaaaaagcta gactttcaaa tactaatttc acttcaagca gggtacgttt 4020
ctggtttgtt tgcttgactt cagtcacaat ttcttatcag accaatggct gacctctttg 4080
agatgtcagg ctaggcttac ctatgtgttc tgtgtcatgt gaatgctgag aagtttgaca 4140
gagatccaac ttcagccttg accccatcag tccctcgggt taactaactg agccaccggt 4200
cctcatggct attttaatga gggtattgat ggttaaatgc atgtctgatc ccttatccca 4260
gccatttgca ctgccagctg ggaactatac cagacctgga tactgatccc aaagtgttaa 4320
attcaactac atgctggaga ttagagatgg tgccaataaa ggacccagaa ccaggatctt 4380
gattgctata gacttattaa taatccaggt caaagagagt gacacacact ctctcaagac 4440
ctggggtgag ggagtctgtg ttatctgcaa ggccatttga ggctcagaaa gtctctcttt 4500
cctatagata tatgcatact ttctgacata taggaatgta tcaggaatac tcaaccatca 4560
caggcatgtt cctacctcag ggcctttaca tgtcctgttt actctgtcta gaatgtcctt 4620
ctgtagatga cctggcttgc ctcgtcaccc ttcaggtcct tgctcaagtg tcatcttctc 4680
ccctagttaa actaccccac accctgtctg ctttccttgc ttatttttct ccatagcatt 4740
ttaccatctc ttacattaga catttttctt atttatttgt agtttataag cttcatgagg 4800
caagtaactt tgctttgttt cttgctgtat ctccagtgcc cagagcagtg cctggtatat 4860
aataaatatt tattgactga gtgaaaaaaa aaaaaaaaaa 4900
<210> 29
<211> 220
<212> PRT
<213> 人(Homo sapiens)
<400> 29
Met Leu Arg Leu Leu Leu Ala Leu Asn Leu Phe Pro Ser Ile Gln Val
1 5 10 15
Thr Gly Asn Lys Ile Leu Val Lys Gln Ser Pro Met Leu Val Ala Tyr
20 25 30
Asp Asn Ala Val Asn Leu Ser Cys Lys Tyr Ser Tyr Asn Leu Phe Ser
35 40 45
Arg Glu Phe Arg Ala Ser Leu His Lys Gly Leu Asp Ser Ala Val Glu
50 55 60
Val Cys Val Val Tyr Gly Asn Tyr Ser Gln Gln Leu Gln Val Tyr Ser
65 70 75 80
Lys Thr Gly Phe Asn Cys Asp Gly Lys Leu Gly Asn Glu Ser Val Thr
85 90 95
Phe Tyr Leu Gln Asn Leu Tyr Val Asn Gln Thr Asp Ile Tyr Phe Cys
100 105 110
Lys Ile Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser
115 120 125
Asn Gly Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro
130 135 140
Leu Phe Pro Gly Pro Ser Lys Pro Phe Trp Val Leu Val Val Val Gly
145 150 155 160
Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr Val Ala Phe Ile Ile
165 170 175
Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met
180 185 190
Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro
195 200 205
Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
210 215 220
<210> 30
<211> 1906
<212> DNA
<213> 人(Homo sapiens)
<400> 30
ccaagtcaca tgattcagga ttcaggggga gaatccttct tggaacagag atgggcccag 60
aactgaatca gatgaagaga gataaggtgt gatgtgggga agactatata aagaatggac 120
ccagggctgc agcaagcact caacggaatg gcccctcctg gagacacagc catgcatgtg 180
ccggcgggct ccgtggccag ccacctgggg accacgagcc gcagctattt ctatttgacc 240
acagccactc tggctctgtg ccttgtcttc acggtggcca ctattatggt gttggtcgtt 300
cagaggacgg actccattcc caactcacct gacaacgtcc ccctcaaagg aggaaattgc 360
tcagaagacc tcttatgtat cctgaaaaga gctccattca agaagtcatg ggcctacctc 420
caagtggcaa agcatctaaa caaaaccaag ttgtcttgga acaaagatgg cattctccat 480
ggagtcagat atcaggatgg gaatctggtg atccaattcc ctggtttgta cttcatcatt 540
tgccaactgc agtttcttgt acaatgccca aataattctg tcgatctgaa gttggagctt 600
ctcatcaaca agcatatcaa aaaacaggcc ctggtgacag tgtgtgagtc tggaatgcaa 660
acgaaacacg tataccagaa tctctctcaa ttcttgctgg attacctgca ggtcaacacc 720
accatatcag tcaatgtgga tacattccag tacatagata caagcacctt tcctcttgag 780
aatgtgttgt ccatcttctt atacagtaat tcagactgaa cagtttctct tggccttcag 840
gaagaaagcg cctctctacc atacagtatt tcatccctcc aaacacttgg gcaaaaagaa 900
aactttagac caagacaaac tacacagggt attaaatagt atacttctcc ttctgtctct 960
tggaaagata cagctccagg gttaaaaaga gagtttttag tgaagtatct ttcagatagc 1020
aggcagggaa gcaatgtagt gtggtgggca gagccccaca cagaatcaga agggatgaat 1080
ggatgtccca gcccaaccac taattcactg tatggtcttg atctatttct tctgttttga 1140
gagcctccag ttaaaatggg gcttcagtac cagagcagct agcaactctg ccctaatggg 1200
aaatgaaggg gagctgggtg tgagtgttta cactgtgccc ttcacgggat acttctttta 1260
tctgcagatg gcctaatgct tagttgtcca agtcgcgatc aaggactctc tcacacagga 1320
aacttcccta tactggcaga tacacttgtg actgaaccat gcccagttta tgcctgtctg 1380
actgtcactc tggcactagg aggctgatct tgtactccat atgaccccac ccctaggaac 1440
ccccagggaa aaccaggctc ggacagcccc ctgttcctga gatggaaagc acaaatttaa 1500
tacaccacca caatggaaaa caagttcaaa gacttttact tacagatcct ggacagaaag 1560
ggcataatga gtctgaaggg cagtcctcct tctccaggtt acatgaggca ggaataagaa 1620
gtcagacaga gacagcaaga cagttaacaa cgtaggtaaa gaaatagggt gtggtcactc 1680
tcaattcact ggcaaatgcc tgaatggtct gtctgaagga agcaacagag aagtggggaa 1740
tccagtctgc taggcaggaa agatgcctct aagttcttgt ctctggccag aggtgtggta 1800
tagaaccaga aacccatatc aagggtgact aagcccggct tccggtatga gaaattaaac 1860
ttgtatacaa aatggttgcc aaggcaacat aaaattataa gaattc 1906
<210> 31
<211> 234
<212> PRT
<213> 人(Homo sapiens)
<400> 31
Met Asp Pro Gly Leu Gln Gln Ala Leu Asn Gly Met Ala Pro Pro Gly
1 5 10 15
Asp Thr Ala Met His Val Pro Ala Gly Ser Val Ala Ser His Leu Gly
20 25 30
Thr Thr Ser Arg Ser Tyr Phe Tyr Leu Thr Thr Ala Thr Leu Ala Leu
35 40 45
Cys Leu Val Phe Thr Val Ala Thr Ile Met Val Leu Val Val Gln Arg
50 55 60
Thr Asp Ser Ile Pro Asn Ser Pro Asp Asn Val Pro Leu Lys Gly Gly
65 70 75 80
Asn Cys Ser Glu Asp Leu Leu Cys Ile Leu Lys Arg Ala Pro Phe Lys
85 90 95
Lys Ser Trp Ala Tyr Leu Gln Val Ala Lys His Leu Asn Lys Thr Lys
100 105 110
Leu Ser Trp Asn Lys Asp Gly Ile Leu His Gly Val Arg Tyr Gln Asp
115 120 125
Gly Asn Leu Val Ile Gln Phe Pro Gly Leu Tyr Phe Ile Ile Cys Gln
130 135 140
Leu Gln Phe Leu Val Gln Cys Pro Asn Asn Ser Val Asp Leu Lys Leu
145 150 155 160
Glu Leu Leu Ile Asn Lys His Ile Lys Lys Gln Ala Leu Val Thr Val
165 170 175
Cys Glu Ser Gly Met Gln Thr Lys His Val Tyr Gln Asn Leu Ser Gln
180 185 190
Phe Leu Leu Asp Tyr Leu Gln Val Asn Thr Thr Ile Ser Val Asn Val
195 200 205
Asp Thr Phe Gln Tyr Ile Asp Thr Ser Thr Phe Pro Leu Glu Asn Val
210 215 220
Leu Ser Ile Phe Leu Tyr Ser Asn Ser Asp
225 230
<210> 32
<211> 1629
<212> DNA
<213> 人(Homo sapiens)
<400> 32
tttcctgggc ggggccaagg ctggggcagg ggagtcagca gaggcctcgc tcgggcgccc 60
agtggtcctg ccgcctggtc tcacctcgct atggttcgtc tgcctctgca gtgcgtcctc 120
tggggctgct tgctgaccgc tgtccatcca gaaccaccca ctgcatgcag agaaaaacag 180
tacctaataa acagtcagtg ctgttctttg tgccagccag gacagaaact ggtgagtgac 240
tgcacagagt tcactgaaac ggaatgcctt ccttgcggtg aaagcgaatt cctagacacc 300
tggaacagag agacacactg ccaccagcac aaatactgcg accccaacct agggcttcgg 360
gtccagcaga agggcacctc agaaacagac accatctgca cctgtgaaga aggctggcac 420
tgtacgagtg aggcctgtga gagctgtgtc ctgcaccgct catgctcgcc cggctttggg 480
gtcaagcaga ttgctacagg ggtttctgat accatctgcg agccctgccc agtcggcttc 540
ttctccaatg tgtcatctgc tttcgaaaaa tgtcaccctt ggacaagctg tgagaccaaa 600
gacctggttg tgcaacaggc aggcacaaac aagactgatg ttgtctgtgg tccccaggat 660
cggctgagag ccctggtggt gatccccatc atcttcggga tcctgtttgc catcctcttg 720
gtgctggtct ttatcaaaaa ggtggccaag aagccaacca ataaggcccc ccaccccaag 780
caggaacccc aggagatcaa ttttcccgac gatcttcctg gctccaacac tgctgctcca 840
gtgcaggaga ctttacatgg atgccaaccg gtcacccagg aggatggcaa agagagtcgc 900
atctcagtgc aggagagaca gtgaggctgc acccacccag gagtgtggcc acgtgggcaa 960
acaggcagtt ggccagagag cctggtgctg ctgctgctgt ggcgtgaggg tgaggggctg 1020
gcactgactg ggcatagctc cccgcttctg cctgcacccc tgcagtttga gacaggagac 1080
ctggcactgg atgcagaaac agttcacctt gaagaacctc tcacttcacc ctggagccca 1140
tccagtctcc caacttgtat taaagacaga ggcagaagtt tggtggtggt ggtgttgggg 1200
tatggtttag taatatccac cagaccttcc gatccagcag tttggtgccc agagaggcat 1260
catggtggct tccctgcgcc caggaagcca tatacacaga tgcccattgc agcattgttt 1320
gtgatagtga acaactggaa gctgcttaac tgtccatcag caggagactg gctaaataaa 1380
attagaatat atttatacaa cagaatctca aaaacactgt tgagtaagga aaaaaaggca 1440
tgctgctgaa tgatgggtat ggaacttttt aaaaaagtac atgcttttat gtatgtatat 1500
tgcctatgga tatatgtata aatacaatat gcatcatata ttgatataac aagggttctg 1560
gaagggtaca cagaaaaccc acagctcgaa gagtggtgac gtctggggtg gggaagaagg 1620
gtctggggg 1629
<210> 33
<211> 277
<212> PRT
<213> 人(Homo sapiens)
<400> 33
Met Val Arg Leu Pro Leu Gln Cys Val Leu Trp Gly Cys Leu Leu Thr
1 5 10 15
Ala Val His Pro Glu Pro Pro Thr Ala Cys Arg Glu Lys Gln Tyr Leu
20 25 30
Ile Asn Ser Gln Cys Cys Ser Leu Cys Gln Pro Gly Gln Lys Leu Val
35 40 45
Ser Asp Cys Thr Glu Phe Thr Glu Thr Glu Cys Leu Pro Cys Gly Glu
50 55 60
Ser Glu Phe Leu Asp Thr Trp Asn Arg Glu Thr His Cys His Gln His
65 70 75 80
Lys Tyr Cys Asp Pro Asn Leu Gly Leu Arg Val Gln Gln Lys Gly Thr
85 90 95
Ser Glu Thr Asp Thr Ile Cys Thr Cys Glu Glu Gly Trp His Cys Thr
100 105 110
Ser Glu Ala Cys Glu Ser Cys Val Leu His Arg Ser Cys Ser Pro Gly
115 120 125
Phe Gly Val Lys Gln Ile Ala Thr Gly Val Ser Asp Thr Ile Cys Glu
130 135 140
Pro Cys Pro Val Gly Phe Phe Ser Asn Val Ser Ser Ala Phe Glu Lys
145 150 155 160
Cys His Pro Trp Thr Ser Cys Glu Thr Lys Asp Leu Val Val Gln Gln
165 170 175
Ala Gly Thr Asn Lys Thr Asp Val Val Cys Gly Pro Gln Asp Arg Leu
180 185 190
Arg Ala Leu Val Val Ile Pro Ile Ile Phe Gly Ile Leu Phe Ala Ile
195 200 205
Leu Leu Val Leu Val Phe Ile Lys Lys Val Ala Lys Lys Pro Thr Asn
210 215 220
Lys Ala Pro His Pro Lys Gln Glu Pro Gln Glu Ile Asn Phe Pro Asp
225 230 235 240
Asp Leu Pro Gly Ser Asn Thr Ala Ala Pro Val Gln Glu Thr Leu His
245 250 255
Gly Cys Gln Pro Val Thr Gln Glu Asp Gly Lys Glu Ser Arg Ile Ser
260 265 270
Val Gln Glu Arg Gln
275
<210> 34
<211> 913
<212> DNA
<213> 人(Homo sapiens)
<400> 34
ccagagaggg gcaggctggt cccctgacag gttgaagcaa gtagacgccc aggagccccg 60
ggagggggct gcagtttcct tccttccttc tcggcagcgc tccgcgcccc catcgcccct 120
cctgcgctag cggaggtgat cgccgcggcg atgccggagg agggttcggg ctgctcggtg 180
cggcgcaggc cctatgggtg cgtcctgcgg gctgctttgg tcccattggt cgcgggcttg 240
gtgatctgcc tcgtggtgtg catccagcgc ttcgcacagg ctcagcagca gctgccgctc 300
gagtcacttg ggtgggacgt agctgagctg cagctgaatc acacaggacc tcagcaggac 360
cccaggctat actggcaggg gggcccagca ctgggccgct ccttcctgca tggaccagag 420
ctggacaagg ggcagctacg tatccatcgt gatggcatct acatggtaca catccaggtg 480
acgctggcca tctgctcctc cacgacggcc tccaggcacc accccaccac cctggccgtg 540
ggaatctgct ctcccgcctc ccgtagcatc agcctgctgc gtctcagctt ccaccaaggt 600
tgtaccattg cctcccagcg cctgacgccc ctggcccgag gggacacact ctgcaccaac 660
ctcactggga cacttttgcc ttcccgaaac actgatgaga ccttctttgg agtgcagtgg 720
gtgcgcccct gaccactgct gctgattagg gttttttaaa ttttatttta ttttatttaa 780
gttcaagaga aaaagtgtac acacaggggc cacccggggt tggggtggga gtgtggtggg 840
gggtagtggt ggcaggacaa gagaaggcat tgagcttttt ctttcatttt cctattaaaa 900
aatacaaaaa tca 913
<210> 35
<211> 193
<212> PRT
<213> 人(Homo sapiens)
<400> 35
Met Pro Glu Glu Gly Ser Gly Cys Ser Val Arg Arg Arg Pro Tyr Gly
1 5 10 15
Cys Val Leu Arg Ala Ala Leu Val Pro Leu Val Ala Gly Leu Val Ile
20 25 30
Cys Leu Val Val Cys Ile Gln Arg Phe Ala Gln Ala Gln Gln Gln Leu
35 40 45
Pro Leu Glu Ser Leu Gly Trp Asp Val Ala Glu Leu Gln Leu Asn His
50 55 60
Thr Gly Pro Gln Gln Asp Pro Arg Leu Tyr Trp Gln Gly Gly Pro Ala
65 70 75 80
Leu Gly Arg Ser Phe Leu His Gly Pro Glu Leu Asp Lys Gly Gln Leu
85 90 95
Arg Ile His Arg Asp Gly Ile Tyr Met Val His Ile Gln Val Thr Leu
100 105 110
Ala Ile Cys Ser Ser Thr Thr Ala Ser Arg His His Pro Thr Thr Leu
115 120 125
Ala Val Gly Ile Cys Ser Pro Ala Ser Arg Ser Ile Ser Leu Leu Arg
130 135 140
Leu Ser Phe His Gln Gly Cys Thr Ile Ala Ser Gln Arg Leu Thr Pro
145 150 155 160
Leu Ala Arg Gly Asp Thr Leu Cys Thr Asn Leu Thr Gly Thr Leu Leu
165 170 175
Pro Ser Arg Asn Thr Asp Glu Thr Phe Phe Gly Val Gln Trp Val Arg
180 185 190
Pro
<210> 36
<211> 723
<212> DNA
<213> 人(Homo sapiens)
<400> 36
atggaggaga gtgtcgtacg gccctcagtg tttgtggtgg atggacagac cgacatccca 60
ttcacgaggc tgggacgaag ccaccggaga cagtcgtgca gtgtggcccg ggtgggtctg 120
ggtctcttgc tgttgctgat gggggccggg ctggccgtcc aaggctggtt cctcctgcag 180
ctgcactggc gtctaggaga gatggtcacc cgcctgcctg acggacctgc aggctcctgg 240
gagcagctga tacaagagcg aaggtctcac gaggtcaacc cagcagcgca tctcacaggg 300
gccaactcca gcttgaccgg cagcgggggg ccgctgttat gggagactca gctgggcctg 360
gccttcctga ggggcctcag ctaccacgat ggggcccttg tggtcaccaa agctggctac 420
tactacatct actccaaggt gcagctgggc ggtgtgggct gcccgctggg cctggccagc 480
accatcaccc acggcctcta caagcgcaca ccccgctacc ccgaggagct ggagctgttg 540
gtcagccagc agtcaccctg cggacgggcc accagcagct cccgggtctg gtgggacagc 600
agcttcctgg gtggtgtggt acacctggag gctggggagg aggtggtcgt ccgtgtgctg 660
gatgaacgcc tggttcgact gcgtgatggt acccggtctt acttcggggc tttcatggtg 720
tga 723
<210> 37
<211> 240
<212> PRT
<213> 人(Homo sapiens)
<400> 37
Met Glu Glu Ser Val Val Arg Pro Ser Val Phe Val Val Asp Gly Gln
1 5 10 15
Thr Asp Ile Pro Phe Thr Arg Leu Gly Arg Ser His Arg Arg Gln Ser
20 25 30
Cys Ser Val Ala Arg Val Gly Leu Gly Leu Leu Leu Leu Leu Met Gly
35 40 45
Ala Gly Leu Ala Val Gln Gly Trp Phe Leu Leu Gln Leu His Trp Arg
50 55 60
Leu Gly Glu Met Val Thr Arg Leu Pro Asp Gly Pro Ala Gly Ser Trp
65 70 75 80
Glu Gln Leu Ile Gln Glu Arg Arg Ser His Glu Val Asn Pro Ala Ala
85 90 95
His Leu Thr Gly Ala Asn Ser Ser Leu Thr Gly Ser Gly Gly Pro Leu
100 105 110
Leu Trp Glu Thr Gln Leu Gly Leu Ala Phe Leu Arg Gly Leu Ser Tyr
115 120 125
His Asp Gly Ala Leu Val Val Thr Lys Ala Gly Tyr Tyr Tyr Ile Tyr
130 135 140
Ser Lys Val Gln Leu Gly Gly Val Gly Cys Pro Leu Gly Leu Ala Ser
145 150 155 160
Thr Ile Thr His Gly Leu Tyr Lys Arg Thr Pro Arg Tyr Pro Glu Glu
165 170 175
Leu Glu Leu Leu Val Ser Gln Gln Ser Pro Cys Gly Arg Ala Thr Ser
180 185 190
Ser Ser Arg Val Trp Trp Asp Ser Ser Phe Leu Gly Gly Val Val His
195 200 205
Leu Glu Ala Gly Glu Glu Val Val Val Arg Val Leu Asp Glu Arg Leu
210 215 220
Val Arg Leu Arg Asp Gly Thr Arg Ser Tyr Phe Gly Ala Phe Met Val
225 230 235 240
<210> 38
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 38
Glu Gly Ser Arg Asn Gln Asp Trp Leu
1 5
<210> 39
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 39
Thr Trp His Arg Tyr His Leu Leu
1 5
<210> 40
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 40
Ser Val Tyr Asp Phe Phe Val Trp Leu
1 5
<210> 41
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成的序列
<400> 41
Ser Ile Ile Asn Phe Glu Lys Leu
1 5
- 基于IL-2的疗法和基于间充质干细胞的疗法的伴随方法和试剂盒
- T细胞疗法和BTK抑制剂的组合疗法
