用于修饰所靶向基因座的方法和组合物
文献发布时间:2023-06-19 12:08:44
分案申请说明
本申请是申请日为2015年06月05日,申请号为201580042190.6,发明名称为“用于修饰所靶向基因座的方法和组合物”的发明专利申请的分案申请。
相关申请的交叉引用
本申请要求2014年6月6日提交的美国临时申请No.62/008,832以及2014年6月27日提交的美国临时申请No.62/017,916的权益,这两个美国临时申请均据此全文以引用的方式并入本文中。
技术领域
所述方法和组合物涉及分子生物学领域。具体地讲,本发明提供了用于修饰细胞中所靶向基因座的方法和组合物。
通过EFS-Web以电子方式将序列表的正式文本作为ASCII格式的序列表提交,该文件名称为461003SEQLIST.TXT,创建日期为2015年6月5日,文件大小为5KB,并且该文件与本说明书同时提交。该ASCII格式文档中所含的序列表是本说明书的一部分,并且全文以引用的方式并入本文中。
背景技术
使用被特别设计成在基因组座位处添加、缺失或取代特定核酸序列的靶向载体进行的同源重组,是在细胞中实现所需基因组修饰的常用方法。可将被特别改造成在靶基因座处或其附近引入切口或双链断裂的核酸酶与靶向载体联合使用,以增强靶基因座处同源重组的效率。
虽然通过同源重组进行靶向修饰的领域在过去二十年内已取得显著进展,但使用靶向载体实现可接受的靶向效率仍然存在困难。需要能够提高产生靶向修饰的功效和效率的方法。
发明内容
本发明提供了用于修饰细胞中一个或多个靶基因座的方法和组合物。
在一些实施例中,提供了用于修饰细胞中靶基因座的方法,所述方法包括:(a)提供包含靶基因座的细胞,所述靶基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,(b)向细胞中引入(i)第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂;和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应;以及(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞。
在一些实施例中,用于修饰细胞中的靶基因座的方法包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,(b)向细胞中引入:(i)编码第一核酸酶试剂并有效连接至细胞中有活性的启动子的一个或多个表达构建体,其中所述第一核酸酶试剂在第一多核苷酸中的第一识别位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性;和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸包含编码第二选择标记并有效连接至第二启动子的第二多核苷酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;以及(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和第二选择标记不同。
在一个实施例中,靶基因座位于细胞的基因组中。在另一个实施例中,靶基因座位于细胞中的载体中。在一个实施例中,第一识别位点处的切口或双链断裂破坏第一选择标记的活性。在又一个实施例中,鉴定步骤(c)包括在允许鉴定没有第一选择标记活性的细胞的条件下培养细胞。在一个实施例中,包含第一选择标记的第一多核苷酸侧接第一靶位点和第二靶位点。在一个实施例中,鉴定步骤(c)包括鉴定包含在第一靶位点和第二靶位点处整合的第一插入多核苷酸的至少一个细胞。在一个实施例中,第一插入多核苷酸包含:(a)第一目标多核苷酸;和(b)编码第二选择标记并有效连接至细胞中有活性的第二启动子的第二多核苷酸,其中所述第二多核苷酸包含第二核酸酶试剂的第二识别位点。
在一个实施例中,所述方法还包括(a)向包含在靶基因座处整合的第一插入多核苷酸的细胞中引入(i)第二核酸酶试剂,其中所述第二核酸酶试剂在第二识别位点处诱导切口或双链断裂;和(ii)包含第二插入多核苷酸的第二靶向载体,所述第二插入多核苷酸侧接第三同源臂和第四同源臂,所述第三同源臂和第四同源臂与位于足够接近第二识别位点处的第三靶位点和第四靶位点相对应;以及(b)鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞。在一个实施例中,第二识别位点处的切口或双链断裂破坏第二选择标记的活性。在一个实施例中,鉴定步骤(b)包括在允许鉴定没有第二选择标记的活性的细胞的条件下培养细胞。在一个实施例中,包含第二选择性标记的第二多核苷酸侧接第三靶位点和第四靶位点。在一个实施例中,鉴定步骤(b)包括鉴定包含在第三靶位点和第四靶位点处整合的第二插入多核苷酸的至少一个细胞。
在一个实施例中,第二插入多核苷酸包含:(a)第二目标多核苷酸;和(b)编码第三选择标记并有效连接至细胞中有活性的第三启动子的第三多核苷酸,其中所述第三多核苷酸包含第三核酸酶试剂的第三识别位点。在一个实施例中,第一核酸酶试剂与第二核酸酶试剂不同。在一个实施例中,第一选择标记与第二选择标记不同。在一个实施例中,第一核酸酶识别位点和第三核酸酶识别位点彼此相同并与第二核酸酶识别位点不同;并且第一核酸酶试剂和第三核酸酶试剂彼此相同并与第二核酸酶试剂不同。在一个实施例中,第一选择标记和第三选择标记相同。在一个实施例中,第一选择标记、第二选择标记或第三选择标记中的一者赋予对抗生素的抗性。在一个实施例中,抗生素包括G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素。在一个实施例中,第一选择标记、第二选择标记或第三选择标记中的一者有效连接至诱导型启动子,并且选择性标记的表达对细胞有毒性。在一个实施例中,第一选择标记、第二选择标记或第三选择标记包括次黄嘌呤-鸟嘌呤磷酸核糖转移酶(HGPRT)或单纯性疱疹病毒的胸苷激酶(HSV-TK)。在一个实施例中,所述细胞为原核细胞。在一个实施例中,所述细胞为真核细胞。在一个实施例中,所述真核细胞为哺乳动物细胞。在一个实施例中,所述哺乳动物细胞为非人哺乳动物细胞。在一个实施例中,所述哺乳动物细胞来自啮齿动物。在一个实施例中,所述啮齿动物为大鼠或小鼠。
在一个实施例中,所述细胞为多能细胞。在一个实施例中,所述哺乳动物细胞为人诱导性多能干(iPS)细胞。在一个实施例中,所述多能细胞为非人胚胎干(ES)细胞。在一个实施例中,所述多能细胞为小鼠胚胎干(ES)细胞或大鼠胚胎干(ES)细胞。在一个实施例中,所述多能细胞为造血干细胞。在一个实施例中,所述多能细胞为神经元干细胞。在一个实施例中,所述哺乳动物细胞为人成纤维细胞。
在一个实施例中,与单独使用第一靶向载体相比,联合使用第一靶向载体与第一核酸酶试剂会提高靶向效率。在一个实施例中,与单独使用第一靶向载体相比,第一靶向载体的靶向效率提高了至少2倍。
在一个实施例中,第一核酸酶试剂或第二核酸酶试剂包括表达构建体,所述表达构建体包含编码核酸酶试剂的核酸序列,并且其中所述核酸有效连接至细胞中有活性的第四启动子。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为编码核酸酶的mRNA。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为锌指核酸酶(ZFN)。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为转录激活因子样效应物核酸酶(TALEN)。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为大范围核酸酶。
在一个实施例中,第一核酸酶试剂或第二核酸酶试剂包括成簇规律间隔短回文重复序列(CRISPR)相关(Cas)蛋白和向导RNA(gRNA)。在一个实施例中,向导RNA(gRNA)包括(a)靶向第一识别位点、第二识别位点或第三识别位点的成簇规律间隔短回文重复序列(CRISPR)RNA(crRNA);和(b)反式激活CRISPR RNA(tracrRNA)。在一个实施例中,第一识别位点或第二识别位点紧密侧接前间区序列邻近基序(PAM)序列。在一个实施例中,目标基因组座位包含SEQ ID NO:1的核苷酸序列。在一个实施例中,Cas蛋白为Cas9。在一个实施例中,gRNA包括:(a)SEQ ID NO:2的核酸序列的嵌合RNA;或(b)SEQ ID NO:3的核酸序列的嵌合RNA。在一个实施例中,crRNA包含SEQ ID NO:4、SEQ ID NO:5或SEQ ID NO:6。在一个实施例中,tracrRNA包含SEQ ID NO:7或SEQ ID NO:8。
在一个实施例中,第一识别位点、第二识别位点和/或第三识别位点位于第一选择标记、第二选择标记或第三选择标记的内含子、外显子、启动子、启动子调控区或增强子区中。在一个实施例中,第一靶位点和第二靶位点紧邻第一识别位点。在一个实施例中,第一靶位点和第二靶位点距第一识别位点约10个核苷酸至约14kb。在一个实施例中,第三靶位点和第四靶位点紧邻第二识别位点。在一个实施例中,第三靶位点和第四靶位点距第二识别位点约10个核苷酸至约14kb。
在一个实施例中,第一同源臂和第二同源臂的总和为至少约10kb。在一个实施例中,第三同源臂和第四同源臂的总和为至少约10kb。在一个实施例中,第一插入多核苷酸的长度在约5kb至约300kb的范围内。在一个实施例中,第二插入多核苷酸的长度在约5kb至约300kb的范围内。
在一个实施例中,将第一插入多核苷酸整合到靶基因座中会导致敲除、敲入、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换,或它们的组合。在一个实施例中,将第二插入多核苷酸整合到靶基因座中会导致敲除、敲入、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换,或它们的组合。
在一个实施例中,第一插入多核苷酸包括含有人多核苷酸的目标多核苷酸。在一个实施例中,第二插入多核苷酸包括含有人多核苷酸的目标多核苷酸。在一个实施例中,第一插入多核苷酸包括含有T细胞受体α基因座区域的目标多核苷酸。
在一个实施例中,第二插入多核苷酸包括含有T细胞受体α基因座区域的目标多核苷酸。在一个实施例中,第一插入多核苷酸或第二插入多核苷酸包括含有T细胞受体α基因座的至少一个可变区基因区段和/或连接区基因区段的目标多核苷酸。在一个实施例中,T细胞受体α基因座区域来源于人。
在一个实施例中,第一插入多核苷酸包括含有有效连接至非人免疫球蛋白重链恒定区核酸序列的非重排人免疫球蛋白重链可变区核酸序列的目标多核苷酸。
在一个实施例中,通过等位基因修饰(MOA)测定法来执行鉴定步骤。在一个实施例中,第一插入多核苷酸包括含有与细胞基因组中的核酸序列同源或直系同源的核酸序列的目标多核苷酸。在一个实施例中,第二插入多核苷酸包含与细胞基因组中的核酸序列同源或直系同源的核酸序列。在一个实施例中,第一插入多核苷酸包括含有外源核酸序列的目标多核苷酸。在一个实施例中,第二插入多核苷酸包括含有外源核酸序列的目标多核苷酸。
在一些实施例中,用于修饰细胞中靶基因座的方法包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的核酸;(b)向细胞中引入(i)编码Cas蛋白和第一向导RNA(gRNA)的一个或多个表达构建体,每个所述表达构建体有效连接至细胞中有活性的启动子,其中所述Cas蛋白在第一核酸中的第一gRNA靶位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性,和(ii)包含第一插入核酸的第一靶向载体,所述第一插入核酸包含编码第二选择标记并有效连接至第二启动子的第二核酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;以及(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和第二选择标记不同。在一个实施例中,第一gRNA不与第一插入核酸杂交。在一个实施例中,目标靶基因座位于细胞的基因组中。在另一个实施例中,目标靶基因座位于细胞中的载体中。在一个实施例中,鉴定步骤(c)包括在允许鉴定具有第二选择标记的活性但没有第一选择标记的活性的修饰细胞的条件下培养细胞。
在一个实施例中,所述方法还包括(d)向在第一靶基因座处包含第一插入核酸的修饰细胞中引入(i)编码Cas蛋白和第二gRNA的一个或多个核酸,每个所述核酸有效连接至修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第二核酸的第一插入核酸中的第二gRNA靶位点处诱导切口或双链断裂,从而破坏第二选择标记的表达或活性,和(ii)包含第二插入核酸的第二靶向载体,所述第二插入核酸包含编码第三选择标记并有效连接至第三启动子的第三核酸,其中所述第二插入核酸侧接第三同源臂和第四同源臂,所述第三同源臂和第四同源臂与位于第二靶基因座中的第三靶位点和第四靶位点相对应;以及(e)鉴定在第二靶基因座处包含第二插入核酸的第二修饰细胞,其中所述第二修饰细胞具有第三选择标记的活性,但不具有第二选择标记的活性,其中所述第二选择标记和第三选择标记不同。在一个实施例中,第一靶基因座和第二靶基因座位于彼此紧邻的位置。在另一个实施例中,第一靶基因座或第二靶基因座位于距第一gRNA靶位点或第二gRNA靶位点约10个核苷酸至约14kb、约10个核苷酸至约100个核苷酸、约100个核苷酸至约500个核苷酸、约500个核苷酸至约1000个核苷酸、约1kb至约5kb、约5kb至约10kb或约10kb至约14kb处。在一个实施例中,第二gRNA不与第二插入核酸杂交。在一个实施例中,鉴定步骤(e)包括在允许鉴定具有第三选择标记的活性但没有第二选择标记的活性的第二修饰细胞的条件下培养修饰细胞。
在一个实施例中,所述方法还包括(f)向在第二靶基因座处包含第二插入核酸的第二修饰细胞中引入:(i)编码Cas蛋白和第三gRNA的一个或多个表达构建体,每个所述表达构建体有效连接至第二修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第三核酸的第二插入核酸中的第三gRNA靶位点处诱导切口或双链断裂,从而破坏第三选择标记的表达或活性,和(ii)包含第三插入核酸的第三靶向载体,所述第三插入核酸包含编码第四选择标记并有效连接至第四启动子的第四核酸,其中所述第三插入核酸侧接第五同源臂和第六同源臂,所述第五同源臂和第六同源臂与位于第三靶基因座中的第五靶位点和第六靶位点相对应;以及(g)鉴定在第三靶基因座处包含第三插入核酸的第三修饰细胞,其中所述第三修饰细胞具有第四选择标记的活性,但不具有第三选择标记的活性,其中所述第三选择标记和第四选择标记不同。在一个实施例中,第二靶基因座和第三靶基因座位于彼此紧邻的位置。在另一个实施例中,第二靶基因座或第三靶基因座位于距第一gRNA靶位点或第二gRNA靶位点约10个核苷酸至约14kb处。
在一个实施例中,第一标记、第二标记、第三标记或第四标记赋予对抗生素的抗性。在一个实施例中,抗生素包括G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素。在一个实施例中,第一选择标记、第二选择标记、第三选择标记或第四选择标记包括次黄嘌呤-鸟嘌呤磷酸核糖转移酶(HGPRT)或单纯性疱疹病毒的胸苷激酶(HSV-TK)。在一个实施例中,第一gRNA、第二gRNA或第三gRNA包括(i)与第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点杂交的核苷酸序列,和(ii)反式激活CRISPR RNA(tracrRNA)。在一个实施例中,第一靶基因座、第二靶基因座或第三靶基因座位于紧邻第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点的位置,使得gRNA靶位点处的切口或双链断裂促进靶基因座处靶向载体的同源重组。在一个实施例中,Cas蛋白为Cas9。在一个实施例中,第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点紧密侧接前间区序列邻近基序(PAM)序列。
在一个实施例中,所述细胞为原核细胞。在另一个实施例中,所述细胞为真核细胞。在一个实施例中,所述真核细胞为哺乳动物细胞。在一个实施例中,所述哺乳动物细胞为成纤维细胞。在一个实施例中,所述哺乳动物细胞为人成纤维细胞。在一个实施例中,所述哺乳动物细胞为非人哺乳动物细胞。在一个实施例中,所述哺乳动物细胞来自啮齿动物。在一个实施例中,所述啮齿动物为大鼠、小鼠或仓鼠。
在一个实施例中,所述真核细胞为多能细胞。在一个实施例中,所述多能细胞为造血干细胞或神经元干细胞。在一个实施例中,所述多能细胞为人诱导性多能干(iPS)细胞。在一个实施例中,所述多能细胞为小鼠胚胎干(ES)细胞或大鼠胚胎干(ES)细胞。
在一个实施例中,第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点位于编码第一选择标记、第二选择标记或第三选择标记的第一核酸、第二核酸或第三核酸中的内含子、外显子、启动子或启动子调控区中。在一个实施例中,第一靶向载体、第二靶向载体或第三靶向载体为至少约10kb。在一个实施例中,第一插入核酸、第二插入核酸或第三插入核酸在约5kb至约300kb的范围内。
在一个实施例中,第一插入核酸、第二插入核酸或第三插入核酸包含人T细胞受体α基因座的基因组区域。在一个实施例中,所述基因组区域包含人T细胞受体α基因座的至少一个可变区基因区段和/或连接区基因区段。
在一个实施例中,第一选择标记和第三选择标记相同。在一个实施例中,第一选择标记和第三选择标记相同,并且第二选择标记和第四选择标记相同。在一个实施例中,第一gRNA和第三gRNA相同。
另外还提供了用于修饰细胞中的靶基因座的方法和组合物。此类方法包括提供包含靶基因座的细胞,所述靶基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点。向细胞中引入第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂。进一步向细胞中引入包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应。然后鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞。
还提供了用于修饰细胞中的靶基因座的方法,所述方法包括:(a)提供包含靶基因座的细胞,所述靶基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,(b)向细胞中引入(i)第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂;和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应;以及(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞。在一个实施例中,靶基因座位于细胞的基因组中。在另一个实施例中,靶基因座位于细胞中的载体中。在一个实施例中,第一识别位点处的切口或双链断裂破坏第一选择标记的活性。在又一个实施例中,鉴定步骤(c)包括在允许鉴定没有第一选择标记活性的细胞的条件下培养细胞。在一个实施例中,包含第一选择标记的第一多核苷酸侧接第一靶位点和第二靶位点。在一个实施例中,鉴定步骤(c)包括鉴定包含在第一靶位点和第二靶位点处整合的第一插入多核苷酸的至少一个细胞。在一个实施例中,第一插入多核苷酸包含:(a)第一目标多核苷酸;和(b)编码第二选择标记并有效连接至细胞中有活性的第二启动子的第二多核苷酸,其中所述第二多核苷酸包含第二核酸酶试剂的第二识别位点。
在一个实施例中,所述方法还包括(a)向包含在靶基因座处整合的第一插入多核苷酸的细胞中引入(i)第二核酸酶试剂,其中所述第二核酸酶试剂在第二识别位点处诱导切口或双链断裂;和(ii)包含第二插入多核苷酸的第二靶向载体,所述第二插入多核苷酸侧接第三同源臂和第四同源臂,所述第三同源臂和第四同源臂与位于足够接近第二识别位点处的第三靶位点和第四靶位点相对应;以及(b)鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞。在一个实施例中,第二识别位点处的切口或双链断裂破坏第二选择标记的活性。在一个实施例中,鉴定步骤(b)包括在允许鉴定没有第二选择标记的活性的细胞的条件下培养细胞。在一个实施例中,包含第二选择标记的第二多核苷酸侧接第三靶位点和第四靶位点。在一个实施例中,鉴定步骤(b)包括鉴定包含在第三靶位点和第四靶位点处整合的第二插入多核苷酸的至少一个细胞。在一个实施例中,第二插入多核苷酸包含:(a)第二目标多核苷酸;和(b)编码第三选择标记并有效连接至细胞中有活性的第三启动子的第三多核苷酸,其中所述第三多核苷酸包含第三核酸酶试剂的第三识别位点。在一个实施例中,第一核酸酶试剂与第二核酸酶试剂不同。在一个实施例中,第一选择标记与第二选择标记不同。在一个实施例中,第一核酸酶识别位点和第三核酸酶识别位点彼此相同并与第二核酸酶识别位点不同;并且第一核酸酶试剂和第三核酸酶试剂彼此相同并与第二核酸酶试剂不同。在一个实施例中,第一选择标记和第三选择标记相同。在一个实施例中,第一选择标记、第二选择标记或第三选择标记中的一者赋予对抗生素的抗性。在一个实施例中,抗生素包括G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素。在一个实施例中,第一选择标记、第二选择标记或第三选择标记中的一者有效连接至诱导型启动子,并且选择标记的表达对细胞有毒性。在一个实施例中,第一选择标记、第二选择标记或第三选择标记包括次黄嘌呤-鸟嘌呤磷酸核糖转移酶(HGPRT)或单纯性疱疹病毒的胸苷激酶(HSV-TK)。在一个实施例中,所述细胞为原核细胞。在一个实施例中,所述细胞为真核细胞。在一个实施例中,所述真核细胞为哺乳动物细胞。在一个实施例中,所述哺乳动物细胞为非人哺乳动物细胞。在一个实施例中,所述哺乳动物细胞来自啮齿动物。在一个实施例中,所述啮齿动物为大鼠或小鼠。在一个实施例中,所述哺乳动物细胞为人成纤维细胞。
在一个实施例中,所述细胞为多能细胞。在一个实施例中,所述哺乳动物细胞为人诱导性多能干(iPS)细胞。在一个实施例中,所述多能细胞为非人胚胎干(ES)细胞。在一个实施例中,所述多能细胞为小鼠胚胎干(ES)细胞或大鼠胚胎干(ES)细胞。在一个实施例中,所述多能细胞为造血干细胞。在一个实施例中,所述多能细胞为神经元干细胞。
在一个实施例中,与单独使用第一靶向载体相比,联合使用第一靶向载体与第一核酸酶试剂会提高靶向效率。在一个实施例中,与单独使用第一靶向载体相比,第一靶向载体的靶向效率提高了至少2倍。
在一个实施例中,第一核酸酶试剂或第二核酸酶试剂包括表达构建体,所述表达构建体包含编码核酸酶试剂的核酸序列,并且所述核酸有效连接至细胞中有活性的第四启动子。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为编码核酸酶的mRNA。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为锌指核酸酶(ZFN)。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为转录激活因子样效应物核酸酶(TALEN)。在一个实施例中,第一核酸酶试剂或第二核酸酶试剂为大范围核酸酶。
在一个实施例中,第一核酸酶试剂或第二核酸酶试剂包括成簇规律间隔短回文重复序列(CRISPR)相关(Cas)蛋白和向导RNA(gRNA)。在一个实施例中,向导RNA(gRNA)包括(a)靶向第一识别位点、第二识别位点或第三识别位点的成簇规律间隔短回文重复序列(CRISPR)RNA(crRNA);和(b)反式激活CRISPR RNA(tracrRNA)。在一个实施例中,第一识别位点或第二识别位点紧密侧接前间区序列邻近基序(PAM)序列。在一个实施例中,目标基因组座位包含SEQ ID NO:1的核苷酸序列。在一个实施例中,Cas蛋白为Cas9。在一个实施例中,gRNA包括:(a)SEQ ID NO:2的核酸序列的嵌合RNA;或(b)SEQ ID NO:3的核酸序列的嵌合RNA。在一个实施例中,crRNA包含SEQ ID NO:4、SEQ ID NO:5或SEQ ID NO:6。在一个实施例中,tracrRNA包含SEQ ID NO:7或SEQ ID NO:8。在一个实施例中,第一识别位点、第二识别位点和/或第三识别位点位于第一选择标记、第二选择标记或第三选择标记的内含子、外显子、启动子、启动子调控区或增强子区中。在一个实施例中,第一靶位点和第二靶位点紧邻第一识别位点。在一个实施例中,第一靶位点和第二靶位点距第一识别位点约10个核苷酸至约14kb。在一个实施例中,第三靶位点和第四靶位点紧邻第二识别位点。在一个实施例中,第三靶位点和第四靶位点距第二识别位点约10个核苷酸至约14kb。在一个实施例中,第一同源臂和第二同源臂的总和为至少约10kb。在一个实施例中,第三同源臂和第四同源臂的总和为至少约10kb。在一个实施例中,第一插入多核苷酸的长度在约5kb至约300kb的范围内。在一个实施例中,第二插入多核苷酸的长度在约5kb至约300kb的范围内。在一个实施例中,将第一插入多核苷酸整合到靶基因座中会导致敲除、敲入、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换,或它们的组合。在一个实施例中,将第二插入多核苷酸整合到靶基因座中会导致敲除、敲入、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换,或它们的组合。在一个实施例中,第一插入多核苷酸包括含有人多核苷酸的目标多核苷酸。在一个实施例中,第二插入多核苷酸包括含有人多核苷酸的目标多核苷酸。在一个实施例中,第一插入多核苷酸包括含有T细胞受体α基因座区域的目标多核苷酸。在一个实施例中,第二插入多核苷酸包括含有T细胞受体α基因座区域的目标多核苷酸。在一个实施例中,第一插入多核苷酸或第二插入多核苷酸包括含有T细胞受体α基因座的至少一个可变区基因区段和/或连接区基因区段的目标多核苷酸。在一个实施例中,T细胞受体α基因座区域来源于人。在一个实施例中,第一插入多核苷酸包括含有有效连接至非人免疫球蛋白重链恒定区核酸序列的非重排人免疫球蛋白重链可变区核酸序列的目标多核苷酸。在一个实施例中,通过等位基因修饰(MOA)测定法来执行鉴定步骤。在一个实施例中,其中第一插入多核苷酸包括含有与细胞基因组中的核酸序列同源或直系同源的核酸序列的目标多核苷酸。在一个实施例中,第二插入多核苷酸包含与细胞基因组中的核酸序列同源或直系同源的核酸序列。在一个实施例中,第一插入多核苷酸包括含有外源核酸序列的目标多核苷酸。在一个实施例中,第二插入多核苷酸包括含有外源核酸序列的目标多核苷酸。
附图说明
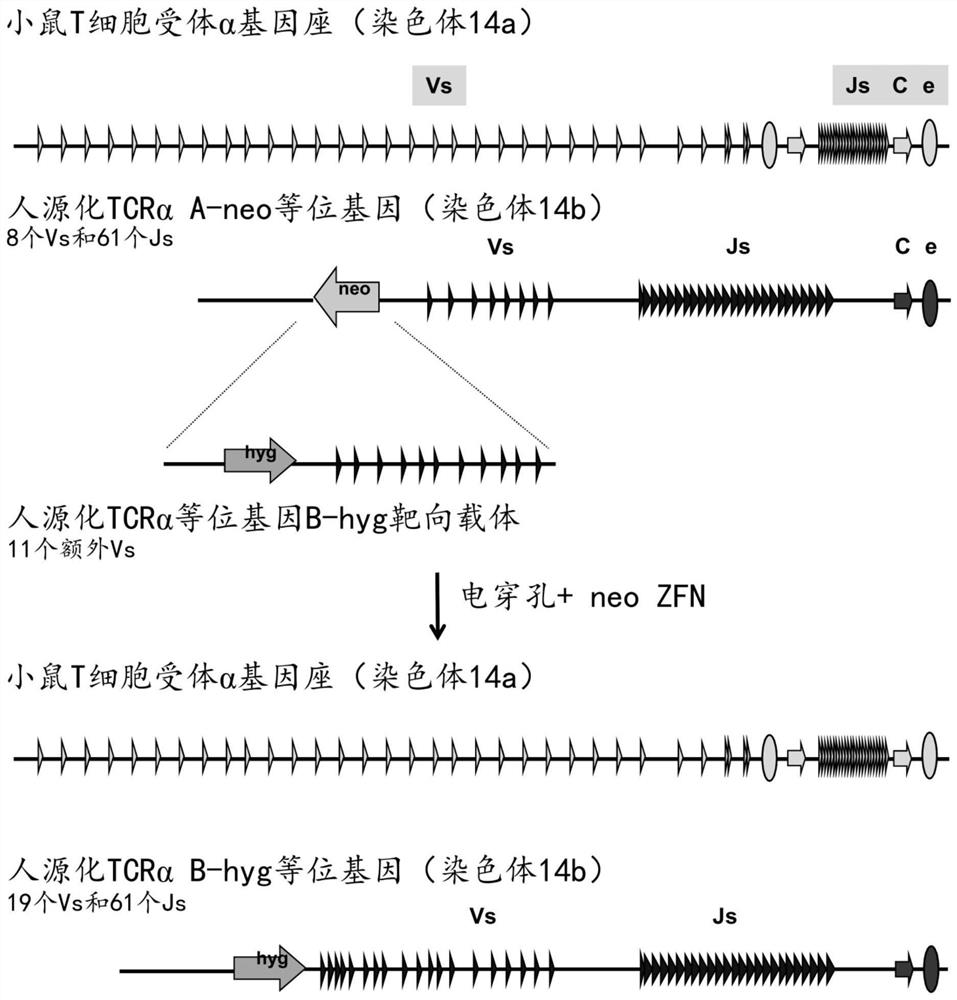
图1提供了基因组靶向事件的示意图,其中使用包含潮霉素选择盒及含11个额外人可变基因区段的大于100kb片段的人源化TCRα等位基因B-hyg靶向载体,来靶向在小鼠14号染色体上具有TCRα基因座的杂合修饰的细胞,该TCRα基因座的一个等位基因是人源化TCRαA-neo等位基因,其包含位于八个人可变(V)基因区段和61个人连接(J)基因区段上游的新霉素选择盒。用表达锌指核酸酶(ZFN)对(其靶向TCRαA-neo等位基因中的新霉素盒)两半的等位基因B-hyg靶向载体和质粒进行电穿孔,生成了经修饰的TCRα基因座(等位基因B-hyg),该经修饰的TCRα基因座从5’到3’包含位于内源恒定区核苷酸序列上游的潮霉素盒、19个人V基因区段和61个人J基因区段。该靶向事件将超过100kb的人TCRα基因序列精确插入到小鼠TCRα基因座中。
图2提供了基因组靶向事件的示意图,其中使用包含新霉素选择盒及含11个额外人可变基因区段的大于100kb片段的人源化TCRα等位基因C-neo靶向载体,来靶向在小鼠14号染色体上具有TCRα基因座的杂合修饰的细胞,该TCRα基因座的一个等位基因是人源化TCRαB-hyg等位基因,其包含位于19个人V基因区段和61个人J基因区段上游的潮霉素选择盒。用表达锌指核酸酶(ZFN)对(其靶向TCRαB-hyg等位基因中的潮霉素盒)两半的等位基因C-neo靶向载体和质粒进行电穿孔,生成了经修饰的TCRα基因座(等位基因C-neo),该经修饰的TCRα基因座从5’到3’包含位于内源恒定区核苷酸序列上游的新霉素盒、30个人V基因区段和61个人J基因区段。该靶向事件将超过100kb的人TCRα基因序列精确插入到小鼠TCRα基因座中。
图3提供了编码新霉素磷酸转移酶的neo
具体实施方式
现在将在下文中参照附图对本发明进行更充分地说明,附图只示出本发明的一些而不是所有的实施例。实际上,这些发明可按照许多不同的形式实施,并且不应理解为局限于本文所示出的实施例;相反,提供这些实施例是为了使得本公开满足适用的法定要求。整个文件中类似的编号指代类似的元素。
在获得以上说明和相关附图中给出的教导益处后,本发明所属领域的技术人员可以想到本发明的许多修改形式和其他实施例。因此,应当理解,本发明并不局限于所公开的具体实施例,并且这些修改形式和其他实施例也包括在所附权利要求的范围内。尽管本文采用了专用术语,但是它们仅用于一般的说明性的意义,而不是为了限制。
本发明提供了用于修饰细胞中的靶基因座(例如,基因组座位)的方法和组合物。所述方法和组合物采用核酸酶试剂和核酸酶试剂识别位点来增强插入多核苷酸在靶基因座中的同源重组事件。本文所提供的各种方法和组合物将核酸酶试剂识别位点策略性地定位在编码选择标记、报告基因或外源蛋白(例如,小鼠细胞中的eGFP或人序列)的多核苷酸内。
本发明还提供了允许在靶基因座(即,基因组座位)处对目标多核苷酸进行连续修饰(即,拼接)的方法。如下文更详细说明,提供了在靶基因座(即,基因组座位)中连续拼接目标多核苷酸的方法,其中所述靶基因座(即,基因组座位)和该方法中所采用的各种靶向载体交替使用包含第一核酸酶试剂的第一识别位点的第一选择标记以及包含第二核酸酶试剂的第二识别位点的第二选择标记。这样,该方法不需要不断供应被改造成识别新识别位点的核酸酶。相反,在具体实施例中,靶向连续拼接仅需要两种核酸酶试剂以及这两种核酸酶试剂的相应识别位点。此外,由于核酸酶试剂靶向外源序列(即,编码选择标记的多核苷酸内的识别位点)并且由于此前已确认任何给定识别位点的功效和脱靶效应,因此可最大程度减少内源基因组序列的非特异性切割,同时提高拼接过程的时间与成本效率。
本发明提供了用于修饰细胞中的靶基因座的方法和组合物。该系统采用核酸酶试剂、核酸酶试剂的识别位点、靶基因座、选择标记、靶向载体以及插入多核苷酸。下文更详细说明这些组分中的每一者。
术语“核酸酶试剂的识别位点”包括核酸酶试剂诱导切口或双链断裂处的DNA序列。核酸酶试剂的识别位点对于细胞可为内源的(或天然的),或者识别位点对于细胞可为外源的。在具体实施例中,识别位点对于细胞为外源的,从而在细胞基因组中不是天然存在的。在更进一步的实施例中,识别位点对于细胞为外源的,并且对于希望定位在靶基因座处的目标多核苷酸为外源的。在进一步的实施例中,外源或内源识别位点在宿主细胞的基因组中仅出现一次。在具体实施例中,鉴定了在基因组内仅出现一次的内源或天然位点。然后可使用这种位点来设计将在内源识别位点处产生切口或双链断裂的核酸酶试剂。
识别位点的长度可有所变化,并且包括例如对于锌指核酸酶(ZFN)对为约30-36bp(即,对于每个ZFN为约15-18bp)、对于转录激活因子样效应物核酸酶(TALEN)为约36bp,或对于CRISPR/Cas9向导RNA为约20bp的识别位点。
可在本文所公开的方法和组合物中使用在所需识别位点中诱导切口或双链断裂的任何核酸酶试剂。可采用天然存在的或天然的核酸酶试剂,只要核酸酶试剂在所需识别位点中诱导切口或双链断裂即可。另选地,可采用经修饰或经改造的核酸酶试剂。“经改造的核酸酶试剂”包括由其天然形式改造(修饰或衍生)成在所需识别位点中特异性识别并诱导切口或双链断裂的核酸酶。因此,经改造的核酸酶试剂可来源于天然的或天然存在的核酸酶试剂,或者可人工生成或合成。核酸酶试剂的修饰在蛋白切割剂中可少至一个氨基酸,或在核酸切割剂中可少至一个核苷酸。在一些实施例中,经改造的核酸酶在识别位点中诱导切口或双链断裂,其中所述识别位点不是会被天然(未经改造的或未经修饰的)核酸酶试剂识别的序列。在识别位点或其他DNA中产生切口或双链断裂在本文中可称为“切断”或“切割”识别位点或其他DNA。
还提供了示例性识别位点的活性变体和片段。此类活性变体可与给定识别位点具有至少65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中所述活性变体保留生物活性,从而能够被核酸酶试剂以序列特异性方式识别并切割。测量核酸酶试剂对识别位点进行双链断裂的测定法是本领域已知的(例如,
在具体实施例中,识别位点定位在编码选择标记的多核苷酸内。这种位置可位于选择标记的编码区内或者位于影响选择标记表达的调控区内。因此,核酸酶试剂的识别位点可位于选择标记的内含子、编码选择标记的多核苷酸的启动子、增强子、调控区或任何非蛋白编码区中。在具体实施例中,识别位点处的切口或双链断裂破坏选择标记的活性。测定功能选择标记存在与否的方法是已知的。
在一个实施例中,核酸酶试剂为转录激活因子样效应物核酸酶(TALEN)。TAL效应物核酸酶是可用于在原核或真核生物基因组中的特异性靶序列处产生双链断裂的一类序列特异性核酸酶。可通过如下方式生成TAL效应物核酸酶:将天然的或经改造的转录激活因子样(TAL)效应物或其功能部分融合到核酸内切酶如FokI的催化结构域。独特的模块化TAL效应物DNA结合结构域允许设计潜在具有任何给定DNA识别特异性的蛋白。因此,TAL效应物核酸酶的DNA结合结构域可被改造成识别特异性DNA靶位点,故可用于在所需靶序列处产生双链断裂。参见WO 2010/079430;Morbitzer et al.(2010)PNAS 10.1073/pnas.1013133107(Morbitzer等人,2010年,《美国国家科学院院刊》,10.1073/pnas.1013133107);Scholze&Boch(2010)Virulence 1:428-432(Scholze和Boch,2010年,《毒力》,第1卷,第428-432页);Christian et al.Genetics(2010)186:757-761(Christian等人,《遗传学》,2010年,第186卷,第757-761页);Li et al.(2010)Nuc.Acids Res.(2010)doi:10.1093/nar/gkq704(Li等人,2010年,《核酸研究》,2010年,doi:10.1093/nar/gkq704);以及Miller et al.(2011)Nature Biotechnology29:143–148(Miller等人,2011年,《自然生物技术》,第29卷,第143–148页);所有这些文献均以引用的方式并入本文。
合适TAL核酸酶的实例以及用于制备合适TAL核酸酶的方法公开于例如美国专利申请No.2011/0239315 A1、No.2011/0269234 A1、No.2011/0145940 A1、No.2003/0232410A1、No.2005/0208489 A1、No.2005/0026157 A1、No.2005/0064474 A1、No.2006/0188987A1和No.2006/0063231 A1中(每一份专利申请均据此以引用的方式并入本文)。在各种实施例中,TAL效应物核酸酶被改造成在例如目标基因座或目标基因组座位中的靶核酸序列中或附近进行切断,其中所述靶核酸序列位于靶向载体将要修饰的序列处或附近。适合与本文所提供的各种方法和组合物一起使用的TAL核酸酶包括被特别设计成在本文所述的靶向载体将要修饰的靶核酸序列处或附近结合的那些TAL核酸酶。
在一个实施例中,TALEN的每个单体包含经由两个高变残基识别单碱基对的33-35个TAL重复序列。在一个实施例中,核酸酶试剂为嵌合蛋白,其包含有效连接至独立核酸酶的基于TAL重复序列的DNA结合结构域。在一个实施例中,独立核酸酶为FokI核酸内切酶。在一个实施例中,核酸酶试剂包含第一基于TAL重复序列的DNA结合结构域和第二基于TAL重复序列的DNA结合结构域,其中所述第一基于TAL重复序列的DNA结合结构域和第二基于TAL重复序列的DNA结合结构域中的每一者均有效连接至FokI核酸酶亚基,其中所述第一基于TAL重复序列的DNA结合结构域和第二基于TAL重复序列的DNA结合结构域识别DNA靶序列每条链中被不同长度(12-20bp)的间隔序列隔开的两条邻接DNA靶序列,并且其中所述FokI核酸酶亚基二聚化以生成能在靶序列处产生双链断裂的活性核酸酶。
在本文所公开的各种方法和组合物中采用的核酸酶试剂还可包括锌指核酸酶(ZFN)。在一个实施例中,ZFN的每个单体包含3个或更多个基于锌指的DNA结合结构域,其中每个基于锌指的DNA结合结构域结合到3bp亚位点。在其他实施例中,ZFN为嵌合蛋白,其包含有效连接至独立核酸酶的基于锌指的DNA结合结构域。在一个实施例中,独立核酸内切酶为FokI核酸内切酶。在一个实施例中,核酸酶试剂包含第一ZFN和第二ZFN,其中所述第一ZFN和第二ZFN中的每一者均有效连接至FokI核酸酶亚基,其中所述第一ZFN和第二ZFN识别DNA靶序列每条链中被约5-7bp间隔序列隔开的两条邻接DNA靶序列,并且其中所述FokI核酸酶亚基二聚化以生成能产生双链断裂的活性核酸酶。参见例如US20060246567;US20080182332;US20020081614;US20030021776;WO/2002/057308A2;US20130123484;US20100291048;WO/2011/017293A2;以及Gaj et al.(2013)Trends in Biotechnology,31(7):397-405(Gaj等人,2013年,《生物技术趋势》,第31卷,第7期,第397-405页);每篇文献均以引用的方式并入本文。
在又一个实施例中,核酸酶试剂为大范围核酸酶。已基于保守序列基序将大范围核酸酶分类为四个家族,这些家族为LAGLIDADG、GIY-YIG、H-N-H和His-Cys框家族。这些基序参与金属离子的配位和磷酸二酯键的水解。大范围核酸酶以其长识别位点以及耐受其DNA底物中的一些序列多态性而著称。大范围核酸酶结构域、结构和功能是已知的,参见例如,Guhan and Muniyappa(2003)Crit Rev Biochem Mol Biol 38:199-248(Guhan和Muniyappa,2003年,《生物化学与分子生物学评论》,第38卷,第199-248页);Lucas et al.,(2001)Nucleic Acids Res 29:960-9(Lucas等人,2001年,《核酸研究》,第29卷,第960-969页);Jurica and Stoddard,(1999)Cell Mol Life Sci 55:1304-26(Jurica和Stoddard,1999年,《细胞和分子生命科学》,第55卷,第1304-1326页);Stoddard,(2006)Q RevBiophys 38:49-95(Stoddard,2006年,《生物物理学季评》,第38卷,第49-95页);以及Moureet al.,(2002)Nat Struct Biol 9:764(Moure等人,2002年,《自然结构生物学》,第9卷,第764页)。在一些实例中,使用天然存在的变体和/或经改造的衍生大范围核酸酶。用于调整动力学、辅因子相互作用、表达、最适条件和/或识别位点特异性以及活性筛选的方法是已知的,参见例如,Epinat et al.,(2003)Nucleic Acids Res 31:2952-62(Epinat等人,2003年,《核酸研究》,第31卷,第2952-2962页);Chevalier et al.,(2002)Mol Cell 10:895-905(Chevalier等人,2002年,《分子细胞》,第10卷,第895-905页);Gimble et al.,(2003)Mol Biol 334:993-1008(Gimble等人,2003年,《分子生物学》,第334卷,第993-1008页);Seligman et al.,(2002)Nucleic Acids Res 30:3870-9(Seligman等人,2002年,《核酸研究》,第30卷,第3870-3879页);Sussman et al.,(2004)J Mol Biol 342:31-41(Sussman等人,2004年,《分子生物学杂志》,第342卷,第31-41页);Rosen et al.,(2006)Nucleic Acids Res 34:4791-800(Rosen等人,2006年,《核酸研究》,第34卷,第4791-4800页);Chames et al.,(2005)Nucleic Acids Res 33:e178(Chames等人,2005年,《核酸研究》,第33卷,第e178页);Smith et al.,(2006)Nucleic Acids Res 34:e149(Smith等人,2006年,《核酸研究》,第34卷,第e149页);Gruen et al.,(2002)Nucleic Acids Res 30:e29(Gruen等人,2002年,《核酸研究》,第30卷,第e29页);Chen and Zhao,(2005)NucleicAcids Res 33:e154(Chen和Zhao,2005年,《核酸研究》,第33卷,第e154页);WO2005105989;WO2003078619;WO2006097854;WO2006097853;WO2006097784;以及WO2004031346。
可在本文中使用任何大范围核酸酶,包括但不限于I-SceI、I-SceII、I-SceIII、I-SceIV、I-SceV、I-SceVI、I-SceVII、I-CeuI、I-CeuAIIP、I-CreI、I-CrepsbIP、I-CrepsbIIP、I-CrepsbIIIP、I-CrepsbIVP、I-TliI、I-PpoI、PI-PspI、F-SceI、F-SceII、F-SuvI、F-TevI、F-TevII、I-AmaI、I-AniI、I-ChuI、I-CmoeI、I-CpaI、I-CpaII、I-CsmI、I-CvuI、I-CvuAIP、I-DdiI、I-DdiII、I-DirI、I-DmoI、I-HmuI、I-HmuII、I-HsNIP、I-LlaI、I-MsoI、I-NaaI、I-NanI、I-NcIIP、I-NgrIP、I-NitI、I-NjaI、I-Nsp236IP、I-PakI、I-PboIP、I-PcuIP、I-PcuAI、I-PcuVI、I-PgrIP、I-PobIP、I-PorI、I-PorIIP、I-PbpIP、I-SpBetaIP、I-ScaI、I-SexIP、I-SneIP、I-SpomI、I-SpomCP、I-SpomIP、I-SpomIIP、I-SquIP、I-Ssp6803I、I-SthPhiJP、I-SthPhiST3P、I-SthPhiSTe3bP、I-TdeIP、I-TevI、I-TevII、I-TevIII、I-UarAP、I-UarHGPAIP、I-UarHGPA13P、I-VinIP、I-ZbiIP、PI-MtuI、PI-MtuHIP PI-MtuHIIP、PI-PfuI、PI-PfuII、PI-PkoI、PI-PkoII、PI-Rma43812IP、PI-SpBetaIP、PI-SceI、PI-TfuI、PI-TfuII、PI-ThyI、PI-TliI、PI-TliII,或其任何活性变体或片段。
在一个实施例中,所述大范围核酸酶识别12至40个碱基对的双链DNA序列。在一个实施例中,所述大范围核酸酶识别基因组中的一个完全匹配的靶序列。在一个实施例中,所述大范围核酸酶为归巢核酸酶。在一个实施例中,所述归巢核酸酶为归巢核酸酶的LAGLIDADG家族。在一个实施例中,归巢核酸酶的LAGLIDADG家族选自I-SceI、I-CreI和I-Dmol。
核酸酶试剂还可包括限制性核酸内切酶,它们包括I型、II型、III型和IV型核酸内切酶。I型和III型限制性核酸内切酶识别特异性识别位点,但通常在距核酸酶结合位点的可变位置处切割,该核酸酶结合位点离切割位点(识别位点)可达数百个碱基对。在II型系统中,限制性酶切活性独立于任何甲基化酶活性,并且通常在结合位点内或附近的特异性位点处发生切割。大多数II型酶切断回文序列,但是IIa型酶识别非回文识别位点并在识别位点之外切割,IIb型酶在识别位点之外的两个位点处切断序列两次,并且IIs型酶识别非对称识别位点并在一侧且距识别位点约1-20个核苷酸的限定距离处切割。IV型限制性酶靶向甲基化DNA。进一步在例如REBASE数据库中说明和分类了限制性酶(地址为rebase.neb.com的网页;Roberts et al.,(2003)Nucleic Acids Res 31:418-20(Roberts等人,2003年,《核酸研究》,第31卷,第418-420页),Roberts et al.,(2003)Nucleic AcidsRes 31:1805-12(Roberts等人,2003年,《核酸研究》,第31卷,第1805-1812页),以及Belfort et al.,(2002)in Mobile DNA II,pp.761-783,Eds.Craigie et al.,(ASMPress,Washington,DC)(Belfort等人,2002年,载于《可移动的DNA II》,第761-783页,Craigie等人编辑,华盛顿特区ASM出版社))。
在各种方法和组合物中采用的核酸酶试剂还可包括CRISPR/Cas系统。此类系统可采用Cas9核酸酶,在某些情况下针对要在其中表达的所需细胞类型进行密码子优化。该系统还采用融合的crRNA-tracrRNA构建体,该构建体与经密码子优化的Cas9一起发挥作用。该单一RNA通常称为向导RNA或gRNA。在gRNA内,crRNA部分被确定为给定识别位点的“靶序列”,并且tracrRNA通常称为“支架”。已证实该系统可在多种真核细胞和原核细胞中发挥作用。简而言之,包含靶序列的短DNA片段被插入到向导RNA表达质粒中。gRNA表达质粒包含靶序列(在一些实施例中约20个核苷酸)、一种形式的tracrRNA序列(支架)以及细胞中有活性的合适启动子及用于在真核细胞中正确加工的必要元件。这些系统中的许多系统依赖于定制的互补寡核苷酸,这些寡核苷酸退火形成双链DNA,接着克隆到gRNA表达质粒中。然后将gRNA表达盒和Cas9表达盒引入细胞中。参见例如Mali P et al.(2013)Science 2013Feb15;339(6121):823-6(Mali P等人,2013年,《科学》,2013年2月15日,第339卷,第6121期,第823-826页);Jinek M et al.Science 2012Aug 17;337(6096):816-21(Jinek M等人,《科学》,2012年8月17日,第337卷,第6096期,第816-821页);Hwang WY et al.Nat Biotechnol2013Mar;31(3):227-9(Hwang WY等人,《自然生物技术》,2013年3月,第31卷,第3期,第227-229页);Jiang W et al.Nat Biotechnol 2013Mar;31(3):233-9(Jiang W等人,《自然生物技术》,2013年3月,第31卷,第3期,第233-239页);以及Cong L et al.Science2013Feb 15;339(6121):819-23(Cong L等人,《科学》,2013年2月15日,第339卷,第6121期,第819-823页),每篇文献均以引用的方式并入本文。
本文所公开的方法和组合物可利用成簇规律间隔短回文重复序列(CRISPR)/CRISPR相关(Cas)系统或此类系统的组分来修饰细胞内的基因组。CRISPR/Cas系统包括参与Cas基因的表达或指导Cas基因的活性的转录物和其他元件。CRISPR/Cas系统可为I型、II型或III型系统。本文所公开的方法和组合物通过利用CRISPR复合物(包含与Cas蛋白复合的向导RNA(gRNA))来采用CRISPR/Cas系统对核酸进行定点切割。
用于本文所公开的方法中的一些CRISPR/Cas系统是非天然存在的。“非天然存在的”系统包括指示受到人工干预的任何系统,诸如系统的一个或多个组分从其天然存在的状态改变或突变,至少基本上不含其在自然界中与之天然相关联的至少一个其他组分,或和其不与之天然相关联的至少一个其他组分相关联。例如,一些CRISPR/Cas系统采用非天然存在的CRISPR复合物,这些复合物包含在天然情况下不会同时存在的gRNA和Cas蛋白。
Cas蛋白通常包含至少一个RNA识别或结合结构域。此类结构域可与向导RNA(gRNA,下文更详细说明)相互作用。Cas蛋白还可包含核酸酶结构域(例如,DNA酶或RNA酶结构域)、DNA结合结构域、解旋酶结构域、蛋白-蛋白相互作用结构域、二聚化结构域以及其他结构域。核酸酶结构域具有用于核酸切割的催化活性。切割包括核酸分子共价键的断裂。切割可产生平头末端或交错末端,并且其可为单链或双链的。
Cas蛋白的实例包括Cas1、Cas1B、Cas2、Cas3、Cas4、Cas5、Cas5e(CasD)、Cas6、Cas6e、Cas6f、Cas7、Cas8a1、Cas8a2、Cas8b、Cas8c、Cas9(Csn1或Csx12)、Cas10、Casl0d、CasF、CasG、CasH、Csy1、Csy2、Csy3、Cse1(CasA)、Cse2(CasB)、Cse3(CasE)、Cse4(CasC)、Csc1、Csc2、Csa5、Csn2、Csm2、Csm3、Csm4、Csm5、Csm6、Cmr1、Cmr3、Cmr4、Cmr5、Cmr6、Csb1、Csb2、Csb3、Csx17、Csx14、Csx10、Csx16、CsaX、Csx3、Csx1、Csx15、Csf1、Csf2、Csf3、Csf4和Cu1966,以及它们的同源物或修饰形式。
Cas蛋白可来自II型CRISPR/Cas系统。例如,Cas蛋白可为Cas9蛋白或来源于Cas9蛋白。这些Cas9蛋白通常共用具有保守架构的四个关键基序。基序1、2和4为RuvC样基序,并且基序3为HNH基序。Cas9蛋白可来自例如酿脓链球菌(Streptococcus pyogenes)、嗜热链球菌(Streptococcus thermophilus)、链球菌属物种(Streptococcus sp.)、金黄色葡萄球菌(Staphylococcus aureus)、达氏拟诺卡氏菌(Nocardiopsis dassonvillei)、始旋链霉菌(Streptomyces pristinaespiralis)、绿色产色链霉菌(Streptomycesviridochromogenes)、绿色产色链霉菌(Streptomyces viridochromogenes)、粉红链孢囊菌(Streptosporangium roseum)、粉红链孢囊菌(Streptosporangium roseum)、酸热脂环酸芽孢杆菌(AlicyclobacHlus acidocaldarius)、假蕈状芽孢杆菌(Bacilluspseudomycoides)、还原硒酸盐芽抱杆菌(Bacillus selenitireducens)、西伯利亚微小杆菌(Exiguobacterium sibiricum)、德氏乳杆菌(Lactobacillus delbrueckii)、唾液乳杆菌(Lactobacillus salivarius)、海洋微颤菌(Microscilla marina)、伯克氏菌(Burkholderiales bacterium)、萘降解极地单胞菌(Polaromonas naphthalenivorans)、极地单胞菌属物种(Polaromonas sp.)、瓦氏鳄球藻(Crocosphaera watsonii)、蓝杆藻属物种(Cyanothece sp.)、铜绿微囊藻(Microcystis aeruginosa)、聚球藻属物种(Synechococcus sp.)、阿拉伯糖醋盐杆菌(Acetohalobium arabaticum)、制氨菌(Ammonifex degensii)、热解纤维素菌(Caldicelulosiruptor becscii)、CandidatusDesulforudis、肉毒梭菌(Clostridium botulinum)、艰难梭菌(Clostridium difficile)、大芬戈尔德菌(Finegoldia magna)、嗜热盐碱厌氧菌(Natranaerobius thermophilus)、丙酸互营细菌(Pelotomaculum thermopropionicum)、喜温嗜酸硫杆菌(Acidithiobacilluscaldus)、嗜酸氧化亚铁硫杆菌(Acidithiobacillus ferrooxidans)、紫色硫细菌(Allochromatium vinosum)、海杆菌属物种(Marinobactersp.)、嗜盐亚硝化球菌(Nitrosococcushalophilus)、瓦氏亚硝化球菌(Nitrosococcuswatsoni)、游海假交替单胞菌(Pseudoalteromonashaloplanktis)、纤线杆菌(Ktedonobacterracemifer)、甲烷盐菌(Methanohalobiumevestigatum)、多变鱼腥藻Anabaenavariabilis)、泡沫节球藻(Nodularia spumigena)、念珠藻属物种(Nostoc sp.)、极大节螺藻(Arthrospiramaxima)、钝顶节螺藻(Arthrospira platensis)、节螺藻属物种(Arthrospira sp.)、鞘丝藻属物种(Lyngbya sp.)、原型微鞘藻(Microcoleus chthonoplastes)、颤藻属物种(Oscillatoria sp.)、运动石袍菌(Petrotoga mobilis)、非洲栖热腔菌(Thermosiphoafricanus)或深海单细胞蓝细菌(Acaryochloris marina)。Cas9家族成员的另外实例在WO2014/131833中有所描述,该专利全文以引用的方式并入本文。来自酿脓链球菌(S.pyogenes)或从其衍生的Cas9蛋白是优选的酶。为来自酿脓链球菌的Cas9蛋白指定了SwissProt登录号Q99ZW2。
Cas蛋白可为野生型蛋白(即,自然界存在的蛋白)、经修饰的Cas蛋白(即,Cas蛋白变体)或者野生型或经修饰的Cas蛋白的片段。Cas蛋白还可以是野生型或经修饰的Cas蛋白的活性变体或片段。活性变体或片段可与野生型或经修饰的Cas蛋白或者其一部分具有至少80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中所述活性变体保留了在所需切割位点处切断的能力,从而保留了切口诱导活性或双链断裂诱导活性。针对切口诱导活性或双链断裂诱导活性的测定法是已知的,并且一般测量Cas蛋白对包含切割位点的DNA底物的总体活性和特异性。
可修饰Cas蛋白以提高或降低核酸结合亲和力、核酸结合特异性和/或酶活性。还可修饰Cas蛋白以改变蛋白的任何其他活性或特性,诸如稳定性。例如,Cas蛋白的一个或多个核酸酶结构域可被修饰、缺失或失活,或者Cas蛋白可被截短以去除对于蛋白质的功能并非必要的结构域,或优化(例如,增强或降低)Cas蛋白的活性。
一些Cas蛋白包含至少两个核酸酶结构域,诸如DNA酶结构域。例如,Cas9蛋白可包含RuvC样核酸酶结构域和HNH样核酸酶结构域。RuvC结构域和HNH结构域各自可切断双链DNA的不同链,从而在DNA中产生双链断裂。参见例如Jinek et al.(2012)Science 337:816-821(Jinek等人,2012年,《科学》,第337卷,第816-821页),该文献全文据此以引用的方式并入本文。
这些核酸酶结构域中的一者或两者可被缺失或突变,使得它们不再发挥功能或具有降低的核酸酶活性。如果这两个核酸酶结构域中的一者被缺失或突变,则所得的Cas蛋白(例如,Cas9)可称为切口酶,并且可在双链DNA内的CRISPR RNA识别序列处生成单链断裂,而不会生成双链断裂(即,其可切割互补链或非互补链,但无法同时切割两者)。如果这两个核酸酶结构域都被缺失或突变,则所得的Cas蛋白(例如,Cas9)将具有降低的切割双链DNA两条链的能力。将Cas9转化为切口酶的突变的实例是来自酿脓链球菌的Cas9的RuvC结构域中的D10A(Cas9的第10位处天冬氨酸至丙氨酸)突变。同样,来自酿脓链球菌的Cas9的HNH结构域中的H939A(氨基酸位置839处组氨酸至丙氨酸)或H840A(氨基酸位置840处组氨酸至丙氨酸)可将Cas9转化为切口酶。将Cas9转化为切口酶的突变的其他实例包括来自嗜热链球菌(S.thermophilus)的Cas9的对应突变。参见例如Sapranauskas et al.(2011)NucleicAcids Research39:9275-9282(Sapranauskas等人,2011年,《核酸研究》,第39卷,第9275-9282页)和WO 2013/141680,每篇文献全文均以引用的方式并入本文。此类突变可使用诸如定点诱变、PCR介导的诱变或全基因合成的方法来生成。形成切口酶的其他突变的实例可见于例如WO/2013/176772A1和WO/2013/142578A1中,这些专利中的每一者均以引用的方式并入本文。
Cas蛋白也可为融合蛋白。例如,Cas蛋白可融合到切割结构域、表观遗传修饰结构域、转录激活结构域或转录阻遏物结构域。参见WO2014/089290,该专利全文以引用的方式并入本文。Cas蛋白也可融合到异源多肽,从而提供增强或减弱的稳定性。融合的结构域或异源多肽可位于N端、C端或Cas蛋白的内部。
Cas蛋白可融合到有助于亚细胞定位的异源多肽。此类异源肽包括例如用于靶向细胞核的核定位信号(NLS)例如SV40 NLS、用于靶向线粒体的线粒体定位信号、ER滞留信号等。参见例如Lange et al.(2007)J.Biol.Chem.282:5101-5105(Lange等人,2007年,《生物化学杂志》,第282卷,第5101-5105页)。此类亚细胞定位信号可位于N端、C端或Cas蛋白内的任何位置处。NLS可包含一段碱性氨基酸,并且可为单分型序列或双分型序列。
Cas蛋白也可连接至细胞穿透结构域。例如,细胞穿透结构域可来源于HIV-1TAT蛋白、来自人乙肝病毒的TLM细胞穿透基序、MPG、Pep-1、VP22、来自单纯性疱疹病毒的细胞穿透肽,或多聚精氨酸肽序列。参见例如WO 2014/089290,该专利全文以引用的方式并入本文。细胞穿透结构域可位于N端、C端或Cas蛋白内的任何位置处。
Cas蛋白还可包含易于示踪或纯化的异源多肽,诸如荧光蛋白、纯化标签或表位标签。荧光蛋白的实例包括绿色荧光蛋白(例如,GFP、GFP-2、tagGFP、turboGFP、eGFP、Emerald、Azami Green、Monomeric Azami Green、CopGFP、AceGFP、ZsGreenl)、黄色荧光蛋白(例如,YFP、eYFP、Citrine、Venus、YPet、PhiYFP、ZsYellowl)、蓝色荧光蛋白(例如,eBFP、eBFP2、Azurite、mKalamal、GFPuv、Sapphire、T-sapphire)、青色荧光蛋白(例如,eCFP、Cerulean、CyPet、AmCyanl、Midoriishi-Cyan)、红色荧光蛋白(mKate、mKate2、mPlum、DsRedmonomer、mCherry、mRFP1、DsRed-Express、DsRed2、DsRed-Monomer、HcRed-Tandem、HcRedl、AsRed2、eqFP611、mRaspberry、mStrawberry、Jred)、橙色荧光蛋白(mOrange、mKO、Kusabira-Orange、Monomeric Kusabira-Orange、mTangerine、tdTomato)以及任何其他合适的荧光蛋白。标签的实例包括谷胱甘肽-S-转移酶(GST)、几丁质结合蛋白(CBP)、麦芽糖结合蛋白、硫氧还蛋白(TRX)、聚(NANP)、串联亲和纯化(TAP)标签、myc、AcV5、AU1、AU5、E、ECS、E2、FLAG、血凝素(HA)、nus、Softag 1、Softag 3、Strep、SBP、Glu-Glu、HSV、KT3、S、S1、T7、V5、VSV-G、组氨酸(His)、生物素羧基载体蛋白(BCCP)以及钙调蛋白。
Cas蛋白可以任何形式提供。例如,Cas蛋白可以蛋白的形式提供,诸如与gRNA复合的Cas蛋白。另选地,Cas蛋白可以编码Cas蛋白的核酸的形式提供,诸如RNA(例如,信使RNA(mRNA))或DNA。任选地,编码Cas蛋白的核酸可进行密码子优化,以便在特定细胞或生物体中有效翻译成蛋白。
编码Cas蛋白的核酸可稳定整合在细胞的基因组中,并有效连接至细胞中有活性的启动子。另选地,编码Cas蛋白的核酸可有效连接至表达构建体中的启动子。表达构建体包括能够指导目标基因或其他核酸序列(例如,Cas基因)的表达并可将这种目标核酸序列转移到靶细胞中的任何核酸构建体。可用于表达构建体的启动子包括例如在大鼠、真核、哺乳动物、非人哺乳动物、人、啮齿动物、小鼠或仓鼠多能细胞中有活性的启动子。其他启动子的实例在本文别处有所描述。
“向导RNA”或“gRNA”包括结合到Cas蛋白并使Cas蛋白靶向靶DNA内特定位置的RNA分子。向导RNA可包含两个区段:“DNA靶向区段”和“蛋白结合区段”。“区段”包括分子的区段、部分或区域,诸如RNA中的一个邻接核苷酸段。一些gRNA包含两个单独的RNA分子:“激活因子-RNA”和“靶向因子-RNA”。其他gRNA为单个RNA分子(单条RNA多核苷酸),其也可称为“单分子gRNA”、“单向导RNA”或“sgRNA”。参见例如WO/2013/176772A1、WO/2014/065596A1、WO/2014/089290A1、WO/2014/093622A2、WO/2014/099750A2、WO/2013142578A1以及WO2014/131833A1,这些专利中的每一者均以引用的方式并入本文。术语“向导RNA”和“gRNA”包括双分子gRNA和单分子gRNA。
示例性双分子gRNA包含crRNA样(“CRISPR RNA”或“靶向因子-RNA”或“crRNA”或“crRNA重复序列”)分子以及对应的tracrRNA样(“反式作用CRISPR RNA”或“激活因子-RNA”或“tracrRNA”或“支架”)分子。crRNA包含gRNA的DNA靶向区段(单链)和一段核苷酸,该段核苷酸形成gRNA的蛋白结合区段的dsRNA双链体的一半。
对应的tracrRNA(激活因子-RNA)包含一段核苷酸,该段核苷酸形成gRNA的蛋白结合区段的dsRNA双链体的另一半。crRNA的一段核苷酸与tracrRNA的一段核苷酸互补并杂交,以形成gRNA的蛋白结合结构域的dsRNA双链体。因此,可以说每个crRNA具有对应的tracrRNA。
crRNA和对应的tracrRNA杂交以形成gRNA。crRNA另外还提供了与CRISPR RNA识别序列杂交的单链DNA靶向区段。如果用于细胞内的修饰,则给定crRNA或tracrRNA分子的准确序列可被设计成对于将在其中使用RNA分子的物种具有特异性。参见例如Mali et al.(2013)Science339:823-826(Mali等人,2013年,《科学》,第339卷,第823-826页);Jinek etal.(2012)Science 337:816-821(Jinek等人,2012年,《科学》,第337卷,第816-821页);Hwang et al.(2013)Nat.Biotechnol.31:227-229(Hwang等人,2013年,《自然生物技术》,第31卷,第227-229页);Jiang et al.(2013)Nat.Biotechnol.31:233-239(Jiang等人,2013年,《自然生物技术》,第31卷,第233-239页);以及Cong et al.(2013)Science339:819-823(Cong等人,2013年,《科学》,第339卷,第819-823页);每篇文献均以引用的方式并入本文。
给定gRNA的DNA靶向区段(crRNA)包含与靶DNA中的序列互补的核苷酸序列。gRNA的DNA靶向区段通过杂交(即,碱基配对)以序列特异性方式与靶DNA相互作用。因此,DNA靶向区段的核苷酸序列可有所变化,并且决定gRNA和靶DNA将与之相互作用的靶DNA内的位置。可修饰对象gRNA的DNA靶向区段,以与靶DNA内的任何所需序列杂交。天然存在的crRNA根据Cas9系统和生物体不同而不同,但通常包含21至72个核苷酸长度的靶向区段,该靶向区段侧接21至46个核苷酸长度的两个正向重复序列(DR)(参见例如WO2014/131833)。就酿脓链球菌而言,DR为36个核苷酸长,并且靶向区段为30个核苷酸长。位于3’的DR与对应的tracrRNA互补并杂交,继而结合于Cas9蛋白。
DNA靶向区段的长度可为约12个核苷酸至约100个核苷酸。例如,DNA靶向区段的长度可为约12个核苷酸(nt)至约80nt、约12nt至约50nt、约12nt至约40nt、约12nt至约30nt、约12nt至约25nt、约12nt至约20nt,或约12nt至约19nt。另选地,DNA靶向区段的长度可为约19nt至约20nt、约19nt至约25nt、约19nt至约30nt、约19nt至约35nt、约19nt至约40nt、约19nt至约45nt、约19nt至约50nt、约19nt至约60nt、约19nt至约70nt、约19nt至约80nt、约19nt至约90nt、约19nt至约100nt、约20nt至约25nt、约20nt至约30nt、约20nt至约35nt、约20nt至约40nt、约20nt至约45nt、约20nt至约50nt、约20nt至约60nt、约20nt至约70nt、约20nt至约80nt、约20nt至约90nt,或约20nt至约100nt。
与靶DNA的核苷酸序列(CRISPR RNA识别序列)互补的DNA靶向区段的核苷酸序列可具有至少约12nt的长度。例如,DNA靶向序列(即,与靶DNA内的CRISPR RNA识别序列互补的DNA靶向区段内的序列)可具有至少约12nt、至少约15nt、至少约18nt、至少约19nt、至少约20nt、至少约25nt、至少约30nt、至少约35nt或至少约40nt的长度。另选地,DNA靶向序列的长度可为约12个核苷酸(nt)至约80nt、约12nt至约50nt、约12nt至约45nt、约12nt至约40nt、约12nt至约35nt、约12nt至约30nt、约12nt至约25nt、约12nt至约20nt、约12nt至约19nt、约19nt至约20nt、约19nt至约25nt、约19nt至约30nt、约19nt至约35nt、约19nt至约40nt、约19nt至约45nt、约19nt至约50nt、约19nt至约60nt、约20nt至约25nt、约20nt至约30nt、约20nt至约35nt、约20nt至约40nt、约20nt至约45nt、约20nt至约50nt,或约20nt至约60nt。在一些情况下,DNA靶向序列可具有至少约20nt的长度。
TracrRNA可为任何形式(例如,全长tracrRNA或活性部分tracrRNA)并具有不同长度。它们可包括初级转录物或加工形式。例如,tracrRNA(作为单向导RNA的一部分或作为双分子gRNA一部分形式的单独分子)可包含以下部分或由以下部分组成:野生型tracrRNA序列的全部或一部分(例如,野生型tracrRNA序列的约或大于约20、26、32、45、48、54、63、67、85个或更多个核苷酸)。来自酿脓链球菌的野生型tracrRNA序列的实例包括171个核苷酸、89个核苷酸、75个核苷酸以及65个核苷酸的形式。参见例如Deltcheva et al.(2011)Nature 471:602-607(Deltcheva等人,2011年,《自然》,第471卷,第602-607页);WO 2014/093661,每篇文献的全文均以引用的方式并入本文。单向导RNA(sgRNA)内的tracrRNA的实例包括存在于+48、+54、+67和+85形式的sgRNA内的tracrRNA区段,其中“+n”指示野生型tracrRNA的至多+n个核苷酸包含在sgRNA中。参见US8,697,359,该专利全文以引用的方式并入本文。
DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比可为至少60%(例如,至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少97%、至少98%、至少99%或100%)。DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比在约20个邻接核苷酸内可为至少60%。例如,在靶DNA的互补链内的CRISPRRNA识别序列的5’端的14个邻接核苷酸内,DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比为100%,并且在其余邻接核苷酸内低至0%。在这种情况下,DNA靶向序列可被视为14个核苷酸长。又如,在靶DNA的互补链内的CRISPR RNA识别序列的5’端的七个邻接核苷酸内,DNA靶向序列与靶DNA内的CRISPR RNA识别序列之间的互补性百分比为100%,并且在其余邻接核苷酸内低至0%。在这种情况下,DNA靶向序列可被视为7个核苷酸长。
gRNA的蛋白结合区段可包含彼此互补的两段核苷酸。蛋白结合区段的两条互补核苷酸杂交形成双链RNA双链体(dsRNA)。对象gRNA的蛋白结合区段与Cas蛋白相互作用,并且gRNA经由DNA靶向区段指导结合的Cas蛋白到达靶DNA内的特异性核苷酸序列。
向导RNA可包括提供额外所需特征(例如,经修饰或调控的稳定性;亚细胞靶向;用荧光标记物示踪;蛋白或蛋白复合物的结合位点;等等)的修饰或序列。此类修饰的实例包括例如5'帽(例如,7-甲基鸟苷酸帽(m7G));3'聚腺苷酸化尾(即,3'聚(A)尾);核糖开关序列(例如,以实现经调控的稳定性和/或经调控的蛋白和/或蛋白复合物可及性);稳定性控制序列;形成dsRNA双链体(即,发夹)的序列);使RNA靶向亚细胞位置(例如,细胞核、线粒体、叶绿体等)的修饰或序列;提供示踪的修饰或序列(例如,与荧光分子的直接缀合、与有利于荧光检测的部分的缀合、允许荧光检测的序列等);为蛋白(例如,作用于DNA的蛋白,包括转录激活因子、转录阻遏物、DNA甲基转移酶、DNA去甲基化酶、组蛋白乙酰转移酶、组蛋白去乙酰化酶等)提供结合位点的修饰或序列;以及它们的组合。
向导RNA可以任何形式提供。例如,gRNA可以RNA的形式(作为两个分子(单独的crRNA和tracrRNA)或作为一个分子(sgRNA))提供,并任选地以与Cas蛋白的复合物形式提供。gRNA还可以编码RNA的DNA形式提供。编码gRNA的DNA可编码单个RNA分子(sgRNA)或单独的RNA分子(例如,单独的crRNA和tracrRNA)。在后一种情况下,编码gRNA的DNA可作为分别编码crRNA和tracrRNA的单独DNA分子提供。
编码gRNA的DNA可稳定整合在细胞的基因组中,并有效连接至细胞中有活性的启动子。另选地,编码gRNA的DNA可有效连接至表达构建体中的启动子。此类启动子可例如在大鼠、真核、哺乳动物、非人哺乳动物、人、啮齿动物、小鼠或仓鼠多能细胞中有活性。在一些情况下,所述启动子为RNA聚合酶III启动子,诸如人U6启动子、大鼠U6聚合酶III启动子或小鼠U6聚合酶III启动子。其他启动子的实例在本文别处有所描述。
另选地,可通过各种其他方法制备gRNA。例如,可通过使用例如T7 RNA聚合酶的体外转录来制备gRNA(参见例如WO 2014/089290和WO 2014/065596)。向导RNA也可为通过化学合成制备的合成产生的分子。
术语“CRISPR RNA识别序列”包括存在于靶DNA中的核酸序列,只要存在结合的充分条件,gRNA的DNA靶向区段就会与所述靶DNA结合。例如,CRISPR RNA识别序列包括向导RNA被设计成与之具有互补性的序列,其中CRISPR RNA识别序列与DNA靶向序列之间的杂交促进CRISPR复合物的形成。不必要求完全互补性,只要存在引起杂交并促进CRISPR复合物形成的充分互补性即可。CRISPR RNA识别序列还包括下文更详细说明的Cas蛋白的切割位点。CRISPR RNA识别序列可包含任何多核苷酸,所述多核苷酸可位于例如细胞的细胞核或细胞质中,或位于细胞的细胞器如线粒体或叶绿体内。
Cas蛋白或gRNA可以靶DNA内的CRISPR RNA识别序列为靶标(即,与之结合,或与之杂交,或与之互补)。合适的DNA/RNA结合条件包括通常存在于细胞中的生理条件。其他合适的DNA/RNA结合条件(例如,无细胞系统中的条件)是本领域已知的(参见例如MolecularCloning:ALaboratory Manual,3rd Ed.(Sambrook et al.,Harbor Laboratory Press2001)(《分子克隆实验指南》,第3版,Sambrook等人,冷泉港实验室出版社,2001年))。与Cas蛋白或gRNA互补并杂交的靶DNA链可称为“互补链”,与该“互补链”互补(并因此不与Cas蛋白或gRNA互补)的靶DNA链可称为“非互补链”或“模板链”。
Cas蛋白可在gRNA的DNA靶向区段将与之结合的靶DNA中存在的、核酸序列之内或之外的位点处切割核酸。“切割位点”包括Cas蛋白产生单链断裂或双链断裂的核酸位置。例如,CRISPR复合物(包含与CRISPR RNA识别序列杂交并与Cas蛋白复合的gRNA)的形成可导致gRNA的DNA靶向区段将与之结合的靶DNA中存在的、核酸序列中或附近(例如,在相距1、2、3、4、5、6、7、8、9、10、20、50个或更多个碱基对内)的一条或两条链切割。如果切割位点位于gRNA的DNA靶向区段将与之结合的核酸序列之外,则切割位点仍被视为在“CRISPR RNA识别序列”内。切割位点可位于核酸的仅一条链上或两条链上。切割位点可位于核酸两条链上的相同位置处(产生平头末端),或可位于每条链上的不同位点处(产生交错末端)。例如可通过使用两种Cas蛋白来产生交错末端,每种Cas蛋白在每条链上的不同切割位点处产生单链断裂,从而产生双链断裂。例如,第一切口酶可在双链DNA(dsDNA)的第一链上形成单链断裂,并且第二切口酶可在dsDNA的第二链上形成单链断裂,从而形成悬垂序列。在一些情况下,第一链上的切口酶的CRISPR RNA识别序列与第二链上的切口酶的CRISPR RNA识别序列相隔至少2、3、4、5、6、7、8、9、10、15、20、25、30、40、50、75、100、250、500或1,000个碱基对。
Cas9对靶DNA的位点特异性切割可在由以下两者决定的位置处发生:(i)gRNA与靶DNA之间的碱基配对互补性,以及(ii)靶DNA中的短基序,称为前间区序列邻近基序(PAM)。PAM可位于CRISPR RNA识别序列的两侧。任选地,CRISPR RNA识别序列可侧接PAM。例如,Cas9的切割位点可为PAM序列上游或下游的约1至约10或者约2至约5个碱基对(例如,3个碱基对)。在一些情况下(例如,当使用来自酿脓链球菌的Cas9或密切相关的Cas9时),非互补链的PAM序列可为5'-N
CRISPR RNA识别序列的实例包括与gRNA的DNA靶向区段互补的DNA序列,或除PAM序列之外还有这种DNA序列。例如,靶基序可为紧接在Cas蛋白所识别的NGG基序前面的20个核苷酸的DNA序列(参见例如WO 2014/165825)。5’端的鸟嘌呤可有利于RNA聚合酶在细胞中进行转录。CRISPR RNA识别序列的其他实例可包括5’端的两个鸟嘌呤核苷酸(例如,GGN
CRISPR RNA识别序列可为细胞内源或外源的任何核酸序列。CRISPR RNA识别序列可为编码基因产物(例如,蛋白)的序列或非编码序列(例如,调控序列),或者可包括两者。在一个实施例中,靶序列紧密侧接前间区序列邻近基序(PAM)序列。在一个实施例中,目标基因座包含SEQ ID NO:1的核苷酸序列。在一个实施例中,gRNA包含编码成簇规律间隔短回文重复序列(CRISPR)RNA(crRNA)和反式激活CRISPR RNA(tracrRNA)的第三核酸序列。在另一个实施例中,大鼠多能细胞的基因组包含与靶序列互补的靶DNA区。在一些此类方法中,Cas蛋白为Cas9。在一些实施例中,gRNA包含(a)SEQ ID NO:2的核酸序列的嵌合RNA;或(b)SEQ ID NO:3的核酸序列的嵌合RNA。在一些此类方法中,crRNA包含SEQ ID NO:4、SEQ IDNO:5或SEQ ID NO:6所示的序列。在一些此类方法中,tracrRNA包含SEQ ID NO:7或SEQ IDNO:8所示的序列。
还提供了核酸酶试剂的活性变体和片段(即,经改造的核酸酶试剂)。此类活性变体可与天然核酸酶试剂具有至少65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的序列同一性,其中所述活性变体保留在所需识别位点处切断的能力,从而保留切口或双链断裂诱导活性。例如,本文所述的任何核酸酶试剂可由天然核酸内切酶序列修饰而成,并且设计成在不被天然核酸酶试剂识别的识别位点处识别并诱导切口或双链断裂。因此,在一些实施例中,经改造的核酸酶具有特异性,以在与对应天然核酸酶试剂识别位点不同的识别位点处诱导切口或双链断裂。针对切口或双链断裂诱导活性的测定法是已知的,并且一般测量核酸内切酶对包含识别位点的DNA底物的总体活性和特异性。
例如,图3示出了选择盒上的ZFN结合位点和切割位点的位置。这些位点如下所示:Neo-ZFN(1,2):核酸酶结合位点/
可通过本领域已知的任何方式将核酸酶试剂引入细胞中。可将编码核酸酶试剂的多肽直接引入细胞中。另选地,可将编码核酸酶试剂的多核苷酸引入细胞中。当将编码核酸酶试剂的多核苷酸引入细胞中时,核酸酶试剂可在细胞内瞬时地、条件性地或组成性地表达。因此,编码核酸酶试剂的多核苷酸可包含在表达盒中,并有效连接至条件启动子、诱导型启动子、组成型启动子或组织特异性启动子。此类目标启动子在本文别处更详细讨论。另选地,将核酸酶试剂作为编码核酸酶试剂的mRNA引入细胞中。
在具体实施例中,编码核酸酶试剂的多核苷酸稳定整合在细胞的基因组中,并有效连接至细胞中有活性的启动子。在其他实施例中,编码核酸酶试剂的多核苷酸位于包含插入多核苷酸的相同靶向载体中,而在其他情况下,编码核酸酶试剂的多核苷酸位于与包含插入多核苷酸的靶向载体分离的载体或质粒中。
当通过引入编码核酸酶试剂的多核苷酸来向细胞提供核酸酶试剂时,可修饰这种编码核酸酶试剂的多核苷酸,以置换与编码核酸酶试剂的天然存在的多核苷酸序列相比在目标细胞中具有更高使用频率的密码子。例如,可修饰编码核酸酶试剂的多核苷酸,以置换与天然存在的多核苷酸序列相比在给定的目标原核或真核细胞(包括细菌细胞、酵母细胞、人细胞、非人细胞、哺乳动物细胞、啮齿动物细胞、小鼠细胞、大鼠细胞或任何其他目标宿主细胞)中具有更高使用频率的密码子。
本文所提供的各种方法和组合物将核酸酶试剂及其对应识别位点与选择标记结合使用。如本文所讨论,编码选择标记的多核苷酸中识别位点的位置可实现在靶基因座处鉴定整合事件的有效方法。此外,本文提供了各种方法,其中采用具有核酸酶识别位点的交替选择标记,以提高多个目标多核苷酸整合在给定所靶向基因座内的效率和功效。
可在本文所公开的方法和组合物中使用各种选择标记。此类选择标记可例如赋予对抗生素如G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素的抗性。此类选择标记包括新霉素磷酸转移酶(neo
编码选择标记的多核苷酸有效连接至细胞中有活性的启动子。此类表达盒及其各种调控组分在本文别处更详细讨论。
提供了可实现在靶基因座处整合至少一个插入多核苷酸的各种方法和组合物。术语“靶基因座”包含希望整合插入多核苷酸的任何DNA区段或区域。在一个实施例中,靶基因座为基因组座位。靶基因座对于细胞可为天然的,或另选地可包含异源或外源DNA区段。此类异源或外源DNA区段可包括转基因、表达盒、编码选择标记的多核苷酸,或者异源或外源DNA区域(即,基因组DNA的异源或外源区域)。靶基因座可包含靶向整合系统中的任一种,包括例如识别位点、选择标记、此前整合的插入多核苷酸、编码核酸酶试剂的多核苷酸、启动子等。另选地,靶基因座可位于适当宿主细胞中所含的酵母人工染色体(YAC)、细菌人工染色体(BAC)、人类人工染色体或任何其他经改造的基因组区域内。因此,在具体实施例中,所靶向基因座可包含来自原核生物、真核生物、酵母、细菌、非人哺乳动物、非人细胞、啮齿动物、人、大鼠、小鼠、仓鼠、兔、猪、牛、鹿、绵羊、山羊、鸡、猫、狗、白鼬、灵长类动物(例如,狨猴、恒河猴)、家养哺乳动物或农业哺乳动物或任何其他目标生物体,或者它们的组合的天然、异源或外源基因组核酸序列。
靶基因座的非限制性实例包括编码在B细胞中表达的蛋白的基因组座位、在未成熟B细胞中表达多肽的基因组座位、在成熟B细胞中表达多肽的基因组座位、免疫球蛋白(Ig)基因座或T细胞受体基因座,包括例如T细胞受体α基因座。此类基因座可来自禽类(例如,鸡)、非人哺乳动物、啮齿动物、人、大鼠、小鼠、仓鼠、兔、猪、牛、鹿、绵羊、山羊、猫、狗、白鼬、灵长类动物(例如,狨猴、恒河猴)、家养哺乳动物或农业哺乳动物或任何其他目标生物体,或者它们的组合。
在进一步的实施例中,在不存在核酸酶试剂诱导的切口或双链断裂的情况下,所靶向基因座无法使用常规方法靶向,或仅可不正确地或仅以显著较低效率靶向。
如上所述,本文所提供的方法和组合物利用核酸酶试剂以及核酸酶试剂的识别位点在选择盒内的策略性定位,同时利用了同源重组事件。此类方法联合采用了识别位点处的切口或双链断裂以及同源重组,从而将插入多核苷酸靶向整合到靶基因座中。“同源重组”常规用于包括在同源区内的交叉位点处两个DNA分子之间的DNA片段交换。
术语“插入多核苷酸”包含希望在靶基因座处整合的DNA区段。在一个实施例中,插入多核苷酸包含一个或多个目标多核苷酸。在其他实施例中,插入多核苷酸可包含一个或多个表达盒。给定表达盒可包含目标多核苷酸、编码选择标记和/或报告基因的多核苷酸,以及影响表达的各种调控组分。可包含在插入多核苷酸内的目标多核苷酸、选择标记和报告基因(例如,eGFP)的非限制性实例在本文别处详细讨论。
在具体实施例中,插入多核苷酸可包含基因组核酸。在一个实施例中,基因组核酸来源于小鼠、人、啮齿动物、非人、大鼠、仓鼠、兔、猪、牛、鹿、绵羊、山羊、鸡、猫、狗、白鼬、灵长类动物(例如,狨猴、恒河猴)、家养哺乳动物或农业哺乳动物或任何其他目标生物体,或者它们的组合。
在进一步的实施例中,插入多核苷酸包含条件等位基因。在一个实施例中,条件等位基因为如US 2011/0104799中所述的多功能等位基因,该专利全文以引用的方式并入本文。在具体实施例中,条件等位基因包含:(a)相对于靶基因的转录呈有义取向的致动序列,以及呈有义或反义取向的药物选择盒;(b)呈反义取向的目标核苷酸序列(NSI)和倒转条件模块(conditional by inversion module)(COIN,其利用外显子断裂内含子和可倒转的基因诱捕样模块;参见,例如US 2011/0104799,该专利全文以引用的方式并入本文);以及(c)在暴露于第一重组酶后重组以形成条件等位基因的可重组单元,所述条件等位基因(i)缺乏致动序列和DSC,并且(ii)含有呈有义取向的NSI和呈反义取向的COIN。
插入多核苷酸可为约5kb至约200kb、约5kb至约10kb、约10kb至约20kb、约20kb至约30kb、约30kb至约40kb、约40kb至约50kb、约60kb至约70kb、约80kb至约90kb、约90kb至约100kb、约100kb至约110kb、约120kb至约130kb、约130kb至约140kb、约140kb至约150kb、约150kb至约160kb、约160kb至约170kb、约170kb至约180kb、约180kb至约190kb,或约190kb至约200kb。
在具体实施例中,插入多核苷酸包含侧接有位点特异性重组靶序列的核酸。已经认识到,虽然整条插入多核苷酸可侧接这种位点特异性重组靶序列,但该插入多核苷酸内的任何区域或单独目标多核苷酸也可侧接此类位点。术语“重组位点”包括由位点特异性重组酶识别且可充当重组事件的底物的核苷酸序列。术语“位点特异性重组酶”包括可促进重组位点之间的重组的一组酶,其中这两个重组位点在单一核酸分子内或在单独的核酸分子上物理隔开。位点特异性重组酶的实例包括但不限于Cre、Flp和Dre重组酶。位点特异性重组酶可通过任何方式引入细胞中,包括将重组酶多肽引入细胞中或将编码位点特异性重组酶的多核苷酸引入宿主细胞中。编码位点特异性重组酶的多核苷酸可位于插入多核苷酸内或单独的多核苷酸内。位点特异性重组酶可有效连接至细胞中有活性的启动子,包括例如诱导型启动子、对于细胞为内源的启动子、对于细胞为异源的启动子、细胞特异性启动子、组织特异性启动子或发育阶段特异性启动子。可位于插入多核苷酸或在插入多核苷酸中的任何目标多核苷酸两侧的位点特异性重组靶序列可包括但不限于loxP、lox511、lox2272、lox66、lox71、loxM2、lox5171、FRT、FRT11、FRT71、attp、att、FRT、rox以及它们的组合。
在其他实施例中,位点特异性重组位点位于插入多核苷酸内所含的编码选择标记和/或报告基因的多核苷酸两侧。在此类情况下,在所靶向基因座处整合插入多核苷酸之后,可除去位点特异性重组位点之间的序列。
在一个实施例中,插入多核苷酸包含编码选择标记的多核苷酸。此类选择标记包括但不限于新霉素磷酸转移酶(neo
插入多核苷酸还可包含有效连接至启动子的报告基因,其中所述报告基因编码报告蛋白,所述报告蛋白选自LacZ、mPlum、mCherry、tdTomato、mStrawberry、J-Red、DsRed、mOrange、mKO、mCitrine、Venus、YPet、增强型黄色荧光蛋白(eYFP)、Emerald、增强型绿色荧光蛋白(EGFP)、CyPet、青色荧光蛋白(CFP)、Cerulean、T-Sapphire、荧光素酶、碱性磷酸酶以及它们的组合。此类报告基因可有效连接至细胞中有活性的启动子。此类启动子可为诱导型启动子、对于报告基因或细胞为内源的启动子、对于报告基因或细胞为异源的启动子、细胞特异性启动子、组织特异性启动子或发育阶段特异性启动子。
靶向载体用于将插入多核苷酸引入所靶向基因座中。靶向载体包含插入多核苷酸,并且还包含上游同源臂和下游同源臂,这些同源臂位于插入多核苷酸两侧。位于插入多核苷酸两侧的同源臂对应于所靶向基因座内的区域。为了便于提及,所靶向基因座内的对应区域在本文中称为“靶位点”。因此,在一个实例中,靶向载体可包含第一插入多核苷酸,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近编码选择标记的多核苷酸内的第一识别位点处的第一靶位点和第二靶位点相对应。因此,靶向载体由此有助于通过在例如细胞的基因组内的同源臂与对应靶位点之间发生的同源重组事件,将插入多核苷酸整合到所靶向基因座中。
靶向载体的同源臂可具有足以促进与对应靶位点的同源重组事件的任何长度,包括例如50-100个碱基、100-1000个碱基、或至少5-10、5-15、5-20、5-25、5-30、5-35、5-40、5-45、5-50、5-55、5-60、5-65、5-70、5-75、5-80、5-85、5-90、5-95、5-100、100-200或200-300千碱基长或更长。如下文进一步详细概述,大靶向载体可采用更大长度的靶向臂。
与靶向载体的上游同源臂和下游同源臂对应的、所靶向基因座内的靶位点位于“足够接近识别位点”处,该识别位点位于编码选择标记的多核苷酸中。靶向载体的上游同源臂和下游同源臂“位于足够接近”识别位点处,其中该距离诸如会促进在识别位点处切口或双链断裂后在靶位点与同源臂之间发生同源重组事件。因此,在具体实施例中,与靶向载体的上游同源臂和/或下游同源臂对应的靶位点位于给定识别位点的至少1个核苷酸内,在给定识别位点的至少10个核苷酸至约14kb内,或在约10个核苷酸至约100个核苷酸内、在给定识别位点的约100个核苷酸至约500个核苷酸内、在约500个核苷酸至约1000个核苷酸内、在约1kb至约5kb内、在约5kb至约10kb内,或在约10kb至约14kb内。在具体实施例中,识别位点紧邻靶位点中的至少一者或两者。
对应于靶向载体的同源臂的靶位点与编码选择标记的多核苷酸内的识别位点的空间关系可有所变化。例如,靶位点可位于识别位点的5’,这两个靶位点都可位于识别位点的3’,或者靶位点可位于识别位点两侧。
当两个区域彼此共有足够水平的序列同一性时,同源臂和靶位点彼此“对应”,从而充当同源重组反应的底物。所谓“同源性”意指DNA序列与对应序列相同或共有序列同一性。给定靶位点与存在于靶向载体上的对应同源臂之间的序列同一性可为允许同源重组发生的任何程度的序列同一性。例如,靶向载体的同源臂(或其片段)与靶位点(或其片段)共有的序列同一性的量可为至少50%、55%、60%、65%、70%、75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性,以使得所述序列经历同源重组。此外,同源臂与对应靶位点之间的对应同源区可具有足以促进在切割的识别位点处同源重组的任何长度。例如,给定同源臂和/或对应靶位点可包含对应同源区,所述对应同源区为至少约50-100个碱基、100-1000个碱基、或5-10、5-15、5-20、5-25、5-30、5-35、5-40、5-45、5-50、5-55、5-60、5-65、5-70、5-75、5-80、5-85、5-90、5-95、5-100、100-200、或200-300千碱基长或更长(诸如,如在本文别处描述的LTVEC载体中所述),以使得同源臂与在细胞基因组内的对应靶位点具有足以经历同源重组的同源性。
为了便于提及,同源臂包括上游同源臂和下游同源臂。该术语涉及靶向载体内的同源臂与插入多核苷酸的相对位置。
靶向载体的同源臂因此被设计成与具有所靶向基因座的靶位点相对应。因此,同源臂可与对细胞为天然的基因座相对应,或者另选地,它们可与整合到细胞基因组中的异源或外源DNA区段的区域相对应,所述区域包括但不限于转基因、表达盒或者异源或外源DNA区域。另选地,靶向载体的同源臂可与酵母人工染色体(YAC)、细菌人工染色体(BAC)、人类人工染色体的区域或在适当宿主细胞中包含的任何其他经改造的区域相对应。更进一步,靶向载体的同源臂可与BAC文库、粘粒文库或P1噬菌体文库的区域相对应,或者可来源于BAC文库、粘粒文库或P1噬菌体文库的区域。因此,在具体实施例中,靶向载体的同源臂与对于以下生物为天然、异源或外源的基因座相对应:原核生物、酵母、禽类(例如,鸡)、非人哺乳动物、啮齿动物、人、大鼠、小鼠、仓鼠、兔、猪、牛、鹿、绵羊、山羊、猫、狗、白鼬、灵长类动物(例如,狨猴、恒河猴)、家养哺乳动物或农业哺乳动物,或任何其他目标生物体。在进一步的实施例中,同源臂与细胞中的基因座相对应,在不存在核酸酶试剂诱导的切口或双链断裂的情况下,该基因座无法使用常规方法靶向,或仅可不正确地或仅以显著较低效率靶向。在一个实施例中,同源臂来源于合成DNA。
在另一些实施例中,上游同源臂和下游同源臂对应于与所靶向基因组相同的基因组。在一个实施例中,同源臂来自相关基因组,例如,所靶向基因组为第一品系的小鼠基因组,且靶向臂来自第二品系的小鼠基因组,其中第一品系与第二品系不同。在其他实施例中,同源臂来自相同动物的基因组或来自相同品系的基因组,例如所靶向基因组为第一品系的小鼠基因组,且靶向臂来自相同小鼠的小鼠基因组或来自相同品系的小鼠基因组。
靶向载体(诸如大靶向载体)还可包含如本文别处所讨论的选择盒或报告基因。选择盒可包含编码选择标记的核酸序列,其中所述核酸序列有效连接至启动子。所述启动子可在目标原核细胞中有活性和/或在目标真核细胞中有活性。此类启动子可为诱导型启动子、对于报告基因或细胞为内源的启动子、对于报告基因或细胞为异源的启动子、细胞特异性启动子、组织特异性启动子或发育阶段特异性启动子。在一个实施例中,选择标记选自新霉素磷酸转移酶(neo
在一个实施例中,靶向载体(诸如大靶向载体)包含有效连接至启动子的报告基因,其中所述报告基因编码报告蛋白,所述报告蛋白选自LacZ、mPlum、mCherry、tdTomato、mStrawberry、J-Red、DsRed、mOrange、mKO、mCitrine、Venus、YPet、增强型黄色荧光蛋白(eYFP)、Emerald、增强型绿色荧光蛋白(EGFP)、CyPet、青色荧光蛋白(CFP)、Cerulean、T-Sapphire、荧光素酶、碱性磷酸酶以及它们的组合。此类报告基因可有效连接至细胞中有活性的启动子。此类启动子可为诱导型启动子、对于报告基因或细胞为内源的启动子、对于报告基因或细胞为异源的启动子、细胞特异性启动子、组织特异性启动子或发育阶段特异性启动子。
在一个实施例中,与单独使用靶向载体相比,联合使用靶向载体(包括例如大靶向载体)与核酸酶试剂会提高靶向效率。在一个实施例中,与单独使用靶向载体时相比,当靶向载体与核酸酶试剂结合使用时,靶向载体的靶向效率提高至少两倍、至少三倍、至少4倍或至少10倍。
术语“大靶向载体”或“LTVEC”包括这样的大靶向载体:其包含对应于且来源于一些核酸序列的同源臂,这些核酸序列比通常由意欲在细胞中进行同源重组的其他方法使用的那些核酸序列大;并且/或者包含插入多核苷酸,所述插入多核苷酸包含的核酸序列比通常由意欲在细胞中进行同源重组的其他方法使用的那些核酸序列大。在具体实施例中,LTVEC的同源臂和/或插入多核苷酸包含真核细胞的基因组序列。LTVEC的尺寸太大,无法通过常规测定法例如Southern印迹和长片段(例如,1kb-5kb)PCR来筛选靶向事件。LTVEC的实例包括但不限于来源于细菌人工染色体(BAC)、人类人工染色体或酵母人工染色体(YAC)的载体。LTVEC及其制备方法的非限制性实例描述于例如美国专利No.6,586,251、6,596,541、7,105,348和WO 2002/036789(PCT/US01/45375)中,这些专利中的每一者均以引用的方式并入本文。
LTVEC可具有任何长度,包括但不限于约20kb至约300kb、约20kb至约30kb、约30kb至约40kb、约40kb至约50kb、约50kb至约75kb、约75kb至约100kb、约100kb至125kb、约125kb至约150kb、约150kb至约175kb、约175kb至约200kb、约200kb至约225kb、约225kb至约250kb、约250kb至约275kb或约275kb至约300kb。
在一个实施例中,LTVEC包含在约5kb至约200kb、约5kb至约10kb、约10kb至约20kb、约20kb至约30kb、约30kb至约40kb、约40kb至约50kb、约60kb至约70kb、约80kb至约90kb、约90kb至约100kb、约100kb至约110kb、约120kb至约130kb、约130kb至约140kb、约140kb至约150kb、约150kb至约160kb、约160kb至约170kb、约170kb至约180kb、约180kb至约190kb或约190kb至约200kb范围内的插入多核苷酸。
在一个实施例中,LTVEC的同源臂来源于BAC文库、粘粒文库或P1噬菌体文库。在其他实施例中,同源臂来源于细胞的所靶向基因座(即,基因组座位),并且在一些情况下,LTVEC被设计用来靶向的靶基因座无法使用常规方法靶向。在另一些实施例中,同源臂来源于合成DNA。在一个实施例中,LTVEC中的上游同源臂和下游同源臂的总和为至少10kb。在一个实施例中,上游同源臂在约1kb至约100kb的范围内。在其他实施例中,上游同源臂在约5kb至约100kb的范围内。在一个实施例中,下游同源臂在约1kb至约100kb的范围内。在一个实施例中,下游同源臂在约5kb至约100kb的范围内。在其他实施例中,上游同源臂和下游同源臂的总和为约1kb至约5kb、约5kb至约10kb、约10kb至约20kb、约20kb至约30kb、约30kb至约40kb、约40kb至约50kb、约50kb至约60kb、约60kb至约70kb、约70kb至约80kb、约80kb至约90kb、约90kb至约100kb、约100kb至约110kb、约110kb至约120kb、约120kb至约130kb、约130kb至约140kb、约140kb至约150kb、约150kb至约160kb、约160kb至约170kb、约170kb至约180kb、约180kb至约190kb或约190kb至约200kb。
在其他实施例中,LTVEC的5’和3’同源臂的总和为约10kb至约30kb、约20kb至约40kb、约40kb至约60kb、约60kb至约80kb、约80kb至约100kb、约100kb至约120kb或约120kb至150kb。在其他情况下,5’和3’同源臂的总和为约16Kb至约150Kb。
在进一步的实施例中,LTVEC和插入多核苷酸被设计成允许在靶基因座处缺失约5kb至约10kb、约10kb至约20kb、约20kb至约40kb、约40kb至约60kb、约60kb至约80kb、约80kb至约100kb、约100kb至约150kb,或约150kb至约200kb、约200kb至约300kb、约300kb至约400kb、约400kb至约500kb、约500kb至约1Mb、约1Mb至约1.5Mb、约1.5Mb至约2Mb、约2Mb至约2.5Mb,或约2.5Mb至约3Mb。
在其他情况下,LTVEC和插入多核苷酸被设计成允许在靶基因座中插入约5kb至约10kb、约10kb至约20kb、约20kb至约40kb、约40kb至约60kb、约60kb至约80kb、约80kb至约100kb、约100kb至约150kb、约150kb至约200kb、约200kb至约250kb、约250kb至约300kb、约300kb至约350kb或约350kb至约400kb范围内的外源核酸序列。在一个实施例中,插入多核苷酸为约130kb或约155kb。
在一个实施例中,LTVEC包含选择盒或报告基因,如本文别处所讨论。
提供了用于修饰细胞中的靶基因座的方法。所述方法包括(a)提供包含第一多核苷酸的细胞,所述第一多核苷酸编码第一选择标记并有效连接至细胞中有活性的第一启动子,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点;(b)向细胞中引入:(i)在第一识别位点处诱导切口或双链断裂的第一核酸酶试剂,和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应;以及(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞。在具体实施例中,包含第一选择标记的第一多核苷酸侧接第一靶位点和第二靶位点,第一靶位点与第一靶向载体中的第一同源臂相对应,第二靶位点与第一靶向载体中的第二同源臂相对应。
可使用各种方法鉴定具有在靶基因座处整合的插入多核苷酸的细胞。在一个实施例中,第一识别位点处的切口或双链断裂破坏第一选择标记的活性。因此,在一个实施例中,通过在一些条件下培养细胞来鉴定此类细胞,所述条件能鉴定没有由多核苷酸编码的选择标记活性的细胞,其中所述多核苷酸具有被核酸酶试剂切断的识别位点。采用此类选择标记并测定其活性的方法是已知的。用于鉴定在靶基因座处具有插入多核苷酸的细胞的其他方法可包括鉴定在所需靶位点处整合有插入多核苷酸的至少一个细胞。此类方法可包括鉴定在其基因组中包含在第一靶位点和第二靶位点处整合的第一插入多核苷酸的至少一个细胞。
还可采用其他方法鉴定具有在靶基因座处整合的插入多核苷酸的细胞。插入多核苷酸在靶基因座处的插入产生“等位基因修饰”。术语“等位基因修饰”或“MOA”包括基因组中的一个或多个基因座位或者染色体座位的一个等位基因的精确DNA序列的修饰。“等位基因修饰(MOA)”的实例包括但不限于缺失、置换或插入少至单个核苷酸,或者跨一个或多个目标基因座位或者目标染色体座位缺失数千碱基,以及介于这两个极端之间的任何和所有可能的修饰。
在各种实施例中,为了方便靶向修饰的鉴定,采用了高通量定量测定法,即,等位基因修饰(MOA)测定法。本文所述的MOA测定法允许在遗传修饰之后大规模筛选亲本染色体中的一个或多个经修饰的等位基因。MOA测定法可经由各种分析技术进行,包括但不限于定量PCR,例如实时PCR(qPCR)。例如,实时PCR包括识别靶基因座的第一引物组和识别非靶向参考基因座的第二引物组。此外,引物组包含识别扩增序列的荧光探针。定量测定法还可经由多种分析技术进行,包括但不限于荧光介导原位杂交(FISH)、比较基因组杂交、等温DNA扩增、定量固定探针杂交、Invader
在各种实施例中,在选择标记内的识别位点中存在切口或双链断裂提高了靶向载体(诸如LTVEC)与所靶向基因座之间重组的效率和/或频率。在一个实施例中,所述重组为同源重组。在各种实施例中,在存在切口或双链断裂的情况下,靶向载体(诸如LTVEC)在靶基因座处的靶向效率是不存在切口或双链断裂(使用例如相同靶向载体和相同同源臂以及目标基因座处的对应靶位点,但不含会造成切口或双链断裂的所添加核酸酶试剂)时的至少约2倍、至少约3倍、至少约4倍、至少约10倍。
本文所提供的各种方法和组合物允许多个目标多核苷酸在给定靶基因座内靶向整合。所述方法采用本文所述的靶向整合系统,其采用核酸酶试剂识别位点在编码选择标记的多核苷酸内的策略性定位。在具体实施例中,选择标记和识别位点在每个插入多核苷酸内交替。这样,顺序插入多核苷酸在给定靶基因座内的拼接以增强的效率和功效进行。
在一个实施例中,用于修饰细胞中的靶基因座的方法包括:(a)提供包含基因座的细胞,所述基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点;(b)向细胞中引入第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂;并且向细胞中引入包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应;并且所述第一插入多核苷酸还包含(1)第一目标多核苷酸;和(2)编码第二选择标记并有效连接至细胞中有活性的第二启动子的第二多核苷酸,其中所述第二多核苷酸包含第二核酸酶试剂的第二识别位点;以及(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞。
在进一步的实施例中,可在靶基因座处整合另外的目标多核苷酸。用于修饰细胞中的靶基因座的此类方法包括:(a)提供包含基因座的细胞,所述基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点;(b)向细胞中引入第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂;并且向细胞中引入包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应;并且所述第一插入多核苷酸还包含(1)第一目标多核苷酸;和(2)编码第二选择标记并有效连接至细胞中有活性的第二启动子的第二多核苷酸,其中所述第二多核苷酸包含第二核酸酶试剂的第二识别位点;(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞;(d)向其基因组中包含在靶基因座处整合的第一插入多核苷酸的细胞中引入:(i)第二核酸酶试剂,其中所述第二核酸酶试剂在第二识别位点处诱导切口或双链断裂;和(ii)包含第二插入多核苷酸的第二靶向载体,所述第二插入多核苷酸侧接第三同源臂和第四同源臂;以及(b)鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞。在具体实施例中,第二识别标记处的切口或双链断裂破坏第二选择标记的活性。在进一步的实施例中,鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞包括在能鉴定没有第二选择标记活性的细胞的条件下培养细胞。在更进一步的实施例中,包含第二选择标记的第二多核苷酸侧接第三靶位点和第四靶位点,第三靶位点与第二靶向载体中的第三同源臂相对应,第四靶位点与第二靶向载体中的第四同源臂相对应。在更进一步的实施例中,鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞包括鉴定包含在第三靶位点和第四靶位点处整合的第二插入多核苷酸的至少一个细胞。
用于修饰细胞中的靶基因座的其他方法包括:(a)提供包含靶基因座的细胞,所述靶基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点;(b)向细胞中引入(i)第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂;和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应,并且所述第一插入多核苷酸还包含(1)第一目标多核苷酸;和(2)编码第二选择标记并有效连接至细胞中有活性的第二启动子的第二多核苷酸,其中所述第二多核苷酸包含第二核酸酶试剂的第二识别位点,并且包含第二选择标记的第二多核苷酸侧接第三靶位点和第四靶位点,第三靶位点与第二靶向载体中的第三同源臂相对应,第四靶位点与第二靶向载体中的第四同源臂相对应;(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞;(d)向包含在靶基因座处整合的第一插入多核苷酸的细胞中引入:(i)第二核酸酶试剂,其中所述第二核酸酶试剂在第二识别位点处诱导切口或双链断裂;和(ii)包含第二插入多核苷酸的第二靶向载体,所述第二插入多核苷酸侧接第三同源臂和第四同源臂,其中所述第二插入多核苷酸包含(1)第二目标多核苷酸;和(2)编码第三选择标记并有效连接至细胞中有活性的第三启动子的第三多核苷酸,其中所述第三多核苷酸包含第三核酸酶试剂的第三识别位点;以及(b)鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞。在具体实施例中,第二识别标记处的切口或双链断裂破坏第二选择标记的活性。在进一步的实施例中,鉴定在其基因组中包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞包括在能鉴定没有第二选择标记活性的细胞的条件下培养细胞。在进一步的实施例中,鉴定在其基因组中包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞包括鉴定在其基因组中包含在第三靶位点和第四靶位点处整合的第二插入多核苷酸的至少一个细胞。
可顺序地重复上述各种方法,以允许任何数量的插入多核苷酸靶向整合到给定的所靶向基因座中。因此,所述各种方法可实现在靶基因座中插入至少1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20个或更多个插入多核苷酸。在特定实施例中,此类顺序拼接方法允许将来自哺乳动物细胞(即,人、非人、啮齿动物、小鼠、猴、大鼠、仓鼠、家养哺乳动物或农业动物)的大基因组区域重建到所靶向基因座(即,基因组座位)中。在此类情况下,包含编码区和非编码区两者的基因组区域的转移和重建允许通过至少部分地保留在天然基因组区域内发现的编码区、非编码区和拷贝数变异来保持给定区的复杂性。因此,所述各种方法提供了例如在任何哺乳动物细胞或目标动物内生成“异源”或“外源”基因组区域的方法。在一个非限制性实例中,生成了在非人动物内的“人源化”基因组区域。
当在给定靶基因座内进行多个插入多核苷酸的整合时,编码选择标记并包含核酸酶试剂识别位点的多核苷酸可在多轮整合之间交替。例如,在具体方法中,第一核酸酶试剂不同于第二核酸酶试剂,并且/或者第一选择标记不同于第二选择标记。在其他实例中,当将三个插入多核苷酸插入到所靶向基因座中时,第一选择标记和第三选择标记可彼此相同,并且在具体实施例中还包含相同识别位点,而第二选择标记可不同于第一选择标记和第三选择标记并包含不同识别位点。按这种方式对选择标记和识别位点进行选择,最大程度减少了必须生成的核酸酶试剂数量,从而提高了整合事件的效率和功效。
提供了利用如本文别处所述的CRISPR/Cas系统来修饰细胞中的一个或多个目标靶基因座的方法和组合物。对于CRISPR/Cas系统而言,术语“靶位点”或“靶序列”可互换使用,并且包括存在于靶DNA中的核酸序列,只要存在充分的结合条件,向导RNA(gRNA)的DNA靶向区段就会与所述靶DNA结合。例如,Cas核酸酶或gRNA以靶DNA内的靶位点(或靶序列)为靶标(或与之结合,或与之杂交,或与之互补)。合适的DNA/RNA结合条件包括通常存在于细胞中的生理条件。其他合适的DNA/RNA结合条件(例如,无细胞系统中的条件)是本领域已知的(参见例如Molecular Cloning:A Laboratory Manual,3rd Ed.(Sambrook et al.,Harbor Laboratory Press 2001)(《分子克隆实验指南》,第3版,Sambrook等人,冷泉港实验室出版社,2001年))。与Cas蛋白或gRNA互补并杂交的靶DNA链称为“互补链”,与该“互补链”互补(并因此不与Cas蛋白或gRNA互补)的靶DNA链称为“非互补链”或“模板链”。
Cas蛋白可在靶序列之内或靶序列之外的位点处切割核酸。“切割位点”包括Cas蛋白在其中产生单链断裂或双链断裂的核酸位置。还可通过使用在每条链上的切割位点处产生单链断裂的两种Cas9蛋白来产生粘性末端。Cas9对靶DNA的位点特异性切割可在由以下两者决定的位置处发生:(i)向导RNA与靶DNA之间的碱基配对互补性,以及(ii)靶DNA中的短基序,称为前间区序列邻近基序(PAM)。例如,Cas9的切割位点可为PAM序列上游的约1至约10或者约2至约5个碱基对(例如,3个碱基对)。在一些实施例中(例如,当使用来自酿脓链球菌的Cas9或密切相关的Cas9时),非互补链的PAM序列可为5'-XGG-3',其中X为任何DNA核苷酸,并且X紧邻靶DNA的非互补链的靶序列的3'。因此,互补链的PAM序列将为5'-CCY-3',其中Y为任何DNA核苷酸,并且Y紧邻靶DNA的互补链的靶序列的5'。在一些此类实施例中,X和Y可互补,并且X-Y碱基对可为任何碱基对(例如,X=C且Y=G;X=G且Y=C;X=A且Y=T,X=T且Y=A)。
因此,在一些实施例中,用于修饰细胞中的目标靶基因座的方法包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的核酸;(b)向细胞中引入(i)编码Cas蛋白和第一向导RNA(gRNA)的一个或多个表达构建体,每个所述表达构建体有效连接至细胞中有活性的启动子,其中所述Cas蛋白在第一核酸中的第一gRNA靶位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性,和(ii)包含第一插入核酸的第一靶向载体,所述第一插入核酸包含编码第二选择标记并有效连接至第二启动子的第二核酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;以及(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和第二选择标记不同。在一个实施例中,第一gRNA不与第一插入核酸杂交。在一个实施例中,目标靶基因座位于细胞的基因组中。在另一个实施例中,目标靶基因座位于细胞中的载体中。在一个实施例中,鉴定步骤(c)包括在允许鉴定具有第二选择标记的活性但没有第一选择标记的活性的修饰细胞的条件下培养细胞。
在一个实施例中,所述方法还包括(d)向在第一靶基因座处包含第一插入核酸的修饰细胞中引入(i)编码Cas蛋白和第二gRNA的一个或多个核酸,每个所述核酸有效连接至修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第二核酸的第一插入核酸中的第二gRNA靶位点处诱导切口或双链断裂,从而破坏第二选择标记的表达或活性,和(ii)包含第二插入核酸的第二靶向载体,所述第二插入核酸包含编码第三选择标记并有效连接至第三启动子的第三核酸,其中所述第二插入核酸侧接第三同源臂和第四同源臂,所述第三同源臂和第四同源臂与位于第二靶基因座中的第三靶位点和第四靶位点相对应;以及(e)鉴定在第二靶基因座处包含第二插入核酸的第二修饰细胞,其中所述第二修饰细胞具有第三选择标记的活性,但不具有第二选择标记的活性,其中所述第二选择标记和第三选择标记不同。在一个实施例中,第一靶基因座和第二靶基因座位于彼此紧邻的位置。在另一个实施例中,第一靶基因座或第二靶基因座位于距第一gRNA靶位点或第二gRNA靶位点约10个核苷酸至约14kb处。在一个实施例中,第二gRNA不与第二插入核酸杂交。在一个实施例中,鉴定步骤(e)包括在允许鉴定具有第三选择标记的活性但没有第二选择标记的活性的第二修饰细胞的条件下培养修饰细胞。
在一个实施例中,所述方法还包括(f)向在第二靶基因座处包含第二插入核酸的第二修饰细胞中引入:(i)编码Cas蛋白和第三gRNA的一个或多个表达构建体,每个所述表达构建体有效连接至第二修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第三核酸的第二插入核酸中的第三gRNA靶位点处诱导切口或双链断裂,从而破坏第三选择标记的表达或活性,和(ii)包含第三插入核酸的第三靶向载体,所述第三插入核酸包含编码第四选择标记并有效连接至第四启动子的第四核酸,其中所述第三插入核酸侧接第五同源臂和第六同源臂,所述第五同源臂和第六同源臂与位于第三靶基因座中的第五靶位点和第六靶位点相对应;以及(g)鉴定在第三靶基因座处包含第三插入核酸的第三修饰细胞,其中所述第三修饰细胞具有第四选择标记的活性,但不具有第三选择标记的活性,其中所述第三选择标记和第四选择标记不同。在一个实施例中,第二靶基因座和第三靶基因座位于彼此紧邻的位置。在另一个实施例中,第二靶基因座或第三靶基因座位于距第一gRNA靶位点或第二gRNA靶位点约10个核苷酸至约14kb处。
在一个实施例中,第一标记、第二标记、第三标记或第四标记赋予对抗生素的抗性。在一个实施例中,抗生素包括G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素。在一个实施例中,第一选择标记、第二选择标记、第三选择标记或第四选择标记包括次黄嘌呤-鸟嘌呤磷酸核糖转移酶(HGPRT)或单纯性疱疹病毒的胸苷激酶(HSV-TK)。在一个实施例中,第一gRNA、第二gRNA或第三gRNA包括(i)与第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点杂交的核苷酸序列,和(ii)反式激活CRISPR RNA(tracrRNA)。在一个实施例中,第一靶基因座、第二靶基因座或第三靶基因座位于紧邻第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点的位置,使得gRNA靶位点处的切口或双链断裂促进靶基因座处靶向载体的同源重组。在一个实施例中,Cas蛋白为Cas9。在一个实施例中,第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点紧密侧接前间区序列邻近基序(PAM)序列。
在具体实施例中,gRNA被设计成靶向第一抗生素选择标记(例如,Hyg
在一个实施例中,所述细胞为原核细胞。在另一个实施例中,所述细胞为真核细胞。在一个实施例中,所述真核细胞为哺乳动物细胞或非人哺乳动物细胞。在一个实施例中,所述哺乳动物细胞为成纤维细胞。在一个实施例中,所述哺乳动物细胞为人成纤维细胞。在一个实施例中,所述哺乳动物细胞为人成体干细胞。在一个实施例中,所述哺乳动物细胞为发育受限的祖细胞。在一个实施例中,所述哺乳动物细胞为发育受限的人祖细胞。
在一个实施例中,所述哺乳动物细胞为非人哺乳动物细胞。在一个实施例中,所述哺乳动物细胞来自啮齿动物。在一个实施例中,所述啮齿动物为大鼠、小鼠或仓鼠。在一个实施例中,所述真核细胞为多能细胞。在一个实施例中,所述多能细胞为造血干细胞或神经元干细胞。在一个实施例中,所述多能细胞为人诱导性多能干(iPS)细胞。在一个实施例中,所述多能细胞为非人ES细胞、人ES细胞、啮齿动物胚胎干(ES)细胞、小鼠胚胎干(ES)细胞或大鼠胚胎干(ES)细胞。
在一个实施例中,第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点位于编码第一选择标记、第二选择标记或第三选择标记的第一核酸、第二核酸或第三核酸中的内含子、外显子、启动子或启动子调控区中。在一个实施例中,第一靶向载体、第二靶向载体或第三靶向载体为至少约10kb。在一个实施例中,第一插入核酸、第二插入核酸或第三插入核酸在约5kb至约300kb的范围内。
在一个实施例中,第一插入核酸、第二插入核酸或第三插入核酸包含人T细胞受体α基因座的基因组区域。在一个实施例中,所述基因组区域包含人T细胞受体α基因座的至少一个可变区基因区段和/或连接区基因区段。
在一个实施例中,第一选择标记和第三选择标记相同。在一个实施例中,第一选择标记和第三选择标记相同,并且第二选择标记和第四选择标记相同。在一个实施例中,第一gRNA和第三gRNA相同。
在一些实施例中,用于修饰细胞中的靶基因座的方法包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的核酸;(b)向细胞中引入(i)编码Cas蛋白和第一gRNA的一个或多个表达构建体,每个所述表达构建体有效连接至细胞中有活性的启动子,其中所述Cas蛋白在第一核酸中的第一gRNA靶位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性,和(ii)包含第一插入核酸的第一靶向载体,所述第一插入核酸包含编码第二选择标记并有效连接至第二启动子的第二核酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和第二选择标记不同;(d)向在第一靶基因座处包含第一插入核酸的修饰细胞中引入:(i)编码Cas蛋白和第二gRNA的一个或多个核酸,每个所述核酸有效连接至修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第二核酸的第一插入核酸中的第二gRNA靶位点处诱导切口或双链断裂,从而破坏第二选择标记的表达或活性,和(ii)包含第二插入核酸的第二靶向载体,所述第二插入核酸包含编码第三选择标记并有效连接至第三启动子的第三核酸,其中所述第二插入核酸侧接第三同源臂和第四同源臂,所述第三同源臂和所述第四同源臂与位于第二靶基因座中的第三靶位点和第四靶位点相对应;以及(e)鉴定在第二靶基因座处包含第二插入核酸的第二修饰细胞,其中所述第二修饰细胞具有第三选择标记的活性,但不具有第二选择标记的活性,其中所述第一选择标记和第三选择标记相同,而所述第二选择标记和第三选择标记不同。
在其他实施例中,用于修饰细胞中的目标靶基因座的方法包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的核酸;(b)向细胞中引入(i)编码Cas蛋白和第一gRNA的一个或多个表达构建体,每个所述表达构建体有效连接至细胞中有活性的启动子,其中所述Cas蛋白在第一核酸中的第一gRNA靶位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性,和(ii)包含第一插入核酸的第一靶向载体,所述第一插入核酸包含编码第二选择标记并有效连接至第二启动子的第二核酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和第二选择标记不同;(d)向在第一靶基因座处包含第一插入核酸的修饰细胞中引入:(i)编码Cas蛋白和第二gRNA的一个或多个核酸,每个所述核酸有效连接至修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第二核酸的第一插入核酸中的第二gRNA靶位点处诱导切口或双链断裂,从而破坏第二选择标记的表达或活性,和(ii)包含第二插入核酸的第二靶向载体,所述第二插入核酸包含编码第三选择标记并有效连接至第三启动子的第三核酸,其中所述第二插入核酸侧接第三同源臂和第四同源臂,所述第三同源臂和所述第四同源臂与位于第二靶基因座中的第三靶位点和第四靶位点相对应;(e)鉴定在第二靶基因座处包含第二插入核酸的第二修饰细胞,其中所述第二修饰细胞具有第三选择标记的活性,但不具有第二选择标记的活性,其中所述第二选择标记和第三选择标记不同;(f)向在第二靶基因座处包含第二插入核酸的第二修饰细胞中引入:(i)编码Cas蛋白和第三gRNA的一个或多个表达构建体,每个所述表达构建体有效连接至第二修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第三核酸的第二插入核酸中的第三gRNA靶位点处诱导切口或双链断裂,从而破坏第三选择标记的表达或活性,和(ii)包含第三插入核酸的第三靶向载体,所述第三插入核酸包含编码第四选择标记并有效连接至第四启动子的第四核酸,其中所述第三插入核酸侧接第五同源臂和第六同源臂,所述第五同源臂和所述第六同源臂与位于第三靶基因座中的第五靶位点和第六靶位点相对应;以及(g)鉴定在第三靶基因座处包含第三插入核酸的第三修饰细胞,其中所述第三修饰细胞具有第四选择标记的活性,但不具有第三选择标记的活性,其中所述第三选择标记和所述第四选择标记不同。在一些实施例中,第一选择标记和第三选择标记相同,并且第二选择标记和第四选择标记相同。在一个实施例中,第一选择标记和第三选择标记相同,第二选择标记和第四选择标记相同,并且第一gRNA和第三gRNA相同。
任何目标多核苷酸都可包含在各种插入多核苷酸中并由此整合在靶基因座处。本文所公开的方法提供整合到所靶向基因座中的至少1、2、3、4、5、6个或更多个目标多核苷酸。
当在靶基因座处整合时,插入多核苷酸内的目标多核苷酸可将一个或多个遗传修饰引入细胞中。所述遗传修饰可包括缺失内源核酸序列和/或将外源或异源或直系同源多核苷酸添加到靶基因座中。在一个实施例中,所述遗传修饰包括在靶基因座处用目标外源多核苷酸替换内源核酸序列。因此,本文所提供的方法允许生成遗传修饰,所述遗传修饰包括敲除、缺失、插入、替换(“敲入”)、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换或它们的组合。此类修饰可在第一、第二、第三、第四、第五、第六、第七或任何后续的插入多核苷酸整合到靶基因座中后发生。
在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸可包括对于其被引入的细胞为天然的或同源的序列;目标多核苷酸对于其被引入的细胞可为异源的;目标多核苷酸对于其被引入的细胞可为外源的;目标多核苷酸对于其被引入的细胞可为直系同源的;或者目标多核苷酸可来自与其被引入的细胞不同的物种。指涉序列的术语“同源”包括所述细胞天然的序列。指涉序列的术语“异源”包括来源于外来物种的序列,或者,如果序列来源于同一物种,则通过有意的人为干预在组成和/或基因座方面从其天然形式进行了实质性修饰。指涉序列的术语“外源”包括源于外来物种的序列。术语“直系同源”包括来自一种物种的在功能上与另一物种中的已知参考序列等效的多核苷酸(即,物种变体)。目标多核苷酸可来自任何目标生物体,包括但不限于非人、啮齿动物、仓鼠、小鼠、大鼠、人、猴、农业哺乳动物或非农业哺乳动物。目标多核苷酸还可包含编码区、非编码区、调控区或基因组DNA。因此,第1、第2、第3、第4、第5、第6、第7和/或任一后续的插入多核苷酸可包含此类序列。
在一个实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸与小鼠核酸序列、人核酸、非人核酸、啮齿动物核酸、大鼠核酸、仓鼠核酸、猴核酸、农业哺乳动物核酸或非农业哺乳动物核酸同源。在更进一步的实施例中,在靶基因座处整合的目标多核苷酸为基因组核酸的片段。在一个实施例中,基因组核酸为小鼠基因组核酸、人基因组核酸、非人核酸、啮齿动物核酸、大鼠核酸、仓鼠核酸、猴核酸、农业哺乳动物核酸或非农业哺乳动物核酸,或者它们的组合。
在一个实施例中,如上所述,目标多核苷酸可在约500个核苷酸至约200kb的范围内。目标多核苷酸可为约500个核苷酸至约5kb、约5kb至约200kb、约5kb至约10kb、约10kb至约20kb、约20kb至约30kb、约30kb至约40kb、约40kb至约50kb、约60kb至约70kb、约80kb至约90kb、约90kb至约100kb、约100kb至约110kb、约120kb至约130kb、约130kb至约140kb、约140kb至约150kb、约150kb至约160kb、约160kb至约170kb、约170kb至约180kb、约180kb至约190kb,或约190kb至约200kb。
在插入多核苷酸内的和/或插入在靶基因座处的目标多核苷酸可编码多肽,可编码miRNA,或者其可包含任何目标调控区或非编码区,包括例如调控序列、启动子序列、增强子序列、转录阻遏物结合序列、或非蛋白编码序列的缺失。另外,在插入多核苷酸内的和/或插入在靶基因座处的目标多核苷酸可编码在神经系统、骨骼系统、消化系统、循环系统、肌肉系统、呼吸系统、心血管系统、淋巴系统、内分泌系统、泌尿系统、生殖系统或它们的组合中表达的蛋白。在一个实施例中,在插入多核苷酸内的和/或插入在靶基因座处的目标多核苷酸编码在骨髓或骨髓来源细胞中表达的蛋白。在一个实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸编码在脾细胞中表达的蛋白。在更进一步的实施例中,在插入多核苷酸内的和/或插入在靶基因座处的目标多核苷酸编码在B细胞中表达的蛋白,编码在未成熟B细胞中表达的蛋白,或编码在成熟B细胞中表达的蛋白。
在一个实施例中,在插入多核苷酸内的和/或插入在靶基因座处的目标多核苷酸包含编码免疫球蛋白重链可变区氨基酸序列的基因组核酸序列。短语“重链”或“免疫球蛋白重链”包括来自任何生物体的免疫球蛋白重链序列,包括免疫球蛋白重链恒定区序列。除非另外指明,否则重链可变结构域包括三个重链CDR和四个骨架(FR)区。重链的片段包括CDR、CDR和FR,以及它们的组合。典型的重链具有在可变结构域之后(从N端到C端)的CH1结构域、铰链、CH2结构域和CH3结构域。重链的功能片段包括能够特异性识别表位(例如,以微摩尔级、纳摩尔级或皮摩尔级的KD识别表位)的片段,其能够表达并从细胞分泌,并且包含至少一个CDR。重链可变结构域由可变区核苷酸序列编码,其一般包含VH、DH和JH区段,所述VH、DH和JH区段来源于存在于种系中的VH、DH和JH区段组库。各种生物体的V、D和J重链区段的序列、位置和命名可见于IMGT数据库,可经由互联网在万维网(www)上以URL“imgt.org”访问该数据库。
在一个实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸包含编码人免疫球蛋白重链可变区氨基酸序列的基因组核酸序列。在一个实施例中,基因组核酸序列包含有效连接至免疫球蛋白重链恒定区核酸序列的非重排人免疫球蛋白重链可变区核酸序列。在一个实施例中,免疫球蛋白重链恒定区核酸序列为小鼠免疫球蛋白重链恒定区核酸序列,或人免疫球蛋白重链恒定区核酸序列,或它们的组合。在一个实施例中,免疫球蛋白重链恒定区核酸序列选自CH1、铰链、CH2、CH3以及它们的组合。在一个实施例中,重链恒定区核酸序列包含CH1-铰链-CH2-CH3。在一个实施例中,基因组核酸序列包含有效连接至免疫球蛋白重链恒定区核酸序列的重排人免疫球蛋白重链可变区核酸序列。在一个实施例中,免疫球蛋白重链恒定区核酸序列为小鼠免疫球蛋白重链恒定区核酸序列,或人免疫球蛋白重链恒定区核酸序列,或它们的组合。在一个实施例中,免疫球蛋白重链恒定区核酸序列选自CH1、铰链、CH2、CH3以及它们的组合。在一个实施例中,重链恒定区核酸序列包含CH1-铰链-CH2-CH3。
在一个实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸包含编码免疫球蛋白轻链可变区氨基酸序列的基因组核酸序列。短语“轻链”包括来自任何生物体的免疫球蛋白轻链序列,并且除非另外指明,否则包括人kappa(κ)和lambda(λ)轻链及VpreB以及替代轻链。除非另外指明,否则轻链可变结构域通常包括三个轻链CDR和四个FR。一般来讲,全长轻链从氨基端到羧基端包括含有FR1-CDR1-FR2-CDR2-FR3-CDR3-FR4的可变结构域,以及轻链恒定区氨基酸序列。轻链可变结构域由轻链可变区核苷酸序列编码,其一般包含轻链VL和轻链JL基因区段,所述轻链VL和轻链JL基因区段来源于存在于种系中的轻链V和J基因区段组库。各种生物体的轻链V和J基因区段的序列、位置和命名可见于IMGT数据库,可经由互联网在万维网(www)上以URL“imgt.org”访问该数据库。轻链包括例如不选择性地结合第一表位或第二表位的那些轻链,所述第一表位或第二表位由在其中呈现这些表位的表位结合蛋白选择性结合。轻链还包括结合并识别一个或多个表位或者帮助重链结合并识别一个或多个表位的那些轻链,所述一个或多个表位由在其中呈现这些表位的表位结合蛋白选择性结合。
在一个实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸包含编码人免疫球蛋白轻链可变区氨基酸序列的基因组核酸序列。在一个实施例中,基因组核酸序列包含非重排人λ和/或κ轻链可变区核酸序列。在一个实施例中,基因组核酸序列包含重排人λ和/或κ轻链可变区核酸序列。在一个实施例中,非重排或重排λ和/或κ轻链可变区核酸序列有效连接至选自λ轻链恒定区核酸序列和κ轻链恒定区核酸序列的小鼠、大鼠或人免疫球蛋白轻链恒定区核酸序列。
在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸可编码胞外蛋白或受体的配体。在具体实施例中,所编码的配体为细胞因子。目标细胞因子包括选自CCL、CXCL、CX3CL和XCL的趋化因子。所述细胞因子还可包括肿瘤坏死因子(TNF)。在另一些实施例中,所述细胞因子为白介素(IL)。在一个实施例中,所述白介素选自IL-1、IL-2、IL-3、IL-4、IL-5、IL-6、IL-7、IL-8、IL-9、IL-10、IL-11、IL-12、IL-13、IL-14、IL-15、IL-16、IL-17、IL-18、IL-19、IL-20、IL-21、IL-22、IL-23、IL-24、IL-25、IL-26、IL-27、IL-28、IL-29、IL-30、IL-31、IL-32、IL-33、IL-34、IL-35以及IL-36。在一个实施例中,所述白介素为IL-2。在具体实施例中,在插入多核苷酸内的和/或在靶基因座处整合的此类目标多核苷酸来自人,并且在更具体的实施例中,可包含人序列。
在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸可编码胞质蛋白或膜蛋白。在一个实施例中,所述膜蛋白为受体,诸如细胞因子受体、白介素受体、白介素2受体α、白介素2受体β或白介素2受体γ。
在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸可包括编码T细胞受体(包括T细胞受体α)至少一个区域的多核苷酸。在具体方法中,所述插入多核苷酸中的每一者包含T细胞受体基因座(即,T细胞受体α基因座)的区域,使得在连续整合完成后,T细胞受体基因座的一部分或全部已经在靶基因座处整合。此类插入多核苷酸可包含T细胞受体基因座(即,T细胞受体α基因座)的可变区段或连接区段中的至少一者或多者。在更进一步的实施例中,编码T细胞受体的区域的目标多核苷酸可来自例如编码突变体蛋白的哺乳动物、非人哺乳动物、啮齿动物、小鼠、大鼠、人、猴、农业哺乳动物或家养哺乳动物多核苷酸。
在其他实施例中,在靶基因座处整合的目标多核苷酸编码核蛋白。在一个实施例中,所述核蛋白为核受体。在具体实施例中,在插入多核苷酸内的和/或在靶基因座处整合的此类目标多核苷酸来自人,并且在更具体的实施例中,可包含人基因组序列。
在插入多核苷酸内的和/或在靶基因组座位处整合的目标多核苷酸可包含编码序列中的遗传修饰。此类遗传修饰包括但不限于编码序列的缺失突变或两个编码序列的融合。
在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸可包含编码突变体蛋白的多核苷酸。在一个实施例中,所述突变体蛋白的特征在于改变的结合特性、改变的定位、改变的表达和/或改变的表达模式。在一个实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸包含至少一种疾病等位基因,包括例如神经疾病等位基因、心血管疾病等位基因、肾脏疾病等位基因、肌肉疾病等位基因、血液疾病等位基因、致癌基因等位基因或免疫系统疾病等位基因。在此类情况下,所述疾病等位基因可为显性等位基因,或者所述疾病等位基因为隐性等位基因。此外,所述疾病等位基因可包括单核苷酸多态性(SNP)等位基因。编码突变体蛋白的目标多核苷酸可来自任何生物体,包括但不限于编码突变体蛋白的哺乳动物、非人哺乳动物、啮齿动物、小鼠、大鼠、人、猴、农业哺乳动物或家养哺乳动物多核苷酸。
在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸还可包含调控序列,包括例如启动子序列、增强子序列或转录阻遏物结合序列。在具体实施例中,在插入多核苷酸内的和/或在靶基因座处整合的目标多核苷酸包括具有非蛋白编码序列缺失但不包含蛋白编码序列缺失的多核苷酸。在一个实施例中,所述非蛋白编码序列的缺失包括调控序列的缺失。在另一个实施例中,所述调控元件的缺失包括启动子序列的缺失。在一个实施例中,所述调控元件的缺失包括增强子序列的缺失。此类目标多核苷酸可来自任何生物体,包括但不限于编码突变体蛋白的哺乳动物、非人哺乳动物、啮齿动物、小鼠、大鼠、人、猴、农业哺乳动物或家养哺乳动物多核苷酸。
如上所概述,本文提供了允许靶向整合一个或多个目标多核苷酸的方法和组合物。此类系统采用多种组分,并且为了便于提及,在本文中术语“靶向整合系统”通常是指整合事件所需的所有组分(即,各种核酸酶试剂、识别位点、插入DNA多核苷酸、靶向载体、靶基因座和目标多核苷酸)。
本文所提供的方法包括向细胞中引入包含靶向整合系统的各种组分的一个或多个多核苷酸或多肽构建体。术语“引入”包括以使得序列(多肽或多核苷酸)能够进入细胞内部的方式将序列呈递到细胞。本文所提供的方法并不取决于将靶向整合系统的任何组分引入细胞中的特定方法,只要使多核苷酸能够进入至少一个细胞内部即可。用于将多核苷酸引入到各种细胞类型中的方法是本领域已知的,并且包括但不限于稳定转染方法、瞬时转染方法和病毒介导的方法。
在一些实施例中,在所述方法和组合物中采用的细胞具有稳定地掺入到其基因组中的DNA构建体。“稳定地掺入”或“稳定地引入”意指将多核苷酸引入到细胞中,使得核苷酸序列整合到细胞的基因组中且能够遗传给其子代。可使用任何方案稳定地掺入DNA构建体或靶向整合系统的各种组分。
转染方案以及将多肽或多核苷酸序列引入到细胞中的方案可有所差别。非限制性转染方法包括基于化学的转染方法,其包括使用:脂质体;纳米粒子;磷酸钙(Graham etal.(1973).Virology 52(2):456–67(Graham等人,1973年,《病毒学》,第52卷,第2期,第456–467页),Bacchetti et al.(1977)Proc Natl Acad Sci USA 74(4):1590–4(Bacchetti等人,1977年,《美国国家科学院院刊》,第74卷,第4期,第1590–1594页),以及Kriegler,M(1991).Transfer and Expression:A Laboratory Manual.New York:W.H.Freeman and Company.pp.96–97(Kriegler,M,1991年,《基因转染和表达实验手册》,纽约W.H.弗里曼公司,第96–97页));树状体;或阳离子聚合物,诸如DEAE-葡聚糖或聚乙烯亚胺。非化学方法包括电穿孔、超声穿孔和光学转染。基于颗粒的转染包括使用基因枪、磁体辅助转染(Bertram,J.(2006)Current Pharmaceutical Biotechnology 7,277–28(Bertram,J.,2006年,《当今药物生物技术》,第7卷,第277–28页))。也可将病毒方法用于转染。
在一个实施例中,将核酸酶试剂随靶向载体或大靶向载体(LTVEC)一起引入到细胞中。另选地,在一段时间内将核酸酶试剂与靶向载体或LTVEC分别引入。在一个实施例中,在引入靶向载体或LTVEC之前引入核酸酶试剂,而在其他实施例中,在引入靶向载体或LTVEC之后引入核酸酶试剂。
可采用本文所公开的各种方法产生非人哺乳动物。此类方法包括(1)采用本文所公开的方法在非人动物多能细胞的靶基因座处整合一个或多个目标多核苷酸,以生成在所靶向基因座中包含插入多核苷酸的经遗传修饰的多能细胞;(2)选择在靶基因座处具有一个或多个目标多核苷酸的经遗传修饰的多能细胞;(3)将经遗传修饰的多能细胞引入到处于桑椹胚前期的非人动物宿主胚胎中;以及(4)将包含经遗传修饰的多能细胞的宿主胚胎植入代孕母体中,以产生来源于经遗传修饰的多能细胞的F0代。所述非人动物可为非人哺乳动物、啮齿动物(例如,小鼠、大鼠、仓鼠)、猴、农业哺乳动物或家养哺乳动物。所述多能细胞可为人ES细胞、人iPS细胞、非人ES细胞、啮齿动物ES细胞(例如,小鼠ES细胞、大鼠ES细胞或仓鼠ES细胞)、猴ES细胞、农业哺乳动物ES细胞或家养哺乳动物ES细胞。参见例如美国专利公开No.2014/0235933、美国专利公开No.2014/0310828以及Tong et al.(2010)Nature,467(7312):211-213(Tong等人,2010年,《自然》,第467卷,第7312期,第211-213页),这些文献中的每一者均全文以引用的方式并入本文。
也可使用核移植技术生成非人哺乳动物。简而言之,用于核移植的方法包括以下步骤:(1)将卵母细胞去核;(2)分离供体细胞或核,以与去核卵母细胞混合;(3)将所述细胞或核插入到所述去核卵母细胞中,以形成重建细胞;(4)将所述重建细胞植入到动物的子宫中,以形成胚胎;以及(5)允许所述胚胎发育。在此类方法中,一般从处死的动物体内取出卵母细胞,但也可从活动物的输卵管和/或卵巢中分离卵母细胞。卵母细胞可在去核之前在本领域普通技术人员已知的多种培养基中成熟。卵母细胞的去核可以本领域普通技术人员所熟知的多种方式进行。通常在融合之前在透明带下显微注射供体细胞来将供体细胞或核插入到去核卵母细胞中以形成重建细胞。融合可通过跨接触/融合平面施加直流电脉冲(电融合)、通过将细胞暴露于促进融合的化学品如聚乙二醇或者借助灭活病毒如仙台病毒来诱导。重建细胞通常在核供体和受体卵母细胞融合之前、期间和/或之后通过电和/或非电方式激活。激活方法包括电脉冲、化学诱导冲击、精子穿透、增加卵母细胞中二价阳离子水平以及减少卵母细胞中细胞蛋白磷酸化(如借助激酶抑制剂)。激活的重建细胞或胚胎通常在本领域普通技术人员所熟知的培养基中培养,然后移植到动物的子宫中。参见例如US20080092249、WO/1999/005266A2、US20040177390、WO/2008/017234A1以及美国专利No.7,612,250,这些专利中的每一者均以引用的方式并入本文。
提供了用于制备在其种系中包含一个或多个如本文所述的遗传修饰的非人动物的其他方法,所述方法包括:(a)采用本文所述的各种方法在原核细胞中修饰非人动物的所靶向基因座;(b)选择在所靶向基因座处包含遗传修饰的经修饰的原核细胞;(c)从经修饰的原核细胞分离经遗传修饰的靶向载体;(d)将经遗传修饰的靶向载体引入到非人动物的多能细胞中,以生成在所靶向基因座处包含插入核酸的经遗传修饰的多能细胞;(e)选择经遗传修饰的多能细胞;(f)将经遗传修饰的多能细胞引入到处于桑椹胚前期的非人动物宿主胚胎中;以及(g)将包含经遗传修饰的多能细胞的宿主胚胎植入代孕母体中,以产生来源于经遗传修饰的多能细胞的F0代。在此类方法中,所述靶向载体可包括大靶向载体。所述非人动物可为非人哺乳动物、啮齿动物、小鼠、大鼠、仓鼠、猴、农业哺乳动物或家养哺乳动物。所述多能细胞可为人ES细胞、人iPS细胞、非人ES细胞、啮齿动物ES细胞(例如,小鼠ES细胞、大鼠ES细胞或仓鼠ES细胞)、猴ES细胞、农业哺乳动物ES细胞或家养哺乳动物ES细胞。
在另外的方法中,分离步骤(c)还包括(c1)使经遗传修饰的靶向载体(即,经遗传修饰的LTVEC)线性化。在更进一步的实施例中,引入步骤(d)还包括(d1)将如本文所述的核酸酶试剂引入到多能细胞中。在其他实施例中,引入步骤(d)还包括(d2),其中非人哺乳动物的多能细胞包含靶基因座,所述靶基因座具有编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,以及将核酸酶试剂引入到所述多能细胞中,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂。进一步向所述多能细胞中引入第一靶向载体,所述第一靶向载体包含来自经修饰的原核细胞基因组的经遗传修饰的靶向载体。所述经修饰的靶向载体包含与第一靶位点和第二靶位点相对应的第一同源臂和第二同源臂,所述第一靶位点和第二靶位点足够接近非人哺乳动物的多能细胞基因组内的第一识别位点。在一个实施例中,通过将如本文所述的选择试剂施加到所述原核细胞或所述多能细胞来执行选择步骤(b)和/或(e)。在一个实施例中,经由如本文所述的等位基因修饰(MOA)测定法执行选择步骤(b)和/或(e)。
提供了经由原核细胞中的细菌同源重组(BHR)来修饰哺乳动物细胞的靶基因座的其他方法,所述方法包括:(a)提供包含靶基因座的原核细胞,所述靶基因座包含编码第一选择标记并有效连接至原核细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,(b)向所述原核细胞中引入包含侧接有第一上游同源臂和第一下游同源臂的插入多核苷酸的靶向载体,其中所述插入多核苷酸包含哺乳动物区,并且向所述原核细胞中引入在第一识别位点处或附近产生切口或双链断裂的核酸酶试剂,以及(c)选择在靶基因座处包含插入多核苷酸的所靶向原核细胞,其中所述原核细胞能够表达介导所述BHR的重组蛋白和重组酶。步骤(a)-(c)可如本文所公开的那样连续重复,以允许在原核细胞中的所靶向基因座处引入多个插入多核苷酸。一旦用原核细胞“构建”所靶向基因座,包含经修饰的靶基因座的靶向载体就可从原核细胞分离并引入到非人哺乳动物细胞内的靶基因座中。随后可将包含经修饰的基因座的哺乳动物细胞制成非人转基因动物。
在一些实施例中,可通过使用细菌人工染色体(BAC)DNA,采用
在一些实施例中,经由
本文所述的各种方法在细胞中采用基因座靶向系统。此类细胞包括:原核细胞,诸如细菌细胞,包括大肠杆菌(E.coli);或者真核细胞,诸如酵母、昆虫、两栖动物、禽类(例如,鸡细胞)、植物,或哺乳动物细胞,包括但不限于小鼠细胞、大鼠细胞、兔细胞、猪细胞、牛细胞、鹿细胞、绵羊细胞、山羊细胞、猫细胞、狗细胞、白鼬细胞、灵长类动物(例如,狨猴、恒河猴)细胞等,以及来自家养哺乳动物的细胞或来自农业哺乳动物的细胞。一些细胞为非人细胞,特别是非人哺乳动物细胞。在一些实施例中,对于不易获得合适的可遗传修饰的多能细胞的那些哺乳动物,采用其他方法使体细胞重新编程为多能细胞,例如经由向体细胞中引入多能诱导性因子的组合来使其重新编程,所述多能诱导性因子包括但不限于Oct3/4、Sox2、KLF4、Myc、Nanog、LIN28和Glis1。
在一个实施例中,所述真核细胞为多能细胞。在一个实施例中,所述多能细胞为胚胎干(ES)细胞。术语“胚胎干细胞”或“ES细胞”包括胚胎起源的全能或多能细胞,其能够在体外进行未分化增殖,并且能够在引入胚胎后促使发育中的胚胎成为任何组织。术语“多能细胞”包括有能力发育成不止一种分化细胞类型的未分化细胞。指涉多核苷酸序列的术语“种系”包括可传给子代的核酸序列。
所述多能细胞可为非人ES细胞或诱导多能干(iPS)细胞。在一个实施例中,所述诱导多能(iPS)细胞来源于成纤维细胞。在具体实施例中,所述诱导多能(iPS)细胞来源于人成纤维细胞。在一些实施例中,所述多能细胞为造血干细胞(HSC)、神经元干细胞(NSC)或外胚层干细胞。所述多能细胞也可为发育受限的祖细胞。在进一步的实施例中,所述多能细胞为啮齿动物多能细胞。在一个实施例中,所述啮齿动物多能细胞为大鼠多能细胞或大鼠ES细胞。在其他实施例中,所述啮齿动物多能细胞为小鼠多能细胞或小鼠ES细胞。
在其他实施例中,所述哺乳动物细胞可为无限增殖化小鼠细胞、大鼠细胞或人细胞。在一个实施例中,所述哺乳动物细胞为人成纤维细胞,而在其他实施例中,所述哺乳动物细胞为癌细胞,包括人癌细胞。
在更进一步的实施例中,所述哺乳动物为人并且所述靶向使用离体人细胞进行。
在一个实施例中,所述哺乳动物细胞为分离自患有疾病的患者的人细胞和/或包含编码突变体蛋白的人多核苷酸。在一个实施例中,所述突变体人蛋白的特征在于改变的结合特性、改变的定位、改变的表达和/或改变的表达模式。在一个实施例中,所述人核酸序列包含至少一个人疾病等位基因。在一个实施例中,所述人核酸序列包含至少一个人疾病等位基因。
在一个实施例中,所述人疾病等位基因为神经疾病等位基因。在一个实施例中,所述人疾病等位基因为心血管疾病等位基因。在一个实施例中,所述人疾病等位基因为肾脏疾病等位基因。在一个实施例中,所述人疾病等位基因为肌肉疾病等位基因。在一个实施例中,所述人疾病等位基因为血液疾病等位基因。在一个实施例中,所述人疾病等位基因为致癌基因的等位基因。在一个实施例中,所述人疾病等位基因为免疫系统疾病等位基因。在一个实施例中,所述人疾病等位基因为显性等位基因。在一个实施例中,所述人疾病等位基因为隐性等位基因。在一个实施例中,所述人疾病等位基因包括单核苷酸多态性(SNP)等位基因。
当所述细胞包括原核细胞时,在具体实施例中,所述原核细胞为大肠杆菌的重组感受态菌株。在一个实施例中,所述原核细胞包含编码重组蛋白和重组酶的核酸。在一个实施例中,所述原核细胞不包含编码重组蛋白和重组酶的核酸,并且将编码重组蛋白和重组酶的核酸引入所述原核细胞中。在一个实施例中,所述核酸包含编码重组蛋白和重组酶的DNA或mRNA。在一个实施例中,编码重组蛋白和重组酶的核酸为pABG。在一个实施例中,所述重组蛋白和重组酶在诱导型启动子的控制下表达。在一个实施例中,所述重组蛋白和重组酶的表达通过阿拉伯糖控制。
本文提供了包含本文所提供的靶向整合系统的各种组分(即,核酸酶试剂、识别位点、插入多核苷酸、目标多核苷酸、靶向载体、选择标记以及其他组分)的多核苷酸或核酸分子。
术语“多核苷酸”、“多核苷酸序列”、“核酸序列”和“核酸片段”可在本文中互换使用。这些术语涵盖核苷酸序列等。多核苷酸可为任选含有合成、非天然或改变的核苷酸碱基的单链或双链的RNA或DNA聚合物。DNA聚合物形式的多核苷酸可由cDNA、基因组DNA、合成DNA或它们的混合物的一个或多个区段构成。多核苷酸可包含脱氧核糖核苷酸和核糖核苷酸,其包括天然存在的分子和合成类似物两者以及这些的任何组合。本文提供的多核苷酸还涵盖所有形式的序列,包括但不限于单链形式、双链形式、发夹、茎-环结构等。
还提供了包含靶向整合系统的各种组分的重组多核苷酸。术语“重组多核苷酸”和“重组DNA构建体”可在本文中互换使用。重组构建体包含核酸序列的人工或异源组合,例如自然界中不共存的调控序列和编码序列。在其他实施例中,重组构建体可包含来自不同来源的调控序列和编码序列,或来自相同来源但以与自然界中的存在方式不同的方式排列的调控序列和编码序列。这种构建体可独自使用或可结合载体使用。如果使用载体,则载体的选择取决于如本领域技术人员众所周知的用以转化宿主细胞的方法。例如,可使用质粒载体。本文提供了成功转化、选择和繁殖宿主细胞所需的且包含任一种分离核酸片段的遗传元件。筛选可尤其通过DNA的Southern分析、mRNA表达的Northern分析、蛋白表达的免疫印迹分析或表型分析来实现。
在具体实施例中,本文所述的靶向整合系统的组分中的一者或多者可提供于表达盒中,以便在原核细胞、真核细胞、细菌、酵母细胞或哺乳动物细胞或其他目标生物体或细胞类型中表达。所述盒可包括有效连接至本文所提供的多核苷酸的5'调控序列和3'调控序列。“有效连接”包括两个或更多个元件之间的功能性连接。例如,目标多核苷酸与调控序列(即,启动子)之间的有效连接为允许表达目标多核苷酸的功能性连接。有效连接的元件可为邻接的或不邻接的。当用以提到两个蛋白编码区的连接时,有效连接意指编码区在同一阅读框中。在另一种情况下,编码蛋白的核酸序列可有效连接至调控序列(例如,启动子、增强子、沉默子序列等)以保持恰当的转录调控。在一种情况下,免疫球蛋白可变区(或V(D)J区段)的核酸序列可有效连接至免疫球蛋白恒定区的核酸序列,以允许在序列之间恰当重组成免疫球蛋白重链或轻链序列。
所述盒可另外含有将共同引入生物体中的至少一个额外目标多核苷酸。或者,所述额外目标多核苷酸可提供于多个表达盒上。这种表达盒提供有多个限制位点和/或重组位点,以使重组多核苷酸的插入处于调控区的转录调控之下。所述表达盒可另外含有选择标记基因。
所述表达盒在5'-3'转录方向上可包含在哺乳动物细胞或目标宿主细胞中起作用的转录和翻译起始区(即,启动子)、本文所提供的重组多核苷酸、以及转录和翻译终止区(即,终止区)。所述调控区(即,启动子、转录调控区和翻译终止区)和/或本文所提供的多核苷酸对于宿主细胞或对于彼此可为天然/类似的。另选地,所述调控区和/或本文所提供的多核苷酸对于宿主细胞或对于彼此可为异源的。例如,有效连接至异源多核苷酸的启动子来自与得到该多核苷酸的物种不同的物种,或者如果来自相同/类似的物种,则对一者或两者由其原始形式和/或基因座进行了实质性修饰,或者启动子不是有效连接的多核苷酸的天然启动子。另选地,所述调控区和/或本文所提供的重组多核苷酸可以是完全合成的。
所述终止区对于转录起始区而言可为天然的,对于有效连接的重组多核苷酸而言可为天然的,对于宿主细胞而言可为天然的,或者可来源于对于启动子、重组多核苷酸、宿主细胞或它们的任何组合而言为另一种的(即,外来的或异源的)来源。
在制备表达盒时,可对各种DNA片段进行操纵,以便提供处于正确取向的DNA序列。为此目的,可采用衔接子或接头将DNA片段连接在一起,或者可涉及其他的操纵以提供便利的限制性位点、去除多余的DNA、去除限制性位点等。出于这个目的,可能涉及体外诱变、引物修复、限制性酶切、退火、再置换(例如转换和颠换)。
多种启动子可用于本文所提供的表达盒中。可根据期望的结果来选择启动子。已经认识到,不同的应用可通过在表达盒中使用不同的启动子来增强,从而调整目标多核苷酸的表达的时机、位置和/或水平。如果需要,此类表达构建体还可含有启动子调控区(例如,赋予可诱导的、组成性的、环境或发育调控的、或细胞或组织特异性/选择性表达的启动子调控区)、转录起始位点、核糖体结合位点、RNA加工信号、转录终止位点和/或多聚腺苷酸化信号。
含有本文所提供的多核苷酸的表达盒还可包含用于选择转化细胞的选择标记基因。利用选择标记基因来选择转化细胞或组织。
在适当的情况下,可优化在所述方法和组合物(即,目标多核苷酸、核酸酶试剂等)中采用的序列,以便增加在细胞中的表达。也就是说,所述基因可使用给定目标细胞中偏好的密码子来合成以便提高表达,所述密码子包括例如哺乳动物偏好密码子、人偏好密码子、啮齿动物偏好密码子、小鼠偏好密码子、大鼠偏好密码子等。
本文所提供的方法和组合物采用靶向整合系统的多种不同组分(即,核酸酶试剂、识别位点、插入多核苷酸、目标多核苷酸、靶向载体、选择标记及其他组分)。在本说明书通篇中已经认识到,靶向整合系统的一些组分可具有活性变体和片段。此类组分包括例如核酸酶试剂(即,经改造的核酸酶试剂)、核酸酶试剂识别位点、目标多核苷酸、靶位点以及靶向载体的对应同源臂。这些组分中的每一者的生物活性在本文别处描述。
如本文所用,在两个多核苷酸或多肽序列的语境中的“序列同一性”或“同一性”指在指定比较窗口内比对以实现最大对应性时这两个序列中相同的残基。当指涉蛋白的情况下使用序列同一性百分比时,已经认识到不相同的残基位置通常差别在于保守氨基酸置换,其中氨基酸残基被置换为具有类似化学特性(例如,电荷或疏水性)的其他氨基酸残基且因此不改变分子的功能特性。当序列差别在于保守置换时,可上调序列同一性百分比以校正置换的保守性质。差别在于此类保守置换的序列被称为具有“序列相似性”或“相似性”。进行这种调节的方式是本领域技术人员众所周知的。通常,这涉及将保守置换作为部分错配而不作为完全错配来评分,从而增加序列同一性百分比。因此,例如,若一个相同氨基酸被给定1的分数且一个非保守置换被给定0的分数,则一个保守置换被给定0至1之间的分数。保守置换的分数例如如在程序PC/GENE(加利福尼亚州山景城的Intelligenetics公司(Intelligenetics,Mountain View,California))中所实现的那样来计算。
如本文所用,“序列同一性百分比”意指通过在比较窗口内比较两个经最佳比对的序列而确定的值,其中与参考序列(其不包含添加或缺失)相比较,多核苷酸序列在比较窗口中的部分可包含添加或缺失(即,空位),以便最佳比对这两个序列。该百分比通过以下方式计算:确定其中相同的核酸碱基或氨基酸残基在两个序列中均出现的位置的数目以得到匹配位置的数目,将匹配位置的数目除以在比较窗口中的位置总数,并且将结果乘以100以得到序列同一性百分比。
除非另作说明,否则本文所提供的序列同一性/相似性值是指使用GAP版本10采用以下参数获得的值:核苷酸序列的同一性%和相似性%使用空位权重(GAP Weight)50和长度权重3及nwsgapdna.cmp评分矩阵;氨基酸序列的同一性%和相似性%使用空位权重8和长度权重2及BLOSUM62评分矩阵;或其任何等同程序。“等同程序”意指任何序列比较程序,其为所考虑的任何两个序列产生这样的比对,当与由GAP版本10产生的对应比对相比较时,该比对具有相同的核苷酸或氨基酸残基匹配和相同的序列同一性百分比。
非限制性实施例包括:
1。一种用于修饰细胞中的靶基因座的方法,包括:(a)提供包含靶基因座的细胞,所述靶基因座包含编码第一选择标记并有效连接至细胞中有活性的第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,(b)向细胞中引入(i)第一核酸酶试剂,其中所述第一核酸酶试剂在第一识别位点处诱导切口或双链断裂;和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸侧接第一同源臂和第二同源臂,所述第一同源臂和第二同源臂与位于足够接近第一识别位点处的第一靶位点和第二靶位点相对应;以及(c)鉴定包含在靶基因座处整合的第一插入多核苷酸的至少一个细胞。
2。一种用于修饰细胞中的靶基因座的方法,包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的第一多核苷酸,其中所述第一多核苷酸还包含第一核酸酶试剂的第一识别位点,(b)向细胞中引入:(i)编码第一核酸酶试剂并有效连接至细胞中有活性的启动子的一个或多个表达构建体,其中所述第一核酸酶试剂在第一多核苷酸中的第一识别位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性;和(ii)包含第一插入多核苷酸的第一靶向载体,所述第一插入多核苷酸包含编码第二选择标记并有效连接至第二启动子的第二多核苷酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和所述第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;以及(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和所述第二选择标记不同。
3。实施例1或2所述的方法,其中靶基因座位于细胞的基因组中。
4。实施例1或2所述的方法,其中靶基因座位于细胞中的载体中。
5。实施例1-4中任一项所述的方法,其中第一识别位点处的切口或双链断裂破坏第一选择标记的活性。
6。实施例1、2、3、4或5所述的方法,其中鉴定步骤(c)包括在允许鉴定没有第一选择标记的活性的细胞的条件下培养细胞。
7。实施例1-6中任一项所述的方法,其中包含第一选择标记的第一多核苷酸侧接第一靶位点和第二靶位点。
8。实施例7所述的方法,其中鉴定步骤(c)包括鉴定包含在第一靶位点和第二靶位点处整合的第一插入多核苷酸的至少一个细胞。
9。实施例1-8中任一项所述的方法,其中第一插入多核苷酸包括:(a)第一目标多核苷酸;和(b)编码第二选择标记并有效连接至细胞中有活性的第二启动子的第二多核苷酸,其中所述第二多核苷酸包含第二核酸酶试剂的第二识别位点。
10。实施例9所述的方法,其中所述方法还包括(a)向包含在靶基因座处整合的第一插入多核苷酸的细胞中引入(i)第二核酸酶试剂,其中所述第二核酸酶试剂在第二识别位点处诱导切口或双链断裂;和(ii)包含第二插入多核苷酸的第二靶向载体,所述第二插入多核苷酸侧接第三同源臂和第四同源臂,所述第三同源臂和第四同源臂与位于足够接近第二识别位点处的第三靶位点和第四靶位点相对应;以及(b)鉴定包含在靶基因座处整合的第二插入多核苷酸的至少一个细胞。
11。实施例10所述的方法,其中第二识别位点处的切口或双链断裂破坏第二选择标记的活性。
12。实施例11所述的方法,其中鉴定步骤(b)包括在允许鉴定没有第二选择标记的活性的细胞的条件下培养细胞。
13。实施例10、11或12所述的方法,其中包含第二选择标记的第二多核苷酸侧接第三靶位点和第四靶位点。
14。实施例13所述的方法,其中鉴定步骤(b)包括鉴定包含在第三靶位点和第四靶位点处整合的第二插入多核苷酸的至少一个细胞。
15。实施例10-14中任一项所述的方法,其中第二插入多核苷酸包括:(a)第二目标多核苷酸;和(b)编码第三选择标记并有效连接至细胞中有活性的第三启动子的第三多核苷酸,其中所述第三多核苷酸包含第三核酸酶试剂的第三识别位点。
16。实施例9-15中任一项所述的方法,其中第一核酸酶试剂与第二核酸酶试剂不同。
17。实施例9-16中任一项所述的方法,其中第一选择标记与第二选择标记不同。
18。实施例15所述的方法,其中第一核酸酶识别位点和第三核酸酶识别位点彼此相同并与第二核酸酶识别位点不同;并且第一核酸酶试剂和第三核酸酶试剂彼此相同并与第二核酸酶试剂不同。
19。实施例15所述的方法,其中第一选择标记和第三选择标记相同。
20。实施例1-19中任一项所述的方法,其中第一选择标记、第二选择标记或第三选择标记中的一者赋予对抗生素的抗性。
21。实施例20所述的方法,其中抗生素包括G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素。
22。实施例1-19中任一项所述的方法,其中第一选择标记、第二选择标记或第三选择标记中的一者有效连接至诱导型启动子,并且选择标记的表达对细胞有毒性。
23。实施例22所述的方法,其中第一选择标记、第二选择标记或第三选择标记包括次黄嘌呤-鸟嘌呤磷酸核糖转移酶(HGPRT)或单纯性疱疹病毒的胸苷激酶(HSV-TK)。
24。实施例1-23中任一项所述的方法,其中所述细胞为原核细胞。
25。实施例1-23中任一项所述的方法,其中所述细胞为真核细胞。
26。实施例25所述的方法,其中所述真核细胞为哺乳动物细胞。
27。实施例26所述的方法,其中所述哺乳动物细胞为非人哺乳动物细胞。
28。实施例27所述的方法,其中所述哺乳动物细胞来自啮齿动物。
29。实施例28所述的方法,其中所述啮齿动物为大鼠或小鼠。
30。实施例26-29中任一项所述的方法,其中所述细胞为多能细胞。
31。实施例26所述的方法,其中所述哺乳动物细胞为人诱导性多能干(iPS)细胞。
32。实施例30所述的方法,其中所述多能细胞为非人胚胎干(ES)细胞。
33。实施例30所述的方法,其中所述多能细胞为小鼠胚胎干(ES)细胞或大鼠胚胎干(ES)细胞。
34。实施例30-33中任一项所述的方法,其中所述多能细胞为造血干细胞。
35。实施例30-33中任一项所述的方法,其中所述多能细胞为神经元干细胞。
36。实施例26所述的方法,其中所述哺乳动物细胞为人成纤维细胞。
37。实施例1或2所述的方法,其中与单独使用第一靶向载体相比,联合使用第一靶向载体与第一核酸酶试剂会提高靶向效率。
38。实施例37所述的方法,其中与单独使用第一靶向载体相比,第一靶向载体的靶向效率提高了至少2倍。
39。实施例1-38中任一项所述的方法,其中第一核酸酶试剂或第二核酸酶试剂包括表达构建体,所述表达构建体包含编码核酸酶试剂的核酸序列,并且其中所述核酸有效连接至细胞中有活性的第四启动子。
40。实施例1-39中任一项所述的方法,其中第一核酸酶试剂或第二核酸酶试剂为编码核酸酶的mRNA。
41。实施例1-39中任一项所述的方法,其中第一核酸酶试剂或第二核酸酶试剂为锌指核酸酶(ZFN)。
42。实施例1-39中任一项所述的方法,其中第一核酸酶试剂或第二核酸酶试剂为转录激活因子样效应物核酸酶(TALEN)。
43。实施例1-39中任一项所述的方法,其中第一核酸酶试剂或第二核酸酶试剂为大范围核酸酶。
44。实施例1-43中任一项所述的方法,其中第一核酸酶试剂或第二核酸酶试剂包括成簇规律间隔短回文重复序列(CRISPR)相关(Cas)蛋白和向导RNA(gRNA)。
45。实施例44所述的方法,其中向导RNA(gRNA)包括(a)靶向第一识别位点、第二识别位点或第三识别位点的成簇规律间隔短回文重复序列(CRISPR)RNA(crRNA);和(b)反式激活CRISPR RNA(tracrRNA)。
46。实施例45所述的方法,其中第一识别位点或第二识别位点紧密侧接前间区序列邻近基序(PAM)序列。
47。实施例44、45或46所述的方法,其中目标基因组座位包含SEQ ID NO:1的核苷酸序列。
48。实施例44、45、46或47所述的方法,其中Cas蛋白为Cas9。
49。实施例44-46中任一项所述的方法,其中gRNA包括:(a)SEQ ID NO:2的核酸序列的嵌合RNA;或(b)SEQ ID NO:3的核酸序列的嵌合RNA。
50。实施例44-46中任一项所述的方法,其中crRNA包含SEQ ID NO:4、SEQ ID NO:5或SEQ ID NO:6。
51。实施例44-46中任一项所述的方法,其中tracrRNA包含SEQ ID NO:7或SEQ IDNO:8。
52。实施例1-51中任一项所述的方法,其中第一识别位点、第二识别位点和/或第三识别位点位于第一选择标记、第二选择标记或第三选择标记的内含子、外显子、启动子、启动子调控区或增强子区中。
53。实施例1-52中任一项所述的方法,其中第一靶位点和第二靶位点紧邻第一识别位点。
54。实施例10-19中任一项所述的方法,其中第一靶位点和第二靶位点距第一识别位点约10个核苷酸至约14kb。
55。实施例10-19中任一项所述的方法,其中第三靶位点和第四靶位点紧邻第二识别位点。
56。实施例10-19中任一项所述的方法,其中第三靶位点和第四靶位点距第二识别位点约10个核苷酸至约14kb。
57。实施例1-56中任一项所述的方法,其中第一同源臂和第二同源臂的总和为至少约10kb。
58。实施例10-57中任一项所述的方法,其中第三同源臂和第四同源臂的总和为至少约10kb。
59。实施例1-58中任一项所述的方法,其中第一插入多核苷酸的长度在约5kb至约300kb的范围内。
60。实施例10-59中任一项所述的方法,其中第二插入多核苷酸的长度在约5kb至约300kb的范围内。
61。实施例1-60中任一项所述的方法,其中将第一插入多核苷酸整合到靶基因座中会导致敲除、敲入、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换,或它们的组合。
62。实施例10-61中任一项所述的方法,其中将第二插入多核苷酸整合到靶基因座中会导致敲除、敲入、点突变、结构域交换、外显子交换、内含子交换、调控序列交换、基因交换,或它们的组合。
63。实施例1-62中任一项所述的方法,其中第一插入多核苷酸包括含有人多核苷酸的目标多核苷酸。
64。实施例8-63中任一项所述的方法,其中第二插入多核苷酸包括含有人多核苷酸的目标多核苷酸。
65。实施例1-64中任一项所述的方法,其中第一插入多核苷酸包括含有T细胞受体α基因座区域的目标多核苷酸。
66。实施例8-65中任一项所述的方法,其中第二插入多核苷酸包括含有T细胞受体α基因座区域的目标多核苷酸。
67。实施例65或66所述的方法,其中第一插入多核苷酸或第二插入多核苷酸包括含有T细胞受体α基因座的至少一个可变区基因区段和/或连接区基因区段的目标多核苷酸。
68。实施例65-67中任一项所述的方法,其中T细胞受体α基因座区域来源于人。
69。实施例1-64中任一项所述的方法,其中第一插入多核苷酸包括含有有效连接至非人免疫球蛋白重链恒定区核酸序列的非重排人免疫球蛋白重链可变区核酸序列的目标多核苷酸。
70。实施例1-69中任一项所述的方法,其中通过等位基因修饰(MOA)测定法来执行鉴定步骤。
71。实施例1-65中任一项所述的方法,其中第一插入多核苷酸包括含有与细胞基因组中的核酸序列同源或直系同源的核酸序列的目标多核苷酸。
72。实施例10-71中任一项所述的方法,其中第二插入多核苷酸包含与细胞基因组中的核酸序列同源或直系同源的核酸序列。
73。实施例1-70中任一项所述的方法,其中第一插入多核苷酸包括含有外源核酸序列的目标多核苷酸。
74。实施例10-70或73中任一项所述的方法,其中第二插入多核苷酸包括含有外源核酸序列的目标多核苷酸。
其他非限制性实施例包括:
1。一种用于修饰细胞中的目标靶基因座的方法,包括:(a)提供包含第一靶基因座的细胞,所述第一靶基因座包含编码第一选择标记并有效连接至第一启动子的核酸,(b)向细胞中引入:(i)编码Cas蛋白和第一向导RNA(gRNA)的一个或多个表达构建体,每个所述表达构建体有效连接至细胞中有活性的启动子,其中所述Cas蛋白在第一核酸中的第一gRNA靶位点处诱导切口或双链断裂,从而破坏第一选择标记的表达或活性;和(ii)包含第一插入核酸的第一靶向载体,所述第一插入核酸包含编码第二选择标记并有效连接至第二启动子的第二核酸,其中所述第一插入核酸侧接第一同源臂和第二同源臂,所述第一同源臂和所述第二同源臂与位于第一靶基因座中的第一靶位点和第二靶位点相对应;以及(c)鉴定在第一靶基因座处包含第一插入核酸的修饰细胞,其中所述修饰细胞具有第二选择标记的活性,但不具有第一选择标记的活性,并且其中所述第一选择标记和所述第二选择标记不同。
2。实施例1所述的方法,其中第一gRNA不与第一插入核酸杂交。
3。实施例1所述的方法,其中目标靶基因座位于细胞的基因组中。
4。实施例1所述的方法,其中目标靶基因座位于细胞中的载体中。
5。实施例1所述的方法,其中鉴定步骤(c)包括在允许鉴定具有第二选择标记的活性但不具有第一选择标记的活性的修饰细胞的条件下培养细胞。
6。实施例1所述的方法,还包括:(d)向在第一靶基因座处包含第一插入核酸的修饰细胞中引入:(i)编码Cas蛋白和第二gRNA的一个或多个核酸,每个所述核酸有效连接至修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第二核酸的第一插入核酸中的第二gRNA靶位点处诱导切口或双链断裂,从而破坏第二选择标记的表达或活性,和(ii)包含第二插入核酸的第二靶向载体,所述第二插入核酸包含编码第三选择标记并有效连接至第三启动子的第三核酸,其中所述第二插入核酸侧接第三同源臂和第四同源臂,所述第三同源臂和所述第四同源臂与位于第二靶基因座中的第三靶位点和第四靶位点相对应;以及(e)鉴定在第二靶基因座处包含第二插入核酸的第二修饰细胞,其中所述第二修饰细胞具有第三选择标记的活性,但不具有第二选择标记的活性,其中所述第二选择标记和所述第三选择标记不同。
7。实施例6所述的方法,其中第一靶基因座和第二靶基因座位于彼此紧邻的位置。
8。实施例6所述的方法,其中第一靶基因座或第二靶基因座位于距第一gRNA靶位点或第二gRNA靶位点约10个核苷酸至约14kb处。
9。实施例8所述的方法,其中第二gRNA不与第二插入核酸杂交。
10。实施例6所述的方法,其中鉴定步骤(e)包括在允许鉴定具有第三选择标记的活性但没有第二选择标记的活性的第二修饰细胞的条件下培养修饰细胞。
11。实施例6所述的方法,还包括:(f)向在第二靶基因座处包含第二插入核酸的第二修饰细胞中引入:(i)编码Cas蛋白和第三gRNA的一个或多个表达构建体,每个所述表达构建体有效连接至第二修饰细胞中有活性的启动子,其中所述Cas蛋白在包含第三核酸的第二插入核酸中的第三gRNA靶位点处诱导切口或双链断裂,从而破坏第三选择标记的表达或活性,和(ii)包含第三插入核酸的第三靶向载体,所述第三插入核酸包含编码第四选择标记并有效连接至第四启动子的第四核酸,其中所述第三插入核酸侧接第五同源臂和第六同源臂,所述第五同源臂和所述第六同源臂与位于第三靶基因座中的第五靶位点和第六靶位点相对应;以及(g)鉴定在第三靶基因座处包含第三插入核酸的第三修饰细胞,其中所述第三修饰细胞具有第四选择标记的活性,但不具有第三选择标记的活性,其中所述第三选择标记和所述第四选择标记不同。
12。实施例11所述的方法,其中第二靶基因座和第三靶基因座位于彼此紧邻的位置。
13。实施例11所述的方法,其中第二靶基因座或第三靶基因座位于距第一gRNA靶位点或第二gRNA靶位点约10个核苷酸至约14kb处。
14。实施例1-13中任一项所述的方法,其中第一标记、第二标记、第三标记或第四标记赋予对抗生素的抗性。
15。实施例14所述的方法,其中抗生素包括G418、潮霉素、杀稻瘟菌素、新霉素或嘌呤霉素。
16。实施例1-13中任一项所述的方法,其中第一选择标记、第二选择标记、第三选择标记或第四选择标记包括次黄嘌呤-鸟嘌呤磷酸核糖转移酶(HGPRT)或单纯性疱疹病毒的胸苷激酶(HSV-TK)。
17。实施例1、6或11所述的方法,其中第一gRNA、第二gRNA或第三gRNA包括(i)与第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点杂交的核苷酸序列,和(ii)反式激活CRISPR RNA(tracrRNA)。
18。实施例1、6或11所述的方法,其中第一靶基因座、第二靶基因座或第三靶基因座位于紧邻第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点的位置,使得gRNA靶位点处的切口或双链断裂促进靶基因座处靶向载体的同源重组。
19。实施例1、6或11所述的方法,其中Cas蛋白为Cas9。
20。实施例19所述的方法,其中第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点紧密侧接前间区序列邻近基序(PAM)序列。
21。实施例1、6或11所述的方法,其中所述细胞为原核细胞。
22。实施例1、6和11所述的方法,其中所述细胞为真核细胞。
23。实施例22所述的方法,其中所述真核细胞为哺乳动物细胞。
24。实施例23所述的方法,其中所述哺乳动物细胞为成纤维细胞。
25。实施例23所述的方法,其中所述哺乳动物细胞为非人哺乳动物细胞。
26。实施例23所述的方法,其中所述哺乳动物细胞来自啮齿动物。
27。实施例26所述的方法,其中所述啮齿动物为大鼠、小鼠或仓鼠。
28。实施例22所述的方法,其中所述真核细胞为多能细胞。
29。实施例28所述的方法,其中所述多能细胞为造血干细胞或神经元干细胞。
30。实施例28所述的方法,其中多能细胞为人诱导性多能干(iPS)细胞。
31。实施例28所述的方法,其中所述多能细胞为小鼠胚胎干(ES)细胞或大鼠胚胎干(ES)细胞。
32。实施例1、6和11中任一项所述的方法,其中第一gRNA靶位点、第二gRNA靶位点或第三gRNA靶位点位于编码第一选择标记、第二选择标记或第三选择标记的第一核酸、第二核酸或第三核酸中的内含子、外显子、启动子或启动子调控区中。
33。实施例1、6或11所述的方法,其中第一靶向载体、第二靶向载体或第三靶向载体为至少约10kb。
34。实施例1、6或11所述的方法,其中第一插入核酸、第二插入核酸或第三插入核酸在约5kb至约300kb的范围内。
35。实施例1、6或11所述的方法,其中第一插入核酸、第二插入核酸或第三插入核酸包含人T细胞受体α基因座的基因组区域。
36。权利要求35所述的方法,其中所述基因组区域包含人T细胞受体α基因座的至少一个可变区基因区段和/或连接区基因区段。
37。实施例6所述的方法,其中第一选择标记和第三选择标记相同。
38。实施例11所述的方法,其中第一选择标记和第三选择标记相同,并且第二选择标记和第四选择标记相同。
39。实施例38所述的方法,其中第一gRNA和第三gRNA相同。
40。实施例1、6、37、38或39所述的方法,其中所述gRNA对潮霉素或新霉素抗性基因具有特异性。
41。实施例40所述的方法,其中对新霉素抗性基因具有特异性的gRNA由包含SEQID NO:13、14、15或16所示的核苷酸序列的核酸编码。
42。实施例40所述的方法,其中对潮霉素抗性基因具有特异性的gRNA由包含SEQID NO:17、18、19或20所示的核苷酸序列的核酸编码。
43。实施例6、37、38或39所述的方法,其中a)第一gRNA由包含SEQ ID NO:13、14、15或16所示的核苷酸序列的核酸编码,并且第二gRNA由包含SEQ ID NO:17、18、19或20所示的核苷酸序列的核酸编码;或b)第一gRNA由包含SEQ ID NO:17、18、19或20所示的核苷酸序列的核酸编码,并且第二gRNA由包含SEQ ID NO:13、14、15或16所示的核苷酸序列的核酸编码。
以下实例以说明性方式而非限制性方式提供。
图1和图2中所示的顺序基因靶向实验展示了将基于BAC的大靶向载体(LTVEC)与锌指核酸酶(ZFN)组合的价值,所述锌指核酸酶被设计成识别并切割药物选择盒中的靶序列。
对于顺序靶向中的第一步(图1),构建LTVEC以形成将136kb编码人T细胞受体α(TCRα)的11个可变(V)结构域的DNA插入到对应小鼠TCRα基因座中的修饰(TCRαB-hyg等位基因)。通过电穿孔法将0.02mg构建的LTVEC导入1千万个小鼠胚胎干(ES)细胞中,所述细胞在TCRα基因座处携带此前形成的修饰(TCRαA-neo等位基因),所述修饰将小鼠可变(V)基因区段和连接(J)基因区段替换为人Vs和Js。在经电穿孔的ES细胞在生长培养基中恢复之后,添加潮霉素以选择来自已在其基因组中掺入了LTVEC的细胞的集落。对分离的集落进行等位基因修饰(MOA)筛选,使得在所筛选的136个潮霉素抗性集落之中鉴定出四个正确靶向的克隆,靶向效率达2.9%(表1,实验1)。除了11个额外Vs的插入之外,正确靶向的克隆还具有替换新霉素(G418)抗性盒(neo
实验2与实验1相同,不同之处在于添加了各0.02mg的两种质粒,这两种质粒各表达Neo-ZFN(1,2)的每一半,Neo-ZFN(1,2)结合于neor基因中的识别序列并催化DNA中的双链断裂。Neo-ZFN(1,2)的加入使得在所筛选的568个潮霉素抗性克隆之中得到55个正确靶向的克隆,靶向效率达9.7%,这表示靶向效率是采用单独LTVEC进行电穿孔时的3.3倍(表1,比较实验1和2)。
实验3与实验2相同,不同之处在于编码Neo-ZFN(3,4)的质粒替换了编码Neo-ZFN(1,2)的质粒。Neo-ZFN(3,4)的加入使得在所筛选的360个潮霉素抗性克隆之中得到42个正确靶向的克隆,靶向效率达11.7%,这表示靶向效率是采用单独LTVEC进行电穿孔时的4倍(表1,比较实验1和3)。
对于顺序靶向中的第二步(图2),0.002mg LTVEC经电穿孔进入1千万个小鼠胚胎干(ES)细胞中而将LTVEC引入ES细胞中,所述LTVEC被设计为形成插入157kb编码11个额外人TCRα可变(V)结构域(与TCRαA-neo或B-hyg等位基因中的那些不同)的DNA的修饰(TCRαC-neo等位基因),所述小鼠胚胎干(ES)细胞携带在顺序靶向的第一步(图1)中形成的TCRαB-hyg等位基因。在经电穿孔的ES细胞在生长培养基中恢复之后,添加G418以选择来自已在其基因组中掺入了LTVEC的细胞的集落。对分离的集落进行MOA筛选,使得在所筛选的192个G418抗性集落之中鉴定出两个正确靶向的克隆,靶向效率达1.0%(表1,实验4)。除了11个额外Vs的插入之外,正确靶向的克隆还具有替换hygr盒的neor盒。
实验5与实验4相同,不同之处在于添加了各0.02mg的两种质粒,这两种质粒各表达Hyg-ZFN(1,2)的每一半,Hyg-ZFN(1,2)结合于hygr中的识别序列并催化DNA中的双链断裂。Hyg-ZFN(1,2)的加入使得在所筛选的192个G418抗性克隆之中得到40个正确靶向的克隆,靶向效率达21%,这表示靶向效率是采用单独LTVEC进行电穿孔时的21倍(表1,比较实验4和5)。
实验6与实验5相同,不同之处在于编码Hyg-ZFN(3,4)的质粒替换了编码Hyg-ZFN(1,2)的质粒。Hyg-ZFN(3,4)的加入使得在所筛选的192个潮霉素抗性克隆之中得到42个正确靶向的克隆,靶向效率达22%,这表示靶向效率是采用单独LTVEC进行电穿孔时的22倍(表1,比较实验4和6)。
表1中汇总的实验确认与仅加入LTVEC的靶向实验相比,在顺序靶向实验中随同LTVEC一起加入靶向neo
本说明书中提到的所有出版物和专利申请都表示本发明所属领域的技术人员的水平。所有出版物和专利申请均以引用的方式并入本文,其程度就如同每篇单独的出版物或专利申请被具体且单独地指明以引用的方式并入。除非从任何实施例的上下文可以显见,否则本发明的方面、步骤或特征可与任何其他方面、步骤或特征组合使用。对范围的提及包括该范围内的任何整数、该范围内的任何子范围。对多个范围的提及包括此类范围的组合。
序列表
<110> 瑞泽恩制药公司
沃基特克·奥尔巴赫
大卫·弗伦杜威
古斯塔沃·德罗格特
安东尼·加戈利亚地
琼科·库诺
大卫·M.·巴伦苏埃拉
<120> 用于修饰所靶向基因座的方法和组合物
<130> 057766-461003
<150> US 62/008,832
<151> 2014-06-06
<150> US 62/017,916
<151> 2014-06-27
<160> 21
<170> 适用于Windows 4.0版的FastSEQ
<210> 1
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 连接至向导RNA (gRNA)的
靶基因座
<220>
<221> misc_feature
<222> 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18,
19, 20, 21
<223> n = A、T、C或G
<400> 1
gnnnnnnnnn nnnnnnnnnn ngg 23
<210> 2
<211> 80
<212> RNA
<213> 人工序列
<220>
<223> 向导RNA (gRNA)
<400> 2
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu 60
ggcaccgagu cggugcuuuu 80
<210> 3
<211> 42
<212> RNA
<213> 人工序列
<220>
<223> 向导RNA (gRNA)
<400> 3
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cg 42
<210> 4
<211> 30
<212> RNA
<213> 人工序列
<220>
<223> crRNA
<400> 4
guuuuagagc uagaaauagc aaguuaaaau 30
<210> 5
<211> 33
<212> RNA
<213> 人工序列
<220>
<223> crRNA
<400> 5
guuuuagagc uagaaauagc aaguuaaaau aag 33
<210> 6
<211> 26
<212> RNA
<213> 人工序列
<220>
<223> crRNA
<400> 6
gaguccgagc agaagaagaa guuuua 26
<210> 7
<211> 12
<212> RNA
<213> 人工序列
<220>
<223> tracrRNA
<400> 7
aaggcuaguc cg 12
<210> 8
<211> 50
<212> RNA
<213> 人工序列
<220>
<223> tracrRNA
<400> 8
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugcuuuu 50
<210> 9
<211> 40
<212> DNA
<213> 人工序列
<220>
<223> Neo-ZFN(1,2):核酸酶结合位点/切割位点
<400> 9
gggcgcccgg ttctttttgt caagaccgac ctgtccggtg 40
<210> 10
<211> 36
<212> DNA
<213> 人工序列
<220>
<223> ZFN(3,4):核酸酶结合位点/切割位点
<400> 10
ccggttcttt ttgtcaagac cgacctgtcc ggtgcc 36
<210> 11
<211> 42
<212> DNA
<213> 人工序列
<220>
<223> ZFN(1,2):核酸酶结合位点/切割位点
<400> 11
tgcgatcgct gcggccgatc ttagccagac gagcgggttc gg 42
<210> 12
<211> 36
<212> DNA
<213> 人工序列
<220>
<223> Hyg-ZFN(3,4):核酸酶结合位点/切割位点
<400> 12
cgctgcggcc gatcttagcc agacgagcgg gttcgg 36
<210> 13
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Neo Crispr#1
<400> 13
ugcgcaagga acgcccgucg 20
<210> 14
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Neo Crispr#2
<400> 14
ggcagcgcgg cuaucguggc 20
<210> 15
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Neo Crispr#3
<400> 15
acgaggcagc gcggcuaucg 20
<210> 16
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Neo Crispr#4
<400> 16
gcucugaugc cgccguguuc 20
<210> 17
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Hyg Crispr#1
<400> 17
acgagcgggu ucggcccauu 20
<210> 18
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Hyg Crispr#2
<400> 18
cuuagccaga cgagcggguu 20
<210> 19
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Hyg Crispr#3
<400> 19
gccgaucuua gccagacgag 20
<210> 20
<211> 20
<212> RNA
<213> 人工序列
<220>
<223> gRNA Hyg Crispr#4
<400> 20
cgaccugaug cagcucucgg 20
<210> 21
<211> 25
<212> DNA
<213> 人工序列
<220>
<223>连接至向导RNA(gRNA)的目标位点
<220>
<221> misc_feature
<222> (3)...(23)
<223> n = A, T, C, or G
<400> 21
ggnnnnnnnn nnnnnnnnnn nnngg 25
- 用于修饰所靶向基因座的方法和组合物
- 用于修饰所靶向基因座的方法和组合物
