用于直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒及使用方法
文献发布时间:2023-06-19 19:28:50
技术领域
本发明涉及DNA化学修饰检测技术领域,特别涉及一种用于直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒及使用方法。
背景技术
BCR-ABL1融合基因是由t(9:22)(q34:11)易位形成,见于90%以上CML、25~30%成人ALL、3%~5%儿童ALL以及少数AML患者,其表达的融合产物具有组成性酪氨酸激酶活性,从而导致不受控的细胞增殖。针对BCR-ABL融合蛋白的致癌机制开发的分子靶向药物酪氨酸激酶抑制剂(一代TKIs伊马替尼)极大地改善了BCR-ABL1阳性患者的治疗和预后,并使得BCR-ABL1融合阳性的CML和ALL成为了可控的慢性疾病。但在治疗过程中,一些患者仍然表现出对TKIs治疗的耐药,其中BCR-ABL1依赖性的耐药机制最为多见,包括ABL1激酶结构域(KD)突变、BCR-ABL1过表达和BCR-ABL1分子上的表观遗传修饰改变。尽管KD突变和BCR-ABL过表达是被研究最多的耐药机制,并直接引导了后续二代(尼洛替尼、达沙替尼、博苏替尼)和三代(博纳替尼)TKIs的研发和应用,但仍有许多病人对二代或三代TKIs治疗不敏感。因此研究其他的耐药机制对于开发新的治疗策略意义重大,例如目前正在研究针对BCR-ABL1启动子甲基化异常的表观遗传修饰改变的耐药患者是否可以考虑使用去甲基化药物(地西他滨)等新的治疗策略。
DNA甲基化是DNA表观遗传学领域最重要的研究内容之一,其中在哺乳动物基因组中丰度最高、亦是被研究最为成熟的修饰类型是DNA胞嘧啶第五位碳原子上的甲基化,即5-甲基胞嘧啶(5-methylcytosine,5mC)修饰。5mC在人类基因组中约占1%,主要发生在基因组中分布不均匀的CpG(C-胞嘧啶、p-磷酸、G-鸟嘌呤)二核苷酸位点中,尤其富含CpG二核苷酸的基因启动子区域的一段DNA片段(CpG岛)。已有大量研究表明,DNA上的5mC修饰是一种抑制基因转录的稳定表观遗传标记物,其水平的维持和改变在个体发育、基因表达调控、机体衰老等过程中发挥重要功能,而其异常改变则与多种疾病,特别是癌症的发生、发展密切相关。在癌组织中,DNA甲基化的异常模式普遍是基因组大范围低甲基化和某些抑癌基因的启动子等局部区域的CpG岛(正常组织中呈现非甲基化状态)的高甲基化,例如异常的低甲基化会引起致癌基因的过度表达,而高甲基化则会导致肿瘤抑制基因的失活,即基因沉默。BCR-ABL1融合基因作为多种白血病发生的驱动因素,其表达主要受BCR基因启动子的调控,因为有研究表明ABL1基因的一个保守的启动子Pa虽然也存在于BCR-ABL1融合基因当中,但是该启动子的高甲基化水平与相关的疾病状态无关。此外,一项关于CML患者的BCR基因启动子甲基化水平与伊马替尼疗效相关性研究还表明,BCR启动子DNA甲基化增加表明患者预后良好,且对伊马替尼的反应性更好。但目前针对于BCR-ABL1启动子甲基化异常是否影响BCR-BAL1基因表达和因BCR-ABL1启动子甲基化异常而耐药的患者是否对去甲基化药物敏感的大数据研究并不多,其中很大的一个因素在于目前传统的5mC检测技术不能同时兼顾检测技术的简便性、特异性、通量和数据分析的简易性。
5mC的检测过程大致可分为样本的预处理和测序这两步。预处理的方式主要包括酶识别法、化学转化法和抗体富集法,其中基于亚硫酸氢盐的化学转化法是最可靠且应用最广泛的样本处理方式。基于亚硫酸氢盐转化的测序技术的基本原理是利用化学试剂亚硫酸氢盐将未甲基化的胞嘧啶(C)转化为尿嘧啶(U),但不改变5mC和其他DNA碱基,由此将5mC和C区分开,最后再通过第一代或二代测序技术即可得到的DNA甲基化修饰图谱。但亚硫酸氢盐的处理过程繁杂,会降解近半数以上的DNA分子,这会造成DNA序列复杂性严重降低,又会增加目的DNA片段扩增的难度。此外,该处理是将占人类基因组95%的未甲基化C转变为了U,因此对DNA序列的碱基平衡影响极大,会导致后续测序数据质量不高。测序技术中,相较于传统的Sanger测序技术(一代),以Illumina测序平台为代表的第二代测序技术(NGS)虽然在测序通量和成本上有较大优势,但因其依赖多轮聚合酶链反应(PCR)对DNA分子进行扩增,这不仅带来了部分修饰信息丢失、碱基错配等问题,而且NGS测序片段较短(约50bp~500bp),这使得测序数据比对和拼接的难度极大。
近些年来,测序市场新兴的第三代测序技术代表之一的纳米孔测序技术是基于核酸分子通过测序芯片上的纳米孔时产生的电流信号差异来识别序列信息,具有对原始DNA样本文库进行直接测序的特性。因此,该技术在原理上使得其能够在DNA不进行任何预处理的情况下在单碱基分辨率水平上直接识别出碱基序列和碱基修饰,且还具有测序通量高、超长读长、操作简便、耗时较少等优点。此外,基于纳米孔基因测序技术的直接靶向测序技术‘nanopore Cas9Targeted-Sequencing’(nCATS,Cas9靶向富集的纳米孔基因测序技术)实现了对单个样本中一个或多个感兴趣基因区域(ROI)的深度测序,这不仅能够矫正测序错误,提高检测度的精确性,并能有效减少其他无关背景基因造成的噪声干扰,减轻了数据分析的难度。但目前该技术并没有开发出用于多个样本中的同一ROI区域的检测方法。
发明内容
基于此,本发明的目的是提供一种用于直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒及使用方法,以至少解决上述相关技术中的不足。
本发明提出一种用于直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒,包括:
源自CRISPR的crRNA探针,所述crRNA探针靶位点位于包含BCR基因启动子在内的CpG岛上游;
反式激活crRNA,所述反式crRNA序列保守,其5’端的序列与crRNA探针3’端序列互补配对,可退火为嵌合的单链向导RNA;
以及,CRISPR相关蛋白Cas9,所述Cas9蛋白具有核酸内切酶活性,与嵌合的单链向导RNA组装成核糖核蛋白复合体RNPs,并在所述嵌合的单链向导RNA的引导下对基因组双链DNA中的所述crRNA靶位点进行切割,使双链DNA断裂;
通过以上试剂构成CRISPR/Cas9靶向富集系统。
进一步的,所述crRNA探针的5’端与靶位点序列互补配对的长度为20个核苷酸,且与所述crRNA探针靶位点相邻的3’端序列为PAM基序,所述crRNA探针序列标识号、碱基序列和相应靶位点的PAM基序如下所示:
BCR_23177704crRNA:5’-CAAGGGAGAAAGCCACTATC-3’;5’-TGG-3’。
进一步的,所述BCR_23177704crRNA探针在每个基因组双链DNA样本中对靶位点的靶向效率可通过实时荧光定量聚合酶链反应实验进行质控。
进一步的,所述实时荧光定量聚合酶链反应实验质控BCR-23177704crRNA探针的靶向效率所用的引物对为BCR_23177704FP/RP,所述引物对序列如下所示:
BCR_23177704FP:5’-CCAACCCAACCCTCCAGAA-3’;
BCR_23177704RP:5’-GTCACAGGTCAGACAACTAAGCA-3’。
进一步的,所述实时荧光定量聚合酶链反应实验的反应程序参数为:95℃预变性2min;95℃变性15sec,55℃退火15sec,72℃延伸20sec,40个循环。
进一步的,所述试剂盒还包括样本基因组DNA提取试剂、基因组DNA去磷酸化试剂、dATP试剂、Taq DNA聚合酶、T4连接酶、Blunt T/A连接酶、AMPure XP磁珠、SPRIselect片段选择磁珠、Barcode标签序列试剂、纳米孔基因测序文库构建试剂和纳米孔基因测序芯片激发试剂。
本发明还提出一种试剂盒的使用方法,应用于上述的用于直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒,所述试剂盒的使用方法包括:
步骤一:提取样本中的基因组DNA;
步骤二:对提取的所述基因组DNA样本进行去磷酸化处理;
步骤三:通过所述CRISPR/Cas9靶向富集系统从已经步骤二处理过的基因组DNA中靶向富集到目的DNA片段;
步骤四:使用BCR_23177704FP/RP引物对,通过实时荧光定量聚合酶链反应实验对BCR_23177704crRNA的靶向效率进行质控,对于质控过关的样本用AMPure XP磁珠进行纯化;
步骤五:添加Barcode标签序列于靶向富集的目的DNA片段上,之后用SPRIselect片段分选磁珠纯化样本,并将纯化后的样本混合到一起;
步骤六:连接纳米孔基因测序接头于barcode标签序列标记好的混和样品后,并用AMPure XP进行纯化,即为制备好的多样本靶向目的DNA测序文库;
步骤七:使用纳米孔基因测序技术对多样本靶向目的DNA测序文库进行测序,并获得靶向测序数据。
进一步的,所述样本选自人外周血样本或骨髓样本、以及人白血病相关细胞系。
本发明的有益效果在于:
本发明提供的用于检测BCR-ABL1融合基因启动子甲基化的试剂盒,首先,所用的BCR_23177704crRNA探针为正向的crRNA探针(该crRNA序列5’到3’的方向与靶向基因区域正链的5’到3’的方向一致,并与靶向该正链区域20bp的碱基序列完全一致,因此称为该类crRNA探针为正向靶向的crRNA探针,反之则称为反向的crRNA探针),且靶向的位点处于包含BCR-ABL1融合基因启动子在内的CpG岛的上游(如图1所示);其次,组装后的RNPs切割相应的靶位点,造成双链DNA断裂,并产生两个平末端断端,但由于位于正向BCR_23177704crRNA探针5’侧的DNA片段断端仍附着有Cas9蛋白,所以Barcode标签序列优先连接到位于BCR_23177704crRNA探针3’侧的DNA片段断端(如图2所示),而添加有Barcode标签序列的ROI片段则在通过与纳米孔基因测序接头连接后直接进行测序。上述两个原因确保了纳米孔基因测序的方向为从切割位点的5’到3’(正链),并使得后续测序范围能够完全覆盖包含融合基因BCR-ABL1融合基因启动子在内的CpG岛(本发明的ROI)。
进一步的,引物对BCR_23177704FP/RP用于对每个样本中BCR_23177704crRNA探针靶位点的靶向效率进行质控(如图3所示),可确保每个样本中对本发明的ROI的富集是有效的,以及后续靶向纳米孔基因测序技术的成功实施。
进一步的,本发明结合CRISPR/Cas9靶向富集系统和纳米孔基因测序技术,所得的测序数据具有目的基因片段的测序深度高、读长长、单碱基分辨率、特异性高等特点;其中测序深度高可确保本发明的ROI测序结果的准确性和可靠性;读长长和单碱基分辨率使得本发明在检测范围和分辨率上均展现出了优越性,表现为本发明能准确评估长度约为2kb的ROI中单碱基水平上的每个CG基序的甲基化状态。
进一步的,本发明是基于能直接对天然DNA进行测序的纳米孔基因测序技术对本发明的ROI进行测序,而无需亚硫酸氢盐处理DNA样本和后续PCR扩增,这不仅在实验过程中极大减少了DNA降解和PCR扩增带来的偏倚和修饰信息的丢失,还极大简化了实验方法和节省了大量时间。
进一步的,本发明引入了Barcode(一段寡核苷酸序列)标签序列试剂,通过在不同样本的ROI上接上特定的Barcode标签序列,用以在后续数据分析时将不同样本的数据分开。因此本发明实现了对多个样本中同一ROI的靶向测序,能极大减少测序成本。
进一步的,样本量的多少取决于测序芯片的DNA测序文库荷载量和测序仪器搭载芯片的数量;其中一张标准FLO-MIN106测序芯片最多可一次性检测6个gDNA样本,而上述芯片可在分别搭载1张芯片和5张芯片的MinION测序仪和GridION测序仪上进行测序;即一次平行测序实验最大可同时满足30个gDNA样本的本发明ROI区域的甲基化检测。
本发明的附加方面和优点将在下面的描述中部分给出,部分将从下面的描述中变得明显,或通过本发明的实践了解到。
附图说明
图1为BCR-ABL1融合基因启动子和CpG岛以及BCR_23177704crRNA探针靶向位点的结构示意图;
图2为CRISPR/Cas9靶向富集系统对靶向位点进行切割的原理示意图;
图3为实时荧光定量聚合酶链反应(qPCR)实验检测crRNA探针的靶向效率的原理示意图;
图4为基于CRISPR/Cas9靶向富集系统的纳米孔基因测序技术对多个样本中ROI进行靶向测序的原理示意图;
图5为甲基化的数据分析流程示意图;
图6为本发明实施例1中K562细胞系gDNA的纯度和片段大小指控图;
图7为本发明实施例1的6个相同的gDNA样本在所测ROI的每个CG基序中发生5mC修饰的百分数占比的散点图;
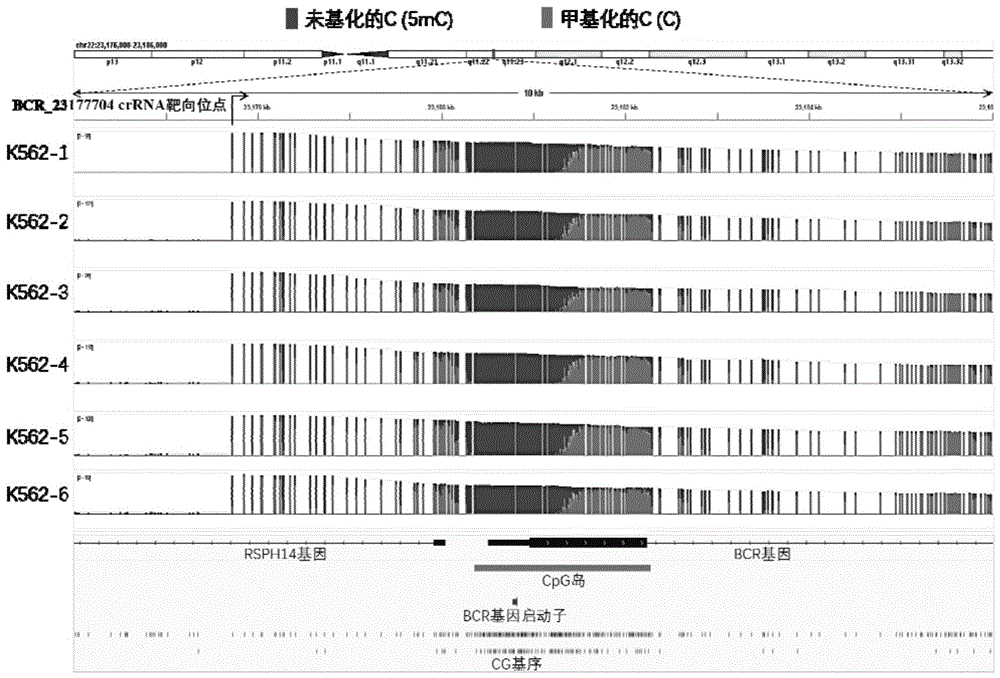
图8为本发明实施例1的6个相同的gDNA样本在所测ROI的5mC测序数据的IGV可视化结果图;
图9为本发明实施例2中6个血液或骨髓gDNA样本的纯度和片段大小指控图;
图10为本发明实施例2的6个血液或骨髓gDNA样本在所测ROI的每个CG基序中发生5mC修饰的百分数占比散点图;
图11为本发明实施例2的6个血液或骨髓gDNA样本在所测ROI的5mC测序数据的IGV可视化结果图。
具体实施方式
为了能够更加详尽地了解本发明的特点与技术内容,下面对本发明的实现进行详细阐述。以下实施例中所描述的实验流程仅用于证明本发明的可行性,本发明的应用并不仅限于此。实施例中所提及的实验操作,如无特殊说明,均为常规实验方法;所提及的试剂耗材,如无特殊说明,均为常规试剂耗材。
除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。本文所使用的术语“及/或”包括一个或多个相关的所列项目的任意的和所有的组合。
本实施例中CRISPR/Cas9靶向富集系统所用试剂购自IDT公司,纳米孔基因测序试剂和测序仪器购自ONT公司,其余所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
下面对本发明实施例的一种基于CRISPR/Cas9靶向富集系统的纳米孔基因测序技术用于直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒及使用方法进行具体、详细地说明。
实施例1:
检测6个相同的K562细胞系gDNA中BCR-ABL1融合基因启动子甲基化状态
本实施例设计了1条crRNA探针(靶向包含BCR-ABL1融合基因启动子在内的CpG岛基因片段)以及所述探针靶向效率质控所用的1对qPCR引物(见表1),进而通过基因组DNA去磷酸化、CRISPR/Cas9靶向富集系统富集目的基因片段和靶向效率质控、多个gDNA样本(2~6)的ROI测序文库构建和纳米孔基因测序,最后通过数据分析实现在单碱基分辨水平上准确评估多个样本中ROI的甲基化状态,完整的实验流程和数据分析分别见图4和图5所示。本实施例1使用6个相同的K562细胞系gDNA作为研究对象,以说明本试剂盒检测BCR-ABL1融合基因启动子5mC甲基化状态检测结果的可重复性,以及5mC修饰检测结果在单碱基分辨率水平上的高准确性。
表1
(1)人慢性粒细胞白血病细胞系(K562)培养及其基因组DNA(gDNA)的提取
K562细胞悬浮生长于含10%胎牛血清、100U/m青霉素和100μg/ml链霉素的RPMI1640培养基中,并在37℃、5% CO2及湿度饱和的培养箱中进行培养。当K562细胞处于对数生长期时,将其移入离心管并进行细胞计数,随后800rpm离心5min,弃上清。随后按照gDNA提取试剂盒(QIAGEN公司,QIAamp DNA Blood Mini Kit)说明书操作提取K562细胞系gDNA:用200μL的PBS重悬细胞沉淀(细胞个数<5×10^6,为该gDNA提取试剂盒的最大起始细胞数量),加入20μL的蛋白酶K和4μL的RNA A酶后混匀;加入200μL裂解液AL并充分涡旋混匀,56℃裂解细胞10min;然后加入200μL无水乙醇,涡旋点震15sec,将反应液转移至过滤柱中,8000rpm离心1min;加500μL缓冲液AW1,8000rpm离心1min;再加500μL缓冲液AW2,14000rpm离心3min;将过滤柱置于新的收集管上,加入50μL~200μL的洗脱液AE,室温静置3min,8000rpm离心1min,收集洗脱的gDNA并储存在-80℃冰箱。在gDNA使用时,用荧光定量仪Qubit测定其浓度,并用全自动毛细管分析仪Qsep100分析其片段大小以及纯度。本实施例1所用的K562细胞系的gDNA用Qubit所测的浓度为216ng/μL,用Qsep100所测的片段大小以及纯度见图6所示,该K562细胞系gDNA的片段主要分布在7kb左右,且20bp~1000bp内的杂峰很少,说明所提K562细胞系gDNA的长度和纯度均可。
(2)BCR-ABL1融合基因启动子和包含其在内的CpG岛的基因组位置关系分析
BCR-ABL1融合基因的表达主要受BCR基因启动子的调控。BCR基因位于第22号染色体长臂的3区4带,其中包含启动子在内的CpG岛片段位于chr22:23180364-23182278(参考基因组版本GRCh38.p13),总长度为1915bp,共有212个CG基序(见图1),其中启动子位于chr22:23180778-23180837。上述区域即为本发明的ROI,并且位于BCR基因的3个断裂位点区的上游,包括长约7kb的主要断裂区(M-bcr),次要断裂区(m-bcr)和第3断裂区(μ-bcr)(见图1)。而ABL1基因可能发生断裂的区域为长约140kb的第1和第2内含子区域内(见图1)。
(3)用于本发明ROI的crRNA探针及该探针靶向效率质控的qPCR引物的设计与合成
本发明基于crRNA探针的设计原则,应用在线设计软件CHOPCHOP在本发明的ROI位置上游约1kb以外的5kb基因区域(在GRCh38.p13参考基因组序列上的位置为chr22:23174364-23179364)设计2条备选的正向靶向切割位点的crRNA探针,以便后续测序方向是沿着BCR基因正链的5’到3’,因此能确保长度长的纳米孔基因测序范围能完全将本发明的ROI覆盖在内。上述设计好的2条备选的正向靶向的crRNA探针交由IDT公司合成,以及组装成RNPs所需的反式激活crRNA(tracrRNA)和Cas9蛋白也均购自IDT公司
2条备选的crRNA探针在每个样本中对靶位点的靶向效率可用qPCR实验进行质控,原理为针对crRNA探针的靶位点设计出扩增产物包括该靶向位点在内的qPCR引物,则包含crRNA探针靶位点在内的基因片段经相应RNPs切割后不能作为模板完成扩增过程。因此,将RNPs切割后的gDNA作为模板的实验组与用未经RNPs切割的gDNA作为模板的对照组相比,实验组的CT值比对照组高,通过计算ΔCT值(实验组-对照组)即可判断出对应crRNA的靶向效率(如图3所示)。所以,针对上述备选的2条crRNA探针的靶位点,用Primer Premier 5软件各设计2对相应的qPCR引物(共4对),并用K562细胞系的gDNA为实验样本,验证各对qPCR引物的特异性和扩增效率,最终选出特异性好、扩增效率高的2对分别用于质控备选的2条crRNA探针靶向效率的qPCR引物。
筛选好的2对qPCR引物即用于对相应2条备选的crRNA探针靶向效率进行验证,此过程可通过简单的体外切割实验和qPCR实验完成。以1条crRNA探针的靶向效率验证实验为例简述验证方法:首先,1μL crRNA探针(20μM)、1μL tracrRNA(20μM)和8μL无酶duplex缓冲液(crRNA和tracrRNA配套的试剂),95℃加热5min后退火为10μL gRNA(2μM),5μL后者与0.8μL Cas9蛋白(6.2μM)、1μL 10×CutSmart缓冲液(NEB公司)以及3.2μL无酶水在室温下孵育30min后组装成10μL RNPs(crRNA终浓度为1μM)(剩余的5μL gRNA可保存在4℃1天);然后,在含有1×CutSmart缓冲液、10μL RNPs的42μL的反应体系中对5μg的K562细胞系gDNA进行简单的体外切割,反应条件为37℃孵育30min;最后,用RNPs切割后和未经RNPs切割的K562细胞系gDNA(用量为15~20ng)分别作为qPCR实验组和对照组的模板,所用引物为筛选好的相应crRNA探针靶向效率质控引物。最终筛选出靶向效率高的1条crRNA探针,为BCR_23177704crRNA(见图1)。所述BCR_23177704crRNA探针20bp长度的序列及相应靶位点的PAM基序如下所示:
BCR_23177704crRNA:5’-CAAGGGAGAAAGCCACTATC-3’;5’-TGG-3’;
以及用于对上述BCR_23177704crRNA探针靶向效率质控的qPCR引物为BCR_23177704FP/RP,引物序列如下所示:
BCR_23177704FP:5’-CCAACCCAACCCTCCAGAA-3’;
BCR_23177704RP:5’-GTCACAGGTCAGACAACTAAGCA-3’;
通过前期靶向测序预实验得出BCR_23177704crRNA探针靶向效率质控合格的样本为:靶向本发明ROI上游的BCR_23177704rRNA探针的qPCR质控结果一定要满足ΔCT≥2,否侧无法保证本发明ROI的靶向测序深度足够用于后续的甲基化数据分析。
(4)组装RNPs和gDNA去磷酸化
①组装RNPs:首先是合成3个10μL gRNA(crRNA终浓度为2μM):3μL BCR-23177704crRNA(20μM)和3μL tracrRNA(20μM),并用24μL无酶duplex缓冲液补充至30μL,95℃加热5min后冷却至4℃;其次是组装6个单独的10μL RNPs(crRNA终浓度为1μM):5μLgRNA、0.8μL Cas9(6.2μM),1μL 10×CutSmart缓冲液和3.2μL无酶水,室温下孵育30min后转至冰上备用,如有多余的RNPs可放置-80℃保存1个月;在RNP孵育期间,继续进行下一步的gDNA去磷酸化。
②gDNA去磷酸化:所需gDNA起始输入量为3~10μg,建议用5μg;单独制备6个30μL的去磷酸化反应体系(6个相同的K562细胞系gDNA分别编号为K562-1,K562-2,K562-3,K562-4,K562-5,K562-6):23μL K562细胞系的gDNA(216ng/μL×23μL≈5μg)、3μL 10×CutSmart缓冲液、3μL快速磷酸酶(NEB公司)和1μL无酶水,37℃反应15min进行gDNA的5’端去磷酸化,然后在80℃下加热2min使快速磷酸酶失活。
此双链gDNA末端中的5’磷酸基团已去除,即使末端中的3’羟基基团可在后续的Taq DNA聚合酶的作用下加上一个脱氧核糖腺苷酸(dATP),但鉴于5’端已去磷酸化,因此基因组双链DNA末端仍不能通过A/T互补配对的方式接上含有T残基末端的Barcode标签序列。此处理有效避免了大量非靶向的基因组背景基因连接上Barcode标签序列,极大地减少了无效测序数据的产出,并减轻了后续数据分析的负担。
(5)RNPs切割BCR_23177704crRNA探针靶位点以及新生断裂末端的制备
将去磷酸化处理的30μL K562细胞系gDNA平衡至室温后,并向其中加入步骤2组装好的10μL RNPs;此外,还需加入1μL 10mM dATP(NEB公司)和1μL Taq DNA聚合酶(NEB公司)。然后,将42μL的反应液在37℃下孵育30min让RNPs切割双链gDNA中相应的crRNA探针靶位点,并让Taq DNA聚合酶在双链gDNA的3’末端加上一个dATP,形成dA尾;最后,在72℃下反应5min使Cas9蛋白失活后,将样本转移至冰上保存。
在上述反应中,RNPs切割双链DNA后所产生的两个DNA片段断端为平末端,但位于crRNA探针5’侧的片段断端因与crRNA探针形成RNA-DNA杂合体,所以附着有RNPs中的Cas9蛋白而未暴露,而位于crRNA探针3’侧的片段断端侧成暴露状态。所以,在后续加dA尾的反应中,RNPs切割后位于crRNA探针5’侧且断端处附着有Cas9的基因片段因无法通过Taq DNA聚合酶的作用加上一个dATP,而不能通过A/T互补配对的方式在加Barcode标签序列的反应中接上含有T残基末端的Barcode标签序列;而位于crRNA探针3’侧的DNA片段因断端裸露则能在加Barcode标签序列的反应中优先连接到Barcode标签序列。此过程实现了对本发明的ROI的富集,即对包含BCR-ABL1融合基因启动子在内的CpG岛基因进行了富集。
(6)qPCR实验质控6样本中BCR_23177704crRNA探针的靶向效率
经RNPs切割后及新生断裂末端制备完成的6个42μL的样品中gDNA的浓度为:(216μg/μL×23μL)÷42μL=118ng/μL。分别取1μL上述6个gDNA样品并稀释10倍至11.8ng/μL后作为实验组模板(RNPs切割后),剩余的6个41μL的gDNA样品放置冰上保存备用;再从原始K562细胞系gDNA样本中取1μL并稀释至11.8ng/μL后作为对照组模板(RNPs切割前)。qPCR反应体系为20μL,每组做2个重复,其中模板1.5μL(10ng~20ng)、10μL的2×SYBR Green荧光染料、2μL的2μM上游及下游引物和4.5μL无酶水,之后根据ΔCT值(实验组CT-对照组CT值)质控6个K562细胞系gDNA样本中BCR_23177704crRNA探针的靶向效率。结果见表2所示,本实施例1中6个K562细胞系gDNA样本中BCR_23177704crRNA探针的靶向效率均合格(ΔCT>2),可进行后续的测序实验。
表2
(7)对靶向效率质控合格的样本进行纯化:
用1×的AMPure XP磁珠(贝克曼公司,4℃储存)纯化末端制备以及靶向效率质控指控合格的gDNA,具体操作为:提前将AMPure XP磁珠平衡至室温,涡旋振荡以充分混匀;将42μL gDNA(1μL用于qPCR质控的gDNA样本可忽略不计)样本从PCR管中转移至低吸附的1.5mL EP管并加入等体积的磁珠(42μL),轻弹混匀后室温静置孵育5min;结束后短暂离心并将EP管放置在磁力上,待液体变澄清后吸去上清并沿管壁加入200μL新鲜配置的70%的酒精,保持试管在磁力架上并转动试管两圈,待磁珠再次成团后吸去清夜,重复该清洗步骤一次;取下EP管短暂离心后放回磁力架上,吸去残余液体,自然风干约1~2min(勿让聚集的磁珠干裂),然后加入25μL无酶水润洗磁珠,并轻弹试管重悬磁珠;室温放置10min,短暂离心后放回磁力架上,待溶液变澄清,吸取23~24μL转移至新的低吸附的1.5mL EP管内,即为ROI富集效率质控合格且末端制备好的纯化gDNA洗脱液;最后,取1μL上述液体用Qubit荧光定量仪进行定量,其中回收率大于40%的gDNA样本可继续进行后续实验。本实施例1在此步的6个K562细胞系gDNA样本定量的具体结果见表3,结果显示该6个gDNA样本回收率均满足要求,可继续进行后续实验。
表3
*用于下一步实验的样本量=纯化后回收的质量-用于Qubit浓度检测的1μL样本质量
(8)构建靶向富集目的片段的测序文库
选用ONT公司的Barcode标签序列试剂盒(EXP-NBD104或EXP-NBD114)和连接法测序建库试剂盒(SQK-LSK109)构建多样本靶向富集ROI的测序文库,具体操作如下:
①连接Barcode标签序列并进行纯化:
在上述纯化好的23~24μL gDNA样本中依次加入试剂:2.5μL的Barcode标签序列试剂(EXP-NBD104或EXP-NBD114,这两种试剂盒均有12管不同的Barcode标签序列试剂,序号分别为NB01~12和NB13~24;每个gDNA样本加入不同的Barcode标签试剂)、25μL的Blunt/TA连接酶混合液(NEB公司),总共约为50μL的Barcode标签序列连接反应混合液;轻弹EP管混匀上述反应液后短暂离心,并放置在室温下静置孵育10min。6个相同的K562细胞系gDNA样本所用的Barcode标签序列为:K561-1+NB07,K561-2+NB08,K561-3+NB09,K561-4+NB10,K561-5+NB11,K561-6+NB12。
在上述Barcode标签序列添加完成后的6管反应液中分别加入涡旋振荡后混合均匀的0.4倍(20μL)的SPRIselect磁珠(贝克曼公司,室温储存),以纯化gDNA样本并去除1kb以下的短gDNA片段,具体操作如下:轻弹混匀上述反应液后室温静置孵育5min;结束后短暂离心并将EP管放置在磁力上,待液体变澄清后吸去上清并沿管壁加入200μL新鲜配置的85%的酒精,保持试管在磁力架上并转动试管两圈,待磁珠再次成团后吸去清夜;取下EP管短暂离心后放回磁力架上,吸去残余液体,自然风干约1~2min(勿让聚集的磁珠干裂),然后加入14μL无酶水润洗磁珠(此处加的无酶水的体积取决于一张测序芯片最多承载的gDNA样本的个数为6个,以及后续用的连接纳米孔基因测序接头的混合gDNA样本的总体积为65μL;则平均到本实施例1的6个样本的体积约为11μL,考虑到纯化回收过程中的损失1~2μL和用于Qubit荧光定量的1μL,所以最终加的无酶水的体积为14μL),并轻弹试管重悬磁珠;室温放置10min,短暂离心后放回磁力架上,待溶液变澄清,吸取12~13μL转移至新的PCR管内,即为Barcode标签序列连接好的纯化gDNA洗脱液;最后,取1μL上述液体用Qubit荧光定量仪进行定量,其中用于下一步实验的样本质量大于500ng的gDNA样本可继续进行后续实验。本实施例1在此步的6个K562细胞系gDNA样本定量的具体结果见表4,结果显示该六个gDNA样本回收量均满足要求,可继续进行后续实验。
表4
*用于下一步实验的样本量=纯化后回收的质量-用于Qubit浓度检测的1μL样本质量
取等质量(Qubit定量浓度×取样体积)的已经连接Barcode标签序列的gDNA样本混合到新的低吸附的1.5mL EP管中,总gDNA样本质量约为~5μg(平均每个样本约800~1000ng),总体积为65μL,以确保有足够多的混合gDNA样本(≥5μg)用于后续纳米孔基因测序接头的连接。
②连接纳米孔基因测序接头并进行纯化:
首先制备35μL的纳米孔基因测序接头混合液,包括10μL的快速T4 DNA连接酶(NEB)、与上述酶配套的5×快速连接反应缓冲液、和5μL纳米孔基因测序接头AMX II(最后加入;注意该测序接头来自EXP-NBD104或EXP-NBD114试剂盒,不是SQK-LSK109试剂盒中的AMX测序接头);然后,将上述测序接头混合液分两次加入65μL的混合gDNA样品中,第一次20μL,第二次加入剩余的15μL;最后,轻弹混匀总体为100μL的测序接头连接反应液,并置于室温下孵育10min。
在上述测序接头添加完成后的100μL测序接头连接反应液中加入等体积(100μL)的TE缓冲液(pH 8.0),轻弹混匀后再加入此时稀释样品总体积0.3倍、且平衡至室温AMPureXP磁珠(0.3×200μL=60μL),以纯化接头连接完成的混合gDNA样本,具体操作为:轻弹混匀上述混合反应液,并放置室温静置孵育10min;结束后短暂离心并将EP管放置在磁力上,待液体变澄清后吸去上清,取下EP管后加入250μL短片段清洗缓冲液SFB(SQK-LSK109),轻弹EP管让磁珠重悬后再将EP管重新置于磁力架中,待溶液再次澄清后弃上清,重复一次此清洗过程;取下EP管短暂离心后放回磁力架上,吸去残余液体,自然风干约1~2min(勿让聚集的磁珠干裂),然后加入14μL的洗脱缓冲液EB(SQK-LSK109),轻弹试管完全重悬磁珠后置于室温孵育10min(如需回收>30kb以上的gDNA片段,可将孵育时间延长至30min);孵育结束后短暂离心并将混合gDNA样品置于磁力架上,待脱去gDNA样品的磁珠成团且溶液变澄清后,吸取13μL含混合gDNA样品的上清液于一个新的低吸附的1.5mL EP管中,即为接上测序接头的混合gDNA样本测序文库;最后,取1μL上述测序文库用Qubit荧光定量仪进行定量,如果总的测序文库回收量大于2.5μg可继续进行后续实验(对于FLO-MIN106测序芯片,gDNA测序文库不宜超过5μg,否侧过载的gDNA文库会造成芯片上纳米孔的堵塞)。本实施例1在此步的6个K562细胞系gDNA混合样品定量的具体结果见表5,结果显示该六个gDNA混合样本回收率均满足要求,可继续进行后续实验。
表5
*用于上机测序的靶向ROI文库质量-=纯化后回收的质量-用于Qubit浓度检测的1μL样本质量
(9)上机测序:
①激活测序芯片:测序芯片选用质检后可用纳米孔大于800个的FLO-MINI106测序芯片,并用ONT公司测序芯片激活试剂盒中的纳米孔基因测序芯片激活缓冲液FLT和FB(EXP-FLP002)对其进行激活,方法为:首先,配制芯片激活混合液,即将30μL的FLT加入到一管FB中,并混合均匀;然后,打开测序芯片的priming激活孔,用枪头排出气泡后取800μL芯片激活混合液从priming激活孔迅速加入至测序芯片中的流通池中,室温孵育5min,此时可进行上机文库的准备。
②准备上机测序文库:制备75μL的上机测序文库,将37.5μL的纳米孔基因测序缓冲液SQB(SQK-LSK109)和25.5μL的纳米孔基因测序芯片上样磁珠(SQK-LSK109)加入到12μL混合的gDNA测序文库中,轻弹试管混匀。
③完成芯片的激活及加载上机测序文库:打开测序芯片的SpotON进样孔的盖子后,取200μL芯片激活混合液从priming激活孔迅速加入至测序芯片的流通池中以完成测序芯片的激活,之后以逐滴的方式将制备好的75μL上机文库从SpotON进样孔加入到测序芯片的流通池中,最后关闭SpotON孔盖和priming激活孔。
④进行测序:将载有测序文库的芯片安装到纳米孔基因测仪MinION或者GridION进行测序,并通过纳米孔基因测序平台的驱动操作软件MinKNOW实时检测测序过程并获取测序数据。
(10)结束实验:
待测序数据达到2Gb左右时或者没有数据产出时即可结束测序。对于使用过的测序芯片,可使用ONT公司的测序芯片清洗试剂盒(Flow Cell Wash Kit)对其进行清洗,然后保存在4℃,下次使用前同样需要进行可用纳米孔孔的检查,如大于800个则可再次利用。
(11)靶向测序数据获取和分析:
①数据获取:在测序时,通过MinKNOW软件控制纳米孔基因测序仪器、监测测序芯片运行状态和收集测序输出的电信号数据(储存形式为Fast5文件)等,具体参照ONT公司官方使用手册。MinKNOW软件会根据所每个样本所对应的Barcode标签序列,将所测的每个样本的fast5数据进行单独储存。本实例1的6个相同的K562细胞系gDNA所得的靶向测序数据分别为barcode07,barcode08,barcode09,barcode10,barcode11和barcode12。
②数据分析:
a.数据分析所需的运行环境:
Megalodon(2.4.1);
Guppy(5.0.11或以上版本);
Minimap2;
Samtools;
NanoPlot;
通过XShell远程连接软件操作Linux系统,将上述5mC数据分析所需的软件安装在一个数据分析环境下,具体安装方式法和使用说明参照官方说明书。
b.数据可视化软件:
IGV;将该软件安装在Windows系统下即可,具体操作参照官方说明书。
③5mC甲基化修饰数据分析过程:
通过XShell远程连接软件操作Linux系统,在安装有所述数据处理软件的环境下,针对样本的电信号测序数据文件Fast5运行一条命令“megalodon Fast5输入文件名/--guppy-param"-d/home/dell/github/rerio/basecall_models/"
--guppy-config res_dna_r941_min_modbases_5mC_CpG_v001.cfg--outputsbasecalls mappings mod_mappings mods per_read_mods--reference
/home/dell/genome/GRCh38.p12.genome.fa--mod-motif m CG 0--devices 0
--processes 20--mod-map-emulate-bisulfite--mod-map-base-conv C T
--mod-map-base-conv m C--guppy-server-path
/home/dell/biosoft/ont-guppy/bin/guppy_basecall_server--output-directory./输出文件名/--write-mods-text”,完成后即可得到能用IGV进行可视化的5mC修饰结果图(bam文件),和所测每个CG基序中发生甲基化修饰C(5mC)的百分数比值(bed文件)。
上述命令运行过程如图5所示,Megalodon软件首先调用Guppy软件对所需分析的Fast5文件进行碱基和修饰识别(basecalling),basecalling是通过神经网络算法将测序所得电信号数据转化为碱基序列数据,同时使用“--trim_barcodes”参数将序列中的barcode标签序列和接头序列去除,最中生成含有5mC修饰的一条条碱基序列(称为reads),储存形式为Fastq文件;其次,是使用Minimap2软件将测序reads比对到人类参考参考基因组GRCh38.p12上;再者,Megalodon利用碱基修饰识别参数(包括5mC、6mA、CpG、dam、dcm等)中的5mC修饰识别模式来检测CG基序中的发生5mC修饰的C,同时计算每个CG位点中发生5mC的百分数占比(5mC在所测C位点测序深度中的百分数占比),使用参数为“--guppy-configres_dna_r941_min_modbases_5mC_CpG_v001.cfg”;然后利用Megalodon的碱基转换参数“--mod-map-emulate-bisulfite--mod-map-base-conv C T
--mod-map-base-conv m C”将纳米孔测序的5mC修饰数据测序数据模拟为亚硫酸氢盐的测序数据,使得发生甲基化的碱基仍然为C碱基(在IGV中可视化为红色的C碱基),而未发生甲基化的C碱基则转变为T碱基(在IGV中因与参考的C碱基比对错误而显示为蓝色的T碱基);最后,使用Samtools对最终生成的可用于5mC修饰分析的bam文件排序,并建立相应的索引文件bai,以便于IGV可视化。
④数据质控:
测序数据质控是针对Fastq文件进行质控,主要分析每个样本的测序数据量、reads数目、reads质量值、reads长度和reads长度N50等。具体使用和文件解读详见软件官方说明书。
(12)数据分析结果
①靶向测序数据汇总和解读:
表6为本实施例1中靶向的reads数和reads长度N50,可见在本实施例1的6个K562细胞系gDNA样本中,BCR_23177704crRNA探针对其靶向富集的目的基因片段(本发明的OI,即包含BCR-ABL1融合基因启动子在内的CpG岛基因片段,该CpG岛和启动子在22号染色体上的位置分别为chr22:23180364-23182278和chr22:23180778-23180837)的测序reads条数基本在100条reads左右(即在该区域的测序深度约为100×),且Reads长度N50(即将所有reads长度相加,然后从最长的开始倒数,数到长度为总长度一半的片段即为Reads长度N50)均大于10kb。
表6
表7为汇总的本发明ROI中每个CG基序中C的测序深度和发生5mC的百分数占比(5mC在所测C位点测序深度中的百分数占比),图7为表7数据对应的散点图。该数据说明,在测序深度满足基本要求(>10×)时,通过测序结果可直接判断本发明ROI中CG基序的C有无发生甲基化以及甲基化的程度(百分比)。
表7
②包含BCR-ABL1融合基因启动子在内的CpG岛甲基化的测序数据可视化:
图8为本发明实施例1的6个相同的gDNA样本在所测ROI的5mC测序数据的IGV可视化结果图。在每个样本中,BCR_23177704crRNA探针靶向的大部分reads均完全涵盖了本发明所检测的ROI(包含BCR-ABL1融合基因启动子在内的CpG岛),因该ROI区域中的任意CG基序中C的甲基化状态都能被检测到,所以可以观察到CpG岛的甲基化状态为:前半部分呈现低甲基化状态(深灰色;其原图在IGV上显示为蓝色),而后半部分为高甲基化状态(浅灰色;其原图在IGV上显示为红色)。因6个相同的K562细胞系gDNA样本的BCR-ABL1基因的启动子在CpG岛上游,所以其整个启动子区域呈现为低甲基化状态。
上述应用靶向纳米孔基因测序技术检测K562细胞系gDNA的ROI(包含BCR-ABL1融合基因启动子在内的CpG岛)的CG基序中C的甲基化修饰水平结果经亚硫酸氢盐扩增子测序(BSAS)检验,结果可靠。
实施例1说明,本发明提供的试剂盒和方法实现了对实施例1中6个相同的K562细胞系gDNA中的ROI进行了高质量的深度测序,且reads长度足够长,基本能完全覆盖本发明的ROI,且能保证一批次所测相同样本5mC检测结果的重复性和准确性,也进一步说明了本发明同时检测多个样本BCR-ABL1融合基因启动子在内的CpG岛甲基化的可行性的。其次,表7和图7表明本发明在检测本发明ROI区域中CG基序的C甲基化修饰水平方面展现出明显的优越性,包括测序深度高、覆盖范围广(测序读长长)、单碱基分辨率、特异性高、结果准确等特点。最后,本发明提供的数据分析方法简便易懂,可满足大样本的数据分析。
实施例2:
检测6个BCR-ABL1融合基因阳性病人的血液或骨髓gDNA样本中BCR-ABL1融合基因启动子甲基化状态
实施例2所用所有试剂和方法均与实施例1相同。
采用实施例1提供的试剂盒和方法检测样本编号为CML-017,CML-028,CML-051,CML-052,ALL-056,CML-057的血液或骨髓gDNA中BCR-ABL1融合基因启动子及其所在的CpG岛的甲基化状态。实施例2中的血液或骨髓样本均来源于临床上确诊的BCR-ABL1融合基因阳性的白血病病人,该样本由本实验室课题合作方赣南医学院附属第一医院检验科提供。本实施例2使用临床上确诊的BCR-ABL1融合基因阳性的白血病病人的血液或骨髓gDNA作为研究对象,以说明本试剂盒能应用于临床研究。
具体方法如下:
(1)血液或骨髓样本的gDNA提取
按照gDNA提取试剂盒(QIAGEN公司,QIAamp DNA Blood Mini Kit)说明书操作提取血液或骨髓样本的gDNA:先取20μL的蛋白酶K和4μL的RNA A酶于1.5mL的EP管中,再加入200μL血液或骨髓样本(有核细胞个数<5×10^6,为该gDNA提取试剂盒的最大起始细胞数量),最后用枪头吹吸混匀;加入200μL裂解液AL,56℃裂解细胞10min(如果未裂解充分,可适当延长孵育时间);然后加入200μL无水乙醇,震荡15sec,将反应液转移至过滤柱中,8000rpm离心1min;加500μL缓冲液AW1,8000rpm离心1min;再加500μL缓冲液AW2,14000rpm离心3min;将过滤柱置于新的收集管上,加入50μL~200μL的洗脱液AE,室温静置3min,8000rpm离心1min,收集洗脱的gDNA并储存在-80℃冰箱。在gDNA使用时,用荧光定量仪Qubit测定其浓度,并用全自动毛细管分析仪Qsep100分析其片段大小以及纯度。本实施例2所用的6例血液或骨髓样本的gDNA用Qubit所测的浓度和基本的临床病例特点见表8和用Qsep100所测的片段大小以及纯度见图9所示,所用6例血液或骨髓样本的gDNA浓度(>210ng/μL)、片段大小(主峰均位于10kb左右)和纯度(无RNA污染)均符合测序要求。
表8
*临床病例特点:疾病名称-编号,疾病状态,BCR-ABL1强合蛋白类型。CML:慢性粒细胞白血病;ALL:急性淋巴细胞白血病
(2)组装RNPs和gDNA去磷酸化
①组装RNPs:首先是合成3个10μL gRNA(crRNA终浓度为2μM):3μL BCR-23177704crRNA(20μM)和3μL tracrRNA(20μM),并用24μL无酶duplex缓冲液补充至30μL,95℃加热5min后冷却至4℃;其次是组装6个单独的10μL RNPs(crRNA终浓度为1μM):5μLgRNA、0.8μL Cas9(6.2μM),1μL 10×CutSmart缓冲液和3.2μL无酶水,室温下孵育30min后转至冰上备用,如有多余的RNPs可放置-80℃保存1个月;在RNP孵育期间,继续进行下一步的gDNA去磷酸化。
②gDNA去磷酸化:所需gDNA起始输入量为3~10μg,建议用5μg。首先在6个PCR管中准备本实施例2中的6个5μg的血液或骨髓gDNA样本(浓度ng/μL×体积μL=5μg):具体见表9所示:
表9
在向上述6个PCR管中均加入3μL 10×CutSmart缓冲液、3μL快速磷酸酶和1μL无酶水,最终去磷酸化体系的总体积为30μL;37℃反应15min进行gDNA的5’端去磷酸化,然后在80℃下加热2min使快速磷酸酶失活。
(3)RNPs切割BCR_23177704crRNA探针靶位点以及新生断裂末端的制备
将6个去磷酸化处理的30μL血液或骨髓gDNA样本平衡至室温后,并向其中加入步骤2组装好的10μL RNPs;此外,还需加入1μL 10mM dATP和1μL Taq DNA聚合酶。然后,将42μL的反应液在37℃下孵育30min让RNPs切割双链gDNA中相应的crRNA探针靶位点,并让TaqDNA聚合酶在双链gDNA的3’末端加上一个dATP,形成dA尾;最后,在72℃下反应5min使Cas9蛋白失活后,将样本转移至冰上保存。
(4)qPCR实验质控6个血液或骨髓gDNA样本中BCR_23177704 crRNA探针的靶向效率
经RNPs切割后及新生断裂末端制备完成的42μL的6个血液或骨髓gDNA样品中gDNA的浓度为:5μg÷42μL=119ng/μL。上述6个样品各取1μL于0.2mL的PCR试管中,用9μL无酶水稀释至11.9ng/μL后作为实验组模板(RNPs切割后),剩余41μL的6个经RNPs切割后及新生断裂末端制备完成的血液或骨髓gDNA样品放置冰上保存备用;再从原始的6个血液或骨髓gDNA样品中各取取1μL并稀释至11.9ng/μL后作为对照组模板(RNPs切割前)。qPCR反应体系为20μL,每组做2个重复,其中模板1.5μL(10ng~20ng)、10μL的2×SYBR Green荧光染料、2μL的2μM上游及下游引物和4.5μL无酶水,之后根据ΔCT值(实验组CT-对照组CT值)质控6个血液或骨髓gDNA样本中BCR_23177704crRNA探针的靶向效率。结果见表10所示,本实施例2中6个血液或骨髓gDNA样本中BCR_23177704crRNA探针的靶向效率均合格(ΔCT>2),可进行后续的测序实验。
表10
(5)对靶向效率质控合格的样本进行纯化:
此步骤和质控要求同实施例1的步骤7。本实施例2的6个血液或骨髓gDNA样本经此步骤纯化后的结果见表11,结果显示该6个血液或骨髓gDNA的回收率均满足要求,可继续进行后续实验。
表11
*用于下一步实验的样本量=纯化后回收的质量-用于Qubit浓躔检测的1μL样本质量
(6)构建靶向富集目的片段的测序文库
此步骤和质控要求同实施例1的步骤8,其中6个血液或骨髓gDNA样本所用的Barcode标签序列为:CML-017+BC07,CML-028+BC08,CML-051+BC09,CML-052+BC10,CML-056+BC11,CML-057+BC12。具体结果如下:
①连接Barcode标签序列并进行纯化:
本实施例2的6个血液或骨髓gDNA样本的在连接Barcode标签序列并纯化后的结果见表12,结果显示该六个血液或骨髓gDNA样本回收量均满足要求,可继续进行后续实验。
表12
*用于下一步实验的样本量=纯化后回收的质量-用于Qubit浓度检测的1μL样本质量
将上述已经连接Barcode标签序列的6个血液或骨髓gDNA样本混合到新的低吸附的1.5mL EP管中,总gDNA样本质量约为~6μg(平均每个样本约1μg),总体积约为65~66μL。
②连接纳米孔基因测序接头并进行纯化:
本实施例2在此步的6个血液或骨髓gDNA混合样本定量的具体结果见表13,结果显示该gDNA混合样本回收量均满足要求,可继续进行后续实验。
表13
*用于上机测序的靶向ROI文库质量-=纯化后回收的质量-用于Qubit浓度检测的1μL样本质量
(7)上机测序:
此步骤和质控要求同实施例1的步骤9。
(8)结束实验:
此步骤和质控要求同实施例1的步骤10。
(9)靶向测序数据获取和分析:
此数据分析过程同实施例1的步骤11。
(10)数据分析结果
表14为本实施例2中靶向的reads数和reads长度N50,可见在本实施例2的6个BCR-ABL1融合基因阳性病人的血液或骨髓gDNA样本中,BCR_23177704crRNA探针对ROI的靶向测序reads条数基本10~20条reads左右(即在该区域的测序深度在10~20×),虽然每个样本的ROI测序深度较低低,但仍能满足甲基化数据分析要求(>10×);每个样本的Reads长度N50约为10kb,该长度的覆盖范围能满足包含BCR-ABL1融合基因启动子在内的CpG岛甲基化的检测要求。而实施例2的数据情况较实施例1差的原因主要考虑为临床血液或骨髓样本复杂,导致提取的gDNA样本的质量低于细胞系的gDNA,所以测序得到的数据质量也较低。
表14
表15为汇总的本发明ROI中每个CG基序中C的测序深度和发生5mC的百分数占比,图10为表15数据对应的散点图。该数据说明,在测序深度满足基本要求(>10×)时,通过测序结果可直接判断BCR-ABL1融合基因阳性的临床血液或骨髓gDNA样本中本发明ROI中CG基序的C有无发生甲基化以及甲基化的程度(百分比)。
表15
图8为本发明实施例2的6个BCR-ABL1融合基因阳性的临床血液或骨髓gDNA样本在所测ROI的5mC测序数据的IGV可视化结果图。如图所示,每个临床gDNA样本的BCR-ABL1基因的CpG岛和启动子均呈现为低甲基化状态(深灰色;其原图在IGV上显示为蓝色)。
本实施例2表明,本发明提供的试剂盒和方法能够做到对临床上确诊的BCR-ABL1融合基因阳性的白血病病人的血液或骨髓gDNA样本中ROI的甲基化程度的检测,即说明本试剂盒能应用于临床大样本研究。
综上,本发明提供了一种直接检测多个样本中BCR-ABL1融合基因启动子甲基化的试剂盒以及一套从实验方法到数据分析的完整流程,通过结合CRISPR/Cas9靶向的纳米孔基因测序技术以及一套简易的数据分析方法,可简单、便捷地对BCR-ABL1融合基因阳性的gDNA样本中的包含BCR-ABL1融合基因启动子在内的CpG岛(本发明ROI)的甲基化程度进行检测,且具有同时检测个多样本、测序深度高、覆盖范围广(测序读长长)、单碱基分辨率、特异性高、结果准确、数据分析方法简易等特点。本发明有望弥补BCR-ABL1融合基因启动子甲基化检测方面存在的局限性,将为进一步揭示该分子事件在BCR-ABL1阳性白血病诊疗过程中的作用提供有力的检测技术支持,例如TKIs耐药相关研究、肿瘤分子标志物相关应用、甲基化药物应用等方面。
以上所述实施例仅表达了本申请的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本申请构思的前提下,还可以做出若干变形和改进,这些都属于本申请的保护范围。因此,本申请专利的保护范围应以所附权利要求为准。
- 用于肺癌检测的DNA甲基化qPCR试剂盒及使用方法
- 检测样本中NonO-TFE3融合基因的寡核苷酸、方法和试剂盒
- 检测样本中ASPL-TFE3融合基因的寡核苷酸、方法和试剂盒
- 检测样本中TFE3融合基因的寡核苷酸、方法和试剂盒
- 检测样本中PSF-TFE3融合基因的寡核苷酸、方法和试剂盒
- 一种用于分析DNA样本中MLH1启动子甲基化状态的试剂盒、方法和引物
- 用于检测样本中的一个或多个目标分析物的试剂盒及其制备和使用方法
