中和SARS-CoV-2的单结构域抗体
文献发布时间:2024-04-18 19:52:40
对以电子方式提交的序列表的引用
根据EFS-Web法律构架和37CFR§§1.821-825(参见MPEP§2442.03(a)),与本申请同时提交了符合ASCII的文本文件形式的序列表(名称为“Sequence_Listing_3000093-005977_ST25.txt”,创建于2022年2月17日,并且大小为132,895字节),并且该序列表的全部内容以引用方式并入本文。
发明背景
1.技术领域
本公开涉及抗SARS-CoV-2的单结构域抗体。所述单结构域抗体阻断SARS-CoV-2感染,并且可用于患者的治疗、预防和/或诊断。
2.背景技术
病毒感染是公共卫生的持续问题。在20世纪和21世纪,已经由新颖病毒引起了流行病。
冠状病毒病(COVID-19)是由新发现的冠状病毒严重急性呼吸系统综合征冠状病毒2(SARS-CoV-2)引起的传染病。SARS-CoV-2于2020年1月12日被世界卫生组织命名。其与2003年鉴定的SARS冠状病毒(SARS CoV)和2012年鉴定的MERS冠状病毒(MERS CoV)一起,属于2003年鉴定的冠状病毒科(Coronaviridae)的β属。SARS-CoV-2基因组与SARS CoV病毒共享约70%的序列同一性,并且与MERS CoV病毒共享约40%的序列相似性。WHO网站(2020)。
大多数感染SARS-CoV-2病毒的人会体验到轻度至中度呼吸道疾病,并且无需特殊治疗即可康复。年龄较大的患者,例如大于60岁的患者,以及患有例如心血管疾病、糖尿病、慢性呼吸道疾病和癌症的潜在医学问题的患者,更有可能发展成严重的疾病。疾病控制中心网站(2020)。在易感群体中,严重急性呼吸系统综合征冠状病毒2(SARS-CoV-2)可能导致致命的人类呼吸系统疾病。COVID-19患者往往表现出急性肺损伤(acute lung injury,ALI)的特性,包括弥漫性肺泡损伤(diffuse alveolar damage,DAD)、上皮坏死以及纤维蛋白和透明素沉积。许多死于COVID-19的患者会发展出急性呼吸窘迫综合征(acuterespiratory distress syndrome,ARDS),一种严重形式的急性肺损伤。Li&Ma
与其他冠状病毒类似,SARS-CoV-2上的刺突糖蛋白(S)同源三聚体在受体结合和病毒进入中发挥着关键作用。刺突糖蛋白分为两个功能亚基,称为S1和S2。所述S1亚基负责经由其C末端受体结合结构域(RBD)与人血管紧张素转换酶2(ACE2)之间的相互作用进行宿主细胞受体的结合。S2亚基在病毒和细胞膜的融合中发挥重要作用。
若干用于预防和治疗COVID-19的药物和疫苗正在开发中。中和SARS-CoV-2-RBD/ACE2相互作用的生物药物尤其令人感兴趣。有至少9种SARS-CoV-2-RBD抗体或抗体组合正在临床试验中进行评估,其中REGN-COV2是最先进的(NCT04452318,第3期)。
本领域存在对抗COVID-19疗法的需求。
发明内容
本公开提供了免疫特异性结合SARS-CoV-2的单结构域抗体(包括但不限于包含或者替代地由抗体片段或其变体组成的抗原结合分子)、其组合物及其使用方法。例如,本公开提供了免疫特异性结合SARS-CoV-2的受体结合结构域(RBD)的单结构域抗体(包括包含或者替代地由抗体片段或其变体组成的抗原结合分子)。本发明还提供了用于在动物,优选地哺乳动物,最优选地人中检测、诊断或预测与SARS-CoV-2相关联的疾病或疾患的方法和组合物,所述方法和组合物包括或替代地由以下组成:使用免疫特异性结合SARS-CoV-2的单结构域抗体(包括但不限于包含或者替代地由抗体片段或其变体组成的抗原结合分子)。可用所公开的单链抗体(包括但不限于包含或者替代地由抗体片段或其变体组成的抗原结合分子)检测、诊断或预测的疾病和疾患包括但不限于COVID-19。本公开还提供了用于在动物,优选地哺乳动物,最优选地人中预防、治疗或改善与SARS-CoV-2相关联的疾病或疾患的方法和组合物,所述方法和组合物包括或替代地由以下组成:向所述动物施用有效量的一种或多种免疫特异性结合SARS-CoV-2的单结构域抗体(包括包含或者替代地由抗体片段或其变体组成的抗原结合分子)。可以通过施用有效量的本文所述的抗体来预防、治疗或改善的疾病和疾患包括但不限于COVID-19。
在实施方式中,本文所述的单结构域抗体免疫特异性地结合SARS-CoV-2,特别是SARS-CoV-2的RBD。本文所述的单结构域抗体可以是包含免疫球蛋白折叠的氨基酸序列,或者可以是在合适的条件(例如,生理条件)下能够形成免疫球蛋白折叠(例如,通过折叠)的氨基酸序列。优选地,当正确折叠以形成免疫球蛋白折叠时,此类氨基酸序列能够免疫特异性地结合SARS-CoV-2;并且更优选地能够免疫特异性地结合SARS-CoV-2的RBD。
在实施方式中,本文所述的抗原结合分子包含或替代地由以下组成:这些单结构域抗体的片段或变体(例如,包括本文所述的那些中任一者的氨基酸序列),所述片段或变体免疫特异性地结合SARS-CoV-2,优选地SARS-CoV-2的RBD。在实施方式中,还提供了编码本文所述的单结构域抗体的核酸分子、包含所述核酸分子的载体和宿主细胞、和/或抗原结合分子。
在实施方式中,本文所述的单结构域抗体包含基本上由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成的氨基酸序列;或此类氨基酸序列的任何合适片段(其通常将包含形成CDR中的至少一个CDR的氨基酸残基中的至少一些氨基酸残基,如本文进一步所述)。
在实施方式中,本文所述的单结构域抗体可以是免疫球蛋白序列或其合适的片段,并且特别地可以是免疫球蛋白可变结构域序列、免疫球蛋白单可变结构域序列或其合适的片段,例如轻链可变结构域序列(例如,VL结构域序列)或其合适的片段;或重链可变结构域序列(例如,VH结构域序列)或其合适的片段。当本文所述的单结构域抗体是重链可变结构域序列时,其可以是来源于常规四链抗体的重链可变结构域序列(例如,来源于人抗体的VH结构域序列)或者是来源于重链抗体(如本文所述)的VHH序列(如本文所述)。
然而,应当注意的是,本文所述的单结构域抗体不限于本文所述的氨基酸序列(或本文所述的用于表达所述氨基酸序列的核苷酸序列)的来源,也不限于生成或获得(或已经生成或获得)本文所述的氨基酸序列或核苷酸序列的方式。因此,本文所述的氨基酸序列可以是天然存在的氨基酸序列(来自任何合适的物种)或合成或半合成的氨基酸序列。在具体但非限制性的实施方式中,本文所述的氨基酸序列是天然存在的免疫球蛋白序列(来自任何合适的物种)或合成或半合成的免疫球蛋白序列,包括但不限于“人源化的”免疫球蛋白序列(包括但不限于部分或完全人源化的小鼠或兔免疫球蛋白序列,特别是部分或完全人源化的VHH序列)、“骆驼化的”免疫球蛋白序列以及已经通过包括但不限于以下的技术获得的免疫球蛋白序列:亲和力成熟(例如,从合成的、随机的或天然存在的免疫球蛋白序列开始)、CDR移植、表面改构、组合来源于不同免疫球蛋白序列的片段、使用重叠引物的PCR装配、以及技术人员熟知的用于工程化免疫球蛋白序列的类似技术;或前述中任一者的任何合适的组合。
类似地,本文所述的核苷酸序列可以是天然存在的核苷酸序列或合成或半合成的序列,并且可以例如是通过PCR从合适的天然存在的模板(例如,从细胞分离的DNA或RNA)分离的序列、已经从文库(例如,表达文库)分离的核苷酸序列、已经通过(使用任何合适的技术,例如错配PCR)将突变引入天然存在的核苷酸序列中而制备的核苷酸序列、已经使用重叠引物通过PCR制备的核苷酸序列、或已经使用任何已知的DNA合成技术制备的核苷酸序列。
在实施方式中,本文所述的单结构域抗体包含或替代地由以下组成:免疫球蛋白单可变结构域序列,例如结构域抗体(或适合用作结构域抗体的氨基酸序列)、单结构域抗体(或适合用作单结构域抗体的氨基酸序列)、“dAb”(或适合用作dAb的氨基酸序列)或VHH序列;其他单可变结构域,或它们中的任一者的任何合适的片段。对于(单)结构域抗体的一般描述,参见EP 0368 684。对于术语“dAb”,例如参见Ward等人(Nature,1989年10月12日;341(6242):544-6);Holt等人,Trends Biotechnol.,2003,21(11):484-490;以及例如WO2006/030220和WO 2006/003388。还应当指出的是,虽然因为它们不是哺乳动物来源的,所以在本文描述的上下文中不太优选,但是单结构域抗体或单可变结构域可以来源于某些鲨鱼物种(例如,“IgNAR结构域”,参见例如WO 2005/18629)。
在实施方式中,本文所述的单结构域抗体包含或替代地由以下组成:具有以下结构的氨基酸序列:FR1-CDR1-FR2-CDR2-FR3-CDR3-FR4,其中FR1至FR4分别指构架区域1至4,并且CDR1至CDR3分别指互补决定区1至3。
在实施方式中,本文所述的单结构域抗体包含或替代地由以下组成:具有以下结构的氨基酸序列:FR1-CDR1-FR2-CDR2-FR3-CDR3-FR4,其中氨基酸序列与SEQ ID NO:1至SEQ ID NO:15所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性。
本公开还提供了包含或替代地由免疫特异性结合SARS-CoV-2的这些单结构域抗体的片段或变体(例如,包括一个或多个具有本文所述的那些中的任一者的氨基酸序列的CDR)组成的抗原结合分子,以及编码这些抗体的核酸分子、包含所述核酸分子的载体和宿主细胞和/或抗原结合分子。
在实施方式中,本文所述的氨基酸序列可以是包含至少一个选自由以下组成的组的氨基酸序列的氨基酸序列:本文所述的CDR1序列、CDR2序列和CDR3序列(或它们的任何合适的组合)。具体地,本文所述的氨基酸序列可以是包含至少一个抗原结合位点的氨基酸序列,其中所述抗原结合位点包含至少一个选自由以下组成的组的氨基酸序列:本文所述的CDR1序列、CDR2序列和CDR3序列(或它们的任何合适的组合)。
在实施方式中,本文所述的氨基酸序列可以是包含至少一段氨基酸残基的任何氨基酸序列,其中该段氨基酸残基具有对应于本文所述的CDR序列中的至少一个CDR序列的序列的氨基酸序列。此类氨基酸序列可包含或可不包含免疫球蛋白折叠。在非限制性示例中,此类氨基酸序列可以是免疫球蛋白序列的合适片段,所述片段包含至少一个CDR序列,但其没有大到足以形成(完整)免疫球蛋白折叠(参见例如描述于WO 2003/050531或WO 2009/127691中的“加速片段(Expedite fragment)”)。或者,此类氨基酸序列可以是合适的“蛋白质支架”,该蛋白质支架包含至少一段对应于CDR序列的氨基酸残基(例如,作为其抗原结合位点的一部分)。用于呈递氨基酸序列的合适支架对于技术人员来说将是清楚的,例如,基于或衍生自免疫球蛋白的结合支架(例如,除了本文所述的免疫球蛋白序列之外)、衍生自蛋白A结构域的蛋白质支架(例如,
包含本文所述的CDR序列中的一个或多个CDR序列的本文所述的任何氨基酸序列优选地免疫特异性地结合至SARS-CoV-2,更优选地结合至SARS-CoV-2的RBD。
在实施方式中,根据本公开的氨基酸序列可以是包含至少一个抗原结合位点的任何氨基酸序列,其中所述抗原结合位点包含选自由以下组成的组的至少两个氨基酸序列:本文所述的CDR1序列、本文所述的CDR2序列和本文所述的CDR3序列,使得(i)当第一氨基酸序列选自本文所述的CDR1序列时,所述第二氨基酸序列选自本文所述的CDR2序列或本文所述的CDR3序列;(ii)当第一氨基酸序列选自本文所述的CDR2序列时,所述第二氨基酸序列选自本文所述的CDR1序列或本文所述的CDR3序列;或者(iii)当第一氨基酸序列选自本文所述的CDR3序列时,所述第二氨基酸序列选自本文所述的CDR1序列或本文所述的CDR3序列。
在实施方式中,本文所述的氨基酸序列可以是包含至少一个抗原结合位点的氨基酸序列,其中所述抗原结合位点包含选自由以下组成的组的至少三个氨基酸序列:本文所述的CDR1序列、本文所述的CDR2序列和本文所述的CDR3序列,使得第一氨基酸序列选自本文所述的CDR1序列,第二氨基酸序列选自本文所述的CDR2序列,并且第三氨基酸序列选自来自本文所述的CDR3序列。
本文描述了CDR1序列、CDR2序列和CDR3序列的组合。包含CDR1、CDR2和CDR3的氨基酸序列优选是免疫球蛋白序列(如本文进一步描述的),但所述免疫球蛋白序列也可以是例如包含用于呈递所述CDR序列的合适支架的任何其他氨基酸序列。
在一个实施方式中,单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)可以免疫特异性地结合SARS-CoV-2的多肽或多肽片段,所述抗体包含或替代地由以下组成:具有VHH结构域的CDR中的任何一个、两个或三个CDR(例如,CDR1、CDR2或CDR3)的氨基酸序列的多肽,所述VHH结构域具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的氨基酸序列。在一个实施方式中,本文所述的单结构域抗体包含具有VHH结构域的CDR1中的任何CDR1的氨基酸序列的多肽,所述VHH结构域具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的氨基酸序列。在实施方式中,本文所述的单结构域抗体包含具有VHH结构域的CDR2中的任何CDR2的氨基酸序列的多肽,所述VHH结构域具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的氨基酸序列。在实施方式中,本文所述的单结构域抗体包含具有VHH结构域的CDR3中的任何CDR3的氨基酸序列的多肽,所述VHH结构域具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的氨基酸序列。本公开还提供了包含或替代地由SEQ ID NO:1至SEQ ID NO:15中任一者所示的VHH结构域的片段或变体(例如,CDR)组成的分子,所述分子免疫特异性地结合SARS-CoV-2,优选地结合SARS-CoV-2的RBD;以及编码这些抗体的核酸分子,和/或分子。
在实施方式中,本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)免疫特异性地结合SARS-CoV-2的多肽或多肽片段,并且包含或替代地由以下组成:具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的VHH结构域的CDR1中的任一个CDR1、具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的VHH结构域的CDR2中的任一个CDR2,和/或具有SEQ ID NO:1至SEQ ID NO:15中任一者所示的VHH结构域的CDR3中的任一个CDR3的氨基酸序列的多肽。在实施方式中,本文所述的单结构域抗体包含SEQ ID NO:1至SEQ ID NO:15中任一者所示的VHH域的CDR1、CDR2和CDR3的氨基酸序列。
在实施方式中,本文所述的单结构域抗体免疫特异性地结合SARS-CoV-2并且包含选自由以下组成的组的一个或多个氨基酸序列:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
d)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
g)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
或它们的任何合适的组合。
当单结构域抗体包含一个或多个根据b)和/或c)的氨基酸序列时:
i)与根据a)的对应氨基酸序列相比,根据b)和/或c)的这种氨基酸序列中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与根据a)的对应氨基酸序列相比,根据b)和/或c)的氨基酸序列优选仅含有氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)根据b)和/或c)的氨基酸序列可以是借助于使用一种或多种已知的亲和力成熟技术进行亲和力成熟而衍生自根据a)的氨基酸序列的氨基酸序列。
类似地,当单结构域抗体包含根据e)和/或f)的一个或多个氨基酸序列时:
i)与根据d)的对应氨基酸序列相比,根据e)和/或f)的这种氨基酸序列中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与根据d)的对应氨基酸序列相比,根据e)和/或f)的氨基酸序列优选仅含有氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)根据e)和/或f)的氨基酸序列可以是借助于使用一种或多种已知的亲和力成熟技术进行亲和力成熟而衍生自根据d)的氨基酸序列的氨基酸序列。
此外,类似地,当单结构域抗体包含根据h)和/或i)的一个或多个氨基酸序列时:
i)与根据g)的对应氨基酸序列相比,根据h)和/或i)的这种氨基酸序列中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与根据g)的对应氨基酸序列相比,根据h)和/或i)的氨基酸序列优选仅含有氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)根据h)和/或i)的氨基酸序列可以是借助于使用一种或多种已知的亲和力成熟技术进行亲和力成熟而衍生自根据g)的氨基酸序列的氨基酸序列。
应当理解,前述段落通常还适用于包含分别根据b)、c)、e)、f)、h)或i)的一个或多个氨基酸序列的任何单结构域抗体。
在实施方式中,本文所述的单结构域抗体优选包含选自由以下组成的组的一个或多个氨基酸序列:
i)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
ii)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;以及
iii)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
或它们的任何合适的组合。
此外,优选地,在此类单结构域抗体中,所述氨基酸序列中的至少一个氨基酸序列形成用于结合SARS-CoV-2的抗原结合位点的一部分。
在实施方式中,本公开涉及一种单结构域抗体,所述单结构域抗体包含选自由以下组成的组的两个或更多个氨基酸序列:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
d)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
g)SEQ ID NO:77至SEQ ID NO:91中至少一者所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
使得(i)当第一氨基酸序列对应于根据a)、b)或c)的氨基酸序列中的一个氨基酸序列时,第二氨基酸序列对应于根据d)、e)、f)、g)、h)或i)的氨基酸序列中的一个氨基酸序列;(ii)当第一氨基酸序列对应于根据d)、e)或f)的氨基酸序列中的一个氨基酸序列时,第二氨基酸序列对应于根据a)、b)、c)、g)、h)或i)的氨基酸序列中的一个氨基酸序列;或者(iii)当第一氨基酸序列对应于根据g)、h)或i)的氨基酸序列中的一个氨基酸序列时,第二氨基酸序列对应于根据a)、b)、c)、d)、e)或f)的氨基酸序列中的一个氨基酸序列。
在实施方式中,本文所述的单结构域抗体优选包含选自由以下组成的组的两个或更多个氨基酸序列:
i)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
ii)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;以及
iii)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
使得,(i)当第一氨基酸序列对应于SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的一个氨基酸序列时,第二氨基酸序列对应于SEQ ID NO:49至SEQ ID NO:62或SEQID NO:77至SEQ ID NO:91所示的氨基酸序列中的一个氨基酸序列;(ii)当第一氨基酸序列对应于SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的一个氨基酸序列时,第二氨基酸序列对应于SEQ ID NO:27至SEQ ID NO:40或SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的一个氨基酸序列;或者(iii)当第一氨基酸序列对应于SEQ ID NO:77至SEQ IDNO:91所示的氨基酸序列中的一个氨基酸序列时,第二氨基酸序列对应于SEQ ID NO:27至SEQ ID NO:40或SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的一个氨基酸序列。
此外,在此类单结构域抗体中,所述至少两个氨基酸序列优选地形成用于结合SARS-CoV-2的抗原结合位点的一部分。
在实施方式中,本公开涉及一种单结构域抗体,所述单结构域抗体包含三个或更多个氨基酸序列,其中第一氨基酸序列选自由以下组成的组:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
第二氨基酸序列选自由以下组成的组:
d)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
并且第三氨基酸序列选自由以下组成的组:
g)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
优选地,第一氨基酸序列选自由SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列组成的组;第二氨基酸序列选自由SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列组成的组;并且第三氨基酸序列选自由SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列组成的组。
同样,优选地,在此类单结构域抗体中,所述至少三个氨基酸序列形成用于结合SARS-CoV-2的抗原结合位点的一部分。
本文描述了此类氨基酸序列的优选组合。
在实施方式中,在此类氨基酸序列中,CDR序列与SEQ ID NO:1至SEQ ID NO:15所示的氨基酸序列中的至少一个氨基酸序列的CDR序列具有至少70%同一性、至少80%同一性、至少90%同一性、至少91%同一性、至少92%同一性、至少93%同一性、至少94%同一性、至少95%同一性、至少96%同一性、至少97%同一性、至少98%同一性、至少99%同一性或约100%同一性。这种氨基酸同一性程度可以例如通过确定所述氨基酸序列与SEQ IDNO:1至SEQ ID NO:15所示的序列中的一个或多个序列之间的氨基酸同一性程度(以本文所述的方式)来确定,其中形成构架区域的氨基酸残基被忽略。
此外,此类氨基酸序列优选使得它们免疫特异性地结合SARS-CoV-2;并且更具体地结合SARS-CoV-2的RBD。
当本公开的单结构域抗体基本上由四个构架区域(分别为FR1至FR4)和三个互补决定区(分别为CDR1至CDR3)组成时:
CDR1优选地选自由以下组成的组:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
和/或
CDR2优选地选自由以下组成的组:
d)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
和/或
CDR3优选地选自由以下组成的组:
g)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
优选地,当本公开的单结构域抗体基本上由四个构架区域(分别为FR1至FR4)和三个互补决定区(分别为CDR1至CDR3)组成时,CDR1选自由SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列组成的组;和/或CDR2选自由SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列组成的组;和/或CDR3选自由SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列组成的组。
特别地,当单结构域抗体基本上由四个构架区域(分别为FR1至FR4)和三个互补决定区(分别为CDR1至CDR3)组成时:
CDR1优选地选自由以下组成的组:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
并且
CDR2优选地选自由以下组成的组:
d)SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
并且
CDR3优选地选自由以下组成的组:
g)SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
优选地,当单结构域抗体基本上由四个构架区域(分别为FR1至FR4)和三个互补决定区(分别为CDR1至CDR3)组成时,CDR1选自由SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列组成的组;CDR2选自由SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列组成的组;并且CDR3选自由SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列组成的组。
再次,本文描述了CDR序列的优选组合。
此外,此类单结构域抗体优选地免疫特异性地结合SARS-CoV-2;并且更优选地结合SARS-CoV-2的RBD。
在一个实施方式中,本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)可以免疫特异性地结合SARS-CoV-2的RBD(例如,包含或替代地由SEQID NO:124或SEQ ID NO:127所示的氨基酸序列组成的多肽)。在实施方式中,本文所述的单结构域抗体免疫特异性地结合SARS-CoV-2的RBD(例如,包含或替代地由SEQ ID NO:124或SEQ ID NO:127所示的氨基酸序列组成的多肽),并且包含或替代地由对应于SEQ ID NO:1至SEQ ID NO:15中的一者或多者或包含在SEQ ID NO:1至SEQ ID NO:15中的一者或多者内的VHH结构域、CDR1、CDR2和/或CDR3组成。
在一个实施方式中,单结构域抗体(包括包含或替代地由抗体片段或其变体(包括衍生物)组成的分子)可包含或替代地由本文所述的VHH结构域和/或CDR组成,所述单结构域抗体免疫特异性地结合SARS-CoV-2(例如,SARS-CoV-2的RBD)并且可以使用本领域中已知的方法(例如,本文所公开的免疫测定)来测定与SARS-CoV-2的免疫特异性结合。
在实施方式中,单结构域抗体基本上由四个构架区域(分别为FR1至FR4)和三个互补决定区(分别为CDR1至CDR3)组成,其中所述氨基酸序列的CDR序列与SEQ ID NO:1至SEQID NO:15所示的氨基酸序列中的至少一个氨基酸序列的CDR序列具有至少70%同一性、至少80%同一性、至少90%同一性、至少91%同一性、至少92%同一性、至少93%同一性、至少94%同一性、至少95%同一性、至少96%同一性、至少97%同一性、至少98%同一性、至少99%同一性或约100%同一性。这种氨基酸同一性程度可以例如通过确定所述氨基酸序列与SEQ ID NO:1至SEQ ID NO:15所示的序列中的一个或多个序列之间的氨基酸同一性程度(以本文所述的方式)来确定,其中形成构架区域的氨基酸残基被忽略。此类单结构域抗体可如本文进一步所述。
在实施方式中,本文所述的单结构域抗体的构架序列可以是任何合适的构架序列。例如,基于标准手册和本文的进一步公开内容,合适的构架序列的示例对于技术人员来说将是清楚的。
构架序列优选地是免疫球蛋白构架序列或衍生自免疫球蛋白构架序列的构架序列(例如,通过人源化或骆驼化)。例如,构架序列可以是衍生自轻链可变结构域(例如,VL结构域序列)和/或重链可变结构域(例如,VH结构域序列)的构架序列。在一些方面中,构架序列是衍生自VH结构域序列的构架序列(其中所述构架序列可以任选地已经部分或完全地人源化)或者是已经骆驼化的常规VH结构域序列。
构架序列优选地使得本公开的单结构域抗体包含或替代地由以下组成:结构域抗体(或适合用作结构域抗体的氨基酸序列);单结构域抗体(或适合用作单结构域抗体的氨基酸序列);“dAb”(或适合用作dAb的氨基酸序列);或VHH序列。
本文公开了此类构架序列(的合适组合)的优选但非限制性示例。
再次,如本文针对本文所述的单结构域抗体一般描述的,还可以使用前述中任一者的合适片段(或片段的组合),例如包含一个或多个CDR序列的片段,所述CDR序列侧接有一个或多个构架序列和/或经由一个或多个构架序列连接(例如,可以以与这些CDR和构架序列相同的次序在所述片段衍生自的全大小免疫球蛋白序列中出现)。此类片段可以包含或可以形成免疫球蛋白折叠,或者替代地使得它们不包含或不能形成免疫球蛋白折叠。
在一个方面中,此类片段包含如本文所述的单一CDR序列(并且特别是CDR3序列),所述单一CDR序列在每一侧上侧接有构架序列(的一部分)(并且特别是所述构架序列的在所述片段所衍生自的免疫球蛋白序列中与所述CDR序列相邻的部分。例如,CDR3序列之前可以是FR3序列(的一部分)并且之后是FR4序列(的一部分))。此类片段还可包含二硫键,并且特别是分别连接CDR序列之前和之后的两个构架区域的二硫键(为了形成此类二硫键的目的,可以使用天然存在于所述构架区域中的半胱氨酸残基,或者可以将半胱氨酸残基合成地添加到或引入到所述构架区域中)。对于这些“加速片段(Expedite fragment)”的进一步描述,再次参见WO 2003/050531或WO 2009/127691。
化合物或构建体,特别是蛋白质或多肽(本文也分别称为“本文所述的化合物”或“本文所述的多肽”)可以包含或基本上由本文所述的一种或多种单结构域抗体(或其合适的片段)组成,并且任选地进一步包含一个或多个其他基团、残基、部分或结合单位。另外的基团、残基、部分、结合单位或氨基酸序列可以或不可以向本文所述的抗体(和/或向所述抗体所存在于的化合物或构建体)提供另外的功能性,并且可以或不可以改变本文所述的单结构域抗体的性质。
例如,此类另外的基团、残基、部分或结合单位可以是一个或多个另外的氨基酸序列,使得化合物或构建体是融合蛋白或融合多肽。在优选但非限制性的方面中,所述一个或多个其他基团、残基、部分或结合单位是免疫球蛋白序列。甚至更优选地,所述一个或多个其他基团、残基、部分或结合单位选自由以下组成的组:结构域抗体、适合用作结构域抗体的氨基酸序列、单结构域抗体、适合用作单结构域抗体的氨基酸序列、dAb、适合用作dAb的氨基酸序列、或VHH序列。
所述基团、残基、部分或结合单位可以例如是化学基团、残基、部分,所述化学基团、残基、部分本身可以具有或可以不具有生物和/或药理学活性。例如但非限制地,此类基团可以连接至本文所述的一种或多种单结构域抗体,以提供本文所述的氨基酸序列或多肽的“衍生物”,如本文进一步所述的。
还提供了化合物或构建体,所述化合物或构建体包含或基本上由本文所述的一种或多种衍生物组成,并且任选地进一步包含任选地经由一个或多个接头连接的一个或多个其他基团、残基、部分或结合单位。优选地,所述一个或多个其他基团、残基、部分或结合单位是氨基酸序列。
在上述化合物或构建体中,本文所述的一种或多种单结构域抗体和一个或多个基团、残基、部分或结合单位可以直接彼此连接和/或经由一个或多个合适的接头或间隔区连接。例如,当所述一个或多个基团、残基、部分或结合单位是氨基酸序列时,所述接头也可以是氨基酸序列,使得所得化合物或构建体是融合蛋白或融合多肽。
本文所述的单结构域抗体可用作形成本文所述的多肽的“结构单位”,例如通过将它们与其他基团、残基、部分或结合单位组合,以形成如本文所述的化合物或构建体(例如,本文所述的双互补位、双/多价和/或双/多特异性多肽),所述化合物或构建体在一个分子内组合一种或多种所需性质或生物学功能。
本文所述的化合物或多肽通常可以通过这样的方法来制备,所述方法包括至少一个将本文所述的一种或多种单结构域抗体与一个或多个另外的基团、残基、部分或结合单位连接,任选地经由一个或多个合适的接头连接,以提供本文所述的化合物或多肽的步骤。
本文所述的多肽还可以通过这样的方法来制备,所述方法通常至少包括以下步骤:提供编码本文所述的多肽的核酸,以合适的方式表达所述核酸,以及回收经表达的本文所述的多肽。此类方法可以通过任何合适的方式执行,这对于技术人员来说将是清楚的,例如基于本文进一步所述的方法和技术。
在实施方式中,与本文所述的对应单结构域抗体相比,本文所述的化合物或多肽可具有延长的半衰期。基于本文的进一步公开内容,此类化合物和多肽的一些优选的但非限制性的示例对于技术人员来说将变得清楚,并且例如包括:本文所述的氨基酸序列或多肽,所述氨基酸序列或多肽已经化学修饰(例如,借助于聚乙二醇化)以延长其半衰期;本文所述的单结构域抗体,所述单结构域抗体包含至少一个用于结合至血清蛋白(例如,血清白蛋白)的附加结合位点;或者包含至少一种本文所述的单结构域抗体的多肽,所述单结构域抗体连接到延长本文所述的单结构域抗体的半衰期的至少一个部分(并且特别是至少一个氨基酸序列)。本文所述的包含此类延长半衰期的部分或氨基酸序列的多肽的示例将基于本文的进一步公开内容对本领域的技术人员变得清楚;并且例如包括但不限于:多肽,在所述多肽中本公开的一种或多种单结构域抗体连接至一种或多种血清蛋白或其片段(例如,(人)血清白蛋白或其合适的片段)或连接至一个或多个可结合血清蛋白的结合单位(例如,结构域抗体、适合用作结构域抗体的氨基酸序列、单结构域抗体、适合用作单结构域抗体的氨基酸序列、dAb、适合用作dAb的氨基酸序列、或可以结合血清蛋白(例如血清白蛋白(例如,人血清白蛋白))、血清免疫球蛋白(例如IgG)或转铁蛋白的VHH序列);多肽,在所述多肽中本文所述的单结构域抗体连接至Fc部分(例如,人Fc)或其合适的部分或片段;或者多肽,在所述多肽中本文所述的一种或多种单结构域抗体适合连接至一个或多个可以结合血清蛋白的小蛋白或肽(例如,WO 91/01743、WO 01/45746和WO 02/076489中所述的蛋白质和肽)。
一般而言,本文所述的具有延长的半衰期的化合物或多肽优选具有为本文所述的对应单结构域抗体的半衰期的至少1.5倍,优选至少2倍,例如至少5倍、例如至少10倍或大于20倍的半衰期。例如,具有延长的半衰期的本文所述的化合物或多肽可以具有与本文所述的对应单结构域抗体相比延长了大于1小时,优选大于2小时,更优选大于6小时,例如大于12小时,或甚至大于24小时、48小时或72小时的半衰期。
本文所述的化合物或多肽可具有与本文所述的对应单结构域抗体相比延长了大于1小时,优选大于2小时,更优选大于6小时,例如大于12小时,或甚至大于24小时、48小时或72小时的血清半衰期。
本文所述的化合物或多肽可表现出在人类中至少约12小时,优选至少24小时,更优选至少48小时,甚至更优选至少72小时或更长时间的血清半衰期。例如,本文所述的化合物或多肽可具有至少5天(例如,约5天至10天),优选至少9天(例如,约9天至14天),更优选至少约10天(例如,约10天至15天),或至少约11天(例如,约11天至16天),更优选至少约12天(例如,约12天至18天或更长时间),或大于14天(例如,约14天至19天)的半衰期。
本文所述的单结构域抗体和抗体片段或变体(包括衍生物)可以包含例如一种或多种氨基酸序列改变(氨基酸残基的添加、缺失、取代和/或插入)。这些改变可以在一个或多个构架区和/或一个或多个CDR中进行。本文所述的单结构域抗体(包括抗体片段、及其变体和衍生物)可以通过本领域已知的方法常规制备。可以根据本公开采用包含或替代地由序列在本文中具体公开的VHH结构域和CDR中的任一者的片段或变体组成的分子。本公开还提供了编码这些单结构域抗体和分子(包括片段、变体和衍生物)的核酸分子。
在一个实施方式中,描述了单结构域抗体(包括包含或替代地由抗体片段或变体组成的分子)的组,其中组成员对应于一个、两个、三个、四个、五个、十个、十五个、二十个或更多个不同的本文描述的抗体(例如,完整抗体、Fab、F(ab')2片段、Fd片段、二硫键连接的Fv(sdFv)、抗独特型(抗Id)抗体和scFv)。本公开还提供了抗体混合物,其中所述混合物对应于本公开的一种、两种、三种、四种、五种、十种、十五种、二十种或更多种不同的抗体(例如,完整抗体、Fab、F(ab')2片段、Fd片段、二硫键连接的Fv(sdFv)、抗独特型(抗Id)抗体和scFv)。
在一个实施方式中,组合物可包含或由一种、两种、三种、四种、五种、十种、十五种、二十种或更多种本文所述的单结构域抗体(包括包含或由抗体片段或其变体组成的分子)组成。本文所述的组合物可包含或替代地由一种或多种单结构域抗体或其片段或变体的一种、两种、三种、四种、五种、十种、十五种、二十种或更多种氨基酸序列组成。或者,本文所述的组合物可包含或替代地由编码本公开的一种或多种单结构域抗体的核酸分子组成。
在一个实施方式中,融合蛋白可包含本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)和异源多肽(例如,与抗体或抗体结构域无关的多肽)。本公开还提供了编码这些融合蛋白的核酸分子。本文所述的组合物可包含或替代地由一种、两种、三种、四种、五种、十种、十五种、二十种或更多种本公开的融合蛋白组成。或者,本文所述的组合物可包含或替代地由编码一种、两种、三种、四种、五种、十种、十五种、二十种或更多种本公开的融合蛋白的核酸分子组成。
在一个实施方式中,提供了重组核酸分子,所述重组核酸分子通常是分离的,编码本文所述的单结构域抗体(包括可包含或由抗体片段或其变体组成的分子)。本公开还提供了用本文所述的核酸分子转化的宿主或宿主细胞及其子代。本公开还提供了一种用于生产本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)的方法。本公开还提供了一种从重组核酸分子表达本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)的方法。本公开的这些和其他方面将在下文进一步详细描述。
在一个实施方式中,用于检测、诊断和/或预测动物,优选地哺乳动物,最优选地人类中的冠状病毒感染,优选地SARS-CoV-2感染(COVID-19)的方法和组合物可以包括使用免疫特异性结合SARS-CoV-2(例如,SARS-CoV-2的RBD)的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)。可用本文所述的单结构域抗体检测、诊断或预测的疾病和疾患包括但不限于COVID-19、急性呼吸衰竭、肺炎、急性呼吸窘迫综合征(acuterespiratory distress syndrome,ARDS)、急性肝损伤、急性心脏损伤、继发感染、急性肾损伤、感染性休克、弥散性血管内凝血、血栓、多系统炎症综合征、慢性疲劳、横纹肌溶解症以及它们的组合。
在一个实施方式中,用于在动物,优选地哺乳动物,最优选地人类中预防、治疗或改善冠状病毒感染,优选地SARS-CoV-2感染(COVID-19)的方法和组合物可包括向所述动物施用有效量的一种或多种免疫特异性结合SARS-CoV-2(例如,SARS-CoV-2的RBD)的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)。可以通过施用有效量的一种或多种本文所述的单结构域抗体或分子来预防、治疗或抑制的疾病和疾患包括但不限于COVID-19、急性呼吸衰竭、肺炎、急性呼吸窘迫综合征(acute respiratory distresssyndrome,ARDS)、急性肝损伤、急性心脏损伤、继发感染、急性肾损伤、感染性休克、弥散性血管内凝血、血栓、多系统炎症综合征、慢性疲劳、横纹肌溶解症以及它们的组合。
附图说明
为了进一步理解本文所述的性质、目的和优点,应当参考结合以下附图阅读的以下详细描述,其中相同的附图标记表示相同的元件。
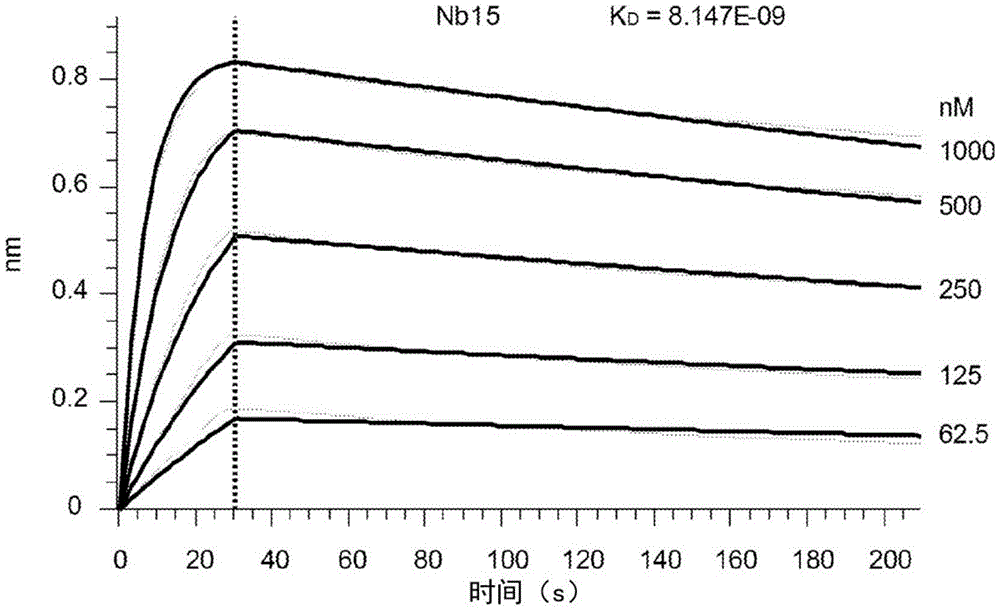
图1A至图1F示出了本文所述的六种示例性单结构域抗体的亲和力测定结果。图1A示出了示例性单结构域抗体Nb15(SEQ ID NO:1)的亲和力测定结果。图1B示出了示例性单结构域抗体msNb12(SEQ ID NO:13)的亲和力测定结果。图1C示出了示例性单结构域抗体Nb17(SEQ ID NO:2)的亲和力测定结果。图1D示出了示例性单结构域抗体Nb19(SEQ ID NO:3)的亲和力测定结果。图1E示出了示例性单结构域抗体Nb56(SEQ ID NO:4)的亲和力测定结果。图1F示出了示例性单结构域抗体mNb30(SEQ ID NO:14)的亲和力测定结果。
图2A至图2F示出了本文所述的六种示例性单结构域抗体Fc融合构建体的亲和力测定结果。图2A示出了mNb30-Fc单体(SEQ ID NO:117)的亲和力测定结果。图2B示出了Nb15-Fc三聚体(SEQ ID NO:118)的亲和力测定结果。图2C示出了Nb17-Fc三聚体(SEQ IDNO:119)的亲和力测定结果。图2D示出了Nb19-Fc三聚体(SEQ ID NO:120)的亲和力测定结果。图2E示出了Nb56-Fc三聚体(SEQ ID NO:121)的亲和力测定结果。图2F示出了msNb12-Fc三聚体(SEQ ID NO:122)的亲和力测定结果。
图3A和图3B示出了来自用SARS-CoV-2 RBD和刺突多肽(分别为SEQ ID NO:129和SEQ ID NO:130)两者免疫的美洲驼的抗体滴度。针对SARS-CoV-2 RBD(图3A)和SARS-CoV-2刺突(图3B)的良好免疫反应是在免疫后24-38天内获得的。
图4A和图4B示出了来自用SARS-CoV-2 RBD和刺突多肽(分别为SEQ ID NO:129和SEQ ID NO:130)(图4A)或用单独的SARS-CoV-2刺突多肽(SEQ ID NO:130)(图4B)免疫的表达骆驼科动物抗体基因的转基因小鼠的抗体滴度。
具体实施方式
在进一步描述主题公开内容之前,应当理解本公开不限于下文描述的本公开的特定实施方式,因为可以对所述特定实施方式进行变型并且仍然落入所附权利要求书的范围内。还应当理解的是,所采用的术语是为了描述特定实施方式的目的,而不是为了限制。相反,本公开的范围将由所附权利要求书确定。
在本说明书和所附权利要求书中,除非上下文另有明确规定,否则单数形式“一”、“一个(种)”和“所述”包括复数个指代物。除非另有定义,否则本文所用的所有技术和科学术语与本公开所属领域的普通技术人员通常理解的含义相同。
定义
如本文所用,除非另有说明,否则术语“SARS-CoV-2”泛指任何SARS-CoV-2序列,包括野生型SARS-CoV-2和任何变体或突变SARS-CoV-2序列。本文提供了某些已知的SARS-CoV-2变体的示例,但除非另有说明,否则本公开不限于这些SARS-CoV-2变体。同样,除非另有说明,否则术语“SARS-COV-1”、“MERS-COV”、“HCOV-OC43”、“HCOV-HKU1”、“HCOV-NL63”和“HCOV-229E”泛指野生型和其任何变体或突变体。
如本文所用,术语“免疫特异性结合SARS-CoV-2的抗体”、“特异性结合SARS-CoV-2的抗体”和“抗SARS-CoV-2抗体”泛指能够结合SARS-CoV-2,优选SARS-CoV-2的受体结合结构域(“RBD”)的抗体。
可以特异性地(例如,免疫特异性地)结合特定抗原决定簇、表位、抗原或蛋白质(或其至少一部分、片段或表位)、对所述特定抗原决定簇、表位、抗原或蛋白质(或其至少一部分、片段或表位)具有亲和力和/或具有特异性的氨基酸序列(例如,单结构域抗体、本文所述的多肽,或通常地抗原结合蛋白或多肽或其片段)被称为“对抗”或“针对”所述抗原决定簇、表位、抗原或蛋白质。
如本文所用,术语“特异性”泛指特定抗原结合分子或抗原结合蛋白(例如,本文所述的单结构域抗体或多肽)分子可结合的不同类型的抗原或抗原决定簇的数量。抗原结合蛋白的特异性可以基于亲和力和/或亲合力来确定,所述亲和力和/或亲合力可以例如使用用于测量抗原结合分子(例如,本文所述的单结构域抗体或多肽)与相关抗原之间的结合的已知技术来测量。通常,抗原结合蛋白(例如,本文所述的单结构域抗体和/或多肽)将以10
如本文所用,免疫特异性结合SARS-CoV-2的RBD的抗体具有结合SARS-CoV-2的RBD,使得冠状病毒,优选地SARS-CoV-2病毒被灭活(“中和”)的性质。可以测试抗SARS-CoV-2抗体候选物的此类活性,例如通过将抗SARS-CoV-2 RBD抗体吸附到固定化的SARS-CoV-2的RBD,然后用过量的分离的ACE2(血管紧张素转换酶2)多肽洗脱所吸附的抗体。如果如通过例如用放射性标记的可溶性SARS-CoV-2 RBD对相应洗脱液执行放射免疫测定所确定,包含过量ACE2多肽的洗脱剂产生包含比在对照洗脱中由“空白”洗脱剂(不包含ACE2的相同洗脱剂)产生的洗脱液中存在的候选抗体浓度更大浓度的候选抗体的洗脱液,则候选抗体与ACE2多肽竞争结合SARS-CoV-2 RBD,并且因此削弱或消除SARS-CoV-2 RBD与ACE2的结合。
如本文所用,具有“中和SARS-CoV-2”性质或能力的抗SARS-CoV-2 RBD的抗体泛指能够降低或抑制冠状病毒,优选地SARS-CoV-2活性的抗SARS-CoV-2 RBD的抗体。可以测试抗SARS-CoV-2 RBD的抗体候选物的此类活性,例如,通过以一种或多种生物测定测量SARS-CoV-2感染和/或活性的预防。抗SARS-CoV-2 RBD的抗体可结合包含SEQ ID NO:124、SEQ IDNO:125、SEQ ID NO:126、SEQ ID NO:127、SEQ ID NO:128或它们的组合的氨基酸序列的表位。
如本文所用,“冠状病毒”泛指作为冠状病毒组的成员的病毒。冠状病毒因其表面上的冠状刺突而得名。存在冠状病毒的四个主要亚组,被称为α、β、γ和δ。优选的冠状病毒包括但不限于SARS-CoV-1、MERS-CoV、SARS-CoV-2、HCoV-OC43、HCoV-HKU1、HCoV-NL63、HCoV-229E或它们的组合。
“抗体”(Ab)和“免疫球蛋白”(Ig)是具有相同结构特性的糖蛋白。虽然抗体表现出对特定抗原的结合特异性,但免疫球蛋白包括抗体和其他缺乏抗原特异性的抗体样分子两者。
“天然抗体和免疫球蛋白”或“常规抗体和免疫球蛋白”通常是由两条相同的轻(L)链和两条相同的重(H)链组成的约150000道尔顿的异四聚糖蛋白。每条轻链通过一个共价二硫键与重链连接,而二硫键的数量在不同免疫球蛋白同种型的重链之间有所不同。每条重链和轻链还具有规则间隔的链内二硫键。每条重链都具有在一个末端的可变结构域(VH),之后是多个恒定结构域。每条轻链都具有在一个末端的可变结构域(VL),并在其另一端具有一个恒定结构域;所述轻链恒定结构域与重链的第一恒定结构域对齐,并且所述轻链可变结构域与重链的可变结构域对齐。特定的氨基酸残基被认为在轻链可变结构域与重链可变结构域之间形成界面(Clothia等人,J.Mol.Biol.186:651(1985);Novotny和Haber,Proc.Natl.Acad.Sci.U.S.A.82:4592(1985))。
术语“可变”泛指以下事实:可变结构域的某些部分在抗体之间在序列上有很大差异并且用于每个特定抗体对其特定抗原的结合和特异性。然而,可变性并未均匀分布在抗体的整个可变结构域中。所述可变性集中在轻链可变结构域和重链可变结构域中被称为互补决定区(CDR)或高变区的三个区段中。可变结构域的更高度保守部分被称为构架(framework,FR)。天然重链和轻链的可变结构域各自包含通过三个CDR连接的主要采用β-折叠结构的四个FR区,所述四个FR区形成环连接,并且在一些情况下形成β-折叠结构的部分。每条链中的CDR被FR区域紧紧地保持在一起,并且与来自另一条链的CDR一起,有助于形成抗体的抗原结合位点(参见Kabat等人,Sequences of Proteins of ImmunologicalInterest,第五版,National Institute of Health,Bethesda,Md.(1991))。恒定结构域不直接参与抗体与抗原的结合,但表现出各种效应子功能,例如抗体在抗体依赖性细胞毒性中的参与。
术语“单可变结构域”或“免疫球蛋白单可变结构域”定义了其中抗原结合位点存在于单免疫球蛋白结构域上并由所述单免疫球蛋白结构域形成的分子。这将单可变结构域与“常规”免疫球蛋白或其片段区分开来,其中两个免疫球蛋白结构域,特别是两个“可变结构域”相互作用以形成抗原结合位点。如上所述,在常规免疫球蛋白中,通常,重链可变结构域(VH)和轻链可变结构域(VL)相互作用以形成抗原结合位点。在这种情况下,VH和VL两者的互补决定区(CDR)将有助于抗原结合位点,例如总共6个CDR将参与抗原结合位点形成。
相反,免疫球蛋白单可变结构域的结合位点由单一VH或VL结构域形成。因此,免疫球蛋白单可变结构域的抗原结合位点由不多于三个CDR形成。术语“免疫球蛋白单可变结构域”确实包含其中抗原结合位点由单可变结构域形成的常规免疫球蛋白的片段。
一般来说,免疫球蛋白单可变结构域将是基本上由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成的氨基酸序列;或此类氨基酸序列的任何合适片段(其则通常将包含形成CDR中的至少一个CDR的氨基酸残基中的至少一些氨基酸残基)。此类免疫球蛋白单可变结构域和片段最优选地使得它们包含免疫球蛋白折叠或能够在合适的条件下形成免疫球蛋白折叠。因此,免疫球蛋白单可变结构域可以例如包含轻链可变结构域序列(例如V,L结构域序列)或其合适的片段;或重链可变结构域序列(例如,VH结构域序列或VHH结构域序列)或其合适的片段;只要它能够形成单抗原结合单位(例如,基本上由免疫球蛋白单可变结构域组成的功能性抗原结合单位,使得单抗原结合结构域不需要与另一可变结构域相互作用来形成功能性抗原结合单位,如对于存在于例如常规抗体中的可变结构域和需要与另一可变结构域相互作用(例如,通过VH/VL相互作用)来形成功能性抗原结合结构域的scFv片段情况而言)即可。
在实施方式中,免疫球蛋白单可变结构域是轻链可变结构域序列(例如,VL结构域序列)或重链可变结构域序列(例如,VH结构域序列)。更具体地,单可变结构域可以是衍生自常规四链抗体的重链可变结构域序列或衍生自重链抗体的重链可变结构域序列。
免疫球蛋白单可变结构域可以是结构域抗体(或适合用作结构域抗体的氨基酸序列)、单结构域抗体(或适合用作单结构域抗体的氨基酸序列)、“dAb”(或适合用作dAb的氨基酸序列)、VHH结构域序列、其他免疫球蛋白单可变结构域,或它们中的任一者的任何合适片段。对于(单)结构域抗体的一般描述参见本文引用的技术以及EP 0 368 684。对于术语“dAb”,参见Ward等人1989(Nature 341:544-546);Holt等人2003(Trends Biotechnol.21:484-490);以及例如WO 04/068820、WO 06/030220和WO 06/003388。还应当指出的是,虽然因为它们不是哺乳动物来源的,所以在本文描述的上下文中不太优选,但是免疫球蛋白单可变结构域可以来源于某些鲨鱼物种(例如,“IgNAR结构域”,参见例如WO 05/18629)。
常规抗体的木瓜蛋白酶消化产生两个相同的抗原结合片段,被称为各自具有单一抗原结合位点的“Fab”片段,以及残留“Fc”片段,所述片段的名称反映了其易于结晶的能力。胃蛋白酶处理产生F(ab')2片段,该片段具有两个抗原结合位点并且仍然能够交联抗原。
“Fv”是包含完整抗原识别和结合位点的最小抗体片段。在双链Fv物质中,这种区域由紧密、非共价缔合的一个重链可变结构域和一个轻链可变结构域的二聚体组成。在单链Fv物质中,一个重链可变结构域和一个轻链可变结构域可以通过柔性肽接头共价连接,使得轻链和重链可以类似于双链Fv物质中的“二聚体”结构缔合。正是在这种配置中,每个可变结构域的三个CDR相互作用以在VH-VL二聚体的表面上定义抗原结合位点。六个CDR共同赋予抗体抗原结合特异性。然而,单可变结构域(或仅包含三个特异于抗原的CDR的Fv的一半)也可以具有识别和结合如本文所述的抗原的能力。
Fab片段还包含轻链的恒定结构域和重链的第一恒定结构域(CH1)。Fab'片段与Fab片段的不同之处在于在重链CH1结构域的羧基末端处添加了几个残基,包括来自抗体铰链区的一个或多个半胱氨酸。Fab'-SH是本文对Fab'的命名,其中恒定结构域的半胱氨酸残基带有游离硫醇基团。F(ab')2抗体片段最初是作为其之间具有铰链半胱氨酸的Fab'片段对产生的。抗体片段的其他化学偶联也是已知的。
来自任何脊椎动物物种的抗体(免疫球蛋白)的“轻链”可以基于其恒定结构域的氨基酸序列被分配两种明显不同的类型中的一种类型,所述两种明显不同的类型被称为卡帕(κ)和拉姆达(λ)。
取决于其重链恒定区的氨基酸序列,免疫球蛋白可被分配不同的类别。存在五种主要类别的免疫球蛋白:IgA、IgD、IgE、IgG和IgM,并且这些免疫球蛋白中的几种免疫球蛋白可以进一步分为亚类(同种型),例如IgG1、IgG2、IgG3、IgG4、IgA1和IgA2。与不同类别的免疫球蛋白对应的重链恒定结构域分别被称为α、δ、ε、γ和μ。不同类别的免疫球蛋白的亚基结构和三维构型是众所周知的。“Therapeutic Antibody Engineering”(第1版)Strohl&Strohl Woodhead Publishing(2012)。
“抗体片段”包含完整抗体的一部分,通常是完整抗体的抗原结合区或可变区。抗体片段的示例包括Fab、Fab'、F(ab')2和Fv片段;双体抗体;单链抗体分子,包括单链Fv(scFv)分子;以及由抗体片段形成的多特异性抗体。“Human Monoclonal Antibodies:Methods and Protocols”(第2版)Steinitz(编辑)Humana Press(2019)。
“人”抗体(也称为“完全人”抗体)是包含人构架区和来自人免疫球蛋白的所有CDR的抗体。在一个示例中,构架和CDR来自相同来源的人类重链和/或轻链氨基酸序列。然而,来自一种人类抗体的构架可以经工程化以包括来自不同人类抗体的CDR。
“人源化”形式的非人(例如,鼠)抗体是嵌合免疫球蛋白、免疫球蛋白链或其片段(例如,Fv、Fab、Fab'、F(ab')2或抗体的其他抗原结合子序列),其包含来自非人免疫球蛋白的最小序列。在大多数情况下,人源化抗体是具有所需的特异性、亲和力和容量的人免疫球蛋白(受体抗体),其中来自受体互补决定区(CDR)的残基被来自非人物种(例如,小鼠、大鼠、兔或骆驼科)的CDR或合成序列(供体抗体)的残基取代。在一些情况下,人免疫球蛋白的Fv构架区(FR)残基被对应的非人残基替代。此外,人源化抗体可包含既不存在于受体抗体中也不存在于导入的CDR或构架序列中的残基。进行这些修饰是为了进一步完善和优化抗体性能。在一个实施方式中,所有CDR都来自人源化免疫球蛋白中的供体免疫球蛋白。不需要存在恒定区,但如果恒定区存在,则它们应该与人免疫球蛋白恒定区基本上相同,例如至少约85-90%,例如约95%或更多同一。因此,除了可能CDR以外的人源化免疫球蛋白的所有部分,与天然人免疫球蛋白序列的对应部分基本上相同。“人源化抗体”是包含人源化轻链免疫球蛋白和人源化重链免疫球蛋白的抗体。人源化抗体与提供CDR的供体抗体结合相同的抗原。人源化免疫球蛋白或抗体的受体构架可能具有有限数量的取自供体构架的氨基酸取代。人源化抗体或其他单克隆抗体可具有附加的保守氨基酸取代,所述附加的保守氨基酸取代基本上对抗原结合或其他免疫球蛋白功能没有影响。可以借助于基因工程来构建人源化免疫球蛋白。参见例如美国专利号5,585,089。
“单链Fv”或“scFv”抗体片段包含抗体的VH结构域和VL结构域,其中这些结构域存在于单个多肽链中。通常,scFv多肽进一步包含在VH结构域与VL结构域之间的多肽接头,这使scFv能够形成抗原结合所需的结构。有关scFv的综述,请参见The Pharmacology ofMonoclonal Antibodies,第113卷,Rosenburg和Moore编辑,Springer-Verlag,New York,第269-315页(1994)。
术语“双体抗体”泛指具有两个抗原结合位点的小抗体片段,所述片段包含在同一多肽链(VH-VL)中连接到轻链可变结构域(VL)的重链可变结构域(VH)。通过使用太短以至于不允许在同一条链上的两个结构域之间配对的接头,这些结构域被迫与另一条链的互补结构域配对并产生两个抗原结合位点。双体抗体在例如EP 404,097;WO 93/11161;和Hollinger等人Proc.Natl.Acad.Sci.USA,90:6444-6448(1993)中有更全面的描述。
“分离的”抗体是已从其自然环境的组分中鉴定出、分离和/或回收的抗体。其自然环境的污染物组分是会干扰抗体的诊断或治疗用途的物质,并且可包括酶、激素和其他蛋白质或非蛋白质溶质。在优选的实施方式中,如通过Lowry方法测定的,抗体将被纯化(1)至抗体的大于95重量%,并且最优选地大于99重量%;(2)至足以获得通过使用旋转杯测序仪获得N末端或内部氨基酸序列的至少15个残基的程度;或(3)至在还原或非还原条件下使用考马斯蓝或优选地银染,通过SDS-PAGE得到同质行。分离的抗体包括重组细胞内的原位抗体,因为抗体的天然环境的至少一种组分将不存在。然而,通常,分离的抗体将通过至少一个纯化步骤来制备。
如本文所用的术语“变体”泛指这样的多肽,所述多肽具有与SARS-CoV-2多肽(例如,SARS-CoV-2 RBD多肽)、抗SARS-CoV-2抗体或其抗体片段相似或相同的功能,但不一定包含SARS-CoV-2多肽、抗SARS-CoV-2抗体或其抗体片段的相似或相同的氨基酸序列,或与SARS-CoV-2多肽、抗SARS-CoV-2抗体或其抗体片段具有相似或相同的结构。具有相似氨基酸同一性的变体泛指满足以下中的至少一项的多肽:(a)包含或替代地由以下组成的多肽:与SARS-CoV-2多肽、抗SARS-CoV-2的抗体或其抗体片段(包括具有本文所述的那些中的任一者的氨基酸序列的VHH结构域或CDR)的氨基酸序列至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%,至少90%、至少95%或至少99%同一的氨基酸序列;(b)由核苷酸序列编码的多肽,所述核苷酸序列的互补序列在严格条件下与具有至少5个氨基酸残基、至少10个氨基酸残基、至少15个氨基酸残基、至少20个氨基酸残基、至少25个氨基酸残基、至少30个氨基酸残基、至少40个氨基酸残基、至少50个氨基酸残基、至少60个氨基酸残基、至少70个氨基酸残基、至少80个氨基酸残基、至少90个氨基酸残基、至少100个氨基酸残基、至少125个氨基酸残基、或至少150个氨基酸残基的编码SARS-CoV-2多肽或其片段、抗SARS-CoV-2的抗体或其抗体片段(包括具有本文所述的那些中的任一者的氨基酸序列的VHH结构域或CDR)的核苷酸序列杂交;以及(c)由与编码SARS-CoV-2多肽或其片段、抗SARS-CoV-2的抗体或其抗体片段(包括具有本文所述的氨基酸序列的VHH结构域或CDR)的核苷酸序列至少30%、至少35%、至少40%、至少45%、至少50%、至少55%、至少60%、至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%、或约100%同一的核苷酸序列编码的多肽。本文所述的与SARS-CoV-2多肽或其片段、抗SARS-CoV-2的抗体或其抗体片段具有相似结构的多肽,泛指与本文所述的SARS-CoV-2多肽或其片段、抗SARS-CoV-2的抗体或其抗体片段具有相似二级、三级或四级结构的多肽。多肽的结构可以通过本领域技术人员已知的方法确定,所述方法包括但不限于X射线晶体学、核磁共振和晶体电子显微镜。为了确定两个氨基酸序列或两个核酸序列的同一性百分比,将序列进行比对以达到最佳比较目的(例如,可以在第一氨基酸或核酸序列的序列中引入空位以与第二氨基酸或核酸序列进行最佳比对)。然后比较在对应氨基酸位置或核苷酸位置的氨基酸残基或核苷酸。当第一序列中的某一位置被第二序列中对应位置处的相同氨基酸残基或核苷酸占据时,则分子在该位置处是相同的。两个序列之间的同一性百分比是序列共享的相同位置数的函数(例如,%同一性=相同重叠位置的数量/位置总数×100%)。在一个实施方式中,两个序列的长度相同。
可以使用本领域技术人员已知的数学算法来完成两个序列之间的同一性百分比的确定。用于比较两个序列的数学算法的示例是Karlin和AltschulProc.Natl.Acad.Sci.USA 87:2264-2268(1990)的算法,如在Karlin和AltschulProc.Natl.Acad.Sci.USA 90:5873-5877(1993)中所修改的。Altschul等人J.Mal.Biol.215:403-410(1990)的BLASTn和BLASTx程序已经引入了这种算法。可以用BLASTn程序执行BLAST核苷酸搜索,得分=100,字长=12,以获得与本文所述的核酸分子同源的核苷酸序列。可以使用BLASTx程序执行BLAST蛋白质搜索,得分=50,字长=3,以获得与本文所述的蛋白质分子同源的氨基酸序列。为了获得用于比较目的的带空位比对,可以利用Gapped BLAST,如Altschul等人Nucleic Acids Res.25:3389-3402(1997)所述。或者,PSI-BLAST可用于执行迭代搜索,所述迭代搜索检测分子之间的远缘关系(出处相同)。当利用BLAST、Gapped BLAST和PSI-BLAST程序时,可以使用相应程序的默认参数(例如,BLASTx和BLASTn)。(参见ncbi.nlm.nih.gov)。
用于比较序列的数学算法的另一个示例是Myers和Miller,CABIOS(1989)的算法。作为GCG序列比对软件包的一部分的ALIGN程序(2.0版)已经并入了此类算法。本领域中已知的用于序列分析的其他算法包括如在Torellis和Robotti Comput.Appl.Biosci.,10:3-5(1994)中所述的ADVANCE和ADAM;以及在Pearson和Lipman Proc.Natl.Acad.Sci.85:2444-8(1988)中所述的FASTA。在FASTA内,ktup是设置搜索的灵敏度和速度的控制选项。
“保守”氨基酸取代是那些不实质上影响或降低蛋白质(例如针对SARS-CoV-2的抗体)亲和力的取代。例如,免疫特异性结合SARS-CoV-2的单结构域抗体可包含至多约1个、至多约2个、至多约5个、至多约10个、至多约15个、至多约20个、或至多约25个保守取代并免疫特异性地结合SARS-CoV-2多肽。术语“保守变体”还包括使用经取代的氨基酸代替未取代的亲本氨基酸,前提条件是抗体免疫特异性地结合SARS-CoV-2。非保守取代是那些降低与SARS-CoV-2结合的活性的取代。
提供功能上相似的氨基酸的保守氨基酸取代表是本领域普通技术人员所众所周知的。以下六组是被认为是彼此的保守取代的氨基酸的示例:
1)丙氨酸(A)、丝氨酸(S)、苏氨酸(T);
2)天冬氨酸(D)、谷氨酸(E);
3)天冬酰胺(N)、谷氨酰胺(Q);
4)精氨酸(R)、赖氨酸(K);
5)异亮氨酸(I)、亮氨酸(L)、甲硫氨酸(M)、缬氨酸(V);和
6)苯丙氨酸(F)、酪氨酸(Y)、色氨酸(W)。
如本文所用的术语“衍生物”泛指本文所述的包含或替代地由SARS-CoV-2多肽或其片段的氨基酸序列组成的变体多肽;或本文所述的免疫特异性地结合SARS-CoV-2的抗体,所述SARS-CoV-2已通过引入氨基酸残基取代、缺失或添加而改变。如本文所用的术语“衍生物”还泛指SARS-CoV-2多肽或其片段、或免疫特异性地结合SARS-CoV-2的抗体,所述SARS-CoV-2已例如通过将任何类型的分子共价附接至所述多肽而修饰。例如但非限制性地,SARS-CoV-2多肽或其片段、或抗SARS-CoV-2的抗体,可以例如通过糖基化、乙酰化、聚乙二醇化、磷酸化、酰胺化、通过已知的保护/封闭基团衍生化、蛋白水解切割、与细胞配体或其他蛋白质连接等进行修饰。SARS-CoV-2多肽或其片段的衍生物、或抗SARS-CoV-2的抗体或其片段的衍生物,可以使用本领域技术人员已知的技术,通过化学修饰来进行修饰,所述技术包括但不限于特异性化学切割、乙酰化、甲酰化、衣霉素的代谢合成等。进一步,SARS-CoV-2多肽或其片段的衍生物,或抗SARS-CoV-2的抗体或其片段的衍生物,可包含一个或多个非经典氨基酸。多肽衍生物具有与本文所述的SARS-CoV-2多肽或其片段、或抗SARS-CoV-2的抗体或其片段相似或相同的功能。
如本文所用的术语“表位”泛指SARS-CoV-2的在动物,优选地哺乳动物中具有抗原性或免疫原性活性的部分。具有免疫原性活性的表位是SARS-CoV-2的在动物体内引起抗体反应的部分。具有抗原活性的表位是SARS-CoV-2的一部分,如通过本领域已知的任何方法,例如通过本文所述的免疫测定法所确定的,抗体免疫特异性地结合至所述部分。抗原表位不一定必须是免疫原性的。
如本文所用的术语“片段”泛指这样的多肽,所述多肽包含具有SARS-CoV-2或免疫特异性地结合SARS-CoV-2的抗体的氨基酸序列的至少5个氨基酸残基、至少10个氨基酸残基、至少15个氨基酸残基、至少20个氨基酸残基、至少25个氨基酸残基,至少30个氨基酸残基、至少35个氨基酸残基、至少40个氨基酸残基、至少45个氨基酸残基、至少50个氨基酸残基、至少60个氨基酸残基、至少70个氨基酸残基残基、至少80个氨基酸残基、至少90个氨基酸残基、至少100个氨基酸残基、至少125个氨基酸残基、至少150个氨基酸残基、至少175个氨基酸残基、至少200个氨基酸残基、或至少250个氨基酸残基的氨基酸序列。
如本文所用的术语“融合蛋白”或“融合多肽”泛指这样的多肽,所述多肽包含或替代地由本文所述的抗SARS-CoV-2的抗体的氨基酸序列和异源多肽(例如,与抗体或抗体结构域无关的多肽)的氨基酸序列组成。
如本文所用的术语“宿主细胞”泛指用核酸分子转染的特定受试者细胞和此类细胞的子代或潜在子代。由于在后续世代中可能发生的突变或环境影响或核酸分子整合到宿主细胞基因组中,子代可能与用核酸分子转染的亲本细胞不同。
“治疗”泛指治疗性治疗和预防性或防护性措施。那些需要治疗的人包括那些已经患有所述疾患的人以及那些需要预防所述疾患的人。如本文所用,术语“治疗”泛指治疗疾病,阻止或减少疾病或其临床症状的发展,和/或缓解疾病,引起疾病或其临床症状的消退。疗法涵盖预防、治疗、补救、减少、缓和疾病、病征和/或疾病的症状,和/或提供疾病、病征和/或疾病的症状的免除。疗法涵盖缓和具有持续疾病病征和/或症状的患者的病征和/或症状。疗法还涵盖“预防”。出于疗法目的,术语“减少的”泛指病征和/或症状的临床上显著减少。疗法包括治疗复发或反复发作的病征和/或症状。疗法涵盖但不限于在任何时候排除病征和/或症状的出现以及减少现有病征和/或症状和消除现有病征和/或症状。疗法包括治疗慢性疾病(“维持”)和急性疾病。例如,治疗包括治疗或预防复发或病征和/或症状的反复发作。
如本文所用的“有效量”泛指化合物、抗体、抗原或细胞当施用于患者以治疗疾病时足以实现对疾病的这种治疗的量。有效量可以是有效预防的量和/或有效防止的量。有效量可以是有效减少的量,有效防止病征/症状的发生、降低病征/症状发生的严重程度、消除病征/症状的发生、减缓病征/症状发生的发展、防止病征/症状发生的发展,和/或有效预防病征/症状发生的量。“有效量”可取决于疾病及其严重程度以及待治疗患者的年龄、体重、病史、易感性和既存病况而变化。出于本公开的目的,术语“有效量”与“治疗有效量”同义。
如本文所用,“哺乳动物”泛指特征在于皮肤上覆盖着毛发并且雌性有用于滋养幼仔的产奶乳腺的哺乳动物纲的任何和所有温血脊椎动物。哺乳动物包括但不限于人类、家畜动物和农场动物以及动物园动物、运动动物或宠物动物。哺乳动物的示例包括但不限于羊驼、犰狳、水豚、猫、骆驼、黑猩猩、毛丝鼠、牛、狗、沙鼠、山羊、大猩猩、豚鼠、仓鼠、马、人类、狐猴、美洲驼、小鼠、非人灵长类动物、猪、大鼠、绵羊、鼩鼱(shrew)、松鼠和貘。哺乳动物包括但不限于牛科动物、犬科动物、马科动物、猫科动物、鼠科动物、绵羊、猪科动物、灵长类动物和啮齿动物物种。哺乳动物还包括由华盛顿哥伦比亚特区(Washington D.C.)的史密森学会国家自然历史博物馆(the National Museum of Natural History,SmithsonianInstitution)维护的《世界哺乳动物物种(the Mammal Species of the World)》中列出的任何和所有哺乳动物。类似地,术语“受试者”或“患者”包括人类和兽医受试者和/或患者。
抗SARS-CoV-2的抗体
除了常规的单克隆抗体(monoclonal antibody,mAb)之外,从骆驼科中分离的纯重链抗体(heavy-chain-only antibody,HCAb)也为开发治疗性抗体提供了替代方案。HCAb仅由两条重链组成,而没有轻链,因此仅包含单可变结构域(VHH),被称为单结构域抗体。除了对抗原具有与传统抗体相似的亲和力和特异性外,单结构域抗体还比大多数抗体尺寸更小并且显示出更高的稳定性。由于其结构,单结构域抗体可以容易地构建为多价或多特异性形式,并用方便的纯化步骤以较低的制造成本进行生产。此外,单结构域抗体可以容易地雾化并经由吸入器直接递送到肺部,这使得所述单结构域抗体特别有希望用于开发靶向呼吸道病原体(例如SARS-CoV-2)的中和抗体。吸入制剂允许在疾病的早期阶段更容易地在医院外施用,这对于对抗COVID-19大流行非常重要。
根据上述本领域中所用的术语,天然存在的重链抗体中所存在的可变结构域在本文中被称为“VHH结构域”,以便将它们与常规四链抗体中所存在的重链可变结构域(其在本文中被称为“VH结构域”)和常规四链抗体中所存在的轻链可变结构域(其在本文中被称为“VL结构域”)区分开。
如本文所述,VHH结构域具有许多不同的结构特性和功能性质,这使得分离的VHH结构域和包含所述分离的VHH结构域的蛋白质非常有利于用作功能性抗原结合结构域或蛋白质。特别地,并且不限于此,VHH结构域(其本质上已经被“设计”为在不存在轻链可变结构域且不与轻链可变结构域发生任何相互作用的情况下功能性地结合抗原)可以充当单个、相对较小的、功能性的抗原结合结构单位、结构域或蛋白质。这将VHH结构域与常规四链抗体的VH结构域和VL结构域区分开来,所述常规四链抗体本身通常不适合作为单抗原结合蛋白或结构域进行实践应用,而是需要以某种形式与另一者组合以提供功能性抗原结合单位(例如,常规抗体片段,例如Fab片段或scFv片段,所述常规抗体片段由共价连接至VL结构域的VH结构域组成)。
由于这些独特的性质,使用VHH结构域作为单抗原结合蛋白或作为抗原结合结构域(例如,作为较大蛋白或多肽的一部分)提供了优于使用常规VH和VL结构域、scFv或常规抗体片段(例如,Fab片段或F(ab')
在实施方式中,本公开提供了免疫特异性结合SARS-CoV-2的单结构域抗体,特别是结合SARS-CoV-2的受体结合结构域(RBD)的单结构域抗体;以及包含至少一种此类单结构域抗体的蛋白质和/或多肽。
在实施方式中,本公开提供了免疫特异性结合SARS-CoV-2的单结构域抗体以及包含所述单结构域抗体的蛋白质和/或多肽,与常规的针对SARS-CoV-2的抗体或其片段相比,与可以基于这种常规抗体或抗体片段的构建体(例如,Fab'片段、F(ab')
-在单价格式、多价格式(例如二价格式)和/或多特异性格式(例如,本文所述的多特异性格式中的一种多特异性格式)时增加的对SARS-CoV-2的亲和力和/或亲合力;
-更适合以多价格式(例如,以二价格式)进行格式化;
-更适合以多特异性格式(例如,本文所述的多特异性格式中的一种多特异性格式)进行格式化;
-提高的对“人源化”取代的适用性或易感性(如本文所述);
-在单价格式、多价格式(例如,二价格式)和/或多特异性格式(例如,本文所述的多特异性格式中的一种多特异性格式)时更低的免疫原性;
-在单价格式、多价格式(例如,二价格式)和/或多特异性格式(例如,本文所述的多特异性格式中的一种多特异性格式)时增加的稳定性;
-在单价格式、多价格式(例如,二价格式)和/或多特异性格式(例如,本文所述的多特异性格式中的一种多特异性格式)时增加的针对SARS-CoV-2的特异性;
-降低的或在需要时增加的与其他病毒(例如其他冠状病毒)的交叉反应性;和/或
-在单价格式、多价格式(例如,二价格式)和/或多特异性格式(例如,本文所述的多特异性格式中的一种多特异性格式)时对于药用(包括预防用途和/或治疗用途)和/或诊断用途(包括但不限于用于诊断测定)而言所需的一种或多种其他改进的性质。
如本文中针对本文所述的氨基酸序列一般描述的,本文所述的单结构域抗体优选地处于基本上分离的形式(如本文所述),或形成蛋白质或多肽的一部分(如本文所述),所述蛋白质或多肽可以包含或基本上由一种或多种本文所述的单结构域抗体组成,并且可以任选地进一步包含一种或多种另外的氨基酸序列(均任选地经由一个或多个合适的接头连接)。例如但非限制,本文所述的一种或多种氨基酸序列可用作此类蛋白质或多肽中的结合单位,所述蛋白质或多肽可任选地包含可充当结合单位的一种或多种另外的氨基酸序列(例如,针对除SARS-CoV-2之外的一种或多种靶标),从而提供本文所述的单价、多价或多特异性多肽,所述单价、多价或多特异性多肽均如本文所述。具体地,此类蛋白质或多肽可以包含或基本上由一种或多种本文所述的单结构域抗体和任选的一种或多种其他单结构域抗体(例如,针对除SARS-CoV-2之外的靶标)组成,所述一种或多种本文所述的单结构域抗体和任选的一种或多种其他单结构域抗体均任选地经由一个或多个合适的接头连接,以便提供单价、多价或多特异性单结构域抗体构建体,如本文进一步所述。此类蛋白质或多肽还可以是基本上分离的形式(如本文所述)。
在本文所述的单结构域抗体中,SARS-CoV-2的结合位点优选地由CDR序列形成。任选地,除了至少一个用于结合SARS-CoV-2的结合位点之外,本文所述的单结构域抗体还可包含一个或多个用于结合其他抗原、蛋白质或靶标的另外的结合位点。对于引入此类第二结合位点的方法和位置,参见例如Keck和Huston,Biophysical Journal,71,1996年10月,2002-2011;EP 0 640 130;和WO 06/07260。
如本文对本文所述的氨基酸序列一般描述的,本文所述的单结构域抗体通常可以针对SARS-CoV-2的任何抗原决定簇、表位、部分、结构域、亚基或构象(在适用情况下)。
如本文已描述的,单结构域抗体的氨基酸序列和结构可以被认为—但不限于—由以下组成:四个构架区域或“FR”(或有时也称为“FW”),其在本领域和本文中被称为“构架区域1”或“FR1”;“构架区域2”或“FR2”;“构架区域3”或“FR3”;和“构架区域4”或“FR4”;所述构架区域被三个互补决定区或“CDR”中断,所述三个互补决定区在本领域中被称为“互补决定区1”或“CDR1”;“互补决定区2”或“CDR2”;和“互补决定区3”或“CDR3”。单结构域抗体中存在的一些优选的构架序列和CDR(以及它们的组合)如本文所述。其他合适的CDR序列可以通过本文所述的方法获得。
根据非限制性但优选的方面,本文所述的单结构域抗体中存在的CDR序列使得:
-单结构域抗体可以以10
和/或使得:
-单结构域抗体可以以介于10
和/或使得:
-单结构域抗体可以以介于1s
优选地,本文所述的单结构域抗体中所存在的CDR序列使得本文所述的单价单结构域抗体(或仅含有一个本文所述的单结构域抗体的多肽)优选地使得其将以小于500nM,优选地小于200nM,更优选地小于10nM,例如小于500pM的亲和力结合SARS-CoV-2。
本文所述的单结构域抗体对SARS-CoV-2的亲和力可以以任何合适的方式确定,例如,使用本文所述的用于测量K
本文描述了用于本文所述的单结构域抗体(和包含所述单结构域抗体的多肽)与SARS-CoV-2的结合的一些优选的IC
在实施方式中,提供了一种免疫特异性结合SARS-CoV-2的单结构域抗体(如本文所述),所述单结构域抗体由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成,其中:
CDR1选自由以下组成的组:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
和/或
CDR2选自由以下组成的组:
d)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列。
和/或
CDR3选自由以下组成的组:
g)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
或此类氨基酸序列的任何合适的片段。
具体地,根据这种优选但非限制性的方面,本公开涉及一种免疫特异性结合SARS-CoV-2的单结构域抗体(如本文所述),所述单结构域抗体由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成,其中:
CDR1选自由以下组成的组:
a)SEQ ID NO:27至SEQ ID NO:40中任一者所示的氨基酸序列;
b)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:27至SEQ ID NO:40所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
并且
CDR2选自由以下组成的组:
d)SEQ ID NO:49至SEQ ID NO:62中任一者所示的氨基酸序列;
e)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:49至SEQ ID NO:62所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
并且
CDR3选自由以下组成的组:
g)SEQ ID NO:77至SEQ ID NO:91中任一者所示的氨基酸序列;
h)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:77至SEQ ID NO:91所示的氨基酸序列中的至少一个氨基酸序列具有3个、2个或1个氨基酸差异的氨基酸序列;
或此类氨基酸序列的任何合适的片段。
如本文一般描述的,当本文描述的单结构域抗体含有一个或多个根据b)和/或c)的CDR1序列时:
i)与根据a)的对应CDR相比,根据b)和/或c)的这种CDR中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与根据a)的对应CDR相比,根据b)和/或c)的CDR优选仅含有氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)根据b)和/或c)的CDR可以是借助于使用一种或多种已知的亲和力成熟技术进行亲和力成熟而衍生自根据a)的CDR的CDR。
类似地,当本文所述的单结构域抗体含有一个或多个根据e)和/或f)的CDR2序列时:
i)与根据d)的对应CDR相比,根据e)和/或f)的这种CDR中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与根据d)的对应CDR相比,根据e)和/或f)的CDR优选仅含有氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)根据e)和/或f)的CDR可以是借助于使用一种或多种已知的亲和力成熟技术进行亲和力成熟而衍生自根据d)的CDR的CDR。
类似地,当本文所述的单结构域抗体含有一个或多个根据h)和/或i)的CDR3序列时:
i)与根据g)的对应CDR相比,根据h)和/或i)的这种CDR中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与根据g)的对应CDR相比,根据h)和/或i)的CDR优选仅含有氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)根据h)和/或i)的CDR可以是借助于使用一种或多种已知的亲和力成熟技术进行亲和力成熟而衍生自根据g)的CDR的CDR。
应当理解,最后三个段落通常适用于包含一个或多个根据b)、c)、e)、f)、h)或i)的CDR1序列、CDR2序列和/或CDR3序列的本文所述的任何单结构域抗体。
包含一个或多个以上列出的CDR的单结构域抗体是优选的;包含两个或更多个以上列出的CDR的单结构域抗体是更优选的;包含三个以上列出的CDR的单结构域抗体是最优选的。
CDR序列的一些优选但非限制性的组合以及CDR序列和构架序列的优选组合在下表1中提到,所述表1列出了本文所述的许多示例性单结构域抗体中存在的CDR序列和构架序列。出现在相同的例示性单结构域抗体中的CDR1序列、CDR2序列和CDR3序列的组合(例如,在表1中的同一行上提到的CDR1序列、CDR2序列和CDR3序列)将通常是优选的,但是本发明在其最广泛的范围内不限于此,并且还包括表1中提到的CDR序列的其他合适的组合。而且,出现在相同的例示性单结构域抗体中的CDR序列和构架序列的组合(例如,在表1中的同一行上提到的CDR序列和构架序列)将通常是优选的,但是本发明在其最广泛的范围内不限于此,并且还包括表1中提及的CDR序列和构架序列的其他合适的组合,以及此类CDR序列和其他合适的构架序列的组合,例如,如本文进一步描述的。
此外,在包含表1中提到的CDR组合的本文所述的单结构域抗体中,每个CDR可以被选自由以下组成的组的CDR替代:与所提及的CDR具有至少80%、至少90%、至少95%或至少99%的序列同一性的氨基酸序列;
其中:
i)与表1中提及的对应CDR序列相比,此类CDR中的任何氨基酸取代优选是保守氨基酸取代;和/或
ii)与表1中提及的对应CDR序列相比,任何此类CDR序列优选仅包含氨基酸取代,并且没有氨基酸缺失或插入;和/或
iii)任何此类CDR序列是借助于已知的亲和力成熟技术衍生的CDR,并且具体地是从表1中提及的对应CDR序列开始。
表1中提及的CDR序列的组合以及CDR序列和构架序列的组合将通常是优选的。
表1.例示性抗SARS-CoV-2的单结构域抗体的构架序列和CDR序列
/>
/>
本文提供了例示性单结构域抗体,所述单结构域抗体以高亲和力免疫特异性地结合SARS-CoV-2并中和SARS-CoV-2和SARS-CoV-2变体。例如,这些单结构域抗体已被证明阻断培养中的细胞的SARS-CoV-2感染和SARS-CoV-2变体感染。
这些例示性单结构域抗体的氨基酸和核苷酸序列分别显示在以下表2和表3中。
表2.例示性抗SARS-CoV-2单结构域抗体的氨基酸序列
/>
表3.编码例示性抗SARS-CoV-2的单结构域抗体的核苷酸序列
/>
/>
/>
/>
在实施方式中,本文所述的单结构域抗体中存在的CDR1序列、CDR2序列和CDR3序列中的至少一者选自由以下组成的组:分别在表1中列出的CDR1序列、CDR2序列和CDR3序列;或者选自分别与分别在表1中列出的CDR1序列、CDR2序列和CDR3序列中的至少一者具有至少80%,优选至少90%,更优选至少95%,甚至更优选至少99%同一性的CDR1序列、CDR2序列和CDR3序列的组;和/或选自由以下组成的组:分别与分别在表1中列出的CDR1序列、CDR2序列和CDR3序列中的至少一者具有3个、2个或仅1个氨基酸差异的CDR1序列、CDR2序列和CDR3序列。
CDR序列优选地经选择为使得本文所述的单结构域抗体以如本文所述的亲和力(测量和/或表达为K
在实施方式中,本文所述的单结构域抗体中存在的至少CDR3序列选自由以下组成的组:在表1中列出的CDR3序列;或者选自与表1中列出的CDR3序列具有至少80%、至少90%、至少95%或至少99%同一性的CDR3序列的组;和/或选自由以下组成的组:与表1中列出的CDR3序列具有3个、2个或仅1个氨基酸差异的CDR3序列。
优选地,本文所述的单结构域抗体中存在的CDR1序列、CDR2序列和CDR3序列中的至少两者选自由以下组成的组:分别在表1中列出的CDR1序列、CDR2序列和CDR3序列;或者选自由以下组成的组:分别与分别在表1中列出的CDR1序列、CDR2序列和CDR3序列中的至少一者具有至少80%、至少90%、至少95%或至少99%同一性的CDR1序列、CDR2序列和CDR3序列;和/或选自由以下组成的组:分别与分别在表1中列出的CDR1序列、CDR2序列和CDR3序列中的至少一者具有3个、2个或仅1个氨基酸差异的CDR1序列、CDR2序列和CDR3序列。
具体地,本文所述的单结构域抗体中存在的至少CDR3序列选自由以下组成的组:表1中列出的CDR3序列,或者分别与表1中列出的CDR3序列中的至少一者具有至少80%、至少90%、至少95%或至少99%序列同一性的CDR3序列的组;并且所存在的CDR1序列和CDR2序列中的至少一者选自由以下组成的组:分别在表1中列出的CDR1序列和CDR2序列,或者分别与分别在表1中列出的CDR1序列和CDR2序列中的至少一者具有至少80%、至少90%、至少95%或至少99%同一性的CDR1序列和CDR2序列的组;和/或选自由以下组成的组:分别与分别在表1中列出的CDR1序列和CDR2序列中的至少一者具有3个、2个或仅1个氨基酸差异的CDR1序列和CDR2序列。
优选地,本文所述的单结构域抗体中存在的所有三个CDR1序列、CDR2序列和CDR3序列选自由以下组成的组:分别在表1中列出的CDR1序列、CDR2序列和CDR3序列;或者选自由以下组成的组:分别与分别在表1中列出的CDR1序列、CDR2序列和CDR3序列中的至少一者具有至少80%、至少90%、至少95%或至少99%同一性的CDR1序列、CDR2序列和CDR3序列;和/或选自由以下组成的组:分别与分别在表1中列出的CDR1序列、CDR2序列和CDR3序列中的至少一者具有3个、2个或仅1个氨基酸差异的CDR1序列、CDR2序列和CDR3序列。
更优选地,本文所述的单结构域抗体中存在的CDR1序列、CDR2序列和CDR3序列中的至少一者选自由以下组成的组:分别在表1中列出的CDR1序列、CDR2序列和CDR3序列。优选地,在这个方面中,所存在的其他两个CDR序列中的至少一者或优选地两者选自与分别在表1中列出的对应CDR序列中的至少一者具有至少80%、至少90%、至少95%或至少99%同一性的CDR序列;和/或选自由以下组成的组:与分别在表1中列出的对应序列中的至少一者具有3个、2个或仅1个氨基酸差异的CDR序列。
具体地,本文所述的单结构域抗体中存在的至少CDR3序列选自由以下组成的组:表1中列出的CDR3序列。优选地,在这个方面中,所存在的CDR1序列和CDR2序列中的至少一者且优选地两者选自由以下组成的组:分别与分别在表1中列出的CDR1序列和CDR2序列具有至少80%、至少90%、至少95%或至少99%同一性的CDR1序列和CDR2序列;和/或选自由以下组成的组:分别与分别在表1中列出的CDR1序列和CDR2序列具有3个、2个或仅1个氨基酸差异的CDR1序列和CDR2序列。
更优选地,本文所述的单结构域抗体中存在的CDR1序列、CDR2序列和CDR3序列中的至少两者选自由以下组成的组:分别在表1中列出的CDR1序列、CDR2序列和CDR3序列。优选地,在这个方面中,所存在的剩余CDR序列选自由以下组成的组:与表1中列出的对应CDR序列中的至少一者具有至少80%、至少90%、至少95%或至少99%同一性的CDR序列;和/或选自由以下组成的组:与表1中列出的对应CDR序列中的至少一者具有3个、2个或仅1个氨基酸差异的CDR序列。
具体地,本文所述的单结构域抗体中存在的至少CDR3序列选自由以下组成的组:表1中列出的CDR3序列,并且CDR1序列或CDR2序列选自由以下组成的组:分别在表1中列出的CDR1序列和CDR2序列。优选地,在这个方面中,所存在的剩余CDR序列选自由以下组成的组:与表1中列出的对应CDR序列中的至少一者具有至少80%、至少90%、至少95%或至少99%同一性的CDR序列;和/或选自由以下组成的组:与表1中列出的对应CDR序列具有3个、2个或仅1个氨基酸差异的CDR序列。
优选地,本文所述的单结构域抗体中存在的所有三个CDR1序列、CDR2序列和CDR3序列选自由以下组成的组:分别在表1中列出的CDR1序列、CDR2序列和CDR3序列。
而且,一般来说,表1中列出的CDR的组合(例如,表1中同一行内提及的那些)是优选的。因此,通常优选的是,当本文所述的单结构域抗体中的CDR是表1中提及的CDR序列或选自由以下组成的组:与表1中列出的CDR序列具有至少80%、至少90%、至少95%或至少99%同一性的CDR序列;和/或选自由以下组成的组:与表1中列出的CDR序列具有3个、2个或仅1个氨基酸差异的CDR序列时,则其他CDR中的至少一者且优选地两者选自属于表1中的相同例示性单结构域抗体序列的CDR序列(例如,在表1的同一行内提及)或选自由以下组成的组:与属于所述相同例示性单结构域抗体序列的CDR序列具有至少80%、至少90%、至少95%或至少99%同一性的CDR序列,和/或选自由以下组成的组:与属于所述相同例示性单结构域抗体序列的CDR序列具有3个、2个或仅1个氨基酸差异的CDR序列。上述段落中指出的其他优选项也适用于表1中提及的CDR的组合。
本文所述的单结构域抗体可包含与表1中提及的CDR1序列中的任一者具有大于80%同一性的CDR1序列、与表1中提及的CDR2序列中的任一者具有3个、2个或1个氨基酸差异的CDR2序列,以及CDR3序列(表1中提及或未提及)。
本文所述的单结构域抗体可以例如包含:(1)与表1中提及的CDR1序列中的任一者具有大于80%同一性的CDR1序列;与表1中提及的CDR2序列中的任一者具有3个、2个或1个氨基酸差异的CDR2序列;与表1中提及的CDR3序列的任一者具有大于80%同一性的CDR3序列;或者(2)与表1中提及的CDR1序列中的任一者具有大于80%同一性的CDR1序列;CDR2序列,以及表1中提及的CDR3序列中的任一者;或者(3)CDR1序列;与表1中提及的CDR2序列中的任一者具有大于80%同一性的CDR2序列;与表1中提及的与所述CDR2序列属于相同例示性单结构域抗体序列的CDR3序列具有3个、2个或1个氨基酸差异的CDR3序列。
本文所述的其他单结构域抗体可以例如包含:(1)与表1中提及的CDR1序列中的任一者具有大于80%同一性的CDR1序列;与表1中提及的属于相同例示性单结构域抗体序列的CDR2序列有3个、2个或1个氨基酸差异的CDR2序列;以及与表1中提及的属于相同例示性单结构域抗体序列的CDR3序列具有大于80%同一性的CDR3序列;(2)CDR1序列;表1中提及的CDR2序列和表1中提及的CDR3序列(其中CDR2序列和CDR3序列可以属于不同的例示性单结构域抗体序列)。
本文所述的其他单结构域抗体可以例如包含:(1)与表1中提及的CDR1序列中的任一者具有大于80%同一性的CDR1序列;表1中提及的属于相同例示性单结构域抗体的CDR2序列;以及表1中提及的属于不同例示性单结构域抗体的CDR3序列;或者(2)表1中提及的CDR1序列;与表1中提及的属于相同例示性单结构域抗体的CDR2序列具有3个、2个或1个氨基酸差异的CDR2序列;以及与表1中提及的CDR3序列中的任一者具有大于80%同一性的CDR3序列。
本文所述的优选单结构域抗体可以例如包含表1中提及的CDR1序列;与表1中提及的属于相同例示性单结构域抗体的CDR2序列具有大于80%同一性的CDR2序列;以及表1中提及的属于相同例示性单结构域抗体的CDR3序列。
最优选地,本文所述的单结构域抗体包含选自分别在表1中列出的的CDR1序列、CDR2序列和CDR3序列的组合中的一个组合的CDR1序列、CDR2序列和CDR3序列。
在实施方式中,(a)CDR1的长度介于1个氨基酸残基与15个氨基酸残基之间,并且通常介于4个氨基酸残基与12个氨基酸残基之间,例如9个或10个氨基酸残基;并且/或者(b)CDR2的长度介于1个氨基酸残基与15个氨基酸残基之间,并且通常介于2个氨基酸残基与12个氨基酸残基之间,例如7个、8个、9个或10个氨基酸残基;并且/或者(c)CDR3的长度介于2个氨基酸残基与35个氨基酸残基之间,并且通常介于3个氨基酸残基与30个氨基酸残基之间,例如介于8个氨基酸残基与22个氨基酸残基之间。
一般而言,具有上述CDR序列的单结构域抗体可以如本文进一步所述,并且优选地具有也如本文进一步所述的构架序列。
在实施方式中,本公开涉及一种免疫特异性结合SARS-CoV-2的单结构域抗体,所述单结构域抗体由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成,其中:
FR1选自由以下组成的组:
a)SEQ ID NO:16至SEQ ID NO:26中任一者所示的氨基酸序列;
b)与SEQ ID NO:16至SEQ ID NO:26所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:16至SEQ ID NO:26所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
和/或
FR2选自由以下组成的组:
d)SEQ ID NO:41至SEQ ID NO:48中任一者所示的氨基酸序列;
e)与SEQ ID NO:41至SEQ ID NO:48所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:41至SEQ ID NO:48所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
和/或
FR3选自由以下组成的组:
g)SEQ ID NO:63至SEQ ID NO:76中任一者所示的氨基酸序列;
h)与SEQ ID NO:63至SEQ ID NO:76所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:63至SEQ ID NO:76所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
和/或
FR4选自由以下组成的组:
j)SEQ ID NO:92至SEQ ID NO:96中任一者所示的氨基酸序列;
k)与SEQ ID NO:92至SEQ ID NO:96所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
l)与SEQ ID NO:92至SEQ ID NO:96所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
或此类氨基酸序列的任何合适的片段。
免疫特异性结合SARS-CoV-2的单结构域抗体可能由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成,其中:
FR1选自由以下组成的组:
a)SEQ ID NO:16至SEQ ID NO:26中任一者所示的氨基酸序列;
b)与SEQ ID NO:16至SEQ ID NO:26所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
c)与SEQ ID NO:16至SEQ ID NO:26所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
并且
FR2选自由以下组成的组:
d)SEQ ID NO:41至SEQ ID NO:48中任一者所示的氨基酸序列;
e)与SEQ ID NO:41至SEQ ID NO:48所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
f)与SEQ ID NO:41至SEQ ID NO:48所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
并且
FR3选自由以下组成的组:
g)SEQ ID NO:63至SEQ ID NO:76中任一者所示的氨基酸序列;
h)与SEQ ID NO:63至SEQ ID NO:76所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
i)与SEQ ID NO:63至SEQ ID NO:76所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
并且
FR4选自由以下组成的组:
j)SEQ ID NO:92至SEQ ID NO:96中任一者所示的氨基酸序列;
k)与SEQ ID NO:92至SEQ ID NO:96所示的氨基酸序列中的至少一个氨基酸序列具有至少80%同一性的氨基酸序列;
l)与SEQ ID NO:92至SEQ ID NO:96所示的氨基酸序列中的至少一个氨基酸序列具有5个、4个、3个、2个或1个氨基酸差异的氨基酸序列;
或此类氨基酸序列的任何合适的片段。
包含以上列出的FR中的一者或多者的单结构域抗体是优选的;包含以上列出的FR中的两者或更多者的单结构域抗体是更优选的;包含以上列出的FR中的三者或更多者的单结构域抗体是更优选的;包含以上列出的FR中的四者的单结构域抗体是最优选的。
FR序列的组合以及CDR序列和FR序列的优选组合在上表1中提及,所述表1列出了在本文所述的许多示例性单结构域抗体中存在的CDR序列和FR序列。出现在同一例示性单结构域抗体中的FR1序列、FR2序列、FR3序列和FR4序列的组合(例如,表1中同一行上提及的FR1序列、FR2序列、FR3序列和FR4序列)可能是优选的,但是本发明在其最广泛的意义上不限于此,并且还包括表1中提及的FR序列的其他合适的组合。而且,出现在相同的例示性单结构域抗体中的CDR序列和FR序列的组合(例如,在表1中的同一行上提到的CDR序列和FR序列)将通常是优选的,但是本发明在其最广泛的范围内不限于此,并且还包括表1中提及的CDR序列和FR序列的其他合适的组合,以及此类CDR序列和其他合适的FR序列的组合,例如,如本文进一步描述的。
此外,在包含表1中提到的FR组合的本文所述的单结构域抗体中,每个FR可以被选自由以下组成的组的FR替代:与所提及的FR具有至少80%、至少90%、至少95%或至少99%的序列同一性的氨基酸序列。
表1中提及的FR序列的组合以及CDR序列和FR序列的组合可为优选的。
本文所述的单结构域抗体可以是天然存在的单结构域抗体(来自任何合适的物种);天然存在的VHH序列(例如,来自骆驼科的合适物种);由能够产生此类单结构域抗体或VHH序列的转基因动物(例如,转基因小鼠)产生和/或衍生的单结构域抗体;或合成或半合成的氨基酸序列或单结构域抗体;包括但不限于部分人源化的单结构域抗体或VHH序列、完全人源化的单结构域抗体或VHH序列、骆驼化的重链可变(VH)结构域序列,以及通过任何合适的技术(例如本文所述的那些)获得的单结构域抗体。
人源化的单结构域抗体可以由四个构架区域(FR1、FR2、FR3、FR4)和三个互补决定区(CDR1、CDR2、CDR3)组成,其中CDR1、CDR2和CDR3如本文所述并且其中所述人源化的单结构域抗体包含至少一个人源化取代,并且具体地是在其构架序列中的至少一个构架序列中的至少一个人源化取代。
单结构域抗体可包含与SEQ ID NO:1至SEQ ID NO:15(参见表2)所示的氨基酸序列中的至少一个氨基酸序列的CDR序列具有至少70%同一性、至少80%同一性、至少90%同一性、至少91%同一性、至少92%同一性、至少93%同一性、至少94%同一性、至少95%同一性、至少96%同一性、至少97%同一性、至少98%同一性、至少99%同一性或100%同一性CDR序列。这种同一性程度可以例如通过测定所述单结构域抗体与SEQ ID NO:1至SEQ IDNO:15所示的序列(表2)中的一个或多个序列之间的氨基酸同一性程度(以本文所述的方式)来测定,其中形成构架区域的氨基酸残基被忽略。此类单结构域抗体可如本文进一步所述。
单结构域抗体可以包含选自由以下组成的组的氨基酸序列:SEQ ID NO:1至SEQID NO:15(参见表2),或选自由以下组成的组的氨基酸序列:与SEQ ID NO:1至SEQ ID NO:15(参见表2)所示的氨基酸序列中的一个或多个氨基酸序列具有至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性的氨基酸序列。
人源化的和/或序列可以包含SEQ ID NO:1至SEQ ID NO:15(参见表2)所示的单结构域抗体的优化变体,所述优化变体与对应的天然VHH序列相比包含至少一种人源化和/或序列优化取代,并且具体地是在其构架序列中的至少一个构架序列中的至少一种人源化和/或序列优化取代。
单结构域抗体或其片段可以包含由SEQ ID NO:97至SEQ ID NO:111(参见表3)中任一者所示的核苷酸序列编码的VHH结构域。
单结构域抗体或其片段包含由SEQ ID NO:97至SEQ ID NO:111(参见表3)中任一者所示的核苷酸序列编码的VHH结构域的一个、两个或全部三个CDR,和/或由SEQ ID NO:97至SEQ ID NO:111(参见表3)中任一者所示的核苷酸序列编码的VHH结构域的一个、两个、三个或全部四个构架区域(FR)。
多肽
本文所述的多肽可以包含或基本上由至少一种本文所述的单结构域抗体组成。本文所述的多肽的示例在SEQ ID NO:112至SEQ ID NO:117(参见表4)和SEQ ID NO:118至SEQID NO:123(参见表5)中给出。
技术人员将清楚,本文中描述为“优选的”(或“更优选的”、“甚至更优选的”,等等)的单结构域抗体对于用于本文所述的多肽中也是优选的(或更优选的,或甚至更优选的,等等)。因此,包含或基本上由一种或多种本文所述的“优选的”单结构域抗体组成的多肽通常将是优选的,并且包含或基本上由一种或多种本文所述的“更优选的”单结构域抗体组成的多肽通常将是更优选的,等等。
一般而言,包含或基本上由仅一种单结构域抗体(例如,仅一种本文所述的单结构域抗体)组成的蛋白质或多肽将在本文中将被称为“单价”蛋白质或多肽或“单价构建体”。包含或基本上由两种或更多种单结构域抗体(例如,至少两种本文所述的单结构域抗体或至少一种本文所述的单结构域抗体和至少一种其他单结构域抗体)组成的蛋白质和多肽将在本文中将被称为“多价”蛋白质或多肽或“多价构建体”,并且与本文所述的对应单价单结构域抗体相比,这些可以提供某些优点。本文描述了此类多价构建体的一些非限制性示例。
本文所述的多肽可包含或基本上由以下组成:至少两种本文所述的单结构域抗体,例如两种或三种本文所述的单结构域抗体。如本文进一步所述,与包含或基本上由仅一种本文所述的单结构域抗体组成的蛋白质或多肽相比,此类多价构建体可以提供某些优点,例如,改进的对SARS-CoV-2的亲合力。
本文所述的多肽可以包含或基本上由至少一种本文描述的单结构域抗体和至少一种其他结合单位(例如,针对另一表位、抗原、靶标、蛋白质或多肽)组成,所述至少一种其他结合单位优选也是单结构域抗体。此类蛋白质或多肽在本文中也称为“多特异性”蛋白质或多肽或“多特异性构建体”,并且与本文所述的对应单价单结构域抗体相比,这些可以提供某些优点。
本文所述的多肽可包含或基本上由以下组成:至少一种本文所述的单结构域抗体、任选的一种或多种另外的单结构域抗体,以及至少一种其他氨基酸序列(例如,蛋白质或多肽),所述至少一种其他氨基酸序列赋予本文所述的单结构域抗体和/或所得融合蛋白至少一种所需特性。同样,与本文所述的对应单价单结构域抗体相比,此类融合蛋白可提供某些优点。本文描述了此类氨基酸序列和此类融合构建体的一些非限制性示例。
还可以组合上述方面中的两个或更多个方面,例如,以提供包含两种本文所述的单结构域抗体和一种其他单结构域抗体以及任选的一种或多种其他氨基酸序列的三价双特异性构建体。本文描述了此类构建体的另外非限制性示例,以及本文所述的上下文中优选的一些构建体。
在上述构建体中,所述一种或多种单结构域抗体和/或其他氨基酸序列可以直接彼此连接和/或经由一个或多个接头序列彼此连接。本文描述了此类接头的一些合适的但非限制性的示例。
本文所述的单结构域抗体或可包含至少一种本文所述的单结构域抗体的化合物、构建体或多肽,与本文所述的对应氨基酸序列相比可具有延长的半衰期。
本文所述的单结构域抗体序列或多肽可经化学修饰以延长其半衰期(例如,借助于聚乙二醇化);是本文所述的包含至少一个附加的用于结合血清蛋白(例如,血清白蛋白)的结合位点的氨基酸序列;或是本文所述的包含至少一种本文所述的单结构域抗体的多肽,所述单结构域抗体连接至至少一个延长本文所述的单结构域抗体的半衰期的部分(并且特别是至少一种氨基酸序列)。本文所述的包含此类半衰期延长部分或氨基酸序列的多肽的示例包括但不限于这样的多肽,在所述多肽中一种或多种本文所述的单结构域抗体连接至一种或多种血清蛋白或其片段(例如,血清白蛋白或其合适的片段)或一个或多个可结合血清蛋白的结合单位(例如,可结合血清蛋白(例如血清白蛋白)、血清免疫球蛋白(例如IgG)或转铁蛋白的单结构域抗体);这样的多肽,在所述多肽中本文所述的单结构域抗体连接至Fc部分(例如,人Fc)或其合适的部分或片段;或者这样的多肽,在所述多肽中一种或多种本文所述的单结构域抗体连接至一种或多种可结合血清蛋白的小蛋白或肽(例如,WO91/01743、WO 01/45746、WO 02/076489和WO 2008/068280中所示的蛋白质和肽)。
再次,单结构域抗体、化合物、构建体或多肽可以包含一种或多种附加的基团、残基、部分或结合单位,例如一种或多种另外的氨基酸序列,并且特别是一种或多种另外的单结构域抗体(例如,不针对SARS-CoV-2),从而提供三特异性或多特异性单结构域抗体构建体。
一般而言,本文所述的具有延长的半衰期的单结构域抗体(或包含所述单结构域抗体的化合物、构建体或多肽)可具有为本文所述的对应氨基酸序列的半衰期的至少1.5倍,优选至少2倍,例如至少5倍、至少10倍、或大于20倍长的半衰期。例如,本文所述的具有延长的半衰期的单结构域抗体、化合物、构建体或多肽可以具有与本文所述的对应氨基酸序列相比延长了大于1小时,优选大于2小时、大于6小时,例如12小时、或大于24小时、48小时或72小时的半衰期。
本文所述的单结构域抗体、化合物、构建体或多肽可以表现出在人中至少约12小时、至少24小时、至少48小时、至少72小时或更长时间的血清半衰期。例如,本文所述的化合物或多肽可以具有至少5天(例如约5天至10天),优选地至少9天(例如约9天至14天)、或至少约10天(例如约10天至15天)、或至少约11天(例如约11天至16天)、或至少约12天(例如约12天至18天)或更长时间、或多于14天(例如约14天至19天)的半衰期。
包含一种或多种本文所述的单结构域抗体的多肽可以结合SARS-CoV-2:
-具有的解离常数(K
和/或
-具有的k
和/或
-具有的k
多肽可包含仅一个本文所述的氨基酸序列,优选地使得它将以小于500nM,优选地小于200nM,更优选地小于10nM,例如小于500pM的亲和力结合SARS-CoV-2。与包含仅一个本文所述的氨基酸序列的多肽相比,包含两个或更多个本文所述的氨基酸序列的多肽可以以增加的亲合力结合SARS-CoV-2。
本文描述了本文所述的氨基酸序列或多肽与SARS-CoV-2的结合的一些优选IC
根据这个方面的多肽可以例如选自由以下组成的组:与SEQ ID NO:112至SEQ IDNO:117(参见表4)和SEQ ID NO:118至SEQ ID NO:123(表5)所示的氨基酸序列中的一个或多个氨基酸序列至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、或至少99%或更高同一的氨基酸序列,其中所述氨基酸序列内包含的单结构域抗体优选地如本文进一步定义。
本文描述了编码本文所述的氨基酸序列(例如,本文所述的单结构域抗体)或包含所述氨基酸序列的多肽的核酸。在实施方式中,编码本文所述的氨基酸序列的核酸可包含SEQ ID NOS:97至SEQ ID NOS:111(参见表3)所示的核苷酸序列中的一个或多个核苷酸序列。再次,如本文针对本公开的核酸一般描述的,此类核酸可以是遗传构建体的形式,如本文所述的。
表达或能够表达本文所述的氨基酸序列(例如,单结构域抗体)和/或包含所述氨基酸序列的多肽的宿主或宿主细胞;和/或含有本公开的核酸的宿主或宿主细胞在本文中进行了描述。
产品或组合物可以包含至少一种本文所述的氨基酸序列、至少一种本文所述的多肽和/或至少一种本文所述的核酸,以及任选的此类组合物的一种或多种本身已知的另外组分,例如,取决于所述组合物的预期用途。此类产品或组合物可以例如是药物组合物、兽医组合物、或用于诊断用途的产品或组合物。
本文描述了用于制备或生成氨基酸序列、化合物、构建体、多肽、核酸、宿主细胞、产品和组合物的方法。
本文描述了所述氨基酸序列、化合物、构建体、多肽、核酸、宿主细胞、产品和组合物的应用和用途,包括用于预防和/或治疗与SARS-CoV-2相关联的疾病和疾患的方法。
一般而言,应当注意的是,如本文所用的术语单结构域抗体在其最广泛的意义上不限于特定的生物来源或特定的制备方法。例如,如下文更详细讨论的,本文所述的单结构域抗体通常可以通过任何合适的技术获得。一类优选的单结构域抗体对应于针对SARS-CoV-2的天然存在的重链抗体的VHH结构域。如本文进一步所述,此类VHH序列通常可以通过以下方式生成或获得:用SARS-CoV-2多肽免疫骆驼科物种(例如,以引发针对SARS-CoV-2的免疫应答和/或重链抗体);从所述骆驼科获得合适的生物样品(例如,血液样品、血清样品或B细胞样品);以及使用任何合适的技术从所述样品开始生成针对SARS-CoV-2的VHH序列。
或者,此类天然存在的针对SARS-CoV-2的VHH结构域可以从骆驼科VHH序列的幼稚文库(naive library)获得,例如,通过使用一种或多种已知的筛选技术,使用SARS-CoV-2或其至少一部分、片段、抗原决定簇或表位来筛选此类文库。此类文库和技术例如描述于WO99/37681、WO 01/90190、WO 03/025020和WO 03/035694中。或者,可以使用衍生自幼稚VHH文库的改进的合成或半合成文库,例如如例如在WO 00/43507中所述的通过例如随机诱变和/或CDR改组的技术从幼稚VHH文库获得的VHH文库。
本文所述的一类单结构域抗体可包含这样的单结构域抗体,所述单结构域抗体具有与天然存在的VHH结构域的氨基酸序列相对应的氨基酸序列,但所述氨基酸序列已“人源化”,例如通过用来自来自人类的常规四链抗体的VH结构域中的对应位置处出现的氨基酸残基中的一个或多个氨基酸残基替换所述天然存在的VHH序列(并且特别是在构架序列中)的氨基酸序列中的一个或多个氨基酸残基。本文所述的另一类单结构域抗体可包含这样的单结构域抗体,所述单结构域抗体具有与天然存在的VH结构域的氨基酸序列相对应的氨基酸序列,但所述氨基酸序列已“骆驼化”,例如通过用重链抗体的VHH结构域中的对应位置处出现的氨基酸残基中的一个或多个氨基酸残基替换来自常规四链抗体的天然存在的VH结构域的氨基酸序列中的一个或多个氨基酸残基。
除了本文所述的人源化或骆驼化取代之外,本文所述的氨基酸序列还可包含一个或多个其他/另外的取代。在实施方式中,此类其他/另外的取代可包括或基本上由以下取代中的一种或多个取代组成:
a)一个或多个保守氨基酸取代;和/或
b)一个或多个取代,在所述取代中某一位置处的“骆驼科”氨基酸残基被出现在所述位置的不同“骆驼科”氨基酸残基替换;和/或
c)一个或多个改善蛋白质的性质的取代,例如改善蛋白质在储存下的长期稳定性和/或性质的取代。
这些取代可以包括但不限于防止或减少(例如,甲硫氨酸残基的)氧化事件的取代;防止或减少焦谷氨酸盐形成的取代;和/或防止或减少(例如,DG、DS、NG或NS基序的)天冬氨酸或天冬酰胺的异构化或脱酰胺化的取代。此类取代通常是本领域普通技术人员已知的。
尽管使用本文所述的单结构域抗体和本公开的多肽是优选的,但显然,基于本文的描述,技术人员将还能够以类似的方式设计和/或生成其他氨基酸序列,特别是针对SARS-CoV-2的单结构域抗体,以及包含此类单结构域抗体的多肽。
例如,对于本领域技术人员来说还将清楚的是,可以将上文针对本文所述的单结构域抗体提及的CDR中的一个或多个CDR“移植”到其他单结构域抗体或其他蛋白质支架,包括但不限于人类支架或非免疫球蛋白支架上。用于此类CDR移植的合适的支架和技术对于技术人员来说是清楚的并且是本领域中众所周知的。例如,已知用于将小鼠或大鼠CDR移植到人构架和支架上的技术可以以类似的方式使用,以提供包含本文所述的单结构域抗体的CDR中的一个或多个CDR和一个或多个人构架区域或序列的嵌合蛋白。
还应当指出的是,当本文所述的单结构域抗体包含除上述优选的CDR序列之外的一个或多个其他CDR序列时,这些CDR序列可以以任何已知的方式获得。
用于从天然存在的VH序列或优选地VHH序列开始获得本文所述的单结构域抗体和/或编码所述单结构域抗体的核酸的其他合适的方法和技术对于技术人员来说将是清楚的。
抗SARS-CoV-2单结构域抗体可用于治疗需要部分或完全阻断和/或中和SARS-CoV-2活性的疾病或疾患。在实施方式中,本文所述的抗SARS-CoV-2的单结构域抗体用于治疗COVID-19和/或与SARS-CoV-2感染相关联或由SARS-CoV-2感染引起的疾病和疾患。在实施方式中,本文所述的抗SARS-CoV-2的单结构域抗体用于治疗急性呼吸衰竭、肺炎、急性呼吸窘迫综合征(ARDS)、急性肝损伤、急性心脏损伤、继发感染、急性肾损伤、感染性休克、弥散性血管内凝血、血栓、多系统炎症综合征、慢性疲劳、横纹肌溶解症以及它们的组合。
在另一方面中,本文所述的抗SARS-CoV-2的单结构域抗体可用作用于检测和分离SARS-CoV-2(例如检测和/或定量各种细胞和/或组织中的SARS-CoV-2表达)的试剂。本文所述的抗SARS-CoV-2的单结构域抗体可在SARS-CoV-2受体结合结构域(RBD)结合测定中使用以筛选将表现出类似药理效应的SARS-CoV-2拮抗剂。
本文所述的单结构域抗体可以免疫特异性地结合包含SEQ ID NO:124、SEQ IDNO:125、SEQ ID NO:126、SEQ ID NO:127、SEQ ID NO:128或它们的组合所示的氨基酸序列的多肽,或包含SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ ID NO:127、SEQ IDNO:128或它们的组合所示的氨基酸序列的一部分(例如片段)的多肽。本文所述的单结构域抗体包括包含或替代地由以下组成的分子:免疫特异性地结合冠状病毒氨基酸序列的受体结合结构域(RBD)的抗体片段或其变体(例如,包含或替代地由以下组成的多肽:SARS-CoV-2刺突蛋白的氨基酸331-524(SEQ ID NO:124)、SARS-CoV刺突蛋白的氨基酸残基318-510(SEQ ID NO:125)、MERS-CoV刺突蛋白的氨基酸残基377-588(SEQ ID NO:126)、和/或SARSCoV-2刺突受体结合结构域的氨基酸319-541(SEQ ID NO:127))。
此外,可由本文所述的单结构域抗体结合的多肽片段的长度可为至少10个、20个、30个、40个、50个、60个、70个、80个、90个、100个、110个、120个、130个、140个、150个、175个或200个氨基酸。在此上下文中,“约”意指具体叙述的范围和在氨基末端和羧基末端中的一者或两者处大或小几、一些、5个、4个、3个、2个或1个氨基酸残基的范围。
结合SARS-CoV-2多肽片段的单结构域抗体可包含或由以下组成:本文所述的多肽的功能区,例如Gamier-Robsonα区、β区、转角区和卷曲区;Chou-Fasmanα区、β区和卷曲区;Kyte-Doolittle亲水区和疏水区;Eisenbergα两亲区和β两亲区;Karplus-Schulz柔性区;Emini表面形成区;和具有高抗原指数的Jameson-Wolf区。在优选的实施方式中,由本文所述的单结构域抗体结合的多肽片段是冠状病毒受体结合结构域多肽(例如,SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ ID NO:127)的抗原性部分(例如,包含四个或更多个具有大于或等于1.5的抗原指数的连续氨基酸,如使用Jameson-Wolf程序的默认参数鉴定的)。
抗体表位
结合多肽的单结构域抗体可包含或替代地由本文所述的多肽的携带表位的部分组成。此多肽部分的表位可以是本文所述的多肽的免疫原性或抗原性表位。“免疫原性表位”被定义为当整个蛋白质是免疫原时所述蛋白质的引发抗体反应的部分。抗体可结合的蛋白质分子区域也可被定义为“抗原表位”。蛋白质的免疫原性表位的数量通常少于抗原性表位的数量。参见例如,Geysen等人Proc.Natl.Acad.Sci.USA 81:3998-4002(1983)。
对于携带抗原性表位(例如,包含抗体可结合的蛋白质分子区域)的多肽的选择,本领域中众所周知的是,模拟蛋白质序列的部分的相对较短的合成肽常规地能够引发与部分模拟的蛋白质反应的抗血清。参见例如,Sutcliffe,J.G.,Shinnick,T.M.,Green,N.和Leamer,R.A.(1983)“Antibodies that react with predetermined sites onproteins”,Science,219:660-666。能够引发蛋白质反应性血清的肽经常以蛋白质的一级序列表示,可以通过一组简单的化学规则来表征,并且既不限于完整蛋白质的免疫显性区域(例如,免疫原性表位)也不限于氨基或羧基末端。因此,本文所述的携带抗原表位的肽和多肽可用于产生特异性地结合本文所述的多肽的抗体,包括单结构域抗体。参见例如,Wilson等人Cell 37:767-778(1984),在第777页。
本文所述的单结构域抗体结合SARS-CoV-2的携带抗原表位的肽和多肽(例如,SARS-CoV-2 RBD),并且优选地包含具有在SARS-CoV-2多肽(例如,SARS-CoV-2 RBD多肽)的氨基酸序列中所包含的至少4个、至少5个、至少6个、至少7个,更优选地至少8个、至少9个、至少10个、至少11个、至少12个、至少13个、至少14个、至少15个、至少20个、至少25个、至少30个、至少40个、至少50个,最优选地在约15个至约30个之间的氨基酸。包含免疫原性或抗原性表位的优选多肽的长度为至少10个、15个、20个、25个、30个、35个、40个、45个、50个、55个、60个、65个、70个、75个、80个、85个、90个、95个或100个氨基酸残基。其他非排他性的优选抗原表位包括本文所公开的抗原表位及其部分。
结合多肽的单结构域抗体可以包含或替代地由冠状病毒氨基酸序列的表位组成。优选地,表位在冠状病毒氨基酸序列的受体结合结构域内。例如,本文所述的单结构域抗体可以结合包含或替代地由在具有SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ IDNO:127、SEQ ID NO:128或它们的组合的氨基酸序列的多肽内所包含的表位组成的多肽。
本文所述的单结构域抗体和抗原结合片段优选地结合由SARS-CoV-2刺突蛋白的氨基酸331-524(SEQ ID NO:124)、SARS-CoV刺突蛋白的氨基酸残基318-510(SEQ ID NO:125)、MERS-CoV刺突蛋白的氨基酸残基377-588(SEQ ID NO:126)的受体结合结构域、SARS-CoV-2刺突蛋白的氨基酸319-541(SEQ ID ON:127)和/或SARS-CoV-2刺突蛋白(SEQ ID NO:128)的受体结合结构域内的6至12个残基组成的表位。
本文所述的单结构域抗体和抗原结合片段优选地结合由SARS CoV-2刺突受体结合结构域的氨基酸319-541(SEQ ID NO:127)的受体结合结构域内的6至12个残基组成的表位:
RVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNF
如本文所用,术语“表位”泛指多肽的在动物,优选地哺乳动物,最优选地人类中具有抗原性或免疫原性活性的部分。在优选的实施方式中,本公开涵盖结合包含表位的多肽的单结构域抗体。如本文所用,“免疫原性表位”被定义为蛋白质的在动物中引发抗体反应的部分,如通过本领域已知的任何方法,例如通过下文所述的用于产生抗体的方法所确定的。(参见例如,Geysen等人Proc.Natl.Acad.Sci.USA 81:3998-4002(1983))。如本文所用,术语“抗原性表位”被定义为抗体可免疫特异性地结合其抗原的蛋白质的一部分,如通过本领域众所周知的任何方法,例如通过本文所述的免疫测定所确定的。免疫特异性结合排除非特异性结合,但不一定排除与其他抗原的交叉反应性。抗原表位不一定必须是免疫原性的。抗原表位可用于例如产生特异性结合所述表位的抗体,包括单结构域抗体。优选的抗原表位包括但不限于本文所公开的抗原表位,以及这些抗原表位中的两种、三种、四种、五种或更多种抗原表位的任意组合。抗原表位可用作免疫测定中的靶分子。(参见例如,Wilson等人Cell 37:767-778(1984);Sutcliffe等人Science 219:660-666(1983))。
类似地,可以使用免疫原性表位,例如以根据本领域众所周知的方法诱导抗体。(参见例如,Sutcliffe等人,出处同上;Wilson等人,出处同上;Chow等人Proc.Natl.Acad.Sci.USA 82:910-914;和Bittle等人J.Gen.Virol.66:2347-2354(1985))。优选的免疫原性表位包括本文所公开的免疫原性表位,以及这些免疫原性表位中的两种、三种、四种、五种或更多种免疫原性表位的任意组合。可以将包含SARS-CoV-2的一种或多种免疫原性表位的多肽与载体蛋白(例如白蛋白)一起呈递给动物系统(例如,兔或小鼠或骆驼科)以引发抗体反应,或者,如果多肽具有足够的长度(至少约25个氨基酸),则所述多肽可以在没有载体的情况下呈递。然而,已显示包含少至8至10个氨基酸的免疫原性表位足以产生能够结合至少变性多肽中的线性表位的抗体(例如,在Western印迹中)。
可通过任何常规手段来产生充当表位的SARS-CoV-2多肽片段。(参见例如,Houghten Proc.Natl.Acad.Sci.USA 82:5131-5135(1985)和美国专利号4,631,211)。
携带表位的SARS-CoV-2多肽可用于根据本领域中众所周知的方法诱导单结构域抗体,所述方法包括但不限于体内免疫、体外免疫和噬菌体展示方法。参见例如,Sutcliffe等人出处同上;Wilson等人出处同上,以及Bittle等人J.Gen.Virol.66:2347-2354(1985)。如果使用体内免疫,可以用游离肽免疫动物;然而,可以通过将肽与大分子载体(例如匙孔血蓝蛋白(keyhole limpet hemocyanin,KLH)或破伤风类毒素)偶联来提高抗肽抗体的效价。例如,包含半胱氨酸残基的肽可以使用例如马来酰亚胺苯甲酰基-N-羟基琥珀酰亚胺酯(MBS)的接头与载体偶联,而其他肽可以使用更一般的连接剂(例如戊二醛)与载体偶联。例如通过腹膜内和/或皮下注射包含约0.5毫克至100毫克的肽或载体蛋白和弗氏佐剂或已知用于刺激免疫应答的任何其他佐剂的乳液来对动物(例如,美洲驼、骆驼和羊驼)进行免疫。可能需要数次加强注射,例如,间隔时间为约两周的数次加强注射,以提供可例如通过使用吸附到固体表面的游离肽的ELISA测定来检测的有用效价的抗肽抗体。免疫动物血清中抗肽抗体的效价可通过选择抗肽抗体来增加,例如通过根据本领域众所周知的方法将肽吸附到固体载体上并洗脱所选抗体。
抗SARS-CoV-2的单结构域抗体融合蛋白
本文还描述了单结构域抗体的衍生物。此类衍生物通常可通过对本文所述的单结构域抗体和/或形成本文所述的单结构域抗体的氨基酸残基中的一个或多个氨基酸残基进行修饰,特别是化学和/或生物(例如,酶促)修饰来获得。
此类修饰的示例,以及单结构域抗体序列内可以以此类方式修饰(例如,在蛋白质主链上,但优选地在侧链上)的氨基酸残基的示例、可用于引入此类修饰的方法和技术,以及此类修饰的潜在用途和优点对于本领域技术人员来说将是清楚的。
例如,此类修饰可以涉及一个或多个官能团、残基或部分至本文所述的单结构域抗体之中或之上的引入(例如,通过共价连接或以任何其他合适的方式),并且特别地赋予本文所述的单结构域抗体一种或多种所需性质或功能的一个或多个官能团、残基或部分的引入。此类官能团的示例对于技术人员来说将是清楚的。
例如,此类修饰可以包括引入(例如,通过共价结合或以任何其他合适的方式)一个或多个官能团,所述官能团延长本文所述的单结构域抗体的半衰期、增加其溶解度和/或吸收;降低本文所述的单结构域抗体的免疫原性和/或毒性;消除或减弱本文所述的单结构域抗体的任何非期望的副作用;和/或赋予本文所述的单结构域抗体和/或多肽其他有利的性质和/或减少其非期望的性质;或前述中的两者或更多者的任意组合。此类官能团和用于引入它们的技术的示例对于技术人员来说将是清楚的,并且包括但不限于用于修饰药物蛋白质,特别是用于修饰抗体或抗体片段(包括scFv和单结构域抗体)的已知官能团和技术,关于所述已知官能团和技术,参考例如Remington’s Pharmaceutical Sciences,第16版,Mack Publishing Co.,Easton,PA(1980)。此类官能团可以例如直接(例如,共价地)或任选地经由合适的接头或间隔区连接至本文所述的单结构域抗体,如技术人员将再次清楚的。
用于延长药物蛋白质的半衰期和/或降低其免疫原性的最广泛使用的技术之一包括附接合适的药理学上可接受的聚合物,例如聚(乙二醇)(PEG)或其衍生物(例如,甲氧基聚(乙二醇)或mPEG)。一般而言,可以使用任何合适形式的聚乙二醇化,例如本领域中用于抗体和抗体片段(包括但不限于(单)结构域抗体和scFv)的聚乙二醇化;参见例如Chapman,Nat.Biotechnol.,54,531-545(2002);Veronese和Harris,Adv.Drug Deliv.Rev.54,453-456(2003);Harris和Chess,Nat.Rev.Drug.Discov.,2,(2003)以及在WO 04/060965中。用于蛋白质聚乙二醇化的各种试剂也可商购获得,例如从Nektar Therapeutics,USA商购获得。
优选地,使用定点聚乙二醇化,特别是经由半胱氨酸残基进行定点聚乙二醇化(参见例如,Yang等人,Protein Engineering,16,10,761-770(2003))。例如,为此目的,PEG可附接至天然存在于本文所述的单结构域抗体中的半胱氨酸残基,本文所述的单结构域抗体可经修饰以引入一个或多个用于附接PEG的半胱氨酸残基,或者包含一个或多个用于附接PEG的半胱氨酸残基的氨基酸序列可融合至本文所述的单结构域抗体的N末端和/或C末端,所有这些均使用已知的蛋白质工程化技术进行。
优选地,对于本文所述的单结构域抗体和蛋白质,使用分子量大于5000,例如大于10000且小于200000,例如小于100000;例如,在20000-80000范围内的PEG。
另一种修饰包括通常作为共翻译和/或翻译后修饰的一部分的N-连接的或O-连接的糖基化,具体取决于用于表达本文所述的单结构域抗体或多肽的宿主细胞。
另一种修饰可包括引入一个或多个可检测标记或其他生成信号的基团或部分,具体取决于经标记的单结构域抗体的预期用途。用于附接、使用和检测它们的合适标记和技术对于技术人员来说是清楚的,并且例如包括但不限于荧光标记、磷光标记、化学发光标记、生物发光标记、放射性同位素、金属、金属螯合物、金属阳离子、发色团和酶。其他合适的标记对于技术人员来说将是清楚的,并且例如包括可以使用NMR或ESR光谱法检测的部分。
本文所述的此类标记的单结构域抗体和多肽可以例如用于体外、体内或原位测定(包括免疫测定,例如ELISA、RIA、EIA和其他“夹心测定”)以及体内诊断和成像目的,具体取决于特定标签的选择。
修饰可以涉及引入螯合基团,例如以螯合上面提及的金属或金属阳离子中的一者。合适的螯合基团包括但不限于二亚乙基三胺五乙酸(DTPA)或乙二胺四乙酸(EDTA)。
另一种修饰可以包括引入作为特异性结合对(例如生物素-(链霉)亲和素结合对)的一部分的官能团。此类官能团可用于将本发明的单结构域抗体连接至与所述结合对的另一半结合的另一种蛋白质、多肽或化学化合物,例如经由形成结合对。例如,本文所述的单结构域抗体可缀合至生物素,并连接至与亲和素或链霉亲和素缀合的另一种蛋白质、多肽、化合物或载体。例如,此类缀合的单结构域抗体可以用作报告分子,例如在其中可检测的产生信号的药剂缀合至亲和素或链霉亲和素的诊断系统中。此类结合对还可以例如用于将本发明的单结构域抗体与载体(包括适合于药物目的的载体)结合。一个非限制性示例是由Cao和Suresh,Journal of Drug Targeting,8,4,257(2000)描述的脂质体制剂。此类结合对还可用于将治疗活性剂连接至本文所述的单结构域抗体。
其他潜在的化学和酶修饰对于技术人员来说将是清楚的。此类修饰也可出于研究目的而引入(例如,以研究功能-活性关系)。
优选地,衍生物使得它们以本文针对本文所述的单结构域抗体所述的亲和力(测量和/或表示为K
蛋白质或多肽可基本上由至少一种本文所述的单结构域抗体组成或包含至少一种本文所述的单结构域抗体。
所述氨基酸残基可以或不可以改变、变更或以其他方式影响单结构域抗体的生物学性质,并且可以或不可以向单结构域抗体添加另外的功能性。例如,此类氨基酸残基可以:
-包含N末端Met残基,例如作为在异源宿主细胞或宿主生物体中表达的结果;
-可以形成指导单结构域抗体在合成后从宿主细胞分泌的信号序列或前导序列。合适的分泌前导肽对于技术人员来说将是清楚的,并且可以如本文进一步所述。通常,此类前导序列将连接至单结构域抗体的N末端,但是本公开内容在其最广泛的意义上不限于此;
-可以形成序列或信号,所述序列或信号允许单结构域抗体被引导至和/或渗透或进入特定器官、组织、细胞、或细胞的部分或区室,和/或允许单结构域抗体渗透或跨越生物屏障,例如细胞膜、细胞层(例如上皮细胞层)、肿瘤(包括实体瘤)或血脑屏障;
-可以形成“标签”,例如这样的氨基酸序列或残基,所述氨基酸序列或残基允许或促进例如使用针对所述序列或残基的亲和技术进行单结构域抗体的纯化。此后,可以去除所述序列或残基(例如,通过化学或酶促切割)以提供单结构域抗体序列(为此目的,标签可以任选地经由可切割接头序列连接至单结构域抗体序列或包含可切割基序)。此类残基的一些优选但非限制性的示例是多个组氨酸残基、谷胱甘肽残基和myc标签;
-可以是一个或多个氨基酸残基,所述氨基酸残基已被官能化和/或可以充当用于官附接能团的位点。合适的氨基酸残基和官能团对于技术人员来说将是清楚的,并且包括但不限于本文针对本文所述的单结构域抗体的衍生物所述的氨基酸残基和官能团。
根据另一方面,本文所述的多肽包含本文所述的单结构域抗体,所述单结构域抗体在其氨基末端、在其羧基末端、或在其氨基末端和其羧基末端两者处与至少一个另外的氨基酸序列融合,例如以便提供包含本文所述的单结构域抗体和一个或多个另外的氨基酸序列的融合蛋白。这种融合体在本文中也将称为“单结构域抗体融合体”。
所述一个或多个另外的氨基酸序列可以是任何合适的和/或期望的氨基酸序列。所述另外的氨基酸序列可以或不可以改变、变更或以其他方式影响单结构域抗体的性质,并且可以或不可以向本文所述的单结构域抗体或多肽添加另外的功能性。优选地,所述另外的氨基酸序列使得其赋予本文所述的单结构域抗体或多肽一种或多种期望的性质或功能。
例如,另外的氨基酸序列还可以提供第二结合位点,所述第二结合位点可以针对任何所需的蛋白质、多肽、抗原、抗原决定簇或表位(包括但不限于与本文所述的单结构域抗体所针对的相同的蛋白质、多肽、抗原、抗原决定簇或表位,或不同的蛋白质、多肽、抗原、抗原决定簇或表位)。
此类氨基酸序列的示例对于技术人员来说将是清楚的,并且通常可以包括用于基于常规抗体及其片段(包括但不限于scFv和单结构域抗体)的肽融合体中的所有氨基酸序列。例如,参考Holliger和Hudson,Nature Biotechnology,23,9,1126-1136(2005)的评论。
例如,此类氨基酸序列可以是这样的氨基酸序列,与本文所述的单结构域抗体相比,所述氨基酸序列延长本文所述的多肽的半衰期、增加其溶解度或增加其吸收、降低其免疫原性或毒性、消除或减弱其非期望的副作用和/或赋予其其他有利性质和/或减少其非期望的性质。此类氨基酸序列的一些非限制性示例是血清蛋白,例如人血清白蛋白(参见例如WO 00/27435)或半抗原分子(例如被循环抗体识别的半抗原,参见例如WO 98/22141)。
具体地,已经描述了可以使用免疫球蛋白片段(例如,VH结构域)与血清白蛋白或其片段的连接来延长半衰期(参见例如WO 00/27435和WO 01/077137)。本文所述的单结构域抗体优选地直接或经由合适的接头,特别是经由合适的连接的肽连接至血清白蛋白(或其合适的片段),使得本文所述的多肽可以表达为基因融合体(蛋白质)。根据一个方面,本文所述的单结构域抗体可以连接至至少包含血清白蛋白的结构域III或其部分的血清白蛋白片段。
另外的氨基酸序列可以提供针对血清蛋白(例如,人血清白蛋白或另一种血清蛋白,例如IgG)的第二结合位点或结合单位,以便提供延长的血清半衰期。此类氨基酸序列例如包括在WO 91/01743、WO 01/45746和WO 02/076489中所述的小肽和结合蛋白以及WO 03/002609和WO 04/003019中所述的dAb。
一种或多种另外的氨基酸序列可以包含常规四链抗体(并且特别是人抗体)和/或重链抗体的一个或多个部分、片段或结构域。例如,本文所述的单结构域抗体可任选地经由接头序列(包括但不限于其他(单)结构域抗体,例如由Ward等人所述的dAb)连接至常规(优选地人)VH或VL结构域,或VH或VL结构域的天然或合成类似物。
所述至少一种单结构域抗体还可以任选地经由接头序列连接至一个或多个(优选地人)重链恒定区1(CH1)、重链恒定区2(CH2)和/或重链恒定区3(CH3)结构域。例如,与合适的CH1结构域连接的单结构域抗体可以例如与合适的轻链一起用于生成类似于常规Fab片段或F(ab')
本文所述的一种或多种单结构域抗体可以连接(任选地经由合适的接头或铰链区)至一个或多个恒定结构域(例如,可以用作Fc部分的一部分/形成Fc部分的2个或3个恒定结构域)、Fc部分,和/或赋予本文所述的多肽一种或多种效应子功能和/或可赋予结合一种或多种Fc受体的能力的一个或多个抗体部分、片段或结构域。例如,为此目的,且不限于此,一个或多个另外的氨基酸序列可以包含抗体的一个或多个CH2结构域和/或CH3结构域,例如来自重链抗体(如本文所述),更优选地来自常规人四链抗体的CH2结构域和/或CH3结构域;和/或可以形成例如来自IgG(例如,来自IgG1、IgG2、IgG3或IgG4)、来自IgE或来自另一人Ig(例如,IgA、IgD或IgM)的Fc区(的一部分)。例如,WO 94/04678描述了包含骆驼科VHH结构域或其人源化衍生物的重链抗体,在所述重链抗体中骆驼科CH2和/或CH3结构域已被人CH2和CH3结构域替代,以便提供由以下组成的免疫球蛋白:2条重链,每条重链包含VHH结构域(例如,单结构域抗体)和人CH2结构域和CH3结构域(但没有CH1结构域),所述免疫球蛋白具有由CH2结构域和CH3结构域提供的效应子功能,并且所述免疫球蛋白可以在没有任何轻链存在的情况下发挥作用。可以连接至本文所述的单结构域抗体以提供效应子功能的其他氨基酸序列对于技术人员来说将是清楚的,并且可以基于期望的效应子功能来选择。例如,参考WO 04/058820、WO 99/42077、WO 02/056910和WO 05/017148,以及由Holliger和Hudson进行的评论,出处同上)。与本文所述的对应单结构域抗体相比,将本文所述的单结构域抗体与Fc部分偶联可导致半衰期延长。对于一些应用,使用赋予延长的半衰期而没有任何生物学上显著的效应子功能的Fc部分和/或恒定结构域(例如,CH2结构域和/或CH3结构域)也可能是合适的或甚至是优选的。具有延长的体内半衰期的包含一个或多个单结构域抗体和一个或多个恒定结构域的其他合适的构建体对于技术人员来说将是清楚的,并且可以例如包含任选地经由接头序列与CH3结构域连接的两个单结构域抗体。一般而言,具有延长的半衰期的任何融合蛋白或衍生物的分子量,即肾吸收的截止值优选大于50kD。
包含本文所述的单结构域抗体和来自IgG的Fc区的例示性构建体如下表4中所示。
表4.例示性抗SARS-CoV-2的单体Fc融合构建体的氨基酸序列。
/>
/>
包含三个本文所述的单结构域抗体和来自IgG的Fc区的例示性构建体如下表5中所示。
表5.例示性抗SARS-CoV-2的三聚Fc融合构建体的氨基酸序列。
/>
/>
/>
为了形成本文所述的多肽,可以将一个或多个本文所述的氨基酸序列连接(任选地经由合适的接头或铰链区)至天然存在的、合成的或半合成的恒定结构域(或其类似物、变体、突变体、部分或片段),所述恒定结构域具有降低的(或基本上没有)自缔合成二聚体的倾向(例如,与常规四链抗体中天然存在的恒定结构域相比)。此类单体(例如,非自缔合的)Fc链变体或其片段对于技术人员来说将是清楚的。例如,Helm等人,J Biol Chem 1996271 7494描述了可用于本文所述的多肽链的单体Fc链变体。
而且,此类单体Fc链变体优选使得它们仍然能够结合补体或相关Fc受体(取决于它们所衍生自的Fc部分),和/或使得它们仍然具有它们所衍生自的Fc部分的效应子功能中的一些或所有效应子功能(或在降低水平下仍适用于预期用途)。或者,在本文所述的此类多肽链中,单体Fc链可用于赋予多肽链延长的半衰期,在这种情况下单体Fc链也可不具有或基本上不具有效应子功能。
本文所述的二价/多价、双特异性/多特异性或双互补位/多互补位多肽还可以连接至Fc部分。
另外的氨基酸序列还可以形成信号序列或前导序列,所述信号序列或前导序列指导本文所述的单结构域抗体或多肽在合成后从宿主细胞分泌(例如,以提供本文所述的多肽的前体形式、原形式或前原形式),具体取决于用于表达本文所述的多肽的宿主细胞。
另外的氨基酸序列还可以形成序列或信号,所述序列或信号允许本文所述的单结构域抗体或多肽被引导朝向和/或渗透或进入特定器官、组织、细胞或细胞的部分或区室,并且/或允许本文所述的单结构域抗体或多肽渗透或跨越生物屏障,例如细胞膜、细胞层(例如上皮细胞层)、肿瘤(包括实体瘤)或血脑屏障。此类氨基酸序列的合适示例对于技术人员来说将是清楚的(参见例如,WO 08/020079)。
一个或多个另外的氨基酸序列包含至少一种另外的单结构域抗体,以便提供本文所述的多肽,所述多肽包含至少两种,例如三种、四种、五种或更多种单结构域抗体,其中所述单结构域抗体可以任选地经由一个或多个接头序列连接。包含两种或更多种单结构域抗体(其中至少一种是本文所述的单结构域抗体)的本文所述的多肽在本文中也称为本文所述的“多价”多肽,并且此类多肽中所存在的单结构域抗体在本文中也被称为“多价格式”。
本文所述的多肽可包含至少两种单结构域抗体,其中至少一种单结构域抗体针对第一抗原(例如针对SARS-CoV-2)并且至少一种单结构域抗体针对第二抗原(例如不同于SARS-CoV-2),所述多肽也被称为本文所述的“多特异性”多肽,并且此类多肽中存在的单结构域抗体在本文中也被称为“多特异性格式”。因此,例如,本文所述的“双特异性”多肽是包含至少一种针对第一抗原(例如,SARS-CoV-2)的单结构域抗体和至少一种针对第二抗原(例如,不同于SARS-CoV-2)的另外的单结构域抗体的多肽,而本文所述的“三特异性”多肽是这样的多肽,所述多肽包含至少一种针对第一抗原(例如,SARS-CoV-2)的单结构域抗体、至少一种针对第二抗原(例如,不同于SARS-CoV-2)的另外的单结构域抗体,以及至少一种针对第三抗原(例如,不同于SARS-CoV-2和第二抗原)的单结构域抗体;等等。
因此,本文所述的双特异性多肽的一种形式是本文所述的二价多肽,所述二价多肽包含针对SARS-CoV-2的第一单结构域抗体和针对第二抗原的第二单结构域抗体,其中所述第一单结构域抗体和第二单结构域抗体可以任选地经由接头序列连接;而本文所述的三特异性多肽以其最简单的形式为本文所述的三价多肽,所述三价多肽包含针对SARS-CoV-2的第一单结构域抗体、针对第二抗原的第二单结构域抗体和针对第三抗原的第三单结构域抗体,其中所述第一、第二和第三单结构域抗体可以任选地经由一个或多个,特别是一个或多个,例如两个接头序列连接。
本文所述的多特异性多肽可包含至少一种针对SARS-CoV-2的单结构域抗体,以及任意数量的针对一种或多种不同于SARS-CoV-2的抗原的单结构域抗体。
此外,尽管本文所述的多肽中的各种单结构域抗体的特定顺序或布置可能对本文所述的最终多肽的性质(包括但不限于对SARS-CoV-2或对一种或多种其他抗原的亲和力、特异性或亲合力)有某种影响,但是所述顺序或布置通常并不重要,并且可以由技术人员任选地在基于本文公开内容的一些有限的常规实验之后选择。因此,当提及本文所述的特异性多价或多特异性多肽时,应当注意,除非另外明确指出,否则这涵盖相关单结构域抗体的任何顺序或布置。
本文所述的多肽可包含两种或更多种单结构域抗体和一种或多种另外的氨基酸序列(如本文所述)。
对于包含一个或多个VHH结构域的多价和多特异性多肽及其制备,还参考Conrath等人,J.Biol.Chem.,第276卷,10.7346-7350,2001;Muyldermans,Reviews in MolecularBiotechnology 74(2001),277-302;以及例如WO 96/34103和WO 99/23221。
本文所述的多特异性多肽的一个非限制性示例包含至少一种本文所述的单结构域抗体和至少一种提供延长的半衰期的单结构域抗体。此类单结构域抗体可以例如是针对血清蛋白,特别是人血清蛋白,例如人血清白蛋白、甲状腺素结合蛋白、(人)转铁蛋白、纤维蛋白原、免疫球蛋白(例如IgG、IgE或IgM),或针对WO 04/003019中列出的血清蛋白之一的单结构域抗体。在这些中,可结合血清白蛋白(例如,人血清白蛋白)或IgG(例如,人IgG)的单结构域抗体是优选的。
一般而言,本文所述的包含一种或多种本文所述的单结构域抗体的具有延长的半衰期的任何多肽,以及本文所述的单结构域抗体的任何衍生物或具有延长的半衰期的此类多肽的任何衍生物,优选具有为本文所述的对应单结构域抗体的半衰期的至少1.5倍、至少2倍、至少5倍、至少10倍或大于20倍长的半衰期。例如,具有延长的半衰期的此类衍生物或多肽可以具有与与本文所述的对应单结构域抗体相比延长大于1小时、大于2小时、大于6小时、大于12小时、或大于24小时、48小时或72小时的半衰期。
衍生物或多肽可以表现出在人中至少约12小时、至少24小时、至少48小时、至少72小时或更长时间的血清半衰期。例如,此类衍生物或多肽的半衰期可以为至少5天(例如,约5天至10天)、至少9天(例如,约9天至14天)、至少约10天(例如,约10天至15天)、或至少约11天(例如,约11天至16天)、至少约12天(例如,约12天至18天或更长时间)、或大于14天(例如,约14天至19天)。
在实施方式中,多肽能够结合一种或多种可以延长多肽的体内半衰期的分子。
本文所述的多特异性多肽可以包含至少一种本文所述的单结构域抗体,以及至少一种引导本文所述的多肽朝向特定器官、组织、细胞、或细胞的部分或区室和/或允许本文所述的多肽渗透或进入特定器官、组织、细胞、或细胞的部分或区室,和/或允许单结构域抗体渗透或跨越生物屏障,例如细胞膜、细胞层(例如上皮细胞层)、肿瘤(包括实体瘤)或血脑屏障的单结构域抗体。此类单结构域抗体的示例包括被引导朝向所需器官、组织或细胞的特异性细胞表面蛋白、标记物或表位的单结构域抗体,以及靶向脑部的单结构域抗体片段。
在本文所述的多肽中,所述一种或多种单结构域抗体和所述一种或多种多肽可以直接彼此连接和/或可以经由一个或多个合适的间隔区或接头或它们的任意组合彼此连接。
用于多价和多特异性多肽的合适的间隔区或接头对于技术人员来说将是清楚的,并且通常可以是本领域中用于连接氨基酸序列的任何接头或间隔区。优选地,所述接头或间隔区适合用于连接旨在用于药物用途的蛋白质或多肽。
一些优选的间隔区包括本领域中用于连接抗体片段或抗体结构域的间隔区和接头。这些包括例如本领域中用于构建双体抗体或scFv片段的接头。然而,在这方面,应该注意的是,在双体抗体和scFv片段中,所使用的接头序列应具有某一长度、一定程度的柔性和允许相关的VH和VL结构域聚集在一起以形成完整抗原结合位点的其他性质,由于每个单结构域抗体本身形成完整的抗原结合位点,因此对本文所述的多肽中所用的接头的长度或柔性没有特别限制。
例如,接头可以是合适的氨基酸序列,特别是具有介于1个至50个之间,优选地介于1个与30个之间,例如介于1个与10个之间的氨基酸残基的氨基酸序列。此类氨基酸序列的一些优选示例包括Gly-Ser接头,例如(Gly
其他合适的接头可包括有机化合物或聚合物,特别是适合用于用于制药用途的蛋白质的那些。例如,聚(乙二醇)部分已被用于连接抗体结构域。
接头和/或间隔区的长度和柔性可以基于所连接的多肽及其所需性质来选择。例如,在本文所述的包含针对相同抗原上的两个或更多个不同抗原决定簇(例如,针对抗原的不同表位和/或针对多聚受体、通道或蛋白质的不同亚基)的单结构域抗体的多特异性多肽中,接头的长度和柔性优选地使得所述接头允许每个单结构域抗体与其预期的抗原决定簇结合。
所使用的接头赋予本文所述的多肽一种或多种其他有利的性质或功能性,和/或提供一个或多个用于形成衍生物和/或用于附接官能团(例如,如本文针对本文所述的单结构域抗体的衍生物所述)的位点。例如,包含一个或多个带电氨基酸残基的接头可提供改善的亲水性质,而形成或包含小表位或标签的接头可用于检测、鉴定和/或纯化的目的。
当在本文所述的多肽中使用两个或更多个接头时,这些接头可以相同或不同。
为了易于表达和生产,本文所述的多肽可以是直链多肽。然而,本公开在其最广泛的意义上不限于此。例如,当本文所述的多肽包含三个或更多个单结构域抗体时,可以通过使用具有三个或更多个“臂”的接头来连接它们,其中每个“臂”连接至单结构域抗体,以便提供“星形”结构。例如也可以使用圆形构建体。
在实施方式中,本文所述的单结构域抗体可以结合包含与其他多肽序列融合的免疫原性或抗原性表位的多肽。例如,SARS-CoV-2多肽(例如,SARS-CoV-2的RBD或其免疫原性或抗原性片段)可以与免疫球蛋白(IgA、IgE、IgG、IgM)或其部分(CH1、CH2、CH3或其任何组合及其部分)或白蛋白(包括但不限于重组人白蛋白或其片段或变体)(参见例如美国专利号5,876,969、欧洲专利号0 413 622和美国专利号5,766,883)的恒定结构域融合,从而产生嵌合多肽。这种融合蛋白可有助于纯化并可延长体内半衰期。对于由人CD4多肽的前两个结构域和哺乳动物免疫球蛋白的重链或轻链的恒定区的各种结构域组成的嵌合蛋白,已经显示了这一点。参见例如,EP 394,827;Traunecker等人Nature 331:84-86(1988)。对于与FcRn结合配偶体(例如IgG或Fc片段)缀合的抗原(例如,胰岛素),已经证明增强了抗原跨上皮屏障到免疫系统的递送(参见例如,PCT公开WO 96/22024和WO 99/04813)。还已发现由于IgG部分二硫键而具有二硫键连接的二聚体结构的IgG融合蛋白在结合和中和其他分子方面比单独的单体多肽或其片段更有效。参见例如,Fountoulakis等人J.Biochem.,270:3958-3964(1995)。编码上述表位的核酸也可以与作为表位标签(例如,血凝素(“HA”)标签或flag标签)的感兴趣的基因重组,以帮助检测和纯化所表达的多肽。例如,由Janknecht等人描述的系统允许在人细胞系中表达的非变性融合蛋白的容易纯化(Janknecht等人991,Proc.Natl.Acad.Sci.USA 88:8972-897)。在这个系统中,将感兴趣的基因亚克隆到牛痘重组质粒中,使得基因的开放阅读框被翻译地融合到由六个组氨酸残基组成的氨基末端标签。该标签用作融合蛋白的基质结合结构域。将来自感染重组牛痘病毒的细胞的提取物加载到次氮基三乙酸镍(NI
在实施方式中,本文所述的单结构域抗体结合与异源抗原(例如多肽、碳水化合物、磷脂或核酸)融合的SARS-CoV-2多肽和/或其携带表位的片段。在特定实施方式中,所述异源抗原是免疫原。
抗SARS-CoV-2的单结构域抗体特异性
本文所述的单结构域抗体对SARS-CoV-2多肽或其片段或变体的结合特异性可以通过任何合适的手段来确定。用于测量结合特异性的合适测定的示例包括但不限于免疫沉淀或体外结合测定,例如放射免疫测定(radioimmunoassay,RIA)或酶联免疫吸附测定(enzyme-linked immunoadsorbent assay,ELISA)。也可以使用其他手段,例如表面等离子体共振。
单结构域抗体的结合亲和力可以例如通过Frankel等人Mol.Immunol.16:101-106,1979所述的Scatchard分析来确定。在实施方式中,通过抗原/抗体解离速率测量结合亲和力。在实施方式中,高结合亲和力通过竞争放射免疫测定测量。在实施方式中,结合亲和力通过ELISA测量。在实施方式中,抗体亲和力通过流式细胞术测量。
“特异性地结合”或“免疫特异性地结合”抗原(例如,SARS-CoV-2或其片段或变体)的单结构域抗体是以高亲和力结合抗原并且不显著结合其他无关抗原的单结构域抗体。
在实施方式中,本文所述的单结构域抗体以约50nM或更低的解离常数(K
本文所述的一些实施方式涉及以约50pM或更低的解离常数(K
本文所述的一些实施方式涉及结合多肽的单结构域抗体,所述多肽包含或替代地由以下组成:与具有SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ ID NO:127或它们的组合中的任一者所示的氨基酸序列的冠状病毒RBD多肽具有至少80%、85%、90%、91%、92%、93%或94同一,更优选地至少95%、96%、97%、98%、99%或100%同一的氨基酸序列的多肽。
本文所述的附加实施方式涉及结合多肽的单结构域抗体,所述多肽包含或替代地由以下组成:与具有SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ ID NO:127或它们的组合中的任一者所示的氨基酸序列的冠状病毒RBD多肽具有约90%至99%的序列同一性的氨基酸序列的多肽。本文所述的单结构域抗体可以选择性地结合与具有SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ ID NO:127或它们的组合中的任一者所示的氨基酸序列的冠状病毒RBD多肽具有至少约91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性的多肽。
本文所述的附加实施方式涉及结合多肽的单结构域抗体,所述多肽所述多肽包含或替代地由以下组成:与具有SEQ ID NO:128的氨基酸序列的SARS-CoV-2刺突多肽具有约90%至99%的序列同一性的氨基酸序列的多肽。本文所述的单结构域抗体可以选择性地结合与具有SEQ ID NO:128的氨基酸序列的SARS-CoV-2刺突多肽具有至少约91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性的多肽。在实施方式中,本文所述的单结构域抗体可以选择性地结合SEQ ID NO:128所示的多肽,所述多肽包含一种或多种突变,例如选自以下的一种或多种突变:R683G、K417N、E484K和/或N510Y。在实施方式中,本文所述的单结构域抗体可以选择性地结合SEQ ID NO:128所示的多肽,所述多肽包含以下突变:K417N、E484K和N510Y。
本文所述的单结构域抗体可以结合SEQ ID NO:124、SEQ ID NO:125、SEQ ID NO:126、SEQ ID NO:127、SEQ ID NO:128或它们的组合中的任一者所示的多肽的片段、变体、衍生物或类似物,所述多肽为例如(i)多肽,在所述多肽中氨基酸残基中的一个或多个氨基酸残基被保守或非保守氨基酸残基(优选地保守氨基酸残基)取代并且这种经取代的氨基酸残基可以是或可以不是由遗传密码编码的氨基酸残基,或(ii)多肽,在所述多肽中氨基酸残基中的一个或多个氨基酸残基包含取代基基团,或(iii)多肽,在所述多肽中所述多肽与另一种化合物(例如用于延长所述多肽的半衰期的化合物(例如,聚乙二醇))融合,或(iv)多肽,在所述多肽中附加氨基酸与多肽(例如IgG Fc融合区肽或前导序列或分泌序列或用于纯化多肽或蛋白原序列的序列)融合。
冠状病毒RBD多肽(例如,冠状病毒RBD,例如SARS-CoV-2 RBD)中功能所必需的氨基酸可以通过本领域已知的方法,例如定点诱变或丙氨酸扫描诱变来鉴定(Cunningham和Wells,Science 244:1081-1085(1989))。后一种工序在分子中的每个残基处引入了单个丙氨酸突变。然后测试所得突变体分子的功能活性,例如配体结合。因此,本文所述的单结构域抗体可以结合冠状病毒多肽中对于功能必需的氨基酸。在实施方式中,本文所述的单结构域抗体结合SARS-CoV-2多肽中对于感染必需的氨基酸,例如通过干扰SARS-CoV-2的RBD与ACE2的结合。在实施方式中,本文所述的单结构域抗体结合SARS-CoV-2多肽中的抑制或减少感染的氨基酸,例如通过干扰SARS-CoV-2的RBD与ACE2的结合。配体-受体结合的关键位点可以例如通过结构分析,例如结晶、核磁共振或光亲和标记来确定(Smith等人J.Mal.Biol.224:899-904(1992)和de Vos等人Science 255:306-312(1992))。
抗SARS-CoV-2的单结构域抗体的活性
本文所述的单结构域抗体中和一种或多种冠状病毒。例如,本文所述的单结构域抗体可以中和SARS-CoV-1、MERS-CoV、SARS-CoV-2、HCoV-OC43、HCoV-HKU1、HCoV-NL63、HCoV-229E或它们的组合。本文所述的抗体可以中和感染任何动物,优选地来自哺乳动物的冠状病毒。最优选地,本文所述的抗体中和感染人类的冠状病毒。
在实施方式中,本文所述的单结构域抗体结合一种或多种冠状病毒的RBD,从而中和所述一种或多种冠状病毒的感染性。例如,本文所述的单结构域抗体优选结合SARS-CoV-2的RBD并抑制RBD与ACE2的结合,从而导致对膜融合和病毒进入宿主细胞的抑制。在实施方式中,本文所述的单结构域抗体结合野生型SARS-CoV-2的RBD和/或SARS-CoV-2变体的RBD并抑制RBD与ACE2的结合,从而导致对膜融合和病毒进入宿主细胞的抑制。在实施方式中,SARS-CoV-2变体包含刺突蛋白中的一个或多个突变,例如在与SEQ ID NO:128中的R683、K417、E484和/或N501对应的位置处的一个或多个突变。在实施方式中,SARS-CoV-2变体在与SEQ ID NO:128对应的刺突蛋白中包含选自以下的一个或多个突变:R683G、K417N、E484K或N501Y。在实施方式中,SARS-CoV-2变体在与SEQ ID NO:128对应的刺突蛋白中包含以下突变:K417N、E484K和N501Y。
可以使用任何合适的方法来测量冠状病毒(例如SARS-CoV-2)的中和作用。例如,其中用SARS-CoV-2刺突蛋白或其RBD假型化慢病毒颗粒的体外中和测定可用于测量本文所述的单结构域抗体的中和作用。此类测定是本领域中众所周知的。通常,将细胞接种在细胞培养板中,并在预定数量单位的所选冠状病毒或假型化病毒加各种浓度的候选单结构域抗体的存在下孵育达某一合适的时段(例如,24-48小时)。抑制冠状病毒感染性的候选单结构域抗体将比在等同浓度的对照抗体存在下测量的冠状病毒感染性的基线水平抑制更多的冠状病毒感染性。
用于测量SARS-CoV-2中和作用的另一种合适方法是例如用于测量本文所述的单结构域抗体阻止SARS-CoV-2 RBD与其宿主细胞受体ACE2的结合的能力的体外受体结合测定。
其他合适的测定对于本领域技术人员来说将是显而易见的。
任选地,在中和测定中,如与在等同浓度的对照抗体存在下测量的基线感染性相比,中和冠状病毒(例如,SARS-CoV-2)的候选单结构域抗体将抑制冠状病毒的感染性的至少和/或约30%、或至少和/或约40%、或至少和/或约50%、或至少和/或约60%、或至少和/或约70%、或至少和/或约80%、或至少和/或约90%、或至少和/或约95%、或至少和/或约96%、或至少和/或约97%、或至少和/或约98%、或至少和/或约99%、或约100%。
如果需要结合特定冠状病毒RBD决定簇的单结构域抗体,则可以筛选候选抗体是否存在对野生型冠状病毒RBD和对在如上所述感兴趣的决定簇处包含Ala取代的突变冠状病毒RBD的差异亲和力。在一个方面中,可以在免疫沉淀或免疫吸附测定中测试候选单结构域抗体与野生型冠状病毒RBD和突变体冠状病毒RBD的结合。例如,可以使用捕获ELISA,其中用给定浓度的野生型冠状病毒RBD或等浓度的突变体冠状病毒RBD包被板,使经包被的板与等浓度的候选单结构域抗体接触,并酶促地检测结合的单结构域抗体,例如,使结合的单结构域抗体与HRP偶联的抗Ig抗体接触并使HRP颜色反应显色。与感兴趣的特定冠状病毒RBD决定簇结合的候选抗体将表现为与野生型冠状病毒RBD的结合活性大于所述候选抗体与对应的经Ala取代的冠状病毒RBD突变体的结合活性(例如,与野生型冠状病毒RBD的结合水平高于与突变体冠状病毒RBD的背景结合水平)。任选地,与感兴趣的特定冠状病毒RBD决定簇结合的候选单结构域抗体将表现为与对应的经Ala取代的冠状病毒RBD突变体的结合活性为所述抗体与野生型冠状病毒RBD的结合活性的小于约50%、或小于约30%、或小于约20%、或小于约10%、或小于约7%、或小于约6%、或小于约5%、或小于约4%、或小于约3%、或小于约2%、或小于约1%、或约0%,例如如通过将针对使用冠状病毒RBD突变体吸附剂的捕获ELISA观察到的HRP颜色反应光密度除以针对使用野生型冠状病毒RBD吸附剂的捕获ELISA观察到的HRP颜色反应光密度所确定的。
本文所述的单结构域抗体可具有本文所述的冠状病毒活性抑制性质和冠状病毒RBD决定簇结合性质的组合。与这些实施方式对应的单结构域抗体可通过使用本文所述的用于选择具有冠状病毒抑制形状的单结构域抗体的冠状病毒竞争性结合和/或活性抑制测定与本文所述的用于选择具有独特冠状病毒RBD决定簇结合性质的单结构域抗体的免疫沉淀或免疫吸附筛选工序的组合来获得。
抗SARS-CoV-2的单结构域抗体的模拟物
进一步,本文所述的单结构域抗体可继而用于使用本领域技术人员众所周知的技术生成“模拟”冠状病毒RBD多肽(例如SARS-CoV-2 RBD)的单结构域抗体。(参见例如,Greenspan和Bona,FASEB J.7(5):437-444(1993);和Nissinoff,J.Immunol.147(8):2429-2438(1991))。例如,本文所述的结合冠状病毒RBD并竞争性抑制冠状病毒与细胞的结合的单结构域抗体(如通过本领域中众所周知的测定,例如下文公开的测定所确定的)可用于产生抗独特型,所述抗独特型“模拟”与ACE2结合的冠状病毒RBD并因此结合并中和冠状病毒。此类中和抗独特型(包括包含或替代地由单结构域抗体片段或变体(例如此类抗独特型的Fab片段)组成的分子)可在治疗方案中用于中和冠状病毒。例如,此类抗独特型抗体可用于结合冠状病毒RBD,从而阻断冠状病毒结合细胞,例如ACE2。
抗SARS-CoV-2的单结构域抗体的交叉反应性
本文所述的单结构域抗体还可以根据其交叉反应性来描述或指定。包括不结合本文所述的多肽的任何其他类似物、直向同源物或同源物的单结构域抗体。本公开还包括这样的单结构域抗体,所述单结构域抗体结合与本文所述的多肽具有至少95%、至少90%、至少85%、至少80%、至少75%、至少70%、至少65%、至少60%、至少55%、和至少50%同一性(如使用本领域已知和本文所述的方法计算的)的多肽。
本文所述的单结构域抗体可以与其他冠状病毒及其对应表位交叉反应。本公开还包括这样的单结构域抗体,所述单结构域抗体不结合与本文所述的多肽具有小于95%、小于90%、小于85%、小于80%、小于75%、小于70%、小于65%、小于60%、小于55%和小于50%的同一性(如使用本领域已知的和本文所述的方法计算的)的多肽。在特定实施方式中,上述交叉反应性是关于任何单一特异性抗原性或免疫原性多肽,或本文所公开的特异性抗原性和/或免疫原性多肽中的2种、3种、4种、5种或更多种特异性抗原性和/或免疫原性多肽的组合。本公开另外包括这样的单结构域抗体,所述单结构域抗体结合由在杂交条件(如本文所述)下与本文所述的多核苷酸杂交的多核苷酸编码的多肽。
在实施方式中,本文所述的单结构域抗体(包括包含或替代地由其变体的抗体片段组成的分子)免疫特异性结合一种或多种SARS-CoV-2变体,包括但不限于包含在刺突蛋白中以下位置处的一个或多个位置处的突变的变体:R683、K417、E484、N501。在实施方式中,本文所述的单结构域抗体(包括包含或替代地由其变体的抗体片段组成的分子)免疫特异性结合一种或多种SARS-CoV-2变体,包括但不限于包含在刺突蛋白中的以下突变中的一个或多个突变的变体:R683G、K417N、E484K和/或N501Y。在实施方式中,本文所述的单结构域抗体(包括包含或替代地由其变体的抗体片段组成的分子)免疫特异性结合一种或多种SARS-CoV-2变体,包括但不限于包含在刺突蛋白中的以下突变的变体:K417N、E484K和N501Y。
在实施方式中,本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)免疫特异性地结合SARS-CoV-2并且不与任何其他抗原交叉反应。
变体和衍生物
还如上所述,本公开还提供了这样的单结构域抗体,所述单结构域抗体包含或替代地由本文所述的VHH结构域和CDR的变体(包括衍生物)组成,所述单结构域抗体免疫特异性地结合SARS-CoV-2。本领域技术人员已知的标准技术可用于在编码本文所述的分子的核苷酸序列中引入突变,包括例如导致氨基酸取代的定点诱变和PCR介导的诱变。优选地,相对于参考VH结构域、CDR1、CDR2或CDR3,变体(包括衍生物)编码少于50个氨基酸取代、少于40个氨基酸取代、少于30个氨基酸取代、少于25个氨基酸取代、少于20个氨基酸取代、少于15个氨基酸取代、少于10个氨基酸取代、少于5个氨基酸取代、少于4个氨基酸取代、少于3个氨基酸取代、或少于2氨基酸取代。在实施方式中,变体在一个或多个预测的非必需氨基酸残基处具有保守氨基酸取代。“保守氨基酸取代”如上文所定义并且通常是其中氨基酸残基被具有带相似电荷的侧链的氨基酸残基取代的氨基酸取代。本领域已经定义了具有带相似电荷的侧链的氨基酸残基家族。这些家族包括具有以下侧链的氨基酸:碱性侧链(例如,赖氨酸、精氨酸、组氨酸)、酸性侧链(例如,天冬氨酸、谷氨酸)、不带电的极性侧链(例如,甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、非极性侧链(例如,丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、β-分支侧链(例如,苏氨酸、缬氨酸、异亮氨酸)和芳香族侧链(例如,酪氨酸、苯丙氨酸、色氨酸、组氨酸)。或者,可以沿着编码序列的全部或部分随机引入突变,例如通过饱和诱变,并且可以筛选所得突变体的生物活性以鉴定出保留活性(例如,结合SARS-CoV-2的能力)的突变体。在诱变后,可以常规地表达所编码的蛋白质,并且可以使用本文所述的技术或通过本领域已知的常规修饰技术来确定所编码的蛋白质的功能和/或生物活性(例如,免疫特异性地结合SARS-CoV-2的能力)。
本文所述的单结构域抗体包括例如通过将任何类型的分子共价连附接至单结构域抗体,使得共价附接不影响抗体免疫特异性地结合SARS-CoV-2的能力而修饰的衍生物(例如,变体)。例如但非限制性地,本文所述的衍生物包括已被修饰(例如通过糖基化、乙酰化、聚乙二醇化、磷酸化、酰胺化、通过已知的保护/封闭基团衍生化、蛋白水解切割、与细胞配体或其他蛋白质连接等进行修饰)的单结构域抗体。许多化学修饰中的任何一种化学修饰都可以通过已知技术进行,包括但不限于特异性化学切割、乙酰化、甲酰化、衣霉素的代谢合成等。此外,该衍生物可包含一种或多种非经典氨基酸。
抗SARS-CoV-2的单结构域抗体的序列和结构
在特定实施方式中,本文所述的免疫特异性地结合SARS-CoV-2的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)包含或替代地由核苷酸序列所编码的氨基酸序列组成,所述核苷酸序列在严格条件(例如,在约45℃下在6x氯化钠/柠檬酸钠(SSC)中与过滤器结合的DNA杂交,随后在约50-65℃下在0.2xSSC/0.1% SDS中洗涤一次或多次)下、在高度严格条件(例如,在约45℃下在6xSSC中与过滤器结合的核酸杂交,随后在约68℃下在0.1xSSC/0.2% SDS中洗涤一次或多次)下、或在本领域技术人员已知的其他严格杂交条件下与同编码本文所公开的VHH结构域中的一者的核苷酸序列互补的核苷酸序列杂交(参见例如,Ausubel,F.M.等人编辑,1989,Current Protocols in MolecularBiology,第I卷,Green Publishing Associates,Inc.和John Wiley&Sons,Inc.,NewYork,第6.3.1-6.3.6页和第2.10.3页)。在实施方式中,本文所述的免疫特异性地结合SARS-CoV-2的单结构域抗体包含或替代地由核苷酸序列编码的氨基酸序列组成,所述核苷酸序列在严格条件下(例如,在如上所述的条件下杂交)或在本领域技术人员已知的其他严格杂交条件下与同编码本文所公开的CDR中的一者的核苷酸序列互补的核苷酸序列杂交。在实施方式中,本文所述的免疫特异性地结合SARS-CoV-2的单结构域抗体包含或替代地由核苷酸序列编码的氨基酸序列组成,所述核苷酸序列在严格条件下(例如,在如上所述的条件下杂交)或在本领域技术人员已知的其他严格杂交条件下与同编码本文所公开的CDR3中的一者的核苷酸序列互补的核苷酸序列杂交。本公开还提供了编码这些单结构域抗体的核酸分子。
本公开还提供了具有与本文所述的抗体中的一种或多种抗体相同的一种或多种生物学特性的单结构域抗体(包括包含或替代地由单结构域抗体片段或其变体组成的分子)。“生物学特性”意指单结构域抗体的体外或体内活性或性质,例如结合SARS-CoV-2(例如,SARS-CoV-2的RBD和/或SARS-CoV-2的抗原和/或表位区域)的能力、实质上阻断SARS-CoV-2/ACE2结合的能力,或阻断SARS-CoV-2感染性的能力。任选地,本文所述的单结构域抗体将与本文具体提及的单结构域抗体中的至少一种单结构域抗体结合相同的表位。这种表位结合可以使用本领域已知的测定常规地确定。
本公开还提供了这样的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子),所述单结构域抗体中和SARS-CoV-2或其变体或片段,所述单结构域抗体包含或替代地由以下组成:具有在SEQ ID NO:1至SEQ ID NO:96和SEQ ID NO:112至SEQ ID NO:123中所包含的氨基酸序列,或具有在由SEQ ID NO:97至SEQ ID NO:111中的任一者编码的多肽内所包含的氨基酸序列的部分(例如,VHH结构域、CDR1、CDR2、或CDR3),或其片段或变体,如上文进一步所定义。
在其他实施方式中,本公开提供了这样的单结构域抗体,所述单结构域抗体竞争性抑制包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合。在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低介于1%与10%之间。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少10%且至多20%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少20%且至多30%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少30%且至多40%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少40%且至多50%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少50%且至多60%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少60%且至多70%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少70%且至多80%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少80%且至多90%。
在优选的实施方式中,本发明提供了这样的单结构域抗体,所述单结构域抗体在竞争性抑制测定中将包含本文所述的片段(例如,VHH结构域、CDR1、CDR2或CDR3)或其变体的单结构域抗体与SARS-CoV-2多肽的结合降低至少90%且至多100%。
本公开还提供了免疫特异性结合SARS-CoV-2的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)的混合物,其中所述混合物具有至少一种、两种、三种、四种、五种或更多种不同的本文所述的单结构域抗体。具体地,本公开提供了免疫特异性结合SARS-CoV-2的RBD的不同单结构域抗体的混合物。在实施方式中,本公开提供了至少2种,优选至少4种、至少6种、至少8种、至少10种、至少12种、至少15种、至少20种或至少25种不同的免疫特异性结合SARS-CoV-2的单结构域抗体的混合物,其中所述混合物中的至少1种、至少2种、至少4种、至少6种或至少10种单结构域抗体是本文所述的单结构域抗体。在实施方式中,混合物中的每种单结构域抗体是本文所述的单结构域抗体。
单结构域抗体组
本公开还提供了免疫特异性结合SARS-CoV-2的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)的组,其中所述组具有至少一种、两种、三种、四种、五种或更多种不同的本文所述的单结构域抗体。具体地,本公开提供了免疫特异性结合SARS-CoV-2的RBD的不同单结构域抗体的组。在实施方式中,本公开提供了具有不同SARS-CoV-2亲和力、不同SARS-CoV-2特异性或不同解离速率的单结构域抗体的组。本公开提供了至少10种,优选地至少25种、至少50种、至少75种、至少100种、至少125种、至少150种、至少175种、至少200种、至少250种、至少300种、至少350种、至少400种、至少450种、至少500种、至少550种、至少600种、至少650种、至少700种、至少750种、至少800种、至少850种、至少900种、至少950种、或至少1000种单结构域抗体的组。单结构域抗体组可在例如96孔板中用于测定,例如ELISA。
组合物
本公开还提供了包含一种或多种单结构域抗体(包括包含或替代地由本文所述的抗体片段或变体组成的分子)的组合物。在一个实施方式中,本文所述的组合物包含一种、两种、三种、四种、五种或更多种单结构域抗体,所述单结构域抗体包含或替代地由以下组成:具有SEQ ID NO:1至SEQ ID NO:15中的任一者中所包含的VHH结构域中的任何一个或多个VHH结构域的氨基酸序列的多肽或其变体。在实施方式中,本文所述的组合物包含一种、两种、三种、四种、五种或更多种单结构域抗体,所述单结构域抗体包含或替代地由以下组成:具有SEQ ID NO:1至SEQ ID NO:15中的任一者中所包含的CDR1中的任何一个或多个CDR1(例如SEQ ID NO:27至SEQ ID NO:40中的任一者)的氨基酸序列的多肽或其变体。在实施方式中,本文所述的组合物包含一种、两种、三种、四种、五种或更多种单结构域抗体,所述单结构域抗体包含或替代地由以下组成:具有SEQ ID NO:1至SEQ ID NO:15中的任一者中所包含的CDR2中的任何一个或多个CDR2(例如SEQ ID NO:49至SEQ ID NO:62中的任一者)的氨基酸序列的多肽或其变体。在优选的实施方式中,本文所述的组合物包含一种、两种、三种、四种、五种或更多种单结构域抗体,所述单结构域抗体包含或替代地由以下组成:具有SEQ ID NO:1至SEQ ID NO:15中的任一者中所包含的CDR3中的任何一个或多个CDR3(例如SEQ ID NO:77至SEQ ID NO:91中的任一者)的氨基酸序列的多肽或其变体。
如下文更详细讨论的,本文所述的组合物可单独使用或与其他组合物组合使用。单结构域抗体(包括包含或替代地由本文所述的抗体片段或变体组成的分子)可以进一步在N末端或C末端处重组融合至异源多肽,或化学缀合(包括共价和非共价缀合)到多肽或其他组合物。例如,本文所述的单结构域抗体可以重组融合或缀合至可作为检测测定中的标记的分子以及效应分子,例如异源多肽、药物、放射性核素或毒素。参见例如,PCT公开WO92/08495;WO 91/14438;WO 89/12624;美国专利号5,314,995;和EP 396,387。
本文所述的组合物可以是药物组合物。组合物,包括药物组合物,可包含本文所述的单结构域抗体或抗原结合片段,以及佐剂、载体、缓冲剂、抗氧化剂、润湿剂、润滑剂、胶凝剂、增稠剂、结合剂、崩解剂、湿润剂、防腐剂、稀释剂、稳定剂、填充剂、赋形剂或它们的组合。
本文所述的单结构域抗体可以被配制用于通过吸入施用,优选地鼻内施用。本文所述的单结构域抗体可以被冻干,优选地稳定化,以用于通过吸入施用,优选地鼻内施用。
本文所述的单结构域抗体(包括包含或替代地由本文所述的抗体片段或变体组成的分子)可用于例如但不限于检测SARS-CoV-2,包括体外和体内诊断和治疗方法两者。例如,单结构域抗体可用于免疫测定以定性和定量地测量生物样品中SARS-CoV-2的水平。参见例如,Harlow等人Antibodies:ALaboratory Manual,(Cold Spring Harbor LaboratoryPress,第2版,1988)。
核酸、载体和宿主细胞
本公开还提供了分离的核酸分子,所述分离的核酸分子编码本文所述的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)。
本文描述了编码本公开的单结构域抗体的核酸分子。所述核酸可以存在于全细胞中、细胞裂解物中,或以部分纯化或基本上纯的形式存在。核酸可以通过标准技术从其他细胞组分或其他污染物(例如,其他细胞核酸或蛋白质)纯化分离,所述标准技术包括碱/SDS处理、CsCl显带(banding)、柱色谱法、琼脂糖凝胶电泳和本领域众所周知的其他技术。参见Ausubel等人(2011)Current Protocols in Molecular Biology John Wiley&Sons,Inc。本文所述的核酸可以是例如DNA或RNA,并且可包含或不包含内含子序列。核酸可以是cDNA分子。本文所述的核酸可使用标准分子生物学技术获得。对于由杂交瘤表达的单结构域抗体(例如,如本文进一步所述的从携带骆驼科免疫球蛋白基因的转基因小鼠制备的杂交瘤),编码由所述杂交瘤制成的重链抗体的cDNA可以通过标准PCR扩增或cDNA克隆技术获得。对于从免疫球蛋白基因文库获得的单结构域抗体(例如,使用噬菌体展示技术),可以从文库中回收编码所述单结构域抗体的核酸。具体地,简并密码子取代可通过产生例如其中一个或多个所选密码子的第三位被混合碱基和/或脱氧肌苷残基取代的序列来实现。Batzer等人(1991)Nucleic Acid Res.19:5081;Ohtsuka等人(1985)J.Biol.Chem.260:2605-08;Rossolini等人(1994)Mol.Cell.Probes 8:91-98。本文所述的核酸还可以是合成的,例如已经适应于在预期宿主细胞或宿主生物体中表达的具有密码子使用的DNA。
本文所述的核酸还可以是载体的形式、存在于载体中和/或作为载体的一部分,所述载体为例如质粒、粘粒或YAC,所述载体也可以是基本上分离的形式。
本文所述的核酸可以基于本文提供的关于本文所述的多肽的氨基酸序列的信息通过任何合适的方式制备或获得,和/或可以从合适的天然来源分离。为了提供类似物,可以例如对编码天然存在的VHH结构域的核苷酸序列进行定点诱变以提供编码所述类似物的本文所述的核酸。此外,若干核苷酸序列,例如编码单结构域抗体的至少一种核苷酸序列和例如编码一种或多种接头的核酸,可以以合适的方式连接在一起以提供本文所述的核酸。
用于生成本文所述的核酸的技术对于技术人员来说将是清楚的,并且可以包括但不限于自动化DNA合成;定点诱变;组合两个或更多个天然存在的和/或合成的序列(或其两个或更多个部分);引入导致截短的表达产物的表达的突变;引入一个或多个限制性位点(例如,以创建可以使用合适的限制性酶容易地消化和/或连接的盒和/或区域);和/或使用例如天然存在形式的VHH结构域序列的序列作为模板,借助于使用一个或多个“错配”引物的PCR反应引入突变。这些和其他技术对于本领域技术人员来说将是清楚的。
本文所述的核酸还可以是遗传构建体的形式、存在于遗传构建体中和/或作为遗传构建体的一部分。此类遗传构建体通常包含至少一种本文所述的核酸,所述核酸任选地连接至本身已知的遗传构建体的一个或多个元件(例如一个或多个合适的调控元件(例如,合适的启动子、增强子、终止子等))和本文提及的遗传构建体的另外元件。包含至少一种本文所述的核酸的此类遗传构建体在本文中也称为“本文所述的遗传构建体”。
本文所述的遗传构建体可以是DNA或RNA,并且优选地是双链DNA。本文所述的遗传构建体还可以是适合于转化预期的宿主细胞或宿主生物体的形式、适合于整合到预期的宿主细胞的基因组DNA中的形式,或适合于在预期宿主生物体中独立复制、维持和/或遗传的形式。例如,本文所述的遗传构建体可以是载体的形式,所述载体为例如质粒、粘粒、YAC、病毒载体或转座子。特别地,载体可以是表达载体,例如可以提供(例如,在合适的宿主细胞、宿主生物体和/或表达系统中)体外和/或体内表达的载体。
在非限制性方面中,本文所述的遗传构建体包含
i)至少一种本文所述的核酸;所述核酸可操作地连接至
ii)一个或多个调控元件,例如启动子和任选的合适的终止子;以及任选地还有
iii)本身已知的遗传构建体的一个或多个另外的元件。
本文所述的核酸和/或本文所述的遗传构建体可用于转化宿主细胞或宿主生物体,例如用于表达和/或产生本文所述的氨基酸序列、单结构域抗体或多肽。应当清楚的是,合适的宿主或宿主细胞可以是例如任何合适的真菌、原核或真核细胞,或细胞系或任何合适的真菌、原核或真核生物体;以及已知用于表达和产生抗体和抗体片段(包括但不限于(单)结构域抗体和scFv片段)的所有其他宿主或宿主细胞,这对于技术人员来说将是清楚的。
本文所述的氨基酸序列、单结构域抗体和多肽还可被引入并表达于多细胞生物体的一个或多个细胞、组织或器官中,例如以用于预防和/或治疗目的(例如,作为基因疗法)。
对于单结构域抗体在细胞中的表达,它们也可以表达为“胞内抗体”,例如如在WO94/02610、WO 95/22618和US 7004940;WO 03/014960;Cattaneo,A.&Biocca,S.(1997)Intracellular Antibodies:Development and Applications.Landes and Springer-Verlag;和Kontermann,Methods 34,(2004),163-170中所述。
本文所述的氨基酸序列、单结构域抗体和多肽还可以例如在转基因哺乳动物的乳汁中产生,例如在兔、牛、山羊或绵羊的乳汁中产生(关于用于将转基因引入哺乳动物中的一般技术,参见例如US 6,741,957、US 6,304,489和US 6,849,992);在植物或植物的各部分,包括但不限于它们的叶、花、果实、种子、根或块茎(例如,在烟草、玉蜀黍、大豆或苜蓿)中产生;或例如在家蚕(Bombix mori)的蛹中产生。
此外,本文所述的氨基酸序列、单结构域抗体和多肽还可以在无细胞表达系统中表达和/或产生,并且此类系统的合适示例对于本领域技术人员来说将是清楚的。一些优选的但非限制性的示例包括在小麦胚芽系统中;在兔网织红细胞裂解物中;或在大肠杆菌(E.coli)Zubay系统中表达。
如上所述,使用单结构域抗体的优点之一是可以通过在合适的细菌系统中表达来制备基于所述单结构域抗体的多肽,并且合适的细菌表达系统、载体、宿主细胞、调控元件等对于本领域技术人员来说将是清楚的。然而,应当注意的是,本公开在其最广泛的意义上并不限于在细菌系统中的表达。
优选地,使用体内或体外表达系统,例如细菌表达系统,所述体内或体外表达系统以适合于制药用途的形式提供本文所述的多肽,并且此类表达系统对于技术人员来说也将是清楚的。如技术人员也将清楚的是,适用于制药用途的本文所述的多肽可以使用肽合成技术来制备。
对于工业规模的生产,用于单结构域抗体或含有单结构域抗体的蛋白质治疗剂的(工业)生产的优选异源宿主包括适合于大规模表达/生产/发酵,并且特别适合于大规模药物(例如,GMP级)表达/生产/发酵的大肠杆菌、毕赤酵母(Pichia pastoris)、酿酒酵母(S.cerevisiae)菌株。此类菌株的合适示例对于本领域技术人员来说将是清楚的。
或者,哺乳动物细胞系,特别是中国仓鼠卵巢(CHO)细胞,可以用于大规模表达/生产/发酵,特别是用于大规模药物表达/生产/发酵。
具体表达系统的选择部分地取决于某些翻译后修饰,更具体地糖基化的要求。期望或需要糖基化的含有单结构域抗体的重组蛋白的生产将需要使用具有糖基化所表达的蛋白的能力的哺乳动物表达宿主。在这方面,本领域的技术人员将清楚所获得的糖基化模式(例如,所附接的残基的种类、数量和位置)将取决于用于表达的细胞或细胞系。优选地,使用人细胞或细胞系(例如,导致基本上具有人糖基化模式的蛋白质)或使用另一种哺乳动物细胞系,所述另一种哺乳动物细胞系可以提供与人糖基化基本上相同和/或在功能上相同或至少模仿人糖基化的糖基化模式。一般来说,原核宿主(例如大肠杆菌)不具有糖基化蛋白质的能力,而使用低等真核生物(例如酵母)通常会导致与人糖基化不同的糖基化模式。然而,应当理解的是,所有前述宿主细胞和表达系统都可以用于本公开,具体取决于要获得的所需氨基酸序列、单结构域抗体或多肽。
因此,本文所述的氨基酸序列、单结构域抗体或多肽可以是糖基化的。本文所述的氨基酸序列、单结构域抗体或多肽可以是非糖基化的。
本文所述的氨基酸序列、单结构域抗体或多肽可以在细菌细胞,特别是适合于大规模药物生产的细菌细胞,例如上述菌株的细胞中产生。
本文所述的氨基酸序列、单结构域抗体或多肽可以在酵母细胞,特别是适合于大规模药物生产的酵母细胞,例如上述物种的细胞中产生。
本文所述的氨基酸序列、单结构域抗体或多肽可以在哺乳动物细胞中,特别是在人细胞中或在人细胞系的细胞中,更特别是在适合于大规模药物生产的人细胞或人细胞系(例如上文所述的细胞系)的细胞中产生。
当宿主细胞中的表达用于产生本文所述的氨基酸序列、单结构域抗体和多肽时,本文所述的氨基酸序列、单结构域抗体和多肽可以在细胞内(例如,在胞质溶胶中、在周质中或在包涵体中)产生,然后从宿主细胞中分离并任选地进一步纯化;或者可以在细胞外(例如,在培养宿主细胞的培养基中)产生,然后从培养基中分离并任选地进一步纯化。因此,根据一个非限制性方面,本文所述的氨基酸序列、单结构域抗体或多肽是已在细胞内产生且已从宿主细胞分离,特别是从细菌细胞或从细菌细胞中的包涵体分离的氨基酸序列、单结构域抗体或多肽。本文所述的氨基酸序列、单结构域抗体或多肽可以是已经在细胞外产生的并且已经从培养宿主细胞的培养基中分离的氨基酸序列、单结构域抗体或多肽。
用于转化本文所述的宿主或宿主细胞的合适技术对于技术人员来说将是清楚的,并且可以取决于预期宿主细胞/宿主生物体和待使用的遗传构建体。在转化后,可以执行用于检测和选择已经用本文所述的核苷酸序列/遗传构建体成功转化的那些宿主细胞或宿主生物体的步骤。例如,这可以是基于本文所述的遗传构建体中存在的选择性标志物的选择步骤,或者是涉及例如使用特异性抗体检测本文所述的氨基酸序列的步骤。
转化的宿主细胞(其可以是稳定细胞系的形式)或宿主生物体(其可以是稳定突变系或菌株的形式)形成本文所述的另外方面。
优选地,这些宿主细胞或宿主生物体使得它们表达或能够表达(例如,在合适的条件下)本文所述的氨基酸序列、单结构域抗体或多肽(并且在宿主生物体的情况下:在所述宿主生物体的至少一个细胞、部分、组织或器官中表达)。本公开还提供了可以例如通过细胞分裂或通过有性或无性繁殖获得的本文所述的宿主细胞或宿主生物体的另外世代、子代和/或后代。
用于生产抗体的方法
本文所述的单结构域抗体(包括包含或替代地由单结构域抗体片段或变体组成的其他分子)可以通过本领域已知的任何用于合成抗体的方法产生,特别是通过化学合成,或优选地通过重组表达技术。
在实施方式中,本公开涉及一种用于生产针对SARS-CoV-2的单结构域抗体的方法。在一个方面中,所述方法至少包括以下步骤:
a)提供单结构域抗体序列的组、集合、集或文库;以及
b)筛选所述单结构域抗体序列的组、集合、集或文库中可结合SARS-CoV-2和/或具有SARS-CoV-2亲和力的单结构域抗体序列;以及
c)分离能够与SARS-CoV-2结合和/或具有SARS-CoV-2亲和力的一种或多种单结构域抗体。
在此类方法中,单结构域抗体序列的组、集合、集或文库可以是单结构域抗体序列的幼稚组、集合、集或文库;单结构域抗体序列的合成或半合成组、集合、集或文库;和/或已进行亲和力成熟的单结构域抗体序列的组、集合、集或文库。
在这种方法的优选方面中,单结构域抗体序列的组、集合、集或文库可以是单结构域抗体序列的免疫组、集合、集或文库,并且特别是来源于已用SARS-CoV-2或用基于所述SARS-CoV-2或来源于所述SARS-CoV-2的合适抗原决定簇(例如其抗原部分、片段、区域、结构域、环或其他表位)免疫的骆驼科物种的VHH序列的免疫组、集合、集或文库。在一个方面中,所述抗原决定簇可以是SARS-CoV-2的受体结合结构域(RBD)和/或刺突蛋白或其片段。
在上述方法中,单结构域抗体或VHH序列的组、集合、集或文库可以展示在噬菌体、噬菌粒、核糖体或合适的微生物(例如,酵母)上,以促进筛选。用于展示和筛选单结构域抗体序列(的组、集合、集或文库)的合适方法、技术和宿主生物体对于本领域技术人员来说将是清楚的,例如基于本文的进一步公开内容。还参见例如WO 03/054016和Hoogenboom(Nature Biotechnology,23(9):1105-1116(2005));
在另一方面中,用于生成单结构域抗体序列的方法至少包括以下步骤:
a)提供来源于表达免疫球蛋白序列的骆驼科物种的细胞集或样品;
b)筛选所述细胞集或样品中的(i)表达可与SARS-CoV-2结合和/或具有SARS-CoV-2亲和力的免疫球蛋白序列的细胞;和(ii)表达重链抗体的细胞,其中子步骤(i)和(ii)可以基本上作为单个筛选步骤执行或以任何合适的顺序作为两个单独的筛选步骤执行,以提供至少一种表达可以与SARS-CoV-2结合和/或具有SARS-CoV-2亲和力的重链抗体的细胞;以及
c)(i)从所述细胞中分离所述重链抗体中所存在的VHH序列;或(ii)从所述细胞中分离编码所述重链抗体中存在的VHH序列的核酸序列,然后表达所述VHH结构域。
在根据这个方面的方法中,所述细胞集或样品可以例如是B细胞集或样品。此外,在这种方法中,细胞样品可以来源于已用SARS-CoV-2或基于所述SARS-CoV-2或来源于所述SARS-CoV-2的合适抗原决定簇(例如其抗原部分、片段、区域、结构域、环、或其他表位)免疫的骆驼科。在一个方面中,所述抗原决定簇可以是SARS-CoV-2的RBD和/或刺突蛋白或其片段。
上述方法可以以任何合适的方式执行。例如参考EP 0 542 810、WO 05/19824、WO04/051268、WO 04/106377和WO 06/079372。步骤b)的筛选优选地使用流式细胞术技术(例如FACS)执行。为此,例如参考Lieby等人,Blood,第97卷,第12期,3820。
在另一方面中,用于生成针对SARS-CoV-2的氨基酸序列的方法可以至少包括以下步骤:
a)提供编码重链抗体或VHH序列的核酸序列的组、集合、集或文库;
b)筛选所述核酸序列的组、集合、集或文库中编码可与SARS-CoV-2结合和/或具有SARS-CoV-2亲和力的重链抗体或VHH序列的核酸序列;
c)分离所述核酸序列,然后分别表达所述重链抗体中存在的VHH序列或表达所述VHH序列。
在此类方法中,编码重链抗体或VHH序列的核酸序列的组、集合、集或文库可以例如是编码重链抗体或VHH序列的幼稚组、集合、集或文库的核酸序列的组、集合、集或文库;编码VHH序列的合成或半合成的组、集合、集或文库的核酸序列的组、集合、集或文库;和/或编码已进行亲和力成熟的VHH序列的组、集合、集或文库的核酸序列的组、集合、集或文库。
在这种方法的优选方面中,所述核酸序列的组、集合、集或文库可以是编码来源于已用SARS-CoV-2或用基于所述SARS-CoV-2或来源于所述SARS-CoV-2的合适抗原决定簇(例如其抗原部分、片段、区域、结构域、环或其他表位)免疫的骆驼科的重链抗体或VHH序列的免疫组、集合、集或文库。在一个方面中,所述抗原决定簇可以是SARS-CoV-2的RBD和/或刺突蛋白或其片段。
在上述方法中,所述核苷酸序列的组、集合、集或文库可以展示在噬菌体、噬菌粒、核糖体或合适的微生物(例如,酵母)上,以促进筛选。用于展示和筛选编码氨基酸序列的核苷酸序列(的组、集合、集或文库)的合适方法、技术和宿主生物体对于本领域技术人员来说将是清楚的,例如基于本文的进一步公开内容。还参见WO 03/054016和Hoogenboom(NatureBiotechnology,23(9):1105-1116(2005))。
本文所述的方法的筛选步骤也可以作为选择步骤执行。因此,如在本说明书中使用的术语“筛选”可包括选择、筛选,或选择和/或筛选技术的任何合适的组合。另外,当使用序列的组、集合、集或文库时,其可包含任何合适数量的序列,例如1个、2个、3个或约5个、10个、50个、100个、500个、1000个、5000个、10
另外,上述氨基酸序列的组、集合、集或文库中的序列中的一个或多个或全部序列可以通过理性或半经验方法(例如计算机建模技术或生物统计学或数据挖掘技术)来获得或定义。
此外,此类组、集合、集或文库可以包含一个、两个或更多个作为彼此的变体的序列(例如,具有设计的点突变或具有随机位置),包含来源于不同的天然多样化序列集合(例如,免疫文库)的多个序列,或多样化序列的任何其他来源(如例如在Hoogenboom等人,NatBiotechnol 23:1105(2005)和Binz等人,Nat Biotechnol 23:1247(2005)中所述)。此类序列的组、集合、集或文库可以展示在噬菌体粒子、核糖体、细菌、酵母细胞、哺乳动物细胞的表面上,并且连接至编码这些载体内的氨基酸序列的核苷酸序列。这使得此类组、集合、集或文库适合于选择工序以分离本文所述的所需氨基酸序列。更一般地,当序列在合适的宿主或宿主细胞上展示时,还可以首先从所述宿主或宿主细胞分离编码所需序列的核苷酸序列,然后通过在合适的宿主生物体中表达所述核苷酸序列来获得所需序列。同样,这可以以任何合适的方式执行。
用于获得针对SARS-CoV-2的VHH序列或单结构域抗体序列的又一技术涉及对能够表达重链抗体的转基因哺乳动物进行免疫(例如,以引发针对SARS-CoV-2的免疫应答和/或重链抗体);从所述转基因哺乳动物获得含有所述VHH序列或单结构域抗体序列(编码所述VHH序列或单结构域抗体序列的核酸序列)的合适生物样品(例如,血液样品、血清样品或B细胞样品);然后使用任何合适的技术(例如,本文所述的方法中的任何方法或杂交瘤技术)从所述样品开始生成针对SARS-CoV-2的VHH序列。例如,为此目的,可以使用在WO 02/085945、WO 04/049794和WO 06/008548以及Janssens等人,Proc.Natl.Acad.Sci.USA.2006年10月10日;103(41):15130-5中描述的表达重链抗体的小鼠和另外的方法和技术。例如,此类表达重链抗体的小鼠可以表达具有任何合适的单可变结构域的重链抗体,所述单可变结构域为例如来自天然来源的单可变结构域(例如,人单可变结构域、骆驼科单可变结构域或鲨鱼单可变结构域),以及例如合成或半合成的单可变结构域。
本公开还涉及VHH序列或单结构域抗体序列,所述VHH序列或单结构域抗体序列是通过以下方式获得的:上述方法;或任选地包括上述方法中的一种方法以及还有至少确定所述VHH序列或单结构域抗体序列的核苷酸序列或氨基酸序列的步骤的方法;以及以已知方式表达或合成所述VHH序列或单结构域抗体序列,例如通过在合适的宿主细胞或宿主生物体中表达或通过化学合成。
一旦本文所述的单结构域抗体分子(包括包含或替代地由抗体片段或其变体组成的分子)已被化学合成或重组地表达,则所述抗体分子可以通过本领域已知的任何用于纯化免疫球蛋白分子,或更通常地,蛋白质分子的方法来纯化,例如通过色谱法(例如,离子交换、亲和力,特别是通过对蛋白A后的特异性抗原的亲和力,以及尺寸分级柱色谱法(sizingcolumn chromatography))、离心、差异溶解度或通过任何其他用于纯化蛋白质的标准技术来纯化。进一步,本文所述的单结构域抗体可融合至本文所述或本领域中以其他方式已知的异源多肽序列,以有助于纯化。
本公开还提供了用于重组地产生本文所述的抗SARS-CoV-2的单结构域抗体的方法。产生本文所述的单结构域抗体的方法是本领域普通技术人员所众所周知的。本文所述的抗SARS-CoV-2的单结构域抗体还可以通过以下方式产生:使用本领域普通技术人员众所周知的常规技术构建包含操纵子和编码本文所述的抗SARS-CoV-2的单结构域抗体的DNA序列的表达载体。此外,本公开涉及载体,特别是质粒、粘粒、病毒、噬菌体和基因工程中常见的其他载体,所述载体包含本文所述的上述核酸分子。载体中所包含的核酸分子可以连接到确保在原核细胞和真核细胞中转录的调控元件。
载体包含有助于在靶宿主细胞内表达外源蛋白的操纵的元件。方便地,首先在细菌宿主(例如,大肠杆菌(E.coli))中进行序列的操纵和用于转化的DNA的产生,并且通常载体将包含有助于此类操纵的序列,包括细菌复制起点和合适的细菌选择标记物。选择标记物编码在选择性培养基中生长的经转化的宿主细胞存活或生长所必需的蛋白质。未用包含选择基因的载体转化的宿主细胞将无法在培养基中存活。典型的选择基因编码赋予对抗生素或其他毒素的抗性、补充营养缺陷型缺陷或供应从复合培养基无法得到的关键营养素的蛋白质。用于转化酵母的示例性载体和方法在本领域中有所描述。参见例如,Burke等人(2000)Methods in Yeast Genetics Cold Spring Harbor Laboratory Press;
编码抗SARS-CoV-2的单结构域抗体的多核苷酸可以可操作地连接到转录和翻译调控序列,所述转录和翻译调控序列提供用于多肽在酵母细胞中的表达。这些载体部件可以包括但不限于以下中的一者或多者:增强子元件、启动子和转录终止序列。也可包括用于分泌多肽的序列(例如,信号序列)。
当核酸被放置为与另一个核酸序列呈功能关系时,所述核酸是“可操作地连接的”。例如,如果用于信号序列的DNA被表达为参与多肽分泌的前蛋白,则所述DNA可操作地连接到所述多肽的DNA;如果启动子或增强子影响序列的转录,则所述启动子或增强子可操作地连接至编码序列。通常,“可操作地连接的”泛指连续连接的DNA序列,并且在分泌前导序列的情况下,泛指连续的且在阅读框中。然而,增强子不必是连续的。
启动子是位于结构基因的起始密码子上游(5')(通常在约100bp至1000bp内)的非翻译序列,所述非翻译序列控制它们可操作地连接至特定核酸序列的转录和翻译。此类启动子分为几类:诱导型、组成型和阻抑型启动子(例如,响应于不存在阻遏物而升高转录水平)。诱导型启动子可以响应于培养条件的一些变化(例如,存在或不存在营养素或温度变化),而在所述诱导型启动子的控制下启动升高水平的DNA转录。
通过本领域普通技术人员众所周知的常规技术将表达载体转染到宿主细胞中以产生转染的宿主细胞,将所述转染的宿主细胞通过本领域普通技术人员众所周知的常规技术培养以产生所述抗SARS-CoV-2的单结构域抗体。
用于表达抗SARS-CoV-2的单结构域抗体的宿主细胞可以是细菌细胞(例如大肠杆菌、酵母(例如,酿酒酵母(S.cerevisiae)))或真核细胞(例如,哺乳动物细胞系)。可使用为此目的明确定义类型的哺乳动物细胞,例如骨髓瘤细胞、3T3、HeLa、C6A2780、Vero、MOCKII、中国仓鼠卵巢(CHO)、Sf9、Sf21、COS、NSO或HEK293细胞系。
可构建载体的一般方法、产生宿主细胞所需的转染方法和从所述宿主细胞产生单结构域抗体及其片段所需的培养方法均包括常规技术。尽管优选地用于产生抗SARS-CoV-2的单结构域抗体的细胞系是哺乳动物细胞系,但是可以使用任何其他合适的细胞系,例如细菌细胞系,例如大肠杆菌源性细菌菌株,或酵母细胞系。
类似地,一旦产生,抗SARS-CoV-2的单结构域抗体就可以根据本领域的标准工序,例如错流过滤、硫酸铵沉淀和亲和柱色谱法进行纯化。
抗SARS-CoV-2的单结构域抗体的诊断用途
本文所述的特异性结合SARS-CoV-2的经标记的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)可用于诊断目的,以检测、诊断、预测或监测与冠状病毒感染相关联的疾病和/或疾患。本公开提供了对SARS-CoV-2的检测,所述检测包括:(a)使用本文所述的一种或多种免疫特异性结合SARS-CoV-2的单结构域抗体测定来自受试者的生物样品中SARS-CoV-2的存在;以及(b)将SARS-CoV-2的水平与对照(例如,没有已知冠状病毒感染的正常生物样品中)进行比较。
“生物样品”旨在指从受试者、体液、身体组织、体细胞、细胞系、组织培养物或可能包含SARS-CoV-2蛋白或mRNA的其他来源获得的任何流体和/或细胞。体液包括但不限于血清、血浆、尿液、滑液、脊髓液、唾液和粘液。组织样品可以取自身体中的几乎任何组织。组织样品也可以从尸检材料中获得。用于从哺乳动物获得组织活检和体液的方法是本领域中众所周知的。当生物样品包含mRNA时,组织活检是优选来源。
此外,本文所述的抗SARS-CoV-2的单结构域抗体可用于在用于检测细胞或组织中的SARS-CoV-2的诊断测定中使用,其中所述单结构域抗体如下文所述进行标记和/或固定在不溶性基质上。抗SARS-CoV-2单结构域抗体还可用于从重组细胞培养物或天然来源亲和纯化SARS-CoV-2多肽,例如SARS-CoV-2RBD多肽。
抗SARS-CoV-2的单结构域抗体可用于以多种众所周知的诊断测定方法中的任何一种诊断测定方法来检测SARS-CoV-2。例如,可以通过以下方式来测定生物样品中的SARS-CoV-2:从所需来源获得样品;将所述样品与抗SARS-CoV-2的单结构域抗体掺混以允许单结构域抗体形成与混合物中存在的任何SARS-COV-2的抗体/SARS-COV-2复合物;以及检测所述混合物中存在的任何抗体/SARS-COV-2复合物。可以通过本领域中已知的适合于特定样品的方法制备生物样品以用于测定。将样品与单结构域抗体掺混的方法和检测抗体/SARS-CoV-2复合物的方法是根据所使用的测定的类型选择的。此类测定包括竞争性和夹心式测定,以及空间阻抑测定。竞争性和夹心式方法采用相分离步骤作为方法的组成部分,而空间阻抑测定是在单一反应混合物中进行的。
用于SARS-CoV-2检测的分析方法使用以下试剂中的一种或多种试剂:标记的SARS-CoV-2类似物、固定化的SARS-CoV-2类似物、标记的抗SARS-CoV-2的单结构域抗体、固定化的抗SARS-CoV-2的单结构域抗体和立体缀合物。经标记的试剂也被称为“示踪剂”。
所使用的标签是任何可检测的功能性,所述功能性不会干扰SARS-CoV-2与抗SARS-CoV-2的单结构域抗体的结合。已知有许多标记用于免疫测定,示例包括可以直接检测的部分,例如荧光染料、化学发光和放射性标记,以及必须反应或衍生化才能检测到的部分(例如酶)。此类标记的示例包括放射性同位素
常规方法可用于将这些标记共价结合到蛋白质或多肽。例如,偶联剂(例如二醛、碳二亚胺、二马来酰亚胺、双亚胺酸酯、双重氮化联苯胺等)可用于用上述荧光、化学发光和酶标记来标记单结构域抗体。参见例如,美国专利号3,940,475(荧光测定法)和3,645,090(酶);Hunter等人Nature,144:945(1962);David等人Biochemistry,13:1014-1021(1974);Pain等人J.Immunol.Methods,40:219-230(1981);以及Nygren,J.Histochem.和Cytochem.,30:407-412(1982)。本文优选的标记是酶,例如辣根过氧化物酶和碱性磷酸酶。
此类标记(包括酶)与单结构域抗体的缀合是免疫测定技术领域的普通技术人员的标准操纵工序。参见例如,O'Sullivan等人“Methods for the Preparation of Enzyme-antibody Conjugates for Use in Enzyme Immunoassay,”Methods in Enzymology,编辑J.J.Langone和H.Van Vunakis,第73卷(Academic Press,New York,N.Y.,1981),第147-166页。
某些测定方法需要试剂的固定化。固定化需要将抗SARS-CoV-2的单结构域抗体与任何在溶液中保持游离的SARS-CoV-2分离。这通常是通过在测定工序之前使抗SARS-CoV-2的抗体或SARS-CoV-2类似物不溶解实现的,如通过吸附到水不溶性基质或表面(Bennich等人,美国专利号3,720,760)、通过共价偶联(例如,使用戊二醛交联)、或通过随后使抗SARS-CoV-2的抗体或SARS-CoV-2类似物不溶解(例如,通过免疫沉淀)。
被称为竞争性或夹心式测定的其他测定方法已在商业诊断行业中良好建立和广泛使用。
竞争性测定依赖于示踪剂SARS-CoV-2类似物与测试样品SARS-CoV-2竞争有限数量的抗SARS-CoV-2的单结构域抗体的抗原结合位点的能力。一般在竞争前或竞争后使抗SARS-CoV-2的单结构域抗体不溶解,然后将与抗SARS-CoV-2的单结构域抗体结合的示踪剂和SARS-CoV-2与未结合的示踪剂和SARS-CoV-2分离。这种分离是通过倾析(在预先使结合配偶体不溶解的情况下)或通过离心(在竞争反应后使结合配偶体沉淀的情况下)来完成的。测试样品SARS-CoV-2的量与如通过标记物质的量测量的结合的示踪剂的量成反比。准备具有已知量的SARS-CoV-2的剂量反应曲线,并与测试结果进行比较以定量地确定测试样品中存在的SARS-CoV-2的量。当酶被用作可检测标记物时,这些测定被称为ELISA系统。
被称为“均相”测定的另一种竞争性测定不需要相分离。在此,制备并使用酶与SARS-CoV-2的缀合物,使得当抗SARS-CoV-2的单结构域抗体与SARS-CoV-2结合时,抗SARS-CoV-2的单结构域抗体的存在会改变酶活性。在这种情况下,SARS-CoV-2或其免疫活性片段与酶(过氧化物酶)的双官能有机桥缀合。缀合物经选择以与抗SARS-CoV-2的单结构域抗体一起使用,以便抗SARS-CoV-2的单结构域抗体的结合抑制或增强标记的酶活性。这种方法本身以EMIT的名称被广泛实践。
空间缀合物在用于均相测定的空间位阻方法中使用。这些缀合物是通过以下方式合成的:将低分子量半抗原共价连接至SARS-CoV-2小片段,使得针对半抗原的抗体基本上不能与抗SARS-CoV-2的单结构域抗体同时结合缀合物。在这种测定工序下,测试样品中存在的SARS-CoV-2将结合抗SARS-CoV-2的单结构域抗体,从而允许抗半抗原与缀合物结合,从而导致缀合物半抗原的特性的变化,例如,当半抗原是荧光团时荧光的变化。
夹心式测定特别可用于SARS-CoV-2或抗SARS-CoV-2的单结构域抗体的测定。在顺序夹心式测定中,使用固定化的抗SARS-CoV-2的单结构域抗体来吸附测试样品SARS-CoV-2,通过洗涤去除测试样品,使用结合的SARS-CoV-2来吸附第二经标记的抗SARS-CoV-2的单结构域抗体,然后将结合的材料与残留的示踪剂分离。结合的示踪剂的量与测试样品SARS-CoV-2成正比。在“同步”夹心测定中,在添加经标记的抗SARS-CoV-2之前,不分离测试样品。使用抗SARS-CoV-2的单结构域抗体作为一种抗体和抗SARS-CoV-2的多克隆抗体作为另一种抗体的顺序夹心式测定可用于测试SARS-CoV-2的样品。
前述仅为用于SARS-CoV-2的示例性诊断测定。现在或以后开发的使用抗SARS-CoV-2的单结构域抗体来测定SARS-CoV-2的其他方法也包括在本发明的范围内,包括上述生物测定。
治疗方法
本文所述的特异性结合SARS-CoV-2的单结构域抗体(包括包含或替代地由抗体片段或其变体组成的分子)可用于诊断目的以检测、诊断、预测或监测冠状病毒感染(优选地SARS-CoV-2感染)和/或与其相关联的疾病或疾患(例如,COVID-19)。
抗SARS-CoV-2的单结构域抗体的治疗组合物和施用
通过将具有所需纯度的所述抗体与任选的生理学上可接受的载体、赋形剂或稳定剂混合来制备本文所述的抗SARS-CoV-2的单结构域抗体的治疗制剂以供以冻干饼或水溶液的形式储存(Remington:The Science and Practice of Pharmacy,第22版,Alfonso,R.,编辑,Mack Publishing Co.(Easton,Pa.:2012))。可接受的载体、赋形剂或稳定剂在所采用的剂量和浓度下对接受者无毒,并且包括缓冲剂,例如磷酸盐、柠檬酸盐和其他有机酸;抗氧化剂,包括抗坏血酸;低分子量(小于约10个残基)多肽;蛋白质,例如血清白蛋白、明胶或免疫球蛋白;亲水性聚合物,例如聚乙烯吡咯烷酮;氨基酸,例如甘氨酸、谷氨酰胺、天冬酰胺、精氨酸或赖氨酸;单糖、双糖和其他糖类物质,包括葡萄糖、甘露糖或糊精;螯合剂,例如EDTA;糖醇,例如甘露醇或山梨糖醇;成盐平衡离子,例如钠;和/或非离子表面活性剂,例如Tween、Pluronics或聚乙二醇(PEG)。
要用于体内施用的抗SARS-CoV-2的单结构域抗体必须是无菌的。这很容易通过在冻干和重构之前或之后经由无菌过滤膜过滤来实现。抗SARS-CoV-2的单结构域抗体通常将冻干形式或以溶液存储。
通常将治疗性抗SARS-CoV-2的单结构域抗体组合物放入具有无菌进入端口的容器(例如静脉内溶液袋或具有皮下注射针可刺穿的塞子的小瓶)中。
抗SARS-CoV-2的单结构域抗体的施用途径根据已知方法,例如通过静脉内、腹膜内、脑内、皮下、肌内、眼内、吸入、任选的鼻内、肺内、动脉内、脑脊髓内、或病灶内途径、口服、鞘内、肠胃外或通过如下所述的缓释系统注射或输注。还可以使用施用途径的任何合适的组合。在实施方式中,全身给予单结构域抗体。
缓释制备物的合适示例包括成形制品形式的半透性聚合物基质,例如膜或微胶囊。缓释基质包括聚酯、水凝胶、聚丙交酯(美国专利号3,773,919、EP 58,481)、L-谷氨酸和γ-乙基-L-谷氨酸的共聚物(Sidman等人Biopolymers,22:547-556(1983))、聚(2-羟乙基-甲基丙烯酸酯)(Langer等人J.Biomed.Mater.Res.,15:167-277(1981)和Langer,Chem.Tech.,12:98-105(1982))、乙烯乙酸乙烯酯(Langer等人,出处同上)或聚-D-(-)-3-羟基丁酸(EP 133,988)。缓释抗SARS-CoV-2的单结构域抗体组合物还包含脂质体包埋的抗体。包含抗体的脂质体是通过本身已知的方法制备的:DE 3,218,121;Epstein等人Proc.Natl.Acad.Sci.USA,82:3688-3692(1985);Hwang等人Proc.Natl.Acad.Sci.USA,77:4030-4034(1980);EP 52,322;EP 36,676;EP 88,046;EP 143,949;EP 142,641;日本专利申请83-118008;美国专利号4,485,045和4,544,545;以及EP 102,324。通常,脂质体是小的(约200-800埃)单层类型,其中脂质含量大于约30mol.%的胆固醇,所选比例经调节以实现最佳抗体疗法。
本文所述的抗SARS-CoV-2的单结构域抗体可以配制为经由鼻内和/或肺内途径,例如经由吸入施用。同样地,本文所述的抗SARS-CoV-2的单结构域抗体可以经由鼻内和/或肺内途径,例如经由吸入施用。
例如,本文所述的抗SARS-CoV-2的单结构域抗体和包含其的组合物可以以蒸气、滴剂、喷雾剂、气溶胶或粉末剂的形式递送。用于液体制剂的商购可得的喷雾器,包括射流喷雾器和超声喷雾器可用于施用。液体制剂可直接雾化,并且冻干粉可在重构后雾化。或者,抗SARS-CoV-2的单结构域抗体可以使用碳氟化合物制剂和定量吸入器雾化,或作为冻干和研磨的粉末吸入。
抗SARS-CoV-2的单结构域抗体和包含其的组合物可以通过被配置用于吸入施用的装置来递送。所述装置被配置用于吸入递送本文所述的单结构域抗体或抗原结合片段或组合物。所述装置可被配置用于经由鼻内、肺内或它们的组合进行吸入递送。
所述装置可以是吹药器、呼吸致动吸入器、机械粉末喷雾器、电力喷雾器(雾化器)、喷雾器、雾化器、气体驱动的喷雾系统、气体驱动的雾化器、或机械泵式喷雾器。
要在治疗上采用的抗SARS-CoV-2的单结构域抗体的“有效量”将取决于例如治疗目标、施用途径、所采用的抗SARS-CoV-2的单结构域抗体的类型和患者的状况。因此,临床医生有必要根据需要滴定剂量并修改施用途径以获得最佳治疗效应。临床医生可以施用抗SARS-CoV-2的单结构域抗体,直到达到实现预期效应的剂量。这种疗法的进展很容易通过常规测定进行监测。
本文所述的抗SARS-CoV-2单结构域抗体可用于中和SARS-CoV-2及其变体,以便阻止或降低SARS-CoV-2与ACE2结合的能力,从而阻止或减少病毒进入宿主细胞。因此,本文所述的抗SARS-CoV-2的单结构域抗体的治疗性施用可以特别可用于治疗和/或预防SARS-CoV-2感染(例如,COVID-19)。
包含本文所述的抗SARS-CoV-2的单结构域抗体的治疗组合物或药物组合物可用于治疗、预防或改善诊断或预后、冠状病毒感染(例如,SARS-CoV-2感染)和/或与其相关联的医学病症(例如,COVID-19、哮喘、急性呼吸衰竭、肺炎、急性呼吸衰竭、肺炎、急性呼吸窘迫综合征(ARDS)、急性肝损伤、急性心脏损伤、继发感染、急性肾损伤、感染性休克、弥散性血管内凝血、血栓、多系统炎症综合征、慢性疲劳、横纹肌溶解症以及它们的组合)。
包含本文所述的抗SARS-CoV-2的单结构域抗体的治疗组合物或药物组合物可以施用于动物(优选地,哺乳动物;更优选地,人)以治疗、预防或改善冠状病毒感染和/或与冠状病毒感染相关联的病症。与冠状病毒感染相关联的病症的示例包括但不限于COVID-19、哮喘、急性呼吸衰竭、肺炎、急性呼吸窘迫综合征(acute respiratory distresssyndrome,ARDS)、急性肝损伤、急性心脏损伤、继发感染、急性肾损伤、感染性休克、弥散性血管内凝血、血栓、多系统炎症综合征、慢性疲劳、横纹肌溶解症以及它们的组合。
作为一般建议,肠胃外施用的单结构域抗体的初始药学有效量将在约0.1mg/kg患者体重/天至500mg/kg患者体重/天的范围内,所用单结构域抗体的典型初始范围为0.2mg/kg/天至100mg/kg/天,更优选地0.3mg/kg/天至20mg/kg/天。在实施方式中,单结构域抗体的有效量介于约1ng与1000ng之间、介于约1μg与1000μg之间、介于约1mg与1000mg之间、或介于约1g与1000g之间。取决于从业者希望实现的药代动力学衰减模式,所需剂量可通过单次推注施用、通过多次推注施用或通过单结构域抗体的连续输注施用来递送。作为非限制性示例,所述单结构域抗体可以施用至少1次、2次、3次、4次、5次、6次、7次、8次、9次或10次。在实施方式中,所述单结构域抗体或其抗原结合片段在1天、2天、3天、4天、5天、6天或7天的过程中施用。在另一实施方式中,所述单结构域抗体或其抗原结合片段在1周、2周、3周或4周的过程中施用。
所述单结构域抗体不需要但任选地与一种或多种目前用于预防或治疗冠状病毒感染或所讨论的与冠状病毒感染相关联的病症的药剂一起配制。此类其他药剂的有效量取决于制剂中存在的抗SARS-CoV-2的单结构域抗体的量、疾患或治疗的类型以及上述其他因素。这些通常以与上文所用相同的剂量和施用途径使用,或者是前文所用剂量的约1%至99%。
试剂盒
药物包装或药物试剂盒可包括一个或多个容器,所述一个或多个容器填充有包含本文所述的抗SARS-CoV-2的单结构域抗体的药物组合物的成分中的一种或多种成分。任选地,与此类容器相关联的可以是管理药物或生物产品的制造、使用或销售的政府机构规定形式的通知,该通知反映了该机构对用于人类施用的制造、使用或销售的批准。
所提供的试剂盒可用于本文所述的方法中。试剂盒可包含在一个或多个容器中的本文所述的单结构域抗体,优选地纯化的单结构域抗体。在替代实施方式中,试剂盒包含免疫特异性地结合SARS-CoV-2的抗体片段。试剂盒可包含基本上分离的SARS-CoV-2多肽作为对照。
试剂盒可进一步包含不与SARS-CoV-2反应的对照抗体。在另一具体实施方式中,本文所述的试剂盒包含用于检测抗体与SARS-CoV-2的结合的装置(例如,单结构域抗体可缀合至可检测的底物,例如荧光化合物、酶底物、放射性化合物或发光化合物,或者识别第一抗体的第二抗体可缀合至可检测的底物)。在具体实施方式中,试剂盒可包含重组产生的或化学合成的SARS-CoV-2多肽。试剂盒中提供的SARS-CoV-2多肽也可以附接至固体载体。在更具体的实施方式中,上述试剂盒的检测装置包括附接有SARS-CoV-2多肽的固体载体。此类试剂盒还可包含经未附接的报告分子标记的抗人抗体。在此实施方式中,单结构域抗体与SARS-CoV-2的结合可以通过所述经报告分子标记的抗体的结合来检测。
公开了用于筛选包含本文所述的多肽的抗原的生物样品的诊断试剂盒。所述诊断试剂盒包含与SARS-CoV-2特异性免疫反应的基本上分离的单结构域抗体,以及用于检测SARS-CoV-2与单结构域抗体的结合的装置。在实施方式中,单结构域抗体附接至固体载体。试剂盒的检测装置可包括第二经标记的抗体。替代地或另外,检测装置可包括经标记的竞争抗原。
在一种诊断配置中,使生物样品与具有通过本文所述的方法获得的表面结合的SARS-CoV-2多肽的固相试剂反应。在SARS-CoV-2与特异性单结构域抗体结合后,通过洗涤去除未结合的血清组分,加入经报告分子标记的抗人抗体,通过洗涤去除未结合的抗人抗体,并使试剂与经报告分子标记的抗人抗体反应,以与固相支持物上结合的抗SARS-CoV-2的单结构域抗体的量成比例地使报告分子与试剂结合。报告分子可以是通过在合适的荧光、发光或比色底物存在下孵育固相来检测的酶。
上述测定中的固体表面试剂是通过用于将蛋白质材料附接到固体支持材料(例如聚合物珠粒、浸渍棒、96孔板或过滤材料)的已知技术制备的。这些附着方法通常包括将蛋白质非特异性吸附到支持物,或通过游离胺基将蛋白质共价附接到固体支持物上的化学反应基团,例如激活的羧基、羟基或醛基。或者,链霉亲和素包被的板可与生物素化抗原结合使用。
因此,本公开提供了用于进行这种诊断方法的测定系统或试剂盒。所述试剂盒一般包含带有表面结合的重组SARS-CoV-2多肽的载体,以及用于检测表面结合的抗SARS-CoV-2的单结构域抗体的经报告分子标记的抗人抗体。
本文所述的另外细节可以在以下实施例中找到,所述实施例进一步定义了本文所述的范围。整个说明书中引用的所有参考文献,以及参考文献中引用的参考文献,据此全文以引用方式明确并入本文。
实施例1
从经免疫的美洲驼中分离针对SARS-CoV-2的受体结合结构域的单结构域抗体
根据下表6中描述的方案,在弗氏佐剂存在下,将一只美洲驼用SARS-CoV-2病毒抗原(RBD(SEQ ID NO:129)和刺突(SEQ ID NO:130)的重组蛋白)进行免疫。
表6.免疫方案
用于免疫的RBD多肽在N末端处包含天然刺突蛋白信号序列,并在C末端处经His标记:
MFVFLVLLPLVSSQRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFHHHHHH(SEQ ID NO:129)
用于免疫的刺突多肽包含在N末端处的其天然信号序列,稳定化突变,在所述稳定化突变中4个氨基酸(天然刺突蛋白中682位至685位处的RRAR)被修饰为单个丙氨酸(A)并且氨基酸K986和V987被改变为PP(以下序列中的983位和984位);并且在C末端处经His标记:
MFVFLVLLPLVSSQCVNLTTRTQLPPAYTNSFTRGVYYPDKVFRSSVLHSTQDLFLPFFSNVTWFHAIHVSGTNGTKRFDNPVLPFNDGVYFASTEKSNIIRGWIFGTTLDSKTQSLLIVNNATNVVIKVCEFQFCNDPFLGVYYHKNNKSWMESEFRVYSSANNCTFEYVSQPFLMDLEGKQGNFKNLREFVFKNIDGYFKIYSKHTPINLVRDLPQGFSALEPLVDLPIGINITRFQTLLALHRSYLTPGDSSSGWTAGAAAYYVGYLQPRTFLLKYNENGTITDAVDCALDPLSETKCTLKSFTVEKGIYQTSNFRVQPTESIVRFPNITNLCPFGEVFNATRFASVYAWNRKRISNCVADYSVLYNSASFSTFKCYGVSPTKLNDLCFTNVYADSFVIRGDEVRQIAPGQTGKIADYNYKLPDDFTGCVIAWNSNNLDSKVGGNYNYLYRLFRKSNLKPFERDISTEIYQAGSTPCNGVEGFNCYFPLQSYGFQPTNGVGYQPYRVVVLSFELLHAPATVCGPKKSTNLVKNKCVNFNFNGLTGTGVLTESNKKFLPFQQFGRDIADTTDAVRDPQTLEILDITPCSFGGVSVITPGTNTSNQVAVLYQDVNCTEVPVAIHADQLTPTWRVYSTGSNVFQTRAGCLIGAEHVNNSYECDIPIGAGICASYQTQTNSPASVASQSIIAYTMSLGAENSVAYSNNSIAIPTNFTISVTTEILPVSMTKTSVDCTMYICGDSTECSNLLLQYGSFCTQLNRALTGIAVEQDKNTQEVFAQVKQIYKTPPIKDFGGFNFSQILPDPSKPSKRSFIEDLLFNKVTLADAGFIKQYGDCLGDIAARDLICAQKFNGLTVLPPLLTDEMIAQYTSALLAGTITSGWTFGAGAALQIPFAMQMAYRFNGIGVTQNVLYENQKLIANQFNSAIGKIQDSLSSTASALGKLQDVVNQNAQALNTLVKQLSSNFGAISSVLNDILSRLDPPEAEVQIDRLITGRLQSLQTYVTQQLIRAAEIRASANLAATKMSECVLGQSKRVDFCGKGYHLMSFPQSAPHGVVFLHVTYVPAQEKNFTTAPAICHDGKAHFPREGVFVSNGTHWFVTQRNFYEPQIITTDNTFVSGNCDVVIGIVNNTVYDPLQPELDSFKEELDKYFKNHTSPDVDLGDISGINASVVNIQKEIDRLNEVAKNLNESLIDLQELGKYEQYIKWPSGRLVPRGSPGSGYIPEAPRDGQAYVRKDGEWVLLSTFLGHHHHHH(SEQ ID NO:130)
如表6中所指示,在第0天、第24天、第38天、第52天、第66天和第80天收集测试血清。通过ELISA测定分析美洲驼的第0天、第24天和第38天血清样品的免疫应答。为此,在Maxisorp 96孔微量滴定板中捕获重组RBD或刺突蛋白。在封闭后,添加连续稀释的血清样品,并通过添加山羊抗美洲驼IgG-HRP(Invitrogen,A16060)来检测结合的美洲驼IgG。结果示出在图3A和图3B中。这些数据表明,经免疫的美洲驼产生了针对RBD的良好免疫应答,并且诱导的抗体也可以识别刺突。
在第80天时,从经免疫的美洲驼收集500ml全血,并根据制造商的使用说明使用Ficoll-Paque plus(GE Healthcare)分离外周血单核细胞(PBMC)。将从PBMC中提取的总RNA用作RT-PCR的起始材料,以通过两步PCR反应扩增VHH基因片段。将这些片段用PstI酶和BstEII酶消化并克隆到pMES4噬菌粒中。将连接的载体电穿孔至TG-1细菌(Lucigen)中以构建VHH结构域文库。文库大小估计为1.04E+10。使用VSCM13辅助噬菌体制备VHH噬菌体文库,并用于SARS-CoV-2 RBD抗体选择。
使用上述VHH噬菌体文库选择单结构域抗体片段,并用SARS-CoV-2的重组RBD(SEQID NO:129)作为靶蛋白执行RBD结合剂的选择。将Maxisorp 96孔板的两个孔用50ul重组RBD蛋白(100ug/ml,在PBS中稀释)在4度下包被过夜,并用封闭缓冲液(5%脱脂乳的PBS溶液)封闭。将添加有PBS的一个孔用作未包被的对照。将噬菌体文库在5%脱脂乳中在室温下封闭1小时,然后添加到各孔中。在室温下孵育2小时后,洗涤各孔(用0.1% Tween-20的PBS溶液洗涤15次)。用TrypLE
在一轮选择后,从RBD包被的孔和未包被的孔中洗脱的噬菌体的比率达到280:1。将回收的TG-1细胞铺板并挑取个别菌落以制备包含粗制单克隆单结构域抗体的周质提取物。简言之,挑取个别菌落并在96深孔板(2xYT培养基、100ug/ml的羧苄西林、0.1%的葡萄糖)中生长。通过添加IPTG(1mM)诱导抗体表达。通过将细菌沉淀重悬于200ul的PBS中并在液氮中快速冷冻来制备周质提取物。将冷冻细胞在室温下缓慢解冻,并以4200rpm离心15分钟。将包含单结构域抗体的上清液用于针对RBD或刺突结合剂的ELISA筛选。为此,在Maxisorp 96孔微量滴定板中捕获重组RBD或刺突蛋白。在封闭后,添加100ul的包含抗体的上清液,并通过添加山羊抗羊驼IgG(VHH结构域)-HRP(JacksonImmunoResearch,128-035-232)来检测结合的美洲驼IgG。
从这次筛选,鉴定出了108种单结构域抗体候选物,并分离出了代表不同CDR3家族的50种候选物进行进一步测试。
实施例2
从表达骆驼、羊驼和单峰骆驼抗体基因的转基因小鼠模型中分离针对SARS-CoV-2的受体结合结构域的单结构域抗体
我们已经生成了表达骆驼、羊驼和单峰骆驼抗体基因代替内源性小鼠抗体基因座的转基因小鼠模型。这个转基因小鼠模型是通过鉴定出来自骆驼、单峰骆驼和羊驼的30种不同的免疫球蛋白可变(VHH)结构域基因而工程化的。这些基因被合成并装配成25Kb的微小基因,然后将所述微小基因用于使用CRISPR-Cas9技术替换胚胎干(ES)细胞中小鼠基因组的整个VH结构域(2.5Mb)。此外,IgM恒定结构域和IgG1恒定结构域的CH1外显子经缺失以废除轻链与重链的配对。使用经修饰的ES细胞来生成嵌合小鼠,并将携带VHH微小基因敲入的F1子代与C57BL/6小鼠回交。通过繁殖杂合小鼠获得携带VHH微小基因敲入的纯合小鼠,并用于免疫。
根据以下表7和表8中描述的方案,在弗氏佐剂存在下,将两组上述纯合转基因小鼠(第1组中5只小鼠,第2组中6只小鼠)用SARS-CoV-2病毒抗原(RBD(SEQ ID NO:129)和刺突(SEQ ID NO:130)的重组蛋白)进行免疫。
表7.免疫方案
表8.免疫方案
如表7和表8中所示,在第0天、第21天、第35天、第49天、第62天收集测试血清。通过ELISA测定分析经免疫的转基因小鼠的第0天、第21天和第35天血清样品的免疫应答。为此,在Maxisorp 96孔微量滴定板中捕获重组RBD或刺突蛋白。封闭后,添加连续稀释的血清样品,并通过添加山羊抗小鼠IgG-HRP来检测结合的小鼠IgG。第1组和第2组的结果分别示出于图4A和图4B中。这些数据表明,经免疫的转基因小鼠生成了不同水平的免疫应答,并挑选最好的(#28436、#29258、#29260)进行单抗体库构建。
在第62天,从经免疫的转基因小鼠中收集脾细胞并提取总RNA用于进行RT-PCR以扩增VHH-D-JH基因片段。将这些片段用SfiI酶消化并克隆到pMES4噬菌粒中。将连接的载体电穿孔至TG-1细菌(Lucigen)中以构建单结构域抗体文库。转基因小鼠#28436、#29258和#29260的文库大小估计分别为9.68E+08、1.35E+09和3.5E+09。使用VSCM13辅助噬菌体制备VHH噬菌体文库,并用于以与美洲驼VHH文库筛选相同的方式进行SARS-CoV-2 RBD和刺突抗体选择。
以RBD蛋白作为靶标选择来自转基因小鼠#28436的文库,并且在一轮选择后,从RBD包被的孔和未包被的孔中洗脱的噬菌体的比率达到900:1。以刺突蛋白作为靶标选择来自转基因小鼠#29258和#29260的文库,并且在两轮选择后,从刺突包被的孔和未包被的孔中洗脱的噬菌体的比率达到1600:1和700:1。
以与美洲驼文库筛选中所述相同的方式,将回收的TG-1细胞铺板并挑取个别菌落以制备包含粗制单克隆单结构域抗体的周质提取物。
通过这些筛选,总共鉴定出了158种独特的单结构域抗体候选物,并分离出了代表不同CDR3家族的63种候选物进行进一步测试。
实施例3
用分离的抗SARS-CoV-2的单结构域抗体中和SARS-CoV-2假型化病毒
假型化SARS-CoV-2病毒是如由Davide F.Robbiani等人(“Convergent antibodyresponses to SARS-CoV-2in convalescent individuals.”Nature.2020Aug;584(7821):437-442.doi:10.1038/s41586-020-2456-9.电子出版2020年6月18日.PMID:32555388)产生的。通过将编码缺乏C末端19个密码子的SARS-CoV-2S的经人类密码子优化的cDNA(Geneart)插入pCR3.1中,来生成表达C末端截短的SARS-CoV-2S蛋白(pSARS-CoV2-Strunc)的质粒。S开放阅读框取自“肺炎病毒分离株”(GenBank:NC_045512)。通过在vpu终止密码子中3'处引入940bp的缺失,导致env发生移码,来从pNL4-332生成env失活的HIV-1报告基因构建体(pNL4-3ΔEnv-nanoluc)。将经人密码子优化的nanoluc荧光素酶报告基因(Nluc,Promega)插入代替nef基因的核苷酸1至100。为了生成假型化病毒原种,使用聚乙烯亚胺,用pNL4-3ΔEnv-nanoluc和pSARS-CoV2-Strunc或pSARS-CoV-S转染293T细胞。pNL4-3ΔEnv-nanoluc和S表达质粒的共转染会导致产生在表面上携带SARS-CoV-2或SARS-CoV S蛋白的基于HIV-1的病毒体。在转染8h后,用PBS洗涤细胞两次并添加新鲜培养基。在转染后48h收集包含病毒体的上清液,过滤并存储于-80℃。
根据制造商的使用说明(GenScript,L00847)执行替代病毒中和测试(sVNT)。
假病毒中和测定如由Davide F.Robbiani等人(“Convergent antibodyresponses to SARS-CoV-2 in convalescent individuals.”Nature.2020Aug;584(7821):437-442.doi:10.1038/s41586-020-2456-9.电子出版2020年6月18日.PMID:32555388)所述。将四倍系列稀释的单结构域抗体与SARS-CoV-2假型化病毒一起在37℃下孵育1h。随后将混合物与293TACE2细胞一起孵育48h,在此之后将细胞用PBS洗涤两次并用荧光素酶细胞培养物裂解5x试剂(Promega)裂解。使用具有Modulus II酶标仪用户界面(TURNER BioSystems)的Nano-Glo荧光素酶测定系统(Promega)测量裂解物中的Nanoluc荧光素酶活性。将获得的相对发光单位归一化为来源于在没有抗体的情况下感染SARS-CoV-2假型化病毒的细胞的相对发光单位。使用四参数非线性回归(GraphPad Prism)确定单结构域抗体的半数最大抑制浓度(IC50)。
表A.通过从美洲驼和表达骆驼科免疫球蛋白基因的转基因小鼠生成的单结构域抗体中和假型化SARS-CoV-2病毒.
nd=不可检测。
表B.来自选定的美洲驼源SARS-CoV-2 RBD抗体的替代病毒中和测试(sVNT)结果.
/>
表C.来自选定的转基因小鼠源SARS-CoV-2 RBD抗体的替代病毒中和测试(sVNT)结果.
/>
表D.来自选定的转基因小鼠源SARS-CoV-2刺突抗体的替代病毒中和测试(sVNT)结果.
/>
来自以上表A至表D的数据表明,针对SARS-CoV-2的RBD/刺突生成的几种单结构域抗体能够中和SARS-CoV-2。特别地,来源于经免疫的美洲驼的最有效的单结构域抗体是Nb15、Nb17、Nb19和Nb56。来源于能够产生骆驼科抗体的经免疫的转基因小鼠的最有效的单结构域抗体是msNb12和mNb30。
实施例4
用分离的抗SARS-CoV-2的单结构域抗体Fc融合体中和SARS-CoV-2假型化病毒
将单体或三聚单结构域抗体与人IgG1分子的Fc区融合,并且在C末端上有附加的6xHis标签。所述Fc区包含铰链区,之后是CH2结构域和CH3结构域。在一些情况下,使用美洲驼IgG2a铰链区代替人IgG1铰链区。Fc融合构建体在Expi293细胞中表达,并且抗体作为二聚Fc分子分泌到培养基中,使用cOmplete His标签纯化树脂(Roche 05893801001)或POROSMabCapture A Select树脂(Thermoscientific,A26457)纯化所述抗体。
下表E提供了6种选定的单结构域抗体及其单体/三聚Fc融合版本的整组假病毒中和测定结果。
表E.SARS-CoV-2假病毒中和结果
实施例5
抗SARS-CoV-2的单结构域抗体对SARS-CoV-2多肽的亲和力
测定如实施例1至实施例4中所述的选定美洲驼源和转基因小鼠源的抗SARS-CoV-2抗体及其Fc融合构建体对SARS-CoV-2 RBD和/或SARS-CoV-2刺突多肽的亲和力。
执行生物层干涉测量(Bio-Layer Interferometry,BLI)测定以确定单结构域抗体对RBD的亲和力。简而言之,将生物素化的RBD固定到链霉亲和素包被的生物传感器上,然后使稀释的单结构域抗体缔合30秒,之后解离2-3分钟。使用传感器数据的全局拟合和经计算以确定缔合和解离常数的稳态分析来应用曲线拟合。
图1A至图1F中提供了六种例示性抗SARS-CoV-2的单结构域抗体(Nb15、msNb12、Nb17、Nb19、Nb56、mNb30)的结果。
图2A至图2F中提供了六种例示性抗SARS-CoV-2的单结构域抗体(mNb30-IgG2a、Nb15-三IgG1、Nb17-三IgG1、Nb19-三IgG1、Nb56-三IgG1、msNb12-三IgG2a)的Fc融合体的结果。
实施例6
使用分离的抗SARS-CoV-2的单结构域抗体中和突变型SARS-CoV-2假型化病毒
使用如上所述的假病毒中和测定,测试如实施例1至实施例4中所述的选定的美洲驼源和转基因小鼠源的抗SARS-CoV-2抗体及其Fc融合构建体的中和SARS-CoV-2变体的能力。所有突变都在刺突蛋白中。结果在下表F中显示。
表F.SARS-CoV-2变体假病毒中和结果
*KEN=K417N+E484K+N501Y
此数据证明本文所公开的单结构域抗体能够中和SARS-CoV-2变体以及野生型SARS-CoV-2。
本说明书中引用的所有参考文献均以引用方式并入本文,就好像每篇参考文献都被具体且单独地指出为以引用方式并入。任何参考文献的引用均为其在申请日之前公开,并且不应解释为承认本公开内容无权凭借在先发明而先于此类参考文献。
应当理解的是,上述要素中的每个要素或者两个或更多个要素一起也可有用地应用于不同于上述类型的其他类型的方法中。在没有进一步分析的情况下,前述内容将如此充分地揭示本公开的要点,以至于其他人可以通过应用当前知识,容易地将其适用于各种应用,而不会遗漏从现有技术的角度来看完全构成所附权利要求书中所述的本公开的通用或特定方面的基本特性的特征。前述实施方式仅以举例方式呈现;本公开的范围仅受以下权利要求书的限制。
序列表
<110>美国卫生及公众服务部代表 (THE UNITED STATES OF AMERICA, ASREPRESENTED BY THE
SECRETARY, DEPARTMENT OF HEALTH AND HUMAN SERVICES)
拉斐尔·卡塞利亚斯 (CASELLAS, Rafael)
徐建良 (XU, Jianliang)
<120>中和SARS-COV-2的单结构域抗体
<130>3000093-005977
<150>US 63/151,530
<151>2021-02-19
<160>130
<170>PatentIn第3.5版
<210>1
<211>124
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb15)
<400>1
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Val Ser Cys Ala Ala Ser Gly Leu Pro Phe Ser Asp Tyr
202530
Leu Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Tyr Val
354045
Ala Ala Ile Ser Gln Asn Gly Gly His Thr Tyr Ala Asp Ser Val Leu
505560
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr Leu
65707580
Gln Met Asn Met Leu Thr Pro Gly Asp Thr Ala Val Tyr Ser Cys Ala
859095
Ala Arg Arg Pro Gly Gly Gly Arg Trp Asp Ala Ala His Asp Tyr Asn
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120
<210>2
<211>119
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb17)
<400>2
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Thr Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Gly Ile Tyr
202530
Arg Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Ile Thr Ser Ser Ala Asp Thr Ala Gln Tyr Arg Asp Ser Val
505560
Lys Gly Arg Phe Ala Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
859095
Ala Ala Arg Asp Pro Thr Thr Leu Glu Tyr Gly Asn Trp Gly Gln Gly
100 105 110
Thr Gln Val Thr Val Ser Ser
115
<210>3
<211>119
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb19)
<400>3
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Gly Phe Ser Ile His
202530
Ala Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Phe Val
354045
Ala Val Val Gly His Lys Thr Asn Tyr Ala Asp Ser Val Lys Gly Arg
505560
Phe Thr Ile Ser Arg Asp Val Gly Lys Asn Thr Val Glu Leu Gln Met
65707580
Asn Ser Leu Lys Val Glu Asp Thr Ala Val Tyr Tyr Cys Tyr Cys Asn
859095
Thr Ile Val Thr Met Thr Gly Val Pro Asp Ala Val Trp Gly Gln Gly
100 105 110
Thr Gln Val Thr Val Ser Ser
115
<210>4
<211>128
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb56)
<400>4
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Val Ala Ser Glu Arg Thr Phe Arg Arg Tyr
202530
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Val Asp Arg Ser His Thr Lys Thr Gly Tyr Ala Asp Phe Val
505560
Lys Gly Arg Phe Thr Ile Ser Thr Asn Tyr Glu Asn Met Val Tyr Leu
65707580
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
859095
Ala Pro Ser Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr Asp
100 105 110
Arg Gly Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120 125
<210>5
<211>114
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb6)
<400>5
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Asn Ile Ala Asn Ser Asn
202530
Ser Met Ala Trp Trp Arg Gln Thr Pro Gly Asn Gln His Glu Arg Val
354045
Ala Ile Ile Ser His Gly Val Thr Asn Tyr Ala Asp Ser Val Lys Gly
505560
Arg Phe Thr Val Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu Gln
65707580
Met Asn Asn Leu Lys Pro Glu Asp Thr Ala Ala Tyr Tyr Cys Tyr Ala
859095
Asp Leu Phe Gly Asn Thr Tyr Trp Gly Gln Gly Thr Gln Val Thr Val
100 105 110
Ser Ser
<210>6
<211>129
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb7)
<400>6
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Leu Ser Ser Glu Arg Thr Phe Arg Arg Tyr
202530
Gly Ile Ala Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Val Asp Arg Ser His Ser Gln Thr Asn Tyr Ala Asp Phe Val
505560
Gln Gly Arg Phe Thr Ile Ser Thr Val Tyr Ala Lys Asn Met Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Ala Ser Tyr Glu Lys Gly Ser Asp Tyr Thr Ser Trp Asn Thr
100 105 110
Asp Arg Gly Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser
115 120 125
Ser
<210>7
<211>128
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb43)
<400>7
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Asp Ser
202530
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Glu Glu Arg Glu Phe Val
354045
Ala Val Ile Thr Trp Asn Gly Gly Thr Thr Tyr Tyr Ala Asp Ser Val
505560
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ala Lys Asn Thr Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Asp Thr Ser Arg Trp Asp Tyr Ser Leu Thr Tyr His Tyr Thr
100 105 110
Arg Glu Tyr Asn Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120 125
<210>8
<211>130
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb47)
<400>8
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Leu Ser Arg Tyr
202530
Ser Met Ser Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Gly Ile Arg Trp Ser Gly Ser Asn Thr Tyr Tyr Ala Asp Ser Met
505560
Lys Gln Arg Phe Thr Ile Ser Gln Asp Asn Val Lys Asn Thr Val His
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Thr Ser Arg Ser Asp His Tyr Leu Asn Ala Val Ala Trp Thr
100 105 110
Leu Pro Asn Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val
115 120 125
Ser Ser
130
<210>9
<211>120
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb50)
<400>9
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Asp Gly Thr
202530
Tyr Arg Met Gly Trp Phe Arg Gln Gly Pro Gly Lys Glu Arg Glu Phe
354045
Val Ala Ala Ile Gly Phe Gly Val Ser Thr Thr Ser Tyr Ala Asp Ser
505560
Val Lys Gly Arg Phe Thr Ile Ser Arg Asn Asn Ala Lys Asn Thr Val
65707580
Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr
859095
Cys Ala Ala Arg Pro Pro Pro Tyr Thr Glu Tyr Asn Tyr Trp Gly Gln
100 105 110
Gly Thr Gln Val Thr Val Ser Ser
115 120
<210>10
<211>124
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb53)
<400>10
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Ser Ser Tyr
202530
Leu Met Ala Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Arg Ile Asn Trp Asn Gly Arg Val Pro Tyr Thr Val Asp Ser Val
505560
Lys Gly Arg Phe Ile Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Trp
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Asp Arg Asn Tyr Gly Thr Gly Gly Ala Glu Thr Val Tyr Glu
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120
<210>11
<211>125
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb55)
<400>11
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly Arg Ala Phe Ser Asp Ser
202530
Ser Ala Met Ala Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe
354045
Val Ala Ala Leu Asn Arg Val Asn Val Ala Tyr Cys Arg Asp Ser Val
505560
Ser Gly Arg Phe Thr Ile Ser Arg Asp Asn Gly Lys Asn Thr Ala Tyr
65707580
Leu Glu Met Asn Ser Val Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
859095
Ala Ser Asp Leu Ile Leu Asn Asp Cys Ser Arg Asn Pro Ala Arg Tyr
100 105 110
Ala Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120 125
<210>12
<211>129
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (Nb60)
<400>12
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Leu Ala Ser Glu Arg Thr Phe Arg Arg Tyr
202530
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Ile Asp Arg Ser His Ser Asn Thr Asp Tyr Ala Asp Phe Val
505560
Lys Gly Arg Phe Thr Ile Ser Thr Val Tyr Ala Lys Asn Met Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Ala Thr Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr
100 105 110
Asp Arg Gly Tyr Asp Val Trp Gly Gln Gly Thr Gln Val Thr Val Ser
115 120 125
Ser
<210>13
<211>121
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (msNb12)
<400>13
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ile Cys Thr Ala Pro Gly Leu Thr His Asn Asn Cys
202530
Gly Leu Asp Trp Tyr Arg Arg Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ser Ser Ile Ser Ala Asp Gly Thr Thr Ser Tyr Ala Asp Ser Val Lys
505560
Gly Arg Phe Thr Ile Ser Lys Asp Lys Val Glu Asp Thr Val Tyr Leu
65707580
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Ser Cys Lys
859095
Thr Ala Phe Pro Tyr Phe Gly Asn Ser Cys Val Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210>14
<211>121
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (mNb30)
<400>14
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Ser Lys Tyr
202530
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Lys Phe Val
354045
Ala Thr Ile Ser Trp Ser Gly Asp Ser Ala Phe Tyr Ala Asp Ser Val
505560
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Ala Ser Ser
115 120
<210>15
<211>121
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体序列 (mNb35)
<400>15
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Lys Tyr
202530
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Lys Phe Val
354045
Ala Thr Ile Ser Trp Ser Gly Asp Ser Ala Phe Tyr Ala Asp Ser Val
505560
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Thr Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210>16
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>16
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Val Ser Cys Ala Ala Ser Gly
2025
<210>17
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>17
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Thr Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
2025
<210>18
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>18
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
2025
<210>19
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>19
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Val Ala Ser Glu
2025
<210>20
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>20
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Leu Ser Ser Glu
2025
<210>21
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>21
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly
2025
<210>22
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>22
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
2025
<210>23
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>23
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Glu Ala Ser Gly
2025
<210>24
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>24
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Leu Ala Ser Glu
2025
<210>25
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>25
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ile Cys Thr Ala Pro Gly
2025
<210>26
<211>26
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR1序列
<400>26
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
2025
<210>27
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>27
Leu Pro Phe Ser Asp Tyr Leu Met Gly
1 5
<210>28
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>28
Arg Thr Phe Gly Ile Tyr Arg Met Gly
1 5
<210>29
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>29
Ser Gly Phe Ser Ile His Ala Met Gly
1 5
<210>30
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>30
Arg Thr Phe Arg Arg Tyr Gly Met Gly
1 5
<210>31
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>31
Asn Ile Ala Asn Ser Asn Ser Met Ala
1 5
<210>32
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>32
Arg Thr Phe Arg Arg Tyr Gly Ile Ala
1 5
<210>33
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>33
Arg Thr Phe Ser Asp Ser Ala Met Gly
1 5
<210>34
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>34
Arg Thr Leu Ser Arg Tyr Ser Met Ser
1 5
<210>35
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>35
Arg Thr Phe Asp Gly Thr Tyr Arg Met Gly
1 5 10
<210>36
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>36
Leu Thr Phe Ser Ser Tyr Leu Met Ala
1 5
<210>37
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>37
Arg Ala Phe Ser Asp Ser Ser Ala Met Ala
1 5 10
<210>38
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>38
Leu Thr His Asn Asn Cys Gly Leu Asp
1 5
<210>39
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>39
Leu Thr Phe Ser Lys Tyr Ala Met Gly
1 5
<210>40
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR1序列
<400>40
Arg Thr Phe Ser Lys Tyr Ala Met Gly
1 5
<210>41
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>41
Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Tyr Val Ala
1 5 10
<210>42
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>42
Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val Ala
1 5 10
<210>43
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>43
Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Phe Val Ala
1 5 10
<210>44
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>44
Trp Trp Arg Gln Thr Pro Gly Asn Gln His Glu Arg Val Ala
1 5 10
<210>45
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>45
Trp Phe Arg Gln Ala Pro Gly Glu Glu Arg Glu Phe Val Ala
1 5 10
<210>46
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>46
Trp Phe Arg Gln Gly Pro Gly Lys Glu Arg Glu Phe Val Ala
1 5 10
<210>47
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>47
Trp Tyr Arg Arg Ala Pro Gly Lys Glu Arg Glu Phe Val Ser
1 5 10
<210>48
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>48
Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Lys Phe Val Ala
1 5 10
<210>49
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>49
Ala Ile Ser Gln Asn Gly Gly His Thr
1 5
<210>50
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>50
Ala Ile Thr Ser Ser Ala Asp Thr Ala Gln
1 5 10
<210>51
<211>7
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>51
Val Val Gly His Lys Thr Asn
1 5
<210>52
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>52
Ala Val Asp Arg Ser His Thr Lys Thr Gly
1 5 10
<210>53
<211>8
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>53
Ile Ile Ser His Gly Val Thr Asn
1 5
<210>54
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>54
Ala Val Asp Arg Ser His Ser Gln Thr Asn
1 5 10
<210>55
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>55
Val Ile Thr Trp Asn Gly Gly Thr Thr Tyr
1 5 10
<210>56
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>56
Gly Ile Arg Trp Ser Gly Ser Asn Thr Tyr
1 5 10
<210>57
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR2序列
<400>57
Ala Ile Gly Phe Gly Val Ser Thr Thr Ser
1 5 10
<210>58
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>58
Arg Ile Asn Trp Asn Gly Arg Val Pro Tyr
1 5 10
<210>59
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>59
Ala Leu Asn Arg Val Asn Val Ala Tyr
1 5
<210>60
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>60
Ala Ile Asp Arg Ser His Ser Asn Thr Asp
1 5 10
<210>61
<211>9
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>61
Ser Ile Ser Ala Asp Gly Thr Thr Ser
1 5
<210>62
<211>10
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR2序列
<400>62
Thr Ile Ser Trp Ser Gly Asp Ser Ala Phe
1 5 10
<210>63
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>63
Tyr Ala Asp Ser Val Leu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
1 5 1015
Lys Asn Thr Val Tyr Leu Gln Met Asn Met Leu Thr Pro Gly Asp Thr
202530
Ala Val Tyr Ser Cys
35
<210>64
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>64
Tyr Arg Asp Ser Val Lys Gly Arg Phe Ala Ile Ser Arg Asp Asn Ala
1 5 1015
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Ile Tyr Tyr Cys
35
<210>65
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>65
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Val Gly
1 5 1015
Lys Asn Thr Val Glu Leu Gln Met Asn Ser Leu Lys Val Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>66
<211>36
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>66
Tyr Ala Asp Phe Val Lys Gly Arg Phe Thr Ile Ser Thr Asn Tyr Glu
1 5 1015
Asn Met Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala
202530
Val Tyr Tyr Cys
35
<210>67
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>67
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Val Ser Arg Asp Asn Ala
1 5 1015
Lys Asn Thr Leu Tyr Leu Gln Met Asn Asn Leu Lys Pro Glu Asp Thr
202530
Ala Ala Tyr Tyr Cys
35
<210>68
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>68
Tyr Ala Asp Phe Val Gln Gly Arg Phe Thr Ile Ser Thr Val Tyr Ala
1 5 1015
Lys Asn Met Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>69
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>69
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ala
1 5 1015
Lys Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>70
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>70
Tyr Ala Asp Ser Met Lys Gln Arg Phe Thr Ile Ser Gln Asp Asn Val
1 5 1015
Lys Asn Thr Val His Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>71
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>71
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asn Asn Ala
1 5 1015
Lys Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>72
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>72
Thr Val Asp Ser Val Lys Gly Arg Phe Ile Ile Ser Arg Asp Asn Ala
1 5 1015
Lys Asn Thr Val Trp Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>73
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>73
Cys Arg Asp Ser Val Ser Gly Arg Phe Thr Ile Ser Arg Asp Asn Gly
1 5 1015
Lys Asn Thr Ala Tyr Leu Glu Met Asn Ser Val Lys Pro Glu Asp Thr
202530
Ala Ile Tyr Tyr Cys
35
<210>74
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>74
Tyr Ala Asp Phe Val Lys Gly Arg Phe Thr Ile Ser Thr Val Tyr Ala
1 5 1015
Lys Asn Met Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>75
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>75
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Lys Asp Lys Val
1 5 1015
Glu Asp Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Ile Tyr Ser Cys
35
<210>76
<211>37
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR3序列
<400>76
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
1 5 1015
Arg Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
202530
Ala Val Tyr Tyr Cys
35
<210>77
<211>17
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>77
Ala Ala Arg Arg Pro Gly Gly Gly Arg Trp Asp Ala Ala His Asp Tyr
1 5 1015
Asn
<210>78
<211>11
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>78
Ala Ala Arg Asp Pro Thr Thr Leu Glu Tyr Gly
1 5 10
<210>79
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>79
Tyr Cys Asn Thr Ile Val Thr Met Thr Gly Val Pro Asp Ala
1 5 10
<210>80
<211>21
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>80
Ala Ala Pro Ser Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr
1 5 1015
Asp Arg Gly Tyr Asp
20
<210>81
<211>8
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>81
Tyr Ala Asp Leu Phe Gly Asn Thr
1 5
<210>82
<211>21
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>82
Ala Ala Ala Ser Tyr Glu Lys Gly Ser Asp Tyr Thr Ser Trp Asn Thr
1 5 1015
Asp Arg Gly Tyr Asp
20
<210>83
<211>20
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>83
Ala Ala Asp Thr Ser Arg Trp Asp Tyr Ser Leu Thr Tyr His Tyr Thr
1 5 1015
Arg Glu Tyr Asn
20
<210>84
<211>22
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>84
Ala Ala Thr Ser Arg Ser Asp His Tyr Leu Asn Ala Val Ala Trp Thr
1 5 1015
Leu Pro Asn Glu Tyr Asp
20
<210>85
<211>11
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>85
Ala Ala Arg Pro Pro Pro Tyr Thr Glu Tyr Asn
1 5 10
<210>86
<211>16
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>86
Ala Ala Asp Arg Asn Tyr Gly Thr Gly Gly Ala Glu Thr Val Tyr Glu
1 5 1015
<210>87
<211>17
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>87
Ala Ser Asp Leu Ile Leu Asn Asp Cys Ser Arg Asn Pro Ala Arg Tyr
1 5 1015
Ala
<210>88
<211>21
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>88
Ala Ala Ala Thr Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr
1 5 1015
Asp Arg Gly Tyr Asp
20
<210>89
<211>14
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>89
Lys Thr Ala Phe Pro Tyr Phe Gly Asn Ser Cys Val Leu Asp
1 5 10
<210>90
<211>13
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>90
Ala Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp
1 5 10
<210>91
<211>13
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>CDR3序列
<400>91
Thr Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp
1 5 10
<210>92
<211>12
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR4序列
<400>92
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
1 5 10
<210>93
<211>12
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR4序列
<400>93
Asn Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
1 5 10
<210>94
<211>12
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR4序列
<400>94
Val Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
1 5 10
<210>95
<211>12
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR4序列
<400>95
Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
1 5 10
<210>96
<211>12
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>FR4序列
<400>96
Tyr Trp Gly Gln Gly Thr Ser Val Thr Ala Ser Ser
1 5 10
<210>97
<211>372
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb15)
<400>97
caggtgcagc tgcaggagtc tgggggaggg ttggtgcagg ctgggggctc tctgagagtc60
tcctgtgcag cgtctggact tcccttcagt gactatttaa tgggctggtt ccgccaggcg 120
ccagggaagg agcgtgagta tgtagccgct attagtcaga atggtggaca cacttatgca 180
gactccgtgc tgggccgatt caccatctcc agagacaacg ccaagaatac ggtgtatctg 240
caaatgaaca tgttgacacc tggggacacg gccgtttata gttgtgctgc ccgaaggccc 300
ggtgggggta ggtgggatgc cgcccatgac tataactact ggggccaggg gacccaggtc 360
accgtctcct ca 372
<210>98
<211>357
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb17)
<400>98
caggtgcagc tgcaggagtc tgggggagga ttggtgcaaa ctgggggctc tctgagactc60
tcctgtgcag cctctggacg caccttcggt atctatcgca tgggctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtagcagct atcacttcga gtgctgatac cgcacagtat 180
cgagactccg tgaagggccg attcgccatc tccagagaca acgccaagaa cacgctgtat 240
ctgcaaatga acagcctgaa acctgaggac acggccattt attattgtgc agcacgggat 300
cccactacat tggagtatgg caactggggc caggggaccc aggtcaccgt ctcctca357
<210>99
<211>357
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb19)
<400>99
caggtgcagc tgcaggagtc tgggggaggc ttggtgcagg ctggggggtc tctgagactg60
tcctgtgcag cctctggaag cggctttagt attcatgcca tgggctggta ccgccaggct 120
ccagggaagc agcgcgagtt cgtcgctgtc gttggccata agacgaacta tgcagactcc 180
gttaagggcc gattcaccat ctccagagac gttggcaaga acacggtgga gctgcaaatg 240
aacagcctga aagttgagga cacagccgtc tattattgtt actgcaatac tatcgtgact 300
atgacagggg ttcctgatgc cgtctggggc caggggaccc aggtcaccgt ctcctca357
<210>100
<211>384
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb56)
<400>100
caggtgcagc tgcaggagtc tgggggagga ttggtgcagg ctggggactc tctgagactc60
tcctgtgtag cctctgaacg cacattcagg cgctatggca tgggctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtagcagct gttgaccgga gtcatactaa gacaggctat 180
gcagacttcg tgaagggccg attcaccatc tccacgaact acgagaacat ggtgtatctg 240
caaatgaaca gcctgaaacc tgaggacacg gccgtttatt actgtgctgc gccgtcgtac 300
gagaaagggt cggaccctac tagttggaac accgacagag ggtatgacta ctggggccag 360
gggacccagg tcaccgtctc ctca384
<210>101
<211>342
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb6)
<400>101
caggtgcagc tgcaggagtc tgggggaggc ttggtgcagg ctggggggtc tctgagactc60
tcctgtgcag cctctggaaa catcgcgaat tccaattcca tggcctggtg gcgccagact 120
ccaggaaacc agcacgagcg ggtcgccatt attagtcatg gtgtaacaaa ctatgcagat 180
tccgtgaagg gccgattcac agtgtccaga gacaacgcca agaatacttt gtatctgcaa 240
atgaacaacc tgaaacctga ggacacagcc gcctattatt gttatgcaga tctcttcgga 300
aacacctact ggggccaggg gacccaggtc accgtctcct ca342
<210>102
<211>387
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb7)
<400>102
caggtgcagc tgcaggagtc tggaggagga ttggtgcagg ctggggactc tctgagactc60
tcctgtctat cctctgaacg cacattcagg cgctatggca tagcctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtagcagct gttgaccgga gtcatagtca gacaaactat 180
gcagacttcg tacagggccg attcaccatc tccacggtct acgccaagaa catggtgtat 240
ctgcaaatga acagcctgaa acctgaggac acggccgttt attactgtgc tgcggcgtcg 300
tacgagaaag ggtcggacta tactagttgg aacaccgaca gagggtatga ctactggggc 360
caggggaccc aggtcaccgt ctcctca 387
<210>103
<211>384
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb43)
<400>103
caggtgcagc tgcaggagtc tgggggagga ttggtgcagg ctggggactc tctgaaactc60
tcctgtgcag cctctggacg caccttcagt gacagtgcca tgggctggtt ccgccaggct 120
ccaggagagg agcgtgagtt tgtagcagtt attacctgga atggtggcac cacatactat 180
gcagactccg tgaagggccg attcaccatc tccagagacg acgccaagaa cacggtgtac 240
ctgcaaatga acagcctgaa acctgaggac acggccgttt attactgtgc agcggacaca 300
agccggtggg actatagtct tacataccac tacacgaggg agtataacta ctggggccag 360
gggacccagg tcaccgtctc ctca384
<210>104
<211>390
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb47)
<400>104
caggtgcagc tgcaggagtc tgggggagga ttggtgcagg ctgggggctc tctgagactc60
tcctgtgcag cctctggacg caccttgagt aggtattcca tgagctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtagcaggt atacggtgga gtggtagtaa cacatactat 180
gcagactcca tgaagcagcg attcaccatc tcccaagaca atgtcaagaa cacggtgcat 240
ctgcaaatga acagcctgaa acctgaggac acggccgttt attactgtgc agccacaagt 300
agaagtgatc attacttgaa tgccgtggct tggacccttc cgaatgagta tgactactgg 360
ggccagggga cccaggtcac cgtctcctca390
<210>105
<211>360
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb50)
<400>105
caggtgcagc tgcaggagtc tgggggagga ttggtgcagg ctgggggatc tctgagactc60
tcctgtgcag cctctggacg caccttcgat ggtacctatc gcatgggctg gttccgccag 120
ggtccaggga aggagcgtga gtttgtagca gctataggct tcggtgttag taccacatcg 180
tatgcagact ccgtgaaggg ccgattcacc atctccagaa acaacgccaa gaacacggtg 240
tatctgcaaa tgaacagcct gaaacctgag gacacggccg tttattactg cgcagcgcgc 300
cccccgcctt acacggagta taactactgg ggccagggga cccaggtcac cgtctcctca 360
<210>106
<211>372
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb53)
<400>106
caggtgcagc tgcaggagtc tggaggagga gtggtgcagg ctggggactc tctgagactc60
tcctgtgcag cctctggact caccttcagt agttatctca tggcctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtagcacgt attaactgga atggtcgtgt gccatacact 180
gtagactctg tgaagggccg attcatcatc tccagagaca atgccaaaaa cacggtgtgg 240
ctgcaaatga acagcctgaa acctgaggac acggccgttt attactgtgc agcagaccgg 300
aactacggca cagggggcgc cgaaacagtg tatgagtact ggggccaggg gacccaggtc 360
accgtctcct ca 372
<210>107
<211>375
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb55)
<400>107
caggtgcagc tgcaggagtc tgggggagga ttggtgcagg ctgggggctc tctgagactc60
tcctgtgaag cctctggacg cgccttcagt gactcgtcgg ccatggcctg gttccgccag 120
gctccaggga aggagcgtga gtttgtagcg gcgcttaaca gagttaatgt tgcatattgt 180
agagactccg tgtcgggccg attcaccatc tccagagaca acggcaagaa tacggcatat 240
ctggaaatga acagtgtgaa acctgaggac acggccattt attactgtgc atcagatcta 300
atcctaaatg attgcagtcg aaaccccgcg aggtatgcct actggggcca ggggacccag 360
gtcaccgtct cctca375
<210>108
<211>387
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (Nb60)
<400>108
caggtgcagc tgcaggagtc tgggggagga ttggtgcagg ctggggactc tctgagactc60
tcctgtctag cctctgaacg cacattcagg cgctatggca tgggctggtt ccgccaggct 120
ccagggaagg agcgtgagtt tgtagcagct attgaccgga gtcatagtaa tacagactat 180
gcagacttcg tgaagggccg attcaccatc tccacggtct acgccaagaa catggtgtat 240
ctgcaaatga acagcctgaa acctgaggac acggccgttt attactgtgc tgcggcgacg 300
tacgagaaag ggtcggaccc tactagttgg aacaccgaca gagggtatga cgtctggggc 360
caggggaccc aggtcaccgt ctcctca 387
<210>109
<211>363
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (msNb12)
<400>109
caagtaaaac ttgaagaatc aggtggtgga agcgttcaag ctggtggatc tctcagactt60
atttgtaccg cacccggctt aacacataat aactgtggct tggactggta cagacgagca 120
ccaggcaagg aacgcgagtt cgtttcatca ataagtgcag atggaaccac ttcatacgca 180
gattccgtca agggacggtt taccattagt aaggacaaag tcgaggacac agtctacctc 240
caaatgaaca gtttgaagcc agaagacact gctatttatt catgcaagac agccttccct 300
tacttcggta atagctgtgt tttggactac tggggtcaag gaacctcagt caccgtctcc 360
tcg 363
<210>110
<211>375
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (mNb30)
<400>110
caggtgcaat tggtagagtc tggcggtgga ctcgttcaag ccggaggctc actgcggttg60
tcttgtgccg catctggtct aaccttctca aaatatgcta tggggtggtt ccggcaggct 120
ccaggtaaag agcggaaatt tgtcgcaaca attagttggt ctggtgatag tgccttctat 180
gctgattcag taaaaggtcg attcactata tcacgggata acgcaagaaa tactgtctat 240
ctccaaatga actctctgaa gcctgaagat actgctgtgt attactgtgc cgcagatcga 300
ggaatggggt atggggattt tatggactac tggggtcaag gaacctcagt caccgcctcc 360
tcggcctcag gggcc375
<210>111
<211>375
<212>DNA
<213>人工序列 (Artificial Sequence)
<220>
<223>单结构域抗体nt序列 (mNb35)
<400>111
caggtccaac tggtagagtc aggcggtgga ctcgttcaag ccggaggctc actgcggttg60
tcttgtgccg catctggtcg aaccttctca aaatatgcta tggggtggtt ccggcaggct 120
ccaggtaaag agcggaaatt tgtcgcaaca attagttggt ctggtgatag tgccttctat 180
gctgattcag taaaaggtcg attcactata tcacgggata acgcaagaaa tactgtctat 240
ctccaaatga actctctgaa gcctgaagat actgctgtgt attactgtac cgcagatcga 300
ggaatggggt atggggattt tatggactac tggggtcaag gtacctcagt caccgtctcc 360
tcggcctcag gggcc375
<210>112
<211>349
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb15-IgG1
<400>112
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Val Ser Cys Ala Ala Ser Gly Leu Pro Phe Ser Asp Tyr
202530
Leu Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Tyr Val
354045
Ala Ala Ile Ser Gln Asn Gly Gly His Thr Tyr Ala Asp Ser Val Leu
505560
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr Leu
65707580
Gln Met Asn Met Leu Thr Pro Gly Asp Thr Ala Val Tyr Ser Cys Ala
859095
Ala Arg Arg Pro Gly Gly Gly Arg Trp Asp Ala Ala His Asp Tyr Asn
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Asp Lys Thr His
115 120 125
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
130 135 140
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
145 150 155 160
Pro Glu Val Thr Cys Val Trp Asp Val Ser His Glu Asp Pro Glu Val
165 170 175
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
180 185 190
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Trp Ser Val Leu
195 200 205
Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys
210 215 220
Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys
225 230 235 240
Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser
245 250 255
Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys
260 265 270
Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln
275 280 285
Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly
290 295 300
Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln
305 310 315 320
Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn
325 330 335
His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
340 345
<210>113
<211>344
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb17-IgG1
<400>113
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Thr Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Gly Ile Tyr
202530
Arg Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Ile Thr Ser Ser Ala Asp Thr Ala Gln Tyr Arg Asp Ser Val
505560
Lys Gly Arg Phe Ala Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
859095
Ala Ala Arg Asp Pro Thr Thr Leu Glu Tyr Gly Asn Trp Gly Gln Gly
100 105 110
Thr Gln Val Thr Val Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys
115 120 125
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
130 135 140
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
145 150 155 160
Val Trp Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
165 170 175
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
180 185 190
Gln Tyr Asn Ser Thr Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln
195 200 205
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
210 215 220
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
225 230 235 240
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
245 250 255
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
260 265 270
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
275 280 285
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
290 295 300
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
305 310 315 320
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
325 330 335
Ser Leu Ser Leu Ser Pro Gly Lys
340
<210>114
<211>344
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb19-IgG1
<400>114
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Gly Phe Ser Ile His
202530
Ala Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Phe Val
354045
Ala Val Val Gly His Lys Thr Asn Tyr Ala Asp Ser Val Lys Gly Arg
505560
Phe Thr Ile Ser Arg Asp Val Gly Lys Asn Thr Val Glu Leu Gln Met
65707580
Asn Ser Leu Lys Val Glu Asp Thr Ala Val Tyr Tyr Cys Tyr Cys Asn
859095
Thr Ile Val Thr Met Thr Gly Val Pro Asp Ala Val Trp Gly Gln Gly
100 105 110
Thr Gln Val Thr Val Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys
115 120 125
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
130 135 140
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
145 150 155 160
Val Trp Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
165 170 175
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
180 185 190
Gln Tyr Asn Ser Thr Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln
195 200 205
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
210 215 220
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
225 230 235 240
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
245 250 255
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
260 265 270
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
275 280 285
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
290 295 300
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
305 310 315 320
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
325 330 335
Ser Leu Ser Leu Ser Pro Gly Lys
340
<210>115
<211>353
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb56-IgG1
<400>115
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Val Ala Ser Glu Arg Thr Phe Arg Arg Tyr
202530
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Val Asp Arg Ser His Thr Lys Thr Gly Tyr Ala Asp Phe Val
505560
Lys Gly Arg Phe Thr Ile Ser Thr Asn Tyr Glu Asn Met Val Tyr Leu
65707580
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
859095
Ala Pro Ser Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr Asp
100 105 110
Arg Gly Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120 125
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
130 135 140
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
145 150 155 160
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser His Glu
165 170 175
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
180 185 190
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
195 200 205
Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
210 215 220
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
225 230 235 240
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
245 250 255
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
260 265 270
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
275 280 285
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
290 295 300
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
305 310 315 320
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
325 330 335
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
340 345 350
Lys
<210>116
<211>371
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>msNb12-IgG2a
<400>116
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ile Cys Thr Ala Pro Gly Leu Thr His Asn Asn Cys
202530
Gly Leu Asp Trp Tyr Arg Arg Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ser Ser Ile Ser Ala Asp Gly Thr Thr Ser Tyr Ala Asp Ser Val Lys
505560
Gly Arg Phe Thr Ile Ser Lys Asp Lys Val Glu Asp Thr Val Tyr Leu
65707580
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Ser Cys Lys
859095
Thr Ala Phe Pro Tyr Phe Gly Asn Ser Cys Val Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser Glu Pro Lys Ile Pro Gln Pro
115 120 125
Gln Pro Lys Pro Gln Pro Gln Pro Gln Pro Gln Pro Lys Pro Gln Pro
130 135 140
Lys Pro Glu Pro Glu Cys Thr Cys Pro Lys Cys Pro Ala Pro Glu Leu
145 150 155 160
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
165 170 175
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser
180 185 190
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
195 200 205
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
210 215 220
Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
225 230 235 240
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
245 250 255
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
260 265 270
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
275 280 285
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
290 295 300
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
305 310 315 320
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
325 330 335
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
340 345 350
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
355 360 365
Pro Gly Lys
370
<210>117
<211>371
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>mNb30-IgG2a
<400>117
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Ser Lys Tyr
202530
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Lys Phe Val
354045
Ala Thr Ile Ser Trp Ser Gly Asp Ser Ala Phe Tyr Ala Asp Ser Val
505560
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Ala Ser Ser Glu Pro Lys Ile Pro Gln Pro
115 120 125
Gln Pro Lys Pro Gln Pro Gln Pro Gln Pro Gln Pro Lys Pro Gln Pro
130 135 140
Lys Pro Glu Pro Glu Cys Thr Cys Pro Lys Cys Pro Ala Pro Glu Leu
145 150 155 160
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
165 170 175
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser
180 185 190
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
195 200 205
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
210 215 220
Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
225 230 235 240
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
245 250 255
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
260 265 270
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
275 280 285
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
290 295 300
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
305 310 315 320
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
325 330 335
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
340 345 350
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
355 360 365
Pro Gly Lys
370
<210>118
<211>627
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb15-三IgG1
<400>118
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Val Ser Cys Ala Ala Ser Gly Leu Pro Phe Ser Asp Tyr
202530
Leu Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Tyr Val
354045
Ala Ala Ile Ser Gln Asn Gly Gly His Thr Tyr Ala Asp Ser Val Leu
505560
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr Leu
65707580
Gln Met Asn Met Leu Thr Pro Gly Asp Thr Ala Val Tyr Ser Cys Ala
859095
Ala Arg Arg Pro Gly Gly Gly Arg Trp Asp Ala Ala His Asp Tyr Asn
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln
130 135 140
Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly Ser Leu Arg Val Ser
145 150 155 160
Cys Ala Ala Ser Gly Leu Pro Phe Ser Asp Tyr Leu Met Gly Trp Phe
165 170 175
Arg Gln Ala Pro Gly Lys Glu Arg Glu Tyr Val Ala Ala Ile Ser Gln
180 185 190
Asn Gly Gly His Thr Tyr Ala Asp Ser Val Leu Gly Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asn Ala Lys Asn Thr Val Tyr Leu Gln Met Asn Met Leu
210 215 220
Thr Pro Gly Asp Thr Ala Val Tyr Ser Cys Ala Ala Arg Arg Pro Gly
225 230 235 240
Gly Gly Arg Trp Asp Ala Ala His Asp Tyr Asn Tyr Trp Gly Gln Gly
245 250 255
Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
260 265 270
Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Gly Gly
275 280 285
Leu Val Gln Ala Gly Gly Ser Leu Arg Val Ser Cys Ala Ala Ser Gly
290 295 300
Leu Pro Phe Ser Asp Tyr Leu Met Gly Trp Phe Arg Gln Ala Pro Gly
305 310 315 320
Lys Glu Arg Glu Tyr Val Ala Ala Ile Ser Gln Asn Gly Gly His Thr
325 330 335
Tyr Ala Asp Ser Val Leu Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
340 345 350
Lys Asn Thr Val Tyr Leu Gln Met Asn Met Leu Thr Pro Gly Asp Thr
355 360 365
Ala Val Tyr Ser Cys Ala Ala Arg Arg Pro Gly Gly Gly Arg Trp Asp
370 375 380
Ala Ala His Asp Tyr Asn Tyr Trp Gly Gln Gly Thr Gln Val Thr Val
385 390 395 400
Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
405 410 415
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
420 425 430
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser
435 440 445
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
450 455 460
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
465 470 475 480
Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
485 490 495
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
500 505 510
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
515 520 525
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
530 535 540
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
545 550 555 560
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
565 570 575
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
580 585 590
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
595 600 605
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
610 615 620
Pro Gly Lys
625
<210>119
<211>612
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb17-三IgG1
<400>119
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Thr Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Gly Ile Tyr
202530
Arg Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Ile Thr Ser Ser Ala Asp Thr Ala Gln Tyr Arg Asp Ser Val
505560
Lys Gly Arg Phe Ala Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
859095
Ala Ala Arg Asp Pro Thr Thr Leu Glu Tyr Gly Asn Trp Gly Gln Gly
100 105 110
Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Gly Gly
130 135 140
Leu Val Gln Thr Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
145 150 155 160
Arg Thr Phe Gly Ile Tyr Arg Met Gly Trp Phe Arg Gln Ala Pro Gly
165 170 175
Lys Glu Arg Glu Phe Val Ala Ala Ile Thr Ser Ser Ala Asp Thr Ala
180 185 190
Gln Tyr Arg Asp Ser Val Lys Gly Arg Phe Ala Ile Ser Arg Asp Asn
195 200 205
Ala Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp
210 215 220
Thr Ala Ile Tyr Tyr Cys Ala Ala Arg Asp Pro Thr Thr Leu Glu Tyr
225 230 235 240
Gly Asn Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly
245 250 255
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
260 265 270
Gln Glu Ser Gly Gly Gly Leu Val Gln Thr Gly Gly Ser Leu Arg Leu
275 280 285
Ser Cys Ala Ala Ser Gly Arg Thr Phe Gly Ile Tyr Arg Met Gly Trp
290 295 300
Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val Ala Ala Ile Thr
305 310 315 320
Ser Ser Ala Asp Thr Ala Gln Tyr Arg Asp Ser Val Lys Gly Arg Phe
325 330 335
Ala Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu Gln Met Asn
340 345 350
Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ala Ala Arg Asp
355 360 365
Pro Thr Thr Leu Glu Tyr Gly Asn Trp Gly Gln Gly Thr Gln Val Thr
370 375 380
Val Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
385 390 395 400
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
405 410 415
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val
420 425 430
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
435 440 445
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
450 455 460
Thr Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
465 470 475 480
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
485 490 495
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
500 505 510
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
515 520 525
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
530 535 540
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
545 550 555 560
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
565 570 575
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
580 585 590
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
595 600 605
Ser Pro Gly Lys
610
<210>120
<211>612
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb19-三IgG1
<400>120
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ser Gly Phe Ser Ile His
202530
Ala Met Gly Trp Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Phe Val
354045
Ala Val Val Gly His Lys Thr Asn Tyr Ala Asp Ser Val Lys Gly Arg
505560
Phe Thr Ile Ser Arg Asp Val Gly Lys Asn Thr Val Glu Leu Gln Met
65707580
Asn Ser Leu Lys Val Glu Asp Thr Ala Val Tyr Tyr Cys Tyr Cys Asn
859095
Thr Ile Val Thr Met Thr Gly Val Pro Asp Ala Val Trp Gly Gln Gly
100 105 110
Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Gln Glu Ser Gly Gly Gly
130 135 140
Leu Val Gln Ala Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
145 150 155 160
Ser Gly Phe Ser Ile His Ala Met Gly Trp Tyr Arg Gln Ala Pro Gly
165 170 175
Lys Gln Arg Glu Phe Val Ala Val Val Gly His Lys Thr Asn Tyr Ala
180 185 190
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Val Gly Lys Asn
195 200 205
Thr Val Glu Leu Gln Met Asn Ser Leu Lys Val Glu Asp Thr Ala Val
210 215 220
Tyr Tyr Cys Tyr Cys Asn Thr Ile Val Thr Met Thr Gly Val Pro Asp
225 230 235 240
Ala Val Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly
245 250 255
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu
260 265 270
Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly Ser Leu Arg Leu
275 280 285
Ser Cys Ala Ala Ser Gly Ser Gly Phe Ser Ile His Ala Met Gly Trp
290 295 300
Tyr Arg Gln Ala Pro Gly Lys Gln Arg Glu Phe Val Ala Val Val Gly
305 310 315 320
His Lys Thr Asn Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
325 330 335
Arg Asp Val Gly Lys Asn Thr Val Glu Leu Gln Met Asn Ser Leu Lys
340 345 350
Val Glu Asp Thr Ala Val Tyr Tyr Cys Tyr Cys Asn Thr Ile Val Thr
355 360 365
Met Thr Gly Val Pro Asp Ala Val Trp Gly Gln Gly Thr Gln Val Thr
370 375 380
Val Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
385 390 395 400
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
405 410 415
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val
420 425 430
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
435 440 445
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
450 455 460
Thr Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
465 470 475 480
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
485 490 495
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
500 505 510
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
515 520 525
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
530 535 540
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
545 550 555 560
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
565 570 575
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
580 585 590
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
595 600 605
Ser Pro Gly Lys
610
<210>121
<211>639
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>Nb56-三IgG1
<400>121
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp
1 5 1015
Ser Leu Arg Leu Ser Cys Val Ala Ser Glu Arg Thr Phe Arg Arg Tyr
202530
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ala Ala Val Asp Arg Ser His Thr Lys Thr Gly Tyr Ala Asp Phe Val
505560
Lys Gly Arg Phe Thr Ile Ser Thr Asn Tyr Glu Asn Met Val Tyr Leu
65707580
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala
859095
Ala Pro Ser Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr Asp
100 105 110
Arg Gly Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
115 120 125
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln
130 135 140
Val Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp Ser
145 150 155 160
Leu Arg Leu Ser Cys Val Ala Ser Glu Arg Thr Phe Arg Arg Tyr Gly
165 170 175
Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val Ala
180 185 190
Ala Val Asp Arg Ser His Thr Lys Thr Gly Tyr Ala Asp Phe Val Lys
195 200 205
Gly Arg Phe Thr Ile Ser Thr Asn Tyr Glu Asn Met Val Tyr Leu Gln
210 215 220
Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala Ala
225 230 235 240
Pro Ser Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr Asp Arg
245 250 255
Gly Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
260 265 270
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gln Val
275 280 285
Gln Leu Gln Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Asp Ser Leu
290 295 300
Arg Leu Ser Cys Val Ala Ser Glu Arg Thr Phe Arg Arg Tyr Gly Met
305 310 315 320
Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val Ala Ala
325 330 335
Val Asp Arg Ser His Thr Lys Thr Gly Tyr Ala Asp Phe Val Lys Gly
340 345 350
Arg Phe Thr Ile Ser Thr Asn Tyr Glu Asn Met Val Tyr Leu Gln Met
355 360 365
Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys Ala Ala Pro
370 375 380
Ser Tyr Glu Lys Gly Ser Asp Pro Thr Ser Trp Asn Thr Asp Arg Gly
385 390 395 400
Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Asp Lys
405 410 415
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
420 425 430
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
435 440 445
Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser His Glu Asp Pro
450 455 460
Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
465 470 475 480
Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Trp Ser
485 490 495
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
500 505 510
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
515 520 525
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
530 535 540
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
545 550 555 560
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
565 570 575
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
580 585 590
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
595 600 605
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
610 615 620
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
625 630 635
<210>122
<211>643
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>msNb12-三IgG2a
<400>122
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ile Cys Thr Ala Pro Gly Leu Thr His Asn Asn Cys
202530
Gly Leu Asp Trp Tyr Arg Arg Ala Pro Gly Lys Glu Arg Glu Phe Val
354045
Ser Ser Ile Ser Ala Asp Gly Thr Thr Ser Tyr Ala Asp Ser Val Lys
505560
Gly Arg Phe Thr Ile Ser Lys Asp Lys Val Glu Asp Thr Val Tyr Leu
65707580
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Ser Cys Lys
859095
Thr Ala Phe Pro Tyr Phe Gly Asn Ser Cys Val Leu Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Lys Leu Glu Glu Ser Gly
130 135 140
Gly Gly Ser Val Gln Ala Gly Gly Ser Leu Arg Leu Ile Cys Thr Ala
145 150 155 160
Pro Gly Leu Thr His Asn Asn Cys Gly Leu Asp Trp Tyr Arg Arg Ala
165 170 175
Pro Gly Lys Glu Arg Glu Phe Val Ser Ser Ile Ser Ala Asp Gly Thr
180 185 190
Thr Ser Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Lys Asp
195 200 205
Lys Val Glu Asp Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu
210 215 220
Asp Thr Ala Ile Tyr Ser Cys Lys Thr Ala Phe Pro Tyr Phe Gly Asn
225 230 235 240
Ser Cys Val Leu Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Val Ser
245 250 255
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
260 265 270
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Ala Gly Gly
275 280 285
Ser Leu Arg Leu Ile Cys Thr Ala Pro Gly Leu Thr His Asn Asn Cys
290 295 300
Gly Leu Asp Trp Tyr Arg Arg Ala Pro Gly Lys Glu Arg Glu Phe Val
305 310 315 320
Ser Ser Ile Ser Ala Asp Gly Thr Thr Ser Tyr Ala Asp Ser Val Lys
325 330 335
Gly Arg Phe Thr Ile Ser Lys Asp Lys Val Glu Asp Thr Val Tyr Leu
340 345 350
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Ser Cys Lys
355 360 365
Thr Ala Phe Pro Tyr Phe Gly Asn Ser Cys Val Leu Asp Tyr Trp Gly
370 375 380
Gln Gly Thr Ser Val Thr Val Ser Ser Glu Pro Lys Ile Pro Gln Pro
385 390 395 400
Gln Pro Lys Pro Gln Pro Gln Pro Gln Pro Gln Pro Lys Pro Gln Pro
405 410 415
Lys Pro Glu Pro Glu Cys Thr Cys Pro Lys Cys Pro Ala Pro Glu Leu
420 425 430
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
435 440 445
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser
450 455 460
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
465 470 475 480
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
485 490 495
Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
500 505 510
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
515 520 525
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
530 535 540
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
545 550 555 560
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
565 570 575
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
580 585 590
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
595 600 605
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
610 615 620
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
625 630 635 640
Pro Gly Lys
<210>123
<211>643
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>mNb30-三IgG2a
<400>123
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 1015
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Ser Lys Tyr
202530
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Lys Phe Val
354045
Ala Thr Ile Ser Trp Ser Gly Asp Ser Ala Phe Tyr Ala Asp Ser Val
505560
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Val Tyr
65707580
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
859095
Ala Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Ala Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Ala Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Leu Thr Phe Ser Lys Tyr Ala Met Gly Trp Phe Arg Gln Ala
165 170 175
Pro Gly Lys Glu Arg Lys Phe Val Ala Thr Ile Ser Trp Ser Gly Asp
180 185 190
Ser Ala Phe Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
195 200 205
Asp Asn Ala Arg Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro
210 215 220
Glu Asp Thr Ala Val Tyr Tyr Cys Ala Ala Asp Arg Gly Met Gly Tyr
225 230 235 240
Gly Asp Phe Met Asp Tyr Trp Gly Gln Gly Thr Ser Val Thr Ala Ser
245 250 255
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
260 265 270
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
275 280 285
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Leu Thr Phe Ser Lys Tyr
290 295 300
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Lys Phe Val
305 310 315 320
Ala Thr Ile Ser Trp Ser Gly Asp Ser Ala Phe Tyr Ala Asp Ser Val
325 330 335
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Arg Asn Thr Val Tyr
340 345 350
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
355 360 365
Ala Ala Asp Arg Gly Met Gly Tyr Gly Asp Phe Met Asp Tyr Trp Gly
370 375 380
Gln Gly Thr Ser Val Thr Ala Ser Ser Glu Pro Lys Ile Pro Gln Pro
385 390 395 400
Gln Pro Lys Pro Gln Pro Gln Pro Gln Pro Gln Pro Lys Pro Gln Pro
405 410 415
Lys Pro Glu Pro Glu Cys Thr Cys Pro Lys Cys Pro Ala Pro Glu Leu
420 425 430
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
435 440 445
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Trp Asp Val Ser
450 455 460
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
465 470 475 480
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
485 490 495
Tyr Arg Trp Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
500 505 510
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
515 520 525
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
530 535 540
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
545 550 555 560
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
565 570 575
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
580 585 590
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
595 600 605
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
610 615 620
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
625 630 635 640
Pro Gly Lys
<210>124
<211>194
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>刺突糖蛋白[严重急性呼吸综合征冠状病毒
2]
<400>124
Asn Ile Thr Asn Leu Cys Pro Phe Gly Glu Val Phe Asn Ala Thr Arg
1 5 1015
Phe Ala Ser Val Tyr Ala Trp Asn Arg Lys Arg Ile Ser Asn Cys Val
202530
Ala Asp Tyr Ser Val Leu Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys
354045
Cys Tyr Gly Val Ser Pro Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn
505560
Val Tyr Ala Asp Ser Phe Val Ile Arg Gly Asp Glu Val Arg Gln Ile
65707580
Ala Pro Gly Gln Thr Gly Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro
859095
Asp Asp Phe Thr Gly Cys Val Ile Ala Trp Asn Ser Asn Asn Leu Asp
100 105 110
Ser Lys Val Gly Gly Asn Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys
115 120 125
Ser Asn Leu Lys Pro Phe Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln
130 135 140
Ala Gly Ser Thr Pro Cys Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe
145 150 155 160
Pro Leu Gln Ser Tyr Gly Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln
165 170 175
Pro Tyr Arg Val Val Val Leu Ser Phe Glu Leu Leu His Ala Pro Ala
180 185 190
Thr Val
<210>125
<211>193
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>刺突糖蛋白S [SARS冠状病毒BJ01]
<400>125
Asn Ile Thr Asn Leu Cys Pro Phe Gly Glu Val Phe Asn Ala Thr Lys
1 5 1015
Phe Pro Ser Val Tyr Ala Trp Glu Arg Lys Lys Ile Ser Asn Cys Val
202530
Ala Asp Tyr Ser Val Leu Tyr Asn Ser Thr Phe Phe Ser Thr Phe Lys
354045
Cys Tyr Gly Val Ser Ala Thr Lys Leu Asn Asp Leu Cys Phe Ser Asn
505560
Val Tyr Ala Asp Ser Phe Val Val Lys Gly Asp Asp Val Arg Gln Ile
65707580
Ala Pro Gly Gln Thr Gly Val Ile Ala Asp Tyr Asn Tyr Lys Leu Pro
859095
Asp Asp Phe Met Gly Cys Val Leu Ala Trp Asn Thr Arg Asn Ile Asp
100 105 110
Ala Thr Ser Thr Gly Asn Tyr Asn Tyr Lys Tyr Arg Tyr Leu Arg His
115 120 125
Gly Lys Leu Arg Pro Phe Glu Arg Asp Ile Ser Asn Val Pro Phe Ser
130 135 140
Pro Asp Gly Lys Pro Cys Thr Pro Pro Ala Leu Asn Cys Tyr Trp Pro
145 150 155 160
Leu Asn Asp Tyr Gly Phe Tyr Thr Thr Thr Gly Ile Gly Tyr Gln Pro
165 170 175
Tyr Arg Val Val Val Leu Ser Phe Glu Leu Leu Asn Ala Pro Ala Thr
180 185 190
Val
<210>126
<211>212
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>刺突糖蛋白[MERS冠状病毒]
<400>126
Gln Ala Glu Gly Val Glu Cys Asp Phe Ser Pro Leu Leu Ser Gly Thr
1 5 1015
Pro Pro Gln Val Tyr Asn Phe Lys Arg Leu Val Phe Thr Asn Cys Asn
202530
Tyr Asn Leu Thr Lys Leu Leu Ser Leu Phe Ser Val Asn Asp Phe Thr
354045
Cys Ser Gln Ile Ser Pro Ala Ala Ile Ala Ser Asn Cys Tyr Ser Ser
505560
Leu Ile Leu Asp Tyr Phe Ser Tyr Pro Leu Ser Met Lys Ser Asp Leu
65707580
Ser Val Ser Ser Ala Gly Pro Ile Ser Gln Phe Asn Tyr Lys Gln Ser
859095
Phe Ser Asn Pro Thr Cys Leu Ile Leu Ala Thr Val Pro His Asn Leu
100 105 110
Thr Thr Ile Thr Lys Pro Leu Lys Tyr Ser Tyr Ile Asn Lys Cys Ser
115 120 125
Arg Leu Leu Ser Asp Asp Arg Thr Glu Val Pro Gln Leu Val Asn Ala
130 135 140
Asn Gln Tyr Ser Pro Cys Val Ser Ile Val Pro Ser Thr Val Trp Glu
145 150 155 160
Asp Gly Asp Tyr Tyr Arg Lys Gln Leu Ser Pro Leu Glu Gly Gly Gly
165 170 175
Trp Leu Val Ala Ser Gly Ser Thr Val Ala Met Thr Glu Gln Leu Gln
180 185 190
Met Gly Phe Gly Ile Thr Val Gln Tyr Gly Thr Asp Thr Asn Ser Val
195 200 205
Cys Pro Lys Leu
210
<210>127
<211>223
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>SARS CoV-2刺突受体结合结构域
<400>127
Arg Val Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn
1 5 1015
Leu Cys Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val
202530
Tyr Ala Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser
354045
Val Leu Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val
505560
Ser Pro Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp
65707580
Ser Phe Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln
859095
Thr Gly Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr
100 105 110
Gly Cys Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly
115 120 125
Gly Asn Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys
130 135 140
Pro Phe Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr
145 150 155 160
Pro Cys Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser
165 170 175
Tyr Gly Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val
180 185 190
Val Val Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly
195 200 205
Pro Lys Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe
210 215 220
<210>128
<211>1253
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>刺突糖蛋白[严重急性呼吸综合征冠状病毒
2]
<400>128
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 1015
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
202530
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
354045
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
505560
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65707580
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
859095
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Arg Arg Ala Ala Ser Val Ala
675 680 685
Ser Gln Ser Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser
690 695 700
Val Ala Tyr Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile
705 710 715 720
Ser Val Thr Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val
725 730 735
Asp Cys Thr Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu
740 745 750
Leu Leu Gln Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr
755 760 765
Gly Ile Ala Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln
770 775 780
Val Lys Gln Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe
785 790 795 800
Asn Phe Ser Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser
805 810 815
Phe Ile Glu Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly
820 825 830
Phe Ile Lys Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp
835 840 845
Leu Ile Cys Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu
850 855 860
Leu Thr Asp Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly
865 870 875 880
Thr Ile Thr Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile
885 890 895
Pro Phe Ala Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr
900 905 910
Gln Asn Val Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn
915 920 925
Ser Ala Ile Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala
930 935 940
Leu Gly Lys Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn
945 950 955 960
Thr Leu Val Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val
965 970 975
Leu Asn Asp Ile Leu Ser Arg Leu Asp Lys Val Glu Ala Glu Val Gln
980 985 990
Ile Asp Arg Leu Ile Thr Gly ArgLeu Gln Ser Leu GlnThr Tyr Val
995 1000 1005
Thr GlnGln Leu Ile Arg AlaAla Glu Ile Arg AlaSer Ala Asn
1010 1015 1020
Leu AlaAla Thr Lys Met SerGlu Cys Val Leu GlyGln Ser Lys
1025 1030 1035
Arg ValAsp Phe Cys Gly LysGly Tyr His Leu MetSer Phe Pro
1040 1045 1050
Gln SerAla Pro His Gly ValVal Phe Leu His ValThr Tyr Val
1055 1060 1065
Pro AlaGln Glu Lys Asn PheThr Thr Ala Pro AlaIle Cys His
1070 1075 1080
Asp GlyLys Ala His Phe ProArg Glu Gly Val PheVal Ser Asn
1085 1090 1095
Gly ThrHis Trp Phe Val ThrGln Arg Asn Phe TyrGlu Pro Gln
1100 1105 1110
Ile IleThr Thr Asp Asn ThrPhe Val Ser Gly AsnCys Asp Val
1115 1120 1125
Val IleGly Ile Val Asn AsnThr Val Tyr Asp ProLeu Gln Pro
1130 1135 1140
Glu LeuAsp Ser Phe Lys GluGlu Leu Asp Lys TyrPhe Lys Asn
1145 1150 1155
His ThrSer Pro Asp Val AspLeu Gly Asp Ile SerGly Ile Asn
1160 1165 1170
Ala SerVal Val Asn Ile GlnLys Glu Ile Asp ArgLeu Asn Glu
1175 1180 1185
Val AlaLys Asn Leu Asn GluSer Leu Ile Asp LeuGln Glu Leu
1190 1195 1200
Gly LysTyr Glu Gln Tyr IleLys Trp Pro Ser GlyArg Leu Val
1205 1210 1215
Pro ArgGly Ser Pro Gly SerGly Tyr Ile Pro GluAla Pro Arg
1220 1225 1230
Asp GlyGln Ala Tyr Val ArgLys Asp Gly Glu TrpVal Leu Leu
1235 1240 1245
Ser ThrPhe Leu Gly
1250
<210>129
<211>243
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>SARS-CoV-2 RBD多肽
<400>129
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Arg Val
1 5 1015
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
202530
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
354045
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
505560
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
65707580
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
859095
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
100 105 110
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
115 120 125
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
130 135 140
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
145 150 155 160
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
165 170 175
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
180 185 190
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
195 200 205
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
210 215 220
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe His His His
225 230 235 240
His His His
<210>130
<211>1256
<212>PRT
<213>人工序列 (Artificial Sequence)
<220>
<223>SARS-CoV-2刺突多肽
<400>130
Met Phe Val Phe Leu Val Leu Leu Pro Leu Val Ser Ser Gln Cys Val
1 5 1015
Asn Leu Thr Thr Arg Thr Gln Leu Pro Pro Ala Tyr Thr Asn Ser Phe
202530
Thr Arg Gly Val Tyr Tyr Pro Asp Lys Val Phe Arg Ser Ser Val Leu
354045
His Ser Thr Gln Asp Leu Phe Leu Pro Phe Phe Ser Asn Val Thr Trp
505560
Phe His Ala Ile His Val Ser Gly Thr Asn Gly Thr Lys Arg Phe Asp
65707580
Asn Pro Val Leu Pro Phe Asn Asp Gly Val Tyr Phe Ala Ser Thr Glu
859095
Lys Ser Asn Ile Ile Arg Gly Trp Ile Phe Gly Thr Thr Leu Asp Ser
100 105 110
Lys Thr Gln Ser Leu Leu Ile Val Asn Asn Ala Thr Asn Val Val Ile
115 120 125
Lys Val Cys Glu Phe Gln Phe Cys Asn Asp Pro Phe Leu Gly Val Tyr
130 135 140
Tyr His Lys Asn Asn Lys Ser Trp Met Glu Ser Glu Phe Arg Val Tyr
145 150 155 160
Ser Ser Ala Asn Asn Cys Thr Phe Glu Tyr Val Ser Gln Pro Phe Leu
165 170 175
Met Asp Leu Glu Gly Lys Gln Gly Asn Phe Lys Asn Leu Arg Glu Phe
180 185 190
Val Phe Lys Asn Ile Asp Gly Tyr Phe Lys Ile Tyr Ser Lys His Thr
195 200 205
Pro Ile Asn Leu Val Arg Asp Leu Pro Gln Gly Phe Ser Ala Leu Glu
210 215 220
Pro Leu Val Asp Leu Pro Ile Gly Ile Asn Ile Thr Arg Phe Gln Thr
225 230 235 240
Leu Leu Ala Leu His Arg Ser Tyr Leu Thr Pro Gly Asp Ser Ser Ser
245 250 255
Gly Trp Thr Ala Gly Ala Ala Ala Tyr Tyr Val Gly Tyr Leu Gln Pro
260 265 270
Arg Thr Phe Leu Leu Lys Tyr Asn Glu Asn Gly Thr Ile Thr Asp Ala
275 280 285
Val Asp Cys Ala Leu Asp Pro Leu Ser Glu Thr Lys Cys Thr Leu Lys
290 295 300
Ser Phe Thr Val Glu Lys Gly Ile Tyr Gln Thr Ser Asn Phe Arg Val
305 310 315 320
Gln Pro Thr Glu Ser Ile Val Arg Phe Pro Asn Ile Thr Asn Leu Cys
325 330 335
Pro Phe Gly Glu Val Phe Asn Ala Thr Arg Phe Ala Ser Val Tyr Ala
340 345 350
Trp Asn Arg Lys Arg Ile Ser Asn Cys Val Ala Asp Tyr Ser Val Leu
355 360 365
Tyr Asn Ser Ala Ser Phe Ser Thr Phe Lys Cys Tyr Gly Val Ser Pro
370 375 380
Thr Lys Leu Asn Asp Leu Cys Phe Thr Asn Val Tyr Ala Asp Ser Phe
385 390 395 400
Val Ile Arg Gly Asp Glu Val Arg Gln Ile Ala Pro Gly Gln Thr Gly
405 410 415
Lys Ile Ala Asp Tyr Asn Tyr Lys Leu Pro Asp Asp Phe Thr Gly Cys
420 425 430
Val Ile Ala Trp Asn Ser Asn Asn Leu Asp Ser Lys Val Gly Gly Asn
435 440 445
Tyr Asn Tyr Leu Tyr Arg Leu Phe Arg Lys Ser Asn Leu Lys Pro Phe
450 455 460
Glu Arg Asp Ile Ser Thr Glu Ile Tyr Gln Ala Gly Ser Thr Pro Cys
465 470 475 480
Asn Gly Val Glu Gly Phe Asn Cys Tyr Phe Pro Leu Gln Ser Tyr Gly
485 490 495
Phe Gln Pro Thr Asn Gly Val Gly Tyr Gln Pro Tyr Arg Val Val Val
500 505 510
Leu Ser Phe Glu Leu Leu His Ala Pro Ala Thr Val Cys Gly Pro Lys
515 520 525
Lys Ser Thr Asn Leu Val Lys Asn Lys Cys Val Asn Phe Asn Phe Asn
530 535 540
Gly Leu Thr Gly Thr Gly Val Leu Thr Glu Ser Asn Lys Lys Phe Leu
545 550 555 560
Pro Phe Gln Gln Phe Gly Arg Asp Ile Ala Asp Thr Thr Asp Ala Val
565 570 575
Arg Asp Pro Gln Thr Leu Glu Ile Leu Asp Ile Thr Pro Cys Ser Phe
580 585 590
Gly Gly Val Ser Val Ile Thr Pro Gly Thr Asn Thr Ser Asn Gln Val
595 600 605
Ala Val Leu Tyr Gln Asp Val Asn Cys Thr Glu Val Pro Val Ala Ile
610 615 620
His Ala Asp Gln Leu Thr Pro Thr Trp Arg Val Tyr Ser Thr Gly Ser
625 630 635 640
Asn Val Phe Gln Thr Arg Ala Gly Cys Leu Ile Gly Ala Glu His Val
645 650 655
Asn Asn Ser Tyr Glu Cys Asp Ile Pro Ile Gly Ala Gly Ile Cys Ala
660 665 670
Ser Tyr Gln Thr Gln Thr Asn Ser Pro Ala Ser Val Ala Ser Gln Ser
675 680 685
Ile Ile Ala Tyr Thr Met Ser Leu Gly Ala Glu Asn Ser Val Ala Tyr
690 695 700
Ser Asn Asn Ser Ile Ala Ile Pro Thr Asn Phe Thr Ile Ser Val Thr
705 710 715 720
Thr Glu Ile Leu Pro Val Ser Met Thr Lys Thr Ser Val Asp Cys Thr
725 730 735
Met Tyr Ile Cys Gly Asp Ser Thr Glu Cys Ser Asn Leu Leu Leu Gln
740 745 750
Tyr Gly Ser Phe Cys Thr Gln Leu Asn Arg Ala Leu Thr Gly Ile Ala
755 760 765
Val Glu Gln Asp Lys Asn Thr Gln Glu Val Phe Ala Gln Val Lys Gln
770 775 780
Ile Tyr Lys Thr Pro Pro Ile Lys Asp Phe Gly Gly Phe Asn Phe Ser
785 790 795 800
Gln Ile Leu Pro Asp Pro Ser Lys Pro Ser Lys Arg Ser Phe Ile Glu
805 810 815
Asp Leu Leu Phe Asn Lys Val Thr Leu Ala Asp Ala Gly Phe Ile Lys
820 825 830
Gln Tyr Gly Asp Cys Leu Gly Asp Ile Ala Ala Arg Asp Leu Ile Cys
835 840 845
Ala Gln Lys Phe Asn Gly Leu Thr Val Leu Pro Pro Leu Leu Thr Asp
850 855 860
Glu Met Ile Ala Gln Tyr Thr Ser Ala Leu Leu Ala Gly Thr Ile Thr
865 870 875 880
Ser Gly Trp Thr Phe Gly Ala Gly Ala Ala Leu Gln Ile Pro Phe Ala
885 890 895
Met Gln Met Ala Tyr Arg Phe Asn Gly Ile Gly Val Thr Gln Asn Val
900 905 910
Leu Tyr Glu Asn Gln Lys Leu Ile Ala Asn Gln Phe Asn Ser Ala Ile
915 920 925
Gly Lys Ile Gln Asp Ser Leu Ser Ser Thr Ala Ser Ala Leu Gly Lys
930 935 940
Leu Gln Asp Val Val Asn Gln Asn Ala Gln Ala Leu Asn Thr Leu Val
945 950 955 960
Lys Gln Leu Ser Ser Asn Phe Gly Ala Ile Ser Ser Val Leu Asn Asp
965 970 975
Ile Leu Ser Arg Leu Asp Pro Pro Glu Ala Glu Val Gln Ile Asp Arg
980 985 990
Leu Ile Thr Gly Arg Leu Gln SerLeu Gln Thr Tyr ValThr Gln Gln
995 1000 1005
Leu IleArg Ala Ala Glu IleArg Ala Ser Ala AsnLeu Ala Ala
1010 1015 1020
Thr LysMet Ser Glu Cys ValLeu Gly Gln Ser LysArg Val Asp
1025 1030 1035
Phe CysGly Lys Gly Tyr HisLeu Met Ser Phe ProGln Ser Ala
1040 1045 1050
Pro HisGly Val Val Phe LeuHis Val Thr Tyr ValPro Ala Gln
1055 1060 1065
Glu LysAsn Phe Thr Thr AlaPro Ala Ile Cys HisAsp Gly Lys
1070 1075 1080
Ala HisPhe Pro Arg Glu GlyVal Phe Val Ser AsnGly Thr His
1085 1090 1095
Trp PheVal Thr Gln Arg AsnPhe Tyr Glu Pro GlnIle Ile Thr
1100 1105 1110
Thr AspAsn Thr Phe Val SerGly Asn Cys Asp ValVal Ile Gly
1115 1120 1125
Ile ValAsn Asn Thr Val TyrAsp Pro Leu Gln ProGlu Leu Asp
1130 1135 1140
Ser PheLys Glu Glu Leu AspLys Tyr Phe Lys AsnHis Thr Ser
1145 1150 1155
Pro AspVal Asp Leu Gly AspIle Ser Gly Ile AsnAla Ser Val
1160 1165 1170
Val AsnIle Gln Lys Glu IleAsp Arg Leu Asn GluVal Ala Lys
1175 1180 1185
Asn LeuAsn Glu Ser Leu IleAsp Leu Gln Glu LeuGly Lys Tyr
1190 1195 1200
Glu GlnTyr Ile Lys Trp ProSer Gly Arg Leu ValPro Arg Gly
1205 1210 1215
Ser ProGly Ser Gly Tyr IlePro Glu Ala Pro ArgAsp Gly Gln
1220 1225 1230
Ala TyrVal Arg Lys Asp GlyGlu Trp Val Leu LeuSer Thr Phe
1235 1240 1245
Leu GlyHis His His His HisHis
1250 1255
- 深部岩石保质取芯装置及其取芯方法
- 深部岩石原位主动保温取芯装置及其保温取芯方法
- 取芯装置、储心机构及取芯方法
- 适用于船用柴油机气缸体的可变径取芯料装置及方法
- 一种船用柴油机气缸体手工脱模芯盒及脱模方法
