用于基因编辑的组合物和方法
文献发布时间:2024-04-18 20:01:23
本申请是申请日为2019年4月19日,申请号为201980039136.4,发明名称为“用于基因编辑的组合物和方法”的专利申请的分案申请。
相关申请的交叉引用
本申请案主张2018年4月19日提交的美国申请案第62/660,023号的优先权,所述申请案的公开内容以全文引用的方式并入本文中。
关于在联邦政府资助的研究和研发下完成的发明的权利的申明
本发明是由国防部、国防部高级研究计划局授予的批准号为HR0011-17-2-0043和美国国家卫生研究院授予的批准号为R01 DA036858在政府支持下完成的。政府在本发明中具有某些权利。
背景技术
尽管基因组编辑被认为是疾病治疗的一种有前景的治疗方法,但其由于双链断裂的潜在基因毒性而携带固有的风险。此外,基因组编辑通常与对靶基因的完全影响或无影响相关(即,它会产生完整的基因敲除)。相比之下,靶向表观基因组工程不携带DSB诱导的基因毒性的风险;此外,它有机会对基因表达产生更多分级影响,并且从而从完全沉默到不太明显的影响之间发挥作用。
本文提供针对本领域的这些和其它需求的解决方案。
发明内容
在一方面中,提供一种融合蛋白,其包括缺乏核酸酶的RNA引导的DNA核酸内切酶、克鲁珀相关盒(Krüppel associated box;KRAB)结构域和DNA甲基转移酶结构域。在一方面中提供SEQ ID NO:1-15中任一者的融合蛋白。
在一方面中提供编码如本文所述的融合蛋白的核酸序列,包括其实施例和方面。
在一方面中提供一种复合物,其包括如本文所述的融合蛋白,包括其实施例和方面,和多核苷酸,其包括:(1)与靶多核苷酸序列互补的DNA靶向序列;和(2)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由所述结合序列与所述多核苷酸结合。
在一方面中提供了一种载体,其包括如本文所述的融合蛋白的核酸序列,包括其实施例和方面。
在一方面中提供一种细胞,其包括如本文所述的融合蛋白,包括其实施例和方面、如本文所述的核酸,包括其实施例和方面、如本文所述的复合物,包括其实施例和方面、或如本文所述的载体,包括其实施例和方面。
在一方面提供了一种沉默细胞中的靶核酸序列的方法,其包括将编码本文所述的融合蛋白(包括其实施例和方面)的第一多核苷酸递送至含有靶核酸的细胞,和将第二多核苷酸递送至细胞,所述第二多核苷酸包括:(i)与所述靶核酸序列互补的DNA靶向序列;和(ii)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列。在不希望受任何理论束缚的情况下,据信融合蛋白通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。因此,在各方面中,融合蛋白通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。
在一方面中提供一种沉默细胞中的靶核酸序列的方法,其包括将本文所述的复合物(包括其实施例和方面)递送至含有靶核酸的细胞,其中所述复合物在所述细胞中使靶核酸序列沉默。在不希望受任何理论束缚的情况下,据信复合物通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。因此,在各方面中,复合物通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。
附图说明
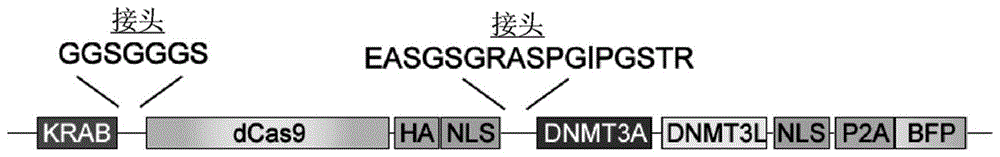
图1A-1F描述用于长期基因沉默的多合一(all-in-one)蛋白的工程改造。图1A为本公开的多合一蛋白(SEQ ID NO:1)的示意图,其具有融合至dCas9的N端(SEQ ID NO:23)的KRAB结构域,由GGSGGGS(SEQ ID NO:17)接头分离,和在dCas9的C端(由EASGSGRASPGIPGSTR(SEQ ID NO:19)接头分离)的Dnmt3A-Dnmt3L。图1B提供针对永久基因沉默测试的dCas9-融合表观遗传调节剂的示意图。dCas9-KRAB蛋白改编自Gilbert等人,《细胞(Cell)》2013年,用于CRISPR干扰(CRISPRi)应用。dCas9-Dnmt3A-Dnmt3L融合改编自Stepper等人,《核酸研究(Nucleic Acids Research)》,2016年。本发明人工程设计了一种新颖的多合一蛋白,其将KRAB结构域(SEQ ID NO:16)、dCas9(D10A,H208A)、Dnmt3A-Dnmt3L(SEQ ID NO:33;其中Dnmt3A为SEQ ID NO:26并且Dnmt3L为SEQ ID NO:28)组合成一种多肽。图1C提供甲基化敏感的GFP报告基因(改编自Stelzer等人,《细胞》2015年)的示意图,所述报告基因用于通过多合一蛋白评估长期沉默。图1D-1E提供在表达图3中所示的GFP报告基因的HEK293T细胞中的肇事逃逸(hit-and-run)实验工作流程的图和结果。图1D显示将两个质粒共转染至细胞中,一个质粒编码肇事逃逸蛋白,并且另一个质粒编码sgRNA。图1E显示对用多合一质粒和sgRNA质粒共转染的细胞进行分选的肇事逃逸分析的结果。图1F显示GFP报告基因沉默的结果取决于sgRNA序列。
图2A-2F描述内源基因的长期沉默。图2A-2C为代表性的流式细胞术数据,显示在转染后22天采集的使用多合一蛋白靶向长期沉默的基因(CD29、CD81、CD151)。象限IV代表基因关闭的细胞,由基因关闭的细胞百分比指示(分别为45%、66%和53%)。图2D提供用三种不同sgRNA对CD29、CD81和CD151的沉默的定量。图2E提供同时对两种或三种基因的沉默的定量,以显示可以通过共同递送靶向不同基因的sgRNA来实现多合一蛋白的多重化。图2F提供表示在转染多合一蛋白和靶向CLTA基因的sgRNA后9个月所采集的时间点的图,其表示大部分细胞已稳定地关闭CLTA基因。
图3A-3I描述内源基因的长期沉默。图3A-3C显示收集的细胞在转染后三十六天失去了CD29(图3A),CD81(图3B)和CD151(图3C)的表达,如通过它们的RNA表达谱所确定。图3D-3F为火山图,其显示靶向基因CD29(图3D)、CD81(图3E)和CD151(图3F)为每个实验中所敲低的仅有重要基因,表明基因沉默的高特异性。图3G-3I提供CD151(图3G),CD81(GIF.3H)和CD29(图3I)的转录本水平的定量,其显示大于96%的靶向基因的基因敲低。
图4A-4H描述不同哺乳动物细胞系中的长期基因沉默。图4A-4F为流式细胞术图,其显示在HeLa(宫颈)(图4A)、U2OS(骨)(图4B)和人诱导的多能细胞(iPSC)(图4C)中BFP表达(其融合至多合一蛋白)。图4D-4F分别为图4A-4C的未转染的对照。图4G显示在用多合一蛋白转染后18天时测量的HeLa和U2OS细胞中所获得的内源基因的稳定沉默。在图4A-4F中,x轴为BFP(融合至多合一蛋白),并且y轴为mCherry。图4H显示在靶向Pcsk9、Npc1、Spcs1和Cd81时,在转染14天后通过qPCR在AML12小鼠肝细胞系中检测到基因沉默。
图5提供分别对应于SEQ ID NO:1-15的融合蛋白p76、p90-p102和p112的示意图。
图6A-6E描述多合一蛋白变异体的基因沉默活性。图6A-6B显示转染至HEK293T细胞中的SEQ ID NOS:1-15的融合蛋白转染后18天的基因沉默结果,以用于靶向沉默CLTA基因。dCas9-KRAB和dCas9-Dnmt3A-Dnmt3L设计显示出长期沉默的短暂和较低效率。图6C-6D提供SEQ ID NO:1(p76)和SEQ ID NO:15(p112)以沉默HIST2H2BE(H2B)内源基因(图6C)和在HEK293T细胞中稳定表达的合成Snrpn-GFP报告基因(图6D)的比较。图6E提供了在50天的时程中关闭HIST2H2BE(H2B)基因的p76和p112蛋白表达(虚线)的图。通过流式细胞术检测BFP来测量蛋白水平,所述BFP与多合一蛋白共表达。
图7A-7B提供多合一蛋白变异体的蛋白质印迹(Western blot)。图7A为使用针对化脓性链球菌Cas9的抗体对多合一蛋白变异体p76和p90-p102的蛋白质印迹分析。顶部条带代表全长蛋白,较小尺寸的条带代表多合一蛋白的蛋白水解。图7B为多合一蛋白变异体的蛋白质印迹分析,以检测从融合蛋白上裂解的游离Dnmt3A。
图8A-8E描述以确定最佳sgRNA的混合筛选。图8A为以确定引起长期基因沉默的最佳sgRNA的混合筛选的示意图。图8B-8E为转染后四周经历基因沉默的细胞百分比的流式细胞术直方图。使用四个HEK293T细胞系,每个细胞系具有带有GFP标签的不同基因,其包括CLTA(图8B)、VIM(图8C)、HIST2H2BE(H2B)(图8D)和RAB11A(图8E)。
图9A-9D为跨越靶向基因的转录起始位点的sgRNA功能图,所述靶向基因包括CLTA(图9A)、H2B(图9B)、RAB11(图9C)和VIM(图9D)。转录起始位点(TSS)和CpG岛标注于每个图上方。每个点代表一个sgRNA,并且将其在长期基因沉默中的功效绘制为sgRNA丰度的log2倍变化。根据微球菌核酸酶(MNase)信号绘制核小体的占有率(底部图)。
图10A-10E描述用于长期基因沉默的功能性sgRNA。图10A为在HEK293T细胞中进行混合筛选以确定多合一蛋白的最佳sgRNA靶向位置的工作流程,所述工作流程改编自K562细胞中先前的蓖麻毒素平铺式筛选以确定dCas9-KRAB的最佳sgRNA(Gilbert,Horlbeck等人,《细胞》2014年)。图10B-10E为代表图,其显示在K562细胞中现有的dCas9-KRAB/CRISPRi数据集的四个基因(所述基因包括ARL1(图10B)、EIF6(图10C)、SMC3(图10D)、HEATR1(图10E))的生长表型(Gilbert,Horlbeck等人,2014年)和多合一蛋白(底部图)。每个点代表一个sgRNA。对于每个基因显示TSS和标注的CpG岛。
图11A-11B提供VPS53(图11A)和VPS54(图11B)的生长表型和核小体定位(来自微球菌核酸酶信号)的比较,显示了功能性sgRNA在核小体耗尽区域的位置。
图12A-12C显示通过mRNA表达递送多合一蛋白。图12A显示两种多合一变异体(p102和p112)的体外转录,显示了每种设计的全长合成。图12B提供流式细胞术图,其显示了mRNA转染至HEK293T细胞中一天后p102和p112的表达。图12C提供转染表达p102和p112多合一变异体的mRNA后,HEK293T细胞中CLTA内源基因沉默的时程。
图13A-13G描述通过多西环素诱导的多合一蛋白的受控表达。图13A提供流式细胞术图,其显示了通过在多西环素诱导型启动子下稳定地编码多合一蛋白的K562细胞中添加多西环素的多合一蛋白的诱导表达。虚线代表未施用多西环素的基线中值BFP荧光。图13B提供在多西环素处理之前和之后细胞的蛋白质印迹以检测多合一蛋白的表达。图13C-13F为多西环素处理后K562细胞14天的CD81(图13C-13D)和CD151(图13E-13F)的基因敲低的流式细胞术图。图13G显示多西环素处理或未经多西环素处理后14天的CD81和CD151基因敲低的定量。
具体实施方式
本文所描述的技术尤其允许哺乳动物细胞中基因的永久沉默而不会在宿主基因组中产生双链DNA断裂。在各实施例中,中心组分为由融合至Dnmt3A、Dnmt3L和KRAB结构域的催化惰性Cas9(dCas9)组成的单个多肽链(本文称为“多合一蛋白”)。本文所提供的这种融合蛋白可以使用单个引导RNA(sgRNA)导向至哺乳动物基因组中的特定位点,并且可以将DNA甲基化和/或向所述位点添加抑制性染色质标记。在各实施例中,结果为在后续细胞分裂中可遗传的基因沉默。在各实施例中,本文所提供的融合蛋白(和sgRNA)仅短暂表达,绕过使用病毒递送方法来诱导永久沉默。
在各实施例中,本文所提供的融合蛋白通过表观基因组编辑而非基因组编辑来提供对内源基因表达的稳健长期或永久沉默。基因的等位基因可以被靶向或单个病原体等位基因可以被选择性靶向。在各实施例中,本文所提供的融合蛋白的优势在于表观遗传编辑是可逆的并且因此本质上比基因组编辑更安全。因此,在各实施例中,本文所提供的融合蛋白可用于预防应用。例如,基因沉默可以实现对感染/生物毒素的急性保护,且接着在感染或中毒风险后不存在逆转。因此,在各实施例中,本文所提供的融合蛋白可用于通过与长期器官功能或内稳态所需的蛋白相互作用而进入细胞的病毒或毒素。在各实施例中,本文所提供的融合蛋白可用于基于基因组编辑的治疗剂。
在各实施例中,哺乳动物细胞中的永久基因沉默可以通过两种组分完成:由融合至三个表观遗传调节剂的dCas9组成的单个多肽链,和将蛋白导向至宿主基因组中的特定位点的单个引导RNA。在各实施例中,组分仅在宿主细胞中短暂表达,从而减少毒性和脱靶事件。
在各实施例中,本文所提供的融合蛋白在宿主细胞中不诱导DNA断裂以用于永久基因沉默。在各实施例中,添加至所关注的基因组位点的表观遗传标记为可逆的,因此允许去除可能发生的任何脱靶事件。
定义
虽然在本文中显示和描述了本发明的各种实施例和方面,但对于本领域的技术人员将显而易见的是,仅借助于实例提供此类实施例和方面。在不脱离本发明的情况下,本领域的技术人员现将意识到许多变化、改变和替代。应理解,本文所描述的本发明的实施例的各个替代方案都可以在实践本发明时采用。
本文所使用的章节标题仅出于组织目的并且不应理解为限制所述主题。本申请中所引用的所有文献或文献的部分,包括但不限于专利、专利申请、文章、书籍、手册以及论文,在此出于任何目的明确地以全文引用的方式并入本文中。
除非另有定义,否则本文所使用的所有技术和科学术语都具有本发明所属领域的技术人员通常所理解的意义。以下参考文献为技术人员提供本发明中所使用的许多术语的一般定义:Singleton等人,《微生物学和分子生物学词典(Dictionary of Microbiologyand Molecular Biology)》(第2版,1994年);《剑桥科学和技术词典(The CambridgeDictionary of Science and Technology)》(Walker编,1988);《遗传学词汇表(TheGlossary of Genetics)》,第5版,R.Rieger等人(编),斯普林格出版社(Springer Verlag)(1991年);和Hale&Marham,《哈珀柯林斯生物学词典(The Harper Collins Dictionary ofBiology)》(1991年)。如本文所使用的,除非另有说明,否则以下术语具有赋予其的含义。
在本公开和所附权利要求书中使用单数不定冠词或定冠词(例如,“一个/种(a)”、“一个/种(an)”、“所述(the)”等)遵循专利中含义为“至少一个/种(at least one)”的传统方法,除非在从上下文中可以明显看出,所述术语在所述特定实例中旨在专门表示一个且仅一个。同样地,术语“包含”为开放式的,不排除额外的项目、特征、组分等。除非另有指出,否则本文中所标识的参考文献通过全文引用的方式明确地并入本文中。
术语“包含”、“包括”和“具有”及其派生词在本文中可作为全面的开放式术语互换使用。例如,“包含”、“包括”或“具有”的使用意味着包含、具有或包括的任何元素并不是涵盖动词的从句主语所含有的唯一元素。
“核酸”是指核苷酸(例如,脱氧核糖核苷酸或核糖核苷酸)和其呈单链、双链或多链形式的聚合物或其互补物。术语“多核苷酸”、“寡核苷酸”、“寡核”或等等在通常和惯用意义上是指核苷酸的直链序列。术语“核苷酸”在通常和惯用意义上是指多核苷酸的单个单元,即单体。核苷酸可以为核糖核苷酸、脱氧核糖核苷酸或其修饰版本。本文所设想的多核苷酸的实例包含单链和双链DNA、单链和双链RNA以及具有单链和双链DNA和RNA的混合物的杂交分子。本文所设想的核酸(例如多核苷酸)的实例,包括但不限于任何类型的RNA,例如mRNA、siRNA、miRNA、sgRNA和引导RNA,以及任何类型的DNA、基因组DNA、质粒DNA和微环DNA以及其任何片段。在各方面中,核酸为信使RNA。在各方面中,信使RNA为信使核糖核蛋白(RNP)。在多核苷酸的情况下,术语“双链体”在通常和惯用意义上是指双链型。核酸可以为直链的或支链的。例如,核酸可以为核苷酸的直链或核酸可以为支链的,例如使得核酸包括核苷酸的一个或多个臂或分支。任选地,支链核酸重复分支以形成更高级的结构,如树枝状大分子等。
如本文可以使用的术语“核酸”、“核酸分子”、“核酸低聚物”、“寡核苷酸”、“核酸序列”、“核酸片段”和“多核苷酸”可互换地使用,并且旨在包括但不限于共价地连接在一起的可以具有各种长度的核苷酸的聚合形式,所述核苷酸为脱氧核糖核苷酸或核糖核苷酸或其类似物、衍生物或其修饰。不同的多核苷酸可以具有不同的三维结构,并且可以执行各种已知或未知的功能。多核苷酸的非限制性实例包括基因、基因片段、外显子、内含子、基因间DNA(包括但不限于异色DNA)、信使RNA(mRNA)、转移RNA、核糖体RNA、核酶、cDNA、重组多核苷酸、支链多核苷酸、质粒、载体、序列的分离DNA、序列的分离RNA、核酸探针和引物。可用于本公开的方法中的多核苷酸可以包括天然核酸序列及其变异体、人工核酸序列或此类序列的组合。
多核苷酸通常由四种核苷酸碱基的特定序列组成:腺嘌呤(A);胞嘧啶(C);鸟嘌呤(G);和胸腺嘧啶(T)(当多核苷酸为RNA时,尿嘧啶(U)为胸腺嘧啶(T))。因此,术语“多核苷酸序列”为多核苷酸分子的字母表示;替代地,所述术语可以应用于多核苷酸分子本身。此字母表示可以输入到具有中央处理单元的计算机中的数据库中,并且用于生物信息学应用,如功能性基因组学和同源性搜索。多核苷酸可以任选地包含一种或多种非标准核苷酸、一种或多种核苷酸类似物和/或修饰核苷酸。
核酸,包括例如具有硫代磷酸酯主链的核酸,可以包括一个或多个反应性部分。如本文所使用的,术语反应性部分包括能够通过共价、非共价或其它相互作用与另一分子(例如核酸或多肽)反应的任何基团。举例来说,核酸可以包括通过共价、非共价或其它相互作用与蛋白或多肽上的氨基酸反应的氨基酸反应性部分。
所述术语还涵盖含有已知核苷酸类似物或经修饰的主链残基或键的核酸,所述核酸为合成的、天然存在的和非天然存在的,其具有与参考核酸类似的结合特性,并且以与参考核苷酸类似的方式代谢。此类类似物的实例包括但不限于磷酸二酯衍生物,所述磷酸二酯衍生物包括例如氨基磷酸酯、二氨基磷酸酯、硫代磷酸酯(也称为具有取代磷酸酯中氧的双键硫的硫代磷酸酯)、二硫代磷酸酯、膦酰羧酸、膦酰羧酸酯、膦酰基乙酸、膦酰甲酸、甲基膦酸酯、硼膦酸酯或O-甲基亚磷酰胺键(参见Eckstein,《寡核苷酸以及类似物:实用方法(Oligonucleotides and Analogues:A Practical Approach)》,牛津大学出版社(OxfordUniversity Press)),以及如在5-甲基胞苷或假尿苷中对核苷酸碱基的修饰;以及肽核酸骨架和键。其它类似核酸包括那些具有正离子骨架的核酸;非离子骨架、经修饰的糖和非核糖骨架(例如本领域已知的二氨基磷酸酯吗啉代寡核苷酸或锁核酸(LNA)),包含美国专利第5,235,033号和第5,034,506号以及《ASC研讨会系列580(ASC Symposium Series 580)》,《反义研究中的碳水化合物修饰(Carbohydrate Modifications in AntisenseResearch)》,Sanghui和Cook编辑的第6章和第7章中描述的那些。含有一个或多个碳环糖的核酸也包括于核酸的一个定义内。可以出于多种原因进行核糖-磷酸骨架的修饰,例如,增加此类分子在生理环境中,或作为生物芯片上的探针的稳定性和半衰期。可以制备天然存在的核酸和类似物的混合物;替代地,可以制备不同核酸类似物的混合物,以及天然存在的核酸和类似物的混合物。在各方面中,DNA中的核苷酸间键为磷酸二酯、磷酸二酯衍生物或两者的组合。
核酸可以包括非特异性序列。如本文中所使用的,术语“非特异性序列”是指含有未被设计成与任何其它核酸序列互补或与任何其它核酸序列仅部分互补的一系列残基的核酸序列。举例来说,非特异性核酸序列为当在与细胞或生物体接触时不充当抑制性核酸的核酸残基的序列。
术语“互补的”或“互补性”是指核酸通过传统的Watson-Crick或其它非传统类型与另一核酸序列形成氢键的能力。例如,序列A-G-T与序列T-C-A互补。互补性百分比指示可以与第二核酸序列形成氢键(例如,Watson-Crick碱基配对)的核酸分子中的残基百分比(例如,10分之5、6、7、8、9、10分别为50%、60%、70%、80%、90%以及100%互补)。“完全互补”是指核酸序列的所有连续残基将与第二核酸序列中相同数目的连续残基氢键键合。如本文所使用,“基本上互补”是指在8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、30、35、40、45、50或更多个核苷酸的区域内,或指在严格条件(即严格杂交条件)下杂交的两个核酸至少有60%、65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%的互补性程度。
短语“严格杂交条件”是指探针将与其靶序列杂交的条件,通常在核酸的复杂混合物中而不是与其它序列杂交。严格条件依赖于序列,并且在不同情况下会有所不同。较长序列在较高温度下特异性地杂交。有关核酸杂交的详尽引导可见于Tijssen,《生物化学和分子生物学技术--用核酸探针杂交(Laboratory Techniques in Biochemistry andMolecular Biology--Hybridization with Nucleic Probes)》,“杂交原理和核酸分析策略综述(Overview of principles of hybridization and the strategy of nucleicacid assays)”(1993年)中。一般来说,选择严格条件比所定义的离子强度pH下的特定序列的热熔点(T
如果核酸所编码的多肽实质上相同,那么在严格条件下并不彼此杂交的所述核酸仍基本上相同。这在例如使用由遗传密码准许的最大密码简并形成核酸拷贝时发生。在此类情况下,核酸通常在适度严格杂交条件下杂交。例示性“适度严格杂交条件”包括在40%甲酰胺、1M NaCl、1% SDS的缓冲液中在37℃下杂交,且在1×SSC中在45℃下洗涤。正杂交为至少两倍背景。普通技术人员将容易认识到替代性杂交和洗涤条件可以用于提供类似严格度的条件。用于确定杂交参数的其它指导原则提供于许多参考文献中,例如《最新分子生物学实验方法汇编(Current Protocols in Molecular Biology)》版,Ausubel等人,同上。
术语“基因”是指设计产生蛋白的DNA片段;其包括编码区之前和之后的区域(前导和尾部)以及各个编码片段(外显子)之间的插入序列(内含子)。前导、尾部和内含子包括基因转录和转译过程中必需的调节元件。此外,“蛋白基因产物”为由特定基因表达的蛋白。
如本文所使用的关于基因的词“表达”或“表达的”是指所述基因的转录和/或转译产物。细胞中DNA分子的表达水平可以根据细胞内存在的相应mRNA的量或细胞产生的DNA编码的蛋白的量来确定。非编码核酸分子(例如sgRNA)的表达水平可以通过本领域众所周知的标准PCR或Northern印迹方法来检测。参见Sambrook等人,1989年《分子克隆:实验指南(Molecular Cloning:A Laboratory Manual)》(18.1-18.88)。
如本文所提供的术语“转录调节序列”是指能够增加或减少生物体内特定基因的转录(例如表达)的DNA片段。转录调节序列的非限制性实例包括启动子、增强子和沉默子。
术语“转录开始位点”和“转录起始位点”可互换地使用以在本文中指基因序列(例如DNA序列)的5'端,其中RNA聚合酶(例如DNA引导的RNA聚合酶)开始合成RNA转录物。转录起始位点可以为转录的DNA序列的第一个核苷酸,其中RNA聚合酶开始合成RNA转录物。技术人员可以通过常规实验和分析确定转录起始位点,例如通过进行失控转录分析或根据FANTOM5数据库的定义。
本文中所使用的术语“启动子”是指启动特定基因转录的DNA区域。启动子通常位于DNA上的基因转录起始位点附近、基因的上游和相同链上(即,有义链上5'处)。启动子的长度可以为约100至约1000个碱基对。
如本文所使用的术语“增强子”是指可以由蛋白(例如转录因子)结合以增加基因转录发生的可能性的DNA区域。增强子的长度可以为约50至约1500个碱基对。增强子可以位于其调节的转录起始位点的下游或上游,并且可以距离转录起始位点数百个碱基对。
如本文所使用的术语“沉默子”是指能够结合被称为阻遏物的转录调节因子的DNA序列,从而对基因的转录产生负面影响。沉默子DNA序列可以在整个DNA的许多不同位置处发现,包括但不限于靶基因上游,其作用是抑制基因的转录(例如沉默基因表达)。
如本文所提供的“引导RNA”或“gRNA”是指与靶多核苷酸序列具有足够互补性以与靶序列杂交并且将CRISPR复合物直接序列特异性结合至靶序列的任何多核苷酸序列。在各方面中,当使用合适的比对算法最佳比对时,引导序列与其对应的靶序列之间的互补性程度为约或大于约50%、60%、75%、80%、85%、90%、95%、97.5%、99%或更高。
在各实施例中,多核苷酸(例如gRNA)为单链核糖核酸。在各方面中,多核苷酸(例如gRNA)的长度为10、20、30、40、50、60、70、80、90、100或更多个核酸残基。在各方面中,多核苷酸(例如gRNA)的长度为10至30个核酸残基。在各方面中,多核苷酸(例如gRNA)的长度为20个核酸残基。在各方面中,多核苷酸的长度(例如gRNA)可以为至少5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27,28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52,53,54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69,70,71,72,73,74,75,76,77,78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100或更多个核酸残基或糖残基的长度。在各方面中,多核苷酸(例如gRNA)的长度为5至50、10至50、15至50、20至50、25至50、30至50、35至50、40至50、45至50、5至75、10至75、15至75、20至75、25至75、30至75、35至75、40至75、45至75、50至75、55至75、60至75、65至75、70至75、5至100、10至100,15至100,20至100,25至100,30至100,35至100,40至100,45至100,50至100,55至100,60至100,65至100,70至100,75至100、80至100、85至100、90至100、95至100或更多个残基。在各方面中,多核苷酸(例如gRNA)的长度为10至15、10至20、10至30、10至40或10至50个残基。
术语“氨基酸”是指天然存在和合成的氨基酸、以及以与天然存在的氨基酸类似的一种方式起作用的氨基酸类似物和氨基酸模拟物。天然存在的氨基酸是由遗传密码编码的氨基酸、以及后来经修饰的那些氨基酸,例如羟基脯氨酸、γ-羧基谷氨酸以及O-磷酸丝氨酸。氨基酸类似物是指具有与天然存在的氨基酸相同的基本化学结构的化合物,即,结合至氢、羧基、氨基和R基的α碳,例如高丝氨酸、正亮氨酸、甲硫氨酸亚砜、甲硫氨酸甲基锍。此类类似物具有经修饰的R基(例如正亮氨酸)或经修饰的肽主链,但保留与天然存在的氨基酸相同的基本化学结构。氨基酸模拟物是指结构不同于氨基酸的一般化学结构但以与天然存在的氨基酸类似的方式起作用的化合物。术语“非天然存在的氨基酸”和“非天然氨基酸”是指自然界中未发现的氨基酸类似物、合成氨基酸和氨基酸模拟物。
氨基酸在本文通过其通常已知的三字母符号或通过IUPAC-IUB生物化学命名委员会(IUPAC-IUB Biochemical Nomenclature Commission)推荐的单字母符号来表示。同样地,核苷酸可以由其通常接受的单字母密码来提及。
术语“多肽”、“肽”和“蛋白”在本文中可互换地使用以指代氨基酸残基的聚合物,其中所述聚合物可以在各方面中结合至不由氨基酸组成的部分。这些术语适用于氨基酸聚合物,其中一个或多个氨基酸残基为相应天然存在的氨基酸的人造化学模拟物,并且适用于天然存在的氨基酸聚合物和非天然存在的氨基酸聚合物。“融合蛋白”是指编码以重组方式表达为单个部分的两个或更多个单独蛋白序列的嵌合蛋白。
“经保守修饰的变异体”适用于氨基酸序列和核酸序列两者。关于特定核酸序列,“经保守修饰的变异体”是指编码相同或基本上相同的氨基酸序列的那些核酸。由于遗传密码的简并,多个核酸序列将编码任何给定的蛋白。例如,密码子GCA、GCC、GCG和GCU都编码氨基酸丙氨酸。因此,在由密码子指定丙氨酸的每个位置,所述密码子可以变为任何所述的对应密码子而不更改所编码的多肽。此类核酸变异为“沉默变异”,其为保守修饰的变异的一个物种。本文中的编码多肽的每个核酸序列还描述核酸的每个可能沉默变异。技术人员将认识到,核酸中的每个密码子(除通常是甲硫氨酸的唯一密码子的AUG和通常是色氨酸的唯一密码子TGG外)可以经修饰以得到功能相同的分子。因此,编码多肽的核酸的每种沉默变异隐含在每个所描述的序列中。
关于氨基酸序列,技术人员将认识到改变、添加或缺失所编码的序列中的单个氨基酸或小百分比的氨基酸的核酸、肽、多肽或蛋白序列的个别取代、缺失或添加为“经保守修饰的变异体”,其中改变会引起氨基酸经化学上类似的氨基酸取代。提供功能上类似的氨基酸的保守取代表在本领域中众所周知。此类经保守修饰的变异体除了本公开的多晶型变异体、种间同源物和等位基因并且不排除所述多晶型变异体、种间同源物和等位基因。以下八组各自含有彼此保守取代的氨基酸:(1)丙氨酸(A)、甘氨酸(G);(2)天冬氨酸(D)、谷氨酸(E);(3)天冬酰胺(N)、谷氨酰胺(Q);(4)精氨酸(R)、赖氨酸(K);(5)异亮氨酸(I)、亮氨酸(L)、甲硫氨酸(M)、缬氨酸(V);(6)苯丙氨酸(F)、酪氨酸(Y)、色氨酸(W);(7)丝氨酸(S)、苏氨酸(T);以及(8)半胱氨酸(C)、甲硫氨酸(M)(参见例如Creighton,《蛋白(Proteins)》(1984年))。
“序列一致性百分比”是通过在比较窗口之上比较两个最佳比对的序列确定的,其中比较窗口中的多核苷酸序列或多肽序列的部分可以包括如相比于用于两个序列的最佳比对的参考序列(其不包括添加或缺失)的添加或缺失(即间隙)。百分比通过以下计算:确定两个序列中出现相同核酸碱基或氨基酸残基的位置数以得到匹配的位置数,将匹配的位置数除以比较窗口中的总位置数并且将结果乘以100以得到序列一致性百分比。
在两个或更多个核酸或多肽序列的情况下,术语“相同”或百分比“一致性”是指以如下所述的默认参数来使用BLAST或BLAST 2.0序列比较算法或通过人工比对和目测检查(参见例如,NCBI网站http://www.ncbi.nlm.nih.gov/BLAST/或等等)测量的,相同的或具有指定百分比的相同氨基酸残基或核苷酸(在比较窗口或指定区域上针对最大对应性进行比较和比对时,在指定区域内具有约60%的一致性,优选65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更高的一致性)的两个或更多个序列或子序列。此类序列然后被称为“基本上相同”。此定义还涉及或可以应用于测试序列的补充。定义还包含具有缺失和/或添加的序列,以及具有取代的序列。如下文所述,优选算法可以解释间隙等。优选地,在长度为至少约25个氨基酸或核苷酸的区域上,或更优选地在长度为50-100个氨基酸或核苷酸的区域上存在一致性。
氨基酸或核苷酸碱基的“位置”由编号表示,所述编号基于其相对于N端(或5'端)的位置顺序地标识参考序列中的每个氨基酸(或核苷酸碱基)。由于在确定最佳比对时必须考虑的缺失、插入、截短、融合等,因此,通常测试序列中的通过仅从N端进行计数而确定的氨基酸残基的编号不一定与其在参考序列中的对应位置的编号相同。例如,在变异体相对于比对的参考序列具有缺失的情况下,变异体中将不存在与参考序列中的缺失位点处的位置相对应的氨基酸。在比对的参考序列中存在插入的情况下,所述插入将不对应于参考序列中的经编号的氨基酸位置。在截短或融合的情况下,参考序列或比对的序列中可能存在不对应于对应序列中的任何氨基酸的氨基酸段。
当在对给定氨基酸或多核苷酸序列进行编号的上下文中使用时,术语“相对于...进行编号”或“对应于...进行编号”是指在将给定氨基酸或多核苷酸序列与参考序列进行比较时,对指定参考序列的残基进行编号。
对于本文所述的特定蛋白(例如KRAB、dCas9、Dnmt3A、Dnmt3L),命名的蛋白包括所述蛋白的任何天然形式或维持所述蛋白活性(例如,与天然蛋白相比,具有至少50%、80%、90%、95%、96%、97%、98%、99%或100%的活性)的变异体或同源物。在各方面中,与天然存在的形式相比,变异体或同源物在整个序列或一部分序列(例如50、100、150或200个连续氨基酸部分)上具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%、99%或100%的氨基酸序列一致性。在各方面中,蛋白为通过其NCBI序列参考所鉴别的蛋白。在各方面中,蛋白为通过其NCBI序列参考或其功能片段或同源物鉴别的蛋白。
如本文所提供的“克鲁珀相关盒结构域”或“KRAB结构域”是指存在于约400个基于人锌指蛋白的转录因子中的转录抑制结构域的类别。KRAB结构域通常包括约45至约75个氨基酸残基。关于KRAB结构域的描述,包括其的功能和用途,可以在例如Ecco,G.,Imbeault,M.,Trono,D.,KRAB锌指蛋白(KRAB zinc finger proteins),《发展(Development)》144,2017年;Lambert等人.人类转录因子(The human transcription factors),《细胞》172,2018年;Gilbert等人,《细胞》(2013年);和Gilbert等人,《细胞》(2014年),其全部以全文引用的方式并入本文中。在各方面中,KRAB结构域为Kox 1的KRAB结构域。在各方面中,KRAB结构域包括SEQ ID NO:16所示的序列。在各方面中,KRAB结构域为SEQ ID NO:16的序列。在各方面中,KRAB结构域包括与SEQ ID NO:16具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,KRAB结构域包括与SEQ ID NO:16具有至少75%序列一致性的氨基酸序列。在各方面中,KRAB结构域包括与SEQ ID NO:16具有至少80%序列一致性的氨基酸序列。在各方面中,KRAB结构域包括与SEQ ID NO:16具有至少85%序列一致性的氨基酸序列。在各方面中,KRAB结构域包括与SEQ ID NO:16具有至少90%序列一致性的氨基酸序列。在各方面中,KRAB结构域包括与SEQ ID NO:16具有至少95%序列一致性的氨基酸序列。
如本文所提供的术语“DNA甲基转移酶”是指催化甲基转移至DNA的酶。DNA甲基转移酶的非限制性实例包括Dnmt1、Dnmt3A、Dnmt3B和Dnmt3L。在各方面中,DNA甲基转移酶为细菌胞嘧啶甲基转移酶和/或细菌非胞嘧啶甲基转移酶。取决于特定的DNA甲基转移酶,DNA的不同区域被甲基化。例如,Dnmt3A通常靶向用于甲基化的CpG二核苷酸。通过DNA甲基化,DNA甲基转移酶可以在不更改DNA序列的情况下修饰DNA片段的活性(例如基因表达)。在各方面中,DNA甲基化引起基因转录的抑制和/或甲基化敏感性转录因子或CTCF的调节。如本文所述,融合蛋白可以包括一个或多个(例如两个)DNA甲基转移酶。当DNA甲基转移酶作为融合蛋白的一部分包括在内时,DNA甲基转移酶可以被称为“DNA甲基转移酶结构域”。在各方面中,DNA甲基转移酶结构域包括一个或多个DNA甲基转移酶。在各方面中,DNA甲基转移酶结构域包括两个DNA甲基转移酶。在各方面中,DNA甲基转移酶结构域为Dnmt3A。在各方面中,DNA甲基转移酶结构域具有SEQ ID NO:26的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少75%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少80%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少85%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少90%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少95%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域为Dnmt3L。在各方面中,DNA甲基转移酶结构域具有SEQ ID NO:28的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:28具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:28具有至少75%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:28具有至少80%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:28具有至少85%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:28具有至少90%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:28具有至少95%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域包括Dnmt3A和Dnmt3L。在各方面中,DNA甲基转移酶结构域具有SEQ ID NO:33的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:33具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:33具有至少75%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:33具有至少80%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:33具有至少85%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ IDNO:33具有至少90%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:33具有至少95%序列一致性的氨基酸序列。关于Dnmt3A-3L结构域结构和用途的描述可以在例如在Siddique等人,通过使用设计的具有提高的DNA甲基化活性的Dnmt3a-Dnmt3L单链融合蛋白,在人细胞中对VEGF-A进行靶向甲基化和基因沉默(Targetedmethylation and gene silencing of VEGF-A in human cells by using a designedDnmt3a-Dnmt3L single-chain fusion protein with increased DNA methylationactivity),《分子生物学杂志(J.Mol.Biol)》425,2013年和Stepper等人,嵌合dCas9-Dnmt3a-Dnmt3L甲基转移酶高效靶向DNA甲基化(Efficient targeted DNA methylationwith chimeric dCas9-Dnmt3a-Dnmt3L methyltransferase),《核酸研究(Nucleic AcidsRes)》45,2017年的文章中找到,其以全文引用的方式并且出于所有目的并入本文中。
本文所提及的“Dnmt3A”、“Dnmt3a”、“DNA(胞嘧啶-5)-甲基转移酶3A”或“DNA甲基转移酶3a”蛋白包括Dnmt3A酶的任何重组或天然存在形式或其保持Dnmt3A酶活性(例如与Dnmt3A相比,至少在50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的活性内)的变异体或同源物。在各方面中,与天然存在的Dnmt3A蛋白相比,变异体或同源物在整个序列或一部分序列(例如50、100、150或200个连续氨基酸部分)上具有至少90%、95%、96%、97%、98%、99%或100%的氨基酸序列一致性。在各方面中,Dnmt3A蛋白与由UniProt参考编号Q9Y6K1鉴别的蛋白或与其具有相当大的一致性的变异体或同源物实质上一致。在各方面中,Dnmt3A多肽由通过NCBI参考序列寄存编号NM_022552鉴别的核酸序列、其同源物或功能片段编码。在各方面中,Dnmt3A包括SEQ ID NO:26所示的序列。在各方面中,Dnmt3A为SEQ ID NO:26所示的序列。在各方面中,Dnmt3A具有与SEQ ID NO:26具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少75%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少80%序列一致性的氨基酸序列。在各方面中,DNA甲基转移酶结构域具有与SEQ ID NO:26具有至少85%序列一致性的氨基酸序列。在各方面中,Dnmt3A具有与SEQ ID NO:26具有至少90%序列一致性的氨基酸序列。在各方面中,Dnmt3A具有与SEQ ID NO:26具有至少95%序列一致性的氨基酸序列。
本文所提及的“Dnmt3L”、“DNA(胞嘧啶-5)-甲基转移酶3L”或“DNA甲基转移酶3L”蛋白包括Dnmt3L酶的任何重组或天然存在形式或其保持Dnmt3L酶活性(例如与Dnmt3L相比,至少在50%,55%,60%,65%,70%,75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%的活性内)的变异体或同源物。在各方面中,与天然存在的Dnmt3L蛋白相比,变异体或同源物在整个序列或一部分序列(例如50、100、150或200个连续氨基酸部分)上具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的氨基酸序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9CWR8鉴别的蛋白或与其具有相当大的一致性的变异体或同源物实质上一致。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9CWR8鉴别的蛋白相同。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9CWR8鉴别的蛋白的氨基酸序列具有至少75%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9CWR8鉴别的蛋白的氨基酸序列具有至少80%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9CWR8鉴别的蛋白的氨基酸序列具有至少85%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9CWR8鉴别的蛋白的氨基酸序列具有至少95%的序列一致性。
在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白或与其具有相当大的一致性的变异体或同源物实质上一致。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白相同。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少50%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白具有至少55%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少60%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少65%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少70%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少75%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少80%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少85%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少90%的序列一致性。在各方面中,Dnmt3L蛋白与由UniProt参考编号Q9UJW鉴别的蛋白的氨基酸序列具有至少95%的序列一致性。在各方面中,Dnmt3L多肽由通过NCBI参考序列寄存编号NM_001081695鉴别的核酸序列或其同源物或功能片段编码。在各方面中,Dnmt3L包括SEQ ID NO:28所示的序列。在各方面中,Dnmt3L为SEQ ID NO:28所示的序列。在各方面中,Dnmt3L具有SEQ ID NO:28具有至少50%,55%,60%,65%,70%,75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQID NO:28具有至少50%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少55%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少60%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少65%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少97%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少75%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少80%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少85%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少90%序列一致性的氨基酸序列。在各方面中,Dnmt3L具有与SEQ ID NO:28具有至少95%序列一致性的氨基酸序列。
术语“RNA引导的DNA核酸内切酶”等在通常和惯常意义上是指在DNA多核苷酸链中裂解磷酸二酯键的酶,其中磷酸二酯键的识别由单独的RNA序列(例如单个引导RNA)促进。
术语“II类CRISPR核酸内切酶”是指具有与Cas9相似的核酸内切酶活性并且参与II类CRISPR系统的核酸内切酶。II类CRISPR系统的实例为化脓性链球菌SF370的II型CRISPR基因座,其含有四个基因Cas9、Cas1、Cas2和Csn1的簇,以及两个非编码RNA元件、tracrRNA和重复序列(直接重复序列)的特性阵列,所述重复序列通过非重复序列的短片段(间隔子,各自约30bp)间隔开。Cpf1酶属于假定的V型CRISPR-Cas系统。II型和V型系统两者均包括于II类CRISPR-Cas系统中。
“核定位序列”或“核定位信号”或“NLS”为将蛋白导向至细胞核的肽。在各方面中,NLS包括五个碱性带正电的氨基酸。NLS可以位于肽链上的任何位置。在各方面中,NLS为衍生自SV40的NLS。在各方面中,NLS包括SEQ ID NO:25所示的序列。在各方面中,NLS为SEQ IDNO:25所示的序列。在各方面中,NLS具有与SEQ ID NO:25至少具有50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少75%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少80%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少85%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少90%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少95%序列一致性的氨基酸序列。在各方面中,NLS具有SEQ IDNO:25的氨基酸序列。
如本文所用,“细胞”是指进行足以保持或复制其基因组DNA的代谢或其它功能的细胞。细胞可通过本领域中众所周知的方法来鉴定,包括例如存在完整膜、特定染料染色、能够产生后代或在配子的情况下能够与第二配子组合以产生活的后代。细胞可以包括原核细胞和真核细胞。原核细胞包括不限于细菌。真核细胞包括不限于酵母细胞和衍生自植物和动物的细胞,例如哺乳动物细胞、昆虫(例如夜蛾)细胞和人细胞。细胞在其为天然地非粘附性的或已经例如通过胰蛋白酶消化进行处理以不粘附于表面时,可以是适用的。
如本文所使用,术语“载体”是指能够转运与其连接的另一核酸的核酸分子。载体的一种类型为“质粒”,其是指直链或环状双链DNA环,其中可以连接额外DNA区段。载体的另一种类型为病毒载体,其中额外的DNA区段可以连接至病毒基因组中。某些载体能够在引入它们的宿主细胞(例如,具有细菌复制起点的细菌载体和游离型哺乳动物载体)中自主复制。其它载体(例如,非游离型哺乳动物载体)在引入宿主细胞后整合至宿主细胞的基因组中,从而与宿主基因组一起复制。此外,某些载体能够指导与其操作性地连接的基因的表达。此类载体在本文中称为“表达载体”。通常,在重组DNA技术中有效用的表达载体通常呈质粒的形式。在本说明书中,“质粒”和“载体”可以互换地使用,因为质粒为最常用形式的载体。然而,本发明旨在包括提供等效功能的此类其它形式的表达载体,如病毒载体(例如复制缺陷型反转录病毒、腺病毒以及腺相关病毒)。另外,一些病毒载体能够特异性地或非特异性地靶向的特定细胞类型。无复制能力的病毒载体或复制缺陷型病毒载体是指能够感染其靶细胞并传递其病毒有效载荷,但随后不能继续导致细胞裂解和死亡的典型裂解途径的病毒载体。
术语“转染”、“转导”、“转染”或“转导”可以互换使用,并且被定义为将核酸分子和/或蛋白引入细胞的过程。可以使用非病毒或基于病毒的方法将核酸引入细胞。核酸分子可为编码完整蛋白或其功能部分的序列。通常,核酸载体,其包含蛋白表达所必需的元件(例如启动子、转录起始位点等)。非病毒转染方法包括不使用病毒DNA或病毒颗粒作为递送系统将核酸分子引入至细胞的任何适当方法。例示性的非病毒转染方法包括编码融合蛋白的核酸的纳米颗粒封装(例如脂质纳米颗粒、金纳米颗粒等)、磷酸钙转染、脂质体转染、核转染、声穿孔、通过热休克转染、磁转染和电穿孔。对于基于病毒的方法,任何有用的病毒载体均可用于本文所述的方法。病毒载体的实例包括但不限于反转录病毒、腺病毒、慢病毒和腺相关病毒载体。在各方面中,按照本领域众所周知的标准程序,使用反转录病毒载体将核酸分子引入至细胞中。术语“转染”或“转导”还指将蛋白从外部环境引入至细胞中。通常,蛋白的转导或转染依赖于能够穿过细胞膜的肽或蛋白与所关注的蛋白的附接。参见,例如,Ford等人.(2001年)《基因疗法(Gene Therapy)》8:1-4和Prochiantz(2007年)《自然方法(Nat.Methods)》4:119-20。
如本文所提供的“肽接头”为包括肽部分的接头。在各实施例中,肽接头为二价肽,如在N端和C端连接至化合物的其余部分的氨基酸序列(例如本文所提供的融合蛋白)。肽接头可以为能够裂解(例如P2A可裂解多肽)的肽部分(二价肽部分)。如本文所提供的肽接头还可互换地称为氨基酸接头。在各方面中,肽接头包括1至约80个氨基酸残基。在各方面中,肽接头包括1至约70个氨基酸残基。在各方面中,肽接头包括1至约60个氨基酸残基。在各方面中,肽接头包括1至约50个氨基酸残基。在各方面中,肽接头包括1至约40个氨基酸残基。在各方面中,肽接头包括1至约30个氨基酸残基。在各方面中,肽接头包括1至约25个氨基酸残基。在各方面中,肽接头包括1至约20个氨基酸残基。在各方面中,肽接头包括约2至约20个氨基酸残基。在各方面中,肽接头包括约2至约19个氨基酸残基。在各方面中,肽接头包括约2至约18个氨基酸残基。在各方面中,肽接头包括约2至约17个氨基酸残基。在各方面中,肽接头包括约2至约16个氨基酸残基。在各方面中,肽接头包括约2至约15个氨基酸残基。在各方面中,肽接头包括约2至约14个氨基酸残基。在各方面中,肽接头包括约2至约13个氨基酸残基。在各方面中,肽接头包括约2至约12个氨基酸残基。在各方面中,肽接头包括约2至约11个氨基酸残基。在各方面中,肽接头包括约2至约10个氨基酸残基。在各方面中,肽接头包括约2至约9个氨基酸残基。在各方面中,肽接头包括约2至约8个氨基酸残基。在各方面中,肽接头包括约2至约7个氨基酸残基。在各方面中,肽接头包括约2至约6个氨基酸残基。在各方面中,肽接头包括约2至约5个氨基酸残基。在各方面中,肽接头包括约2至约4个氨基酸残基。在各方面中,肽接头包括约2至约3个氨基酸残基。在各方面中,肽接头包括约3至约19个氨基酸残基。在各方面中,肽接头包括约3至约18个氨基酸残基。在各方面中,肽接头包括约3至约17个氨基酸残基。在各方面中,肽接头包括约3至约16个氨基酸残基。在各方面中,肽接头包括约3至约15个氨基酸残基。在各方面中,肽接头包括约3至约14个氨基酸残基。在各方面中,肽接头包括约3至约13个氨基酸残基。在各方面中,肽接头包括约3至约12个氨基酸残基。在各方面中,肽接头包括约3至约11个氨基酸残基。在各方面中,肽接头包括约3至约10个氨基酸残基。在各方面中,肽接头包括约3至约9个氨基酸残基。在各方面中,肽接头包括约3至约8个氨基酸残基。在各方面中,肽接头包括约3至约7个氨基酸残基。在各方面中,肽接头包括约3至约6个氨基酸残基。在各方面中,肽接头包括约3至约5个氨基酸残基。在各方面中,肽接头包括约3至约4个氨基酸残基。在各方面中,肽接头包括约10至约20个氨基酸残基。在各方面中,肽接头包括约15至约20个氨基酸残基。在各方面中,肽接头包括约2个氨基酸残基。在各方面中,肽接头包括约3个氨基酸残基。在各方面中,肽接头包括约4个氨基酸残基。在各方面中,肽接头包括约5个氨基酸残基。在各方面中,肽接头包括约6个氨基酸残基。在各方面中,肽接头包括约7个氨基酸残基。在各方面中,肽接头包括约8个氨基酸残基。在各方面中,肽接头包括约9个氨基酸残基。在各方面中,肽接头包括约10个氨基酸残基。在各方面中,肽接头包括约11个氨基酸残基。在各方面中,肽接头包括约12个氨基酸残基。在各方面中,肽接头包括约13个氨基酸残基。在各方面中,肽接头包括约14个氨基酸残基。在各方面中,肽接头包括约15个氨基酸残基。在各方面中,肽接头包括约16个氨基酸残基。在各方面中,肽接头包括约17个氨基酸残基。在各方面中,肽接头包括约18个氨基酸残基。在各方面中,肽接头包括约19个氨基酸残基。在各方面中,肽接头包括约20个氨基酸残基。在各方面中,肽接头包括约21个氨基酸残基。在各方面中,肽接头包括约22个氨基酸残基。在各方面中,肽接头包括约23个氨基酸残基。在各方面中,肽接头包括约24个氨基酸残基。在各方面中,肽接头包括约25个氨基酸残基。
在各方面中,肽接头包括SEQ ID NO:17所示的序列。在各方面中,肽接头为SEQ IDNO:17所示的序列。在各方面中,肽接头包括SEQ ID NO:18所示的序列。在各方面中,肽接头为SEQ ID NO:18所示的序列。在各方面中,肽接头包括SEQ ID NO:19所示的序列。在各方面中,肽接头为SEQ ID NO:19所示的序列。在各方面中,肽接头包括SEQ ID NO:20所示的序列。在各方面中,肽接头为SEQ ID NO:20所示的序列。在各方面中,肽接头包括SEQ ID NO:21所示的序列。在各方面中,肽接头为SEQ ID NO:21所示的序列。在各方面中,肽接头包括SEQ ID NO:22所示的序列。在各方面中,肽接头为SEQ ID NO:22所示的序列。在各方面中,肽接头包括SEQ ID NO:27所示的序列。在各方面中,肽接头为SEQ ID NO:27所示的序列。在各方面中,肽接头包括SEQ ID NO:24所示的序列。在各方面中,肽接头为SEQ ID NO:24所示的序列。在各方面中,肽接头包括SEQ ID NO:29所示的序列。在各方面中,肽接头为SEQ IDNO:29所示的序列。在各方面中,肽接头为XTEN多肽。在各方面中,肽接头具有与SEQ ID NO:17、18、19、20、21、22、24、27或29具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:17、18、19、20、21、22、24、27或29具有至少90%序列一致性的氨基酸序列。
在各方面中,肽接头具有与SEQ ID NO:17具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:18具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:19具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:20具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:21具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:22具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ IDNO:24具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:27具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:29具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:17、18、19、20、21、22、24、27或29具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ IDNO:17具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:18具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:19具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:20具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:21具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:22具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:24具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:27具有至少95%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:29具有至少95%序列一致性的氨基酸序列。
如本文所使用的术语“XTEN”、“XTEN接头”或“XTEN多肽”是指缺乏疏水性氨基酸残基的重组多肽(例如非结构化重组肽)。XTEN的开发和使用可以在例如Schellenberger等人,《自然生物技术(Nature Biotechnology)》27,1186-1190(2009)中找到,其以全文引用的方式并且出于所有目的并入本文中。在各方面中,XTEN接头包括SEQ ID NO:31所示的序列。在各方面中,XTEN接头为SEQ ID NO:31所示的序列。在各方面中,XTEN接头包括SEQ IDNO:32所示的序列。在各方面中,XTEN接头为SEQ ID NO:32所示的序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少90%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少95%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少90%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少95%序列一致性的氨基酸序列。
“可检测剂”或“可检测部分”是可以通过适当方式检测的组合物,所述方式为例如光谱、光化学、生物化学、免疫化学、化学、磁共振成像或其它物理方式。例如,有用的可检测试剂包括
可检测部分是单价可检测剂或能够与另一组合物形成键的可检测剂。在各方面中,可检测试剂为HA标签。在各方面中,HA标签包括SEQ ID NO:24所示的序列。在各方面中,HA标签为SEQ ID NO:24所示的序列。在各方面中,HA标签具有与SEQ ID NO:24具有至少80%序列一致性的氨基酸序列。在各方面中,HA标签具有与SEQ ID NO:24具有至少85%序列一致性的氨基酸序列。在各方面中,HA标签具有与SEQ ID NO:24具有至少90%序列一致性的氨基酸序列。在各方面中,HA标签具有与SEQ ID NO:24具有至少95%序列一致性的氨基酸序列。在各方面中,可检测试剂为蓝色荧光蛋白(BFP)。在各方面中,BFP包括SEQ IDNO:30所示的序列。在各方面中,BFP为SEQ ID NO:30所示的序列。在各方面中,BFP具有与SEQ ID NO:30具有至少80%序列一致性的氨基酸序列。在各方面中,BFP具有与SEQ ID NO:30具有至少85%序列一致性的氨基酸序列。在各方面中,BFP具有与SEQ ID NO:30具有至少90%序列一致性的氨基酸序列。在各方面中,BFP具有与SEQ ID NO:30具有至少95%序列一致性的氨基酸序列。
根据本公开的各方面,可以用作成像剂和/或标记剂的放射性物质(例如放射性同位素)包括但不限于
“接触”是根据其通常含义使用,并且是指使至少两种不同物质变得足够接近以进行反应、相互作用或物理触摸的过程。然而,应了解,所得反应产物可以直接由所添加试剂之间的反应或由来自可以在反应混合物中产生的所添加试剂中的一个或多个的中间物来产生。
术语“接触”可以包括允许两种物质反应、相互作用或物理接触,其中所述两种物质可以为例如本文所提供的融合蛋白和核酸序列(例如靶DNA序列)。
如本文所定义,当参考如本文所提供的组合物(例如融合蛋白、复合物、核酸、载体)使用时,术语“抑制(inhibition/inhibit/inhibiting)、“阻遏(repression/repressing)”、“沉默(silencing/silence)”等是指相对于核酸序列的活性(例如基因的转录)在无组合物(例如融合蛋白、复合物、核酸、载体)存在的情况下,对核酸序列的活性(例如转录)产生负面影响(例如降低)。在各方面中,抑制是指疾病或疾病症状(例如癌症)的减少。因此,抑制包括至少部分地、部分地或完全地阻断核酸序列的活化(例如转录),或降低、预防或延迟核酸序列的活化(例如转录)。抑制活性(例如转录)可以为对照中的90%、80%、70%、60%、50%、40%、30%、20%、10%或更低。在各方面中,与对照相比,抑制为1.5倍、2倍、3倍、4倍、5倍、10倍或更高。
“对照”样品或值是指充当参考(通常为已知参考)以用于与测试样品比较的样品。例如,测试样品可以例如在存在测试化合物的情况下从测试条件中获取,并且与已知条件的样品进行比较,例如在不存在测试化合物的情况下(阴性对照)或在存在已知化合物的情况下(阳性对照)进行比较。对照还可以表示从多个测试或结果中收集的平均值。本领域技术人员将认识到,对照可以经设计以用于评估任何数目的参数。例如,可以设计一种对照以比较基于药理学数据(例如半衰期)或治疗措施(例如副作用的比较)的治疗益处。本领域技术人员将理解,哪些对照在给定情形下是有价值的,并且能够基于与对照值的比较来分析数据。对照对于确定数据的重要性也很有价值。例如,如果对照中给定参数的值广泛变化,那么测试样品的变化将不被视为显著的。
融合蛋白
本文尤其提供了融合蛋白,其可以使用基于CRISPR的表观基因组编辑在哺乳动物细胞中永久地(例如不可逆地)和可逆地关闭基因。在各实施例中,融合蛋白包括可以在细胞中瞬时表达的四种蛋白(例如,催化失活的Cas9(例如dCas9)、KRAB结构域、Dnmt3A和Dnmt3L)的单个多肽融合物。可以使用与靶核酸序列(例如DNA序列)互补的多核苷酸将融合蛋白导向至哺乳动物基因组中的特定位点,并且所述多核苷酸进一步包括能够结合融合蛋白的序列(即,结合序列)。一旦恰当地定位并且不希望受到理论的束缚,融合蛋白将DNA甲基化和/或阻抑性染色质标记添加至靶核酸,从而引起在后续细胞分裂中可遗传的基因沉默。通过这种方式,融合蛋白可以进行表观基因组编辑,而无需在宿主基因组中生成DNA双链断裂,从而使其成为操作活生物体的基因组的安全并且可逆的方式。
在各实施例中,融合蛋白包含缺乏核酸酶的RNA引导的DNA核酸内切酶;KRAB结构域和DNA甲基转移酶结构域。在各方面中,融合蛋白包含从N端至C端的DNA甲基转移酶结构域、缺乏核酸酶的RNA引导的DNA核酸内切酶和KRAB结构域。在各方面中,融合蛋白包含从N端至C端的KRAB结构域、缺乏核酸酶的RNA引导的DNA核酸内切酶和DNA甲基转移酶结构域。在各实施例中,融合蛋白进一步包含一个或多个肽接头。在各方面中,融合蛋白进一步包含一个或多个可检测标签。在各方面中,融合蛋白胶进一步包含一个或多个核定位序列。在各方面中,融合蛋白进一步包含一个或多个肽接头、一个或多个可检测标签、一个或多个核定位序列或前述中的两种或更多种的组合。当融合蛋白包含一个或多个肽接头时,每个肽衬里可以相同或不同。当融合蛋白包含一个或多个可检测标签时,每个可检测标签可以相同或不同。在各方面中,融合蛋白包含1至10个可检测标签。在各方面中,融合蛋白包含1至9个可检测标签。在各方面中,融合蛋白包含1至8个可检测标签。在各方面中,融合蛋白包含1至7个可检测标签。在各方面中,融合蛋白包含1至6个可检测标签。在各方面中,融合蛋白包含1至5个可检测标签。在各方面中,融合蛋白包含1至4个可检测标签。在各方面中,融合蛋白包含1至3个可检测标签。在各方面中,融合蛋白包含1至2个可检测标签。在各方面中,融合蛋白包含1个可检测标签。在各方面中,融合蛋白包含2个可检测标签。在各方面中,融合蛋白包含3个可检测标签。在各方面中,融合蛋白包含4个可检测标签。在各方面中,融合蛋白包含5个可检测标签。
在各实施例中,融合蛋白包含以下结构:A-B-C、或B-A-C或C-A-B、或C-B-A、或B-C-A、或A-C-B;其中A包含缺乏核酸酶的RNA引导的DNA核酸内切酶;B包含KRAB结构域,C包含DNA甲基转移酶结构域;其中左侧的分量为N端,右侧的分量为C端。在各方面中,融合蛋白进一步包含一个或多个肽接头和一个或多个可检测标签。在各方面中,A-B、B-A、B-C、C-B、A-C和C-A各自独立地经由共价键、肽接头、可检测标签、核定位序列或其两种或更多种的组合连接在一起。肽接头可以为本领域中已知的任何接头(例如,P2A可裂解肽、XTEN接头等)。在各方面中,融合蛋白包含其它组分,如可检测标签(例如HA标签、蓝色荧光蛋白等)。
在各实施例中,融合蛋白包含以下结构:A-L
在各实施例中,融合蛋白包含以下结构:B-L
在各实施例中,融合蛋白包含以下结构:B-L
在各实施例中,融合蛋白包含以下结构:C-L
术语“缺乏核酸酶的RNA引导的DNA核酸内切酶”等在通常和惯用意义上是指将DNA多核苷酸内的特定磷酸二酯键靶向的RNA引导的DNA核酸内切酶(例如天然存在的RNA引导的DNA核酸内切酶的突变形式),其中磷酸二酯键的识别通过单独的多核苷酸序列(例如,RNA序列(例如单个引导RNA(sgRNA)))促进,但不能将目标磷酸二酯键裂解为显著程度(例如在生理条件下没有可测量的磷酸二酯键裂解)。因此,当与多核苷酸(例如,sgRNA)复合时,缺乏核酸酶的RNA引导的DNA核酸内切酶保留了DNA结合能力(例如,与靶序列的特异性结合),但是缺乏显著的核酸内切酶活性(例如,任何量的可检测核酸内切酶活性)。在各方面中,缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9、ddCpf1、缺乏核酸酶的Cas9变异体或缺乏核酸酶的II类CRISPR核酸内切酶。
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9。如本文提及的术语“dCas9”或“dCas9蛋白”是其中核酸内切酶活性的两个催化位点都有缺陷或缺乏活性的Cas9蛋白。在各方面中,dCas9蛋白在对应于化脓性链球菌Cas9的D10A和H840A的位置处具有突变。在各方面中,由于野生型Cas9的核酸内切酶催化位点(RuvC和HNH)处的点突变,dCas9蛋白缺乏核酸内切酶活性。点突变可以为D10A和H840A。在各方面中,dCas9基本上不具有可检测核酸内切酶(例如,脱氧核糖核酸内切酶)活性。在各方面中,dCas9包括SEQ IDNO:23的氨基酸序列。在各方面中,dCas9具有SEQ ID NO:23的氨基酸序列。在各方面中,dCas9具有与SEQ ID NO:23具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,dCas9具有与SEQ ID NO:23具有至少75%序列一致性的氨基酸序列。在各方面中,dCas9具有与SEQ ID NO:23具有至少80%序列一致性的氨基酸序列。在各方面中,dCas9具有与SEQ ID NO:23具有至少85%序列一致性的氨基酸序列。在各方面中,dCas9具有与SEQ ID NO:23具有至少90%序列一致性的氨基酸序列。在各方面中,dCas9具有与SEQ IDNO:23具有至少95%序列一致性的氨基酸序列。
如本文所提及的“CRISPR相关蛋白9”、“Cas9”、“Csn1”或“Cas9蛋白”包括Cas9核酸内切酶的任何重组或天然存在形式或其维持Cas9核酸内切酶活性(例如,与Cas9相比,活性至少在50%、80%、90%、95%、96%、97%、98%、99%或100%之内)的变异体或同源物。在一些方面,与天然存在的Cas9蛋白相比,变异体或同源物在整个序列或一部分序列(例如50、100、150或200个连续氨基酸部分)上具有至少90%、95%、96%、97%、98%、99%或100%氨基酸序列一致性。在各方面中,Cas9蛋白与由UniProt参考编号Q99ZW2鉴别的蛋白或与其具有相当大的一致性的变异体或同源物实质上一致。在各方面中,Cas9蛋白与由UniProt参考编号Q99ZW2鉴别的蛋白的氨基酸序列具有至少75%的序列一致性。在各方面中,Cas9蛋白与由UniProt参考编号Q99ZW2鉴别的蛋白的氨基酸序列具有至少80%的序列一致性。在各方面中,Cas9蛋白与由UniProt参考编号Q99ZW2鉴别的蛋白的氨基酸序列具有至少85%的序列一致性。在各方面中,Cas9蛋白与由UniProt参考编号Q99ZW2鉴别的蛋白的氨基酸序列具有至少90%的序列一致性。在各方面中,Cas9蛋白与由UniProt参考编号Q99ZW2鉴别的蛋白的氨基酸序列具有至少95%的序列一致性。
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为“ddCpf1”或“ddCas12a”。术语“DNAse-dead Cpf1”或“ddCpf1”是指突变的氨基酸球菌属种Cpf1(AsCpf1),其导致Cpf1 DNA酶活性失活。在各方面中,ddCpf1包括在AsCpf1的RuvC结构域中的E993A突变。在各方面中,ddCpf1基本上不具有可检测核酸内切酶(例如,脱氧核糖核酸内切酶)活性。在各方面中,ddCpf1包括SEQ ID NO:34的氨基酸序列。在各方面中,ddCpf1具有SEQ ID NO:34的氨基酸序列。在各方面中,ddCpf1具有与SEQ ID NO:34具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,ddCpf1具有与SEQ ID NO:34具有至少75%序列一致性的氨基酸序列。在各方面中,ddCpf1具有与SEQ ID NO:34具有至少80%序列一致性的氨基酸序列。在各方面中,ddCpf1具有与SEQ ID NO:34具有至少85%序列一致性的氨基酸序列。在各方面中,ddCpf1具有与SEQ ID NO:34具有至少90%序列一致性的氨基酸序列。在各方面中,ddCpf1具有与SEQ ID NO:34具有至少95%序列一致性的氨基酸序列。
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为dLbCpf1。术语“dLbCpf1”是指缺乏DNA酶活性的来自毛螺旋菌科细菌ND2006(LbCpf1)的突变Cpf1。在各方面中,dLbCpf1包括D832A突变。在各方面中,dLbCpf1基本上不具有可检测核酸内切酶(例如,脱氧核糖核酸内切酶)活性。在各方面中,dLbCpf1包括SEQ ID NO:35的氨基酸序列。在各方面中,dLbCpf1具有SEQ ID NO:35的氨基酸序列。在各方面中,dLbCpf1具有与SEQ IDNO:35具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,dLbCpf1具有与SEQ ID NO:35具有至少75%序列一致性的氨基酸序列。在各方面中,dLbCpf1具有与SEQ ID NO:35具有至少80%序列一致性的氨基酸序列。在各方面中,dLbCpf1具有与SEQ IDNO:35具有至少85%序列一致性的氨基酸序列。在各方面中,dLbCpf1具有与SEQ ID NO:35具有至少90%序列一致性的氨基酸序列。在各方面中,dLbCpf1具有与SEQ ID NO:35具有至少95%序列一致性的氨基酸序列。
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为dFnCpf1。术语“dFnCpf1”是指缺乏DNA酶活性的来自新凶手弗朗西斯菌(Francisella novicida)U112(FnCpf1)的突变Cpf1。在各方面中,dFnCpf1包括D917A突变。在各方面中,dFnCpf1基本上不具有可检测核酸内切酶(例如,脱氧核糖核酸内切酶)活性。在各方面中,dFnCpf1包括SEQID NO:36的氨基酸序列。在各方面中,dFnCpf1具有SEQ ID NO:36的氨基酸序列。在各方面中,dFnCpf1具有与SEQ ID NO:36具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,dFnCpf1具有与SEQ ID NO:36具有至少75%序列一致性的氨基酸序列。在各方面中,dFnCpf1具有与SEQ ID NO:36具有至少80%序列一致性的氨基酸序列。在各方面中,dFnCpf1具有与SEQ ID NO:36具有至少85%序列一致性的氨基酸序列。在各方面中,dFnCpf1具有与SEQ ID NO:36具有至少90%序列一致性的氨基酸序列。在各方面中,dFnCpf1具有与SEQ ID NO:36具有至少95%序列一致性的氨基酸序列。
如本文所提及的“Cpf1”或“Cpf1蛋白”包括Cpf1(来自普雷沃菌属(Prevotella)和弗朗西斯菌属(Francisella)1类的CRISPR)核酸内切酶的任何重组或天然存在形式或其维持Cpf1核酸内切酶活性(例如,与Cpf1相比,活性至少在50%,80%,90%,95%,96%,97%,98%,99%或100%之内)的变异体或同源物。在各方面中,与天然存在的Cpf1蛋白相比,变异体或同源物在整个序列或一部分序列(例如50、100、150或200个连续氨基酸部分)上具有至少90%、95%、96%、97%、98%、99%或100%氨基酸序列一致性。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白或与其具有相当大的一致性的变异体或同源物实质上一致。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白相同。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白的氨基酸序列具有至少75%的序列一致性。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白的氨基酸序列具有至少80%的序列一致性。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白相同。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白的氨基酸序列具有至少85%的序列一致性。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白相同。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白的氨基酸序列具有至少90%的序列一致性。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白相同。在各方面中,Cpf1蛋白与由UniProt参考编号U2UMQ6鉴别的蛋白的氨基酸序列具有至少95%的序列一致性。
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为缺乏核酸酶的Cas9变异体。术语“缺乏核酸酶的Cas9变异体”是指具有与野生型Cas9相比增加其对PAM的结合特异性的一种或多种突变的Cas9蛋白,并且还包括使所述蛋白不能或具有严重受损的核酸内切酶活性的突变。不希望受理论所束缚,据信靶序列应与PAM(前间隔序列邻近基序)相关;即,由CRISPR复合物识别的短序列。PAM的精确序列和长度要求取决于所使用的CRISPR酶不同,但是PAM通常为与前间隔序列邻近的2-5个碱基对序列(即靶序列)。缺乏核酸酶的Cas9变异体对PAM的结合特异性可以通过本领域中已知的任何方法来确定。已知Cas9变异体的描述和用途可以在例如Shmakov等人,2类CRISPR-Cas系统的多样性和进化(Diversity andevolution of class 2CRISPR-Cas systems.)《自然评论·微生物学(Nat.Rev.Microbiol)》15,2017和Cebrian-Serrano等人,CRISPR-Cas直向同源物和变异体:优化基因组工程工具的组库、特异性和传递(CRISPR-Cas orthologues and variants:optimizing the repertoire,specificity and delivery of genome engineeringtools.)《哺乳动物基因组(Mamm.Genome)》7-8,2017年中找到,其以全文引用的方式并且出于所有目的并入本文中。例示性的Cas9变异体于以下表4中列出。
表4
/>
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为缺乏核酸酶的II类CRISPR核酸内切酶。如本文所使用的术语“缺乏核酸酶的II类CRISPR核酸内切酶”是指具有导致降低、受损或失活核酸内切酶活性的突变的任何II类CRISPR核酸内切酶。
在各实施例中,DNA甲基转移酶结构域为Dnmt3A-3L结构域。如本文所提供的“Dnmt3A-3L结构域”是指包括Dnmt3A和Dnmt3L两者的蛋白。在各方面中,Dnmt3A和Dnmt3L共价连接。在各方面中,Dnmt3A通过肽接头共价连接至Dnmt3L。在各方面中,肽接头包括SEQID NO:27所示的序列。在各方面中,肽接头为SEQ ID NO:27所示的序列。在各方面中,肽接头具有与SEQ ID NO:27具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:27具有至少90%序列一致性的氨基酸序列。在各方面中,肽接头具有与SEQ ID NO:27具有至少95%序列一致性的氨基酸序列。在各方面中,Dnmt3A-3L结构域包括SEQ ID NO:33所示的序列。在各方面中,Dnmt3A-3L结构域为SEQ IDNO:33所示的序列。在各方面中,Dnmt3A-3L结构域具有与SEQ ID NO:33具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,Dnmt3A-3L结构域具有与SEQ IDNO:33具有至少75%序列一致性的氨基酸序列。在各方面中,Dnmt3A-3L结构域具有与SEQID NO:33具有至少80%序列一致性的氨基酸序列。在各方面中,Dnmt3A-3L结构域具有与SEQ ID NO:33具有至少85%序列一致性的氨基酸序列。在各方面中,Dnmt3A-3L结构域具有与SEQ ID NO:33具有至少90%序列一致性的氨基酸序列。在各方面中,Dnmt3A-3L结构域具有与SEQ ID NO:33具有至少95%序列一致性的氨基酸序列。
在各实施例中,肽接头为XTEN接头。在各方面中,XTEN接头包括约16至约80个氨基酸残基。在各方面中,XTEN接头包括约17至约80个氨基酸残基。在各方面中,XTEN接头包括约18至约80个氨基酸残基。在各方面中,XTEN接头包括约19至约80个氨基酸残基。在各方面中,XTEN接头包括约20至约80个氨基酸残基。在各方面中,XTEN接头包括约30至约80个氨基酸残基。在各方面中,XTEN接头包括约40至约80个氨基酸残基。在各方面中,XTEN接头包括约50至约80个氨基酸残基。在各方面中,XTEN接头包括约60至约80个氨基酸残基。在各方面中,XTEN接头包括约70至约80个氨基酸残基。在各方面中,XTEN接头包括约16至约70个氨基酸残基。在各方面中,XTEN接头包括约16至约60个氨基酸残基。在各方面中,XTEN接头包括约16至约50个氨基酸残基。在各方面中,XTEN接头包括约16至约40个氨基酸残基。在各方面中,XTEN接头包括约16至约35个氨基酸残基。在各方面中,XTEN接头包括约16至约30个氨基酸残基。在各方面中,XTEN接头包括约16至约25个氨基酸残基。在各方面中,XTEN接头包括约16至约20个氨基酸残基。在各方面中,XTEN接头包括约16个氨基酸残基。在各方面中,XTEN接头包括约17个氨基酸残基。在各方面中,XTEN接头包括约18个氨基酸残基。在各方面中,XTEN接头包括约19个氨基酸残基。在各方面中,XTEN接头包括约20个氨基酸残基。
在各实施例中,XTEN接头包括SEQ ID NO:31所示的序列。在各方面中,XTEN接头为SEQ ID NO:31所示的序列。在各方面中,XTEN接头包括SEQ ID NO:32所示的序列。在各方面中,XTEN接头为SEQ ID NO:32所示的序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQID NO:31具有至少85%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ IDNO:31具有至少90%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少95%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少50%,55%,60%,65%,70%,75%,80%,85%,90%,91%,92%,93%,94%,95%,96%,97%,98%,99%或100%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQID NO:32具有至少75%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ IDNO:32具有至少80%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少85%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少90%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少95%序列一致性的氨基酸序列。
融合蛋白可以包括用于将融合蛋白靶向至细胞的特定区域(例如,细胞质、细胞核)的氨基酸序列。因此,在各方面中,融合蛋白进一步包括核定位信号(NLS)肽。在各方面中,NLS包括SEQ ID NO:25所示的序列。在各方面中,NLS为SEQ ID NO:25所示的序列。在各方面中,NLS具有与SEQ ID NO:25具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少75%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少80%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少85%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ IDNO:25具有至少90%序列一致性的氨基酸序列。在各方面中,NLS具有与SEQ ID NO:25具有至少95%序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括从N端至C端的KRAB结构域、缺乏核酸酶的RNA引导的DNA核酸内切酶和DNA甲基转移酶结构域。
在各实施例中,缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9,并且DNA甲基转移酶结构域为Dnmt3A-3L结构域。
在各实施例中,dCas9经由肽接头共价连接至KRAB结构域,并且其中dCas9经由肽接头共价连接至Dnmt3A-3L结构域。
在各实施例中,肽接头为XTEN接头。在各方面中,XTEN接头包括SEQ ID NO:31所示的序列。在各方面中,XTEN接头为SEQ ID NO:31所示的序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少75%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQID NO:31具有至少80%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ IDNO:31具有至少85%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少90%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:31具有至少95%序列一致性的氨基酸序列。在各方面中,XTEN接头包括SEQ ID NO:32所示的序列。在各方面中,XTEN接头为SEQ ID NO:32所示的序列。在各方面中,XTEN接头具有与SEQ IDNO:32具有至少75%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少80%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少85%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少90%序列一致性的氨基酸序列。在各方面中,XTEN接头具有与SEQ ID NO:32具有至少95%序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:1的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:1的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:2的氨基酸序列。在各方面中,所述融合蛋白为SEQ ID NO:2的氨基酸序列。在各方面中,所述融合蛋白包括SEQ ID NO:3的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:3的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:4的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:4的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:5的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:5的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:6的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:6的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:7的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:7的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:8的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:8的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:9的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:9的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:10的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:10的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:11的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:11的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:12的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:12的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:13的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:13的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:14的氨基酸序列。在各方面中,融合蛋白为SEQ IDNO:14的氨基酸序列。在各方面中,融合蛋白包括SEQ ID NO:15的氨基酸序列。在各方面中,融合蛋白为SEQ ID NO:15的氨基酸序列。
在各实施例中、融合蛋白包括与SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:1具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:2具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:3具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQID NO:4具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:5具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:6具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:7具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:8具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:9具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:10具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:11具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:12具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:13具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:14具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQID NO:15具有至少75%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括与SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:1具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:2具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:3具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:4具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:5具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:6具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:7具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:8具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:9具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:10具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:11具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:12具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:13具有至少75%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQID NO:14具有至少75%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ IDNO:15具有至少75%序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括与SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:1具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:2具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:3具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:4具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:5具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:6具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:7具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:8具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:9具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:10具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:11具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:12具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:13具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQID NO:14具有至少80%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ IDNO:15具有至少80%序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括与SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15具有至少85%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:1具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:2具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:3具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:4具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:5具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:6具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:7具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:8具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:9具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:10具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:11具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:12具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:13具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:14具有至少85%的序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:15具有至少85%的序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括与SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:1具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:2具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:3具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:4具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:5具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:6具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:7具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:8具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:9具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:10具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:11具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:12具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:13具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQID NO:14具有至少90%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ IDNO:15具有至少90%序列一致性的氨基酸序列。
在各实施例中,融合蛋白包括与SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:1具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:2具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:3具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:4具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:5具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:6具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:7具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:8具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:9具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:10具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:11具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:12具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ ID NO:13具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQID NO:14具有至少95%序列一致性的氨基酸序列。在各方面中,融合蛋白包括与SEQ IDNO:15具有至少95%序列一致性的氨基酸序列。
复合物
为了使融合蛋白进行表观基因组编辑,融合蛋白与多核苷酸(例如,sgRNA)相互作用(例如,非共价结合),即与靶多核苷酸序列(例如,待鉴定的靶DNA序列)互补,并且进一步包括可以与本文所述的融合蛋白的缺乏核酸酶的RNA引导的DNA核酸内切酶结合的序列(即,结合序列)。通过形成此复合物,融合蛋白被适当地定位以进行表观基因组编辑。术语“复合物”是指包括两种或更多种组分的组合物,其中所述组分结合在一起以形成功能单元。在各方面中,本文所述的复合物包括本文所述的融合蛋白和本文所述的多核苷酸。因此,在一方面中提供了如本文所述的融合蛋白,包括其实施例和方面,和多核苷酸,其包括:(1)与靶多核苷酸序列互补的DNA靶向序列;和(2)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列,其中缺乏核酸酶的RNA引导的DNA核酸内切酶经由结合序列(例如,能够结合至DNA靶向序列的氨基酸序列)与多核苷酸结合。
DNA靶向序列是指包括与靶多核苷酸序列(DNA或RNA)互补的核苷酸序列的多核苷酸。在各方面中,DNA靶向序列可以为单个RNA分子(单个RNA多核苷酸),其可以包括“单个引导RNA”或“sgRNA”。在各方面中,DNA靶向序列包括两个RNA分子(例如,经由在结合序列(例如,dCas9-结合序列)处的杂交而结合在一起)。在各方面中,DNA靶向序列(例如sgRNA)与靶多核苷酸序列至少互补50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%或99%。在各方面中,DNA靶向序列(例如sgRNA)与细胞基因序列至少互补50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、96%、97%、98%或99%。在各方面中,DNA靶向序列(例如sgRNA)结合细胞基因序列。在各方面中,DNA靶向序列(例如sgRNA)与细胞基因序列至少互补75%。在各方面中,DNA靶向序列(例如sgRNA)与细胞基因序列至少互补80%。在各方面中,DNA靶向序列(例如sgRNA)结合细胞基因序列。在各方面中,DNA靶向序列(例如sgRNA)与细胞基因序列至少互补85%。在各方面中,DNA靶向序列(例如sgRNA)结合细胞基因序列。在各方面中,DNA靶向序列(例如sgRNA)与细胞基因序列至少互补90%。在各方面中,DNA靶向序列(例如sgRNA)结合细胞基因序列。在各方面中,DNA靶向序列(例如sgRNA)与细胞基因序列至少互补95%。在各方面中,DNA靶向序列(例如sgRNA)结合细胞基因序列。
如本文所提供的“靶多核苷酸序列”为存在于细胞中或由细胞表达的核酸序列,引导序列(或DNA靶向序列)经设计以具有互补性,其中靶序列与引导序列(或DNA靶向序列)之间的杂交促进CRISPR复合物的形成。如果存在足够的互补性以引起杂交并且促进CRISPR复合物的形成,则不一定需要完全互补性。在各方面中,靶多核苷酸序列为外源核酸序列。在各方面中,靶多核苷酸序列为内源核酸序列。
靶多核苷酸序列可以为适用于表观基因组编辑的多核苷酸(例如DNA序列)的任何区域。在各方面中,靶多核苷酸序列为基因的一部分。在各方面中,靶多核苷酸序列为转录调节序列的一部分。在各方面中,靶多核苷酸序列为启动子、增强子或沉默子的一部分。在各方面中,靶多核苷酸序列为启动子一部分。在各方面中,靶多核苷酸序列为增强子的一部分。在各方面中,靶多核苷酸序列为沉默子的一部分。
在各实施例中,靶多核苷酸序列为低甲基化的核酸序列。根据本领域中的标准含义,本文所使用的“低甲基化的核酸序列”,并且是指5-甲基胞嘧啶核苷酸上(例如,在CpG中)甲基的丢失或缺乏。甲基的丢失或缺乏可能是相对于标准对照而言的。甲基化可以分别相对于年轻细胞或非癌细胞在例如老化细胞中或在癌症(例如,瘤形成早期)中进行。因此,复合物可用于重新建立正常的(例如未患病的未老化的)甲基化水平。
在各实施例中,靶多核苷酸序列在转录起始位点两侧约3000个碱基对(bp)内。在各方面中,靶多核苷酸序列在转录起始位点两侧约3000、2900、2800、2700、2600、2500、2400、2300、2200、2100、2000、1900、1800、1700、1600、1500、1400、1300、1200、1100,1000、900、800、700、600、500、400、300、200或100个碱基对(bp)内。
在各实施例中,靶多核苷酸序列在启动子序列处、附近或之内。在各方面中,靶多核苷酸序列在CpG岛内。在各方面中,已知靶多核苷酸序列与以DNA低甲基化为特征的疾病或病况相关。
在各实施例中,例示性靶多核苷酸序列包括表1和2中描述的那些。在各方面中,靶多核苷酸序列包括SEQ ID NO:37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93或95的序列。在各方面中,靶多核苷酸序列包括与SEQ ID NO:37、39、41、43、45、47、49、51、53、55、57、59、61、63、65、67、69、71、73、75、77、79、81、83、85、87、89、91、93或95具有至少80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%序列一致性的氨基酸序列。在各方面中,靶多核苷酸序列为SEQ ID NO:37。在各方面中,靶多核苷酸序列为SEQ ID NO:39。在各方面中,靶多核苷酸序列为SEQ IDNO:41。在各方面中,靶多核苷酸序列为SEQ ID NO:43。在各方面中,靶多核苷酸序列为SEQID NO:45。在各方面中,靶多核苷酸序列为SEQ ID NO:47。在各方面中,靶多核苷酸序列为SEQ ID NO:49。在各方面中,靶多核苷酸序列为SEQ ID NO:51。在各方面中,靶多核苷酸序列为SEQ ID NO:53。在各方面中,靶多核苷酸序列为SEQ ID NO:55。在各方面中,靶多核苷酸序列为SEQ ID NO:57。在各方面中,靶多核苷酸序列为SEQ ID NO:59。在各方面中,靶多核苷酸序列为SEQ ID NO:61。在各方面中,靶多核苷酸序列为SEQ ID NO:63。在各方面中,靶多核苷酸序列为SEQ ID NO:65。在各方面中,靶多核苷酸序列为SEQ ID NO:67。在各方面中,靶多核苷酸序列为SEQ ID NO:69。在各方面中,靶多核苷酸序列为SEQ ID NO:71。在各方面中,靶多核苷酸序列为SEQ ID NO:73。在各方面中,靶多核苷酸序列为SEQ ID NO:75。在各方面中,靶多核苷酸序列为SEQ ID NO:77。在各方面中,靶多核苷酸序列为SEQ ID NO:79。在各方面中,靶多核苷酸序列为SEQ ID NO:81。在各方面中,靶多核苷酸序列为SEQ IDNO:83。在各方面中,靶多核苷酸序列为SEQ ID NO:85。在各方面中,靶多核苷酸序列为SEQID NO:87。在各方面中,靶多核苷酸序列为SEQ ID NO:89。在各方面中,靶多核苷酸序列为SEQ ID NO:91。在各方面中,靶多核苷酸序列为SEQ ID NO:93。在各方面中,靶多核苷酸序列为SEQ ID NO:95。
在各方面中,靶多核苷酸序列与SEQ ID NO:37具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:39具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:41具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:43具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列为SEQ ID NO:45。在各方面中,靶多核苷酸序列与SEQ ID NO:47具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:49具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:51具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:53具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:55具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:57具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:59具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:61具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:63具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:65具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:67具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:69具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:71具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:73具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:75具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:77具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:79具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:81具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:83具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:85具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:87具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:89具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:91具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ IDNO:93具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:95具有至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的序列一致性。
在各方面中,靶多核苷酸序列与SEQ ID NO:37具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:39具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:41具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:43具有至少90%的序列一致性。在各方面中,靶多核苷酸序列为SEQ ID NO:45。在各方面中,靶多核苷酸序列与SEQ ID NO:47具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:49具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:51具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:53具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:55具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:57具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:59具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:61具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:63具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:65具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:67具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:69具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:71具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:73具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:75具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:77具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:79具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:81具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:83具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:85具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:87具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:89具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:91具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:93具有至少90%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:95具有至少90%的序列一致性。
在各方面中,靶多核苷酸序列与SEQ ID NO:37具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:39具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:41具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:43具有至少95%的序列一致性。在各方面中,靶多核苷酸序列为SEQ ID NO:45。在各方面中,靶多核苷酸序列与SEQ ID NO:47具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:49具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:51具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:53具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:55具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:57具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:59具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:61具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:63具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:65具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:67具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:69具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:71具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:73具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:75具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:77具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:79具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:81具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:83具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:85具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQID NO:87具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:89具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:91具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:93具有至少95%的序列一致性。在各方面中,靶多核苷酸序列与SEQ ID NO:95具有至少95%的序列一致性。
在各实施例中,复合物包括通过结合多核苷酸的结合序列而与多核苷酸结合并且由此形成核糖核蛋白复合物的dCas9。在各方面中,结合序列形成发夹结构。在各方面中,结合序列的长度为30-100nt、35-50nt、37-47nt或42nt。
在各实施例中,结合序列(例如,Cas9结合序列)与Cas9蛋白(例如,dCas9蛋白)相互作用或结合,并且它们一起结合至由DNA靶向序列识别的靶多核苷酸序列。结合序列(例如,Cas9-结合序列)包括两个互补的核苷酸片段,其彼此杂交以形成双链RNA双链体(dsRNA双链体)。核苷酸的这两个互补片段可以通过称为接头或接头核苷酸(例如在单分子多核苷酸的情况下)的中间核苷酸共价连接,并且杂交形成结合序列(例如Cas9-结合序列)的双链RNA双链体(dsRNA双链体或“Cas9-结合发夹”,因此产生茎-环结构。替代地,在各方面中,核苷酸的两个互补片段可以不共价连接,而是通过互补序列((例如两个分子的多核苷酸)之间的杂交而保持在一起。
结合序列(例如Cas9结合序列)可以具有10个核苷酸至100个核苷酸的长度,例如10个核苷酸(nt)至20nt、20nt至30nt、30nt至40nt、40nt至50nt、50nt至60nt、60nt至70nt、70nt至80nt、80nt至90nt、或90nt至100nt。在各方面中,结合序列具有15个核苷酸(nt)至80nt的长度。在各方面中,结合序列具有15nt至50nt的长度。在各方面中,结合序列具有15nt至40nt的长度。在各方面中,结合序列具有15nt至30nt的长度。在各方面中,结合序列具有37nt至47nt的长度(例如42nt)。在各方面中,结合序列具有15nt到25nt的长度。
结合序列(例如Cas9结合序列)的dsRNA双链体可以具有6个碱基对(bp)至50bp的长度。例如,结合序列(例如Cas9结合序列)的dsRNA双链体可以具有6bp至40bp,6bp至30bp,6bp至25bp,6bp至20bp,6bp至15bp,8bp至40bp,8bp至30bp,8bp至25bp,8bp至20bp或8bp至15bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有8bp至10bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有10bp至15bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有15bp至18bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有18bp至20bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有20bp至25bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有25bp至30bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有30bp至35bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有35bp至40bp的长度。在各方面中,结合序列(例如Cas9结合序列)的dsRNA双链体具有40bp至50bp的长度。
在各实施例中,与本文所述的融合蛋白形成复合物的例示性多核苷酸包括表1和2中描述为sgRNA的那些。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92或94的序列或它们相对应的RNA序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括与SEQ ID NO:38、40、42、44、46、48、50、52、54、56、58、60、62、64、66、68、70、72、74、76、78、80、82、84、86、88、90、92或94的序列或它们相对应的RNA序列具有至少80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98、或99%的序列一致性。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:38的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:40的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:42的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:44的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:46的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:48的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:50的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:52的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:54的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:56的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:58的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:60的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:62的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:64的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:66的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:68的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:70的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:72的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:74的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:76的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:78的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:80的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:82的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:84的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:86的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:88的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:90的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:92的序列。在各方面中,与本文所述的融合蛋白形成复合物的多核苷酸包括SEQ ID NO:94的序列。
核酸和载体
本文所述的融合蛋白,包括其实施例和方面,可以作为编码融合蛋白的核酸序列提供。因此,在一方面中提供了编码本文所述的融合蛋白的核酸序列,包括其实施例和方面。因此,在一方面中提供了编码本文所述的融合蛋白的核酸序列(包含DNA靶向序列),包括其实施例和方面。在各方面中,核酸序列编码本文所述的融合蛋白,所述的融合蛋白包括具有本文所述的具有某些%序列一致性的氨基酸序列的融合蛋白。在各方面中,核酸为RNA。在各方面中,核酸为信使RNA。在各方面中,信使RNA为信使RNP。在各方面中,核酸序列编码本文所述的融合蛋白,包括其实施例和方面。在各方面中,核酸序列编码SEQ ID NO:1的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:2的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:3的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:4的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:5的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:6的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:7的融合蛋白。在各方面中,核酸序列编码SEQID NO:8的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:9的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:10的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:11的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:12的融合蛋白。在各方面中,核酸序列编码SEQID NO:13的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:14的融合蛋白。在各方面中,核酸序列编码SEQ ID NO:15的融合蛋白。
进一步考虑了编码本文所述的融合蛋白的核酸序列,包括其实施例和方面,可以包括在载体中。因此,在一方面中提供了包括如本文所述的核酸序列的载体,包括其实施例和方面。在各方面中,载体包含编码本文所述的融合蛋白的核酸序列,所述的融合蛋白包括具有本文所述的具有某些%序列一致性的氨基酸序列的融合蛋白。在各方面中,核酸为信使RNA。在各方面中,信使RNA为信使RNP。在各方面中,载体包含编码SEQ ID NO:1的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:2的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:3的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ IDNO:4的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:5的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:6的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:7的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:8的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:9的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:10的融合蛋白的核酸序列。在各方面中,载体包含编码SEQID NO:11的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:12的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:13的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ ID NO:14的融合蛋白的核酸序列。在各方面中,载体包含编码SEQ IDNO:15的融合蛋白的核酸序列。
在各实施例中,载体进一步包括多核苷酸,其中多核苷酸包括:(1)与靶多核苷酸序列互补的DNA靶向序列;和(2)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列。因此,一个或多个载体可以包括用于预形成表观基因组编辑的所有必要组分。
细胞
本文所述的组合物可以并入细胞中。在细胞内部,如本文所述的组合物,包括其实施例和方面,可以进行表观基因组编辑。因此,在一方面中提供了一种细胞,其包括如本文所述的融合蛋白,包括其实施例和方面、如本文所述的核酸,包括其实施例和方面、如本文所述的复合物,包括其实施例和方面、或如本文所述的载体,包括其实施例和方面。在各方面中,提供了一种细胞,其包括如本文所述的融合蛋白,包括其实施例和方面。在各方面中,提供了一种细胞,其包括如本文所述的核酸,包括其实施例和方面。在各方面中,提供了一种细胞,其包括如本文所述的复合物,包括其实施例和方面。在各方面中,提供了一种细胞,其包括如本文所述的载体,包括其实施例和方面。在各方面中,细胞为真核细胞。在各方面中,细胞为哺乳动物细胞。
方法
预期本文所述的组合物可以用于表观基因组编辑,并且更具体地,用于引起靶核酸序列(例如基因)的阻遏或沉默的表观基因组编辑。在不希望受任何理论束缚的情况下,沉默可能由于抑制性染色质标记的甲基化和/或在含有靶核酸序列的染色质上引入(例如特定组蛋白(例如H3K9、H3K27)的单甲基化、二甲基化或三甲基化、去乙酰化、乙酰化、磷酸化、泛素化)引起的。在不希望受任何理论束缚的情况下,所述方法可以用于通过例如经由甲基化封闭染色质或在含有靶核酸序列(例如基因)的染色质上引入抑制性染色质标记来改变表观遗传状态。在不希望受任何理论束缚的情况下,预期Dnmt3A-3L融合功能在CpG岛中发现的CG DNA位点添加甲基标记并且KRAB结构域募集通过引入抑制性标记改变组蛋白的表观遗传因素。在不希望受任何理论束缚的情况下,DNA在CpG岛中发现的CG序列的C核苷酸处被甲基化(即,在CpG岛中的CG DNA位点的C核苷酸处添加甲基标记)。
在一方面,提供了一种沉默细胞中的靶核酸序列的方法,其包括将编码本文所述的融合蛋白(包括其实施例和方面)的第一多核苷酸递送至含有靶核酸的细胞;和将第二多核苷酸递送至细胞,所述第二多核苷酸包括:(i)与靶核酸序列互补的DNA靶向序列;和(ii)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列。在不希望受任何理论束缚的情况下,融合蛋白通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。在不希望受任何理论束缚的情况下,染色质甲基化是指DNA在CpG岛中发现的CG序列的C核苷酸处被甲基化(即,在CpG岛中的CGDNA位点的C核苷酸处添加甲基标记)。在各方面中,靶核酸序列的约3000个碱基对内的序列被甲基化。在各方面中,靶核酸序列的约3000、2900、2800、2700、2600、2500、2400、2300、2200、2100、2000、1900、1800、1700、1600、1500、1400、1300、1200、1100、1000、900、800、700、600、500、400、300、200或100个碱基对内的序列被甲基化。
如本文所使用的术语“抑制性染色质标记”是指对染色质进行的修饰,其引起靶核酸序列(例如基因)的沉默(例如转录的降低或抑制)。抑制性染色质标记的实例包括但不限于组蛋白(例如,H3K9、H3K27、H3K79、H2BK5)的单甲基化、二甲基化和/或三甲基化、乙酰化/去乙酰化、磷酸化和泛素化。
在各实施例中,沉默是指转录的完全抑制。在各方面中,沉默是指与对照水平的转录相比,转录显著降低。
在各实施例中,第一多核苷酸含于第一载体内。在各方面中,第一多核苷酸含于第二载体内。在各方面中,第一载体与第二载体相同。在各方面中,第一载体与第二载体不同。
在各实施例中,通过本领域已知的任何方法,例如通过转染、电穿孔或转导将本文所述的多核苷酸递送至细胞中。
替代地,在一方面中提供了一种沉默细胞中靶核酸序列的方法,包括将如本文所述的复合物(包括其实施例和方面)递送至含有靶核酸的细胞。在不希望受任何理论束缚的情况下,复合物通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。
在各实施例中,细胞为哺乳动物细胞。
在各实施例中,所述方法的特异性比非靶核酸序列的特异性高2倍。在各方面中,所述方法的特异性比非靶核酸序列的特异性高至少2倍(例如2倍、3倍、4倍、5倍、6倍、7倍、8倍、9倍、10倍、15倍、20倍、25倍)。确定特异性的方法为本领域众所周知的,并且包括但不限于RNA测序(RNA-seq)、亚硫酸氢盐测序、染色质免疫沉淀、流式细胞术和qPCR。因此,在各方面中,通过RNA-seq确定特异性。在各方面中,通过亚硫酸氢盐测序确定特异性。在各方面中,通过染色质免疫沉淀确定特异性。在各方面中,通过流式细胞术确定特异性。在各方面中,通过qPCR确定特异性。
在各方面中,通过本领域已知的任何方法,例如通过核糖核蛋白(RNP)递送将复合物递送至细胞中。
实施例N1-N41
实施例N1。一种融合蛋白,其包含缺乏核酸酶的RNA引导的DNA核酸内切酶、克鲁珀相关盒结构域和DNA甲基转移酶结构域。
实施例N2。根据实施例N1所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9、ddCpf1、缺乏核酸酶的Cas9变异体或缺乏核酸酶的II类CRISPR核酸内切酶。
实施例N3。根据实施例N1或N2所述的融合蛋白,其中所述DNA甲基转移酶结构域为Dnmt3A-3L结构域。
实施例N4。根据实施例N1所述的融合蛋白,其中所述融合蛋白包含从N端至C端的DNA甲基转移酶结构域、缺乏核酸酶的RNA引导的DNA核酸内切酶和克鲁珀相关盒结构域
实施例N5。根据实施例N4所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9,并且所述DNA甲基转移酶结构域为Dnmt3A-3L结构域
实施例N6。根据实施例N5所述的融合蛋白,其中缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9,并且DNA甲基转移酶结构域为Dnmt3A-3L结构域
实施例N7。根据实施例N6所述的融合蛋白,其中肽接头为XTEN接头。
实施例N8。根据实施例N1所述的融合蛋白,其中所述融合蛋白包含从N端至C端的克鲁珀相关盒结构域、缺乏核酸酶的RNA引导的DNA核酸内切酶和DNA甲基转移酶结构域
实施例N9。根据实施例N8所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9,并且所述DNA甲基转移酶结构域为Dnmt3A-3L结构域。
实施例N10。根据实施例N9所述的融合蛋白,其中所述dCas9经由肽接头共价连接至所述Dnmt3A-3L结构域,并且其中所述克鲁珀相关盒结构域经由肽接头共价连接至所述dCas9。
实施例N11。根据实施例N10所述的融合蛋白,其中所述肽接头为XTEN接头。
实施例N12。根据实施例N1至N3中任一项所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由肽接头共价连接至所述克鲁珀相关盒结构域。
实施例N13。根据实施例N1至N3中任一项所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由肽接头共价连接至所述DNA甲基转移酶结构域。
实施例N14。根据实施例N1至N3中任一项所述的融合蛋白,其中所述克鲁珀相关盒结构域经由肽接头共价连接至所述DNA甲基转移酶结构域。
实施例N15。根据实施例N12至N14中任一项所述的融合蛋白,其中所述肽接头为XTEN接头。
实施例N16。根据实施例N15所述的融合蛋白,其中所述XTEN接头包含约16至80个氨基酸残基。
实施例N17。根据实施例N1至N16中任一项所述的融合蛋白,其进一步包含核定位信号肽。
实施例N18。根据实施例N1所述的融合蛋白,其中所述融合蛋白包含SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15的氨基酸序列。
实施例N19。一种核酸序列,其编码根据实施例N1至N18中任一项所述的融合蛋白。
实施例N20。根据实施例N19所述的核酸序列,其中所述核酸序列为信使RNA。
实施例N21。一种复合物,其包含:(i)根据实施例N1至N18中任一项所述的融合蛋白;和(ii)多核苷酸,其包含:(a)与靶多核苷酸序列互补的DNA靶向序列;和(b)所述缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由所述结合序列与所述多核苷酸结合。
实施例N22。根据实施例N21所述的复合物,其中所述靶多核苷酸序列为基因的一部分。
实施例N23。根据实施例N21所述的复合物,其中所述靶多核苷酸序列为转录调节序列的一部分。
实施例N24。根据实施例N21所述的复合物,其中所述目标多核苷酸序列为启动子、增强子或沉默子的一部分。
实施例N25。根据实施例N21所述的复合物,其中所述靶多核苷酸序列在转录起始位点两侧约3000bp内。
实施例N26。一种载体,其包含根据实施例N19或N20所述的核酸序列。
实施例N27。根据实施例N26所述的载体,其进一步包含多核苷酸,其中所述多核苷酸包含:(a)与靶多核苷酸序列互补的DNA靶向序列;和(b)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列。
实施例N28。一种细胞,其包含根据实施例N1至N18中任一项所述的融合蛋白;根据实施例N19或N20所述的核酸;根据实施例N21到N25中任一项所述的复合物,或根据实施例N26或N27所述的载体。
实施例N29。根据实施例N28所述的细胞,其中所述细胞为真核细胞。
实施例N30。根据实施例N28所述的细胞,其中所述细胞为哺乳动物细胞。
实施例N31。一种沉默细胞中的靶核酸序列的方法,其包含:(i)将编码根据权根据实施例N1至N18中任一项所述的融合蛋白的第一多核苷酸递送至含有所述靶核酸的细胞;和(ii)将第二多核苷酸递送至所述细胞,所述第二多核苷酸包含:(a)与所述靶核酸序列互补的DNA靶向序列;和(b)所述缺乏核酸酶的RNA引导DNA核酸内切酶的结合序列。
实施例N32。根据实施例N31所述的方法,其中所述融合蛋白通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。
实施例N33。根据实施例N31或N32所述的方法,其中所述第一多核苷酸含于第一载体内。
实施例N34。根据实施例N31到N33中任一项所述的方法,其中所述第一多核苷酸含于第二载体内。
实施例N35。根据实施例N34所述的方法,其中所述第一载体与第二载体相同。
实施例N36。根据实施例N34所述的方法,其中所述第一载体与所述第二载体不同。
实施例N37。根据实施例N31所述的方法,其中所述细胞为哺乳动物细胞。
实施例N38。根据实施例N31所述的方法,其中所述方法的特异性比非靶核酸序列的特异性高2倍。
实施例N39。一种沉默细胞中的靶核酸序列的方法,所述方法包含将实施例N21至N25中任一项所述的复合物递送至含有所述靶核酸的细胞。
实施例N40。根据实施例N39所述的方法,其中所述复合物通过使含有靶核酸序列的染色质甲基化和/或通过向含有靶核酸序列的染色质引入抑制性染色质标记来沉默细胞中的靶核酸序列。
实施例N41。根据实施例N39或N40所述的方法,其中所述细胞为哺乳动物细胞。
实施例N42。根据实施例N39至N41中任一项所述的方法,其中所述方法的特异性比非靶核酸序列的特异性高2倍。
实施例1至36
实施例1。一种融合蛋白,其包含缺乏核酸酶的RNA引导的DNA核酸内切酶、克鲁珀相关盒(KRAB)结构域和DNA甲基转移酶结构域。
实施例2。根据实施例1所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9、ddCpf1、缺乏核酸酶的Cas9变异体或缺乏核酸酶的II类CRISPR核酸内切酶。
实施例3。根据实施例1或2所述的融合蛋白,其中所述DNA甲基转移酶结构域为Dnmt3A-3L结构域。
实施例4。根据实施例1至3中任一实施例所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由肽接头共价连接至所述KRAB结构域。
实施例5。根据实施例1至4中任一项所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由肽接头共价连接至所述DNA甲基转移酶结构域。
实施例6。根据实施例1至5中任一项所述的融合蛋白,其中所述KRAB结构域经由肽接头共价连接至所述DNA甲基转移酶结构域。
实施例7。根据实施例4至6中任一项所述的融合蛋白,其中所述肽接头为XTEN接头。
实施例8。根据实施例7所述的融合蛋白,其中所述XTEN接头包含约16至80个氨基酸残基。
实施例9。根据实施例1至8中任一项所述的融合蛋白,其进一步包含核定位信号肽。
实施例10。根据实施例1至9中任一项所述的融合蛋白,其中所述融合蛋白包括从N端至C端的KRAB结构域、缺乏核酸酶的RNA引导的DNA核酸内切酶和DNA甲基转移酶结构域。
实施例11。根据实施例1至10所述的融合蛋白,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶为dCas9,并且所述DNA甲基转移酶结构域为Dnmt3A-3L结构域。
实施例12。根据实施例11所述的融合蛋白,其中所述dCas9经由肽接头共价连接至所述KRAB结构域,并且其中所述dCas9经由肽接头共价连接至所述Dnmt3A-3L结构域。
实施例13。根据实施例12所述的融合蛋白,其中所述肽接头为XTEN接头。
实施例14。根据实施例1至13中任一项所述的融合蛋白,其中所述融合蛋白包含SEQ ID NO:1、2、3、4、5、6、7、8、9、10、11、12、13、14或15的氨基酸序列。
实施例15。一种核酸序列,其编码根据实施例1至14中任一项所述的融合蛋白。
实施例16。一种复合物,其包含:(i)根据实施例1至18中任一项所述的融合蛋白;和(ii)多核苷酸,其包含:(a)与靶多核苷酸序列互补的DNA靶向序列;和(b)所述缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列,其中所述缺乏核酸酶的RNA引导的DNA核酸内切酶经由所述结合序列与所述多核苷酸结合。
实施例17。根据实施例21所述的复合物,其中所述靶多核苷酸序列为基因的一部分。
实施例18。根据实施例21所述的复合物,其中所述靶多核苷酸序列为转录调节序列的一部分。
实施例19。根据实施例21所述的复合物,其中所述目标多核苷酸序列为启动子、增强子或沉默子的一部分。
实施例20。根据实施例21所述的复合物,其中所述靶多核苷酸序列为低甲基化的核酸序列。
实施例21。根据实施例21所述的复合物,其中所述靶多核苷酸序列在转录起始位点两侧约3000bp内。
实施例22。一种载体,其包含根据实施例19所述的核酸序列。
实施例23。根据实施例26所述的载体,其进一步包含多核苷酸,其中所述多核苷酸包含:(a)与靶多核苷酸序列互补的DNA靶向序列;和(b)缺乏核酸酶的RNA引导的DNA核酸内切酶的结合序列。
实施例24。一种细胞,其包含根据实施例1至14中任一实施例所述的融合蛋白;根据实施例15所述的核酸;根据实施例16至21中任一实施例所述的复合物;或根据实施例22或23所述的载体。
实施例25。根据实施例28所述的细胞,其中所述细胞为真核细胞。
实施例26。根据实施例28所述的细胞,其中所述细胞为哺乳动物细胞。
实施例27。一种沉默细胞中的靶核酸序列的方法,其包含:(i)将编码根据实施例1至14所述的融合蛋白的第一多核苷酸递送至含有所述靶核酸的细胞;和(ii)将第二多核苷酸递送至所述细胞,所述第二多核苷酸包含:(a)与所述靶核酸序列互补的DNA靶向序列;和(b)所述缺乏核酸酶的RNA引导DNA核酸内切酶的结合序列,其中所述融合蛋白通过使含有所述靶核酸序列的染色质甲基化和/或通过向含有所述靶核酸序列的染色质引入抑制性染色质标记来沉默所述细胞中的所述靶核酸序列。
实施例28。根据实施例27所述的方法,其中所述第一多核苷酸含于第一载体内。
实施例29。根据实施例27所述的方法,其中所述第一多核苷酸含于第二载体内。
实施例30。根据实施例28或29所述的方法,其中所述第一载体与第二载体相同。
实施例31。根据实施例28或29所述的方法,其中所述第一载体与所述第二载体不同。
实施例32。根据实施例27至31中任一项所述的方法,其中所述细胞为哺乳动物细胞。
实施例33。根据实施例27至32中任一项所述的方法,其中所述方法的特异性比非靶核酸序列的特异性高2倍。
实施例34。一种沉默细胞中的靶核酸序列的方法,所述方法包含将根据实施例16至20中任一项所述的复合物递送至含有所述靶核酸的细胞中,其中所述复合物通过以下方式使细胞中的靶核酸序列沉默:(i)使含有所述靶核酸序列的染色质甲基化,(ii)向含有所述靶核酸序列的染色质引入抑制性染色质标记,或(iii)使含有所述靶核酸序列的染色质甲基化并且向含有所述靶核酸序列的染色质引入抑制性染色质标记。
实施例35。根据实施例34所述的方法,其中所述细胞为哺乳动物细胞。
实施例36。根据实施例34或35所述的方法,其中所述方法的特异性比非靶核酸序列的特异性高2倍。
实例
实施例和方面通过以下实例进一步说明。实例仅意图说明实施例和方面,并且不应被解释为限制本文中的范围。
实例1
针对永久基因沉默测试的dCas9-融合表观遗传调节剂。多合一蛋白的初始版本(V1,p76(SEQ ID NO:1))(图1A)具有融合至dCas9的N端(SEQ ID NO:23)的KRAB结构域,由GGSGGGS(SEQ ID NO:17)接头分离,和在dCas9的C端(由EASGSGRASPGIPGSTR(SEQ ID NO:19)接头分离)的Dnmt3A-Dnmt3L。另一种多合一蛋白将KRAB结构域(SEQ ID NO:16)、dCas9(D10A,H208A)、Dnmt3A-Dnmt3L(SEQ ID NO:33;其中SEQ ID NO:26为Dnmt3A并且SEQ IDNO:28为Dnmt3L)组合成一种多肽(图1B)。参考图1B,dCas9-KRAB蛋白改编自Gilbert等人,《细胞》2013年用于CRISPR干扰(CRISPRi)应用,并且dCas9-Dnmt3A-Dnmt3L融合改编自Stepper等人,《核酸研究(Nucleic Acids Research)》,2016年。
在HEK293T细胞中使用DNA甲基化敏感的GFP报告基因(改编自Stelzer等人,《细胞》2015年)测试V1表观遗传编辑器的活性,以通过多合一蛋白评估长期沉默(图1C)。将普遍存在的染色质开放元件(UCOE)添加至GAPDH CpG岛(CGI)的上游以预防哺乳动物细胞中慢病毒载体的背景沉默。当GAPDH CGI甲基化时,gfp基因关闭。A、B和C表示我们编码单个引导RNA(sgRNA)在启动子中的靶向的位置。这些靶序列和相对应的sgRNA序列在下表1中列出。将两个质粒共转染至细胞中,一个质粒编码肇事逃逸蛋白,并且另一个质粒编码sgRNA(图1D)。转染两天后,对表达肇事逃逸蛋白和sgRNA表达载体的细胞进行分选。通过流式细胞术来评估GFP荧光随时间的变化。当用sgRNA表达多合一蛋白时,观察到经历GFP报告基因的长期沉默的细胞群(图1E)。经历长期沉默的细胞数高于dCas9-Dnmt3A-Dnmt3L(缺乏KRAB结构域)。
表1
/>
GFP报告基因的沉默取决于sgRNA序列,其中引导件C引起所测试的三个sgRNA序列中的沉默水平最高。混合编码不同序列的sgRNA在基因沉默中没有显著变化。
实例2
使用肇事逃逸融合蛋白将三个基因(CD29、CD81、CD151)用于长期沉默。所有这三种蛋白均在细胞表面定位,并且通过细胞的细胞表面抗体染色和随后的流式细胞术评估基因敲低。转染后22天采集的代表性的流式细胞术数据于图2A-2C所示。象限IV代表基因关闭的细胞,由基因关闭的细胞百分比指示。第一象限和第二象限中细胞的缺少表明细胞中不再存在肇事逃逸蛋白(由BFP标记)。图2D提供了用三种不同sgRNA序列或所有三种sgRNA的池的CD29、CD81和CD151沉默的定量。用于此实验的靶向DNA序列和其sgRNA概述于表2中。
表2
同时靶向两个或三个基因,以显示可以通过共同递送靶向不同基因的sgRNA来实现多合一蛋白的多重化。NT sgRNA是指非靶向sgRNA对照。结果于图2E中所示。
追踪从单个克隆开始的细胞的基因沉默,观察到大多数细胞保持了靶向CLTA基因关闭(39个克隆中有37个)。图2F中的图表示转染多合一蛋白和靶向CLTA基因的sgRNA后9个月的时间点。
本文所述的系统可以靶向哺乳动物基因组中的任何基因,尤其是在基因启动子处含有CpG岛的那些基因。Dnmt3A-Dnmt3L规范地靶向CpG二核苷酸。可以靶向的基因的实例包括但不限于CXCR4、CD4、CD8、CD45、PD-1、CLTA-4、TGFBR、TCRa、TCRb、B2M。
实例3
收集在转染后三十六天失去ITGB1(CD29)、CD81和CD151表达的细胞,并且分析其RNA表达谱。如图3A-3C所示,与非靶向sgRNA对照相比,检测到靶向基因的成功的基因敲低。图3D-3F为火山图,其显示靶向基因为每个实验中所敲低的仅有重要基因,表明基因沉默的高特异性。图3G-3I为显示大于96%的靶向基因的基因敲低的转录本水平的定量。
实例4
多合一蛋白可以在HeLa(子宫颈)、U2OS(骨)和人诱导多能干细胞(iPSC)中转染并表达。图4A-4F中的流式细胞术图显示与蛋白融合的BFP表达。将HeLa和U2OS细胞中的三个内源基因(即,CD29、CD81和CD151)靶向。如图4G所示,检测到转染后18天测量的稳定沉默。当靶向Pcsk9、Npc1、Spcs1和Cd81时在AML12小鼠肝细胞细胞系中检测到基因沉默。如图4H所示,在转染后14天通过qPCR检测沉默。本实验中所使用的sgRNA序列概述于表3中。
表3
/>
实例5
图5提供了针对基因沉默而设计和测试的多合一蛋白构建体的示意图。SEQ IDNO:1的初始设计(p76,V1)经修饰以在dCas9(SEQ ID NO:29)的N或C端处编码XTEN接头(例如16个氨基酸(SEQ ID NO:31)或80个氨基酸(SEQ ID NO:32))。所有载体在dCas9的C端处含有HA标签(SEQ ID NO:24)。在各方面中,使用CAG启动子,因为其例如在构建体p76和p90-102、p112(V2)中提供了良好的表达。参考图5,p90至p102的蛋白构建体分别对应于SEQ IDNO:2-14,并且蛋白构建体p112对应于SEQ ID NO:15。
实例6
测试图5中所示的蛋白构建体转染后18天在HEK293T细胞中CLTA基因的沉默(图6A-6B)。检测到可变水平的基因沉默活性,包括一组与p76(V1)设计相比具有更持久的基因沉默的变异体,例如p99(SEQ ID NO:11)、p100(SEQ ID NO:12)和p112(SEQ ID NO:15)。图6A和6B显示dCas9-KRAB和dCas9-Dnmt3A-Dnmt3L构建体显示出长期沉默的瞬时和较低效率。
测试p76(SEQ ID NO:1)、p112(SEQ ID NO:15)以沉默HIST2H2BE(H2B)内源基因和在HEK293T细胞中稳定表达的合成Snrpn-GFP报告基因(图6C-6D)。转染后追踪细胞50天。p112变异体以比p76(V1)设计更高的效率保持基因沉默。dCas9-Dnmt3A-Dnmt3L和dCas9-KRAB融合蛋白具有长期沉默的瞬时和较低效率。图6E提供了在50天的时程中关闭HIST2H2BE(H2B)基因的p76和p112蛋白表达的曲线图。通过流式细胞术检测BFP来测量蛋白水平,所述BFP与多合一蛋白共表达。
实例7
使用针对化脓性链球菌Cas9的抗体对多合一蛋白变异体p76、p90-p102进行蛋白质印迹分析。参考图7A,顶部条带代表全长蛋白,较小尺寸的条带代表多合一蛋白的蛋白水解。显示出极少蛋白水解的变异体,如p99(SEQ ID NO:11)、p100(SEQ ID NO:12)和p102(SEQ ID NO:14),表现出更高的基因沉默效率。具有高水平蛋白水解的变异体,如p96(SEQID NO:8)和p97(SEQ ID NO:9),引起持续基因沉默的效率降低。
用多合一蛋白变异体进行蛋白质印迹分析,以检测从融合蛋白上裂解的游离Dnmt3A。如图7B所示,相比于具有可检测裂解Dnmt3A的变异体,即p76(SEQ ID NO:1)、p91(SEQ ID NO:3)、p96(SEQ ID NO:8)、p98(SEQ ID NO:10)具有极少或无可检测自由Dnmt3的变异体,如p92(SEQ ID NO:4)、p100(SEQ ID NO:12)、p101(SEQ ID NO:13)和p102(SEQ IDNO:14)具有更高的持续基因沉默效率。
实例8
如图8A所示,分析混合筛选以确定引起长期基因沉默的最佳sgRNA。使用四个HEK293T细胞系,每个细胞系具有带有GFP标签的不同基因(CLTA、VIM、HIST2H2BE(H2B)和RAB11A)。由距各基因的转录起始位点(TSS)跨越+/-2.5kb的sgRNA组成的平铺式库通过慢病毒递送稳定地在细胞中表达,接着瞬时表达出表达多合一蛋白的质粒DNA。转染后四周,对保持基因沉默的细胞进行分选以确定sgRNA标识。图8B-8E为显示转染后四周经历基因沉默的细胞百分比的流式细胞术直方图。
图9A-9D为跨越靶向基因(CLTA、H2B、RAB11、VIM)的转录起始位点的sgRNA功能图。转录起始位点(TSS)和CpG岛标注于每个图上方。每个点代表一个sgRNA,并且将其在长期基因沉默中的功效绘制为sgRNA丰度的log2倍变化。根据微球菌核酸酶信号绘制核小体的占有率(底部图)。
实例9
图10A显示了在HEK293T细胞中进行混合筛选以确定多合一蛋白的最佳sgRNA靶向位置的工作流程,所述工作流程改编自K562细胞中先前的蓖麻毒素平铺式筛选以确定dCas9-KRAB的最佳sgRNA(Gilbert,Horlbeck等人,《细胞》2014年)。sgRNA首先通过慢病毒递送在HEK293T细胞中稳定地表达,随后瞬时转染编码多合一蛋白的质粒(第0天)。将表达多合一蛋白的细胞(第2天)进行分选,并且使其生长三天。在第5天分裂细胞,从中收集一半作为初始时间点,另一半在最后一个时间点传代十天(第15天)。生长表型(γ)计算为log2sgRNA富集度除以T(初始)和T(最终)之间的细胞倍增数。
图10B-10E为代表图,其显示了在K562细胞中现有的dCas9-KRAB/CRISPRi数据集的四个基因(ARL1、EIF6、SMC3、HEATR1)的生长表型(Gilbert,Horlbeck等人,2014年)和多合一蛋白(底部图)。每个点代表一个sgRNA。对于每个基因显示TSS和标注的CpG岛。使用多合一蛋白的功能性sgRNA的范围比功能性sgRNA的范围跨越更广,这意味着更广范围的有效靶向。
实例10
图11A-11B提供VPS53和VPS54的生长表型和核小体定位(来自微球菌核酸酶信号)的比较,并且显示功能性sgRNA在核小体耗尽区域的位置。此外,与dCas9-KRAB/CRISPRi相比,使用多合一蛋白时功能性sgRNA的范围更广。
实例11
两种多合一变异体(p102(SEQ ID NO:14)和p112(SEQ ID NO:15))的体外转录显示了每种设计的全长合成(图12A)。图12B提供流式细胞术图,其显示了mRNA转染至HEK293T细胞中一天后p102和p112的表达。图12C显示了转染表达p102和p112多合一变异体的mRNA后,HEK293T细胞中CLTA内源基因沉默的时程。
实例12
图13A提供流式细胞术图,其显示了通过在多西环素诱导型启动子下稳定地编码多合一蛋白的K562细胞中添加多西环素的多合一蛋白的诱导表达。在多西环素诱导后追踪蛋白表达四天。图13A中的面板中的虚线代表未施用多西环素的基线中值BFP荧光。在多西环素处理之前和之后进行细胞的蛋白质印迹法以检测多合一蛋白的表达(图13B)。诱导后96小时无法检测到多合一蛋白的存在。多西环素处理后K562细胞14天的CD81和CD151的基因敲低的流式细胞术图显示于图13C-13F中。显示了靶基因的基因敲低的细胞百分比。由于BFP+象限中没有细胞存在,因此无法检测到多合一蛋白的表达。多西环素处理或未经多西环素处理后14天的CD81和CD151基因敲低的定量显示于图13G中。
参考文献
Ecco等人,《发展(Development)》144,2017年。Lambert等人,《细胞》172,2018年。Siddique等人,《分子生物学杂志(J.Mol.Biol)》,425,2013年。Stepper等人,《核酸研究(Nucleic Acids Research)》45,2017年。Shmakov等人,《自然评论·微生物学(Nat.Rev.Microbiol)》15,2017年。Cebrian-Serrano等人,《哺乳动物基因组(Mamm.Genome)》7-8,2017年。Pulecio等人,《细胞·干细胞(Cell Stem Cell)》21,2017年。
非正式序列表
SEQ ID NO:1(p76(多合一蛋白序列,版本1):KRAB(粗体;来自Gilbert等人,《细胞》,2013年,2014年)、接头(带下划线)、dCas9(斜体)、HA标签(小写)、SV40NLS(小写斜体)、Dnmt3A(粗体斜体;残基612-912;来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线;来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线;来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
SEQ ID NO:2(p90(KRAB-dCas9-XTEN16-Dnmt3A-Dnmt3L-P2A-BFP):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年);接头(带下划线)、dCas9(斜体)、HA标签(小写)、SV40 NLS(小写斜体)、XTEN16(大写,16个氨基酸序列)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线;来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
SEQ ID NO:3(p91(KRAB-dCas9-Dnmt3A-Dnmt3L-
SEQ ID NO:4(p92(KRAB-dCas9-XTEN16-Dnmt3A-Dnmt3L-P2A-BFP):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年)、接头(带下划线)、dCas9(斜体)、HA标签(小写)、SV40 NLS(小写斜体)、XTEN16(16个氨基酸序列)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线,来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
SEQ ID NO:5(p93(KRAB-dCas9-
SEQ ID NO:6(p94(KRAB-dCas9-
SEQ ID NO:7(p95(KRAB-
SEQ ID NO:8(p96(KRAB-XTEN16-dCas9-Dnmt3A-Dnmt3L-P2A-P2A-BFP):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年)、接头(带下划线)、XTEN16(16个氨基酸序列)、dCas9(斜体)、HA标签(小写)、SV40 NLS(小写斜体)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线,来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
SEQ ID NO:9(p97(KRAB-
SEQ ID NO:10(p98(KRAB-
SEQ ID NO:11(p99(KRAB-XTEN16-dCas9-XTEN80-Dnmt3A-Dnmt3L-P2A-BFP):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年)、XTEN16(16个氨基酸序列)、dCas9(斜体)、HA标签(小写)、接头(带下划线)、SV40 NLS(小写斜体)、XTEN80(小写斜体粗体,80个氨基酸序列)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线,来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
/>
SEQ ID NO:12(p100(KRAB-XTEN16-dCas9-XTEN80-Dnmt3A-Dnmt3L-P2A-P2A-BFP):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年)、XTEN16(16个氨基酸序列)、dCas9(斜体)、HA标签(小写)、接头(带下划线)、SV40 NLS(小写斜体)、XTEN80(小写斜体粗体,80个氨基酸序列)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线,来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
/>
SEQ ID NO:13(p101(KRAB-
/>
SEQ ID NO:14(p102(KRAB-XTEN80-dCas9-XTEN16-Dnmt3A-Dnmt3L-P2A-BFP):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年)、接头(带下划线)、XTEN80(小写斜体粗体,80个氨基酸序列)、dCas9(斜体)、HA标签(小写)、SV40 NLS(小写斜体)、XTEN16(16个氨基酸序列)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线,来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、P2A肽裂解序列(小写粗体)、BFP(小写带下划线)
/>
SEQ ID NO:15(p112(Dnmt3A-Dnmt3L-XTEN80-dCas9-BFP-KRAB):KRAB(粗体,来自Gilbert等人,《细胞》,2013年,2014年)、接头(带下划线)、XTEN80(小写斜体粗体,80个氨基酸序列)、dCas9(斜体)、HA标签(小写)、SV40 NLS(小写斜体)、Dnmt3A(粗体斜体,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、27个氨基酸接头(斜体带下划线,来自Siddique等人,分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、Dnmt3L(粗体带下划线,来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)、BFP(小写带下划线)
/>
SEQ ID NO:16(KRAB;来自Gilbert等人,《细胞》,2013年,2014年)DAKSLTAWSRTLVTFKDVFVDFTREEWKLLDTAQQIVYRNVMLENYKNLVSLGYQLTKPDVI LRLEKGEEP
SEQ ID NO:17(接头)GGSGGGS
SEQ ID NO:18(接头)GGSGGGS
SEQ ID NO:19(接头)EASGSGRASPGIPGSTR
SEQ ID NO:20(接头)SRAD
SEQ ID NO:21(接头)GSG
SEQ ID NO:22(接头)SPG
SEQ ID NO:23(dCas9)
MDKKYSIGLAIGTNSVGWAVITDEYKVPSKKFKVLGNTDRHSIKKNLIGALLFDSGETAEAT
RLKRTARRRYTRRKNRICYLQEIFSNEMAKVDDSFFHRLEESFLVEEDKKHERHPIFGNIVD
EVAYHEKYPTIYHLRKKLVDSTDKADLRLIYLALAHMIKFRGHFLIEGDLNPDNSDVDKLFI
QLVQTYNQLFEENPINASGVDAKAILSARLSKSRRLENLIAQLPGEKKNGLFGNLIALSLGL
TPNFKSNFDLAEDAKLQLSKDTYDDDLDNLLAQIGDQYADLFLAAKNLSDAILLSDILRVNT
EITKAPLSASMIKRYDEHHQDLTLLKALVRQQLPEKYKEIFFDQSKNGYAGYIDGGASQEEF
YKFIKPILEKMDGTEELLVKLNREDLLRKQRTFDNGSIPHQIHLGELHAILRRQEDFYPFLK
DNREKIEKILTFRIPYYVGPLARGNSRFAWMTRKSEETITPWNFEEVVDKGASAQSFIERMT
NFDKNLPNEKVLPKHSLLYEYFTVYNELTKVKYVTEGMRKPAFLSGEQKKAIVDLLFKTNRK
VTVKQLKEDYFKKIECFDSVEISGVEDRFNASLGTYHDLLKIIKDKDFLDNEENEDILEDIV
LTLTLFEDREMIEERLKTYAHLFDDKVMKQLKRRRYTGWGRLSRKLINGIRDKQSGKTILDF
LKSDGFANRNFMQLIHDDSLTFKEDIQKAQVSGQGDSLHEHIANLAGSPAIKKGILQTVKVV
DELVKVMGRHKPENIVIEMARENQTTQKGQKNSRERMKRIEEGIKELGSQILKEHPVENTQLQNEKLYLYYLQNGRDMYVDQELDINRLSDYDVDAIVPQSFLKDDSIDNKVLTRSDKNRGKSDNVPSEEVVKKMKNYWRQLLNAKLITQRKFDNLTKAERGGLSELDKAGFIKRQLVETRQITKHVAQILDSRMNTKYDENDKLIREVKVITLKSKLVSDFRKDFQFYKVREINNYHHAHDAYLNAVVGTALIKKYPKLESEFVYGDYKVYDVRKMIAKSEQEIGKATAKYFFYSNIMNFFKTEITLANGEIRKRPLIETNGETGEIVWDKGRDFATVRKVLSMPQVNIVKKTEVQTGGFSKESILPKRNSDKLIARKKDWDPKKYGGFDSPTVAYSVLVVAKVEKGKSKKLKSVKELLGITIMERSSFEKNPIDFLEAKGYKEVKKDLIIKLPKYSLFELENGRKRMLASAGELQKGNELALPSKYVNFLYLASHYEKLKGSPEDNEQKQLFVEQHKHYLDEIIEQISEFSKRVILADANLDKVLSAYNKHRDKPIREQAENIIHLFTLTNLGAPAAFKYFDTTIDRKRYTSTKEVLDATLIHQSITGLYETRIDLSQLGGD
SEQ ID NO:24(HA标签)YPYDVPDYA
SEQ ID NO:25(SV40 NLS)PKKKRKV
SEQ ID NO:26(Dnmt3A;残基612-912;来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)
NHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACV
SEQ ID NO:27(27个氨基酸接头;来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)
SSGNSNANSRGPSFSSGLVPLSLRGSH
SEQ ID NO:28(Dnmt3L;来自Siddique等人,《分子生物学杂志(JMB)》,2013年;Stepper等人,《自然评论·微生物学(NAR)》,2016年)
MGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPL
SEQ ID NO:29(P2A肽裂解序列)ATNFSLLKQAGDVEENPGP
SEQ ID NO:30(BFP)
SELIKENMHMKLYMEGTVDNHHFKCTSEGEGKPYEGTQTMRIKVVEGGPLPFAFDILATSFLYGSKTFINHTQGIPDFFKQSFPEGFTWERVTTYEDGGVLTATQDTSLQDGCLIYNVKIRGVNFTSNGPVMQKKTLGWEAFTETLYPADGGLEGRNDMALKLVGGSHLIANIKTTYRSKKPAKNLKMPGVYYVDYRLERIKEANNETYVEQHEVAVARYCDLPSKLGHKLN*
SEQ ID NO:31(XTEN16(16个氨基酸序列))SGSETPGTSESATPES
SEQ ID NO:32(XTEN80(80个氨基酸序列))
GGPSSGAPPPSGGSPAGSPTSTEEGTSESATPESGPGTSTEPSEGSAPGSPAGSPTSTEEGTSTEPSEGSAPGTSTEPSE
SEQ ID NO:33(Dnmt3A-Dnmt3L结构域)
NHDQEFDPPKVYPPVPAEKRKPIRVLSLFDGIATGLLVLKDLGIQVDRYIASEVCEDSITVGMVRHQGKIMYVGDVRSVTQKHIQEWGPFDLVIGGSPCNDLSIVNPARKGLYEGTGRLFFEFYRLLHDARPKEGDDRPFFWLFENVVAMGVSDKRDISRFLESNPVMIDAKEVSAAHRARYFWGNLPGMNRPLASTVNDKLELQECLEHGRIAKFSKVRTITTRSNSIKQGKDQHFPVFMNEKEDILWCTEMERVFGFPVHYTDVSNMSRLARQRLLGRSWSVPVIRHLFAPLKEYFACVSSGNSNANSRGPSFSSGLVPLSLRGSHMGPMEIYKTVSAWKRQPVRVLSLFRNIDKVLKSLGFLESGSGSGGGTLKYVEDVTNVVRRDVEKWGPFDLVYGSTQPLGSSCDRCPGWYMFQFHRILQYALPRQESQRPFFWIFMDNLLLTEDDQETTTRFLQTEAVTLQDVRGRDYQNAMRVWSNIPGLKSKHAPLTPKEEEYLQAQVRSRSKLDAPKVDLLVKNCLLPLREYFKYFSQNSLPL
SEQ ID NO:34(ddAsCfp1)
MTQFEGFTNLYQVSKTLRFELIPQGKTLKHIQEQGFIEEDKARNDHYKELKPIIDRIYKTYA
DQCLQLVQLDWENLSAAIDSYRKEKTEETRNALIEEQATYRNAIHDYFIGRTDNLTDAINKR
HAEIYKGLFKAELFNGKVLKQLGTVTTTEHENALLRSFDKFTTYFSGFYENRKNVFSAEDIS
TAIPHRIVQDNFPKFKENCHIFTRLITAVPSLREHFENVKKAIGIFVSTSIEEVFSFPFYNQ
LLTQTQIDLYNQLLGGISREAGTEKIKGLNEVLNLAIQKNDETAHIIASLPHRFIPLFKQIL
SDRNTLSFILEEFKSDEEVIQSFCKYKTLLRNENVLETAEALFNELNSIDLTHIFISHKKLE
TISSALCDHWDTLRNALYERRISELTGKITKSAKEKVQRSLKHEDINLQEIISAAGKELSEA
FKQKTSEILSHAHAALDQPLPTTLKKQEEKEILKSQLDSLLGLYHLLDWFAVDESNEVDPEF
SARLTGIKLEMEPSLSFYNKARNYATKKPYSVEKFKLNFQMPTLASGWDVNKEKNNGAILFV
KNGLYYLGIMPKQKGRYKALSFEPTEKTSEGFDKMYYDYFPDAAKMIPKCSTQLKAVTAHFQ
THTTPILLSNNFIEPLEITKEIYDLNNPEKEPKKFQTAYAKKTGDQKGYREALCKWIDFTRD
FLSKYTKTTSIDLSSLRPSSQYKDLGEYYAELNPLLYHISFQRIAEKEIMDAVETGKLYLFQ
IYNKDFAKGHHGKPNLHTLYWTGLFSPENLAKTSIKLNGQAELFYRPKSRMKRMAHRLGEKM
LNKKLKDQKTPIPDTLYQELYDYVNHRLSHDLSDEARALLPNVITKEVSHEIIKDRRFTSDK
FFFHVPITLNYQAANSPSKFNQRVNAYLKEHPETPIIGIDRGERNLIYITVIDSTGKILEQR
SLNTIQQFDYQKKLDNREKERVAARQAWSVVGTIKDLKQGYLSQVIHEIVDLMIHYQAVVVL
ANLNFGFKSKRTGIAEKAVYQQFEKMLIDKLNCLVLKDYPAEKVGGVLNPYQLTDQFTSFAK
MGTQSGFLFYVPAPYTSKIDPLTGFVDPFVWKTIKNHESRKHFLEGFDFLHYDVKTGDFILH
FKMNRNLSFQRGLPGFMPAWDIVFEKNETQFDAKGTPFIAGKRIVPVIENHRFTGRYRDLYP
ANELIALLEEKGIVFRDGSNILPKLLENDDSHAIDTMVALIRSVLQMRNSNAATGEDYINSP
VRDLNGVCFDSRFQNPEWPMDADANGAYHIALKGQLLLNHLKESKDLKLQNGISNQDWLAYI
QELRN
SEQ ID NO:35(ddLbCfp1)
MSKLEKFTNCYSLSKTLRFKAIPVGKTQENIDNKRLLVEDEKRAEDYKGVKKLLDRYYLSFI
NDVLHSIKLKNLNNYISLFRKKTRTEKENKELENLEINLRKEIAKAFKGNEGYKSLFKKDII
ETILPEFLDDKDEIALVNSFNGFTTAFTGFFDNRENMFSEEAKSTSIAFRCINENLTRYISN
MDIFEKVDAIFDKHEVQEIKEKILNSDYDVEDFFEGEFFNFVLTQEGIDVYNAIIGGFVTES
GEKIKGLNEYINLYNQKTKQKLPKFKPLYKQVLSDRESLSFYGEGYTSDEEVLEVFRNTLNK
NSEIFSSIKKLEKLFKNFDEYSSAGIFVKNGPAISTISKDIFGEWNVIRDKWNAEYDDIHLK
KKAVVTEKYEDDRRKSFKKIGSFSLEQLQEYADADLSVVEKLKEIIIQKVDEIYKVYGSSEK
LFDADFVLEKSLKKNDAVVAIMKDLLDSVKSFENYIKAFFGEGKETNRDESFYGDFVLAYDI
LLKVDHIYDAIRNYVTQKPYSKDKFKLYFQNPQFMGGWDKDKETDYRATILRYGSKYYLAIM
DKKYAKCLQKIDKDDVNGNYEKINYKLLPGPNKMLPKVFFSKKWMAYYNPSEDIQKIYKNGT
FKKGDMFNLNDCHKLIDFFKDSISRYPKWSNAYDFNFSETEKYKDIAGFYREVEEQGYKVSF
ESASKKEVDKLVEEGKLYMFQIYNKDFSDKSHGTPNLHTMYFKLLFDENNHGQIRLSGGAEL
FMRRASLKKEELVVHPANSPIANKNPDNPKKTTTLSYDVYKDKRFSEDQYELHIPIAINKCP
KNIFKINTEVRVLLKHDDNPYVIGIARGERNLLYIVVVDGKGNIVEQYSLNEIINNFNGIRI
KTDYHSLLDKKEKERFEARQNWTSIENIKELKAGYISQVVHKICELVEKYDAVIALADLNSG
FKNSRVKVEKQVYQKFEKMLIDKLNYMVDKKSNPCATGGALKGYQITNKFESFKSMSTQNGF
IFYIPAWLTSKIDPSTGFVNLLKTKYTSIADSKKFISSFDRIMYVPEEDLFEFALDYKNFSR
TDADYIKKWKLYSYGNRIRIFRNPKKNNVFDWEEVCLTSAYKELFNKYGINYQQGDIRALLC
EQSDKAFYSSFMALMSLMLQMRNSITGRTDVDFLISPVKNSDGIFYDSRNYEAQENAILPKN
ADANGAYNIARKVLWAIGQFKKAEDEKLDKVKIAISNKEWLEYAQTSVKH
SEQ ID NO:36(ddFnCfp1)
MYPYDVPDYASGSGMSIYQEFVNKYSLSKTLRFELIPQGKTLENIKARGLILDDEKRAKDYK
KAKQIIDKYHQFFIEEILSSVCISEDLLQNYSDVYFKLKKSDDDNLQKDFKSAKDTIKKQIS
EYIKDSEKFKNLFNQNLIDAKKGQESDLILWLKQSKDNGIELFKANSDITDIDEALEIIKSF
KGWTTYFKGFHENRKNVYSSNDIPTSIIYRIVDDNLPKFLENKAKYESLKDKAPEAINYEQI
KKDLAEELTFDIDYKTSEVNQRVFSLDEVFEIANFNNYLNQSGITKFNTIIGGKFVNGENTK
RKGINEYINLYSQQINDKTLKKYKMSVLFKQILSDTESKSFVIDKLEDDSDVVTTMQSFYEQ
IAAFKTVEEKSIKETLSLLFDDLKAQKLDLSKIYFKNDKSLTDLSQQVFDDYSVIGTAVLEY
ITQQIAPKNLDNPSKKEQELIAKKTEKAKYLSLETIKLALEEFNKHRDIDKQCRFEEILANF
AAIPMIFDEIAQNKDNLAQISIKYQNQGKKDLLQASAEDDVKAIKDLLDQTNNLLHKLKIFHISQSEDKANILDKDEHFYLVFEECYFELANIVPLYNKIRNYITQKPYSDEKFKLNFENSTLANGWDKNKEPDNTAILFIKDDKYYLGVMNKKNNKIFDDKAIKENKGEGYKKIVYKLLPGANKMLPKVFFSAKSIKFYNPSEDILRIRNHSTHTKNGSPQKGYEKFEFNIEDCRKFIDFYKQSISKHPEWKDFGFRFSDTQRYNSIDEFYREVENQGYKLTFENISESYIDSVVNQGKLYLFQIYNKDFSAYSKGRPNLHTLYWKALFDERNLQDVVYKLNGEAELFYRKQSIPKKITHPAKEAIANKNKDNPKKESVFEYDLIKDKRFTEDKFFFHCPITINFKSSGANKFNDEINLLLKEKANDVHILSIARGERHLAYYTLVDGKGNIIKQDTFNIIGNDRMKTNYHDKLAAIEKDRDSARKDWKKINNIKEMKEGYLSQVVHEIAKLVIEYNAIVVFEDLNFGFKRGRFKVEKQVYQKLEKMLIEKLNYLVFKDNEFDKTGGVLRAYQLTAPFETFKKMGKQTGIIYYVPAGFTSKICPVTGFVNQLYPKYESVSKSQEFFSKFDKICYNLDKGYFEFSFDYKNFGDKAAKGKWTIASFGSRLINFRNSDKNHNWDTREVYPTKELEKLLKDYSIEYGHGECIKAAICGESDKKFFAKLTSVLNTILQMRNSKTGTELDYLISPVADVNGNFFDSRQAPKNMPQDADANGAYHIGLKGLMLLGRIKNNQEGKKLNLVIKNEEYFEFVQNRNN
- 一种压机料车用于瓷抛砖精准布料装置
- 一种小型无人机空中精准发射救援物品的装置及方法
- 一种预制构件生产预埋定位装置
- 一种用于在预制构件中预埋箱体的预埋装置及施工方法
- 一种小型预制构件精准布料装置
- 一种小型预制构件的精准定位装置
