使用酶辅助纳米技术对核酸的视觉和模块检测
文献发布时间:2023-06-19 11:35:49
技术领域
本发明涉及使用酶辅助纳米技术的核酸检测。更具体地,本发明提供了用于对核酸进行特异性检测的方法和装置,其使用两个独立的酶-DNA纳米结构(即易于调节的识别元件和灵敏的通用信号传导元件)的集成回路,以使靶标识别与视觉信号放大解耦。
背景技术
病原体核酸的检测在感染诊断和管理中具有广泛的应用。作为需要长处理时间(即数天)并需要物种特异性方案(例如细菌与病毒)的常规病原体培养的替代方法,核酸技术已在临床实验室中被越来越多地采用,以提供有关(及超出)感染的前所未有的分子信息[Niemz,A.,Ferguson,T.M.和Boyle,D.S.Trends Biotechnol 29:240-250(2011);Nong,R.Y.等人,Expert Rev Proteomics 9:21-32(2012);Zumla,A.等人Lancet Infect Dis14:1123-1135(2014)]。例如,基于核酸的人乳头瘤病毒(HPV)测试对于当代宫颈癌测试是必不可少的。HPV是最常见的性传播感染,是宫颈癌的主要原因[Crosbie,E.J.等人,Lancet382:889-899(2013)]。HPV有多于100种亚型,其中15种被认为具有高恶性风险[Bouvard,V.等人Lancet Oncol 10:321-322(2009)]。HPV感染是一种全球流行病;尽管大部分是良性的,但这些感染中的一些感染可能会发展成致命的宫颈癌。这种复杂的病因、致癌作用和疾病进展主要与以下两个因素有关:1)特定HPV分子亚型的感染,和2)感染的持续性[Bodily,J.和Laimins,L.Trends Microbiol 19:33-39(2011);Schiffman,M.等人Nat Rev DisPrimers 2:16086(2016)]。
当前对病原体核酸的检测几乎仅在大型集中式临床实验室中进行。这种有限的成就源于与常规技术相关的高复杂性和成本。在HPV检测的情况下,商业测定主要利用聚合酶链式反应(PCR,例如Cobas HPV)来扩增和检测特定DNA靶标[Rao,A.等人J Clin Microbiol51:1478-1484(2013);Cui,M.等人J Clin Microbiol 52:2210-2211(2014)]。此类系统不仅需要大型专用设备来进行PCR热循环和荧光测量,而且还需要经过培训的人员进行操作。已经开发出先进的等温扩增测定法来减轻仪器对温度循环的需求,但是,这些测定法有其自身的局限性。例如,环介导的等温扩增(LAMP)具有严格的序列要求并且不易推广[Zhao,Y.等人,Chem Rev 115:12491-12545(2015)]。重要的是,与其他核酸扩增方法一样,LAMP易于产生假阳性(例如,来自引物二聚体形成)。可替代地,可以使用序列特异性信号传导探针(例如荧光Taqman报告物)来提高检测准确性;然而,这些探针昂贵且用法复杂[Gardner,S.N.等人,J Clin Microbiol 41:2417-2427(2003)]。由于每段DNA靶标在靶标扩增过程中的偶联信号传导都需要专用的序列特异性探针,因此所述方法变得成本越来越高,并且难以进行多重化或执行复杂计算[Juskowiak,B.Anal Bioanal Chem 399:3157-3176(2011)]。
需要一种分子平台来使得能够对核酸进行快速的视觉和模块检测。
发明内容
不依赖于靶核酸扩增,本发明的技术以指数方式增强了来自直接和独立靶标杂交的视觉信号。本发明被称为用于核酸的视觉鉴定的酶辅助纳米技术(enVision),其由两个独立的酶-DNA纳米结构(易于调节的识别元件和灵敏的通用信号传导元件)的集成回路组成,使靶标识别与视觉信号放大解耦。选择DNA纳米结构作为功能元件,因为它们可以被设计为具有稳定三维构象以促进各种酶活性,并且即使紧密包装串扰也极小,从而使得能够进行独立操作[Lee,H.等人Nat Nanotechnol 7:389-393(2012);Wang,L.等人,NucleicAcids Res 45:12090-12099(2017);Li,J.等人,Nat Chem 9:1056-1067(2017)]。
在第一方面,提供了一种检测样品中的靶核酸的方法,所述方法包括以下步骤:
(a)提供包含核酸的样品;
(b)提供包含至少一种DNA聚合酶和至少一种识别纳米结构的组合物,其中所述识别纳米结构包含具有保守序列区和可变序列区的DNA聚合酶特异性DNA适体,其中所述可变序列区包含与所述样品中的靶核酸互补的至少10个核苷酸的突出端区段;或者
(c)提供包含至少一种DNA聚合酶和至少一种识别纳米结构的组合物,其中所述识别纳米结构包含DNA聚合酶特异性DNA适体和反向体(inverter)寡核苷酸,其中所述适体具有保守序列区和可变序列区,其中所述可变序列区包含与反向体寡核苷酸的一部分互补并与其形成双链体的至少10个核苷酸的突出端区段,其中所述反向体寡核苷酸比所述适体-反向体双链体长至少一个核苷酸,从而产生反向体突出端,并具有与样品中的靶核酸互补的超过10个核苷酸;
(d)使包含核酸的样品与(b)或(c)的组合物接触,其中所述靶核酸结合至:
(i)(b)中适体的可变序列区促进稳定的适体-DNA聚合酶复合物的形成,从而抑制DNA聚合酶的活性;或者
(ii)(c)中的反向体寡核苷酸使识别纳米结构不稳定,从而使所述DNA聚合酶从所述DNA适体的抑制中释放出来;
(e)提供可与来自步骤(d)的活性DNA聚合酶反应的信号传导纳米结构,其中所述信号传导纳米结构包含可响应DNA聚合酶的自引发部分;
(f)在标记寡核苷酸(dNTP)和信号显影试剂存在下使信号传导纳米结构与步骤(d)的活性DNA聚合酶接触,其中所述活化的DNA聚合酶将标记的寡核苷酸添加到信号传导纳米结构中,并且信号显影试剂结合至并入自引发部分中的标记寡核苷酸;
(g)检测信号显影,其中信号强度指示;
(i)在使用组合物(b)时样品中不存在靶核酸;或者
(ii)在使用组合物(c)时样品中存在靶核酸。
在一些实施方案中,DNA聚合酶是Taq聚合酶,它是一种热稳定DNA聚合酶,按最初分离出它的嗜热细菌栖热水生菌(Thermus aquaticus)来命名。
在一些实施方案中,DNA适体保守序列区包含核酸序列5'-CAATGTACAGTATTG-3'(SEQ ID NO:153)。
在一些实施方案中,所述反向体寡核苷酸比所述适体双链体区长至少一个核苷酸。优选地,所述反向体寡核苷酸的长度为所述适体双链体区的约两倍。
在一些实施方案中,约一半反向体寡核苷酸长度形成适体-反向体双链体,并且约一半形成突出端区段。
在一些实施方案中,根据本发明的任何方面的方法进一步包括提供与靶核酸互补的第二识别纳米结构用于双链体检测,所述靶核酸与样品中第一识别纳米结构的靶核酸不同。
在一些实施方案中,所述方法提供一个或多个另外的识别纳米结构用于多重检测,其各自具有与靶核酸互补的反向体序列,所述靶核酸与样品中其他识别纳米结构的靶核酸不同。表4示出了用于多基因座HPV检测的合适适体和反向体寡核苷酸序列的例子。
在一些实施方案中,错配被引入到反向体寡核苷酸突出端中,以适应靶核酸序列的可变性,其可用于泛检测。表2列出了用于HPV泛检测的合适的适体和反向体寡核苷酸序列的例子。
在一些实施方案中,错配被引入到可变序列区双链体中以赋予强序列特异性,其可用于紧密相关的靶核酸的多重检测,例如用于对病毒进行亚分型。表2列出了用于对HPV进行亚分型的合适的适体和反向体寡核苷酸序列的例子。
在一些实施方案中,所述方法进一步包括提供与一个或多个靶核酸互补的一个或多个另外的识别纳米结构用于多重检测,所述靶核酸与样品中第一识别纳米结构的靶核酸不同。
在一些实施方案中,每个识别纳米结构包含每个识别纳米结构的DNA适体:反向体寡核苷酸:DNA聚合酶比率的组合,以形成选自包括“与”(AND)、“或”(OR)、“非”(NOT)、“与非”(NAND)和“或非”(NOR)的组的逻辑门。表5列出了用于HPV逻辑门检测的合适的适体和反向体寡核苷酸序列的例子。
在一些实施方案中,识别纳米结构的组合以及每个识别纳米结构的DNA适体:反向体寡核苷酸:DNA聚合酶比率选自包括以下的组(i)至(v):
(i)两个纳米结构,每个纳米结构具有1:1:0.5的DNA适体:反向体寡核苷酸:DNA聚合酶比率,以形成“与”逻辑门;
(ii)两个纳米结构,每个纳米结构具有1:1:1的DNA适体:反向体寡核苷酸:DNA聚合酶比率,以形成“或”逻辑门;
(iii)一个纳米结构,其具有1:0:1,以形成“非”逻辑门;
(iv)两个纳米结构,每个纳米结构具有1:0:1的DNA适体:反向体寡核苷酸:DNA聚合酶比率,以形成“与非”门;以及
(v)两个纳米结构,每个纳米结构具有1:0:0.5的DNA适体:反向体寡核苷酸:DNA聚合酶比率,以形成“或非”门。
在一些实施方案中,如上文所述的逻辑门组(i)至(v)的组合允许检测多于两种靶标。
在一些实施方案中,逻辑函数可以用于进行分子计算,例如其中“或”函数改善检测覆盖度和/或“与”函数改善检测特异性。
在一些实施方案中,信号传导纳米结构的自引发部分包含核酸序列:
5'-AGCAGGCAGTTACGGGCTGGTGCGATGAGAGACGCGGAGTGTGGCGGCCGGATAGTAATGACTGCGACCGGTGTACCAGTGGCGTGAGGCAGGTCGTGAGGCGGCGTACGTAGAGCGTTGAGCAGGATGCCAACAGTCGATCAGGACGAGTGCTAACGCATTGTCGATAGCTCAGCTGTCTGAGCTATCGACAATGCGTT-3'(SEQ ID NO:5)。
在一些实施方案中,靶标是至少一种选自包括DNA、RNA、PNA和其他核酸类似物的组的核酸。
在一些实施方案中,靶标是至少一种与非人类或人类疾病、遗传变体、法医学、菌株鉴定、环境和/或食物污染相关的核酸。
在一些实施方案中,靶标是病原体。在一些实施方案中,病原体是病毒。病毒的非限制性例子是如本文所示的HPV,其可以根据本发明的方法检测和/或表征。
在一些实施方案中,在信号传导步骤中所用的dNTP标记是生物素。
在一些实施方案中,信号显影试剂包含融合蛋白,所述融合蛋白包含抗生物素蛋白或其衍生物和酶以及相应底物,所述酶选自包括但不限于HRP、β-内酰胺酶、淀粉酶、β-半乳糖苷酶的组,所述底物选自包括但不限于DAB、TMB、ABTS、头孢硝噻、鲁米诺、淀粉和碘的组,其中信号可以作为但不限于颜色、荧光、发光或电化学变化进行测量和定量。
在一些实施方案中,在步骤(a)之前扩增样品中的靶核酸。
在一些实施方案中,样品中的靶核酸的扩增通过嵌套不对称PCR或等温扩增方法进行。
在一些实施方案中,检测和信号传导步骤在物理上和/或空间上是分开的。
在一些实施方案中,检测和信号传导纳米结构连接到底物。
在一些实施方案中,信号传导纳米结构连接至珠。
在一些实施方案中,检测和信号传导纳米结构连接到微流体装置或侧流装置。
在一些实施方式中,方法步骤a)至e)中的一个或多个在16℃至40℃范围的温度下进行。在一些实施方案中,步骤d)至g)在16℃至40℃范围的温度下进行。
在一些优选实施方案中,方法步骤a)至e)中的一个或多个在环境温度下进行。在一些优选实施方案中,步骤d)至g)在环境温度下进行。
在一些实施方案中,步骤a)至d)独立于步骤e)至g)。在步骤a)至d)中可以使用响应于刺激来释放酶活性的替代性机制。替代性机制可包括通过与酶的抑制剂的接近、温度变化、替代性DNA酶等来抑制酶。在步骤e)至g)中可以使用读出酶活性的替代性机制来检测步骤a)至d)活化后的不同刺激物。
根据本发明的另一方面,提供了一种分离的信号传导纳米结构核酸,其包含含有以下核酸序列的自引发部分:
5'-AGCAGGCAGTTACGGGCTGGTGCGATGAGAGACGCGGAGTGTGGCGGCCGGATAGTAATGACTGCGACCGGTGTACCAGTGGCGTGAGGCAGGTCGTGAGGCGGCGTACGTAGAGCGTTGAGCAGGATCCAACAGTCGATCAGGACGAGTGCTAACGCATTGTCGATAGCTCAGCTGTCTGAGCTATCGACAATGCGTT-3'(SEQ ID NO:5)。
根据本发明的另一方面,提供了一种装置,其包括在第1位置的如上文所定义的所述组合物b)或组合物c)、在第2位置处连接的如上文所定义的所述信号传导纳米结构,以及用于混合所述检测纳米结构和样品核酸以将活性酶释放到所述第2位置的中间级。
在一些实施方案中,中间级是连接第1位置和第2位置的流体通道。
根据上文的装置改善了根据本发明的检测样品中的特定核酸的方法的反应动力学和灵敏度,部分原因是优化了不同酶活性的持续时间。
根据本发明的替代性装置配置包括在第一位置中的所述检测和所述信号传导纳米结构,其中所述信号传导纳米结构连接至珠并与第一位置中的检测纳米结构混合,之后可以将珠捕获在该位置或第2位置的适当位置以在其给定位置释放活性酶。
在一些实施方案中,所述装置选自包括微流体装置和侧流装置的组。
优选地,所述装置是如上文所述的微流体装置。此种装置的例子在图1、图6和图7中示出。参考图1和图6,将理解的是,在一些实施方案中,第1位置可为待测试样品被加载到微流体装置上并与预加载的检测纳米结构接触的位置。然后样品和检测纳米结构可以混合并相互作用,同时穿过中间流体通道,从而使得阳性检测释放出活性聚合酶。然后,活性酶进入预加载有固定的通用信号传导纳米结构的第2位置(反应室),其中所述活性酶聚合生成包含标记寡核苷酸的信号分子,并且信号显影试剂结合至并入的标记寡核苷酸。
根据本发明的另一方面,提供了一种核酸检测试剂盒,其包括:
(a)包含至少一种DNA聚合酶和至少一种识别纳米结构的组合物,其中所述识别纳米结构包含具有保守序列区和可变序列区的DNA聚合酶特异性DNA适体,其中所述可变序列区包含与靶核酸互补的至少10个核苷酸的突出端区段;和/或
(b)包含至少一种DNA聚合酶和至少一种识别纳米结构的组合物,其中所述识别纳米结构包含DNA聚合酶特异性DNA适体和反向体寡核苷酸,其中所述适体具有保守序列区和可变序列区,其中所述可变序列区包含与所述反向体寡核苷酸的一部分互补并与其形成双链体的至少10个核苷酸的突出端区段,其中所述反向体寡核苷酸比所述适体-反向体双链体长至少一个核苷酸并且具有与靶核酸互补的超过10个核苷酸;和/或
(c)可与活性DNA聚合酶反应的信号传导纳米结构,其中所述信号传导纳米结构包含可响应DNA聚合酶的自引发部分;和/或
(d)标记寡核苷酸(dNTP)和信号显影试剂,其中活性DNA聚合酶将标记寡核苷酸添加至信号传导纳米结构,并且信号显影试剂结合至并入自引发部分中的标记寡核苷酸。
在一些实施方案中,本发明的核酸检测试剂盒包括根据本发明的任何方面如上文所定义的(a)至(d)。
在一些实施方案中,本发明的核酸检测试剂盒被配置到根据本发明的任何方面如上文所定义的装置中。
根据本发明的另一方面,提供了一种诊断受试者的疾病的方法,所述方法包括以下步骤:
(a)提供来自受试者的包含核酸的样品;
(b)提供包含至少一种DNA聚合酶和至少一种识别纳米结构的组合物,其中所述识别纳米结构包含具有保守序列区和可变序列区的DNA聚合酶特异性DNA适体,其中所述可变序列区包含与所述样品中的靶核酸互补的至少10个核苷酸的突出端区段;或者
(c)提供包含至少一种DNA聚合酶和至少一种识别纳米结构的组合物,其中所述识别纳米结构包含DNA聚合酶特异性DNA适体和反向体(inverter)寡核苷酸,其中所述适体具有保守序列区和可变序列区,其中所述可变序列区包含与反向体寡核苷酸的一部分互补并与其形成双链体的至少10个核苷酸的突出端区段,其中所述反向体寡核苷酸比所述适体-反向体双链体长至少一个核苷酸,从而产生反向体突出端,并具有与样品中的靶核酸互补的超过10个核苷酸;
(d)使包含核酸的样品与(b)或(c)的组合物接触,其中所述靶核酸结合至:
(i)(b)中适体的可变序列区促进稳定的适体-DNA聚合酶复合物的形成,从而抑制DNA聚合酶的活性;或者
(ii)(c)中的反向体寡核苷酸使识别纳米结构不稳定,从而使所述DNA聚合酶从所述DNA适体的抑制中释放出来;
(e)提供可与来自步骤(d)的活性DNA聚合酶反应的信号传导纳米结构,其中所述信号传导纳米结构包含可响应DNA聚合酶的自引发部分;
(f)在标记寡核苷酸(dNTP)和信号显影试剂存在下使信号传导纳米结构与步骤(d)的活性DNA聚合酶接触,其中所述活化的DNA聚合酶将标记的寡核苷酸添加到信号传导纳米结构中,并且信号显影试剂结合至并入自引发部分中的标记寡核苷酸;
(g)检测信号显影,其中信号强度指示;
(i)使用组合物(b)时样品中不存在靶核酸;或者
(ii)使用组合物(c)时样品中存在靶核酸
(h)当检测到样品中存在靶核酸时诊断受试者患有疾病。
在一些实施方案中,所述疾病是致病性疾病。在一些实施方案中,所述疾病是HPV相关疾病。
在一些实施方案中,使用表2、表4和表5中列出的一种或多种适体和/或反向体寡核苷酸检测HPV。
根据本发明的任何方面,适体和/或反向体和/或信号传导纳米结构寡核苷酸中的至少一者是从其天然核酸在结构上和/或化学上修饰的。
在一些实施方案中,所述结构和/或化学修饰选自包括以下的组:在合成期间添加硫代磷酸酯(PS)键、2'-O-甲基修饰、亚磷酰胺C3间隔基和5'添加例如氨基、硫醇、亚磷酰胺(acryldite)或叠氮化物基团。
附图说明
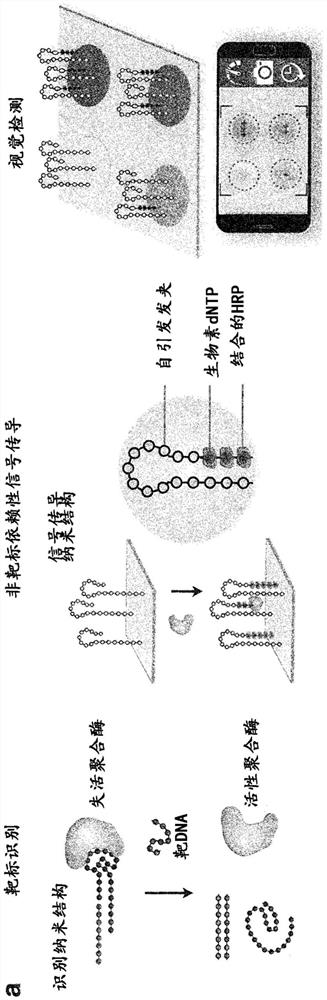
图1示出病原体核酸的视觉和模块检测。(a)enVision系统由一系列酶-DNA纳米结构组成,以实现靶标识别、非靶标依赖性信号传导和视觉检测。纳米结构被设计来将识别与信号传导解耦。识别纳米结构是杂合复合物,由失活适体和Taq DNA聚合酶构成。在互补靶DNA的存在下,复合物解离以活化聚合酶活性。活性聚合酶以非靶标依赖性方式前进以延长通用的自引发信号传导纳米结构。并入修饰的脱氧核苷酸(dNTP),以将辣根过氧化物酶(HRP)固定在信号传导纳米结构上。添加光学底物后,视觉信号可以被酶促增强,通过肉眼检测到并通过智能手机相机进行定量。照片(插图)示出在智能手机应用程序中在无(-)和变化(+)量的靶DNA的存在下,实际视觉读出的例子。(b)enVision微流体系统的示意图。所述平台被设计以补充模块enVision工作流。在入口处预加载有特异性识别纳米结构的独立测定盒可以按需要安装在共用信号传导盒上。共用盒装有通用信号传导纳米结构,所述纳米结构固定在嵌入的膜上,用于进行非靶标依赖性信号传导和视觉检测。盒滑动方向由箭头指示。(c)为多功能测定集成和并行处理而开发的微流体enVision原型的照片。
图2示出使用enVision进行核酸定量。(a)识别纳米结构的组装和活性。组装识别纳米结构,并将其与互补或乱序靶DNA序列一起孵育,以确定所得的聚合酶缔合和活性。(上图)分子结合的实时传感图。进行了一系列操作,即适体固定、聚合酶添加以及与靶DNA序列一起孵育。通过生物层干涉术原位监测分子结合,以确定聚合酶缔合。(下图)在每次操作结束时,通过平行实验使用Taqman测定法(对Taqman探针的5'核酸外切酶降解的荧光测量)确定对应的聚合酶活性。注意到与互补靶DNA一起孵育后聚合酶活性完全恢复。(b)信号传导纳米结构活性。在比较实验中,用等浓度的活性DNA聚合酶处理自引发信号传导纳米结构及其大小相似的线性模板。通过差异性放置的Taqman探针(位置如具有黑色中心的灰色点所指示)的5'核酸外切酶降解,在远离起始引发位点的不同核苷酸位置处确定聚合酶活性。(c)enVision系统的检测灵敏度。通过滴定已知量的靶DNA并测量其相关视觉信号来确定检测限(虚线)。所有视觉信号都通过智能手机获取。(d)enVision和关于不同数量的靶DNA的荧光测量之间的相关性。视觉信号与荧光信号匹配良好(R
图3示出enVision的可编程性。(a)可编程识别纳米结构的示意图。杂合结构由保守序列区和可变区(双链体和突出段区段)组成,所述保守序列区结合以使DNA聚合酶(pol)失活,可以使所述可变区与靶DNA互补。没有按比例绘制。(b)可变区中靶标错配的影响。将设计成具有不同数量的与可变区的错配的合成DNA靶标与识别纳米结构一起孵育。所有信号均针对互补DNA靶标的信号进行归一化(0个错配)。针对双链体区的错配产生显著更低的信号(*P<0.05,**P<0.005,***P<0.0005,n.s.不显著,Student氏t检验)。(c)泛HPV识别。根据HPV共有基因组开发两个泛HPV识别纳米结构,以具有不同数量的与从六种HPV亚型获得的DNA靶标的错配。所有错配都映射到双链体和突出端区域。纳米结构1在突出端区域中容纳更多的错配,表现出更好的泛识别能力。所有信号均针对互补DNA靶标(阳性)进行归一化。(d)针对特定HPV亚分型的enVision和qPCR测量的比较。根据HPV基因组的高度可变区设计特定的纳米结构,并在敏感双链体区内包含序列变异。通过enVision智能手机平台得到颜色强度(左图)以及通过
图4示出用于多基因座覆盖度的多重enVision。(a)HPV基因组图中的新探针基因座。HPV基因组由七个早期表达(E)基因和两个衣壳蛋白(L)基因构成。针对每种HPV亚型设计新纳米结构识别探针,以分别鉴定E1、L1和L2基因座。(b)用于高覆盖度、多基因座检测的多重enVision测定。将从CaSki细胞(左图,HPV 16阳性)和HeLa细胞(右图,HPV 18阳性)获得的基因组DNA直接与单独识别探针(分别为E1、L1和L2)或三种探针的集合(组合的)一起孵育,以确定细胞HPV感染状态。与任何个体化测定法相比,组合探针显示出显著更高的信号(*P<0.0005,n.s.不显著,Student氏t检验;n.d.未检测)。所有信号均归一化为相对于其相应组合信号的百分比。(c)细胞系中的HPV亚分型。使用多重enVision测定法针对不同HPV亚型直接剖析细胞系的基因组DNA。如先前文献所报道,所述测量与已知的细胞系HPV感染密切相关(深灰色:存在,浅灰色:不存在)。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图5示出患者样品的分子剖析。(a)从临床宫颈管刷样品测量HPV 16和HPV 18信号(n=35)。示出L1特异性信号,用于与临床金标准进行比较。有关所有临床样本的多基因座测量,请参见图19。(b)使用HPV 16和HPV 18L1基因座测定的接受者工作特征(ROC)曲线确定检测准确性。在Youden氏指数截止点下,HPV 16测定显示出92.9%灵敏度(13/14)和90.5%特异性(19/21),并且HPV 18测定显示出83.3%灵敏度(5/6)和100%特异性(29/29)。(c)所有患者均进行基因座特异性HPV 16enVision测定(L1、L2和E1基因座测定)。示出L1阳性(左图)和L1阴性(右图)患者的代表性例子。注意在L1阴性患者的亚组中,包含L2和E1基因座测定可以提高检测覆盖度,以鉴定先前无法检测到的感染。通过独立的
图6示出enVision微流体平台的示意图。(a)所述平台的分解示意图和(b)截面侧视图。所述平台包括独特的测定盒和共用信号传导盒。每个测定盒预加载有独特的识别纳米结构,并含有蛇形微通道以改善混合。将聚碳酸酯膜嵌入共用盒中,以固定信号传导纳米结构。由抽取隔片驱动的从样品入口到共用出口的流体流动由箭头表示。
图7示出根据本发明的装置的操作的示意图。
图8示出识别纳米结构的活性。(a)将不同量的抑制性适体添加到固定量的聚合酶(5个单位)中,以确定在使抑制效果最大化同时使识别纳米结构复合的最佳比率。(b)对于优化的纳米结构复合物,孵育不同量的互补DNA靶标以及作为对照的乱序寡核苷酸序列。注意,仅互补靶标导致聚合酶活性(条形图)强且成比例的增加,而乱序寡核苷酸序列产生的活性可忽略不计(在基线时)(*P<0.05、***P<0.0005、****P<0.00005,Student氏t检验)。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图9示出信号传导纳米结构的退火。(a)发夹信号传导纳米结构及其等同大小的线性对应物(具有过量引物)在室温下通过8%天然凝胶电泳进行拆分。分析了每个样品中引发和未引发部分的谱带强度(*****P<0.000005,Student氏t检验)。(b)自引发信号传导纳米结构及其线性对应物的熔解曲线分析。随着反应温度升高记录
图10示出视觉和荧光读出。(a)酶反应的优化。在对照(水)和不同数量的DNA靶标存在下,测量DNA聚合酶(DNA pol,上图)和HRP(下图)的实时活性。通过
图11示出嵌套式不对称扩增。(a)嵌套扩增的示意图。为了显著扩大微量样品的单链DNA群体,采用嵌套式不对称PCR扩增。首先在均等浓缩的双引物存在下对样品进行指数扩增,并且随后使用过量的单引物进行线性扩增。(b)嵌套式不对称扩增的效率。在8%PAGE凝胶上分析来自(1)1pmol、(2)100fmol、(3)10fmol、(4)1fmol、(5)100amol、(6)10amol、(7)1amol和(8)无合成HPV16序列的模板对照的扩增产物。上箭头指示较大的双链产物,而下箭头对应于单链产物。
图12示出基于核酸序列的扩增(NASBA)。(a)NASBA的示意图。首先通过T7 RNA聚合酶针对RNA转录和单链RNA扩增引发DNA靶标。然后将RNA产物逆转录成cDNA,然后进行RNA消化以产生单链DNA产物。(b)NASBA的效率。在8%PAGE凝胶上分析来自(1)1pmol、(2)100fmol、(3)10fmol、(4)1fmol、(5)100amol、(6)10amol、(7)1amol和(8)无合成HPV 16序列的模板对照的扩增产物。箭头表示DNA产物的预期大小。
图13示出
图14示出直接RNA检测。测试了开发的enVision测定法(即HPV 16和HPV 18测定法),以用于直接检测不同HPV亚型的DNA(左图)和RNA(右图)。所有RNA靶标均无需任何cDNA转化即可直接使用。靶标被认为是互补靶标或脱靶。enVision测定显示对RNA靶标的特异性和直接检测,其信号与DNA靶标的信号相当,而两种靶标的脱靶信号均接近基线。所有信号均针对每种测定的阳性DNA信号进行归一化,以进行相对比较。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图15示出enVision逻辑门。通过改变识别纳米结构的组合以及每个纳米结构中不同组分(即适体、反向体和聚合酶)的比率,对以下逻辑计算进行编程:(a)“与”门、(b)“或”门、(c)HPV 16的“非”门、(d)HPV 18的“非”门、(e)“与非”门和(f)“或非”门。对于设计的每个门,都说明了用于建立配置的组分(每个图的右上图)。用从HPV 16(靶标1)和HPV 18(靶标2)分离的DNA靶标的不同组合测试每个门。所有靶标组合及其预期计算输出都汇总在相应的真值表中(每个图的左下图)。观察到的enVision信号(每个图的右下图)显示出与预期输出的良好一致性。如先前所述,将所有信号相对于适当对照(无靶标对照)进行归一化。高于检测阈值(即比背景信号高3x s.d.)的归一化信号被视为真实信号(灰色条形);否则,称为假信号(黑色条形)。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图16示出使用enVision“或”和“与”逻辑门来检测多个靶标的组合。通过改变识别纳米结构的组合以及每个纳米结构中不同组分(即适体、反向体和聚合酶)的比率,展示了使用逻辑门同时检测多个靶标。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图17示出基因组DNA的多重扩增。来自CaSki基因组DNA的扩增产物的完整凝胶电泳图。在以下不同HPV 16基因座引物的存在下扩增基因组DNA:(1)E1;(2)L1;(3)L2;(4)E1、L1和L2的组合引物;以及(5)无引物。泳道7加载有15bp DNA梯。箭头指示50bp谱带的位置。
图18示出基因组DNA的多重enVision检测。直接将等量的细胞基因组DNA(上图:CaSki,中图:HeLa,下图:C33-a)与针对不同HPV亚型(左图:HPV 16,右图:HPV 18)的特异性识别纳米结构一起孵育。用单独的识别纳米结构(即E1、L1和L2)处理样品,或者用三种结构的集合(组合的)同时处理样品。将所有信号归一化为对于每种HPV亚型观察到的最大信号的百分比。如先前文献所报道,所述多重测量与已知的细胞系HPV感染密切相关(水平灰色条形:存在,水平白色条形:不存在)。注意,单基因座测量可能会遗漏阳性感染(例如,HeLa,HPV 18基因座L2)。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图19示出enVision和LAMP设计和性能的比较。(a)在不同HPV亚型的E1、L1和L2基因座的高度可变区域中发现的探针选项的比较。鉴定用于enVision系统(左图)和LAMP(右图)的探针选项。enVision平台不仅产生更多的探针选择,而且为所有测试区域提供了全面覆盖度。(b)在细胞基因组DNA中针对HPV亚分型的enVision性能(左图)与排名最高的LAMP引物集的性能(右图)的比较。EnVision具有83.3%灵敏度(5/6)和100%特异性(12/12),而LAMP具有50.0%灵敏度(3/6)和75.0%特异性(9/12)。在已知感染的不同细胞系中进行多基因座测量。如先前所述,将所有信号相对于适当对照(无靶标对照)进行归一化。注意,与已知细胞感染状态相比,仅enVision技术显示出准确的HPV亚分型(水平灰色条形:存在,水平白色条形:不存在)。LAMP显示明显的假阳性(例如CaSki细胞,HPV 18;HeLa细胞,HPV16)。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图20示出临床样品中的多重enVision检测。在临床宫颈管刷样品(n=35例患者)中进行了高覆盖度的多基因座enVision测定(同时检测E1、L1和L2),以对HPV 16(上图)和HPV 18(下图)进行分子亚分型。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
图21示出使用
图22示出在双链体区段中具有不同数量的核苷酸的适体的DNA聚合酶抑制的效率。所述效率在双链体区段中约20个核苷酸处开始进入平台期。所有测量均一式三份进行,并且数据显示为平均值±s.d.。
具体实施方式
本发明的优点是多方面的。首先,感测机制-所述技术在靶标杂交后立即活化,从而转导显著信号增强。通过并入双酶信号传导级联(例如DNA聚合酶和辣根过氧化物酶),所述技术得益于两种酶的活性,用于各种不同靶标的视觉检测。即使不存在靶核酸扩增,enVision平台也产生快速且灵敏的颜色读出,其为肉眼可见并可通过智能手机定量。由于这种感测机制,所述技术可应用于准确且直接地检测不同类型的核酸(例如DNA和RNA,无需cDNA转化),而无需大量设备。其次,测定可编程性-通过将识别与信号传导解耦,所述技术能够实现模块检测和多功能集成。新的测定可以通过仅修改高度可编程识别元件中的单个序列区域来容易地设计,并配置为进行逻辑计算。通用信号传导元件可用于所有视觉测量。当在可配置的微流体平台上实施时,enVision技术因此显示出高检测灵敏度和可编程性,从而能够在室温下对感染细胞的多种病原体核酸进行视觉剖析。通过在单个反应中进行多基因座多路复用,进一步开发一种高覆盖度的enVision系统,以询问各种病毒-宿主基因组整合基因座。使用HPV作为临床模型,证实了所述技术通过改善亚型间分化以及亚型内检测覆盖度用于患者宫颈内样品中的感染的分子分型的临床实用性。
为了方便起见,将在本说明书中提及的文后参考书目以参考文献列表的形式列出并且附加在实施例的末尾处。将此类文后参考书目的全部内容通过引用并入本文。
除非另外定义,否则本文中使用的所有技术术语和科学术语具有与本发明所属领域的技术人员通常所理解的相同的含义。为方便起见,在此收集了在说明书、实施例和所附权利要求中使用的某些术语。
必须指出的是,除非上下文另外清楚地指出,否则如在本文以及在所附权利要求书中所用的,单数形式“一个/一种(a)”、“一个/一种(an)”以及“所述(the)”包括复数指示物。因此,例如,提及“一个靶序列”包括多个此类靶序列,并且提及“一种酶”是提及本领域技术人员已知的一种或多种酶及其等同物,等等。
如本文所用,术语“适体”是指单链DNA或RNA分子。适体能够以高亲和力和特异性结合各种分子。例如,如本文所用,在不存在靶DNA的情况下,DNA适体与聚合酶牢固结合以抑制聚合酶活性。
如本文所用,短语“核酸”或“核酸序列”是指寡核苷酸、核苷酸、多核苷酸或其任何片段;基因组或合成起源的DNA或RNA(其可以是单链或双链的并且可以表示正义链或反义链);肽核酸(PNA);或任何DNA样或RNA样材料。
如本文所用,术语“寡核苷酸”是指至少约6个核苷酸至60个核苷酸、优选地约15至30个核苷酸、最优选地约20至25个核苷酸的核酸序列,其可用于PCR扩增或在杂交测定或微阵列中。如本文所用,术语“寡核苷酸”基本上等同于术语“扩增子”、“引物”、“寡聚体”和“探针”,与这些术语在本领域中通常被定义一样。
如本文所用,术语“反向体序列”或“反向体寡核苷酸”是指与靶核酸序列互补的寡核苷酸,其一部分参与形成双链体并且一部分参与突出端。本文显示,双链体和突出端序列各自具有20个核苷酸的较长序列通过稳定适体与DNA聚合酶的结合来稳健地产生其抑制作用。可在环境温度下在互补靶标存在下去除这种抑制作用。反向体序列的存在/不存在确定识别元件的功能状态(例如,开或关状态)。在反向体序列存在下,聚合酶活性用靶标开启;在反向体序列不存在下,聚合酶活性可以用靶标关闭。图3a示出聚合酶、适体和反向体排列的示意图。
如本文所用,术语“可变序列区”是指决定对靶序列的序列特异性(即,定义可以被识别的靶序列)的区域。反向体序列和适体序列的一部分被包含在此可变序列区内。可以更改此区域以能够检测新靶标。当不使用反向体时,“可变序列区”是指适体上与靶核酸互补的突出端区段。
如本文所用,术语“样品”以其最广泛含义使用。例如,疑似含有HPV基因组序列(包括但不限于HPV 6、16、18、31、33和5)的生物样品可包括体液;细胞提取物、从细胞中分离的染色体、细胞器或膜;细胞;基因组DNA、RNA或cDNA(在溶液中或与固体支持物结合);组织;组织印迹;等。
应当理解,可以对本发明中使用的寡核苷酸进行结构和/或化学修饰,以例如在执行本发明的方法期间延长其在可能含有核酸酶的样品中的活性,或改善在试剂盒中的贮藏期限。因此,可对根据本发明使用的适体和/或反向体和/或信号传导纳米结构或任何寡核苷酸引物或探针进行化学修饰。在一些实施方案中,所述结构和/或化学修饰包括添加标签,例如荧光标签、放射性标签、生物素、5'尾部;在合成期间添加硫代磷酸酯(PS)键、2'-O-甲基修饰和/或亚磷酰胺C3间隔基。
例如,对信号传导寡核苷酸进行修饰,以连接具有5'氨基的化学物质。可以在5'端进行其他连接修饰,例如硫醇、亚磷酰胺、叠氮化物等。
如本文所用,术语“包含”或“包括”应解释为详细说明如所提到的所述特征、整数、步骤或组分的存在,但不排除一个或多个特征、整数、步骤或组分或其组的存在或添加。然而,在本公开文本的上下文中,术语“包含”或“包括”还包括“由……组成”。词语“包含(comprising)”的变体(诸如“包含(comprise)”和“包含(comprises)”)以及“包括(including)”的变体(诸如“包括(include)”和“包括(includes)”)具有相应变化的含义。
实施例
大体上遵循本领域已知的并且未具体描述的标准分子生物学技术,如Green和Sambrook,Molecular Cloning:A Laboratory Manual,Cold Spring Harbor Laboratory,纽约(2012)中所述。
实施例1
方法
识别纳米结构表征
所有序列都可在表1-7中找到,并购自Integrated DNA Technologies(IDT)。为了制备识别纳米结构,在50mM NaCl、1.5mM MgCl
信号传导纳米结构表征
与相似大小的线性模板的稳定性相比,信号传导纳米结构的稳定性以两种不同的方式确定。首先,在室温下对两个反应(即纳米结构和线性模板)进行退火,并通过凝胶电泳分析每个反应中的引发和未引发部分。其次,通过以下方式进行熔解曲线分析:将寡核苷酸与10,000X稀释的
enVision装置制造
包括4个测定盒和共用盒的原型enVision装置分别由聚二甲基硅氧烷(PDMS,DowCorning)和聚(甲基丙烯酸甲酯)(PMMA)制造。通过将两个PDMS片层等离子粘合在一起(50mTorr,50W,1min)来制造每个测定盒。通过常规光刻法使用SU-8光刻胶和硅晶片来制备200μm厚的铸模。通过将未固化的PDMS(10:1弹性体基质与固化剂的比率)倒在铸模上来复制反应室和微通道。聚合物固化(75℃持续30分钟)后,将两个PDMS片组装在一起。通过CO
信号传导纳米结构固定
将胺修饰的寡核苷酸(IDT)用于功能化。为了将寡核苷酸固定在微流体装置上,使用聚苯乙烯珠来增加用于锚定寡核苷酸的表面积。将3μm羧酸修饰的聚苯乙烯珠(Spherotech)在PBS缓冲液中洗涤,然后将其重悬在MES缓冲液(Thermo FisherScientific)中。将所述珠在室温下用EDC/磺基-NHS(Pierce Biotechnology)活化15分钟,并在室温下与过量5'胺修饰的寡核苷酸在PBS缓冲液中孵育2小时。然后通过离心洗涤所述珠并将其重悬于10mM Tris-EDTA缓冲液中。
装置准备
为了准备装置进行操作,将珠固定的信号传导纳米结构冻干并将修饰的dNTP反应混合物冻干到共用盒上。在冻干前,将所有装置均用乙醇和PBS缓冲液冲洗。简言之,通过混合dATP、dGTP、dTTP和25%dCTP:75%生物素-16-氨基烯丙基-2'-dCTP(Trilinkbiotechnologies)来制备4mM储备dNTP反应混合物。将50μg DNA功能化的聚苯乙烯珠(3μm,如上制备)在20μl dNTP混合物中引入装置的聚碳酸酯膜上,并使其在真空下干燥30分钟(Labconco FreeZone)。
enVision平台的操作
操作步骤如图7所示。将核酸样品添加到单独测定盒的入口,每个测定盒在pH 8.5的含有50mM NaCl、1.5mM MgCl
由于enVision工作流中的关键步骤主要是孵育和洗涤,其中不需要精确的流速,因此可以使用抽取隔片(Thermo Fisher)来通过负压驱动流体运动。所有反应均在室温下完成,并包括适当的阴性对照。直接使用移动智能手机(Samsung)对样品和对照的视觉读出进行成像。将彩色图像转换为灰度级,并使用具有捕获的信号传导DNA珠的每个膜区域的平均(均值)黑色像素强度用于信号的数字定量。
数据归一化
其中
I(靶标或对照,t
I(靶标或对照,t
将样品的信号强度差(视觉颜色或荧光)计算为在给定时间点(t
序列设计
HPV基因组序列通过以下参考编号从GenBank获得(HPV 6:AF092932.1,HPV 11:FR872717.1,HPV 16:K02718.1,HPV 18:AY262282.1,HPV 31:J04353.1,HPV 33:M12732.1,HPV 58:D90400.1,HPV 66:U31794.1)。使用Clustal Omega软件进行多序列比对[Sievers,F.等人Mol Syst Biol 7:539(2011)]。从所产生的序列比对,定义了高度保守序列和发散序列,以分别设计泛识别和亚型特异性识别纳米结构。简言之,确定高度保守性区域(20bp,<3个不相同的碱基对),其上游或下游紧密侧接保守性较低的区域(20bp,<6个不相同的碱基对)。选择最保守的基序来形成泛HPV识别纳米结构的识别结构域。为了设计亚型特异性识别纳米结构,定位高发散区(40bp,<12个相同的碱基对)。使用类似的方法来定义HPV基因组的L1、L2和E1基因中的发散区,并设计每种HPV亚型的基因座特异性识别纳米结构,以提高检测覆盖度。所有设计的序列都可以在表中找到。
错配表征
为了评价识别纳米结构中双链体和突出端区域的错配灵敏度,在互补DNA靶标中以每两个核苷酸间隔使碱基对随机突变,并使用enVision平台测量所产生的信号变化。为了确定识别纳米结构的泛HPV检测能力,使用六种HPV亚型(HPV 6、16、18、31、33和58)的序列以及完全互补序列(阳性对照)作为靶序列。将错配核苷酸分别映射到双链体和突出端区域,并测量所产生的信号变化。
不对称扩增
在微量样品的情况下,通过嵌套不对称PCR扩增制备单链DNA。扩增可以在<1小时内完成。对于指数扩增,使用0.8μM正向和反向引物(IDT)和在含有2.5mM dNTP的1X
等温不对称扩增
为了实现等温不对称扩增,采用基于核酸序列的扩增(NASBA)。整个过程可以在<1小时内完成。使用在含有2.5mM dNTP、5mM NTP、6mM MgCl
使用HPV亚型特异性序列进行此比较。如上文所述,在enVision平台上对合成寡核苷酸进行亚型特异性检测。对于qPCR实验(Applied Biosystems),设计特定PCR引物对,以跨越每种HPV亚型的相同靶序列。简言之,使用以下热循环方案将100fmol的合成模板与5单位的
直接RNA检测
通过T7 RNA聚合酶(New England Biolabs)制备RNA靶标。简言之,将具有T7启动子引物(表3)(1pmol)的退火DNA模板与1X缓冲液中的T7RNA聚合酶在37℃下孵育2小时,然后添加2单位无RNA酶的DNA酶I(Promega)并在41℃下孵育20分钟。使产生的RNA在0.3M乙酸钠中沉淀。将此混合物用1体积的异丙醇处理,并在4℃下孵育1小时,之后离心(在4℃下12,000g持续30分钟)。将沉淀以冷却的乙醇(75%)洗涤两次,如先前所述离心,并在室温下风干。当溶于无核酸酶水中时,通过吸光度测量(Nanodrop,Fisher Scientific)来确定最终的RNA浓度。对于直接RNA检测,在1单位的SUPERase RNA酶抑制剂(SUPERase.In
构建enVision逻辑门
图15和表5分别汇总了所有逻辑门配置和组分。简言之,使用预组装的识别纳米结构来演示“与”、“或”、“非”、“非与”和“或与”函数。在构建不同门时,改变所使用的识别纳米结构的组合以及每种杂合纳米结构中组分的比率(即DNA适体:DNA反向体:Taq聚合酶)以编程不同的计算函数。具体而言,为了准备“与”门,使用两种识别纳米结构,并且每种纳米结构的组分以比率(1:1:0.5)混合;“或”门:两种结构(1:1:1);“非”门:一种结构(1:0:1);“与非”门:两种结构(1:0:1);并且“或非”门:两种结构(1:0:0.5)。如先前所述,预先组装所有识别纳米结构。通用信号传导纳米结构通常用于所有逻辑门。如真值表所述,用DNA靶标的不同组合测试门配置,并将enVision信号与预期输出进行比较。如上文所述将信号归一化。高于检测阈值(即,背景信号的>3X s.d.)的归一化信号被视为真实信号;否则称为假信号。
细胞培养和DNA提取
所有人类细胞系均获自美国典型培养物保藏中心;CaSki(ATCC CRL-1550)、SiHa(ATCC HTB-35)、HeLa(ATCC CCL-2)、PZ-HPV-7(ATCC CRL2221)。CaSki细胞在RMPI-1640中生长。SiHa、HeLa和C33-a在补充有10%FBS和青霉素-链霉素的伊格尔最低基础培养基中(Corning)。PZ-HPV-7在补充牛垂体提取物和人重组表皮生长因子的角质形成细胞无血清培养基中(ATCC)。对所有细胞系进行测试,并且其均无支原体污染(
与LAMP的比较
在“序列设计”部分中鉴定的HPV基因组的相同发散区上,使用自动化在线LAMP引物设计服务器(PrimerExplorer.jp)以默认设置[Tomita,N.等人,Nat Protoc 3:877-882(2008)]鉴定可能的LAMP扩增引物。将由软件返回的LAMP引物集数量与可在同一区域内设计的enVision识别纳米结构数量进行比较。对于随后的功能性测试,进一步将排名最高的LAMP引物集的性能与enVision系统的性能进行比较。对于每个LAMP扩增,使用0.2μM的引物集中的每个引物和100ng从已知HPV感染的细胞系中提取的纯化的基因组DNA。将此混合物与1X等温扩增缓冲液II、6mM MgSO
临床样品处理
从35名个体收集宫颈管刷样品,在BD SurePath
凝胶电泳
将DNA样品用适量的6X上样染料(Thermo Fisher)稀释,并在100V下在具有TAE缓冲液的8%聚丙烯酰胺凝胶(Thermo Scientific)上运行。将所述凝胶用TAE缓冲液中10,000X稀释的
统计分析
所有测量均一式三份进行,并且数据显示为平均值±标准差。所有显著性检验均通过两尾Student氏t检验进行。对于样品间的比较,各自测试多对样品,并使用Bonferroni校正来调整多重假设测试的所得P值。调整后P<0.05的值被确定为显著。临床研究的接受者工作特征(ROC)曲线由患者剖析数据生成,并通过绘制灵敏度对比(1-特异性)的曲线来构建,并使用梯形法则计算曲线下面积(AUC)的值。使用临床报告(即Cobas HPV)作为分类器(真阳性和真阴性)。从最接近相应ROC曲线左上部分(完美的灵敏度或特异性)的点确立每种标记物的最佳阈值。使用标准公式计算检测灵敏度、特异性和准确性。使用R程序包(版本3.4.2)进行统计分析。
实施例2
enVision平台
enVision平台由一系列酶辅助的DNA纳米结构组成,以可实现三个功能步骤:DNA靶标识别、非靶标依赖性信号增强和视觉检测(图1a)。通过纳米结构的正交序列设计,靶标识别与信号增强解耦。在识别步骤中,识别元件是独特的杂合纳米结构。它由结合至TaqDNA聚合物的修饰的适体组成[Dang,C.&Jayasena,S.D.J Mol Biol 264:268-278(1996);Park,K.S.等人Sci Adv 2:e1600300(2016)]。在不存在靶DNA的情况下,适体与聚合酶牢固结合以抑制聚合酶活性。在互补靶DNA的存在下,靶标杂交后,杂合物解离以活化聚合酶活性。在信号传导步骤中,活性聚合酶以非靶标依赖性方式延长通用的自引发纳米结构。通过并入具有(dNTP)的修饰寡核苷酸以将辣根过氧化物酶(HRP)固定到信号传导结构中,视觉信号可以酶促产生用于肉眼检测,并且易于通过智能手机进行定量。插页(图1a,右图)示出通过智能手机成像的实际测定输出的例子。
为了补充enVision测定的检测可编程性和模块性,实施可配置的微流体平台,所述平台可以容纳例如4个测定盒(图1b)。具体而言,集成以下两个组件:(i)独立的测定盒和(ii)共用信号传导盒。每个测定盒预先加载有特定的DNA识别纳米结构,并且可以按需插入以实现多功能测定集成。共用支架盒装有通用信号传导元件,这些信号传导元件固定在聚碳酸酯膜上,用于非靶标依赖性信号传导和视觉检测。流体流动和样品混合通过微通道的平行回路进行(图6),并在所述盒上的共用出口处通过负压驱动,以简化测定程序(图7)。图1c示出开发用于临床检测病原体DNA靶标的原型装置。对于临床应用,此系统区室化提高了测定模块性(即按需集成)并简化了实施方式(即统一的表面固定和单流驱动)[Shao,H.等人Nat Commun 6:6999(2015)]。
实施例3
用于核酸的视觉定量的优化的测定
分别评价DNA纳米结构作为功能识别和信号传导元件的性能。对于识别纳米结构,聚合酶与修饰的适体缔合以形成杂合复合物(图2a,上图)。这种优化的复合物表现出可忽略不计的聚合酶活性(图8a)。在互补靶DNA存在下,杂合结构解离,并且聚合酶活性完全恢复(图2a,下图)。发现这种识别和活化表现出很高的序列特异性,因为只有互补靶标才导致强聚合酶活性,而乱序寡核苷酸序列产生的活性却可忽略不计(图8b)。
进一步将通用识别元件设计为自引发纳米结构。与其相似大小的线性对应物相比,所述结构显示出增强的聚合酶占有率和活性(图2b),这可能是由于其在室温下具有优越的引发效率(图9)。用于优化纳米结构的组装和表征的所有序列均可在表1中找到。
表1.用于活性和灵敏度表征的寡核苷酸。
*
为了能够进行视觉检测,使用活化的聚合酶来延长信号传导纳米结构,并通过生物素化核苷酸(生物素-16-AA-dCTP)并入辣根过氧化物酶(HRP)。在光学底物存在下,HRP活性迅速显影为褐色沉淀。在滴定分析中,观察到的颜色强度可能与靶DNA的量增加相关。当用智能手机相机成像时,视觉测定表明检测限(LOD)为0.260fmol靶DNA(图10c),并且所述测定可在室温下在短至30分钟内完成。通过与有效的不对称扩增整合(图11),可以将LOD进一步提高到7.205amol靶DNA(图2c)。也可并入通过基于核酸序列的扩增(NASBA)进行的等温不对称扩增[Pardee,K.等人Cell 165:1255-1266(2016)]以改善实际应用(图12)。用于扩增的所有引物和靶标都可在表1中找到。检测灵敏度通过定点照护(point-of-care)智能手机读数来确定;所有过程(包括不对称扩增和NASBA)均在<2小时内完成。为了使视觉信号与标准荧光信号相关,用FAM修饰的核苷酸进行类似的实验(图10d)。enVision视觉测定与解释荧光信号饱和的逻辑函数密切相关(R
实施例4
enVision的可编程性
为了设计用于核酸的视觉检测的测定,研究了识别纳米结构的可编程性。杂合结构由折叠以结合并抑制DNA聚合酶的保守序列区和可与靶DNA互补的可变区(即双链体和突出端区段)组成(图3a)。通过诱导识别纳米结构的可变区与靶DNA之间的错配,鉴定出与突出端区域相比,双链体区显示更高的对序列错配的灵敏度(图3b)。因此,双链体区可赋予强序列特异性,而突出端区域可容纳更多的序列变异性,所述序列变异性是可用于泛检测的特征。
通过开发两个泛HPV识别纳米结构来验证此设计原理(图3c)。通过与HPV共有基因组匹配来设计所述结构,并且其含有不同数量的针对来自六种HPV亚型的DNA靶标的错配(表2)。通过绘制所有错配区域的图谱,发现在突出端区域容纳更多错配的泛HPV纳米结构1显示出更好的对共有基因组的泛检测能力。在双链体区中含有更多错配的泛HPV纳米结构2显示出大体上降低的信号,这表明双链体区在赋予测定特异性方面的重要性。
接下来,设计用于HPV亚分型的特定纳米结构。鉴定HPV基因组的高度可变区域,以设计用于使HPV亚型(即HPV 6、11、16、18、31、33、58和66)特异性分化的识别纳米结构的新组(图3d,左图);将序列变异性设计为包含在灵敏双链体区内。与需要用于靶标扩增和检测的引物对的常规qPCR分析相比,enVision识别纳米结构可以容易地适应,因为其特异性取决于单个探针序列(即可变区)。使用这种可编程性来快速设计新的特异性HPV亚分型纳米结构(表2),并将其与enVision技术集成在一起。
表2.用于HPV泛检测和特异性亚分型的纳米结构。
在设计PCR引物以检测相同HPV DNA靶标(表3)的比较分析中,enVision平台显示出更好的特异性(图3d,左图),而基于
表3.
实施例5
用于增强检测覆盖度的多重分析
为了提高检测覆盖度,接下来确定是否可以在单个室中同时进行多重enVision测定。尽管临床HPV测定通常检测病毒基因组的单个区域(L1基因座)以进行感染确定,但也可以将病毒基因组部分整合到宿主细胞中,并且这可能导致癌性生长[Williams,V.M.等人,Future Virol 6:45-57(2011);McBride,A.A.&Warburton,A.PLoS Pathog 13:e1006211(2017)]。使用HPV病毒基因组中的非保守区域,设计各种enVision识别纳米结构来检测E1、L1和L2基因座,以提高检测覆盖度(图4a;表4)。
表4.用于多基因座HPV检测的纳米结构。
这些模块识别纳米结构可以在单个反应室中容易配置为不同的逻辑门(例如“或”、“与”、“非”、“与非”和“或非”),以进行可编程计算并实现来自DNA靶标的不同组合的视觉读出(图15;表5)。模块识别纳米结构也可容易地配置以检测呈各种逻辑门配置的多个靶标(图16;表2)
表5.用于enVision逻辑门的纳米结构。
为了增强检测覆盖度,对于每种HPV亚型(例如HPV 16和HPV 18),将其三种类型的基因座特异性纳米结构(即E1、L1和L2基因座)混合以在单个反应中创建“或”逻辑门配置。直接使用宫颈癌细胞系的基因组DNA测试这种高覆盖度的多基因座HPV检测,所述细胞系是通过PCR分析独立验证的(图17)。数据表明,与分别使用E1、L1和L2识别探针进行个体化检测相比,多重enVision测定可以进一步增强检测信号(图4b)。重要的是,尽管真阳性显示出显著信号增强,但阴性样品通过这种多重方法表现出最小背景信号变化(图18)。
使用多重enVision测定,接下来在人宫颈癌细胞系中进行HPV剖析(图4c)。所有实验均在室温下直接从细胞基因组DNA进行,而无需任何预扩增。与任何单个基因座检测相比,多重enVision分析不仅提高信号增强,而且扩大检测覆盖度,以准确鉴定细胞的感染状态和亚型(图18)。所得测量与已发表的文献研究密切相关[Adey,A.等人Nature 500:207-211(2013);Meissner,J.D.J Gen Virol 80:1725-1733(1999);Yee,C.等人,Am J Pathol119:361-366(1985)]:CaSki和SiHa细胞仅显示HPV 16的显著信号,而HeLa和PZ-HPV-7仅显示HPV 18的阳性信号。
将enVision性能针对另一种等温检测技术即LAMP进一步基准化,用于对人类癌细胞系进行HPV剖析。在HPV基因组的相同发散区上(图4a),应用已发布的标准[Tomita,N.等人,Nat Protoc 3:877-882(2008);Song,J.等人Anal Chem 88:7289-7294(2016)],以分别针对每种HPV亚型的E1、L1和L2基因座设计LAMP引物集(详细内容请参见方法部分)。所有LAMP序列信息都可在表6中找到。
表6.用于不同HPV基因座的排名最高的LAMP引物。
根据LAMP引物的ΔG,即在LAMP反应期间自由能的变化,确定排名最高的LAMP引物。
与LAMP的有限引物选项相比,enVision技术不仅产生显著更多的探针选择,而且还提供所测试的所有亚型和基因座的全面覆盖度(图19a)。使用从已知感染的细胞系中分离的基因组DNA,进一步比较enVision技术与排名最高的LAMP引物集的性能(图19b)。仅enVision技术显示出准确的HPV亚分型,而LAMP表现出显著的假阳性(例如CaSki细胞,HPV18;HeLa细胞,HPV 16)。
实施例6
临床样品的HPV剖析
为了测试enVision平台在检测HPV感染并对其进行亚分型中的临床效用,进行一项旨在解决以下两个问题的可行性研究:(1)enVision平台在检测HPV感染方面的准确性如何,以及(2)可提高测定覆盖度,从而鉴定先前无法检测的感染。
使用临床宫颈管刷样品,进行enVision测定以确定HPV感染状态。获取患者样品(n=35),并使用enVision平台测量患者基因组中的HPV 16和HPV18L1基因座(图5a),以便与常规金标准(即Cobas HPV,其仅通过qPCR分析测试L1基因座[Gardner,S.N.等人,J ClinMicrobiol 41:2417-2427(2003)])直接比较。enVision平台通过临床报告(HPV 16,AUC=0.965;HPV 18,AUC=0.944)实现高检测准确度(图5b)。
除了这些临床样品中的L1基因座、L2和E1基因座整合以外,还使用高覆盖度的多基因座enVision测定法来进行测量(图20)。在HPV 16L1阳性临床样品中,对于其他检测基因座仍观察到增强的视觉信号。有趣的是,在L1阴性样品的子集中,高覆盖度enVision系统检测到特异性L2和/或E1基因座整合(图5c)。通过设计独立的
在本说明书中对明显先前已公开的文件的任何列示或讨论都不应一定被视为承认这个文件是最先进技术的一部分或者是公知常识。
讨论
通过整合一系列酶辅助DNA纳米结构,enVision平台使得能够通过肉眼进行多种病原体核酸的灵敏和多功能检测。与核酸扩增技术相比,enVision通过直接靶标杂交和独立的视觉信号增强进行检测。因此,所述技术非常适合于以下临床应用:(1)级联信号增强在靶标杂交后(无需靶标扩增)产生对于肉眼快速且灵敏的颜色读出。因此,整个测定可以在室温下进行,并易于通过智能手机进行定量;(2)DNA纳米结构有效地将识别功能与信号传导功能解耦,从而实现高度可编程的检测和逻辑计算;以及(3)微流体集成补充测定模块性并加快反应。与临床金标准和其他等温检测技术相比,enVision平台不仅展示了优异的灵敏度和特异性,而且还以最少设备要求提供多功能能力(例如,直接RNA检测、逻辑计算和测定模块性)(表7)。
表7.检测技术的比较。
CI,置信区间。a:Rao,A.等人J Clin Microbiol 51:1478-1484(2013);b:Lucchi,N.W.等人Sci Rep 6:36808(2016);c:Lee,J.S.等人,Biomed Res Int 2013,8(2013);d:Kang,L.N.等人J Clin Microbiol 52:1954-1961(2014)。
所开发技术的科学和临床应用可能很广泛。识别纳米结构可以容易地以非常低的成本进行适配,以用相似的效率检测不同靶序列(即,仅需对由规则核苷酸构成的单个探针序列进行适配,而不需要常规引物的新集合或专用的化学修饰的报告物)。信号传导纳米结构是通用的,并且可用于所有靶序列。这种模块性不仅简化了新测定开发,而且还使可编程配置能够进行分子计算。因此预期,所述技术对于研究逃避免疫检测的快速病毒突变(即RNA病毒)[Schotte,L.等人,Antimicrob Agents Chemother 59:4695-4706(2015)]以及多重病毒-宿主基因组整合[Cheung,J.L.等人,J Infect Dis 194:1706-1712(2006)]将特别有用。临床上,enVision技术的视觉检测和低设备要求使所述技术非常适合于社区诊所和资源有限的环境中的定点照护检测工作流。
进一步预期可以进行若干技术改进以增强当前技术。首先,可以容易地修改本发明流控技术(即,四个测定盒)以容纳更多且更小的室。由于当前核酸处理是在芯片外进行,因此还可以集成其他样品制备模块以使得能够进行核酸提取和处理,从而增强平台的临床实用性[Adey,A.等人Nature 500:207-211(2013)]。此种新设计将使得能够对多种病原体核酸进行实用的阵列型视觉检测。其次,在此当前检测平台中,使用智能手机和校正的照明系统来成像并分析视觉读出。由于智能手机变得越来越普及并且具有更多的分析能力[Laksanasopin,T.等人Sci Transl Med 7:273re1(2015);Nemiroski,A.等人Proc NatlAcad Sci U S A 111:11984-11989(2014);Paterson,A.S.等人Lab Chip 17:1051-1059(2017)],可预见到的是,可以直接在智能手机中实现进一步的图像校正和分析算法,以自动校正照明差异并定量色彩强度用于实际应用。
参考文献
1.Adey,A.et al.The haplotype-resolved genome and epigenome of theaneuploid HeLa cancer cell line.Nature 500,207-211(2013).
2.Bodily,J.&Laimins,L.A.Persistence of human papillomavirusinfection:keys to malignant progression.Trends Microbiol 19,33-39(2011).
3.Bouvard,V.et al.A review of human carcinogens--Part B:biologicalagents.Lancet Oncol 10,321-322(2009).
4.Cheung,J.L.,Lo,K.W.,Cheung,T.H.,Tang,J.W.&Chan,P.K.Viral load,E2gene disruption status,and lineage of human papillomavirus type 16infectionin cervical neoplasia.J Infect Dis 194,1706-1712(2006).
5.Crosbie,E.J.,Einstein,M.H.,Franceschi,S.&Kitchener,H.C.Humanpapillomavirus and cervical cancer.Lancet 382,889-899(2013).
6.Cui,M.et al.Clinical performance of Roche Cobas 4800HPV Test.J ClinMicrobiol 52,2210-2211(2014).
7.Dang,C.&Jayasena,S.D.Oligonucleotide inhibitors of Taq DNApolymerase facilitate detection of low copy number targets by PCR.J Mol Biol264,268-278(1996).
8.Gardner,S.N.,Kuczmarski,T.A.,Vitalis,E.A.&Slezak,T.R.Limitations ofTaqMan PCR for detecting divergent viral pathogens illustrated by hepatitisA,B,C,and E viruses and human immunodeficiency virus.J Clin Microbiol 41,2417-2427(2003).
9.Juskowiak,B.Nucleic acid-based fluorescent probes and theiranalytical potential.Anal Bioanal Chem 399,3157-3176(2011).
10.Kang,L.N.et al.Optimal positive cutoff points for care HPV testingof clinician-and self-collected specimens in primary cervical cancerscreening:an analysis from rural China.J Clin Microbiol 52,1954-1961(2014).
11.Laksanasopin,T.et al.A smartphone dongle for diagnosis ofinfectious diseases at the point of care.Sci Transl Med 7,273re1(2015).
12.Lee,H.et al.Molecularly self-assembled nucleic acid nanoparticlesfor targeted in vivo siRNA delivery.Nat Nanotechnol 7,389-393(2012).
13.Lee,J.S.,Song,J.J.,Deaton,R.&Kim,J.-W.Assessing the DetectionCapacity of Microarrays as Bio/Nanosensing Platforms.Biomed Res Int 2013,8(2013).
14.Li,J.,Green,A.A.,Yan,H.&Fan,C.Engineering nucleic acidstructuresfor programmable molecular circuitry and intracellularbiocomputation.Nat Chem 9,1056-1067(2017).
15.Lucchi,N.W.et al.Evaluation of the Illumigene Malaria LAMP:ARobust Molecular Diagnostic Tool for Malaria Parasites.Sci Rep 6,36808(2016).
16.McBride,A.A.&Warburton,A.The role of integration in oncogenicprogression of HPV-associated cancers.PLoS Pathog 13,e1006211(2017).
17.Meissner,J.D.Nucleotide sequences and further characterization ofhuman papillomavirus DNA present in the CaSki,SiHa and HeLa cervicalcarcinoma cell lines.J Gen Virol 80,1725-1733(1999).
18.Nemiroski,A.et al.Universal mobile electrochemical detectordesigned for use in resource-limited applications.Proc Natl Acad Sci U S A111,11984-11989(2014).
19.Niemz,A.,Ferguson,T.M.&Boyle,D.S.Point-of-care nucleic acidtesting for infectious diseases.Trends Biotechnol 29,240-250(2011).
20.Nong,R.Y.,Gu,J.,Darmanis,S.,Kamali-Moghaddam,M.&Landegren,U.DNA-assisted protein detection technologies.Expert Rev Proteomics 9,21-32(2012).
21.Pardee,K.et al.Rapid,Low-Cost Detection of Zika Virus UsingProgrammable Biomolecular Components.Cell 165,1255-1266(2016).
22.Park,K.S.et al.Rapid identification of health care-associatedinfections with an integrated fluorescence anisotropy system.Sci Adv 2,e1600300(2016).
23.Paterson,A.S.et al.A low-cost smartphone-based platform for highlysensitive point-of-care testing with persistent luminescent phosphors.LabChip 17,1051-1059(2017).
24.Rao,A.et al.Development and Characterization of the cobas HumanPapillomavirus Test.J Clin Microbiol 51,1478-1484(2013).
25.Schiffman,M.et al.Carcinogenic human papillomavirus infection.NatRev Dis Primers 2,16086(2016).
26.Schotte,L.et al.Characterization of Poliovirus NeutralizationEscape Mutants of Single-Domain Antibody Fragments(VHHs).Antimicrob AgentsChemother 59,4695-4706(2015).
27.Shao,H.et al.Chip-based analysis of exosomal mRNA mediating drugresistance in glioblastoma.Nat Commun 6,6999(2015).
28.Sievers,F.et al.Fast,scalable generation of high-quality proteinmultiple sequence alignments using Clustal Omega.Mol Syst Biol 7,539(2011).
29.Song,J.et al.Instrument-Free Point-of-Care Molecular Detection ofZika Virus.Anal Chem 88,7289-7294(2016).
30.Tomita,N.,Mori,Y.,Kanda,H.&Notomi,T.Loop-mediated isothermalamplification(LAMP)of gene sequences and simple visual detection ofproducts.Nat Protoc 3,877-882(2008).
31.Wang,L.,Meng,Z.,Martina,F.,Shao,H.&Shao,F.Fabrication of circularassemblies with DNA tetrahedrons:from static structures to a dynamic rotarymotor.Nucleic Acids Res 45,12090-12099(2017).
32.Williams,V.M.,Filippova,M.,Soto,U.&Duerksen-Hughes,P.J.HPV-DNAintegration and carcinogenesis:putative roles for inflammation and oxidativestress.Future Virol 6,45-57(2011).
33.Yee,C.,Krishnan-Hewlett,I.,Baker,C.C.,Schlegel,R.&Howley,P.M.Presence and expression of human papillomavirus sequences in humancervical carcinoma cell lines.Am J Pathol 119,361-366(1985).
34.Zhao,Y.,Chen,F.,Li,Q.,Wang,L.&Fan,C.Isothermal Amplification ofNucleic Acids.Chem Rev 115,12491-12545(2015).
35.Zumla,A.et al.Rapid point of care diagnostic tests for viral andbacterial respiratory tract infections--needs,advances,and futureprospects.Lancet Infect Dis 14,1123-1135(2014).
序列表
<110> 新加坡国立大学
新加坡科技研究局
<120> 使用酶辅助纳米技术对核酸的视觉和模块检测
<130> SP101228WO
<150> 10201805745P
<151> 2018-07-03
<160> 153
<170> PatentIn 3.5版
<210> 1
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 识别纳米结构表征适体序列
<400> 1
aagtatctgt aataaagtca caatgtacag tattg 35
<210> 2
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 识别纳米结构表征反向体序列
<400> 2
tgactttatt acagatactt ctacaacccc ggtaccatct 40
<210> 3
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 识别纳米结构表征互补靶标
<400> 3
agatggtacc ggggttgtag aagtatctgt aataaagtca 40
<210> 4
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 识别纳米结构表征乱序靶标
<400> 4
agtagaacgc gatggtacag gcactgcagg gtccatgtca 40
<210> 5
<211> 200
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 信号传导纳米结构表征自引发模板
<400> 5
agcaggcagt tacgggctgg tgcgatgaga gacgcggagt gtggcggccg gatagtaatg 60
actgcgaccg gtgtaccagt ggcgtgaggc aggtcgtgag gcggcgtacg tagagcgttg 120
agcaggatgc caacagtcga tcaggacgag tgctaacgca ttgtcgatag ctcagctgtc 180
tgagctatcg acaatgcgtt 200
<210> 6
<211> 174
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 信号传导纳米结构表征线性模板
<400> 6
agcaggcagt tacgggctgg tgcgatgaga gacgcggagt gtggcggccg gatagtaatg 60
actgcgaccg gtgtaccagt ggcgtgaggc aggtcgtgag gcggcgtacg tagagcgttg 120
agcaggatgc caacagtcga tcaggacgag tgctaacgca ttgtcgatag ctca 174
<210> 7
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 信号传导纳米结构表征线性模板引物
<400> 7
tgagctatcg acaatgcgtt 20
<210> 8
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 10 bp Taqman探针
<400> 8
ctgatcgact gttggcatcc 20
<210> 9
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 20 bp Taqman探针
<400> 9
ttggcatcct gctcaacgct 20
<210> 10
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 30 bp Taqman探针
<400> 10
tgctcaacgc tctacgtacg 20
<210> 11
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 50 bp Taqman探针
<400> 11
gccgcctcac gacctgcctc 20
<210> 12
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 100 bp Taqman探针
<400> 12
tatccggccg ccacactccg 20
<210> 13
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 130 bp Taqman探针
<400> 13
cgcaccagcc cgtaactgcc 20
<210> 14
<211> 100
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 5'胺通用信号传导纳米结构
<400> 14
gcggcgtacg tagagcgttg agcaggatgc caacagtcga tcaggacgag tgctaacgca 60
ttgtcgatag ctcagctgtc tgagctatcg acaatgcgtt 100
<210> 15
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标突出端2错配
<400> 15
agatggtacc gcggttgtat aagtatctgt aataaagtca 40
<210> 16
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标突出端4错配
<400> 16
agatagtgcc gcggttgtat aagtatctgt aataaagtca 40
<210> 17
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标突出端6错配
<400> 17
agatagtgca gcggttatat aagtatctgt aataaagtca 40
<210> 18
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标突出端8错配
<400> 18
acatagtgca gcggctatat aagtatctgt aataaagtca 40
<210> 19
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标突出端10错配
<400> 19
acgtagtgca gcagctatat aagtatctgt aataaagtca 40
<210> 20
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标突出端12错配
<400> 20
acgtagtgta gcagctatct aagtatctgt aataaagtca 40
<210> 21
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标双链体2错配
<400> 21
agatggtacc ggggttgtag aattatctgt aatagagtca 40
<210> 22
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标双链体4错配
<400> 22
agatggtacc ggggttgtag aattagctgt actagagtca 40
<210> 23
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标双链体6错配
<400> 23
agatggtacc ggggttgtag tattagctct actagagtca 40
<210> 24
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标双链体8错配
<400> 24
agatggtacc ggggttgtag tattcgctct actagagtaa 40
<210> 25
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标双链体10错配
<400> 25
agatggtacc ggggttgtag tattcgctct acaagattaa 40
<210> 26
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 互补靶标双链体12错配
<400> 26
agatggtacc ggggttgtag tattcgcact acaagattac 40
<210> 27
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16正向引物
<400> 27
atggattata tgatatttat gc 22
<210> 28
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16反向引物
<400> 28
ctgataaaga tgtagagg 18
<210> 29
<211> 87
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16产物
<400> 29
ctgataaaga tgtagagggt acagatggta ccggggttgt agaagtatct gtaataaagt 60
catctgcata aatatcatat aatccat 87
<210> 30
<211> 53
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 NASBA正向引物
<400> 30
aattctaata cgactcacta tagggagaag ggcagcctca cctacttcta tta 53
<210> 31
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 NASBA反向引物
<400> 31
aaagatgtag agggtacaga 20
<210> 32
<211> 107
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 NASBA产物
<400> 32
gcagcctcac ctacttctat taataatgga ttatatgata tttatgcaga tgactttatt 60
acagatactt ctacaacccc ggtaccatct gtaccctcta catcttt 107
<210> 33
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1适体
<400> 33
tttaaataat ctggatattt caatgtacag tattg 35
<210> 34
<211> 30
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1反向体
<400> 34
aaatatccag attatttaaa aatggctgca 30
<210> 35
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1互补阳性靶标
<400> 35
cataaggatc tgcagccatt tttaaataat ctggatattt 40
<210> 36
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1 HPV 06靶标
<400> 36
catatgggtc tgcagccatt tgtaaataat ctggatattt 40
<210> 37
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1 HPV 16靶标
<400> 37
catatggttc tgacaccatt ttaatataat ctggatattt 40
<210> 38
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1 HPV 18靶标
<400> 38
cataaggatc tgcagacatt tgtaaataat caggatattt 40
<210> 39
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1 HPV 31靶标
<400> 39
catatggctc agcaaccatt ttaagataat ctggatattt 40
<210> 40
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1 HPV 33靶标
<400> 40
catatggctc agcaaccatt ttaagataat ctggatattt 40
<210> 41
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构1 HPV 58靶标
<400> 41
cataaggttc actggccatt tttaaataat ctggatattt 40
<210> 42
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2适体
<400> 42
aaataattgt gcctcagagg caatgtacag tattg 35
<210> 43
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2反向体
<400> 43
cctctgaggc acaattattt aataaaccat attggctaca 40
<210> 44
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2互补阳性靶标
<400> 44
tgtagccaat atggtttatt aaataattgt gcctcagagg 40
<210> 45
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2 HPV 06靶标
<400> 45
tgtagccaat atggcttatt aaacaattgt gcctcagagg 40
<210> 46
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2 HPV 16靶标
<400> 46
tgtaaccaat aaggtttatt gaatatttgg gcatcagagg 40
<210> 47
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2 HPV 18靶标
<400> 47
tgtaaccaat atggtttatt aaacaactgg gagtcagagg 40
<210> 48
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2 HPV 31靶标
<400> 48
tgcatccaat atggtttatt aaaaatttgt gcatctgaag 40
<210> 49
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2 HPV 33靶标
<400> 49
tgtagccaat atggcttatt aaataactga gattcggaag 40
<210> 50
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 泛HPV纳米结构2 HPV 58靶标
<400> 50
tgtagccaat aaggcttatt aaataattgt gattctgagg 40
<210> 51
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 06适体
<400> 51
taatgtcagg ttcaaaagat caatgtacag tattg 35
<210> 52
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 06反向体
<400> 52
atcttttgaa cctgacatta accctaccca acaccctgtt 40
<210> 53
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 06靶标
<400> 53
aacagggtgt tgggtagggt taatgtcagg ttcaaaagat 40
<210> 54
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 11适体
<400> 54
cagggatagg gtcaaatggt caatgtacag tattg 35
<210> 55
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 11反向体
<400> 55
accatttgac cctatccctg accctgtcca acattctgtt 40
<210> 56
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 11靶标
<400> 56
aacagaatgt tggacagggt cagggatagg gtcaaatggt 40
<210> 57
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16适体
<400> 57
aagtatctgt aataaagtca caatgtacag tattg 35
<210> 58
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16反向体
<400> 58
tgactttatt acagatactt ctacaacccc ggtaccatct 40
<210> 59
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16靶标
<400> 59
agatggtacc ggggttgtag aagtatctgt aataaagtca 40
<210> 60
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18适体
<400> 60
gcactgcagg gtccatgtca caatgtacag tattg 35
<210> 61
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18反向体
<400> 61
tgacatggac cctgcagtgc ctgtaccatc gcgttctact 40
<210> 62
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18靶标
<400> 62
agtagaacgc gatggtacag gcactgcagg gtccatgtca 40
<210> 63
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 31适体
<400> 63
tatccacagt aaaatcagtg caatgtacag tattg 35
<210> 64
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 31反向体
<400> 64
cactgatttt actgtggata cacctgccac acataatgtt 40
<210> 65
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 31靶标
<400> 65
aacattatgt gtggcaggtg tatccacagt aaaatcagtg 40
<210> 66
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 33适体
<400> 66
tgtgtacatt atccacatcg caatgtacag tattg 35
<210> 67
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 33反向体
<400> 67
cgatgtggat aatgtacaca ccccaatgca acactcatac 40
<210> 68
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 33靶标
<400> 68
gtatgagtgt tgcattgggg tgtgtacatt atccacatcg 40
<210> 69
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 58适体
<400> 69
catgtatagt atcagcatcg caatgtacag tattg 35
<210> 70
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 58反向体
<400> 70
cgatgctgat actatacatg attttcagag tcctctgcac 40
<210> 71
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 58靶标
<400> 71
gtgcagagga ctctgaaaat catgtatagt atcagcatcg 40
<210> 72
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 66适体
<400> 72
tgggtgcctc atcatcaata caatgtacag tattg 35
<210> 73
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 66反向体
<400> 73
tattgatgat gaggcaccca tttcatttcg tcagtctggt 40
<210> 74
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 66靶标
<400> 74
accagactga cgaaatgaaa tgggtgcctc atcatcaata 40
<210> 75
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 06正向引物
<400> 75
gaagatacat ttgatattta tgc 23
<210> 76
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 06反向引物
<400> 76
attaggtgtg gaagttaa 18
<210> 77
<211> 91
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 06产物
<400> 77
gaagatacat ttgatattta tgctgaatct tttgaacctg acattaaccc tacccaacac 60
cctgttacaa atatatcaga tacatattta a 91
<210> 78
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 11正向引物
<400> 78
acacgtttga tatttatgc 19
<210> 79
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 11反向引物
<400> 79
tattaggtgt ggaggta 17
<210> 80
<211> 93
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 11产物
<400> 80
acacgtttga tatttatgct gaaccatttg accctatccc tgaccctgtc caacattctg 60
ttacacagtc ttatcttacc tccacaccta ata 93
<210> 81
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18正向引物
<400> 81
acttgtttga tatatatgca 20
<210> 82
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18反向引物
<400> 82
gcgaatattt aaaaaatgc 19
<210> 83
<211> 90
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18产物
<400> 83
acttgtttga tatatatgca gatgacatgg accctgcagt gcctgtacca tcgcgttcta 60
ctacctcctt tgcatttttt aaatattcgc 90
<210> 84
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 31正向引物
<400> 84
ggcttatatg acatttatgc 20
<210> 85
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 31反向引物
<400> 85
actgtacagc agtagaa 17
<210> 86
<211> 85
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 31产物
<400> 86
ggcttatatg acatttatgc agacactgat tttactgtgg atacacctgc cacacataat 60
gtttcccctt ctactgctgt acagt 85
<210> 87
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 33正向引物
<400> 87
gtttgtatga tgtttatgc 19
<210> 88
<211> 17
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 33反向引物
<400> 88
ttgcaaacgt actgtat 17
<210> 89
<211> 75
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 33产物
<400> 89
gtttgtatga tgtttatgct gacgatgtgg ataatgtaca caccccaatg caacactcat 60
acagtacgtt tgcaa 75
<210> 90
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 58正向引物
<400> 90
tggactttat gatatttatg c 21
<210> 91
<211> 15
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 58反向引物
<400> 91
gcaaaggacg tatgt 15
<210> 92
<211> 81
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 58产物
<400> 92
tggactttat gatatttatg ctgacgatgc tgatactata catgattttc agagtcctct 60
gcactcacat acgtcctttg c 81
<210> 93
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 66正向引物
<400> 93
gcctatatga tatttatgca 20
<210> 94
<211> 16
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 66反向引物
<400> 94
aggtaattgt gcagaa 16
<210> 95
<211> 86
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 66产物
<400> 95
gcctatatga tatttatgca aatattgatg atgaggcacc catttcattt cgtcagtctg 60
gtgctacacc ttctgcacaa ttacct 86
<210> 96
<211> 60
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2模板
<400> 96
tctaatacga ctcactatag agatggtacc ggggttgtag aagtatctgt aataaagtca 60
<210> 97
<211> 60
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2模板互补体
<400> 97
tgactttatt acagatactt ctacaacccc ggtaccatct ctatagtgag tcgtattaga 60
<210> 98
<211> 60
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2模板
<400> 98
tctaatacga ctcactatag agtagaacgc gatggtacag gcactgcagg gtccatgtca 60
<210> 99
<211> 60
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2模板互补体
<400> 99
tgacatggac cctgcagtgc ctgtaccatc gcgttctact ctatagtgag tcgtattaga 60
<210> 100
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1适体
<400> 100
caaccacccc cacttccacc caatgtacag tattg 35
<210> 101
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1反向体
<400> 101
ggtggaagtg ggggtggttg cagtcagtac agtagtggaa 40
<210> 102
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1靶标
<400> 102
ttccactact gtactgactg caaccacccc cacttccacc 40
<210> 103
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> L1适体
<400> 103
gtttctgaag tagatatggc caatgtacag tattg 35
<210> 104
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1反向体
<400> 104
gccatatcta cttcagaaac tacatataaa aatactaact 40
<210> 105
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1靶标
<400> 105
agttagtatt tttatatgta gtttctgaag tagatatggc 40
<210> 106
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2适体
<400> 106
aagtatctgt aataaagtca caatgtacag tattg 35
<210> 107
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> L2反向体
<400> 107
tgactttatt acagatactt ctacaacccc ggtaccatct 40
<210> 108
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2靶标
<400> 108
agatggtacc ggggttgtag aagtatctgt aataaagtca 40
<210> 109
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1适体
<400> 109
tgtgcccccg ttgtctatag caatgtacag tattg 35
<210> 110
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1反向体
<400> 110
ctatagacaa cgggggcaca gagggcaaca acagcagtgt 40
<210> 111
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1靶标
<400> 111
acactgctgt tgttgccctc tgtgcccccg ttgtctatag 40
<210> 112
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1适体
<400> 112
aggtacagga gactgtgtag caatgtacag tattg 35
<210> 113
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1反向体
<400> 113
ctacacagtc tcctgtacct gggcaatatg atgctaccaa 40
<210> 114
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1靶标
<400> 114
ttggtagcat catattgccc aggtacagga gactgtgtag 40
<210> 115
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2适体
<400> 115
gcactgcagg gtccatgtca caatgtacag tattg 35
<210> 116
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2反向体
<400> 116
tgacatggac cctgcagtgc ctgtaccatc gcgttctact 40
<210> 117
<211> 40
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2靶标
<400> 117
agtagaacgc gatggtacag gcactgcagg gtccatgtca 40
<210> 118
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1正向引物
<400> 118
caacgtgttg cgattggtgt 20
<210> 119
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1反向引物
<400> 119
accattcccc atgaacatgc ta 22
<210> 120
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1 Taqman探针
<400> 120
acacccagta tagctgacag 20
<210> 121
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV L1正向引物
<400> 121
cacctaatgg ctgaccacga 20
<210> 122
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1反向引物
<400> 122
acttgcagtt ggacatccct 20
<210> 123
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1 Taqman探针
<400> 123
cacctacaca ggcccaaacc 20
<210> 124
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2正向引物
<400> 124
ttggaacagg gtcgggtaca 20
<210> 125
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2反向引物
<400> 125
gaagggccca caggatctac 20
<210> 126
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2 Taqman探针
<400> 126
tgggaacaag gcctcccaca 20
<210> 127
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16“非”适体
<400> 127
tgactttatt acagatactt caatgtacag tattg 35
<210> 128
<211> 35
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18“非”适体
<400> 128
tgacatggac cctgcagtgc caatgtacag tattg 35
<210> 129
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1 LAMP正向内部引物
<400> 129
tggcgccctt ctacctgtaa acagcgggta tggcaata 38
<210> 130
<211> 43
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1 LAMP反向内部引物
<400> 130
caccatgtag tcagtatagt ggtgtttcac taacaccctc tcc 43
<210> 131
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1 LAMP正向引物3
<400> 131
agagctgcaa aaaggaga 18
<210> 132
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 E1 LAMP反向引物3
<400> 132
gtgtttggca tatagtgtgt c 21
<210> 133
<211> 48
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1 LAMP正向内部引物
<400> 133
tggcagcaca taatgacata tttgtggtaa ccaactattt gttactgt 48
<210> 134
<211> 47
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1 LAMP反向内部引物
<400> 134
aactttaagg agtacctacg acatgagtta aggttatttt gcacagt 47
<210> 135
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1 LAMP正向引物3
<400> 135
ccacaataat ggcatttgtt g 21
<210> 136
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L1 LAMP反向引物3
<400> 136
atgtatgtat gtcataacgt ctg 23
<210> 137
<211> 50
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2 LAMP正向内部引物
<400> 137
ccggggttgt agaagtatct gtaatctcac ctacttctat taataatgga 50
<210> 138
<211> 46
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2 LAMP反向内部引物
<400> 138
taccatctgt accctctaca tctttggaat attgtatgca ccacca 46
<210> 139
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2 LAMP正向引物3
<400> 139
catatactac cacttcacat gc 22
<210> 140
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 16 L2 LAMP反向引物3
<400> 140
aatgggtata tcaggacctg 20
<210> 141
<211> 44
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1 LAMP正向内部引物
<400> 141
tgaatctgtg ttgcttccac ttcgcggctg tttacaatat caga 44
<210> 142
<211> 42
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1 LAMP反向内部引物
<400> 142
aacatggcgg caatgtatgt agtgtctaca ctgctgttgt tg 42
<210> 143
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1 LAMP正向引物3
<400> 143
tagtgggcag aaaaaggc 18
<210> 144
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 E1 LAMP反向引物3
<400> 144
attgctattg tcacttgtac c 21
<210> 145
<211> 43
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1 LAMP正向内部引物
<400> 145
aatcatattc ctcaacatgt ctgctctcct gtacctgggc aat 43
<210> 146
<211> 45
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1 LAMP反向内部引物
<400> 146
actttaactg cagatgttat gtcctaccaa agttccaatc ctcta 45
<210> 147
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1 LAMP正向引物3
<400> 147
caatatgtgc ttctacacag t 21
<210> 148
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L1 LAMP反向引物3
<400> 148
tccaccaaac tagtagttgg 20
<210> 149
<211> 47
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2 LAMP正向内部引物
<400> 149
aaggaggtag tagaacgcga tgcttgtttg atatatatgc agatgac 47
<210> 150
<211> 43
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2 LAMP反向内部引物
<400> 150
tgcatttttt aaatattcgc ccactggagg ttaaagggac cgt 43
<210> 151
<211> 19
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2 LAMP正向引物3
<400> 151
ctttagtatc tgccacgga 19
<210> 152
<211> 18
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> HPV 18 L2 LAMP反向引物3
<400> 152
tacaggcaca tcccaaga 18
<210> 153
<211> 15
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> DNA适体保守序列
<400> 153
caatgtacag tattg 15
- 使用酶辅助纳米技术对核酸的视觉和模块检测
- 使用内切核酸酶剪切/连接酶重新密封反应和毛细管电泳或微阵列检测核酸差异
