新型OMNI-50 CRISPR核酸酶-RNA复合物
文献发布时间:2024-01-17 01:26:37
本申请要求于2020年11月4日提交的美国临时申请第63/109,835号的权益,其
内容在此引入作为参考。
在整个本申请中,参考了各种出版物,包括括号中参考的出版物。本申请中所提及的所有出版物的公开内容在此以引用的方式整体并入本申请中,以便提供对本发明所涉及的技术和可以与本发明一起采用的技术中的特征的附加描述。
本申请通过引用并入了存在于名为“211103_91628-A-PCT_Sequence_Listing_AWG.txt”的文件中的核苷酸序列,其大小为88千字节,并且其在2021年10月25日以IBM-PC机器格式创建,具有与MS-Windows的操作系统兼容性,该文件含在作为本申请的一部分的2021年11月3日提交的文本文件中。
技术领域
本发明尤其涉及用于基因组编辑的组合物和方法。
背景技术
细菌和古细菌适应性免疫的成簇规律间隔短回文重复序列(ClusteredRegularly Interspaced Short Palindromic Repeat,CRISPR)系统示出蛋白质组成和基因组基因座架构(genomic loci architecture)的极端多样性。CRISPR系统已经成为研究和基因组工程的重要工具。然而,CRISPR系统的许多细节尚未确定,并且CRISPR核酸酶的适用性可能受到序列特异性要求、表达或递送挑战的限制。不同的CRISPR核酸酶具有不同的特点,诸如:大小、PAM位点、中靶活性、特异性、切割模式(例如,平端(blunt)、交错末端)和切割后形成的突出的得失位(indel)模式。不同的特点集合可以用于不同的应用。例如,一些CRISPR核酸酶可能能够靶向其他CRISPR核酸酶由于PAM位点的限制而无法靶向的特定基因组基因座。另外,目前在使用中的一些CRISPR核酸酶展现预免疫性,这可能会限制体内适用性。参见Charlesworth等人,《自然医学(Nature Medicine)》(2019)和Wagner等人,《自然医学》(2019)。因此,新型CRISPR核酸酶和激活并且靶向它们的RNA分子的发现、工程化和改进是重要的。
发明内容
本发明提供了包含非天然存在的RNA分子的组合物,该RNA分子包含crRNA重复序列部分和引导序列部分,其中RNA分子在tracrRNA序列的存在下与OMNI-50核酸酶形成复合物并且将该OMNI-50核酸酶靶向到DNA靶位点,其中该tracrRNA序列由RNA分子的tracrRNA部分或第二RNA分子的tracrRNA部分编码。
本发明还提供了包含非天然存在的RNA分子的组合物,该RNA分子包含crRNA重复序列部分、引导序列部分和tracrRNA部分,其中RNA分子与OMNI-50核酸酶形成复合物并且将该OMNI-50核酸酶靶向到与RNA分子的引导序列部分具有互补性的DNA靶位点。
本发明还提供了包含非天然存在的RNA分子的组合物,该RNA分子包含RNA支架部分,该RNA支架部分具有以下结构:
crRNA重复序列部分-接头部分-tracrRNA部分;
其中该RNA支架部分与OMNI-50 CRISPR核酸酶形成复合物并且将该OMNI-50CRISPR核酸酶靶向到与RNA分子的引导序列部分互补的DNA靶位点。
本文公开了可以用于基因组工程化、表观基因组工程化、基因组打靶、细胞的基因组编辑和/或体外诊断的组合物和方法,该组合物和方法使用OMNI-50 CRISPR核酸酶和非天然存在的RNA分子,该非天然存在的RNA分子包含能够特异性结合并且激活OMNI-50CRISPR核酸酶的支架部分,以基于RNA分子的引导序列部分(也称为RNA间隔区部分),靶向DNA靶位点。
所公开的组合物可以用于修饰基因组DNA序列。如本文所用,基因组DNA是指存在于一个或多个目的细胞中的线性和/或染色体DNA和/或质粒或其他染色体外DNA序列。在一些实施方案中,目的细胞为真核细胞。在一些实施方案中,目的细胞为原核细胞。在一些实施方案中,该方法在基因组DNA序列中的预先确定靶位点处产生双链断裂(DSB),引起基因组中靶位点处DNA序列的突变、插入和/或缺失。
附图说明
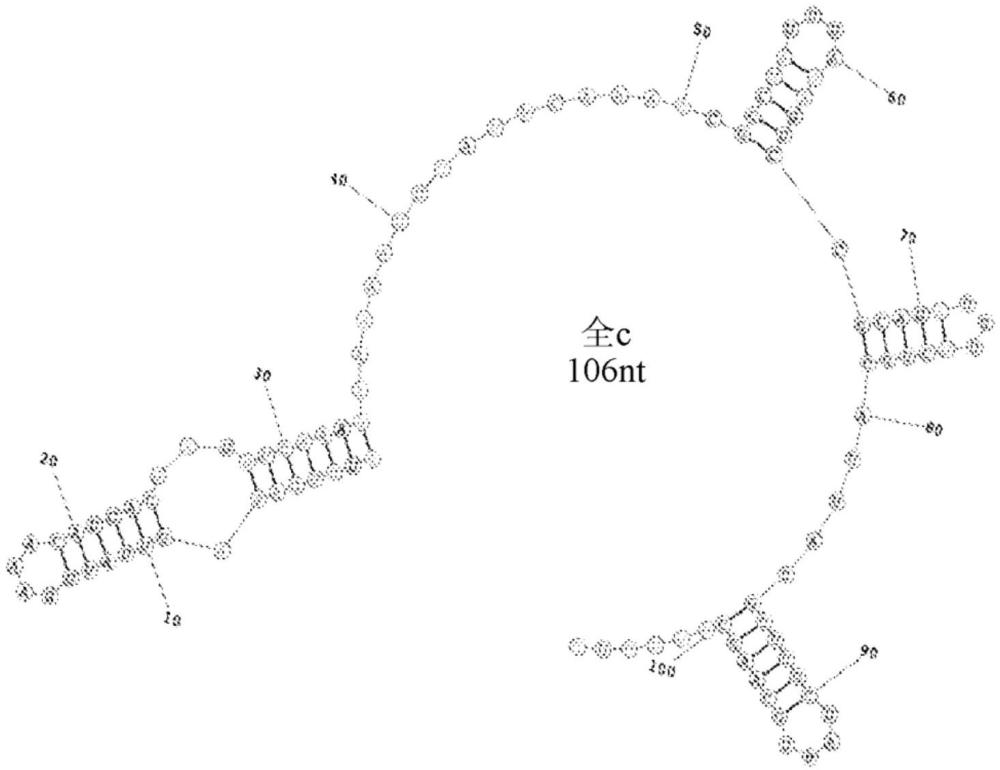
图1A至图1G:表3中列出的3'修剪的sgRNA的预测二级结构。图1A:“全c”RNA结构。图1B:“短1”RNA结构。图1C:“短2”RNA结构。图1D:“短3”RNA结构。图1E:“短4”RNA结构。图1F:“短5”RNA结构。图1G:“短6”RNA结构。
图2:RNP中3'修剪的引导物(表3)的活性测定。纯化的OMNI-50与IVT转录的短引导物反应。对于体外测定,RNP与5'-FAM标记的线性底物反应。通过将切割片段的荧光除以切割片段和未切割片段的总和来计算切割效率。对于体内测定,用RNP电穿孔U2OS细胞,并且通过二代测序(NGS)以得失位频率确定活性。
图3A至图3M:表4中列出的完整支架(full scaffold)sgRNA版本f和版本c以及表5中列出的高级别的(high ranked)短sgRNA的预测二级结构。图3A:“全f”RNA结构。图3B:“全c”RNA结构。图3C:“NGS13”RNA结构。图3D:“NGS14”RNA结构。图3E:“NGS15”RNA结构。图3F:“NGS16”RNA结构。图3G:“NGS17”RNA结构。图3H:“NGS18”RNA结构。图3I:“NGS40”RNA结构。图3J:“NGS41”RNA结构。图3K:“NGS42”RNA结构。图3L:“NGS43”RNA结构。图3M:“NGS44”RNA结构。
图4A至图4F:表6中列出的中级别(medium ranked)的短sgRNA的预测二级结构。图4A:“NGS9”RNA结构。图4B:“NGS2”RNA结构。图4C:“NGS3”RNA结构。图4D:“NGS12”RNA结构。图4E:“NGS1”RNA结构。图4F:“NGS6”RNA结构。
图5A至图5E:在具有许可性引导物(permissive guides)(g35、g62)的U2OS哺乳动物细胞系中的RNP活性。用OMNI-50的RNP与所示的sgRNA的对U2OS细胞进行电穿孔,并且通过NGS将活性确定为靶向的原型间隔区(protospacer)及其已知的脱靶上的得失位频率(g35、g62,表8、表9)。图5A:具有NGS1、NGS2、NGS3、NGS6或NGS9支架的“g35-ON”或“g35-OT2”引导物(间隔区),以及“无处理”(NT)和“无引导物”对照。图5B:具有NGS1、NGS12、NGS13、NGS14、NGS15、NGS16、NGS17、NGS18、NGS2、NGS3、NGS6或NGS9支架的g35中靶或g35脱靶引导物(间隔区),以及“无处理”(NT)对照。图5C:具有NGS1、NGS6、NGS12、NGS17或全f支架的“g35-ON”或“g35-OT2”引导物(间隔区),以及“无处理”(NT)对照。图5D:具有全f或NGS1支架的“g62-ON”、“g62-OT1”或“g62-OT2”引导物(间隔区),以及“无处理”(NT)和“无引导物”对照。图5E:具有NGS9或NGS17支架的“g62-ON”、“g62-OT1”或“g62-OT2”引导物(间隔区),以及“无引导物”和“无处理”(NT)对照。
图6A至图6C:在具有挑战性的引导物(g58)的U2OS哺乳动物细胞系中的活性。用具有所指示的sgRNA的OMNI-50的RNP电穿孔U2OS细胞,并且通过NGS将活性确定为靶向原型间隔区及其已知的脱靶上的得失位频率(g58,表8、表9)。图6A:具有全f或NGS1支架的“g58-ON”或“g58-OT2”引导物(间隔区),以及“无处理”(NT)对照和“无引导物”对照。图6B:具有NGS12、NGS9或NGS17支架的“g58-ON”或“g58-OT2”引导物(间隔区),以及“无处理”(NT)对照和“无引导物”对照。图6C:具有全f、NGS12、NGS40、NGS41、NGS42、NGS43或NGS44支架的“g58”或“g58-OT”引导物(间隔区),以及“无处理”(NT)对照和“无引导物”对照。
图7A至图7B:在具有短sgRNA的HSC500和LCL细胞中的活性。图7A:用具有所指示的sgRNA的OMNI-50的RNP电穿孔HSC500细胞,并且通过NGS将活性确定为靶向原型间隔区及其已知的脱靶上的得失位频率(g58和g35,表8、表9)。图7B:用具有所指示的sgRNA的OMNI-50的RNP电穿孔LCL细胞,并且通过NGS将活性确定为靶向原型间隔区及其已知的脱靶上的得失位频率(g58和g35,表8、表9)。
图8:RNA支架的代表性实例。示例性的RNA支架部分包含通过四环(tetraloop)连接至tracrRNA部分的crRNA部分。crRNA部分包含crRNA重复序列。tracrRNA部分包含tracrRNA反重复序列(anti-repeat sequence)和附加的tracrRNA区段(section)。RNA分子还可以包含与crRNA重复序列连接的引导序列部分(即RNA间隔区),使得RNA分子充当单引导RNA分子。
具体实施方式
本发明提供了包含非天然存在的RNA分子的组合物,该RNA分子包含crRNA重复序列部分和引导序列部分,其中RNA分子在tracrRNA序列的存在下与OMNI-50核酸酶形成复合物并且将该OMNI-50核酸酶靶向到DNA靶位点,其中该tracrRNA序列由RNA分子的tracrRNA部分或第二RNA分子的tracrRNA部分编码。
包含crRNA重复序列部分和引导序列部分的RNA分子和tracrRNA分子可以是单独的分子。替代地,包含crRNA重复序列部分和引导序列部分的RNA分子可以与tracrRNA分子连接以形成具有crRNA重复序列部分、引导序列部分和tracrRNA部分的单个RNA分子。
在一些实施例中,crRNA重复序列部分的长度小于17个核苷酸,优选长度为12至16个核苷酸,或crRNA重复序列部分的长度为17个或更多个核苷酸,优选长度为18至24个核苷酸。
在一些实施例中,crRNA重复序列部分与由秘鲁江崎氏菌(Ezakiella peruensis)菌株M6.X2编码的成熟OMNI-50crRNA重复序列具有至少60%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,crRNA重复序列部分与SEQ ID NO:23具有至少60%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,crRNA重复序列部分与SEQ ID NO:8、23、24和25中的任一个具有至少95%的序列同一性。
在一些实施例中,crRNA重复序列不是SEQ ID NO:8或23。
在一些实施例中,包含crRNA重复序列部分和引导序列部分的RNA分子进一步包含tracrRNA部分。
在一些实施例中,crRNA重复序列部分通过多核苷酸接头部分与tracrRNA部分共价连接。
在一些实施例中,该组合物进一步包含包含tracrRNA部分的第二RNA分子。
在一些实施例中,OMNI-50核酸酶与SEQ ID NO:1的氨基酸序列具有至少95%的序列同一性。
在一些实施例中,引导序列部分的长度为17至30个核苷酸,优选22个核苷酸。
本发明还提供了包含非天然存在的RNA分子的组合物,该RNA分子包含tracrRNA部分,其中RNA分子在crRNA重复序列部分和引导序列部分的存在下与OMNI-50核酸酶形成复合物并且将该OMNI-50核酸酶靶向到DNA靶位点,其中crRNA重复序列部分和引导序列部分由RNA分子或第二RNA分子编码。
包含crRNA重复序列部分和引导序列部分的RNA分子和包含tracrRNA部分的RNA分子可以是单独的分子。替代地,包含crRNA重复序列部分和引导序列部分的RNA分子可以与tracrRNA分子连接以形成具有crRNA重复序列部分、引导序列部分和tracrRNA部分的单个RNA分子。
在一些实施例中,tracrRNA部分的长度小于91个核苷酸,优选长度为90至80、89至80、79至70、69至60、59至50、49至40或39至28个核苷酸,或者tracrRNA部分的长度为91个或更多个核苷酸,优选长度为91至112个核苷酸。
在一些实施例中,tracrRNA部分与由秘鲁江崎氏菌菌株M6.X2编码的成熟OMNI-50tracrRNA序列具有至少30%至40%、41%至50%、51%至60%、61%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,tracrRNA部分与SEQ ID NO:5的tracrRNA部分具有至少30%至40%、41%至50%、51%至60%、61%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,tracrRNA部分与SEQ ID NO:4、5、16至21、29至31、74至126、132至137和148至167中的任一个的tracrRNA部分具有至少95%的序列同一性。
在一些实施例中,tracrRNA部分不同于SEQ ID NO:4或5的tracrRNA部分。
在一些实施例中,tracrRNA部分包含长度小于19个核苷酸,优选长度为14至18个核苷酸的tracrRNA反重复序列部分(anti-repeat sequence portion)。
在一些实施例中,tracrRNA部分包含与SEQ ID NO:26具有至少60%至70%、71%至80%、81%至90%、91%至95%或96%至99%序列同一性的tracrRNA反重复序列部分。
在一些实施例中,tracrRNA部分包含与SEQ ID NO:9、26至28和138中的任一个具有至少95%序列同一性的tracrRNA反重复序列部分。
在一些实施例中,tracrRNA部分包含不同于SEQ ID NO:9或26的tracrRNA反重复序列部分。
在一些实施例中,RNA分子包含tracrRNA部分并且进一步包含crRNA重复序列部分和引导序列部分。
在一些实施例中,tracrRNA部分通过多核苷酸接头部分与crRNA重复序列共价连接。
在一些实施例中,多核苷酸接头部分的长度为4至10个核苷酸。
在一些实施例中,多核苷酸接头具有GAAA序列。
在一些实施例中,该组合物进一步包含第二RNA分子,该第二RNA分子包含crRNA重复序列部分和引导序列部分。
在一些实施例中,OMNI-50核酸酶与SEQ ID NO:1的氨基酸序列具有至少95%的序列同一性。
在一些实施例中,引导序列部分的长度为17至30个核苷酸,优选长度为22个核苷酸。
本发明提供了包含非天然存在的RNA分子的组合物,该RNA分子包含RNA支架部分,该RNA支架部分具有以下结构:
crRNA重复序列部分-接头部分-tracrRNA部分;
其中该RNA支架部分与OMNI-50 CRISPR核酸酶形成复合物并且将该OMNI-50CRISPR核酸酶靶向到与RNA分子的引导序列部分互补的DNA靶位点。
在一些实施例中,RNA支架部分的长度为112、111至110、109至105、104至100、99至95、94至90、89至85、84至80、79至75、74至70、69至50或49至45个核苷酸。
在一些实施例中,RNA支架部分与SEQ ID NO:5具有至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少85%、至少90%或至少95%的序列同一性。
在一些实施例中,crRNA重复序列部分的长度小于17个核苷酸,优选长度为12至16个核苷酸,或crRNA重复序列部分的长度为17个或更多个核苷酸,优选长度为18至24个核苷酸。
在一些实施例中,crRNA重复序列部分与由秘鲁江崎氏菌菌株M6.X2编码的成熟OMNI-50crRNA重复序列具有至少60%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,crRNA重复序列部分与SEQ ID NO:23具有至少60%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,crRNA重复序列部分与SEQ ID NO:8、23、24和25中的任一个具有至少95%的序列同一性。
在一些实施例中,crRNA重复序列不是SEQ ID NO:8或23。
在一些实施例中,tracrRNA部分的长度小于91个核苷酸,优选长度为90至80、89至80、79至70、69至60、59至50、49至40或39至28个核苷酸,或者tracrRNA部分的长度为91个或更多个核苷酸,优选长度为91至112个核苷酸。
在一些实施例中,tracrRNA部分与由秘鲁江崎氏菌菌株M6.X2编码的成熟OMNI-50tracrRNA序列具有至少30%至40%、41%至50%、51%至60%、61%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,tracrRNA部分与SEQ ID NO:5的tracrRNA部分具有至少30%至40%、41%至50%、51%至60%、61%至70%、71%至80%、81%至90%、91%至95%或96%至99%的序列同一性。
在一些实施例中,tracrRNA部分与SEQ ID NO:4、5、16至21、29至31、74至126、132至137和148至167中的任一个的tracrRNA部分具有至少95%的序列同一性。
在一些实施例中,tracrRNA部分不同于SEQ ID NO:4或5的tracrRNA部分。
在一些实施例中,tracrRNA部分包含tracrRNA反重复序列部分,其中crRNA重复序列和tracrRNA反重复序列部分通过接头部分共价连接。
在一些实施例中,接头部分是长度为4至10个核苷酸的多核苷酸接头。
在一些实施例中,多核苷酸接头具有GAAA序列。
在一些实施例中,tracrRNA反重复序列部分的长度小于19个核苷酸,优选长度为14至18个核苷酸。
在一些实施例中,tracrRNA部分包含与SEQ ID NO:5具有至少60%至70%、71%至80%、81%至90%、91%至95%或96%至99%序列同一性的tracrRNA反重复序列部分。
在一些实施例中,tracrRNA反重复序列部分与SEQ ID NO:9、26至28和138中的任一个具有至少95%的序列同一性。
在一些实施例中,tracrRNA反重复序列部分不同于SEQ ID NO:9或26。
在一些实施例中,tracrRNA部分包含与tracrRNA反重复部分连接的第一核苷酸区段(a first section of nucleotide),并且该第一核苷酸区段与SEQ ID NO:10、11、127、128、139至143、AAC、A、AA、AAA和ACAAACC中的任一个具有至少95%的序列同一性。
在一些实施例中,tracrRNA部分包含与第一核苷酸区段连接的第二核苷酸区段(asecond section of nucleotide),并且该第二核苷酸区段与SEQ ID NO:12、32、144至146、GCCUAUU、GCCUAU、AAUGGC、AAAGGC、UAUAGGC、AUAGGC和GCCU中的任一个具有至少95%的序列同一性。
在一些实施例中,tracrRNA部分包含与第二核苷酸区段连接的第三核苷酸区段(athird section of nucleotide),并且该第三核苷酸区段与SEQ ID NO:13、33、34、129、130、147、CGCAG、CGC、CGCAGG、C、CUUCUGC和CGCAGUUG中的任一个具有至少95%的序列同一性。
在一些实施例中,tracrRNA部分包含与第三核苷酸区段连接的第四核苷酸部分(afourth portion of nucleotide),并且该第四核苷酸区段与SEQ ID NO:14、15、35至73、131、AUU、AUUAUUU、AUUU、AUUUUUUUU、AGCUUUUUU、UUUU、UUUUU和UUU中的任一个具有至少95%的序列同一性。
在一些实施例中,RNA支架部分与SEQ ID NO:4、5、16至21、29至31、74至126、132至137和148至167的核苷酸序列具有至少95%的同一性。
在一些实施例中,RNA支架部分具有全F、全C、短1、短2、短3、短4、短5、短6、NGS13、NGS14、NGS15、NGS16、NGS17、NGS18、NGS40、NGS41、NGS42、NGS43、NGS44、NGS9、NGS2、NGS3、NGS12、NGS1或NGS6 RNA支架中的任一个的预测结构。
在一些实施例中,RNA支架部分不同于SEQ ID NO:4或5。
在一些实施例中,引导序列部分与RNA分子的crRNA重复序列部分共价连接,形成具有以下结构的单引导RNA分子:
引导序列部分-crRNA重复序列部分-接头部分-tracrRNA部分。
在一些实施例中,引导序列部分的长度为17至30个核苷酸,更优选20至23个核苷酸,更优选22个核苷酸。
在一些实施例中,该组合物进一步包含OMNI-50 CRISPR核酸酶,其中该OMNI-50CRISPR核酸酶与SEQ ID NO:1的氨基酸序列具有至少95%的同一性。
在一些实施例中,RNA分子通过体外转录(in vitro transcription,IVT)或固相人工寡核苷酸合成(solid-phase artificial oligonucleotide synthesis)形成。
在一些实施例中,RNA分子包含修饰的核苷酸。
本发明还提供了编码本文所描述的实施例中的任一个的RNA分子的多核苷酸分子。
本发明提供了包含至少一种特异性结合、激活和/或靶向OMNI-50核酸酶的非天然存在的RNA分子的组合物。
本发明还提供了在无细胞系统或细胞基因组中的DNA靶位点处修饰核苷酸序列的方法,该方法包含向该系统或细胞中引入本文所描述的实施例中的任一个的组合物和OMNI-50 CRISPR核酸酶。DNA靶位点由组合物的RNA分子编码的RNA间隔区确定,使得RNA间隔区在序列上与DNA靶位点互补。
在一些实施例中,细胞是真核细胞或原核细胞。
本发明还提供了用于修饰无细胞系统或细胞基因组中的DNA靶位点处的核苷酸序列的试剂盒,该试剂盒包括向该系统或细胞中引入本文所描述的实施例中的任一个的组合物、OMNI-50 CRISPR核酸酶,以及用于将该组合物和该OMNI-50 CRISPR核酸酶递送至该细胞的说明书。
在一些实施例中,非天然存在的RNA分子包含不同于秘鲁江崎氏菌菌株M6.X2的野生型crRNA序列的crRNA序列部分。在一些实施例中,非天然存在的RNA分子包含比秘鲁江崎氏菌菌株M6.X2的野生型crRNA序列更短的crRNA序列部分。
在一些实施例中,非天然存在的RNA分子包含不同于秘鲁江崎氏菌菌株M6.X2的野生型tracrRNA序列的tracrRNA序列部分。在一些实施例中,非天然存在的RNA分子包含比秘鲁江崎氏菌菌株M6.X2的野生型tracrRNA序列更短的tracrRNA序列部分。
在本发明的实施例中,非天然存在的RNA分子包含“间隔区”或“引导序列”部分。RNA分子的“间隔区部分”或“引导序列部分”是指能够与特定靶DNA序列杂交的核苷酸序列,例如引导序列部分具有与沿引导序列部分的长度方向与被靶向的DNA序列完全互补的核苷酸序列。在一些实施例中,引导序列部分的长度为17、18、19、20、21、22、23、24、25、26、27、28、29或30个核苷酸,或长度为大约17至30、17至29、17至28、17至27、17至26、17至25、17至24、18至22、19至22、18至20、17至20或21至22个核苷酸。优选地,引导序列部分的全长与沿引导序列部分的长度被靶向的DNA序列完全互补。该引导序列部分可以是具有“支架部分”的RNA分子的一部分,该支架部分可以与CRISPR核酸酶形成复合物并且激活该CRISPR核酸酶,其中该RNA分子的引导序列部分充当该CRISPR复合物的DNA靶向部分。当具有支架部分和引导序列部分的RNA分子与CRISPR分子同时存在时,RNA分子能够将CRISPR核酸酶靶向到特定的靶DNA序列。每种可能性都代表单独的实施例。可以定制设计RNA分子间隔区部分以靶向任何期望的序列。
在实施例中,核酸酶结合的RNA核苷酸序列(nuclease-binding RNAnucleotidesequence)和DNA靶向的RNA核苷酸序列(DNA-targeting RNA nucleotide sequence)(例如间隔区或引导序列部分)在单引导RNA分子(sgRNA)上,其中sgRNA分子可以与OMNI-50CRISPR核酸酶形成复合物并且充当DNA靶向模块(DNA targeting module)。
在实施例中,核酸酶结合的RNA核苷酸序列在第一RNA分子上并且DNA靶向的RNA核苷酸序列在第二RNA分子上,并且第一RNA分子和第二RNA分子通过碱基配对相互作用并且与CRISPR核酸酶复合(complex),以充当靶向模块。
根据本发明的一些方面,所公开的方法包含修饰无细胞系统或细胞基因组中靶位点处的核苷酸序列的方法,该方法包含将本文所描述的实施例中的任一个的组合物引入到细胞中。
本发明还提供了本发明的任何组合物或方法用于修饰细胞中DNA靶位点处的核苷酸序列的用途。
本发明提供了一种修饰真核细胞的基因组中靶位点处的核苷酸序列的方法。
本发明提供了一种修饰哺乳动物细胞的基因组中靶位点处的核苷酸序列的方法。
本发明提供了一种修饰植物细胞的基因组中靶位点处的核苷酸序列的方法。
在一些实施例中,该方法离体(ex vivo)进行。在一些实施例中,该方法在体内进行。在一些实施例中,该方法的一些步骤离体进行并且一些步骤在体内进行。在一些实施例中,哺乳动物细胞为人类细胞。
本发明还提供了通过本文所描述的任一种方法获得的一种或多种修饰的细胞。在实施例中,这一种或多种修饰的细胞能够产生子代细胞(progeny cell)。在实施例中,这一种或多种修饰的细胞能够在移植(engraftment)后产生子代细胞。
本发明还提供一种组合物,其包含这些修饰的细胞和药学上可接受的载体。还提供了一种制备此组合物的体外或离体方法,其包含将细胞与药学上可接受的载体混合。
根据本发明的一些方面,所公开的方法包含将本文所描述的组合物中的任一者用于治疗罹患与基因组突变相关的疾病的受试者(subject)的用途,其包含修饰该受试者的基因组中靶位点处的核苷酸序列。
根据本发明的一些方面,所公开的方法包含一种治疗患有突变病症(mutationdisorder)的受试者的方法,其包含将本文所描述的组合物中的任一者靶向到与该突变病症相关的等位基因。
在一些实施例中,突变病症与选自以下中的任一者的疾病或病症相关:瘤形成(neoplasia);老年性黄斑变性(age-related macular degeneration);精神分裂症;神经无序、神经退行性障碍或运动障碍;脆性X综合征;分泌酶相关病症;朊病毒相关病症;ALS;成瘾;自闭症;阿尔茨海默病(Alzheimer's Disease);中性粒细胞减少症;炎症相关病症;帕金森病(Parkinson's Disease);血液和凝血疾病和病症;细胞失调和肿瘤疾病和病症;炎症和免疫相关疾病和病症;代谢、肝脏、肾脏和蛋白质疾病和病症;肌肉和骨骼疾病和病症;皮肤疾病和病症;神经系统和神经元疾病和病症以及眼部疾病和病症。
本发明的某些实施例将核酸酶靶向到与疾病或病症相关的特定基因座,作为基因编辑、治疗方法或疗法的形式。例如,为了诱导基因的编辑或敲除,可以使用定制设计的引导RNA分子将本文所公开的组合物特异性靶向基因的致病突变等位基因。引导RNA分子优选通过首先考虑核酸酶的PAM要求来设计,如本文所示,其还取决于其中进行基因编辑的系统。例如,设计用于将OMNI-50核酸酶靶向到靶位点的引导RNA分子被设计成含有与邻近OMNI-50PAM序列“NGG”的区域互补的间隔区。引导RNA分子被进一步优选设计成含有足够并且优选最佳长度的间隔区(即引导RNA分子中与靶等位基因互补的区域),以增加核酸酶的比活性(specific activity)并且降低脱靶效应。例如,设计成靶向OMNI-50核酸酶的引导RNA分子可以被设计成含有22个核苷酸的间隔区,以获得高的中靶切割活性(on-targetcleavage activity)。
作为非限制性实例,引导RNA分子可以被设计成将核酸酶靶向到突变等位基因的特定区域,例如起始密码子附近,使得当核酸酶引起DNA损伤后,诱导非同源末端连接(non-homologous end joining,NHEJ)途径,并且通过引入移码突变导致突变等位基因沉默。这种引导RNA分子设计的方式特别适用于改变显性负效突变(dominant negativemutation)的效应,并且从而治疗受试者。作为单独的非限制性实例,引导RNA分子可以被设计成靶向突变等位基因的特定致病突变,使得在由核酸酶引起的DNA损伤后,诱导同源定向修复(homology directed repair,HDR)途径并且导致突变等位基因的模板介导的校正。这种引导RNA分子设计的方式特别适用于改变突变等位基因的单倍剂量不足效应(haploinsufficiency effect),并且从而治疗受试者。
可以靶向改变以治疗疾病或病症的特定基因的非限制性实例呈现于下文中。文献中描述了诱导突变病症的特定疾病相关基因和突变。此类突变可以用于设计DNA靶向的RNA分子(DNA-targeting RNA molecule)以将CRISPR组合物靶向到疾病相关基因的等位基因,其中CRISPR组合物引起DNA损伤并且诱导DNA修复途径以改变等位基因并且从而治疗突变病症。
ELANE基因中的突变与中性粒细胞减少症相关。因此,非限制性地,本发明的靶向ELANE的实施例可以用于治疗罹患中性粒细胞减少症的受试者的方法。
CXCR4是人类免疫缺陷病毒1型(HIV-1)感染的共受体。因此,非限制性地,本发明的靶向CXCR4的实施例可以用于治疗罹患HIV-1的受试者或赋予受试者对HIV-1感染的抵抗力(resistance)的方法。
程序性细胞死亡蛋白1(PD-1)的破坏增强了CAR-T细胞介导的肿瘤细胞杀伤,并且PD-1可以是其它癌症疗法中的靶。因此,非限制性地,本发明的靶向PD-1的实施例可以用于治疗罹患癌症的受试者的方法。在实施例中,治疗为CAR-T细胞治疗,其中T细胞已根据本发明修饰为PD-1缺陷型。
另外,BCL11A是在抑制血红蛋白产生中起作用的基因。可以通过抑制BCL11A来增加珠蛋白产生以治疗诸如地中海贫血或镰状细胞贫血等疾病。参见例如PCT国际公布号WO2017/077394A2;美国公开号US2011/0182867A1;Humbert等人《科学·转化医学(Sci.Transl.Med.)》(2019);和Canver等人《自然(Nature)》(2015)。因此,非限制性地,本发明的靶向BCL11A的增强子的实施例可以用于治疗罹患β地中海贫血或镰状细胞贫血的受试者的方法。
本发明的实施例还可以用于靶向任何疾病相关基因,用于研究、改变或治疗以下表A或表B中列出的任何疾病或病症。实际上,通过使用本文所公开的核酸酶靶向适当的疾病相关基因,例如美国公开号2018/0282762A1和欧洲专利号EP3079726B1中所列的那些疾病相关基因,可以研究、改变或治疗与基因座相关的任何疾病。
除非另外定义,否则本文所使用的所有技术和/或科学术语都具有与本发明所涉及领域的技术人员通常所理解的含义相同的含义。尽管与本文所描述的方法和材料类似或相当的方法和材料可以用于本发明的实施例的实践或测试,但下文将描述示范性的方法和/或材料。倘若有冲突,将以本专利说明书,包括定义为准。另外,材料、方法和实例仅是说明性的,而不旨在必然是限制性的。
在讨论中,除非另有说明,否则诸如“基本上”和“约”等修饰本发明的实施例的一个或多个特征的条件或关系特点的形容词应理解为意指该条件或特点被定义为在该实施例用于其预期应用的操作的可接受的容限(tolerance)内。除非另有指示,否则说明书和权利要求书中的词语“或”被认为是包括性的“或”而不是排他性的或,并且指示其连接的项目的至少一个和任何组合。
应理解,如上文和本文其它地方所用,术语“一(a)”和“一个(an)”是指“一个或多个”所列举的组分。本领域普通技术人员将清楚,单数的使用包括复数,除非另有具体陈述。因此,术语“一(a)”、“一个(an)”和“至少一个”在本申请中可互换使用。
为了更好地理解本教导而不以任何方式限制本教导的范围,除非另有指示,否则在说明书和权利要求书中使用的表示数量、百分比或比例的所有数字和其它数值应理解为在所有情况下均由术语“约”修饰。因此,除非有相反的指示,否则以下说明书和所附权利要求中阐述的数字参数是近似值,该近似值可以根据试图获得的期望特性而变化。
至少,每个数值参数至少应根据报告的有效数字的数量并且通过应用普通的舍入技术来解释。
应理解,在本文记载的数值范围的情况下,除非另有说明,否则本发明涵盖上限和下限之间的每个整数,并且包括上限和下限。
在本申请的说明书和权利要求书中,动词“包含”、“包括”和“具有”及其同根词中的每一者用于指示动词的一个或多个客体不一定是动词的一个或多个主体的组分、要素或部件的完整列表。本文所使用的其它术语意在通过其在本领域中熟知的含义来定义。
术语“多核苷酸”、“核苷酸”、“核苷酸序列”、“核酸”和“寡核苷酸”可互换使用。
它们是指任何长度的核苷酸的聚合形式,其中核苷酸可以是脱氧核糖核苷酸或核糖核苷酸,或它们的类似物。多核苷酸可以具有任何三维结构,并且可以执行任何已知或未知的功能。以下是多核苷酸的非限制性实例:基因或基因片段的编码区或非编码区、由连锁分析定义的基因座、外显子、内含子、信使RNA(mRNA)、转移RNA、核糖体RNA、短干扰RNA(siRNA)、短发夹RNA(shRNA)、微RNA(miRNA)、核酶、cDNA、重组多核苷酸、分支多核苷酸、质粒、载体(vector)、任何序列的分离DNA、任何序列的分离RNA、核酸探针和引物。多核苷酸可以包含一种或多种修饰的核苷酸,诸如甲基化核苷酸和核苷酸类似物。如果存在,对核苷酸结构的修饰可以在聚合物组装之前或之后赋予。核苷酸序列可以被非核苷酸组分中断。可以在聚合后进一步修饰多核苷酸,诸如通过与标记组分缀合。
术语“核苷酸类似物”或“修饰的核苷酸”是指在核苷的含氮碱基(例如,胞嘧啶(C)、胸腺嘧啶(T)或尿嘧啶(U)、腺嘌呤(A)或鸟嘌呤(G))中或其上、核苷的糖基(sugarmoiety)(例如,核糖、脱氧核糖、修饰的核糖、修饰的脱氧核糖、六元糖类似物或开链糖类似物)或磷酸酯中或其上含有一个或多个化学修饰(例如取代)的核苷酸。
本文所描述的RNA序列中的每一个可以包含一个或多个核苷酸类似物。
如本文所用,以下核苷酸标识符用于表示引用的核苷酸碱基:
如本文所用,术语“靶向序列”或“靶向分子”是指包含能够与特定靶序列杂交的核苷酸序列的核苷酸序列或分子,例如靶向序列具有沿靶向序列的长度方向与被靶向的序列至少部分地互补的核苷酸序列。靶向序列或靶向分子可以是可以例如经由支架部分与CRISPR核酸酶形成复合物的靶向RNA分子的一部分,其中该靶向序列充当CRISPR复合物的靶向部分,例如间隔区部分。当具有靶向序列的RNA分子与CRISPR核酸酶同时存在时,该RNA分子单独或与附加的一种或多种RNA分子(例如tracrRNA分子)组合,能够将CRISPR核酸酶靶向到特定靶序列。作为非限制性实例,CRISPR RNA分子或单引导RNA分子的引导序列部分可以充当靶向分子。每种可能性都代表一个单独的实施例。可以定制设计靶向序列以靶向任何期望的序列。
如本文所用,术语“靶”是指靶向序列或靶向分子与具有被靶向的核苷酸序列的核酸的优先杂交。应理解,术语“靶”涵盖可变的杂交效率,使得存在对具有被靶向的核苷酸序列的核酸的优先靶向,但也可能发生除中靶杂交之外的无意的脱靶杂交。应理解,在一个RNA分子靶向一个序列的情况下,该RNA分子和CRISPR核酸酶分子的复合物靶向该序列以提供(for)核酸酶活性。
RNA分子的“引导序列部分”是指能够与特定靶DNA序列杂交的核苷酸序列,例如,引导序列部分具有沿引导序列部分的长度方向与被靶向的DNA序列部分地或完全地互补的核苷酸序列。在一些实施例中,引导序列部分的长度为17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49或50个核苷酸,或长度为大约17至50、17至49、17至48、17至47、17至46、17至45、17至44、17至43、17至42、17至41、17至40、17至39、17至38、17至37、17至36、17至35、17至34、17至33、17至31、17至30、17至29、17至28、17至27、17至26、17至25、17至24、17至22、17至21、18至25、18至24、18至23、18至22、18至21、19至25、19至24、19至23、19至22、19至21、19至20、20至22、18至20、20至21、21至22或17至20个核苷酸。优选地,引导序列部分的全长沿引导序列部分的长度方向与被靶向的DNA序列完全互补。引导序列部分可以是可以与CRISPR核酸酶形成复合物的RNA分子的一部分,其中引导序列部分充当CRISPR复合物的DNA靶向部分。当具有引导序列部分的RNA分子与CRISPR分子同时存在时,该RNA分子单独或与附加的一种或多种RNA分子(例如tracrRNA分子)组合,能够将CRISPR核酸酶靶向到特定靶DNA序列。因此,CRISPR复合物可以通过将具有引导序列部分的RNA分子与CRISPR核酸酶的直接结合或通过将具有引导序列部分的RNA分子和附加的一种或多种RNA分子与CRISPR核酸酶的结合来形成。每种可能性代表一个单独的实施例。可以定制设计引导序列部分以靶向任何期望的序列。因此,包含“引导序列部分”的分子是一种类型的靶向分子。在本申请中,术语“引导分子”、“RNA引导分子”、“引导RNA分子”和“gRNA分子”与包含引导序列部分的分子同义。
在靶向多个细胞中存在的DNA序列的情况下,应理解,靶向包括RNA分子的引导序列部分与一个或多个细胞中的序列的杂交,并且还包括RNA分子与多个细胞中少于所有细胞中的靶序列的杂交。因此,应理解,在RNA分子靶向多个细胞中的序列的情况下,RNA分子和CRISPR核酸酶的复合物应理解为与一或多个细胞中的靶序列的杂交,并且还可以与少于所有细胞中的靶序列杂交。因此,应理解,RNA分子和CRISPR核酸酶的复合物在一个或多个细胞中与靶序列杂交时引入相关的双链断裂,并且还可以在少于所有细胞中与靶序列杂交时引入相关的双链断裂。如本文所用,术语“修饰的细胞”是指由于与靶序列杂交的结果,即中靶杂交的结果,双链断裂受RNA分子和CRISPR核酸酶的复合物影响的细胞。
如本文所用,术语“野生型”是本领域技术人员所理解的术语,并且意指生物体、菌株、基因或特征的典型形式,因为其天然存在,而与突变体或变体形式相区别。因此,如本文所用,在氨基酸或核苷酸序列是指野生型序列的情况下,变体是指序列的变体,例如包含取代、缺失、插入。在本发明的实施例中,工程化CRISPR核酸酶是变体CRISPR核酸酶,其与表1中所指示的OMNI-50 CRISPR核酸酶相比包含至少一个氨基酸修饰(例如,取代、缺失和/或插入)。
术语“非天然存在”或“工程化”可互换使用并且指示人为操纵。当涉及核酸分子或多肽时,这些术语可以意指核酸分子或多肽至少基本上不含至少一种与其在自然界中天然相关的和在自然界中发现的其它组分。
如本文所用,术语“氨基酸”包括天然和/或非天然或合成氨基酸,包括甘氨酸和D型或I型旋光异构体两者,以及氨基酸类似物和肽模拟物。
如本文所用,“基因组DNA”是指存在于一个或多个目的细胞中的线性和/或染色体DNA和/或质粒或其它染色体外(extrachromosomal)DNA序列。在一些实施例中,目的细胞为真核细胞。在一些实施例中,目的细胞为原核细胞。在一些实施例中,这些方法在基因组DNA序列中的预先确定的靶位点处产生双链断裂(DSB),引起基因组中靶位点处DNA序列的突变、插入和/或缺失。
“真核”细胞包括但不限于真菌细胞(诸如酵母)、植物细胞、动物细胞、哺乳动物细胞和人类细胞。
如本文所用,术语“核酸酶”是指能够切割核酸的核苷酸亚基之间的磷酸二酯键的酶。核酸酶可以分离自或衍生自天然来源。天然来源可以是任何活生物体。替代地,核酸酶可以是保留磷酸二酯键切割活性的修饰的或合成的蛋白质。
如本文所用,术语“PAM”是指位于被靶向的DNA序列附近并且被CRISPR核酸酶识别的靶DNA的核苷酸序列。PAM序列可以根据核酸酶特性(identity)而不同。
如本文所用,术语“突变病症”或“突变疾病”是指与由突变引起的基因功能异常相关的任何病症或疾病。表现为突变病症的功能异常基因在其等位基因中的至少一者中含有突变并且该功能异常基因被称为“疾病相关基因”。突变可以在疾病相关基因的任何部分中,例如在调控、编码或非编码部分中。突变可以是任何类别的突变,诸如取代、插入或缺失。根据任何类型的突变机制,诸如隐性、显性负性、功能获得、功能丧失或导致基因产物单倍剂量不足的突变,疾病相关基因的突变可以表现为病症或疾病。
本领域技术人员将理解,本发明的实施例公开了包含支架部分的RNA分子,该支架部分能够与OMNI-50 CRISPR核酸酶复合并且能够激活OMNI-50 CRISPR核酸酶以使其靶向到与原型间隔区相邻基序(PAM)相邻的目的靶DNA位点。OMNI-50 CRISPR核酸酶通过具有与目的靶DNA位点互补的引导序列部分(即RNA间隔区)靶向到目的DNA位点。然后核酸酶介导靶DNA的切割以在原型间隔区靶位点内产生双链断裂。
术语“蛋白结合序列”或“核酸酶结合序列”是指能够与CRISPR核酸酶结合以形成CRISPR复合物的序列。技术人员将理解,能够与CRISPR核酸酶结合以形成CRISPR复合物的支架RNA或tracrRNA包含蛋白结合序列或核酸酶结合序列。
CRISPR核酸酶的“RNA结合部分”是指CRISPR核酸酶的可以结合RNA分子以形成CRISPR复合物的部分,例如,sgRNA的RNA支架部分的核酸酶结合序列。CRISPR核酸酶的“活性部分(activity portion)”或“活性部分(active portion)”是指例如当与靶向DNA的RNA分子(DNA-targeting RNA molecule)复合时,在DNA分子中影响双链断裂的CRISPR核酸酶的一部分。
术语“RNA支架”或“支架”是指包含与tracrRNA部分共价连接的crRNA部分的非天然存在的分子的一部分。如本文所用,“crRNA部分”包含crRNA重复序列。如本文所用,“tracrRNA部分”包含tracrRNA反重复序列。tracrRNA部分可以进一步包含与tracrRNA反重复序列连接的附加的tracrRNA序列。此类序列可以包括但不限于融合(nexus)、发夹或融合、发夹或tracrRNA反重复序列上游或下游的其它tracrRNA序列。因此,RNA支架的tracrRNA部分包含反重复序列,其任选地与附加的tracrRNA区段连接。
如本文所用,包含RNA支架部分和RNA引导序列部分(或RNA间隔区部分)的RNA分子充当单引导RNA(sgRNA)分子。sgRNA的RNA支架部分特异性结合并且激活CRISPR核酸酶,并且sgRNA的RNA间隔区部分将CRISPR核酸酶靶向到DNA靶位点。例如,sgRNA分子可以通过引导序列部分与RNA支架的crRNA重复序列部分的共价连接形成。
因此,在本发明的实施例中,RNA分子可以被设计为支架部分和间隔区部分的合成融合体,一起形成能够结合并且靶向OMNI-50 CRISPR核酸酶的单引导RNA(sgRNA)。参见Jinek等人,《科学(Science)》(2012)。
本发明的实施例还可以利用单独的crRNA分子和单独的tracrRNA分子形成CRISPR复合物。在此类实施例中,crRNA分子可以经由crRNA分子的crRNA重复序列部分和tracrRNA分子的tracrRNA反重复序列部分之间的至少部分杂交而与tracrRNA分子杂交。此类部分杂交也可以含有典型的凸起(bulge),该凸起将杂交的RNA核苷酸分离成“上部”和“下部”茎。独立的(separate)crRNA和tracrRNA分子在本文所描述的本发明的某些应用中可能是有利的。
在本发明的实施例中,RNA分子的支架部分可以包含“融合”区和/或“发夹”区,“融合”区和/或“发夹”区可以进一步限定RNA分子的结构。(参见Briner等人,《分子细胞(Molecular Cell)》(2014))。
如本文所用,术语“同向重复序列(direct repeat sequence)”是指特定氨基酸序列或核苷酸序列的两次或更多次重复。
如本文所用,能够与CRISPR核酸酶“相互作用”或“结合”的RNA序列或分子是指RNA序列或分子与CRISPR核酸酶形成CRISPR复合物的能力。
如本文所用,术语“可操作地连接”是指两个序列或分子之间的允许它们以其预期方式起作用的关系(即融合、杂交)。在本发明的实施例中,当RNA分子以可操作方式连接到启动子时,RNA分子和启动子两者都被允许以其预期方式起作用。
如本文所用,术语“异源启动子”是指不与被启动的分子或途径一起天然存在的启动子。
如本文所用,如果分子序列之间有X%的核苷酸或氨基酸相同并且处于相同的相对位置,则一个序列或分子与另一序列或分子具有X%的“序列同一性”。例如,与第二核苷酸序列具有至少95%的序列同一性的第一核苷酸序列将在相同的相对位置上与另一序列的至少95%的核苷酸是相同的。作为非限制性实例,序列同一性可以通过建立第一核苷酸序列与第二核苷酸序列的比对来确定,例如通过应用尼德勒曼-翁施算法(Needleman–Wunsch algorithm)。
本文所描述的CRISPR核酸酶或CRISPR组合物可以包括并且作为蛋白质、DNA分子、RNA分子、核糖核蛋白(RNP)、核酸载体或它们的任何组合递送。在一些实施例中,RNA分子包含化学修饰。合适的化学修饰的非限制性实例包括2'-0-甲基(M)、2'-0-甲基、3'硫代磷酸酯(MS)或2'-0-甲基、3'硫代PACE(MSP)、假尿苷和1-甲基假尿苷。每种可能性都代表本发明的一个单独的实施例。
本文所描述的CRISPR核酸酶和/或编码该CRISPR核酸酶的多核苷酸以及任选的附加蛋白质(例如,ZFP、TALEN、转录因子、限制酶)和/或诸如引导RNA的核苷酸分子可以通过任何合适手段递送至靶细胞。靶细胞可以是任何类型的细胞,例如,真核细胞或原核细胞,在任何环境中,例如分离的或未分离的,维持在培养物中的、体外的、离体的、体内的或在植物中的。
在一些实施例中,待递送的组合物包括核酸酶的mRNA和引导分子的RNA。在一些实施例中,待递送的组合物包括核酸酶的mRNA、引导物的RNA和供体模板。在一些实施例中,待递送的组合物包括CRISPR核酸酶和引导RNA。在一些实施例中,待递送的组合物包括CRISPR核酸酶、引导RNA和用于经由例如同源定向修复进行基因编辑的供体模板。在一些实施例中,待递送的组合物包括核酸酶的mRNA、DNA靶向的RNA和tracrRNA。在一些实施例中,待递送的组合物包括核酸酶的mRNA、DNA靶向的RNA和tracrRNA以及供体模板。在一些实施例中,待递送的组合物包括CRISPR核酸酶、DNA靶向的RNA和tracrRNA。在一些实施例中,待递送的组合物包括CRISPR核酸酶、DNA靶向的RNA和tracrRNA以及用于经由例如同源定向修复进行基因编辑的供体模板。
任何合适的病毒载体系统可以用于递送RNA组合物。常规的基于病毒和非病毒的基因转移方法可以用于将核酸和/或CRISPR核酸酶引入细胞(例如,哺乳动物细胞、植物细胞等)和靶组织中。此类方法也可以用于在体外向细胞施用编码核酸和/或CRISPR核酸酶蛋白。在某些实施例中,施用核酸和/或CRISPR核酸酶用于体内或离体基因治疗用途。非病毒载体递送系统包括裸核酸和与递送载体诸如脂质体或泊洛沙姆(poloxamer)复合的核酸。关于基因治疗程序的综述,参见Anderson,《科学》(1992);Nabel和Felgner,《生物技术趋势(TIBTECH)》(1993);Mitani和Caskey,《生物技术趋势(TIBTECH)》(1993);Dillon,《生物技术趋势(TIBTECH)》(1993);Miller,《自然》(1992);Van Brunt,《生物技术(Biotechnology)》(1988);Vigne等人,《恢复性神经病学和神经科学(RestorativeNeurology and Neuroscience)》8:35-36(1995);Kremer和Perricaudet,《英国医学公报(British Medical Bulletin)》(1995);Haddada等人,《微生物学和免疫学的最新主题(Current Topics in Microbiology and Immunology)》(1995);
和Yu等人,《基因治疗(Gene Therapy)》1:13-26(1994)。
核酸和/或蛋白质的非病毒递送方法包括电穿孔、脂质转染、显微注射、基因枪法(biolistics)、粒子枪加速(particle gun acceleration)、病毒体、脂质体、免疫脂质体、聚阳离子或脂质:核酸缀合物、人工病毒体(artificial virion)和试剂增强的核酸摄取(agent-
enhanced uptake of nucleic acid)或可以通过细菌或病毒(例如农杆菌(Agrobacterium)、根瘤菌属种(Rhizobium sp.)NGR234、苜蓿中华根瘤菌(Sinorhizoboiummeliloti)、百脉根中慢生根瘤菌(Mesorhizobium loti)、烟草花叶病毒(tobacco mosaicvirus)、马铃薯X病毒、花椰菜花叶病毒(cauliflower mosaic virus)和木薯叶脉花叶病毒(cassava veinmosaic virus))递送至植物细胞。参见,例如,Chung等人《植物科学趋势(Trends PlantSci.)》(2006)。使用例如Sonitron 2000系统(Rich-Mar)进行的声致穿孔(sonoporation)也可以用于递送核酸。阳离子脂质介导的蛋白质和/或核酸递送也被认为是用于体内或体外递送方法。参见Zuris等人,《自然生物技术(Nat.Biotechnol.)》(2015),Coelho等人,《新英格兰医学杂志(N.Engl.J.Med.)》(2013);Judge等人,《分子治疗(Mol.Ther.)》
(2006);和Basha等人,《分子治疗》(2011)。
额外的示范性核酸递送系统包括由
RNAiMAX)。适用于多核苷酸的高效受体识别脂质转染的阳离子和中性脂质包括PCT国际公布号WO/1991/017424和WO/1991/016024中公开的那些阳离子和中性脂质。可以递送至细胞(离体施用)或靶组织(体内施用)。
脂质:核酸复合物的制备,包括靶向脂质体(targeted liposome)诸如免疫脂质复合物的制备是本领域技术人员熟知的(参见,例如,Crystal,《科学》(1995);Blaese等人,
《癌症基因治疗(Cancer Gene Ther.)》(1995);Behr等人,《生物缀合物化学(BioconjugateChem.)》(1994);Remy等人,《生物缀合物化学》(1994);Gao和Huang,《基因治疗》
(1995);Ahmad和Allen,《癌症研究(Cancer Res.)》(1992);美国专利号4,186,183;
4,217,344;4,235,871;4,261,975;4,485,054;4,501,728;4,774,085;4,837,028;和
4,946,787)。
其他的递送方法包括将待递送的核酸包装到EnGeneIC递送载体(EDV)中的使用。
这些EDV使用双特异性抗体特异性递送至靶组织,其中抗体的一个臂对靶组织具有特异性并且另一个臂对EDV具有特异性。抗体将EDV带到靶细胞表面,并且然后EDV通过内吞作用被带入细胞。EDV一旦进入细胞中,内容物就释放(参见MacDiamid等人,《自然生物技术(Nature Biotechnology)》(2009))。
使用基于RNA或DNA病毒的系统来递送核酸利用了将病毒靶向到体内特定细胞和将病毒有效负载(viral payload)运输到细胞核的高度进化的过程。病毒载体可以直接向患者施用(体内)或其可以用于体外处理细胞并且向患者施用修饰后的细胞(离体)。常规的用于递送核酸的基于病毒的系统包括但不限于用于基因转移的重组逆转录病毒、慢病毒、腺病毒、腺相关病毒、牛痘苗病毒和单纯疱疹病毒载体。然而,RNA病毒优选用于递送本文所描述的RNA组合物。另外,在许多不同的细胞类型和靶组织中已经观察到高转导效率。本发明的核酸可以通过非整合慢病毒(non-integrating lentivirus)递送。任选地,利用慢病毒递送RNA。任选地,慢病毒包括核酸酶的mRNA、引导物的RNA。任选地,慢病毒包括核酸酶的mRNA、引导物的RNA和供体模板。任选地,慢病毒包括核酸酶蛋白、引导RNA。任选地,慢病毒包括核酸酶蛋白、引导RNA和/或用于经由例如同源定向修复进行基因编辑的供体模板。任选地,慢病毒包括核酸酶的mRNA、DNA靶向的RNA和tracrRNA。任选地,慢病毒包括核酸酶的mRNA、DNA靶向的RNA和tracrRNA,以及供体模板。任选地,慢病毒包括核酸酶蛋白、DNA靶向的RNA和tracrRNA。任选地,慢病毒包括核酸酶蛋白、DNA靶向的RNA和tracrRNA,以及用于经由例如同源定向修复进行基因编辑的供体模板。
如上所提及的,本文所描述的组合物可以使用非整合慢病毒颗粒方法,例如
逆转录病毒的嗜性(tropism)可以通过掺入外源包膜蛋白来改变,从而扩大靶细胞的潜在靶群体。慢病毒载体是能够转导或感染非分裂细胞并且通常产生高病毒滴度的逆转录病毒载体。逆转录病毒基因转移系统的选择取决于靶组织。逆转录病毒载体包含具有至多6kb至10kb的外源序列包装能力的顺式作用长末端重复序列(cis-actinglongterminal repeat,LTR)。最小顺式作用LTR对于载体的复制和包装是足够的,其然后用于将治疗基因整合到靶细胞中以提供永久转基因表达。广泛使用的逆转录病毒载体包括基于鼠白血病病毒(MuLV)、长臂猿白血病病毒(GaLV)、猿免疫缺陷病毒(SIV)、人免疫缺陷病毒(HIV)及它们的组合的那些载体(参见,例如Buchscher Panganiban,《病毒学杂志(J.Virol.)》(1992);Johann等人,《病毒学杂志》(1992);Sommerfelt等人,《病毒学(Virol.)》(1990);Wilson等人,《病毒学杂志》(1989);Miller等人,《病毒学杂志》(1991);PCT国际公布号WO/1994/026877A1)。
目前,至少六种病毒载体方法可以用于临床试验中的基因转移,这些方法利用涉及通过插入到辅助细胞系中的基因来补充缺陷载体以生成转导剂的方式。
pLASN和MFG-S是已经用于临床试验的逆转录病毒载体的实例(Dunbar等人,《血液(Blood)》(1995);Kohn等人,《自然医学(Nat.Med.)》(1995);Malech等人,《美国国家科学院院刊(PNAS)》(1997))。PA317/pLASN是用于基因治疗试验的第一种治疗性载体。(Blaese等人,《科学》(1995))。已经观察到MFG-S包装的载体的转导效率为50%以上。(Ellem等人,《免疫学免疫治疗(Immunol Immunother.)》(1997);Dranoff等人,《人类基因治疗(Hum.GeneTher.)》(1997))。
包装细胞用于形成能够感染宿主细胞的病毒颗粒。此类细胞包括包装腺病毒、AAV的293细胞和包装逆转录病毒的psi.2细胞或PA317细胞。用于基因治疗的病毒载体通常由将核酸载体包装到病毒颗粒中的生产细胞系生成。载体通常含有包装和随后整合到宿主(如果适用)中所需的最小病毒序列,其它病毒序列被编码待表达的蛋白质的表达盒替换。缺失病毒功能由包装细胞系反式提供。例如,用于基因治疗的AAV载体通常仅具有来自AAV基因组的反向末端重复(ITR)序列,该反向末端重复序列对于包装和整合到宿主基因组中是必需的。病毒DNA包装在细胞系中,该细胞系含有编码其它AAV基因(即rep和cap)但缺乏ITR序列的辅助质粒。该细胞系还感染了腺病毒作为辅助病毒。辅助病毒促进AAV载体的复制和AAV基因从辅助质粒的表达。由于缺乏ITR序列,辅助质粒没有以显著量包装。可以通过例如热处理来减少腺病毒污染,腺病毒比AAV对热处理更敏感。另外,可以使用杆状病毒系统以临床规模生产AAV(参见美国专利号7,479,554)。
在许多基因治疗应用中,期望对特定组织类型以高度特异性递送基因治疗载体。因此,病毒载体可以通过将配体表达为具有病毒的外表面上的病毒衣壳蛋白的融合蛋白来进行修饰以使其对给定细胞类型具有特异性。选择对已知存在于目的细胞类型上的受体具有亲和力的配体。例如,Han等人,《美国国家科学院院刊(Proc.Natl.Acad.Sci.USA)》
(1995)报道了莫洛尼鼠白血病病毒可以被修饰以表达与gp70融合的人类调蛋白(heregulin),并且重组病毒感染表达人类表皮生长因子受体的某些人类乳腺癌细胞。该原理可以扩展到其它病毒-靶细胞对,其中靶细胞表达受体,并且病毒表达包含用于细胞表面受体的配体的融合蛋白。例如,可以将丝状噬菌体工程化以展示对几乎任何选择的细胞受体具有特异性结合亲和力的抗体片段(例如,FAB或Fv)。尽管以上描述主要适用于病毒载体,但相同原理可以适用于非病毒载体。此类载体可以被工程化以含有有利于被特定靶细胞摄取的特定摄取序列。
基因治疗载体可以通过施用给个体患者体内递送,通常通过全身施用(例如静脉内、腹膜内、肌内、皮下或颅内输注)或局部施用,如下文所描述。替代地,可以将载体离体递送至细胞,诸如从个体患者取出(explanted)的细胞(例如淋巴细胞、骨髓穿刺液、组织活检)或全适供体造血干细胞,随后通常在选择已并入载体的细胞后将细胞再植入到患者中。在一些实施例中,可以利用mRNA的体内和离体递送以及RNP递送。
用于诊断、研究或基因治疗的离体细胞转染(例如,通过将转染的细胞再输注到宿主生物体中)是所属领域的技术人员熟知的。在优选的实施例中,细胞分离自受试者生物体,用RNA组合物转染,并且再输注回受试者生物体(例如患者)中。适于离体转染的各种细胞类型是本领域技术人员熟知的(参见,例如,Freshney,《动物细胞培养—
—基本技术和特殊应用指南(Culture of Animal Cells,A Manual of BasicTechnique andSpecialized Applications)》(第6版,2010))和其中引用的用于讨论如何从患者分离和培养细胞的参考文献。
合适的细胞包括但不限于真核细胞和原核细胞和/或细胞系。此类细胞或由此类细胞生成的细胞系的非限制性实例包括COS、CHO(例如CHO--S、CHO-K1、CHO-DG44、CHO-DUXB11、CHO-DUKX、CHOK1SV)、VERO、MDCK、WI38、V79、B14AF28-G3、BHK、HaK、NSO、SP2/0-Ag14、HeLa、HEK293(例如HEK293-F、HEK293-H、HEK293-
T)和perC6细胞、任何植物细胞(分化的或未分化的)以及昆虫细胞,诸如草地贪夜蛾(Spodopterafugiperda,Sf),或真菌细胞,诸如酵母菌属(Saccharomyces)、毕赤酵母属(Pichia)和裂殖酵母属(Schizosaccharomyces)。在某些实施例中,该细胞系是CHO
-K1、MDCK或HEK293细胞系。另外,可以分离原代细胞并且在用核酸酶(例如ZFN或TALEN)或核酸酶系统(例如CRISPR)处理后离体用于再引入待治疗的受试者中。
合适的原代细胞包括外周血单核细胞(PBMC)和其它血细胞亚类,诸如但不限于CD4+
T细胞或CD8+T细胞。合适的细胞还包括干细胞,诸如例如胚胎干细胞、诱导多能干细胞、造血干细胞(CD34+)、神经元干细胞和间充质干细胞。
在一个实施例中,干细胞用于细胞转染和基因治疗的离体程序中。使用干细胞的优势在于干细胞可以在体外分化成其它细胞类型或可以引入哺乳动物(诸如细胞的供体)中,其中它们将在骨髓中植入(engraft)。使用细胞因子诸如GM-CSF、IFN-γ和TNF-α将CD34+细胞体外分化为临床重要的免疫细胞类型的方法是已知的(作为非限制性实例参见Inaba等人,《实验医学杂志(J.Exp.Med.)》(1992))。
使用已知方法分离干细胞用于转导和分化。例如,通过用结合不需要的细胞诸如CD4+和CD8+(T细胞)、CD45+(panB细胞)、GR-1(粒细胞)和Iad(分化的抗原呈递细胞)的抗体对骨髓细胞进行淘选,从骨髓细胞中分离干细胞(作为非限制性实例,参见Inaba等人,《实验医学杂志》(1992))。已经修饰的干细胞也可以用于一些实施例中。
值得注意的是,本文所描述的组合物可以适用于在有丝分裂后细胞或不活跃分裂的任何细胞(例如停滞细胞)中的基因组编辑。可以使用本发明的CRISPR核酸酶进行编辑的有丝分裂后细胞的实例包括但不限于肌细胞、心肌细胞、肝细胞、骨细胞和神经元。
还可以将含有治疗性RNA组合物的载体(例如逆转录病毒、脂质体等)直接施用至生物体,用于体内转导细胞。替代地,可以施用裸RNA或mRNA。施用是通过通常用于将分子引入最终与血液或组织细胞接触的任何途径,所述途径包括但不限于注射、输注、局部应用和电穿孔。施用此类核酸的合适的方法是可用并且所属领域的技术人员熟知的,并且尽管可以使用超过一种途径施用具体组合物,但特定途径通常可以提供比另一途径更迅速并且更有效的反应。
适合于将转基因引入免疫细胞(例如T细胞)的载体包括非整合慢病毒载体。参见例如美国专利公开号2009/0117617。
药学上可接受的载体部分地由所施用的特定组合物以及用于施用该组合物的特定方法确定。因此,存在多种可用的药物组合物的合适制剂,如下所描述的(参见,例如,雷明顿制药科学(Remington's Pharmaceutical Sciences),第17版,1989)。
术语“同源定向修复”或“HDR”是指用于修复细胞中DNA损伤的机制,例如在修复DNA中的双链和单链断裂期间。HDR需要核苷酸序列同源性并且使用“核酸模板”(核酸模板或供体模板在本文中可互换使用)来修复发生双链或单链断裂的序列(例如DNA靶序列)。这导致遗传信息从例如核酸模板转移到DNA靶序列。如果核酸模板序列不同于DNA靶序列并且部分或全部核酸模板多核苷酸或寡核苷酸并入DNA靶序列中,那么HDR可以导致DNA靶序列的改变(例如插入、缺失、突变)。在一些实施例中,整个核酸模板多核苷酸、核酸模板多核苷酸的一部分或核酸模板的拷贝在DNA靶序列的位点处整合。
术语“核酸模板”和“供体”是指插入或复制到基因组中的核苷酸序列。核酸模板包含核苷酸序列(例如一个或多个核苷酸的核苷酸序列)该核苷酸序列将被添加到靶核酸中或将该核苷酸序列作为模板来改变靶核酸,或可以用于修饰靶序列。核酸模板序列可以具有任何长度,例如长度在2与10,000个核苷酸之间(或其间或以上的任何整数值),优选长度在约100与1,000个核苷酸之间(或其间的任何整数),更优选长度在约200与500个核苷酸之间。核酸模板可以是单链核酸、双链核酸。在一些实施例中,核酸模板包含对应于例如靶位置处的靶核酸的野生型序列的例如一个或多个核苷酸的核苷酸序列。在一些实施例中,核酸模板包含核糖核苷酸序列,例如一个或多个核糖核苷酸的核糖核苷酸序列,该序列对应于靶核酸的野生型序列,例如靶位置处的野生型序列。在一些实施例中,核酸模板包含修饰的核糖核苷酸。
例如,用于校正突变基因或用于增加野生型基因的表达的外源序列(也称为“供体序列”、“供体模板”或“供体”)也可以进行插入。将显而易见的是,供体序列通常与其所位于的基因组序列不完全相同。供体序列可以含有侧接两个同源区的非同源序列,以允许在目的位置处进行有效的HDR。另外,供体序列可以包含含有与细胞染色质中的目的区域不同源的序列的载体分子。供体分子可以含有几个与细胞染色质同源的不连续区域。例如,为了靶向插入通常不存在于目的区域中的序列,所述序列可以存在于供体核酸分子中并且侧接与目的区域中的序列同源的区域。
供体多核苷酸可以是单链的和/或双链的DNA或RNA,并且可以以线性或环状形式引入细胞中。参见,例如,美国专利公开号2010/0047805;2011/0281361;2011/0207221;和2019/0330620。如果以线性形式引入,可以通过本领域技术人员已知的方法保护供体序列的末端(例如,免受核酸外切降解)。例如,将一个或多个双脱氧核苷酸残基添加到线性分子的3'末端和/或将自互补寡核苷酸连接到一个末端或两个末端。参见,例如,Chang和Wilson,《美国国家科学院院刊》(1987);Nehls等人,《科学》(1996)。用于保护外源多核苷酸免于降解的其他方法包括但不限于添加末端氨基基团以及使用修饰的核苷酸间键,诸如例如,硫代磷酸酯、氨基磷酸酯(phosphoramidate)和O-甲基核糖或脱氧核糖残基。
因此,本发明的使用供体模板进行修复的实施例可以使用可以以线性或环状形式引入细胞中的DNA或RNA、单链的和/或双链的供体模板。在本发明的实施例中,基因编辑组合物包含:(1)RNA分子,其包含引导序列以在修复之前影响基因中的双链断裂,和(2)用于修复的供体RNA模板,包含引导序列的RNA分子是第一RNA分子并且供体RNA模板是第二RNA分子。在一些实施例中,引导RNA分子和模板RNA分子作为单个分子的一部分连接。
供体序列也可以是寡核苷酸并且用于内源序列的基因校正或靶向改变。寡核苷酸可以在载体上被引入到细胞中、可以被电穿孔到细胞中,或可以经由本领域已知的其它方法引入。寡核苷酸可以用于‘校正’内源基因中的突变序列(例如,β珠蛋白中的镰状突变),或可以用于将具有期望目的的序列插入内源基因座中。
多核苷酸可以作为具有额外序列(诸如例如,复制起点、启动子和编码抗生素抗性的基因)的载体分子的一部分引入到细胞中。此外,供体多核苷酸可以作为裸核酸引入,作为与诸如脂质体或泊洛沙姆等剂复合的核酸引入,或可以通过重组病毒(例如腺病毒、AAV、疱疹病毒、逆转录病毒、慢病毒和整合酶缺陷慢病毒(IDLV))进行递送。
一般插入供体,使得其表达由整合位点处的内源启动子(即驱动插入供体的内源基因表达的启动子)驱动。然而,将显而易见的是,供体可以包含启动子和/或增强子,例如组成型启动子或诱导型或组织特异性启动子。
供体分子可以插入内源基因中,使得内源基因中的全部、一些被表达或全都不被表达。例如,本文所描述的转基因可以插入内源基因座,使得内源序列中的一些(转基因的N末端和/或C末端)被表达为例如与转基因的融合体或全都不被表达,。在其它实施例中,转基因(例如,具有或不具有诸如用于内源基因的额外编码序列)整合于任何内源基因座中,例如安全港基因座,例如CCR5基因、CXCR4基因、PPP1R12c(也称为AAVS1)基因、白蛋白基因或Rosa基因。参见,例如,美国专利号7,951,925和8,110,379;美国公开号2008/0159996;20100/0218264;2010/0291048;2012/0017290;2011/0265198;
2013/0137104;2013/0122591;2013/0177983和2013/0177960以及美国临时申请号61/823,689)。
当内源序列(转基因的内源序列或一部分)与转基因一起表达时,内源序列可以是全长序列(野生型或突变体)或部分序列。优选地,内源序列是功能性的。这些全长或部分序列的功能的非限制性实例包括增加由转基因(例如,治疗基因)表达的多肽的血清半衰期和/或充当载体(carrier)。
此外,尽管不是表达必需的,但外源序列也可以包括转录或翻译调控序列,例如启动子、增强子、绝缘子(insulator)、内部核糖体进入位点、编码2A肽和/或聚腺苷酸化信号的序列。
在某些实施例中,供体分子包含选自由以下组成的组的序列:编码蛋白质的基因(例如,编码细胞或个体中缺乏的蛋白质的编码序列或编码蛋白质的基因的替代版本)、调控序列和/或编码诸如微RNA或siRNA等结构核酸的序列。
对于前述实施例,本文所公开的每个实施例被认为适用于其它所公开的实施例中的每一个。例如,应理解本发明的任何RNA分子或组合物可以被用于本发明的任何方法中。
如本文所用,所有标题仅用于组织(organization),并且不旨在以任何方式限制本发明。任何个别部分的内容可以同等地适用于所有部分。
本发明的其他目的、优点和新颖特征对于本领域普通技术人员而言在检查以下实例之后将变得明显,这些实例不旨在是限制性的。另外,如上文所描绘并且如所附权利要求书部分中所要求保护的本发明的各种实施例和方面中的每一者在以下实例中都能找到实验支持。
应理解,为了清楚起见,在单独实施例的上下文中描述的本发明的某些特征也可以在单个实施例中组合提供。相反地,为了简洁起见,在单个实施例的上下文中描述的本发明的各种特征也可以单独地或以任何合适的子组合或如在本发明的任何其它描述的实施例中合适地提供。在各种实施例的上下文中描述的某些特征不应被认为是那些实施例的基本特征,除非该实施例在不具有那些要素的情况下不可操作。
通常,本文所用的命名法(nomenclature)和本发明所用的实验室程序包括分子技术、生物化学技术、微生物学技术和重组DNA技术。此类技术在文献中得到了充分解释。
参见,例如,Sambrook等人,《分子克隆:实验室手册(Molecular Cloning:AlaboratoryManual)》(1989);Ausubel,R.M.(编辑)的,《分子生物学现行方案(CurrentProtocolsin Molecular Biology)》第I至III卷(1994);Ausubel等人,《分子生物学现行方案》,约翰·威利父子出版公司(John Wiley and Sons,Baltimore,Maryland),巴尔的摩,马里兰州(1989);Perbal,《分子克隆实用指南(APractical Guide to MolecularCloning)》,约翰·威利父子出版公司(John Wiley&Sons,New York),纽约(1988);Watson等人,《重组DNA(Recombinant DNA)》,《科学美国人书籍(Scientific American Books)》,纽约;Birren等人(编辑)的,《基因组分析:实验室手册系列(Genome Analysis:ALaboratoryManual Series)》,第1至4卷,冷泉港实验室出版社(Cold Spring HarborLaboratory Press,New York),纽约(1998);美国专利号4,666,828;4,683,202;4,801,531;5,192,659和5,272,057中阐述的方法;Cellis,J.E.(编辑)的,《细胞生物学:实验室手册(Cell Biology:
ALaboratory Handbook)》,第I至III卷(1994);Freshney,《动物细胞培养-基本技术手册(Culture of Animal Cells-AManual of Basic Technique)》第三版,Wiley-Liss,纽约(1994);Coligan J.E.(编辑)的,《免疫学现行方案(Current Protocols inImmunology)》
第I至III卷(1994);Stites等人(编辑)的,《基础和临床免疫学(Basic andClinicalImmunology)》(第8版),阿普尔顿&兰格公司(Appleton&Lange,Norwalk,CT)诺瓦克,康涅狄克州(1994);Mishell和Shiigi(编辑)的,《蛋白质纯化和表征策略-实验室课程手册(Strategies for Protein Purification and Characterization-A LaboratoryCourseManual)》CSHL出版社(1996);Clokie和Kropinski(编辑)的,《噬菌体方法和方案(Bacteriophage Methods and Protocols)》,第1卷:分离、表征和相互作用(2009),其全部通过引用并入本文。贯穿本文件提供了其它的一般参考资料。
下面提供实施例以便于更完整地理解本发明。以下实例说明了制备和实践本发明的示范性模式。然而,本发明的范围不限于这些实施例中公开的具体实施方式,这些实施例仅用于说明的目的。
实验细节
下面提供实施例以便于更全面地理解本发明。以下实施例说明了制备和实践本发明的示范性模式。然而,本发明的范围不限于这些实施例中公开的具体实施方式,这些实施例仅用于说明的目的。
OMNI-50序列,诸如OMNI-50 CRISPR重复(crRNA)序列、OMNI-50反式激活crRNA(transactivating crRNA,tracrRNA)序列和OMNI-50核酸酶多肽序列,是从秘鲁江崎氏菌菌株M6.X2中预测的。
在PCT国际申请号PCT/US2020/030782中公开了具有间隔区优化的全长支架、OMNI-50“NGG”PAM识别和哺乳动物细胞中的活性,其全部内容通过引用并入本文。OMNI-50元件(element)的概述可以在下表1中找到。
OMNI-50蛋白表达
在PCT国际申请号PCT/US2020/030782中描述了用于蛋白质生产和用于RNP组装的合成引导物生产的表达方法。简言之,针对人类细胞对核酸酶开放阅读框进行密码子优化(表1),并且将其克隆到具有以下元件的修饰的pET9a质粒中:SV40 NLS–OMNI-50ORF(来自第二个氨基酸,针对人类进行优化的)–HA标签-SV40 NLS-8His-标签。序列可以在表2中找到。OMNI-50构建体在T7表达细胞(NEB公司)中表达。细胞在TB中生长,并且通过添加1mMIPTG同时将培养物的温度降低至18℃而在对数中期进行表达。使用超声法裂解细胞,并且使用Ni-NTA柱纯化得到澄清的裂解物。收集含有OMNI蛋白的级分并且在HiLoad 16/600Superdex 200pg-SEC,AKTA Pure(GE医疗生命科学公司(GE Healthcare LifeSciences))上通过SEC进一步纯化。将含有OMNI蛋白的级分汇集并且浓缩成10mg/ml储备液以及在液氮中快速冷冻并且储存在-80℃下。
所用的合成sgRNA
OMNI-50的所有合成sgRNA均在3'端和5'端用3个2'-O-甲基3'-硫代磷酸酯合成(Agilent或Synthego)。
体外RNP活性
通过将1mg/mL蛋白质与1μM IVT-转录的或合成制造的sgRNA在室温下孵育10分钟来形成RNP。OMNI-50RNP在推荐的切割缓冲液中与100ng含有g35原型间隔区的线性5'-FAM-标记的DNA底物反应,该g35原型间隔区由与OMNI的PAM序列相邻的sgRNA靶向(表8)。通过切割片段的荧光强度除以切割和未切割片段的荧光强度的总和来计算切割效率(图2)。
在哺乳动物细胞系中的活性
此处测定的支架的不同间隔区和靶列于表8中。通过将100uM核酸酶与120uM合成引导物和100uM Cas9电穿孔增强子(IDT)混合来组装RNP。在室温下孵育10分钟后,将RNP复合物与200,000个预洗涤的U2OS、LCL或HSC细胞混合,并且根据制造商的方案(分别)使用龙沙集团(Lonza)SE、SG或P3细胞系4D-Nucleofector
3'修剪的引导物的活性
通过产生较短的支架部分来优化OMNI-50引导RNA。包含最小程度地牺牲OMNI-50核酸酶特异性和活性的短的、非天然存在的支架部分的RNA分子是高度期望的,因为它们降低了CRISPR系统的复杂性并且允许RNA分子的可靠的人工制造和生产。首先,基于‘全c’双链体版本测试3'修剪的sgRNA支架。设计六(6)个短引导物,其中3'端从OMNI-50双链体版本c的全长引导物开始修剪12个核苷酸(表3,图1A至图1G)。使用IVT试剂盒(GeneJet RNA清理和浓缩微提试剂盒(GeneJet RNA cleanupand concentration micro kit),ThermoScientific公司)与具有T7启动子的DNA模板,随后是g35 22-核苷酸间隔区(表8)和缩短的支架设计,转录引导物并且进行牛痘苗加帽。每个引导RNA分子与纯化的OMNI-50核酸酶反应以生成RNP,在体外测试g35线性模板的切割,并且在体内测试U2OS细胞系中内源性g35位点处的编辑。从图2可以看出,具有长达46个核苷酸的缩短的支架RNA分子保留了体外活性。此外,在短至70个核苷酸的支架中也检测到基于细胞的编辑水平。
用于对引导物进行评级(ranking)的引导物选择
设计测定用于基于支架的活性对支架进行评级。测定了沿支架序列的四个部分具有缺失的各种支架。支架变体根据它们在测定中的表现分为三类(高、中和低;分别见表5至表7)。还使用如下所描述的独立测定测试了来自高评分和中评分列表(图3和图4)的引导物变体的活性。
跨基因组位点和细胞类型的短引导物的活性
用不同的间隔区测试短引导RNA分子在基因组位点上和不同的细胞系上的活性(表8至表9,图5至图8)。选择几种任选的间隔区,特别是g58,其是具有挑战性的靶(challenging target);和g35或g62,它们是具有许可性的靶(permissive target)。中级别的引导物在大多数许可性位点上是有活性的(g35和g62,图5)。然而,高级别的引导物,特别是NGS17和NGS40-44,在挑战性位点(g58,图6)和在挑战性细胞系(HSC和LCL,图7和图8)中被证明是有功能性的。检测到使用一些短引导RNA分子的脱靶编辑增加,这意味着使用短引导RNA分子的RNP活性增加。如可以参见表5和表9,在第一部分、第二部分和第三部分中具有最小缺失的支架在不同的基因组位点和细胞类型中都保持高活性。
参考文献
1.Ahmad和Allen(1992)“抗体介导的脂质体包覆阿霉素对肺癌细胞的体外特异性结合和细胞毒性(Antibody-mediated Specific Binging and Cytotoxicity ofLipsome-entrapped Doxorubicin to Lung Cancer Cells in Vitro)”,《癌症研究(Cancer Research)》52:4817-20。
2.Anderson(1992)“人类基因疗法(Human gene therapy)”,《科学》256:808-13。
3.Basha等人(2011)“阳离子脂质组成对抗原呈递细胞中siRNA脂质纳米颗粒制剂的基因沉默特性的影响(Influence of Cationic Lipid Composition on GeneSilencing Properties of Lipid Nanoparticle Formulations of siRNAin Antigen-Presenting Cells)”,《分子治疗》19(12):2186-200。
4.Behr(1994)“用合成阳离子两亲物进行的基因转移:基因治疗展望(Genetransfer with synthetic cationic amphiphiles:Prospects for gene therapy)”,《生物缀合物化学》5:382-89。
5.Blaese等人(1995)“癌症治疗中的载体:它们将如何递送(Vectors in cancertherapy:how will they deliver)”《癌症基因治疗》2:291-97。
6.Blaese等人(1995)“用于ADA-SCID的T淋巴细胞导向基因治疗:4年后的初步试验结果(T lympocyte-directed gene therapy for ADA-SCID:initial trial resultsafter 4years)”,《科学》270(5235):475-80。
7.Briner等人(2014)“引导RNA功能模块指导Cas9活性和正交性(Guide RNAfunctional modules direct Cas9 activity and orthognality)”,《分子细胞》56:333-39。
8.Buchschacher和Panganiban(1992)“外源基因诱导表达的人类免疫缺陷病毒载体(Human immunodeficiency virus vectors for inducible expression of foreigngenes)”,《病毒学杂志》66:2731-39。
9.Burstein等人(2017)“来自未培养微生物的新型CRISPR-Cas系统(New CRISPR-Cas systems from uncultivated microbes)”,《自然》542:237-41。
10.Canver等人,(2015)“由Cas9介导的原位饱和诱变法分离BCL11A增强子(BCL11A enhancer dissection by Cas9-mediated in situ saturatingmutagenesis)”,《自然》第527卷,第192-214页。
11.Chang和Wilson(1987)“在哺乳动物细胞中,DNA末端的修饰相对于同源重组可以减少末端连接(Modification of DNA ends can decrease end-joining relative tohomologous recombination in mammalian cells)”,《美国国家科学院院刊》84:4959-4963。
12.Charlesworth等人(2019)“识别人类对Cas9蛋白预先存在的适应性免疫(Identification of preexisting adaptive immunity to Cas9 proteins inhumans)”,《自然医学》,25(2),249。
13.Chung等人(2006)“农杆菌并不孤单:基因通过病毒和其它细菌转移到植物中(Agrobacterium is not alone:gene transfer to plants by viruses and otherbacteria)”,《植物科学趋势》11(1):1-4。
14.Coelho等人(2013)“RNAi治疗转甲状腺素蛋白淀粉样变性的安全性和功效(Safety and efficacy of RNAi therapy for transthyretin amyloidosis)”《新英格兰医学杂志》369,819-829。
15.Crystal(1995)“将基因转移到人类:早期教训和成功的障碍(Transfer ofgenes to humans:early lessons and obstacles to success)”,《科学》270(5235):404-10。
16.Dillon(1993)“基因治疗中的调节基因表达(Regulation gene expressionin gene therapy)”《生物技术趋势(Trends in Biotechnology)》11(5):167-173。
17.Dranoff等人(1997)“用被工程化以分泌人类粒细胞巨噬细胞集落刺激因子的自体辐射黑素瘤细胞接种的I期研究(Aphase I study of vaccination withautologous,irradiated melanoma cells engineered to secrete human granulocytemacrophage colony stimulating factor)”,《人类基因治疗》8(1):111-23。
18.Dunbar等人(1995)“逆转录病毒标记的CD34富集的外周血和骨髓细胞有助于自体移植后的长期植入(Retrovirally marked CD34-enriched peripheral blood andbone marrow cells contribute to long-term engraftment after autologoustransplantation)”,《血液》85:3048-57。
19.Ellem等人(1997)“病例报告:人类首次使用粒细胞/巨噬细胞集落刺激因子诱导的自体黑素瘤细胞进行免疫治疗的免疫应答和临床过程(A case report:immuneresponses and clinical course of the first human use of ganulocyte/macrophage-colony-stimulating-factor-tranduced autologous melanoma cells forimmunotherapy)”,《癌症免疫学与免疫治疗》44:10-20。
20.Gao和Huang(1995)“阳离子脂质体介导的基因转移(Cationic liposome-mediated gene transfer)”《基因治疗(Gene Ther.)》2(10):710-22。
21.Haddada等人(1995)“使用腺病毒载体的基因治疗(Gene Therapy UsingAdenovirus Vectors)”,存在于:《腺病毒的分子库III:生物学和发病机制(The MolecularRepertoire of Adenoviruses III:Biology and Pathogenesis)》中,Doerfler和
22.Han等人(1995)“配体导向的逆转录病毒靶向人类乳腺癌细胞(Ligand-directed retro-viral targeting of human breast cancer cells)”,《美国国家科学院院刊》92(21):9747-51。
23.Humbert等人,(2019)“在非人灵长类动物中具有HbF再激活的CRISPR-Cas9–编辑的HSC富集群体的治疗相关植入(Therapeutically relevant engraftment of aCRISPR-Cas9–edited HSC-enriched population with HbF reactivation in nonhumanprimates)”,《科学·转化医学》第11卷,第1-13页。
24.Inaba等人(1992)“从补充有粒细胞/巨噬细胞集落刺激因子的小鼠骨髓培养物中生成大量树突细胞(Generation of large numbers of dendritic cells frommouse bone marrow cultures supplemented with granulocyte/macrophage colony-stimulating factor)”,《实验医学杂志》176(6):1693-702。
25.Jiang和Doudna(2017)“CRISPR-Cas9结构和机制(CRISPR-Cas9 Structuresand Mechanisms)”,《生物物理学年评(Annual Review of Biophysics)》46:505-29。
26.Jinek等人(2012)“在适应性细菌免疫中的可编程双RNA引导的DNA内切核酸酶(Aprogrammable dual-RNA-guided DNA endonuclease in adaptive bacterialimmunity)”,《科学》337(6096):816-21。
27.Johan等人(1992)“GLVR1,长臂猿白血病病毒的受体,与粗糙脉孢菌的磷酸盐通透酶同源并且在脑和胸腺中以高水平表达(GLVR1,a receptor for gibbon apeleukemia virus,is homologous to a phosphate permease of Neurospora crassa andis expressed at high levels in the brain and thymus)”,《病毒学杂志(J Virol)》66(3):1635-40。
28.Judge等人(2006)“非炎性合成siRNA介导体内强效基因沉默的设计(Designof noninflammatory synthetic siRNA mediating potent gene silencing in vivo)”,《分子治疗》13(3):494-505。
29.Kohn等人(1995)“基因修饰的脐带血细胞在患有腺苷脱氨酶缺乏的新生儿中的植入(Engraftment of gene-modified umbilical cord blood cells in neonateswith adnosine deaminase deficiency)”,《自然医学》1:1017-23。
30.Kremer和Perricaudet(1995)“腺病毒和腺相关病毒介导的基因转移(Adenovirus and adeno-associated virus mediated gene transfer)”,《英国医学公报(Br.Med.Bull.)》51(1):31-44。
31.Macdiarmid等人(2009)“用含有siRNA或细胞毒性药物的靶向微细胞序贯治疗耐药性肿瘤(Sequential treatment of drug-resistant tumors with targetedminicells containing siRNAor a cytotoxic drug)”,《自然生物技术》27(7):643-51。
32.Malech等人(1997)“慢性肉芽肿病的基因治疗后NADPH氧化酶校正的粒细胞的长期产生(Prolonged production ofNADPH oxidase-corrected granulocyes aftergene therapy of chronic granulomatous disease)”,《美国国家科学院院刊(PNAS)》94(22):12133-38。
33.Maxwell等人(2018)“基于转录-翻译的详细无细胞测定以破译CRISPR原型间隔区相邻基序(Adetailed cell-free transcription-translation-based assay todecipher CRISPR protospacer adjacent motifs)”,《方法(Methods)》14348-57
34.Miller等人(1991)“基于长臂猿白血病病毒的逆转录病毒包装细胞的构建和特性(Construction and properties of retrovirus packaging cells based ongibbon ape leukemia virus)”,《病毒学杂志》65(5):2220-24。
35.Miller(1992)“人类基因治疗走向成熟(Human gene therapy comes ofage)”,《自然》357:455-60。
36.Mir等人(2019)“II-C型CRISPR-Cas9生物学、机制和应用(Type II-C CRISPR-Cas9 Biology,Mechanism and Application)”,《ACS化学生物学(ACS Chem.Biol.)》13(2):357-365。
37.Mitani和Caskey(1993)“递送治疗性基因-匹配方式和应用(Deliveringtherapeutic genes–matching approach and application)”,《生物技术趋势》11(5):162-66。
38.Nabel和Felgner(1993)“用于免疫治疗和免疫接种的直接基因转移(Directgene transfer for immunotherapy and immunization)”,《生物技术趋势》11(5):211-15。
39.Nehls等人(1996)“胸腺上皮细胞分化中的两个遗传上可分离的步骤(Twogenetically separable steps in the differentiation of thymic epithelium)”《科学》272:886-889。
40.Nishimasu等人“Cas9与引导RNA和靶DNA复合物的晶体结构(Crystalstructure of Cas9 in complex with guide RNAand target DNA)”(2014)《细胞(Cell)》156(5):935-49。
41.Nishimasu等人(2015)“金黄色葡萄球菌Cas9的晶体结构(Crystal Structureof Staphylococcus aureus Cas9)”《细胞》162(5):1113-26。
42.Palermo等人(2018)“通过‘传感’、‘调节’和‘锁定’催化性HNH结构域在CRISPR-Cas9活化期间REC瓣的关键作用(Key role of the REC lobe during CRISPR-Cas9activation by‘sensing’,‘regulating’,and‘locking’the catalytic HNHdomain)”,《生物物理学季刊(Quarterly Reviews of Biophysics)》51,e9,1-11。
43.Remy等人(1994)“一系列亲脂性DNA结合分子的基因转移(Gene Transferwith a Series of Lipphilic DNA-Binding Molecules)”,《生物缀合物化学》5(6):647-54。
44.Sentmanat等人(2018)“CRISPR-Cas9编辑的验证策略概述(A Survey ofValidation Strategies for CRISPR-Cas9 Editing)”,《科学报告(ScientificReports)》8:888,doi:10.1038/s41598-018-19441-8。
45.Sommerfelt等人(1990)“类人猿D型逆转录病毒受体基因在人类19号染色体上的定位(Localization of the receptor gene for type D simian retroviruses onhuman chromosome 19)”,《病毒学杂志》64(12):6214-20。
46.Van Brunt(1988)“分子框架:作为生物行为者的转基因动物(Molecularframing:transgenic animals as bioactors)”《生物技术》6:1149-54。
47.Vigne等人(1995)“用于基因治疗的第三代腺载体(Third-generationadenovectors for gene therapy)”,《恢复性神经病学和神经科学》8(1、2):35-36。
48.Wagner等人(2019)“成人群体中酿脓链球菌Cas9反应性T细胞的高发率(Highprevalence of Streptococcus pyogenes Cas9-reactive T cells within the adulthuman population)”《自然医学》,25(2),242
49.Wilson等人(1989)“长臂猿白血病病毒和人类T细胞白血病病毒逆转录病毒包膜糖蛋白与莫洛尼鼠白血病病毒的gag和pol蛋白形成的传染性杂交病毒(Formation ofinfectious hybrid virion with gibbon ape leukemia virus and human T-cellleukemia virus retroviral envelope glycoproteins and the gag and pol proteinsof Moloney murine leukemia virus)”,《病毒学杂志》63:2374-78。
50.Yu等人(1994)“HIV感染基因治疗的进展(Progress towards gene therapyfor HIV infection)”,《基因治疗》1(1):13-26。
51.Zetsche等人(2015)“Cpf1是2类CRIPSR-Cas系统的单个RNA引导的内切核酸酶(Cpf1 is a single RNA-guided endonuclease of a class 2CRIPSR-Cas system)”《细胞》163(3):759-71。
52.Zuris等人(2015)“阳离子脂质介导的蛋白质递送能够在体外和体内高效地进行基于蛋白质的基因组编辑(Cationic lipid-mediated delivery of proteins enablesefficient protein based genome editing in vitro and in vivo)”《自然生物技术》33(1):73-80。
序列表
<110> 埃门多生物公司
<120> 新型OMNI-50 CRISPR核酸酶-RNA复合物
<130> 91628-A-PCT/GJG/AWG
<150> 63/109,835
<151> 2020-11-04
<160> 190
<170> PatentIn版本3.5
<210> 1
<211> 1370
<212> PRT
<213> 秘鲁江崎氏菌(Ezakiella peruensis)菌株M6.X2
<400> 1
Met Thr Lys Val Lys Asp Tyr Tyr Ile Gly Leu Asp Ile Gly Thr Ser
1 5 10 15
Ser Val Gly Trp Ala Val Thr Asp Glu Ala Tyr Asn Val Leu Lys Phe
20 25 30
Asn Ser Lys Lys Met Trp Gly Val Arg Leu Phe Asp Asp Ala Lys Thr
35 40 45
Ala Glu Glu Arg Arg Gly Gln Arg Gly Ala Arg Arg Arg Leu Asp Arg
50 55 60
Lys Lys Glu Arg Leu Ser Leu Leu Gln Asp Phe Phe Ala Glu Glu Val
65 70 75 80
Ala Lys Val Asp Pro Asn Phe Phe Leu Arg Leu Asp Asn Ser Asp Leu
85 90 95
Tyr Met Glu Asp Lys Asp Gln Lys Leu Lys Ser Lys Tyr Thr Leu Phe
100 105 110
Asn Asp Lys Asp Phe Lys Asp Lys Asn Phe His Lys Lys Tyr Pro Thr
115 120 125
Ile His His Leu Leu Met Asp Leu Ile Glu Asp Asp Ser Lys Lys Asp
130 135 140
Ile Arg Leu Val Tyr Leu Ala Cys His Tyr Leu Leu Lys Asn Arg Gly
145 150 155 160
His Phe Ile Phe Glu Gly Gln Lys Phe Asp Thr Lys Ser Ser Phe Glu
165 170 175
Asn Ser Leu Asn Glu Leu Lys Val His Leu Asn Asp Glu Tyr Gly Leu
180 185 190
Asp Leu Glu Phe Asp Asn Glu Asn Leu Ile Asn Ile Leu Thr Asp Pro
195 200 205
Lys Leu Asn Lys Thr Ala Lys Lys Lys Glu Leu Lys Ser Val Ile Gly
210 215 220
Asp Thr Lys Phe Leu Lys Ala Val Ser Ala Ile Met Ile Gly Ser Ser
225 230 235 240
Gln Lys Leu Val Asp Leu Phe Glu Asn Pro Glu Asp Phe Asp Asp Ser
245 250 255
Ala Ile Lys Ser Val Asp Phe Ser Thr Thr Ser Phe Asp Asp Lys Tyr
260 265 270
Ser Asp Tyr Glu Leu Ala Leu Gly Asp Lys Ile Ala Leu Val Asn Ile
275 280 285
Leu Lys Glu Ile Tyr Asp Ser Ser Ile Leu Glu Asn Leu Leu Lys Glu
290 295 300
Ala Asp Lys Ser Lys Asp Gly Asn Lys Tyr Ile Ser Asn Ala Phe Val
305 310 315 320
Lys Lys Tyr Asn Lys His Gly Gln Asp Leu Lys Glu Phe Lys Arg Leu
325 330 335
Val Arg Gln Tyr His Lys Ser Ala Tyr Phe Asp Ile Phe Arg Ser Glu
340 345 350
Lys Val Asn Asp Asn Tyr Val Ser Tyr Thr Lys Ser Ser Ile Ser Asn
355 360 365
Asn Lys Arg Val Lys Ala Asn Lys Phe Thr Asp Gln Glu Ala Phe Tyr
370 375 380
Lys Phe Ala Lys Lys His Leu Glu Thr Ile Lys Tyr Lys Ile Asn Lys
385 390 395 400
Val Asn Gly Ser Lys Ala Asp Leu Glu Leu Ile Asp Gly Met Leu Arg
405 410 415
Asp Met Glu Phe Lys Asn Phe Met Pro Lys Ile Lys Ser Ser Asp Asn
420 425 430
Gly Val Ile Pro Tyr Gln Leu Lys Leu Met Glu Leu Asn Lys Ile Leu
435 440 445
Glu Asn Gln Ser Lys His His Glu Phe Leu Asn Val Ser Asp Glu Tyr
450 455 460
Gly Ser Val Cys Asp Lys Ile Ala Ser Ile Met Glu Phe Arg Ile Pro
465 470 475 480
Tyr Tyr Val Gly Pro Leu Asn Pro Asn Ser Lys Tyr Ala Trp Ile Lys
485 490 495
Lys Gln Lys Asp Ser Glu Ile Thr Pro Trp Asn Phe Lys Asp Val Val
500 505 510
Asp Leu Asp Ser Ser Arg Glu Glu Phe Ile Asp Ser Leu Ile Gly Arg
515 520 525
Cys Thr Tyr Leu Lys Asp Glu Lys Val Leu Pro Lys Ala Ser Leu Leu
530 535 540
Tyr Asn Glu Tyr Met Val Leu Asn Glu Leu Asn Asn Leu Lys Leu Asn
545 550 555 560
Asp Leu Pro Ile Thr Glu Glu Met Lys Lys Lys Ile Phe Asp Gln Leu
565 570 575
Phe Lys Thr Arg Lys Lys Val Thr Leu Lys Ala Val Ala Asn Leu Leu
580 585 590
Lys Lys Glu Phe Asn Ile Asn Gly Glu Ile Leu Leu Ser Gly Thr Asp
595 600 605
Gly Asp Phe Lys Gln Gly Leu Asn Ser Tyr Asn Asp Phe Lys Ala Ile
610 615 620
Val Gly Asp Lys Val Asp Ser Asp Asp Tyr Arg Asp Lys Ile Glu Glu
625 630 635 640
Ile Ile Lys Leu Ile Val Leu Tyr Gly Asp Asp Lys Ser Tyr Leu Gln
645 650 655
Lys Lys Ile Lys Ala Gly Tyr Gly Lys Tyr Phe Thr Asp Ser Glu Ile
660 665 670
Lys Lys Met Ala Gly Leu Asn Tyr Lys Asp Trp Gly Arg Leu Ser Lys
675 680 685
Lys Leu Leu Thr Gly Leu Glu Gly Ala Asn Lys Ile Thr Gly Glu Arg
690 695 700
Gly Ser Ile Ile His Phe Met Arg Glu Tyr Asn Leu Asn Leu Met Glu
705 710 715 720
Leu Met Ser Ala Ser Phe Thr Phe Thr Glu Glu Ile Gln Lys Leu Asn
725 730 735
Pro Val Asp Asp Arg Lys Leu Ser Tyr Glu Met Val Asp Glu Leu Tyr
740 745 750
Leu Ser Pro Ser Val Lys Arg Met Leu Trp Gln Ser Leu Arg Ile Val
755 760 765
Asp Glu Ile Lys Asn Ile Met Gly Thr Asp Ser Lys Lys Ile Phe Ile
770 775 780
Glu Met Ala Arg Gly Lys Glu Glu Val Lys Ala Arg Lys Glu Ser Arg
785 790 795 800
Lys Asn Gln Leu Leu Lys Phe Tyr Lys Asp Gly Lys Lys Ala Phe Ile
805 810 815
Ser Glu Ile Gly Glu Glu Arg Tyr Ser Tyr Leu Leu Ser Glu Ile Glu
820 825 830
Gly Glu Glu Glu Asn Lys Phe Arg Trp Asp Asn Leu Tyr Leu Tyr Tyr
835 840 845
Thr Gln Leu Gly Arg Cys Met Tyr Ser Leu Glu Pro Ile Asp Ile Ser
850 855 860
Glu Leu Ser Ser Lys Asn Ile Tyr Asp Gln Asp His Ile Tyr Pro Lys
865 870 875 880
Ser Lys Ile Tyr Asp Asp Ser Ile Glu Asn Arg Val Leu Val Lys Lys
885 890 895
Asp Leu Asn Ser Lys Lys Gly Asn Ser Tyr Pro Ile Pro Asp Glu Ile
900 905 910
Leu Asn Lys Asn Cys Tyr Ala Tyr Trp Lys Ile Leu Tyr Asp Lys Gly
915 920 925
Leu Ile Gly Gln Lys Lys Tyr Thr Arg Leu Thr Arg Arg Thr Gly Phe
930 935 940
Thr Asp Asp Glu Leu Val Gln Phe Ile Ser Arg Gln Ile Val Glu Thr
945 950 955 960
Arg Gln Ala Thr Lys Glu Thr Ala Asn Leu Leu Lys Thr Ile Cys Lys
965 970 975
Asn Ser Glu Ile Val Tyr Ser Lys Ala Glu Asn Ala Ser Arg Phe Arg
980 985 990
Gln Glu Phe Asp Ile Val Lys Cys Arg Ala Val Asn Asp Leu His His
995 1000 1005
Met His Asp Ala Tyr Ile Asn Ile Ile Val Gly Asn Val Tyr Asn
1010 1015 1020
Thr Lys Phe Thr Lys Asp Pro Met Asn Phe Val Lys Lys Gln Glu
1025 1030 1035
Lys Ala Arg Ser Tyr Asn Leu Glu Asn Met Phe Lys Tyr Asp Val
1040 1045 1050
Lys Arg Gly Gly Tyr Thr Ala Trp Ile Ala Asp Asp Glu Lys Gly
1055 1060 1065
Thr Val Lys Asn Ala Ser Ile Lys Arg Ile Arg Lys Glu Leu Glu
1070 1075 1080
Gly Thr Asn Tyr Arg Phe Thr Arg Met Asn Tyr Ile Glu Ser Gly
1085 1090 1095
Ala Leu Phe Asn Ala Thr Leu Gln Arg Lys Asn Lys Gly Ser Arg
1100 1105 1110
Pro Leu Lys Asp Lys Gly Pro Lys Ser Ser Ile Glu Lys Tyr Gly
1115 1120 1125
Gly Tyr Thr Asn Ile Asn Lys Ala Cys Phe Ala Val Leu Asp Ile
1130 1135 1140
Lys Ser Lys Asn Lys Ile Glu Arg Lys Leu Met Pro Val Glu Arg
1145 1150 1155
Glu Ile Tyr Ala Lys Gln Lys Asn Asp Lys Lys Leu Ser Asp Glu
1160 1165 1170
Ile Phe Ser Lys Tyr Leu Lys Asp Arg Phe Gly Ile Glu Asp Tyr
1175 1180 1185
Arg Val Val Tyr Pro Val Val Lys Met Arg Thr Leu Leu Lys Ile
1190 1195 1200
Asp Gly Ser Tyr Tyr Phe Ile Thr Gly Gly Ser Asp Lys Thr Leu
1205 1210 1215
Glu Leu Arg Ser Ala Leu Gln Leu Ile Leu Pro Lys Lys Asn Glu
1220 1225 1230
Trp Ala Ile Lys Gln Ile Asp Lys Ser Ser Glu Asn Asp Tyr Leu
1235 1240 1245
Thr Ile Glu Arg Ile Gln Asp Leu Thr Glu Glu Leu Val Tyr Asn
1250 1255 1260
Thr Phe Asp Ile Ile Val Asn Lys Phe Lys Thr Ser Val Phe Lys
1265 1270 1275
Lys Ser Phe Leu Asn Leu Phe Gln Asp Asp Lys Ile Glu Asn Ile
1280 1285 1290
Asp Phe Lys Phe Lys Ser Met Asp Phe Lys Glu Lys Cys Lys Thr
1295 1300 1305
Leu Leu Met Leu Val Lys Ala Ile Arg Ala Ser Gly Val Arg Gln
1310 1315 1320
Asp Leu Lys Ser Ile Asp Leu Lys Ser Asp Tyr Gly Arg Leu Ser
1325 1330 1335
Ser Lys Thr Asn Asn Ile Gly Asn Tyr Gln Glu Phe Lys Ile Ile
1340 1345 1350
Asn Gln Ser Ile Thr Gly Leu Phe Glu Asn Glu Val Asp Leu Leu
1355 1360 1365
Lys Leu
1370
<210> 2
<211> 4113
<212> DNA
<213> 秘鲁江崎氏菌(Ezakiella peruensis)菌株M6.X2
<400> 2
atgacaaaag taaaagatta ttatatcgga cttgatatag gtacatcatc agttggctgg 60
gcagtaacag acgaggctta caatgttcta aaattcaact ccaagaagat gtggggagtt 120
cgtctttttg atgatgccaa aactgctgaa gaaagacgag ggcaaagagg ggccaggaga 180
agacttgacc gcaaaaaaga acgcttaagt ctcttgcaag atttttttgc agaggaagtt 240
gctaaagtag atccaaattt ctttttgcgt ctagataaca gcgaccttta tatggaggac 300
aaagatcaaa agttaaagtc caagtacact ttatttaatg ataaagattt taaagacaag 360
aacttccaca aaaaatatcc gactatccac catctcctta tggacttgat tgaagatgat 420
agcaaaaaag atattagact ggtttattta gcttgccatt acttacttaa aaatcgtggc 480
cactttattt ttgaaggaca aaaatttgat acaaagagct cctttgaaaa ttctctaaat 540
gaattaaagg tccacttaaa tgatgaatac ggtcttgatc ttgagtttga taatgaaaat 600
ttgataaata tacttacaga tcctaagtta aacaagaccg caaaaaagaa agaacttaaa 660
agtgttattg gagatacaaa atttctaaag gcagtatctg ctattatgat tggtagctct 720
caaaagctag tagatctatt tgaaaatcct gaagactttg atgattcggc aatcaaatca 780
gtggattttt ctacgacgag ttttgatgat aaatatagcg attacgagtt agcccttggg 840
gataaaattg cccttgtaaa tatattaaaa gaaatctatg actcatctat acttgaaaat 900
ttattaaaag aagccgataa atcaaaagat ggcaataagt acatttctaa cgcctttgta 960
aaaaaatata acaagcatgg ccaggacctc aaggaattta agcgcctagt tagacagtac 1020
cataaatcag cctacttcga catctttagg agtgaaaaag taaacgataa ctatgtttca 1080
tataccaagt caagtatatc caataacaag agagtgaagg cgaataagtt tacagaccaa 1140
gaagcttttt ataagtttgc taaaaagcac ctagaaacta taaaatacaa aattaataaa 1200
gttaatggta gcaaagctga ccttgaacta atagatggaa tgctaaggga tatggaattt 1260
aaaaatttca tgccaaagat aaaatcttct gataatggag ttatacctta tcaattgaaa 1320
cttatggagc taaataagat ccttgaaaac caatccaaac accatgaatt tttaaacgta 1380
tccgatgaat atggaagcgt ttgcgacaag attgcttcga ttatggaatt taggattcca 1440
tattatgttg ggcctttaaa tcctaactca aaatatgctt ggattaagaa gcaaaaggac 1500
agcgaaatca cgccatggaa ttttaaagat gtagttgatt tggattcttc aagggaagag 1560
tttatagata gcttaattgg caggtgcaca tatttaaaag atgaaaaagt tctaccaaag 1620
gcctcgcttc tctacaatga gtatatggtt ttaaatgaac tcaacaattt aaaattaaat 1680
gatcttccta ttactgaaga aatgaagaag aaaatcttcg atcaactctt taagaccagg 1740
aaaaaagtaa cattaaaggc tgtcgctaat cttctcaaaa aagaatttaa tataaatgga 1800
gaaatcctat tgtccggcac agatggggat tttaaacaag ggctaaactc ttataacgat 1860
tttaaggcca ttgttgggga caaggttgac agcgacgact atagggataa aatcgaagaa 1920
attatcaagc taatcgtcct ctatggagat gacaaatctt acttgcaaaa gaaaataaag 1980
gcgggatacg gcaagtattt tacagattca gaaatcaaaa agatggctgg cctaaattat 2040
aaagactggg gcagattaag taaaaaacta ctcacaggtt tagaaggcgc caataaaatt 2100
acaggcgaaa gaggatctat aatccatttt atgcgtgagt acaatttaaa cttaatggaa 2160
ttaatgagcg ccagcttcac ttttacagag gaaattcaaa agttaaatcc agttgacgat 2220
agaaaactct cctatgagat ggttgatgag ctttatttat caccttcagt taagagaatg 2280
ttatggcaaa gtctaagaat agttgatgaa attaaaaata taatgggcac tgattccaag 2340
aaaatcttta ttgaaatggc caggggcaaa gaagaagtca aggctagaaa agaatctaga 2400
aaaaatcagc tcttaaaatt ttacaaggat ggcaaaaaag cctttatatc agaaatcggc 2460
gaagaaagat atagctatct tttaagtgaa atcgaaggag aagaggaaaa caaattcaga 2520
tgggacaatc tttatctcta ctacacccag cttggcaggt gtatgtatag tcttgagcca 2580
attgatattt cagaactctc atcgaaaaac atctatgacc aagaccacat ttatccaaag 2640
tcaaaaatct atgatgattc aattgaaaac agagttttgg ttaagaaaga tttaaatagc 2700
aagaaaggca attcataccc aataccggat gagattttaa ataaaaattg ctatgcttat 2760
tggaaaattc tatatgacaa gggactaatt ggtcaaaaga aatataccag acttacacgt 2820
aggacaggat ttactgatga tgaacttgtc caatttatat ccaggcaaat agttgagacc 2880
aggcaggcta ccaaagaaac agcaaatctc ttaaaaacca tttgcaaaaa ttcagaaata 2940
gtttactcta aggcagaaaa tgctagcaga ttcagacagg aatttgatat agtaaaatgc 3000
cgtgcagtca atgacctcca ccacatgcat gacgcttata taaatataat cgttggcaat 3060
gtctacaata caaaatttac caaagacccc atgaactttg tcaaaaaaca agagaaagct 3120
agaagttata acttggaaaa catgtttaaa tatgacgtaa agcgcggggg ctatacagca 3180
tggatagcag acgatgaaaa aggcactgtt aaaaatgcta gcatcaagag aataagaaaa 3240
gaactagagg ggaccaacta cagatttact cgcatgaatt atatagaaag tggtgcacta 3300
tttaatgcta ccctgcaaag aaaaaacaaa ggaagtcgcc ctctaaaaga taaggggcct 3360
aagagctcaa tagaaaaata tggtggatat actaatataa acaaggcttg ctttgcagtg 3420
ttggatatta aatcaaaaaa taaaatagaa agaaaattaa tgccagttga aagagaaata 3480
tacgctaagc aaaagaatga taaaaaattg agtgatgaaa tatttagcaa atatttgaaa 3540
gatagattcg gaattgaaga ttatagagta gtatatcctg tagtaaagat gagaactttg 3600
ttaaaaatag atggatctta ttattttata actggtggaa gtgacaaaac attggaatta 3660
aggagtgcac ttcaattaat attaccaaag aaaaatgaat gggcaataaa gcaaattgat 3720
aaatccagtg agaatgatta cctaacaatt gaaaggatac aagatttaac ggaagaactt 3780
gtatacaata cgtttgatat aatagtgaat aaatttaaaa catctgtatt taaaaaatca 3840
tttttgaatt tattccaaga tgataaaatc gaaaatatag attttaaatt caaatcaatg 3900
gattttaaag aaaagtgtaa aactctattg atgctagtaa aagccatcag agcttctggt 3960
gtacgccaag acttaaaatc tatagattta aaatcagact atggtagatt gagctccaag 4020
actaataata taggaaacta tcaagaattt aaaatcataa accaatcaat tacaggcctc 4080
tttgaaaacg aagtggactt gttaaaatta tga 4113
<210> 3
<211> 4107
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 针对人类优化的OMNI-50 DNA序列
<400> 3
accaaggtga aggactacta cataggcttg gacatcggca cctctagcgt cgggtgggcc 60
gtcaccgatg aagcctataa cgtgcttaag tttaatagca agaaaatgtg gggcgtgcgg 120
ctgttcgacg acgctaagac ggcagaggag cgtaggggcc agcgaggagc aagacgacgt 180
ctggatcgga agaaggagag actcagcctg ctgcaggact tcttcgccga agaggtagca 240
aaggtcgacc ccaacttctt cctcaggctg gacaattccg atctgtacat ggaagataag 300
gaccagaaac tgaaaagcaa atatacactg ttcaacgaca aggacttcaa ggataagaat 360
tttcataaga agtaccccac aatacatcac ctgctgatgg atctgatcga ggacgacagt 420
aagaaggaca tccggctcgt ctacctggcc tgtcactatt tgctcaagaa caggggtcat 480
ttcatcttcg agggccagaa gttcgacact aaatcaagct tcgagaacag tttgaacgag 540
ctcaaagttc atttgaacga cgagtatgga ctggacctcg aatttgacaa cgagaacctg 600
attaacatct tgactgaccc aaaactcaat aaaacggcca agaagaagga gctgaagtcc 660
gtaatcggcg acaccaagtt cctcaaagcc gtttccgcga taatgatcgg ctctagccag 720
aaactcgtcg acttgttcga gaaccccgag gatttcgacg actctgcgat aaagtccgtt 780
gacttctcaa ctacctcttt cgacgacaag tactctgact atgaactcgc tctgggtgac 840
aagatcgctc tggtcaacat ccttaaggaa atttacgata gctccatcct cgagaacctg 900
ctcaaagagg cagacaagtc taaggacggt aacaaatata tcagtaatgc attcgtgaag 960
aagtacaata aacacggaca agatctgaaa gagttcaaac gtctggtacg acaatatcac 1020
aagagtgcgt attttgatat tttcagatcc gagaaggtga atgacaatta cgtcagctac 1080
actaaaagct caattagcaa caataaacgc gtcaaagcaa acaagttcac tgatcaagag 1140
gccttctaca aattcgccaa gaaacatctg gagacaatca agtataagat caacaaggta 1200
aacggctcca aggcagatct ggagctgatt gacgggatgc tgcgggacat ggagttcaag 1260
aactttatgc ccaaaattaa gtccagtgac aacggggtga ttccatacca gctcaagctg 1320
atggaattga acaaaatact cgagaatcag tcaaagcatc acgagttcct caatgtcagc 1380
gacgagtacg gctccgtgtg tgataaaatc gcatctatca tggagttccg tatcccctac 1440
tacgtgggac ccctgaaccc caatagcaag tacgcctgga tcaagaagca gaaagatagt 1500
gagattactc cctggaactt caaggacgtc gtggaccttg actccagcag agaggagttc 1560
attgactcac tgatcggacg ctgtacttac cttaaggacg agaaggtcct tcccaaagct 1620
tctttgctgt ataacgaata catggtgctg aacgagctga ataacctgaa gttgaacgac 1680
cttcccatca ccgaggagat gaagaagaag atatttgacc agttgttcaa aacaagaaag 1740
aaggtcaccc ttaaagcggt ggcaaacctg ctgaagaagg agttcaacat caacggcgag 1800
attctgctct ctgggaccga cggtgacttc aagcagggct tgaactcata caatgacttc 1860
aaagctatcg tgggcgataa agtcgattcc gatgattacc gggacaagat tgaggagatc 1920
attaaactga tagttcttta cggtgacgat aagagttacc ttcagaagaa gattaaagct 1980
gggtatggaa aatacttcac cgacagtgag attaagaaaa tggcggggct gaactacaag 2040
gattggggaa ggctctcaaa gaagctgctg acgggactcg agggtgcaaa caagatcact 2100
ggagagcggg gctccattat tcacttcatg agggaatata accttaatct gatggagctt 2160
atgtcagctt catttacgtt caccgaagag atacagaaac ttaaccccgt ggatgaccgc 2220
aagctgtcat acgaaatggt ggacgaactg tacctttctc ccagtgtgaa acggatgctc 2280
tggcagtccc tgcgcatcgt cgacgagata aagaacatca tgggaaccga cagtaagaag 2340
attttcatcg agatggctcg gggtaaggaa gaggtgaaag cccgcaagga gtcaaggaag 2400
aaccaactgc tgaagttcta taaagacgga aagaaggcat tcatcagcga gattggcgag 2460
gagaggtact cttacttgct ttctgagata gagggtgagg aagagaataa gtttcgatgg 2520
gataacctgt acctttatta tactcaactg ggtcgctgca tgtactcttt ggaacctatc 2580
gacatatctg agctgtcttc aaagaatatt tacgatcagg atcatatcta ccccaaaagc 2640
aagatttacg acgacagtat cgagaatagg gtgctggtga agaaggacct taactccaag 2700
aagggtaaca gctatcctat cccagacgaa atcctgaaca agaactgtta cgcctactgg 2760
aagatcctgt acgataaagg tcttatcggg cagaagaagt acactcggct gacccggaga 2820
actggcttca cggacgacga gctcgttcag ttcatctcaa gacagatcgt ggaaactaga 2880
caagcaacaa aggagactgc taacctgctc aagacaatat gtaagaactc cgagatcgtg 2940
tattccaaag ccgagaacgc aagtcggttt aggcaagagt tcgacatcgt gaagtgtagg 3000
gcggtgaacg atcttcatca tatgcacgat gcctacatca acatcatagt ggggaacgtg 3060
tataacacca agttcacgaa ggaccctatg aatttcgtaa agaagcagga aaaggcgcgg 3120
agctacaatc tcgagaatat gttcaagtac gatgtgaaac gtggcggata caccgcttgg 3180
atcgccgatg acgagaaggg caccgtgaag aacgcgagta ttaaacgtat ccggaaggag 3240
ctggaaggca caaattatag gttcacaaga atgaactaca ttgagtctgg agcgcttttc 3300
aacgccactc tccagcggaa gaataagggc tccagacccc tgaaggacaa aggcccgaaa 3360
tcttccatcg agaagtacgg cggctacaca aacatcaata aagcctgttt cgctgttctt 3420
gacatcaagt ctaagaacaa gattgagagg aagctgatgc ccgtcgagcg tgagatctat 3480
gccaaacaga agaacgacaa gaagctgtcc gacgagattt tctcaaagta cctcaaggac 3540
cgatttggca tcgaggacta cagggttgtc tacccagtgg tgaaaatgcg cacactgctc 3600
aagatcgacg gcagctacta cttcatcaca ggcggttctg ataagaccct ggagttgcga 3660
tctgctctgc agctgattct ccctaagaag aacgagtggg cgatcaaaca gatcgacaag 3720
tcttccgaaa acgactatct gacgatcgag cgtatccagg acctgaccga ggagctggtg 3780
tataacactt tcgacatcat cgtcaacaag ttcaagacca gtgtcttcaa gaagtctttc 3840
cttaacttgt ttcaggacga caagattgag aacattgact tcaagtttaa gtccatggac 3900
ttcaaggaga aatgcaagac acttctcatg ctggtcaagg cgattcgggc atccggcgtg 3960
aggcaggatc tcaagtccat cgacctcaag tctgattacg gacggctcag ttcaaagacc 4020
aacaacatcg gcaattacca ggagttcaag attattaatc agtccatcac tggactgttc 4080
gagaatgagg tcgatctcct gaagctg 4107
<210> 4
<211> 106
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> sgRNA V1支架
<400> 4
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuugc uauugcaagc uuuuuu 106
<210> 5
<211> 112
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> sgRNA V2支架
<400> 5
guuugagagu uauguaagaa auuacaugac gaguucaaau aaaaauuuau ucaaaccgcc 60
uauuuauagg ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 112
<210> 6
<211> 1410
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> pET9a OMNI50-His标签编码ORF蛋白序列
<400> 6
Met Pro Lys Lys Lys Arg Lys Val Gly Thr Thr Lys Val Lys Asp Tyr
1 5 10 15
Tyr Ile Gly Leu Asp Ile Gly Thr Ser Ser Val Gly Trp Ala Val Thr
20 25 30
Asp Glu Ala Tyr Asn Val Leu Lys Phe Asn Ser Lys Lys Met Trp Gly
35 40 45
Val Arg Leu Phe Asp Asp Ala Lys Thr Ala Glu Glu Arg Arg Gly Gln
50 55 60
Arg Gly Ala Arg Arg Arg Leu Asp Arg Lys Lys Glu Arg Leu Ser Leu
65 70 75 80
Leu Gln Asp Phe Phe Ala Glu Glu Val Ala Lys Val Asp Pro Asn Phe
85 90 95
Phe Leu Arg Leu Asp Asn Ser Asp Leu Tyr Met Glu Asp Lys Asp Gln
100 105 110
Lys Leu Lys Ser Lys Tyr Thr Leu Phe Asn Asp Lys Asp Phe Lys Asp
115 120 125
Lys Asn Phe His Lys Lys Tyr Pro Thr Ile His His Leu Leu Met Asp
130 135 140
Leu Ile Glu Asp Asp Ser Lys Lys Asp Ile Arg Leu Val Tyr Leu Ala
145 150 155 160
Cys His Tyr Leu Leu Lys Asn Arg Gly His Phe Ile Phe Glu Gly Gln
165 170 175
Lys Phe Asp Thr Lys Ser Ser Phe Glu Asn Ser Leu Asn Glu Leu Lys
180 185 190
Val His Leu Asn Asp Glu Tyr Gly Leu Asp Leu Glu Phe Asp Asn Glu
195 200 205
Asn Leu Ile Asn Ile Leu Thr Asp Pro Lys Leu Asn Lys Thr Ala Lys
210 215 220
Lys Lys Glu Leu Lys Ser Val Ile Gly Asp Thr Lys Phe Leu Lys Ala
225 230 235 240
Val Ser Ala Ile Met Ile Gly Ser Ser Gln Lys Leu Val Asp Leu Phe
245 250 255
Glu Asn Pro Glu Asp Phe Asp Asp Ser Ala Ile Lys Ser Val Asp Phe
260 265 270
Ser Thr Thr Ser Phe Asp Asp Lys Tyr Ser Asp Tyr Glu Leu Ala Leu
275 280 285
Gly Asp Lys Ile Ala Leu Val Asn Ile Leu Lys Glu Ile Tyr Asp Ser
290 295 300
Ser Ile Leu Glu Asn Leu Leu Lys Glu Ala Asp Lys Ser Lys Asp Gly
305 310 315 320
Asn Lys Tyr Ile Ser Asn Ala Phe Val Lys Lys Tyr Asn Lys His Gly
325 330 335
Gln Asp Leu Lys Glu Phe Lys Arg Leu Val Arg Gln Tyr His Lys Ser
340 345 350
Ala Tyr Phe Asp Ile Phe Arg Ser Glu Lys Val Asn Asp Asn Tyr Val
355 360 365
Ser Tyr Thr Lys Ser Ser Ile Ser Asn Asn Lys Arg Val Lys Ala Asn
370 375 380
Lys Phe Thr Asp Gln Glu Ala Phe Tyr Lys Phe Ala Lys Lys His Leu
385 390 395 400
Glu Thr Ile Lys Tyr Lys Ile Asn Lys Val Asn Gly Ser Lys Ala Asp
405 410 415
Leu Glu Leu Ile Asp Gly Met Leu Arg Asp Met Glu Phe Lys Asn Phe
420 425 430
Met Pro Lys Ile Lys Ser Ser Asp Asn Gly Val Ile Pro Tyr Gln Leu
435 440 445
Lys Leu Met Glu Leu Asn Lys Ile Leu Glu Asn Gln Ser Lys His His
450 455 460
Glu Phe Leu Asn Val Ser Asp Glu Tyr Gly Ser Val Cys Asp Lys Ile
465 470 475 480
Ala Ser Ile Met Glu Phe Arg Ile Pro Tyr Tyr Val Gly Pro Leu Asn
485 490 495
Pro Asn Ser Lys Tyr Ala Trp Ile Lys Lys Gln Lys Asp Ser Glu Ile
500 505 510
Thr Pro Trp Asn Phe Lys Asp Val Val Asp Leu Asp Ser Ser Arg Glu
515 520 525
Glu Phe Ile Asp Ser Leu Ile Gly Arg Cys Thr Tyr Leu Lys Asp Glu
530 535 540
Lys Val Leu Pro Lys Ala Ser Leu Leu Tyr Asn Glu Tyr Met Val Leu
545 550 555 560
Asn Glu Leu Asn Asn Leu Lys Leu Asn Asp Leu Pro Ile Thr Glu Glu
565 570 575
Met Lys Lys Lys Ile Phe Asp Gln Leu Phe Lys Thr Arg Lys Lys Val
580 585 590
Thr Leu Lys Ala Val Ala Asn Leu Leu Lys Lys Glu Phe Asn Ile Asn
595 600 605
Gly Glu Ile Leu Leu Ser Gly Thr Asp Gly Asp Phe Lys Gln Gly Leu
610 615 620
Asn Ser Tyr Asn Asp Phe Lys Ala Ile Val Gly Asp Lys Val Asp Ser
625 630 635 640
Asp Asp Tyr Arg Asp Lys Ile Glu Glu Ile Ile Lys Leu Ile Val Leu
645 650 655
Tyr Gly Asp Asp Lys Ser Tyr Leu Gln Lys Lys Ile Lys Ala Gly Tyr
660 665 670
Gly Lys Tyr Phe Thr Asp Ser Glu Ile Lys Lys Met Ala Gly Leu Asn
675 680 685
Tyr Lys Asp Trp Gly Arg Leu Ser Lys Lys Leu Leu Thr Gly Leu Glu
690 695 700
Gly Ala Asn Lys Ile Thr Gly Glu Arg Gly Ser Ile Ile His Phe Met
705 710 715 720
Arg Glu Tyr Asn Leu Asn Leu Met Glu Leu Met Ser Ala Ser Phe Thr
725 730 735
Phe Thr Glu Glu Ile Gln Lys Leu Asn Pro Val Asp Asp Arg Lys Leu
740 745 750
Ser Tyr Glu Met Val Asp Glu Leu Tyr Leu Ser Pro Ser Val Lys Arg
755 760 765
Met Leu Trp Gln Ser Leu Arg Ile Val Asp Glu Ile Lys Asn Ile Met
770 775 780
Gly Thr Asp Ser Lys Lys Ile Phe Ile Glu Met Ala Arg Gly Lys Glu
785 790 795 800
Glu Val Lys Ala Arg Lys Glu Ser Arg Lys Asn Gln Leu Leu Lys Phe
805 810 815
Tyr Lys Asp Gly Lys Lys Ala Phe Ile Ser Glu Ile Gly Glu Glu Arg
820 825 830
Tyr Ser Tyr Leu Leu Ser Glu Ile Glu Gly Glu Glu Glu Asn Lys Phe
835 840 845
Arg Trp Asp Asn Leu Tyr Leu Tyr Tyr Thr Gln Leu Gly Arg Cys Met
850 855 860
Tyr Ser Leu Glu Pro Ile Asp Ile Ser Glu Leu Ser Ser Lys Asn Ile
865 870 875 880
Tyr Asp Gln Asp His Ile Tyr Pro Lys Ser Lys Ile Tyr Asp Asp Ser
885 890 895
Ile Glu Asn Arg Val Leu Val Lys Lys Asp Leu Asn Ser Lys Lys Gly
900 905 910
Asn Ser Tyr Pro Ile Pro Asp Glu Ile Leu Asn Lys Asn Cys Tyr Ala
915 920 925
Tyr Trp Lys Ile Leu Tyr Asp Lys Gly Leu Ile Gly Gln Lys Lys Tyr
930 935 940
Thr Arg Leu Thr Arg Arg Thr Gly Phe Thr Asp Asp Glu Leu Val Gln
945 950 955 960
Phe Ile Ser Arg Gln Ile Val Glu Thr Arg Gln Ala Thr Lys Glu Thr
965 970 975
Ala Asn Leu Leu Lys Thr Ile Cys Lys Asn Ser Glu Ile Val Tyr Ser
980 985 990
Lys Ala Glu Asn Ala Ser Arg Phe Arg Gln Glu Phe Asp Ile Val Lys
995 1000 1005
Cys Arg Ala Val Asn Asp Leu His His Met His Asp Ala Tyr Ile
1010 1015 1020
Asn Ile Ile Val Gly Asn Val Tyr Asn Thr Lys Phe Thr Lys Asp
1025 1030 1035
Pro Met Asn Phe Val Lys Lys Gln Glu Lys Ala Arg Ser Tyr Asn
1040 1045 1050
Leu Glu Asn Met Phe Lys Tyr Asp Val Lys Arg Gly Gly Tyr Thr
1055 1060 1065
Ala Trp Ile Ala Asp Asp Glu Lys Gly Thr Val Lys Asn Ala Ser
1070 1075 1080
Ile Lys Arg Ile Arg Lys Glu Leu Glu Gly Thr Asn Tyr Arg Phe
1085 1090 1095
Thr Arg Met Asn Tyr Ile Glu Ser Gly Ala Leu Phe Asn Ala Thr
1100 1105 1110
Leu Gln Arg Lys Asn Lys Gly Ser Arg Pro Leu Lys Asp Lys Gly
1115 1120 1125
Pro Lys Ser Ser Ile Glu Lys Tyr Gly Gly Tyr Thr Asn Ile Asn
1130 1135 1140
Lys Ala Cys Phe Ala Val Leu Asp Ile Lys Ser Lys Asn Lys Ile
1145 1150 1155
Glu Arg Lys Leu Met Pro Val Glu Arg Glu Ile Tyr Ala Lys Gln
1160 1165 1170
Lys Asn Asp Lys Lys Leu Ser Asp Glu Ile Phe Ser Lys Tyr Leu
1175 1180 1185
Lys Asp Arg Phe Gly Ile Glu Asp Tyr Arg Val Val Tyr Pro Val
1190 1195 1200
Val Lys Met Arg Thr Leu Leu Lys Ile Asp Gly Ser Tyr Tyr Phe
1205 1210 1215
Ile Thr Gly Gly Ser Asp Lys Thr Leu Glu Leu Arg Ser Ala Leu
1220 1225 1230
Gln Leu Ile Leu Pro Lys Lys Asn Glu Trp Ala Ile Lys Gln Ile
1235 1240 1245
Asp Lys Ser Ser Glu Asn Asp Tyr Leu Thr Ile Glu Arg Ile Gln
1250 1255 1260
Asp Leu Thr Glu Glu Leu Val Tyr Asn Thr Phe Asp Ile Ile Val
1265 1270 1275
Asn Lys Phe Lys Thr Ser Val Phe Lys Lys Ser Phe Leu Asn Leu
1280 1285 1290
Phe Gln Asp Asp Lys Ile Glu Asn Ile Asp Phe Lys Phe Lys Ser
1295 1300 1305
Met Asp Phe Lys Glu Lys Cys Lys Thr Leu Leu Met Leu Val Lys
1310 1315 1320
Ala Ile Arg Ala Ser Gly Val Arg Gln Asp Leu Lys Ser Ile Asp
1325 1330 1335
Leu Lys Ser Asp Tyr Gly Arg Leu Ser Ser Lys Thr Asn Asn Ile
1340 1345 1350
Gly Asn Tyr Gln Glu Phe Lys Ile Ile Asn Gln Ser Ile Thr Gly
1355 1360 1365
Leu Phe Glu Asn Glu Val Asp Leu Leu Lys Leu Gly Ser Tyr Pro
1370 1375 1380
Tyr Asp Val Pro Asp Tyr Ala Ala Ala Pro Lys Lys Lys Arg Lys
1385 1390 1395
Val Ala Ala Ser His His His His His His His His
1400 1405 1410
<210> 7
<211> 6439
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> pET9a OMNI50-His标签DNA序列
<400> 7
taatacgact cactataggg agaccacaac ggtttccctc tagagagaca ataaccctga 60
taatgcttca ataatattga aaaaggaaga gtatgcctaa gaagaagaga aaggtgggta 120
ccaccaaggt gaaggactac tacataggct tggacatcgg cacctctagc gtcgggtggg 180
ccgtcaccga tgaagcctat aacgtgctta agtttaatag caagaaaatg tggggcgtgc 240
ggctgttcga cgacgctaag acggcagagg agcgtagggg ccagcgagga gcaagacgac 300
gtctggatcg gaagaaggag agactcagcc tgctgcagga cttcttcgcc gaagaggtag 360
caaaggtcga ccccaacttc ttcctcaggc tggacaattc cgatctgtac atggaagata 420
aggaccagaa actgaaaagc aaatatacac tgttcaacga caaggacttc aaggataaga 480
attttcataa gaagtacccc acaatacatc acctgctgat ggatctgatc gaggacgaca 540
gtaagaagga catccggctc gtctacctgg cctgtcacta tttgctcaag aacaggggtc 600
atttcatctt cgagggccag aagttcgaca ctaaatcaag cttcgagaac agtttgaacg 660
agctcaaagt tcatttgaac gacgagtatg gactggacct cgaatttgac aacgagaacc 720
tgattaacat cttgactgac ccaaaactca ataaaacggc caagaagaag gagctgaagt 780
ccgtaatcgg cgacaccaag ttcctcaaag ccgtttccgc gataatgatc ggctctagcc 840
agaaactcgt cgacttgttc gagaaccccg aggatttcga cgactctgcg ataaagtccg 900
ttgacttctc aactacctct ttcgacgaca agtactctga ctatgaactc gctctgggtg 960
acaagatcgc tctggtcaac atccttaagg aaatttacga tagctccatc ctcgagaacc 1020
tgctcaaaga ggcagacaag tctaaggacg gtaacaaata tatcagtaat gcattcgtga 1080
agaagtacaa taaacacgga caagatctga aagagttcaa acgtctggta cgacaatatc 1140
acaagagtgc gtattttgat attttcagat ccgagaaggt gaatgacaat tacgtcagct 1200
acactaaaag ctcaattagc aacaataaac gcgtcaaagc aaacaagttc actgatcaag 1260
aggccttcta caaattcgcc aagaaacatc tggagacaat caagtataag atcaacaagg 1320
taaacggctc caaggcagat ctggagctga ttgacgggat gctgcgggac atggagttca 1380
agaactttat gcccaaaatt aagtccagtg acaacggggt gattccatac cagctcaagc 1440
tgatggaatt gaacaaaata ctcgagaatc agtcaaagca tcacgagttc ctcaatgtca 1500
gcgacgagta cggctccgtg tgtgataaaa tcgcatctat catggagttc cgtatcccct 1560
actacgtggg acccctgaac cccaatagca agtacgcctg gatcaagaag cagaaagata 1620
gtgagattac tccctggaac ttcaaggacg tcgtggacct cgactccagc agagaggagt 1680
tcattgactc actgatcgga cgctgtactt accttaagga cgagaaggtc cttcccaaag 1740
cttctttgct gtataacgaa tacatggtgc tgaacgagct gaataacctg aagttgaacg 1800
accttcccat caccgaggag atgaagaaga agatatttga ccagttgttc aaaacaagaa 1860
agaaggtcac ccttaaagcg gtggcaaacc tgctgaagaa ggagttcaac atcaacggcg 1920
agattctgct ctctgggacc gacggtgact tcaagcaggg cttgaactca tacaatgact 1980
tcaaagctat cgtgggcgat aaagtcgatt ccgatgatta ccgggacaag attgaggaga 2040
tcattaaact gatagttctt tacggtgacg ataagagtta ccttcagaag aagattaaag 2100
ctgggtatgg aaaatacttc accgacagtg agattaagaa aatggcgggg ctgaactaca 2160
aggattgggg aaggctctca aagaagctgc tgacgggact cgagggtgca aacaagatca 2220
ctggagagcg gggctccatt attcacttca tgagggaata taaccttaat ctgatggagc 2280
ttatgtcagc ttcatttacg ttcaccgaag agatacagaa acttaacccc gtggatgacc 2340
gcaagctgtc atacgaaatg gtggacgaac tgtacctttc tcccagtgtg aaacggatgc 2400
tctggcagtc cctgcgcatc gtcgacgaga taaagaacat catgggaacc gacagtaaga 2460
agattttcat cgagatggct cggggtaagg aagaggtgaa agcccgcaag gagtcaagga 2520
agaaccaact gctgaagttc tataaagacg gaaagaaggc attcatcagc gagattggcg 2580
aggagaggta ctcttacttg ctttctgaga tagagggtga ggaagagaat aagtttcgat 2640
gggataacct gtacctttat tatactcaac tgggtcgctg catgtactct ttggaaccta 2700
tcgacatatc tgagctgtct tcaaagaata tttacgatca ggatcatatc taccccaaaa 2760
gcaagattta cgacgacagt atcgagaata gggtgctggt gaagaaggac cttaactcca 2820
agaagggtaa cagctatcct atcccagacg aaatcctgaa caagaactgt tacgcctact 2880
ggaagatcct gtacgataaa ggtcttatcg ggcagaagaa gtacactcgg ctgacccgga 2940
gaactggctt cacggacgac gagctcgttc agttcatctc aagacagatc gtggaaacta 3000
gacaagcaac aaaggagact gctaacctgc tcaagacaat atgtaagaac tccgagatcg 3060
tgtattccaa agccgagaac gcaagtcggt ttaggcaaga gttcgacatc gtgaagtgta 3120
gggcggtgaa cgatcttcat catatgcacg atgcctacat caacatcata gtggggaacg 3180
tgtataacac caagttcacg aaggacccta tgaatttcgt aaagaagcag gaaaaggcgc 3240
ggagctacaa tctcgagaat atgttcaagt acgatgtgaa acgtggcgga tacaccgctt 3300
ggatcgccga tgacgagaag ggcaccgtga agaacgcgag tattaaacgt atccggaagg 3360
agctggaagg cacaaattat aggttcacaa gaatgaacta cattgagtct ggagcgcttt 3420
tcaacgccac tctccagcgg aagaataagg gctccagacc cctgaaggac aaaggcccga 3480
aatcttccat cgagaagtac ggcggctaca caaacatcaa taaagcctgt ttcgctgttc 3540
ttgacatcaa gtctaagaac aagattgaga ggaagctgat gcccgtcgag cgtgagatct 3600
atgccaaaca gaagaacgac aagaagctgt ccgacgagat tttctcaaag tacctcaagg 3660
accgatttgg catcgaggac tacagggttg tctacccagt ggtgaaaatg cgcacactgc 3720
tcaagatcga cggcagctac tacttcatca caggcggttc tgataagacc ctggagttgc 3780
gatctgctct gcagctgatt ctccctaaga agaacgagtg ggcgatcaaa cagatcgaca 3840
agtcttccga aaacgactat ctgacgatcg agcgtatcca ggacctgacc gaggagctgg 3900
tgtataacac tttcgacatc atcgtcaaca agttcaagac cagtgtcttc aagaagtctt 3960
tccttaactt gtttcaggac gacaagattg agaacattga cttcaagttt aagtccatgg 4020
acttcaagga gaaatgcaag acacttctca tgctggtcaa ggcgattcgg gcatccggcg 4080
tgaggcagga tctcaagtcc atcgacctca agtctgatta cggacggctc agttcaaaga 4140
ccaacaacat cggcaattac caggagttca agattattaa tcagtccatc actggactgt 4200
tcgagaatga ggtcgatctc ctgaagctgg gatcctaccc atacgatgtt ccagattacg 4260
cggccgctcc aaaaaagaaa agaaaagttg cggctagcca tcatcaccat caccatcatc 4320
attaaggctg ctaacaaagc ccgaaaggaa gctgagttgg ctgctgccac cgctgagcaa 4380
taactagcat aaccccttgg ggcctctaaa cgggtcttga ggggtttttt gctgaaagga 4440
ggaactatat ccggatatcc acaggacggg tgtggtcgcc atgatcgcgt agtcgatagt 4500
ggctccaagt agcgaagcga gcaggactgg gcggcggcca aagcggtcgg acagtgctcc 4560
gagaacgggt gcgcatagaa attgcatcaa cgcatatagc gctagcagca cgccatagtg 4620
actggcgatg ctgtcggaat ggacgatatc ccgcaagagg cccggcagta ccggcataac 4680
caagcctatg cctacagcat ccagggtgac ggtgccgagg atgacgatga gcgcattgtt 4740
agatttcata cacggtgcct gactgcgtta gcaatttaac tgtgataaac taccgcatta 4800
aagcttatcg atgataagct gtcaaacatg agaattctta gaaaaactca tcgagcatca 4860
aatgaaactg caatttattc atatcaggat tatcaatacc atatttttga aaaagccgtt 4920
tctgtaatga aggagaaaac tcaccgaggc agttccatag gatggcaaga tcctggtatc 4980
ggtctgcgat tccgactcgt ccaacatcaa tacaacctat taatttcccc tcgtcaaaaa 5040
taaggttatc aagtgagaaa tcaccatgag tgacgactga atccggtgag aatggcaaaa 5100
gcttatgcat ttctttccag acttgttcaa caggccagcc attacgctcg tcatcaaaat 5160
cactcgcatc aaccaaaccg ttattcattc gtgattgcgc ctgagcgaga cgaaatacgc 5220
gatcgctgtt aaaaggacaa ttacaaacag gaatcgaatg caaccggcgc aggaacactg 5280
ccagcgcatc aacaatattt tcacctgaat caggatattc ttctaatacc tggaatgctg 5340
ttttcccggg gatcgcagtg gtgagtaacc atgcatcatc aggagtacgg ataaaatgct 5400
tgatggtcgg aagaggcata aattccgtca gccagtttag tctgaccatc tcatctgtaa 5460
catcattggc aacgctacct ttgccatgtt tcagaaacaa ctctggcgca tcgggcttcc 5520
catacaatcg atagattgtc gcacctgatt gcccgacatt atcgcgagcc catttatacc 5580
catataaatc agcatccatg ttggaattta atcgcggcct cgagcaagac gtttcccgtt 5640
gaatatggct cataacaccc cttgtattac tgtttatgta agcagacagt tttattgttc 5700
atgaccaaaa tcccttaacg tgagttttcg ttccactgag cgtcagaccc cgtagaaaag 5760
atcaaaggat cttcttgaga tccttttttt ctgcgcgtaa tctgctgctt gcaaacaaaa 5820
aaaccaccgc taccagcggt ggtttgtttg ccggatcaag agctaccaac tctttttccg 5880
aaggtaactg gcttcagcag agcgcagata ccaaatactg tccttctagt gtagccgtag 5940
ttaggccacc acttcaagaa ctctgtagca ccgcctacat acctcgctct gctaatcctg 6000
ttaccagtgg ctgctgccag tggcgataag tcgtgtctta ccgggttgga ctcaagacga 6060
tagttaccgg ataaggcgca gcggtcgggc tgaacggggg gttcgtgcac acagcccagc 6120
ttggagcgaa cgacctacac cgaactgaga tacctacagc gtgagctatg agaaagcgcc 6180
acgcttcccg aagggagaaa ggcggacagg tatccggtaa gcggcagggt cggaacagga 6240
gagcgcacga gggagcttcc agggggaaac gcctggtatc tttatagtcc tgtcgggttt 6300
cgccacctct gacttgagcg tcgatttttg tgatgctcgt caggggggcg gagcctatgg 6360
aaaaacgcca gcaacgcggc ctttttacgg ttcctggcct tttgctggcc ttttgctcac 6420
atgttcgatc ccgcgaaat 6439
<210> 8
<211> 14
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 8
guuugagagu uaug 14
<210> 9
<211> 16
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 9
caugacgagu ucaaau 16
<210> 10
<211> 17
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 10
aaaaauuuau ucaaacc 17
<210> 11
<211> 12
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 11
aaaaauuuau uc 12
<210> 12
<211> 14
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 12
gccuauuuau aggc 14
<210> 13
<211> 14
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 13
cgcagauguu cugc 14
<210> 14
<211> 27
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 14
auuaugcuug cuauugcaag cuuuuuu 27
<210> 15
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 15
auuaugcuug cuauu 15
<210> 16
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 16
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuugc uauu 94
<210> 17
<211> 82
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 17
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uu 82
<210> 18
<211> 70
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 18
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag 70
<210> 19
<211> 58
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 19
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauu 58
<210> 20
<211> 46
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 20
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauuc 46
<210> 21
<211> 34
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 21
guuugagagu uauggaaaca ugacgaguuc aaau 34
<210> 22
<211> 54
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 22
guuugagagu uauguaauuu cauauaggac uaaaacgaaa gcuauaauau guau 54
<210> 23
<211> 17
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 23
guuugagagu uauguaa 17
<210> 24
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 24
guuugagagu uau 13
<210> 25
<211> 12
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 25
guuugagagu ua 12
<210> 26
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 26
uuacaugacg aguucaaau 19
<210> 27
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 27
augacgaguu caaau 15
<210> 28
<211> 14
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 28
ugacgaguuc aaau 14
<210> 29
<211> 149
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 29
guuugagagu uauguaauuu cauauaggac uaaaacgaaa gcuauaauau guaugaaauu 60
acaugacgag uucaaauaaa aauuuauuca aaccgccuau uuauaggccg cagauguucu 120
gcauuaugcu ugcuauugca agcuuuuuu 149
<210> 30
<211> 104
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 30
guuugagagu uaugaaaaug acgaguucaa auaaaaauuu auucaaaccg ccuauuuaua 60
ggccgcagau guucugcauu augcuugcua uugcaagcuu uuuu 104
<210> 31
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 31
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 32
<211> 10
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 32
gccuauaggc 10
<210> 33
<211> 14
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 33
cgcagauguu cugu 14
<210> 34
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 34
cgcagauguu cug 13
<210> 35
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 35
agcuugcuau ugcaauuuu 19
<210> 36
<211> 11
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 36
auuauuuuuu u 11
<210> 37
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 37
auuaugcuuu ugcaauuuu 19
<210> 38
<211> 17
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 38
auuuugcaag cuuuuuu 17
<210> 39
<211> 17
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 39
cuauugcaag cuuuuuu 17
<210> 40
<211> 11
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 40
caagcuuuuu u 11
<210> 41
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 41
auuaugcuau ugcauuuuu 19
<210> 42
<211> 16
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 42
uauugcaagc uuuuuu 16
<210> 43
<211> 11
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 43
aaagcuuuuu u 11
<210> 44
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 44
auuuugcuau ugcuuuuuu 19
<210> 45
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 45
auugcaagcu uuuuu 15
<210> 46
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 46
agcaagcuuu uuu 13
<210> 47
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 47
auuaauugca agcuuuuuu 19
<210> 48
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 48
auuaugcgca agcuuuuuu 19
<210> 49
<211> 11
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 49
auagcuuuuu u 11
<210> 50
<211> 21
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 50
auuacuauug caagcuuuuu u 21
<210> 51
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 51
auuacaagcu uuuuu 15
<210> 52
<211> 21
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 52
auugcuauug caagcuuuuu u 21
<210> 53
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 53
ugcuugcuau agcuuuuuu 19
<210> 54
<211> 14
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 54
uugcaagcuu uuuu 14
<210> 55
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 55
aucuauugca agcuuuuuu 19
<210> 56
<211> 23
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 56
aucuugcuau ugcaagcuuu uuu 23
<210> 57
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 57
aucaagcuuu uuu 13
<210> 58
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 58
auuaugcucu uuuuu 15
<210> 59
<211> 17
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 59
auauugcaag cuuuuuu 17
<210> 60
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 60
auuaugcuuu uuu 13
<210> 61
<211> 21
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 61
auuaugcuug cuauagcuuu u 21
<210> 62
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 62
ugcuugcuau ugcaauuuu 19
<210> 63
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 63
auuauaagcu uuuuu 15
<210> 64
<211> 11
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 64
auuacuuuuu u 11
<210> 65
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 65
auuaagcuuu uuu 13
<210> 66
<211> 21
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 66
auuauuauug caagcuuuuu u 21
<210> 67
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 67
auuuauugca agcuuuuuu 19
<210> 68
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 68
ugcuugcuca agcuuuuuu 19
<210> 69
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 69
auuuuuugca agcuuuuuu 19
<210> 70
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 70
ugcuuuugca agcuuuuuu 19
<210> 71
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 71
auuuugugca agcuuuuuu 19
<210> 72
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 72
auuaugcuua agcuuuuuu 19
<210> 73
<211> 19
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 73
ugcuauugca agcuuuuuu 19
<210> 74
<211> 96
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 74
guuugagagu uaugaaaaug acgaguucaa auaaaaauuu auucaaaccg ccuauuuaua 60
ggccgcagau guucugcagc uugcuauugc aauuuu 96
<210> 75
<211> 86
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 75
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau uuuuuu 86
<210> 76
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 76
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuuuu gcaauuuu 98
<210> 77
<211> 96
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 77
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuuugcaagc uuuuuu 96
<210> 78
<211> 96
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 78
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcc uauugcaagc uuuuuu 96
<210> 79
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 79
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcc aagcuuuuuu 90
<210> 80
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 80
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuauu gcauuuuu 98
<210> 81
<211> 84
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 81
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcua uugcaagcuu uuuu 84
<210> 82
<211> 82
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 82
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau uu 82
<210> 83
<211> 79
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 83
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuu 79
<210> 84
<211> 87
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 84
guuugagagu uaugaaauga cgaguucaaa uaaaaauuua uucaaaccgc cuauuuauag 60
gccgcagaug uucugcaaag cuuuuuu 87
<210> 85
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 85
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuuugcuauu gcuuuuuu 98
<210> 86
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 86
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca gcuugcuauu gcaauuuu 98
<210> 87
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 87
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugua uugcaagcuu uuuu 94
<210> 88
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 88
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca gcaagcuuuu uu 92
<210> 89
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 89
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaauugcaa gcuuuuuu 98
<210> 90
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 90
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcgcaagcuu uuuu 94
<210> 91
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 91
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uagcuuuuuu 90
<210> 92
<211> 100
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 92
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuacuauugc aagcuuuuuu 100
<210> 93
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 93
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuacaagcuu uuuu 94
<210> 94
<211> 100
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 94
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uugcuauugc aagcuuuuuu 100
<210> 95
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 95
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuauuuuuuu 90
<210> 96
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 96
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcu gcuugcuaua gcuuuuuu 98
<210> 97
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 97
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucuguu gcaagcuuuu uu 92
<210> 98
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 98
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca ucuauugcaa gcuuuuuu 98
<210> 99
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 99
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauagg 60
ccgcagaugu ucugcaucuu gcuauugcaa gcuuuuuu 98
<210> 100
<211> 89
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 100
guuugagagu uaugaaauga cgaguucaaa uaaaaauuua uucaaaccgc cuauuuauag 60
gccgcagaug uucugcauca agcuuuuuu 89
<210> 101
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 101
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcucuu uuuu 94
<210> 102
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 102
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca ucaagcuuuu uu 92
<210> 103
<211> 96
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 103
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uauugcaagc uuuuuu 96
<210> 104
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 104
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca aagcuuuuuu 90
<210> 105
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 105
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuuuu uu 92
<210> 106
<211> 84
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 106
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuau 84
<210> 107
<211> 100
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 107
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuugc uauagcuuuu 100
<210> 108
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 108
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcu gcuugcuauu gcaauuuu 98
<210> 109
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 109
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuauaagcuu uuuu 94
<210> 110
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 110
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuacuuuuuu 90
<210> 111
<211> 88
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 111
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuuuuuuu 88
<210> 112
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 112
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaagcuuuu uu 92
<210> 113
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 113
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauugc aagcuuuuuu 90
<210> 114
<211> 100
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 114
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuauuauugc aagcuuuuuu 100
<210> 115
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 115
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuuauugcaa gcuuuuuu 98
<210> 116
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 116
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcu gcuugcucaa gcuuuuuu 98
<210> 117
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 117
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuuuuugcaa gcuuuuuu 98
<210> 118
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 118
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcu gcuuuugcaa gcuuuuuu 98
<210> 119
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 119
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuuugugcaa gcuuuuuu 98
<210> 120
<211> 88
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 120
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag gcuauugcaa gcuuuuuu 88
<210> 121
<211> 88
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 121
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca gcuuuuuu 88
<210> 122
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 122
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugca uuaugcuuaa gcuuuuuu 98
<210> 123
<211> 82
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 123
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucuguu uu 82
<210> 124
<211> 84
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 124
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcu uuuu 84
<210> 125
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 125
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggccgcag auguucugcu gcuauugcaa gcuuuuuu 98
<210> 126
<211> 80
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 126
guuugagagu uaugaaaaug acgaguucaa auaaaaauuu auucaaaccg ccuauuuaua 60
ggccgcagau guucugcuuu 80
<210> 127
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 127
auuuauucaa acc 13
<210> 128
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 128
aaauauucaa acc 13
<210> 129
<211> 10
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 129
gauguucugc 10
<210> 130
<211> 10
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 130
cgcguucugc 10
<210> 131
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 131
ugcaagcuuu uuu 13
<210> 132
<211> 78
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 132
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccuauugcaa gcuuuuuu 78
<210> 133
<211> 97
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 133
guuugagagu uaugaaaaug acgaguucaa auaaaaauuu auucaaaccg ccuauuuaua 60
ggccuucugc auuaugcuug cuauugcaag cuuuuuu 97
<210> 134
<211> 100
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 134
guuugagagu uauguaagaa auuacaugac gaguucaaau aaaaauuuau ucaaaccgcc 60
uaugauguuc ugcauuaugc uugcuauugc aagcuuuuuu 100
<210> 135
<211> 80
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 135
guuugagagu uagaaaugac gaguucaaau aaaaauuuau ucaaaccgcc uauuuauagg 60
ccgcaguugc aagcuuuuuu 80
<210> 136
<211> 84
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 136
guuugagagu uagaaaugac gaguucaaau auuuauucaa accgccuauu uauaggccgc 60
agauguucug cugcaagcuu uuuu 84
<210> 137
<211> 98
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 137
guuugagagu uauggaaaca ugacgaguuc aaauaaauau ucaaaccgcc uauuuauagg 60
ccgcguucug cauuaugcuu gcuauugcaa gcuuuuuu 98
<210> 138
<211> 15
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 138
ugacgaguuc aaaua 15
<210> 139
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 139
aaaaauucaa acc 13
<210> 140
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 140
aauuauucaa acc 13
<210> 141
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 141
aaaaauuuaa acc 13
<210> 142
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 142
aaaaauuuau ucc 13
<210> 143
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 143
aaaaauuuau uca 13
<210> 144
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 144
ccuauuuaua ggc 13
<210> 145
<211> 12
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 145
cuauuuauag gc 12
<210> 146
<211> 13
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 146
gccuauuuau agg 13
<210> 147
<211> 11
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 147
agauguucug c 11
<210> 148
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 148
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 149
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 149
guuugagagu uauggaaaca ugacgaguuc aaauaauuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 150
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 150
guuugagagu uauggaaaca ugacgaguuc aaauauuuau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 151
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 151
guuugagagu uauggaaaca ugacgaguuc aaauaacgcc uauuuauagg ccgcagaugu 60
ucugcauuau gcuugcuauu gcaagcuuuu uu 92
<210> 152
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 152
guuugagagu uauggaaaca ugacgaguuc aaauaaauau ucaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 153
<211> 84
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 153
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau ggccgcagau guucugcauu 60
augcuugcua uugcaagcuu uuuu 84
<210> 154
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 154
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuaaaccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 155
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 155
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauuccgcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 156
<211> 78
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 156
guuugagagu uagaaaugac gaguucaaau aaaaggccgc agauguucug cauuaugcuu 60
gcuauugcaa gcuuuuuu 78
<210> 157
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 157
guuugagagu uauggaaaca ugacgaguuc aaauaaccua uuuauaggcc gcagauguuc 60
ugcauuaugc uugcuauugc aagcuuuuuu 90
<210> 158
<211> 94
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 158
guuugagagu uauggaaaca ugacgaguuc aaauaaaacg ccuauuuaua ggccgcagau 60
guucugcauu augcuugcua uugcaagcuu uuuu 94
<210> 159
<211> 88
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 159
guuugagagu uauggaaaca ugacgaguuc aaauacuauu uauaggccgc agauguucug 60
cauuaugcuu gcuauugcaa gcuuuuuu 88
<210> 160
<211> 84
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 160
guuugagagu uauggaaaca ugacgaguuc aaauaauaua ggccgcagau guucugcauu 60
augcuugcua uugcaagcuu uuuu 84
<210> 161
<211> 82
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 161
guuugagagu uauggaaaca ugacgaguuc aaauaauagg ccgcagaugu ucugcauuau 60
gcuugcuauu gcaagcuuuu uu 82
<210> 162
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 162
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucagcc uauuuauagg 60
ccgcagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 163
<211> 90
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 163
guuugagagu uaugaaaaug acgaguucaa auaccgccua uuuauaggcc gcagauguuc 60
ugcauuaugc uugcuauugc aagcuuuuuu 90
<210> 164
<211> 96
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 164
guuugagagu uauggaaaca ugacgaguuc aaauacaaac cgccuauuua uaggccgcag 60
auguucugca uuaugcuugc uauugcaagc uuuuuu 96
<210> 165
<211> 102
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 165
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccuauuua 60
uaggagaugu ucugcauuau gcuugcuauu gcaagcuuuu uu 102
<210> 166
<211> 78
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 166
guuugagagu uagaaaugac gaguucaaau aauaggccgc agauguucug cauuaugcuu 60
gcuauugcaa gcuuuuuu 78
<210> 167
<211> 92
<212> RNA
<213> 人工序列(Artificial Sequence)
<220>
<223> OMNI-50支架或支架部分
<400> 167
guuugagagu uauggaaaca ugacgaguuc aaauaaaaau uuauucaaac cgccugaugu 60
ucugcauuau gcuugcuauu gcaagcuuuu uu 92
<210> 168
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 168
gcagtccggg ctgggagcgg gt 22
<210> 169
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 169
cagctgcggg aaagggattc cc 22
<210> 170
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 170
cagctgcggg aatgggattc cc 22
<210> 171
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 171
gtgtcaagcc ccagaggcca ca 22
<210> 172
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 172
gtgtcaagcc ccagaggaca ca 22
<210> 173
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 173
cacagcgggt gtagactccg ag 22
<210> 174
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 174
cacagcaggt gtagactccg ag 22
<210> 175
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 175
gcagtccggg ctgggagcgg gtggg 25
<210> 176
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 176
cagctgcggg aaagggattc ccagg 25
<210> 177
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 177
cagctgcggg aatgggattc ccagg 25
<210> 178
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 178
gtgtcaagcc ccagaggcca caggg 25
<210> 179
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 179
gtgtcaagcc ccagaggaca caggg 25
<210> 180
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 180
cacagcgggt gtagactccg agggg 25
<210> 181
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 181
cacagcaggt gtagactccg agggg 25
<210> 182
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 182
gcagacaggg ccaggagcgg gaggg 25
<210> 183
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 183
gtggggcggg aaagggagtc ccagg 25
<210> 184
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 184
cagctgaggg aaaggaattc ccagg 25
<210> 185
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 185
aagccaaacc ccaaaggcca cacgg 25
<210> 186
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 186
aagacaagac ccagaggaca caggg 25
<210> 187
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 187
acagtcctgg ctgggagcag gtggg 25
<210> 188
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 188
cgcctgtggg aatgggcttc cccgg 25
<210> 189
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 189
tggccaagcc tcagaggcca caggg 25
<210> 190
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 间隔区或靶序列
<400> 190
tcttcaagcc ccagaggaca ctggg 25
- 新型OMNI-50 CRISPR核酸酶
- 使用空肠弯曲杆菌CRISPR/CAS系统衍生的RNA引导的工程化核酸酶的基因编辑
