基于表型语义关联基因聚类调控网络预测单基因遗传病潜在致病机制的分析方法
文献发布时间:2024-04-18 19:54:10
技术领域
本发明属于计算机技术领域,具体涉及一种基于表型语义关联基因聚类调控网络预测单基因遗传病潜在致病机制的分析方法。
背景技术
随着对疾病与基因关系研究的不断深入,标准化的人类表型可以越来越准确地描述已知疾病所呈现的异常表型及其层级关系。这使得通过对表型之间的语义相似性进行自动量化,进而为疾病诊断及发现致病基因提供了新的可能性。自2009年以来,先后出现了一系列基于基因型—表型—疾病三者关系,进行遗传病辅助诊断的工具及应用。它们分别采用了不同的数学模型及算法,用于对三者关系进行分析。例如,有的通过自然语言处理(Natural Language Processing,NLP)方法优化对于罕见遗传疾病表型的查询;有的通过随机漫步(Random Walk,RW)方法,确定表型相关致病基因的优先级。
对以上提及的多种基于不同数学模型的基因型——表型相关性预测算法进行分析及试用后可知,这些算法仅基于已知的人源基因型表型知识图谱建立预测模型,没有收录来源于模式动物的研究成果。近年来,基于各类模式动物,构建出大量具有典型人类疾病特征的动物模型,基于更有力的证据证实了大量基因型-表型之间的相关性。已知模型均未包含此类基因型表型关系研究的最新科研成果,将会造成模型的偏倚和预测结果的不准确。因此,亟待开发一个涉及多个物种,包含多个数据来源,基于证据可信度加权评分的基因型——表型相关性预测算法,已满足对于遗传病致病机制研究的实际需求。
发明内容
有鉴于此,本发明的目的在于提供一种适用于单基因遗传病致病机制研究的语义关联聚类调控网络建模方法,使用基于语义相似度构建的基因型——表型预测网络发现单基因遗传病潜在致病机制。基于基因型—表型关系,以及各表型本体之间的相互关系,计算各基因与目标表型之间的关联系数,从而推测与特定表型相关的致病基因。可通过该方法的运用,为开发单基因遗传病的机制研究和诊疗方案提供数据支撑和算法支撑。
为达到上述目的,本发明提供如下技术方案:
一种基于表型语义关联基因聚类调控网络预测单基因遗传病潜在致病机制的分析方法,包括以下步骤:
S1:基于实时更新的公开数据库创建人类及模式动物的表型—基因型关系数据集;
S2:构建基于表型语义关联基因的聚类调控网络预测模型SSGPN(SemanticSimilarity of Genotype-Phenotype Network);
S3:利用所述数据集训练所述SSGPN模型;
S4:将待查询的表型术语输入训练好的SSGPN模型中,计算关系数据集的表型信息含量和语义相关性,倒序输出与所述待查询的表型术语相关的基因及对应的语义相似度值。
进一步,步骤S1中,使用实时更新的公开数据库中的数据资源,分别提取基因—疾病,疾病—表型,基因型—表型之间的数据关系,并将其相互整合,构建一个完整的基因型—表型关系数据集。
在该数据集中,p值代表与某个特定表型及其子表型相关的基因数量,特定表型对应的基因数量来源于所有使用的参考数据库中的基因型—表型关系,根据不同物种的证据强度程度赋予权重,进行加和计算:
p=avg[f
其中f
Ω代表与模型中收录的与单基因遗传病相关的所有基因的数量,根据不同物种对应人类的同源基因取并集计算:
Ω=(Ω
其中Ω
特定表型的IC值,是由特定表型与致病基因的关联程度所决定的,如公式所示:
IC
其中IC
进一步,步骤S2中所述的SSGPN模型用于计算输入的查询表型术语与数据集中标注了关联基因的表型术语之间的相似度值,其计算方法如下:
S21:输入一个或多个待查询表型术语Qp;
S22:将每一个输入的表型术语Qp,与数据集中各个基因的所有相关表型术语Dp做比对,找到其中相似度值最大的表型MICA(Q
其中CA(D
S23:将获得所述表型MICA(Q
S24:对步骤S23中的公式进行优化,使用其对称版本,分别计算待查询表型术语Q与特定基因对应表型表型术语集D的语义相似度,及该基因对应表型术语集D与待查询表型术语Q的语义相似度,并取其平均值,公式如下:
sim
其中,sim
S25:将输入表型与数据集中所有的基因进行对称语义相似度计算;
S26:按对称语义相似度的值从高到低排序输出与输入表型相关的基因,并得到其对应的语义相似度值。
本发明的有益效果在于:基于本体论结构化的表型和基因型数据存在层级关系,对其语义相似性进行量化分析可实现对两者相关性的评估,并可根据量化相关系数研究功能相近的基因之间的聚类调控网络机制,本发明对多个基因型表型数据库的数据进行结构化整合,建立综合性的疾病—基因型—表型数据集,创新性地将语义相似度理论与基因聚类网络调控相结合,建立相关预测模型,从数据关系层面为开发单基因遗传病的机制研究提供数据支撑和算法支撑,提高了准确度和易用性。
本发明的其他优点、目标和特征将在随后的说明书中进行阐述,并且在某种程度上对本领域技术人员而言是显而易见的,或者本领域技术人员可以从本发明的实践中得到教导。本发明的目标和其他优点可以通过下面的说明书来实现和获得。
附图说明
为了使本发明的目的、技术方案和有益效果更加清楚,本发明提供如下附图进行说明:
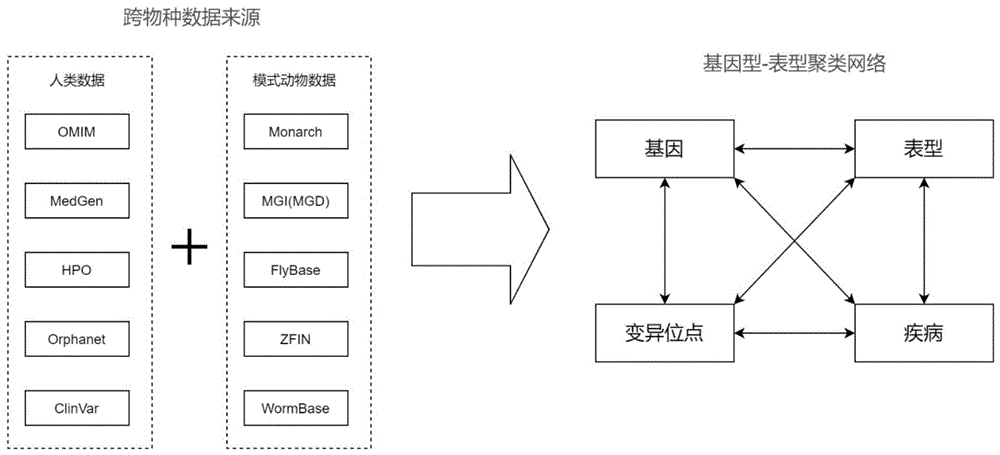
图1为不同数据库间的数据相互关联结构图;
图2为最大信息量共同父表型示意图;
图3为语言相似度计算示意图;
图4为SSGPN模型的构建与分析流程示意图;
图5为利用SSGPN模型对先天性糖基化障碍的致病基因进行预测的实际效果图,其中A为样本的相似度得分分布情况,B为SSGPN模型的ROC性能评估曲线。
具体实施方式
步骤一:创建表型—基因型关系数据集
想要根据遗传病异常表型预测新的潜在靶点基因和突变位点,首要基础就是尽可能完整地掌握现有疾病—表型—致病基因之间的本体结构关系、相互作用关系、以及基因和致病变异之间的数据关系。为此,可分别使用ClinVar、MedGen、Orphanet、HPO、OMIM等实时更新的公开数据库中的数据资源,分别提取了基因—疾病,疾病—表型,基因型—表型之间的数据关系,并将其相互整合,构建一个相对完整的基因型—表型关系数据集,作为训练SSGPN模型的基础数据集(训练集)。不同数据库间的数据相互关联结构如图1所示。
步骤二:利用SSGPN模型计算关系数据集的表型信息含量和语义相关性:
(1)临床诊断中,异常表型对于鉴定疾病的重要程度往往决定于其特异性。根据本体论的概念,特异性是由术语的信息含量(information content,IC)所决定的。术语的频率被定义为由该术语及其派生术语所注释对象的出现比例。IC值则是该频率的负自然对数。因此,随着从根部术语逐渐向更具体的叶部术语变化,IC值是在逐渐升高的。在针对遗传疾病表型—基因型相关性的研究中,特定表型的IC值,是由特定表型与致病基因的关联程度所决定的。|p|值代表与某个特定表型及其子表型相关的基因数量,|Ω|代表与模型中收录的与单基因遗传病相关的所用基因的数量,如公式1所示:
IC
(2)对于两个不同的表型,它们之间的最大信息量共同父表型(most informativecommon ancestor,MICA)的IC值,可被认为是这两个表型之间的相似度,具体如公式2所示。例如,对于先天性糖基化障碍疾病CDG,如图2所示,CDG的两个典型表型:大脑皮层萎缩(Cerebral cortical atrophy,HP:0002120)和小脑发育缺陷/发育不全(Aplasia/Hypoplasia of the cerebellum,HP:0007360),其最近的共同父系节点表型是脑形态学异常(Abnormality of brain morphology,HP:0012443),则HP:0002120和HP:0007360的相似度值就等于HP:0012443的IC值。
其中CA(D
基于以上方法,可以计算输入的待查询表型术语Q与数据集中标注了关联基因的表型术语D之间的相似度值。具体来说,首先可将每一个输入的查询表型Qp,与数据集中各个基因的所有相关表型Dp做比对,找到其中相似度值最大的表型(MICA(QpDp)),并获得该相似度值IC(MICA(QpDp))。之后,将所有相似度最大的值取平均数,就能得到查询表型与数据集中表型相关基因的语义相似度(semantic similarity)。具体如公式3所示:
例如,使用淋巴管扩张(HP:0031842)和肝硬化(HP:0001394)作为输入表型,查询它们与PMM2基因和MPI基因的语义相似度。根据训练集可知,MPI有58个相关表型,其中包含输入的HP:0031842和HP:0001394。因此,根据公式,MPI和输入表型的语义相似度,就是这两个表型的IC值的平均数,计算可得sim(QP,MPI)=5.155。而对于PMM2来说,其有20个相关表型,其中包含HP:0031842,但并不包含HP:0001394。根据HPO结构关系可知,HP:0001394与MPI相关表型中的肝纤维化(HP:0001395)相似度最高,拥有共同父表型异常肝脏形态(HP:0410042)。则HP:0410042的IC值就是两者的MICA值。根据公式计算可得,输入表型与PMM2的语义相似度sim(QP,PMM2)=4.245。具体计算过程如图2所示。
(3)观察公式可知,查询表型与数据集中表型的匹配度越高,返回的术语间相似度值也就越大。图2展示了该方法的基本步骤,这种方法可称为本体相似度查询法(Ontological Similarity Search,OSS)。然而,上方公式并没有考虑到除了与查询表型有最大相似度的表型之外,每个基因还会有其他的相关表型。例如如下情况:数据集中两个基因(GeneA and GeneB)的相关表型中,都正好有与查询表型一致的表型,但GeneA比GeneB还存在更多的与查询表型相似,但不完全一样的表型。根据经验可知,在这种情况下,GeneA比GeneB与查询表型更相关。但如只按照上述公式的方法计算,则查询表型与两个基因之间的语言相似度是一样的,这显然与事实情况不符。因此可对上述公式进行优化,使用其对称版本,分别计算待查询表型术语Q与特定基因对应表型表型术语集D的语义相似度,及该基因对应表型术语集D与待查询表型术语Q的语义相似度,并取其平均值。这样就能得到更准确的对称语义相似度(symmetric semantic similarity)。具体如公式4所示:
sin
其中,sin
由此可根据对称语义相似度来评价输入表型与特定基因之间的相关性。语义相似度的值越大,就代表两者的相关程度越高。将输入表型与数据集中所有的基因进行语义相似度计算,结果按倒序排序,就能得到与输入表型最相关基因,并得到其对应的语义相似度值(valueof symmetric semantic similarity,Sim
作为评价输入表型与基因相关程度的对称语言相似度Sim
所述构建SSGPN模型的理论方法的具体流程包括:
(1)收集并整合有关遗传病——基因型——表型的多个公开数据资源;利用收集的多来源数据资源构建一个完整的表型-基因型关系数据集;
(2)通过信息量及语义相似度理论计算各表型之间的语义相似度值;
(3)计算特定表型与基因之间的相关性,建立基于输入表型对潜在致病基因进行预测的SSGPN模型;
(4)将多个疾病亚型的典型表型作为模型输入表型,预测致病基因,量化评价SSGPN模型的预测能力;
(5)根据疾病的综合表型组合,利用SSGPN预测潜在新致病基因,通过第三方数据对预测结构进一步验证及研究。实验流程如图4所示。
对于模型的评价标准,除了常用的混淆矩阵外,还可以使用ROC曲线作为对该模型的评估方法。图5展示了利用该模型对先天性糖基化障碍(Congenital disorder ofglycosylation,CDG)的致病基因进行预测的实际效果,其中A为样本的相似度得分分布情况,B为SSGPN模型的ROC性能评估曲线。先天性糖基化障碍是一系列会导致糖基化异常的代谢类遗传疾病的统称。CDG的患者往往都是缺少糖基化过程中所必须的酶,导致糖基化过程出现异常。患者的CDG类型取决于缺少哪种类型的酶。根据最新的研究成果,现已发现的CDG类型已超过130种,且随着每年新的糖基化障碍病例的发现,该数字还在不断增长中。CDG大多是单基因遗传疾病,遗传模式通常为常染色体隐性遗传,但也有少部分呈现常染色体显性遗传和性伴遗传模式。按照实验流程,为了对SSGPN模型的有效性进行评价,我们首先将不同CDG亚型的临床特征作为输入表型,使用SSGPN模型预测其对应的致病基因。具体来说,我们将Orphanet数据库中收录的共计28个CDG亚型的表型数据,分别作为28个患有CDG亚型的模拟患者样本数据,合并9例非CDG患者亚型数据,输入SSGPN模型,计算各样本分别对应的基因语义相似度列表,并查询其真实致病基因的语义相似度值,以及计算归一化得分。将各样本数据分别进行计算后,结果如图5A所示。28个CDG亚型的归一化系数平均值为0.848。另一方面,CDG亚型样本的预测结果和非CDG样本的差距也进一步明显,这意味着模型可以更容易的区分正样本和负样本。通过ROC曲线对模型预测结果评价后可知(图5B),对于表型数量大于5的CDG亚型样本及非CDG样本,ROC曲线下的面积(Area under curve,AUC)为0.893,属于较为理想的预测模型。
最后说明的是,以上优选实施例仅用以说明本发明的技术方案而非限制,尽管通过上述优选实施示例已经对本发明进行了详细的描述,但本领域技术人员应当理解,可以在形式上和细节上对其作出各种各样的改变,而不偏离本发明权利要求书所限定的范围。
- 基于基因变异与疾病表型自动关联匹配的遗传病预测系统
- 基于病人临床症状数据和全外显子组测序数据筛选单基因遗传病致病基因的分析检测系统
