外泌体mRNA在乳腺疾病诊断中的应用
文献发布时间:2023-06-19 10:00:31
技术领域
本发明涉及生物医学诊断领域,具体而言,涉及一种外泌体mRNA在乳腺疾病诊断中的应用。
背景技术
乳腺癌(mammary cancer)是女性最常见的恶性肿瘤之一,发病率占全身各种恶性肿瘤的7-10%,在妇女仅次于子宫癌,已成为威胁妇女健康的主要病因。它的发病常与遗传有关,以及40-60岁之间,绝经期前后的妇女发病率较高。它是一种通常发生在乳房腺上皮组织,严重影响妇女身心健康甚至危及生命的最常见的恶性肿瘤之一。乳腺癌男性罕见,仅约1-2%的乳腺患者是男性。
近年来我国乳腺癌的发病率呈迅速上升趋势,在女性各肿瘤中居首位。由于乳腺癌病因尚不明确,早期诊断和早期综合治疗是防治乳腺癌的最有效的手段。有研究表明,其原发灶越小,预后越好。T4、T3、T2、T1期乳腺癌的10年生存率分别为19.7%,46.0%,62.6%,87.8%。直径小于1cm的微小癌,其10年总体生存率在90%-99%。
目前,我国对乳腺癌的预警和早期诊断的技术主要有以下四种:
1.临床体格检查:定期接受临床体格检查是早期发现乳腺癌的有效方法之一。我国也开展了一系列乳腺癌的预防工作,但是,乳腺癌普查耗资巨大,成本/效果比高。
2.影像学诊断法:影像学检查是以钼靶X线摄片为主要手段,辅以超声扫描和MRI扫描。钼靶X线摄片方法能发现细小钙化点,但对于不典型的病变,尤其是致密型乳腺内的病变及近胸壁的病变易漏诊。2014年2月11日发表在著名的《英国医学杂志》上一项研究报告40~59岁女性的每年乳腺钼靶X线检查并不能降低乳腺癌的死亡率,也不优于普通体检的诊断率,而且有22%的女性因为乳腺钼靶X线检查被过度诊断了(BritishMedicalJournal,2014,348.)。另外,我国女性乳腺小而致密,这也会降低患者接受钼靶检查的阳性率。
3.生物靶标早期检测:包括乳腺癌相关基因普查,例如BRCA1和BRCA2的突变分析,但目前分子诊断研究大多关注基因多态性与乳腺癌的关系,只能给出患者的易感风险,不适用于临床疾病的诊断。另外临床上最常用的乳腺癌血清肿瘤标志物有CA-153、CEA、CA-125等,虽然可以一定程度上反应疾病的状态,但是特异性并不高,在很多其他肿瘤,如卵巢癌、盆腔肿瘤、胃癌、结肠癌、肝癌等都会相应存在。据报道CA-153的敏感性仅仅为50%左右,在早期乳腺癌患者中的表达水平往往较低,而且特异性也不高,对于乳腺癌的早期筛查方面有一定的局限性。
4.穿刺活检:乳腺组织穿刺活检的病理学检查准确性较高,但为有创性操作,还会因标本取材不足而产生假阴性结果,亦或因取材部位的不同、乳腺癌瘤内异质性的影响而产生不同的结果。
总的来说:对于乳腺结节的筛查有着以下不足:(1)临床体检耗资大;(2)影像学诊断法阳性漏检率高;(3)目前现在的较为成熟的生物靶标,只能做风险评估,不适于临床诊断;(4)穿刺活检首先是有创性,另外对于取样者及穿刺部位要求较高,容易因外在因素的影响造成不同的结果。因此,本领域迫切需要建立新的乳腺结节检查技术,能够早期客观、灵敏和稳定的诊断乳腺癌,以便进行及时、有针对性的治疗,提高患者的生存质量和生存率,降低医疗成本,节约社会资源。
发明内容
外泌体一般认为是一类直径30-150nm,具有完整膜结构的细胞外囊泡,主要负责细胞间的物质运输和信息传递,其中包裹有细胞特异性的蛋白质、脂类和核酸。现有的研究表明外泌体在很多生理病理过程中扮演重要的角色,如抗原呈递、肿瘤生长和转移等。所有的细胞都会分泌外泌体,而肿瘤细胞分泌的外泌体多于正常细胞。
现在,外泌体与癌症的相关性研究报道,大多都基于外泌体MicroRNA和LncRNA的研究,也有部分文献报道了外泌体蛋白与卵巢癌、前列腺癌的相关性
ENAH基因家族成员参与肌动蛋白的运动,属于Ena/VASP蛋白家族的成员。其在肌动蛋白聚合中起着关键调控作用,参与调节肌动蛋白丝的组装并调节细胞粘附和运动。ENAH基因有不同的剪切体和转录本,其中研究最多的是MenaINV和11a两种。Sumanta
现有关于ENAH mRNA的报道,主要为体外培养的肿瘤细胞、模式生物(种植肿瘤细胞的小鼠),鲜少采用获取的人乳腺癌组织或乳头吸引液,在外泌体中的表达情况以及与乳腺癌的相关性更未见报道。
本发明研究了外泌体的ENAH mRNA表达水平,同时研究了其总型和MenaINV、11a两种亚型与乳腺疾病的相关性。在外泌体中两种亚型的表达量较低,样本检出率低、检测效果无检测意义,而总型检测具有诊断学意义。
为了实现本发明的上述目的,特采用以下技术方案:
检测剂在制备乳腺癌诊断、病程监控、预后评估试剂或试剂盒中的应用;
所述检测剂包括第一检测剂,所述第一检测剂用于检测ENAH mRNA,其中增加的ENAHmRNA表达量是乳腺癌严重程度的指征;
所述ENAH mRNA为总mRNA,其为待检测外泌体中ENAH不同转录本mRNA的总和。
根据本发明的再一方面,还涉及一种乳腺癌诊断试剂盒,其含有如上所定义的检测剂。
本发明的有益效果为:
1.本发明可以完成乳腺结节良恶性鉴别、乳腺癌辅助诊断和病程监控。
2.利用本发明可以对疑似病灶患者进行筛查鉴别,可以解决影像学无法确认的早期乳腺恶性结节。
3.首次发现上述外泌体ENAH mRNA标志物可用于人体乳腺结节良恶性鉴别、乳腺癌辅助诊断和病程监控。
参考文献:
[1]Jayanthi Lea,Detection of phosphatidylserine-positive exosomes as adiagnostic marker for ovarian malignancies: a proof of conceptstudy.Oncotarget, 2017, Vol. 8, (No. 9), 14395-14407
[2]Mariantonia Logozzi.Increased Plasmatic Levels of PSA-ExpressingExosomes Distinguish Prostate Cancer Patients from Benign ProstaticHyperplasia:A Prospective Study Cancers 2019,11,1449;doi:10.3390/cancers11101449
[3]James McKiernan.A Prospective Adaptive Utility Trial to ValidatePerformance of a Novel Urine Exosome Gene Expression Assay to Predict High-grade Prostate Cancer in Patients with Prostate-specific Antigen 2–10 ng/mlat Initial Biopsy.EURURO-7980; No. of Pages 8
[4]Sumanta Goswami,Identification of invasion specific splice variants ofthe cytoskeletal protein Mena present in mammary tumor cells during invasionin vivo.Clin Exp Metastasis (2009) 26:153-159
[5]Evanthia T.Mena invasive (MenaINV) and Mena11a isoforms play distinctroles in breast cancer cell cohesion and association with TMEM.Clin ExpMetastasis (2011) 28:515-527。
附图说明
为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
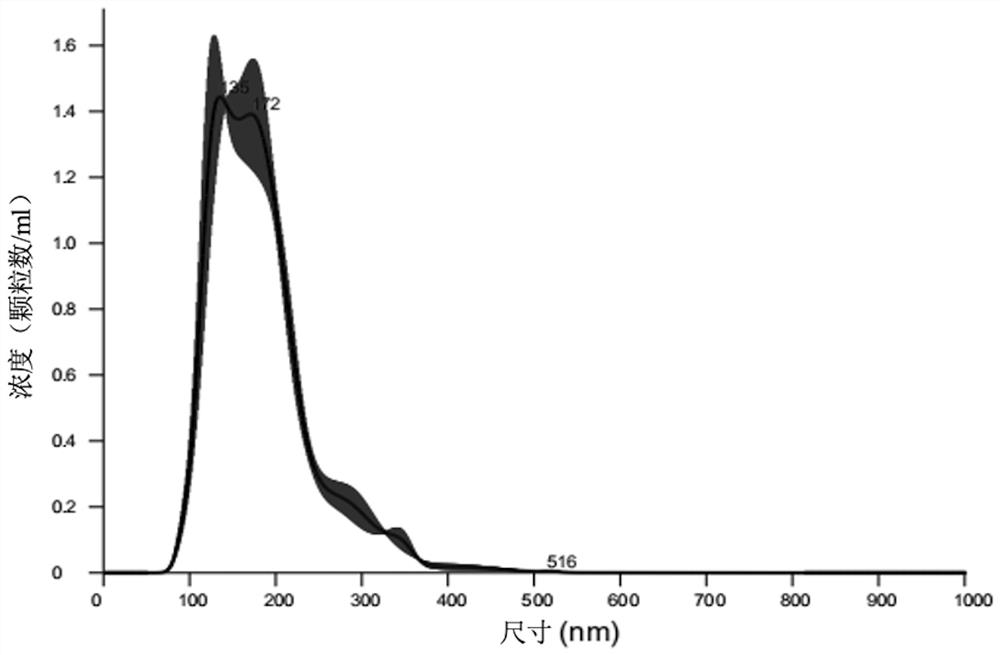
图1为本发明一个实施例中血浆外泌体NTA结果图;
图2为本发明一个实施例中术前术后靶标表达水平变化图。
具体实施方式
现将详细地提供本发明实施方式的参考,其一个或多个实例描述于下文。提供每一实例作为解释而非限制本发明。实际上,对本领域技术人员而言,显而易见的是,可以对本发明进行多种修改和变化而不背离本发明的范围或精神。例如,作为一个实施方式的部分而说明或描述的特征可以用于另一实施方式中,来产生更进一步的实施方式。
除非另有定义,本文所使用的所有的技术和科学术语与属于本发明的技术领域的技术人员通常理解的含义相同。本文中在本发明的说明书中所使用的术语只是为了描述具体的实施例的目的,不是旨在于限制本发明。本文所使用的术语“和/或”包括一个或多个相关的所列项目的任意的和所有的组合。
本发明涉及检测剂在制备乳腺癌诊断、病程监控、预后评估试剂或试剂盒中的应用;
所述检测剂包括第一检测剂,所述第一检测剂用于检测ENAH mRNA,其中增加的ENAHmRNA表达量是乳腺癌严重程度的指征;
所述ENAH mRNA为总mRNA,其为待检测外泌体中ENAH不同转录本mRNA的总和。
任何包含外泌体的样本都可作为本申请的检测样本,示例性的样本可以为细胞培养物上清液、血液样本(全血、血清、血浆)、乳汁、乳头吸出物、乳头溢出液、组织液、淋巴液,或者从生物组织样品得到的澄清的裂解液。
生物组织样本优选乳腺组织,例如癌组织或癌旁组织。血液样本优选为外周血样本。
含有外泌体的样本可预先进行富集,富集方式可通过密度梯度离心、超速离心、超滤、聚乙二醇沉淀中的任一种方法进行。
在本发明中,检测剂可以是定性或者定量的检测剂。例如,在一些情况下,健康或者良性乳腺癌通过PCR方式检测ENAH mRNA时,在一定循环数下不会出峰,因此也可以通过定性的方式与恶性乳腺癌进行区分。
在一些实施方式中,所述检测剂中还含有第二检测剂,所述第二检测剂用于检测SEPT9 mRNA,其中降低的SEPT9 mRNA表达量是乳腺癌病情加重的指征,所述SEPT9 mRNA为总mRNA,其为待检测外泌体中SEPT9不同转录本mRNA的总和。
本发明发现了乳腺癌诊断的新的标志物,外泌体中ENAH mRNA,以及任选的SEPT9mRNA。
本发明还提供了ENAH mRNA及SEPT9 mRNA二者联合进行乳腺癌诊断的技术方案,其能提供更好的诊断准确度。
利用本技术方案可以对疑似病灶患者进行筛查鉴别,可以解决影像学无法确认的早期乳腺恶性结节。
本文使用的术语“标志物”或“生化标志物”指要用作分析患者实验样品的靶标的分子。如本领域技术人员显而易见的,mRNA的检测不应当解释为限于上述基因直接转录得到的特定mRNA序列,例如,作为替代性的mRNA变体、短链或前-mRNA处理的结果。变体的核苷酸序列与对应的标志物序列具有95%、96%、97%、98%、99%或更高的同一性;短链则只要能代表全长mRNA本身的特异性序列都可胜任。显然地,mRNA水平的检测也可通过检测cDNA以进行。
诊断可以为辅助诊断。本发明中,术语“预后”具有本领域技术人员知悉的含义。在本发明的一个实施方案中,预后是指生存概率(PFS)。其中,如果对象是一个群体(2个或2个以上患者),预后可以是指中位生存概率或平均生存概率。
在本发明中,“总mRNA”或“总型”或“total mRNA”意味着检测对象为待检测外泌体中ENAH或SEPT9不同转录本mRNA的总和。
ENAH不同转录本mRNA为本领域技术人员所公知,例如NCBI上记录的26个不同的转录本,转录本号分别为:XM_017001747.1、XM_017001746.2、XM_024448307.1、XM_024448305.1、XM_017001750.1、XM_024448306.1、XM_024448318.1、XM_011544229.2、XM_024448308.1、XM_024448319.1、XM_017001748.1、XM_017001752.1、XM_024448316.1、XM_024448314.1、XM_024448309.1、XM_024448310.1、XM_017001749.1、XM_024448313.1、XM_024448311.1、XM_024448315.1、XM_017001751.1、XM_024448317.1、NM_001377481.1、NM_001377482.1、NM_001008493.3、NM_018212.6、NM_001377483.1。
SEPT9不同转录本mRNA为本领域技术人员所公知,例如NCBI上记录的11个不同的转录本,转录本号分别为:NM_006640.5、NM_001113491.2、NM_001113492.2、NM_001113493、NM_001113494.1、NM_001113495.2、NM_001113496.2、NM_001293695.2、NM_001293696.2、NM_001293697.2、NM_001293698.2。
检测total mRNA的常规方法显然通常有2种:
(1)检测所有转录本共有的mRNA序列;
(2)分别检测上述转录本各自的表达量,然后加和。
在一些实施方式中,通过检测ENAH mRNA不同转录本mRNA的共有序列检测所述ENAH mRNA。
在一些实施方式中,通过检测SEPT9不同转录本mRNA的共有序列检测所述SEPT9mRNA。
本领域技术人员可通过BLAST等常规技术手段获得共有序列,以上述转录本为例,通过分析容易获知ENAH mRNA共有转录本可以检测的外显子有3个,SEPT9 mRNA共有转录本可以检测的外显子有9个,本领域技术人员可以采用跨不同外显子的检测区段设计检测引物、探针用于检测。也可以在任意外显子上单独设计检测引物、探针用于检测。
在一些实施方式中,所述检测剂用于执行如下方法中的至少一种:
PCR法、一代测序、二代测序、核酸芯片、电泳、HPLC、共振光散射法和生物质谱法。
在一些实施方式中,所述PCR法为实时荧光定量PCR和/或数字PCR。
在一些实施方式中,所述第一检测剂选自l~p中至少一种引物对:
l:SEQ ID NO:34和SEQ ID NO:35;
m:SEQ ID NO:36和SEQ ID NO:37;
n:SEQ ID NO:38和SEQ ID NO:39;
o:SEQ ID NO:40和SEQ ID NO:41;
p:SEQ ID NO:42和SEQ ID NO:43。
在一些实施方式中,所述第二检测剂选自a~f中至少一种引物对:
a:SEQ ID NO:1和SEQ ID NO:2;
b:SEQ ID NO:3和SEQ ID NO:4;
c:SEQ ID NO:5和SEQ ID NO:6;
d:SEQ ID NO:7和SEQ ID NO:8;
e:SEQ ID NO:9和SEQ ID NO:10;
f:SEQ ID NO:11和SEQ ID NO:12。
在一些实施方式中,所述检测剂还含有外泌体提取试剂、逆转录酶、随机引物、双链特异性荧光染料、dNTP、DNA聚合酶、内参引物以及水中的一种或多种。
外泌体提取试剂可用于执行如下任一种方法:密度梯度离心、超速离心、超滤、聚乙二醇沉淀。
在一些实施方式中,所述双链特异性荧光染料定量选自溴化乙锭、SYBR Green、PicoGreen、RiboGreen中的任一种。
在一些实施方式中,所述水通常为核酸和/或无核酸酶的水。水可以为蒸馏水(Distilled Water)、去离子水(Deionized Water)或反渗水(Reverse osmosis Water)。
在一些实施方式中,所述DNA聚合酶选自Taq、Bst、Vent、Phi29、Pfu、Tru、Tth、Tl1、Tac、Tne、Tma、Tih、Tf1、Pwo、Kod、Sac、Sso、Poc、Pab、Mth、Pho、ES4 DNA聚合酶、Klenow片段中的任一种。
内参引物即为用于检测参考标准品的引物,还可以理解为进一步包括mRNA测量标准化所需的样本和/或试剂。例如,RNA测量可通过一种或多种“管家”mRNA作为标准化的基准,这对于本领域技术人员是熟悉的。
在一些实施方式中,该“管家”mRNA为泛素mRNA和/或肌动蛋白 mRNA。
在一些实施方式中,所述内参引物(泛素mRNA)选自g~k中至少一种引物对:
g:SEQ ID NO:13和SEQ ID NO:14;
h:SEQ ID NO:15和SEQ ID NO:16;
i:SEQ ID NO:17和SEQ ID NO:18;
j:SEQ ID NO:19和SEQ ID NO:20;
k:SEQ ID NO:21和SEQ ID NO:22。
在一些实施方式中,所述内参引物(肌动蛋白 mRNA)选自q~t中至少一种引物对:
q:SEQ ID NO:49和SEQ ID NO:50;
r:SEQ ID NO:51和SEQ ID NO:52;
s:SEQ ID NO:53和SEQ ID NO:54;
t:SEQ ID NO:55和SEQ ID NO:56。
在一些实施方式中,所述检测剂还包括探针。
在一些实施方式中,所述探针包括核苷酸序列为SEQ ID NO:44~ SEQ ID NO:48中的至少一种所示的探针,且SEQ ID NO: 44~ SEQ ID NO:48与l~p中的引物对依次对应、配合使用。
在一些实施方式中,所述探针包括核苷酸序列为SEQ ID NO:23~ SEQ ID NO:28中的至少一种所示的探针,且SEQ ID NO: 23~ SEQ ID NO:28与a~f中的引物对依次对应、配合使用。
在一些实施方式中,所述探针包括核苷酸序列为SEQ ID NO:29~ SEQ ID NO:33中的至少一种所示的探针,且SEQ ID NO: 29~ SEQ ID NO:33与g~k中的引物对依次对应、配合使用。
在一些实施方式中,所述探针包括核苷酸序列为SEQ ID NO:57~ SEQ ID NO:60中的至少一种所示的探针,且SEQ ID NO:57~ SEQ ID NO:60与q~t中的引物对依次对应、配合使用。
在一些实施方式中,所述探针带有可检测的标记。
如本文所用的术语“标记”指可用于提供可检测的(优选可定量的)效果且可以连接至核酸或蛋白的任何原子或分子。标记包括但不限于染料;放射性标记,诸如
在一些实施方式中,所述标记是荧光团、比色标记、量子点、生物素以及其他可以用于探测的标签分子(如用于拉曼衍射成像的炔烃基团,用于click反应的环烯烃,用于聚合物标记的引发集团),也可以选自多肽/蛋白分子,LNA/PNA,非天然氨基酸及其类似物(比如拟肽),非天然核酸及其类似物(拟核苷酸)和纳米结构(包括无机纳米颗粒,NV-center,聚集/组装诱导发光分子,稀土离子配体分子,多金属氧簇等)。
在一些实施方式中,所述探针为自身淬灭探针,5′端标记荧光发射基团,3′端标记淬灭基团。
在一些实施方式中,所述荧光发射基团选自FAM、HEX、TET、NED、ROX、CY5、CY3、Texas Red、TFAM、SYBR Green I、VIC以及JOE中的任一种;
在一些实施方式中,所述淬灭基团选自TAMRA、BHQ、Dabcyl、Eclipse以及NFQ-MGB中的任一种。
根据本发明的再一方面,还涉及一种乳腺癌诊断试剂盒,其含有如上所定义的检测剂。
根据本发明的再一方面,还涉及一种乳腺癌的诊断方法,其包括使用如上所定义的检测剂或试剂盒定量检测血液样本外泌体中ENAH mRNA和/或SEPT9 mRNA。
升高或者降低通常是显著性的,确定受试者和健康群体/良性肿瘤对照组/受试者的初始状态(baseline)相比,是否具有显著性的差异可用本领域公知的统计学方法进行,并使用置信区间和/或
容易理解,由于增加的ENAH mRNA表达量和降低的SEPT9 mRNA表达量都可以作为乳腺癌病情加重的指征,因此ENAH mRNA表达量与SEPT9 mRNA表达量的比值将使得数据的分析变得更为显著;因此优选通过检测ENAH mRNA表达量与SEPT9 mRNA表达量的比值进行乳腺癌病情诊断。
进一步地,根据不同检测样本和检测目的的不同,可以设置ENAH mRNA表达量与SEPT9 mRNA表达量的比值(以下简称R)的阈值,例如,R≥0.5、0.6、0.7、0.8、0.9、1.0、1.1、1.2、1.3、1.4、1.5时为阳性样本(例如恶性乳腺癌样本),优选R≥1.19为阳性。
下面将结合实施例对本发明的实施方案进行详细描述。
在下述实施例中,当采用荧光定量PCR时,若通过双链特异性荧光染料进行定量,则仅使用ENAH mRNA(l~p中的至少一种)和/或SEPT9 mRNA(a~f中的至少一种),和mRNA内参引物(g~k、q~t中的至少一种)即可;若采用探针法进行检测,则还需要使用相应探针。引物和探针的序列如下所示:
a:
SEPT9 mRNA上游引物序列:AGCAGGGCTTCGAGTTCAAC(SEQ ID NO:1);
SEPT9 mRNA下游引物序列:CGGCTGATTTTGGATTTGAA (SEQ ID NO:2);
SEPT9 mRNA探针序列:ATCCACCTTAATCAACACCC(SEQ ID NO:23);
b:
SEPT9 mRNA上游引物序列:ATCAAGTCCATCACGCACGA (SEQ ID NO:3);
SEPT9 mRNA下游引物序列:CCCGAACCCTGGTGTGTC (SEQ ID NO:4);
SEPT9 mRNA探针序列:CCGGACGCCTTTCTCCTC(SEQ ID NO:24);
c:
SEPT9 mRNA上游引物序列:CGGGGACCACATCAACAAC (SEQ ID NO:5);
SEPT9 mRNA下游引物序列:TGATGTTGACCTCCTCCTGC (SEQ ID NO:6);
SEPT9 mRNA探针序列:CAGCCCATCATGAAGTTCATC(SEQ ID NO:25);
d:
SEPT9 mRNA上游引物序列:GCGTCCACTGCTGCCTCTA(SEQ ID NO:7);
SEPT9 mRNA下游引物序列:ACCTTGCTCAGGCGTTTCA(SEQ ID NO:8);
SEPT9 mRNA探针序列:CCACCGGCCACCCTCAG(SEQ ID NO:26);
e:
SEPT9 mRNA上游引物序列:CCACTTCAAACAGCGGATCA(SEQ ID NO:9);
SEPT9 mRNA下游引物序列:CCGAGTCCTCATCAAATTCCT(SEQ ID NO:10);
SEPT9 mRNA探针序列:CAGACCTGCTGTCCAACGGC(SEQ ID NO:27);
f:
SEPT9 mRNA上游引物序列:TACCAGGTCAACGGCAAGAG(SEQ ID NO:11);
SEPT9 mRNA下游引物序列:CGCAGGTAGGCAAACTCACA(SEQ ID NO:12);
SEPT9 mRNA探针序列:CCATCGAAGTTGAAAACACCAC(SEQ ID NO:28);
g:
UBC mRNA上游引物序列:TGGGTCGCAGTTCTTGTTTG(SEQ ID NO:13);
UBC mRNA下游引物序列:ACCTCGAGGGTGATGGTCTT(SEQ ID NO:14);
UBC mRNA探针序列:TGCAGATCTTCGTGAAGACTCTG(SEQ ID NO:29);
h:
UBC mRNA上游引物序列:GGATTTGGGTCGCAGTTCTTG(SEQ ID NO:15);
UBC miRNA下游引物序列:GTGTCACTGGGCTCAACCTC(SEQ ID NO:16);
UBC mRNA探针序列:TCACTTGACAATGCAGATCTT(SEQ ID NO:30);
i:
UBC mRNA上游引物序列:GGGTCGCAGTTCTTGTTTGT(SEQ ID NO:17);
UBC mRNA下游引物序列:AACCTCGAGGGTGATGGTCT(SEQ ID NO:18);
UBC mRNA探针序列:TCACTTGACAATGCAGATC(SEQ ID NO:31);
j:
UBC mRNA上游引物序列:GGTCGCAGTTCTTGTTTG(SEQ ID NO:19);
UBC mRNA下游引物序列:TCGAGGGTGATGGTCTTA(SEQ ID NO:20);
UBC mRNA探针序列:ACTTGACAATGCAGATCTT(SEQ ID NO:32);
k:
UBC mRNA上游引物序列:TCGCAGTTCTTGTTTGTG(SEQ ID NO:21);
UBC mRNA下游引物序列:TCGAGGGTGATGGTCTTA(SEQ ID NO:22);
UBC mRNA探针序列:AATGCAGATCTTCGTGAAGACTC(SEQ ID NO:33);
l:
ENAH mRNA上游引物序列:ATCACCATACAGGCAACAACA (SEQ ID NO:34);
ENAH mRNA下游引物序列:CAACCCTTTAGGAATGGCA (SEQ ID NO:35);
ENAH mRNA探针序列:TCCTGCCCACCACTCTGAA(SEQ ID NO:44);
m:
ENAH mRNA上游引物序列:ACTGGATTCAGCAGAGTTCAT (SEQ ID NO:36);
ENAH mRNA下游引物序列:AGTTTATCACGACCTGATGGT (SEQ ID NO:37);
ENAH mRNA探针序列:CTATCACCATACAGGCAACAA(SEQ ID NO:45);
n:
ENAH mRNA上游引物序列:CTATCACCATACAGGCAACAA (SEQ ID NO:38);
ENAH mRNA下游引物序列:ATGGCACAGTTTATCACGAC (SEQ ID NO:39);
ENAH mRNA探针序列:TGCCCACCACTCTGAATGT(SEQ ID NO:46);
o:
ENAH mRNA上游引物序列:ACATTCAGAGTGGTGGGCA(SEQ ID NO:40);
ENAH mRNA下游引物序列:GATTGTACTTCAACCCTTTAGGA(SEQ ID NO:41);
ENAH mRNA探针序列:ACCATCAGGTCGTGATAAACT(SEQ ID NO:47);
p:
ENAH mRNA上游引物序列:GGTGCCAGCTGGTGGCTCAACTGGA(SEQ ID NO:42);
ENAH mRNA下游引物序列:TGTAGCTTGATTGTACTTCAACC(SEQ ID NO:43);
ENAH mRNA探针序列:TGGGCAGGAAGATTCAGGA(SEQ ID NO:48)。
q:
ACTB mRNA上游引物序列:CGCCAGCTCACCATGGAT(SEQ ID NO:49);
ACTB mRNA下游引物序列:GATGGAGGGGAAGACGGC(SEQ ID NO:50);
ACTB mRNA探针序列:CATGTGCAAGGCCGGCT(SEQ ID NO:57)。
r:
ACTB mRNA上游引物序列:AGGCACCAGGGCGTGAT(SEQ ID NO:51);
ACTB mRNA下游引物序列:CCGTGCTCGATGGGGTAC(SEQ ID NO:52);
ACTB mRNA探针序列:GCCCAGAGCAAGAGAGGCA(SEQ ID NO:58)。
s:
ACTB mRNA上游引物序列:CGCGAGAAGATGACCCAGA(SEQ ID NO:53);
ACTB mRNA下游引物序列:GTACGGCCAGAGGCGTACA(SEQ ID NO:54);
ACTB mRNA探针序列:AGCCATGTACGTTGCTATC(SEQ ID NO:59)。
t:
ACTB mRNA上游引物序列:CAGCCTTCCTTCCTGGGC(SEQ ID NO:55);
ACTB mRNA下游引物序列:GCGTACAGGTCTTTGCGGA(SEQ ID NO:56);
ACTB mRNA探针序列:CAACTCCATCATGAAGT(SEQ ID NO:60)。
实施例1
在以下实施例中,通过ENAH mRNA测定来分析,该测定被设计在荧光定量PCR检测平台上运行,检测组织外泌体和血浆外泌体中ENAH mRNA的表达水平。采用Lightcycle480平台检测。
实验方法如下:
1,血浆处理:
使用EDTA采集全血,室温2小时内离心分离得到血浆。离心条件:1500g,10分钟,吸取上清,再次离心:15000g,离心10分钟,吸取上清,即为分离得到的血浆。
2,外泌体鉴定:
2mL血浆,用0.2μm的滤膜进行过滤,采用QIAGEN试剂盒(exo-RNeasy Serum/PlasmaMidi Kit)富集外泌体后,使用外泌体洗脱液将外泌体洗脱下来,得到结构完整的外泌体。用颗粒跟踪分析技术鉴定提取的外泌体,血浆外泌体NTA见图1。由图可以看出:粒径大小在40-200nm,主峰在140nm左右,符合外泌体粒径范。
3,提取血浆外泌体RNA:
2mL血浆,用0.2μm的滤膜进行过滤,采用QIAGEN试剂盒(exo-RNeasy Serum/PlasmaMidi Kit)提取血浆外泌体RNA,具体操作按照试剂盒说明书进行。
注:建议使用商业化的外泌体mRNA提取试剂盒,避免引入DNA污染,防止后续检测产生非特异性扩增。必要时可使用DNase I进行DNA消化,再进行RT-PCR检测。
4,PCR检测:
使用SuperScript™ III Platinum™ One-Step qRT-PCR Kit进行一步法检测,具体PCR反应体系和程序见表1,同时设立阳性对照(阳性细胞系:MCF-7提取的总RNA)和阴性对照(未逆转录的RNA)对检测过程进行监控。
5,结果分析:
以ACTB为内参,用1000*2
表1 one-step Probe PCR 反应体系
表2 one-step Probe PCR 反应程序
实施例2
本实施例检测样本类型为:血浆。共入组39例患者,影像学检测显示均有结节存在。19例乳腺恶性结节样本,具体为11例导管原位癌伴微浸润、7例导管原位癌、1例小叶原位癌;20例乳腺良性结节,具体为13例纤维腺瘤、5例乳头状瘤、2例导管上皮增生。
血浆外泌体提取,RNA提取及检测见实施例1。检测结果见表3。
表3:乳腺结节良恶性鉴别
结果:19例乳腺恶性结节,14例判读为阳性,灵敏度为:73.68%;20例乳腺良性结节,18例判读为阴性,特异性为:90%。
结论:血浆外泌体中ENAH mRNA在乳腺恶性结节中的表达水平高于良性结节,可用于乳腺结节的良恶性鉴别。
实施例3
本实施例检测样本类型为:血浆,共入组91例样本。其中37例乳腺癌:3例中级别导管原位癌、5例高级别导管原位癌、17例浸润性导管癌、3例小叶原位癌,9例浸润性小叶癌;54非乳腺癌:13例肺癌、10例结直肠癌、13例乳腺良性疾病(7例乳腺炎、4例乳头状瘤、2例导管上皮增生)和18例健康人。
血浆外泌体提取,RNA提取及检测见实施例1。检测结果见表4。
表4:乳腺癌辅助诊断
结果:37例乳腺癌,28例检测阳性,灵敏度为:75.68%;54例非乳腺癌,47例检测阴性,特异性为:87.04%,其中:13例肺癌,10例阴性,特异性为:76.92%,10例结直肠癌,8例阳性,特异性为:80%,13例乳腺良性疾病,12例阴性,特异性为92.31%:18例健康人,17例阴性,特异性为:94.44%。
结论:血浆外泌体中ENAH mRNA在乳腺癌的表达水平不仅高于乳腺良性疾病,也高于其他肿瘤,具有一定的乳腺肿瘤特异性和乳腺组织特异性,表明血浆外泌体中ENAH mRNA表达水平可以用于乳腺癌的辅助诊断。
实施例4
本实施例检测样本类型为:血浆,入组了7例乳腺癌患者(2例导管原位癌、1例中级别导管原位癌伴小灶区浸润、1例导管原位癌伴微浸润、1例小叶原位癌、2例浸润性小叶癌),收集每个患者术前、术后7-10天的样本。通过检测7例患者术前术后配对血浆外泌体中ENAHmRNA表达水平的变化,考察靶标对乳腺癌治疗效果的监控能力。
血浆外泌体提取,RNA提取及检测见实施例1。检测结果见表5。检测结果趋势图见图2。
表5 乳腺癌病程监控
结果:入组7例乳腺癌术前术后配对样本,术后外泌体中的ENAH mRNA含量都有下降,其中有6例下降幅度超过50%。
结论:这说明外泌体中ENAH mRNA表达水平可有效监控乳腺癌治疗效果。
实施例5
入组样本:实施例4入组的91例血浆样本,同时检测血浆外泌体中ENAH和SEPT9 mRNA的表达,考察两个基因联合检测(ENAH的相对含量值/SEPT9的相对含量值)的临床检测性能。
血浆外泌体提取,RNA提取及检测见实施例1。检测结果见表6。
表6
结果:两个基因联合检测,进行相对定量值的比值进行判读,37例乳腺癌,31例检测阳性,灵敏度为:83.78%;54例非乳腺癌,49例检测阴性,特异性为:90.74%,其中:13例肺癌,11例阴性,特异性为:84.62%,10例结直肠癌,9例阳性,特异性为:90%,13例乳腺良性疾病,12例阴性,特异性为92.31%:18例健康人,17例阴性,特异性为:94.44%。
结论:由结果可知,血浆外泌体ENAH和SEPT9 联合检测,使用两基因表达量的比值(ENAH的相对含量值/SEPT9的相对含量值)对样本进行判读,检测性能比单靶标(ENAH)大大提升,乳腺癌的检测灵敏度从75.68%提升至83.78%;检测特异性从87.04%提升至90.74%。
以上所述实施例的各技术特征可以进行任意的组合,为使描述简洁,未对上述实施例中的各个技术特征所有可能的组合都进行描述,然而,只要这些技术特征的组合不存在矛盾,都应当认为是本说明书记载的范围。
以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明专利范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明专利的保护范围应以所附权利要求为准。
序列表
<110> 江苏为真生物医药技术股份有限公司;苏州普瑞迈德医学检验所有限公司
<120> 外泌体mRNA在乳腺疾病诊断中的应用
<160> 60
<170> SIPOSequenceListing 1.0
<210> 1
<211> 20
<212> DNA
<213> artificial sequence
<400> 1
agcagggctt cgagttcaac 20
<210> 2
<211> 20
<212> DNA
<213> artificial sequence
<400> 2
cggctgattt tggatttgaa 20
<210> 3
<211> 20
<212> DNA
<213> artificial sequence
<400> 3
atcaagtcca tcacgcacga 20
<210> 4
<211> 18
<212> DNA
<213> artificial sequence
<400> 4
cccgaaccct ggtgtgtc 18
<210> 5
<211> 19
<212> DNA
<213> artificial sequence
<400> 5
cggggaccac atcaacaac 19
<210> 6
<211> 20
<212> DNA
<213> artificial sequence
<400> 6
tgatgttgac ctcctcctgc 20
<210> 7
<211> 19
<212> DNA
<213> artificial sequence
<400> 7
gcgtccactg ctgcctcta 19
<210> 8
<211> 19
<212> DNA
<213> artificial sequence
<400> 8
accttgctca ggcgtttca 19
<210> 9
<211> 20
<212> DNA
<213> artificial sequence
<400> 9
ccacttcaaa cagcggatca 20
<210> 10
<211> 21
<212> DNA
<213> artificial sequence
<400> 10
ccgagtcctc atcaaattcc t 21
<210> 11
<211> 20
<212> DNA
<213> artificial sequence
<400> 11
taccaggtca acggcaagag 20
<210> 12
<211> 20
<212> DNA
<213> artificial sequence
<400> 12
cgcaggtagg caaactcaca 20
<210> 13
<211> 20
<212> DNA
<213> artificial sequence
<400> 13
tgggtcgcag ttcttgtttg 20
<210> 14
<211> 20
<212> DNA
<213> artificial sequence
<400> 14
acctcgaggg tgatggtctt 20
<210> 15
<211> 21
<212> DNA
<213> artificial sequence
<400> 15
ggatttgggt cgcagttctt g 21
<210> 16
<211> 20
<212> DNA
<213> artificial sequence
<400> 16
gtgtcactgg gctcaacctc 20
<210> 17
<211> 20
<212> DNA
<213> artificial sequence
<400> 17
gggtcgcagt tcttgtttgt 20
<210> 18
<211> 20
<212> DNA
<213> artificial sequence
<400> 18
aacctcgagg gtgatggtct 20
<210> 19
<211> 18
<212> DNA
<213> artificial sequence
<400> 19
ggtcgcagtt cttgtttg 18
<210> 20
<211> 18
<212> DNA
<213> artificial sequence
<400> 20
tcgagggtga tggtctta 18
<210> 21
<211> 18
<212> DNA
<213> artificial sequence
<400> 21
tcgcagttct tgtttgtg 18
<210> 22
<211> 18
<212> DNA
<213> artificial sequence
<400> 22
tcgagggtga tggtctta 18
<210> 23
<211> 20
<212> DNA
<213> artificial sequence
<400> 23
atccacctta atcaacaccc 20
<210> 24
<211> 18
<212> DNA
<213> artificial sequence
<400> 24
ccggacgcct ttctcctc 18
<210> 25
<211> 21
<212> DNA
<213> artificial sequence
<400> 25
cagcccatca tgaagttcat c 21
<210> 26
<211> 17
<212> DNA
<213> artificial sequence
<400> 26
ccaccggcca ccctcag 17
<210> 27
<211> 20
<212> DNA
<213> artificial sequence
<400> 27
cagacctgct gtccaacggc 20
<210> 28
<211> 22
<212> DNA
<213> artificial sequence
<400> 28
ccatcgaagt tgaaaacacc ac 22
<210> 29
<211> 23
<212> DNA
<213> artificial sequence
<400> 29
tgcagatctt cgtgaagact ctg 23
<210> 30
<211> 21
<212> DNA
<213> artificial sequence
<400> 30
tcacttgaca atgcagatct t 21
<210> 31
<211> 19
<212> DNA
<213> artificial sequence
<400> 31
tcacttgaca atgcagatc 19
<210> 32
<211> 19
<212> DNA
<213> artificial sequence
<400> 32
acttgacaat gcagatctt 19
<210> 33
<211> 23
<212> DNA
<213> artificial sequence
<400> 33
aatgcagatc ttcgtgaaga ctc 23
<210> 34
<211> 21
<212> DNA
<213> artificial sequence
<400> 34
atcaccatac aggcaacaac a 21
<210> 35
<211> 19
<212> DNA
<213> artificial sequence
<400> 35
caacccttta ggaatggca 19
<210> 36
<211> 21
<212> DNA
<213> artificial sequence
<400> 36
actggattca gcagagttca t 21
<210> 37
<211> 21
<212> DNA
<213> artificial sequence
<400> 37
agtttatcac gacctgatgg t 21
<210> 38
<211> 21
<212> DNA
<213> artificial sequence
<400> 38
ctatcaccat acaggcaaca a 21
<210> 39
<211> 20
<212> DNA
<213> artificial sequence
<400> 39
atggcacagt ttatcacgac 20
<210> 40
<211> 19
<212> DNA
<213> artificial sequence
<400> 40
acattcagag tggtgggca 19
<210> 41
<211> 23
<212> DNA
<213> artificial sequence
<400> 41
gattgtactt caacccttta gga 23
<210> 42
<211> 25
<212> DNA
<213> artificial sequence
<400> 42
ggtgccagct ggtggctcaa ctgga 25
<210> 43
<211> 23
<212> DNA
<213> artificial sequence
<400> 43
tgtagcttga ttgtacttca acc 23
<210> 44
<211> 19
<212> DNA
<213> artificial sequence
<400> 44
tcctgcccac cactctgaa 19
<210> 45
<211> 21
<212> DNA
<213> artificial sequence
<400> 45
ctatcaccat acaggcaaca a 21
<210> 46
<211> 19
<212> DNA
<213> artificial sequence
<400> 46
tgcccaccac tctgaatgt 19
<210> 47
<211> 21
<212> DNA
<213> artificial sequence
<400> 47
accatcaggt cgtgataaac t 21
<210> 48
<211> 19
<212> DNA
<213> artificial sequence
<400> 48
tgggcaggaa gattcagga 19
<210> 49
<211> 18
<212> DNA
<213> artificial sequence
<400> 49
cgccagctca ccatggat 18
<210> 50
<211> 18
<212> DNA
<213> artificial sequence
<400> 50
gatggagggg aagacggc 18
<210> 51
<211> 17
<212> DNA
<213> artificial sequence
<400> 51
aggcaccagg gcgtgat 17
<210> 52
<211> 18
<212> DNA
<213> artificial sequence
<400> 52
ccgtgctcga tggggtac 18
<210> 53
<211> 19
<212> DNA
<213> artificial sequence
<400> 53
cgcgagaaga tgacccaga 19
<210> 54
<211> 19
<212> DNA
<213> artificial sequence
<400> 54
gtacggccag aggcgtaca 19
<210> 55
<211> 18
<212> DNA
<213> artificial sequence
<400> 55
cagccttcct tcctgggc 18
<210> 56
<211> 19
<212> DNA
<213> artificial sequence
<400> 56
gcgtacaggt ctttgcgga 19
<210> 57
<211> 17
<212> DNA
<213> artificial sequence
<400> 57
catgtgcaag gccggct 17
<210> 58
<211> 19
<212> DNA
<213> artificial sequence
<400> 58
gcccagagca agagaggca 19
<210> 59
<211> 19
<212> DNA
<213> artificial sequence
<400> 59
agccatgtac gttgctatc 19
<210> 60
<211> 17
<212> DNA
<213> artificial sequence
<400> 60
caactccatc atgaagt 17
- 外泌体mRNA在乳腺疾病诊断中的应用
- 外泌体中SEPT9 mRNA在诊断乳腺癌中的应用
