作为结直肠肿瘤标志物的DNA甲基化位点组合及其应用
文献发布时间:2024-04-18 20:01:23
技术领域
本说明书涉及生物技术领域,特别涉及作为结直肠肿瘤标志物的DNA甲基化位点组合及其应用。
背景技术
结直肠肿瘤(Colorectal tumor)是发生在结肠(大肠)和直肠部位的肿瘤,包括结直肠癌、结直肠腺瘤等。结肠腺瘤是起源于结直肠黏膜腺上皮的良性肿瘤,其与结直肠癌的发生关系密切,被认为是一种癌前病变。
随着人们生活习惯的改变,结直肠肿瘤,特别是结直肠癌的发病率和死亡率均明显上升。结直肠肿瘤的早期诊断和治疗(例如,息肉切除)可以预防恶性肿瘤的发生发展,降低死亡率。因此,检测结直肠肿瘤和/或预测结直肠肿瘤的患病风险是提高患者存活率的有效手段。
目前,诊断结直肠肿瘤常用粪便潜血试验和结肠镜检查。其中,粪便潜血试验易受食物、药物和其他因素的影响,可能导致假阳性结果。粪便潜血试验的敏感度在30-80%之间。而结肠镜检查属于入侵性检查,在结肠镜下取出肿瘤样本进行病理学检查活性,该检查有多种禁忌症,如严重的心脏疾病、心肺功能不全、急性腹泻等。另外,结肠镜检查的敏感度也只在60-70%之间。因此,亟待提出一种敏感度和特异性更高的生物标志物,例如用于实现具有较强适用性的检测结直肠肿瘤的方法和预测结直肠肿瘤患病风险的方法。
发明内容
本说明书一个或多个实施例提供了作为生物标志物用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的DNA甲基化位点组合,其特征在于,所述DNA甲基化位点组合包括以下组中的一个或多个:位于NDRG4基因上染色体坐标为chr16:58497406的位点NDRG4_40;位于THBD基因上染色体坐标为chr20:23031082的位点THBD_102;位于WIF1基因上染色体坐标为chr12:65515031的位点WIF1_68;位于SDC2-2基因上染色体坐标为chr8:97505785的位点SDC2-2_56;位于DNAAF9基因上染色体坐标为chr20:3388892的位点DNAAF9_41;位于LIFR基因上染色体坐标为chr5:38557321的位点LIFR_42;位于ZNF304基因上染色体坐标为chr19:57862624的位点ZNF304_71;其中,对应位点的染色体坐标来源于人类参考基因组GRCh37。
在一些实施例中,所述DNA甲基化位点组合包括NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71。
在一些实施例中,所述DNA甲基化位点组合由NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71组成。
在一些实施例中,所述检测试剂包括用于扩增DNA甲基化位点组合的引物组,其中:用于扩增NDRG4_40的引物对,如SEQ ID NO:1和SEQ ID NO:2所示;用于扩增THBD_102的引物对,如SEQ ID NO:3和SEQ ID NO:4所示;用于扩增WIF1_68的引物对,如SEQ ID NO:5和SEQ ID NO:6所示;用于扩增SDC2-2_56的引物对,如SEQ ID NO:7和SEQ ID NO:8所示;用于扩增DNAAF9_41的引物对,如SEQ ID NO:9和SEQ ID NO:10所示;用于扩增LIFR_42的引物对,如SEQ ID NO:11和SEQ ID NO:12所示;用于扩增ZNF304_71的引物对,如SEQ ID NO:13和SEQ ID NO:14所示。
在一些实施例中,所述检测结直肠肿瘤或预测结直肠肿瘤患病风险的方法包括:获取受试者生物样本中所述DNA甲基化位点组合的甲基化水平;基于所述DNA甲基化位点组合的甲基化水平,使用筛查模型评估所述受试者是否可能患有结直肠肿瘤或所述受试者发展结直肠肿瘤的风险。
在一些实施例中,所述筛查模型为基于所述DNA甲基化位点组合的甲基化阈值的模型。
在一些实施例中,所述评估包括:对于所述DNA甲基化位点组合中的每个DNA甲基化位点,对比所述DNA甲基化位点的甲基化率与对应所述DNA甲基化位点的甲基化阈值,确定所述DNA甲基化位点组合的阳性位点数量;基于所述阳性位点数量获得评估结果,其中,所述阳性位点数量≥1指示所述受试者可能患有结直肠肿瘤或所述受试者存在发展结直肠肿瘤的风险。
在一些实施例中,所述DNA甲基化位点的甲基化阈值通过以下方法确定:获取训练样本集,所述训练样本集包括已知的结直肠肿瘤患者和非结直肠肿瘤患者的所述DNA甲基化位点的甲基化率;使用ROC曲线对所述训练样本集进行分析,确定用于区分所述结直肠肿瘤患者和所述非结直肠肿瘤患者的截止值,以所述截止值作为所述DNA甲基化位点的甲基化阈值;其中,所述截止值选自特异性在95%~100%时的甲基化率。
在一些实施例中,NDRG4_40的甲基化阈值为0.1857;THBD_102的甲基化阈值为0.1094;WIF1_68的甲基化阈值为0.2983;SDC2-2_56的甲基化阈值为0.0566;DNAAF9_41的甲基化阈值为0.0172;LIFR_42的甲基化阈值为0.0407;ZNF304_71的甲基化阈值为0.0959。
在一些实施例中,所述结直肠肿瘤包括结直肠癌和结直肠腺瘤。
在一些实施例中,所述生物样本来自于所述受试者的结直肠灌洗液。
本说明书一个或多个实施例还提供了一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的试剂盒,所述试剂盒包含如前文所述的检测试剂。
本说明书一个或多个实施例还提供了一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的装置,所述装置包括存储器、处理器以及存储在所述存储器上并可在所述处理器上运行的计算机程序,其特征在于,所述处理器执行所述程序时实现以下方法:获取受试者生物样本中DNA甲基化位点组合的甲基化水平;基于所述DNA甲基化位点组合的甲基化水平,使用筛查模型评估所述受试者是否可能患有结直肠肿瘤或所述受试者发展结直肠肿瘤的风险。
附图说明
本说明书将以示例性实施例的方式进一步说明,这些示例性实施例将通过附图进行详细描述。这些实施例并非限制性的,在这些实施例中,相同的编号表示相同的结构,其中:
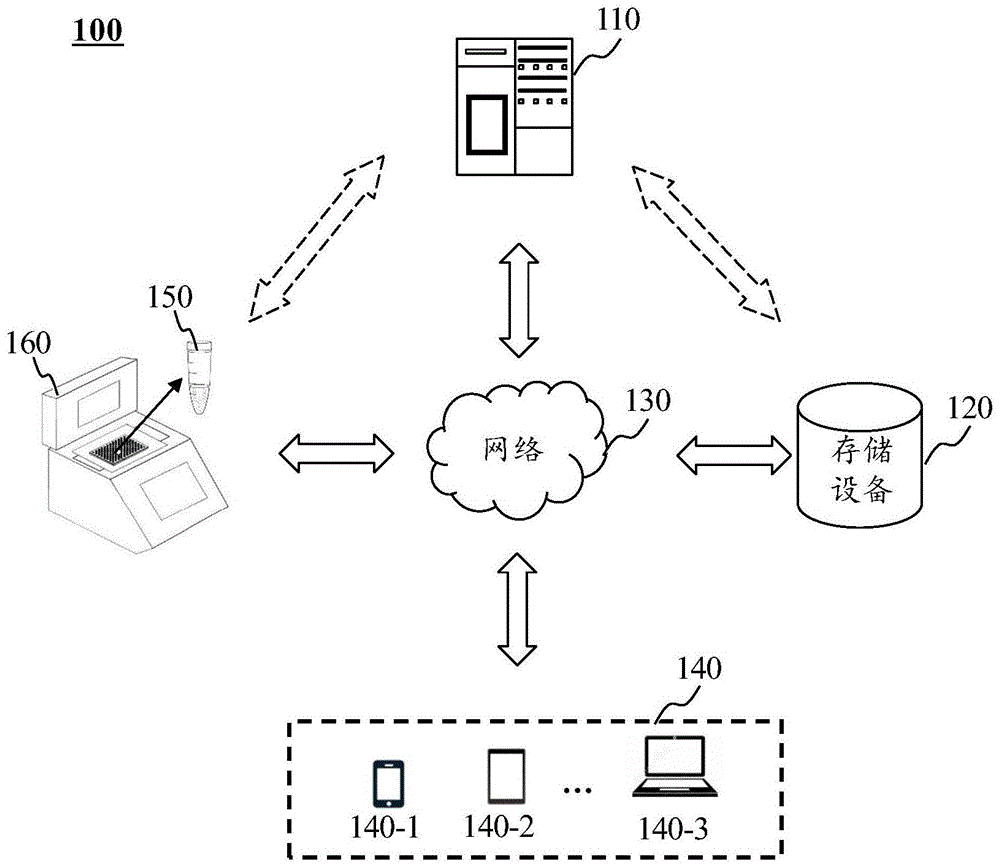
图1是根据本说明书一些实施例所示的一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统的应用场景图;
图2是根据本说明书一些实施例所示的计算设备的架构的示意图;
图3是根据本说明书一些实施例所示的用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统的模块图;
图4是根据本说明书一些实施例所示的检测结直肠肿瘤或预测结直肠肿瘤患病风险的方法的流程示意图;
图5是根据本说明书一些实施例所示的确定DNA甲基化位点的甲基化阈值的流程示意图;
图6是根据本说明书一些实施例所示的甲基化位点NDRG4_40在训练样本集中的ROC曲线图;
图7是根据本说明书一些实施例所示的甲基化位点SDC2_101在训练样本集中的ROC曲线图;
图8是根据本说明书一些实施例所示的甲基化位点SEP9-1_69在训练样本集中的ROC曲线图;
图9是根据本说明书一些实施例所示的甲基化位点SEP9-2_61在训练样本集中的ROC曲线图;
图10是根据本说明书一些实施例所示的甲基化位点SFRP1_40在训练样本集中的ROC曲线图;
图11是根据本说明书一些实施例所示的甲基化位点TFPI2_117在训练样本集中的ROC曲线图;
图12是根据本说明书一些实施例所示的甲基化位点THBD_102在训练样本集中的ROC曲线图;
图13是根据本说明书一些实施例所示的甲基化位点BCAT1_38在训练样本集中的ROC曲线图;
图14是根据本说明书一些实施例所示的甲基化位点WIF1_68在训练样本集中的ROC曲线图;
图15是根据本说明书一些实施例所示的甲基化位点WIF1_88在训练样本集中的ROC曲线图;
图16是根据本说明书一些实施例所示的甲基化位点WNT5A_47在训练样本集中的ROC曲线图;
图17是根据本说明书一些实施例所示的甲基化位点TFPI2-2_91在训练样本集中的ROC曲线图;
图18是根据本说明书一些实施例所示的甲基化位点SDC2-2_56在训练样本集中的ROC曲线图;
图19是根据本说明书一些实施例所示的甲基化位点SDC2-2_73在训练样本集中的ROC曲线图;
图20是根据本说明书一些实施例所示的甲基化位点DNAAF9_41在训练样本集中的ROC曲线图;
图21是根据本说明书一些实施例所示的甲基化位点LIFR_42在训练样本集中的ROC曲线图;
图22是根据本说明书一些实施例所示的甲基化位点ZNF304_71在训练样本集中的ROC曲线图。
具体实施方式
为了更清楚地说明本说明书实施例的技术方案,下面将对实施例描述中所需要使用的附图作简单的介绍。显而易见地,下面描述中的附图仅仅是本说明书的一些示例或实施例,对于本领域的普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图将本说明书应用于其它类似情景。除非从语言环境中显而易见或另做说明,图中相同标号代表相同结构或操作。
应当理解,本文使用的“系统”、“装置”、“单元”和/或“模块”是用于区分不同级别的不同组件、元件、部件、部分或装配的一种方法。然而,如果其他词语可实现相同的目的,则可通过其他表达来替换所述词语。
如本说明书和权利要求书中所示,除非上下文明确提示例外情形,“一”、“一个”、“一种”和/或“该”等词并非特指单数,也可包括复数。一般说来,术语“包括”与“包含”仅提示包括已明确标识的步骤和元素,而这些步骤和元素不构成一个排它性的罗列,方法或者设备也可能包含其它的步骤或元素。
本说明书中使用了流程图用来说明根据本说明书的实施例的系统所执行的操作。应当理解的是,前面或后面操作不一定按照顺序来精确地执行。相反,可以按照倒序或同时处理各个步骤。同时,也可以将其他操作添加到这些过程中,或从这些过程移除某一步或数步操作。
DNA甲基化与癌症的发生发展关系密切,相关基因位点的甲基化过程是肿瘤极早期发生的关键性事件,并且肿瘤甲基化模式具有癌种/组织/时空特异性的特点。DNA甲基化主要出现在基因启动子区域的CpG岛中的胞嘧啶,通过阻碍转录因子的结合从而导致基因特别是抑癌基因的表达降低,导致肿瘤事件的发生。发明人们发现,DNA甲基化发展进程的研究对鉴别/预测结直肠肿瘤的发生具有重要意义。本说明书提出可以以DNA甲基化位点组合作为结直肠肿瘤标志物,进行结直肠肿瘤检测、结直肠肿瘤患病风险预测。该DNA甲基化位点组合的检测样本可广泛来源于受试者的体液、细胞、组织和器官,特别是受试者的结直肠灌洗液,能够用于实现准确、快速、无创的结直肠肿瘤检测、患病风险预测。
本说明书提供了一种检测结直肠肿瘤或预测结直肠肿瘤患病风险的方法,及其系统和装置,所述方法、系统和装置基于前述DNA甲基化位点组合的相关甲基化水平评估受试者患结直肠肿瘤的可能性或发展结直肠癌的风险。
本说明书还提供了一种DNA甲基化位点组合的检测试剂,包括扩增前述DNA甲基化位点组合的试剂,能够在包括检测结直肠肿瘤、预测结直肠肿瘤患病风险等在内的多个方面进行广泛应用。
本说明书还提供了一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的试剂盒。
本说明书还提供了DNA甲基化位点组合作为生物标志物的相关用途,以及DNA甲基化位点组合的检测试剂的相关用途。所述用途包括但不限于在制备用于检测结直肠肿瘤的试剂盒中的用途,在制备用于预测结直肠肿瘤患病风险的试剂盒中的用途等,能够兼顾及提高筛查、预测、筛选的敏感度和特异性。
根据本说明书的一方面,提供了一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统。图1是根据本说明书一些实施例所示的一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统的应用场景图。在一些实施例中,场景100包括处理设备110、存储设备120和网络130。
处理设备110用于处理数据和/或信息。在一些实施例中,处理设备110可以从存储设备120或场景100的其他组件(例如,用户终端140、检测设备160)处获得数据和/或信息,并基于这些信息和/或数据执行程序指令,以执行一个或多个本说明书中描述的功能。例如,处理设备110可以从存储设备120处获取训练样本集,并基于训练样本集构建筛查模型。又例如,处理设备110可以获取检测设备160测得的受试者生物样本150的DNA甲基化位点组合的甲基化水平相关信息,并调用存储在存储设备120处的筛查模型处理该甲基化水平相关信息,以评估受试者是否患有结直肠肿瘤的可能性或发展结直肠肿瘤的风险。在一些实施例中,处理设备110可以为服务器或中央处理器。
存储设备120用于存储数据和/或信息。在一些实施例中,存储设备120可以存储从处理设备110或场景100的其他组件(例如,用户终端140、检测设备160)处获得数据和/或信息。例如,存储设备120可以存储筛查模型,以备处理设备110调用。又例如,存储设备120可以从检测设备160处获取并存储受试者生物样本150的DNA甲基化位点组合的甲基化水平相关信息。再例如,存储设备120可以接收并存储用户终端140上传的信息,如受试者的身份信息等。
网络130用于提供信息交换的渠道。在一些实施例中,处理设备110和场景100的其他组件(例如,存储设备120、用户终端140、检测设备160)之间可以通过网络130交换信息。例如,处理设备110可以通过网络130接收存储设备120中的数据。又例如,检测设备160测得的受试者生物样本150的DNA甲基化位点组合的甲基化水平相关信息可以通过网络传输至处理设备110。在一些实施例中,网络130可以是有线网络或无线网络中的任意一种或多种。例如,网络130可以包括电缆网络、光纤网络等。在一些实施例中,网络130可以是点对点的、共享的、中心式的等各种拓扑结构或者多种拓扑结构的组合。在一些实施例中,网络130可以包括一个或以上网络接入点。例如,通过基站和/或一个或多个网络交换点等进出点,场景100的一个或多个组件可连接到网络130上以交换数据和/或信息。
在一些实施例中,场景100还包括用户终端140。用户终端140用于实现场景100向用户提供的服务。例如,用户可以通过用户终端140向处理设备110发送受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息。又例如,用户可以通过用户终端140接收处理设备110发送的受试者的评估结果。再例如,用户可以通过用户终端140向处理设备110发送受试者的临床检测结果,以使处理设备110基于受试者的临床检测结果更新训练样本集,并进行筛查模型的迭代。在一些实施例中,用户终端140可以包括智能手机140-1、平板计算机140-2、膝上型计算机140-3等或其他具有输入和/或输出功能的设备中的一种或其任意组合。
在一些实施例中,场景100还包括检测设备160。检测设备160用于检测生物样本150的DNA甲基化位点组合的甲基化水平。作为示例,检测设备可以包括实现以下一种或多种方法的装置:WGBS、RRBS、oxBS-seq、MethylCap-seq、MBD-seq、MeDIP-seq、HPLC、MSRF、MASP、甲基化芯片法(如805k芯片)、焦磷酸测序法、dPCR和MS-PCR。
根据本说明书的又一方面,提供了一种计算设备。图2是根据本说明书一些实施例所示的计算设备的架构的示意图。如图2所示,计算设备200包括处理器210、存储器220、输入/输出接口230和通信端口240。在一些实施例中,计算设备200可以实现处理设备110和/或存储设备120。例如,处理设备110可以在计算设备200上实现,并且计算设备200被配置为执行本说明书描述的处理设备110的功能。在一些实施例中,用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的装置可以在计算设备200中实现。
处理器210用于执行计算指令(程序代码)并执行本说明书描述的处理设备110的功能。计算指令可以包括程序、对象、组件、数据结构、过程、模块和功能(功能指本申请中描述的特定功能)。例如,处理器210可以处理用户输入的结直肠肿瘤检测或预测结直肠肿瘤患病风险的可能性的指令。在一些实施例中,计算设备200可以包括一个或多个处理器210;处理器210可以包括中央处理器(CPU)、专用集成电路(ASIC)以及能够执行一个或多个功能的任何电路和处理器等,或者任意组合。
存储器220用于存储从场景100的任何组件处获得的数据/信息。在一些实施例中,存储器220可以包括随机存取存储器(RAM)、只读存储器(ROM)等,或其任意组合。
输入输出接口230用于输入或输出信号、数据或信息。在一些实施例中,输入输出接口230可以用于实现用户(例如,受试者、操作者等)与处理设备210的交互行为。在一些实施例中,用户可以通过输入输出接口230输入受试者的相关信息(例如,DNA甲基化位点组合的甲基化水平相关信息,以及姓名、年龄等基础身份信息)。在一些实施例中,输入输出接口230可以包括输入装置和输出装置。例如,键盘、鼠标、显示设备、麦克风和扬声器等。
通信端口240用于连接到网络130以便进行数据通信。连接可以是有线连接、无线连接或两者的组合,例如通过电缆、光缆、移动网络、WIFI、WLAN或蓝牙等连接。在一些实施例中,通信端口240可以是标准化端口,如RS232、RS485等。在一些实施例中,通信端口240可以是专门设计的端口。
图3是根据本说明书一些实施例所示的用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统的模块图。如图3所示,用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统300包括获取模块310、分析模块320和确定模块330。
获取模块310用于获取受试者生物样本中DNA甲基化位点组合的甲基化水平,例如,所述DNA甲基化位点组合可以包括NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71中的一个或多个位点。
在一些实施例中,获取模块310可以包括检测单元和信息处理单元。检测单元用于对受试者的生物样本进行DNA甲基化检测。示例性的,检测单元可以包括实现以下一种或多种方法的装置:WGBS、RRBS、oxBS-seq、MethylCap-seq、MBD-seq、MeDIP-seq、HPLC、MSRF、MASP、甲基化芯片法(如805k芯片)、焦磷酸测序法、dPCR和MS-PCR。信息处理单元用于处理检测单元的检测数据,以获得受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息。
分析模块320用于基于受试者生物样本的DNA甲基化位点组合的甲基化水平,使用筛查模型评估受试者是否可能患有结直肠肿瘤或受试者发展结直肠肿瘤的风险。在一些实施例中,分析模块320可以用于使用基于DNA甲基化位点组合的甲基化阈值的模型进行评估。在一些实施例中,分析模块320可以用于使用基于机器学习算法或深度学习算法构建的模型进行评估。
确定模块330用于获取训练样本集,训练样本集包括已知的结直肠肿瘤患者和非结直肠肿瘤患者的DNA甲基化位点的甲基化率;以及使用ROC曲线对训练样本集进行分析,确定用于区分结直肠肿瘤患者和非结直肠肿瘤患者的截止值,以该截止值作为所述DNA甲基化位点的甲基化阈值。
关于系统300各模块实现其功能的更多内容可以参见图4、图5及其相关描述。
应当理解,图3所示的用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的系统300及其模块可以利用各种方式来实现。例如,在一些实施例中,系统300及其模块可以通过硬件、软件或者软件和硬件的结合来实现。其中,硬件部分可以利用专用逻辑来实现;软件部分则可以存储在存储器中,由适当的指令执行系统,例如微处理器或者专用设计硬件来执行。本领域技术人员可以理解上述的方法和系统可以使用计算机可执行指令和/或包含在处理器控制代码中来实现,例如在诸如磁盘、CD或DVD-ROM的载体介质、诸如只读存储器(固件)的可编程的存储器或者诸如光学或电子信号载体的数据载体上提供了这样的代码。本说明书的系统及其模块不仅可以有诸如超大规模集成电路或门阵列、诸如逻辑芯片、晶体管等的半导体、或者诸如现场可编程门阵列、可编程逻辑设备等的可编程硬件设备的硬件电路实现,也可以用例如由各种类型的处理器所执行的软件实现,还可以由上述硬件电路和软件的结合(例如,固件)来实现。
需要注意的是,以上对于系统300及其模块的描述,仅为描述方便,并不能把本说明书限制在所举实施例范围之内。可以理解,对于本领域的技术人员来说,在了解该系统的原理后,可能在不背离这一原理的情况下,对各个模块进行任意组合,或者构成子系统与其他模块连接。在一些实施例中,图3中披露的获取模块、分析模块和确定模块可以是一个系统中的不同模块,也可以是一个模块实现上述的两个或两个以上模块的功能。例如,各个模块可以共用一个存储模块,各个模块也可以分别具有各自的存储模块。诸如此类的变形,均在本说明书的保护范围之内。
根据本说明书的又一方面,提供了一种检测结直肠肿瘤或预测结直肠肿瘤患病风险的方法。图4是根据本说明书一些实施例所示的检测结直肠肿瘤或预测结直肠肿瘤患病风险的方法的流程示意图。如图4所示,流程400包括步骤401和步骤403。在一些实施例中,流程400中的至少一部分步骤(例如,步骤401和/或步骤403)可以由计算设备(如图2所示的计算设备200,图1所示的处理设备110)完成。例如,流程400中的至少一部分步骤可以被实现为存储在存储设备120、存储器220中的一个指令(例如,应用程序)。图1中的处理设备110,图2中的处理器210和/或模块可以执行该指令,并且在执行指令时,处理设备110、处理器210和/或模块可以被配置为执行流程400。以下所示过程的操作仅出于说明的目的。在一些实施例中,流程400可以利用未描述的一个或以上附加操作和/或未描述的一个或以上操作来完成。另外,图4所示和以下描述的过程的操作顺序并非旨在限制。
步骤401,获取受试者生物样本中DNA甲基化位点组合的甲基化水平。在一些实施例中,步骤401可以由计算设备(例如,图1的处理设备110、图3的获取模块310)执行。
在一些实施例中,患有结直肠肿瘤的受试者的生物样本中DNA甲基化位点组合的甲基化水平可区别于非结直肠肿瘤受试者(或称正常受试者)的生物样本中DNA甲基化位点组合的甲基化水平。
术语“受试者”是指接收观察、检测或实验的对象。在一些实施例中,受试者可以是哺乳动物。例如,哺乳动物可以包括人、小鼠和大鼠等。在一些实施例中,受试者可以是人。
术语“生物样本”是指分离自受试者的器官、组织、细胞和/或体液的组合物。在一些实施例中,该组合物包含一种或多种目标分析物。例如,目标分析物可以为核酸和代谢物等。在一些实施例中,生物样本由受试者的体液中获得。例如,体液可以包括灌洗液、全血、血浆、血清、组织液、唾液、尿液、粪便等。在一些实施例中,生物样本可以来自于受试者的结直肠灌洗液。
DNA甲基化位点组合包括一个或多个DNA甲基化位点。术语“DNA甲基化位点”是指在基因组DNA的CpG二核苷酸的胞嘧啶第5’碳位共价结合一个甲基基团,成为5-甲基胞嘧啶(5mC)。
DNA甲基化位点组合适于区分患结直肠肿瘤群体和正常群体,能够用于检测或预测不同类型的结直肠肿瘤。在一些实施例中,结直肠肿瘤包括结直肠癌。例如,按病理类型分,结直肠癌可以包括腺癌、腺鳞癌和未分化癌;按解剖部位分,结直肠癌可以包括直肠癌、左半结肠癌(例如,左侧横结肠癌、降结肠癌或乙状结肠癌)和右半结肠癌(例如,盲肠癌、升结肠癌或右侧横结肠癌)。在一些实施例中,结直肠肿瘤包括结直肠腺瘤。例如,直结肠腺瘤可以包括结肠腺瘤和直肠腺瘤。在一些实施例中,结直肠肿瘤还包括类癌(Carcinoid)、阑尾肿瘤和结肠间质瘤等。
在一些较优的实施例中,DNA甲基化位点组合适于检测或预测的结直肠肿瘤包括结直肠癌和结直肠腺瘤。在一些实施例中,DNA甲基化位点组合适于检测或预测的结直肠癌,其分期包括Ⅰ期、Ⅱ期、Ⅲ期和Ⅳ期。在一些实施例中,DNA甲基化位点组合适于检测或预测的结直肠腺瘤,其分级包括一级、二级(例如,低度型和/或高度型)和三级。
在一些实施例中,DNA甲基化位点组合的DNA甲基化位点可位于结直肠肿瘤相关基因(例如,已知的或可能潜在的结直肠肿瘤基因)上。结直肠肿瘤相关基因的非限制性实例可以包括但不限于NDRG4(染色体坐标为chr16:58497369-58497501)、SDC2(染色体坐标为chr8:97506318-97506450)、SDC2-2(染色体坐标为chr8:97505730-97505866)、SFRP1(染色体坐标为chr8:41166970-41167048)、TFPI2(染色体坐标为chr7:93519985-93520149)、TFPI2-2(染色体坐标为chr7:93519341-93519462)、THBD(染色体坐标为chr20:23030981-23031129)、BCAT1(染色体坐标为chr12:25101973-25102116)、SEP9-1(染色体坐标为chr17:75369564-75369660)、SEP9-2(染色体坐标为chr17:75369559-75369649)、WIF1(染色体坐标为chr12:65514965-65515076)、WNT5A(染色体坐标为chr3:55521225-55521340)、DNAAF9(染色体坐标为Chr20:3388932-3388804)、LIFR(染色体坐标为Chr5:38557362-38557228)和ZNF304(染色体坐标为Chr19:57862581-57862691)。
需要说明的是,本文所用染色体坐标信息来源于人类参考基因组GRCh37。
在一些实施例中,作为生物标志物用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的DNA甲基化位点组合可以包括位于NDRG4、SDC2、SDC2-2、SFRP1、TFPI2、TFPI2-2、THBD、BCAT1、SEP9-1、SEP9-2、WIF1、WNT5A、DNAAF9、LIFR和ZNF304上的一个或多个DNA甲基化位点。
在一些实施例中,作为生物标志物用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的DNA甲基化位点组合可以包括NDRG4_40(位于NDRG4上,染色体坐标为chr16:58497406)、SDC2_101(位于SDC2上,染色体坐标为chr8:97506415)、SEP9-1_69(位于SEP9-1上,染色体坐标为chr17:75369592)、SEP9-2_61(位于SEP9-2上,染色体坐标为chr17:75369591)、SFRP1_40(位于SFRP1上,染色体坐标为chr8:41167007)、TFPI2_117(位于TFPI2上,染色体坐标为chr7:93520101)、THBD_102(位于THBD上,染色体坐标为chr20:23031082)、BCAT1_38(位于BCAT1上,染色体坐标为chr12:25102010)、WIF1_68(位于WIF1上,染色体坐标为chr12:65515031)、WIF1_88(位于WIF1上,染色体坐标为chr12:65515051)、WNT5A_47(位于WNT5A上,染色体坐标为chr3:55521260)、TFPI2-2_91(位于TFPI2-2上,染色体坐标为chr7:93519373)、SDC2-2_56(位于SDC2-2上,染色体坐标为chr8:97505785)、SDC2-2_73(位于SDC2-2上,染色体坐标为chr8:97505802)、DNAAF9_41(位于DNAAF9上,染色体坐标为chr20:3388892)、LIFR_42(位于LIFR上,染色体坐标为chr5:38557321)和ZNF304_71(位于ZNF304上,染色体坐标为chr19:57862624)中的一个或多个DNA甲基化位点。
在一些实施例中,作为生物标志物用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的DNA甲基化位点组合可以包括以下组中的至少1个、2个、3个、4个、5个或6个位点:NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71。
在一些较优的实施例中,所述DNA甲基化位点组合可以包括NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71。可选的,所述DNA甲基化位点组合还可以包括一个或多个其他结直肠肿瘤相关基因上的DNA甲基化位点。
在一些实施例中,所述DNA甲基化位点组合可以由以下组中的至少4个、5个或6个位点组成:NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71。例如,所述DNA甲基化位点组合可以由SDC2-2_56、ZNF304_71、NDRG4_40和LIFR_42组成。又例如,所述DNA甲基化位点组合可以由SDC2-2_56、ZNF304_71、NDRG4_40、THBD_102和LIFR_42组成。又例如,所述DNA甲基化位点组合可以由NDRG4_40、SDC2-2_56、DNAAF9_41、ZNF304_71、THBD_102和LIFR_42组成。在一些较优的实施例中,所述DNA甲基化位点组合可以由NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71组成。
本说明书一些实施例提供的DNA甲基化位点组合的甲基化水平与结直肠肿瘤之间存在显著的相关性。该DNA甲基化位点组合的甲基化状态可以被量化,并用于衡量DNA甲基化位点组合的甲基化水平。包含该DNA甲基化位点组合的样本可广泛地采集自受试者的器官、组织、细胞和体液等,特别是可以采集自受试者的结直肠灌洗液,样本采集和检测的舒适性较高。该DNA甲基化位点组合作为结直肠肿瘤标志物在检测结直肠肿瘤、预测结直肠肿瘤患病风险等方面的应用可实现筛查/诊断、预测、评估的敏感度和特异性的提高。
在一些实施例中,DNA甲基化位点组合的甲基化水平可以通过使用DNA甲基化位点组合的检测试剂检测受试者生物样本而获得。DNA甲基化位点组合的检测试剂用于实现DNA甲基化位点组合甲基化水平的检测。关于DNA甲基化位点组合的检测试剂的更多内容可以在本说明的其他部分找到。
计算设备可通过多种方式实现步骤401的执行。在一些实施例中,处理设备110可调用存储在存储设备120中的受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息。例如,受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息由用户终端140经网络130上传至存储设备120,处理设备110可调用并获取该甲基化水平相关信息用于进一步的分析评估。在一些实施例中,处理设备110可以接收检测设备160检测获得的受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息。例如,处理设备110向检测设备160(例如,PCR仪和/或NGS测序仪)发送检测指令,检测设备160基于该检测指令检测获得受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息,并将该甲基化水平相关信息发送至处理设备110。在一些实施例中,处理设备110可基于用户输入获得受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息。
步骤403,基于受试者生物样本中DNA甲基化位点组合的甲基化水平,使用筛查模型评估受试者是否可能患有结直肠肿瘤或受试者发展结直肠肿瘤的风险。在一些实施例中,步骤403可以由计算设备(例如,图1的处理设备110、图3的分析模块320)执行。
在一些实施例中,筛查模型可以是基于DNA甲基化位点组合的甲基化阈值的模型(或称阈值模型)。阈值模型可以通过阈值判断划分受试者生物样本的类型,实现对患病可能性或发展疾病的风险的评估。在一些实施例中,使用阈值模型进行评估可包括阳性位点判断步骤和综合评估步骤。
在一些实施例中,步骤403进一步包括:对于DNA甲基化位点组合中的每个DNA甲基化位点,对比DNA甲基化位点的甲基化率与对应DNA甲基化位点的甲基化阈值,确定DNA甲基化位点组合的阳性位点数量。
在一些实施例中,DNA甲基化位点组合的甲基化水平可通过甲基化率来定量描述,甲基化率的确定方式可以基于具体的甲基化检测方法来设置。
在一些实施例中,受试者生物样本的DNA甲基化位点组合的甲基化水平通过甲基化转化、特异性扩增和测序进行检测。例如,甲基化转化可以包括使用甲基化转化试剂使DNA甲基化位点中的非甲基化胞嘧啶转化为胸腺嘧啶,甲基化胞嘧啶不发生转化。对于DNA甲基化位点组合的每个DNA甲基化位点,其甲基化率可以通过以下公式(1)确定:
甲基化率=NumC/(NumC+NumT)(1)
其中,NumC代表包含特定DNA甲基化位点的所有测序读段(reads)中,该特定DNA甲基化位点为胞嘧啶的读段数量;NumT代表包含特定DNA甲基化位点的所有测序读段中,该特定DNA甲基化位点为胸腺嘧啶的读段数量。
甲基化阈值是指用于作为评价DNA甲基化位点的甲基化水平的界限。在一些实施例中,DNA甲基化位点组合中单个DNA甲基化位点的甲基化率大于或等于对应该DNA甲基化位点的甲基化阈值,则可以判断该DNA甲基化位点为阳性位点。反之则为阴性位点。
关于确定甲基化阈值的更多内容可以参见图5及其相关描述。
在一些实施例中,步骤403还进一步包括:基于受试者生物样本的DNA甲基化位点组合的阳性位点数量获得评估结果。其中,若阳性位点数量≥1,则可以判断该受试者可能患有结直肠肿瘤,或者该受试者可能存在发展结直肠肿瘤的风险。反之,则可以排除该受试者患有结直肠肿瘤的可能性,或者排除该受试者发展结直肠肿瘤的风险。例如,DNA甲基化位点组合包括n(>2)个甲基化位点,上述甲基化位点中任意一个或多个在受试者样本中的甲基化率大于对应甲基化阈值,均指示该受试者可能患有结直肠肿瘤或存在发展结直肠肿瘤的风险。
在一些实施例中,筛查模型可以是机器学习模型或深度学习模型。例如,机器学习模型可以包括线性回归模型(Linear Regression)、逻辑回归模型(LogisticRegression)、支持向量机(Support Vector Machines)、K近邻(K-Nearest Neighbors)和朴素贝叶斯(Naive Bayes)等;深度学习模型可以包括人工神经网络(Artificial NeuralNetworks)、卷积神经网络(Convolutional Neural Networks)、循环神经网络(RecurrentNeural Networks)、长短时记忆网络(Long Short-Term Memory)和深度强化学习(Deep ReinforcementLearning)等。
在一些实施例中,筛查模型的输入可以是受试者生物样本的DNA甲基化位点组合的甲基化率,筛查模型的输出可以是受试者患有结直肠肿瘤的概率或受试者发展结直肠肿瘤的概率。
在一些实施例中,筛查模型可以基于第一训练样本和第一标签训练得到。第一训练样本可以是一个或多个已知的结直肠肿瘤患者样本的DNA甲基化位点组合的甲基化率和非结直肠肿瘤患者样本的DNA甲基化位点组合的甲基化率,第一标签可以是对应于第一训练样本的样本对象是否患有结直肠肿瘤。
术语“已知的结直肠肿瘤患者”是指对象或个体具有结直肠肿瘤临床症状且获得临床诊断验证。术语“非结直肠肿瘤患者”是指对象或个体未罹患结直肠肿瘤且日常生活无障碍。
计算设备可通过多种方式实现步骤403的执行。在一些实施例中,处理设备110可以调用存储在存储设备120中的筛查模型,并使用该筛查模型处理受试者生物样本的DNA甲基化位点组合的甲基化水平相关信息,以获得评估结果。在另一些实施例中,处理设备110可以基于用户指令更新存储在存储设备120中的筛查模型,并使用更新的筛查模型获得评估结果。其中,处理设备110可以通过网络130从公开或非公开的数据库收集结直肠癌群体和正常群体的关联DNA甲基化位点组合的甲基化水平相关信息,用于更新训练样本集并进行筛查模型的优化。处理设备110还可以基于用户输入或基于用户终端140上传的数据/信息更新训练样本集,并进行筛查模型的优化。
在一些实施例中,流程400还包括:基于评估结果,对罹患结直肠肿瘤的患者施用药物。在一些实施例中,结直肠肿瘤患者适于施用的药物包括但不限于奥沙利铂(Oxaliplatin)、伊立替康(Irinotecan)、5-氟尿嘧啶(5-FU)、亚叶酸钙、替吉奥(Tegafur)、抗EGFR抗体药物(例如,西妥昔单抗和希罗达肌单抗)、VEGF抑制剂(例如,贝伐珠单抗)和抗PD-1/PD-L1抑制剂等。
应当注意的是,上述有关流程400的描述仅仅是为了示例和说明,而不限定本说明书的适用范围。对于本领域技术人员来说,在本说明书的指导下可以对流程400进行各种修正和改变。然而,这些修正和改变仍在本说明书的范围之内。
图5是根据本说明书一些实施例所示的确定DNA甲基化位点的甲基化阈值的流程示意图。如图5所示,流程500包括步骤501和步骤503。在一些实施例中,流程500可以由计算设备(例如,图1的处理设备110、图3的确定模块330)执行。
步骤501,获取训练样本集,训练样本集包括已知的结直肠肿瘤患者和非结直肠肿瘤患者的DNA甲基化位点的甲基化率。
在一些实施例中,已知的结直肠肿瘤患者可以包括结直肠癌患者和结直肠腺瘤患者。已知的结直肠肿瘤患者可以是确诊后未接受治疗的个体,也可以是确诊后已接受治疗的个体。在一些实施例中,结直肠癌患者可选自结直肠癌Ⅰ期、结直肠癌Ⅱ期、结直肠癌Ⅲ期和结直肠癌Ⅳ期患者。在一些实施例中,结直肠腺瘤患者可选自结直肠腺瘤一级患者、结直肠腺瘤二级患者和结直肠腺瘤三级患者。
步骤503,使用ROC曲线对训练样本集进行分析,确定用于区分结直肠肿瘤患者和非结直肠肿瘤患者的截止值,以截止值作为DNA甲基化位点的甲基化阈值。
截止值是用于划分或区分结直肠肿瘤群体和非结直肠群体的可量化的值。在一些实施例中,截止值可作为甲基化阈值,通过衡量受试者的特定DNA甲基化位点组合的甲基化水平来检测或预测结直肠肿瘤。
术语“ROC曲线”是指以实验敏感度为纵坐标,以1-特异性为横坐标绘制的曲线。ROC曲线可以用于选择最佳截止值,以及评价模型性能。在一些实施例中,可以针对单个DNA甲基化位点,使用训练样本集的甲基化率数据制作ROC曲线,并基于适应应用需求的截止值选取方式确定适合的甲基化阈值。
在一些实施例中,截止值选取方式可影响截止值划分或区分结直肠肿瘤群体和非结直肠群体的效果。在一些实施例中,截止值选取方式可以是使用特异性设置值对应的甲基化率数值作为截止值,例如,特异性设置值为95%~100%。在一些实施例中,截止值选取方式可以是使用敏感度设置值对应的甲基化率数值作为截止值,例如,敏感度设置值为95%~100%。在另一些实施例中,截止值选取方式可以是使用约登指数最大值对应的甲基化率数值作为截止值。其中,约登指数可以是敏感度+特异性-1。
在一些较优的实施例中,使用特异性设置值对应的甲基化率数值作为截止值,旨在减少筛查模型引起的过度诊断问题,平衡筛查模型的特异性和敏感度。例如,特异性设定值为95%、96%、97%、98%、99%或100%。
在一些更优的实施例中,截止值可以为特异性在100%时的甲基化率。在一些实施例中,位点NDRG4_40的甲基化阈值可以为0.1857;位点THBD_102的甲基化阈值可以为0.1094;位点WIF1_68的甲基化阈值可以为0.2983;位点SDC2-2_56的甲基化阈值可以为0.0566;位点DNAAF9_41的甲基化阈值可以为0.0172;位点LIFR_42的甲基化阈值可以为0.0407;位点ZNF304_71的甲基化阈值可以为0.0959。
在一些实施例中,本说明书提供的筛选模型在检测结直肠肿瘤或预测结直肠肿瘤患病风险中的敏感度可以大于86%、88%、90%、92%、94%、96%或98%。在一些实施例中,本说明书提供的筛选模型在检测结直肠肿瘤或预测结直肠肿瘤患病风险中的特异性可以大于90%、92%、94%、96%、98%或99%。
计算设备可通过多种方式实现流程500的执行。在一些实施例中,处理设备110可以调用存储在存储设备120中的训练样本集,并基于预设的截止值选取方式确定DNA甲基化位点的甲基化阈值。在一些实施例中,处理设备110可以基于用户修改截止策略的指令,调用存储在存储设备120中的训练样本集重新确定DNA甲基化位点的甲基化阈值。在一些实施例中,处理设备110可以基于用户指令更新存储在存储设备120中的筛查模型,并使用更新的筛查模型获得评估结果。其中,处理设备110可以实时或定期地通过网络130从公开或非公开的数据库收集结直肠癌群体和正常群体的关联DNA甲基化位点组合的甲基化水平相关信息,用于存储设备120中的训练样本集,并使用更新的训练样本集优化DNA甲基化位点的甲基化阈值。
应当注意的是,上述有关流程500的描述仅仅是为了示例和说明,而不限定本说明书的适用范围。对于本领域技术人员来说,在本说明书的指导下可以对流程500进行各种修正和改变。然而,这些修正和改变仍在本说明书的范围之内。
根据本说明书的又一方面,提供了一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的装置,装置可以包括存储器、处理器以及存储在存储器上并可在处理器上运行的计算机程序,处理器执行程序时可实现获取受试者生物样本中DNA甲基化位点组合的甲基化水平;基于DNA甲基化位点组合的甲基化水平,使用筛查模型评估受试者是否可能患有结直肠肿瘤或受试者发展结直肠肿瘤的风险。
关于检测结直肠肿瘤或预测结直肠肿瘤患病风险的方法的更多内容可以参见图4、图5及其相关描述。
根据本说明书的又一方面,提供了一种检测试剂,其用于检测DNA甲基化位点组合。该DNA甲基化位点组合可作为检测结直肠肿瘤的生物标志物,包括NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71中的一个或多个位点。
在一些实施例中,检测试剂包括用于扩增DNA甲基化位点组合的引物组,该引物组用于获得包含DNA甲基化位点组合的特异性扩增片段,并放大检测信息。
在一些实施例中,用于扩增DNA甲基化位点组合的引物组包括用于扩增NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71中的一个或多个位点的引物对。
在一些较优的实施例中,用于扩增DNA甲基化位点组合的引物组包括用于扩增NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71中的全部位点的引物对。
在一些实施例中,用于扩增NDRG4_40的引物对如SEQ ID NO:1和SEQ ID NO:2所示。在另一些实施例中,用于扩增NDRG4_40的引物对的序列分别与SEQ ID NO:1和SEQ IDNO:2所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,用于扩增THBD_102的引物对如SEQ ID NO:3和SEQ ID NO:4所示。在另一些实施例中,用于扩增THBD_102的引物对的序列分别与SEQ ID NO:3和SEQ IDNO:4所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,用于扩增WIF1_68的引物对如SEQ ID NO:5和SEQ ID NO:6所示。在另一些实施例中,用于扩增WIF1_68的引物对的序列分别与SEQ ID NO:5和SEQ ID NO:6所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,用于扩增SDC2-2_56的引物对如SEQ ID NO:7和SEQ ID NO:8所示。在另一些实施例中,用于扩增SDC2-2_56的引物对的序列分别与SEQ ID NO:7和SEQ IDNO:8所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,用于扩增DNAAF9_41的引物对如SEQ ID NO:9和SEQ ID NO:10所示。在另一些实施例中,用于扩增DNAAF9_41的引物对的序列分别与SEQ ID NO:9和SEQ IDNO:10所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,用于扩增LIFR_42的引物对如SEQ ID NO:11和SEQ ID NO:12所示。在另一些实施例中,用于扩增LIFR_42的引物对的序列分别与SEQ ID NO:11和SEQ IDNO:12所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,用于扩增ZNF304_71的引物对如SEQ ID NO:13和SEQ ID NO:14所示。在另一些实施例中,用于扩增ZNF304_71的引物对的序列分别与SEQ ID NO:13和SEQID NO:14所示序列具有至少95%、96%、97%、98%或99%的相似度。
在一些实施例中,检测试剂还包括检测DNA甲基化位点组合的甲基化水平的其他试剂。其他试剂可以包括选自以下方法中的一种或多种所用的试剂:全基因组重亚硫酸盐测序法(WGBS)、简化基因组重亚硫酸盐测序法(RRBS)、氧化-重亚硫酸盐测序法(oxBS-seq)、甲基化DNA捕捉测序法(MethylCap-seq)、甲基结合蛋白测序法(MBD-seq)、甲基化DNA免疫共沉淀测序法(MeDIP-seq)、高效液相色谱法(HPLC)、甲基化敏感的限制性指纹法(MSRF)、甲基化敏感扩增多态性法(MASP)、甲基化芯片法(如805k芯片)、焦磷酸测序法、数字PCR法(dPCR)和甲基化特异PCR法(MS-PCR)。在一些较优的实施例中,其他试剂可以为WGBS或RRBS所用的试剂。
根据本说明书的又一方面,提供了一种用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的试剂盒,试剂盒包含本说明书一些实施例所示的DNA甲基化位点组合的检测试剂。
根据本说明书的又一方面,提供了一种检测试剂在制备用于检测结直肠肿瘤或预测结直肠肿瘤患病风险的试剂盒中的用途,该检测试剂用于检测本说明书一些实施例所示的DNA甲基化位点组合的甲基化水平。
下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂公司购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
实施例
方法与步骤
收集用于DNA甲基化检测分析的灌洗液样本组
采集94名受试者的结直肠灌洗液样本作为训练样本集,具体的包括结直肠肿瘤组(共81名结直肠肿瘤患者)和正常对照组(共13名健康的正常人),其中结直肠肿瘤组进一步包括结直肠癌组(共56名结直肠癌患者,包括I期患者15名、II期患者20名、III期患者15名和IV期患者6名)和结肠腺瘤组(共25名结肠腺瘤患者)。样本采集后均保存于50mL灌洗液DNA保存管中,保存管内含7.5mL添加剂,以4000rpm离心10min,弃上清,用1×PBS洗涤沉淀。
灌洗液样本组的DNA提取
对于灌洗液样本组的DNA提取,向上述灌洗液沉淀中加入180μL Buffer GTL,重悬沉淀;再加入20μL蛋白酶K,涡旋震荡混匀。于56℃孵育1h,直到样品完全溶解,继续于90℃孵育1h。短暂离心,使管壁上的溶液收集到管底。管中加入200μL Buffer GL,涡旋震荡彻底混匀。加入200μL无水乙醇,涡旋震荡彻底混匀。短暂离心,使管壁上的溶液收集到管底。
将管中溶液转移至已放置硅基质材料膜的离心管中,向硅基质材料膜上加入500μL已加入无水乙醇的Buffer GW1,12000rpm离心1min,倒掉收集管中的废液,将硅基质材料膜重新放回收集管中。向硅基质材料膜上加入500μL已加入无水乙醇的Buffer GW2,12000rpm离心1min,倒掉收集管中的废液,将硅基质材料膜重新放回收集管中。12000rpm离心2min,倒掉收集管中的废液,将硅基质材料膜置于室温数分钟以彻底晾干。
将硅基质材料膜转移至新离心管中,加入50~200μL Buffer GE,室温放置2~5min,12000rpm离心1min,收集DNA溶液,-20℃保存以备进一步使用。使用微量分光光度计Nano-300和Qubit测定DNA浓度(浓度应不小于1ng/μL)。
灌洗液样本组的DNA甲基化转化
对灌洗液样本组进行亚硫酸盐转化处理:在PCR管中加入50μL灌洗液沉淀DNA样本、150μLBisulfite Mix和25μL MBuffer B-保护液;短暂离心后,将PCR管置于PCR仪上,85℃恒温孵育50min后冷却至室温,短暂离心。其中,灌洗液沉淀DNA样本取自前述DNA溶液,50μL灌洗液沉淀DNA样本中DNA含量为20~1000ng。Bisulfite Mix的配制包括向内含亚硫酸氢钠的干粉管中加入1.2mL MBuffer A-转化液,震荡混匀直至干粉完全溶解。
亚硫酸盐处理后的DNA纯化处理:PCR管中溶液全部导入1.5mL离心管中。离心管中加入285μLMBuffer C-结合液、115μL异丙醇、10μL磁珠悬液(使用前充分混匀),震荡10min。短暂离心后放入磁力架上吸附2min,弃上清。离心管中加入1000μL MBuffer D-洗涤液,勿离开磁力架,孵育30s,弃上清。离心管中加入1000μL MBuffer E-孵育液,室温孵育15min,短暂离心后放入磁力架上吸附2min,弃上清。离心管中加入1000μL MBuffer D-洗涤液,勿离开磁力架,孵育30s,弃上清,本步骤重复操作一次。将离心管中多余的洗涤液吸干净后,置于超净工作台,吹干5min。
对于灌洗液样本组的DNA纯化与回收:向离心管中加入50μL MBuffer F-洗脱液,56℃温润,有助于提高洗脱效率,漩涡震荡使其充分混匀,静候5min。短暂离心,放于磁力架上吸附2min。吸取上清于干净的新离心管中,收集DNA溶液作为DNA转化样本,于-20℃保存以备进一步使用。
多重PCR-NGS检测
第一轮PCR,使用如表1所示的结直肠肿瘤甲基化特异性引物对94个DNA转化样本进行PCR反应。
表1.结直肠肿瘤甲基化特异性引物
/>
第一轮PCR的反应体系包括:10×ACE buffer,3μL;dNTP Mix(10mM),1μL;Primer混合引物,5μL;TMAC 600mm,2.5μL;50%甘油,6μL;5×Enhancer,2μL;灭菌水,5μL;Ace Taq酶,0.5μL;DNA转化样本(即亚硫酸盐处理后的DNA),5μL。
第一轮PCR的反应条件为:1)循环数1:95℃5min;2)循环数35:95℃30s,50℃1min,72℃30s;3)循环数1:72℃5min。
第二轮PCR的反应体系包括:10×ACE buffer,3μL;dNTP Mix(10mM),1μL;引物AP5(5μM),2μL;引物Index(5μM),2μL;50%甘油,6μL;灭菌水,10.5μL;AceTaq酶,0.5μL;第一轮PCR反应产物,5μL。其中:引物AP5的序列为AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT(SEQ ID NO:31);引物index的序列为CAAGCAGAAGACGGCATACGAGATNNNNNNNNGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT(SEQ ID NO:32)。需要说明的是,N为A、T、C或G,“NNNNNNNN”代表用于区分不同样本的索引index。
第二轮PCR的反应条件为:1)循环数1:95℃10min;2)循环数20:95℃30s,55℃30s,72℃30s;3)循环数1:72℃5min。
扩增产物经核酸纯化试剂纯化后得到测序文库,然后使用测序试剂Miniseq
数据处理
基于NGS测序结果,使用如前文式(1)所示的公式计算94个结直肠灌洗液样本中174个DNA甲基化位点的甲基化率。
实例1:结直肠肿瘤组和正常对照组的甲基化率的相关数据分析揭示差异甲基化位点的甲基化水平在结直肠肿瘤患者中显著改变
对94个结直肠灌洗液样本中的174个DNA甲基化位点的甲基化率进行检验分析。采用F检验验证各甲基化位点的甲基化率在结直肠肿瘤组与正常对照组之间的分布是否具备方差齐性。对于甲基化率在结直肠肿瘤组与正常对照组之间属于方差齐性分布的甲基化位点,采用独立样本双尾学生t检验验证结直肠肿瘤组与正常对照组间各位点的甲基化率平均值是否存在显著性差异。对于甲基化率在结直肠肿瘤组与正常对照组之间属于方差不齐性分布的甲基化位点,采用独立样本双尾学生t’检验验证结直肠肿瘤组与正常对照组间各位点的甲基化率平均值是否存在显著性差异。以P<0.001,组间差异倍数大于2倍作为评价显著性的标准,从174个甲基化位点中选取17点作为差异甲基化位点,分别为NDRG4_40、SDC2_101、SEP9-1_69、SEP9-2_61、SFRP1_40、TFPI2_117、THBD_102、BCAT1_38、WIF1_68、WIF1_88、WNT5A_47、TFPI2-2_91、SDC2-2_56、SDC2-2_73、DNAAF9_41、LIFR_42和ZNF304_71。
实例2:差异甲基化位点及其组合在结直肠肿瘤预测中的应用分析
使用ROC曲线分析单个差异甲基化位点预测结直肠肿瘤的能力。具体的,以各差异甲基化位点在结直肠肿瘤组(例如,标记为1)中的甲基化率和在正常对照组(例如,标记为0)中的甲基化率制作ROC曲线。其中,位点NDRG4_40对应的AUC值为0.771(如图6所示),位点SDC2_101对应的AUC值为0.724(如图7所示),位点SEP9-1_69对应的AUC值为0.837(如图8所示),位点SEP9-2_61对应的AUC值为0.782(如图9所示),位点SFRP1_40对应的AUC值为0.791(如图10所示),位点TFPI2_117对应的AUC值为0.776(如图11所示),位点THBD_102对应的AUC值为0.751(如图12所示),位点BCAT1_38对应的AUC值为0.639(如图13所示),位点WIF1_68对应的AUC值为0.729(如图14所示),位点WIF1_88对应的AUC值为0.757(如图15所示),位点WNT5A_47对应的AUC值为0.758(如图16所示),位点TFPI2-2_91对应的AUC值为0.827(如图17所示),位点SDC2-2_56对应的AUC值为0.880(如图18所示),位点SDC2-2_73对应的AUC值为0.878(如图19所示),位点DNAAF9_41对应的AUC值为0.908(如图20所示),位点LIFR_42对应的AUC值为0.915(如图21所示),位点ZNF304_71对应的AUC值为0.886(如图22所示)。
针对单个差异甲基化位点,考虑选取合适的甲基化阈值,基于该差异甲基化位点的甲基化阈值对训练样本集中的正常人和结直肠肿瘤患者进行区分。按照特异性为100%时的甲基化率设定差异甲基化位点的甲基化阈值。选取的阈值如下:位点NDRG4_40的阈值为0.1875,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为30.4%;位点SDC2_101的阈值为0.7297,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为28.6%;位点SEP9-1_69的阈值为0.3713,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为51.8%;位点SEP9-2_61的阈值为0.4613,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为26.8%;位点SFRP1_40的阈值为0.5920,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为19.6%;位点TFPI2_117的阈值为0.7110,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为12.5%;位点THBD_102的阈值为0.1094,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为46.4%;位点BCAT1_38的阈值为0.3462,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为21.4%;位点WIF1_68的阈值为0.2983,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为39.3%;位点WIF1_88的阈值为0.2726,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为42.9%;位点WNT5A_47的阈值为0.0678,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为21.4%;位点TFPI2-2_91的阈值为0.2144,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为55.4%;位点SDC2-2_56的阈值为0.0566,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为69.6%;位点SDC2-2_73的阈值为0.0582,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为66.1%;位点DNAAF9_41的阈值为0.0172,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为50.0%;位点LIFR_42的阈值为0.0407,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为62.5%;位点ZNF304_71的阈值为0.0959,使用前述阈值对训练样本集进行结直肠肿瘤预测的敏感度为53.6%。
在选取的17个差异甲基化位点的甲基化阈值的基础上,考虑以2~17个差异甲基化位点组合的方式建立筛查模型,以进一步提升敏感度。具体的,针对模型相关联的每个差异甲基化位点的甲基化率,如果样本中该位点的甲基化率不低于对应阈值,则该位点判定为阳性,如果样本中该位点的甲基化率低于对应阈值,则该位点判定为阴性;针对模型相关联的全部差异甲基化位点,若其中一个或以上的差异甲基化位点被判定为阳性位点,则预测该样本对应受试者可能患有结直肠肿瘤或存在发展结直肠肿瘤的风险,反之则可排除可能性或风险。
以2~17个位点组合的方式建立筛查模型,基于训练样本集中的结直肠癌组样本和正常对照组样本,使用上述筛查模型进行结直肠癌预测。表2示出了2~17个位点组合建立的各筛查模型在预测结直肠癌中的敏感度范围,以及敏感度最大和敏感度最小对应的位点组合。
表2.2~17位点组合对应筛查模型在预测结直肠癌中的敏感度范围
/>
/>
由表2可以看出,在筛查模型预测结直肠癌的相关分析结果中,随着组合的位点数量增加,模型的敏感度范围的最小值呈增长趋势,模型的敏感度范围的最大值在组合的位点数量增加至5时达到峰值。特别地,NDRG4_40、WIF1_68、SDC2.2_56、DNAAF9_41和ZNF304_71的5位点组合建立的筛查模型1在使用位点数最少的情况下即可达到94.64%的敏感度。
以2~17个位点组合的方式建立筛查模型,基于训练样本集中的结肠腺瘤组样本和正常对照组样本,使用上述筛查模型进行结肠腺瘤预测。表3示出了2~17个位点组合建立的各筛查模型在预测结肠腺瘤中的敏感度范围,以及敏感度最大和敏感度最小对应的位点组合。
表3.2~17位点组合对应筛查模型在预测结肠腺瘤中的敏感度范围
/>
/>
由表3可以看出,在筛查模型预测结肠腺瘤的相关分析结果中,随着组合的位点数量增加,模型的敏感度范围的最小值呈增长趋势,模型的敏感度范围的最大值在组合的位点数量增加至5时达到峰值。特别地,NDRG4_40、THBD_102、SDC2.2_56、DNAAF9_41和LIFR_42的5位点组合建立的筛查模型2在使用位点数最少的情况下即可达到91.3%的敏感度。
考虑将筛查模型1关联的位点与筛查模型2关联的位点进行组合并建立新的筛查模型,以进一步提升模型预测的全面性。具体的,选取NDRG4_40、THBD_102、WIF1_68、SDC2-2_56、DNAAF9_41、LIFR_42和ZNF304_71作为目标甲基化位点,建立基于7位点组合的筛查模型3。
在对训练样本集中结直肠肿瘤的预测中,筛查模型3的总体敏感度为93.8%,总体特异性为100%。
基于训练样本集中的结直肠癌组样本和正常对照组样本,使用筛查模型3进行结直肠癌预测。结果显示,筛查模型3从56个结直肠癌样本中鉴别出53个阳性样本,敏感度为94.6%,特异性为100%。其中,从15个I期结直肠癌样本中鉴别出13个阳性样本,敏感度为86.67%;从20个II期结直肠癌样本中鉴别出19个阳性样本,敏感度为95%;从15个III期结直肠癌样本中鉴别出15个阳性样本,敏感度为100%;从6个IV期结直肠癌患者样本中鉴别出6个阳性样本,敏感度为100%。
基于训练样本集中的结肠腺瘤组样本和正常对照组样本,使用筛查模型3进行结肠腺瘤预测。结果显示,筛查模型3从25个结肠腺瘤样本中鉴别出23个阳性样本,敏感度为92%,特异性为100%。
综上所述,本说明书实施例的DNA甲基化位点组合及对应筛查模型在对结直肠肿瘤的预测中具备良好的敏感度和特异性,可用于实现结直肠肿瘤的准确、快速、无创临床筛查或预测。
上文已对基本概念做了描述,显然,对于本领域技术人员来说,上述详细披露仅仅作为示例,而并不构成对本说明书的限定。虽然此处并没有明确说明,本领域技术人员可能会对本说明书进行各种修改、改进和修正。该类修改、改进和修正在本说明书中被建议,所以该类修改、改进、修正仍属于本说明书示范实施例的精神和范围。
同时,本说明书使用了特定词语来描述本说明书的实施例。如“一个实施例”、“一实施例”、和/或“一些实施例”意指与本说明书至少一个实施例相关的某一特征、结构或特点。因此,应强调并注意的是,本说明书中在不同位置两次或多次提及的“一实施例”或“一个实施例”或“一个替代性实施例”并不一定是指同一实施例。此外,本说明书的一个或多个实施例中的某些特征、结构或特点可以进行适当的组合。
一些实施例中使用了描述成分、属性数量的数字,应当理解的是,此类用于实施例描述的数字,在一些示例中使用了修饰词“大约”、“近似”或“大体上”来修饰。除非另外说明,“大约”、“近似”或“大体上”表明所述数字允许有±20%的变化。相应地,在一些实施例中,说明书和权利要求中使用的数值参数均为近似值,该近似值根据个别实施例所需特点可以发生改变。在一些实施例中,数值参数应考虑规定的有效数位并采用一般位数保留的方法。尽管本说明书一些实施例中用于确认其范围广度的数值域和参数为近似值,在具体实施例中,此类数值的设定在可行范围内尽可能精确。
针对本说明书引用的每个专利、专利申请、专利申请公开物和其他材料,如文章、书籍、说明书、出版物、文档等,特此将其全部内容并入本说明书作为参考。与本说明书内容不一致或产生冲突的申请历史文件除外,对本说明书权利要求最广范围有限制的文件(当前或之后附加于本说明书中的)也除外。需要说明的是,如果本说明书附属材料中的描述、定义、和/或术语的使用与本说明书所述内容有不一致或冲突的地方,以本说明书的描述、定义和/或术语的使用为准。
最后,应当理解的是,本说明书中所述实施例仅用以说明本说明书实施例的原则。其他的变形也可能属于本说明书的范围。因此,作为示例而非限制,本说明书实施例的替代配置可视为与本说明书的教导一致。相应地,本说明书的实施例不仅限于本说明书明确介绍和描述的实施例。
