长牡蛎Cgsh1p基因表达量变化在预测壬基酚污染方面的应用
文献发布时间:2024-01-17 01:16:56
技术领域
本发明属于基因技术领域,具体涉及长牡蛎Cgsh1p基因表达量变化在预测壬基酚污染方面的应用。
背景技术
长牡蛎(Crassostrea gigas),也叫太平洋牡蛎,属于瓣鳃纲(Bivalvia),珍珠贝目(Pterioida),牡蛎科,是冠轮动物(Lophotrochozoa)中软体动物门(Mollusca)的代表物种。其肉质肥美,具有极高的营养及经济价值,是我国乃至世界上重要的水产养殖品种。
壬基酚(Nonylphenol,NP)是一种类性激素的内分泌干扰物。有报道显示,壬基酚在我国近海养殖水体中广泛存在,且在生物体内具有蓄积性,不易降解。一方面,壬基酚可以通过与激素受体相互作用来引起水生生物的内分泌紊乱,影响其健康状态及养殖产业的发展;另一方面,壬基酚的积累可通过食物链传递,最终危害人体健康。
传统的壬基酚检测方法一般使用高效液相色谱-质谱联用的办法,但该方法存在样品处理繁琐,检测仪器要求高,且对操作人员的要求较高。因此开发一种快速、简便、有效的指示壬基酚污染的办法势在必行。
利用荧光定量PCR(qRT-PCR)检测基因的表达具有成本低,操作简便,且灵敏度高的优势。壬基酚对水生生物的刺激必然会导致相关基因的表达变化,因此选取合适的基因,以其表达的变化指示环境激素壬基酚的污染是一种行之有效的方法。
发明内容
本发明的目的之一在于提供一种长牡蛎Cgsh1p基因表达量变化在预测壬基酚污染方面的应用。
本发明的目的之二在于提供一种预测壬基酚污染的方法,通过Cgsh1p基因显著的表达变化指示壬基酚的污染。
为了实现上述目的,本发明的技术方案概述如下:
本发明找到一个长牡蛎Cgsh1p基因,该基因在有壬基酚刺激时表达发生显著变化(表达量变化大),因此能够作为长牡蛎受到壬基酚污染的指示基因。所述Cgsh1p基因的核苷酸序列如SEQ ID NO.1所示,基因编号XM_034480938.1。
所述Cgsh1p基因的特异性引物对为:
Cgsh1p-F:5′-CAGGAAAGAGGAGAACTGCCGT-3′;
Cgsh1p-R:5′-TTTAGTGAATCAACCCGAACCG-3′。
另外,本发明还公开了一种预测壬基酚污染的方法,所述方法包括如下步骤:
(1)选择该水域内生长的长牡蛎,提取待测长牡蛎样品鳃组织的总RNA,并反转为cDNA;
(2)以步骤(1)中所合成cDNA为模板,利用权利要求2所述的引物对,进行荧光定量PCR扩增;获得Cgsh1p基因的表达量;
(3)如果Cgsh1p基因表达量升高,指示该水域内有壬基酚污染风险。
具体地,所述荧光定量PCR反应程序为:①95℃预变性30sec;②95℃,5sec,③60℃,30sec,②③步进行40次循环。
更加具体地,所述方法为:
a.候选指示基因序列的获得:基于高通量转录组(RNA-seq)测序结果,我们筛选出长牡蛎在壬基酚刺激后,表达量发生极显著升高的Cgsh1p基因作为指示壬基酚污染的基因,并进一步使用荧光定量PCR验证该基因在壬基酚应激期间的表达量变化。
b.引物设计:根据获得的候选基因序列设计该基因的特异扩增引物(熔解曲线验证PCR产物峰单一,琼脂糖胶检测条带单一)。
c.PCR模板的准备:收集样品后,用TRIzol(试剂,市购于TaKaRa公司)提取总RNA,用Evo M-MLV反转录试剂盒(含去除gDNA试剂,用于qPCR)(试剂盒,市购于艾科瑞公司),按照说明书反转成双链cDNA。作为荧光定量PCR的模板。
d.荧光定量PCR反应:以反转的cDNA为模版,用自行设计Cgsh1p基因特异引物在ABI 7500real-time PCR仪中进行扩增。获得该基因的表达量,验证该基因是否发生明显的表达变化,通过Cgsh1p基因表达量的变化,预测壬基酚污染的可能性,为改善养殖水质奠定基础。
本发明的优点:
1.本发明首次利用高通量转录组RNA-seq技术筛选壬基酚刺激后,表达量发生极显著变化的Cgsh1p基因,数据真实可靠。
2.筛选出的长牡蛎Cgsh1p基因适用于指征海区壬基酚污染。如果长牡蛎Cgsh1p基因发生明显的表达量升高,提示该区域可能有壬基酚污染,能够及时对其进行有效的预测。其应用较为普遍,操作相对简单,成本较低,灵敏度高。
3.本研究筛选出的长牡蛎Cgsh1p基因是经过转录组RNA-seq高通量筛选,并进一步的使用荧光定量PCR技术进行验证,是海区壬基酚污染的有效指示基因。
附图说明
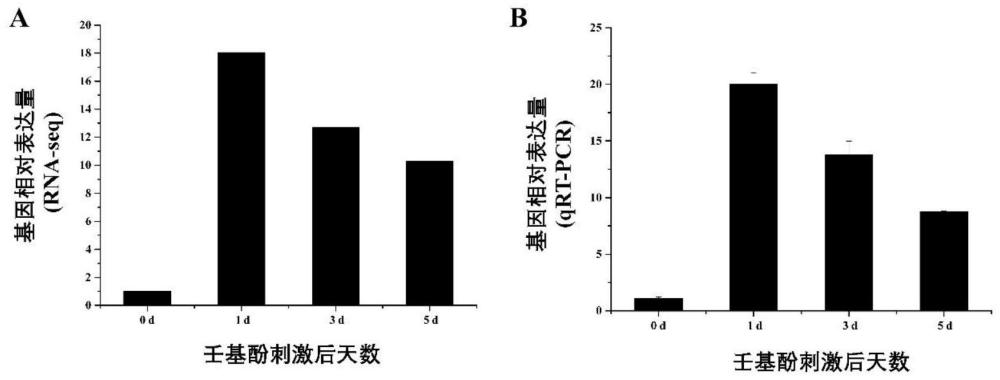
图1是转录组RNA-seq技术及荧光定量PCR技术验证壬基酚应激后长牡蛎Cgsh1p基因的表达量变化。
具体实施方式
下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但下述实施例中所涉及的具体实验方法如无特殊说明,均为常规方法或按照制造厂商说明书建议的条件实施。
若未特别指明,实施例中所用技术手段为本领域技术人员所熟知的常规手段。下述实施例中的试验方法,如无特别说明,均为常规方法。如无特殊说明,所采用的试剂及材料,均可以从市场中购买获得。
除非另行定义,文中所使用的所有专业与科学用语与本领域熟练人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法及材料皆可应用于本发明中。文中所述的较佳实施方法与材料仅作示范之用。
实施例1
候选壬基酚污染指示基因的选择:基于长牡蛎壬基酚刺激后的高通量转录组数据,我们选取长牡蛎在壬基酚刺激后表达量发生极显著变化的Cgsh1p基因,转录组数据显示,该基因在壬基酚刺激后的表达量变化如图1A所示,所选取的Cgsh1p基因序列如SEQ IDNO.1所示。
经过荧光定量PCR验证候选指示基因表达变化:根据候选基因序列设计出特异的引物对。Cgsh1p基因的特异性引物为:
Cgsh1p-F:5′-CAGGAAAGAGGAGAACTGCCGT-3′;
Cgsh1p-R:5′-TTTAGTGAATCAACCCGAACCG-3′;
在壬基酚应激样品中进行荧光定量PCR,获得该基因在壬基酚应激后1天,3天及5天的的表达量。其具体实施方案如下:
(1)实验材料:实验用健康长牡蛎(平均壳高80mm)采集于烟台当地海区。实验前在充气过滤海水中暂养一周,水温19℃±1℃。壬基酚应激实验:取30只牡蛎暴露于20μg/L的含壬基酚的海水中,在壬基酚暴露后0天(对照),1天,3天和5天后分别取样。取样时,随机抽取5只长牡蛎,选取鳃组织进行取样。样品经液氮速冻后放于-80℃长时间保存。
(2)RNA提取:使用TRIzol试剂,按照说明书的步骤提取总RNA。
(3)第一链cDNA的合成:用Evo M-MLV反转录试剂盒(含去除gDNA试剂,用于qPCR)(试剂盒,市购于艾科瑞公司)。依照说明书将上步提取的总RNA反转为cDNA模板。
1)反转前需去除基因组DNA。按表1成分在冰上配制混合液。
表1反转前基因组DNA去除体系
PCR仪42℃反应2min,然后放置于4℃。
2)反转录cDNA反应。按表2成分于冰上配制20μl的反应体系:
表2反转录cDNA体系
在PCR仪中37℃反应30min,之后85℃反应5sec(灭活酶),4℃保存反转好的cDNA。需长期保存时,放于-20℃。
(4)引物设计:根据所选指示基因全长序列,使用引物设计软件Primer Premier5,设计出荧光定量实验所用的特异性引物。设计好后交由上海生工公司合成。
(5)定量PCR反应:在ABI 7500 real-time PCR仪(荧光定量PCR仪,市购于ABI公司)中进行。每个定量PCR反应需进行3个重复。反应结束后根据该基因的CT值计算出该基因的表达量。按表3含量在冰上配制定量PCR反应体系。
表3荧光定量PCR体系
定量PCR反应程序为:①95℃预变性30sec;②95℃,5sec,③60℃,30sec,②③步需要进行40次循环;反应完成以后,确认扩增曲线及熔解曲线。一方面通过观察熔解曲线为单一峰判断无非特异扩增;另一方面,吸取2μl PCR产物,在3%的琼脂糖胶中进行电泳,之后观察到PCR产物条带单一,从而进一步确定扩增产物特异且单一。
(6)根据上步中获得的CT值,通过使用2
实施例2一种预测壬基酚污染的方法
对于长牡蛎生长区域,我们想知道该区域内是否存在壬基酚污染,具体的方法为:
(1)选择该水域内生长的长牡蛎,提取待测长牡蛎样品鳃组织的总RNA,并反转为cDNA;
(2)以步骤(1)中合成的cDNA为模板,利用如下引物对进行荧光定量PCR扩增;所述PCR反应程序为:①95℃预变性30sec;②95℃,5sec,③60℃,30sec,②③步进行40次循环。获得Cgsh1p基因的表达量;
Cgsh1p-F:5′-CAGGAAAGAGGAGAACTGCCGT-3′;
Cgsh1p-R:5′-TTTAGTGAATCAACCCGAACCG-3′;
(3)如果Cgsh1p基因表达量发生显著升高(差异显著性**p<0.01),指示该水域内有壬基酚污染风险。如果想继续确认该预测,可以采用现有的比较成熟的高效液相色谱-质谱联用的办法对其进行验证。这种方法操作简单,为改善养殖水质奠定基础。
以上所述之实施例,只是本发明的较佳实施例而已,仅仅用以解释本发明,并非限制本发明实施范围,对于本技术领域的技术人员来说,当然可根据本说明书中所公开的技术内容,通过置换或改变的方式轻易做出其它的实施方式,故凡在本发明的原理上所作的变化和改进等,均应包括于本发明申请专利范围内。
