用于靶向富集病原的探针组、方法以及试剂盒
文献发布时间:2024-04-18 19:58:21
技术领域
本发明属于分子生物学和医学领域,特别是涉及一种NGS靶向捕获法检测临床常见病原菌的试剂盒及方法。
背景技术
感染性疾病的病原体种类繁多,临床表现复杂,快速、全面、准确地鉴别病原体,可以为疾病的诊断、治疗及流行病调查提供有效的依据。
传统的病原菌分离培养及表型鉴定方法检测周期耗时长,且操作繁琐、敏感性低、特异性差。研究表明,约70%的感染患者因传统检测方法无法确定病原体,未能得到及时有效的治疗,预后较差。而分子诊断技术可有效弥补传统方法的不足,对指导临床预防、诊断、治疗及疗效评价起到重要作用。
核酸检测技术已成病原体检测的主流发展趋势,摆脱了传统方法对于病原体分离培养的依赖,显著提高了病原检测的敏感性,缩短检测时间。基于多重PCR技术开展的多重病原体检测体系在疑似病毒感染患者的病原体筛查和确认中具有较高的临床价值。此外,PCR技术在检测病原菌耐药也发挥了重要作用。由于PCR技术的敏感度高,极微量的靶基因污染也会造成结果判断失误,所以假阳性问题较为突出。宏基因组二代测序技术(mNGS)通过对临床样本中微生物核酸的测序分析,无偏倚地检测多种病原微生物,逐渐应用于临床感染性疾病病原诊断,在鉴定疑难、罕见感染病原及非典型病原体等常规检测技术鉴定难度大的病原体具有重要的临床价值。而宏基因组测序技术面临缺乏标准流程、报告解读、价格昂贵等问题。
病原靶向测序(tNGS)通过超多重PCR扩增与高通量测序两种技术结合,能够对待测样本中的几十种至几百种已知病原微生物及其毒力和/或耐药基因进行检测。与mNGS相比,tNGS具有病原谱范围明确,其灵敏度与病原载量相关,不受人源含量影响,对数据量要求不高,成本较低,故在临床检验受到越倒越多的关注。
发明内容
本发明的目的是提供一种临床常见病原菌检测的捕获探针、试剂盒及方法。通过针对临床常见的26种病原菌基因组序列设计特异性的捕获探针,用于临床标本中26种病原菌检测,该方法具有特异性强,灵敏度高,成本低等优点。
本发明首先提供了一种靶向探针捕获法检测26种病原菌的探针组,能够与常见病原菌的遗传物质特异性结合,包括有用于扩增如下病原菌的探针:
产单核细胞李斯特菌,探针数20条,核苷酸序列如SEQ ID NO. 1-20;
EB病毒,探针数11条,核苷酸序列如SEQ ID NO. 21-31;
水痘带状疱疹病毒,探针数10条,核苷酸序列如SEQ ID NO. 32-41;
人巨细胞病毒,探针数12条,核苷酸序列如SEQ ID NO. 42-53;
脑膜炎奈瑟菌,探针数19条,核苷酸序列如SEQ ID NO. 54-72;
肺炎克雷伯菌,探针数23条,核苷酸序列如SEQ ID NO. 73-95;
流感嗜血杆菌,探针数19条,核苷酸序列如SEQ ID NO. 96-114;
金黄色葡萄球菌,探针数20条,核苷酸序列如SEQ ID NO. 115-134;
无乳链球菌,探针数19条,核苷酸序列如SEQ ID NO. 135-153;
肺炎链球菌,探针数19条,核苷酸序列如SEQ ID NO. 154-172;
屎肠球菌,探针数20条,核苷酸序列如SEQ ID NO. 173-192;
结核分枝杆菌复合群,探针数22条,核苷酸序列如SEQ ID NO. 193-214;
铜绿假单胞菌,探针数24条,核苷酸序列如SEQ ID NO. 215-238;
鲍曼不动杆菌,探针数22条,核苷酸序列如SEQ ID NO. 239-260;
大肠埃希菌,探针数18条,核苷酸序列如SEQ ID NO. 261-278;
粪肠球菌,探针数21条,核苷酸序列如SEQ ID NO. 279-299;
黄曲霉,探针数32条,核苷酸序列如SEQ ID NO. 300-331;
新型隐球菌,探针数28条,核苷酸序列如SEQ ID NO. 332-359;
白色念珠菌,探针数27条,核苷酸序列如SEQ ID NO. 360-386;
近平滑念珠菌,探针数27条,核苷酸序列如SEQ ID NO. 387-413;
格特隐球菌,探针数28条,核苷酸序列如SEQ ID NO. 414-441;
烟曲霉,探针数33条,核苷酸序列如SEQ ID NO. 442-474;
人类疱疹病毒1,探针数11条,核苷酸序列如SEQ ID NO. 475-485;
人类疱疹病毒2,探针数11条,核苷酸序列如SEQ ID NO. 486-496;
人类疱疹病毒6A,探针数10条,核苷酸序列如SEQ ID NO. 497-506;
人类疱疹病毒6B,探针数10条,核苷酸序列如SEQ ID NO. 507-516。
上述的靶向探针捕获法检测26种病原菌的探针组在制备病原菌检测试剂盒中的应用。
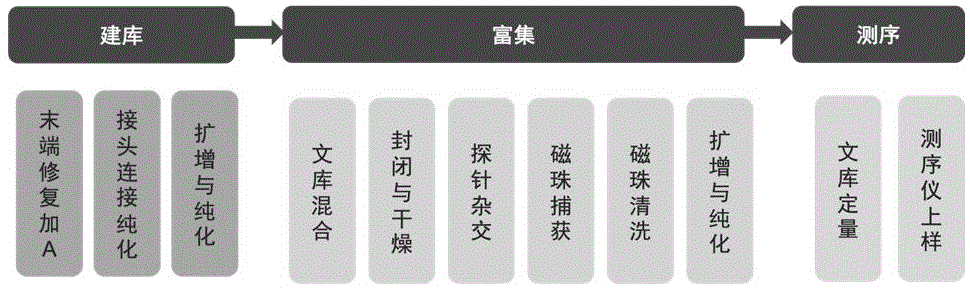
所述的应用中还包括如下步骤:1)待测样本DNA提取;2)文库构建;3)探针富集;4)上机测序。
所述的步骤2)中的步骤包括:对DNA进行片段化处理,再进行末端修复,3'端加上“A”尾,片段两端再加上文库接头,然后进行PCR扩增。
DNA进行片段化处理、末端修复或者3'端加上“A”尾的步骤中,在PCR仪中的反应程序为:4℃1分钟;20℃ 20分钟,72℃ 20分钟。
PCR扩增步骤中,在PCR仪中的反应程序为98℃ 1分钟;98℃ 15秒,60℃ 15秒,72℃ 30秒,共8个循环;72℃10分钟。
所述的步骤3)中的探针富集中的探针杂交过程的PCR仪程序是:95℃ 10分钟;从95℃ 开始梯度降温,每个循环中3分钟降3℃,共10个循环;65℃1小时。
杂交后文库通过与链霉亲和素磁珠结合捕获,磁珠捕获文库进行清洗,获得捕获文库磁珠悬浮液,洗脱后进行PCR扩增,程序是:98℃ 10分钟;98℃ 15秒,60℃ 30秒,72℃1分钟,共10个循环;72℃ 5分钟。
本发明的有益效果是:基于NGS靶向探针捕获技术,针对26种常见的人体致病微生物,设计了能够与其特异性结合的探针组合,可以靶向捕获待测的病原菌的遗传物质,从而将其检测出来。通过临床样本测试,确定了其检测性能以及合适的探针用量。本发明的试剂盒特异性强,灵敏度高,成本低。
附图说明
图1为本发明的检测流程图。
图2为本发明的生信分析流程图。
图3为实施例1中不同探针用量中靶率比较。
具体实施方式
本专利中的检测方法主要面向以下的病原体:
产单核细胞李斯特菌,EB病毒,水痘带状疱疹病毒、人巨细胞病毒,脑膜炎奈瑟菌,肺炎克雷伯菌,流感嗜血杆菌,金黄色葡萄球菌,无乳链球菌,肺炎链球菌,屎肠球菌,结核分枝杆菌复合群,铜绿假单胞菌,鲍曼不动杆菌,大肠埃希菌,粪肠球菌,黄曲霉,新型隐球菌,白色念珠菌,近平滑念珠菌,格特隐球菌,烟曲霉,人类疱疹病毒1,人类疱疹病毒2,人类疱疹病毒6A,人类疱疹病毒6B。
针对上述的特定的病原体,本专利的液相杂交捕获法可以有针对性地将这些病原体的特征序列进行富集,一方面避免了人源细胞的干扰,另一方面也可以显著提高了检测灵敏度,具有了更强的特异性,避免了传统mNGS检测中存在的问题。
本专利的方法中,通过捕获法获得各个病原的特异性区域,在探针设计方法中,需要保证具有较高的特异性,不受其他物种的影响,同时探针之间也不应该存在相互干扰,我们按照以下设计原则,对目标病原体设计能够靶向捕获的探针序列:下载病原体物种的全部基因组序列,按照窗口滑动长度为80-120bp,筛选出候选探针序列,使用分析流程进行分析、鉴定及注释,确保其保守性>90%,特异性>95%。除此之外,还需要满足引物设计的基本原则:GC含量在30-60%之间;在进行设计过程中,还需要综合考虑到探针的容错性,以及探针相互之间不能结合,以及杂交过程反应的均一性,另外,还需要合理根据通常情况下的病原体的载量设计合理的探针覆盖数量,若通常病原体的载量偏小时,需要设定出合理的探针覆盖区域和覆盖量;如果载量较大时,也需要进行合理调整,使捕获得到的读段数量更加均一,也避免对其他的病原体的捕获和检测产生干扰。本专利的方法中,对于同一个病原微生物,设计的探针均为多条,这样能够确保覆盖到的物种基因组范围更大,提高检测灵敏度。
通过上述的设计探针思路后,经过大量测试,本专利得到的用于富集相应的病原的探针序列如下所示:
产单核细胞李斯特菌,探针数20条,核苷酸序列如SEQ ID NO. 1-20;
EB病毒,探针数11条,核苷酸序列如SEQ ID NO. 21-31;
水痘带状疱疹病毒,探针数10条,核苷酸序列如SEQ ID NO. 32-41;
人巨细胞病毒,探针数12条,核苷酸序列如SEQ ID NO. 42-53;
脑膜炎奈瑟菌,探针数19条,核苷酸序列如SEQ ID NO. 54-72;
肺炎克雷伯菌,探针数23条,核苷酸序列如SEQ ID NO. 73-95;
流感嗜血杆菌,探针数19条,核苷酸序列如SEQ ID NO. 96-114;
金黄色葡萄球菌,探针数20条,核苷酸序列如SEQ ID NO. 115-134;
无乳链球菌,探针数19条,核苷酸序列如SEQ ID NO. 135-153;
肺炎链球菌,探针数19条,核苷酸序列如SEQ ID NO. 154-172;
屎肠球菌,探针数20条,核苷酸序列如SEQ ID NO. 173-192;
结核分枝杆菌复合群,探针数22条,核苷酸序列如SEQ ID NO. 193-214;
铜绿假单胞菌,探针数24条,核苷酸序列如SEQ ID NO. 215-238;
鲍曼不动杆菌,探针数22条,核苷酸序列如SEQ ID NO. 239-260;
大肠埃希菌,探针数18条,核苷酸序列如SEQ ID NO. 261-278;
粪肠球菌,探针数21条,核苷酸序列如SEQ ID NO. 279-299;
黄曲霉,探针数32条,核苷酸序列如SEQ ID NO. 300-331;
新型隐球菌,探针数28条,核苷酸序列如SEQ ID NO. 332-359;
白色念珠菌,探针数27条,核苷酸序列如SEQ ID NO. 360-386;
近平滑念珠菌,探针数27条,核苷酸序列如SEQ ID NO. 387-413;
格特隐球菌,探针数28条,核苷酸序列如SEQ ID NO. 414-441;
烟曲霉,探针数33条,核苷酸序列如SEQ ID NO. 442-474;
人类疱疹病毒1,探针数11条,核苷酸序列如SEQ ID NO. 475-485;
人类疱疹病毒2,探针数11条,核苷酸序列如SEQ ID NO. 486-496;
人类疱疹病毒6A,探针数10条,核苷酸序列如SEQ ID NO. 497-506;
人类疱疹病毒6B,探针数10条,核苷酸序列如SEQ ID NO. 507-516。
本发明中的方法进行检测时,所采用的样本可以是指痰液、口腔分泌液、鼻腔分泌液、肺泡灌洗液、脑脊液等。在进行检测前,需要对样本进行DNA提取后进行后续的靶向富集和NGS检测。
具体的检测过程如下所示:
实施例1 合适的探针用量
1、待测样本准备:待测样本DNA浓度在0-5 ng/μL之间,可直接取25 μL进入后续反应;若浓度>5 ng/μL,需用无酶水将DNA稀释至0-5 ng/μL,至体积25μL。
2、文库构建
使用Hieff NGS OnePot II DNA Library Prep Kit for Illumina(YEASEN;12204ED96)建库。
取5μL Smerase mix加入25μL样本中,在PCR仪运行DNA片段化/末端修复/加A反应,其反应程序为4℃ 1分钟;20℃ 20分钟,72℃ 20分钟。结束后,配制连接反应体系,FastT4 DNA Ligase 2.5μL,Ligation Enhancer 15μL,Adapter 2.5μL,于PCR仪上运行20℃ 15分钟。连接产物磁珠纯化。取接头连接及纯化后产物10μL,加入2×Ultima AmplificationMix 12.5μL,Primer Mix 2.5μL,于PCR仪运行98℃ 1分钟;98℃ 15秒,60℃ 15秒,72℃ 30秒,8个循环;72℃ 10分钟。PCR产物磁珠纯化,收集纯化产物待用。
3、探针富集
使用xGen Lockdown Reagents及定制26种病原菌探针组合(IDT)进行探针富集。
10个样本,等质量进行文库混合,混合文库总量为1 μg。混合文库池中加入封闭混合液(终浓度为0.25mM),放入真空干燥机进行干燥,直至文库完全干燥。文库干粉中加入杂交溶液13μL,加入4μL探针杂交,探针用量分为0.1pmol、0.3pmol、1pmol 3个梯度。置于PCR仪中运行hyb-t程序,95℃ 10分钟;从95℃ 开始梯度降温,每个循环3分钟降3℃,10个循环,65℃1小时。杂交后文库通过与链霉亲和素磁珠结合捕获,使用清洗液对磁珠捕获文库进行清洗,获得捕获文库磁珠悬浮液。配制PCR反应体系,于PCR仪运行98℃ 10分钟;98℃15秒,60℃ 30秒,72℃ 1分钟,10个循环;72℃ 5分钟。PCR产物纯化测定其浓度后用于上机测序。
表1 xGen Lockdown Reagents试剂盒封闭液配制
表2 xGen Lockdown Reagents试剂盒杂交溶液配制
4、上机测序
富集后的文库进行定量,文库得量如下表3,上机测序。
表3 xGen Lockdown Reagents试剂盒杂交溶液配制
5、数据分析
数据分析流程见附图2。
探针用量会降低人源reads的占比,提升病原reads占比,提升病原序列中靶率(见图3),提升覆盖区域的测序深度与覆盖均一度。
表4 不同探针用量获得的病原菌reads数比较
实施例2 临床样本检测性能
用本发明的探针靶向富集方法检测临床10份感染患者的样本,样本类型包括肺泡灌洗液、痰液和脑脊液。操作步骤与实施例1相同,探针用量为1 pmol。结果见表5,结果显示本发明所建立的方法在1M的测序通量下,靶向富集可以鉴定临床样本中的探针组合覆盖的微生物,与mNGS(其测序通量为20M)测序结果完全一致,本发明准确、可靠。
表5 临床样本RPM(Reads per million)结果展示
实施例3 最低检出限
病原微生物标准品:ZymoBIOMICS™ Microbial Community DNA Standard(ZymoResearch),包含本发明探针覆盖的病原铜绿假单胞菌、大肠埃希菌、粪肠球菌、金黄色葡萄球菌、单核细胞增多李斯特菌。
标准品与临床阴性样本(不含本发明和标准品中覆盖病原)DNA混合,形成高(1000-50000 copies)、中(100-5000 copies)、低(10-500 copies)、极低(1-50 copies)
用本发明的探针靶向富集方法平行检测不同拷贝数的标准品5次,操作步骤与实施例1相同,探针用量为1 pmol。结果见表6,本发明所建立的方法对于细菌的检出限低于16-40 copies。本发明检测灵敏度高。
表6 不同微生物最低拷贝数及检出的reads数
实施例4 与mNGS检出差异比较
病原微生物标准品:ZymoBIOMICS™ Microbial Community DNA Standard(ZymoResearch),包含本发明探针覆盖的病原铜绿假单胞菌、大肠埃希菌、粪肠球菌、金黄色葡萄球菌、单核细胞增多李斯特菌。
标准品与临床阴性样本(不含本发明和标准品中覆盖病原)DNA混合,极低(1-50copies)下,用本发明的探针靶向富集方法平行检测不同拷贝数的标准品5次,同时行mNGS检测,操作步骤与实施例1相同,探针用量为1 pmol。结果见表7和表8,本发明所建立的方法检出的reads数是mNGS的4-10倍,并且在1M reads测试数据量下,本发明检出微生物的RPM显著优于mNGS。本发明检测灵敏度高,成本低。
表7 mNGS与tNGS检出的reads数比较
表8 mNGS与tNGS检出的RPM比较
实施例4 探针覆盖范围对比
从烟曲霉、新型隐球菌和白色念珠菌的探针中分别选出15条探针按照探针,分别是SEQ ID NO.442-456、SEQ ID NO. 332-346和SEQ ID NO.360-374,并等摩尔数混合成新的CSF panel2。
筛选出12例临床样本,用本发明的探针靶向富集方法进行平行检测,操作步骤与实施例1相同,测试其对样本中烟曲霉、新型隐球菌和白色念珠菌的检测效果。结果见表9,本发明所建立的方法检出的reads数明显高于CSF panle2的检测结果,并且在1M reads测试数据量下,本发明检出微生物的RPM显著优于探针覆盖范围少的产品。本发明检测灵敏度高。
表9 tNGS探针覆盖范围检出的RPM/ Reads比较
实施例5 特异性验证
采用其他的常见病原标准品,混合后进行特异性验证,采用本专利中的方法进行检测,判断是否检出相应捕获区域数据,具体结果如下:
表10 特异性验证结果
可见,本专利的检测方法特异性良好。
- 用于靶向病原体基因RNA的gRNA及基于C2c2的病原体基因的检测方法、检测试剂盒
- 捕获探针的设计方法、捕获探针、捕获探针组和试剂盒
- 一种用于无创检测待测胎儿染色体是否为非整倍体的试剂盒及其专用探针组
- 用于检测T790M和C797S顺反式突变类型的探针组、试剂盒、反应体系及系统
- 用于检测自身炎症性疾病致病基因的探针组及试剂盒
- 用于检测病原微生物的捕获探针组、方法、试剂盒及应用
- 用于检测病原微生物的捕获探针组、方法、试剂盒及应用
