利用CRISPR-Cas12a蛋白检测艰难梭菌的方法
文献发布时间:2023-06-19 19:18:24
技术领域
本发明涉及分子生物学检测技术领域,是一种利用CRISPR-Cas12a蛋白检测艰难梭菌的方法。
背景技术
艰难梭菌(
目前临床上对于艰难梭菌感染的实验诊断主要采用检查病原体的微生物培养法和检测细菌毒素的免疫学方法。微生物培养法包括培养初筛、菌落形态观察、纯化培养、血清型鉴定和生化鉴定等,操作步骤复杂、繁琐,一般用时4天至7天,费时费力。不能满足临床腹泻快速诊断的要求。免疫学方法可直接检测标本中的艰难梭菌A 毒素和B毒素,特异性高,操作简单,但是敏感性低。分子诊断,诸如PCR方法在检测艰难梭菌上已经有较好的应用,大大缩短了检测的时间,但是这些方法需要使用复杂的仪器和装备精良的实验室。等温扩增方法,如LAMP方法克服了PCR技术需要昂贵的设备仪器等缺点,具有操作简便,结果判断简单等优点。但LAMP技术要求多对引物且对靶基因的要求较高,设计比较繁琐,限制了其进一步的应用。重组酶聚合酶扩增(RPA)是一种快速兴起的恒温扩增技术,在恒温(37℃)条件下即可获得可检测级别的扩增核酸。但在实际检测过程中,需要筛选出很多的引物才能达到较高的灵敏度。
是“Clustered regularly interspaced short palindromic repeats”的缩写,指规律成簇的间隔短回文重复。Cas是“CRISPR-associated”缩写,为CRISPR相关。CRISPR-Cas系统是一个细菌及古细菌进化出来用以抵御噬菌体入侵的适应性机制,后被发现并发展成为一种由引导RNA指引Cas核酸酶对靶向基因进行特定核酸编辑的技术。目前,基于CRISPR的核酸快速检测技术主要分为两大类,即2020年诺贝尔化学奖得主JenniferDoudna开发的依赖于Cas12a的称为DETECTR的核酸恒温检测技术,以及CRISPR专利所有者张锋开发的依赖于Cas13a的称为SHERLOCK的核酸恒温检测技术。其原理都是通过恒温扩增方法如RPA对目的片段进行扩增富集,扩增后的目的片段会被Cas蛋白通过一段crRNA的引导靶向识别,激活Cas蛋白成为一个DNA切割机(Cas12a)或者RNA切割机(Cas13a),将附近所有的单链DNA(Cas12a)或者单链RNA(cas13a)进行切割。这一特性作用于单链核酸荧光探针则可用于报告检测结果。
发明内容
本发明提供了一种利用CRISPR-Cas12a蛋白检测艰难梭菌的方法,克服了上述现有技术之不足,其能在37℃下进行等温扩增,即可快速准确的实现对艰难梭菌特异基因的扩增和检测,无需像PCR一样通过变温仪器来实现扩增检测。
本发明的技术方案是通过以下措施来实现的:一种利用CRISPR-Cas12a蛋白检测艰难梭菌的方法,按照下述步骤进行:步骤一,制备Cas12a蛋白;步骤二,设计并合成适合RPA扩增的引物;步骤三,设计并合成适合Cas12a蛋白的crRNA;步骤四,设计并合成DNA报告分子;步骤五,设计并合成阳性质粒;步骤六,制备阳性质粒;步骤七,配制扩增检测体系;步骤八,CRISPR-Cas12a蛋白检测。
下面是对上述发明技术方案的进一步优化或/和改进:
上述Cas12a蛋白为LbCas12a蛋白。
上述步骤二中,设计并合成适合RPA扩增的引物的具体过程为:根据保守的艰难梭菌tcdB基因,设计特异检测艰难梭菌的上游引物和下游引物,将设计好的所述上游引物和所述下游引物在NCBI中进行blast,显示是特异的艰难梭菌序列,其中,上游引物的核苷酸序列如SEQ ID NO.1所示,下游引物的核苷酸序列如SEQ ID NO.2所示,艰难梭菌tcdB基因的序列如SEQ ID NO.4所示。
上述步骤三中,设计并合成适合Cas12a蛋白的crRNA的具体过程为:根据Cas12a蛋白的特点,在扩增的基因片段序列直接设计特异识别艰难梭菌的crRNA,在NCBI中进行blast,显示是特异的tcdB基因序列,其中,crRNA的核酸序列如SEQ ID NO.3所示。
上述步骤四中,DNA报告分子的序列为荧光报告基团-TTATT-荧光淬灭基团,其中,荧光报告基团为FAM、HEX、TET、JOE和VIC中的一种,荧光淬灭基团为BHQ1、BHQ2和BHQ中的一种。
上述步骤五中,设计并合成阳性质粒的具体过程为:将艰难梭菌tcdB基因的核酸序列克隆到pUc57质粒中,克隆位点为SamI,抗性为氨苄抗性,克隆菌株为DH5α。
上述步骤六中,制备阳性质粒的具体过程为:以1%接种量,接种到含有100μg/ml氨苄的LB培养基中,在摇床中37℃,220rpm过夜培养,吸取菌液,采用天根质粒提取试剂盒提取质粒,使用Nanodrop2000测浓度,计算拷贝数,进行系列梯度稀释,以备后续使用。
上述步骤七中,扩增检测体系为50μL反应体系,扩增检测体系中的组分与用量包括:
RPA缓冲液 29.5μL;
上游引物 10μM 2.1μL;
下游引物 10μM 2.1μL;
核酸模板+双蒸水 13.8μL;
醋酸镁 280mM 2.5μL。
上述步骤八中,CRISPR-Cas12a蛋白检测时,检测体系中的组分与用量包括:NEBufferr2.1 10× 2μL;
Cas12a蛋白 10mg/ml 0.5μL;
DNA报告分子 10μM 1μL;
crRNA 5μM 0.4μL;
检测产物+双蒸水 16.1μL。
上述CRISPR-Cas12a蛋白检测的具体过程为:将NEBufferr2.1、Cas12a蛋白、DNA报告分子、crRNA、检测产物+双蒸水混合均匀后,通过荧光检测仪测试反应并观察结果,其中,荧光检测仪的温度设置为37℃。
本发明在等温条件下(37℃)进行,10分钟至60分钟内,即可实现对艰难梭菌特异基因的扩增和检测检测,检测灵敏度可以达到1.0×10
附图说明
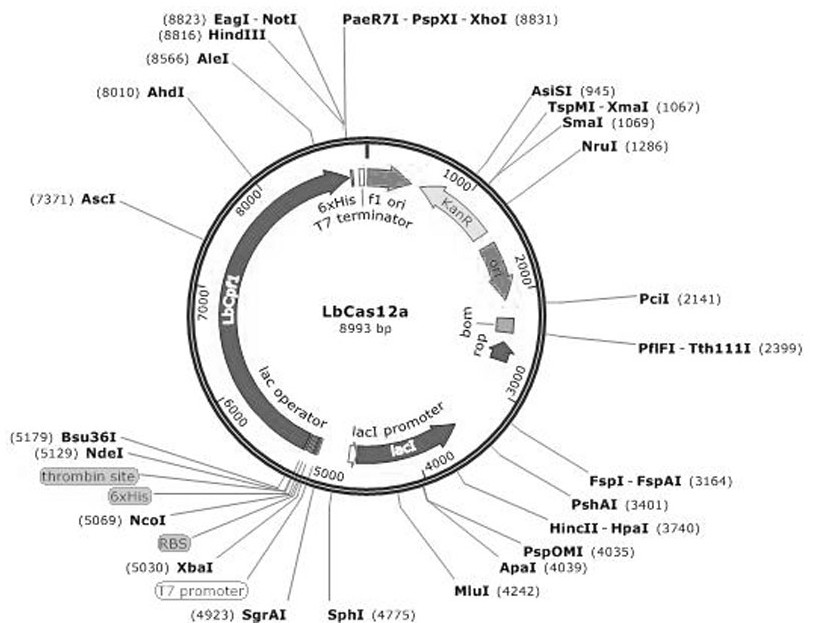
附图1为本发明实施例11中表达载体pET-28a-LbCas12a的质粒图谱。
附图2为本发明实施例12中对不同浓度艰难梭菌质粒DNA灵敏度检测结果图。
具体实施方式
本发明不受下述实施例的限制,可根据本发明的技术方案与实际情况来确定具体的实施方式。本发明中所提到各种化学试剂和化学用品如无特殊说明,均为现有技术中公知公用的化学试剂和化学用品。
下面结合实施例对本发明作进一步描述:
实施例1:该利用CRISPR-Cas12a蛋白检测艰难梭菌的方法,按照下述步骤进行:步骤一,制备Cas12a蛋白;步骤二,设计并合成适合RPA扩增的引物;步骤三,设计并合成适合Cas12a蛋白的crRNA;步骤四,设计并合成DNA报告分子;步骤五,设计并合成阳性质粒;步骤六,制备阳性质粒;步骤七,配制扩增检测体系;步骤八,CRISPR-Cas12a蛋白检测。
实施例2:作为上述实施例的优化,Cas12a蛋白为LbCas12a蛋白。
实施例3:作为上述实施例的优化,步骤二中,设计并合成适合RPA扩增的引物的具体过程为:根据保守的艰难梭菌tcdB基因,设计特异检测艰难梭菌的上游引物和下游引物,将设计好的所述上游引物和所述下游引物在NCBI中进行blast,显示是特异的艰难梭菌序列,其中,上游引物的核苷酸序列如SEQ ID NO.1所示,下游引物的核苷酸序列如SEQ IDNO.2所示,艰难梭菌tcdB基因的序列如SEQ ID NO.4所示。
实施例4:作为上述实施例的优化,步骤三中,设计并合成适合Cas12a蛋白的crRNA的具体过程为:根据Cas12a蛋白的特点,在扩增的基因片段序列直接设计特异识别艰难梭菌的crRNA,在NCBI中进行blast,显示是特异的tcdB基因序列,其中,crRNA的核酸序列如SEQ ID NO.3所示。
实施例5:作为上述实施例的优化,步骤四中,DNA报告分子的序列为荧光报告基团-TTATT-荧光淬灭基团,其中,荧光报告基团为FAM、HEX、TET、JOE和VIC中的一种,荧光淬灭基团为BHQ1、BHQ2和BHQ中的一种。
实施例6:作为上述实施例的优化,步骤五中,设计并合成阳性质粒的具体过程为:将艰难梭菌tcdB基因的核酸序列克隆到pUc57质粒中,克隆位点为SamI,抗性为氨苄抗性,克隆菌株为DH5α。
实施例7:作为上述实施例的优化,步骤六中,制备阳性质粒的具体过程为:以1%接种量,接种到含有100μg/ml氨苄的LB培养基中,在摇床中37℃,220rpm过夜培养,吸取菌液,采用天根质粒提取试剂盒提取质粒,使用Nanodrop2000测浓度,计算拷贝数,进行系列梯度稀释,以备后续使用。
实施例8:作为上述实施例的优化,步骤七中,扩增检测体系为50μL反应体系,扩增检测体系中的组分与用量包括:
RPA缓冲液 29.5μL;
上游引物(10μM) 2.1μL;
下游引物(10μM) 2.1μL;
核酸模板+双蒸水 13.8μL;
醋酸镁(280mM) 2.5μL。
实施例9:作为上述实施例的优化,检测体系中的组分与用量包括:
NEBufferr2.1(10×) 2μL;
Cas12a蛋白(10mg/ml) 0.5μL;
DNA报告分子(10μM) 1μL;
crRNA(5μM) 0.4μL;
检测产物+双蒸水 16.1μL。
实施例10:作为上述实施例的优化,CRISPR-Cas12a蛋白检测的具体过程为:将NEBufferr2.1、Cas12a蛋白、DNA报告分子、crRNA、检测产物+双蒸水混合均匀后,通过荧光检测仪测试反应并观察结果,其中,荧光检测仪的温度设置为37℃。
本发明通过重组酶聚合酶等温核酸扩增体系、制备Cas12a蛋白,设计并合成RPA扩增引物,设计并合成crRNA,设计并合成DNA报告分子,制备阳性对照,可方便快速准确地鉴定艰难梭菌DNA,操作简便,无需像PCR一样通过变温仪器来实现扩增检测,只需在37℃下进行等温扩增即可完成检测。
实施例11:Cas12a蛋白的获取
(1)在NCBI上查找Cas12a蛋白(Lachnospiraceae bacterium,LbCas12a)的基因序列,GenBank序列号为:MW477886.1。Cas12a基因的核苷酸序列如SEQ ID No:5所示,该基因编码的蛋白的氨基酸序列如SEQ ID No:6所示。人工合成Cas12a基因并克隆到T载体上,然后用NdeI-HindIII双酶切,并将此片段克隆至pET-28a表达载体的NdeI和HindIII酶切位点之间,构建表达载体pET-28a-Cas12a,表达载体pET-28a-Cas12a的质粒图谱如附图1所示。
(2)将步骤(1)中所述构建表达载体pET-28a-Cas12a转化到大肠杆菌BL21(DE3)中,并涂于含30μg/ml Kan平板上,37℃培养12h至16h,挑取单克隆菌株,加入1L终浓度为30μg/ml的Kan的LB液体培养基中培养4h,再加入终浓度为0.7mM的IPTG诱导,30℃过夜培养,收取菌液,5000rpm离心30min,得菌体;
(3)用缓冲液对步骤(2)中得到的所述菌体进行超声破碎,取上清,10000rpm离心30min,纯化后用缓冲液冲洗至基线水平,然后用含咪唑浓度为250mM的缓冲液洗脱蛋白,洗脱后的蛋白经过sephadex G25色谱柱脱盐得到蛋白,-80℃保存,得到纯化后的Cas12a蛋白。
实施例12:灵敏度实验
(1)阳性质粒的合成
将核酸序列(如SEQ ID NO.4所示)克隆到pUc57质粒中(可委托上海生工操作),克隆位点为SamI,用Nanodrop2000测浓度,计算拷贝数。
(2)制备重组质粒工作标准品,分别为:
工作标准品1,含有1.0×10
工作标准品2,含有1.0×10
工作标准品3,含有1.0×10
工作标准品4,含有1.0×10
(3)配制反应体系
RPA扩增体系(50μL反应体系)包括如下组分与用量:
RPA缓冲液 29.5μL;
上游引物(10μM) 2.1μL;
下游引物(10μM) 2.1μL;
核酸模板+双蒸水 13.8μL;
醋酸镁(280mM) 2.5μL。
按所述扩增体系配制反应液(按6个反应进行配制),在1.5mlEP管中混合均匀,吸取46.5μL分别加入到5个RPA反应单元(乐尚生物(无锡)科技有限公司,试剂盒可实现DNA扩增。包含DNA扩增所需的所有酶和试剂,用户只需提供引物和模板中,使冻干粉溶解,向以上5个配制好的反应试剂试管中分别加入1μL阴性质控品、1μL标准工作品4、1μL标准工作品3、1μL标准工作品2、1μL标准工作品1为模板,并向管盖上加入2.5μL280 mM醋酸镁,加好样后每个反应管进行充分混匀,每个反应管总体积为50μL,37℃反应15分钟。
(4)CRISPR-Cas12a检测体系(20μL反应体系)包括如下组分与用量:
NEBufferr2.1(10×) 2μL;
Cas12a蛋白(10mg/ml) 0.5μL;
DNA报告分子(10μM) 1μL;
crRNA(5μM) 0.4μL;
检测产物+双蒸水 16.1μL;
吸取2ul步骤(3)的反应产物,按以上体系进行配制,放入常温等温扩增荧光检测仪(T8便携式常温等温扩增荧光检测仪,TwistDx),设定反应温度为37℃,反应时间60分钟。
检测结果如附图2所示,由附图2可知,结果显示在60分钟内,均产生扩增信号,检测灵敏度可以达到1.0×10
综上所述,本发明在等温条件下(37℃)进行,10分钟至60分钟内,即可实现对艰难梭菌特异基因的扩增和检测检测,检测灵敏度可以达到1.0×10
以上技术特征构成了本发明的实施例,其具有较强的适应性和实施效果,可根据实际需要增减非必要的技术特征,来满足不同情况的需求。
序列表
<110> 新疆维吾尔自治区疾病预防控制中心
<120> 利用CRISPR-Cas12a蛋白检测艰难梭菌的方法
<130> 2010
<160> 6
<170> PatentIn version 3.5
<210> 1
<211> 33
<212> DNA
<213> 人工序列
<400> 1
ctatcttagc ttctatttct tgtcttaata atg 33
<210> 2
<211> 33
<212> DNA
<213> 人工序列
<400> 2
ctggtttaaa tactattaca gatgcagcca aag 33
<210> 3
<211> 41
<212> RNA
<213> 人工序列
<400> 3
uaauuucuac uaaguguaga uaucuaaugc aguugauacu a 41
<210> 4
<211> 293
<212> DNA
<213> 人工序列
<400> 4
ttactgccat tatacctatc ttagcttcta tttcttgtct taataatggg tcacttgttt 60
cacttagctc tttgattgct gcacctaaac ttacaccatc tatgatagtt gcaattacag 120
gtaatccttc agataatgta ggaagtaaat ctatagtttc atctaatgca gttgatacta 180
attcaacaac tttggctgca tctgtaatag tatttaaacc agtactaaat aattgagcat 240
aaacttgaac tttcattgct acacttaaat tactaagaga ttctttagaa cta 293
<210> 5
<211> 3687
<212> DNA
<213> 人工序列
<400> 5
atgagcaagc tggagaagtt tacaaactgc tactccctgt ctaagaccct gaggttcaag 60
gccatccctg tgggcaagac ccaggagaac atcgacaata agcggctgct ggtggaggac 120
gagaagagag ccgaggatta taagggcgtg aagaagctgc tggatcgcta ctatctgtct 180
tttatcaacg acgtgctgca cagcatcaag ctgaagaatc tgaacaatta catcagcctg 240
ttccggaaga aaaccagaac cgagaaggag aataaggagc tggagaacct ggagatcaat 300
ctgcggaagg agatcgccaa ggccttcaag ggcaacgagg gctacaagtc cctgtttaag 360
aaggatatca tcgagacaat cctgccagag ttcctggacg ataaggacga gatcgccctg 420
gtgaacagct tcaatggctt taccacagcc ttcaccggct tctttgataa cagagagaat 480
atgttttccg aggaggccaa gagcacatcc atcgccttca ggtgtatcaa cgagaatctg 540
acccgctaca tctctaatat ggacatcttc gagaaggtgg acgccatctt tgataagcac 600
gaggtgcagg agatcaagga gaagatcctg aacagcgact atgatgtgga ggatttcttt 660
gagggcgagt tctttaactt tgtgctgaca caggagggca tcgacgtgta taacgccatc 720
atcggcggct tcgtgaccga gagcggcgag aagatcaagg gcctgaacga gtacatcaac 780
ctgtataatc agaaaaccaa gcagaagctg cctaagttta agccactgta taagcaggtg 840
ctgagcgatc gggagtctct gagcttctac ggcgagggct atacatccga tgaggaggtg 900
ctggaggtgt ttagaaacac cctgaacaag aacagcgaga tcttcagctc catcaagaag 960
ctggagaagc tgttcaagaa ttttgacgag tactctagcg ccggcatctt tgtgaagaac 1020
ggccccgcca tcagcacaat ctccaaggat atcttcggcg agtggaacgt gatccgggac 1080
aagtggaatg ccgagtatga cgatatccac ctgaagaaga aggccgtggt gaccgagaag 1140
tacgaggacg atcggagaaa gtccttcaag aagatcggct ccttttctct ggagcagctg 1200
caggagtacg ccgacgccga tctgtctgtg gtggagaagc tgaaggagat catcatccag 1260
aaggtggatg agatctacaa ggtgtatggc tcctctgaga agctgttcga cgccgatttt 1320
gtgctggaga agagcctgaa gaagaacgac gccgtggtgg ccatcatgaa ggacctgctg 1380
gattctgtga agagcttcga gaattacatc aaggccttct ttggcgaggg caaggagaca 1440
aacagggacg agtccttcta tggcgatttt gtgctggcct acgacatcct gctgaaggtg 1500
gaccacatct acgatgccat ccgcaattat gtgacccaga agccctactc taaggataag 1560
ttcaagctgt attttcagaa ccctcagttc atgggcggct gggacaagga taaggagaca 1620
gactatcggg ccaccatcct gagatacggc tccaagtact atctggccat catggataag 1680
aagtacgcca agtgcctgca gaagatcgac aaggacgatg tgaacggcaa ttacgagaag 1740
atcaactata agctgctgcc cggccctaat aagatgctgc caaaggtgtt cttttctaag 1800
aagtggatgg cctactataa ccccagcgag gacatccaga agatctacaa gaatggcaca 1860
ttcaagaagg gcgatatgtt taacctgaat gactgtcaca agctgatcga cttctttaag 1920
gatagcatct cccggtatcc aaagtggtcc aatgcctacg atttcaactt ttctgagaca 1980
gagaagtata aggacatcgc cggcttttac agagaggtgg aggagcaggg ctataaggtg 2040
agcttcgagt ctgccagcaa gaaggaggtg gataagctgg tggaggaggg caagctgtat 2100
atgttccaga tctataacaa ggacttttcc gataagtctc acggcacacc caatctgcac 2160
accatgtact tcaagctgct gtttgacgag aacaatcacg gacagatcag gctgagcgga 2220
ggagcagagc tgttcatgag gcgcgcctcc ctgaagaagg aggagctggt ggtgcaccca 2280
gccaactccc ctatcgccaa caagaatcca gataatccca agaaaaccac aaccctgtcc 2340
tacgacgtgt ataaggataa gaggttttct gaggaccagt acgagctgca catcccaatc 2400
gccatcaata agtgccccaa gaacatcttc aagatcaata cagaggtgcg cgtgctgctg 2460
aagcacgacg ataaccccta tgtgatcggc atcgataggg gcgagcgcaa tctgctgtat 2520
atcgtggtgg tggacggcaa gggcaacatc gtggagcagt attccctgaa cgagatcatc 2580
aacaacttca acggcatcag gatcaagaca gattaccact ctctgctgga caagaaggag 2640
aaggagaggt tcgaggcccg ccagaactgg acctccatcg agaatatcaa ggagctgaag 2700
gccggctata tctctcaggt ggtgcacaag atctgcgagc tggtggagaa gtacgatgcc 2760
gtgatcgccc tggaggacct gaactctggc tttaagaata gccgcgtgaa ggtggagaag 2820
caggtgtatc agaagttcga gaagatgctg atcgataagc tgaactacat ggtggacaag 2880
aagtctaatc cttgtgcaac aggcggcgcc ctgaagggct atcagatcac caataagttc 2940
gagagcttta agtccatgtc tacccagaac ggcttcatct tttacatccc tgcctggctg 3000
acatccaaga tcgatccatc taccggcttt gtgaacctgc tgaaaaccaa gtataccagc 3060
atcgccgatt ccaagaagtt catcagctcc tttgacagga tcatgtacgt gcccgaggag 3120
gatctgttcg agtttgccct ggactataag aacttctctc gcacagacgc cgattacatc 3180
aagaagtgga agctgtactc ctacggcaac cggatcagaa tcttccggaa tcctaagaag 3240
aacaacgtgt tcgactggga ggaggtgtgc ctgaccagcg cctataagga gctgttcaac 3300
aagtacggca tcaattatca gcagggcgat atcagagccc tgctgtgcga gcagtccgac 3360
aaggccttct actctagctt tatggccctg atgagcctga tgctgcagat gcggaacagc 3420
atcacaggcc gcaccgacgt ggattttctg atcagccctg tgaagaactc cgacggcatc 3480
ttctacgata gccggaacta tgaggcccag gagaatgcca tcctgccaaa gaacgccgac 3540
gccaatggcg cctataacat cgccagaaag gtgctgtggg ccatcggcca gttcaagaag 3600
gccgaggacg agaagctgga taaggtgaag atcgccatct ctaacaagga gtggctggag 3660
tacgcccaga ccagcgtgaa gcactaa 3687
<210> 6
<211> 1228
<212> PRT
<213> 人工序列
<400> 6
MSKLEKFTNC YSLSKTLRFK AIPVGKTQEN IDNKRLLVED EKRAEDYKGV KKLLDRYYLS 60
FINDVLHSIK LKNLNNYISL FRKKTRTEKE NKELENLEIN LRKEIAKAFK GNEGYKSLFK 120
KDIIETILPE FLDDKDEIAL VNSFNGFTTA FTGFFDNREN MFSEEAKSTS IAFRCINENL 180
TRYISNMDIF EKVDAIFDKH EVQEIKEKIL NSDYDVEDFF EGEFFNFVLT QEGIDVYNAI 240
IGGFVTESGE KIKGLNEYIN LYNQKTKQKL PKFKPLYKQV LSDRESLSFY GEGYTSDEEV 300
LEVFRNTLNK NSEIFSSIKK LEKLFKNFDE YSSAGIFVKN GPAISTISKD IFGEWNVIRD 360
KWNAEYDDIH LKKKAVVTEK YEDDRRKSFK KIGSFSLEQL QEYADADLSV VEKLKEIIIQ 420
KVDEIYKVYG SSEKLFDADF VLEKSLKKND AVVAIMKDLL DSVKSFENYI KAFFGEGKET 480
NRDESFYGDF VLAYDILLKV DHIYDAIRNY VTQKPYSKDK FKLYFQNPQF MGGWDKDKET 540
DYRATILRYG SKYYLAIMDK KYAKCLQKID KDDVNGNYEK INYKLLPGPN KMLPKVFFSK 600
KWMAYYNPSE DIQKIYKNGT FKKGDMFNLN DCHKLIDFFK DSISRYPKWS NAYDFNFSET 660
EKYKDIAGFY REVEEQGYKV SFESASKKEV DKLVEEGKLY MFQIYNKDFS DKSHGTPNLH 720
TMYFKLLFDE NNHGQIRLSG GAELFMRRAS LKKEELVVHP ANSPIANKNP DNPKKTTTLS 780
YDVYKDKRFS EDQYELHIPI AINKCPKNIF KINTEVRVLL KHDDNPYVIG IDRGERNLLY 840
IVVVDGKGNI VEQYSLNEII NNFNGIRIKT DYHSLLDKKE KERFEARQNW TSIENIKELK 900
AGYISQVVHK ICELVEKYDA VIALEDLNSG FKNSRVKVEK QVYQKFEKML IDKLNYMVDK 960
KSNPCATGGA LKGYQITNKF ESFKSMSTQN GFIFYIPAWL TSKIDPSTGF VNLLKTKYTS 1020
IADSKKFISS FDRIMYVPEE DLFEFALDYK NFSRTDADYI KKWKLYSYGN RIRIFRNPKK 1080
NNVFDWEEVC LTSAYKELFN KYGINYQQGD IRALLCEQSD KAFYSSFMAL MSLMLQMRNS 1140
ITGRTDVDFL ISPVKNSDGI FYDSRNYEAQ ENAILPKNAD ANGAYNIARK VLWAIGQFKK 1200
AEDEKLDKVK IAISNKEWLE YAQTSVKH 1228
- 检测和分析艰难梭菌菌株的组合物和试剂盒及用该组合物和试剂盒检测艰难梭菌菌株的方法
- 一种检测艰难梭菌菌属基因及毒素基因的多重荧光PCR试剂盒和方法
