小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法
文献发布时间:2023-06-19 19:35:22
技术领域
本发明涉及基因组学及生物信息学技术领域,尤其涉及小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法。
背景技术
小麦是世界上最重要的粮食作物之一,实现高产,稳产,优质是小麦育种的三个目标。遗传基础的狭窄限制了小麦育种目标的实现,通过远缘杂交的方式丰富小麦育种基因池是打破小麦育种基因资源瓶颈的有效方式。为了能够有效地利用外源基因,摆脱遗传累赘,渐渗系材料被广泛地应用于小麦遗传改良,但是传统的原位杂交方法无法有效地检测渐渗系材料。
发明内容
本发明提供小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,用以解决现有技术中无法有效检测渐渗系材料的缺陷,本发明能够简单而快速地检测渐渗系材料,并且能高效并准确地挖掘外源目的基因。
本发明提供一种小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,包括以下步骤:
A、获得小麦远缘杂交后代渐渗系和小麦受体的全基因组序列;
B、将小麦参考基因组序列和小麦近缘植物参考基因组序列作为参考序列,将小麦受体的reads以及渐渗系的reads分别与所述参考序列进行比对,挑选出覆盖度大于等于基因全长2/3且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,作为候选基因。
根据本发明提供的小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,还包括以下步骤:
C、将所述候选基因与小麦参考基因组序列进行对比得到两者的SNP差异,根据SNP差异位点开发分子标记,利用所述分子标记验证所述候选基因的可靠性。本发明提供小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,该方法只需对小麦远缘杂交衍生的渐渗系及其小麦受体两个样品进行重测序,结合生物信息学分析、比较基因组学和功能标记开发验证,可实现从小麦远缘杂交渐渗系中快速挖掘外源功能候选基因。本发明能够简单而快速地检测渐渗系材料,并且能高效并准确地挖掘外源候选基因,为功能基因的克隆奠定基础。
在本发明的一些实施例中,步骤A包括对原始测序数据进行质控、去污染和接头,得到高质量的测序数据;具体方法为:去除两端测序质量值小于20的碱基,小于25bp的测序读长将被去除,过滤使用Trimmomatic软件执行。
在本发明的一些实施例中,步骤B中,将小麦参考基因组序列与野生近缘植物基因组序列进行整合作为总的参考序列。
根据本发明提供的小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,步骤B覆盖度查看的具体方法为,用IGV软件查看覆盖度挑选覆盖度大于等于基因全长2/3的并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因。
在本发明的一些实施例中,步骤B中,小麦近缘植物参考基因组序列与小麦参考基因组序列进行整合作为总的参考序列;和/或
利用BWA将小麦受体的重测序数据以及渐渗系的重测序数据分别与所述总的参考进行序列比对,和/或
然后用hts-nim-tools软件筛选可靠度较高的reads;和/或
最后用IGV软件查看覆盖度。
在本发明的一些实施例中,步骤B中包括,利用BWA将小麦受体的reads以及渐渗系的reads分别与所述参考序列进行比对;
和/或,用hts-nim-tools软件筛选reads;
和/或,用IGV软件查看覆盖度。
在本发明的一些实施例中,步骤B包括利用BWA软件将小麦受体重测序数据以及渐渗系的重测序数据与所述总的参考序列进行比对,然后用hts-nim-tools筛选可靠度较高的reads,用IGV软件比对,找出覆盖度高并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,获得渐渗系中外源染色体片段中特异的基因,作为候选基因。
在本发明的一些实施例中,步骤C中,将获得的候选基因序列通过BLAST与小麦基因序列进行比对。
在本发明的一些实施例中,步骤C中,将获得的候选基因序列通过BLAST与小麦参考基因组序列进行比对。
根据本发明提供的小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,还包括以下步骤:
D、对筛选出的候选基因利用KEGG、GO、TO中的一个或几个进行功能注释。
在本发明的一些实施例中,步骤C中,所开发的分子标记包括EST、KASP标记。
在本发明的一些实施例中,本发明所述小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,包括以下步骤:
A、小麦远缘杂交后代渐渗系和小麦受体的重测序:分别获取小麦远缘杂交后代渐渗系和小麦受体植株的叶片,分别提取DNA;利用第二代测序技术对各自的样品进行建库和测序,分别得到小麦受体和渐渗系的重测序数据;
B、渐渗系中外源染色体片段中特异的基因:将小麦参考基因组序列和小麦近缘植物参考基因组序列进行整合作为总的参考序列;将小麦受体的reads以及渐渗系的reads分别与所述总的参考序列进行比对,排序并去除重复,然后筛选可靠度较高的reads,最后将reads映射到注释文件上相对应的基因,相比较小麦受体的结果,用IGV软件比对,找出覆盖度高并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,获得渐渗系中外源染色体片段中特异的基因,作为候选基因;
C、分子标记开发:将获得的候选基因序列与小麦参考基因序列进行比对,根据比对结果找出候选基因与小麦基因组之间的SNP差异,根据SNP差异位点开发分子标记;通过聚丙烯凝胶电泳验证标记的可靠性;
D、候选基因功能预测:对于步骤C中通过标记筛选的候选基因,通过KEGG,GO,TO结合进行功能注释。
在一些实施例中,本发明提供小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法,包括以下步骤:
A、小麦远缘杂交后代渐渗系和小麦受体的重测序:分别获取小麦远缘杂交后代渐渗系和小麦受体植株的叶片,分别提取DNA;利用第二代测序技术和平台对各自的样品进行建库和测序,分别得到小麦受体和渐渗系的重测序数据;
B、渐渗系中冰草外源染色体片段中特异的基因:将小麦参考基因组序列和小麦近缘植物参考基因组序列进行整合作为总的参考序列;将小麦受体的reads以及渐渗系的reads分别与所述总的参考序列进行比对,排序并去除重复,然后筛选可靠度较高的reads,最后将reads映射到注释文件上相对应的基因,相比较小麦受体的结果,用IGV软件比对,找出覆盖度高并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,获得渐渗系中外源染色体片段中特异的基因,作为候选基因;
C、分子标记开发:将获得的候选基因序列与小麦参考基因序列进行比对,根据比对结果找出候选基因与小麦基因组上基因之间的SNP差异,根据SNP差异位点开发分子标记并进行PCR检测,利用设计的引物在小麦近缘植物、小麦受体和渐渗系中进行PCR验证,确定所获得的外源染色体片段中特异表达的基因的真实性。
D、候选基因预测:对于步骤C中通过标记筛选的候选基因,通过KEGG,GO,TO结合进行功能注释。
本发明还提供一种小麦-冰草渐渗系中发掘外源功能候选基因的方法,包括利用上述的小麦远缘杂交后代渐渗系中发掘外源功能候选基因的方法。
根据本发明提供的小麦-冰草渐渗系中发掘外源功能候选基因的方法,所述小麦-冰草渐渗系为普冰3504。
根据本发明提供的小麦-冰草渐渗系中发掘外源功能候选基因的方法,所述小麦受体为Fukuhokumugi;
和/或,所述小麦近缘植物为冰草Z559。
在一些实施例中,步骤S1还包括对原始测序数据进行质控、去污染和接头,得到高质量的测序数据,具体方法为:去除两端测序质量值小于20的碱基,小于25bp的测序读长将被去除,过滤使用Trimmomatic软件执行。
在一些实施例中,步骤S2中挑选出仅在小麦冰草渐渗系与小麦近缘植物冰草中存在的基因,具体方法为:通过对小麦冰草渐渗系与小麦受体Fukuhokumugi的重测序数据进行分析,用hts-nim-tools筛选可靠度较高的reads,然后用IGV软件查看覆盖度高且仅在小麦冰草渐渗系与小麦近缘植物冰草中存在的基因。
在一些实施例中,步骤S3中所述标记包括7720-2和501600-1。
在一些实施例中,所述小麦-冰草渐渗系中发掘外源功能候选基因的方法,包括以下步骤:
S1、小麦受体Fukuhokumugi和渐渗系的重测序:分别获取Fukuhokumugi和渐渗系植株的叶片,然后分别提取DNA,对Fukuhokumugi和渐渗系各自样品构建高通量测序文库,并使用Illumina HiSeq 2500高通量测序平台进行重测序;
S2、渐渗系中冰草外源染色体片段中特异的基因:整合冰草基因组序列和小麦参考基因组序列作为总的参考序列,用BWA软件将Fukuhokumugi和渐渗系的覆盖全基因组的reads与所述总的参考序列进行比对,排序并去除重复,然后用hts-nim-tools筛选可靠度较高的reads,用IGV软件比对,找出覆盖度高并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,获得渐渗系中外源染色体片段中特异的基因,作为候选基因;
S3、分子标记开发:将获得的候选基因序列与小麦参考基因序列进行比对,并根据比对结果找出候选基因与小麦A/B/D基因组上基因之间的SNP差异,根据SNP差异位点开发分子标记;利用开发的标记在冰草Z559、Fukuhokumugi和渐渗系中进行PCR验证,从而验证外源染色体片段中特异的基因的真实性;
S4、候选基因预测:对于步骤S3中通过标记筛选的候选基因,通过KEGG,GO,TO结合进行功能注释。
根据本发明提供的小麦-冰草渐渗系中发掘外源功能候选基因的方法,步骤C中利用以下引物验证候选基因的可靠性:
7720-2_F:5′-GGTCACTCCCAACTTGTGCAG-3′(SEQ ID NO:1),
7720-2_R:5′-GCATGCATTGTGCCTGCTA-3′(SEQ ID NO:2);
或
501600-1_F:5′-TTCACCCAAGGAAACTGAAACAG-3′(SEQ ID NO:3),
501600-1_R:5′-GCTCTTTCAGTGGGAAGCTGC-3′(SEQ ID NO:4)。
本发明还提供一种分子标记501600-1,扩增所述分子标记501600-1的引物为501600-1_F和501600-1_R。
本发明还提供上述的分子标记在小麦-冰草渐渗系中发掘外源功能候选基因中的应用。
上述的分子标记在分子标记辅助育种或小麦种质资源改良中的应用,所述分子标记辅助育种或小麦种质改良的目标为如下任一种或几种:
(1)增加小麦的小穗数;
(2)增加小麦的小穗粒数;
(3)增加小麦的穗粒数;
(4)提高小麦的结实性能;
(5)增加小麦的产量。
本发明还提供基因Ac3P01G501600.1,所述Ac3P01G501600.1的核苷酸序列为如下任一种:
(1)如SEQ ID NO 6所示;
(2)与SEQ ID NO 6限定的DNA序列杂交且编码具有相同功能的蛋白的DNA分子;
(3)与(1)或(2)中的DNA序列具有90%以上同源性且编码具有相同功能的蛋白的DNA分子。
本发明还提供基因Ac3P01G501600.1在如下任一种或几种中的应用:
(1)增加小麦的小穗数;
(2)增加小麦的小穗粒数;
(3)增加小麦的穗粒数;
(4)提高小麦的结实性能;
(5)增加小麦的产量。
借由上述技术方案,本发明至少具有下列优点及有益效果:
本发明提供一种只需对小麦远缘杂交衍生的渐渗系及其小麦受体两个样品进行重测序,结合生物信息学分析、比较基因组学和功能标记开发验证,可实现从小麦远缘杂交渐渗系中快速挖掘外源功能候选基因。首先将小麦野生近缘种的基因组序列与小麦参考基因组序列进行整合作为总的参考序列;其次对小麦远缘杂交衍生的渐渗系和小麦受体进行重测序,鉴定在渐渗系中特异表达的基因;再次进行与小麦基因组的比较基因组学分析,开发功能标记;最后结合功能注释确定与目的性状相关的外源功能候选基因。本发明能够简单而快速地检测渐渗系材料,并且能高效并准确地挖掘外源功能候选基因,为功能基因的克隆奠定基础。
附图说明
为了更清楚地说明本发明或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作一简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1为本发明实施例1中根据候选基因Ac1P04G477200.1开发的多态性标记7720-2和候选基因Ac3P01G501600.1开发的多态性标记501600-1电泳图。其中,M:DNA Marker;1:冰草Z559;2:普冰3504;3:Fukuhokumugi。
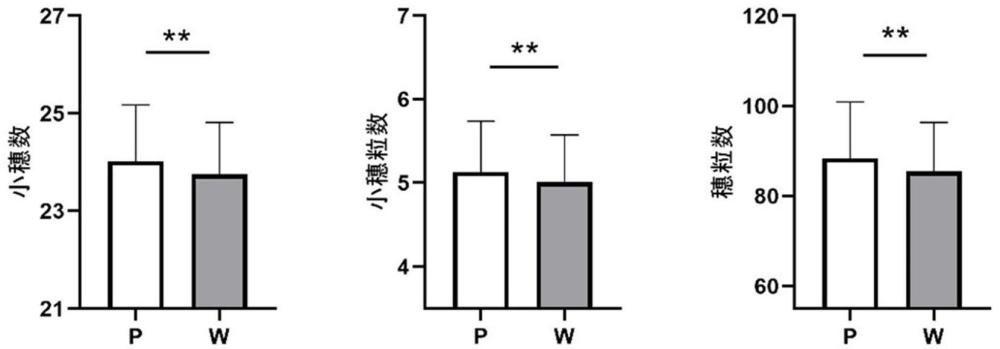
图2为本发明实施例1中根据候选基因Ac3P01G501600.1开发的多态性标记501600-1在渐渗系3504后代群体中的数据分析结果,P表示阳性植株,W表示阴性植株。
具体实施方式
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如Sambrook等分子克隆实验手册(Sambrook J&Russell DW,Molecular Cloning:a Laboratory Manual,2001),或按照制造厂商说明书建议的条件。
为使本发明的目的、技术方案和优点更加清楚,下面将结合本发明中的附图,对本发明中的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。
基于本发明中的实施例,本领域普通技术人员在没有作出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
在本发明的一个具体实施方式中,提供小麦-冰草渐渗系中发掘外源功能候选基因的方法,包括以下步骤:
S1、小麦受体Fukuhokumugi和渐渗系的全基因组重测序:分别获取Fukuhokumugi和渐渗系植株的叶片,然后分别提取DNA,对Fukuhokumugi和渐渗系各自样品构建高通量测序文库,并使用Illumina HiSeq 2500高通量测序平台进行全基因组重测序;
S2、渐渗系中冰草外源染色体片段中特异的基因:整合冰草全基因组序列和小麦参考基因组序列作为总的参考序列,用BWA软件将Fukuhokumugi和渐渗系的reads分别与所述总的参考序列进行比对,然后用hts-nim-tools筛选出符合参数且可靠度较高的reads,然后用picard去重复,然后用IGV软件查看覆盖度挑选覆盖度大于等于基因全长2/3的并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,获得渐渗系中外源染色体片段中特异的基因,作为候选基因;
S3、分子标记开发:将获得的候选基因序列与小麦参考基因序列进行比对,并根据比对结果找出候选基因与小麦A/B/D基因组上基因之间的SNP差异,根据SNP差异位点设计分子标记;利用设计的分子标记在冰草Z559、Fukuhokumugi和渐渗系中进行PCR验证,分别对每个候选基因开发了相对应的多态性分子标记(图1),从而验证外源染色体片段中特异表达的候选基因的真实性。
步骤S3中分子标记包括7720-2和501600-1,扩增所述分子标记的引物对分别如下:
7720-2_F:5′-GGTCACTCCCAACTTGTGCAG-3′;
7720-2_R:5′-GCATGCATTGTGCCTGCTA-3′。
501600-1_F:5′-TTCACCCAAGGAAACTGAAACAG-3′;
501600-1_R:5′-GCTCTTTCAGTGGGAAGCTGC-3′。
实施例1小麦-冰草渐渗系中发掘外源功能候选基因的方法
前期研究表明,普冰3504为小麦-冰草渐渗系,其来自小麦近缘植物冰草渐渗片段能够显著提高小麦的穗粒数。本实施例提供了该小麦背景下外源染色体片段中功能候选基因快速发掘的方法,在筛选小麦-冰草渐渗系普冰3504中与穗粒数相关功能基因的实例。
具体方法为:
A、小麦受体Fukuhokumugi和渐渗系普冰3504的重测序:
分别获取小麦远缘杂交后代渐渗系和小麦受体植株的叶片,分别提取DNA,最后对小麦受体Fukuhokumugi和普冰3504的样品构建高通量测序文库,使用Illumina HiSeq2500高通量测序平台进行重测序。对原始测序数据进行质控、去污染和接头,去除两端测序质量值小于20的碱基,小于25bp的测序读长将被去除,过滤使用Trimmomatic软件执行,得到高质量的测序数据。
B、普冰3504中冰草外源染色体片段中特异表达的基因分析:
整合冰草全基因组序列和小麦参考基因组(IWGSC RefSeq v2.0)作为总的参考序列,使用BWA软件将小麦Fukuhokumugi和普冰3504的重测序数据与参考序列进行比对,只保留唯一比对的序列比对结果,然后用hts-nim-tools软件提取符合参数且可靠度较高的reads序列,然后用picard去重复,然后用IGV软件查看覆盖度挑选覆盖度大于等于基因全长2/3的并且仅在小麦远缘杂交后代渐渗系与小麦近缘植物存在的基因,获得渐渗系中外源染色体片段中特异的基因,作为候选基因。
C、分子标记开发与利用
根据获得的候选基因序列,与小麦基因序列进行比对,并依据比对的结果找出候选基因与小麦A/B/D基因组上基因之间的SNP差异,根据SNP差异位点设计分子标记。对设计的分子标记在冰草Z559、Fukuhokumugi和渐渗系普冰3504中进行PCR验证,分别对每个候选基因(Ac1P04G477200.1、Ac3P01G501600.1)开发了相对应的多态性分子标记(图1),从而验证外源染色体片段中特异表达的基因的真实性。
基因Ac1P04G477200.1的核苷酸序列如SEQ ID NO:5所示,基因Ac3P01G501600.1的核苷酸序列如SEQ ID NO:6所示。
所述分子标记包括7720-2和501600-1,它们的检测引物分别如下:
7720-2_F:5′-GGTCACTCCCAACTTGTGCAG-3′;
7720-2_R:5′-GCATGCATTGTGCCTGCTA-3′。
501600-1_F:5′-TTCACCCAAGGAAACTGAAACAG-3′;
501600-1_R:5′-GCTCTTTCAGTGGGAAGCTGC-3′。
将Ac3P01G501600.1基因标记501600-1在渐渗系3504后代群体中检测发现,阳性植株与阴性植株相比,小穗数,小穗粒数,穗粒数均显著增加(图2)。该标记能够应用于分子标记辅助育种。
最后应说明的是:以上实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述实施例对本发明进行了详细的说明,本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的精神和范围。
- 小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法
- 小麦远缘杂交后代小片段易位系中发掘外源功能候选基因的方法
