一种右旋糖酐蔗糖酶及其制备方法和应用
文献发布时间:2023-06-19 11:59:12
技术领域
本发明属于酶制备领域,具体涉及一种右旋糖酐蔗糖酶及其制备方法和应用。
背景技术
右旋糖酐(dextran,dex)又叫α右旋糖酐,是葡萄糖单元经脱水后所构成的高分子多糖化合物。微分子右旋糖酐能改善微循环,消除血管内红细胞聚集,防止血栓形成及渗透利尿的作用,可用于治疗急性失血性休克、心肌梗塞、脑血栓、脑供不足、周围血管病及防止弥散性血管内凝血和肾功能衰竭等功效。微分子量右旋糖酐主要存在以下缺陷:(1)目前双酶法或多酶法生产微分子量右旋糖酐成本高、纯化困难,需要两步才能完成(2)胞内表达的蛋白质易在菌体内形成包涵体,且纯化前还需要经过耗时且成本较高的细胞破碎步骤。因此,构建右旋糖酐蔗糖酶突变体的大肠杆菌高效分泌表达菌株能一步催化生产微分子量右旋糖酐,同时解决该酶胞内表达时存在的问题。
发明内容
本发明通过对右旋糖酐蔗糖酶氨基酸序列进行突变改造,再在其编码基因上连接分泌表达信号肽,以利于其在大肠杆菌中实现分泌表达,从而高效快速地获得可生产单一分子量(4000~5000)右旋糖酐的右旋糖酐蔗糖酶。
本发明的第一个目的是提供一种右旋糖酐蔗糖酶,具体为一种催化生产微分子量右旋糖酐的右旋糖酐蔗糖酶突变体DexT1,其氨基酸序列如SEQ ID NO.1所示。
本发明的第二个目的是提供上述右旋糖酐蔗糖酶的编码基因,所述编码基因的核苷酸序列如SEQ ID NO.2所示。
本发明的第三个目的是提供上述右旋糖酐蔗糖酶的编码基因的分泌表达信号肽,所述分泌表达信号肽氨基酸序列如SEQ ID NO.3所示,所述信号肽编码基因的核苷酸序列如SEQ ID NO.4所示。
本发明的第四目的是提供一种含有上述右旋糖酐蔗糖酶编码基因以及信号肽编码基因的重组载体。
优选的,所述重组载体所用的表达载体为pET28a(+)。
本发明的第五目的是提供一种基因工程菌,以大肠杆菌BL21为宿主细胞,通过载体表达连接上述右旋糖酐蔗糖酶编码基因以及信号肽编码基因。
本发明的第六目的是提供上述右旋糖酐蔗糖酶的制备方法,步骤如下:将右旋糖酐蔗糖酶及其分泌表达信号肽的编码基因和表达载体pET28a(+)相连接,并将连接产物转化至大肠杆菌BL21,获得重组菌株;培养重组菌株,诱导重组突变内右旋糖酐蔗糖酶的表达;离心获得含权利要求1所述右旋糖酐蔗糖酶的酶液;对酶进行活性测定。
本发明的有益技术效果是:与一般右旋糖酐蔗糖酶相比,右旋糖酐蔗糖酶突变体DexT1可以直接一步酶催化生产分子量在4000~5000之间的右旋糖酐,且催化较为稳定,分子量分布较为集中,免去了先催化生成高分子量右旋糖酐再水解生产微分子量右旋糖酐的步骤,同时在右旋糖酐蔗糖酶的N端加入DsbAT信号肽实现了右旋糖酐蔗糖酶在胞外的高效分泌,。本发明的突变右旋糖酐蔗糖酶DexT1可应用于药物行业等生物技术领域。
附图说明
图1 DsbAT-DexT1的PCR扩增结果。
图2工程菌pET28a-DsbAT-DexT1菌落PCR结果
图3质粒pET28a-DsbAT-DexT1的构建。
图4质粒pET28a-DsbAT-DexT1双酶切鉴定结果。
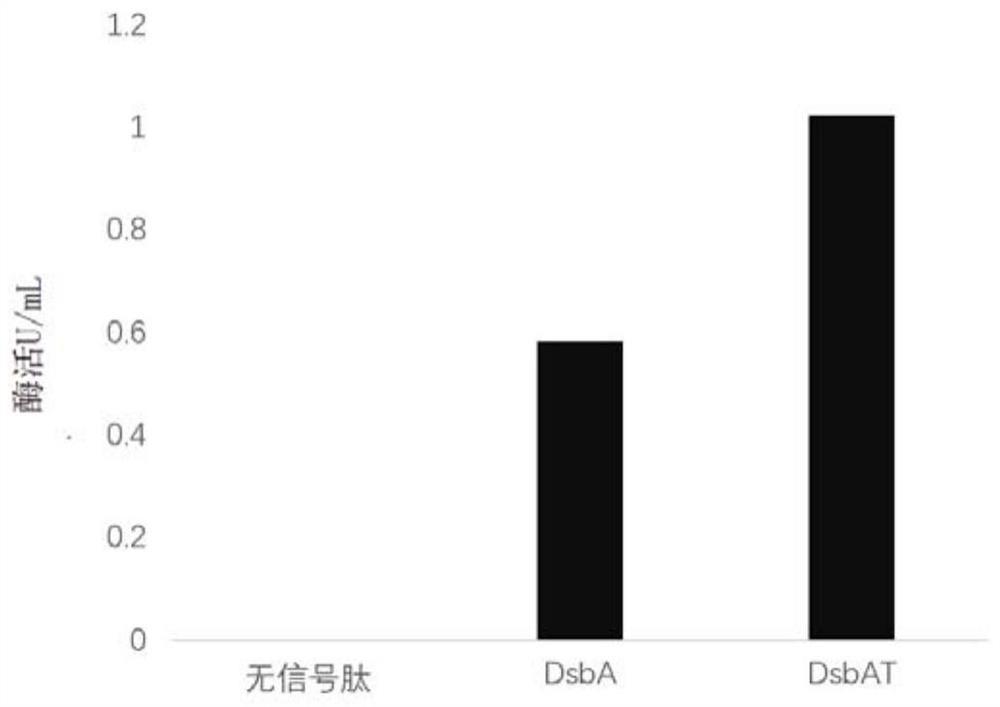
图5发酵上清液酶活检测。
图6产物右旋糖酐的GPC图谱。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行完整的描述,显然,所描述的实施例仅是本发明一部分实施例。基于本发明的实施例,本领域的普通技术人员在没有作出创造性劳动的前提下所获得的所有其他实施例,都属于本发明的保护范围。
实验试剂和材料
1.菌株及载体:大肠杆菌E.coli BL21(DE3)和表达载体pET28a由南京工业大学提供,该材料也可通过商业途径购买。
2.酶类及其他生化试剂:DNA聚合酶及dNTP购自南京诺唯赞生物科技股份有限公司,其他都为国产试剂(均可从普通生化试剂公司买到)
3.培养基:LB培养基:Peptone 10g/L,Yeast extract 5g/L,NaCl 10g/L,蒸馏水溶解。固体培养基在此基础上加2%(w/v)琼脂。
说明:以下实施例中未作具体说明的分子生物学实验方法,均参照《分子克隆实验指南》(第三版)一书中所列具体方法实行,或按照试剂盒和产品说明书进行。
实施例1所述右旋糖酐蔗糖酶的编码基因的分泌表达信号肽
本发明中所提供的的信号肽其核苷酸序列如SEQ ID NO.4所示,其是对信号肽DsbA信号肽进行改造而得来。该信号肽的N区使用的是信号肽DsbA的前三个氨基酸残基MKKK,中间H区序列为DsbA的疏水序列穿插强疏水性氨基酸亮氨酸和异亮氨酸IWLLALGLVLALAFI,C区使用的是DsbA的后四个氨基酸残基SASA。此信号肽序列通过化学合成或PCR的方法获得,经过signalP4.1对其可用性进行分析。
此信号肽片段长度较短,故通过引物自连合成得到该片段,同时在下游引物中加入DexT1的同源臂序列,反应条件:预变性95℃,3min;变性95℃,15s,退火63℃,15s;延伸72℃,15s;33个循环;彻底延伸72℃,5min。DexT1胞外酶活达到1U/ml,相对于原始出发菌种效果明显。
扩增体系:
DsbAT上游引物:5’-ATGGATGAAAAAAAAAATTTGGCTGCTGGCTCTGGGTCTGGTACTGGCC-3’
DsbAT下游引物:
5’-GGCTTTTTCAAAGCTTGCCATTGCGCTTGCGGAGATGAACGCCAGGGCCAGTACCAGACCC-3’
实施例2本发明所述右旋糖酐蔗糖酶的构建
以来源于柑橘明串珠菌NRRL B-1299的基因组为模板,设计含有EcoRI和SalI的突变体引物,利用overlap PCR扩增得到含突变基因的质粒。PCR扩增产物经1%琼脂糖凝胶电泳检验测定后回收。对质粒pET28a用EcoRI和SalI进行双酶切线性化处理,并纯化回收。PCR产物和线性化的质粒用一步克隆的方法做连接反应,体系如下:DexT1纯化产物3μL,线性化pET28a质粒4μL,CE buffer4μL,ExnaseII2μL,去离子水7μL,于37℃下反应30分钟,构建重组质粒pET28a-DexT1。取所得纯化产物10μL与大肠杆菌DH5α感受态混合,置于冰上30min,于42℃下热击90s,迅速置于冰上5min。将上步所得的感受态细胞加入至900μL LB培养基中,在37℃180rpm下活化50min。12000rpm离心1min,弃部分上清,留100μL左右活化后感受态细胞μL均匀涂布于含有100mg/L的卡那霉素LB固体培养基平板上,37℃培养箱静置过夜,挑取平板上的单菌落接种于5mL LB培养基中,于37℃180rpm条件下过夜培养,经基因测序确定扩增结果后提取质粒备用。
DexT1上游引物:5’-ATGGGTCGCGGATCCGAATTCATGGCAAGCTTTGAAAAAGC-3’
DexT1下游引物:
5’-TGCGGCCGCAAGCTTGTCGACTTTTGCCATCGTACCATCGTT-3’
实施例3右旋糖酐蔗糖酶突变体的表达
将DsbAT片段与DexT1片段通过overlap进行连接。以前期构建的DexT1-pET28a质粒为模板进行PCR扩增得到DexT1片段。反应条件:预变性95℃,3min;变性95℃,15s,退火52℃,15s;延伸72℃,4min;33个循环;彻底延伸72℃,5min。扩增体系:
DexT1片段引物设计中,在下游引物加入带有SalI的同源臂。
DsbAT上游引物:
5’-ATGGGTCGCGGATCCGAATTCATGGGTCGCGGATCCGAATTC-3’
DexT1下游引物:
5’-TGCGGCCGCAAGCTTGTCGACTTTTGCCATCGTACCATCGTT-3’
PCR扩增产物与线性化载体质粒用一步克隆方法进行连接反应,体系如下:DsbAT-DexT1纯化产物3μL,线性化pET28a质粒4μL,CE buffer 4μL,ExnaseII2μL,去离子水7μL,于37℃下反应30分钟,构建重组质粒pET28a-DsbAT-DexT1。取所得纯化产物10μL与大肠杆菌DH5α感受态混合,置于冰上30min,于42℃下热击90s,迅速置于冰上5min。将上步所得的感受态细胞加入至900μL LB培养基中,在37℃180rpm下活化50min。12000rpm离心1min,弃部分上清,留100μL左右活化后感受态细胞μL均匀涂布于含有100mg/L的卡那霉素LB固体培养基平板上,37℃培养箱静置过夜,挑取平板上的单菌落接种于5mL LB培养基中,于37℃180rpm条件下过夜培养。
提取上述构建的分泌表达质粒pET28a-DsbAT-DexT1,进行PCR验证,最后在37℃下使用DNA限制性内切酶EcoRI和SalI对pET28a-DsbAT-DexT1分泌表达质粒进行双酶切,酶切5h后对酶切产物进行核酸电泳检测确认。随后将PCR产物进行纯化,使用无菌水进行最终洗脱,将纯化产物化转至大肠杆菌BL21(DE3)中,转化液涂布于LB平板中37℃培养12h,长出的单菌落为表达菌株。挑取单菌落接种至5mL LB培养基中37℃200rpm下培养12h,再进一步接种到200mL LB培养基中扩大培养,当菌液OD600长至0.6~0.8时,加入0.1mM IPTG诱导培养24h。
实施例4右旋糖酐蔗糖酶突变体表达酶活性的测定
发酵结束后,离心取上清液,取1mL和测量酶活用的酶活反应体系2.5mL混合反应30min,反应后加入定量DNS试剂,煮沸5min,使用分光光度计在540nm波长下测量,代入果糖标准曲线计算反应后果糖含量,判断酶活。
酶活反应体系为:乙酸钠1.46g/L,氯化钙0.05g/L,蔗糖120g/L,用20%盐酸调节pH至5.5。
酶活力的定义:在一定条件下,每分钟催化1umol蔗糖反应生成果糖所需要的酶量为1个酶活单位,用1U表示。酶活检测结果如图5所示。
实施案例5酶催化产物分子量变化的测定
将离心后上清以1:4的体积比与蔗糖反应体系混合,蔗糖反应体系配制方法为:蔗糖240g/L,乙酸钠0.73g/L,氯化钙0.025g/L,体系pH为5.5。在30℃下反应2h。反应结束后使用凝胶渗透色谱法(GPC)测量溶液中右旋糖酐的平均分子量,利用HPLC,以ΜLtrahydrogel250Column为色谱柱配备示差折光检测器检测,ddH
序列表
<110> 南京工业大学
<120> 一种右旋糖酐蔗糖酶及其制备方法和应用
<160> 4
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1267
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 1
Phe Glu Lys Ala Pro Asp Ser Val Ile Thr Gly Gly Arg Tyr Ser Leu
1 5 10 15
Lys Asp Gly Tyr Tyr Val Tyr Leu Asp Lys Gln Gly Lys Gln Val Val
20 25 30
Gly Pro Lys Asn Ile Asp Asn His Leu Gln Tyr Phe Asp Glu Thr Thr
35 40 45
Gly Lys Gln Val Lys Gly Asp Phe Arg Ser Val Asn Gly Lys Arg Ile
50 55 60
Tyr Phe Asn Ala Asn Leu Gly Tyr Ala Asp Asp Tyr Thr Thr Asp Val
65 70 75 80
Ala Gly Lys Leu Val Gly Tyr Asp Ser Asn Gly Asn Gln Val Lys Ala
85 90 95
Gly Tyr Val Thr Asn Ser Gln Gly Lys Thr Tyr Tyr Phe Asn Asn Gln
100 105 110
Gly Glu Ala Ile Ile Gly Leu Lys Thr Asp Asn Asn Lys Thr Gln Tyr
115 120 125
Phe Gly Pro Asp Gly Ala Gln Val Lys Gly Ala Phe Gln Gln Val Asn
130 135 140
Gly Lys Asn Ile Tyr Phe Asp Ala Gln Thr Gly Tyr Ala Arg Gln Asn
145 150 155 160
Val Gly Phe Leu Asp Gly Thr Ala Lys Gly Phe Asp Glu Gln Gly Asn
165 170 175
Gln Ile Lys Ser Gly Ile Ala Thr Asp Leu Ser Gly Asn Val Tyr Tyr
180 185 190
Phe Asp Ala Ser Gly Lys Met Leu Thr Gly Val Gln Asn Ile Asp Gly
195 200 205
Lys Lys Tyr Tyr Phe Asp Glu Gln Gly His Arg Arg Arg Asn Tyr Ala
210 215 220
Gly Val Phe Asn Asn Glu Phe Ile Tyr Phe Gly Leu Asp Gly Val Gly
225 230 235 240
Gln Ser Ala Ile Glu Tyr Gln Phe Glu Lys Gly Leu Thr Ser Gln Asn
245 250 255
Ser Val Ala Thr Ser His Asn Ala Ala Lys Ser Tyr Asp Thr Lys Ser
260 265 270
Phe Thr Asn Val Asp Gly Phe Leu Thr Ala Asn Ser Trp Tyr Arg Pro
275 280 285
Thr Asp Ile Leu Arg Asn Gly Thr Lys Trp Glu Pro Ser Thr Glu Thr
290 295 300
Asp Phe Arg Pro Leu Leu Met Thr Trp Trp Pro Asp Lys Glu Val Gln
305 310 315 320
Ala Asn Tyr Leu Asn Tyr Met Ser Ala Leu Gly Leu Gly Asp Gln Lys
325 330 335
Ile Tyr Thr Gly Ala Ser Ser Gln Leu Asp Leu Asn Asn Ala Ala Leu
340 345 350
Ile Val Gln Glu Ala Ile Glu Lys Lys Ile Ser Leu Glu Lys Ser Thr
355 360 365
Lys Trp Leu Asp Asp Ser Ile Lys Ser Phe Ile Lys Ser Lys Arg Lys
370 375 380
Asp Ile Gln Gly Asn Leu Val Asp Thr Asn Pro Gly Trp Thr Ile Asp
385 390 395 400
Ser Glu Thr Gly Ser Thr Asn His Leu Gln Asn Gly Ala Phe Ile Phe
405 410 415
Thr Asn Ser Pro Leu Val Pro Glu Ala Asn Ala Ala Glu Gly Asn Arg
420 425 430
Leu Ile Asn Arg Thr Pro Ser Gln Gln Thr Gly Asn His Ile Ser Tyr
435 440 445
Ala Ser Gln Pro Tyr Ser Gly Asp Asp Trp Gly Tyr Glu Leu Leu Leu
450 455 460
Gly Asn Asp Val Asp Asn Ser Asn Pro Ile Val Gln Ala Glu Gln Leu
465 470 475 480
Asn Trp Ile His Tyr Leu Met Asn Phe Gly Thr Ile Thr Ala Pro Gln
485 490 495
Asp Pro Asp Ala His Leu Ala Asn Phe Asp Ser Ile Arg Ile Asp Ala
500 505 510
Val Asp Asn Val Asp Ala Asp Leu Leu Gln Ile Ala Gly Asp Tyr Phe
515 520 525
Lys Ala Ala Tyr Gln Val Gly Glu Asn Asp Lys Asn Ala Asn Gln His
530 535 540
Ile His Ile Leu Glu Asp Trp Ser Pro Asn Asp Val Trp Tyr Asn Gln
545 550 555 560
Gln Val Asn Gly Asn Ser Gln Leu Thr Met Asp Ala Thr Met Gln Asn
565 570 575
Gln Leu Leu Ala Ser Leu Thr Arg Pro Ile Thr Ser Arg Asp Ser Met
580 585 590
Lys Ser Phe Thr Lys Asp Ala Leu Leu Val His Arg Thr Ala Asp Asn
595 600 605
Ser Tyr Asn Gln Ala Val Pro Asn Tyr Ser Phe Ile Arg Ala His Asp
610 615 620
Ser Glu Val Gln Thr Ile Ile Ala Lys Ile Ile Ser Asp Lys His Pro
625 630 635 640
Asp Leu Tyr Pro Thr Val Asp Lys Ala Leu Leu Ala Lys Asp Ser Ala
645 650 655
Leu Tyr Asp Glu Ala Phe Thr Glu Tyr Asn Ala Asp Met Gln Lys Ile
660 665 670
Ser Ser Gln Lys Gln Tyr Thr His Asn Asn Met Pro Ser Ala Tyr Ala
675 680 685
Ile Leu Leu Thr Asn Lys Asp Thr Val Pro Arg Val Tyr Tyr Gly Asp
690 695 700
Leu Phe Thr Asp Asn Gly Glu Tyr Met Ala Asn Lys Thr Pro Tyr Tyr
705 710 715 720
Asp Ala Ile Thr Ser Leu Leu Thr Ala Arg Thr Lys Phe Val Ser Gly
725 730 735
Gly Gln Ser Leu Ser Val Asp Lys Asn Asp Val Leu Thr Ser Val Arg
740 745 750
Tyr Gly Lys Gly Ala Leu Ser Ala Thr Asp Asn Gly Ser Ser Asp Thr
755 760 765
Arg Asn Gln Gly Ile Gly Val Ile Val Ser Asn Asn Pro Asn Leu Asp
770 775 780
Leu Asn Asn Asp Lys Val Thr Leu Ser Met Gly Ile Ser His Ala His
785 790 795 800
Gln Ala Tyr Arg Pro Leu Leu Leu Thr Asn Ser Gln Gly Ile Val Ala
805 810 815
Tyr Ala Thr Asp Ser Glu Val Pro Gln Asn Leu Tyr Lys Thr Thr Asn
820 825 830
Asp Lys Gly Glu Leu Thr Phe Asp Ala Ser Glu Ile Lys Gly Tyr Asp
835 840 845
Thr Val Gln Thr Ser Gly Tyr Leu Ala Val Trp Val Pro Val Gly Ala
850 855 860
Ser Asp Glu Gln Asp Ala Arg Thr Ile Ala Ser Thr Glu Lys Asn Asn
865 870 875 880
Gly Asn Ser Val Tyr His Ser Asn Ala Ala Leu Asp Ser Gln Leu Ile
885 890 895
Tyr Glu Gly Phe Ser Asn Phe Gln Thr Val Pro Ser Lys Asn Ala Ser
900 905 910
Ala Asp Glu Tyr Ala Asn Val Ile Ile Ala Lys His Ala Ala Asp Phe
915 920 925
Asn Lys Trp Gly Val Thr Ser Phe Gln Met Ala Pro Gln Tyr Arg Ser
930 935 940
Ser Thr Asp Gly Ser Phe Leu Asp Ala Val Asp Thr Val Gln Asn Gly
945 950 955 960
Tyr Ala Phe Thr Asp Arg Tyr Asp Leu Gly Phe Asn Ala Ala Asp Gly
965 970 975
Ser Lys Asn Pro Thr Lys Tyr Gly Thr Asp Glu Asp Leu Arg Asn Ala
980 985 990
Ile Lys Ser Leu His Ala Gln Lys Thr Tyr Asp Gly Ser Ser Ile Gln
995 1000 1005
Val Met Ala Asp Phe Val Pro Asp Gln Leu Tyr Asn Met Pro Leu Glu
1010 1015 1020
Gln Ala Val Ser Val Ile Arg Thr Asp Lys Tyr Gly Val Asn Ser Glu
1025 1030 1035 1040
Asn Pro Asp Ile Gln Asn Ile Ile Tyr Ala Ala Asn Ile Lys Ser Ser
1045 1050 1055
Gly Thr Asp Tyr Gln Ser Ile Tyr Gly Gly Lys Tyr Leu Ala Glu Leu
1060 1065 1070
Gln Lys Asn Pro Leu Phe Lys Ser Leu Phe Asp Arg Ile Gln Ile Ser
1075 1080 1085
Thr Lys Lys Thr Ile Asp Pro Asn Thr Arg Ile Thr Gln Trp Ser Ala
1090 1095 1100
Lys Tyr Phe Asn Gly Ser Asn Ile Gln Gly Lys Gly Ile Asn Tyr Val
1105 1110 1115 1120
Leu Lys Asp Trp Ala Ser Asn Lys Tyr Phe Asn Val Ser Ser Asn Asp
1125 1130 1135
Asp Met Tyr Ser Arg Leu Pro Lys Gln Leu Met Asn Gln Glu Ser Asn
1140 1145 1150
Thr Gly Phe Ile Val Asp Asp Ile Gly Val Lys Tyr Tyr Ser Ile Ser
1155 1160 1165
Gly Tyr Gln Ala Lys Asn Thr Phe Val Glu Asp Gly Asn Gly Glu Trp
1170 1175 1180
Tyr Tyr Phe Asp Asn Asp Gly Tyr Met Val Lys Ser Thr Glu Glu Ser
1185 1190 1195 1200
Gly Pro Leu Arg Thr Val Asn Ala Ser Ser Lys Lys Tyr Tyr Ile Leu
1205 1210 1215
Pro Asn Gly Val Glu Ile Arg Asn Ser Phe Gly Gln Asp Ile Gln Gly
1220 1225 1230
Asn Thr Tyr Tyr Phe Asp Ala Arg Gly Glu Met Val Thr Ser Gln Tyr
1235 1240 1245
Ile Ser Asp Asp Thr Gln Asn Ile Tyr Tyr Phe Asn Asn Asp Gly Thr
1250 1255 1260
Met Ala Lys
1265
<210> 2
<211> 3810
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
tttgaaaaag cgccggattc tgtccctgaa actatcactg gcggccgcta ttccctgaag 60
gacggctact acgtttacct ggataaacaa ggtaaacagg tggtgggtcc gaaaaacatc 120
gacaaccatc tgcagtactt cgacgaaacc actggtaaac aggttaaagg cgacttccgc 180
tccgttaacg gtaaacgtat ctattttaac gcgaacctgg gctacgcgga cgactacact 240
accgacgttg cgggcaagct ggtgggctac gacagcaacg gtaaccaggt gaaagcaggc 300
tacgtgacca actctcaggg taaaacctac tactttaaca accagggtga agctatcatt 360
ggcctgaaga ccgataacaa caaaactcag tattttggtc cggacggtgc gcaggttaaa 420
ggtgcattcc agcaggtaaa cggcaaaaac atttacttcg acgctcaaac cggctacgcg 480
cgtcagaacg ttggcttcct ggacggtacg gcaaagggtt ttgacgaaca gggcaaccag 540
atcaaatctg gtattgcgac ggacctgtcc ggtaacgttt attatttcga tgcttccggt 600
aagatgctga ctggtgtgca gaacatcgac ggcaaaaaat attacttcga cgaacaaggt 660
caccgccgtc gtaactatgc aggtgttttc aacaacgaat tcatctattt cggtctggat 720
ggtgttggcc agtccgccat cgaataccag ttcgaaaaag gtctgaccag ccagaacagc 780
gtggccacta gccacaacgc cgctaagtcc tatgatacca aatccttcac caatgttgat 840
ggcttcctga ccgccaactc ctggtaccgt ccgaccgata tcctgcgtaa cggcactaaa 900
tgggaaccgt ctactgagac cgattttcgt ccgctgctga tgacttggtg gccggataaa 960
gaagtccagg cgaactatct gaactacatg agcgcgctgg gcctgggtga tcagaaaatc 1020
tacaccggtg catcttccca gctggacctg aacaacgctg ctctgatcgt ccaggaagcc 1080
atcgagaaaa aaatctctct ggaaaagagc accaagtggc tggacgacag catcaaatct 1140
ttcattaaaa gcaaacgtaa ggacattcag ggtaacctgg tagatactaa tccgggctgg 1200
accatcgact ccgaaactgg ctctactaac cacctgcaga acggtgcttt catctttacc 1260
aactctccgc tggtgcctga agctaacgcc gctgaaggta atcgtctgat taaccgtacc 1320
ccgtctcagc agactggtaa ccacatcagc tacgcttctc agccgtactc cggtgatgat 1380
tggggctacg aactgctgct gggcaatgac gtggataaca gcaacccgat cgttcaggcc 1440
gagcagctga actggattca ctacctgatg aacttcggca ccatcacggc accacaggat 1500
cctgatgcgc acctggccaa cttcgacagc attcgtatcg acgcagtgga taacgttgat 1560
gcggatctgc tgcagatcgc aggtgattac ttcaaagctg cttaccaggt aggtgaaaac 1620
gacaaaaacg ccaaccaaca cattcacatc ctggaagatt ggtctccgaa cgacgtctgg 1680
tacaaccagc aagtaaacgg caattctcag ctgaccatgg acgcaaccat gcagaaccag 1740
ctgctggcct ccctgacgcg tccgatcact tctcgtgaca gcatgaagtc ttttactaaa 1800
gatgccctgc tggtccaccg taccgcagac aactcctaca accaggcagt tccgaactac 1860
agctttatcc gcgcccacga ttccgaagtt cagacgatta tcgctaaaat tatcagcgac 1920
aaacatccgg acctgtaccc gaccgtggac aaagctctgc tggcgaaaga ctccgctctg 1980
tacgatgaag ctttcaccga gtataacgcg gatatgcaga aaatcagcag ccagaaacag 2040
tacacccata ataacatgcc ttctgcttac gcgatcctgc tgactaacaa ggacaccgtc 2100
ccgcgtgtat actacggtga cctgttcact gataacggtg aatatatggc caacaaaacc 2160
ccttactacg atgccattac tagcctgctg acggcgcgta ccaaattcgt tagcggcggt 2220
cagtccctga gcgtggataa aaatgacgtt ctgacctctg tccgttacgg taaaggtgca 2280
ctgtctgcga ccgacaacgg ttcctccgat acccgtaacc agggcatcgg tgtaattgtt 2340
tccaacaacc caaacctgga cctgaacaac gacaaagtta ccctgtctat gggtatctct 2400
cacgcgcatc aggcctatcg tccgctgctg ctgaccaact ctcagggcat tgtggcctac 2460
gcaaccgatt ccgaagtgcc gcagaacctg tacaaaacca cgaacgacaa aggcgaactg 2520
accttcgatg cctccgaaat caaaggctac gatacggtcc agacgtctgg ctatctggca 2580
gtttgggtcc cagtgggcgc ttctgacgaa caggacgcac gcactattgc atccacggaa 2640
aaaaacaacg gcaatagcgt ttaccactct aacgcggctc tggatagcca actgatttac 2700
gaaggtttca gcaacttcca gaccgttccg tctaaaaacg cgagcgcgga cgaatacgca 2760
aacgttatca ttgcgaaaca tgctgcggac ttcaataaat ggggtgttac cagcttccag 2820
atggcaccgc agtaccgttc tagcaccgac ggctctttcc tggacgccgt agatactgtt 2880
cagaacggct acgcgttcac cgatcgttac gacctgggct tcaacgcggc ggatggttcc 2940
aaaaacccga ctaaatacgg cacggacgag gacctgcgta acgcaattaa atctctgcat 3000
gcgcagaaaa cttatgatgg ctctagcatc caggtcatgg ctgattttgt tccggaccag 3060
ctgtacaaca tgccgctgga acaggcagtg tctgtgatcc gcaccgataa gtacggcgtt 3120
aattccgaga acccggacat tcagaatatc atttacgcag ctaatatcaa aagctccggt 3180
actgactacc agagcattta cggtggcaaa tacctggcag aactgcagaa aaacccgctg 3240
ttcaagtccc tgttcgatcg tattcagatt agcactaaaa aaaccatcga ccctaatact 3300
cgcattaccc agtggtccgc gaagtacttc aacggcagca atatccaggg caagggtatc 3360
aattacgttc tgaaagactg ggctagcaac aaatatttca atgttagctc taacgacgac 3420
atgtatagcc gtctgccgaa acaactgatg aaccaggagt ccaacactgg cttcatcgtg 3480
gacgacattg gcgtaaaata ttactctatt agcggttacc aagcgaaaaa caccttcgtg 3540
gaggatggca acggtgaatg gtactacttc gacaatgatg gttacatggt aaaatctacc 3600
gaagaatctg gtccactgcg taccgttaac gcctctagca aaaaatacta catcctgcca 3660
aacggtgttg aaatccgtaa ctctttcggc caggacatcc agggtaacac ttattacttc 3720
gatgcacgcg gcgagatggt gacttcccag tacatttctg acgatacgca gaacatctac 3780
tacttcaaca acgacggtac catggctaaa 3810
<210> 3
<211> 23
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 3
Met Lys Lys Lys Ile Trp Leu Leu Ala Leu Gly Leu Val Leu Ala Leu
1 5 10 15
Ala Phe Ile Ser Ala Ser Ala
20
<210> 4
<211> 69
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
atgaaaaaaa agatctggct gctggctctg ggcctggttc tggcgctggc cttcatcagc 60
gcttccgcg 69
- 一种耐热右旋糖酐蔗糖酶突变体及其制备方法与应用
- 一种右旋糖酐蔗糖酶突变体及其制备方法与应用
