测量低聚半乳糖的方法
文献发布时间:2024-04-18 19:58:53
技术领域
本发明涉及一种在制备具有高GOS纤维含量的乳制品和/或其中乳糖含量也显著降低的富含GOS纤维的乳制品的同时进行低聚半乳糖(GOS)定量的在线(in-line)方法。
背景技术
低聚半乳糖(GOS)是定义为具有通过糖苷键连接的两个或更多个半乳糖部分(包括多达九个)的碳水化合物。人和动物无法消化GOS。GOS还可以包括一个或多个非半乳糖糖部分,包括葡萄糖。摄入GOS的有益作用之一是它能够充当益生元,通过选择性地刺激有益的结肠微生物(例如细菌)的增殖来提供生理益处。已确定的健康作用使得对作为食物的GOS的兴趣日益增加。
β-半乳糖苷酶(EC 3.2.1.23)可以催化两种类型的反应。对于大多数β-半乳糖苷酶,将乳糖水解为单糖D-葡萄糖和D-半乳糖是优选的反应。在该反应的催化过程中,该酶水解乳糖并瞬时结合半乳糖-酶复合物中的半乳糖单糖,该半乳糖-酶复合物将半乳糖转移到水的羟基基团上,从而释放D-半乳糖和D-葡萄糖。然而,在某些条件下,例如高乳糖浓度和/或高温,β-半乳糖苷酶可以将半乳糖转移到D-半乳糖或D-葡萄糖的羟基基团上。这种反应称为转半乳糖基化。转半乳糖基化的主要产物是GOS。
已经开发了用于产生高水平GOS的酶和方法。参见,例如,具有转半乳糖基化活性的多肽,WO 2013/182686。另外,已经开发了产生高水平GOS同时还进一步减少剩余乳糖的方法。参见,例如,使用乳糖酶提供高GOS纤维水平和低乳糖水平,WO 2020/117548。在酸奶、奶酪、奶饮料和奶粉等奶基产品的乳品应用的上下文中,虽然GOS的产生会减少内源性乳糖,但对于乳糖不耐症个体来说,乳糖水平可能仍然过高。据估计,世界上约70%的人口患有乳糖不耐症,即如果他们摄入乳糖,就会患上消化系统障碍。
如上所述,某些β-半乳糖苷酶在正确的条件下可以驱动乳糖转化为GOS。然而,这些相同的酶在正确的条件下可以催化GOS水解为葡萄糖和半乳糖(取决于GOS的组成)。通常,这发生在GOS形成峰值时或接近峰值时。GOS水解可以通过酶灭活(例如通过加热)来防止。然而,了解何时灭活需要关于转半乳糖基化程度的可靠且准确的信息,特别是何时达到最佳GOS浓度。
本领域持续需要测定最佳GOS产率的方法。在工业乳品环境中尤其如此,在该环境中,奶生产商在短时间内处理数十万升奶,并且没有先进的实验室设备,也没有经过训练的现场(on premises)化学家。
发明内容
提出了一种用于测定样品中碳水化合物含量的方法,该方法具有以下步骤:获得对应于该样品的FTIR(傅里叶变换红外光谱术)光谱数据;提供该FTIR光谱数据的至少一部分作为经过训练的机器学习模型的输入;以及使用该经过训练的机器学习模型处理该FTIR光谱数据的至少一部分,以生成碳水化合物含量值,从而提供该样品中碳水化合物含量水平的定量指示。
任选地,碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。任选地,DP2是乳糖。任选地,碳水化合物是DP3+GOS。任选地,样品是奶基底物。任选地,作为经过训练的机器学习模型的输入提供的FTIR光谱数据部分是有限光谱范围内的FTIR光谱数据。任选地,有限光谱范围包括1046cm
任选地,经过训练的机器学习模型是使用训练数据集训练的监督学习模型,对于多个训练样品中的每一个,该训练数据集具有对应于该训练样品的FTIR光谱数据和该训练样品中碳水化合物含量水平的测量指示。
任选地,经过训练的机器学习模型是偏最小二乘回归(PLSR)模型。
任选地,经过训练的机器学习模型是神经网络回归模型。
任选地,经过训练的机器学习模型包括多元线性回归(MLR)。
任选地,经过训练的机器学习模型是主成分回归(PCR)。
任选地,经过训练的机器学习模型包括经典最小二乘法CLS。
任选地,经过训练的机器学习模型包括决策树算法。
任选地,FTIR光谱数据是从基于服务器的数据存储中获得的,该FTIR光谱数据由客户端设备上传到该数据存储中。
任选地,使用经过训练的机器学习模型对FTIR光谱数据的至少一部分进行的处理是使用从客户端设备获得的FTIR光谱数据在服务器设备上进行的。
任选地,碳水化合物含量值可供客户端设备访问。
提出了一种用于训练机器学习模型以预测奶基底物中碳水化合物含量的方法,该方法具有以下步骤:获得训练数据集,对于多个训练样品中的每一个,该训练数据集具有对应于该训练样品的FTIR(傅里叶变换红外光谱术)光谱数据和该训练样品中碳水化合物含量水平的测量指示;以及使用该训练数据集进行监督学习,以确定该机器学习模型的经过训练的模型系数。
任选地,碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。任选地,DP2是乳糖。任选地,碳水化合物是DP3+GOS。
在本发明的另一方面,提出了一种计算机程序,当在数据处理装置上执行时,该计算机程序控制该数据处理装置以进行上述方法。
在本发明的另一方面,提出了一种用于制备含有碳水化合物的奶产品的方法,该方法具有以下步骤:用转半乳糖基化酶处理奶基底物;对该奶基底物的样品进行FTIR(傅里叶变换红外光谱术),以获得对应于该样品的FTIR光谱数据;基于使用经过训练的机器学习模型对该FTIR光谱数据的至少一部分进行的处理,获得碳水化合物含量值,从而提供该样品中碳水化合物含量水平的定量指示;以及基于该碳水化合物含量值,确定何时通过对该奶基进行巴氏杀菌来灭活该转半乳糖基化酶。
任选地,碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。任选地,DP2是乳糖。任选地,碳水化合物是DP3+GOS。
29.
任选地,FTIR光谱数据的至少一部分由客户端设备上传到服务器,用于由经过训练的机器学习模型进行基于服务器的处理以生成碳水化合物含量值,并且碳水化合物含量值由客户端设备作为基于服务器的处理的结果而获得。
任选地,用于制备奶基碳水化合物的方法具有优于10%的准确度,表示为平均值的预测标准误差(3.75%),在含有至少0.1%脂肪、至少0.5%溶解乳糖和至少1%蛋白质的奶基中,GOS的浓度在0-7.5%的浓度范围内。
任选地,用于制备奶基碳水化合物的方法具有高于0.9(不太优选的是0.85、0.8、0.75等)的PLS回归模型的线性度(R2),以验证GOS含量。
任选地,转半乳糖基化酶衍生自两歧双歧杆菌(Bifidobacterium bifidum)。任选地,转半乳糖基化酶是来自两歧双歧杆菌的截短的β-半乳糖苷酶。任选地,来自两歧双歧杆菌的截短的β-半乳糖苷酶在C末端被截短。任选地,来自两歧双歧杆菌的截短的β-半乳糖苷酶是与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少70%序列同一性的多肽。
任选地,该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQID NO:5或其转半乳糖基酶活性片段具有至少80%序列同一性。任选地,该多肽与SEQID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少90%序列同一性。任选地,该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少95%序列同一性。任选地,该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少99%序列同一性。任选地,该多肽是根据SEQ ID.NO:1、SEQID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段的序列。任选地,该多肽是根据SEQ ID NO:1的序列。
附图说明
图1:通过珀金埃尔默公司(Perkin Elmer)的Spectrum One FTIR仪器在波数范围650-4400cm
图2.预测误差(RMSECV(+),RMSC(o))相对于模型#1中使用的PLS成分/潜在变量的数量作图。
图3:模型系数:模型#1中DP3+(Y1)预测的回归向量作为波数(cm-1)的函数。
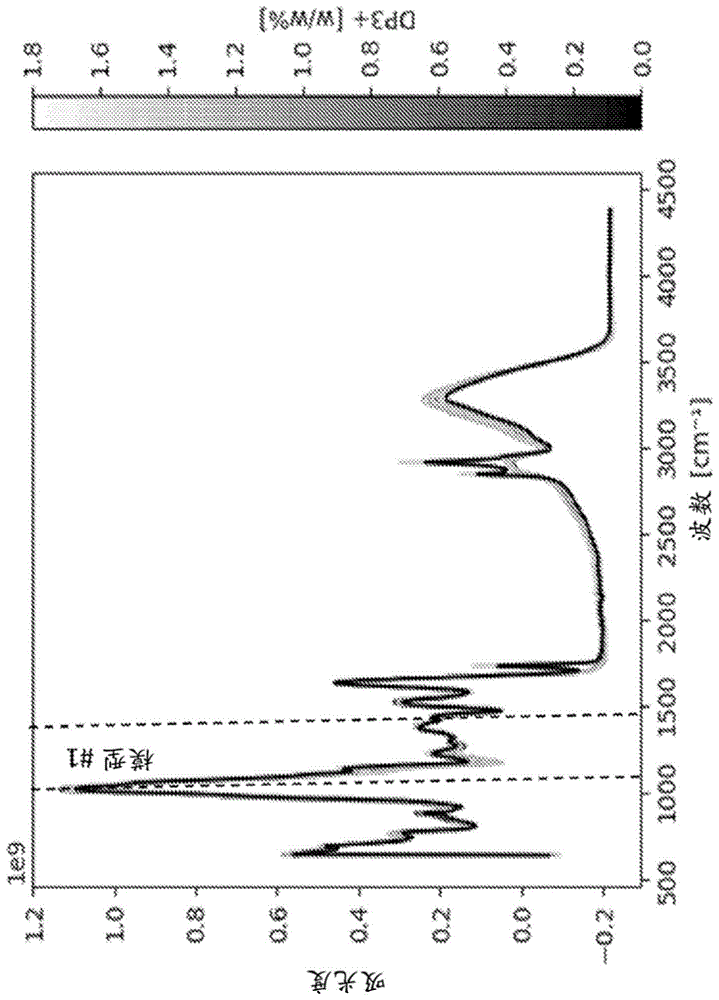
图4:通过MilkoScan FT2仪器分析的476个(119×4个重复)含GOS的奶样品的光谱数据,以中红外吸光度作为波数的函数表示。颜色条根据HPLC测量的GOS(%(w/w)DP3+)水平进行着色,如第二轴上以%所示。
图5:通过MilkoScan FT2仪器分析的476个(119×4个重复)含GOS(DP3+)的奶样品的光谱数据的放大区域(波数范围:1038cm
图6:DP3+的模型预测(以w/w%为单位)相对于测量的DP3+(以w/w%为单位)。来自训练集的数据为黑色,来自测试集的数据为灰色。灰线表示直线对测量值与预测值的最小二乘回归拟合,而灰线表示预测值等于测量值的完美模型。
图7:模型#1范围内训练数据集的平均吸光度光谱(左侧轴),以及模型#1的模型系数(右侧轴)。
具体实施方式
本发明的详细说明:
除非另有说明,否则本发明教导的实践将使用在本领域技术范围内的分子生物学(包括重组技术)、微生物学、细胞生物学和生物化学的常规技术。此类技术在以下文献中得到充分解释,例如,
除非在本文中另有定义,否则本文所用的全部技术术语和科学术语具有与本发明教导所属领域的普通技术人员通常所理解的相同含义。Singleton等人,
本文提供的数值范围包括限定该范围的数值在内。
定义:
“β-半乳糖苷酶”是催化β-半乳糖苷酶(包括乳糖)水解为单糖的糖苷水解酶。β-半乳糖苷酶有时也称为“乳糖酶”。
术语“低聚半乳糖”在本文中也称为“GOS”,是指由2至20个主要为半乳糖分子组成的不可消化的低聚糖。GOS通常由β-半乳糖苷酶(也称为乳糖酶)通过降解例如奶和/或奶基产品中的乳糖而形成。
术语“GOS纤维”等同于本文中的“DP3+GOS”,是指具有3个或更多个主要为半乳糖分子的聚合度的不可消化的低聚半乳糖。
本文中的DP2是指二糖的种类,包括乳糖、GOS二糖(例如别乳糖)。
关于多肽,术语“野生型”、“亲本”或“参照”是指在一个或多个氨基酸位置处不包含人为取代、插入或缺失的天然存在的多肽。类似地,关于多核苷酸,术语“野生型”、“亲本”或“参照”是指不包含人为核苷酸变化的天然存在的多核苷酸。然而,注意编码野生型、亲本、或参照多肽的多核苷酸不限于天然存在的多核苷酸,并且涵盖编码野生型、亲本、或参照多肽的任何多核苷酸。
对野生型多肽的提及应理解为包括多肽的成熟形式。“成熟”多肽或其变体是其中不存在信号序列的多肽或变体,例如,在多肽表达期间或之后从未成熟形式的多肽切割。
关于多肽,术语“变体”是指与指定的野生型、亲本或参照多肽不同的多肽,因为它包括一个或多个天然存在的或人为的氨基酸取代、插入或缺失。类似地,关于多核苷酸,术语“变体”是指在核苷酸序列方面与指定的野生型、亲本或参照多核苷酸不同的多核苷酸。野生型、亲本或参照多肽或多核苷酸的特性将从上下文中显而易见。
术语“重组”当用于提及主题细胞、核酸、蛋白质或载体时,表明受试者已经从其天然状态被修饰。因此,例如,重组细胞表达在天然(非重组)形式的细胞内没有发现的基因,或者以不同于在自然界发现的水平或在不同于在自然界发现的条件下表达天然基因。重组核酸与天然序列相差一个或多个核苷酸,和/或可操作地连接到异源序列,例如,表达载体中的异源启动子。重组蛋白可以与天然序列相差一个或多个氨基酸,和/或与异源序列融合。包含编码β-半乳糖苷酶的核酸的载体是重组载体。
术语“回收的”、“分离的”和“单独的”是指从自然界发现的与其天然相关的至少一种其他材料或成分中除去的化合物、蛋白质(多肽)、细胞、核酸、氨基酸、或者其他指定的材料或成分。其“分离的”多肽包括但不限于含有在异源宿主细胞中表达的分泌多肽的培养液。
术语“聚合物”是指连接在一起的一系列单体基团。聚合物由单个单体的多个单元组成。如本文所使用的术语“葡萄糖聚合物”是指作为聚合物连接在一起的葡萄糖单元。只要存在至少三个葡萄糖单元,葡萄糖聚合物就可以含有非葡萄糖的糖,例如乳糖或半乳糖。
术语“氨基酸序列”与术语“多肽”、“蛋白质”和“肽”同义,并且可互换地使用。当此类氨基酸序列显示出活性时,它们可以被称为“酶”。使用针对氨基酸残基的常规单字母或三字母密码,其中氨基酸序列以标准氨基端-至-羧基端取向(即N→C)呈现。
术语“核酸”涵盖能够编码多肽的DNA、RNA、异源双链体、以及合成分子。核酸可以是单链的或双链的,并且可以被化学修饰。术语“核酸”和“多核苷酸”可互换地使用。由于遗传密码是简并的,因此可以使用多于一个密码子来编码特定氨基酸,并且本发明的组合物和方法涵盖编码特定氨基酸序列的核苷酸序列。除非另有说明,否则核酸序列以5′-至-3′取向呈现。
关于细胞使用的术语“转化”、“稳定转化”和“转基因”意指细胞包含整合到其基因组中或作为通过多代维系的附加体而携带的非天然(例如,异源)核酸序列。
在将核酸序列插入细胞的上下文中,术语“引入”意指本领域已知的“转染”、“转化”或“转导”。
“宿主菌株”或“宿主细胞”是已经引入了表达载体、噬菌体、病毒或其他DNA构建体,包括编码目的多肽(例如,β-半乳糖苷酶)的多核苷酸的生物。示例性宿主菌株是能够表达目的多肽的微生物细胞(例如,细菌、丝状真菌和酵母)。术语“宿主细胞”包括从细胞产生的原生质体。
关于多核苷酸或蛋白质的术语“异源”是指不是天然存在于宿主细胞中的多核苷酸或蛋白质。
关于多核苷酸或蛋白质的术语“内源”是指天然存在于宿主细胞中的多核苷酸或蛋白质。
术语“表达”是指基于核酸序列产生多肽的过程。该过程包括转录和翻译两者。
“选择性标记”或“可选择标记”是指能够在宿主中被表达以促进选择携带该基因的宿主细胞的基因。可选择标记的实例包括但不限于在宿主细胞上赋予代谢优势(如营养优势)的抗微生物剂(例如,潮霉素、博来霉素或氯霉素)和/或基因。
“载体”是指设计用于将核酸引入一种或多种细胞类型中的多核苷酸序列。载体包括克隆载体、表达载体、穿梭载体、质粒、噬菌体颗粒、盒等。
“表达载体”是指包含编码目的多肽的DNA序列的DNA构建体,该编码序列可操作地连接到能够在合适的宿主中实现DNA表达的合适的控制序列。此类控制序列可包括实现转录的启动子、控制转录的任选操纵子序列、编码mRNA上合适的核糖体结合位点的序列、增强子以及控制转录和翻译终止的序列。
术语“可操作地连接”意指指定成分处于允许它们以预期方式起作用的关系(包括但不限于并列)。例如,调控序列可操作地连接到编码序列,使得编码序列的表达受调控序列的控制。
“信号序列”是附接到蛋白质N末端部分的氨基酸序列,该氨基酸序列有利于蛋白质在细胞外的分泌。成熟形式的细胞外蛋白质缺乏在分泌过程中被切除的信号序列。
“生物学活性的”是指具有指定生物活性,例如酶活性的序列。
术语“比活性”是指在特定条件下,每单位时间可通过酶或酶制剂转化为产物的底物的摩尔数。比活性通常表示为单位(U)/mg蛋白质。
如本文所使用的,“序列同一性百分比”意指当使用具有默认参数的CLUSTAL W算法比对时,特定序列具有与指定的参照序列中的氨基酸残基相同的至少一定百分比的氨基酸残基。参见Thompson等人(1994)Nucleic Acids Res.[核酸研究]22:4673-4680。CLUSTALW算法的默认参数是:
空位开放罚分:10.0
空位延伸罚分:0.05
蛋白质权重矩阵:BLOSUM系列
DNA权重矩阵:IUB
延迟发散序列%:40
空位分隔距离:8
DNA转换权重:0.50
列表亲水残基:GPSNDQEKR
使用负性矩阵:关
切换特殊残基罚分:开
切换亲水罚分:开
切换结束空位分隔罚分关。
与参照序列相比,缺失被视为不相同的残基。包括在任一末端发生的缺失。例如,相对于成熟多肽,具有成熟617残基多肽的C末端的五个氨基酸缺失的变体将具有99%的序列同一性百分比(612/617相同的残基×100,四舍五入到最接近的整数)。这种变体将被与成熟多肽具有“至少99%序列同一性”的变体所涵盖。
“融合”多肽序列通过两个主题多肽序列之间的肽键连接,即可操作地连接。
术语“丝状真菌”是指所有丝状形式的真菌亚门(Eumycotina),特别是子囊菌亚门(Pezizomycotina)物种。
术语“约”是指参照值的±5%。
“乳糖酶处理的奶(lactase treated milk)”意指经乳糖酶处理以减少乳糖量的奶。
“降乳糖奶(reduced lactose milk)”意指其中乳糖的百分比为约2%或更低的奶。
“无乳糖奶(lactose free milk)”意指其中乳糖百分比低于0.1%(w/v)的奶,并且在一些情况下,意指其中乳糖百分比低于0.01%(w/v)的奶。
在本发明的上下文中,术语“奶”应理解为从任何哺乳动物如牛、绵羊、山羊、水牛或骆驼获得的乳状分泌物。
在本发明的上下文中,术语“奶基底物”意指任何生的和/或加工的奶材料或衍生自奶组分的材料。有用的奶基底物包括但不限于包括乳糖的任何奶或奶样产品的溶液/悬浮液,如全脂或低脂奶、脱脂奶、白脱奶(buttermilk)、复原奶粉、炼奶、奶粉溶液、UHT奶、乳清、乳清渗透物、酸乳清或奶油。优选地,奶基底物是奶或脱脂奶粉的水溶液。奶基底物可以比生奶更浓。在一个实施例中,奶基底物的蛋白质与乳糖的比率为至少0.2、优选至少0.3、至少0.4、至少0.5、至少0.6、或最优选至少0.7。可以根据本领域已知的方法对奶基底物进行均化和/或巴氏杀菌。
序列
来自两歧双歧杆菌的成熟截短形式的B-半乳糖苷酶BIF917的氨基
酸序列如SEQ ID NO:1所示:
VEDATRSDSTTQMSSTPEVVYSSAVDSKQNRTSDFDANWKFMLSDSVQAQDPAFDDSAWQQVDLPHDYSITQKYSQSNEAESAYLPGGTGWYRKSFTIDRDLAGKRIAINFDGVYMNATVWFNGVKLGTHPYGYSPFSFDLTGNAKFGGENTIVVKVENRLPSSRWYSGSGIYRDVTLTVTDGVHVGNNGVAIKTPSLATQNGGDVTMNLTTKVANDTEAAANITLKQTVFPKGGKTDAAIGTVTTASKSIAAGASADVTSTITAASPKLWSIKNPNLYTVRTEVLNGGKVLDTYDTEYGFRWTGFDATSGFSLNGEKVKLKGVSMHHDQGSLGAVANRRAIERQVEILQKMGVNSIRTTHNPAAKALIDVCNEKGVLVVEEVFDMWNRSKNGNTEDYGKWFGQAIAGDNAVLGGDKDETWAKFDLTSTINRDRNAPSVIMWSLGNEMMEGISGSVSGFPATSAKLVAWTKAADSTRPMTYGDNKIKANWNESNTMGDNLTANGGVVGTNYSDGANYDKIRTTHPSWAIYGSETASAINSRGIYNRTTGGAQSSDKQLTSYDNSAVGWGAVASSAWYDVVQRDFVAGTYVWTGFDYLGEPTPWNGTGSGAVGSWPSPKNSYFGIVDTAGFPKDTYYFYQSQWNDDVHTLHILPAWNENVVAKGSGNNVPVVVYTDAAKVKLYFTPKGSTEKRLIGEKSFTKKTTAAGYTYQVYEGSDKDSTAHKNMYLTWNVPWAEGTISAEAYDENNRLIPEGSTEGNASVTTTGKAAKLKADADRKTITADGKDLSYIEVDVTDANGHIVPDAANRVTFDVKGAGKLVGVDNGSSPDHDSYQADNRKAFSGKVLAIVQSTKEAGEITVTAKADGLQSSTVKIATTAVPGTSTEKT
来自两歧双歧杆菌的成熟截短形式的B-半乳糖苷酶BIF995的氨基酸序列如SEQID NO:2所示:
VEDATRSDSTTQMSSTPEVVYSSAVDSKQNRTSDFDANWKFMLSDSVQAQDPAFDDSAWQQVDLPHDYSITQKYSQSNEAESAYLPGGTGWYRKSFTIDRDLAGKRIAINFDGVYMNATVWFNGVKLGTHPYGYSPFSFDLTGNAKFGGENTIVVKVENRLPSSRWYSGSGIYRDVTLTVTDGVHVGNNGVAIKTPSLATQNGGDVTMNLTTKVANDTEAAANITLKQTVFPKGGKTDAAIGTVTTASKSIAAGASADVTSTITAASPKLWSIKNPNLYTVRTEVLNGGKVLDTYDTEYGFRWTGFDATSGFSLNGEKVKLKGVSMHHDQGSLGAVANRRAIERQVEILQKMGVNSIRTTHNPAAKALIDVCNEKGVLVVEEVFDMWNRSKNGNTEDYGKWFGQAIAGDNAVLGGDKDETWAKFDLTSTINRDRNAPSVIMWSLGNEMMEGISGSVSGFPATSAKLVAWTKAADSTRPMTYGDNKIKANWNESNTMGDNLTANGGVVGTNYSDGANYDKIRTTHPSWAIYGSETASAINSRGIYNRTTGGAQSSDKQLTSYDNSAVGWGAVASSAWYDVVQRDFVAGTYVWTGFDYLGEPTPWNGTGSGAVGSWPSPKNSYFGIVDTAGFPKDTYYFYQSQWNDDVHTLHILPAWNENVVAKGSGNNVPVVVYTDAAKVKLYFTPKGSTEKRLIGEKSFTKKTTAAGYTYQVYEGSDKDSTAHKNMYLTWNVPWAEGTISAEAYDENNRLIPEGSTEGNASVTTTGKAAKLKADADRKTITADGKDLSYIEVDVTDANGHIVPDAANRVTFDVKGAGKLVGVDNGSSPDHDSYQADNRKAFSGKVLAIVQSTKEAGEITVTAKADGLQSSTVKIATTAVPGTSTEKTVRSFYYSRNYYVKTGNKPILPSDVEVRYSDGTSDRQNVTWDAVSDDQIAKAGSFSVAGTVAGQKISVRVTMIDEIGAL
来自两歧双歧杆菌的成熟截短形式的B-半乳糖苷酶BIF1068的氨基酸序列如SEQID NO:3所示:
VEDATRSDSTTQMSSTPEVVYSSAVDSKQNRTSDFDANWKFMLSDSVQAQDPAFDDSAWQQVDLPHDYSITQKYSQSNEAESAYLPGGTGWYRKSFTIDRDLAGKRIAINFDGVYMNATVWFNGVKLGTHPYGYSPFSFDLTGNAKFGGENTIVVKVENRLPSSRWYSGSGIYRDVTLTVTDGVHVGNNGVAIKTPSLATQNGGDVTMNLTTKVANDTEAAANITLKQTVFPKGGKTDAAIGTVTTASKSIAAGASADVTSTITAASPKLWSIKNPNLYTVRTEVLNGGKVLDTYDTEYGFRWTGFDATSGFSLNGEKVKLKGVSMHHDQGSLGAVANRRAIERQVEILQKMGVNSIRTTHNPAAKALIDVCNEKGVLVVEEVFDMWNRSKNGNTEDYGKWFGQAIAGDNAVLGGDKDETWAKFDLTSTINRDRNAPSVIMWSLGNEMMEGISGSVSGFPATSAKLVAWTKAADSTRPMTYGDNKIKANWNESNTMGDNLTANGGVVGTNYSDGANYDKIRTTHPSWAIYGSETASAINSRGIYNRTTGGAQSSDKQLTSYDNSAVGWGAVASSAWYDVVQRDFVAGTYVWTGFDYLGEPTPWNGTGSGAVGSWPSPKNSYFGIVDTAGFPKDTYYFYQSQWNDDVHTLHILPAWNENVVAKGSGNNVPVVVYTDAAKVKLYFTPKGSTEKRLIGEKSFTKKTTAAGYTYQVYEGSDKDSTAHKNMYLTWNVPWAEGTISAEAYDENNRLIPEGSTEGNASVTTTGKAAKLKADADRKTITADGKDLSYIEVDVTDANGHIVPDAANRVTFDVKGAGKLVGVDNGSSPDHDSYQADNRKAFSGKVLAIVQSTKEAGEITVTAKADGLQSSTVKIATTAVPGTSTEKTVRSFYYSRNYYVKTGNKPILPSDVEVRYSDGTSDRQNVTWDAVSDDQIAKAGSFSVAGTVAGQKISVRVTMIDEIGALLNYSASTPVGTPAVLPGSRPAVLPDGTVTSANFAVHWTKPADTVYNTAGTVKVPGTATVFGKEFKVTATIRVQ
来自两歧双歧杆菌的成熟截短形式的B-半乳糖苷酶BIF1172的氨基酸序列如SEQID NO:4所示:
VEDATRSDSTTQMSSTPEVVYSSAVDSKQNRTSDFDANWKFMLSDSVQAQDPAFDDSAWQQVDLPHDYSITQKYSQSNEAESAYLPGGTGWYRKSFTIDRDLAGKRIAINFDGVYMNATVWFNGVKLGTHPYGYSPFSFDLTGNAKFGGENTIVVKVENRLPSSRWYSGSGIYRDVTLTVTDGVHVGNNGVAIKTPSLATQNGGDVTMNLTTKVANDTEAAANITLKQTVFPKGGKTDAAIGTVTTASKSIAAGASADVTSTITAASPKLWSIKNPNLYTVRTEVLNGGKVLDTYDTEYGFRWTGFDATSGFSLNGEKVKLKGVSMHHDQGSLGAVANRRAIERQVEILQKMGVNSIRTTHNPAAKALIDVCNEKGVLVVEEVFDMWNRSKNGNTEDYGKWFGQAIAGDNAVLGGDKDETWAKFDLTSTINRDRNAPSVIMWSLGNEMMEGISGSVSGFPATSAKLVAWTKAADSTRPMTYGDNKIKANWNESNTMGDNLTANGGVVGTNYSDGANYDKIRTTHPSWAIYGSETASAINSRGIYNRTTGGAQSSDKQLTSYDNSAVGWGAVASSAWYDVVQRDFVAGTYVWTGFDYLGEPTPWNGTGSGAVGSWPSPKNSYFGIVDTAGFPKDTYYFYQSQWNDDVHTLHILPAWNENVVAKGSGNNVPVVVYTDAAKVKLYFTPKGSTEKRLIGEKSFTKKTTAAGYTYQVYEGSDKDSTAHKNMYLTWNVPWAEGTISAEAYDENNRLIPEGSTEGNASVTTTGKAAKLKADADRKTITADGKDLSYIEVDVTDANGHIVPDAANRVTFDVKGAGKLVGVDNGSSPDHDSYQADNRKAFSGKVLAIVQSTKEAGEITVTAKADGLQSSTVKIATTAVPGTSTEKTVRSFYYSRNYYVKTGNKPILPSDVEVRYSDGTSDRQNVTWDAVSDDQIAKAGSFSVAGTVAGQKISVRVTMIDEIGALLNYSASTPVGTPAVLPGSRPAVLPDGTVTSANFAVHWTKPADTVYNTAGTVKVPGTATVFGKEFKVTATIRVQRSQVTIGSSVSGNALRLTQNIPADKQSDTLDAIKDGSTTVDANTGGGANPSAWTNWAYSKAGHNTAEITFEYATEQQLGQIVMYFFRDSNAVRFPDAGKTKIQI
来自两歧双歧杆菌的成熟截短形式的B-半乳糖苷酶BIF1241的氨基酸序列如SEQID NO:5所示:
VEDATRSDSTTQMSSTPEVVYSSAVDSKQNRTSDFDANWKFMLSDSVQAQDPAFDDSAWQQVDLPHDYSITQKYSQSNEAESAYLPGGTGWYRKSFTIDRDLAGKRIAINFDGVYMNATVWFNGVKLGTHPYGYSPFSFDLTGNAKFGGENTIVVKVENRLPSSRWYSGSGIYRDVTLTVTDGVHVGNNGVAIKTPSLATQNGGDVTMNLTTKVANDTEAAANITLKQTVFPKGGKTDAAIGTVTTASKSIAAGASADVTSTITAASPKLWSIKNPNLYTVRTEVLNGGKVLDTYDTEYGFRWTGFDATSGFSLNGEKVKLKGVSMHHDQGSLGAVANRRAIERQVEILQKMGVNSIRTTHNPAAKALIDVCNEKGVLVVEEVFDMWNRSKNGNTEDYGKWFGQAIAGDNAVLGGDKDETWAKFDLTSTINRDRNAPSVIMWSLGNEMMEGISGSVSGFPATSAKLVAWTKAADSTRPMTYGDNKIKANWNESNTMGDNLTANGGVVGTNYSDGANYDKIRTTHPSWAIYGSETASAINSRGIYNRTTGGAQSSDKQLTSYDNSAVGWGAVASSAWYDVVQRDFVAGTYVWTGFDYLGEPTPWNGTGSGAVGSWPSPKNSYFGIVDTAGFPKDTYYFYQSQWNDDVHTLHILPAWNENVVAKGSGNNVPVVVYTDAAKVKLYFTPKGSTEKRLIGEKSFTKKTTAAGYTYQVYEGSDKDSTAHKNMYLTWNVPWAEGTISAEAYDENNRLIPEGSTEGNASVTTTGKAAKLKADADRKTITADGKDLSYIEVDVTDANGHIVPDAANRVTFDVKGAGKLVGVDNGSSPDHDSYQADNRKAFSGKVLAIVQSTKEAGEITVTAKADGLQSSTVKIATTAVPGTSTEKTVRSFYYSRNYYVKTGNKPILPSDVEVRYSDGTSDRQNVTWDAVSDDQIAKAGSFSVAGTVAGQKISVRVTMIDEIGALLNYSASTPVGTPAVLPGSRPAVLPDGTVTSANFAVHWTKPADTVYNTAGTVKVPGTATVFGKEFKVTATIRVQRSQVTIGSSVSGNALRLTQNIPADKQSDTLDAIKDGSTTVDANTGGGANPSAWTNWAYSKAGHNTAEITFEYATEQQLGQIVMYFFRDSNAVRFPDAGKTKIQISADGKNWTDLAATETIAAQESSDRVKPYTYDFAPVGATFVKVTVTNADTTTPSGVVCAGLTEIELKTAT
额外的突变
在一些实施例中,本发明的β-半乳糖苷酶进一步包括可提供另外的性能或稳定性益处的一个或多个突变。示例性性能益处包括但不限于增加的热稳定性、增加的储存稳定性、增加的溶解度、改变的pH曲线、增加的比活性、经修饰的底物特异性、经修饰的底物结合、经修饰的pH依赖性活性、经修饰的pH依赖性稳定性、增加的氧化稳定性、和增加的表达。在一些情况下,性能益处是在相对较低的温度下实现的。在一些情况下,性能益处是在相对较高的温度下实现的。
此外,本发明的β-半乳糖苷酶可以包含任何数量的保守氨基酸取代。下表中列出了示例性保守氨基酸取代。
保守氨基酸取代
/>
读者将理解,一些前述保守突变可以通过遗传操作产生,而其他通过用遗传或其他方式将合成的氨基酸引入多肽中产生。
本发明的β-半乳糖苷酶可以是“前体”、“未成熟”或“全长”的,在这种情况下,它们包含信号序列;或“成熟”的,在这种情况下,它们缺乏信号序列。成熟形式的多肽通常是最有用的。除非另有说明,否则本文使用的氨基酸残基编号是指相应β-半乳糖苷酶多肽的成熟形式。只要所得的多肽保留β-半乳糖苷酶活性,本发明的β-半乳糖苷酶多肽也可以被截短以除去N-末端或C-末端。
本发明的β-半乳糖苷酶可以是“嵌合”或“杂合”多肽,因为其包括第一β-半乳糖苷酶多肽的至少一部分和第二β-半乳糖苷酶多肽的至少一部分。本发明的β-半乳糖苷酶可进一步包含异源信号序列,即允许跟踪或纯化等的表位。示例性异源信号序列来自地衣芽孢杆菌(B.licheniformis)淀粉酶(LAT)、枯草芽孢杆菌(B.subtilis)(AmyE或AprE)、和链霉菌属(Streptomyces)CelA。
β-半乳糖苷酶的产生
本发明的β-半乳糖苷酶可以在宿主细胞中产生,例如通过分泌或细胞内表达。在将β-半乳糖苷酶分泌到细胞培养基中后,可以获得包含β-半乳糖苷酶的培养的细胞材料(例如,全细胞培养液)。任选地,β-半乳糖苷酶可以从宿主细胞中分离,或甚至从细胞培养液中分离,这取决于最终β-半乳糖苷酶所需的纯度。可以根据本领域熟知的方法克隆和表达编码β-半乳糖苷酶的基因。合适的宿主细胞包括细菌、真菌(包括酵母和丝状真菌)和植物细胞(包括藻类)。特别有用的宿主细胞包括黑曲霉(Aspergillus niger)、米曲霉(Aspergillus oryzae)或里氏木霉(Trichoderma reesei)。其他宿主细胞包括细菌细胞,例如枯草芽孢杆菌或地衣芽孢杆菌、以及链霉菌属和大肠杆菌(E.Coli)。
宿主细胞还可以表达编码同源或异源β-半乳糖苷酶(即,与宿主细胞不同物种的β-半乳糖苷酶)或一种或多种其他酶的核酸。β-半乳糖苷酶可以是变体β-半乳糖苷酶。另外,宿主可以表达一种或多种辅酶、蛋白质、肽。
载体
可以构建包含编码β-半乳糖苷酶的核酸的DNA构建体,以在宿主细胞中表达。由于遗传密码中熟知的简并性,编码相同氨基酸序列的变体多核苷酸可以用常规技术进行设计和制备。针对特定宿主细胞优化密码子使用也是本领域熟知的。编码β-半乳糖苷酶的核酸可以掺入载体中。可以使用熟知的转化技术(例如以下公开的那些)将载体转移至宿主细胞中。
载体可以是可被转化到宿主细胞中并在其中复制的任何载体。例如,包含编码β-半乳糖苷酶的核酸的载体可以在细菌宿主细胞中转化和复制,作为使载体繁殖和扩增的手段。载体也可以被转化到表达宿主中,使得编码核酸可以被表达为功能性β-半乳糖苷酶。用作表达宿主的宿主细胞可以包括,例如,丝状真菌。
编码β-半乳糖苷酶的核酸可以可操作地连接到合适的启动子,从而允许在宿主细胞中进行转录。启动子可以是在所选宿主细胞中显示转录活性的任何DNA序列,并且可以衍生自编码与宿主细胞同源或异源的蛋白质的基因。用于指导编码β-半乳糖苷酶的DNA序列的转录(尤其是在细菌宿主中)的示例性启动子是大肠杆菌的lac操纵子的启动子、天蓝色链霉菌(Streptomyces coelicolor)琼脂酶基因dagA或celA启动子、地衣芽孢杆菌α-淀粉酶基因(amyL)的启动子、嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)生麦芽糖淀粉酶基因(amyM)的启动子、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)α-淀粉酶(amyQ)的启动子、枯草芽孢杆菌xylA和xylB基因的启动子等。对于在真菌宿主中的转录,有用的启动子的实例是衍生自编码米曲霉TAKA淀粉酶、米黑根毛霉(Rhizomucor miehei)天冬氨酸蛋白酶、黑曲霉中性α-淀粉酶、黑曲霉酸稳定性α-淀粉酶、黑曲霉葡糖淀粉酶、米黑根毛霉脂肪酶、米曲霉碱性蛋白酶、米曲霉磷酸三糖异构酶或构巢曲霉(A.nidulans)乙酰胺酶的基因的那些启动子。当编码β-半乳糖苷酶的基因在细菌物种(如大肠杆菌)中表达时,可以从,例如,包括T7启动子和噬菌体λ启动子的细菌噬菌体启动子中选择合适的启动子。用于在酵母物种中表达的适合的启动子的实例包括但不限于酿酒酵母(Saccharomycescerevisiae)的Gal 1和Gal 10启动子以及毕赤酵母(Pichia pastoris)AOX1或AOX2启动子。cbh1是来自里氏木霉的内源诱导型启动子。参见Liu等人(2008)“Improvedheterologous gene expression in Trichoderma reesei by cellobiohydrolase Igene(cbh1)promoter optimization[纤维二糖水解酶I基因(cbh1)启动子优化改善里氏木霉中的异源基因表达],”Acta Biochim.Biophys.Sin(Shanghai)[生物化学与生物物理学报(上海)]40(2):158-65。
编码序列可以可操作地连接到信号序列。编码信号序列的DNA可以是与待表达的β-半乳糖苷酶基因天然相关或来自不同属或物种的DNA序列。可以将包含DNA构建体或载体的信号序列和启动子序列引入真菌宿主细胞,并且它们可以衍生自相同的来源。例如,信号序列是可操作地连接到cbh1启动子的cbh1信号序列。
表达载体还可以包含合适的转录终止子,以及在真核生物中,包含可操作地连接到编码变体β-半乳糖苷酶的DNA序列的聚腺苷酸化序列。终止序列和聚腺苷酸化序列可以适当地衍生自与启动子相同的来源。
载体可以进一步包含使载体能够在宿主细胞中复制的DNA序列。此类序列的实例是质粒pUC19、pACYC177、pUB110、pE194、pAMB1和pIJ702的复制起点。
载体还可以包含可选择标记,例如其产物弥补分离的宿主细胞中的缺陷的基因,例如来自枯草芽孢杆菌或地衣芽孢杆菌的dal基因,或者赋予抗生素抗性(例如氨苄青霉素、卡那霉素、氯霉素或四环素抗性)的基因。此外,载体可以包含曲霉属(Aspergillus)选择标记,如amdS、argB、niaD和xxsC,引起潮霉素抗性的标记,或者选择可以通过共转化(如本领域已知的)实现。参见,例如,国际PCT申请WO 91/17243。
细胞内表达在一些方面可能是有利的,例如,当使用某些细菌或真菌作为宿主细胞产生大量β-半乳糖苷酶用于随后的富集或纯化时。β-半乳糖苷酶的细胞外分泌至培养基中还可用于制备包含分离的β-半乳糖苷酶的培养的细胞材料。
表达载体通常包括克隆载体的成分,例如允许载体在所选宿主生物中自主复制的元件和用于选择目的的一个或多个表型可检测的标记。表达载体通常包含控制核苷酸序列,例如启动子、操纵子、核糖体结合位点、翻译起始信号以及任选地抑制子基因或一个或多个激活子基因。另外,表达载体可包含编码氨基酸序列的序列,该氨基酸序列能够将β-半乳糖苷酶靶向宿主细胞细胞器(如过氧化物酶体)或靶向特定宿主细胞区室。这种靶向序列包括但不限于序列SKL。对于在控制序列的指导下进行表达,将β-半乳糖苷酶的核酸序列以关于表达的适当方式可操作地连接到控制序列。
用于分别连接编码β-半乳糖苷酶的DNA构建体、启动子、终止子和其他元件,并将它们插入到含有复制所需信息的合适的载体中的程序是本领域技术人员熟知的(参见,例如,Sambrook等人,Molecular Cloning:A Laboratory Manual[分子克隆:实验室手册],第2版,Cold Spring Harbor[冷泉港实验室],1989,和第3版,2001)。
宿主细胞的转化和培养
包含DNA构建体或表达载体的分离的细胞有利地用作重组产生β-半乳糖苷酶的宿主细胞。通过将DNA构建体(以一或多个拷贝)整合到宿主染色体中,可以方便地用编码酶的DNA构建体转化细胞。通常认为这种整合是有利的,因为DNA序列更可能在细胞中稳定保持。可以根据常规方法,例如,通过同源或异源重组,将DNA构建体整合到宿主染色体中。可替代地,可以用上述与不同类型的宿主细胞相关的表达载体转化细胞。
合适的细菌宿主生物的实例是革兰氏阳性细菌物种,例如芽孢杆菌科(Bacillaceae),包括枯草芽孢杆菌、地衣芽孢杆菌、迟缓芽孢杆菌(Bacillus lentus)、短芽孢杆菌(Bacillus brevis)、嗜热脂肪地芽孢杆菌(Geobacillus stearothermophilus)(原嗜热脂肪芽孢杆菌)、嗜碱芽孢杆菌(Bacillus alkalophilus)、解淀粉芽孢杆菌、凝结芽孢杆菌(Bacillus coagulans)、灿烂芽孢杆菌(Bacillus lautus)、巨大芽孢杆菌(Bacillus megaterium)和苏云金芽孢杆菌(Bacillus thuringiensis);链霉菌属物种,如鼠灰链霉菌(Streptomyces murinus);乳酸细菌物种,包括乳球菌属物种(Lactococcussp.),如乳酸乳球菌(Lactococcus lactis);乳杆菌属物种(Lactobacillus sp.),包括罗伊氏乳杆菌(Lactobacillus reuteri);明串珠菌属物种(Leuconostoc sp.);片球菌属物种(Pediococcus sp.);和链球菌属物种(Streptococcus sp.)。可替代地,可以选择属于肠杆菌科(Enterobacteriaceae)(包括大肠杆菌)或属于假单胞菌科(Pseudomonadaceae)的革兰氏阴性细菌物种的菌株作为宿主生物。
合适的酵母宿主生物可以选自生物技术相关的酵母物种,例如但不限于酵母物种,如毕赤酵母属物种(Pichia sp.)、汉逊酵母属物种(Hansenula sp.)或克鲁维酵母属(Kluyveromyces)、耶氏酵母属(Yarrowinia)、裂殖酵母属(Schizosaccharomyces)物种或者酵母属(Saccharomyces)的物种,包括酿酒酵母,或者属于裂殖酵母属的物种,例如,粟酒裂殖酵母(S.pombe)物种。甲基营养型酵母物种菌株毕赤酵母可以用作宿主生物。可替代地,宿主生物可以是汉逊酵母属物种。丝状真菌中合适的宿主生物包括曲霉属的物种,例如,黑曲霉、米曲霉、塔宾曲霉(Aspergillus tubigensis)、泡盛曲霉(Aspergillusawamori)或构巢曲霉。可替代地,镰刀菌属(Fusarium)物种(例如,尖孢镰刀菌(Fusariumoxysporum))的菌株或根毛霉属(Rhizomucor)物种(如米黑根毛霉)的菌株可以用作宿主生物。其他合适的菌株包括嗜热菌属(Thermomyces)和毛霉菌属(Mucor)物种。此外,木霉属物种(Trichoderma sp.)可以用作宿主。转化曲霉属宿主细胞的合适的程序包括,例如,EP238023中描述的程序。真菌宿主细胞表达的β-半乳糖苷酶可以被糖基化,即,将包含糖基部分。糖基化模式可以与野生型β-半乳糖苷酶中存在的相同或不同。糖基化的类型和/或程度可能改变酶和/或生物化学特性。
从表达宿主缺失基因是有利的,其中可以通过转化的表达载体治愈基因缺陷。已知方法可用于获得具有一种或多种失活基因的真菌宿主细胞。可通过完全或部分缺失、通过插入失活或通过使基因对其预期目的不起作用的任何其他方式(使得阻止基因表达功能性蛋白)来完成基因灭活。已克隆的、来自木霉属物种或其他丝状真菌宿主的基因可以缺失,例如,cbh1、cbh2、egl1和egl2基因。基因缺失可通过本领域已知方法通过将待失活的所需基因的形式插入质粒中来完成。
将DNA构建体或载体引入宿主细胞中包括以下技术,例如转化;电穿孔;核显微注射;转导;转染,例如脂质转染介导的和DEAE-糊精介导的转染;与磷酸钙DNA沉淀一起孵育;用DNA包被的微粒高速轰击;以及原生质体融合。通用转化技术是本领域已知的。参见,例如Sambrook等人(2001),同上。异源蛋白质在木霉属中的表达描述于例如美国专利号6,022,725中。对于曲霉属菌株的转化,还参考了Cao等人(2000)Science[科学]9:991-1001。可以用载体系统构建遗传稳定的转化体,由此将编码β-半乳糖苷酶的核酸稳定整合到宿主细胞染色体中。然后通过已知技术选择并纯化转化体。
表达
产生β-半乳糖苷酶的方法可以包括在有利于产生该酶的条件下培养上述宿主细胞,并从细胞和/或培养基中回收该酶。
用于培养细胞的培养基可以是适合于所考虑的宿主细胞生长并获得β-半乳糖苷酶表达的任何常规培养基。合适的培养基和培养基成分可从商业供应商获得或可以根据公开的配方(例如,如在美国典型培养物保藏中心(American Type Culture Collection)的目录中所述)制备。
从宿主细胞中分泌的酶可以用于全培养液制剂中。在本发明的方法中,可以使用本领域已知的任何培养方法来实现重组微生物的用过的(spent)全发酵液的制备,从而导致β-半乳糖苷酶的表达。因此,发酵可以理解为包括在实验室或工业发酵罐中在合适的培养基和允许β-半乳糖苷酶被表达或分离的条件下进行的摇瓶培养、小规模或大规模发酵(包括连续发酵、分批发酵、补料分批发酵或固态发酵)。术语“用过的全发酵液”在本文中被定义为包括培养基、细胞外蛋白质(例如,酶)和细胞生物质的发酵材料的未分级内容物。应当理解,术语“用过的全发酵液”还涵盖已使用本领域熟知的方法裂解或透化的细胞生物质。
从宿主细胞中分泌的酶可方便地通过熟知的程序从培养基中回收,这些程序包括通过离心或过滤从培养基中分离细胞,并借助于盐(例如硫酸铵)沉淀培养基的蛋白质成分,然后使用色谱程序,例如离子交换色谱法、亲和色谱法等。
可以将在载体中编码β-半乳糖苷酶的多核苷酸可操作地连接到控制序列,该控制序列能够通过宿主细胞提供编码序列的表达,即该载体是表达载体。可以例如通过添加其他转录调控元件来修饰控制序列,以使由控制序列指导的转录水平对转录调节因子更有应答。控制序列可特别地包含启动子。
宿主细胞可以在允许表达β-半乳糖苷酶的合适条件下培养。这些酶的表达可以是组成型的,使得它们能被连续产生,或诱导型的,需要刺激来启动表达。在诱导型表达的情况下,当需要时可通过例如向培养基中添加诱导物质,例如地塞米松或IPTG或槐糖来启动蛋白质生产。多肽也可以在体外无细胞系统(例如TNT
富集和纯化β-半乳糖苷酶的方法
发酵、分离和浓缩技术是本领域熟知的,并且可使用常规方法来制备含β-半乳糖苷酶多肽的溶液。
发酵后,获得发酵液,通过常规分离技术去除微生物细胞和各种悬浮固体(包括残留的粗发酵材料),以便获得β-半乳糖苷酶溶液。通常使用过滤、离心、微滤、旋转真空鼓式过滤、超滤、离心后超滤、萃取或色谱法等。
期望浓缩含β-半乳糖苷酶多肽的溶液以优化回收。使用未浓缩的溶液需要增加孵育时间以便收集经富集或纯化的酶沉淀物。
使用常规浓缩技术浓缩含酶溶液,直至获得期望的酶水平。含酶溶液的浓缩可通过在本文中所讨论的技术中的任一种来实现。富集和纯化的示例性方法包括但不限于旋转鼓式真空过滤和/或超滤。
例如,在本发明的某些实施例中,β-半乳糖苷酶可以衍生自链球菌属物种、明串珠菌属物种或乳杆菌属物种。可衍生β-半乳糖苷酶的链球菌属物种的实例包括唾液链球菌(S.salivarius)、表兄链球菌(S.sobrinus)、牙链球菌(S.dentirousetti)、汗毛链球菌(S.downei)、变异链球菌(S.mutans)、口腔链球菌(S.oralis)、解没食子酸链球菌(S.gallolyticus)和血链球菌(S.sanguinis)。可衍生β-半乳糖苷酶的明串珠菌属物种的实例包括肠膜明串珠菌(L.mesenteroides)、阿米巴氏明串珠菌(L.amelibiosum)、阿根廷明串珠菌(L.argentinum)、肉明串珠菌(L.carnosum)、柠檬明串珠菌(L.citreum)、乳脂明串珠菌(L.cremoris)、葡聚糖明串珠菌(L.dextranicum)和果糖明串珠菌(L.fructosum)。可衍生β-半乳糖苷酶的乳杆菌属物种的实例包括嗜酸乳杆菌(L.acidophilus)、德氏乳杆菌(L.delbrueckii)、瑞士乳杆菌(L.helveticus)、唾液乳杆菌(L.salivarius)、干酪乳杆菌(L.casei)、弯曲乳杆菌(L.curvatus)、植物乳杆菌(L.plantarum)、清酒乳杆菌(L.sakei)、短乳杆菌(L.brevis)、布赫内氏乳杆菌(L.buchneri)、发酵乳杆菌(L.fermentum)和罗伊氏乳杆菌(L.reuteri)。
巴氏杀菌时间和温度
奶基的巴氏杀菌:超高温(UHT)通常在135℃-150℃下进行2-15秒,高热/更短时间(HHST)通常在85℃-115℃下进行0.5-9秒,并且高温/短时间(HTST)巴氏杀菌通常在70℃-85℃下进行15-30秒。酸奶奶基的巴氏杀菌通常在85℃-96℃下进行5-10min。
本发明的优选的实施例
提出了一种用于测定样品中碳水化合物含量的方法,该方法具有以下步骤:获得对应于该样品的FTIR(傅里叶变换红外光谱术)光谱数据;提供该FTIR光谱数据的至少一部分作为经过训练的机器学习模型的输入;以及使用该经过训练的机器学习模型处理该FTIR光谱数据的至少一部分,以生成碳水化合物含量值,从而提供该样品中碳水化合物含量水平的定量指示。
优选地,碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。优选地,DP2是乳糖。更优选地,碳水化合物是DP3+GOS。
优选地,样品是奶基底物。优选地,作为经过训练的机器学习模型的输入提供的FTIR光谱数据部分是有限光谱范围内的FTIR光谱数据。优选地,有限光谱范围包括1046cm
优选地,经过训练的机器学习模型是使用训练数据集训练的监督学习模型,对于多个训练样品中的每一个,该训练数据集具有对应于该训练样品的FTIR光谱数据和该训练样品中碳水化合物含量水平的测量指示。
优选地,经过训练的机器学习模型是偏最小二乘回归(PLSR)模型。
优选地,经过训练的机器学习模型是神经网络回归模型。
优选地,经过训练的机器学习模型包括多元线性回归(MLR)。
优选地,经过训练的机器学习模型是主成分回归(PCR)。
优选地,经过训练的机器学习模型包括经典最小二乘法CLS。
优选地,经过训练的机器学习模型包括决策树算法。
优选地,FTIR光谱数据是从基于服务器的数据存储中获得的,该FTIR光谱数据由客户端设备上传到该数据存储中。
优选地,使用经过训练的机器学习模型对FTIR光谱数据的至少一部分进行的处理是使用从客户端设备获得的FTIR光谱数据在服务器设备上进行的。
优选地,碳水化合物含量值可供客户端设备访问。
提出了一种用于训练机器学习模型以预测奶基底物中碳水化合物含量的方法,该方法具有以下步骤:获得训练数据集,对于多个训练样品中的每一个,该训练数据集具有对应于该训练样品的FTIR(傅里叶变换红外光谱术)光谱数据和该训练样品中碳水化合物含量水平的测量指示;以及使用该训练数据集进行监督学习,以确定该机器学习模型的经过训练的模型系数。
优选地,碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。优选地,DP2是乳糖。优选地,碳水化合物是DP3+GOS。
在本发明的另一方面,提出了一种计算机程序,当在数据处理装置上执行时,该计算机程序控制该数据处理装置以进行上述方法。
在本发明的另一方面,提出了一种用于制备含有碳水化合物的奶产品的方法,该方法具有以下步骤:用转半乳糖基化酶处理奶基底物;对该奶基底物的样品进行FTIR(傅里叶变换红外光谱术),以获得对应于该样品的FTIR光谱数据;基于使用经过训练的机器学习模型对该FTIR光谱数据的至少一部分进行的处理,获得碳水化合物含量值,从而提供该样品中碳水化合物含量水平的定量指示;以及基于该碳水化合物含量值,确定何时通过对该奶基进行巴氏杀菌来灭活该转半乳糖基化酶。
优选地,碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。优选地,DP2是乳糖。优选地,碳水化合物是DP3+GOS。
29.
优选地,FTIR光谱数据的至少一部分由客户端设备上传到服务器,用于由经过训练的机器学习模型进行基于服务器的处理以生成碳水化合物含量值,并且碳水化合物含量值由客户端设备作为基于服务器的处理的结果而获得。
优选地,用于制备奶基碳水化合物的方法具有优于10%的准确度,表示为平均值的预测标准误差(3.75%),在含有至少0.1%脂肪、至少0.5%溶解乳糖和至少1%蛋白质的奶基中,GOS的浓度在0-7.5%的浓度范围内。
优选地,用于制备奶基碳水化合物的方法具有高于0.9(不太优选的是0.85、0.8、0.75等)的PLS回归模型的线性度(R2),以验证GOS含量。
优选地,转半乳糖基化酶衍生自两歧双歧杆菌。更优选地,转半乳糖基化酶是来自两歧双歧杆菌的截短的β-半乳糖苷酶。又更优选地,来自两歧双歧杆菌的截短的β-半乳糖苷酶在C末端被截短。仍更优选地,来自两歧双歧杆菌的截短的β-半乳糖苷酶是与SEQID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少70%序列同一性的多肽。
优选地,该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQID NO:5或其转半乳糖基酶活性片段具有至少80%序列同一性。更优选地,该多肽与SEQID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少90%序列同一性。仍更优选地,该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ IDNO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少95%序列同一性。在又更优选的实施例中,该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少99%序列同一性。仍更优选地,该多肽是根据SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段的序列。在最优选的实施例中,该多肽是根据SEQ ID NO:1的序列。
在用于制备含有碳水化合物的奶产品的方法中,优选地在巴氏杀菌之前应用线性度(R2)高于0.9(不太优选的是0.85、0.8、0.75等)的PLS模型。
在用于制备含有碳水化合物的奶产品的方法中,优选在应用红外吸收测量技术之前,将具有超过20%总固体的奶基底物在水中稀释。
优选地,奶基底物具有1%-60%(w/w);或2%-50%(w/w);或3%-40%(w/w);或4%-30%(w/w)之间的乳糖浓度。
优选地,转半乳糖基化酶的失活是通过热处理进行。更优选地,热处理为约70℃至95℃并持续约5分钟至30分钟。仍更优选地,热处理在约95℃下持续5至30分钟。在其他优选的实施例中,热处理为约135℃至约150℃持续约2秒至约15秒。
优选地,具有GOS纤维的奶基产品是酸奶、冰淇淋、UHT奶、风味奶产品、浓缩奶/炼奶产品、奶粉或奶酪。优选地,奶基产品中的GOS纤维是稳定的,在28天内具有小于约10%的变化。优选地,具有GOS纤维的奶基产品含有超过约1.5%(w/w)的GOS纤维。更优选地,具有GOS纤维的奶基产品含有超过约3.2%(w/w)的GOS纤维。仍更优选地,具有GOS纤维的奶基产品含有超过约4%(w/w)的GOS纤维。在又更优选的实施例中,具有GOS纤维的奶基产品含有超过约7%(w/w)的GOS纤维。仍更优选地,具有GOS纤维的奶基产品含有超过约14%(w/w)的GOS纤维。在又更优选的实施例中,具有GOS纤维的奶基产品含有超过约30%(w/w)的GOS纤维。
优选地,该方法通过使用全中红外光谱仪器来进行,该仪器被设置用于记录包括约400-6000、更具体地至少900-1500cm
优选地,傅里叶变换红外(FTIR)光谱术通过使用全中红外光谱仪器来进行,以获得包含足够信息的记录的光谱数据。优选地,记录的光谱包括约900-1500cm
在本发明的其他优选的实施例中,该方法还具有使低乳糖奶基产品脱水以提供粉末以及将该粉末溶解在水中的步骤。
本公开在以下实例中进一步详细描述,这些实例不意欲以任何方式限制本公开所要求保护的范围。附图意在被视为本公开的说明书和描述的组成部分。提供以下实例以说明但不限制所要求保护的公开内容。
还在以下方面中描述了本发明:
1.一种用于测定样品中碳水化合物含量的方法,该方法包括:
获得对应于该样品的FTIR(傅里叶变换红外光谱术)光谱数据;
提供该FTIR光谱数据的至少一部分作为经过训练的机器学习模型的输入;以及
使用该经过训练的机器学习模型处理该FTIR光谱数据的至少一部分,以生成碳水化合物含量值,从而提供该样品中碳水化合物含量水平的定量指示。
2.根据方面1所述的方法,其中该碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。
3.根据方面2所述的方法,其中该DP2是乳糖。
4.根据方面2所述的方法,其中该碳水化合物是DP3+GOS。
5.根据前述方面中任一项所述的方法,其中该样品是奶基底物。
6.根据前述方面中任一项所述的方法,其中作为该经过训练的机器学习模型的输入提供的该FTIR光谱数据部分包括有限光谱范围内的FTIR光谱数据。
7.根据方面6所述的方法,其中该有限光谱范围包括1046cm
8.根据方面6所述的方法,其中该有限光谱范围包括下限在900cm
9.根据方面8所述的方法,其中该有限光谱范围包括下限在1008cm
10.根据方面9所述的方法,其中该有限光谱范围包括波数区域1037:1450cm
11.根据前述方面中任一项所述的方法,其中该经过训练的机器学习模型包括使用训练数据集训练的监督学习模型,对于多个训练样品中的每一个,该训练数据集包括对应于该训练样品的FTIR光谱数据和该训练样品中碳水化合物含量水平的测量指示。
12.根据方面1至10中任一项所述的方法,其中该经过训练的机器学习模型包括偏最小二乘回归(PLSR)模型。
13.根据方面1至10中任一项所述的方法,其中该经过训练的机器学习模型包括神经网络回归模型。
14.根据方面1至10中任一项所述的方法,其中该经过训练的机器学习模型包括多元线性回归(MLR)。
15.根据方面1至10中任一项所述的方法,其中该经过训练的机器学习模型包括主成分回归(PCR)。
16.根据方面1至10中任一项所述的方法,其中该经过训练的机器学习模型包括经典最小二乘法CLS。
17.根据前述方面中任一项所述的方法,其中该FTIR光谱数据是从基于服务器的数据存储中获得的,该FTIR光谱数据由客户端设备上传到该数据存储中。
18.根据前述方面中任一项所述的方法,其中使用该经过训练的机器学习模型对该FTIR光谱数据的至少一部分进行的处理是使用从客户端设备获得的FTIR光谱数据在服务器设备上进行的。
19.根据方面17和18中任一项所述的方法,其中该碳水化合物含量值可供该客户端设备访问。
20.一种用于训练机器学习模型以预测奶基底物中碳水化合物含量的方法,该方法包括:
获得训练数据集,对于多个训练样品中的每一个,该训练数据集包括对应于该训练样品的FTIR(傅里叶变换红外光谱术)光谱数据和该训练样品中碳水化合物含量水平的测量指示;以及
使用该训练数据集进行监督学习,以确定该机器学习模型的经过训练的模型系数。
21.根据方面20所述的方法,其中该碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。
22.根据方面21所述的方法,其中该DP2是乳糖。
23.根据方面21所述的方法,其中该碳水化合物是DP3+GOS。
24.一种计算机程序,当在数据处理装置上执行时,该计算机程序控制该数据处理装置以进行根据前述权利要求中任一项所述的方法。
25.一种用于制备含有碳水化合物的奶产品的方法,该方法包括:
用转半乳糖基化酶处理奶基底物;
对该奶基底物的样品进行FTIR(傅里叶变换红外光谱术),以获得对应于该样品的FTIR光谱数据;
基于使用经过训练的机器学习模型对该FTIR光谱数据的至少一部分进行的处理,获得碳水化合物含量值,从而提供该样品中碳水化合物含量水平的定量指示;以及
基于该碳水化合物含量值,确定何时通过对该奶基进行巴氏杀菌来灭活该转半乳糖基化酶。
26.根据方面25所述的方法,其中该碳水化合物是GOS、DP3+GOS、葡萄糖、半乳糖和DP2中的一种或多种。
27.根据方面26所述的方法,其中该DP2是乳糖。
28.根据方面26所述的方法,其中该碳水化合物是DP3+GOS。
29.根据方面25至28中任一项所述的方法,其中该FTIR光谱数据的至少一部分由客户端设备上传到服务器,用于由该经过训练的机器学习模型进行基于服务器的处理以生成该碳水化合物含量值,并且该碳水化合物含量值由该客户端设备作为该基于服务器的处理的结果而获得。
30.根据方面25至29中任一项所述的方法,该方法具有优于10%的准确度,表示为平均值的预测标准误差(3.75%),在含有至少0.1%脂肪、至少0.5%溶解乳糖和至少1%蛋白质的奶基中,GOS的浓度在0-7.5%的浓度范围内。
31.根据方面25至29中任一项所述的方法,该方法具有高于0.9(不太优选的是0.85、0.8、0.75等)的PLS回归模型的线性度(R2),以验证GOS含量。
32.根据方面25至31中任一项所述的方法,其中该转半乳糖基化酶衍生自两歧双歧杆菌。
33.根据方面32所述的方法,其中该转半乳糖基化酶是来自两歧双歧杆菌的截短的β-半乳糖苷酶。
34.根据方面33所述的方法,其中该来自两歧双歧杆菌的截短的β-半乳糖苷酶在C末端被截短。
35.根据方面34所述的方法,其中该来自两歧双歧杆菌的截短的β-半乳糖苷酶包含与SEQ ID.NO:1、SEQ ID NO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少70%序列同一性的多肽。
36.根据方面35所述的方法,其中该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ IDNO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少80%序列同一性。
37.根据方面36所述的方法,其中该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ IDNO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少90%序列同一性。
38.根据方面37所述的方法,其中该多肽与SEQ ID.NO:1、SEQ IDNO:2、SEQ ID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少95%序列同一性。
39.根据方面38所述的方法,其中该多肽与SEQ ID.NO:1、SEQ ID NO:2、SEQ IDNO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段具有至少99%序列同一性。
40.根据方面39所述的方法,其中该多肽包含根据SEQ ID.NO:1、SEQ ID NO:2、SEQID NO:3、SEQ ID NO:4或SEQ ID NO:5或其转半乳糖基酶活性片段的序列。
41.根据方面40所述的方法,其中该多肽包含根据SEQ ID.NO:1的序列。
实例
实例1:HPLC乳糖测定
以下方法描述了实例4-8的奶样品中乳糖定量的程序。对样品进行衍生化,并通过带UV和FLD检测的HPLC进行分析。
化学品:二甲亚砜,DMSO(CAS:67-68-5西格玛公司(Sigma)A8418);磷酸,H3PO4(CAS:7664-38-2,西格玛公司P5811);乙酸,≥99%(CAS:64-19-7,西格玛公司A6283);磷酸钠,NaH
衍生化试剂通过将12.3%w/v 4-氨基苯甲酸和10%w/v 2-甲基吡啶硼烷在DMSO/乙酸(70/30%v/v)中混合而获得。样品溶剂为10mM Na
利用ThermoScientific UltiMate 3000 HPLC系统,该系统配备有在线真空脱气器、泵、自动进样器、柱温控制和UV/VIS检测器。使用ThermoScientific Chomeleon 7.2.9软件进行定量。柱:Knauer Prontosil RP-C18 SH(150×4.6mm,3μm),来自Mikrolab公司(KN 15EF180PSG/15VH185PSJ)。将四丁基硫酸氢铵用作洗脱液系统中的离子对试剂。进样体积为20μL,柱温为20℃,等度:MP A流速:0.8mL/min,A:含有20mM四丁基硫酸氢铵的10mM磷酸钠缓冲液(pH 2.0)。在每个进样柱之间用50/50%v/v乙腈/水洗涤,压力:250巴,运行时间:100min,检测器在303nm处测量吸光度。用于校准标准品的乳糖在ddH
样品制备:使用在pH 2.5的10mM NaH
实例2:用于对GOS纤维进行定量的HPLC方法
在双蒸馏水(ddH
仪器:
通过HPLC进行高级低聚半乳糖(GOS,即DP3及以上)、DP2、葡萄糖和半乳糖的定量。样品分析在配备有DGP-3600SD双梯度分析泵、WPS-3000TSL恒温自动进样器、TCC-3000SD恒温柱温箱和RI-101折光率检测器(昭和公司(Shodex),JM科学公司(JM Science))的DionexUltimate 3000HPLC系统(赛默飞世尔科技公司(Thermo Fisher Scientific))上进行。使用Chromeleon数据系统软件(6.80版,DU10ABuild 2826,171948)进行数据采集和分析。
色谱条件:
使用配备有在70℃下操作的分析保护柱(Carbo-Ag
在整个分析中保持0.3ml/min的等度流速,总运行时间为45分钟。进样体积设置为10μL。在恒温自动进样器区室中将样品保持在30℃,以确保所有成分的溶解。通过折光率检测器(RI-101,昭和公司,JM科学公司)监测洗脱液,并通过相对于上述乳糖标准品的峰面积的峰面积进行定量。将DP 3及以上(DP3+)的峰定量为低聚半乳糖(DP3、DP4、DP5等)。用质量平衡证实了所有DP3+低聚半乳糖成分都提供相同的响应这一假设。包括乳糖、葡萄糖和半乳糖的DP2种类均分别根据上述乳糖标准品进行定量。
实例3:总GOS
总GOS(TGOS)定义为转半乳糖基化分子的总和。该值的计算依据是TGOS分子中结合的半乳糖量乘以因子1.4,其中因子1.4表示半乳糖与葡萄糖的比率。这将包括更大部分的转半乳糖基化二糖,如别乳糖(β-D-吡喃半乳糖基(1→6)-D-葡萄糖)、(β-D-吡喃半乳糖基(1→3)-D-葡萄糖)或(β-D-吡喃半乳糖基(1→4)-D-半乳糖)。TGOS分子中结合的半乳糖是通过从总半乳糖(即游离半乳糖、TGOS中结合的半乳糖和乳糖中结合的半乳糖之和)中减去游离半乳糖和乳糖中结合的半乳糖来计算的。该计算方法符合AOAC 2001.02方法。
基于从实例1获得的值和从实例2的方法获得的输出值,如下计算了TGOS:
TGOS=(总碳水化合物(%w/w)/1.9-半乳糖(%w/w)-乳糖(%w/w)/1.9)×1.4
总碳水化合物(%w/w)是DP3+(%w/w)、DP2(%w/w)、葡萄糖(%w/w)和半乳糖(%w/w)之和。
总碳水化合物(%w/w)/1.9表示样品中的总半乳糖。
葡萄糖(%w/w)是样品中的游离葡萄糖。
半乳糖(%w/w)是样品中的游离半乳糖。
乳糖(%w/w)/1.9是乳糖分子中结合的半乳糖。
实例4:GOS在3%蛋白质的低脂奶基中的原位产生
制备奶基用于与产生GOS的两歧双歧杆菌B-半乳糖苷酶(SEQ IDNo.1)进行酶反应,以进行GOS的原位产生。将脱脂奶(阿拉福兹公司(Arla Foods),丹麦,0.1%脂肪,4.7%乳糖,3.66%蛋白质)用自来水标准化为3%蛋白质并按原样使用(产生3.8%乳糖)或用
表1
实例5:GOS在6%蛋白质的奶基中的原位产生
制备奶基用于与产生GOS的两歧双歧杆菌B-半乳糖苷酶(SEQ ID No.1)进行酶反应,以进行GOS的原位产生。将脱脂奶UF浓缩物(10.07%w/v蛋白质和0.09%w/v脂肪)用自来水标准化为5.7%w/v蛋白质并用
表2
/>
实例6:GOS在具有不同乳糖水平的脱脂奶中的原位产生
制备奶基用于与产生GOS的两歧双歧杆菌B-半乳糖苷酶(SEQ ID No.1)进行酶反应,以进行GOS的原位产生。将Arla脱脂奶(4.7%乳糖,3.66%蛋白质和0.09%脂肪)通过自来水稀释标准化为3%乳糖,或用
表3
/>
实例7:GOS在3%蛋白质的全脂奶基中的原位产生
制备奶基用于与产生GOS的两歧双歧杆菌B-半乳糖苷酶(SEQ IDNo.1)进行酶反应,以进行GOS的原位产生。将Arla脱脂奶(4.7%乳糖,3.66%蛋白质和0.09%脂肪)通过添加自来水和添加奶油(阿拉福兹公司,丹麦,38%脂肪)标准化为3%乳糖和3.5%脂肪。奶基按原样使用或用
表4
实例8:GOS在3%蛋白质的高脂奶基中的原位产生
制备奶基用于与产生GOS的两歧双歧杆菌B-半乳糖苷酶(SEQ ID No.1)进行酶反应,以进行GOS的原位产生。将脱脂奶UF浓缩物(10.07%蛋白质和0.09%脂肪)通过自来水稀释和添加奶油(阿拉福兹公司,丹麦,38%脂肪)标准化为6%蛋白质和3.5%脂肪。奶基按原样使用或用
表5
实例9:
傅里叶变换红外(FTIR)光谱术-珀金埃尔默公司的Spectrum One仪器:
红外光谱法基于记录电磁光谱的中红外(MIR)区域的仪器。通常,MIR红外吸光度在650-4400cm
将10个含GOS的奶样品(选自实例4-8)的样品组在真空中冷冻干燥过夜,并使用配备有通用衰减全反射(UATR)部件的Spectrum One FTIR仪器(珀金埃尔默公司,美国马萨诸塞州沃尔瑟姆)对粉末一式三份进行分析,使用4cm
图1显示了通过珀金埃尔默公司的Spectrum One FTIR仪器在波数范围650-4400cm
实例10
推导出用于预测奶样品中GOS浓度的偏最小二乘回归(PLSR)模型算法。该算法的原理是使用FTIR光谱的选定区域来预测通过HPLC测量的GOS浓度(如实例2中所述)。该算法基于称为偏最小二乘(PLS)回归模型的多元数学模型,其中FTIR光谱中的许多波数用于创建HPLC结果的最佳预测。使用类似的方法来预测实例1和2测量的葡萄糖、半乳糖、DP2和乳糖,但是下面仅详细描述DP3+GOS预测。通过预测实例1和2中测量的所有碳水化合物,将能够应用实例3来预测总GOS浓度。
需要这种类型的模型,因为FTIR光谱中的单个/或几个波数都不能用于对GOS含量进行定量。FTIR光谱报告所有奶组分(奶、脂肪和糖),但通过使用PLS回归,发现了DP3+GOS预测的最佳区域。
数学模型PLS回归在其他地方有描述(Martens,Izquierdo,Thomassen,和Martens,1986)。
PLSR模型是使用Matlab 2019a中的PLS-Toolbox 8.7构建和测试的。这一实施情况(implementation)使用NIPALS算法来估计权重,即PLS负载。
收集包含含GOS的奶样品的光谱信息的30个FTIR光谱的样品集(如实例9中所述记录),并与相干HPLC DP3+GOS结果(如实例2中所述)一起制表。
使用五个波数区域进行建模,从而产生模型#1至#5(表6)。使用范围为1037-1445cm
该模型使用交叉验证(百叶窗(Venetian blinds),10个分割)进行验证,并且表5中报告的模型性能是交叉验证的结果。DP3+GOS预测的模型#1性能显示出R平方R
表6:
与使用完整光谱(模型#2)相比,所发现的具有最佳光谱范围的模型(模型#1)具有优越的模型性能。该性能通过应尽可能接近1.00的模型R平方和应尽可能低的误差(RMECV)测量。模型#1中的光谱范围尤其涵盖GOS等低聚糖中存在的C-O-C(1157和1250cm
最后,光谱范围低于和超出模型#1范围的另一个模型(模型#3)的性能要低得多-说明所发现的模型#1涵盖了GOS酶活性预测的最佳光谱范围。表6中报告的所有模型都有4个PLS成分。
模型#1的PLS模型系数如图3所示。描绘了Y1(=DP3+)的回归向量,并将模型系数作为波数的函数。每个波数对DP3+GOS预测的重要性与模型系数成正比。模型系数的绝对值表示每个波数的相对重要性,而系数的符号表示对DP3+GOS值的贡献的符号。
实例11:基于Foss MilkoScan FT2仪器的傅里叶变换红外(FTIR)光谱术:
为了测试一般实施情况,使用了另一种FTIR光谱仪器。使用由Foss Intergrator2软件控制的Foss MilkoScan FT2仪器进行分析。在每次运行前,通过嵌入式清洁程序对装置进行清洁。Foss Integrator 2程序的调零选项用于控制每个清洁步骤的成功。为了进行测量,将100mL样品放置在仪器喷嘴下方。每个样品分析4次。将奶样品放置在仪器上,流管入口系统采样两次(每次11mL),并且每个样品一式两份进行测量。
数据通过两种方式收集。首先,提取仪器提供的预评估标准数据的汇总输出。该输出用作脂肪、蛋白质和乳糖含量的对照。
其次,从Intergator2软件中提取每次仪器运行的未处理原始数据(FTIR光谱)。图4给出了来自MilkoScan FT2分析含有GOS的奶样品的光谱数据的实例(如实例4-8中所述生成)。来自仪器软件的FTIR光谱以数据点而非波数给出。FOSS MilkoScan光谱在926:5010cm
实例12:使用MilkoScan FTIR光谱预测奶样品中的DP3+GOS的PLS回归模型。
从Milkoscan数据文件推导出用于预测奶样品中DP3+GOS浓度的偏最小二乘回归(PLSR)模型算法。该算法的原理是使用FTIR光谱的选定区域来预测通过HPLC测量的DP3+GOS浓度(如实例2中所述)。该算法基于称为偏最小二乘(PLS)回归模型的多元数学模型,其中FTIR光谱中的许多波数/数据点用于创建HPLC结果的最佳预测。使用类似的方法来预测实例1和2测量的葡萄糖、半乳糖、DP2和乳糖,但是下面仅详细描述DP3+GOS预测。通过预测实例1和2中测量的所有碳水化合物,将能够应用实例3来预测总GOS浓度。
需要这种类型的模型,因为FTIR光谱中的单个/或几个波数都不能用于对DP3+GOS含量进行定量。FTIR光谱报告所有奶组分(奶、脂肪和糖),但通过使用PLS回归,发现了DP3+GOS预测的最佳区域。
数学模型PLS回归在其他地方有描述(Martens等人,1986)。
PLSR模型是使用scikit学习包v.0.22.1的python函数sklearn.pls.PLSRegression构建和测试的。这一实施情况使用NIPALS算法来估计权重,即PLS负载。
收集包含含GOS的奶样品的光谱信息的119个数据文件的样品集(如实例4-8中产生),并与相干HPLC GOS(DP3+)结果(如实例2中所述)一起制表。将119个含GOS的奶样品随机分为测试集和训练集。训练集用于构建模型,而测试集用于验证模型。训练集包含95个含GOS的奶样品,而24个含GOS的奶样品用于验证PLSR模型。
使用范围为270-376的数据点找到了最佳模型(模型#6)。通过换算,该数据点范围对应于波数1041cm
在表7中,比较了三种不同的PLS回归模型。与使用完整光谱(模型#7)相比,具有最佳光谱范围的所发现的模型(模型#6)具有优越的模型性能。
测试范围低于和超出模型#6范围的另一个模型(模型#8)的性能要低得多-说明所发现的模型#6涵盖了最佳光谱范围。表7中报告的所有模型最多允许8个PLS成分,通过交叉验证找到了此最大值内的最佳成分数并在表中报告。
通过交叉验证找到的模型#6中PLS成分的最佳数量为13。然而,13个PLS成分会产生一个略微过拟合的模型,对原始FTIR光谱中非常小的变化敏感。因此,选择了具有8个潜在变量的次优模型,因为经过评估可以在模型准确度和模型稳健性之间给出最佳权衡。
使用24个含GOS的奶样品的测试集验证了该模型。DP3+GOS预测的模型#6性能显示出R平方R
表7.
模型#6的PLS模型系数如图7所示。每个数据点对DP3+GOS预测的重要性与模型系数成正比。模型系数的绝对值表示每个数据点的相对重要性,而系数的符号表示对DP3+GOS值的贡献的符号。图7显示,大多数模型系数具有相同的数量级,但符号存在区域变化。即,大多数数据点对DP3+GOS预测具有相似的重要性,但根据区域与DP3+GOS呈正或负相关。由于后续波数处的吸光度高度相关,我们预计模型系数的符号和大小都会出现这种“区域”行为,这反映出后续数据点对DP3+GOS预测具有类似的影响。
实例13
通过将4.1%、9.8%、20.5%或30.76%脱脂奶粉在自来水中稀释来制备复原奶样品。以0.7g/L、1.5g/L、3g/L、6g/L、12g/L或18g/L的剂量添加Zymstar GOS(材料A15017,批次4863445828),然后在5℃、10℃或45℃下孵育。在2、4、7、16和24小时后,提取样品并在95℃下孵育12分钟以灭活酶。对所得样品进行傅里叶变换红外(FTIR)光谱术,并根据实例1和2对样品中的各个碳水化合物进行定量。对于含有30.75%脱脂奶粉的样品,在水中1:3稀释之前和之后均进行FTIR测量。
实例14:
除(PLSR)模型外的几种其他模型类型也可用于预测从Milkoscan数据文件推导出的奶样品中GOS的浓度。如果有足够的数据,一个明显的选择是神经网络。FTIR光谱(或可替代地一些特定光谱区域)可以用作输入数据,其中通过HPLC测量的DP3+GOS浓度作为目标值,并且DP3+GOS预测作为神经网络的输出。神经网络由一组称为节点的单元组成。通常,这些节点按层排列,从一层连接到下一层,使得信号从输入层(FTIR光谱)传输到输出层(DP3+GOS预测),中间可能有多层。每个节点将传入连接的加权和作为输入,并基于非线性激活函数计算输出。该算法通过调整权重进行优化,通常是为了使目标和输出之间的均方误差最小化。通常还实施正则化(regularization)以防止过拟合。一个实例是L2正则化,其中损失函数受到权重平方和乘以正则化参数α的惩罚。神经网络有多种形式和结构,概述可以在(Schmidhuber,2015)中找到。通常,神经网络的一些结构比其他结构更适合特定类别的问题。在化学计量学中使用神经网络的多个实例尤其可以在文献(Cui和Fearn,2018)中找到。
Python提供了几种用于实现如下sklearn.neural_network和tensorflow.kereas的神经网络的框架。通常,可以调整许多超参数以找到当前问题的最佳模型。对于以下实例,使用scikitlearn v.0.22.1。
在包含244个新鲜奶样品和377个复原奶样品的数据集上训练和评估神经网络,这些复原奶样品使用初始乳糖含量范围为2%至15%的奶基以与实例4-8和实例13类似的方式制备。80%的数据集用于训练,而20%用于评估模型。仅使用光谱范围1041cm
发现与具有少量固体的复原奶相比,具有大量固体的复原奶的原始FTIR光谱与数据集的新鲜奶样品的差异更大。而且,由于人们可能不想在高固体含量的样品上使用所有FTIR仪器,因此研究了模型是否因包含稀释样品而受到损害。在模型#9包括实例13中的所有样品的情况下,稀释样品从模型#10中排除。超参数设置为与模型#9的超参数匹配,即将它们设置为{层大小=(106);优化算法=LBFGS;α=5*10
使用类似的方法来预测实例1和2测量的葡萄糖、半乳糖、DP2和乳糖。通过预测实例1和2中测量的所有碳水化合物,将能够应用实例3来预测总GOS浓度。
在整个波数范围926:5010cm
表8
/>
表9:
参考文献:
Cui,C.,&Fearn,T.(2018).Modern practical convolutional neural networksfor multivariate regression:Applications to NIR calibration.Chemometrics andIntelligent Laboratory Systems,182,9-20.
Grelet,C.,Fernández Pierna,J.A.,Dardenne,P.,Baeten,V.,&Dehareng,F.(2015).Standardization of milk mid-infrared spectra from a European dairynetwork.Journal of Dairy Science,98,2150-2160.
Martens,H.,Izquierdo,L.,Thomassen,M.,&Martens,M.(1986).Partial least-squares regression on design variables as an alternative to analysis ofvariance.Analytica Chimica Acta,191,133-148.
Schmidhuber,J.(2015).Deep learning in neural networks:Anoverview.Neural Networks,61,85-117.
Ref.5.See SK Learn Neural network at the website:
scikit-learn.org/stable/modules/generated/sklearn.neural_network.MLPRegressor.html。
序列表
<110>杜邦营养生物科学有限公司
<120>测量低聚半乳糖的方法
<130>P122370EP
<140>EP 21167494.0
<141>2021-04-08
<160>5
<170>PatentIn 3.5版
<210>1
<211>887
<212>PRT
<213>两歧双歧杆菌(Bifidobacterium bifidum)
<400>1
Val Glu Asp Ala Thr Arg Ser Asp Ser Thr Thr Gln Met Ser Ser Thr
1 5 1015
Pro Glu Val Val Tyr Ser Ser Ala Val Asp Ser Lys Gln Asn Arg Thr
202530
Ser Asp Phe Asp Ala Asn Trp Lys Phe Met Leu Ser Asp Ser Val Gln
354045
Ala Gln Asp Pro Ala Phe Asp Asp Ser Ala Trp Gln Gln Val Asp Leu
505560
Pro His Asp Tyr Ser Ile Thr Gln Lys Tyr Ser Gln Ser Asn Glu Ala
65707580
Glu Ser Ala Tyr Leu Pro Gly Gly Thr Gly Trp Tyr Arg Lys Ser Phe
859095
Thr Ile Asp Arg Asp Leu Ala Gly Lys Arg Ile Ala Ile Asn Phe Asp
100 105 110
Gly Val Tyr Met Asn Ala Thr Val Trp Phe Asn Gly Val Lys Leu Gly
115 120 125
Thr His Pro Tyr Gly Tyr Ser Pro Phe Ser Phe Asp Leu Thr Gly Asn
130 135 140
Ala Lys Phe Gly Gly Glu Asn Thr Ile Val Val Lys Val Glu Asn Arg
145 150 155 160
Leu Pro Ser Ser Arg Trp Tyr Ser Gly Ser Gly Ile Tyr Arg Asp Val
165 170 175
Thr Leu Thr Val Thr Asp Gly Val His Val Gly Asn Asn Gly Val Ala
180 185 190
Ile Lys Thr Pro Ser Leu Ala Thr Gln Asn Gly Gly Asp Val Thr Met
195 200 205
Asn Leu Thr Thr Lys Val Ala Asn Asp Thr Glu Ala Ala Ala Asn Ile
210 215 220
Thr Leu Lys Gln Thr Val Phe Pro Lys Gly Gly Lys Thr Asp Ala Ala
225 230 235 240
Ile Gly Thr Val Thr Thr Ala Ser Lys Ser Ile Ala Ala Gly Ala Ser
245 250 255
Ala Asp Val Thr Ser Thr Ile Thr Ala Ala Ser Pro Lys Leu Trp Ser
260 265 270
Ile Lys Asn Pro Asn Leu Tyr Thr Val Arg Thr Glu Val Leu Asn Gly
275 280 285
Gly Lys Val Leu Asp Thr Tyr Asp Thr Glu Tyr Gly Phe Arg Trp Thr
290 295 300
Gly Phe Asp Ala Thr Ser Gly Phe Ser Leu Asn Gly Glu Lys Val Lys
305 310 315 320
Leu Lys Gly Val Ser Met His His Asp Gln Gly Ser Leu Gly Ala Val
325 330 335
Ala Asn Arg Arg Ala Ile Glu Arg Gln Val Glu Ile Leu Gln Lys Met
340 345 350
Gly Val Asn Ser Ile Arg Thr Thr His Asn Pro Ala Ala Lys Ala Leu
355 360 365
Ile Asp Val Cys Asn Glu Lys Gly Val Leu Val Val Glu Glu Val Phe
370 375 380
Asp Met Trp Asn Arg Ser Lys Asn Gly Asn Thr Glu Asp Tyr Gly Lys
385 390 395 400
Trp Phe Gly Gln Ala Ile Ala Gly Asp Asn Ala Val Leu Gly Gly Asp
405 410 415
Lys Asp Glu Thr Trp Ala Lys Phe Asp Leu Thr Ser Thr Ile Asn Arg
420 425 430
Asp Arg Asn Ala Pro Ser Val Ile Met Trp Ser Leu Gly Asn Glu Met
435 440 445
Met Glu Gly Ile Ser Gly Ser Val Ser Gly Phe Pro Ala Thr Ser Ala
450 455 460
Lys Leu Val Ala Trp Thr Lys Ala Ala Asp Ser Thr Arg Pro Met Thr
465 470 475 480
Tyr Gly Asp Asn Lys Ile Lys Ala Asn Trp Asn Glu Ser Asn Thr Met
485 490 495
Gly Asp Asn Leu Thr Ala Asn Gly Gly Val Val Gly Thr Asn Tyr Ser
500 505 510
Asp Gly Ala Asn Tyr Asp Lys Ile Arg Thr Thr His Pro Ser Trp Ala
515 520 525
Ile Tyr Gly Ser Glu Thr Ala Ser Ala Ile Asn Ser Arg Gly Ile Tyr
530 535 540
Asn Arg Thr Thr Gly Gly Ala Gln Ser Ser Asp Lys Gln Leu Thr Ser
545 550 555 560
Tyr Asp Asn Ser Ala Val Gly Trp Gly Ala Val Ala Ser Ser Ala Trp
565 570 575
Tyr Asp Val Val Gln Arg Asp Phe Val Ala Gly Thr Tyr Val Trp Thr
580 585 590
Gly Phe Asp Tyr Leu Gly Glu Pro Thr Pro Trp Asn Gly Thr Gly Ser
595 600 605
Gly Ala Val Gly Ser Trp Pro Ser Pro Lys Asn Ser Tyr Phe Gly Ile
610 615 620
Val Asp Thr Ala Gly Phe Pro Lys Asp Thr Tyr Tyr Phe Tyr Gln Ser
625 630 635 640
Gln Trp Asn Asp Asp Val His Thr Leu His Ile Leu Pro Ala Trp Asn
645 650 655
Glu Asn Val Val Ala Lys Gly Ser Gly Asn Asn Val Pro Val Val Val
660 665 670
Tyr Thr Asp Ala Ala Lys Val Lys Leu Tyr Phe Thr Pro Lys Gly Ser
675 680 685
Thr Glu Lys Arg Leu Ile Gly Glu Lys Ser Phe Thr Lys Lys Thr Thr
690 695 700
Ala Ala Gly Tyr Thr Tyr Gln Val Tyr Glu Gly Ser Asp Lys Asp Ser
705 710 715 720
Thr Ala His Lys Asn Met Tyr Leu Thr Trp Asn Val Pro Trp Ala Glu
725 730 735
Gly Thr Ile Ser Ala Glu Ala Tyr Asp Glu Asn Asn Arg Leu Ile Pro
740 745 750
Glu Gly Ser Thr Glu Gly Asn Ala Ser Val Thr Thr Thr Gly Lys Ala
755 760 765
Ala Lys Leu Lys Ala Asp Ala Asp Arg Lys Thr Ile Thr Ala Asp Gly
770 775 780
Lys Asp Leu Ser Tyr Ile Glu Val Asp Val Thr Asp Ala Asn Gly His
785 790 795 800
Ile Val Pro Asp Ala Ala Asn Arg Val Thr Phe Asp Val Lys Gly Ala
805 810 815
Gly Lys Leu Val Gly Val Asp Asn Gly Ser Ser Pro Asp His Asp Ser
820 825 830
Tyr Gln Ala Asp Asn Arg Lys Ala Phe Ser Gly Lys Val Leu Ala Ile
835 840 845
Val Gln Ser Thr Lys Glu Ala Gly Glu Ile Thr Val Thr Ala Lys Ala
850 855 860
Asp Gly Leu Gln Ser Ser Thr Val Lys Ile Ala Thr Thr Ala Val Pro
865 870 875 880
Gly Thr Ser Thr Glu Lys Thr
885
<210>2
<211>965
<212>PRT
<213>两歧双歧杆菌(Bifidobacterium bifidum)
<400>2
Val Glu Asp Ala Thr Arg Ser Asp Ser Thr Thr Gln Met Ser Ser Thr
1 5 1015
Pro Glu Val Val Tyr Ser Ser Ala Val Asp Ser Lys Gln Asn Arg Thr
202530
Ser Asp Phe Asp Ala Asn Trp Lys Phe Met Leu Ser Asp Ser Val Gln
354045
Ala Gln Asp Pro Ala Phe Asp Asp Ser Ala Trp Gln Gln Val Asp Leu
505560
Pro His Asp Tyr Ser Ile Thr Gln Lys Tyr Ser Gln Ser Asn Glu Ala
65707580
Glu Ser Ala Tyr Leu Pro Gly Gly Thr Gly Trp Tyr Arg Lys Ser Phe
859095
Thr Ile Asp Arg Asp Leu Ala Gly Lys Arg Ile Ala Ile Asn Phe Asp
100 105 110
Gly Val Tyr Met Asn Ala Thr Val Trp Phe Asn Gly Val Lys Leu Gly
115 120 125
Thr His Pro Tyr Gly Tyr Ser Pro Phe Ser Phe Asp Leu Thr Gly Asn
130 135 140
Ala Lys Phe Gly Gly Glu Asn Thr Ile Val Val Lys Val Glu Asn Arg
145 150 155 160
Leu Pro Ser Ser Arg Trp Tyr Ser Gly Ser Gly Ile Tyr Arg Asp Val
165 170 175
Thr Leu Thr Val Thr Asp Gly Val His Val Gly Asn Asn Gly Val Ala
180 185 190
Ile Lys Thr Pro Ser Leu Ala Thr Gln Asn Gly Gly Asp Val Thr Met
195 200 205
Asn Leu Thr Thr Lys Val Ala Asn Asp Thr Glu Ala Ala Ala Asn Ile
210 215 220
Thr Leu Lys Gln Thr Val Phe Pro Lys Gly Gly Lys Thr Asp Ala Ala
225 230 235 240
Ile Gly Thr Val Thr Thr Ala Ser Lys Ser Ile Ala Ala Gly Ala Ser
245 250 255
Ala Asp Val Thr Ser Thr Ile Thr Ala Ala Ser Pro Lys Leu Trp Ser
260 265 270
Ile Lys Asn Pro Asn Leu Tyr Thr Val Arg Thr Glu Val Leu Asn Gly
275 280 285
Gly Lys Val Leu Asp Thr Tyr Asp Thr Glu Tyr Gly Phe Arg Trp Thr
290 295 300
Gly Phe Asp Ala Thr Ser Gly Phe Ser Leu Asn Gly Glu Lys Val Lys
305 310 315 320
Leu Lys Gly Val Ser Met His His Asp Gln Gly Ser Leu Gly Ala Val
325 330 335
Ala Asn Arg Arg Ala Ile Glu Arg Gln Val Glu Ile Leu Gln Lys Met
340 345 350
Gly Val Asn Ser Ile Arg Thr Thr His Asn Pro Ala Ala Lys Ala Leu
355 360 365
Ile Asp Val Cys Asn Glu Lys Gly Val Leu Val Val Glu Glu Val Phe
370 375 380
Asp Met Trp Asn Arg Ser Lys Asn Gly Asn Thr Glu Asp Tyr Gly Lys
385 390 395 400
Trp Phe Gly Gln Ala Ile Ala Gly Asp Asn Ala Val Leu Gly Gly Asp
405 410 415
Lys Asp Glu Thr Trp Ala Lys Phe Asp Leu Thr Ser Thr Ile Asn Arg
420 425 430
Asp Arg Asn Ala Pro Ser Val Ile Met Trp Ser Leu Gly Asn Glu Met
435 440 445
Met Glu Gly Ile Ser Gly Ser Val Ser Gly Phe Pro Ala Thr Ser Ala
450 455 460
Lys Leu Val Ala Trp Thr Lys Ala Ala Asp Ser Thr Arg Pro Met Thr
465 470 475 480
Tyr Gly Asp Asn Lys Ile Lys Ala Asn Trp Asn Glu Ser Asn Thr Met
485 490 495
Gly Asp Asn Leu Thr Ala Asn Gly Gly Val Val Gly Thr Asn Tyr Ser
500 505 510
Asp Gly Ala Asn Tyr Asp Lys Ile Arg Thr Thr His Pro Ser Trp Ala
515 520 525
Ile Tyr Gly Ser Glu Thr Ala Ser Ala Ile Asn Ser Arg Gly Ile Tyr
530 535 540
Asn Arg Thr Thr Gly Gly Ala Gln Ser Ser Asp Lys Gln Leu Thr Ser
545 550 555 560
Tyr Asp Asn Ser Ala Val Gly Trp Gly Ala Val Ala Ser Ser Ala Trp
565 570 575
Tyr Asp Val Val Gln Arg Asp Phe Val Ala Gly Thr Tyr Val Trp Thr
580 585 590
Gly Phe Asp Tyr Leu Gly Glu Pro Thr Pro Trp Asn Gly Thr Gly Ser
595 600 605
Gly Ala Val Gly Ser Trp Pro Ser Pro Lys Asn Ser Tyr Phe Gly Ile
610 615 620
Val Asp Thr Ala Gly Phe Pro Lys Asp Thr Tyr Tyr Phe Tyr Gln Ser
625 630 635 640
Gln Trp Asn Asp Asp Val His Thr Leu His Ile Leu Pro Ala Trp Asn
645 650 655
Glu Asn Val Val Ala Lys Gly Ser Gly Asn Asn Val Pro Val Val Val
660 665 670
Tyr Thr Asp Ala Ala Lys Val Lys Leu Tyr Phe Thr Pro Lys Gly Ser
675 680 685
Thr Glu Lys Arg Leu Ile Gly Glu Lys Ser Phe Thr Lys Lys Thr Thr
690 695 700
Ala Ala Gly Tyr Thr Tyr Gln Val Tyr Glu Gly Ser Asp Lys Asp Ser
705 710 715 720
Thr Ala His Lys Asn Met Tyr Leu Thr Trp Asn Val Pro Trp Ala Glu
725 730 735
Gly Thr Ile Ser Ala Glu Ala Tyr Asp Glu Asn Asn Arg Leu Ile Pro
740 745 750
Glu Gly Ser Thr Glu Gly Asn Ala Ser Val Thr Thr Thr Gly Lys Ala
755 760 765
Ala Lys Leu Lys Ala Asp Ala Asp Arg Lys Thr Ile Thr Ala Asp Gly
770 775 780
Lys Asp Leu Ser Tyr Ile Glu Val Asp Val Thr Asp Ala Asn Gly His
785 790 795 800
Ile Val Pro Asp Ala Ala Asn Arg Val Thr Phe Asp Val Lys Gly Ala
805 810 815
Gly Lys Leu Val Gly Val Asp Asn Gly Ser Ser Pro Asp His Asp Ser
820 825 830
Tyr Gln Ala Asp Asn Arg Lys Ala Phe Ser Gly Lys Val Leu Ala Ile
835 840 845
Val Gln Ser Thr Lys Glu Ala Gly Glu Ile Thr Val Thr Ala Lys Ala
850 855 860
Asp Gly Leu Gln Ser Ser Thr Val Lys Ile Ala Thr Thr Ala Val Pro
865 870 875 880
Gly Thr Ser Thr Glu Lys Thr Val Arg Ser Phe Tyr Tyr Ser Arg Asn
885 890 895
Tyr Tyr Val Lys Thr Gly Asn Lys Pro Ile Leu Pro Ser Asp Val Glu
900 905 910
Val Arg Tyr Ser Asp Gly Thr Ser Asp Arg Gln Asn Val Thr Trp Asp
915 920 925
Ala Val Ser Asp Asp Gln Ile Ala Lys Ala Gly Ser Phe Ser Val Ala
930 935 940
Gly Thr Val Ala Gly Gln Lys Ile Ser Val Arg Val Thr Met Ile Asp
945 950 955 960
Glu Ile Gly Ala Leu
965
<210>3
<211>1038
<212>PRT
<213>两歧双歧杆菌(Bifidobacterium bifidum)
<400>3
Val Glu Asp Ala Thr Arg Ser Asp Ser Thr Thr Gln Met Ser Ser Thr
1 5 1015
Pro Glu Val Val Tyr Ser Ser Ala Val Asp Ser Lys Gln Asn Arg Thr
202530
Ser Asp Phe Asp Ala Asn Trp Lys Phe Met Leu Ser Asp Ser Val Gln
354045
Ala Gln Asp Pro Ala Phe Asp Asp Ser Ala Trp Gln Gln Val Asp Leu
505560
Pro His Asp Tyr Ser Ile Thr Gln Lys Tyr Ser Gln Ser Asn Glu Ala
65707580
Glu Ser Ala Tyr Leu Pro Gly Gly Thr Gly Trp Tyr Arg Lys Ser Phe
859095
Thr Ile Asp Arg Asp Leu Ala Gly Lys Arg Ile Ala Ile Asn Phe Asp
100 105 110
Gly Val Tyr Met Asn Ala Thr Val Trp Phe Asn Gly Val Lys Leu Gly
115 120 125
Thr His Pro Tyr Gly Tyr Ser Pro Phe Ser Phe Asp Leu Thr Gly Asn
130 135 140
Ala Lys Phe Gly Gly Glu Asn Thr Ile Val Val Lys Val Glu Asn Arg
145 150 155 160
Leu Pro Ser Ser Arg Trp Tyr Ser Gly Ser Gly Ile Tyr Arg Asp Val
165 170 175
Thr Leu Thr Val Thr Asp Gly Val His Val Gly Asn Asn Gly Val Ala
180 185 190
Ile Lys Thr Pro Ser Leu Ala Thr Gln Asn Gly Gly Asp Val Thr Met
195 200 205
Asn Leu Thr Thr Lys Val Ala Asn Asp Thr Glu Ala Ala Ala Asn Ile
210 215 220
Thr Leu Lys Gln Thr Val Phe Pro Lys Gly Gly Lys Thr Asp Ala Ala
225 230 235 240
Ile Gly Thr Val Thr Thr Ala Ser Lys Ser Ile Ala Ala Gly Ala Ser
245 250 255
Ala Asp Val Thr Ser Thr Ile Thr Ala Ala Ser Pro Lys Leu Trp Ser
260 265 270
Ile Lys Asn Pro Asn Leu Tyr Thr Val Arg Thr Glu Val Leu Asn Gly
275 280 285
Gly Lys Val Leu Asp Thr Tyr Asp Thr Glu Tyr Gly Phe Arg Trp Thr
290 295 300
Gly Phe Asp Ala Thr Ser Gly Phe Ser Leu Asn Gly Glu Lys Val Lys
305 310 315 320
Leu Lys Gly Val Ser Met His His Asp Gln Gly Ser Leu Gly Ala Val
325 330 335
Ala Asn Arg Arg Ala Ile Glu Arg Gln Val Glu Ile Leu Gln Lys Met
340 345 350
Gly Val Asn Ser Ile Arg Thr Thr His Asn Pro Ala Ala Lys Ala Leu
355 360 365
Ile Asp Val Cys Asn Glu Lys Gly Val Leu Val Val Glu Glu Val Phe
370 375 380
Asp Met Trp Asn Arg Ser Lys Asn Gly Asn Thr Glu Asp Tyr Gly Lys
385 390 395 400
Trp Phe Gly Gln Ala Ile Ala Gly Asp Asn Ala Val Leu Gly Gly Asp
405 410 415
Lys Asp Glu Thr Trp Ala Lys Phe Asp Leu Thr Ser Thr Ile Asn Arg
420 425 430
Asp Arg Asn Ala Pro Ser Val Ile Met Trp Ser Leu Gly Asn Glu Met
435 440 445
Met Glu Gly Ile Ser Gly Ser Val Ser Gly Phe Pro Ala Thr Ser Ala
450 455 460
Lys Leu Val Ala Trp Thr Lys Ala Ala Asp Ser Thr Arg Pro Met Thr
465 470 475 480
Tyr Gly Asp Asn Lys Ile Lys Ala Asn Trp Asn Glu Ser Asn Thr Met
485 490 495
Gly Asp Asn Leu Thr Ala Asn Gly Gly Val Val Gly Thr Asn Tyr Ser
500 505 510
Asp Gly Ala Asn Tyr Asp Lys Ile Arg Thr Thr His Pro Ser Trp Ala
515 520 525
Ile Tyr Gly Ser Glu Thr Ala Ser Ala Ile Asn Ser Arg Gly Ile Tyr
530 535 540
Asn Arg Thr Thr Gly Gly Ala Gln Ser Ser Asp Lys Gln Leu Thr Ser
545 550 555 560
Tyr Asp Asn Ser Ala Val Gly Trp Gly Ala Val Ala Ser Ser Ala Trp
565 570 575
Tyr Asp Val Val Gln Arg Asp Phe Val Ala Gly Thr Tyr Val Trp Thr
580 585 590
Gly Phe Asp Tyr Leu Gly Glu Pro Thr Pro Trp Asn Gly Thr Gly Ser
595 600 605
Gly Ala Val Gly Ser Trp Pro Ser Pro Lys Asn Ser Tyr Phe Gly Ile
610 615 620
Val Asp Thr Ala Gly Phe Pro Lys Asp Thr Tyr Tyr Phe Tyr Gln Ser
625 630 635 640
Gln Trp Asn Asp Asp Val His Thr Leu His Ile Leu Pro Ala Trp Asn
645 650 655
Glu Asn Val Val Ala Lys Gly Ser Gly Asn Asn Val Pro Val Val Val
660 665 670
Tyr Thr Asp Ala Ala Lys Val Lys Leu Tyr Phe Thr Pro Lys Gly Ser
675 680 685
Thr Glu Lys Arg Leu Ile Gly Glu Lys Ser Phe Thr Lys Lys Thr Thr
690 695 700
Ala Ala Gly Tyr Thr Tyr Gln Val Tyr Glu Gly Ser Asp Lys Asp Ser
705 710 715 720
Thr Ala His Lys Asn Met Tyr Leu Thr Trp Asn Val Pro Trp Ala Glu
725 730 735
Gly Thr Ile Ser Ala Glu Ala Tyr Asp Glu Asn Asn Arg Leu Ile Pro
740 745 750
Glu Gly Ser Thr Glu Gly Asn Ala Ser Val Thr Thr Thr Gly Lys Ala
755 760 765
Ala Lys Leu Lys Ala Asp Ala Asp Arg Lys Thr Ile Thr Ala Asp Gly
770 775 780
Lys Asp Leu Ser Tyr Ile Glu Val Asp Val Thr Asp Ala Asn Gly His
785 790 795 800
Ile Val Pro Asp Ala Ala Asn Arg Val Thr Phe Asp Val Lys Gly Ala
805 810 815
Gly Lys Leu Val Gly Val Asp Asn Gly Ser Ser Pro Asp His Asp Ser
820 825 830
Tyr Gln Ala Asp Asn Arg Lys Ala Phe Ser Gly Lys Val Leu Ala Ile
835 840 845
Val Gln Ser Thr Lys Glu Ala Gly Glu Ile Thr Val Thr Ala Lys Ala
850 855 860
Asp Gly Leu Gln Ser Ser Thr Val Lys Ile Ala Thr Thr Ala Val Pro
865 870 875 880
Gly Thr Ser Thr Glu Lys Thr Val Arg Ser Phe Tyr Tyr Ser Arg Asn
885 890 895
Tyr Tyr Val Lys Thr Gly Asn Lys Pro Ile Leu Pro Ser Asp Val Glu
900 905 910
Val Arg Tyr Ser Asp Gly Thr Ser Asp Arg Gln Asn Val Thr Trp Asp
915 920 925
Ala Val Ser Asp Asp Gln Ile Ala Lys Ala Gly Ser Phe Ser Val Ala
930 935 940
Gly Thr Val Ala Gly Gln Lys Ile Ser Val Arg Val Thr Met Ile Asp
945 950 955 960
Glu Ile Gly Ala Leu Leu Asn Tyr Ser Ala Ser Thr Pro Val Gly Thr
965 970 975
Pro Ala Val Leu Pro Gly Ser Arg Pro Ala Val Leu Pro Asp Gly Thr
980 985 990
Val Thr Ser Ala Asn Phe Ala ValHis Trp Thr Lys ProAla Asp Thr
995 1000 1005
Val TyrAsn Thr Ala Gly ThrVal Lys Val Pro GlyThr Ala Thr
1010 1015 1020
Val PheGly Lys Glu Phe LysVal Thr Ala Thr IleArg Val Gln
1025 1030 1035
<210>4
<211>1142
<212>PRT
<213>两歧双歧杆菌(Bifidobacterium bifidum)
<400>4
Val Glu Asp Ala Thr Arg Ser Asp Ser Thr Thr Gln Met Ser Ser Thr
1 5 1015
Pro Glu Val Val Tyr Ser Ser Ala Val Asp Ser Lys Gln Asn Arg Thr
202530
Ser Asp Phe Asp Ala Asn Trp Lys Phe Met Leu Ser Asp Ser Val Gln
354045
Ala Gln Asp Pro Ala Phe Asp Asp Ser Ala Trp Gln Gln Val Asp Leu
505560
Pro His Asp Tyr Ser Ile Thr Gln Lys Tyr Ser Gln Ser Asn Glu Ala
65707580
Glu Ser Ala Tyr Leu Pro Gly Gly Thr Gly Trp Tyr Arg Lys Ser Phe
859095
Thr Ile Asp Arg Asp Leu Ala Gly Lys Arg Ile Ala Ile Asn Phe Asp
100 105 110
Gly Val Tyr Met Asn Ala Thr Val Trp Phe Asn Gly Val Lys Leu Gly
115 120 125
Thr His Pro Tyr Gly Tyr Ser Pro Phe Ser Phe Asp Leu Thr Gly Asn
130 135 140
Ala Lys Phe Gly Gly Glu Asn Thr Ile Val Val Lys Val Glu Asn Arg
145 150 155 160
Leu Pro Ser Ser Arg Trp Tyr Ser Gly Ser Gly Ile Tyr Arg Asp Val
165 170 175
Thr Leu Thr Val Thr Asp Gly Val His Val Gly Asn Asn Gly Val Ala
180 185 190
Ile Lys Thr Pro Ser Leu Ala Thr Gln Asn Gly Gly Asp Val Thr Met
195 200 205
Asn Leu Thr Thr Lys Val Ala Asn Asp Thr Glu Ala Ala Ala Asn Ile
210 215 220
Thr Leu Lys Gln Thr Val Phe Pro Lys Gly Gly Lys Thr Asp Ala Ala
225 230 235 240
Ile Gly Thr Val Thr Thr Ala Ser Lys Ser Ile Ala Ala Gly Ala Ser
245 250 255
Ala Asp Val Thr Ser Thr Ile Thr Ala Ala Ser Pro Lys Leu Trp Ser
260 265 270
Ile Lys Asn Pro Asn Leu Tyr Thr Val Arg Thr Glu Val Leu Asn Gly
275 280 285
Gly Lys Val Leu Asp Thr Tyr Asp Thr Glu Tyr Gly Phe Arg Trp Thr
290 295 300
Gly Phe Asp Ala Thr Ser Gly Phe Ser Leu Asn Gly Glu Lys Val Lys
305 310 315 320
Leu Lys Gly Val Ser Met His His Asp Gln Gly Ser Leu Gly Ala Val
325 330 335
Ala Asn Arg Arg Ala Ile Glu Arg Gln Val Glu Ile Leu Gln Lys Met
340 345 350
Gly Val Asn Ser Ile Arg Thr Thr His Asn Pro Ala Ala Lys Ala Leu
355 360 365
Ile Asp Val Cys Asn Glu Lys Gly Val Leu Val Val Glu Glu Val Phe
370 375 380
Asp Met Trp Asn Arg Ser Lys Asn Gly Asn Thr Glu Asp Tyr Gly Lys
385 390 395 400
Trp Phe Gly Gln Ala Ile Ala Gly Asp Asn Ala Val Leu Gly Gly Asp
405 410 415
Lys Asp Glu Thr Trp Ala Lys Phe Asp Leu Thr Ser Thr Ile Asn Arg
420 425 430
Asp Arg Asn Ala Pro Ser Val Ile Met Trp Ser Leu Gly Asn Glu Met
435 440 445
Met Glu Gly Ile Ser Gly Ser Val Ser Gly Phe Pro Ala Thr Ser Ala
450 455 460
Lys Leu Val Ala Trp Thr Lys Ala Ala Asp Ser Thr Arg Pro Met Thr
465 470 475 480
Tyr Gly Asp Asn Lys Ile Lys Ala Asn Trp Asn Glu Ser Asn Thr Met
485 490 495
Gly Asp Asn Leu Thr Ala Asn Gly Gly Val Val Gly Thr Asn Tyr Ser
500 505 510
Asp Gly Ala Asn Tyr Asp Lys Ile Arg Thr Thr His Pro Ser Trp Ala
515 520 525
Ile Tyr Gly Ser Glu Thr Ala Ser Ala Ile Asn Ser Arg Gly Ile Tyr
530 535 540
Asn Arg Thr Thr Gly Gly Ala Gln Ser Ser Asp Lys Gln Leu Thr Ser
545 550 555 560
Tyr Asp Asn Ser Ala Val Gly Trp Gly Ala Val Ala Ser Ser Ala Trp
565 570 575
Tyr Asp Val Val Gln Arg Asp Phe Val Ala Gly Thr Tyr Val Trp Thr
580 585 590
Gly Phe Asp Tyr Leu Gly Glu Pro Thr Pro Trp Asn Gly Thr Gly Ser
595 600 605
Gly Ala Val Gly Ser Trp Pro Ser Pro Lys Asn Ser Tyr Phe Gly Ile
610 615 620
Val Asp Thr Ala Gly Phe Pro Lys Asp Thr Tyr Tyr Phe Tyr Gln Ser
625 630 635 640
Gln Trp Asn Asp Asp Val His Thr Leu His Ile Leu Pro Ala Trp Asn
645 650 655
Glu Asn Val Val Ala Lys Gly Ser Gly Asn Asn Val Pro Val Val Val
660 665 670
Tyr Thr Asp Ala Ala Lys Val Lys Leu Tyr Phe Thr Pro Lys Gly Ser
675 680 685
Thr Glu Lys Arg Leu Ile Gly Glu Lys Ser Phe Thr Lys Lys Thr Thr
690 695 700
Ala Ala Gly Tyr Thr Tyr Gln Val Tyr Glu Gly Ser Asp Lys Asp Ser
705 710 715 720
Thr Ala His Lys Asn Met Tyr Leu Thr Trp Asn Val Pro Trp Ala Glu
725 730 735
Gly Thr Ile Ser Ala Glu Ala Tyr Asp Glu Asn Asn Arg Leu Ile Pro
740 745 750
Glu Gly Ser Thr Glu Gly Asn Ala Ser Val Thr Thr Thr Gly Lys Ala
755 760 765
Ala Lys Leu Lys Ala Asp Ala Asp Arg Lys Thr Ile Thr Ala Asp Gly
770 775 780
Lys Asp Leu Ser Tyr Ile Glu Val Asp Val Thr Asp Ala Asn Gly His
785 790 795 800
Ile Val Pro Asp Ala Ala Asn Arg Val Thr Phe Asp Val Lys Gly Ala
805 810 815
Gly Lys Leu Val Gly Val Asp Asn Gly Ser Ser Pro Asp His Asp Ser
820 825 830
Tyr Gln Ala Asp Asn Arg Lys Ala Phe Ser Gly Lys Val Leu Ala Ile
835 840 845
Val Gln Ser Thr Lys Glu Ala Gly Glu Ile Thr Val Thr Ala Lys Ala
850 855 860
Asp Gly Leu Gln Ser Ser Thr Val Lys Ile Ala Thr Thr Ala Val Pro
865 870 875 880
Gly Thr Ser Thr Glu Lys Thr Val Arg Ser Phe Tyr Tyr Ser Arg Asn
885 890 895
Tyr Tyr Val Lys Thr Gly Asn Lys Pro Ile Leu Pro Ser Asp Val Glu
900 905 910
Val Arg Tyr Ser Asp Gly Thr Ser Asp Arg Gln Asn Val Thr Trp Asp
915 920 925
Ala Val Ser Asp Asp Gln Ile Ala Lys Ala Gly Ser Phe Ser Val Ala
930 935 940
Gly Thr Val Ala Gly Gln Lys Ile Ser Val Arg Val Thr Met Ile Asp
945 950 955 960
Glu Ile Gly Ala Leu Leu Asn Tyr Ser Ala Ser Thr Pro Val Gly Thr
965 970 975
Pro Ala Val Leu Pro Gly Ser Arg Pro Ala Val Leu Pro Asp Gly Thr
980 985 990
Val Thr Ser Ala Asn Phe Ala ValHis Trp Thr Lys ProAla Asp Thr
995 1000 1005
Val TyrAsn Thr Ala Gly ThrVal Lys Val Pro GlyThr Ala Thr
1010 1015 1020
Val PheGly Lys Glu Phe LysVal Thr Ala Thr IleArg Val Gln
1025 1030 1035
Arg SerGln Val Thr Ile GlySer Ser Val Ser GlyAsn Ala Leu
1040 1045 1050
Arg LeuThr Gln Asn Ile ProAla Asp Lys Gln SerAsp Thr Leu
1055 1060 1065
Asp AlaIle Lys Asp Gly SerThr Thr Val Asp AlaAsn Thr Gly
1070 1075 1080
Gly GlyAla Asn Pro Ser AlaTrp Thr Asn Trp AlaTyr Ser Lys
1085 1090 1095
Ala GlyHis Asn Thr Ala GluIle Thr Phe Glu TyrAla Thr Glu
1100 1105 1110
Gln GlnLeu Gly Gln Ile ValMet Tyr Phe Phe ArgAsp Ser Asn
1115 1120 1125
Ala ValArg Phe Pro Asp AlaGly Lys Thr Lys IleGln Ile
1130 1135 1140
<210>5
<211>1211
<212>PRT
<213>两歧双歧杆菌(Bifidobacterium bifidum)
<400>5
Val Glu Asp Ala Thr Arg Ser Asp Ser Thr Thr Gln Met Ser Ser Thr
1 5 1015
Pro Glu Val Val Tyr Ser Ser Ala Val Asp Ser Lys Gln Asn Arg Thr
202530
Ser Asp Phe Asp Ala Asn Trp Lys Phe Met Leu Ser Asp Ser Val Gln
354045
Ala Gln Asp Pro Ala Phe Asp Asp Ser Ala Trp Gln Gln Val Asp Leu
505560
Pro His Asp Tyr Ser Ile Thr Gln Lys Tyr Ser Gln Ser Asn Glu Ala
65707580
Glu Ser Ala Tyr Leu Pro Gly Gly Thr Gly Trp Tyr Arg Lys Ser Phe
859095
Thr Ile Asp Arg Asp Leu Ala Gly Lys Arg Ile Ala Ile Asn Phe Asp
100 105 110
Gly Val Tyr Met Asn Ala Thr Val Trp Phe Asn Gly Val Lys Leu Gly
115 120 125
Thr His Pro Tyr Gly Tyr Ser Pro Phe Ser Phe Asp Leu Thr Gly Asn
130 135 140
Ala Lys Phe Gly Gly Glu Asn Thr Ile Val Val Lys Val Glu Asn Arg
145 150 155 160
Leu Pro Ser Ser Arg Trp Tyr Ser Gly Ser Gly Ile Tyr Arg Asp Val
165 170 175
Thr Leu Thr Val Thr Asp Gly Val His Val Gly Asn Asn Gly Val Ala
180 185 190
Ile Lys Thr Pro Ser Leu Ala Thr Gln Asn Gly Gly Asp Val Thr Met
195 200 205
Asn Leu Thr Thr Lys Val Ala Asn Asp Thr Glu Ala Ala Ala Asn Ile
210 215 220
Thr Leu Lys Gln Thr Val Phe Pro Lys Gly Gly Lys Thr Asp Ala Ala
225 230 235 240
Ile Gly Thr Val Thr Thr Ala Ser Lys Ser Ile Ala Ala Gly Ala Ser
245 250 255
Ala Asp Val Thr Ser Thr Ile Thr Ala Ala Ser Pro Lys Leu Trp Ser
260 265 270
Ile Lys Asn Pro Asn Leu Tyr Thr Val Arg Thr Glu Val Leu Asn Gly
275 280 285
Gly Lys Val Leu Asp Thr Tyr Asp Thr Glu Tyr Gly Phe Arg Trp Thr
290 295 300
Gly Phe Asp Ala Thr Ser Gly Phe Ser Leu Asn Gly Glu Lys Val Lys
305 310 315 320
Leu Lys Gly Val Ser Met His His Asp Gln Gly Ser Leu Gly Ala Val
325 330 335
Ala Asn Arg Arg Ala Ile Glu Arg Gln Val Glu Ile Leu Gln Lys Met
340 345 350
Gly Val Asn Ser Ile Arg Thr Thr His Asn Pro Ala Ala Lys Ala Leu
355 360 365
Ile Asp Val Cys Asn Glu Lys Gly Val Leu Val Val Glu Glu Val Phe
370 375 380
Asp Met Trp Asn Arg Ser Lys Asn Gly Asn Thr Glu Asp Tyr Gly Lys
385 390 395 400
Trp Phe Gly Gln Ala Ile Ala Gly Asp Asn Ala Val Leu Gly Gly Asp
405 410 415
Lys Asp Glu Thr Trp Ala Lys Phe Asp Leu Thr Ser Thr Ile Asn Arg
420 425 430
Asp Arg Asn Ala Pro Ser Val Ile Met Trp Ser Leu Gly Asn Glu Met
435 440 445
Met Glu Gly Ile Ser Gly Ser Val Ser Gly Phe Pro Ala Thr Ser Ala
450 455 460
Lys Leu Val Ala Trp Thr Lys Ala Ala Asp Ser Thr Arg Pro Met Thr
465 470 475 480
Tyr Gly Asp Asn Lys Ile Lys Ala Asn Trp Asn Glu Ser Asn Thr Met
485 490 495
Gly Asp Asn Leu Thr Ala Asn Gly Gly Val Val Gly Thr Asn Tyr Ser
500 505 510
Asp Gly Ala Asn Tyr Asp Lys Ile Arg Thr Thr His Pro Ser Trp Ala
515 520 525
Ile Tyr Gly Ser Glu Thr Ala Ser Ala Ile Asn Ser Arg Gly Ile Tyr
530 535 540
Asn Arg Thr Thr Gly Gly Ala Gln Ser Ser Asp Lys Gln Leu Thr Ser
545 550 555 560
Tyr Asp Asn Ser Ala Val Gly Trp Gly Ala Val Ala Ser Ser Ala Trp
565 570 575
Tyr Asp Val Val Gln Arg Asp Phe Val Ala Gly Thr Tyr Val Trp Thr
580 585 590
Gly Phe Asp Tyr Leu Gly Glu Pro Thr Pro Trp Asn Gly Thr Gly Ser
595 600 605
Gly Ala Val Gly Ser Trp Pro Ser Pro Lys Asn Ser Tyr Phe Gly Ile
610 615 620
Val Asp Thr Ala Gly Phe Pro Lys Asp Thr Tyr Tyr Phe Tyr Gln Ser
625 630 635 640
Gln Trp Asn Asp Asp Val His Thr Leu His Ile Leu Pro Ala Trp Asn
645 650 655
Glu Asn Val Val Ala Lys Gly Ser Gly Asn Asn Val Pro Val Val Val
660 665 670
Tyr Thr Asp Ala Ala Lys Val Lys Leu Tyr Phe Thr Pro Lys Gly Ser
675 680 685
Thr Glu Lys Arg Leu Ile Gly Glu Lys Ser Phe Thr Lys Lys Thr Thr
690 695 700
Ala Ala Gly Tyr Thr Tyr Gln Val Tyr Glu Gly Ser Asp Lys Asp Ser
705 710 715 720
Thr Ala His Lys Asn Met Tyr Leu Thr Trp Asn Val Pro Trp Ala Glu
725 730 735
Gly Thr Ile Ser Ala Glu Ala Tyr Asp Glu Asn Asn Arg Leu Ile Pro
740 745 750
Glu Gly Ser Thr Glu Gly Asn Ala Ser Val Thr Thr Thr Gly Lys Ala
755 760 765
Ala Lys Leu Lys Ala Asp Ala Asp Arg Lys Thr Ile Thr Ala Asp Gly
770 775 780
Lys Asp Leu Ser Tyr Ile Glu Val Asp Val Thr Asp Ala Asn Gly His
785 790 795 800
Ile Val Pro Asp Ala Ala Asn Arg Val Thr Phe Asp Val Lys Gly Ala
805 810 815
Gly Lys Leu Val Gly Val Asp Asn Gly Ser Ser Pro Asp His Asp Ser
820 825 830
Tyr Gln Ala Asp Asn Arg Lys Ala Phe Ser Gly Lys Val Leu Ala Ile
835 840 845
Val Gln Ser Thr Lys Glu Ala Gly Glu Ile Thr Val Thr Ala Lys Ala
850 855 860
Asp Gly Leu Gln Ser Ser Thr Val Lys Ile Ala Thr Thr Ala Val Pro
865 870 875 880
Gly Thr Ser Thr Glu Lys Thr Val Arg Ser Phe Tyr Tyr Ser Arg Asn
885 890 895
Tyr Tyr Val Lys Thr Gly Asn Lys Pro Ile Leu Pro Ser Asp Val Glu
900 905 910
Val Arg Tyr Ser Asp Gly Thr Ser Asp Arg Gln Asn Val Thr Trp Asp
915 920 925
Ala Val Ser Asp Asp Gln Ile Ala Lys Ala Gly Ser Phe Ser Val Ala
930 935 940
Gly Thr Val Ala Gly Gln Lys Ile Ser Val Arg Val Thr Met Ile Asp
945 950 955 960
Glu Ile Gly Ala Leu Leu Asn Tyr Ser Ala Ser Thr Pro Val Gly Thr
965 970 975
Pro Ala Val Leu Pro Gly Ser Arg Pro Ala Val Leu Pro Asp Gly Thr
980 985 990
Val Thr Ser Ala Asn Phe Ala ValHis Trp Thr Lys ProAla Asp Thr
995 1000 1005
Val TyrAsn Thr Ala Gly ThrVal Lys Val Pro GlyThr Ala Thr
1010 1015 1020
Val PheGly Lys Glu Phe LysVal Thr Ala Thr IleArg Val Gln
1025 1030 1035
Arg SerGln Val Thr Ile GlySer Ser Val Ser GlyAsn Ala Leu
1040 1045 1050
Arg LeuThr Gln Asn Ile ProAla Asp Lys Gln SerAsp Thr Leu
1055 1060 1065
Asp AlaIle Lys Asp Gly SerThr Thr Val Asp AlaAsn Thr Gly
1070 1075 1080
Gly GlyAla Asn Pro Ser AlaTrp Thr Asn Trp AlaTyr Ser Lys
1085 1090 1095
Ala GlyHis Asn Thr Ala GluIle Thr Phe Glu TyrAla Thr Glu
1100 1105 1110
Gln GlnLeu Gly Gln Ile ValMet Tyr Phe Phe ArgAsp Ser Asn
1115 1120 1125
Ala ValArg Phe Pro Asp AlaGly Lys Thr Lys IleGln Ile Ser
1130 1135 1140
Ala AspGly Lys Asn Trp ThrAsp Leu Ala Ala ThrGlu Thr Ile
1145 1150 1155
Ala AlaGln Glu Ser Ser AspArg Val Lys Pro TyrThr Tyr Asp
1160 1165 1170
Phe AlaPro Val Gly Ala ThrPhe Val Lys Val ThrVal Thr Asn
1175 1180 1185
Ala AspThr Thr Thr Pro SerGly Val Val Cys AlaGly Leu Thr
1190 1195 1200
Glu IleGlu Leu Lys Thr AlaThr
1205 1210
- 来自饲料类芽孢杆菌(Paenibacillus pabuli)的能够制造低聚半乳糖的酶及制造低聚半乳糖的方法
- 合成低聚半乳糖的专用菌株及用其合成低聚半乳糖的方法
