一种新型抗菌肽及其工程菌的构建方法和应用
文献发布时间:2024-04-18 20:00:50
技术领域
本发明属于基因工程领域,涉及一种新型抗菌肽及其工程菌的构建方法和应用。
背景技术
伊枯草菌素(Iturin)主要是由芽孢杆菌合成的环脂肽类化合物,其结构为7个α-氨基酸残基组成的环肽骨架与含有14-17个碳原子的β-氨基脂肪酸侧链连接而成。研究表明iturin具有广谱的抗菌活性,尤其对病原丝状真菌具有较强的抑制作用。此外,它还具有高稳定性、低毒、低残留等特点,在生物防治、果蔬贮藏和生物医药等领域具有广阔的应用前景。然而其结构复杂,难以化学直接合成,且大多数野生型菌株iturin产量低(30-430mg·L
启动子替换能够促进iturin、surfactin等环脂肽化合物的高效生物合成,然而,微生物代谢系统是由相互关联的代谢路径构成的复杂网络,这些基于启动子替换的静态调控策略,容易超载或破坏代谢网络失衡,导致有毒中间体的积累,底物转化率降低,产品得率降低。与静态调控相比,动态途径调控可平衡细胞生长和目的产物合成所需的物质流和能量流资源,可对目标产物进行精准调控,并可动态自主适应细胞内外环境的变化,如温度、渗透压和碳源的变化。
群体感应(quorum-sensing,QS)是随细胞培养密度变化而调控目标产物的动态调控系统,将细胞生长与基因表达耦合,以减少细胞“生长”和“生产”之间的细胞资源竞争,避免有毒代谢物的积累,动态调控目标化合物的生物合成。研究表明,AprE和NprE等胞外蛋白酶可降解ComX,并可能影响ComQXPA系统对群体感应系统启动子P
发明内容
本发明为构建高产iturin菌株,同时合成高抑菌活性,并具有新型结构的抗菌肽iturin,提供了一种新型抗菌肽的生产与应用技术。
为实现上述目的,本发明采用以下技术方案:
本发明的第一个目的是提供抗菌肽Iturin,所述Iturin化学结构式如式Ⅰ所示:
其中R为(CH
进一步的,前述的抗菌肽Iturin,所述R为(CH
或者所述R代表(CH
或者所述R代表(CH
或者所述R代表(CH
本发明的第二个目的是提供单产前述的抗菌肽iturin的菌株,所述前述的抗菌肽iturin的菌株采用以下方法构建得到:
(1)以Bacillus subtilis 1A751Δpps为出发菌株,敲除surfactin基因簇中的srfAB与srfAC之间接口区域的基因片段,构建得到不产生环脂肽的底盘工程菌株B.subtilis 1A751 WR;
在某个特殊的实施例中,所述surfactin基因簇中的srfAB与srfAC之间接口区域的基因片段如SQ ID NO.1所示;
(2)将iturin基因簇转入B.subtilis 1A751 WR,构建单产iturin菌株。
在某个特殊的实施例中,所述Iturin基因簇登录号为:GenBank OQ410964。
进一步的,所述方法还包括步骤(3):所述步骤(3)为将iturin基因簇的天然启动子P
本发明的第三个目的是提供一种构建前述菌株的方法,所述构建方法包括以下步骤:
(1)以Bacillus subtilis 1A751Δpps为出发菌株,敲除surfactin基因簇中的srfAB与srfAC之间接口区域的基因片段,构建得到不产生环脂肽的底盘工程菌株B.subtilis 1A751 WR;
在某个特殊的实施例中,所述surfactin基因簇中的srfAB与srfAC之间接口区域的基因片段如SQ ID NO.1所示;
(2)将iturin基因簇转入B.subtilis 1A751 WR,构建单产iturin菌株。
在某个特殊的实施例中,所述Iturin基因簇登录号为:GenBank OQ410964。
进一步的,所述方法还包括步骤(3):所述步骤(3)为将iturin基因簇的天然启动子P
本发明的第四个目的是提供前述一种提高抗菌肽Iturin产量的方法,所述方法为将前述菌株的iturin基因簇的天然启动子P
本发明的第五个目的是提供前述的抗菌肽Iturin或前述的单产iturin菌株或前述的单产iturin菌株的发酵提取物在制备抑菌剂或抑菌药物中的应用。
进一步的,所述抑菌剂或抑菌药物能够抑制病原细菌和/或病原真菌。
进一步的,所述病原细菌包括Clostridium perfringens、Bacillus anthracis、Staphylococcus aureus、Micrococcus tetragenus、Escherichia coli中的一种或多种的组合;所述病原真菌包括Fusarium matsutum,Botrytis cinerea,Fusarium tricinctum和Colletotrichum capsica中的一种或多种的组合。
进一步的,对病原细菌半数有效抑菌浓度(IC
本发明技术方案相对于现有技术具有以下有益效果:
本发明提供抗菌肽Iturin,以及产生所述的抗菌肽iturin的菌株。所述菌株能产生8种iturins,包括4种已知iturins和4种新型iturins,化学结构式如式Ⅰ所示。所述抗菌肽Iturin及该菌株发酵提取物对多种病原细菌和真菌具有较强的抑制作用,可用于生物防治、果蔬贮藏和生物医药等领域。本发明还提供提高所述菌株产量的方法,能够将所得菌株iturin产量增加到16.7g·L
附图说明
图1B.subtilis 1A751 WR的DNA测序图
图2利用TAR技术捕获iturin合成基因簇,其中图2A中所示1~5分别为酵母菌转化板上随机挑取的5个克隆子,图2B中由上至下分别为Kpn I和BamH I酶切含有iturin基因簇的重组质粒;
图3PCR验证B.subtilis 1A751 WR基因组中整合的iturin基因簇
图4利用CRISPR/Cas9技术替换菌株1A751 WR-itu中的iturin原始启动子;其中
图4A由左到右为PCR扩增的上游同源臂和融合三个不同启动子片段(P
图4B由上至下分别为PCR验证含有三个不同启动子片段的修复模板是否插入基因编辑载体,
图4C由左至右分别为PCR验证三个不同启动子片段是否成功替换iturin基因簇天然启动子;
图5利用CRISPRi技术单抑制和双抑制菌株1A751 WR-P
图5A为验证dCas9基因是否整合至目的宿主基因组,
图5B为验证靶向AprE基因的sgRNA1是否插入基因编辑载体,
图5C为验证靶向NprE基因的sgRNA2是否插入基因编辑载体,
图5D为验证sgRNA1和sgRNA2是否同时插入基因编辑载体;
图6HPLC分析启动子替换对iturin生产的影响
图7HPLC分析AprE和NprE基因在不同木糖终浓度和添加时间下的单抑制AprE基因对iturin生产的影响,其中
图7A为发酵24h添加不同终浓度(0/5/10/15g·L
图7B为发酵48h添加不同终浓度(0/5/10/15g·L
图7C为发酵72h添加不同终浓度(0/5/10/15g·L
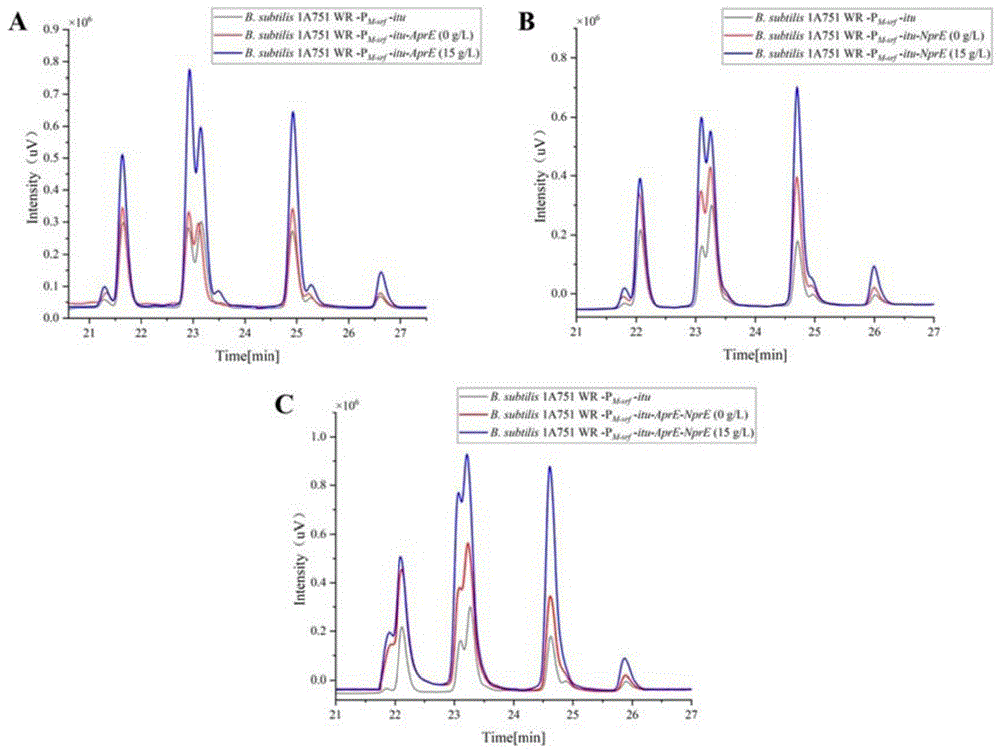
图8HPLC分析单抑制和双抑制AprE和NprE基因对菌株1A751 WR-P
图8A为发酵72h添加不同终浓度(0/15g/L)木糖下的单抑制AprE基因对iturin生产的影响,
图8B为发酵72h添加不同终浓度(0/15g/L)木糖下的单抑制NprE基因对iturin生产的影响,
图8C为发酵72h添加不同终浓度(0/15g/L)木糖下的双抑制AprE和NprE基因对iturin生产的影响;
图9利用UPLC-HRESI-MS和GC-MS鉴定新iturin结构C
图9A为C
图9B为C
图10利用UPLC-HRESI-MS和GC-MS鉴定新iturin结构C
图10A为C
图10B为C
图11利用UPLC-HRESI-MS和GC-MS鉴定新iturin结构C
图11A为C
图11B为C
图12利用UPLC-HRESI-MS和GC-MS鉴定新iturin结构C
图12A为C
图12B为C
图13C
图14利用外标法分析iturin的产量,其中
图14A为不同C
图14B为C
图15单产iturin(a),surfactin(b)和plipastatin(c)菌株的抗菌活性比较,其中
图15A为三种环脂肽提取物对五种病原细菌的抑制作用,所述1~5分别为Clostridium perfringens、Bacillus anthracis、Staphylococcus aureus、Micrococcustetragenus和Escherichia coli,
图15B为三种环脂肽及其提取物对四种病原真菌的抑制作用,所述1~4分别为Fusariummatsutum、Botrytis cinerea、Fusarium tricinctum和Colletotrichumcapsici,上排为三种环脂肽对四种病原真菌的平板对峙,下排为三种环脂肽提取物对四种病原真菌的抑制作用,M为甲醇;
图16单产iturin,surfactin和plipastatin菌株提取物在50%抑制浓度下的抗菌活性比较,其中
图16A为C.perfringens的剂量反应曲线,
图16B为B.anthracis的剂量反应曲线,
图16C为S.aureus的剂量反应曲线,
图16D为M.tetragenus的剂量反应曲线,
图16E为E.coli的剂量反应曲线;
图17单产iturin(a),surfactin(b)和plipastatin(c)菌株提取物和两性霉素B(AmB)的抗真菌活性的比较,其中
图17A由左至右分别为培养1d、2d和3d下的抗F.matsutum活性比较,
图17B由左至右分别为培养1d、2d和3d下的抗B.cinerea活性比较,
图17C由左至右分别为培养1d、2d和3d下的抗F.tricinctum活性比较,
图17D由左至右分别为培养1d、2d和3d下的抗C.capsici活性比较。
具体实施方式
以下结合实施例来进一步解释本发明,但实施例并不对本发明做任何形式的限定。
实施例1利用TAR技术构建单产iturin菌株
1、实验方法
基因编辑质粒构建:以E.coli/B.subtilis穿梭载体pJOE8999作为起始质粒;合成两条反向互补的sgRNA(SEQ ID NO.2、3所示sgRNA-Δsrf-F/R)单链核酸并退火,采用Bsa I介导的金门组装插入质粒pJOE8999中,转化至E.coli DH5α,在含有10μL 0.5M异丙基-β-D-硫代半乳糖苷(IPTG,0.5M)、40μL 20mg/mL5-溴-4-氯-3-吲哚-β-D-吡喃半乳糖苷(X-Gal,20mg·mL
底盘工程菌株的构建:将B.subtilis 1A751Δpps感受态细胞置于冰上融化10min,加入500ng基因编辑质粒pJOE8999-srfAB-AC并轻轻混匀,45℃热激90秒后,冰上静置2分钟。随后将混合液置于37℃慢摇孵育30分钟,添加5μL 30%D-甘露糖以诱导启动子P
捕获载体构建:以B.amyloliquefaciens HYM-12基因组DNA为模板,分别用引物itu-front-F/R(SEQ ID NO.10、11所示)和itu-back-F/R PCR(SEQ ID NO.12、13所示)扩增两个1kb的上下游同源臂;Sfp(0.8kb)和degQ(0.3kb)基因也分别使用引物sfp-F/R(SEQ IDNO.14、15所示)和degQ-F/R(SEQ ID NO.16、17所示)扩增而成;随后用引物itu-front-F(SEQ ID NO.10所示)和degQ-R(SEQ ID NO.17所示)通过重叠延伸PCR将这四个片段组装成大小为3kb左右的片段;其中,引物itu-front-F、itu-front-R和degQ-R分别含有Sal I、NheI和Sph I限制性酶切位点及相应的保护碱基。用Sal I和Sph I双酶切消化组装片段和pCAPB2载体骨架,利用T4连接酶进行连接,得到iturin基因簇特异性捕获载体pCAPB02-FBSD。
利用TAR技术捕获iturin生物合成基因簇(GenBank OQ410964):S.cerevisiaeVL6-48在添加1%腺嘌呤的YPD液体培养基中30℃培养,直至OD
2、实验结果
将基因编辑质粒转化至B.subtilis 1A751Δpps中,经DNA测序验证,发现srfAB和srfAC之间的基因组片段从8kb减少到2kb,与预期设计结果一致,说明成功敲除了srfAB-AC之间接口区域的基因片段(图1),得到了不产生环脂肽的底盘工程菌株B.subtilis 1A751WR。
如图2A所示,3号为潜在含有重组质粒pCAPB02-FBSD-iturin的酵母阳性克隆子,提取该质粒并用限制性内切酶Kpn I和BamH I酶切质粒,所得图谱各片段大小均与预期结果一致(图2B),表明iturin基因簇被成功捕获。通过枯草芽孢杆菌自然转化将其转入B.subtilis 1A751WR,随机选择5个转化子进行5’和3’接口PCR验证,同时得到1107bp和1313bp的目的条带(图3),表明iturin基因簇已成功整合到菌株1A751 WR基因组,获得单产iturin菌株B.subtilis1A751 WR-itu。
敲除序列如SEQ ID NO.1所示:
AATAGAAGCGAAAAAACATTTGCTGCTTTTAGATATGCATCATATCATCGCAGACGGCGTCTCAAGAGGCATCTTTGTAAAAGAATTGGCGCTGCTTTACAAAGGAGAGCAGCTTCCGGAGCCGACGCTTCATTATAAAGATTTCGCCGTTTGGCAAAATGAAGCTGAGCAAAAAGAACGGATGAAGGAGCATGAGGCGTACTGGATGTCAGTTCTTTCAGGCGAGCTGCCAGAGCTTGATCTCCCGCTCGATTATGCCCGTCCGCCTGTGCAAAGCTTTAAAGGAGATACGATCCGTTTCCGTACGGGAAGTGAGACGGCAAAGGCGGTAGAAAAACTGCTTGCCGAAACCGGAACGACCTTGCACATGGTGCTCCATGCTGTTTTCCACGTCTTTTTAAGCAAAATTTCCGGACAGCGGGATATCGTCATCGGCTCCGTTACTGCCGGCCGGACGAATGCTGATGTTCAGGACATGCCGGGAATGTTCGTCAATACACTTGCCCTGAGAATGGAAGCGAAAGAACAGCAAACATTTGCGGAGCTTTTGGAGCTAGCAAAGCAGACGAACCTGTCAGCCCTTGAGCATCAGGAGTATCCGTTTGAAGATCTGGTTAATCAGCTTGATCTCCCTCGGGATATGAGCCGAAACCCATTGTTTAATGTGATGGTGACGACAGAAAACCCTGATAAAGAACAGCTTACATTGCAAAATCTGAGCATTTCACCTTATGAGGCTCATCAGGGAACTTCTAAGTTTGATCTGACACTGGGCGGATTTACTGATGAAAATGGCATTGGCTTGCAGCTCGAATATGCGACAGATCTGTTCGCAAAAGAAACAGCTGAAAAATGGAGCGAATACGTTCTGCGGCTACTAAAGGCTGTTGCGGATAACCCGAACCAGCCGCTTTCCAGTCTGTTACTGGTCACCGAGACAGAAAAGCAAGCGCTTCTTGAGGCATGGAAGGGCAAAGCGCTGCCTGTGCCGACAGACAAAACGGTTCATCAGCTATTCGAAGAGACTGTCCAGCGCCACAAAGACCGCCCGGCTGTCACATACAACGGCCAATCTTGGACGTACGGCGAGCTGAACGCGAAGGCAAACCGCCTCGCCCGGATTCTGATGGACTGCGGCATCAGCCCGGATGACCGCGTCGGCGTTCTCACGAAGCCGTCGCTTGAAATGTCCGCCGCGGTGCTCGGCGTCTTGAAAGCCGGAGCGGCGTTTGTGCCGATTGATCCTGACTACCCGGATCAGCGGATTGAGTATATTTTACAGGACAGCGGCGCGAAGCTTCTCTTGAAACAGGAAGGCATTTCAGTGCCGGACAGCTATACGGGAGATGTCATTCTTCTCGACGGAAGCCGCACGATTCTAAGCCTGCCGCTTGATGAAAACGACGAGGGAAATCCAGAAACCGCTGTAACCGCGGAGAACTTGGCGTACATGATTTACACGTCTGGAACGACCGGACAGCCGAAGGGTGTCATGGTCGAGCACCATGCGCTTGTGAACCTGTGCTTCTGGCACCACGACGCGTTCAGCATGACAGCGGAGGACCGCAGTGCGAAGTACGCGGGCTTCGGGTTCGACGCTTCCATTTGGGAGATGTTCCCGACCTGGACGATCGGCGCTGAACTTCACGTCATTGATGAAGCGATCCGCCTCGATATCGTCCGCCTGAACGATTATTTTGAAACGAACGGCGTAACGATCACGTTCCTGCCGACACAGCTTGCGGAACAGTTCATGGAGCTTGAGAACACATCACTTCGCGTCCTCTTGACCGGAGGAGACAAGCTGAAGCGGGCAGTGAAAAAGCCGTACACACTCGTCAACAACTACGGGCCGACAGAAAATACGGTCGTTGCCACAAGCGCAGAAATCCATCCGGAGGAAGGCTCGCTTTCCATCGGACGGGCCATTGCCAATACGAGAGTATACATTCTCGGCGAGGGCAATCAGGTGCAGCCGGAAGGCGTAGCCGGAGAGCTTTGCGTGGCGGGGCGCGGACTGGCACGAGGCTATCTGAATCGAGAAGACGAAACCGCGAAGCGGTTTGTCGCTGATCCGTTTGTGCCGGGTGAACGCATGTACCGCACCGGCGACTTGGTGAAGTGGGTGAACGGCGGCATCGAATACATCGGCCGGATCGACCAGCAGGTCAAGGTCCGCGGCTACCGGATCGAGCTCTCAGAAATTGAAGTCCAGCTCGCCCAGCTTTCTGAGGTGCAGGATGCGGCGGTGACAGCTGTCAAAGATAAAGGCGGCAATACAGCGATCGCGGCGTATGTCACACCGGAAACAGCTGACATAGAAGCACTAAAATCAACACTAAAGGAAACCCTGCCGGATTACATGATCCCGGCGTTCTGGGTGACGCTGAACGAGCTTCCGGTTACGGCAAACGGCAAAGTCGACCGCAAAGCCTTGCCTGAGCCGGACATCGAAGCGGGAAGCGGAGAATACAAAGCGCCGACGACCGACATGGAAGAGCTGCTTGCCGGCATCTGGCAGGACGTGCTTGGAATGTCTGAAGTCGGTGTCACCGACAATTTCTTCTCGCTTGGCGGAGATTCCATCAAAGGAATTCAAATGGCGAGCCGCTTGAATCAGCACGGCTGGAAGCTGGAAATGAAAGATCTCTTCCAGCATCCGACGATCGAAGAGCTCACCCAGTACGTAGAGCGTGCCGAAGGCAAACAGGCAGACCAAGGCCCGGTGGAGGGCGAAGTCATCCTGACGCCGATCCAGCGCTGGTTCTTTGAAAAGAACTTCACGAACAAGCACCACTGGAACCAATCGGTGATGCTTCACGCCAAAAAGGGCTTTGATCCTGAACGGGTGGAGAAAACATTGCAGGCGCTGATCGAGCATCATGACGCGCTCCGCATGGTCTACCGCGAGGAAAACGGGGACATCGTTCAGGTGTATAAACCGATCGGTGAGAGCAAGGTCAGCTTCGAAATCGTGGATCTGTACGGCTCCGATGAAGAGATGCTGAGAAGCCAGATTAAGCTTCTCGCGAACAAGCTGCAAAGCAGTCTCGATCTGCGAAACGGGCCGCTTTTAAAGGCGGAGCAATATCGCACAGAAGCTGGGGATCACCTGCTCATTGCTGTACACCATCTCGTGGTCGACGGTGTGTCATGGCGGATTTTGCTTGAAGACTTTGCTTCAGGCTACATGCAGGCTGAGAAAGAAGAGAGCCTTGTCTTCCCGCAAAAAACAAACTCCTTCAAGGATTGGGCGGAAGAACTGGCGGCATTCAGCCAATCAGCGCATCTTTTACAGCAGGCTGAATACTGGTCGCAAATTGCCGCTGAACAGGTTTCTCCTTTACCTAAGGATTGTGAAACAGAGCAGCGGATCGTCAAGGATACATCATCTGTCCTATGTGAATTAACGGCAGAAGACACTAAGCATCTTTTAACAGATGTTCATCAGCCATATGGAACTGAAATCAACGATATTCTTCTCAGCGCGCTCGGTTTGACAATGAAAGAATGGACAAAGGGGGCCAAAATTGGCATTAACCTTGAGGGACACGGCCGGGAGGACATTATCCCGAATGTGAATATCTCCAGAACGGTCGGCTGGTTTACGGCACAATACCCTGTTGTGCTCGACATATCTGACGCAGATGCCTCAGCTGTGATCAAAACAGTCAAAGAAAACCTGCGCCGCATTCCGGACAAAGGTGTTGGCTACGGCATTCTTCGTTATTTCACAGAAACAGCGGAAACAAAGGGCTTCACACCGGAGATCAGCTTCAACTATTTGGGCCAATTCGACAGTGAAGTCAAAACCGATTTCTTTGAACCGTCCGCTTTCGATATGGGGCGCCAAGTAAGCGGAGAATCAGAGGCGCTGTACGCATTAAGCTTCAGCGGCATGATCAGAAACGGCCGGTTTGTGCTTTCCTGCTCCTACAATGAGAAGGAGTTTGAAAGAGCTACAGTCGAGGAGCAAATGGAACGGTTTAAAGAAAACCTCCTGATGCTAATCCGCCATTGCACGGAAAAAGAAGACAAGGAATTCACACCAAGCGACTTCAGCGCCGAAGACCTTGAAATGGACGAGATGGGAGATATCTTTGACATGCTTGAGGAGAATTTAAAATAAAACGCAAGGGAATTACAGAAGGCGGGAGCGAAACATATGAGTCAATTTAGCAAGGATCAGGTTCAAGATATGTATTACCTATCGCCGATGCAGGAAGGGATGCTTTTTCATGCCATCCTGAATCCCGGCCAAAGCTTTTACCTTGAACAAATCACGATGAAAGTAAAAGGCAGCTTGAATATCAAATGTCTTGAAGAAAGCATGAATGTGATCATGGACCGGTACGATGTATTTCGTACCGTGTTCATTCACGAAAAAGTAAAAAGGCCTGTCCAAGTCGTATTGAAAAAACGGCAGTTCCATATAGAAGAAATCGATCTGACACACTTAACGGGCAGCGAGCAAACAGCCAAAATCAATGAGTACAAAGAACAGGATAAGATCAGGGGTTTTGATTTGACGCGGGATATTCCGATGCGGGCAGCCATTTTCAAGAAAGCTGAAGAAAGCTTTGAATGGGTGTGGAGCTACCACCACATTATTTTGGACGGATGGTGCTTCGGCATCGTCGTACAGGATCTATTTAAGGTATACAATGCTCTGCGCGAACAAAAGCCGTACAGCCTGCCGCCCGTCAAACCGTATAAAGACTACATCAAGTGGCTTGAAAAGCAGGATAAACAAGCATCACTGCGTTACTGGCGCGAATATTTAGAGGGCTTTGAAGGACAAACGACGTTTGCGGAGCAAAGAAAGAAACAAAAGGACGGCTATGAGCCGAAAGAGCTGCTCTTTTCACTGTCGGAGGCGGAAACAAAGGCCTTTACCGAGCTTGCAAAATCGCAGCATACCACTTTGAGTACGGCGCTGCAGGCAGTCTGGAGCGTATTGATCAGCCGCTATCAGCAGTCTGGCGATTTGGCCTTCGGTACAGTTGTTTCAGGGCGTCCCGCGGAAATCAAAGGCGTTGAACATATGGTCGGGCTGTTTATCAACGTTGTCCCGAGACGTGTGAAGCTGTCTGAGGGTATCACATTTAACGGCTTGCTCAAGCGGCTGCAGGAGCAATCGCTGCAGTCCGAGCCGCATCAATATGTGCCGCTTTATGACATCCAAAGCCAGGCCGATCAGCCGAAACTGATTGACCACATCATTGTTTTTGAGAACTATCCGCTTCAGGATGCGAAAAATGAAGAAAGCAGTGAAAACGGCTTTGATATGGTGGATGTTCATGTTTTTGAGAAGTCGAATTATGATCTCAACCTGATGGCTTCCCCGGGAGATGAGATGCTGATTAAGCTTGCCTATAATGAGAATGTGTTTGATGAGGCGTTTATCCTGCGCTTGAAATCTCAGCTTCTTACAGCAATTCAGCAGCTCATCCAGAATCCTGATCAGCCTGTCAGCACGATCAACCTCGTTGACGACAGGGAGAGAGAATTTTTGCTAACCGGCTTAAACCCGCCGGCTCAAGCTCATGAAACAAAGCCTCTGACGTATTGGTTCAAGGAAGCAGTGAACGCCAATCCGGATGCACCGGCGCTTACGTATTCCGGCCAGACCCTGTCTTATCGCGAATTAGATGAGGAAGCGAACCGCATTGCACGCCGGCTGCAAAAACACGGTGCGGGCAAAGGCTCTGTTGTAGCGCTGTACACGAAGCGCTCACTTGAACTGGTGATCGGCATTCTCGGTGTATTAAAGGCGGGAGCAGCCTATCTGCCGGTTGATCCGAAGCTGCCAGAGGACCGAATCTCGTATATGCTGGCTGACAGTGCGGCAGCCTGTCTTCTGACGCATCAGGAGATGAAAGAACAAGCGGCTGAGCTGCCGTATACAGGCACAACGCTCTTCATTGATGATCAAACACGGTTTGAAGAACAGGCAAGCGATCCTGCAACCGCGATTGATCCTAATGATCCGGCATATA(5999 bp)
实施例2利用CRISPR/Cas9技术构建iturin启动子替换菌株
1、实验方法
合成两条反向互补的sgRNA(SEQ ID NO.18、19所示P-sgRNA-F/R)单链核酸并退火,采用BsaI-HFv2介导的金门组装插入质粒pJOE8999中,转化至E.coli DH5α,在含有IPTG(0.5M)、X-Gal(20mg·mL
利用芽孢杆菌自然感受态转化,将三个启动子(P
2、实验结果
PCR分别扩增得到了P
经菌落PCR筛选获得启动子成功替换的阳性单克隆:启动子P
实施例3利用CRISPRi技术构建AprE和NprE基因抑制菌株
1、实验方法
利用芽孢杆菌自然感受态转化,将质粒pJMP1-loxCm转入实施例2构建的菌株B.subtilis1A751 WR-P
利用金门组装将靶向AprE基因的sgRNA1(SEQ ID NO.37、38所示AprE-sgRNA-F/R)和靶向NprE基因的sgRNA2(SEQ ID NO.39、40所示NprE-sgRNA-F/R)分别插入质粒PsgRNA中,利用PCR验证(SEQ ID NO.45所示Verify-F/SEQ ID NO.42所示PsgRNA-AprE-R和SEQ IDNO.44所示PsgRNA-NprE-R,843bp)以得到质粒PsgRNA-AprE和PsgRNA-NprE。以它们为模板,利用引物(SEQ ID NO.41、42所示PsgRNA-AprE-F/R和SEQ ID NO.43、44所示PsgRNA-NprE-F/R)分别PCR扩增得到长度为3158和235bp的两条片段,再次利用金门组装,PCR验证(SEQID NO.46、47所示YZ-sgRNA-F/R,970bp)得到双sgRNA质粒PsgRNA-AprE-NprE。随后,利用芽孢杆菌自然感受态转化,将三个质粒分别转入菌株1A751 WR-itu-dCas9中,利用相同引物进行PCR验证质粒是否成功转入。
2、实验结果
经菌落PCR筛选获得dCas9基因成功整合的阳性单克隆,其中整合片段与目的基因组的5’和3’接口和部分dCas9基因片段均扩增得到预期条带大小,分别为1025bp、1199bp和1113bp(图5A),说明dCas9基因成功整合至目的基因组lacA位点。
采用金门组装将靶向AprE基因的sgRNA1和靶向NprE基因的sgRNA2分别插入质粒PsgRNA中,经菌落PCR验证均得到了843bp的目的条带,将两个单基因抑制质粒分别命名为PsgRNA-AprE和PsgRNA-NprE。以它们为模板,经PCR扩增并结合金门组装,成功构建了双基因抑制质粒PsgRNA-AprE-NprE。单抑制和双抑制菌株分别得到大小为843bp(图5B、5C)和970bp(图5D)的目的条带,将单抑制菌株命名为B.subtilis 1A751 WR-P
表1本发明所用菌株和质粒
/>
/>
表2本发明所用引物
/>
/>
实施例4分析工程菌株的iturin产量
1、实验方法
三株不同启动子替换菌株发酵:将实施例2获得的三株不同启动子替换菌分别接种在含有Spec抗性的5mL种子培养基中,37℃、180r·min
基因抑制菌株发酵:将实施例3获得的单抑制菌株1A751 WR-P
HPLC分析(Shimadzu LC-20AD,Kyoto,Japan):采用Aglilent RT C18 column(4.6mm×150mm,5μm,Aglilent,Santa Clara,California,USA),进样量20μL,流速0.75mL·min
UPLC-HRESI-MS分析(G2-XS QTof,Waters,Milford,Massachusetts,USA):通过UPLC柱(2.1mm×100mm,1.7μm,Waters)进行,MS系统以0.3mL·min
GC-MS分析(TSQ 9000,Thermo Fisher,Waltham,Massachusetts,USA):将获得的衍生物溶解在正己烷中,并通过配备有Agilent J&W HP-5MS柱(30m×0.25mm×0.25μm,Agilent)的GC-MS进行分析。柱烘箱在140℃下启动并保持3分钟,然后在4℃·min
环脂肽iturin的分离、纯化和制备:采用制备液相色谱仪(Shimadzu LC-20AT),Aglilent ZORBAX SB-C18柱(9.4mm×250mm,5μm,Aglilent),样品浓度20mg·mL
2、实验结果
HPLC图谱显示(图6),在启动子P
HPLC图谱显示(图7),不加入木糖的单抑制AprE菌株iturin产量均与母体菌株相当。在不同添加时间下,终浓度为5g·L
通过UPLC-HRESI-MS/MS分析菌株的发酵提取物,结果表明该菌株合成了8种iturins,包括4种可能新的iturins和4种已知的iturins。m/z 987.4921(1)、m/z1001.5081(2)、m/z1015.5223(3)和m/z 1029.5393(4)处的分子离子不同于所有已知iturin,推测它们是含有10-13个碳原子脂肪酸侧链的新iturin衍生物的分子离子。m/z987.4921处的信号归属于分子式C
为定量分析启动子替换和基因抑制工程菌的iturin产量,对单产iturin菌株1A751 WR-itu发酵产物进行分离、纯化和制备。HPLC图谱显示(图13),C
表3不同工程菌株的iturin产量
结果显示:在启动子P
实施例5工程菌株抑菌活性研究
1、实验方法
在改良Landy培养基中培养5天后,经酸沉获得发酵提取物,方法同上。利用对峙实验比较iturin,surfactin和plipastatin单产菌株对病原丝状真菌的抑制活性。通过凝胶扩散法比较不同提取物对病原细菌和真菌抑制活性的差异。采用肉汤稀释法测定不同提取物的50%抑制浓度(IC
2、实验结果
Surfactin和plipastatin对5种致病菌表现出较强的抑制活性,而iturin的抑制活性相对较弱(图15A),最大抑制直径分别为24、25和22mm。Iturin,surfactin和plipastatin对S.aureus的IC
表4单产iturin,surfactin和plipastatin菌株提取物的50%抑菌浓度的统计分析
结果表明,iturin,surfactin和plipastatin具有较强的抗细菌作用,对S.aureus和B.anthracis的抑制作用最为显著。
对于病原真菌,对峙实验表明,只有单产iturin菌株表现出强大的抑制作用(图15B),而单产surfactin和plipastatin菌株的抑制作用较弱。提取物的抗真菌作用与对峙实验结果相同,iturin对四种病原真菌均有较强的抑制作用,其最大抑制直径为25mm。含有iturin的培养基在很长一段时间内始终保持明显的抑菌圈,而其他两种环脂肽的抑菌圈仅为8mm,AmB的抑菌圈也在第三天基本消失(图17),说明iturin对真菌表现出更有效和持久的抑制作用。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
