用于生产乙醇的表达酶的酵母
文献发布时间:2023-06-19 12:16:29
序列表的引用
本申请含有计算机可读形式的序列表,将其通过引用并入本文。
背景技术
从含淀粉材料和含纤维素材料生产乙醇是本领域中熟知的。
对于含淀粉材料,工业上最常使用的商业方法(通常被称作“传统方法”)包括在高温(约85℃)下典型地使用细菌α-淀粉酶液化经糊化的淀粉,随后典型地在葡糖淀粉酶和酿酒酵母(Saccharomyces cerevisae)的存在下厌氧进行同时糖化和发酵(SSF)。
用于生产用作燃料的乙醇的酵母,如在玉米乙醇工业中,要求几种特性,以确保乙醇有效生产的成本。这些特性包括乙醇耐受性、低副产品产量、快速发酵、和限制残留在发酵中的剩余糖量的能力。这些特性对工业方法的可行性具有明显的作用。
酵母菌属的酵母表现出生产乙醇所需的许多特性。具体地,酿酒酵母的菌株在燃料乙醇工业中广泛用于乙醇生产。酿酒酵母的菌株被广泛用于燃料乙醇工业,具有在发现于,例如,玉米醪发酵中的发酵条件下生产高产量乙醇的能力。这种菌株的实例是在称为
酿酒酵母已经被基因工程化以表达α-淀粉酶和/或葡糖淀粉酶从而提高产量并减少SSF期间必需的外源添加的酶的量(例如,WO 2018/098381、WO 2017/087330、WO 2017/037614、WO 2011/128712、WO 2011/153516、US 2018/0155744)。酵母也已经被工程化以表达海藻糖酶,从而试图通过分解残留的海藻糖来增加发酵产量(例如,WO 2017/077504)。
WO 2008/135547涉及在用于通过使包含发酵生物的发酵培养基与选自由磷脂酶、溶血磷脂酶和脂肪酶组成的组中的脂解酶、以及金属盐接触而产生发酵产物的方法中减少泡沫。
WO 2014/147219涉及来自雷塞氏篮状菌(Talaromyces leycettanus)的磷脂酶A。
WO 2015/140275披露了来自苏云金芽孢杆菌的磷脂酶C。
尽管在过去数十年乙醇生产方法有显著的改善,但是仍然希望并需要以经济和商业相关的规模,提供改善的从含淀粉材料和含纤维素材料发酵乙醇的方法。
例如,乙醇发酵过程中的泡沫产生是主要问题,尤其是其中将含淀粉材料在糖化和发酵之前使用α-淀粉酶和蛋白酶进行液化的乙醇生产过程中。另外,在发酵期间使用氮补充剂(例如尿素)是增加的费用。因此,尤其需要在乙醇发酵中减少泡沫和/或减少补充氮的需求。
发明内容
本文尤其描述了从含淀粉材料或含纤维素材料生产发酵产物(如乙醇)的方法,以及适用于此类方法的酵母。申请人惊奇地发现,表达磷脂酶的酵母在发酵过程中提供有益的特性,例如减少起泡,提高油提取产率和提高乙醇产率。
第一方面涉及从含淀粉材料或含纤维素材料生产发酵产物的方法,这些方法包括:(a)糖化所述含淀粉材料或含纤维素材料;和(b)用发酵生物发酵步骤(a)的经糖化的材料;其中所述发酵生物包含编码磷脂酶的异源多核苷酸。在一些实施例中,该磷脂酶是磷脂酶A或磷脂酶C。
在这些方法的一些实施例中,在同时糖化和发酵(SSF)中同时进行发酵和糖化。在其他实施例中,顺序进行发酵和糖化(SHF)。
在这些方法的一些实施例中,该方法包括从该发酵中(例如,通过蒸馏)回收该发酵产物。
在这些方法的一些实施例中,该发酵产物是乙醇。
在这些方法的一些实施例中,发酵在减氮条件下(例如,少于1000ppm尿素或氢氧化铵,如少于750ppm、少于500ppm、少于400ppm、少于300ppm、少于250ppm、少于200ppm、少于150ppm、少于100ppm、少于75ppm、少于50ppm、少于25ppm、或少于10ppm)进行。
在所述方法的一些实施例中,在相同条件(例如,在发酵约54小时时或在发酵54小时后,如实例3或4中描述的条件)下,当与使用没有编码磷脂酶的异源多核苷酸的相同细胞的相同方法相比时,所述方法导致发酵产物(例如,乙醇)的更高产率和/或减少的泡沫积累。在一些实施例中,所述方法导致发酵产物的产率高出至少0.25%(例如0.5%、0.75%、1.0%、1.25%、1.5%、1.75%、2%、3%或5%)。
在这些方法的一些实施例中,磷脂酶具有与SEQ ID NO:235-242和252-342中任一个的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。在这些方法的一些实施例中,磷脂酶具有与SEQID NO:235、236、237、238、239、240、241和242中任一个的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码磷脂酶,所述磷脂酶具有与SEQ IDNO:235-242和252-342中任一个(例如,SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在这些方法的一些实施例中,该异源多核苷酸编码磷脂酶,该磷脂酶具有包含SEQ IDNO:235-242和252-342中任一个(例如SEQ ID NO:SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列或由其组成的成熟多肽序列。
在这些方法的一些实施例中,糖化步骤发生在含淀粉材料上,并且其中该含淀粉材料是糊化的或未糊化的淀粉。
在这些方法的一些实施例中,该方法包括在糖化之前,通过使该含淀粉材料与α-淀粉酶接触来液化该材料。
在这些方法的一些实施例中,液化含淀粉材料和/或糖化含淀粉材料在外源添加的蛋白酶存在下进行。
在这些方法的一些实施例中,发酵生物包含编码葡糖淀粉酶的异源多核苷酸,例如具有成熟多肽序列的葡糖淀粉酶,所述成熟多肽序列与密孔菌属葡糖淀粉酶(例如SEQID NO:229的血红密孔菌葡糖淀粉酶)、粘褶菌属葡糖淀粉酶(例如SEQ ID NO:8的篱边粘褶菌)、或SEQ ID NO:102-113中任一个的葡糖淀粉酶(例如SEQ ID NO:103或104的扣囊复膜酵母菌葡糖淀粉酶或SEQ ID NO:230的里氏木霉葡糖淀粉酶)的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性。
在这些方法的一些实施例中,发酵生物包含编码α-淀粉酶的异源多核苷酸,例如具有与SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列的α-淀粉酶。在这些方法的一些实施例中,异源多核苷酸编码α-淀粉酶,所述α-淀粉酶具有与SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码具有成熟多肽序列的α-淀粉酶,所述成熟多肽序列包含SEQ ID NO:SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列或由其组成。
在这些方法的一些实施例中,发酵生物包含编码海藻糖酶的异源多核苷酸,例如具有与SEQ ID NO:175-226中任一个的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列的海藻糖酶。在这些方法的一些实施例中,异源多核苷酸编码海藻糖酶,所述海藻糖酶具有与SEQ IDNO:175-226中任一个的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码具有成熟多肽序列的海藻糖酶,所述成熟多肽序列包含SEQ ID NO:SEQ ID NO:175-226中任一个的氨基酸序列或由其组成。
在这些方法的一些实施例中,发酵生物包含编码蛋白酶的异源多核苷酸,该蛋白酶具有与SEQ ID NO:9-73中任一个(例如SEQ ID NO:9、14、16、21、22、33、41、45、61、62、66、67和69中任一个;如SEQ NO:9、14、16和69中任一个)的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在这些方法的一些实施例中,糖化步骤发生在含纤维素材料上,并且其中该含纤维素材料经过预处理(例如,稀酸预处理)。
在这些方法的一些实施例中,对含纤维素材料进行糖化,并且其中该酶组合物包含一种或多种选自以下的酶:纤维素酶(例如,内切葡聚糖酶、纤维二糖水解酶、或β-葡糖苷酶),AA9多肽,半纤维素酶(例如,木聚糖酶、乙酰木聚糖酯酶、阿魏酸酯酶、阿拉伯呋喃糖苷酶、木糖苷酶、或葡糖醛酸糖苷酶),CIP,酯酶,棒曲霉素,木质素分解酶,氧化还原酶,果胶酶,蛋白酶、以及膨胀素。
在这些方法的一些实施例中,该发酵生物是酵母属(Saccharomyces)、红酵母属(Rhodotorula)、裂殖酵母属(Schizosaccharomyces)、克鲁维酵母属(Kluyveromyces)、毕赤酵母属(Pichia)、汉逊酵母属(Hansenula)、红冬孢酵母属(Rhodosporidium)、假丝酵母属(Candida)、耶氏酵母属(Yarrowia)、油脂酵母属(Lipomyces)、隐球菌属(Cryptococcus)或德克拉酵母属(Dekkera)菌种细胞。在一些实施例中,该发酵生物是酿酒酵母细胞。
另一个方面涉及包含编码磷脂酶的异源多核苷酸的重组酵母细胞。在一些实施例中,该磷脂酶是磷脂酶A或磷脂酶C。
在所述酵母细胞的一些实施例中,在相同条件(例如,在发酵约54小时时或在发酵54小时后,如实例3或4中描述的条件)下,当与使用相同过程和没有编码磷脂酶的异源多核苷酸的相同细胞相比时,所述细胞导致发酵产物的更高产率和/或减少的泡沫积累。在一些实施例中,所述细胞导致发酵产物的产率高出至少0.25%(例如0.5%、0.75%、1.0%、1.25%、1.5%、1.75%、2%、3%或5%)。
在一些实施例中,该重组酵母细胞是酵母属、红酵母属、裂殖酵母属、克鲁维酵母属、毕赤酵母属、汉逊酵母属、红冬孢酵母属、假丝酵母属、耶氏酵母属、油脂酵母属、隐球菌属或德克拉酵母属菌种细胞。在一些实施例中,该重组酵母细胞是酿酒酵母细胞。
在酵母细胞的一些实施例中,发酵生物包含编码葡糖淀粉酶的异源多核苷酸,例如具有成熟多肽序列的葡糖淀粉酶,所述成熟多肽序列与密孔菌属葡糖淀粉酶(例如SEQID NO:229的血红密孔菌葡糖淀粉酶)、粘褶菌属葡糖淀粉酶(例如SEQ ID NO:8的篱边粘褶菌)、或SEQ ID NO:102-113中任一个的葡糖淀粉酶(例如SEQ ID NO:103或104的扣囊复膜酵母菌葡糖淀粉酶或SEQ ID NO:230的里氏木霉葡糖淀粉酶)的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性。
在酵母细胞的一些实施例中,发酵生物包含编码α-淀粉酶的异源多核苷酸,其中所述α-淀粉酶具有与SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码α-淀粉酶,所述α-淀粉酶具有与SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码具有成熟多肽序列的α-淀粉酶,所述成熟多肽序列包含SEQ ID NO:SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列或由其组成。
在酵母细胞的一些实施例中,发酵生物包含编码海藻糖酶的异源多核苷酸,其中所述海藻糖酶具有与SEQ ID NO:175-226中任一个的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码海藻糖酶,所述海藻糖酶具有与SEQ IDNO:175-226中任一个的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在这些方法的一些实施例中,异源多核苷酸编码具有成熟多肽序列的海藻糖酶,所述成熟多肽序列包含SEQ ID NO:SEQ ID NO:175-226中任一个的氨基酸序列或由其组成。
在酵母细胞的一些实施例中,发酵生物包含编码蛋白酶的异源多核苷酸,例如具有与SEQ ID NO:9-73中任一个(例如SEQ ID NO:9、14、16、21、22、33、41、45、61、62、66、67和69中任一个;如SEQ NO:9、14、16和69中任一个)的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列的蛋白酶。
附图说明
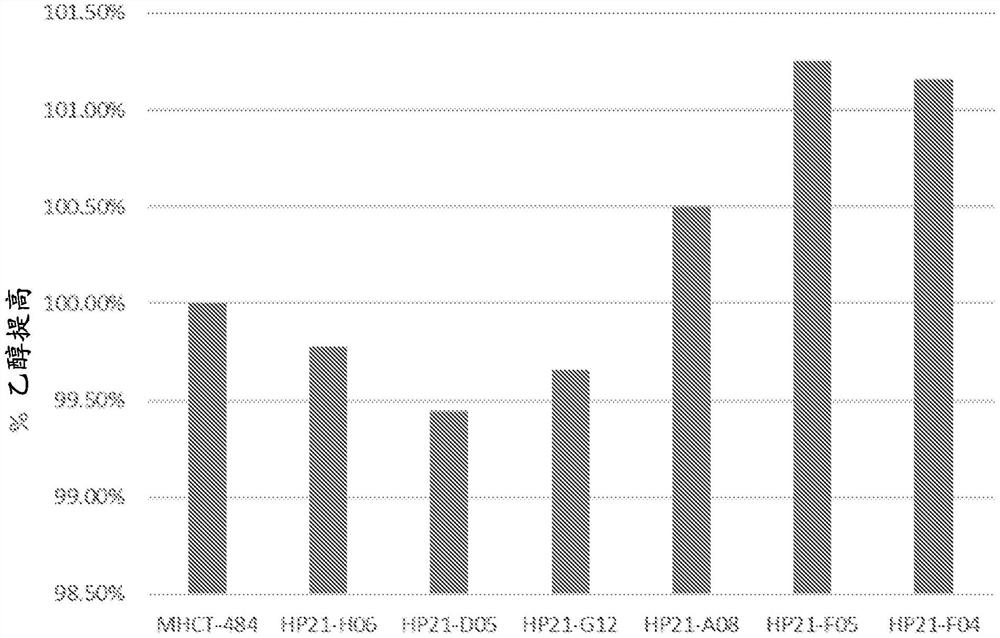
图1显示了实例3中所述的表达磷脂酶的酵母菌株和对照菌株MHCT-484的乙醇提高%。
图2显示了如实例4中所述的在0和150ppm尿素下AMP醪发酵54小时后的标准化平均乙醇提高。
图3显示了如实例4中所述的在0和300ppm尿素下非AMP醪发酵54小时后的标准化平均乙醇提高。
图4显示了如实例9所述,与对照菌株yMHCT48(左)相比,表达磷脂酶的酵母菌株HP21-FO4(右)的发酵过程中提高的消泡能力。
定义
除非由上下文以另外的方式定义或清楚表明,本文使用的所有技术术语和科学术语具有如本领域普通技术人员所通常理解的相同的含义。
等位基因变体:术语“等位基因变体”意指占据同一染色体基因座的基因的两种或更多种替代形式中的任一种。等位基因变异通过突变而自然产生,并且可以导致群体内部的多态性。基因突变可以是沉默的(所编码的多肽无变化)或可以编码具有改变的氨基酸序列的多肽。多肽的等位基因变体是由基因的等位基因变体编码的多肽。
α-淀粉酶:术语“α淀粉酶”意指1,4-α-D-葡聚糖葡聚糖水解酶(EC.3.2.1.1),其催化淀粉以及其他直链和支链1,4-糖苷寡糖和多糖的水解。出于本发明的目的,可以使用以下实例部分中描述的α-淀粉酶测定来测定α淀粉酶活性。
辅助活性9:术语“辅助活性9”或“AA9”意指分类为溶解性多糖单加氧酶(Quinlan等人,2011,Proc.Natl.Acad.Sci.USA[美国国家科学院院刊]208:15079-15084;Phillips等人,2011,ACS Chem.Biol.[ACS化学生物学]6:1399-1406;Lin等人,2012,Structure[结构]20:1051-1061)的多肽。根据Henrissat,1991,Biochem.J.[生物化学杂志]280:309-316以及Henrissat和Bairoch,1996,Biochem.J.[生物化学杂志]316:695-696,AA9多肽之前被分类为糖苷水解酶家族61(GH61)。
AA9多肽通过具有纤维素分解活性的酶增强含纤维素材料的水解。可以通过测量在以下条件下来自由纤维素分解酶水解含纤维素材料的还原糖的增加或纤维二糖与葡萄糖总量的增加来确定纤维素分解增强活性:1-50mg总蛋白/g预处理的玉米秸秆(PCS)中的纤维素,其中总蛋白包括50-99.5%w/w纤维素分解酶蛋白和0.5-50%w/w AA9多肽蛋白,在适合的温度(例如40℃-80℃,例如,50℃、55℃、60℃、65℃、或70℃)、和适合的pH(例如4-9,例如,4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、或8.5)下持续1-7天,与不具有纤维素分解增强活性的相等的总蛋白负载的对照水解(1-50mg纤维素分解蛋白/g PCS中的纤维素)进行比较。
可以使用
AA9多肽增强活性还可以通过以下来确定:在40℃,将AA9多肽与0.5%磷酸溶胀纤维素(PASC)、100mM乙酸钠(pH 5)、1mM MnSO
还可以根据WO 2013/028928确定高温组合物的AA9多肽增强活性。
AA9多肽通过将达到相同的水解程度所需要的纤维素分解酶的量降低优选至少1.01倍,例如至少1.05倍、至少1.10倍、至少1.25倍、至少1.5倍、至少2倍、至少3倍、至少4倍、至少5倍、至少10倍、或至少20倍,来增强由具有纤维素分解活性的酶催化的含纤维素材料的水解。
β-葡糖苷酶:术语“β-葡糖苷酶”意指β-D-葡糖苷葡糖水解酶(beta-D-glucosideglucohydrolase)(E.C.3.2.1.21),其催化末端非还原β-D-葡萄糖残基的水解,并释放β-D-葡萄糖。可以根据Venturi等人,2002,J.Basic Microbiol.[基础微生物学杂志]42:55-66的程序使用对硝基苯基-β-D-吡喃葡萄糖苷作为底物来确定β-葡糖苷酶活性。一个单位的β-葡糖苷酶定义为在25℃,pH 4.8下,在含有0.01%
β-木糖苷酶:术语“β-木糖苷酶”意指β-D-木糖苷木糖水解酶(β-D-xylosidexylohydrolase)(E.C.3.2.1.37),其催化短β(1→4)-低聚木糖的外切水解,以将连续的D-木糖残基从非还原端移除。可以在含有0.01%
过氧化氢酶:术语“过氧化氢酶”意指过氧化氢:过氧化氢氧化还原酶(EC1.11.1.6),该酶催化2H
催化结构域:术语“催化结构域”意指含有该酶的催化机构的酶的区域。
纤维二糖水解酶:术语“纤维二糖水解酶”意指1,4-β-D-葡聚糖纤维二糖水解酶(E.C.3.2.1.91和E.C.3.2.1.176),其催化纤维素、纤维寡糖、或任何含β-1,4-连接的葡萄糖的聚合物中的1,4-β-D-糖苷键的水解,从该链的还原端(纤维二糖水解酶I)或非还原端(纤维二糖水解酶II)释放纤维二糖(Teeri,1997,Trends in Biotechnology[生物技术趋势]15:160-167;Teeri等人,1998,Biochem.Soc.Trans.[生化学会会刊]26:173-178)。可以根据由以下所述的程序来确定纤维二糖水解酶活性:Lever等人,1972,Anal.Biochem.[分析生物化学]47:273-279;van Tilbeurgh等人,1982,FEBS Letters[欧洲生化学会联合会快报]149:152-156;van Tilbeurgh和Claeyssens,1985,FEBS Letters[欧洲生化学会联合会快报]187:283-288;和Tomme等人,1988,Eur.J.Biochem.[欧洲生物化学杂志],170:575-581。
纤维素分解酶或纤维素酶:术语“纤维素分解酶”或“纤维素酶”意指水解含纤维素材料的一种或多种(例如,若干种)酶。此类酶包括一种或多种内切葡聚糖酶、一种或多种纤维二糖水解酶、一种或多种β-葡糖苷酶、或其组合。用于测量纤维素分解酶活性的两种基本方法包括:(1)测量总纤维素分解酶活性,以及(2)测量个体纤维素分解酶活性(内切葡聚糖酶、纤维二糖水解酶和β-葡糖苷酶),如在Zhang等人,2006,Biotechnology Advances[生物技术进展]24:452-481中所述的。可使用不溶性底物,包括沃特曼(Whatman)№1滤纸、微晶纤维素、细菌纤维素、藻类纤维素、棉花、预处理的木质纤维素等,测量总纤维素分解酶活性。最常见的总纤维素分解活性测定是将沃特曼№1滤纸用作底物的滤纸测定。该测定是由国际纯粹与应用化学联合会(IUPAC)建立的(Ghose,1987,Pure Appl.Chem.[纯粹与应用化学]59:257-68)。
可以通过测量在以下条件下,由一种或多种纤维素分解酶进行的含纤维素材料水解期间,糖的产生/释放的增加来确定纤维素分解酶活性:1-50mg纤维素分解酶蛋白/g预处理的玉米秸秆(PCS)中的纤维素(或其他预处理的含纤维素材料),在适合的温度(例如40℃-80℃,例如,50℃、55℃、60℃、65℃、或70℃)下,以及在适合的pH(例如4-9,例如,5.0、5.5、6.0、6.5、或7.0)下持续3-7天,与未添加纤维素分解酶蛋白的对照水解相比。典型条件为:1mL反应,洗涤或未洗涤的PCS,5%不溶性固体(干重),50mM乙酸钠(pH 5),1mM MnSO
编码序列:术语“编码序列”或“编码区”意指指定多肽的氨基酸序列的多核苷酸序列。编码序列的边界一般由开放阅读框决定,该开放阅读框通常以ATG起始密码子或替代起始密码子(如GTG和TTG)开始,并且以终止密码子(如TAA、TAG、和TGA)结束。编码序列可以是基因组DNA、cDNA、合成的多核苷酸、和/或重组多核苷酸的序列。
控制序列:术语“控制序列”意指多肽表达所必需的核酸序列。控制序列对于编码多肽的多核苷酸可以是天然的或外源的,并且彼此可以是天然的或外源的。此类控制序列包括但不限于,前导子序列、多腺苷酸化序列、前肽序列、启动子序列、信号肽序列和转录终止子序列。出于引入有利于将控制序列与编码多肽的多核苷酸的编码区连接的特异性限制位点的目的,这些控制序列可以提供有多个接头。
破坏:术语“破坏”意指参照基因的编码区和/或控制序列被部分或完全修饰(如通过缺失、插入和/或取代一个或多个核苷酸),从而使得编码的多肽的表达不存在(失活)或降低,和/或编码的多肽的酶活性不存在或降低。可以使用本领域已知的技术来测量破坏的效果,例如使用本文引用的无细胞提取物测量来检测酶活性的缺乏或降低;或通过相应的mRNA的缺失或降低(例如降低至少25%、降低至少50%、降低至少60%、降低至少70%、降低至少80%、或降低至少90%);具有酶活性的相应多肽的量的缺失或降低(例如降低至少25%、降低至少50%、降低至少60%、降低至少70%、降低至少80%、或降低至少90%);或具有酶活性的相应多肽的特定活性的缺失或降低(例如降低至少25%、降低至少50%、降低至少60%、降低至少70%、降低至少80%、或降低至少90%)。可以通过本领域已知的方法破坏具体目的基因,例如通过定向同源重组(directed homologous recombination)(参见Methods in Yeast Genetics[酵母遗传学方法](1997版),Adams,Gottschling,Kaiser和Stems,冷泉港出版社(Cold Spring Harbor Press),(1998))。
内源基因:术语“内源基因”意指对参照宿主细胞而言天然的基因。“内源基因表达”意指内源基因的表达。
内切葡聚糖酶:术语“内切葡聚糖酶”意指4-(1,3;1,4)-β-D-葡聚糖4-葡聚糖水解酶(E.C.3.2.1.4),其催化纤维素、纤维素衍生物(如羧甲基纤维素和羟乙基纤维素)、地衣多糖中的1,4-β-D-糖苷键,混合β-1,3-1,4葡聚糖如谷类β-D-葡聚糖或木葡聚糖以及含有纤维素组分的其他植物材料中的β-1,4键的内切水解。可以通过测量底物粘度的降低或通过还原糖测定所确定的还原性末端的增加来确定内切葡聚糖酶活性(Zhang等人,2006,Biotechnology Advances[生物技术进展]24:452-481)。还可以根据Ghose,1987,Pure andAppl.Chem.[纯粹与应用化学]59:257-268的程序,在pH 5,40℃下,使用羧甲基纤维素(CMC)作为底物来确定内切葡聚糖酶活性。
表达:术语“表达”包括在多肽的生产中涉及的任何步骤,包括但不限于,转录、转录后修饰、翻译、翻译后修饰、以及分泌。可以对表达进行测量—例如,来检测增加的表达—通过本领域已知的技术,如测量mRNA和/或翻译的多肽的水平。
表达载体:术语“表达载体”意指直链或环状DNA分子,其包含编码多肽的多核苷酸并且可操作地连接至提供用于其表达的控制序列。
可发酵的培养基:术语“可发酵的培养基”或“发酵培养基”是指包含一种或多种(例如,两种、几种)糖的培养基,如葡萄糖、果糖、蔗糖、纤维二糖、木糖、木酮糖、阿拉伯糖、甘露糖、半乳糖和/或可溶性低聚糖,其中该培养基能够部分地被宿主细胞转化(发酵)为希望的产物,如乙醇。在一些情况下,该发酵培养基衍生自天然来源,如甘蔗、淀粉、或纤维素;并且可以来自这种来源的酶水解(糖化)的预处理。术语发酵培养基在本文理解为指在添加发酵生物之前的介质,例如,由糖化过程产生的介质,以及在同时糖化和发酵过程(SSF)中使用的介质。
葡糖淀粉酶:术语“葡糖淀粉酶”(1,4-α-D-葡聚糖葡糖水解酶,EC 3.2.1.3)被定义为催化D-葡萄糖从淀粉或相关寡糖和多糖分子的非还原端释放的酶。出于本发明的目的,葡糖淀粉酶活性可以根据本领域已知的方法来测定,例如在2018年7月25日提交的美国临时专利申请号62/703,103的实例中描述的那些方法。
半纤维素分解酶或半纤维素酶:术语“半纤维素分解酶”或“半纤维素酶”意指可对半纤维素材料进行水解的一种或多种(例如,几种)酶。参见,例如Shallom和Shoham,2003,Current Opinion In Microbiology[微生物学当前观点]6(3):219-228)。半纤维素酶是植物生物质降解中的关键组分。半纤维素酶的实例包括但不限于:乙酰甘露聚糖酯酶、乙酰木聚糖酯酶、阿拉伯聚糖酶、阿拉伯呋喃糖苷酶、香豆酸酯酶、阿魏酸酯酶、半乳糖苷酶、葡糖醛酸糖苷酶、葡糖醛酸酯酶、甘露聚糖酶、甘露糖苷酶、木聚糖酶、和木糖苷酶。这些酶的底物(半纤维素)是支链和直链多糖的异质性组,这些多糖经由氢键与植物细胞壁中的纤维素微纤维相结合,从而将它们交联成稳健的网络。半纤维素还共价附接至木质素,从而与纤维素一起形成高度复杂的结构。半纤维素的可变结构和组织要求许多酶的协同作用以使其完全降解。半纤维素酶的催化模块是水解糖苷键的糖苷水解酶(GH),或是水解乙酸或阿魏酸侧基团的酯键的碳水化合物酯酶(CE)。这些催化模块基于其一级序列的同源性,可以分配到GH和CE家族。一些家族(具有总体上类似的折叠)可以进一步分组为宗族(clan),以字母标记(例如,GH-A)。在碳水化合物活性酶(CAZy)数据库中可得到这些以及其他碳水化合物活性酶的最详实和最新的分类。半纤维素分解酶活性可根据Ghose和Bisaria,1987,Pure&AppI.Chem.[理论与应用化学]59:1739-1752,在适宜温度例如40℃-80℃,例如50℃、55℃、60℃、65℃或70℃,以及适宜pH例如4-9,例如5.0、5.5、6.0、6.5或7.0下进行测量。
异源多核苷酸:术语“异源多核苷酸”在本文定义为对宿主细胞不是天然的多核苷酸;天然多核苷酸,其中对编码区已进行了结构修饰;天然多核苷酸,其表达由于通过重组DNA技术(例如不同的(外源)启动子)操纵DNA而被定量改变;或宿主细胞中的一种天然多核苷酸,该宿主细胞具有该多核苷酸的一个或多个额外拷贝以定量改变表达。“异源基因”是包含异源多核苷酸的基因。
高严格条件:术语“高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃、于5X SSPE、0.3%SDS、200微克/mL剪切并变性的鲑鱼精子DNA和50%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2X SSC、0.2%SDS,在65℃下洗涤三次,每次15分钟。
宿主细胞:术语“宿主细胞”意指对包含本文所述多核苷酸(例如,编码α-淀粉酶和/或海藻糖酶的多核苷酸)的核酸构建体或表达载体的转化、转染、转导等敏感的任何细胞类型。术语“宿主细胞”涵盖由于复制期间出现的突变而与亲本细胞不完全相同的任何亲本细胞子代。术语“重组细胞”在本文中定义为包含一种或多种(例如,两种、几种)异源多核苷酸的非天然存在的宿主细胞。
低严格条件:术语“低严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃、于5X SSPE、0.3%SDS、200微克/mL剪切并变性的鲑鱼精子DNA和25%甲酰胺中预杂交和杂交12至24小时。运载体材料最终使用0.2X SSC,0.2%SDS,在50℃下洗涤三次,每次15分钟。
成熟多肽:术语“成熟多肽”在本文中定义为具有生物学活性的多肽,其在翻译和任何翻译后修饰(例如N-末端加工、C-末端截短、糖基化、磷酸化等)之后处于其最终形式。成熟多肽序列缺乏信号序列,其可以使用本领域已知的技术来测定(参见,例如Zhang和Henzel,2004,Protein Science[蛋白质科学]13:2819-2824)。
中严格条件:术语“中严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃、于5X SSPE、0.3%SDS、200微克/mL剪切并变性的鲑鱼精子DNA和35%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2X SSC、0.2%SDS,在55℃下洗涤三次,每次15分钟。
中-高严格条件:术语“中-高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃、于5X SSPE、0.3%SDS、200微克/mL剪切并变性的鲑鱼精子DNA和35%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2X SSC、0.2%SDS,在60℃下洗涤三次,每次15分钟。
核酸构建体:术语“核酸构建体”意指包含一个或多个(例如,两个、若干个)控制序列的多核苷酸。多核苷酸可以是单链的或双链的,并且可以分离自天然存在的基因、可以被修饰成以另外的不会在自然界中存在的方式包含核酸的区段,或可以是合成的。
可操作地连接:术语“可操作地连接”意指如下构型,在该构型中,控制序列被放置在相对于多核苷酸的编码序列适当的位置处,使得该控制序列指导该编码序列的表达。
磷脂酶:术语“磷脂酶”是指催化磷脂转化为脂肪酸和其他亲脂性物质的酶,例如磷脂酶A(EC号3.1.1.4、3.1.1.5和3.1.1.32)或磷脂酶C(EC号3.1.4.3和3.1.4.11)。出于本发明的目的,可以使用本领域已知的活性测定或根据本文实例(实例2)中所述的方法来测定磷脂酶活性。
预处理的玉米秸秆:术语“预处理的玉米秸秆”或“PCS”意指通过热和稀硫酸处理、碱预处理、中性预处理、或本领域已知的任何预处理从玉米秸秆得到的含纤维素材料。
蛋白酶:术语“蛋白酶”在本文中定义为水解肽键的酶。它包括属于EC 3.4酶组的任何酶(包括其13个亚类中的每一个)。EC编号参考加利福尼亚州(California)的圣迭戈(San Diego)的NC-IUBMB,学术出版社(Academic Press)的1992年酶命名法,分别包括出版于以下中的增刊1-5:Eur.J.Biochem.[欧洲生物化学杂志]223:1-5(1994);Eur.J.Biochem.[欧洲生物化学杂志]232:1-6(1995);Eur.J.Biochem.[欧洲生物化学杂志]237:1-5(1996);Eur.J.Biochem.[欧洲生物化学杂志]250:1-6(1997);和Eur.J.Biochem.[欧洲生物化学杂志]264:610-650(1999)。术语“枯草杆菌酶”是指根据Siezen等人,1991,Protein Engng.[蛋白质工程]4:719-737和Siezen等人,1997,ProteinScience[蛋白质科学]6:501-523的丝氨酸蛋白酶亚组。丝氨酸蛋白酶或丝氨酸肽酶是蛋白酶亚组,其特征为在活性位点上具有丝氨酸,与底物形成了共价加合物。另外,枯草杆菌酶(和丝氨酸蛋白酶)的特征为除了丝氨酸以外,还具有两个活性位点氨基酸残基,即组氨酸和天冬氨酸残基。枯草杆菌酶可以被划分为6个亚类,即,枯草杆菌蛋白酶家族、嗜热蛋白酶家族、蛋白酶K家族、羊毛硫氨酸抗生素肽酶家族、Kexin家族和Pyrolysin家族。术语“蛋白酶活性”意指蛋白水解活性(EC 3.4)。蛋白酶活性可以使用本领域所述的方法(例如,US2015/0125925)或使用可商购的测定试剂盒(例如,西格玛奥德里奇公司(Sigma-Aldrich))来测定。
支链淀粉酶:术语“支链淀粉酶”意指具有支链淀粉6-葡聚糖-水解酶活性的淀粉去分支酶(EC 3.2.1.41),其催化支链淀粉中α-1,6-糖苷键的水解,从而释放具有还原碳水化合物端的麦芽三糖。出于本发明的目的,支链淀粉酶活性可以根据WO 2016/087237中所述的PHADEBAS测定或甘薯淀粉测定来测定。
序列同一性:两个氨基酸序列之间或两个核苷酸序列之间的关联度通过参数“序列同一性”来描述。
出于本文描述的目的,使用尼德曼-翁施算法(Needleman-Wunsch algorithm)(Needleman和Wunsch,J.Mol.Biol.[分子生物学杂志]1970,48,443-453)来确定两个氨基酸序列之间的序列同一性的程度,该算法如在EMBOSS软件包(EMBOSS:欧洲分子生物学开放软件套件(The European Molecular Biology Open Software Suite),Rice等人,TrendsGenet[遗传学趋势]2000,16,276-277)(优选3.0.0版或更新版本)的Needle程序中所实施的。所使用的任选参数是空位开放罚分10、空位延伸罚分0.5、以及EBLOSUM62(BLOSUM62的EMBOSS版本)取代矩阵。将标记为“最长同一性”的Needle的输出(使用非简化选项(nobriefoption)获得)用作同一性百分比并且计算如下:
(相同的残基X 100)/(参照序列的长度-比对中的空位总数)
出于本文描述的目的,使用尼德曼-翁施算法(Needleman和Wunsch,1970,见上文)来确定两个脱氧核糖核苷酸序列之间的序列同一性的程度,该算法如在EMBOSS软件包(EMBOSS:欧洲分子生物学开放软件套件,Rice等人,2000,见上文)(优选3.0.0版或更新版本)的Needle程序中所实施的。所使用的任选参数是空位开放罚分10、空位延伸罚分0.5、以及EDNAFULL(NCBI NUC4.4的EMBOSS版本)取代矩阵。将标记为“最长同一性”的Needle的输出(使用非简化选项(nobrief option)获得)用作同一性百分比并且计算如下:
(相同的脱氧核糖核苷酸X 100)/(参照序列的长度-比对中的空位总数)
信号肽:术语“信号肽”在本文中定义为以符合阅读框的方式连接(融合)至具有生物活性的多肽的氨基末端且指导该多肽进入细胞的分泌途径的肽。信号序列可以使用本领域已知的技术来测定(参见,例如,Zhang和Henzel,2004,Protein Science[蛋白质科学]13:2819-2824)。本文所述的多肽可包含本领域已知的任何合适的信号肽,或本文所述的任何信号肽(例如,SEQ ID NO:7的酿酒酵母MFα1信号肽,SEQ ID NO:227的啤酒酵母EXG1信号肽,或SEQ ID NO:234的酿酒酵母AG2信号肽,或其具有至少80%、85%、90%、95%、96%、97%、98%或99%序列同一性的信号肽)。
海藻糖酶:术语“海藻糖酶”意指将海藻糖降解成其单元单糖(即,葡萄糖)的酶。将海藻糖酶分类于EC 3.2.1.28(α,α-海藻糖酶)和EC。3.2.1.93(α,α-磷酸海藻糖酶)。EC类别基于国际生物化学与分子生物学联合会(IUBMB)的命名委员会(Nomenclature Committee)的推荐。EC类别的描述可见于互联网,例如,在“http:
出于本发明的目的,海藻糖酶活性可根据本文实验部分中所述的海藻糖酶活性测定法来测定。
非常高严格条件:术语“非常高严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃、于5X SSPE、0.3%SDS、200微克/mL剪切并变性的鲑鱼精子DNA和50%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2X SSC、0.2%SDS,在70℃下洗涤三次,每次15分钟。
非常低严格条件:术语“非常低严格条件”意指对于长度为至少100个核苷酸的探针而言,遵循标准DNA印迹程序,在42℃、于5X SSPE、0.3%SDS、200微克/mL剪切并变性的鲑鱼精子DNA和25%甲酰胺中预杂交和杂交12至24小时。载体材料最终使用0.2X SSC、0.2%SDS,在45℃下洗涤三次,每次15分钟。
木聚糖酶:术语“木聚糖酶”意指1,4-β-D-木聚糖-木糖水解酶(1,4-β-D-xylan-xylohydrolase)(E.C.3.2.1.8),其催化木聚糖中1,4-β-D-木糖苷键的内水解。木聚糖酶活性可以在37℃在0.01%
木糖异构酶:术语“木糖异构酶”或“XI”意指可以在体内将D-木糖催化为D-木酮糖,并且在体外将D-葡萄糖转化为D-果糖的酶。木糖异构酶也称为“葡萄糖异构酶”,并且被分类为E.C.5.3.1.5。由于该酶的结构非常稳定,因此木糖异构酶是研究蛋白质结构和功能之间关系的良好模型(Karimaki等人,Protein Eng Des Sel[蛋白质工程、设计与选择],12004,17(12):861-869)。木糖异构酶活性可以使用本领域已知的技术来测定(例如,使用D-山梨醇脱氢酶的偶联酶测定法,如Verhoeven等人,2017,Sci Rep[科学报告]7,46155所述)。
本文提及“约”值或参数包括指向该值或参数本身的实施例。例如,提及“约X”的描述包括实施例“X”。当与测量值组合使用时,“约”包括涵盖至少与测量该具体数值的方法相关的不确定性的范围,并且可以包括在给定的数值附近正或负两个标准差的范围。
同样,提及“源自”另一个基因或多肽X的基因或多肽包括该基因或多肽X。
如本文和所附权利要求书中所用的,单数形式“一种/个(a/an)”、“或”以及“该(the)”包括复数指代物,除非上下文另外清楚表明。
应该理解的是本文所述的实施例包括“由……实施例组成”和/或“基本由……实施例组成”。如本文所用,除了由于表达语言或必需含义情况下另有要求外,词语“包含(comprise)”或变形如“包含(comprises)”或“包含(comprising)”以包容性意义使用,即指定所述特征的存在,但不排除各种实施例中存在或加入了其他特征。
具体实施方式
本文尤其描述了从含淀粉材料或含纤维素材料生产发酵产物如乙醇的方法。
在工业规模发酵期间,酵母遇到各种各样的生理挑战,这些挑战包括可变浓度的糖,高浓度的酵母代谢产物如乙醇、甘油、有机酸、渗透胁迫、以及来自污染微生物(如野生酵母和细菌)的潜在竞争。因此,许多酵母不适合用于工业发酵。酵母属的最广泛使用的可商购的工业菌株(即,用于工业规模发酵)是,例如,在产品
申请人惊奇地发现,表达磷脂酶的酵母提供了可用于乙醇发酵的有益特性,例如减少了对补充氮的需求。
在一方面是从含淀粉材料或含纤维素材料生产发酵产物的方法,该方法包括:
(a)糖化所述含淀粉材料或含纤维素材料;以及
(b)用发酵生物发酵步骤(a)的经糖化的材料;
其中所述发酵生物包含编码磷脂酶的异源多核苷酸。
糖化和发酵步骤可以顺序或同时(SSF)进行。在一个实施例中,糖化和发酵步骤同时(SSF)进行。在另一个实施例中,糖化和发酵步骤顺序进行。
本文所述的发酵生物可以衍生自本领域技术人员已知的能够生产发酵产物(如乙醇)的任何宿主细胞。如本文所用的,菌株的“衍生物”衍生自参照菌株,如通过诱变、重组DNA技术、交配、细胞融合、或在酵母菌株之间的胞导。本领域的技术人员应该理解,遗传改变(包括本文示例的代谢修饰)可以参考适合的宿主生物及其相应的代谢反应或用于希望的遗传材料(如希望的代谢途径的基因)的适合的来源生物加以描述。然而,考虑到多种多样的生物的全基因组测序以及基因组学领域中高水平的技能,本领域的技术人员可以将本文提供的教导和指导应用于其他生物中。例如,可以通过掺入相同的或来自不同于参照物种的物种的类似编码核酸而容易地将本文示例的代谢改变应用于其他物种中。
用于制备本文所述的重组细胞的宿主细胞可以来自任何适合的宿主,如酵母菌株,包括但不限于,酵母属、红酵母属、裂殖酵母属、克鲁维酵母属、毕赤酵母属、汉逊酵母属、红冬孢酵母属、假丝酵母属、耶氏酵母属、油脂酵母属、隐球菌属、或德克拉酵母属菌种细胞。具体地,考虑了酵母属宿主细胞,如酿酒酵母、贝酵母或卡尔酵母细胞。优选地,该酵母细胞是酿酒酵母细胞。适合的细胞可以例如,衍生自可商购的菌株和多倍体或非整倍体工业菌株,包括但不限于,来自Superstart
发酵生物可以是酵母属菌株,例如是使用美国专利号8,257,959-BB中所描述和涉及的方法产生的酿酒酵母菌株。
该菌株也可以是酿酒酵母菌株NMI V14/004037(参见,WO 2015/143324和WO2015/143317,各自通过引用并入本文),菌株号V15/004035、V15/004036和V15/004037(参见,WO 2016/153924,通过引用并入本文),菌株号V15/001459、V15/001460、V15/001461(参见,WO 2016/138437,通过引用并入本文),菌株号NRRL Y67342(参见,WO 2017/063159,通过引用并入本文)或描述于WO 2017/087330(通过引用并入本文)的任何菌株的衍生物。
已经产生了根据本发明的发酵生物,以例如通过降低酶的成本来提高发酵产率并改善工艺经济性,因为提高方法性能所需的部分或全部必需酶是由发酵生物产生的。
本文所述的发酵生物可以利用以下表达载体,这些表达载体包含一个或多个(例如,两个、几个)异源基因的编码序列,这些编码序列被连接至一个或多个控制序列,这一个或多个控制序列指导在与这一个或多个控制序列相容的条件下在适合的细胞中的表达。可以在任何本文所述的细胞和方法中使用此类表达载体。本文所述的多核苷酸可以按多种方式操纵,以提供希望的多肽的表达。取决于表达载体,在多核苷酸插入载体之前对其进行操作可能是理想的或必需的。用于利用重组DNA方法修饰多核苷酸的技术是本领域熟知的。
可以将构建体或载体(或多个构建体或载体)引入细胞中,这样使得构建体或载体被维持作为染色体整合体或作为自主复制的染色体外载体,如早前所述;构建体或载体(或这些构建体或载体)包含一个或多个(例如,两个、几个)异源基因。
各种核苷酸和控制序列可以连接在一起以产生重组表达载体,该重组表达载体可以包括一个或多个(例如,两个、几个)合宜的限制位点以允许在此类位点插入或取代该多核苷酸。可替代地,可以通过将一种或多种多核苷酸或者包含该序列的核酸构建体插入用于表达的适当载体中而表达该一种或多种多核苷酸。在产生表达载体时,编码序列如此位于载体中,使得编码序列与用于表达的适当控制序列可操作地连接。
重组表达载体可以是可以方便地经受重组DNA程序并且可以引起该多核苷酸表达的任何载体(例如,质粒或病毒)。载体的选择将典型地取决于载体与待引入载体的宿主细胞的相容性。载体可以是直链或闭合环状质粒。
载体可以是自主复制载体,即作为染色体外实体存在的载体,其复制独立于染色体复制,例如质粒、染色体外元件、微染色体或人工染色体。载体可以含有用于确保自我复制的任何手段。可替代地,载体可以是这样的载体,当它引入宿主细胞中时整合入基因组中并与其中已整合了它的一个或多个染色体一起复制。此外,可以使用单一载体或质粒或两个或更多个载体或质粒(这些载体或质粒共同含有待引入到细胞的基因组中的总DNA)或转座子。
表达载体可以含有任何适合的启动子序列,该启动子序列可被细胞识别以表达本文所述的基因。启动子序列含有转录控制序列,这些转录控制序列介导该多肽的表达。启动子可以是在选择的细胞中显示出转录活性的任何多核苷酸,包括突变型、截短型及杂合型启动子,并且可以是由编码与该细胞同源或异源的细胞外或细胞内多肽的基因获得。
本文所述的每种异源多核苷酸都可以被可操作地连接至对于该多核苷酸而言外源的启动子上。例如,在一个实施例中,编码己糖转运体的异源多核苷酸被可操作地连接至对于该多核苷酸而言外源的启动子上。这些启动子可以与所选的天然启动子相同或与其具有高度序列同一性(例如,至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%)。
用于指导核酸构建体在酵母细胞中的转录的适合的启动子的实例包括但不限于获得自以下的基因的启动子:烯醇酶(例如,酿酒酵母烯醇酶或东方伊萨酵母烯醇酶(ENO1))、半乳糖激酶(例如,酿酒酵母半乳糖激酶或东方伊萨酵母半乳糖激酶(GAL1))、醇脱氢酶/甘油醛-3磷酸脱氢酶(例如,酿酒酵母醇脱氢酶/甘油醛-3磷酸脱氢酶或东方伊萨酵母醇脱氢酶/甘油醛-3磷酸脱氢酶(ADH1、ADH2/GAP))、磷酸甘油醛异构酶(例如,酿酒酵母磷酸甘油醛异构酶或东方伊萨酵母磷酸甘油醛异构酶(TPI))、金属硫蛋白(例如,酿酒酵母金属硫蛋白或东方伊萨酵母金属硫蛋白(CUP1))、3-磷酸甘油酸激酶(例如,酿酒酵母3磷酸甘油酸激酶或东方伊萨酵母3-磷酸甘油酸激酶(PGK))、PDC1、木糖还原酶(XR)、木糖醇脱氢酶(XDH)、L-(+)-乳酸-细胞色素C氧化还原酶(CYB2)、翻译延长因子-1(TEF1)、翻译延长因子-2(TEF2)、甘油醛-3-磷酸脱氢酶(GAPDH)、和乳清酸核苷5'-磷酸脱羧酶(URA3)基因。其他合适的启动子可以获自啤酒酵母TDH3、HXT7、PGK1、RPL18B和CCW12基因。酵母宿主细胞的其他有用的启动子由Romanos等人,1992,Yeast[酵母]8:423-488描述。
控制序列也可以是被宿主细胞识别以终止转录的适合的转录终止子序列。终止子序列被可操作地连接至编码多肽的多核苷酸的3’-末端。可以使用在选择的酵母细胞中具有功能的任何终止子。终止子可以与所选的天然终止子相同或与其具有高度序列同一性(例如,至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98%或至少约99%)。
酵母宿主细胞的适合的终止子可以获得自以下的基因:烯醇酶(例如,酿酒酵母或东方伊萨酵母烯醇酶)、细胞色素C(例如,酿酒酵母或东方伊萨酵母细胞色素(CYC1))、甘油醛-3磷酸脱氢酶(例如,酿酒酵母或东方伊萨酵母甘油醛-3-磷酸脱氢酶(gpd))、PDC1、XR、XDH、转醛醇酶(TAL)、转酮醇酶(TKL)、核糖5-磷酸-酮醇异构酶(RKI)、CYB2、以及半乳糖基因家族(尤其是GAL10终止子)。其他合适的终止子可以获自啤酒酵母ENO2或TEF1基因。酵母宿主细胞的其他有用的终止子由Romanos等人,1992,同上描述。
控制序列还可以是启动子下游和基因的编码序列上游的mRNA稳定子区域,其增加该基因的表达。
适合的mRNA稳定子区域的实例从以下获得:苏云金芽孢杆菌cryIIIA基因(WO 94/25612)和枯草芽孢杆菌SP82基因(Hue等人,1995,Journal of Bacteriology[细菌学杂志]177:3465-3471)。
控制序列也可以是适合的前导子序列,其中转录时,该前导子序列是对由宿主细胞翻译重要的mRNA的非翻译区。前导子序列可操作地连接至编码该多肽的多核苷酸的5’-末端。可以使用在选择的酵母细胞中具有功能的任何前导子序列。
酵母宿主细胞的适合的前导子获得自以下的基因:烯醇酶(例如,酿酒酵母或东方伊萨酵母烯醇酶(ENO-1))、3-磷酸甘油酸激酶(例如,酿酒酵母或东方伊萨酵母3-磷酸甘油酸激酶)、α-因子(例如,酿酒酵母或东方伊萨酵母α-因子)、以及醇脱氢酶/甘油醛-3-磷酸脱氢酶(例如,酿酒酵母或东方伊萨酵母醇脱氢酶/甘油醛-3磷酸脱氢酶(ADH2/GAP))。
控制序列还可以是多腺苷酸化序列;与多核苷酸3’末端可操作地连接并在转录时由宿主细胞识别为向转录的mRNA添加聚腺苷酸残基的信号的序列。可以使用在选择的宿主细胞中具有功能的任何多腺苷酸化序列。对于酵母细胞有用的多腺苷酸化序列描述于以下文献:Guo和Sherman,1995,Mol.Cellular Biol.[分子细胞生物学]15:5983-5990。
也可能令人希望的是添加调节序列,该调节序列允许相对于宿主细胞的生长而调节多肽的表达。调节系统的实例是引起基因表达响应化学或物理刺激物,包括调节化合物的存在而开启或关闭的那些系统。原核系统中的调节系统包括lac、tac和trp操纵子系统。在酵母中,可以使用ADH2系统或GAL1系统。
这些载体可以含有一个或多个(例如,两个、几个)允许方便地选择转化细胞、转染细胞、转导细胞等细胞的选择性标记。选择性标记是一种基因,其产物提供了杀生物剂抗性或病毒抗性、对重金属抗性、对营养缺陷型的原养型等。酵母宿主细胞的适合的标记包括但不限于:ADE2、HIS3、LEU2、LYS2、MET3、TRP1和URA3。
这些载体可以含有一个或多个(例如,两个、几个)允许将该载体整合进宿主细胞的基因组中或在该细胞中独立于基因组而自主复制的元件。
对于整合到宿主细胞基因组中,载体可以依靠编码该多肽的多核苷酸序列或用于通过同源或非同源重组整合到该基因组中的该载体的任何其他元件。可替代地,载体可以含有用于指导通过同源重组而整合入宿主细胞基因组中的染色体中的精确位置处的另外的多核苷酸。为了增加在精确位置处整合的可能性,整合元件应当含有足够数目的核酸,例如100至10,000个碱基对、400至10,000个碱基对和800至10,000个碱基对,这些核酸与相应的靶序列具有高度序列同一性以增强同源重组的概率。整合元件可以是与宿主细胞基因组内的靶序列同源的任何序列。此外,整合元件可以是非编码或编码的多核苷酸。另一方面,载体可以通过非同源重组整合入宿主细胞的基因组中。潜在整合位点包括本领域所描述的那些(例如,参见US2012/0135481)。
对于自主复制,载体可以进一步包含使该载体能够在酵母细胞中自主复制的复制起点。复制起点可以是在细胞中发挥作用的介导自主复制的任何质粒复制子。术语“复制起点”或“质粒复制子”意指使质粒或载体能够在体内复制的多核苷酸。用于酵母宿主细胞中的复制起点的实例是2微米复制起点、ARS1、ARS4、ARS1与CEN3的组合、及ARS4与CEN6的组合。
可以将本文所述的多核苷酸的超过一个的拷贝插入到宿主细胞中以增加多肽的产生。通过将序列的至少一个另外的拷贝整合到酵母细胞基因组中或者通过包括与该多核苷酸一起的可扩增的选择性标记基因可以获得多核苷酸的增加的拷贝数目,其中通过在适当的选择性试剂的存在下培养细胞可以选择含有选择性标记基因的经扩增的拷贝的细胞、以及由此该多核苷酸的另外的拷贝。
用于连接以上所描述的元件以构建本文所述的重组表达载体的程序是本领域技术人员所熟知的(参见例如,Sambrook等人,1989,Molecular Cloning,A LaboratoryManual[分子克隆:实验手册],第2版,冷泉港(Cold Spring Harbor),纽约)。
本领域已知的用于制备用于乙醇发酵的重组细胞的另外的程序和技术描述于例如WO 2016/045569中,将其内容通过引用特此并入。
该发酵生物可以呈组合物的形式,该组合物包含发酵生物(例如,本文所述的酵母菌株)和天然存在和/或非天然存在的组分。
本文所述的发酵生物可以是任何活的形式,包括粉碎的、干燥的,包括活性干燥和速溶的、压缩的、膏状(液体)形式等。在一个实施例中,发酵生物(例如,酿酒酵母菌株)是干酵母,例如活性干酵母或速溶酵母。在一个实施例中,发酵生物(例如,酿酒酵母菌株)是粉碎酵母。在一个实施例中,发酵生物(例如,酿酒酵母菌株)是压缩酵母。在一个实施例中,发酵生物(例如,酿酒酵母菌株)是膏状酵母。
在一个实施例中是组合物,组合物包含本文所述的发酵生物(例如,酿酒酵母菌株)和选自下组的一种或多种组分,该组由以下组成:表面活性剂、乳化剂、树胶、溶胀剂、以及抗氧化剂和其他加工助剂。
本文所述的组合物可包含本文所述的发酵生物(例如,酿酒酵母菌株)和任何适合的表面活性剂。在一个实施例中,一种或多种表面活性剂是阴离子表面活性剂、阳离子表面活性剂、和/或非离子表面活性剂。
本文所述的组合物可包含本文所述的发酵生物(例如,酿酒酵母菌株)和任何适合的乳化剂。在一个实施例中,乳化剂是山梨聚糖的脂肪酸酯。在一个实施例中,乳化剂选自下组:山梨聚糖单硬脂酸酯(SMS)、单双甘酯的柠檬酸酯、聚甘油酯、丙二醇的脂肪酸酯。
在一个实施例中,组合物包含本文所述的发酵生物(例如,酿酒酵母菌株)和Olindronal SMS、Olindronal SK、或Olindronal SPL,包括欧洲专利号1,724,336(将其通过引用特此并入)中涉及的组合物。针对活性干酵母,这些产品可以从奥地利的布塞蒂公司(Bussetti)商购获得。
本文所述的组合物可包含本文所述的发酵生物(例如,酿酒酵母菌株)和任何适合的树胶。在一个实施例中,树胶选自下组:槐树豆胶、瓜尔胶、黄芪胶、阿拉伯胶、黄原胶和阿拉伯树胶,具体地针对膏状、压缩和干酵母。
本文所述的组合物可包含本文所述的发酵生物(例如,酿酒酵母菌株)和任何适合的溶胀剂。在一个实施例中,溶胀剂是甲基纤维素或羧甲基纤维素。
本文所述的组合物可包含本文所述的发酵生物(例如,酿酒酵母菌株)和任何适合的抗氧化剂。在一个实施例中,抗氧化剂是丁羟茴醚(BHA)和/或丁羟甲苯(BHT)、或抗坏血酸(维生素C),具体地针对活性干酵母。
本文所述的发酵生物还可以包含一个或多个(例如,两个、若干个)基因破坏,例如以将糖代谢从不希望的产物转移至乙醇。在一些方面,当在相同条件下培养时,与没有这一个或多个破坏的细胞相比,重组宿主细胞产生更大量的乙醇。在一些方面,使被破坏的内源基因中的一个或多个失活。
在某些实施例中,本文提供的发酵生物包含一个或多个内源基因的破坏,这一个或多个内源基因编码在生产替代发酵产物(如甘油)或其他副产物(如乙酸或二醇)中涉及的酶。例如,本文提供的细胞可以包含以下一个或多个中的破坏:甘油3-磷酸脱氢酶(GPD,催化二羟丙酮磷酸反应为甘油3-磷酸)、甘油3-磷酸酶(GPP,催化甘油-3磷酸转化为甘油)、甘油激酶(催化甘油3-磷酸转化为甘油)、二羟丙酮激酶(催化二羟丙酮磷酸转化为二羟丙酮)、甘油脱氢酶(催化二羟丙酮转化为甘油)、和醛脱氢酶(ALD,例如,将乙醛转化为乙酸)。
可以使用模型分析来设计另外的优化途径利用的基因破坏。用于鉴定并设计有利于生物合成希望的产物的代谢改变的一种示例性计算方法是OptKnock计算框架(OptKnockcomputational framework),Burgard等人,2003,Biotechnol.Bioeng.[生物技术与生物工程]84:647-657。
可以使用本领域熟知的方法(包括本文描述的那些方法)构建包含基因破坏的发酵生物。可以破坏该基因的一部分,如编码区或为编码区的表达所需的控制序列。该基因的这样一种控制序列可以是启动子序列或其功能部分,即足以影响该基因的表达的部分。例如,可以将启动子序列失活从而无表达或可以将天然启动子替换为较弱的启动子以减少编码序列的表达。可修饰的其他控制序列包括但不限于前导子、前肽序列、信号序列、转录终止子以及转录激活因子。
可以通过基因缺失技术来构建包含基因破坏的发酵生物,以消除或减少该基因的表达。基因缺失技术使得可以部分或完全除去该基因,从而消除其表达。这类方法中,使用已经构建为邻接地含有侧翼于该基因的5'和3'区的质粒,通过同源重组完成该基因的缺失。
还可以通过引入、取代和/或除去该基因中的或为其转录或翻译所需的其控制序列中的一个或多个(例如,两个、若干个)核苷酸来构建包含基因破坏的发酵生物。例如,可以插入或除去核苷酸,用于引入终止密码子、除去起始密码子或移码开放阅读框。可以根据本领域已知的方法,通过定点诱变或PCR产生的诱变完成这样的修饰。参见,例如Botstein和Shortle,1985,Science[科学]229:4719;Lo等人,1985,Proc.Natl.Acad.Sci.U.S.A.[美国国家科学院院刊]81:2285;Higuchi等人,1988,Nucleic Acids Res[核酸研究]16:7351;Shimada,1996,Meth.Mol.Biol.[分子生物学方法]57:157;Ho等人,1989,Gene[基因]77:61;Horton等人,1989,Gene[基因]77:61;以及Sarkar和Sommer,1990,BioTechniques[生物技术]8:404。
还可以通过将破坏性核酸构建体插入该基因中而构建包含基因破坏的发酵生物,该破坏性核酸构建体包含与该基因同源的核酸片段,该片段将产生具有同源性的区域的重复并且在重复的区域之间掺入构建体DNA。这样的一种基因破坏可以消除基因表达,如果插入的构建体将该基因的启动子与编码区分离或打断编码序列,这样使得产生无功能性基因产物。破坏构建体可以简单地是伴有与该基因同源的5’和3’区的选择性标记基因。该选择性标记使得可以鉴定含有破坏的基因的转化株。
还可以通过基因转化过程构建包含基因破坏的发酵生物(参见,例如Iglesias和Trautner,1983,Molecular General Genetics[分子普通遗传学]189:73-76)。例如,在基因转化方法中,将对应于该基因的核苷酸序列体外诱变,以产生缺陷核苷酸序列,然后将其转化进重组菌株中以产生缺陷基因。通过同源重组,该缺陷核苷酸序列替换该内源基因。该缺陷核苷酸序列还包含用于选择含有缺陷基因的转化株的标记可以是令人希望的。
可以使用本领域熟知的方法(包括但不限于化学诱变),通过随机或特异诱变进一步构建包含基因破坏的发酵生物(参见,例如Hopwood,The Isolation of Mutants inMethods in Microbiology[微生物学方法中的突变体分离](J.R.Norris和D.W.Ribbons,编辑)第363-433页,Academic Press[学术出版社],纽约,1970)。可以通过使亲本菌株经受诱变并筛选其中该基因的表达已经被减少或失活的突变菌株来修饰该基因。诱变可以是特异的或随机的,例如通过使用适合的物理或化学诱变剂、使用适合的寡核苷酸或使DNA序列经受PCR产生的诱变来进行。此外,诱变可以通过使用这些诱变方法的任何组合来进行。
适合本发明目的的物理或化学诱变剂的实例包括紫外线(UV)照射,羟胺,N-甲基-N'-硝基-N-亚硝基胍(MNNG),N-甲基-N'-亚硝基胍(NTG)邻甲基羟胺,亚硝酸,乙基甲磺酸(EMS),亚硫酸氢钠,甲酸和核苷酸类似物。当使用此类试剂时,诱变典型地是在适合条件下在所选的诱变剂的存在下通过孵育有待诱变的亲本菌株并选择展现出该基因的减少表达或无表达的突变体来进行的。
可以使用来自其他微生物来源的与本文所述的基因同源或互补的核苷酸序列来破坏所选的重组菌株中的对应基因。
在一方面,重组细胞中的基因修饰未用选择性标记加以标记。可以通过将突变体在反向选择培养基中进行培养来除去选择性标记基因。在该选择性标记基因含有侧翼于其5'和3'端的重复序列的情况下,当该突变体菌株经受反向选择时,这些重复序列将有助于该选择性标记基因通过同源重组而环出。还可以通过向该突变体菌株中引入核酸片段,该核酸片段包含缺陷基因的5'和3'区但是缺乏该选择性标记基因,随后在反向选择培养基上进行选择,通过同源重组来除去该选择性标记基因。通过同源重组,含有该选择性标记基因的缺陷基因被缺乏该选择性标记基因的核酸片段替换。还可以使用本领域已知的其他方法。
在一些方面,本文所述的方法从含淀粉材料生产发酵产物。含淀粉材料在本领域中是熟知的,其含有两种类型的同聚多糖(直链淀粉和支链淀粉)并且通过α-(1-4)-D-糖苷键连接。可以使用任何适合的含淀粉起始材料。通常基于希望的发酵产物(如乙醇)来选择起始材料。含淀粉起始材料的实例包括谷类、块茎或谷物。具体地,该含淀粉材料可以是玉米、小麦、大麦、黑麦、西非高粱(milo)、西米、木薯(cassava)、木薯淀粉(tapioca)、高粱、燕麦、水稻、豌豆、豆类、或甘薯、或其混合物。还考虑了糯型(waxy type)与非糯型(non-waxytype)的玉米与大麦。
在一个实施例中,该含淀粉起始材料是玉米。在一个实施例中,该含淀粉起始材料是小麦。在一个实施例中,该含淀粉起始材料是大麦。在一个实施例中,该含淀粉起始材料是黑麦。在一个实施例中,该含淀粉起始材料是西非高粱。在一个实施例中,该含淀粉起始材料是西米。在一个实施例中,该含淀粉起始材料是木薯。在一个实施例中,该含淀粉起始材料是木薯淀粉。在一个实施例中,该含淀粉起始材料是高粱。在一个实施例中,该含淀粉起始材料是水稻。在一个实施例中,该含淀粉起始材料是豌豆。在一个实施例中,该含淀粉起始材料是豆类。在一个实施例中,该含淀粉起始材料是甘薯。在一个实施例中,该含淀粉起始材料是燕麦。
使用含淀粉材料的方法可以包括常规方法(例如,包括以下更详细描述的液化步骤)或粗淀粉水解方法。在使用含淀粉材料的一些实施例中,该含淀粉材料的糖化在高于初始糊化温度的温度下进行。在使用含淀粉材料的一些实施例中,该含淀粉材料的糖化在低于初始糊化温度的温度下进行。
在使用含淀粉材料的方面,这些方法可以进一步包括液化步骤,该液化步骤是通过在高于初始糊化温度的温度下使含淀粉材料经受α-淀粉酶和任选地蛋白酶和/或葡糖淀粉酶来进行的。液化中还可以存在和/或添加其他酶如支链淀粉酶和植酸酶。在一些实施例中,在所述方法的步骤a)和b)之前进行该液化步骤。
液化步骤可以进行0.5-5小时,如1-3小时,如典型地约2小时。
术语“初始糊化温度”意指含淀粉材料糊化开始的最低温度。通常,在水中加热的淀粉在约50℃与75℃之间开始糊化;糊化的准确温度依赖于具体的淀粉,并能够由本领域的技术人员容易地确定。因此,初始糊化温度可以根据植物物种、植物物种的具体品种、以及生长条件而变化。给定的含淀粉材料的初始糊化温度可以是使用Gorinstein和Lii,1992,
液化典型地是在从70℃-100℃范围内的温度下进行的。在一个实施例中,液化中的温度是在75℃-95℃之间,如在75℃-90℃之间,在80℃-90℃之间,或在82℃-88℃之间,如约85℃。
喷射蒸煮步骤可以在液化步骤之前进行,例如,在110℃-145℃、120℃-140℃、125℃-135℃之间、或约130℃的温度下进行约1-15分钟,进行约3-10分钟,或约5分钟。
液化期间的pH可以在4与7之间,如pH 4.5-6.5,pH 5.0-6.5,pH 5.0-6.0,pH 5.2-6.2,或约5.2、约5.4、约5.6或约5.8。
在一个实施例中,在液化之前,该方法进一步包括以下步骤:
i)优选地通过干磨来减小该含淀粉材料的粒度;
ii)形成包含该含淀粉材料和水的浆料。
该含淀粉起始材料(如全谷物)可例如通过磨制减少粒度,以打开结构、增大表面积并且允许进一步加工。通常存在两种类型的方法:湿磨和干磨。在干磨中,整个谷粒被碾磨并且使用。湿磨使胚芽与粗粉(淀粉颗粒和蛋白质)良好分离。湿磨常常应用于使用淀粉水解物产生例如糖浆的场合(location)。干磨和湿磨在淀粉加工领域中都是众所周知的。在一个实施例中,使该含淀粉材料经受干磨。在一个实施例中,将粒度减小至在0.05至3.0mm之间、例如0.1-0.5mm,或使得至少30%、至少50%、至少70%、或至少90%的含淀粉材料适合通过具有0.05至3.0mm筛网、例如0.1-0.5mm筛网的筛子。在另一个实施例中,至少50%,例如至少70%、至少80%、或至少90%的含淀粉材料适合通过具有#6筛网的筛子。
该水性浆料可以包含含淀粉材料的从10-55w/w-%干固体(DS),例如25-45w/w-%干固体(DS)、或30-40w/w-%干固体(DS)。
最初,可以将α-淀粉酶,任选地蛋白酶和任选地葡糖淀粉酶添加到该水性浆料中来开始液化(稀化)。在一个实施例中,这些酶中仅有一部分(例如,约1/3)被添加到该水性浆料中,而这些酶中的剩余部分(例如,约2/3)在液化步骤过程中被添加。
液化中使用的α-淀粉酶的非穷尽性列表可以在下面的“α-淀粉酶”部分中找到。液化中使用的适合的蛋白酶的实例包括上文“蛋白酶”部分中描述的任何蛋白酶。液化中使用的适合的葡糖淀粉酶的实例包括在“葡糖淀粉酶”部分中找到的任何葡糖淀粉酶。
在使用含淀粉材料的方面,在糖化步骤a)和/或发酵步骤b)或同时糖化和发酵(SSF)中可存在和/或添加葡糖淀粉酶。糖化步骤a)和/或发酵步骤b)或同时糖化和发酵(SSF)的葡糖淀粉酶典型地不同于任选地在上述任何液化步骤中添加的葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶是与真菌α-淀粉酶一起存在和/或添加的。
在一些方面,该发酵生物包含编码葡糖淀粉酶的异源多核苷酸,例如,如WO 2017/087330中所披露,将其内容通过引用特此并入。
葡糖淀粉酶的实例可在以下的“葡糖淀粉酶”部分中找到。
当依序进行糖化和发酵时,糖化步骤a)可以在本领域中熟知的条件下进行。例如,糖化步骤a)可持续长达从约24至约72小时。在一个实施例中,进行预糖化。预糖化在30℃-65℃,典型地约60℃的温度典型地进行40-90分钟。在一个实施例中,在同时糖化和发酵(SSF)中,预糖化之后是发酵过程中的糖化。糖化典型地在从20℃-75℃、优选地从40℃-70℃,典型地约60℃的温度下并且典型地在4与5之间的pH下、如约pH 4.5下进行。
如本领域已知并且例如如本文所述,在发酵培养基中进行发酵。发酵培养基包括发酵底物,即被发酵生物代谢的碳水化合物源。利用本文所述的方法,该发酵培养基可以包含用于一种或多种发酵生物的营养素和一种或多种生长刺激剂。营养素和生长刺激剂在发酵领域中广泛使用,并且包括氮源例如氨;尿素、维生素和矿物质或其组合。
通常,发酵生物如酵母(包括酿酒酵母)需要充分的氮源用于增殖和发酵。如果必要的话,可以使用许多补充氮源,且这些氮源是本领域熟知的。该氮源可以是有机的,如尿素、DDG、湿滤饼或玉米醪,或无机的,如氨或氢氧化铵。在一个实施例中,该氮源是尿素。
发酵可以在低氮条件下进行,例如当使用表达蛋白酶的酵母时。在一些实施例中,该发酵步骤在以下条件下进行:少于1000ppm补充氮(例如,尿素或氢氧化铵),如少于750ppm、少于500ppm、少于400ppm、少于300ppm、少于250ppm、少于200ppm、少于150ppm、少于100ppm、少于75ppm、少于50ppm、少于25ppm、或少于10ppm补充氮。在一些实施例中,该发酵步骤在没有补充氮的条件下进行。
同时糖化和发酵(“SSF”)广泛地用于工业规模的发酵产物生产方法中,尤其是乙醇生产方法中。当进行SSF时,同时进行糖化步骤a)和发酵步骤b)。不存在针对糖化的保持阶段,意指可以将发酵生物(如酵母)以及一种或多种酶一同添加。然而,还考虑了分开添加发酵生物以及一种或多种酶。SSF典型地在从25℃至40℃、如从28℃至35℃、如从30℃至34℃、或约32℃的温度下进行。在一个实施例中,发酵进行6至120小时,特别是24至96小时。在一个实施例中,该pH在4-5之间。
在一个实施例中,在糖化、发酵或同时糖化和发酵(SSF)中存在和/或添加纤维素分解酶组合物。此类纤维素分解酶组合物的实例可以在以下的“纤维素分解酶和组合物”部分中找到。该纤维素分解酶组合物可以与葡糖淀粉酶一起存在和/或添加,如以下“葡糖淀粉酶”部分中所披露的。
表达的磷脂酶可以是适用于本文所述的宿主细胞和/或方法的任何磷脂酶,如天然存在的磷脂酶(例如,来自另一个物种的天然磷脂酶或由修饰的表达载体表达的内源磷脂酶)或其保留磷脂酶活性的变体。
在一些实施例中,该发酵生物包含编码磷脂酶的异源多核苷酸,例如,如WO 2018/075430中所披露,将其内容通过引用特此并入。在一些实施例中,磷脂酶被分类为磷脂酶A。在其他实施例中,磷脂酶被分类为磷脂酶C。考虑本文描述或参照的任何磷脂酶用于在发酵生物中表达。
在一些实施例中,当在相同条件下培养时,与不具有编码磷脂酶的异源多核苷酸的宿主细胞相比,包含编码该磷脂酶的异源多核苷酸的发酵生物具有增加的磷脂酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码磷脂酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的磷脂酶活性水平。
可以与本文所述的宿主细胞和/或方法一起使用的示例性磷脂酶包括细菌、酵母或丝状真菌磷脂酶,例如,衍生自本文描述或参照的任何微生物。
可以用发酵生物表达并与本文所述方法一起使用的其他磷脂酶在实例中描述,包括但不限于表1所示的磷脂酶(或其衍生物)。
表1.
考虑与本发明一起使用的另外的磷脂酶可以在WO 2018/075430(将其内容并入本文)中找到。
编码适合的磷脂酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(
磷脂酶可以是细菌磷脂酶。例如,该磷脂酶可以衍生自革兰氏阳性细菌,如芽孢杆菌属、梭菌属(Clostridium)、肠球菌属(Enterococcus)、土芽孢杆菌属(Geobacillus)、乳杆菌属(Lactobacillus)、乳球菌属(Lactococcus)、海洋芽孢杆菌属(Oceanobacillus)、葡萄球菌属(Staphylococcus)、链球菌属(Streptococcus)或链霉菌属;或者革兰氏阴性细菌,如弯曲杆菌属(Campylobacter)、大肠杆菌(E.coli)、黄杆菌属(Flavobacterium)、梭杆菌属(Fusobacterium)、螺杆菌属(Helicobacter)、泥杆菌属(Ilyobacter)、奈瑟氏菌属(Neisseria)、假单胞菌属(Pseudomonas)、沙门氏菌属(Salmonella)或脲原体属(Ureaplasma)。
在一个实施例中,该磷脂酶衍生自嗜碱芽孢杆菌(Bacillus alkalophilus)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)、短芽孢杆菌(Bacillus brevis)、环状芽孢杆菌(Bacillus circulans)、克劳氏芽孢杆菌(Bacillus clausii)、凝结芽孢杆菌(Bacilluscoagulans)、坚强芽孢杆菌(Bacillus firmus)、灿烂芽孢杆菌(Bacillus lautus)、迟缓芽孢杆菌(Bacillus lentus)、地衣芽孢杆菌(Bacillus licheniformis)、巨大芽孢杆菌(Bacillus megaterium)、短小芽孢杆菌(Bacillus pumilus)、嗜热脂肪芽孢杆菌(Bacillus stearothermophilus)、枯草芽孢杆菌(Bacillus subtilis)、或苏云金芽孢杆菌(Bacillus thuringiensis)。
在另一个实施例中,该磷脂酶衍生自似马链球菌(Streptococcus equisimilis)、酿脓链球菌(Streptococcus pyogenes)、乳房链球菌(Streptococcus uberis)、或马链球菌兽疫亚种(Streptococcus equi subsp.Zooepidemicus)。
在另一个实施例中,该磷脂酶衍生自不产色链霉菌(Streptomycesachromogenes)、除虫链霉菌(Streptomyces avermitilis)、天蓝链霉菌(Streptomycescoelicolor)、灰色链霉菌(Streptomyces griseus)或浅青紫链霉菌(Streptomyceslividans)。
磷脂酶可以是真菌磷脂酶。例如,该磷脂酶可以衍生自酵母,如假丝酵母属(Candida)、克鲁维酵母属、毕赤酵母属、酵母属、裂殖酵母、耶氏酵母属或伊萨酵母属(Issatchenkia);或者衍生自丝状真菌,如枝顶孢霉属(Acremonium)、伞菌属(Agaricus)、链格孢属(Alternaria)、曲霉属、短梗霉属(Aureobasidium)、葡萄座腔菌属(Botryospaeria)、拟蜡菌属(Ceriporiopsis)、毛喙壳属(Chaetomidium)、金孢子菌属(Chrysosporium)、麦角菌属(Claviceps)、旋孢腔菌属(Cochliobolus)、鬼伞属(Coprinopsis)、乳白蚁属(Coptotermes)、棒囊壳属(Corynascus)、隐丛赤壳菌属(Cryphonectria)、隐球菌属(Cryptococcus)、色二孢属(Diplodia)、黑耳属(Exidia)、线黑粉酵母属(Filibasidium)、镰孢属(Fusarium)、赤霉属(Gibberella)、全鞭毛虫属(Holomastigotoides)、腐质霉属(Humicola)、耙齿菌属(Irpex)、香菇属(Lentinula)、小腔球菌属(Leptospaeria)、梨孢菌属(Magnaporthe)、黑果菌属(Melanocarpus)、亚灰树花菌属、毛霉属(Mucor)、毁丝霉属(Myceliophthora)、新美鞭菌属(Neocallimastix)、链孢菌属(Neurospora)、拟青霉属(Paecilomyces)、青霉菌属、平革菌属(Phanerochaete)、瘤胃壶菌属(Piromyces)、Poitrasia、假黑盘菌属(Pseudoplectania)、假披发虫属(Pseudotrichonympha)、根毛霉属(Rhizomucor)、裂褶菌属(Schizophyllum)、柱顶孢属(Scytalidium)、篮状菌属(Talaromyces)、嗜热子囊菌属(Thermoascus)、梭孢壳霉属(Thielavia)、弯颈霉属(Tolypocladium)、木霉属(Trichoderma)、长毛盘菌属(Trichophaea)、轮枝孢属(Verticillium)、小包脚菇属(Volvariella)、或炭角菌属(Xylaria)。
在另一个实施例中,该磷脂酶衍生自卡尔酵母、酿酒酵母、糖化酵母(Saccharomyces diastaticus)、道格拉氏酵母(Saccharomyces douglasii)、克鲁弗酵母(Saccharomyces kluyveri)、诺地酵母(Saccharomyces norbensis)、或卵形酵母(Saccharomyces oviformis)。
在另一个实施例中,该磷脂酶衍生自解纤维枝顶孢霉(Acremoniumcellulolyticus)、棘孢曲霉(Aspergillus aculeatus)、泡盛曲霉(Aspergillusawamori)、臭曲霉(Aspergillus foetidus)、烟曲霉、日本曲霉(Aspergillus japonicus)、构巢曲霉(Aspergillus nidulans)、黑曲霉、米曲霉、狭边金孢子菌(Chrysosporiuminops)、嗜角质金孢子菌(Chrysosporium keratinophilum)、卢克诺文思金孢子菌(Chrysosporium lucknowense)、粪状金孢子菌(Chrysosporium merdarium)、毡金孢子菌(Chrysosporium pannicola)、昆士兰金孢子菌(Chrysosporium queenslandicum)、热带金孢子菌(Chrysosporium tropicum)、带纹金孢子菌(Chrysosporium zonatum)、杆孢状镰孢(Fusarium bactridioides)、谷类镰孢(Fusarium cerealis)、库威镰孢(Fusariumcrookwellense)、大刀镰孢(Fusarium culmorum)、禾谷镰孢(Fusarium graminearum)、禾赤镰孢(Fusarium graminum)、异孢镰孢(Fusarium heterosporum)、合欢木镰孢(Fusariumnegundi)、尖孢镰孢(Fusarium oxysporum)、多枝镰孢(Fusarium reticulatum)、粉红镰孢(Fusarium roseum)、接骨木镰孢(Fusarium sambucinum)、肤色镰孢(Fusariumsarcochroum)、拟分枝孢镰孢(Fusarium sporotrichioides)、硫色镰孢(Fusariumsulphureum)、圆镰孢(Fusarium torulosum)、拟丝孢镰孢(Fusarium trichothecioides)、镶片镰孢(Fusarium venenatum)、灰腐质霉(Humicola grisea)、特异腐质霉(Humicolainsolens)、疏棉状腐质霉(Humicola lanuginosa)、白耙齿菌(Irpex lacteus)、米黑根毛霉(Mucor miehei)、嗜热毁丝霉(Myceliophthora thermophila)、粗糙链孢菌(Neurosporacrassa)、绳状青霉菌(Penicillium funiculosum)、产紫青霉菌(Penicilliumpurpurogenum)、黄孢原毛平革菌(Phanerochaete chrysosporium)、无色梭孢壳霉(Thielavia achromatica)、成层梭抱壳菌(Thielavia albomyces)、白毛梭孢壳霉(Thielavia albopilosa)、澳洲梭孢壳霉(Thielavia australeinsis)、菲美蒂梭抱壳菌(Thielavia fimeti)、小孢梭孢壳霉(Thielavia microspora)、卵孢梭孢壳霉(Thielaviaovispora)、秘鲁梭孢壳霉(Thielavia peruviana)、毛梭孢壳霉(Thielavia setosa)、瘤孢梭孢壳霉(Thielavia spededonium)、耐热梭孢壳(Thielavia subthermophila)、土生梭孢壳霉(Thielavia terrestris)、哈茨木霉(Trichoderma harzianum)、康宁木霉(Trichoderma koningii)、长枝木霉(Trichoderma longibrachiatum)、里氏木霉、或绿色木霉(Trichoderma viride)。
应理解的是对于前述的种,本发明涵盖完全和不完全阶段(perfect andimperfect states),和其他分类学的等同物(equivalent),例如无性型(anamorph),而与它们已知的种名无关。本领域的技术人员会容易地识别适当等同物的身份。
这些物种的菌株可容易地在许多培养物保藏中心为公众所获得,例如美国典型培养物保藏中心(ATCC)、德国微生物菌种保藏中心(Deutsche Sammlung vonMikroorganismen und Zellkulturen GmbH,DSMZ)、荷兰菌种保藏中心(CentraalbureauVoor Schimmelcultures,CBS)和美国农业研究服务专利培养物保藏中心北方地区研究中心(Agricultural Research Service Patent Culture Collection,Northern RegionalResearch Center,NRRL)。
可以使用本文描述或参照的磷脂酶编码序列或其子序列,以及本文描述或参照的磷脂酶或其片段来设计核酸探针以根据本领域熟知的方法来鉴定并克隆编码来自不同属或物种的菌株的磷脂酶的DNA。特别地,可以遵循标准DNA印迹程序,使用此类探针来与目的细胞的基因组DNA或cDNA杂交,以便鉴定和分离其中的相应基因。此类探针可明显短于完整序列,但是长度应为至少15,例如至少25、至少35、或至少70个核苷酸。优选地,该核酸探针长度为至少100个核苷酸,例如长度为至少200个核苷酸、至少300个核苷酸、至少400个核苷酸、至少500个核苷酸、至少600个核苷酸、至少700个核苷酸、至少800个核苷酸、或至少900个核苷酸。DNA和RNA探针两者都可使用。典型地将探针进行标记(例如,用
可以针对与上文所述的探针杂交并编码亲本的DNA来筛选由此类其他菌株制备的基因组DNA或cDNA文库。来自此类其他菌株的基因组DNA或其他DNA可通过琼脂糖或聚丙烯酰胺凝胶电泳或其他分离技术来分离。可将来自文库的DNA或分离的DNA转移到并固定在硝化纤维素或其他适合的运载体材料上。为了鉴定与编码序列或其子序列杂交的克隆或DNA,在DNA印迹中使用该运载体材料。
在一个实施例中,该核酸探针是编码SEQ ID NO:235-242和252-342中任一个的磷脂酶(例如,分别为SEQ ID NO:244-251和343-433的编码序列)或其片段的多核苷酸或其子序列。
在一个实施例中,该核酸探针是编码SEQ ID NO:235、236、237、238、239、240、241和242中任一个的磷脂酶(例如,分别为SEQ ID NO:244、245、246、247、248、249、250或251的编码序列)或其片段的多核苷酸或其子序列。
出于上述探针的目的,杂交表明多核苷酸与标记的核酸探针,或其全长互补链或前述的子序列杂交;杂交是在非常低的至非常高的严格条件下进行。可使用例如X射线片(X-ray film)检测在这些条件下与核酸探针杂交的分子。严格性和洗涤条件如上述所定义。
在一个实施例中,磷脂酶由多核苷酸编码,该多核苷酸在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与本文描述或参照的磷脂酶中任一种的编码序列(例如,编码SEQ ID NO:235-242和252-342中任一个的编码序列;例如分别为SEQ ID NO:244-251或343-433的相应编码序列,或编码SEQ ID NO:235、236、237、238、239、240、241和242中任一个的编码序列;例如分别为SEQ ID NO:244、245、246、247、248、249、250或251的相应编码序列)的全长互补链杂交。(Sambrook等人,1989,Molecular Cloning,A Laboratory Manual[分子克隆:实验室手册],第2版Cold SpringHarbor,New York[冷泉港,纽约])。
也可以使用以上提到的探针从其他来源,包括从自然界(例如,土壤、堆肥、水、青贮等)分离的微生物或直接从天然材料(例如,土壤、堆肥、水、青贮等)获得的DNA样品鉴定并获得磷脂酶。用于从天然生境中直接分离微生物和DNA的技术是本领域熟知的。随后可以通过类似地筛选另一种微生物的基因组或cDNA文库或混合DNA样品而得到编码磷脂酶的多核苷酸。
一旦已经用如在此描述的适合的探针检测到编码磷脂酶的多核苷酸,则可以通过利用本领域的普通技术人员已知的技术来分离或克隆该序列(参见,例如,Sambrook等人,1989,Molecular Cloning,A Laboratory Manual[分子克隆实验手册],第2版,冷泉港(Cold Spring Harbor),纽约)。用于分离或克隆编码α-淀粉酶的多核苷酸的技术包括从基因组DNA分离、从cDNA制备或其组合。可以例如通过使用熟知的聚合酶链式反应(PCR)或表达文库的抗体筛选来检测具有共有结构特征的克隆DNA片段,实现从这样的基因组DNA克隆多核苷酸。参见例如,Innis等人,1990,PCR:A Guide to Methods and Application[PCR:方法和应用指南],Academic Press[学术出版社],纽约。还可以使用其他核酸扩增程序,如连接酶链式反应(LCR)、连接激活转录(LAT)和基于核苷酸序列的扩增(NASBA)。
在一个实施例中,磷脂酶具有成熟多肽序列,其包含本文描述或参照的磷脂酶中任一种(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列或由其组成。在另一个实施例中,磷脂酶具有成熟多肽序列,其是本文描述或参照的磷脂酶中任一种(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的片段。在一个实施例中,片段中氨基酸残基的数目是参照的全长磷脂酶(例如SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)中氨基酸残基数目的至少75%,例如至少80%、85%、90%或95%。在其他实施例中,磷脂酶可以包含本文描述或参照的任何磷脂酶的催化结构域(例如,SEQ ID NO:235-242和252-342中任一个的催化结构域,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)。
磷脂酶可以是上述磷脂酶中任一种的变体(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)。在一个实施例中,磷脂酶具有与上述磷脂酶中任一种(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在一个实施例中,磷脂酶具有与上述磷脂酶中任一种(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在一个实施例中,磷脂酶具有上述磷脂酶中任一种(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列的一个或多个(例如,两个,若干个)的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
氨基酸改变的性质通常较小,也就是说不会显著影响蛋白质折叠和/或活性的保守氨基酸取代或插入;典型地为一个至约30个氨基酸的小缺失;小氨基末端或羧基末端延伸,如氨基末端甲硫氨酸残基;多至约20-25个残基的小接头肽;或小的延伸,其通过改变净电荷或另一功能(例如聚组氨酸段、抗原表位或结合结构域)来促进纯化。
保守取代的实例在下组之内:碱性氨基酸(精氨酸、赖氨酸和组氨酸)、酸性氨基酸(谷氨酸和天冬氨酸)、极性氨基酸(谷氨酰胺和天冬酰胺)、疏水氨基酸(亮氨酸、异亮氨酸和缬氨酸)、芳族氨基酸(苯丙氨酸、色氨酸和酪氨酸)、以及小氨基酸(甘氨酸、丙氨酸、丝氨酸、苏氨酸和甲硫氨酸)。通常不会改变比活性的氨基酸取代是本领域已知的并且例如由H.Neurath和R.L.Hill,1979,于The Proteins[蛋白质],Academic Press[学术出版社],纽约中描述。最普遍发生的交换是Ala/Ser、Val/Ile、Asp/Glu、Thr/Ser、Ala/Gly、Ala/Thr、Ser/Asn、Ala/Val、Ser/Gly、Tyr/Phe、Ala/Pro、Lys/Arg、Asp/Asn、Leu/Ile、Leu/Val、Ala/Glu和Asp/Gly。
可替代地,这些氨基酸改变具有这样一种性质:改变多肽的物理化学特性。例如,氨基酸改变可以提高磷脂酶的热稳定性、改变底物特异性、改变最适pH,等等。
可以根据本领域已知的程序来鉴定必需氨基酸,如定点诱变或丙氨酸扫描诱变(Cunningham和Wells,1989,Science[科学]244:1081-1085)。在后一项技术中,在该分子中的每个残基处引入单个丙氨酸突变,并且对所得突变体分子的活性进行测试以鉴定对于该分子的活性至关重要的氨基酸残基。还参见,Hilton等人,1996,J.Biol.Chem.[生物化学杂志]271:4699-4708。活性部位或其他生物学相互作用还可通过对结构的物理分析来确定,如由下述技术确定:核磁共振、晶体学(crystallography)、电子衍射、或光亲和标记,连同对推定的接触位点氨基酸进行突变。参见例如,de Vos等人,1992,Science[科学]255:306-312;Smith等人,1992,J.Mol.Biol.[分子生物学杂志]224:899-904;Wlodaver等人,1992,FEBS Lett.[欧洲生化学会联合会快报]309:59-64。还可以从与参照磷脂酶相关的其他磷脂酶的同一性分析推断必需氨基酸的同一性。
可以使用本领域熟知的多重序列比对(MSA)技术来确定另外的有关本文多肽的结构-活性关系的指导。基于本文的教导,本领域的技术人员可以与本文所述的或本领域已知的任何数目的磷脂酶做相似的比对。此类比对帮助本领域的技术人员测定潜在相关的结构域(例如,结合结构域或催化结构域),并且在不同的α-淀粉酶序列中哪些氨基酸残基是保守的和非保守的。本领域中应理解的是,改变在所披露的多肽之间的特定位置处是保守的氨基酸将更有可能导致生物活性的改变(Bowie等人,1990,Science[科学]247:1306-1310:“Residues that are directly involved in protein functions such as binding orcatalysis will certainly be among the most conserved[直接涉及到蛋白功能如结合或催化的残基将一定是在最保守的残基中]”)。相比之下,取代在多肽之间不是高度保守的氨基酸将不太可能或不显著地改变生物活性。
本领域的技术人员可以在本领域已知的公开的X射线晶体学研究中找到甚至另外的关于结构-活性关系的指导。
使用已知的诱变、重组和/或改组方法,随后进行相关的筛选程序可以做出单或多氨基酸取代、缺失和/或插入并对其进行测试,该相关的筛选程序例如由Reidhaar-Olson和Sauer,1988,Science[科学]241:53-57;Bowie和Sauer,1989,Proc.Natl.Acad.Sci.USA[美国国家科学院院刊]86:2152-2156;WO 95/17413;或WO 95/22625披露的那些。其他可以使用的方法包括易错PCR、噬菌体展示(例如Lowman等人,1991,Biochemistry[生物化学]30:10832-10837;美国专利号5,223,409;WO 92/06204)以及区域定向诱变(Derbyshire等人,1986,Gene[基因]46:145;Ner等人,1988,DNA 7:127)。
诱变/改组方法可以与高通量、自动化的筛选方法组合以检测由宿主细胞表达的克隆的、诱变的多肽的活性(Ness等人,1999,Nature Biotechnology[自然生物技术]17:893-896)。可以从宿主细胞回收编码活性α-淀粉酶的诱变的DNA分子,并使用本领域的标准方法对其进行快速测序。这些方法允许快速确定多肽中各个氨基酸残基的重要性。
在一些实施例中,磷脂酶在相同条件下具有本文描述或参照的任一种磷脂酶(例如,SEQ ID NO:235-242和252-342中任一个,例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的磷脂酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,磷脂酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任一种磷脂酶(例如,SEQ ID NO:235-242和252-342中任一个的磷脂酶的编码序列;例如分别为SEQ IDNO:244-251或343-433的相应编码序列;或SEQ ID NO:235、236、237、238、239、240、241或242中任一个的磷脂酶;例如分别为SEQ ID NO:244、245、246、247、248、249、250或251的相应编码序列)的编码序列的全长互补链杂交。在一个实施例中,磷脂酶编码序列与来自本文描述或参照的任一种磷脂酶(例如,SEQ ID NO:235-242和252-342中任一个的磷脂酶的编码序列;例如分别为SEQ ID NO:244-251或343-433的相应编码序列;或SEQ ID NO:235、236、237、238、239、240、241或242中任一个的磷脂酶;例如分别为SEQ ID NO:244、245、246、247、248、249、250或251的相应编码序列)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,磷脂酶包含本文描述或参照的任何磷脂酶(例如,SEQ ID NO:235-242和252-342中任一个的磷脂酶的编码序列;例如分别为SEQ ID NO:244-251或343-433的相应编码序列;或SEQ ID NO:235、236、237、238、239、240、241或242中任一个的磷脂酶;例如分别为SEQ ID NO:244、245、246、247、248、249、250或251的相应编码序列)的编码序列。在一个实施例中,磷脂酶包含编码序列,该编码序列是来自本文描述或参照的任何磷脂酶的编码序列的子序列,其中该子序列编码具有磷脂酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
本文所述的任何相关方面或实施例的参照编码序列可以是天然编码序列或简并序列,例如设计用于特定宿主细胞的密码子优化的(例如,针对在酿酒酵母中表达而优化的)编码序列。
磷脂酶可以是融合多肽或可切割的融合多肽,其中另一个多肽融合在α-淀粉酶的N-末端或C-末端处。通过将编码另一种多肽的多核苷酸与编码磷脂酶的多核苷酸融合而产生融合多肽。用于产生融合多肽的技术是本领域已知的,并包括连接编码多肽的编码序列,使得它们在阅读框中,并且使该融合多肽的表达在相同的一个或多个启动子和终止子的控制下。还可使用内含肽技术构建融合多肽,其中在翻译后产生融合物(Cooper等人,1993,EMBO J.[欧洲分子生物学学会杂志]12:2575-2583;Dawson等人,1994,Science[科学]266:776-779)。
表达的α-淀粉酶和/或外源α-淀粉酶可以是适用于本文所述的宿主细胞和/或方法的任何α-淀粉酶,如天然存在的α-淀粉酶(例如,来自另一个物种的天然α-淀粉酶或由修饰的表达载体表达的内源α-淀粉酶)或其保留α-淀粉酶活性的变体。对于本发明涉及α-淀粉酶的外源添加的方面,还考虑了预期由下述发酵生物表达的任何α-淀粉酶。
在一些实施例中,该发酵生物包含编码α-淀粉酶的异源多核苷酸,例如,如WO2017/087330中所披露,将其内容通过引用特此并入。考虑本文描述或参照的任何α-淀粉酶用于在发酵生物中表达。
在一些实施例中,当在相同条件下培养时,与不具有编码α-淀粉酶的异源多核苷酸的宿主细胞相比,包含编码该α-淀粉酶的异源多核苷酸的发酵生物具有增加的α-淀粉酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码α-淀粉酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的α-淀粉酶活性水平。
可以与本文所述的宿主细胞和/或方法一起使用的示例性α-淀粉酶包括细菌、酵母或丝状真菌α-淀粉酶,例如,衍生自本文描述或参照的任何微生物。
术语“细菌α-淀粉酶”意指在EC 3.2.1.1项下分类的任何细菌α-淀粉酶。本文所用的细菌α-淀粉酶可例如源自芽孢杆菌属(Bacillus)(有时也称作地芽孢杆菌属(Geobacillus))的菌株。在一个实施例中,该芽孢杆菌属α-淀粉酶源自解淀粉芽孢杆菌、地衣芽孢杆菌、嗜热脂肪芽孢杆菌或枯草芽孢杆菌的菌株,但是也可以源自其他芽孢杆菌属菌种。
细菌α-淀粉酶的具体实例包括WO 99/19467中的SEQ ID NO:3的嗜热脂肪芽孢杆菌α-淀粉酶(BSG),WO 99/19467中的SEQ ID NO:5的解淀粉芽孢杆菌α-淀粉酶(BAN),以及WO 99/19467中的SEQ ID NO:4的地衣芽孢杆菌α-淀粉酶(BLA)(所有序列都通过引用特此并入)。在一个实施例中,该α-淀粉酶可以是具有成熟多肽序列的酶,所述成熟多肽序列与WO 99/19467中的SEQ ID NO:3、4或5所示的任何序列具有至少60%,例如至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%或至少99%同一性。
在一个实施例中,该α-淀粉酶源自嗜热脂肪芽孢杆菌。嗜热脂肪芽孢杆菌α-淀粉酶可为成熟野生型或其成熟变体。该成熟嗜热脂肪芽孢杆菌α-淀粉酶可以在重组产生过程中是天然地截短的。例如,该嗜热脂肪芽孢杆菌α-淀粉酶可以在C-末端处被截短,使得其为480-495个氨基酸长,如约491个氨基酸长,例如使得其缺乏功能性淀粉结合结构域(与WO99/19467中的SEQ ID NO:3相比)。
芽孢杆菌α-淀粉酶还可为变体和/或杂合体。这样的变体的实例可见于以下任一者中:WO 96/23873、WO 96/23874、WO 97/41213、WO 99/19467、WO 00/60059、以及WO 02/10355(各自通过引用特此并入)。具体的α-淀粉酶变体在美国专利号6,093,562、6,187,576、6,297,038和7,713,723中披露(通过引用特此并入)并包括具有以下改变的嗜热脂肪芽孢杆菌α-淀粉酶(常常称作BSGα-淀粉酶)变体:在位置R179、G180、I181和/或G182处的一个或两个氨基酸的缺失,优选WO 96/23873中披露的双缺失–参见例如第20页,第1-10行(通过引用特此并入),如与WO 99/19467中披露的SEQ ID NO:3所示的嗜热脂肪芽孢杆菌α-淀粉酶的氨基酸序列相比对应于位置I181和G182的缺失,或使用WO 99/19467(该参考文献通过引用特此并入)中的SEQ ID NO:3用于编号的氨基酸R179和G180的缺失。在一些实施例中,该芽孢杆菌α-淀粉酶(如嗜热脂肪芽孢杆菌α-淀粉酶)具有与在WO 99/19467中披露的SEQ ID NO:3所示的野生型BSGα-淀粉酶氨基酸序列相比对应于位置181和182的缺失的双缺失,并且还任选地包含N193F取代(也表示为I181*+G182*+N193F)。该细菌α-淀粉酶还可以在对应于WO 99/19467中的SEQ ID NO:4所示的地衣芽孢杆菌α-淀粉酶、或WO 99/19467中的SEQ ID NO:3的嗜热脂肪芽孢杆菌α-淀粉酶的S242和/或E188P变体中的S239的位置处具有取代。
在一个实施例中,该变体是嗜热脂肪芽孢杆菌α-淀粉酶的S242A、E或Q变体,例如S242Q变体。
在一个实施例中,该变体是嗜热脂肪芽孢杆菌α-淀粉酶的位置E188变体,例如E188P变体。
在一个实施例中,该细菌α-淀粉酶可以是截短的芽孢杆菌属α-淀粉酶。在一个实施例中,截短是这样的,例如,以使得WO 99/19467中的SEQ ID NO:3所示的嗜热脂肪芽孢杆菌α-淀粉酶是约491个氨基酸长,如从480至495个氨基酸长,或者使得其缺乏功能性淀粉结合结构域。
该细菌α-淀粉酶也可以是杂合细菌α-淀粉酶,例如包含地衣芽孢杆菌α-淀粉酶(WO 99/19467的SEQ ID NO:4中所示)的445个C-末端氨基酸残基和源自解淀粉芽孢杆菌的α-淀粉酶(WO 99/19467的SEQ ID NO:5中所示)的37个N-末端氨基酸残基的α-淀粉酶。在一个实施例中,该杂合体具有以下取代中的一个或多个,尤其是全部:G48A+T49I+G107A+H156Y+A181T+N190F+I201F+A209V+Q264S(使用WO 99/19467的SEQ ID NO:4中的地衣芽孢杆菌编号方式)。在一些实施例中,这些变体具有以下突变(或在其他芽孢杆菌α-淀粉酶中的对应突变)中的一个或多个:H154Y、A181T、N190F、A209V以及Q264S和/或在位置176与179之间的两个残基的缺失,例如E178和G179的缺失(使用WO 99/19467的SEQ ID NO:5进行位置编号)。
在一个实施例中,该细菌α-淀粉酶是嵌合α-淀粉酶的成熟部分,其披露于Richardson等人(2002),The Journal of Biological Chemistry[生化杂志],277卷,29期,7月19日发表,267501-26507页中,称作BD5088或其变体。此α-淀粉酶与WO 2007/134207中的SEQ ID NO:2所示的相同。成熟酶序列在位置1处的起始“Met”氨基酸之后开始。
该α-淀粉酶可以是热稳定α-淀粉酶,如热稳定细菌α-淀粉酶,例如来自嗜热脂肪芽孢杆菌。在一个实施例中,如WO 2018/098381的实例1中所述确定的,在本文所述的方法中使用的α-淀粉酶在pH4.5、85℃、0.12mM CaCl
在一个实施例中,该热稳定α-淀粉酶在pH 4.5、85℃、0.12mM CaCl
在一个实施例中,该热稳定α-淀粉酶在pH 4.5、85℃、0.12mM CaCl
在一个实施例中,该α-淀粉酶是细菌α-淀粉酶,例如衍生自芽孢杆菌属,如嗜热脂肪芽孢杆菌的菌株,例如,如在WO 99/019467中披露为SEQ ID NO:3的嗜热脂肪芽孢杆菌,其中在位置R179、G180、I181和/或G182处具有一个或两个氨基酸缺失,特别是R179和G180缺失、或具有I181和G182缺失,具有如下突变列表中的突变。
在一些实施例中,该嗜热脂肪芽孢杆菌α-淀粉酶具有双缺失I181+G182、以及任选的取代N193F,进一步包含以下取代或取代的组合中的一个:
V59A+Q89R+G112D+E129V+K177L+R179E+K220P+N224L+Q254S;
V59A+Q89R+E129V+K177L+R179E+H208Y+K220P+N224L+Q254S;
V59A+Q89R+E129V+K177L+R179E+K220P+N224L+Q254S+D269E+D281N;
V59A+Q89R+E129V+K177L+R179E+K220P+N224L+Q254S+I270L;
V59A+Q89R+E129V+K177L+R179E+K220P+N224L+Q254S+H274K;
V59A+Q89R+E129V+K177L+R179E+K220P+N224L+Q254S+Y276F;
V59A+E129V+R157Y+K177L+R179E+K220P+N224L+S242Q+Q254S;
V59A+E129V+K177L+R179E+H208Y+K220P+N224L+S242Q+Q254S;
V59A+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S;
V59A+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+H274K;
V59A+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+Y276F;
V59A+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+D281N;
V59A+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+M284T;
V59A+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+G416V;
V59A+E129V+K177L+R179E+K220P+N224L+Q254S;
V59A+E129V+K177L+R179E+K220P+N224L+Q254S+M284T;
A91L+M96I+E129V+K177L+R179E+K220P+N224L+S242Q+Q254S;
E129V+K177L+R179E;
E129V+K177L+R179E+K220P+N224L+S242Q+Q254S;
E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+Y276F+L427M;
E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+M284T;
E129V+K177L+R179E+K220P+N224L+S242Q+Q254S+N376*+I377*;
E129V+K177L+R179E+K220P+N224L+Q254S;
E129V+K177L+R179E+K220P+N224L+Q254S+M284T;
E129V+K177L+R179E+S242Q;
E129V+K177L+R179V+K220P+N224L+S242Q+Q254S;
K220P+N224L+S242Q+Q254S;
M284V;
V59A+Q89R+E129V+K177L+R179E+Q254S+M284V;以及
V59A+E129V+K177L+R179E+Q254S+M284V;
在一个实施例中,该α-淀粉酶选自下组:嗜热脂肪芽孢杆菌α-淀粉酶变体,其具有双缺失I181*+G182*、以及任选的取代N193F,并且进一步具有以下取代或取代的组合中的一个:
E129V+K177L+R179E;
V59A+Q89R+E129V+K177L+R179E+H208Y+K220P+N224L+Q254S;
V59A+Q89R+E129V+K177L+R179E+Q254S+M284V;
V59A+E129V+K177L+R179E+Q254S+M284V;以及
E129V+K177L+R179E+K220P+N224L+S242Q+Q254S(使用本文的SEQ ID NO:1进行编号)。
应理解的是,当提及嗜热脂肪芽孢杆菌α-淀粉酶及其变体时,它们通常以截短的形式产生。具体地,截短可以是这样的,以使得WO99/19467中的SEQ ID NO:3所示的嗜热脂肪芽孢杆菌α-淀粉酶或其变体在C-末端处截短并且典型地是约从480-495个氨基酸长,如约491个氨基酸长,例如使得其缺乏功能性淀粉结合结构域。
在一个实施例中,该α-淀粉酶变体可以是如下酶,该酶具有与WO 99/19467中的SEQ ID NO:3所示的序列至少60%,例如至少70%、至少80%、至少90%、至少95%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%或至少99%,但小于100%同一性程度的成熟多肽序列。
在一个实施例中,将该细菌α-淀粉酶(例如芽孢杆菌属α-淀粉酶,如尤其是嗜热脂肪芽孢杆菌α-淀粉酶或其变体)以在0.01-10KNU-A/g DS之间的浓度给出到液化中,例如,在0.02和5KNU-A/g DS,如0.03和3KNU-A,优选地0.04和2KNU-A/g DS,如尤其地0.01和2KNU-A/g DS之间。在一个实施例中,将该细菌α-淀粉酶(例如芽孢杆菌属α-淀粉酶,如尤其是嗜热脂肪芽孢杆菌α-淀粉酶或其变体)以在0.0001-1mg EP(酶蛋白)/g DS之间的浓度给出到液化中,例如0.0005-0.5mg EP/g DS,如0.001-0.1mg EP/g DS。
在一个实施例中,该细菌α-淀粉酶衍生自SEQ ID NO:76的枯草芽孢杆菌α-淀粉酶、SEQ ID NO:82的枯草芽孢杆菌α-淀粉酶、SEQ ID NO:83的枯草芽孢杆菌α-淀粉酶、SEQID NO:84的枯草芽孢杆菌α-淀粉酶、或SEQ ID NO:85的地衣芽孢杆菌α-淀粉酶、SEQ IDNO:89的发酵植物多糖梭菌(Clostridium phytofermentans)α-淀粉酶、SEQ ID NO:90的发酵植物多糖梭菌α-淀粉酶、SEQ ID NO:91的发酵植物多糖梭菌α-淀粉酶、SEQ ID NO:92的发酵植物多糖梭菌α-淀粉酶、SEQ ID NO:93的发酵植物多糖梭菌α-淀粉酶、SEQ ID NO:94的发酵植物多糖梭菌α-淀粉酶、SEQ ID NO:95的热纤维梭菌(Clostridium thermocellum)α-淀粉酶、SEQ ID NO:96的嗜热裂孢菌(Thermobifida fusca)α-淀粉酶、SEQ ID NO:97的嗜热裂孢菌α-淀粉酶、SEQ ID NO:98的嗜热厌氧菌、SEQ ID NO:99的嗜热厌氧菌(Anaerocellum thermophilum)、SEQ ID NO:100的嗜热厌氧菌、SEQ ID NO:101的除虫链霉菌、或SEQ ID NO:88的除虫链霉菌。
在一个实施例中,α-淀粉酶衍生自解淀粉芽孢杆菌,例如SEQ ID NO:231的解淀粉芽孢杆菌α-淀粉酶(例如,如WO 2018/002360中所述,或如WO 2017/037614中所述的其变体)。
在一个实施例中,该α-淀粉酶衍生自酵母α-淀粉酶,如SEQ ID NO:77的扣囊复膜酵母菌α-淀粉酶、SEQ ID NO:78的西方德巴利酵母α-淀粉酶、SEQ ID NO:79的西方德巴利酵母α-淀粉酶、SEQ ID NO:80的橘林油脂酵母(Lipomyces kononenkoae)α-淀粉酶、SEQ IDNO:81的橘林油脂酵母α-淀粉酶。
在一个实施例中,该α-淀粉酶衍生自丝状真菌α-淀粉酶,如SEQ ID NO:86的黑曲霉α-淀粉酶、或SEQ ID NO:87的黑曲霉α-淀粉酶。
可以用发酵生物表达并与本文所述方法一起使用的其他α-淀粉酶在实例中描述,包括但不限于表2所示的α-淀粉酶(或其衍生物)。
表2.
考虑与本发明一起使用的另外的α-淀粉酶可以在WO 2011/153516(将其内容并入本文)中找到。
编码适合的α-淀粉酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(
α-淀粉酶编码序列也可用于设计核酸探针以从不同属或种的菌株鉴定和克隆编码海藻糖酶的DNA,如上所述。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码α-淀粉酶的多核苷酸,如上文所述的。
用于分离或克隆编码α-淀粉酶的多核苷酸的技术在上文中描述。
在一个实施例中,α-淀粉酶具有成熟多肽序列,其包含本文描述或参照的α-淀粉酶中任一种(例如,SEQ ID NO:76-101、121-174和231中任一个)的氨基酸序列或由其组成。在另一个实施例中,α-淀粉酶具有成熟多肽序列,其是本文描述或参照的α-淀粉酶中任一种(例如,SEQ ID NO:76-101、121-174和231中任一个)的片段。在一个实施例中,片段中氨基酸残基的数目是参照的全长α-淀粉酶(例如SEQ ID NO:76-101、121-174和231中任一个)中氨基酸残基数目的至少75%,例如至少80%、85%、90%或95%。在其他实施例中,α-淀粉酶可以包含本文描述或参照的任何α-淀粉酶的催化结构域(例如,SEQ ID NO:76-101、121-174和231中任一个的催化结构域)。
α-淀粉酶可以是上述α-淀粉酶中任一种(例如,SEQ ID NO:76-101、121-174和231中任一个)的变体。在一个实施例中,α-淀粉酶具有与上述α-淀粉酶中任一种(例如,SEQ IDNO:76-101、121-174和231中任一个)具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在一个实施例中,α-淀粉酶具有与上述α-淀粉酶中任一种(例如,SEQ ID NO:76-101、121-174和231中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在一个实施例中,α-淀粉酶具有上述α-淀粉酶中任一种(例如,SEQ ID NO:76-101、121-174和231中任一个)的氨基酸序列的一个或多个(例如,两个,若干个)的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,α-淀粉酶在相同条件下具有本文描述或参照的任一种α-淀粉酶(例如,SEQ ID NO:76-101、121-174和231中任一个)的α-淀粉酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,α-淀粉酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任一种α-淀粉酶(例如,SEQ ID NO:76-101、121-174和231中任一个)的编码序列的全长互补链杂交。在一个实施例中,α-淀粉酶编码序列与来自本文描述或参照的任一种α-淀粉酶(例如,SEQID NO:76-101、121-174和231中任一个)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,α-淀粉酶包含本文描述或参照的任一种α-淀粉酶(SEQ ID NO:76-101、121-174和231中任一个)的编码序列。在一个实施例中,α-淀粉酶包含编码序列,该编码序列是来自本文描述或参照的任何α-淀粉酶的编码序列的子序列,其中该子序列编码具有α-淀粉酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
本文所述的任何相关方面或实施例的参照编码序列可以是天然编码序列或简并序列,例如设计用于特定宿主细胞的密码子优化的(例如,针对在酿酒酵母中表达而优化的)编码序列。
该α-淀粉酶还可以包括融合多肽或可切割的融合多肽,如上文所述的。
表达的海藻糖酶和/或外源海藻糖酶可以是适用于本文所述的发酵生物和/或其使用方法的任何海藻糖酶,如天然存在的海藻糖酶或其保留海藻糖酶活性的变体。对于本发明涉及海藻糖酶的外源添加的方面,还考虑了预期由下述发酵生物表达的任何海藻糖酶(例如,在液化和/或糖化之前、期间或之后添加)。
在一些实施例中,当在相同条件下培养时,与不具有编码海藻糖酶的异源多核苷酸的宿主细胞相比,包含编码该海藻糖酶的异源多核苷酸的发酵生物具有增加的海藻糖酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码海藻糖酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的海藻糖酶活性水平。
可以用发酵生物表达并用于本文所述方法的海藻糖酶包括但不限于表3所示的海藻糖酶(或其衍生物)。
表3.
编码适合的海藻糖酶的另外的多核苷酸可以衍生自任何适合属的微生物,包括在UniProtKB数据库(
海藻糖酶编码序列也可用于设计核酸探针以从不同属或种的菌株鉴定和克隆编码海藻糖酶的DNA,如上所述。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码海藻糖酶的多核苷酸,如上文所述的。
用于分离或克隆编码海藻糖酶的多核苷酸的技术在上文中描述。
在一个实施例中,海藻糖酶具有成熟多肽序列,其包含本文描述或参照的海藻糖酶中任一种(例如,SEQ ID NO:175-226中任一个)的氨基酸序列或由其组成。在另一个实施例中,海藻糖酶具有成熟多肽序列,其是本文描述或参照的海藻糖酶中任一种(例如,SEQID NO:175-226中任一个)的片段。在一个实施例中,片段中的氨基酸残基数目是参照全长海藻糖酶(例如,SEQ ID NO:175-226中任一个)中氨基酸残基数目的至少75%,例如至少80%、85%、90%或95%。在其他实施例中,海藻糖酶可包含本文描述或参照的任何海藻糖酶的催化结构域(例如,SEQ ID NO:175-226中任一个的催化结构域)。
海藻糖酶可以是上述海藻糖酶中任一种(例如,SEQ ID NO:175-226中任一个)的变体。在一个实施例中,海藻糖酶具有与上述海藻糖酶中任一种(例如,SEQ ID NO:175-226中任一个)具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在一个实施例中,海藻糖酶具有与上述海藻糖酶中任一种(例如,SEQ ID NO:175-226中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在一个实施例中,海藻糖酶具有上述海藻糖酶中任一种(例如,SEQ ID NO:175-226中任一个)的氨基酸序列的一个或多个(例如,两个,若干个)的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,海藻糖酶在相同条件下具有本文描述或参照的任何海藻糖酶(例如,SEQ ID NO:175-226中任一个)的海藻糖酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,海藻糖酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任何海藻糖酶(例如,SEQ ID NO:175-226中任一个)的编码序列的全长互补链杂交。在一个实施例中,海藻糖酶编码序列与来自本文描述或参照的任何海藻糖酶(例如,SEQ ID NO:175-226中任一个)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,海藻糖酶包含本文描述或参照的任何海藻糖酶(SEQ ID NO:175-226中任一个)的编码序列。在一个实施例中,海藻糖酶包含编码序列,该编码序列是来自本文描述或参照的任何海藻糖酶的编码序列的子序列,其中该子序列编码具有海藻糖酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
本文所述的任何相关方面或实施例的参照编码序列可以是天然编码序列或简并序列,例如设计用于特定宿主细胞的密码子优化的(例如,针对在酿酒酵母中表达而优化的)编码序列。
海藻糖酶还可以包括融合多肽或可切割的融合多肽,如上文所述。
表达的葡糖淀粉酶和/或外源葡糖淀粉酶可以是适用于本文所述的发酵生物和/或其使用方法的任何葡糖淀粉酶,如天然存在的葡糖淀粉酶或其保留葡糖淀粉酶活性的变体。对于本发明涉及葡糖淀粉酶的外源添加的方面,还考虑了预期由下述发酵生物表达的任何葡糖淀粉酶(例如,在液化和/或糖化之前、期间或之后添加)。
在一些实施例中,该发酵生物包含编码葡糖淀粉酶的异源多核苷酸,例如,如WO2017/087330中所披露,将其内容通过引用特此并入。考虑本文描述或参照的任何葡糖淀粉酶用于在发酵生物中表达。
在一些实施例中,当在相同条件下培养时,与不具有编码葡糖淀粉酶的异源多核苷酸的宿主细胞相比,包含编码该葡糖淀粉酶的异源多核苷酸的发酵生物具有增加的葡糖淀粉酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码葡糖淀粉酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的葡糖淀粉酶活性水平。
可以与本文所述的宿主细胞和/或方法一起使用的示例性葡糖淀粉酶包括细菌、酵母或丝状真菌葡糖淀粉酶,例如,获得自本文描述或参照的任何微生物,如上文在与α-淀粉酶有关的部分下描述的。
该葡糖淀粉酶可以衍生自任何适合的来源,例如衍生自微生物或植物。优选的葡糖淀粉酶是真菌或细菌来源的,选自由以下组成的组:曲霉属葡糖淀粉酶,特别是黑曲霉G1或G2葡糖淀粉酶(Boel等人,1984,EMBO J.[欧洲分子生物学学会杂志]3(5),第1097-1102页),或其变体,如WO 92/00381、WO 00/04136和WO 01/04273中披露的那些(来自诺维信公司,丹麦);WO 84/02921中披露的泡盛曲霉葡糖淀粉酶;米曲霉葡糖淀粉酶(Agric.Biol.Chem.[农业与生物化学](1991),55(4),第941-949页),或其变体或片段。其他曲霉属葡糖淀粉酶变体包括具有增强的热稳定性的变体:G137A和G139A(Chen等人(1996),Prot.Eng.[蛋白质工程]9,499-505);D257E和D293E/Q(Chen等人.(1995),Prot.Eng.[蛋白质工程]8,575-582);N182(Chen等人(1994),Biochem.J.301[生物化学杂志],275-281);二硫键、A246C(Fierobe等人,1996,Biochemistry[生物化学],35:8698-8704);和在A435和S436位置引入Pro残基(Li等人,1997,Protein Engng.[蛋白质工程]10,1199-1204)。
其他葡糖淀粉酶包括罗耳阿太菌(Athelia rolfsii)(以前命名为罗尔伏革菌(Corticium rolfsii))葡糖淀粉酶(参见美国专利号4,727,026和Nagasaka等人(1998)“Purification and properties of the raw-starch-degrading glucoamylases fromCorticium rolfsii[来自罗尔伏革菌的粗淀粉降解葡糖淀粉酶的纯化及性质]”Appl.Microbiol.Biotechnol.[应用微生物学与生物技术]50:323-330),篮状菌属葡糖淀粉酶,特别是源自埃默森篮状菌(WO 99/28448)、雷塞氏篮状菌(美国专利号Re.32,153)、杜氏篮状菌(Talaromyces duponti)、以及嗜热篮状菌(美国专利号4,587,215)。在一个实施例中,糖化和/或发酵期间使用的葡糖淀粉酶是WO 99/28448中披露的埃默森篮状菌葡糖淀粉酶。
涵盖的细菌葡糖淀粉酶包括来自梭菌属(Clostridium),特别是热解淀粉梭菌(C.thermoamylolyticum)(EP 135,138)和热硫化氢梭菌(C.thermohydrosulfuricum)(WO86/01831)的葡糖淀粉酶。
涵盖的真菌葡糖淀粉酶包括均在WO 2006/069289中披露的瓣环栓菌,纸质大纹饰孢(Pachykytospora papyracea);和大白桩蘑(Leucopaxillus giganteus);或WO 2007/124285中披露的红边隔孢伏革菌(Peniophora rufomarginata);或其混合物。还考虑了杂合葡糖淀粉酶。实例包括WO 2005/045018中披露的杂合葡糖淀粉酶。
在一个实施例中,该葡糖淀粉酶衍生自密孔菌属的菌株,特别是如描述于WO2011/066576中的密孔菌属的菌株(其中的SEQ ID NO:2、4或6),包括血红密孔菌葡糖淀粉酶,或者衍生自粘褶菌属的菌株,如篱边粘褶菌或密粘褶菌的菌株,特别是如描述于WO2011/068803中的粘褶菌属的菌株(其中的SEQ ID NO:2、4、6、8、10、12、14或16)。在一个实施例中,该葡糖淀粉酶是WO 2011/068803中的SEQ ID NO:2(即,篱边粘褶菌葡糖淀粉酶)。在一个实施例中,该葡糖淀粉酶是SEQ ID NO:8的篱边粘褶菌葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶是SEQ ID NO:229的血红密孔菌葡糖淀粉酶。
在一个实施例中,该葡糖淀粉酶是密粘褶菌葡糖淀粉酶(在WO 2014/177546中披露为SEQ ID NO:3)。在另一个实施例中,该葡糖淀粉酶衍生自黑层孔属(Nigrofomes)的菌株,特别是披露于WO 2012/064351中的黑层孔属物种的菌株(其中的SEQ ID NO:2所披露)。
还考虑了具有成熟多肽序列的葡糖淀粉酶,该成熟多肽序列与任何上述葡糖淀粉酶表现出高度同一性,即,与上述成熟多肽序列中的任一个表现出至少60%,如至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或甚至100%同一性。
葡糖淀粉酶能以如下量添加到糖化和/或发酵中:0.0001-20AGU/g DS,例如0.001-10AGU/g DS、0.01-5AGU/g DS、或0.1-2AGU/g DS。
葡糖淀粉酶能以如下量添加到糖化和/或发酵中:1-1,000μg EP/g DS,例如10-500μg/g DS、或25-250μg/g DS。
葡糖淀粉酶能以0.1-100μg EP/g DS,例如0.5-50μg EP/g DS、1-25μg EP/g DS、或2-12μg EP/g DS的量添加到液化中。
在一个实施例中,葡糖淀粉酶作为进一步包含α-淀粉酶(例如,文中所述的任何α-淀粉酶)的共混物添加。在一个实施例中,该α-淀粉酶是真菌α-淀粉酶,尤其是酸性真菌α-淀粉酶。α-淀粉酶典型地具有副活性。
在一个实施例中,该葡糖淀粉酶是共混物,其包含WO 99/28448中披露为SEQ IDNO:34的埃默森篮状菌葡糖淀粉酶和WO 06/069289中披露为SEQ ID NO:2的瓣环栓菌葡糖淀粉酶。
在一个实施例中,葡糖淀粉酶是共混物,其包含WO 99/28448中披露的埃默森篮状菌葡糖淀粉酶、WO 06/69289中披露为SEQ ID NO:2的瓣环栓菌葡糖淀粉酶、以及α-淀粉酶。
在一个实施例中,该葡糖淀粉酶是共混物,其包含披露于WO99/28448中的埃默森篮状菌葡糖淀粉酶、披露于WO 06/69289中的瓣环栓菌葡糖淀粉酶、以及在WO 2006/069290的表5中披露为V039的具有黑曲霉葡糖淀粉酶接头和SBD的微小根毛霉(Rhizomucorpusillus)α-淀粉酶。
在一个实施例中,该葡糖淀粉酶是共混物,其包含如在WO 2011/068803中的SEQID NO:2所示的篱边粘褶菌葡糖淀粉酶和α-淀粉酶,特别是WO 2013/006756中的SEQ IDNO:3披露的、具有黑曲霉葡糖淀粉酶接头和淀粉结合结构域(SBD)的微小根毛霉α-淀粉酶,特别地具有以下取代:G128D+D143N。
在一个实施例中,该α-淀粉酶可以源自根毛霉属的菌株,优选微小根毛霉的菌株,如WO 2013/006756中的SEQ ID NO:3所示的,或亚灰树花菌属(Meripilus),优选大型亚灰树花菌的菌株。在一个实施例中,该α-淀粉酶衍生自在WO 2006/069290的表5中披露为V039的具有黑曲霉葡糖淀粉酶接头和淀粉结合结构域(SBD)的微小根毛霉。
在一个实施例中,该微小根毛霉α-淀粉酶或具有黑曲霉葡糖淀粉酶接头和淀粉结合结构域(SBD)的微小根毛霉α-淀粉酶具有以下取代或取代组合中的至少一个:D165M;Y141W;Y141R;K136F;K192R;P224A;P224R;S123H+Y141W;G20S+Y141W;A76G+Y141W;G128D+Y141W;G128D+D143N;P219C+Y141W;N142D+D143N;Y141W+K192R;Y141W+D143N;Y141W+N383R;Y141W+P219C+A265C;Y141W+N142D+D143N;Y141W+K192R V410A;G128D+Y141W+D143N;Y141W+D143N+P219C;Y141W+D143N+K192R;G128D+D143N+K192R;Y141W+D143N+K192R+P219C;和G128D+Y141W+D143N+K192R;或G128D+Y141W+D143N+K192R+P219C(使用WO 2013/006756中的SEQ ID NO:3进行编号)。
在一个实施例中,该葡糖淀粉酶共混物包含篱边粘褶菌葡糖淀粉酶(例如,WO2011/068803中的SEQ ID NO:2)和微小根毛霉α-淀粉酶。
在一个实施例中,该葡糖淀粉酶共混物包含如在WO2011/068803中的SEQ ID NO:2所示的篱边粘褶菌葡糖淀粉酶以及WO 2013/006756的SEQ ID NO:3披露的、具有黑曲霉葡糖淀粉酶接头和淀粉结合结构域(SBD)的微小根毛霉,具有以下取代:G128D+D143N。
包含葡糖淀粉酶的可商购组合物包括AMG 200L;AMG 300L;SAN
在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:102的西方德巴利酵母葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:103的扣囊复膜酵母菌葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:104的扣囊复膜酵母菌葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:105的酿酒酵母葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:106的黑曲霉葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:107的米曲霉葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:108的米根霉(Rhizopus oryzae)葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:109的热纤维梭菌葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:110的热纤维梭菌葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ IDNO:111的Arxula adeninivorans葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQID NO:112的树脂枝孢霉(Hormoconis resinae)葡糖淀粉酶。在一个实施例中,该葡糖淀粉酶衍生自SEQ ID NO:113的出芽短梗霉菌(Aureobasidium pullulans)葡糖淀粉酶。
在一个实施例中,该葡糖淀粉酶是里氏木霉葡糖淀粉酶,例如SEQ ID NO:230的里氏木霉葡糖淀粉酶。
在一个实施例中,该葡糖淀粉酶在85℃下具有至少20%、至少30%、或至少35%的相对活性热稳定性,如WO 2018/098381的实例4(热稳定性)中描述的进行测定。
在一个实施例中,如WO 2018/098381的实例4(pH最佳值)中所述确定的,该葡糖淀粉酶在pH 5.0下具有至少90%,例如至少95%、至少97%、或100%的相对活性pH最佳值。
在一个实施例中,如WO 2018/098381的实例4(pH稳定性)中所述确定的,该葡糖淀粉酶在pH 5.0下具有至少80%、至少85%、至少90%的pH稳定性。
在一个实施例中,在液化中使用的葡糖淀粉酶,如草酸青霉菌葡糖淀粉酶变体,具有在pH 4.0下如WO 2018/098381的实例15中所描述的确定为DSC Td的至少70℃,优选地至少75℃、如至少80℃、如至少81℃、如至少82℃、如至少83℃、如至少84℃、如至少85℃、如至少86℃、如至少87%、如至少88℃、如至少89℃、如至少90℃的热稳定性。在一个实施例中,该葡糖淀粉酶(如草酸青霉菌葡糖淀粉酶变体)具有在pH 4.0下如WO 2018/098381的实例15中所描述的确定为DSC Td的在70℃和95℃之间范围内的(如在80℃和90℃之间)热稳定性。
在一个实施例中,液化中使用的葡糖淀粉酶(如草酸青霉菌葡糖淀粉酶变体)在pH4.8下具有确定为DSC Td的至少70℃,优选地至少75℃、如至少80℃、如至少81℃、如至少82℃、如至少83℃、如至少84℃、如至少85℃、如至少86℃、如至少87%、如至少88℃、如至少89℃、如至少90℃、如至少91℃的热稳定性,如WO2018/098381的实例15中所述。在一个实施例中,该葡糖淀粉酶(如草酸青霉菌葡糖淀粉酶变体)在pH 4.8下具有确定为DSC Td的在70℃和95℃之间范围内(如在80℃和90℃之间)的热稳定性,如WO 2018/098381的实例15中所述。
在一个实施例中,液化中使用的葡糖淀粉酶,如草酸青霉菌葡糖淀粉酶变体,具有如WO 2018/098381的实例16中描述所确定的至少100%,如至少105%、如至少110%、如至少115%、如至少120%、如至少125%的残余活性。在一个实施例中,该葡糖淀粉酶(如草酸青霉菌葡糖淀粉酶变体)具有如WO 2018/098381的实例16中所描述的确定为残余活性的在100%和130%之间范围内的热稳定性。
在一个实施例中,该葡糖淀粉酶(例如真菌来源的,如丝状真菌)是来自青霉属(Penicillium)的菌株,例如草酸青霉菌(Penicillium oxalicum)的菌株,特别是在WO2011/127802(将其通过引用特此并入)中披露为SEQ ID NO:2的草酸青霉菌葡糖淀粉酶。
在一个实施例中,该葡糖淀粉酶具有与WO 2011/127802中的SEQ ID NO:2所示的成熟多肽至少80%,例如至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%同一性的成熟多肽序列。
在一个实施例中,该葡糖淀粉酶是WO 2011/127802中披露为SEQ ID NO:2的草酸青霉菌葡糖淀粉酶的变体,具有K79V取代。如WO 2013/036526(将其通过引用特此并入)中披露的,K79V葡糖淀粉酶变体相对于亲本对蛋白酶降解具有降低的敏感度。
在一个实施例中,该葡糖淀粉酶源自草酸青霉菌。
在一个实施例中,该葡糖淀粉酶是WO 2011/127802中披露为SEQ ID NO:2的草酸青霉菌葡糖淀粉酶的变体。在一个实施例中,该草酸青霉菌葡糖淀粉酶是WO 2011/127802中作为SEQ ID NO:2披露的,在位置79处具有Val(V)。
涵盖的草酸青霉菌葡糖淀粉酶变体披露于WO 2013/053801(其通过引用由此并入)中。
在一个实施例中,这些变体对蛋白酶降解具有降低的敏感度。
在一个实施例中,这些变体与亲本相比具有改善的热稳定性。
在一个实施例中,该葡糖淀粉酶具有对应于PE001变体的K79V取代(使用WO 2011/127802的SEQ ID NO:2进行编号),并且进一步包含以下改变之一或改变的组合:
T65A;Q327F;E501V;Y504T;Y504*;T65A+Q327F;T65A+E501V;T65A+Y504T;T65A+Y504*;Q327F+E501V;Q327F+Y504T;Q327F+Y504*;E501V+Y504T;E501V+Y504*;T65A+Q327F+E501V;T65A+Q327F+Y504T;T65A+E501V+Y504T;Q327F+E501V+Y504T;T65A+Q327F+Y504*;T65A+E501V+Y504*;Q327F+E501V+Y504*;T65A+Q327F+E501V+Y504T;T65A+Q327F+E501V+Y504*;E501V+Y504T;T65A+K161S;T65A+Q405T;T65A+Q327W;T65A+Q327F;T65A+Q327Y;P11F+T65A+Q327F;R1K+D3W+K5Q+G7V+N8S+T10K+P11S+T65A+Q327F;P2N+P4S+P11F+T65A+Q327F;P11F+D26C+K33C+T65A+Q327F;P2N+P4S+P11F+T65A+Q327W+E501V+Y504T;R1E+D3N+P4G+G6R+G7A+N8A+T10D+P11D+T65A+Q327F;P11F+T65A+Q327W;P2N+P4S+P11F+T65A+Q327F+E501V+Y504T;P11F+T65A+Q327W+E501V+Y504T;T65A+Q327F+E501V+Y504T;T65A+S105P+Q327W;T65A+S105P+Q327F;T65A+Q327W+S364P;T65A+Q327F+S364P;T65A+S103N+Q327F;P2N+P4S+P11F+K34Y+T65A+Q327F;P2N+P4S+P11F+T65A+Q327F+D445N+V447S;P2N+P4S+P11F+T65A+I172V+Q327F;P2N+P4S+P11F+T65A+Q327F+N502*;P2N+P4S+P11F+T65A+Q327F+N502T+P563S+K571E;P2N+P4S+P11F+R31S+K33V+T65A+Q327F+N564D+K571S;P2N+P4S+P11F+T65A+Q327F+S377T;P2N+P4S+P11F+T65A+V325T+Q327W;P2N+P4S+P11F+T65A+Q327F+D445N+V447S+E501V+Y504T;P2N+P4S+P11F+T65A+I172V+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+S377T+E501V+Y504T;P2N+P4S+P11F+D26N+K34Y+T65A+Q327F;P2N+P4S+P11F+T65A+Q327F+I375A+E501V+Y504T;P2N+P4S+P11F+T65A+K218A+K221D+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+S103N+Q327F+E501V+Y504T;P2N+P4S+T10D+T65A+Q327F+E501V+Y504T;P2N+P4S+F12Y+T65A+Q327F+E501V+Y504T;K5A+P11F+T65A+Q327F+E501V+Y504T;P2N+P4S+T10E+E18N+T65A+Q327F+E501V+Y504T;P2N+T10E+E18N+T65A+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+E501V+Y504T+T568N;P2N+P4S+P11F+T65A+Q327F+E501V+Y504T+K524T+G526A;P2N+P4S+P11F+K34Y+T65A+Q327F+D445N+V447S+E501V+Y504T;P2N+P4S+P11F+R31S+K33V+T65A+Q327F+D445N+V447S+E501V+Y504T;P2N+P4S+P11F+D26N+K34Y+T65A+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+F80*+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+K112S+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+E501V+Y504T+T516P+K524T+G526A;P2N+P4S+P11F+T65A+Q327F+E501V+N502T+Y504*;P2N+P4S+P11F+T65A+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+S103N+Q327F+E501V+Y504T;K5A+P11F+T65A+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+E501V+Y504T+T516P+K524T+G526A;P2N+P4S+P11F+T65A+V79A+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+V79G+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+V79I+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+V79L+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+V79S+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+L72V+Q327F+E501V+Y504T;S255N+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+E74N+V79K+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+G220N+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Y245N+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Q253N+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+D279N+Q327F+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+S359N+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+D370N+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+V460S+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+V460T+P468T+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+T463N+E501V+Y504T;P2N+P4S+P11F+T65A+Q327F+S465N+E501V+Y504T;和P2N+P4S+P11F+T65A+Q327F+T477N+E501V+Y504T。
在一个实施例中,该草酸青霉菌葡糖淀粉酶变体具有对应于PE001变体的K79V取代(使用WO 2011/127802的SEQ ID NO:2进行编号),并且进一步包含以下取代或取代的组合中的一个:
P11F+T65A+Q327F;
P2N+P4S+P11F+T65A+Q327F;
P11F+D26C+K33C+T65A+Q327F;
P2N+P4S+P11F+T65A+Q327W+E501V+Y504T;
P2N+P4S+P11F+T65A+Q327F+E501V+Y504T;以及
P11F+T65A+Q327W+E501V+Y504T。
考虑与本发明一起使用的另外的葡糖淀粉酶可以在WO2011/153516(将其内容并入本文)中找到。
编码适合的葡糖淀粉酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(www.uniprot.org)内可容易获得的那些。
葡糖淀粉酶编码序列也可用于设计核酸探针以鉴定和克隆编码来自不同属或种的菌株的葡糖淀粉酶的DNA,如上文所述的。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码葡糖淀粉酶的多核苷酸,如上文所述的。
用于分离或克隆编码葡糖淀粉酶的多核苷酸的技术在上文中描述。
在一个实施例中,葡糖淀粉酶具有成熟多肽序列,其包含本文描述或参照的葡糖淀粉酶中任一种(例如,SEQ ID NO:8、102-113、229和230中任一个)的氨基酸序列或由其组成。在另一个实施例中,葡糖淀粉酶具有成熟多肽序列,其是本文描述或参照的葡糖淀粉酶中任一种(例如,SEQ ID NO:8、102-113、229和230中任一个)的片段。在一个实施例中,片段中氨基酸残基的数目是参照的全长葡糖淀粉酶(例如SEQ ID NO:8、102-113、229和230中任一个)中氨基酸残基数目的至少75%,例如至少80%、85%、90%或95%。在其他实施例中,葡糖淀粉酶可以包含本文描述或参照的任何葡糖淀粉酶的催化结构域(例如,SEQ ID NO:8、102-113、229和230中任一个的催化结构域)。
葡糖淀粉酶可以是上述葡糖淀粉酶中任一种(例如,SEQ ID NO:8、102-113、229和230中任一个)的变体。在一个实施例中,葡糖淀粉酶具有与上述葡糖淀粉酶中任一种(例如,SEQ ID NO:8、102-113、229和230中任一个)具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在一个实施例中,葡糖淀粉酶具有与上述葡糖淀粉酶中任一种(例如,SEQ ID NO:8、102-113、229和230中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在一个实施例中,葡糖淀粉酶具有上述葡糖淀粉酶中任一种(例如,SEQ ID NO:8、102-113、229和230中任一个)的氨基酸序列的一个或多个(例如,两个,若干个)的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,葡糖淀粉酶在相同条件下具有本文描述或参照的任一种葡糖淀粉酶(例如,SEQ ID NO:8、102-113、229和230中任一个)的葡糖淀粉酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,葡糖淀粉酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任一种葡糖淀粉酶(例如,SEQ ID NO:8、102-113、229和230中任一个)的编码序列的全长互补链杂交。在一个实施例中,葡糖淀粉酶编码序列与来自本文描述或参照的任一种葡糖淀粉酶(例如,SEQ ID NO:8、102-113、229和230中任一个)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,葡糖淀粉酶包含本文描述或参照的任一种葡糖淀粉酶(SEQ IDNO:8、102-113、229和230中任一个)的编码序列。在一个实施例中,葡糖淀粉酶包含编码序列,该编码序列是来自本文描述或参照的任一种葡糖淀粉酶的编码序列的子序列,其中该子序列编码具有葡糖淀粉酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
本文所述的任何相关方面或实施例的参照编码序列可以是天然编码序列或简并序列,例如设计用于特定宿主细胞的密码子优化的(例如,针对在酿酒酵母中表达而优化的)编码序列。
该葡糖淀粉酶还可以包括融合多肽或可切割的融合多肽,如上文所述的。
表达的蛋白酶和/或外源蛋白酶可以是适用于本文所述的发酵生物和/或其使用方法的任何蛋白酶,如天然存在的蛋白酶或其保留蛋白酶活性的变体。对于本发明涉及蛋白酶的外源添加的方面,还考虑了由以下描述的发酵生物表达的任何蛋白酶。
蛋白酶根据其催化机制分为以下几组:丝氨酸蛋白酶(S)、半胱氨酸蛋白酶(C)、天冬氨酸蛋白酶(A)、金属蛋白酶(M)以及未知或还未分类的蛋白酶(U),参见Handbook ofProteolytic Enzymes[蛋白水解酶手册],A.J.Barrett,N.D.Rawlings,J.F.Woessner(编辑),Academic Press[学术出版社](1998),特别是概述部分。
可以使用任何适合的测定来测量蛋白酶活性,其中采用一种底物,该底物包括与所讨论的蛋白酶的特异性相关的肽键。测定pH值和测定温度同样适用于所讨论的蛋白酶。测定pH值的实例是pH 6、7、8、9、10或11。测定温度的实例是30℃、35℃、37℃、40℃、45℃、50℃、55℃、60℃、65℃、70℃或80℃。
在一些方面,当在相同条件下培养时,与不具有编码蛋白酶的异源多核苷酸的发酵生物相比,包含编码蛋白酶的异源多核苷酸的发酵生物具有增加的蛋白酶活性水平。在一些方面,当在相同条件下培养时,与不具有编码蛋白酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的蛋白酶活性水平。
可以用发酵生物表达并用于本文所述方法的示例性蛋白酶包括但不限于表4所示的蛋白酶(或其衍生物)。
表4.
编码适合的蛋白酶的另外的多核苷酸可以衍生自任何适合属的微生物,包括在UniProtKB数据库(
在一个实施例中,该蛋白酶衍生自曲霉属,如SEQ ID NO:9的黑曲霉蛋白酶、SEQID NO:41的溜曲霉蛋白酶、或SEQ ID NO:45的齿状曲霉蛋白酶。在一个实施例中,该蛋白酶衍生自叉丝孔菌属,如SEQ ID NO:12的污叉丝孔菌蛋白酶。在一个实施例中,该蛋白酶衍生自青霉属,如SEQ ID NO:14的简青霉蛋白酶、SEQ ID NO:66的南极青霉蛋白酶、或SEQ IDNO:67的苏门答腊青霉蛋白酶。在一方面,该蛋白酶衍生自亚灰树花菌属,如SEQ ID NO:16的大型亚灰树花菌蛋白酶。在一方面,该蛋白酶衍生自篮状菌属,如SEQ ID NO:21的利亚尼篮状菌蛋白酶。在一方面,该蛋白酶衍生自嗜热子囊菌属,如SEQ ID NO:22的嗜热嗜热子囊菌蛋白酶。在一方面,该蛋白酶衍生自灵芝属,如SEQ ID NO:33的灵芝蛋白酶。在一方面,该蛋白酶衍生自半内果菌属,如SEQ ID NO:61的栖土半内果菌蛋白酶。在一方面,该蛋白酶衍生自木霉属,如SEQ ID NO:69的脐孢木霉蛋白酶。
蛋白酶编码序列也可用于设计核酸探针以从不同属或种的菌株鉴定和克隆编码蛋白酶的DNA,如上所述。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码蛋白酶的多核苷酸,如上文所述。
用于分离或克隆编码蛋白酶的多核苷酸的技术在上文中描述。
在一个实施例中,蛋白酶具有成熟多肽序列,其包含SEQ ID NO:9-73中任一个(例如,SEQ ID NO:9、14、16、21、22、33、41、45、61、62、66、67、和69中任一个;例如SEQ NO:9、14、16、和69中任一个)的氨基酸序列或由其组成。在另一个实施例中,该蛋白酶具有成熟多肽序列,该成熟多肽序列是SEQ ID NO:9-73中任一个的蛋白酶的片段(例如,其中该片段具有蛋白酶活性)。在一个实施例中,片段中的氨基酸残基数目是参照全长蛋白酶(例如,SEQ IDNO:9-73中任一个)中氨基酸残基数目的至少75%,例如至少80%、85%、90%或95%。在其他实施例中,该蛋白酶可包含本文描述或参照的任何蛋白酶的催化结构域(例如,SEQ IDNO:9-73中任一个的催化结构域)。
该蛋白酶可以是上述蛋白酶中任一种(例如,SEQ ID NO:9-73中任一个)的变体。在一个实施例中,该蛋白酶具有与上述蛋白酶中任一种(例如,SEQ ID NO:9-73中任一个)至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在一个实施例中,该蛋白酶具有与上述蛋白酶中任一种(例如,SEQ ID NO:9-73中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在一个实施例中,该蛋白酶具有上述蛋白酶中任一种(例如,SEQ ID NO:9-73中任一个)的氨基酸序列的一个或多个(例如,两个,若干个)的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一个实施例中,蛋白酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任一种蛋白酶(例如,SEQ ID NO:9-73中任一个)的编码序列的全长互补链杂交。在一个实施例中,蛋白酶编码序列与来自本文描述或参照的任一种蛋白酶(例如,SEQ ID NO:9-73中任一个)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,蛋白酶包含本文描述或参照的任一种蛋白酶(SEQ ID NO:9-73中任一个)的编码序列。在一个实施例中,蛋白酶包含编码序列,该编码序列是来自本文描述或参照的任何蛋白酶的编码序列的子序列,其中该子序列编码具有蛋白酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
本文所述的任何相关方面或实施例的参照编码序列可以是天然编码序列或简并序列,例如设计用于特定宿主细胞的密码子优化的(例如,针对在酿酒酵母中表达而优化的)编码序列。
该蛋白酶还可以包括融合多肽或可切割的融合多肽,如上文所述。
在一个实施例中,根据本文所述的方法使用的蛋白酶是丝氨酸蛋白酶。在一个具体的实施例中,该蛋白酶是属于家族53的丝氨酸蛋白酶,例如内切蛋白酶,如来自大型亚灰树花菌、污叉丝孔菌、变色栓菌、漏斗多孔菌、桦革褶菌、灵芝、洁丽香菇、或芽孢杆菌属物种19138的S53蛋白酶,在从含淀粉材料生产乙醇的方法中,当糊化的或未糊化的淀粉的糖化和/或发酵期间存在/或添加该S53蛋白酶时,乙醇产率得到提高。在一个实施例中,该蛋白酶选自:(a)属于EC3.4.21酶组的蛋白酶;和/或(b)属于EC 3.4.14酶组的蛋白酶;和/或(c)肽酶S53家族的丝氨酸蛋白酶,其包含两种不同类型的肽酶:三肽基氨肽酶(外切型)和内切肽酶;如在1993,Biochem.J.[生物化学杂志]290:205-218和在MEROPS蛋白酶数据库,发行9.4(2011年1月31日)(www.merops.ac.uk)中所述的。该数据库描述于Rawlings,N.D.,Barrett,A.J.和Bateman,A.,2010,“MEROPS:the peptidase database[MEROPS:肽酶数据库]”,Nucl.Acids Res.[核酸研究]38:D227-D233中。
为了确定给定的蛋白酶是否为丝氨酸蛋白酶和S53家族蛋白酶,参照上述手册以及其中指示的原则。可以对所有类型的蛋白酶进行这样的确定,而不论其是天然存在的或野生型蛋白酶;还是基因工程化的或合成的蛋白酶。
肽酶S53家族含有酸起作用的内肽酶和三肽基-肽酶。催化三联体的残基是Glu、Asp、Ser,并且在氧阴离子穴中存在另外的酸性残基Asp。残基的顺序是Glu、Asp、Asp、Ser。该Ser残基在枯草杆菌蛋白酶的Asp、His、Ser三联体中是等同于Ser的亲核体,并且该三联体的Glu是枯草杆菌蛋白酶中的广义碱基His的代替物。
该S53家族的肽酶倾向于在酸性pH下最有活性(不像同源的枯草杆菌蛋白酶),并且这可归因于羧基残基(尤其是Asp)在氧阴离子穴中的功能重要性。这些氨基酸序列不与S8家族(即丝氨酸内肽酶枯草杆菌蛋白酶和同系物)中的那些密切相似,并且这一点与完全不同的活性位点残基以及关于最大活性的所得的较低的pH一起,为该家族提供了实质性差异。肽酶单元的蛋白质折叠对于这个家族的成员来说与枯草杆菌蛋白酶的类似,具有宗族类型SB。
在一个实施例中,根据本文所述的方法使用的蛋白酶是半胱氨酸蛋白酶。
在一个实施例中,根据本文所述的方法使用的蛋白酶是天冬氨酸蛋白酶。天冬氨酸蛋白酶描述在例如Hand-book of Proteolytic En-zymes[蛋白水解酶手册],A.J.Barrett,N.D.Rawlings和J.F.Woessner编辑,美国学术出版社,圣迭戈,1998,第270章中。天冬氨酸蛋白酶的合适实例包括,例如,R.M.Berka等人Gene[基因],96,313(1990));(R.M.Berka等人Gene[基因],125,195-198(1993));和Gomi等人Biosci.Biotech.Biochem.[生物科学生物技术生物化学]57,1095-1100(1993)中披露的那些,这些文献通过引用并入文中。
该蛋白酶还可以是金属蛋白酶,将其定义为选自下组的蛋白酶,该组由以下组成:
(a)属于EC 3.4.24的蛋白酶(金属内肽酶);优选EC 3.4.24.39(酸性金属蛋白酶);
(b)属于上述手册的M组的金属蛋白酶;
(c)尚未指定宗族的金属蛋白酶(指定:宗族MX),或属于宗族MA、MB、MC、MD、ME、MF、MG、MH中任一种的金属蛋白酶(如上述手册的第989-991页所定义的);
(d)其他家族的金属蛋白酶(如上述手册的第1448-1452页所定义的);
(e)具有HEXXH基序的金属蛋白酶;
(f)具有HEFTH基序的金属蛋白酶;
(g)属于家族M3、M26、M27、M32、M34、M35、M36、M41、M43或M47中任一种的金属蛋白酶(如上述手册的第1448-1452页所定义的);
(h)属于M28E家族的金属蛋白酶;以及
(i)属于家族M35的金属蛋白酶(如上述手册的第1492-1495页所定义的)。
在其他具体实施例中,金属蛋白酶是其中肽键上的亲核攻击由被二价金属阳离子活化的水分子介导的水解酶。二价阳离子的实例是锌、钴或锰。可以通过氨基酸配体将金属离子保持在适当位置。配体的数目可以是五、四、三、二、一或零。在具体实施例中,数目是二或三,优选是三。
对于在本发明的方法中使用的金属蛋白酶的起源没有限制。在一个实施例中,将该金属蛋白酶分类为EC 3.4.24、优选EC 3.4.24.39。在一个实施例中,该金属蛋白酶是酸稳定的金属蛋白酶,例如真菌酸稳定的金属蛋白酶,如衍生自嗜热子嚢菌属的菌株,优选橙色嗜热子囊菌的菌株,尤其是橙色嗜热子囊菌CGMCC No.0670(分类为EC3.4.24.39)的金属蛋白酶。在另一个实施例中,该金属蛋白酶衍生自曲霉属的菌株,优选米曲霉的菌株。
在一个实施例中,该金属蛋白酶与WO 2010/008841的SEQ ID NO:1(一种金黄色嗜热子囊菌金属蛋白酶)的氨基酸-178至177、-159至177,或优选氨基酸1至177(成熟多肽)具有至少80%、至少82%、至少85%、至少90%、至少95%、或至少97%的序列同一性程度;并且该金属蛋白酶具有金属蛋白酶活性。在具体实施例中,该金属蛋白酶由与上述SEQ IDNO:1具有一定同一性程度的氨基酸序列组成。
橙色嗜热子囊菌金属蛋白酶是适于在本发明的方法中使用的金属蛋白酶的优选实例。另一种金属蛋白酶来源于米曲霉并且包括披露于WO 2003/048353中的SEQ ID NO:11的序列,或其氨基酸-23-353;-23-374;-23-397;1-353;1-374;1-397;177-353;177-374;或177-397,以及披露于WO 2003/048353中的SEQ ID NO:10。
另一种适于在本发明的方法中使用的金属蛋白酶是包括WO 2010/008841的SEQID NO:5的米曲霉金属蛋白酶,或金属蛋白酶是与SEQ ID NO:5具有至少约80%、至少82%、至少85%、至少90%、至少95%或至少97%同一性程度的分离多肽;并且该金属蛋白酶具有金属蛋白酶活性。在具体实施例中,该金属蛋白酶由WO 2010/008841的SEQ ID NO:5的氨基酸序列组成。
在具体实施例中,金属蛋白酶具有如下氨基酸序列,该氨基酸序列与金黄色嗜热子囊菌或米曲霉金属蛋白酶的氨基酸序列的氨基酸-178至177、-159至177或+1至177相差四十个、三十五个、三十个、二十五个、二十个或相差十五个氨基酸。
在另一个实施例中,金属蛋白酶具有如下氨基酸序列,该氨基酸序列与这些金属蛋白酶的氨基酸序列的氨基酸-178至177、-159至177或+1至177相差十个、或相差九个、或相差八个、或相差七个、或相差六个、或相差五个氨基酸,例如,相差四个、相差三个、相差两个、或相差一个氨基酸。
在具体实施例中,该金属蛋白酶a)包含以下或者b)由以下组成:
i)WO 2010/008841的SEQ ID NO:1的氨基酸-178至177、-159至177或+1至177的氨基酸序列;
ii)WO 2010/008841的SEQ ID NO:3的氨基酸-23-353、-23-374、-23-397、1-353、1-374、1-397、177-353、177-374、或177-397的氨基酸序列;
iii)WO 2010/008841的SEQ ID NO:5的氨基酸序列;或
i)、ii)和iii)的具有蛋白酶活性的序列的等位基因变体或片段。
WO 2010/008841的SEQ ID NO:1的氨基酸-178至177、-159至177、或+1至177或WO2010/008841的SEQ ID NO:3的氨基酸-23-353、-23-374、-23-397、1-353、1-374、1-397、177-353、177-374、或177-397的片段;是在这些氨基酸序列的氨基和/或羧基端缺失了一个或多个氨基酸的多肽。在一个实施例中,片段含有至少75个氨基酸残基、或至少100个氨基酸残基、或至少125个氨基酸残基、或至少150个氨基酸残基、或至少160个氨基酸残基、或至少165个氨基酸残基、或至少170个氨基酸残基、或至少175个氨基酸残基。
为了确定给定的蛋白酶是否是金属蛋白酶,参照上述“Handbook of ProteolyticEnzymes[蛋白水解酶手册]”以及其中指示的原则。可以对所有类型的蛋白酶进行这样的确定,而不论其是天然存在的或野生型蛋白酶;还是基因工程化的或合成的蛋白酶。
该蛋白酶可以是例如野生型蛋白酶的变体,该蛋白酶具有本文定义的热稳定性特性。在一个实施例中,该热稳定蛋白酶是金属蛋白酶的变体。在一个实施例中,在本文所述的方法中使用的热稳定蛋白酶是真菌来源的,如真菌金属蛋白酶,如源自嗜热子囊菌属的菌株、优选金黄色嗜热子囊菌的菌株、尤其是金黄色嗜热子囊菌CGMCC No.0670(分类为EC3.4.24.39)的真菌金属蛋白酶。
在一个实施例中,该热稳定蛋白酶是披露于以下的变体:WO 2003/048353中披露的SEQ ID NO:2所示的金属蛋白酶的成熟部分或WO 2010/008841中的SEQ ID NO:1的成熟部分,该变体进一步具有以下取代或取代的组合中的一个:
S5*+D79L+S87P+A112P+D142L;
D79L+S87P+A112P+T124V+D142L;
S5*+N26R+D79L+S87P+A112P+D142L;
N26R+T46R+D79L+S87P+A112P+D142L;
T46R+D79L+S87P+T116V+D142L;
D79L+P81R+S87P+A112P+D142L;
A27K+D79L+S87P+A112P+T124V+D142L;
D79L+Y82F+S87P+A112P+T124V+D142L;
D79L+Y82F+S87P+A112P+T124V+D142L;
D79L+S87P+A112P+T124V+A126V+D142L;
D79L+S87P+A112P+D142L;
D79L+Y82F+S87P+A112P+D142L;
S38T+D79L+S87P+A112P+A126V+D142L;
D79L+Y82F+S87P+A112P+A126V+D142L;
A27K+D79L+S87P+A112P+A126V+D142L;
D79L+S87P+N98C+A112P+G135C+D142L;
D79L+S87P+A112P+D142L+T141C+M161C;
S36P+D79L+S87P+A112P+D142L;
A37P+D79L+S87P+A112P+D142L;
S49P+D79L+S87P+A112P+D142L;
S50P+D79L+S87P+A112P+D142L;
D79L+S87P+D104P+A112P+D142L;
D79L+Y82F+S87G+A112P+D142L;
S70V+D79L+Y82F+S87G+Y97W+A112P+D142L;
D79L+Y82F+S87G+Y97W+D104P+A112P+D142L;
S70V+D79L+Y82F+S87G+A112P+D142L;
D79L+Y82F+S87G+D104P+A112P+D142L;
D79L+Y82F+S87G+A112P+A126V+D142L;
Y82F+S87G+S70V+D79L+D104P+A112P+D142L;
Y82F+S87G+D79L+D104P+A112P+A126V+D142L;
A27K+D79L+Y82F+S87G+D104P+A112P+A126V+D142L;
A27K+Y82F+S87G+D104P+A112P+A126V+D142L;
A27K+D79L+Y82F+D104P+A112P+A126V+D142L;
A27K+Y82F+D104P+A112P+A126V+D142L;
A27K+D79L+S87P+A112P+D142L;以及
D79L+S87P+D142L。
在一个实施例中,该热稳定蛋白酶是披露为以下的金属蛋白酶的变体:WO 2003/048353中披露的SEQ ID NO:2的成熟部分或WO2010/008841中SEQ ID NO:1的成熟部分,该变体具有以下取代或取代的组合中的一个:
D79L+S87P+A112P+D142L;
D79L+S87P+D142L;以及
A27K+D79L+Y82F+S87G+D104P+A112P+A126V+D142L。
在一个实施例中,该蛋白酶变体与披露于WO 2003/048353中的SEQ ID NO:2的多肽的成熟部分或披露于WO 2010/008841中的SEQ ID NO:1的成熟部分具有至少75%同一性,优选地至少80%、更优选地至少85%、更优选地至少90%、更优选地至少91%、更优选地至少92%、甚至更优选地至少93%、最优选地至少94%、以及甚至最优选地至少95%、如甚至至少96%、至少97%、至少98%、至少99%,但小于100%同一性。
该热稳定蛋白酶还可以源自任何细菌,只要该蛋白酶具有热稳定性特性。
在一个实施例中,该热稳定蛋白酶源自细菌火球菌属的菌株,如强烈火球菌的菌株(pfu蛋白酶)。
在一个实施例中,该蛋白酶是如美国专利号6,358,726-B1(宝酒造公司(TakaraShuzo Company))的SEQ ID NO:1所示的一种。
在一个实施例中,该热稳定蛋白酶是如下蛋白酶,该蛋白酶具有与美国专利号6,358,726-B1中的SEQ ID NO:1至少80%同一性、如至少85%、如至少90%、如至少95%、如至少96%、如至少97%、如至少98%、如至少99%同一性的成熟多肽序列。强烈火球菌蛋白酶可以购买自日本宝酒造生物公司(Takara Bio,Japan)。
该强烈火球菌蛋白酶可以是热稳定蛋白酶,如WO 2018/098381的SEQ ID NO:13中所述。发现此蛋白酶(PfuS)在确定的pH 4.5下具有110%(80℃/70℃)和103%(90℃/70℃)的热稳定性。
在一个实施例中,在本文所述的方法中使用的热稳定性蛋白酶具有确定为在80℃/70℃下的相对活性的超过20%的热稳定性值,如WO 2018/098381的实例2中描述所确定的。
在一个实施例中,该蛋白酶具有确定为在80℃/70℃下的相对活性的超过30%、超过40%、超过50%、超过60%、超过70%、超过80%、超过90%、超过100%,如超过105%,如超过110%,如超过115%,如超过120%的热稳定性。
在一个实施例中,该蛋白酶具有确定为在80℃/70℃下的相对活性的在20%与50%之间,如在20%与40%之间,如20%与30%之间的热稳定性。在一个实施例中,该蛋白酶具有确定为在80℃/70℃下的相对活性的在50%与115%之间,如在50%与70%之间,如在50%与60%之间,如在100%与120%之间,如在105%与115%之间的热稳定性。
在一个实施例中,该蛋白酶具有确定为在85℃/70℃下的相对活性的超过10%的热稳定性值,如WO 2018/098381的实例2中描述所确定的。
在一个实施例中,该蛋白酶具有确定为在85℃/70℃下的相对活性的超过10%,如超过12%、超过14%、超过16%、超过18%、超过20%、超过30%、超过40%、超过50%、超过60%、超过70%、超过80%、超过90%、超过100%、超过110%的热稳定性。
在一个实施例中,该蛋白酶具有确定为在85℃/70℃下的相对活性的在10%与50%之间,如在10%与30%之间,如在10%与25%之间的热稳定性。
在一个实施例中,该蛋白酶具有确定为在80℃下的残余活性的超过20%、超过30%、超过40%、超过50%、超过60%、超过70%、超过80%、超过90%;和/或该蛋白酶具有确定为在84℃下的残余活性的超过20%、超过30%、超过40%、超过50%、超过60%、超过70%、超过80%、超过90%。
“相对活性”以及“残余活性”的测定是如在WO 2018/098381的实例2中所述进行的。
在一个实施例中,蛋白酶在85℃下可以具有高于90,例如高于100的热稳定性,如使用WO 2018/098381的实例3中披露的Zein-BCA测定法所确定的。
在一个实施例中,蛋白酶在85℃下具有高于60%,例如高于90%,例如高于100%,例如高于110%的热稳定性,如使用WO 2018/098381的Zein-BCA测定法所确定的。
在一个实施例中,蛋白酶在85℃下具有在60%-120%之间,例如在70%-120%之间,例如在80%-120%之间,例如在90%-120%之间,例如在100%-120%之间,例如110%-120%的热稳定性,如使用WO 2018/098381的Zein-BCA测定法确定的。
在一个实施例中,通过WO 2018/098381和本文所述的AZCL-酪蛋白测定确定的,该热稳定蛋白酶具有JTP196蛋白酶变体或蛋白酶Pfu的活性的至少20%,如至少30%、如至少40%、如至少50%、如至少60%、如至少70%、如至少80%、如至少90%、如至少95%、如至少100%。
在一个实施例中,通过WO 2018/098381和本文所述的AZCL-酪蛋白测定确定的,该热稳定蛋白酶具有蛋白酶196变体或蛋白酶Pfu的蛋白酶活性的至少20%,如至少30%、如至少40%、如至少50%、如至少60%、如至少70%、如至少80%、如至少90%、如至少95%、如至少100%。
在一些实施例中,支链淀粉酶是在液化步骤和/或糖化步骤或同时糖化和发酵(SSF)中存在的和/或添加的。
普鲁兰酶(E.C.3.2.1.41,普鲁兰糖6-葡聚糖-水解酶)是去分支酶,其特征在于它们在例如支链淀粉和普鲁兰糖中水解α-1,6-糖苷键的能力。
在一些实施例中,该发酵生物包含编码支链淀粉酶的异源多核苷酸。考虑本文描述或参照的任何支链淀粉酶用于在发酵生物中表达。
该支链淀粉酶可以是适合宿主细胞和/或本文所述的方法的任何支链淀粉酶,如天然存在的支链淀粉酶或其保留支链淀粉酶活性的变体。
在一些实施例中,当在相同条件下培养时,与不具有编码支链淀粉酶的异源多核苷酸的宿主细胞相比,包含编码该支链淀粉酶的异源多核苷酸的发酵生物具有增加的支链淀粉酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码支链淀粉酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的支链淀粉酶活性水平。
可以与本文所述的宿主细胞和/或方法一起使用的示例性支链淀粉酶包括细菌、酵母或丝状真菌支链淀粉酶,例如,获得自本文描述或参照的任何微生物,如上文在与α-淀粉酶有关的部分下描述的。
考虑的支链淀粉酶包括来自美国专利号4,560,651(通过引用特此并入)中披露的淀粉脱支芽孢杆菌(Bacillus amyloderamificans)的支链淀粉酶、WO 01/151620(通过引用特此并入)中披露为SEQ ID NO:2的支链淀粉酶、WO 01/151620(通过引用特此并入)中披露为SEQ ID NO:4的脱支芽孢杆菌(Bacillus deramificans)的支链淀粉酶,以及来自WO01/151620(通过引用特此并入)中披露为SEQ ID NO:6的嗜酸性支链淀粉芽孢杆菌(Bacillus acidopullulyticus)的支链淀粉酶,以及还有描述于FEMS Mic.Let.[FEMS微生物学通讯](1994)115,97-106中的支链淀粉酶。
考虑的另外的支链淀粉酶包括来自沃斯氏火球菌(Pyrococcus woesei)、特别是来自WO 92/02614中披露的沃斯氏火球菌DSM号3773的支链淀粉酶。
在一个实施例中,该支链淀粉酶是GH57家族支链淀粉酶。在一个实施例中,该支链淀粉酶包括X47结构域,如披露于作为WO2011/087836公开的US 61/289,040(将其通过引用特此并入)中。更具体地,该支链淀粉酶可以衍生自热球菌属的菌株,包括嗜热高温球菌(Thermococcus litoralis)和热水高温球菌(Thermococcus hydrothermalis),如热水高温球菌支链淀粉酶刚好在X47结构域之后的X4位点截短(即,氨基酸1-782)。该支链淀粉酶还可以是嗜热高温球菌和热水高温球菌支链淀粉酶的杂合体或具有截短位点X4的、披露于作为WO 2011/087836公开的US 61/289,040(将其通过引用特此并入)中的热水高温球菌/嗜热高温球菌杂合酶。
在另一个实施例中,该支链淀粉酶是包含披露于WO 2011/076123(诺维信公司)中的X46结构域的支链淀粉酶。
该支链淀粉酶能以有效量添加,该有效量包括约0.0001-10mg酶蛋白/克DS的优选量,优选0.0001-0.10mg酶蛋白/克DS,更优选0.0001-0.010mg酶蛋白/克DS。支链淀粉酶活性可以确定为NPUN。用于确定NPUN的测定描述于WO 2018/098381中。
适合的可商购支链淀粉酶产品包括PROMOZYME D、PROMOZYME
在一个实施例中,该支链淀粉酶衍生自SEQ ID NO:114的枯草芽孢杆菌支链淀粉酶。在一个实施例中,该支链淀粉酶衍生自SEQ ID NO:115的地衣芽孢杆菌支链淀粉酶。在一个实施例中,该支链淀粉酶衍生自SEQ ID NO:116的水稻支链淀粉酶。在一个实施例中,该支链淀粉酶衍生自SEQ ID NO:117的小麦支链淀粉酶。在一个实施例中,该支链淀粉酶衍生自SEQ ID NO:118的发酵植物多糖梭菌支链淀粉酶。在一个实施例中,该支链淀粉酶衍生自SEQ ID NO:119的除虫链霉菌支链淀粉酶。在一个实施例中,该支链淀粉酶衍生自SEQ IDNO:120的肺炎克雷柏氏菌(Klebsiella pneumoniae)支链淀粉酶。
考虑与本发明一起使用的另外的支链淀粉酶可以在WO 2011/153516(将其内容并入本文)中找到。
编码适合的支链淀粉酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(www.uniprot.org)内可容易获得的那些。
支链淀粉酶编码序列也可用于设计核酸探针以鉴定和克隆编码来自不同属或种的菌株的支链淀粉酶的DNA,如上文所述的。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码支链淀粉酶的多核苷酸,如上文所述的。
用于分离或克隆编码支链淀粉酶的多核苷酸的技术在上文中描述。
在一个实施例中,支链淀粉酶具有成熟多肽序列,其包含本文描述或参照的支链淀粉酶中任一种(例如,SEQ ID NO:114-120中任一个)的氨基酸序列或由其组成。在另一个实施例中,支链淀粉酶具有成熟多肽序列,其是本文描述或参照的支链淀粉酶中任一种(例如,SEQ ID NO:114-120中任一个)的片段。在一个实施例中,片段中氨基酸残基的数目是参照的全长支链淀粉酶中氨基酸残基数目的至少75%,例如至少80%、85%、90%或95%。在其他实施例中,支链淀粉酶可以包含本文描述或参照的任何支链淀粉酶(例如,SEQ ID NO:114-120中任一个)的催化结构域。
支链淀粉酶可以是上述支链淀粉酶中任一种(例如,SEQ ID NO:114-120中任一个)的变体。在一个实施例中,支链淀粉酶具有与上述支链淀粉酶中任一种(例如,SEQ IDNO:114-120中任一个)具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
在一个实施例中,支链淀粉酶具有与上述支链淀粉酶中任一种(例如,SEQ ID NO:114-120中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。在一个实施例中,支链淀粉酶具有上述支链淀粉酶中任一种(例如,SEQID NO:114-120中任一个)的氨基酸序列的一个或多个(例如,两个,若干个)的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,支链淀粉酶在相同条件下具有本文描述或参照的任何支链淀粉酶(例如,SEQ ID NO:114-120中任一个)的支链淀粉酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,支链淀粉酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任何支链淀粉酶(例如,SEQ ID NO:114-120中任一个)的编码序列的全长互补链杂交。在一个实施例中,支链淀粉酶编码序列与来自本文描述或参照的任何支链淀粉酶(例如,SEQ ID NO:114-120中任一个)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,支链淀粉酶包含本文描述或参照的任何支链淀粉酶(例如,SEQID NO:114-120中任一个)的编码序列。在一个实施例中,支链淀粉酶包含编码序列,该编码序列是来自本文描述或参照的任何支链淀粉酶的编码序列的子序列,其中该子序列编码具有支链淀粉酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
本文所述的任何相关方面或实施例的参照编码序列可以是天然编码序列或简并序列,例如设计用于特定宿主细胞的密码子优化的(例如,针对在酿酒酵母中表达而优化的)编码序列。
该支链淀粉酶还可以包括融合多肽或可切割的融合多肽,如上文所述的。
在一些方面,本文所述的方法从含纤维素材料生产发酵产物。生物质的初生细胞壁中的主要多糖是纤维素,第二丰富的是半纤维素,而第三丰富的是果胶。细胞停止生长后产生的次生细胞壁也含有多糖,并且它通过与半纤维素共价交联的聚合木质素得到强化。纤维素是脱水纤维二糖的均聚物,因此是线性β-(1-4)-D-葡聚糖,而半纤维素包括多种化合物,例如具有一系列取代基以复杂支链结构的木聚糖、木葡聚糖、阿拉伯糖基木聚糖、以及甘露聚糖。尽管纤维素一般为多形态的,但发现其在植物组织中主要作为平行葡聚糖链的不溶性晶体基质存在。半纤维素通常氢键结合至纤维素以及其他半纤维素,这有助于稳定细胞壁基质。
纤维素通常见于例如植物的茎、叶、壳、皮和穗轴或树的叶、枝和木材(wood)中。该含纤维素材料可以是,但不限于:农业废弃物、草本材料(包括能量作物)、城市固体废物、纸浆和造纸厂废弃物、废纸、和木材(包括林业废弃物)(参见,例如Wiselogel等人,1995,于Handbook on Bioethanol[生物乙醇手册](Charles E.Wyman编辑),第105-118页,Taylor&Francis[泰勒-弗朗西斯出版集团],华盛顿特区;Wyman,1994,Bioresource Technology[生物资源技术]50:3-16;Lynd,1990,Applied Biochemistry and Biotechnology[应用生物化学与生物技术]24/25:695-719;Mosier等人,1999,Recent Progress inBioconversion of Lignocellulosics[木质纤维素的生物转化的最近进展],Advances inBiochemical Engineering/Biotechnology[生物化学工程/生物技术的进展],T.Scheper主编,第65卷,第23-40页,纽约斯普林格出版社(Springer-Verlag),纽约)。在本文中应理解的是,纤维素可为任何形式的木质纤维素,在混合基质中含有木质素、纤维素和半纤维素的植物细胞壁材料。在一个实施例中,该含纤维素材料是任何生物质材料。在另一个实施例中,该含纤维素材料是木质纤维素,该木质纤维素包含纤维素、半纤维素、以及木质素。
在一个实施例中,该含纤维素材料是农业废弃物、草本材料(包括能源作物)、城市固体废物、纸浆和造纸厂废弃物、废纸、或木材(包括林业废弃物)。
在另一个实施例中,该含纤维素材料是芦竹、甘蔗渣、竹子、玉米芯、玉米纤维、玉米秸秆、芒属、稻秸、柳枝稷或麦秸。
在另一个实施例中,该含纤维素材料是山杨、桉树、冷杉、松树、白杨、云杉或柳树。
在另一个实施例中,该含纤维素材料是海藻纤维素、细菌纤维素、棉短绒、滤纸、微晶纤维素(例如,
在另一个实施例中,该含纤维素材料是水生生物质(aquatic biomass)。如本文所用的,术语“水生生物质”意指在水生环境中通过光合作用过程产生的生物质。水生生物质可以是藻类、挺水植物、浮叶植物、或沉水植物。
该含纤维素材料可按原样使用或可使用本领域已知的常规方法进行预处理,如本文所述的。在一个优选的实施例中,该含纤维素材料进行了预处理。
可以使用本领域常规的方法来完成使用含纤维素材料的方法。此外,这些方法可以使用配置为实施这些方法的任何常规生物质加工装置来执行。
在一个实施例中,在糖化之前对该含纤维素材料进行预处理。
在实践文中所述的工艺中,可以使用本领域中已知的任何预处理工艺来破坏纤含维素材料的植物细胞壁组分(Chandra等人,2007,Adv.Biochem.Engin./Biotechnol.[生化工程/生物技术进展]108:67-93;Galbe和Zacchi,2007,Adv.Biochem.Engin./Biotechnol.[生化工程/生物技术进展]108:41-65;Hendriks和Zeeman,2009,Bioresource Technology[生物资源技术]100:10-18;Mosier等人,2005,Bioresource Technology[生物资源技术]96:673-686;Taherzadeh和Karimi,2008,Int.J.Mol.Sci.[国际分子科学杂志]9:1621-1651;Yang和Wyman,2008,Biofuels Bioproducts and Biorefining-Biofpr.[生物燃料、生物产品与生物精炼]2:26-40)。
该含纤维素材料也可以在预处理之前使用本领域中已知的方法进行粒度减小、筛分、预浸泡、润湿、洗涤和/或调理。
常规预处理包括但不限于:蒸汽预处理(伴随或不伴随爆破)、稀酸预处理、热水预处理、碱预处理、石灰预处理、湿氧化、湿爆破、氨纤维爆破、有机溶剂预处理、以及生物预处理。另外的预处理包括氨渗滤、超声、电穿孔、微波、超临界CO
在一个实施例中,在糖化(即水解)和/或发酵之前对该含纤维素材料进行预处理。预处理优选在水解前进行。可替代地,预处理可以与酶水解同时进行,以释放可发酵糖,如葡萄糖、木糖、和/或纤维二糖。在多数情况下,预处理步骤自身导致将生物质转化为可发酵糖(甚至在不存在酶的情况下)。
在一个实施例中,将该含纤维素材料用蒸汽预处理。在蒸汽预处理中,加热该含纤维素材料以破坏植物细胞壁组分,包括木质素、半纤维素、以及纤维素,以使纤维素和其他级分(例如半纤维素)可接近酶。该含纤维素材料经过或穿过反应容器,将蒸汽注入该反应容器以增加温度至所需温度和压力,并且将蒸汽保持在其中持续希望的反应时间。优选在140℃-250℃(例如,160℃-200℃或170℃-190℃)进行蒸汽预处理,其中最佳温度范围取决于化学催化剂的任选添加。蒸汽预处理的停留时间优选是1-60分钟,例如1-30分钟、1-20分钟、3-12分钟、或4-10分钟,其中最佳停留时间取决于温度和化学催化剂的任选添加。蒸汽预处理允许相对高的固体加载量,这样使得该含纤维素材料在预处理过程中通常仅变得潮湿。蒸汽预处理经常与预处理后的材料的爆发放料(explosive discharge)合并,这被称为蒸汽爆炸,即,快速急骤蒸发至大气压和材料的湍流,以通过破碎增加可接触的表面积(Duff和Murray,1996,Bioresource Technology[生物资源技术]855:1-33;Galbe和Zacchi,2002,Appl.Microbiol.Biotechnol.[应用微生物学与生物技术]59:618-628;美国专利申请号2002/0164730)。在蒸汽预处理过程中,半纤维素乙酰基基团被切割,并且所得的酸自催化半纤维素部分水解成单糖和低聚糖。仅在有限的程度上去除木质素。
在一个实施例中,使该含纤维素材料经受化学预处理。术语“化学处理”是指能促进纤维素、半纤维素和/或木质素分离和/或释放的任何化学预处理。这种预处理可以将结晶纤维素转化为无定形纤维素。适合的化学预处理方法的实例包括例如稀酸预处理、石灰预处理、湿氧化、氨纤维/冷冻爆破(AFEX)、氨渗滤(APR)、离子液体、以及有机溶剂预处理。
有时在蒸汽预处理之前添加化学催化剂(如H
还可以使用碱性条件下的若干种预处理方法。这些碱性预处理包括但不限于:氢氧化钠、石灰、湿氧化、氨渗滤(APR)、以及氨纤维/冷冻爆破(AFEX)预处理。
用氧化钙或氢氧化钙在85-150℃的温度下进行石灰预处理,并且停留时间从1小时到若干天(Wyman等人,2005,Bioresource Technology[生物资源技术]96:1959-1966;Mosier等人,2005,Bioresource Technology[生物资源技术]96:673-686)。WO 2006/110891、WO 2006/110899、WO 2006/110900、和WO 2006/110901披露了使用氨的预处理方法。
湿氧化是一种热预处理,其典型地在添加氧化剂(如过氧化氧或过压氧)的情况下在180-200℃下持续5-15分钟进行(Schmidt和Thomsen,1998,Bioresource Technology[生物资源技术]64:139-151;Palonen等人,2004,Appl.Biochem.Biotechnol.[应用生物化学与生物技术]117:1-17;Varga等人,2004,Biotechnol.Bioeng.[生物技术与生物工程]88:567-574;Martin等人,2006,J.Chem.Technol.Biotechnol.[化工技术与生物技术杂志]81:1669-1677)。优选地在1%-40%干物质(例如2%-30%干物质或5%-20%干物质)下进行预处理,并且通常通过添加碱(如碳酸钠)提高初始pH。
被称为湿爆破(湿氧化和蒸汽爆炸的组合)的湿氧化预处理方法的修改方案能够处理高达30%的干物质。在湿爆破中,在某一停留时间后,在预处理期间引入氧化剂。然后通过闪变至大气压结束预处理(WO 2006/032282)。
氨纤维爆炸(AFEX)涉及在温和温度如90℃-150℃和高压如17-20巴,用液体或气体氨将含纤维素材料处理5-10分钟,其中干物质含量可以高达60%(Gollapalli等人,2002,Appl.Biochem.Biotechnol.[应用生物化学与生物技术]98:23-35;Chundawat等人,2007,Biotechnol.Bioeng.[生物技术与生物工程]96:219-231;Alizadeh等人,2005,Appl.Biochem.Biotechnol.[应用生物化学与生物技术]121:1133-1141;Teymouri等人,2005,Bioresource Technology[生物资源技术]96:2014-2018)。在AFEX预处理期间,纤维素和半纤维素保持相对完整。木质素-碳水化合物复合物被切割。
有机溶剂预处理通过用含水乙醇(40-60%乙醇)在160℃-200℃提取30-60分钟而将含纤维素材料去木质素化(Pan等人,2005,Biotechnol.Bioeng.[生物技术与生物工程]90:473-481;Pan等人,2006,Biotechnol.Bioeng.[生物技术与生物工程]94:851-861;Kurabi等人,2005,Appl.Biochem.Biotechnol.[应用生物化学与生物技术]121:219-230)。通常添加硫酸作为催化剂。在有机溶剂预处理中,大部分半纤维素和木质素被去除。
适合的预处理方法的其他实例由Schell等人,2003,Appl.Biochem.Biotechnol.[应用生物化学与生物技术]105-108:69-85,和Mosier等人,2005,BioresourceTechnology[生物资源技术]96:673-686,以及美国专利申请2002/0164730进行了描述。
在一个实施例中,化学预处理作为稀酸处理,并且更优选作为连续稀酸处理进行。酸典型地是硫酸,但也可以使用其他酸,如乙酸、柠檬酸、硝酸、磷酸、酒石酸、琥珀酸、氯化氢、或其混合物。弱酸处理优选地在1-5(例如,1-4或1-2.5)的pH范围内进行。在一方面,酸浓度优选地在从0.01wt.%至10wt.%酸(例如,0.05wt.%至5wt.%酸或0.1wt.%至2wt.%酸)的范围内。使酸与该含纤维素材料相接触,并且保持在优选地140℃-200℃(例如165℃-190℃)范围内的温度下,持续从1至60分钟范围内的时间。
在另一个实施例中,预处理在水性浆料中进行。在优选方面,在预处理过程中该含纤维素材料以优选在10wt.%-80wt.%之间,例如20wt.%-70wt.%或30wt.%-60wt.%,如约40wt.%的量存在。预处理的含纤维素材料可以不洗涤或使用本领域已知的任何方法洗涤,例如,用水洗涤。
在一个实施例中,使该含纤维素材料经受机械或物理预处理。术语“机械预处理”或“物理预处理”是指促进颗粒粒度减小的任何预处理。例如,这种预处理可以涉及多种类型的研磨或碾磨(例如,干磨、湿磨、或振动球磨)。
该含纤维素材料可以物理地(机械地)且化学地预处理。机械或物理预处理可以与蒸汽/蒸汽爆炸、水热解(hydrothermolysis)、稀酸或弱酸处理、高温、高压处理、辐射(例如,微波福射)或其组合相结合。在一方面,高压意指在优选约100至约400psi(例如约150至约250psi)的范围内的压力。在另一方面,高温意指在约100℃至约300℃(例如约140℃至约200℃)的范围内的温度。在一个优选的方面,机械或物理预处理在分批期间使用蒸汽枪水解器系统,例如从瑞典的顺智公司(Sunds Defibrator AB)可获得的顺智水解器(SundsHydrolyzer)来进行,该系统使用如上所定义的高压和高温。根据需要,可以顺序或同时进行物理和化学预处理。
因此,在一个实施例中,使该含纤维素材料经受物理(机械)或化学预处理、或其任何组合,以促进纤维素、半纤维素和/或木质素的分离和/或释放。
在一个实施例中,使该含纤维素材料经受生物预处理。术语“生物预处理”是指促进纤维素、半纤维素、和/或木质素从该含纤维素材料中分离和/或释放的任何生物预处理。生物预处理技术可以涉及应用溶解木质素的微生物和/或酶(参见例如,Hsu,T.-A.,1996,Pretreatment of biomass[生物质的预处理],Handbook on Bioethanol:Production andUtilization[生物乙醇:生产与利用手册],Wyman,C.E.,编辑,Taylor&Francis,Washington,DC,179-212;Ghosh和Singh,1993,Adv.Appl.Microbiol.[应用微生物学进展]39:295-333;McMillan,J.D.,1994,Pretreating lignocellulosic biomass:a review[预处理木质纤维素生物质:综述],Enzymatic Conversion of Biomass for FuelsProduction[生物质的酶促转化用于燃料生产],Himmel,M.E.,Baker,J.O.,和Overend,R.P.,编著,ACS Symposium Series 566,American Chemical Society,Washington,DC,chapter 15[ACS研讨会系列566,美国化学会,华盛顿特区,第15章];Gong,C.S.,Cao,N.J.,Du,J.以及Tsao,G.T.,1999,Ethanol production from renewable resources[利用可再生资源生产乙醇],于Advances in Biochemical Engineering/Biotechnology[生化工程/生物技术进展],Scheper,T.编,海德堡,德国,65:207-241;Olsson和Hahn-Hagerdal,1996,Enz.Microb.Tech.[酶微生物技术]18:312-331;以及Vallander和Eriksson,1990,Adv.Biochem.Eng./Biotechnol.[生物化学工程/生物技术的进展]42:63-95)。
分开的或同时的糖化(即水解)和发酵包括但不限于:分开的水解和发酵(SHF);同时糖化和发酵(SSF);同时糖化和共发酵(SSCF);混合的水解和发酵(HHF);分离的水解和共发酵(SHCF);杂合的水解和共发酵(HHCF)。
SHF使用分开的处理步骤,以首先将该含纤维素材料酶促水解为可发酵糖(例如,葡萄糖、纤维二糖、以及戊糖单体),并且然后将可发酵糖发酵为乙醇。在SSF中,该含纤维素材料的酶水解和糖发酵成乙醇被组合在一个步骤中(Philippidis,G.P.,1996,Cellulosebioconversion technology[纤维素生物转化技术],于Handbook on Bioethanol:Production and Utilization[生物乙醇手册:生产和利用],Wyman,C.E.编辑,[泰勒-弗朗西斯出版集团],华盛顿特区,179-212)。SSCF涉及多种糖的共发酵(Sheehan和Himmel,1999,Biotechnol.Prog.[生物技术进展]15:817-827)。HHF涉及分离的水解步骤并且另外涉及同时糖化和水解步骤,其可在同一反应器中进行。HHF过程中的步骤可以在不同的温度下进行,即高温酶糖化,随后在发酵生物耐受的更低温度下进行SSF。本文应理解的是,本领域中已知的包含预处理、酶水解(糖化)、发酵、或其组合的任何方法,可以用于实施本文所述的方法。
常规的装置可以包括分批补料搅拌反应器、批式搅拌反应器、具有超滤作用的连续流搅拌反应器、和/或连续活塞流柱式反应器(de Castilhos Corazza等人,2003,ActaScientiarum.Technology[技术学报]25:33-38;Gusakov和Sinitsyn,1985,Enz.Microb.Technol.[酶与微生物技术]7:346-352)、磨耗反应器(Ryu和Lee,1983,Biotechnol.Bioeng.[生物技术与生物工程]25:53-65)。另外的反应器类型包括:用于水解和/或发酵的流化床、升流式(upflow blanket)反应器、固定化反应器、以及挤出机型反应器。
在糖化步骤(即,水解步骤)中,该含纤维素材料和/或含淀粉材料(例如预处理的)被水解,来分解纤维素、半纤维素和/或淀粉为可发酵糖,如葡萄糖、纤维二糖、木糖、木酮糖、阿拉伯糖、甘露糖、半乳糖、和/或可溶性低聚糖。水解由例如纤维素分解酶组合物酶促进行。这些组合物的酶可以同时或顺序添加。
酶水解可以在易于由本领域技术人员确定的条件下,在适合的含水环境中进行。在一方面,水解在适于一种或多种酶的活性的条件,即对于这种或这些酶来说最佳条件下进行。水解能以分批补料或连续的过程进行,其中将该含纤维素材料和/或含淀粉材料逐渐补入,例如,含有酶的水解溶液中。
糖化通常在搅拌釜反应器或发酵罐中,在受控的pH、温度、和混合条件下进行。适合的处理时间、温度以及pH条件可以由本领域技术人员容易地确定。例如,糖化可以持续长达200小时,但是典型地进行优选约12至约120小时,例如约16至约72小时或约24至约48小时。温度优选约25℃至约70℃,例如约30℃至约65℃,约40℃至约60℃,或约50℃至55℃的范围。pH优选约3至约8,例如约3.5至约7,约4至约6,或约4.5至约5.5的范围。干固体含量在约5wt.%至约50wt.%,例如约10wt.%至约40wt.%,或约20wt.%至约30wt.%的范围内。
可以使用纤维素分解酶组合物进行糖化。这样的酶组合物在以下的“纤维素分解酶组合物”部分中进行了描述。这些纤维素分解酶组合物可包含有用于降解该含纤维素材料的任何蛋白质。在一方面,该纤维素分解酶组合物包含或进一步包含选自下组的一种或多种(例如,若干种)蛋白质,该组由以下组成:纤维素酶、AA9(GH61)多肽、半纤维素酶、酯酶、棒曲霉素、木质素分解酶、氧化还原酶、果胶酶、蛋白酶、以及膨胀素。
在另一个实施例中,该纤维素酶优选是选自下组的一种或多种(例如,若干种)酶,该组由以下组成:内切葡聚糖酶、纤维二糖水解酶、以及β-葡糖苷酶。
在另一个实施例中,该半纤维素酶优选是选自下组的一种或多种(例如,若干种)酶,该组由以下组成:乙酰基甘露聚糖酯酶、乙酰基木聚糖酯酶、阿拉伯聚糖酶、阿拉伯呋喃糖苷酶、香豆酸酯酶、阿魏酸酯酶、半乳糖苷酶、葡萄糖醛酸酶、葡糖醛酸酯酶、甘露聚糖酶、甘露糖苷酶、木聚糖酶、以及木糖苷酶。在另一个实施例中,该氧化还原酶是选自下组的一种或多种(例如,若干种)酶,该组由以下组成:过氧化氢酶、漆酶、以及过氧化物酶。在本发明的方法中使用的酶或酶组合物可以是以任何适于使用的形式存在的,例如像发酵液配制品或细胞组合物、具有或不具有细胞碎片的细胞裂解物、半纯化或纯化的酶制剂、或作为酶的来源的宿主细胞。酶组合物可以是干粉或颗粒、无粉尘的颗粒、液体、稳定化液体或稳定化受保护的酶。可以根据已建立的方法例如通过添加稳定剂(如糖、糖醇或其他多元醇)、和/或乳酸或另一种有机酸,对液体酶制剂进行稳定化。
在一个实施例中,纤维素分解酶组合物或半纤维素分解酶组合物对于该含纤维素材料的有效量是约0.5mg至约50mg,例如,约0.5mg至约40mg、约0.5mg至约25mg、约0.75mg至约20mg、约0.75mg至约15mg、约0.5mg至约10mg、或约2.5mg至约10mg/g的该含纤维素材料。
在一个实施例中,以这样一种化合物对纤维素的葡糖基单元的以下摩尔比添加该化合物:约10
术语“液体(liquor)”意指在如WO 2012/021401中所述的条件下,由处理浆料中的木质纤维素和/或半纤维素材料、或其单糖(例如,木糖、阿拉伯糖、甘露糖等)所产生的溶液相(水相、有机相或其组合)、及其可溶性内容物。可以通过对木质纤维素或半纤维素材料(或原料)加热和/或加压进行处理,任选地是在催化剂例如酸的存在下、任选地是在有机溶剂的存在下、和任选地与材料的物理破坏相结合,然后将溶液与残余固形物分离,来产生用于加强AA9多肽(GH61多肽)纤维素分解的液体。在由纤维素分解酶制剂对纤维素底物的水解期间,从液体与AA9多肽的组合中可得到纤维素分解增强的程度是由此类条件确定的。可以使用本领域的标准方法,如过滤、沉淀或离心,而将液体与经过处理的材料进行分离。
在一个实施例中,对于纤维素来说的液体的有效量是约10
在发酵步骤中,例如作为预处理和酶水解步骤的结果由该含纤维素材料释放的糖,由发酵生物(如本文所述的酵母)发酵为乙醇。水解(糖化)和发酵可以是分开的或同时的。
在实施本文所述的方法的发酵步骤中可以使用任何适合的经水解的含纤维素材料。这类原料包括但不限于碳水化合物(例如,木质纤维素、木聚糖、纤维素、淀粉等)。该材料通常是基于经济学,即,每当量糖势的成本,以及对酶致转变的难降解性而进行选择。
使用含纤维素材料通过发酵生物生产乙醇是由糖(单糖)的代谢产生的。经水解的含纤维素材料的糖组成和发酵生物利用不同糖的能力对工艺产率具有直接影响。在申请人在本文的披露之前,本领域已知的菌株有效地利用葡萄糖但不(或非常有限地)代谢戊糖(像木糖,其为通常在水解材料中发现的单糖)。
发酵培养基的组成和发酵条件取决于发酵生物,并且可以由本领域技术人员容易地确定。通常,发酵在已知适于产生发酵产物的条件下进行。在一些实施例中,发酵过程在有氧或微需氧条件下(即,氧气浓度小于空气中的氧气浓度)或厌氧条件下进行。在一些实施例中,发酵在厌氧条件下(即,没有可检测的氧气)或小于约5、约2.5或约1mmol/L/h的氧气中进行。在没有氧的情况下,糖酵解中产生的NADH不能通过氧化磷酸化氧化。在厌氧条件下,宿主细胞可利用丙酮酸或其衍生物作为电子和氢受体以产生NAD+。
发酵过程通常在对重组真菌细胞最佳的温度下进行。例如,在一些实施例中,发酵过程在约25℃至约42℃的范围内的温度下进行。通常,该方法在低于约38℃,低于约35℃,低于约33℃,或低于约38℃,但至少约20℃、22℃或25℃的温度下进行。
发酵刺激剂可以用在本文所描述的方法中,以进一步改善发酵,并且特别是改善发酵生物的性能,例如速率增加和产物产率(例如乙醇产率)。“发酵刺激剂”是指用于发酵生物(特别是酵母)生长的刺激剂。用于生长的优选发酵刺激剂包括维生素和矿物质。维生素的实例包括多种维生素、生物素、泛酸、烟酸、内消旋肌醇、硫胺素、吡哆醇、对氨基苯酸、叶酸、核黄素、以及维生素A、B、C、D和E。例如,参见Alfenore等人,Improving ethanolproduction and viability of Saccharomyces cerevisia by a vitamin feedingstrategy during fed-batch process[通过在进料分批方法过程中的一种维生素进料策略改进乙醇产生和酿酒酵母(Saccharomyces cerevisiae)的存活力],Springer-Verlag[施普林格出版社](2002),将其通过引用由此并入。矿物质的实例包括可以供应包含P、K、Mg、S、Ca、Fe、Zn、Mn、和Cu营养素的矿物质和矿物盐。
在糖化过程中可以存在和/或添加纤维素分解酶或纤维素分解酶组合物。纤维素分解酶组合物是包含水解含纤维素材料的一种或多种(例如,若干种)酶的酶制剂。此类酶包括内切葡聚糖酶、纤维二糖水解酶、β-葡糖苷酶、和/或其组合。
在一些实施例中,该发酵生物包含编码可水解含纤维素材料的酶(例如,内切葡聚糖酶、纤维二糖水解酶、β-葡糖苷酶或其组合)的一种或多种(例如,若干种)异源多核苷酸。考虑本文描述或参照的任何酶(可水解含纤维素材料)用于在发酵生物中表达。
该纤维素分解酶可以是适合宿主细胞和/或本文所述的方法的任何纤维素分解酶(例如,内切葡聚糖酶、纤维二糖水解酶、β-葡糖苷酶),如天然存在的纤维素分解酶或其保留纤维素分解酶活性的变体。
在一些实施例中,当在相同条件下培养时,与不具有编码纤维素分解酶的异源多核苷酸的宿主细胞相比,包含编码该纤维素分解酶的异源多核苷酸的发酵生物具有增加的纤维素分解酶(例如,增加的内切葡聚糖酶、纤维二糖水解酶、和/或β-葡糖苷酶)活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码纤维素分解酶的异源多核苷酸的发酵生物相比,该发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的纤维素分解酶活性水平。
可以与本文所述的宿主细胞和/或方法一起使用的示例性纤维素分解酶包括细菌、酵母或丝状真菌纤维素分解酶,例如,获得自本文描述或参照的任何微生物,如上文在与蛋白酶有关的部分下描述的。
该纤维素分解酶可以是任何来源的。在一个实施例中,该纤维素分解酶衍生自木霉属的菌株,如里氏木霉的菌株;腐质霉属的菌株,如特异腐质霉的菌株,和/或金孢子菌属的菌株,如卢克诺文思金孢子菌的菌株。在一个优选的实施例中,该纤维素分解酶衍生自里氏木霉的菌株。
该纤维素分解酶组合物可进一步包含以下多肽(如酶)中的一种或多种:具有纤维素分解增强活性的AA9多肽(GH61多肽)、β-葡糖苷酶、木聚糖酶、β-木糖苷酶、CBH I、CBHII、或其两种、三种、四种、五种、或六种的混合物。
另外的一种或多种多肽(例如,AA9多肽)和/或一种或多种酶(例如,β-葡糖苷酶、木聚糖酶、β-木糖苷酶、CBH I和/或CBH II)对于该纤维素分解酶组合物生产生物(例如,里氏木霉)可以是外源的。
在一个实施例中,该纤维素分解酶组合物包含具有纤维素分解增强活性的AA9多肽和β-葡糖苷酶。
在另一个实施例中,该纤维素分解酶组合物包含具有纤维素分解增强活性的AA9多肽、β-葡糖苷酶、以及CBH I。
在另一个实施例中,该纤维素分解酶组合物包含具有纤维素分解增强活性的AA9多肽、β-葡糖苷酶、CBH I以及CBH II。
其他酶(如内切葡聚糖酶),还可以包含在该纤维素分解酶组合物中。
如以上提到的,该纤维素分解酶组合物可以包含多种不同的多肽,包括酶。
在一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的橙色嗜热子囊菌AA9(GH61A)多肽(例如,WO 2005/074656),以及米曲霉β-葡糖苷酶融合蛋白(例如,披露于WO2008/057637中的一种,特别是如SEQ ID NO:59和60中所示)。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的橙色嗜热子囊菌AA9(GH61A)多肽(例如,WO 2005/074656中的SEQ ID NO:2),以及烟曲霉β-葡糖苷酶(例如,WO2005/047499的SEQ ID NO:2)。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的埃默森青霉(Penicillium emersonii)AA9(GH61A)多肽,特别是披露于WO 2011/041397中的一种,以及烟曲霉β-葡糖苷酶(例如,WO 2005/047499的SEQ ID NO:2)。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的埃默森青霉AA9(GH61A)多肽,特别是披露于WO 2011/041397中的一种,以及烟曲霉β-葡糖苷酶(例如,WO2005/047499的SEQ ID NO:2),或披露于WO 2012/044915(通过引用特此并入)的变体,特别是包含一个或多个(例如所有)的以下取代的变体:F100D、S283G、N456E、F512Y。
在一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的AA9(GH61A)多肽,特别是衍生自埃默森青霉菌株的一种(例如,WO 2011/041397中的SEQ ID NO:2),烟曲霉β-葡糖苷酶(例如,WO 2005/047499中的SEQ ID NO:2)变体,该变体具有一个或多个(特别是所有)的以下取代:F100D、S283G、N456E、F512Y且披露于WO 2012/044915中;烟曲霉Cel7A CBH1,例如在WO2011/057140中披露为SEQ ID NO:6的一种和烟曲霉CBH II,例如在WO 2011/057140中披露为SEQ ID NO:18的一种。
在一个优选的实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含半纤维素酶或半纤维素分解酶组合物,如烟曲霉木聚糖酶和烟曲霉β-木糖苷酶。
在一个实施例中,纤维素分解酶组合物还包括木聚糖酶(例如,衍生自曲霉属,特别是棘孢曲霉或烟曲霉的菌株;或篮状菌属,特别是Talaromyces leycettanus的菌株)和/或β-木糖苷酶(例如,衍生自曲霉属,特别是烟曲霉,或篮状菌属,特别是埃默森篮状菌(Talaromyces emersonii)的菌株)。
在一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的橙色嗜热子囊菌AA9(GH61A)多肽(例如,WO 2005/074656),米曲霉β-葡糖苷酶融合蛋白(例如,披露于WO 2008/057637中的一种,特别是如SEQ ID NO:59和60),以及棘孢曲霉木聚糖酶(例如,在WO 94/21785中的Xyl II)。
在另一个实施例中,该纤维素分解酶组合物包含里氏木霉纤维素分解制剂,该里氏木霉纤维素分解制剂进一步包含具有纤维素分解增强活性的橙色嗜热子囊菌GH61A多肽(例如,WO 2005/074656中的SEQ ID NO:2)、烟曲霉β-葡糖苷酶(例如,WO 2005/047499的SEQ ID NO:2)、以及棘孢曲霉木聚糖酶(披露于WO 94/21785中的Xyl II)。
在另一个实施例中,该纤维素分解酶组合物包含里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的橙色嗜热子囊菌AA9(GH61A)多肽(例如,WO 2005/074656中的SEQ ID NO:2)、烟曲霉β-葡糖苷酶(例如,WO2005/047499的SEQ ID NO:2)、以及棘孢曲霉木聚糖酶(例如,披露于WO 94/21785中的XylII)。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的埃默森青霉AA9(GH61A)多肽(特别是披露于WO 2011/041397中的一种)、烟曲霉β-葡糖苷酶(例如,WO2005/047499的SEQ ID NO:2)、以及烟曲霉木聚糖酶(例如,WO 2006/078256中的Xyl III)。
在另一个实施例中,该纤维素分解酶组合物包含里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的埃默森青霉AA9(GH61A)多肽,特别是披露于WO 2011/041397中的一种、烟曲霉β-葡糖苷酶(例如,WO 2005/047499的SEQ ID NO:2)、烟曲霉木聚糖酶(例如,WO 2006/078256中的Xyl III)、以及来自烟曲霉的CBH I,特别是在WO 2011/057140中披露为SEQ ID NO:2的Cel7A CBH1。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的埃默森青霉AA9(GH61A)多肽,特别是披露于WO 2011/041397中的一种、烟曲霉β-葡糖苷酶(例如,WO 2005/047499的SEQ ID NO:2)、烟曲霉木聚糖酶(例如,WO 2006/078256中的Xyl III)、来自烟曲霉的CBH I,特别是在WO 2011/057140中披露为SEQ ID NO:2的Cel7A CBH1,以及衍生自烟曲霉的CBH II,特别是在WO 2013/028928中披露为SEQ ID NO:4的一种。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物进一步包含具有纤维素分解增强活性的埃默森青霉AA9(GH61A)多肽(特别是披露于WO 2011/041397中的一种)、烟曲霉β-葡糖苷酶(例如,WO2005/047499的SEQ ID NO:2)或其变体,该变体具有一个或多个(特别是所有)的以下取代:F100D、S283G、N456E、F512Y;烟曲霉木聚糖酶(例如,WO 2006/078256中的Xyl III)、来自烟曲霉的CBH I(特别是在WO 2011/057140中披露为SEQ ID NO:2的Cel7A CBH I),以及衍生自烟曲霉的CBH II(特别是在WO 2013/028928中披露的一种)。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物包含CBH I(GENSEQP登录号AZY49536(WO 2012/103293);CBHII(GENSEQP登录号AZY49446(WO 2012/103288);β-葡糖苷酶变体(GENSEQP登录号AZU67153(WO 2012/44915)),特别是具有一个或多个(特别是所有)的以下取代:F100D、S283G、N456E、F512Y;以及AA9(GH61多肽)(GENSEQP登录号BAL61510(WO 2013/028912))。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物包含CBH I(GENSEQP登录号AZY49536(WO 2012/103293));CBH II(GENSEQP登录号AZY49446(WO 2012/103288);GH10木聚糖酶(GENSEQP登录号BAK46118(WO 2013/019827));以及β-木糖苷酶(GENSEQP登录号AZI04896(WO 2011/057140))。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物包含CBH I(GENSEQP登录号AZY49536(WO 2012/103293));CBH II(GENSEQP登录号AZY49446(WO 2012/103288));以及AA9(GH61多肽;GENSEQP登录号BAL61510(WO 2013/028912))。
在另一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物包含CBH I(GENSEQP登录号AZY49536(WO 2012/103293));CBH II(GENSEQP登录号AZY49446(WO2012/103288)),AA9(GH61多肽;GENSEQP登录号BAL61510(WO 2013/028912)),和过氧化氢酶(GENSEQP登录号BAC11005(WO 2012/130120))。
在一个实施例中,该纤维素分解酶组合物是里氏木霉纤维素分解酶组合物,该里氏木霉纤维素分解酶组合物包含CBH I(GENSEQP登录号AZY49446(WO 2012/103288);CBHII(GENSEQP登录号AZY49446(WO 2012/103288)),β-葡糖苷酶变体(GENSEQP登录号AZU67153(WO 2012/44915)),具有一个或多个(特别是所有)的以下取代:F100D、S283G、N456E、F512Y;AA9(GH61多肽;GENSEQP登录号BAL61510(WO 2013/028912)),GH10木聚糖酶(GENSEQP登录号BAK46118(WO 2013/019827)),以及β-木糖苷酶(GENSEQP登录号AZI04896(WO 2011/057140))。
在一个实施例中,该纤维素分解组合物是里氏木霉纤维素分解酶制剂,该里氏木霉纤维素分解酶制剂包含EG I(Swissprot登录号P07981)、EG II(EMBL登录号M19373)、CBHI(见上文);CBH II(见上文);具有以下取代的β-葡糖苷酶变体(见上文):F100D、S283G、N456E、F512Y;AA9(GH61多肽;见上文),GH10木聚糖酶(见上文);以及β-木糖苷酶(见上文)。
也考虑披露于WO 2013/028928中的所有的纤维素分解酶组合物并且通过引用特此并入。
该纤维素分解酶组合物包含或可以进一步包含选自下组的一种或多种(若干种)蛋白质,该组由以下组成:纤维素酶、具有纤维素分解增强活性的AA9(即GH61)多肽、半纤维素酶、棒曲霉素、酯酶、漆酶、木质素分解酶、果胶酶、过氧化物酶、蛋白酶、以及膨胀素。
在一个实施例中,该纤维素分解酶组合物是商业纤维素分解酶组合物。适合用于本发明的方法的商业纤维素分解酶组合物的实例包括:
另外的酶及其组合物可在WO 2011/153516和WO 2016/045569(将其内容并入本文)中找到。
编码适合的纤维素分解酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(www.uniprot.org)内可容易获得的那些。
纤维素分解酶编码序列也可用于设计核酸探针以鉴定和克隆编码来自不同属或种的菌株的纤维素分解酶的DNA,如上文所述的。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码纤维素分解酶的多核苷酸,如上文所述的。
用于分离或克隆编码纤维素分解酶的多核苷酸的技术在上文中描述。
在一个实施例中,该纤维素分解酶具有与本文描述或参照的任何纤维素分解酶(例如,任何内切葡聚糖酶、纤维二糖水解酶、或β-葡糖苷酶)至少60%,例如至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性的成熟多肽序列。在一方面,该纤维素分解酶具有如下成熟多肽序列,该序列与本文描述或参照的任何纤维素分解酶相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸。在一个实施例中,该纤维素分解酶具有如下成熟多肽序列,该序列包含以下或由其组成:本文描述或参照的任何纤维素分解酶的氨基酸序列、等位基因变体、或其具有纤维素分解酶活性的片段。在一个实施例中,该纤维素分解酶具有一个或多个(例如,两个、若干个)氨基酸的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,该纤维素分解酶在相同条件下具有本文描述或参照的任何纤维素分解酶(例如,任何内切葡聚糖酶、纤维二糖水解酶、或β-葡糖苷酶)的纤维素分解酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,该纤维素分解酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任何纤维素分解酶(例如,任何内切葡聚糖酶、纤维二糖水解酶、或β-葡糖苷酶)的编码序列的全长互补链杂交。在一个实施例中,该纤维素分解酶编码序列与本文描述或参照的任何纤维素分解酶的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,编码该纤维素分解酶的多核苷酸包含文描述或参照的任何纤维素分解酶(例如,任何内切葡聚糖酶、纤维二糖水解酶、或β-葡糖苷酶)的编码序列。在一个实施例中,编码该纤维素分解酶的多核苷酸包含来自本文描述或参照的任何纤维素分解酶的编码序列的子序列,其中该子序列编码具有纤维素分解酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
该纤维素分解酶还可以包括融合多肽或可切割的融合多肽,如上文所述的。
在一方面,该发酵生物(例如,酵母细胞)进一步包含编码木糖异构酶(XI)的异源多核苷酸。该木糖异构酶可以是适合宿主细胞和本文所述的方法的任何木糖异构酶,如天然存在的木糖异构酶或其保留木糖异构酶活性的变体。在一个实施例中,该木糖异构酶存在于宿主细胞的胞液中。
在一些实施例中,当在相同条件下培养时,与不具有编码木糖异构酶的异源多核苷酸的宿主细胞相比,包含编码该木糖异构酶的异源多核苷酸的发酵生物具有增加的木糖异构酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码木糖异构酶的异源多核苷酸的宿主细胞相比,发酵生物具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的木糖异构酶活性水平。
可以与本文所述的重组宿主细胞和使用方法一起使用的示例性木糖异构酶包括但不限于,来自真菌瘤胃壶菌属菌种(WO 2003/062430)或其他来源(Madhavan等人,2009,Appl Microbiol Biotechnol.[应用微生物学与生物技术]82(6),1067-1078)的XI,已在酿酒酵母宿主细胞中表达。适用于酵母中表达的另外的其他XI已在US 2012/0184020(来自黄化瘤胃球菌(Ruminococcus flavefaciens)的XI)、WO 2011/078262(来自黄胸散白蚁(Reticulitermes speratus)和达尔文澳白蚁(Mastotermes darwiniensis)的若干种XI)、以及WO 2012/009272(含有来自软弱贫养菌(Abiotrophia defectiva)的XI的构建体和真菌细胞)中描述。US 8,586,336描述了表达通过牛瘤胃液获得的XI(在本文显示为SEQ IDNO:74)的酿酒酵母宿主细胞。
编码适合的木糖异构酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(www.uniprot.org)内可容易获得的那些。在一个实施例中,该木糖异构酶是细菌、酵母或丝状真菌木糖异构酶,例如,获得自本文描述或参照的任何微生物,如上文所述的。
木糖异构酶编码序列也可用于设计核酸探针以鉴定和克隆编码来自不同属或种的菌株的木糖异构酶的DNA,如上文所述的。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码木糖异构酶的多核苷酸,如上文所述的。
用于分离或克隆编码木糖异构酶的多核苷酸的技术在上文中描述。
在一个实施例中,该木糖异构酶具有与本文描述或参照的任何木糖异构酶(例如,SEQ ID NO:74的木糖异构酶)具有至少60%,例如至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性的成熟多肽序列。在一方面,该木糖异构酶具有如下成熟多肽序列,该序列与本文描述或参照的任何木糖异构酶(例如,SEQ ID NO:74的木糖异构酶)相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸。在一个实施例中,该木糖异构酶具有如下成熟多肽序列,该序列包含以下或由其组成:本文描述或参照的任何木糖异构酶(例如,SEQ ID NO:74的木糖异构酶)的氨基酸序列、等位基因变体、或其具有木糖异构酶活性的片段。在一个实施例中,该木糖异构酶具有一个或多个(例如,两个、若干个)氨基酸的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,该木糖异构酶在相同条件下具有本文描述或参照的任何木糖异构酶(例如,SEQ ID NO:74的木糖异构酶)的木糖异构酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,该木糖异构酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任何木糖异构酶(例如,SEQ ID NO:74的木糖异构酶)的编码序列的全长互补链杂交。在一个实施例中,该木糖异构酶编码序列与来自本文描述或参照的任何木糖异构酶(例如,SEQ IDNO:74的木糖异构酶)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,编码该木糖异构酶的异源多核苷酸包含本文描述或参照的任何木糖异构酶(例如,SEQ ID NO:74的木糖异构酶)的编码序列。在一个实施例中,编码该木糖异构酶的异源多核苷酸包含来自本文描述或参照的任何木糖异构酶的编码序列的子序列,其中该子序列编码具有木糖异构酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
这些木糖异构酶还可以包括融合多肽或可切割的融合多肽,如上文所述的。
在一方面,该发酵生物(例如,酵母细胞)进一步包含编码木酮糖激酶(XK)的异源多核苷酸。如本文所用的,木酮糖激酶提供了将D-木酮糖转化为木酮糖5-磷酸的酶活性。该木酮糖激酶可以是适合宿主细胞和本文所述的方法的任何木酮糖激酶,如天然存在的木酮糖激酶或其保留木酮糖激酶活性的变体。在一个实施例中,该木酮糖激酶存在于宿主细胞的胞液中。
在一些实施例中,当在相同条件下培养时,与不具有编码木酮糖激酶的异源多核苷酸的宿主细胞相比,包含编码该木酮糖激酶的异源多核苷酸的发酵生物具有增加的木酮糖激酶活性水平。在一些实施例中,当在相同条件下培养时,与不具有编码木酮糖激酶的异源多核苷酸的宿主细胞相比,宿主细胞具有增加至少5%,例如至少10%、至少15%、至少20%、至少25%、至少50%、至少100%、至少150%、至少200%、至少300%或至少500%的木糖异构酶活性水平。
可以与本文所述的发酵生物和使用方法一起使用的示例性木酮糖激酶包括但不限于,SEQ ID NO:75的酿酒酵母木酮糖激酶。编码适合的木酮糖激酶的另外的多核苷酸可以获得自任何属的微生物,包括在UniProtKB数据库(www.uniprot.org)内可容易获得的那些。在一个实施例中,该木酮糖激酶是细菌、酵母或丝状真菌木酮糖激酶,例如,获得自本文描述或参照的任何微生物,如上文所述的。
木酮糖激酶编码序列也可用于设计核酸探针以鉴定和克隆编码来自不同属或种的菌株的木酮糖激酶的DNA,如上文所述的。
还可以从其他来源,包括从自然界(例如,土壤、堆肥、水等)分离的微生物或直接从自然材料(例如,土壤、堆肥、水等)获得的DNA样品鉴定和获得编码木酮糖激酶的多核苷酸,如上文所述的。
用于分离或克隆编码木酮糖激酶的多核苷酸的技术在上文中描述。
在一个实施例中,该木酮糖激酶具有与本文描述或参照的任何木酮糖激酶(例如,SEQ ID NO:75的酿酒酵母木酮糖激酶)至少60%,例如至少65%、至少70%、至少75%、至少80%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性的成熟多肽序列。在一个实施例中,该木酮糖激酶具有如下成熟多肽序列,该序列与本文描述或参照的任何木酮糖激酶(例如,SEQID NO:75的酿酒酵母木酮糖激酶)相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸相差不超过三个氨基酸相差不超过两个氨基酸、或相差一个氨基酸。在一个实施例中,该木酮糖激酶具有如下成熟多肽序列,该序列包含以下或由其组成:本文描述或参照的任何木酮糖激酶(例如,SEQ ID NO:75的酿酒酵母木酮糖激酶)的氨基酸序列、等位基因变体、或其具有木酮糖激酶活性的片段。在一个实施例中,该木酮糖激酶具有一个或多个(例如,两个、若干个)氨基酸的氨基酸取代、缺失和/或插入。在一些实施例中,氨基酸取代、缺失和/或插入的总数不超过10,例如不超过9、8、7、6、5、4、3、2或1。
在一些实施例中,该木酮糖激酶在相同条件下具有本文描述或参照的任何木酮糖激酶(例如,SEQ ID NO:75的酿酒酵母木酮糖激酶)的木酮糖激酶活性的至少20%,例如至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%或100%。
在一个实施例中,该木酮糖激酶编码序列在至少低严格条件下,例如中严格条件下、中-高严格条件下、高严格条件下、或非常高严格条件下与来自本文描述或参照的任何木酮糖激酶(例如,SEQ ID NO:75的酿酒酵母木酮糖激酶)的编码序列的全长互补链杂交。在一个实施例中,该木酮糖激酶编码序列与来自本文描述或参照的任何木酮糖激酶(例如,SEQ ID NO:75的酿酒酵母木酮糖激酶)的编码序列具有至少65%,例如至少70%、至少75%、至少80%、至少85%、至少85%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%序列同一性。
在一个实施例中,编码该木酮糖激酶的异源多核苷酸包含本文描述或参照的任何木酮糖激酶(例如,SEQ ID NO:75的酿酒酵母木酮糖激酶)的编码序列。在一个实施例中,编码该木酮糖激酶的异源多核苷酸包含来自本文描述或参照的任何木酮糖激酶的编码序列的子序列,其中该子序列编码具有木酮糖激酶活性的多肽。在一个实施例中,子序列中的核苷酸残基的数目是参照编码序列的数目的至少75%,例如至少80%、85%、90%或95%。
这些木酮糖激酶还可以包括融合多肽或可切割的融合多肽,如上文所述的。
在一方面,该发酵生物(例如,酵母细胞)进一步包含编码核酮糖5磷酸3-差向异构酶(RPE1)的异源多核苷酸。如本文所用的,核酮糖5磷酸3-差向异构酶提供了将L-核酮糖5-磷酸转化为L-木酮糖5-磷酸的酶活性(EC 5.1.3.22)。该RPE1可以是适合宿主细胞和本文所述的方法的任何RPE1,如天然存在的RPE1或其保留RPE1活性的变体。在一个实施例中,该RPE1存在于宿主细胞的胞液中。
在一个实施例中,该重组细胞包含编码核酮糖5磷酸3-差向异构酶(RPE1)的异源多核苷酸,其中该RPE1是酿酒酵母RPE1,或者是与酿酒酵母RPE1具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的RPE1。
在一方面,该发酵生物(例如,酵母细胞)进一步包含编码核酮糖5磷酸异构酶(RKI1)的异源多核苷酸。如本文所用的,核酮糖5磷酸异构酶提供了将核糖-5-磷酸转化为核酮糖-5-磷酸的酶活性。该RKI1可以是适合宿主细胞和本文所述的方法的任何RKI1,如天然存在的RKI1或其保留RKI1活性的变体。在一个实施例中,该RKI1存在于宿主细胞的胞液中。
在一个实施例中,该发酵生物包含编码核酮糖5磷酸异构酶(RKI1)的异源多核苷酸,其中该RKI1是酿酒酵母RKI1,或者是具有与酿酒酵母RKI1至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列的RKI1。
在一方面,该发酵生物(例如,酵母细胞)进一步包含编码转酮酶(TKL1)的异源多核苷酸。该TKL1可以是适合宿主细胞和本文所述的方法的任何TKL1,如天然存在的TKL1或其保留TKL1活性的变体。在一个实施例中,该TKL1存在于宿主细胞的胞液中。
在一个实施例中,该发酵生物包含编码转酮酶(TKL1)的异源多核苷酸,其中该TKL1是酿酒酵母TKL1,或者是具有与酿酒酵母TKL1至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列的TKL1。
在一方面,该发酵生物(例如,酵母细胞)进一步包含编码转醛酶(TAL1)的异源多核苷酸。该TAL1可以是适合宿主细胞和本文所述的方法的任何TAL1,如天然存在的TAL1或其保留TAL1活性的变体。在一个实施例中,该TAL1存在于宿主细胞的胞液中。
在一个实施例中,该发酵生物包含编码转醛酶(TAL1)的异源多核苷酸,其中该TAL1是酿酒酵母TAL1,或者是具有与酿酒酵母TAL1至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列的TAL1。
发酵产物可以是由发酵得到的任何物质。发酵产物可以是,但不限于:醇(例如,阿拉伯糖醇、正丁醇、异丁醇、乙醇、甘油、甲醇、乙二醇、1,3-丙二醇[丙二醇]、丁二醇、丙三醇、山梨醇和木糖醇);烷烃(例如,戊烷、己烷、庚烷、辛烷、壬烷、癸烷、十一烷、以及十二烷)、环烷烃(例如,环戊烷、环己烷、环庚烷、以及环辛烷)、烯烃(例如,戊烯、己烯、庚烯、以及辛烯);氨基酸(例如,天冬氨酸、谷氨酸、甘氨酸、赖氨酸、丝氨酸、以及苏氨酸);气体(例如,甲烷、氢气(H
在一方面,发酵产物是醇。术语“醇”涵盖含有一个或多个羟基部分的物质。醇可以是但不限于:正丁醇、异丁醇、乙醇、甲醇、阿拉伯糖醇、丁二醇、乙二醇、甘油、丙三醇、1,3-丙二醇、山梨醇、木糖醇。参见例如,Gong等人,1999,Ethanol生产from renewableresources[由可再生资源生产乙醇],在Biochemical Engineering/Biotechnology[生化工程/生物技术进展]中,Scheper,T.,编辑,Springer-Verlag Berlin Heidelberg,Germany[施普林格出版社柏林海德堡,德国],65:207-241;Silveira和Jonas,2002,Appl.Microbiol.Biotechnol.[应用微生物学与生物技术]59:400-408;Nigam和Singh,1995,Process Biochemistry[生物化学工艺]30(2):117-124;Ezeji等人,2003,WorldJournal of Microbiology and Biotechnology[世界微生物学和生物技术杂志]19(6):595-603。在一个实施例中,该发酵产物是乙醇。
在另一方面,发酵产物是烷烃。烷烃可以是非支链或支链烷烃。烷烃可以是但不限于:戊烷、己烷、庚烷、辛烷、壬烷、癸烷、十一烷、或十二烷。
在另一方面,发酵产物是环烷烃。环烷烃可以是但不限于:环戊烷、环己烷、环庚烷或环辛烷。
在另一方面,发酵产物是烯烃。烯烃可以是非支链或支链烯烃。烯烃可以是但不限于:戊烯、己烯、庚烯或辛烯。
在另一方面,发酵产物是氨基酸。有机酸可以是但不限于:天冬氨酸、谷氨酸、甘氨酸、赖氨酸、丝氨酸、或苏氨酸。参见,例如Richard和Margaritis,2004,Biotechnology andBioengineering[生物技术和生物工程]87(4):501-515。
在另一方面,发酵产物是气体。气体可以是但不限于:甲烷、H
在另一方面,发酵产物是异戊二烯。
在另一方面,发酵产物是酮。术语“酮”涵盖含有一个或多个酮部分的物质。酮可以是但不限于:丙酮。
在另一方面,发酵产物是有机酸。有机酸可以是但不限于:乙酸、醋酮酸、己二酸、抗坏血酸、柠檬酸、2,5-二酮-D-葡糖酸、甲酸、富马酸、葡糖二酸、葡糖酸、葡糖醛酸、戊二酸、3-羟基丙酸、衣康酸、乳酸、苹果酸、丙二酸、草酸、丙酸、琥珀酸、或木糖酸。参见,例如Chen和Lee,1997,Appl.Biochem.Biotechnol.[应用生物化学与生物技术]63-65:435-448。
在另一方面,发酵产物是聚酮化合物。
在一些方面中,在相同条件(例如,在发酵约54小时时或在发酵54小时后,如实例3或4中描述的条件)下,当与使用没有编码磷脂酶的异源多核苷酸的相同细胞的相同方法相比时,发酵生物(或其方法)提供发酵产物(例如,乙醇)的更高产率。在一些实施例中,所述方法导致发酵产物(例如,乙醇)的产率高出至少0.25%,例如0.5%、0.75%、1.0%、1.25%、1.5%、1.75%、2%、3%或5%。
可以使用本领域已知的任何方法,任选地从发酵培养基中回收发酵产物(例如,乙醇),该方法包括但不限于:层析法、电泳程序、差别溶解度、蒸馏或萃取。例如,通过常规蒸馏方法从发酵的纤维素材料中分离和纯化醇。可以获得纯度高达约96vol.%的乙醇,其能用作例如燃料乙醇、饮用乙醇(即,可饮用的中性含酒精饮料),或工业乙醇。
在这些方法的一些方面,回收后的发酵产物是基本上纯的。关于本文的这些方法,“基本上纯的”意指回收的制剂包含不超过15%的杂质,其中杂质意指除发酵产物(例如,乙醇)以外的化合物。在一种变体中,提供了基本上纯的制剂,其中该制剂包含不超过25%的杂质、或不超过20%的杂质、或不超过10%的杂质、或不超过5%的杂质、或不超过3%的杂质、或不超过1%的杂质、或不超过0.5%的杂质。
可以使用本领域已知的方法实施适合的测定来测试乙醇和污染物的产生以及糖消耗。例如,可以通过方法(如HPLC(高效液相层析法)、GC-MS(气相层析-质谱法)和LC-MS(液相层析-质谱法))或使用本领域熟知的常规程序的其他适合的分析方法对乙醇产物以及其他有机化合物进行分析。还可以用培养上清液测试发酵液中的乙醇的释放。可以使用例如针对葡萄糖和醇类的折光率检测器、以及针对有机酸的UV检测器通过HPLC(Lin等人,Biotechnol.Bioeng.[生物技术与生物工程]90:775-779(2005))、或使用本领域熟知的其他适合的测定和检测方法量化在发酵培养基中的副产物和残余的糖(例如,葡萄糖或木糖)。
可在以下编号的段落中进一步描述本发明:
段落[1].一种从含淀粉材料或含纤维素材料生产发酵产物的方法,所述方法包括:
(a)糖化所述含淀粉材料或含纤维素材料;以及
(b)用发酵生物发酵步骤(a)的经糖化的材料;
其中所述发酵生物包含编码磷脂酶的异源多核苷酸。
段落[2].根据段落[1]所述的方法,其中所述磷脂酶是磷脂酶A或磷脂酶C。
段落[3].根据段落[1]或[2]所述的方法,其中所述磷脂酶具有与SEQ ID NO:235-242和252-342中任一个(例如,SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[4].根据段落[1]-[3]中任一项所述的方法,其中所述异源多核苷酸编码磷脂酶,所述磷脂酶具有与SEQ ID NO:235-242和252-342中任一个(例如,SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。
段落[5].根据段落[1]-[4]中任一项所述的方法,其中所述异源多核苷酸编码磷脂酶,所述磷脂酶具有包含SEQ ID NO:235-242和252-342中任一个(例如SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列或由其组成的成熟多肽序列。
段落[6].根据段落[1]-[5]中任一项所述的方法,其中对含淀粉材料进行步骤(a)的糖化,其中所述含淀粉材料是糊化或未糊化淀粉。
段落[7].根据段落[6]所述的方法,所述方法包括在糖化之前,通过使所述含淀粉材料与α-淀粉酶接触来液化所述材料。
段落[8].根据段落[7]所述的方法,其中液化所述含淀粉材料和/或糖化所述含淀粉材料在外源添加的蛋白酶存在下进行。
段落[9].根据段落[1]-[8]中任一项所述的方法,其中发酵在减氮条件下(例如,少于1000ppm尿素或氢氧化铵,如少于750ppm、少于500ppm、少于400ppm、少于300ppm、少于250ppm、少于200ppm、少于150ppm、少于100ppm、少于75ppm、少于50ppm、少于25ppm、或少于10ppm)进行。
段落[10].根据段落[1]-[9]中任一项所述的方法,其中发酵和糖化在同时糖化和发酵(SSF)中同时进行。
段落[11].根据段落[1]-[9]中任一项所述的方法,其中发酵和糖化是顺序进行的(SHF)。
段落[12].根据段落[1]-[11]中任一项所述的方法,所述方法包括从所述发酵中回收所述发酵产物。
段落[13].根据段落[12]所述的方法,其中从所述发酵中回收所述发酵产物包括蒸馏。
段落[14].根据段落[1]-[13]中任一项所述的方法,其中所述发酵产物是乙醇。
段落[15].根据段落[1]-[14]中任一项所述的方法,其中所述发酵生物包含编码葡糖淀粉酶的异源多核苷酸。
段落[16].根据段落[15]所述的方法,其中所述葡糖淀粉酶具有成熟多肽序列,所述成熟多肽序列与密孔菌属葡糖淀粉酶(例如SEQ ID NO:229的血红密孔菌葡糖淀粉酶)、粘褶菌属葡糖淀粉酶(例如SEQ ID NO:8的篱边粘褶菌)、或SEQ ID NO:102-113中任一个的葡糖淀粉酶(例如SEQ ID NO:103或104的扣囊复膜酵母菌葡糖淀粉酶或SEQ ID NO:230的里氏木霉葡糖淀粉酶)的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性。
段落[17].根据段落[1]-[16]中任一项所述的方法,其中所述发酵生物包含编码α-淀粉酶的异源多核苷酸。
段落[18].根据段落[17]所述的方法,其中所述α-淀粉酶具有与SEQ ID NO:76-101、121-174和231中任一个的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[19].根据段落[1]-[18]中任一项所述的方法,其中所述发酵生物包含编码蛋白酶的异源多核苷酸。
段落[20].根据段落[19]所述的方法,其中所述蛋白酶具有与SEQ ID NO:9-73中任一个(例如SEQ ID NO:9、14、16、21、22、33、41、45、61、62、66、67和69中任一个;如SEQ NO:9、14、16和69中任一个)的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[21].根据段落[1]-[20]中任一项所述的方法,其中所述发酵生物包含编码海藻糖酶的异源多核苷酸。
段落[22].根据段落[21]所述的方法,其中所述海藻糖酶具有与SEQ ID NO:175-226中任一个的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[23].根据段落[1]-[22]中任一项所述的方法,其中糖化步骤发生在含纤维素材料上,并且其中所述含纤维素材料进行了预处理。
段落[24].根据段落[23]所述的方法,其中所述预处理是稀酸预处理。
段落[25].根据段落[1]-[24]中任一项所述的方法,其中对含纤维素材料进行糖化,并且其中所述酶组合物包含选自以下的一种或多种酶:纤维素酶、AA9多肽、半纤维素酶、CIP、酯酶、棒曲霉素、木质素分解酶、氧化还原酶、果胶酶、蛋白酶和膨胀素。
段落[26].根据段落[25]所述的方法,其中所述纤维素酶是选自以下的一种或多种酶:内切葡聚糖酶、纤维二糖水解酶、以及β-葡糖苷酶。
段落[27].根据段落[25]或[26]所述的方法,其中所述半纤维素酶是选自以下的一种或多种酶:木聚糖酶、乙酰木聚糖酯酶、阿魏酸酯酶、阿拉伯呋喃糖苷酶、木糖苷酶、以及葡糖醛酸糖苷酶。
段落[28].根据段落[1]-[27]中任一项所述的方法,其中所述发酵生物是酵母属、红酵母属、裂殖酵母属、克鲁维酵母属、毕赤酵母属、汉逊酵母属、红冬孢酵母属、假丝酵母属、耶氏酵母属、油脂酵母属、隐球菌属或德克拉酵母属菌种细胞。
段落[29].根据段落[1]-[28]中任一项所述的方法,其中所述发酵生物是酿酒酵母细胞。
段落[30].根据段落[1]-[29]中任一项所述的方法,其中在相同条件(例如,在发酵约54小时时或在发酵54小时后,如实例3或4中描述的条件)下,当与使用没有编码磷脂酶的异源多核苷酸的相同细胞的相同方法相比时,所述方法导致发酵产物的更高产率和/或减少的泡沫积累。
段落[31].根据段落[30]所述的方法,其中所述方法导致发酵产物的产率高出至少0.25%(例如0.5%、0.75%、1.0%、1.25%、1.5%、1.75%、2%、3%或5%)。
段落[32].一种重组酵母细胞,所述重组酵母细胞包含编码磷脂酶的异源多核苷酸。
段落[33].根据段落[32]所述的重组酵母细胞,其中所述磷脂酶是磷脂酶A或磷脂酶C。
段落[34].根据段落[32]或[33]所述的重组酵母细胞,其中所述磷脂酶具有与SEQID NO:235-242和252-342中任一个(例如,SEQ ID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[35].根据段落[32]-[34]中任一项所述的重组酵母细胞,其中所述异源多核苷酸编码磷脂酶,所述磷脂酶具有与SEQ ID NO:235-242和252-342中任一个(例如,SEQ IDNO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列相差不超过十个氨基酸,例如相差不超过五个氨基酸、相差不超过四个氨基酸、相差不超过三个氨基酸、相差不超过两个氨基酸、或相差一个氨基酸的成熟多肽序列。
段落[36].根据段落[32]-[35]中任一项所述的重组酵母细胞,其中所述异源多核苷酸编码磷脂酶,所述磷脂酶具有包含SEQ ID NO:235-242和252-342中任一个(例如SEQID NO:235、236、237、238、239、240、241和242中任一个)的氨基酸序列或由其组成的成熟多肽序列。
段落[37].根据段落[32]-[36]中任一项所述的重组酵母细胞,其中所述发酵生物包含编码葡糖淀粉酶的异源多核苷酸。
段落[38].根据段落[37]所述的重组酵母细胞,其中所述葡糖淀粉酶具有成熟多肽序列,所述成熟多肽序列与密孔菌属葡糖淀粉酶(例如SEQ ID NO:229的血红密孔菌葡糖淀粉酶)、粘褶菌属葡糖淀粉酶(例如SEQ ID NO:8的篱边粘褶菌)、或SEQ ID NO:102-113中任一个的葡糖淀粉酶(例如SEQ ID NO:103或104的扣囊复膜酵母菌葡糖淀粉酶或SEQ IDNO:230的里氏木霉葡糖淀粉酶)的氨基酸序列具有60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性。
段落[39].根据段落[32]-[38]中任一段所述的重组酵母细胞,其中所述发酵生物包含编码α-淀粉酶的异源多核苷酸。
段落[40].根据段落[39]所述的重组酵母细胞,其中所述α-淀粉酶具有与SEQ IDNO:76-101、121-174和231中任一个的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[41].根据段落[32]-[40]中任一项所述的重组酵母细胞,其中所述发酵生物包含编码蛋白酶的异源多核苷酸。
段落[42].根据段落[41]所述的重组酵母细胞,其中所述蛋白酶具有与SEQ IDNO:9-73中任一个(例如SEQ ID NO:9、14、16、21、22、33、41、45、61、62、66、67和69中任一个;如SEQ NO:9、14、16和69中任一个)的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[43].根据段落[32]-[42]中任一项所述的重组酵母细胞,其中所述发酵生物包含编码海藻糖酶的异源多核苷酸。
段落[44].根据段落[43]所述的重组酵母细胞,其中所述海藻糖酶具有与SEQ IDNO:175-226中任一个的氨基酸序列具有至少60%,例如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的成熟多肽序列。
段落[45].根据段落[32]-[44]中任一项所述的重组酵母,其中所述细胞是酵母属、红酵母属、裂殖酵母属、克鲁维酵母属、毕赤酵母属、汉逊酵母属、红冬孢酵母属、假丝酵母属、耶氏酵母属、油脂酵母属、隐球菌属或德克拉酵母属菌种细胞。
段落[46].根据段落[45]所述的重组酵母,其中所述细胞是酿酒酵母细胞。
段落[47].根据段落[32]-[46]中任一项所述的重组细胞,其中在相同条件(例如,在发酵约54小时时或在发酵54小时后,如实例3或4中描述的条件)下,当与使用相同方法和没有编码磷脂酶的异源多核苷酸的相同细胞进行发酵相比时,所述方法导致发酵产物的更高产率和/或减少的泡沫积累。
段落[48].根据段落[47]所述的重组细胞,其中所述细胞导致发酵产物的产率高出至少0.25%(例如0.5%、0.75%、1.0%、1.25%、1.5%、1.75%、2%、3%或5%)。
本文描述和要求保护的本发明不限于本文披露的具体方面的范围,因为这些方面旨在作为本发明几方面的说明。任何等同方面旨在处于本发明的范围之内。实际上,除了本文所示和描述的那些之外,本发明的各种修改因前述说明而对本领域的技术人员变得清楚。此类修改也旨在落入所附权利要求的范围内。在冲突的情况下,以包括定义的本披露为准。所有参考文献针对描述通过引用具体结合。
提供以下实例以说明本发明的某些方面,但不旨在以任何方式限制所要求保护的本发明的范围。
实例
用作缓冲液和底物的化学品是至少试剂级的商业产品。
ETHANOL
YPD+clonNAT平板是由10g酵母萃取物、20g蛋白胨、20g细菌培养用琼脂、以及加至960ml的去离子水组成,然后进行高压釜处理。加入40mL无菌50%葡萄糖和1mL clonNAT储备液,然后混合并倒入。
clonNAT储备液由2g硫酸诺尔丝菌素和加至20ml的去离子水组成。
该实例描述了在酿酒酵母TDH3、TEF2、PGK1、ADH1或RPL18B启动子的控制下,含有异源磷脂酶的酵母细胞的构建。设计含有启动子,基因和终止子的三片DNA,以允许这3个DNA片段之间的同源重组并进入酵母MHCT-484的X-3位点(WO 2018/222990)。所得的菌株具有包含一个启动子的片段(左片段)、包含一个基因的片段(中片段)、以及一个PRM9终止子片段(右片段),将三者整合到X-3位点处的酿酒酵母基因组中。
通过赛默飞世尔科技公司(马萨诸塞州沃尔瑟姆)合成含有与X-3位点具有60bp同源性、酿酒酵母启动子TDH3、TEF2、PGK1、ADH1或RPL18B(分别为SEQ ID NO:1、2、4、5和6)以及酿酒酵母MF1α信号肽(SEQ ID NO:7)的合成线性未克隆DNA。为了产生用于转化到酵母中的另外的线性DNA,将含有上述左盒的五个线性DNA中的每一个PCR扩增。
通过赛默飞世尔科技公司合成含有酿酒酵母PRM9终止子(SEQ ID NO:243)和与X-3位点具有300bp同源性的合成线性未克隆DNA。
通过赛默飞世尔科技公司合成包含酿酒酵母MF1α信号肽的编码序列,成熟多肽的编码序列和50bp PRM9终止子的合成线性未克隆DNA。
用上述左、中和右整合片段转化酵母MHCT-484(WO 2018/222990)。在每个转化库中,使用固定的左片段和右片段以及包含磷脂酶基因的固定的中间片段,每个片段为100ng。为了帮助左片段、中片段和右片段在基因组X-3位点处的同源重组,还在转化中使用含有Cas9和对X-3(pMcTs442)特异的指导RNA的质粒。按照酵母电穿孔方案,将这四种组分转化到酿酒酵母菌株MHCT-484中。在YPD+cloNAT上选择转化体以选择含有CRISPR/Cas9质粒pMcTs442的转化体。使用Q-pix Colony Picking System(分子仪器公司;加利福尼亚州圣何塞)挑选转化体,以接种1孔含有YPD+cloNAT培养基的96孔板。使板生长2天,然后添加甘油至20%的终浓度,并将这些板储存在-80℃下直至需要。通过使用基因座特异性引物的PCR和随后的测序来验证特异性磷脂酶构建体的整合。此实例中产生的菌株示于表5中。
表5.
将酵母菌株在含有2%葡萄糖的标准YPD培养基中培养24小时。培养后,将样品离心,并如下所述测定上清液的酶活性。
通过使用来自Invitrogen(加利福尼亚州卡尔斯巴德)的
用于PLA1活性测定的溶液的初步制备:
1.反应缓冲液:100mM MOPS+0.5mM Zn pH 7
2.PLA底物:
a.将40ul DMSO(组分E)加入一瓶PLA底物(组分A)中
b.避光
c.用于100rxns的足够体积
3.阳性对照PLA(组分D)的500U/mL原液
a.将组分D小瓶溶于100uL反应缓冲液中
4.10mM DOPC(组分B)
a.将组分B小瓶溶于100ul ETOH中
5.10mM DOPG(组分C)
a.将组分C小瓶溶于100ul ETOH中
用于PLA1活性测定的溶液和样品的最终制备:
1.通过在反应缓冲液中稀释500U/mL原液阳性对照制成10U/mL阳性对照
a.实例:向980mL反应缓冲液中添加20ul 500U/mL阳性对照
2.连续稀释阳性对照,获得8点标准曲线,起始浓度从10U/ml开始
a.加入底物时,最终阳性对照浓度降低两倍(5U/ml)
3.必要时,将样品在反应缓冲液中稀释
4.脂质混合物:30ul 10mM DOPC,30ul 10mM DOPG,30ul1mM PLA底物
5.PLA底物
a.在装有搅拌棒的小烧杯中,将50ul脂质缓慢混合至5mL反应缓冲液中。
b.搅拌约2-5分钟
PLA1活性测定的测定方案:
1.50ul 8点标准曲线用于第10、11、12列
a.最后一行留空用于缓冲液
2.向剩余的孔中添加50ul样品
3.向包含样品和对照的孔中添加50ul PLA-脂质底物。混合均匀,没有引入气泡。
4.在505EX/515EM上读取T0(也可以使用450EX/515EM)
5.覆盖平板并避光孵育
6.孵育5小时后读数
表6.PLA1活性测定条件
通过使用来自Invitrogen的
用于PLC活性测定的溶液的初步制备:
1.反应缓冲液:100mM NaAc+0.5mM Zn pH 5
2.PLC底物的200X原液
a.将100ul DMSO(组分B)加入一瓶PLC底物中
b.避光
c.用于125rxns的足够体积
3.阳性对照PC-PLC(组分E)的40U/mL原液
a.将组分E小瓶溶于200uL反应缓冲液中
用于PLC活性测定的溶液和样品的最终二次制备:
1.通过稀释40U/mL原液阳性对照40倍制备1U/mL阳性对照a.实例:向0.975mL反应缓冲液中添加25ul 40U/mL阳性对照
2.连续稀释阳性对照,获得8点标准曲线,起始浓度从0.125U/ml开始
a.加入底物时,最终阳性对照浓度降低两倍(0.0625U/ml)
3.必要时,将样品在反应缓冲液中稀释
4.PLC底物:将40ul卵磷脂(组分D)和100ul PLC底物200X原液(在先前步骤中制备)添加至9.86mL反应缓冲液中。
PLC活性测定的测定方案:
1.75ul 8点标准曲线用于第10、11、12列
a.最后一行留空用于缓冲液
2.向剩余的孔中添加75ul样品
3.向包含样品和对照的孔中添加75ul PLC底物。混合均匀,没有引入气泡。
4.在509EX/516EM上读取T0(也可以使用490EX/520EM)
5.覆盖平板并避光孵育
6.5小时后读数
表7.PLC活性测定条件
使用工业玉米醪(
表8.微量滴定板发酵反应条件
表9.来自SSF的磷脂酶酵母菌株酶活性测量和乙醇滴度
该实验的目的是确定酵母中表达磷脂酶是否会在发酵结束时提高乙醇产量。酵母菌株(HP21-H06、HP21-D05、HP21-G12、HP21-A08、HP21-F05和HP21-F04)繁殖过夜,然后用于发酵工业液化醪。在发酵结束时对发酵醪进行采样,将上清液过滤并通过HPLC分析,以确定最终的乙醇滴度和残余糖。
将酵母菌株作为甘油原液接收,并保持在-80℃冷冻直至使用。
按照以下步骤繁殖酵母菌株:
1.从-80℃冷冻器中取出酵母的冷冻小瓶。
2.对于每个样品,获得2x 50mL无菌锥形瓶并标记。
3.将烧瓶与6%YPD培养基,移液管和吸头以及冷冻管一起放入无菌罩中。
4.使用无菌技术,将约50mL 6%YPD培养基倒入每个瓶中。
5.使用200uL无菌移液管和无菌吸头将150uL适当的酵母样品添加到每个烧瓶中。
6.每次添加后将烧瓶加盖以保持无菌溶液。
7.将烧瓶置于32℃振荡培养箱中,并以约150rpm混合过夜。
然后根据以下程序测试上述繁殖浆液以测定用于加入的酵母细胞的数目:
1.从培养箱中取出繁殖的酵母样品。
2.将50mL样品倒入50mL倒装falcon管中。
3.将样品以3,500rpm离心7分钟。
4.将上清液丢弃到旧的50mL烧瓶中以进行高压灭菌。
5.向每个管中加入10mL去离子水。
6.将管涡旋混合以悬浮沉淀。
7.将重复样品合并到单个50mL falcon管中。
8.将样品以3,500rpm离心7分钟。
9.将上清液丢弃到最初的50mL烧瓶中以进行高压灭菌。
10.向每个剩余管中加入10mL去离子水。
11.将管涡旋混合以悬浮沉淀。
a.保存该浆液,并将其用于发酵。
12.通过向9.9mL去离子水中添加100uL,将酵母浆液100X稀释到15mL falcon管中。
13.使用
a.将50uL 100X浆液稀释液添加到450uL裂解缓冲液中。
b.将样品涡旋并放置约5分钟以发生细胞裂解。
c.使用核盒对裂解溶液进行采样,然后将其置于
d.
e.将结果数值乘以1000,得到用于电子表格的最终细胞计数。
14.该实验中使用的酵母以及根据上述程序获得的酵母计数显示在下表10中。
表10.
15.以下计算用于确定添加到每个管中的酵母浆液的mL数。
工业液化醪的发酵按照以下程序进行:
1.获得工业液化醪并冷冻保存,以供将来分析。本实验中使用的醪来自使用
2.在开始该研究之前,将两升的醪解冻约2小时。
3.使用200g/L尿素和1g/L青霉素的原液将醪调节至500ppm尿素和3ppm青霉素。
4.使用40%v/v H
5.将50.0+/-0.05g制备的醪称重到250mL培养基瓶中。
6.将瓶子封盖并在冰箱中储存过夜。
7.早上使用重复移液管向瓶中添加酶和酵母。
8.测试的条件包括
9.用钻孔盖将瓶子盖住。将孔用标记带覆盖。使用0.24规格的注射器针头刺穿均匀的通气孔,以允许CO2气体在发酵过程中逸出。
10.将瓶子置于设定为150rpm的32℃空气振荡器中以开始发酵。
11.发酵进行54小时。
1.发酵52小时后,用配备有切割尖端的5mL移液管将5g发酵醪移液到15mL锥形falcon管中。
2.向每个管中加入50uL 40%H
3.用涡旋混合器将管短暂混合,并以3,500rpm离心7分钟。
4.将上清液倒入装有0.45um过滤器的注射器中,并使用注射器的柱塞将样品推过过滤器,进入预先编号的HPLC小瓶中。
5.将HPLC小瓶加盖,并将样品进行HPLC以分析发酵过程中产生的乙醇和糖。HPLC设置如表11所示。
表11.
该方法使用用于糊精(DP4+)、麦芽三糖、麦芽糖、葡萄糖、果糖、乙酸、乳酸、甘油以及乙醇的校准标准品对分析物进行定量。使用4点校准(包括原点)。
在图1中显示了测试菌株的乙醇提高。菌株HP21-F05和HP21-F04显示出乙醇比对照菌株MHCT-484提高约1.1%,而菌株HP21-A08显示出比对照的乙醇滴度提高约0.5%。
该实验的目的是检测氮负荷(尿素水平)对表达磷脂酶的酵母的乙醇产量的影响。将酵母菌株(HP21-H08、HP21-F04和HP21-F05)繁殖过夜,然后用于发酵工业液化醪。在发酵结束时对发酵醪进行取样,将上清液过滤并在HPLC上运行,以测定最终乙醇滴度和残余糖。
如上文实例3中所述繁殖酵母菌株。
然后根据上文实例3中所述程序测试上述繁殖浆液以测定用于加入的酵母细胞的数目。该实验中使用的酵母以及相应的酵母计数显示于表12中。
表12.
工业液化醪的发酵按照以下程序进行:
1.获得工业液化醪并冷冻保存,以供将来分析。本实验中使用的两种醪来自使用
2.在开始该研究之前,将两升的每种醪解冻约2小时。
3.使用1g/L青霉素原液向醪中加入3ppm青霉素。使用100和20g/L的原液,在酶加入过程中将尿素添加到发酵中。
4.使用40%v/v H
5.将约5g制备的醪加入到预先称重的15mL管中
6.将管重新称重,并在计算酶,酵母和尿素的剂量中使用醪的重量。
7.将管封盖并在冰箱中储存过夜。
8.早上使用Biomek FX液体处理机器人向管中加入酶,酵母和尿素。
9.在该实验中测试的条件显示在下表13中。在该实验中的酵母剂量为每次发酵7,000,000个细胞。
10.使管盖上有孔,以允许从发酵释放出CO
11.将管置于32℃培养箱中开始发酵。
12.将管用涡旋混合器每天混合两次,持续52小时。
表13.
1.发酵52小时后,向每个管中加入50uL 40%H
2.用涡旋混合器将管短暂混合,并以3,500rpm离心7分钟。
3.将管置于Biomek液体处理器上,该处理器用于将200uL上清液从管中移液到96孔0.22μm滤板中。
4.将滤板置于圆底96孔聚丙烯板的顶部。
5.将过滤器和板置于在桶上具有生物密封盖的地面离心机中,并以3,500rpm旋转7分钟,或者直到液体通过96孔过滤板的所有孔为止。
6.将板从离心机中移出,并将具有过滤样品的圆底板加热密封并送至HPLC以分析发酵过程中产生的乙醇和糖。HPLC设置如表14所示。
表14.
该方法使用用于糊精(DP4+)、麦芽三糖、麦芽糖、葡萄糖、果糖、乙酸、乳酸、甘油以及乙醇的校准标准品对分析物进行定量。使用4点校准(包括原点)。图2显示了在0和150ppm尿素下AMP醪发酵54小时后的标准化平均乙醇值。
所有测试的表达磷脂酶的菌株在没有尿素负载的情况下显示出更大的乙醇滴度增加。图3显示了在0和150ppm尿素下非AMP醪发酵54小时后的标准化平均乙醇值。所有测试的表达磷脂酶的菌株在没有尿素负载的情况下显示出更大的乙醇滴度增加。
该实例描述了在酿酒酵母PGK1启动子的控制下,含有表1的剩余异源磷脂酶的酵母细胞的构建。设计含有启动子,基因和终止子的三片DNA,以允许4个DNA片段之间的同源重组并进入菌株MeJi797(表达α-淀粉酶和葡糖淀粉酶的MBG5012的衍生物;WO2019/161227)的XII-2位点。所得的菌株具有包含一个启动子的片段(左片段)、包含一个基因的片段(中片段)、以及一个PRM9终止子片段(右片段),将三者整合到XII-2位点处的酿酒酵母基因组中。
用引物1229945(5’-TCTTT TCGCG CCCTG GAAAG G-3’;SEQ ID NO:434)和1227122(5’-TGTTT TATAT TTGTT GTAAA AAGTA GATAA TTACT TCCTT GATGA TCTG-3’;SEQ ID NO:435)从HP27质粒DNA中PCR扩增含有与XII-2位点具有500bp同源性和酿酒酵母pPGK1启动子的线性DNA。各50皮摩尔的正向和反向引物用于PCR反应,该反应含有5ng质粒DNA作为模板,各0.1mM dATP、dGTP、dCTP、dTTP,1X Phusion HF缓冲液(赛默飞世尔科技公司),以及2个单位的Phusion热启动DNA聚合酶,最终体积为50μL。在T100
用引物1229946(5’-GTGAC AACAA CAGCC TGTTC TC-3’;SEQ ID NO:436)和1222995(5’-AGCTA ATGCG GAGGA TGCTG C-3’;SEQ ID NO:437)从GeneArt合成的DNA中PCR扩增含有与酿酒酵母pPGK1启动子(SEQ ID NO:4)具有243bp同源性和MF1α信号肽(SEQ IDNO:7)的线性DNA。各50皮摩尔的正向和反向引物用于PCR反应,该反应含有5ng质粒DNA作为模板,各0.1mM dATP、dGTP、dCTP、dTTP,1X Phusion HF缓冲液(赛默飞世尔科技公司),以及2个单位的Phusion热启动DNA聚合酶,最终体积为50μL。在T100
用引物1221473(5’-ACAGA AGACG GGAGA CACTA GC-3’;SEQ ID NO:438)和1229949(5’-GGGGT CGCAA CTTTT CCC-3’;SEQ ID NO:439)从TH12质粒DNA(GeneArt)中PCR扩增含有PRM9终止子的250bp和500bp XII-2 3'-末端同源性的DNA。各50皮摩尔的正向和反向引物用于PCR反应,该反应含有5ng质粒DNA作为模板,各0.1mM dATP、dGTP、dCTP、dTTP,1X Phusion HF缓冲液(赛默飞世尔科技公司),以及2个单位的Phusion热启动DNA聚合酶,最终体积为50μL。在T100
含有MF1α信号肽,磷脂酶基因和50bp PRM9终止子的合成线性未克隆DNA获自Geneart或Twist Bioscience。
用上述左、中和右整合片段转化酵母MeJi797。在每个转化池中,使用固定的左片段和右片段,每个片段100ng。中间片段由信号肽和磷脂酶基因组成,每个片段约100ng(总共700ng)。为了帮助左片段、中片段和右片段在基因组XII-2位点处的同源重组,还在转化中使用含有MAD7和对XII-2特异的指导RNA的质粒(pMlBa638)。按照酵母电穿孔方案,将这4种组分转化到酿酒酵母菌株MeJi797中。在YPD+cloNAT上选择转化体以选择含有Mad7质粒pMlBa638的转化体。使用Q-pix Colony Picking System(分子仪器公司)挑选转化体,以接种1孔含有YPD+cloNAT培养基的96孔板。使板生长2天,然后添加甘油至20%的终浓度,并将这些板储存在-80℃下直至需要。通过使用基因座特异性引物的PCR和随后的测序来验证特异性磷脂酶构建体的整合。产生的菌株用于以下实例。
使用下文描述的96孔玉米醪发酵测试实例5中描述的菌株的乙醇产量和残余葡萄糖。通过将10μL每种菌株接种到每孔含有150uL YP+2%葡萄糖培养基的96孔种子板中来制备繁殖板。将板在30℃和300RPM下孵育过夜。第二天,将10μL种子培养物转移到含有500μL补充有100ppm尿素和0.42AGU/g Spirizyme Excel的East Kansas Agri-EnergyLiquefact Amp玉米醪的96深孔板中。用EnzyScreen板盖将板密封,并紧紧夹住以限制氧转移。将玉米醪板在32℃下静态孵育56小时。发酵完成后,将板置于-80℃约10分钟,然后将100μL的8%H
表15.
从Trenton Agri获得商业Amp玉米醪,其干固体含量为35.9%(w/w),并用自来水稀释至32.0%(w/w)。稀释后,用39%(w/v)NaOH溶液将醪的pH值调节至5.1。将尿素和lactrol添加到pH调节的Amp醪中,使其最终浓度分别为150ppm和3ppm。将制得的玉米醪等分到250mL烧瓶中(100g/瓶)。
为了繁殖酵母,将50mL的6%YPD和100μl的酵母-甘油原液在125mL烧瓶中混合,然后在32℃下孵育过夜。孵育后,将45mL繁殖物转移至50mL离心管中,并以3500rpm离心10分钟。倾析出液体部分,用去离子水两次洗涤细胞。将细胞重悬于10mL去离子水中,并使用
根据DOE,将外源性α-葡糖淀粉酶(Spirizyme Achieve-T
与对照菌株MHCT-484相比,两种表达磷脂酶的酵母菌株均显示出最终乙醇产量的显著提高(对照菌株MHCT-484为13.30%,菌株HP21-F04和HP21-F05分别为13.43%和13.42%)。
从Trenton Agri获得商业Amp玉米醪,其干固体含量为35.9%(w/w),并用水稀释至32.0%(w/w)。稀释后,用39%(w/v)NaOH溶液将醪的pH值调节至5.1。将尿素和lactrol添加到pH调节的Amp醪中,使其最终浓度分别为150ppm和3ppm。将制得的玉米醪等分到250mL烧瓶中(100g/瓶)。
为了繁殖酵母,将50mL的6%YPD和100μL的酵母-甘油原液在125mL烧瓶中混合,然后在32℃下孵育过夜。孵育后,将45mL繁殖物转移至50mL离心管中,并以3500rpm离心10分钟。倾析出液体部分,用去离子水两次洗涤细胞。将细胞重悬于10mL去离子水中,并使用
根据DOE,将外源性α-葡糖淀粉酶(Spirizyme Achieve-T
提取油(g/烧瓶)=第一提取油(g/烧瓶)+第二提取油(g/烧瓶)
与对照菌株MHCT-484相比,表达脂肪酶的酵母菌株HP21-F04和HP21-F05均提高了最终油产量(菌株HP21-F04和HP21-F05分别为0.518g/烧瓶和0.966g/烧瓶,而对照菌株MHCT-484为0.483g/烧瓶)。
从Trenton Agri获得商业Amp玉米醪,其干固体含量为35.9%(w/w),并用自来水稀释至32.0%(w/w)。稀释后,用39%(w/v)NaOH溶液将醪的pH值调节至5.1。将尿素和lactrol添加到pH调节的Amp醪中,使其最终浓度分别为150ppm和3ppm。将制得的玉米醪等分到250mL烧瓶中(100g/瓶)。
为了繁殖酵母,将50mL的6%YPD和100μL的酵母-甘油原液在125mL烧瓶中混合,然后在32℃下孵育过夜。孵育后,将45mL繁殖物转移至50mL离心管中,并以3500rpm离心10分钟。倾析出液体部分,用去离子水两次洗涤细胞。将细胞重悬于10mL去离子水中,并使用
根据DOE,将外源性α-葡糖淀粉酶(Spirizyme Achieve-T
发酵12小时后,使用摄像机监测对照MHCT-484和表达脂肪酶的酵母HP21-F04的消泡能力。与对照yMHCT48相比,表达脂肪酶的酵母HP21-F04具有显著的消泡能力(图4)。
- 共表达外源糖化酶的酵母菌株、获得所述酵母菌株的方法及其用于生产生物乙醇的用途
- 用于生产乙醇的表达酶的酵母
