冠状病毒的检测方法及试剂盒
文献发布时间:2023-06-19 13:48:08
技术领域
本发明属于分子生物学检测技术领域,具体而言,涉及一种可以检测多种冠状病毒的检测方法并提供一种适用于所述检测方法的试剂盒,所述多种冠状病毒包括新型冠状病毒SARS-CoV-2分子分型与变异株。
背景技术
冠状病毒(Coronaviruses,CoVs)属于冠状病毒科,是自然界广泛存在、能够感染人和动物的一大类病毒家族,在电镜下观察其病毒粒子表面有类似日冕状的纤突而被命名。冠状病毒的基因组较大,其单股正链基因序列长达26~32kb,是已知自然界序列最长的RNA病毒。由严重急性呼吸综合征冠状病毒2型(Severe acute respiratory syndromecoronavirus 2,SARS-CoV-2)引起的持续流行性新型冠状病毒肺炎(Coronavirus disease2019,COVID-19)疫情最初爆发于2019年12月下旬,如今对全球公共卫生和经济构成了前所未有的威胁。SARS-CoV-2感染主要引起以肺部病变为主的呼吸道传染病,还可引起急性心肌损伤和急性呼吸窘迫综合征等严重的并发症,甚至导致死亡。尽管2003年出现的严重急性呼吸综合征冠状病毒(Severe acute respiratory syndrome coronavirus,SARS-CoV)和2012年出现中东呼吸道综合征冠状病毒(Middle east respiratory syndromecoronavirus,MERS-CoV)感染的死亡率比SARS-CoV-2高,但SARS-CoV-2的传播速度远高于MERS-CoV和SARS-CoV。截至2021年08月17日,已有200多个国家受到该病毒的影响,尽管采取了所有干预和控制措施,实验室确诊的COVID-19病例数仍在增加,有超过2亿感染确诊例,死亡人数超过400万(
在全球疫情爆发的情况下,尽早(最好是在症状出现的最早阶段)地快速检测SARS-CoV-2的感染对控制病毒的传播具有至关重要的作用,尤其是SARS-CoV-2感染者的临床表现与流感等其他季节性呼吸道感染患者的临床表现非常相似,难以区分。
因此,迫切需要一种特异、敏感的SARS-CoV-2的检测、鉴定和筛查方法,以便在患者出现临床症状前的早期确定COVID-19阳性病例。目前,SARS-CoV-2的检测主要采用两种诊断策略:一是通过分子诊断方法检测呼吸道标本(鼻咽拭子、咽拭子、唾液、痰液和下呼吸道分泌物)中的病毒RNA;二是使用血液样本检测特定抗体的血清学或抗体测试。血清学检测可监测患者在急性期(发病第一周)和恢复期(发病后2-4周)的免疫反应和抗体生成。因此血清型检测特别适用于对COVID-19感染的回顾性研究,而不适用于SARS-CoV-2早期感染的鉴定。自SARS-CoV-2基因组序列信息首次公布以来,一系列核酸扩增方法(Nucleic acidamplification testing,NAAT)凭借耗时短、操作简便、自动化等优点快速建立起来用于SARS-CoV-2病毒RNA的检测,包括下一代测序(NGS)、实时定量逆转录聚合酶链反应(RT-PCR)、环介导等温扩增(LAMP)、重组酶聚合酶扩增(RPA)和基于规则间隔短回文重复序列(CRISPR)机制的方法。在新冠疫情爆发初期,在不清楚致病病原体的情况下,NGS技术及时地识别和发现了病原体SARS-CoV-2。除了用于确认疑似COVID-19病例外,NGS技术还可以帮助我们监测SARS-CoV-2基因组中可能发生的突变,这对于了解SARS-CoV-2病毒的致病机制和进化具有重要意义。虽然NGS的价格逐年在降低,但是成本仍然很高,而且整个运行程序繁琐(包括耗时复杂的文库制备和数据处理),需要专业的人员进行数据分析,并且一次可处理的样本有限,这些因素极大地限制了其在大规模爆发疫情中的应用。实时RT-PCR检测SARS-CoV-2是一种特异性强、灵敏度高的检测方法,被世界卫生组织推荐为有效、简便的方法,同时也是当前研究和临床实验室鉴定COVID-19大流行的金标准方法。虽然实时RT-PCR检测已得到广泛应用,但全球范围内检测需求的增加导致试剂和相关耗材的短缺,从而影响到越来越多疑似COVID-19感染的诊断。为了满足全球对SARS-CoV-2诊断的空前需求,许多替代方法,如LAMP、RPA和CRISPR,已经被开发出来,以实现对病毒RNA的同等的检测水平。虽然与NGS相比,这些检测方法能更快、更简便、成本更低廉和使用更少的时间来确诊COVID-19,但它们不能提供SARS-CoV-2的亚型和突变信息。因此,迫切需要开发和建立一种综合的诊断方法,不仅可用于病毒鉴定,而且还可直接用于SARS-CoV-2基因组的分型和突变检测,及时为疫情防控提供信息。
质谱法(mass spectrometry,MS)是一种通过测量待测物离子的质荷比(m/z)从而实现对样品的定性和定量分析与鉴定的方法。质谱仪的基本工作原理是:首先样品中各组分的分子在离子源中发生电离,生成不同质荷比的离子(带正电荷或负电荷);然后经过加速引出和传输调制电场,形成离子束,进入质量分析器;在质量分析器中,再利用电场和磁场对不同质荷比的离子的作用力不同,将他们按照质荷比的大小分别聚焦到离子探测器上,最后由信号记录系统得到这些离子的质谱图。随着电喷雾电离(electrosprayionization,ESI)和基质辅助激光解吸电离(matrix-assisted laser desorption/ionization,MALDI)离子源的发明,质谱在有机生物分子的鉴定方面发挥出越来越重要的作用。现已逐步走入临床,给临床检验带来了一场新的技术革命。临床医学质谱相比于传统的免疫与生化法,具有检测速度快,样本分析能力强,平台拓展性强等优势,因此广泛应用于序列分析、基因分型、单核苷酸多态性分析以及临床检测等领域。
发明内容
本发明的目的是提供一种特异性强、灵敏度高,快速,准确并可检测多种冠状病毒的检测方法以及相应的试剂盒。本发明进一步提供与本发明检测方法相关的引物和探针,这些引物和探针具有高的特异性。
本发明所述多种冠状病毒的检测方法,包括以下步骤:
1)、样品采集;
2)、提取样品中的病毒RNA;
3)、以病毒RNA为模板,进行PCR扩增;
4)、对PCR扩增产物使用质谱仪进行检测,并对结果进行判定。
其中,步骤1)所述样品采集中的样品即待测样本,其来源包括,人鼻咽拭子、唾液、痰液样本、肺泡灌洗液样本、血液样本、下呼吸道分泌物、粪便样本或环境样本。
其中,步骤2)、提取样品中的病毒RNA,可以采用任何一种已知的提取方法,如:Qiagen试剂盒提取法,Trizol LS有机溶剂提取法,磁珠提取法。
其中,步骤3)所述PCR扩增,需要使用引物和探针,为避免漏检,本发明给出了可检测多种冠状病毒的引物和探针,这些引物和探针可用于检测7种人冠状病毒,它们是:人冠状病毒229E、NL63、HKU1、OC43、SARS-CoV、MERS-CoV以及SARS-CoV-2。
其中,SARS-CoV-2病毒包括S、L、V、G、GH、GR、GV与GRY分型,以及英国变异株、南非变异株、巴西变异株。
本发明所述引物与探针,其序列为SEQ ID NO.1-SEQ ID NO.63所示。
其中,用于检测人冠状病毒229E的专用引物与探针,为SEQ ID NO.28-SEQ IDNO.30所示。
其中,用于检测人冠状病毒NL63的专用引物与探针,为SEQ ID NO.25-SEQ IDNO.27所示。
其中,用于检测人冠状病毒HKU1的专用引物与探针,为SEQ ID NO.55-SEQ IDNO.57所示。
其中,用于检测人冠状病毒OC43的专用引物与探针,为SEQ ID NO.58-SEQ IDNO.60所示。
其中,用于检测严重急性呼吸综合征冠状病毒(SARS-CoV)的专用引物与探针,为SEQ ID NO.52-SEQ ID NO.54所示。
其中,用于检测中东呼吸综合征冠状病毒(MERS-CoV)的专用引物与探针,为SEQID NO.40-SEQ ID NO.42所示。
其中,用于检测新型冠状病毒(SARS-CoV-2)的专用引物与探针,为SEQ ID NO.13-SEQ ID NO.15及SEQ ID NO.49-SEQ ID NO.51所示。
其中,用于检测SARS-CoV-2分子分型与重要变异株的专用引物与探针,为SEQ IDNO.1-SEQ ID NO.9,SEQ ID NO.16-SEQ ID NO.24,SEQ ID NO.31-SEQ ID NO.39,SEQ IDNO.43-SEQ ID NO.48,SEQ ID NO.61-SEQ ID NO.63所示。
其中,检测SARS-CoV-2的分子分型与重要变异株,是指能准确将SARS-CoV-2分成S、L、V、G、GH、GR、GV和GRY型别,以及3种重要的变异株,分别为南非变异株、巴西变异株,英国变异株。
本发明涉及21个靶点,每个靶点涉及一套引物组,每个引物组由两条引物组成,一条为正向引物,一条为反向引物。由此,本发明共有21套引物组与探针,分别针对21个检测靶点实现多重检测反应。
21个靶点和引物及探针的对应关系如下:
引物名称及序列信息见下表。
表1引物与探针序列信息
本发明步骤3)所述PCR扩增,采用一步法多重PCR技术,可实现多重检测反应,基本方法如下:
RNA逆转录酶对提取的RNA样本进行逆转录之后,使用Taq酶对逆转录产物进行PCR扩增。使用虾碱性磷酸(SAP)对PCR产物进行处理,经单碱基延伸反应后进行样本点样。
优选的,方法如下:
使用含有RNA逆转录酶与Taq酶的混合反应体系,对提取的病毒RNA进行含7种人冠状病毒,及新型冠状病毒的型别与变异株的一步法检测。同时与上述处理方法一致,使用SAP对扩增的PCR产物进行处理后,经单碱基延伸反应后进行样本点样。
最优选的,方法如下:
根据QIAamp Viral RNA Mini kit试剂盒操作说明提取样本中的病毒RNA,同时向RNA保存管中加入RNA酶抑制剂。(以上步骤可用其他核酸提取试剂盒或方法完成病毒RNA的提取)。多重PCR反应:使用以上步骤获得的病毒RNA样本作为模板,加入10μL多重PCR反应体系包括:2.6μL的各个引物最佳扩增浓度的混合体系,5μL的2×Reaction Mix,0.4μL
反应产物点样:反应产物点样:核酸质谱点样的基质配制:使用乙腈溶液将3-羟基-2-吡啶甲酸(3-hydroxypicolinic acid,3HPA)配制成50mg/mL;柠檬酸氢二胺(Ammonium citrate dibasic,ACD)配制成终浓度250mM/mL;最后,3HPA:ACD按照10:1的体积比配制为最终使用的核酸质谱点样基质溶液。基质包被:将约1μL基质溶液包被在质谱检测靶板中央,室温静置约2分钟。核酸样本点样:将1μL(3)中收集获得的反应产物,点样至已包被基质的靶板,室温静置约2分钟后,准备使用质谱仪进行检测。
本发明步骤4)、对PCR扩增产物使用质谱仪进行检测,并对结果进行判定,包括使用质谱仪检测PCR反应产物,检测方法如下:
经上述处理获得的PCR反应产物,与核酸质谱点样基质充分混合静置。使用质谱仪检测样本中各组分的分子在离子源中发生电离,生成不同质荷比的例子;通过质谱仪的质量分析器分析经加速引出和传输调制电场形成的电子束;利用电场和磁场对不同质荷比的离子作用不同,将其按照质荷比大小聚焦到离子探测器上,由质谱仪的信号记录系统得到这些离子的质谱图。优选的,方法如下:
AXIMA质谱仪对处理的核酸样本进行检测,使用仪器配套软件进行质谱检测结果分析。
最优选的,方法如下:
使用AXIMA质谱仪对经多重PCR反应,SAP反应,单碱基延伸反应,与树脂纯化后的病毒RNA核酸样本进行检测。同时使用本发明设计的MALDI-MS软件分析检测结果;若检测结果为阳性,则会在相应靶基因的阳性产物荷质比处出现特异的产物峰;若检测结果为阴性,则会在相应靶基因的初始荷质比处出现特异的产物峰;综合各个靶标的阳性特异产物峰的检测情况,判定样本的人冠状病毒检测情况,确定SARS-CoV-2的分子型别与位点突变情况。本发明在上述检测方法的基础上,进一步提供用于检测冠状病毒的试剂盒。
本发明的试剂盒,可以检测7种人冠状病毒,并且可以对其中的SARS-CoV-2病毒的分型及变异株进行检测。本发明所述的试剂盒,包括本发明所述的引物和探针。
本发明所述的试剂盒,根据需要,还可以包括检测用具和试剂,如PCR所需的试剂如溶剂,酶,对照品,所述用具可以包括,使用说明书,棉签,支架,试剂瓶,试纸等检测工作中必要的配套产品,具体如:反应buffer混合液,RNA逆转录酶与聚合酶,虾碱性磷酸酶反应(Shrimp alkaline phosphatase,SAP)组分(SAP buffer和SAP酶),单碱基延伸反应组分(单碱基延伸buffer,单碱基延伸酶);
阳性对照和阴性对照,所述阳性对照为包含各检测靶点的阳性样本,或包含检测靶点的野生型或突变型的阳性样本,所述阴性对照为正常人RNA核酸样本。
本发明的试剂盒中,所述引物和探针,包括用于检测人冠状病毒229E的引物和探针,其序列为SEQ ID NO.28-SEQ ID NO.30。
本发明所述的试剂盒,还包括用于检测人冠状病毒NL63的引物和探针,其序列为SEQ ID NO.25-SEQ ID NO.27。
本发明所述的试剂盒,还包括用于检测人冠状病毒HKU1的引物和探针,其序列为SEQ ID NO.55-SEQ ID NO.57所示。
本发明所述的试剂盒,还包括用于检测人冠状病毒OC43的引物和探针,其序列为SEQ ID NO.58-SEQ ID NO.60所示。
本发明所述的试剂盒,还包括用于检测人冠状病毒严重急性呼吸综合征冠状病毒(SARS-CoV)的引物和探针,其序列为SEQ ID NO.52-SEQ ID NO.54所示。
本发明所述的试剂盒,还包括用于检测中东呼吸综合征冠状病毒(MERS-CoV)的引物和探针,其序列为SEQ ID NO.40-SEQ ID NO.42所示。
本发明所述的试剂盒,还包括用于检测SARS-CoV-2病毒的引物和探针,其序列为SEQ ID NO.13-SEQ ID NO.15及SEQ ID NO.49-SEQ ID NO.51所示。
本发明所述的试剂盒,还包括用于检测SARS-CoV-2病毒分子分型与变异株的引物和探针,其序列为SEQ ID NO.1-SEQ ID NO.9,SEQ ID NO.16-SEQ ID NO.24,SEQ IDNO.31-SEQ ID NO.39,SEQ ID NO.43-SEQ ID NO.48,SEQ ID NO.61-SEQ ID NO.63所示。
本发明所述的试剂盒,包括SEQ ID NO.1-SEQ ID NO.63所示的引物及探针。
本发明所述的试剂盒,其中的引物及探针,以及所述试剂和用具,可以是满足检测1人次样本的用量,也可以是,满足多人次样本的用量,如5人次,10人次,20人次,50人次,100人次,1000人次等,以可以满足筛查需要来确定具体剂量。
以1人次样本的用量为例,其中:
SEQ ID NO.1-SEQ ID NO.63所示的引物及探针,各自的用量及配制方法如下:
1)引物配制:将每一条引物稀释成50μM,每一条引物取10μL上述浓度的引物进行混合,使用无核酸水补齐至1mL,即为1人次样本使用的多重PCR反应中所使用的引物混合液,每人次用量为2.6μL。
2)延伸探针配制:将每一条探针稀释成250μM,每一条探针加入体积按照如下公式进行计算:V=(InM-7.82)*9/0.94*4,其中M为探针的分子量,V为计算得到的每一种探针的理论加入量,单位为μL。对于分子量小于6000dalton的探针,按照理论加入量混合;分子量大于6000dalton的探针,按照理论加入量的1.5倍进行混合。混合完成的探针,使用无核酸酶水补齐至1mL,即为1人次样本使用的单碱基延伸反应使用的探针混合液,每人次用量为0.94μL。
本发明进一步包括本发明的试剂盒的应用,本发明所述的应用,包括以下步骤:
使用病毒采集管收集病人呼吸道标本(鼻咽拭子、咽拭子、唾液、痰液和下呼吸道分泌物)。根据QIAamp Viral RNA Mini kit试剂盒操作说明提取样本中的病毒RNA,洗脱体积为试剂盒推荐体积50μL,同时向RNA保存管中加入RNA酶抑制剂。(以上步骤可用其他核酸提取试剂盒或方法完成病毒RNA的提取)。
多重PCR反应:使用以上步骤获得的病毒RNA样本作为模板,加入10μL多重PCR反应体系包括:2.6μL的各个引物最佳扩增浓度的混合体系,5μL的2×Reaction Mix,0.4μL
质谱检测:使用AXIMA质谱仪检测上述反应产物,MALDI-MS软件分析检测结果。
结果判读:检测结果的判定。若检测结果为阳性,则会在相应靶基因的阳性产物荷质比处出现特异的产物峰(阳性产物荷质比见表2);若检测结果为阴性,则会在相应靶基因的初始荷质比处出现特异的产物峰(初始产物荷质比见表2)。综合各个靶标的阳性特异产物峰的检测情况,判定样本的人冠状病毒检测情况,确定SARS-CoV-2的分子型别与位点突变情况。
表2检测靶标的初始及阳性荷质比
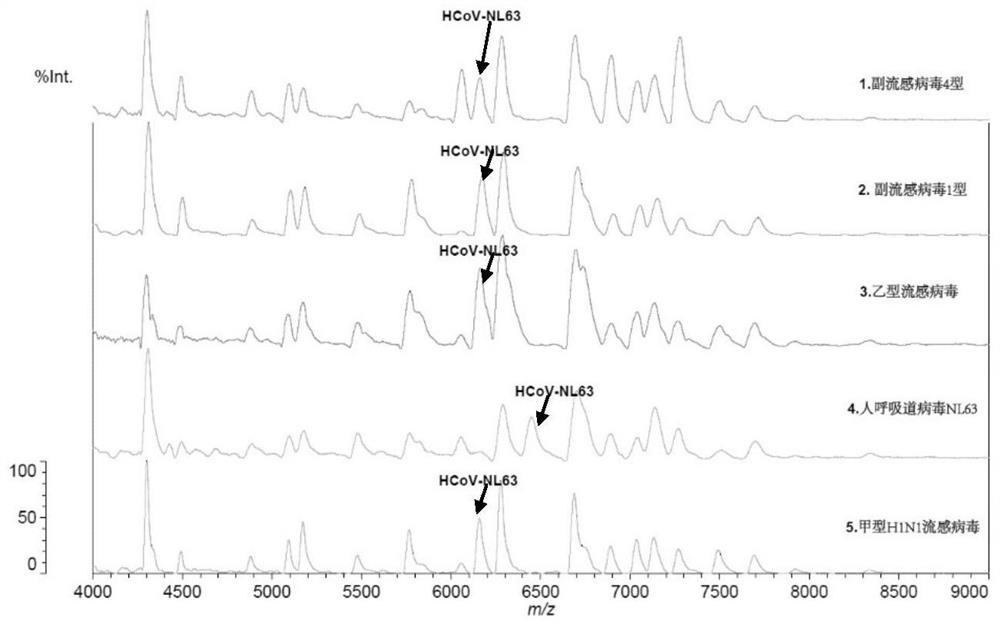
本发明进一步提供一种同时展示多个样本质谱检测峰图的分析方法,包括本发明的MALDI-MS质谱结果分析软件,可同时呈现不同样本中7种人冠状病毒及SARS-CoV-2的型别及变异株的检出情况,便于同时呈现多个样本的分析结果(图1)。
本发明的MALDI-MS质谱分析软件,包括以下计算模块:
1、使用AXIMA质谱仪器配套的结果分析软件MALDI-MS,进行质谱检测结果分析;
2、使用软件中的“Load Data”模块,选取待展示的结果(可同时展示10个质谱检测结果);
3、使用结果分析中的“Display contents”程序,对展示的结果进行统一化处理,设置程序为“Process”,“Stack”与“Set 1-5”,即可实现多个样本的同一坐标的拟合分析。
这些模块的集合应用,可实现多个样本拟合为同一坐标的比较分析,用于样本与阴性对照或其他样本的比较(图2)。
本发明的引物和探针是经过筛选,设计,合成,应用,并取得良好效果后最终确定的,是选取种间特异、种内保守的基因或区域作为7种人冠状病毒检测的靶基因(包括SARS-CoV-2的ORF1a基因和N基因,及另外6种人冠状病毒的RdRp基因),选取能准确将SARS-CoV-2分成S、L、V、G、GH、GR、GV、与GRY型别,鉴定新型冠状病毒重要变异株(英国变异株、南非变异株、巴西变异株)的相关突变位点区域(
作为SARS-CoV-2病毒分子分型及变异株鉴定的检测靶标,同时选择人RNase P基因作为内对照用于监测样本核酸提取情况。首先从GenBank数据库(https://www.ncbi.nlm.nih.gov/genbank/)和GISAID数据库(https://www.gisaid.org)中下载经过完整注释的7种人冠状病毒作为参考序列的代表株基因序列,将参考序列与NCBI的nr数据库进行核酸序列BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi),下载比对得到的结果,获取更多检测靶基因序列。使用引物设计软件针对上述分析得到的靶基因保守区域序列设计特异性的扩增引物与探针,设计结果必须满足每一个靶基因,检测试剂各项标准的罚分不能够超过软件设定界值的条件。如果不满足该条件,需要根据软件提示,重新对设计不理想靶基因的保守区进行选择。经过多次尝试,最终获得能准确检测7种人冠状病毒及用于SARS-CoV-2分型的突变位点与变异位点的最佳引物与探针。使用NCBI提供的在线引物验证工具Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer blast/)对试剂进行评价,验证引物的特异性。
引物及探针设计:使用引物设计软件针对上述分析得到的靶基因保守区域序列设计特异性的扩增引物与探针,设计结果必须满足每一个靶基因,检测试剂各项标准的罚分不能够超过软件设定界值的条件。如果不满足该条件,需要根据软件提示,重新对设计不理想靶基因的保守区进行选择。经过多次尝试,最终获得能准确检测7种人冠状病毒及用于SARS-CoV-2分型的突变位点的最佳引物与探针。
多重检测引物组及探针的确定:使用NCBI提供的在线引物验证工具Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer blast/)对试剂进行评价,验证引物的特异性。选择能准确鉴定7种人冠状病毒,区分SARS-CoV-2野生型和突变型的最佳引物对与探针组合,形成最终的单孔21重的质谱分析体系。
本发明的引物及探针,其序列是全新的,它们的设计是根据以上描述的方法设计的,它们的制备方法可以采用例如Oligo DNA的人工化学合成制备方法,或采用β-乙腈亚磷酰胺化学合成Oligo DNA的制备方法。
这些制备方法本身是常规的,可以参考分子克隆实验指南教科书。
本发明所述的21种特异的检测靶标包括:取种间特异、种内保守的区域(RdRp基因)作为用于人冠状病毒229E,NL63,HKU1,OC43,严重急性呼吸综合征冠状病毒(SARS-CoV),以及中东呼吸综合征冠状病毒(MERS-CoV)6种冠状病毒检测的靶基因;种间特异、种内保守的基因或区域(包括ORF1a基因和N基因)作为SARS-CoV-2病毒检测的靶基因;12个不同的靶向区域覆盖T8782G,C28144T,G11083T,G26144T,C241T,C3037T,A23043G,G28882A,G25563T,A23063T,G23012A,C22227T 12个重要的突变位点用于SARS-CoV-2的分子分型及重要突变病毒株检测,实现SARS-CoV-2病毒S、L、V、G、GH、GV、GR和GRY共8种型别的准确区分及3种重要变异株的检测(英国变异株,南非变异株,与巴西变异株);在检测体系中纳入人RNase P基因作为内参对照,用于监测样本核酸提取情况。本发明选取种间特异、种内保守的基因或区域作为7种人冠状病毒检测的靶基因(包括SARS-CoV-2的ORF1a基因和N基因,及另外6种人冠状病毒的RdRp基因),选取能准确将SARS-CoV-2分成S、L、V、G、GH、GV、GR、GRY型别,鉴定新型冠状病毒重要变异株(英国变异株、南非变异株、巴西变异株)的相关突变位点区域(
作为SARS-CoV-2病毒分子分型的检测靶标,同时选择人RNase P基因作为内对照用于监测样本核酸提取情况。
本发明方法,能够快速、特异和灵敏地从临床样本中检测7种人冠状病毒,并对SARS-CoV-2病毒进行分子分型与重要变异株检测。与现有检测人冠状病毒与SARS-CoV-2的其它技术相比,本发明方案具有如下优点:
首先,该方法首次对整个检测反应进行了改良和优化,使用一步法多重PCR技术与核酸质谱检测分析联用,实现从临床样本核酸提取后直接进行检测和分型,不需要额外进行RNA的逆转录,极大的缩短了检测时间和减小试剂的消耗;本次使用的SAP处理PCR反应产物,消化掉剩余的dNTP,相比以往使用试剂,节约了反应时间,降低了反应成本;其次,与传统的PCR或其他分子检测技术相比,质谱鉴定依据的是分析物的分子量,通过合理的选择和设计,适当添加修饰碱基,可以将延伸探针的质量加以区分,从而达到一次性检测多重靶基因的目的,有效的将提高了目的基因检出效率;该方法可根据需求对检测靶标进行组合搭配,根据需要灵活地增减试剂的设计方案,为后续SARS-CoV-2病毒其他变异株的检测提供了极大的便利;使用MALDI-MS软件分析质谱检测结果,突破性的实现多个核酸样本检测结果的呈现与同一坐标的分析比较,为样本间检测结果的比较带来了便利。
以下对于文中出现的术语作进一步的解释和说明:
引物:用于扩增目的基因的两段寡核苷酸序列
探针:单碱基延伸反应中用于目的基因检测的核酸序列
PCR扩增:聚合酶链式反应,一种用于放大扩增特定的DNA片段的分子技术
一步法多重PCR技术:包含RNA逆转录酶和DNA聚合酶的反应体系,可以实现一个反应中同时完成RNA的逆转录与多重PCR扩增反应的技术
RNA的逆转录:以RNA为模板合成DNA,合成的DNA链为与RNA互补的DNA SAP处理PCR反应产物,消化掉剩余的dNTP:使用虾碱性磷酸酶清除一步法多重PCR反应体系中多余的单核苷酸,避免影响后续反应
适当添加修饰碱基,可以将延伸探针的质量加以区分:扩增引物中添加修饰碱基(ACGTTGGATG),避免引物对检测结果分析的干扰
Rpm:转速
PCR:聚合酶链式反应
SAP:虾碱性磷酸酶
扩增buffer:一步法扩增反应缓冲液
Reaction Mix:反应预混液
QIAamp Viral RNA Mini kit:病毒RNA提取试剂盒
dNTP:一步法扩增反应的寡核苷酸
单碱基延伸buffer:单碱基延伸反应使用缓冲液
附图说明:
图1、多个样本的7种人冠状病毒及SARS-CoV-2分子分型及变异株检测结果。
图2、同一坐标展示7种人冠状病毒及SARS-CoV-2分子分型及变异株检测结果。注:红色:含SARS-CoV-2病毒株突变位点的RNA核酸样本;蓝色:正常人RNA核酸样本。
具体实施方式
以下通过实施例进一步说明本发明。实施例是基于本发明的前提下进行实施,给出了详细的实施方式和具体的操作过程。此处所描述的具体实施方式和操作过程仅用于解释本发明,但本发明的保护范围不限于下述的实施例。下面就临床核酸样本使用本发明进行7种人冠状病毒检测,及SARS-CoV-2病毒分子分型与变异株检测实施例的实施方式和具体操作过程阐述如下。
实施例1,检测案例:
样品采集:
使用专用的病毒采集管收集病人呼吸道标本(鼻咽拭子、咽拭子、唾液、痰液和下呼吸道分泌物)。
RNA提取:
根据QIAamp Viral RNA Mini kit试剂盒操作说明提取样本中的病毒RNA,洗脱体积为试剂盒推荐体积50μL,同时向RNA保存管中加入RNA酶抑制剂。(以上步骤可用其他核酸提取试剂盒或方法完成病毒RNA的提取)
多重PCR反应(使用本发明实施例2的试剂盒),使用以上步骤获得的病毒RNA样本作为模板,加入10μL多重PCR反应体系包括:2.6μL的各个引物最佳扩增浓度的混合体系,5μL的2×Reaction Mix,0.4μL
反应产物点样:核酸质谱点样的基质配制:使用乙腈溶液将
质谱检测:使用AXIMA质谱仪检测上述反应产物,MALDI-MS软件分析检测结果。
结果判读:检测结果的判定。若检测结果为阳性,则会在相应靶基因的阳性产物荷质比处出现特异的产物峰(阳性产物荷质比见表2);若检测结果为阴性,则会在相应靶基因的初始荷质比处出现特异的产物峰(初始产物荷质比见表2)。综合各个靶标的阳性特异产物峰的检测情况,判定样本的人冠状病毒检测情况,确定SARS-CoV-2的分子型别与位点突变情况(如图2)。
其中,所述引物与探针在多重PCR反应体系中的终浓度为各引物的最终浓度为50μM,各个探针的终浓度为250μM。
实施例2,
试剂盒的组成案例:
1、引物与探针混合液:将21对引物均稀释成50μM,每一条引物取10μL上述浓度进行混合,使用无核酸水补充至1mL。,每一条探针稀释成250μM,每一条探针按照公式:V=(InM-7.82)*9/0.94*4算得每一种探针的理论加入量,单位为μL,M为分子质量,单位为dalton。其中分子量小于6000dalton的探针,按照理论加入量混合;分子量大于6000dalton的探针,按照理论加入量的1.5倍进行混合,并使用无核酸水补充至1mL。混合后的引物及探针分别在多重PCR与单碱基延伸反应中使用。
2、反应体系组成:一步法多重PCR反应体系包含2×Reaction Mix,
3、核酸点样基质:50mg/mL的
实施例3、
试剂盒的性能测试
1、准确性验证
选择5株感染细胞后的SARS-CoV-2分离株和2份阳性患者临床标本中提取的RNA,参照上述实施例1的方法和步骤进行测试,结果显示,这些样本可以准确鉴定出两个SARS-CoV-2检测靶基因,并判定这些样本为SARS-CoV-2阳性;而根据同孔用于检测SARS-CoV-2分型与变异株的12个突变位点检测结果,将这些样本分为不同的型别。同时使用PCR-测序方法对这些样本的12个标记突变位点进行测序,结果显示该方法的分型结果与PCR-测序方法获得的分型结果一致。综上所述,说明该方法具有良好的检测和分型性能。
2、特异性验证
选取呼吸道感染常见的病原体评估该方法的特异性,选择的病原体包括腺病毒、肠道病毒、博卡病毒、偏肺病毒A型和B型、鼻病毒、甲型H1N1流感、甲型H3N2流感、乙型流感、副流感病毒(1型至4型)、呼吸道合胞病毒(A型和B型)、嗜肺军团菌、百日咳杆菌、肺炎支原体、肺炎衣原体、流感嗜血杆菌、金黄色葡萄球菌、卡他莫拉菌、肺炎克雷伯菌、铜绿假单胞菌、鲍曼不动杆菌、大肠埃希菌、肺炎链球菌和脑膜炎奈瑟菌,参照上述实施例1的方法和步骤进行测试,结果显示,该方法包含的21重检测引物组与探针,对所有测试的病原体无交叉反应,表明该方法的特异性好。
3、灵敏度检测
使用倍比稀释的梯度阳性质粒来进行灵敏度的评估,参照上述实施例1的方法和步骤进行测试,每个稀释度重复测试3次,结果显示,该方法检测的所有靶点均在10拷贝/反应时可稳定检出。
序列表
<110> 中国医学科学院病原生物学研究所
<120> 一种新型冠状病毒SARS-CoV-2检测和分子分型方法及试剂盒
<130>
<160> 4
<210> 1
<211> 30
<212> RNA
<213> SARS-CoV-2_N-28882
<400> 1
acgttggatg caaagcaaga gcagcatcac 30
<210> 2
<211> 35
<212> RNA
<213> SARS-CoV-2_N-28882
<400> 2
Acgttggatg ttcgcccaca tgagggacaa ggaca 35
<210> 3
<211> 14
<212> RNA
<213> SARS-CoV-2_N-28882
<400> 3
Ccaggcagca gtag 14
<210> 4
<211> 30
<212> RNA
<213> SARS-CoV-2_S-23063
<400> 1
Acgttggatg acagttgctg gtgcatgtag 30
<210> 5
<211> 31
<212> RNA
<213> SARS-CoV-2_S-23063
<400> 2
acgttggatg ctttacaatc atatggtttc c 31
<210> 6
<211> 15
<212> RNA
<213> SARS-CoV-2_S-23063
<400> 3
Ggtttccaac ccact 15
<210> 7
<211> 30
<212> RNA
<213> SARS-CoV-2_S-22227
<400> 1
Acgttggatg tacctattgg caaatctacc 30
<210> 8
<211> 30
<212> RNA
<213> SARS-CoV-2_S-22227
<400> 2
Acgttggatg tttagtgcgt gatctccctc 30
<210> 9
<211> 16
<212> RNA
<213> SARS-CoV-2_S-22227
<400> 3
Cctcagggtt tttcgg 16
<210> 10
<211> 30
<212> RNA
<213> RNase_P
<400> 1
Acgttggatg tgagcggctg tctccacaag 30
<210> 11
<211> 30
<212> RNA
<213> RNase_P
<400> 2
Acgttggatg tggcggtgtt tgcagatttg 30
<210> 12
<211> 16
<212> RNA
<213> SARS-CoV-2_ORF1a
<400> 3
Cagagcgggt tctgac 16
<210> 13
<211> 30
<212> RNA
<213>
<400> 1
Acgttggatg aattggaacg ccttgtcctc 30
<210> 14
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF1a
<400> 2
Acgttggatg ttggttcacc gctctcactc 30
<210> 15
<211> 17
<212> RNA
<213> SARS-CoV-2_ORF1a
<400> 3
Ctctcactca acatggc 17
<210> 16
<211> 30
<212> RNA
<213> SARS-CoV-2_S-23403
<400> 1
Acgttggatg ttctaaccag gttgctgttc 30
<210> 17
<211> 30
<212> RNA
<213> SARS-CoV-2_S-23403
<400> 2
Acgttggatg acacgccaag taggagtaag 30
<210> 18
<211> 18
<212> RNA
<213> SARS-CoV-2_S-23403
<400> 3
Acttctgtgc agttaaca 18
<210> 19
<211> 30
<212> RNA
<213> SARS-CoV-2_5'UTR-241
<400> 1
Acgttggatg ccatcttacc tttcggtcac 19
<210> 20
<211> 30
<212> RNA
<213> SARS-CoV-2_5'UTR-241
<400> 2
Acgttggatg actcgtctat cttctgcagg 30
<210> 21
<211> 19
<212> RNA
<213> SARS-CoV-2_5'UTR-241
<400> 3
Catcagcaca tctaggttt 19
<210> 22
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF1ab-3037
<400> 1
Acgttggatg ggagtatggc tacatactac 30
<210> 23
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF1ab-3037
<400> 2
Acgttggatg tcttcttcat cctcatctgg 30
<210> 24
<211> 20
<212> RNA
<213> SARS-CoV-2_ORF1ab-3037
<400> 3
Cccccctcat ctggagggta 20
<210> 25
<211> 30
<212> RNA
<213> HCoV-NL63
<400> 1
Acgttggatg ctgaacttgt ttatgagaac 30
<210> 26
<211> 30
<212> RNA
<213> HCoV-NL63
<400> 2
Acgttggatg tgtcaacctg tacattaccc 30
<210> 27
<211> 20
<212> RNA
<213> HCoV-NL63
<400> 3
Ttactagcag gtttaacagg 20
<210> 28
<211> 29
<212> RNA
<213> HCoV-229E
<400> 1
Acgttggatg tctgttttta catgcttcc 29
<210> 29
<211> 30
<212> RNA
<213> HCoV-229E
<400> 2
Acgttggatg taagtaaccg tatcagaacc 30
<210> 30
<211> 21
<212> RNA
<213> HCoV-229E
<400> 3
Ctttctcaaa cacaaactca c 21
<210> 31
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF3a-26144
<400> 1
Acgttggatg tgagcctgaa gaacatgtcc 30
<210> 32
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF3a-26144
<400> 2
Acgttggatg gtagtcgtcg tcggttcatc 30
<210> 33
<211> 22
<212> RNA
<213> SARS-CoV-2_ORF3a-26144
<400> 3
Ccgcttaaca actccggatg aa 22
<210> 34
<211> 31
<212> RNA
<213> SARS-CoV-2_ORF1ab-8782
<400> 1
Acgttggatg gtcattagta taactaccac c 31
<210> 35
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF1ab-8782
<400> 2
Acgttggatg gtcactcgtg acatagcatc 30
<210> 36
<211> 22
<212> RNA
<213> SARS-CoV-2_ORF1ab-8782
<400> 3
Ctgattttga cacatggttt ag22
<210> 37
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF3a-25563
<400> 1
Acgttggatg agagtgctag ttgccatctc 30
<210> 38
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF3a-25563
<400> 2
Acgttggatg ccctttcgga tggcttattg 30
<210> 39
<211> 23
<212> RNA
<213> SARS-CoV-2_ORF3a-25563
<400> 3
Ccccaacttc ttgctgtttt tca 23
<210> 40
<211> 30
<212> RNA
<213> MERS-CoV
<400> 1
Acgttggatg aagtccatgg ctgcaactcg 30
<210> 41
<211> 30
<212> RNA
<213> MERS-CoV
<400> 2
Acgttggatg aagcatgaaa tcccagccac 30
<210> 42
<211> 23
<212> RNA
<213> MERS-CoV
<400> 3
Gtatccaccg tagaactttg tag 23
<210> 43
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF8-28144
<400> 1
Acgttggatg aggctggttc taaatcaccc 30
<210> 44
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF8-28144
<400> 2
Acgttggatg acccaattta ggttcctggc 30
<210> 45
<211> 23
<212> RNA
<213> SARS-CoV-2_ORF8-28144
<400> 3
Aatggcaatt aattgtaaaa ggt 23
<210> 46
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF1ab-11083
<400> 1
Acgttggatg acatcattgc aaaagcagac 30
<210> 47
<211> 30
<212> RNA
<213> SARS-CoV-2_ORF1ab-11083
<400> 2
Acgttggatg gtccagagta ctcaatggtc 30
<210> 48
<211> 24
<212> RNA
<213> SARS-CoV-2_ORF1ab-11083
<400> 3
Ccaatggtct ttgttctttt tttt 24
<210> 49
<211> 30
<212> RNA
<213> SARS-CoV-2_N
<400> 1
Acgttggatg ctacctggcg tggtttgtat 30
<210> 50
<211> 31
<212> RNA
<213> SARS-CoV-2_N
<400> 2
Acgttggatg gaaatgctgg tattgttggt g 31
<210> 51
<211> 24
<212> RNA
<213> SARS-CoV-2_N
<400> 3
Ggggtgtaac tggtatgatt tcgg 24
<210> 52
<211> 30
<212> RNA
<213> SARS-CoV
<400> 1
Acgttggatg tggagaaatg tttacgcagg 30
<210> 53
<211> 29
<212> RNA
<213> SARS-CoV
<400> 2
Acgttggatg caggctctat gagtgtctc 29
<210> 54
<211> 25
<212> RNA
<213> HCoV-NL63
<400> 3
Ggctctatga gtgtctctat agaaa 25
<210> 55
<211> 30
<212> RNA
<213> HCoV-HKU1
<400> 1
Acgttggatg tggcctgtaa tggccataag 30
<210> 56
<211> 29
<212> RNA
<213> HCoV-HKU1
<400> 2
Acgttggatg cataatctgt acgataaac 29
<210> 57
<211> 26
<212> RNA
<213> HCoV-HKU1
<400> 3
Cccctaagcg tttttgtaaa ttgcgt 26
<210> 58
<211> 30
<212> RNA
<213> HCoV-OC43
<400> 1
Acgttggatgt gatacacaag tggataagc 30
<210> 59
<211> 30
<212> RNA
<213> HCoV-OC43
<400> 2
Acgttggatg gttaaagact gatagtgttc 30
<210> 60
<211> 27
<212> RNA
<213> HCoV-OC43
<400> 3
Tggtctttta atagaacgat ttgtaag 27
<210> 61
<211> 30
<212> RNA
<213> SARS-CoV-2_S-23012
<400> 1
Acgttggatg acgttggatg gtgggttgga aaccatatga 30
<210> 62
<211> 42
<212> RNA
<213> SARS-CoV-2_S-23012
<400> 2
Acgttggatg acgttggatg gaaatctatc agaggccggt ag 42
<210> 63
<211> 19
<212> RNA
<213> SARS-CoV-2_S-23012
<400> 3
Gacaccttgt aatggtgtt 19
- 检测冠状病毒抗体的试剂盒、冠状病毒抗体的检测方法
- 检测冠状病毒抗体的试剂盒、冠状病毒抗体的检测方法
