使用合成的mRNA将尿细胞直接重编程为神经干细胞的方法
文献发布时间:2023-06-19 11:32:36
技术领域
本发明涉及用于将尿细胞直接重编程为神经干细胞的方法以及用于治疗神经损伤疾病的药物组合物,该药物组合物包括通过该方法制备的重编程的神经干细胞。
背景技术
干细胞是指对自我更新或分化具有无限潜力的细胞,这些自我更新或分化与体内所需的特定细胞和组织有关。干细胞分为三种类型:从早期胚胎分离的胚胎干细胞(ES细胞)、从胚胎原始生殖细胞分离的胚胎生殖细胞(EG细胞)和从成体组织分离的多能成体祖细胞(MAPC细胞)。
由于干细胞具有分化为带特定功能的细胞的潜力,因此干细胞已成为恢复各种器官的功能、作为细胞治疗剂的研究对象,近来,干细胞的应用已扩展到整形外科和美容领域。
可以以两种方式大致概括成体干细胞在体内的作用。第一,干细胞本身分化为我们体内的组织和细胞,并使受损的组织和细胞再生;以及第二,干细胞在其寿命期间不断分泌生长因子和蛋白质(例如细胞因子),以帮助邻近细胞的生长和再生。
直接重编程(Direct reprogramming)是一种允许根据需要将体细胞转化为其他类型的细胞的技术。由于直接重编程不像日本山中(Yamanaka)教授的重编程那样经历多能干细胞状态,因此直接重编程可以节省时间并展示出高效率水平,同时解决了与肿瘤发生有关的风险以及由多能干细胞进一步分化导致的成本。然而,当前进行的直接重编程方法主要使用小鼠细胞作为细胞源,因此在人类体细胞中以相同方式再生的可能性不高。此外,在人类体细胞中经常使用皮肤细胞(成纤维细胞),但是在这种情况下,需要一种收集细胞来源的侵入性方法,并且这给供体带来了疼痛和安全风险,并且便利性变差。另外,临床应用是困难的,因为通过在直接重编程过程中引入所需的重编程因子(基因)的方法主要使用病毒,并且使用病毒的基因整合系统不适用于治疗,因为该系统会因不加选择地插入基因组中而诱发突变。
因此,本发明人使用容易收集的源自人尿的细胞作为细胞来源并使用合成的mRNA的转染以将重编程因子引入细胞中来解决上述问题,并确认了从尿细胞高效直接重编程为神经干细胞所需的最佳培养条件,从而完成了本发明。
相关技术文献
韩国专利申请特许号10-2013-0087020
发明内容
技术问题
本发明的目的是提供用于将尿细胞直接重编程为神经干细胞的组合物。
本发明的另一个目的是提供用于将尿细胞直接重编程为神经干细胞的方法。
本发明的又一个目的是提供用于预防或治疗神经损伤疾病的方法,该方法包括施用作为活性成分的从尿细胞重编程的神经干细胞。
本发明的又一个目的是提供用于预防或治疗神经损伤疾病的药物组合物,该药物组合物包含作为活性成分的从尿细胞重编程的神经干细胞。
技术方案
本发明提供用于将尿细胞直接重编程为神经干细胞的组合物,该组合物包含:
(i)Oct4蛋白或编码该Oct4蛋白的核酸;
(ii)Sox2蛋白或编码该Sox2蛋白的核酸;
(iii)Klf4蛋白或编码该Klf4蛋白的核酸;以及
(iv)Glis1蛋白或编码该Glis1蛋白的核酸。
如本文所用,术语“重编程的神经干细胞”包括通过直接重编程制成的、用于建立具有与神经干细胞相似或相同的多能能力的未分化干细胞的神经干细胞。诱导的神经干细胞具有与神经干细胞相同或相似的特征,具体地可以显示相似的细胞形态,具有相似的基因和蛋白质表达模式,并且在体内和体外具有多能能力。因此,本发明的重编程的神经干细胞可以是可以分化为神经细胞(神经元)、星形细胞、少突胶质细胞、GABA能神经细胞、多巴胺能神经细胞等的神经干细胞。
如本文所用,术语“尿细胞(Urine cell,UC)”被称为体细胞,其可以在没有任何不便和痛苦的情况下,在任何时间容易且重复地获得。
本发明人发现,当在尿细胞中表达重编程因子Oct4、Sox2、Klf4和Glis1时,已经分化细胞类型的尿细胞被重编程为具有分化能力的神经干细胞。
术语“重编程因子”始于重编程,是Yamanaka在2006年引入的一个概念。成体的所有细胞在经历正常的发育过程中都以未分化的状态逐渐分化,并由此变成了各自功能被特化的细胞。其中,受精细胞是全能的,并且当受精细胞随着后续发育步骤发展而变成囊胚时,可分为内细胞团和外部细胞,在这种情况下,内细胞团细胞可产生被称为多能性的胚胎细胞和生殖细胞。胚胎干细胞表现出多能特异性基因表达方面,并且其代表性实例包括Oct4、Sox2、Nanog、Lin28等。重编程可以说是一种在体细胞中诱导这种特异性基因表达以恢复与未分化细胞(例如胚胎干细胞和成体干细胞)相似的特性的技术。本发明人使用Oct4、Sox2、Klf4和Glis1将尿细胞重编程为神经干细胞,作为与重编程有关的一系列研究中使用的各种因素的一部分。
在本发明的组合物中使用的Oct4、Sox2、Klf4和Glis1包括所有源自人和动物(例如马、绵羊、猪、山羊、骆驼、羚羊和狗)的Oct4、Sox2、Klf4和Glis1,并且优选人的Oct4、Sox2、Klf4和Glis1。此外,用于重编程为神经干细胞的本发明的Oct4、Sox2、Klf4和Glis1蛋白可以包括具有其野生型氨基酸序列的蛋白,还可以包括Oct4、Sox2、Klf4和Glis1蛋白的变体。
在具体的示例性实施方式中,使用了首次在重编程中使用的并在Nature.2011Jun8;474(7350):225-9中公开的Oct4、Sox2、Klf4和Glis1的基因序列。
Oct4、Sox2、Klf4和Glis1蛋白的变体是指通过缺失、插入、非保守或保守置换或其组合而使至少一个氨基酸残基与Oct4、Sox2、Klf4和Glis1的天然氨基酸序列不同的蛋白。变体可以是显示出与天然蛋白质相同的生物活性的功能等同物,或者可以是根据需要对蛋白质的物理性质和化学性质进行修饰的变体。该变体是在某些物理或化学条件下结构稳定性增加或生理活性增加的变体。
包括编码Oct4、Sox2、Klf4和Glis1蛋白的核苷酸序列的核酸是包括编码野生型Oct4、Sox2、Klf4和Glis1蛋白或如上所述的变体形式的Oct4、Sox2、Klf4和Glis1蛋白的核苷酸序列的核酸,可以通过一个或多个碱基的取代、缺失和插入或其组合来修饰,并且可以是天然分离的或使用化学合成方法来制备。
具有编码Oct4、Sox2、Klf4和Glis1蛋白的核苷酸序列的核酸可以是单链或双链,并且可以是DNA分子(基因组、cDNA)或RNA分子。在本发明的特定示例性实施方式中,使用合成的信使RNA(mRNA)作为编码Oct4、Sox2、Klf4和Glis1蛋白的核酸,并且可以使用引入了表达Oct4、Sox2、Klf4和Glis1的DNA的合成的mRNA表达载体来制备合成的信使RNA。
本发明提供了用于将尿细胞直接重编程为神经干细胞的方法,该方法包括:将Oct4、Sox2、Klf4和Glis1蛋白或编码该蛋白的核酸引入尿细胞。
更具体地,该方法可以包括:
(a)从尿中分离尿细胞并培养该尿细胞;
(b)将权利要求1或3的组合物引入培养的尿细胞中;
(c)通过在神经干细胞诱导培养基中培养已经引入该组合物的尿细胞,来诱导直接重编程为神经干细胞;以及
(d)从已被神经干细胞诱导培养基诱导直接重编程的细胞中选择具有与神经干细胞相似特征的神经干细胞系。
在步骤(a)中,可以在含有胎牛血清(Fetal bovine serum,FBS)、碱性成纤维细胞生长因子(basic fibroblast growth factor,bFGF)和上皮生长因子(Epithelial growthfactor,EGF)的培养基中培养尿细胞。
如本文所用,术语“培养基”是指能够支持细胞体外生长和存活的培养基,并且包括本领域中适用于诱导和培养尿细胞和重编程的神经干细胞的所有典型的培养基。根据细胞的类型,可以选择培养基和培养条件。用于培养的培养基优选为细胞培养基础培养基(Cell culture minimum medium,CCMM),并且通常包括碳源、氮源和微量元素组分。这种细胞培养基础培养基的示例包括:Dulbecco改良的Eagle培养基(Dulbecco's ModifiedEagle's Medium,DMEM)、最小必需培养基(Minimal Essential Medium,MEM)、基础培养基Eagle(Basal Medium Eagle,BME)、RPMI1640、F-10、F-12、α最小必需培养基(αMinimalEssential Medium,αMEM)、Glasgow最小必需培养基(Glasgow's Minimal EssentialMedium,GMEM)、Iscove改良的Dulbecco培养基(Iscove's Modified Dulbecco's Medium,IMEM)等,但不限于此。此外,培养基可以包括抗生素,例如青霉素、链霉素或庆大霉素。
在本发明的具体实例中,可以通过在包含FBS、bFGF和EGF的基础培养基中培养从尿中分离的细胞来获得培养基,具体地,可以通过将bFGF和EGF添加到高葡萄糖DMEM和包括FBS的肾上皮细胞生长培养基(Renal epithelial cell growth medium,REGM)来获得培养基,并在该培养基中培养从尿中分离的细胞。更具体地,在本发明中,高葡萄糖DMEM和REGM培养基可以另外包括L-谷氨酰胺和青霉素-链霉素。
在步骤(b)中,作为将重编程因子引入尿细胞的方法,可以使用本领域通常使用的将核酸或蛋白质提供于细胞内的方法而没有限制,并且优选可以使用向分化细胞的培养液中施用重编程因子的方法或将重编程因子直接注入分化细胞的方法,在这种情况下使用的重编程因子可以以从用已插入相应因子的基因的病毒载体转染的包装细胞获得的病毒、通过体外转录产生的信使RNA、在各种细胞系中产生的蛋白质等的形式使用、。在本发明的特定示例性实施方式中,在将重编程因子引入尿细胞中时,使用了通过编码Oct4、Sox2、Klf4和Glis1的DNA的转录产生的信使RNA。
对于将合成的信使RNA直接注入分化细胞的方法,可以选择和使用本领域中已知的任何方法,并且可以从使用显微注射、电穿孔、粒子轰击、直接肌肉注射、绝缘子和转座子的方法中适当地选择和应用一种方法。具体地,在本发明的示例性实施方式中,使用电穿孔将重编程因子的信使RNA引入尿细胞中。
在步骤(c)中,将已引入编码Oct4、Sox2、Klf4和Glis1蛋白的核酸的尿细胞诱导为神经干细胞的培养基(神经干细胞诱导培养基)可以是通过以下获得的培养基:将DME/MF12培养基和神经基础培养基以1:1的体积比混合,并向该培养基混合物中添加1×N2、1×B27、人LIF、TGF-β抑制剂和GSK3-β抑制剂。更具体地,该培养基可以进一步包括以下中的一种或多种:Shh激动剂、腺苷酸环化酶激活剂、组蛋白去乙酰化酶抑制剂和抗坏血酸2-磷酸酯。在本发明的特定示例性实施方式中,证实了在补充有Shh激动剂、腺苷酸环化酶激活剂、组蛋白去乙酰化酶抑制剂和抗坏血酸2-磷酸酯以上全部的培养基中,神经干细胞转化率是最佳的。
此外,在低氧(O
在步骤(d)中,对诱导的神经干细胞进行选择是为了收集在进行步骤(c)之后产生的神经干细胞集落,并且所选择的神经干细胞可以在神经干细胞培养基中培养。神经干细胞培养基可以是通过向培养基(其中DMEM/F12培养基和神经基础培养基以1:1的体积比混合)中添加1×N2、1×B27、人LIF、TGF-β抑制剂和GSK3-β抑制剂而获得的培养基。此外,可以使用0.5mM EDTA溶液或Accutase溶液对诱导的神经干细胞进行传代培养。
此外,本发明提供了用于预防或治疗神经损伤疾病的药物组合物,该药物组合物包含作为活性成分的通过所述方法诱导的神经干细胞。
此外,本发明提供了用于预防或治疗神经损伤疾病的方法,该方法包括施用通过该方法制备的作为活性成分的直接重编程的神经干细胞。
神经干细胞是多能细胞,其可以分化为神经细胞(神经元)、星形细胞、少突胶质细胞、GABA能神经细胞、多巴胺能神经细胞等,并且可以修复或恢复受损或失去的神经细胞,因此有可能治疗由神经细胞的损伤或消失引起的疾病而不受限制。
具体地,由神经细胞损伤引起的疾病可以选自由以下组成的组:帕金森氏病、阿尔茨海默氏病、匹克氏病、亨廷顿氏病、肌萎缩性侧索硬化症、缺血性脑病(中风)、脱髓鞘疾病、多发性硬化症、癫痫、退行性神经疾病和脊髓损伤。
有利效果
本发明是一种利用源自人尿的细胞作为细胞来源以使获得细胞来源的便利性最大化并且通过使用合成的mRNA的非整合性直接重编程方法来增强临床可行性的技术。
附图说明
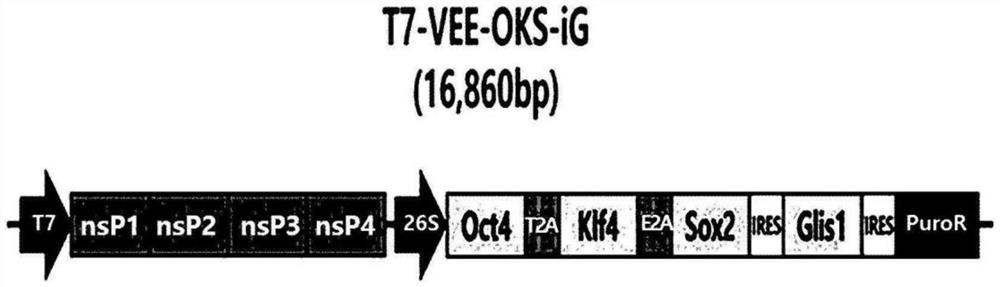
图1是用于制备OCT4、KLF4、SOX2和GLIS1基因的合成的mRNA的T7-VEE-OKS-iG载体的示意图;
图2示出了通过FACS分析来证实与GFP(荧光基因)结合的OCT4、KLF4、SOX2和GLIS1基因的合成的mRNA是否被引入尿细胞的结果;
图3示出了使用直接重编程技术获得源自尿细胞的神经干细胞集落的整个过程;
图4示出了使用源自H9胚胎干细胞的神经干细胞作为阳性对照,通过用RT-PCR进行mRNA水平分析来确认诱导的神经干细胞是否表达神经干细胞标志基因(例如SOX1、SOX2、PAX6和PLZF)的结果;
图5示出了通过免疫测定在蛋白质水平上来确认诱导的神经干细胞是否表达神经干细胞标志基因(例如SOX1、SOX2、PAX6和PLZF)的结果;
图6示出了通过Ki67的免疫染色来确认诱导的神经干细胞是否具有分化能力的结果;
图7示出了用qRT-PCR通过mRNA定量分析来确认与源自H9胚胎干细胞的神经干细胞相比,诱导的神经干细胞是否表达神经干细胞标志基因(例如SOX1、SOX2、PAX6和PLZF)的结果;
图8示出了用RT-PCR通过mRNA水平分析来确认诱导的神经干细胞是否表达发育前脑、中脑、后脑和脊髓特异性标志基因的结果;
图9和图10示出了通过在全局基因表达水平上进行总RNA测序,来确认诱导的神经干细胞是否表现出相比原始尿源性细胞更类似于源自H9胚胎干细胞的神经干细胞的mRNA和长非编码RNA(Inc-RNA)表达模式的结果;
图11示出了通过VEE基因的RT-PCR和基因组DNA PCR分析来确认引入神经干细胞中的合成的mRNA是否保留在宿主中以及合成的mRNA是否整合到宿主基因组中的结果;
图12示出了通过STR分析来确认诱导的神经干细胞是否源自人尿细胞的结果;
图13示出了通过核型分析来确认诱导的神经干细胞是否保留了正常染色体的结果;
图14示出了通过免疫筛选法来确认神经细胞基因标志TUJ1和MAP2的表达以验证诱导的神经干细胞分化为神经细胞的能力的结果;
图15示出了通过免疫染色来确认诱导的神经干细胞可以分化为GABA神经细胞、运动神经细胞和多巴胺神经细胞的结果;
图16示出了通过免疫筛选法来确认S100β和GFAP的表达的结果,所述S100β和GFAP是用于验证诱导的神经干细胞分化为星形细胞(星形细胞为胶质细胞)的能力的标志;
图17示出了通过免疫筛选法来确认PDGFR、OLIG2和O4的表达的结果,所述PDGFR、OLIG2和O4是用于验证诱导的神经干细胞分化为少突胶质细胞(少突胶质细胞为胶质细胞)的能力的标志;
图18示出了在从尿细胞诱导为神经干细胞的过程中按日期来确认OCT4、REX4和NANOG的相对mRNA表达水平的结果;
图19示出了确认是否表达了多能阶段的H9胚胎干细胞和诱导的神经干细胞的标志基因的结果;
图20示出了使用碱性条件(21%O
图21示出了当在培养基中进一步包括Shh激动剂、腺苷酸环化酶激活剂、组蛋白去乙酰化酶抑制剂和抗坏血酸2-磷酸酯中的一种或多种时,确认神经干细胞的诱导效率提高的结果,该培养基是通过以下获得的:以1:1的体积比混合DMEMF12培养基和神经基础培养基,并向培养基混合物中加入1×N2、1×B27、人LIF、TGF-β抑制剂和GSK3-β抑制剂。证实了在补充有Shh激动剂、腺苷酸环化酶激活剂、组蛋白去乙酰化酶抑制剂和抗坏血酸2-磷酸酯以上全部的培养基中,神经干细胞转化率是最佳的。
具体实施方式
下文将参考实施例对本发明进行更详细的描述。然而,提供以下实施例仅是为了更容易理解本发明,并且本发明不受以下实施例的限制。
实施例1:从尿中分离尿细胞
基于Sutherland和Bain于1972年在英国开发的技术,详细信息如下。首先,将供体捐赠的尿以1000g离心10分钟。除去上清液后,用20ml含有1%青霉素/链霉素/两性霉素B抗生素的PBS溶液稀释留在下层中的沉淀。然后将稀释的PBS+沉淀溶液以1000g离心10分钟。再次除去上清液后,用1ml基础培养基(基于DMEMF12的包含1%青霉素/链霉素/两性霉素B抗生素、1%L-谷氨酰胺和10%FBS的培养基)稀释留在下层的沉淀,并接种到涂有明胶的12孔细胞培养皿上。接着,在通过添加1ml基础培养基培养细胞3天后,将该培养基更换为生长培养基(基于通过将DMEM和REGM以1:1混合而获得的培养基的包含1%青霉素/链霉素抗生素、1%L-谷氨酰胺,5%FBS、10ng/ml bFGF和10g/ml EGF的培养基)来培养细胞。
实施例2:使用电穿孔将重编程因子引入尿细胞
通过电穿孔将合成的mRNA引入实施例1中培养的尿源性细胞的方法具体如下。合成的mRNA通过典型的体外转录试剂盒(RiboMAX◎大规模RNA生产系统,Promega)合成的,而成为要合成的mRNA的骨架的DNA是T7-VEE-OKS-iG(Steven Dowdy,Addgene),T7-VEE-OKS-iG包括OCT4、KLF4、SOX2和GLIS1的基因(图1)。作为OCT4、KLF4、SOX2和GLIS1基因的序列,使用在Nature.2011Jun 8;474(7350):225-9中公开的那些序列,而对于相应的基因序列和mRNA应用方法,参考Cell Stem Cell.2013Aug1;13(2):246-54和美国专利第9,862,930B2号。
通过以1600V、10ms电穿孔3次,持续1小时,用0.5μg合成的mRNA对以0.2ug/mlB18R蛋白预处理的1×10
本发明人通过同时引入包括GFP荧光蛋白的合成的mRNA来可视化、确定和验证通过电穿孔引入的效率,通过FACS分析证实了表达GPF荧光蛋白的人尿源性细胞的效率。通过验证确认,在处理后约48小时,通过电穿孔,合成的mRNA的人尿源性引入效率达到了72.2%,确定了这足以进行下一步的神经干细胞诱导实验(图2)。
实施例3:源自尿的重编程的神经干细胞的诱导
在将导入了mRNA的尿源性细胞接种到涂有Matrigel
此外,在进一步补充有1×N2、1×B27、人LIF、TGF-β抑制剂和GSK3-β抑制剂的培养基中进一步包括Shh激动剂、腺苷酸环化酶激活剂、组蛋白去乙酰化酶抑制剂和抗坏血酸2-磷酸酯中的一种或多种时,证实了对神经干细胞诱导效率的影响。证实了在补充有Shh激动剂、腺苷酸环化酶激活剂、组蛋白去乙酰化酶抑制剂和抗坏血酸2-磷酸酯以上全部的培养基中,神经干细胞转化率是最佳的(图21)。
收集诱导的神经干细胞集落,并通过将神经干细胞培养基置于涂有Matrigel的细胞培养皿中来进行培养。接着可以使用0.5mM EDTA溶液或Accutase溶液对诱导的神经干细胞进行传代培养,然后在以下实施例中进行验证诱导的神经干细胞的各种特性的实验。
实施例4:诱导的神经干细胞的分子生物学特性分析
进行实验以验证源自本发明的神经干细胞的分子生物学特性。
<4-1>
使用源自H9胚胎干细胞的神经干细胞作为阳性对照,用RT-PCR通过mRNA水平分析确认了诱导的神经干细胞表达了神经干细胞标志基因,例如SOX1、SOX2、PAX6和PLZF(图4)。本实验中使用的引物序列如下:SOX1(正向-ACACTTGAAGCCCAGATGG;SEQ ID NO:1,反向-ATAGGCTCACTTTTGGACGG;SEQ ID NO:2)、SOX2(正向-TCAGGAGTTGTCAAGGCAGAGA;SEQ ID NO:3,反向-CCGCCGCCGATGATTGTTATTA;SEQ ID NO:4)、PAX6(正向-GGCTCAAATGCGACTTCAG;SEQID NO:5,反向-CCCTTCGATTAGAAAACCATACC;SEQ ID NO:6)、PLZF(正向-TATACAGCCACGCTGCAAGCCA;SEQ ID NO:7,反向-TGGTCTCCAGCATCTTCAGGCA;SEQ ID NO:8)。
<4-2>
还使用免疫染色通过蛋白质水平分析证实了诱导的神经干细胞表达了神经干细胞标志基因,例如SOX1、NESTIN、SSEA1、SOX2、PAX6和PLZF(图5)。本实验中使用的抗体如下:SOX1(AF3369,R&D Systems)、NESTIN(MAB5326,Millipore)、SSEA1(MAB4301,Millipore)、SOX2(SC-20088,Santacruz)、PAX6(PAX6,DSHB)、PLZF(AF2944,R&D Systems)。
<4-3>
通过Ki67的免疫染色证实了诱导的神经干细胞具有细胞分裂能力(图6)。本实验中使用的抗体如下:Ki67(Ab9260,Millipore)。
<4-4>
还用qRT-PCR通过对mRNA进行定量分析证实了,与源自H9胚胎干细胞的神经干细胞相比,诱导的神经干细胞表达了神经干细胞标志基因,例如SOX1、SOX2、PLZF和PAX6(图7)。
<4-5>
还用RT-PCR通过mRNA水平分析证实了诱导的神经干细胞表达了发育前脑、中脑、后脑和脊髓特异性标志基因(图8)。本实验中使用的引物序列如下:EMX1(正向-CAGGCCCAGGTAGTTCAATGGG;SEQ ID NO:9,反向-GCTCAGCCTTAAGCCCTGTCTC;SEQ ID NO:10)、FOXG1(正向-ACCCGTCAATGACTTCGCAGAG;SEQ ID NO:11,反向-AGGGTTGGAAGAAGACCCCTGA;SEQ ID NO:12)、OTX1(正向-CTCAAACAACCCCCATACGGCA;SEQ ID NO:13,反向-GGTAGCGAGTCTTGGCGAAGAG;SEQ ID NO:14)、OTX2(正向-TTCAGGGCTGTGTGAATTGTGTGA;SEQID NO:15,反向-CAGAGGTGGAGTTCAAGGTTGCAT;SEQ ID NO:16)、EN1(正向-GAGTTCCAGGCAAACCGCTACA;SEQ ID NO:17,反向-GTTGTACAGTCCCTGGGCCATG;SEQ ID NO:18)、GBX2(正向-CGGTTAGCAGCCACCTTTCCAT;SEQ ID NO:19,反向-GACGTGCTTCACATGGCTCAGA;SEQ ID NO:20)、HOXB2(正向-GAGACCCAGGAGCCAAAAGC;SEQ IDNO:21,反向-GAAGGAGACGTGGCGGATTG;SEQ ID NO:22)、HOXB4(正向-CTGAGGGCCAGAATGACTGCTC;SEQ ID NO:23,反向-CAGAACTCAACTGGCCCCTCAC;SEQ ID NO:24)。
<4-6>
通过在全局基因表达水平上进行总RNA测序证实了,诱导的神经干细胞表现出相比原始尿源性细胞更类似于源自H9胚胎干细胞的神经干细胞的mRNA和Inc-RNA表达模式(图9和图10)。
<4-7>
通过VEE基因的RT-PCR和基因组DNA PCR分析证实了,引入神经干细胞的合成的mRNA不再保留在宿主中,并且不整合到宿主基因组中(图11)。本实验中使用的引物序列如下:VEE(正向-ACGAAGGGCAAGTCGCTGTT;SEQ ID NO:25,反向-TTTCGTCGGCCCAGTTGGTA;SEQID NO:26)。
<4-8>
通过STR分析证实了,人尿细胞和诱导的神经干细胞源自同一个体(图12)。
<4-9>
通过核型分析证实了诱导的神经干细胞保留了正常染色体(图13)。
实施例5:诱导的神经干细胞分化能力的分析
<5-1>分化为神经细胞的能力的分析
通过免疫染色证实了,在诱导的神经干细胞分化为神经细胞时,表达了神经细胞标志基因TUJ1和MAP2(图14)。神经细胞分化方法如下。在神经细胞分化培养基中培养14天后对神经干细胞进行分析,该培养基是通过向DMEM/F12培养基中添加1×N2、1×B27、300ng/ml cAMP、0.2mM维生素C、10ng/ml BDNF和10ng/ml GDNF而获得的。本实验中使用的抗体如下。Tuj1(801202,BioLegend)、MAP2(AB5622,Millipore)。
此外,可以证实,通过免疫染色分别对GABA、HB8和TH进行染色,诱导的神经干细胞可以分化为GABA神经细胞、运动神经细胞和多巴胺神经细胞(图15)。本实验中使用的抗体如下:GABA(A2052,Sigma)、HB9(81.5C10,DSHB)、TH(AB152,Millipore)。
<5-2>分化为胶质细胞的能力的分析
通过免疫染色,凭借S100β和GFAP染色证实了诱导的神经干细胞可以分化为星形细胞(星形细胞为胶质细胞中的一种)(图16)。星形细胞分化方法如下。将诱导的神经干细胞培养在星形细胞分化培养基中培养14天或更长时间,然后进行分析,该星形细胞分化培养基在含有1×N2和1×B27的DMEM/F12培养基中补充有2.5%FBS或20ng/ml CNTF和10ng/ml BMP4。本实验中使用的抗体如下:S100β(S2535,Sigma)、GFAP(SAB4501162,Sigma)。
通过免疫染色,凭借PDGFR、OLIG2和O4染色证实了诱导的神经干细胞可以分化为少突胶质细胞(少突胶质细胞为胶质细胞中的一种)(图17)。少突胶质细胞分化方法如下。在将诱导的神经干细胞在通过向DMEM/F12培养基中添加1×N2、1×B27、25ug/ml胰岛素、1uM Shh激动剂和100nM视黄酸获得的培养基中培养5天或更长时间后,将细胞在通过向DMEM/F12培养基添加1×N2、1×B27、25ug/ml胰岛素、100ng/ml生物素、60ng/ml T3、10ng/ml PDGF-AA、10ng/ml IGF-1、5ng/ml HGF、10ng/ml NT-3和1uM cAMP获得的培养基中培养7天或更长时间,然后进行分析。本实验中使用的抗体如下:PDGFR(SC338,Santa Cruz)、OLIG2(AB9610,Millipore)、O4(MAB345,Millipore)。
实施例6:不经历多能阶段的神经干细胞诱导方法的验证与分析
已知在本发明中使用的重编程因子OCT4、SOX2、KLF4和GLIS1基因的组合可以在特定条件下建立诱导的多能干细胞(美国专利第09862930号)。
然而,验证了由于以下原因,在本发明中诱导的神经干细胞没有经过多能阶段。
第一,使用了不包含bFGF和B18R蛋白的神经干细胞诱导培养基,bFGF对于建立诱导的人多能干细胞是必不可少的,该B18R蛋白是表达内源基因的mRNA所必需的,因此根据迄今报道的研究结果不可能建立诱导的人多能干细胞。
第二,源自本发明的神经干细胞的时间是在根据方案(图3)的诱导过程后的8天左右形成,并且已知在诱导过程之后诱导的人多能干细胞需要3周或更长时间,因此经历多能阶段的时间太短了。
第三,当通过基于日期的qRT-PCR分析观察时,多能阶段标志基因OCT4、REX1和NANOG的表达从第0天到第12天保持低表达(图18)。本实验中使用的引物序列如下:OCT4(正向-GACAGGGGGAGGGGAGGAGCTAGG;SEQ ID NO:27,反向-CTTCCCTCCAACCAGTTGCCCCAAAC;SEQID NO:28)、REX1(正向-CTGAAGAAACGGGCAAAGAC;SEQ ID NO:29,反向-GAACATTCAAGGGAGCTTGC;SEQ ID NO:30)、NANOG(正向-CAGCCCTGATTCTTCCACCAGTCCC;SEQID NO:31,反向-GGAAGGTTCCCAGTCGGGTTCACC;SEQ ID NO:32)。
第四,证实了当通过免疫染色验证诱导的神经干细胞时,与H9胚胎干细胞不同,未检测到OCT4和NANOG的表达(图19)。本实验中使用的抗体如下:OCT4(SC5279,圣克鲁斯)、NANOG(AF1997,R&D Systems)。
基于上述要点,可以确认本发明的方法无需经历多能干细胞阶段就可以立即将尿细胞诱导为神经干细胞。
实施例7:提高神经干细胞诱导效率的实验
进一步确认了能够改善从尿细胞向神经干细胞转化的效率的培养条件等。
使用一般氧气条件(21%O
序列表
<110> 思特科技公司
<120> 使用合成的mRNA将尿细胞直接重编程为神经干细胞的方法
<130> G21U13C0007P/CN
<150> KR1020180101208
<151> 2018-08-28
<160> 32
<170> PatentIn version 3.5
<210> 1
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> SOX1 forward
<400> 1
acacttgaag cccagatgg 19
<210> 2
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> SOX1 reverse
<400> 2
ataggctcac ttttggacgg 20
<210> 3
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> SOX2 forward
<400> 3
tcaggagttg tcaaggcaga ga 22
<210> 4
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> SOX2 reverse
<400> 4
ccgccgccga tgattgttat ta 22
<210> 5
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> PAX6 forward
<400> 5
ggctcaaatg cgacttcag 19
<210> 6
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> PAX6 reverse
<400> 6
cccttcgatt agaaaaccat acc 23
<210> 7
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> PLZF forward
<400> 7
tatacagcca cgctgcaagc ca 22
<210> 8
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> PLZF reverse
<400> 8
tggtctccag catcttcagg ca 22
<210> 9
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> EMX1 forward
<400> 9
caggcccagg tagttcaatg gg 22
<210> 10
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> EMX1 reverse
<400> 10
gctcagcctt aagccctgtc tc 22
<210> 11
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> FOXG1 forward
<400> 11
acccgtcaat gacttcgcag ag 22
<210> 12
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> FOXG1 reverse
<400> 12
agggttggaa gaagacccct ga 22
<210> 13
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> OTX1 forward
<400> 13
ctcaaacaac ccccatacgg ca 22
<210> 14
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> OTX1 reverse
<400> 14
ggtagcgagt cttggcgaag ag 22
<210> 15
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> OTX2 forward
<400> 15
ttcagggctg tgtgaattgt gtga 24
<210> 16
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> OTX2 reverse
<400> 16
cagaggtgga gttcaaggtt gcat 24
<210> 17
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> EN1 forward
<400> 17
gagttccagg caaaccgcta ca 22
<210> 18
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> EN1 reverse
<400> 18
gttgtacagt ccctgggcca tg 22
<210> 19
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> GBX2 forward
<400> 19
cggttagcag ccacctttcc at 22
<210> 20
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> GBX2 reverse
<400> 20
gacgtgcttc acatggctca ga 22
<210> 21
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> HOXB2 forward
<400> 21
gagacccagg agccaaaagc 20
<210> 22
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> HOXB2 reverse
<400> 22
gaaggagacg tggcggattg 20
<210> 23
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> HOXB4 forward
<400> 23
ctgagggcca gaatgactgc tc 22
<210> 24
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> HOXB4 reverse
<400> 24
cagaactcaa ctggcccctc ac 22
<210> 25
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> VEE forward
<400> 25
acgaagggca agtcgctgtt 20
<210> 26
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> VEE reverse
<400> 26
tttcgtcggc ccagttggta 20
<210> 27
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> OCT4 forward
<400> 27
gacaggggga ggggaggagc tagg 24
<210> 28
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> OCT4 reverse
<400> 28
cttccctcca accagttgcc ccaaac 26
<210> 29
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> REX1 forward
<400> 29
ctgaagaaac gggcaaagac 20
<210> 30
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> REX1 reverse
<400> 30
gaacattcaa gggagcttgc 20
<210> 31
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> NANOG forward
<400> 31
cagccctgat tcttccacca gtccc 25
<210> 32
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> NANOG reverse
<400> 32
ggaaggttcc cagtcgggtt cacc 24
- 使用合成的mRNA将尿细胞直接重编程为神经干细胞的方法
- 一种高效快速重编程体细胞为神经干细胞的方法及其应用
