酶促DNA修复
文献发布时间:2023-06-19 11:00:24
背景技术
合成的脱氧核糖核酸(DNA)具有作为用于数字信息的存储介质的用途。DNA可以比其他存储介质以更高的密度和更长的寿命存储数字信息。然而,DNA易受到来自各种来源的损伤,诸如紫外线(UV)辐射、水解、热损伤、毒素、诱变化学物质和病毒。对DNA分子本身的损伤可能使恢复在核苷酸碱基序列中存储的数字信息变得更加困难或不可能恢复在核苷酸碱基序列中存储的数字信息。存储条件和存储时间长度可以影响损伤的类型和程度。针对极长期存储,诸如数千年,即使在最防护的存储条件下,DNA也有可能遭受损伤。针对短期存储,修复错误的能力可以使较少鲁棒存储选项可行。
有助于准确恢复在受损DNA中编码的数字信息的技术可以提高DNA作为用于数字信息的存储介质的可用性。
发明内容
本公开提供了用于修复DNA以改进在DNA核苷酸序列中存储的数字信息的恢复的技术。DNA通过暴露于包含一种或多种酶(诸如DNA聚合酶、DNA连接酶和(多种)核酸内切酶)的修复混合物来被修复。酶促修复使更多的DNA可用于测序,这转而改进了存储的数字信息从其中取回的序列数据。
恢复存储在DNA中的数字信息可以包括:通过聚合酶链反应(PCR)来扩增存储的DNA分子的池,并且利用DNA测序仪对PCR产物进行测序以生成序列数据。表示在池中的单个DNA分子的核苷酸碱基序列的序列数据被解码以再生数字信息。如果池中太多的DNA分子被损伤,则可能无法从足够数目的DNA分子中获得序列数据,这转而可以使再生数字信息变得困难或不可能。错误校正技术和存储冗余可以减轻一些而非全部类型的损伤的影响。
对受损DNA执行酶促修复可以显着地增加能够通过PCR成功扩增的单个DNA分子的数目。来自DNA的池的更多数目的不同分子的扩增许更多的数字信息被重建。在一些实施方式中,在酶促修复之后,可以通过PCR来恢复的DNA的量可以变成四倍。这可以在能否恢复所有数字信息和不可能恢复所有数字信息之间形成差异。
不同的酶可以被使用以修复DNA,并且每种酶适合于修复不同类型的损伤。可以对修复DNA起作用的酶的示例包括DNA聚合酶、DNA连接酶和核酸内切酶。诸如酶浓度、温度和暴露于修复混合物的时长的不同反应条件也影响修复效率。在合适的反应条件下将受损DNA暴露于适当的酶增加了来自能够通过PCR来扩增的DNA的池的不同的DNA分子的数目,并且因此增加了可用于解码和重生数字信息的序列数据的量。
提供本发明内容是为了以简化的形式介绍选择的概念,这些概念将在下面的具体实施方式中进一步描述。本发明内容既不旨在标识所要求保护的主题的关键特征或必要特征,也不旨在用于限制所要求保护的主题的范围。例如,术语“技术”可以涉及被上述内容和整个文档所允许的(多个)系统和/或(多个)方法。
附图说明
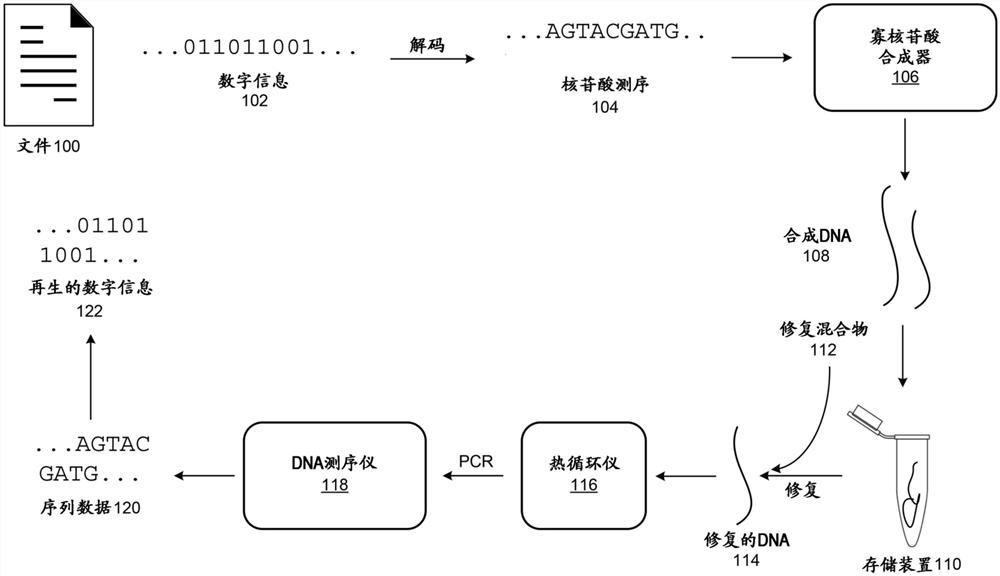
图1是图示可以被用于将来自计算机文件的数字信息编码在DNA中并且之后再生成该数字信息的设备和技术的图。
图2是示出用于在DNA中编码数字信息、存储DNA、以及然后从DNA中解码序列数据以恢复数字信息的说明性过程的流程图。
图3是示出在由PCR和测序的扩增之前修复合成DNA的说明性过程的流程图。
图4是示出用于如果存在小于完整DNA的阈值数量,将DNA与修复混合物接触的说明性过程的流程图。
图5是示出单个双链DNA分子的示意图,单个双链DNA分子包括五个单独的引物位置,引物位置被用于生成三个不同长度的扩增产物。
图6是示出与在没有酶促修复的相同条件下存储的DNA相比,DNA分子的酶促修复使DNA的量加倍的条形图。
图7是示出利用修复混合物的四个小时的孵化比更短的孵化时间产生更高的DNA浓度的条形图。
图8是示出利用修复混合物的四个小时的孵化比更长的孵化时间产生更高的DNA浓度的条形图。
具体实施方式
诸如DNA的多核苷酸可以被用于通过设计核苷酸碱基(腺嘌呤(A)、胞嘧啶(C)、鸟嘌呤(G)和胸腺嘧啶(T))的序列来存储数字信息,该核苷酸碱基的序列编码数字信息的零和一。存在本领域技术人员已知的用于使用核苷酸碱基来代表数字信息的各种技术和编码方案。参见Grass等人的Robust Chemical Preservation of Digital Information onDNA in Silica with Error-correcting Codes,54Angew.Chem.Int.Ed.2552(2015)以及Organick等人的Random access in large-scale DNA data storage,36:3Nat.Biotech.243(2018)。使用DNA而不是另一存储介质存储数字信息的优点包括信息密度和寿命。在本公开中所描述的DNA修复技术可以提高从原本无法恢复的受损DNA中恢复序列信息的能力、以及降低为了能够再生编码的数字信息而必须被测序的DNA的量的能力。
本公开内容可以与任何类型的双链(ds)多核苷酸一起使用,因此对“DNA”的引用是说明性的,并不旨在将本申请限制于特定类型的多核苷酸。核糖核酸(RNA)、DNA-RNA杂合体、包括诸如dNaM和dTPT3的非天然碱基的核苷酸、以及包括少于全部四个天然碱基的核苷酸的使用也被包括在本公开所描述的技术范围内。因此,在本文中所使用的“多核苷酸”可以包括具有天然碱基和/或非天然碱基的ds-DNA和ds-RNA。在本文中所使用的DNA也包括具有非天然碱基和/或包括少于天然碱基的全部的DNA链。
图1图示了使用DNA来存储数字信息并从DNA中再生数字信息的示意表示。计算机文件100代表可以被传统计算机访问的任何类型的文件或文档。例如,文件100可以为视频文件或文字处理文档。文件100是数字信息102的一个示例。然而,数字信息102也可以包括其他类型的二进制序列,诸如不对计算机文件100编码的序列。
数字信息102可以被转换为一串核苷酸碱基。用于对数字信息102的整体进行编码所需的核苷酸碱基的数目可以超过单个合成DNA分子的长度。因此,数字信息102的核苷酸编码被分成多个核苷酸序列104。可以存在表示单个文件100所需的数百、数千或数百万个核苷酸序列104。在此阶段,核苷酸序列104不是DNA分子,只有核苷酸碱基中的字符串(例如,大约80-150个字符长的字母序列)当被重组和被正确解码时产生数字信息102。这些字母字符串提供用于建立DNA分子的池的指令,DNA分子将共同存储数字信息102。核苷酸序列104可以包括实现除编码数字信息102以外的目的的子序列,诸如,例如,文件100的标识以及单独的核苷酸序列104中的每个核苷酸序列应该如何被相互重组的标识。
一旦核苷酸序列104被设计,这些核苷酸串的每个核苷酸串被提供到寡核苷酸合成器106,该寡核苷酸合成器106创建具有指定序列的合成DNA 108的池。寡核苷酸合成器106通过将核苷酸连接在由核苷酸序列104所指定的序列中来执行多核苷酸的化学合成。利用当前的多核苷酸合成技术,链沿3'至5'方向生长,相对于自然生物合成,该方向是向后的。作为化学过程,可能发生不正确的相互作用,导致有缺陷的产品。所合成的多核苷酸序列越长,存在的缺陷就越多,因此,就目前的技术而言,这个过程仅实用于产生相对较短的核苷酸序列。针对具有足够质量的DNA分子,当前的实际限制为大约100-200个碱基对(bp)。因此,数字信息102被分成多个核苷酸序列104,每个核苷酸序列具有在约100至200bp的范围内的长度。核苷酸序列104中的每个核苷酸序列可以由寡核苷酸合成器106多次合成以创建多个合成DNA分子,该多个合成DNA分子各自具有相同序列。
由寡核苷酸合成器106产生的合成DNA 108为独立的DNA分子的集合,独立的DNA分子具有由核苷酸序列104指示并且合起来编码数字信息的序列102(包括可以由合成过程引入的任何错误)。在合成DNA 108的池中,可以存在冗余,因为多个分子具有相同的序列、以及由于包括多个不同的核苷酸序列104的多样性。
合成DNA 108可以被放置进存储装置110中,直到需要取回文件100为止。存储装置110可以为诸如在缓冲水溶液中的短期存储装置。存储装置110可以为诸如档案存储装置的长期存储装置,其中合成DNA 108在干燥或其他保藏的状态下被保持数十年、数百年或者甚至数千年。不管存储技术如何,合成DNA 108当在存储装置110中时都可能遭受损伤。
DNA可以发生多种类型的损伤,诸如水解损伤、氧化性损伤、辐射损伤和化学损伤。水解DNA损伤涉及单个碱基的脱氨基或完全去除。AP(无嘌呤/无嘧啶)位点是化学不牢固的并且容易单链断裂。水解损伤可以由各种代谢物的生化反应以及活性氧类的过剩造成。氧化性DNA损伤是指特定碱基的氧化。8-羟基脱氧鸟苷(8-OHdG)为用于氧化性DNA损伤的最常见标志。8-OHdG最常由化学致癌物来形成和增强。紫外线和其他类型的辐射可以以DNA链断裂的形式损伤DNA。这涉及一条或两条DNA链的切割。紫外线引起的损伤也可以导致嘧啶二聚体的产生,其中共价交联发生在胞嘧啶和胸腺嘧啶残基中。最常见的嘧啶二聚体为环丁烷嘧啶二聚体(CPD)和嘧啶(6-4)嘧啶酮光产物(6-4PP)。化学DNA损伤可以由暴露于多环芳烃(PAH)而造成。PAH为通常与石油、煤炭、香烟烟雾和汽车尾气相关联的强有力的、普遍存在的大气污染物。由于PAHs的针对DNA损伤的常见标志为苯并(a)芘二醇环氧化合物(BPDE)。BPDE被发现非常活泼并且已知与DNA的蛋白质、脂质和鸟嘌呤残基共价结合以产生BPDE加合物。损伤的DNA可以存在于存储装置110中,并且损伤的类型和程度可以阻止对DNA的分析,DNA的分析转而阻止数字信息102的恢复。
双链断裂难以修复,因为DNA链的两个部分可以变得彼此分离并且无法重新连接。单链断裂较容易修复,因为互补支架的主链使两条链彼此退火并且保留DNA链的整体结构。如果损伤造成异常受损碱基(例如8-OHdG)或单链断裂,则包括在本公开中的技术是有用的。
从存储装置110中提取的DNA分子可以利用包含一种或多种酶的修复混合物112来修复。包括在修复混合物112中的酶可以为聚合酶、连接酶、核酸内切酶和/或糖基化酶。DNA聚合酶为从脱氧核糖核苷酸(DNA的构建块)合成DNA分子的酶。DNA聚合酶将核苷酸添加到DNA链的3'端,一次添加一个核苷酸。DNA连接酶为特定类型的酶(连接酶),其通过催化磷酸二酯键的形成来共同促进DNA链的连接。连接酶为可以通过形成新的化学键来催化两个大分子连接的酶。核酸内切酶为在多核苷酸链内使磷酸二酯键裂解的酶。核酸内切酶在DNA修复中起作用。AP核酸内切酶特别地仅在AP位点催化DNA的切开并且因此针对随后的切除、修复合成和连接反应来准备DNA。糖基化酶为水解糖基化合物的酶。DNA糖基化酶为参与碱基切除修复的一族酶。碱基切除修复为去除和替换在DNA中受损碱基的机制。DNA糖基化酶催化这个过程的第一步。糖基化酶在使糖-磷酸主链保持完整、创建AP位点的同时去除受损含氮碱基。
从存储装置110中移除的DNA可以在许多不同条件下利用修复混合物112来孵化。例如,来自存储装置110的DNA可以在37℃处与修复混合物112结合多个小时,诸如两个、三个、四个、五个或六个小时。
在将来自存储装置110的DNA与修复混合物112接触后,修复的DNA 114可以被放进热循环仪116中并通过PCR来扩增。PCR为在分子生物学中使用的已知技术,以指数式扩增DNA的特定片段的单个拷贝或几个拷贝,以生成该特殊DNA片段的数千个到数百万个拷贝。许多PCR方法依赖于热循环。热循环使反应物展露于反复加热和冷却的循环中,以允许不同温度依赖性反应,特别是DNA解链和酶驱动的DNA复制。PCR采用两种主要试剂:引物(为短的单链DNA片段,被称为寡核苷酸,该寡核苷酸为目标DNA区域的互补序列)以及DNA聚合酶。在PCR的第一步中,DNA双螺旋的两条链在被称为DNA解链的过程中的高温下被物理分离。在第二步中,温度被降低,并且引物结合到DNA的互补序列上。然后,两条DNA链变成用于DNA聚合酶的模板以从自由核苷酸(DNA的A、C、G和T)中酶促地组装新的DNA。随着PCR继续进行,生成的DNA本身被用作用于复制的模板,启动链式反应,在该链式反应中原始DNA模板被指数式扩增。热循环仪116为通常被用于经由PCR来扩增DNA的片段的机器。大多数热循环仪116的设计包括具有孔的热块,其中容纳反应混合物的管可以被插入。然后,热循环仪116在离散的、预编程的步骤中升高和降低块的温度,以提供热循环。
PCR扩增增加了可用于DNA测序仪118分析的DNA链的数目。然而,PCR可以引入错误,所以由PCR产生的DNA分子不是修复的DNA 114的全部相同的拷贝是可能的。并非所有类型的DNA测序都需要热循环仪116的使用和通过PCR的修复的DNA 114的扩增。因此,在本公开中提供的技术同样可适用于不将PCR用作预测序步骤的实现。
DNA测序仪118检测在DNA样本中的单个核苷酸的序列,并且生成代表该序列的序列数据120。考虑到DNA的样本,诸如PCR产物,DNA测序仪118确定核苷酸碱基的顺序。然后这个被报告为序列数据120,该序列数据可以是被称为“读取”的文本字符串。该读取可以处于任何合适的格式,诸如纯文本、FASTQ、EMBL或FASTA。存在许多不同类型的DNA测序仪118和测序技术。本公开的内容可以与能够确定在DNA的链中核苷酸碱基的顺序的任何方法或技术一起使用。
能够被使用的测序技术为合成测序(
能够被使用的另一测序技术为纳米孔测序。纳米孔为直径约一个纳米级别的小孔。由于离子穿过纳米孔的传导,纳米孔在导电流体中的沉浸以及跨纳米孔的电势的应用引起微小的电流。流经纳米孔的电流量对纳米孔的尺寸敏感。当多核苷酸分子经过纳米孔时,在多核苷酸分子上的每个核苷酸以不同程度阻塞纳米孔。因此,当多核苷酸分子经过纳米孔时,经过纳米孔的电流的变化表示多核苷酸序列的读取。纳米孔测序比合成测序具有更高的错误率(例如,超过10%)。然而,纳米孔测序的读取长度更长-长达800000bp。
由DNA测序仪118输出的序列数据120可以被解码以创建再生的数字信息122。序列数据120的解码可以为被用于对核苷酸序列104进行编码的过程的逆向。该编码/解码过程可以包括错误校正和冗余,从而即使存在诸如由寡核苷酸合成器106、存储时的损伤、PCR扩增或DNA测序仪118引入的错误,数字信息102也正确地再生。由寡核苷酸合成、PCR扩增和DNA测序引入的错误通常具有能够由错误校正和冗余来解决的类型和范围。在数字信息恢复的上下文中,对合成DNA 108的损伤必须依据单个DNA分子的池来考虑。如果通过该受损DNA分子来编码的序列数据120能够从在池中的其他DNA分子中被获得,则对单个DNA分子的损伤可以对创建再生数字信息122的最终能力没有影响。然而,对在合成DNA 108的池中的大量单个DNA分子在存储装置110期间的大量损伤可以引起恢复的数字信息122不准确,或者甚至可以阻止原始数字信息102的恢复。因此,在一些实例中,为了获得合成DNA 108的足够代表性样本以恢复所存储的数字信息102,在测序之前对合成DNA 108的酶促修复可以是必要的。
说明性过程
为了易于理解,本公开中所讨论的操作被描绘为作为独立框的单独操作。然而,这些分离地描绘的操作不应当被解释为其执行中所依赖的必需顺序。过程被描述的顺序不旨在被解释为限制,并且任何数目的所述过程框可以以任何顺序来组合以实施过程或备选过程。此外,还有可能所提供的操作的一个或多个被修改或省略。图2-4可以通过图1所示的系统和装置来被全部或部分实施。
图2示出了用于修复存储的编码数字信息的DNA的过程200。
在202处,数字信息被编码为核苷酸的序列。该数字信息可以表示计算机文件或可执行计算机程序的全部或部分。编码可以使用本领域技术人员已知的任何编码技术,以用于将二进制信息翻译成包括核苷酸碱基的代码。编码模式可以包括错误校正,诸如,例如,汉明(Hamming)码、里德-所罗门(Reed-Solomon)码、多维奇偶校验、Bose-Chaudhuri-Hocquenghem码或任何其他合适的错误校正码。
在204处,合成DNA分子被合成。该合成DNA分子包括编码数字信息的核苷酸的序列的至少部分。合成DNA分子也可以包括编码其他信息的核苷酸。合成DNA分子可以通过寡核苷酸合成器(诸如图1中所介绍的寡核苷酸合成器106)来合成。合成DNA分子的长度可以在大约80–250bp之间、大约100–180bp之间或大约120–160bp之间。
在206处,合成DNA分子被存储。合成DNA分子可以使用任何用于存储DNA的技术来存储。例如,合成DNA分子可以被存储在缓冲水溶液(例如,Tris-EDTA)中,并在约4℃下冷藏。另外,合成DNA分子可以在-20℃下被冷冻或在-80℃下作为沉淀被存储在乙醇中。DNA也可以在-196℃下被存储在液氮中。合成DNA分子可以在室温下被存储,诸如通过干燥DNA以及将其存储在纸卡(例如,
在208处,合成DNA分子与修复混合物接触。修复混合物可以为在图1中介绍的修复混合物112。修复混合物包括至少一种酶,并且可以包括DNA聚合酶、DNA连接酶或核酸内切酶中的任一或全部。针对在本公开中所描述的所有酶,源自天然来源的酶或从重组来源获得的酶可以被使用。
可以被使用的一种合适的DNA聚合酶为Bst DNA聚合酶。Bst聚合酶源自于革兰氏阳性(Gram-positive)细菌的杆状嗜热芽孢杆菌(Bacillus stearothermophilus)。Bst聚合酶具有类似解旋酶的活性,使其能够解链DNA链。
可以被使用的一种合适的DNA连接酶为Taq DNA连接酶。Taq DNA连接酶为耐热性的连接酶,该连接酶催化在两条相邻DNA链的5'-磷酸和3'-羟基之间的磷酸二酯键的形成。该酶源自于嗜热细菌水生栖热菌(Thermus aquaticus)。
可以被包括在修复混合物中的一种合适的核酸内切酶为最初从肠杆菌噬菌体T4分离的T4核酸内切酶V。该酶具有DNA糖基化酶和APlyase活性两者。T4核酸内切酶V识别由紫外线照射引起的顺式-顺式-环丁烷嘧啶二聚体(cis-syn-cyclobutane pyrimidinedimers)。该酶裂解嘧啶二聚体的5'-端的糖基键,并且内切核苷酸的活性裂解在AP位点的磷酸二酯键。
可以被用于DNA修复的一种合适的核酸内切酶为核酸内切酶IV。核酸内切酶IV可以作用于在DNA中的多种氧化性损伤。该酶为无嘌呤的/无嘧啶的(AP)核酸内切酶,该酶将水解在DNA中的完整的AP位点。AP位点在为5'的第一磷酸二酯键处被裂解到病变(lesion),在3'终端处留下羟基并且在5'终端处留下脱氧核糖5'-磷酸盐。该酶还具有3'-二酯酶活性并且可以从DNA的3'-端释放磷酸甘油醛、完整的脱氧核糖5-磷酸盐和磷酸盐。
可以被包括在修复混合物中的另一种合适的核酸内切酶为核酸内切酶VIII。该核酸内切酶来自充当N-糖基化酶和AP-裂解酶的E.coli。N-糖基化酶活性从双链DNA中释放受损嘧啶,生成无嘌呤(AP位点)。AP-裂解酶活性裂解3'和5'到AP位点,留下5'-磷酸盐和3'-磷酸盐。被核酸内切酶VIII识别并去除的受损碱基包括尿素、5,6-二羟基胸腺嘧啶、胸腺嘧啶二醇、5-羟基-5-甲基乙内酰脲、尿嘧啶二醇、6-羟基-5、6-二氢胸腺嘧啶和甲基酒石酰脲(1,2)。当核酸内切酶VIII类似于核酸内切酶III时,核酸内切酶VIII具有β和δ裂解酶活性,而核酸内切酶III仅具有β裂解酶活性。
将合成DNA分子与修复混合物接触可以在多种反应条件下被执行。合成DNA分子可以在20-60℃、30-50℃、35-45℃或大约37℃的温度下利用反应混合物来孵化。孵化的时长可以为2-8小时、3-5小时或大约4小时。值得注意的是,在将合成DNA与修复混合物接触之前,合成DNA可以被维持在约室温或更冷的温度下。因此,过程200的先前步骤可以在小于25℃、小于22℃、小于19℃、小于16℃或更低的温度下被执行。如在本文中所使用的,“大约(approximately)”或“约(about)”表示所述值的±10%的范围。
在210处,在与修复混合物接触之后,合成DNA被测序以生成序列数据。测序可以通过用于DNA测序的任何合适的技术来被执行。例如,测序可以通过在图1中介绍的DNA测序仪118来被执行。
在212处,在210处获得的序列数据被解码以获得再生的数字信息。如果合成DNA已被充分修复,并且在过程中引入的任何其他错误已被缓解,则再生的数字信息将包含与最初在核苷酸中编码的数字信息相同或几乎相同的二进制序列。
图3示出了用于包括酶促修复的DNA的预测序处理的过程300。
在302处,合成DNA与修复混合物接触。该修复混合物可以为在图1中介绍的修复混合物112。该修复混合物包含一种或多种适合于修复DNA的酶。在实施方式中,修复混合物包括Bst DNA聚合酶、Taq DNA连接酶、核酸内切酶IV、T4核酸内切酶V或核酸内切酶VIII中的至少一种。在实施方式中,修复混合物包括Bst DNA聚合酶、Taq DNA连接酶、核酸内切酶IV、T4核酸内切酶V和核酸内切酶VIII。修复混合物还可以被配制,使其仅包括单一酶。例如,修复混合物可以仅包含酶T4核酸内切酶V或核酸内切酶IV。利用单一酶配制的修复混合物可以包含其他非酶成分,诸如水、缓冲液、dNTPs和氧化的烟酰胺腺嘌呤二核苷酸(NAD
合成DNA可以为由诸如寡核苷酸合成器106的寡核苷酸合成器合成的DNA。合成DNA具有至少部分通过数字信息的编码来预定的序列。合成DNA的长度可以大约在50-200bp、80-180bp或120-160bp之间。合成DNA与修复混合物的接触可以在约37℃的温度下被执行约四个小时。
在304处,合成DNA通过PCR来扩增。PCR可以通过诸如在图1中介绍的热循环仪116的热循环仪的使用来被执行。通过与修复混合物接触来修复合成DNA增加了能够通过被用于PCR的引物来扩增的DNA的完整链的数目。与不使用修复混合物来扩增合成DNA相比,这引起来自DNA的池的不同DNA链的增加数目的扩增。因此,在修复之后的扩增产物提供了在DNA的池中存储的多种DNA序列的更好表示。与在相同条件下未经修复的合成DNA的PCR扩增相比,由于利用修复混合物来准备DNA而扩增的不同DNA链的数目的增长可以为大约1.5倍、1.8倍、2.0倍、4.0倍或更多的增长。因此,在一些实现中,在302处使合成DNA与修复混合物接触可以使在304处通过PCR产生的DNA的量加倍。
在306处,在PCR扩增之后的合成DNA被测序。测序可以通过任何合适的技术来执行,以读取DNA分子的核苷酸序列,诸如在图1中介绍的DNA测序仪118。由于使用修复混合物,完整DNA链的数目的增长提供了更多待测序的DNA不同分子。更多数目的不同DNA分子允许测序以捕获更多的在DNA池序列中存储的多种DNA,并且在不严格的测序条件下可以允许序列数据数字信息被解码。测序条件可以被放松的一种方式为通过减小覆盖范围或读取深度。DNA测序的覆盖范围(或深度)为在重建序列中包括给定核苷酸的唯一读取的数目。
图4示出了用于从受损DNA链中恢复数字信息的过程400。从被用于数据存储的DNA中恢复数字信息的目的是能够无错误地重建原始数字信息。这在图1中通过与数字信息102相同的再生数字信息122的序列来说明。在存储期间对DNA链造成的损伤可以被认为是在通信通道中的“噪声”。DNA修复是用于降低噪声的技术。
在402,在合成DNA的样本中的完整DNA链的数量小于阈值水平是确定的。在样本中的DNA的完整链的数量可以通过定量PCR(qPCR)来测量。qPCR使用DNA扩增的线性度来确定在样本中的DNA的数量。通过在反应中使用荧光报告分子,随着PCR反应继续进行,在qPCR试验中实时测量DNA生成是可能的。荧光超过检测阈值(例如,0.2个荧光单位)并且变得可测量的PCR循环的数目被称为定量循环(Cq)。包括大量可扩增的DNA的起始样本(即,具有引物结合位点的完整DNA链)利用比具有更少量的DNA的样本的更少的PCR循环来达到该阈值。针对具有最佳PCR效率(100%)的序列,每个PCR循环使DNA的数量加倍,所以1的Cq值的增长相当于检测到DNA的初始数量的50%减少。因此,Cq值与在样本中的目标DNA的量成反比。
阈值水平可以基于成功恢复数字信息所需的DNA的数量。DNA的数量与恢复数字信息的能力没有直接关联。大量的仅有几条不同的DNA链可以提供DNA的合理量值,但是由DNA链编码的序列将不会包括再生数字信息所需的数据的全部。但是,假设在存储期间造成的损伤大致随机分布在DNA分子的池的各处,计算可以被进行以标识可能包含足够多样的单个DNA分子的DNA的量值。
阈值水平也可以基于存储前后针对合成DNA的Cq值的变化。在合成DNA的合成后,池的试样可以被采用,并且DNA的数量使用qPCR来测量。这为在存储之前的DNA的量提供了起始值。存储后,Cq值被再次测量。如上所述,Cq值减少一大概指示可用DNA的减半。如果Cq值之差大于阈值数(例如,1.0、2.0、3.0)。阈值数可以基于从DNA的其他池中恢复数字信息的实验来实验式地确定。用于在合成DNA中编码数字信息的技术以及在合成DNA分子的合成中的冗余水平可以影响阈值。
在404处,合成DNA与修复混合物接触。修复混合物包括DNA聚合酶、DNA连接酶或核酸内切酶中的至少一种。修复混合物可以与在图1中所介绍的修复混合物112相同。备选地,如果存在足够数量的完整DNA链以继续进行是确定的,则合成DNA可以在不首先使用修复混合物的情况下被测序。
在406处,合成DNA被测序以生成序列数据。合成DNA可以通过在图1中所介绍的DNA测序仪118来测序。取决于DNA的数量和所使用的测序技术,测序可以前面是修复的DNA的PCR扩增。
在408处,序列数据被解码以获得再生的数字信息。
示例
图5示出了合成dsDNA链500和结合到DNA链500的五种不同引物的示意图。DNA链500具有113bp(SEQ ID:1)的长度。该DNA链500在以下示例中被使用。
第一正向引物(Primer1F 502(SEQ ID:2))结合到DNA链500的一端。当与结合到DNA链500的另一端的第一反向引物(Primer1R 504(SEQ ID:3))配对时,扩增产物为DNA链500的整个113bp长度。第二正向引物(Primer2F 506(SEQ ID:4))结合到DNA链500,该DNA链500部分地与Primer1F 502的结合位点重叠。使用Primer2F 506作为正向引物的PCR扩增产物以及作为反向引物的第二反向引物(Primer2R 508(SEQ ID:5))为来自DNA链500的中间的83bp长片段。当与第二正向引物(Primer2F 506)一起使用时,位于靠近DNA链500的中间的第三引物(Primer3R 508(SEQ ID:6))创建为53bp长的扩增产物。
可以从DNA链500创建的这三个不同长度的序列被用于测试链长度对DNA修复和恢复的影响。
链长和酶促修复对DNA恢复的影响
图6示出了条形图600,比较了在具有酶促修复和不具有酶促修复的样本中的完整DNA的量。三个不同长度53bp、83bp和113bp的DNA样本使用上述引物由图5的DNA链500来创建。DNA样本被暴露在高热和高湿下以模拟老化。在该示例中,DNA样本被存储在70℃和50%相对湿度(RH)下的烘箱中两个小时。这模拟了在室温和20%RH下大约存储15年。暴露于高热量和高湿气相信会对DNA样本主要造成水解损伤。不受理论的束缚,相信对DNA样本的损伤包括DNA链的主链的断裂。
在条形图600的纵轴上示出为100%的DNA样本的初始浓度通过在暴露于高热和高湿之前测量针对每个样本的DNA浓度来确定。在该示例中,定量PCR(qPCR)被使用以测量在暴露于高热和高湿之前和之后的在样本中的DNA的量。使用
在没有酶促修复的情况下,可用DNA的超过一半在暴露于高热和高湿期间被损失。DNA损失的百分比随DNA样本的长度增加而增加。针对53bp DNA链,原始DNA的约55%被损失,以及针对113bp DNA链,原始DNA的约65%被损失。
然而,利用酶促修复,可以被恢复的完整DNA的量大约为从未修复样本中的恢复的两倍。针对53bp DNA链和83bp DNA链,在利用修复混合物的酶促修复之后,原始DNA数量的大约85%被恢复。针对更长的113bp DNA样本,原始DNA数量的超过70%被恢复。
在该示例中,修复混合物为可来自于New England BioLabs Inc.(登记#M0309S)的可用的
孵化时间的比较
图7和图8为条形图700和800,示出了修复时间对从受损DNA样本中恢复的DNA的量的影响。在图7和图8中被分析的样本为在图5中所介绍的全长113bp DNA链500。修复的样本在上述条件下利用
在图7的条形图700中所示的样本被暴露于70℃温度和50%RH下三个小时。被标记为“参考”的列表示在暴露于高热和高湿之前样本中的DNA的数量。在没有修复的情况下,DNA的原始数量的少于40%被恢复。随着利用修复混合物的孵化时长的增加,被恢复的DNA的量逐渐增加。与利用修复混合物的四个小时的孵化后,大约70%的最高恢复被获得。
图8的条形图800所示的样本值在被用于生成图7所示的数据的相同条件下被生成,除了暴露于70℃温度和50%RH的时长为四个小时。在暴露于高热和高湿的该时长之后,未修复的DNA样本的仅约20%保留下来。有趣的是,将利用修复混合物的孵化的时长增加到四个小时以上降低了DNA恢复的量。在这组样本中,在经过四个小时的利用修复混合物的孵化后,约为原始量的45%的DNA恢复的最高量被获得。在图7和图8之间的孵化的四个小时后,被恢复的DNA的量差可能是由于针对图8中的样本在高热和高湿下的增加的暴露。
修复混合物的比较
下表1比较了不同酶组合有效修复受损DNA的能力。被测试的酶为Bst DNA聚合酶、T4核酸内切酶V、核酸内切酶IV、核酸内切酶VIII、DNA连接酶和尿嘧啶-DNA糖基化酶(UDG)。所有酶从New England BioLabs Inc.来获得,并且根据包括在提供的缓冲液中的标准稀释的制造商的规范来准备。DNA样本为来自图5的DNA链500的113bp全长序列。来自长期存储的损伤通过将溶液中的DNA样本暴露在70℃和50%RH下24小时来模拟。
除了预混溶液,每个样本通过将5ng DNA与50μL超纯水混合来准备。预混溶液包括5μL的可来自于New England BioLabs.Inc.(登记#B9004S)的
修复后可用的DNA的量通过使用Roche
“未受损”的样本为未经受高热和高湿的DNA链的样本。意料中的,该样本比任何其他样本具有更低的Cq值。“无修复”样本为未利用修复混合物来被孵化的DNA的样本。该样本的Cq值为31.11。因此,低于31.11的Cq值表明由酶促修复带来的益处。高于31.11的值表明在这些条件下修复混合物没有产生任何益处。
“对照”样本为在没有任何酶的情况下利用预混溶液来孵化的DNA的样本。27.59的用于对照样本的Cq值比用于许多包括酶的反应混合物的Cq值低。不受理论的束缚,相信当处理非常低的DNA浓度时,修复损伤的预混溶液的成功是人为存在。在更高的DNA浓度下类似的益处未被预期。
表1各种酶组合修复受损DNA的能力的比较
出人意料地,各自仅包含单个核酸内切酶、T4核酸内切酶V和核酸内切酶IV的修复混合物17和18比具有多种酶的修复混合物表现更好。不受理论的束缚,相信多种酶可以彼此干扰并且降低修复受损DNA的能力。虽然包含多种酶的修复混合物可以被用于修复已遭受许多不同类型损伤的DNA链的损伤,但如果DNA链以相同的方式受到损伤,酶的混合物可以降低修复功效。
表2比较了具有单一核酸内切酶的修复混合物对
表2包含单个核酸内切酶的修复混合物与商业DNA修复混合物的比较
这些比较表明,DNA链的长度对DNA被修复的能力具有较小的影响。此外,在
表3比较了酶浓度和孵化时间对修复功效的影响。
“未受损”的样本为未经受高热和高湿的DNA链的样本。然而,该样本可以具有来自测试之前的短期存储的一定程度的损伤。“无修复”样本为未利用修复混合物来孵化的DNA样本。该样本的Cq值为7.91。因此,在该比较中,低于7.91的Cq值表明由酶促修复带来的益处。
表3酶浓度和孵化时间的比较。
在该比较中的所有样本示出了相对于“无修复”样本的改进。用于四个小时的孵化产生比用于三个小时或用于五个小时的孵化更低的Cq值,因此产生更高的DNA数量。将
最好的结果,具有6.04的Cq值的样本号7,利用两倍
说明性实施例
以下条款描述了用于实现在本公开中所描述的特征的多个可能的实施例。在本文中所描述的各种实施例不是限制性的,来自任何给定实施例的每个特征也不要求在其他实施例中存在。除非上下文另外明确指示,否则任何两个或更多个实施例可以被组合在一起。如在此文档的本文中所使用的,“或”意味着和/或。例如,“A或B”意味着没有B的A、没有A的B或A和B。如在本文中所使用的,“包括”意味着包括所有列出的特征并且可能包括未列出的其他特征的添加。“基本上由……组成”意味着包括所列出的特征和不会实质性影响所列出特征的基本特性和新颖特性的那些附加特征。“由...组成”仅意味着所列出的特征而排除未列出的任何特征。
条款1。在脱氧核糖核酸(DNA)中存储数字信息的方法,包括:将数字信息编码为核苷酸的序列;合成具有核苷酸的序列的合成DNA分子;存储合成DNA分子;将合成DNA分子与修复混合物接触,该修复混合物包括以下中的至少一项:DNA聚合酶、DNA连接酶或核酸内切酶;在与修复混合物的接触之后,对合成DNA分子进行测序以生成序列数据;以及对序列数据进行解码以获得再生的数字信息。
条款2。根据条款1所述的方法,其中对数字信息进行编码使用包括错误校正的编码方案。
条款3。根据条款1或条款2所述的方法,其中合成该合成DNA分子通过寡核苷酸合成器来执行,并且该DNA分子为大约在100-180个碱基对(bp)长之间。
条款4。根据条款1-3中的任一项所述的方法,其中将合成DNA分子与修复混合物的接触是在大约37℃下被执行大约4小时。
条款5。根据条款1-4中的任一项所述的方法,其中,继合成该合成DNA分子之后,合成DNA分子被维持在不大于约25℃的温度下,直到将合成DNA分子与修复混合物接触。
条款6。根据条款1-5中的任一项所述的方法,其中修复混合物包括Bst DNA聚合酶。
条款7。根据条款1-6中的任一项所述的方法,其中修复混合物包括Taq DNA连接酶。
条款8。根据条款1-7中的任一项所述的方法,其中修复混合物包括以下中的至少一项:T4核酸内切酶V、核酸内切酶IV或核酸内切酶VIII中。
条款9。一种修复对数字信息进行编码的合成DNA的方法,包括:将合成DNA与修复混合物进行接触,该修复混合物包括以下中的至少一项:DNA聚合酶、DNA连接酶或核酸内切酶;以及通过聚合酶链反应(PCR)来扩增合成DNA,其中与不与修复混合物接触的合成DNA的PCR扩增相比,在与修复混合物的接触之后的合成DNA的PCR扩增将Cq循环数降低至少1.5。
条款10。根据条款9所述的方法,其中合成DNA具有约100-180bp的长度,并且根据数字信息的预定编码通过寡核苷酸合成器来被合成。
条款11。根据条款9或10所述的方法,其中合成DNA与修复混合物的接触在约37℃下被执行约4小时。
条款12。根据条款9-11中的任一项所述的方法,其中修复混合物包括以下中的至少一项:Bst DNA聚合酶、Taq DNA连接酶、核酸内切酶IV、T4核酸内切酶V或核酸内切酶VIII。
条款13。根据条款9-12中的任一项所述的方法,其中修复混合物包括Bst DNA聚合酶、Taq DNA连接酶、核酸内切酶IV、T4核酸内切酶V和核酸内切酶VIII。
条款14。根据条款9-13中的任一项所述的方法,其中修复混合物基本上由T4核酸内切酶V组成。
条款15。根据条款9-13中的任一项所述的方法,其中修复混合物基本上由核酸内切酶IV组成。
条款16。根据条款9-15中的任一项所述的方法,其中与未与修复混合物接触的合成DNA的PCR扩增相比,在与修复混合物的接触之后的合成DNA的PCR扩增将Cq循环数降低至少2.0。
条款17。根据条款9-16中的任一项所述的方法,还包括在PCR扩增之后对合成DNA进行测序。
条款18。恢复存储在合成DNA中的数字信息的方法包括:确定在合成DNA中的完整DNA链的数量小于阈值水平;将合成DNA与修复混合物进行接触,该修复混合物包括以下中的至少一项:DNA聚合酶、DNA连接酶或核酸内切酶;以及对合成DNA进行测序以生成序列数据。
条款19。根据条款18的方法,其中阈值水平是基于存储之前的合成DNA的Cq值和存储之后的合成DNA的Cq值之差。
条款20。根据条款18或19的所述方法,还包括对序列数据进行解码以获得再生的数字信息。
结论
尽管已经用特定于结构特征和/或方法动作的语言描述了主题,但应该理解,在所附权利要求书中定义的主题不必限于上述特定的特征或动作。而是,特定的特征和动作被公开作为实施权利要求的示例形式。
在描述本发明的上下文中所使用的术语“一”、“一个”、“该”和类似指示物(尤其是在以下权利要求的上下文中)应解释为涵盖单数和复数,除非在本文中另有指示或与上下文明显矛盾。术语“基于”、“根据”和类似指示物应解释为含义“至少部分基于”,其包括为“部分基于”和“整体基于”,除非另有指示或与上下文明显矛盾。
在本文中描述了某些实施例,包括发明人已知的用于实施本发明的最佳模式。当然,在阅读了前面的描述之后,这些所描述的实施例的变型对于本领域普通技术人员而言将变得明显。技术人员将知道如何适当地采用这样的变型,并且在本文中所公开的实施例可以不同于具体所描述的来实践。因此,在所附权利要求书中记载的主题的所有修改和等价都包括在本公开的范围内。而且,除非在本文中另外指示或与上下文明显矛盾,否则本发明涵盖了上述元素在其所有可能的变化中的任何组合。
- 酶促DNA修复
- 通过酶促水解及随后的化学/酶促酯化生产生物柴油
