一种长吻鮠的DNA指纹图谱及其构建方法和应用
文献发布时间:2024-05-31 01:29:11
技术领域
本发明属于分子生物学领域,具体涉及一种长吻鮠的DNA指纹图谱及其构建方法和应用。
背景技术
长吻鮠(L.longirostris)隶属于鲇形目(Siluriformes)、鲿科(Bagridae)、鮠属(Leiocassis),因其具有肉嫩刺少、营养价值高等优点,深受消费者的喜爱,是我国重要的经济鱼。长吻鮠主要分布在长江、淮河、珠江等水域,由于长期的地理隔绝,不同地理位置的长吻鮠的遗传性状存在差异,为了研究长吻鮠的遗传背景、遗传结构及不同地理群体之间的关系,实现长吻鮠种质资源的精准化管理和高效利用十分必要。
目前,现有的长吻鮠种质资源或者群体鉴定依然主要采用传统的外观评定方法。DNA指纹图谱技术在水产养殖品种上的应用并不多,以SNP分子标记来构建长吻鮠DNA指纹图谱的更是未见报道,所以构建长吻鮠的DNA指纹图谱十分有必要。
目前长吻鮠品种鉴定及区域分布技术中主要存在的问题:1、现有形态学方法对长吻鮠种质资源进行鉴定,受人为因素和环境条件影响较大。2、现有技术中利用微卫星标记技术(SSR)或随机扩增多态性DNA技术(RAPD)分析不同群体长吻鮠的遗传多样性,根据亲缘关系远近进行群体鉴定,但这些分子标记技术,所需实验周期长,精确度低。3、已有利用第三代分子标记技术,定位长吻鮠与其他品种鱼类在基因组上的插入缺失,能够鉴定待测样本是否为长吻鮠群体,但此方法无法更进一步鉴定其区域分布。
发明内容
发明目的:针对现有技术存在的问题,本发明提供了用于鉴定长吻鮠的DNA指纹图谱,利用该DNA指纹图谱可以快速、高效和准确的鉴定未知群体是否为长吻鮠,并明确所属群体及其具体生活地区。本发明还提供所述长吻鮠的DNA指纹图谱的构建方法和应用。本发明首次提出了用于鉴定长吻鮠的DNA指纹图谱并且准确度高。
技术方案:为了实现上述目的,本发明所述一种长吻鮠的DNA指纹图谱,所述DNA指纹图谱包括121个SNP位点,所述SNP位点及该处的特异性核苷酸如下:
本发明所述的长吻鮠的DNA指纹图谱,所述长吻鮠的指纹图谱包括广东群体长吻鮠的DNA指纹图谱、湖北群体长吻鮠的DNA指纹图谱、四川群体长吻鮠的DNA指纹图谱和江苏群体长吻鮠的DNA指纹图谱,四种DNA指纹图谱均包括121个SNP位点,所述SNP位点及该处的特异性核苷酸如下,所述广东群体长吻鮠的DNA指纹图谱的SNP位点及特异性核苷酸如下:
所述湖北群体长吻鮠的DNA指纹图谱的SNP位点及特异性核苷酸如下:
所述四川群体长吻鮠的DNA指纹图谱的SNP位点及特异性核苷酸如下:
所述江苏群体长吻鮠的DNA指纹图谱的SNP位点及特异性核苷酸如下:
本发明所述的长吻鮠的DNA指纹图谱的构建方法,包括如下步骤:
(1)广泛收集不同地理群体长吻鮠的尾鳍;
(2)对收集到的尾鳍组织使用DNA提取试剂盒进行提取及质量检测;
(3)对质控合格后的片段进行重测序;
(4)对重测序数据进行检测及分析质量分布检测、碱基分布检测及污染监测;(5)选择长吻鮠全基因组作为参考基因组,将简化后的Clean Reads比对到参考基组上,位置排序和标记重复reads,最终统计各项比对指标,比对率以及覆盖率指标能反映样本、建库及测序以及参考序列的质量;
(6)使用Sentieon对每个样本进行变异位点检测,获得每个样本的gVCF,在后续的joint-calling步骤中,再对所有样本的gVCF进行联合分析,得到群体中每个个体的变异结果,构建长吻鮠的特异性SNP位点数据库;
(7)根据缺少率、MAF值、单拷贝、位点杂合度和深度进一步过滤,筛选高多态性SNP位点,用于构建DNA指纹图谱。
其中,步骤(2)所述对收集到的尾鳍组织使用DNA提取试剂盒进行提取及质量检测为:进行DNA提取,测OD值及浓度,并跑电泳检测质量后使用,其中OD
其中,步骤(4)中对重测序数据进行质量分布检测、碱基分布检测及污染监测,保证数据质量。
其中,步骤(5)中参考基因组为NCBI上的PRJNA692071长吻鮠全基因组。
其中,步骤(6)中变异位点检测,为保证SNP准确性,需要进行质控,对于合并后得到的SNP位点通过过滤参数进行初步过滤,过滤参数为QD<2.0||FS>60.0||MQ<40.0||SOR>3.0||MQRankSum<-12.5||ReadPosRankSum<-8.0。
其中,步骤(7)中根据位点缺失率为0和MAF值大于等于0.1进行过滤;再提取位点上下游100bp的序列进行拷贝数分析,保留位点上下游序列在基因组上是唯一的这种位点;其次通过过滤位点杂合率小于0.1和平均深度大于5的位点,然后按照>4Mb的间隔进行筛选位点,保证位点间距,进行进一步过滤。
本发明所述的长吻鮠的DNA指纹图谱的构建方法在不同群体长吻鮠鉴定以及长吻鮠具体生活地区鉴定中的应用。
其中,所述应用的过程为:
(1)对待测长吻鮠的尾鳍组织样本进行DNA提取及重测序;
(2)对重测序后数据进行SNP位点筛选;所述筛选条件为设置位点缺失率为0和MAF值≥0.1进行过滤;然后再提取位点上下游100bp的序列进行拷贝数分析,保留位点上下游序列在基因组上是唯一的位点;再通过过滤位点杂合率<0.1和平均深度>5的位点;最后按照间隔>4Mb进行筛选位点,得到待测鱼样本的SNP位点;
(3)将步骤(2)中得到的SNP位点处的核苷酸与长吻鮠的DNA指纹图谱中SNP位点相比较,符合率≥95%时,将待测样本鉴定为长吻鮠群体。
本发明提供了所述长吻鮠的DNA指纹图谱在鉴定长吻鮠中的应用以及一种具体的长吻鮠的鉴定方法,包括以下步骤:检测待测鱼样本的基因组DNA中121个SNP位点处的核苷酸,并与权利要求1中所述的长吻鮠的DNA指纹图谱中对应的121个SNP位点处的特异性核苷酸相比较,符合率≥95%时,将待测鱼样本鉴定为长吻鮠。
作为优选,所述待测鱼样本为鱼的尾鳍组织。
进一步地,所述长吻鮠生活地区包括广东、四川、湖北和江苏。
进一步地,待测鱼样品鉴定为长吻鮠群体后,还包括鉴定长吻鮠具体生活地区的步骤,所述鉴定长吻鮠具体生活地区的步骤为计算样本间遗传距离,与该发明中样本进行亲缘关系比较分析,最终鉴定长吻鮠具体生活地区为广东、四川、湖北或江苏。这四个地区有着优质长吻鮠鱼种质资源,能为长吻鮠新种质创制提供很好的育种材料。因此,本发明选择了利用这4个地理群体的长吻鮠的DNA建立长吻鮠的DNA指纹图谱。
本发明首次利用高多态性SNP位点构建长吻鮠DNA指纹图谱。本发明公开了长吻鮠的DNA指纹图谱及其应用。本发明所述的长吻鮠的DNA指纹图谱包括121个特异性的SNP位点,本发明提供的DNA指纹图谱库以及特定群体(广东群体、四川群体、湖北群体和江苏群体)长吻鮠品种的DNA指纹图谱基于重测序得到,用于长吻鮠品种鉴定时,突破了传统的外观评定方法,可以更准确的追朔长吻鮠的群体来源,提供更加客观、准确的鉴定结果。
有益效果:本发明与现有技术相比,其显著优点是:
1、本发明首次利用121个高多态性SNP位点构建了长吻鮠的DNA指纹图谱,能够更加有效且精准的鉴别不同群体长吻鮠以及长吻鮠具体生活地区。
2、本发明通过对四个不同地区(广东、湖北、四川和江苏)的长吻鮠进行重测序,构建的长吻鮠的DNA指纹图谱更加全面,能够更加有效的进行长吻鮠种质识别,与传统的群体鉴定方法相比,本方法更加客观、准确和可靠,可以实现长吻鮠种质资源的精准化管理和高效利用。
3、本发明能够同时实现鉴别未知群体是否为长吻鮠以及明确其所属地区,更加高效。
4、本发明利用高多态性SNP位点作为长吻鮠不同群体的鉴定指标,SNP位点属于第三代分子标记技术,与前两代分子标记相比,其具有测序时间短、性价比高和测序通量高等优势。
附图说明
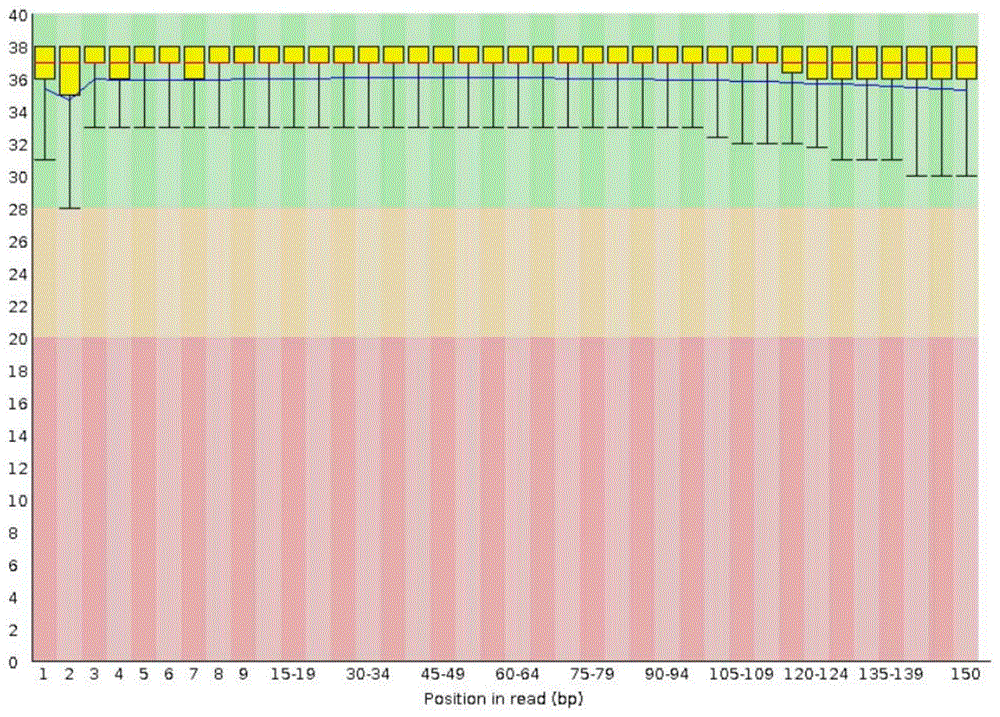
图1为样本测序数据碱基质量分布图;
图2为样本测序碱基含量分布图;
图3为121个SNP在染色体上的存在情况;
图4为四个不同地理群体长吻鮠的遗传距离关系;
图5为验证未知地理群体长吻鮠的遗传距离关系。
具体实施方式
下面结合附图对本发明的技术方案作进一步说明。
本发明的研究过程及原理在于:广泛收集不同地理群体长吻鮠,构建基于重测序后的长吻鮠DNA数据库,在该数据库中筛选和过滤长吻鮠特异性位点,基于特异性位点计算样本间亲缘关系并作系统进化树。具体步骤包括:1、广泛收集广东、湖北、四川和江苏群体长吻鮠;2、对收集的不同地理群体的长吻鮠分别进行DNA提取,并对提取的DNA进行质量分析;3、对质控合格后的片段进行重测序;4、对重测序数据进行质量分布检测、碱基分布检测、污染检测;5、采用软件将各个样本的reads与参考基因组进行比对,设置合理的筛选过滤参数,构建长吻鮠的特异位点(SNP位点)数据库;6、对SNP位点进行变异检测及统计。7、进行指纹图谱位点筛选;8、计算样本间亲缘关系并构建系统进化树;9、构建长吻鮠SNP分子身份证。
实施例1
长吻鮠的DNA指纹图谱的建立
1、广泛收集不同地理群体长吻鮠的尾鳍,其中广东群体16条,记为G1-G16;湖北群体20条,记为H1-H20;四川群体20条,记为S1-S20;江苏群体的长吻鮠20条,记为J1-J20。
2、对收集到的尾鳍组织使用DNA提取试剂盒进行提取及质量检测。
使用诺维赞公司的DNA提取试剂盒(FastPure Cell/Tissue DNA Isolation MiniKit,DC102-01)进行DNA提取,根据说明书测OD值及浓度,并跑电泳检测质量后使用。其中OD260/OD280应在1.8-2.0之间;样本量为2μl时,样本浓度需≥50ng/μl。DNA质量检测结果均合格。
3、对质控合格后的片段进行重测序。
使用MGISEQ T7测序仪进行测序,对检测合格的DNA样品随机打断,筛选符合要求的、适合大小的DNA片段。经纯化的DNA片段连接测序接头,滚环扩增制备DNA,然后在阵列化芯片上进行测序。
4、对重测序数据进行检测及分析质量分布检测、碱基分布检测及污染监测。保证数据质量,要在信息分析前对原始数据进行过滤和质量评估。
为了反映测序数据过程中测序质量的稳定性,以Clean Reads的碱基位置作为横坐标,每个位置的平均测序质量值作为纵坐标,得到每个样本测序的质量分布图。从该图上可以看出质量值均在30以上,说明测序质量良好。结果如图1所示。
碱基分布用来检测有无AT、GC分离现象,而这种现象可能是测序或者建库所带来的,并且会影响后续的定量分析。理论上G和C碱基及A和T碱基含量每个测序循环上应分别相等,且整个测序过程稳定不变,呈水平线。以Clean Reads中的碱基位置作为横坐标,以每个位置的ATCGN碱基的比例作为纵坐标,得到相应的分布图。结果如图2所示。
5、选择NCBI项目PRJNA692071长吻鮠全基因组作为参考基因组。将简化后的CleanReads比对到参考基组上,进行位置排序和标记重复reads,最终统计各项比对指标。比对率以及覆盖率指标能反映样本、建库及测序以及参考序列等的质量。
6、对SNP位点进行变异检测及统计。使用Sentieon对每个样本进行变异位点检测,获得每个样本的gVCF。在后续的joint-calling步骤中,再对所有样本的gVCF进行联合分析,得到群体中每个个体的变异结果。为保证SNP准确性,需要进行质控,对于合并后得到的SNP位点通过GATK推荐的过滤参数进行初步过滤:QD<2.0||FS>60.0||MQ<40.0||SOR>3.0||MQRankSum<-12.5||ReadPosRankSum<-8.0,获得5,023,513个SNP。
7、基于上述SNP位点,再进一步通过缺失率、MAF值、单拷贝、位点杂合率、位点深度进行过滤。首先根据位点缺失率为0和MAF值大于等于0.1进行过滤;再提取位点上下游100bp的序列进行拷贝数分析,保留位点上下游序列在基因组上是唯一的这种位点,获得570,949个SNP位点。其次通过过滤位点杂合率小于0.1和平均深度大于5的位点,保留600个位点。然后按照>4Mb的间隔进行筛选位点,保证位点间距。最后得到如表1的121个SNP位点,121个SNP在染色体上的存在情况如图3。
表1长吻鮠的DNA指纹图谱
/>
/>
8、计算所有样本间亲缘关系。
基于表1的121个SNP位点,计算样本间遗传距离来进行样本区分。对121个位点,使用Plink软件计算样本间遗传距离,根据遗传距离能够分开所有样本,遗传距离矩阵数值的大小表示遗传距离的远近,数值越小,表示样本间的亲缘关系越近,结果如图4所示。
9、构建不同地理群体长吻鮠DNA指纹图谱。
根据筛选出的SNP位点,构建不同地理群体长吻鮠DNA指纹图谱。不同地区的长吻鮠DNA指纹图谱如下表2-5:
表2广东群体长吻鮠的DNA指纹图谱
/>
/>
表3湖北群体长吻鮠的DNA指纹图谱
/>
表4四川群体长吻鮠的DNA指纹图谱
/>
/>
表5江苏群体长吻鮠的DNA指纹图谱
/>
实施例2
按实施例1中的方法,取江苏地区的长吻鮠样本(A1),进行DNA提取,取尾鳍使用诺维赞公司的DNA提取试剂盒(FastPure Cell/Tissue DNA Isolation Mini Kit)进行DNA提取,根据说明书测OD值及浓度,并跑电泳检测质量,符合要求后存放于-20℃冰箱备用。
使用MGISEQ T7测序仪进行测序,对检测合格的DNA样品随机打断,筛选符合要求的、适合大小的DNA片段。经纯化的DNA片段连接测序接头,滚环扩增制备DNA,然后在阵列化芯片上进行测序。
测序结果表明,该样品121个核心SNP位点有117个符合表5中江苏群体长吻鮠指纹图谱,符合率96.7%。并且对121个位点,使用Plink软件计算未知样本与本发明中已知的四个群体长吻鮠之间的遗传距离。聚类图结果表明该未知样本与江苏群体聚为一类(图5),因此判断该样品为江苏群体长吻鮠,符合取样结果。
本发明通过实施例1和2证明本发明的可行性和精准性,本发明实施例1广泛收集4个地理群体长吻鮠的尾鳍,并提取DNA,通过重测序技术获得121个高多态性SNP位点构建鉴别不同群体长吻鮠的DNA指纹图谱。实施例2用于证明本发明构建的DNA指纹图谱的可行性,随机挑选一个江苏群体样本,对该121个高多态性SNP位点的基因型进行聚类分析,发现该样本与江苏群体的DNA指纹图谱的符合率达到96.7%,可以鉴定为江苏群体。同时分别随机挑选广东、湖北、四川的群体,发现该样本与其对应群体的DNA指纹图谱的符合率≥95%,可以鉴定为广东、湖北或四川群体。即本发明中的DNA指纹图谱能够有效鉴别江苏、广东、湖北、四川不同群体长吻鮠,并且准确度高。
- 一种长吻鮠幼鱼活性菌饲料及其制备方法
- 一种长吻鮠稚鱼专用开口饲料及其制备方法
- 一种穿山龙高效液相色谱指纹图谱的建立方法及其标准指纹图谱和应用
- 一种灵芝孢子油的指纹图谱及其构建方法和应用
- 一种假俭草种质的指纹图谱的构建方法及应用
- 一套用于构建桃DNA指纹图谱的SSR引物、应用及构建方法
- 一套用于构建桃DNA指纹图谱的SSR引物、应用及构建方法
