一种基于多任务和多模态的癌症生存分析系统
文献发布时间:2023-06-19 10:41:48
技术领域
本发明涉及癌症生存分析和分级的技术领域,尤其是指一种基于多任务和多模态的癌症生存分析系统。
背景技术
随着下一代测序技术的发展,病人组学数据的获取越来越方便,例如基因组学、转录组学、蛋白组学、表观组学等多组学数据。不同组学是对病人不同分子层次的描述,多组学的融合能够更好地了解病人的疾病发展状态,从而提供更精准和高效的诊断和预后结果。影像数据一直以来被广泛用于分析癌症病人的病情。随着这几十年影像技术和计算机技术的发展,病理学图片通过各种工具、技术被保存在电子设备中,使得随时读取病人病理图成为可能,但这些病理图分辨率非常高,通过病理学家进行分析耗时耗力。而近年,欧洲放射和肿瘤治疗学会提出放射基因组学的概念,旨在通过计算机的方式将基因组学和医学影像联系起来,从而更好地为癌症病人进行诊断、治疗、预后。现在放射基因组学已经从癌症范畴扩大到精神疾病例如阿尔兹海默症和神经分裂等多种疾病领域,但针对多组学数据和病理图这样高度异构数据的融合依然面临着困难;
多任务学习是目前机器学习中一个热门的研究领域,目的是基于共享表示,通过利用不同任务之间的关联信息,提升不同任务各自的学习器的准确率。从多任务的定义可以将要求分为两点,一点是任务之间应该具备一定的关联信息,从而可以促进两个任务相互利用其中的信息,提高预测准确率。另一点是定义任务,明确要完成的任务是监督学习任务还是非监督学习任务。癌症病人的生存分析和分级符合多任务学习的关联要求的有监督学习任务,理论上能够分别提升这两个任务的预测效果;
综上所述,我们考虑将多任务与多模态融合应用到癌症病人的生存分析和分级中,目的是提高这两个任务的预测准确率。
发明内容
本发明的目的在于克服现有技术的缺点与不足,提出了一种基于多任务和多模态的癌症生存分析系统,突破传统单组学/单影像系统信息源单一的局限性,充分捕获多组学与影像等多模态间的异构与互补特性,挖掘多任务间的关联性,从而增强癌症的生存分析与分级能力。
为实现上述目的,本发明所提供的技术方案为:一种基于多任务和多模态的癌症生存分析系统,包括:
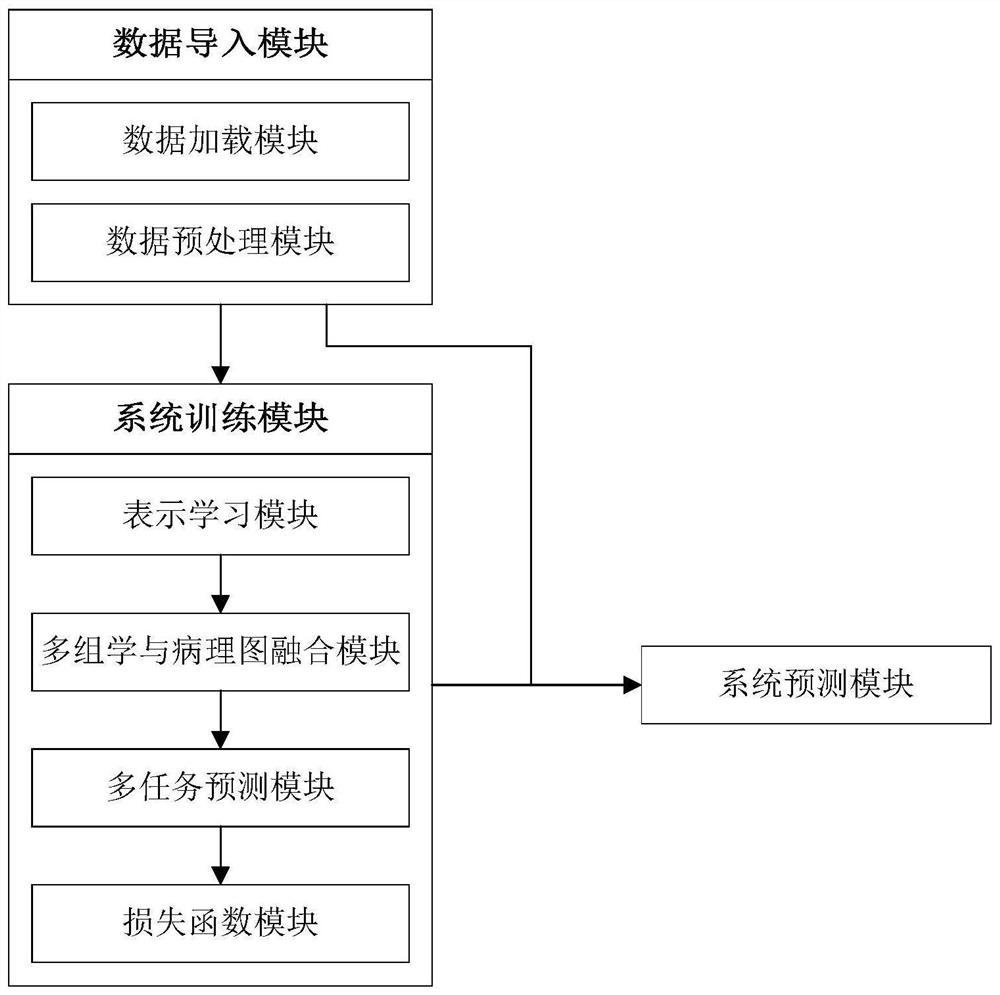
数据导入模块,用于加载多组学数据、病理图和临床数据,并对多组学数据、病理图和临床数据进行预处理,从而为后续系统训练模块和系统预测模块提供标准的输入;
系统训练模块,利用ResNet网络模型对病理图进行表示学习,得到病理图的表征,利用稀疏图卷积神经网络对多组学数据进行表示学习,得到多组学数据的表征,利用全连接神经网络对病理图和多组学数据的表征进行融合,利用全连接神经网络对融合后的表征进行多任务预测,通过反向传播进行模型参数学习,并最终得到最优预测模型,该模型用于同时进行癌症生存分析和癌症分级;
系统预测模块,基于系统训练模块训练出的最优预测模型,依据新的癌症病人的多组学数据和病理数据进行生存分析和癌症分级。
进一步,所述数据导入模块包括数据加载模块和数据预处理模块,其中:
所述数据加载模块从存储设备中读取多组学数据、病理图和临床数据;所述多组学数据包括基因组学数据、转录组学数据、蛋白质组学数据和表观基因组学数据,所述病理图是指经过伊红染色法染色的组织病理图,所述临床数据包括性别、年龄、生存时间、删失和癌症等级;
所述数据预处理模块用于对多组学数据与病理图进行预处理;针对训练样本中多组学数据中的每个组学数据,即需要输入到系统训练模块中的数据,去除空缺样本数超过阈值的特征,使用特征均值填补空缺值,去除均值低于阈值的特征和方差低于阈值的特征;针对待预测样本中多组学据中的每个组学数据,即需要输入到系统预测模块中的数据,则分别保留与训练样本中对应组学的特征相同的特征,并用特征均值填补空缺值;针对每个病人的病理图,先使用OpenSlide工具将其裁剪为z个r
进一步,所述系统训练模块包括表示学习模块、多组学与病理图融合模块、多任务预测模块和损失函数模块,其中:
所述表示学习模块用于对每个样本的多组学数据和病理图进行表示学习,以提取多组学数据和病理图中的关键特征,降低多组学数据和病理图的维度,从而便于后续的融合,包括以下步骤:
a、基于ResNet对r
其中,ResNet(·)为ResNet152网络模型,所采用的ResNet152网络模型已在ImageNet数据集上进行预训练,X
b、基于稀疏图卷积神经网络对多组学数据进行表示学习:
其中,σ(·)为激活函数ReLU(·)=max(0,·),max(0,·)表示取0和·中的较大值,
所述多组学与病理图融合模块用于对多组学和病理图表征进行融合,包括以下步骤:
a、多组学表征与病理图表征的拼接:
其中,F
b、对拼接后的特征利用四层的全连接神经网络进行的深层的融合表征:
其中,
所述多任务预测模块用于对多组学和病理图的融合表征进行多任务预测,包括以下步骤:
a、使用两层的前馈神经网络进行生存分析任务预测:
其中,
b、使用两层的全连接神经网络进行癌症分级任务预测,癌症分级的标签取决于细胞在显微镜下的外观,低等级表示癌症的生长慢,高等级表示癌症的生长快:
其中,
所述损失函数模块用于对表示学习模块、多组学与病理图融合模块和多任务预测模块中的参数进行训练,包括以下步骤:
a、设定多任务损失函数,生存分析的损失函数为Cox局部对数似然:
其中,l
分级任务的损失函数为负对数似然:
l
其中,l
b、通过最小化损失函数l和优化器Adam训练表示学习模块、多组学与病理图融合模块和多任务预测模块中的参数,经过d次迭代后获得最优预测模型。
进一步,所述模型预测模块依据多组学数据和病理图进行癌症病人的生存分析和分级,包含以下步骤:
a、使用数据导入模块中的数据加载模块加载多组学数据和病理图,再使用数据导入模块中的数据预处理模块处理多组学数据和病理图;
b、经过步骤a处理后的多组学数据和病理图输入由系统训练模块训练出的最优诊断模型中,对癌症病人的样本进行生存分析和分级;
c、每个病人由多个样本构成,针对生存分析任务,每个病人的风险值由其样本的均值计算而来,而针对分级任务,每个病人的分级结果由其样本结果投票所得,若出现同票情况则同时输出多种结果。
本发明与现有技术相比,具有如下优点与有益效果:
1、充分利用现有的病理图表征技术,即经过ImageNet预训练的ResNet152网络模型,提取病理图的深度特征,从而提升后续多模态融合和多任务学习的能力。
2、通过引入外部知识库,即基因-基因相互作用网络,增强系统对多组学数据的表征能力,从而提升后续多模态融合和多任务学习的能力。
3、通过深度学习方法充分挖掘多组学与病理图之间的互补特性,增强癌症生存分析和分级能力,同时这种将分子水平的多种基因数据与病理图相结合的方法,有利于进一步揭示癌症的致病机理。
4、通过多任务学习的方式捕获多任务间的共享关联性,同时增强各任务的性能。
附图说明
图1为本发明系统各个模块的关系示意图。
图2为本发明系统训练和预测的流程图。
图3为本发明系统所使用稀疏图卷积神经网络的结构示意图。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
本实施例公开了一种基于多任务和多模态的癌症生存分析系统,是使用Python语言开发的可在Windows,Linux和Mac设备上运行,系统各模块间关系如图1所示,系统训练和预测的流程如图2所示。它包括有:
数据导入模块,用于加载多组学数据,即mRNA表达量数据和DNA甲基化数据、病理图和临床数据,并对mRNA表达量数据和DNA甲基化数据、病理图和临床数据进行预处理,从而为后续系统训练模块和系统预测模块提供标准的输入;
系统训练模块,利用ResNet网络模型对病理图进行表示学习,得到病理图的表征,利用稀疏图卷积神经网络对分别对mRNA表达量数据和DNA甲基化数据进行表示学习,得到mRNA表达量数据和DNA甲基化数据的表征,利用全连接神经网络对病理图,mRNA表达量数据和DNA甲基化数据的表征进行融合,利用全连接神经网络对融合后的表征进行多任务预测,即生存分析任务和癌症分级任务,通过反向传播进行模型参数学习,并最终得到最优的模型,该模型可用于同时进行癌症生存分析和癌症分级;
系统预测模块,基于系统训练模块训练出的最优模型,依据新的癌症病人的mRNA表达量数据,DNA甲基化数据和病理数据进行生存分析和癌症分级。
所述数据导入模块包括数据加载模块、数据预处理模块,其中:
所述数据加载模块从存储设备中读取癌症病人(例如,胶质瘤)的CSV格式的mRNA表达量数据和DNA甲基化数据,读取病人的SVS格式的病理图数据,读取病人的CSV格式的临床数据,包括生存时间、删失和癌症等级;
所述数据预处理模块用于对mRNA表达量数据,DNA甲基化数据和病理图的数据进行预处理,针对训练样本的mRNA表达量数据和DNA甲基化数据,分别去除空缺样本数超20%的特征、使用特征均值填补空缺值、去均值最低的5%的特征和方差最低的5%的特征,针对待预测样本中的mRNA表达量数据和DNA甲基化数据,则分别保留与其对应组学的特征相同的特征,并用特征均值填补空缺值,针对每个病人的病理图数据先使用OpenSlide工具将其裁剪为z个1024×1024像素大小的病理图小块,每个小块为一个样本,最终每个病人由z个样本构成,z大于或等于1,每个样本中对应一个1024×1024像素大小的病理图和该病人经预处理后的mRNA表达量数据和DNA甲基化数据。
所述系统训练模块包括表示学习模块、多组学与病理图融合模块、多任务预测模块、损失函数模块,其中:
所述表示学习模块用于对每个样本的多组学数据和病理图进行表示学习,以提取多组学数据和病理图数据中的关键特征,降低多组学数据和病理图数据的维度,从而便于后续的融合,包括以下步骤:
a、基于ResNet对1024×1024像素大小病理图进行表示学习:
其中,ResNet(·)为ResNet152网络模型,本系统所采用的ResNet152网络模型已在ImageNet数据集上进行预训练,X
b、基于稀疏图卷积神经网络对mRNA表达量和DNA甲基化数据进行表示学习:
其中,
所述多组学与病理图融合模块用于对mRNA表达量,DNA甲基化数据和病理图表征进行融合,包括以下步骤:
a、mRNA表达量表征,DNA甲基化表征和病理图表征的拼接:
其中,F
b、对拼接后的特征利用四层的全连接神经网络进行的深层的融合表征:
其中,
所述多任务预测模块用于对mRNA表达量,DNA甲基化和病理图的融合表征进行生存分析任务和分级任务的预测,包括以下步骤:
a、使用两层的前馈神经网络进行生存分析任务预测:
其中,
b、使用两层的前馈神经网络进行癌症分级任务预测,癌症分级的标签一般取决于细胞在显微镜下的外观,通常较低的等级表示癌症的生长较慢,较高的等级表示癌症的生长较快:
其中,
所述损失函数模块用于对表示学习模块、多组学与病理图融合模块和多任务预测模块中的参数进行训练,包括以下步骤:
a、设定多任务损失函数,生存分析的损失函数为Cox局部对数似然:
其中,l
分级任务的损失函数为负对数似然:
l
其中,l
b、通过最小化损失函数l和优化器Adam训练表示学习模块、多组学与病理图融合模块和多任务预测模块中的参数,经过d次迭代后获得最优预测模型。
所述模型预测模块依据mRNA表达量,DNA甲基化数据和病理图进行癌症病人的生存分析和分级,包含以下步骤:
a、使用数据加载模块加载mRNA表达量、DNA甲基化数据和病理图,使用数据预处理模块处理mRNA表达量、DNA甲基化数据和病理图;
b、经过步骤a处理后的mRNA表达量、DNA甲基化数据和病理图输入由系统训练模块训练出的最优预测模型中对病人的样本进行的生存分析和分级;
c、每个病人由多个样本构成,针对生存分析任务,每个病人的风险值由其样本的均值计算而来,而针对分级任务,每个病人的分级结果由其样本结果投票所得,若出现同票情况则同时输出多种结果。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
- 一种基于多任务和多模态的癌症生存分析系统
- 一种基于Transformer和多任务学习的多模态情感分析方法及系统
