贵州黑山羊多羔主效基因应用及引物对和试剂盒
文献发布时间:2023-06-19 12:18:04
技术领域
本发明属于分子遗传育种领域,具体涉及贵州黑山羊多羔主效基因应用及引物对和试剂盒。
背景技术
贵州黑山羊是贵州特有地方品种,主产于贵州省毕节地区,具有被毛纯黑、肉质优良、体型较大、肉质细嫩、膻味小和抗逆性强等特点,但其繁殖力较低,平均产羔率为152%。科研单位为提高贵州黑山羊的繁殖性能而做了大量研究工作。
产羔性状是羊的重要经济性状,其遗传机制是增加产羔数和提高养羊生产效率的重要基础,也是近年来研究的焦点之一。目前,国内外研究者多通过PCR-SSCP和RFLP等技术对影响繁殖性状候选基因进行分析,通过对不同基因型与繁殖性状进行关联性分析,进而判断候选基因是否为主效基因。近年来,虽然用候选基因和转录组方法筛选了一些对山羊产羔性能有影响的候选基因,但未从根本上确定生产性能的主效基因。因此,利用高通量测序技术研究差异表达基因成为一种趋势,已成为研究繁殖遗传机制的一个重要的新途径。
近年来,随着基因组研究的发展,转录组学、蛋白质组学、代谢组学等组学技术相继出现,其中转录组学(transcriptome)是率先发展起来并应用最广泛的技术,主要是研究转录产物的分类,基因转录本的结构功能、剪接模式和其他转录后修饰,并计算所有转录本的表达情况,已应用到猪、绵羊、山羊等相关基因的筛选。
在绵羊上,利用转录组方法已成功筛选到了影响绵羊产仔数的主效基因。如Pokhareld等利用转录组鉴定卵巢中mRNA和miRNA对绵羊繁殖性状的研究,发现绵羊卵巢中差异表达基因和miRNAs对绵羊的繁殖性状有一定的影响,且发现了79个新的miRNAs。Mahdavi等发现FecB基因和BMPR-IB基因对Kalehkoohi羊的产羔数有一定影响。Chantepie等研究表明,BMP15变异的等位基因FecX能控制绵羊排卵率和繁殖能力,携带纯合子FecX的母羊繁殖能力强,降低了BMP15启动子活性。Dag I
发明内容
本发明的目的是提供贵州黑山羊多羔主效基因应用及引物对和试剂盒,贵州黑山羊产羔性的主效基因对研究贵州黑山羊多胎性状具有重要意义,明确贵州黑山羊产羔性的主效基因,在世代选育中,可以逐步淘汰具有单羔繁殖性能的基因个体,选留具有多羔繁殖性能的个体,从而组建贵州黑山羊多羔繁殖性能专门化新品系,这对贵州地方品种羊的种业提升及,新品系的培育具有重要意义,对贵州黑山羊多胎基因专门化品系的开发利用,乃至新品种培育等奠定良好的基础。
为了达到上述目的,本发明提供了一种贵州黑山羊多羔主效基因应用,所述贵州黑山羊多羔主效基因包含:上调基因AMH(抗苗勒氏激素,NCBI Reference Sequence:XM_018050766.1)、CYP19A1(芳香化酶,NCBI Reference Sequence:XM_013967046.2)、FOXL2(叉头状L2,NCBI Reference Sequence:XM_018049897.1)、FSHR(NCBI ReferenceSequence:NM_001285636.1)、GATA4(锌指转录因子4,NCBI Reference Sequence:XM_018051833.1)、INHBA(NCBI Reference Sequence:NM_001285581.1)、LHB(NCBI ReferenceSequence:XM_018062755.1)、LHX9(NCBI Reference Sequence:XM_018060812.1)、ZP3(透明带蛋白3,NCBI Reference Sequence:XM_018041042.1),和下调基因CNOT1(NCBIReference Sequence:XM_005692051.3)、DMRTA1(NCBI Reference Sequence:XM_013965867.2)、GHRHR(NCBI Reference Sequence:XM_005679238.3)、Hox-All(GenBank:JK747534.1)、Oviductin(输卵管蛋白,NCBI Reference Sequence:NM_001285584.1)、PCYT1B(NCBI Reference Sequence:XM_018044011.1)、SLC34A2(NCBI ReferenceSequence:XM_005681484.3)、WDR19(NCBI Reference Sequence:XM_018049377.1)、Wnt-7A(NCBI Reference Sequence:XM_018066672.1);所述贵州黑山羊产羔性的主效基因在选育具有单羔或多羔性的贵州黑山羊方面的应用,所述上调基因和下调基因分别在多羔性的贵州黑山羊中表达量上调和下调。
本发明的另一目的是提供一种选育单羔或多羔贵州黑山羊的方法,该方法包含鉴定贵州黑山羊繁殖性能的主效基因,所述主效基因为所述的贵州黑山羊多羔主效基因。
优选地,针对上调基因,鉴定AMH采用PCR扩增的引物对的核苷酸序列如SEQ IDNO.1和SEQ ID NO.2所示;鉴定CYP19A1采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.3和SEQ ID NO.4所示;鉴定FOXL2采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.5和SEQID NO.6所示;鉴定FSHR采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.7和SEQ ID NO.8所示;鉴定GATA4采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.9和SEQ ID NO.10所示;鉴定INHBA采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.11和SEQ ID NO.12所示;鉴定LHB采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.13和SEQ ID NO.14所示;鉴定LHX9采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.15和SEQ ID NO.16所示;鉴定ZP3采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.17和SEQ ID NO.18所示;针对下调基因,鉴定CNOT1采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.19和SEQ ID NO.20所示;鉴定DMRTA1采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.21和SEQ ID NO.22所示;鉴定GHRHR采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.21和SEQ ID NO.22所示;鉴定Hox-All采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.23和SEQ ID NO.24所示;鉴定Oviductin采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.25和SEQ ID NO.26所示;鉴定PCYT1B采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.27和SEQ ID NO.28所示;鉴定SLC34A2采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.29和SEQ ID NO.30所示;鉴定WDR19采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.31和SEQ ID NO.32所示;鉴定Wnt-7A采用PCR扩增的引物对的核苷酸序列如SEQ ID NO.33和SEQ ID NO.34所示。
本发明的另一目的是提供一种鉴定所述的主效基因的引物对,该引物对为所述的引物对。
本发明的另一目的是提供一种鉴定所述的主效基因的试剂盒,该试剂盒中的引物对为所述的引物对。
本发明贵州黑山羊多羔主效基因应用及引物对和试剂盒,具有以下优点:
本发明贵州黑山羊产羔性的主效基因,上调基因和下调基因都参与子宫、卵巢、输卵管中的细胞增殖、分化,蛋白结合,信号传导等作用,从测序结果中筛选了与产羔相关的基因进行了转录组数据验证,结果其表达趋势与转录组结果一致。而目前尚未有关于山羊产羔性状的主效基因或有效分子标记,本发明针对贵州黑山羊,筛选并分析出贵州黑山羊产羔性的主效基因,为研究贵州黑山羊产羔性状提供基础,在世代选育中,确定选育多羔繁殖性能的基因个体为育种目标,逐渐淘汰单羔性状繁殖母羊,选留多羔繁殖性能的贵州黑山羊基因个体,组建贵州黑山羊多羔繁殖性能专门化新品系,对提高贵州黑山羊种羊场的制种供种能力及经济效益具有重要指导意义。
附图说明
图1为差异基因GO功能富集结果。
图2为单、多羔组卵巢的差异基因KEGG富集分析结果。
图3为单、多羔组输卵管的差异基因KEGG富集分析结果。
图4为单、多羔组子宫的差异基因KEGG富集分析结果。
图5为单、多羔组子宫组织上调基因验证结果。
图6为单、多羔组子宫组织下调基因验证结果。
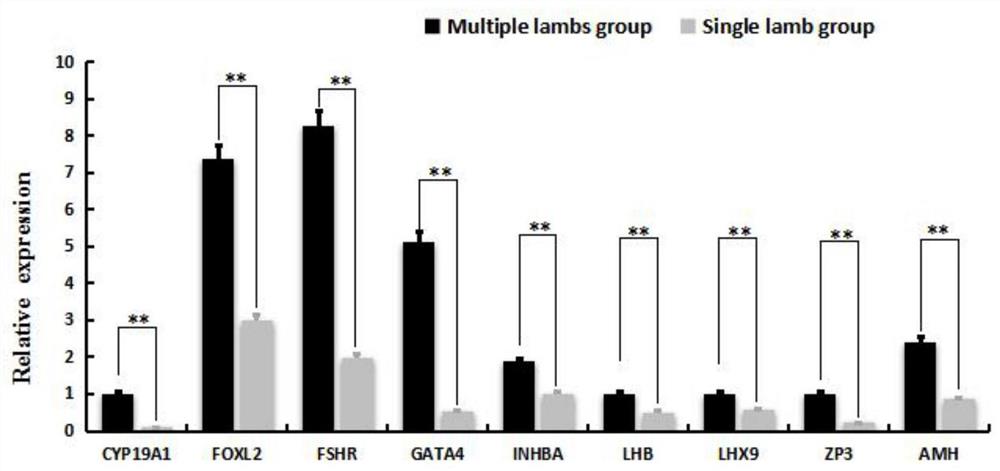
图7为单、多羔组卵巢上调基因验证结果。
图8为单、多羔组卵巢下调基因验证结果。
图9为单、多羔组输卵管上调基因验证结果。
图10为单、多羔组输卵管下调基因验证结果。
具体实施方式
下面将对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
以下实验例采用的材料具体如下:
1、具有产羔记录、体况健康的单、多羔贵州黑山羊(各3只)的子宫、卵巢、输卵管组织;
2、主要试剂:Trizol试剂(购自Invitrogen,Carlsbad,CA,USA);RNeasy RNA纯化试剂盒(购自Qiagen,Valencia,CA,USA);Agilent 2100Bioanalyzer(购自Agilent RNA6000Nano Kit);氯仿;无水乙醇;反转录试剂盒;2×Taq PCRMasterMix;DL2000 Marker。
实验例1总RNA的提取以及测序文库的建立
根据Trizol试剂和RNeasy RNA纯化试剂盒分别提取贵州黑山羊子宫、输卵管、卵巢组织总RNA,使用Agilent 2100Bioanalyzer检测总RNA的浓度、RIN值(RNA完整值)、28S/18S和片断大小。通过带有Oligo(dT)的磁珠富集有polyA结构的mRNA,采用离子打断的方式将RNA打断成短片段,以短片段RNA为模板,用6碱基随机引物和逆转录酶合成cDNA第一链,并加入缓冲液、dNTPs、RNase和DNA Polymerase合成第二链cDNA。将双链DNA末端补平并5'端磷酸化,3'端形成突出一个“A”的粘性末端,连接成一个3'端带有凸出“T”的鼓泡状的接头,连接的产物通过特异引物进行PCR扩增成单链,用一段桥式引物将单链DNA环化得到单链环状DNA文库。
实验例2Illumina测序
1、Illumina测序过程
利用Illumina HiSeq测序平台进行文库测序,由深圳华大基因股份有限公司测序,所得原始下机序列(Raw Reads)处理,通过去除接头污染的Reads(Reads中接头污染的碱基数大于5bp。对于双端测序,若一端受到接头污染,则去掉两端的Reads);去除低质量的Reads(质量值低于10的碱基占该reads总碱基数的比例大于20%的reads为低质量的reads);去除含N比例大于5%的Reads(对于双端测序,若一端含N比例大于5%,则会去掉两端Reads)。
2、测序结果
测序结果显示,多羔组和单羔组子宫、输卵管、卵巢18个样,平均每个样共检测到8.38Gb碱基数,与参考基因组比对率平均每个样品达到91.03%,样品间均匀的比对率表明样品之间的数据具有可比性,测序结果见表2。
表2测序结果初步分析
注:ML表示多羔;SL表示单羔;ovary为卵巢;oviduct为输卵管;uterus为子宫;TotalCleanBases表示原始数据过滤后剩余总碱基数;CleanReads Q20表示过滤后数据中质量值≥20的碱基占比;Clean Reads Q30表示过滤后数据中质量值≥30的碱基占比;CleanReadsRatio表示占总的比例;Total Clean Reads表示原始数据过滤后剩余总序列数。
对测序结果分析发现,在子宫、输卵管、卵巢组织中,多羔组平均每个样检测出总转录本为40415,未知的转录本为19185,其中总基因数为26722,未知基因为1190;单羔组平均每个样检测出总转录本为41117,未知的转录本为19366,其中总基因数为27093,未知基因为1201,结果参见表3。
表3转录组结果分析
实验例3基因组比对及表达量分析
使用HISAT软件将clean reads映射到山羊(Capra hircus)参考基因组(https://www.ncbi.nlm.nih.gov/assembly/GCF_001704415.1),得到转录组测序结果。然后使用RSEM计算基因和转录本的表达水平,得到FPKM值,即为基因的表达量。
基因表达量分析结果:对所有样品的基因表达量分析,发现各个样本表达量分布整体趋势一致,且未完全重叠,表明各样本之间存在一定的差异。
对于多个样品的表达量数据,利用维恩图展示了基因在组间的表达情况,结果表明在单、多羔组的卵巢组织共有表达基因为29522个,输卵管共有表达基因为28741个,子宫共有表达基因为28948个。
实验例4鉴定差异表达基因
将得到的FPKM值,用DEGseq进行差异分析,根据正态分布计算P值,通过Benjamini,Y.and Hochberg,Y.和Storey,J.and Tibshirani,R.两种策略将P值矫正为Q值。利用PossionDis方法计算FDR值,以FDR≤0.001,q≤0.001的基因为显著差异表达基因。
通过采用DEGseq方法对基因表达进行差异分析,筛选差异表达基因条件为表达差异倍数|logFoldChange|≥2和q≤0.001。如表4所示,结果显示,在卵巢组织中差异表达基因共8120个,多羔组对单羔组上调基因3205个,下调基因4915个;在输卵管组织中差异表达基因共5255个,多羔组对单羔组上调基因1317个,下调基因3888个;在子宫组织中差异表达基因共5180个,多羔组对单羔组上调基因2597个,下调基因2583个。
表4筛选的差异表达基因数目
注:SL:单羔;ML:多羔。
实验例5差异基因GO和KEGG功能分析
将所有基因映射到Gene Ontology数据库的各个Term,应用phyper函数进行GO和Pathway通路富集分析,计算每个Term的差异基因数目,以FDR≤0.01和q≤0.001为显著富集Pathway。
1、差异表达基因GO功能富集分析
为分析差异基因显著富集的GO功能条目,采用R软件中phyper函数计算,通过FDR校正,以|logFoldChange|≥2和q≤0.001的功能视为显著富集,从而展示样本中存在差异的基因功能。如图1所示(A:SL_ovary-vs-ML_ovary;B:SL_oviduct-vs-ML_oviduct;C:SL_uterus-vs-ML_uterus),为差异基因GO功能富集结果,从图1可知,卵巢、输卵管、子宫组织中分别有2858条、1855条和1836条注释到分子功能、细胞组成和生物过程。在生物过程中主要参与细胞分化、生殖过程、生物调节作用,在细胞组成中主要参与细胞器、细胞膜、细胞的结构组成,在分子功能中主要与各种蛋白结合、催化活性、细胞增殖相关。
2、差异表达基因KEGG富集分析
将单、多羔组卵巢、子宫、输卵管组织中所有转录本注释到KEGG数据库,统计各个Pathway不同层级上包含的差异表达基因数目,以|logFoldChange|≥2和q≤0.001为条件,进而确定差异表达基因主要参与的代谢途径和信号通路。如图2所示,为差异基因KEGG富集分析结果,从图2可知,卵巢的转录本注释到334个KEGG通路中,其中差异表达基因个数为4469;输卵管转录本注释到331个KEGG通路中,其中差异表达基因个数为2789;子宫转录本注释到332个KEGG通路中,其中差异表达基因个数为2893。这些差异基因主要参与信号传导、生殖系统、内分泌系统和癌症相关的通路,其中cGMP-PKG signaling pathway、cAMPsignaling pathway、TGF-beta signalingpathway、mTOR signalingpathway在影响贵州黑山羊单、多羔中可能发挥重要作用。
实验例6差异基因qRT-PCR验证
1、差异基因qRT-PCR实验
从测序结果中选取表达上调和下调的基因各9个,利用实时荧光定量PCR(qRT-PCR)进行验证。设计荧光定量PCR引物(参见表5),以组织的cDNA为模板,以羊的GAPDH基因为内参,每个样品进行3次重复检测,使用2
表5差异基因荧光引物信息
qRT-PCR体系(20μL):2×UltraSYBR Mixture 10μL;Forward Primer 0.5μL;Reverse Primer 0.5μL;cDNA 0.5μL;ddH2O 8.5μL。qRT-PCR程序:95℃预变性3min;95℃变性30s,退火(见表1)30s,72℃延伸30s,40个循环;95℃变性5s;最后从60℃到95℃按0.5℃增值进行溶解曲线分析,荧光采集时间为5s。
运用2
2、差异表达基因qRT-PCR结果
从子宫、卵巢、输卵管转录组中筛选上调基因AMH、CYP19A1、FOXL2、FSHR、GATA4、INHBA、LHB、LHX9、ZP3,下调基因CNOT1、DMRTA1、GHRHR、Hox-All、Oviductin、PCYT1B、SLC34A2、WDR19、Wnt-7A进行组织qRT-PCR验证,结果表明,qRT-PCR结果与转录组测序(RNA-seq)结果一致(见图5~10)。上调基因对卵泡的生长发育、卵巢颗粒细胞增殖、性腺发育具有重要影响,而这些功能都与多羔性状息息相关。下调基因具有破坏、抑制胚胎活率、细胞活力等作用,其高表达会影响山羊的产羔率。为后续研究差异基因在细胞水平上的分子调控机制奠定基础。
尽管本发明的内容已经通过上述优选实施例作了详细介绍,但应当认识到上述的描述不应被认为是对本发明的限制。在本领域技术人员阅读了上述内容后,对于本发明的多种修改和替代都将是显而易见的。因此,本发明的保护范围应由所附的权利要求来限定。
序列表
<110> 贵州省种畜禽种质测定中心
<120> 贵州黑山羊产羔性的主效基因的应用及引物对和试剂盒
<160> 38
<170> SIPOSequenceListing 1.0
<210> 1
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 1
ctctgcgtga gctgagcgt 19
<210> 2
<211> 18
<212> DNA
<213> Artificial Sequence
<400> 2
ttgccgtagc gtgggttg 18
<210> 3
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 3
tggacaggtt ggaggaggt 19
<210> 4
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 4
ccaggaagag gatgttagag g 21
<210> 5
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 5
cgcagaagcc cccatactc 19
<210> 6
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 6
cggcaccttg atgaagcact 20
<210> 7
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 7
atcatgttgg tgggctgggt ct 22
<210> 8
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 8
tgcgcttggc tatcttggtg tc 22
<210> 9
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 9
ccagagcgaa gaaggcaaac aag 23
<210> 10
<211> 24
<212> DNA
<213> Artificial Sequence
<400> 10
gggactgacc acaggggaag agaa 24
<210> 11
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 11
gggtacgtgg agatagagga c 21
<210> 12
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 12
ttgaaagaga cggatggtga 20
<210> 13
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 13
ttcaccacca gcatctgcg 19
<210> 14
<211> 18
<212> DNA
<213> Artificial Sequence
<400> 14
gagaccattg ggtccacg 18
<210> 15
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 15
agactccgtc taccacctga 20
<210> 16
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 16
tgcaagaggg tctcgaagtg 20
<210> 17
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 17
tgtggttagg tttgtggtcg g 21
<210> 18
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 18
tgggtgtcat cttctcggcg 20
<210> 19
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 19
taaggcttgc agaggtaggg 20
<210> 20
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 20
taagttggcg gattgaggga 20
<210> 21
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 21
tcaggagggg aagagagtc 19
<210> 22
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 22
tgtgaagggg agagaaagc 19
<210> 23
<211> 25
<212> DNA
<213> Artificial Sequence
<400> 23
tgaccgcctc tcatttcgct accat 25
<210> 24
<211> 25
<212> DNA
<213> Artificial Sequence
<400> 24
ccagccacca gaagaccctc cttgt 25
<210> 25
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 25
agcgtggtcc ctgctcctct aac 23
<210> 26
<211> 23
<212> DNA
<213> Artificial Sequence
<400> 26
gggctcaatg gcgtattctc tga 23
<210> 27
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 27
gggaagtaca ccaagcaagc 20
<210> 28
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 28
tgtaaccgaa gctgatggca 20
<210> 29
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 29
acttgctggt aggagtttg 19
<210> 30
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 30
ttctgttctc tgtgttgga 19
<210> 31
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 31
tgatgagtcg gtccaaaaca 20
<210> 32
<211> 19
<212> DNA
<213> Artificial Sequence
<400> 32
ggatgacgcc aacaatagc 19
<210> 33
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 33
attgcatacc tcacctccct 20
<210> 34
<211> 20
<212> DNA
<213> Artificial Sequence
<400> 34
gattattcat tcccacagcc 20
<210> 35
<211> 22
<212> DNA
<213> Artificial Sequence
<400> 35
gcctggacga gtgtcagttt ca 22
<210> 36
<211> 21
<212> DNA
<213> Artificial Sequence
<400> 36
atagcggatg tcggcagagc a 21
<210> 37
<211> 18
<212> DNA
<213> Artificial Sequence
<400> 37
gcaagttcca cggcacag 18
<210> 38
<211> 18
<212> DNA
<213> Artificial Sequence
<400> 38
tcagcaccag catcaccc 18
- 贵州黑山羊多羔主效基因应用及引物对和试剂盒
- 一组用于检测小麦赤霉病主效抗病基因基因型的引物组合、试剂或试剂盒及应用和方法
