酪氨酸脱羧酶CtTyDC、编码基因、试剂盒及制备酪胺的方法
文献发布时间:2023-06-19 18:32:25
技术领域
本发明涉及一种酪氨酸脱羧酶CtTyDC、编码基因、试剂盒及制备酪胺的方法。
背景技术
酪氨酸脱羧酶(TyDC)是一种氨基酸脱羧酶(amino acid decarboxylases,AADCs),它可以催化酪氨酸脱羧生成酪胺,是连接初生代谢和各种次生代谢产物之间的桥梁,在控制最终产物生物合成方面具有关键的调控作用。
5′-磷酸吡哆醛(pyridoxal 5'-phosphate,PLP)在许多酶促过程中充当辅酶,包括脱羧、脱氨、转氨作用、外消旋化等。需要PLP的酶通常被称为PLP依赖性酶,它们广泛参与到许多的关键细胞代谢途径中。目前已报道的来自植物(如罂粟、水稻、唐松草、拟南芥、红景天、黄花石蒜等)的酪氨酸脱羧酶TyDC大多属于PLP依赖型酶家族,它们催化酪氨酸脱羧生成酪胺时都需要依赖辅因子PLP,加大了原料的投入,增加了生产成本。PLP的催化机制为:PLP辅因子通过形成席夫碱键与蛋白质结合,PLP分子中的醛基与蛋白质中高度保守的活性位点赖氨酸残基的ε-氨基结合生成内部醛亚胺。底物进入酶的活性位点后,底物的未质子化的氨基基团会攻击内部醛亚胺的席夫碱键,从而导致内部醛亚胺的分解并在底物和PLP之间形成新的席夫碱键,被称为外部醛亚胺。其失去α-羧基,形成醌型中间体。醌型中间体通过与蛋白中的酸性酪氨酸残基相互作用,使得碳环质子化,然后导致亚胺络合物的再生以及芳香胺和CO
可见,现有已知酪氨酸脱羧酶TyDC大多属于PLP依赖型酶,催化酪氨酸脱羧生成酪胺时均需要依赖辅因子PLP,加大了原料的投入,增加了生产成本,降低了生产效率。
发明内容
有鉴于此,本发明的一个目的在于提供一种酪氨酸脱羧酶CtTyDC。该酪氨酸脱羧酶CtTyDC催化酪氨酸脱羧生产酪胺时不需要PLP的参与,大大简化了反应步骤,节约了反应成本,在工业生产和实际应用中具有良好的应用前景。
本发明的另一个目的在于提供一种编码上述酪氨酸脱羧酶CtTyDC的基因。
本发明的再一个目的在于提供一种应用上述酪氨酸脱羧酶CtTyDC制备得到的试剂盒。
本发明的又一个目的在于提供一种制备酪胺的方法。该方法利用酪氨酸脱羧酶CtTyDC体外催化酪氨酸脱羧生成酪胺,反应不需要添加辅因子PLP。
本发明采用如下技术方案实现上述目的。
本发明提供一种酪氨酸脱羧酶CtTyDC,所述酪氨酸脱羧酶CtTyDC的氨基酸序列如SEQ ID NO:1所示,或者该序列经替换、缺失或添加一个或几个氨基酸形成的具有同等功能的氨基酸序列。
根据本发明的酪氨酸脱羧酶CtTyDC,优选地,所述酪氨酸脱羧酶CtTyDC来源于管花肉苁蓉。
本发明还提供一种编码上述酪氨酸脱羧酶CtTyDC的基因,其核苷酸序列如SEQ IDNO:2所示。
本发明再提供一种编码上述酪氨酸脱羧酶CtTyDC的基因,其核苷酸序列的全长cDNA序列如SEQ ID NO:3所示。
本发明又提供一种应用如上述酪氨酸脱羧酶CtTyDC制备得到的试剂盒。
本发明另提供一种制备酪胺的方法,包括使包含上述的酪氨酸脱羧酶CtTyDC和酪氨酸底物的反应体系在适宜条件下进行反应,从而获得酪胺。
根据本发明的方法,优选地,所述酪氨酸底物包括L-酪氨酸和L-DOPA中的至少一种。
根据本发明的方法,优选地,所述反应体系进一步包含缓冲液。
根据本发明的方法,优选地,所述反应体系以150μL计,包含1~8mM酪氨酸底物、10~70μg酪氨酸脱羧酶CtTyDC和80~120mM磷酸钠缓冲盐溶液,磷酸钠缓冲盐溶液补足至150μL。
根据本发明的方法,优选地,所述适宜条件包括20~40℃的反应温度和10~50小时的反应时间。
本发明从管花肉苁蓉中克隆得到了酪氨酸脱羧酶CtTyDC,通过酶促催化反应发现,该酶能够催化酪氨酸脱羧生成酪胺,且反应的发生不依赖辅因子PLP。通过紫外及蛋白质谱测定,进一步排除了内源PLP参与的可能性,从而进一步证明了该酶催化酪氨酸脱羧反应的非PLP依赖性。通过结构指导下的定点突变,提供了酪氨酸脱羧酶CtTyDC不依赖PLP特性的关键氨基酸位点。利用本发明酪氨酸脱羧酶CtTyDC催化反应制备酪胺,能够减少原料的投入,有效的节约了成本,简化反应步骤,提高效率。
附图说明
图1为本发明的酪氨酸脱羧酶CtTyDC基因编码区琼脂糖凝胶电泳图。
图2为本发明异源表达得到的酪氨酸脱羧酶CtTyDC的SDS-PAGE电泳图。
图3为本发明的酪氨酸脱羧酶CtTyDC催化L-酪氨酸反应的HPLC-HR-ESI-MS图。
图4为本发明的酪氨酸脱羧酶CtTyDC催化L-酪氨酸、D-酪氨酸反应的高效液相色谱图。
图5(A)、图5(B)、图5(C)分别为本发明的酪氨酸脱羧酶CtTyDC催化L-酪氨酸时,不同的反应时间、温度、pH所对应的酪氨酸脱羧酶CtTyDC的相对活性图。
图6为本发明的酪氨酸脱羧酶CtTyDC经同源模建构建得到的蛋白三维结构。
图7为本发明的酪氨酸脱羧酶CtTyDC突变体蛋白的活性图。
具体实施方式
下面结合具体实施例对本发明作进一步的说明,但本发明的保护范围并不限于此。
本发明首次从富含苯乙醇苷类化合物的中药管花肉苁蓉中克隆获得一种具有催化酪氨酸脱羧功能的酶,经进一步筛选、鉴定以及优化得到活性高且稳定的酪氨酸脱羧酶CtTyDC。基于此,完成了本发明。
本发明制备酪胺的方法,包括使包含酪氨酸脱羧酶CtTyDC和酪氨酸底物的反应体系在适宜条件下进行反应,从而获得酪胺。其中,“酪氨酸脱羧酶CtTyDC和酪氨酸底物的反应体系”有时简称为“反应体系”。
<酪氨酸脱羧酶CtTyDC>
一种酪氨酸脱羧酶CtTyDC,所述酪氨酸脱羧酶CtTyDC的氨基酸序列如SEQ ID NO:1所示,或者该序列经替换、缺失或添加一个或几个氨基酸形成的具有同等功能的氨基酸序列。
本发明首次从植物管花肉苁蓉(学名:Cistanche tubulosa(Schenk)Wight)中筛选鉴定得到一种酪氨酸脱羧酶,命名为CtTyDC。本发明的酪氨酸脱羧酶CtTyDC可为天然蛋白酶或包含突变且仍具有催化酪氨酸脱羧活性的酶。优选地,本发明的酪氨酸脱羧酶CtTyDC包含SEQ ID NO:1所示的氨基酸序列,或者该序列经替换、缺失或添加一个或几个氨基酸形成的具有同等功能的氨基酸序列。在某些实施方案中,酪氨酸脱羧酶CtTyDC的氨基酸序列与SEQ ID NO:1所示序列具有90%以上,优选92%以上,更优选95%以上,进一步优选98%以上,还优选99%以上的同源性且来源于相同的物种管花肉苁蓉。本申请中“同源性”是指两个序列之间的相似性,可通过本领域已知的任何算法来确定。例如,两个氨基酸序列之间的同一性程度可使用Needleman-Wunsch算法确定。优选地,本发明的酪氨酸脱羧酶CtTyDC的分子量为56.33KDa;理论等电点为6.68,表明其为酸性蛋白;不稳定系数(instability index)为48.27,表明该蛋白可能不稳定;疏水性平均值(Grand average ofhydropathicity,GRAVY)为-0.041,表明其为亲水性蛋白,蛋白由509个氨基酸组成。
<编码基因>
一种编码上述酪氨酸脱羧酶CtTyDC的基因,其核苷酸序列如SEQ ID NO:2所示,该核苷酸序列长度为1,530bp。该核苷酸序列的全长cDNA序列如SEQ ID NO:3所示,该全长cDNA序列长度为1,882bp,5′端非翻译区长度为209bp,3′端非翻译区长度为143bp。
本发明通过蛋白同源模建、分子对接以及结构指导下的定点突变,对CtTyDC蛋白催化活性的关键位点进行了研究,发现F100、P101、S102、T166、T261及L324这几个位点突变后,酶活性明显降低,但在反应体系中添加辅酶PLP后,活性又会恢复,进而推测这几个关键位点导致了酪氨酸脱羧酶CtTyDC具有催化反应不依赖外源辅因子PLP的特性。
<试剂盒>
一种应用上述酪氨酸脱羧酶CtTyDC制备得到的试剂盒。现有研究表明,酪氨酸脱羧酶与多种次生代谢产物的合成息息相关,而这些代谢产物对人类某些疾病具有治疗作用,如阿尔茨海默病、肿瘤、炎症等。因此,将酪氨酸脱羧酶CtTyDC制成试剂盒的产品形式,能够更好的应用于临床,具有更好的实际应用价值。
<制备酪胺的方法>
一种制备酪胺的方法,包括使包含酪氨酸脱羧酶CtTyDC和酪氨酸底物的反应体系在适宜条件下进行反应,从而获得酪胺。
本发明中,酪氨酸底物包括L-酪氨酸和L-DOPA中的至少一种。本发明通过对酪氨酸脱羧酶CtTyDC体外催化能力的研究发现,酪氨酸脱羧酶CtTyDC表现出了显著的底物选择特异性及立体催化特异性,对L-酪氨酸和L-DOPA表现出明显的催化特性,而以L-苯丙氨酸、L-色氨酸、L-组氨酸为底物时,则不能催化脱羧反应的发生。表明酪氨酸脱羧酶CtTyDC对底物的构象具有严格的选择性,只能接受L-酪氨酸、L-DOPA为底物。
在某些实施方案中,本发明反应体系还可以包含缓冲液。优选地,缓冲液的pH值在6.5~8.5范围内,优选为8.0。优选地,缓冲液可为磷酸盐缓冲液,例如,磷酸钾缓冲液或磷酸钠缓冲液等。缓冲液中盐类物质的浓度不特别限定,只要能够提供本发明所述的pH值范围即可。优选地,盐类物质的浓度为50~150mM,优选为100mM。
在某些实施方案中,反应体系以150μL计,包含1~8mM酪氨酸底物、10~70μg酪氨酸脱羧酶CtTyDC和50~150mM磷酸钠缓冲盐溶液,磷酸钠缓冲盐溶液补足至150μL,及按等比例放大的反应体系。
根据本发明一个优选的实施方式,反应体系以150μL计,包含4mM L-酪氨酸底物、50μg酪氨酸脱羧酶CtTyDC和100mM磷酸钠缓冲盐溶液,磷酸钠缓冲盐溶液补足至150μL。
本发明所述的“适宜条件”是指能够保证酪氨酸脱羧酶CtTyDC和酪氨酸底物进行反应的条件。所述条件包括反应温度为20~40℃,优选为25~35℃,更优选为30℃。所述条件还包括反应时间为10~50小时,优选为11~48小时,更优选为12小时。
优选地,本发明的反应体系中可进一步包含反应所需的其他物质。
选取管花肉苁蓉植株新鲜肉质茎,采用液氮速冻研磨法,根据OMEGA RNA Plantkit试剂盒的具体操作提取总RNA。琼脂糖凝胶电泳检测质量,同时利用Nanodrop 2000分光光度计测定RNA浓度。选择无基因组DNA残留且A260/280的值在2.0的RNA按照SMARTer RACE5′/3′Kit(ClonTech)试剂盒的具体操作分别进行反转录获得5′-RACE-cDNA,3′-RACE-cDNA及总cDNA。
5′-RACE-cDNA的获得:取0.2~2.0μg RNA,与1.0μL 5′-CDS-primer A,ddH
3′-RACE-cDNA的获得:取0.2~2.0μg RNA,与1.0μL 3′-CDS-primer A,ddH
RT-cDNA的获得:取0.2~2.0μg RNA,与10μM oligo dT
2.RACE法快速扩增cDNA末端
目的基因5′端和3′端序列的克隆参照SMARTer
RACE扩增的反应体系为:5′/3′-RACE-cDNA:40ng,UPM(10μM):1.0μL,5′/3′-RACE特异引物(10μM):0.5μL,10×KOD buffer:1.0μL,MgSO
PCR反应程序为:94℃预变性2min;之后94℃变性15s,起始65℃退火,30s,每个循环降低0.5℃,68℃延伸,50s,共30个循环;紧接着94℃变性15s,54℃退火,30s,68℃延伸,50s,共25个循环;最后72℃延伸1min。
将测序所得的5′RACE和3′RACE序列进行拼接,得到全长cDNA序列如SEQ ID NO:3所示。分析5′-RACE和3′-RACE所得到的cDNA片段序列,设计一对带有酶切位点的特异性引物,序列如SEQ ID NO:6、7所示,其中SEQ ID NO:6通过引物设计引入EcoR I酶切位点,SEQID NO:7通过引物设计引入Xho I酶切位点。利用一对特异性引物扩增得到约1530bp的编码区序列全长,序列如SEQ ID NO:2所示,编码氨基酸序列如SEQ ID NO:1所示,琼脂糖凝胶电泳如图1所示。图1中,M指的是DNA Marker,左边数字(比如3000bp)表示的是Marker中DNA的大小。
基于获得的CtTyDC全长序列,利用ORF Finder分析基因的开放阅读框;利用NCBI中的blastx对CtTyDC编码的氨基酸序列进行同源性比较分析;利用ExPASy在线服务器(http://web.expasy.org/protparam/)预测基因编码蛋白的理化性质。将CtTyDC编码的氨基酸序列进行BLAST序列分析,结果显示两条序列与已报道的芳香族氨基酸脱羧酶相似度可达89%。利用Expasy对Ct TyDC所编码蛋白的理化性质进行预测,显示其分子量为56.33kDa;理论等电点为6.68,表明其为酸性蛋白;疏水性平均值(Grand average ofhydropathicity,GRAVY)为-0.041,表明此蛋白为亲水性蛋白。
为了验证获得的CtTyDC序列是否具有酪氨酸脱羧酶的功能。基于pET28a表达载体酶切位点信息及获得的CtTyDC全长序列,设计合适的带酶切位点的正反向引物,以普通法反转录获得的RT-cDNA为模板,用KOD-Plus-Neo DNA Polymerase进行PCR扩增。选取片段大小与预测值一致的PCR产物进行胶回收所得回收产物与
用设计好的酶切位点双酶切所提取的CtTyDC质粒及pET28a载体。37℃酶切5h,凝胶电泳回收产物。利用NEB T4 DNA ligase 0.5μL将目的基因(53ng/1.4kb)连接于pET-28a载体(66ng/5.3kb),加10×T4buffer 1.0μL,ddH
HR-MS检测
CtTyDC蛋白体外酶促反应体系如下:100mM磷酸钠缓冲液(pH8.0),4mM L-酪氨酸,50μg CtTyDC蛋白,反应体系150μL。28℃水浴反应12h。加入2倍体积的甲醇终止反应,涡旋混匀,15000×g,4℃离心1h,上清液进行液相检测。用Agilent 1260液相进行检测,
色谱柱:XBridge BEH Amide Column(4.6mm×150mm,3.5μm),流速1.0mL/min,柱温30℃,检测波长为280nm,流动相为乙腈(A)和0.1%甲酸水(B),洗脱程序如下:A:B(v/v):0min(100:0)-10min(85:15)-22min(55:45)。
利用岛津LCMS-IT-TOF离子阱飞行时间质谱仪进行高分辨质谱分析。其中,色谱柱、液相条件等同上。
质谱条件参数分别为:正、负离子模式,自动多级MS
通过高效液相紫外特征吸收谱,结合图3所示的高分辨质谱分子量分析和分子式预测确定酪胺的生成。酪氨酸脱羧酶CtTyDC催化L-酪氨酸的反应式如下所示。
为了探究获得的酪氨酸脱羧酶CtTyDC催化底物的选择性,选择了酪氨酸结构类似物如L-DOPA、L-苯丙氨酸、L-色氨酸、L-组氨酸为底物进行了体外酶促反应,结果发现酪氨酸脱羧酶CtTyDC对L-DOPA也表现出明显的催化特性,而不能催化其他类型底物发生脱羧反应。此外,考察了酪氨酸脱羧酶CtTyDC催化的立体选择性,结果发现当以D-酪氨酸为底物时,酪氨酸脱羧酶CtTyDC不能催化脱羧反应的发生,而以L-酪氨酸为底物时,酪氨酸脱羧酶CtTyDC却能催化脱羧,如图4所示,表明酪氨酸脱羧酶CtTyDC对底物的构象具有严格的选择性,这一结果也与自然界中生物利用的天然氨基酸均为左旋相一致。
在鉴定了酪氨酸脱羧酶CtTyDC功能的基础上,进一步摸索了酪氨酸脱羧酶CtTyDC催化体外酶促反应的最适条件。以L-酪氨酸为底物,探究了CtTyDC催化反应的转化率随时间的变化趋势,以及温度和pH值对酶催化活性的影响。
考察转化率随时间的变化趋势,具体操作如下:于磷酸钠缓冲体系中(pH8.0),先加入底物L-酪氨酸,再加入CtTyDC蛋白并开始计时。分别于反应的第5min、15min、30min、1h、2h、4h、6h、8h、12h、24h后取样,加入2倍体积的甲醇终止反应。高速离心取上清进行HPLC分析。结果发现,0~4h,转化率呈线性增长;4~12h,转化率增长趋势放缓;12h后,转化率趋于平稳,并在反应48h后接近100%。
考察温度对酪氨酸脱羧酶CtTyDC酶活力的影响,具体操作如下:在冰上加入反应体系,之后分别置于0℃、4℃、16℃、25℃、30℃、37℃、42℃、50℃、65℃进行反应,反应3h后加入2倍体积的甲醇终止反应。高速离心取上清进行HPLC分析。温度对酶活性的影响表现为:0~30℃之间,酶活性随温度升高而增强,在30℃时酶活性最强,之后随温度升高酶活性下降。因此确定30℃为该反应的最适温度。
考察pH值对酪氨酸脱羧酶CtTyDC酶活力的影响,具体操作如下:将L-酪氨酸和CtTyDC蛋白分别加入以下不同pH值的缓冲液中,包括:0.1M醋酸-醋酸钠缓冲液(pH4.0~5.0),0.2M磷酸盐缓冲液(pH6.0~8.0),0.2MTris-HCl缓冲液(pH8.0~9.0),0.1M碳酸钠-碳酸氢钠缓冲液(pH9.0~11.0)。于30℃反应3h后加入2倍体积的甲醇终止反应。高速离心取上清进行HPLC分析。pH对酶活性的影响表现为:当pH值小于6.5时,酶活性丧失,pH值在6.5~8.0之间,酶活性随pH值升高而增强,在pH值为8.0时酶活性最强,之后随着pH值升高酶活性降低。因此,确定pH=8为该反应的最适pH值,结果如图5(A)、图5(B)和图5(C)所示。
由于PLP作为生物体中最基本的一类辅因子,存在于多个细胞器中,且蛋白序列中的赖氨酸易与PLP结合,从而引入内源性的PLP。因此,为了排除CtTyDC蛋白催化酪氨酸脱羧反应不需要PLP的添加是由于混入了内源性的PLP所致,实验对CtTyDC蛋白进行了紫外及质谱分析。
利用NanoDrop 2000对CtTyDC蛋白进行紫外检测,结果发现CtTyDC蛋白只在280nm处有吸收峰,而没有PLP的特征吸收峰,提示CtTyDC蛋白没有内源结合PLP。
为了进一步对结果进行确证,实验在CtTyDC蛋白(7.60mM)中加入了一定量的NaBH
为揭示CtTyDC催化酪氨酸脱羧反应不依赖PLP的分子机制,实验对CtTyDC蛋白进行了同源模建和分子对接研究。选择源自P.somniferum的酪氨酸脱羧酶(PDB ID:6EEM)为模板,使用Modeller 9.14软件构建CtTyDC蛋白三维结构,模建的结构采用PROCHECK程序的Ramachandran plot以及PROFILE 3D进行打分评价,以概率密度函数(probabilitydensity function,PDF)最低、DOPE统计势最低及GA341打分最接近1.0者作为模建结果,并应用于后续分子对接中。最优结构如图6所示,其PDF总能量为2542.99561molpdf,DOPE分数为-59838.97656,GA341得分为1.0000。酪氨酸脱羧酶催化脱羧反应需要底物酪氨酸和辅酶PLP的参与,故需要分别分析两个配体的结合位点。通过NCBI BLAST比对分析,选择同源性较高且同时含有两个配体的蛋白晶体复合物Dopa decarboxylase(PDB ID:1JS3)作为比对模板,分子对接采用AutoDock vina程序进行,网格大小设为40×40×40,格点间隔为0.375nm,利用AutoDock Tools 1.5.6完成PLP、酪氨酸配体及目的蛋白受体的对接前准备。PLP配体结合口袋三维中心坐标为x=45.86,y=35.322,z=66.377;酪氨酸配体结合口袋三维中心坐标为x=40.974,y=36.952,z=67.83。优化采用拉马克遗传算法(Lamarckiangenetic algorithm,LGA),设定对接参数等完成对接。选择结合自由(K
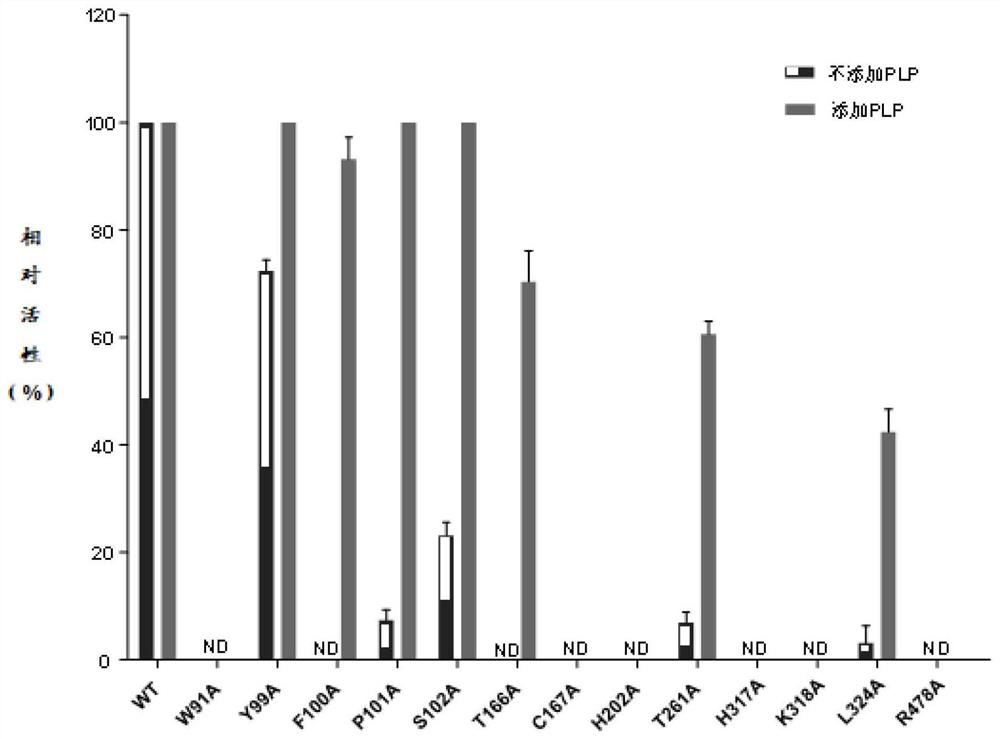
根据同源模建及分子对接结果,本申请选择了CtTyDC氨基酸序列中14个关键位点进行突变,包括位点W91、Y99、F100、P101、S102、T165、T166、C167、H202、T261、H317、K318、L324、R478,对上述位点进行了丙氨酸扫描突变。
以pET28a-CtTyDC质粒为模板,以KOD-Plus-Neo高保真酶以及突变引物进行PCR扩增。PCR产物加1μLDMT酶于37℃孵育1h进行去甲基化,之后依据BIOMIG Gel/PCRExtraction Kit说明书进行PCR纯化,转化至大肠杆菌DMT感受态细胞,涂布于含Kana抗性的LB固体培养平板上,37℃培养过夜。菌落PCR筛选阳性克隆,用T7F、T7R引物测序。突变成功的质粒重新导入Transetta(DE3)菌株中进行诱导表达,并将各个表达产物进行纯化。结果发现T165A突变体蛋白没有表达,其余突变体蛋白正常表达。
为进一步研究这些氨基酸位点突变对CtTyDC催化活性的影响,我们以L-酪氨酸为底物进行了体外酶促反应。酶促反应体系如下:4mM L-酪氨酸,50μg纯化的CtTyDC突变体蛋白,100mM磷酸钠缓冲盐(pH8.0)补足至150μL。30℃反应12小时。此外,还考察了辅酶PLP的存在对突变体蛋白活性的影响,反应体系为:4mM L-酪氨酸,50μg纯化的CtTyDC突变体蛋白,1mM PLP,100mM磷酸钠缓冲盐(pH8.0)补足至150μL。30℃反应12小时。结果如图7所示,其中,W91、C167、H202、H317、K318、R478突变成丙氨酸(Ala)后,不管反应体系中是否添加辅酶PLP,蛋白活性都完全丧失,表明这几个位点是CtTyDC催化脱羧反应的关键位点。F100、P101、S102、T166、T261、L324这几个位点突变后酶活性都降低,但在反应体系中添加辅酶PLP后,活性又会恢复,推测这几个位点与CtTyDC催化反应不依赖PLP的特性有关。图7中,横坐标的位点后缀有A,表示氨基酸位点突变,例如W91A代表位点W91突变成丙氨酸(Ala)。
本发明并不限于上述实施方式,在不背离本发明的实质内容的情况下,本领域技术人员可以想到的任何变形、改进、替换均落入本发明的范围。
序列表
<110> 北京中医药大学
<120> 酪氨酸脱羧酶CtTyDC、编码基因、试剂盒及制备酪胺的方法
<160> 7
<170> SIPOSequenceListing 1.0
<210> 1
<211> 509
<212> PRT
<213> Cistanche tubulosa
<400> 1
Met Gly Ser Leu Gln Asn Lys Lys Pro Asp Gly Ala Ala Ser Ala Gly
1 5 10 15
Ile Ile Lys Pro Met Asp Pro Glu Glu Phe Arg Arg Gln Gly His Leu
20 25 30
Val Ile Asp Phe Ile Ala Asp Tyr Tyr Lys Asn Val Glu Lys Tyr Pro
35 40 45
Val Arg Ser Gln Val Glu Pro Gly Tyr Leu Lys Lys Arg Met Pro Asp
50 55 60
Ser Ala Pro His Gly Pro Glu Pro Ile Glu Glu Ile Leu Arg Asp Val
65 70 75 80
Gln Asn Asp Ile Val Pro Gly Ile Thr His Trp Gln Ser Pro Asn Tyr
85 90 95
Tyr Ala Tyr Phe Pro Ser Ser Gly Ser Ile Ala Gly Phe Leu Gly Glu
100 105 110
Met Leu Ser Thr Gly Phe Asn Ile Val Gly Phe Asn Trp Met Ser Ser
115 120 125
Pro Ala Ala Thr Glu Leu Glu Ser Ile Val Met Asp Trp Leu Gly Lys
130 135 140
Met Leu Lys Leu Pro Ser Glu Phe Leu Phe Ser Gly Gly Gly Gly Gly
145 150 155 160
Val Leu Gln Gly Thr Thr Cys Glu Ala Ile Leu Cys Thr Leu Val Ala
165 170 175
Ala Arg Asp Gln Met Leu Arg Lys Ile Gly Arg Glu Asn Ile Asn Lys
180 185 190
Leu Val Val Tyr Gly Ser Asp Gln Thr His Ser Ala Leu Gln Lys Ala
195 200 205
Ala Gln Ile Ala Gly Ile Asn Pro Asn Asn Phe Arg Ala Val Ser Thr
210 215 220
Thr Lys Glu Thr Ala Phe Gly Leu Thr Gly Lys Ala Leu Arg Ala Ala
225 230 235 240
Ile Glu Ser Asp Val Asp Leu Gly Leu Val Pro Leu Phe Leu Cys Ala
245 250 255
Thr Val Gly Thr Thr Ser Ser Thr Ala Val Asp Pro Leu Glu Pro Leu
260 265 270
Cys Asp Val Ala Glu Glu Tyr Gly Val Trp Val His Val Asp Ala Ala
275 280 285
Tyr Ala Gly Ser Val Cys Ile Cys Pro Glu Tyr Arg His Phe Ile Asp
290 295 300
Gly Val Glu Lys Ala His Ser Phe Ser Phe Asn Ala His Lys Trp Phe
305 310 315 320
Leu Thr Thr Leu Asp Cys Cys Cys Leu Trp Val Lys Asp Pro Gly Ala
325 330 335
Leu Val Lys Ala Leu Ser Thr Tyr Pro Glu Tyr Leu Arg Asn Lys Ala
340 345 350
Ser Glu Thr Lys Gln Val Val Asp Tyr Lys Asp Trp Gln Ile Thr Leu
355 360 365
Ser Arg Arg Phe Arg Ser Leu Lys Leu Trp Met Val Leu Arg Ser Tyr
370 375 380
Gly Val Ala Asn Leu Arg Lys Phe Leu Arg Ser His Val Lys Met Ala
385 390 395 400
Met Asn Phe Glu Gly Leu Val Gly Met Asp Lys Arg Phe Glu Val Val
405 410 415
Val Pro Arg Asn Phe Ala Thr Val Cys Phe Arg Ile Ser Pro Val Glu
420 425 430
Ile Ala Gly Asn His Gln Met Ile Ala Ser Arg Glu Glu Ala Ala Asn
435 440 445
Ser Phe Asn Ala Arg Leu Leu Glu Met Ile Asn Glu Ser Gly Glu Val
450 455 460
Tyr Met Thr His Ala Val Val Gly Gly Val Tyr Val Met Arg Phe Ala
465 470 475 480
Val Gly Ala Ser Leu Thr Glu Asn Arg His Val Ile Leu Ala Trp Lys
485 490 495
Val Val Gln Glu His Ala Asn Ala Leu Leu Ala Ser Ser
500 505
<210> 2
<211> 1530
<212> DNA
<213> Cistanche tubulosa
<400> 2
atgggcagcc ttcagaataa aaaaccagac ggtgccgcct ccgccggcat cattaaacct 60
atggaccccg aagagttcag gcggcagggc catcttgtca tcgacttcat cgccgattac 120
tacaagaacg tagagaaata ccctgtccga agccaagtcg aaccgggcta tctaaagaaa 180
cggatgccag attccgctcc acacggccca gaaccaatcg aggaaatcct ccgcgacgtc 240
caaaacgaca tcgttccggg catcacacac tggcagagcc ccaactatta tgcttacttc 300
ccgtccagtg ggagtatagc aggatttctc ggagaaatgc tgagcactgg ttttaacatc 360
gttggattta actggatgtc gtctccggcc gccactgagc tggagagcat cgtcatggac 420
tggctcggga aaatgctcaa gctcccgtcg gagtttttgt tctccggcgg aggcggcgga 480
gtcttgcagg gaaccacctg cgaggccatt ctgtgcactc ttgtcgccgc ccgagaccag 540
atgctgagaa agatcggcag agagaatatt aacaagttgg tcgtgtacgg gtcggatcag 600
acccactccg cgttgcaaaa ggcggcccag atcgctggca tcaacccgaa taatttccgg 660
gccgtatcca ccacgaagga aacagcattc gggctgacgg ggaaggccct ccgcgccgcg 720
atcgagtccg atgtggatct cgggctggtg ccgctgtttt tatgcgccac cgttgggacg 780
acatcgtcga ccgccgtgga cccgctggaa ccgctgtgcg acgtggcgga ggagtacggg 840
gtttgggtcc acgtggacgc ggcctacgcc ggcagcgttt gtatttgccc tgagtatcgc 900
catttcatcg atggggttga aaaagcgcat tcgttcagct ttaacgcgca taagtggttt 960
ttgacgactt tagattgctg ctgcctgtgg gttaaggacc caggcgccct ggtaaaagcg 1020
ctatcaactt atcccgagta tttgagaaac aaagcctcgg agacaaaaca ggtcgtcgat 1080
tacaaagact ggcaaatcac tctcagccga cgattccgat cactgaagct ctggatggtc 1140
ctccgtagct atggcgtggc caacctcagg aaattcctcc gcagccacgt gaaaatggcg 1200
atgaatttcg aaggtctggt cggaatggac aagcggttcg aggtggtggt gccgagaaac 1260
ttcgccaccg tctgcttccg gatctcgccg gtggagatcg ccggaaacca ccagatgatc 1320
gcttcgaggg aggaggcggc caatagtttt aacgcgagat tgctggagat gataaatgaa 1380
tcgggtgagg tttacatgac gcacgcggtg gttggcggcg tttacgtgat gcgtttcgcc 1440
gtcggtgcga gcttgacgga aaacaggcac gttattttgg cctggaaagt tgtgcaggaa 1500
catgcaaacg ctctgttagc cagttcgtga 1530
<210> 3
<211> 1882
<212> DNA
<213> Cistanche tubulosa
<400> 3
ctaatacgac tcactatagg gcaagcagtg gtatcaacgc agagtacatg ggggcaaacc 60
ccccccaaaa aaaaaggaaa atatattact acatattata catatataca tataccacag 120
tatcttcttt tctatttcct gacaagctaa caagtgaaag ataccaacat aaaagataat 180
atttgcacca ggcttcaaat ttaacatcga tgggcagcct tcagaataaa aaaccagacg 240
gtgccgcctc cgccggcatc attaaaccta tggaccccga agagttcagg cggcagggcc 300
atcttgtcat cgacttcatc gccgattact acaagaacgt agagaaatac cctgtccgaa 360
gccaagtcga accgggctat ctaaagaaac ggatgccaga ttccgctcca cacggcccag 420
aaccaatcga ggaaatcctc cgcgacgtcc aaaacgacat cgttccgggc atcacacact 480
ggcagagccc caactattat gcttacttcc cgtccagtgg gagtatagca ggatttctcg 540
gagaaatgct gagcactggt tttaacatcg ttggatttaa ctggatgtcg tctccggccg 600
ccactgagct ggagagcatc gtcatggact ggctcgggaa aatgctcaag ctcccgtcgg 660
agtttttgtt ctccggcgga ggcggcggag tcttgcaggg aaccacctgc gaggccattc 720
tgtgcactct tgtcgccgcc cgagaccaga tgctgagaaa gatcggcaga gagaatatta 780
acaagttggt cgtgtacggg tcggatcaga cccactccgc gttgcaaaag gcggcccaga 840
tcgctggcat caacccgaat aatttccggg ccgtatccac cacgaaggaa acagcattcg 900
ggctgacggg gaaggccctc cgcgccgcga tcgagtccga tgtggatctc gggctggtgc 960
cgctgttttt atgcgccacc gttgggacga catcgtcgac cgccgtggac ccgctggaac 1020
cgctgtgcga cgtggcggag gagtacgggg tttgggtcca cgtggacgcg gcctacgccg 1080
gcagcgtttg tatttgccct gagtatcgcc atttcatcga tggggttgaa aaagcgcatt 1140
cgttcagctt taacgcgcat aagtggtttt tgacgacttt agattgctgc tgcctgtggg 1200
ttaaggaccc aggcgccctg gtaaaagcgc tatcaactta tcccgagtat ttgagaaaca 1260
aagcctcgga gacaaaacag gtcgtcgatt acaaagactg gcaaatcact ctcagccgac 1320
gattccgatc actgaagctc tggatggtcc tccgtagcta tggcgtggcc aacctcagga 1380
aattcctccg cagccacgtg aaaatggcga tgaatttcga aggtctggtc ggaatggaca 1440
agcggttcga ggtggtggtg ccgagaaact tcgccaccgt ctgcttccgg atctcgccgg 1500
tggagatcgc cggaaaccac cagatgatcg cttcgaggga ggaggcggcc aatagtttta 1560
acgcgagatt gctggagatg ataaatgaat cgggtgaggt ttacatgacg cacgcggtgg 1620
ttggcggcgt ttacgtgatg cgtttcgccg tcggtgcgag cttgacggaa aacaggcacg 1680
ttattttggc ctggaaagtt gtgcaggaac atgcaaacgc tctgttagcc agttcgtgat 1740
ttgctttttg tcaacttttc agatttttgt tttaaatgtc ccttgttctg gaaaatatcc 1800
catgtttttg gtttgtgaaa ttattaaata attgaaatat tactattatg ggcaaaaaaa 1860
aaaaaaaaaa aaaaaaaaaa aa 1882
<210> 4
<211> 28
<212> DNA
<213> Artifical sequence
<400> 4
gaacgaatgc gctttttcaa ccccatcg 28
<210> 5
<211> 26
<212> DNA
<213> Artifical sequence
<400> 5
atgcgccacc gttgggacga catcgt 26
<210> 6
<211> 31
<212> DNA
<213> Artifical sequence
<400> 6
gaattcatgg gcagccttca gaataaaaaa c 31
<210> 7
<211> 29
<212> DNA
<213> Artifical sequence
<400> 7
ctcgagcgaa ctggctaaca gagcgtttg 29
- 一种用于人类BRCA1和BRCA2基因全编码序列突变位点检测的引物、方法和试剂盒
- 华法林个体化用药相关基因多态性检测试剂盒及其制备方法与应用
- 一种检测KRAS基因外显子2密码子12/13突变的试剂盒及其制备方法
- 一种Nucyep酶的编码基因、重组表达载体、重组工程菌及其制备方法和应用
- 用于生产偶姻的方法,其适用的丙酮酸脱羧酶,以及两者的制备,和编码PDC基因的DNA序列
- 使造血干细胞移植后附着生长稳定的组合物、用于获取该组合物的试剂盒、使造血干细胞移植后附着生长稳定的方法、人单克隆抗体和多克隆抗体及其制备方法、编码人单克隆抗体的基因以及导入了该基因的重组细胞
