一株高产FR901379的菌株及其构建方法与应用
文献发布时间:2024-04-18 19:58:26
技术领域
本发明属于基因工程技术领域,具体涉及过表达目的基因使FR901379产量提高的鞘茎点霉属的基因工程菌及其构建方法与应用。
背景技术
棘白菌素类抗真菌药物是一类环脂肽类天然产物的衍生物,能够选择性地抑制真菌细胞壁中的β-1,3葡聚糖合成酶活性,从而影响真菌细胞壁的合成,导致真菌细胞裂解死亡。棘白菌素类药物作用机制独特,安全性高,抗菌谱宽且对耐药菌有效。随着真菌耐药性加剧,棘白菌素类药物在全球抗真菌药物市场中所占比例成呈逐年上升趋势。目前,临床上应用的棘白菌素类抗真菌药物有卡泊芬净,米卡芬净和阿尼芬净。其中,米卡芬净具有其独特性,其前体FR901379具有磺酰基基团,从而具有极好的水溶性,进而提高了其生物利用度。
米卡芬净的工业生产包括三步:首先由Coleophoma empetri发酵产生FR901379,然后经游他游动链霉菌发酵将脂肪酸侧链水解掉,最后通过化学修饰加上4-(5-(4-(戊基氧基)苯基)-3-异噁唑基)苯甲酸甲酯侧链最终生成米卡芬净。其中C.empetri发酵产生FR901379是生产工艺中重要的一步,对于生产成本控制和后处理工艺至关重要。目前对于C.empetri生产性能的优化一般通过发酵培养基优化,物理诱变等方式,未有通过遗传改造提高菌株生产性能的相关报道。C.empetri发酵产物中除了FR901379外,还有另外两个副产物WF11899B和WF11899C,副产物与FR901379最大的区别在于L-鸟氨酸C-4位与L-高酪氨酸C-4位的羟基化修饰程度不同。在C.empetri发酵过程中存在产量低,副产物多的现象,导致后续分离纯化困难,导致米卡芬净的生产成本一直居高不下。目前米卡芬净工业生产上迫切需要解决上述问题,但是负责L-鸟氨酸C-4位与L-高酪氨酸C-4位羟化的酶的编码基因尚不可知,这极大地限制了相关代谢工程改造。因此,鉴定负责L-鸟氨酸C-4位和L-高酪氨酸C-4位羟化的氧化酶将为代谢工程改造提供改造靶点,降低副产物积累,提高FR901379产量,促进米卡芬净生产技术提质增效。
发明内容
本发明提供了一种基因工程菌株,所述基因工程菌株的出发菌株为鞘茎点霉属真菌。
所述鞘茎点霉属真菌包括Coleophoma sp.或C.empetri。
在一个具体的实施方式中,鞘茎点霉属真菌为鞘茎点霉(Coleophoma sp.)MEFC009,其保藏于中国微生物菌种保藏管理委员会普通微生物中心(CGMCC),保藏编号为CGMCC No.21058,保藏日期为2020年11月18日,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,电话:010-64807355。
本发明中,针对鞘茎点霉(Coleophoma sp.)MEFC009中的细胞色素P450单加氧酶McfF和McfH进行了遗传改造,获得了不同的基因工程菌株,某些基因工程菌株可以高产米卡芬净前体FR901379。
本发明中,所述McfF为细胞色素P450单加氧酶(cytochrome P450monooxygenase),其氨基酸序列如SEQ ID No.2所示,其核酸序列如SEQ ID No.1所示。所述McfH为细胞色素P450单加氧酶(cytochrome P450 monooxygenase),其氨基酸序列如SEQID No.4所示,其核酸序列如SEQ ID No.3所示。
本发明中,所述细胞色素P450单加氧酶又称为P450酶。
一方面,本发明提供了一种细胞色素P450单加氧酶。在一个实施方式中,所述细胞色素P450单加氧酶与SEQ ID No.2或4相比具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性;优选的,所述细胞色素P450单加氧酶来源于鞘茎点霉属真菌;更优选的,所述细胞色素P450单加氧酶的氨基酸序列与SEQ ID No.2或4相比具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性,并且所述细胞色素P450单加氧酶来源于鞘茎点霉属真菌。所述鞘茎点霉属真菌包括Coleophoma sp.或Coleophoma empetri,例如,鞘茎点霉(Coleophoma sp.)MEFC009。
在优选的实施方式中,所述细胞色素P450单加氧酶的氨基酸序列如SEQ ID No.2或SEQ ID No.4所示。
另一方面,本发明还提供了包含上述细胞色素P450单加氧酶或其编码基因的生物材料。所述生物材料选自:包含上述细胞色素P450单加氧酶的载体,或者,包含上述细胞色素P450单加氧酶的宿主细胞。
另一方面,本发明还提供了编码上述细胞色素P450单加氧酶的基因。
另一方面,本发明还提供了包含上述基因的载体,或者,包含所述载体的宿主细胞。
在一个实施方式中,所述载体包括克隆载体和表达载体,例如,pET系列载体(如,pET-14、pET-21、pET-22、pET-28、pET-30、pET-42、pET-GST、pET-His、pET-Trx、pET-GST、pET-CKS、pET-DsbA),pMAL系列载体(如pMAL-2c)、pGEX系列载体(如pGEX-4T-2、pGEX-6T-1)、pBAD系列载体(如pBAD-His、pBAD-Myc)、pMBP系列载体(pMBP-P、pMBP-C)、pTYB2、pQE-9、pACYCDuet-1、pCDFDuet-1、pColADuet-1、pRSFDuet-1、pllP-OmpA、pUC系列载体(如,pUC18、pUC19),pQE-30、pXH2-1,pXH-43,pTRII,pGSF957。
在一个实施方式中,所述宿主细胞选自大肠杆菌(例如,大肠杆菌DH5α、大肠杆菌BL21(DE3)、Rosetta(DE3)、Codon Plus(DE3)-RIPL、BL21 Codon plus(DE3)、Top 10、JM109)、酵母菌(例如,酿酒酵母、毕赤酵母、解酯耶氏酵母)、鞘茎点霉真菌。
另一方面,本发明还提供了上述细胞色素P450单加氧酶、其编码基因、包含基因的载体、上述宿主细胞、或上述生物材料在制备米卡芬净前体FR901379中的应用。
另一方面,本发明还提供了上述细胞色素P450单加氧酶、其编码基因、包含基因的载体、上述宿主细胞、或上述生物材料在制备高产米卡芬净前体FR901379的基因工程菌株中的应用;优选的,所述基因工程菌株的出发菌株为鞘茎点霉属真菌。
所述鞘茎点霉属真菌包括Coleophoma sp.或C.empetri。
在一个具体的实施方式中,鞘茎点霉属真菌为鞘茎点霉(Coleophoma sp.)MEFC009,其保藏于中国微生物菌种保藏管理委员会普通微生物中心(CGMCC),保藏编号为CGMCC No.21058,保藏日期为2020年11月18日,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,电话:010-64807355。
上述制备高产米卡芬净前体FR901379的基因工程菌株,为在出发菌株中引入上述细胞色素P450单加氧酶;优选的,所述引入为过表达。
所述的“引入”包括将上述目的基因在出发菌株中进行表达的步骤,优选,过表达。例如,将目的基因构建到表达载体上,将表达载体转入到宿主细胞中以表达目的基因,优选,过表达。在其他的实施方式中,所述的“引入”包括将目的基因插入到宿主细胞的基因组中;优选的,所述插入到宿主细胞的基因组中可以采用同源重组双交换的方法;在一个实施方式中,可以采用将目的基因以及同源臂插入到载体上,然后将载体转入到宿主细胞中,利用同源臂与宿主细胞基因组发生同源重组双交换从而将目的基因插入到合适的基因组的位置;在其他的实施方式中,还可以采用基因编辑的方式,例如,利用CRIspR/Cas系统在期望的基因组位点上进行切割,同时将目的基因作为外源供体插入到切割位点上。
另一方面,本发明还提供了上述基因工程菌株在生产米卡芬净前体FR901379中的应用。
另一方面,本发明还提供了一种制备米卡芬净前体FR901379的方法,所述方法包括利用上述基因工程菌株进行发酵的步骤;任选的,所述方法还包括分离/纯化FR901379的步骤。
本发明中,FR901379为米卡芬净前体,其结构式如式(I)所示:
本发明中,WF11899B和WF11899C的结构式分别如式(II)和式(III)所示:
本发明中还提供了一种化合物,其结构式如式(IV)所示:
另一方面,本发明还提供了一种高产米卡芬净前体FR901379的基因工程菌,所述基因工程菌为过表达上述细胞色素P450单加氧酶的基因工程菌,所述基因工程菌的出发菌株为鞘茎点霉属真菌。
在一个实施方式中,所述基因工程菌中过表达第一细胞色素P450单加氧酶;所述第一细胞色素P450单加氧酶与SEQ ID No.2相比具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性;优选的,所述第一细胞色素P450单加氧酶来源于鞘茎点霉属真菌;更优选的,所述第一细胞色素P450单加氧酶的氨基酸序列与SEQ IDNo.2相比具有至少70%的序列同一性,并且所述第一细胞色素P450单加氧酶来源于鞘茎点霉属真菌。
在一个实施方式中,所述基因工程菌中过表达第二细胞色素P450单加氧酶;所述第二细胞色素P450单加氧酶与SEQ ID No.4相比具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性;优选的,所述第二细胞色素P450单加氧酶来源于鞘茎点霉属真菌;更优选的,所述第二细胞色素P450单加氧酶的氨基酸序列与SEQ IDNo.4相比具有至少70%的序列同一性,并且所述第二细胞色素P450单加氧酶来源于鞘茎点霉属真菌。
在一个实施方式中,所述基因工程菌中同时过表达上述第一细胞色素P450单加氧酶和上述第二细胞色素P450单加氧酶。
本发明中的“过表达”是指基因工程菌与野生型的出发菌株相比,所述目的基因的表达量或活性要高于野生型的出发菌株。在一个实施方式中,上述过表达可以通过引入表达载体来过表达目的基因来实现;其他的实施方式中,上述过表达也可以通过在出发菌株中引入额外的目的基因的拷贝、通过增加目的基因的拷贝数来实现;其他的实施方式中,还可以通过对目的基因的启动子优化来实现,比如,通过将目的基因的原始启动子替换为启动子活性更高的启动子来实现目的基因的过表达。
另一方面,本发明还提供了利用上述基因工程菌株制备/生产米卡芬净前体FR901379的方法,所述方法包括对上述基因工程菌株进行培养的步骤;或者,上述基因工程菌株在制备/生产米卡芬净前体FR901379中的应用。
另一方面,本发明还提供了一种制备/生产米卡芬净前体FR901379的方法,所述方法包括利用上述基因工程菌株进行培养的步骤;优选的,所述方法还包括分离/纯化FR901379的步骤。
另一方面,本发明还提供了一种高产WF11899B的基因工程菌株,所述WF11899B如上述式II所示,所述基因工程菌的出发菌株为鞘茎点霉属真菌。进一步的,所述高产WF11899B的基因工程菌株为将出发菌株中的细胞色素P450单加氧酶进行突变所得到的基因工程菌株;所述细胞色素P450单加氧酶与SEQ ID No.2相比具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性。
另一方面,本发明还提供了利用上述基因工程菌株制备/生产WF11899B的方法,所述方法包括对上述基因工程菌株进行培养的步骤;或者,上述基因工程菌株在制备/生产WF11899B中的应用。
另一方面,本发明还提供了一种制备/生产WF11899B的方法,所述方法包括利用上述基因工程菌株进行培养的步骤;优选的,所述方法还包括分离/纯化WF11899B的步骤。
另一方面,本发明还提供了一种高产式IV所示化合物的基因工程菌株,所述基因工程菌的出发菌株为鞘茎点霉属真菌。进一步的,所述高产式IV所示化合物的基因工程菌株为将出发菌株中的细胞色素P450单加氧酶突变所得到的基因工程菌株;所述细胞色素P450单加氧酶与SEQ ID No.4相比具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性。
另一方面,本发明还提供了利用上述基因工程菌株制备/生产式IV所示化合物的方法,所述方法包括对上述基因工程菌株进行培养的步骤;或者,上述基因工程菌株在制备/生产式IV所示化合物中的应用。
另一方面,本发明还提供了一种制备/生产式IV所示化合物的方法,所述方法包括利用上述基因工程菌株进行培养的步骤;优选的,所述方法还包括分离/纯化式IV所示化合物的步骤。
本发明所述的突变包括通过基因缺失、基因插入或基因取代的方式导致基因功能或活性丧失。
在优选的实施方式中,基因突变为将目标基因进行基因敲除。
在一个实施方式中,所述基因突变可以采用本领域常规的技术操作来实现,例如,通过同源重组的方式进行基因敲入或基因敲除从而导致基因功能或活性丧失;或者,采用基因编辑的方式,如锌指核酸内切酶(ZFN)、类转录激活因子效应物核酸酶(TALEN)或CRIspR技术对上述基因进行突变从而导致基因功能或活性丧失。
另一方面,本发明还提供了上述基因工程菌的构建方法。
附图说明
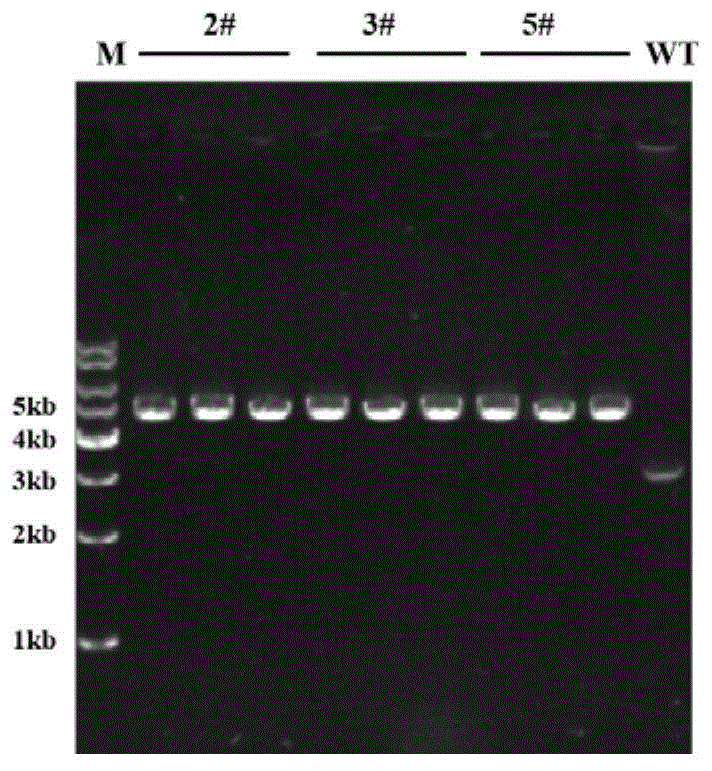
图1为敲除mcfF基因所获转化子的基因组PCR验证结果;其中2#,3#,5#是基因mcfF缺失的转化子,WT为对照菌株Coleophoma sp.MEFC009。
图2为基因mcfF缺失菌株Coleophoma sp.-ΔmcfF发酵产物HPLC分析结果;其中Coleophoma sp.-ΔmcfF为基因mcfF缺失菌株,WT为对照菌株Coleophoma sp.MEFC009;1:FR901379;2:WF11899B;3:WF11899C。
图3为化合物FR901379,WF11899B和WF11899C的化学结构;
图4为敲除mcfH基因所获转化子的基因组PCR验证结果;其中6#,8#,9#是基因mcfH基因缺失的转化子,WT为对照菌株Coleophoma sp.MEFC009。
图5为基因mcfH缺失菌株Coleophoma sp.-ΔmcfH发酵产物HPLC分析结果;其中Coleophoma sp.-ΔmcfH为基因mcfH缺失菌株,WT为对照菌株Coleophoma sp.MEFC009;1:FR901379;2:WF11899B;3:WF11899C。
图6为基因mcfH缺失菌株Coleophoma sp.-ΔmcfH发酵产物中化合物4的LC-MS分析结果。
图7为基因mcfH缺失菌株Coleophoma sp.-ΔmcfH发酵产物中化合物4的化学结构;
图8为过表达P450酶McfF所获得的转化子基因组PCR验证;其中1-23为转化子,WT为野生型Coleophoma sp.MEFC009。
图9为过表达P450酶McfF的工程菌株和Coleophoma sp.MEFC009发酵产物HPLC分析结果;其中1:FR901379;2:WF11899B;3:WF11899C。
图10为过表达P450酶McfF的工程菌株和Coleophoma sp.MEFC009发酵液中FR901379(WF11899A)、WF11899B和WF11899C产量分析;
图11为过表达P450酶McfH所获得的转化子基因组PCR验证;其中1-23为转化子,WT为野生型Coleophoma sp.MEFC009。
图12为过表达P450酶McfH的工程菌株和Coleophoma sp.MEFC009发酵产物HPLC分析结果;其中1:FR901379;2:WF11899B;3:WF11899C。
图13为过表达P450酶McfH的工程菌株和Coleophoma sp.MEFC009发酵液中FR901379(WF11899A)、WF11899B和WF11899C产量分析。
图14为同时过表达P450酶McfF和McfH所获得的转化子基因组PCR验证;其中1-18为转化子,WT为野生型Coleophoma sp.MEFC009。
图15为同时过表达P450酶McfF和McfH的工程菌株和Coleophoma sp.MEFC009发酵液中FR901379(WF11899A、WF11899B和WF11899C产量分析。
具体实施方式
下面结合具体实施例对本发明做进一步说明,但本发明不受实施例的限制。以下实施例中所用材料、试剂、仪器和方法,未经特殊说明,均为本领域中的常规材料、试剂、仪器和方法,均可通过商业渠道获得。
本发明中质粒提取采用OMEGA公司的Plasmid Mini Kit I试剂(D6942-01),PCR片段纯化采用OMEGA公司DNA片段回收Cycle-Pure Kit试剂盒(D6492-01)。一步克隆酶
种子培养基:15g/L可溶性淀粉,10g/L蔗糖,5g/L棉籽饼粉,10g/L蛋白胨,1g/LKH
发酵培养基:30g/L玉米淀粉,30g/L蛋白胨,6g/L(NH
STC:1M山梨醇,50mM Tris-HCl(pH 8.0),50mM CaCl
PSTC:40%PEG4000,1M山梨醇,50mM Tris-HCl(pH 8.0),50mM CaCl
顶层琼脂:PDB、1M山梨醇和4g/L琼脂糖,灭菌后45-48℃保温。
再生筛选培养基平板PDA-SH:PDA平板、1M山梨醇和100mg/L潮霉素B。
筛选培养基PDA-H:PDA平板和100mg/L潮霉素B。
质粒pXH2-1记载在Xuenian Huang,Xuefeng Lu,Jian-Jun Li.Cloning,characterization and application of a glyceraldehyde-3-phosphatedehydrogenase promoter from Aspergillus terreus,J Ind Microbiol Biotechnol(2014)41:585–592。
本实施方式中,所利用的出发菌株为鞘茎点霉属真菌,鞘茎点霉(Coleophomasp.)MEFC009,上述菌株保藏于中国微生物菌种保藏管理委员会普通微生物中心(CGMCC),保藏编号为CGMCC No.21058,保藏日期为2020年11月18日,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,电话:010-64807355。
实施例1.构建敲除基因mcfF的工程菌株Coleophoma sp.-ΔmcfF
以野生型Coleophoma sp.MEFC009的基因组为模板,采用pfu DNA聚合酶(Fermentas,Catalog No.:EP0501)进行PCR扩增,用引物UmcfF-F(5’-cacctctaagatagtctatc-3’)和UmcfF-R(5’-ctttacgcttgcgatcccgaaTGTATAAGATGCATCAGTGCC-3’)可以扩增获得大小约为1.2kb的mcfF的上游序列U-mcfF,用引物DmcfF-F(5’-cctgggttcgcaaagataattgCTCGAACGTTGGATATATAGC-3’)和DmcfF-R(5’-ttgccaaaacaggctctgata-3’)可以扩增获得大小为1.2kb的mcfF的下游序列D-mcfF。以质粒pXH2-1为模板,用引物hph-F(5’-ttcgggatcgcaagcgtaaag-3’)和hph-R(5’-caattatctttgcgaacccagg-3’)进行PCR扩增,获得大小约为2.2kb的潮霉素抗性筛选片段hph;通过融合PCR将hph片段、上游序列U-mcfF和下游序列D-mcfF进行融合,再以融合产物为模板,用巢式引物UmcfF-CS-F(5’-ggatactttcaattatgcggcc-3’)和DmcfF-CS-R(5’-aattgagggacagtcattct-3’)通过PCR扩增出大小为4.4kb的敲除打靶元件UmcfF-hph-DmcfF。
以Coleophoma sp.MEFC009为出发菌,首先从PDA平板上取少量菌丝,用手持匀浆器破碎,取1ml种子液接种到50ml的种子培养基,于250ml的三角瓶中,220rpm,25℃进行摇床培养。2天后,离心收集菌丝。5000rpm,4℃,5min。将菌丝体再次用匀浆器破碎,取0.5ml-2ml种子液接种到50ml的种子培养基,相同的条件下培养1天,将培养基与菌丝体一起倒入50ml的无菌离心管中,5000rpm,离心收集菌丝。用0.6M MgSO
向140μl上述原生质体悬液中加入10μl的UmcfF-hph-DmcfF片段,再加入50μl的PSTC,轻轻混匀,冰浴30min。加入1ml PSTC,混匀后室温放置20min;然后与10ml顶层琼脂混合后倾注于3块再生筛选培养基平板PDA-SH上,在30℃、黑暗条件下培养5-7天,得到转化子。
从转化筛选平板上挑取具有潮霉素抗性转化子转移至PDA-H上,在25℃培养5-7天进行传代培养,连续传代3代。选取稳定传代的3个转化子2#,3#,5#进行单孢分离纯化,并提取单孢分离之后的转化子基因组。利用外部引物UmcfF-F(5’-cacctctaagatagtctatc-3’)和DmcfF-R(5’-ttgccaaaacaggctctgata-3’)对转化子基因组进行PCR验证,能扩增出大小约为4.7kb的条带的为阳性转化子,而Coleophoma sp.MEFC009只能扩增出大小约为3.2kb的条带,图1结果表明在2#,3#,5#转化子为阳性转化子,说明在基因mcfF位置发生了同源重组,整合了外源片段UmcfF-hph-DmcfF,将阳性菌株定义为Coleophoma sp.-ΔmcfF。
实施例2.基因mcfF缺失工程菌株Coleophoma sp.-ΔmcfF发酵验证
将3株阳性工程菌株2#,3#,5#和对照菌株Coleophoma sp.MEFC009接种于PDA固体平板上,25℃培养5-7天。挑取少量的菌丝,利用核酸提取仪(
HPLC分析方法为:液相色谱柱为Agilent C-18反向柱883975-902(4.6×150mm,5μm);流动相为A:0.05%(体积比)三氟乙酸水溶液,流动相B:0.05%(体积比)三氟乙酸乙腈溶液,流速为1ml/min,紫外检测波长:210nm,30℃,总洗脱时间为37min。梯度洗脱条件:0-5min,流动相B占流动相的体积由5%线性上升到24%,5-35min,流动相B占流动相的体积由24%线性上升到62%,35-37min,流动相B占流动相的体积由62%线性上升到100%。结果如图2所示;与出发菌株相比,Coleophoma sp.-ΔmcfF中的化合物1(FR901379)消失,相应的化合物2(WF11899B)的产量是出发菌株Coleophoma sp.中化合物1和化合物2的总和且化合物WF11899B与FR901379相比,在L-高酪氨酸的C-4位缺少一个羟基(如图3所示),说明基因mcfF编码的P450酶负责L-高酪氨酸C-4位的羟基化,该基因的核酸序列如SEQ ID No.1所示,其编码的氨基酸序列如SEQ ID No.2所示。
实施例3.构建敲除基因mcfH的工程菌株Coleophoma sp.-ΔmcfH
以野生型Coleophoma sp.MEFC009的基因组为模板,采用pfu DNA聚合酶(Fermentas,Catalog No.:EP0501)进行PCR扩增,用引物UmcfH-F(5’-gtgagtgttcctcaaggcag-3’)和UmcfH-R(5’-ctttacgcttgcgatcccgaaATCACCGATCAGACCATCTC-3’)可以扩增获得大小约为1.5kb的mcfH的上游序列U-mcfH,用引物DmcfH-F(5’-cctgggttcgcaaagataattgCGCCAAGTTGTCAGCCCAAA-3’)和DmcfH-R(5’-ccgctttaatcaacttggca-3’)可以扩增获得大小为1.4kb的mcfH的下游序列D-mcfH。以质粒pXH2-1为模板,用引物hph-F(5’-ttcgggatcgcaagcgtaaag-3’)和hph-R(5’-caattatctttgcgaacccagg-3’)进行PCR扩增,获得大小约为2.2kb的潮霉素抗性筛选片段hph;通过融合PCR将hph片段、上游序列U-mcfH和下游序列D-mcfH进行融合,再以融合产物为模板,用巢式引物UmcfH-CS-F(5’-atagcctattcatgatttct-3’)和DmcfH-CS-R(5’-tacgcccgagcgacccgagt-3’)通过PCR扩增出大小为4.8kb的敲除打靶元件UmcfH-hph-DmcfH。
以Coleophoma sp.MEFC009为出发菌,首先从PDA平板上取少量菌丝,用手持匀浆器破碎,取1ml种子液接种到50ml的种子培养基,于250ml的三角瓶中,220rpm,25℃进行摇床培养。2天后,离心收集菌丝。5000rpm,4℃,5min。将菌丝体再次用匀浆器破碎,取0.5ml-2ml种子液接种到50ml的种子培养基,相同的条件下培养1天,将培养基与菌丝体一起倒入50ml的无菌离心管中,5000rpm,离心收集菌丝。用0.6M MgSO
向140μl上述原生质体悬液中加入10μl的UmcfH-hph-DmcfH片段,再加入50μl的PSTC,轻轻混匀,冰浴30min。加入1ml PSTC,混匀后室温放置20min;然后与10ml顶层琼脂混合后倾注于3块再生筛选培养基平板PDA-SH上,在30℃、黑暗条件下培养5-7天,得到转化子。
从转化筛选平板上挑取具有潮霉素抗性转化子转移至PDA-H上,在25℃培养5-7天进行传代培养,连续传代3代。选取稳定传代的3个转化子6#,8#,9#进行单孢分离纯化,并提取单孢分离之后的转化子基因组。利用外部引物UmcfH-F(5’-gtgagtgttcctcaaggcag-3’)和DmcfH-R(5’-ccgctttaatcaacttggca-3’)对转化子基因组进行PCR验证,能扩增出大小约为5.0kb的条带的为阳性转化子,而Coleophoma sp.MEFC009只能扩增出大小约为3.7kb的条带,图4说明在6#,8#,9#转化子为阳性转化子,说明在基因mcfH位置发生了同源重组,整合了外源片段UmcfH-hph-DmcfH,将阳性菌株定义为Coleophoma sp.-ΔmcfH。
实施例4.基因mcfH缺失工程菌株Coleophoma sp.-ΔmcfH发酵验证
将3株工程菌株6#,8#,9#和对照菌株Coleophoma sp.MEFC009接种于PDA固体平板上,25℃培养5-7天。挑取少量的菌丝,利用核酸提取仪(
HPLC分析方法为:液相色谱柱为Agilent C-18反向柱883975-902(4.6×150mm,5μm);流动相为A:0.05%(体积比)三氟乙酸水溶液,流动相B:0.05%(体积比)三氟乙酸乙腈溶液,流速为1ml/min,紫外检测波长:210nm,30℃,总洗脱时间为37min。梯度洗脱条件:0-5min,流动相B占流动相的体积由5%线性上升到24%,5-35min,流动相B占流动相的体积由24%线性上升到62%,35-37min,流动相B占流动相的体积由62%线性上升到100%。结果如图5所示;Coleophoma sp.-ΔmcfH与出发菌株Coleophoma sp.MEFC009相比,化合物1,2,3消失,相应的出现了化合物4。
通过半制备液相色谱(HITACHI Primaide)对化合物4进行分离纯化。制备方法:流动相为A:0.05%(体积比)三氟乙酸水溶液,流动相B:0.05%(体积比)三氟乙酸乙腈溶液,流速为2ml/min,紫外检测波长:210nm,30℃;等度进行洗脱,流动相B占流动相的体积为55%,总洗脱时间为20min。并通过液质联用(LC-MS)和核磁共振(NMR)对化合物4进行分析。LC-MS分析方法为:Agilent公司1290高效液相色谱,色谱柱为Agilent Zorbax Extend-C18的C18柱(2.1×50mm,1.8μm);流动相总流速为0.6mL/min;流动相A:0.05%(体积比)甲酸水溶液,流动相B:0.05%(体积比)甲酸乙腈溶液,总洗脱时间为7.0min;洗脱条件为:梯度洗脱条件:0-1min,流动相B占流动相的体积由5%线性上升到20%,1-6min,流动相B占流动相的体积由20%线性上升到60%,6-7min,流动相B占流动相的体积由60%线性上升到100%。结果如图6所示,可知化合物4的质荷比[M+H]
实施例5.过表达P450酶McfF重组菌株的构建
5.1过表达P450酶McfF表达盒构建
以质粒pXH2-1为模板,利用引物PgpdAt-F(5’-ccctgggttcgcaaagataattggttacactctgggaggatcc-3’)和PgpdAt-R(5’-gttgtgatgattgatgagttg-3’)进行PCR扩增,获得大小约为0.7kb的启动子片段PgpdAt;以Coleophoma sp.MEFC009的基因组为模板,利用引物mcfF-F(5’-caactcatcaatcatcacaacATGCTTTCAGACACGACGGC-3’)和mcfF-R(5’-gatttcagtaacgttaagtggCTATTCCGTCCGCCTTCTTA-3’)进行PCR扩增,获得大小约为1.7kb大小的片段mcfF;以质粒pXH2-1为模板,利用引物TtrpC-F(5’-ccacttaacgttactgaaat-3’)和TtrpC-R(5’-tacctctaaacaagtgtacc-3’)为引物进行PCR扩增,获得大小约为0.7kb大小的终止子片段TtrpC;通过融合PCR,将片段PgpdAt,mcfF和TtrpC进行融合,得到表达盒PgpdAt-mcfF-TtrpC。以质粒pXH2-1为模板,利用引物hph-F(5’-ttcgggatcgcaagcgtaaag-3’)和hph-R(5’-caattatctttgcgaacccagg-3’)进行PCR扩增,获得大小约为2.2kb的潮霉素抗性筛选片段hph。
5.2共转化构建P450酶McfF过表达的重组菌株
以Coleophoma sp.MEFC009为出发菌,首先从PDA平板上取少量菌丝,用手持匀浆器破碎,取1ml种子液接种到50ml的种子培养基,于250ml的三角瓶中,220rpm,25℃进行摇床培养。2天后,离心收集菌丝。5000rpm,4℃,5min。将菌丝体再次用匀浆器破碎,取0.5ml-2ml种子液接种到50ml的种子培养基,相同的条件下培养1天,将培养基与菌丝体一起倒入50ml的无菌离心管中,5000rpm,离心收集菌丝。用0.6M MgSO
向140μl上述原生质体悬液中加入PgpdAt-mcfF-TtrpC片段和hph片段,再加入50μl的PSTC,轻轻混匀,冰浴30min。加入1ml PSTC,混匀后室温放置20min;然后与10ml顶层琼脂混合后倾注于3块再生筛选培养基平板PDA-SH上,在30℃、黑暗条件下培养5-7天,得到转化子。
从转化筛选平板上挑取具有潮霉素抗性转化子转移至PDA-H上,在25℃培养5-7天进行传代培养,连续传代3次。选取23个转化子进行分离纯化,对纯化后的转化子基因组利用引物PgpdAt-F(5’-ccctgggttcgcaaagataattggttacactctgggaggatcc-3’)和TtrpC-R(5’-tacctctaaacaagtgtacc-3’)进行PCR验证,能扩增出大小约为3.2kb的条带为阳性转化子,如图8所示;在23个转化子中,除了转化子21#,其他的转化子都为阳性转化子,基因组上整合了表达元件PgpdAt-mcfF-TtrpC。
实施例6.过表达P450酶McfF重组菌株的发酵验证
将实施例5中得到的过表达mcfF的工程菌株和对照菌株Coleophoma sp.MEFC009接种于PDA固体平板上,25℃培养5-7天。挑取少量的菌丝,利用核酸提取仪(
HPLC分析方法为:液相色谱柱为Agilent C-18反向柱883975-902(4.6×150mm,5μm);流动相为A:0.05%(体积比)三氟乙酸水溶液,流动相B:0.05%(体积比)三氟乙酸乙腈溶液,流速为1ml/min,紫外检测波长:210nm,30℃,总洗脱时间为37min。梯度洗脱条件:0-5min,流动相B占流动相的体积由5%线性上升到24%,5-35min,流动相B占流动相的体积由24%线性上升到62%,35-37min,流动相B占流动相的体积由62%线性上升到100%。HPLC分析结果如图9所示,从中可以看出,与野生型Coleophoma sp.MEFC009相比,工程菌株Coleophoma sp.::mcfF中化合物FR901379的产量上升,副产物WF11899B的产量降低。如图10所示,过表达mcfF的工程菌株中FR901379的产量高于对照菌株Coleophoma sp.MEFC009,过表达mcfF的工程菌株中FR901379的产量为706mg/L,与对照菌株Coleophoma sp.MEFC009相比提高了130%。除了鞘茎点霉(Coleophoma sp.)MEFC009外,菌株Coleophoma empetriF-11899同样可以产生FR901379。我们同样在C.empetri F-11899中过表达mcfF的同源基因BAN91490.1(Sequence ID in NCBI),FR901379的产量与对照菌株C.empetri F-11899相比提高了100%。
实施例7.过表达P450酶McfH重组菌株的构建
以Coleophoma sp.MEFC009的基因组为模板,利用引物mcfH-F(5’-caactcatcaatcatcacaacATGGTTCCATCAATGATCTC-3’)和mcfH-R(5’-gatttcagtaacgttaagtggTCACAGGGCTACTTTCGATC-3’)进行PCR扩增,获得大小约为1.7kb大小的片段mcfH;利用融合PCR,将实施例5中的片段PgpdAt,TtrpC和片段mcfH进行融合,得到表达盒PgpdAt-mcfH-TtrpC。
以Coleophoma sp.MEFC009为出发菌,首先从PDA平板上取少量菌丝,用手持匀浆器破碎,取1ml种子液接种到50ml的种子培养基,于250ml的三角瓶中,220rpm,25℃进行摇床培养。2天后,离心收集菌丝。5000rpm,4℃,5min。将菌丝体再次用匀浆器破碎,取0.5ml-2ml种子液接种到50ml的种子培养基,相同的条件下培养1天,将培养基与菌丝体一起倒入50ml的无菌离心管中,5000rpm,离心收集菌丝。用0.6M MgSO
向140μl上述原生质体悬液中加入PgpdAt-mcfH-TtrpC片段和hph片段,再加入50μl的PSTC,轻轻混匀,冰浴30min。加入1ml PSTC,混匀后室温放置20min;然后与10ml顶层琼脂混合后倾注于3块再生筛选培养基平板PDA-SH上,在30℃、黑暗条件下培养5-7天,得到转化子。
从转化筛选平板上挑取具有潮霉素抗性转化子转移至PDA-H上,在25℃培养5-7天进行传代培养,连续传代3次。选取23个转化子进行分离纯化,对纯化后的转化子基因组利用引物PgpdAt-F(5’-ccctgggttcgcaaagataattggttacactctgggaggatcc-3’)和TtrpC-R(5’-tacctctaaacaagtgtacc-3’)进行PCR验证,能扩增出大小约为3.2kb的条带为阳性转化子,如图11所示;这23个转化子中都为阳性转化子,基因组上整合了表达元件PgpdAt-mcfH-TtrpC。
实施例8.过表达P450酶McfH重组菌株的发酵验证
将实施例7中的过表达P450酶McfH的工程菌株和对照菌株Coleophomasp.MEFC009接种于PDA固体平板上,25℃培养5-7天。挑取少量的菌丝,利用核酸提取仪(
HPLC分析方法为:液相色谱柱为Agilent C-18反向柱883975-902(4.6×150mm,5μm);流动相为A:0.05%(体积比)三氟乙酸水溶液,流动相B:0.05%(体积比)三氟乙酸乙腈溶液,流速为1ml/min,紫外检测波长:210nm,30℃,总洗脱时间为37min。梯度洗脱条件:0-5min,流动相B占流动相的体积由5%线性上升到24%,5-35min,流动相B占流动相的体积由24%线性上升到62%,35-37min,流动相B占流动相的体积由62%线性上升到100%。HPLC分析结果如图12所示,从中可以看出,与野生型Coleophoma sp.MEFC009相比,工程菌株Coleophoma sp.::mcfH中化合物FR901379的产量上升,副产物WF11899C积累减少,几乎不再产生。如图13所示,过表达mcfH的工程菌株中FR901379的产量比对照菌株Coleophomasp.MEFC009要高,过表达mcfH的工程菌株中FR901379的产量为436mg/L,比对照菌株Coleophoma sp.MEFC009提高了44%。我们同样在C.empetri F-11899中过表达mcfH的同源基因BAN91489.1(Sequence ID in NCBI),FR901379的产量与对照菌株C.empetri F-11899相比提高了32.8%。
实施例9.同时过表达P450酶McfF和McfH重组菌株的构建
以Coleophoma sp.MEFC009为出发菌,首先从PDA平板上取少量菌丝,用手持匀浆器破碎,取1ml种子液接种到50ml的种子培养基,于250ml的三角瓶中,220rpm,25℃进行摇床培养。2天后,离心收集菌丝。5000rpm,4℃,5min。将菌丝体再次用匀浆器破碎,取0.5ml-2ml种子液接种到50ml的种子培养基,相同的条件下培养1天,将培养基与菌丝体一起倒入50ml的无菌离心管中,5000rpm,离心收集菌丝。用0.6M MgSO
将前述实施例中的构建的表达盒PgpdAt-mcfF-TtrpC,PgpdAt-mcfH-TtrpC和hph,添加到200μl上述原生质体悬液,再加入50μl的PSTC,轻轻混匀,冰浴30min。加入1ml PSTC,混匀后室温放置20min;然后与10ml顶层琼脂混合后倾注于3块再生筛选培养基平板PDA-SH上,在30℃、黑暗条件下培养5-7天,得到转化子。
从转化筛选平板上挑取具有潮霉素抗性转化子转移至PDA-H上,在25℃培养5-7天进行传代培养,连续传代3次。选取18个转化子进行分离纯化,对纯化后的转化子基因组分别利用引物PgpdAt-F(5’-ccctgggttcgcaaagataattggttacactctgggaggatcc-3’)和mcfF-R(5’-gatttcagtaacgttaagtggCTATTCCGTCCGCCTTCTTA-3’),PgpdAt-F(5’-ccctgggttcgcaaagataattggttacactctgggaggatcc-3’)和mcfH-R(5’-gatttcagtaacgttaagtggTCACAGGGCTACTTTCGATC-3’)进行PCR验证,能同时扩增出大小约为2.5kb的条带为阳性转化子,如图14所示;这18个转化子中除了转化子1#,16#,17#外,其他的都为阳性转化子,基因组上同时整合了表达元件PgpdAt-mcfF-TtrpC和PgpdAt-mcfH-TtrpC。
实施例10.同时过表达P450酶McfF和McfH重组菌株的发酵验证
将实施例9中同时过表达P450酶McfF和McfH的重组菌株和对照菌株Coleophomasp.MEFC009接种于PDA固体平板上,25℃培养5-7天。挑取少量的菌丝,利用核酸提取仪(
虽然本发明已以较佳的实施例公开如上,但其并非用以限定本发明,在不脱离本发明精神和范围内,本领域技术人员都可以在此基础上做出各种改动与变型,因此,本发明的保护范围应该以权利要求书所界定的为准。
SEQUENCE LISTING
<110> 中国科学院青岛生物能源与过程研究所
<120> 一株高产FR901379的菌株及其构建方法与应用
<130> 111
<160> 4
<170> PatentIn version 3.5
<210> 1
<211> 1641
<212> DNA
<213> Artificial Sequence
<220>
<223> mcfF
<400> 1
atgctttcag acacgacggc tcgcatagag cgcatcatta gcgaacagac tttattcagc 60
gctgtactca gtttgtttat gataggcctc atggctcatc tcgtgcttgc acgcttctcg 120
atacataacc aattctggag tgctcaagta tggacaggag ttcgtgcaga atggtttccc 180
aagataagag caaaattccg caccattggc ggtatacgcc aaatgttaag cgatggctat 240
aaatgctttt caaaacagga gagagcattt gttttgccca tgctcggcga gaaaccgtgg 300
ctcgtgctac ctccttccag cattcctgag cttcttgcaa agtctgattc agaggttgat 360
atgcgcatag tccacgagca acagctgcaa catgagtata cgcaaggcgc cctcggtcgc 420
catgttgtcg acgtgcccat tcaatacgat gttatccatc gtcaattgaa tcgaaagctt 480
cctcatttga tagacccctt taatgatgag tttgataaaa gcttcagaaa atactggggc 540
actgatgcat cttatacaga tgtaaaagtc tcagcaacat gcgaaaagat aatcgctcaa 600
gttgcaaacc gaattttcgc aggcccagaa atctgtcgga atgaggattt cttggaacac 660
tctaggctct attcagcagg agtagggagg tgtgcaatca tcctacgtat gcttccacag 720
gtaatacgat cattagttgc accgcttgtt acatacccaa accgcaaaca ccacgatgtt 780
tgcttgagag tctgcctccc agttgtaaga gacaggctac aacgaacctc tgagaagaga 840
ggcaatcttc aatctgagtg ggaaccgccg gtggatatgc ttcaatggat tatcgaagaa 900
gcttttaatc gcaatgaacc aaaagagctt gatgctcacc tgatcactca acggatactt 960
aagcttaact ttgtctccat cgaaactatt cacatgtcta tgacccatgc cattctcgat 1020
ctttaccgct cacctcattc cgagagattt gtagctggcc ttcgtcaaga atgtgatcgc 1080
gtacttgaag cgaataatgg ccaatggacc aagagcgggc tcgatgacct cttgtgcatt 1140
gactcgacaa tccgcgagtc gatgagatac tcgaacgttg gatatatagc actgactcga 1200
atggttgtcg acccgcatgg gacccaattt catgctaatg gcaaaggcaa cagcagtcca 1260
atgtccatac cttctggcat ccgagtctgc gtgcccgccc atgcaatcca cagagaccct 1320
gagttttatt cttccccaca tgaatttcaa gccttccgct ttgctgaagc ttatgaaaag 1380
aatagaaaca taggaaacga atcttacgag gccaagatat ctatcgtcac cacgacagat 1440
aaatttctgc ccttcggcca cggccgccac gcatgcccag gccgcttttt tgccgctcag 1500
atgatgaagc taatgctggt ttacttggtg caaaattacg acgtggaaaa gctatccaca 1560
ggagtagaaa acaaagtcac ggtagggacc gcgaagcctg acagtaattt gagtttaaga 1620
gtaagaaggc ggacggaata g 1641
<210> 2
<211> 546
<212> PRT
<213> Artificial Sequence
<220>
<223> McfF
<400> 2
Met Leu Ser Asp Thr Thr Ala Arg Ile Glu Arg Ile Ile Ser Glu Gln
1 5 1015
Thr Leu Phe Ser Ala Val Leu Ser Leu Phe Met Ile Gly Leu Met Ala
202530
His Leu Val Leu Ala Arg Phe Ser Ile His Asn Gln Phe Trp Ser Ala
354045
Gln Val Trp Thr Gly Val Arg Ala Glu Trp Phe Pro Lys Ile Arg Ala
505560
Lys Phe Arg Thr Ile Gly Gly Ile Arg Gln Met Leu Ser Asp Gly Tyr
65707580
Lys Cys Phe Ser Lys Gln Glu Arg Ala Phe Val Leu Pro Met Leu Gly
859095
Glu Lys Pro Trp Leu Val Leu Pro Pro Ser Ser Ile Pro Glu Leu Leu
100 105 110
Ala Lys Ser Asp Ser Glu Val Asp Met Arg Ile Val His Glu Gln Gln
115 120 125
Leu Gln His Glu Tyr Thr Gln Gly Ala Leu Gly Arg His Val Val Asp
130 135 140
Val Pro Ile Gln Tyr Asp Val Ile His Arg Gln Leu Asn Arg Lys Leu
145 150 155 160
Pro His Leu Ile Asp Pro Phe Asn Asp Glu Phe Asp Lys Ser Phe Arg
165 170 175
Lys Tyr Trp Gly Thr Asp Ala Ser Tyr Thr Asp Val Lys Val Ser Ala
180 185 190
Thr Cys Glu Lys Ile Ile Ala Gln Val Ala Asn Arg Ile Phe Ala Gly
195 200 205
Pro Glu Ile Cys Arg Asn Glu Asp Phe Leu Glu His Ser Arg Leu Tyr
210 215 220
Ser Ala Gly Val Gly Arg Cys Ala Ile Ile Leu Arg Met Leu Pro Gln
225 230 235 240
Val Ile Arg Ser Leu Val Ala Pro Leu Val Thr Tyr Pro Asn Arg Lys
245 250 255
His His Asp Val Cys Leu Arg Val Cys Leu Pro Val Val Arg Asp Arg
260 265 270
Leu Gln Arg Thr Ser Glu Lys Arg Gly Asn Leu Gln Ser Glu Trp Glu
275 280 285
Pro Pro Val Asp Met Leu Gln Trp Ile Ile Glu Glu Ala Phe Asn Arg
290 295 300
Asn Glu Pro Lys Glu Leu Asp Ala His Leu Ile Thr Gln Arg Ile Leu
305 310 315 320
Lys Leu Asn Phe Val Ser Ile Glu Thr Ile His Met Ser Met Thr His
325 330 335
Ala Ile Leu Asp Leu Tyr Arg Ser Pro His Ser Glu Arg Phe Val Ala
340 345 350
Gly Leu Arg Gln Glu Cys Asp Arg Val Leu Glu Ala Asn Asn Gly Gln
355 360 365
Trp Thr Lys Ser Gly Leu Asp Asp Leu Leu Cys Ile Asp Ser Thr Ile
370 375 380
Arg Glu Ser Met Arg Tyr Ser Asn Val Gly Tyr Ile Ala Leu Thr Arg
385 390 395 400
Met Val Val Asp Pro His Gly Thr Gln Phe His Ala Asn Gly Lys Gly
405 410 415
Asn Ser Ser Pro Met Ser Ile Pro Ser Gly Ile Arg Val Cys Val Pro
420 425 430
Ala His Ala Ile His Arg Asp Pro Glu Phe Tyr Ser Ser Pro His Glu
435 440 445
Phe Gln Ala Phe Arg Phe Ala Glu Ala Tyr Glu Lys Asn Arg Asn Ile
450 455 460
Gly Asn Glu Ser Tyr Glu Ala Lys Ile Ser Ile Val Thr Thr Thr Asp
465 470 475 480
Lys Phe Leu Pro Phe Gly His Gly Arg His Ala Cys Pro Gly Arg Phe
485 490 495
Phe Ala Ala Gln Met Met Lys Leu Met Leu Val Tyr Leu Val Gln Asn
500 505 510
Tyr Asp Val Glu Lys Leu Ser Thr Gly Val Glu Asn Lys Val Thr Val
515 520 525
Gly Thr Ala Lys Pro Asp Ser Asn Leu Ser Leu Arg Val Arg Arg Arg
530 535 540
Thr Glu
545
<210> 3
<211> 1593
<212> DNA
<213> Artificial Sequence
<220>
<223> mcfH
<400> 3
atggttccat caatgatctc ggcagtggtc ccaataatca cctccacaaa tctggttttt 60
tatggagcaa ctggacttgt cctatttgct atcctcgcct actcacttaa cagattgaca 120
acttgggaat actcgatccc aaacgaagta caatggattg atcgtcgcaa agagtctttc 180
tcttatcttc gcgcaaaagc ccgatcgctt gttagaaaca aagaaaatgt gcttgaagct 240
tatttcaagt tcaacaaact tggtaatgca gcagcatgtg cggttgcttt cggtcgtcca 300
ttgttgcttc tgccgcctac tttcatccgc tggattgttg atcaacctga atctaccatc 360
agtttggatc cgatacacga tgacttccat gcatttgttg gagatggtct gatcggtgat 420
cataccgtgc aggagctgtt acgccgagag ttgactctta acctggacaa gttgacgcct 480
atgatcaacg aagagattgt ttccgcttta gatgacgtat tgggaaactc tactgaatgg 540
aaaaccactt ctcttgctga tgatttgaaa acaatcattg cgcggacctc taacagagta 600
tttatgggca aagacttaag ccaaaacgaa gactacatca acactgccaa agggttggca 660
atggttgtta tgccagaaac cgtgctccaa gatctcgttc cacaaattct gaaaggacca 720
ctttcaaaga taactagagt gttcaacaac atctatgcga tgaaaagaat cacgtcacat 780
ttgcttcctg tagtaaggca acggtatatt gatgttaaga acgtattcga tggttctgga 840
gacaaagaac agcttcctga caacttgctg acatggatgg tgcaaaagtc aattcgtcga 900
ggagagtcta cagcaaatat cgataaagta ttggtagcgc gtatcggaat ggttaacctg 960
gcagctatcg aaactaccac tgctgccatg actaaaagca tcctagactt ggtcactgtg 1020
ggtgttgaag gaggcttcct ggaagctgtt caggaagaag cattggccgt gttggaagga 1080
tgcaactatc aaccagaaaa gagtgatgtt tcgaaattga atttcaccga aaatgcaatc 1140
aaggagtcac tccgccttca agtcgccttt ccaggtctca tgcgccaagt tgtcagccca 1200
aaaggtgtta cgctagacaa tggtctacac gtgccttacg gtacccgtgt tggtgtatct 1260
gcagctggaa tccatgttga cgactcggtc tatgaaaacg ccacgaccta caatcctggc 1320
agattcatgg tcatagattt agacgctcga ggcaaaccta atccgttatg gaaaggtact 1380
gagaactacc tagcctttgg ccttgggaga cgatcgtgtc ccggccggtg gtatgtgacc 1440
gatcagttaa aactcacact tgcccatatc ttctcgaagt acgagattaa atttgagaag 1500
gctgcaaaga agacaagtgc gctaagaaaa atcctacctg gtgctcctca agatcacgtt 1560
atgattcgac gtagatcgaa agtagccctg tga 1593
<210> 4
<211> 530
<212> PRT
<213> Artificial Sequence
<220>
<223> McfH
<400> 4
Met Val Pro Ser Met Ile Ser Ala Val Val Pro Ile Ile Thr Ser Thr
1 5 1015
Asn Leu Val Phe Tyr Gly Ala Thr Gly Leu Val Leu Phe Ala Ile Leu
202530
Ala Tyr Ser Leu Asn Arg Leu Thr Thr Trp Glu Tyr Ser Ile Pro Asn
354045
Glu Val Gln Trp Ile Asp Arg Arg Lys Glu Ser Phe Ser Tyr Leu Arg
505560
Ala Lys Ala Arg Ser Leu Val Arg Asn Lys Glu Asn Val Leu Glu Ala
65707580
Tyr Phe Lys Phe Asn Lys Leu Gly Asn Ala Ala Ala Cys Ala Val Ala
859095
Phe Gly Arg Pro Leu Leu Leu Leu Pro Pro Thr Phe Ile Arg Trp Ile
100 105 110
Val Asp Gln Pro Glu Ser Thr Ile Ser Leu Asp Pro Ile His Asp Asp
115 120 125
Phe His Ala Phe Val Gly Asp Gly Leu Ile Gly Asp His Thr Val Gln
130 135 140
Glu Leu Leu Arg Arg Glu Leu Thr Leu Asn Leu Asp Lys Leu Thr Pro
145 150 155 160
Met Ile Asn Glu Glu Ile Val Ser Ala Leu Asp Asp Val Leu Gly Asn
165 170 175
Ser Thr Glu Trp Lys Thr Thr Ser Leu Ala Asp Asp Leu Lys Thr Ile
180 185 190
Ile Ala Arg Thr Ser Asn Arg Val Phe Met Gly Lys Asp Leu Ser Gln
195 200 205
Asn Glu Asp Tyr Ile Asn Thr Ala Lys Gly Leu Ala Met Val Val Met
210 215 220
Pro Glu Thr Val Leu Gln Asp Leu Val Pro Gln Ile Leu Lys Gly Pro
225 230 235 240
Leu Ser Lys Ile Thr Arg Val Phe Asn Asn Ile Tyr Ala Met Lys Arg
245 250 255
Ile Thr Ser His Leu Leu Pro Val Val Arg Gln Arg Tyr Ile Asp Val
260 265 270
Lys Asn Val Phe Asp Gly Ser Gly Asp Lys Glu Gln Leu Pro Asp Asn
275 280 285
Leu Leu Thr Trp Met Val Gln Lys Ser Ile Arg Arg Gly Glu Ser Thr
290 295 300
Ala Asn Ile Asp Lys Val Leu Val Ala Arg Ile Gly Met Val Asn Leu
305 310 315 320
Ala Ala Ile Glu Thr Thr Thr Ala Ala Met Thr Lys Ser Ile Leu Asp
325 330 335
Leu Val Thr Val Gly Val Glu Gly Gly Phe Leu Glu Ala Val Gln Glu
340 345 350
Glu Ala Leu Ala Val Leu Glu Gly Cys Asn Tyr Gln Pro Glu Lys Ser
355 360 365
Asp Val Ser Lys Leu Asn Phe Thr Glu Asn Ala Ile Lys Glu Ser Leu
370 375 380
Arg Leu Gln Val Ala Phe Pro Gly Leu Met Arg Gln Val Val Ser Pro
385 390 395 400
Lys Gly Val Thr Leu Asp Asn Gly Leu His Val Pro Tyr Gly Thr Arg
405 410 415
Val Gly Val Ser Ala Ala Gly Ile His Val Asp Asp Ser Val Tyr Glu
420 425 430
Asn Ala Thr Thr Tyr Asn Pro Gly Arg Phe Met Val Ile Asp Leu Asp
435 440 445
Ala Arg Gly Lys Pro Asn Pro Leu Trp Lys Gly Thr Glu Asn Tyr Leu
450 455 460
Ala Phe Gly Leu Gly Arg Arg Ser Cys Pro Gly Arg Trp Tyr Val Thr
465 470 475 480
Asp Gln Leu Lys Leu Thr Leu Ala His Ile Phe Ser Lys Tyr Glu Ile
485 490 495
Lys Phe Glu Lys Ala Ala Lys Lys Thr Ser Ala Leu Arg Lys Ile Leu
500 505 510
Pro Gly Ala Pro Gln Asp His Val Met Ile Arg Arg Arg Ser Lys Val
515 520 525
Ala Leu
530
- 一株高产甘露醇的肠膜明串珠菌突变菌株及其应用方法
- 一株高产甘露醇的肠膜明串珠菌突变菌株及其应用方法
- 一株硫脲降解菌株及应用该菌株处理含硫脲废水的方法
- 一株米卡芬净前体FR901379高产菌株及其应用
- 一株米卡芬净前体FR901379高产菌株及其应用
