一种基于高通量测序技术检测拷贝数变异的文库构建方法
文献发布时间:2023-06-19 19:30:30
技术领域
本发明涉及文库构建技术领域,具体为一种基于高通量测序技术检测拷贝数变异的文库构建方法。
背景技术
拷贝数变异(CNV)检测类型包括:非整倍体、重复、缺失、嵌合、杂合性丢失(lossof heterozygosity,LOH)、单亲二体(uniparentaldisomy,UPD)、单倍体、三倍体和多倍体等。染色体非整倍体是指体外受精-胚胎移植周期中发生胚胎染色体非整倍体(aneuploidy)异常,表现为在正常二倍体基础上,染色体数目增加或减少一条甚至多条;重复是指染色体上增加了相同的某个区段因而引起变异的现象;缺失是指染色体某一区段丢失的现象;嵌合体在遗传学上用以指不同遗传性状嵌合或混杂表现的个体;杂合性丢失(LOH)是指一对同源染色体上特定位点的等位基因,一侧带有突变(有害),一侧正常,由于某种原因,正常的一侧对应序列发生缺失或突变,致使该基因座位变为半合或纯合;单亲二体(UPD)指来自父母一方的染色体片段被另一方的同源部分取代,或一个个体的两条同源染色体都来自同一亲体;单倍体是指体细胞含有一套染色体组,三倍体则是指每一个细胞中具有三套完整的染色体组。基因组拷贝数变异的检测可应用到组织检测如肿瘤组织、羊水、流产物组织,液体活检如血液、尿液的细胞、游离核酸,单细胞领域如胚胎植入前遗传学诊断、胚胎植入遗传学筛查、癌症患者血液中游离的循环肿瘤细胞、孕妇外周血中游离的胎儿细胞、干细胞、单细胞或几个细胞的微生物。
目前基因组拷贝数变异检测的主要方法有:比较基因组杂交,荧光定量PCR,荧光原位杂交,多重连接探针扩增技术,高通量测序。其中,比较基因组杂交分辨率比较低,Mb级,通量低,成本高;荧光定量PCR同样通量低,成本高,一次只能测一个拷贝数变异;荧光原位杂交,只针对特定位置,分辨率低,探针杂交效率不稳定;多重连接探针扩增技术,操作复杂,通量低,成本高,覆盖度小,易造成PCR污染;鉴于此,我们提出了一种基于高通量测序技术检测拷贝数变异的文库构建方法。
发明内容
针对现有技术的不足,本发明提供了一种基于高通量测序技术检测拷贝数变异的文库构建方法,解决了上述背景技术提到的问题。
为实现以上目的,本发明通过以下技术方案予以实现:一种基于高通量测序技术检测拷贝数变异的文库构建方法,所述构建方法包括以下步骤:
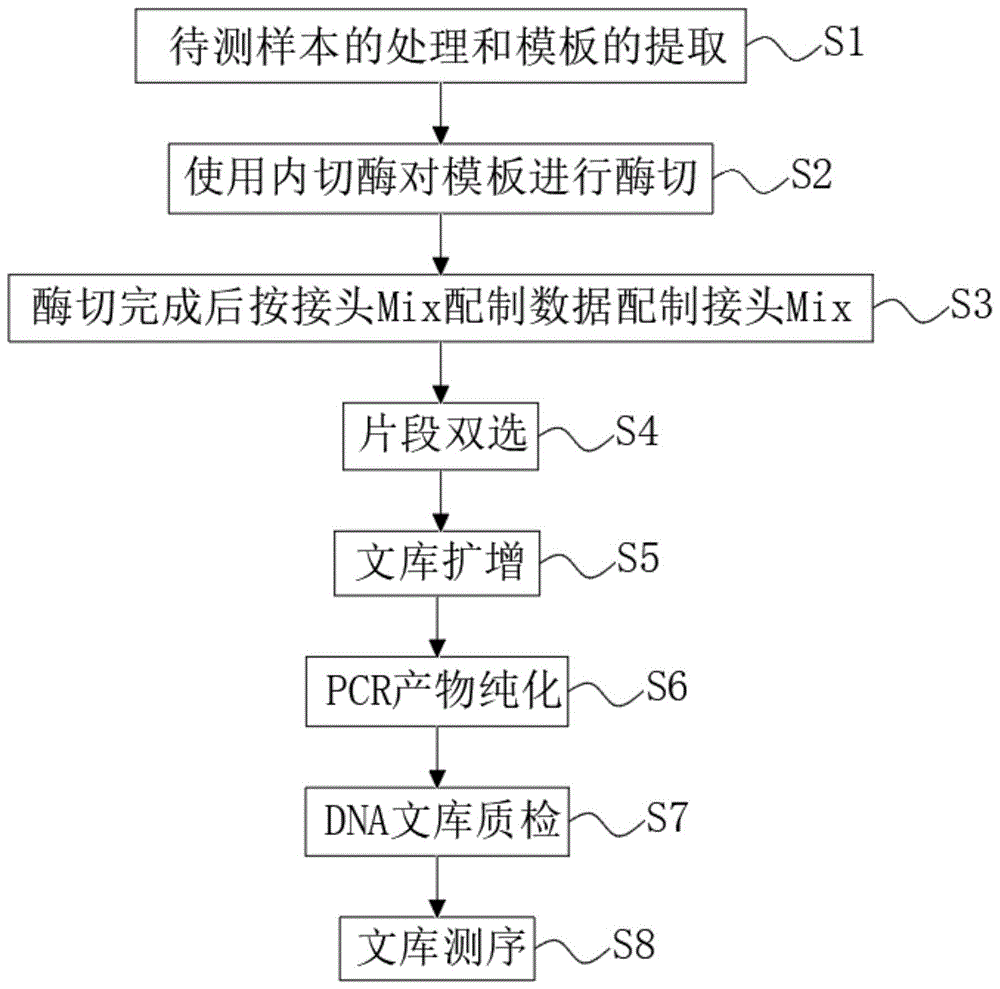
S1、待测样本的处理和模板的提取;
S2、使用内切酶对模板进行酶切;
S3、酶切完成后按接头Mix配制数据配制接头Mix;
S4、片段双选;
S5、文库扩增;
S6、PCR产物纯化;
S7、DNA文库质检;
S8、文库测序。
可选的,所述S1进一步的包括以下步骤:
样品适用范围包括手术切除的新鲜病理组织,甲醛固定石蜡包埋病理组织,石蜡切片及血液等标本;
蜡块样品切成5~8μM切片,取5片,或已经制成的5~8μM切片取5片,经过脱蜡剂脱蜡后,使用石蜡包埋DNA提取试剂盒提取基因组DNA;
新鲜病理组织使用组织DNA提取试剂盒提取基因组DNA;
血液样本使用提取试剂盒提取基因组DNA;
所提DNA溶于1×TE,使用Qubit 4.0对提取好的DNA进行定量,确认模板浓度后使用1×TE溶液调整DNA浓度到10ng/μL作为PCR扩增的模板。
可选的,所述S4进一步的包括以下步骤:
将连接产物转移至1.5mL离心管中,加入70μL无核酸酶水补足至100μL,加入60μLDNA纯化磁珠,混匀后室温放置5分钟;
放置到磁力架上,然后磁力架上放置4分钟至液体澄清,转移上清至新的1.5mL离心管中;
加入18μL DNA纯化磁珠,混匀后室温放置5分钟,放置到磁力架上,等液体清亮,去上清,用200μL的80%酒精清洗;
室温干燥后用22μL Low TE提取DNA,取20μL溶液于新的0.2mL PCR管中。
可选的,所述S5进一步的包括以下步骤:
S51、按照配制表配制反应体系;
S52、按扩增程序数据扩增程序进行PCR扩增。
可选的,所述S6进一步的包括以下步骤:
S61、向PCR产物管中加入50uL磁珠,用移液器吹打混匀,室温孵育2min。
S62、将孵育完成的产物管放置到磁力架上进行磁珠吸附,待溶液澄清后,小心吸取上清,弃上清保留磁珠。
S63、向上步PCR管中加入200uL80%的乙醇(当日配制),用磁力架快速在PCR管不同的面吸附2-3次,充分洗涤磁珠,静置15s后,用移液器小心吸取上清,弃上清保留磁珠。
S64、重复步骤S63一次。
S65、将含有磁珠的PCR管置于磁力架上室温静置,待到管中乙醇完全挥发后,向PCR管中加入20uL无核酸酶水,充分混匀洗涤磁珠后静置2min。
S66、将孵育完成的产物管放置到磁力架上进行磁珠吸附,待溶液澄清后,小心吸取上清转移至新的PCR管中,即为洗脱得到的DNA文库,弃去磁珠。
可选的,所述S7进一步的包括:使用Qubit 4.0对文库浓度进行测定,使用labchip对文库的片段大小进行测定。
可选的,所述S8进一步的包括:
测序使用Miseq测序仪进行测序,使用V2芯片,测序读长PE150;
测序完成后得到目标序列信息,即fastq文件;
然后将上述获得的fastq文件经生信分析处理,通过与野生型序列信息进行比对得到基因突变信息。
本发明提供了一种基于高通量测序技术检测拷贝数变异的文库构建方法。具备以下有益效果:
(1)、该基于高通量测序技术检测拷贝数变异的文库构建方法,可高效特异捕获全基因组SNP用于CNV分析,提高CNV检测类型和准确性;CNV检测方法与CNV-Seq相比,可以检测CNV-seq能够检测的非整倍体、重复、缺失,也能检测CNV-seq技术无法检测的单倍体、三倍体、多倍体和LOH等。
(2)、该基于高通量测序技术检测拷贝数变异的文库构建方法,CNV检测方法对全基因组部分代表性区域进行高深度测序,相比于全基因组测序技术,显著降低测序成本(降低80%)。
附图说明
图1为本发明的流程结构示意图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
请参阅图1,本发明提供一种技术方案:一种基于高通量测序技术检测拷贝数变异的文库构建方法,构建方法包括以下步骤:
S1、待测样本的处理和模板的提取,待测样本的处理和模板的提取包括以下步骤:
样品适用范围包括手术切除的新鲜病理组织,甲醛固定石蜡包埋病理组织,石蜡切片及血液等标本;
蜡块样品切成5~8μM切片,取5片,或已经制成的5~8μM切片取5片,经过脱蜡剂脱蜡后,使用石蜡包埋DNA提取试剂盒提取基因组DNA,具体步骤按试剂操作说明;
新鲜病理组织使用组织DNA提取试剂盒提取基因组DNA,具体步骤按试剂盒操作说明;
血液样本使用提取试剂盒提取基因组DNA,具体步骤按试剂盒操作说明;
所提DNA溶于1×TE,使用Qubit 4.0对提取好的DNA进行定量,确认模板浓度后使用1×TE溶液调整DNA浓度到10ng/μL作为PCR扩增的模板。
S2、使用内切酶对模板进行酶切,酶切反应体系配制如下表所示
体系配制完成后混匀离心后按反应程序进行酶切反应,反应程序如下表所示:
S3、酶切完成后按接头Mix配制数据配制接头Mix,加入至酶切产物之中,接头Mix配制数如下表所示:
体系配制完成后混匀离心后按反应程序进行连接反应,反应程序如下表所示:
S4、片段双选
将连接产物转移至1.5mL离心管中,加入70μL无核酸酶水补足至100μL,加入60μLDNA纯化磁珠,混匀后室温放置5分钟;
放置到磁力架上,然后磁力架上放置4分钟至液体澄清,转移上清至新的1.5mL离心管中;
加入18μL DNA纯化磁珠,混匀后室温放置5分钟,放置到磁力架上,等液体清亮,去上清,用200μL的80%酒精清洗;
室温干燥后用22μL Low TE提取DNA,取20μL溶液于新的0.2mL PCR管中。
S5、文库扩增,文库扩增包括以下步骤:
S51、按照配制表配制反应体系,配制表数据如下:
S52、按扩增程序数据扩增程序进行PCR扩增,扩增程序数据如下表所示:
S6、PCR产物纯化,PCR产物纯化包括以下步骤:
S61、向PCR产物管中加入50uL磁珠,用移液器吹打混匀,室温孵育2min。
S62、将孵育完成的产物管放置到磁力架上进行磁珠吸附,待溶液澄清后,小心吸取上清,弃上清保留磁珠。
S63、向上步PCR管中加入200uL80%的乙醇(当日配制),用磁力架快速在PCR管不同的面吸附2-3次,充分洗涤磁珠,静置15s后,用移液器小心吸取上清,弃上清保留磁珠。
S64、重复步骤S63一次。
S65、将含有磁珠的PCR管置于磁力架上室温静置,待到管中乙醇完全挥发后,向PCR管中加入20uL无核酸酶水,充分混匀洗涤磁珠后静置2min。
S66、将孵育完成的产物管放置到磁力架上进行磁珠吸附,待溶液澄清后,小心吸取上清转移至新的PCR管中,即为洗脱得到的DNA文库,弃去磁珠。
S7、DNA文库质检:
使用Qubit 4.0对文库浓度进行测定,使用labchip对文库的片段大小进行测定。
S8、文库测序:
测序使用Miseq测序仪进行测序,使用V2芯片,测序读长PE150。测序完成后得到目标序列信息,即fastq文件。然后将上述获得的fastq文件经生信分析处理,通过与野生型序列信息进行比对得到基因突变信息。
本文库构建方法的原理是基于高通量测序技术,采用核酸内切酶对样本基因组DNA进行切割得到酶切产物,将酶切物与测序接头连接,得到连接产物;对连接产物进行PCR扩增,得到测序文库。
本发明中,针对多种CNV变异类型的检测需求,开发出一套适合CNV检测的文库构建流程,充分分析CNV对均一性和重复性的需求,控制核酸内切酶的酶切识别位点,能够在人类全基因组上稳定均匀的覆盖,将基因组采用核酸内切酶进行打断,捕获固定片段范围的DNA序列构建文库,能够使用较少的数据量(测序成本)对全基因组部分代表性区域进行高深度测序,获得高密度和覆盖均一的足量能进行CNV分析的SNP位点。
本发明提供了一种基于高通量测序技术检测拷贝数变异的引物,包括特异修饰性扩增引物和接头扩增引物,特异修饰性扩增引物的序列如SEQ ID NO.1~SEQ ID NO.4所示,接头扩增引物的序列如SEQ ID NO.5~SEQ ID NO.6所示。
该基于高通量测序技术检测拷贝数变异的文库构建方法,可高效特异捕获全基因组SNP用于CNV分析,提高CNV检测类型和准确性;CNV检测方法与CNV-Seq相比,可以检测CNV-seq能够检测的非整倍体、重复、缺失,也能检测CNV-seq技术无法检测的单倍体、三倍体、多倍体和LOH等。
尽管已经示出和描述了本发明的实施例,对于本领域的普通技术人员而言,可以理解在不脱离本发明的原理和精神的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由所附权利要求及其等同物限定。
