一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法
文献发布时间:2023-06-19 11:22:42
技术领域
本发明涉及生物技术领域,尤其涉及一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法。
背景技术
牛角是牛科动物特有的皮肤衍生物结构,承担负重、攻击和防卫功能,在现代规模化、高密度养殖环境下,牛角的存在则更具有攻击性,不仅容易造成牛只之间的意外伤害(乳房损伤、流产等),降低母牛的终身产奶效益,还会增加饲养人员受伤的风险。为有效缓解这一问题,规模化牧场已将人工去角作为常规工作,目前常见的去角方法包括物理去角(割角,高温灼烧)和化学去角(化学腐蚀)等。但是上述方法不仅会给犊牛带来严重的应激反应,增加犊牛感染细菌或病毒的概率,而且还会影响犊牛的育成,影响奶牛和肉牛养殖的经济效益。不仅如此,随着我国奶肉牛存栏量、规模化的不断提高,牧场的牛角去除工作将更加繁重,因此无角牛的培育工作愈发重要。
牛角性状属于质量形状,可分为有角性状、无角性状(polled)两种,同时也存在介于有角牛和无角牛之间的突变类型畸形角(scurs)。自然界中一直存在着自然突变的无角牛,随着研究的深入,人们将无角性状定位在1号染色体。近年来,科学家对自然突变的无角牛进行了测序分析,研究发现不同品种无角牛的无角性状主要起源于3种遗传变异,分别是是202 bp片段的重复(P
目前,CRISPR/Cas9基因编辑技术广泛应用于农业动物新品种培育和动物疾病模型的建立。2020年,谷明娟等利用CRISPR/Cas9 技术编辑了蒙古牛Pc位点,获得了4 个杂合突变的定点整合阳性单克隆细胞株(谷明娟等.蒙古牛无角POLLED位点的定点编辑[J].农业生物技术学报,2020,28(02):242-250.)。然而,利用杂合突变体培育后代会发生等位基因分离,导致无法确保后代无角性状的稳定遗传,因此,需要研究开发出一种Pc位点纯合突变体作为无角牛的育种材料。
发明内容
为了解决现有技术中存在的问题,本发明的目的是提供一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法
为了实现本发明目的,本发明的技术方案如下:
第一方面,本发明提供一种无角Pc位点定点整合的纯合无角基因型细胞的制备方法,利用连接有sgRNA的pX458载体与含有Pc片段和靶位点两侧约800bp的同源序列左臂和右臂的同源重组片段载体,将sgRNA和同源重组片段共转染至荷斯坦种公牛的离体成纤维细胞,对成纤维细胞基因组进行Pc片段插入,筛选获得发生双等位Pc位点插入的纯合无角基因型体细胞株;
所述sgRNA是由如下单链寡核苷酸退火合成的带有粘性末端的双链DNA分子:
sgRNA1-F:caccgGTCTATCCCAAAAGTGTGGG;
sgRNA1-R:aaacCCCACACTTTTGGGATAGACc;
或由如下单链寡核苷酸退火合成的带有粘性末端的双链DNA分子:
sgRNA3-F:caccgATAAGAGATAGAAATAGAAG;
sgRNA3-R:aacCTTCTATTTCTATCTCTTATc。
优选采用以上sgRNA1-F和sgRNA1-R合成的sgRNA。
作为优选,所述同源重组片段的核苷酸序列如SEQ ID NO.1所示。
进一步优选,所述同源重组片段载体的骨架载体为pUC57载体。
进一步地,本发明采用牛耳缘成纤维细胞作为所述荷斯坦种公牛的离体成纤维细胞。试验发现,牛耳缘成纤维细胞相对于现有技术中所使用的具有更高转染效率、甚至更容易出现阳性的胎儿成纤维细胞系,在本发明技术方案中反而可表现出更好的重组效果,并可获得纯合基因型体细胞株。
更进一步地,本发明所述制备方法还包括对所获得的纯合无角基因型体细胞株进行外源基因残留检测,获得无Cas9基因残留的纯合无角基因型体细胞株。
本发明前述制备方法中,所述的筛选包括:利用流式分选筛选GFP阳性细胞,对Pc片段进行PCR扩增;为更好的确保筛选准确性,还可进行基因组测序。
需要说明的是,本发明方法中采用的共转染方法包括但不限于电转染或脂质体转染。
第二方面,本发明提供一种由前述制备方法制备得到的无角Pc位点定点整合的纯合无角基因型体细胞。
以及本发明前述制备方法及所述纯合无角基因型体细胞在制备具有无角Pc位点定点整合的纯合无角基因型基因编辑牛中的应用。
所述应用具体可体现为一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法,以本发明所获得的纯合无角基因型体细胞为核供体细胞,通过体细胞核移植技术获得纯合基因型胚胎并移植,生产的子代即为具有无角Pc位点纯合基因型的荷斯坦牛。
本发明涉及到的原料或试剂均为普通市售产品,涉及到的操作如无特殊说明均为本领域常规操作。
在符合本领域常识的基础上,上述各优选条件,可以相互组合,得到具体实施方式。
本发明的有益效果在于:
本发明以Pc位点为靶位点,筛选获得定点插入效率最佳的sgRNA,并利用Tild-CRISPR/Cas9技术将无角Pc位点整合到有角荷斯坦种公牛耳缘成纤维细胞系基因组中,获得了P
附图说明
此处的附图被并入说明书中并构成本说明书的一部分,示出了符合本发明的实施例,并与说明书一起用于解释本发明的原理。
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,对于本领域普通技术人员而言,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
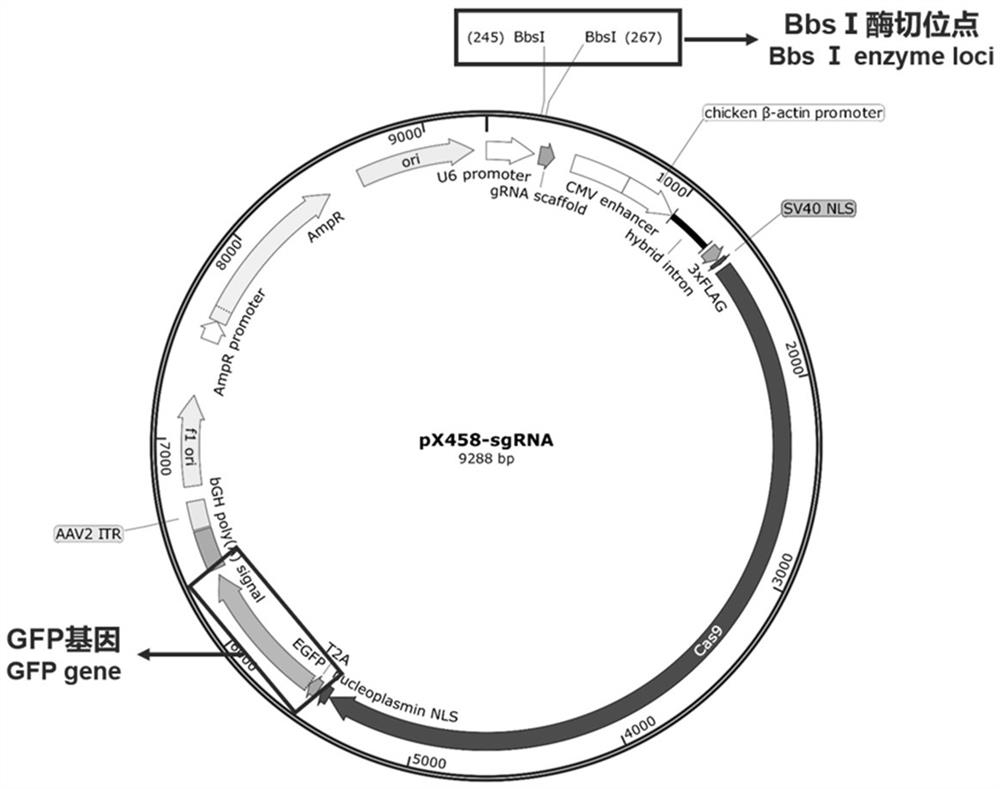
图1为pX458-sgRNA载体模式图。
图2为6个pX458-sgRNA经脂质体转染24小时荧光蛋白表达结果。
图3为采用流式细胞仪分选GFP阳性细胞的结果。
图4为T7E1酶切鉴定sgRNA突变效率的聚丙烯凝胶电泳结果;图中减号为对照,加号为酶切后结果,方框圈出的即为被T7E1切开后所得片段。
图5为sgRNA1和sgRNA3的测序峰图;图中方框圈出的为因发生突变产生的峰。
图6为菌落克隆使用Hind III和BamH I进行双酶切产物的琼脂糖凝胶电泳结果;图中,(A)10个菌落单克隆使用Hind III和BamH I进行双酶切产物琼脂糖凝胶电泳结果,1-10即10个挑选的菌落单克隆,箭头指示的即为所构建的同源重组片段;(B)测序正确的菌落单克隆扩增后使用Hind III和BamH I进行双酶切产物琼脂糖凝胶电泳结果,a为PUC57-LA1-PC-LA2 ;b为使用Hind Ⅲ和BamHⅠ进行双酶产物,箭头指示的即为所构建的同源重组片段。
图7为建立突变细胞系所用组织样品。
图8为显微镜下的牛耳成纤维细胞。
图9为pX458-sgRNA1和pX458-sgRNA3经脂质体转染24小时荧光蛋白表达结果。
图10为T7E1酶切鉴定sgRNA1和sgRNA3突变效率的聚丙烯凝胶电泳结果;图中减号为对照,加号为酶切后结果,方框圈出的即为被T7E1切开后所得片段。
图11为流式细胞仪分选的GFP阳性细胞。
图12为部分鉴定纯合突变细胞系琼脂糖凝胶电泳部分实验结果;图中为1-23、67-112号细胞系琼脂糖凝胶电泳,箭头指示为杂合突变细胞系,星星表示纯合突变细胞系(106号)。
图13为纯合突变细胞系测序结果。
图14为PCR鉴定纯合细胞系(106号)Cas9基因残留的凝胶电泳结果。
具体实施方式
为了能够更清楚地理解本发明的上述目的、特征和优点,下面将对本发明的方案进行进一步描述。需要说明的是,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
除非另有说明,本文中所用的专业与科学术语与本领域熟练人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法或材料也可应用于本发明中。
在下面的描述中阐述了很多具体细节以便于充分理解本发明,但本发明还可以采用其他不同于在此描述的方式来实施;显然,说明书中的实施例只是本发明的一部分实施例,而不是全部的实施例。
下面将结合实施例对本发明的优选实施方式进行详细说明。需要理解的是以下实施例的给出仅是为了起到说明的目的,并不是用于对本发明的范围进行限制。本领域的技术人员在不背离本发明的宗旨和精神的情况下,可以对本发明进行各种修改和替换。
下述实施例中所用的试验材料包括:荷斯坦牛雄性耳成纤维细胞系为本实验室所有,牛耳组织取自北京奶牛中心种公牛,牛只编号信息为11119680-7#和11119683-8#。
下述实施例中所用的试验试剂包括:大肠杆菌Trans 5a感受态细胞购买自全式金;pX458 载体质粒、去内毒素质粒小提试剂盒购买自Omega;细胞组织血液全基因组DNA提取试剂盒购买自天根;PCR产物和胶回收纯化试剂盒购买自Vazyme;Taq Mix购买自金普来;2×PhantaR Max Master Mix(Dye Plus)、Taq DNA聚合酶购买自Neb;限制性内切酶、LipofectamineTM 3000试剂盒、FBS、DMEM购买自Gibco;PBS、0.25% Trypsin-EDTA、DMSO、硅胶购买自Sigma。
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
实施例1 :sgRNA的合成及突变效率的验证
一、sgRNA设计及载体构建
针对靶位点在网站(MIT CRISPR design tool)设计sgRNA,挑选得分排列前6的sgRNA进行合成。合成的sgRNA进行99℃,10 min退火,连接到经由BbsI酶切处理的pX458-sgRNA载体中(图1)(pX458载体可表达绿色荧光蛋白,可用于具有指示cas 9蛋白的表达情况)。随后使用连接产物转染Trans 5α感受态细胞,挑选细菌克隆进行测序。将测序正确的细菌克隆扩大培养,利用去内毒素质粒提取试剂盒进行质粒提取,提取的质粒可用于转染成纤维细胞。
二、T7E1酶切及测序鉴定sgRNA突变效率
将雄性荷斯坦种公牛耳缘成纤维细胞复苏培养,接种到六孔板中进行培养,直至细胞汇合度达到70%以上即可进行脂质体转染处理,使用4μg pX458-sgRNA质粒转染1×10
根据sgRNA靶位点设计的上下游的引物:
PcH-F4:5 ʹ-AATGGCTACCCACTCCAGTATT-3 ʹ;
PcH-R1: 5 ʹ-GGCAGAGATGTTGGTCTTGGCTGT-3 ʹ。
进行PCR扩增,50 μL PCR反应体系如下:
Q5 Buffer(5×) 10 μL,Q5 high GC Buffer 10 μL,dNTP 4 μL,模板(提取获得的耳缘成纤维细胞全基因组) 4 μL,上、下游引物各1 μL,Q5酶 0.5 μL,ddH
反应条件:98℃预变性30 s;98℃变性10 s,60℃退火20 s,72℃延伸30 s,循环33次;72℃ 2 min;4℃保存。
随后使用PCR产物和胶回收纯化试剂盒回收PCR产物。将400 ng产物进行变性及程序性退火,20 μL反应体系如下:产物 400 ng,LA Buffer 1μL,ddH
对退火产物使用T7E1进行酶切处理,25 μL反应体系如下:退火产物 20 μL,Buffer 2 2.5 μL,T7E1 0.5 μL,ddH
本发明针对Pc位点设计了6对引物,引物序列见表1。随后构建pX458-sgRNA载体。待培养牛耳成纤维细胞至汇合度达75%,6个pX458-sgRNA质粒分别转染牛纤维细,24 h后GFP荧光蛋白表达结果如图2所示。在转染72 h后,流式细胞仪检测,结果如图3所示。T7E1酶切处理后的PCR回收产物进行聚丙烯酰氨凝胶电泳,发生突变的sgRNA产物可被T7E1切割成两条带,进而得到pX458-sgRNA的突变效率,T7E1酶切结果如图4所示,结果表明pX458-sgRNA1和pX458-sgRNA3的突变效率较高。为保证实验的准确性,对突变效率较高的pX458-sgRNA1和pX458-sgRNA3进行测序,如果测序结果中有错峰出现则说明发生突变,测序结果如图5所示,结果表明pX458-sgRNA1和pX458-sgRNA3均发生突变,与T7E1酶切产物聚丙烯酰氨凝胶电泳结果相符。
表1 6 对sgRNA序列
实施例2 :同源重组片段的构建与验证
一、构建同源重组片段
本发明使用Tild-CRISPR Cas9技术进行基因编辑,即在构建Pc同源重组片断时在目的片断两侧增加经由PCR扩增获得的约800 bp的同源臂。
针对Pc靶位点、左右同源臂LA1、LA2、Pc片段分别设计引物,引物序列见表2:
表2 构建同源重组片段所需引物序列
使用Q5 PCR反应体系(50 μL)分别扩增LA1、LA2、Pc片段,反应体系如下:Q5Buffer(10×) 10 μL,dNTP 4 μL,酶切后pUC57载体 1 μL,上、下游引物各1 μL(100 mM),Q5酶 0.5 μL,ddH
LA1与Pc片段的反应条件为:98℃预变性30 s;98℃变性10 s,58℃退火20 s,72℃延伸30 s,循环33次;72℃ 2 min;4℃保存。
LA2片段的反应条件为:98℃预变性30 s;98℃变性10 s,58℃退火20 s,72℃延伸40 s,循环33次;72℃ 2 min;4℃保存。
随后使用BamHⅠ/HindⅢ双酶切pUC57载体,反应体系如下:cutsmart Buffer 10 μL,pUC57载体 10 μg,BamH Ⅰ和Hind Ⅲ各5 μL,ddH
之后,将扩增获得的左、右同源臂以及Pc片段共同连接到经由BamHⅠ和Hind Ⅲ酶切后的pUC57载体上,10 μL反应体系如下:LA1 0.25 μL,LA2 0.25 μL,Pc 0.3 μL,pUC57载体 200 ng,A红embly Mix 5 μL,ddH
所构建的同源打靶载体包括Pc位点及两侧各约800 bp同源臂,共1901 bp,同源重组片段的序列如SEQ ID No.1所示。
将构建的质粒转染Trans 5a感受态细胞,挑选10个菌落克隆,使用Hind III和BamH I进行双酶切处理鉴定连接效率,10 μL反应体系如下:cutsmart Buffer(10×)1 μL;质粒(1-10号)500 ng;NheⅠ酶和KpnⅠ酶各0.2 μL;ddH
二、同源重组片段的验证
挑选的10个菌落克隆双酶切处理后的琼脂糖凝胶电泳结果如图6A所示,结果表明10个菌落克隆均已成功连接(连接效率达100%)。随后将10个菌落克隆送公司进行测序,结果表明所构建同源重组片段基因序列完全正确,可用于后续实验。随后大量扩增测序正确的菌落克隆,使用去内素质粒小提试剂盒提取质粒。使用Hind III和BamH I进行双酶切处理,酶切产物进行琼脂糖凝胶电泳,结果如图6B所示。对1901 bp的同源重组片段进行胶回收用于后续实验,至此同源重组片段正式构建完毕。
实施例3:纯合突变细胞系的制备
一、荷斯坦种公牛耳缘成纤维细胞系的建立
首先使用耳缺钳收集身体健康、遗传性能良好、生产性能优良的荷斯坦种公牛耳缘下部组织,组织块大小在1-2cm
二、荷斯坦种公牛牛耳成纤维细胞系冻存
当细胞汇合度达到95%以上,即可进行细胞冻存。使用0.25% 的胰酶在5% CO
共计获得并冻存成纤维细胞200余管,并已通过复苏培养初步验证细胞活性良好,显微镜下成纤维细胞如图8所示,细胞呈现典型的成纤维细胞形态即梭形或不规则三角形,胞质呈现放射状。
三、无角牛纯合突变细胞系的制备和鉴定
1、制备纯合突变细胞系
首先于6孔板中复苏冻存的荷斯坦种公牛牛耳成纤维细胞系,当细胞汇合度达到70%以上时,使用Lip3000脂质体转染试剂盒将4 μg pX458-sgRNA
其中,pX458-sgRNA
在制备纯合突变细胞系实验中转染24 h后使用流式细胞仪分选表达绿色荧光蛋白的细胞8000个,流式细胞分选结果如图11所示。随后将细胞稀释培养筛选获得单细胞克隆共计147个。
2、纯合突变细胞系的鉴定
收取的单细胞克隆1000 rap离心5 min后弃上清液,使用细胞组织血液全基因组DNA提取试剂盒提取单细胞克隆的全基因组作为模版,进行目的片段的PCR扩增,使用在Pc片段前后设计的引物,引物序列如下:
New-F2:5 ʹ-CCATGAGAATTAGAGTGGGATGC-3 ʹ;
New-R2:5 ʹ-ATTGGCTGGCAGATTCTTTACCA-3 ʹ。
25 μL反应体系如下:Phanta Max Master Mix (2×) 12.5 μL,提取获得的单克隆全基因组 1 μL,上、下游引物各1 μL(100 mM),ddH
反应条件如下:93℃预变性30 s;93℃变性20 s,58℃退火30 s,72℃延伸1 min10s,循环33次;72℃ 2 min;产物置于4℃保存。
对PCR扩增目的片段进行琼脂糖凝胶电泳,结果如图12所示。结果表明,共获得杂合突变细胞系8个,纯合突变细胞系1个(106号)。同时,为保证实验结果的准确性,对筛选获得的纯合突变细胞系进行测序,如图13所示,结果与PCR鉴定结果相符,可用于后续实验。
需要说明的是,本发明具体实施方式采用脂质体转染作为示例性说明,但在实际操作中并不局限于此。本发明技术方案在同源重组效率等方面仍存在可继续优化的空间。
实施例4:纯合突变细胞系外源基因残留检测
为检测纯合突变细胞系中是否有外源基因残留,使用细胞组织血液全基因组DNA提取试剂盒提取单细胞克隆的全基因组作为模版进行目的片段的PCR扩增,引物序列如下:
PXCas9-F:5 ʹ-AAGATATCGTGCTGACCCTGA-3 ʹ ;
PXCas9-R:5 ʹ-ATTTCGATCACGATGTTCTCG-3 ʹ。
25 μL反应体系如下:Taq Mix(2×) 12.5 μL,模板(提取获得的纯合体变细胞系的全基因组)1 μL,上、下游引物各1 μL(100 mM),ddH
反应条件:94℃预变性30 s;94℃变性30 s,58℃退火30 s,72℃延伸40 s,循环33次;72℃ 2 min;4℃保存。
对PCR扩增目的片段进行琼脂糖凝胶电泳,结果如图14所示。结果表明,结果表明,纯合突变细胞系中没有Cas9外源基因残留。
细菌基因组的残留对于无角牛的实际生产应用造成了极大限制,为有效避免细菌基因组的残留,本发明特别采用流式分选的方法进行Pc位点同源重组细胞克隆的筛选,该方法可以很大程度上减少细菌DNA在细胞中停留的时间,随着细胞的不断分裂,pX458-sgRNA会逐渐的丢失。为进一步验证是否存在外源基因残留,试验还对pX458-sgRNA骨架中的Cas9基因进行了鉴定,结果发现试验最终获得纯合阳性细胞克隆中不含Cas9基因。综上,Pc位点可以作为后续无角荷斯坦奶牛培育的分子设计育种靶位点。
以上所述仅是本发明的具体实施方式,使本领域技术人员能够理解或实现本发明。对这些实施例的多种修改对本领域的技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所述的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
序列表
<110> 中国农业科学院北京畜牧兽医研究所
<120> 一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法
<160> 25
<170> SIPOSequenceListing 1.0
<210> 1
<211> 1901
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
atcgaacctg ggtcttctgc attggctggc agattcttta ccactgagcc accacaccct 60
agagtgcaaa gggggcttag caccacagga ggttcacaaa gatgtaagct gttcttatta 120
aggctgaggt gggggttggg agaaggggga gaaaaagttt tgtaagttgt taatttataa 180
taaatcccca aagaaatggt ctttcaagta catacttatc taaaactttg tcaatagggg 240
aaatgttctt aggagagaaa aggaattttt tcttttagca taaagctgac tttctaatat 300
gggcttccct agtagctcag ctggtgaaga acccgcctgc aatgtgggaa acctgggttt 360
gagccctggg ttgggaagat cccctggaga aaagaatggc tacccactcc agtattctgg 420
cctggagaat tccatggact gtataccatg gggttgcaaa gagttggaca cgactgagtg 480
actttcattt tcactttctg actttctaat attcgaggaa tgcttagaag tgtggccggt 540
agaaaatagt cctttgtgcc tgggacttca agaaggcggc actatcttga tggaactcag 600
tctcatcacc tgtgaaatga agagtacgtg gtaccaacta ctttctgagc tcacgcacag 660
ctggacgtct gcgcctttct tgttatactg cagatgaaaa cattttatca gatgtttgcc 720
taagtatgga ttacatttaa gatacatatt tttctttctt gtctgaaagt ctttgtagtg 780
agagcaggct ggaattatgt ctggggtgag atagttttct tggtaggctg tgaaatgaag 840
agtacgtggt accaactact ttctgagctc acgcacagct ggacgtctgc gcctttcttg 900
ttatactgca gatgaaaaca ttttatcaga tgtttgccta agtatggatt acatttaaga 960
tacatatttt tctttcttgt ctgaaagtct ttgtagtgag agcaggctgg aattatgtct 1020
ggggtgagat agttttcttt gctctttaga tcaaaactct cttttcattt ttaagtctat 1080
cccaaaagtg tgggaggtgt ccttgatgtt gaattatagg cagagggtca gtttatcaac 1140
acccaagacc aacatctctg cctttgataa gagatagaaa tagaagtgga gagagaggag 1200
gaaaaacatg actcacgata cattctgggt tgtttgtttt tgtttttatt tttgttttgg 1260
aaggagcggg tgggggaacg tgctgattaa agaaagtcta gagagaacaa gattcttaaa 1320
aataaatcca cagtgaagcc cagcgggtgg ggatttccca cagattttca gggctttttt 1380
gtgttgtcat ggggatatta gtcaatgttg gtgtcttatt ttggagtcac tatgagtgaa 1440
ccatgtttaa ggagctatgg ctcagctgct aaactatttt caaaaggaaa atggtgtgtt 1500
acggtttccg agcagtgggg ccctggtaca ggtaatatca tactcaaaag cactcttttg 1560
tgctacatga atgaatcctg ctaaaccatg cggattaatt ttagctgaaa ttttttaggt 1620
taaagtgaag tttgtgaagt gacatctgag aaaaatatgt tggaaatcta gggagaacta 1680
gagtcaatgg gatttttaag aaactgactc tttccattat ctttcttttt ttccttcccc 1740
acaactgttt ttcttttttt acactgagtc tgtataagaa aacaggactt gccatgatat 1800
gaagtctact ggtagattaa acagctcaag tttactcaat catttactcc atatcttttt 1860
cattaaaaac actaggtaag tcttatttat cattctagtc a 1901
<210> 2
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
aatggctacc cactccagta tt 22
<210> 3
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
ggcagagatg ttggtcttgg ctgt 24
<210> 4
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
caccggtcta tcccaaaagt gtggg 25
<210> 5
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
aaaccccaca cttttgggat agacc 25
<210> 6
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
caccgtgatg ttgaattata ggcag 25
<210> 7
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
aaacctgcct ataattcaac atcac 25
<210> 8
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
caccgataag agatagaaat agaag 25
<210> 9
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
aaaccttcta tttctatctc ttatc 25
<210> 10
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
caccgagaag gcggcactat cttga 25
<210> 11
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
aaactcaaga tagtgccgcc ttctc 25
<210> 12
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
caccgttctg actttctaat attcg 25
<210> 13
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
aaaccgaata ttagaaagtc agaac 25
<210> 14
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
caccgtcgag gaatgcttag aagtg 25
<210> 15
<211> 25
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
aaaccacttc taagcattcc tcgac 25
<210> 16
<211> 51
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
aatgcatcta gatatcggat ccgcggccgc atcgaacctg ggtcttctgc a 51
<210> 17
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
ccatcaagat agtgccgcct tcttgaagtc ccaggcac 38
<210> 18
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 18
tgggaggtgt ccttgatgtt gaattatagg cagagggt 38
<210> 19
<211> 49
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 19
catgcaggcc tctgcagtcg acgctagctg actagaatga taaataaga 49
<210> 20
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 20
gaaggcggca ctatcttgat ggaactcagt ctcatcac 38
<210> 21
<211> 38
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 21
tcaacatcaa ggacacctcc cacacttttg ggatagac 38
<210> 22
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 22
ccatgagaat tagagtggga tgc 23
<210> 23
<211> 23
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 23
attggctggc agattcttta cca 23
<210> 24
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 24
aagatatcgt gctgaccctg a 21
<210> 25
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 25
atttcgatca cgatgttctc g 21
- 一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法
- 一种具有无角Pc位点纯合基因型的荷斯坦牛的制备方法
