一种微生物培养过程中多种物质实时检测的方法
文献发布时间:2024-04-18 19:58:30
技术领域
本发明涉及光谱检测分析技术领域,尤其涉及一种微生物培养过程中多种物质实时检测的方法。
背景技术
生物制药领域,过程分析技术(PAT)利用先进的分析手段实时监测和控制生物制药生产过程中的多个参数和指标。以确保产品的质量、一致性和生产效率,减少产品失败率并降低生产成本。拉曼检测技术作为一种典型的过程分析技术,以非侵入性、高灵敏度等特点在药物生产过程和质量分析具有广泛的应用。
2010年,众多学者利用拉曼技术结合化学计量学方法来实现大肠杆菌微生物的快速检测、分离和识别,同时也尝试采用更先进的拉曼成像技术来对微生物内的化学成分进行可视化分析,推动了该技术在微生物研究领域的应用。2011年NR Abu-Absi,BM Kenty等首次实现了对生物反应器中多项参数葡萄糖、乳酸、铵、谷氨酰胺和存活细胞密度的同时监测。到目前为止,培养过程中如滴定度、16种蛋白氨基酸浓度等更多关键指标被证明可以通过拉曼技术结合化学计量学的方式实时监测。
2018年Shuai He,Yi Mon Ei Kyaw研究团队提出了一种新的无标记定量测量细胞外蛋白激酶A(PKA)活性的方法。通过对SERS谱进行主成分分析(PCA),研究团队成功识别到了725和1395cm
偏最小二乘回归(PLSR)是目前使用最为普遍的一种化学计量学算法,常用与多元统计分析,处理多变量数据之间的相关性和预测。PLSR能够降低数据维度,在保留原始数据的信息的同时,把多个自变量X和因变量Y之间的线性关系提取出来,并用少量的“潜在变量”来映射回归关系。PLSR在满足朗伯-比尔定律的线性回归模型中效果表现优异,但最近研究表明在处理非线性问题以及多批次数据适用情况下导致的模型泛化能力表现不佳。由于训练的模型过于依赖于当前输入数据,使得模型鲁棒性没有取得令人的满意结果。
随着机器学习的发展,将其他回归算法如支持向量回归(SVR)、随机森林回归(RFR)与神经网络回归(ANNR)等非线性算法结合拉曼光谱能够较好的克服上述线性模型的缺点。
生物多批次培养具有明显的特异性,提高模型的鲁棒性需要使用到不同批次的过程数据进行模型的训练。一定程度上数据集的丰富性扩大,模型的鲁棒性也会随之增长,但过多的数据会使得模型的准确性趋于低饱和。且由于间断采样导致数据灾难,数据集存在分布不均的偏差。部分的样本数量过多,使得模型拟合不准确而出现错误的预测结果。
深度网络中神经元之间的复杂联系,以及多层深度能够容纳海量的数据,使之成为更为理想的高参数回归模型。2012年卷积神经网络(CNN)结构AlexNet,在ImageNet图像分类大赛上获得第一名。CNN的局部连接和权值共享特性,减小了网络的参数数量和计算复杂度,同时增加模型的稳定性。CNN在分类和回归任务上都取得良好结果。
细菌培养过程中某一时刻的所有信息都分布在同一条拉曼谱图不同波数的峰位上。由于生物大分子之间的耦合效应以及生物荧光干扰,很难通过常规的谱图识别定位特征峰。
发明内容
针对现有技术中所存在的不足,本发明提供了一种微生物培养过程中多种物质实时检测的方法。其解决了现有技术中存在的微生物培养中物质实时检测不准确的问题。
本发明第一方面,提供一种微生物培养过程中多种物质实时检测的方法,包括以下步骤:
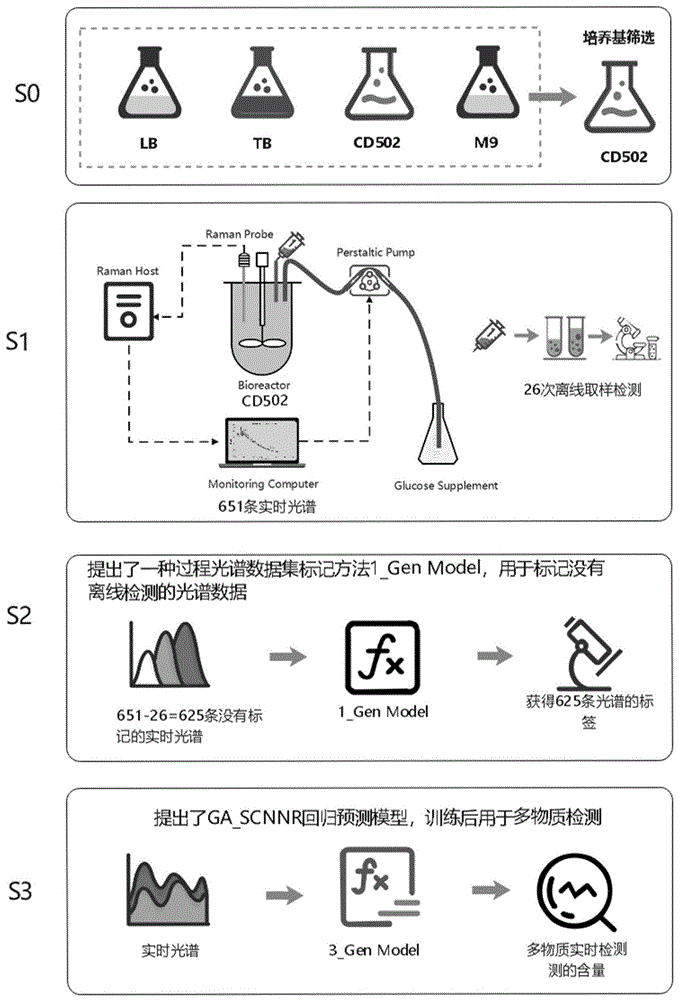
S0将微生物进行发酵培养,得到发酵样品;
S1建立数据集:在培养过程中连续采集发酵样品的拉曼光谱数据得到过程数据集;同时间断取样并检测发酵样品中物质浓度得到离线数据集;
S2标记数据集:根据离线数据集,将过程数据集划分为有标签的过程数据集和无标签的过程数据集,并对无标签的过程数据集进行标记,得到无标签的过程标记数据集;
S3建立回归模型:有标签的过程数据集与无标签的过程标记数据集共同组成半监督数据集,用来训练遗传算法-半监督卷积神经网络模型,建立拉曼光谱与物质浓度之间的回归模型。
本发明一实施例中,步骤S1中,连续采集发酵样品的拉曼光谱数据的方法包括:将拉曼光谱仪与发酵样品连接,实时检测发酵样品的拉曼光谱数据;
间断取样并检测发酵样品中物质浓度的方法包括:每间隔一定时间,采集发酵样品并检测吸光值和物质浓度;
优选间隔时间为1h;
优选物质包括葡萄糖、乳酸、铵离子、OD600和目标蛋白。
本发明一实施例中,步骤S2中,根据离线数据集,将过程数据集划分为有标签的过程数据集和无标签的过程数据集的方法包括:将过程数据集中与取样时间对应的拉曼光谱数据划分为有标签的过程数据集,其余拉曼光谱数据划分为无标签的过程数据集;
优选地,对无标签的过程数据集进行标记的方法包括模型计算法和插值法;
更优选地,所述模型计算法为利用有标签的过程数据集训练完成的模型对无标签的过程数据集进行标记;
更优选地,所述插值法为利用三样条插值法对无标签的过程数据集进行标记。
本发明一具体实施例中,利用有标签的过程数据集训练完成模型的方法包括:对有标签的过程数据集进行扩充和预处理,用于训练PLSR和SVR模型;通过模型集成方式计算PLSR和SVR模型的回归参数,通过boosting模型的迭代方法得到1_Gen模型;
优选地,所述扩充的方法包括:添加基线斜率、引入高斯噪声和比例数法;
优选地,所述预处理的方法包括利用基线校正法、Savitzky-Golay滤波法、标准正态变量变换法、归一化对拉曼光谱数据进行处理;
优选地,所述三样条插值法包括利用插值函数对过程数据进行标记,所述插值函数如式(1)所示:
其中,S
本发明一实施例中,步骤S3中,训练遗传算法-半监督卷积神经网络模型的方法包括:通过遗传算法对半监督数据集进行物质的特征波段提取,并对特征波段进行强化;构成训练样本集,用于训练半监督卷积神经网络模型。
本发明一具体实施例中,所述特征波段提取的方法包括:以目标函数的均方根误差(RMSE)取反的方式作为适应度函数,利用遗传算法对有监督过程数据集的特征波段进行提取;
所述对特征波段进行强化的方法包括:采用横向三次样条插值的方式对特征波段进行强化,使之还原为原始光谱长度。
本发明一实施例中,还包括S4迁移学习:根据深度卷积神经网络,在训练完成的遗传算法-半监督卷积神经网络模型基础上,引入少量目标物质的拉曼光谱数据和离线数据,即得目标物质的半监督卷积神经网络模型;
优选地,所述目标物质为目标蛋白;
优选地,在训练完成的遗传算法-半监督卷积神经网络模型基础上,引入少量目标物质的拉曼光谱数据和离线数据的方法包括:在吸光值的半监督数据集预训练SCNNR模型为基础,通过添加全连接层,引入少量的目标蛋白拉曼光谱数据集和离线数据集,即可得到目标蛋白的SCNNR;
优选地,利用目的蛋白的数据集对预训练SCNNR模型参数进行调整。
本发明第二方面,提供一种微生物培养过程中多种物质实时检测装置,包括:数据集标记模块、回归模型建立模块和迁移学习模块;
所述数据集标记模块用于标记过程数据集;
所述回归模型建立模块用于建立拉曼光谱与物质浓度之间的回归模型;
所述迁移学习模块用于建立少量目标物质的半监督卷积神经网络回归模型。
本发明第三方面,提供一种微生物培养过程中多种物质实时检测装置,其特征在于,包括处理器、存储器以及存储在所述存储器中且被配置为由所述处理器执行的计算机程序,所述处理器执行所述计算机程序时实现上述的微生物培养过程中多种物质实时检测的方法。
本发明第四方面,提供一种计算机可读存储介质,其特征在于,所述计算机可读存储介质包括存储的计算机程序,其中,在所述计算机程序运行时控制所述计算机可读存储介质所在设备执行上述的微生物培养过程中多种物质实时检测的方法。
本发明的技术原理为:
在发明以大肠杆菌培养过程中多种物质检测为目标,在深度学习模型CNN的基础上提出了更加具有适用性的遗传、半监督卷积神经网络回归模型(GA_SCNNR),用于重组大肠杆菌培养的拉曼模型建立。
本发明使用遗传算法关联回归模型,以均方根误差(RMSE)取反的方式作为适应度函数,实现了在同一光谱对不同检测物质进行特征位置划分,从2048个输入光谱变量(X1,X2,……,X2048)提取不同的最佳适应特征波段。
CNN的网络参数训练通常需要大量的数据,为克服采样数据集的不足,本发明分别使用了有标签的离线数据集和无标签的过程数据,两者共同构成半监督数据集来训练模型。由于离线检测数据的稀有,使用了添加基线斜率、引入高斯噪声和插值计算三种一维数据增强方式扩充标签数据集,强化了拉曼光谱信息与被测物质含量的关联性。因生物反应器的过程拉曼光谱采集是具有时间序列的,可通过时间序列模型回归、插值拟合等方式标记无标签数据集,从而更加细致地捕捉被检测物质的连续变化过程,契合实际情况中物质含量是模拟量的变化特点。
同时,GA_SCNNR可通过迁移学习的方式增强模型的实际适应力,在保留第N-1批次训练完成的模型前提下,添加额外的全连接层,便能构成第N批次的新模型,以适应新的应用场景,形成(训练-应用-训练)的动态模型结构。具体来说,本发明在已完成训练的SCNNR(OD600)模型的基础上,添加了两个空白全连接层,分别包含128和64个神经元,构建了迁移学习模型。收集了20条目的蛋白参考数据,并通过数据增强技术将数据集扩大到320条,扩充了16倍。接着,使用目的蛋白数据集对预训练模型进行微调,进一步在目标任务上进行训练,调整模型的参数以适应该任务。通过以上方法,成功构建了适用于目的蛋白表达任务的迁移学习模型。该模型利用了SCNNR(OD600)模型的先验知识,并通过微调操作,在目标任务上取得了更好的性能和适应性。这种方法为解决目的蛋白表达任务中难以获取离线参考值的问题提供了一种有效的解决方案。
相比于现有技术,本发明具有如下有益效果:
(1)本发明提出了遗传半监督卷积神经网络(GA_SCNNR)框架,以实时拉曼光谱为基础,在重组大肠杆菌培养过程中建立了多参数回归模型,可实现大量的无标记的过程光谱数据用于模型的训练,训练集样本从52个扩增到1302个,极大程度上提升了过程检测的实时性和鲁棒性。
(2)本发明提出来一种光谱标记方法,以过程谱标记方法以扩充模型的训练数据集,利用了大量无标签的过程光谱数据,使得模型有更大的数据集用于训练,在少量离线样本的前提下也能训练出高精度的模型。
(3)本发明针对不同的检测物质遗传算法的引入,可准确的在同一条拉曼光谱数据中解析出最适的特征位置,提升了多参数目标检测的准确性。
(4)本发明使用了深度卷积神经网络利用迁移学习,能够在少量数据的情况下依然能训练出高精度的模型,在及其少量目标蛋白数据集的情况下,成功实现了目标蛋白表达的检测。
(5)本发明模型预测的实用性得到了验证,可用于指导培养过程的产量与趋势检测。
因此,与传统的化学计量学模型相比,GA_SCNNR框架还拥有以下优势:CNN网络独特的卷积结构,可代替光谱数据预处理过程并且具有更高的精确度。实际的生物培养过程,检测参数和培养体系时常变化,GA_SCNNR框架通过迁移学习拥有可扩展的能力,在保留之前训练的参数的前提下用少量样本下便可重新适应新的检测任务,为生物检测通用型模型的开发提供了新的思路。
附图说明
图1为本发明微生物培养过程中多种物质实时检测方法的流程图。
图2为本发明实施例4中原始光谱通过不同技术增强的情况。
图3为本发明实施例6中遗传算法提取特征波段流程图。
图4为发明实施例6中提取的最佳适应波段。
图5为本发明实施例7中1_Gen Mode计算过程图。
图6为本发明实施例7中光谱标记过程与CNNR模型训练。
图7为本发明实施例7中SCNNR结构与目的蛋白的迁移学习。
图8为本发明实施例7中GA_SCNNR训练过程图。
图9(a)为本发明实施例7不同目的蛋白浓度下的三维PCA分布,9(b)为第二维度和第三维度视角。
图10(a)为本发明实施例8中四种培养产物的离线参考数据集在PLSR、SVR与CNNR不同模型上决定系数;10(b)为625条葡萄糖浓度的过程标记数据,将其以不同占比加入参考数据集中,构建半监督数据集。
图11为本发明实施例8中线数据集经过预处理(包括Savitzky-Golay滤波(6,21)、AirPLS基线校正和SNV),并与未经处理的原始数据集分别在PLSR、SVR和CNNR模型上进行模型决定系数的验证图。
图12为本发明实施例8中两种过程标记方法在1_Gen Mode的交叉验证性能。
图13为本发明实施例8中五种物质的10倍交叉验证的箱线图,红色十字符号代表了在交叉验证时决定系数的异常值。
图14为本发明实施例8中模型的多目标训练过程。
图15为本发明实施例8中重组蛋白培养过程中参数实时检测。
图16为本发明实施例8中验证实时参数检测过程准确性,蓝色曲线为GA_SCNNR计算实时输出值,红色曲线是在蓝色曲线基础上进行的多项式平滑化,黑色空心圆圈是通过离线测量的参数真实值。
具体实施方式
下文将结合具体实施例对本发明的技术方案做更进一步的详细说明。应当理解,下列实施例仅为示例性地说明和解释本发明,而不应被解释为对本发明保护范围的限制。凡基于本发明上述内容所实现的技术均涵盖在本发明旨在保护的范围内。
除非另有说明,以下实施例中使用的原料和试剂均为市售商品,或者可以通过已知方法制备。
术语定义
“有标签”、“有监督”代表的含义相同,“无标签”、“无监督”代表的含义相同。
本发明中“物质”并不局限于葡萄糖、乳酸、铵离子、目标蛋白、OD600,实时拉曼探头中能检测出特征变化的物质均适用于本方法。通常拉曼探头可以检测到蛋白质、碳水化合物、脂质、代谢产物,具体物质需要根据实际使用的硬件探头决定。
符号说明
本发明所用主要仪器:电子天平(sartorius),高速冷冻离心机(cence湘仪),恒温摇床(radobio),紫外可见分光光度计(ThermoFisher),自动式压力蒸汽灭菌锅(天美),超净工作台(苏净安泰),5L发酵罐(霍尔斯),烘箱(bluepard),自动生化分析仪(西尔曼),激光探头式拉曼光谱仪(MARQMETRIX)。
本发明所用的培养基和试剂:采用锥形瓶进行分批发酵,研究了不同培养基组分对重组大肠杆菌生长和生物量的影响,确定CD502作为发酵罐培养的主要培养基。下表中展示了培养基以及补料中使用的主要材料。
CD502培养基由无机物质构成,具有组成成分精确、清楚,重复性强等优点,且能达到较高的蛋白和生物量。
实施例1发酵罐高密度培养
1、一级种子培养:将在LB中的甘油菌接种于CD502培养基,37℃培养过夜。
2、二级种子培养:将适应在CD502的菌液接种于含有50ml培养基的250mL锥形瓶中,37℃培养过夜。
3、5L全自动发酵罐:配备pH、溶氧和拉曼电极。5L发酵罐中注入2.0L CD502培养基,接种50mL过夜培养的菌液,添加卡那霉素至50mg/L,控制温度37℃,用30%氨水和30%磷酸控制pH6.8,初始通气量为2L/min压缩空气,初始搅拌转速200r·min
其中,μ为比生长速率值,Pset为设定比生长速率值;Xo和Vo为补料起始时细胞干重(g/L)和发酵液体积(L):SF为补料培养基葡萄糖的浓度(g/L):m为保持系数[g/(g-h)-0.025],Yx/s为产出系数(g/g),即每g葡母糖可产生多少g细胞干重。
4、在发酵过程中,对培养基中的菌液进行采样,600nm处的吸光度值。培养至一定时间后进行IPTG诱导(终浓度为1mmol·L
实施例2离线分析检测
在诱导前期每一个小时取一次离线样本测定离线分析值,用于模型的建立与验证。
取1mL发酵液,稀释至适当倍数,于600nm波长处测定吸光值。将发酵液离心洗涤两遍,收集菌体,在-40℃下冷冻干燥至恒重,测定细菌干重。1.0OD_600相当于0.54g/L细胞干重。葡萄糖、乳酸、铵离子浓度测定,采用生化分析仪(西尔曼),菌液离心,收集上清,并用水稀释至合适的倍数进行测定。定量测定目标蛋白浓度,BCA试剂盒(碧云天)。采用Octet一步法定量测定,利用HISK Sensor,将标准品和样品用相同的稀释缓冲液(PBST(1X PBS+0.02%吐温20))稀释至合适倍数,来测定样品的目的蛋白的浓度,利用再生缓冲液(pH=1.7 10mM甘氨酸)再生传感器。
实施例3过程光谱采集
激光探头式拉曼光谱仪激发波长785nm,采用侵入式不锈钢探头放入生物反应器培养液面以下,通过光纤传输获取的散射光谱数据,将信号传输至拉曼光谱仪。利用局域网进行通信,光谱仪的信号被传送至终端计算机,进行光谱可视化和参数设置,信号传递以及硬件流程如图1中S1所示。参数设置integration time 1000mS,Averages 50,Timebetween samples 0mS,Number of sample 1,Laser power 300mW。每1.5min可获得一条培养过程中的时间序列光谱,波数范围为(42.215-3288.678cm
实施例4数据集构成
CNN高精度和强鲁棒性依赖于大规模的数据训练,丰富的数据集使得模型能充分学习到拉曼光谱的特征变化与物质浓度之间的回归关系。大规模数据集提供丰富的数据分布和更多的数据样本,可有效促进CNN模型的泛化性能,避免出现过拟合现象。同时,更大规模的数据集有助于增加训练量和数据多样性,使模型更全面地学习到特征,避免欠拟合问题。并且数据集缺乏多样性会导致数据分布不平衡,并产生数据偏差。为减少这种风险和错误率,采用大规模数据集,以提供覆盖更广泛的样本分布,降低数据集偏差,减少模型在数据分布不均衡下的过拟合风险和误判,从而提高模型性能和准确性。
GA_SCNNR的数据集使用了通过一维数据增强的有监督数据集和无标签的时间序列过程数据集共同构成。
1、有监督一维数据增强
大肠杆菌培养过程中连续多次取样,会影响菌落数量和生长以及交叉污染,同时离线样本的检测也会消耗大量的资源及时间。受此约束想要获取大量的离线检测值是并不容易的,与回归模型训练需要大规模数据集构成了矛盾。
本实施例中,总共培养了三个批次的重组大肠杆菌,取前两次的数据用于模型的训练,最后一个批次的数据用于验证模型。每个批次可获得葡萄糖、铵离子、乳酸、OD_600样本个数是26、重组蛋白含量样本10个,作为模型回归预测的离线参考值Yi,分别选取了与取样时间所对应的26条时间序列拉曼光谱值作为Xi。使用以下三种一维数据增强(1D DataAugmentation)对已经标记的过程光谱进行扩充。
(1)在拉曼光谱中,基线斜率指光谱中的一个趋势线或拟合线的斜率,用于去除或减少背景信号对于样品信号的干扰。通常使用多项式拟合方法来绘制一个类似于斜率为零的趋势线,也称为基线。这个基线可以移动、增加或减少,以更好地拟合样品信号并去除背景噪声。在26组原始拉曼光谱的基础上,施加四种不同的基线斜率扩充数据集。五类检测物质对应的拉曼光谱数据由26条分别扩充到104条、目的蛋白扩充至40条。
(2)高斯白噪声是一种随机噪声信号,其频率范围包括所有可测量的频率,并且其谱密度在所有频率上都是相等的,因此称为“白噪声”。高斯白噪声具有其随机性和普遍性。在拉曼光谱数据中添加强度不等的随机高斯白噪声,将原有标签的光谱数据集规模扩大四倍,由104条扩充到416条、目的蛋白扩充至160条。
(3)在离散样本序列所对应的拉曼光谱情况下,通过相邻光谱乘以比例系数总和为1的权重系数,对滑动窗口内包含的5条光谱乘以对应比例系数后再相加得到新的过程光谱。而与之对应的标签值也乘以对应比例系数,使得拉曼光谱的序列变化更加连续,从而获得更加密集的序列光谱。使用比例数法再次扩充数据集,将随机白噪声扩充的拉曼光谱扩大2倍,拉曼光谱扩大至832条、目的蛋白扩充至320条。
原始光谱通过不同技术增强的情况如图2所示,(a)为原始的离线样本值对应的拉曼光谱,(b)为添加基线拉曼光谱扩大四倍,(c)为添加高斯噪声拉曼光谱扩大四倍,(d)为添加比例权重拉曼光谱扩大两倍。
2、无监督序列标记
重组大肠杆菌培养过程的相关指标具有模拟量的变化特性,即除了在人为外界条件的干预下,各种代谢指标的含量不会发生明显瞬时突变。每种参数的含量变化有十分明显的曲线趋势。因此在设计实验时对每次补料这种人工干预前后都会取样,在正常光滑趋势线上获取到突变值。为此本发明设计了两种过程光谱标记方案,以便全过程光谱都可作检测值趋势曲线的关系。在有限的大间距离线检测值的基础上通过插值拟合的方式,构造一个光滑的插值函数来逼近更为密集实际数据点,得到对应全过程时间序列光谱的连续标签值曲线。
三次样条插值是一种光滑插值的方法,在区间内将插值函数表示为三次多项式,插值区间[X
方案一,全过程光谱具有时间序列,物质浓度也会随着时间改变,因此借助时间这个变量为纽带建立时间序列光谱与离线的插值函数
式(2)中分别说明,插值函数在插值点上的函数值等于原函数在该点上的函数值,相邻的两个三次多项式在节点处函数值相等,相邻的两个三次多项式在节点处一阶导数值相等,插值函数在区间首尾两个点上的二阶导数为0,即自然边界条件。
方案二,使用有标签的原始数据集训练完成的预测模型。将现有时间序列光谱作为输入,求得标签输出。这种模式适合需要频繁干扰的情况应用,连续补料下依旧能保持较为可靠的结果。因为有标记的数据集有限,GA_SCNNR模型的无标签数据标记使用PLSR,SVR两种小样本预测模型的结果。采用Boosting方法,训练两个弱回归模型,构建出一个更强和鲁棒的预测模型,用来给过程数据集添加代谢产物浓度的标签。
实施例5光谱预处理
光谱预处理作为传统模型中不可或缺的环节,拉曼光谱在采集过程中环境复杂多变,容易受到荧光背景、环境辐射等干扰因素的影响,采集到的拉曼光谱信号存在着噪声严重、基线漂移的现象,很大程度上影响了拉曼光谱分析的准确度。因此,传统回归算法在对拉曼光谱进行建模分析前需要对拉曼光谱进行预处理操作,通常包括平滑滤波和基线校正,从而提升模型预测的准确率。
采用迭代自适应重加权惩罚最小二乘法(Adaptive Iteratively ReweightedPenalized Least Squares,AirPLS)的基线校正方法,该算法核心是依据前一次迭代的拟合基线和原始信号残差平方和的权重,完成惩罚最小二乘的权重的自适应调整。airPLS算法每进行一次迭代过程,都是在对加权惩罚最小二乘函数求最小值。airPLS算法快速准确地找到不规则变化的基线并对其进行扣除,代价函数如式(3)。
式(3)中,
Savitzky-Golay滤波法(窗口大小21,三阶多项式)用来去除拉曼光谱的噪音。对窗口内的数据进行加权滤波,加权的权重是高阶多项式进行最小二乘拟合得到。窗口拟合函数如式(4)
式(4)中,m为奇数表示窗口大小,a
标准正态变量变换(Standard Normal Variate Transformation,SNV)用来消除细菌体积大小与表面散射以及光程变化对NIR漫反射的影响。SNV是基于光谱阵列的行进行的如(5)所示:
式(5)中,
拉曼光谱基线产生的原因十分复杂,主要包括以下几个方面:首先是样品的杂质、光源强度、波长等因素都可能导致荧光的产生,从而对光谱的基线产生干扰;其次,来自仪器、环境和样品的多种散射噪声会遮盖光谱的拉曼信号,进而影响光谱基线的形态和位置;此外,光谱背景噪声通常来自各种环境因素,如运动强度、温度变化、湿度变化等;光源强度不稳定和激发波长不准确等仪器因素也可能会对拉曼光谱基线产生影响。噪音是由于光源的不稳定性和波长漂移、检测器的噪声以及光谱仪的机械振动,以及采集时间、激光功率和焦距等原因都可能会对拉曼光谱引起噪声影响。
原始拉曼光谱经过SNV散射处理方法能够消除不同光谱之间的强度差异,同时保留散射光谱的特征信息。其次使用基线校正可以消除基线漂移的影响,使光谱更加平滑和连续。最后,S-G滤波可以平滑光谱中存在的噪声,同时保留光谱的信号特征,从而提高光谱的信噪比。经过光谱预处理后,所得到的拉曼光谱信号能够增强样品的信号对峰,并优化相应的波长和强度指标,使光谱数据更加准确和可靠。
实施例6最优拉曼波段提取
本实施例的拉曼光谱数据,其包含2048个特征波段,而不是所有波段都对目标值有贡献。因此在回归时需要对光谱进行特征波段提取,选择与目标变量相关性较高的特征波段,来建立预测模型。在本项研究中预测了多种物质,不同的物质对应的特征峰也在不同的位置。
遗传算法(Genetic Algorithm,GA)是基于自然选择和遗传进化过程的优化算法。通过种群中个体的适应度评估来驱动进化的过程,遗传算法将潜在解表示为一串二进制编码,这些编码在整个搜索过程中按照类似于基因的方式进行交叉和变异。在每一代中,算法通过循环计算个体的“适应度”,然后从中选择复制或交叉高适应度的个体,同时执行一定的变异操作以引入种群的多样性,不断迭代进化直到满足停止条件为止。
拉曼光谱使用遗传算法进行特征提选取,是一个反复选取不同的波段进行回归运算以满足最大适应度的过程。本文对2048个拉曼特征波段进行二进制编码操作。使用2048个二进制位表示拉曼波段的选择与否,区间范围为[000……0000,111……1111]。1代表选取该位置的波段,0代表该位置未被选取,如图3表明了遗传算法提取特征波段流程。
优化的目标函数是PLS或者SVR模型的RMSE,需要说明的是目标函数是求最小值,而适应度函数是按照最大值进行计算,本发明中使用将RMSE取反数的方式解决此问题。最佳适应波段如图4所示,原始波段长度为2048提取出的最佳波段,途中灰色线与光谱相交处即为最佳的适应波段,其中(a)葡萄糖浓度的最佳波段,(b)乳酸含量的最佳波段,(c)铵离子含量的最佳波段,(d)OD_600含量的最佳波段。
实施例7建立回归模型
1、模型评价指标
回归预测模型的训练结果使用均方根误差RMSE、和决定系数(Coefficient ofDetermination,R
/>
式(6)、(7)中为第i个真实值的取值,为模型的第i个预测值,为真实值的均值,m为样本数量。RMSE提供了对预测值中误差的平均大小进行度量。较小的RMSE值表示更高的预测准确性。是一个介于0到1之间的值,用于表示模型解释因变量变异性的比例。当值接近1时,表示模型对因变量的解释能力较强,拟合效果较好;而值接近0时,表示模型对因变量的解释能力较弱,拟合效果较差。
通过交叉验证来检验模型的鲁棒性,数据集分成k个不重叠的子集,进行k次模型训练和测试,每次挑选其中一个子集作为测试集,剩余的k-1个子集作为训练集进行模型训练,如此反复进行,从而得到k次模型的评估结果。在这个过程中,每个子集都能够做一次测试集,从而提高了数据的利用率,并且由于k次的评估结果会取平均值,因此可以得到更加准确的模型评估结果。
2、回归预测模型建立
2.1第一代拉曼回归预测模型
根据数据结构的特点,首先利用26组有标签的葡萄糖、乳酸、OD_600数据集,所有光谱通过实施例4步骤1的方法进行处理,包括添加基线、高斯噪声,扩充数据集到416条有标签的数据集,以增加数据的多样性和覆盖范围。
为了消除光谱中的噪声和散射影响,采用了传统模型中的预处理方法。比如使用实施例5中的Savitzky-Golay平滑(窗口大小为17个数据)等预处理方法消除噪声散射的影响。此外,对光谱数据进行归一化处理,将光谱强度放缩至0-1的范围内,以消除不同批次数据的差异性影响。
通过模型集成的方式,得到了PLSR和SVR模型的回归参数,并通过Boosting模型的迭代方法得到更准确和适应性更强的1_Gen模型。训练好的1_Gen模型可以用于2_Gen模型中,对更大量的过程光谱数据进行检测参数的标记。
图5展示了1_Gen Mode计算过程。PLSR模型是基于MTALAB R2020a的Plsregress()函数开发,PLSR模型的主成分数量设定为3。SVR模型则是基于Fitrsvm()函数开发,使用常态径向基核函数(Radial Basis Function,RBF)为核函数做非线性回归拟合。
2.2第二代拉曼回归预测模型
第二代模型旨在标记625条没有标签过程光谱数据集,用于GA_SCNNR模型的训练。训练完成的1_Gen Mode可为过程光谱数据集“贴上标签”。为了添加时序标签,使用了三次样条插值法将原始的26个离散标签值密集化为625个点,以表示四种培养产物(葡萄糖、乳酸、铵离子、OD_600)的浓度。使用1_Gen Model法添加的标签,由于拉曼光谱散射误差的影响,导致预测的参数曲线并不平滑,会出现小强度的锯齿数据的情况,如图6所示。为了平滑化数据,可以使用多项式拟合的方法。通过这样的两种标签添加方法,并乘以适当的权重系数,可以获得合理的有标签数据。
还针对四种不同的检测物质(包括葡萄糖、乳酸、铵离子、OD_600)使用提及的遗传算法进行了处理,提取出与每种物质对应的特征波段。通过遗传算法,从原始光谱长度(2048个点)中提取了与物质浓度对应的特征位置,该操作使得光谱长度缩减为与物质浓度相关的特征。为了保持模型的一致性并增强光谱特征,我们使用横向三次样条插值的方法将光谱还原为原始长度(2048个点),不同物质的峰长变化如表1所示。图6展示了2_Gen模型的计算过程。
表1四种物质的特征波段提取剂特征强化
第一批次的“贴标签”后数据集(625条)与标签数据集(26条)共同构成半监督数据集(651条)训练关于四种培养物质的SCNNR模型。实现了拉曼光谱全过程的采样预测,模型能更好反映出物质含量的过程数据变化。
2.3第三代拉曼回归预测模型
基于迁移学习的第三代模型,用于解决目的蛋白表达检测的问题。目的蛋白的表达需要在重组大肠杆菌培养到20个小时后,再加入诱导剂才能表达。蛋白表达的周期约占整个培养周期的1/5,因表达时间短,目的蛋白的离线常考值的大量获得比起其余四种物质更加困难。
首先,首先预训练基于SCNNR的模型使用OD_600的半监督数据集。在预训练阶段,模型作为特征提取器需要充分捕捉光谱特征,因此未使用遗传算法进行光谱特征提取。图7所示为迁移学习用于求解目标蛋白含量。
其次,冻结SCNNR(OD_600)模型,基于此模型再引入两个全连接层,每层分别包含128和64个神经元。从第一个批次中获取了8条目的蛋白参考数据,通过数据增强后数据集扩大8倍达到64条。然后,利用目的蛋白的数据集微调预训练模型,针对目标任务对模型参数进行调整,以提高模型在目的蛋白表达任务上的性能。
采用二维卷积神经网络框架对一维拉曼光谱数据进行训练。卷积网络有一个四维张量层、两个卷积层、两个池化层、两个全连接层、一个Dropout层构成;迁移模型在卷积网络上新增加了两个空白的全连接层。
由于拉曼光谱数据的长度为2048,为了适应二维卷积网络的输入要求,使用了reshape函数将光谱数据转换为一个四维张量[x_transform,1,1,n],其中‘x_transform’表示一维拉曼光谱数据行数,第二个参数表示列数为1,第三个参数表示通道数为1,第四个参数表示输入数据的样本数量。到此可将一维光谱数据视为二维的灰度图片,并可直接将其应用于二维卷积网络进行训练。
卷积层用于特征提取,前三层有64、32和32个卷积核,卷积核大小为(5*1)、(3*1)和(3*1),最大池化层从过滤器覆盖的特征图区域中提取最大的元素,池化核的大小为(2*1),进行特征图的缩放提高训练期间的稳定性,卷积核和池化核均被扁平化为宽度为1的过滤器。并通过256个神经元的全连接层来接收到卷积层和池化层的特征,通过权重参数的组合与整合,对特征进行更加复杂和高维的表达。再引入Dropout(0.7)随机地关闭70%的神经元防止模型过拟合。
在训练配置方面选择基于梯度的优化的Adam优化器,为降低内存使用率,每次数据的最小批次‘MiniBatchSize'设置为50,所有数据重复训练100遍。学习率降低周期为20轮,初始学习(0.01)率将会在每个指定周期进行降低(0.001),且以RMSE作为损失函数。在没训练的50轮引入30%的验证集做验证,可直观的观察模型的欠与过拟合。所使用的开发环境基于Python3.10与MATLAB R2020a中的"Deep Learning Toolbox",以TensorFlow2.10.0作为二维CNN开发框架完成CNNR和迁移学习,图8为GA_SCNNR训练过程。
由训练过程图可知,数据在迭代100轮时不管是训练集还是验证集都能达到一个较低的loss,通过构造四维张量的的一维数据用二维模型框架来训练数据很快便能得到收敛。
2.4、拉曼检测目的蛋白表达
当拉曼光谱被应用于重组大肠杆菌中的目的蛋白含量的实时监测时,表达周期短以及每次采样时间长之间的矛盾是难以获得大量数据的根本原因。离线样本数量的有限性建模难以精确拟合预测标签,本研究尝试用全光谱建立OD600的CNNR模型上以迁移学习的方式训练少量的目的蛋白表达数据集。分别在目的蛋白含量为0、0.10、0.52、1.20mg/L的四个时刻,每个时刻取相近时间序列的5条光谱,用于对拉曼光谱进行主成分分析(PCA),图9表明激光探头拉曼可用于目的蛋白含量的定量分析。
通过三个主成分可以将四类不同的目的蛋白浓度的拉曼光谱区分开来,其中在第一个维度包含了光谱90%以上的信息基本就能做到浓度的定性区分,PCA证明了拉曼光谱在目的蛋白ProA-5m检测的可行性。结合迁移后的CNNR(OD_600)便可实现定量检测。
实施例8预测模型的应用
1、预测模型的对比
在前两批培养实验中,每次可获取26条关于葡萄糖、乳酸、铵离子和OD_600浓度的离线参考数据。为了验证标记的625条过程光谱的合理性,在葡萄糖的离线参考数据基础上,分别加入了0%、10%、30%、50%、80%五个梯度的过程光谱数据,用于训练PLSR、SVR和SCNNR预测模型。通过计算每个模型的RMSEcv的均值,结果如图10a所示。在过程光谱引入量较少时,PLSR和SVR的性能优于SCNNR。然而随着过程数据量的增加,PLSR模型达到了数据饱和,SVR模型的计算复杂度也大幅增加,此时SCNNR对于大数据的拟合处理能力的得到体现。具体的模型性能如图10b所示。
为了验证模型在不同情况下的鲁棒性,使用了两批次共52个葡萄糖浓度的离线数据集进行模型训练。离线数据集经过预处理(包括Savitzky-Golay滤波(6,21)、AirPLS基线校正和SNV),并与未经处理的原始数据集分别在PLSR、SVR和CNNR模型上进行模型决定系数的验证。具体结果如图11所示,其中,(a)为原始光谱;(b)为S-G滤波;(c)为SNV散射处理;(d)为air PLS基线校正;(e)为归一化;(f)为R2预处理影响。
根据上述分析可知,PLSR与SVR在对光谱数据进行回归预测时,非常依赖数据的预处理步骤。因此需要对光谱进行预处理以提取有效的特征。然而,使用CNNR可以通过学习特征提取器来自动识别光谱中的相关特征,从而避免了传统方法中的预处理步骤。在CNNR中的卷积和池化层中,网络可以自动学习滤波器和特征映射,从而提取光谱数据中的相关特征。这些操作基于数据本身的模式和结构,减少了人为干预和预处理过程,简化了分析流程。其原因是,由于CNNR中的卷积核和池化核自身可以充当滤波器的作用,因此在预处理前后对模型的决定系数影响不大。因此,CNNR卷积网络可以作为一种替代方法,用于光谱数据的滤波、基线校正和SNV(Standard Normal Variate)处理。
2、半监督卷积神经网络
SCNNR相较于CNNR的区别主要在于数据集上使用了标记过程光谱,两个批次实验共计获得1250条以贴标签形式的标记过程光谱。标记过程光谱与标记光谱共同构成1302条半监督数据集用于卷积神经网络的回归训练,并且对所有分析物包括目的蛋白表达含量的数据进行了扩充改进。
根据实施例4中的两类数据贴标签的方法,模型计算法与插值法这两种方法都可以对过程光谱进行标记。以葡萄糖的浓度为例,使用1_Gen Mode模型计算法计算出的参考值,先使用标记光谱计算模型的回归参数,再用来标记过程光谱,通过计算的方式可以在一定程度上反映真实的浓度变化趋势。临近时刻前后采样的拉曼光谱由于散射噪声等因素的误差波动,导致在相邻时刻的计算值并不光滑,为解决此类问题可以在计算浓度值的基础上用多项式拟合的的方式拟合出光滑过渡自然的浓度变化曲线。此外使用三样条插值法在现有离散点的基础上根据过程光谱的数量为之添加过程值,所得到的浓度变化曲线更为自然。通过模型计算法和插值法的组合使用,可以较好地处理噪声波动和过程光谱的不连续性,得到相对平滑且准确的浓度变化曲线,图12展示了两种过程标记方法在1_Gen Mode的交叉验证性能,(a)wi插值法与1_Gen Mode计算的葡萄糖浓度的标签,(b)两种标记方法的数据集在10_fold cross validation下的SCNNR的RMSE箱线图。
使用两种不同方式标记的标记过程光谱与标记光谱作为半监督数据集,进行SCCNR的归回预测。使用REMS验证模型拟合的准确性,为衡量模型的鲁棒性使用10倍交叉验证重复5次的方式,产生50个随机划分的数据集训练测试集。图中显示了葡萄糖含量在两个不同标记数据集的RESMcv的分布。如图12(b)所示,两种标记方案在10倍交叉验证中均没有出现离群值,通过模型计算法比插值法得到的箱线图数据分布更加稳定。物质浓度会在培养过程任意时刻领域下呈现出稳定的变化趋势的客观规律下,由拉曼光谱计算出的浓度值也应该稳定,故不应单纯考虑交叉验证的稳定性。模型计算法表明了物质浓度的实时波动性,而插值法突显了浓度变化的稳定性,故应该结合两类标记方法的特点施加不同对应权重标记为更加可靠的过程标签。
3、模型鲁棒性
同一条拉曼光谱对应了多种物质的回归标签,本发明使用了经典的光谱回归模型PLSR(标记光谱数据集)、CNNR(标记光谱数据集)回归模型与GA_SCNNR(标记光谱数据+标记过程光谱数据集)做对比,测试了在葡萄糖浓度、乳酸含量、铵离子浓度、目的蛋白表达含量时的模型准确度。每一类数据集都按照训练集和测试集按照7:3划分,重复10遍取其RMSEP均值作为检测结果,如表2所示。
表2RMSEP均值检测结果
GA_SCNNR由于在数据集中加入了大量标记过程光谱,在五类物质含量标签的回归上都相较PLSR有明显的提升。并行在训练完成的SCNNR(OD_600)的基础上以迁移学习的方式实现了目的蛋白表达含量的预测,解决了在少量数据集中也能准确预测目标蛋白的浓度的问题,图12展示了不同的物质在GA_SCNNR上交叉验证性能,数据集分为10份重复10遍,用决定系数R2作为不同检测物质的统一衡量指标。
每类检测物质代表了重组大肠杆菌生长过程中的不同属性,每类物质的量纲不一致单纯考察RMSEcv不具有直观意义。决定系数范围在0到1之间,可忽略量纲带来的差异性。分析图13可知,由中心趋势和离散程度来看除了铵离子以外,GA_SCNNR在其他四种物质的检测中具有良好的稳定性且决定系数都趋近于1,特别是与大肠杆菌密度相关的OD_600检测上。数据表明SCNNR具有良好的准确性以及鲁棒性,可用于实际的生物培养体系的多变量回归模型建立。此外铵离子的光谱特征可能与其他物质的光谱特征有重叠,导致在光谱图谱中无法明确地辨别铵离子的信号。
4、实时过程检测
实时监测可以帮助评估大肠杆菌培养的稳定性,并及时检测异常情况,如突变或不良的生长特征。有助于及时调整培养条件,保证培养过程的稳定性和一致性,在稳定性评估和过程控制具有重要意义。并且本发明预测大肠杆菌培养产物的产量和趋势。这有助于优化培养策略,提高目标产物的产量和质量。
GA_SCNNR在上述结果测试中,展现出良好的准确性与鲁棒性。本发明共进行了三批次的重组大肠杆菌培养,取前两次的结果作为训练集,用于训练GA_SCNNR框架,模型的多目标训练过程如图14所示。在第三次实验中训练完成的GA_SCNNR用于实时检测过程产生指导第三次培养,同时也进行了离线取样检测用于验证模型结果。
通过前两个批次获取的半监督数据集,训练完成的GA_SCNNR多参数回归模型,在葡萄糖、乳酸、OD_600和目的蛋白的训练中模型的结果表现出较高的准确性和稳定性。然而在铵离子的检测过程中,模型的表现相对较差。
前两批数据已经完成了GA_SCNNR的训练,第三批数据被用于验证。每1.5分钟可以获取一条拉曼光谱,并通过GA_SCNNR模型进行计算。在重组蛋白培养过程中,计算得到的实时参数指标可以在线显示多参数的动态曲线,如图15所示,可用于重组蛋白培养过程中参数实时检测。
在培养过程中,也采用了取样离线检测的方式用于验证模型的准确性。如图16所示。拉曼实时检测在领域的范围内会呈现振荡性性质,引入了五阶多项式曲线拟合的方式使得曲线更为光滑,在实现同一时刻的领域位置参数检测的稳定唯一性。及其重要的是通过迁移学习的重组蛋白预测模型也有较高的准确性如图16(e)所示,第250个样本之前,由于未加入诱导剂重组蛋白的离线检测值也趋近于0,实际预测值也较为平稳的约等于0。第250个样本时刻加入诱导剂,产生重组蛋白模型的实时值也能快速响应。
由此可知,经过实际验证拉曼光谱检测技术与GA_SCNNR的结合,可准确快速的检测出重组大肠杆菌培养过程中的多参数指标。实现培养过程中关键参数检测,并且可以检测出目标产物的含量的数值。
最后说明的是,以上实施例用以说明本发明的技术方案而非限制,尽管参照较佳实施例对本发明进行了详细说明,本领域的普通技术人员应当理解,可以对本发明的技术方案进行修改或者等同替换,而不脱离本发明技术方案的宗旨和范围,其均应涵盖在本发明的权利要求范围当中。
- 一种涡轮叶片热障涂层模拟试验过程中损伤实时检测方法
- 一种高效环保型浮游微生物检测装置及其微生物检测方法
- 在合成气发酵过程中用于在多种底物浓度降低或不存在下维持微生物培养物的方法
- 在合成气发酵过程中用于在多种底物浓度降低或不存在下维持微生物培养物的方法
