基于改进深度神经网络的多模态癌症数据生存分析系统
文献发布时间:2024-04-18 20:01:23
技术领域
本发明涉及癌症患者生存分析的技术领域,尤其是指一种以多种模态数据为依据,通过改进深度神经网络进行数据挖掘,进行癌症患者确诊后生存时间预测的多模态癌症数据生存分析系统。
背景技术
在各类高风险疾病频发的今天,深入研究更加适合患者的个性化治疗方案十分有意义。精准医疗作为一种个性化的疾病预防、诊断和治疗思路,结合了多种生物信息技术,综合考虑每个人在基因、环境等生理状态和个人生活方式方面的个体差异。随着高通量测序技术以及各类生物数据采集设备的快速发展,精准医疗的进步被推向了一个新的阶段。精准医疗的研究中,生存分析是较为重要的研究方向,它可以预测癌症患者确诊后的生存周期,可以客观综合地量化患者的生理状况,是主治医生制定治疗方案的重要参考指标。生存分析可以利用到各种不同类型的数据,即多种模态的数据,例如图片、序列、文本和数值模态等。
病理学图片和放射学影像都属于图片模态,作为有效的诊断工具应用于精准医学。放射学影像由于其非侵入式的特点通常作为疾病诊断的手段,主治医生可借助放射学影像研判病灶的初步情况,宏观把握病患的发病情况;而由活体穿刺手术产生的病理学标本图片则能够更加准确地在微观反应病患的癌症发展情况,亦可用于手术治疗的实时情况参考。上述图片可反映患者癌症发展程度,程度越深则意味着病患的生存时间会越短。
以各类分子组学数据为代表的数据属于序列模态。分子组学是生物学中系统地研究一组分子的领域,被广泛用于研究癌症的发生机制,其背后的遗传学、转录学、表达与生物过程原理与患者的生存时间密切相关。
人口统计学数据、临床记录都属于文本和数值模态。此类数据的特点是数据量小,信息密度大,与癌症生存时间密切相关。年龄、性别、体质指数是最常见的三种人口统计学数据,与病患预后生存情况有密切的联系。临床记录通常指记录在病患电子病历中的疾病史和生活习惯,基础病记录、过敏史、吸烟史等项目也会影响到癌症生存时间的评估。
近些年的研究表明,多模态数据融合可以从多角度挖掘病例的生物医学信息,能够更加准确地完成生存分析等临床任务。但目前存在样本数量少、样本原始数据质量差、维度高、多模态数据异构导致难以进行数据挖掘和融合的应用难题,使得多模态的数据综合利用在生存分析等临床任务时效果不佳,缺少一种有效的面向多模态癌症数据的生存分析系统。
发明内容
本发明的目的在于克服现有技术的缺点与不足,提出一种基于改进深度神经网络的多模态癌症数据生存分析系统,它以癌症确诊患者的原始多模态数据作为输入数据,能够输出对患者的生存预测结果。
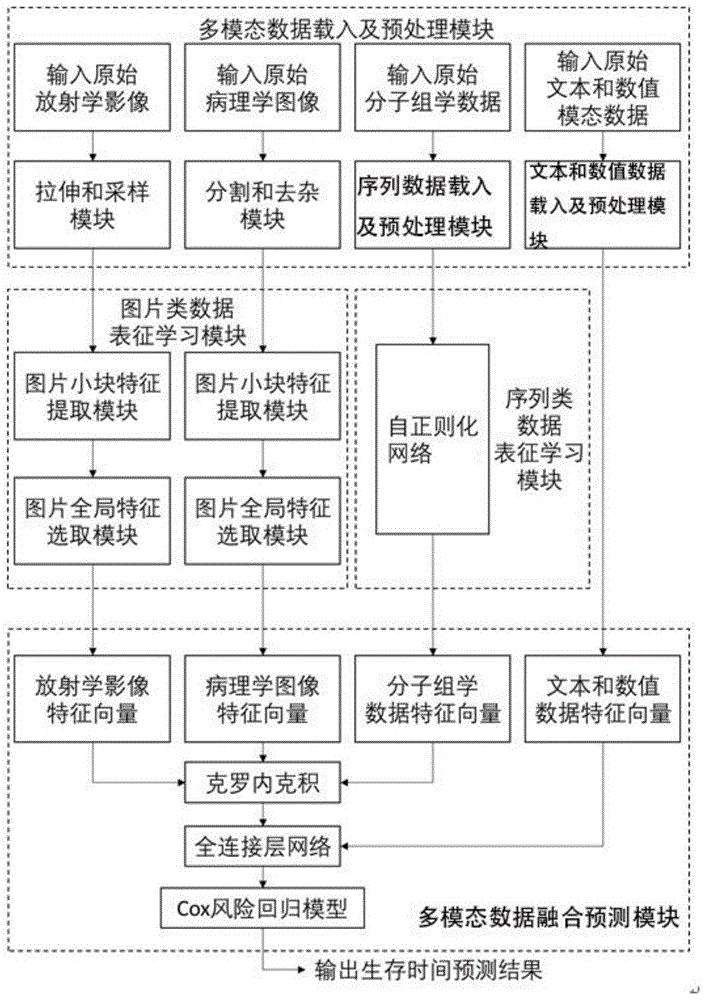
为实现上述目的,本发明所提供的技术方案为:基于改进深度神经网络的多模态癌症数据生存分析系统,它以癌症确诊患者的原始多模态数据作为输入数据,自动进行数据载入和预处理,使用改进深度神经网络分别有针对性地挖掘图片、序列、文本和数值类型的多模态数据的内涵信息,以特征的形式表达出来,使用克罗内克积将多模态数据的特征有机融合,并最终输入到Cox风险回归模型中,完成生存分析的任务,输出对病患生存时间的预测结果,其具体包括以下功能模块:
多模态数据载入及预处理模块,用于导入图片、序列、文本和数值模态数据,并针对导入的数据依据其模态类型进行特定的预处理,针对数据质量差、维度高的难题,能够得到便于使用的预处理后的各模态数据;
图片类数据表征学习模块,即放射学影像和病理学图片表征学习,用于对预处理后的图片类数据进行表征学习,针对模态数据挖掘困难的问题,能够得到图片类数据的特征向量;
序列类数据表征学习模块,即分子组学数据表征学习,用于对预处理后的分子组学数据进行表征学习,针对样本数量少、模态数据挖掘困难的问题,能够得到序列类数据的特征向量;
多模态数据融合预测模块,用于对各模态数据特征向量进行克罗内克积融合和后续全连接层网络,以及使用Cox风险回归模型进行预测,针对多模态数据异构难以融合的问题,能够输出对病患生存时间的预测结果。
进一步,所述多模态数据载入及预处理模块包括图片数据载入及预处理模块、序列数据载入及预处理模块、文本和数值数据载入及预处理模块;
所述图片数据载入及预处理模块包括针对超大像素规模图片的分割和去杂模块、针对3D图片的拉伸和采样模块;所述分割和去杂模块读取超大像素规模图片为RGB矩阵的形式,等距选取规定数值为r
所述序列数据载入及预处理模块包括特征化数据表示模块、共表达特征降维模块;所述特征化数据表示模块利用TCGA数据库读取病患的分子组学数据为特征的形式,具体为病例编号-特征名-特征值的形式,并剔除了在样本比例为p
所述文本和数值数据载入及预处理模块包括数值模态数据载入模块、针对文本信息的转化和载入模块;所述数值模态数据载入模块读取患者的编号和数值模态数据信息,生成数据矩阵,对缺失的数据按空值处理;所述针对文本信息的转化和载入模块按关键词检索病患的非数值文本数据,将病患的文本信息转化为布尔值,即检索到有相关疾病史或生活习惯时值为1,未知或无相关记录则为0;所述文本和数值数据载入及预处理模块汇总数值模态数据载入模块和针对文本信息的转化和载入模块生成的数据,此类数据属于低维数据,无需进行数据挖掘,即可作为文本和数值模态数据特征向量h
进一步,所述图片类数据表征学习模块包括基于改进深度神经网络的图片小块特征提取模块和图片全局特征选取模块;
所述基于改进深度神经网络的图片小块特征提取模块将通用图片处理领域常用的VGG卷积神经网络改进为ResNet网络,对预处理后生成的小图进行表征学习,第l+1层的ResNet网络
其中,
所述图片全局特征选取模块采用两阶段筛选的方法;第一阶段具体是指:设通过图片小块特征提取模块后获得的某一小图的局部生存判别特征向量有N个元素,设X矩阵表示这N个生存判别特征映射,即X=[X
其中,z和w是特征映射的行列长度,随后将特征映射从左至右从上至下排成一行,得到向量
第二阶段具体是指:设一张完整的图片被分成了M个小块,而在上一个阶段已经得到了每一个小块上面每一个局部生存判别特征的得分f
其中,f
进一步,所述序列类数据表征学习模块采用自正则化网络SNN,它用于处理前一阶段传入的经过预处理的低维的特征矩阵的一种网络结构;对于癌症分子组学数据这种训练样本相对少的学习场景,深度神经网络容易过度拟合,与其它类型的神经网络结构相比,前馈网络中的权值是共享的,因此更容易受到低质量样本数据的扰动和优化技术的影响而产生训练的不稳定性;为了减少对低样本量组学数据的过拟合,在训练前馈网络时采用更强大的正则化技术——自正则化网络SNN;在自正则化网络SNN中,用比例指数线性单元SeLU代替校正线性单元ReLU激活,使每一层的输出趋于零均值和单位方差,借此能够训练良好正则化的前馈网络,克服深度神经网络容易过度拟合的难题,避免出现梯度消失或梯度爆炸等的不稳定现象,获得更好的分子组学特征表示;网络主体架构由l层全连接层构成,最终会输出n维的特征信息h
进一步,所述多模态数据融合预测模块对各模态数据特征向量进行克罗内克积融合和后续全连接层网络,以及使用Cox风险回归模型进行预测;所述克罗内克积融合是一种有效的融合异构程度大的多模态数据的方法,它允许各类模态的数据以特征向量的形式融合到一起,并且完全保留原始每个单模态的特征信息,融合后的特征h
融合后的特征h
本发明与现有技术相比,具有如下优点与有益效果:
1、提出了一套完善的系统,该系统以采集到的原始医学数据作为输入,以生存时间临床任务预测结果作为输出,全程由本系统自动完成,无需任何额外的专业人士的标注和挑选,是一种端到端的生物医学预测系统,实现了预测全过程的自动化。
2、本发明提出有效的数据载入和预处理技术,完成了对数据质量差、维度高的多模态原始数据的前期处理工作,使得后续对多模态数据进行数据挖掘和融合成为可能。
3、本发明提出有效的图片类数据表征学习和分子组学数据表征学习,使用改进深度神经网络和自正则化网络,充分挖掘患者多角度的生理信息,克服了样本数量少、多模态数据挖掘困难的难题。
4、本发明将异构的多模态数据转化为高度同构的特征向量,在特征层面通过克罗内克积的方法实现癌症患者多角度生物信息的融合,不丢失单模态的原始信息,克服了多模态数据异构难以融合的难题。
附图说明
图1为本发明系统的架构图。
具体实施方式
下面结合具体实施例对本发明作进一步详细的描述,但本发明的实施方式不限于此。
本实施例公开了一种基于改进深度神经网络的多模态癌症数据生存分析系统,其架构如图1所示,目的是通过融合癌症患者各类形式医学图片、分子组学信息、人口统计学信息和电子病历等多种模态的异构数据来提高对癌症临床生存分析预测的准确度。该系统无需任何额外的专业人士的标注和挑选,是一种端到端的生物医学预测系统,以采集到的原始医学数据作为输入,利用公开数据库和开源软件包进行预处理,通过所提出算法对数据进行有效的表征学习,充分发掘数据中隐含的特征,各模态的特征向量过克罗内克积的方法实现癌症患者多角度生物信息的融合,不丢失单模态的原始信息,能够完成癌症生存分析的临床任务。其具体包括以下功能模块:
多模态数据载入及预处理模块,用于导入图片、序列、文本和数值模态数据,并针对导入的数据依据其模态类型进行特定的预处理,针对数据质量差、维度高的难题,能够得到便于使用的预处理后的各模态数据;
图片类数据表征学习模块,即放射学影像和病理学图片表征学习,用于对预处理后的图片类数据进行表征学习,针对模态数据挖掘困难的问题,能够得到图片类数据的特征向量;
序列类数据表征学习模块,即分子组学数据表征学习,用于对预处理后的分子组学数据进行表征学习,针对样本数量少、模态数据挖掘困难的问题,能够得到序列类数据的特征向量;
多模态数据融合预测模块,用于对各模态数据特征向量进行克罗内克积融合和后续全连接层网络,以及使用Cox风险回归模型进行预测,针对多模态数据异构难以融合的问题,能够输出对病患生存时间的预测结果。
具体地,所述多模态数据载入及预处理模块包括图片数据载入及预处理模块、序列数据载入及预处理模块、文本和数值数据载入及预处理模块。
所述图片数据载入及预处理模块包括针对超大像素规模图片的分割和去杂模块、针对3D图片的拉伸和采样模块;所述分割和去杂模块读取超大像素规模图片为RGB矩阵的形式,等距选取规定数值为r
所述序列数据载入及预处理模块包括特征化数据表示模块、共表达特征降维模块;所述特征化数据表示模块利用TCGA数据库读取病患的分子组学数据为特征的形式,具体为病例编号-特征名-特征值的形式,并剔除了在样本比例为p
所述文本和数值数据载入及预处理模块包括数值模态数据载入模块、针对文本信息的转化和载入模块;所述数值模态数据载入模块读取患者的编号和数值模态数据信息,生成数据矩阵,对缺失的数据按空值处理;所述针对文本信息的转化和载入模块按关键词检索病患的非数值文本数据,将病患的文本信息转化为布尔值,即检索到有相关疾病史或生活习惯时值为1,未知或无相关记录则为0;所述文本和数值数据载入及预处理模块汇总数值模态数据载入模块和针对文本信息的转化和载入模块生成的数据,此类数据属于低维数据,无需进行数据挖掘,即可作为文本和数值模态数据特征向量h
在本实施例中,所述多模态数据载入及预处理模块的具体实施方式。使用公开癌症数据集GBMLGG脑癌(glioma)项目数据作为实施例的数据来源,对本发明所述系统的生存分析预测性能进行评估,从存储设备载入脑癌患者的放射学影像数据(MRI,3D灰度图片),病理学图片(WSI,超大像素规模2D彩色图片),分子组学数据(主要为mRNA-Seq数据和IDH信息)、人口统计学和电子病历数据(主要为性别、年龄、入院时生理状况评估等),样本共769例。
在本实施例中,所述图片数据载入及预处理模块的具体实施方式。读取超大像素规模图片为RGB矩阵的形式,等距选取规定数值为224的行列作为子阵,随后考察其灰度矩阵,设定有效区域占比阈值为0.8,设定均值阈值为230,三是方差阈值30,只有灰度符合阈值的小图会被保留,以达到分割和去杂的目的。读取3D图片为三维数组的形式,使用均值填充的方式统一拉伸为224×224×224的像素尺寸,再沿空间某一特定坐标轴等距采样生成数量为224的224×224的小图,以便后续利用。
在本实施例中,所述序列数据载入及预处理模块的分子组学数据预处理具体实施方式包括利用TCGA数据库读取病患的分子组学数据为特征的形式,具体为病例编号-特征名-特征值的形式,数据维度为769×20531。剔除在80%样本中无表达或缺失数据的特征数据,各余下特征按照样本数量排序截取前5000个特征。利用lmQCM软件包,使用病例编号-特征名-特征值的数据生成共表达特征矩阵,根据软件包统计的相关性截取前200个共表达特征,作为后续算法的输入。
具体地,所述图片类数据表征学习模块包括基于改进深度神经网络的图片小块特征提取模块和图片全局特征选取模块。
所述基于改进深度神经网络的图片小块特征提取模块将通用图片处理领域常用的VGG卷积神经网络改进为ResNet网络,对预处理后生成的小图进行表征学习,第l=1层的ResNet网络
其中,
在本实施例中,使用ResNet对预处理后保留下的小图进行表征学习,放射学影像产生的小图和病理学图片产生的小图的表征学习和特征选择将单独进行,但采用相同的算法。本实施例选择最佳的网络结构ResNet-50,即50层堆叠的ResNet网络,其输出的特征即为小图的局部特征。
所述图片全局特征选取模块,采用两阶段筛选并通过全连接层合并的方法。第一阶段具体是指,设通过表征学习模块获得的某一小图的局部生存判别特征设置为50个,设X矩阵表示这50个生存判别特征映射,即X=[X
其中z和w是特征映射的行列长度,设定为z=w=18,随后将这些特征映射从左至右从上至下排成一行得到向量
第二阶段具体是指,一张完整的组织病理学图片分割筛选后保留前224个得分最高的小图,把所有的小块聚在一起形成全局生存判别特征映射评分矩阵:
其中f
具体地,序列类数据表征学习模块采用自正则化网络SNN,它用于处理前一阶段传入的经过预处理的低维的特征矩阵的一种网络结构;对于癌症分子组学数据这种训练样本相对少的学习场景,深度神经网络容易过度拟合,与其它类型的神经网络结构相比,前馈网络中的权值是共享的,因此更容易受到低质量样本数据的扰动和优化技术的影响而产生训练的不稳定性;为了减少对低样本量组学数据的过拟合,在训练前馈网络时采用更强大的正则化技术——自正则化网络SNN;在自正则化网络SNN中,用比例指数线性单元SeLU代替校正线性单元ReLU激活,使每一层的输出趋于零均值和单位方差,借此能够训练良好正则化的前馈网络,克服深度神经网络容易过度拟合的难题,避免出现梯度消失或梯度爆炸等的不稳定现象,获得更好的分子组学特征表示;网络主体架构由l层全连接层构成,最终会输出n维的特征信息h
在本实施例中,所述序列类数据表征学习模块设定为4层全连接层网络,并在训练前馈网络时采用自正则化网络。在网络中,用比例指数线性单元(SeLU)代替校正线性单元(ReLU)激活,使每一层的输出趋于零均值和单位方差。借此能够训练良好正则化的前馈网络,最终会输出64维的特征信息h
具体地,所述多模态数据融合预测模块对各模态数据特征向量进行克罗内克积(Kronecker product)融合和后续全连接层网络,以及使用Cox风险回归模型进行预测;所述克罗内克积融合是一种有效的融合异构程度大的多模态数据的方法,它允许各类模态的数据以特征向量的形式融合到一起,并且完全保留原始每个单模态的特征信息,融合后的特征h
融合后的特征h
本实施例的软件环境为Ubuntu 20.04.2+Python 3.6.12+Pytorch 1.9.0+CUDA102,769例样本按照8:2的比例随机分成训练集(615例)和测试集(154例),训练过程为避免过拟合采用dropout机制,退出率设置为0.5。经过训练后的系统网络在测试集上进行测试,输出最终的生存分析算法性能评估结果用于网络参数的调整。算法性能评估标准为一致性索引(Concordance index,C-index),它常用于生存分析算法的性能对比,能综合反映生存分析算法在预测样本生存周期时的准确度,其取值范围为[0,1],取值越大越好,其计算公式为:
其中
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
- 一种三元复合驱采油废水的处理及原油回收方法及其回收装置
- 一种机载电磁式无人机回收方法及回收装置
- 一种机载气囊式无人机回收方法及回收装置
- 一种机载吸盘式无人机回收方法及回收装置
- 一种成品电缆破皮回收装置及其回收方法
- 一种电缆金属回收冶炼用的回收装置以及回收方法
