合并的敲入筛选和在内源基因座控制下共表达的异源多肽
文献发布时间:2023-06-19 13:48:08
在先相关申请
本申请要求于2019年3月14日提交的美国临时申请第62/818,535号、2019年3月14日提交的美国临时申请第62/818,578号、2019年7月8日提交的美国临时申请第62/871,309号、2019年7月8日提交的美国临时申请第62/871,467号的权益,所有这些申请都通过引用整体并入本文。
发明背景
免疫细胞疗法已经发展了三十多年。从传统的随机整合病毒基因修饰方法到靶向非病毒整合的演变为进一步释放细胞免疫疗法的潜力提供了广阔的前景。然而,靶向整合所特有的关键工程挑战仍然存在,例如预测不同靶位点的效率和开发高通量筛选平台以快速测试被靶向以插入细胞的基因组基因座中的合并的DNA序列。快速鉴定细胞中的靶向基因组整合的选择有限。
此外,用于治疗用途的离体或活体基因编辑细胞修饰的当前技术集中在校正现有突变、限制由导致功能错误基因的单个突变引起的疾患的治疗适用性、或整合全新的合成基因,需要广泛的研究和开发来产生新的治疗有用的合成DNA序列。因此,基因组修饰的选择有限。鉴于T细胞在过继细胞疗法中的重要性,获得人类T细胞并对其进行修饰以产生具有所需功能的编辑T细胞的能力可有利于过继T细胞疗法的开发和应用。
发明内容
合并的敲入筛选(Pooled Knock-In Screening)
本公开涉及用于鉴定细胞基因组中的靶向插入的组合物和方法。本发明人已经发现了一种合并的敲入筛选方法,以快速测定合并的细胞群体中的许多靶向敲入。通过DNA测序策略可鉴定靶向整合,所述策略选择性地扩增中靶敲入(on-target knockin)(构建体,任选地编码异源多肽,插入所需基因座),同时避免未整合到细胞基因组中的构建体。由于(同源定向修复)HDR模板的同源臂用于与靶基因座进行互补碱基配对,但自身不会复制到靶位点中,因此可将与靶基因组基因座错配的短DNA碱基对区域引入HDR模板侧翼的一个或两个同源臂中。在HDR后不会将错配区域引入靶位点中,从而产生一个通过扩增(例如PCR)容易检测到的序列,该序列对于中靶敲入是独特的(那些未敲入的构建体将含有模板错配,因此不会被扩增)。参见例如图15a。对所得扩增子的测序提供了有关不同敲入的丰度的信息(特定敲入的更多序列表明具有该敲入的细胞相对于其他敲入的丰度更高,从而提供有关敲入在生物系统中的影响的信息)。在一些实施方案中,添加对于每个HDR模板来说独特的条形码能够基于条形码的身份对合并群体中每个单独插入物的丰度进行DNA读出。本文提供的组合物和方法可用于鉴定任何细胞例如T细胞中的靶向基因组整合。例如,如下文所论述,在一些实施方案中,可使用所述方法来测定T细胞受体(TCR)基因座处的不同异源敲入的影响,任选地与内源或异源TCR蛋白共表达为单一蛋白质,其随后自切割以生成单独的异源敲入多肽和内源或异源TCR蛋白。相同的策略可应用于细胞中任何所需的基因座。
本文提供了一种用于鉴定细胞基因组中的靶向插入的方法。在一些实施方案中,所述方法包括(a)向细胞群体中引入(i)切割所述细胞基因组中的靶区域以产生靶插入位点的靶向核酸酶;和(ii)序列彼此不同的多个DNA模板,其中每个DNA模板包含:i.异源编码或非编码核酸序列;ii.指示所述异源编码或非编码核酸序列的身份的独特条形码核苷酸序列;和iii.共同引物结合序列,其中每个DNA模板的5′端和3′端包含与所述插入位点侧翼的基因组序列同源的核苷酸序列,并且其中一个或两个同源核苷酸序列与基因组序列中的同源序列相比包含错配核苷酸序列,其中所述错配核苷酸序列在重组期间不插入到靶插入位点中;(b)允许重组发生,从而产生修饰细胞群体;(c)用一对引物从所述细胞扩增DNA以形成扩增的DNA,其中第一引物与所述共同引物结合序列互补,并且其中第二引物与插入位点侧翼的基因组序列中的同源序列结合并且不与DNA模板中的错配核苷酸序列结合;或者其中第一引物与插入位点侧翼的5′基因组区域中的第一同源序列结合并且不与DNA模板中与第一同源序列在相同位置的错配序列结合,而第二引物与插入位点侧翼的3′基因组区域结合并且不与DNA模板中与第二同源序列在相同位置的错配核苷酸序列结合;以及(f)对所述扩增的DNA进行测序以鉴定插入细胞的靶插入位点中的DNA模板。
在一些实施方案中,所述错配核苷酸序列的长度为约3至40个核苷酸。在一些实施方案中,所述条形码序列在扩增的DNA中并且被测序。
在一些实施方案中,所述方法还包括确定所述群体中具有插入靶插入位点中的不同DNA模板的细胞的相对数量。在一些实施方案中,所述方法还包括向修饰细胞群体施加选择压力。
在一些实施方案中,所述方法还包括比较在对细胞施加选择压力之前和之后所述群体中具有插入靶插入位点中的不同DNA模板的细胞的相对数量。
在一些实施方案中,通过将包含DNA模板的病毒载体引入细胞中来插入DNA模板。
在一些实施方案中,所述群体是哺乳动物细胞群体。在一些实施方案中,所述哺乳动物细胞是人类细胞。在一些实施方案中,所述人类细胞是T细胞、B细胞、自然杀伤(NK)细胞、髓系细胞或其他免疫细胞。在一些实施方案中,所述T细胞是调节性T细胞、效应T细胞或原初T细胞。在一些实施方案中,所述效应T细胞是CD8+T细胞或CD4+T细胞。在一些实施方案中,所述效应T细胞是CD8+CD4+T细胞。在一些实施方案中,所述细胞是原代细胞。
在一些实施方案中,所述DNA模板包含编码异源多肽的核酸。在一些实施方案中,所述DNA模板包含本文所述的核酸构建体中的任一者。
在一些实施方案中,所述靶插入位点在TCR-α亚基恒定基因(TRAC)的外显子1或TCR-β亚基恒定基因(TRBC)的外显子1中。在一些实施方案中,所述基因组序列是人T细胞TCR基因座序列。
在一些实施方案中,所述靶向核酸酶选自由以下组成的组:RNA指导的核酸酶结构域、转录激活因子样效应物核酸酶(TALEN)、锌指核酸酶(ZFN)和megaTAL。在一些实施方案中,将靶向核酸酶、指导RNA和DNA模板作为核糖核蛋白复合物(RNP)-DNA模板复合物引入细胞中,其中所述RNP-DNA模板复合物包含:(i)RNP,其中所述RNP包含靶向核酸酶和指导RNA;和(ii)DNA模板。
本文还提供了一种包含编码多肽的编码核苷酸序列的核酸构建体,其中每个DNA模板的5′端和3′端包含与细胞基因组中的插入位点侧翼的基因组序列同源的核苷酸序列,其中一个或两个同源核苷酸序列与细胞中的同源基因组序列相比包含错配核苷酸序列;并且其中所述错配核苷酸序列的长度足以防止与对应于错配核苷酸序列的基因组序列特异性结合的引物的结合。
在一些实施方案中,所述编码核苷酸序列包含由自切割肽的编码序列的编码序列连接的两个异源编码序列。在一些实施方案中,所述错配核苷酸序列的长度为约3至约40个核苷酸。在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)多肽;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中所述核酸构建体包含条形码序列,其中插入序列是T细胞的TCR基因座,其中一个或两个同源核苷酸序列包含错配核苷酸序列,并且其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)多肽;(iii)第二自切割肽序列;(iv)第一异源TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中所述核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座,其中一个或两个同源核苷酸序列包含错配核苷酸序列,并且其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)第二异源TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;(v)第三自切割肽序列;(vi)多肽;和(vii)第四自切割肽序列或polyA序列,其中所述核酸构建体包含条形码序列,插入序列是人T细胞的TCR基因座,其中一个或两个同源核苷酸序列包含错配核苷酸序列,并且其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)合成抗原受体;(iii)第二自切割肽序列;(iv)异源多肽;和(v)第三自切割肽序列或polyA序列,其中所述核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)多肽;(iii)第二自切割肽序列;(iv)合成抗原受体;和(v)第三自切割肽序列或polyA序列,其中所述核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一TCRβ或α亚基链,其中所述TCR亚基链包含TCR亚基链的可变区和恒定区;(iii)第二自切割肽序列;(iv)第二TCRβ或α亚基链,其中所述第二TCR亚基链不同于所述第一TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;或者所述TCR亚基包含所述亚基的可变区;和(v)第三自切割肽序列或polyA序列,其中所述核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,所述核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)合成抗原受体;和(v)第二自切割肽序列或polyA序列,其中所述核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,所述核酸构建体编码合成抗原受体,其中所述合成抗原受体是嵌合抗原受体(CAR)或SynNotch受体。
在一些实施方案中,本文所述的核酸构建体中的任一者包含指示多肽身份的条形码序列。在一些实施方案中,所述核酸构建体包含一对独特条形码,其在编码多肽的核苷酸序列的侧翼(即,条形码序列位于编码多肽的核苷酸序列的任一侧,其中每个条形码具有不同的序列)。在一些实施方案中,一个或多个条形码位于自切割肽序列或polyA序列之前、之后或之中。
在一些实施方案中,所述核酸构建体包含一个或多个将核酸构建体的组分隔开的接头序列。在一些实施方案中,一个或多个接头序列具有相同的序列。
还提供了包含两个或更多个本文所述的核酸构建体的文库,其中每个构建体编码不同的多肽。
还提供了包含本文所述的任何文库的细胞群体。进一步提供了包含一种或多种本文所述的核酸构建体的细胞。在一些实施方案中,所述细胞是人T细胞。
还提供了一种用于确定具有特定DNA模板的细胞的转录组的方法,所述方法包括:
(a)向细胞群体中引入
(i)切割所述细胞基因组中的靶区域以产生靶插入位点的靶向核酸酶;和
(ii)序列彼此不同的多个DNA模板,其中每个DNA模板包含:
i.异源编码或非编码核酸序列;
ii.指示所述异源编码或非编码核酸序列的身份的独特条形码核苷酸序列;和
iii.共同引物结合序列,
其中每个DNA模板的5′端和3′端包含与所述靶插入位点侧翼的基因组序列同源的核苷酸序列,并且其中无一个、一个或两个同源核苷酸序列与基因组序列中的同源序列相比包含错配核苷酸序列,其中所述错配核苷酸序列在重组期间不插入到靶插入位点中;
(b)允许重组发生,从而产生修饰细胞群体;
(c)在所述引入、所述允许或两者之前或之后,将细胞分成多个分区,其中至少大多数分区含有单个细胞;
(d)在所述分区中,通过延伸与所述细胞中的mRNA互补的寡核苷酸来从所述mRNA生成cDNA,其中所述寡核苷酸包含分区特异性条形码,从而形成与分区特异性条形码连接的cDNA的池;
(e)组合所述分区的内含物以形成来自多个细胞的cDNA混合物;
(f)从所述cDNA混合物的第一等分试样,至少扩增所述cDNA的包含所述独特条形码和所述分区特异性条形码的双条形码部分;
(g)对所述双条形码部分进行核苷酸测序以生成包含所述独特条形码和所述分区特异性条形码的测序读段;
(h)从所述cDNA池的第一等分试样或第二等分试样,进行对所述池中的cDNA的核苷酸测序,从而生成包含分区特异性条形码和来自多个cDNA的序列的测序读段,
(i)基于双条形码部分测序读段将独特条形码序列与分区特异性条形码序列相关联,从而形成特定DNA模板与分区特异性条形码的关联;以及
(j)使用(i)的所述关联将来自第二等分试样的测序读段与特定模板相关联,从而提供具有特定DNA模板的细胞的转录组。
在一些实施方案中,分区的内含物在执行之前和扩增之前或之后被组合。
在一些实施方案中,所述方法还包括确定所述群体中具有插入靶插入位点中的不同DNA模板的细胞的相对数量。
在一些实施方案中,所述方法还包括向所述修饰细胞群体施加选择压力。
在一些实施方案中,所述方法还包括比较在对细胞施加选择压力之前和之后所述群体中具有插入靶插入位点中的不同DNA模板的细胞的相对数量。
在一些实施方案中,通过将包含DNA模板的病毒载体引入细胞中来插入DNA模板。
在一些实施方案中,所述群体是哺乳动物细胞群体。在一些实施方案中,所述哺乳动物细胞是人类细胞。
在一些实施方案中,所述人类细胞是T细胞、B细胞、自然杀伤(NK)细胞、髓系细胞或其他免疫细胞。
在一些实施方案中,所述T细胞是调节性T细胞、效应T细胞或原初T细胞。
在一些实施方案中,所述效应T细胞是CD8+T细胞或CD4+T细胞。
在一些实施方案中,所述效应T细胞是CD8+CD4+T细胞。
在一些实施方案中,所述细胞是原代细胞。
在一些实施方案中,所述DNA模板包含编码异源多肽的核酸。
在一些实施方案中,所述靶插入位点在TCR-α亚基恒定基因(TRAC)的外显子1或TCR-β亚基恒定基因(TRBC)的外显子1中。
在一些实施方案中,所述基因组序列是人T细胞TCR基因座序列。
在一些实施方案中,所述靶向核酸酶选自由以下组成的组:RNA指导的核酸酶结构域、转录激活因子样效应物核酸酶(TALEN)、锌指核酸酶(ZFN)和megaTAL。
在一些实施方案中,将靶向核酸酶、指导RNA和DNA模板作为核糖核蛋白复合物(RNP)-DNA模板复合物引入细胞中,其中所述RNP-DNA模板复合物包含:(i)RNP,其中所述RNP包含靶向核酸酶和指导RNA;和(ii)DNA模板。
在内源基因座控制下共表达的异源多肽
本公开还涉及用于修饰T细胞基因组的组合物和方法。本发明人已发现可修饰人T细胞以改变T细胞特异性和功能。通过将编码多肽和异源T细胞受体(TCR)或合成抗原受体(例如嵌合抗原受体(CAR))的核酸插入T细胞基因组中的特定内源位点(例如,TCR基因座),可制备具有所需的TCR或CAR抗原特异性和多肽功能的人T细胞。此外,本文所述的组合物和方法可用于生成具有改变的特异性和功能性的人T细胞,同时限制与T细胞疗法相关的副作用。
本文提供异源表达多肽的人T细胞,其中所述多肽由插入细胞TCR基因座中的核酸构建体编码。在一些实施方案中,所述多肽是截短的人PD-1蛋白,其包含人PD-1胞外结构域和跨膜结构域并且缺少80-90个(例如87个)羧基末端PD-1氨基酸。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人4-1BB胞内结构域的人PD-1胞外结构域或其至少120或130个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个(例如11个)氨基酸)。在一些实施方案中,所述多肽包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地PD-1胞内结构域的1-10个氨基酸)的人PD-1胞外结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人ICOS胞内结构域的人PD-1胞外结构域。在一些实施方案中,所述跨膜结构域是人ICOS或PD-1跨膜结构域。
在一些实施方案中,所述多肽是截短的人CTLA4蛋白,其包含人CTLA4胞外结构域和跨膜结构域并且缺少30-40个(例如34个)羧基末端CTLA4氨基酸。在一些实施方案中,所述截短的人CTLA4蛋白包含人CTLA4胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人CTLA4蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地CTLA4胞内结构域的1-10个氨基酸)的人CTLA4胞外结构域。
在一些实施方案中,所述多肽是截短的人CD200R蛋白,其包含人CD200R胞外结构域和跨膜结构域并且缺少50-60个羧基末端CD200R氨基酸。在一些实施方案中,所述截短的人CD200R蛋白包含人CD200R胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人CD200R蛋白胞内结构域。
在一些实施方案中,所述多肽是截短的人BTLA蛋白,其包含人BTLA胞外结构域和跨膜结构域并且缺少100-110个(例如104个)羧基末端BTLA氨基酸。在一些实施方案中,所述截短的人BTLA蛋白包含人BTLA胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人BTLA蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人CD28胞内结构域的人BTLA胞外结构域或其至少110或120个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)。
在一些实施方案中,所述多肽是截短的人TIM-3蛋白,其包含人TIM-3胞外结构域和跨膜结构域并且缺少65-75个(例如71个)羧基末端TIM-3氨基酸。在一些实施方案中,所述截短的人TIM-3蛋白包含人TIM-3胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人TIM-3蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人CD28胞内结构域的人TIM-3胞外结构域或其至少160或170个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)。
在一些实施方案中,所述多肽是截短的人TIGIT蛋白,其包含人TIGIT胞外结构域和跨膜结构域并且缺少70-80个(例如75个)羧基末端TIGIT氨基酸。在一些实施方案中,所述截短的人TIGIT蛋白包含人TIGIT胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人TIGIT蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人CD28胞内结构域的人TIGIT胞外结构域或其至少100或110个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)。在一些实施方案中,所述跨膜结构域是人CD28或TIGIT跨膜结构域。
在一些实施方案中,所述多肽是截短的人TGFβR2蛋白,其包含人TGFβR2胞外结构域和跨膜结构域并且缺少360-370个(例如366个)羧基末端TGFβR2氨基酸。在一些实施方案中,所述截短的人TGFβR2蛋白包含人TGFβR2胞内结构域的前1-20个(例如13个)氨基酸但缺少剩余的人TGFβR2蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人4-1BB胞内结构域的人TGFβR2胞外结构域或其至少130或140个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个氨基酸)。在一些实施方案中,所述跨膜结构域是人4-1BB或TGFβR2跨膜结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人Myd88胞内结构域或其至少90或100个氨基酸的部分(和任选地TGFβR2胞内结构域的1-20个氨基酸)的人TGFβR2胞外结构域。
在一些实施方案中,所述多肽包含截短的人IL-10RA蛋白,其包含人IL-10RA胞外结构域和跨膜结构域并且缺少310-320个(例如315个)羧基末端IL-10RA氨基酸。在一些实施方案中,所述截短的人IL-10RA蛋白包含人IL-10RA胞内结构域的前1-20个(例如13个)氨基酸但缺少剩余的人IL-10RA蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-10RA胞外结构域。在一些实施方案中,所述跨膜结构域包含人IL-7RA或IL-10RA跨膜结构域或其至少20个氨基酸长的部分。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-4RA胞外结构域。在一些实施方案中,所述跨膜结构域包含人IL-7RA或IL-4RA跨膜结构域或其至少20个氨基酸长的部分。
在一些实施方案中,所述多肽是包含人Fas胞外结构域和跨膜结构域并且缺少132-142个(例如138个)羧基末端Fas氨基酸的截短的人Fas蛋白。在一些实施方案中,所述截短的人Fas蛋白包含人Fas胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人Fas蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,所述跨膜结构域是人Fas或CD28跨膜结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人41BB胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,所述多肽包含SEQ ID NO:62或由SEQ ID NO:62组成。在一些实施方案中,所述跨膜结构域是人Fas或MyD88跨膜结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人ICOS胞内结构域或其至少25或35个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,所述跨膜结构域是人Fas或ICOS跨膜结构域。
在一些实施方案中,所述多肽是截短的人TRAIL-R2蛋白,其包含人TRAIL-R2胞外结构域和跨膜结构域并且缺少196-206个(例如202个)羧基末端TRAIL-R2氨基酸。在一些实施方案中,所述截短的人TRAIL-R2蛋白包含人TRAIL-R2胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人TRAIL-R2蛋白胞内结构域。
在一些实施方案中,所述多肽包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地TRAIL-R2胞内结构域的1-20个氨基酸)的人TRAIL-R2胞外结构域。在一些实施方案中,所述跨膜结构域是人TRAIL-R2或CD28跨膜结构域。在一些实施方案中,所述多肽包含全长CCR10、MCT4、SOD1、TCF7、IL-2RA、IL-7RA或41BB蛋白。
在一些实施方案中,所述T细胞异源表达包含与选自由以下组成的组的氨基酸序列具有至少95%同一性的氨基酸序列的多肽:SEQ ID NO:42、SEQ ID NO:45、SEQ ID NO:47、SEQ ID NO:50、SEQ ID NO:51、SEQ ID NO:52、SEQ ID NO:56、SEQ ID NO:57、SEQ IDNO:59、SEQ ID NO:61、SEQ ID NO:62、SEQ ID NO:63、SEQ ID NO:67和SEQ ID NO:69,如表3所示。
在一些实施方案中,所述靶插入位点在TCR-α亚基恒定基因(TRAC)的外显子1中。在一些实施方案中,所述靶插入位点在TCR-β亚基恒定基因(TRBC)的外显子1中。
在一些实施方案中,所述异源核酸构建体包含与选自由以下组成的组的核酸序列具有至少95%同一性的核酸序列:SEQ ID NO:6、SEQ ID NO:9、SEQ ID NO:11、SEQ ID NO:14、SEQ ID NO:15、SEQ ID NO:16、SEQ ID NO:20、SEQ ID NO:21、SEQ ID NO:23、SEQ IDNO:25、SEQ ID NO:26、SEQ ID NO:27、SEQ ID NO:31和SEQ ID NO:33,如表3所示。
在一些实施方案中,所述T细胞表达识别靶抗原的抗原特异性T细胞受体(TCR)。在一些实施方案中,所述T细胞是调节性T细胞、效应T细胞或原初T细胞。在一些实施方案中,所述效应T细胞是CD8+T细胞或CD4+T细胞。在一些实施方案中,所述效应T细胞是CD8+CD4+T细胞。在一些实施方案中,所述T细胞是原代细胞。
在一些实施方案中,所述异源核酸构建体编码(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)本文所述的任何多肽;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中,如果细胞的内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果细胞的内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,由所述核酸构建体编码的多肽序列与选自由以下组成的组的氨基酸序列具有至少95%同一性:SEQ ID NO:42、SEQ ID NO:45、SEQ ID NO:47、SEQ IDNO:50、SEQ ID NO:51、SEQ ID NO:52、SEQ ID NO:56、SEQ ID NO:57、SEQ ID NO:59、SEQ IDNO:61、SEQ ID NO:62、SEQ ID NO:63、SEQ ID NO:67和SEQ ID NO:69。
还提供了包含编码多肽的核酸序列的核酸,所述多肽包含与选自由以下组成的组的蛋白质具有至少95%同一性的氨基酸序列:SEQ ID NO:38、SEQ ID NO:40、SEQ ID NO:45、SEQ ID NO:46、SEQ ID NO:47、SEQ ID NO:48、SEQ ID NO:49、SEQ ID NO:51、SEQ IDNO:52、SEQ ID NO:53、SEQ ID NO:54、SEQ ID NO:60、SEQ ID NO:61和SEQ ID NO:62、SEQID NO:63、SEQ ID NO:64和SEQ ID NO:65。
在一些实施方案中,所述核酸构建体包含与人TCR基因座具有同源性的侧翼同源臂序列。
还提供了包含本文所述的任何核酸构建体的T细胞。
进一步提供了一种核酸构建体,其按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中所述TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)与选自由以下组成的组的氨基酸序列具有至少95%同一性的多肽序列:SEQID NO:42、SEQ ID NO:45、SEQ ID NO:47、SEQ ID NO:50、SEQ ID NO:51、SEQ ID NO:52、SEQID NO:56、SEQ ID NO:57、SEQ ID NO:59、SEQ ID NO:61、SEQ ID NO:62、SEQ ID NO:63、SEQID NO:67和SEQ ID NO:69;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源T细胞TCR亚基的N末端的一部分,其中,如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,所述核酸构建体包含与选自由以下组成的组的核酸序列具有至少95%同一性的核酸序列:SEQ ID NO:42、SEQ ID NO:45、SEQ ID NO:47、SEQ ID NO:50、SEQ ID NO:51、SEQID NO:52、SEQ ID NO:56、SEQ ID NO:57、SEQ ID NO:59、SEQID NO:61、SEQ ID NO:62、SEQ ID NO:63、SEQ ID NO:67和SEQ ID NO:69。
还提供了一种修饰人T细胞的方法,所述方法包括(a)向所述人T细胞中引入(i)切割人T细胞的TCR基因座中的靶区域以在细胞基因组中产生靶插入位点的靶向核酸酶;和(ii)编码多肽的核酸构建体,多肽选自由以下组成的组:包含人PD-1胞外结构域和跨膜结构域并且缺少80-90个(例如87个)羧基末端PD-1氨基酸的截短的人PD-1蛋白;包含经由跨膜结构域连接至人4-1BB胞内结构域的人PD-1胞外结构域或其至少120或130个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个(例如11个)氨基酸)的多肽;包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地PD-1胞内结构域的1-10个氨基酸)的人PD-1胞外结构域的多肽;包含经由跨膜结构域连接至人ICOS胞内结构域的人PD-1胞外结构域的多肽;包含人CTLA4胞外结构域和跨膜结构域并且缺少30-40个(例如34个)羧基末端CTLA4氨基酸的截短的人CTLA4蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地CTLA4胞内结构域的1-10个氨基酸)的人CTLA4胞外结构域的多肽;包含人CD200R胞外结构域和跨膜结构域并且缺少50-60个羧基末端CD200R氨基酸的截短的人CD200R蛋白;包含人BTLA胞外结构域和跨膜结构域并且缺少100-110个(例如104个)羧基末端BTLA氨基酸的截短的人BTLA蛋白。在一些实施方案中,所述截短的人BTLA蛋白包含人BTLA胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人BTLA蛋白胞内结构域;包含经由跨膜结构域连接至人CD28胞内结构域的人BTLA胞外结构域或其至少110或120个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TIM-3胞外结构域和跨膜结构域并且缺少65-75个(例如71个)羧基末端TIM-3氨基酸的截短的人TIM-3蛋白;包含经由跨膜结构域连接至人CD28胞内结构域的人TIM-3胞外结构域或其至少160或170个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TIGIT胞外结构域和跨膜结构域并且缺少70-80个(例如75个)羧基末端TIGIT氨基酸的截短的人TIGIT蛋白;包含经由跨膜结构域连接至人CD28胞内结构域的人TIGIT胞外结构域或其至少100或110个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TGFβR2胞外结构域和跨膜结构域并且缺少360-370个(例如366个)羧基末端TGFβR2氨基酸的截短的人TGFβR2蛋白;包含经由跨膜结构域连接至人4-1BB胞内结构域的人TGFβR2胞外结构域或其至少130或140个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个氨基酸)的多肽;包含经由跨膜结构域连接至人Myd88胞内结构域或其至少90或100个氨基酸的部分(和任选地TGFβR2胞内结构域的1-20个氨基酸)的人TGFβR2胞外结构域的多肽;包含人IL-10RA胞外结构域和跨膜结构域并且缺少310-320个(例如315个)羧基末端IL-10RA氨基酸的截短的人IL-10RA蛋白;包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-10RA胞外结构域的多肽;包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-4RA胞外结构域的多肽;包含人Fas胞外结构域和跨膜结构域并且缺少132-142个(例如138个)羧基末端Fas氨基酸的截短的人Fas蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人4-1BB胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人ICOS胞内结构域或其至少25或35个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含人TRAIL-R2胞外结构域和跨膜结构域并且缺少196-206个(例如202个)羧基末端TRAIL-R2氨基酸的截短的人TRAIL-R2蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地TRAIL-R2胞内结构域的1-20个氨基酸)的人TRAIL-R2胞外结构域的多肽;和包含IL2RA蛋白、IL7RA蛋白、MCT4蛋白或TCF7蛋白的多肽;以及(b)允许重组发生,从而将所述核酸构建体插入靶插入位点中以生成修饰的人T细胞。
在一些方法中,所述靶插入位点在TCR-α亚基恒定基因(TRAC)的外显子1中或在TCR-β亚基恒定基因(TRBC)的外显子1中。在一些方法中,通过将包含核酸构建体的病毒载体引入细胞中来插入核酸构建体。在一些方法中,所述靶向核酸酶选自由以下组成的组:RNA指导的核酸酶结构域、转录激活因子样效应物核酸酶(TALEN)、锌指核酸酶(ZFN)和megaTAL。在一些方法中,将靶向核酸酶、指导RNA和DNA模板作为核糖核蛋白复合物(RNP)-DNA模板复合物引入细胞中,其中所述RNP-DNA模板复合物包含:(i)RNP,其中所述RNP包含靶向核酸酶和指导RNA;和(ii)核酸构建体。
在一些方法中,所述T细胞表达识别靶抗原的抗原特异性T细胞受体(TCR)。在一些实施方案中,所述T细胞是调节性T细胞、效应T细胞或原初T细胞。在一些实施方案中,所述效应T细胞是CD8+T细胞或CD4+T细胞。在一些实施方案中,所述效应T细胞是CD8+CD4+T细胞。在一些实施方案中,所述T细胞是原代细胞。
还提供了通过本文所述的任何方法产生的修饰的T细胞。
进一步提供了一种增强人类受试者中的免疫应答的方法,所述方法包括施用本文所述的任何T细胞。在一些实施方案中,所述T细胞表达识别受试者中的靶抗原的抗原特异性TCR。在一些实施方案中,所述人类受试者患有癌症并且所述靶抗原是癌症特异性抗原。在一些实施方案中,所述人类受试者患有自身免疫性病症并且所述抗原是与所述自身免疫性病症相关的抗原。在一些实施方案中,所述受试者患有感染并且所述靶抗原是与所述感染相关的抗原。在一些实施方案中,所述T细胞是自体的。在一些实施方案中,所述T细胞是同种异体的。
附图说明
本申请包括以下附图。所述附图旨在说明组合物和方法的某些实施方案和/或特征,并补充组合物和方法的任何描述。所述附图不限制组合物和方法的范围,除非书面说明明确指出情况如此。
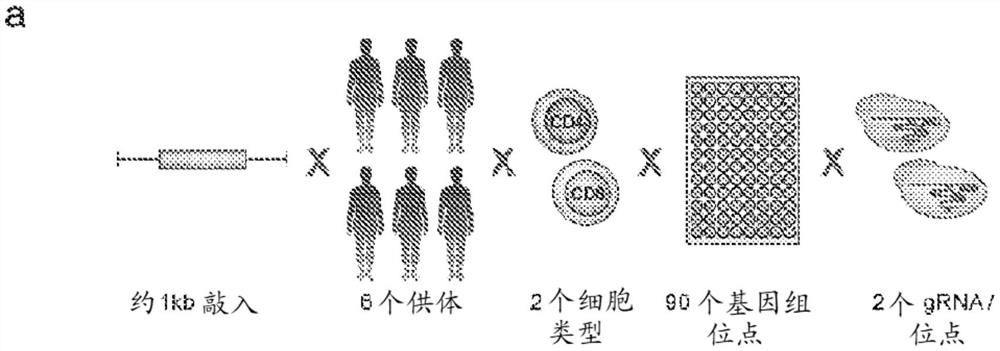
图1a-1f显示跨内源基因座的阵列敲入揭示了原代人T细胞中有效非病毒基因靶向的规则。(a)进行阵列敲入筛选,将大型DNA模板(GFP或tNGFR,约800bp)靶向整合到91个独特的基因组位点。对于每个位点选择了两个gRNA,并通过在来自6个独特健康人类供血者的CD4和CD8 T细胞中执行阵列敲入来测定细胞类型和生物供体之间的差异。(b)靶基因表达、靶位点可及性、gRNA切割效率和敲入百分比观测值的阵列敲入筛选时间线和读出。RNA-Seq在第0天(活化前)、第2天(电穿孔时间)和第4天(扩增期间)进行。ATAC-Seq在第0天和第2天进行。确定每个指导物的实际切割效率的扩增子测序在第6天进行(使用单独的仅RNP板,其中没有对HDR模板进行电穿孔)。在第6天(对于没有第二次刺激的样品(“-刺激”))或在第7天(对于第二次刺激后24小时的样品(“+刺激”),通过流式细胞术在细胞水平上分析了GFP或tNGFR表达的实际敲入百分比。(c)在90个独特基因组靶位点敲入大型HDR模板的敲入百分比观测值,每个位点测试两个gRNA。观测到广泛范围的敲入效率,从低于CX3CR1和ELOB等基因的可检测水平到B2M、IL2RA、RAB11A和STAT1的供体中的平均约50%。在各测试位点,中靶RNP的敲入观测值比不特异于任何人类基因组序列的乱序RNP要高得多。(d)gRNA和靶基因组位点参数与敲入百分比观测值的相关性。切割位点和整合位点之间的相对距离(“切割距离”)或Cas9相对于整合位点的定向(“切割方向”)的相关性很小(尽管主要使用切割距离<约25bp的仅gRNA)。与gRNA切割分数预测值相比,每个gRNA的NHEJ切割%实际观测值(在不存在HDRT的情况下)与敲入%观测值(当包括HDRT时)更相关。靶基因的RNA表达与敲入%更相关,尤其是在更接近蛋白质水平读出的日子里(应注意,由于敲入GFP或tNGFR的表达是由每个基因内源启动子驱动的,因此实际敲入%可能比低表达基因的敲入%观测值更高)。gRNA切割位点的DNA可及性与敲入百分比观测值类似地相关。(e)gRNA和靶基因组位点参数的多元线性回归比任何单个参数更能预测敲入%观测值。基因组位点、gRNA和细胞类型(CD4或CD8)的每种组合的敲入%预测值相对于平均敲入%观测值(n=6个独特供体的平均值)进行绘图。(f)检查多元线性回归模型对单个参数的加权,揭示了gRNA观测切割效率、靶基因的RNA表达水平和gRNA靶位点的DNA可及性对敲入%观测值有很大的独立贡献。因此,在原代T细胞中大量敲入的理想靶基因组基因座高度表达,在电穿孔时可及,并且含有有效切割的gRNA的靶序列。
图2a-2g显示了基因工程化内源蛋白(GEEP)及其特性。(a)我们验证在内源基因座处对细胞表面蛋白进行工程化的所有不同方法的示意性描述。在任何给定的细胞表面蛋白的基因座内,我们可修饰(从左到右)5′非编码区以超越利用合成/外源启动子进行的内源基因调控,添加或置换在特定内源启动子下表达的蛋白质,通过将编码新型胞外结构域的序列靶向编码跨膜结构域的外显子来置换受体特异性,并通过敲入新的信号传导结构域来改变受体的信号传导。(b)为了测试我们是否可通过敲入合成启动子来调整基因表达,我们将SFFV启动子靶向IL2RA和PDCD1的5′非编码区。当我们在电穿孔后7天通过流式细胞术分析未经再刺激培养的编辑T细胞时,我们看到成功的敲入导致任一蛋白质的持续表达。(顶部)我们表明,在针对IL2RA的中靶条件(IL2RA RNP+SFFV HDR DNA模板)下编辑的T细胞保持CD25的高表达,而在对照条件(乱序RNP+SFFV HDR DNA模板)下编辑的T细胞可见CD25表达水平返回到基线。(底部)类似地,在针对PDCD1的中靶条件(PDCD1 RNP+SFFV HDR DNA模板)下编辑的T细胞保持高水平的PD1,而在对照条件下编辑的T细胞可见PD1表达水平返回到基线。(c)为了测试我们是否可将合成产物置于内源启动子的调控之下,我们将编码tNGFR的插入物和2A序列或PolyA尾靶向PD1的N末端编码区,以使得tNGFR将在PD1启动子的调控下分别在有或没有PD1的情况下表达。当我们再刺激经编辑的T细胞并在48小时后通过流式细胞术对其进行分析时,我们看到PD1和tNGFR与tNGFR-2A插入物(顶部)的高度共表达以及tNGFR和PD1 KO与tNGFR-PolyA插入物的高度表达(底部)。(d)为了测试我们是否可改变受体的胞外特异性,我们测试了是否可改变TCR特异性。使用先前描述的靶向策略,我们能够以非常高的敲入效率将1G4 TCR受体敲入内源TRAC基因座中。(e)为了测试我们是否可敲入额外的或替代的信号传导结构域来产生合成信号级联,我们设计了将CD28或41BB胞内结构域结合到CD3亚基之一的C末端的构建体。为了更容易读出胞内结构域敲入,我们还在构建体中包括了以2A序列开头的荧光蛋白,作为成功敲入的标志物。成功的整合将产生表达CD3链的多顺反子序列,该CD3链包含新的信号传导结构域,其同时融合到C末端和荧光蛋白。(顶部)我们展示了CD28胞内结构域在CD3ε的C末端的成功整合,如通过GFP+细胞的百分比来衡量。(底部)此外,我们展示了41BB胞内结构域在CD3ε的C末端的成功整合,如通过mCherry+细胞的百分比来衡量。(f)为了测试将合成产物置于内源启动子之下是否真正模拟了相应的内源蛋白质的表达动力学,我们在5天的过程中通过流式细胞术分析了具有敲入IL2RAN末端的tNGFR-2A的T细胞,并将tNGFR表达动力学与IL2RA进行比较。在CD8和CD4子集中,在不存在再刺激的情况下,IL2RA和tNGFR表达均随时间的推移而降低。类似地,在再刺激的细胞中,CD8和CD4细胞同时可见IL2RA和tNGFR的上调。(g)测试合成CD3信号传导优势的竞争性混合增殖测定的结果。我们将未分选的编辑T细胞与CD28IC-2A-GFP、41BBIC-2A-mCherry或2A-BFP敲入合并到相同的CD3复合物成员的基因座。然后我们在无刺激、仅CD3刺激、仅CD28刺激或CD3/CD28刺激的情况下培养混合细胞群体。培养4天后,通过流式细胞术分析样品中GFP+和mCherry+亚群相对于BFP+亚群的相对生长。然后我们将比例相对于在相应的未刺激条件下测得的比例归一化。
图3a-3e显示了T细胞特异性和功能的同时工程化。(a)我们用于将新的置换TCR和另一种目标蛋白质同时框内整合在内源TCR-α基因座的策略的示意图描述。我们设计了一种单一HDR DNA模板,其(按顺序)包括Furin-间隔子-T2A序列、新的全长TCR-β链的序列、Furin-间隔子-E2A序列、目标蛋白质的序列、Furin-间隔子-P2A序列,以及TCR-α链的新可变区的序列。这些外源序列的侧翼是与内源TCR-α基因座外显子1区域同源的同源臂。成功的敲入产生了表达三种独立蛋白质的多顺反子mRNA。(b)来自我们TCR+有效载荷敲入的流式细胞仪读出的代表性数据。对于最初的测试,我们的TCR替代物是1G4 TCR,其靶向NY-ESO-1癌睾丸抗原,并且我们的其他目标蛋白质是截短的神经生长因子受体(tNGFR)。该构建体在内源TCR-α基因座的适当整合将产生NY-ESO-1 TCR+tNGFR+T细胞。左侧的流图说明了敲入效率,由使用NY-ESO-1 dextramer染色阳性的T细胞百分比确定。右侧的直方图表明NY-ESO-1 TCR+群体一致表达tNGFR。(c)我们在内源TRAC基因座上的TCR+有效载荷敲入策略导致新型TCR-β链、TCR-α链和目标蛋白质的协调基因表达。然而,这三种蛋白质应保持独立的蛋白质水平调控。为了测试这一点,我们对NYESO-1+tNGFR+CD4/CD8 T细胞进行了分选,并将在稳态下相对于TCR刺激后的NY-ESO-1 TCR表达水平与tNGFR表达水平进行了比较。已知TCR刺激会导致TCR内化,并且在刺激24小时后,我们通过dextramer染色观测到NY-ESO-1 TCR表达降低。相比之下,tNGFR蛋白表达在刺激24小时后仍保持高水平。(d)在验证我们的TCR+有效载荷敲入策略后,我们设计了第二种构建体,其将tNGFR置换为显性阴性TGFβ受体2(dnTGFβR2)作为我们的额外目标蛋白质。我们假设添加dnTGFβR2不仅使我们能够将T细胞靶向特定的癌抗原,而且在由TGFβ1介导的免疫抑制性肿瘤微环境中为T细胞提供功能优势。为了测试这一点,我们合并了两个未分选的、经编辑的T细胞群体,它们含有NY-ESO-1 TCR+dnTGFβR2+T细胞或NY-ESO-1 TCR+tNGFR+T细胞,并在各种培养条件(+/-刺激+/-TGFβ1)下扩增了这个混合群体。我们在图3b和图3c中的观测结果使我们能够区分合并样品内的两个目标群体,并通过流式细胞术测定它们的相对扩增。5天后,我们发现在25ng/mL TGFβ1中培养的经刺激合并样品可见NY-ESO-1 TCR+dnTGFβR2+T细胞超过NY-ESO-1 TCR+tNGFR+T细胞的显著扩增。(e)为了验证重新编程的T细胞特异性和增强的功能,我们利用了一种杀伤测定法,其中表达NY-ESO-1抗原的黑色素瘤细胞系与NY-ESO-1 TCR+dnTGFβR2+T细胞或NY-ESO-1 TCR+tNGFR+T细胞在存在或不存在额外TGFβ1的情况下在各种效应物/靶标比率下共培养。我们发现NY-ESO-1 TCR+tNGFR+在存在TGFβ1的情况下表现相对较差,但NY-ESO-1 TCR+dnTGFBR2+T细胞能够克服TGFβ1的抑制力。在Incucyte上监测癌细胞的生长/杀伤,并使用图像分析软件进行量化。清除%计算为(仅癌细胞的汇合%-共培养条件的汇合%)/(仅癌细胞的汇合%),并且所有值均取自共培养96小时拍摄的图像。
图4a-h显示了原代人T细胞中的靶向合并敲入筛选。
(a)使用非病毒基因组靶向的靶向合并敲入筛选的通用方法。将各自包含独特插入序列的HDR模板文库电穿孔到原代人T细胞中,以产生修饰的T细胞文库。在对T细胞文库施加选择压力后,每个插入物的独特条形码可仅为通过PCR利用在同源定向修复期间未整合的HDRT 3′同源臂中的恒定短变异序列的序列。
(b)设计了36成员合并敲入文库,其含有先前描述和新型的嵌合基因和治疗基因,并靶向原代人T细胞中的TCRα基因座以及新TCR特异性(对于NY-ESO-1,总插入物大小为约2-3kB)。用CD3/CD38磁珠进行TCR刺激后修饰的T细胞文库的比较揭示了源自在人类供体中具有高度可重现性的凋亡介体FAS的四种嵌合蛋白的显著相对扩增。
(c)将多种体外选择压力施加到治疗性T细胞合并敲入文库。文库内的单个功能基因在特定的选择性环境中而不是在所有条件下显示出更大的相对增殖。与仅刺激的比较进一步阐明了个别治疗性敲入构建体的独特功能贡献。两种新型TGFBR2衍生嵌合蛋白,以及先前描述的dnTGFBR2,在外源TGFB存在下选择性地增加增殖。转录因子TCF7在过量TCR刺激存在下选择性富集(抗CD3/CD28刺激是仅刺激条件的5倍)。具有各种免疫检查点的新型和描述的CD28嵌合转换受体在仅CD3刺激的情形下选择性地增加增殖。显示了每个条件下n=4个独立健康供体的平均值。
(d)在人黑色素瘤实体肿瘤异种移植模型中的体内合并敲入筛选。A375人黑色素瘤系表达靶NY-ESO-1肽/MHC,其通过与治疗构建体文库一起敲入TRAC基因座的新TCR特异性来识别。扩增后,将含有约200万个敲入阳性NY-ESO-1 TCR表达细胞的1000万个T细胞的批量群体I.V.转移到荷瘤小鼠中,并保存输入对照T细胞群体。收获转移后四天的肿瘤,并且分选出体内选择后的修饰T细胞文库并相对于输入对照进行分析。
(e)在体内黑色素瘤异种移植模型中验证的体外合并敲入筛选中鉴定的各种命中,包括TGFBR2衍生构建体和转录因子TCF7。显示了n=2个独立健康供体的平均值。
(f)将单个HDRT敲入TRAC基因座允许置换具有新特异性的内源TCR以及新基因修饰功能的表达。合并的靶向敲入筛选允许快速鉴定在特定环境中修改T细胞功能的新构建体。对来自体外合并敲入筛选的命中进行额外的个别验证。与dnTGFBR2构建体或具有对照tNGFR插入物的TCR敲入相比,在不存在或存在外源TGFB的情况下,具有TGFBR2胞外结构域和41BB胞内结构域的嵌合蛋白显示出更大的抗原特异性癌细胞杀伤。
(g)与具有对照GFP插入物的TCR敲入相比,新TCR特异性加上FAS细胞外41BB细胞间嵌合体或转录因子TCF7的单个敲入类似地显示出更大的抗原特异性杀伤。
图5a-5f显示了在原代人T细胞中91个独特基因组基因座的大型非病毒敲入的结果。
a)在91个独特的基因组基因座上进行了非病毒阵列敲入筛选。GFP融合蛋白有效敲入TCR-α的C末端和CD3复合物的四个成员都得到实现。无HDR模板对照在GFP通道中显示出最小的背景荧光水平。
b)对于其他靶标,将tNGFR-2A多顺反子盒敲入靶基因的N末端。在另外24个靶向的表面受体中的许多受体上实现了有效的敲入。在GFP融合构建体和tNGFR-2A靶向构建体中,观测到的GFP或tNGFR表达由每个基因的内源启动子驱动,在靶基因座上产生不同的表达水平。例如,注意到靶向B2M或CD45基因座的tNGFR的极高表达,而CXCR4处的表达相对较低。在一些靶位点未观测到敲入,例如CX3CR1和LTK,而在其他位点,超过50%的细胞被成功靶向,例如IL2RA和CD28。
c)与未刺激的细胞相比,靶向各种检查点抑制剂在刺激时显示出敲入百分比观测值更高。应注意,所有细胞在分离时和电穿孔之前两天接受初始CD3/CD28活化,以实现有效的非病毒基因组靶向,并且在无需额外刺激(“无刺激”或“未刺激”)下电穿孔后四天或在CD3/CD28珠粒刺激24小时后在电穿孔后五天进行流式细胞术(图1b)。
d)靶向16种不同转录因子的非病毒基因组。一些靶基因座如JunD显示出低的敲入百分比观测值,但敲入基因的表达水平高,而其他位点如NCOA3显示出高的敲入百分比观测值但总体表达水平低。
e)有效靶向七种独特的细胞骨架元件。同样不是在不同内源启动子下整合靶基因的可变表达水平。
f)额外32个靶基因的大型敲入。所有呈现均来自同一健康供血者,并代表在阵列敲入筛选期间测试的总共n=6个供体。呈现显示对于每个基因座测试的两个gRNA的效率更高。除非在CD8和CD4 T细胞之间或在刺激和未刺激条件之间可见敲入%观测值有显著差异(图6),否则显示的是未刺激的CD8 T细胞条件。在所有图中,X轴是GFP荧光或tNGFR染色,而Y轴显示细胞大小(FSC-A)。
图6a-e显示了在多种细胞类型和刺激条件下对91个靶基因座的敲入百分比观测值的分析。
a)CD8对CD4 T细胞中的相对敲入百分比观测值。两种细胞类型中敲入观测值差异最大的分别是它们的标志性表面受体CD8A和CD4。CD8 T细胞中41BB(TNFRSF9)和LAG3的敲入要高得多,而细胞因子IL2的敲入观测值在CD4 T细胞中更高。绝大多数靶向位点在两种细胞类型之间没有显示出很大的差异。n=6个供体在91个靶基因组位点的敲入%观测值,每个基因座2个gRNA。
b)刺激对未刺激CD8 T细胞中的相对敲入百分比观测值(图1b)。在电穿孔后四天的第二次刺激后,通过流式细胞术观测到的各种活化/耗竭标志物如PD1、41BB和OX40(TNFRSF4)的敲入量更高。相比之下,在其他位点如FBL、CCR2和IL7R的敲入观测值在没有第二次刺激(“未刺激”)的情况下更高。n=6个供体,在91个靶基因组基因座,每个基因座2个gRNA。
c)对91个独特HDR模板中每一者的脱靶敲入%观测值的分析,这些HDR模板含有GFP或tNGFR敲入序列以及它们的靶基因组基因座特异性的同源臂。在阵列敲入筛选中的所有6个供体中,所有91个HDR模板都用乱序gRNA进行电穿孔(形成对人类基因组中的任何位点都不具特异性的RNP)。虽然绝大多数HDR模板显示出很少甚至没有观测到的脱靶敲入,但少数HDR模板(靶向基因FBL、IL2RG和STAT2)显示出更高的数量。未来对这些模板的DNA序列的分析可得到对脱靶整合模式的进一步深入了解。
d)所有模板、供体和细胞类型中的敲入阳性细胞的MFI观测值与针对靶基因、供体和细胞类型的每个组合记录的RNA-Seq表达值相关。来自n=6个独特人类供血者的汇总数据。
e)阵列敲入筛选中使用的每个gRNA(91个靶位点x每个位点2个gRNA=182个总gRNA)的预测切割分数与在执行阵列敲入筛选的6个供体中的每一者中的观测切割效率的相关性。在不存在HDR模板的情况下,所有182个gRNA被单独电穿孔到所有6个供体的大量CD3+T细胞中,并通过扩增子测序分析每个靶基因座的编辑%。可能是由于原代人T细胞中基于RNP的敲除效率高(绝大多数gRNA根据扩增子测序显示>95%NHEJ编辑),未观察到预测切割分数与这些条件下的切割观测值相关。
图7a-7g显示了gRNA和靶DNA基因座参数与敲入效率观测值的相关性。
a)在所有供体中所测试gRNA的切割位点与其相关HDR模板的整合位点之间的距离(“切割距离”,以bp计)与敲入效率观测值相关。切割位点和整合位点之间短距离的效用已经得到很好的描述,但在小于大约25bp的切割距离窗口内,与敲入观测值的相关性较低。
b)gRNA可识别DNA序列并在相对于整合位点的5′或3′方向切割。朝向5′方向的切割被定义为当gRNA的NGG PAM在5′到3′方向上面向整合位点时,并被赋值为-1。朝向3′方向的切割被定义为当gRNA的NGG PAM在5′到3′方向上背离整合位点时,并被赋值为1。在91个靶向基因座中未观测到与切割方向性的相关性。
c)每个指导物的预测中靶切割分数与中靶敲入百分比观测值不相关。
d)在所测试的6个供体中的每一者中每个gRNA的NHEJ效率观测值(图6e)显示与敲入效率观测值正相关。X轴显示具有NHEJ编辑的等位基因的比例。
e)大量RNA-Seq在6个测试的健康供体、2种细胞类型(CD4和CD8)和三个时间点的所有组合中进行。确定91个靶基因在T细胞分离时和活化前(“第0天”)、CD3/CD28刺激后两天电穿孔时(“第2天”)或在电穿孔后的扩增阶段期间(“第4天”)的表达水平。所有三个时间点的RNA表达水平与敲入%观测值相关,其中最高相关性是最接近蛋白质水平流式细胞术读出时间的时间点(第4天)。应注意,每个基因座的实际敲入效率可能高于观测效率,因为阵列敲入筛选中每个构建体的表达是由靶基因的内源启动子驱动的。以低于流式细胞仪读出检测限的水平表达的基因可能具有更高的实际敲入百分比,这是由于蛋白质表达水平低而不可见的。X轴显示log10每百万读段的转录本数(TPM)。
f)ATAC-Seq在6个测试的健康供体、2种细胞类型(CD4和CD8)和两个时间点(活化前的第0天和电穿孔前的第2天)的所有组合中进行。DNA可及性是针对1kb窗口确定的,该窗口以91个靶基因座的每个gRNA的切割位点为中心。在两个时间点,靶基因座的可及性与敲入效率观测值相关。X轴显示log10每百万读段的读段数(RPM)。g)包括每个gRNA参数(预测切割除外)、RNA表达和DNA可及性的多元线性回归模型(图1e、图1f)显示出比孤立的任何单个参数更大的相关性。
图8a-8e显示了对具有不同敲入效率预测值和观测值的敲入靶位点的检查结果。
a)在2种细胞类型(CD4和CD8)中对于91个独特基因组靶位点中的每一者,通过多元线性回归模型预测的敲入效率与实际观测到的敲入效率之间的差异的分析,每个位点2个gRNA。绝大多数基因的敲入效率预测值在实际观测量的一倍变化范围内,但少数基因的敲入效率预测值比实际观测值高得多(ELOB、JUND),而一些基因的预测敲入值比观测值低得多(DDX20、STAT4、ITGB1)。
b)敲入%预测值高于观测值的顶部6个基因靶标。两个测试的gRNA是着色的,并且每个指导物的两条线代表CD4和CD8 T细胞。
c)敲入%预测值低于观测值的底部6个基因靶标。由于这些位点显示出比预期值更高的敲入效率,因此进一步检查这些靶标及其序列背景可能会揭示可提高跨靶位点的整体敲入效率的设计特征。
d)在该位点测试的两个gRNA之间预测精度方差最高的6个靶基因座。对于这些位点中的至少两个(SATB1、CCR7),由于DNAHDRT序列中的设计误差,发现显示出比实际观测值高得多的预测敲入的gRNA实际上切割其相关HDR模板(所有gRNA的gRNA结合序列和/或PAM位点在其相应的HDR模板中被破坏,以防止在整合之前游离地或在同源定向修复后的第二轮切割中切割HDR模板)。
e)所测试的两种细胞类型(CD4和CD8 T细胞)之间预测精度方差最高的前6个靶基因座。显示了来自n=6个独特健康供体的平均值(a-e)。
图9a-9d显示了具有内源产物的合成调控的基因工程化内源蛋白的示意图和结果。
a)描述我们将新型启动子靶向目标基因N末端的敲入策略的示意图,有或没有额外的选择标志物。
b)我们的敲入策略的代表性流数据,其中我们(按5′到3′顺序)整合了SFFV启动子、选择标志物tNGFR和2A序列,使得产生两种蛋白质、tNGFR和内源蛋白质的多顺反子mRNA在限定的内源基因座由SFFV启动子表达。我们靶向三种免疫受体PD1、Lag3和IL2RA的N末端,它们的表达在T细胞活化时高度上调。在顶行,我们观测到在没有再刺激的情况下电穿孔后培养7天的细胞中每种相应免疫受体的表达水平。一致地,我们观测到在对照条件(乱序RNP+HDRDNA模板)下表达水平或免疫受体相对较低。在中靶条件(中靶RNP+HDR DNA模板)下,我们看到tNGFR+细胞(也敲入了SFFV启动子)具有每种免疫受体的高表达水平,而tNGFR-细胞具有与对照相似或低于对照的表达水平,后者最有可能归因于在没有HDR DNA模板整合的情况下与中靶RNP一起发生的KO。当我们再刺激这些细胞时,我们看到对照样品中每种免疫受体的表达水平都有所增加。在再刺激的中靶样品中,tNGFR+细胞保持每个相应免疫受体的高表达水平,而tNGFR-细胞上调表达水平,但程度较轻。
c)当我们将对照和未经再刺激的中靶编辑细胞中tNGFR表达水平相对于相应免疫受体的表达水平进行比较时,我们看到中靶细胞具有tNGFR及其相应免疫受体的高表达水平(由线性关系展示),而对照细胞具有相应免疫受体的较低表达水平和可忽略的tNGFR表达。
d)在验证了我们将新型/合成启动子与选择标志物整合在一起的敲入策略后,我们将我们的敲入策略应用于一系列转录因子,这些转录因子的过表达可能有益于T细胞增殖和长期功能。为了读出我们构建体的成功整合,我们检查了中靶样品中四种不同转录因子的tNGFR表达水平,并且发现我们能够实现10-25%的敲入效率。该策略对于能够有效调节转录因子表达和后续T细胞功能具有重要意义。
图10a-10e显示了在PDCD1基因座处具有合成产物的内源调控的基因工程化内源蛋白的示意图和结果。
a)描述我们用于将新型蛋白质靶向目标基因的N末端以在内源基因调控下协调表达新型蛋白质和内源蛋白质或表达敲除内源蛋白质的新型蛋白质的敲入策略的示意图。
b)验证我们在PD1的内源基因调控下协调表达新型蛋白质和PD1的策略的代表性流图。在静息细胞(顶行)中,PD1和tNGFR表达最少。然而,在用CD3/CD28 Dynabeads再刺激48小时后,我们看到tNGFR和PD1的协调上调。
c)验证我们在PDCD1的内源基因调控下同时表达新型蛋白质和敲除PD1的策略的代表性流图。在静息细胞(顶行)中,PD1和tNGFR表达最少。然而,在用CD3/CD28 Dynabeads再刺激48小时后,我们看到tNGFR上调,而PD1没有上调。
d)验证我们在PDCD1的内源基因调控下协调表达多种新型蛋白质和PD1的策略的代表性流图。基于tNGFR读出,我们能够在PDCD1基因座成功地整合我们的新型构建体。
e)验证我们在PDCD1的内源基因调控下同时表达多种新型蛋白质和敲除PD1的策略的代表性流图。基于tNGFR读出,我们能够在PDCD1基因座成功地整合我们的新型构建体。
图11a-11d显示了具有合成产物的内源调控的基因工程化内源蛋白的示意图和结果。
a)描述我们用于将新型蛋白质靶向目标基因的N末端以在内源基因调控下协调表达新型蛋白质和内源蛋白质的敲入策略的示意图。
b)来自实验的代表性流数据,其中我们在IL2RA的N末端整合了tNGFR-2A构建体。我们证实tNGFR表达水平因整合位点、时间和细胞培养条件而有所不同,并且重要的是,反映了其启动子控制表达的内源蛋白质的表达水平。在靶位点是IL2RA的细胞中,我们在电穿孔后第3天看到线性IL2RA高、tNGFR高的群体,表明两者的协调表达。在电穿孔后第7天,在没有再刺激的情况下培养的细胞可见IL2RA和tNGFR的表达逐渐且协调地降低,而在再刺激的细胞中,我们看到IL2RA高、tNGFR高群体的维持。
c)来自实验的代表性流数据,其中我们在CD28的N末端整合了tNGFR-2A构建体。我们类似地在第3天观测到线性CD28高tNGFR高群体。CD28表达水平在没有再刺激的情况下保持高水平,这反映在我们的第7天分析中。在没有再刺激的情况下培养的细胞中,我们看到持续的CD28高tNGFR高群体,而在再刺激的细胞中,我们看到CD28和tNGFR表达的同时调节。CD28表达的更急剧下降可能是由于基因表达调节和蛋白质内化的组合,而tNGFR没有被内化。
d)来自实验的代表性流数据,其中我们在Lag3的N末端整合了tNGFR-2A构建体。在第3天,Lag3和tNGFR的表达可忽略不计,并且在第7天没有再刺激的情况下,两种表达水平都保持低水平。然而,当我们在第7天再刺激细胞并对其进行分析时,我们看到Lag3和tNGFR的同时上调。
图12a-12b显示了在CD3复合物成员中具有内源特异性和合成信号传导的基因工程化内源蛋白的示意图和结果。
a)描述我们设计用于修饰TCR复合物中的不同CD3亚基中的每一者的C末端的三种不同构建体的示意图,所述CD3亚基包括CD3δ链、CD3ε链、CD3γ链和CD3ζ链。对于初始测试,我们设计了一种构建体,其将在不同CD3亚基中的每一者的C末端敲入2A-BFP。2A-BFP整合将产生多顺反子mRNA,其产生两种不同的蛋白质:未修饰的CD3链和BFP。一旦验证了2A-BFP整合,我们就修饰了构建体以在2A序列之前包括激活免疫受体的细胞质结构域,因此CD3亚基链的C末端现在包含一个额外的信号传导结构域/基序。
b)为了读出信号传导结构域的成功整合,我们通过流式细胞术分析了表达荧光蛋白的T细胞的百分比。添加额外的信号传导结构域对敲入效率没有显著/一致的影响。额外信号传导结构域相对于内源CD3信号基序的定位没有优化,但修饰单个CD3亚基的胞内结构域的能力为调整TCR信号传导提供了一个有前途的平台。
图13a-13b显示了四组分多顺反子(multi-cistronic/polycistronic)盒对人TCR-α基因座的敲入。
a)我们用于在内源TCR-α基因座上同时框内整合一个新的替代TCR和两个额外目标蛋白质的策略的示意图描述。我们设计了单一HDR DNA模板,其(按顺序)包括Furin-间隔子-T2A序列、新的全长TCR-β链的序列、Furin-间隔子-E2A序列、第一目标蛋白质的序列、Furin-间隔子-F2A序列、第二目标蛋白质的序列、Furin-间隔子-P2A序列,以及TCR-α链的新可变区的序列。这些外源序列在与内源TCR-α基因座外显子1区域同源的同源臂侧翼。成功的敲入将产生表达四种不同蛋白质的多顺反子mRNA。
b)来自我们的敲入策略的流式细胞仪读出的代表性数据。对于初始测试,我们的TCR替代物是1G4 TCR,并且我们的额外目标蛋白质是tNGFR和GFP。将该构建体在内源TCR-α基因座适当整合将产生NY-ESO-1 TCR+tNGFR+GFP+T细胞。左上的流图说明了敲入效率,由用NY-ESO-1 dextramer染色阳性的T细胞百分比确定。NY-ESO-1+细胞都一致地表达GFP和tNGFR(右上流图),而NY-ESO-1-TCR-细胞不表达(左下流图)。相对较小百分比的TCR+NY-ESO-1-细胞同时表达GFP和tNGFR,但不单独表达(右下流图)。这种观察结果很可能是通过我们的构建体在具有活性表达的基因座的脱靶整合或我们的构建体与1G4 TCR-α链、TCR-β链或两者的不当表达的中靶整合来解释的。
图14a-14e显示了在敲入新TCR特异性以及dnTGFβR2功能基因后T细胞功能的表征结果。
a)图3d(合并的增殖测定)的构建体设计和实验装置的示意图描述。NY-ESO-1 TCR+dnTGFβR2+T细胞和NY-ESO-1 TCR+tNGFR+T细胞经过独立编辑和扩增。扩增后,将两个批量编辑的样品合并在一起。合并群体包括NY-ESO-1 TCR+dnTGFβR2+T细胞、NY-ESO-1 TCR+tNGFR+、TCR KO T细胞和NY-ESO-1-TCR+T细胞的异质群体。NY-ESO-1 TCR+dnTGFβR2+T细胞和NY-ESO-1 TCR+tNGFR+的输入数目基于观测到的敲入百分比大致归一化。在有或没有免疫培养刺激和存在或不存在TGFβ1的情况下,合并群体的重复进一步扩增。
b)确定NY-ESO-1 TCR+dnTGFβR2+T细胞相对于NY-ESO-1TCR+tNGFR+的相对扩增的门控策略。在实验的这个阶段(初始分离后19天,2轮刺激,在500U/mL IL-2中连续培养)的大多数T细胞是CD8+T细胞。因此,我们完成了对CD8+T细胞的流动分析。对NY-ESO-1+CD3+CD8+T细胞进行门控,我们在检查tNGFR表达时看到细胞的双峰分布。tNGFR-NY-ESO-1+CD3+CD8+T细胞的比例代表NY-ESO-1 TCR+dnTGFβR2+T细胞并用于下游分析。
c)另一个独立的健康供体中的重复合并增殖测定的结果。5天后,我们再次发现在25ng/mL TGFβ1中培养的经刺激合并样品可见NY-ESO-1 TCR+dnTGFβR2+T细胞超过NY-ESO-1 TCR+tNGFR+T细胞的显著扩增。
d)另外两个独立健康供体中的重复杀伤测定结果。此外,我们发现NY-ESO-1 TCR+tNGFR+在存在TGFβ1的情况下表现相对较差,但NY-ESO-1 TCR+dnTGFBR2+T细胞能够克服TGFβ1的抑制力并在该测定中表现最佳。
e)共培养108小时后,从上图中的杀伤测定中回收T细胞,并通过流式细胞术分析CD8+T细胞上的激活标志物/检查点分子。在只有T细胞而没有癌细胞的样品中,PD1高群体可忽略不计,这表明处于稳态的T细胞并未处于激活或耗竭状态。在降低的效应物/靶标比率下,我们看到所有变体和培养条件下PD1高群体普遍增加,这表明因癌细胞的持续清除而持续激活或由于无法有效清除癌细胞而开始耗竭。在1∶2的效应物/靶标比率下,NY-ESO-1TCR+dnTGFBR2+T细胞的PD1高T细胞百分比显著降低,这一观察结果与TGFβ1添加无关。这可能是因为NY-ESO-1 TCR+dnTGFBR2+T细胞通常更有效地清除癌细胞。TGFβ1已被证明可增加抗原诱导的PD1表达。因此,NY-ESO-1 TCR+dnTGFBR2+T细胞中PD1高T细胞的百分比较低也可归因于显性阴性受体的直接下游效应。
图15a-15c描绘了选择性检测中靶敲入的DNA测序策略。
a)同源定向修复结果的DNA测序由于大量HDRT引入细胞并保持游离状态而变得复杂。成功的中靶敲入可与野生型或NHEJ修饰的基因组基因座、非整合型游离模板和nhej介导的脱靶整合区分开来。为了克服这一挑战,同源定向修复的两个方面可用于特异性地在中靶敲入处产生独特的可扩增序列。首先,在同源定向修复期间只有HDRT同源臂的一小部分区域(连同插入区域的整个长度)被复制到基因组中,而当基因组基因座交叉时,大多数同源臂用于互补碱基配对。其次,在交叉期间可容忍同源臂中的小错配,只要绝大多数同源臂与基因组靶位点保持互补即可。这实现了一种策略,其中将一小段错配引入同源臂(在这种情况下,与3′HA错配约10bp),因此将包括在任何游离模板中。这些错配也将包括在任何脱靶整合中,因为整个同源臂在NHEJ介导的随机dsDNA断裂的脱靶位点的整合期间被整合。然而,在中靶基因座,错配不会复制到基因组中。这使得简单的PCR能够通过使用包含在插入区域内的一个引物从中靶基因座扩增(因此无法从非整合基因组基因座引发),并且结合于基因组序列的第二个引物与引入HDRT中的同源臂错配位点重叠。只有中靶敲入具有两个引物结合位点。
b)将三顺反子HDRT敲入TRAC基因座,用新特异性(NY-ESO-1)以及具有标准未改变同源臂和在同源臂序列中包含相对于约100bp的靶基因组位点的约10bp错配的3′HA的另一种基因(tNGFR)替换内源TCR。
c)与未改变的同源臂相比,约2.5kb NY-ESO-1 TCR+tNGFR的敲入在同源臂错配的情况下效率稍低,但仍易于检测。
图16a-6h显示了在合并的敲入筛选中具有不同合并阶段的模板转换的分析结果。
a)样品合并可在非病毒基因组靶向方案的每个不同步骤进行:包含合并的敲入文库的独特成员的dsDNA片段可在组装成已包含恒定元件(例如同源臂)的DNA质粒之前合并(“合并的组装”);包含每个独特文库成员的完整HDRT序列的DNA质粒可在PCR反应之前合并以生成大量dsDNA HDR模板(“合并的PCR”);每个独特文库成员的dsDNA HDR模板可在电穿孔进入最终细胞之前合并(“合并的电穿孔”);或者,使用每个独特文库成员单独电穿孔的细胞可在电穿孔后但在最终读出之前合并(“合并的培养”)。
b)两个成员即GFP或RFP模板的文库,每个模板都包含在编码TRAC外显子1的新TCR特异性(NY-ESO-1特异性1G4克隆)的敲入盒中,用于分析合并阶段。敲入阳性原代人T细胞可根据新TCR特异性(TCR+NY-ESO-1+)的表达进行鉴定。
c)分析敲入阳性细胞的GFP和RFP表达。仅具有GFP或RFP模板的细胞显示每个相应fluor的表达,而合并的培养条件仅显示相等的GFP和RFP阳性细胞群体,没有任何双重GFP+RFP+细胞。电穿孔步骤之前的合并条件(合并的组装、合并的PCR或合并的电穿孔)均显示单一GFP+或RFP+细胞以及双重GFP+RFP+细胞,这可能是由于两个TRAC基因座的双等位基因敲入,因为T细胞通常从两个等位基因表达功能性TCR-α链。多个群体被分选用于条形码测序,包括批量敲入阴性细胞(NY-ESO-1-)、批量敲入阳性细胞(NY-ESO-1+)和RFP+GFP-或RFP-GFP+细胞的个别群体。使用转化为cDNA的分离mRNA或使用2步PCR的分离基因组DNA进行中靶敲入的下一代DNA测序。初始PCR使用在HDR模板的3′HA中错配重叠的反向引物(图15)和插入序列内的恒定正向引物(总扩增区域约140bp)扩增条形码区域。然后在合并测序用样品之前进行第二次索引PCR。
d)为了分析选择性中靶敲入PCR测序策略的选择性,相对于在第一次PCR之前使用恒定数量的输入基因组DNA并读出测序读段的总相对数量的编辑细胞的批量群体分析了分选的敲入阳性(NY-ESO-1+)对敲入阴性(NY-ESO-1-)细胞的扩增总量(在任何方案步骤的样品之间均未使用浓度归一化)。敲入阳性细胞显示出相对于批量编辑群体,包含条形码的敲入HDRT区域的扩增增强,而敲入阴性细胞显示出很少或没有成功扩增,证明了中靶敲入相对于非整合游离HDRT或脱靶整合的扩增和测序选择性(图15)。
e)跨合并阶段分析内源基因组基因座在条形码测序PCR期间扩增的程度,并比较分离的mRNA与基因组DNA。所有条件都显示出没有条形码序列的少量读段(例如,在基因组基因座包含野生型序列),尽管在对mRNA进行测序时,量始终略高(占总读段数的约1%)。对mRNA进行测序具有扩增每一个别细胞中可测序条形码数量的优势,但需要在要表达的编码区(例如TCRα基因座)中进行合并的敲入筛选,并且条形码要整合到编码序列中的简并碱基中。相比之下,基因组DNA的测序具有可推广到任何可进行成功敲入的基因组基因座的优势(图1),但与使用低细胞数量的mRNA(转化为cDNA)测序相比,其信噪比可能较低。
f)包含GFP HDR模板条形码的测序读段的百分比与通过流式细胞术在合并条件下观测到的表达GFP蛋白的细胞百分比相对应,并且在对基因组DNA或mRNA进行测序时保持恒定,证明了合并的敲入筛选测序策略通过对其DNA条形码进行测序来准确评估细胞群体频率的能力。
g)显示了在对基因组DNA进行测序时在合并条件下包含正确条形码的已分选GFP+或RFP+细胞中已测序条形码的百分比。仅GFP或RFP模板的敲入产生100%包含正确条形码的读段,并且电穿孔后细胞的合并的培养产生>99%正确条形码。然而,在不同供体之间并且无论分析分选的GFP+或RFP+细胞,在早期实验阶段的合并产生了高度一致的模板转换量增加。
h)使用在合并阶段观测到的用于合并的敲入筛选的同源臂错配引发策略对模板转换量进行量化。观测到的模板转换量在基因组DNA或mRNA测序之间高度一致。最早的合并阶段(合并的组装)显示出最大的模板转换量,但是在合并的PCR和合并的电穿孔条件下观测到一致的模板转换量,表明在Gibson组装反应、产生HDR模板的PCR期间以及甚至可能在同源定向修复期间的细胞内可能发生了交叉或模板转换事件。鉴于在具有两个成员(GFP和RFP)的合并的敲入文库中,大约一半的实际模板转换量将产生具有相同序列的条形码,任意大型文库中的预测模板转换量将更高。鉴于当前的合并的敲入文库设计的参数(独特的文库插入物与其相应条形码之间的约400bp,由新敲入的TCR-a特异性隔开),合并的组装反应的预测模板转换量为约50%,而合并的电穿孔仅为约10%。所有实验均显示一名代表性供体(b、c)或来自n=2个独特健康供体的一个或多个技术重复(d-h)。
图17a-17e显示了36成员合并敲入文库改变T细胞功能的设计和对其筛选后的结果。
a)构建了36个潜在治疗基因的合并的敲入文库,可使用单一HDR模板将其与新TCR特异性(NY-ESO-1)整合在一起。所述文库被设计成包含先前公开的和新成员,这些成员可能会改变各种广泛类别的免疫治疗T细胞功能:胞内结构域被截短(“tPD1”或“tCTLA4”)或被激活的结构域代替(嵌合转换受体,“CTLA4-CD28”)的免疫检查点;类似地被截短或胞内结构域被转换的凋亡介体;参与细胞增殖的基因;趋化因子;转录因子;参与与肿瘤环境中存活相关的代谢途径的基因;和作为截短/显性阴性受体(“dnTGFβR2”)或具有转换的胞内结构域的抑制性细胞因子受体。
b)合成所有36个构建体并置于TCR插入盒中,所述盒将用新特异性(NY-ESO-1TCR)替换内源T细胞受体,以及驱动可能改变内源TCR-α启动子的T细胞功能的新基因的表达。每个文库成员都在阵列敲入筛选中单独测试,并测定了表面表达的TCR的敲入百分比和MFI,以测定单个插入物对TCR表达的任何潜在影响。
c)所有36个构建体都成功地显示了功能性TCR表达,如通过新NY-ESO-1 TCR的表面dextramer染色所分析。
d)总插入物大小范围为约2,000-3,000bp(不包括同源臂序列),并且在模板大小和敲入效率之间几乎没有观测到相关性。
e)个别地敲入所有36个文库成员后的NY-ESO-1 TCR表达的MFI观测值。在文库成员中观测到高度一致的TCR表达水平。
图18a-18k显示了在原代人T细胞中合并的敲入筛选的技术验证结果。
a)对36成员HDR模板文库的合并的敲入筛选,其中每个成员都包含恒定的新特异性(NY-ESO-1特异性TCR)以及带有条形码的独特基因,所述基因可能会改变T细胞功能,所有这些基因都靶向在TCR-α基因座进行整合(TRAC外显子1)。电穿孔后,生成修饰T细胞文库,然后可对其进行测定,例如通过添加第二次TCR刺激(初始刺激用于敲入构建体)。然后通过DNA测序确定每个文库成员的独特条形码的频率。然后可将条形码频率与输入群体进行比较,以查看每个文库成员对该测定中T细胞行为的相对影响。
b)36成员文库中的两个基因可通过流式细胞术容易地检测到,GFP和RFP的对照敲入。对获得新NY-ESO-1特异性TCR的敲入阳性细胞进行门控,揭示同时具有GFP+或RFP+的细胞比例大致相当。
c)36成员文库的合并的电穿孔七天后,修饰T细胞文库中条形码的分布。每个文库成员的总读段百分比在四个独特的健康人类T细胞供体中是一致的,并且文库显示出相对均匀的分布(基尼系数(Gini coefficient)=0.048)。
d)通过流式细胞术在蛋白质水平上观测到的群体频率与通过合并的敲入测序方法在DNA水平上检测到的条形码频率之间的对应关系。对于通过流式细胞术容易观测到的蛋白质GFP和RFP,在蛋白质水平上阳性细胞的比例与具有相应GFP和RFP条形码的读段的比例相似。
e)插入序列的大小与修饰T细胞文库中检测到的频率之间的关系。NY-ESO-1-β和NY-ESO-1-α VJ区段及其相关的2A元件为约1.5kb,而在同一构建体中敲入的额外功能基因的大小在约0.5-1.5kb之间变化,产生的总插入物大小在2-3kb之间。对于文库中以略低的频率存在的较大的插入物观测到轻微的相关性(R
f)在36个合并的敲入构建体的合并的电穿孔后七天,修饰T细胞文库以1∶1 CD3/CD28珠粒∶细胞比率进行刺激,或者作为输入群体进行分离。显示了体外TCR刺激5天后条形码频率相对于输入群体的log2倍数变化。源自凋亡介体FAS细胞表面蛋白的构建体在四个独特的健康T细胞供体中显示出相对增殖的显著增加。
g)在技术重复之间和对于不同合并阶段检查合并的敲入筛选结果的可再现性(图16a)。同一生物供体中TCR刺激筛选的技术重复显示出高度相关性(R
h)敲入阳性活细胞的数量对于执行大型合并的筛选很重要。电穿孔后10天测定了合并的敲入后原代人T细胞的扩增。假设分离出100万个原代人T细胞,电穿孔后四天回收了平均约50万个敲入阳性细胞(平均敲入效率为10%-20%),并且在四个健康的人类供体中这些细胞在培养的额外天数中继续稳健扩增。
i)敲入实验生成混合细胞群体,一些具有包含所需敲入的等位基因,一些具有敲除等位基因,而一些具有未编辑的等位基因(图18b)。当分选不切实际或不可行时,可对分选的敲入阳性细胞(此处根据NY-ESO-1 dextramer染色进行分选)以及未分选的批量编辑细胞群体进行合并的敲入筛选。合并的敲入后的测序条形码频率在分选和未分选的批量群体之间高度相关(R
j-k)对于大多数合并的敲入实验,在施加选择压力之前,电穿孔后T细胞扩增7-10天。除了IL2RA丰度相对较大的增加外,这段时期的培养扩增(仅包含培养基+IL-2)并未显示文库成员的丰度有任何大的变化。
实验显示或代表n=2个(d、g、i)或n=4个(c、e-f、h、j-k)独特的健康人类T细胞供体。虚线表示非功能性对照文库成员的最大和最小丰度。
图19a-19d显示合并的敲入筛选鉴定了在模拟肿瘤环境的不同体外选择压力下不同的功能序列。
a)编码潜在功能修饰蛋白的36成员DNA序列文库与恒定的新TCR特异性(NY-ESO-1)的合并的电穿孔,生成修饰原代人T细胞的合并文库。然后可应用模拟肿瘤环境的各种体外选择压力,并且可将修饰T细胞池中独特条形码的分布与输入T细胞群体或在给定的选择压力之间进行比较,揭示在每个特定环境中赋予T细胞增殖变化的文库序列。
b)在TGFB中体外培养5天后文库成员的分布,表示为相对于输入群体的log2倍数变化的排序列表。在电穿孔后第7天取出输入细胞,并在培养基中用25ng/mL的外源TGFB应用1∶1 CD3/CD28珠粒∶细胞刺激。相对于输入,多种FAS衍生的抗凋亡受体以及TGFBR2衍生的抗抑制性受体增加了相对增殖。然而,与仅基于珠粒的刺激相比,FAS衍生受体显示出丰度的相对减少(但仍然是绝对增加),这表明对TGFB介导的抑制的敏感性可能增强。相比之下,TGFBR2衍生受体在TGFB存在下显示出迄今为止最大的相对增殖。先前公布的显性阴性TGFBR2受体仅为一种新型嵌合TGFBR2细胞外-41BB细胞内构建体。
c)在过量TCR刺激的情况下(5:1CD3/CD28珠粒∶细胞比率,而不是标准的1∶1比率),与刺激前的输入群体相比,FAS衍生构建体再次显示出相对丰度增加。当将抑制性过度刺激群体与标准刺激进行比较时,FAS构建体在抑制条件下再次显示出更大的相对抑制,而与标准量的CD3/CD28刺激相比,在所有四个供体中表达转录因子TCF7的构建体在过度刺激下显示出更大的相对增殖。
d)在不存在CD28接合共刺激信号下仅通过TCR刺激修饰T细胞文库(通过与NY-ESO-1特异性dextramer一起孵育)显示一些但不是全部的CD28嵌合转换受体的选择性增加。例如CTLA4、TIM3和BTLA的多种免疫检查点蛋白的胞外结构域与CD28的胞内结构域融合。与CD3/CD28刺激相比,仅通过TCR(CD3)的刺激显示出CTLA4-CD28、TIM3-CD28和BTLA-CD28构建体的增殖相对增加。所有图表都显示出与修饰T细胞文库输入相比的log2倍数变化,或与CD3/CD28刺激相比的相对log2倍数变化。显示了n=4个独特健康供体的平均值并用于对构建体进行排序。虚线表示非功能性对照文库成员的最大和最小丰度。
图20a-20d显示了实体肿瘤异种移植模型中的体内合并敲入筛选的结果。
a)36成员潜在治疗性敲入构建体文库的合并的敲入,该文库赋予新的独特功能修饰蛋白以及恒定的新TCR特异性(NY-ESO-1特异性TCR,1G4克隆)。生成和扩增10天后,将修饰T细胞文库(2.5e6个NY-ESO-1+T细胞)过继转移到带有在转移前7天皮下注射的实体人黑色素瘤异种移植物(表达NY-ESO-1 TCR的靶肽/MHC的A375黑色素瘤细胞)的免疫缺陷NSG小鼠中。在实体肿瘤环境中经过5天的体内选择压力后,解剖肿瘤,分选T细胞,并通过DNA测序分析条形码的相对丰度。
b)体内实体肿瘤合并敲入筛选的生物重复显示出比体外合并敲入筛选更大的文库间差异(图4b),但始终显示相同的顶部文库命中。
c)同一供体内的体内合并敲入筛选的技术重复类似地显示出比体外合并敲入筛选更大的差异(图18g)。
d)体外合并敲入筛选的多次命中类似地显示出实体肿瘤异种移植环境中增殖和/或持久性增加。转录因子TCF7以及TGFβR2衍生的嵌合受体均显示出相对丰度的稳健且可再现的增加。在进行的任何体外筛选中未鉴定的其他文库成员,例如代谢物转运蛋白MCT4,在体内肿瘤环境中显示出强烈的相对富集。实验显示或代表n=2个(b-d)独特的健康人类T细胞供体。虚线表示非功能性对照文库成员的最大和最小丰度。
图21a-21h显示了来自合并的敲入筛选的命中的个体验证数据。
a)带有抑制性细胞因子受体TGFβR2的胞外结构域和增殖性受体41BB的胞内结构域的TGFβR2-41BB嵌合受体的个体功能验证。使用单一HDR模板,原代人T细胞被工程化以表达新TCR特异性(NY-ESO-1)以及抗抑制性TGFβR2-41BB受体。
b)在存在TGFβ的情况下,TGFβR2-41BB修饰细胞概括了观测到的与仅刺激相比更大的相对增殖的表型(图19)。电穿孔后7天,用CD3/CD28珠粒(珠粒/细胞比率1∶1)刺激也表达TGFβR2-41BB或GFP对照的分选NY-ESO-1+T细胞,并通过每个指定日期的绝对细胞计数测定增殖。刺激后6天进行活化和耗竭标志物的表面染色。
c)TGFβR2-41BB修饰细胞相比于GFP对照显示出更大的抗原特异性体外肿瘤杀伤,并且当在指定的T细胞/癌细胞比率范围内添加外源TGF-β下与A375人黑色素瘤细胞共培养时,杀伤力与dnTGFβR2的表达至少相当。在开始共培养杀伤测定后5天,取出T细胞并针对PD1的表面表达进行染色。
d)带有凋亡受体FAS的胞外结构域和增殖性受体41BB的胞内结构域的FAS-41BB嵌合受体的个体功能验证。使用单一HDR模板,原代人T细胞被工程化以表达新TCR特异性(NY-ESO-1)以及抗凋亡FAS-41BB受体。
e)在抗原非依赖性增殖测定(电穿孔后7天的CD3/CD28珠粒刺激)中,FAS-41BB嵌合受体的表达与GFP对照受体的表达(两者以及新TCR特异性)相比大大增加了相对增殖,验证了在合并的筛选中通过刺激可见的增殖增加(图4c)。至关重要的是,仅在刺激时才观测到FAS-41BB受体的增殖增加,而在没有刺激的情况下IL-2的持续扩增与对照相比没有显示出相对增殖优势。在珠粒刺激后6天还观测到活化和耗竭标志物的表面表达降低。
f)FAS-41BB修饰的T细胞显示出更大的抗原特异性体外肿瘤杀伤。
g)TCF7表达构建体的个体功能验证。使用单一HDR模板,原代人T细胞被工程化以表达新TCR特异性(NY-ESO-1)以及通过TCR-α启动子的TCF7表达而改变的转录程序。
h)与标准刺激(1∶1珠粒∶细胞比率)相比,在过度刺激条件(5∶1CD3/CD28珠粒∶细胞比率)下,TCF7的表达概括了与TCR+GFP对照敲入相比更高的相对增殖观测值。指定活化和耗竭标志物的表达在各条件之间没有变化。应注意,在这些个体验证实验中,与FAS-41BB嵌合体的增殖效应相比,TCF7表达的相对增殖改变的效应大小类似地概括了合并的敲入筛选中观测到的效应大小(图4c)。
i)表达TCF7的修饰T细胞显示出更大的抗原特异性体外肿瘤杀伤。实验显示或代表n=2个(b-c、e-fh-i)独特的健康人类T细胞供体。
图22显示了可用于本文所述筛选方法中的核酸构建体的示例性示意图。在图22所示的任一构建体中,一个或多个条形码可包含在2A序列之前、2A序列内部(任选地具有简并碱基)、或2A序列之后。在图22所示的任一构建体中,一对独特条形码,即具有不同序列的条形码,可在任一侧侧接基因X即目标基因。
图23a-e显示了与单细胞RNA测序配对的合并的敲入筛选,用于治疗性原代T细胞修饰的快速表型分析。
a)对照和潜在治疗性构建体的36成员文库被敲入原代人T细胞的TCRα基因座,同时用NY-ESO-1癌症抗原特异性TCR替换它们的内源TCR。在仅体外扩增(输入)或过继转移到体内抗原特异性黑色素瘤异种移植模型后四天后,对活T细胞进行分选并生成单细胞液滴。每个细胞的特定敲入构建体通过扩增子测序确定(图24a-e)并与每个单细胞的转录组相关联。
b)在合并的敲入筛选中从两个供体鉴定的所有单细胞并结合两个供体中的单细胞RNA测序的UMAP表示。
c)UMAP表示上的归一化基因表达(Z分数)揭示了输入群体和体内群体之间在激活状态标志物(CCR7和MK167)和效应子功能标志物(GZMB和IFNG)方面的表达差异。
d)批量细胞合并敲入筛选(图4d)和单细胞合并敲入筛选中每个文库成员的体内丰度的相关性。
e)NY-ESO-1 TCR加对照、TCF7或TGFβR2-41BB多顺反子构建体的体内表型特征。三种敲入构建体之间在体内差异表达的基因的相对基因表达热图揭示了不同的基因特征。
图24a-e:单细胞RNAseq联合合并的敲入筛选的分子和分析管线。
a)在联合合并敲入加单细胞RNAseq实验期间,将细胞与基因敲入相关联的测序管线的分子图。细胞表达的特定敲入构建体的条形码(“敲入条形码”)在HDR期间整合到细胞基因组DNA中(图4a),并存在于整合的TCRαVJ区域的编码区的简并碱基中。在液滴中转录和单细胞分离后,来自单个细胞的TCR+基因X mRNA转录物与含有poly(dT)引物和独特细胞条形码的珠粒结合。逆转录后,直接与敲入条形码上游的引物结合产生包含敲入条形码以及细胞条形码的扩增子。该扩增子两端的下一代测序产生一对匹配的敲入条形码和细胞条形码,以及通用分子标识符(UMI)。应注意,只有在基于液滴的polyA下拉期间分离出的一部分cDNA用于对条形码进行测序,并且单独的一部分cDNA可用于生成单细胞转录组。
b)用于将敲入条形码与联合合并敲入加单细胞RNAseq实验中的单个细胞相关联的计算分析管线。
c)与敲入条形码和细胞条形码的每个测序组合相关的唯一分子标识符(UMI)数量的直方图。在逆转录步骤(a)期间添加UMI,并且每个UMI代表一个独特的mRNA转录物。从进一步分析中过滤出仅具有单一UMI的敲入条形码/细胞条形码组合。
d)与每个已测序细胞条形码相关的敲入条形码数量的直方图。正如预期的那样,绝大多数细胞条形码只有单一与其相关的敲入条形码。具有两个关联的敲入条形码的细胞条形码可代表真正的双等位基因敲入或文库制备期间模板转换的结果。具有两个以上相关敲入条形码的细胞条形码很少见,并且可能代表模板转换事件。从进一步分析中过滤出具有两个或更多个相关敲入的少数细胞条形码。
e)超过75%的分配有敲入条形码的细胞条形码也具有通过质量过滤器的单细胞转录组(参见实施例)。大量已对转录组进行测序但未分配敲入条形码的细胞条形码可能是由于文库制备过程中的效率低下,具有双等位基因基因敲入的细胞被过滤掉,或没有中靶敲入的细胞存在于分选和测序的样品中。
图25a-e提供的数据显示合并的敲入筛选揭示了改善体外和体内抗原特异性肿瘤控制的治疗性敲入盒。
(a)将单个多顺反子敲入TRAC基因座允许同时替换内源抗原特异性和天然或合成基因产物的共表达以改变细胞功能。互补的体外和体内合并敲入筛选允许快速鉴定增强环境特异性T细胞功能的新构建体,包括编码新型TGFβR2-41BB和FAS-41BB嵌合受体或TCF7转录因子的多顺反子。
(b)编码NY-ESO-1抗原特异性的多顺反子加上FAS细胞外41BB转换受体或转录因子TCF7,类似地在扩增筛选中被鉴定为命中,与TCR敲入和对照GFP插入物相比显示出增强的体外NY-ESO-1+癌细胞杀伤。
(c)在体外和体内扩增筛选中鉴定为命中的具有TGFβR2转换受体或dnTGFβR2的多顺反子,增强了体外NY-ESO-1+癌细胞杀伤。在不存在或存在外源TGFβ1的情况下,与dnTGFβR2构建体或具有对照tNGFR插入物的TCR敲入相比,具有TGFβR2胞外结构域和41BB胞内结构域的嵌合蛋白显示出更大的抗原特异性癌细胞杀伤。代表n=2个独立健康供体(b、c)。
(d)黑色素瘤小鼠异种移植模型。NSG小鼠、非肥胖糖尿病(NOD)/严重联合免疫缺陷(SCID)/Il2rg
(e)过继转移仅媒介物(盐水,灰色)或NY-ESO-1 TCR细胞与额外的多顺反子构建体后的肿瘤大小测定:tNGFR对照(黑色)、转录因子TCF7(橙色)或嵌合TGFβR2-41BB受体(红色)。与单独的媒介物相比,三种多顺反子NY-ESO-1 TCR构建体显示肿瘤大小在统计学上显著减小,但只有TGFβR2-41BB构建体导致肿瘤清除。在n=2(TCF7,图25)或n=4(tNGFR、TGFβR2-41BB,图26)个独特的健康人类供体中显示的每个条件具有n=8+小鼠的一个代表性供体。**P<0.01,***P<0.001,****P<0.0001(使用Holm-Sidak多重比较检验的双向方差分析(ANOVA))。
图26a-e显示了来自合并的敲入筛选的FAS-41BB嵌合受体命中的体外验证。
(a)带有凋亡受体FAS的胞外结构域和增殖性受体41BB的胞内结构域的Fas-41BB嵌合受体的个体功能验证。使用多顺反子HDR模板,原代人T细胞被工程化以表达新TCR特异性(NY-ESO-1抗原)以及嵌合Fas-41BB受体。(b)不依赖于抗原的验证测定。在不依赖于抗原的增殖测定(电穿孔后7天抗CD3/CD28珠粒再刺激)中,Fas-41BB嵌合受体的表达与GFP对照受体的表达相比增加了相对扩增(以及新TCR特异性),从而验证了在合并的筛选中在刺激下可见的扩增观测值增加。与合并的筛选类似地,仅在再刺激时才可见Fas-41BB受体的扩增增加,而在没有再刺激的情况下,IL-2的持续扩增与对照相比没有显示出相对扩增优势。在珠粒刺激后还观测到一些活化和耗竭标志物的表面表达降低。(c)抗原特异性验证测定。以NY-ESO-1 TCR/Fas-41BB构建体靶向的T细胞在各种T细胞/癌细胞比率下相比于使用对照NY-ESO-1 TCR构建体靶向的那些显示出更大的体外NY-ESO-1+癌细胞杀伤。在以1∶4T细胞/癌细胞比率共培养后96小时,在多个生物供体中观测到抗原特异性体外杀伤增加(n=5个独特的健康人类T细胞供体,每个供体2个技术重复)。**P<0.01,Wilcoxon配对符号秩检验。共培养杀伤测定开始后5天,取出T细胞并针对活化和耗竭标志物的表面表达进行染色。(d)合并的敲入加单细胞RNAseq数据揭示了体外扩增后不同FAS衍生嵌合蛋白的丰度变化。(e)五种不同FAS衍生嵌合蛋白的基因表达分析揭示了不同的基因表达特征。应注意FAS-41BB构建体中与增殖相关的基因的丰富表达,这在合并的刺激筛选中显示出最大的相对增殖潜力。除非另有说明,否则实验显示或代表n=2个(b-c)独特的健康人类T细胞供体。
图27a-d显示了合并的敲入筛选命中TCF7和体内肿瘤对照实验的体外验证。
(a)TCF7表达构建体的个体功能验证。使用多顺反子HDR模板,原代人T细胞被工程化以表达新TCR特异性(NY-ESO-1抗原)以及通过内源TCR-α基因调控控制的TCF7而改变的转录程序。
(b)不依赖于抗原的验证测定。TCF7的表达概括了在过度刺激条件下(5∶1抗CD3/CD28珠粒/细胞比率)相对于标准刺激(1∶1珠粒/细胞比率)与NY-ESO-1 TCR+GFP+对照敲入相比较高的相对扩增观测值。指定的活化和耗竭标志物的表达在修饰之间没有出现变化。
(c)抗原特异性验证测定。在各种T细胞/癌细胞比率下,以NY-ESO-1 TCR/TCF7构建体靶向的T细胞相比于以对照NY-ESO-1 TCR构建体靶向的T细胞显示出更大的体外NY-ESO-1+癌细胞杀伤。在以1∶4 T细胞/癌细胞比率共培养后96小时,在多个生物供体中观测到抗原特异性体外杀伤增加,尽管效应的量值强烈依赖于供体(n=5个独特的健康人类T细胞供体,每个供体2个技术重复;**P<0.01,Wilcoxon配对符号秩检验)。共培养杀伤测定开始后5天,取出T细胞并针对活化和耗竭标志物的表面表达进行染色。
(d)A375黑色素瘤异种移植模型中体内肿瘤生长的个体肿瘤追踪。在肿瘤接种后第9天,1.5e6个分选的NY-ESO-1 TCR/tNGFR对照T细胞(黑色)或NY-ESO-1 TCR/TCF7 T细胞(橙色),或没有T细胞(灰色,仅媒介物)被过继转移。虽然tNGFR对照和TCF7细胞均显示出肿瘤大小相对于仅媒介物有统计学显著减少(图23e),但TCF7表达相对于tNGFR对照T细胞没有显示出统计学显著改善。除非另有说明,否则实验显示或代表n=2个(b-c)独特的健康人类T细胞供体。
图28a-e显示了TGFβR2-41BB嵌合受体的体外和体内验证。
(a)带有抑制性细胞因子受体TGFβR2的胞外结构域和增殖性受体41BB的胞内结构域的TGFβR2-41BB嵌合受体的个体功能验证。使用多顺反子HDR模板,原代人T细胞被工程化以表达新TCR特异性(NY-ESO-1)以及TGFβR2-41BB嵌合转换受体。
b)不依赖于抗原的验证测定。在存在TGFβ的情况下,TGFβR2-41BB修饰细胞概括了观测到的与仅刺激相比更大的相对扩增表型。也表达TGFβR2-41BB或GFP对照的分选的NY-ESO-1+T细胞,在电穿孔后7天用抗CD3/CD28珠粒(珠粒/细胞比率1∶1)再刺激,并通过量化每个指定日期的绝对细胞计数来测定扩增。刺激后6天进行活化和耗竭标志物的表面染色。
(c)在外源TGFβ存在下,体外抗原非依赖性TCR刺激后24小时,细胞因子IFNγ、IL-2和TNFα的产生增加。*P<0.05,**P<0.01(使用Holm-Sidak多重比较检验的单向方差分析(ANOVA))。
(d)抗原特异性验证测定。TGFβR2-41BB修饰细胞相比于tNGFR对照显示出更大的体外NY-ESO-1+癌细胞杀伤,并且在指定范围的T细胞/癌细胞比率下添加外源TGFβ下与A375人黑色素瘤细胞共培养时,与dnTGFβR2修饰细胞的杀伤相似。在外源TGFβ存在下以1∶1T细胞/癌细胞比率共培养后72小时,在多个生物供体中观测到抗原特异性体外杀伤增加(n=4个独特的健康人类T细胞供体,每个供体2个技术重复;**P<0.01,Wilcoxon配对符号秩检验)。在共培养杀伤测定开始后5天,取出T细胞并针对PD1的表面表达进行染色。
(e)A375黑色素瘤异种移植模型中体内肿瘤生长的个体肿瘤追踪。在肿瘤接种后第9天,1.5e6个分选的NY-ESO-1 TCR/tNGFR对照T细胞(黑色)或NY-ESO-1 TCR/TGFβR2-41BB T细胞(红色),或无T细胞(灰色,仅媒介物)被过继转移。虽然在测试的四个供体中观测到可变性,但TGFβR2-41BB细胞显示肿瘤负担在统计学上显著减少(图25e,来自供体1的汇总数据)。在多个供体的许多情况下,TGFβR2-41BB细胞清除了肿瘤,这在任何对照小鼠中均未观测到。与第一对(供体1和2)或第二对(供体3和4)独特的健康人类供体同时检查仅媒介物对照小鼠的单独群组。注意到供体1和供体2tNGFR和仅媒介物对照迹线从图25d再现。除非另有说明,否则实验显示或代表n=2个(b-d)独特的健康人类T细胞供体。
图29A-G显示大型DNA插入物的多重文库的合并的敲入筛选。
(A)将36成员构建体文库非病毒靶向合并敲入到原代人T细胞的TRAC基因座中,并在电穿孔后7天对敲入条形码进行后续测序。36成员文库中的所有构建体条形码始终充分表现为具有均匀的文库分布(n=4,独立人类供体,基尼系数=0.048)。
(B)观测到敲入效率和插入物大小之间的弱负相关(R
(C)流式细胞术鉴定了所有对于NY-ESO-1 TCR染色的敲入阳性细胞(引入TRAC基因座;脱靶整合不应产生NY-ESO-1 TCR+细胞)。可评估表达GFP(NY-ESO-1 TCR+GFP+)或RFP蛋白(NY-ESO-1TCR+RFP+)的敲入阳性细胞的百分比,并且可对这些细胞进行FACS分选。
(D)在四名供血者的实验中表达GFP(NY-ESO-1 TCR+GFP+)或RFP蛋白(NY-ESO-1TCR+RFP+)的敲入细胞百分比与相应GFP或RFP模板条形码的频率紧密对应。ns=不显著(配对双向T检验)。
(E)利用36成员大型敲入文库的同源臂(HA)错配引发策略的验证。根据NY-ESO-1TCR表达以及GFP+、RFP+或两者皆无对敲入阳性细胞进行分选。当使用与基因组序列匹配(并且缺少引入同源臂中的错配)的引物对中靶敲入进行测序时,在相应群体中具有GFP或RFP条形码的测序读段的百分比与预期模板转换和双等位基因整合校正后的预测百分比密切匹配。然而,正如预期的那样,使用结合模板同源臂(包含错配序列)的引物进行测序并没有强烈地富集GFP+或RFP+分选群体的中靶敲入。
(F-G)文库成员的分布(基于条形码频率)在IL2电穿孔后的离体培养10天内,在整个T细胞扩增过程中基本一致。由于培养条件,IL2RA编码构建体显示出比输入增加的丰度。虚线代表对照文库成员的最大和最小丰度(编码GFP、RFP和tNGFR)。*P<0.05,****P<0.0001(使用Holm-Sidak多重比较检验的双向方差分析(ANOVA))。除非另有说明,否则所有实验均在来自n=4个(B-D、F-G)或n=2个(E)个别健康人类供体的原代T细胞电穿孔后七天进行分析。
图30A-F显示了通过合并的敲入筛选鉴定的新基因构建体的功能验证和改进的体外癌细胞杀伤。
(A)阵列敲入实验验证了所选文库成员(FAS-41BB、TGFBR2-41BB、IL2RA、TIM3-CD28、CTLA-CD28)在合并的敲入筛选中改进的环境相关适应性。对照构建体(tNGFR)、在测试环境中不会导致统计学显著适应性改善的中性构建体(TCF7、PD1-41BB、tBTLA)以及来自筛选的阴性命中(截短的CTLA4;tCTLA4)也包括在阵列实验中。
(B)流式细胞术证实了敲入构建体中编码的预期蛋白质产物相对于用相同刺激条件处理的对照细胞的过表达。在敲入阳性细胞(对NY-ESO-1 TCR+进行门控)中,与电穿孔后七天的对照细胞(TIM3-CD28,在10天测量)相比,所有测试的八种构建体都显示出预期的转基因蛋白质产物的表达增加。蛋白质表达的时间过程如图32A所示。
(C)在多种条件下对八种个别敲入构建体测定了扩增、活力和增殖效应。FAS-41BB敲入构建体在刺激后增加了扩增,而TGFβR2-41BB构建体在将外源TGFβ添加到测定中时显示出扩增和增殖(通过CFSE稀释)的最大相对增加。
(D)用八种选定的个别敲入构建体进行体外癌细胞杀伤测定。在分选的NY-ESO-1+T细胞与每个指定的敲入构建体共培养后72小时,显示了在不同的T效应物(E)与癌细胞靶标(T)的比率(x轴)下,A375人类黑色素瘤靶细胞的百分比(y轴)。TGFβR2-41BB(红色),与对照细胞(tNGFR,绿色)相比,显著改善了靶细胞杀伤。相比之下,tCTLA4(黑色)损害杀伤。在更高的E∶T比率下,额外的构建体在细胞杀伤方面显示出更温和的改善(也参见图32C)。
(E)D中癌细胞杀伤数据的时间过程数据,对来自四个独立健康供血者的细胞中进行的实验取平均值。
(F)与具有对照tNGFR构建体的敲入细胞相比,TGFβR2-41BB敲入构建体在不存在和存在外源TGFβ1的情况下均增强了体外NY-ESO-1+癌细胞杀伤。n=4个独立的健康供血者。在n=4个(B-F)独立健康人类供体中进行的实验。*P<0.05,**P<0.01,***P<0.001,****P<0.0001(配对双尾T检验)。也参见图32。
图31A-I显示了PoKI-Seq合并敲入筛选与单细胞RNA测序的结合。
(A)与单细胞RNA测序配对的合并敲入实验的设计,称为PoKI-seq。该平台提供了由每个敲入构建体引起的细胞表型的高维评估(详情也参见图33A)。整合在每个细胞中的敲入构建体因而可与对细胞转录组的影响相关联。
(B)为了验证敲入模板条形码对每一个别细胞的分子分配,对表达整合NY-ESO-1TCR(均为TCR+)的批量敲入阳性细胞进行分选,还表达GFP+或RFP+的NY-ESO-1阳性细胞也是如此。在分选的NY-ESO-1 TCR+GFP+和NY-ESO-1 TCR+RFP+群体中,绝大多数模板条形码对应于预期蛋白质产物的表达。
(C)PoKI-seq也准确鉴定了具有双等位基因整合的细胞。观测到的具有双等位基因敲入构建体的细胞的频率与基于2成员GFP/RFP文库敲入实验预测的那些密切匹配。正如预期的那样,在具有双等位基因整合的分选GFP+和RFP+细胞中,一个条形码分别对应于GFP或RFP。值得注意的是,相同敲入构建体的双等位基因整合(总双等位基因整合的1/36)无法与单等位基因整合区分开来。
(D)来自两个人类供血者的合并的敲入T细胞群体在体外鉴定的所有单细胞状态的UMAP表示。在合并的敲入编辑后七天,在存在或不存在外源TGFβ的情况下,用CD3/CD28珠粒以1∶1的比率刺激分选的敲入阳性T细胞(NY-ESO-1 TCR+)。
(E)覆盖在UMAP表示上的最近邻聚类(Louvain)揭示了对应于不同细胞状态的单细胞群体。呈现了在选定簇中显示富集或耗竭的标志基因。
(F)对于D中的每个单细胞分配敲入构建体。超过58%的细胞被分配敲入构建体。大约3.4%的细胞被分配了3个或更多个敲入条形码,这可能是由于测序细胞双联体、多模板的罕见不完美整合或模板转换。与成功分配条形码的细胞转录组相比,无法分配敲入构建条形码的细胞往往质量较低,具有较少调用基因和独特UMI(图33B)。
(G)在TGFβ处理条件下具有指定敲入构建体的细胞的密度图(在单细胞状态的UMAP表示中)。与对照和其他敲入构建体相比,观测到TGFβR2衍生构建体的明显差异。
(H)如通过卡方残差观测值对预期值所测量的限定单细胞簇中具有选择敲入构建体的细胞的过表达分析。在仅刺激(顶行)的情况下,FAS-41BB构建体在增殖簇8中富集。随着外源抑制性细胞因子TGFβ的加入,具有TGFβR2衍生的敲入构建体的细胞在与增殖性状态(簇8)和效应物状态(簇12)对应的簇中表现出强烈的富集,以及与TGFβ反应相关的簇(簇2、4、6)的消耗。
(I)PoKI-seq实验中选定敲入构建体的基因表达热图。基因列表是从H中检查的簇中的基因生成的,与所有其他簇相比,绝对log倍数变化>0.8。在外源TGFβ存在下,TGFβR2衍生构建体的转录效应彼此强烈相关,但在仅刺激条件下则不然。TGFβR2衍生构建体改变了对TGFB的转录反应,维持了与仅刺激条件相关的基因如增殖标志物MKI67和TOP2A的表达。也参见图33。
图32A-C显示了与图30相关的合并的敲入筛选命中的阵列体外验证。
(A)在门控NY-ESO-1TCR+细胞中,与对照敲入相比,每个指定敲入构建体在电穿孔后5、7和10天的蛋白质表达时间过程(NY-ESO-1 TCR+tNGFR用于除tNGFR本身之外的所有构建体,其中TCR+tBTLA构建体用作对照)。检测到一些内源基因产物(Fas、IL2RA、Tim-3)的表达,但在除TIM3-CD28的第5天和第7天之外的所有时间点添加敲入构建体后都观测到表达增加。在第10天,观测到TIM3-CD28构建体表达高于内源水平,这可能是由于TCR启动子相对于内源Tim-3的活化依赖性表达一致的高表达。
(B)在分选细胞中使用单个敲入构建体的其他的活力(在总淋巴细胞群体中的活细胞染色%)、增殖(CFSE低%)和扩增(与输入相比的总细胞数)测定,如图30C所示。Fas-41BB嵌合受体在刺激后显示出最高的活力,以及在刺激后4天通过CFSE稀释染色测量的最大量的增殖。当仅提供CD3刺激时,TIM3-CD28嵌合受体显示出最大量的增殖,类似于合并的敲入筛选。在过度刺激(5∶1 CD3/CD28珠粒∶细胞比率)的情况下,Fas-41BB嵌合受体再次显示出最大的相对扩增。显示了n=4个独立健康供体的三个技术重复。
(C)分选的NY-ESO-1 TCR+T细胞与靶RFP+A375黑色素瘤在电穿孔后9天开始以指定的效应物/靶细胞比率共培养,并通过Incucyte延时显微镜成像72小时。所测试的八个个别敲入构建体中的每一者的靶细胞杀伤百分比相对于对照显示(类似测试的TCR+tNGFR和TCR+GFP构建体的平均值)。显示了n=4个独立健康供体中的每一者的三个技术重复的平均值+SEM。
图33A-F显示了与图31相关的合并的敲入构建体的PoKI-Seq分子管线、质量控制指标和单细胞表型。
(A)使用PoKI-Seq将细胞的转录组与其敲入构建体相关联的分子测序管线图。细胞中特定敲入构建体的条形码(“敲入条形码”)在整合的TCRαVJ区的编码区的简并碱基中编码。在液滴中转录和单细胞分离后,来自个别细胞的TCR+基因XmRNA转录物与含有poly(dT)引物和独特细胞条形码的珠粒结合。逆转录后,直接与敲入条形码上游的引物结合产生包含敲入条形码以及细胞条形码的扩增子。该扩增子两端的下一代测序产生一对匹配的敲入条形码和细胞条形码,以及通用分子标识符(UMI)。在基于液滴的polyA下拉期间仅分离出一部分cDNA用于生成单细胞转录组(25%),并且其余cDNA(75%)可用于对敲入条形码进行测序。
(B)离体原代人T细胞中来自PoKI-Seq的质量控制指标。每个细胞都调用了大量独特基因和独特UMI。值得注意的是,通过Cell Ranger(10X)分配转录组但未分配敲入构建体(“0”)的单细胞显示QC指标明显较差。在测试的两个供体和两种条件(刺激+/-TGFβ)中的每一者中,平均覆盖率(每个敲入构建体具有单等位基因整合的个别细胞的数目)为约136X。至少3个都含有相同敲入条形码的UMI被用于将细胞分配给特定的敲入构建体,其中大多数细胞拥有超过3个。
(C)包含选择的敲入构建体的敲入序列的转录物的归一化基因表达值的热图。敲入构建体由内源TCR启动子驱动,生成比包含敲入构建体序列部分的内源基因更高的表达水平(例如,从TCR启动子驱动的Fas-41BB的表达水平高于内源Fas,参见图30B和图32A)。转录物在10X文库制备期间被片段化,因此无法将内源基因的转录物与敲入构建体产生的转录物区分开来。对于许多敲入构建体,观察到相关的预期mRNA丰度增加,类似于在图30B和图32A中关于预期蛋白质产物所见。
(D)在指定的单细胞簇中使用PoKI-Seq检查的每个敲入构建体的富集(卡方残差)。TGFβR2转换受体或显性阴性受体在存在外源TGFβ的情况下在特定簇中显示出强烈的富集,这与它们对细胞状态的环境依赖性的特定生物学效应一致。颜色指示卡方残差值并且大小指示卡方残差的量值。
(E)每个定义的单细胞簇内的GO术语富集分析。GO术语进一步支持对个别细胞状态簇的功能解释。颜色是与所有其他簇相比,指定簇内与指定GO术语相关的基因集的平均log倍数变化。大小是超几何富集检验的p值。
(F)在任何单细胞簇中鉴定的所有差异表达基因的平均表达的成对Pearson相关性,针对指定的敲入构建体和对照在仅刺激和刺激+TGFβ体外条件下计算。显性转录差异是由暴露于TGFβ驱动的,但在刺激条件内,促进最大增殖优势的敲入构建体(Fas转换受体和IL2RA,但明显不是Fas-CD28构建体)显示出最相似的转录谱。相比之下,在存在TGFβ的情况下,所有三种TGFβR2衍生受体彼此之间都显示出比其他敲入构建体更相关的转录变化。
图34是用于合并的敲入筛选的示例性构建体的图。多顺反子构建体包括三个2A片段、目标基因(转录因子和治疗构建体的文库)和NY-ESO特异性T细胞受体(TCR)链。为了防止由于模板转换而导致不正确的条形码/基因分配,用于构建体鉴定的条形码从TRAV区域的3′端转移到最接近目标基因(5′和3′端)。在基因的每一侧插入一个独特条形码并添加恒定接头序列允许组合策略(在一个多顺反子构建体中组合两种不同的目标基因)。
图35a-d显示了使用图34中描绘的构建体进行模板转换的结果。使用两个示例构建体(mCherry对GFP,在上述多顺反子盒中)评价模板转换。质粒池(n=2)通过合并的组装构建。HDR模板是从质粒池中生成的,并被电穿孔到两个个别健康供体的原代T细胞中。根据NY-ESO-1 TCR和GFP或mCherry表达对细胞进行分选。通过cDNA的扩增子测序分析正确条形码读段的数目。将正确分配读段的百分比与用mCherry/GFP模板分别电穿孔并在培养期间合并的T细胞和仅用一种构建体电穿孔的T细胞进行比较(图35a和图35b)。计算了2成员文库的模板转换(图35c)并对N成员文库进行预测(图2d)。使用新的条形码策略,N成员文库的预测模板转换从之前设计中的50%降低到改进型合并敲入文库设计中的平均7.6%。改进型合并敲入文库设计的模板转换观察值和预测值:(a)包含GFP或(b)mCherry HDR模板条形码的测序读段的百分比与不同合并条件下通过流式细胞术观测到的表达GFP或mCherry蛋白的细胞百分比相对应。计算了(c)2成员文库的模板转换观测量和(d)N成员文库的模板转换预测值。在合并的组装阶段,新文库设计的模板转换预测值为7.6%。所有实验均在n=2个独特健康供体中进行。
定义
在本说明书和所附权利要求书中使用的单数形式“一个”、“一种”和“所述”包括复数个所指物,除非上下文另有明确规定。
术语“核酸”或“核苷酸”是指单链或双链形式的脱氧核糖核酸(DNA)或核糖核酸(RNA)及其聚合物。除非特别限制,否则所述术语涵盖含有天然核苷酸的已知类似物的核酸,其具有与参考核酸相似的结合特性并且以类似于天然存在的核苷酸的方式代谢。除非另有说明,否则特定的核酸序列还隐含地涵盖其保守修饰变体(例如,简并密码子取代)、等位基因、直向同源物、SNP和互补序列以及明确指出的序列。具体而言,简并密码子取代可通过生成其中一个或多个所选(或所有)密码子的第三个位置被混合碱基和/或脱氧肌苷残基取代的序列来实现(Batzer等人,Nucleic Acid Res.19:5081(1991);Ohtsuka等人,J.Biol.Chem.260:2605-2608(1985);和Rossolini等人,Mol.Cell.Probes 8:91-98(1994))。
术语“基因”可以指参与产生或编码多肽链的DNA区段。它可包括编码区之前和之后的区域(前导和尾部)以及各个编码区段(外显子)之间的插入序列(内含子)。或者,术语“基因”可以指参与产生或编码非翻译RNA的DNA区段,例如rRNA、tRNA、指导RNA(例如,单一指导RNA)或微RNA。
如本文所用,关于细胞中的核酸(例如基因)或蛋白质的术语“内源”是在特定细胞中出现的核酸或蛋白质,如其在自然界中发现的,例如,在其天然基因组位置或基因座。此外,“内源表达”核酸或蛋白质的细胞表达在自然界中发现的核酸或蛋白质。
“启动子”定义为指导核酸转录的一个或多个核酸控制序列。如本文所用,启动子包括转录起始位点附近的核酸序列,例如在聚合酶II型启动子的情况下为TATA元件。启动子还任选地包括远端增强子或阻遏子元件,其可位于距转录起始位点多达数千个碱基对处。
当核酸被置于与另一核酸序列的功能关系中时,它被“可操作地连接”。例如,如果启动子或增强子影响序列的转录,则启动子或增强子与编码序列可操作地连接;或者如果核糖体结合位点被定位以促进翻译,则其与编码序列可操作地连接。
“多肽”、“肽”和“蛋白质”在本文中可互换使用以指氨基酸残基的聚合物。如本文所用,所述术语涵盖任何长度的氨基酸链,包括全长蛋白质,其中氨基酸残基通过共价肽键连接。
如本文所用,术语“互补”或“互补性”是指核苷酸或核酸之间的特定碱基配对。互补核苷酸通常是A和T(或A和U),以及G和C。本文所述的指导RNA可包含这样的序列,例如,与基因组序列完全互补或基本互补(例如,具有1-4个错配)的DNA靶向序列。
“CRISPR/Cas”系统是指一类广泛用于防御外来核酸的细菌系统。CRISPR/Cas系统存在于广泛多种真细菌和古细菌生物中。CRISPR/Cas系统包括I型、II型和III型亚型。野生型II型CRISPR/Cas系统利用RNA介导的核酸酶,例如Cas9,与指导物和激活RNA复合以识别并切割外源核酸。具有指导RNA和活化RNA两者的活性的指导RNA也是本领域已知的。在一些情况下,此类双重活性指导RNA被称为单指导RNA(sgRNA)。
Cas9同源物存在于广泛多种真细菌中,包括但不限于以下分类群的细菌:放线菌门(Actinobacteria)、产水菌门(Aquificae)、拟杆菌门(Bacteroidetes)-绿菌门(Chlorobi)、衣原体门(Chlamydiae)-疣微菌门(Verrucomicrobia)、绿弯菌门(Chlroflexi)、蓝藻门(Cyanobacteria)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、螺旋体门(Spirochaetes)和热袍菌门(Thermotogae)。示例性的Cas9蛋白是化脓性链球菌(Streptococcus pyogenes)Cas9蛋白。额外的Cas9蛋白及其同源物描述于例如Chylinksi等人,RNA Biol.2013年5月1日;10(5):726-737;Nat.Rev.Microbiol.2011年6月;9(6):467-477;Hou等人,Proc Natl Acad Sci U SA.2013年9月24日;110(39):15644-9;Sampson等人,Nature.2013年5月9日;497(7448):254-7;和Jinek等人,Science.2012年8月17日;337(6096):816-21。本文提供的任何Cas9核酸酶的变体可优化以用于宿主细胞中的有效活性或增强的稳定性。因此,还考虑了工程化的Cas9核酸酶。参见例如“Slaymaker等人,“Rationally engineered Cas9 nucleaseswith improved specificity,”Science 351(6268):84-88(2016))。
如本文所用,术语“Cas9”是指RNA介导的核酸酶(例如,细菌或古细菌来源的,或源自其的)。示例性的RNA介导的核酸酶包括上述Cas9蛋白及其同源物。其他RNA介导的核酸酶包括Cpf1(参见例如Zetsche等人,Cell,第163卷,第3期,第759-771页,2015年10月22日)及其同源物。
如本文所用,术语“核糖核蛋白”复合物等是指如下各物的混合物:靶向核酸酶例如Cas9和crRNA(例如指导RNA或单指导RNA)、Cas9蛋白和反式激活crRNA的混合物(tracrRNA)、Cas9蛋白和指导RNA,或它们的组合(例如,Cas9蛋白、tracrRNA和crRNA指导RNA混合在一起)。应当理解,在本文所述的任何实施方案中,Cas9核酸酶可被Cpf1核酸酶或任何其他引导的核酸酶取代。
如本文所用,在修饰细胞基因组的上下文中的短语“修饰”是指中靶基因组区域诱导基因组序列的结构变化。例如,修饰可采取将核苷酸序列插入细胞基因组的形式。例如,可将编码多肽的核苷酸序列插入到T细胞的TCR基因座的基因组序列中。通篇使用的“TCR基因座”是基因组中编码TCRα亚基、TCRβ亚基、TCRγ亚基或TCRδ亚基的基因所在的位置。例如,可通过中靶基因组区域内诱导双链断裂或在相反链上和靶基因组区域侧翼的一对单链切口来进行此类修饰。用于中靶基因组区域处或内诱导单链或双链断裂的方法包括使用Cas9核酸酶结构域或其衍生物,以及针对靶基因组区域的指导RNA或指导RNA对。
如本文所用,在引入核酸或包含核酸的复合物例如RNP-DNA模板复合物的上下文中的短语“引入”是指核酸序列或RNP-DNA模板复合体从细胞外到细胞内的易位。在一些情况下,引入是指核酸或复合物从细胞外易位到细胞核内。考虑了这种易位的各种方法,包括但不限于电穿孔、与纳米线或纳米管接触、受体介导的内化、经由细胞穿透肽的易位、脂质体介导的易位等。
如本文所用,短语“异源的”是指自然界中通常不存在的。术语“异源核苷酸序列”是指在自然界中通常不会在给定细胞中发现的核苷酸序列。因此,异源核苷酸序列可以是:(a)对其宿主细胞是外来的(即,对于细胞是外源的);(b)在宿主细胞中天然存在(即,内源)但在细胞中以非自然量存在(即,比宿主细胞中天然存在的量多或少);或(c)天然存在于宿主细胞中,但位于其天然基因座之外。
如本文所用,“细胞”可以是真核细胞、原核细胞、动物细胞、植物细胞、真菌细胞等。任选地,细胞是哺乳动物细胞,例如人类细胞。在一些情况下,细胞是人T细胞或能够分化成表达TCR受体分子的T细胞的细胞。这些包括造血干细胞和源自造血干细胞的细胞。
如本文所用,术语“可选择标志物”是指允许选择包含标志物的宿主细胞例如T细胞的基因。可选择标志物可包括但不限于:荧光标志物、发光标志物和药物可选择标志物、细胞表面受体等。在一些实施方案中,选择可以是阳性选择;也就是说,表达标志物的细胞是从一个群体中分离出来的,例如以产生表达可选择标志物的富集细胞群体。可通过适合于所用可选择标志物的任何适宜的分离技术进行分离。例如,如果使用荧光标志物,则细胞可通过荧光激活细胞分选进行分离,而如果插入了细胞表面标志物,则细胞可通过亲和分离技术从异质群体中分离出来,例如磁性分离、亲和色谱、将亲和试剂附着在固体基质上的“淘选”、荧光激活细胞分选或其他适宜的技术。
如本文所用,短语“造血干细胞”是指可产生血细胞的一类干细胞。造血干细胞可产生髓系或淋巴系细胞,或其组合。造血干细胞主要存在于骨髓中,尽管它们可从外周血或其一部分中分离出来。各种细胞表面标志物可用于鉴定、分选或纯化造血干细胞。在一些情况下,造血干细胞被鉴定为c-kit
如本文所用,短语“造血细胞”是指源自造血干细胞的细胞。造血细胞可通过从生物体、系统、器官或组织(例如,血液或其部分)分离来获得或提供。或者,可分离造血干细胞并通过分化干细胞来获得或提供造血细胞。造血细胞包括分化成其他细胞类型的潜力有限的细胞。此类造血细胞包括但不限于多能祖细胞、谱系限制性祖细胞、常见髓系祖细胞、粒细胞-巨噬细胞祖细胞或巨核细胞-红系祖细胞。造血细胞包括淋巴和髓系细胞,例如淋巴细胞、红细胞、粒细胞、单核细胞和血小板。在一些实施方案中,造血细胞是免疫细胞,例如T细胞、B细胞、巨噬细胞、自然杀伤(NK)细胞或树突细胞。在一些实施方案中,细胞是先天免疫细胞。
如本文所用,短语“T细胞”是指表达T细胞受体分子的淋巴细胞。T细胞包括人αβT细胞和人γδT细胞。T细胞包括但不限于原初T细胞、刺激T细胞、原代T细胞(例如未培养的)、培养的T细胞、永生化T细胞、辅助T细胞、细胞毒性T细胞、记忆T细胞、调节性T细胞、自然杀伤T细胞、其组合或其亚群。T细胞可以是CD4
如本文所用,原代细胞上下文中的短语“原代”是尚未转化或永生化的细胞。此类原代细胞可被培养、继代培养或传代有限次数(例如,培养0、1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19或20次)。在一些情况下,原代细胞适应体外培养条件。在一些情况下,原代细胞从生物体、系统、器官或组织中分离,任选地分选并直接利用,无需培养或继代培养。在一些情况下,原代细胞被刺激、激活或分化。例如,原代T细胞可通过接触CD3、CD28激动剂、IL-2、IFN-γ或其组合(例如,在其存在下培养)来激活。
如本文所用,术语“同源定向修复”或HDR是指通过来自同源模板核酸的聚合来修复DNA链的切割或切口端的细胞过程。因此,原始序列被模板的序列替换。在一些情况下,可引入外源模板核酸,例如DNA模板,以获得靶位点的特定HDR诱导的序列变化。以这种方式,可在切割位点,例如由靶向核酸酶产生的切割位点引入特定的突变。单链DNA模板或双链DNA模板可被细胞用作模板来编辑或修饰细胞的基因组,例如通过HDR。通常,单链DNA模板或双链DNA模板具有至少一个与靶位点同源的区域。在一些情况下,单链DNA模板或双链DNA模板具有两个同源区域,例如5′端和3′端,位于包含要插入在靶切割或插入位点的DNA模板的区域的侧翼。
如在多核苷酸或多肽序列的上下文中使用的术语“基本同一性”或“基本同一”是指与参考序列具有至少60%序列同一性的序列。或者,同一性百分比可以是从60%到100%的任何整数。示例性实施方案至少包括:60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%,相比于使用本文所述程序的参考序列;优选使用标准参数的BLAST,如下所述。本领域技术人员将认识到,通过考虑密码子简并性、氨基酸相似性、阅读框定位等,可适当地调整这些值以确定由两个核苷酸序列编码的蛋白质的相应身份。
对于序列比较,通常一个序列充当与测试序列进行比较的参考序列。当使用序列比较算法时,将测试序列和参考序列输入计算机,必要时指定子序列坐标,并指定序列算法程序参数。可使用默认程序参数,或者可指定替代参数。然后,序列比较算法根据程序参数计算测试序列相对于参考序列的序列同一性百分比。
如本文所用,“比较窗口”包括对选自由20至600、通常约50至约200、更通常约100至约150中的多个连续位置中的任一个的区段的提及,其中在两个序列最佳比对后,可将序列与具有相同数量连续位置的参考序列进行比较。用于比较的序列比对方法是本领域公知的。用于比较的序列的最佳比对可通过Smith和Waterman Add.APL.Math.2:482(1981)的局部同源性算法,通过Needleman和Wunsch J.Mol.Biol.48:443(1970)的同源比对算法,通过Pearson和Lipman Proc.Natl.Acad.Sci.(U.S.A.)85:2444(1988)的相似性搜索方法,通过这些算法(例如,BLAST)的计算机化实现,或通过手动对齐和目视检查来进行。
适合确定百分比序列同一性和序列相似性的算法是BLAST和BLAST 2.0算法,其分别描述于Altschul等人,(1990)J.Mol.Biol.215:403-410和Altschul等人,(1977)NucleicAcids Res.25:3389-3402中。执行BLAST分析的软件可通过国家生物技术信息中心(NCBI)网站公开获得。所述算法包括首先通过识别查询序列中长度为W的短字词来识别高评分序列对(HsP),当与数据库序列中相同长度的字词对齐时,这些字词匹配或满足某个正值阈值分数T。T被称为邻域字词得分阈值(Altschul等人,同上)。这些最初的邻域字词命中充当种子,用于启动搜索以找到包含它们的更长的HSP。然后沿着每个序列在两个方向上扩展字词命中,直到累积对齐分数可以增加。对于核苷酸序列,使用参数M(一对匹配残基的奖励分数;总是>0)和N(错配残基的惩罚分数;总是<0)计算累积分数。对于氨基酸序列,使用评分矩阵来计算累积分数。在以下情况下,字词命中在每个方向的扩展将停止:累积对齐分数从其最大实现值下降了数量X;由于一个或多个负评分残基比对的累积,累积分数变为零或更低;或到达任一序列的末尾。BLAST算法参数W、T和X决定比对的灵敏度和速度。BLASTN程序(用于核苷酸序列)使用字长(W)为28、期望值(E)为10、M=1、N=-2以及两条链的比较作为默认值。对于氨基酸序列,BLASTP程序使用字长(W)为3、期望值(E)为10和BLOSUM62评分矩阵作为默认值(参见Henikoff和Henikoff,Proc.Natl.Acad.Sci.USA 89:10915(1989))。
BLAST算法还对两个序列之间的相似性进行统计分析(参见例如Karlin和Altschul,Proc.Nat′l.Acad.Sci.USA 90:5873-5787(1993))。BLAST算法提供的一种相似性度量是最小总和概率(P(N)),它提供了两个核苷酸或氨基酸序列之间偶然发生匹配的概率的指示。例如,如果测试核酸与参考核酸的比较中的最小总和概率小于约0.01,更优选小于约10
具体实施方式
以下描述叙述了本发明组合物和方法的各个方面和实施方案。没有特定的实施方案旨在限定组合物和方法的范围。相反,实施方案仅提供至少包括在所公开的组合物和方法的范围内的各种组合物和方法的非限制性实例。将从本领域普通技术人员的角度阅读描述;因此,技术人员熟知的信息不一定包括在内。
合并的敲入筛选
本公开涉及用于鉴定细胞基因组中的靶向插入的组合物和方法。本发明人已经发现了一种合并的敲入筛选方法,以快速测定合并的细胞群体中的许多靶向敲入。
筛选方法
本文提供了用于鉴定细胞基因组中的靶向插入的方法。在本文提供的方法中,将(i)切割细胞基因组中的靶区域以产生靶插入位点的靶向核酸酶;和(ii)序列彼此不同的多个DNA模板引入到细胞群体中。所述DNA模板可包含:i.异源编码或非编码核酸序列;ii.任选地指示所述异源编码或非编码核酸序列的身份的独特条形码核苷酸序列;和iii.共同引物结合序列,其中每个DNA模板的5′端和3′端包含与插入位点侧翼的基因组序列同源的核苷酸序列,并且其中一个或两个同源核苷酸序列与基因组序列中的同源序列相比包含错配核苷酸序列,其中所述错配核苷酸序列在重组期间不插入到靶插入位点中。
如本文所用,“多个DNA模板”是指序列不同的两个或更多个DNA模板。在一些实施方案中,所述多个包括至少10、20、30、40、50、60、70、80、90或100个序列不同的DNA模板。在一些实施方案中,存在多个序列不同的一种或多种DNA模板的多个拷贝。
在本文所述的组合物和方法中,一种或两种同源序列的长度为至少约50、100、150、200、250、300、350、400或450个核苷酸。在一些情况下,与基因组序列同源的核苷酸序列与基因组序列至少80%、90%、95%、99%或100%互补。在一些实施方案中,同源序列与人T细胞TCR基因座中的基因组序列同源。通篇使用的“TCR基因座”是基因组中编码TCRα亚基、TCRβ亚基、TCRγ亚基或TCRδ亚基的基因所在的位置。
在本文所述的组合物和方法中,错配核苷酸序列被设计为与细胞基因组序列中的相应序列不互补。参见例如图4a。错配序列是足够非互补的,以在后续扩增期间最小化或消除错配核苷酸序列与细胞基因组序列中的相应序列之间的碱基配对。因此,当使用如本文所述的“结合插入位点侧翼的基因组序列但不结合模板中的错配核苷酸”的引物进行扩增时,这意味着当基因组序列和模板都存在于同一扩增反应中时,所述引物与基因组序列充分互补以启动从基因组序列扩增,但与模板中的错配序列的互补性不足以启动模板的扩增。引物靶向与模板中错配序列位于相同位置的基因组序列部分。也就是说,当模板的同源“臂”序列与靶细胞中的基因组DNA对齐(例如,通过BLAST)时,引物结合的基因组DNA中的序列将对应于模板中错配序列的位置,在错配序列的任一侧在模板和基因组序列之间存在比对序列。
在本文所述的组合物和方法中,DNA模板侧翼的一个或两个同源序列(臂)中错配核苷酸序列的长度足以允许大部分同源序列与基因组中的插入位点侧翼的基因组序列保持互补。在一些实施方案中,同源序列(臂)各自的长度为50-500,例如200-400,例如250-350,例如300个核苷酸。可选择同源臂的长度以优化靶基因组位点的同源重组。选择错配核苷酸序列的长度足以防止与错配核苷酸序列对应的基因组序列特异性结合的引物的结合,使得当重组发生时,一对引物(一个结合对应于错配核苷酸序列的基因组序列的引物和一个结合DNA模板中的共同引物结合位点的引物),与野生型基因座、非同源末端连接(NHEJ)修饰的基因组位点、非整合的游离模板或NHEJ介导的脱靶整合相比,可用于选择性扩增中靶插入。在一些实施方案中,错配核苷酸序列的长度为约3至约50个核苷酸,例如长度为约5、10、15、20、25、30、35、40、45或50个核苷酸。
在本文提供的组合物和方法中,错配核苷酸序列被插入到同源序列中的某个位置,使得当发生同源重组时,错配核苷酸序列不会插入到具有DNA模板的基因组中。在一些实施方案中,将错配核苷酸序列插入到距DNA模板或同源臂序列任一端约25、50、75、100、125或更多个核苷酸处。在一些实施方案中,将错配核苷酸序列插入到距DNA模板或同源臂序列任一端约25、50、75、100、125个或处。在一些实施方案中,错配序列可以插入到DNA模板或同源臂序列3′端下游约25、50、75、100、125或更多个核苷酸处。在一些实施方案中,错配序列可以插入到DNA模板或同源臂序列5′端上游约25、50、75、100、125或更多个核苷酸处。在一些实施方案中,将错配序列插入到DNA模板或同源臂序列5′端上游约25、50、75、100、125或更多个核苷酸处,并且将错配序列插入到DNA模板或同源臂序列3′端下游约25、50、75、100、125或更多个核苷酸处。由于在重组时错配序列未整合到细胞的基因组中,因此可以选择性地扩增和鉴定不包含错配序列的中靶插入。参见例如图15a。
在将靶向核酸酶和多个DNA模板引入细胞群体后,允许发生重组,从而产生修饰细胞群体。一旦细胞被修饰,就用一对引物从细胞中扩增DNA,例如通过聚合酶链反应(PCR)或其他扩增方法。在一些实施方案中,第一引物与共同引物结合序列互补,而第二引物与插入位点侧翼的基因组序列结合并且不与DNA模板中的错配核苷酸序列结合。在另一个实施方案中,第一引物与插入位点侧翼的5′基因组区域结合并且不与DNA模板中相应的第一错配序列结合,而第二引物与插入位点侧翼的3′基因组区域结合并且不与DNA模板中相应的第二错配核苷酸序列结合。
在一些实施方案中,DNA模板中的共同引物结合位点在相对于条形码序列的DNA模板中的核酸序列中,使得当来自细胞的DNA用结合共同引物结合位点的第一引物和结合于插入位点侧翼的基因组区域的第二引物扩增时,条形码序列也被扩增。可根据需要设计引物序列以靶向模板的任一端。因此,在一些情况下,例如,错配序列位于DNA模板的5′端,或者其位于DNA模板的3′端(或两者),并且相应地设计引物以与相对于错配在DNA模板内部适当定位的共同引物结合序列的引物组合来扩增条形码序列。
在第一引物与插入位点侧翼的5′基因组区域结合而不与DNA模板中的错配序列结合并且第二引物与插入位点侧翼的3′基因组区域结合而不与DNA模板中的错配核苷酸序列结合的实施方案中,整个DNA模板(包括条形码)可被扩增。
扩增后,对DNA进行测序以鉴定插入细胞靶插入位点中的DNA模板。在一些实施方案中,对DNA模板进行测序以鉴定DNA模板。在一些实施方案中,对条形码序列进行测序以鉴定DNA模板(其基于条形码序列,可基于模板序列和条形码序列的已知相关性来预测DNA模板序列)。
通常将使用测序方法,以便可确定不同序列的绝对或相对数量。测序方法包括但不限于Sanger测序(包括微流控Sanger测序)、焦磷酸测序、大规模平行特征测序、纳米孔DNA测序、单分子实时测序(SMRT)(Pacific Biosciences,Menlo Park,CA)、离子半导体测序、连接测序、边合成边测序(Illumina,San Diego,Ca)、Polony测序、454测序、固相测序、DNA纳米球测序、heliscope单分子测序、质谱测序、焦磷酸测序、支持的寡核苷酸连接检测(SOLiD)测序、DNA微阵列测序、RNAP测序和隧道电流DNA测序,仅举几例。本文所述的一种或多种测序方法可用于高通量测序方法。如本文所用,术语“高通量测序”是指与核酸测序相关的所有方法,其中在给定时间对多于一种核酸序列进行测序。
在一些实施方案中,修饰的细胞在允许异源多肽表达的条件下培养。在其他实施方案中,细胞在有效扩增修饰细胞群体的条件下培养。
在一些实施方案中,所述方法还包括确定具有插入靶插入位点中的不同DNA模板的群体中的细胞的相对数量。
在一些实施方案中,在确定具有插入靶插入位点中的不同DNA模板的群体中的细胞的相对数量之前,对修饰细胞群体施加选择压力。通过对细胞施加选择压力,可鉴定赋予细胞(例如T细胞)所需功能的编码或非编码序列。在一些实施方案中,鉴定了在存在或不存在选择压力的情况下编码赋予细胞所需功能的多肽的DNA模板。在一些实施方案中,比较在对修饰的细胞施加选择压力之前和之后具有插入靶插入位点中的不同DNA模板的群体中的细胞的相对数量。以这种方式,可鉴定合并群体中每个单独插入物的丰度,包括那些在特定条件下富集的插入物。在一些实施方案中,选择压力是细胞刺激。在一些实施方案中,选择压力可以是但不限于使细胞与免疫抑制性细胞因子接触、在不利的代谢条件下培养细胞、细胞的过度刺激、细胞的部分刺激(例如仅CD3或CD28刺激)。
在一些实施方案中,对细胞进行体外或体内表型选择或富集以将修饰与所需表型相关联。本文所述的任何筛选方法都可以在体外、离体或体内进行。在一些实施方案中,可使用各种条件下的细胞状态标志物进行基于FACS的选择。应当理解,可在各种体外和体内环境中测试细胞群体。
在一些实施方案中,在细胞修饰后,可分析表达可检测表型的细胞的一个或多个亚群以确定所述亚群中具有插入靶插入位点中的不同DNA模板的细胞的相对数量。在一些实施方案中,DNA模板任选地编码可用于离析或分离修饰细胞亚群的可选择标志物。
在一些实施方案中,结合如上所述的监测细胞增殖,或者代替监测细胞增殖,可监测随着模板插入物而变的细胞的mRNA。参见例如图24a。例如,这可使用单细胞RNA-seq进行,即在分区中,其可包括液滴或其他类型的分区。从细胞得到的cDNA读段可与基于分区特异性条形码的特定细胞相关联。为了将每个分区特异性条形码与特定模板插入相关联,可在反应中扩增一部分cDNA以形成双条形码扩增子,该扩增子包含连接到cDNA的分区特异性条形码以及指示模板插入物的身份的独特条形码。通过对这些扩增子进行测序,可将分区特异性条形码(代表特定细胞)与指示插入这些相同细胞中的模板的独特条形码相关联。因此,来自RNA-seq的cDNA读段可根据分区特异性条形码分选为来自包含相同模板插入物的细胞的读段(由双条形码扩增子中独特条形码和分区特异性条形码的关联决定)。参见例如图24b和实施例2。因此,在一些实施方案中,所述方法包括生成双条形码扩增子,其包含连接至cDNA的分区特异性条形码以及指示来自包含如本文所述的分区特异性条形码的cDNA的模板插入物的身份的独特条形码。
在一些实施方案中,通过将包含DNA模板的病毒载体引入细胞来插入DNA模板文库。病毒载体的实例包括但不限于腺相关病毒(AAV)载体、逆转录病毒载体或慢病毒载体。在一些实施方案中,慢病毒载体是整合酶缺陷型慢病毒载体。
在一些实施方案中,通过将包含核酸的非病毒载体引入细胞来插入DNA模板文库。在非病毒递送方法中,核酸可以是裸DNA,或者在非病毒质粒或载体中。对于非病毒递送方法,可使用基于Cas9‘穿梭’系统和阴离子聚合物的非病毒基因组靶向方案来插入DNA模板。转座子传递系统也可用于将DNA模板文库插入细胞中。
在一些实施方案中,通过将以下各物引入T细胞中来将核酸插入T细胞:(a)切割TCR-α亚基恒定基因(TRAC)的外显子1中的靶区域以在T细胞基因组中产生插入位点的靶向核酸酶;和(b)DNA模板,其中通过同源定向修复(HDR)将核酸序列并入插入位点。在一些实施方案中,通过将以下各物引入T细胞中来将核酸插入T细胞:(a)切割TCR-β亚基恒定基因(TRBC)的外显子1中的靶区域以在T细胞基因组中产生插入位点的靶向核酸酶;和(b)DNA模板,其中通过同源定向修复(HDR)将核酸序列并入插入位点。在一些实施方案中,将核酸插入到T细胞的TRAC外显子2、TRAC外显子3、TRAC外显子4、TRBC1外显子1、TRBC1外显子2、TRBC1外显子3、TRBC1外显子4、TRBC2外显子1、TRBC2外显子2、TRBC2外显子3或TRBC2外显子4中。
在一些情况下,核酸序列作为线性DNA模板被引入细胞。在一些情况下,核酸序列作为双链DNA模板被引入细胞。在一些情况下,DNA模板是单链DNA模板。在一些情况下,单链DNA模板是纯单链DNA模板。如本文所用,“纯单链DNA”是指基本上缺乏另一条或相反DNA链的单链DNA。“基本上缺乏”是指纯单链DNA中一条DNA链相比于另一条DNA链的缺乏程度为至少100倍。在一些情况下,DNA模板是双链或单链质粒或小环。
在一些实施方案中,靶向核酸酶选自由以下组成的组:RNA指导的核酸酶结构域、转录激活因子样效应物核酸酶(TALEN)、锌指核酸酶(ZFN)和megaTAL(参见例如Merkert和Martin“Site-Specific Genome Engineering in Human Pluripotent Stem Cells,”Int.J.Mol.Sci.18(7):1000(2016))。在一些实施方案中,RNA指导的核酸酶是Cas9核酸酶并且所述方法还包括将指导RNA引入细胞中,所述指导RNA与细胞基因组中的靶区域(例如T细胞中的TRAC基因的外显子1中的靶区域)特异性杂交。在其他实施方案中,RNA指导的核酸酶是Cas9核酸酶并且所述方法还包括将与TRBC基因的外显子1中的靶区域特异性杂交的指导RNA引入细胞中。
如通篇所用,指导RNA(gRNA)序列是与位点特异性或靶向核酸酶相互作用并与细胞基因组内的靶核酸特异性结合或杂交的序列,使得gRNA和靶向核酸酶共定位于细胞基因组中的靶核酸。每个gRNA包括长度为约10至50个核苷酸的DNA靶向序列或原型间隔区序列,其与基因组中的靶DNA序列特异性结合或杂交。例如,DNA靶向序列的长度为约10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49或50个核苷酸。在一些实施方案中,gRNA包含crRNA序列和反式激活crRNA(tracrRNA)序列。在一些实施方案中,gRNA不包含tracrRNA序列。
通常,DNA靶向序列被设计为互补(例如,完全互补)或基本上互补靶DNA序列。在一些情况下,DNA靶向序列可结合摆动或简并碱基以结合多个遗传元件。在一些情况下,结合区域的3′或5′端的19个核苷酸与一个或多个靶遗传元件完全互补。在一些情况下,可改变结合区域以增加稳定性。例如,可并入非天然核苷酸以增加RNA对降解的抗性。在一些情况下,可改变或设计结合区域以避免或减少结合区域中二级结构的形成。在一些情况下,可设计结合区域以优化G-C含量。在一些情况下,G-C含量优选在约40%和约60%之间(例如,40%、45%、50%、55%、60%)。在一些实施方案中,Cas9蛋白可以是活性核酸内切酶形式,使得当作为与指导RNA的复合物的一部分或与DNA模板的复合物的一部分与靶核酸结合时,双链断裂被引入到靶核酸中。在本文提供的方法中,可将Cas9多肽或编码Cas9多肽的核酸引入细胞中。双链断裂可通过HDR修复以将DNA模板插入细胞的基因组中。多种Cas9核酸酶可用于本文所述的方法中。例如,可利用Cas9核酸酶,其需要紧邻由指导RNA靶向的区域的3′的NGG原型间隔区相邻基序(PAM)。此类Cas9核酸酶可被靶向例如包含NGG序列的TRAC的外显子1或TRAB的外显子1中的区域。作为另一个实例,具有正交PAM基序要求的Cas9蛋白可用于靶向不具有相邻NGG PAM序列的序列。具有正交PAM序列特异性的示例性Cas9蛋白包括但不限于在Esvelt等人,Nature Methods 10:1116-1121(2013)中描述的那些。
在一些情况下,Cas9蛋白是切口酶,因此当作为与指导RNA的复合物的一部分与靶核酸结合时,单链断裂或切口被引入靶核酸中。一对Cas9切口酶(每个都与结构不同的指导RNA结合)可靶向靶基因组区域的两个近端位点,并因此将一对近端单链断裂引入靶基因组区域,例如TRAC基因的外显子1或TRBC基因的外显子1。切口酶对可提供增强的特异性,因为脱靶效应可能导致单个切口,这些切口通常通过碱基切除修复机制进行修复而不会造成损伤。示例性的Cas9切口酶包括具有D10A或H840A突变的Cas9核酸酶(参见例如Ran等人,“Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editingspecificity,”Cell 154(6):1380-1389(2013))。
在一些实施方案中,将Cas9核酸酶、指导RNA和核酸序列作为核糖核蛋白复合物(RNP)-DNA模板复合物引入细胞,其中所述RNP-DNA模板复合物包含:(i)RNP,其中RNP包含Cas9核酸酶和指导RNA;(ii)DNA模板。
在一些实施方案中,RNP与DNA模板的摩尔比可为约3∶1至约100∶1。例如,摩尔比可为约5∶1至10∶1、约5∶1至约15∶1、5∶1至约20∶1、5∶1至约25∶1、约8∶1至约12∶1、约8∶1至约15∶1、约8∶1至约20∶1、或约8∶1至约25∶1。
在一些实施方案中,RNP-DNA模板复合物中的DNA模板的浓度为约2.5pM至约25pM。在一些实施方案中,DNA模板的量为约1μg至约10μg。
在一些情况下,RNP-DNA模板复合物是通过将RNP与DNA模板在约20℃至约25℃的温度下孵育少于约一分钟至约三十分钟而形成的。在一些实施方案中,在将RNP-DNA模板复合物引入细胞之前,将RNP-DNA模板复合物和细胞混合。
在一些实施方案中,通过电穿孔将核酸序列或RNP-DNA模板复合物引入细胞。用于电穿孔细胞以引入RNP-DNA模板复合物的方法、组合物和装置可包括本文实施例中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括在WO/2006/001614或Kim,J.A.等人,Biosens.Bioelectron.23,1353-1360(2008)中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括美国专利申请公开第2006/0094095号、第2005/0064596号或第2006/0087522号中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括Li,L.H.等人,Cancer Res.Treat.1,341-350(2002);美国专利第6,773,669号;第7,186,559号;第7,771,984号;第7,991,559号;第6485961号;第7029916号;以及美国专利申请公开第2014/0017213号;和第2012/0088842号中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括在Geng,T.等人,J.Control Release144,91-100(2010);和Wang,J.等人,Lab.Chip 10,2057-2061(2010)中描述的那些。
在一些实施方案中,RNP在阴离子聚合物的存在下被递送至细胞。在一些实施方案中,阴离子聚合物是阴离子多肽或阴离子多糖。在一些实施方案中,阴离子聚合物是阴离子多肽(例如,聚谷氨酸(PGA)、聚天冬氨酸或聚羧基谷氨酸)。在一些实施方案中,阴离子聚合物是阴离子多糖(例如,透明质酸(HA)、肝素、硫酸肝素或糖胺聚糖)。在一些实施方案中,阴离子聚合物是聚(丙烯酸)(PAA)、聚(甲基丙烯酸)(PMAA)、聚(苯乙烯磺酸盐)或聚磷酸盐。在一些实施方案中,阴离子聚合物具有至少15kDa(例如,在15kDa和50kDa之间)的分子量。在一些实施方案中,阴离子聚合物和Cas蛋白的摩尔比分别在10∶1和120∶1之间(例如,10∶1、20∶1、30∶1、40∶1、50∶1、60∶1、70∶1、80∶1、90∶1、100∶1、110∶1或120∶1)。在该方面的一些实施方案中,sgRNA∶Cas蛋白的摩尔比在0.25∶1和4∶1之间(例如,0.25∶1、0.5∶1、1∶1、1.2∶1、1.4∶1、1.6∶1、1.8∶1、2∶1、2.2∶1、2.4∶1、2.6∶1、2.8∶1、3∶1、3.2∶1、3.4∶1、3.6∶1、3.8∶1或4∶1)。
在一些实施方案中,供体模板包含同源定向修复(HDR)模板和一个或多个DNA结合蛋白靶序列。在一些实施方案中,供体模板具有一个DNA结合蛋白靶序列和一个或多个原型间隔区相邻基序(PAM)。包含DNA结合蛋白(例如,RNA指导的核酸酶)、供体gRNA和供体模板的复合物可将供体模板穿梭(例如,不切割DNA结合蛋白靶序列)到所需的细胞内位置(例如细胞核),使得HDR模板可整合到切割的靶核酸中。在一些实施方案中,DNA结合蛋白靶序列和PAM位于HDR模板的5′末端。特别地,在一些实施方案中,PAM可位于DNA结合蛋白靶序列的5′末端。在其他实施方案中,PAM可位于DNA结合蛋白靶序列的3′末端。在一些实施方案中,DNA结合蛋白靶序列和PAM位于HDR模板的3′末端。特别地,在一些实施方案中,PAM可位于DNA结合蛋白靶序列的5′末端。在其他实施方案中,PAM位于DNA结合蛋白靶序列的3′末端。在一些实施方案中,供体模板具有两个DNA结合蛋白靶序列和两个PAM。特别地,在一些实施方案中,第一DNA结合蛋白靶序列和第一PAM位于HDR模板的5′末端,而第二DNA结合蛋白靶序列和第二PAM位于HDR模板的3′末端。在一些实施方案中,第一PAM位于第一DNA结合蛋白靶序列的5′末端并且第二PAM位于第二DNA结合蛋白靶序列的5′。在其他实施方案中,第一PAM位于第一DNA结合蛋白靶序列的5′末端并且第二PAM位于第二DNA结合蛋白靶序列的3′。在其他实施方案中,第一PAM位于第一DNA结合蛋白靶序列的3′末端并且第二PAM位于第二DNA结合蛋白靶序列的5′。在其他实施方案中,第一PAM位于第一DNA结合蛋白靶序列的3′末端并且第二PAM位于第二DNA结合蛋白靶序列的3′。
在一些实施方案中,将核酸序列或RNP-DNA模板复合物引入约1×10
在一些实施方案中,细胞是哺乳动物细胞,例如人类细胞。细胞也可以是细胞系。在一些实施方案中,人类细胞是造血细胞,例如免疫细胞,例如造血干细胞、T细胞、B细胞、巨噬细胞、自然杀伤(NK)细胞或树突细胞。
在本文提供的方法和组合物中,人T细胞可以是原代T细胞。在一些实施方案中,T细胞是调节性T细胞、效应T细胞或原初T细胞。在一些实施方案中,效应T细胞是CD8
组合物
本文还提供了包含编码多肽的编码核苷酸序列的核酸构建体,其中每个DNA模板的5′端和3′端包含与细胞基因组中插入位点侧翼的基因组序列同源的核苷酸序列,其中一个或两个同源核苷酸序列与细胞中的同源基因组序列相比包含错配核苷酸序列;并且其中错配核苷酸序列的长度足以防止与对应于错配核苷酸序列的基因组序列特异性结合的引物的结合。细胞中插入位点的示例性基因组序列可包括例如人TCR基因座内的序列。
在一些实施方案中,编码核苷酸序列包含由自切割肽的编码序列连接的两个异源编码序列。自切割肽的实例包括但不限于自切割病毒2A肽,例如,猪捷申病毒-1(P2A)肽、明脉扁刺蛾(Thosea asigna)病毒(T2A)肽、马鼻炎A病毒(E2A)肽或口蹄疫病毒(F2A)肽。自切割2A肽允许从单个构建体表达多个基因产物。(参见例如Chng等人,“Cleavage efficient2A peptides for high level monoclonal antibody expression in CHO cells,”MAbs7(2):403-412(2015))。在一些实施方案中,核酸构建体包含两个或更多个自切割肽。在一些实施方案中,两个或更多个自切割肽都是相同的。在其他实施方案中,两个或更多个自切割肽中的至少一者是不同的。
在一些实施方案中,一个或多个接头序列将核酸构建体的组分隔开。接头序列的长度可以是二、三、四、五、六、七、八、九、十个氨基酸或更长。在一些实施方案中,构建体中的一个或多个接头序列具有所述序列。在一些实施方案中,构建体中的一个或多个接头序列具有不同的序列。在一些实施方案中,接头是GSG接头或SGSG接头。
在一些实施方案中,错配核苷酸序列的长度为约3至约40个核苷酸。在一些实施方案中,核酸构建体是图22中所示的构建体。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)多肽;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中核酸构建体包含条形码序列,其中插入序列是T细胞的TCR基因座,其中一个或两个同源核苷酸序列包含错配核苷酸序列,并且其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。如通篇所用,术语“内源TCR亚基”是TCR亚基,例如,由引入核酸构建体的细胞内源表达的TCR-α或TCR-β。在一些实施方案中,在将核酸构建体插入细胞的TCR基因座后,构建体处于内源TCR启动子例如TRAC1启动子或TRBC启动子的控制下。一旦构建体通过HDR并入T细胞的基因组中,并且在内源启动子的控制下,T细胞可在允许插入的构建体转录成编码融合多肽的单个mRNA序列的条件下培养。插入本文所述的编码异源T细胞受体和异源多肽组分的任何核酸构建体将产生具有异源TCR受体特异性和异源多肽功能的T细胞。类似地,插入本文所述的编码合成抗原受体和异源多肽的任何核酸构建体将产生具有异源TCR受体特异性和异源多肽功能的T细胞。
在一些实施方案中,条形码可插入到编码内源TCR亚基的N末端的一部分的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)多肽;(iii)第二自切割肽序列;(iv)第一异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座,其中一个或两个同源核苷酸序列包含错配核苷酸序列,并且其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,条形码可插入到编码内源TCR亚基的N末端的一部分的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)第二异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(v)第三自切割肽序列;(vi)多肽;和(vii)第四自切割肽序列或poly A序列,其中核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座,其中一个或两个同源核苷酸序列包含错配核苷酸序列,并且其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,条形码可插入在编码第四自切割肽或polyA序列的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)合成抗原受体;(iii)第二自切割肽序列;(iv)异源多肽;和(v)第三自切割肽序列或polyA序列,其中核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,条形码可插入在编码第三自切割肽或polyA序列的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)多肽;(iii)第二自切割肽序列;(iv)合成抗原受体;和(v)第三自切割肽序列或polyA序列,其中核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,条形码可插入在编码第三自切割肽或polyA序列的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一TCRβ或α亚基链,其中TCR亚基链包含TCR亚基链的可变区和恒定区;(iii)第二自切割肽序列;(iv)第二TCRβ或α亚基链,其中第二TCR亚基链不同于第一TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;或者TCR亚基包含所述亚基的可变区;和(v)第三自切割肽序列或polyA序列,其中核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,条形码可插入在编码第三自切割肽或polyA序列的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)合成抗原受体;和(v)第二自切割肽序列或polyA序列,其中核酸构建体包含条形码序列,其中插入序列是人T细胞的TCR基因座。
在一些实施方案中,条形码可插入在编码第三自切割肽或polyA序列的核酸序列之中、之前或之后。在一些实施方案中,条形码可插入在编码第一自切割肽的核酸序列之中、之前或之后。
在任何编码poly A序列的构建体中,poly A序列用作终止子序列,可被另一种合适的编码停止或终止转录的终止子序列的核酸取代。
在一些实施方案中,核酸构建体编码合成抗原受体,其中合成抗原受体是嵌合抗原受体(CAR)或SynNotch受体。参见例如Sadelain等人,Cancer Discov.3(4):388-398(2013));Srivastava Trends Immunol.36(8):494-502(2015));Toda等人,Science 361(6398):156-162(2018);和Cho等人,Scientific Reports 8:3846(2018),关于CAR和SynNotch设计和使用)。
在一些实施方案中,本文所述的任何一种核酸构建体包含一个或多个指示多肽身份的条形码序列。在一些实施方案中,本文所述的核酸构建体中的任一者包含一对独特条形码,其位于编码多肽的核苷酸序列的侧翼(即,编码多肽的核苷酸序列任一端的不同条形码)。在一些实施方案中,本文所述的核酸构建体中的任一者包含位于自切割肽序列或polyA序列之前、之后或之中的一个或多个条形码。
在一些实施方案中,核酸构建体包含一个或多个将核酸构建体的组分隔开的接头序列。在一些实施方案中,一个或多个接头序列具有相同的序列。关于示例性构建体,参见图22和图34。
还提供了包含两个或更多个本文所述的核酸构建体的文库,其中每个构建体编码不同的多肽。还提供了包含本文所述的任何文库的细胞群体。进一步提供了包含一种或多种本文所述的核酸构建体的细胞。在一些实施方案中,细胞是人T细胞。
在内源基因座控制下共表达的异源多肽
本文提供异源表达多肽的人T细胞,其中所述多肽由插入细胞TCR基因座的核酸构建体编码。本文所述的任何多肽均可在人T细胞中异源表达。示例性多肽包括但不限于如SEQ ID No:37-72所示的氨基酸序列。可异源表达的其他多肽包括包含如SEQ ID No:73-116所示的氨基酸序列的多肽。包含与SEQ ID No:37-116所示的任一氨基酸序列具有至少80%、85%、90%、99%或100%同一性的氨基酸序列的多肽也可在人T细胞中异源表达。
在一些实施方案中,多肽是截短的人PD-1蛋白,其包含人PD-1胞外结构域和跨膜结构域并且缺少80-90个(例如87个)羧基末端PD-1氨基酸。在一些实施方案中,截短的人PD-1蛋白包含人PD-1胞内结构域的前1-20个(例如12个)氨基酸但缺少剩余的人PD-1蛋白胞内结构域。在一些实施方案中,截短的人PD-1蛋白包含SEQ ID NO:37或由SEQ ID NO:37组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人4-1BB胞内结构域的人PD-1胞外结构域或其至少120或130个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个(例如11个)氨基酸)。在一些实施方案中,跨膜结构域是人4-1BB或PD-1跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:38或由SEQ ID NO:38组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地PD-1胞内结构域的1-10个氨基酸)的人PD-1胞外结构域。在一些实施方案中,跨膜结构域是人PD-1或MyD88跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:39或由SEQ ID NO:39组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人ICOS胞内结构域的人PD-1胞外结构域。在一些实施方案中,跨膜结构域是人ICOS或PD-1跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:40或由SEQ ID NO:40组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人CTLA4胞外结构域和跨膜结构域并且缺少30-40个(例如34个)羧基末端CTLA4氨基酸的截短的人CTLA4蛋白。在一些实施方案中,截短的人CTLA4蛋白包含人CTLA4胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人CTLA4蛋白胞内结构域。在一些实施方案中,截短的CTLA4蛋白包含SEQ ID NO:41或由SEQ ID NO:41组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地CTLA4胞内结构域的1-10个氨基酸)的人CTLA4胞外结构域。在一些实施方案中,跨膜结构域是人CTLA4或CD28跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:42或由SEQ ID NO:42组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人CD200R胞外结构域和跨膜结构域并且缺少50-60个羧基末端CD200R氨基酸的截短的人CD200R蛋白。在一些实施方案中,截短的人CD200R蛋白包含人CD200R胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人CD200R蛋白胞内结构域。在一些实施方案中,截短的人CD200R蛋白包含SEQ ID NO:43或由SEQ ID NO:43组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人BTLA胞外结构域和跨膜结构域并且缺少100-110个(例如104个)羧基末端BTLA氨基酸的截短的人BTLA蛋白。在一些实施方案中,截短的人BTLA蛋白包含人BTLA胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人BTLA蛋白胞内结构域。在一些实施方案中,截短的人BTLA4蛋白包含SEQ ID NO:44或由SEQ ID NO:44组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人CD28胞内结构域的人BTLA胞外结构域或其至少110或120个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)。在一些实施方案中,跨膜结构域是人CD28或BTLA跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:45或由SEQ ID NO:45组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人TIM-3胞外结构域和跨膜结构域并且缺少65-75个(例如71个)羧基末端TIM-3氨基酸的截短的人TIM-3蛋白。在一些实施方案中,截短的人TIM-3蛋白包含人TIM-3胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人TIM-3蛋白胞内结构域。在一些实施方案中,多肽包含SEQ ID NO:46或由SEQ ID NO:46组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人CD28胞内结构域的人TIM-3胞外结构域或其至少160或170个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)。在一些实施方案中,跨膜结构域是人CD28或TIM-3跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:47或由SEQ ID NO:47组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人TIGIT胞外结构域和跨膜结构域并且缺少70-80个(例如75个)羧基末端TIGIT氨基酸的截短的人TIGIT蛋白。在一些实施方案中,截短的人TIGIT蛋白包含人TIGIT胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人TIGIT蛋白胞内结构域。在一些实施方案中,多肽包含SEQ ID NO:48或由SEQ ID NO:48组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人CD28胞内结构域的人TIGIT胞外结构域或其至少100或110个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)。在一些实施方案中,跨膜结构域是人CD28或TIGIT跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:49或由SEQ ID NO:49组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人TGFβR2胞外结构域和跨膜结构域并且缺少360-370个(例如366个)羧基末端TGFβR2氨基酸的截短的人TGFβR2蛋白。在一些实施方案中,截短的人TGFβR2蛋白包含人TGFβR2胞内结构域的前1-20个(例如13个)氨基酸但缺少剩余的人TGFβR2蛋白胞内结构域。在一些实施方案中,多肽包含SEQ ID NO:50或由SEQ ID NO:50组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人4-1BB胞外结构域的人TGFβR2胞外结构域或其至少130或140个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个氨基酸)。在一些实施方案中,跨膜结构域是人4-1BB或TGFβR2跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:51或由SEQ ID NO:51组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人Myd88胞内结构域或其至少90或100个氨基酸的部分(和任选地TGFβR2胞内结构域的1-20个氨基酸)的人TGFβR2胞外结构域。在一些实施方案中,跨膜结构域是人TGFβR2或Myd88跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:52或由SEQ ID NO:52组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含含有人IL-10RA胞外结构域和跨膜结构域并且缺少310-320个(例如315个)羧基末端IL-10RA氨基酸的截短的人IL-10RA蛋白。在一些实施方案中,截短的人IL-10RA蛋白包含人IL-10RA胞内结构域的前1-20个(例如13个)氨基酸但缺少剩余的人IL-10RA蛋白胞内结构域。在一些实施方案中,多肽包含SEQ ID NO:53或由SEQ IDNO:53组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-10RA胞外结构域。在一些实施方案中,跨膜结构域包含人IL-7RA或IL-10RA跨膜结构域或其至少20个氨基酸长的部分。在一些实施方案中,多肽包含SEQ ID NO:54或由SEQ IDNO:54组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-4RA胞外结构域。在一些实施方案中,跨膜结构域包含人IL-7RA或IL-4RA跨膜结构域或其至少20个氨基酸长的部分。在一些实施方案中,多肽包含SEQ ID NO:55或由SEQ ID NO:55组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人Fas胞外结构域和跨膜结构域并且缺少132-142个(例如138个)羧基末端Fas氨基酸的截短的人Fas蛋白。在一些实施方案中,截短的人Fas蛋白包含人Fas胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人Fas蛋白胞内结构域。在一些实施方案中,多肽包含SEQ ID NO:59或由SEQ ID NO:59组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,跨膜结构域是人Fas或CD28跨膜结构域。在一些实施方案中,多肽包含SEQID NO:60或由SEQ ID NO:60组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人4-1BB胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,跨膜结构域是人Fas或4-1BB跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:61或由SEQ ID NO:61组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,多肽包含SEQ ID NO:62或由SEQ ID NO:62组成。在一些实施方案中,跨膜结构域是人Fas或MyD88跨膜结构域。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人ICOS胞内结构域或其至少25或35个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域。在一些实施方案中,跨膜结构域是人Fas或ICOS跨膜结构域。在一些实施方案中,多肽包含SEQID NO:63或由SEQ ID NO:63组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽是包含人TRAIL-R2胞外结构域和跨膜结构域并且缺少196-206个(例如202个)羧基末端TRAIL-R2氨基酸的截短的人TRAIL-R2蛋白。在一些实施方案中,截短的人TRAIL-R2蛋白包含人TRAIL-R2胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人TRAIL-R2蛋白胞内结构域。在一些实施方案中,多肽包含SEQ ID NO:64或由SEQ ID NO:64组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地TRAIL-R2胞内结构域的1-20个氨基酸)的人TRAIL-R2胞外结构域。在一些实施方案中,跨膜结构域是人TRAIL-R2或CD28跨膜结构域。在一些实施方案中,多肽包含SEQ ID NO:65或由SEQ ID NO:65组成。在一些实施方案中,相关结构域包含与表1中列出的序列具有至少95%或100%同一性的氨基酸序列。
在一些实施方案中,多肽包含全长CCR10、MCT4、SOD1、TCF7、IL-2RA、IL-7RA或41BB蛋白。
在一些实施方案中,多肽包含与选自由以下组成的组的氨基酸序列具有至少95%同一性的氨基酸序列:SEQ ID NO:42、SEQ ID NO:45、SEQ ID NO:47、SEQ ID NO:50、SEQ IDNO:51、SEQ ID NO:52、SEQ ID NO:56、SEQ ID NO:57、SEQ ID NO:59、SEQ ID NO:61、SEQ IDNO:62、SEQ ID NO:64、SEQ ID NO:67和SEQ ID NO:69。
表1
本文所述的核酸序列,例如SEQ ID No:1-36,以及编码本文所述的任何多肽的核酸序列,可插入到T细胞基因组的任何基因座,例如,T细胞的TCR基因座。在一些实施方案中,将编码SEQ ID No:37-116中任一者的核酸序列插入到T细胞的TCR基因座中。在一些实施方案中,将与如SEQ ID No:1-36所示的任一核酸序列或核酸序列具有至少80%、85%、90%、99%或100%同一性的核酸序列或编码SEQ ID No:37-116中任一者的核酸序列插入到T细胞的TCR基因座中。
在一些实施方案中,核酸序列或构建体包含与选自由以下组成的组的核酸序列具有至少95%同一性的核酸序列:SEQ ID NO:6、SEQ ID NO:9、SEQ ID NO:11、SEQ ID NO:14、SEQ ID NO:15、SEQ ID NO:16、SEQ ID NO:20、SEQ ID NO:21、SEQ ID NO:23、SEQ ID NO:25、SEQ ID NO:26、SEQ ID NO:27、SEQ ID NO:31和SEQ ID NO:33。与选自由SEQ ID NO:6、SEQ ID NO:9、SEQ ID NO:11、SEQ ID NO:14、SEQ ID NO:15、SEQ ID NO:16、SEQ ID NO:20、SEQ ID NO:21、SEQ ID NO:23、SEQ ID NO:25、SEQ ID NO:26、SEQ ID NO:27、SEQ ID NO:31和SEQ ID NO:33组成的组的核酸序列具有至少95%同一性的核酸序列可插入T细胞基因组中的任何基因座,例如T细胞的TCR基因座。
本发明人已经发现,本文所述的核酸构建体可插入到T细胞中以改变T细胞的功能。在一些实施方案中,构建体编码融合蛋白,所述融合蛋白包含经由跨膜结构域连接到第二蛋白质的胞内结构域的第一蛋白质的胞外结构域(表2)。在一些实施方案中,融合蛋白可通过表达插入TCR或其他T细胞基因座的异源编码序列而在T细胞中表达,如本文别处所述。然而,鉴于发现第二蛋白质的胞内结构域改变了第一蛋白质的功能(例如,信号传导),其他选择也是可能的。例如,在一些实施方案中,可将编码第二蛋白质的胞内结构域的异源核酸构建体插入到T细胞的基因组中以修饰细胞中的内源蛋白质(即,具有所需的胞外结构域)。例如,可将异源胞内结构域连接到由内源基因座编码的内源蛋白质的胞质结构域或其片段,以产生具有胞内结构域活性的修饰的内源(融合)蛋白质。内源蛋白质可以是本发明人测试的任何构建体中的第一蛋白质或不同的蛋白质。或者,内源蛋白质可以是任何构建体中的第二蛋白质,在这种情况下,将融合物的异源胞外结构域的编码序列引入内源基因座,从而在内源基因座的调控下生成融合物。异源胞内或胞外结构域可插入内源蛋白质的胞内结构域中,如图2所示。
例如,包含经由跨膜结构域连接至人4-1BB胞内结构域的人PD-1胞外结构域或其至少120或130个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个(例如11个)氨基酸)的多肽可从PD-1或4-BB内源基因座表达,其中另一个成员如图2所示被引入(例如,4-1BB胞内结构域与内源PD-1基因座中的内源PD-1胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人ICOS胞内结构域的人PD-1胞外结构域的多肽可从PD-1或ICOS内源基因座表达,其中另一个成员如图2所示被引入(例如,ICOS胞内结构域与内源PD-1胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地CTLA4胞内结构域的1-10个氨基酸)的人CTLA4胞外结构域的多肽可从CTLA4或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,CD28胞内结构域与内源CTLA4基因座中的内源CTLA4胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人CD28胞内结构域的人BTLA胞外结构域或其至少110或120个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽可从BTLA或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,CD28胞内结构域与内源BTLA基因座中的内源BTLA胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人CD28胞内结构域的人TIM-3胞外结构域或其至少160或170个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽可从TIM-3或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,CD28胞内结构域与内源Tim-3基因座中的内源TIM-3胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人CD28胞内结构域的人TIGIT胞外结构域或其至少100或110个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽可从TIGIT或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,CD28胞内结构域与内源TIGIT基因座中的内源TIGIT胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人4-1BB胞内结构域的人TGFβR2胞外结构域或其至少130或140个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个氨基酸)的多肽可从TGFβR2或41BB内源基因座表达,其中另一个成员如图2所示被引入(例如,41BB胞内结构域与内源TGFβR2基因座中的内源TGFβR2胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人Myd88胞内结构域或其至少90或100个氨基酸的部分(和任选地TGFβR2胞内结构域的1-20个氨基酸)的多肽可从TGFβR2或Myd88内源基因座表达,其中另一个成员如图2所示被引入(例如,Myd88胞内结构域与内源TGFβR2基因座中的内源TGFβR2胞外结构域融合)。
在另一个实例中,包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-10RA胞外结构域的多肽可从IL-10RA或IL-7RA内源基因座表达,其中另一个成员如图2所示被引入(例如,IL-7RA胞内结构域与内源IL-10RA基因座中的内源IL-10RA胞外结构域融合)。
在一些实例中,包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-4RA胞外结构域的多肽可从IL-4RA或IL-7RA内源基因座表达,其中另一个成员如图2所示被引入(例如,IL-7RA胞内结构域与内源IL-4RA基因座中的内源IL-4RA胞外结构域融合)。
在一些实例中,包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽可从Fas或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,CD28胞内结构域与内源Fas基因座中的内源Fas胞外结构域融合)。
在一些实例中,包含经由跨膜结构域连接至人41BB胞内结构域或其至少30或40个氨基酸(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽可从Fas或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,41BB胞内结构域与内源Fas基因座中的内源Fas胞外结构域融合)。
在一些实例中,包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽可从Fas或MyD88内源基因座表达,其中另一个成员如图2所示被引入(例如,MyD88胞内结构域与内源Fas基因座中的内源Fas胞外结构域融合)。
在一些实例中,包含经由跨膜结构域连接至人ICOS胞内结构域或其至少25或35个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽可从Fas或ICOS内源基因座表达,其中另一个成员如图2所示被引入(例如,ICOS胞内结构域与内源Fas基因座中的内源Fas胞外结构域融合)。
在一些实例中,包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地TRAIL-R2胞内结构域的1-20个氨基酸)的人TRAIL-R2胞外结构域的多肽可从TRAIL-R2或CD28内源基因座表达,其中另一个成员如图2所示被引入(例如,CD28胞内结构域与内源TRAIL-R2基因座中的内源TRAIL-R2胞外结构域融合)。
在已显示截短的多肽具有活性(例如,和Fas)的实施方案中,这些截短的蛋白质可从异源表达盒(即,与编码序列可操作地连接的启动子)表达或者T细胞中的内源基因座可如本文所述被修饰以表达截短的形式。也可表达其他截短的多肽(例如,PD-1、CTL4、CD200R、BTLA、TIM-3、TIGIT、IL-10RA、Fas)(例如,整合的或例如从病毒载体表达)。
最后,本文显示以下全长基因产物对T细胞增殖有影响(例如MCT4和TCF7)。这些基因产物和其他全长基因(例如CCR10、SOD1、IL-2RA、IL-7RA、4lBB)可从引入T细胞的异源表达盒(整合的或例如从病毒载体表达)表达,或者它们的内源基因座可被修饰以具有异源启动子序列(例如,如图2中一般性所示),导致与内源启动子相比基因产物的更高表达。
可要求保护任何多肽序列、核酸序列、包含多肽或核酸序列的T细胞,或使用本文所述的T细胞、多肽或核酸序列的方法。
将异源编码序列插入TCR基因座意味着异源蛋白的表达将受内源TCR启动子控制,并且在一些实施方案中将作为与TCR多肽的更大融合蛋白的一部分表达,随后将其切割形成单独的TCR和异源多肽。如前所述,TCR多肽可以是内源的,或者也可添加到TCR基因座以提供对T细胞的新型TCR亲和力(例如但不限于对癌抗原)。在一些实施方案中,将核酸构建体插入TCR-α亚基恒定基因(TRAC)的外显子1中的靶插入位点。在一些实施方案中,将核酸构建体插入TCR-β亚基恒定基因(TRBC)的外显子1中的靶插入位点。在将核酸构建体插入细胞的TCR基因座后,构建体处于内源TCR启动子例如TRAC1启动子或TRBC启动子的控制下。如下所述,本文提供的核酸构建体编码与多肽共表达的TCR或合成抗原受体。一旦构建体通过HDR并入T细胞的基因组中,并且在内源启动子的控制下,T细胞就可在允许插入的构建体转录成编码融合多肽的单个mRNA序列的条件下培养,然后加工成单独的异源多肽(例如,通过切割连接多肽的肽序列)。插入本文所述的编码异源T细胞受体和异源多肽的组分的任何核酸构建体将产生具有异源TCR受体特异性和异源多肽功能的T细胞。在一些实施方案中,T细胞表达识别靶抗原的抗原特异性TCR。类似地,插入本文所述的编码合成抗原受体和异源多肽的任何核酸构建体将产生具有异源TCR受体特异性和异源多肽功能的T细胞。在一些实施方案中,T细胞表达识别靶抗原的合成抗原受体。
在一些实施方案中,插入人T细胞中的异源核酸按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)如本文所述的异源多肽;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中,如果细胞的内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果细胞的内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在本文所述的组合物和方法中,如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链。在一些方法中,如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
如通篇所用,术语“内源TCR亚基”是TCR亚基,例如,由引入核酸构建体的细胞内源表达的TCR-α或TCR-β。如上所述,本文所述的核酸构建体编码表达为多顺反子序列的多个氨基酸序列,所述多顺反子序列被加工(即自切割)以产生两个或更多个氨基酸序列,例如TCR-α亚基、TCR-β亚基和由构建体编码的多肽,或合成抗原受体(例如CAR或SynNotch受体)和由构建体编码的多肽。
在一些核酸构建体中,编码内源TCR亚基的N末端部分的核酸的大小将取决于内源TRAC或TRBC核酸序列中在TRAC外显子1或TRBC外显子1的起始端与靶向插入位点之间的核苷酸数量。例如,如果TRAC外显子1的起始端与插入位点之间的核苷酸数量小于或大于25个核苷酸,则编码内源TCR-α亚基的N末端部分的小于或大于25个核苷酸的核酸可以在构建体中。
在上面的实例中,从构建体转录的mRNA序列的翻译导致一种蛋白质的表达,该蛋白质自切割成四个单独的多肽序列,即缺乏跨膜结构域的无活性的内源可变区肽(其可以例如在内质网中降解或在翻译后分泌)、全长异源抗原特异性TCR-β链或TCR-α链、如本文所述的多肽序列和全长异源抗原特异性TCR-α链或TCR-β链。全长抗原特异性TCR-β链和全长抗原特异性TCR-α链形成具有所需抗原特异性的TCR。在一些实施方案中,多肽增强或赋予T细胞中所需的功能。从本文所述的任何其他核酸构建体转录的mRNA在T细胞中类似地加工。在一些实施方案中,构建体编码两个、三个、四个、五个、六个、七个或更多个多肽序列,任选地由编码自切割序列的核酸序列隔开。
在一些实施方案中,异源核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)多肽;(iii)第二自切割肽序列;(iv)第一异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(v)第三自切割肽序列;(vi)第二异源TCR亚基链的可变区;和(vii)内源TCR亚基的N末端的一部分,其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)第一异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(iii)第二自切割肽序列;(iv)第二异源TCR亚基链,其中TCR亚基链包含TCR亚基的可变区和恒定区;(v)第三自切割肽序列;(vi)多肽;和(vii)第四自切割肽序列或poly A序列,其中如果内源TCR亚基是TCR-alpha(TCR-α)亚基,则第一异源TCR亚基链是异源TCR-beta(TCR-β)亚基链并且第二异源TCR亚基链是异源TCR-α亚基链,而其中如果内源TCR亚基是TCR-β亚基,则第一异源TCR亚基链是异源TCR-α亚基链并且第二异源TCR亚基链是异源TCR-β亚基链。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)合成抗原受体;(iii)第二自切割肽序列;(iv)多肽;和(v)第三自切割肽序列或polyA序列。
在一些实施方案中,核酸构建体按以下顺序编码:(i)第一自切割肽序列;(ii)多肽;(iii)第二自切割肽序列;(iv)合成抗原受体;和(v)第三自切割肽序列或polyA序列。
在一些实施方案中,核酸构建体编码合成抗原受体,其中所述合成抗原受体是嵌合抗原受体(CAR)或SynNotch受体。参见例如Sadelain等人,Cancer Discov.3(4):388-398(2013));Srivastava Trends Immunol.36(8):494-502(2015));Toda等人,Science 361(6398):156-162(2018);和Cho等人,Scientific Reports 8:3846(2018),关于CAR和SynNotch设计和使用)。
在任何编码poly A序列的构建体中,所述poly A序列用作终止子序列,可被另一种编码停止或终止转录的终止子序列的合适核酸取代。
自切割肽的实例包括但不限于自切割病毒2A肽,例如,猪捷申病毒-1(P2A)肽、明脉扁刺蛾病毒(T2A)肽、马鼻炎A病毒(E2A)肽或口蹄疫病毒(F2A)肽。自切割2A肽允许从单个构建体表达多个基因产物。(参见例如Chng等人,“Cleavage efficient 2A peptidesfor high level monoclonal antibody expression in CHO cells,”MAbs 7(2):403-412(2015))。在一些实施方案中,核酸构建体包含两个或更多个自切割肽。在一些实施方案中,两个或更多个自切割肽都是相同的。在其他实施方案中,两个或更多个自切割肽中的至少一者是不同的。
在一些实施方案中,一个或多个接头序列将核酸构建体的组分隔开。接头序列的长度可以是二、三、四、五、六、七、八、九、十个氨基酸或更长。
在一些实施方案中,核酸构建体包含与人TCR基因座具有同源性的侧翼同源臂序列。在本文所述的组合物和方法中,一个或两个同源臂序列的长度为至少约50、100、150、200、250、300、350、400或450个核苷酸。在一些情况下,与基因组序列同源的核苷酸序列与基因组序列至少80%、90%、95%、99%或100%互补。在一些实施方案中,与TCR基因座中插入位点侧翼的TCR基因座中的基因组序列中的同源序列相比,一个或两个同源臂序列任选地包含错配核苷酸序列。
在一些实施方案中,核酸构建体任选地编码可用于离析或分离修饰的T细胞亚群的可选择标志物。在一些实施方案中,核酸构建体任选地包含指示多肽身份的条形码序列。
本文所述的任何多肽可由本文所述的任何核酸构建体编码。在一些实施方案中,由异源核酸构建体编码的多肽序列与选自由以下组成的组的氨基酸序列具有至少95%同一性:SEQ ID NO:42、SEQ ID NO:45、SEQ ID NO:47、SEQ ID NO:50、SEQ ID NO:51、SEQ IDNO:52、SEQ ID NO:56、SEQ ID NO:57、SEQ ID NO:59、SEQ ID NO:61、SEQ ID NO:62、SEQ IDNO:64、SEQ ID NO:67和SEQ ID NO:69。在一些实施方案中,核酸构建体包含编码多肽的核酸序列,所述多肽包含与选自由以下组成的组的蛋白质具有至少95%同一性的氨基酸序列:SEQ ID NO:38、SEQ ID NO:40、SEQ ID NO:45、SEQ ID NO:46、SEQ ID NO:47、SEQ IDNO:48、SEQ ID NO:49、SEQ ID NO:51、SEQ ID NO:52、SEQ ID NO:53、SEQ ID NO:54、SEQ IDNO:60、SEQ ID NO:61、SEQ ID NO:62、SEQ ID NO:63、SEQ ID NO:64和SEQ ID NO:65。
还提供了包含本文所述的任何核酸序列的人T细胞。还提供了包含本文所述的任何核酸序列的人T细胞群体(例如多个)。
修饰T细胞的方法
编码本文所述的任何多肽的任何核酸构建体可用于制备修饰的T细胞。在一些实施方案中,所述方法包括(a)向人T细胞中引入(i)靶向核酸酶,所述核酸酶切割人T细胞的TCR基因座中的靶区域以在细胞基因组中产生靶插入位点;和(ii)编码本文所述的任何多肽的核酸构建体,例如:包含人PD-1胞外结构域和跨膜结构域并且缺少80-90个(例如87个)羧基末端PD-1氨基酸的截短的人PD-1蛋白;包含经由跨膜结构域连接至人4-1BB胞内结构域的人PD-1胞外结构域或其至少120或130个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个(例如11个)氨基酸)的多肽;包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地PD-1胞内结构域的1-10个氨基酸)的人PD-1胞外结构域的多肽;包含经由跨膜结构域连接至人ICOS胞内结构域的人PD-1胞外结构域的多肽;包含人CTLA4胞外结构域和跨膜结构域并且缺少30-40个(例如34个)羧基末端CTLA4氨基酸的截短的人CTLA4蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地CTLA4胞内结构域的1-10个氨基酸)的人CTLA4胞外结构域的多肽;包含人CD200R胞外结构域和跨膜结构域并且缺少50-60个羧基末端CD200R氨基酸的截短的人CD200R蛋白;包含人BTLA胞外结构域和跨膜结构域并且缺少100-110个(例如104个)羧基末端BTLA氨基酸的截短的人BTLA蛋白。在一些实施方案中,所述截短的人BTLA蛋白包含人BTLA胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人BTLA蛋白胞内结构域;包含经由跨膜结构域连接至人CD28胞内结构域的人BTLA胞外结构域或其至少110或120个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TIM-3胞外结构域和跨膜结构域并且缺少65-75个(例如71个)羧基末端TIM-3氨基酸的截短的人TIM-3蛋白;包含经由跨膜结构域连接至人CD28胞内结构域的人TIM-3胞外结构域或其至少160或170个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TIGIT胞外结构域和跨膜结构域并且缺少70-80个(例如75个)羧基末端TIGIT氨基酸的截短的人TIGIT蛋白;包含经由跨膜结构域连接至人CD28胞内结构域的人TIGIT胞外结构域或其至少100或110个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TGFβR2胞外结构域和跨膜结构域并且缺少360-370个(例如366个)羧基末端TGFβR2氨基酸的截短的人TGFβR2蛋白;包含经由跨膜结构域连接至人4-1BB胞内结构域的人TGFβR2胞外结构域或其至少130或140个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个氨基酸)的多肽;包含经由跨膜结构域连接至人Myd88胞内结构域或其至少90或100个氨基酸的部分(和任选地TGFβR2胞内结构域的1-20个氨基酸)的人TGFβR2胞外结构域的多肽;包含人IL-10RA胞外结构域和跨膜结构域并且缺少310-320个(例如315个)羧基末端IL-10RA氨基酸的截短的人IL-10RA蛋白;包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-10RA胞外结构域的多肽;包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-4RA胞外结构域的多肽;包含人Fas胞外结构域和跨膜结构域并且缺少132-142个(例如138个)羧基末端Fas氨基酸的截短的人Fas蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人4-1BB胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人ICOS胞内结构域或其至少25或35个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含人TRAIL-R2胞外结构域和跨膜结构域并且缺少196-206个(例如202个)羧基末端TRAIL-R2氨基酸的截短的人TRAIL-R2蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地TRAIL-R2胞内结构域的1-20个氨基酸)的人TRAIL-R2胞外结构域的多肽;包含IL2RA蛋白、IL7RA蛋白、MCT4蛋白或TCF7蛋白的多肽;或包含与选自由SEQ ID NO:6、SEQ ID NO:9、SEQ ID NO:11、SEQ ID NO:14、SEQ ID NO:15、SEQ ID NO:16、SEQ ID NO:20、SEQ ID NO:21、SEQ ID NO:23、SEQ ID NO:25、SEQ ID NO:26、SEQ ID NO:28、SEQ ID NO:31和SEQ ID NO:33组成的组的蛋白质具有至少95%同一性的氨基酸序列的多肽;以及(b)允许重组发生,从而将核酸构建体插入靶插入位点以产生修饰的人T细胞。
在一些实施方案中,通过将以下各物引入T细胞中来将核酸插入T细胞中:(a)切割TCR-α亚基恒定基因(TRAC)的外显子1中的靶区域以产生T细胞基因组中的插入位点的靶向核酸酶;和(b)核酸构建体,其中所述核酸构建体通过同源定向修复(HDR)并入插入位点中。在一些实施方案中,通过将以下各物引入T细胞中来将核酸构建体插入T细胞中:(a)切割TCR-β亚基恒定基因(TRBC)的外显子1中的靶区域以产生T细胞基因组中的插入位点的靶向核酸酶;和(b)核酸构建体,其中核酸序列通过同源定向修复(HDR)并入插入位点中。
在一些实施方案中,通过将包含核酸构建体的病毒载体引入细胞来插入核酸构建体。病毒载体的实例包括但不限于腺相关病毒(AAV)载体、逆转录病毒载体或慢病毒载体。在一些实施方案中,慢病毒载体是整合酶缺陷型慢病毒载体。
在一些实施方案中,通过将包含核酸构建体的非病毒载体引入细胞来插入核酸构建体。在非病毒递送方法中,核酸可以是裸DNA,或者在非病毒质粒或载体中。对于非病毒递送方法,可使用基于Cas9‘穿梭’系统和阴离子聚合物的非病毒基因组靶向方案插入DNA模板。
在一些情况下,核酸序列作为线性DNA模板被引入细胞。在一些情况下,核酸序列作为双链DNA模板被引入细胞。在一些情况下,使用转座子传递系统将DNA模板引入细胞。在一些情况下,DNA模板是单链DNA模板。在一些情况下,单链DNA模板是纯单链DNA模板。如本文所用,“纯单链DNA”是指基本上缺乏另一条或相反DNA链的单链DNA。“基本上缺乏”是指纯单链DNA中一条DNA链相比于另一条DNA链的缺乏程度为至少100倍。在一些情况下,DNA模板是双链或单链质粒或小环。
在一些实施方案中,靶向核酸酶选自由以下组成的组:RNA指导的核酸酶结构域、转录激活因子样效应物核酸酶(TALEN)、锌指核酸酶(ZFN)和megaTAL(参见例如Merkert和Martin“Site-Specific Genome Engineering in Human Pluripotent Stem Cells,”Int.J.Mol.Sci.18(7):1000(2016))。在一些实施方案中,RNA指导的核酸酶是Cas9核酸酶并且所述方法还包括将指导RNA引入细胞中,所述指导RNA与细胞基因组中的靶区域(例如T细胞中的TRAC基因的外显子1中的靶区域)特异性杂交。在其他实施方案中,RNA指导的核酸酶是Cas9核酸酶并且所述方法还包括将与TRBC基因的外显子1中的靶区域特异性杂交的指导RNA引入细胞中。
如通篇所用,指导RNA(gRNA)序列是与位点特异性或靶向核酸酶相互作用并与细胞基因组内的靶核酸特异性结合或杂交的序列,使得gRNA和靶向核酸酶共定位于细胞基因组中的靶核酸。每个gRNA包括长度为约10至50个核苷酸的DNA靶向序列或原型间隔区序列,其与基因组中的靶DNA序列特异性结合或杂交。例如,DNA靶向序列的长度为约10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49或50个核苷酸。在一些实施方案中,gRNA包含crRNA序列和反式激活crRNA(tracrRNA)序列。在一些实施方案中,gRNA不包含tracrRNA序列。
通常,DNA靶向序列被设计为互补(例如,完全互补)或基本上互补靶DNA序列。在一些情况下,DNA靶向序列可结合摆动或简并碱基以结合多个遗传元件。在一些情况下,结合区域的3′或5′端的19个核苷酸与一个或多个靶遗传元件完全互补。在一些情况下,可改变结合区域以增加稳定性。例如,可并入非天然核苷酸以增加RNA对降解的抗性。在一些情况下,可改变或设计结合区域以避免或减少结合区域中二级结构的形成。在一些情况下,可设计结合区域以优化G-C含量。在一些情况下,G-C含量优选在约40%和约60%之间(例如,40%、45%、50%、55%、60%)。在一些实施方案中,Cas9蛋白可以是活性核酸内切酶形式,使得当作为与指导RNA的复合物的一部分或与DNA模板的复合物的一部分与靶核酸结合时,双链断裂被引入到靶核酸中。在本文提供的方法中,可将Cas9多肽或编码Cas9多肽的核酸引入细胞中。双链断裂可通过HDR修复以将DNA模板插入细胞的基因组中。多种Cas9核酸酶可用于本文所述的方法中。例如,可利用Cas9核酸酶,其需要紧邻由指导RNA靶向的区域的3′的NGG原型间隔区相邻基序(PAM)。此类Cas9核酸酶可被靶向例如包含NGG序列的TRAC的外显子1或TRAB的外显子1中的区域。作为另一个实例,具有正交PAM基序要求的Cas9蛋白可用于靶向不具有相邻NGG PAM序列的序列。具有正交PAM序列特异性的示例性Cas9蛋白包括但不限于在Esvelt等人,Nature Methods 10:1116-1121(2013)中描述的那些。
在一些情况下,Cas9蛋白是切口酶,因此当作为与指导RNA的复合物的一部分与靶核酸结合时,单链断裂或切口被引入靶核酸中。一对Cas9切口酶(每个都与结构不同的指导RNA结合)可靶向靶基因组区域的两个近端位点,并因此将一对近端单链断裂引入靶基因组区域,例如TRAC基因的外显子1或TRBC基因的外显子1。切口酶对可提供增强的特异性,因为脱靶效应可能导致单个切口,这些切口通常通过碱基切除修复机制进行修复而不会造成损伤。示例性的Cas9切口酶包括具有D10A或H840A突变的Cas9核酸酶(参见例如Ran等人,“Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editingspecificity,”Cell 154(6):1380-1389(2013))。
在一些实施方案中,将Cas9核酸酶、指导RNA和核酸序列作为核糖核蛋白复合物(RNP)-核酸序列(例如DNA模板)复合物引入细胞,其中所述RNP-核酸序列复合物包含:(i)RNP,其中RNP包含Cas9核酸酶和指导RNA;和(ii)核酸序列或构建体。
在一些实施方案中,RNP与DNA模板的摩尔比可为约3∶1至约100∶1。例如,摩尔比可为约5∶1至10∶1、约5∶1至约15∶1、5∶1至约20∶1、5∶1至约25∶1、约8∶1至约12∶1、约8∶1至约15∶1、约8∶1至约20∶1、或约8∶1至约25∶1。
在一些实施方案中,RNP-DNA模板复合物中的DNA模板的浓度为约2.5pM至约25pM。在一些实施方案中,DNA模板的量为约1μg至约10μg。
在一些情况下,RNP-DNA模板复合物是通过将RNP与DNA模板在约20℃至约25℃的温度下孵育少于约一分钟至约三十分钟而形成的。在一些实施方案中,在将RNP-DNA模板复合物引入细胞之前,将RNP-DNA模板复合物和细胞混合。
在一些实施方案中,通过电穿孔将核酸序列或RNP-DNA模板复合物引入细胞。用于电穿孔细胞以引入RNP-DNA模板复合物的方法、组合物和装置可包括本文实施例中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括在WO/2006/001614或Kim,J.A.等人,Biosens.Bioelectron.23,1353-1360(2008)中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括美国专利申请公开第2006/0094095号、第2005/0064596号或第2006/0087522号中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括Li,L.H.等人,Cancer Res.Treat.1,341-350(2002);美国专利第6,773,669号;第7,186,559号;第7,771,984号;第7,991,559号;第6485961号;第7029916号;以及美国专利申请公开第2014/0017213号;和第2012/0088842号中描述的那些。用于电穿孔细胞以引入RNP-DNA模板复合物的额外或替代方法、组合物和装置可包括在Geng,T.等人,J.Control Release144,91-100(2010);和Wang,J.等人,Lab.Chip 10,2057-2061(2010)中描述的那些。
在一些实施方案中,RNP在阴离子聚合物的存在下被递送至细胞。在一些实施方案中,阴离子聚合物是阴离子多肽或阴离子多糖。在一些实施方案中,阴离子聚合物是阴离子多肽(例如,聚谷氨酸(PGA)、聚天冬氨酸或聚羧基谷氨酸)。在一些实施方案中,阴离子聚合物是阴离子多糖(例如,透明质酸(HA)、肝素、硫酸肝素或糖胺聚糖)。在一些实施方案中,阴离子聚合物是聚(丙烯酸)(PAA)、聚(甲基丙烯酸)(PMAA)、聚(苯乙烯磺酸盐)或聚磷酸盐。在一些实施方案中,阴离子聚合物具有至少15kDa(例如,在15kDa和50kDa之间)的分子量。在一些实施方案中,阴离子聚合物和Cas蛋白的摩尔比分别在10∶1和120∶1之间(例如,10∶1、20∶1、30∶1、40∶1、50∶1、60∶1、70∶1、80∶1、90∶1、100∶1、110∶1或120∶1)。在该方面的一些实施方案中,sgRNA∶Cas蛋白的摩尔比在0.25∶1和4∶1之间(例如,0.25∶1、0.5∶1、1∶1、1.2∶1、1.4∶1、1.6∶1、1.8∶1、2∶1、2.2∶1、2.4∶1、2.6∶1、2.8∶1、3∶1、3.2∶1、3.4∶1、3.6∶1、3.8∶1或4∶1)。
在一些实施方案中,供体模板包含同源定向修复(HDR)模板和一个或多个DNA结合蛋白靶序列。在一些实施方案中,供体模板具有一个DNA结合蛋白靶序列和一个或多个原型间隔区相邻基序(PAM)。包含DNA结合蛋白(例如,RNA指导的核酸酶)、供体gRNA和供体模板的复合物可将供体模板穿梭(不切割DNA结合蛋白靶序列)到所需的细胞内位置(例如细胞核),使得HDR模板可整合到切割的靶核酸中。在一些实施方案中,DNA结合蛋白靶序列和PAM位于HDR模板的5′末端。特别地,在一些实施方案中,PAM可位于DNA结合蛋白靶序列的5′末端。在其他实施方案中,PAM可位于DNA结合蛋白靶序列的3′末端。在一些实施方案中,DNA结合蛋白靶序列和PAM位于HDR模板的3′末端。特别地,在一些实施方案中,PAM可位于DNA结合蛋白靶序列的5′末端。在其他实施方案中,PAM位于DNA结合蛋白靶序列的3′末端。在一些实施方案中,供体模板具有两个DNA结合蛋白靶序列和两个PAM。特别地,在一些实施方案中,第一DNA结合蛋白靶序列和第一PAM位于HDR模板的5′末端,而第二DNA结合蛋白靶序列和第二PAM位于HDR模板的3′末端。在一些实施方案中,第一PAM位于第一DNA结合蛋白靶序列的5′末端并且第二PAM位于第二DNA结合蛋白靶序列的5′。在其他实施方案中,第一PAM位于第一DNA结合蛋白靶序列的5′末端并且第二PAM位于第二DNA结合蛋白靶序列的3′。在其他实施方案中,第一PAM位于第一DNA结合蛋白靶序列的3′末端并且第二PAM位于第二DNA结合蛋白靶序列的5′。在其他实施方案中,第一PAM位于第一DNA结合蛋白靶序列的3′末端并且第二PAM位于第二DNA结合蛋白靶序列的3′。
在一些实施方案中,将核酸序列或RNP-DNA模板复合物引入约1×10
在本文提供的方法和组合物中,人T细胞可以是原代T细胞。在一些实施方案中,T细胞是调节性T细胞、效应T细胞或原初T细胞。在一些实施方案中,效应T细胞是CD8
治疗方法
本文所述的任何方法和组合物均可用于修饰从人类受试者获得的T细胞。本文所述的任何方法和组合物可用于修饰从人类受试者获得的T细胞以增强受试者中的免疫应答。本文所述的任何方法和组合物均可用于修饰从人类受试者获得的T细胞以治疗或预防疾病(例如,癌症、感染性疾病、自身免疫性疾病、移植排斥、移植物抗宿主病或受试者中的其他炎症性病症)。
本文提供了一种增强人类受试者中的免疫应答的方法,所述方法包括施用本文所述的任何修饰的T细胞,即异源表达本文所述多肽的T细胞,例如:包含人PD-1胞外结构域和跨膜结构域并且缺少80-90个(例如87个)羧基末端PD-1氨基酸的截短的人PD-1蛋白;包含经由跨膜结构域连接至人4-1BB胞内结构域的人PD-1胞外结构域或其至少120或130个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个(例如11个)氨基酸)的多肽;包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地PD-1胞内结构域的1-10个氨基酸)的人PD-1胞外结构域的多肽;包含经由跨膜结构域连接至人ICOS胞内结构域的人PD-1胞外结构域的多肽;包含人CTLA4胞外结构域和跨膜结构域并且缺少30-40个(例如34个)羧基末端CTLA4氨基酸的截短的人CTLA4蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地CTLA4胞内结构域的1-10个氨基酸)的人CTLA4胞外结构域的多肽;包含人CD200R胞外结构域和跨膜结构域并且缺少50-60个羧基末端CD200R氨基酸的截短的人CD200R蛋白;包含人BTLA胞外结构域和跨膜结构域并且缺少100-110个(例如104个)羧基末端BTLA氨基酸的截短的人BTLA蛋白。在一些实施方案中,所述截短的人BTLA蛋白包含人BTLA胞内结构域的前1-12个(例如6个)氨基酸但缺少剩余的人BTLA蛋白胞内结构域;包含经由跨膜结构域连接至人CD28胞内结构域的人BTLA胞外结构域或其至少110或120个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TIM-3胞外结构域和跨膜结构域并且缺少65-75个(例如71个)羧基末端TIM-3氨基酸的截短的人TIM-3蛋白;包含经由跨膜结构域连接至人CD28胞内结构域的人TIM-3胞外结构域或其至少160或170个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TIGIT胞外结构域和跨膜结构域并且缺少70-80个(例如75个)羧基末端TIGIT氨基酸的截短的人TIGIT蛋白;包含经由跨膜结构域连接至人CD28胞内结构域的人TIGIT胞外结构域或其至少100或110个氨基酸的部分(和任选地CD28胞外结构域的1-20个氨基酸)的多肽;包含人TGFβR2胞外结构域和跨膜结构域并且缺少360-370个(例如366个)羧基末端TGFβR2氨基酸的截短的人TGFβR2蛋白;包含经由跨膜结构域连接至人4-1BB胞内结构域的人TGFβR2胞外结构域或其至少130或140个氨基酸的部分(和任选地4-1BB胞外结构域的1-20个氨基酸)的多肽;包含经由跨膜结构域连接至人Myd88胞内结构域或其至少90或100个氨基酸的部分(和任选地TGFβR2胞内结构域的1-20个氨基酸)的人TGFβR2胞外结构域的多肽;包含人IL-10RA胞外结构域和跨膜结构域并且缺少310-320个(例如315个)羧基末端IL-10RA氨基酸的截短的人IL-10RA蛋白;包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-10RA胞外结构域的多肽;包含经由跨膜结构域连接至人IL-7RA胞内结构域的人IL-4RA胞外结构域的多肽;包含人Fas胞外结构域和跨膜结构域并且缺少132-142个(例如138个)羧基末端Fas氨基酸的截短的人Fas蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人4-1BB胞内结构域或其至少30或40个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人MyD88胞内结构域或其至少90或100个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含经由跨膜结构域连接至人ICOS胞内结构域或其至少25或35个氨基酸的部分(和任选地Fas胞内结构域的1-20个氨基酸)的人Fas胞外结构域的多肽;包含人TRAIL-R2胞外结构域和跨膜结构域并且缺少196-206个(例如202个)羧基末端TRAIL-R2氨基酸的截短的人TRAIL-R2蛋白;包含经由跨膜结构域连接至人CD28胞内结构域或其至少30或40个氨基酸的部分(和任选地TRAIL-R2胞内结构域的1-20个氨基酸)的人TRAIL-R2胞外结构域的多肽;包含IL2RA蛋白、IL7RA蛋白、MCT4蛋白或TCF7蛋白的多肽;或包含选自由SEQ ID NO:37-SEQ ID NO:116组成的组的一个或多个氨基酸序列的多肽。
在一些实施方案中,从受试者获得T细胞并使用本文提供的任何方法对其进行修饰以表达抗原特异性TCR或合成抗原受体,然后向受试者施用修饰的T细胞。在一些实施方案中,受试者患有癌症并且靶抗原是癌症特异性抗原。在一些实施方案中,受试者患有自身免疫性病症并且抗原是与自身免疫性病症相关的抗原。在一些实施方案中,受试者患有感染并且靶抗原是与感染相关的抗原。
还提供了一种治疗人类受试者的癌症的方法,所述方法包括:a)从受试者获得T细胞;b)使用本文提供的任何方法修饰T细胞以表达抗原特异性TCR或识别受试者中靶抗原的合成抗原受体;以及c)向受试者施用修饰的T细胞,其中人类受试者患有癌症并且靶抗原是癌症特异性抗原。如通篇所用,短语“癌症特异性抗原”是指癌细胞所特有的或在癌细胞中比在非癌细胞中更丰富表达的抗原。在一些实施方案中,癌症特异性抗原是肿瘤特异性抗原。
在一些实施方案中,肿瘤浸润淋巴细胞、异质性和癌症特异性T细胞群体是从癌症受试者获得并离体扩增。患者癌症的特征决定了一组定制的细胞修饰,并且使用本文所述的任何方法将这些修饰应用于肿瘤浸润淋巴细胞。
本文还提供了一种治疗人类受试者的自身免疫性疾病的方法,所述方法包括:a)从受试者获得T细胞;b)使用本文提供的任何方法修饰T细胞以表达识别受试者中靶抗原的抗原特异性TCR或合成抗原受体;以及c)向受试者施用修饰的T细胞,其中人类受试者患有自身免疫性病症并且靶抗原是与自身免疫性病症相关的抗原。在一些实施方案中,T细胞是调节性T细胞。
本文还提供了一种治疗人类受试者的感染的方法,所述方法包括:a)从受试者获得T细胞;b)使用本文提供的任何方法修饰T细胞以表达抗原特异性TCR或识别受试者中靶抗原的合成抗原受体;以及c)向受试者施用修饰的T细胞,其中受试者患有感染并且靶抗原是与受试者中的感染相关的抗原。
本文提供的任何治疗方法还可包括在修饰T细胞之前扩增T细胞群体。本文提供的任何治疗方法还可包括在修饰T细胞之后和施用于受试者之前扩增T细胞群体。
公开了可用于、可结合使用、可用于制备所公开的方法和组合物或者是其产物的材料、组合物和组分。这些和其他材料在本文中公开,并且应当理解,当公开这些材料的组合、子集、相互作用、组等时,虽然可能没有明确公开这些化合物的每个不同个体和集体组合和排列的具体参考,但每一个都在本文中被明确考虑和描述。例如,如果公开和讨论了一种方法并且讨论了可对包括在所述方法中的一个或多个分子进行的许多修改,则明确考虑所述方法的每一种组合和排列以及可能的修改,除非有相反的明确说明。同样,这些的任何子集或组合也被明确地考虑和公开。该概念适用于本公开的所有方面,包括但不限于使用所公开组合物的方法中的步骤。因此,如果存在可执行的多种额外步骤,应理解这些额外步骤中的每一者都可用所公开方法的任何特定方法步骤或方法步骤的组合来执行,并且每个这样的组合或组合子集被明确考虑并且应该被认为是公开的。
本文引用的出版物和引用它们的材料特此明确地通过引用整体并入。
实施例
实施例1
本文描述了非病毒基因组靶向作为大型治疗性内源遗传修饰的发现平台。在原代人T细胞的91个独特基因组位点进行了大型DNA有效载荷的阵列敲入筛选,并确定了用于预测可有效靶向的基因组基因座的规则集。这些高效工具可有效产生基因工程化内源蛋白(GEEP),其通过将合成修饰与内源遗传元件无缝结合来改变细胞输入、输出和调节控制。最后,基于同源定向修复的独特特征,开发了一种用于大型合并敲入的通用技术。T细胞受体基因座的靶向内源敲入的高通量合并筛选揭示了新型功能蛋白嵌合体,其结合了新TCR特异性以在存在肿瘤抑制信号的情况下增强T细胞功能,包括在体内实体肿瘤模型中。总之,本文提供了一个强大的发现平台,用于通过非病毒基因组靶向实现的下一代细胞疗法。
FDA于2017年批准了两种基于T细胞的B细胞白血病和淋巴瘤疗法,为工程化T细胞疗法的30年发展画上了句点。虽然这项工程的基础技术自最早的工程化T细胞临床试验以来已经取得了进步,但两个核心方面仍保持不变,即病毒感染的需要,以及病毒基因组随机整合到细胞DNA中。最近开发了一种有效的非病毒基因组靶向方法,所述方法在递送大型新DNA序列时不需要病毒载体(Roth等人,Nature 559:405-409(2018))。此外,随着可靶向核酸酶如CRISPR/Cas9的应用,这些DNA序列可通过同源定向修复以单碱基对分辨率靶向整合到特定基因组位点。通过用CAR或新TCR特异性替代内源T细胞受体,将新抗原受体置于内源调控控制之下,已经显示出将治疗基因靶向特定基因组位点的优势。
将大型新DNA序列靶向特定位点的能力为内源基因组基因座的工程化带来了各种特定的问题。与随机病毒载体整合不同,每个靶基因座都是独特的,需要新的gRNA组合来引发dsDNA断裂,以及在同源定向修复期间将新DNA序列靶向该位点的同源臂。实际上,靶向不同基因组基因座的基因产生截然不同的效率。为了确定适合非病毒基因组靶向的内源基因组基因座谱,进行大型阵列敲入筛选,将GFP或tNGFR模板(约800bp)整合到六个健康人类供体中的91个独特基因组基因座中(图1a、图1b)。靶向多种基因,包括TCR复合物成员、免疫表面受体、检查点受体、转录因子以及许多细胞骨架和管家基因,显示出广泛多种敲入效率观测值(图1c和图5)。筛选在CD4和CD8 T细胞中进行,其中记录静息和再刺激细胞中的GFP或tNGFR敲入百分比(图6)。靶基因的RNA表达和gRNA切割位点的DNA可及性均与敲入效率观测值相关(图1d)。多元线性回归显示出比单独的任何gRNA、RNA表达或DNA可及性参数更高的预测值(图1d和图7、图8),并证明gRNA切割效率、靶基因RNA表达和靶位点DNA可及性独立地有助于敲入效率观测值(图1e)。
确定可被有效修饰的基因组基因座的能力,以及向特定位点添加大型新DNA序列的能力,开启了如下问题:特异性地添加到内源基因座的新合成遗传指令如何独特地改变细胞功能(图2a)。随机整合的新遗传材料赋予正交功能性,但靶向敲入可将合成元件与内源序列整合以产生基因工程化内源蛋白(GEEP)。使用基于Cas9‘穿梭’系统和阴离子聚合物的改进型非病毒基因组靶向方案。在靶基因组基因座内的多个位置有效地产生GEEP(图2a)。
将新的病毒启动子整合到内源基因的转录起始位点会产生具有驱动内源基因产物表达的合成启动子的‘启动子GEEP’(图2b)。在TCR刺激后9天,IL2RA和PDCD1处的启动子GEEP在静息细胞中显示IL2RA和PD1的持续高表达,而这些激活依赖性基因的内源调控回路显示出低表达水平(图2b和图9)。相比之下,新基因产物在同一位点的整合产生了具有驱动新合成基因产物表达的内源调控回路的‘产物GEEP’(图2c)。在PDCD1基因座处产生产物GEEP,其包含2A肽以维持内源PD1基因的表达或包含polyA序列以去除内源PD1基因表达(图2c和图10)。在IL2RA、CD28和LAG3基因座产生的产物GEEP都反映了其相应内源基因的表达动力学(图2d和图11)。在靶表面受体跨膜结构域之前特异性整合一个新的胞外结构域,产生了具有合成特异性驱动内源信号传导的‘特异性GEEP’(图2e),例如在内源TCRα基因座上,其中能够置换胞外TCR特异性,同时保持内源恒定信号传导结构域。最后,在跨膜结构域之后向表面受体整合一个新的信号传导结构域,产生了‘信号传导GEEP’,其中内源特异性驱动合成信号传导(图2f)。在所有四个CD3基因座(CD3D、CD3E、CD3G、CD3Z)上产生了信号传导GEEP,其中附加了CD28胞内结构域或41BB胞内结构域(图12)。虽然与对照敲入细胞相比,CD28胞内结构域融合在存在CD3刺激的情况下都没有显示出增殖增加(图12),但在没有CD28共刺激的情况下,CD3ζ-41BB信号传导GEEP特异性地显示出响应于CD3刺激的增殖增加(图2g)。
已经制定了确定可靶向基因组基因座的指南和基因工程化内源蛋白的设计以将合成元件与内源序列相结合,确定将多个大DNA序列组合到单个治疗性内源敲入基因盒中是否可增强免疫治疗环境中的T细胞功能性。T细胞的功效是其抗原特异性和功能性的产物。首先,证明了三个基因盒可整合在内源TCR-α基因座上,以新特异性替换内源TCR以及驱动高表达内源TCR启动子表达新基因(图3a)。将TCRβ-tNGFR-TCRα盒敲入TRAC外显子1显示,几乎所有成功敲入新TCR(NY-ESO-1黑色素瘤癌抗原特异性1G4克隆)的细胞也显示了额外tNGFR基因的表达(图3b)。将四个基因盒敲入TCR-α基因座也同样成功(图13)。
为了确定这种新基因产物是否可改变T细胞功能,tNGFR被替换为先前描述的显性阴性TGFβR2受体,该受体可最大限度地减少TGFβ信号传导对T细胞的抑制作用(Ishigame等人,J.Immunol.190(12):6340-6350(2013))。头对头增殖测定显示,与添加新TCR和tNGFR对照相比,在外源TGFβ存在下选择性地递送具有dnTGFβR2的新NY-ESO-1 TCR特异性后相对增殖增加(图3c和图14)。与新NY-ESO-1 TCR+tNGFR相比,在1G4 TCR的特定MHC-A2等位基因上表达NY-ESO-1抗原的靶黑色素瘤细胞系(A375细胞)的抗原特异性杀伤类似地显示在新NY-ESO-1 TCR+dnTGFβR2递送下的改进的杀伤(图3d和图14)。因此,内源TCR-α基因座的非病毒敲入可通过单个基因盒有效地改变T细胞特异性以及T细胞功能性。
小分子和生物药物的开发显著地取决于高通量筛选方法的应用,以便能够同时测定许多潜在的治疗候选物。然而,类似的筛选方法,即大DNA序列的合并的敲入,尚未应用于加速基于细胞的疗法的开发。为了克服这一限制,如本文所述,开发了一种通用的非病毒合并敲入筛选方法,以快速测定合并细胞群体中的许多靶向敲入(图4a)。基于新TCR特异性和新功能改变基因的单一敲入(图3),假设HDR模板文库的合并的敲入,其中每个成员都包含恒定的新TCR特异性以及独特的第三基因产物,可快速鉴定在特定治疗相关环境中修改T细胞功能的新DNA序列(图4a)。
首先,开发了一种DNA测序策略,其与NHEJ编辑或野生型靶基因组基因座、游离型非整合HDR模板或脱靶整合相比,选择性地扩增中靶敲入(图15)。由于HDR模板的同源臂用于与靶基因座进行互补碱基配对,但自身并未复制到靶位点,因此与引入3′同源臂的靶基因组基因座错配的一小段DNA碱基对区域产生了中靶敲入所特有的PCR扩增子(图15a),而敲入效率没有大幅降低(图15b、图15c)。在TCR+功能基因盒的TCR-α VJ区域的简并碱基内为每个插入物添加独特条形码,从而能够对合并群体中每个单独插入物的丰度进行DNA读出。使用双成员文库(新NY-ESO-1TCR+GFP或RFP)进行详细实验,包括在DNA组装之前测试文库合并、通过PCR产生HDRT、HDRT电穿孔或细胞培养/扩增,然后对分选的GFP+或RFP+细胞进行测序,揭示了当电穿孔前合并时最小的模板转换量,其在早期合并时会增加(图16)。再现观测到的GFP+和RFP+比例的条形码测序可通过分离的基因组DNA和转化为cDNA的mRNA来实现(图16)。因此,展示了一种简单的合并的敲入方法,其可将许多新的大DNA序列靶向特定的靶基因组基因座,并通过DNA测序容易地确定它们在细胞群体中的相对丰度。
使用两个示例构建体(图34中所示的多顺反子盒中的mCherry与GFP)评价模板转换。质粒池(n=2)通过合并的组装构建。HDR模板是从质粒池中生成的,并被电穿孔到两个健康供体的原代T细胞中。根据NY-ESO-1 TCR和GFP或mCherry表达对细胞进行分选。通过cDNA的扩增子测序分析正确条形码读段的数目。将正确分配读段的百分比与用mCherry/GFP模板单独电穿孔并在培养期间合并的T细胞和仅用一种构建体电穿孔的T细胞进行比较(图35a和图35b)。计算了2成员文库的模板转换(图35c)并对N成员文库进行预测(图2d)。使用图34的条形码策略,N成员文库的预测模板转换从之前设计中的50%降低到改进型合并敲入文库设计中的平均7.6%。改进型合并敲入文库设计的模板转换观测值和预测值:(图35a)包含GFP或(图35b)mCherry HDR模板的条形码的测序读段的百分比与在不同合并条件下通过流式细胞术观测到的表达GFP或mCherry蛋白的细胞百分比相对应。图35c显示了2成员文库的模板转换观测量,并且图35d显示了N成员文库的模板转换预测值。在合并的组装阶段,文库设计的模板转换预测值为7.6%。所有实验均在n=2个独特健康供体中进行。使用图34中所示的示例性构建体减少了在文库组装的早期步骤的合并期间可能发生的模板转换量。由于合并可在早期方案步骤(例如在文库组装期间)变得可行,因此可将方法从数十个扩展到数百个测试构建体。
接下来将合并的敲入筛选应用于发现原代人T细胞中内源遗传基因座的潜在治疗相关修饰。设计了先前公开的以及新型的蛋白质嵌合体的36成员文库,它们可重新连接抑制性或阻遏性信号以向T细胞提供激活或刺激性信号,同时引入新TCR特异性(图17)。使用这个更大的文库进行合并的敲入筛选的技术验证显示每个文库成员的有效敲入,并且对独特条形码进行测序仍然准确地反映了它们在细胞群体中的比例(图18a-e)。对于初始应用,合并的修饰T细胞文库经受刺激,并将群体丰度与输入进行比较。值得注意的是,与大多数文库成员相比,基于具有多种免疫调节胞内结构域的FAS凋亡基因的嵌合受体显示出增殖的相对急剧增加(图4b和图18f)。这些大型合并的敲入筛选结果具有高度可重现性,可在早期合并阶段和批量编辑或分选的细胞中进行,并且不会阻止电穿孔后的稳健细胞扩增(图18g-k)。
利用合并的敲入筛选在给定测定法中快速确定许多基因产物的功能效应的能力,将一系列不同的体外选择压力应用于用36成员TCR+功能修饰基因文库修饰的原代人T细胞(图4c和图18a)。在没有再刺激的情况下,表达IL2RA的T细胞相对扩增(图18k),而在刺激下,Fas衍生的嵌合体显示出更大的相对扩增(图14f)。相比之下,添加免疫抑制细胞因子TGFβ赋予dnTGFβR2构建体选择性增殖优势,并且新型嵌合TGFβR2-41BB显示出更大的增殖(图19b)。反映肿瘤环境中抗原丰度的过度刺激揭示了转录因子TCF7的选择性增殖优势(图19c)。并且在没有CD28共刺激的情况下通过TCR的刺激显示出多种CD28嵌合受体如TIM3-CD28嵌合体和CTLA4-CD28嵌合体的增殖优势(图19d)。
接下来,使用抗原特异性人类黑色素瘤异种移植模型进行了体内合并敲入筛选(Roth等人,Nature 559:405-409(2018))。将合并的修饰T细胞文库转移到带有表达NY-ESO-1抗原的人类黑色素瘤的免疫缺陷NSG小鼠中,并在五天后从肿瘤中提取T细胞(图4d)。来自体外筛选的各种命中也显示在体内肿瘤环境中的选择性增殖富集,包括TCF7和TGFβR2-41BB嵌合体(图4d和图20)。来自体内筛选的一些命中,例如代谢蛋白MCT4,在进行的任何体外筛选中都没有显示出富集。
合并的敲入筛选迅速揭示了许多新的DNA序列,这些序列在整合到内源TCR-α基因座时可增强T细胞功能,以及在单一盒内的新TCR特异性(图4e)。对这些序列中的三个进行了单独验证,即抗抑制性TGFβR2-41BB嵌合体、抗凋亡FAS-41BB嵌合体和改变TCF7的转录程序。TGFβR2-41BB嵌合体在有和没有外源TGFβ的情况下改善了抗原特异性癌细胞杀伤(图4f和图21a-c)。Fas-41BB嵌合体在TCR刺激后显示出急剧增加的增殖,以及更强的抗原特异性靶细胞杀伤(图4g和图21d-f)。事实上,最近的一项独立研究发现了与显性负性FAS蛋白类似的效应。最后,新TCR特异性与转录因子TCF7的敲入概括了在合并的筛选中可见的过量刺激导致的增殖轻度增加以及类似地增加抗原特异性靶细胞杀伤(图4g和图21g-i)。
如上所述,为了验证我们的合并的敲入筛选的命中,我们首先对原始增殖表型进行了单独验证,以及对TCF7、TGFβR2-41BB和强烈体外命中FAS-41BB进行了体外癌症杀伤测定(使用A375黑色素瘤细胞)(图25a)。抗凋亡FAS-41BB嵌合体(图26)和改变TCF7的转录程序(图27)各自改善了环境依赖性扩增以及NY-ESO-1+癌细胞的体外杀伤(图25b)。抗抑制性TGFβR2-41BB嵌合体类似地显示出对NY-ESO-1+癌细胞的体外杀伤改善,尤其是在存在外源TGFβ的情况下(图25c)。
我们进一步检查了TCF7或TGFβR2-41BB在实体肿瘤异种移植模型中的体内功能能力(图25d)。用表达NY-ESO-1特异性TCR(1G4克隆)的多顺反子盒与对照构建体(tNGFR)、转录因子TCF7或嵌合TGFβR2-41BB受体工程化的T细胞均显示出相对于仅媒介物的统计学显著的肿瘤大小减小(图25e)。虽然TCF7和TGFβR2-41BB在体内筛选中均显示出丰度增加,但通过单细胞RNA测序测量的它们的转录特征显示出显著差异,其中TGFβR2-41BB显示出效应细胞因子如IFN-γ比TCF7更高的表达。与这些数据一致,相对于tNGFR对照,TCF7没有显示出增强的肿瘤控制(图25e和图27),而TGFβR2-41BB受体显示出肿瘤大小的急剧减小并导致在四个人类T细胞供体中测试的许多小鼠的肿瘤清除(图25e和图28)。因此,TGFβR2-41BB嵌合体在体内实体肿瘤模型中提高了抗肿瘤功效。
总体而言,本文所述的非病毒基因组靶向平台是用于修改T细胞特异性和功能的适应性发现平台。通过能够实现有效的基因靶向的内源基因座的大型阵列敲入筛选特征,确定了从随机整合病毒基因传递过渡到靶向非病毒方法时的关键指标。开发了用于在内源基因座整合合成DNA元件以产生基因工程化内源蛋白(GEEP)的框架。此外,将多个基因产物整合到特定内源位点,即TCRα基因座,允许使用单个基因盒同时操纵T细胞特异性以及功能性。
CRISPR技术极大地提高了在治疗相关细胞类型中操纵人类基因组的能力。但是高通量筛选方法用于探索有效的无限数量的可能与治疗相关的潜在操纵。开发了一种合并的敲入筛选方法,所述方法允许在限定的基因组靶位点对大DNA序列池进行普遍敲入。体外和体内合并敲入筛选的应用揭示了新的基因嵌合体,当与新TCR特异性一起引入时,这些基因嵌合体在具有挑战性的肿瘤环境中增强了T细胞功能。细胞疗法承诺细胞本身可以与小分子和生物制剂一起成为治疗药物的新支柱。合并的敲入筛选将实现基于高通量筛选的相同药物发现过程,该过程产生的绝大多数小分子和生物治疗剂可应用于基于细胞的疗法。使用非病毒基因组靶向的合并的敲入筛选是为下一代细胞疗法修改T细胞特异性和功能的理想平台。
方法
用于基因靶向的人原代T细胞的分离
从健康供体的新鲜全血或Trima单采(太平洋血液中心)后去白细胞室的残留物中分离出原代人T细胞。外周血单核细胞(PBMC)通过Ficoll离心使用SepMate管(STEMCELL(Vancouver,CA),根据制造商的说明)从全血样品中分离。使用EasySep人类T细胞分离试剂盒(STEMCELL,根据制造商的说明)通过磁性负选择从所有细胞来源的PBMC中分离T细胞。分离的T细胞在分离后立即用于电穿孔实验,或根据制造商的说明在Bambanker冷冻培养基(Bulldog Bio)中冷冻以备后用。如下所述刺激新鲜分离的T细胞。先前冷冻的T细胞被解冻,在没有刺激的情况下在培养基中培养1天,然后如对于新鲜分离样品所述进行刺激和处理。根据UCSF人类研究委员会(CHR#13-11950)批准的方案,从健康人类供体获取新鲜血液。
原代人T细胞培养
使用补充有5%胎牛血清、50μM 2-巯基乙醇和10μM N-乙酰基L-胱氨酸的XVivo15培养基(STEMCELL)来培养原代人T细胞。在准备电穿孔时,使用抗人CD3/CD28磁性Dynabeads(ThermoFisher)以1∶1的珠粒/细胞比率以每毫升培养基约100万个细胞的起始密度刺激T细胞2天,并在含有IL-2(500U ml
RNP产生
RNP是通过将双组分gRNA与Cas9复合而产生的。双组分gRNA由crRNA和tracrRNA组成,两者都是化学合成的(Dharmacon(Lafayette,CO0,IDT(Coralville,IA))和冻干的。收到后,将冻干的RNA以160μM的浓度重悬于含150mM KCl的10mM Tris-HCL(7.4pH)中,并在-80℃下以等分试样储存。Cas9-NLS(QB3 Macrolab)重组产生、纯化,并以40μM储存在20mMHEPES-KOH pH 7.5、150mM KCl、10%甘油、1mM DTT中。为了产生RNP,将crRNA和tracrRNA等分试样解冻,按体积1∶1混合,并通过在37℃下孵育30分钟进行退火以形成80μM gRNA溶液。接下来,gRNA溶液与Cas9-NLS按体积1∶1混合(gRNA与Cas9的摩尔比为2∶1),并在37℃下孵育15分钟以形成20μM RNP溶液。RNP在复合后立即进行电穿孔。
双链HDR DNA模板产生
每个双链同源定向修复DNA模板(HDRT)都包含一个新型/合成的DNA插入物,侧接同源臂。我们使用Gibson Assemblies来构建包含HDRT的质粒,然后将这些质粒用作高输出PCR扩增的模板(Kapa热启动聚合酶(Kapa Biosystems,Basel,Switzerland)。所得PCR扩增子/HDRT经SPRI纯化(1.0x)并洗脱到H2O中。通过NanoDrop使用1∶20稀释来确定洗脱的HDRT的浓度,然后相对于1μg/μL归一化。扩增的HDRT的大小通过在1.0%琼脂糖凝胶中的凝胶电泳确认。
原代T细胞电穿孔
对于所有电穿孔实验,如上所述制备和培养原代T细胞。刺激48-56小时后,从其培养容器中收集T细胞,并将抗CD3/抗CD28 Dynabeads与T细胞磁性分离。电穿孔前立即将去珠细胞以90g离心10min,吸移并重悬于Lonza电穿孔缓冲液P3中。每个实验条件接受750,000-100万范围内的重悬于20uL P3缓冲液中的活化T细胞,并且所有电穿孔实验均以96孔格式进行。
对于阵列敲入筛选(图1),首先将HDRT等分到96孔聚丙烯V型底板的孔中。如所述,在RNP组装期间,在gRNA和Cas9复合步骤之间添加了聚谷氨酸。然后将复合的RNP添加到HDRT中,并在室温下使其一起孵育至少30s。对于GEEP敲入和合并的敲入筛选(图2-4),截短的Cas9靶序列(tCTS)被额外添加到HDR模板的5′端和3′端,从而实现Cas9‘穿梭’。对于所有变化,将重悬于电穿孔缓冲液中的T细胞加入到RNP和HDRT混合物中,简单混合,然后转移到96孔电穿孔比色板中。
所有电穿孔均使用带有脉冲代码EH115的Lonza 4D 96孔电穿孔系统进行。除非另有说明,否则将3.5μl RNP(包含50pmol的总RNP)以及浓度为1μg μl-1的1-3μl HDR模板(总共1-3μg HDR模板)电穿孔。在所有电穿孔后立即将80μl预热的培养基(不合细胞因子)加入每个孔中,并且使细胞在细胞培养箱中在37℃下静置15min,同时留在电穿孔比色皿中。15min后,将细胞移至最终培养容器中。
阵列敲入筛选
对于6个独特健康供体中的每一者,将原代人T细胞的5 X 96孔板电穿孔。在三个板中,将靶向91个独特基因组基因座中的每一者的HDR模板与两个中靶gRNA之一或一个乱序gRNA一起电穿孔。最后两个板仅使用中靶gRNA(与Cas9复合形成RNP)进行电穿孔,但没有用于扩增子测序的HDR模板。在电穿孔后四天,以及另外在电穿孔后五天以1∶1 CD3/CD28dynabeads∶细胞比率再刺激24小时后,通过流式细胞术对于使用HDR模板的中靶和乱序RNP板分析技术重复中观测到的敲入效率。电穿孔后四天,仅在电穿孔四天后从仅中靶gRNA板上分离基因组DNA。
初始分离后(第0天)、电穿孔前不久(第2天)和电穿孔后扩增期间(第4天),来自每个供体的约1e6个CD4和CD8T细胞通过FACS进行分选,用于RNA-Seq和ATAC-Seq分析(图1b)。分选细胞的一半冷冻在Bambanker冷冻培养基(Bulldog Bio)中用于ATAC测序,并且一半冷冻在RNAlater(QIAGEN)中用于批量RNA测序。
为了构建敲入率的预测模型,我们采用了多元线性回归方法。简而言之,该模型将观测到的测量参数与观测到的敲入率拟合,并描述为:
Y
其中对于gRNA位点,Yi是观测到的敲入率,β0是常见截距并且∈i是估计误差。β1至βk是回归权重(或系数),其衡量测量参数(Xki)和敲入率之间的关联估计。为了构建模型,我们对每个gRNA和细胞类型下各供体的测量值取平均值。对于基因表达和染色质可及性,将数值进行了对数转换。用于生成模型的参数如图1f中所描述。所得模型用于预测对于所有位点、个别供体和细胞类型的敲入率观测值。将所有回归权重的绝对值相加,并确定每个参数的回归权重占总数的百分比。
扩增子测序
基因组DNA是从原代人T细胞中分离出来的,其在不存在其同源HDR模板的情况下,用阵列敲入筛选中使用的每个gRNA单独编辑。吸移出上清液后,每种条件下约100,000个细胞重悬于20μl Quickextract DNA提取溶液(Epicenter)中,至浓度为每μl 5,000个细胞。根据制造商的方案,将Quickextract中的基因组DNA加热至65℃持续6min,然后加热至98℃持续2min。使用NEB Q5 2X Master Mix热启动聚合酶和制造商推荐的热循环仪条件,将1μl混合物(包含来自5,000个细胞的基因组DNA)用作两步PCR扩增子测序方法中的模板。在使用引物扩增以预测的gRNA切割位点为中心的大约200bp区域的初始18个循环PCR反应后,进行1.0X SPRI纯化,并将每个生物供体的一半样品合并用于索引(每个供体有两个gRNA,在每个插入位点切割,每个位点一个gRNA的样品被合并,每个供体产生两个独特池)。执行10个循环的PCR以附加P5和P7 Illumina测序衔接头和供体特异性条形码,然后再次进行1.0XSPRI纯化。对于供体/gRNA索引将浓度归一化,合并样品,并在Illumina Mini-Seq上以2X150bp读段运行模式对文库进行测序。
在基因组模式下使用CRISPRessoPooled命令和默认参数,利用CRISPResso处理扩增子。我们使用了hg19人类参考基因组组装。得到的扩增子区域与每个样品的gRNA位点相匹配。消除了具有通过CRISPResso比对检测为没有插入缺失的单一突变碱基的潜在测序误差的读段。剩余的读段用于计算NHEJ百分比,或“切割百分比观测值”。
批量RNA测序
根据制造商的方案,使用RNeasy Mini试剂盒(Qiagen)从冷冻样品中提取总RNA。使用Qubit和Nanodrop 2000进行RNA定量,并通过Bioanalyzer RNA 6000Nano试剂盒(Agilent Technologies)对10个随机样品确定RNA的质量。我们确认样品的平均RNA完整性数目(RIN)>9,并且迹线揭示完整、未降解的总RNA的特征性大小分布。根据制造商的方案,使用Illumina TruSeq RNA样品制备试剂盒v2(目录号RS-122-2001)构建RNA文库。来自每个样品的总RNA(500ng)用于建立cDNA文库。在高灵敏度DNA试剂盒(Agilent)上对36个最终文库中的10个的随机集合进行了质量检查,揭示平均片段大小为400bp。构建总共36个富集文库(3个池的12个独特索引文库)并使用Illumina HiSeq
使用智人(Homo sapiens)ENSEMBL GRCh37(hg19)cDNA参考基因组注释,利用kallisto处理RNA-Seq读段。转录物计数在基因水平上合计。从归一化基因水平计数表中对目标基因进行子集化,并作为每百万读段的转录本数(TPM)进行分析。
ATAC-测序
ATAC-seq文库是按照Omni-ATAC方案[REF-方法1]制备的。简而言之,使用Ghost-Dye 710(Tonbo Biosciences,CA,USA)将冷冻细胞解冻并对活细胞染色。50,000个活细胞经FACS分选并用冷PBS洗涤一次。对大多数样品进行了技术重复。将细胞沉淀重悬在含有0.1%NP40(Life Technologies,Carlsbad,CA)、0.1%Tween-20(Sigma Aldrich)和0.01%洋地黄皂苷(Promega,WI,USA)的50μl冷ATAC-重悬缓冲液(10mM Tris-HCl(SigmalAldrich,MO,USA)pH 7.4、10mM NaCl、3mM MgCl2(Sigma Aldrich)持续3min。将样品在含有0.1%Tween 20的冷重悬缓冲液中洗涤一次,并4C离心10min。将提取的细胞核重悬在50μlTn5反应缓冲液(1 x TD缓冲液(Illumina,CA,USA)、100nM Tn5转座酶(Ilumina)、0.01%洋地黄皂苷、0.1%Tween-20、PBS和H20)中,并在37C下以300rpm孵育30min。根据制造商的方案,使用MinElute PCR纯化柱(Qiagen,Germany)纯化转座样品。使用定制的Nextera条形码编码PCR引物对纯化的样品进行扩增和索引,如[REF-方法2]中所述。使用MinElute柱纯化DNA文库并以等摩尔浓度合并。为了去除引物二聚体,使用AmPure珠粒(Beckman Coulter,CA,USA)将合并的文库进一步净化。ATAC文库在NovaSeq上以配对端X循环模式进行测序。
使用cutadapt v1.18修剪ATAC-seq读段以去除Nextera转座酶序列,然后使用Bowtie2 v2.3.4.3与hg19对齐。使用samtools v1.9view功能(samtools view-F 1804-f2-q 30-h-b)删除了低质量读段。使用picard v2.18.26删除重复项,然后使用bedtoolsbamtobed功能将读段转换为BED格式,并相对于每百万读段的读段数进行归一化。映射在CRISPR切割位点周围的1kb窗口内的ATAC-seq读段使用bedtoolsintersect功能进行计数。
流式细胞术和细胞分选
所有流式细胞术分析均在Attune NxT声聚焦细胞仪(ThermoFisher)上进行。FACS在FACSAria平台(BD)上进行。用于流式细胞术和细胞分选的细胞表面染色通过在25uLFACS缓冲液(PBS中的2%FBS)中沉淀和重悬来进行,其中抗体在室温下在黑暗中相应稀释20min。在重悬和分析之前,细胞在FACS缓冲液中洗涤一次。
合成产物+内源产物动力学流式细胞术分析
将非病毒编辑的T细胞分成多个重复,并从电穿孔后的第3天开始在5天时期内每天通过流式细胞术进行分析。在这5天时期期间,T细胞每2天用额外的培养基和IL-2装满,至最终浓度为500U/mL,有或没有1∶1分裂。在电穿孔后第5天,一组细胞用CD3/CD28Dynabeads刺激,而另一组细胞保持不受刺激。
体外增殖测定
在测定之前,非病毒编辑的T细胞在独立的培养物中扩增。大约两周的扩增(每2-3天补充500U/mL IL-2)后,将未分选的、经编辑的群体合并,用于竞争性混合增殖测定。
对于CD3竞争性混合增殖测定,我们将未分选的具有CD28IC-2A-GFP、41BBIC-2A-mCherry或2A-BFP敲入的样品合并到相同的CD3复合物成员的基因座。为了确定用于合并的输入数目,我们考虑了各个群体中活GFP+、mCherry+或BFP+的数目(敲入%*总活细胞计数),如通过流式细胞术分析所确定的。然后以50,000的起始总细胞计数将合并的样品分布到圆底96孔板中。然后在无刺激、仅CD3刺激、仅CD28刺激或CD3/CD28刺激下培养分布的样品。CD3和/或CD28刺激是用板结合抗体进行的。所有样品均在补充有IL-2(50U/mL)的XVivo15培养基中培养。培养4天后,通过流式细胞术分析样品中GFP+和mCherry+亚群相对于BFP+亚群的相对生长。
对于NY-ESO-1竞争性混合增殖测定,我们将未分选的样品与1G4+dnTGFβR2+或1G4+tNGFR+T细胞合并。为了确定每个群体的输入数目,考虑了如通过流式细胞术分析确定的任一群体中的活1G4+TCR+数目(敲入%*总活细胞计数)。然后将合并的样品以总起始细胞计数为50,000分布到圆底96孔板中。然后在没有刺激的情况下或用Immunocult(CD3/CD28/CD2)培养分布的样品。所有样品均在添加或不添加TGFβ1的情况下用补充有IL-2(500U/mL)的XVivo15培养基培养。培养5天后,利用流式细胞术分析样品,并量化1G4+dnTGFβR2+和1G4+tNGFR+亚群的相对生长。
体外抗原特异性杀伤测定
在开始共培养前大约24h接种经稳定转导以表达核RFP(Zaretsky 2016NEJM)的A375-nRFP(NY-ESO-1+HLA-A*0201+)黑色素瘤细胞系(每孔接种约1,500个细胞)。以指定的E∶T比率添加修饰的T细胞。杀伤测定在具有IL-2和葡萄糖的cRPMI中进行。样品另外装满TGFβ1或等体积的对照培养基。使用IncuCyte ZOOM(Essen,Ann Arbor,MI)通过nRFP实时成像测量癌细胞清除率持续4-5天,并通过以下等式确定:(仅A375孔中的汇合%-共培养孔中的汇合%)/(仅A375孔中的汇合%)。在测定结束时,回收细胞,并通过流式细胞术分析表达各种耗竭标志物的T细胞的百分比。
合并的敲入筛选
使用如所述的非病毒基因组靶向方法进行靶向合并敲入筛选,不同之处在于在TRAC外显子1靶向HDR模板的3′同源臂中引入约10bp的DNA错配用于替换内源TCR。敲入文库每个成员的独特条形码也被引入到HDR模板的TCRαVJ区域的3′端的约6个简并碱基中(图4a)。合并的敲入文库中包括的36个构建体是使用Benchling DNA序列编辑器设计的,作为dsDNA基因块(IDT)商业合成,并使用Gibson Assemblies单独克隆到包含NY-ESO-1 TCR替换HDR序列的pUC19质粒中(除了合并的组装条件,而文库中的所有基因块在组装之前合并)。构建体中包括的36种多肽的设计示于表2中。每个构建体的胞外结构域、跨膜结构域和胞内结构域的大小(蛋白质大小)分别在表2的第6、7和8列(在蛋白质大小[aa]下)中描述。在图例中描述的不同阶段合并文库(图16),但除非另有说明,否则在电穿孔之前合并HDR模板。
表2
由合并的敲入生成的修饰T细胞文库如所述进行电穿孔、培养和扩增,然后进行各种体外测定,在电穿孔后第7天开始并且在电穿孔后第12天结束。在刺激测定中,修饰T细胞文库用CD3/CD28 dynabeads刺激,珠粒/细胞比率为1∶1,并且对于过度刺激条件,珠粒/细胞比率为5∶1。在TGFβ测定中,将25ng/mL的人TGFβ添加到培养基。对于仅CD3刺激条件,1G4TCR(NY-ESO-1特异性)结合dextramer(Immudex)与50uL的1∶50稀释的细胞(总共500,000个细胞)在室温下结合12分钟,然后返回到培养基。除非另有说明,否则所有体外测定均以500,000个分选的NY-ESO-1+T细胞开始。
在体外或体内测定结束时,将T细胞沉淀并提取基因组DNA(QuickExtract)或在Trizol中稳定mRNA。根据制造商的说明使用Zymo Direct-zol离心柱纯化mRNA,并根据制造商的说明使用Maxima H RT First Strand cDNA Synthesis(Thermo)将mRNA转化为cDNA。除非另有说明,否则文库由分离的mRNA/cDNA制成。对分离的基因组DNA或cDNA进行两步PCR。第一次PCR(PCR1)包括插入物的TCRαVJ区域中的正向引物结合和与3′同源臂中错配位点重叠的基因组区域中的反向引物结合(图15),并使用Kapa Hifi Hotstart聚合酶进行12个循环,然后进行1.0X SPRI纯化。第二次PCR使用NEB Next Ultra II Q5聚合酶进行10个循环,以附加P5和P7 Illumina测序衔接头和样品特异性条形码,然后再次进行1.0X SPRI纯化。将归一化文库跨样品合并,并在Illumina Mini-Seq上以2X150bp读段运行模式进行测序。使用PDict在R中确定来自质量过滤读段的条形码计数。
体内异种移植模型
NY-ESO-1黑色素瘤肿瘤异种移植模型如前所述使用(Roth等人,Nature 559:405-409(2018))。所有小鼠实验均在UCSF机构动物护理和使用委员会协议下完成。我们使用8至12周龄的NOD/SCID/IL-2Rγ-null(NSG)雄性小鼠(Jackson Laboratory)进行所有实验。通过将肿瘤以1x106个A375人黑色素瘤细胞(ATCC CRL-1619)皮下注射到经剃毛的右侧腹,向小鼠接种肿瘤。在肿瘤接种后七天,评估肿瘤大小,并将肿瘤体积在20-40mm3之间的小鼠用于后续实验。使用电子卡尺测量肿瘤的长度和宽度,并且体积计算为v=1/6*π*长度*宽度*(长度+宽度)/2。将指定数量的具有合并的敲入文库的T细胞重悬于100μl无血清RPMI中并经眼眶后注射。批量编辑的T细胞群体(10x106)在转移时包含至少10%NY-ESO-1敲入阳性细胞。T细胞转移后五天,通过70μm过滤器机械分离组织产生来自肿瘤和脾脏的单细胞悬液,并通过FACS从大量肿瘤细胞中分选T细胞(CD45+TCR+)。所有动物实验均按照经批准的IACUC协议(UCSF)的相关伦理法规进行,包括任何维度的肿瘤大小限度为2.0cm。
实施例2
我们敲入编码多顺反子基因程序的大DNA序列的条形码编码池,并将合并的敲入筛选与单细胞RNA测序相结合,以快速确定每个构建体的高维表型信息。
传统的合并的筛选方法的一个主要限制是仅测量群体内给定文库成员的丰度,从而限制了对细胞状态和功能性的更详细分析。合并的扰动与高维表型读出的结合提供了一种快速方法来增加获得的关于每一个体扰动的信息。单细胞RNA测序生成了这样的表型,我们最近将其与原代T细胞中的合并的敲除筛选相结合(Utzschneider,D.T.等人,T CellFactor 1-Expressing Memory-like CD8+T Cells Sustain the Immune Response toChronic Viral Infections.Immunity(2016))。我们接下来测试了合并的敲入筛选是否可类似地与单细胞RNA测序相结合,以显著扩大单个合并实验中生成的表型信息的数量。
我们在A375体内实体肿瘤模型中进行了合并的敲入筛选(图4d),将输入群体与T细胞转移后五天的体内群体进行比较(图23a)。与丰度筛选相反,在从肿瘤或输入群体中分选活TCR+T细胞后,我们进行了单细胞液滴分离(10X),然后对单细胞转录组进行测序,并与靶向扩增子测序相结合,以确定与每个细胞相关的敲入构建体(图3a和图24a)。来自两个独特供体的单细胞转录组的UMAP表示显示供体之间的高度重叠以及输入细胞群体与体内5天后从实体肿瘤中分离的T细胞之间的巨大差异(图23b)。输入和体内簇与已知的增殖和效应基因特征重叠(图23c)。对敲入构建体条形码的靶向扩增子测序的计算过滤能够将单细胞转录组与单个敲入相关联(图24b)。来自单细胞体内合并敲入筛选的具有个别敲入条形码的细胞比例与批量细胞体内合并敲入筛选相关(R
然而,群体中丰度的增加可能并不总是对应于功能功效。在一致的单细胞RNA seq中的合并的敲入筛选揭示了文库成员的丰度以及它们的个体转录特征。我们将来自合并的敲入筛选的两个命中的体内单细胞转录组与对照进行了比较:转录因子TCF7,在过度刺激后在体外富集(图19)和在体内富集;和转换受体TGFβR2-41BB,它是体内命中以及TGFβ抑制后最强的体外命中(图19)。虽然TCF7和TGFβR2-41BB均显示出基因如TNFSFR9(41BB)的表达相对于对照增加,但TGFβR2-41BB构建体显示出效应细胞因子如IFN-γ的表达增加,这可能会在测试的黑色素瘤异种移植模型中驱动肿瘤清除。
合并的敲入筛选加单细胞RNA测序
根据制造商的方案,使用Chromium Single Cell 3′Reagent Kit,v3 chemistry(10x Genomics,PN-1000092)对8个单独的样品(2个供体,每个供体2个接受者,匹配的植入前和植入后细胞)进行单细胞RNA测序。简而言之,TCR阳性细胞通过FACS(BD FACS Aria)分选,并以1000个细胞/ul重悬于PBS+1%FBS中,以达到每个条件6000个细胞的目标回收率。我们在GEM回收后进行了11个PCR循环以进行cDNA扩增,并且每个cDNA样品的25%继续用于转录组文库制备。我们进行了13个PCR循环以引入Chromium i7多重索引(10x Genomics,PN-120262)。cDNA在缓冲液EB中按1∶5稀释,并通过生物分析仪DNA高灵敏度(Agilent,5067-4626)和/或Qubit dsDNA高灵敏度(Thermo Fisher,Q32854)试剂进行定量。将样品等量合并并在NovaSeq S4流动池(Illumina)上使用读段参数28x8x91进行测序。使用CellRanger(10x Genomics,3.0.2版)将原始fastq文件映射到人类转录组(GRCh38),并使用Seurat(3.0.1版)进一步分析。
在上述最初的11个循环cDNA扩增步骤后,使用1uM p5正向引物(AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTC)(SEQ.ID NO.117)和1uM的定制TCRa-read2反向引物(GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTGAGGAACCAGCCTTATTGTTCATCCGTA)(SEQ IDNO:118)将50%(20ul)的每个cDNA样品加载到KAPA HIFI 2x PCR反应中并使用以下参数运行:98℃持续45″,[98℃持续20″,67℃持续30″,72℃持续30″]x10,72℃持续60″,保持在4℃。PCR产物用0.8x AMPure XP(Be ckman Coulter,A63882)纯化并在45ul缓冲液EB(Qiagen,1014608)中洗脱。我们进行了9个PCR循环以引入Chromium i7多重索引(10 xGenomics,PN-120262)。cDNA在缓冲液EB中按1∶5稀释,并通过Tapestation DNA高灵敏度(Agilent,5067-5593))和/或Qubit dsD NA高灵敏度(Thermo Fisher,Q32854)试剂进行定量。将样品等量合并并在具有25%PhiX的NovaSeq SP流动池(Illumina)上使用读段参数28x8x98进行测序。如图24b中所述分析测序数据并分配条形码。
实施例3
大型DNA构建体的多重文库的合并的敲入
我们接下来检查了36个合并模板的更大文库是否也可通过其DNA条形码引入和跟踪。定量条形码测序证明,即使使用更大的文库,所有敲入构建体在四个独立人类供体中进行的多个合并敲入实验中也得到充分表现(图29A)。所述文库包含范围从2kb到3kb的大型敲入构建体,甚至最大的构建体在这些生物重复中也得到充分表现(图29B)。36成员文库包含先前在2成员文库中测试的GFP和RFP模板,并且当对敲入阳性细胞进行门控时(通过对引入的NY-ESO-1TCR进行dextramer染色),可鉴定GFP+和RFP+细胞(图29C)。正如预期的那样,具有GFP或RFP测序条形码的读段百分比与四个人类供体在蛋白质水平上观测到的GFP或RFP阳性细胞的百分比紧密对应(图29D)。
我们接下来直接验证了同源臂错配测序策略,以使用更大的36成员合并敲入文库选择性地扩增中靶条形码。对于gDNA和cDNA测序条件,当从具有成功的中靶敲入(NY-ESO-1TCR+)的细胞中分选GFP+或RFP+细胞时,当使用在考虑双等位基因整合和模板转换时预测的速率下结合基因组序列的引物时对正确条形码选择性测序(图29E)。然而,正如预期的那样,与模板同源臂错配互补的引物应该优先扩增非HDR整合模板,其在分选的群体中没有显示相同的正确条形码富集(图29E)。最后,我们能够很容易地跟踪在白细胞介素2(IL-2)中培养的扩增T细胞群体中,敲入构建体条形码随时间的相对丰度。条形码丰度在所有构建体的整个扩增过程中都得以保持,但编码IL2RA的敲入构建体是一个显著的例外,IL2RA是白细胞介素(IL-2)的高亲和力受体,随着时间的推移,其在培养物中富集,与其已知的促进T细胞适应性的作用一致(图29F-G)。这些结果表明,可生成大型合并敲入文库,并且可在治疗相关的原代人T细胞群体中对它们的条形码进行定量测序。
合并的敲入命中单独验证和改进体外癌细胞杀伤
合并的敲入筛选鉴定了赋予靶向群体中敲入细胞竞争优势的基因构建体。我们想验证合并的敲入筛选平台,并确认敲入构建体“命中”将类似地提高单个敲入实验中的T细胞适应性(图30A)。首先,我们进行了测试以确保DNA敲入构建体导致预期的蛋白质产物的表达。具有八种个别敲入构建体的细胞的表面和细胞内染色揭示了每种预期蛋白质的稳健表达,包括在经刺激的T细胞中高于内源表达水平的Fas和CD25(IL2RA)表达增加(图30B和图32A)。我们接下来直接验证了八种个别敲入构建体在环境依赖性体外合并筛选中确定的适应性益处。事实上,抗凋亡FAS-41BB嵌合体和抗抑制性TGFβR2-41BB嵌合体均促进相对于对照的环境依赖性增加的扩增(图30C和图32B)。由TGFβR2-41BB构建体诱导的细胞扩增增加与在TGFβ存在下细胞增殖增加的证据相关(通过相对于对照细胞的CFSE稀释测量)(图30C和图32B)。这些发现验证了基于合并文库的方法来鉴定个别敲入序列以设计T细胞功能。
我们接下来进行了测试,以查看已鉴定的促进环境依赖性T细胞离体扩增的敲入构建体是否也可增强体外癌细胞杀伤。尽管这不是最初在合并的筛选中测试的表型,但在一系列效应子/靶细胞比率下,TGFβR2-41BB增加了来自四名人类供血者的T细胞中的靶标体外癌细胞杀伤(图30D-E)。相比之下,截短的CTLA4构建体显示出细胞杀伤减少(图30D-E),与其在合并的敲入筛选中的适应性受损一致(尽管使用该个别构建体未观测到通过CFSE评估的增殖减少,图32B)。最后,我们检查了TGFβR2-41BB嵌合受体是否在体外癌症杀伤方面也显示出进一步的环境依赖性改进。在存在外源TGFβ的情况下,TGFβR2-41BB构建体在四个健康人类供体进行的实验中成功挽救了受损的癌细胞杀伤(图30F)。尽管合并的筛选侧重于对T细胞适应性的细胞内在影响,但它们也成功地鉴定了可增强体外抗癌细胞功效的新型基因构建体。
PoKI-Seq:合并的敲入与单细胞转录组分析来评估丰度和细胞状态
合并的筛选方法揭示了影响群体中细胞丰度的修饰。然而,细胞丰度仅衡量细胞功能的一个方面,并且理想的筛选方法也将允许系统地评估修饰细胞状态。最近,新的条形码策略克服了这个问题,并允许通过scRNA-seq评估CRISPR敲除细胞的合并群体(Adamson等人,2016;Datlinger等人,2017;Dixit等人,2016;Jaitin等人,2016),包括原代人T细胞(Shifrut等人,2018)。我们接下来测试了合并的敲入筛选是否可类似地与scRNA-seq结合以生成关于修饰细胞状态的高维表型信息,同时还记录每个细胞的特定敲入构建体条形码,我们将这种方法称为PoKI-Seq(
首先,为了验证在整个实验管线中保持PoKI-Seq模板条形码的保真度,我们使用单细胞平台重复了GFP和RFP分选实验。从合并的敲入阳性群体(NY-ESO-1 TCR+)中分选GFP+或RFP+细胞使预期模板条形码强烈富集,证实了PoKI-Seq将特定敲入构建体准确分配给细胞的能力(图31B)。PoKI-Seq相对于合并敲入的批量扩增子测序的一个优势是能够将具有单个敲入构建体的细胞与接受了多于一个(最有可能来自双等位基因整合)的细胞区分开来。分配了两种敲入构建体的细胞的总体比例(图31C)与基于GFP/RFP 2成员文库实验的预测频率密切对应。正如预期的那样,当分选的GFP和RFP阳性细胞被分配两个敲入构建体时,所分配的敲入构建体中的至少一者几乎总是分别为GFP或RFP(图31C)。这些实验证实了PoKI-seq的分子生物学,并且还证明了它能够用单个或多个敲入构建体对个别细胞进行表型分析。
我们接下来测试了PoKI-Seq是否可将模板条形码分配给具有完全36成员合并敲入文库的大量细胞群体中的单细胞转录组。在存在或不存在外源TGFβ的情况下,在离体刺激后,我们对来自两个人类供血者的细胞进行了PoKI-Seq。出现了不同的细胞状态簇,尤其是添加了外源TGFβ(图31D-E)。对超过40,000个个别细胞进行了测序,其中约58%被成功分配了一个(单等位基因)或两个(双等位基因)条形码(图31F)。PoKI-Seq之后的质量控制指标证实了每个细胞的稳健的基因和UMI识别率以及分配给每个敲入构建体(每个供血者和每个离体测试条件)的平均>130X细胞覆盖率(图33B)。单细胞模板条形码分配得到进一步证实,因为由相应的敲入构建体编码的转录物倾向于以增加的水平表达,类似于在递送个别敲入构建体时在蛋白质水平上观测到的情况(图30B和图33C)。这些指标共同为PoKI-Seq平台建立了条形码分配到单细胞转录组的保真度。
接下来,我们检查了PoKI-Seq是否可测量由特定敲入构建体引起的离体人T细胞的细胞状态变化。在TCR刺激和刺激加TGFβ条件下,每个敲入构建体在个别细胞簇中引起不同的富集模式,这大致对应于体外合并敲入筛选的结果(图33D)。个别敲入构建体的密度图揭示,在刺激加TGFβ培养条件下,与对照和非TGFBR2衍生构建体相比,用TGFβR2衍生构建体修饰的T细胞的细胞状态存在显著差异(图31G)。具体而言,TGFβR2衍生构建体在仅刺激条件下与细胞另外相关的簇中显示出显著富集,所述簇包括以与细胞增殖相关的基因为特征的簇8和以与细胞杀伤相关的基因为特征的簇12(图31E、图31H和图33D、图33E)。类似地,TGFβR2衍生的敲入构建体从以其他方式通过TGFβ处理促进的簇(簇2、4和6)中的细胞耗尽(图31H)。在鉴定的单细胞簇中差异表达的所有基因的敲入构建体的聚类表明,在存在TGFβ的情况下,TGFβR2衍生构建体之间具有很强的相似性,并揭示了通过受体调节的下游靶基因。对于没有TGFβ刺激的细胞,最突出的基因表达相似性是在FAS衍生受体和IL2RA中(图33F)。分层聚类揭示,暴露于TGFβ驱动了最大的转录变化。例如,在存在外源TGFβ的情况下,TGFβR2衍生的嵌合受体促进了各种标志性增殖基因如MKI67和TOP2A的持续表达,而这些基因在表达其他KI构建体的细胞中被TGFβ抑制。PoKI-Seq证实了多种TGFβR2衍生受体的共同生物学效应,这些受体将抑制信号转化为促进细胞增殖和效应细胞状态的信号。更通常地,PoKI-Seq能够揭示由个别敲入构建体调节的细胞状态中的靶基因通路和环境依赖性变化。
应当理解,本文描述的实施例和实施方案仅用于说明目的,并且将向本领域技术人员建议根据其进行的各种修改或改变,并且将被包括在本申请的精神和范围内以及所附权利要求的范围内。本文引用的所有出版物、专利和专利申请均特此通过引用将其引用的内容并入本文。
在所附权利要求中,术语“一个”或“一种”旨在表示“一个或多个/一种或多种”。术语“包含(comprise)”及其变体如“包含(comprises)”和“包含(comprising)”,当在步骤或元件的叙述之前时,旨在表示其他步骤或元件的添加是可选的且不排除。本说明书中引用的所有专利、专利申请和其他公开的参考资料均通过引用整体并入本文。
SEQ ID No:73-116
PD-1胞外结构域SEQ ID NO:73
PGWFLDSPDRPWNPPTFSPALLVVTEGDNATFTCSFSNTSESFVLNWYRMSPSNQTDKLAAFPEDRSQPGQDCRFRVTQLPNGRDFHMSVVRARRNDSGTYLCGAISLAPKAQIKESLRAELRVTERRAEVPTAHPSPSPRPAGQFQTLV
PD-1跨膜结构域SEQ ID NO:74
VGVVGGLLGSLVLLVWVLAVI
PD-1胞内结构域SEQ ID NO:75
CSRAARGTIGARRTGQPLKEDPSAVPVFSVDYGELDFQWREKTPEPPVPCVPEQTEYATIVFPSGMGTSSPARRGSADGPRSAQPLRPEDGHCSWPL
4-1BB胞外结构域SEQ ID NO:76
LQDPCSNCPAGTFCDNNRNQICSPCPPNSFSSAGGQRTCDICRQCKGVFRTRKECSSTSNAECDCTPGFHCLGAGCSMCEQDCKQGQELTKKGCKDCCFGTFNDQKRGICRPWTNCSLDGKSVLVNGTKERDVVCGPSPADLSPGASSVTPPAPAREPGHSPQ
4-1BB跨膜结构域SEQ ID NO:77
IISFFLALTSTALLFLLFFLTLRFSVV
4-1BB胞内结构域SEQ ID NO:78
KRGRKKLLYIFKQPFMRPVQTTQEEDGCSCRFPEEEEGGCEL
ICOS胞外结构域SEQ ID NO:79
EINGSANYEMFIFHNGGVQILCKYPDIVQQFKMQLLKGGQILCDLTKTKGSGNTVSIKSLKFCHSQLSNNSVSFFLYNLDHSHANYYFCNLSIFDPPPFKVTLTGGYLHIYESQLCCQLK
ICOS TM SEQ ID NO:80
FWLPIGCAAFVVVCILGCILI
ICOS胞内结构域SEQ ID NO:81
CWLTKKKYSSSVHDPNGEYMFMRAVNTAKKSRLTDVTL
CTLA4胞外结构域SEQ ID NO:82
KAMHVAQPAVVLASSRGIASFVCEYASPGKATEVRVTVLRQADSQVTEVCAATYMMGNELTFLDDSICTGTSSGNQVNLTIQGLRAMDTGLYICKVELMYPPPYYLGIGNGTQIYVIDPEPCPDSD
CTLA4跨膜结构域SEQ ID NO:83
FLLWILAAVSSGLFFYSFLIT
CTLA4胞内结构域SEQ ID NO:84
AVSLSKMLKKRSPLTTGVYVKMPPTEPECEKQFQPYFIPIN
CD28胞外结构域SEQ ID NO:85
NKILVKQSPMLVAYDNAVNLSCKYSYNLFSREFRASLHKGLDSAVEVCVVYGNYSQQLQVYSKTGFNCDGKLGNESVTFYLQNLYVNQTDIYFCKIEVMYPPPYLDNEKSNGTIIHVKGKHLCPSPLFPGPSKP
CD28跨膜结构域SEQ ID NO:86
FWVLVVVGGVLACYSLLVTVAFIIFWV
CD28胞内结构域SEQ ID NO:87
RSKRSRLLHSDYMNMTPRRPGPTRKHYQPYAPPRDFAAYRS
CD200R胞外结构域SEQ ID NO:88
QPNNSLMLQTSKENHALASSSLCMDEKQITQNYSKVLAEVNTSWPVKMATNAVLCCPPIALRNLIIITWEIILRGQPSCTKAYKKETNETKETNCTDERITWVSRPDQNSDLQIRTVAITHDGYYRCIMVTPDGNFHRGYHLQVLVTPEVTLFQNRNRTAVCKAVAGKPAAHISWIPEGDCATKQEYWSNGTVTVKSTCHWEVHNVSTVTCHVSH
CD200R跨膜结构域SEQ ID NO:89
LTGNKSLYIELLPVPGAKKSA
CD200R胞内结构域SEQ ID NO:90
KVNGCRKYKLNKTESTPVVEEDEMQPYASYTEKNNPLYDTTNKVKASEALQSEVDTDLHTL
BTLA胞外结构域SEQ ID NO:91
KESCDVQLYIKRQSEHSILAGDPFELECPVKYCANRPHVTWCKLNGTTCVKLEDRQTSWKEEKNISFFILHFEPVLPNDNGSYRCSANFQSNLIESHSTTLYVTDVKSASERPSKDEMASRPWLLYR
BTLA跨膜结构域SEQ ID NO:92
LLPLGGLPLLITTCFCLFCCL
BTLA胞内结构域SEQ ID NO:93
RRHQGKQNELSDTAGREINLVDAHLKSEQTEASTRQNSQVLLSETGIYDNDPDLCFRMQEGSEVYSNPCLEENKPGIVYASLNHSVIGPNSRLARNVKEAPTEYASICVRS
TIM-3胞外结构域SEQ ID NO:94
SEVEYRAEVGQNAYLPCFYTPAAPGNLVPVCWGKGACPVFECGNVVLRTDERDVNYWTSRYWLNGDFRKGDVSLTIENVTLADSGIYCCRIQIPGIMNDEKFNLKLVIKPAKVTPAPTRQRDFTAAFPRMLTTRGHGPAETQTLGSLPDINLTQISTLANELRDSRLANDLRDSGATIRIG
TIM-3跨膜结构域SEQ ID NO:95
IYIGAGICAGLALALIFGALI
TIM-3胞内结构域SEQ ID NO:96
FKWYSHSKEKIQNLSLISLANLPPSGLANAVAEGIRSEENIYTIEENVYEVEEPNEYYCYVSSRQQPSQPLGCRFAMP
TIGIT胞外结构域SEQ ID NO:97
MMTGTIETTGNISAEKGGSIILQCHLSSTTAQVTQVNWEQQDQLLAICNADLGWHISPSFKDRVAPGPGLGLTLQSITVNDTGEYFCIYHTYPDGTYTGRIFLEVLESSVAEHGARFQIP
TIGIT跨膜结构域SEQ ID NO:98
LLGAMAATLVVICTAVIVVVA
TIGIT胞内结构域SEQ ID NO:99
LTRKKKALRIHSVEGDLRRKSAGQEEWSPSAPSPPGSCVQAEAAPAGLCGEQRGEDCAELHDYFNVLSYRSLGNCSFFTETG
TGFβR2胞外结构域SEQ ID NO:100
TIPPHVQKSVNNDMIVTDNNGAVKFPQLCKFCDVRFSTCDNQKSCMSNCSITSICEKPQEVCVAVWRKNDENITLETVCHDPKLPYHDFILEDAASPKCIMKEKKKPGETFFMCSCSSDECNDNIIFSEEYNTSNPDLLLVIFQ
TGFβR2跨膜结构域SEQ ID NO:101
VTGISLLPPLGVAISVIIIFY
TGFβR2胞内结构域SEQ ID NO:102
CYRVNRQQKLSSTWETGKTRKLMEFSEHCAIILEDDRSDISSTCANNINHNTELLPIELDTLVGKGRFAEVYKAKLKQNTSEQFETVAVKIFPYEEYASWKTEKDIFSDINLKHENILQFLTAEERKTELGKQYWLITAFHAKGNLQEYLTRHVISWEDLRKLGSSLARGIAHLHSDHTPCGRPKMPIVHRDLKSSNILVKNDLTCCLCDFGLSLRLDPTLSVDDLANSGQVGTARYMAPEVLESRMNLENVESFKQTDVYSMALVLLWEMTSRCNAVGEVKDYEPPFGSKVREHPCVESMKDNVLRDRGRPEIPSFWLNHQGIQMVCETLTECWDHDPEARLTAQCVAERFSELEHLDRLSGRSCSEEKIPEDGSLNTTK
IL-10RA胞外结构域SEQ ID NO:103
HGTELPSPPSVWFEAEFFHHILHWTPIPNQSESTCYEVALLRYGIESWNSISNCSQTLSYDLTAVTLDLYHSNGYRARVRAVDGSRHSNWTVTNTRFSVDEVT[JVGSVNLEIHNGFILGKIQLPRPKMAPANDTYESIFSHFREYEIAIRKVPGNFTFTHKKVKHENFSLLTSGEVGEFCVQVKPSVASRSNKGMWSKEECISLTRQYFTVTN
IL-10RA跨膜结构域SEQ ID NO:104
VIIFFAFVLLLSGALAYCLAL
IL-10RA胞内结构域SEQ ID NO:105
QLYVRRRKKLPSVLLFKKPSPFIFISQRPSPETQDTIHPLDEEAFLKVSPELKNLDLHGSTDSGFGSTKPSLQTEEPQFLLPDPHPQADRTLGNREPPVLGDSCSSGSSNSTDSGICLQEPSLSPSTGPTWEQQVGSNSRGQDDSGIDLVQNSEGRAGDTQGGSALGHHSPPEPEVPGEEDPAAVAFQGYLRQTRCAEEKATKTGCLEEESPLTDGLGPKFGRCLVDEAGLHPPALAKGYLKQDPLEMTLASSGAPTGQWNQPTEEWSLLALSSCSDLGISDWSFAHDLAPLGCVAAPGGLLGSFNSDLVTLPLISSLQSSE
IL-4RA胞外结构域SEQ ID NO:106
MKVLQEPTCVSDYMSISTCEWKMNGPTNCSTELRLLYQLVFLLSEAHTCIPENNGGAGCVCHLLMDDVVSADNYTLDLWAGQQLLWKGSFKPSEHVKPRAPGNLTVHTNVSDTLLLTWSNPYPPDNYLYNHLTYAVNIWSENDPADFRIYNVTYLEPSLRIAASTLKSGISYRARVRAWAQCYNTTWSEWSPSTKWHNSYREPFEQH
IL-4RA跨膜结构域SEQ ID NO:107
LLLGVSVSCIVILAVCLLCYVSIT
IL-4RA胞内结构域SEQ ID NO:108
KIKKEWWDQIPNPARSRLVAIIIQDAQGSQWEKRSRGQEPAKCPHWKNCLTKLLPCFLEHNMKRDEDPHKAAKEMPFQGSGKSAWCPVEISKTVLWPESISVVRCVELFEAPVECEEEEEVEEEKGSFCASPESSRDDFQEGREGIVARLTESLFLDLLGEENGGFCQQDMGESCLLPPSGSTSAHMPWDEFPSAGPKEAPPWGKEQPLHLEPSPPASPTQSPDNLTCTETPLVIAGNPAYRSFSNSLSQSPCPRELGPDPLLARHLEEVEPEMPCVPQLSEPTTVPQPEPETWEQILRRNVLQHGAAAAPVSAPTSGYQEFVHAVEQGGTQASAVVGLGPPGEAGYKAFSSLLASSAVSPEKCGFGASSGEEGYKPFQDLIPGCPGDPAPVPVPLFTFGLDREPPRSPQSSHLPSSSPEHLGLEPGEKVEDMPKPPLPQEQATDPLVDSLGSGIVYSALTCHLCGHLKQCHGQEDGGQTPVMASPCCGCCCGDRSSPPTTPLRAPDPSPGGVPLEASLCPASLAPSGISEKSKSSSSFHPAPGNAQSSSQTPKIVNFVSVGPTYMRVS
IL-7RA跨膜结构域SEQ ID NO:109
PILLTISILSFFSVALLVILACVLW
IL-7RA胞内结构域SEQ ID NO:110
KKRIKPIVWPSLPDHKKTLEHLCKKPRKNLNVSFNPESFLDCQIHRVDDIQARDEVEGFLQDTFPQQLEESEKQRLGGDVQSPNCPSEDVVITPESFGRDSSLTCLAGNVSACDAPILSSSRSLDCRESGKNGPHVYQDLLLSLGTTNSTLPPPFSLQSGILTLNPVAQGQPILTSLGSNQEEAYVTMSSFYQNQ
Fas胞外结构域SEQ ID NO:111
QVTDINSKGLELRKTVTTVETQNLEGLHHDGQFCHKPCPPGERKARDCTVNGDEPDCVPCQEGKEYTDKAHFSSKCRRCRLCDEGHGLEVEINCTRTQNTKCRCKPNFFCNSTVCEHCDPCTKCEHGIIKECTLTSNTKCKEEGSRSN
Fas跨膜结构域SEQ ID NO:112
LGWLCLLLLPIPLIVWV
Fas胞内结构域SEQ ID NO:113
KRKEVQKTCRKHRKENQGSHESPTLNPETVAINLSDVDLSKYITTIAGVMTLSQVKGFVRKNGVNEAKIDEIKNDNVQDTAEQKVQLLRNWHQLHGKKEAYDTLIKDLKKANLCTLAEKIQTIILKDITSDSENSNFRNEIQSLV
TRAILR2胞外结构域SEQ ID NO:114
ITQQDLAPQQRAAPQQKRSSPSEGLCPPGHHISEDGRDCISCKYGQDYSTHWNDLLFCLRCTRCDSGEVELSPCTTTRNTVCQCEEGTFREEDSPEMCRKCRTGCPRGMVKVGDCTPWSDIECVHKESGTKHSGEVPAVEETVTSSPGTPASPCS
TRAILR2跨膜结构域SEQ ID NO:115
LSGIIIGVTVAAVVLIVAVFV
TRAILR2胞内结构域SEQ ID NO:116
CKSLLWKKVLPYLKGICSGGGGDPERVDRSSQRPGAEDNVLNEIVSILQPTQVPEQEMEVQEPAEPTGVNMLSPGESEHLLEPAEAERSQRRRLLVPANEGDPTETLRQCFDDFADLVPFDSWEPLMRKLGLMDNEIKVAKAEAAGHRDTLYTMLIKWVNKTGRDASVHTLLDALETLGERLAKQKIEDHLLSSGKFMYLEGNADSAMS。
序列表
<110> 加利福尼亚大学董事会(The Regents of the University of California)
T·L·罗斯 (Roth, Theodore L)
P-Y·J·李(Li, Po-Yi Jonathan)
A·曼森(Marson, Alexander)
J·奈斯(Nies, Jasper)
C·莫厄尔(Mowery, Cody)
E·士夫洛特(Shifrut, Eric)
F·布里斯彻克(Blaeschke, Franziska)
R·阿帕熙(Apathy, Ryan)
<120> 合并的敲入筛选和在内源基因座控制下共表达的异源多肽
<130> 081906-235410WO-1176415
<150> 62/818,535
<151> 2019-03-14
<150> 62/818,578
<151> 2019-03-14
<150> 62/871,467
<151> 2019-07-08
<150> 62/871,309
<151> 2019-07-08
<160> 118
<170> PatentIn version 3.5
<210> 1
<211> 603
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 1
atgcagatcc cacaggcgcc ctggccagtc gtctgggcgg tgctacaact gggctggcgg 60
ccaggatggt tcttagactc cccagacagg ccctggaacc ccccaacctt ctccccagcc 120
ctgctcgtgg tgaccgaagg ggacaacgcc accttcacct gcagcttctc caacacatcg 180
gagagcttcg tgctaaactg gtaccgcatg agccccagca accagacgga caagctggca 240
gcattccctg aggaccgtag ccagcctggc caggactgcc gtttccgtgt cacacaactg 300
cccaacgggc gtgacttcca catgagcgtg gtcagggccc ggcgcaatga cagcggcaca 360
tacctttgtg gggccatctc cctggccccc aaggcgcaga tcaaagagag cctgcgggca 420
gagctcaggg tgacagagag aagggcagaa gtgcccacag cccaccctag cccctcacct 480
agaccagccg gccagttcca aacactggtg gttggtgtcg tgggcggcct gctgggcagc 540
ctggtgctgc tagtctgggt cctggccgtc atctgctccc gggccgcacg tgggacaata 600
gga 603
<210> 2
<211> 708
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 2
atgcagatcc cacaggcgcc ctggccagtc gtctgggcgg tgctacaact gggctggcgg 60
ccaggatggt tcttagactc cccagacagg ccctggaacc ccccaacctt ctccccagcc 120
ctgctcgtgg tgaccgaagg ggacaacgcc accttcacct gcagcttctc caacacatcg 180
gagagcttcg tgctaaactg gtaccgcatg agccccagca accagacgga caagctggca 240
gcattccctg aggaccgtag ccagcctggc caggactgcc gtttccgtgt cacacaactg 300
cccaacgggc gtgacttcca catgagcgtg gtcagggccc ggcgcaatga cagcggcaca 360
tacctttgtg gggccatctc cctggccccc aaggcgcaga tcaaagagag cctgcgggca 420
gagctcaggg tgacagagag aagggcagaa gtgcccacag cccaccctgc ccctgcgaga 480
gagccaggac actctccgca gatcatctcc ttctttcttg cgctgacgtc gactgcgttg 540
ctcttcctgc tgttcttcct cacgctccgt ttctctgttg ttaaacgggg cagaaagaaa 600
ctcctgtata tattcaaaca accatttatg agaccagtac aaactactca agaggaagat 660
ggctgtagct gccgatttcc agaagaagaa gaaggaggat gtgaactg 708
<210> 3
<211> 900
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 3
atgcagatcc cacaggcgcc ctggccagtc gtctgggcgg tgctacaact gggctggcgg 60
ccaggatggt tcttagactc cccagacaga ccctggaacc ctcctacctt ctccccagcc 120
ctgcttgtgg tgaccgaagg ggacaacgcc accttcacct gcagcttctc caacacatcg 180
gagagcttcg tgctaaactg gtaccgcatg agccctagca accagacgga caagctggca 240
gcattccctg aggaccgtag ccagcctggc caggactgcc gtttccgtgt cacacaactg 300
cctaacgggc gtgacttcca catgagcgtg gtcagagccc ggcgcaatga cagcggcaca 360
tacctttgtg gggccatctc cctggcccct aaggcgcaga tcaaagagag cctgcgggca 420
gagcttaggg tgacagagag aagagcagaa gtgcctacag cccaccctag cccttcacct 480
agaccagccg gccagttcca aacactggtg gttggtgtcg tgggcggcct gctgggcagc 540
ctggtgctgc tagtctgggt cctggccgtc atctgctctc gggccgcacg aggaacacag 600
gtggcggctg actggaccgc gctggcggag gagatggatt ttgagtactt ggagatccgg 660
caactggaga cacaagcgga ccccactggc agactgctgg acgcctggca gggacgccct 720
ggcgcctctg taggccgact gcttgagctg cttaccaagc tgggccgcga cgacgtgctg 780
ctggagctgg gacccagcat tgaggaggat tgccaaaagt atatcttgaa gcagcagcag 840
gaggaggctg agaagccttt acaggtggcc gctgtagaca gtagtgtccc acggacagca 900
<210> 4
<211> 678
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 4
atgcagatcc cacaggcgcc ctggccagtc gtctgggcgg tgctacaact gggctggcgg 60
ccaggatggt tcttagactc cccagacagg ccctggaacc ccccaacctt ctccccagcc 120
ctgctcgtgg tgaccgaagg ggacaacgcc accttcacct gcagcttctc caacacatcg 180
gagagcttcg tgctaaactg gtaccgcatg agccccagca accagacgga caagctggca 240
gcattccctg aggaccgtag ccagcctggc caggactgcc gtttccgtgt cacacaactg 300
cccaacgggc gtgacttcca catgagcgtg gtcagggccc ggcgcaatga cagcggcaca 360
tacctttgtg gggccatctc cctggccccc aaggcgcaga tcaaagagag cctgcgggca 420
gagctcaggg tgacagagag aagggcagaa gtgcccacag cccaccatat ttatgaatca 480
caactttgtt gccagctgaa gttctggtta cccataggat gtgcagcctt tgttgtagtc 540
tgcattttgg gatgcatact tatttgttgg cttacaaaaa agaagtattc atccagtgtg 600
cacgacccta acggtgaata catgttcatg agagcagtga acacagccaa aaaatctaga 660
ctcacagatg tgacccta 678
<210> 5
<211> 567
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 5
atggcttgcc ttggatttca gcggcacaag gctcagctga acctggctac caggacctgg 60
ccctgcactc tcctgttttt tcttctcttc atccctgtct tctgcaaagc aatgcacgtg 120
gcccagcctg ctgtggtact ggccagcagc cgaggcatcg ccagctttgt gtgtgagtat 180
gcatctccag gcaaagccac tgaggtccgg gtgacagtgc ttcggcaggc tgacagccag 240
gtgactgaag tctgtgcggc aacctacatg atggggaatg agttgacctt cctagatgat 300
tccatctgca cgggcacctc cagtggaaat caagtgaacc tcactatcca aggactgagg 360
gccatggaca cgggactcta catctgcaag gtggagctca tgtacccacc gccatactac 420
ctgggcatag gcaacggaac ccagatttat gtaattgatc cagaaccgtg cccagattct 480
gacttcctcc tctggatcct tgcagcagtt agttcggggt tgttttttta tagctttctc 540
ctcacagctg tttctttgag caaaatg 567
<210> 6
<211> 690
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 6
atggcttgcc ttggatttca gcggcacaag gctcagctga acctggctac caggacctgg 60
ccctgcactc tcctgttttt tcttctcttc atccctgtct tctgcaaagc aatgcacgtg 120
gcccagcctg ctgtggtact ggccagcagc cgaggcatcg ccagctttgt gtgtgagtat 180
gcatctccag gcaaagccac tgaggtccgg gtgacagtgc ttcggcaggc tgacagccag 240
gtgactgaag tctgtgcggc aacctacatg atggggaatg agttgacctt cctagatgat 300
tccatctgca cgggcacctc cagtggaaat caagtgaacc tcactatcca aggactgagg 360
gccatggaca cgggactcta catctgcaag gtggagctca tgtacccacc gccatactac 420
ctgggcatag gcaacggaac ccagatttat gtaattgatc cagaaccgtg cccagattct 480
gacttcctcc tctggatcct tgcagcagtt agttcggggt tgttttttta tagctttctc 540
ctcacagctg tttctttgag caaaatgagg agtaagagga gcaggctcct gcacagtgac 600
tacatgaaca tgactccccg ccgccccggg cccacccgca agcattacca gccctatgcc 660
ccaccacgcg acttcgcagc ctatcgctcc 690
<210> 7
<211> 813
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 7
atgctctgcc cttggagaac tgctaaccta gggctactgt tgattttgac tatcttctta 60
gtggccgaag cggagggtgc tgctcaacca aacaactcat taatgctgca aactagcaag 120
gagaatcatg ctttagcttc aagcagttta tgtatggatg aaaaacagat tacacagaac 180
tactcgaaag tactcgcaga agttaacact tcatggcctg taaagatggc tacaaatgct 240
gtgctttgtt gccctcctat cgcattaaga aatttgatca taataacatg ggaaataatc 300
ctgagaggcc agccttcctg cacaaaagcc tacaagaaag aaacaaatga gaccaaggaa 360
accaactgta ctgatgagag aataacctgg gtctccagac ctgatcagaa ttcggacctt 420
cagattcgta ccgtggccat cactcatgac gggtattaca gatgcataat ggtaacacct 480
gatgggaatt tccatcgtgg atatcacctc caagtgttag ttacacctga agtgaccctg 540
tttcaaaaca ggaatagaac tgcagtatgc aaggcagttg cagggaagcc agctgcgcat 600
atctcctgga tcccagaggg cgattgtgcc actaagcaag aatactggag caatggcaca 660
gtgactgtta agagtacatg ccactgggag gtccacaatg tgtctaccgt gacctgccac 720
gtctcccatt tgactggcaa caagagtctg tacatagagc tacttcctgt tccaggtgcc 780
aaaaaatcag caaaattata tattccatat atc 813
<210> 8
<211> 555
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 8
atgaagacat tgcctgccat gcttggaact gggaaattat tttgggtctt cttcttaatc 60
ccatatctgg acatctggaa catccatggg aaagaatcat gtgatgtaca gctttatata 120
aagagacaat ctgaacactc catcttagca ggagatccct ttgaactaga atgccctgtg 180
aaatactgtg ctaacaggcc tcatgtgact tggtgcaagc tcaatggaac aacatgtgta 240
aaacttgaag atagacaaac aagttggaag gaagagaaga acatttcatt tttcattcta 300
cattttgaac cagtgcttcc taatgacaat gggtcatacc gctgttctgc aaattttcag 360
tctaatctca ttgaaagcca ctcaacaact ctttatgtga cagatgtaaa aagtgcctca 420
gaacgaccct ccaaggacga aatggcaagc agaccctggc tcctgtatcg tttacttcct 480
ttggggggat tgcctctact catcactacc tgtttctgcc tgttctgctg cctgagaagg 540
caccaaggaa agcaa 555
<210> 9
<211> 684
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 9
atgaagacat tgcctgccat gcttggaact gggaaattat tttgggtctt cttcttaatc 60
ccatatctgg acatctggaa catccatggg aaagaatcat gtgatgtaca gctttatata 120
aagagacaat ctgaacactc catcttagca ggagatccct ttgaactaga atgccctgtg 180
aaatactgtg ctaacaggcc tcatgtgact tggtgcaagc tcaatggaac aacatgtgta 240
aaacttgaag atagacaaac aagttggaag gaagagaaga acatttcatt tttcattcta 300
cattttgaac cagtgcttcc taatgacaat gggtcatacc gctgttctgc aaattttcag 360
tctaatctca ttgaaagcca ctcaacaact ctttatgtga cagatgtaaa aagtgcctca 420
gaacgaccct ccaaggacga aatgtgtcca agtcccctat ttcccggacc ttctaagccc 480
ttttgggtgc tggtggtggt tggtggagtc ctggcttgct atagcttgct agtaacagtg 540
gcctttatta ttttctgggt gaggagtaag aggagcaggc tcctgcacag tgactacatg 600
aacatgactc cccgccgccc cgggcccacc cgcaagcatt accagcccta tgccccacca 660
cgcgacttcg cagcctatcg ctcc 684
<210> 10
<211> 690
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 10
atgttttcac atcttccctt tgactgtgtc ctgctgctgc tgctgctact acttacaagg 60
tcctcagaag tggaatacag agcggaggtc ggtcagaatg cctatctgcc ctgcttctac 120
accccagccg ccccagggaa cctcgtgccc gtctgctggg gcaaaggagc ctgtcctgtg 180
tttgaatgtg gcaacgtggt gctcaggact gatgaaaggg atgtgaatta ttggacatcc 240
agatactggc taaatgggga tttccgcaaa ggagatgtgt ccctgaccat agagaatgtg 300
actctagcag acagtgggat ctactgctgc cggatccaaa tcccaggcat aatgaatgat 360
gaaaaattta acctgaagtt ggtcatcaaa ccagccaagg tcacccctgc accgactcgg 420
cagagagact tcactgcagc ctttccaagg atgcttacca ccaggggaca tggcccagca 480
gagacacaga cactggggag cctccctgat ataaatctaa cacaaatatc cacattggcc 540
aatgagttac gggactctag attggccaat gacttacggg actctggagc aaccatcaga 600
ataggcatct acatcggagc agggatctgt gctgggctgg ctctggctct tatcttcggc 660
gctttaattt tcaaatggta ttctcatagc 690
<210> 11
<211> 819
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 11
atgttttcac atcttccctt tgactgtgtc ctgctgctgc tgctgctact acttacaagg 60
tcctcagaag tggaatacag agcggaggtc ggtcagaatg cctatctgcc ctgcttctac 120
accccagccg ccccagggaa cctcgtgccc gtctgctggg gcaaaggagc ctgtcctgtg 180
tttgaatgtg gcaacgtggt gctcaggact gatgaaaggg atgtgaatta ttggacatcc 240
agatactggc taaatgggga tttccgcaaa ggagatgtgt ccctgaccat agagaatgtg 300
actctagcag acagtgggat ctactgctgc cggatccaaa tcccaggcat aatgaatgat 360
gaaaaattta acctgaagtt ggtcatcaaa ccagccaagg tcacccctgc accgactcgg 420
cagagagact tcactgcagc ctttccaagg atgcttacca ccaggggaca tggcccagca 480
gagacacaga cactggggag cctccctgat ataaatctaa cacaaatatc cacattggcc 540
aatgagttac gggactctag attggccaat gacttacggt gtccaagtcc cctatttccc 600
ggaccttcta agcccttttg ggtgctggtg gtggttggtg gagtcctggc ttgctatagc 660
ttgctagtaa cagtggcctt tattattttc tgggtgagga gtaagaggag caggctcctg 720
cacagtgact acatgaacat gactcctcgc cgccctgggc ccacccgcaa gcattaccag 780
ccctatgccc caccacgcga cttcgcagcc tatcgctcc 819
<210> 12
<211> 507
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 12
atgcgctggt gtctcctcct gatctgggcc caggggctga ggcaggctcc cctcgcctca 60
ggaatgatga caggcacaat agaaacaacg gggaacattt ctgcagagaa aggtggctct 120
atcatcttac aatgtcacct ctcctccacc acggcacaag tgacccaggt caactgggag 180
cagcaggacc agcttctggc catttgtaat gctgacttgg ggtggcacat ctccccatcc 240
ttcaaggatc gagtggcccc aggtcccggc ctgggcctca ccctccagtc gctgaccgtg 300
aacgatacag gggagtactt ctgcatctat cacacctacc ctgatgggac gtacactggg 360
agaatcttcc tggaggtcct agaaagctca gtggctgagc acggtgccag gttccagatt 420
ccattgcttg gagccatggc cgcgacgctg gtggtcatct gcacagcagt catcgtggtg 480
gtcgcgttga ctagaaagaa gaaagcc 507
<210> 13
<211> 636
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 13
atgcgctggt gtctcctcct gatctgggcc caggggctga ggcaggctcc cctcgcctca 60
ggaatgatga caggcacaat agaaacaacg gggaacattt ctgcagagaa aggtggctct 120
atcatcttac aatgtcacct ctcctccacc acggcacaag tgacccaggt caactgggag 180
cagcaggacc agcttctggc catttgtaat gctgacttgg ggtggcacat ctccccatcc 240
ttcaaggatc gagtggcccc aggtcccggc ctgggcctca ccctccagtc gctgaccgtg 300
aacgatacag gggagtactt ctgcatctat cacacctacc ctgatgggac gtacactggg 360
agaatcttcc tggaggtcct agaaagctca gtggcttgtc caagtcccct atttcccgga 420
ccttctaagc ccttttgggt gctggtggtg gttggtggag tcctggcttg ctatagcttg 480
ctagtaacag tggcctttat tattttctgg gtgaggagta agaggagcag gctcctgcac 540
agtgactaca tgaacatgac tccccgccgc cccgggccca cccgcaagca ttaccagccc 600
tatgccccac cacgcgactt cgcagcctat cgctcc 636
<210> 14
<211> 603
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 14
atgggtcggg ggctgctcag gggcctgtgg ccgctgcaca tcgtcctgtg gacgcgtatc 60
gccagcacga tcccaccgca cgttcagaag tcggttaata acgacatgat agtcactgac 120
aacaacggtg cagtcaagtt tccacaactg tgtaaatttt gtgatgtgag attttccacc 180
tgtgacaacc agaaatcctg catgagcaac tgcagcatca cctccatctg tgagaagcca 240
caggaagtct gtgtggctgt atggagaaag aatgacgaga acataacact agagacagtt 300
tgccatgacc ccaagctccc ctaccatgac tttattctgg aagatgctgc ttctccaaag 360
tgcattatga aggaaaaaaa aaagcctggt gagactttct tcatgtgttc ctgtagctct 420
gatgagtgca atgacaacat catcttctca gaagaatata acaccagcaa tcctgacttg 480
ttgctagtca tatttcaagt gacaggcatc agcctcctgc caccactggg agttgccata 540
tctgtcatca tcatcttcta ctgctaccgc gttaaccggc agcagaagct gagttcatcc 600
gga 603
<210> 15
<211> 708
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 15
atgggtcggg ggctgctcag gggcctgtgg ccgctgcaca tcgtcctgtg gacgcgtatc 60
gccagcacga tcccaccgca cgttcagaag tcggttaata acgacatgat agtcactgac 120
aacaacggtg cagtcaagtt tccacaactg tgtaaatttt gtgatgtgag attttccacc 180
tgtgacaacc agaaatcctg catgagcaac tgcagcatca cctccatctg tgagaagcca 240
caggaagtct gtgtggctgt atggagaaag aatgacgaga acataacact agagacagtt 300
tgccatgacc ccaagctccc ctaccatgac tttattctgg aagatgctgc ttctccaaag 360
tgcattatga aggaaaaaaa aaagcctggt gagactttct tcatgtgttc ctgtagctct 420
gatgagtgca atgacaacat catcttctca gaagaatata acaccagcaa tcctgacttg 480
ttgctagtca tatttcaagt gacaggcatc agcctcctgc caccactggg agttgccata 540
tctgtcatca tcatcttcta ctgctaccgc gttaaccggc agaaacgggg cagaaagaaa 600
ctcctgtata tattcaaaca accatttatg agaccagtac aaactactca agaggaagat 660
ggctgtagct gccgatttcc agaagaagaa gaaggaggat gtgaactg 708
<210> 16
<211> 888
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 16
atgggtcggg ggctgctcag gggcctgtgg ccgctgcaca tcgtcctgtg gacgcgtatc 60
gccagcacga tcccaccgca cgttcagaag tcggttaata acgacatgat agtcactgac 120
aacaacggtg cagtcaagtt tccacaactg tgtaaatttt gtgatgtgag attttccacc 180
tgtgacaacc agaaatcctg catgagcaac tgcagcatca cctccatctg tgagaagcca 240
caggaagtct gtgtggctgt atggagaaag aatgacgaga acataacact agagacagtt 300
tgccatgacc ccaagctccc ctaccatgac tttattctgg aagatgctgc ttctccaaag 360
tgcattatga aggaaaaaaa aaagcctggt gagactttct tcatgtgttc ctgtagctct 420
gatgagtgca atgacaacat catcttctca gaagaatata acaccagcaa tcctgacttg 480
ttgctagtca tatttcaagt gacaggcatc agcctcctgc caccactggg agttgccata 540
tctgtcatca tcatcttcta ctgctaccgc gttaaccggc agacacaggt ggcggccgac 600
tggaccgcgc tggcggagga gatggacttt gagtacttgg agatccggca actggagaca 660
caagcggacc ccactggcag gctgctggac gcctggcagg gacgccctgg cgcctctgta 720
ggccgactgc tcgagctgct taccaagctg ggccgcgacg acgtgctgct ggagctggga 780
cccagcattg aggaggattg ccaaaagtat atcttgaagc agcagcagga ggaggctgag 840
aagcctttac aggtggccgc tgtagacagc agtgtcccac ggacagca 888
<210> 17
<211> 789
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 17
atgctgccgt gcctcgtagt gctgctggcg gcgctcctca gcctccgtct tggctcagac 60
gctcatggga cagagctgcc cagccctccg tctgtgtggt ttgaagcaga atttttccac 120
cacatcctcc actggacacc catcccaaat cagtctgaaa gtacctgcta tgaagtggcg 180
ctcctgaggt atggaataga gtcctggaac tccatctcca actgtagcca gaccctgtcc 240
tatgacctta ccgcagtgac cttggacctg taccacagca atggctaccg ggccagagtg 300
cgggctgtgg acggcagccg gcactccaac tggaccgtca ccaacacccg cttctctgtg 360
gatgaagtga ctctgacagt tggcagtgtg aacctagaga tccacaatgg cttcatcctc 420
gggaagattc agctacccag gcccaagatg gcccccgcaa atgacacata tgaaagcatc 480
ttcagtcact tccgagagta tgagattgcc attcgcaagg tgccgggaaa cttcacgttc 540
acacacaaga aagtaaaaca tgaaaacttc agcctcctaa cctctggaga agtgggagag 600
ttctgtgtcc aggtgaaacc atctgtcgct tcccgaagta acaaggggat gtggtctaaa 660
gaggagtgca tctccctcac caggcagtat ttcaccgtga ccaacgtcat catcttcttt 720
gcctttgtcc tgctgctctc cggagccctc gcctactgcc tggccctcca gctgtatgtg 780
cggcgccga 789
<210> 18
<211> 1368
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 18
atgctgccgt gcctcgtagt gctgctggcg gcgctcctca gcctccgtct tggctcagac 60
gctcatggga cagagctgcc cagccctccg tctgtgtggt ttgaagcaga atttttccac 120
cacatcctcc actggacacc catcccaaat cagtctgaaa gtacctgcta tgaagtggcg 180
ctcctgaggt atggaataga gtcctggaac tccatctcca actgtagcca gaccctgtcc 240
tatgacctta ccgcagtgac cttggacctg taccacagca atggctaccg ggccagagtg 300
cgggctgtgg acggcagccg gcactccaac tggaccgtca ccaacacccg cttctctgtg 360
gatgaagtga ctctgacagt tggcagtgtg aacctagaga tccacaatgg cttcatcctc 420
gggaagattc agctacccag gcccaagatg gcccccgcaa atgacacata tgaaagcatc 480
ttcagtcact tccgagagta tgagattgcc attcgcaagg tgccgggaaa cttcacgttc 540
acacacaaga aagtaaaaca tgaaaacttc agcctcctaa cctctggaga agtgggagag 600
ttctgtgtcc aggtgaaacc atctgtcgct tcccgaagta acaaggggat gtggtctaaa 660
gaggagtgca tctccctcac caggcagtat ttcaccgtga ccaacgtccc tatcttacta 720
accatcagca ttttgagttt tttctctgtc gctctgttgg tcatcttggc ctgtgtgtta 780
tggaaaaaaa ggattaagcc tatcgtatgg cccagtctcc ccgatcataa gaagactctg 840
gaacatcttt gtaagaaacc aagaaaaaat ttaaatgtga gtttcaatcc tgaaagtttc 900
ctggactgcc agattcatag ggtggatgac attcaagcta gagatgaagt ggaaggtttt 960
ctgcaagata cgtttcctca gcaactagaa gaatctgaga agcagaggct tggaggggat 1020
gtgcagagcc ccaactgccc atctgaggat gtagtcatca ctccagaaag ctttggaaga 1080
gattcatccc tcacatgcct ggctgggaat gtcagtgcat gtgacgcccc tattctctcc 1140
tcttccaggt ccctagactg cagggagagt ggcaagaatg ggcctcatgt gtaccaggac 1200
ctcctgctta gccttgggac tacaaacagc acgctgcccc ctccattttc tctccaatct 1260
ggaatcctga cattgaaccc agttgctcag ggtcagccca ttcttacttc cctgggatca 1320
aatcaagaag aagcatatgt caccatgtcc agcttctacc aaaaccag 1368
<210> 19
<211> 1359
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 19
atggggtggc tttgctctgg gctcctgttc cctgtgagct gcctggtcct gctgcaggtg 60
gcaagctctg ggaacatgaa ggtcttgcag gagcccacct gcgtctccga ctacatgagc 120
atctctactt gcgagtggaa gatgaatggt cccaccaatt gcagcaccga gctccgcctg 180
ttgtaccagc tggtttttct gctctccgaa gcccacacgt gtatccctga gaacaacgga 240
ggcgcggggt gcgtgtgcca cctgctcatg gatgacgtgg tcagtgcgga taactataca 300
ctggacctgt gggctgggca gcagctgctg tggaagggct ccttcaagcc cagcgagcat 360
gtgaaaccca gggccccagg aaacctgaca gttcacacca atgtctccga cactctgctg 420
ctgacctgga gcaacccgta tccccctgac aattacctgt ataatcatct cacctatgca 480
gtcaacattt ggagtgaaaa cgacccggca gatttcagaa tctataacgt gacctaccta 540
gaaccctccc tccgcatcgc agccagcacc ctgaagtctg ggatttccta cagggcacgg 600
gtgagggcct gggctcagtg ctataacacc acctggagtg agtggagccc cagcaccaag 660
tggcacaact cctacaggga gcccttcgag cagcacctcc ctatcttact aaccatcagc 720
attttgagtt ttttctctgt cgctctgttg gtcatcttgg cctgtgtgtt atggaaaaaa 780
aggattaagc ctatcgtatg gcccagtctc cccgatcata agaagactct ggaacatctt 840
tgtaagaaac caagaaaaaa tttaaatgtg agtttcaatc ctgaaagttt cctggactgc 900
cagattcata gggtggatga cattcaagct agagatgaag tggaaggttt tctgcaagat 960
acgtttcctc agcaactaga agaatctgag aagcagaggc ttggagggga tgtgcagagc 1020
cccaactgcc catctgagga tgtagtcatc actccagaaa gctttggaag agattcatcc 1080
ctcacatgcc tggctgggaa tgtcagtgca tgtgacgccc ctattctctc ctcttccagg 1140
tccctagact gcagggagag tggcaagaat gggcctcatg tgtaccagga cctcctgctt 1200
agccttggga ctacaaacag cacgctgccc cctccatttt ctctccaatc tggaatcctg 1260
acattgaacc cagttgctca gggtcagccc attcttactt ccctgggatc aaatcaagaa 1320
gaagcatatg tcaccatgtc cagcttctac caaaaccag 1359
<210> 20
<211> 816
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 20
atggattcat acctgctgat gtggggactg ctcacgttca tcatggtgcc tggctgccag 60
gcagagctct gtgacgatga cccgccagag atcccacacg ccacattcaa agccatggcc 120
tacaaggaag gaaccatgtt gaactgtgaa tgcaagagag gtttccgcag aataaaaagc 180
gggtcactct atatgctctg tacaggaaac tctagccact cgtcctggga caaccaatgt 240
caatgcacaa gctctgccac tcggaacaca acgaaacaag tgacacctca acctgaagaa 300
cagaaagaaa ggaaaaccac agaaatgcaa agtccaatgc agccagtgga ccaagcgagc 360
cttccaggtc actgcaggga acctccacca tgggaaaatg aagccacaga gagaatttat 420
catttcgtgg tggggcagat ggtttattat cagtgcgtcc agggatacag ggctctacac 480
agaggtcctg ctgagagcgt ctgcaaaatg acccacggga agacaaggtg gacccagccc 540
cagctcatat gcacaggtga aatggagacc agtcagtttc caggtgaaga gaagcctcag 600
gcaagccccg aaggccgtcc tgagagtgag acttcctgcc tcgtcacaac aacagatttt 660
caaatacaga cagaaatggc tgcaaccatg gagacgtcca tatttacaac agagtaccag 720
gtagcagtgg ccggctgtgt tttcctgctg atcagcgtcc tcctcctgag tgggctcacc 780
tggcagcgga gacagaggaa gagtagaaga acaatc 816
<210> 21
<211> 1377
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 21
atgacaattc taggtacaac ttttggcatg gttttttctt tacttcaagt cgtttctgga 60
gaaagtggct atgctcaaaa tggagacttg gaagatgcag aactggatga ctactcattc 120
tcatgctata gccagttgga agtgaatgga tcgcagcact cactgacctg tgcttttgag 180
gacccagatg tcaacatcac caatctggaa tttgaaatat gtggggccct cgtggaggta 240
aagtgcctga atttcaggaa actacaagag atatatttca tcgagacaaa gaaattctta 300
ctgattggaa agagcaatat atgtgtgaag gttggagaaa agagtctaac ctgcaaaaaa 360
atagacctaa ccactatagt taaacctgag gctccttttg acctgagtgt cgtctatcgg 420
gaaggagcca atgactttgt ggtgacattt aatacatcac acttgcaaaa gaagtatgta 480
aaagttttaa tgcacgatgt agcttaccgc caggaaaagg atgaaaacaa atggacgcat 540
gtgaatttat ccagcacaaa gctgacactc ctgcagagaa agctccaacc ggcagcaatg 600
tatgagatta aagttcgatc catccctgat cactatttta aaggcttctg gagtgaatgg 660
agtccaagtt attacttcag aactccagag atcaataata gctcagggga gatggatcct 720
atcttactaa ccatcagcat tttgagtttt ttctctgtcg ctctgttggt catcttggcc 780
tgtgtgttat ggaaaaaaag gattaagcct atcgtatggc ccagtctccc cgatcataag 840
aagactctgg aacatctttg taagaaacca agaaaaaatt taaatgtgag tttcaatcct 900
gaaagtttcc tggactgcca gattcatagg gtggatgaca ttcaagctag agatgaagtg 960
gaaggttttc tgcaagatac gtttcctcag caactagaag aatctgagaa gcagaggctt 1020
ggaggggatg tgcagagccc caactgccca tctgaggatg tagtcatcac tccagaaagc 1080
tttggaagag attcatccct cacatgcctg gctgggaatg tcagtgcatg tgacgcccct 1140
attctctcct cttccaggtc cctagactgc agggagagtg gcaagaatgg gcctcatgtg 1200
taccaggacc tcctgcttag ccttgggact acaaacagca cgctgccccc tccattttct 1260
ctccaatctg gaatcctgac attgaaccca gttgctcagg gtcagcccat tcttacttcc 1320
ctgggatcaa atcaagaaga agcatatgtc accatgtcca gcttctacca aaaccag 1377
<210> 22
<211> 765
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 22
atgggaaaca gctgttacaa catagtagcc actctgttgc tggtcctcaa ctttgagagg 60
acaagatcat tgcaggatcc ttgtagtaac tgcccagctg gtacattctg tgataataac 120
aggaatcaga tttgcagtcc ctgtcctcca aatagtttct ccagcgcagg tggacaaagg 180
acctgtgaca tatgcaggca gtgtaaaggt gttttcagga ccaggaagga gtgttcctcc 240
accagcaatg cagagtgtga ctgcactcca gggtttcact gcctgggggc aggatgcagc 300
atgtgtgaac aggattgtaa acaaggtcaa gaactgacaa aaaaaggttg taaagactgt 360
tgctttggga catttaacga tcagaaacgt ggcatctgtc gaccctggac aaactgttct 420
ttggatggaa agtctgtgct tgtgaatggg acgaaggaga gggacgtggt ctgtggacca 480
tctccagccg acctctctcc gggagcatcc tctgtgaccc cgcctgcccc tgcgagagag 540
ccaggacact ctccgcagat catctccttc tttcttgcgc tgacgtcgac tgcgttgctc 600
ttcctgctgt tcttcctcac gctccgtttc tctgttgtta aacggggcag aaagaaactc 660
ctgtatatat tcaaacaacc atttatgaga ccagtacaaa ctactcaaga ggaagatggc 720
tgtagctgcc gatttccaga agaagaagaa ggaggatgtg aactg 765
<210> 23
<211> 591
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 23
atgctgggca tctggaccct cctacctctg gttcttacgt ctgttgctag attatcgtcc 60
aaaagtgtta atgcccaagt gactgacatc aactccaagg gattggaatt gaggaagact 120
gttactacag ttgagactca gaacttggaa ggcctgcatc atgatggcca attctgccat 180
aagccctgtc ctccaggtga aaggaaagct agggactgca cagtcaatgg ggatgaacca 240
gactgcgtgc cctgccaaga agggaaggag tacacagaca aagcccattt ttcttccaaa 300
tgcagaagat gtagattgtg tgatgaagga catggcttag aagtggaaat aaactgcacc 360
cggacccaga ataccaagtg cagatgtaaa ccaaactttt tttgtaactc tactgtatgt 420
gaacactgtg acccttgcac caaatgtgaa catggaatca tcaaggaatg cacactcacc 480
agcaacacca agtgcaaaga ggaaggatcc agatctaact tggggtggct ttgtcttctt 540
cttttgccaa ttccactaat tgtttgggtg aagagaaagg aagtacagaa a 591
<210> 24
<211> 714
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 24
atgctgggca tctggaccct cctacctctg gttcttacgt ctgttgctag attatcgtcc 60
aaaagtgtta atgcccaagt gactgacatc aactccaagg gattggaatt gaggaagact 120
gttactacag ttgagactca gaacttggaa ggcctgcatc atgatggcca attctgccat 180
aagccctgtc ctccaggtga aaggaaagct agggactgca cagtcaatgg ggatgaacca 240
gactgcgtgc cctgccaaga agggaaggag tacacagaca aagcccattt ttcttccaaa 300
tgcagaagat gtagattgtg tgatgaagga catggcttag aagtggaaat aaactgcacc 360
cggacccaga ataccaagtg cagatgtaaa ccaaactttt tttgtaactc tactgtatgt 420
gaacactgtg acccttgcac caaatgtgaa catggaatca tcaaggaatg cacactcacc 480
agcaacacca agtgcaaaga ggaaggatcc agatctaact tggggtggct ttgtcttctt 540
cttttgccaa ttccactaat tgtttgggtg aagagaaagg aagtacagaa aaggagtaag 600
aggagcaggc tcctgcacag tgactacatg aacatgactc ctcgccgccc tgggcctacc 660
cgcaagcatt accagcccta tgccccacca cgcgacttcg cagcctatcg ctcc 714
<210> 25
<211> 717
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 25
atgctgggca tctggaccct cctacctctg gttcttacgt ctgttgctag attatcgtcc 60
aaaagtgtta atgcccaagt gactgacatc aactccaagg gattggaatt gaggaagact 120
gttactacag ttgagactca gaacttggaa ggcctgcatc atgatggcca attctgccat 180
aagccctgtc ctccaggtga aaggaaagct agggactgca cagtcaatgg ggatgaacca 240
gactgcgtgc cctgccaaga agggaaggag tacacagaca aagcccattt ttcttccaaa 300
tgcagaagat gtagattgtg tgatgaagga catggcttag aagtggaaat aaactgcacc 360
cggacccaga ataccaagtg cagatgtaaa ccaaactttt tttgtaactc tactgtatgt 420
gaacactgtg acccttgcac caaatgtgaa catggaatca tcaaggaatg cacactcacc 480
agcaacacca agtgcaaaga ggaaggatcc agatctaact tggggtggct ttgtcttctt 540
cttttgccaa ttccactaat tgtttgggtg aagagaaagg aagtacagaa aaaacggggc 600
agaaagaaac tcctgtatat attcaaacaa ccatttatga gaccagtaca aactactcaa 660
gaggaagatg gctgtagctg ccgatttcca gaagaagaag aaggaggatg tgaactg 717
<210> 26
<211> 897
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 26
atgctgggca tctggaccct cctacctctg gttcttacgt ctgttgctag attatcgtcc 60
aaaagtgtta atgcccaagt gactgacatc aactccaagg gattggaatt gaggaagact 120
gttactacag ttgagactca gaacttggaa ggcctgcatc atgatggcca attctgccat 180
aagccctgtc ctccaggtga aaggaaagct agggactgca cagtcaatgg ggatgaacca 240
gactgcgtgc cctgccaaga agggaaggag tacacagaca aagcccattt ttcttccaaa 300
tgcagaagat gtagattgtg tgatgaagga catggcttag aagtggaaat aaactgcacc 360
cggacccaga ataccaagtg cagatgtaaa ccaaactttt tttgtaactc tactgtatgt 420
gaacactgtg acccttgcac caaatgtgaa catggaatca tcaaggaatg cacactcacc 480
agcaacacca agtgcaaaga ggaaggatcc agatctaact tggggtggct ttgtcttctt 540
cttttgccaa ttccactaat tgtttgggtg aagagaaagg aagtacagaa aacacaggtg 600
gcggccgact ggaccgcgct ggcggaggag atggactttg agtacttgga gatccggcaa 660
ctggagacac aagcggaccc cactggcagg ctgctggacg cctggcaggg acgccctggc 720
gcctctgtag gccgactgct cgagctgctt accaagctgg gccgcgacga cgtgctgctg 780
gagctgggac ccagcattga ggaggattgc caaaagtata tcttgaagca gcagcaggag 840
gaggctgaga agcctttaca ggtggccgct gtagacagca gtgtcccacg gacagca 897
<210> 27
<211> 705
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 27
atgctgggca tctggaccct cctacctctg gttcttacgt ctgttgctag attatcgtcc 60
aaaagtgtta atgcccaagt gactgacatc aactccaagg gattggaatt gaggaagact 120
gttactacag ttgagactca gaacttggaa ggcctgcatc atgatggcca attctgccat 180
aagccctgtc ctccaggtga aaggaaagct agggactgca cagtcaatgg ggatgaacca 240
gactgcgtgc cctgccaaga agggaaggag tacacagaca aagcccattt ttcttccaaa 300
tgcagaagat gtagattgtg tgatgaagga catggcttag aagtggaaat aaactgcacc 360
cggacccaga ataccaagtg cagatgtaaa ccaaactttt tttgtaactc tactgtatgt 420
gaacactgtg acccttgcac caaatgtgaa catggaatca tcaaggaatg cacactcacc 480
agcaacacca agtgcaaaga ggaaggatcc agatctaact tggggtggct ttgtcttctt 540
cttttgccaa ttccactaat tgtttgggtg aagagaaagg aagtacagaa atgttggctt 600
acaaaaaaga agtattcatc cagtgtgcac gaccctaacg gtgaatacat gttcatgaga 660
gcagtgaaca cagccaaaaa atctagactc acagatgtga cccta 705
<210> 28
<211> 714
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 28
atggaacaac ggggacagaa tgccccggcc gcttcggggg cccggaaaag acacggccca 60
ggacctagag aggcgcgggg agccaggcct gggccccggg tccccaagac ccttgtgctc 120
gttgtcgccg cggtcctgct gttggtctca gctgagtctg ctctgatcac ccaacaagac 180
ctagctcccc agcagagagc ggccccacaa caaaagaggt ccagcccctc agagggattg 240
tgtccacctg gacaccatat ctcagaagac ggtagagatt gcatctcctg caaatatgga 300
caggactata gcactcactg gaatgacctc cttttctgct tgcgctgcac caggtgtgat 360
tcaggtgaag tggagctaag tccctgcacc acgaccagaa acacagtgtg tcagtgcgaa 420
gaaggcacct tccgggaaga agattctcct gagatgtgcc ggaagtgccg cacagggtgt 480
cccagaggga tggtcaaggt cggtgattgt acaccctgga gtgacatcga atgtgtccac 540
aaagaatcag gtacaaagca cagtggggaa gtcccagctg tggaggagac ggtgacctcc 600
agcccaggga ctcctgcctc tccctgttct ctctcaggca tcatcatagg agtcacagtt 660
gcagccgtag tcttgattgt ggctgtgttt gtttgcaagt ctttactgtg gaag 714
<210> 29
<211> 837
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 29
atggaacaac ggggacagaa tgccccggcc gcttcggggg cccggaaaag acacggccca 60
ggacctagag aggcgcgggg agccaggcct gggccccggg tccccaagac ccttgtgctc 120
gttgtcgccg cggtcctgct gttggtctca gctgagtctg ctctgatcac ccaacaagac 180
ctagctcccc agcagagagc ggccccacaa caaaagaggt ccagcccctc agagggattg 240
tgtccacctg gacaccatat ctcagaagac ggtagagatt gcatctcctg caaatatgga 300
caggactata gcactcactg gaatgacctc cttttctgct tgcgctgcac caggtgtgat 360
tcaggtgaag tggagctaag tccctgcacc acgaccagaa acacagtgtg tcagtgcgaa 420
gaaggcacct tccgggaaga agattctcct gagatgtgcc ggaagtgccg cacagggtgt 480
cccagaggga tggtcaaggt cggtgattgt acaccctgga gtgacatcga atgtgtccac 540
aaagaatcag gtacaaagca cagtggggaa gtcccagctg tggaggagac ggtgacctcc 600
agcccaggga ctcctgcctc tccctgttct ctctcaggca tcatcatagg agtcacagtt 660
gcagccgtag tcttgattgt ggctgtgttt gtttgcaagt ctttactgtg gaagaggagt 720
aagaggagca ggctcctgca cagtgactac atgaacatga ctcctcgccg ccctgggccc 780
acccgcaagc attaccagcc ctatgcccca ccacgcgact tcgcagccta tcgctcc 837
<210> 30
<211> 1086
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 30
atggggacca agcccacaga gcaggtctcc tggggacttt actccgggta cgatgaggag 60
gcctattcgg ttgggccgct gccagagctc tgttacaagg ctgatgtcca ggctttcagt 120
cgggccttcc aacccagtgt ctccctgatg gtggctgtac tgggtctggc tggcaatggc 180
ctagtcttgg ccacccatct ggctgctagg cgaactaccc gatctcccac ctccgttcac 240
ctgctccagt tggccctggc tgacctttta ttggccctga ctttgccttt tgctgcagca 300
ggggctcttc agggctggaa tctaggaagt accacctgcc gtgccatctc aggcctctac 360
tcggcctctt tccacgctgg cttcctcttc ctagcctgta tcagcgccga ccgctatgtg 420
gccatcgcac gagctctccc agccgggcag cggccctcaa cgcctagccg agcgcacttg 480
gtttcagtct tcgtgtggct gttgtcgctg tttctggctc tacctgcgct ccttttcagc 540
cgggacgggc cacgtgaagg ccaacgacgc tgtcggctca tttttcccga aagcctcacg 600
cagactgtga aaggagcaag cgcagtggcg caggtggtcc tcggcttcgc gctccctctg 660
ggcgtcatgg cagcctgtta tgcgcttctg ggccgaacgc ttctggctgc cagagggcca 720
gagcggcggc gtgcactgcg cgtcgtggtg gctttggtgg tggccttcgt ggtgctgcag 780
ttgccctaca gccttgccct gctgctggat acagccgatc tactggcagc ccgcgagcgg 840
agctgctcct ccagcaagcg caaggatcta gctttgctgg tcaccggcgg cttgaccctg 900
gtccgttgca gcctcaatcc ggtgctttat gcctttttgg gcctgcgttt ccgacgggat 960
ctgcggagac tgctccaggg cggaggatgc agcccgaagc ctaaccctcg tggccgctgc 1020
cctcgtcgac tccgcctttc ttcctgttct gctcctactg agacccacag tctctcttgg 1080
gacaac 1086
<210> 31
<211> 1395
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 31
atgggaggag ccgtggtgga tgagggacct acaggagtca aggcccctga tggaggatgg 60
ggatgggctg tgctcttcgg ctgtttcgtc atcactggct tctcctacgc cttccccaag 120
gccgtcagtg tcttcttcaa ggagctcata caggagtttg ggatcggcta cagcgacaca 180
gcctggatct cctccatcct gctggccatg ctctacggga caggtccgct ctgcagtgtg 240
tgcgtgaacc gctttggctg ccggcccgtc atgcttgtgg gaggtctctt tgcgtcgctg 300
ggcatggtgg ctgcgtcctt ttgccggagc atcatccagg tctacctcac cactggggtc 360
atcacggggt tgggtttggc actcaacttc cagccctcgc tcatcatgct gaaccgctac 420
ttcagcaagc ggcgccctat ggccaacgga ctggcggcag caggtagtcc tgtcttcctg 480
tgtgccctga gtccgctggg acagctgctg caggatcgct acggctggcg gggcggcttc 540
ctcatcctgg gcggcctgct gcttaactgt tgtgtgtgtg ccgcactcat gaggcctctg 600
gtggtcacgg cccagccggg ctcgggaccg ccgcgacctt cccgacgact gctagatctg 660
agtgtcttcc gagatcgcgg ctttgtgctt tacgccgtgg ccgcctcggt catggtgctg 720
ggactcttcg tcccgcccgt gttcgtggtg agctacgcca aggacctggg cgtgcccgac 780
accaaggccg ccttcctgct caccatcctg ggcttcattg acatcttcgc gcgaccggcc 840
gcaggcttcg tggcgggact tggaaaggtg cggccttact ccgtctacct cttcagcttc 900
tccatgttct tcaacggcct cgcggacctg gcgggttcta cggcgggcga ctacggcggc 960
ctcgtggtct tctgcatctt ctttggcatc tcctacggca tggtgggagc cctgcagttc 1020
gaggtgctca tggccatcgt gggcacccac aagttctcca gtgccattgg cctggtgctg 1080
ctgatggagg cggtggccgt gctcgtcggg cctccttcgg gaggcaaact cctggatgcg 1140
acccacgtct acatgtacgt gttcatcctg gcgggagccg aggtgctcac ctcctccctg 1200
attttgctgc tgggcaactt cttctgcatt aggaagaagc ccaaagagcc acagcctgag 1260
gtggcggccg cggaggagga gaagctccac aagcctcctg cagactcggg ggtggacttg 1320
cgggaggtgg agcatttcct gaaggctgag cctgagaaaa acggggaggt ggttcacacc 1380
ccggaaacaa gtgtc 1395
<210> 32
<211> 462
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 32
atggcgacga aggccgtgtg cgtgctgaag ggcgacggcc cagtgcaggg catcatcaat 60
ttcgagcaga aggaaagtaa tggaccagtg aaggtgtggg gaagcattaa aggactgact 120
gaaggcctgc atggattcca tgttcatgag tttggagata atacagcagg ctgtaccagt 180
gcaggtcctc actttaatcc tctatccaga aaacacggtg ggccaaagga tgaagagagg 240
catgttggag acttgggcaa tgtgactgct gacaaagatg gtgtggccga tgtgtctatt 300
gaagattctg tgatctcact ctcaggagac cattgcatca ttggccgcac actggtggtc 360
catgaaaaag cagatgactt gggcaaaggt ggaaatgaag aaagtacaaa gacaggaaac 420
gctggaagtc gtttggcttg tggtgtaatt gggatcgccc aa 462
<210> 33
<211> 1152
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 33
atgccgcagc tggattctgg cggaggagga gcgggcggag gagatgatct cggcgcgccg 60
gatgagctgc tggccttcca ggatgaaggc gaggagcagg atgacaagag ccgagatagc 120
gccgccggtc ctgagcgcga cctggccgag ctcaagtcgt cgcttgtgaa cgaatccgag 180
ggagcagccg gaggagcagg aatcccggga gttccgggag ccggcgccgg agcccgaggc 240
gaggccgaag ctcttggacg agaacacgct gcgcagagac tcttcccgga caaacttcca 300
gagcccctgg aggacggcct gaaggccccg gagtgcacca gcggcatgta caaagagacc 360
gtctactccg ccttcaatct gctcatgcat tacccacctc cttcgggagc aggacagcac 420
cctcagccgc agcctccgct gcacaaggcc aatcagcctc ctcacggtgt ccctcaactc 480
tctctctacg aacatttcaa cagcccacat cccacccctg cacctgcgga catcagccag 540
aagcaagttc acaggcctct gcagacccct gacctctctg gcttctactc cctgacctca 600
ggcagcatgg ggcagctccc ccacactgtg agctggttca cccacccatc cttgatgcta 660
ggttctggtg tacctggtca cccagcagcc atccctcacc cggccattgt gcctccttca 720
gggaagcagg agctgcagcc tttcgaccgc aacctgaaga cacaagcaga gtccaaggca 780
gagaaggagg ccaagaagcc aaccatcaag aagcccctca atgccttcat gctgtacatg 840
aaggagatga gagccaaggt cattgcagag tgcacactta aggagagcgc tgccatcaac 900
cagatcctgg gccgcaggtg gcacgcgctg tcgcgagaag agcaggccaa gtactatgag 960
ctggcccgca aggagaggca gctgcacatg cagctatacc caggctggtc agcgcgggac 1020
aactacggga agaagaagag gcggtcgagg gaaaagcacc aagaatccac cacaggagga 1080
aaaagaaatg cattcggtac ttacccggag aaggccgctg ccccagcccc gttccttccg 1140
atgacagtgc tc 1152
<210> 34
<211> 831
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 34
atgggggcag gtgccactgg ccgcgccatg gacgggccgc gcctgctgct gttgctgctt 60
ctgggggtgt cccttggagg tgccaaggag gcatgcccca caggcctgta cacacacagc 120
ggtgagtgct gcaaagcctg caacctgggc gagggtgtgg cccagccttg tggagccaac 180
cagaccgtgt gtgagccctg cctggacagc gtgacgttct ccgacgtggt gagcgcgacc 240
gagccgtgca agccgtgcac cgagtgcgtg gggctccaga gcatgtcggc gccatgcgtg 300
gaggccgacg acgccgtgtg ccgctgcgcc tacggctact accaggatga gacgactggg 360
cgctgcgagg cgtgccgcgt gtgcgaggcg ggctcgggcc tcgtgttctc ctgccaggac 420
aagcagaaca ccgtgtgcga ggagtgcccc gacggcacgt attccgacga ggccaaccac 480
gtggacccgt gcctgccctg caccgtgtgc gaggacaccg agcgccagct ccgcgagtgc 540
acacgctggg ccgacgccga gtgcgaggag atccctggcc gttggattac acggtccaca 600
cccccagagg gctcggacag cacagccccc agcacccagg agcctgaggc acctccagaa 660
caagacctca tagccagcac ggtggcaggt gtggtgacca cagtgatggg cagctcccag 720
cccgtggtga cccgaggcac caccgacaac ctcatccctg tctattgctc catcctggct 780
gctgtggttg tgggtcttgt ggcctacata gccttcaaga ggtggaacag c 831
<210> 35
<211> 729
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 35
ggatcgggtg ggactagtgg cagcaagggc gaggagctgt tcaccggggt ggtgcccatc 60
ctggtcgagc tggacggcga cgtaaacggc cacaagttca gcgtgcgcgg cgagggcgag 120
ggcgatgcca ccaacggcaa gctgaccctg aagttcatct gcaccaccgg caagctgccc 180
gtgccctggc ccaccctcgt gaccaccctg acctacggcg tgcagtgctt cagccgctac 240
cccgaccaca tgaagcgcca cgacttcttc aagtccgcca tgcccgaagg ctacgtccag 300
gagcgcacca tcagcttcaa ggacgacggc acctacaaga cccgcgccga ggtgaagttc 360
gagggcgaca ccctggtgaa ccgcatcgag ctgaagggca tcgacttcaa ggaggacggc 420
aacatcctgg ggcacaagct ggagtacaac ttcaacagcc acaacgtcta tatcaccgcc 480
gacaagcaga agaacggcat caaggccaac ttcaagatcc gccacaacgt ggaggacggc 540
agcgtgcagc tcgccgacca ctaccagcag aacaccccca tcggcgacgg ccccgtgctg 600
ctgcccgaca accactacct gagcacccag tccgtgctga gcaaagaccc caacgagaag 660
cgcgatcaca tggtcctgct ggagttcgtg accgccgccg ggatcactgg aaccggtgct 720
ggaagtggt 729
<210> 36
<211> 747
<212> DNA
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 36
ggatcgggtg ggactagtgg cgtgagcaag ggcgaggagg ataacatggc catcatcaag 60
gagttcatgc gcttcaaggt gcacatggag ggctccgtga acggccacga gttcgagatc 120
gagggcgagg gcgagggccg cccctacgag ggcacccaga ccgccaagct gaaggtgacc 180
aagggtggcc ccctgccctt cgcctgggac atcctgtccc ctcagttcat gtacggctcc 240
aaggcctacg tgaagcaccc cgccgacatc cccgactact tgaagctgtc cttccccgag 300
ggcttcaagt gggagcgcgt gatgaacttc gaggacggcg gcgtggtgac cgtgacccag 360
gactcctccc tgcaggacgg cgagttcatc tacaaggtga agctgcgcgg caccaacttc 420
ccctccgacg gccccgtaat gcagaagaag accatgggct gggaggcctc ctccgagcgg 480
atgtaccccg aggacggcgc cctgaagggc gagatcaagc agaggctgaa gctgaaggac 540
ggcggccact acgacgctga ggtcaagacc acctacaagg ccaagaagcc cgtgcagctg 600
cccggcgcct acaacgtcaa catcaagttg gacatcacct cccacaacga ggactacacc 660
atcgtggaac agtacgaacg cgccgagggc cgccactcca ccggcggcat ggacgagctg 720
tacaagggaa ccggtgctgg aagtggt 747
<210> 37
<211> 201
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 37
Met Gln Ile Pro Gln Ala Pro Trp Pro Val Val Trp Ala Val Leu Gln
1 5 10 15
Leu Gly Trp Arg Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp
20 25 30
Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp
35 40 45
Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val
50 55 60
Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala
65 70 75 80
Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg
85 90 95
Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg
100 105 110
Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu
115 120 125
Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val
130 135 140
Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro
145 150 155 160
Arg Pro Ala Gly Gln Phe Gln Thr Leu Val Val Gly Val Val Gly Gly
165 170 175
Leu Leu Gly Ser Leu Val Leu Leu Val Trp Val Leu Ala Val Ile Cys
180 185 190
Ser Arg Ala Ala Arg Gly Thr Ile Gly
195 200
<210> 38
<211> 236
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 38
Met Gln Ile Pro Gln Ala Pro Trp Pro Val Val Trp Ala Val Leu Gln
1 5 10 15
Leu Gly Trp Arg Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp
20 25 30
Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp
35 40 45
Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val
50 55 60
Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala
65 70 75 80
Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg
85 90 95
Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg
100 105 110
Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu
115 120 125
Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val
130 135 140
Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His Pro Ala Pro Ala Arg
145 150 155 160
Glu Pro Gly His Ser Pro Gln Ile Ile Ser Phe Phe Leu Ala Leu Thr
165 170 175
Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu Arg Phe Ser
180 185 190
Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
195 200 205
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
210 215 220
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
225 230 235
<210> 39
<211> 300
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 39
Met Gln Ile Pro Gln Ala Pro Trp Pro Val Val Trp Ala Val Leu Gln
1 5 10 15
Leu Gly Trp Arg Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp
20 25 30
Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp
35 40 45
Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val
50 55 60
Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala
65 70 75 80
Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg
85 90 95
Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg
100 105 110
Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu
115 120 125
Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val
130 135 140
Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro
145 150 155 160
Arg Pro Ala Gly Gln Phe Gln Thr Leu Val Val Gly Val Val Gly Gly
165 170 175
Leu Leu Gly Ser Leu Val Leu Leu Val Trp Val Leu Ala Val Ile Cys
180 185 190
Ser Arg Ala Ala Arg Gly Thr Gln Val Ala Ala Asp Trp Thr Ala Leu
195 200 205
Ala Glu Glu Met Asp Phe Glu Tyr Leu Glu Ile Arg Gln Leu Glu Thr
210 215 220
Gln Ala Asp Pro Thr Gly Arg Leu Leu Asp Ala Trp Gln Gly Arg Pro
225 230 235 240
Gly Ala Ser Val Gly Arg Leu Leu Glu Leu Leu Thr Lys Leu Gly Arg
245 250 255
Asp Asp Val Leu Leu Glu Leu Gly Pro Ser Ile Glu Glu Asp Cys Gln
260 265 270
Lys Tyr Ile Leu Lys Gln Gln Gln Glu Glu Ala Glu Lys Pro Leu Gln
275 280 285
Val Ala Ala Val Asp Ser Ser Val Pro Arg Thr Ala
290 295 300
<210> 40
<211> 226
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 40
Met Gln Ile Pro Gln Ala Pro Trp Pro Val Val Trp Ala Val Leu Gln
1 5 10 15
Leu Gly Trp Arg Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp
20 25 30
Asn Pro Pro Thr Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp
35 40 45
Asn Ala Thr Phe Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val
50 55 60
Leu Asn Trp Tyr Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala
65 70 75 80
Ala Phe Pro Glu Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg
85 90 95
Val Thr Gln Leu Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg
100 105 110
Ala Arg Arg Asn Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu
115 120 125
Ala Pro Lys Ala Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val
130 135 140
Thr Glu Arg Arg Ala Glu Val Pro Thr Ala His His Ile Tyr Glu Ser
145 150 155 160
Gln Leu Cys Cys Gln Leu Lys Phe Trp Leu Pro Ile Gly Cys Ala Ala
165 170 175
Phe Val Val Val Cys Ile Leu Gly Cys Ile Leu Ile Cys Trp Leu Thr
180 185 190
Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro Asn Gly Glu Tyr Met
195 200 205
Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser Arg Leu Thr Asp Val
210 215 220
Thr Leu
225
<210> 41
<211> 189
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 41
Met Ala Cys Leu Gly Phe Gln Arg His Lys Ala Gln Leu Asn Leu Ala
1 5 10 15
Thr Arg Thr Trp Pro Cys Thr Leu Leu Phe Phe Leu Leu Phe Ile Pro
20 25 30
Val Phe Cys Lys Ala Met His Val Ala Gln Pro Ala Val Val Leu Ala
35 40 45
Ser Ser Arg Gly Ile Ala Ser Phe Val Cys Glu Tyr Ala Ser Pro Gly
50 55 60
Lys Ala Thr Glu Val Arg Val Thr Val Leu Arg Gln Ala Asp Ser Gln
65 70 75 80
Val Thr Glu Val Cys Ala Ala Thr Tyr Met Met Gly Asn Glu Leu Thr
85 90 95
Phe Leu Asp Asp Ser Ile Cys Thr Gly Thr Ser Ser Gly Asn Gln Val
100 105 110
Asn Leu Thr Ile Gln Gly Leu Arg Ala Met Asp Thr Gly Leu Tyr Ile
115 120 125
Cys Lys Val Glu Leu Met Tyr Pro Pro Pro Tyr Tyr Leu Gly Ile Gly
130 135 140
Asn Gly Thr Gln Ile Tyr Val Ile Asp Pro Glu Pro Cys Pro Asp Ser
145 150 155 160
Asp Phe Leu Leu Trp Ile Leu Ala Ala Val Ser Ser Gly Leu Phe Phe
165 170 175
Tyr Ser Phe Leu Leu Thr Ala Val Ser Leu Ser Lys Met
180 185
<210> 42
<211> 230
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 42
Met Ala Cys Leu Gly Phe Gln Arg His Lys Ala Gln Leu Asn Leu Ala
1 5 10 15
Thr Arg Thr Trp Pro Cys Thr Leu Leu Phe Phe Leu Leu Phe Ile Pro
20 25 30
Val Phe Cys Lys Ala Met His Val Ala Gln Pro Ala Val Val Leu Ala
35 40 45
Ser Ser Arg Gly Ile Ala Ser Phe Val Cys Glu Tyr Ala Ser Pro Gly
50 55 60
Lys Ala Thr Glu Val Arg Val Thr Val Leu Arg Gln Ala Asp Ser Gln
65 70 75 80
Val Thr Glu Val Cys Ala Ala Thr Tyr Met Met Gly Asn Glu Leu Thr
85 90 95
Phe Leu Asp Asp Ser Ile Cys Thr Gly Thr Ser Ser Gly Asn Gln Val
100 105 110
Asn Leu Thr Ile Gln Gly Leu Arg Ala Met Asp Thr Gly Leu Tyr Ile
115 120 125
Cys Lys Val Glu Leu Met Tyr Pro Pro Pro Tyr Tyr Leu Gly Ile Gly
130 135 140
Asn Gly Thr Gln Ile Tyr Val Ile Asp Pro Glu Pro Cys Pro Asp Ser
145 150 155 160
Asp Phe Leu Leu Trp Ile Leu Ala Ala Val Ser Ser Gly Leu Phe Phe
165 170 175
Tyr Ser Phe Leu Leu Thr Ala Val Ser Leu Ser Lys Met Arg Ser Lys
180 185 190
Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg
195 200 205
Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp
210 215 220
Phe Ala Ala Tyr Arg Ser
225 230
<210> 43
<211> 271
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 43
Met Leu Cys Pro Trp Arg Thr Ala Asn Leu Gly Leu Leu Leu Ile Leu
1 5 10 15
Thr Ile Phe Leu Val Ala Glu Ala Glu Gly Ala Ala Gln Pro Asn Asn
20 25 30
Ser Leu Met Leu Gln Thr Ser Lys Glu Asn His Ala Leu Ala Ser Ser
35 40 45
Ser Leu Cys Met Asp Glu Lys Gln Ile Thr Gln Asn Tyr Ser Lys Val
50 55 60
Leu Ala Glu Val Asn Thr Ser Trp Pro Val Lys Met Ala Thr Asn Ala
65 70 75 80
Val Leu Cys Cys Pro Pro Ile Ala Leu Arg Asn Leu Ile Ile Ile Thr
85 90 95
Trp Glu Ile Ile Leu Arg Gly Gln Pro Ser Cys Thr Lys Ala Tyr Lys
100 105 110
Lys Glu Thr Asn Glu Thr Lys Glu Thr Asn Cys Thr Asp Glu Arg Ile
115 120 125
Thr Trp Val Ser Arg Pro Asp Gln Asn Ser Asp Leu Gln Ile Arg Thr
130 135 140
Val Ala Ile Thr His Asp Gly Tyr Tyr Arg Cys Ile Met Val Thr Pro
145 150 155 160
Asp Gly Asn Phe His Arg Gly Tyr His Leu Gln Val Leu Val Thr Pro
165 170 175
Glu Val Thr Leu Phe Gln Asn Arg Asn Arg Thr Ala Val Cys Lys Ala
180 185 190
Val Ala Gly Lys Pro Ala Ala His Ile Ser Trp Ile Pro Glu Gly Asp
195 200 205
Cys Ala Thr Lys Gln Glu Tyr Trp Ser Asn Gly Thr Val Thr Val Lys
210 215 220
Ser Thr Cys His Trp Glu Val His Asn Val Ser Thr Val Thr Cys His
225 230 235 240
Val Ser His Leu Thr Gly Asn Lys Ser Leu Tyr Ile Glu Leu Leu Pro
245 250 255
Val Pro Gly Ala Lys Lys Ser Ala Lys Leu Tyr Ile Pro Tyr Ile
260 265 270
<210> 44
<211> 185
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 44
Met Lys Thr Leu Pro Ala Met Leu Gly Thr Gly Lys Leu Phe Trp Val
1 5 10 15
Phe Phe Leu Ile Pro Tyr Leu Asp Ile Trp Asn Ile His Gly Lys Glu
20 25 30
Ser Cys Asp Val Gln Leu Tyr Ile Lys Arg Gln Ser Glu His Ser Ile
35 40 45
Leu Ala Gly Asp Pro Phe Glu Leu Glu Cys Pro Val Lys Tyr Cys Ala
50 55 60
Asn Arg Pro His Val Thr Trp Cys Lys Leu Asn Gly Thr Thr Cys Val
65 70 75 80
Lys Leu Glu Asp Arg Gln Thr Ser Trp Lys Glu Glu Lys Asn Ile Ser
85 90 95
Phe Phe Ile Leu His Phe Glu Pro Val Leu Pro Asn Asp Asn Gly Ser
100 105 110
Tyr Arg Cys Ser Ala Asn Phe Gln Ser Asn Leu Ile Glu Ser His Ser
115 120 125
Thr Thr Leu Tyr Val Thr Asp Val Lys Ser Ala Ser Glu Arg Pro Ser
130 135 140
Lys Asp Glu Met Ala Ser Arg Pro Trp Leu Leu Tyr Arg Leu Leu Pro
145 150 155 160
Leu Gly Gly Leu Pro Leu Leu Ile Thr Thr Cys Phe Cys Leu Phe Cys
165 170 175
Cys Leu Arg Arg His Gln Gly Lys Gln
180 185
<210> 45
<211> 228
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 45
Met Lys Thr Leu Pro Ala Met Leu Gly Thr Gly Lys Leu Phe Trp Val
1 5 10 15
Phe Phe Leu Ile Pro Tyr Leu Asp Ile Trp Asn Ile His Gly Lys Glu
20 25 30
Ser Cys Asp Val Gln Leu Tyr Ile Lys Arg Gln Ser Glu His Ser Ile
35 40 45
Leu Ala Gly Asp Pro Phe Glu Leu Glu Cys Pro Val Lys Tyr Cys Ala
50 55 60
Asn Arg Pro His Val Thr Trp Cys Lys Leu Asn Gly Thr Thr Cys Val
65 70 75 80
Lys Leu Glu Asp Arg Gln Thr Ser Trp Lys Glu Glu Lys Asn Ile Ser
85 90 95
Phe Phe Ile Leu His Phe Glu Pro Val Leu Pro Asn Asp Asn Gly Ser
100 105 110
Tyr Arg Cys Ser Ala Asn Phe Gln Ser Asn Leu Ile Glu Ser His Ser
115 120 125
Thr Thr Leu Tyr Val Thr Asp Val Lys Ser Ala Ser Glu Arg Pro Ser
130 135 140
Lys Asp Glu Met Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser Lys Pro
145 150 155 160
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
165 170 175
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
180 185 190
Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
195 200 205
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
210 215 220
Ala Tyr Arg Ser
225
<210> 46
<211> 230
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 46
Met Phe Ser His Leu Pro Phe Asp Cys Val Leu Leu Leu Leu Leu Leu
1 5 10 15
Leu Leu Thr Arg Ser Ser Glu Val Glu Tyr Arg Ala Glu Val Gly Gln
20 25 30
Asn Ala Tyr Leu Pro Cys Phe Tyr Thr Pro Ala Ala Pro Gly Asn Leu
35 40 45
Val Pro Val Cys Trp Gly Lys Gly Ala Cys Pro Val Phe Glu Cys Gly
50 55 60
Asn Val Val Leu Arg Thr Asp Glu Arg Asp Val Asn Tyr Trp Thr Ser
65 70 75 80
Arg Tyr Trp Leu Asn Gly Asp Phe Arg Lys Gly Asp Val Ser Leu Thr
85 90 95
Ile Glu Asn Val Thr Leu Ala Asp Ser Gly Ile Tyr Cys Cys Arg Ile
100 105 110
Gln Ile Pro Gly Ile Met Asn Asp Glu Lys Phe Asn Leu Lys Leu Val
115 120 125
Ile Lys Pro Ala Lys Val Thr Pro Ala Pro Thr Arg Gln Arg Asp Phe
130 135 140
Thr Ala Ala Phe Pro Arg Met Leu Thr Thr Arg Gly His Gly Pro Ala
145 150 155 160
Glu Thr Gln Thr Leu Gly Ser Leu Pro Asp Ile Asn Leu Thr Gln Ile
165 170 175
Ser Thr Leu Ala Asn Glu Leu Arg Asp Ser Arg Leu Ala Asn Asp Leu
180 185 190
Arg Asp Ser Gly Ala Thr Ile Arg Ile Gly Ile Tyr Ile Gly Ala Gly
195 200 205
Ile Cys Ala Gly Leu Ala Leu Ala Leu Ile Phe Gly Ala Leu Ile Phe
210 215 220
Lys Trp Tyr Ser His Ser
225 230
<210> 47
<211> 273
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 47
Met Phe Ser His Leu Pro Phe Asp Cys Val Leu Leu Leu Leu Leu Leu
1 5 10 15
Leu Leu Thr Arg Ser Ser Glu Val Glu Tyr Arg Ala Glu Val Gly Gln
20 25 30
Asn Ala Tyr Leu Pro Cys Phe Tyr Thr Pro Ala Ala Pro Gly Asn Leu
35 40 45
Val Pro Val Cys Trp Gly Lys Gly Ala Cys Pro Val Phe Glu Cys Gly
50 55 60
Asn Val Val Leu Arg Thr Asp Glu Arg Asp Val Asn Tyr Trp Thr Ser
65 70 75 80
Arg Tyr Trp Leu Asn Gly Asp Phe Arg Lys Gly Asp Val Ser Leu Thr
85 90 95
Ile Glu Asn Val Thr Leu Ala Asp Ser Gly Ile Tyr Cys Cys Arg Ile
100 105 110
Gln Ile Pro Gly Ile Met Asn Asp Glu Lys Phe Asn Leu Lys Leu Val
115 120 125
Ile Lys Pro Ala Lys Val Thr Pro Ala Pro Thr Arg Gln Arg Asp Phe
130 135 140
Thr Ala Ala Phe Pro Arg Met Leu Thr Thr Arg Gly His Gly Pro Ala
145 150 155 160
Glu Thr Gln Thr Leu Gly Ser Leu Pro Asp Ile Asn Leu Thr Gln Ile
165 170 175
Ser Thr Leu Ala Asn Glu Leu Arg Asp Ser Arg Leu Ala Asn Asp Leu
180 185 190
Arg Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser Lys Pro Phe Trp Val
195 200 205
Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu Leu Val Thr
210 215 220
Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser Arg Leu Leu
225 230 235 240
His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg
245 250 255
Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg
260 265 270
Ser
<210> 48
<211> 169
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 48
Met Arg Trp Cys Leu Leu Leu Ile Trp Ala Gln Gly Leu Arg Gln Ala
1 5 10 15
Pro Leu Ala Ser Gly Met Met Thr Gly Thr Ile Glu Thr Thr Gly Asn
20 25 30
Ile Ser Ala Glu Lys Gly Gly Ser Ile Ile Leu Gln Cys His Leu Ser
35 40 45
Ser Thr Thr Ala Gln Val Thr Gln Val Asn Trp Glu Gln Gln Asp Gln
50 55 60
Leu Leu Ala Ile Cys Asn Ala Asp Leu Gly Trp His Ile Ser Pro Ser
65 70 75 80
Phe Lys Asp Arg Val Ala Pro Gly Pro Gly Leu Gly Leu Thr Leu Gln
85 90 95
Ser Leu Thr Val Asn Asp Thr Gly Glu Tyr Phe Cys Ile Tyr His Thr
100 105 110
Tyr Pro Asp Gly Thr Tyr Thr Gly Arg Ile Phe Leu Glu Val Leu Glu
115 120 125
Ser Ser Val Ala Glu His Gly Ala Arg Phe Gln Ile Pro Leu Leu Gly
130 135 140
Ala Met Ala Ala Thr Leu Val Val Ile Cys Thr Ala Val Ile Val Val
145 150 155 160
Val Ala Leu Thr Arg Lys Lys Lys Ala
165
<210> 49
<211> 212
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 49
Met Arg Trp Cys Leu Leu Leu Ile Trp Ala Gln Gly Leu Arg Gln Ala
1 5 10 15
Pro Leu Ala Ser Gly Met Met Thr Gly Thr Ile Glu Thr Thr Gly Asn
20 25 30
Ile Ser Ala Glu Lys Gly Gly Ser Ile Ile Leu Gln Cys His Leu Ser
35 40 45
Ser Thr Thr Ala Gln Val Thr Gln Val Asn Trp Glu Gln Gln Asp Gln
50 55 60
Leu Leu Ala Ile Cys Asn Ala Asp Leu Gly Trp His Ile Ser Pro Ser
65 70 75 80
Phe Lys Asp Arg Val Ala Pro Gly Pro Gly Leu Gly Leu Thr Leu Gln
85 90 95
Ser Leu Thr Val Asn Asp Thr Gly Glu Tyr Phe Cys Ile Tyr His Thr
100 105 110
Tyr Pro Asp Gly Thr Tyr Thr Gly Arg Ile Phe Leu Glu Val Leu Glu
115 120 125
Ser Ser Val Ala Cys Pro Ser Pro Leu Phe Pro Gly Pro Ser Lys Pro
130 135 140
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
145 150 155 160
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val Arg Ser Lys Arg Ser
165 170 175
Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg Arg Pro Gly
180 185 190
Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala
195 200 205
Ala Tyr Arg Ser
210
<210> 50
<211> 201
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 50
Met Gly Arg Gly Leu Leu Arg Gly Leu Trp Pro Leu His Ile Val Leu
1 5 10 15
Trp Thr Arg Ile Ala Ser Thr Ile Pro Pro His Val Gln Lys Ser Val
20 25 30
Asn Asn Asp Met Ile Val Thr Asp Asn Asn Gly Ala Val Lys Phe Pro
35 40 45
Gln Leu Cys Lys Phe Cys Asp Val Arg Phe Ser Thr Cys Asp Asn Gln
50 55 60
Lys Ser Cys Met Ser Asn Cys Ser Ile Thr Ser Ile Cys Glu Lys Pro
65 70 75 80
Gln Glu Val Cys Val Ala Val Trp Arg Lys Asn Asp Glu Asn Ile Thr
85 90 95
Leu Glu Thr Val Cys His Asp Pro Lys Leu Pro Tyr His Asp Phe Ile
100 105 110
Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile Met Lys Glu Lys Lys Lys
115 120 125
Pro Gly Glu Thr Phe Phe Met Cys Ser Cys Ser Ser Asp Glu Cys Asn
130 135 140
Asp Asn Ile Ile Phe Ser Glu Glu Tyr Asn Thr Ser Asn Pro Asp Leu
145 150 155 160
Leu Leu Val Ile Phe Gln Val Thr Gly Ile Ser Leu Leu Pro Pro Leu
165 170 175
Gly Val Ala Ile Ser Val Ile Ile Ile Phe Tyr Cys Tyr Arg Val Asn
180 185 190
Arg Gln Gln Lys Leu Ser Ser Ser Gly
195 200
<210> 51
<211> 236
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 51
Met Gly Arg Gly Leu Leu Arg Gly Leu Trp Pro Leu His Ile Val Leu
1 5 10 15
Trp Thr Arg Ile Ala Ser Thr Ile Pro Pro His Val Gln Lys Ser Val
20 25 30
Asn Asn Asp Met Ile Val Thr Asp Asn Asn Gly Ala Val Lys Phe Pro
35 40 45
Gln Leu Cys Lys Phe Cys Asp Val Arg Phe Ser Thr Cys Asp Asn Gln
50 55 60
Lys Ser Cys Met Ser Asn Cys Ser Ile Thr Ser Ile Cys Glu Lys Pro
65 70 75 80
Gln Glu Val Cys Val Ala Val Trp Arg Lys Asn Asp Glu Asn Ile Thr
85 90 95
Leu Glu Thr Val Cys His Asp Pro Lys Leu Pro Tyr His Asp Phe Ile
100 105 110
Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile Met Lys Glu Lys Lys Lys
115 120 125
Pro Gly Glu Thr Phe Phe Met Cys Ser Cys Ser Ser Asp Glu Cys Asn
130 135 140
Asp Asn Ile Ile Phe Ser Glu Glu Tyr Asn Thr Ser Asn Pro Asp Leu
145 150 155 160
Leu Leu Val Ile Phe Gln Val Thr Gly Ile Ser Leu Leu Pro Pro Leu
165 170 175
Gly Val Ala Ile Ser Val Ile Ile Ile Phe Tyr Cys Tyr Arg Val Asn
180 185 190
Arg Gln Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro
195 200 205
Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys
210 215 220
Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
225 230 235
<210> 52
<211> 296
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 52
Met Gly Arg Gly Leu Leu Arg Gly Leu Trp Pro Leu His Ile Val Leu
1 5 10 15
Trp Thr Arg Ile Ala Ser Thr Ile Pro Pro His Val Gln Lys Ser Val
20 25 30
Asn Asn Asp Met Ile Val Thr Asp Asn Asn Gly Ala Val Lys Phe Pro
35 40 45
Gln Leu Cys Lys Phe Cys Asp Val Arg Phe Ser Thr Cys Asp Asn Gln
50 55 60
Lys Ser Cys Met Ser Asn Cys Ser Ile Thr Ser Ile Cys Glu Lys Pro
65 70 75 80
Gln Glu Val Cys Val Ala Val Trp Arg Lys Asn Asp Glu Asn Ile Thr
85 90 95
Leu Glu Thr Val Cys His Asp Pro Lys Leu Pro Tyr His Asp Phe Ile
100 105 110
Leu Glu Asp Ala Ala Ser Pro Lys Cys Ile Met Lys Glu Lys Lys Lys
115 120 125
Pro Gly Glu Thr Phe Phe Met Cys Ser Cys Ser Ser Asp Glu Cys Asn
130 135 140
Asp Asn Ile Ile Phe Ser Glu Glu Tyr Asn Thr Ser Asn Pro Asp Leu
145 150 155 160
Leu Leu Val Ile Phe Gln Val Thr Gly Ile Ser Leu Leu Pro Pro Leu
165 170 175
Gly Val Ala Ile Ser Val Ile Ile Ile Phe Tyr Cys Tyr Arg Val Asn
180 185 190
Arg Gln Thr Gln Val Ala Ala Asp Trp Thr Ala Leu Ala Glu Glu Met
195 200 205
Asp Phe Glu Tyr Leu Glu Ile Arg Gln Leu Glu Thr Gln Ala Asp Pro
210 215 220
Thr Gly Arg Leu Leu Asp Ala Trp Gln Gly Arg Pro Gly Ala Ser Val
225 230 235 240
Gly Arg Leu Leu Glu Leu Leu Thr Lys Leu Gly Arg Asp Asp Val Leu
245 250 255
Leu Glu Leu Gly Pro Ser Ile Glu Glu Asp Cys Gln Lys Tyr Ile Leu
260 265 270
Lys Gln Gln Gln Glu Glu Ala Glu Lys Pro Leu Gln Val Ala Ala Val
275 280 285
Asp Ser Ser Val Pro Arg Thr Ala
290 295
<210> 53
<211> 263
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 53
Met Leu Pro Cys Leu Val Val Leu Leu Ala Ala Leu Leu Ser Leu Arg
1 5 10 15
Leu Gly Ser Asp Ala His Gly Thr Glu Leu Pro Ser Pro Pro Ser Val
20 25 30
Trp Phe Glu Ala Glu Phe Phe His His Ile Leu His Trp Thr Pro Ile
35 40 45
Pro Asn Gln Ser Glu Ser Thr Cys Tyr Glu Val Ala Leu Leu Arg Tyr
50 55 60
Gly Ile Glu Ser Trp Asn Ser Ile Ser Asn Cys Ser Gln Thr Leu Ser
65 70 75 80
Tyr Asp Leu Thr Ala Val Thr Leu Asp Leu Tyr His Ser Asn Gly Tyr
85 90 95
Arg Ala Arg Val Arg Ala Val Asp Gly Ser Arg His Ser Asn Trp Thr
100 105 110
Val Thr Asn Thr Arg Phe Ser Val Asp Glu Val Thr Leu Thr Val Gly
115 120 125
Ser Val Asn Leu Glu Ile His Asn Gly Phe Ile Leu Gly Lys Ile Gln
130 135 140
Leu Pro Arg Pro Lys Met Ala Pro Ala Asn Asp Thr Tyr Glu Ser Ile
145 150 155 160
Phe Ser His Phe Arg Glu Tyr Glu Ile Ala Ile Arg Lys Val Pro Gly
165 170 175
Asn Phe Thr Phe Thr His Lys Lys Val Lys His Glu Asn Phe Ser Leu
180 185 190
Leu Thr Ser Gly Glu Val Gly Glu Phe Cys Val Gln Val Lys Pro Ser
195 200 205
Val Ala Ser Arg Ser Asn Lys Gly Met Trp Ser Lys Glu Glu Cys Ile
210 215 220
Ser Leu Thr Arg Gln Tyr Phe Thr Val Thr Asn Val Ile Ile Phe Phe
225 230 235 240
Ala Phe Val Leu Leu Leu Ser Gly Ala Leu Ala Tyr Cys Leu Ala Leu
245 250 255
Gln Leu Tyr Val Arg Arg Arg
260
<210> 54
<211> 456
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 54
Met Leu Pro Cys Leu Val Val Leu Leu Ala Ala Leu Leu Ser Leu Arg
1 5 10 15
Leu Gly Ser Asp Ala His Gly Thr Glu Leu Pro Ser Pro Pro Ser Val
20 25 30
Trp Phe Glu Ala Glu Phe Phe His His Ile Leu His Trp Thr Pro Ile
35 40 45
Pro Asn Gln Ser Glu Ser Thr Cys Tyr Glu Val Ala Leu Leu Arg Tyr
50 55 60
Gly Ile Glu Ser Trp Asn Ser Ile Ser Asn Cys Ser Gln Thr Leu Ser
65 70 75 80
Tyr Asp Leu Thr Ala Val Thr Leu Asp Leu Tyr His Ser Asn Gly Tyr
85 90 95
Arg Ala Arg Val Arg Ala Val Asp Gly Ser Arg His Ser Asn Trp Thr
100 105 110
Val Thr Asn Thr Arg Phe Ser Val Asp Glu Val Thr Leu Thr Val Gly
115 120 125
Ser Val Asn Leu Glu Ile His Asn Gly Phe Ile Leu Gly Lys Ile Gln
130 135 140
Leu Pro Arg Pro Lys Met Ala Pro Ala Asn Asp Thr Tyr Glu Ser Ile
145 150 155 160
Phe Ser His Phe Arg Glu Tyr Glu Ile Ala Ile Arg Lys Val Pro Gly
165 170 175
Asn Phe Thr Phe Thr His Lys Lys Val Lys His Glu Asn Phe Ser Leu
180 185 190
Leu Thr Ser Gly Glu Val Gly Glu Phe Cys Val Gln Val Lys Pro Ser
195 200 205
Val Ala Ser Arg Ser Asn Lys Gly Met Trp Ser Lys Glu Glu Cys Ile
210 215 220
Ser Leu Thr Arg Gln Tyr Phe Thr Val Thr Asn Val Pro Ile Leu Leu
225 230 235 240
Thr Ile Ser Ile Leu Ser Phe Phe Ser Val Ala Leu Leu Val Ile Leu
245 250 255
Ala Cys Val Leu Trp Lys Lys Arg Ile Lys Pro Ile Val Trp Pro Ser
260 265 270
Leu Pro Asp His Lys Lys Thr Leu Glu His Leu Cys Lys Lys Pro Arg
275 280 285
Lys Asn Leu Asn Val Ser Phe Asn Pro Glu Ser Phe Leu Asp Cys Gln
290 295 300
Ile His Arg Val Asp Asp Ile Gln Ala Arg Asp Glu Val Glu Gly Phe
305 310 315 320
Leu Gln Asp Thr Phe Pro Gln Gln Leu Glu Glu Ser Glu Lys Gln Arg
325 330 335
Leu Gly Gly Asp Val Gln Ser Pro Asn Cys Pro Ser Glu Asp Val Val
340 345 350
Ile Thr Pro Glu Ser Phe Gly Arg Asp Ser Ser Leu Thr Cys Leu Ala
355 360 365
Gly Asn Val Ser Ala Cys Asp Ala Pro Ile Leu Ser Ser Ser Arg Ser
370 375 380
Leu Asp Cys Arg Glu Ser Gly Lys Asn Gly Pro His Val Tyr Gln Asp
385 390 395 400
Leu Leu Leu Ser Leu Gly Thr Thr Asn Ser Thr Leu Pro Pro Pro Phe
405 410 415
Ser Leu Gln Ser Gly Ile Leu Thr Leu Asn Pro Val Ala Gln Gly Gln
420 425 430
Pro Ile Leu Thr Ser Leu Gly Ser Asn Gln Glu Glu Ala Tyr Val Thr
435 440 445
Met Ser Ser Phe Tyr Gln Asn Gln
450 455
<210> 55
<211> 453
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 55
Met Gly Trp Leu Cys Ser Gly Leu Leu Phe Pro Val Ser Cys Leu Val
1 5 10 15
Leu Leu Gln Val Ala Ser Ser Gly Asn Met Lys Val Leu Gln Glu Pro
20 25 30
Thr Cys Val Ser Asp Tyr Met Ser Ile Ser Thr Cys Glu Trp Lys Met
35 40 45
Asn Gly Pro Thr Asn Cys Ser Thr Glu Leu Arg Leu Leu Tyr Gln Leu
50 55 60
Val Phe Leu Leu Ser Glu Ala His Thr Cys Ile Pro Glu Asn Asn Gly
65 70 75 80
Gly Ala Gly Cys Val Cys His Leu Leu Met Asp Asp Val Val Ser Ala
85 90 95
Asp Asn Tyr Thr Leu Asp Leu Trp Ala Gly Gln Gln Leu Leu Trp Lys
100 105 110
Gly Ser Phe Lys Pro Ser Glu His Val Lys Pro Arg Ala Pro Gly Asn
115 120 125
Leu Thr Val His Thr Asn Val Ser Asp Thr Leu Leu Leu Thr Trp Ser
130 135 140
Asn Pro Tyr Pro Pro Asp Asn Tyr Leu Tyr Asn His Leu Thr Tyr Ala
145 150 155 160
Val Asn Ile Trp Ser Glu Asn Asp Pro Ala Asp Phe Arg Ile Tyr Asn
165 170 175
Val Thr Tyr Leu Glu Pro Ser Leu Arg Ile Ala Ala Ser Thr Leu Lys
180 185 190
Ser Gly Ile Ser Tyr Arg Ala Arg Val Arg Ala Trp Ala Gln Cys Tyr
195 200 205
Asn Thr Thr Trp Ser Glu Trp Ser Pro Ser Thr Lys Trp His Asn Ser
210 215 220
Tyr Arg Glu Pro Phe Glu Gln His Leu Pro Ile Leu Leu Thr Ile Ser
225 230 235 240
Ile Leu Ser Phe Phe Ser Val Ala Leu Leu Val Ile Leu Ala Cys Val
245 250 255
Leu Trp Lys Lys Arg Ile Lys Pro Ile Val Trp Pro Ser Leu Pro Asp
260 265 270
His Lys Lys Thr Leu Glu His Leu Cys Lys Lys Pro Arg Lys Asn Leu
275 280 285
Asn Val Ser Phe Asn Pro Glu Ser Phe Leu Asp Cys Gln Ile His Arg
290 295 300
Val Asp Asp Ile Gln Ala Arg Asp Glu Val Glu Gly Phe Leu Gln Asp
305 310 315 320
Thr Phe Pro Gln Gln Leu Glu Glu Ser Glu Lys Gln Arg Leu Gly Gly
325 330 335
Asp Val Gln Ser Pro Asn Cys Pro Ser Glu Asp Val Val Ile Thr Pro
340 345 350
Glu Ser Phe Gly Arg Asp Ser Ser Leu Thr Cys Leu Ala Gly Asn Val
355 360 365
Ser Ala Cys Asp Ala Pro Ile Leu Ser Ser Ser Arg Ser Leu Asp Cys
370 375 380
Arg Glu Ser Gly Lys Asn Gly Pro His Val Tyr Gln Asp Leu Leu Leu
385 390 395 400
Ser Leu Gly Thr Thr Asn Ser Thr Leu Pro Pro Pro Phe Ser Leu Gln
405 410 415
Ser Gly Ile Leu Thr Leu Asn Pro Val Ala Gln Gly Gln Pro Ile Leu
420 425 430
Thr Ser Leu Gly Ser Asn Gln Glu Glu Ala Tyr Val Thr Met Ser Ser
435 440 445
Phe Tyr Gln Asn Gln
450
<210> 56
<211> 272
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 56
Met Asp Ser Tyr Leu Leu Met Trp Gly Leu Leu Thr Phe Ile Met Val
1 5 10 15
Pro Gly Cys Gln Ala Glu Leu Cys Asp Asp Asp Pro Pro Glu Ile Pro
20 25 30
His Ala Thr Phe Lys Ala Met Ala Tyr Lys Glu Gly Thr Met Leu Asn
35 40 45
Cys Glu Cys Lys Arg Gly Phe Arg Arg Ile Lys Ser Gly Ser Leu Tyr
50 55 60
Met Leu Cys Thr Gly Asn Ser Ser His Ser Ser Trp Asp Asn Gln Cys
65 70 75 80
Gln Cys Thr Ser Ser Ala Thr Arg Asn Thr Thr Lys Gln Val Thr Pro
85 90 95
Gln Pro Glu Glu Gln Lys Glu Arg Lys Thr Thr Glu Met Gln Ser Pro
100 105 110
Met Gln Pro Val Asp Gln Ala Ser Leu Pro Gly His Cys Arg Glu Pro
115 120 125
Pro Pro Trp Glu Asn Glu Ala Thr Glu Arg Ile Tyr His Phe Val Val
130 135 140
Gly Gln Met Val Tyr Tyr Gln Cys Val Gln Gly Tyr Arg Ala Leu His
145 150 155 160
Arg Gly Pro Ala Glu Ser Val Cys Lys Met Thr His Gly Lys Thr Arg
165 170 175
Trp Thr Gln Pro Gln Leu Ile Cys Thr Gly Glu Met Glu Thr Ser Gln
180 185 190
Phe Pro Gly Glu Glu Lys Pro Gln Ala Ser Pro Glu Gly Arg Pro Glu
195 200 205
Ser Glu Thr Ser Cys Leu Val Thr Thr Thr Asp Phe Gln Ile Gln Thr
210 215 220
Glu Met Ala Ala Thr Met Glu Thr Ser Ile Phe Thr Thr Glu Tyr Gln
225 230 235 240
Val Ala Val Ala Gly Cys Val Phe Leu Leu Ile Ser Val Leu Leu Leu
245 250 255
Ser Gly Leu Thr Trp Gln Arg Arg Gln Arg Lys Ser Arg Arg Thr Ile
260 265 270
<210> 57
<211> 459
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 57
Met Thr Ile Leu Gly Thr Thr Phe Gly Met Val Phe Ser Leu Leu Gln
1 5 10 15
Val Val Ser Gly Glu Ser Gly Tyr Ala Gln Asn Gly Asp Leu Glu Asp
20 25 30
Ala Glu Leu Asp Asp Tyr Ser Phe Ser Cys Tyr Ser Gln Leu Glu Val
35 40 45
Asn Gly Ser Gln His Ser Leu Thr Cys Ala Phe Glu Asp Pro Asp Val
50 55 60
Asn Ile Thr Asn Leu Glu Phe Glu Ile Cys Gly Ala Leu Val Glu Val
65 70 75 80
Lys Cys Leu Asn Phe Arg Lys Leu Gln Glu Ile Tyr Phe Ile Glu Thr
85 90 95
Lys Lys Phe Leu Leu Ile Gly Lys Ser Asn Ile Cys Val Lys Val Gly
100 105 110
Glu Lys Ser Leu Thr Cys Lys Lys Ile Asp Leu Thr Thr Ile Val Lys
115 120 125
Pro Glu Ala Pro Phe Asp Leu Ser Val Val Tyr Arg Glu Gly Ala Asn
130 135 140
Asp Phe Val Val Thr Phe Asn Thr Ser His Leu Gln Lys Lys Tyr Val
145 150 155 160
Lys Val Leu Met His Asp Val Ala Tyr Arg Gln Glu Lys Asp Glu Asn
165 170 175
Lys Trp Thr His Val Asn Leu Ser Ser Thr Lys Leu Thr Leu Leu Gln
180 185 190
Arg Lys Leu Gln Pro Ala Ala Met Tyr Glu Ile Lys Val Arg Ser Ile
195 200 205
Pro Asp His Tyr Phe Lys Gly Phe Trp Ser Glu Trp Ser Pro Ser Tyr
210 215 220
Tyr Phe Arg Thr Pro Glu Ile Asn Asn Ser Ser Gly Glu Met Asp Pro
225 230 235 240
Ile Leu Leu Thr Ile Ser Ile Leu Ser Phe Phe Ser Val Ala Leu Leu
245 250 255
Val Ile Leu Ala Cys Val Leu Trp Lys Lys Arg Ile Lys Pro Ile Val
260 265 270
Trp Pro Ser Leu Pro Asp His Lys Lys Thr Leu Glu His Leu Cys Lys
275 280 285
Lys Pro Arg Lys Asn Leu Asn Val Ser Phe Asn Pro Glu Ser Phe Leu
290 295 300
Asp Cys Gln Ile His Arg Val Asp Asp Ile Gln Ala Arg Asp Glu Val
305 310 315 320
Glu Gly Phe Leu Gln Asp Thr Phe Pro Gln Gln Leu Glu Glu Ser Glu
325 330 335
Lys Gln Arg Leu Gly Gly Asp Val Gln Ser Pro Asn Cys Pro Ser Glu
340 345 350
Asp Val Val Ile Thr Pro Glu Ser Phe Gly Arg Asp Ser Ser Leu Thr
355 360 365
Cys Leu Ala Gly Asn Val Ser Ala Cys Asp Ala Pro Ile Leu Ser Ser
370 375 380
Ser Arg Ser Leu Asp Cys Arg Glu Ser Gly Lys Asn Gly Pro His Val
385 390 395 400
Tyr Gln Asp Leu Leu Leu Ser Leu Gly Thr Thr Asn Ser Thr Leu Pro
405 410 415
Pro Pro Phe Ser Leu Gln Ser Gly Ile Leu Thr Leu Asn Pro Val Ala
420 425 430
Gln Gly Gln Pro Ile Leu Thr Ser Leu Gly Ser Asn Gln Glu Glu Ala
435 440 445
Tyr Val Thr Met Ser Ser Phe Tyr Gln Asn Gln
450 455
<210> 58
<211> 255
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 58
Met Gly Asn Ser Cys Tyr Asn Ile Val Ala Thr Leu Leu Leu Val Leu
1 5 10 15
Asn Phe Glu Arg Thr Arg Ser Leu Gln Asp Pro Cys Ser Asn Cys Pro
20 25 30
Ala Gly Thr Phe Cys Asp Asn Asn Arg Asn Gln Ile Cys Ser Pro Cys
35 40 45
Pro Pro Asn Ser Phe Ser Ser Ala Gly Gly Gln Arg Thr Cys Asp Ile
50 55 60
Cys Arg Gln Cys Lys Gly Val Phe Arg Thr Arg Lys Glu Cys Ser Ser
65 70 75 80
Thr Ser Asn Ala Glu Cys Asp Cys Thr Pro Gly Phe His Cys Leu Gly
85 90 95
Ala Gly Cys Ser Met Cys Glu Gln Asp Cys Lys Gln Gly Gln Glu Leu
100 105 110
Thr Lys Lys Gly Cys Lys Asp Cys Cys Phe Gly Thr Phe Asn Asp Gln
115 120 125
Lys Arg Gly Ile Cys Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Lys
130 135 140
Ser Val Leu Val Asn Gly Thr Lys Glu Arg Asp Val Val Cys Gly Pro
145 150 155 160
Ser Pro Ala Asp Leu Ser Pro Gly Ala Ser Ser Val Thr Pro Pro Ala
165 170 175
Pro Ala Arg Glu Pro Gly His Ser Pro Gln Ile Ile Ser Phe Phe Leu
180 185 190
Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu
195 200 205
Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
210 215 220
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
225 230 235 240
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
245 250 255
<210> 59
<211> 197
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 59
Met Leu Gly Ile Trp Thr Leu Leu Pro Leu Val Leu Thr Ser Val Ala
1 5 10 15
Arg Leu Ser Ser Lys Ser Val Asn Ala Gln Val Thr Asp Ile Asn Ser
20 25 30
Lys Gly Leu Glu Leu Arg Lys Thr Val Thr Thr Val Glu Thr Gln Asn
35 40 45
Leu Glu Gly Leu His His Asp Gly Gln Phe Cys His Lys Pro Cys Pro
50 55 60
Pro Gly Glu Arg Lys Ala Arg Asp Cys Thr Val Asn Gly Asp Glu Pro
65 70 75 80
Asp Cys Val Pro Cys Gln Glu Gly Lys Glu Tyr Thr Asp Lys Ala His
85 90 95
Phe Ser Ser Lys Cys Arg Arg Cys Arg Leu Cys Asp Glu Gly His Gly
100 105 110
Leu Glu Val Glu Ile Asn Cys Thr Arg Thr Gln Asn Thr Lys Cys Arg
115 120 125
Cys Lys Pro Asn Phe Phe Cys Asn Ser Thr Val Cys Glu His Cys Asp
130 135 140
Pro Cys Thr Lys Cys Glu His Gly Ile Ile Lys Glu Cys Thr Leu Thr
145 150 155 160
Ser Asn Thr Lys Cys Lys Glu Glu Gly Ser Arg Ser Asn Leu Gly Trp
165 170 175
Leu Cys Leu Leu Leu Leu Pro Ile Pro Leu Ile Val Trp Val Lys Arg
180 185 190
Lys Glu Val Gln Lys
195
<210> 60
<211> 238
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 60
Met Leu Gly Ile Trp Thr Leu Leu Pro Leu Val Leu Thr Ser Val Ala
1 5 10 15
Arg Leu Ser Ser Lys Ser Val Asn Ala Gln Val Thr Asp Ile Asn Ser
20 25 30
Lys Gly Leu Glu Leu Arg Lys Thr Val Thr Thr Val Glu Thr Gln Asn
35 40 45
Leu Glu Gly Leu His His Asp Gly Gln Phe Cys His Lys Pro Cys Pro
50 55 60
Pro Gly Glu Arg Lys Ala Arg Asp Cys Thr Val Asn Gly Asp Glu Pro
65 70 75 80
Asp Cys Val Pro Cys Gln Glu Gly Lys Glu Tyr Thr Asp Lys Ala His
85 90 95
Phe Ser Ser Lys Cys Arg Arg Cys Arg Leu Cys Asp Glu Gly His Gly
100 105 110
Leu Glu Val Glu Ile Asn Cys Thr Arg Thr Gln Asn Thr Lys Cys Arg
115 120 125
Cys Lys Pro Asn Phe Phe Cys Asn Ser Thr Val Cys Glu His Cys Asp
130 135 140
Pro Cys Thr Lys Cys Glu His Gly Ile Ile Lys Glu Cys Thr Leu Thr
145 150 155 160
Ser Asn Thr Lys Cys Lys Glu Glu Gly Ser Arg Ser Asn Leu Gly Trp
165 170 175
Leu Cys Leu Leu Leu Leu Pro Ile Pro Leu Ile Val Trp Val Lys Arg
180 185 190
Lys Glu Val Gln Lys Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp
195 200 205
Tyr Met Asn Met Thr Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr
210 215 220
Gln Pro Tyr Ala Pro Pro Arg Asp Phe Ala Ala Tyr Arg Ser
225 230 235
<210> 61
<211> 239
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 61
Met Leu Gly Ile Trp Thr Leu Leu Pro Leu Val Leu Thr Ser Val Ala
1 5 10 15
Arg Leu Ser Ser Lys Ser Val Asn Ala Gln Val Thr Asp Ile Asn Ser
20 25 30
Lys Gly Leu Glu Leu Arg Lys Thr Val Thr Thr Val Glu Thr Gln Asn
35 40 45
Leu Glu Gly Leu His His Asp Gly Gln Phe Cys His Lys Pro Cys Pro
50 55 60
Pro Gly Glu Arg Lys Ala Arg Asp Cys Thr Val Asn Gly Asp Glu Pro
65 70 75 80
Asp Cys Val Pro Cys Gln Glu Gly Lys Glu Tyr Thr Asp Lys Ala His
85 90 95
Phe Ser Ser Lys Cys Arg Arg Cys Arg Leu Cys Asp Glu Gly His Gly
100 105 110
Leu Glu Val Glu Ile Asn Cys Thr Arg Thr Gln Asn Thr Lys Cys Arg
115 120 125
Cys Lys Pro Asn Phe Phe Cys Asn Ser Thr Val Cys Glu His Cys Asp
130 135 140
Pro Cys Thr Lys Cys Glu His Gly Ile Ile Lys Glu Cys Thr Leu Thr
145 150 155 160
Ser Asn Thr Lys Cys Lys Glu Glu Gly Ser Arg Ser Asn Leu Gly Trp
165 170 175
Leu Cys Leu Leu Leu Leu Pro Ile Pro Leu Ile Val Trp Val Lys Arg
180 185 190
Lys Glu Val Gln Lys Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
195 200 205
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
210 215 220
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
225 230 235
<210> 62
<211> 299
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 62
Met Leu Gly Ile Trp Thr Leu Leu Pro Leu Val Leu Thr Ser Val Ala
1 5 10 15
Arg Leu Ser Ser Lys Ser Val Asn Ala Gln Val Thr Asp Ile Asn Ser
20 25 30
Lys Gly Leu Glu Leu Arg Lys Thr Val Thr Thr Val Glu Thr Gln Asn
35 40 45
Leu Glu Gly Leu His His Asp Gly Gln Phe Cys His Lys Pro Cys Pro
50 55 60
Pro Gly Glu Arg Lys Ala Arg Asp Cys Thr Val Asn Gly Asp Glu Pro
65 70 75 80
Asp Cys Val Pro Cys Gln Glu Gly Lys Glu Tyr Thr Asp Lys Ala His
85 90 95
Phe Ser Ser Lys Cys Arg Arg Cys Arg Leu Cys Asp Glu Gly His Gly
100 105 110
Leu Glu Val Glu Ile Asn Cys Thr Arg Thr Gln Asn Thr Lys Cys Arg
115 120 125
Cys Lys Pro Asn Phe Phe Cys Asn Ser Thr Val Cys Glu His Cys Asp
130 135 140
Pro Cys Thr Lys Cys Glu His Gly Ile Ile Lys Glu Cys Thr Leu Thr
145 150 155 160
Ser Asn Thr Lys Cys Lys Glu Glu Gly Ser Arg Ser Asn Leu Gly Trp
165 170 175
Leu Cys Leu Leu Leu Leu Pro Ile Pro Leu Ile Val Trp Val Lys Arg
180 185 190
Lys Glu Val Gln Lys Thr Gln Val Ala Ala Asp Trp Thr Ala Leu Ala
195 200 205
Glu Glu Met Asp Phe Glu Tyr Leu Glu Ile Arg Gln Leu Glu Thr Gln
210 215 220
Ala Asp Pro Thr Gly Arg Leu Leu Asp Ala Trp Gln Gly Arg Pro Gly
225 230 235 240
Ala Ser Val Gly Arg Leu Leu Glu Leu Leu Thr Lys Leu Gly Arg Asp
245 250 255
Asp Val Leu Leu Glu Leu Gly Pro Ser Ile Glu Glu Asp Cys Gln Lys
260 265 270
Tyr Ile Leu Lys Gln Gln Gln Glu Glu Ala Glu Lys Pro Leu Gln Val
275 280 285
Ala Ala Val Asp Ser Ser Val Pro Arg Thr Ala
290 295
<210> 63
<211> 235
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 63
Met Leu Gly Ile Trp Thr Leu Leu Pro Leu Val Leu Thr Ser Val Ala
1 5 10 15
Arg Leu Ser Ser Lys Ser Val Asn Ala Gln Val Thr Asp Ile Asn Ser
20 25 30
Lys Gly Leu Glu Leu Arg Lys Thr Val Thr Thr Val Glu Thr Gln Asn
35 40 45
Leu Glu Gly Leu His His Asp Gly Gln Phe Cys His Lys Pro Cys Pro
50 55 60
Pro Gly Glu Arg Lys Ala Arg Asp Cys Thr Val Asn Gly Asp Glu Pro
65 70 75 80
Asp Cys Val Pro Cys Gln Glu Gly Lys Glu Tyr Thr Asp Lys Ala His
85 90 95
Phe Ser Ser Lys Cys Arg Arg Cys Arg Leu Cys Asp Glu Gly His Gly
100 105 110
Leu Glu Val Glu Ile Asn Cys Thr Arg Thr Gln Asn Thr Lys Cys Arg
115 120 125
Cys Lys Pro Asn Phe Phe Cys Asn Ser Thr Val Cys Glu His Cys Asp
130 135 140
Pro Cys Thr Lys Cys Glu His Gly Ile Ile Lys Glu Cys Thr Leu Thr
145 150 155 160
Ser Asn Thr Lys Cys Lys Glu Glu Gly Ser Arg Ser Asn Leu Gly Trp
165 170 175
Leu Cys Leu Leu Leu Leu Pro Ile Pro Leu Ile Val Trp Val Lys Arg
180 185 190
Lys Glu Val Gln Lys Cys Trp Leu Thr Lys Lys Lys Tyr Ser Ser Ser
195 200 205
Val His Asp Pro Asn Gly Glu Tyr Met Phe Met Arg Ala Val Asn Thr
210 215 220
Ala Lys Lys Ser Arg Leu Thr Asp Val Thr Leu
225 230 235
<210> 64
<211> 238
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 64
Met Glu Gln Arg Gly Gln Asn Ala Pro Ala Ala Ser Gly Ala Arg Lys
1 5 10 15
Arg His Gly Pro Gly Pro Arg Glu Ala Arg Gly Ala Arg Pro Gly Pro
20 25 30
Arg Val Pro Lys Thr Leu Val Leu Val Val Ala Ala Val Leu Leu Leu
35 40 45
Val Ser Ala Glu Ser Ala Leu Ile Thr Gln Gln Asp Leu Ala Pro Gln
50 55 60
Gln Arg Ala Ala Pro Gln Gln Lys Arg Ser Ser Pro Ser Glu Gly Leu
65 70 75 80
Cys Pro Pro Gly His His Ile Ser Glu Asp Gly Arg Asp Cys Ile Ser
85 90 95
Cys Lys Tyr Gly Gln Asp Tyr Ser Thr His Trp Asn Asp Leu Leu Phe
100 105 110
Cys Leu Arg Cys Thr Arg Cys Asp Ser Gly Glu Val Glu Leu Ser Pro
115 120 125
Cys Thr Thr Thr Arg Asn Thr Val Cys Gln Cys Glu Glu Gly Thr Phe
130 135 140
Arg Glu Glu Asp Ser Pro Glu Met Cys Arg Lys Cys Arg Thr Gly Cys
145 150 155 160
Pro Arg Gly Met Val Lys Val Gly Asp Cys Thr Pro Trp Ser Asp Ile
165 170 175
Glu Cys Val His Lys Glu Ser Gly Thr Lys His Ser Gly Glu Val Pro
180 185 190
Ala Val Glu Glu Thr Val Thr Ser Ser Pro Gly Thr Pro Ala Ser Pro
195 200 205
Cys Ser Leu Ser Gly Ile Ile Ile Gly Val Thr Val Ala Ala Val Val
210 215 220
Leu Ile Val Ala Val Phe Val Cys Lys Ser Leu Leu Trp Lys
225 230 235
<210> 65
<211> 279
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 65
Met Glu Gln Arg Gly Gln Asn Ala Pro Ala Ala Ser Gly Ala Arg Lys
1 5 10 15
Arg His Gly Pro Gly Pro Arg Glu Ala Arg Gly Ala Arg Pro Gly Pro
20 25 30
Arg Val Pro Lys Thr Leu Val Leu Val Val Ala Ala Val Leu Leu Leu
35 40 45
Val Ser Ala Glu Ser Ala Leu Ile Thr Gln Gln Asp Leu Ala Pro Gln
50 55 60
Gln Arg Ala Ala Pro Gln Gln Lys Arg Ser Ser Pro Ser Glu Gly Leu
65 70 75 80
Cys Pro Pro Gly His His Ile Ser Glu Asp Gly Arg Asp Cys Ile Ser
85 90 95
Cys Lys Tyr Gly Gln Asp Tyr Ser Thr His Trp Asn Asp Leu Leu Phe
100 105 110
Cys Leu Arg Cys Thr Arg Cys Asp Ser Gly Glu Val Glu Leu Ser Pro
115 120 125
Cys Thr Thr Thr Arg Asn Thr Val Cys Gln Cys Glu Glu Gly Thr Phe
130 135 140
Arg Glu Glu Asp Ser Pro Glu Met Cys Arg Lys Cys Arg Thr Gly Cys
145 150 155 160
Pro Arg Gly Met Val Lys Val Gly Asp Cys Thr Pro Trp Ser Asp Ile
165 170 175
Glu Cys Val His Lys Glu Ser Gly Thr Lys His Ser Gly Glu Val Pro
180 185 190
Ala Val Glu Glu Thr Val Thr Ser Ser Pro Gly Thr Pro Ala Ser Pro
195 200 205
Cys Ser Leu Ser Gly Ile Ile Ile Gly Val Thr Val Ala Ala Val Val
210 215 220
Leu Ile Val Ala Val Phe Val Cys Lys Ser Leu Leu Trp Lys Arg Ser
225 230 235 240
Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr Pro Arg
245 250 255
Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro Pro Arg
260 265 270
Asp Phe Ala Ala Tyr Arg Ser
275
<210> 66
<211> 362
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 66
Met Gly Thr Lys Pro Thr Glu Gln Val Ser Trp Gly Leu Tyr Ser Gly
1 5 10 15
Tyr Asp Glu Glu Ala Tyr Ser Val Gly Pro Leu Pro Glu Leu Cys Tyr
20 25 30
Lys Ala Asp Val Gln Ala Phe Ser Arg Ala Phe Gln Pro Ser Val Ser
35 40 45
Leu Met Val Ala Val Leu Gly Leu Ala Gly Asn Gly Leu Val Leu Ala
50 55 60
Thr His Leu Ala Ala Arg Arg Thr Thr Arg Ser Pro Thr Ser Val His
65 70 75 80
Leu Leu Gln Leu Ala Leu Ala Asp Leu Leu Leu Ala Leu Thr Leu Pro
85 90 95
Phe Ala Ala Ala Gly Ala Leu Gln Gly Trp Asn Leu Gly Ser Thr Thr
100 105 110
Cys Arg Ala Ile Ser Gly Leu Tyr Ser Ala Ser Phe His Ala Gly Phe
115 120 125
Leu Phe Leu Ala Cys Ile Ser Ala Asp Arg Tyr Val Ala Ile Ala Arg
130 135 140
Ala Leu Pro Ala Gly Gln Arg Pro Ser Thr Pro Ser Arg Ala His Leu
145 150 155 160
Val Ser Val Phe Val Trp Leu Leu Ser Leu Phe Leu Ala Leu Pro Ala
165 170 175
Leu Leu Phe Ser Arg Asp Gly Pro Arg Glu Gly Gln Arg Arg Cys Arg
180 185 190
Leu Ile Phe Pro Glu Ser Leu Thr Gln Thr Val Lys Gly Ala Ser Ala
195 200 205
Val Ala Gln Val Val Leu Gly Phe Ala Leu Pro Leu Gly Val Met Ala
210 215 220
Ala Cys Tyr Ala Leu Leu Gly Arg Thr Leu Leu Ala Ala Arg Gly Pro
225 230 235 240
Glu Arg Arg Arg Ala Leu Arg Val Val Val Ala Leu Val Val Ala Phe
245 250 255
Val Val Leu Gln Leu Pro Tyr Ser Leu Ala Leu Leu Leu Asp Thr Ala
260 265 270
Asp Leu Leu Ala Ala Arg Glu Arg Ser Cys Ser Ser Ser Lys Arg Lys
275 280 285
Asp Leu Ala Leu Leu Val Thr Gly Gly Leu Thr Leu Val Arg Cys Ser
290 295 300
Leu Asn Pro Val Leu Tyr Ala Phe Leu Gly Leu Arg Phe Arg Arg Asp
305 310 315 320
Leu Arg Arg Leu Leu Gln Gly Gly Gly Cys Ser Pro Lys Pro Asn Pro
325 330 335
Arg Gly Arg Cys Pro Arg Arg Leu Arg Leu Ser Ser Cys Ser Ala Pro
340 345 350
Thr Glu Thr His Ser Leu Ser Trp Asp Asn
355 360
<210> 67
<211> 465
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 67
Met Gly Gly Ala Val Val Asp Glu Gly Pro Thr Gly Val Lys Ala Pro
1 5 10 15
Asp Gly Gly Trp Gly Trp Ala Val Leu Phe Gly Cys Phe Val Ile Thr
20 25 30
Gly Phe Ser Tyr Ala Phe Pro Lys Ala Val Ser Val Phe Phe Lys Glu
35 40 45
Leu Ile Gln Glu Phe Gly Ile Gly Tyr Ser Asp Thr Ala Trp Ile Ser
50 55 60
Ser Ile Leu Leu Ala Met Leu Tyr Gly Thr Gly Pro Leu Cys Ser Val
65 70 75 80
Cys Val Asn Arg Phe Gly Cys Arg Pro Val Met Leu Val Gly Gly Leu
85 90 95
Phe Ala Ser Leu Gly Met Val Ala Ala Ser Phe Cys Arg Ser Ile Ile
100 105 110
Gln Val Tyr Leu Thr Thr Gly Val Ile Thr Gly Leu Gly Leu Ala Leu
115 120 125
Asn Phe Gln Pro Ser Leu Ile Met Leu Asn Arg Tyr Phe Ser Lys Arg
130 135 140
Arg Pro Met Ala Asn Gly Leu Ala Ala Ala Gly Ser Pro Val Phe Leu
145 150 155 160
Cys Ala Leu Ser Pro Leu Gly Gln Leu Leu Gln Asp Arg Tyr Gly Trp
165 170 175
Arg Gly Gly Phe Leu Ile Leu Gly Gly Leu Leu Leu Asn Cys Cys Val
180 185 190
Cys Ala Ala Leu Met Arg Pro Leu Val Val Thr Ala Gln Pro Gly Ser
195 200 205
Gly Pro Pro Arg Pro Ser Arg Arg Leu Leu Asp Leu Ser Val Phe Arg
210 215 220
Asp Arg Gly Phe Val Leu Tyr Ala Val Ala Ala Ser Val Met Val Leu
225 230 235 240
Gly Leu Phe Val Pro Pro Val Phe Val Val Ser Tyr Ala Lys Asp Leu
245 250 255
Gly Val Pro Asp Thr Lys Ala Ala Phe Leu Leu Thr Ile Leu Gly Phe
260 265 270
Ile Asp Ile Phe Ala Arg Pro Ala Ala Gly Phe Val Ala Gly Leu Gly
275 280 285
Lys Val Arg Pro Tyr Ser Val Tyr Leu Phe Ser Phe Ser Met Phe Phe
290 295 300
Asn Gly Leu Ala Asp Leu Ala Gly Ser Thr Ala Gly Asp Tyr Gly Gly
305 310 315 320
Leu Val Val Phe Cys Ile Phe Phe Gly Ile Ser Tyr Gly Met Val Gly
325 330 335
Ala Leu Gln Phe Glu Val Leu Met Ala Ile Val Gly Thr His Lys Phe
340 345 350
Ser Ser Ala Ile Gly Leu Val Leu Leu Met Glu Ala Val Ala Val Leu
355 360 365
Val Gly Pro Pro Ser Gly Gly Lys Leu Leu Asp Ala Thr His Val Tyr
370 375 380
Met Tyr Val Phe Ile Leu Ala Gly Ala Glu Val Leu Thr Ser Ser Leu
385 390 395 400
Ile Leu Leu Leu Gly Asn Phe Phe Cys Ile Arg Lys Lys Pro Lys Glu
405 410 415
Pro Gln Pro Glu Val Ala Ala Ala Glu Glu Glu Lys Leu His Lys Pro
420 425 430
Pro Ala Asp Ser Gly Val Asp Leu Arg Glu Val Glu His Phe Leu Lys
435 440 445
Ala Glu Pro Glu Lys Asn Gly Glu Val Val His Thr Pro Glu Thr Ser
450 455 460
Val
465
<210> 68
<211> 154
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 68
Met Ala Thr Lys Ala Val Cys Val Leu Lys Gly Asp Gly Pro Val Gln
1 5 10 15
Gly Ile Ile Asn Phe Glu Gln Lys Glu Ser Asn Gly Pro Val Lys Val
20 25 30
Trp Gly Ser Ile Lys Gly Leu Thr Glu Gly Leu His Gly Phe His Val
35 40 45
His Glu Phe Gly Asp Asn Thr Ala Gly Cys Thr Ser Ala Gly Pro His
50 55 60
Phe Asn Pro Leu Ser Arg Lys His Gly Gly Pro Lys Asp Glu Glu Arg
65 70 75 80
His Val Gly Asp Leu Gly Asn Val Thr Ala Asp Lys Asp Gly Val Ala
85 90 95
Asp Val Ser Ile Glu Asp Ser Val Ile Ser Leu Ser Gly Asp His Cys
100 105 110
Ile Ile Gly Arg Thr Leu Val Val His Glu Lys Ala Asp Asp Leu Gly
115 120 125
Lys Gly Gly Asn Glu Glu Ser Thr Lys Thr Gly Asn Ala Gly Ser Arg
130 135 140
Leu Ala Cys Gly Val Ile Gly Ile Ala Gln
145 150
<210> 69
<211> 384
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 69
Met Pro Gln Leu Asp Ser Gly Gly Gly Gly Ala Gly Gly Gly Asp Asp
1 5 10 15
Leu Gly Ala Pro Asp Glu Leu Leu Ala Phe Gln Asp Glu Gly Glu Glu
20 25 30
Gln Asp Asp Lys Ser Arg Asp Ser Ala Ala Gly Pro Glu Arg Asp Leu
35 40 45
Ala Glu Leu Lys Ser Ser Leu Val Asn Glu Ser Glu Gly Ala Ala Gly
50 55 60
Gly Ala Gly Ile Pro Gly Val Pro Gly Ala Gly Ala Gly Ala Arg Gly
65 70 75 80
Glu Ala Glu Ala Leu Gly Arg Glu His Ala Ala Gln Arg Leu Phe Pro
85 90 95
Asp Lys Leu Pro Glu Pro Leu Glu Asp Gly Leu Lys Ala Pro Glu Cys
100 105 110
Thr Ser Gly Met Tyr Lys Glu Thr Val Tyr Ser Ala Phe Asn Leu Leu
115 120 125
Met His Tyr Pro Pro Pro Ser Gly Ala Gly Gln His Pro Gln Pro Gln
130 135 140
Pro Pro Leu His Lys Ala Asn Gln Pro Pro His Gly Val Pro Gln Leu
145 150 155 160
Ser Leu Tyr Glu His Phe Asn Ser Pro His Pro Thr Pro Ala Pro Ala
165 170 175
Asp Ile Ser Gln Lys Gln Val His Arg Pro Leu Gln Thr Pro Asp Leu
180 185 190
Ser Gly Phe Tyr Ser Leu Thr Ser Gly Ser Met Gly Gln Leu Pro His
195 200 205
Thr Val Ser Trp Phe Thr His Pro Ser Leu Met Leu Gly Ser Gly Val
210 215 220
Pro Gly His Pro Ala Ala Ile Pro His Pro Ala Ile Val Pro Pro Ser
225 230 235 240
Gly Lys Gln Glu Leu Gln Pro Phe Asp Arg Asn Leu Lys Thr Gln Ala
245 250 255
Glu Ser Lys Ala Glu Lys Glu Ala Lys Lys Pro Thr Ile Lys Lys Pro
260 265 270
Leu Asn Ala Phe Met Leu Tyr Met Lys Glu Met Arg Ala Lys Val Ile
275 280 285
Ala Glu Cys Thr Leu Lys Glu Ser Ala Ala Ile Asn Gln Ile Leu Gly
290 295 300
Arg Arg Trp His Ala Leu Ser Arg Glu Glu Gln Ala Lys Tyr Tyr Glu
305 310 315 320
Leu Ala Arg Lys Glu Arg Gln Leu His Met Gln Leu Tyr Pro Gly Trp
325 330 335
Ser Ala Arg Asp Asn Tyr Gly Lys Lys Lys Arg Arg Ser Arg Glu Lys
340 345 350
His Gln Glu Ser Thr Thr Gly Gly Lys Arg Asn Ala Phe Gly Thr Tyr
355 360 365
Pro Glu Lys Ala Ala Ala Pro Ala Pro Phe Leu Pro Met Thr Val Leu
370 375 380
<210> 70
<211> 277
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 70
Met Gly Ala Gly Ala Thr Gly Arg Ala Met Asp Gly Pro Arg Leu Leu
1 5 10 15
Leu Leu Leu Leu Leu Gly Val Ser Leu Gly Gly Ala Lys Glu Ala Cys
20 25 30
Pro Thr Gly Leu Tyr Thr His Ser Gly Glu Cys Cys Lys Ala Cys Asn
35 40 45
Leu Gly Glu Gly Val Ala Gln Pro Cys Gly Ala Asn Gln Thr Val Cys
50 55 60
Glu Pro Cys Leu Asp Ser Val Thr Phe Ser Asp Val Val Ser Ala Thr
65 70 75 80
Glu Pro Cys Lys Pro Cys Thr Glu Cys Val Gly Leu Gln Ser Met Ser
85 90 95
Ala Pro Cys Val Glu Ala Asp Asp Ala Val Cys Arg Cys Ala Tyr Gly
100 105 110
Tyr Tyr Gln Asp Glu Thr Thr Gly Arg Cys Glu Ala Cys Arg Val Cys
115 120 125
Glu Ala Gly Ser Gly Leu Val Phe Ser Cys Gln Asp Lys Gln Asn Thr
130 135 140
Val Cys Glu Glu Cys Pro Asp Gly Thr Tyr Ser Asp Glu Ala Asn His
145 150 155 160
Val Asp Pro Cys Leu Pro Cys Thr Val Cys Glu Asp Thr Glu Arg Gln
165 170 175
Leu Arg Glu Cys Thr Arg Trp Ala Asp Ala Glu Cys Glu Glu Ile Pro
180 185 190
Gly Arg Trp Ile Thr Arg Ser Thr Pro Pro Glu Gly Ser Asp Ser Thr
195 200 205
Ala Pro Ser Thr Gln Glu Pro Glu Ala Pro Pro Glu Gln Asp Leu Ile
210 215 220
Ala Ser Thr Val Ala Gly Val Val Thr Thr Val Met Gly Ser Ser Gln
225 230 235 240
Pro Val Val Thr Arg Gly Thr Thr Asp Asn Leu Ile Pro Val Tyr Cys
245 250 255
Ser Ile Leu Ala Ala Val Val Val Gly Leu Val Ala Tyr Ile Ala Phe
260 265 270
Lys Arg Trp Asn Ser
275
<210> 71
<211> 243
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 71
Gly Ser Gly Gly Thr Ser Gly Ser Lys Gly Glu Glu Leu Phe Thr Gly
1 5 10 15
Val Val Pro Ile Leu Val Glu Leu Asp Gly Asp Val Asn Gly His Lys
20 25 30
Phe Ser Val Arg Gly Glu Gly Glu Gly Asp Ala Thr Asn Gly Lys Leu
35 40 45
Thr Leu Lys Phe Ile Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro
50 55 60
Thr Leu Val Thr Thr Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr
65 70 75 80
Pro Asp His Met Lys Arg His Asp Phe Phe Lys Ser Ala Met Pro Glu
85 90 95
Gly Tyr Val Gln Glu Arg Thr Ile Ser Phe Lys Asp Asp Gly Thr Tyr
100 105 110
Lys Thr Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg
115 120 125
Ile Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly
130 135 140
His Lys Leu Glu Tyr Asn Phe Asn Ser His Asn Val Tyr Ile Thr Ala
145 150 155 160
Asp Lys Gln Lys Asn Gly Ile Lys Ala Asn Phe Lys Ile Arg His Asn
165 170 175
Val Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr
180 185 190
Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser
195 200 205
Thr Gln Ser Val Leu Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met
210 215 220
Val Leu Leu Glu Phe Val Thr Ala Ala Gly Ile Thr Gly Thr Gly Ala
225 230 235 240
Gly Ser Gly
<210> 72
<211> 249
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 72
Gly Ser Gly Gly Thr Ser Gly Val Ser Lys Gly Glu Glu Asp Asn Met
1 5 10 15
Ala Ile Ile Lys Glu Phe Met Arg Phe Lys Val His Met Glu Gly Ser
20 25 30
Val Asn Gly His Glu Phe Glu Ile Glu Gly Glu Gly Glu Gly Arg Pro
35 40 45
Tyr Glu Gly Thr Gln Thr Ala Lys Leu Lys Val Thr Lys Gly Gly Pro
50 55 60
Leu Pro Phe Ala Trp Asp Ile Leu Ser Pro Gln Phe Met Tyr Gly Ser
65 70 75 80
Lys Ala Tyr Val Lys His Pro Ala Asp Ile Pro Asp Tyr Leu Lys Leu
85 90 95
Ser Phe Pro Glu Gly Phe Lys Trp Glu Arg Val Met Asn Phe Glu Asp
100 105 110
Gly Gly Val Val Thr Val Thr Gln Asp Ser Ser Leu Gln Asp Gly Glu
115 120 125
Phe Ile Tyr Lys Val Lys Leu Arg Gly Thr Asn Phe Pro Ser Asp Gly
130 135 140
Pro Val Met Gln Lys Lys Thr Met Gly Trp Glu Ala Ser Ser Glu Arg
145 150 155 160
Met Tyr Pro Glu Asp Gly Ala Leu Lys Gly Glu Ile Lys Gln Arg Leu
165 170 175
Lys Leu Lys Asp Gly Gly His Tyr Asp Ala Glu Val Lys Thr Thr Tyr
180 185 190
Lys Ala Lys Lys Pro Val Gln Leu Pro Gly Ala Tyr Asn Val Asn Ile
195 200 205
Lys Leu Asp Ile Thr Ser His Asn Glu Asp Tyr Thr Ile Val Glu Gln
210 215 220
Tyr Glu Arg Ala Glu Gly Arg His Ser Thr Gly Gly Met Asp Glu Leu
225 230 235 240
Tyr Lys Gly Thr Gly Ala Gly Ser Gly
245
<210> 73
<211> 150
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 73
Pro Gly Trp Phe Leu Asp Ser Pro Asp Arg Pro Trp Asn Pro Pro Thr
1 5 10 15
Phe Ser Pro Ala Leu Leu Val Val Thr Glu Gly Asp Asn Ala Thr Phe
20 25 30
Thr Cys Ser Phe Ser Asn Thr Ser Glu Ser Phe Val Leu Asn Trp Tyr
35 40 45
Arg Met Ser Pro Ser Asn Gln Thr Asp Lys Leu Ala Ala Phe Pro Glu
50 55 60
Asp Arg Ser Gln Pro Gly Gln Asp Cys Arg Phe Arg Val Thr Gln Leu
65 70 75 80
Pro Asn Gly Arg Asp Phe His Met Ser Val Val Arg Ala Arg Arg Asn
85 90 95
Asp Ser Gly Thr Tyr Leu Cys Gly Ala Ile Ser Leu Ala Pro Lys Ala
100 105 110
Gln Ile Lys Glu Ser Leu Arg Ala Glu Leu Arg Val Thr Glu Arg Arg
115 120 125
Ala Glu Val Pro Thr Ala His Pro Ser Pro Ser Pro Arg Pro Ala Gly
130 135 140
Gln Phe Gln Thr Leu Val
145 150
<210> 74
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 74
Val Gly Val Val Gly Gly Leu Leu Gly Ser Leu Val Leu Leu Val Trp
1 5 10 15
Val Leu Ala Val Ile
20
<210> 75
<211> 97
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 75
Cys Ser Arg Ala Ala Arg Gly Thr Ile Gly Ala Arg Arg Thr Gly Gln
1 5 10 15
Pro Leu Lys Glu Asp Pro Ser Ala Val Pro Val Phe Ser Val Asp Tyr
20 25 30
Gly Glu Leu Asp Phe Gln Trp Arg Glu Lys Thr Pro Glu Pro Pro Val
35 40 45
Pro Cys Val Pro Glu Gln Thr Glu Tyr Ala Thr Ile Val Phe Pro Ser
50 55 60
Gly Met Gly Thr Ser Ser Pro Ala Arg Arg Gly Ser Ala Asp Gly Pro
65 70 75 80
Arg Ser Ala Gln Pro Leu Arg Pro Glu Asp Gly His Cys Ser Trp Pro
85 90 95
Leu
<210> 76
<211> 163
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 76
Leu Gln Asp Pro Cys Ser Asn Cys Pro Ala Gly Thr Phe Cys Asp Asn
1 5 10 15
Asn Arg Asn Gln Ile Cys Ser Pro Cys Pro Pro Asn Ser Phe Ser Ser
20 25 30
Ala Gly Gly Gln Arg Thr Cys Asp Ile Cys Arg Gln Cys Lys Gly Val
35 40 45
Phe Arg Thr Arg Lys Glu Cys Ser Ser Thr Ser Asn Ala Glu Cys Asp
50 55 60
Cys Thr Pro Gly Phe His Cys Leu Gly Ala Gly Cys Ser Met Cys Glu
65 70 75 80
Gln Asp Cys Lys Gln Gly Gln Glu Leu Thr Lys Lys Gly Cys Lys Asp
85 90 95
Cys Cys Phe Gly Thr Phe Asn Asp Gln Lys Arg Gly Ile Cys Arg Pro
100 105 110
Trp Thr Asn Cys Ser Leu Asp Gly Lys Ser Val Leu Val Asn Gly Thr
115 120 125
Lys Glu Arg Asp Val Val Cys Gly Pro Ser Pro Ala Asp Leu Ser Pro
130 135 140
Gly Ala Ser Ser Val Thr Pro Pro Ala Pro Ala Arg Glu Pro Gly His
145 150 155 160
Ser Pro Gln
<210> 77
<211> 27
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 77
Ile Ile Ser Phe Phe Leu Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu
1 5 10 15
Leu Phe Phe Leu Thr Leu Arg Phe Ser Val Val
20 25
<210> 78
<211> 42
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 78
Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe Lys Gln Pro Phe Met
1 5 10 15
Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly Cys Ser Cys Arg Phe
20 25 30
Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
35 40
<210> 79
<211> 120
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 79
Glu Ile Asn Gly Ser Ala Asn Tyr Glu Met Phe Ile Phe His Asn Gly
1 5 10 15
Gly Val Gln Ile Leu Cys Lys Tyr Pro Asp Ile Val Gln Gln Phe Lys
20 25 30
Met Gln Leu Leu Lys Gly Gly Gln Ile Leu Cys Asp Leu Thr Lys Thr
35 40 45
Lys Gly Ser Gly Asn Thr Val Ser Ile Lys Ser Leu Lys Phe Cys His
50 55 60
Ser Gln Leu Ser Asn Asn Ser Val Ser Phe Phe Leu Tyr Asn Leu Asp
65 70 75 80
His Ser His Ala Asn Tyr Tyr Phe Cys Asn Leu Ser Ile Phe Asp Pro
85 90 95
Pro Pro Phe Lys Val Thr Leu Thr Gly Gly Tyr Leu His Ile Tyr Glu
100 105 110
Ser Gln Leu Cys Cys Gln Leu Lys
115 120
<210> 80
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 80
Phe Trp Leu Pro Ile Gly Cys Ala Ala Phe Val Val Val Cys Ile Leu
1 5 10 15
Gly Cys Ile Leu Ile
20
<210> 81
<211> 38
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 81
Cys Trp Leu Thr Lys Lys Lys Tyr Ser Ser Ser Val His Asp Pro Asn
1 5 10 15
Gly Glu Tyr Met Phe Met Arg Ala Val Asn Thr Ala Lys Lys Ser Arg
20 25 30
Leu Thr Asp Val Thr Leu
35
<210> 82
<211> 126
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 82
Lys Ala Met His Val Ala Gln Pro Ala Val Val Leu Ala Ser Ser Arg
1 5 10 15
Gly Ile Ala Ser Phe Val Cys Glu Tyr Ala Ser Pro Gly Lys Ala Thr
20 25 30
Glu Val Arg Val Thr Val Leu Arg Gln Ala Asp Ser Gln Val Thr Glu
35 40 45
Val Cys Ala Ala Thr Tyr Met Met Gly Asn Glu Leu Thr Phe Leu Asp
50 55 60
Asp Ser Ile Cys Thr Gly Thr Ser Ser Gly Asn Gln Val Asn Leu Thr
65 70 75 80
Ile Gln Gly Leu Arg Ala Met Asp Thr Gly Leu Tyr Ile Cys Lys Val
85 90 95
Glu Leu Met Tyr Pro Pro Pro Tyr Tyr Leu Gly Ile Gly Asn Gly Thr
100 105 110
Gln Ile Tyr Val Ile Asp Pro Glu Pro Cys Pro Asp Ser Asp
115 120 125
<210> 83
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 83
Phe Leu Leu Trp Ile Leu Ala Ala Val Ser Ser Gly Leu Phe Phe Tyr
1 5 10 15
Ser Phe Leu Leu Thr
20
<210> 84
<211> 41
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 84
Ala Val Ser Leu Ser Lys Met Leu Lys Lys Arg Ser Pro Leu Thr Thr
1 5 10 15
Gly Val Tyr Val Lys Met Pro Pro Thr Glu Pro Glu Cys Glu Lys Gln
20 25 30
Phe Gln Pro Tyr Phe Ile Pro Ile Asn
35 40
<210> 85
<211> 134
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 85
Asn Lys Ile Leu Val Lys Gln Ser Pro Met Leu Val Ala Tyr Asp Asn
1 5 10 15
Ala Val Asn Leu Ser Cys Lys Tyr Ser Tyr Asn Leu Phe Ser Arg Glu
20 25 30
Phe Arg Ala Ser Leu His Lys Gly Leu Asp Ser Ala Val Glu Val Cys
35 40 45
Val Val Tyr Gly Asn Tyr Ser Gln Gln Leu Gln Val Tyr Ser Lys Thr
50 55 60
Gly Phe Asn Cys Asp Gly Lys Leu Gly Asn Glu Ser Val Thr Phe Tyr
65 70 75 80
Leu Gln Asn Leu Tyr Val Asn Gln Thr Asp Ile Tyr Phe Cys Lys Ile
85 90 95
Glu Val Met Tyr Pro Pro Pro Tyr Leu Asp Asn Glu Lys Ser Asn Gly
100 105 110
Thr Ile Ile His Val Lys Gly Lys His Leu Cys Pro Ser Pro Leu Phe
115 120 125
Pro Gly Pro Ser Lys Pro
130
<210> 86
<211> 27
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 86
Phe Trp Val Leu Val Val Val Gly Gly Val Leu Ala Cys Tyr Ser Leu
1 5 10 15
Leu Val Thr Val Ala Phe Ile Ile Phe Trp Val
20 25
<210> 87
<211> 41
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 87
Arg Ser Lys Arg Ser Arg Leu Leu His Ser Asp Tyr Met Asn Met Thr
1 5 10 15
Pro Arg Arg Pro Gly Pro Thr Arg Lys His Tyr Gln Pro Tyr Ala Pro
20 25 30
Pro Arg Asp Phe Ala Ala Tyr Arg Ser
35 40
<210> 88
<211> 215
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 88
Gln Pro Asn Asn Ser Leu Met Leu Gln Thr Ser Lys Glu Asn His Ala
1 5 10 15
Leu Ala Ser Ser Ser Leu Cys Met Asp Glu Lys Gln Ile Thr Gln Asn
20 25 30
Tyr Ser Lys Val Leu Ala Glu Val Asn Thr Ser Trp Pro Val Lys Met
35 40 45
Ala Thr Asn Ala Val Leu Cys Cys Pro Pro Ile Ala Leu Arg Asn Leu
50 55 60
Ile Ile Ile Thr Trp Glu Ile Ile Leu Arg Gly Gln Pro Ser Cys Thr
65 70 75 80
Lys Ala Tyr Lys Lys Glu Thr Asn Glu Thr Lys Glu Thr Asn Cys Thr
85 90 95
Asp Glu Arg Ile Thr Trp Val Ser Arg Pro Asp Gln Asn Ser Asp Leu
100 105 110
Gln Ile Arg Thr Val Ala Ile Thr His Asp Gly Tyr Tyr Arg Cys Ile
115 120 125
Met Val Thr Pro Asp Gly Asn Phe His Arg Gly Tyr His Leu Gln Val
130 135 140
Leu Val Thr Pro Glu Val Thr Leu Phe Gln Asn Arg Asn Arg Thr Ala
145 150 155 160
Val Cys Lys Ala Val Ala Gly Lys Pro Ala Ala His Ile Ser Trp Ile
165 170 175
Pro Glu Gly Asp Cys Ala Thr Lys Gln Glu Tyr Trp Ser Asn Gly Thr
180 185 190
Val Thr Val Lys Ser Thr Cys His Trp Glu Val His Asn Val Ser Thr
195 200 205
Val Thr Cys His Val Ser His
210 215
<210> 89
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 89
Leu Thr Gly Asn Lys Ser Leu Tyr Ile Glu Leu Leu Pro Val Pro Gly
1 5 10 15
Ala Lys Lys Ser Ala
20
<210> 90
<211> 61
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 90
Lys Val Asn Gly Cys Arg Lys Tyr Lys Leu Asn Lys Thr Glu Ser Thr
1 5 10 15
Pro Val Val Glu Glu Asp Glu Met Gln Pro Tyr Ala Ser Tyr Thr Glu
20 25 30
Lys Asn Asn Pro Leu Tyr Asp Thr Thr Asn Lys Val Lys Ala Ser Glu
35 40 45
Ala Leu Gln Ser Glu Val Asp Thr Asp Leu His Thr Leu
50 55 60
<210> 91
<211> 127
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 91
Lys Glu Ser Cys Asp Val Gln Leu Tyr Ile Lys Arg Gln Ser Glu His
1 5 10 15
Ser Ile Leu Ala Gly Asp Pro Phe Glu Leu Glu Cys Pro Val Lys Tyr
20 25 30
Cys Ala Asn Arg Pro His Val Thr Trp Cys Lys Leu Asn Gly Thr Thr
35 40 45
Cys Val Lys Leu Glu Asp Arg Gln Thr Ser Trp Lys Glu Glu Lys Asn
50 55 60
Ile Ser Phe Phe Ile Leu His Phe Glu Pro Val Leu Pro Asn Asp Asn
65 70 75 80
Gly Ser Tyr Arg Cys Ser Ala Asn Phe Gln Ser Asn Leu Ile Glu Ser
85 90 95
His Ser Thr Thr Leu Tyr Val Thr Asp Val Lys Ser Ala Ser Glu Arg
100 105 110
Pro Ser Lys Asp Glu Met Ala Ser Arg Pro Trp Leu Leu Tyr Arg
115 120 125
<210> 92
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 92
Leu Leu Pro Leu Gly Gly Leu Pro Leu Leu Ile Thr Thr Cys Phe Cys
1 5 10 15
Leu Phe Cys Cys Leu
20
<210> 93
<211> 111
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 93
Arg Arg His Gln Gly Lys Gln Asn Glu Leu Ser Asp Thr Ala Gly Arg
1 5 10 15
Glu Ile Asn Leu Val Asp Ala His Leu Lys Ser Glu Gln Thr Glu Ala
20 25 30
Ser Thr Arg Gln Asn Ser Gln Val Leu Leu Ser Glu Thr Gly Ile Tyr
35 40 45
Asp Asn Asp Pro Asp Leu Cys Phe Arg Met Gln Glu Gly Ser Glu Val
50 55 60
Tyr Ser Asn Pro Cys Leu Glu Glu Asn Lys Pro Gly Ile Val Tyr Ala
65 70 75 80
Ser Leu Asn His Ser Val Ile Gly Pro Asn Ser Arg Leu Ala Arg Asn
85 90 95
Val Lys Glu Ala Pro Thr Glu Tyr Ala Ser Ile Cys Val Arg Ser
100 105 110
<210> 94
<211> 181
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 94
Ser Glu Val Glu Tyr Arg Ala Glu Val Gly Gln Asn Ala Tyr Leu Pro
1 5 10 15
Cys Phe Tyr Thr Pro Ala Ala Pro Gly Asn Leu Val Pro Val Cys Trp
20 25 30
Gly Lys Gly Ala Cys Pro Val Phe Glu Cys Gly Asn Val Val Leu Arg
35 40 45
Thr Asp Glu Arg Asp Val Asn Tyr Trp Thr Ser Arg Tyr Trp Leu Asn
50 55 60
Gly Asp Phe Arg Lys Gly Asp Val Ser Leu Thr Ile Glu Asn Val Thr
65 70 75 80
Leu Ala Asp Ser Gly Ile Tyr Cys Cys Arg Ile Gln Ile Pro Gly Ile
85 90 95
Met Asn Asp Glu Lys Phe Asn Leu Lys Leu Val Ile Lys Pro Ala Lys
100 105 110
Val Thr Pro Ala Pro Thr Arg Gln Arg Asp Phe Thr Ala Ala Phe Pro
115 120 125
Arg Met Leu Thr Thr Arg Gly His Gly Pro Ala Glu Thr Gln Thr Leu
130 135 140
Gly Ser Leu Pro Asp Ile Asn Leu Thr Gln Ile Ser Thr Leu Ala Asn
145 150 155 160
Glu Leu Arg Asp Ser Arg Leu Ala Asn Asp Leu Arg Asp Ser Gly Ala
165 170 175
Thr Ile Arg Ile Gly
180
<210> 95
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 95
Ile Tyr Ile Gly Ala Gly Ile Cys Ala Gly Leu Ala Leu Ala Leu Ile
1 5 10 15
Phe Gly Ala Leu Ile
20
<210> 96
<211> 78
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 96
Phe Lys Trp Tyr Ser His Ser Lys Glu Lys Ile Gln Asn Leu Ser Leu
1 5 10 15
Ile Ser Leu Ala Asn Leu Pro Pro Ser Gly Leu Ala Asn Ala Val Ala
20 25 30
Glu Gly Ile Arg Ser Glu Glu Asn Ile Tyr Thr Ile Glu Glu Asn Val
35 40 45
Tyr Glu Val Glu Glu Pro Asn Glu Tyr Tyr Cys Tyr Val Ser Ser Arg
50 55 60
Gln Gln Pro Ser Gln Pro Leu Gly Cys Arg Phe Ala Met Pro
65 70 75
<210> 97
<211> 120
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 97
Met Met Thr Gly Thr Ile Glu Thr Thr Gly Asn Ile Ser Ala Glu Lys
1 5 10 15
Gly Gly Ser Ile Ile Leu Gln Cys His Leu Ser Ser Thr Thr Ala Gln
20 25 30
Val Thr Gln Val Asn Trp Glu Gln Gln Asp Gln Leu Leu Ala Ile Cys
35 40 45
Asn Ala Asp Leu Gly Trp His Ile Ser Pro Ser Phe Lys Asp Arg Val
50 55 60
Ala Pro Gly Pro Gly Leu Gly Leu Thr Leu Gln Ser Leu Thr Val Asn
65 70 75 80
Asp Thr Gly Glu Tyr Phe Cys Ile Tyr His Thr Tyr Pro Asp Gly Thr
85 90 95
Tyr Thr Gly Arg Ile Phe Leu Glu Val Leu Glu Ser Ser Val Ala Glu
100 105 110
His Gly Ala Arg Phe Gln Ile Pro
115 120
<210> 98
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 98
Leu Leu Gly Ala Met Ala Ala Thr Leu Val Val Ile Cys Thr Ala Val
1 5 10 15
Ile Val Val Val Ala
20
<210> 99
<211> 82
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 99
Leu Thr Arg Lys Lys Lys Ala Leu Arg Ile His Ser Val Glu Gly Asp
1 5 10 15
Leu Arg Arg Lys Ser Ala Gly Gln Glu Glu Trp Ser Pro Ser Ala Pro
20 25 30
Ser Pro Pro Gly Ser Cys Val Gln Ala Glu Ala Ala Pro Ala Gly Leu
35 40 45
Cys Gly Glu Gln Arg Gly Glu Asp Cys Ala Glu Leu His Asp Tyr Phe
50 55 60
Asn Val Leu Ser Tyr Arg Ser Leu Gly Asn Cys Ser Phe Phe Thr Glu
65 70 75 80
Thr Gly
<210> 100
<211> 144
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 100
Thr Ile Pro Pro His Val Gln Lys Ser Val Asn Asn Asp Met Ile Val
1 5 10 15
Thr Asp Asn Asn Gly Ala Val Lys Phe Pro Gln Leu Cys Lys Phe Cys
20 25 30
Asp Val Arg Phe Ser Thr Cys Asp Asn Gln Lys Ser Cys Met Ser Asn
35 40 45
Cys Ser Ile Thr Ser Ile Cys Glu Lys Pro Gln Glu Val Cys Val Ala
50 55 60
Val Trp Arg Lys Asn Asp Glu Asn Ile Thr Leu Glu Thr Val Cys His
65 70 75 80
Asp Pro Lys Leu Pro Tyr His Asp Phe Ile Leu Glu Asp Ala Ala Ser
85 90 95
Pro Lys Cys Ile Met Lys Glu Lys Lys Lys Pro Gly Glu Thr Phe Phe
100 105 110
Met Cys Ser Cys Ser Ser Asp Glu Cys Asn Asp Asn Ile Ile Phe Ser
115 120 125
Glu Glu Tyr Asn Thr Ser Asn Pro Asp Leu Leu Leu Val Ile Phe Gln
130 135 140
<210> 101
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 101
Val Thr Gly Ile Ser Leu Leu Pro Pro Leu Gly Val Ala Ile Ser Val
1 5 10 15
Ile Ile Ile Phe Tyr
20
<210> 102
<211> 380
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 102
Cys Tyr Arg Val Asn Arg Gln Gln Lys Leu Ser Ser Thr Trp Glu Thr
1 5 10 15
Gly Lys Thr Arg Lys Leu Met Glu Phe Ser Glu His Cys Ala Ile Ile
20 25 30
Leu Glu Asp Asp Arg Ser Asp Ile Ser Ser Thr Cys Ala Asn Asn Ile
35 40 45
Asn His Asn Thr Glu Leu Leu Pro Ile Glu Leu Asp Thr Leu Val Gly
50 55 60
Lys Gly Arg Phe Ala Glu Val Tyr Lys Ala Lys Leu Lys Gln Asn Thr
65 70 75 80
Ser Glu Gln Phe Glu Thr Val Ala Val Lys Ile Phe Pro Tyr Glu Glu
85 90 95
Tyr Ala Ser Trp Lys Thr Glu Lys Asp Ile Phe Ser Asp Ile Asn Leu
100 105 110
Lys His Glu Asn Ile Leu Gln Phe Leu Thr Ala Glu Glu Arg Lys Thr
115 120 125
Glu Leu Gly Lys Gln Tyr Trp Leu Ile Thr Ala Phe His Ala Lys Gly
130 135 140
Asn Leu Gln Glu Tyr Leu Thr Arg His Val Ile Ser Trp Glu Asp Leu
145 150 155 160
Arg Lys Leu Gly Ser Ser Leu Ala Arg Gly Ile Ala His Leu His Ser
165 170 175
Asp His Thr Pro Cys Gly Arg Pro Lys Met Pro Ile Val His Arg Asp
180 185 190
Leu Lys Ser Ser Asn Ile Leu Val Lys Asn Asp Leu Thr Cys Cys Leu
195 200 205
Cys Asp Phe Gly Leu Ser Leu Arg Leu Asp Pro Thr Leu Ser Val Asp
210 215 220
Asp Leu Ala Asn Ser Gly Gln Val Gly Thr Ala Arg Tyr Met Ala Pro
225 230 235 240
Glu Val Leu Glu Ser Arg Met Asn Leu Glu Asn Val Glu Ser Phe Lys
245 250 255
Gln Thr Asp Val Tyr Ser Met Ala Leu Val Leu Trp Glu Met Thr Ser
260 265 270
Arg Cys Asn Ala Val Gly Glu Val Lys Asp Tyr Glu Pro Pro Phe Gly
275 280 285
Ser Lys Val Arg Glu His Pro Cys Val Glu Ser Met Lys Asp Asn Val
290 295 300
Leu Arg Asp Arg Gly Arg Pro Glu Ile Pro Ser Phe Trp Leu Asn His
305 310 315 320
Gln Gly Ile Gln Met Val Cys Glu Thr Leu Thr Glu Cys Trp Asp His
325 330 335
Asp Pro Glu Ala Arg Leu Thr Ala Gln Cys Val Ala Glu Arg Phe Ser
340 345 350
Glu Leu Glu His Leu Asp Arg Leu Ser Gly Arg Ser Cys Ser Glu Glu
355 360 365
Lys Ile Pro Glu Asp Gly Ser Leu Asn Thr Thr Lys
370 375 380
<210> 103
<211> 214
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 103
His Gly Thr Glu Leu Pro Ser Pro Pro Ser Val Trp Phe Glu Ala Glu
1 5 10 15
Phe Phe His His Ile Leu His Trp Thr Pro Ile Pro Asn Gln Ser Glu
20 25 30
Ser Thr Cys Tyr Glu Val Ala Leu Leu Arg Tyr Gly Ile Glu Ser Trp
35 40 45
Asn Ser Ile Ser Asn Cys Ser Gln Thr Leu Ser Tyr Asp Leu Thr Ala
50 55 60
Val Thr Leu Asp Leu Tyr His Ser Asn Gly Tyr Arg Ala Arg Val Arg
65 70 75 80
Ala Val Asp Gly Ser Arg His Ser Asn Trp Thr Val Thr Asn Thr Arg
85 90 95
Phe Ser Val Asp Glu Val Thr Leu Thr Val Gly Ser Val Asn Leu Glu
100 105 110
Ile His Asn Gly Phe Ile Leu Gly Lys Ile Gln Leu Pro Arg Pro Lys
115 120 125
Met Ala Pro Ala Asn Asp Thr Tyr Glu Ser Ile Phe Ser His Phe Arg
130 135 140
Glu Tyr Glu Ile Ala Ile Arg Lys Val Pro Gly Asn Phe Thr Phe Thr
145 150 155 160
His Lys Lys Val Lys His Glu Asn Phe Ser Leu Leu Thr Ser Gly Glu
165 170 175
Val Gly Glu Phe Cys Val Gln Val Lys Pro Ser Val Ala Ser Arg Ser
180 185 190
Asn Lys Gly Met Trp Ser Lys Glu Glu Cys Ile Ser Leu Thr Arg Gln
195 200 205
Tyr Phe Thr Val Thr Asn
210
<210> 104
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 104
Val Ile Ile Phe Phe Ala Phe Val Leu Leu Leu Ser Gly Ala Leu Ala
1 5 10 15
Tyr Cys Leu Ala Leu
20
<210> 105
<211> 322
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 105
Gln Leu Tyr Val Arg Arg Arg Lys Lys Leu Pro Ser Val Leu Leu Phe
1 5 10 15
Lys Lys Pro Ser Pro Phe Ile Phe Ile Ser Gln Arg Pro Ser Pro Glu
20 25 30
Thr Gln Asp Thr Ile His Pro Leu Asp Glu Glu Ala Phe Leu Lys Val
35 40 45
Ser Pro Glu Leu Lys Asn Leu Asp Leu His Gly Ser Thr Asp Ser Gly
50 55 60
Phe Gly Ser Thr Lys Pro Ser Leu Gln Thr Glu Glu Pro Gln Phe Leu
65 70 75 80
Leu Pro Asp Pro His Pro Gln Ala Asp Arg Thr Leu Gly Asn Arg Glu
85 90 95
Pro Pro Val Leu Gly Asp Ser Cys Ser Ser Gly Ser Ser Asn Ser Thr
100 105 110
Asp Ser Gly Ile Cys Leu Gln Glu Pro Ser Leu Ser Pro Ser Thr Gly
115 120 125
Pro Thr Trp Glu Gln Gln Val Gly Ser Asn Ser Arg Gly Gln Asp Asp
130 135 140
Ser Gly Ile Asp Leu Val Gln Asn Ser Glu Gly Arg Ala Gly Asp Thr
145 150 155 160
Gln Gly Gly Ser Ala Leu Gly His His Ser Pro Pro Glu Pro Glu Val
165 170 175
Pro Gly Glu Glu Asp Pro Ala Ala Val Ala Phe Gln Gly Tyr Leu Arg
180 185 190
Gln Thr Arg Cys Ala Glu Glu Lys Ala Thr Lys Thr Gly Cys Leu Glu
195 200 205
Glu Glu Ser Pro Leu Thr Asp Gly Leu Gly Pro Lys Phe Gly Arg Cys
210 215 220
Leu Val Asp Glu Ala Gly Leu His Pro Pro Ala Leu Ala Lys Gly Tyr
225 230 235 240
Leu Lys Gln Asp Pro Leu Glu Met Thr Leu Ala Ser Ser Gly Ala Pro
245 250 255
Thr Gly Gln Trp Asn Gln Pro Thr Glu Glu Trp Ser Leu Leu Ala Leu
260 265 270
Ser Ser Cys Ser Asp Leu Gly Ile Ser Asp Trp Ser Phe Ala His Asp
275 280 285
Leu Ala Pro Leu Gly Cys Val Ala Ala Pro Gly Gly Leu Leu Gly Ser
290 295 300
Phe Asn Ser Asp Leu Val Thr Leu Pro Leu Ile Ser Ser Leu Gln Ser
305 310 315 320
Ser Glu
<210> 106
<211> 207
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 106
Met Lys Val Leu Gln Glu Pro Thr Cys Val Ser Asp Tyr Met Ser Ile
1 5 10 15
Ser Thr Cys Glu Trp Lys Met Asn Gly Pro Thr Asn Cys Ser Thr Glu
20 25 30
Leu Arg Leu Leu Tyr Gln Leu Val Phe Leu Leu Ser Glu Ala His Thr
35 40 45
Cys Ile Pro Glu Asn Asn Gly Gly Ala Gly Cys Val Cys His Leu Leu
50 55 60
Met Asp Asp Val Val Ser Ala Asp Asn Tyr Thr Leu Asp Leu Trp Ala
65 70 75 80
Gly Gln Gln Leu Leu Trp Lys Gly Ser Phe Lys Pro Ser Glu His Val
85 90 95
Lys Pro Arg Ala Pro Gly Asn Leu Thr Val His Thr Asn Val Ser Asp
100 105 110
Thr Leu Leu Leu Thr Trp Ser Asn Pro Tyr Pro Pro Asp Asn Tyr Leu
115 120 125
Tyr Asn His Leu Thr Tyr Ala Val Asn Ile Trp Ser Glu Asn Asp Pro
130 135 140
Ala Asp Phe Arg Ile Tyr Asn Val Thr Tyr Leu Glu Pro Ser Leu Arg
145 150 155 160
Ile Ala Ala Ser Thr Leu Lys Ser Gly Ile Ser Tyr Arg Ala Arg Val
165 170 175
Arg Ala Trp Ala Gln Cys Tyr Asn Thr Thr Trp Ser Glu Trp Ser Pro
180 185 190
Ser Thr Lys Trp His Asn Ser Tyr Arg Glu Pro Phe Glu Gln His
195 200 205
<210> 107
<211> 24
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 107
Leu Leu Leu Gly Val Ser Val Ser Cys Ile Val Ile Leu Ala Val Cys
1 5 10 15
Leu Leu Cys Tyr Val Ser Ile Thr
20
<210> 108
<211> 569
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 108
Lys Ile Lys Lys Glu Trp Trp Asp Gln Ile Pro Asn Pro Ala Arg Ser
1 5 10 15
Arg Leu Val Ala Ile Ile Ile Gln Asp Ala Gln Gly Ser Gln Trp Glu
20 25 30
Lys Arg Ser Arg Gly Gln Glu Pro Ala Lys Cys Pro His Trp Lys Asn
35 40 45
Cys Leu Thr Lys Leu Leu Pro Cys Phe Leu Glu His Asn Met Lys Arg
50 55 60
Asp Glu Asp Pro His Lys Ala Ala Lys Glu Met Pro Phe Gln Gly Ser
65 70 75 80
Gly Lys Ser Ala Trp Cys Pro Val Glu Ile Ser Lys Thr Val Leu Trp
85 90 95
Pro Glu Ser Ile Ser Val Val Arg Cys Val Glu Leu Phe Glu Ala Pro
100 105 110
Val Glu Cys Glu Glu Glu Glu Glu Val Glu Glu Glu Lys Gly Ser Phe
115 120 125
Cys Ala Ser Pro Glu Ser Ser Arg Asp Asp Phe Gln Glu Gly Arg Glu
130 135 140
Gly Ile Val Ala Arg Leu Thr Glu Ser Leu Phe Leu Asp Leu Leu Gly
145 150 155 160
Glu Glu Asn Gly Gly Phe Cys Gln Gln Asp Met Gly Glu Ser Cys Leu
165 170 175
Leu Pro Pro Ser Gly Ser Thr Ser Ala His Met Pro Trp Asp Glu Phe
180 185 190
Pro Ser Ala Gly Pro Lys Glu Ala Pro Pro Trp Gly Lys Glu Gln Pro
195 200 205
Leu His Leu Glu Pro Ser Pro Pro Ala Ser Pro Thr Gln Ser Pro Asp
210 215 220
Asn Leu Thr Cys Thr Glu Thr Pro Leu Val Ile Ala Gly Asn Pro Ala
225 230 235 240
Tyr Arg Ser Phe Ser Asn Ser Leu Ser Gln Ser Pro Cys Pro Arg Glu
245 250 255
Leu Gly Pro Asp Pro Leu Leu Ala Arg His Leu Glu Glu Val Glu Pro
260 265 270
Glu Met Pro Cys Val Pro Gln Leu Ser Glu Pro Thr Thr Val Pro Gln
275 280 285
Pro Glu Pro Glu Thr Trp Glu Gln Ile Leu Arg Arg Asn Val Leu Gln
290 295 300
His Gly Ala Ala Ala Ala Pro Val Ser Ala Pro Thr Ser Gly Tyr Gln
305 310 315 320
Glu Phe Val His Ala Val Glu Gln Gly Gly Thr Gln Ala Ser Ala Val
325 330 335
Val Gly Leu Gly Pro Pro Gly Glu Ala Gly Tyr Lys Ala Phe Ser Ser
340 345 350
Leu Leu Ala Ser Ser Ala Val Ser Pro Glu Lys Cys Gly Phe Gly Ala
355 360 365
Ser Ser Gly Glu Glu Gly Tyr Lys Pro Phe Gln Asp Leu Ile Pro Gly
370 375 380
Cys Pro Gly Asp Pro Ala Pro Val Pro Val Pro Leu Phe Thr Phe Gly
385 390 395 400
Leu Asp Arg Glu Pro Pro Arg Ser Pro Gln Ser Ser His Leu Pro Ser
405 410 415
Ser Ser Pro Glu His Leu Gly Leu Glu Pro Gly Glu Lys Val Glu Asp
420 425 430
Met Pro Lys Pro Pro Leu Pro Gln Glu Gln Ala Thr Asp Pro Leu Val
435 440 445
Asp Ser Leu Gly Ser Gly Ile Val Tyr Ser Ala Leu Thr Cys His Leu
450 455 460
Cys Gly His Leu Lys Gln Cys His Gly Gln Glu Asp Gly Gly Gln Thr
465 470 475 480
Pro Val Met Ala Ser Pro Cys Cys Gly Cys Cys Cys Gly Asp Arg Ser
485 490 495
Ser Pro Pro Thr Thr Pro Leu Arg Ala Pro Asp Pro Ser Pro Gly Gly
500 505 510
Val Pro Leu Glu Ala Ser Leu Cys Pro Ala Ser Leu Ala Pro Ser Gly
515 520 525
Ile Ser Glu Lys Ser Lys Ser Ser Ser Ser Phe His Pro Ala Pro Gly
530 535 540
Asn Ala Gln Ser Ser Ser Gln Thr Pro Lys Ile Val Asn Phe Val Ser
545 550 555 560
Val Gly Pro Thr Tyr Met Arg Val Ser
565
<210> 109
<211> 25
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 109
Pro Ile Leu Leu Thr Ile Ser Ile Leu Ser Phe Phe Ser Val Ala Leu
1 5 10 15
Leu Val Ile Leu Ala Cys Val Leu Trp
20 25
<210> 110
<211> 195
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 110
Lys Lys Arg Ile Lys Pro Ile Val Trp Pro Ser Leu Pro Asp His Lys
1 5 10 15
Lys Thr Leu Glu His Leu Cys Lys Lys Pro Arg Lys Asn Leu Asn Val
20 25 30
Ser Phe Asn Pro Glu Ser Phe Leu Asp Cys Gln Ile His Arg Val Asp
35 40 45
Asp Ile Gln Ala Arg Asp Glu Val Glu Gly Phe Leu Gln Asp Thr Phe
50 55 60
Pro Gln Gln Leu Glu Glu Ser Glu Lys Gln Arg Leu Gly Gly Asp Val
65 70 75 80
Gln Ser Pro Asn Cys Pro Ser Glu Asp Val Val Ile Thr Pro Glu Ser
85 90 95
Phe Gly Arg Asp Ser Ser Leu Thr Cys Leu Ala Gly Asn Val Ser Ala
100 105 110
Cys Asp Ala Pro Ile Leu Ser Ser Ser Arg Ser Leu Asp Cys Arg Glu
115 120 125
Ser Gly Lys Asn Gly Pro His Val Tyr Gln Asp Leu Leu Leu Ser Leu
130 135 140
Gly Thr Thr Asn Ser Thr Leu Pro Pro Pro Phe Ser Leu Gln Ser Gly
145 150 155 160
Ile Leu Thr Leu Asn Pro Val Ala Gln Gly Gln Pro Ile Leu Thr Ser
165 170 175
Leu Gly Ser Asn Gln Glu Glu Ala Tyr Val Thr Met Ser Ser Phe Tyr
180 185 190
Gln Asn Gln
195
<210> 111
<211> 148
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 111
Gln Val Thr Asp Ile Asn Ser Lys Gly Leu Glu Leu Arg Lys Thr Val
1 5 10 15
Thr Thr Val Glu Thr Gln Asn Leu Glu Gly Leu His His Asp Gly Gln
20 25 30
Phe Cys His Lys Pro Cys Pro Pro Gly Glu Arg Lys Ala Arg Asp Cys
35 40 45
Thr Val Asn Gly Asp Glu Pro Asp Cys Val Pro Cys Gln Glu Gly Lys
50 55 60
Glu Tyr Thr Asp Lys Ala His Phe Ser Ser Lys Cys Arg Arg Cys Arg
65 70 75 80
Leu Cys Asp Glu Gly His Gly Leu Glu Val Glu Ile Asn Cys Thr Arg
85 90 95
Thr Gln Asn Thr Lys Cys Arg Cys Lys Pro Asn Phe Phe Cys Asn Ser
100 105 110
Thr Val Cys Glu His Cys Asp Pro Cys Thr Lys Cys Glu His Gly Ile
115 120 125
Ile Lys Glu Cys Thr Leu Thr Ser Asn Thr Lys Cys Lys Glu Glu Gly
130 135 140
Ser Arg Ser Asn
145
<210> 112
<211> 17
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 112
Leu Gly Trp Leu Cys Leu Leu Leu Leu Pro Ile Pro Leu Ile Val Trp
1 5 10 15
Val
<210> 113
<211> 145
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 113
Lys Arg Lys Glu Val Gln Lys Thr Cys Arg Lys His Arg Lys Glu Asn
1 5 10 15
Gln Gly Ser His Glu Ser Pro Thr Leu Asn Pro Glu Thr Val Ala Ile
20 25 30
Asn Leu Ser Asp Val Asp Leu Ser Lys Tyr Ile Thr Thr Ile Ala Gly
35 40 45
Val Met Thr Leu Ser Gln Val Lys Gly Phe Val Arg Lys Asn Gly Val
50 55 60
Asn Glu Ala Lys Ile Asp Glu Ile Lys Asn Asp Asn Val Gln Asp Thr
65 70 75 80
Ala Glu Gln Lys Val Gln Leu Leu Arg Asn Trp His Gln Leu His Gly
85 90 95
Lys Lys Glu Ala Tyr Asp Thr Leu Ile Lys Asp Leu Lys Lys Ala Asn
100 105 110
Leu Cys Thr Leu Ala Glu Lys Ile Gln Thr Ile Ile Leu Lys Asp Ile
115 120 125
Thr Ser Asp Ser Glu Asn Ser Asn Phe Arg Asn Glu Ile Gln Ser Leu
130 135 140
Val
145
<210> 114
<211> 155
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 114
Ile Thr Gln Gln Asp Leu Ala Pro Gln Gln Arg Ala Ala Pro Gln Gln
1 5 10 15
Lys Arg Ser Ser Pro Ser Glu Gly Leu Cys Pro Pro Gly His His Ile
20 25 30
Ser Glu Asp Gly Arg Asp Cys Ile Ser Cys Lys Tyr Gly Gln Asp Tyr
35 40 45
Ser Thr His Trp Asn Asp Leu Leu Phe Cys Leu Arg Cys Thr Arg Cys
50 55 60
Asp Ser Gly Glu Val Glu Leu Ser Pro Cys Thr Thr Thr Arg Asn Thr
65 70 75 80
Val Cys Gln Cys Glu Glu Gly Thr Phe Arg Glu Glu Asp Ser Pro Glu
85 90 95
Met Cys Arg Lys Cys Arg Thr Gly Cys Pro Arg Gly Met Val Lys Val
100 105 110
Gly Asp Cys Thr Pro Trp Ser Asp Ile Glu Cys Val His Lys Glu Ser
115 120 125
Gly Thr Lys His Ser Gly Glu Val Pro Ala Val Glu Glu Thr Val Thr
130 135 140
Ser Ser Pro Gly Thr Pro Ala Ser Pro Cys Ser
145 150 155
<210> 115
<211> 21
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 115
Leu Ser Gly Ile Ile Ile Gly Val Thr Val Ala Ala Val Val Leu Ile
1 5 10 15
Val Ala Val Phe Val
20
<210> 116
<211> 209
<212> PRT
<213> 人工序列(Artificial Sequence)
<220>
<223> 合成构建体
<400> 116
Cys Lys Ser Leu Leu Trp Lys Lys Val Leu Pro Tyr Leu Lys Gly Ile
1 5 10 15
Cys Ser Gly Gly Gly Gly Asp Pro Glu Arg Val Asp Arg Ser Ser Gln
20 25 30
Arg Pro Gly Ala Glu Asp Asn Val Leu Asn Glu Ile Val Ser Ile Leu
35 40 45
Gln Pro Thr Gln Val Pro Glu Gln Glu Met Glu Val Gln Glu Pro Ala
50 55 60
Glu Pro Thr Gly Val Asn Met Leu Ser Pro Gly Glu Ser Glu His Leu
65 70 75 80
Leu Glu Pro Ala Glu Ala Glu Arg Ser Gln Arg Arg Arg Leu Leu Val
85 90 95
Pro Ala Asn Glu Gly Asp Pro Thr Glu Thr Leu Arg Gln Cys Phe Asp
100 105 110
Asp Phe Ala Asp Leu Val Pro Phe Asp Ser Trp Glu Pro Leu Met Arg
115 120 125
Lys Leu Gly Leu Met Asp Asn Glu Ile Lys Val Ala Lys Ala Glu Ala
130 135 140
Ala Gly His Arg Asp Thr Leu Tyr Thr Met Leu Ile Lys Trp Val Asn
145 150 155 160
Lys Thr Gly Arg Asp Ala Ser Val His Thr Leu Leu Asp Ala Leu Glu
165 170 175
Thr Leu Gly Glu Arg Leu Ala Lys Gln Lys Ile Glu Asp His Leu Leu
180 185 190
Ser Ser Gly Lys Phe Met Tyr Leu Glu Gly Asn Ala Asp Ser Ala Met
195 200 205
Ser
<210> 117
<211> 49
<212> DNA
<213> 人工序列(Artificial sequence)
<220>
<223> 合成构建体
<400> 117
aatgatacgg cgaccaccga gatctacact ctttccctac acgacgctc 49
<210> 118
<211> 62
<212> DNA
<213> 人工序列(Artificial sequence)
<220>
<223> 合成构建体
<400> 118
gtgactggag ttcagacgtg tgctcttccg atctgaggaa ccagccttat tgttcatccg 60
ta 62
- 合并的敲入筛选和在内源基因座控制下共表达的异源多肽
- 一种通过催化内源糖转化成异源糖来提高总碳水化合物或可溶性碳水化合物含量或内源碳水化合物甜度的方法
